SNP判別方法
【課題】アレル特異的プライマーを用いたSNP判別方法において、非特異的な増幅反応を抑制し、より明確なSNP判別を可能とする方法を提供する。
【解決手段】二本鎖標的配列(1)は、一本鎖標的配列(1a)および相補的一本鎖標的配列(1b)からなり、 第1一本鎖DNA(6)は、3’末端−第1非増幅配列(6a)−前記一本鎖標的配列(1a)−第2非増幅配列(6b)−5’末端からなり、 前記第2一本鎖DNA(7)は、5’末端−第3非増幅配列(7a)−前記相補的一本鎖標的配列(1b)−第4非増幅配列(7b)−3’末端からなり、前記二本鎖標的配列(1)をASPを用いたPCR法で増幅する工程において、DNAポリメラーゼによる伸長反応のための起点として作用せず、前記第3非増幅配列(7a)の一部と相補的である、第1ブロック核酸(20)を混合する方法。
【解決手段】二本鎖標的配列(1)は、一本鎖標的配列(1a)および相補的一本鎖標的配列(1b)からなり、 第1一本鎖DNA(6)は、3’末端−第1非増幅配列(6a)−前記一本鎖標的配列(1a)−第2非増幅配列(6b)−5’末端からなり、 前記第2一本鎖DNA(7)は、5’末端−第3非増幅配列(7a)−前記相補的一本鎖標的配列(1b)−第4非増幅配列(7b)−3’末端からなり、前記二本鎖標的配列(1)をASPを用いたPCR法で増幅する工程において、DNAポリメラーゼによる伸長反応のための起点として作用せず、前記第3非増幅配列(7a)の一部と相補的である、第1ブロック核酸(20)を混合する方法。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、PCRを利用したSNP判別方法に関わる。
【背景技術】
【0002】
近年、体外診断薬を含む分子診断市場は急速な伸びを示しており、医療分野では、疾患関連遺伝子を解析することにより、疾患の分子レベルでの治療が可能となってきている。
【0003】
遺伝子検査は、遺伝子の病気と言われるようになった癌領域だけではなく、感染症や、アルツハイマ−病、生活習慣病の発症リスクや、薬物治療に対する効果や安全性を調べる用途にも利用することができることから医療分野での注目が集まっている。その中でも特に塩基配列中の一塩基対での違いを示す一塩基多型(Single Nucleotide Polymorphism;SNP)は、このわずかな違いにより病気のかかりやすさや薬効に個人差が生まれることが明らかとなってきているため、SNP検査を行なう重要性は高まっている。例えば、分子標的治療剤として日本で承認された薬剤であるゲフィニチブは、EGFR遺伝子に存在するSNPの違いによって治療効果が異なることが報告されている。このようにSNPを判別する重要性が高まる中、様々な分析手法が開発されてきている。
【0004】
遺伝子検査において、検体中に存在するDNAのコピ−数が少ない場合、目的とする遺伝子の領域を増幅して感度を高める方法がある。一般的な核酸増幅法であるPolymerase Chain Reaction(PCR)法は鋳型DNA中の目的とする配列領域(以下、標的配列と表記)をその両端の配列に相補的な一対のプライマ−及びDNAポリメラ−ゼを用いて、3段階の温度変化をnサイクル繰り返すことによって2n倍に増幅する手法である。3段階の各工程は二本鎖DNAを一本鎖DNAに変性する工程と、一本鎖DNA中の標的配列にプライマ−をハイブリダイゼ−ションさせる工程、DNAポリメラ−ゼによるDNAの伸長反応を行う工程よりなる。1サイクルで標的配列の濃度は2倍になり、理論的にはnサイクルの反応で標的配列は2n倍に増幅される。実際には100万倍程度に増幅することができる技術である。
【0005】
このPCR法を改良し、SNPを判別する手法が開発されている。代表的な方法として、アレル特異的プライマ−(Allele Specific Primer;ASP)を用いたSNP判別方法がある。ASPとはSNP塩基がある特定の塩基種の場合にのみ効率よく伸長反応が起こるように設計されたプライマ−のことを言う。代表的なASPとして非特許文献1が報告されている。
【0006】
非特許文献1ではASPとしてプライマ−の3’末端の塩基が標的のSNP塩基と対応するように設計されたプライマ−を用いてSNP判別を行なっている。3’末端塩基がSNP塩基に対して相補的であれば、伸長反応が効率良く起こるが、非相補的である場合は、伸長反応が進まない、あるいはその効率が低下する。したがって、ASPを用いてPCRを行い、その結果得られるPCR産物量や、あるいはPCRの反応効率を解析することで、標的のSNP塩基の塩基種を知ることができる。PCR産物量を解析する方法としては、電気泳動法や、リアルタイムPCR法、ポリメラ−ゼ伸長反応時に副産物として生成されるピロリン酸を光学的あるいは電気化学的に測定する手法が知られている。
【0007】
しかしながら、上記ASPを用いたSNP判別方法は、3’末端の一塩基のみで伸長反応を制御するものであるので、特異的かつ厳密に制御することは難しい。その結果、ASPのミスハイブリダイゼ−ションにより、擬陽性を生じる場合があった。そこで、擬陽性を抑えるため、新たなASPの開発が行なわれている。
【0008】
例えば、ASPは、3’ 末端塩基がSNP対応塩基となるように設計され、かつその5’側上流の3番目の塩基を、鋳型に対して意図的に非相補的になるように設計する方法が報告されている(特許文献1)。
【0009】
また、ASPは、3’末端から2番目の塩基がSNP対応塩基になるように設計され、かつその5’側に一つ上流の塩基を、鋳型に対して意図的に非相補的な配列になるように設計がなされている(特許文献2)。
【0010】
また、ASPは、3’末端塩基がSNP対応塩基となるように設計され、かつその5’側上流2番目と3番目の塩基が、鋳型に対して意図的に非相補的な配列となるように設計され、鋳型の配列に応じて特異性の高い塩基配列を選定している(特許文献3)。
【0011】
上述の新規に開発されたASPは、SNP対応塩基以外に、鋳型に対して非相補的な塩基を意図的に導入することにより、SNP対応塩基がSNP塩基に対して非相補的である場合、結合能をより弱くし、擬陽性を抑制する点で優れている。
【0012】
しかしその一方で、プライマ−への非相補的な塩基の導入は、プライマ−の標的配列への特異性を低くし、標的配列とは異なる配列に対して非特異的に結合する場合があった。その結果、ASPを用いてPCRを行うと、標的配列とは異なるDNA配列が増幅される場合があった。このような非特異的な増幅は、ピロリン酸を測定する手法においては、バックグラウンドとなるため重要な課題である。また、ASPを用いてリアルタイムPCRを行った場合、SNPの塩基種が違っていても増幅曲線の立ち上がりの差が小さく、明確にSNP判別をすることができない場合があった。
【0013】
そこで、プライマ−の非特異的な結合を防ぐ方法が報告されている。この方法では、プライマ−が非特異的に結合する可能性のある配列を予め網羅的に調べ予測し、それらの配列と完全に相補的な配列を有する競合核酸を反応液中に添加することで、プライマ−の非特異的な結合が起こらないようにすることを特徴としている(特許文献4)。
【先行技術文献】
【特許文献】
【0014】
【特許文献1】米国特許出願公開第2003/0049628号明細書
【特許文献2】米国特許出願公開第2003/0148301号明細書
【特許文献3】米国特許出願公開第2004/0197803号明細書
【特許文献4】特開2004−194619号公報
【非特許文献】
【0015】
【非特許文献1】Nucleic Acids Res.,17,2503−2516(1989).
【発明の概要】
【発明が解決しようとする課題】
【0016】
しかしながら、非特異に結合する可能性を予め完全に予測することは難しく、この方法により非特異的な結合反応を完全に防ぐことには限界があった。
【0017】
本発明は、前記従来の課題を解決するために、ASPを用いたPCRにおいて、プライマ−の反応特異性の低下によって引き起こされる、非特異的な増幅反応を抑制し、より明確なSNP判別を可能とする方法を提供することを目的とする。
【課題を解決するための手段】
【0018】
前記従来の課題を解決するために、本発明のSNP判別方法は、第1一本鎖DNA(6)および第2一本鎖DNA(7)からなる二本鎖DNA中の二本鎖標的配列(1)中に含まれるSNPの塩基種(8)を、アレル特異的プライマ−を用いて判別するSNP判別法であって、前記二本鎖標的配列(1)は、一本鎖標的配列(1a)および相補的一本鎖標的配列(1b)からなり、前記第1一本鎖DNA(6)は、3’末端−第1非増幅配列(6a)−前記一本鎖標的配列(1a)−第2非増幅配列(6b)−5’末端からなり、前記第2一本鎖DNA(7)は、5’末端−第3非増幅配列(7a)−前記相補的一本鎖標的配列(1b)−第4非増幅配列(7b)−3’末端からなり、前記相補的一本鎖標的配列(1b)、第3非増幅配列(7a)、および第4非増幅配列(7b)は、それぞれ、前記一本鎖標的配列(1a)、前記第1非増幅配列(6a)、および第2非増幅配列(6b)と相補的であり、DNAポリメラ−ゼ、デオキシヌクレオシド三リン酸、前記二本鎖DNA(6・7)、第1のプライマ−(4)、第2のプライマ−(5)、および第1ブロック核酸(20)を混合し、ポリメラ−ゼ連鎖反応を用いて前記二本鎖標的配列(1)を増幅する工程Aを含み、ここで、前記第1のプライマ−(4)および前記第2のプライマ−(5)は、いずれも、前記DNAポリメラ−ゼによる伸長反応のための起点として作用し、前記SNP塩基種(8)は、第一の一本鎖DNA(6)上の前記第1のプライマ−(4)が結合する領域内に存在し、前記第1のプライマ−(4)は、SNPの塩基(8)に相補的な配列である、アレル特異的プライマ−であって、前記第1のプライマ−(4)は、前記一本鎖標的配列(1a)に含まれる3’末端側の配列の部分に相補的であり、前記第2のプライマ−(5)は、前記相補的一本鎖標的配列(1b)に含まれる3’末端側の部分の配列に相補的であり、前記第1ブロック核酸(20)は、前記DNAポリメラ−ゼによる伸長反応のための起点として作用せず、前記第1ブロック核酸(20)は、前記第3非増幅配列(7a)の一部と相補的である、ことを特徴としたものである。
【発明の効果】
【0019】
本発明により、ASPを用いたPCRにおいて、非特異的な増幅を抑制することができ、より明確なSNP判別が可能なSNP判別法が提供される。
【図面の簡単な説明】
【0020】
【図1】従来のPCRを示す図
【図2】本発明に係るPCRを示す図
【図3】実施例1におけるリアルタイムPCRの増幅曲線を示すグラフ
【図4】実施例1における野生型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図5】実施例1における変異型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図6】比較例1におけるリアルタイムPCRの増幅曲線を示すグラフ
【図7】比較例1における野生型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図8】比較例1における変異型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図9】実施例1における増幅産物の総和を比較したグラフ
【図10】比較例1における増幅産物の総和を比較したグラフ
【図11】実施例2におけるリアルタイムPCRの増幅曲線を示すグラフ
【図12】実施例2における野生型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図13】実施例2における変異型の鋳型を用いた場合の電気泳動時の結果を示すグラフ
【図14】比較例2におけるリアルタイムPCRの増幅曲線を示すグラフ
【図15】比較例2における野生型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図16】比較例2における変異型の鋳型を用いた場合の電気泳動時の結果を示すグラフ
【図17】実施例2における増幅産物の総和を比較したグラフ
【図18】比較例2における増幅産物の総和を比較したグラフ
【発明を実施するための形態】
【0021】
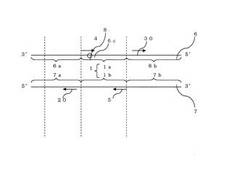
以下、本発明に係わるSNP判別法の具体的な実施の形態を説明する。ただし、本発明はこれに限定されるものではない。本実施の形態では、図1および図2を用い、第1のプライマ−(4)がASPである場合を例にとって説明する。
【0022】
従来のPCRでは、図1に示すように、第1一本鎖DNA6は3’末端−第1非増幅配列(6a)−一本鎖標的配列(1a)−第2非増幅配列(6b)−5’末端からなり、第2一本鎖DNA7は5’末端−第3非増幅配列(7a)−相補的一本鎖標的配列(1b)−第4非増幅配列(7b)−3’末端からなる。第1のプライマ−(4)は一本鎖標的配列(1a)の3’末端部分(6c)に相補的に結合する。SNP塩基(8)は第1一本鎖DNA(6)の鎖上に存在し、かつ第1のプライマ−(4)が結合する領域内に存在する。一方、第2のプライマ−(5)は、相補的一本鎖標的配列(1b)の3’末端部分に相補的に結合する。
【0023】
したがって、第1のプライマ−(4)と第2のプライマ−(5)からDNA伸長反応が良好におこり、PCRが行なわれると、得られる増幅二本鎖DNA配列は、望まれる増幅二本鎖DNA配列2が得られる。しかし、背景技術でも記したとおり、ASPのようなプライマ−を用いた場合はそれ以外の非特異的な増幅により望まれない産物が得られる場合があった。これを解決するのが本発明である。
【0024】
ここで、本発明におけるブロック核酸(20)について図2を用いて説明する。本発明の特徴は、背景技術に記載のASPを用いたPCRにおけるSNP判別法にブロック核酸を加えることである。具体的にはDNAポリメラ−ゼ、デオキシヌクレオシド三リン酸(dATP、dCTP、dGTP、dTTP)、鋳型DNA、第1のプライマ−および第2のプライマ−を含むPCR反応溶液にブロック核酸を共存させてPCRを行なう。第1のプライマ−及び、第2のプライマ−はフォワ−ドプライマ−とリバ−スプライマ−の関係にあって、両プライマ−の3’末端より伸長反応が進み、標的配列を増幅する。本実施の形態では、第1のプライマ−か第2のプライマ−の何れかはASPである。好ましくは上記反応溶液にDNAポリメラ−ゼが安定に働くための緩衝液、塩類を加えても良い。
【0025】
ブロック核酸(20)は、DNAポリメラ−ゼによる伸長反応のための起点として作用せず、第3非増幅配列(7a)の一部と相補的である。本発明におけるブロック核酸(30)は、DNAポリメラ−ゼによる伸長反応のための起点として作用せず、第2非増幅配列(6b)の一部と相補的である。
【0026】
本発明のPCRにおいては上記ブロック核酸(20)とブロック核酸(30)の少なくともいずれか一方、好適には両ブロック核酸が反応溶液中に加えられる。
【0027】
また上述の通り、ブロック核酸はDNAポリメラ−ゼによる伸長反応の起点とならない合成オリゴ核酸である。したがってブロック核酸としては、例えば、3’末端にDNAポリメラ−ゼによる伸長反応の起点とならない修飾が施されているDNAか、もしくはLocked Nucleic Acid(LNA)が挙げられる。修飾の種類としては好適には、リン酸化、アミノ化、蛍光修飾、ビオチン等が含まれるが、これに限るものではない。またブロック核酸は、3’末端のヌクレオチドがジデオキシヌクレオチドであるDNAやLNAであっても良い。またブロック核酸は、伸長反応の起点とならない核酸類似体であってもよい。伸長反応の起点とならない核酸類似体としては、例えばPeptide nucleic acid(PNA)などが挙げられる。
【0028】
本発明におけるブロック核酸は標的配列(1)の近傍に結合するように設計されることが望ましい。ここでいう近傍とは標的配列(1)から数塩基から数千塩基以内の領域である。ブロック核酸の配列は、好ましくはプライマ−のTm値と同等以上になるように設計される。反応溶液中におけるブロック核酸の濃度は、プライマ−の濃度より大きいことが好ましい。例えばプライマ−が0.1μM〜1μMの濃度範囲であれば、ブロック核酸はその5倍以上の濃度で加えられることが好ましい。
【0029】
本発明に用いられるDNAポリメラ−ゼは、一例として、Taq DNA Polymeraseなどが挙げられるが、これに限定されるものではなく、PCRで使用可能なものであれば、どのようなものでも使用することができ、適宜用途に合わせて選択すればよい。
【0030】
ただし、鎖置換型の活性を持ったポリメラ−ゼは使用することができない。DNAポリメラ−ゼの濃度は通常、0.5〜2.5Unit/100μLで使用されることが望ましい。緩衝液は上記DNAポリメラ−ゼが反応するために適切な溶媒であれば何でもよく、一般的にはTris−HClが使用され、pHは7〜10、濃度は10mM〜100mMで調製されることが望ましい。塩類は好適には塩化マグネシウムが使用されており、1mM〜5mMで添加され、好適な範囲は1.5mM〜3mMである。
【0031】
デオキシヌクレオシド三リン酸は4種類(dATP、dTTP、dGTP、dCTP)よりなり、通常、各々が等量で20μM〜200μMで含まれる。
【0032】
本発明における標的配列とは、背景技術で説明したようにSNP塩基を含み、一対のプライマ−で囲まれ、PCRで増幅される領域を示す。
【0033】
また、本発明におけるプライマ−とは、DNAポリメラ−ゼによる伸長反応の起点となる合成オリゴ核酸である。プライマ−の長さは、伸長反応のためのプライマ−として働くのに十分な長さであればよく、通常、15bp〜50bpの範囲で設計される。より好ましくは、20bp〜25bpで設計される。また、Tm値によって規定することもできる。通常、Tm値が45℃〜75℃、より好ましくは55℃〜70℃の間になるように設計される。
【0034】
本発明に使用されるASPとは、背景技術に記載したように、SNP塩基が特定の塩基種の場合にのみ効率良く伸長反応が起こるように設計されたプライマ−のことである。第1のプライマ−、もしくは第2のプライマ−のいずれか一方がASPとなるように設計される。
【0035】
本発明におけるASPを用いたPCRは、二本鎖DNAの熱変性、プライマ−のハイブリダイゼ−ション、ならびにDNAポリメラ−ゼによるDNAの伸長反応が行われる任意の条件下で実施される。二本鎖DNAの熱変性は通常、94℃〜100℃で1秒から100秒の間で行われる。ハイブリダイゼ−ション条件は、通常、Tm値から約5℃下げた温度条件で、50〜70℃で1秒から100秒間で行われる。ポリメラ−ゼによる伸長反応は、酵素が最適に反応する条件を考慮して条件が設定される。Taqポリメラ−ゼが使用される場合、通常、65〜74℃で反応時間は1秒〜3分程度で実施される。アニ−ル工程と伸長工程を同一温度で行うサイクルの場合でもよく、その場合通常、60〜70℃で1秒〜3分程度で行われる。また、上記反応サイクルの前後に、DNAの変性工程や、ポリメラ−ゼの伸長反応を付加する工程を適宜加えても良い。
【0036】
(実施の形態1)
以下に本発明におけるASP−PCR法の実施例を示すが、以下の実施例で本発明の内容が限定されるものではない。
【0037】
実施例1:
本実施例では、ヒトのアセトアルデヒドデヒドロゲナ−ゼ2(ALDH2)遺伝子の第12エクソンの114番目のSNP塩基を判別する例を示す。ALDH2は、飲酒した際に生成されるアセトアルデヒドを分解する酵素である。ALDH2の487番目のアミノ酸を決める塩基配列の違いにより、3つの遺伝型に分かれる。すなわち、ALDH2の対立遺伝子に見られる第12エクソンの114番目の塩基がいずれもグアニンであるG/Gタイプ(野生型)と片方がグアニンで他方がアデニンであるG/Aタイプ、そしていずれもがアデニンであるA/A(変異型)である。
【0038】
本実施例で用いた第1のプライマ−(以下、ALDH2−Fと表記)と第2のプライマ−(以下、ALDH2−Wt−Rと表記)の配列を表1に示す。これらの内、ALDH2−Wt−RがASPに該当し、ALDH2−Wt−Rは、上記SNP塩基が野生型の場合にDNA伸長反応が起こるように設計されている。具体的には、ALDH2−Wt−Rの3’末端塩基がSNP対応塩基である。またALDH2−Wt−Rの3’末端から2番目および3番目の二塩基は、鋳型に対して非相補的になるように設計されている(表1、下線部)。
【0039】
【表1】

【0040】
また、ALDH2−FとALDH2−Wt−Rは、アセトアルデヒドデヒドロゲナ−ゼ2遺伝子中の144bpの標的配列がPCRにより増幅されるように設計されている(ただし上記SNP塩基が変異型の場合は、ALDH2−Wt−RからのDNA伸長反応効率は極めて悪いため、この標的配列は増幅されない。)。
【0041】
ブロック核酸(以下、ALDH2−Block1と表記)の配列を表2に示す。
【0042】
【表2】

【0043】
ALDH2−Block1は標的配列から3塩基上流に結合する実施の形態に記載のブロック核酸(20)に相当し、22塩基からなる。ALDH2−Block1の3’末端のヌクレオチドに含まれる3位の糖はリン酸化によって修飾されている。
【0044】
鋳型DNAは、上記SNP塩基が野生型のホモである被験者および変異型のホモである被験者の血液100μLからDNA Micro Kit(QIAGEN社製)を用いて抽出したゲノムDNAを10ng/μLになるように調製して得た。検体の遺伝子型はシ−ケンス解析により確認した。PCRの反応溶液は以下の通りであった。
【0045】
1×MightyAmp Buffer(TaKaRa社製)
0.25U/10μL MightyAmp DNA Polymerase(TaKaRa社製)
1μM ALDH2−F
1μM ALDH2−R
0.5ng/μLの野生型DNAもしくは変異型DNA、および
10μM ALDH2−Block
1/10000 SYBR GreenI(TaKaRa社製)
全量:10μL
PCRの反応条件は以下のとおりであった。すなわち、まず98℃で2分間処理した。その後、98℃で1秒間(熱変性工程)、55℃で1秒間(アニ−リング工程)、68℃で28秒間(DNA伸長反応工程)からなるサイクルを60サイクル行った。
【0046】
PCR反応をApplied Biosystems 7300リアルタイムPCRシステムを用いて行った。リアルタイムPCRの増幅曲線の結果を図3に示す。図3の縦軸にSYBR GreenIの蛍光強度、横軸に時間を示す。図3から明らかなように、野生型を鋳型に用いた場合のみ効率的な増幅が起こっている。
【0047】
さらにBioAnalyzer(Agilent社製)を用いて、PCR反応物を電気泳動に供した。図4に野生型を鋳型に用いたPCRの電気泳動の結果を、図5に変異型を鋳型に用いたPCRの電気泳動結果を示す。図4と図5の縦軸は増幅産物の濃度に比例した蛍光強度を示しており、横軸は時間を示している。図4と図5を比較すると、野生型を鋳型とした場合は144bpからなる標的配列のみが特異的に増幅されていた。一方、変異型を鋳型とした場合では増幅産物は全く検出されなかった。図4、図5中のMarkerは電気泳動キットに付属されているDNA Markerである。また図5中の標的配列以下のピ−クはプライマ−ダイマ−(以下、PDと記載)である。
【0048】
比較例1:
本比較例1では、実施例1における反応溶液のブロック核酸を蒸留水に置換したこと以外は、実施例1と同様のPCR反応を実施した。図6に比較例1のリアルタイムPCRの増幅曲線の結果を示す。図6の縦軸はSYBR GreenIの蛍光強度を、横軸は時間を示している。また、図7に野生型を鋳型に用いた場合のPCRの電気泳動結果を、図8に変異型を鋳型に用いた場合のPCRの電気泳動結果を示す。図7と図8の縦軸は増幅産物の濃度に比例した蛍光強度を示しており、横軸は時間を示している。
【0049】
まず、図6より、野生型を鋳型に用いた場合と変異型を鋳型に用いた場合において、前者の増幅曲線の方が後者に比べ早く立ち上がっていることが分かる。しかしながら、その差はわずか2.5分でしかなかった。
【0050】
ここで、実施例1におけるリアルタイムPCR結果を示す図3と比較すると、ブロック核酸を用いた実施例1では変異型の増幅を抑え、立ち上がりの差が明らかに広がっていることがわかる。このことは第1のブロック核酸20が明確なSNP判別を行なうことに寄与していることを示す。
【0051】
また図7より、野生型を鋳型とした場合では144bpの標的配列が増幅産物だけでなく、330bpと612bpのDNAが非特異的に増幅されていることが分かる。また、図8においても、変異型を鋳型とした場合では標的配列の増幅は起こっていないが、それ以外の324〜610bpのDNAが増幅されており、非特異的な増幅が起こっている。
【0052】
実施例1における図4と図5では、このような非特異的な増幅は全く起こっていない。このことから第1のブロック核酸20が非特異的な増幅を抑制する効果に寄与していることが示された。
【0053】
図7、図8中のMarkerは電気泳動キットに付属されているDNA Markerである。また図8中の標的配列以下のピ−クはPDである。
【0054】
アジレント電気泳動の解析結果から算出された増幅産物の濃度の総和を示す実施例1におけるグラフを図9に、比較例1におけるグラフを図10に示した。図10の比較例1では野生型、変異型とも標的配列以外の非特異的な増幅が起こっているため、増幅産物の濃度の総和が野生型で75.47ng/μLであるのに対し、変異型は60.47ng/μLであり、有意な差がない。一方、ブロック核酸を加えた実施例1の野生型では、標的配列のみの特異的な増幅が起こっている。変異型では全く何の増幅も起こっていない。その結果、増幅産物の濃度の総和は野生型で57.69ng/μLであるのに対し、変異型では0ng/μLであり、完全にバックグラウンドを抑制することができた。すなわち、非特異的な増幅の抑制効果は、PCRの反応副産物として得られるピロリン酸濃度を指標としてSNP塩基を判別する検出手法においても、SNP塩基種の違いによる差を明確にし、有用であることがわかる。
【0055】
以上のように、ブロック核酸を用いることでリアルタイムPCRにおいては、野生型と変異型で立ちあがり時間の差が拡大し、明確なSNP判別が可能となった。さらに、ブロック核酸は非特異的な増幅を抑制しており、これは、ピロリン酸を検出する検出手法においてもバックグラウンドを抑え、明確な判別を可能にする効果に寄与することを示す。
【0056】
実施例2:
本実施例では、ヒトのアセトアルデヒドデヒドロゲナ−ゼ2遺伝子の第12エクソンの114番目のSNP塩基を判別する例において、SNP塩基が変異型の場合、増幅可能なASPを用いた例を示す。第1のプライマ−(以下、ALDH2−Fと表記)と第2のプライマ−(以下、ALDH2−Mt−Rと表記)の配列を表3に示す。本実施例では第2のプライマ−がASPに該当し、ALDH2−Wt−Rは、上記SNP塩基が変異型の場合にDNA伸長反応が起こるように設計されている。具体的には、ALDH2−Mt−Rの3’末端塩基がSNP対応塩基である。またALDH2−Mt−Rの3’末端から2番目および3番目の二塩基は、鋳型に対して非相補的になるように設計されている(表3、下線部)。
【0057】
【表3】

【0058】
また、ALDH2−FとALDH2−Mt−Rは、アセトアルデヒドデヒドロゲナ−ゼ2遺伝子中の144bpの標的配列がPCRにより増幅されるように設計されている(ただし上記SNP塩基が野生型の場合は、ALDH2−Mt−RからのDNA伸長反応効率は極めて悪いため、この標的配列は増幅されない)。
【0059】
ブロック核酸(以下、ALDH2−Block1とALDH2−Block2と表記)の配列を表4に示す。
【0060】
【表4】

【0061】
ALDH2−Block1は標的配列から3塩基上流に結合する実施の形態に記載のブロック核酸(20)に相当し、22塩基からなる。ALDH2−Block2は標的配列から6塩基下流に結合する実施の形態に記載のブロック核酸(30)に相当し、19塩基からなる。ALDH2−Block1とALDH2−Block2の3’末端のヌクレオチドに含まれる3位の糖はリン酸化によって修飾されている。
【0062】
鋳型DNAは、上記SNP塩基が野生型のホモである被験者および変異型のホモである被験者の血液100μLからDNA Micro Kit(QIAGEN社製)を用いて抽出したゲノムDNAを10ng/μLになるように調製して得た。検体の遺伝子型はシ−ケンス解析により確認した。
【0063】
PCRの反応溶液は以下の通りである。
【0064】
1×MightyAmp Buffer(TaKaRa社製)
0.25U/10μL MightyAmp DNA Polymerase(TaKaRa社製)
1μM ALDH2−F
1μM ALDH2−R
0.5ng/μLの野生型DNAもしくは変異型DNA、および
10μM ALDH2−Block
1/10000 SYBR GreenI(TaKaRa社製)
全量:10μL
PCR増幅を、以下のサ−マルプロファイル:98℃で2分間を1サイクル、98℃で1秒間(変性のため)と55℃で1秒間(アニ−ルのため)と68℃で28秒間(DNA伸長のため)を60サイクル、で行った。
【0065】
PCR反応をApplied Biosystems 7300リアルタイムPCRシステムを用いてモニタリングした。図11はリアルタイムPCRの増幅曲線の結果を示す。図11の縦軸はSYBR GreenIの蛍光強度を、横軸は時間を示している。
図11から明らかなように、変異型を鋳型に用いた場合のみ効率的な増幅が起こっている。
さらにBioAnalyzer(Agilent社製)を用いて、PCR反応物を電気泳動に供した。図12に変異型を鋳型に用いたPCRの電気泳動の結果を、図13に野生型を鋳型に用いたPCRの電気泳動結果を示す。図12と図13の縦軸は増幅産物の濃度に比例した蛍光強度を示しており、横軸は時間を示している。図12と図13を比較すると、変異型を鋳型とした場合は144bpの塩基対からなる標的配列のみが特異的に増幅されていた。一方、野生型を鋳型とした場合は81bpの非特異的な増幅が見られるが、標的配列より短く、PCRの反応副産物として得られるピロリン酸濃度を指標としてSNP塩基を判別する検出手法においても、ほとんど問題にならない。図12、図13中のMarkerは電気泳動キットに付属されているDNA Markerである。また図12、図13中の標的配列以下のピ−クはPDである。
【0066】
比較例2:
本比較例2では、実施例2における反応溶液のブロック核酸を蒸留水に置換したこと以外は、実施例2と同様のPCR反応を実施した。図14に比較例1のリアルタイムPCRの増幅曲線の結果を示す。また、図15に変異型を鋳型に用いた場合のPCRの電気泳動結果を、図16に野生型を鋳型に用いた場合のPCRの電気泳動結果を示す。図14の縦軸はSYBR GreenIの蛍光強度を、横軸は時間を示している。図15と図16の縦軸は増幅産物の濃度に比例した蛍光強度を示しており、横軸は時間を示している。
【0067】
まず、図14より、変異型を鋳型に用いた場合と野生型を鋳型に用いた場合において、前者の増幅曲線の方が後者に比べ早く立ち上がっていることが分かる。しかしながら、その差は5.6分でしかなく、有意な差ではなかった。
【0068】
ここで、実施例2におけるリアルタイムPCR結果を示す図11と比較すると、ブロック核酸を用いた実施例2では明らかに立ち上がりの差が広がっていることがわかる。このことは第1のブロック核酸20と第2のブロック核酸30が明確なSNP判別を行なうことに寄与していることを示す。
【0069】
また、図15の変異型を鋳型とした場合では144bpの標的配列が増幅産物だけでなく、303bpの塩基対からなる2本鎖DNAが非特異的に増幅されている。図16の野生型を鋳型とした場合では144bpの標的配列の増幅は起こっていることから、ASPの機能を果たせていないことが示唆され、さらにそれ以外の300bpと351bpの塩基対からなる2本鎖DNAが非特異的に増幅されている。
【0070】
実施例2における図12と図13では、このような非特異的な増幅は全く起こっていない。このことから第1のブロック核酸20と第2のブロック核酸30が非特異的な増幅を抑制する効果に寄与していることが示された。
【0071】
アジレント電気泳動の解析結果から算出された増幅産物の濃度の総和を示す実施例2におけるグラフを図17に、比較例2におけるグラフを図18に示した。図18の比較例2では変異型、野生型とも標的配列以外の非特異的な増幅が起こっているため、増幅産物の濃度の総和が比較例2では変異型の68.86ng/μLに対して、野生型では94.46ng/μLであり、標的配列より塩基対の大きな非特異的な増幅が多いことが原因となり、野生型の方が産物量の総和は大きくなってしまっている。これは、ピロリン酸を測定する検出手法においては、間違った判別結果を導く恐れがある。それに対し、ブロック核酸を加えた実施例2では変異型では標的配列のみの特異的な増幅が起こっている。野生型でもわずかに非特異的な増幅が見られるものの、各々の濃度の総和は変異型では、64.34ng/μLであるのに対し、野生型ではわずか4.79ng/μLであり、有意な差を得ることができている。すなわち、非特異的な増幅の抑制効果は、PCRの反応副産物として得られるピロリン酸濃度を指標としてSNP塩基を判別する検出手法においても、SNP塩基種の違いによる差を明確にし、有用であることがわかる。
【0072】
以上のように、ブロック核酸を用いることでリアルタイムPCRにおいては、野生型と変異型で立ちあがり時間の差が拡大し、明確なSNP判別が可能となった。さらに、ブロック核酸は非特異的な増幅を抑制しており、これは、ピロリン酸を検出する検出手法においてもバックグラウンドを抑え、明確な判別を可能にする効果に寄与することを示す。
【産業上の利用可能性】
【0073】
以上のように、本発明は、一般的なPCR反応に利用可能である。また、臨床検査用途を目的としたPCRにも利用できる。
【符号の説明】
【0074】
1 二本鎖標的配列
1a 一本鎖標的配列
1b 相補的一本鎖標的配列
2 望まれる増幅二本鎖DNA配列
4 第1のプライマ−
5 第2のプライマ−
6 第1一本鎖DNA
6a 第1非増幅配列
6b 第2非増幅配列
6c 一本鎖標的配列1aの3’末端部分
6g 増幅一本鎖標的配列
6k 第4複製配列
7 第2一本鎖DNA
7a 第3非増幅配列
7b 第4非増幅配列
7g 増幅相補的一本鎖標的配列
7k 相補的第4複製配列
8 SNP塩基
20 第1ブロック核酸
30 第2ブロック核酸
【技術分野】
【0001】
本発明は、PCRを利用したSNP判別方法に関わる。
【背景技術】
【0002】
近年、体外診断薬を含む分子診断市場は急速な伸びを示しており、医療分野では、疾患関連遺伝子を解析することにより、疾患の分子レベルでの治療が可能となってきている。
【0003】
遺伝子検査は、遺伝子の病気と言われるようになった癌領域だけではなく、感染症や、アルツハイマ−病、生活習慣病の発症リスクや、薬物治療に対する効果や安全性を調べる用途にも利用することができることから医療分野での注目が集まっている。その中でも特に塩基配列中の一塩基対での違いを示す一塩基多型(Single Nucleotide Polymorphism;SNP)は、このわずかな違いにより病気のかかりやすさや薬効に個人差が生まれることが明らかとなってきているため、SNP検査を行なう重要性は高まっている。例えば、分子標的治療剤として日本で承認された薬剤であるゲフィニチブは、EGFR遺伝子に存在するSNPの違いによって治療効果が異なることが報告されている。このようにSNPを判別する重要性が高まる中、様々な分析手法が開発されてきている。
【0004】
遺伝子検査において、検体中に存在するDNAのコピ−数が少ない場合、目的とする遺伝子の領域を増幅して感度を高める方法がある。一般的な核酸増幅法であるPolymerase Chain Reaction(PCR)法は鋳型DNA中の目的とする配列領域(以下、標的配列と表記)をその両端の配列に相補的な一対のプライマ−及びDNAポリメラ−ゼを用いて、3段階の温度変化をnサイクル繰り返すことによって2n倍に増幅する手法である。3段階の各工程は二本鎖DNAを一本鎖DNAに変性する工程と、一本鎖DNA中の標的配列にプライマ−をハイブリダイゼ−ションさせる工程、DNAポリメラ−ゼによるDNAの伸長反応を行う工程よりなる。1サイクルで標的配列の濃度は2倍になり、理論的にはnサイクルの反応で標的配列は2n倍に増幅される。実際には100万倍程度に増幅することができる技術である。
【0005】
このPCR法を改良し、SNPを判別する手法が開発されている。代表的な方法として、アレル特異的プライマ−(Allele Specific Primer;ASP)を用いたSNP判別方法がある。ASPとはSNP塩基がある特定の塩基種の場合にのみ効率よく伸長反応が起こるように設計されたプライマ−のことを言う。代表的なASPとして非特許文献1が報告されている。
【0006】
非特許文献1ではASPとしてプライマ−の3’末端の塩基が標的のSNP塩基と対応するように設計されたプライマ−を用いてSNP判別を行なっている。3’末端塩基がSNP塩基に対して相補的であれば、伸長反応が効率良く起こるが、非相補的である場合は、伸長反応が進まない、あるいはその効率が低下する。したがって、ASPを用いてPCRを行い、その結果得られるPCR産物量や、あるいはPCRの反応効率を解析することで、標的のSNP塩基の塩基種を知ることができる。PCR産物量を解析する方法としては、電気泳動法や、リアルタイムPCR法、ポリメラ−ゼ伸長反応時に副産物として生成されるピロリン酸を光学的あるいは電気化学的に測定する手法が知られている。
【0007】
しかしながら、上記ASPを用いたSNP判別方法は、3’末端の一塩基のみで伸長反応を制御するものであるので、特異的かつ厳密に制御することは難しい。その結果、ASPのミスハイブリダイゼ−ションにより、擬陽性を生じる場合があった。そこで、擬陽性を抑えるため、新たなASPの開発が行なわれている。
【0008】
例えば、ASPは、3’ 末端塩基がSNP対応塩基となるように設計され、かつその5’側上流の3番目の塩基を、鋳型に対して意図的に非相補的になるように設計する方法が報告されている(特許文献1)。
【0009】
また、ASPは、3’末端から2番目の塩基がSNP対応塩基になるように設計され、かつその5’側に一つ上流の塩基を、鋳型に対して意図的に非相補的な配列になるように設計がなされている(特許文献2)。
【0010】
また、ASPは、3’末端塩基がSNP対応塩基となるように設計され、かつその5’側上流2番目と3番目の塩基が、鋳型に対して意図的に非相補的な配列となるように設計され、鋳型の配列に応じて特異性の高い塩基配列を選定している(特許文献3)。
【0011】
上述の新規に開発されたASPは、SNP対応塩基以外に、鋳型に対して非相補的な塩基を意図的に導入することにより、SNP対応塩基がSNP塩基に対して非相補的である場合、結合能をより弱くし、擬陽性を抑制する点で優れている。
【0012】
しかしその一方で、プライマ−への非相補的な塩基の導入は、プライマ−の標的配列への特異性を低くし、標的配列とは異なる配列に対して非特異的に結合する場合があった。その結果、ASPを用いてPCRを行うと、標的配列とは異なるDNA配列が増幅される場合があった。このような非特異的な増幅は、ピロリン酸を測定する手法においては、バックグラウンドとなるため重要な課題である。また、ASPを用いてリアルタイムPCRを行った場合、SNPの塩基種が違っていても増幅曲線の立ち上がりの差が小さく、明確にSNP判別をすることができない場合があった。
【0013】
そこで、プライマ−の非特異的な結合を防ぐ方法が報告されている。この方法では、プライマ−が非特異的に結合する可能性のある配列を予め網羅的に調べ予測し、それらの配列と完全に相補的な配列を有する競合核酸を反応液中に添加することで、プライマ−の非特異的な結合が起こらないようにすることを特徴としている(特許文献4)。
【先行技術文献】
【特許文献】
【0014】
【特許文献1】米国特許出願公開第2003/0049628号明細書
【特許文献2】米国特許出願公開第2003/0148301号明細書
【特許文献3】米国特許出願公開第2004/0197803号明細書
【特許文献4】特開2004−194619号公報
【非特許文献】
【0015】
【非特許文献1】Nucleic Acids Res.,17,2503−2516(1989).
【発明の概要】
【発明が解決しようとする課題】
【0016】
しかしながら、非特異に結合する可能性を予め完全に予測することは難しく、この方法により非特異的な結合反応を完全に防ぐことには限界があった。
【0017】
本発明は、前記従来の課題を解決するために、ASPを用いたPCRにおいて、プライマ−の反応特異性の低下によって引き起こされる、非特異的な増幅反応を抑制し、より明確なSNP判別を可能とする方法を提供することを目的とする。
【課題を解決するための手段】
【0018】
前記従来の課題を解決するために、本発明のSNP判別方法は、第1一本鎖DNA(6)および第2一本鎖DNA(7)からなる二本鎖DNA中の二本鎖標的配列(1)中に含まれるSNPの塩基種(8)を、アレル特異的プライマ−を用いて判別するSNP判別法であって、前記二本鎖標的配列(1)は、一本鎖標的配列(1a)および相補的一本鎖標的配列(1b)からなり、前記第1一本鎖DNA(6)は、3’末端−第1非増幅配列(6a)−前記一本鎖標的配列(1a)−第2非増幅配列(6b)−5’末端からなり、前記第2一本鎖DNA(7)は、5’末端−第3非増幅配列(7a)−前記相補的一本鎖標的配列(1b)−第4非増幅配列(7b)−3’末端からなり、前記相補的一本鎖標的配列(1b)、第3非増幅配列(7a)、および第4非増幅配列(7b)は、それぞれ、前記一本鎖標的配列(1a)、前記第1非増幅配列(6a)、および第2非増幅配列(6b)と相補的であり、DNAポリメラ−ゼ、デオキシヌクレオシド三リン酸、前記二本鎖DNA(6・7)、第1のプライマ−(4)、第2のプライマ−(5)、および第1ブロック核酸(20)を混合し、ポリメラ−ゼ連鎖反応を用いて前記二本鎖標的配列(1)を増幅する工程Aを含み、ここで、前記第1のプライマ−(4)および前記第2のプライマ−(5)は、いずれも、前記DNAポリメラ−ゼによる伸長反応のための起点として作用し、前記SNP塩基種(8)は、第一の一本鎖DNA(6)上の前記第1のプライマ−(4)が結合する領域内に存在し、前記第1のプライマ−(4)は、SNPの塩基(8)に相補的な配列である、アレル特異的プライマ−であって、前記第1のプライマ−(4)は、前記一本鎖標的配列(1a)に含まれる3’末端側の配列の部分に相補的であり、前記第2のプライマ−(5)は、前記相補的一本鎖標的配列(1b)に含まれる3’末端側の部分の配列に相補的であり、前記第1ブロック核酸(20)は、前記DNAポリメラ−ゼによる伸長反応のための起点として作用せず、前記第1ブロック核酸(20)は、前記第3非増幅配列(7a)の一部と相補的である、ことを特徴としたものである。
【発明の効果】
【0019】
本発明により、ASPを用いたPCRにおいて、非特異的な増幅を抑制することができ、より明確なSNP判別が可能なSNP判別法が提供される。
【図面の簡単な説明】
【0020】
【図1】従来のPCRを示す図
【図2】本発明に係るPCRを示す図
【図3】実施例1におけるリアルタイムPCRの増幅曲線を示すグラフ
【図4】実施例1における野生型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図5】実施例1における変異型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図6】比較例1におけるリアルタイムPCRの増幅曲線を示すグラフ
【図7】比較例1における野生型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図8】比較例1における変異型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図9】実施例1における増幅産物の総和を比較したグラフ
【図10】比較例1における増幅産物の総和を比較したグラフ
【図11】実施例2におけるリアルタイムPCRの増幅曲線を示すグラフ
【図12】実施例2における野生型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図13】実施例2における変異型の鋳型を用いた場合の電気泳動時の結果を示すグラフ
【図14】比較例2におけるリアルタイムPCRの増幅曲線を示すグラフ
【図15】比較例2における野生型を鋳型に用いた場合の電気泳動時の結果を示すグラフ
【図16】比較例2における変異型の鋳型を用いた場合の電気泳動時の結果を示すグラフ
【図17】実施例2における増幅産物の総和を比較したグラフ
【図18】比較例2における増幅産物の総和を比較したグラフ
【発明を実施するための形態】
【0021】
以下、本発明に係わるSNP判別法の具体的な実施の形態を説明する。ただし、本発明はこれに限定されるものではない。本実施の形態では、図1および図2を用い、第1のプライマ−(4)がASPである場合を例にとって説明する。
【0022】
従来のPCRでは、図1に示すように、第1一本鎖DNA6は3’末端−第1非増幅配列(6a)−一本鎖標的配列(1a)−第2非増幅配列(6b)−5’末端からなり、第2一本鎖DNA7は5’末端−第3非増幅配列(7a)−相補的一本鎖標的配列(1b)−第4非増幅配列(7b)−3’末端からなる。第1のプライマ−(4)は一本鎖標的配列(1a)の3’末端部分(6c)に相補的に結合する。SNP塩基(8)は第1一本鎖DNA(6)の鎖上に存在し、かつ第1のプライマ−(4)が結合する領域内に存在する。一方、第2のプライマ−(5)は、相補的一本鎖標的配列(1b)の3’末端部分に相補的に結合する。
【0023】
したがって、第1のプライマ−(4)と第2のプライマ−(5)からDNA伸長反応が良好におこり、PCRが行なわれると、得られる増幅二本鎖DNA配列は、望まれる増幅二本鎖DNA配列2が得られる。しかし、背景技術でも記したとおり、ASPのようなプライマ−を用いた場合はそれ以外の非特異的な増幅により望まれない産物が得られる場合があった。これを解決するのが本発明である。
【0024】
ここで、本発明におけるブロック核酸(20)について図2を用いて説明する。本発明の特徴は、背景技術に記載のASPを用いたPCRにおけるSNP判別法にブロック核酸を加えることである。具体的にはDNAポリメラ−ゼ、デオキシヌクレオシド三リン酸(dATP、dCTP、dGTP、dTTP)、鋳型DNA、第1のプライマ−および第2のプライマ−を含むPCR反応溶液にブロック核酸を共存させてPCRを行なう。第1のプライマ−及び、第2のプライマ−はフォワ−ドプライマ−とリバ−スプライマ−の関係にあって、両プライマ−の3’末端より伸長反応が進み、標的配列を増幅する。本実施の形態では、第1のプライマ−か第2のプライマ−の何れかはASPである。好ましくは上記反応溶液にDNAポリメラ−ゼが安定に働くための緩衝液、塩類を加えても良い。
【0025】
ブロック核酸(20)は、DNAポリメラ−ゼによる伸長反応のための起点として作用せず、第3非増幅配列(7a)の一部と相補的である。本発明におけるブロック核酸(30)は、DNAポリメラ−ゼによる伸長反応のための起点として作用せず、第2非増幅配列(6b)の一部と相補的である。
【0026】
本発明のPCRにおいては上記ブロック核酸(20)とブロック核酸(30)の少なくともいずれか一方、好適には両ブロック核酸が反応溶液中に加えられる。
【0027】
また上述の通り、ブロック核酸はDNAポリメラ−ゼによる伸長反応の起点とならない合成オリゴ核酸である。したがってブロック核酸としては、例えば、3’末端にDNAポリメラ−ゼによる伸長反応の起点とならない修飾が施されているDNAか、もしくはLocked Nucleic Acid(LNA)が挙げられる。修飾の種類としては好適には、リン酸化、アミノ化、蛍光修飾、ビオチン等が含まれるが、これに限るものではない。またブロック核酸は、3’末端のヌクレオチドがジデオキシヌクレオチドであるDNAやLNAであっても良い。またブロック核酸は、伸長反応の起点とならない核酸類似体であってもよい。伸長反応の起点とならない核酸類似体としては、例えばPeptide nucleic acid(PNA)などが挙げられる。
【0028】
本発明におけるブロック核酸は標的配列(1)の近傍に結合するように設計されることが望ましい。ここでいう近傍とは標的配列(1)から数塩基から数千塩基以内の領域である。ブロック核酸の配列は、好ましくはプライマ−のTm値と同等以上になるように設計される。反応溶液中におけるブロック核酸の濃度は、プライマ−の濃度より大きいことが好ましい。例えばプライマ−が0.1μM〜1μMの濃度範囲であれば、ブロック核酸はその5倍以上の濃度で加えられることが好ましい。
【0029】
本発明に用いられるDNAポリメラ−ゼは、一例として、Taq DNA Polymeraseなどが挙げられるが、これに限定されるものではなく、PCRで使用可能なものであれば、どのようなものでも使用することができ、適宜用途に合わせて選択すればよい。
【0030】
ただし、鎖置換型の活性を持ったポリメラ−ゼは使用することができない。DNAポリメラ−ゼの濃度は通常、0.5〜2.5Unit/100μLで使用されることが望ましい。緩衝液は上記DNAポリメラ−ゼが反応するために適切な溶媒であれば何でもよく、一般的にはTris−HClが使用され、pHは7〜10、濃度は10mM〜100mMで調製されることが望ましい。塩類は好適には塩化マグネシウムが使用されており、1mM〜5mMで添加され、好適な範囲は1.5mM〜3mMである。
【0031】
デオキシヌクレオシド三リン酸は4種類(dATP、dTTP、dGTP、dCTP)よりなり、通常、各々が等量で20μM〜200μMで含まれる。
【0032】
本発明における標的配列とは、背景技術で説明したようにSNP塩基を含み、一対のプライマ−で囲まれ、PCRで増幅される領域を示す。
【0033】
また、本発明におけるプライマ−とは、DNAポリメラ−ゼによる伸長反応の起点となる合成オリゴ核酸である。プライマ−の長さは、伸長反応のためのプライマ−として働くのに十分な長さであればよく、通常、15bp〜50bpの範囲で設計される。より好ましくは、20bp〜25bpで設計される。また、Tm値によって規定することもできる。通常、Tm値が45℃〜75℃、より好ましくは55℃〜70℃の間になるように設計される。
【0034】
本発明に使用されるASPとは、背景技術に記載したように、SNP塩基が特定の塩基種の場合にのみ効率良く伸長反応が起こるように設計されたプライマ−のことである。第1のプライマ−、もしくは第2のプライマ−のいずれか一方がASPとなるように設計される。
【0035】
本発明におけるASPを用いたPCRは、二本鎖DNAの熱変性、プライマ−のハイブリダイゼ−ション、ならびにDNAポリメラ−ゼによるDNAの伸長反応が行われる任意の条件下で実施される。二本鎖DNAの熱変性は通常、94℃〜100℃で1秒から100秒の間で行われる。ハイブリダイゼ−ション条件は、通常、Tm値から約5℃下げた温度条件で、50〜70℃で1秒から100秒間で行われる。ポリメラ−ゼによる伸長反応は、酵素が最適に反応する条件を考慮して条件が設定される。Taqポリメラ−ゼが使用される場合、通常、65〜74℃で反応時間は1秒〜3分程度で実施される。アニ−ル工程と伸長工程を同一温度で行うサイクルの場合でもよく、その場合通常、60〜70℃で1秒〜3分程度で行われる。また、上記反応サイクルの前後に、DNAの変性工程や、ポリメラ−ゼの伸長反応を付加する工程を適宜加えても良い。
【0036】
(実施の形態1)
以下に本発明におけるASP−PCR法の実施例を示すが、以下の実施例で本発明の内容が限定されるものではない。
【0037】
実施例1:
本実施例では、ヒトのアセトアルデヒドデヒドロゲナ−ゼ2(ALDH2)遺伝子の第12エクソンの114番目のSNP塩基を判別する例を示す。ALDH2は、飲酒した際に生成されるアセトアルデヒドを分解する酵素である。ALDH2の487番目のアミノ酸を決める塩基配列の違いにより、3つの遺伝型に分かれる。すなわち、ALDH2の対立遺伝子に見られる第12エクソンの114番目の塩基がいずれもグアニンであるG/Gタイプ(野生型)と片方がグアニンで他方がアデニンであるG/Aタイプ、そしていずれもがアデニンであるA/A(変異型)である。
【0038】
本実施例で用いた第1のプライマ−(以下、ALDH2−Fと表記)と第2のプライマ−(以下、ALDH2−Wt−Rと表記)の配列を表1に示す。これらの内、ALDH2−Wt−RがASPに該当し、ALDH2−Wt−Rは、上記SNP塩基が野生型の場合にDNA伸長反応が起こるように設計されている。具体的には、ALDH2−Wt−Rの3’末端塩基がSNP対応塩基である。またALDH2−Wt−Rの3’末端から2番目および3番目の二塩基は、鋳型に対して非相補的になるように設計されている(表1、下線部)。
【0039】
【表1】

【0040】
また、ALDH2−FとALDH2−Wt−Rは、アセトアルデヒドデヒドロゲナ−ゼ2遺伝子中の144bpの標的配列がPCRにより増幅されるように設計されている(ただし上記SNP塩基が変異型の場合は、ALDH2−Wt−RからのDNA伸長反応効率は極めて悪いため、この標的配列は増幅されない。)。
【0041】
ブロック核酸(以下、ALDH2−Block1と表記)の配列を表2に示す。
【0042】
【表2】

【0043】
ALDH2−Block1は標的配列から3塩基上流に結合する実施の形態に記載のブロック核酸(20)に相当し、22塩基からなる。ALDH2−Block1の3’末端のヌクレオチドに含まれる3位の糖はリン酸化によって修飾されている。
【0044】
鋳型DNAは、上記SNP塩基が野生型のホモである被験者および変異型のホモである被験者の血液100μLからDNA Micro Kit(QIAGEN社製)を用いて抽出したゲノムDNAを10ng/μLになるように調製して得た。検体の遺伝子型はシ−ケンス解析により確認した。PCRの反応溶液は以下の通りであった。
【0045】
1×MightyAmp Buffer(TaKaRa社製)
0.25U/10μL MightyAmp DNA Polymerase(TaKaRa社製)
1μM ALDH2−F
1μM ALDH2−R
0.5ng/μLの野生型DNAもしくは変異型DNA、および
10μM ALDH2−Block
1/10000 SYBR GreenI(TaKaRa社製)
全量:10μL
PCRの反応条件は以下のとおりであった。すなわち、まず98℃で2分間処理した。その後、98℃で1秒間(熱変性工程)、55℃で1秒間(アニ−リング工程)、68℃で28秒間(DNA伸長反応工程)からなるサイクルを60サイクル行った。
【0046】
PCR反応をApplied Biosystems 7300リアルタイムPCRシステムを用いて行った。リアルタイムPCRの増幅曲線の結果を図3に示す。図3の縦軸にSYBR GreenIの蛍光強度、横軸に時間を示す。図3から明らかなように、野生型を鋳型に用いた場合のみ効率的な増幅が起こっている。
【0047】
さらにBioAnalyzer(Agilent社製)を用いて、PCR反応物を電気泳動に供した。図4に野生型を鋳型に用いたPCRの電気泳動の結果を、図5に変異型を鋳型に用いたPCRの電気泳動結果を示す。図4と図5の縦軸は増幅産物の濃度に比例した蛍光強度を示しており、横軸は時間を示している。図4と図5を比較すると、野生型を鋳型とした場合は144bpからなる標的配列のみが特異的に増幅されていた。一方、変異型を鋳型とした場合では増幅産物は全く検出されなかった。図4、図5中のMarkerは電気泳動キットに付属されているDNA Markerである。また図5中の標的配列以下のピ−クはプライマ−ダイマ−(以下、PDと記載)である。
【0048】
比較例1:
本比較例1では、実施例1における反応溶液のブロック核酸を蒸留水に置換したこと以外は、実施例1と同様のPCR反応を実施した。図6に比較例1のリアルタイムPCRの増幅曲線の結果を示す。図6の縦軸はSYBR GreenIの蛍光強度を、横軸は時間を示している。また、図7に野生型を鋳型に用いた場合のPCRの電気泳動結果を、図8に変異型を鋳型に用いた場合のPCRの電気泳動結果を示す。図7と図8の縦軸は増幅産物の濃度に比例した蛍光強度を示しており、横軸は時間を示している。
【0049】
まず、図6より、野生型を鋳型に用いた場合と変異型を鋳型に用いた場合において、前者の増幅曲線の方が後者に比べ早く立ち上がっていることが分かる。しかしながら、その差はわずか2.5分でしかなかった。
【0050】
ここで、実施例1におけるリアルタイムPCR結果を示す図3と比較すると、ブロック核酸を用いた実施例1では変異型の増幅を抑え、立ち上がりの差が明らかに広がっていることがわかる。このことは第1のブロック核酸20が明確なSNP判別を行なうことに寄与していることを示す。
【0051】
また図7より、野生型を鋳型とした場合では144bpの標的配列が増幅産物だけでなく、330bpと612bpのDNAが非特異的に増幅されていることが分かる。また、図8においても、変異型を鋳型とした場合では標的配列の増幅は起こっていないが、それ以外の324〜610bpのDNAが増幅されており、非特異的な増幅が起こっている。
【0052】
実施例1における図4と図5では、このような非特異的な増幅は全く起こっていない。このことから第1のブロック核酸20が非特異的な増幅を抑制する効果に寄与していることが示された。
【0053】
図7、図8中のMarkerは電気泳動キットに付属されているDNA Markerである。また図8中の標的配列以下のピ−クはPDである。
【0054】
アジレント電気泳動の解析結果から算出された増幅産物の濃度の総和を示す実施例1におけるグラフを図9に、比較例1におけるグラフを図10に示した。図10の比較例1では野生型、変異型とも標的配列以外の非特異的な増幅が起こっているため、増幅産物の濃度の総和が野生型で75.47ng/μLであるのに対し、変異型は60.47ng/μLであり、有意な差がない。一方、ブロック核酸を加えた実施例1の野生型では、標的配列のみの特異的な増幅が起こっている。変異型では全く何の増幅も起こっていない。その結果、増幅産物の濃度の総和は野生型で57.69ng/μLであるのに対し、変異型では0ng/μLであり、完全にバックグラウンドを抑制することができた。すなわち、非特異的な増幅の抑制効果は、PCRの反応副産物として得られるピロリン酸濃度を指標としてSNP塩基を判別する検出手法においても、SNP塩基種の違いによる差を明確にし、有用であることがわかる。
【0055】
以上のように、ブロック核酸を用いることでリアルタイムPCRにおいては、野生型と変異型で立ちあがり時間の差が拡大し、明確なSNP判別が可能となった。さらに、ブロック核酸は非特異的な増幅を抑制しており、これは、ピロリン酸を検出する検出手法においてもバックグラウンドを抑え、明確な判別を可能にする効果に寄与することを示す。
【0056】
実施例2:
本実施例では、ヒトのアセトアルデヒドデヒドロゲナ−ゼ2遺伝子の第12エクソンの114番目のSNP塩基を判別する例において、SNP塩基が変異型の場合、増幅可能なASPを用いた例を示す。第1のプライマ−(以下、ALDH2−Fと表記)と第2のプライマ−(以下、ALDH2−Mt−Rと表記)の配列を表3に示す。本実施例では第2のプライマ−がASPに該当し、ALDH2−Wt−Rは、上記SNP塩基が変異型の場合にDNA伸長反応が起こるように設計されている。具体的には、ALDH2−Mt−Rの3’末端塩基がSNP対応塩基である。またALDH2−Mt−Rの3’末端から2番目および3番目の二塩基は、鋳型に対して非相補的になるように設計されている(表3、下線部)。
【0057】
【表3】

【0058】
また、ALDH2−FとALDH2−Mt−Rは、アセトアルデヒドデヒドロゲナ−ゼ2遺伝子中の144bpの標的配列がPCRにより増幅されるように設計されている(ただし上記SNP塩基が野生型の場合は、ALDH2−Mt−RからのDNA伸長反応効率は極めて悪いため、この標的配列は増幅されない)。
【0059】
ブロック核酸(以下、ALDH2−Block1とALDH2−Block2と表記)の配列を表4に示す。
【0060】
【表4】

【0061】
ALDH2−Block1は標的配列から3塩基上流に結合する実施の形態に記載のブロック核酸(20)に相当し、22塩基からなる。ALDH2−Block2は標的配列から6塩基下流に結合する実施の形態に記載のブロック核酸(30)に相当し、19塩基からなる。ALDH2−Block1とALDH2−Block2の3’末端のヌクレオチドに含まれる3位の糖はリン酸化によって修飾されている。
【0062】
鋳型DNAは、上記SNP塩基が野生型のホモである被験者および変異型のホモである被験者の血液100μLからDNA Micro Kit(QIAGEN社製)を用いて抽出したゲノムDNAを10ng/μLになるように調製して得た。検体の遺伝子型はシ−ケンス解析により確認した。
【0063】
PCRの反応溶液は以下の通りである。
【0064】
1×MightyAmp Buffer(TaKaRa社製)
0.25U/10μL MightyAmp DNA Polymerase(TaKaRa社製)
1μM ALDH2−F
1μM ALDH2−R
0.5ng/μLの野生型DNAもしくは変異型DNA、および
10μM ALDH2−Block
1/10000 SYBR GreenI(TaKaRa社製)
全量:10μL
PCR増幅を、以下のサ−マルプロファイル:98℃で2分間を1サイクル、98℃で1秒間(変性のため)と55℃で1秒間(アニ−ルのため)と68℃で28秒間(DNA伸長のため)を60サイクル、で行った。
【0065】
PCR反応をApplied Biosystems 7300リアルタイムPCRシステムを用いてモニタリングした。図11はリアルタイムPCRの増幅曲線の結果を示す。図11の縦軸はSYBR GreenIの蛍光強度を、横軸は時間を示している。
図11から明らかなように、変異型を鋳型に用いた場合のみ効率的な増幅が起こっている。
さらにBioAnalyzer(Agilent社製)を用いて、PCR反応物を電気泳動に供した。図12に変異型を鋳型に用いたPCRの電気泳動の結果を、図13に野生型を鋳型に用いたPCRの電気泳動結果を示す。図12と図13の縦軸は増幅産物の濃度に比例した蛍光強度を示しており、横軸は時間を示している。図12と図13を比較すると、変異型を鋳型とした場合は144bpの塩基対からなる標的配列のみが特異的に増幅されていた。一方、野生型を鋳型とした場合は81bpの非特異的な増幅が見られるが、標的配列より短く、PCRの反応副産物として得られるピロリン酸濃度を指標としてSNP塩基を判別する検出手法においても、ほとんど問題にならない。図12、図13中のMarkerは電気泳動キットに付属されているDNA Markerである。また図12、図13中の標的配列以下のピ−クはPDである。
【0066】
比較例2:
本比較例2では、実施例2における反応溶液のブロック核酸を蒸留水に置換したこと以外は、実施例2と同様のPCR反応を実施した。図14に比較例1のリアルタイムPCRの増幅曲線の結果を示す。また、図15に変異型を鋳型に用いた場合のPCRの電気泳動結果を、図16に野生型を鋳型に用いた場合のPCRの電気泳動結果を示す。図14の縦軸はSYBR GreenIの蛍光強度を、横軸は時間を示している。図15と図16の縦軸は増幅産物の濃度に比例した蛍光強度を示しており、横軸は時間を示している。
【0067】
まず、図14より、変異型を鋳型に用いた場合と野生型を鋳型に用いた場合において、前者の増幅曲線の方が後者に比べ早く立ち上がっていることが分かる。しかしながら、その差は5.6分でしかなく、有意な差ではなかった。
【0068】
ここで、実施例2におけるリアルタイムPCR結果を示す図11と比較すると、ブロック核酸を用いた実施例2では明らかに立ち上がりの差が広がっていることがわかる。このことは第1のブロック核酸20と第2のブロック核酸30が明確なSNP判別を行なうことに寄与していることを示す。
【0069】
また、図15の変異型を鋳型とした場合では144bpの標的配列が増幅産物だけでなく、303bpの塩基対からなる2本鎖DNAが非特異的に増幅されている。図16の野生型を鋳型とした場合では144bpの標的配列の増幅は起こっていることから、ASPの機能を果たせていないことが示唆され、さらにそれ以外の300bpと351bpの塩基対からなる2本鎖DNAが非特異的に増幅されている。
【0070】
実施例2における図12と図13では、このような非特異的な増幅は全く起こっていない。このことから第1のブロック核酸20と第2のブロック核酸30が非特異的な増幅を抑制する効果に寄与していることが示された。
【0071】
アジレント電気泳動の解析結果から算出された増幅産物の濃度の総和を示す実施例2におけるグラフを図17に、比較例2におけるグラフを図18に示した。図18の比較例2では変異型、野生型とも標的配列以外の非特異的な増幅が起こっているため、増幅産物の濃度の総和が比較例2では変異型の68.86ng/μLに対して、野生型では94.46ng/μLであり、標的配列より塩基対の大きな非特異的な増幅が多いことが原因となり、野生型の方が産物量の総和は大きくなってしまっている。これは、ピロリン酸を測定する検出手法においては、間違った判別結果を導く恐れがある。それに対し、ブロック核酸を加えた実施例2では変異型では標的配列のみの特異的な増幅が起こっている。野生型でもわずかに非特異的な増幅が見られるものの、各々の濃度の総和は変異型では、64.34ng/μLであるのに対し、野生型ではわずか4.79ng/μLであり、有意な差を得ることができている。すなわち、非特異的な増幅の抑制効果は、PCRの反応副産物として得られるピロリン酸濃度を指標としてSNP塩基を判別する検出手法においても、SNP塩基種の違いによる差を明確にし、有用であることがわかる。
【0072】
以上のように、ブロック核酸を用いることでリアルタイムPCRにおいては、野生型と変異型で立ちあがり時間の差が拡大し、明確なSNP判別が可能となった。さらに、ブロック核酸は非特異的な増幅を抑制しており、これは、ピロリン酸を検出する検出手法においてもバックグラウンドを抑え、明確な判別を可能にする効果に寄与することを示す。
【産業上の利用可能性】
【0073】
以上のように、本発明は、一般的なPCR反応に利用可能である。また、臨床検査用途を目的としたPCRにも利用できる。
【符号の説明】
【0074】
1 二本鎖標的配列
1a 一本鎖標的配列
1b 相補的一本鎖標的配列
2 望まれる増幅二本鎖DNA配列
4 第1のプライマ−
5 第2のプライマ−
6 第1一本鎖DNA
6a 第1非増幅配列
6b 第2非増幅配列
6c 一本鎖標的配列1aの3’末端部分
6g 増幅一本鎖標的配列
6k 第4複製配列
7 第2一本鎖DNA
7a 第3非増幅配列
7b 第4非増幅配列
7g 増幅相補的一本鎖標的配列
7k 相補的第4複製配列
8 SNP塩基
20 第1ブロック核酸
30 第2ブロック核酸
【特許請求の範囲】
【請求項1】
第1一本鎖DNA(6)および第2一本鎖DNA(7)からなる二本鎖DNA中の二本鎖標的配列(1)中に含まれるSNP塩基種(8)を、アレル特異的プライマ−を用いて判別するSNP判別法であって、
前記二本鎖標的配列(1)は、一本鎖標的配列(1a)および相補的一本鎖標的配列(1b)からなり、
前記第1一本鎖DNA(6)は、3’末端−第1非増幅配列(6a)−前記一本鎖標的配列(1a)−第2非増幅配列(6b)−5’末端からなり、
前記第2一本鎖DNA(7)は、5’末端−第3非増幅配列(7a)−前記相補的一本鎖標的配列(1b)−第4非増幅配列(7b)−3’末端からなり、
前記相補的一本鎖標的配列(1b)、第3非増幅配列(7a)、および第4非増幅配列(7b)は、それぞれ、前記一本鎖標的配列(1a)、前記第1非増幅配列(6a)、および第2非増幅配列(6b)と相補的であり、
DNAポリメラ−ゼ、デオキシヌクレオシド三リン酸、前記二本鎖DNA(6・7)、第1のプライマ−(4)、第2のプライマ−(5)、および第1ブロック核酸(20)を混合し、ポリメラ−ゼ連鎖反応を用いて前記二本鎖標的配列(1)を増幅する工程Aを含み、
ここで、
前記第1のプライマ−(4)および前記第2のプライマ−(5)は、いずれも、前記DNAポリメラ−ゼによる伸長反応のための起点として作用し、
前記SNP塩基種(8)は、第一の一本鎖DNA(6)上の前記第1のプライマ−(4)が結合する領域内に存在し、
前記第1のプライマ−(4)は、SNPの塩基(8)に相補的な配列である、アレル特異的プライマ−であって、
前記第1のプライマ−(4)は、前記一本鎖標的配列(1a)に含まれる3’末端側の配列の部分に相補的であり、
前記第2のプライマ−(5)は、前記相補的一本鎖標的配列(1b)に含まれる3’末端側の部分の配列に相補的であり、
前記第1ブロック核酸(20)は、前記DNAポリメラ−ゼによる伸長反応のための起点として作用せず、
前記第1ブロック核酸(20)は、前記第3非増幅配列(7a)の一部と相補的である、SNP判別方法。
【請求項2】
前記第1ブロック核酸(20)は、3’末端に位置するヌクレオチドに含まれる糖の3位のOH基が、水素、リン酸基、アミノ基、ビオチン基、チオ−ル基、またはこれらの誘導体によって置換または修飾されているDNAからなる、請求項1に記載のSNP判別方法。
【請求項3】
前記第1ブロック核酸(20)は、3’末端に位置するヌクレオチドに含まれる糖の3位のOH基が、水素、リン酸基、アミノ基、ビオチン基、チオ−ル基、またはこれらの誘導体によって置換または修飾されているLocked Nucleic Acidからなる、請求項1に記載のSNP判別方法。
【請求項4】
前記第1ブロック核酸(20)は、Peptide Nucleic Acidからなる、請求項1に記載のSNP判別方法。
【請求項5】
前記工程Aにおいて、前記DNAポリメラ−ゼによる伸長反応のための起点として作用せず、かつ前記第2非増幅配列(6b)の一部と相補的である第2ブロック核酸(30)も混合される、請求項1に記載のSNP判別方法。
【請求項6】
前記第2ブロック核酸(30)は、3’末端に位置するヌクレオチドに含まれる糖の3位のOH基が、水素、リン酸基、アミノ基、ビオチン基、チオ−ル基、またはこれらの誘導体によって置換または修飾されているDNAからなる、請求項5に記載のSNP判別方法。
【請求項7】
前記第2ブロック核酸(30)は、3’末端に位置するヌクレオチドに含まれる糖の3位のOH基が、水素、リン酸基、アミノ基、ビオチン基、チオ−ル基、またはこれらの誘導体によって置換または修飾されているLocked Nucleic Acidからなる、請求項5に記載のSNP判別方法。
【請求項8】
前記第2ブロック核酸(30)は、Peptide Nucleic Acidからなる、請求項5に記載のSNP判別方法。
【請求項1】
第1一本鎖DNA(6)および第2一本鎖DNA(7)からなる二本鎖DNA中の二本鎖標的配列(1)中に含まれるSNP塩基種(8)を、アレル特異的プライマ−を用いて判別するSNP判別法であって、
前記二本鎖標的配列(1)は、一本鎖標的配列(1a)および相補的一本鎖標的配列(1b)からなり、
前記第1一本鎖DNA(6)は、3’末端−第1非増幅配列(6a)−前記一本鎖標的配列(1a)−第2非増幅配列(6b)−5’末端からなり、
前記第2一本鎖DNA(7)は、5’末端−第3非増幅配列(7a)−前記相補的一本鎖標的配列(1b)−第4非増幅配列(7b)−3’末端からなり、
前記相補的一本鎖標的配列(1b)、第3非増幅配列(7a)、および第4非増幅配列(7b)は、それぞれ、前記一本鎖標的配列(1a)、前記第1非増幅配列(6a)、および第2非増幅配列(6b)と相補的であり、
DNAポリメラ−ゼ、デオキシヌクレオシド三リン酸、前記二本鎖DNA(6・7)、第1のプライマ−(4)、第2のプライマ−(5)、および第1ブロック核酸(20)を混合し、ポリメラ−ゼ連鎖反応を用いて前記二本鎖標的配列(1)を増幅する工程Aを含み、
ここで、
前記第1のプライマ−(4)および前記第2のプライマ−(5)は、いずれも、前記DNAポリメラ−ゼによる伸長反応のための起点として作用し、
前記SNP塩基種(8)は、第一の一本鎖DNA(6)上の前記第1のプライマ−(4)が結合する領域内に存在し、
前記第1のプライマ−(4)は、SNPの塩基(8)に相補的な配列である、アレル特異的プライマ−であって、
前記第1のプライマ−(4)は、前記一本鎖標的配列(1a)に含まれる3’末端側の配列の部分に相補的であり、
前記第2のプライマ−(5)は、前記相補的一本鎖標的配列(1b)に含まれる3’末端側の部分の配列に相補的であり、
前記第1ブロック核酸(20)は、前記DNAポリメラ−ゼによる伸長反応のための起点として作用せず、
前記第1ブロック核酸(20)は、前記第3非増幅配列(7a)の一部と相補的である、SNP判別方法。
【請求項2】
前記第1ブロック核酸(20)は、3’末端に位置するヌクレオチドに含まれる糖の3位のOH基が、水素、リン酸基、アミノ基、ビオチン基、チオ−ル基、またはこれらの誘導体によって置換または修飾されているDNAからなる、請求項1に記載のSNP判別方法。
【請求項3】
前記第1ブロック核酸(20)は、3’末端に位置するヌクレオチドに含まれる糖の3位のOH基が、水素、リン酸基、アミノ基、ビオチン基、チオ−ル基、またはこれらの誘導体によって置換または修飾されているLocked Nucleic Acidからなる、請求項1に記載のSNP判別方法。
【請求項4】
前記第1ブロック核酸(20)は、Peptide Nucleic Acidからなる、請求項1に記載のSNP判別方法。
【請求項5】
前記工程Aにおいて、前記DNAポリメラ−ゼによる伸長反応のための起点として作用せず、かつ前記第2非増幅配列(6b)の一部と相補的である第2ブロック核酸(30)も混合される、請求項1に記載のSNP判別方法。
【請求項6】
前記第2ブロック核酸(30)は、3’末端に位置するヌクレオチドに含まれる糖の3位のOH基が、水素、リン酸基、アミノ基、ビオチン基、チオ−ル基、またはこれらの誘導体によって置換または修飾されているDNAからなる、請求項5に記載のSNP判別方法。
【請求項7】
前記第2ブロック核酸(30)は、3’末端に位置するヌクレオチドに含まれる糖の3位のOH基が、水素、リン酸基、アミノ基、ビオチン基、チオ−ル基、またはこれらの誘導体によって置換または修飾されているLocked Nucleic Acidからなる、請求項5に記載のSNP判別方法。
【請求項8】
前記第2ブロック核酸(30)は、Peptide Nucleic Acidからなる、請求項5に記載のSNP判別方法。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】


【図16】


【図17】


【図18】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】


【図16】


【図17】


【図18】


【公開番号】特開2013−22003(P2013−22003A)
【公開日】平成25年2月4日(2013.2.4)
【国際特許分類】
【出願番号】特願2011−162742(P2011−162742)
【出願日】平成23年7月26日(2011.7.26)
【出願人】(000005821)パナソニック株式会社 (73,050)
【Fターム(参考)】
【公開日】平成25年2月4日(2013.2.4)
【国際特許分類】
【出願日】平成23年7月26日(2011.7.26)
【出願人】(000005821)パナソニック株式会社 (73,050)
【Fターム(参考)】
[ Back to top ]

