イヌ科動物血統の同定のための方法および材料
【課題】イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を提供すること。
【解決手段】この方法は、以下の工程を包含する:(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および(b)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程。工程(b)において、各イヌ科動物ゲノム個体群プロファイルは、上記イヌ科動物個体群における上記マーカーセットについての遺伝子型情報を含む。
【解決手段】この方法は、以下の工程を包含する:(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および(b)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程。工程(b)において、各イヌ科動物ゲノム個体群プロファイルは、上記イヌ科動物個体群における上記マーカーセットについての遺伝子型情報を含む。
【発明の詳細な説明】
【技術分野】
【0001】
(関連出願の相互参照)
本出願は、2003年12月17日に出願された米国仮特許出願第60/530,464号の利益を主張する。
【0002】
(政府のライセンス権に関する記載)
米国政府は、本発明における支払い済みのライセンスを有し、そして限定された状況において、特許権者が米国国立衛生研究所によって与えられたHG300035の条件で規定されるような妥当な条件でライセンスを他者に供与するよう要求する権利を有する。
【0003】
(発明の分野)
本発明は、多型マーカーを用いてイヌ科動物ゲノムへの1種以上のイヌ科動物個体群の寄与度を決定することに関する。
【背景技術】
【0004】
(発明の背景)
Canis familiaris(イエイヌ)は、表現型が分化し遺伝的に隔離された400種以上の血統に分岐した単一種であり、これらのうちの152種は、米国でアメリカンケンネルクラブ公認である(非特許文献1)。別個の犬血統は、形態学、挙動、および疾患罹患率の特有の集まりによって特徴付けられる(非特許文献2)。種々のイヌの形態学は、1000年前から存在し、そしてイヌ間での生殖隔離は、19世紀中頃において血統クラブおよび血統標準の出現とともに形式化された。その時以来、「品種の境界(breed barrier)」規則(すなわち、イヌの雌親と雄親の両方が登録された一員でなければ、いずれのイヌも品種登録された一員になり得ないという規則)が発布され、各犬種間で比較的閉じた遺伝子プール(genetic pool)を保証している。
【0005】
350種類以上の遺伝性障害は、純血種イヌ個体群を分離する(非特許文献3)。これらの多くはヒトの一般的な障害と類似しており、特有の形態を生成するために使用された積極的な同系交配プログラムの結果として、特定の品種または品種群に限定されている。
【0006】
個々の犬種を客観的に決定すること(例えば、特定品種に帰属するイヌであるという証明)のための多くの可能な使用が存在する。歴史的記録物は信頼度において品種ごとに変動するので、前の個体群情報に基づかない遺伝分析が、個体群構造を決定する最も直接的かつ正確な方法である。過去10年間にわたり、分子生物学的方法(molecular method)が、野生イヌ科動物種についての我々の理解を深めるために、そしてイエイヌとそれら野生種との関係を決定するために使用されてきた。ミトコンドリアDNA配列分析は、イエイヌとオオカミとの間の関係を説明し、4万〜10万年前に起こった複数の家畜化事象を解明する(非特許文献4;非特許文献5;非特許文献6)。しかし、ミトコンドリアDNAの進化は、現代の犬種間での関係を推論することを可能にするには遅すぎる。ほとんどの現代の犬種は、400年未満の間ずっと存在してきた。加えて、系統学的距離尺度および系統樹作成プログラムは、イヌ個体群において一般に観察されるような網目状進化を扱うための機能がない(非特許文献7;非特許文献8;非特許文献9)。ある先行研究は、核のマイクロサテライトの座位が5種からそれらの起源種へとイヌを割り当てるために使用され得そしてこれらの品種間で大きな遺伝的距離を示すことを示した(非特許文献10)。別の研究は、28種の収集物中の2種のペアの関連性を検出するためにマイクロサテライトを使用したが、品種間の広範な系統学的関係を証明し得なかった(非特許文献9)。このような関連性を見出すことの失敗は、マイクロサテライトの座位の特質(非特許文献9)、検査した限定された数の品種、またはこの研究において使用された分析方法を反映し得る。その代わりに、この失敗は、大部分の品種の最も近い起源およびそれらの生物における先祖型の混合によって、純血種イヌ個体群における複雑な構造を反映し得る。
【先行技術文献】
【非特許文献】
【0007】
【非特許文献1】CrowleyおよびAdelman編、American Kennel Club「The Complete Dog Book」、Howell Book Hues,New York,NY、1998年
【非特許文献2】Ostranderら、「Trends in Genetics」、2000年、第16号、p.117−23
【非特許文献3】Pattersonら、J.Am.Vet.Med.Assoc.、1988年、第193号、p.1131
【非特許文献4】Vilaら、Science、1997年、第276号、p.1687−9
【非特許文献5】Savolainenら、Science、2002年、第298号、p.1610−3
【非特許文献6】Leonardら、Science、2002年、第298号、p.1613−6
【非特許文献7】Zajcら、Mamm.Genome、1997年 第8巻、第3号、p.182−5
【非特許文献8】KoskinenおよびBredbacka、Animal Genetics、2000年、第31号、p.310−17
【非特許文献9】Irionら、J.Hered.、2003年、第94巻、第1号、p81−7
【非特許文献10】Koskinen、Anim.Genet,、2003年、第34号、p297
【発明の概要】
【発明が解決しようとする課題】
【0008】
関連する品種群を規定する方法、および個々のイヌゲノムへの品種の寄与度を明確に同定する方法の必要性が存在する。本発明は、この必要性および他の必要性に取り組む。
【課題を解決するための手段】
【0009】
(発明の要旨)
一局面において、本発明は、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を提供する。この方法は、以下の工程を包含する:(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および(b)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程。工程(b)において、各イヌ科動物ゲノム個体群プロファイルは、上記イヌ科動物個体群におけるマーカーセットについての遺伝子型情報を含む。このマーカーセットは、少なくとも約5個のマーカー(例えば、イヌ科動物ゲノムの地図上に示される少なくとも約5個のマーカー)を含み得る。本発明の方法における使用に適する例示的なマーカーとしては、例えば、マイクロサテライトマーカー、一塩基多型(SNP)、ミトコンドリアマーカー、および制限酵素断片長多型が挙げられる。例えば、上記マーカーセットは、表2に示すSNPマーカーのうちの少なくとも5個、および/または、表1に示すマイクロサテライトマーカーのうちの少なくとも5個を含み得る。上記マーカーセットは、1つ以上の個体群特異的マーカー(例えば、1つ以上の個体群特異的SNPマーカー、または1つ以上の個体群特異的マイクロサテライトマーカー)を含み得る。例えば、1つ以上のSNPマーカーは、372c5t−82、372e13t−57、372m6t−88、372m23t−76、373a15t−112、373e1t−50、373elt−130、373g19t−246、373i8s−224、373k8s−181、372c5s−168、372C15S−196、372e15s−71、および373a21t−93からなる群より選択され得る。
【0010】
上記マーカーセットの各々についての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性は、当該分野において標準的な方法(例えば、ハイブリダイゼーション法、ポリメラーゼ連鎖反応法、サイズ分画法、DNA塩基配列決定法など)を用いて得られ得る。例えば、上記方法の工程(a)は、上記マーカーセットの各々に特異的なプライマーを用いて、上記試験イヌ科動物のゲノムDNAを増幅する工程、およびこの増幅産物のサイズを決定する工程を包含し得る。工程(a)はまた、上記マーカーセットの各々に特異的なプライマーを用いて、上記試験イヌ科動物のゲノムDNAを増幅する工程、およびこの増幅産物のヌクレオチド配列を決定する工程を包含し得る。いくつかの実施形態において、上記プライマーは、配列番号1〜200からなる群より選択される。いくつかの実施形態において、上記プライマーは、配列番号1〜244〜327からなる群より選択される。
【0011】
イヌ科動物個体群プロファイルにおける遺伝子型情報は、例えば、そのイヌ科動物個体群の一員である1種以上のイヌ科動物におけるマーカーセット中のほとんどまたはすべてのマーカーの片方または両方の対立遺伝子の同一性、および/または、そのイヌ科動物個体群におけるマーカーセット中のほとんどまたはすべてのマーカーの少なくとも1つの対立遺伝子について推定された対立遺伝子頻度のような情報を含み得る。イヌ科動物個体群プロファイル中の推定された対立遺伝子頻度の各々は、代表的には、このイヌ科動物個体群の一員である少なくとも2つのイヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性に基づいている。イヌ科動物個体群プロファイルデータベースは、約5個と約数百個のとの間のイヌ科動物個体群プロファイル(例えば、少なくとも100個のイヌ科動物個体群プロファイル)を含み得る。いくつかの実施形態において、上記イヌ科動物個体群プロファイルは、登録種(例えば、アメリカンケンネルクラブに登録された品種)のプロファイルを含む。
【0012】
いくつかの実施形態において、上記マーカーセットは、約1500個より少ないSNPマーカーを含み、そして上記方法は、上記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する。いくつかの実施形態において、上記マーカーセットは、約200個より少ないSNPマーカー(例えば、約100個のSNPマーカー、または約50個のSNPマーカー)を含み、そして上記方法は、上記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する。
【0013】
上記方法の工程(b)において、1種以上のイヌ科動物個体群が上記試験イヌ科動物ゲノムへ寄与した可能性は、任意の適切なアルゴリズム(例えば、ベイジアンモデルベースのクラスタリングアルゴリズムまたは割り当てアルゴリズム)を用いて決定され得る。いくつかの実施形態において、工程(b)は、特定のイヌ科動物個体群が上記試験イヌ科動物ゲノムへ寄与した確率を決定する工程を包含し、この確率は、この試験イヌ科動物ゲノム中の対立遺伝子がこの特定のイヌ科動物個体群中で生じる条件付き確率を、この試験イヌ科動物ゲノム中の対立遺伝子が上記データベース内の、各イヌ科動物個体群中で生じる条件付き確率の合計で割って決定することにより決定される。いくつかの実施形態において、工程(b)は、上記試験イヌ科動物ゲノムへの2つ以上の遺伝的に関連するイヌ科動物個体群の寄与度の間を、この試験イヌ科動物ゲノム中の対立遺伝子をこの2つ以上の遺伝的に関連するイヌ科動物個体群プロファイルを含むデータベースと比較することによって識別する工程を包含する。例示的な遺伝的に関連したイヌ科動物個体群としては、ベルジアンシープドッグおよびベルジアンタービュレン;コリーおよびシェトランドシープドッグ;ウィペットおよびグレイハウンド;シベリアンハスキーおよびアラスカンマラミュート;マスチフおよびブルマスチフ;グレータースイスマウンテンドックおよびバーニーズマウンテンドッグ;ウェストハイランドホワイトテリアおよびケアーンテリア;ならびにラサアプソ、シーズー、およびペキニーズが挙げられるが、これらに限定されない。いくつかの実施形態において、本発明の方法は、上記試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する文書を提供する工程をさらに包含する。この文書は、上記試験イヌ科動物ゲノムまたは上記試験イヌ科動物へ寄与した1種以上のイヌ科動物個体群に関する情報(例えば、健康に関する情報(例えば、疾患の素因)、保険情報、または任意の他の種類の情報)を提供し得る。上記文書はまた、上記試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度についての証明書を提供し得る。いくつかの実施形態において、上記文書は、上記試験イヌ科動物のゲノムへ寄与した1種以上のイヌ科動物個体群についての表示(representation)(例えば、写真、図、または他の描写)を提供する。
【0014】
いくつかの実施形態において、本発明は、1種以上のイヌ科動物個体群を規定する方法を提供する。この方法は、以下の工程を包含する:(a)各イヌ科動物ゲノムセットについて、各マーカーセットのついての片方または両方の対立遺伝子の同一性を得る工程;および、(b)上記イヌ科動物ゲノムセットの1つ以上の一員が、統計的モデリングを用いて各マーカーについて1セットの対立遺伝子頻度によって特徴付けられる別個のイヌ科動物個体群を規定する可能性を決定することによって、1種以上のイヌ科動物個体群を規定する工程。
【0015】
別の局面において、本発明は、各マーカーセットについてイヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得るために、核酸配列を含む基材を提供する。
【0016】
さらなる局面において、本発明は、イヌ科動物個体群の区別に使用するために、媒体上に保存されたデータ構造を含むコンピュータ読み取り可能な媒体を提供し、このデータ構造は:(a)マーカーの名称またはこのマーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および(b)イヌ科動物個体群におけるそのマーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、そのマーカーフィールドのインスタンス化およびこの遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールドを備える。例えば、この遺伝子型情報フィールドは、イヌ科動物個体群におけるマーカー(例えば、SNPマーカー)の対立遺伝子頻度の推定値を保存することが可能であり得る。上記遺伝子型情報フィールドはまた、そのイヌ科動物個体群の一員である1種以上のイヌ科動物における各マーカーセットの片方または両方の対立遺伝子の同一性を保存することが可能であり得る。いくつかの実施形態において、上記コンピュータに読み取り可能媒体は、以下をこの媒体上に保存したものを有する基板を備える:
(a)イヌ科動物個体群の区別に使用するためのデータ構造であって、このデータ構造は;
(i)マーカーの名称またはこのマーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(ii)イヌ科動物個体群におけるそのマーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、このマーカーフィールドのインスタンス化およびこの遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、データ構造;ならびに
(b)イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を実施するための、コンピュータが実行可能な命令であって、以下の工程:
(i)各マーカーセットについて試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(ii)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物プロファイルを含むデータベースとを比較することによって決定する工程であって、イヌ科動物個体群プロファイルの各々は、そのイヌ科動物個体群における上記マーカーセットについての遺伝子型情報を含む、工程
を包含する、コンピュータが実行可能な命令を備える、コンピュータに読み取り可能な情報を含む。
【0017】
本発明の上述の局面および付随する多くの利益は、添付の図面と一緒に以下の詳細な説明を参照することによってより良く理解されるので、より容易に理解される。
【0018】
例えば、本発明は、以下の項目を提供する。
(項目1)
イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法であって、以下の工程:
(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(b)該試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、該試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程であって、各イヌ科動物個体群プロファイルは、該イヌ科動物個体群におけるマーカーセットに関する遺伝子型情報を含む、工程
を包含する、方法。
(項目2)
項目1に記載の方法であって、前記マーカーセットは、少なくとも約5個のマーカーを含む、方法。
(項目3)
項目1に記載の方法であって、前記マーカーセットは、マイクロサテライトマーカーを含む、方法。
(項目4)
項目3に記載の方法であって、前記マイクロサテライトマーカーは、表1に示すマイクロサテライトマーカーのうちの少なくとも5個を含む、方法。
(項目5)
項目1に記載の方法であって、前記マーカーセットは、一塩基多型(SNP)を含む、方法。
(項目6)
項目5に記載の方法であって、前記SNPマーカーは、表2に示すSNPマーカーのうちの少なくとも5個を含む、方法。
(項目7)
項目1に記載の方法であって、前記マーカーセットは、1つ以上の個体群特異的マーカーを含む、方法。
(項目8)
項目7に記載の方法であって、前記1つ以上の個体群特異的マーカーは、1つ以上のSNPマーカーを含む、方法。
(項目9)
項目8に記載の方法であって、前記1つ以上のSNPマーカーは、372c5t−82、372e13t−57、372m6t−88、372m23t−76、373a15t−112、373e1t−50、373elt−130、373g19t−246、373i8s−224、373k8s−181、372c5s−168、372C15S−196、372e15s−71、および373a21t−93からなる群より選択される、方法。
(項目10)
項目1に記載の方法であって、イヌ科動物個体群プロファイルにおける前記遺伝子型情報は、前記マーカーセットの各々の片方または両方の対立遺伝子の同一性を含む、方法。
(項目11)
項目1に記載の方法であって、イヌ科動物個体群プロファイルにおける前記遺伝子型情報は、前記マーカーセットの各々の少なくとも片方の対立遺伝子についての対立遺伝子頻度を含む、方法。
(項目12)
項目1に記載の方法であって、前記イヌ科動物個体群プロファイルデータベースは、約5個と約500個と間のイヌ科動物個体群プロファイルを含む、方法。
(項目13)
項目1に記載の方法であって、前記イヌ科動物個体群プロファイルデータベースは、少なくとも約5種のアメリカンケンネルクラブ公認血統種についてプロファイルを含む、方法。
(項目14)
項目1に記載の方法であって、前記マーカーセットは、約1500個よりも少ないSNPマーカーを含み、そして該方法は、前記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する、方法。
(項目15)
項目1に記載の方法であって、前記マーカーセットは、約200個よりも少ないSNPマーカーを含み、そして該方法は、前記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する、方法。
(項目16)
項目1に記載の方法であって、工程(a)は、前記マーカーセットの各々に特異的なプライマーを用いて、前記試験イヌ科動物のゲノムDNAを増幅する工程、および該増幅産物のサイズを決定する工程を包含する、方法。
(項目17)
項目1に記載の方法であって、工程(b)は、遺伝子型クラスタリングプログラムを用いる工程を包含する、方法。
(項目18)
項目1に記載の方法であって、工程(b)は、割当てアルゴリズムを用いる工程を包含する、方法。
(項目19)
項目1に記載の方法であって、工程(b)は、特定のイヌ科動物個体群が前記試験イヌ科動物ゲノムへ寄与した確率を決定する工程を包含し、該確率は、該試験イヌ科動物ゲノム中の対立遺伝子が該特定のイヌ科動物個体群中で生じる条件付き確率を、該試験イヌ科動物ゲノム中の対立遺伝子が前記データベース内の、各イヌ科動物個体群中で生じる条件付き確率の合計で割って決定することにより決定される、方法。
(項目20)
項目1に記載の方法であって、工程(b)は、前記試験イヌ科動物ゲノムへの2つ以上の遺伝的に関連するイヌ科動物個体群の寄与度の間を、該試験イヌ科動物ゲノム中の対立遺伝子を該2つ以上の遺伝的に関連するイヌ科動物個体群プロファイルを含むデータベースと比較することによって識別する工程を包含する、方法。
(項目21)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ベルジアンシープドッグおよびベルジアンタービュレンを含む、方法。
(項目22)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、コリーおよびシェトランドシープドッグを含む、方法。
(項目23)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ウィペットおよびグレイハウンドを含む、方法。
(項目24)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、シベリアンハスキーおよびアラスカンマラミュートを含む、方法。
(項目25)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、マスチフおよびブルマスチフを含む、方法。
(項目26)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、グレータースイスマウンテンドックおよびバーニーズマウンテンドッグを含む、方法。
(項目27)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ウェストハイランドホワイトテリアおよびケアーンテリアを含む、方法。
(項目28)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ラサアプソ、シーズー、およびペキニーズを含む、方法。
(項目29)
項目1に記載の方法であって、前記方法は、前記試験イヌ科動物ゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する文書を提供する工程をさらに包含する、方法。
(項目30)
項目29に記載の方法であって、前記文書は、前記試験イヌ科動物ゲノムまたは前記イヌ科動物へ寄与した前記1種以上のイヌ科動物個体群に関する情報を提供する、方法。
(項目31)
項目30に記載の方法であって、前記情報は、健康に関する情報である、方法。
(項目32)
項目30に記載の方法であって、前記情報は、保険情報である、方法。
(項目33)
項目29に記載の方法であって、前記文書は、1種以上のイヌ科動物個体群の前記試験イヌ科動物ゲノムのゲノムへの寄与度の証明書を提供する、方法。
(項目34)
項目29に記載の方法であって、前記文書は、前記試験イヌ科動物ゲノムへ寄与した1種以上のイヌ科動物個体群の表示を提供する、方法。
(項目35)
1種以上のイヌ科動物個体群を規定する方法であって、以下の工程:
(a)各イヌ科動物ゲノムセットについて、各マーカーセットのついての片方または両方の対立遺伝子の同一性を得る工程;および
(b)イヌ科動物ゲノムセットの1つ以上の一員が、統計的モデリングを用いて各マーカーについて1セットの対立遺伝子頻度によって特徴付けられる別個のイヌ科動物個体群を規定する可能性を決定することによって、1種以上のイヌ科動物個体群を規定する工程を包含する、方法。
(項目36)
コンピュータに読み取り可能な媒体であって、イヌ科動物個体群の区別に使用するために、該媒体上に保存されたデータ構造を含み、該データ構造は:
(a)マーカーの名称または該マーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(b)イヌ科動物個体群における該マーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、該マーカーフィールドのインスタンス化および該遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、コンピュータに読み取り可能な媒体。
(項目37)
コンピュータに読み取り可能な媒体であって、以下:
(a)イヌ科動物個体群の区別に使用するために該媒体上に保存されたデータ構造であって、該データ構造は;
(i)マーカーの名称または該マーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(ii)イヌ科動物個体群における該マーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、該マーカーフィールドのインスタンス化および該遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、データ構造;ならびに
(b)イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を実施するための、コンピュータが実行可能な命令であって、以下の工程:
(i)各マーカーセットについて試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(ii)該試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、該試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物プロファイルを含むデータベースとを比較することによって決定する工程であって、イヌ科動物個体群プロファイルの各々は、該イヌ科動物個体群における該マーカーセットについての遺伝子型情報を含む、工程
を包含する、コンピュータが実行可能な命令;
を該媒体上に保存したものを備える、コンピュータに読み取り可能な媒体。
【図面の簡単な説明】
【0019】
【図1】図1は、2種のイヌ科動物個体群に対する疾患素因に関する情報とともに、試験イヌ科動物(ファイド(Fido))ゲノムへの2種のイヌ科動物個体群(ボーダーコリーおよびブルマスチフ)の寄与度を表示する例示的な文書を示す。
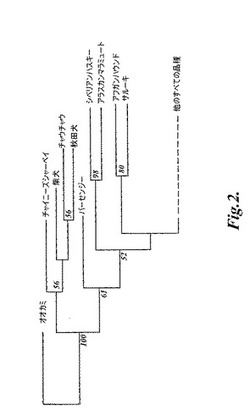
【図2】図2は、実施例4に記載されるように、85種の犬種とハイイロオオカミとのコンセンサス近隣結合木(consensus neighbor−joining tree)を示す。統計学的支持によって枝を形成する9種が示される。残りの76種は、わずかに系統学的構造を示し、そして簡略化のために「他のすべての品種」と標識した1つの枝にまとめている。このコンセンサスを形成した木は、コード距離尺度に基づいている。このデータについて500回のブートストラップ複製が行われ、そして各枝を支持するブートストラップの比が、複製で50%を超える場合には、支持された枝に関して百分率で、対応するノードにて示される。木の根にあるオオカミ個体群は、8個体から構成される。この個体は、以下の各国々(中国、オマーン、イラン、スウェーデン、イタリア、メキシコ、カナダ、および米国)に由来する1つの個体である。枝の長さは、ブートストラップ値に比例している。
【発明を実施するための形態】
【0020】
(好ましい実施形態の詳細な説明)
本明細書は、本明細書とともに提出された2枚のコンパクトディスクに含まれるファイルを、それらの全体を参考して本明細書により援用する。第1のコンパクトディスクは表3および表4を含み、第2のコンパクトディスクは、配列表を含む。
【0021】
本明細書中で具体的に規定されない限り、本明細書中で使用される全ての用語は、本発明の当業者が使用するのと同じ意味を有する。
【0022】
第一の局面において、本発明は、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を提供する。この方法は、以下の工程:
(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(b)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程であって、この工程において各イヌ科動物個体群プロファイルは、このイヌ科動物個体群におけるマーカーセットに関する遺伝子型情報を含む、工程
を包含する。
【0023】
本明細書中で使用される場合、用語「イヌ科動物個体群の寄与度を決定する」とは、1種以上のイヌ科動物個体群が試験イヌ科動物ゲノムへ寄与したか否かについて推論をするために、イヌ科動物個体群の寄与度を統計学的方法を使用して推定または推測することをいう。
【0024】
本明細書中で使用される場合、用語「イヌ科動物」とは、オオカミ、ジャッカル、キツネ、コヨーテ、およびイエイヌを含む、Canidae科の一員である動物をいう。例えば、イヌ科動物は、イエイヌ、オオカミ、またはCanidae科の一種以上からの何らかの遺伝的寄与度を有する動物であり得る。用語「イヌ科動物個体群」は、血統関係にあるイヌ科動物の一群(例えば、イエイヌ種)をいう。用語「品種(breed)」とは、ヒトによって制御された状況下で選択されている比較的均一な表現型の形質を有する種内の動物の一群をいう。例えば、アメリカンケンネルクラブ(AKC)は、7種の群(ハーディング、ハウンド、ノンスポーティング、スポーティング、テリア、トイ、およびワーキング)に分かれる152種を認めている(American Kennel Club(1998)「The Complete Dog Book」、CrowleyおよびAdelman編、Howell Book Hues、New York、NY)。本発明の方法は、任意の犬種の遺伝的寄与度を推定するために使用され得る。この任意の犬種としては、アフガンハウンド、エアデールテリア、秋田犬、アラスカンマラミュート、アメリカンエスキモードッグ、アメリカンフォックスハウンド、アメリカンへアレスラットテリア、アメリカンスタッフォードシャーテリア、アメリカンウォータースパニエル、オーストラリアンキャトルドッグ、オーストラリアンシェパード、オーストラリアンテリア、バセニー、バセットハウンド、ビーグル、ビアデッドコリー、ベドリントンテリア、ベルジアンラケノア、ベルジアンマリノア、ベルジアンシープドッグ、ベルジアンタービュレン、バーニーズマウンテンドッグ、ビションフリーゼ、ブラットハウンド、ボーダーコリー、ボーダーテリア、ボルゾイ、ボストンテリア、ブーヴィエデフランドル、ボイキンスパニエル、ボクサー、ブリアード、ブリタニー、ブルドッグ、ブラッセルグリフォン、ブルマスチフ、ブルテリア、ケアーンテリア、カーディガンウェルシュコーギー、キャバリアキングチャールズスパニエル、チェサピークベイレトリバー、チワワ、チャイニーズクレステッド、チャイニーズシャーペイ、チャウチャウ、クライバースパニエル、コッカースパニエル、コリー、カーリーコーテッドレトリバー、ダックスフンド、ダルメシアン、ダンディーディンモントテリア、ドーベルマンピンシェル、ドゴカナリオ、イングリッシュコッカースパニエル、イングリッシュフォックスハウンド、イングリッシュセッター、イングリッシュスプリンガースパニエル、エントレブッハーマウンテンドッグ、フィールドスパニエル、フラットコーテッドレトリバー、フレンチブルドッグ、ジャーマンロングヘアードポインター、ジャーマンシェパードドッグ、ジャーマンショートヘアードポインター、ジャーマンワイヤーヘアードポインター、ジャイアントシュナウザー、ゴールデンレトリバー、ゴードンセッター、グレートデーン、グレートピレニーズ、グレートスミスマウンテンドッグ、グレイハウンド、ハリヤー、ハバニーズ、イビザンハウンド、アイリッシュセッター、アイリッシュテリア、アイリッシュウォータースパニエル、アイリッシュウルフハウンド、イタリアングレイハウンド、ジャックラッセルテリア、キースホンド、ケリーブルーテリア、コモンドール、クバーズ、ラブラドールレトリバー、レオンベルガー、ラサアプソ、ローシェン、マルチーズ、スタンダードマンチェスターテリア、トイマンチェスターテリア、マスチフ、ミニチュアブルテリア、ミニチュアピンシャー、ミニチュアプードル、ミニチュアシュナウザー、ミュンスターレンダー、ナポリタンマスチフ、ニューファンドランド、ニューギニアシンギングドッグ、ノルウェジアンエルクハウンド、ノーリッチテリア、オールドイングリッシュシープドッグ、パピヨン、ペキニーズ、ウェルシュコーギーペンブローク、プチバセットグリフォンヴァンデーン、ファラオハウンド、ポインター、ポリッシュローランドシープドッグ、ポメラニアン、ポルトガルウォータードッグ、プレサカナリオ、パグ、プーリー、プーミー、ローデシアンリッジバック、ロットワイラー、セントバーナード、サルーキ、サモエド、スキッパーキ、スコティッシュディアハウンド、スコティッシュテリア、シルキーテリア、シェトランドシープドッグ、柴犬、シーズー、シベリアンハスキー、スムースフォックステリア、ソフトコーテッドウィートンテリア、スピノニイタリアーノ、スタッフォードシャーブルテリア、スタンダードプードル、スタンダードシュナウザー、サセックススパニエル、チベタンスパニエル、チベタンテリア、トイフォックステリア、トイプードル、ビズラ、ワイマラナー、ウェルシュスプリンガースパニエル、ウェルシュテリア、ウェストハイランドホワイトテリア、ワイヤーヘアードポインティンググリフォン、ウィペット、ヨークシャーテリアが挙げられるが、これらに限定されない。
【0025】
本発明の方法はまた、イヌ科動物個体群からの遺伝的寄与度を決定するために使用され得る。この個体群は、認められた品種のサブセット(例えば、特定のブリーダーが起源であるダルマシアンの一群)または品種として認められていないか未だ認められていないイヌ科動物の一群である。同様に、本発明の方法は、イエイヌではないイヌ科動物個体群からの遺伝的寄与度を決定するために使用され得る。
【0026】
本発明の方法の第1工程は、各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程を包含する。用語「マーカー」は、試験イヌ科動物ゲノムへのこれらのイヌ科動物個体群の遺伝的寄与度を推定するために有用である、本発明の方法において使用されたイヌ科動物個体群にわたって十分に情報価値のある、任意の多型のゲノム座(genomic locus)をいう。ゲノム座は、それが少なくとも2つの対立遺伝子を有する場合、多型である。用語「対立遺伝子」とは、ゲノム座の他の形態からその核酸配列によって区別され得るゲノム座の特定の形態をいう。従って、ゲノム座の異なる対立遺伝子は、その座位(locus)で代替的な核酸配列を表す。任意の個体のイヌ科動物ゲノムにおいて、各マーカーについて2つの対立遺伝子が存在する。両方の対立遺伝子が同じである場合、ゲノムは、そのマーカーにホモ接合性である。逆に、その2つの対立遺伝子が異なる場合、ゲノムは、そのマーカーにヘテロ接合性である。
【0027】
個体群特異的対立遺伝子は、あるイヌ科動物個体群においていくらかの頻度で存在する対立遺伝子であるが、比較イヌ科動物個体群からサンプル抽出されたイヌ科動物中で観察されていない(しかし、それらは、実に低い頻度で存在し得る)。個体群特異的対立遺伝子は、特定の個体群に個体を割り当てるために使用され得る。従って、個体群間の対立遺伝子頻度における差異は、遺伝的寄与度を決定するために使用され得る。
【0028】
「マーカーセット」とは、試験イヌ科動物ゲノムへの本発明の方法に使用されたイヌ科動物個体群の遺伝的寄与度を決定するために十分なマーカーの最小数をいう。この必要とされるマーカーの最小数は、以下にさらに記載されるように、使用されている特定のイヌ科動物個体群についてのマーカーの情報性(informativeness)に依存する。マーカーセットは、少なくとも約5個のマーカー、少なくとも約10個のマーカー、少なくとも約50個のマーカー、または約100個より多いマーカーを含み得る。
【0029】
本発明に従って使用され得る代表的なマーカーとしては、マイクロサテライトマーカー、ミトコンドリアマーカー、制限酵素断片長多型、および一塩基多型(SNP)が挙げられる。有用なイヌ科動物のマイクロサテライトマーカーとしては、ジヌクレオチドリピート(例えば、(CA)n)、トリヌクレオチドリピート、およびテトラヌクレオチドリピート(例えば、(GAAA)n)が挙げられるが、これらに限定されない(Franciscoら、(1996)Mamm.Genome 7:359−62;Ostranderら、(1993)Genomics 16:207−13)。本発明の方法における使用のための例示的なマーカーとしては、表1に示すマイクロサテライトマーカー、表2に示すSNPマーカー、およびGuyonら(2003)Proc.Natl.Acad.Sci U.S.A.100(9):5296−5301に記載されるマーカーが挙げられる。本発明の方法において使用されるマーカーセットは、表1のマイクロサテライトマーカーからの少なくとも約5個のマーカー、および/または、表2のSNPマーカーからの少なくとも約5個のマーカーを含み得る。いくつかの実施形態において、マーカーセットは、372c5t−82、372e13t−57、372m6t−88、372m23t−76、373a15t−112、373e1t−50、373elt−130、373g19t−246、373i8s−224、373k8s−181、372c5s−168、372C15S−196、372e15s−71、および373a21t−93からなる群より選択される。いくつかの実施形態において、約1500個よりも少ない数のSNPマーカーを含む1セットのマーカーは、試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定するために使用される。いくつかの実施形態において、約200個よりも少ない数のSNPマーカーを含む1セットのマーカーは、試験イヌ科動物ゲノムへの少なくとも約87種のイヌ科動物個体群の寄与度を決定するために使用される。
【0030】
本発明の方法に従って、各マーカーの片方または両方の対立遺伝子の同一性が、得られ得る。いくつかの実施形態において、試験イヌ科動物における片方または両方の対立遺伝子の同一性は、当該分野において標準的な方法を使用して実験的に決定され得る。例えば、ゲノムマーカーの片方または両方の対立遺伝子の同一性は、当該分野で公知の任意の遺伝子型決定方法を使用して決定され得る。例示的な遺伝子型決定方法としては、ハイブリダイゼーション法、ポリメラーゼ連鎖反応法(PCR)、サイズ分画法、DNA塩基配列決定法、DNAマイクロアレイ法、ビーズの高密度光ファイバーアレイ法(high density fiber−optic arrays of beads)(例えば、Jianbingら、(2003)Chin.Sci.Bull.48(18):1903−5を参照のこと)、プライマー伸長法、質量分析法(例えば、Jurinkeら、(2002)Meth.Mol.Biol.187:179−92を参照のこと)、およびホールゲノムサンプリング分析法(whole−genome sampling analysis)(例えば、Kennedyら、(2003)Nat.Biotechnol.21(10):1233−7を参照のこと)の使用が挙げられるが、これらに限定されない。試験イヌ科動物におけるマーカーの対立遺伝子の同一性はまた、既に決定されており、かつ例えば刊行された文献のような供給源から利用可能であり得る。
【0031】
いくつかの実施形態において、試験イヌ科動物のゲノムDNAは、マーカーに特異的なプライマーを使用して増幅され得、その後にこの増幅産物のサイズ分析または配列決定が続く。イヌ科動物ゲノム中のマーカーの片方または両方の対立遺伝子の同一性を得るための例示的な方法は、実施例1に記載される。いくつかの実施形態において、マイクロサテライトマーカーを含むゲノムDNAを増幅するために使用されたプライマーは、配列番号1〜200からなる群より選択されるが、他のプライマーおよび他のマイクロサテライトマーカーも使用され得る。いくつかの実施形態において、SNPマーカーを含むゲノムDNAを増幅するために使用されたプライマーは、配列番号244〜327からなる群より選択されるが、他のプライマーおよび他のSNPマーカーも使用され得る。422匹のイヌ科動物(85品種を表す414匹のイヌと8匹のオオカミとを含む)における68個〜100個のマイクロサテライトの対立遺伝子の同一性は、(同封して提出したコンパクトディスク中の)表3に示される。189種のイヌ科動物(67品種を表す186匹のイヌと2匹のオオカミと1匹のコヨーテとを含む)における100個のSNPマーカーの対立遺伝子の同一性は、(同封して提出したコンパクトディスク中の)表4に示される。
【0032】
本発明の方法の第1工程において使用されるマーカーセット中に含まれるマーカーの最小数は、使用される特定のイヌ科動物個体群のついてのマーカーの情報性に依存する。このマーカーの情報は、本発明の方法において使用されるイヌ科動物個体群内かつこれら個体群間で異なる対立遺伝子数の関数、これらの対立遺伝子の頻度、および座位における変異率の割合である。ゲノム座の多型の程度(degree of polymorphism)は、多型情報内容(PIC)の推定によって評価され得る。この多型情報量は、対立遺伝子数およびそれらの分配頻度の関数である。本発明の方法における使用に適切なマイクロサテライトマーカーについての例示的なPIC値は、表1に示される。本発明の方法における使用に適切なマーカーは、実施例1に示されるように、約0.65%の平均PIC値を有し得る。
【0033】
異なるイヌ科動物個体群中のマーカーの対立遺伝子数、およびイヌ科動物個体群内およびこれら個体群間での対立遺伝子頻度を決定する方法は、実施例1に記載される。例えば、マーカーごとの対立遺伝子の平均数、(ハーディワインバーグ平衡の推定に基づいて)予測されたヘテロ接合性、観察されたヘテロ接合性、および94匹のイヌ科動物(18品種を表す90匹のイヌと、4匹のオオカミとを含む)における95個のマイクロサテライトマーカー間での推定された同系交配係数は、実施例1に記載される。品種の境界の存在は、同種由来のイヌが異種由来のイヌよりも遺伝学的により類似しているはずであることを予測する。この予測を試験するために、品種の構成員(membership)に起因し得る個々のイヌ間における遺伝的多様性の割合が、推定された。マイクロサテライトデータ(68品種を表す328匹のイヌにおける96個のマーカーを含む)についての分子変動の分析は、実施例1に記載されるように、遺伝分散の合計の27%よりも多くの割合を占める品種間の分散を示した。同様に、SNPマーカーデータ(60品種を表す120匹のイヌにおける75個のSNPを含む)から計算された品種間の遺伝学的距離は、実施例1に記載されるように、FST=0.36であった。これらの観察は、少数の犬種を分析したこれまでの報告(Koskinen(2003)Anim.Genet.34:297;Irionら、(2003)J.Hered.94:81)と一致し、品種の境界が品種間で顕著な遺伝的隔離をもたらしていたということを予測し、そしてヒト個体群の間で見出されたより低い遺伝学的差異(代表的には、5%〜10%の範囲内)とは著しく異なる(Rosenbergら、(2002)Science 298:2381−5;Cavelli−Sforzaら、(1994)The History and Geography of Human Genes、Princeton University Press,Princeton)。イヌにおける品種間での変動は、家畜個体群について報告された範囲よりも高い(MacHughら、(1998)Anim.Genet.29:333;Lavalら、(2000)Gen.Sel.Evol.32:187)。犬種間での顕著な遺伝学的差異は、品種の構成員が、個々のイヌ科動物についての遺伝子型情報から決定され得ることを示す。
【0034】
マーカーの情報性に関するデータセットにおけるマーカーの別個の対立遺伝子の数の影響は、実施例2に示される。例えば、19種のイヌ科動物個体群および95個のマイクロサテライトマーカーの分析において、イヌ科動物の86%が、10個よりも多い別個の対立遺伝子をそれぞれ有した5個のマーカーを使用して、イヌ科動物の品種に正確に割当てられ、そしてイヌ科動物の95%が、10個よりも多い別個の対立遺伝子をそれぞれ有した10個のマーカーを使用して、イヌ科動物の品種に正確に割当てられた。1個〜3個の別個の対立遺伝子を有するマーカーについて、イヌ科動物の46%が、5個のマーカーを使用してイヌ科動物の品種に正確に割当てられ、そしてイヌ科動物の62%が、10個以上のマーカーを使用して正確に割当てられた。
【0035】
イヌ科動物個体群ごとに4種もしくは5種のイヌ科動物についての95個のマーカーに関する遺伝子型情報を使用して、19種のイヌ科動物個体群間で識別する能力に対する使用されたマーカー数の影響は、実施例2に示される。例えば、個体を正しいイヌ科動物個体群に100%首尾よく割当てるために必要とされるマーカーの最小数は、イヌ科動物個体群に依存して2個(ペキニーズ)と52個(アメリカンへアレステリア)との間に及んだ。100%の精度を有する選択されたイヌ科動物個体群を有する、19種のイヌ科動物個体群にわたって試験された94匹のすべての個体にうちの少なくとも90%をうまく割当てるために必要とされるマイクロサテライトマーカーの最小数は、8個(ペキニーズについて)と95個(プレサカナリオ、チワワ、およびアメリカンへアレステリアについて)との間に及んだ。
【0036】
本発明の第一局面の方法の第2工程は、試験イヌ科動物ゲノム中の対立遺伝子とイヌ科動物プロファイルを含むデータベースとを比較することによって、試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する工程を包含する。第2工程において、各イヌ科動物個体群プロファイルは、イヌ科動物個体群におけるマーカーセット中のマーカーの対立遺伝子に関する遺伝子型情報を含む。本明細書中において使用される「イヌ科動物個体群プロファイル」とは、イヌ科動物個体群におけるマーカーセットに関する遺伝子型情報の収集物をいう。従って、イヌ科動物個体群プロファイルは、イヌ科動物個体群におけるマーカーセット中のほとんどまたは全てのマーカーのほとんどまたは全ての対立遺伝子に関する遺伝子型情報を含み得る。例えば、イヌ科動物個体群プロファイルは、イヌ科動物個体群におけるマーカーセット中の各マーカーの各対立遺伝子に関する遺伝子型情報を含み得る。イヌ科動物個体群プロファイル中の遺伝子型情報は、例えば、そのイヌ科動物個体群の一員である1種以上のイヌ科動物におけるマーカーセット中のほとんどもしくは全てのマーカーの片方もしくは両方の対立遺伝子の同一性、および/または、そのイヌ科動物個体群におけるマーカーセット中のほとんどもしくは全てのマーカーの少なくとも1つの対立遺伝子について推定された対立遺伝子頻度のような情報を含み得る。「対立遺伝子頻度」とは、個体群中の対立遺伝子の発生率をいう。対立遺伝子頻度は、代表的には直接計数(direct counting)によって推定される。一般に、イヌ科動物個体群における対立遺伝子頻度は、そのイヌ科動物個体群の少なくとも約5種の一員中の各マーカーセットについての片方または両方の対立遺伝子の同一性を得ることによって推定される。「イヌ科動物個体群プロファイルデータベース」とは、本発明の例示的な方法において使用される全てのイヌ科動物個体群についてのイヌ科動物個体群プロファイルの収集物をいう。いくつかの実施形態において、イヌ科動物個体群プロファイルデータベースは、約5個と約500個の間のイヌ科動物プロファイル(例えば、約20個のイヌ科動物プロファイル、約50個のイヌ科動物プロファイル、または約100個のイヌ科動物プロファイル)を含む。
【0037】
試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する工程は、イヌ科ゲノムを特定のイヌ科動物個体群へ割当てる工程、および1種以上のイヌ科動物個体群に由来したイヌ科動物ゲノムの比を決定する工程の両方を包含する。この方法のいくつかの実施形態において、ベイジアンモデルベースのクラスタリングアプローチが使用される。個体を個体群に割り当てるために使用されるクラスタリング法の2つの広範なクラスが存在する(Pritchardら(2000)Genetics 155:945−59)。距離に基づく方法は、個体のすべてのペアの間の距離を提供するためにペアワイズ距離行列を計算する。各クラスターからの知見を考慮することによって進められるモデルベースの方法は、いくつかのパラメーターモデルからランダムに抽出される;次いで、各クラスターに対応するパラメーターについての推論が、標準的な統計学的方法を使用して、各個体のクラスターの構成員についての推論と一緒に行われる。任意の標準的な統計学的方法は、本発明の方法(最大尤度法、ブートストラップ手法、ベイジアン法、および遺伝子型データを分析するために使用され得る任意の他の統計学的手法を含む)において使用され得る。これらの統計学的方法は、当該分野において周知である。集団遺伝学研究のための多くのソフトウェアプログラムが開発されており、本発明の方法において使用され得る。このプログラムとしては、TFPGA、Arlequin、GDA、GENEPOP、GeneStrut、POPGENE(Labate(2000)Crop.Sci.40:1521−1528)、およびstructure(Pritchardら、(2000)Genetics 155:945−59)が挙げられるが、これらに限定されない。
【0038】
例示的なベイジアンモデルベースのクラスタリングアプローチは、遺伝子型クラスタリングプログラムであるstructure(Pritchardら、(2000)Genetics 155:945−59)によって提供される、このプログラムは、種内で個体群を規定するために有用であると証明されている(Rosenburgら、(2001)Genetics 159:699−713;Rosenburgら、(2002)Science 298:2381−5;Falushら、(2003)Genetics 164(4):1567−87)。structureによって使用されるクラスタリング方法は、個体群中で個体または関連する個体のセットを正確に配置するために遺伝子型または遺伝学的起源のいずれに関する以前の情報も必要としない。
【0039】
任意のマルチローカス(multi−locus)遺伝子型分析に有用なアルゴリズム(例えば従来の割り当てアルゴリズム)は、本発明の方法において使用され得る。適切なアルゴリズムとしては、RannalaおよびMountain、(1997)Proc.Natl.Acad.Sci.U.S.A.94:9197−9201と、Cornuetら、(1999)Genetics 153:1989−2000とに記載されるアルゴリズム、ならびにそれらの変形物が挙げられる。マルチプルローカス遺伝子型分析について利用可能な例示的なプログラムとしては、Doh(www2.biology.ualberta.ca/jbrzusto/Doh.phpにて利用可能)およびGeneClass(www.montpellier.inra.fr/URLB/geneclass/genecass.htmにて利用可能)が挙げられる。
【0040】
いくつかの実施形態において、本発明の方法は、特定のイヌ科動物個体群が試験イヌ科動物ゲノムへ寄与した条件付き確率を決定する工程を包含し、この条件付き確率は、試験イヌ科動物ゲノム中の対立遺伝子が特定のイヌ科動物個体群中で生じる条件付き確率を、試験イヌ科動物ゲノム中の対立遺伝子がデータベース内の、各イヌ科動物個体群中で生じる条件付き確率の合計で割って決定することにより決定される。
【0041】
本発明の方法のいくつかの実施形態は、試験イヌ科動物ゲノム中の対立遺伝子と遺伝学的に関連する2種以上のイヌ科動物個体群のプロファイルを含むデータベースとを比較することによって、試験イヌ科動物ゲノムへの遺伝学的に関連する2種以上のイヌ科動物個体群の寄与度の間を識別する工程を包含する。この遺伝学的に関連する2種以上のイヌ科動物個体群は、ベルジアンシープドッグおよびベルジアンタービュレン;コリーおよびシェトランドシープドッグ;ウィペットおよびグレイハウンド;シベリアンハスキーおよびアラスカンマラミュート;マスチフおよびブルマスチフ;グレータースイスマウンテンドックおよびバーニーズマウンテンドッグ;ウェストハイランドホワイトテリアおよびケアーンテリア;またはラサアプソ、シーズー、およびペキニーズを含み得る。
【0042】
94匹のイヌ科動物(18品種を表す90匹のイヌ科動物と4匹のオオカミとを含む)からの95個のマイクロサテライトマーカーについての遺伝子型情報に割り当てアルゴリズムを使用することにより、本発明の方法が、実施例2に記載されるように、99%の精度で各個々のイヌ科動物をその品種へと割り当てるために使用されている。実施例3に記載されるように、同じ遺伝子型情報に使用したクラスタリングアルゴリズムは、20種のイヌ科動物個体群を予測し、そして各イヌ科動物を99%の精度で1種の個体群へと割り当てた。
【0043】
72品種を表す341匹のイヌ科動物からの68個のマイクロサテライトマーカーについての遺伝子型情報に割り当てアルゴリズムを使用することにより、本発明の方法が、実施例2に記載されるように、イヌ科動物の96%を正しい品種へと割り当てるために使用されている。85品種を表す414匹のイヌ科個体群からの96個のマイクロサテライトマーカーについての遺伝子型情報に割り当てアルゴリズムを使用することにより、本発明の方法が、実施例4に記載されるように、イヌ科動物の99%を正しい品種へと割り当てるために使用されている。同様の結果が、クラスタリングアルゴリズムを使用して得られた。67品種を表す189匹のイヌ科動物からの100個のSNPマーカーについての遺伝子型情報に割り当てアルゴリズムを使用することにより、本発明の方法が、実施例6に記載されるように、99%よりも高い確率でイヌ科動物の80%を正しい品種へと割り当てるために使用されている。
【0044】
本発明の方法はまた、雑種イヌ科動物へのイヌ科動物個体群の寄与度を決定するために有用である。交配された(admixed)個体は、イヌ科動物個体群のおよそ50%を表す。個体の交配された(admixed)状態を検出するモデルは、群を以下の2つのクラスに分けて考えられ得る:先祖個体群の考えられる交配種(mixture)の各々について独特の対立遺伝子の組み合わせセットを必要とするモデル(NasonおよびEllstrand(1993)J.Hered.84:1−12;Epifanio&Philipp(1997)J.Hered.88:62−5)と、先祖個体群は特有の対立遺伝子を説明する組み合わせを含む必要がないがその代わりに個体間の対立遺伝子頻度における差異に確率論的に基づいて交配された(admixed)状態へと個体を割り当てる、ベイジアン法のモデル(Coranderら(2003)Genetics 163(1):367−74;AndersonおよびThompson(2002)Genetics 160:1217−29;Pritchardら(2000)Genetics 155:945−59;RannalaおよびMountain(1997)Proc.Natl.Acad.Sci.U.S.A.94:9197−9201)。後者のモデルセットが、各々の個体群/世代の組み合わせについてのベイジアン事後確率割り当てベクトルを考慮にいれ、それによって、その割り当てベクトルに組み入れられる不確定度分析を考慮に入れる際、それら後者のモデルセットは、ほとんどの個体群およびデータセット関して、より情報価値があるが、複数種の先祖個体群からの個体の正確な近い世代で交配された種(recent admixture)の割り当てのための既存のモデルは、それらモデルが開発されてきた際の範囲内(2世代の予測のみ、および数種の先祖個体群のみを考慮する程度まで)に制限される。例えば、AndersonおよびThompson(2002)の方法は、非連鎖マイクロサテライトデータを有する、2世代の2種の個体群のために開発されている。ナイーブベイジアン分類モデルは、先祖の部分個体群への交配種(mixture)への個体の確率論的割り当てのために、連鎖マイクロサテライト座位および非連鎖マイクロサテライト座位の情報、高次元の先祖個体群、ならびに高度に秩序だった(higher−ordered)世代系図を組み入れ、このモデルは、実施例7に記載される。このモデルは、実施例7に記載されるように、既存モデルの世代、部分個体群(subpopulation)、および連鎖の制限に対して同時に取り組み、そして2世代モデルおよび3世代モデルは、正確な交配種(admixture)の検出および割り当てのために実施されている。
【0045】
18品種の81匹のイヌ科動物と4匹のオオカミから構成される85匹のイヌ科動物からの95個のマーカーについての遺伝子型情報のインシリコでの交配(mix)に、クラスタリングアルゴリズムを使用することにより、本発明の方法は、実施例5に記載されるように、100%の精度で両親レベルにおけるインシリコでの交配を同定するために使用されている。本発明の方法また、実施例5に記載されるように、祖父母レベルおけるインシリコでの交配の検出においても非常に正確であり、そして曽祖父母レベルにおけるインシリコでの交配を検出においてもかなり正確であった。従って、本発明の方法は、純血種のイヌ(イヌ由来の1/2オオカミ交配種および1/4オオカミ交配種も同様)から、両親レベルおよび祖父母レベルにおける交配を識別するために使用され得、そして、雑種のイヌのゲノムにおける品種の寄与度を同定し得る。
【0046】
88品種の429匹のイヌ科動物からの96個のマーカーについての遺伝子型情報のインシリコでの交配にベイジアン分類モデルを使用することにより、本発明の方法は、実施例7に記載されるように、98%より高いF1交配種(mix)および94%より高いF2交配種の正確な割り当てのために使用されている。この160匹の既知の雑種イヌ科動物からの72個のマーカーについての遺伝子型情報に関するモデルを使用して、本発明の方法は、実施例7に記載されるように、96%より高いF1交配種および91%より高いF2交配種を正確に割り当てるために使用されている。
【0047】
本発明の方法は、試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する文書を提供する工程をさらに包含し得る。用語「文書」は、図表、証明書、カード、または任意の他の種類の書類をいう。文書は、試験イヌ科動物ゲノムへの1種以上のイヌ科動物個体群の寄与度を、数値形式または図画形式で表示し得る。例えば、文書としては、1種以上のイヌ科動物個体群の写真もしくは他の描写、図、または表示が挙げられ得る。文書はまた、決定された寄与度についての信頼値(例えば、80%、85%、90%、95%、または99%の信頼度)を提供し得る。いくつかの実施形態において、文書は、試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度の証明書を提供する。
【0048】
いくつかの実施形態において、文書は、試験イヌ科動物ゲノムまたは試験イヌ科動物へ寄与した1種以上のイヌ科動物個体群に関する情報をさらに提供する。試験イヌ科動物ゲノムへ寄与したイヌ科動物個体群に関する情報としては、イヌ科動物個体群の特徴および起源に関連する情報、または試験イヌ科動物の所有者にとって有用である任意の他の種類の情報が挙げられ得る。いくつかの実施形態において、情報は、健康に関する情報を含む。多くのイヌ科動物個体群は、特定の疾患または状態になりやすい素因を有する。例えば、アフガンハウンドは、緑内障、肝炎、および甲状腺機能低下症に罹りやすく;バセニーは、大腸菌腸炎およびピルビン酸キナーゼ欠乏症に罹りやすく;ビーグルは、膀胱癌および難聴に罹りやすく;バーニーズマウンテンドッグは、小脳変性症に罹りやすく;ボーダーテリアは、乏突起細胞腫に罹りやすく;そしてラブラドールレトリバーは、食物アレルギーに罹りやすい(Dr.Bob‘s All Creatures Site,Breed Predisposition to Disease and Congenital Conditions,http://www.petdoc.ws/BreedPre.htm; Pattersonら、(1998)J.Am.Vet.Med.Assoc.193:1131を参照のこと)。イヌにおいて発見された遺伝疾患に関して、46%は、1種または数種において、支配的または排他的に生じると考えられる(Pattersonら(1998)J.Am.Vet.Med.Assoc.193:1131)。従って、試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度に関する情報は、試験された個々の動物について健康上のリスクを前向きに検討する目的のために、雑種のイヌ科動物の所有者または世話人(専門家および非専門家の両方)にとって、特に有益である。例えば、ニューファンドランドとバーニーズマウンテンドッグとの交配種(mixture)であると見出される雑種のイヌは、一般的なイヌ個体群においてはまれな頻度で生じるがこれら特定の品種においてはかなりの頻度で生じる遺伝疾患について、活発にモニターされるべきである。従って、このタイプの雑種個体は、悪性組織球増殖症についてのスクリーニングから恩恵を受け(disease heritability of .298 in Bernese Mountain dogs,Padgettら、1995 J.Small Anim.Pract.36(3):93−8)、加えて、I型シスチン尿症の遺伝的スクリーニングからも恩恵を受ける(nonsense mutation isolated in Newfoundlands at exon 2 of SLC3A1 gene,Henthornら、(2000)Hum.Genet.107(4):295−303)。
【0049】
健康に関する情報としてはまた、可能性のある処置、特別食または製品、診断情報、および保険情報が挙げられ得る。試験イヌ科動物のゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する例示的な文書は、図1に示される。
【0050】
いくつかの実施形態において、本発明は、1種以上のイヌ科動物個体群を規定する方法を提供する。この方法は、(a)各イヌ科動物ゲノムセットについて、各マーカーセットのついての片方または両方の対立遺伝子の同一性を得る工程;および(b)イヌ科動物ゲノムセットの1つ以上の一員が、統計的モデリングを用いて各マーカーについて1セットの対立遺伝子頻度によって特徴付けられる別個のイヌ科動物個体群を規定する可能性を決定することによって、1種以上のイヌ科動物個体群を規定する工程を包含する。1種以上のイヌ科動物個体群を規定するための本発明の例示的な方法は、実施例3および実施例4に記載される。
【0051】
別の局面において、本発明は、各マーカーセットについてイヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を決定するための核酸配列を含む基材を、提供する。基材は、マーカーの対立遺伝子の同一性を決定するために適切な任意の形態であり得る。例えば、基材は、マイクロアレイまたはビーズの集合体の形態であり得る。
【0052】
さらなる局面において、本発明は、イヌ科動物個体群の区別に使用するために、媒体上に保存されたデータ構造を含むコンピュータ読み取り可能な媒体を提供し、このデータ構造は:マーカーの名称(例えば、SNPマーカー)またはこのマーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および、そのマーカーについての遺伝子型情報(例えば、イヌ科動物ゲノムにおけるマーカーの片方または両方の対立遺伝子の同一性、あるいはイヌ科動物個体群におけるマーカーの対立遺伝子頻度の推定値)を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、そのマーカーフィールドのインスタンス化およびこの遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールドを備える。
【0053】
「コンピュータに読み取り可能な媒体」とは、コンピュータによってアクセスされ得る任意の利用可能な媒体をいい、そして揮発性媒体および非揮発性媒体の両方の、取り外し可能な媒体および取り外しができない媒体が挙げられる。一例であり限定はしないが、コンピュータに読み取り可能な媒体は、コンピュータ記憶媒体および通信媒体を備え得る。コンピュータ記憶媒体としては、情報(例えば、コンピュータに読み取り可能な命令、データ構造、プログラムモジュール、または他のデータ)を保存するための任意の方法または技術で実施される、揮発性および非揮発性の両方の、取り外し可能な媒体および取り外しができない媒体が挙げられる。コンピュータ記憶媒体としては、RAM、ROM、EEPROM、フラッシュメモリまたは他のメモリー技術、CD−ROM、デジタル多用途ディスク(DVD)または他の光ディスク記憶装置、磁気カセット、磁気テープ、磁気ディスク記憶装置または他の磁気記憶デバイス、あるいは任意の他のコンピュータ記憶媒体が挙げられるが、それらに限定されない。通信媒体は、代表的には、コンピュータに読み取り可能な命令、データ構造、プログラムモジュール、または変調されたデータ信号(例えば、搬送波または、任意の情報伝達媒体を含む他の転送機構)で具体化する。用語「変調されたデータ信号」は、その信号の特性セットを1セット以上有する信号か、またはその信号のコード化情報のような方式で変更された信号を意味する。一例であり限定されないが、通信媒体としては、有線媒体(例えば、有線ネットワーク通信または直接配線された(direct−wired)通信)および無線媒体(例えば、音響媒体、RF赤外線媒体、および他の無線媒体)が挙げられる。上述の任意の組み合わせがまた、コンピュータに読み取り可能な媒体の範囲内に含まれるはずである。
【0054】
「データ構造」とは、データの概念的配置をいい、そして代表的には、列および行によって特徴付けられる。そのデータは、列と行の交差(row−column intersection)によって形成された各セルを占めるかまたは潜在的に占めている。本発明のコンピュータに読み取り可能な媒体中のデータ構造は、上述のように、マーカーフィールドおよび遺伝子型情報フィールドを含む。マーカーフィールドおよび遺伝子型情報フィールドのインスタンス化は、1つのレコードを提供し、そして1セットのレコードは、1つのイヌ科動物個体群プロファイルを提供する。従って、データ構造は、イヌ科動物個体群プロファイルのデータベースを作製するために使用され得る。
【0055】
いくつかの実施形態において、コンピュータに読み取り可能な媒体は、以下をこの媒体上に保存されたものを有する基板を備える:
(a)イヌ科動物個体群の区別に使用するためのデータ構造であって、このデータ構造は;
(i)マーカーの名称またはこのマーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(ii)そのマーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、このマーカーフィールドのインスタンス化およびこの頻度フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、データ構造;ならびに
(b)イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を実施するための、コンピュータが実行可能な命令であって、以下の工程:
(i)各マーカーセットについて試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(ii)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物プロファイルを含むデータベースとを比較することによって決定する工程であって、イヌ科動物個体群プロファイルの各々は、そのイヌ科動物個体群における上記マーカーセットについての遺伝子型情報を含む、工程
を包含する、コンピュータが実行可能な命令。
【0056】
以下の実施例は、本発明を実施するための現在考えられている最良の形態を単に例示するが、本発明を限定するために構成されるべきではない。
【実施例】
【0057】
(実施例1)
この実施例は、1セットのマーカーについての片方または両方の対立遺伝子の同一性を得るため、かつイヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定するのに適切なマーカーを選択するための、本発明の代表的な方法を説明する。
【0058】
(A.方法)
(1.サンプル収集およびDNA抽出)
8ヶ国(中国、オマーン、イタリア、イラン、米国(アラスカ)、カナダ(ケベック)、スウェーデン、メキシコ)からの、103品種の513匹のアメリカンケンネルクラブに登録されたイヌ、および8匹のハイイロオオカミからのイヌ科動物DNAサンプルを、ドッグショーおよびドッグクラブの特別イベントで、ボランティアから口内(頬)塗抹標本および/または血液サンプルを収集することで得た。同様に、これらは、郵送による提供によっても得られた。アメリカンケンネルクラブ登録番号および詳細な系統情報が、全てのイヌについて要求される。なぜなら、この実施例への参加は、共通の祖父母を有さない無関係のイヌには制限されたからである。系統情報がまた、サンプル抽出された個体の84%について収集された。多くの場合、5世代の系図が得られ、イヌは時折、曽祖父母レベルまたそれより高いレベルで、重複して現れるが、その一方で、完全連鎖検査は、同犬種間での高い無関連度(high degree of unrelatedness)を示している。系図が利用可能でなかった場合、これら個体について、無関連性(unrelatedness)を血統クラブの代表犬(representative)で検証した。個体イヌ科動物の各々に、イヌ科動物識別番号を与えた。品種および他のイヌ科動物個体群について使用される略称は、表5に示される。加えて、20種のAKC品種からの交配コンポーネント(admixture component)を含む160匹の雑種イヌ科動物からのDNAサンプルを、口内塗抹標本を収集することによって得た。
【0059】
口内塗抹標本を、アメリカンケンネルクラブ(AKC)ウェブサイト(http://www.akc.org/)より提案されるものと類似する様式で、細胞学的ブラシ(Medical Packaging Corp.,Camarillo,CA)を使用して収集した。DNAを、製造元(Qiagen,Valencia,CA)のプロトコルに従い、QiaAmp血液キットを使用して口内塗抹標本から抽出した。血液からのDNA抽出は、以前に記載されたように行った(Comstockら、(2002)Mol.Ecol.11:2489−98)。
【0060】
(2.マイクロサテライトマーカーの分析)
100個のジヌクレオチドマイクロサテライトマーカーを、イヌの3300個のマーカー地図上に現在位置決めされている1596個のマイクロサテライトから選択した(Guyonら、(2003)Proc.Natl.Acad.Sci U.S.A.100(9):5296−5301)(表1)。マーカーを情報性(informativeness)に基づいて選択し、PIC値として計算し、そして、38種すべての常染色体にわたって分布させた。選択されたマーカーは、0.65%(36%〜86%の範囲)の平均PIC値、および29.5Mb(21.5Mb〜50.9Mbの範囲)の平均間隔を有した。テトラヌクテオチドマイクロサテライトよりもジヌクレオチドマイクロサテライトを、品種同定を妨害し得る、観察された偽の変異(spurious mutation)の数を減らすように選択した。
【0061】
DNAサンプルを96ウェルプレート上に並べた。ポジティブコントロールを、一貫した対立遺伝子のビニングを保証するために各プレート上に含めた。PCRを、1ngのゲノムDNAおよび最終濃度の以下の試薬を含有する10μlの反応液中で行った。以下の試薬とは、16mMの硫酸アンモニウム、67mMのTris−HCl pH8.8、2.0mMのMgCl2、0.1mMのdNTP、300nMの順方向プライマー(配列番号1〜100)、逆方向プライマー(配列番号101〜200)、および色素標識したM13プライマー(PE Applied Biosystems,Foster City,CA USA)である。順方向プライマーを、5’末端上に19塩基のM13順方向(−29)配列(5’−CACGACGTTGTAAAACGAC−3’)(配列番号201)を含むように再設計した。6FAMTM、VICTM、NEDTM、またはPETTM(ABI,Foster City,CA)色素のいずれかで標識された0.25pmolのM13プライマー(配列番号201)を各反応液に加えることで、サンプルを標識した。PCRのインキュベーションを、標準的なプロトコル(例えば、Loweら、(2003)Genomics 82:86−95;http://www.fhcrc.org/science/dog_genome/dog.htmlを参照のこと)に従って行った。使用されたアニ−リング温度は、表1に示される。異なる色素で標識された4つのサンプルを、1枚の96ウェルプレート内で3μlの各反応混合液を混ぜ合わせることにより、PCRの終了後に、多重化した。サンプルを、16pmolのGeneScanTM−500LIZTMサイズ標準(ABI,Foster City,CA)を含有する2容量のHi−DiTMホルムアミド中で、製造元のプロトコルに従って変性させた。全てのサンプルを、対立遺伝子を分離するために、ABI 3730 DNA AnalyzerTM(PE applied Biosystems)キャピラリー電気泳動装置上にロードした。遺伝子型を、GeneMapperTMv3.0ソフトウェア(ABI,Foster City,CA)を使用してコールした。全てのコールを、手動で確認し、そして次に続く各実行(run)をビン(bin)外にある新たな対立遺伝子の出現についてスキャンした。4個のマーカーを、一貫して増幅できなかったので切り捨てた。
【0062】
(3.SNPの発見および遺伝子型決定)
50種のイヌ動物の細菌性人工染色体(BAC)を、イヌ科動物の放射線ハイブリッド地図(Guyonら(2003)Proc.Natl.Acad.Sci U.S.A.100(9):5296−5301)からランダムに選択した。Primer3プログラム(http://www.genome.wi.mit.edu/sci−bin/primer/primer3_www.cgiにて利用可能)を、BACの各末端配列から、プライマーを設計するために使用した。結果として平均334塩基対の単位複製配列が得られた。プライマーを、67種のイエイヌ種の189匹のイヌと、コヨーテとハイイロオオカミの19867塩基対の非連続ゲノム配列を増幅するために使用した。結果として得られたPCR産物を、標準的なABI色素ターミネ−ター化学を用いてABI3700キャピラリーシーケンサー(ABI,Foster City,CA)上で、標準的な方法を使用して配列決定し、そして再度、配列決定した。読み取った全ての配列を、Phred、Phrap、およびConsed(EwingおよびGreen(1998)Genome Res.8:186−94;Ewingら(1998)Genome Res.8:175−85;www.genome.washington.eduにて利用可能)を使用して整列させ、表示した。コンピュータプログラムPolyphredを、読み取った配列内およびその配列間で、多型領域(SNPおよび挿入/欠失の両方)を同定するために使用した(Nickersonら(1997)Nucl.Acids Res.25:2745−51、droog.mbt.washington.eduにて利用可能)。全ての対立遺伝子のコールを、手動で確認し、そのトレースを目視検査によって確認した。
【0063】
(4.統計分析)
分子分散分析(AMOVA)を、ハーディワインバーグ平衡の仮定の下で、GDA(LewisおよびZaykin(2001)Genetic Data Analysis:Computer Program for the Analysis of Allelic Data,Version 1.0(d16c)、http://lewis.eeb.uconn.edu/lewishome/softare.htmlにて利用可能)を使用して行った。分析において同系交配が考慮された場合、品種間の遺伝的変動の比について、同様の結果が得られた。
【0064】
各品種についての予測したヘテロ接合性を、Tajima不変推定量(Tajima(1989)Genetics 123:585−95)を使用して、対立遺伝子頻度から計算した。
【0065】
(B.結果)
(1.ジヌクレオチドマイクロサテライトの情報性)
422匹のイヌ科動物(85品種の414匹のイヌと8匹のオオカミとを含む)における68個〜100個のマイクロサテライトの対立遺伝子(増幅した領域の長さ)の同一性は、(本明細書とともに提出されたコンパクトディスク中の)表3に示される。148個の対立遺伝子が、特定のイヌ科動物個体群に特有であることが見出されている:1個は各々、ACKR、AUST、BORD、BOX、BULD、DACH、GOLD、GSHP、GSMD、IBIZ、KEES、NELK、PEKE、POM、ROTT、SFXT、TERVおよびWHIPに特有であり、2個は各々、BEAG、CAIR、HUSK、IRSE、MAST、OES、SCHP、SCWT、SPOOおよびSSHPに特有であり、3個は各々、AMAL、BMD、KOMO、NEWF、STBDおよびWSSPに特有であり、4個は各々、KUVZ、PNTRおよびPRESに特有であり、5個は各々、それぞれBSIJおよびSHARに特有であり、6個は、AKITに特有であり、そして、64個の対立遺伝子は、WOLFに特有である。
【0066】
6つの異なるデータセットを、実施例2〜5、および実施例7にさらに記載されるように、後の分析に使用した。第1のデータセットは、94匹のイヌ科動物(18品種の90匹のイヌ科動物と4匹のオオカミとを含む)における、95個のマイクロサテライトマーカー(マイクロサテライトマーカー1〜14、16、18〜21、23〜26、39〜100;表1参照のこと)についての遺伝子型情報を含んだ(データセット1;表6)。 第2のデータセットは、72品種の341匹のイヌ科動物における68個のマイクロサテライトマーカー(マイクロサテライトマーカー2〜8、11、12、14〜16、18〜21、23、24、26〜32、34〜36、38、41、42、44〜46、50、51、53、54、56、60〜64、67、68、70〜74、78、79、81〜83、85、87〜91、93〜98;表1参照のこと)についての遺伝型情報を含んだ(データセット2;表7)。第3のデータセットは、85品種の414匹のイヌ科動物における96個のマクロサテライトマーカー(マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100;表1参照のこと)についての遺伝型情報を含んだ(データセット3;表8)。第4のデータセットは、85匹のイヌ科動物(18品種の81匹のイヌと4種のオオカミとを含む)における96個のマイクロサテライトマーカー(マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100;表1参照のこと)についての遺伝型情報を含んだ(データセット4;表9)。第5のデータセットは、88品種の429匹のイヌ科動物における96個のマイクロサテライトマーカー(マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100;表1参照のこと)についての遺伝子型情報を含んだ。第6のデータセットは、(本明細書とともに提出されたコンパクトディスク中の)表3に示されるような、160種の雑種イヌ科動物における、表1中の72個のマイクロサテライトマーカーについての遺伝子型情報を含んだ。
【0067】
多型マーカーの比、マーカーごとの対立遺伝子の平均数、多型マーカーごとの対立遺伝子の平均数、(ハーディワインバーグ平衡の推定に基づいて)予測されたヘテロ接合性、観察されたヘテロ接合性、およびデータセット1中の95個のマイクロサテライトマーカーにわたる推定された同系交配係数は、表10に示される。Tajima不変推定量を使用して、96個のマイクロサテライト(データセット3)にわたって平均された85種のイヌ科動物個体群の予測されたヘテロ接合性は、表11に示される。
【0068】
品種の境界の存在は、同種由来のイヌが異種由来のイヌよりも遺伝学的により類似しているはずであることを予測する。この予測を試験するために、品種の構成員(membership)に起因し得る個々のイヌ間における遺伝的多様性の割合が、推定された。マイクロサテライトデータ(85品種の414匹のイヌにおける96個のマーカーを含む)(データセット3、表8)についての分子変動の分析は、遺伝分散の合計の27%よりも多くの割合を占める品種間の分散を示した。
【0069】
(2.SNPマーカーの情報性)
67種のイエイヌの189匹のイヌ科動物、コヨーテ、およびオオカミを使用して、およそ20Kbの非連続のイヌ科動物ゲノム配列中の100個の多型部位を、表2に記載されるように同定した。これらは、92個の一塩基置換、および11個の挿入または欠失変異(長さ1ヌクレオチド〜8ヌクレオチドの範囲)を含む。189匹のイヌ科動物(67品種の186匹のイヌ、2匹のオオカミ、および1匹のコヨーテを含む)における100個のSNPマーカーについての対立遺伝子の同一性は、(本明細書とともに提出されたコンパクトディスク中の)表4に示される。60品種の120匹のイヌからの75個のSNPにおけるマイナー対立遺伝子頻度は、表2に示されるように、0.4%〜48%の範囲に及んだ。これらのうちの14個のSNPは、以下の品種に特異的であった:372c5t−82(イングリッシュシェパード)、372e13t−57(コッカースパニエル)、372m6t−88(イングリッシュシェパード)、372m23t−76(アラスカンマラミュート)、373a15t−112(チェサピークベイレトリバー)、373e1t−50(スピノニイタリアーノ)、373elt−130(スコティッシュディアハウンド)、373g19t−246(ボルゾイ)、373i8s−224(チェサピークベイレトリバー)、373k8s−181(チベタンテリア)、372c5s−168(秋田犬)、372C15S−196(ラブラドールレトリバー)、372e15s−71(フィールドスパニエル)、および373a21t−93(イタリアングレイハウンド)。
【0070】
すべてのイヌが1個体群として考慮された場合、観察されたヘテロ接合性(Tajima&Nei(1984)Mol.Biol.Evol.1:269−85)は、8×10−4であった。これは、ヒト個体群において見られる値と基本的に同じである(Sachidanandamら、(2001)Nature 409:928−33;Venterら(2001)Science 291:3104−51)。しかし、品種が分岐される場合、最小非近交系(スコティッシュディアハウンド;2.5×10−4)から最大非近交系(イングリッシュスパニエル;1.0×10−3)の間のヘテロ接合性における範囲は、4倍である。60品種の120匹のイヌにおける75個のSNPについてのSNPデータから計算された品種間の遺伝的距離は、FST=0.36であった。
【0071】
Tajima不変推定量を使用し、75個のSNP座位における対立遺伝子頻度(データセット3)に基づいて予測された60種のイヌ科動物個体群のヘテロ接合性は、表12に示される。各品種は、2匹のイヌで表される。
【0072】
(実施例2)
この実施例は、94匹のイヌ科動物からの95個のマイクロサテライトマーカーについての遺伝子型情報、および341匹のイヌ科動物からの68個のマイクロサテライトマーカーについての遺伝子型情報に関して、割り当て試験計算器を使用する、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を推定するための本発明の代表的な方法を説明する。
【0073】
(A.方法)
(1.データセット)
データセット1は、18品種の90匹のイヌと4匹のオオカミを含む、94匹のイヌ科動物(AHRT、AKIT、BEAG、BMD、BOX、BULD、BULM、CHIH、DACH、GOLD、IBIZ、MAST、NEWF、PEKE、POM、PRES、PUG、ROTT、WOLF;イヌ科動物個体群の略称については表5を参照のこと)からの95個のマイクロサテライトマーカーについての遺伝子型情報を含んだ。95個のマイクロサテライトマーカーは、マイクロサテライトマーカー1〜14、16、18〜21、23〜26、39〜100であった(表1)。このデータセットは、各品種についての5匹のイヌ科動物と4匹のオオカミからの遺伝子型情報を含んだ(表6)。データセット1中のイヌ科動物についての遺伝子型情報は、(本明細書とともに提出されたコンパクトディスク中の)表3に示される。
【0074】
データセット2は、72品種の341匹のイヌ科動物(ACKR、AFGH、AHRT、AIRT、AKIT、AMAL、AMWS、AUSS、AUST、BASS、BEAG、BEDT、BELS、BLDH、BMD、BORD、BORZ、BOX、BSJI、BULD、BULM、CAIR、CHBR、CHIH、CKCS、CLSP、COLL、DACH、DANE、DNDT、DOBP、ECKR、FCR、GOLD、GREY、GSD、GSHP、GSMD、HUSK、IBIZ、IRSE、IRTR、IWOF、KEES、KOMO、KUVZ、LAB、MAST、MBLT、MNTY、NELK、NEWF、OES、PEKE、PNTR、POM、PRES、PTWD、PUG、RHOD、ROTT、SCHP、SCWT、SFXT、SHAR、SPOO、SSHP、STBD、TERV、WHIP、WHWT、WSSP;イヌ科動物個体群の略称については表5を参照のこと)からの68個のマーカーについての遺伝子型情報を含んだ。68個のマイクロサテライトマーカーは、マイクロサテライトマーカー2〜8、11、12、14〜16、18〜21、23、24、26〜32、34〜36、38、41、42、44〜46、50、51、53、54、56、60〜64、67、68、70〜74、78、79、81〜83、85、87〜91、93〜98であった(表1)。このデータセットは、SFXT(2匹のイヌ科動物)、ACKR、AFGH、DNDT、OES(各々3匹のイヌ科動物)、AIRT、BASS、BEDT、IRTR、MNTY、SCHP、SCWT、および、TERV(各々4匹のイヌ科動物)を除き、各品種についての5匹のイヌ科動物からの遺伝子型情報を含んだ(表7)。データセット2中のイヌ科動物についての遺伝子型情報は、(本明細書とともに提出されたコンパクトディスク中の)表3に示される。
【0075】
(2.Doh分析)
割り当て試験計算器Doh(www2.biology.ualberta.ca/jbrzusto/Doh.phpにて利用可能)を、遺伝子型情報の2つのデータセットの分析のために使用した。個々のイヌ科動物すべてを、試験されるイヌ科動物を除いたそれらの公知の個体群で指定した。次いでこれを、試験イヌ科動物の遺伝子型を生成する最も高い可能性を有するイヌ科動物個体群へと、プログラムを使用して割り当てた。このプログラムは、この手順を、試験イヌ科動物としての各イヌ科動物に対して繰り返す。
【0076】
(B.結果)
(1.データセット1を使用するDoh分析)
94匹のイヌ科動物(18品種の90匹のイヌと4匹のオオカミ)における95個のマイクロサテライトマーカーについての遺伝子型情報を含む、データセット1中の遺伝子型情報にDohを使用して、イヌ科動物の99%を、正確なイヌ科動物個体群へと割り当てた。イヌ科動物の100%を、以下の品種(AHRT、AKIT、BEAG、BMD、BOX、BULD、CHIH、DACH、GOLD、IBIZ、MAST、NEWF、PEKE、POM、PUG、ROTT、WOLF)について、正確に割り当てた。誤って割り当てられた唯一のイヌ科動物は、(5匹のイヌ中の)プレサカナリオ種のイヌ一匹であった。誤って割り当てられたプレサカナリオ犬は、チワワに割り当てられていた。
【0077】
対立遺伝子パターンの識別能力は、個々のマイクロサテライトマーカー座位の数、各座位における対立遺伝子の多様性、および各品種からサンプル抽出された個体の数に、依存したことが見出された。マーカーの対立遺伝子の数およびマーカーの数の、そのマーカーの情報性への影響を評価するために、最初の19品種についてのDoh割り当て分析を、5個、10個、15個、および20個のマーカー、データセット中に見出された1〜3個の別個の対立遺伝子を有するビニングマーカー、4〜6個の別個の対立遺伝子、7〜10個の別個の対立遺伝子、および10個より多い別個の対立遺伝子を使用して、行った。20個のマーカーを含まなかったビンについては、最大数のマーカーを使用した。10個より多い個別の対立遺伝子を有するマーカーについては、イヌ科動物の86%を、5個のマーカーを使用してそれらの品種へと正確に割り当て、そしてイヌ科動物の95%を、10個、15個、または20個のマーカーを使用して正確に割り当てた。7〜10個の別個の対立遺伝子を含むマーカーについては、イヌ科動物の84%を、5個のマーカーを使用してそれらの品種へと正確に割り当て、そしてイヌ科動物の91%を、10個のマーカーを使用して正確に割り当て、そしてイヌ科動物の94%を、15個、または20個のマーカーを使用して正確に割り当てた。4〜6個の別個の対立遺伝子を含むマーカーについては、イヌ科動物の62%を、5個のマーカーを使用してそれらの品種へと正確に割り当て、そしてイヌ科動物の71%を、10個、15個、または20個のマーカーを使用して正確に割り当てた。1〜3個の別個の対立遺伝子を含むマーカーについては、イヌ科動物の46%を、5個のマーカーを使用してそれらの品種へと正確に割り当て、そしてイヌ科動物の62%を、10個、15個、または20個のマーカーを使用して正確に割り当てた。正しいイヌ科動物個体群へと個体の100%をうまく割り当てるのに必要な(他のどの品種への割り当ても誤った割り当てである)、95個のマーカー内の対立遺伝子頻度の探索に向けられた2つのクラス(0―1)において見出されたマイクロサテライトマーカーの最小数とは、PEKEについて2個、BOX、POM、およびWOLFについて3個、AKIT、MAST、およびPUGについて4個、NEWFおよびROTTについて5個、BMDについて6個、BEAGについて8個、IBIZについて11個、GOLDについて12個、DACHについて17個、BULDについて19個、BULMについて26個、PRESについて44個、CHIHについて49個、そしてAHRTについて52個であった。100%(0−1)の識別に必要なマイクロサテライトマーカーの最小数と、19品種のイヌ科動物において試験された94匹のイヌ科動物についての95個のマイクロサテライトマーカーにわたる対立遺伝子の平均数(表10参照のこと)との間に、正の相関が存在する。100%の精度を有する選択されたイヌ科動物個体群を有す、19種のイヌ科動物個体群にわたって試験された94匹すべての個体の少なくとも90%をうまく割り当てるのに必要な、95個のマーカー内の対立遺伝子頻度の探索の向けた複数のクラス(0、1、2、...18)において見出されたマイクロサテライトマーカーの最小数とは、PEKE、BOX、POM、WOLF、AKIT、MAST、PUG、NEWF、ROTT、およびBMDについて8個、BEAGについて11個、GOLDについて14個、DACHについて23個、BULDについて24個、BULMについて28個、そしてPRES、CHIH、およびAHRTについて95個であった。
【0078】
予想どおり、識別能力は、各品種において観察される同系交配のレベルを反映する。例えば、ある品種は、品種の対立遺伝子変動の平均の3倍未満の対立遺伝子変動を有し、これらの品種は、高い識別能力と、個体群の長いボトルネックおよび小さな有効個体群サイズといった独特な個体群の動態との両方を有する。
【0079】
(2.データセット2を使用するDoh分析)
72品種の341匹のイヌ科動物からの68個についての遺伝子型情報を含む、データセット2中の遺伝子型情報にDoh分析を使用して、試験されたイヌの96%を、表13に記載されるように、正しい品種へと割り当てた。両方のベルジアン種(ベルジアンシープドッグおよびベルジアンタービュレン)を1つの品種として数えた場合、試験されたイヌの98%を、正しい品種へと割り当てた。
【0080】
(実施例3)
この実施例は、94匹のイヌ科動物からの95匹のマイクロサテライトマーカーについての遺伝子型情報にクラスター分析を使用して、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を推定するための本発明の代表的な方法を説明する。
【0081】
(A.方法)
(1.データセット)
データセット1は、実施例2に記載されるように、18品種の90匹のイヌおよび4匹のオオカミを含む、94匹のイヌ科動物からの95個のマイクロサテライトマーカーについての遺伝子型情報を含んだ。
【0082】
(2.クラスター分析)
クラスター分析を、マルチローカス遺伝子型クラスタリングプログラムであるstructure(Pritchardら(2000)Genetics 155:945〜59;Falushら(2003)Science 299:1582〜5)を使用して、行った。このプログラムは、対立遺伝子頻度のパターンに基づいて遺伝的に別個の部分個体群を同定するために、ベイジアンモデルベースのクラスタリングアルゴリズムを利用する。複数の実行を、バーンインの長さが10,000工程で、かつGibbsサンプラーを100,000回繰り返す設定状態で、各K値(遺伝クラスター数)について完了した。相関対立遺伝子頻度モデルを、許容された非対称な交配種(admixture)に使用した。2〜80のK値すべてを試験し、そして最も高い尤度を生じたクラスタリング解を、さらなる検証のために保持した。データセットについての全体的に最良のクラスタリング解を選択するために、全ペアWilcoxon2サンプル試験(all−pair Wilcoxon two−sample test)を、最も高い尤度の5つのK値について行った。
【0083】
(3.ネスト化した(nested)セットのクラスタリング)
完全なデータセットで開始して、すべての個体を、サブクラスターへ階層的に分けた。この場合において、各(K+1)番目のサブクラスターを、10回の実行にわたって観察された最も高い尤度の値に基づいて、前のKクラスターのうちの1つを分岐させることにより作製した。個体のクラスターを派生させるための階層的方法を利用することにより、部分個体群間の遺伝的多様性が、修正された交配種(admixture)の量が原因で減少される場合に、個体群の系統発生を解明するための合理的な方法論を推測し得る。
【0084】
(B.結果)
structureを使用する最大尤度の計算は、データセット1(19匹のイヌ科動物個体群中の95個のマーカー)中の20個体群を予測し、そして、表14に示されるように、各個体を99%の精度で1つの群に割り当てた。個体の品種群に割り当てられなかった1個体とは、たった1匹のプレサカナリオだけであった。この種は、ブルドッグ群とブルマチフ郡との間に配置された。プレサカナリオは、種々のマスチフ型の交配(admixture)を介して育成されてきた再現種である。誤って割り当てられたイヌは、具体的には、最近の12世代内のブルドッグおよびブルマスチフの両者まで、その遺伝形質を辿り得る。
【0085】
クラスタリングの割り当てでは、この分析レベルにおいては、ブルマスチフとマスチフとの間を区別することができないが、これは、表15A〜Dに示されるように、ネスト化(nested)分析によって解決される。ネスト化分析において、同様のクラスタリングアルゴリズムを、段階的様式で適用した。まず、全体のセットを、2つの個体群へと分けた。最大尤度に基づき、次いでこれら2つの個体群の一方を、合計3つの個体群を提供するために2つに分けた。この手順を、すべての個体群が決定されるまで繰り返した。5〜9つの群からの分岐は、マスチフ型品種の間の関係性を明確に示す。この関係性および予想された階層は、品種の説明書(account)から予測された関係性と完全に一致する。
【0086】
(実施例4)
この実施例は、85匹のイヌ科動物からの96個のマイクロサテライトマーカーについての遺伝子型情報にクラスター分析を使用して、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を推定するための本発明の代表的な方法を説明する。
【0087】
(A.方法)
(1.データセット)
データセット3は、85品種の414匹のイヌ科動物(ACKR、AFGH、AHRT、AIRT、AKIT、AMAL、AMWS、AUSS、AUST、BASS、BEAG、BEDT、BELS、BICH、BLDH、BMD、BORD、BORZ、BOX、BSJI、BULD、BULM、CAIR、CHBR、CHIH、CHOW、CKCS、CLSP、COLL、DACH、DANE、DOBP、ECKR、FBLD、FCR、GOLD、GREY、GSD、GSHP、GSMD、GSNZ、HUSK、IBIZ、IRSE、IRTR、ITGR、IWOF、KEES、KERY、KOMO、KUVZ、LAB、LHSA、MAST、MBLT、MNTY、MSNZ、NELK、NEWF、OES、PEKE、PHAR、PNTR、POM、PRES、PTWD、PUG、RHOD、ROTT、SALU、SAMO、SCHP、SCWT、SHAR、SHIB、SHIH、SPOO、SSHP、SSNZ、STBD、TIBT、TERV、WHIP、WHWT、WSSP;イヌ科動物個体群の略称については表5を参照のこと)からの96個のマーカーについての遺伝子型情報を含んだ。96個のマイクロサテライトマーカーは、マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100であった(表1)。このデータセットは、AIRT、BASS、BEDT、BICH、FBLD、IRTR、MNTY、PHAR、SCHP、SCWT、TERV(それぞれ4匹のイヌ科動物)を除き、全品種についての5匹のイヌ科動物についての遺伝子型情報を含んだ(表8)。このデータセット中のイヌ科動物についての遺伝子型情報は、(本明細書とともに提出したコンパクトディスク中)表3に示される。
【0088】
(2.統計分析)
structureを、20,000回繰り返すバーンインの後、Gibbsサンプラーを100,000回繰り返す間、実行した。相関対立遺伝子頻度モデルを、許容された非対称の交配種(admixture)に使用した。structureの実行全体にわたる類似度係数を、(Rosenbergら、(2002)Science 298:2381−5)に記載されるようにコンピュータで計算した。このプログラムを68品種の部分的なデータセットに実行した場合、40を超えるK値において、プログラムは、個体が何も割り当てられなかったクラスターを生じ、このクラスターは実行から実行への間、不安定であったことに注目のこと。このことは、2〜3の個体群に分岐するよう初期設定されたアルゴリズムが、このような多数の個体群を同時に扱うことが不可能であるため、最も起こりやすい。structureは、信頼できる別々の20の個体群を既に示しているので(Rosenbergら(2001)Genetics 159:699〜713)、このデータは、10〜11品種のそれぞれを8個のサブセットへ分けたセットであり、これらのサブセットの可能性のあるすべてのペアを分析した。歴史的に関連がある品種、または形態学的に類似する品種を、同じサブセット中に保持した。
【0089】
次いで、structureを、各Kにおいて15回実行する設定で、K=2〜K=10において全体のデータに適用した。Kが増加するほど、structureはまず、最も多岐した群をクラスターへと分け、その後に、より近縁種の分岐を続ける(Rosenbergら(2002)Science 298:2381)。分析において、尤度は、Kが増加するとともに増加し、各Kにおいて見出されたさらなる構造を反映するが、複数の異なるクラスタリング解が、K>4で見出され、そしてそれ故に、K=2〜4を、包括的な品種構造を記述するために使用した。これは、サブグループの系統発生分析およびクラスター分析を近縁種の一群(constellation)を規定するために使用した。K=2〜5におけるstructureの実行を、類似する結果を有する交配モデル(admixtrue model)がない状態下で繰り返した。別の分析において、8匹のオオカミを、K=2におけるstructureの実行に加えた。このオオカミを、8ヶ国(中国、オマーン、イラン、イタリア、スウェーデン、メキシコ、カナダ(オンタリオ)、および米国(アラスカ))からサンプル抽出した。すべてのオオカミを、表16に示される犬種の第1のクラスターとともにクラスター形成した。
【0090】
各品種を、品種の平均的な多数派に基づいて4つの群のうちの1つに割り当て、structureを、K=2〜4において各郡に実行した。さらなる一貫したパターンは、報告された品種ペアおよび品種トリオ以外の個々の群内では、観察されなかった。異常値分析を、http://www.rubic.rdg.ac.uk/〜mab/software.htmlにて利用可能なソフトウェアパッケージfdist2を使用して行った。11個のマーカーを、85種の個体群を仮定した無限対立遺伝子モデルおよび1個体群あたり10個のハプロタイプ遺伝子型の平均のもとでの、シミュレーションによって達成された、95番目のパーセント点を超えるFst値を有する潜在的な「異常値」として同定した(BeaumontおよびNichols(Dec.22,1996)Proceedings:Biological Sciences 263:1619)。これらの除かれたマーカーで行われた割り当ておよび構造分析は、有意な変化を生じなかった。
【0091】
系統樹分析について、個々のイヌおよびオオカミを、品種または種に基づいて、86種の個体群のうちの1つに割り当てた。個体群間の距離を、コード距離尺度(chord distance measure)を用い、プログラムMicrosat(E.Minch、A.Ruiz−Linares、D.Goldstein、M.Feldman,L.L.Cavalli−Sforza(1995、1996))を使用して、コンピュータで計算した。500回のブートストラップ複製を作成した。このプログラムは、ウェブサイトhttp://hpgl.stanford.edu/projects/microsat/microsat.htmlからダウンロードされ得る。近接結合樹を、プログラムNeighborを使用して各複製について構築し、そしてプログラムConsenseを、多数派支配型(majority−rule)コンセンサス樹を作成するため使用した。これらプログラムは両方とも、http://evolution.genetics.washington.edu/phylip.htmlにて利用可能な、Phylipパッケージ(Felsenstein(1989)Cladistics 5:164)の一部である。オオカミの個体群を、その樹の基礎を成す(root)ために外群(outgroup)として指定した。異なる8つの国々からのオオカミを、図2に示される樹において、簡略化のために1個体群にまとめた。個体として考えられる場合、すべてのオオカミは、枝の根が同じ場所にある1つの枝から分岐され。系統分析における分岐の順番は、ヘテロ接合性と相関し(表11)、一番よく似ている(closely mirrored)最初のクラスターに分岐する12品種が、structureによって同定された。これらの知見は、この分析が、異常に同系交配であるかまたは最近野生のイヌ科動物と交配された特異体質品種に分岐するのではなく、遺伝的に関連する品種の別個の部分群を同定したことを、主張する。
【0092】
割り当て試験を、J.Brzustowski(http://www2.biology.ualberta.ca/jbrzusto/Doh.php)から利用可能なDoh割り当て試験計算器を用いて行った。すべてのイヌを、試験される1種のイヌを除いて、それらの公知の品種で指定した。次いでこれを、試験イヌの遺伝子型を生成する可能性が最も高い品種へと、プログラムを使用して割り当てた。このプログラムは、この手順を、試験イヌとしての各イヌに対して繰り返す。ベルジアンシープドッグ種およびベルジアンタービュレン種を、この分析のために1つの記号表示にまとめた;それらが別個の品種として扱われる場合、個々のイヌを、基本的にランダムにどちらか一方に割り当てる。
【0093】
(B.結果)
structureを20〜22品種のオーバーラップするサブセットに同時に適用した場合、表17に示されるように、ほとんどの品種がその品種からのすべてのイヌだけから構成される別個のクラスターを形成することが、観察された。以下の4品種のイヌだけが、一貫して、他の同種とクラスター形成できなかった:ペロデプレサカナリオ、ジャーマンショートヘアードポインター、オーストラリアンシェパード、およびチワワ。加えて、以下の品種の6つのペアが、実行の大部分において、ともにクラスター形成した:ベルジアンシープドッグおよびベルジアンタービュレン、コリーおよびシェトランドシープドック、ウィペットおよびグレイハウンド、シベリアハスキーおよびアラスカンマラミュート、マスチフおよびブルマスチフ、グレートスミスマウンテンドックおよびバーニーズマウンテンドッグ。これらの組み合わせは、公知の品種の歴史に基づいて予測される。これらの近縁種のペアがそれでもなお遺伝的に別個であるかどうかを試験するために、structureをこれらの各クラスターに適用した。1つだけを除いてすべてのクラスターは、表18に示されるように、個々の品種に対応する2種の個体群へと分岐した。1つの例外は、ベルジアンシーブドックとベルジアンタービュレンであった。ヨーロッパケンネルクラブおよび日本ケンネルクラブは、それらを、1品種の毛色および毛長の変種として分類し(YamazakiおよびYamazaki(1995)Legacy of the Dog:The Ultimate Illustrated Guide to Over 200 Breeds,Chronicle Books,San Fracisco,CA;WilcoxおよびWalkowicz(1995)Atlas of Dog Breeds of the World,T.F.H.Publications,Neptune City,NJ)、そして、その一方で、アメリカンケンネルクラブが、これらを別個の品種として認識するとはいえ、品種の境界は、遺伝的差異を結果としてもたらすには、明らかに近い世代(recent)でありまたは厳密さが不十分である。これらの例は、アルゴリズムは真の遺伝的差異を有する群に分けるだけに過ぎないことを確認する(Falshら(2003)Science 299:1582−5;PritchardおよびRosenberg(1999)Am.J.Hum.Genet.65:200−8)。
【0094】
イヌが遺伝子型データだけを基にしてその品種へと割り当てられ得るかどうかを試験するために、leave one−out分析を用いる直接割り当て方法(Paetkauら(1995)Mol.Ecol.4:347−54)を、使用した。99%の個々のイヌが、正しい品種へと正確に割り当てられた。414匹のうちの4匹のイヌのみが、誤って割り当てられた:1匹は(ペロデプレサカナリオに割り当てられた)ビーグルであり、1匹は(ケーアンテリアに割り当てられた)チワワであり、そして2匹は、(クバーズおよびスタンダードプードルそれぞれに割り当てられた)ジャーマンショートヘアードポインターである。品種に関連する4つエラーは、structure分析において単独の品種クラスターを形成しなかった品種を含んだ。
【0095】
現代のイヌの品種が別個の遺伝単位を形成することが示されると、品種間の広範な歴史的関係性を規定することが試みられた。最初に、標準的な近隣結合法を、コード距離尺度(Cavalli−SforzaおよびEdwards(1967)Evolution 32:550)を使用して計算された距離を用い、品種の多数派支配型コンセンサス樹を構築するために使用した(図2)。これは、特定の変異モデルを仮定せず、そして近縁の分類群に対して良好に機能すると考えられる(Goldsteinら(1995)Genetics 139:463)。この樹を、オオカミサンプルを使用して基礎を成した(root)。樹の最も深い分岐は、4種のアジアスピッツ型品種に分かれ、そしてこの枝内でシャーペイが最初に分岐し、その後に柴犬が続き、秋田犬およびチャウチャウは一緒のグループになった。第2の分岐は、バセンジー(古代アフリカ品種)に分かれた。第3の分岐は、2種の北極スピッツ型品種(アラスカンマラミュートとシベリアンハスキー)に分かれ、そして第4の分岐は、残りの品種から、2種の中東品種のハウンド(アフガンとサルーキ)に分かれた。
【0096】
最初の4つの分岐は、半分より多くのブートストラップ複製が示すように、「多数派支配」基準を超えた。対照的に、残りの品種は、わずかなに系統学的に一致する関係性を示した。これは、structure分析でともにクラスター形成される5品種のペアの近縁群を除いて、近縁関係にあるウェストハイランドホワイトテリアとケアーンテリアとの新たな1つのペアであり、そして類似する外見を有する仲間の品種(companion breed)であるアジアの3種(ラサアプソ、シーズー、およびペキニーズ)の有意な分類である。これらの3種の間の近縁関係はまた、実行の大部分において3つのうちの少なくとも2つが一緒にクラスター形成することで、structure分析において観察された。樹のフラットトポロジーは、おそらく、より大きな共通の創始系統(founder stock)ならびに、血統クラブおよび品種の境界規則の出現より前の、表現型的に異なるイヌ間での広範な遺伝子流動の発生を反映する。加えて、これは、表現型的に類似するか歴史的に関連するイヌ由来の系統を使用した、飢饉(19世紀および20世紀の不況および戦争)の間に絶滅したいくつかの歴史的に古い品種の再生を示す可能性がある。
【0097】
系統発生分析は、現代のヨーロッパ血統と推定されたより大きな品種群からの古代血統を有するいくつかの品種の分岐を示したが、更なる部分個体群は、少なくとも2つの理由によってこのアプローチでは検出されない後者の郡内に、存在し得る(Rosenbergら、(2001)Genetics 159:699)。第一に、犬種の真の進化の歴史は、この方法によって仮定された二分岐樹モデルでは、十分には表されないが、新種を生成するための現存種の交配(現在まで続くプロセス)に関しては、かなり関与した。第二に、遺伝的距離マトリックスに基づいた方法は、品種のペアについてのすべての遺伝子型データを、1つの数字へと激減させることで情報を失う。
【0098】
structureに組み込まれたクラスタリングアルゴリズムは、これらの制限を克服するために明示的に設計され(Pritchardら(2000)Am.J.Hum.Genet.67:170−81;Falushら(2003)Genetics 164:1567;Rosenbergら(2001)Genetics 159:69−713)、そして、いくつかの種の遺伝学的構造を推測するために適用されている(Rosenbergら(2002)Scinece 298:2181−5;Falushら(2003)Science 299:1582−5;Rosenbergら(2001)Genetics 159:699−713)。structureを、先祖の個体群源を同定するためにKの増加値(プログラムが発見しようと試みた部分個体群の数)を使用して、全体のデータセットに対して実行した。この分析において、現代種は、1つの先祖個体群と厳密に酷似し得るか、または2つ以上の先祖タイプの交配種を表し得る。
【0099】
K=2において、1つのクラスターが、系統発生分析において分岐するために、最初の7品種によって支えられたが、一方で、他のクラスターは、フラットな系統発生のトポロジーで多数の品種を含んだ(表19A)。プログラムの5つ実行は、実行にわたって0.99の類似する係数で、ほぼ同一の結果を生成した(Rosenbergら(2002)Science 298:2381)。7種の他の品種は、それらの祖先の相当大きな部分を、最初のクラスターと共有する。これら14種のすべては古代から始まり、そしてそれらの祖先をアジアまたはアフリカまでさかのぼる。8つの異なる国々からのオオカミの多様なセットが分析に含まれた場合は、これらは、完全にこのクラスター内にある(表20)。オオカミの外群へとつながる枝もまた、系統発生分析においてこの群の品種内にある(図2)。
【0100】
K=3において、系統樹からは容易には明白に理解されなかったさらなる構造が検出された(表19B)。新たな3つクラスターは、マスチフの遺伝形質および外見において関連した品種から主に構成され、そして、それらの近縁種(ブルマスチフ、フレンチブルドッグ、ミニチュアブルテリア、およびペロデプレサカナリオ)とともに、マスチフ、ブルドック、およびボクサーによって支えられる。クラスター内に含まれるものはまた、古代マスチフタイプの祖先からそれらの大きさを獲得したと報告されている大型品種である、ロットワイラー、ニューファンドランド、およびバーニーズマウンテンドッグである。ジャーマンシェパードドッグを含むことはあまり期待されない。この品種の正確な起源は未知であるが、この結果は、作業犬タイプ(例えば、ボクサー)の前で軍用犬および警察犬として費やした年数が、この有名な品種の遺伝学的背景の形成に関与することを示唆する。他の3つの品種は、structureの実行にわたってこのクラスター中に部分的でかつ不定な構成員を示した(表16)。これは、類似係数を0.84まで低下させた。
【0101】
K=4において、4つのクラスターが観察された。これは、牧畜犬として使用された数品種:ベルジアンシープドッグ、ベルジアンタービュレン、コリー、およびシェットランドシープドッグを含んだ(表19C)。アイリッシュウルフハウンド、グレイハウンド、ボルゾイ、およびセントバーナードもまた、頻繁にこのクラスターへと割り当てられた。歴史的記録物は、これらのイヌがこれまでに家畜を集めるために使用されていたとは示唆しないが、得られた結果は、これらの品種が牧畜タイプの祖先かまたは子孫であるかのいずれかであることを示唆する。残りのクラスター中の品種は、主に比較的最近のヨーロッパ血統からなり、そして、主には異なるタイプの猟犬(セントハウンド、テリア、スパニエル、ポインター、およびレトリバー)である。K=4におけるクラスタリングは、0.61の類似係数を示した。これは、ほとんどの品種についての類似クラスター構成員の割り当てを反映するが、実行にわたって他の品種についての割り当て変数を反映しない(表16)。K=5において、類似係数は、0.26まで下がり、さらに一致する部分個体群は推測されなかった。これは、サンプル抽出された純血種のイヌ個体群においてさらに高レベルの部分構造が欠けていることを示唆している。
【0102】
得られた結果は、イエイヌ種にわたる関係性の下記の構図を描く。異なる品種は、遺伝学的に別個であり、そして個体は、それらの遺伝子型に基づいて品種へと容易に割り当てられ得る。この分岐レベルは、ほとんどの品種の起源が祖先血統から交配されたときからの短い時間を考慮すると驚くべきものであり、そして、品種の境界規則の結果として生じる、各品種内の強力な生殖隔離を支持する。この結果は、別個の「適応放散」を表す少なくともの4つの別個の品種分離を支持する。アジア血統およびアフリカ血統を有する品種の部分セットは、残りの品種から分かれ、そして共有の対立遺伝子頻度パターンを示す。一見したところ、1つの遺伝クラスター内に、中央アフリカ由来種(バセンジー)、中東由来種(サルーキおよびアフガン)、同様にチベット由来種(チベタンテリア、ラサアプソ)、中国由来種(チャウチャウ、ペキニーズ、シャーペイ、シーズー)、日本由来種(秋田犬、柴犬)、ならびに北極由来種(アラスカンマラミュート、シベリアンハスキー、サモエド)を包含するようであり、これは驚くべきことである。しかし、初期の雑種の野良犬は、アジアに由来し、そして遊牧民族とともに、南はアフリカまでそして北は北極まで移動し、結果としてアジアの至るところに移動したという仮説が立てられている(Savolainenら(2002)Science 298:1610;Leonardら(2002)Science 298:1613;SablinおよびKhlopachev(2002)Current Anthropology 43:795)。このクラスターは、オオカミに表現型的に似ている北欧種(例えば、アラスカンマラミュートおよびシベリアンハスキー)を含み、そして、イエイヌの直接の祖先であるオオカミに対して最も近い遺伝学的関係性を示す。従って、これらの品種由来のイヌは、祖先イヌの遺伝子プールの最も代表的な生物であり得る。古代起源の品種であると一般に考えられている数種(例えば、ファラオハウンドおよびイビザンハウンド)は、この群に含まれないことは、注目すべきことである。これらは、すべての犬種のうち最も古い種であるとしばしば考えられている。この種は、5000年以上前に墓の壁に描かれた古代エジプトのイヌから直接的に由来する種である。しかし、この結果は、これら2つの種が、より近代において、他の種の組み合わせから再生されていることを示す。したがって、それらの外見は古代エジプト種のハウンドと一致するが、それらのゲノムは一致しない。同様の結論をノルウェジアンエルクハウンドへと適用すると、これは、5000年前以上にスカンジナビア起源種から直接的に伝わったという報告(American Kennel Club(1998)「The Complete Dog Book」、CrowleyおよびAdelman編、Howell Book Houes、New York、NY;WilcoxおよびWalkowicz(1995)Atlas of Dog Breeds of the World,T.F.H.Publications,Neptune City,NJ)とは異なり、他の北極犬よりも近代のヨーロッパ種でクラスター形成する。
【0103】
大多数の品種は、共通のヨーロッパ血統由来の、より最近の放散(radiation)を表すようである。個々の品種が遺伝学的に分化される間、それらは、基本的に同時期に分化されたようである。この放散は、1800年代のヨーロッパにおける血統の概念の導入および血統クラブの創設の後に、あまり体系化されてなかった表現型的多様性からの別個の品種の繁殖をおそらくは反映する。より高感度なクラスター分析は、この群内の3つの部分個体群のさらなる遺伝学的構造を見分けることが可能である。あるクラスターは、マスチフ様品種を含み、そして共通祖先由来の共通する形態を反映するように見える。別のクラスターは、シェットランドシープドッグ、2匹のベルジアンシープドッグ、およびコリーを含み、そして、共通する祖先の牧畜習性を反映し得る。残りの個体群は、狩猟の種々の局面に献身的な品種の繁殖によって、主に占められている。これらの品種について、歴史的記録および血統クラブの記録は、高度に絡み合った血統を示唆し、これは、得られた結果と一致する。
【0104】
犬種は、伝統的に、人間の活動におけるイヌの役割、身体的な表現型、および歴史的記録に基づいて、分類されてきた。上に記載された結果は、遺伝学的多様性のパターンに基づいて、個々の分類を提供する。この分類は、伝統的な分類の部分セットを支持し、そしてまた、品種間の以前は認識されていなかった関係を解明する。品種間の遺伝学的関係性の正確な理解は、形態、習性、および疾患罹患率における品種の差異の複雑な遺伝学的基礎を解明することを目的とした研究のための基礎を築く。
【0105】
(実施例5)
この実施例は、マイクロサテライトマーカーを用いて、交配されたイヌ科動物の子孫のゲノムへの、異なるイヌ科動物個体群からの親、祖父母、および曽祖父母の寄与度を推定する、インシリコでの方法を説明する。
【0106】
(A.方法)
(1.データセット)
データセット4は、18種の異なる犬種の81匹のイヌと4匹のオオカミからなる、85匹のイヌ科動物(AHRT、AKIT、BEAG、BMD、BOX、BULD、BULM、CHIH、DACH、GOLD、IBIZ、MAST、NEWF、PEKE、POM、PRES、PUG、ROTT、WOLF;イヌ科動物個体群の略称については表5を参照のこと)からの95のマーカーについての遺伝子型情報を含んだ。95個のマイクロサテライトマーカーは、マイクロサテライトマーカー1〜14、16、18〜21、23〜26、39〜100であった(表1)。このデータセットは、85匹のイヌ科動物の各々の90%よりも多くが、正確な品種へと割り当てられた事実に基づいて選択された。4匹のオオカミは、1種のイヌ科動物個体群として設計された。12品種は、5匹のイヌで、3品種は、4匹のイヌで、そして3品種は3匹のイヌで、表9に示されるように表された。各イヌ科動物において使用されたマイクロサテライトマーカーの各々についての遺伝子型は、(本明細書とともに提出されたコンパクトディスク中の)表3に示される。
【0107】
(2.クラスター分析)
インシリコでのイヌ科動物の交配種を、各座位における各親からの2つの対立遺伝子のうちの1つをランダムに引き出し、そしてそれらをその座位における交配種の対立遺伝子として設計することにより、作製した。F1交配種を、81匹の原種イヌ科動物の2つの対立遺伝子をインシリコで交配することによって作製した。次いで、N2交配種を、それらの2つの親のうちの1つとF1とをインシリコで交配することによって作製し、そして、N3交配種を、同じ親とN2とをインシリコで交配することによって作製した。
【0108】
3つのタイプの交配種が形成された(試験交配種、コントロール交配種、祖父母交配種)。試験交配種において、2つの親を、ランダムに選択された異なる2種から選択した。100個のF1交配種、N2交配種、およびN3交配種が、形成された。F1交配種が、異なる品種からの親を2つ有し、N2交配種が、1種からの祖父母および別の種から祖父母の4つのうちの3つを有し、そしてN3交配種が、1種からの曽祖父母および別の種から曽祖父母の8つのうちの7つを有することに注目のこと。
【0109】
コントロール交配種において、2つの親を同種から選択し、そして100個のF1混合種、N2混合種、およびN3混合種を、同じ手順によって形成した。これらすべての混合種が選択された品種からの純血種のイヌに対応することに注目のこと。
【0110】
いくつかの祖父母交配種をまた、異なる4種の品種から4つの祖父母を選択することにより形成した。
【0111】
300個すべての試験混合種を、選択された85匹のイヌ科動物を用いるstructureの実行において、同時に実行した。同様の分析を、コントロール交配種について、および4つの祖父母交配種について行った。プログラムを、以下のパラメーター設定で実行した:#define NUMINDS 395;#define NUMLOCI 95;#define LABEL 1;#define POPDATA 1;#define POPFLAG 1;#define PHENOTYPE 0;#define MARKERNAMES 0;#define MAPDISTANCES 0;#define ONEROWPERIND 1;#define PHASEINFO 0;#define PHASED 0;#define EXTRACOLS 0;#define MISSING 0;#define PLOIDY 2;#define MAXPOPS 19;#define BURNIN 5000;#define NUMREPS 5000;#define USEPOINFO 1;#define GENSBACK 0;#define MIGRPRIOR 0.0;#defineNOADMIX 0;#define LINKAGE 0;#define INFERALPHA 1;#define ALPHA 1.0;#define POPALPHAS 0;#define UNIFPRIOPALPHA 1;#define ALPHAMAX 10.0;#define ALPHAPROPSD 0.025;#define FREQSCORR 1;#define ONEFST 0;#define FPRIORMEAN 0.01;#define FPRIORSD 0.05;#define INFERLAMBDA 0;#define LAMBDA 1;#define COMPUTEPROB 1;#define PFROMPOPFLAGONLY 0;#define ANCESTDIST 1;#define NUMBOXES 1000;#define ANCESTPINT 0.95;#define STARTATPOPINFO 1;#define METROFREQ 10;#define UPDATEFREQ 1;#define PRINTQHAT 1。
【0112】
85匹のイヌ科動物の各々は、それらの適切な品種に属するように設計され、そして、その交配種は、どの品種にも割り当てられなかった。
【0113】
(B.結果)
コントロール交配種について、各交配種は、常に、プログラムによって正しい品種へと割り当てられた。そして、その品種へと割り当てられたゲノムの比率は、300回のケースすべてにおいて95%を超え(最小値は95.75%であった)、297回のケースにおいては98%を超え、そして266回のケースにおいては99%を超えた。従って、単一の種への95%を超えるゲノムの割り当ては、試験交配種についての交配の明白な検出を提供した。そして98%を超える割り当ては、0.99の信頼度における交配の強固な証拠を提供する。
【0114】
F1試験交配種について、100個の交配種すべては、各品種の寄与度が28%〜70%の範囲で正確に割り当てられた、2つの親種からゲノム寄与度であった。100回のケースのうち82回において、2つの親種の各々は、40%より高い寄与度、および60%より低い寄与度で割り当てられた。このことは、2種間での交配種が、親レベルの時点で100%確実に同定され得ることを示している。
【0115】
N2試験交配種について、100回のケースのうちの99回は、1つの品種へと割り当てられたゲノムのうちの98%未満を有し、100回のケースのうちの97回は、1つの品種へと割り当てられたゲノムのうちの95%未満を有した。これは、祖父母レベルにおける交配を検出する高度に正確な能力を示した。交配が検出されたケースの1つを除いて、交配に寄与する両方の品種は、正確に割り当てられた(1つのケースにおいて、4つの祖父母のうちの1つに寄与する品種は、有意に寄与するものとしては検出されなかった)。80〜85%のケースにおいて、N2交配種は、F1交配種から確実に識別され得る(すなわち、親レベルではなく祖父母レベルで生じた交配が検出され得る)。
【0116】
N3試験交配種について、100回のケースのうちの85回は、1つの品種へと割り当てられたゲノムのうちの98%未満を有し、100回のケースのうちの77回は、1つの品種へと割り当てられたゲノムのうちの95%未満を有した。これは、祖父母レベルにおける交配を検出するかなり良好な能力を示した。交配が検出されたケースの1つを除いて、交配に寄与する両方の品種は、正確に割り当てられた。すべてのケースにおいて、N3交配種は、F1交配種から確実に識別され得る(すなわち、親レベルではなく祖父母レベルで生じた交配が検出され得る)が、祖父母レベルおよび曽祖父母レベルにおける交配の間で区別する能力は低かった。
【0117】
最終的に、4つの異なる祖父母を有する交配種について、4つの祖父母種すべてが、20〜30%の範囲と推定された各品種の交配種のゲノムへの寄与度で、正確に同定された。
【0118】
これらの結果は、いくらかが曽祖父母レベルにおける交配を識別する能力で、純血種のイヌからの(イヌ由来の1/2オオカミ交配種および1/4オオカミ交配種も同様)、親レベルおよび祖父母レベルにおける交配を識別する方法の能力を明白に証明している。この方法また、雑種イヌのゲノムにおける品種の寄与度を正確に同定する。各品種からのより多くのイヌ、および本明細書に他で記載された基準に従って選択された追加にマーカーおよび最適化されたマーカーセットを含む、より大きなデータベースは、曽祖父母レベルにおける交配をより正確に識別することを可能にし、そして、単純な拡張によって、より遠い祖先において生じる交配も正確に識別することを可能にする。
【0119】
(実施例6)
この実施例は、SNPマーカーを使用する、試験イヌ科動物のゲノムへのイヌ科動物の寄与度を推定するための本発明の代表的な方法を説明する。
【0120】
(A.方法)
(1.データセット)
種々の犬種における一塩基多型(SNP)のデータセットを、各品種における各対立遺伝子頻度を計算するために使用した。データベースは、実施例1に記載されるように、品種ごとに11純血種のうちの2種に関しての、67品種の189匹のイヌ科動物からの100個のSNPについての遺伝子型情報を含んだ。イヌ科動物における対立遺伝子の同一性は、(本明細書とともに提出されたコンパクトディスク中の)表4に示される。
【0121】
(2.Doh分析)
leave−one−out手順を使用して、各イヌをデータベースから一時的に除き、各品種の対立遺伝子頻度とイヌの遺伝子型との比較に基づいて、品種へと割り当てた。ベイズの定理を、割り当てについて使用した:所与の品種から生じた確率とは、その品種のイヌにおいて生じる観察された遺伝子型を、データベース内のすべての品種について生じる観察された遺伝子型である条件付き確率の合計で割った条件付き確率である(基本的に、Cornuetら(1999)Genetics 153:1989〜2000において記載される)。ソフトウェアを、このアルゴリズムを実施するために開発した。2個体のみを有する品種をデータベース内に含んだが、それらの一員を分類する試みはしなかった。なぜなら、2つの一員のうちの1つを一時的に除くことは、信頼できる対立遺伝子頻度を計算するための十分な情報を残さなかったからである。
【0122】
(B.結果)
この分析の出力結果は、表21に示されるような、各イヌについての、データベース中の各品種から生じた確率のリストである。80%のイヌは、99%以上の確率で正確な品種へと割り当てられた。遺伝子型が5個以上の個体について得られた品種について、88%のイヌが、99%の確率で正確な品種へと割り当てられた。14匹のイヌ(試験された合計のうちの16%)は、65%よりも良好な確率で正確な品種へと割り当てられた。これらについて、13匹は、50%よりも良好な確率(90%よりも高い確率のほぼ1/3)で誤って割り当てられた。残りのイヌは、数種から生じた20〜45%の確率で割り当てられ、これらのうちの1つは、正しかった。
【0123】
これらの結果は、SNPマーカーに基づいた品種割り当ての実行可能性を実証している。性能は、より多く(各品種について5匹以上)のイヌについてのSNP遺伝子型プロファイルを、より大きなSNPセットを使用して生成すること、および最大限の情報性となるSNPを選択することによって増強され得る。SNPは、品種にわたる高いヘテロ接合性を有すること(すなわち、高頻度で生じる両方の対立遺伝子)に基づくパネル、および品種間の頻度における大きな差異に基づくパネルの両方を包含して選択され得る。
【0124】
(実施例7)
この実施例は、マイクロサテライトマーカーを用いて、交配された子孫イヌ科動物のゲノムへの、異なるイヌ科動物個体群からの親および祖父母イヌ科動物の寄与度を推定するためのナイーブベイジアン分類モデルを説明する。
【0125】
(A.方法)
(1.データセット)
データセット5は、88品種の429匹のイヌ科動物(ACKR、AFGH、AHRT、AIRT、AKIT、AMAL、AMWS、ASBT、AUSS、AUST、BASS、BEAG、BEDT、BELS、BICH、BLDH、BMD、BORD、BORZ、BOX、BRIA、BSJI、BULD、BULM、CAIR、CHBR、CHIH、CHOW、CKCS、CLSP、COLL、DACH、DANE、DOBP、ECKR、FBLD、FCR、GOLD、GREY、GSD、GSHP、GSMD、GSNZ、HUSK、IBIZ、IRSE、IRTR、ITGR、IWOF、KEES、KERY、KOMO、KUVZ、LAB、LHSA、MAST、MBLT、MNTY、MSNZ、NELK、NEWF、OES、PEKE、PHAR、PNTR、POM、PRES、PTWD、PUG、RHOD、ROTT、SALU、SAMO、SCHP、SCWT、SHAR、SHIB、SHIH、SPOO、SSHP、SSNZ、STBD、TIBT、TERV、TPOO、WHIP、WHWT、WSSP;イヌ科動物個体群の略称については表5を参照のこと)からの96個のマーカーについての遺伝子型情報を含んだ。96個のマイクロサテライトマーカーは、マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100であった(表1)。このデータセット中のイヌ科動物についての遺伝子型情報は、(本明細書とともに提出したコンパクトディスク中)表3に示される。
【0126】
データセット6は、既知の交配種構成員を有する160匹の雑種イヌ科動物からの、表1における72個のマーカーについての遺伝子型情報を含んだ。このデータセット中の雑種イヌ科動物についての遺伝子型情報は、(本明細書とともに提出したコンパクトディスク中)表3に示される。
【0127】
(2.分析)
ナイーブベイジアンモデルを構築した。これは、先祖の部分個体群の交配種への個体の確率論的割り当てのために、連鎖および非連鎖マイクロサテライト座位情報、高次元の先祖個体群、および高度に秩序だった世代系図を組み込んでいる。2世代および3世代のモデルを、既存モデルの世代、部分個体群、および連鎖の制限を同時に対処し、正確に交配種を検出しかつ割り当てるために、組み込んだ。
【0128】
2世代モデルは、「純血の」部分個体群の2つのクラスよりもより大きく拡張して、AndersonおよびThompson(2002)Genetics 160:1217−29に概説されたモデルに、厳密に従っている。非連鎖座位(L)について、部分個体群(N)(品種とみなされる)、および第1番目の座位における対立遺伝子(j1)を有する。座位(L)における各個体について、遺伝子型:(g1(0)、g1(1))を含めた。部分個体群対立遺伝子情報を集めることは、任意の所与の対立遺伝子に頻度(fij(i)で表される)についての情報を提供する。したがって個体について、交配されなかった(non−admixed)部分個体群の割り当てには、以下:
【0129】
【数1】

【0130】
を与える。ここで親の交配種の割り当てについては、以下:
【0131】
【数2】

【0132】
を有する。ここで上付き添え字(0)は、父親と子との関係、そして(1)は、母親と子との関係を、(明白で交換可能なオプションとともに)表す。
【0133】
3世代モデルは、モデルの拡張が、部分個体群(N)にわたる4個体群の2世代の表示(representation)を考慮することを可能にする:
【0134】
【数3】

【0135】
最も高い後方確率での交配種の徹底的な探索は、2世代および3世代モデルについて可能である。
【0136】
インシリコでの個体について、モデルの評価は、leave−one−outクロス検定によって行われた。この場合、インシリコ雑種個体の生成に使用した、サンプル抽出された対立遺伝子は、先祖個体群から除かれ、そして対立遺伝子頻度は、最大尤度の交配比率の割り当ての前に、更新される。
【0137】
(B.結果)
96個すべてのジヌクレオチドマーカーのわたるインシリコ雑種個体群についての分析は、2世代および3世代におけるモデルが、品種特異的に不足するというような明らかな傾向がない状態で、F1交配種の98.4%およびF2交配種の94.3%を正しく非常に良好に割り当てることを示している。96個のジヌクレオチドマーカーのうちの72個において遺伝子型決定された既知の雑種個体160匹についての分析は、2世代および3世代におけるモデルが、ほぼ正確に、F1交配種の96.2%およびF2交配種の91.8%を正しく割り当てることを示している。
【0138】
本発明の好ましい実施形態が例示されそして説明されているが、種々の変更が、本発明の精神および範囲を逸脱することなく、本発明において行われ得ることが理解される。
【0139】
【表1】

【0140】
【表2】

【0141】
【表3】

【0142】
【表4】

【0143】
【表5】

【0144】
【表6】

【0145】
【表7】

【0146】
【数4】

【0147】

【0148】

【0149】

【0150】

【0151】

【0152】

【0153】

【0154】

【0155】

【0156】

【0157】

【0158】

【0159】

【0160】

【0161】

【0162】

【0163】

【0164】

【0165】

【0166】

【0167】

【0168】

【0169】

【0170】

【0171】

【0172】

【0173】

【0174】

【0175】

【0176】

【0177】

【0178】

【0179】

【0180】

【0181】

【0182】

【0183】

【0184】

【0185】

【0186】

【0187】

【0188】

【0189】

【0190】

【0191】

【0192】

【0193】

【0194】

【0195】

【0196】

【0197】

【0198】

【0199】

【0200】

【0201】

【0202】

【0203】

【0204】

【0205】

【0206】

【0207】

【0208】

【0209】

【0210】

【0211】

【0212】

【0213】

【0214】

【0215】

【0216】

【0217】

【0218】

【0219】

【0220】

【0221】

【0222】

【0223】

【0224】

【0225】

【0226】

【0227】

【0228】

【0229】

【0230】

【0231】

【0232】

【0233】

【0234】

【0235】

【0236】

【0237】

【0238】

【0239】

【0240】

【0241】

【0242】

【0243】

【0244】

【0245】

【0246】

【0247】

【0248】

【0249】

【0250】

【0251】

【0252】

【0253】

【0254】

【0255】

【0256】

【0257】

【0258】

【0259】

【0260】

【0261】

【0262】

【0263】

【0264】

【0265】

【0266】

【0267】

【0268】

【0269】

【0270】

【0271】

【0272】

【0273】

【0274】

【0275】

【0276】

【0277】

【0278】

【0279】

【0280】

【0281】

【0282】

【0283】

【0284】

【0285】

【0286】

【0287】

【0288】

【0289】

【0290】

【0291】

【0292】

【0293】

【0294】

【0295】

【0296】

【0297】

【0298】

【0299】

【0300】

【0301】

【0302】

【0303】

【0304】

【0305】

【0306】

【0307】

【0308】

【0309】

【0310】

【0311】

【0312】

【0313】

【0314】

【0315】

【0316】

【0317】

【0318】

【0319】

【0320】

【0321】

【0322】

【0323】

【0324】

【0325】

【0326】

【0327】

【0328】

【0329】

【0330】

【0331】

【0332】

【0333】

【0334】

【0335】

【0336】

【0337】

【0338】

【0339】

【0340】

【0341】

【0342】

【0343】

【0344】

【0345】

【0346】

【0347】

【0348】

【0349】

【0350】

【0351】

【0352】

【0353】

【0354】

【0355】

【0356】

【0357】

【0358】

【0359】

【0360】

【0361】

【0362】

【0363】

【0364】

【0365】

【0366】
【数5】

【0367】

【0368】

【0369】

【0370】

【0371】

【0372】

【0373】

【0374】

【0375】

【0376】

【0377】

【0378】

【0379】

【0380】

【0381】

【0382】

【0383】

【0384】

【0385】

【0386】

【0387】

【0388】

【0389】

【0390】

【0391】

【0392】

【0393】

【0394】

【0395】

【0396】

【0397】

【0398】

【0399】

【0400】

【0401】

【0402】

【0403】

【0404】

【0405】

【0406】

【0407】

【0408】

【0409】

【0410】

【0411】

【0412】

【0413】

【0414】
【表8】

【0415】
【表9】

【0416】
【表10】

【0417】
【表11】

【0418】
【表12】

【0419】
【表13】

【0420】
【表14】

【0421】
【表15】

【0422】
【表16】

【0423】
【表17】

【0424】
【表18】

【0425】
【表19】

【0426】
【表20】

【0427】
【表21】

【0428】
【表22】

【0429】
【表23】

【0430】
【表24】

【0431】
【表25】

【0432】
【表26】

【0433】
【表27】

【0434】
【表28】

【0435】
【表29】

【0436】
【表30】

【0437】
【表31】

【0438】
【表32】

【0439】
【表33】

【0440】
【表34】

【0441】
【表35】

【0442】
【表36】

【0443】
【表37】

【0444】
【表38】

【0445】
【表39】

【0446】
【表40】

【0447】
【表41】

【0448】
【表42】

【0449】
【表43】

【0450】
【表44】

【0451】
【表45】

【0452】
【表46】

【0453】
【表47】

【0454】
【表48】

【0455】
【表49】

【0456】
【表50】

【0457】
【表51】

【0458】
【表52】

【0459】
【表53】

【0460】
【表54】

【0461】
【表55】

【0462】
【表56】

【0463】
【表57】

【0464】
【表58】

【0465】
【表59】

【0466】
【表60】

【0467】
【表61】

【0468】
【表62】

【0469】
【表63】

【0470】
【表64】

【0471】
【表65】

【0472】
【表66】

【0473】
【表67】

【0474】
【表68】

【0475】
【表69】

【0476】
【表70】

【0477】
【表71】

【0478】
【表72】

【0479】
【表73】

【0480】
【表74】

【0481】
【表75】

【0482】
【表76】

【0483】
【表77】

【0484】
【表78】

【0485】
【表79】

【0486】
【表80】

【0487】
【表81】

【0488】
【表82】

【0489】
【表83】

【0490】
【表84】

【0491】
【表85】

【0492】
【表86】

【0493】
【表87】

【0494】
【表88】

【0495】
【表89】

【0496】
【表90】

【0497】
【表91】

【0498】
【表92】

【0499】
【表93】

【0500】
【表94】

【0501】
【表95】

【0502】
【表96】

【0503】
【表97】

【技術分野】
【0001】
(関連出願の相互参照)
本出願は、2003年12月17日に出願された米国仮特許出願第60/530,464号の利益を主張する。
【0002】
(政府のライセンス権に関する記載)
米国政府は、本発明における支払い済みのライセンスを有し、そして限定された状況において、特許権者が米国国立衛生研究所によって与えられたHG300035の条件で規定されるような妥当な条件でライセンスを他者に供与するよう要求する権利を有する。
【0003】
(発明の分野)
本発明は、多型マーカーを用いてイヌ科動物ゲノムへの1種以上のイヌ科動物個体群の寄与度を決定することに関する。
【背景技術】
【0004】
(発明の背景)
Canis familiaris(イエイヌ)は、表現型が分化し遺伝的に隔離された400種以上の血統に分岐した単一種であり、これらのうちの152種は、米国でアメリカンケンネルクラブ公認である(非特許文献1)。別個の犬血統は、形態学、挙動、および疾患罹患率の特有の集まりによって特徴付けられる(非特許文献2)。種々のイヌの形態学は、1000年前から存在し、そしてイヌ間での生殖隔離は、19世紀中頃において血統クラブおよび血統標準の出現とともに形式化された。その時以来、「品種の境界(breed barrier)」規則(すなわち、イヌの雌親と雄親の両方が登録された一員でなければ、いずれのイヌも品種登録された一員になり得ないという規則)が発布され、各犬種間で比較的閉じた遺伝子プール(genetic pool)を保証している。
【0005】
350種類以上の遺伝性障害は、純血種イヌ個体群を分離する(非特許文献3)。これらの多くはヒトの一般的な障害と類似しており、特有の形態を生成するために使用された積極的な同系交配プログラムの結果として、特定の品種または品種群に限定されている。
【0006】
個々の犬種を客観的に決定すること(例えば、特定品種に帰属するイヌであるという証明)のための多くの可能な使用が存在する。歴史的記録物は信頼度において品種ごとに変動するので、前の個体群情報に基づかない遺伝分析が、個体群構造を決定する最も直接的かつ正確な方法である。過去10年間にわたり、分子生物学的方法(molecular method)が、野生イヌ科動物種についての我々の理解を深めるために、そしてイエイヌとそれら野生種との関係を決定するために使用されてきた。ミトコンドリアDNA配列分析は、イエイヌとオオカミとの間の関係を説明し、4万〜10万年前に起こった複数の家畜化事象を解明する(非特許文献4;非特許文献5;非特許文献6)。しかし、ミトコンドリアDNAの進化は、現代の犬種間での関係を推論することを可能にするには遅すぎる。ほとんどの現代の犬種は、400年未満の間ずっと存在してきた。加えて、系統学的距離尺度および系統樹作成プログラムは、イヌ個体群において一般に観察されるような網目状進化を扱うための機能がない(非特許文献7;非特許文献8;非特許文献9)。ある先行研究は、核のマイクロサテライトの座位が5種からそれらの起源種へとイヌを割り当てるために使用され得そしてこれらの品種間で大きな遺伝的距離を示すことを示した(非特許文献10)。別の研究は、28種の収集物中の2種のペアの関連性を検出するためにマイクロサテライトを使用したが、品種間の広範な系統学的関係を証明し得なかった(非特許文献9)。このような関連性を見出すことの失敗は、マイクロサテライトの座位の特質(非特許文献9)、検査した限定された数の品種、またはこの研究において使用された分析方法を反映し得る。その代わりに、この失敗は、大部分の品種の最も近い起源およびそれらの生物における先祖型の混合によって、純血種イヌ個体群における複雑な構造を反映し得る。
【先行技術文献】
【非特許文献】
【0007】
【非特許文献1】CrowleyおよびAdelman編、American Kennel Club「The Complete Dog Book」、Howell Book Hues,New York,NY、1998年
【非特許文献2】Ostranderら、「Trends in Genetics」、2000年、第16号、p.117−23
【非特許文献3】Pattersonら、J.Am.Vet.Med.Assoc.、1988年、第193号、p.1131
【非特許文献4】Vilaら、Science、1997年、第276号、p.1687−9
【非特許文献5】Savolainenら、Science、2002年、第298号、p.1610−3
【非特許文献6】Leonardら、Science、2002年、第298号、p.1613−6
【非特許文献7】Zajcら、Mamm.Genome、1997年 第8巻、第3号、p.182−5
【非特許文献8】KoskinenおよびBredbacka、Animal Genetics、2000年、第31号、p.310−17
【非特許文献9】Irionら、J.Hered.、2003年、第94巻、第1号、p81−7
【非特許文献10】Koskinen、Anim.Genet,、2003年、第34号、p297
【発明の概要】
【発明が解決しようとする課題】
【0008】
関連する品種群を規定する方法、および個々のイヌゲノムへの品種の寄与度を明確に同定する方法の必要性が存在する。本発明は、この必要性および他の必要性に取り組む。
【課題を解決するための手段】
【0009】
(発明の要旨)
一局面において、本発明は、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を提供する。この方法は、以下の工程を包含する:(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および(b)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程。工程(b)において、各イヌ科動物ゲノム個体群プロファイルは、上記イヌ科動物個体群におけるマーカーセットについての遺伝子型情報を含む。このマーカーセットは、少なくとも約5個のマーカー(例えば、イヌ科動物ゲノムの地図上に示される少なくとも約5個のマーカー)を含み得る。本発明の方法における使用に適する例示的なマーカーとしては、例えば、マイクロサテライトマーカー、一塩基多型(SNP)、ミトコンドリアマーカー、および制限酵素断片長多型が挙げられる。例えば、上記マーカーセットは、表2に示すSNPマーカーのうちの少なくとも5個、および/または、表1に示すマイクロサテライトマーカーのうちの少なくとも5個を含み得る。上記マーカーセットは、1つ以上の個体群特異的マーカー(例えば、1つ以上の個体群特異的SNPマーカー、または1つ以上の個体群特異的マイクロサテライトマーカー)を含み得る。例えば、1つ以上のSNPマーカーは、372c5t−82、372e13t−57、372m6t−88、372m23t−76、373a15t−112、373e1t−50、373elt−130、373g19t−246、373i8s−224、373k8s−181、372c5s−168、372C15S−196、372e15s−71、および373a21t−93からなる群より選択され得る。
【0010】
上記マーカーセットの各々についての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性は、当該分野において標準的な方法(例えば、ハイブリダイゼーション法、ポリメラーゼ連鎖反応法、サイズ分画法、DNA塩基配列決定法など)を用いて得られ得る。例えば、上記方法の工程(a)は、上記マーカーセットの各々に特異的なプライマーを用いて、上記試験イヌ科動物のゲノムDNAを増幅する工程、およびこの増幅産物のサイズを決定する工程を包含し得る。工程(a)はまた、上記マーカーセットの各々に特異的なプライマーを用いて、上記試験イヌ科動物のゲノムDNAを増幅する工程、およびこの増幅産物のヌクレオチド配列を決定する工程を包含し得る。いくつかの実施形態において、上記プライマーは、配列番号1〜200からなる群より選択される。いくつかの実施形態において、上記プライマーは、配列番号1〜244〜327からなる群より選択される。
【0011】
イヌ科動物個体群プロファイルにおける遺伝子型情報は、例えば、そのイヌ科動物個体群の一員である1種以上のイヌ科動物におけるマーカーセット中のほとんどまたはすべてのマーカーの片方または両方の対立遺伝子の同一性、および/または、そのイヌ科動物個体群におけるマーカーセット中のほとんどまたはすべてのマーカーの少なくとも1つの対立遺伝子について推定された対立遺伝子頻度のような情報を含み得る。イヌ科動物個体群プロファイル中の推定された対立遺伝子頻度の各々は、代表的には、このイヌ科動物個体群の一員である少なくとも2つのイヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性に基づいている。イヌ科動物個体群プロファイルデータベースは、約5個と約数百個のとの間のイヌ科動物個体群プロファイル(例えば、少なくとも100個のイヌ科動物個体群プロファイル)を含み得る。いくつかの実施形態において、上記イヌ科動物個体群プロファイルは、登録種(例えば、アメリカンケンネルクラブに登録された品種)のプロファイルを含む。
【0012】
いくつかの実施形態において、上記マーカーセットは、約1500個より少ないSNPマーカーを含み、そして上記方法は、上記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する。いくつかの実施形態において、上記マーカーセットは、約200個より少ないSNPマーカー(例えば、約100個のSNPマーカー、または約50個のSNPマーカー)を含み、そして上記方法は、上記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する。
【0013】
上記方法の工程(b)において、1種以上のイヌ科動物個体群が上記試験イヌ科動物ゲノムへ寄与した可能性は、任意の適切なアルゴリズム(例えば、ベイジアンモデルベースのクラスタリングアルゴリズムまたは割り当てアルゴリズム)を用いて決定され得る。いくつかの実施形態において、工程(b)は、特定のイヌ科動物個体群が上記試験イヌ科動物ゲノムへ寄与した確率を決定する工程を包含し、この確率は、この試験イヌ科動物ゲノム中の対立遺伝子がこの特定のイヌ科動物個体群中で生じる条件付き確率を、この試験イヌ科動物ゲノム中の対立遺伝子が上記データベース内の、各イヌ科動物個体群中で生じる条件付き確率の合計で割って決定することにより決定される。いくつかの実施形態において、工程(b)は、上記試験イヌ科動物ゲノムへの2つ以上の遺伝的に関連するイヌ科動物個体群の寄与度の間を、この試験イヌ科動物ゲノム中の対立遺伝子をこの2つ以上の遺伝的に関連するイヌ科動物個体群プロファイルを含むデータベースと比較することによって識別する工程を包含する。例示的な遺伝的に関連したイヌ科動物個体群としては、ベルジアンシープドッグおよびベルジアンタービュレン;コリーおよびシェトランドシープドッグ;ウィペットおよびグレイハウンド;シベリアンハスキーおよびアラスカンマラミュート;マスチフおよびブルマスチフ;グレータースイスマウンテンドックおよびバーニーズマウンテンドッグ;ウェストハイランドホワイトテリアおよびケアーンテリア;ならびにラサアプソ、シーズー、およびペキニーズが挙げられるが、これらに限定されない。いくつかの実施形態において、本発明の方法は、上記試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する文書を提供する工程をさらに包含する。この文書は、上記試験イヌ科動物ゲノムまたは上記試験イヌ科動物へ寄与した1種以上のイヌ科動物個体群に関する情報(例えば、健康に関する情報(例えば、疾患の素因)、保険情報、または任意の他の種類の情報)を提供し得る。上記文書はまた、上記試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度についての証明書を提供し得る。いくつかの実施形態において、上記文書は、上記試験イヌ科動物のゲノムへ寄与した1種以上のイヌ科動物個体群についての表示(representation)(例えば、写真、図、または他の描写)を提供する。
【0014】
いくつかの実施形態において、本発明は、1種以上のイヌ科動物個体群を規定する方法を提供する。この方法は、以下の工程を包含する:(a)各イヌ科動物ゲノムセットについて、各マーカーセットのついての片方または両方の対立遺伝子の同一性を得る工程;および、(b)上記イヌ科動物ゲノムセットの1つ以上の一員が、統計的モデリングを用いて各マーカーについて1セットの対立遺伝子頻度によって特徴付けられる別個のイヌ科動物個体群を規定する可能性を決定することによって、1種以上のイヌ科動物個体群を規定する工程。
【0015】
別の局面において、本発明は、各マーカーセットについてイヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得るために、核酸配列を含む基材を提供する。
【0016】
さらなる局面において、本発明は、イヌ科動物個体群の区別に使用するために、媒体上に保存されたデータ構造を含むコンピュータ読み取り可能な媒体を提供し、このデータ構造は:(a)マーカーの名称またはこのマーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および(b)イヌ科動物個体群におけるそのマーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、そのマーカーフィールドのインスタンス化およびこの遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールドを備える。例えば、この遺伝子型情報フィールドは、イヌ科動物個体群におけるマーカー(例えば、SNPマーカー)の対立遺伝子頻度の推定値を保存することが可能であり得る。上記遺伝子型情報フィールドはまた、そのイヌ科動物個体群の一員である1種以上のイヌ科動物における各マーカーセットの片方または両方の対立遺伝子の同一性を保存することが可能であり得る。いくつかの実施形態において、上記コンピュータに読み取り可能媒体は、以下をこの媒体上に保存したものを有する基板を備える:
(a)イヌ科動物個体群の区別に使用するためのデータ構造であって、このデータ構造は;
(i)マーカーの名称またはこのマーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(ii)イヌ科動物個体群におけるそのマーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、このマーカーフィールドのインスタンス化およびこの遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、データ構造;ならびに
(b)イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を実施するための、コンピュータが実行可能な命令であって、以下の工程:
(i)各マーカーセットについて試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(ii)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物プロファイルを含むデータベースとを比較することによって決定する工程であって、イヌ科動物個体群プロファイルの各々は、そのイヌ科動物個体群における上記マーカーセットについての遺伝子型情報を含む、工程
を包含する、コンピュータが実行可能な命令を備える、コンピュータに読み取り可能な情報を含む。
【0017】
本発明の上述の局面および付随する多くの利益は、添付の図面と一緒に以下の詳細な説明を参照することによってより良く理解されるので、より容易に理解される。
【0018】
例えば、本発明は、以下の項目を提供する。
(項目1)
イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法であって、以下の工程:
(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(b)該試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、該試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程であって、各イヌ科動物個体群プロファイルは、該イヌ科動物個体群におけるマーカーセットに関する遺伝子型情報を含む、工程
を包含する、方法。
(項目2)
項目1に記載の方法であって、前記マーカーセットは、少なくとも約5個のマーカーを含む、方法。
(項目3)
項目1に記載の方法であって、前記マーカーセットは、マイクロサテライトマーカーを含む、方法。
(項目4)
項目3に記載の方法であって、前記マイクロサテライトマーカーは、表1に示すマイクロサテライトマーカーのうちの少なくとも5個を含む、方法。
(項目5)
項目1に記載の方法であって、前記マーカーセットは、一塩基多型(SNP)を含む、方法。
(項目6)
項目5に記載の方法であって、前記SNPマーカーは、表2に示すSNPマーカーのうちの少なくとも5個を含む、方法。
(項目7)
項目1に記載の方法であって、前記マーカーセットは、1つ以上の個体群特異的マーカーを含む、方法。
(項目8)
項目7に記載の方法であって、前記1つ以上の個体群特異的マーカーは、1つ以上のSNPマーカーを含む、方法。
(項目9)
項目8に記載の方法であって、前記1つ以上のSNPマーカーは、372c5t−82、372e13t−57、372m6t−88、372m23t−76、373a15t−112、373e1t−50、373elt−130、373g19t−246、373i8s−224、373k8s−181、372c5s−168、372C15S−196、372e15s−71、および373a21t−93からなる群より選択される、方法。
(項目10)
項目1に記載の方法であって、イヌ科動物個体群プロファイルにおける前記遺伝子型情報は、前記マーカーセットの各々の片方または両方の対立遺伝子の同一性を含む、方法。
(項目11)
項目1に記載の方法であって、イヌ科動物個体群プロファイルにおける前記遺伝子型情報は、前記マーカーセットの各々の少なくとも片方の対立遺伝子についての対立遺伝子頻度を含む、方法。
(項目12)
項目1に記載の方法であって、前記イヌ科動物個体群プロファイルデータベースは、約5個と約500個と間のイヌ科動物個体群プロファイルを含む、方法。
(項目13)
項目1に記載の方法であって、前記イヌ科動物個体群プロファイルデータベースは、少なくとも約5種のアメリカンケンネルクラブ公認血統種についてプロファイルを含む、方法。
(項目14)
項目1に記載の方法であって、前記マーカーセットは、約1500個よりも少ないSNPマーカーを含み、そして該方法は、前記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する、方法。
(項目15)
項目1に記載の方法であって、前記マーカーセットは、約200個よりも少ないSNPマーカーを含み、そして該方法は、前記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する、方法。
(項目16)
項目1に記載の方法であって、工程(a)は、前記マーカーセットの各々に特異的なプライマーを用いて、前記試験イヌ科動物のゲノムDNAを増幅する工程、および該増幅産物のサイズを決定する工程を包含する、方法。
(項目17)
項目1に記載の方法であって、工程(b)は、遺伝子型クラスタリングプログラムを用いる工程を包含する、方法。
(項目18)
項目1に記載の方法であって、工程(b)は、割当てアルゴリズムを用いる工程を包含する、方法。
(項目19)
項目1に記載の方法であって、工程(b)は、特定のイヌ科動物個体群が前記試験イヌ科動物ゲノムへ寄与した確率を決定する工程を包含し、該確率は、該試験イヌ科動物ゲノム中の対立遺伝子が該特定のイヌ科動物個体群中で生じる条件付き確率を、該試験イヌ科動物ゲノム中の対立遺伝子が前記データベース内の、各イヌ科動物個体群中で生じる条件付き確率の合計で割って決定することにより決定される、方法。
(項目20)
項目1に記載の方法であって、工程(b)は、前記試験イヌ科動物ゲノムへの2つ以上の遺伝的に関連するイヌ科動物個体群の寄与度の間を、該試験イヌ科動物ゲノム中の対立遺伝子を該2つ以上の遺伝的に関連するイヌ科動物個体群プロファイルを含むデータベースと比較することによって識別する工程を包含する、方法。
(項目21)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ベルジアンシープドッグおよびベルジアンタービュレンを含む、方法。
(項目22)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、コリーおよびシェトランドシープドッグを含む、方法。
(項目23)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ウィペットおよびグレイハウンドを含む、方法。
(項目24)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、シベリアンハスキーおよびアラスカンマラミュートを含む、方法。
(項目25)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、マスチフおよびブルマスチフを含む、方法。
(項目26)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、グレータースイスマウンテンドックおよびバーニーズマウンテンドッグを含む、方法。
(項目27)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ウェストハイランドホワイトテリアおよびケアーンテリアを含む、方法。
(項目28)
項目20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ラサアプソ、シーズー、およびペキニーズを含む、方法。
(項目29)
項目1に記載の方法であって、前記方法は、前記試験イヌ科動物ゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する文書を提供する工程をさらに包含する、方法。
(項目30)
項目29に記載の方法であって、前記文書は、前記試験イヌ科動物ゲノムまたは前記イヌ科動物へ寄与した前記1種以上のイヌ科動物個体群に関する情報を提供する、方法。
(項目31)
項目30に記載の方法であって、前記情報は、健康に関する情報である、方法。
(項目32)
項目30に記載の方法であって、前記情報は、保険情報である、方法。
(項目33)
項目29に記載の方法であって、前記文書は、1種以上のイヌ科動物個体群の前記試験イヌ科動物ゲノムのゲノムへの寄与度の証明書を提供する、方法。
(項目34)
項目29に記載の方法であって、前記文書は、前記試験イヌ科動物ゲノムへ寄与した1種以上のイヌ科動物個体群の表示を提供する、方法。
(項目35)
1種以上のイヌ科動物個体群を規定する方法であって、以下の工程:
(a)各イヌ科動物ゲノムセットについて、各マーカーセットのついての片方または両方の対立遺伝子の同一性を得る工程;および
(b)イヌ科動物ゲノムセットの1つ以上の一員が、統計的モデリングを用いて各マーカーについて1セットの対立遺伝子頻度によって特徴付けられる別個のイヌ科動物個体群を規定する可能性を決定することによって、1種以上のイヌ科動物個体群を規定する工程を包含する、方法。
(項目36)
コンピュータに読み取り可能な媒体であって、イヌ科動物個体群の区別に使用するために、該媒体上に保存されたデータ構造を含み、該データ構造は:
(a)マーカーの名称または該マーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(b)イヌ科動物個体群における該マーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、該マーカーフィールドのインスタンス化および該遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、コンピュータに読み取り可能な媒体。
(項目37)
コンピュータに読み取り可能な媒体であって、以下:
(a)イヌ科動物個体群の区別に使用するために該媒体上に保存されたデータ構造であって、該データ構造は;
(i)マーカーの名称または該マーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(ii)イヌ科動物個体群における該マーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、該マーカーフィールドのインスタンス化および該遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、データ構造;ならびに
(b)イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を実施するための、コンピュータが実行可能な命令であって、以下の工程:
(i)各マーカーセットについて試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(ii)該試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、該試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物プロファイルを含むデータベースとを比較することによって決定する工程であって、イヌ科動物個体群プロファイルの各々は、該イヌ科動物個体群における該マーカーセットについての遺伝子型情報を含む、工程
を包含する、コンピュータが実行可能な命令;
を該媒体上に保存したものを備える、コンピュータに読み取り可能な媒体。
【図面の簡単な説明】
【0019】
【図1】図1は、2種のイヌ科動物個体群に対する疾患素因に関する情報とともに、試験イヌ科動物(ファイド(Fido))ゲノムへの2種のイヌ科動物個体群(ボーダーコリーおよびブルマスチフ)の寄与度を表示する例示的な文書を示す。
【図2】図2は、実施例4に記載されるように、85種の犬種とハイイロオオカミとのコンセンサス近隣結合木(consensus neighbor−joining tree)を示す。統計学的支持によって枝を形成する9種が示される。残りの76種は、わずかに系統学的構造を示し、そして簡略化のために「他のすべての品種」と標識した1つの枝にまとめている。このコンセンサスを形成した木は、コード距離尺度に基づいている。このデータについて500回のブートストラップ複製が行われ、そして各枝を支持するブートストラップの比が、複製で50%を超える場合には、支持された枝に関して百分率で、対応するノードにて示される。木の根にあるオオカミ個体群は、8個体から構成される。この個体は、以下の各国々(中国、オマーン、イラン、スウェーデン、イタリア、メキシコ、カナダ、および米国)に由来する1つの個体である。枝の長さは、ブートストラップ値に比例している。
【発明を実施するための形態】
【0020】
(好ましい実施形態の詳細な説明)
本明細書は、本明細書とともに提出された2枚のコンパクトディスクに含まれるファイルを、それらの全体を参考して本明細書により援用する。第1のコンパクトディスクは表3および表4を含み、第2のコンパクトディスクは、配列表を含む。
【0021】
本明細書中で具体的に規定されない限り、本明細書中で使用される全ての用語は、本発明の当業者が使用するのと同じ意味を有する。
【0022】
第一の局面において、本発明は、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を提供する。この方法は、以下の工程:
(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(b)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程であって、この工程において各イヌ科動物個体群プロファイルは、このイヌ科動物個体群におけるマーカーセットに関する遺伝子型情報を含む、工程
を包含する。
【0023】
本明細書中で使用される場合、用語「イヌ科動物個体群の寄与度を決定する」とは、1種以上のイヌ科動物個体群が試験イヌ科動物ゲノムへ寄与したか否かについて推論をするために、イヌ科動物個体群の寄与度を統計学的方法を使用して推定または推測することをいう。
【0024】
本明細書中で使用される場合、用語「イヌ科動物」とは、オオカミ、ジャッカル、キツネ、コヨーテ、およびイエイヌを含む、Canidae科の一員である動物をいう。例えば、イヌ科動物は、イエイヌ、オオカミ、またはCanidae科の一種以上からの何らかの遺伝的寄与度を有する動物であり得る。用語「イヌ科動物個体群」は、血統関係にあるイヌ科動物の一群(例えば、イエイヌ種)をいう。用語「品種(breed)」とは、ヒトによって制御された状況下で選択されている比較的均一な表現型の形質を有する種内の動物の一群をいう。例えば、アメリカンケンネルクラブ(AKC)は、7種の群(ハーディング、ハウンド、ノンスポーティング、スポーティング、テリア、トイ、およびワーキング)に分かれる152種を認めている(American Kennel Club(1998)「The Complete Dog Book」、CrowleyおよびAdelman編、Howell Book Hues、New York、NY)。本発明の方法は、任意の犬種の遺伝的寄与度を推定するために使用され得る。この任意の犬種としては、アフガンハウンド、エアデールテリア、秋田犬、アラスカンマラミュート、アメリカンエスキモードッグ、アメリカンフォックスハウンド、アメリカンへアレスラットテリア、アメリカンスタッフォードシャーテリア、アメリカンウォータースパニエル、オーストラリアンキャトルドッグ、オーストラリアンシェパード、オーストラリアンテリア、バセニー、バセットハウンド、ビーグル、ビアデッドコリー、ベドリントンテリア、ベルジアンラケノア、ベルジアンマリノア、ベルジアンシープドッグ、ベルジアンタービュレン、バーニーズマウンテンドッグ、ビションフリーゼ、ブラットハウンド、ボーダーコリー、ボーダーテリア、ボルゾイ、ボストンテリア、ブーヴィエデフランドル、ボイキンスパニエル、ボクサー、ブリアード、ブリタニー、ブルドッグ、ブラッセルグリフォン、ブルマスチフ、ブルテリア、ケアーンテリア、カーディガンウェルシュコーギー、キャバリアキングチャールズスパニエル、チェサピークベイレトリバー、チワワ、チャイニーズクレステッド、チャイニーズシャーペイ、チャウチャウ、クライバースパニエル、コッカースパニエル、コリー、カーリーコーテッドレトリバー、ダックスフンド、ダルメシアン、ダンディーディンモントテリア、ドーベルマンピンシェル、ドゴカナリオ、イングリッシュコッカースパニエル、イングリッシュフォックスハウンド、イングリッシュセッター、イングリッシュスプリンガースパニエル、エントレブッハーマウンテンドッグ、フィールドスパニエル、フラットコーテッドレトリバー、フレンチブルドッグ、ジャーマンロングヘアードポインター、ジャーマンシェパードドッグ、ジャーマンショートヘアードポインター、ジャーマンワイヤーヘアードポインター、ジャイアントシュナウザー、ゴールデンレトリバー、ゴードンセッター、グレートデーン、グレートピレニーズ、グレートスミスマウンテンドッグ、グレイハウンド、ハリヤー、ハバニーズ、イビザンハウンド、アイリッシュセッター、アイリッシュテリア、アイリッシュウォータースパニエル、アイリッシュウルフハウンド、イタリアングレイハウンド、ジャックラッセルテリア、キースホンド、ケリーブルーテリア、コモンドール、クバーズ、ラブラドールレトリバー、レオンベルガー、ラサアプソ、ローシェン、マルチーズ、スタンダードマンチェスターテリア、トイマンチェスターテリア、マスチフ、ミニチュアブルテリア、ミニチュアピンシャー、ミニチュアプードル、ミニチュアシュナウザー、ミュンスターレンダー、ナポリタンマスチフ、ニューファンドランド、ニューギニアシンギングドッグ、ノルウェジアンエルクハウンド、ノーリッチテリア、オールドイングリッシュシープドッグ、パピヨン、ペキニーズ、ウェルシュコーギーペンブローク、プチバセットグリフォンヴァンデーン、ファラオハウンド、ポインター、ポリッシュローランドシープドッグ、ポメラニアン、ポルトガルウォータードッグ、プレサカナリオ、パグ、プーリー、プーミー、ローデシアンリッジバック、ロットワイラー、セントバーナード、サルーキ、サモエド、スキッパーキ、スコティッシュディアハウンド、スコティッシュテリア、シルキーテリア、シェトランドシープドッグ、柴犬、シーズー、シベリアンハスキー、スムースフォックステリア、ソフトコーテッドウィートンテリア、スピノニイタリアーノ、スタッフォードシャーブルテリア、スタンダードプードル、スタンダードシュナウザー、サセックススパニエル、チベタンスパニエル、チベタンテリア、トイフォックステリア、トイプードル、ビズラ、ワイマラナー、ウェルシュスプリンガースパニエル、ウェルシュテリア、ウェストハイランドホワイトテリア、ワイヤーヘアードポインティンググリフォン、ウィペット、ヨークシャーテリアが挙げられるが、これらに限定されない。
【0025】
本発明の方法はまた、イヌ科動物個体群からの遺伝的寄与度を決定するために使用され得る。この個体群は、認められた品種のサブセット(例えば、特定のブリーダーが起源であるダルマシアンの一群)または品種として認められていないか未だ認められていないイヌ科動物の一群である。同様に、本発明の方法は、イエイヌではないイヌ科動物個体群からの遺伝的寄与度を決定するために使用され得る。
【0026】
本発明の方法の第1工程は、各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程を包含する。用語「マーカー」は、試験イヌ科動物ゲノムへのこれらのイヌ科動物個体群の遺伝的寄与度を推定するために有用である、本発明の方法において使用されたイヌ科動物個体群にわたって十分に情報価値のある、任意の多型のゲノム座(genomic locus)をいう。ゲノム座は、それが少なくとも2つの対立遺伝子を有する場合、多型である。用語「対立遺伝子」とは、ゲノム座の他の形態からその核酸配列によって区別され得るゲノム座の特定の形態をいう。従って、ゲノム座の異なる対立遺伝子は、その座位(locus)で代替的な核酸配列を表す。任意の個体のイヌ科動物ゲノムにおいて、各マーカーについて2つの対立遺伝子が存在する。両方の対立遺伝子が同じである場合、ゲノムは、そのマーカーにホモ接合性である。逆に、その2つの対立遺伝子が異なる場合、ゲノムは、そのマーカーにヘテロ接合性である。
【0027】
個体群特異的対立遺伝子は、あるイヌ科動物個体群においていくらかの頻度で存在する対立遺伝子であるが、比較イヌ科動物個体群からサンプル抽出されたイヌ科動物中で観察されていない(しかし、それらは、実に低い頻度で存在し得る)。個体群特異的対立遺伝子は、特定の個体群に個体を割り当てるために使用され得る。従って、個体群間の対立遺伝子頻度における差異は、遺伝的寄与度を決定するために使用され得る。
【0028】
「マーカーセット」とは、試験イヌ科動物ゲノムへの本発明の方法に使用されたイヌ科動物個体群の遺伝的寄与度を決定するために十分なマーカーの最小数をいう。この必要とされるマーカーの最小数は、以下にさらに記載されるように、使用されている特定のイヌ科動物個体群についてのマーカーの情報性(informativeness)に依存する。マーカーセットは、少なくとも約5個のマーカー、少なくとも約10個のマーカー、少なくとも約50個のマーカー、または約100個より多いマーカーを含み得る。
【0029】
本発明に従って使用され得る代表的なマーカーとしては、マイクロサテライトマーカー、ミトコンドリアマーカー、制限酵素断片長多型、および一塩基多型(SNP)が挙げられる。有用なイヌ科動物のマイクロサテライトマーカーとしては、ジヌクレオチドリピート(例えば、(CA)n)、トリヌクレオチドリピート、およびテトラヌクレオチドリピート(例えば、(GAAA)n)が挙げられるが、これらに限定されない(Franciscoら、(1996)Mamm.Genome 7:359−62;Ostranderら、(1993)Genomics 16:207−13)。本発明の方法における使用のための例示的なマーカーとしては、表1に示すマイクロサテライトマーカー、表2に示すSNPマーカー、およびGuyonら(2003)Proc.Natl.Acad.Sci U.S.A.100(9):5296−5301に記載されるマーカーが挙げられる。本発明の方法において使用されるマーカーセットは、表1のマイクロサテライトマーカーからの少なくとも約5個のマーカー、および/または、表2のSNPマーカーからの少なくとも約5個のマーカーを含み得る。いくつかの実施形態において、マーカーセットは、372c5t−82、372e13t−57、372m6t−88、372m23t−76、373a15t−112、373e1t−50、373elt−130、373g19t−246、373i8s−224、373k8s−181、372c5s−168、372C15S−196、372e15s−71、および373a21t−93からなる群より選択される。いくつかの実施形態において、約1500個よりも少ない数のSNPマーカーを含む1セットのマーカーは、試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定するために使用される。いくつかの実施形態において、約200個よりも少ない数のSNPマーカーを含む1セットのマーカーは、試験イヌ科動物ゲノムへの少なくとも約87種のイヌ科動物個体群の寄与度を決定するために使用される。
【0030】
本発明の方法に従って、各マーカーの片方または両方の対立遺伝子の同一性が、得られ得る。いくつかの実施形態において、試験イヌ科動物における片方または両方の対立遺伝子の同一性は、当該分野において標準的な方法を使用して実験的に決定され得る。例えば、ゲノムマーカーの片方または両方の対立遺伝子の同一性は、当該分野で公知の任意の遺伝子型決定方法を使用して決定され得る。例示的な遺伝子型決定方法としては、ハイブリダイゼーション法、ポリメラーゼ連鎖反応法(PCR)、サイズ分画法、DNA塩基配列決定法、DNAマイクロアレイ法、ビーズの高密度光ファイバーアレイ法(high density fiber−optic arrays of beads)(例えば、Jianbingら、(2003)Chin.Sci.Bull.48(18):1903−5を参照のこと)、プライマー伸長法、質量分析法(例えば、Jurinkeら、(2002)Meth.Mol.Biol.187:179−92を参照のこと)、およびホールゲノムサンプリング分析法(whole−genome sampling analysis)(例えば、Kennedyら、(2003)Nat.Biotechnol.21(10):1233−7を参照のこと)の使用が挙げられるが、これらに限定されない。試験イヌ科動物におけるマーカーの対立遺伝子の同一性はまた、既に決定されており、かつ例えば刊行された文献のような供給源から利用可能であり得る。
【0031】
いくつかの実施形態において、試験イヌ科動物のゲノムDNAは、マーカーに特異的なプライマーを使用して増幅され得、その後にこの増幅産物のサイズ分析または配列決定が続く。イヌ科動物ゲノム中のマーカーの片方または両方の対立遺伝子の同一性を得るための例示的な方法は、実施例1に記載される。いくつかの実施形態において、マイクロサテライトマーカーを含むゲノムDNAを増幅するために使用されたプライマーは、配列番号1〜200からなる群より選択されるが、他のプライマーおよび他のマイクロサテライトマーカーも使用され得る。いくつかの実施形態において、SNPマーカーを含むゲノムDNAを増幅するために使用されたプライマーは、配列番号244〜327からなる群より選択されるが、他のプライマーおよび他のSNPマーカーも使用され得る。422匹のイヌ科動物(85品種を表す414匹のイヌと8匹のオオカミとを含む)における68個〜100個のマイクロサテライトの対立遺伝子の同一性は、(同封して提出したコンパクトディスク中の)表3に示される。189種のイヌ科動物(67品種を表す186匹のイヌと2匹のオオカミと1匹のコヨーテとを含む)における100個のSNPマーカーの対立遺伝子の同一性は、(同封して提出したコンパクトディスク中の)表4に示される。
【0032】
本発明の方法の第1工程において使用されるマーカーセット中に含まれるマーカーの最小数は、使用される特定のイヌ科動物個体群のついてのマーカーの情報性に依存する。このマーカーの情報は、本発明の方法において使用されるイヌ科動物個体群内かつこれら個体群間で異なる対立遺伝子数の関数、これらの対立遺伝子の頻度、および座位における変異率の割合である。ゲノム座の多型の程度(degree of polymorphism)は、多型情報内容(PIC)の推定によって評価され得る。この多型情報量は、対立遺伝子数およびそれらの分配頻度の関数である。本発明の方法における使用に適切なマイクロサテライトマーカーについての例示的なPIC値は、表1に示される。本発明の方法における使用に適切なマーカーは、実施例1に示されるように、約0.65%の平均PIC値を有し得る。
【0033】
異なるイヌ科動物個体群中のマーカーの対立遺伝子数、およびイヌ科動物個体群内およびこれら個体群間での対立遺伝子頻度を決定する方法は、実施例1に記載される。例えば、マーカーごとの対立遺伝子の平均数、(ハーディワインバーグ平衡の推定に基づいて)予測されたヘテロ接合性、観察されたヘテロ接合性、および94匹のイヌ科動物(18品種を表す90匹のイヌと、4匹のオオカミとを含む)における95個のマイクロサテライトマーカー間での推定された同系交配係数は、実施例1に記載される。品種の境界の存在は、同種由来のイヌが異種由来のイヌよりも遺伝学的により類似しているはずであることを予測する。この予測を試験するために、品種の構成員(membership)に起因し得る個々のイヌ間における遺伝的多様性の割合が、推定された。マイクロサテライトデータ(68品種を表す328匹のイヌにおける96個のマーカーを含む)についての分子変動の分析は、実施例1に記載されるように、遺伝分散の合計の27%よりも多くの割合を占める品種間の分散を示した。同様に、SNPマーカーデータ(60品種を表す120匹のイヌにおける75個のSNPを含む)から計算された品種間の遺伝学的距離は、実施例1に記載されるように、FST=0.36であった。これらの観察は、少数の犬種を分析したこれまでの報告(Koskinen(2003)Anim.Genet.34:297;Irionら、(2003)J.Hered.94:81)と一致し、品種の境界が品種間で顕著な遺伝的隔離をもたらしていたということを予測し、そしてヒト個体群の間で見出されたより低い遺伝学的差異(代表的には、5%〜10%の範囲内)とは著しく異なる(Rosenbergら、(2002)Science 298:2381−5;Cavelli−Sforzaら、(1994)The History and Geography of Human Genes、Princeton University Press,Princeton)。イヌにおける品種間での変動は、家畜個体群について報告された範囲よりも高い(MacHughら、(1998)Anim.Genet.29:333;Lavalら、(2000)Gen.Sel.Evol.32:187)。犬種間での顕著な遺伝学的差異は、品種の構成員が、個々のイヌ科動物についての遺伝子型情報から決定され得ることを示す。
【0034】
マーカーの情報性に関するデータセットにおけるマーカーの別個の対立遺伝子の数の影響は、実施例2に示される。例えば、19種のイヌ科動物個体群および95個のマイクロサテライトマーカーの分析において、イヌ科動物の86%が、10個よりも多い別個の対立遺伝子をそれぞれ有した5個のマーカーを使用して、イヌ科動物の品種に正確に割当てられ、そしてイヌ科動物の95%が、10個よりも多い別個の対立遺伝子をそれぞれ有した10個のマーカーを使用して、イヌ科動物の品種に正確に割当てられた。1個〜3個の別個の対立遺伝子を有するマーカーについて、イヌ科動物の46%が、5個のマーカーを使用してイヌ科動物の品種に正確に割当てられ、そしてイヌ科動物の62%が、10個以上のマーカーを使用して正確に割当てられた。
【0035】
イヌ科動物個体群ごとに4種もしくは5種のイヌ科動物についての95個のマーカーに関する遺伝子型情報を使用して、19種のイヌ科動物個体群間で識別する能力に対する使用されたマーカー数の影響は、実施例2に示される。例えば、個体を正しいイヌ科動物個体群に100%首尾よく割当てるために必要とされるマーカーの最小数は、イヌ科動物個体群に依存して2個(ペキニーズ)と52個(アメリカンへアレステリア)との間に及んだ。100%の精度を有する選択されたイヌ科動物個体群を有する、19種のイヌ科動物個体群にわたって試験された94匹のすべての個体にうちの少なくとも90%をうまく割当てるために必要とされるマイクロサテライトマーカーの最小数は、8個(ペキニーズについて)と95個(プレサカナリオ、チワワ、およびアメリカンへアレステリアについて)との間に及んだ。
【0036】
本発明の第一局面の方法の第2工程は、試験イヌ科動物ゲノム中の対立遺伝子とイヌ科動物プロファイルを含むデータベースとを比較することによって、試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する工程を包含する。第2工程において、各イヌ科動物個体群プロファイルは、イヌ科動物個体群におけるマーカーセット中のマーカーの対立遺伝子に関する遺伝子型情報を含む。本明細書中において使用される「イヌ科動物個体群プロファイル」とは、イヌ科動物個体群におけるマーカーセットに関する遺伝子型情報の収集物をいう。従って、イヌ科動物個体群プロファイルは、イヌ科動物個体群におけるマーカーセット中のほとんどまたは全てのマーカーのほとんどまたは全ての対立遺伝子に関する遺伝子型情報を含み得る。例えば、イヌ科動物個体群プロファイルは、イヌ科動物個体群におけるマーカーセット中の各マーカーの各対立遺伝子に関する遺伝子型情報を含み得る。イヌ科動物個体群プロファイル中の遺伝子型情報は、例えば、そのイヌ科動物個体群の一員である1種以上のイヌ科動物におけるマーカーセット中のほとんどもしくは全てのマーカーの片方もしくは両方の対立遺伝子の同一性、および/または、そのイヌ科動物個体群におけるマーカーセット中のほとんどもしくは全てのマーカーの少なくとも1つの対立遺伝子について推定された対立遺伝子頻度のような情報を含み得る。「対立遺伝子頻度」とは、個体群中の対立遺伝子の発生率をいう。対立遺伝子頻度は、代表的には直接計数(direct counting)によって推定される。一般に、イヌ科動物個体群における対立遺伝子頻度は、そのイヌ科動物個体群の少なくとも約5種の一員中の各マーカーセットについての片方または両方の対立遺伝子の同一性を得ることによって推定される。「イヌ科動物個体群プロファイルデータベース」とは、本発明の例示的な方法において使用される全てのイヌ科動物個体群についてのイヌ科動物個体群プロファイルの収集物をいう。いくつかの実施形態において、イヌ科動物個体群プロファイルデータベースは、約5個と約500個の間のイヌ科動物プロファイル(例えば、約20個のイヌ科動物プロファイル、約50個のイヌ科動物プロファイル、または約100個のイヌ科動物プロファイル)を含む。
【0037】
試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する工程は、イヌ科ゲノムを特定のイヌ科動物個体群へ割当てる工程、および1種以上のイヌ科動物個体群に由来したイヌ科動物ゲノムの比を決定する工程の両方を包含する。この方法のいくつかの実施形態において、ベイジアンモデルベースのクラスタリングアプローチが使用される。個体を個体群に割り当てるために使用されるクラスタリング法の2つの広範なクラスが存在する(Pritchardら(2000)Genetics 155:945−59)。距離に基づく方法は、個体のすべてのペアの間の距離を提供するためにペアワイズ距離行列を計算する。各クラスターからの知見を考慮することによって進められるモデルベースの方法は、いくつかのパラメーターモデルからランダムに抽出される;次いで、各クラスターに対応するパラメーターについての推論が、標準的な統計学的方法を使用して、各個体のクラスターの構成員についての推論と一緒に行われる。任意の標準的な統計学的方法は、本発明の方法(最大尤度法、ブートストラップ手法、ベイジアン法、および遺伝子型データを分析するために使用され得る任意の他の統計学的手法を含む)において使用され得る。これらの統計学的方法は、当該分野において周知である。集団遺伝学研究のための多くのソフトウェアプログラムが開発されており、本発明の方法において使用され得る。このプログラムとしては、TFPGA、Arlequin、GDA、GENEPOP、GeneStrut、POPGENE(Labate(2000)Crop.Sci.40:1521−1528)、およびstructure(Pritchardら、(2000)Genetics 155:945−59)が挙げられるが、これらに限定されない。
【0038】
例示的なベイジアンモデルベースのクラスタリングアプローチは、遺伝子型クラスタリングプログラムであるstructure(Pritchardら、(2000)Genetics 155:945−59)によって提供される、このプログラムは、種内で個体群を規定するために有用であると証明されている(Rosenburgら、(2001)Genetics 159:699−713;Rosenburgら、(2002)Science 298:2381−5;Falushら、(2003)Genetics 164(4):1567−87)。structureによって使用されるクラスタリング方法は、個体群中で個体または関連する個体のセットを正確に配置するために遺伝子型または遺伝学的起源のいずれに関する以前の情報も必要としない。
【0039】
任意のマルチローカス(multi−locus)遺伝子型分析に有用なアルゴリズム(例えば従来の割り当てアルゴリズム)は、本発明の方法において使用され得る。適切なアルゴリズムとしては、RannalaおよびMountain、(1997)Proc.Natl.Acad.Sci.U.S.A.94:9197−9201と、Cornuetら、(1999)Genetics 153:1989−2000とに記載されるアルゴリズム、ならびにそれらの変形物が挙げられる。マルチプルローカス遺伝子型分析について利用可能な例示的なプログラムとしては、Doh(www2.biology.ualberta.ca/jbrzusto/Doh.phpにて利用可能)およびGeneClass(www.montpellier.inra.fr/URLB/geneclass/genecass.htmにて利用可能)が挙げられる。
【0040】
いくつかの実施形態において、本発明の方法は、特定のイヌ科動物個体群が試験イヌ科動物ゲノムへ寄与した条件付き確率を決定する工程を包含し、この条件付き確率は、試験イヌ科動物ゲノム中の対立遺伝子が特定のイヌ科動物個体群中で生じる条件付き確率を、試験イヌ科動物ゲノム中の対立遺伝子がデータベース内の、各イヌ科動物個体群中で生じる条件付き確率の合計で割って決定することにより決定される。
【0041】
本発明の方法のいくつかの実施形態は、試験イヌ科動物ゲノム中の対立遺伝子と遺伝学的に関連する2種以上のイヌ科動物個体群のプロファイルを含むデータベースとを比較することによって、試験イヌ科動物ゲノムへの遺伝学的に関連する2種以上のイヌ科動物個体群の寄与度の間を識別する工程を包含する。この遺伝学的に関連する2種以上のイヌ科動物個体群は、ベルジアンシープドッグおよびベルジアンタービュレン;コリーおよびシェトランドシープドッグ;ウィペットおよびグレイハウンド;シベリアンハスキーおよびアラスカンマラミュート;マスチフおよびブルマスチフ;グレータースイスマウンテンドックおよびバーニーズマウンテンドッグ;ウェストハイランドホワイトテリアおよびケアーンテリア;またはラサアプソ、シーズー、およびペキニーズを含み得る。
【0042】
94匹のイヌ科動物(18品種を表す90匹のイヌ科動物と4匹のオオカミとを含む)からの95個のマイクロサテライトマーカーについての遺伝子型情報に割り当てアルゴリズムを使用することにより、本発明の方法が、実施例2に記載されるように、99%の精度で各個々のイヌ科動物をその品種へと割り当てるために使用されている。実施例3に記載されるように、同じ遺伝子型情報に使用したクラスタリングアルゴリズムは、20種のイヌ科動物個体群を予測し、そして各イヌ科動物を99%の精度で1種の個体群へと割り当てた。
【0043】
72品種を表す341匹のイヌ科動物からの68個のマイクロサテライトマーカーについての遺伝子型情報に割り当てアルゴリズムを使用することにより、本発明の方法が、実施例2に記載されるように、イヌ科動物の96%を正しい品種へと割り当てるために使用されている。85品種を表す414匹のイヌ科個体群からの96個のマイクロサテライトマーカーについての遺伝子型情報に割り当てアルゴリズムを使用することにより、本発明の方法が、実施例4に記載されるように、イヌ科動物の99%を正しい品種へと割り当てるために使用されている。同様の結果が、クラスタリングアルゴリズムを使用して得られた。67品種を表す189匹のイヌ科動物からの100個のSNPマーカーについての遺伝子型情報に割り当てアルゴリズムを使用することにより、本発明の方法が、実施例6に記載されるように、99%よりも高い確率でイヌ科動物の80%を正しい品種へと割り当てるために使用されている。
【0044】
本発明の方法はまた、雑種イヌ科動物へのイヌ科動物個体群の寄与度を決定するために有用である。交配された(admixed)個体は、イヌ科動物個体群のおよそ50%を表す。個体の交配された(admixed)状態を検出するモデルは、群を以下の2つのクラスに分けて考えられ得る:先祖個体群の考えられる交配種(mixture)の各々について独特の対立遺伝子の組み合わせセットを必要とするモデル(NasonおよびEllstrand(1993)J.Hered.84:1−12;Epifanio&Philipp(1997)J.Hered.88:62−5)と、先祖個体群は特有の対立遺伝子を説明する組み合わせを含む必要がないがその代わりに個体間の対立遺伝子頻度における差異に確率論的に基づいて交配された(admixed)状態へと個体を割り当てる、ベイジアン法のモデル(Coranderら(2003)Genetics 163(1):367−74;AndersonおよびThompson(2002)Genetics 160:1217−29;Pritchardら(2000)Genetics 155:945−59;RannalaおよびMountain(1997)Proc.Natl.Acad.Sci.U.S.A.94:9197−9201)。後者のモデルセットが、各々の個体群/世代の組み合わせについてのベイジアン事後確率割り当てベクトルを考慮にいれ、それによって、その割り当てベクトルに組み入れられる不確定度分析を考慮に入れる際、それら後者のモデルセットは、ほとんどの個体群およびデータセット関して、より情報価値があるが、複数種の先祖個体群からの個体の正確な近い世代で交配された種(recent admixture)の割り当てのための既存のモデルは、それらモデルが開発されてきた際の範囲内(2世代の予測のみ、および数種の先祖個体群のみを考慮する程度まで)に制限される。例えば、AndersonおよびThompson(2002)の方法は、非連鎖マイクロサテライトデータを有する、2世代の2種の個体群のために開発されている。ナイーブベイジアン分類モデルは、先祖の部分個体群への交配種(mixture)への個体の確率論的割り当てのために、連鎖マイクロサテライト座位および非連鎖マイクロサテライト座位の情報、高次元の先祖個体群、ならびに高度に秩序だった(higher−ordered)世代系図を組み入れ、このモデルは、実施例7に記載される。このモデルは、実施例7に記載されるように、既存モデルの世代、部分個体群(subpopulation)、および連鎖の制限に対して同時に取り組み、そして2世代モデルおよび3世代モデルは、正確な交配種(admixture)の検出および割り当てのために実施されている。
【0045】
18品種の81匹のイヌ科動物と4匹のオオカミから構成される85匹のイヌ科動物からの95個のマーカーについての遺伝子型情報のインシリコでの交配(mix)に、クラスタリングアルゴリズムを使用することにより、本発明の方法は、実施例5に記載されるように、100%の精度で両親レベルにおけるインシリコでの交配を同定するために使用されている。本発明の方法また、実施例5に記載されるように、祖父母レベルおけるインシリコでの交配の検出においても非常に正確であり、そして曽祖父母レベルにおけるインシリコでの交配を検出においてもかなり正確であった。従って、本発明の方法は、純血種のイヌ(イヌ由来の1/2オオカミ交配種および1/4オオカミ交配種も同様)から、両親レベルおよび祖父母レベルにおける交配を識別するために使用され得、そして、雑種のイヌのゲノムにおける品種の寄与度を同定し得る。
【0046】
88品種の429匹のイヌ科動物からの96個のマーカーについての遺伝子型情報のインシリコでの交配にベイジアン分類モデルを使用することにより、本発明の方法は、実施例7に記載されるように、98%より高いF1交配種(mix)および94%より高いF2交配種の正確な割り当てのために使用されている。この160匹の既知の雑種イヌ科動物からの72個のマーカーについての遺伝子型情報に関するモデルを使用して、本発明の方法は、実施例7に記載されるように、96%より高いF1交配種および91%より高いF2交配種を正確に割り当てるために使用されている。
【0047】
本発明の方法は、試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する文書を提供する工程をさらに包含し得る。用語「文書」は、図表、証明書、カード、または任意の他の種類の書類をいう。文書は、試験イヌ科動物ゲノムへの1種以上のイヌ科動物個体群の寄与度を、数値形式または図画形式で表示し得る。例えば、文書としては、1種以上のイヌ科動物個体群の写真もしくは他の描写、図、または表示が挙げられ得る。文書はまた、決定された寄与度についての信頼値(例えば、80%、85%、90%、95%、または99%の信頼度)を提供し得る。いくつかの実施形態において、文書は、試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度の証明書を提供する。
【0048】
いくつかの実施形態において、文書は、試験イヌ科動物ゲノムまたは試験イヌ科動物へ寄与した1種以上のイヌ科動物個体群に関する情報をさらに提供する。試験イヌ科動物ゲノムへ寄与したイヌ科動物個体群に関する情報としては、イヌ科動物個体群の特徴および起源に関連する情報、または試験イヌ科動物の所有者にとって有用である任意の他の種類の情報が挙げられ得る。いくつかの実施形態において、情報は、健康に関する情報を含む。多くのイヌ科動物個体群は、特定の疾患または状態になりやすい素因を有する。例えば、アフガンハウンドは、緑内障、肝炎、および甲状腺機能低下症に罹りやすく;バセニーは、大腸菌腸炎およびピルビン酸キナーゼ欠乏症に罹りやすく;ビーグルは、膀胱癌および難聴に罹りやすく;バーニーズマウンテンドッグは、小脳変性症に罹りやすく;ボーダーテリアは、乏突起細胞腫に罹りやすく;そしてラブラドールレトリバーは、食物アレルギーに罹りやすい(Dr.Bob‘s All Creatures Site,Breed Predisposition to Disease and Congenital Conditions,http://www.petdoc.ws/BreedPre.htm; Pattersonら、(1998)J.Am.Vet.Med.Assoc.193:1131を参照のこと)。イヌにおいて発見された遺伝疾患に関して、46%は、1種または数種において、支配的または排他的に生じると考えられる(Pattersonら(1998)J.Am.Vet.Med.Assoc.193:1131)。従って、試験イヌ科動物ゲノムのゲノムへの1種以上のイヌ科動物個体群の寄与度に関する情報は、試験された個々の動物について健康上のリスクを前向きに検討する目的のために、雑種のイヌ科動物の所有者または世話人(専門家および非専門家の両方)にとって、特に有益である。例えば、ニューファンドランドとバーニーズマウンテンドッグとの交配種(mixture)であると見出される雑種のイヌは、一般的なイヌ個体群においてはまれな頻度で生じるがこれら特定の品種においてはかなりの頻度で生じる遺伝疾患について、活発にモニターされるべきである。従って、このタイプの雑種個体は、悪性組織球増殖症についてのスクリーニングから恩恵を受け(disease heritability of .298 in Bernese Mountain dogs,Padgettら、1995 J.Small Anim.Pract.36(3):93−8)、加えて、I型シスチン尿症の遺伝的スクリーニングからも恩恵を受ける(nonsense mutation isolated in Newfoundlands at exon 2 of SLC3A1 gene,Henthornら、(2000)Hum.Genet.107(4):295−303)。
【0049】
健康に関する情報としてはまた、可能性のある処置、特別食または製品、診断情報、および保険情報が挙げられ得る。試験イヌ科動物のゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する例示的な文書は、図1に示される。
【0050】
いくつかの実施形態において、本発明は、1種以上のイヌ科動物個体群を規定する方法を提供する。この方法は、(a)各イヌ科動物ゲノムセットについて、各マーカーセットのついての片方または両方の対立遺伝子の同一性を得る工程;および(b)イヌ科動物ゲノムセットの1つ以上の一員が、統計的モデリングを用いて各マーカーについて1セットの対立遺伝子頻度によって特徴付けられる別個のイヌ科動物個体群を規定する可能性を決定することによって、1種以上のイヌ科動物個体群を規定する工程を包含する。1種以上のイヌ科動物個体群を規定するための本発明の例示的な方法は、実施例3および実施例4に記載される。
【0051】
別の局面において、本発明は、各マーカーセットについてイヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を決定するための核酸配列を含む基材を、提供する。基材は、マーカーの対立遺伝子の同一性を決定するために適切な任意の形態であり得る。例えば、基材は、マイクロアレイまたはビーズの集合体の形態であり得る。
【0052】
さらなる局面において、本発明は、イヌ科動物個体群の区別に使用するために、媒体上に保存されたデータ構造を含むコンピュータ読み取り可能な媒体を提供し、このデータ構造は:マーカーの名称(例えば、SNPマーカー)またはこのマーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および、そのマーカーについての遺伝子型情報(例えば、イヌ科動物ゲノムにおけるマーカーの片方または両方の対立遺伝子の同一性、あるいはイヌ科動物個体群におけるマーカーの対立遺伝子頻度の推定値)を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、そのマーカーフィールドのインスタンス化およびこの遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールドを備える。
【0053】
「コンピュータに読み取り可能な媒体」とは、コンピュータによってアクセスされ得る任意の利用可能な媒体をいい、そして揮発性媒体および非揮発性媒体の両方の、取り外し可能な媒体および取り外しができない媒体が挙げられる。一例であり限定はしないが、コンピュータに読み取り可能な媒体は、コンピュータ記憶媒体および通信媒体を備え得る。コンピュータ記憶媒体としては、情報(例えば、コンピュータに読み取り可能な命令、データ構造、プログラムモジュール、または他のデータ)を保存するための任意の方法または技術で実施される、揮発性および非揮発性の両方の、取り外し可能な媒体および取り外しができない媒体が挙げられる。コンピュータ記憶媒体としては、RAM、ROM、EEPROM、フラッシュメモリまたは他のメモリー技術、CD−ROM、デジタル多用途ディスク(DVD)または他の光ディスク記憶装置、磁気カセット、磁気テープ、磁気ディスク記憶装置または他の磁気記憶デバイス、あるいは任意の他のコンピュータ記憶媒体が挙げられるが、それらに限定されない。通信媒体は、代表的には、コンピュータに読み取り可能な命令、データ構造、プログラムモジュール、または変調されたデータ信号(例えば、搬送波または、任意の情報伝達媒体を含む他の転送機構)で具体化する。用語「変調されたデータ信号」は、その信号の特性セットを1セット以上有する信号か、またはその信号のコード化情報のような方式で変更された信号を意味する。一例であり限定されないが、通信媒体としては、有線媒体(例えば、有線ネットワーク通信または直接配線された(direct−wired)通信)および無線媒体(例えば、音響媒体、RF赤外線媒体、および他の無線媒体)が挙げられる。上述の任意の組み合わせがまた、コンピュータに読み取り可能な媒体の範囲内に含まれるはずである。
【0054】
「データ構造」とは、データの概念的配置をいい、そして代表的には、列および行によって特徴付けられる。そのデータは、列と行の交差(row−column intersection)によって形成された各セルを占めるかまたは潜在的に占めている。本発明のコンピュータに読み取り可能な媒体中のデータ構造は、上述のように、マーカーフィールドおよび遺伝子型情報フィールドを含む。マーカーフィールドおよび遺伝子型情報フィールドのインスタンス化は、1つのレコードを提供し、そして1セットのレコードは、1つのイヌ科動物個体群プロファイルを提供する。従って、データ構造は、イヌ科動物個体群プロファイルのデータベースを作製するために使用され得る。
【0055】
いくつかの実施形態において、コンピュータに読み取り可能な媒体は、以下をこの媒体上に保存されたものを有する基板を備える:
(a)イヌ科動物個体群の区別に使用するためのデータ構造であって、このデータ構造は;
(i)マーカーの名称またはこのマーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(ii)そのマーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、このマーカーフィールドのインスタンス化およびこの頻度フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、データ構造;ならびに
(b)イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を実施するための、コンピュータが実行可能な命令であって、以下の工程:
(i)各マーカーセットについて試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(ii)この試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、この試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物プロファイルを含むデータベースとを比較することによって決定する工程であって、イヌ科動物個体群プロファイルの各々は、そのイヌ科動物個体群における上記マーカーセットについての遺伝子型情報を含む、工程
を包含する、コンピュータが実行可能な命令。
【0056】
以下の実施例は、本発明を実施するための現在考えられている最良の形態を単に例示するが、本発明を限定するために構成されるべきではない。
【実施例】
【0057】
(実施例1)
この実施例は、1セットのマーカーについての片方または両方の対立遺伝子の同一性を得るため、かつイヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定するのに適切なマーカーを選択するための、本発明の代表的な方法を説明する。
【0058】
(A.方法)
(1.サンプル収集およびDNA抽出)
8ヶ国(中国、オマーン、イタリア、イラン、米国(アラスカ)、カナダ(ケベック)、スウェーデン、メキシコ)からの、103品種の513匹のアメリカンケンネルクラブに登録されたイヌ、および8匹のハイイロオオカミからのイヌ科動物DNAサンプルを、ドッグショーおよびドッグクラブの特別イベントで、ボランティアから口内(頬)塗抹標本および/または血液サンプルを収集することで得た。同様に、これらは、郵送による提供によっても得られた。アメリカンケンネルクラブ登録番号および詳細な系統情報が、全てのイヌについて要求される。なぜなら、この実施例への参加は、共通の祖父母を有さない無関係のイヌには制限されたからである。系統情報がまた、サンプル抽出された個体の84%について収集された。多くの場合、5世代の系図が得られ、イヌは時折、曽祖父母レベルまたそれより高いレベルで、重複して現れるが、その一方で、完全連鎖検査は、同犬種間での高い無関連度(high degree of unrelatedness)を示している。系図が利用可能でなかった場合、これら個体について、無関連性(unrelatedness)を血統クラブの代表犬(representative)で検証した。個体イヌ科動物の各々に、イヌ科動物識別番号を与えた。品種および他のイヌ科動物個体群について使用される略称は、表5に示される。加えて、20種のAKC品種からの交配コンポーネント(admixture component)を含む160匹の雑種イヌ科動物からのDNAサンプルを、口内塗抹標本を収集することによって得た。
【0059】
口内塗抹標本を、アメリカンケンネルクラブ(AKC)ウェブサイト(http://www.akc.org/)より提案されるものと類似する様式で、細胞学的ブラシ(Medical Packaging Corp.,Camarillo,CA)を使用して収集した。DNAを、製造元(Qiagen,Valencia,CA)のプロトコルに従い、QiaAmp血液キットを使用して口内塗抹標本から抽出した。血液からのDNA抽出は、以前に記載されたように行った(Comstockら、(2002)Mol.Ecol.11:2489−98)。
【0060】
(2.マイクロサテライトマーカーの分析)
100個のジヌクレオチドマイクロサテライトマーカーを、イヌの3300個のマーカー地図上に現在位置決めされている1596個のマイクロサテライトから選択した(Guyonら、(2003)Proc.Natl.Acad.Sci U.S.A.100(9):5296−5301)(表1)。マーカーを情報性(informativeness)に基づいて選択し、PIC値として計算し、そして、38種すべての常染色体にわたって分布させた。選択されたマーカーは、0.65%(36%〜86%の範囲)の平均PIC値、および29.5Mb(21.5Mb〜50.9Mbの範囲)の平均間隔を有した。テトラヌクテオチドマイクロサテライトよりもジヌクレオチドマイクロサテライトを、品種同定を妨害し得る、観察された偽の変異(spurious mutation)の数を減らすように選択した。
【0061】
DNAサンプルを96ウェルプレート上に並べた。ポジティブコントロールを、一貫した対立遺伝子のビニングを保証するために各プレート上に含めた。PCRを、1ngのゲノムDNAおよび最終濃度の以下の試薬を含有する10μlの反応液中で行った。以下の試薬とは、16mMの硫酸アンモニウム、67mMのTris−HCl pH8.8、2.0mMのMgCl2、0.1mMのdNTP、300nMの順方向プライマー(配列番号1〜100)、逆方向プライマー(配列番号101〜200)、および色素標識したM13プライマー(PE Applied Biosystems,Foster City,CA USA)である。順方向プライマーを、5’末端上に19塩基のM13順方向(−29)配列(5’−CACGACGTTGTAAAACGAC−3’)(配列番号201)を含むように再設計した。6FAMTM、VICTM、NEDTM、またはPETTM(ABI,Foster City,CA)色素のいずれかで標識された0.25pmolのM13プライマー(配列番号201)を各反応液に加えることで、サンプルを標識した。PCRのインキュベーションを、標準的なプロトコル(例えば、Loweら、(2003)Genomics 82:86−95;http://www.fhcrc.org/science/dog_genome/dog.htmlを参照のこと)に従って行った。使用されたアニ−リング温度は、表1に示される。異なる色素で標識された4つのサンプルを、1枚の96ウェルプレート内で3μlの各反応混合液を混ぜ合わせることにより、PCRの終了後に、多重化した。サンプルを、16pmolのGeneScanTM−500LIZTMサイズ標準(ABI,Foster City,CA)を含有する2容量のHi−DiTMホルムアミド中で、製造元のプロトコルに従って変性させた。全てのサンプルを、対立遺伝子を分離するために、ABI 3730 DNA AnalyzerTM(PE applied Biosystems)キャピラリー電気泳動装置上にロードした。遺伝子型を、GeneMapperTMv3.0ソフトウェア(ABI,Foster City,CA)を使用してコールした。全てのコールを、手動で確認し、そして次に続く各実行(run)をビン(bin)外にある新たな対立遺伝子の出現についてスキャンした。4個のマーカーを、一貫して増幅できなかったので切り捨てた。
【0062】
(3.SNPの発見および遺伝子型決定)
50種のイヌ動物の細菌性人工染色体(BAC)を、イヌ科動物の放射線ハイブリッド地図(Guyonら(2003)Proc.Natl.Acad.Sci U.S.A.100(9):5296−5301)からランダムに選択した。Primer3プログラム(http://www.genome.wi.mit.edu/sci−bin/primer/primer3_www.cgiにて利用可能)を、BACの各末端配列から、プライマーを設計するために使用した。結果として平均334塩基対の単位複製配列が得られた。プライマーを、67種のイエイヌ種の189匹のイヌと、コヨーテとハイイロオオカミの19867塩基対の非連続ゲノム配列を増幅するために使用した。結果として得られたPCR産物を、標準的なABI色素ターミネ−ター化学を用いてABI3700キャピラリーシーケンサー(ABI,Foster City,CA)上で、標準的な方法を使用して配列決定し、そして再度、配列決定した。読み取った全ての配列を、Phred、Phrap、およびConsed(EwingおよびGreen(1998)Genome Res.8:186−94;Ewingら(1998)Genome Res.8:175−85;www.genome.washington.eduにて利用可能)を使用して整列させ、表示した。コンピュータプログラムPolyphredを、読み取った配列内およびその配列間で、多型領域(SNPおよび挿入/欠失の両方)を同定するために使用した(Nickersonら(1997)Nucl.Acids Res.25:2745−51、droog.mbt.washington.eduにて利用可能)。全ての対立遺伝子のコールを、手動で確認し、そのトレースを目視検査によって確認した。
【0063】
(4.統計分析)
分子分散分析(AMOVA)を、ハーディワインバーグ平衡の仮定の下で、GDA(LewisおよびZaykin(2001)Genetic Data Analysis:Computer Program for the Analysis of Allelic Data,Version 1.0(d16c)、http://lewis.eeb.uconn.edu/lewishome/softare.htmlにて利用可能)を使用して行った。分析において同系交配が考慮された場合、品種間の遺伝的変動の比について、同様の結果が得られた。
【0064】
各品種についての予測したヘテロ接合性を、Tajima不変推定量(Tajima(1989)Genetics 123:585−95)を使用して、対立遺伝子頻度から計算した。
【0065】
(B.結果)
(1.ジヌクレオチドマイクロサテライトの情報性)
422匹のイヌ科動物(85品種の414匹のイヌと8匹のオオカミとを含む)における68個〜100個のマイクロサテライトの対立遺伝子(増幅した領域の長さ)の同一性は、(本明細書とともに提出されたコンパクトディスク中の)表3に示される。148個の対立遺伝子が、特定のイヌ科動物個体群に特有であることが見出されている:1個は各々、ACKR、AUST、BORD、BOX、BULD、DACH、GOLD、GSHP、GSMD、IBIZ、KEES、NELK、PEKE、POM、ROTT、SFXT、TERVおよびWHIPに特有であり、2個は各々、BEAG、CAIR、HUSK、IRSE、MAST、OES、SCHP、SCWT、SPOOおよびSSHPに特有であり、3個は各々、AMAL、BMD、KOMO、NEWF、STBDおよびWSSPに特有であり、4個は各々、KUVZ、PNTRおよびPRESに特有であり、5個は各々、それぞれBSIJおよびSHARに特有であり、6個は、AKITに特有であり、そして、64個の対立遺伝子は、WOLFに特有である。
【0066】
6つの異なるデータセットを、実施例2〜5、および実施例7にさらに記載されるように、後の分析に使用した。第1のデータセットは、94匹のイヌ科動物(18品種の90匹のイヌ科動物と4匹のオオカミとを含む)における、95個のマイクロサテライトマーカー(マイクロサテライトマーカー1〜14、16、18〜21、23〜26、39〜100;表1参照のこと)についての遺伝子型情報を含んだ(データセット1;表6)。 第2のデータセットは、72品種の341匹のイヌ科動物における68個のマイクロサテライトマーカー(マイクロサテライトマーカー2〜8、11、12、14〜16、18〜21、23、24、26〜32、34〜36、38、41、42、44〜46、50、51、53、54、56、60〜64、67、68、70〜74、78、79、81〜83、85、87〜91、93〜98;表1参照のこと)についての遺伝型情報を含んだ(データセット2;表7)。第3のデータセットは、85品種の414匹のイヌ科動物における96個のマクロサテライトマーカー(マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100;表1参照のこと)についての遺伝型情報を含んだ(データセット3;表8)。第4のデータセットは、85匹のイヌ科動物(18品種の81匹のイヌと4種のオオカミとを含む)における96個のマイクロサテライトマーカー(マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100;表1参照のこと)についての遺伝型情報を含んだ(データセット4;表9)。第5のデータセットは、88品種の429匹のイヌ科動物における96個のマイクロサテライトマーカー(マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100;表1参照のこと)についての遺伝子型情報を含んだ。第6のデータセットは、(本明細書とともに提出されたコンパクトディスク中の)表3に示されるような、160種の雑種イヌ科動物における、表1中の72個のマイクロサテライトマーカーについての遺伝子型情報を含んだ。
【0067】
多型マーカーの比、マーカーごとの対立遺伝子の平均数、多型マーカーごとの対立遺伝子の平均数、(ハーディワインバーグ平衡の推定に基づいて)予測されたヘテロ接合性、観察されたヘテロ接合性、およびデータセット1中の95個のマイクロサテライトマーカーにわたる推定された同系交配係数は、表10に示される。Tajima不変推定量を使用して、96個のマイクロサテライト(データセット3)にわたって平均された85種のイヌ科動物個体群の予測されたヘテロ接合性は、表11に示される。
【0068】
品種の境界の存在は、同種由来のイヌが異種由来のイヌよりも遺伝学的により類似しているはずであることを予測する。この予測を試験するために、品種の構成員(membership)に起因し得る個々のイヌ間における遺伝的多様性の割合が、推定された。マイクロサテライトデータ(85品種の414匹のイヌにおける96個のマーカーを含む)(データセット3、表8)についての分子変動の分析は、遺伝分散の合計の27%よりも多くの割合を占める品種間の分散を示した。
【0069】
(2.SNPマーカーの情報性)
67種のイエイヌの189匹のイヌ科動物、コヨーテ、およびオオカミを使用して、およそ20Kbの非連続のイヌ科動物ゲノム配列中の100個の多型部位を、表2に記載されるように同定した。これらは、92個の一塩基置換、および11個の挿入または欠失変異(長さ1ヌクレオチド〜8ヌクレオチドの範囲)を含む。189匹のイヌ科動物(67品種の186匹のイヌ、2匹のオオカミ、および1匹のコヨーテを含む)における100個のSNPマーカーについての対立遺伝子の同一性は、(本明細書とともに提出されたコンパクトディスク中の)表4に示される。60品種の120匹のイヌからの75個のSNPにおけるマイナー対立遺伝子頻度は、表2に示されるように、0.4%〜48%の範囲に及んだ。これらのうちの14個のSNPは、以下の品種に特異的であった:372c5t−82(イングリッシュシェパード)、372e13t−57(コッカースパニエル)、372m6t−88(イングリッシュシェパード)、372m23t−76(アラスカンマラミュート)、373a15t−112(チェサピークベイレトリバー)、373e1t−50(スピノニイタリアーノ)、373elt−130(スコティッシュディアハウンド)、373g19t−246(ボルゾイ)、373i8s−224(チェサピークベイレトリバー)、373k8s−181(チベタンテリア)、372c5s−168(秋田犬)、372C15S−196(ラブラドールレトリバー)、372e15s−71(フィールドスパニエル)、および373a21t−93(イタリアングレイハウンド)。
【0070】
すべてのイヌが1個体群として考慮された場合、観察されたヘテロ接合性(Tajima&Nei(1984)Mol.Biol.Evol.1:269−85)は、8×10−4であった。これは、ヒト個体群において見られる値と基本的に同じである(Sachidanandamら、(2001)Nature 409:928−33;Venterら(2001)Science 291:3104−51)。しかし、品種が分岐される場合、最小非近交系(スコティッシュディアハウンド;2.5×10−4)から最大非近交系(イングリッシュスパニエル;1.0×10−3)の間のヘテロ接合性における範囲は、4倍である。60品種の120匹のイヌにおける75個のSNPについてのSNPデータから計算された品種間の遺伝的距離は、FST=0.36であった。
【0071】
Tajima不変推定量を使用し、75個のSNP座位における対立遺伝子頻度(データセット3)に基づいて予測された60種のイヌ科動物個体群のヘテロ接合性は、表12に示される。各品種は、2匹のイヌで表される。
【0072】
(実施例2)
この実施例は、94匹のイヌ科動物からの95個のマイクロサテライトマーカーについての遺伝子型情報、および341匹のイヌ科動物からの68個のマイクロサテライトマーカーについての遺伝子型情報に関して、割り当て試験計算器を使用する、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を推定するための本発明の代表的な方法を説明する。
【0073】
(A.方法)
(1.データセット)
データセット1は、18品種の90匹のイヌと4匹のオオカミを含む、94匹のイヌ科動物(AHRT、AKIT、BEAG、BMD、BOX、BULD、BULM、CHIH、DACH、GOLD、IBIZ、MAST、NEWF、PEKE、POM、PRES、PUG、ROTT、WOLF;イヌ科動物個体群の略称については表5を参照のこと)からの95個のマイクロサテライトマーカーについての遺伝子型情報を含んだ。95個のマイクロサテライトマーカーは、マイクロサテライトマーカー1〜14、16、18〜21、23〜26、39〜100であった(表1)。このデータセットは、各品種についての5匹のイヌ科動物と4匹のオオカミからの遺伝子型情報を含んだ(表6)。データセット1中のイヌ科動物についての遺伝子型情報は、(本明細書とともに提出されたコンパクトディスク中の)表3に示される。
【0074】
データセット2は、72品種の341匹のイヌ科動物(ACKR、AFGH、AHRT、AIRT、AKIT、AMAL、AMWS、AUSS、AUST、BASS、BEAG、BEDT、BELS、BLDH、BMD、BORD、BORZ、BOX、BSJI、BULD、BULM、CAIR、CHBR、CHIH、CKCS、CLSP、COLL、DACH、DANE、DNDT、DOBP、ECKR、FCR、GOLD、GREY、GSD、GSHP、GSMD、HUSK、IBIZ、IRSE、IRTR、IWOF、KEES、KOMO、KUVZ、LAB、MAST、MBLT、MNTY、NELK、NEWF、OES、PEKE、PNTR、POM、PRES、PTWD、PUG、RHOD、ROTT、SCHP、SCWT、SFXT、SHAR、SPOO、SSHP、STBD、TERV、WHIP、WHWT、WSSP;イヌ科動物個体群の略称については表5を参照のこと)からの68個のマーカーについての遺伝子型情報を含んだ。68個のマイクロサテライトマーカーは、マイクロサテライトマーカー2〜8、11、12、14〜16、18〜21、23、24、26〜32、34〜36、38、41、42、44〜46、50、51、53、54、56、60〜64、67、68、70〜74、78、79、81〜83、85、87〜91、93〜98であった(表1)。このデータセットは、SFXT(2匹のイヌ科動物)、ACKR、AFGH、DNDT、OES(各々3匹のイヌ科動物)、AIRT、BASS、BEDT、IRTR、MNTY、SCHP、SCWT、および、TERV(各々4匹のイヌ科動物)を除き、各品種についての5匹のイヌ科動物からの遺伝子型情報を含んだ(表7)。データセット2中のイヌ科動物についての遺伝子型情報は、(本明細書とともに提出されたコンパクトディスク中の)表3に示される。
【0075】
(2.Doh分析)
割り当て試験計算器Doh(www2.biology.ualberta.ca/jbrzusto/Doh.phpにて利用可能)を、遺伝子型情報の2つのデータセットの分析のために使用した。個々のイヌ科動物すべてを、試験されるイヌ科動物を除いたそれらの公知の個体群で指定した。次いでこれを、試験イヌ科動物の遺伝子型を生成する最も高い可能性を有するイヌ科動物個体群へと、プログラムを使用して割り当てた。このプログラムは、この手順を、試験イヌ科動物としての各イヌ科動物に対して繰り返す。
【0076】
(B.結果)
(1.データセット1を使用するDoh分析)
94匹のイヌ科動物(18品種の90匹のイヌと4匹のオオカミ)における95個のマイクロサテライトマーカーについての遺伝子型情報を含む、データセット1中の遺伝子型情報にDohを使用して、イヌ科動物の99%を、正確なイヌ科動物個体群へと割り当てた。イヌ科動物の100%を、以下の品種(AHRT、AKIT、BEAG、BMD、BOX、BULD、CHIH、DACH、GOLD、IBIZ、MAST、NEWF、PEKE、POM、PUG、ROTT、WOLF)について、正確に割り当てた。誤って割り当てられた唯一のイヌ科動物は、(5匹のイヌ中の)プレサカナリオ種のイヌ一匹であった。誤って割り当てられたプレサカナリオ犬は、チワワに割り当てられていた。
【0077】
対立遺伝子パターンの識別能力は、個々のマイクロサテライトマーカー座位の数、各座位における対立遺伝子の多様性、および各品種からサンプル抽出された個体の数に、依存したことが見出された。マーカーの対立遺伝子の数およびマーカーの数の、そのマーカーの情報性への影響を評価するために、最初の19品種についてのDoh割り当て分析を、5個、10個、15個、および20個のマーカー、データセット中に見出された1〜3個の別個の対立遺伝子を有するビニングマーカー、4〜6個の別個の対立遺伝子、7〜10個の別個の対立遺伝子、および10個より多い別個の対立遺伝子を使用して、行った。20個のマーカーを含まなかったビンについては、最大数のマーカーを使用した。10個より多い個別の対立遺伝子を有するマーカーについては、イヌ科動物の86%を、5個のマーカーを使用してそれらの品種へと正確に割り当て、そしてイヌ科動物の95%を、10個、15個、または20個のマーカーを使用して正確に割り当てた。7〜10個の別個の対立遺伝子を含むマーカーについては、イヌ科動物の84%を、5個のマーカーを使用してそれらの品種へと正確に割り当て、そしてイヌ科動物の91%を、10個のマーカーを使用して正確に割り当て、そしてイヌ科動物の94%を、15個、または20個のマーカーを使用して正確に割り当てた。4〜6個の別個の対立遺伝子を含むマーカーについては、イヌ科動物の62%を、5個のマーカーを使用してそれらの品種へと正確に割り当て、そしてイヌ科動物の71%を、10個、15個、または20個のマーカーを使用して正確に割り当てた。1〜3個の別個の対立遺伝子を含むマーカーについては、イヌ科動物の46%を、5個のマーカーを使用してそれらの品種へと正確に割り当て、そしてイヌ科動物の62%を、10個、15個、または20個のマーカーを使用して正確に割り当てた。正しいイヌ科動物個体群へと個体の100%をうまく割り当てるのに必要な(他のどの品種への割り当ても誤った割り当てである)、95個のマーカー内の対立遺伝子頻度の探索に向けられた2つのクラス(0―1)において見出されたマイクロサテライトマーカーの最小数とは、PEKEについて2個、BOX、POM、およびWOLFについて3個、AKIT、MAST、およびPUGについて4個、NEWFおよびROTTについて5個、BMDについて6個、BEAGについて8個、IBIZについて11個、GOLDについて12個、DACHについて17個、BULDについて19個、BULMについて26個、PRESについて44個、CHIHについて49個、そしてAHRTについて52個であった。100%(0−1)の識別に必要なマイクロサテライトマーカーの最小数と、19品種のイヌ科動物において試験された94匹のイヌ科動物についての95個のマイクロサテライトマーカーにわたる対立遺伝子の平均数(表10参照のこと)との間に、正の相関が存在する。100%の精度を有する選択されたイヌ科動物個体群を有す、19種のイヌ科動物個体群にわたって試験された94匹すべての個体の少なくとも90%をうまく割り当てるのに必要な、95個のマーカー内の対立遺伝子頻度の探索の向けた複数のクラス(0、1、2、...18)において見出されたマイクロサテライトマーカーの最小数とは、PEKE、BOX、POM、WOLF、AKIT、MAST、PUG、NEWF、ROTT、およびBMDについて8個、BEAGについて11個、GOLDについて14個、DACHについて23個、BULDについて24個、BULMについて28個、そしてPRES、CHIH、およびAHRTについて95個であった。
【0078】
予想どおり、識別能力は、各品種において観察される同系交配のレベルを反映する。例えば、ある品種は、品種の対立遺伝子変動の平均の3倍未満の対立遺伝子変動を有し、これらの品種は、高い識別能力と、個体群の長いボトルネックおよび小さな有効個体群サイズといった独特な個体群の動態との両方を有する。
【0079】
(2.データセット2を使用するDoh分析)
72品種の341匹のイヌ科動物からの68個についての遺伝子型情報を含む、データセット2中の遺伝子型情報にDoh分析を使用して、試験されたイヌの96%を、表13に記載されるように、正しい品種へと割り当てた。両方のベルジアン種(ベルジアンシープドッグおよびベルジアンタービュレン)を1つの品種として数えた場合、試験されたイヌの98%を、正しい品種へと割り当てた。
【0080】
(実施例3)
この実施例は、94匹のイヌ科動物からの95匹のマイクロサテライトマーカーについての遺伝子型情報にクラスター分析を使用して、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を推定するための本発明の代表的な方法を説明する。
【0081】
(A.方法)
(1.データセット)
データセット1は、実施例2に記載されるように、18品種の90匹のイヌおよび4匹のオオカミを含む、94匹のイヌ科動物からの95個のマイクロサテライトマーカーについての遺伝子型情報を含んだ。
【0082】
(2.クラスター分析)
クラスター分析を、マルチローカス遺伝子型クラスタリングプログラムであるstructure(Pritchardら(2000)Genetics 155:945〜59;Falushら(2003)Science 299:1582〜5)を使用して、行った。このプログラムは、対立遺伝子頻度のパターンに基づいて遺伝的に別個の部分個体群を同定するために、ベイジアンモデルベースのクラスタリングアルゴリズムを利用する。複数の実行を、バーンインの長さが10,000工程で、かつGibbsサンプラーを100,000回繰り返す設定状態で、各K値(遺伝クラスター数)について完了した。相関対立遺伝子頻度モデルを、許容された非対称な交配種(admixture)に使用した。2〜80のK値すべてを試験し、そして最も高い尤度を生じたクラスタリング解を、さらなる検証のために保持した。データセットについての全体的に最良のクラスタリング解を選択するために、全ペアWilcoxon2サンプル試験(all−pair Wilcoxon two−sample test)を、最も高い尤度の5つのK値について行った。
【0083】
(3.ネスト化した(nested)セットのクラスタリング)
完全なデータセットで開始して、すべての個体を、サブクラスターへ階層的に分けた。この場合において、各(K+1)番目のサブクラスターを、10回の実行にわたって観察された最も高い尤度の値に基づいて、前のKクラスターのうちの1つを分岐させることにより作製した。個体のクラスターを派生させるための階層的方法を利用することにより、部分個体群間の遺伝的多様性が、修正された交配種(admixture)の量が原因で減少される場合に、個体群の系統発生を解明するための合理的な方法論を推測し得る。
【0084】
(B.結果)
structureを使用する最大尤度の計算は、データセット1(19匹のイヌ科動物個体群中の95個のマーカー)中の20個体群を予測し、そして、表14に示されるように、各個体を99%の精度で1つの群に割り当てた。個体の品種群に割り当てられなかった1個体とは、たった1匹のプレサカナリオだけであった。この種は、ブルドッグ群とブルマチフ郡との間に配置された。プレサカナリオは、種々のマスチフ型の交配(admixture)を介して育成されてきた再現種である。誤って割り当てられたイヌは、具体的には、最近の12世代内のブルドッグおよびブルマスチフの両者まで、その遺伝形質を辿り得る。
【0085】
クラスタリングの割り当てでは、この分析レベルにおいては、ブルマスチフとマスチフとの間を区別することができないが、これは、表15A〜Dに示されるように、ネスト化(nested)分析によって解決される。ネスト化分析において、同様のクラスタリングアルゴリズムを、段階的様式で適用した。まず、全体のセットを、2つの個体群へと分けた。最大尤度に基づき、次いでこれら2つの個体群の一方を、合計3つの個体群を提供するために2つに分けた。この手順を、すべての個体群が決定されるまで繰り返した。5〜9つの群からの分岐は、マスチフ型品種の間の関係性を明確に示す。この関係性および予想された階層は、品種の説明書(account)から予測された関係性と完全に一致する。
【0086】
(実施例4)
この実施例は、85匹のイヌ科動物からの96個のマイクロサテライトマーカーについての遺伝子型情報にクラスター分析を使用して、イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を推定するための本発明の代表的な方法を説明する。
【0087】
(A.方法)
(1.データセット)
データセット3は、85品種の414匹のイヌ科動物(ACKR、AFGH、AHRT、AIRT、AKIT、AMAL、AMWS、AUSS、AUST、BASS、BEAG、BEDT、BELS、BICH、BLDH、BMD、BORD、BORZ、BOX、BSJI、BULD、BULM、CAIR、CHBR、CHIH、CHOW、CKCS、CLSP、COLL、DACH、DANE、DOBP、ECKR、FBLD、FCR、GOLD、GREY、GSD、GSHP、GSMD、GSNZ、HUSK、IBIZ、IRSE、IRTR、ITGR、IWOF、KEES、KERY、KOMO、KUVZ、LAB、LHSA、MAST、MBLT、MNTY、MSNZ、NELK、NEWF、OES、PEKE、PHAR、PNTR、POM、PRES、PTWD、PUG、RHOD、ROTT、SALU、SAMO、SCHP、SCWT、SHAR、SHIB、SHIH、SPOO、SSHP、SSNZ、STBD、TIBT、TERV、WHIP、WHWT、WSSP;イヌ科動物個体群の略称については表5を参照のこと)からの96個のマーカーについての遺伝子型情報を含んだ。96個のマイクロサテライトマーカーは、マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100であった(表1)。このデータセットは、AIRT、BASS、BEDT、BICH、FBLD、IRTR、MNTY、PHAR、SCHP、SCWT、TERV(それぞれ4匹のイヌ科動物)を除き、全品種についての5匹のイヌ科動物についての遺伝子型情報を含んだ(表8)。このデータセット中のイヌ科動物についての遺伝子型情報は、(本明細書とともに提出したコンパクトディスク中)表3に示される。
【0088】
(2.統計分析)
structureを、20,000回繰り返すバーンインの後、Gibbsサンプラーを100,000回繰り返す間、実行した。相関対立遺伝子頻度モデルを、許容された非対称の交配種(admixture)に使用した。structureの実行全体にわたる類似度係数を、(Rosenbergら、(2002)Science 298:2381−5)に記載されるようにコンピュータで計算した。このプログラムを68品種の部分的なデータセットに実行した場合、40を超えるK値において、プログラムは、個体が何も割り当てられなかったクラスターを生じ、このクラスターは実行から実行への間、不安定であったことに注目のこと。このことは、2〜3の個体群に分岐するよう初期設定されたアルゴリズムが、このような多数の個体群を同時に扱うことが不可能であるため、最も起こりやすい。structureは、信頼できる別々の20の個体群を既に示しているので(Rosenbergら(2001)Genetics 159:699〜713)、このデータは、10〜11品種のそれぞれを8個のサブセットへ分けたセットであり、これらのサブセットの可能性のあるすべてのペアを分析した。歴史的に関連がある品種、または形態学的に類似する品種を、同じサブセット中に保持した。
【0089】
次いで、structureを、各Kにおいて15回実行する設定で、K=2〜K=10において全体のデータに適用した。Kが増加するほど、structureはまず、最も多岐した群をクラスターへと分け、その後に、より近縁種の分岐を続ける(Rosenbergら(2002)Science 298:2381)。分析において、尤度は、Kが増加するとともに増加し、各Kにおいて見出されたさらなる構造を反映するが、複数の異なるクラスタリング解が、K>4で見出され、そしてそれ故に、K=2〜4を、包括的な品種構造を記述するために使用した。これは、サブグループの系統発生分析およびクラスター分析を近縁種の一群(constellation)を規定するために使用した。K=2〜5におけるstructureの実行を、類似する結果を有する交配モデル(admixtrue model)がない状態下で繰り返した。別の分析において、8匹のオオカミを、K=2におけるstructureの実行に加えた。このオオカミを、8ヶ国(中国、オマーン、イラン、イタリア、スウェーデン、メキシコ、カナダ(オンタリオ)、および米国(アラスカ))からサンプル抽出した。すべてのオオカミを、表16に示される犬種の第1のクラスターとともにクラスター形成した。
【0090】
各品種を、品種の平均的な多数派に基づいて4つの群のうちの1つに割り当て、structureを、K=2〜4において各郡に実行した。さらなる一貫したパターンは、報告された品種ペアおよび品種トリオ以外の個々の群内では、観察されなかった。異常値分析を、http://www.rubic.rdg.ac.uk/〜mab/software.htmlにて利用可能なソフトウェアパッケージfdist2を使用して行った。11個のマーカーを、85種の個体群を仮定した無限対立遺伝子モデルおよび1個体群あたり10個のハプロタイプ遺伝子型の平均のもとでの、シミュレーションによって達成された、95番目のパーセント点を超えるFst値を有する潜在的な「異常値」として同定した(BeaumontおよびNichols(Dec.22,1996)Proceedings:Biological Sciences 263:1619)。これらの除かれたマーカーで行われた割り当ておよび構造分析は、有意な変化を生じなかった。
【0091】
系統樹分析について、個々のイヌおよびオオカミを、品種または種に基づいて、86種の個体群のうちの1つに割り当てた。個体群間の距離を、コード距離尺度(chord distance measure)を用い、プログラムMicrosat(E.Minch、A.Ruiz−Linares、D.Goldstein、M.Feldman,L.L.Cavalli−Sforza(1995、1996))を使用して、コンピュータで計算した。500回のブートストラップ複製を作成した。このプログラムは、ウェブサイトhttp://hpgl.stanford.edu/projects/microsat/microsat.htmlからダウンロードされ得る。近接結合樹を、プログラムNeighborを使用して各複製について構築し、そしてプログラムConsenseを、多数派支配型(majority−rule)コンセンサス樹を作成するため使用した。これらプログラムは両方とも、http://evolution.genetics.washington.edu/phylip.htmlにて利用可能な、Phylipパッケージ(Felsenstein(1989)Cladistics 5:164)の一部である。オオカミの個体群を、その樹の基礎を成す(root)ために外群(outgroup)として指定した。異なる8つの国々からのオオカミを、図2に示される樹において、簡略化のために1個体群にまとめた。個体として考えられる場合、すべてのオオカミは、枝の根が同じ場所にある1つの枝から分岐され。系統分析における分岐の順番は、ヘテロ接合性と相関し(表11)、一番よく似ている(closely mirrored)最初のクラスターに分岐する12品種が、structureによって同定された。これらの知見は、この分析が、異常に同系交配であるかまたは最近野生のイヌ科動物と交配された特異体質品種に分岐するのではなく、遺伝的に関連する品種の別個の部分群を同定したことを、主張する。
【0092】
割り当て試験を、J.Brzustowski(http://www2.biology.ualberta.ca/jbrzusto/Doh.php)から利用可能なDoh割り当て試験計算器を用いて行った。すべてのイヌを、試験される1種のイヌを除いて、それらの公知の品種で指定した。次いでこれを、試験イヌの遺伝子型を生成する可能性が最も高い品種へと、プログラムを使用して割り当てた。このプログラムは、この手順を、試験イヌとしての各イヌに対して繰り返す。ベルジアンシープドッグ種およびベルジアンタービュレン種を、この分析のために1つの記号表示にまとめた;それらが別個の品種として扱われる場合、個々のイヌを、基本的にランダムにどちらか一方に割り当てる。
【0093】
(B.結果)
structureを20〜22品種のオーバーラップするサブセットに同時に適用した場合、表17に示されるように、ほとんどの品種がその品種からのすべてのイヌだけから構成される別個のクラスターを形成することが、観察された。以下の4品種のイヌだけが、一貫して、他の同種とクラスター形成できなかった:ペロデプレサカナリオ、ジャーマンショートヘアードポインター、オーストラリアンシェパード、およびチワワ。加えて、以下の品種の6つのペアが、実行の大部分において、ともにクラスター形成した:ベルジアンシープドッグおよびベルジアンタービュレン、コリーおよびシェトランドシープドック、ウィペットおよびグレイハウンド、シベリアハスキーおよびアラスカンマラミュート、マスチフおよびブルマスチフ、グレートスミスマウンテンドックおよびバーニーズマウンテンドッグ。これらの組み合わせは、公知の品種の歴史に基づいて予測される。これらの近縁種のペアがそれでもなお遺伝的に別個であるかどうかを試験するために、structureをこれらの各クラスターに適用した。1つだけを除いてすべてのクラスターは、表18に示されるように、個々の品種に対応する2種の個体群へと分岐した。1つの例外は、ベルジアンシーブドックとベルジアンタービュレンであった。ヨーロッパケンネルクラブおよび日本ケンネルクラブは、それらを、1品種の毛色および毛長の変種として分類し(YamazakiおよびYamazaki(1995)Legacy of the Dog:The Ultimate Illustrated Guide to Over 200 Breeds,Chronicle Books,San Fracisco,CA;WilcoxおよびWalkowicz(1995)Atlas of Dog Breeds of the World,T.F.H.Publications,Neptune City,NJ)、そして、その一方で、アメリカンケンネルクラブが、これらを別個の品種として認識するとはいえ、品種の境界は、遺伝的差異を結果としてもたらすには、明らかに近い世代(recent)でありまたは厳密さが不十分である。これらの例は、アルゴリズムは真の遺伝的差異を有する群に分けるだけに過ぎないことを確認する(Falshら(2003)Science 299:1582−5;PritchardおよびRosenberg(1999)Am.J.Hum.Genet.65:200−8)。
【0094】
イヌが遺伝子型データだけを基にしてその品種へと割り当てられ得るかどうかを試験するために、leave one−out分析を用いる直接割り当て方法(Paetkauら(1995)Mol.Ecol.4:347−54)を、使用した。99%の個々のイヌが、正しい品種へと正確に割り当てられた。414匹のうちの4匹のイヌのみが、誤って割り当てられた:1匹は(ペロデプレサカナリオに割り当てられた)ビーグルであり、1匹は(ケーアンテリアに割り当てられた)チワワであり、そして2匹は、(クバーズおよびスタンダードプードルそれぞれに割り当てられた)ジャーマンショートヘアードポインターである。品種に関連する4つエラーは、structure分析において単独の品種クラスターを形成しなかった品種を含んだ。
【0095】
現代のイヌの品種が別個の遺伝単位を形成することが示されると、品種間の広範な歴史的関係性を規定することが試みられた。最初に、標準的な近隣結合法を、コード距離尺度(Cavalli−SforzaおよびEdwards(1967)Evolution 32:550)を使用して計算された距離を用い、品種の多数派支配型コンセンサス樹を構築するために使用した(図2)。これは、特定の変異モデルを仮定せず、そして近縁の分類群に対して良好に機能すると考えられる(Goldsteinら(1995)Genetics 139:463)。この樹を、オオカミサンプルを使用して基礎を成した(root)。樹の最も深い分岐は、4種のアジアスピッツ型品種に分かれ、そしてこの枝内でシャーペイが最初に分岐し、その後に柴犬が続き、秋田犬およびチャウチャウは一緒のグループになった。第2の分岐は、バセンジー(古代アフリカ品種)に分かれた。第3の分岐は、2種の北極スピッツ型品種(アラスカンマラミュートとシベリアンハスキー)に分かれ、そして第4の分岐は、残りの品種から、2種の中東品種のハウンド(アフガンとサルーキ)に分かれた。
【0096】
最初の4つの分岐は、半分より多くのブートストラップ複製が示すように、「多数派支配」基準を超えた。対照的に、残りの品種は、わずかなに系統学的に一致する関係性を示した。これは、structure分析でともにクラスター形成される5品種のペアの近縁群を除いて、近縁関係にあるウェストハイランドホワイトテリアとケアーンテリアとの新たな1つのペアであり、そして類似する外見を有する仲間の品種(companion breed)であるアジアの3種(ラサアプソ、シーズー、およびペキニーズ)の有意な分類である。これらの3種の間の近縁関係はまた、実行の大部分において3つのうちの少なくとも2つが一緒にクラスター形成することで、structure分析において観察された。樹のフラットトポロジーは、おそらく、より大きな共通の創始系統(founder stock)ならびに、血統クラブおよび品種の境界規則の出現より前の、表現型的に異なるイヌ間での広範な遺伝子流動の発生を反映する。加えて、これは、表現型的に類似するか歴史的に関連するイヌ由来の系統を使用した、飢饉(19世紀および20世紀の不況および戦争)の間に絶滅したいくつかの歴史的に古い品種の再生を示す可能性がある。
【0097】
系統発生分析は、現代のヨーロッパ血統と推定されたより大きな品種群からの古代血統を有するいくつかの品種の分岐を示したが、更なる部分個体群は、少なくとも2つの理由によってこのアプローチでは検出されない後者の郡内に、存在し得る(Rosenbergら、(2001)Genetics 159:699)。第一に、犬種の真の進化の歴史は、この方法によって仮定された二分岐樹モデルでは、十分には表されないが、新種を生成するための現存種の交配(現在まで続くプロセス)に関しては、かなり関与した。第二に、遺伝的距離マトリックスに基づいた方法は、品種のペアについてのすべての遺伝子型データを、1つの数字へと激減させることで情報を失う。
【0098】
structureに組み込まれたクラスタリングアルゴリズムは、これらの制限を克服するために明示的に設計され(Pritchardら(2000)Am.J.Hum.Genet.67:170−81;Falushら(2003)Genetics 164:1567;Rosenbergら(2001)Genetics 159:69−713)、そして、いくつかの種の遺伝学的構造を推測するために適用されている(Rosenbergら(2002)Scinece 298:2181−5;Falushら(2003)Science 299:1582−5;Rosenbergら(2001)Genetics 159:699−713)。structureを、先祖の個体群源を同定するためにKの増加値(プログラムが発見しようと試みた部分個体群の数)を使用して、全体のデータセットに対して実行した。この分析において、現代種は、1つの先祖個体群と厳密に酷似し得るか、または2つ以上の先祖タイプの交配種を表し得る。
【0099】
K=2において、1つのクラスターが、系統発生分析において分岐するために、最初の7品種によって支えられたが、一方で、他のクラスターは、フラットな系統発生のトポロジーで多数の品種を含んだ(表19A)。プログラムの5つ実行は、実行にわたって0.99の類似する係数で、ほぼ同一の結果を生成した(Rosenbergら(2002)Science 298:2381)。7種の他の品種は、それらの祖先の相当大きな部分を、最初のクラスターと共有する。これら14種のすべては古代から始まり、そしてそれらの祖先をアジアまたはアフリカまでさかのぼる。8つの異なる国々からのオオカミの多様なセットが分析に含まれた場合は、これらは、完全にこのクラスター内にある(表20)。オオカミの外群へとつながる枝もまた、系統発生分析においてこの群の品種内にある(図2)。
【0100】
K=3において、系統樹からは容易には明白に理解されなかったさらなる構造が検出された(表19B)。新たな3つクラスターは、マスチフの遺伝形質および外見において関連した品種から主に構成され、そして、それらの近縁種(ブルマスチフ、フレンチブルドッグ、ミニチュアブルテリア、およびペロデプレサカナリオ)とともに、マスチフ、ブルドック、およびボクサーによって支えられる。クラスター内に含まれるものはまた、古代マスチフタイプの祖先からそれらの大きさを獲得したと報告されている大型品種である、ロットワイラー、ニューファンドランド、およびバーニーズマウンテンドッグである。ジャーマンシェパードドッグを含むことはあまり期待されない。この品種の正確な起源は未知であるが、この結果は、作業犬タイプ(例えば、ボクサー)の前で軍用犬および警察犬として費やした年数が、この有名な品種の遺伝学的背景の形成に関与することを示唆する。他の3つの品種は、structureの実行にわたってこのクラスター中に部分的でかつ不定な構成員を示した(表16)。これは、類似係数を0.84まで低下させた。
【0101】
K=4において、4つのクラスターが観察された。これは、牧畜犬として使用された数品種:ベルジアンシープドッグ、ベルジアンタービュレン、コリー、およびシェットランドシープドッグを含んだ(表19C)。アイリッシュウルフハウンド、グレイハウンド、ボルゾイ、およびセントバーナードもまた、頻繁にこのクラスターへと割り当てられた。歴史的記録物は、これらのイヌがこれまでに家畜を集めるために使用されていたとは示唆しないが、得られた結果は、これらの品種が牧畜タイプの祖先かまたは子孫であるかのいずれかであることを示唆する。残りのクラスター中の品種は、主に比較的最近のヨーロッパ血統からなり、そして、主には異なるタイプの猟犬(セントハウンド、テリア、スパニエル、ポインター、およびレトリバー)である。K=4におけるクラスタリングは、0.61の類似係数を示した。これは、ほとんどの品種についての類似クラスター構成員の割り当てを反映するが、実行にわたって他の品種についての割り当て変数を反映しない(表16)。K=5において、類似係数は、0.26まで下がり、さらに一致する部分個体群は推測されなかった。これは、サンプル抽出された純血種のイヌ個体群においてさらに高レベルの部分構造が欠けていることを示唆している。
【0102】
得られた結果は、イエイヌ種にわたる関係性の下記の構図を描く。異なる品種は、遺伝学的に別個であり、そして個体は、それらの遺伝子型に基づいて品種へと容易に割り当てられ得る。この分岐レベルは、ほとんどの品種の起源が祖先血統から交配されたときからの短い時間を考慮すると驚くべきものであり、そして、品種の境界規則の結果として生じる、各品種内の強力な生殖隔離を支持する。この結果は、別個の「適応放散」を表す少なくともの4つの別個の品種分離を支持する。アジア血統およびアフリカ血統を有する品種の部分セットは、残りの品種から分かれ、そして共有の対立遺伝子頻度パターンを示す。一見したところ、1つの遺伝クラスター内に、中央アフリカ由来種(バセンジー)、中東由来種(サルーキおよびアフガン)、同様にチベット由来種(チベタンテリア、ラサアプソ)、中国由来種(チャウチャウ、ペキニーズ、シャーペイ、シーズー)、日本由来種(秋田犬、柴犬)、ならびに北極由来種(アラスカンマラミュート、シベリアンハスキー、サモエド)を包含するようであり、これは驚くべきことである。しかし、初期の雑種の野良犬は、アジアに由来し、そして遊牧民族とともに、南はアフリカまでそして北は北極まで移動し、結果としてアジアの至るところに移動したという仮説が立てられている(Savolainenら(2002)Science 298:1610;Leonardら(2002)Science 298:1613;SablinおよびKhlopachev(2002)Current Anthropology 43:795)。このクラスターは、オオカミに表現型的に似ている北欧種(例えば、アラスカンマラミュートおよびシベリアンハスキー)を含み、そして、イエイヌの直接の祖先であるオオカミに対して最も近い遺伝学的関係性を示す。従って、これらの品種由来のイヌは、祖先イヌの遺伝子プールの最も代表的な生物であり得る。古代起源の品種であると一般に考えられている数種(例えば、ファラオハウンドおよびイビザンハウンド)は、この群に含まれないことは、注目すべきことである。これらは、すべての犬種のうち最も古い種であるとしばしば考えられている。この種は、5000年以上前に墓の壁に描かれた古代エジプトのイヌから直接的に由来する種である。しかし、この結果は、これら2つの種が、より近代において、他の種の組み合わせから再生されていることを示す。したがって、それらの外見は古代エジプト種のハウンドと一致するが、それらのゲノムは一致しない。同様の結論をノルウェジアンエルクハウンドへと適用すると、これは、5000年前以上にスカンジナビア起源種から直接的に伝わったという報告(American Kennel Club(1998)「The Complete Dog Book」、CrowleyおよびAdelman編、Howell Book Houes、New York、NY;WilcoxおよびWalkowicz(1995)Atlas of Dog Breeds of the World,T.F.H.Publications,Neptune City,NJ)とは異なり、他の北極犬よりも近代のヨーロッパ種でクラスター形成する。
【0103】
大多数の品種は、共通のヨーロッパ血統由来の、より最近の放散(radiation)を表すようである。個々の品種が遺伝学的に分化される間、それらは、基本的に同時期に分化されたようである。この放散は、1800年代のヨーロッパにおける血統の概念の導入および血統クラブの創設の後に、あまり体系化されてなかった表現型的多様性からの別個の品種の繁殖をおそらくは反映する。より高感度なクラスター分析は、この群内の3つの部分個体群のさらなる遺伝学的構造を見分けることが可能である。あるクラスターは、マスチフ様品種を含み、そして共通祖先由来の共通する形態を反映するように見える。別のクラスターは、シェットランドシープドッグ、2匹のベルジアンシープドッグ、およびコリーを含み、そして、共通する祖先の牧畜習性を反映し得る。残りの個体群は、狩猟の種々の局面に献身的な品種の繁殖によって、主に占められている。これらの品種について、歴史的記録および血統クラブの記録は、高度に絡み合った血統を示唆し、これは、得られた結果と一致する。
【0104】
犬種は、伝統的に、人間の活動におけるイヌの役割、身体的な表現型、および歴史的記録に基づいて、分類されてきた。上に記載された結果は、遺伝学的多様性のパターンに基づいて、個々の分類を提供する。この分類は、伝統的な分類の部分セットを支持し、そしてまた、品種間の以前は認識されていなかった関係を解明する。品種間の遺伝学的関係性の正確な理解は、形態、習性、および疾患罹患率における品種の差異の複雑な遺伝学的基礎を解明することを目的とした研究のための基礎を築く。
【0105】
(実施例5)
この実施例は、マイクロサテライトマーカーを用いて、交配されたイヌ科動物の子孫のゲノムへの、異なるイヌ科動物個体群からの親、祖父母、および曽祖父母の寄与度を推定する、インシリコでの方法を説明する。
【0106】
(A.方法)
(1.データセット)
データセット4は、18種の異なる犬種の81匹のイヌと4匹のオオカミからなる、85匹のイヌ科動物(AHRT、AKIT、BEAG、BMD、BOX、BULD、BULM、CHIH、DACH、GOLD、IBIZ、MAST、NEWF、PEKE、POM、PRES、PUG、ROTT、WOLF;イヌ科動物個体群の略称については表5を参照のこと)からの95のマーカーについての遺伝子型情報を含んだ。95個のマイクロサテライトマーカーは、マイクロサテライトマーカー1〜14、16、18〜21、23〜26、39〜100であった(表1)。このデータセットは、85匹のイヌ科動物の各々の90%よりも多くが、正確な品種へと割り当てられた事実に基づいて選択された。4匹のオオカミは、1種のイヌ科動物個体群として設計された。12品種は、5匹のイヌで、3品種は、4匹のイヌで、そして3品種は3匹のイヌで、表9に示されるように表された。各イヌ科動物において使用されたマイクロサテライトマーカーの各々についての遺伝子型は、(本明細書とともに提出されたコンパクトディスク中の)表3に示される。
【0107】
(2.クラスター分析)
インシリコでのイヌ科動物の交配種を、各座位における各親からの2つの対立遺伝子のうちの1つをランダムに引き出し、そしてそれらをその座位における交配種の対立遺伝子として設計することにより、作製した。F1交配種を、81匹の原種イヌ科動物の2つの対立遺伝子をインシリコで交配することによって作製した。次いで、N2交配種を、それらの2つの親のうちの1つとF1とをインシリコで交配することによって作製し、そして、N3交配種を、同じ親とN2とをインシリコで交配することによって作製した。
【0108】
3つのタイプの交配種が形成された(試験交配種、コントロール交配種、祖父母交配種)。試験交配種において、2つの親を、ランダムに選択された異なる2種から選択した。100個のF1交配種、N2交配種、およびN3交配種が、形成された。F1交配種が、異なる品種からの親を2つ有し、N2交配種が、1種からの祖父母および別の種から祖父母の4つのうちの3つを有し、そしてN3交配種が、1種からの曽祖父母および別の種から曽祖父母の8つのうちの7つを有することに注目のこと。
【0109】
コントロール交配種において、2つの親を同種から選択し、そして100個のF1混合種、N2混合種、およびN3混合種を、同じ手順によって形成した。これらすべての混合種が選択された品種からの純血種のイヌに対応することに注目のこと。
【0110】
いくつかの祖父母交配種をまた、異なる4種の品種から4つの祖父母を選択することにより形成した。
【0111】
300個すべての試験混合種を、選択された85匹のイヌ科動物を用いるstructureの実行において、同時に実行した。同様の分析を、コントロール交配種について、および4つの祖父母交配種について行った。プログラムを、以下のパラメーター設定で実行した:#define NUMINDS 395;#define NUMLOCI 95;#define LABEL 1;#define POPDATA 1;#define POPFLAG 1;#define PHENOTYPE 0;#define MARKERNAMES 0;#define MAPDISTANCES 0;#define ONEROWPERIND 1;#define PHASEINFO 0;#define PHASED 0;#define EXTRACOLS 0;#define MISSING 0;#define PLOIDY 2;#define MAXPOPS 19;#define BURNIN 5000;#define NUMREPS 5000;#define USEPOINFO 1;#define GENSBACK 0;#define MIGRPRIOR 0.0;#defineNOADMIX 0;#define LINKAGE 0;#define INFERALPHA 1;#define ALPHA 1.0;#define POPALPHAS 0;#define UNIFPRIOPALPHA 1;#define ALPHAMAX 10.0;#define ALPHAPROPSD 0.025;#define FREQSCORR 1;#define ONEFST 0;#define FPRIORMEAN 0.01;#define FPRIORSD 0.05;#define INFERLAMBDA 0;#define LAMBDA 1;#define COMPUTEPROB 1;#define PFROMPOPFLAGONLY 0;#define ANCESTDIST 1;#define NUMBOXES 1000;#define ANCESTPINT 0.95;#define STARTATPOPINFO 1;#define METROFREQ 10;#define UPDATEFREQ 1;#define PRINTQHAT 1。
【0112】
85匹のイヌ科動物の各々は、それらの適切な品種に属するように設計され、そして、その交配種は、どの品種にも割り当てられなかった。
【0113】
(B.結果)
コントロール交配種について、各交配種は、常に、プログラムによって正しい品種へと割り当てられた。そして、その品種へと割り当てられたゲノムの比率は、300回のケースすべてにおいて95%を超え(最小値は95.75%であった)、297回のケースにおいては98%を超え、そして266回のケースにおいては99%を超えた。従って、単一の種への95%を超えるゲノムの割り当ては、試験交配種についての交配の明白な検出を提供した。そして98%を超える割り当ては、0.99の信頼度における交配の強固な証拠を提供する。
【0114】
F1試験交配種について、100個の交配種すべては、各品種の寄与度が28%〜70%の範囲で正確に割り当てられた、2つの親種からゲノム寄与度であった。100回のケースのうち82回において、2つの親種の各々は、40%より高い寄与度、および60%より低い寄与度で割り当てられた。このことは、2種間での交配種が、親レベルの時点で100%確実に同定され得ることを示している。
【0115】
N2試験交配種について、100回のケースのうちの99回は、1つの品種へと割り当てられたゲノムのうちの98%未満を有し、100回のケースのうちの97回は、1つの品種へと割り当てられたゲノムのうちの95%未満を有した。これは、祖父母レベルにおける交配を検出する高度に正確な能力を示した。交配が検出されたケースの1つを除いて、交配に寄与する両方の品種は、正確に割り当てられた(1つのケースにおいて、4つの祖父母のうちの1つに寄与する品種は、有意に寄与するものとしては検出されなかった)。80〜85%のケースにおいて、N2交配種は、F1交配種から確実に識別され得る(すなわち、親レベルではなく祖父母レベルで生じた交配が検出され得る)。
【0116】
N3試験交配種について、100回のケースのうちの85回は、1つの品種へと割り当てられたゲノムのうちの98%未満を有し、100回のケースのうちの77回は、1つの品種へと割り当てられたゲノムのうちの95%未満を有した。これは、祖父母レベルにおける交配を検出するかなり良好な能力を示した。交配が検出されたケースの1つを除いて、交配に寄与する両方の品種は、正確に割り当てられた。すべてのケースにおいて、N3交配種は、F1交配種から確実に識別され得る(すなわち、親レベルではなく祖父母レベルで生じた交配が検出され得る)が、祖父母レベルおよび曽祖父母レベルにおける交配の間で区別する能力は低かった。
【0117】
最終的に、4つの異なる祖父母を有する交配種について、4つの祖父母種すべてが、20〜30%の範囲と推定された各品種の交配種のゲノムへの寄与度で、正確に同定された。
【0118】
これらの結果は、いくらかが曽祖父母レベルにおける交配を識別する能力で、純血種のイヌからの(イヌ由来の1/2オオカミ交配種および1/4オオカミ交配種も同様)、親レベルおよび祖父母レベルにおける交配を識別する方法の能力を明白に証明している。この方法また、雑種イヌのゲノムにおける品種の寄与度を正確に同定する。各品種からのより多くのイヌ、および本明細書に他で記載された基準に従って選択された追加にマーカーおよび最適化されたマーカーセットを含む、より大きなデータベースは、曽祖父母レベルにおける交配をより正確に識別することを可能にし、そして、単純な拡張によって、より遠い祖先において生じる交配も正確に識別することを可能にする。
【0119】
(実施例6)
この実施例は、SNPマーカーを使用する、試験イヌ科動物のゲノムへのイヌ科動物の寄与度を推定するための本発明の代表的な方法を説明する。
【0120】
(A.方法)
(1.データセット)
種々の犬種における一塩基多型(SNP)のデータセットを、各品種における各対立遺伝子頻度を計算するために使用した。データベースは、実施例1に記載されるように、品種ごとに11純血種のうちの2種に関しての、67品種の189匹のイヌ科動物からの100個のSNPについての遺伝子型情報を含んだ。イヌ科動物における対立遺伝子の同一性は、(本明細書とともに提出されたコンパクトディスク中の)表4に示される。
【0121】
(2.Doh分析)
leave−one−out手順を使用して、各イヌをデータベースから一時的に除き、各品種の対立遺伝子頻度とイヌの遺伝子型との比較に基づいて、品種へと割り当てた。ベイズの定理を、割り当てについて使用した:所与の品種から生じた確率とは、その品種のイヌにおいて生じる観察された遺伝子型を、データベース内のすべての品種について生じる観察された遺伝子型である条件付き確率の合計で割った条件付き確率である(基本的に、Cornuetら(1999)Genetics 153:1989〜2000において記載される)。ソフトウェアを、このアルゴリズムを実施するために開発した。2個体のみを有する品種をデータベース内に含んだが、それらの一員を分類する試みはしなかった。なぜなら、2つの一員のうちの1つを一時的に除くことは、信頼できる対立遺伝子頻度を計算するための十分な情報を残さなかったからである。
【0122】
(B.結果)
この分析の出力結果は、表21に示されるような、各イヌについての、データベース中の各品種から生じた確率のリストである。80%のイヌは、99%以上の確率で正確な品種へと割り当てられた。遺伝子型が5個以上の個体について得られた品種について、88%のイヌが、99%の確率で正確な品種へと割り当てられた。14匹のイヌ(試験された合計のうちの16%)は、65%よりも良好な確率で正確な品種へと割り当てられた。これらについて、13匹は、50%よりも良好な確率(90%よりも高い確率のほぼ1/3)で誤って割り当てられた。残りのイヌは、数種から生じた20〜45%の確率で割り当てられ、これらのうちの1つは、正しかった。
【0123】
これらの結果は、SNPマーカーに基づいた品種割り当ての実行可能性を実証している。性能は、より多く(各品種について5匹以上)のイヌについてのSNP遺伝子型プロファイルを、より大きなSNPセットを使用して生成すること、および最大限の情報性となるSNPを選択することによって増強され得る。SNPは、品種にわたる高いヘテロ接合性を有すること(すなわち、高頻度で生じる両方の対立遺伝子)に基づくパネル、および品種間の頻度における大きな差異に基づくパネルの両方を包含して選択され得る。
【0124】
(実施例7)
この実施例は、マイクロサテライトマーカーを用いて、交配された子孫イヌ科動物のゲノムへの、異なるイヌ科動物個体群からの親および祖父母イヌ科動物の寄与度を推定するためのナイーブベイジアン分類モデルを説明する。
【0125】
(A.方法)
(1.データセット)
データセット5は、88品種の429匹のイヌ科動物(ACKR、AFGH、AHRT、AIRT、AKIT、AMAL、AMWS、ASBT、AUSS、AUST、BASS、BEAG、BEDT、BELS、BICH、BLDH、BMD、BORD、BORZ、BOX、BRIA、BSJI、BULD、BULM、CAIR、CHBR、CHIH、CHOW、CKCS、CLSP、COLL、DACH、DANE、DOBP、ECKR、FBLD、FCR、GOLD、GREY、GSD、GSHP、GSMD、GSNZ、HUSK、IBIZ、IRSE、IRTR、ITGR、IWOF、KEES、KERY、KOMO、KUVZ、LAB、LHSA、MAST、MBLT、MNTY、MSNZ、NELK、NEWF、OES、PEKE、PHAR、PNTR、POM、PRES、PTWD、PUG、RHOD、ROTT、SALU、SAMO、SCHP、SCWT、SHAR、SHIB、SHIH、SPOO、SSHP、SSNZ、STBD、TIBT、TERV、TPOO、WHIP、WHWT、WSSP;イヌ科動物個体群の略称については表5を参照のこと)からの96個のマーカーについての遺伝子型情報を含んだ。96個のマイクロサテライトマーカーは、マイクロサテライトマーカー1〜9、11〜38、40〜42、44〜75、77〜100であった(表1)。このデータセット中のイヌ科動物についての遺伝子型情報は、(本明細書とともに提出したコンパクトディスク中)表3に示される。
【0126】
データセット6は、既知の交配種構成員を有する160匹の雑種イヌ科動物からの、表1における72個のマーカーについての遺伝子型情報を含んだ。このデータセット中の雑種イヌ科動物についての遺伝子型情報は、(本明細書とともに提出したコンパクトディスク中)表3に示される。
【0127】
(2.分析)
ナイーブベイジアンモデルを構築した。これは、先祖の部分個体群の交配種への個体の確率論的割り当てのために、連鎖および非連鎖マイクロサテライト座位情報、高次元の先祖個体群、および高度に秩序だった世代系図を組み込んでいる。2世代および3世代のモデルを、既存モデルの世代、部分個体群、および連鎖の制限を同時に対処し、正確に交配種を検出しかつ割り当てるために、組み込んだ。
【0128】
2世代モデルは、「純血の」部分個体群の2つのクラスよりもより大きく拡張して、AndersonおよびThompson(2002)Genetics 160:1217−29に概説されたモデルに、厳密に従っている。非連鎖座位(L)について、部分個体群(N)(品種とみなされる)、および第1番目の座位における対立遺伝子(j1)を有する。座位(L)における各個体について、遺伝子型:(g1(0)、g1(1))を含めた。部分個体群対立遺伝子情報を集めることは、任意の所与の対立遺伝子に頻度(fij(i)で表される)についての情報を提供する。したがって個体について、交配されなかった(non−admixed)部分個体群の割り当てには、以下:
【0129】
【数1】

【0130】
を与える。ここで親の交配種の割り当てについては、以下:
【0131】
【数2】

【0132】
を有する。ここで上付き添え字(0)は、父親と子との関係、そして(1)は、母親と子との関係を、(明白で交換可能なオプションとともに)表す。
【0133】
3世代モデルは、モデルの拡張が、部分個体群(N)にわたる4個体群の2世代の表示(representation)を考慮することを可能にする:
【0134】
【数3】

【0135】
最も高い後方確率での交配種の徹底的な探索は、2世代および3世代モデルについて可能である。
【0136】
インシリコでの個体について、モデルの評価は、leave−one−outクロス検定によって行われた。この場合、インシリコ雑種個体の生成に使用した、サンプル抽出された対立遺伝子は、先祖個体群から除かれ、そして対立遺伝子頻度は、最大尤度の交配比率の割り当ての前に、更新される。
【0137】
(B.結果)
96個すべてのジヌクレオチドマーカーのわたるインシリコ雑種個体群についての分析は、2世代および3世代におけるモデルが、品種特異的に不足するというような明らかな傾向がない状態で、F1交配種の98.4%およびF2交配種の94.3%を正しく非常に良好に割り当てることを示している。96個のジヌクレオチドマーカーのうちの72個において遺伝子型決定された既知の雑種個体160匹についての分析は、2世代および3世代におけるモデルが、ほぼ正確に、F1交配種の96.2%およびF2交配種の91.8%を正しく割り当てることを示している。
【0138】
本発明の好ましい実施形態が例示されそして説明されているが、種々の変更が、本発明の精神および範囲を逸脱することなく、本発明において行われ得ることが理解される。
【0139】
【表1】

【0140】
【表2】

【0141】
【表3】

【0142】
【表4】

【0143】
【表5】

【0144】
【表6】

【0145】
【表7】

【0146】
【数4】

【0147】

【0148】

【0149】

【0150】

【0151】

【0152】

【0153】

【0154】

【0155】

【0156】

【0157】

【0158】

【0159】

【0160】

【0161】

【0162】

【0163】

【0164】

【0165】

【0166】

【0167】

【0168】

【0169】

【0170】

【0171】

【0172】

【0173】

【0174】

【0175】

【0176】

【0177】

【0178】

【0179】

【0180】

【0181】

【0182】

【0183】

【0184】

【0185】

【0186】

【0187】

【0188】

【0189】

【0190】

【0191】

【0192】

【0193】

【0194】

【0195】

【0196】

【0197】

【0198】

【0199】

【0200】

【0201】

【0202】

【0203】

【0204】

【0205】

【0206】

【0207】

【0208】

【0209】

【0210】

【0211】

【0212】

【0213】

【0214】

【0215】

【0216】

【0217】

【0218】

【0219】

【0220】

【0221】

【0222】

【0223】

【0224】

【0225】

【0226】

【0227】

【0228】

【0229】

【0230】

【0231】

【0232】

【0233】

【0234】

【0235】

【0236】

【0237】

【0238】

【0239】

【0240】

【0241】

【0242】

【0243】

【0244】

【0245】

【0246】

【0247】

【0248】

【0249】

【0250】

【0251】

【0252】

【0253】

【0254】

【0255】

【0256】

【0257】

【0258】

【0259】

【0260】

【0261】

【0262】

【0263】

【0264】

【0265】

【0266】

【0267】

【0268】

【0269】

【0270】

【0271】

【0272】

【0273】

【0274】

【0275】

【0276】

【0277】

【0278】

【0279】

【0280】

【0281】

【0282】

【0283】

【0284】

【0285】

【0286】

【0287】

【0288】

【0289】

【0290】

【0291】

【0292】

【0293】

【0294】

【0295】

【0296】

【0297】

【0298】

【0299】

【0300】

【0301】

【0302】

【0303】

【0304】

【0305】

【0306】

【0307】

【0308】

【0309】

【0310】

【0311】

【0312】

【0313】

【0314】

【0315】

【0316】

【0317】

【0318】

【0319】

【0320】

【0321】

【0322】

【0323】

【0324】

【0325】

【0326】

【0327】

【0328】

【0329】

【0330】

【0331】

【0332】

【0333】

【0334】

【0335】

【0336】

【0337】

【0338】

【0339】

【0340】

【0341】

【0342】

【0343】

【0344】

【0345】

【0346】

【0347】

【0348】

【0349】

【0350】

【0351】

【0352】

【0353】

【0354】

【0355】

【0356】

【0357】

【0358】

【0359】

【0360】

【0361】

【0362】

【0363】

【0364】

【0365】

【0366】
【数5】

【0367】

【0368】

【0369】

【0370】

【0371】

【0372】

【0373】

【0374】

【0375】

【0376】

【0377】

【0378】

【0379】

【0380】

【0381】

【0382】

【0383】

【0384】

【0385】

【0386】

【0387】

【0388】

【0389】

【0390】

【0391】

【0392】

【0393】

【0394】

【0395】

【0396】

【0397】

【0398】

【0399】

【0400】

【0401】

【0402】

【0403】

【0404】

【0405】

【0406】

【0407】

【0408】

【0409】

【0410】

【0411】

【0412】

【0413】

【0414】
【表8】

【0415】
【表9】

【0416】
【表10】

【0417】
【表11】

【0418】
【表12】

【0419】
【表13】

【0420】
【表14】

【0421】
【表15】

【0422】
【表16】

【0423】
【表17】

【0424】
【表18】

【0425】
【表19】

【0426】
【表20】

【0427】
【表21】

【0428】
【表22】

【0429】
【表23】

【0430】
【表24】

【0431】
【表25】

【0432】
【表26】

【0433】
【表27】

【0434】
【表28】

【0435】
【表29】

【0436】
【表30】

【0437】
【表31】

【0438】
【表32】

【0439】
【表33】

【0440】
【表34】

【0441】
【表35】

【0442】
【表36】

【0443】
【表37】

【0444】
【表38】

【0445】
【表39】

【0446】
【表40】

【0447】
【表41】

【0448】
【表42】

【0449】
【表43】

【0450】
【表44】

【0451】
【表45】

【0452】
【表46】

【0453】
【表47】

【0454】
【表48】

【0455】
【表49】

【0456】
【表50】

【0457】
【表51】

【0458】
【表52】

【0459】
【表53】

【0460】
【表54】

【0461】
【表55】

【0462】
【表56】

【0463】
【表57】

【0464】
【表58】

【0465】
【表59】

【0466】
【表60】

【0467】
【表61】

【0468】
【表62】

【0469】
【表63】

【0470】
【表64】

【0471】
【表65】

【0472】
【表66】

【0473】
【表67】

【0474】
【表68】

【0475】
【表69】

【0476】
【表70】

【0477】
【表71】

【0478】
【表72】

【0479】
【表73】

【0480】
【表74】

【0481】
【表75】

【0482】
【表76】

【0483】
【表77】

【0484】
【表78】

【0485】
【表79】

【0486】
【表80】

【0487】
【表81】

【0488】
【表82】

【0489】
【表83】

【0490】
【表84】

【0491】
【表85】

【0492】
【表86】

【0493】
【表87】

【0494】
【表88】

【0495】
【表89】

【0496】
【表90】

【0497】
【表91】

【0498】
【表92】

【0499】
【表93】

【0500】
【表94】

【0501】
【表95】

【0502】
【表96】

【0503】
【表97】

【特許請求の範囲】
【請求項1】
イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法であって、以下の工程:
(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(b)該試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、該試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程であって、各イヌ科動物個体群プロファイルは、該イヌ科動物個体群におけるマーカーセットに関する遺伝子型情報を含む、工程
を包含する、方法。
【請求項2】
請求項1に記載の方法であって、前記マーカーセットは、少なくとも約5個のマーカーを含む、方法。
【請求項3】
請求項1に記載の方法であって、前記マーカーセットは、マイクロサテライトマーカーを含む、方法。
【請求項4】
請求項3に記載の方法であって、前記マイクロサテライトマーカーは、表1に示すマイクロサテライトマーカーのうちの少なくとも5個を含む、方法。
【請求項5】
請求項1に記載の方法であって、前記マーカーセットは、一塩基多型(SNP)を含む、方法。
【請求項6】
請求項5に記載の方法であって、前記SNPマーカーは、表2に示すSNPマーカーのうちの少なくとも5個を含む、方法。
【請求項7】
請求項1に記載の方法であって、前記マーカーセットは、1つ以上の個体群特異的マーカーを含む、方法。
【請求項8】
請求項7に記載の方法であって、前記1つ以上の個体群特異的マーカーは、1つ以上のSNPマーカーを含む、方法。
【請求項9】
請求項8に記載の方法であって、前記1つ以上のSNPマーカーは、372c5t−82、372e13t−57、372m6t−88、372m23t−76、373a15t−112、373e1t−50、373elt−130、373g19t−246、373i8s−224、373k8s−181、372c5s−168、372C15S−196、372e15s−71、および373a21t−93からなる群より選択される、方法。
【請求項10】
請求項1に記載の方法であって、イヌ科動物個体群プロファイルにおける前記遺伝子型情報は、前記マーカーセットの各々の片方または両方の対立遺伝子の同一性を含む、方法。
【請求項11】
請求項1に記載の方法であって、イヌ科動物個体群プロファイルにおける前記遺伝子型情報は、前記マーカーセットの各々の少なくとも片方の対立遺伝子についての対立遺伝子頻度を含む、方法。
【請求項12】
請求項1に記載の方法であって、前記イヌ科動物個体群プロファイルデータベースは、約5個と約500個と間のイヌ科動物個体群プロファイルを含む、方法。
【請求項13】
請求項1に記載の方法であって、前記イヌ科動物個体群プロファイルデータベースは、少なくとも約5種のアメリカンケンネルクラブ公認血統種についてプロファイルを含む、方法。
【請求項14】
請求項1に記載の方法であって、前記マーカーセットは、約1500個よりも少ないSNPマーカーを含み、そして該方法は、前記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する、方法。
【請求項15】
請求項1に記載の方法であって、前記マーカーセットは、約200個よりも少ないSNPマーカーを含み、そして該方法は、前記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する、方法。
【請求項16】
請求項1に記載の方法であって、工程(a)は、前記マーカーセットの各々に特異的なプライマーを用いて、前記試験イヌ科動物のゲノムDNAを増幅する工程、および該増幅産物のサイズを決定する工程を包含する、方法。
【請求項17】
請求項1に記載の方法であって、工程(b)は、遺伝子型クラスタリングプログラムを用いる工程を包含する、方法。
【請求項18】
請求項1に記載の方法であって、工程(b)は、割当てアルゴリズムを用いる工程を包含する、方法。
【請求項19】
請求項1に記載の方法であって、工程(b)は、特定のイヌ科動物個体群が前記試験イヌ科動物ゲノムへ寄与した確率を決定する工程を包含し、該確率は、該試験イヌ科動物ゲノム中の対立遺伝子が該特定のイヌ科動物個体群中で生じる条件付き確率を、該試験イヌ科動物ゲノム中の対立遺伝子が前記データベース内の、各イヌ科動物個体群中で生じる条件付き確率の合計で割って決定することにより決定される、方法。
【請求項20】
請求項1に記載の方法であって、工程(b)は、前記試験イヌ科動物ゲノムへの2つ以上の遺伝的に関連するイヌ科動物個体群の寄与度の間を、該試験イヌ科動物ゲノム中の対立遺伝子を該2つ以上の遺伝的に関連するイヌ科動物個体群プロファイルを含むデータベースと比較することによって識別する工程を包含する、方法。
【請求項21】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ベルジアンシープドッグおよびベルジアンタービュレンを含む、方法。
【請求項22】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、コリーおよびシェトランドシープドッグを含む、方法。
【請求項23】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ウィペットおよびグレイハウンドを含む、方法。
【請求項24】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、シベリアンハスキーおよびアラスカンマラミュートを含む、方法。
【請求項25】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、マスチフおよびブルマスチフを含む、方法。
【請求項26】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、グレータースイスマウンテンドックおよびバーニーズマウンテンドッグを含む、方法。
【請求項27】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ウェストハイランドホワイトテリアおよびケアーンテリアを含む、方法。
【請求項28】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ラサアプソ、シーズー、およびペキニーズを含む、方法。
【請求項29】
請求項1に記載の方法であって、前記方法は、前記試験イヌ科動物ゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する文書を提供する工程をさらに包含する、方法。
【請求項30】
請求項29に記載の方法であって、前記文書は、前記試験イヌ科動物ゲノムまたは前記イヌ科動物へ寄与した前記1種以上のイヌ科動物個体群に関する情報を提供する、方法。
【請求項31】
請求項30に記載の方法であって、前記情報は、健康に関する情報である、方法。
【請求項32】
請求項30に記載の方法であって、前記情報は、保険情報である、方法。
【請求項33】
請求項29に記載の方法であって、前記文書は、1種以上のイヌ科動物個体群の前記試験イヌ科動物ゲノムのゲノムへの寄与度の証明書を提供する、方法。
【請求項34】
請求項29に記載の方法であって、前記文書は、前記試験イヌ科動物ゲノムへ寄与した1種以上のイヌ科動物個体群の表示を提供する、方法。
【請求項35】
1種以上のイヌ科動物個体群を規定する方法であって、以下の工程:
(a)各イヌ科動物ゲノムセットについて、各マーカーセットのついての片方または両方の対立遺伝子の同一性を得る工程;および
(b)イヌ科動物ゲノムセットの1つ以上の一員が、統計的モデリングを用いて各マーカーについて1セットの対立遺伝子頻度によって特徴付けられる別個のイヌ科動物個体群を規定する可能性を決定することによって、1種以上のイヌ科動物個体群を規定する工程を包含する、方法。
【請求項36】
コンピュータに読み取り可能な媒体であって、イヌ科動物個体群の区別に使用するために、該媒体上に保存されたデータ構造を含み、該データ構造は:
(a)マーカーの名称または該マーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(b)イヌ科動物個体群における該マーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、該マーカーフィールドのインスタンス化および該遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、コンピュータに読み取り可能な媒体。
【請求項37】
コンピュータに読み取り可能な媒体であって、以下:
(a)イヌ科動物個体群の区別に使用するために該媒体上に保存されたデータ構造であって、該データ構造は;
(i)マーカーの名称または該マーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(ii)イヌ科動物個体群における該マーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、該マーカーフィールドのインスタンス化および該遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、データ構造;ならびに
(b)イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を実施するための、コンピュータが実行可能な命令であって、以下の工程:
(i)各マーカーセットについて試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(ii)該試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、該試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物プロファイルを含むデータベースとを比較することによって決定する工程であって、イヌ科動物個体群プロファイルの各々は、該イヌ科動物個体群における該マーカーセットについての遺伝子型情報を含む、工程
を包含する、コンピュータが実行可能な命令;
を該媒体上に保存したものを備える、コンピュータに読み取り可能な媒体。
【請求項1】
イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法であって、以下の工程:
(a)各マーカーセットについての試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(b)該試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、該試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物個体群プロファイルを含むデータベースとを比較することによって決定する工程であって、各イヌ科動物個体群プロファイルは、該イヌ科動物個体群におけるマーカーセットに関する遺伝子型情報を含む、工程
を包含する、方法。
【請求項2】
請求項1に記載の方法であって、前記マーカーセットは、少なくとも約5個のマーカーを含む、方法。
【請求項3】
請求項1に記載の方法であって、前記マーカーセットは、マイクロサテライトマーカーを含む、方法。
【請求項4】
請求項3に記載の方法であって、前記マイクロサテライトマーカーは、表1に示すマイクロサテライトマーカーのうちの少なくとも5個を含む、方法。
【請求項5】
請求項1に記載の方法であって、前記マーカーセットは、一塩基多型(SNP)を含む、方法。
【請求項6】
請求項5に記載の方法であって、前記SNPマーカーは、表2に示すSNPマーカーのうちの少なくとも5個を含む、方法。
【請求項7】
請求項1に記載の方法であって、前記マーカーセットは、1つ以上の個体群特異的マーカーを含む、方法。
【請求項8】
請求項7に記載の方法であって、前記1つ以上の個体群特異的マーカーは、1つ以上のSNPマーカーを含む、方法。
【請求項9】
請求項8に記載の方法であって、前記1つ以上のSNPマーカーは、372c5t−82、372e13t−57、372m6t−88、372m23t−76、373a15t−112、373e1t−50、373elt−130、373g19t−246、373i8s−224、373k8s−181、372c5s−168、372C15S−196、372e15s−71、および373a21t−93からなる群より選択される、方法。
【請求項10】
請求項1に記載の方法であって、イヌ科動物個体群プロファイルにおける前記遺伝子型情報は、前記マーカーセットの各々の片方または両方の対立遺伝子の同一性を含む、方法。
【請求項11】
請求項1に記載の方法であって、イヌ科動物個体群プロファイルにおける前記遺伝子型情報は、前記マーカーセットの各々の少なくとも片方の対立遺伝子についての対立遺伝子頻度を含む、方法。
【請求項12】
請求項1に記載の方法であって、前記イヌ科動物個体群プロファイルデータベースは、約5個と約500個と間のイヌ科動物個体群プロファイルを含む、方法。
【請求項13】
請求項1に記載の方法であって、前記イヌ科動物個体群プロファイルデータベースは、少なくとも約5種のアメリカンケンネルクラブ公認血統種についてプロファイルを含む、方法。
【請求項14】
請求項1に記載の方法であって、前記マーカーセットは、約1500個よりも少ないSNPマーカーを含み、そして該方法は、前記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する、方法。
【請求項15】
請求項1に記載の方法であって、前記マーカーセットは、約200個よりも少ないSNPマーカーを含み、そして該方法は、前記試験イヌ科動物ゲノムへの少なくとも87種のイヌ科動物個体群の寄与度を決定する、方法。
【請求項16】
請求項1に記載の方法であって、工程(a)は、前記マーカーセットの各々に特異的なプライマーを用いて、前記試験イヌ科動物のゲノムDNAを増幅する工程、および該増幅産物のサイズを決定する工程を包含する、方法。
【請求項17】
請求項1に記載の方法であって、工程(b)は、遺伝子型クラスタリングプログラムを用いる工程を包含する、方法。
【請求項18】
請求項1に記載の方法であって、工程(b)は、割当てアルゴリズムを用いる工程を包含する、方法。
【請求項19】
請求項1に記載の方法であって、工程(b)は、特定のイヌ科動物個体群が前記試験イヌ科動物ゲノムへ寄与した確率を決定する工程を包含し、該確率は、該試験イヌ科動物ゲノム中の対立遺伝子が該特定のイヌ科動物個体群中で生じる条件付き確率を、該試験イヌ科動物ゲノム中の対立遺伝子が前記データベース内の、各イヌ科動物個体群中で生じる条件付き確率の合計で割って決定することにより決定される、方法。
【請求項20】
請求項1に記載の方法であって、工程(b)は、前記試験イヌ科動物ゲノムへの2つ以上の遺伝的に関連するイヌ科動物個体群の寄与度の間を、該試験イヌ科動物ゲノム中の対立遺伝子を該2つ以上の遺伝的に関連するイヌ科動物個体群プロファイルを含むデータベースと比較することによって識別する工程を包含する、方法。
【請求項21】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ベルジアンシープドッグおよびベルジアンタービュレンを含む、方法。
【請求項22】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、コリーおよびシェトランドシープドッグを含む、方法。
【請求項23】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ウィペットおよびグレイハウンドを含む、方法。
【請求項24】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、シベリアンハスキーおよびアラスカンマラミュートを含む、方法。
【請求項25】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、マスチフおよびブルマスチフを含む、方法。
【請求項26】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、グレータースイスマウンテンドックおよびバーニーズマウンテンドッグを含む、方法。
【請求項27】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ウェストハイランドホワイトテリアおよびケアーンテリアを含む、方法。
【請求項28】
請求項20に記載の方法であって、前記2つ以上の遺伝的に関連するイヌ科動物個体群は、ラサアプソ、シーズー、およびペキニーズを含む、方法。
【請求項29】
請求項1に記載の方法であって、前記方法は、前記試験イヌ科動物ゲノムへの1種以上のイヌ科動物個体群の寄与度を表示する文書を提供する工程をさらに包含する、方法。
【請求項30】
請求項29に記載の方法であって、前記文書は、前記試験イヌ科動物ゲノムまたは前記イヌ科動物へ寄与した前記1種以上のイヌ科動物個体群に関する情報を提供する、方法。
【請求項31】
請求項30に記載の方法であって、前記情報は、健康に関する情報である、方法。
【請求項32】
請求項30に記載の方法であって、前記情報は、保険情報である、方法。
【請求項33】
請求項29に記載の方法であって、前記文書は、1種以上のイヌ科動物個体群の前記試験イヌ科動物ゲノムのゲノムへの寄与度の証明書を提供する、方法。
【請求項34】
請求項29に記載の方法であって、前記文書は、前記試験イヌ科動物ゲノムへ寄与した1種以上のイヌ科動物個体群の表示を提供する、方法。
【請求項35】
1種以上のイヌ科動物個体群を規定する方法であって、以下の工程:
(a)各イヌ科動物ゲノムセットについて、各マーカーセットのついての片方または両方の対立遺伝子の同一性を得る工程;および
(b)イヌ科動物ゲノムセットの1つ以上の一員が、統計的モデリングを用いて各マーカーについて1セットの対立遺伝子頻度によって特徴付けられる別個のイヌ科動物個体群を規定する可能性を決定することによって、1種以上のイヌ科動物個体群を規定する工程を包含する、方法。
【請求項36】
コンピュータに読み取り可能な媒体であって、イヌ科動物個体群の区別に使用するために、該媒体上に保存されたデータ構造を含み、該データ構造は:
(a)マーカーの名称または該マーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(b)イヌ科動物個体群における該マーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、該マーカーフィールドのインスタンス化および該遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、コンピュータに読み取り可能な媒体。
【請求項37】
コンピュータに読み取り可能な媒体であって、以下:
(a)イヌ科動物個体群の区別に使用するために該媒体上に保存されたデータ構造であって、該データ構造は;
(i)マーカーの名称または該マーカーの対立遺伝子の名称を保存することが可能である、マーカーフィールド;および
(ii)イヌ科動物個体群における該マーカーについての遺伝子型情報を保存することが可能である遺伝子型情報フィールドであって、1つのレコードは、該マーカーフィールドのインスタンス化および該遺伝子型情報フィールドのインスタンス化を含み、そして1セットのレコードは、1つのイヌ科動物プロファイルを表す、遺伝子型情報フィールド
を備える、データ構造;ならびに
(b)イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を決定する方法を実施するための、コンピュータが実行可能な命令であって、以下の工程:
(i)各マーカーセットについて試験イヌ科動物ゲノム中の片方または両方の対立遺伝子の同一性を得る工程;および
(ii)該試験イヌ科動物ゲノムへのイヌ科動物個体群の寄与度を、該試験イヌ科動物ゲノム中の対立遺伝子と、イヌ科動物プロファイルを含むデータベースとを比較することによって決定する工程であって、イヌ科動物個体群プロファイルの各々は、該イヌ科動物個体群における該マーカーセットについての遺伝子型情報を含む、工程
を包含する、コンピュータが実行可能な命令;
を該媒体上に保存したものを備える、コンピュータに読み取り可能な媒体。
【図1】


【図2】




【図2】


【公開番号】特開2011−115174(P2011−115174A)
【公開日】平成23年6月16日(2011.6.16)
【国際特許分類】
【出願番号】特願2011−30110(P2011−30110)
【出願日】平成23年2月15日(2011.2.15)
【分割の表示】特願2006−545432(P2006−545432)の分割
【原出願日】平成16年12月15日(2004.12.15)
【出願人】(506139369)フレッド ハッチンソン キャンサー リサーチ センター (11)
【Fターム(参考)】
【公開日】平成23年6月16日(2011.6.16)
【国際特許分類】
【出願日】平成23年2月15日(2011.2.15)
【分割の表示】特願2006−545432(P2006−545432)の分割
【原出願日】平成16年12月15日(2004.12.15)
【出願人】(506139369)フレッド ハッチンソン キャンサー リサーチ センター (11)
【Fターム(参考)】
[ Back to top ]

