コアα1,3−フコース転移酵素をコードするDNA、発現ベクター、形質転換体、組換えタンパク質、及び組換えタンパク質を生産する方法
【課題】カイコにおけるコアα1,3−フコース転移酵素活性を有するタンパク質をコードする遺伝子DNAを含む発現ベクター、及びコアα1,3−フコース転移酵素活性を有する組換えタンパク質を提供する。
【解決手段】鱗翅目、カイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNA、および上記DNAを含む発現ベクター、上記のDNAを含む発現ベクターを宿主細胞又は宿主個体に導入し、得られる形質転換体を培養又は飼育し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法。
【解決手段】鱗翅目、カイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNA、および上記DNAを含む発現ベクター、上記のDNAを含む発現ベクターを宿主細胞又は宿主個体に導入し、得られる形質転換体を培養又は飼育し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、コアα1,3−フコース転移酵素活性を有するタンパク質をコードする遺伝子DNAに関し、より具体的にはカイコに由来する該DNAに関する。本発明はさらに、該DNAを含むベクター、形質転換体、さらに、コアα1,3−フコース転移酵素活性を有する組換えタンパク質、及び該タンパク質を生産する方法に関する。
【背景技術】
【0002】
従来、遺伝子操作技術を利用して、ヒトや哺乳動物の治療に用いる糖タンパク質を生産することが検討され、治療用抗体等、実用化された例もある。糖鎖付加の無いタンパク質を大量に生産させる系としては、大腸菌を用いた生産系が主に挙げられるが、この生産系では糖タンパク質への糖鎖付加が生じないことから糖タンパク質の生産においては有効であるとは云い難い。糖タンパク質は生産されたタンパク質に糖鎖が付加しない場合、糖タンパク質本来の機能および活性の低下や生体内半減期の減少を招く場合があることが知られており、現行での糖タンパク質の生産は哺乳動物の細胞を使用した系が主流となっている。しかしながらこの系では細胞あたりの糖タンパク質生産量は限られており、生産コストや手間の面で優れた方法とは云い難い。従って、タンパク質に糖鎖を付加する機能を有する植物体もしくは昆虫細胞等が生産宿主として検討されている。
【0003】
タンパク質の糖鎖構造は、その結合様式により2種類に分類される。1つは、タンパク質のアスパラギン残基に結合するN−結合型糖鎖であって、他方は、タンパク質のセリン残基あるいはスレオニン残基に結合するO−結合型糖鎖である。このうちN−結合型糖鎖について注目すると、動物、植物、昆虫、酵母などでは高マンノース型糖鎖、複合型糖鎖、およびそのハイブリッド型糖鎖が存在する。これらの型は、マンノース3 分子、N−アセチルグルコサミン2 分子からなる、共通する5糖のコア糖鎖構造を糖鎖の還元末端側にもっている。
糖タンパク質のN−結合型糖鎖は、細胞の小胞体において、まず脂質との複合体の形態で合成され、タンパク質に転移する。そして、転移した糖鎖を有するタンパク質は、小胞体からゴルジ体に輸送され、その過程において糖鎖の刈り込みや伸長を受ける。ゴルジ体におけるこの糖鎖の伸長は、末端部糖鎖合成と呼ばれ、生物種に依存して大きく異なることが知られている。さらに、コア糖鎖内の還元末端部位に存在するN-アセチルグルコサミン残基に付加するフコース残基の結合様式も生物種により異なることから知られている。
【0004】
植物体もしくは昆虫細胞等を哺乳類型糖タンパク質の生産宿主とするときに、弊害となるのは、植物もしくは昆虫によって生産された糖タンパク質は哺乳類(例えばヒト)の糖鎖構造と異なる部分があり、よって、ヒトに対して抗原性を有する場合が多いということである。この抗原性は、糖タンパク質のコアα1,3−フコースを含む糖鎖部分に起因する場合がある。すなわち、植物もしくは昆虫は、ヒトとは異なり、糖鎖コア構造の還元末端部位に存在するN−アセチルグルコサミン残基へのフコースのα1,3−結合を有するタンパク質を生産するということである。なお、昆虫ではα1,3−結合及びα1,6−結合の両方が見出されている。
従って、上述の抗原性の原因を熟知し、抗原性を消失または抑制するためには、まず、コアα1,3−フコース転移反応を司る遺伝子である、コアα1,3−フコース転移酵素(コアα1,3−フコシルトランスフェラーゼ)遺伝子を同定する必要がある。
植物では、リョクトウ(Vigna radiata)でα1,3−フコシルトランスフェラーゼ遺伝子を単離・同定したことが報告されている(非特許文献1及び特許文献1参照)。また、昆虫では、ショウジョウバエ(Drosophila melanogaster)でα1,3−フコシルトランスフェラーゼを単離・同定したことが報告されている(非特許文献2参照)。
【0005】
昆虫の中でもカイコを用いたタンパク質の生産系は、その生産量、および生産されたタンパク質の安定性や生産コスト、簡便性などの面で従来から優れた方法として利用されている。しかしながら、上述のコアα1,3−フコースによる抗原性の問題から、ヒトへの利用を目的とした糖タンパク質の生産系としての改良が求められてきた。
特許文献2では、カイコのコアα1,3−フコシルトランスフェラーゼのDNA配列(1,486塩基対)を、カイコのゲノムDNAからPCR法で増幅している。
カイコを用いた糖タンパク質生産系の利便性が注目される中、コアα1,3−フコシルトランスフェラーゼ活性を有するタンパク質をコードする、完全長ORF(open reading frame)cDNAの探索と同定が求められている。
【先行技術文献】
【特許文献】
【0006】
【特許文献1】特表2002−536978号公報
【特許文献2】特開2010−29102号公報
【非特許文献】
【0007】
【非特許文献1】J.Biol.Chem.1999,July 30;274(31);p.21830−21839
【非特許文献2】J.Biol.Chem.2001,July 27;276(30);p.28058−28067
【発明の概要】
【発明が解決しようとする課題】
【0008】
本発明は、カイコにおけるコアα1,3−フコース転移酵素活性を有するタンパク質をコードする遺伝子DNAを同定することによって、さらに、該コアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAを含む発現ベクター、コアα1,3−フコース転移酵素活性を有する組換えタンパク質、及び該タンパク質の生産方法を提供することを目的とする。
【課題を解決するための手段】
【0009】
本発明者らは上記目的を達成するために鋭意研究を重ねた結果、カイコ(具体的には、Bombyx mori)のゲノムデータベースにおいて7番染色体上に、ショウジョウバエのコアα1,3−フコース転移酵素活性を有するアミノ酸配列の一部をコードする相同な配列を見出し、本発明を完成させるに至った。
従って本発明は先ず、鱗翅目のコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAに向けられている。ここで鱗翅目動物の例としては、アゲハチョウ科(Papilionidae)、タテハチョウ科(Nymphalidae)、ヤガ科(Noctuidae)、スズメガ科(Sphingidae)、カイコガ科(Bombycidae)等が挙げられ、ギフチョウ、オオムラサキ、スズメガ、カイコガ等が挙げられる。
本発明は、より具体的には、カイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAに向けられている。本発明はより具体的に、下記の配列番号1の塩基配列で表されるカイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAである。
【0010】
配列番号1:
atgcgggtga gggcggctcg ggccctgcgt gctttaagac gcgtggcact cttgtttccg
ctagtattga ctgcgctggc gctaatcctg ctgccgcgcc cgggacctct cacacccctt
gcacagaatc ctaccattgc cgcacagatc cactcgctca aagacgacca gcatattcaa
gatcgttcca atgaacttca agaagaggtg ctgcacgagg aaggtgacac cgtatcccgt
cgccactggt tcatgcgcgg aggtgaacaa cggccttgga aacacgatcc tagtgcacaa
atattccctg aggatgcccc tggggatgac agaatcgttc aacagctatc atacatatta
ccgaacgacg aggaggcgcc tattaaaaaa atactacttg ccaatggcct cggggcgtgg
ggcgtaaccg gtggtcgtac agaattcctc cgaaacaagt gccccataga ccggtgtcat
cttacggcgg actcgaggga tgccgcctct gcagacgcta tactcttcaa agatcaccat
acgcctttta acgtcaaaag atctcctaat cagatttgga ttctctacta cttggaatgt
ccttaccaca cggcttcctt gcggtcttca tccttcgacg tatttaactg gactgcaact
taccgcagag actctgatat agtagcacct tacgaaagat gggtgtatta tgataatttg
attcctgaaa aagatataga aaggaactat gcagctaata aaacaaagaa ggtagcgtgg
tttgtatcaa actgccacgc tcgcaaccgt cggctccagt acgcgcggca actcagcaaa
ttcatccaag tggacatata cggcgcgtgc ggctctcatc actgcccacg aaccgacccc
aactgcttgg aaatgttgga caaagaatac aagttttatc tggcttttga aaattcaaac
tgtcgggatt acataacgga aaaattcttc gtcaatggat tacaacacaa cgttttaccg
atcgtgatgg gagcccggcc atcggagtat gcagcagtgg ctccacgtaa ttcgtacatt
catgtagaag agtttgccgg tcctgaagaa ctcgctgcgt acttacatcg ccttgatgaa
gaccaagatc tatacaactc ttactttaaa tggaagggaa caggagaatt cattaacaca
tatttctttt gccgcgtatg tgcgatgatt cacgcgaacg aaagacggca aaggaatact
cactacagcg acgtgcaggc atggtggcgg gactccacgt gcacgcgagg cgaatggcga
gccatggaat ctgatactaa agataatgga taa
【0011】
本発明はまた、下記(a)又は(b)のタンパク質をコードするDNAに向けられている。
(a)下記の配列番号2のアミノ酸配列からなるタンパク質
(b)下記の配列番号2のアミノ酸配列において1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
【0012】
配列番号2:
Met Arg Val Arg Ala Ala Arg Ala Leu Arg Ala Leu Arg Arg Val Ala
Leu Leu Phe Pro Leu Val Leu Thr Ala Leu Ala Leu Ile Leu Leu Pro
Arg Pro Gly Pro Leu Thr Pro Leu Ala Gln Asn Pro Thr Ile Ala Ala
Gln Ile His Ser Leu Lys Asp Asp Gln His Ile Gln Asp Arg Ser Asn
Glu Leu Gln Glu Glu Val Leu His Glu Glu Gly Asp Thr Val Ser Arg
Arg His Trp Phe Met Arg Gly Gly Glu Gln Arg Pro Trp Lys His Asp
Pro Ser Ala Gln Ile Phe Pro Glu Asp Ala Pro Gly Asp Asp Arg Ile
Val Gln Gln Leu Ser Tyr Ile Leu Pro Asn Asp Glu Glu Ala Pro Ile
Lys Lys Ile Leu Leu Ala Asn Gly Leu Gly Ala Trp Gly Val Thr Gly
Gly Arg Thr Glu Phe Leu Arg Asn Lys Cys Pro Ile Asp Arg Cys His
Leu Thr Ala Asp Ser Arg Asp Ala Ala Ser Ala Asp Ala Ile Leu Phe
Lys Asp His His Thr Pro Phe Asn Val Lys Arg Ser Pro Asn Gln Ile
Trp Ile Leu Tyr Tyr Leu Glu Cys Pro Tyr His Thr Ala Ser Leu Arg
Ser Ser Ser Phe Asp Val Phe Asn Trp Thr Ala Thr Tyr Arg Arg Asp
Ser Asp Ile Val Ala Pro Tyr Glu Arg Trp Val Tyr Tyr Asp Asn Leu
Ile Pro Glu Lys Asp Ile Glu Arg Asn Tyr Ala Ala Asn Lys Thr Lys
Lys Val Ala Trp Phe Val Ser Asn Cys His Ala Arg Asn Arg Arg Leu
Gln Tyr Ala Arg Gln Leu Ser Lys Phe Ile Gln Val Asp Ile Tyr Gly
Ala Cys Gly Ser His His Cys Pro Arg Thr Asp Pro Asn Cys Leu Glu
Met Leu Asp Lys Glu Tyr Lys Phe Tyr Leu Ala Phe Glu Asn Ser Asn
Cys Arg Asp Tyr Ile Thr Glu Lys Phe Phe Val Asn Gly Leu Gln His
Asn Val Leu Pro Ile Val Met Gly Ala Arg Pro Ser Glu Tyr Ala Ala
Val Ala Pro Arg Asn Ser Tyr Ile His Val Glu Glu Phe Ala Gly Pro
Glu Glu Leu Ala Ala Tyr Leu His Arg Leu Asp Glu Asp Gln Asp Leu
Tyr Asn Ser Tyr Phe Lys Trp Lys Gly Thr Gly Glu Phe Ile Asn Thr
Tyr Phe Phe Cys Arg Val Cys Ala Met Ile His Ala Asn Glu Arg Arg
Gln Arg Asn Thr His Tyr Ser Asp Val Gln Ala Trp Trp Arg Asp Ser
Thr Cys Thr Arg Gly Glu Trp Arg Ala Met Glu Ser Asp Thr Lys Asp
Asn Gly
【0013】
本発明はまた、下記(a)又は(b)のDNAである。
(a)配列番号1の塩基配列からなるDNA
(b)配列番号1に示す塩基配列からなるDNAと相補的な塩基配列からなるDNAと、ストリンジェントな条件下でハイブリダイズし、かつコアα1,3−フコース転移酵素活性を有するタンパク質をコードするカイコ由来のDNA
【0014】
本発明によるDNAを用いて組換えタンパク質を生産することができる。従って、本発明はさらに、上記のDNAを含む発現ベクター、及び該発現ベクターを宿主細胞に導入して得られる形質転換体に向けられている。宿主として例えば哺乳類細胞株が挙げられる。
本発明はまた、下記(a)又は(b)の組換えタンパク質に向けられている。
(a)配列番号2のアミノ酸配列からなるタンパク質。
(b)配列番号2のアミノ酸配列の1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
本発明によるDNAを用いて組換えタンパク質を生産する方法として、上記のDNAを含む発現ベクターを宿主細胞に導入し、得られる形質転換体を培養し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法がある。
本発明によるDNAを用いて組換えタンパク質を生産する方法としてまた、上記のDNAを含む発現ベクターを宿主個体に導入し、得られる形質転換体を飼育し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法がある。
なお、本明細書中において、フコース転移酵素を“FucT”と略記し、例えばコアα1,3−フコース転移酵素を“コアα1,3-FucT”と略記する。
【発明の効果】
【0015】
本発明による、コアα1,3−フコース転移酵素活性を有するタンパク質(コアα1,3−フコース転移酵素、あるいはコアα1,3−フコシルトランスフェラーゼ)をコードするDNAを利用して得られる組換えタンパク質、すなわちコアα1,3−フコース転移酵素活性を有する組換えタンパク質は、試薬として有用である。より具体的には、該組換えタンパク質は、N-結合型糖鎖に関して、コアα1,3−フコースを有する糖鎖、及びその糖鎖を有する糖タンパク質を合成するための合成用酵素として有用である。
【図面の簡単な説明】
【0016】
【図1】クローニングにおける3’RACEでのPCR結果を表す電気泳動の写真である。
【図2】クローニングにおける3’RACEでのPCR結果を表す電気泳動の写真である。
【図3】クローニングにおける5’RACEでのPCR結果を表す電気泳動の写真である。
【図4】クローニングにおける5’RACEでのPCR結果を表す電気泳動の写真である。
【図5】FucTAはDrosophila melanogaster FucTA (core alpha(1,3)-fucosyltransferase)遺伝子(NCBI Number: AJ302045)にコードされるタンパク質のアミノ酸配列。FucTは本実施例で単離されたカイコのcore alpha(1,3)-fucosyltransferase遺伝子にコードされるタンパク質のアミノ酸配列。FucTAとFucTのタンパク質のアミノ酸配列上の相同性を比較した。
【図6】Genome上でのエクソン-イントロン構造、及び翻訳開始コドンを概略的に表す。
【図7】CHO細胞株の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した結果を表す。
【図8】CHO細胞のコアα1,3-FucT形質転換体の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した結果を表す。コントロールとしてCHO細胞株の粗抽出液を用いた。
【図9】CHO細胞のコアα1,3-FucT形質転換体の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した結果を表す。GDP-フコースを添加しない検体をコントロールとした。
【図10】CHO細胞株の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をN−グリコシダーゼ処理に供し、生成物をHPLCにて分析した結果を表す。
【図11】CHO細胞のコアα1,3-FucT形質転換体の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をN−グリコシダーゼ処理に供し、生成物をHPLCにて分析した結果を表す。
【図12】CHO細胞株の粗抽出液又はCHO細胞のコアα1,3-FucT形質転換体の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させる実験において、反応時間を変動させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した結果を表す。
【発明を実施するための形態】
【0017】
本発明者らは、カイコ(Bombyx mori)の幼虫から、コアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAを同定した。
本明細書中において、コアα1,3−フコース転移酵素活性を有するタンパク質は、コアα1,3−フコース転移酵素又はコアα1,3−フコシルトランスフェラーゼとも呼称される。コアα1,3−フコシルトランスフェラーゼとは、糖タンパク質のタンパク質部分の合成後、N-結合型糖鎖が付加したペプチド鎖に最も近い、糖鎖の還元末端のN-アセチルグルコサミン残基に、GDP−フコースを糖供与体として、α1,3−結合でフコース残基を付加する酵素である。
【0018】
本発明は、より具体的には配列番号1の塩基配列で表されるカイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAを提供する。
本発明はまた、下記(a)又は(b)のタンパク質をコードするDNAを提供する。
(a)配列番号2のアミノ酸配列からなるタンパク質
(b)配列番号2のアミノ酸配列において1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
ここで、「1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列」は、そのアミノ酸配列からなるタンパク質が所望の機能を失わない限り、その欠失、置換もしくは付加されるアミノ酸の数は限定されるものではない。例えば、置換などされるアミノ酸の数は、30個以下、好ましくは10個以下、より好ましくは5個以下、特に好ましくは3個以下である。
【0019】
本発明はまた、具体的に下記(a)又は(b)のDNAを提供する。
(a)配列番号1の塩基配列からなるDNA
(b)配列番号1に示す塩基配列からなるDNAと相補的な塩基配列からなるDNAと、ストリンジェントな条件下でハイブリダイズし、かつコアα1,3−フコース転移酵素活性を有するタンパク質をコードするカイコ由来のDNA
ここで、「ストリンジェントな条件」とは、相同遺伝子をコードするDNAを単離するために適したハイブリダイゼーション反応条件を意味し、すなわち特異的なハイブリッドが形成され、非特異的なハイブリッドが形成されない条件を言い、当業者にとって公知の方法によって決定することができる。「ストリンジェントな条件でハイブリダイズするDNA」として、プローブとして使用するDNAの塩基配列と一定以上の相同性を有するDNAが挙げられ、例えば70%以上、好ましくは80%以上、より好ましくは90%以上、更に好ましくは95%以上、最も好ましくは98%以上の相同性を有するDNAである。
本発明のDNAは、コアα1,3−フコシルトランスフェラーゼをコードするものであれば、その形態に制限がなく、cDNA由来のもの、ゲノムDNA由来のもの、化学合成DNA、遺伝暗号の縮重に基づく任意の塩基配列を有するDNAなどを包含する。
【0020】
本発明のDNAを同定するまでの経緯について、以下のとおりである。
カイコ染色体の塩基配列は未だ不明瞭な領域が存在する。カイコのコアα1,3−フコシルトランスフェラーゼ遺伝子を同定する際、その候補遺伝子が得られた段階でカイコ染色体上での塩基配列を確認したところ、報告されていない部分と重なったため、得られた候補遺伝子がカイコ染色体上に実在するかといった問題を簡単には解決することができなかった。従ってカイコよりゲノムDNAを抽出し、候補遺伝子が存在すると仮定されるゲノムDNA部分の塩基配列を解読することにより、本遺伝子がexon-intron構造をとることやデータベースにおいて7番染色体上に存在することなどを確認することで遺伝子として実在するか検証をおこなう必要があった。
【0021】
他の生物種との相同性について、以下のとおりである。
コアα1,3−フコシルトランスフェラーゼ遺伝子は、リョクトウやショウジョウバエなど、幾つかの種において同定されている。従って通常は種間で保存されたアミノ酸配列領域を基に相同性検索をおこなうことで、カイコにおける本タンパク質の候補配列を得ることができると想定されるが、昆虫であるショウジョウバエのコアα1,3−フコース転移酵素と、同定したカイコのコアα1,3−フコース転移酵素との相同性は、相同性の高い部分でもわずか49%(全長では47.3%)であって、相同性が高いとは云い難い。また、タンパク質の長さはリョクトウが510アミノ酸であり、ショウジョウバエが503アミノ酸であるのに対し、カイコは450アミノ酸であった。
染色体上でもカイコのコアα1,3−フコース転移酵素遺伝子の特徴は、他の生物種の遺伝子と異なっており、リョクトウやショウジョウバエにおいては約2Kbpのゲノム塩基配列上に4つのエクソンから構成されるexon-intron構造を持つのに対し、カイコでは約10Kbpのゲノム塩基配列上に7つのエクソンから構成されるexon-intron構造を持つ。またコアα1,3−フコース転移酵素のアミノ酸配列において、コア酵素に特有な、種を超えて保存性の高い領域(モチーフ)は報告されておらず、よって、遺伝子の全長を単離し、そのコードするタンパク質の酵素活性を確認するまでは本遺伝子がコアα1,3−フコース転移酵素であるか、同定するのは容易でなかったという経緯がある。すなわちカイコから得られた遺伝子が他の生物種の当該遺伝子と同様の機能を有することを確定することは容易ではなかった。そこで我々は鋭意研究を重ねた結果、カイコのRNAから完全長ORFを持つcDNAを単離し、その配列から予測されるアミノ酸配列の中にこれまで報告された他の生物種のα1,3−フコース転移酵素に共通する4つのモチーフを見出した。さらに、得られたcDNAのコードするタンパク質を発現させ、その酵素活性を測定し、本遺伝子がカイコのコアα1,3−フコース転移酵素であることを同定し、本発明を完成するに至った。
【0022】
本発明によるDNAを用いて組換えベクターを構築し、適当な宿主へ該ベクターを導入し、形質転換体を得て、下記の(a)又は(b)の組換えタンパク質を生産させることができる。
(a)配列番号2のアミノ酸配列からなるタンパク質。
(b)配列番号2のアミノ酸配列の1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
本発明のDNAを用いて、組換えタンパク質を生産するには、例えば、Molecular Cloning(出版社:Cold spring harbor laboratory press)等の多くの教科書や文献に基づいて実施することができる。具体的には、転写を制御するプロモーター配列(例えば、trp、lac、T7、SV40初期プロモーター)などの制御遺伝子を付加し、適当なベクター(例えば、pBR322、pUC19、pSV・SPORT1など)に組み込むことにより、宿主細胞内で複製し、機能する発現ベクター(例えば発現プラスミド)を作製する。そのようなベクターは一般的に複製開始点、選択マーカー、プロモーターを含み、必要に応じてエンハンサー、ターミネーター、リボソーム結合部位、ポリアデニル化シグナルなどを含んでいてもよい。
【0023】
発現ベクターとして、導入遺伝子が宿主細胞に導入されたか否か、若しくは宿主細胞中で確実に発現しているか否かを確認するために、各種選択マーカーを含むものが好ましい。このようなマーカーの例として、テトラサイクリン耐性遺伝子、アンピシリン耐性遺伝子、カナマイシン耐性遺伝子、ハイグロマイシン耐性遺伝子などの抗生物質耐性マーカー遺伝子がある。
また、組換えタンパク質を、形質転換体の細胞外へ分泌可能な状態で得ることもできる。例えば発現ベクターにシグナルペプチドをコードする配列を適当に組み込むことにより、あるいはシグナルペプチドをコードする市販の適当なベクターを用いることにより、組換えタンパク質を形質転換体の細胞外へ分泌させることができる。
実際に、市販されている種々の発現用ベクターから適宜選択し、該発現用ベクターのプロトコルに従って、本発明のDNAを含む発現ベクターを構築することができる。
市販されている発現用ベクターの例として、宿主細胞が大腸菌である場合は、例えばpETベクター(Novagen社製)、pTrxFUSベクター(Invitrogen社製)、pCYBベクター(NEW ENGLAMD Bio Labs社製)などが、宿主細胞が酵母である場合は、例えばpESP-1発現ベクター(STRATAGENE社製)、pAUR123ベクター(宝酒造社製)、pPICベクター(Invitrogen社製)などが、また宿主細胞が動物細胞である場合は、例えばpMAM-neo発現ベクター(CLONTECH社製)、pcDNA3.1ベクター(Invitrogen社製)、pBK-CMVベクター(STRATAGENE社製)などが、宿主細胞が昆虫細胞である場合は、例えばpBacPAKベクター(CLONTECH社製)、pAcUW31ベクター(CLONTECH社製)、pAcP(+)IE1ベクター(Novagen社製)などが、それぞれ挙げられる。
【0024】
こうして構築された発現ベクターは、例えばポリエチレングリコール法、エレクトロポレーション法、アグロバクテリウム法、パーティクルガン法、塩化カルシウム法、プロトプラスト法、インジェクション法などの当業者に公知の方法によって、宿主細胞に導入することができる。
こうして、発現ベクターを適当な宿主細胞に導入して、形質転換体を得る。宿主細胞としては、大腸菌などの原核生物、酵母のような単細胞真核生物、昆虫細胞、植物細胞、哺乳類などの多細胞生物の細胞や個体などが挙げられる。好ましくは、昆虫の細胞や個体であり、昆虫の細胞や個体としては、sf9細胞、BmN4細胞、カイコ幼虫(品種:w1-pnd)などが例示できる。また好ましくは、哺乳類の細胞であり、哺乳類の細胞としては、CHO細胞、293細胞、COS−7細胞などが例示できる。
また、形質転換体に導入されたDNAは、ハイブリダイゼーションやPCRなどの当業者に公知の方法によって、転写産物発現を確認することができる。
本発明によるDNAを用いて組換えタンパク質を生産する方法において、形質転換体である細胞や個体を、培地又は餌、温度、日数、雰囲気などの適当な条件下で培養又は飼育する。形質転換体により生産された組換えタンパク質を回収する実施態様として、形質転換体を破砕し、その上清から組換えタンパク質を抽出する方法、あるいは形質転換体外、すなわち培養培地や繭などに分泌された組換えタンパク質を回収する方法などが挙げられる。さらに、精製工程を設けて、精製された組換えタンパク質を得ることもできる。
【実施例】
【0025】
以下に本発明を、詳細に説明する。
[コアα1,3−フコース転移酵素のクローニング]
野生型蚕(品種:w1-pnd)の組織由来RNAから、3'および5'RACE法にてα1,3−フコース転移酵素遺伝子をクローニングし、その塩基配列を決定した。
1.相同性検索
Drosophila melanogaster FucTA (core alpha(1,3)-fucosyltransferase)遺伝子(NCBI Number: AJ302045)を基に、アミノ酸配列相同性検索(NCBI Blast)を行い、Bombyx mori genome*上で相同性の高い下記の配列を得た。この配列をカイコのコアα1,3-FucT cDNA断片とした。
(* NCBI number: AADK01016505; Bombyx mori strain Dazao whole genome shotgun seq.)
Drosophila melanogaster FucTA (core alpha(1,3)-fucosyltransferase)遺伝子(NCBI Number: AJ302045)を基に、アミノ酸配列相同性検索(NCBI Blast)を行い、Bombyx mori genome上で相同性のある配列(NCBI number: AADK01016505; Bombyx mori strain Dazao whole genome shotgun seq.)を得た。この相同性のある領域は5箇所あり、それぞれの領域における同一のアミノ酸の割合はDrosophilaのアミノ酸の数を分母に、カイコのアミノ酸の数を分子にして記載すると、57/79、40/59、40/68、40/88、30/72であり必ずしも相同性が高いとは判断されなかった。しかしながら、このカイコのゲノム配列から想定されるタンパク質をコードするcDNA配列の断片(下記)をカイコのコアα1,3-FucT cDNA断片として、当該タンパク質の全長ORF(Open reading frame、タンパク質の読み取り枠)のクローニングと当該酵素活性の測定を試みることとした。
agaatcgttcaacagctatcatacatattaccgaacgacgaggaggcgcctattaaaaaaatactacttgccaatggcctcggggcgtggggcgtaaccggtggtcgtacagaattcctccgaaacaagtgccccatagaccggtgtcatcttacggcggactcgagggatgccgcctctgcagacgctatactcttcaaagatcaccatacgccttttaacgtcaaaagatctcctaatcaggtaacggtacagatttggattctctactacttggaatgtccttaccacacggcttccttgcggtcttcatccttcgacgtatttaactggactgcaacttaccgcagagactctgatatagtagcaccttacgaaagatgggtgtattatgataatttgattcctgaaaaagatatagaaaggaactatgcagctaataaaacaaagaaggtacaggtagcgtggtttgtatcaaactgccacgctcgcaaccgtcggctccagtacgcgcggcaactcagcaaattcatccaagtggacatatacggcgcgtgcggctctcatcactgcccacgaaccgaccccaactgcttggaaatgttggacaaagaatacaagttttatctggcttttgaaaattcaaactgtcgggattacataacggaaaaattcttcgtcaatggattattaagacacaacgttttaccgatcgtgatgggagcccggccatcggagtatgcagcagtggctccacgtaattcgtacattcatgtagaagagtttgccggtcctgaagaactcgctgcgtacttacatcgccttgatgaagaccaagatctatacaactcttactttaaatggaag
【0026】
2.クローニング
上記カイコのコアα1,3-FucT cDNA断片は3'末端側、5'末端側共にORF全長の末端配列を含んではいないため、それぞれ以下の方法で延長させた。
2−1.3'RACE
カイコ幼虫(品種:w1-pnd)より、Invitrogen社製の「TRIzol」のプロトコルに従って、total RNAを抽出した。
上記total RNA 1.5μg分をtemplate とし、Oligo dT primerを用いて逆転写反応を行いcDNAを合成した。コアα1,3-FucT cDNA断片内、既知配列特異的上流primer A1(gcagcagtggctccacgtaattcg:配列番号3)を用いてPCR(反応条件として、94℃2分、「94℃30秒、55℃30秒、72℃2分」を35サイクル、72℃5分にて行った)を行った。
そのPCRの結果を、図1に示す。
その結果、PCR産物が複数得られたので、それぞれのバンドを0.8% アガロースにて分離・精製後(Promega社製「Wizard SV Gel and PCR Clean-up System」のプロトコル参照)、既知配列特異的上流配列および制限酵素切断部位を含むprimer A2(ctgatctagaggtaccggatccggtcctgaagaactcgctgcgtacttacatcg:配列番号4)を用いてPCRを行い(反応条件は上記同様)制限酵素BamHIで切断し、pBluescript II SK(+)に導入することによってcDNA断片を得た(TAKARA社製「3'-Full RACE Core set」のプロトコル参照)。
そのPCRの結果を図2に示す。
【0027】
得られたcDNA配列をシークエンスし、下記の目的とする3'ORF末端配列を得た。
ggaacaggagaattcattaacacatatttcttttgccgcgtatgtgcgatgattcacgcgaacgaaagacggcaaaggaatactcactacagcgacgtgcaggcatggtggcgggactccacgtgcacgcgaggcgaatggcgagccatggaatctgatactaaagataatggataa
【0028】
2−2. 5'RACE
カイコ幼虫(品種:w1-pnd)よりtotal RNAを抽出した(Invitrogen社製「TRIzol」のプロトコル参照)。
次いでtotal RNAよりmRNAを精製した(Invitrogen社製「FastTrack MAG mRNA Isolation Kits」のプロトコル参照)。
次に、上記mRNA 0.5μgをtemplateとして、5'末端既知配列特異的リン酸化primer B1(gtaaggtgctac:配列番号5)を用いて逆転写反応を行い、1st strand cDNAを合成した。RNAを分解した後、一本鎖cDNAを環化させてコアα1,3-FucT cDNA断片内既知配列特異的primer B2(cttggaatgtccttaccacacg:配列番号6)、B3(gtctatggggcacttgtttc:配列番号7)を用いてPCRを行い(反応条件として、94℃2分、「94℃30秒、55℃30秒、72℃2分」を37サイクル、72℃5分にて行った)複数のcDNA断片を得た。
そのPCRの結果を図3に示す。
その結果、PCR 産物が複数得られたので其々のバンドを3% アガロースにて分離・精製後、既知配列特異的primer B4(ggactgcaacttaccgcagagac:配列番号8)、B3(gtctatggggcacttgtttc:配列番号9)を用いてPCRを行い(反応条件として、94℃2分、「94℃30秒、55℃30秒、72℃2分」を37サイクル、72℃5分にて行った)、cDNA断片を得た。
そのPCRの結果を図4に示す。
これらcDNA断片をTA cloning Vector pCR2.1へ導入することによって、目的とする5'ORF末端配列を含むcDNA断片を得た(Invitrogen社製「FastTrack MAG mRNA Isolation Kits」のプロトコル参照)。
【0029】
得られたcDNA配列をシークエンスし、以下の目的とする5'ORF末端配列を得た。
atgcgggtgagggcggctcgggccctgcgtgctttaagacgcgtggcactcttgtttccgctagtattgactgcgctggcgctaatcctgctgccgcgcccgggacctctcacaccccttgtacagaatcctaccattgccgcacagatccactcgctcaaagacgaccagcatattcaagatcgttccaatgaacttcaagaagaggtgctgcacgaggaaggtgacaccgtatcccgtcgccactggttcatgcgcggaggtgaacaacggccttggaaacacgatcctagtgcacaaatattccctgaggatgcccctggggatgac
【0030】
2−3.cDNAの取得
得られたORF領域の配列情報より、primer C1(atgcgggtgagggcggctcg:配列番号10)、C2(ttatccattatctttagtatcagattcc:配列番号11)、およびtemplate としてカイコ幼虫(品種:w1-pnd)由来total RNAを用いてPCRを行い(反応条件として、94℃2分、「94℃30秒、55℃30秒、72℃2分」を37サイクル、72℃5分にて行った)、カイコのコアα1,3-FucTcDNAを得た。これをTA cloning Vector pCR2.1へ導入し、カイコのコアα1,3-FucTcDNAの全配列を確認した(コアα1,3-FucT ORF 全長cDNA:配列番号1)。
情報として得られたカイコのコアα1,3-FucT cDNA断片部分(上記「1.相同性検索」で得たコアα1,3-FucT cDNA断片の配列)と、実際に得られた配列(配列番号1)を比較することにより、3箇所塩基配列が異なる部位が見受けられた。この3箇所は、上記「1.相同性検索」で得たコアα1,3-FucT cDNA断片の配列中、下線で示した箇所である。この領域はいずれもexon-intronの境界部分であり、相同性検索時の解析ソフトの影響を受ける。D. melanogaster FucTAと比較した結果、配列番号1の配列を、目的とするカイコのコアα1,3-FucTの遺伝子配列とした。
カイコのコアα1,3-FucT ORF 全長cDNA配列(配列番号1)を基にタンパク質のアミノ酸配列が決定された(配列番号2)。
【0031】
3.D. melanogaster FucTAとのタンパク質のアミノ酸配列上の相同性
得られたカイコのコアα1,3-FucTの全長部位とD. melanogaster FucTA とでタンパク質のアミノ酸配列上の相同性を比較した(図5参照)。図5中、FucTAは、D. melanogaster FucTA(core alpha(1,3)-fucosyltransferase)遺伝子(NCBI Number: AJ302045)を表し、FucTは、配列番号2のコアα1,3-FucT タンパク質のアミノ酸配列を表す。なお、相同性の比較には、ClustalW2(http://www.ebi.ac.uk/Tools/clustalw2/index.html)を使用した。
【0032】
4.Genome上での塩基配列確認
配列番号1の配列を基にBlast検索をおこなうことによって、得られた配列が二つのBombyx mori genome shotgun sequence上に存在することが判明したことから、exon-intron構造を決定し、およびORFのfirst ATG(予想される開始コドン)が、得られた配列上に存在することを確認した(図6参照)。図6中、6−1.はカイコのコアα1,3-FucT遺伝子のexon-intron構造を表す。黒色のexonはORF領域を表し、淡色のexonは開始コドンより上流の非翻訳領域(5'UTR部位)を表す。6−1において、AADK01016505は Bombyx mori strain Dazao whole genome shotgun seq.を意味し、AADK01011744は Bombyx mori strain Dazao, whole genome shotgun seq.を意味する。
図6中、6−2において、濃い下線部分は得られた5'ORF領域を表し、及びその上流にある淡い下線部分は5'UTR部位のゲノム塩基配列である。ATG(枠付き)より15塩基上流部位に終止コドン(枠付きTAA)が存在することから、このATGが翻訳開始コドンであると予想される。
【0033】
また、2つのgenome shotgun seq.のgapを埋めるため、カイコ幼虫(品種:w1-pnd) 5齢3日目より得られた脂肪体よりgenomic DNAを抽出した(三光純薬製「Sepa Gene」のプロトコル参照)。2種類のBombyx mori genome shotgun seq.末端部分primer C1(cgaggagtgaagtggtgtga:配列番号12)、C2(ccggaacaaatattccgttgt:配列番号13)よりgenomic PCRを行い、得られた断片の配列をシークエンスすることで、上記gap配列は約2.2Kbpであることが判明した。また、図6中、6−1.に2つのgenome shotgun seq.とgap配列との関係を記載した。
その他、Bombyx mori genome*上の塩基配列と実際に得られたカイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)とを比較したところ、配列は完全に一致した。しかしながら、上記2−2において得た5’ORF末端の配列とは1箇所異なる塩基が見受けられた(該5’ORF末端配列の下線部分参照)。そこでさらに、抽出したgenomic DNAの配列を調べたところ、Bombyx mori genome*上の塩基配列と一致したことから、上記2−2において違いの見受けられた塩基はチミン(上記2−2表記上のt)ではなくシトシン(c)であることが確定した。よってカイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)))がBombyx mori genome*上の塩基配列と一致した。
(* NCBI number: AADK01016505; Bombyx mori strain Dazao whole genome shotgun seq.)
【0034】
なお、配列番号1の配列の上流にある配列を含めたDNA配列を、以下に配列番号14として示す。
配列番号14:
atagccgggc attacacata cgattctaat tccattgaaa tggatataat aataaagaag
tgcatctgga ccggggttca gggcggataa ccgcgagtcg agatgcgggt gagggcggct
cgggccctgc gtgctttaag acgcgtggca ctcttgtttc cgctagtatt gactgcgctg
gcgctaatcc tgctgccgcg cccgggacct ctcacacccc ttgcacagaa tcctaccatt
gccgcacaga tccactcgct caaagacgac cagcatattc aagatcgttc caatgaactt
caagaagagg tgctgcacga ggaaggtgac accgtatccc gtcgccactg gttcatgcgc
ggaggtgaac aacggccttg gaaacacgat cctagtgcac aaatattccc tgaggatgcc
cctggggatg acagaatcgt tcaacagcta tcatacatat taccgaacga cgaggaggcg
cctattaaaa aaatactact tgccaatggc ctcggggcgt ggggcgtaac cggtggtcgt
acagaattcc tccgaaacaa gtgccccata gaccggtgtc atcttacggc ggactcgagg
gatgccgcct ctgcagacgc tatactcttc aaagatcacc atacgccttt taacgtcaaa
agatctccta atcagatttg gattctctac tacttggaat gtccttacca cacggcttcc
ttgcggtctt catccttcga cgtatttaac tggactgcaa cttaccgcag agactctgat
atagtagcac cttacgaaag atgggtgtat tatgataatt tgattcctga aaaagatata
gaaaggaact atgcagctaa taaaacaaag aaggtagcgt ggtttgtatc aaactgccac
gctcgcaacc gtcggctcca gtacgcgcgg caactcagca aattcatcca agtggacata
tacggcgcgt gcggctctca tcactgccca cgaaccgacc ccaactgctt ggaaatgttg
gacaaagaat acaagtttta tctggctttt gaaaattcaa actgtcggga ttacataacg
gaaaaattct tcgtcaatgg attacaacac aacgttttac cgatcgtgat gggagcccgg
ccatcggagt atgcagcagt ggctccacgt aattcgtaca ttcatgtaga agagtttgcc
ggtcctgaag aactcgctgc gtacttacat cgccttgatg aagaccaaga tctatacaac
tcttacttta aatggaaggg aacaggagaa ttcattaaca catatttctt ttgccgcgta
tgtgcgatga ttcacgcgaa cgaaagacgg caaaggaata ctcactacag cgacgtgcag
gcatggtggc gggactccac gtgcacgcga ggcgaatggc gagccatgga atctgatact
aaagataatg gataa
【0035】
[カイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)の発現によるコアα1,3−フコース転移酵素活性の測定]
上記のとおりカイコより単離したcDNAがコアα1,3−FucTをコードすることを確認するため、翻訳産物の酵素活性を測定した。
1.対照実験
CHO細胞株の粗抽出液を用いて、ダブシル化グリコペプチドであるダブシル化GnGn−ペプチド(Dabsylated tetrapeptide asialo agalacto biantennery)を基質として、GDP-フコース(GDP-fucose)を反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した。基質であるダブシル化GnGn−ペプチドをdabsylated GnGn peptideと略記し、以下にその構造を示す。
【化1】

【0036】
なお、コアα−フコース転移酵素(core alpha FucT)存在下の、ダブシル化GnGn−ペプチドとGDP-フコースの反応は一般的に次のように表される。
[ダブシル化GnGn−ペプチドとGDP-フコースの反応]
Core alpha FucT dabsylated GnGnF6 peptide 及び/又は
GDP-fucose+dabsylated GnGn -−−--→ dabsylated GnGnF3 peptide及び/又は
peptide dabsylated GnGnF3F6 peptide
F6:糖鎖コア構造の還元末端部位に存在するN-アセチルグルコサミン(GlcNAc)残基にα1,6-結合によりフコースが付加していることを表す。
F3:糖鎖コア構造の還元末端部位に存在するN-アセチルグルコサミン残基にα1,3-結合によりフコースが付加していることを表す。
F3F6:糖鎖コア構造の還元末端部位に存在するN-アセチルグルコサミン残基にα1,6-結合及び1,3-結合によりフコースが付加していることを表す。
【0037】
対照実験における操作手順及び条件は次のとおりである。
<CHO細胞の粗抽出液の調製>
CHO細胞を10% Fetal Bovine Serum(FBS)の存在下でIscove's Modified Dulbecco's Medium培地(Gibco-BRL)を用いて37℃で5%CO2条件下で培養し10cmの培養皿へ1/10希釈で継代し、3日間培養した。底面に付着している細胞をピペッティングにより剥し、遠心分離して上清を廃棄し、再びPBSバッファーに懸濁することで洗浄し、再度遠心分離して上清を廃棄後PBSバッファーを加え、細胞数を計測した。1.0×107cellsを1.5mlのサンプリングチューブへ加え、遠心にて上清を除去し、試料とした。
試料を1mM PMSF、0.1% NP40存在下のPBS(-)1mL、へ加え、sonicatorで30秒2回、氷上にて超音波破砕をおこない、その後19000xgで4℃10分にて遠心分離して上清を回収し、粗抽出液とした。粗抽出液中のタンパク質量はBioRad社製Protein Assay試薬を用いてブラッドフォード法により測定した。その際コントロールとしてBovine serum albuminを標準にして測定し、粗抽出液タンパク質量:22.5μgとした。
その後以下の試薬と混合し、反応温度37度にて所定の時間反応させ、定まった時間経過の後、60℃で5分加熱して反応を停止させた。MilliQ水を110μL添加して、0.45μmフィルターにてろ過し、ろ液をHPLC分析した。なお、GDP-Fucoseを加えない反応では、4μLの蒸留水を加え同様の操作を行った。
【0038】
<反応条件>
反応液の組成
1M MES-NaOH pH7.0 2μL
150mM MnCl2 2μL
1% Triton X-100 2μL
2.5mM GDP-Fucose 4μL
5nmol/μL 基質* 1μL
粗抽出液+MilliQ水 9μL
計 20μL
*dabsylated GnGn peptide(EMD社製)
【0039】
<HLPCの条件>
HLPC分析機器として、Gilson社製HPLCシステムを用いて分析した。
カラムはDevelosil ODS-HG-3 φ3.0×250mm(NOMURA CHEMICAL CO. LTD.)カラムを用い
流速:0.4mL/分、カラム温度:40℃の条件でおこなった。ダブシル化GnGn−ペプチドの検出は465nmの吸光度にておこない、分析したサンプル量を60μLとした。用いたsolventおよび溶出分離条件を以下に示す。
SolventA
1M リン酸ナトリウム(pH6.4) 10mL
アセトニトリル 50mL
Milli Q水 940mL
SolventB
1M リン酸ナトリウム(pH6.4) 10mL
アセトニトリル 500mL
Milli Q水 490mL
溶出分離条件
0分−4分 (B 30%)
4分−35分 (B 30%からB37%までの、直線濃度勾配)
35分−40分 (B 100%)
40分−60分 (B 30%)
【0040】
得られた結果を図7に示す(反応時間:17時間)。図7において、GDP-fucoseあり、と示されているクロマトグラムが表すように、ピークが2つ検出された。これは元の反応基質であるdabsylated GnGn(図7中、GnGnDABSと示される)、CHO細胞中に存在するコアα1,6-FucTによるフコース転移によって生じたdabsylated GnGnF6(図7中、GnGnF6と示されている)とである。一方で、GDP-fucoseを添加しなかったサンプルからは、反応基質以外のピークは検出されなかった。
このようにして、CHO細胞の内在性のコアα1,6-FucT活性を測定することができた。
【0041】
2.発現ベクター及びコアα1,3-FucT形質転換体の作製
カイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)を用いて、哺乳類細胞であるCHO細胞へ形質転換し、発現させた。
<コアα1,3-FucT形質転換体の作製手順>
Invitrogen社製の「pcDNA3.1/Hygro」を用いて、そのプロトコルに従って、全長コアα1,3-FucTのcDNA(ORF全長を含む)を、BamHI-XbaI siteを用いて導入した。CHO細胞へトランスフェクションし、ハイグロマイシンBによる300μg/mLでの選択の後、約1ヶ月選択し、その後細胞を回収し、RT-PCRによってRNAレベルでの転写産物発現の確認を行った。
【0042】
3.CHO細胞のコアα1,3-FucT形質転換体におけるコアα1,3−フコース転移酵素活性の分析
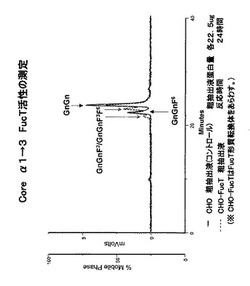
(1)上記のようにして得たCHO細胞のカイコのコアα1,3-FucT形質転換体(CHO-FucTと略記する)を1.0×107cells用いて、上記1.対照試験で記述した<粗抽出液の調製>に従ってCHO-FucT 粗抽出液を調製した(粗抽出液タンパク質量:22.5μg)。このCHO-FucT 粗抽出液を用いて対照試験と同様の<反応条件>及び<HLPCの条件>に従って、FucT活性を調べた。同時に、上記1.対照試験で記述したCHO細胞の粗抽出液をコントロールとして同様に調べた。
その結果を図8に示す(反応時間:24時間)。図8の破線のクロマトグラムが示しているように、カイコのコアα1,3-FucT候補cDNAを発現させたCHO細胞の粗抽出液を用いたサンプルからは、4本のピークが検出され、コントロールと比較してピーク数が増加していた。これはコントロールのCHO細胞と同様、内在性のコアα1,6-FucTによるGnGnF6生成に加えて、カイコのコアα1,3-FucTにより更に生じたGnGnF3およびGnGnF3F6であると推測することができる。
このデータは、得られたCHO-FucTの抽出液において、コアα1,3−フコース転移酵素活性が認められることを示唆している。
【0043】
(2)上記(1)の実験において、CHO-FucTの抽出液を用いるが但し反応液においてGDP-Fucoseを加えないものをコントロールとし、同様の実験を行った(粗抽出液タンパク質量:22.5μg、反応時間:24時間)。その結果を図9に示す。図9のグラフに示されるようにコントロール(GDP-fucose不在)では反応基質以外のピークは検出されなかった。GDP-fucose不在下では新たなピークが生じないことから、ピークの生成にはフコースの転移が関与していることが示唆される。
【0044】
4.N−グリコシダーゼ処理を用いた反応生成物の確認
上記3.で確認されたピークが、上記ダブシル化GnGn−ペプチドとGDP-フコースとの反応式に示すdabsylated GnGnF3 peptide及び/又はdabsylated GnGnF3F6peptideであることを確かめるために、図8に示される反応生成物に、N−グリコシダーゼA又はN−グリコシダーゼFを作用させ、糖鎖の切断を試みた。N−グリコシダーゼAは、上記反応式に示す、dabsylated GnGn peptide、dabsylated GnGnF6 peptide、dabsylated GnGnF3 peptide, dabsylated GnGnF3F6peptideの何れの糖鎖も切断することが可能であり、その結果dabsylated peptideが、その反応生成物として検出される。
しかし、N−グリコシダーゼFは、α1,3-結合したコアフコースを含む糖鎖は、切断することができないため、その反応性生物として、dabsylated peptide、dabsylated GnGnF3 peptide、dabsylated GnGnF3F6 peptideが検出される。
【0045】
試験条件は以下のとおりである。
<反応条件>
CHO細胞およびCHO細胞のFucT形質転換体細胞を其々1.0×107cells調整し、粗抽出液を回収した。粗抽出液中のタンパク質量はBioRad社製Protein Assay試薬を用いてブラッドフォード法により測定した。その際コントロールとしてBovine serum albuminを標準にして測定し、粗抽出液タンパク質量:46.2μgとした。
各粗抽出液を用いて以下の組成にて試薬と混合し、反応温度37℃にて24時間反応させ、その後、60℃5分加熱して反応を停止させた。
組成
1M MES-NaOH pH7.0 2μL
150mM MnCl2 2μL
1% Triton X-100 2μL
2.5mM GDP-Fucose 4μL
5nmol/μL 基質* 1μL
粗抽出液+Milli Q水 9μL
計 20μL
*N-Glycan Acceptor Substrate, Dabsylated tetrapeptide asialo agalacto biantennery EMD社製
こうして得た調製液に、MilliQ水を110μL添加して130μLの反応液とし、N−グリコシダーゼ処理をおこなった。
【0046】
CHO細胞及びカイコのコアα1,3- FucT発現CHO細胞(CHO-FucT)のそれぞれの粗抽出液について、上記反応を行ったのち、各々130μLの反応液から80μLをとり、以下、2種類のN−グリコシダーゼ処理を各々おこなった。N−グリコシダーゼF処理では、反応液80μLへN−グリコシダーゼF(1U)を1μL加え、37℃24時間反応させた後、90℃ 5分処理することで酵素を失活させた。その後0.45μmフィルターろ過を行い、HPLCサンプルとした。N−グリコシダーゼAではpHを調整する為、80μLの反応液に1M MES (pH5.1) を3.3μL加え、N−グリコシダーゼA(1U)を1μL加え、37℃24時間反応させた後、90℃ 5分処理することで酵素を失活させた。
その後1M MES pH7.0を5μL加えることでpHを調整し、0.45μmフィルターろ過を行い、HPLCサンプルとした。HPLCの条件は、上記の1.対照試験で記述したものと同じである。
【0047】
<N−グリコシダーゼ処理>
<N−グリコシダーゼF>
反応液 80μL
N−グリコシダーゼF(1U) 1μL
計 81μL
37℃ 24時間
90℃ 5分
0.45μmフィルターろ過を行い、HPLCサンプルとした。
<N−グリコシダーゼA>
反応液 80μL
1M MES pH5.1 3.3μL
N−グリコシダーゼA(1U) 1μL
計 84.4μL
37℃ 24時間
90℃ 5分
1M MES pH7.0 5μL
計 89.4μL
0.45μmフィルターろ過を行い、HPLCサンプルとした。
【0048】
CHO細胞の粗抽出液による結果を図10に示し、及びカイコのコアα1,3-FucT発現CHO細胞(CHO-FucT)の粗抽出液による結果を図11に示す。
CHO細胞の粗抽出液による図10のクロマトグラムから、N−グリコシダーゼA、及びN−グリコシダーゼFのどちらの処理においても、糖鎖部位の切断が生じたことから、コアα1,3-フコース転移以外のフコース転移であることが判る。一方カイコのコアα1,3-FucT発現CHO細胞の粗抽出液による図11のクロマトグラムから、N−グリコシダーゼAでは全ての糖鎖が切断されるのに対して、N−グリコシダーゼFでは糖鎖切断が生じない2つのピークが残り、この2つのピークはCHO細胞の粗抽出液を用いたN−グリコシダーゼF処理のデータ(図10参照)には見られないピークである。よって、これらのピークが、GnGnF3及びGnGnF3F6であると示唆される。
【0049】
5.反応時間と生成物の変化
反応時間を0〜24時間の間で変動させて、上記3.(1)の実験を行った(粗抽出液タンパク質量:25.5μg)。その結果を図12に表す。図12に示されるクロマトグラムは、経時的に基質量が減少し、その分、反応生成物が増加することを示しており、よって、新たに生成したピークは基質(dabsylated GnGn)の反応が関与していることが判る。
【0050】
以上の実験結果から、形質転換されていないCHO細胞株と比較することで、カイコのコアα1,3-FucTのcDNAで形質転換されたCHO−FucT細胞に特異的なピークの増加を確認することができた。そして、GDP−フコースの有無の実験、及び反応時間を変動させた実験から、各ピークの生成は、基質が関与したフコース転移反応であることが言える。そして、N−グリコシダーゼ処理を用いた反応生成物の確認結果から、コアα1,3-FucTのcDNAで形質転換されたCHO細胞の抽出液の反応により生成されたピークが、コアα1,3-フコース転移に起因するものであるといえる。
こうして、カイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)が、コアα1,3−フコース転移酵素をコードすることを確認することができた。
【技術分野】
【0001】
本発明は、コアα1,3−フコース転移酵素活性を有するタンパク質をコードする遺伝子DNAに関し、より具体的にはカイコに由来する該DNAに関する。本発明はさらに、該DNAを含むベクター、形質転換体、さらに、コアα1,3−フコース転移酵素活性を有する組換えタンパク質、及び該タンパク質を生産する方法に関する。
【背景技術】
【0002】
従来、遺伝子操作技術を利用して、ヒトや哺乳動物の治療に用いる糖タンパク質を生産することが検討され、治療用抗体等、実用化された例もある。糖鎖付加の無いタンパク質を大量に生産させる系としては、大腸菌を用いた生産系が主に挙げられるが、この生産系では糖タンパク質への糖鎖付加が生じないことから糖タンパク質の生産においては有効であるとは云い難い。糖タンパク質は生産されたタンパク質に糖鎖が付加しない場合、糖タンパク質本来の機能および活性の低下や生体内半減期の減少を招く場合があることが知られており、現行での糖タンパク質の生産は哺乳動物の細胞を使用した系が主流となっている。しかしながらこの系では細胞あたりの糖タンパク質生産量は限られており、生産コストや手間の面で優れた方法とは云い難い。従って、タンパク質に糖鎖を付加する機能を有する植物体もしくは昆虫細胞等が生産宿主として検討されている。
【0003】
タンパク質の糖鎖構造は、その結合様式により2種類に分類される。1つは、タンパク質のアスパラギン残基に結合するN−結合型糖鎖であって、他方は、タンパク質のセリン残基あるいはスレオニン残基に結合するO−結合型糖鎖である。このうちN−結合型糖鎖について注目すると、動物、植物、昆虫、酵母などでは高マンノース型糖鎖、複合型糖鎖、およびそのハイブリッド型糖鎖が存在する。これらの型は、マンノース3 分子、N−アセチルグルコサミン2 分子からなる、共通する5糖のコア糖鎖構造を糖鎖の還元末端側にもっている。
糖タンパク質のN−結合型糖鎖は、細胞の小胞体において、まず脂質との複合体の形態で合成され、タンパク質に転移する。そして、転移した糖鎖を有するタンパク質は、小胞体からゴルジ体に輸送され、その過程において糖鎖の刈り込みや伸長を受ける。ゴルジ体におけるこの糖鎖の伸長は、末端部糖鎖合成と呼ばれ、生物種に依存して大きく異なることが知られている。さらに、コア糖鎖内の還元末端部位に存在するN-アセチルグルコサミン残基に付加するフコース残基の結合様式も生物種により異なることから知られている。
【0004】
植物体もしくは昆虫細胞等を哺乳類型糖タンパク質の生産宿主とするときに、弊害となるのは、植物もしくは昆虫によって生産された糖タンパク質は哺乳類(例えばヒト)の糖鎖構造と異なる部分があり、よって、ヒトに対して抗原性を有する場合が多いということである。この抗原性は、糖タンパク質のコアα1,3−フコースを含む糖鎖部分に起因する場合がある。すなわち、植物もしくは昆虫は、ヒトとは異なり、糖鎖コア構造の還元末端部位に存在するN−アセチルグルコサミン残基へのフコースのα1,3−結合を有するタンパク質を生産するということである。なお、昆虫ではα1,3−結合及びα1,6−結合の両方が見出されている。
従って、上述の抗原性の原因を熟知し、抗原性を消失または抑制するためには、まず、コアα1,3−フコース転移反応を司る遺伝子である、コアα1,3−フコース転移酵素(コアα1,3−フコシルトランスフェラーゼ)遺伝子を同定する必要がある。
植物では、リョクトウ(Vigna radiata)でα1,3−フコシルトランスフェラーゼ遺伝子を単離・同定したことが報告されている(非特許文献1及び特許文献1参照)。また、昆虫では、ショウジョウバエ(Drosophila melanogaster)でα1,3−フコシルトランスフェラーゼを単離・同定したことが報告されている(非特許文献2参照)。
【0005】
昆虫の中でもカイコを用いたタンパク質の生産系は、その生産量、および生産されたタンパク質の安定性や生産コスト、簡便性などの面で従来から優れた方法として利用されている。しかしながら、上述のコアα1,3−フコースによる抗原性の問題から、ヒトへの利用を目的とした糖タンパク質の生産系としての改良が求められてきた。
特許文献2では、カイコのコアα1,3−フコシルトランスフェラーゼのDNA配列(1,486塩基対)を、カイコのゲノムDNAからPCR法で増幅している。
カイコを用いた糖タンパク質生産系の利便性が注目される中、コアα1,3−フコシルトランスフェラーゼ活性を有するタンパク質をコードする、完全長ORF(open reading frame)cDNAの探索と同定が求められている。
【先行技術文献】
【特許文献】
【0006】
【特許文献1】特表2002−536978号公報
【特許文献2】特開2010−29102号公報
【非特許文献】
【0007】
【非特許文献1】J.Biol.Chem.1999,July 30;274(31);p.21830−21839
【非特許文献2】J.Biol.Chem.2001,July 27;276(30);p.28058−28067
【発明の概要】
【発明が解決しようとする課題】
【0008】
本発明は、カイコにおけるコアα1,3−フコース転移酵素活性を有するタンパク質をコードする遺伝子DNAを同定することによって、さらに、該コアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAを含む発現ベクター、コアα1,3−フコース転移酵素活性を有する組換えタンパク質、及び該タンパク質の生産方法を提供することを目的とする。
【課題を解決するための手段】
【0009】
本発明者らは上記目的を達成するために鋭意研究を重ねた結果、カイコ(具体的には、Bombyx mori)のゲノムデータベースにおいて7番染色体上に、ショウジョウバエのコアα1,3−フコース転移酵素活性を有するアミノ酸配列の一部をコードする相同な配列を見出し、本発明を完成させるに至った。
従って本発明は先ず、鱗翅目のコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAに向けられている。ここで鱗翅目動物の例としては、アゲハチョウ科(Papilionidae)、タテハチョウ科(Nymphalidae)、ヤガ科(Noctuidae)、スズメガ科(Sphingidae)、カイコガ科(Bombycidae)等が挙げられ、ギフチョウ、オオムラサキ、スズメガ、カイコガ等が挙げられる。
本発明は、より具体的には、カイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAに向けられている。本発明はより具体的に、下記の配列番号1の塩基配列で表されるカイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAである。
【0010】
配列番号1:
atgcgggtga gggcggctcg ggccctgcgt gctttaagac gcgtggcact cttgtttccg
ctagtattga ctgcgctggc gctaatcctg ctgccgcgcc cgggacctct cacacccctt
gcacagaatc ctaccattgc cgcacagatc cactcgctca aagacgacca gcatattcaa
gatcgttcca atgaacttca agaagaggtg ctgcacgagg aaggtgacac cgtatcccgt
cgccactggt tcatgcgcgg aggtgaacaa cggccttgga aacacgatcc tagtgcacaa
atattccctg aggatgcccc tggggatgac agaatcgttc aacagctatc atacatatta
ccgaacgacg aggaggcgcc tattaaaaaa atactacttg ccaatggcct cggggcgtgg
ggcgtaaccg gtggtcgtac agaattcctc cgaaacaagt gccccataga ccggtgtcat
cttacggcgg actcgaggga tgccgcctct gcagacgcta tactcttcaa agatcaccat
acgcctttta acgtcaaaag atctcctaat cagatttgga ttctctacta cttggaatgt
ccttaccaca cggcttcctt gcggtcttca tccttcgacg tatttaactg gactgcaact
taccgcagag actctgatat agtagcacct tacgaaagat gggtgtatta tgataatttg
attcctgaaa aagatataga aaggaactat gcagctaata aaacaaagaa ggtagcgtgg
tttgtatcaa actgccacgc tcgcaaccgt cggctccagt acgcgcggca actcagcaaa
ttcatccaag tggacatata cggcgcgtgc ggctctcatc actgcccacg aaccgacccc
aactgcttgg aaatgttgga caaagaatac aagttttatc tggcttttga aaattcaaac
tgtcgggatt acataacgga aaaattcttc gtcaatggat tacaacacaa cgttttaccg
atcgtgatgg gagcccggcc atcggagtat gcagcagtgg ctccacgtaa ttcgtacatt
catgtagaag agtttgccgg tcctgaagaa ctcgctgcgt acttacatcg ccttgatgaa
gaccaagatc tatacaactc ttactttaaa tggaagggaa caggagaatt cattaacaca
tatttctttt gccgcgtatg tgcgatgatt cacgcgaacg aaagacggca aaggaatact
cactacagcg acgtgcaggc atggtggcgg gactccacgt gcacgcgagg cgaatggcga
gccatggaat ctgatactaa agataatgga taa
【0011】
本発明はまた、下記(a)又は(b)のタンパク質をコードするDNAに向けられている。
(a)下記の配列番号2のアミノ酸配列からなるタンパク質
(b)下記の配列番号2のアミノ酸配列において1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
【0012】
配列番号2:
Met Arg Val Arg Ala Ala Arg Ala Leu Arg Ala Leu Arg Arg Val Ala
Leu Leu Phe Pro Leu Val Leu Thr Ala Leu Ala Leu Ile Leu Leu Pro
Arg Pro Gly Pro Leu Thr Pro Leu Ala Gln Asn Pro Thr Ile Ala Ala
Gln Ile His Ser Leu Lys Asp Asp Gln His Ile Gln Asp Arg Ser Asn
Glu Leu Gln Glu Glu Val Leu His Glu Glu Gly Asp Thr Val Ser Arg
Arg His Trp Phe Met Arg Gly Gly Glu Gln Arg Pro Trp Lys His Asp
Pro Ser Ala Gln Ile Phe Pro Glu Asp Ala Pro Gly Asp Asp Arg Ile
Val Gln Gln Leu Ser Tyr Ile Leu Pro Asn Asp Glu Glu Ala Pro Ile
Lys Lys Ile Leu Leu Ala Asn Gly Leu Gly Ala Trp Gly Val Thr Gly
Gly Arg Thr Glu Phe Leu Arg Asn Lys Cys Pro Ile Asp Arg Cys His
Leu Thr Ala Asp Ser Arg Asp Ala Ala Ser Ala Asp Ala Ile Leu Phe
Lys Asp His His Thr Pro Phe Asn Val Lys Arg Ser Pro Asn Gln Ile
Trp Ile Leu Tyr Tyr Leu Glu Cys Pro Tyr His Thr Ala Ser Leu Arg
Ser Ser Ser Phe Asp Val Phe Asn Trp Thr Ala Thr Tyr Arg Arg Asp
Ser Asp Ile Val Ala Pro Tyr Glu Arg Trp Val Tyr Tyr Asp Asn Leu
Ile Pro Glu Lys Asp Ile Glu Arg Asn Tyr Ala Ala Asn Lys Thr Lys
Lys Val Ala Trp Phe Val Ser Asn Cys His Ala Arg Asn Arg Arg Leu
Gln Tyr Ala Arg Gln Leu Ser Lys Phe Ile Gln Val Asp Ile Tyr Gly
Ala Cys Gly Ser His His Cys Pro Arg Thr Asp Pro Asn Cys Leu Glu
Met Leu Asp Lys Glu Tyr Lys Phe Tyr Leu Ala Phe Glu Asn Ser Asn
Cys Arg Asp Tyr Ile Thr Glu Lys Phe Phe Val Asn Gly Leu Gln His
Asn Val Leu Pro Ile Val Met Gly Ala Arg Pro Ser Glu Tyr Ala Ala
Val Ala Pro Arg Asn Ser Tyr Ile His Val Glu Glu Phe Ala Gly Pro
Glu Glu Leu Ala Ala Tyr Leu His Arg Leu Asp Glu Asp Gln Asp Leu
Tyr Asn Ser Tyr Phe Lys Trp Lys Gly Thr Gly Glu Phe Ile Asn Thr
Tyr Phe Phe Cys Arg Val Cys Ala Met Ile His Ala Asn Glu Arg Arg
Gln Arg Asn Thr His Tyr Ser Asp Val Gln Ala Trp Trp Arg Asp Ser
Thr Cys Thr Arg Gly Glu Trp Arg Ala Met Glu Ser Asp Thr Lys Asp
Asn Gly
【0013】
本発明はまた、下記(a)又は(b)のDNAである。
(a)配列番号1の塩基配列からなるDNA
(b)配列番号1に示す塩基配列からなるDNAと相補的な塩基配列からなるDNAと、ストリンジェントな条件下でハイブリダイズし、かつコアα1,3−フコース転移酵素活性を有するタンパク質をコードするカイコ由来のDNA
【0014】
本発明によるDNAを用いて組換えタンパク質を生産することができる。従って、本発明はさらに、上記のDNAを含む発現ベクター、及び該発現ベクターを宿主細胞に導入して得られる形質転換体に向けられている。宿主として例えば哺乳類細胞株が挙げられる。
本発明はまた、下記(a)又は(b)の組換えタンパク質に向けられている。
(a)配列番号2のアミノ酸配列からなるタンパク質。
(b)配列番号2のアミノ酸配列の1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
本発明によるDNAを用いて組換えタンパク質を生産する方法として、上記のDNAを含む発現ベクターを宿主細胞に導入し、得られる形質転換体を培養し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法がある。
本発明によるDNAを用いて組換えタンパク質を生産する方法としてまた、上記のDNAを含む発現ベクターを宿主個体に導入し、得られる形質転換体を飼育し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法がある。
なお、本明細書中において、フコース転移酵素を“FucT”と略記し、例えばコアα1,3−フコース転移酵素を“コアα1,3-FucT”と略記する。
【発明の効果】
【0015】
本発明による、コアα1,3−フコース転移酵素活性を有するタンパク質(コアα1,3−フコース転移酵素、あるいはコアα1,3−フコシルトランスフェラーゼ)をコードするDNAを利用して得られる組換えタンパク質、すなわちコアα1,3−フコース転移酵素活性を有する組換えタンパク質は、試薬として有用である。より具体的には、該組換えタンパク質は、N-結合型糖鎖に関して、コアα1,3−フコースを有する糖鎖、及びその糖鎖を有する糖タンパク質を合成するための合成用酵素として有用である。
【図面の簡単な説明】
【0016】
【図1】クローニングにおける3’RACEでのPCR結果を表す電気泳動の写真である。
【図2】クローニングにおける3’RACEでのPCR結果を表す電気泳動の写真である。
【図3】クローニングにおける5’RACEでのPCR結果を表す電気泳動の写真である。
【図4】クローニングにおける5’RACEでのPCR結果を表す電気泳動の写真である。
【図5】FucTAはDrosophila melanogaster FucTA (core alpha(1,3)-fucosyltransferase)遺伝子(NCBI Number: AJ302045)にコードされるタンパク質のアミノ酸配列。FucTは本実施例で単離されたカイコのcore alpha(1,3)-fucosyltransferase遺伝子にコードされるタンパク質のアミノ酸配列。FucTAとFucTのタンパク質のアミノ酸配列上の相同性を比較した。
【図6】Genome上でのエクソン-イントロン構造、及び翻訳開始コドンを概略的に表す。
【図7】CHO細胞株の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した結果を表す。
【図8】CHO細胞のコアα1,3-FucT形質転換体の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した結果を表す。コントロールとしてCHO細胞株の粗抽出液を用いた。
【図9】CHO細胞のコアα1,3-FucT形質転換体の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した結果を表す。GDP-フコースを添加しない検体をコントロールとした。
【図10】CHO細胞株の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をN−グリコシダーゼ処理に供し、生成物をHPLCにて分析した結果を表す。
【図11】CHO細胞のコアα1,3-FucT形質転換体の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をN−グリコシダーゼ処理に供し、生成物をHPLCにて分析した結果を表す。
【図12】CHO細胞株の粗抽出液又はCHO細胞のコアα1,3-FucT形質転換体の粗抽出液を用いて、ダブシル化GnGn−ペプチドを基質として、GDP-フコースを反応させる実験において、反応時間を変動させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した結果を表す。
【発明を実施するための形態】
【0017】
本発明者らは、カイコ(Bombyx mori)の幼虫から、コアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAを同定した。
本明細書中において、コアα1,3−フコース転移酵素活性を有するタンパク質は、コアα1,3−フコース転移酵素又はコアα1,3−フコシルトランスフェラーゼとも呼称される。コアα1,3−フコシルトランスフェラーゼとは、糖タンパク質のタンパク質部分の合成後、N-結合型糖鎖が付加したペプチド鎖に最も近い、糖鎖の還元末端のN-アセチルグルコサミン残基に、GDP−フコースを糖供与体として、α1,3−結合でフコース残基を付加する酵素である。
【0018】
本発明は、より具体的には配列番号1の塩基配列で表されるカイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNAを提供する。
本発明はまた、下記(a)又は(b)のタンパク質をコードするDNAを提供する。
(a)配列番号2のアミノ酸配列からなるタンパク質
(b)配列番号2のアミノ酸配列において1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
ここで、「1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列」は、そのアミノ酸配列からなるタンパク質が所望の機能を失わない限り、その欠失、置換もしくは付加されるアミノ酸の数は限定されるものではない。例えば、置換などされるアミノ酸の数は、30個以下、好ましくは10個以下、より好ましくは5個以下、特に好ましくは3個以下である。
【0019】
本発明はまた、具体的に下記(a)又は(b)のDNAを提供する。
(a)配列番号1の塩基配列からなるDNA
(b)配列番号1に示す塩基配列からなるDNAと相補的な塩基配列からなるDNAと、ストリンジェントな条件下でハイブリダイズし、かつコアα1,3−フコース転移酵素活性を有するタンパク質をコードするカイコ由来のDNA
ここで、「ストリンジェントな条件」とは、相同遺伝子をコードするDNAを単離するために適したハイブリダイゼーション反応条件を意味し、すなわち特異的なハイブリッドが形成され、非特異的なハイブリッドが形成されない条件を言い、当業者にとって公知の方法によって決定することができる。「ストリンジェントな条件でハイブリダイズするDNA」として、プローブとして使用するDNAの塩基配列と一定以上の相同性を有するDNAが挙げられ、例えば70%以上、好ましくは80%以上、より好ましくは90%以上、更に好ましくは95%以上、最も好ましくは98%以上の相同性を有するDNAである。
本発明のDNAは、コアα1,3−フコシルトランスフェラーゼをコードするものであれば、その形態に制限がなく、cDNA由来のもの、ゲノムDNA由来のもの、化学合成DNA、遺伝暗号の縮重に基づく任意の塩基配列を有するDNAなどを包含する。
【0020】
本発明のDNAを同定するまでの経緯について、以下のとおりである。
カイコ染色体の塩基配列は未だ不明瞭な領域が存在する。カイコのコアα1,3−フコシルトランスフェラーゼ遺伝子を同定する際、その候補遺伝子が得られた段階でカイコ染色体上での塩基配列を確認したところ、報告されていない部分と重なったため、得られた候補遺伝子がカイコ染色体上に実在するかといった問題を簡単には解決することができなかった。従ってカイコよりゲノムDNAを抽出し、候補遺伝子が存在すると仮定されるゲノムDNA部分の塩基配列を解読することにより、本遺伝子がexon-intron構造をとることやデータベースにおいて7番染色体上に存在することなどを確認することで遺伝子として実在するか検証をおこなう必要があった。
【0021】
他の生物種との相同性について、以下のとおりである。
コアα1,3−フコシルトランスフェラーゼ遺伝子は、リョクトウやショウジョウバエなど、幾つかの種において同定されている。従って通常は種間で保存されたアミノ酸配列領域を基に相同性検索をおこなうことで、カイコにおける本タンパク質の候補配列を得ることができると想定されるが、昆虫であるショウジョウバエのコアα1,3−フコース転移酵素と、同定したカイコのコアα1,3−フコース転移酵素との相同性は、相同性の高い部分でもわずか49%(全長では47.3%)であって、相同性が高いとは云い難い。また、タンパク質の長さはリョクトウが510アミノ酸であり、ショウジョウバエが503アミノ酸であるのに対し、カイコは450アミノ酸であった。
染色体上でもカイコのコアα1,3−フコース転移酵素遺伝子の特徴は、他の生物種の遺伝子と異なっており、リョクトウやショウジョウバエにおいては約2Kbpのゲノム塩基配列上に4つのエクソンから構成されるexon-intron構造を持つのに対し、カイコでは約10Kbpのゲノム塩基配列上に7つのエクソンから構成されるexon-intron構造を持つ。またコアα1,3−フコース転移酵素のアミノ酸配列において、コア酵素に特有な、種を超えて保存性の高い領域(モチーフ)は報告されておらず、よって、遺伝子の全長を単離し、そのコードするタンパク質の酵素活性を確認するまでは本遺伝子がコアα1,3−フコース転移酵素であるか、同定するのは容易でなかったという経緯がある。すなわちカイコから得られた遺伝子が他の生物種の当該遺伝子と同様の機能を有することを確定することは容易ではなかった。そこで我々は鋭意研究を重ねた結果、カイコのRNAから完全長ORFを持つcDNAを単離し、その配列から予測されるアミノ酸配列の中にこれまで報告された他の生物種のα1,3−フコース転移酵素に共通する4つのモチーフを見出した。さらに、得られたcDNAのコードするタンパク質を発現させ、その酵素活性を測定し、本遺伝子がカイコのコアα1,3−フコース転移酵素であることを同定し、本発明を完成するに至った。
【0022】
本発明によるDNAを用いて組換えベクターを構築し、適当な宿主へ該ベクターを導入し、形質転換体を得て、下記の(a)又は(b)の組換えタンパク質を生産させることができる。
(a)配列番号2のアミノ酸配列からなるタンパク質。
(b)配列番号2のアミノ酸配列の1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
本発明のDNAを用いて、組換えタンパク質を生産するには、例えば、Molecular Cloning(出版社:Cold spring harbor laboratory press)等の多くの教科書や文献に基づいて実施することができる。具体的には、転写を制御するプロモーター配列(例えば、trp、lac、T7、SV40初期プロモーター)などの制御遺伝子を付加し、適当なベクター(例えば、pBR322、pUC19、pSV・SPORT1など)に組み込むことにより、宿主細胞内で複製し、機能する発現ベクター(例えば発現プラスミド)を作製する。そのようなベクターは一般的に複製開始点、選択マーカー、プロモーターを含み、必要に応じてエンハンサー、ターミネーター、リボソーム結合部位、ポリアデニル化シグナルなどを含んでいてもよい。
【0023】
発現ベクターとして、導入遺伝子が宿主細胞に導入されたか否か、若しくは宿主細胞中で確実に発現しているか否かを確認するために、各種選択マーカーを含むものが好ましい。このようなマーカーの例として、テトラサイクリン耐性遺伝子、アンピシリン耐性遺伝子、カナマイシン耐性遺伝子、ハイグロマイシン耐性遺伝子などの抗生物質耐性マーカー遺伝子がある。
また、組換えタンパク質を、形質転換体の細胞外へ分泌可能な状態で得ることもできる。例えば発現ベクターにシグナルペプチドをコードする配列を適当に組み込むことにより、あるいはシグナルペプチドをコードする市販の適当なベクターを用いることにより、組換えタンパク質を形質転換体の細胞外へ分泌させることができる。
実際に、市販されている種々の発現用ベクターから適宜選択し、該発現用ベクターのプロトコルに従って、本発明のDNAを含む発現ベクターを構築することができる。
市販されている発現用ベクターの例として、宿主細胞が大腸菌である場合は、例えばpETベクター(Novagen社製)、pTrxFUSベクター(Invitrogen社製)、pCYBベクター(NEW ENGLAMD Bio Labs社製)などが、宿主細胞が酵母である場合は、例えばpESP-1発現ベクター(STRATAGENE社製)、pAUR123ベクター(宝酒造社製)、pPICベクター(Invitrogen社製)などが、また宿主細胞が動物細胞である場合は、例えばpMAM-neo発現ベクター(CLONTECH社製)、pcDNA3.1ベクター(Invitrogen社製)、pBK-CMVベクター(STRATAGENE社製)などが、宿主細胞が昆虫細胞である場合は、例えばpBacPAKベクター(CLONTECH社製)、pAcUW31ベクター(CLONTECH社製)、pAcP(+)IE1ベクター(Novagen社製)などが、それぞれ挙げられる。
【0024】
こうして構築された発現ベクターは、例えばポリエチレングリコール法、エレクトロポレーション法、アグロバクテリウム法、パーティクルガン法、塩化カルシウム法、プロトプラスト法、インジェクション法などの当業者に公知の方法によって、宿主細胞に導入することができる。
こうして、発現ベクターを適当な宿主細胞に導入して、形質転換体を得る。宿主細胞としては、大腸菌などの原核生物、酵母のような単細胞真核生物、昆虫細胞、植物細胞、哺乳類などの多細胞生物の細胞や個体などが挙げられる。好ましくは、昆虫の細胞や個体であり、昆虫の細胞や個体としては、sf9細胞、BmN4細胞、カイコ幼虫(品種:w1-pnd)などが例示できる。また好ましくは、哺乳類の細胞であり、哺乳類の細胞としては、CHO細胞、293細胞、COS−7細胞などが例示できる。
また、形質転換体に導入されたDNAは、ハイブリダイゼーションやPCRなどの当業者に公知の方法によって、転写産物発現を確認することができる。
本発明によるDNAを用いて組換えタンパク質を生産する方法において、形質転換体である細胞や個体を、培地又は餌、温度、日数、雰囲気などの適当な条件下で培養又は飼育する。形質転換体により生産された組換えタンパク質を回収する実施態様として、形質転換体を破砕し、その上清から組換えタンパク質を抽出する方法、あるいは形質転換体外、すなわち培養培地や繭などに分泌された組換えタンパク質を回収する方法などが挙げられる。さらに、精製工程を設けて、精製された組換えタンパク質を得ることもできる。
【実施例】
【0025】
以下に本発明を、詳細に説明する。
[コアα1,3−フコース転移酵素のクローニング]
野生型蚕(品種:w1-pnd)の組織由来RNAから、3'および5'RACE法にてα1,3−フコース転移酵素遺伝子をクローニングし、その塩基配列を決定した。
1.相同性検索
Drosophila melanogaster FucTA (core alpha(1,3)-fucosyltransferase)遺伝子(NCBI Number: AJ302045)を基に、アミノ酸配列相同性検索(NCBI Blast)を行い、Bombyx mori genome*上で相同性の高い下記の配列を得た。この配列をカイコのコアα1,3-FucT cDNA断片とした。
(* NCBI number: AADK01016505; Bombyx mori strain Dazao whole genome shotgun seq.)
Drosophila melanogaster FucTA (core alpha(1,3)-fucosyltransferase)遺伝子(NCBI Number: AJ302045)を基に、アミノ酸配列相同性検索(NCBI Blast)を行い、Bombyx mori genome上で相同性のある配列(NCBI number: AADK01016505; Bombyx mori strain Dazao whole genome shotgun seq.)を得た。この相同性のある領域は5箇所あり、それぞれの領域における同一のアミノ酸の割合はDrosophilaのアミノ酸の数を分母に、カイコのアミノ酸の数を分子にして記載すると、57/79、40/59、40/68、40/88、30/72であり必ずしも相同性が高いとは判断されなかった。しかしながら、このカイコのゲノム配列から想定されるタンパク質をコードするcDNA配列の断片(下記)をカイコのコアα1,3-FucT cDNA断片として、当該タンパク質の全長ORF(Open reading frame、タンパク質の読み取り枠)のクローニングと当該酵素活性の測定を試みることとした。
agaatcgttcaacagctatcatacatattaccgaacgacgaggaggcgcctattaaaaaaatactacttgccaatggcctcggggcgtggggcgtaaccggtggtcgtacagaattcctccgaaacaagtgccccatagaccggtgtcatcttacggcggactcgagggatgccgcctctgcagacgctatactcttcaaagatcaccatacgccttttaacgtcaaaagatctcctaatcaggtaacggtacagatttggattctctactacttggaatgtccttaccacacggcttccttgcggtcttcatccttcgacgtatttaactggactgcaacttaccgcagagactctgatatagtagcaccttacgaaagatgggtgtattatgataatttgattcctgaaaaagatatagaaaggaactatgcagctaataaaacaaagaaggtacaggtagcgtggtttgtatcaaactgccacgctcgcaaccgtcggctccagtacgcgcggcaactcagcaaattcatccaagtggacatatacggcgcgtgcggctctcatcactgcccacgaaccgaccccaactgcttggaaatgttggacaaagaatacaagttttatctggcttttgaaaattcaaactgtcgggattacataacggaaaaattcttcgtcaatggattattaagacacaacgttttaccgatcgtgatgggagcccggccatcggagtatgcagcagtggctccacgtaattcgtacattcatgtagaagagtttgccggtcctgaagaactcgctgcgtacttacatcgccttgatgaagaccaagatctatacaactcttactttaaatggaag
【0026】
2.クローニング
上記カイコのコアα1,3-FucT cDNA断片は3'末端側、5'末端側共にORF全長の末端配列を含んではいないため、それぞれ以下の方法で延長させた。
2−1.3'RACE
カイコ幼虫(品種:w1-pnd)より、Invitrogen社製の「TRIzol」のプロトコルに従って、total RNAを抽出した。
上記total RNA 1.5μg分をtemplate とし、Oligo dT primerを用いて逆転写反応を行いcDNAを合成した。コアα1,3-FucT cDNA断片内、既知配列特異的上流primer A1(gcagcagtggctccacgtaattcg:配列番号3)を用いてPCR(反応条件として、94℃2分、「94℃30秒、55℃30秒、72℃2分」を35サイクル、72℃5分にて行った)を行った。
そのPCRの結果を、図1に示す。
その結果、PCR産物が複数得られたので、それぞれのバンドを0.8% アガロースにて分離・精製後(Promega社製「Wizard SV Gel and PCR Clean-up System」のプロトコル参照)、既知配列特異的上流配列および制限酵素切断部位を含むprimer A2(ctgatctagaggtaccggatccggtcctgaagaactcgctgcgtacttacatcg:配列番号4)を用いてPCRを行い(反応条件は上記同様)制限酵素BamHIで切断し、pBluescript II SK(+)に導入することによってcDNA断片を得た(TAKARA社製「3'-Full RACE Core set」のプロトコル参照)。
そのPCRの結果を図2に示す。
【0027】
得られたcDNA配列をシークエンスし、下記の目的とする3'ORF末端配列を得た。
ggaacaggagaattcattaacacatatttcttttgccgcgtatgtgcgatgattcacgcgaacgaaagacggcaaaggaatactcactacagcgacgtgcaggcatggtggcgggactccacgtgcacgcgaggcgaatggcgagccatggaatctgatactaaagataatggataa
【0028】
2−2. 5'RACE
カイコ幼虫(品種:w1-pnd)よりtotal RNAを抽出した(Invitrogen社製「TRIzol」のプロトコル参照)。
次いでtotal RNAよりmRNAを精製した(Invitrogen社製「FastTrack MAG mRNA Isolation Kits」のプロトコル参照)。
次に、上記mRNA 0.5μgをtemplateとして、5'末端既知配列特異的リン酸化primer B1(gtaaggtgctac:配列番号5)を用いて逆転写反応を行い、1st strand cDNAを合成した。RNAを分解した後、一本鎖cDNAを環化させてコアα1,3-FucT cDNA断片内既知配列特異的primer B2(cttggaatgtccttaccacacg:配列番号6)、B3(gtctatggggcacttgtttc:配列番号7)を用いてPCRを行い(反応条件として、94℃2分、「94℃30秒、55℃30秒、72℃2分」を37サイクル、72℃5分にて行った)複数のcDNA断片を得た。
そのPCRの結果を図3に示す。
その結果、PCR 産物が複数得られたので其々のバンドを3% アガロースにて分離・精製後、既知配列特異的primer B4(ggactgcaacttaccgcagagac:配列番号8)、B3(gtctatggggcacttgtttc:配列番号9)を用いてPCRを行い(反応条件として、94℃2分、「94℃30秒、55℃30秒、72℃2分」を37サイクル、72℃5分にて行った)、cDNA断片を得た。
そのPCRの結果を図4に示す。
これらcDNA断片をTA cloning Vector pCR2.1へ導入することによって、目的とする5'ORF末端配列を含むcDNA断片を得た(Invitrogen社製「FastTrack MAG mRNA Isolation Kits」のプロトコル参照)。
【0029】
得られたcDNA配列をシークエンスし、以下の目的とする5'ORF末端配列を得た。
atgcgggtgagggcggctcgggccctgcgtgctttaagacgcgtggcactcttgtttccgctagtattgactgcgctggcgctaatcctgctgccgcgcccgggacctctcacaccccttgtacagaatcctaccattgccgcacagatccactcgctcaaagacgaccagcatattcaagatcgttccaatgaacttcaagaagaggtgctgcacgaggaaggtgacaccgtatcccgtcgccactggttcatgcgcggaggtgaacaacggccttggaaacacgatcctagtgcacaaatattccctgaggatgcccctggggatgac
【0030】
2−3.cDNAの取得
得られたORF領域の配列情報より、primer C1(atgcgggtgagggcggctcg:配列番号10)、C2(ttatccattatctttagtatcagattcc:配列番号11)、およびtemplate としてカイコ幼虫(品種:w1-pnd)由来total RNAを用いてPCRを行い(反応条件として、94℃2分、「94℃30秒、55℃30秒、72℃2分」を37サイクル、72℃5分にて行った)、カイコのコアα1,3-FucTcDNAを得た。これをTA cloning Vector pCR2.1へ導入し、カイコのコアα1,3-FucTcDNAの全配列を確認した(コアα1,3-FucT ORF 全長cDNA:配列番号1)。
情報として得られたカイコのコアα1,3-FucT cDNA断片部分(上記「1.相同性検索」で得たコアα1,3-FucT cDNA断片の配列)と、実際に得られた配列(配列番号1)を比較することにより、3箇所塩基配列が異なる部位が見受けられた。この3箇所は、上記「1.相同性検索」で得たコアα1,3-FucT cDNA断片の配列中、下線で示した箇所である。この領域はいずれもexon-intronの境界部分であり、相同性検索時の解析ソフトの影響を受ける。D. melanogaster FucTAと比較した結果、配列番号1の配列を、目的とするカイコのコアα1,3-FucTの遺伝子配列とした。
カイコのコアα1,3-FucT ORF 全長cDNA配列(配列番号1)を基にタンパク質のアミノ酸配列が決定された(配列番号2)。
【0031】
3.D. melanogaster FucTAとのタンパク質のアミノ酸配列上の相同性
得られたカイコのコアα1,3-FucTの全長部位とD. melanogaster FucTA とでタンパク質のアミノ酸配列上の相同性を比較した(図5参照)。図5中、FucTAは、D. melanogaster FucTA(core alpha(1,3)-fucosyltransferase)遺伝子(NCBI Number: AJ302045)を表し、FucTは、配列番号2のコアα1,3-FucT タンパク質のアミノ酸配列を表す。なお、相同性の比較には、ClustalW2(http://www.ebi.ac.uk/Tools/clustalw2/index.html)を使用した。
【0032】
4.Genome上での塩基配列確認
配列番号1の配列を基にBlast検索をおこなうことによって、得られた配列が二つのBombyx mori genome shotgun sequence上に存在することが判明したことから、exon-intron構造を決定し、およびORFのfirst ATG(予想される開始コドン)が、得られた配列上に存在することを確認した(図6参照)。図6中、6−1.はカイコのコアα1,3-FucT遺伝子のexon-intron構造を表す。黒色のexonはORF領域を表し、淡色のexonは開始コドンより上流の非翻訳領域(5'UTR部位)を表す。6−1において、AADK01016505は Bombyx mori strain Dazao whole genome shotgun seq.を意味し、AADK01011744は Bombyx mori strain Dazao, whole genome shotgun seq.を意味する。
図6中、6−2において、濃い下線部分は得られた5'ORF領域を表し、及びその上流にある淡い下線部分は5'UTR部位のゲノム塩基配列である。ATG(枠付き)より15塩基上流部位に終止コドン(枠付きTAA)が存在することから、このATGが翻訳開始コドンであると予想される。
【0033】
また、2つのgenome shotgun seq.のgapを埋めるため、カイコ幼虫(品種:w1-pnd) 5齢3日目より得られた脂肪体よりgenomic DNAを抽出した(三光純薬製「Sepa Gene」のプロトコル参照)。2種類のBombyx mori genome shotgun seq.末端部分primer C1(cgaggagtgaagtggtgtga:配列番号12)、C2(ccggaacaaatattccgttgt:配列番号13)よりgenomic PCRを行い、得られた断片の配列をシークエンスすることで、上記gap配列は約2.2Kbpであることが判明した。また、図6中、6−1.に2つのgenome shotgun seq.とgap配列との関係を記載した。
その他、Bombyx mori genome*上の塩基配列と実際に得られたカイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)とを比較したところ、配列は完全に一致した。しかしながら、上記2−2において得た5’ORF末端の配列とは1箇所異なる塩基が見受けられた(該5’ORF末端配列の下線部分参照)。そこでさらに、抽出したgenomic DNAの配列を調べたところ、Bombyx mori genome*上の塩基配列と一致したことから、上記2−2において違いの見受けられた塩基はチミン(上記2−2表記上のt)ではなくシトシン(c)であることが確定した。よってカイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)))がBombyx mori genome*上の塩基配列と一致した。
(* NCBI number: AADK01016505; Bombyx mori strain Dazao whole genome shotgun seq.)
【0034】
なお、配列番号1の配列の上流にある配列を含めたDNA配列を、以下に配列番号14として示す。
配列番号14:
atagccgggc attacacata cgattctaat tccattgaaa tggatataat aataaagaag
tgcatctgga ccggggttca gggcggataa ccgcgagtcg agatgcgggt gagggcggct
cgggccctgc gtgctttaag acgcgtggca ctcttgtttc cgctagtatt gactgcgctg
gcgctaatcc tgctgccgcg cccgggacct ctcacacccc ttgcacagaa tcctaccatt
gccgcacaga tccactcgct caaagacgac cagcatattc aagatcgttc caatgaactt
caagaagagg tgctgcacga ggaaggtgac accgtatccc gtcgccactg gttcatgcgc
ggaggtgaac aacggccttg gaaacacgat cctagtgcac aaatattccc tgaggatgcc
cctggggatg acagaatcgt tcaacagcta tcatacatat taccgaacga cgaggaggcg
cctattaaaa aaatactact tgccaatggc ctcggggcgt ggggcgtaac cggtggtcgt
acagaattcc tccgaaacaa gtgccccata gaccggtgtc atcttacggc ggactcgagg
gatgccgcct ctgcagacgc tatactcttc aaagatcacc atacgccttt taacgtcaaa
agatctccta atcagatttg gattctctac tacttggaat gtccttacca cacggcttcc
ttgcggtctt catccttcga cgtatttaac tggactgcaa cttaccgcag agactctgat
atagtagcac cttacgaaag atgggtgtat tatgataatt tgattcctga aaaagatata
gaaaggaact atgcagctaa taaaacaaag aaggtagcgt ggtttgtatc aaactgccac
gctcgcaacc gtcggctcca gtacgcgcgg caactcagca aattcatcca agtggacata
tacggcgcgt gcggctctca tcactgccca cgaaccgacc ccaactgctt ggaaatgttg
gacaaagaat acaagtttta tctggctttt gaaaattcaa actgtcggga ttacataacg
gaaaaattct tcgtcaatgg attacaacac aacgttttac cgatcgtgat gggagcccgg
ccatcggagt atgcagcagt ggctccacgt aattcgtaca ttcatgtaga agagtttgcc
ggtcctgaag aactcgctgc gtacttacat cgccttgatg aagaccaaga tctatacaac
tcttacttta aatggaaggg aacaggagaa ttcattaaca catatttctt ttgccgcgta
tgtgcgatga ttcacgcgaa cgaaagacgg caaaggaata ctcactacag cgacgtgcag
gcatggtggc gggactccac gtgcacgcga ggcgaatggc gagccatgga atctgatact
aaagataatg gataa
【0035】
[カイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)の発現によるコアα1,3−フコース転移酵素活性の測定]
上記のとおりカイコより単離したcDNAがコアα1,3−FucTをコードすることを確認するため、翻訳産物の酵素活性を測定した。
1.対照実験
CHO細胞株の粗抽出液を用いて、ダブシル化グリコペプチドであるダブシル化GnGn−ペプチド(Dabsylated tetrapeptide asialo agalacto biantennery)を基質として、GDP-フコース(GDP-fucose)を反応させ、フコースがダブシル化GnGn−ペプチドへ結合した生成物をHPLCにて分析した。基質であるダブシル化GnGn−ペプチドをdabsylated GnGn peptideと略記し、以下にその構造を示す。
【化1】

【0036】
なお、コアα−フコース転移酵素(core alpha FucT)存在下の、ダブシル化GnGn−ペプチドとGDP-フコースの反応は一般的に次のように表される。
[ダブシル化GnGn−ペプチドとGDP-フコースの反応]
Core alpha FucT dabsylated GnGnF6 peptide 及び/又は
GDP-fucose+dabsylated GnGn -−−--→ dabsylated GnGnF3 peptide及び/又は
peptide dabsylated GnGnF3F6 peptide
F6:糖鎖コア構造の還元末端部位に存在するN-アセチルグルコサミン(GlcNAc)残基にα1,6-結合によりフコースが付加していることを表す。
F3:糖鎖コア構造の還元末端部位に存在するN-アセチルグルコサミン残基にα1,3-結合によりフコースが付加していることを表す。
F3F6:糖鎖コア構造の還元末端部位に存在するN-アセチルグルコサミン残基にα1,6-結合及び1,3-結合によりフコースが付加していることを表す。
【0037】
対照実験における操作手順及び条件は次のとおりである。
<CHO細胞の粗抽出液の調製>
CHO細胞を10% Fetal Bovine Serum(FBS)の存在下でIscove's Modified Dulbecco's Medium培地(Gibco-BRL)を用いて37℃で5%CO2条件下で培養し10cmの培養皿へ1/10希釈で継代し、3日間培養した。底面に付着している細胞をピペッティングにより剥し、遠心分離して上清を廃棄し、再びPBSバッファーに懸濁することで洗浄し、再度遠心分離して上清を廃棄後PBSバッファーを加え、細胞数を計測した。1.0×107cellsを1.5mlのサンプリングチューブへ加え、遠心にて上清を除去し、試料とした。
試料を1mM PMSF、0.1% NP40存在下のPBS(-)1mL、へ加え、sonicatorで30秒2回、氷上にて超音波破砕をおこない、その後19000xgで4℃10分にて遠心分離して上清を回収し、粗抽出液とした。粗抽出液中のタンパク質量はBioRad社製Protein Assay試薬を用いてブラッドフォード法により測定した。その際コントロールとしてBovine serum albuminを標準にして測定し、粗抽出液タンパク質量:22.5μgとした。
その後以下の試薬と混合し、反応温度37度にて所定の時間反応させ、定まった時間経過の後、60℃で5分加熱して反応を停止させた。MilliQ水を110μL添加して、0.45μmフィルターにてろ過し、ろ液をHPLC分析した。なお、GDP-Fucoseを加えない反応では、4μLの蒸留水を加え同様の操作を行った。
【0038】
<反応条件>
反応液の組成
1M MES-NaOH pH7.0 2μL
150mM MnCl2 2μL
1% Triton X-100 2μL
2.5mM GDP-Fucose 4μL
5nmol/μL 基質* 1μL
粗抽出液+MilliQ水 9μL
計 20μL
*dabsylated GnGn peptide(EMD社製)
【0039】
<HLPCの条件>
HLPC分析機器として、Gilson社製HPLCシステムを用いて分析した。
カラムはDevelosil ODS-HG-3 φ3.0×250mm(NOMURA CHEMICAL CO. LTD.)カラムを用い
流速:0.4mL/分、カラム温度:40℃の条件でおこなった。ダブシル化GnGn−ペプチドの検出は465nmの吸光度にておこない、分析したサンプル量を60μLとした。用いたsolventおよび溶出分離条件を以下に示す。
SolventA
1M リン酸ナトリウム(pH6.4) 10mL
アセトニトリル 50mL
Milli Q水 940mL
SolventB
1M リン酸ナトリウム(pH6.4) 10mL
アセトニトリル 500mL
Milli Q水 490mL
溶出分離条件
0分−4分 (B 30%)
4分−35分 (B 30%からB37%までの、直線濃度勾配)
35分−40分 (B 100%)
40分−60分 (B 30%)
【0040】
得られた結果を図7に示す(反応時間:17時間)。図7において、GDP-fucoseあり、と示されているクロマトグラムが表すように、ピークが2つ検出された。これは元の反応基質であるdabsylated GnGn(図7中、GnGnDABSと示される)、CHO細胞中に存在するコアα1,6-FucTによるフコース転移によって生じたdabsylated GnGnF6(図7中、GnGnF6と示されている)とである。一方で、GDP-fucoseを添加しなかったサンプルからは、反応基質以外のピークは検出されなかった。
このようにして、CHO細胞の内在性のコアα1,6-FucT活性を測定することができた。
【0041】
2.発現ベクター及びコアα1,3-FucT形質転換体の作製
カイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)を用いて、哺乳類細胞であるCHO細胞へ形質転換し、発現させた。
<コアα1,3-FucT形質転換体の作製手順>
Invitrogen社製の「pcDNA3.1/Hygro」を用いて、そのプロトコルに従って、全長コアα1,3-FucTのcDNA(ORF全長を含む)を、BamHI-XbaI siteを用いて導入した。CHO細胞へトランスフェクションし、ハイグロマイシンBによる300μg/mLでの選択の後、約1ヶ月選択し、その後細胞を回収し、RT-PCRによってRNAレベルでの転写産物発現の確認を行った。
【0042】
3.CHO細胞のコアα1,3-FucT形質転換体におけるコアα1,3−フコース転移酵素活性の分析
(1)上記のようにして得たCHO細胞のカイコのコアα1,3-FucT形質転換体(CHO-FucTと略記する)を1.0×107cells用いて、上記1.対照試験で記述した<粗抽出液の調製>に従ってCHO-FucT 粗抽出液を調製した(粗抽出液タンパク質量:22.5μg)。このCHO-FucT 粗抽出液を用いて対照試験と同様の<反応条件>及び<HLPCの条件>に従って、FucT活性を調べた。同時に、上記1.対照試験で記述したCHO細胞の粗抽出液をコントロールとして同様に調べた。
その結果を図8に示す(反応時間:24時間)。図8の破線のクロマトグラムが示しているように、カイコのコアα1,3-FucT候補cDNAを発現させたCHO細胞の粗抽出液を用いたサンプルからは、4本のピークが検出され、コントロールと比較してピーク数が増加していた。これはコントロールのCHO細胞と同様、内在性のコアα1,6-FucTによるGnGnF6生成に加えて、カイコのコアα1,3-FucTにより更に生じたGnGnF3およびGnGnF3F6であると推測することができる。
このデータは、得られたCHO-FucTの抽出液において、コアα1,3−フコース転移酵素活性が認められることを示唆している。
【0043】
(2)上記(1)の実験において、CHO-FucTの抽出液を用いるが但し反応液においてGDP-Fucoseを加えないものをコントロールとし、同様の実験を行った(粗抽出液タンパク質量:22.5μg、反応時間:24時間)。その結果を図9に示す。図9のグラフに示されるようにコントロール(GDP-fucose不在)では反応基質以外のピークは検出されなかった。GDP-fucose不在下では新たなピークが生じないことから、ピークの生成にはフコースの転移が関与していることが示唆される。
【0044】
4.N−グリコシダーゼ処理を用いた反応生成物の確認
上記3.で確認されたピークが、上記ダブシル化GnGn−ペプチドとGDP-フコースとの反応式に示すdabsylated GnGnF3 peptide及び/又はdabsylated GnGnF3F6peptideであることを確かめるために、図8に示される反応生成物に、N−グリコシダーゼA又はN−グリコシダーゼFを作用させ、糖鎖の切断を試みた。N−グリコシダーゼAは、上記反応式に示す、dabsylated GnGn peptide、dabsylated GnGnF6 peptide、dabsylated GnGnF3 peptide, dabsylated GnGnF3F6peptideの何れの糖鎖も切断することが可能であり、その結果dabsylated peptideが、その反応生成物として検出される。
しかし、N−グリコシダーゼFは、α1,3-結合したコアフコースを含む糖鎖は、切断することができないため、その反応性生物として、dabsylated peptide、dabsylated GnGnF3 peptide、dabsylated GnGnF3F6 peptideが検出される。
【0045】
試験条件は以下のとおりである。
<反応条件>
CHO細胞およびCHO細胞のFucT形質転換体細胞を其々1.0×107cells調整し、粗抽出液を回収した。粗抽出液中のタンパク質量はBioRad社製Protein Assay試薬を用いてブラッドフォード法により測定した。その際コントロールとしてBovine serum albuminを標準にして測定し、粗抽出液タンパク質量:46.2μgとした。
各粗抽出液を用いて以下の組成にて試薬と混合し、反応温度37℃にて24時間反応させ、その後、60℃5分加熱して反応を停止させた。
組成
1M MES-NaOH pH7.0 2μL
150mM MnCl2 2μL
1% Triton X-100 2μL
2.5mM GDP-Fucose 4μL
5nmol/μL 基質* 1μL
粗抽出液+Milli Q水 9μL
計 20μL
*N-Glycan Acceptor Substrate, Dabsylated tetrapeptide asialo agalacto biantennery EMD社製
こうして得た調製液に、MilliQ水を110μL添加して130μLの反応液とし、N−グリコシダーゼ処理をおこなった。
【0046】
CHO細胞及びカイコのコアα1,3- FucT発現CHO細胞(CHO-FucT)のそれぞれの粗抽出液について、上記反応を行ったのち、各々130μLの反応液から80μLをとり、以下、2種類のN−グリコシダーゼ処理を各々おこなった。N−グリコシダーゼF処理では、反応液80μLへN−グリコシダーゼF(1U)を1μL加え、37℃24時間反応させた後、90℃ 5分処理することで酵素を失活させた。その後0.45μmフィルターろ過を行い、HPLCサンプルとした。N−グリコシダーゼAではpHを調整する為、80μLの反応液に1M MES (pH5.1) を3.3μL加え、N−グリコシダーゼA(1U)を1μL加え、37℃24時間反応させた後、90℃ 5分処理することで酵素を失活させた。
その後1M MES pH7.0を5μL加えることでpHを調整し、0.45μmフィルターろ過を行い、HPLCサンプルとした。HPLCの条件は、上記の1.対照試験で記述したものと同じである。
【0047】
<N−グリコシダーゼ処理>
<N−グリコシダーゼF>
反応液 80μL
N−グリコシダーゼF(1U) 1μL
計 81μL
37℃ 24時間
90℃ 5分
0.45μmフィルターろ過を行い、HPLCサンプルとした。
<N−グリコシダーゼA>
反応液 80μL
1M MES pH5.1 3.3μL
N−グリコシダーゼA(1U) 1μL
計 84.4μL
37℃ 24時間
90℃ 5分
1M MES pH7.0 5μL
計 89.4μL
0.45μmフィルターろ過を行い、HPLCサンプルとした。
【0048】
CHO細胞の粗抽出液による結果を図10に示し、及びカイコのコアα1,3-FucT発現CHO細胞(CHO-FucT)の粗抽出液による結果を図11に示す。
CHO細胞の粗抽出液による図10のクロマトグラムから、N−グリコシダーゼA、及びN−グリコシダーゼFのどちらの処理においても、糖鎖部位の切断が生じたことから、コアα1,3-フコース転移以外のフコース転移であることが判る。一方カイコのコアα1,3-FucT発現CHO細胞の粗抽出液による図11のクロマトグラムから、N−グリコシダーゼAでは全ての糖鎖が切断されるのに対して、N−グリコシダーゼFでは糖鎖切断が生じない2つのピークが残り、この2つのピークはCHO細胞の粗抽出液を用いたN−グリコシダーゼF処理のデータ(図10参照)には見られないピークである。よって、これらのピークが、GnGnF3及びGnGnF3F6であると示唆される。
【0049】
5.反応時間と生成物の変化
反応時間を0〜24時間の間で変動させて、上記3.(1)の実験を行った(粗抽出液タンパク質量:25.5μg)。その結果を図12に表す。図12に示されるクロマトグラムは、経時的に基質量が減少し、その分、反応生成物が増加することを示しており、よって、新たに生成したピークは基質(dabsylated GnGn)の反応が関与していることが判る。
【0050】
以上の実験結果から、形質転換されていないCHO細胞株と比較することで、カイコのコアα1,3-FucTのcDNAで形質転換されたCHO−FucT細胞に特異的なピークの増加を確認することができた。そして、GDP−フコースの有無の実験、及び反応時間を変動させた実験から、各ピークの生成は、基質が関与したフコース転移反応であることが言える。そして、N−グリコシダーゼ処理を用いた反応生成物の確認結果から、コアα1,3-FucTのcDNAで形質転換されたCHO細胞の抽出液の反応により生成されたピークが、コアα1,3-フコース転移に起因するものであるといえる。
こうして、カイコのコアα1,3-FucT ORF 全長cDNA(配列番号1)が、コアα1,3−フコース転移酵素をコードすることを確認することができた。
【特許請求の範囲】
【請求項1】
鱗翅目のコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNA。
【請求項2】
カイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNA。
【請求項3】
配列番号1の塩基配列で表されるカイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNA。
【請求項4】
下記(a)又は(b)のタンパク質をコードするDNA。
(a)配列番号2のアミノ酸配列からなるタンパク質
(b)配列番号2のアミノ酸配列において1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
【請求項5】
下記(a)又は(b)のDNA。
(a)配列番号1の塩基配列からなるDNA
(b)配列番号1に示す塩基配列からなるDNAと相補的な塩基配列からなるDNAと、ストリンジェントな条件下でハイブリダイズし、かつコアα1,3−フコース転移酵素活性を有するタンパク質をコードするカイコ由来のDNA
【請求項6】
請求項1〜5のいずれか1項記載のDNAを含む発現ベクター。
【請求項7】
請求項6記載の発現ベクターを宿主細胞に導入して得られる形質転換体。
【請求項8】
該宿主細胞が哺乳類細胞株である、請求項7記載の形質転換体。
【請求項9】
下記(a)又は(b)の組換えタンパク質。
(a)配列番号2のアミノ酸配列からなるタンパク質。
(b)配列番号2のアミノ酸配列の1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
【請求項10】
請求項6記載の発現ベクターを宿主細胞に導入し、得られる形質転換体を培養し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法。
【請求項11】
請求項6記載の発現ベクターを宿主個体に導入し、得られる形質転換体を飼育し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法。
【請求項1】
鱗翅目のコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNA。
【請求項2】
カイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNA。
【請求項3】
配列番号1の塩基配列で表されるカイコのコアα1,3−フコース転移酵素活性を有するタンパク質をコードするDNA。
【請求項4】
下記(a)又は(b)のタンパク質をコードするDNA。
(a)配列番号2のアミノ酸配列からなるタンパク質
(b)配列番号2のアミノ酸配列において1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
【請求項5】
下記(a)又は(b)のDNA。
(a)配列番号1の塩基配列からなるDNA
(b)配列番号1に示す塩基配列からなるDNAと相補的な塩基配列からなるDNAと、ストリンジェントな条件下でハイブリダイズし、かつコアα1,3−フコース転移酵素活性を有するタンパク質をコードするカイコ由来のDNA
【請求項6】
請求項1〜5のいずれか1項記載のDNAを含む発現ベクター。
【請求項7】
請求項6記載の発現ベクターを宿主細胞に導入して得られる形質転換体。
【請求項8】
該宿主細胞が哺乳類細胞株である、請求項7記載の形質転換体。
【請求項9】
下記(a)又は(b)の組換えタンパク質。
(a)配列番号2のアミノ酸配列からなるタンパク質。
(b)配列番号2のアミノ酸配列の1若しくは数個のアミノ酸が欠失、置換もしくは付加されたアミノ酸配列からなり、かつコアα1,3−フコース転移酵素活性を有するタンパク質
【請求項10】
請求項6記載の発現ベクターを宿主細胞に導入し、得られる形質転換体を培養し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法。
【請求項11】
請求項6記載の発現ベクターを宿主個体に導入し、得られる形質転換体を飼育し、及び生産された組換えタンパク質を回収することを含む、コアα1,3−フコース転移酵素活性を有する組換えタンパク質を生産する方法。
【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図1】


【図2】


【図3】


【図4】




【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図1】


【図2】


【図3】


【図4】


【公開番号】特開2012−29589(P2012−29589A)
【公開日】平成24年2月16日(2012.2.16)
【国際特許分類】
【出願番号】特願2010−170078(P2010−170078)
【出願日】平成22年7月29日(2010.7.29)
【出願人】(000231637)日本製粉株式会社 (144)
【出願人】(399032282)株式会社 免疫生物研究所 (14)
【Fターム(参考)】
【公開日】平成24年2月16日(2012.2.16)
【国際特許分類】
【出願日】平成22年7月29日(2010.7.29)
【出願人】(000231637)日本製粉株式会社 (144)
【出願人】(399032282)株式会社 免疫生物研究所 (14)
【Fターム(参考)】
[ Back to top ]

