シトクロムP4501A2、2A6及び2D6、PXR及びUDP−グルクロノシルトランスフェラーゼ1A族遺伝子の遺伝型分析のためのHTSNPS、及びそれを用いた遺伝子多重分析法
【課題】シトクロムP450 1A2、2A6及び2D6、PXR及びUDP−グルクロノシルトランスフェラーゼ1A族遺伝子の遺伝型分析のためのhtSNPs、及びそれを用いた遺伝子多重分析法を提供する。
【解決手段】本発明は、シトクロムP4501A2(CYP1A2)、2A6(CYP2A6)及び2D6(CYP2D6)、PXR及びUDP−グルクロノシルトランスフェラーゼ1A族(UGT1A)遺伝子の遺伝型分析のためのhtSNP及びこれを用いた遺伝子分析チップに関し、より詳細には、ヒトCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A遺伝子のハプロタイプ分析のためのhtSNPの選別方法、前記htSNPを用いて前記遺伝子の遺伝型を決定する方法及びそのための遺伝子分析チップに関するものである。
【解決手段】本発明は、シトクロムP4501A2(CYP1A2)、2A6(CYP2A6)及び2D6(CYP2D6)、PXR及びUDP−グルクロノシルトランスフェラーゼ1A族(UGT1A)遺伝子の遺伝型分析のためのhtSNP及びこれを用いた遺伝子分析チップに関し、より詳細には、ヒトCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A遺伝子のハプロタイプ分析のためのhtSNPの選別方法、前記htSNPを用いて前記遺伝子の遺伝型を決定する方法及びそのための遺伝子分析チップに関するものである。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、シトクロムP450 1A2(CYP1A2)、2A6(CYP2A6)及び2D6(CYP2D6)、PXR及びUDP−グルクロノシルトランスフェラーゼ1A族(UGT1A)遺伝子の遺伝型分析のためのhtSNP、及びそれを用いた遺伝子分析チップに関し、より詳しくは、ヒトCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子のハプロタイプ分析のためのhtSNPの選別方法、前記htSNPを用いて前記遺伝子の遺伝型を決定する方法及びそのための遺伝子分析チップに関するものである。
【背景技術】
【0002】
個体間の遺伝的多様性は薬物の毒性及び効能において個体間の差異をもたらす。従って、薬物開発の初期段階でこのような薬剤学的に重要な蛋白質の遺伝的多様性に関する効果を考慮するのは、薬物開発の失敗に対する危険性を低めることができる重要な要素である。このような個体間遺伝的多様性を測定する一つの調査集団となるのが“ハプロタイプ(haplotype)”である。ハプロタイプとは、一つの調査集団内に存在する各遺伝子配列の多型性の組み合わせを意味し、これは個別的な多型性よりも遺伝的多様性に関するより正確かつ信頼できる情報を提供する。
【0003】
一方、ヒトシトクロムP450は、薬物、発癌原及び毒素のような外来化学物質とステロイド、脂肪酸及びビタミンのような内部気質の酸化を促進するヘム蛋白質の一員である(Nelson et al.,Pharmacogenetics 6:1-42,1996)。肝を含む腎臓、腸管及び肺のような器官で多様なシトクロムP450の亜型が発見された。
【0004】
ヒトシトクロムP450 1A2(Cytochrome P450 1A2、以下‘CYP1A2’という)はCYP1A1、CYP1B1と共にCYP1族に属する薬物代謝酵素である。CYP1A2は肝で主に生産され、総シトクロムP450酵素量の15%を占める。この酵素はカフェイン(caffeine)、クロザピン(clozapine)、イミパラミン(imiparamine)、プロプラノロール(propranolol)を含む医学的に重要な薬物の代謝に関与する。この酵素は、また17β−エストラジオール(estradiol)、ウロポルフィリノーゲン(uroporphyrinogen)IIIのような内部合成物質とポリサイクリック芳香族炭化水素エポキシ化(polycyclic aromatic hydrocarbon epoxidation)と芳香族/ヘテロサイクリックアミンN−ヒドロキシ化(aromatic/heterocyclic amine N-hydroxylation)のような発癌物質生活性化(carcinogen bioactivation)反応を触媒する(Brosen K.,Clinical Pharmacokinetic, 1995,29(suppl1):20-25;Josephy PD.,Environ. Mol. Mutagen,2001,38:12-18)。
【0005】
また、ヒトシトクロムP450 2A6(Cytochrome P450 2A6、以下‘CYP2A6’という)遺伝子は19番目染色体に位置しており、CYP2A6遺伝子の上流には塩基配列類似性の高い類似遺伝子(pseudogene)であるCYP2A7が位置している。
【0006】
CYP2A6酵素はニコチン(nicotine)をコチニン(cotinine)に変換する機能を有し、ニコチン代謝の約80%に関与する重要な酵素であり、生体内で抗癌剤であるテガフール(tegafur)を活性型である5−フルオロウラシル(5-fluorouracil、5-FU)に変換させる過程に関与する酵素である。前記酵素は肝で主に生産され、肺、大腸、乳房、腎腸及び子宮のような臓器では少量発現する(Drug Metab Dispos.,29(2):91-5,2001;Adv Drug Deliv Rev.,18;54(10):1245-56,2002)。
【0007】
また、ヒトシトクロムP450 2D6(Cytochrome P450 2D6、以下‘CYP2D6’という)遺伝子は22番目染色体に位置しており、CYP2D6遺伝子の一方の面には類似遺伝子(pseudogenes)であるCYP2D7とCYP2D8が位置している。前記遺伝子によりコーディングされる酵素は精神活性薬物(psychoactive drugs)、心血管系薬物、モルヒネ系薬物など、100種類以上の臨床的に重要な薬物を代謝することが知られている。
【0008】
CYP2D6遺伝子によりコーディングされる酵素は、肝で主に生産され、シトクロムP450酵素の総量の約2%を占めるが、30%程度の薬物代謝に関与する重要な酵素である。
【0009】
個体内で前記酵素の活性は非常に多様であり、これらはその活性程度によりそれぞれPM(poor metabolizers)、IM(intermediate metabolizers)、EM(extensive metabolizers)及びUM(ultrarapid metaboilzers)群に分類されている。前記のように酵素の活性がそれぞれ異なるように現れるのは、前記遺伝子の遺伝的多型性による原因もある。CYP1A2の遺伝子は遺伝的多型性を現わすことが知られているが、現在までこの遺伝子のプロモーター、エクソン及びイントロンで24種以上の変異が発見され、これら変異遺伝子の組み合わせであるハプロタイプは2007年6月現在36種類(http://www.cypalleles.ki.se/cyp1a2.htm)、CYP2A6の遺伝型は50種類(http://www.cypalleles.ki.se/cyp2a6.htm)、CYP2D6の遺伝的多型性は約80種類知られており(www.imm.ki.se/cypalleles/cyp2d6.htm)、種族間の明確な差異を示している。しかしながら、このような遺伝子変異とハプロタイプに対して多様な種類が報告されているため、分析時間と費用の効率性を増大するために最小限の単一塩基多型(single nucleotide polymorphism、SNP)を通じて正確なハプロタイプを決定することが重要である。
【0010】
一方、多様な種類の薬物が体内に入って排出されるまで体内では代謝と輸送が起こるが、ここに関与する酵素系としては、シトクロムP450(CYP)と薬物輸送蛋白質がある。この中で薬物の酸化代謝に関与するCYP酵素に対する研究が多く行われており、現在15種類程度のCYP酵素が遺伝的多型性を示すことが報告されており、代表的なものとしてCYP2D6、CYP2C9、CYP3A4、CYP2B6、MDR1及びCYP2C19などがある。遺伝的な多型性はこれら酵素の基質薬物の臨床効果、治療効果及び副作用の発生に影響を与える重要な因子として作用するようになる。これらの中で一部の変異遺伝子は酵素喪失を招いて薬物代謝能が全くない形態で現れ、一部の変異遺伝子は酵素活性度が一部減少する形態で現れることもある。例えば、CYP2D6及びCYP2C9などのような酵素は変異遺伝子により表現型が変わり、比較的に遺伝型と表現型間に高い相関性を示す反面、CYP3A4、CYP2B6及びMDR1などの遺伝子は機能性変異遺伝子の有無から表現型を予測するのに困難がある。
【0011】
ヒトの場合、服用する薬物の50%以上を代謝させるCYP3A4は個体間に多くの活性差異を示し、CYP2B6の場合も個体間の差異が最大270倍程度生じることが知られている。このような個人差にもかかわらず、個人による活性の差異は遺伝型から直ちに予測し難いが、これはこれら遺伝型と表現型の相関性が低い薬物代謝酵素や薬物輸送蛋白質の場合、蛋白質の発現が外部因子により顕著に変わることがあるためである。従って、これら遺伝子の場合、変異遺伝子の有無よりは蛋白質の発現調節が代謝活性の個人差を招くより重要な要素になるだろう。また、酵素の発現誘導により酵素の生産量自体が多くなり、活性が増加する結果を示すことがあるが、これら発現誘導メカニズムは外部物質が薬物受容体と結合して標的遺伝子のプロモーターに結合することによって行われる。このような薬物受容体の代表的なものとして、プレグナンX受容体(Pregnane X receptor;PXR)がある。PXR受容体はNR1I2という遺伝子で発現することが知られている。
【0012】
PXRは個人間発現量の差異が報告されており、興味深い点は受容体の発現量がCYP3A4とCYP2B6のような薬物代謝酵素の発現量と高い相関性を示すということである(Current Drug Metabolism, 2005,6:369-383)。従って、個体間の薬物代謝酵素の発現量差異は変異型蛋白質によるものというよりはPXR遺伝子の発現量や活性差異に起因する。
【0013】
このような脈絡で、最近、個人別PXR遺伝子の遺伝的多型性(polymorphism)研究や発現差異に対する研究が関心を引いているが、アミノ酸の配列に変化がないにもかかわらず、人体でエリスロマイシン呼吸検査法(erythromycin breath test)またはリファムピン(rifampin)によるCYP3A4活性増加などの薬物反応に個人間の差異を招く一部の変異遺伝子が報告されたことがある(Pharmacogenetics,2001,11:555-572)。このようなPXRの変異は標的遺伝子の発現変化に応じた活性変化を招くため、薬物や生体分子の体内動態の差異を招くこともでき、PXRの結合子である併用薬物による薬物相互作用の個人差にも大きく寄与する。
【0014】
一方、UDP−グルクロノシルトランスフェラーゼ(UDP-glucuronosyltransferase;UGT)は生体内で耐因性及び外因性物質にグルクロン酸が接合する反応を触媒する酵素として、フェノール、アルコール、アミン及び脂肪酸化合物などのように生体毒性を有する多様な物質のグルクロン酸接合体を生成させることによって、水溶性物質に転換させて胆汁や尿で排出されるようにする(Parkinson A,Toxicol Pathol.,24:48-57,1996)。
【0015】
このようなUGTは肝細胞の小胞体及び核膜に主に存在することが報告されており、腎腸及び皮膚のような他の組織でもその発現が報告されたことがある。UGT酵素は一次アミノ酸配列間の類似性に基づいて大きくUGT1及びUGT2の亜族に区分することができ、この中でヒトUGT1A族の場合には9種類(UGT1A1、及びUGT1A3乃至UGT1A10)の異性体が報告されており、その中の5種類(UGT1A1、UGT1A3、UGT1A4、UGT1A6、及びUGT1A9)が肝で発現することが知られている。
【0016】
このようなUGT1A族遺伝子は個人毎に遺伝的多型性を有することが知られており、最近までUGT1A族の遺伝的多型性はUGT1A1、及びUGT1A3乃至UGT1A10のそれぞれにより多様な種類が存在することが知られている(http://galien.pha.ulaval.ca/alleles/alleles.html)。UGT1A族遺伝子群の多型性は特に種族間に明確な差異を見せており、このような多型性により酵素の活性が異なるように現れることが確認され、薬物治療などに対する感受性を決定する要因として重要視されている。また、UGT1A1*6とUGT1A1*28はギルバート(Gilbert)症候群と関連があり(Monaghan G,Lancet,347:578-81,1996)、その他にも多様な疾病と関連した多様な機能的な変異型が報告されている。
【0017】
ところで、このような遺伝子変異とハプロタイプは多様な種類が報告されているため、検索方法の効率性を考えざるを得ない。ハプロタイプはArlequin、SNPAlyzeまたはこれらと類似するソフトウェアを用いて分析が可能である。各ハプロタイプに対する遺伝子変異検索を全ての単一塩基多型に対して分析するとすれば、費用及び時間の面で効率性を期待し難い。
【0018】
効率性増大のための方法として、ハプロタイプタグ単一塩基多型(haplotype-tagging SNP;htSNP)選別技術を挙げることができる。htSNP選別技術は、それぞれのハプロタイプの正確な表示のために最小限の標識セットの選択を助けるための方法として、選別された単一塩基多型(SNP)のみを確認する場合、全てのハプロタイプを予測できる。
【0019】
多くの遺伝子の場合に遺伝的多型性の分布は種族による差異を示すことが知られているため、前記遺伝子の場合にも韓国人に頻繁に発生される固有遺伝子変異及びハプロタイプはないか、あるとすれば頻度はどのくらいか、それぞれのハプロタイプに応じたhtSNPの選別はどのようになるか、確認する必要がある。しかしながら、韓国人において前記遺伝子の変異、これによるハプロタイプ及びこれを分析するためのhtSNPに対する研究は非常に不十分であるのが実情である。
【0020】
また、最近西洋人で主に発見されるCYP2D6遺伝子変異を中心にスナップショット(SNaPshot)分析を用いて11個のSNPを分析する方法(Sistonen J et al.,Clin Chem. 2005 Jul;51(7):1291-5)、及びロシュ社やジュリーラブ社のCYP2D6診断チップが報告された。しかしながら、これらはCYP2D6変異遺伝子の組み合わせが西洋人で発見される変異を中心に形成されているため、韓国人を含む東洋人に対するCYP2D6遺伝子変異診断に関する研究は非常に不十分であるのが実情である。
【0021】
そこで、本発明者等は韓国人で主に発見されるヒトCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子の変異を短時間内に正確に確認できる方法を開発するために研究を行い、韓国人で主に発見されるヒトCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子の変異に対するhtSNP選別方法及び前記選別されたhtSNPの有用性を確認することによって本発明を完成した。
【発明の概要】
【発明が解決しようとする課題】
【0022】
従って、本発明の目的は、韓国人で発見されるCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子のハプロタイプを分析できるhtSNPを選別する方法を提供することにある。
また、本発明の他の目的は、前記htSNPを用いてヒトCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子の遺伝型を決定する方法を提供することにある。
【0023】
また、本発明のさらに他の目的は、遺伝子分析チップを含むキットを用いてヒトCYP2D6遺伝子の遺伝型を決定する方法を提供することにある。
【課題を解決するための手段】
【0024】
前記のような目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A遺伝子からなる群より選択された遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記(c)工程で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;(e)前記(d)工程で変異が存在すると確認されたPCR産物の塩基配列において、ハプロタイプ(haplotype)を分析する工程;及び(f)前記(e)工程で分析されたハプロタイプの塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析し、htSNPを選別する工程を含む、ヒトCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1Aからなる群より選択される遺伝子のhtSNPを選別する方法を提供する。
【0025】
本発明の他の目的を達成するために、本発明は、(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;(c)前記(b)工程のゲノムDNAの鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G、−163C>A、1514G>A、2159G>A、2321G>C、3613T>C、5347C>T及び5521A>Gからなる群より選択される少なくとも11個のCYP1A2遺伝子の変異の存在有無を調査する工程を含む、ヒトCYP1A2遺伝子の遺伝型を決定する方法を提供する。
【0026】
本発明のさらに他の目的を達成するために、本発明は、(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;(c)前記(b)工程のゲノムDNAを鋳型とし、ヒトCYP1A2遺伝子のプロモーター領域を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G及び163C>Aを含むCYP1A2遺伝子の変異の存在有無を調査する工程を含む、CYP1A2プロモーター遺伝子の変異を検出する方法を提供する。
【0027】
本発明のさらに他の目的を達成するために、本発明は、(a)被験者から生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び(d)前記工程(c)で得られたPCR産物の塩基配列において、−48T>G、13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>T、及び6091C>Tからなる群より選択されるCYP2A6遺伝子変異の存在有無を調査する工程を含む、ヒトCYP2A6遺伝子の遺伝型を決定する方法を提供する。
【0028】
本発明のさらに他の目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D欠失(deletion);及び2D6重複(duplication)を含む少なくとも11個のCYP2D6遺伝子変異の存在有無をSNaPshot技法を用いて調査する工程を含む、ヒトCYP2D6遺伝子の遺伝型を決定する方法を提供する。
【0029】
本発明のさらに他の目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記工程(c)で得られたPCR産物の塩基配列において、−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cからなる群より選択されたPXR遺伝子の遺伝子変異の存在有無を調査する工程を含む、PXR遺伝子の遺伝型を分析する方法を提供する。
【0030】
本発明の他の目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)で採取された試料から核酸を抽出する工程;(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族個別遺伝子増幅する工程;及び(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での−39(TA)6>(TA)7、211G>A、233C>T及び686C>A;UGT1A3での31T>C、133C>T及び140T>C;UGT1A4での31C>T、142T>G及び292C>T;UGT1A6での19T>G、541A>G及び552A>C;UGT1A7での387T>G、391C>A、392G<A、622T>C及び701T>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子群の機能的変異型の存在有無を確認する工程を含む、UGT1A族遺伝子群の機能的変異型を決定する方法を提供する。
【0031】
本発明のさらに他の目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)で採取された試料から核酸を抽出する工程;(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族遺伝子を増幅する工程;及び(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での211G>A、233C>T及び686C>A;UGT1A6での19T>G、541A>G及び552A>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子変異型の存在有無を確認する工程を含む、イリノテカン(irinotecan)に対する感受性と関連したGT1A族遺伝子の多型性を決定する方法を提供する。
【0032】
本発明のさらに他の目的を達成するために、本発明は、(a)検査しようとする遺伝子を抽出した後、多重(multiplex)PCRを遂行して同定しようとするSNP周辺を含むPCR産物を得る工程;(b)各対立遺伝子(allele)の特異的な塩基を同定できるASPE(allele specific primer extension)プライマーを用いてASPE反応を遂行する工程;(c)前記反応産物を遺伝子チップに混成化させる工程;及び(d)前記チップを分析する工程を含む、遺伝子分析チップを用いてヒトCYP2D6遺伝子の遺伝型を決定する方法を提供する。
【0033】
また、本発明は、CYP2D6遺伝型の判別のためのSNaPshot方法の遺伝型分析用キット及びSNP検査用ジップ・コード(ZiP Code)オリゴ塩基チップを含む、遺伝型分析用チップを提供する。
【0034】
本発明で“生物学的試料”は、被験者の血液、皮膚細胞、粘膜細胞及び毛髪を含み、好ましくは血液であっても良い。
【0035】
本発明で核酸は、DNAまたはRNAであっても良く、好ましくはDNA、より好ましくはゲノム(genomic)DNAであっても良い。
【0036】
以下、本明細書に記載された変異について説明する。
本発明で用語“aN>M”または“NaM”(この時、aは整数、N及びMはそれぞれ独立的にA、C、TまたはGである。)は、遺伝子塩基配列でa番目N塩基がM塩基に置換されたことを意味し、“ainsN”または“adelN”(この時、aは整数、NはA、C、TまたはGである。)は、遺伝子塩基配列でa番目にN塩基がもう一つ挿入され、または欠失されたことを意味する。
【0037】
例えば、“−1584C>T”変異とは、ヒトCYP2D6遺伝子の塩基配列で−1584番目塩基がCからTに置換されたことをいう。
【0038】
“2573insC”変異とは、ヒトCYP2D6遺伝子の塩基配列で2573番目塩基にCが挿入(付加)されたことをいい、“4125〜4133insGTGCCCACT”変異とは、ヒトCYP2D6遺伝子の4125番目塩基から4133番目塩基の位置にGTGCCCACTの9個の塩基が挿入されたことをいう。
【0039】
また、“2D6欠失(deletion)”変異とは、ヒトCYP2D6遺伝子全体が染色体上から欠失されたことをいう。
【0040】
ひいては、本発明で“2D6重複(duplication)”変異とは、ヒトCYP2D6遺伝子が二以上同一の染色体上に重複していることをいう。
【図面の簡単な説明】
【0041】
【図1】CYP1A2遺伝子の塩基配列において、本発明で初めで糾明したCYP1A2遺伝子の1個の変異の位置を示したものである。
【図2】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの一例である。
【図3】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの多の一例である。
【図4】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの他の一例である。
【図5】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの他の一例である。
【図6】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの他の一例である。
【図7】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの全ての単一塩基多型が野生型である遺伝子の変異を検索した結果を示す(X軸は各プライマーの分子量による移動程度、Y軸は各ピークの高さである。以下図14まで同様)。
【図8】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−3860G>A(CYP1A2*1C)、−2467delT(CYP1A2*1D)及び163C>A(CYP1A2*1F)の変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(hetero)変異遺伝子の場合の結果を示す。
【図9】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの3860G>A(CYP1A2*1C)、−2467delT(CYP1A2*1D)及び163C>A(CYP1A2*1F)の位置が二本のDNAの全てが変異型を有している同型接合型(homo)変異遺伝子である場合の結果を示す。
【図10】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)及び2808A>Cの位置が異型接合型変異遺伝子である場合の結果を示す。
【図11】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)は同型接合型変異、−2467delT(CYP1A2*1D)、−739T>G(CYP1A2*1E)、−3598G>T、−3113G>A、−2847T>C及び1708T>Cの位置は異型接合型変異遺伝子である場合の結果を示す。
【図12】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)は同型接合型変異、−2467delT(CYP1A2*1D)、−3598G>T及び2847T>Cの位置は異型接合型変異遺伝子である場合の結果を示す。
【図13】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)、−2467delT(CYP1A2*1D)及び3594T>Gの位置は異型接合型変異遺伝子である場合の結果を示す。
【図14】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)は同型接合型変異、−3860G>A(CYP1A2*C)、−2467delT(CYP1A2*1D)及び2603insAの位置は異型接合型変異遺伝子である場合の結果を示す。
【図15】本発明でhtSNP組み合わせを選別するために用いたCYP2A6の韓国人でのハプロタイプの種類及び頻度を示す。
【図16】本発明による、機能性変異6種類と機能性が疑われる3種類の変異遺伝子を遺伝子変異検査対象に含むCYP2A6遺伝子のハプロタイプを分析するためのhtSNP組み合わせの選別を例示したものである。
【図17】本発明による、アミノ酸置変異が8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含むCYP2A6遺伝子のハプロタイプを分析するためのhtSNP組み合わせの選別を例示したものである。
【図18】本発明による、アミノ酸置換変異8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含むCYP2A6遺伝子のハプロタイプを分析するための他のhtSNP組み合わせの選別を例示したものである。
【図19】本発明による、アミノ酸置換変異8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含むCYP2A6遺伝子のハプロタイプを分析するための他のhtSNP組み合わせの選別を例示したものである。
【図20】本発明による、アミノ酸置換変異8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含んでCYP2A6遺伝子のハプロタイプを分析するための他のhtSNP組み合わせの選別を例示したものである。
【図21】本発明による、アミノ酸置換変異8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含んでCYP2A6遺伝子のハプロタイプを分析するための他のhtSNP組み合わせの選別を例示したものである。
【図22】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の−48T>G、2134A>G及び6558T>Cの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(hetero)変異遺伝子対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図23】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の567C>Tの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図24】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の6458A>T及び6558T>Cの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図25】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の−48T>G、13G>A及び6558T>Cの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図26】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の3391T>Cの変異位置が一本は変異型、他の一本は欠失されたタイプの異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図27】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の−48T>G及び2134A>Gの変異位置が一本は変異型、他の一本は欠失されたタイプの異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図28】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の−48T>G、6558T>C、及び6600G>Tの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図29】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の6458A>Tの変異位置が一本は変異型、他の一本は欠失されたタイプの異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図30】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の6558T>C及び6582G>Tの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図31】CYP2A6遺伝子が相同染色体に正常に存在する遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図32】CYP2A6遺伝子が一つの染色体には存在せず、一つの遺伝子だけを有する遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図33】CYP2A6遺伝子が欠失されてCYP2A6遺伝子の一部の残った部分とCYP2A7遺伝子の接合形態にある遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図34】本発明で選別されたCYP2D6のhtSNP組み合わせの例を示す。
【図35】本発明で選別されたCYP2D6のhtSNP組み合わせの他の例を示す。
【図36】本発明で選別されたCYP2D6のhtSNP組み合わせの他の例を示す。
【図37】本発明で選別されたCYP2D6のhtSNP組み合わせの他の例を示す。
【図38】本発明で選別されたCYP2D6のhtSNP組み合わせの例を示す。示したものである。
【図39】本発明で選別されたCYP2D6のhtSNP組み合わせの例を示す。
【図40】本発明で選別されたCYP2D6のhtSNP組み合わせの1種類に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図41】本発明で選別されたCYP2D6のhtSNP組み合わせの1種類に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図42】遺伝子分析チップを用いてCYP2D6の遺伝型を分析する過程を示す。
【図43】CYP2D6の遺伝子分析チップ上のプローブを示す。
【図44】ロング(long)PCRを用いたCYP2D6遺伝子の増幅を示す。
【図45】ASPE反応過程を示す。
【図46】実施例12によりCYP2D6遺伝子の変異を分析した結果を示す遺伝子チップである。
【図47】本発明で選別されたPXR遺伝子のhtSNP組み合わせの例を示す。
【図48】本発明の方法により選別された−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cが全て野生型であるPXR遺伝子の機能的変異型を検索した結果を示す(X軸は各プライマーの分子量による移動程度、Y軸は各ピークの高さを示す。)。
【図49】本発明の方法により選別された−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(hetero)変異遺伝子であるPXR遺伝子の機能的変異型を検索した結果を示す。
【図50】本発明の方法により選別された−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cの位置が二本のDNAの全てが変異型を有している同型接合型(homo)変異遺伝子であるPXR遺伝子の機能的変異型を検索した結果を示す。
【図51】韓国人50名を対象としてUGT1A1(a)及びUGT1A3(b)族遺伝子の機能的変異型を分析した結果を示す(図中、Tはチアミン、Cはシトシン、Gはグアニン、Aはアデニンを意味する。)。
【図52】韓国人50名を対象としてUGT1A4(a)及びUGT1A6(b)族遺伝子の機能的変異型を分析した結果を示す。
【図53】韓国人50名を対象としてUGT1A7族遺伝子の機能的変異型を分析した結果を示す。
【図54】韓国人50名を対象としてUGT1A9族遺伝子の機能的変異型を分析した結果を示す。
【図55】韓国人50名を対象としてUGT1A1、UGT1A6及びUGT1A9族遺伝子のイリノテカン感受性関連多型性を分析した結果であり、(a)は、UGT1A1で211G>A、233C>T及び686C>A;UGT1A6で19T>G、541A>G及び552A>C;及びUGT1A9で726T>G及び766G>Aが全て野生型でC>Aが異型(hetero);UGT1A6で19T>G及び552A>Cが異型、541A>Gが野生型;及びUGT1A9で726T>G及び766G>Aが野生型である場合の、(c)は、UGT1A1で211G>A、233C>T及び686C>Aが野生型;UGT1A6で19T>G、541A>G及び552A>Cが異型;及びUGT1A9で726T>Gが異型、766G>Aが野生型である場合の、(d)は、UGT1A1で211G>A及び233C>Tが異型、686C>Aが野生型;UGT1A6で19T>G、541A>G及び552A>Cが異型;及びUGT1A9で726T>G及び766G>Aが野生型である場合の結果である。
【発明を実施するための形態】
【0042】
本発明の上記した特徴及び/または他の特徴は添付図面を参照した下記の実施態様の説明により明らかになり、また容易に理解できるであろう。
【0043】
1.CYP2A6
本発明は、韓国人CYP1A2遺伝子の変異分析を通じて韓国人において発見されるCYP1A2遺伝子の遺伝型を糾明し、これに基づいてそれぞれのハプロタイプの最適の標識セットであるhtSNPを選別し、その有用性を確認したという点に特徴がある。また、本発明は、ヒトCYP1A2遺伝子の新規なハプロタイプを糾明したという点に特徴がある。
【0044】
本発明によるヒトCYP1A2遺伝子のhtSNPを選別する方法は次の工程を含む:
(a)被験者から生物学的試料を採取する工程;
(b)前記(a)工程で採取された試料から核酸を抽出する工程;
(c)前記(b)工程の核酸を鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;
(d)前記(c)工程で得られたPCR産物の塩基配列を分析して変異の存在有無を確認する工程;及び
(e)前記(d)工程で変異が存在すると確認されたPCR産物の塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析する工程。
【0045】
前記(a)工程で採取された試料から核酸を抽出する方法は、特に限定されず、当業界に公知となった技術または市販されている抽出用キットを使用することができる。例えば、DNAまたはRNA抽出用キットはQiagen(米国)及びStratagene(米国)で購入できる。前記でRNAを抽出して使用する場合には逆転写によりcDNAを製造して使用する。
【0046】
前記(c)工程で前記ヒトCYP1A2遺伝子の断片は、ヒトCYP1A2遺伝子の公知となった変異、例えば単一塩基多型(single nucleotide polymorphism;SNP)を含んでいる断片をいう。前記ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーは、ヒトCYP1A2遺伝子またはその断片の塩基配列に基づいてデザインすることができ、例えば、配列番号2乃至配列番号31のプライマーからなる群より選択されることができるが、これに制限されない。
【0047】
前記(d)工程で変異は、単一塩基多型、遺伝子の欠失及び遺伝子の重複を含むが、これに制限されず、例えば、表5に示す17個の変異を含むことができる。
【0048】
また、塩基配列を分析する方法としては、特に限定されず、当業界に公知となった方法を使用することができる。例えば、その配列部分を直接決定するために、自動塩基配列分析器を使用するか、またはパイロシーケンシング(pyrosequencing)を遂行できる。前記パイロシーケンシングはDNAシーケンシングに利用されることもある公知のSNP分析方法として、DNAが重合される間に放出されるPPi(inorganic pyrophosphate)の光の発現を検出する方法である。前記塩基配列分析は、例えば、配列番号32乃至61のプライマーからなる群より選択されたプライマーを用いて遂行されることができるが、これに制限されない。
【0049】
また、前記(d)工程で変異の存在有無は、野生型CYP1A2遺伝子の塩基配列と比較して確認できる。野生型CYP1A2遺伝子の塩基配列、例えば、配列番号1(GenBank accession No.:NT_010194)の塩基配列、または当業界に公知となったCYP1A2遺伝型の各塩基配列に基づいて比較して確認できる(Drug Metab. Pharmacokinet,2005,20(1):24-33)。
【0050】
前記でハプロタイプの頻度及び種類を予測することは当業界で公知の技術プログラムまたは市販されるプログラムを使用して分析できる。例えば、Haploviewプログラムは無料で配布されるプログラムで利用でき、SNPAlyzeのような商用化されたプログラムを使用することもできる。前記 Haploviewソフトウェアは当業界に公知となっており、好ましくはインターネットウェブサイトである http://www.broad.mit.edu/mpg/haploview を利用することができる。
【0051】
本発明の方法は前記(a)乃至(d)工程を反復する工程を追加的に含むことができる。種族や患者などのようなある特定集団におけるCYP1A2遺伝子の変異様相及びこれに対するハプロタイプを分析しようとする場合には、各CYP1A2遺伝型の頻度を調査して前記集団で多く発見されるCYP1A2遺伝型を選定した後、これを対象として以降の(e)工程を遂行することができる。
【0052】
前記(e)工程では、前記(e)工程で予測されたCYP1A2のハプロタイプ資料をSNPタガーソフトウェアで分析してhtSNPを選別する。前記SNPタガーソフトウェアは当業界に公知となっており、例えば、Genehunter、 Merlin、Allegro、SNPHAP、htSNP finder(PCA based)などが挙げられ、好ましくは、http://www.well.ox.ac.uk/〜xiayi/haplotypeまたは http://slack.ser.man.ac.uk/progs/htsnp.htmlのウェブサイトを利用することができる。
【0053】
このように選別されたhtSNPはディプロタイプ(diplotype)決定時に正確度を高めるために検証され得る。人体の遺伝型は二本の染色体により決定されるので、遺伝型を判読すれば二つのハプロタイプ組み合わせで判定される。しかしながら、複数個のSNPを同時に分析した場合、特定のハプロタイプの組み合わせが他のハプロタイプの組み合わせと同一な変異分析結果を示す可能性がある。従って、本発明により開発された診断法を用いて遺伝型が判読された場合、正確な遺伝型を決定できることが検証されなければならない。このような検証は、Matlab(The Math Works Inc., 米国)プログラムを用いて遺伝子分析結果から正確な遺伝型判読が可能である否かが分析される。
【0054】
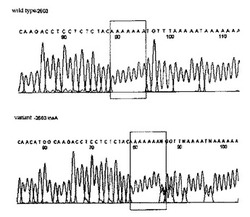
本発明の一実施例では、韓国人で発見されるCYP1A2遺伝型に対するhtSNPを選別するために、まず韓国人におけるCYP1A2遺伝子の変異を調査した。その結果、韓国人のCYP1A2遺伝子で総17個の単一塩基多型を発見した(表5参照)。その中の1個の単一塩基多型(−2603insA)は新規なものである。
【0055】
本発明で初めて提供される前記1個の単一塩基多型は二本のDNAのうち、一本は変異型、他の一本は野生型を有していることが示された(図1参照)。
【0056】
本発明の他の実施例では、韓国人で発見される17個の単一塩基多型によるハプロタイプを分析した。その結果、本発明では今まで明らかになったことのないCYP1A2遺伝子に対する韓国人のハプロタイプ(表6参照)及びこれに基づいた遺伝型を糾明した。例えば、表6に記載されたCYP1A2遺伝子のハプロタイプ2(CYP1A2*1L)は、CYP1A2遺伝子の塩基配列において−3860、−2467及び163塩基位置に単一塩基多型がある遺伝型をいうものとして、より具体的には−3860G>A、−2467T>delT(−2467delT)及び163C>Aの単一塩基多型を有することを示す。
【0057】
本発明の他の実施例では、韓国人で発見されたCYP1A2遺伝子の変異による17種のハプロタイプに対する最小マーカであるhtSNPを選別するために、CYP1A2遺伝子の変異を有している塩基配列を用いて分析されたハプロタイプに基づいてSNPタガーソフトウェアを用いて分析した。本発明で選別されたhtSNP組み合わせの例を図2乃至図6に示した。
【0058】
前記方法により本発明で選別されたhtSNP組み合わせは、ヒトCYP1A2遺伝子のハプロタイプを分析することに使用することができる。従って、本発明はヒトCYP1A2遺伝子のハプロタイプを決定する方法を提供する。前記方法は次の工程を含む:
【0059】
(a)被験者から生物学的試料を採取する工程;
(b)前記(a)工程の採取された試料から核酸を抽出する工程;
(c)前記(b)工程の核酸を鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び
(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G、−163C>A、1514G>A、2159G>A、2321G>C、3613T>C、5347C>T及び5521A>Gからなる群より選択されるCYP1A2遺伝子の変異の存在有無を調査する工程。
【0060】
前記(b)工程で核酸を抽出する方法は前記の通りである。
前記ヒトCYP1A2遺伝子の断片は、前記の通り、ヒトCYP1A2遺伝子の公知となった単一塩基多型を含む断片をいう。前記(c)工程で使用され得るプライマーは、これに制限されないが、配列番号2乃至配列番号31からなる群より選択される。
【0061】
前記(d)工程で調査される単一塩基多型は、図4乃至図6に示すhtSNPの中で選択することができ、図4に示す−3860G>A、−3598G>T、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型;図5に示す−3860G>A、−3113G>A、−2808A>C、−2603insA、−2467delT、−739T>G、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型;及び図6に示す−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型または−3860G>A、−3598G>T2321G>C、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査することもできる。
【0062】
このように前記(d)工程で調査される単一塩基多型は、韓国人で発見されるCYP1A2遺伝子変異に関するものであるため、韓国人CYP1A2遺伝子のハプロタイプと遺伝型の決定に非常に特異的である。
【0063】
前記(d)工程で前記PCR産物の塩基配列にCYP1A2遺伝子の単一塩基多型が存在するか否かの調査は、当業界で公知の多型性分析方法を用いて遂行することができる。好ましくはスナップショット分析([Peter M. Vallone,et al.,Int J Legal Med,2004,118:147-157]参照)、電気泳動分析またはこれらの組み合わせ、より好ましくはスナップショット分析を用いて遂行することができる。
【0064】
前記スナップショット分析はSNP位置の隣接部位にアニーリング(annealing)される配列(前記SNP部位は含まない)を有するプライマーとddNTPを用いたPCR反応を通じて遺伝子型を分析する方法である。本発明に使用されるスナップショット分析は、前記工程(c)で調査されるCYP1A2遺伝子のSNPに基づいて公知の方法により設計及び製作したものを使用することができ、SNP位置のすぐそばの塩基が3'末端となり、前記SNP位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたものであれば制限なしに使用されることができ、好ましくは配列番号64乃至74のプライマーからなる群より選択されるプライマーを使用することができる。この時、前記SNP位置の隣接部位にアニーリングされる配列は、約20bpの長さを有することが好ましく、同時に複数個のSNPを決定しようとする場合、各SNPに対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計、例えばT塩基を5個ずつ5'位置にさらに添加し、プライマー間にサイズ差異を作り合成してPCR産物の長さをそれぞれ異なるようにすることができる。このように作られたスナップショットプライマーに各SNPに相補的なddNTPが結合するようになり、これら合成物はSNPにより長さの差異が発生することによって同時に複数個のSNPの決定が可能である。
【0065】
その後、スナップショット分析で判定された遺伝型診断結果が正確であるかを検査するために、遺伝型が分かる他の遺伝子分析法を通じた結果との一致性を調査することができるが、ここで他の遺伝子分析法とは、特別に限定されないが、好ましくは自動塩基配列分析法またはパイロシーケンシング法であっても良い。
【0066】
本発明により韓国人で発見されたCYP1A2遺伝子の変異総17個の中で11個の単一塩基多型はプロモーター領域に位置する。前記11個の単一塩基多型には本発明で初めて糾明した−2603insA変異が含まれる。従って、本発明ではヒトCYP1A2プロモーター遺伝子の変異を分析する方法を提供する。前記方法は次の工程を含む:
【0067】
(a)被験者から生物学的試料を採取する工程;
(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;
(c)前記(b)工程のゲノムDNAを鋳型とし、ヒトCYP1A2遺伝子のプロモーター領域を増幅できるプライマーを用いてPCRを遂行する工程;及び
(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G及び163C>AからなるCYP1A2遺伝子の変異の存在有無を調査する工程。
【0068】
前記(b)工程で核酸を抽出する方法は前記の通りである。
前記(c)工程でヒトCYP1A2遺伝子のプロモーター領域を増幅できるプライマーは、配列番号1の−3860G>Aから−163C>Aまでの単一塩基多型を増幅できるプライマーであれば、制限なしに使用することができ、好ましくは配列番号62及び配列番号63のプライマーを使用することができる。
【0069】
前記(d)工程で前記PCR産物の塩基配列にCYP1A2遺伝子の単一塩基多型が存在するか否かの調査は、当業界に公知となった多型性分析方法を用いて遂行することができる。好ましくはスナップショット分析を用いて遂行することができる。本発明に使用されるスナップショット分析は、前記11個のCYP1A2遺伝子の単一塩基多型に基づいてデザインされたプライマーを用いて遂行することができる。本発明に使用されるスナップショットプライマーは、単一塩基多型を含まない隣接した配列の塩基配列が含まれるようにデザインされたものであれば、制限なしに使用することができる。好ましくは配列番号64乃至配列番号74からなる群より選択される塩基配列を有するプライマーを使用することができる。
【0070】
本発明の他の実施例では、CYP1A2酵素活性に影響を与えるプロモーター部分の11個の単一塩基多型を用いてCYP1A2プロモーター遺伝子の変異をSNaPshot分析により検出した。その結果、本発明の方法によりCYP1A2プロモーター遺伝子の変異を高速に正確に検出できることが確認された(図7乃至図14参照)。
【0071】
2.CYP2A6
本発明は韓国人CYP2A6遺伝子の変異分析を通じて韓国人で主に発見されるCYP2A6遺伝子の遺伝型を糾明し、これに基づいてそれぞれのハプロタイプの最適の標識セットであるhtSNPを選別し、その有用性を確認したという点に特徴がある。
本発明によるヒトCYP2A6遺伝子のhtSNPを選別する方法は次の工程を含む:
【0072】
(a)被験者から生物学的試料を採取する工程;
(b)前記工程(a)の採取された試料から核酸を抽出する工程;
(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;
(d)前記工程(c)で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;
(e)前記工程(d)で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプを分析する工程;及び
(f)前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype/)を用いて分析してhtSNPを選別する工程。
【0073】
前記工程(a)で採取された試料から核酸を抽出する方法は特に限定されず、当業界に公知となった技術または市販されている抽出用キットを使用することができる。例えば、DNAまたはRNA抽出用キットはQiagen(米国)及びStratagene(米国)などで購入できる。前記でRNAを抽出して使用する場合には逆転写によりcDNAを製造して使用する。
【0074】
前記工程(c)で前記ヒトCYP2A6遺伝子の断片は、ヒトCYP2A6遺伝子の公知の変異、例えば単一塩基多型(single nucleotide polymorphism;SNP)を含む断片をいう。ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーはヒトCYP2A6遺伝子またはその断片の塩基配列に基づいてデザインすることができ、例えば、配列番号76乃至89のプライマーからなる群より選択することができるが、これに制限されない。
【0075】
前記工程(d)で確認される変異は、SNP、遺伝子の欠失及び遺伝子の重複を含むが、これに制限されず、例えば、表15に示す30個の変異を含むことができる。
【0076】
また、前記工程(d)で変異の存在有無の確認は当業界に公知となった変異検出方法により遂行されることができ、好ましくは配列分析、電気泳動分析を用いて当業界に公知となっている野生型CYP2A6遺伝子の塩基配列、例えば、配列番号75(GenBank accession No.:NC_000019)の塩基配列、または当業界に公知のCYP2A6遺伝型の各塩基配列に基づいて比較することによって遂行したり、RFLP分析などを用いて野生型CYP2A6遺伝子の制限酵素切断様相と比較することによって遂行することができる。また、CYP2A6遺伝子の欠失または重複変異の場合にはPCR産物の電気泳動分析を通じて確認できる。前記配列分析は自動塩基配列分析器を使用して、またはパイロシーケンシング(pyrosequencing)を用いて遂行できる。
【0077】
前記工程(e)で変異が存在することが確認されたPCR産物の塩基配列中のハプロタイプは、SNPAlyzeやHaplotyper、Arlequinなどのプログラムを使用して分析できる。
【0078】
本発明の方法は、前記工程(a)乃至(e)を反復する工程を追加的に含むことができる。種族や患者などのようなある特定集団におけるCYP2A6遺伝子の変異様相を分析しようとする場合には、各CYP2A6遺伝型の頻度を調査して前記集団で多く発見されるCYP2A6遺伝型を選定した後、これを対象として以降の工程(f)を遂行できる。
【0079】
前記工程(f)では前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアで分析してhtSNPを選別し、htSNPの選別に用いられるソフトウェアとしてはSNPtagger以外にも、HapBlock、LDSelect、Haploview、htSNP、TagIT、tagSNPsなど多様なものが使用され得る。前記SNPtaggerソフトウェアは当業界に公知となっており、好ましくはhttp://www.well.ox.ac.uk/〜xiayi/haplotype/を用いることができる。このように選別されたhtSNPは、正確度を高めディプロタイプ(diplotype)を決定できることが検証された。前記検証はMatlab(The Math Works Inc., 米国)を用いて遂行することができる。
【0080】
本発明の一実施例では、韓国人のCYP2A6遺伝型に対するhtSNPを選別するために、まず韓国人で発見されるCYP2A6遺伝子の変異を調査した。その結果、韓国人のCYP2A6遺伝子で総30個のSNPを発見した(表15参照)。
【0081】
本発明の他の実施例では、前記で選別された30個のSNPのうち、機能性遺伝子変異を含むアミノ酸置換を招く変異8個及び頻度が高い変異6個を含む14個SNPに対してDYNACOM社のSNPAlyzeプログラムを用いてハプロタイプを分析し、総19個の1%以上頻度のハプロタイプを確認することができた。ハプロタイプの分析に用いられるプログラムは、前記SNPAlyzeプログラムに制限されず、Haplotyper(http://www.people.fas.harvard.edu/〜junliu/Haplo/docMain.htm)、Arlequin(http://lgb.unife.ch/arlequin)、イズテック社のSNP Analyzer(http://www.istech21.com/)など、多くの類似ソフトウェアが当業界に公知となっている
【0082】
本発明のさらに他の実施例では、韓国人で主に発見されるCYP2A6遺伝子の遺伝型を容易に決定できる最小マーカであるhtSNPを選別するために、前記19個のハプロタイプと遺伝子欠失の場合を含む20種類のハプロタイプに対する塩基配列及び頻度をSNPタガーソフトウェアを用いて分析してhtSNPを選別し、その例を図16乃至21に示した。
【0083】
前記方法により本発明で選別されたhtSNP組み合わせはヒトCYP2A6遺伝子の遺伝型を分析することに使用され得る。従って、本発明はヒトCYP2A6遺伝子の遺伝型を決定する方法を提供する。前記方法は次の工程を含む:
【0084】
(a)被験者から生物学的試料を採取する工程;
(b)前記工程(a)の採取された試料から核酸を抽出する工程;
(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び
(d)前記工程(c)で得られたPCR産物の塩基配列において、−48T>G、13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>T、及び6091C>Tからなる群より選択されるCYP2A6遺伝子変異の存在有無を調査する工程。
【0085】
前記工程(b)で核酸を抽出する方法は前記の通りである。
前記ヒトCYP2A6遺伝子の断片は、前記の通り、ヒトCYP2A6遺伝子の公知となった変異、例えばSNPを含んでいる断片をいう。前記工程(c)で用いられるプライマーは、これに制限されないが、配列番号90、91、120及び103のプライマーであっても良い。
【0086】
前記工程(d)で調査される変異は、図16乃至図21に示すhtSNPの中で選択され得る。例えば、前記工程(d)では図16に示す−48T>G;22C>T;567C>T;2134A>G;3391T>C;6458A>T;6558T>C;6582G>T;6600G>T;6091C>T、5971G>A及び5983T>Gの中の一つ;及び13G>A;51G>A;1620T>C;及び1836G>Tからなる変異の存在有無を調査でき、
【0087】
図17に示す−48T>G;22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6458A>T;6558T>C;6600G>T;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、
【0088】
図18に示す22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6354T>C;6458A>T;6558T>C;6600G>T;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、
【0089】
図19に示す−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>C;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、
【0090】
図20に示す−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6458A>T;6558T>C;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、
【0091】
図21に示す−48T>G;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>C;6600G>T;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、好ましくは図16に例示した変異の存在有無を調査できる。
【0092】
図16に例示した変異の中で機能性が立証されるか、機能性が最も有力に予想される変異は、アミノ酸を置換する変異または遺伝子の欠失を招く変異である。従って、図16の変異の中で機能性CYP2A6変異を検出するためには、最も好ましくは、図16の例示変異の中でアミノ酸置換変異や遺伝子欠失変異が調査される。遺伝子欠失の場合、単一塩基多型により判別し難いため、遺伝子欠失を標識できる変異を探さなければならないが、この過程で6091C>T変異を発見した。6091C>T変異は、前記工程(c)で増幅されるPCR産物の中でCYP2A6遺伝子が欠失された染色体で特異的に発見される単一塩基多型であり、遺伝子欠失に対する標識変異として用いることができる。このような機能性の判別目的により選定された変異組み合わせは、−48T>G;13G>A;567C>T;2134A>G;3391T>C;6458A>T;6558T>C;6582G>T;6600G>T;及び6091C>Tの10個の変異である。6091C>T変異の代わりに遺伝子欠失を標識する他の変異として、CYP2A6遺伝子基準に5971G>A及び5983T>Gを用いても良い。
【0093】
前記工程(d)で調査される変異は、韓国人で主に発見されるCYP2A6遺伝子変異に対するものであるので、韓国人のCYP2A6遺伝子のハプロタイプと遺伝型の決定に非常に特異的である。
【0094】
前記工程(d)で前記PCR産物の塩基配列にCYP2A6遺伝子の変異が存在するか否かの調査は、当業界に公知となった多型性分析方法を用いて遂行することができる。スナップショット分析([Peter M. Vallone,et al.,Int J Legal Med,2004,118:147-157]参照)、電気泳動分析またはこれらの組み合わせ、最も好ましくはスナップショット分析が変異を調査するために用いられる。
【0095】
前記スナップショット分析はSNP位置の隣接部位にアニーリング(annealing)される配列(前記SNP部位は含まない)を有するプライマーとddNTPを用いたPCR反応を通じて遺伝子型を分析する方法である。本発明に使用されるスナップショット分析は、前記工程(c)で調査されるCYP2A6遺伝子のSNPに基づいて公知となった方法により設計及び製作したものを使用することができ、SNP位置のすぐそばの塩基が3'末端となり、前記SNP位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたものであれば制限なしに使用されることができ、好ましくは配列番号97乃至102のプライマーからなる群より選択されるプライマーを使用することができる。この時、前記SNP位置の隣接部位にアニーリングされる配列は約20bpの長さを有することが好ましく、同時に複数個のSNPを決定しようとする場合、各SNPに対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計、例えばT塩基を5個ずつ5'位置にさらに添加してプライマー間にサイズ差異を作って合成し、PCR産物の長さをそれぞれ異なるようにすることができる。このように作られたスナップショットプライマーに各SNPに相補的なddNTPが結合するようになり、これら合成物はSNPにより長さの差異が発生することによって同時に複数個のSNPの決定が可能である。
【0096】
その後、スナップショット分析のために増幅されたPCR産物の塩基配列を当業界に公知となった配列分析法で分析でき、特別に限定されないが、好ましくは自動塩基配列分析法であっても良い。
【0097】
例えば、前記工程(c)で図16に示すhtSNP組み合わせを調査する場合には、配列番号92乃至配列番号101からなる群より選択される塩基配列を有するプライマーを使用することができ、好ましくは配列番号92乃至配列番号101のプライマーを全て使用することができるが、これに限定されない。
【0098】
その後、スナップショット分析で増幅されたPCR産物の塩基配列を当業界に公知となった配列分析法で分析でき、特別に限定されないが、好ましくは自動塩基配列分析法であっても良い。
【0099】
本発明のさらに他の実施例では、本発明で選別されたhtSNP組み合わせの有用性を確認した。このために図16に示すhtSNP組み合わせの中で10個の機能性または機能性予想CYP2A6変異を選定してスナップショット分析を実施するために、工程(c)で得られたPCR産物の塩基配列を分析した。その結果、本発明の方法が韓国人で発見されるCYP2A6遺伝型を同時に高速に分析できることを確認した(図22乃至図32参照)。
【0100】
本発明の方法により分析されることができるCYP2A6遺伝子の遺伝型は、−48T>G、13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>T、及び6091C>Tを含み、各遺伝型及びこれによる変異について図22乃至32に示した。例えば、図22を参照して説明すれば、−48T>G、6558T>C、2134A>Gの3個の位置で変異を有しており、残りの7個の位置では野生型を示す遺伝型である。図23は567C>T位置だけで変異を有しており、残りの9個の位置では野生型を有する遺伝型である。また、CYP2A6*4遺伝型は2A6欠失変異を含む遺伝型であり、CYP2A6遺伝子がヒト染色体上で欠失されて酵素生産が全く生じない。CYP2A6遺伝子が欠失された場合、遺伝子の形態はCYP2A6遺伝子の一部とCYP2A7の一部が互いに連結された形態をしているので、この部分を調査して欠失に特異的な変異を探すことができる。
【0101】
3.CYP2D6
本発明は韓国人CYP2D6遺伝子の変異分析を通じて韓国人で主に発見されるCYP2D6遺伝子の遺伝型を糾明し、これに基づいてそれぞれのハプロタイプの最適の標識セットであるhtSNPを選別し、その有用性を確認したという点に特徴がある。
【0102】
本発明によるヒトCYP2D6遺伝子のhtSNPを選別する方法は次の工程を含む:
(a)ヒトから生物学的試料を採取する工程;
(b)前記(a)工程の採取された試料から核酸を抽出する工程;
(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;
(d)前記(c)工程で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;
(e)前記(d)工程で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプ(haplotype)を分析する工程;及び
(f)前記(e)工程で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアを用いて分析してhtSNPを選別する工程。
【0103】
前記(a)工程で採取された試料から核酸を抽出する方法は、特に限定されず、当業界に公知となった技術または市販されている抽出用キットを使用することができる。例えば、DNAまたはRNA抽出用キットは、Qiagen(米国)及びStratagene(米国)で購入できる。前記でRNAを抽出して使用する場合には逆転写によりcDNAを製造して使用する。
【0104】
前記(c)工程で前記ヒトCYP2D6遺伝子の断片は、ヒトCYP2D6遺伝子の公知となった変異、例えば単一塩基多型(single nucleotide polymorphism ;SNP)を含んでいる断片をいう。前記ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーはヒトCYP2D6遺伝子またはその断片の塩基配列に基づいてデザインすることができ、例えば、配列番号106、配列番号107、配列番号121乃至配列番号127、配列番号129乃至配列番号136、配列番号138、配列番号139、配列番号149及び配列番号150からなる群より選択される塩基配列を有するものであっても良いが、これに制限されない。
【0105】
前記(d)工程で確認される変異は、単一塩基多型、遺伝子の欠失及び遺伝子の重複を含むが、これに制限されず、例えば、表34に示す33個の変異を含むことができる。
【0106】
また、前記(d)工程で変異の存在有無の確認は、当業界に公知となった変異検出方法により遂行することができ、好ましくは配列分析、電気泳動分析、RFLP分析などを用いて遂行することができる。前記配列分析は自動塩基配列分析器を使用するかパイロシーケンシング(pyrosequencing)を用いて遂行することができる。前記パイロシーケンシングはDNAシーケンシングに利用されることもある公知のSNP分析方法によりDNAが重合される間に放出されるPPi(inorganic pyrophosphate)の光の発現を検出する方法である。
【0107】
変異の存在有無は野生型CYP2D6遺伝子の塩基配列と比較して確認できる。野生型CYP2D6遺伝子の塩基配列は当業界に公知となっている。例えば、配列番号105(GenBank accession No. AY545216)の塩基配列、または当業界に公知となったCYP2D6遺伝型の各塩基配列または情報に基づいて比較して確認できる(GenBank accession No. M33388,http://www.cypalleles.ki.se/cyp2d6.htm)。または、RFLPを遂行して野生型CYP2D6遺伝子の制限酵素切断様相と比較して確認することもできる。CYP2D6遺伝子の欠失または重複変異の場合にはPCR産物の電気泳動分析を通じて確認できる。
【0108】
前記(d)工程で変異が存在することが確認されたPCR産物の塩基配列中のハプロタイプの分析は全長塩基配列を通じて遂行できる。
【0109】
本発明の方法は前記(a)乃至(e)工程を反復する工程を追加的に含むことができる。種族や患者などのようなある特定集団でCYP2D6遺伝子の変異様相を分析しようとする場合には、各CYP2D6遺伝型の頻度を調査して前記集団で多く発見されるCYP2D6遺伝型を選定した後、これを対象として以下の(f)工程を遂行できる。
【0110】
前記(f)工程では、前記(e)工程で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアで分析してhtSNPを選別する。前記SNPタガーソフトウェアは当業界に公知となっており、例えば Genehunter、Merlin、Allegro、SNPHAP、htSNP finder(PCA based)などが挙げられ、好ましくは、http://www.well.ox.ac.uk/〜xiayi/haplotypeまたは http://slack.ser.man.ac.uk/progs/htsnp.htmlのウェブサイトを利用することができる。
【0111】
このように選別されたhtSNPはディプロタイプ(diplotype)決定時に正確度を高めるために検証され得る。人体の遺伝型は二本の染色体により決定されるので、遺伝型を判読すれば二つのハプロタイプ組み合わせで判定される。しかしながら、複数個のSNPを同時に分析した場合、特定のハプロタイプの組み合わせが他のハプロタイプの組み合わせと同一の変異分析結果を示す可能性がある。従って、本発明により開発された診断法を用いて遺伝型が判読された場合、正確な遺伝型を決定できることが検証されなければならない。このような検証は、Matlab(The Math Works Inc., 米国)プログラムを用いた遺伝子分析結果から正確な遺伝型判読が可能であるか否かを分析することによって遂行される。
【0112】
本発明の一実施例では韓国人で発見されるCYP2D6遺伝型に対するhtSNPを選別するために、まず韓国人で発見されるCYP2D6遺伝子の変異を調査した。その結果、韓国人で主に発見される33個の変異とこれによる12種のハプロタイプ(遺伝型)を糾明した(表34及び35参照)。
【0113】
本発明の他の実施例では韓国人で主に発見されるCYP2D6遺伝型を容易に決定できる最小マーカであるhtSNPを選別するために、12種のCYP2D6遺伝型の塩基配列をSNPtaggerソフトウェアを用いて分析した。本発明で選別されたhtSNP組み合わせの例を図34乃至図39に示した。
【0114】
前記方法により本発明で選別されたhtSNP組み合わせは、ヒトCYP2D6遺伝子の遺伝型を分析することに使用され得る。従って、本発明はヒトCYP2D6遺伝子の遺伝型を決定する方法を提供する。前記方法は次の工程を含む:
【0115】
(a)ヒトから生物学的試料を採取する工程;
(b)前記(a)工程の採取された試料から核酸を抽出する工程;
(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び
(d)前記(c)工程で得られたPCR産物の塩基配列において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複からなる群より選択される一つを含む少なくとも11個のCYP2D6遺伝子変異の存在有無を調査する工程。
【0116】
前記(b)工程で核酸を抽出する方法は前記の通りである。
前記ヒトCYP2D6遺伝子の断片は、前記の通り、ヒトCYP2D6遺伝子の公知となった変異、例えば単一塩基多型を含んでいる断片をいう。前記(c)工程で使用され得るプライマーは、これに制限されないが、配列番号106、配列番号107、配列番号121乃至配列番号127、配列番号129乃至配列番号136、配列番号138、配列番号139、配列番号149及び配列番号150からなる群より選択される塩基配列を有するものであっても良い。
【0117】
前記(d)工程で調査される変異は、図34乃至図39に示すhtSNPの中で選択され得る。前記(d)工程では、図34に示す−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複からなる一つを含む変異の存在有無を調査できる。
【0118】
また、図35に示す−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>C、3877G>A、4388C>T及び4401C>Tからなる群より一つ;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査することもできる。
【0119】
また、図36に示す−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1584C>G;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複から選択される一つを含む変異の存在有無を調査することもできる。
【0120】
また、図37に示す−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;4125〜4133insGTGCCCACT;−1235A>G;1887insTA;2D6欠失;及び2D6重複を含む変異の存在有無を調査することもできる。
【0121】
また、図38に示す−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;1611T>A;1661G>C及び4180G>Cからなる群より一つ;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;−1235A>G;1887insTA;2D6欠失;及び2D6重複から選択される一つを含む変異の存在有無を前記(d)工程で調査することもできる。
【0122】
また、図39に示す−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;1887insTA;2988G>A;4125−4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査することもできる。
【0123】
好ましくは図34に例示した変異の存在有無を調査できる。このように前記(d)工程で調査される変異は、韓国人で主に発見されるCYP2D6遺伝子変異に関するものであるので、韓国人のCYP2D6遺伝子のハプロタイプと遺伝型の決定に非常に特異的である。
【0124】
前記(d)工程で前記PCR産物の塩基配列にCYP2D6遺伝子の変異が存在するか否かの調査は、当業界に公知となった多型性分析方法を用いて遂行できる。好ましくはスナップショット分析([Peter M. Vallone,et al.,Int J Legal Med,2004,118:147-157]参照)、電気泳動分析またはこれらの組み合わせを用いて遂行できる。前記CYP2D6遺伝子の変異が単一塩基多型である場合にはスナップショット分析を利用することができる。
【0125】
前記スナップショット分析は、SNP位置の隣接部位にアニーリング(annealing)される配列(前記SNP部位は含まない)を有するプライマーとddNTPを用いたPCR反応を通じて遺伝子型を分析する方法である。本発明に使用されるスナップショット分析は、前記工程(c)で調査されるCYP2D6遺伝子のSNPに基づいて公知の方法により設計及び製作したものを使用することができ、SNP位置のすぐそばの塩基が3'末端となり、前記SNP位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたものであれば制限なしに使用することができる。この時、前記SNP位置の隣接部位にアニーリングされる配列は約20bpの長さを有することが好ましく、同時に複数個のSNPを決定しようとする場合、各SNPに対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計、例えばT塩基を5個ずつ5'位置にさらに添加してプライマー間のサイズに差を作って合成し、PCR産物の長さをそれぞれ異なるようにすることができる。このように作られたスナップショットプライマーに各SNPに相補的なddNTPが結合するようになり、これら合成物はSNPにより長さの差異が発生することによって同時に複数個のSNPの決定が可能である。
【0126】
例えば、前記(c)工程で図34に示すhtSNP組み合わせを調査する場合には、配列番号141乃至配列番号148、配列番号152及び配列番号153からなる群より選択される塩基配列を有するプライマーを使用することができ、好ましくは配列番号141乃至配列番号148、配列番号152及び配列番号153のプライマーを全て使用することができる。その後、スナップショット分析で増幅されたPCR産物の塩基配列を公知となった配列分析法で分析できる。前記配列分析法としては当業界に公知となった方法であれば制限なしに使用されることができ、好ましくは自動塩基配列分析法であっても良い。
【0127】
本発明のさらに他の実施例では、本発明で選別されたhtSNP組み合わせの有用性を確認した。このために図34に示すhtSNP組み合わせを用いてスナップショット分析を実施した後、得られたPCR産物の塩基配列を分析した。その結果、本発明の方法が韓国人で発見されるCYP2D6遺伝型を同時に高速で分析できることを確認した(図40及び41参照)。
【0128】
本発明の方法により分析することができるCYP2D6遺伝子の遺伝型は、CYP2D6*1A、CYP2D6*2A、CYP2D6*5、CYP2D6*2N、CYP2D6*10B、CYP2D6*14B、CYP2D6*18、CYP2D6*21B、CYP2D6*41A、CYP2D6*49、CYP2D6*52及びCYP2D6*60を含む。各遺伝型及びこれによる変異を表34に示した。表34を参照して説明すれば、例えばCYP2D6*1A遺伝型は野生型であり、CYP2D6*2A遺伝型は野生型CYP2D6遺伝子の塩基配列でSNP1、SNP5、SNP8、SNP9、SNP12−SNP18、SNP21、SNP25及びSNP28位置に変異を含む遺伝型である。また、CYP2D6*5遺伝型は2D6欠失変異を含む遺伝型として、CYP2D6遺伝子がヒト染色体上で完全欠如したものであって酵素生産が全く生じない。CYP2D6*2N遺伝型は2D6重複変異を含む遺伝型として、CYP2D6遺伝子が2個以上同一染色体に存在する。
【0129】
また、本発明は遺伝子分析チップを用いてヒトCYP2D6遺伝子の遺伝型を決定する方法を提供する。前記方法は次の工程を含む:
【0130】
(a)検査しようとする遺伝子を抽出した後、多重(multiplex)PCRを遂行して同定しようとするSNP周辺を含むPCR産物を得る工程;
(b)各対立遺伝子(allele)の特異的な塩基を同定できるASPE(allele specific primer extension)プライマーを用いてASPE反応を遂行する工程;
(c)前記反応産物を遺伝子チップに混成化させる工程;及び
(d)前記チップを分析する工程。
また、本発明はSNP検査用ジップ・コード(ZiP Code)オリゴ塩基チップを含む遺伝型分析用キットを提供する(図42参照)。
【0131】
前記工程(b)で、ASPE反応のためにはそれぞれのSNPに合う一対のプライマーを製作するようになるが、前記ASPEプライマーは多型性を示す塩基(SNP site)を3’末端に含んで対立遺伝子(allele)と特異的に結合するシークエンスで製作され、5’方向には24bpのオリゴヌクレオチドであるZiP Codeが含まれており、前記ZiP Codeはそれぞれの対立遺伝子ごとに異なる配列を有するように製作される。
【0132】
本発明では、論文などを通じて公開された配列及び生命情報学技法を通じて選別され設計された配列のうち、実験的検証を通じて他の試料と相互交差反応が起こらない最適のZiP Code配列を選別し、選別された配列はTM値が61℃±2であり、ZiP Codeの相互間に干渉がないように製作され、ヘアピン二次構造のΔG値が−2以上である配列だけが選択された。
【0133】
前記のように構成されたASPEプライマーを用いてASPE反応を遂行すれば、プライマーの3’末端に該当する対立遺伝子を有している試料だけがプライマーと反応して対立遺伝子特異的延長反応が生じるようになる。この時、シアニン5(Cyanine 5、CY5)蛍光物質が共有結合されたdUTP(Cy5-dUTP)を混合して延長反応を遂行すれば、各対立遺伝子を有している試料だけがCy5蛍光物質を標識するようになる(図45参照)。この時、蛍光物質はCy5に制限されず、Cy3、TAMRA、Texas−Red、Cy3.5、Rhodamin 6G、SyBR Greenなど、当該分野で使用可能な種々のものが代替して使用され得る。
【0134】
本発明の分析チップ上には、前記ZiP Codeと相補的に結合するオリゴヌクレオチドをプローブ(cZip Code)が植えられており、前記のようにZiP Codeプライマーを用いて延長された試料に含まれている各対立遺伝子を同定できるようになる(図43参照)。
【0135】
前記プローブは3’方向に10bpの塩基配列をスペーサで挿入してターゲットと交雑反応がよく生じるように誘導し、例えば、前記スペーサ配列は5’−CAG GCCA AGT−3’であることが好ましい。
【0136】
また、本発明において、前記プローブは配列番号158乃至配列番号184の塩基配列を有することが好ましい。
【0137】
前記工程(c)及び(d)で前記反応産物を遺伝子チップに混成化させ、混成化されたチップを分析する方法は、当業界に公知となった方法により遂行されることができ、一般的なDNAチップスキャナーは如何なるものでも使用することができる。より好ましくは、Axon社のGenePix 4100Bスキャナーを用い、スキャニングされたイメージをGenePix Pro 6.0 softwareを用いて分析できる。
【0138】
本発明の遺伝子分析チップを用いてCYP2D6遺伝子の変異を分析すれば、配列分析を通じて確認された結果と同一の結果を得ることができる。従って、本発明の遺伝子分析チップによれば多様な遺伝子の変異を経済的に容易に分析することができる。
【0139】
4.PXR
本発明の方法は韓国人のPXR遺伝子の変異に基づいて選別された、htSNPを用いてPXR遺伝子の機能的変異型を分析することを特徴とする。
本発明によるヒトPXR遺伝子のhtSNPを選別する方法は次の工程を含む:
【0140】
(a)ヒトから生物学的試料を採取する工程;
(b)前記工程(a)の採取された試料から核酸を抽出する工程;
(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;
(d)前記工程(c)で得られたPCR産物の塩基配列を分析して変異の存在有無を確認する工程;
(e)前記工程(d)で変異が存在することが確認されたPCR産物の塩基配列中のハプロタイプを分析する工程;及び
(f)前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアを用いて分析してhtSNPを選別する工程。
【0141】
前記工程(a)で採取された試料から核酸を抽出する方法は、特別に限定されず、当業界に公知となった技術または市販されている抽出用キットを使用することができる。例えば、DNAまたはRNA抽出用キットは、Qiagen(米国)及びStratagene(米国)などで購入できる。前記でRNAを抽出して使用する場合には逆転写によりcDNAを製造して使用する。
【0142】
前記工程(c)で前記ヒトPXR遺伝子の断片は、ヒトPXR遺伝子の公知となった変異、例えば単一塩基多型(single nucleotide polymorphism;SNP)を含んでいる断片をいう。前記ヒトPXR遺伝子またはその断片を増幅できるプライマーは、ヒトPXR遺伝子またはその断片の塩基配列に基づいてデザインすることができ、例えば、配列番号221乃至240のプライマーからなる群より選択することができるが、これに制限されない。
【0143】
前記工程(d)で確認される変異は、単一塩基多型、遺伝子の欠失及び遺伝子の重複を含むが、これに制限されず、例えば、表48に示す22個の変異を含むことができる。
【0144】
また、前記工程(d)で変異の存在有無の確認は、当業界に公知となった変異検出方法により遂行でき、好ましくは配列分析、電気泳動分析、RFLP分析などを用いて遂行できる。前記配列分析は自動塩基配列分析器を使用するかパイロシーケンシング(pyrosequencing)を用いて遂行できる。
【0145】
また、前記工程(d)で変異の存在有無は、当業界に公知となっている野生型PXR遺伝子の塩基配列、例えば、配列番号2200(GenBank accession No.:NT_005612)の塩基配列、または当業界に公知となったPXR遺伝型の各塩基配列に基づいて比較して確認したり、RFLPを遂行して野生型PXR遺伝子の制限酵素切断様相と比較して確認することもできる。PXR遺伝子の欠失または重複変異の場合にはPCR産物の電気泳動分析を通じて確認できる。
【0146】
前記工程(d)で変異が存在することが確認されたPCR産物の塩基配列でハプロタイプの頻度及び種類を予測することは、当業界に公知となった技術プログラムまたは市販されるプログラムを使用して分析できる。例えば、Haploviewプログラムは無料で配布されるプログラムとして利用でき、SNPAlyzeのような商用化されたプログラムを使用することもできる。前記 Haploviewソフトウェアは当業界に公知となっており、好ましくはインターネットウェブサイトである http://www.broad.mit.edu/mpg/haploviewを利用することができる。
本発明の方法は前記工程(a)乃至(e)を反復する工程を追加的に含むことができる。種族や患者などのようなある特定集団におけるPXR遺伝子の変異様相及びこれに対するハプロタイプを分析しようとする場合には、各PXR遺伝型の頻度を調査して前記集団で多く発見されるPXR遺伝型を選定した後、これを対象として工程(f)を遂行できる。
【0147】
前記工程(f)では前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアで分析してhtSNPを選別する。前記SNPタガーソフトウェアは当業界に公知となっており、例えば Genehunter、Merlin、Allegro、SNPHAP、htSNP finder(PCA based)などが挙げられ、好ましくは、http://www.well.ox.ac.uk/〜xiayi/haplotypeまたは http://slack.ser.man.ac.uk/progs/htsnp.htmlのウェブサイトを利用することができる。
【0148】
このように選別されたhtSNPはディプロタイプ(diplotyp)決定時に正確度を高めるために検証することができる。人体の遺伝型は二本の染色体により決定されるので、遺伝型は二つのハプロタイプ組み合わせで判読される。しかしながら、複数個のSNPを同時に分析した場合、特定のハプロタイプの組み合わせが他のハプロタイプの組み合わせと同一の変異分析結果を示す可能性がある。従って、本発明により開発された診断法を用いて遺伝型が判読られた場合、正確な遺伝型を決定できることが検証されなければならない。このような検証は、Matlab(The Math Works Inc., 米国)プログラムを用いた遺伝子分析結果から遺伝型が判読される分析によって遂行される。
【0149】
本発明の一実施例では、韓国人のPXR遺伝子の機能的変異型に対するhtSNPを選別するために、まず韓国人でPXR遺伝子の変異を調査した。その結果、韓国人のPXR遺伝子で総22個のSNPを発見した(表48参照)。
本発明の他の実施例では、前記で選別された22個のSNPの中で6個の機能的変異によるハプロタイプをDYNACOM社のSNPAlyzeプログラムを用いて分析し、総14個のハプロタイプを確認した(表49参照)。
【0150】
本発明のさらに他の実施例では、韓国人で発見されたPXR遺伝子の機能的変異型を容易に決定できる最小マーカであるhtSNPを選別するために、前記14個のハプロタイプの塩基配列をSNPタガーソフトウェアを用いて分析してhtSNPを選別した(図47参照)。
【0151】
前記方法で本発明で選別されたhtSNP組み合わせは、ヒトPXR遺伝子の機能的変異型を分析することに使用することができる。従って、本発明はヒトPXR遺伝子の機能的変異型を決定する方法を提供する。前記方法は次の工程を含む:
【0152】
(a)ヒトから生物学的試料を採取する工程;
(b)前記工程(a)の採取された試料から核酸を抽出する工程;
(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び
(d)前記工程(c)で得られたPCR産物の塩基配列において、−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cからなる群より選択されたPXR遺伝子の機能的変異の存在有無を調査する工程。
【0153】
前記工程(b)で核酸を抽出する方法は前記の通りである。
前記ヒトPXR遺伝子の断片は、前記の通り、ヒトPXR遺伝子の公知となった変異、例えばSNPを含む断片をいう。前記工程(c)で使用されるプライマーは、これに制限されないが、配列番号242乃至247のプライマーからなる群より選択され得る。
【0154】
このように前記工程(d)で調査されるSNPは韓国人で発見されるPXRの機能的変異遺伝子に関するものであり、韓国人PXR遺伝子の機能的変異のハプロタイプ及と機能的変異型の決定に非常に特異的である。
【0155】
前記工程(d)で前記PCR産物の塩基配列にPXR遺伝子の変異が存在するか否かの調査は、当業界に公知となった多型性分析方法を用いて遂行できる。好ましくはスナップショット分析([Peter M. Vallone,et al.,Int J Legal Med,2004,118:147-157]参照)、電気泳動分析またはこれらの組み合わせ、より好ましくはスナップショット分析を用いて遂行できる。
【0156】
前記スナップショット分析は、SNP位置の隣接部位にアニーリング(annealing)される配列(前記SNP部位は含まない)を有するプライマーとddNTPを用いたPCR反応を通じて遺伝子型を分析する方法である。本発明に使用されるスナップショット分析は、前記工程(c)で調査されるPXR遺伝子のSNPに基づいて公知の方法により設計及び製作したものを使用することができ、SNP位置のすぐそばの塩基が3'末端となり、前記SNP位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたものであれば制限なしに使用することができ、好ましくは配列番号242乃至2457のプライマーからなる群より選択されるプライマーを使用することができる。この時、前記SNP位置の隣接部位にアニーリングされる配列は約20bpの長さを有することが好ましく、同時に複数個のSNPを決定しようとする場合、各SNPに対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計、例えばT塩基を5個ずつ5'位置にさらに添加してプライマー間にサイズ差異を作って合成し、PCR産物の長さをそれぞれ異なるようにすることができる。このように作られたスナップショットプライマーに各SNPに相補的なddNTPが結合するようになり、これら合成物はSNPにより長さの差異が発生することによって同時に複数個のSNPの決定が可能である。
【0157】
その後、スナップショット分析で判定された遺伝型診断結果が正確であることを検査するために、遺伝型が分かる他の遺伝子分析法を通じた結果との一致性を調査することができる。ここで他の遺伝子分析法は、特に限定されないが、好ましくは自動塩基配列分析法またはパイロシーケンシング法であっても良い。
【0158】
本発明の他の実施例では、本発明で選別されたhtSNP組み合わせの有用性を確認した。このために図47に示すhtSNP組み合わせを用いてスナップショット分析を実施した後、得られたPCR産物の塩基配列を分析した。その結果、本発明の方法が韓国人で発見されるPXR遺伝子の機能的変異型を同時に高速に分析できることを確認した(図48乃至図50参照)。
本発明の方法により分析され得るPXR遺伝子の機能的変異型は−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cを含む。
【0159】
5.UGT1A
本発明によるヒトUGT1A族遺伝子群の機能的変異型を決定する方法は次の工程を含む:
(a)ヒトから生物学的試料を採取する工程;
(b)前記工程(a)で採取された試料から核酸を抽出する工程;
(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族個別遺伝子を増幅する工程;及び
(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での−39(TA)6>(TA)7、211G>A、233C>T及び686C>A;UGT1A3での31T>C、133C>T及び140T>C;UGT1A4での31C>T、142T>G及び292C>T;UGT1A6での19T>G、541A>G及び552A>C;UGT1A7での387T>G、391C>A、392G<A、622T>C及び701T>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子群の機能的変異型の存在有無を確認する工程。
【0160】
また、本発明によるイリノテカン(Irinotecan)に対する感受性と関連したUGT1A族遺伝子の多型性を決定する方法は次の工程を含む:
(a)ヒトから生物学的試料を採取する工程;
(b)前記工程(a)で採取された試料から核酸を抽出する工程;
(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族遺伝子を増幅する工程;及び
(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での211G>A、233C>T及び686C>A;UGT1A6での19T>G、541A>G及び552A>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子変異型の存在有無を確認する工程。
【0161】
本発明の方法は、韓国人で主に発見されるUGT1A族遺伝子群の多型性に基づいて選別された、最適のUGT1A族遺伝子群の機能的変異型または薬物感受性を決定する多型性標識セットを用いることを特徴とするため、既存の方法と比較して韓国人を対象として時間及び費用の面で効率的に分析できる効果を奏する。
【0162】
本発明の工程(a)では、ヒト、好ましくは韓国人、中国人及び日本人などのアジア人、さらに好ましくは韓国人から生物学的試料を採取する。前記生物学的試料は、血液、皮膚細胞、粘膜細胞または毛髪などであり、好ましくは血液であっても良い。
【0163】
本発明の工程(b)では、前記工程(a)で採取された生物学的試料から核酸を抽出する。前記核酸はDNAまたはRNAなどであっても良く、好ましくはDNA、より好ましくはゲノム(genomic)DNAであっても良い。また、採取された試料から核酸を抽出する工程は特に限定されないが、当業界に公知となった技術により遂行したり、市販されている抽出用キット、例えばQiagen社(米国)またはStratagene社(米国)で市販されているDNAまたはRNA抽出用キットを使用して遂行できる。
【0164】
本発明の工程(c)では、前記工程(b)で抽出された核酸を鋳型とし、ヒトUGT1A族の各遺伝子を増幅できるプライマーを使用してUGT1A族遺伝子が増幅される。この時、前記工程(b)で抽出された核酸がRNAである場合には、逆転写を用いてcDNAに転換してこれを鋳型として使用する。前記各プライマーはヒトUGT1A族遺伝子またはその断片の塩基配列に基づいて公知の方法により設計し製作される。
【0165】
本発明の工程(c)で、UGT1A族遺伝子群の機能的変異型を決める方法の場合には、UGT1A1、UGT1A3、UGT1A4、UGT1A6、UGT1A7及びUGT1A9族の各遺伝子を増幅することが好ましく、イリノテカン(Irinotecan)に対する感受性を決定するUGT1A族遺伝子群の多型性を決定する方法の場合には、UGT1A1、UGT1A6及びUGT1A9族の各遺伝子を増幅することが好ましい。
【0166】
本発明の工程(d)では、前記工程(c)で増幅された各UGT1A族遺伝子を使用してUGT1A族遺伝子群の機能的変異型または薬物感受性と関連した多型を分析する。この時、当該分野で公知の多型性分析法を用いて機能的変異型または多型が分析される。例えば、スナップショット(SNaPshot)分析、電気泳動分析、パイロシーケンシング(pyrosequencing)またはこれらの組み合わせが行われる。
【0167】
具体的には、分析するUGT1A遺伝子の変異型が単一塩基多型である場合には、スナップショット分析を遂行することが好ましい。、この時のスナップショット分析では単一塩基多型(SNP)位置の隣接部位にアニーリング(annealing)されるプライマー及びddNTPを用いてPCR反応を遂行する。この時、スナップショット分析で使用されるプライマーは、UGT1A族遺伝子の単一塩基多型に基づいて公知の方法により設計及び製作したものである。例えば、単一塩基多型位置のすぐそばの塩基が3'末端となり、隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されるように設計及び製作される。この時、アニーリングされる配列は約20bpの長さを有することが好ましく、同時に複数個の単一塩基多型を決定しようとする場合は、各単一塩基多型に対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計し、PCR産物の長さをそれぞれ異なるようにする。
【0168】
本発明の一実施様態によれば、UGT1A族遺伝子群の機能的変異型を確認するスナップショット分析では、配列番号:2905乃至314の配列を有するプライマーを使用することができ、UGT1A族遺伝子群のイリノテカン感受性関連多型性を確認するスナップショット分析には配列番号:315乃至322の配列を有するプライマーを使用することができる。
【0169】
一方、スナップショット分析で増幅されたPCR産物の塩基配列を公知の配列分析法で分析することができる。好ましくは自動塩基配列分析法により分析することができるがこれに限定されない。
【0170】
また、分析するUGT1A遺伝子の変異型が単一塩基多型ではない場合(例えば、UGT1A1での−39(TA)6>(TA)7型)にはスナップショット分析の代わりに公知のパイロシーケンシング(pyrosequencing)分析を遂行でき、この時のパイロシーケンシングは、公知の通り、DNAが重合される間に放出されるPPi(無機ピロリン酸塩)の発光程度を測定して分析を遂行できる。本発明の一実施様態によれば、UGT1A1族遺伝子の−39(TA)6>(TA)7型を確認するパイロシーケンシング分析には、配列番号:292乃至294の配列を有するプライマーを使用することができる。
【実施例】
【0171】
以下、本発明を実施例により詳しく説明する。但し、下記の実施例は本発明を例示するものに過ぎず、本発明の内容が下記の実施例に限定されるものではない。
【0172】
<CYP1A2>
実施例1:韓国人におけるCYP1A2遺伝子の遺伝型分析
<1−1>CYP1A2遺伝子の増幅
48名の健康な被験者から血液を分離した後、Qiagen社のゲノムDNA分離キットを使用してDNAをそれぞれ分離した。CYP1A2遺伝子は7個のエクソンを含み総遺伝子の長さが約11kbである。従って、CYP1A2遺伝子を15個の断片に分けてPCRを遂行した。各PCRに使用したプライマーは下記の表1の通りである。本明細書に記載された塩基配列でA、T、G及びCはそれぞれアデニン、チミン、グアニン及びシトシンを意味する。
【0173】
【表1】

【0174】
各プライマーの位置とPCR産物の大きさは下記の表2の通りである。また、ヌクレオチドの位置はCytochrome P450(CYP) Allele Nomenclature Committee(http://www.cypalleles.ki.se/cyp1a2.htm)の命名法により記載した。
【0175】
【表2】

【0176】
各PCR断片に対する反応条件は下記の表3の通りである。
【0177】
【表3】

【0178】
<1−2>PCR産物の塩基配列分析
前記実施例<1−1>で得られた各PCR産物の塩基配列を自動配列分析器を用いて分析した。この時に使用したプライマーは下記の表4の通りである。
【0179】
【表4】

【0180】
前記実施例<1−1>で増幅されたCYP1A2遺伝子の全体塩基配列を自動塩基配列分析器を用いて分析した。その後、野生型CYP1A2遺伝子の塩基配列(配列番号1)と比較した結果、総17個の単一塩基多型を発見し、その結果を下記の表5に示した。その中で単一塩基多型−2603insAは新規なものであることを確認した。
【0181】
【表5】

【0182】
その後、本発明者等は前記新規な1個の単一塩基多型がCYP1A2の一本に位置するか否かと、同一なDNA本に他の遺伝子変異はないか、染色体の他の部分に位置した類似遺伝子から起因したものではないかどうかを調査するために、前記単一塩基多型が発見された変異遺伝子を含む被験者のDNAを鋳型とし、前記配列番号38及び39のプライマーを使用してPCRを遂行し、増幅された産物の配列を前記と同様な方法で分析した。
その結果、前記1個の単一塩基多型は全てプロモーター部位−2603insAに位置した。この変異型の単一塩基多型は二本のDNAのうち、一本は変異型、他の一本は野生型を有していることが示された(図1)。
【0183】
実施例2:CYP1A2変異型のハプロタイプ分析
前記実施例1で糾明された17個のCYP1A2遺伝子の変異は、その組み合わせによりCYP1A2の酵素活性に影響を与える可能性があり、幾つかのハプロタイプに対する酵素活性変異は既に報告されている。従って、本発明者等は実施例1で確認した変異によるハプロタイプをDYNACOM社のSNPAlyzeプログラムを使用して分析した。その結果、下記の表6に示した通り、他種族で発見されない韓国型の新たなハプロタイプが確認された。
【0184】
【表6】

□:各単一塩基多型の変異
【0185】
実施例3:htSNPの選別及び検証
CYP1A2遺伝子の単一塩基多型の組み合わせであるハプロタイプがCYP1A2の酵素活性に影響を与える可能性が多くのハプロタイプで報告されたことがある。このように作られた詳細なハプロタイプの情報は最小マーカで確認することができる。これら最小マーカをhtSNPといい、前記htSNPはそれぞれのハプロタイプの正確な表示のために必要なマーカであり、色々な組み合わせを構成するようになる。この最適化された標識セットであるhtSNP組み合わせをSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype)を用いて選別した。選別されたhtSNP組み合わせの例を図2乃至図6に示し、各選別されたhtSNP組み合わせは最適の標識セットのうちの一つとして、「1」は野生型を、「2」は変異型、そして「V」表示は選択されたそれぞれのhtSNPを示す。htSNPの選別は図2乃至6の組み合わせ以外にも他の組み合わせが可能である。
その後、探し出した組み合わせの中でディプロタイプを考慮して互いに重ならずにディプロタイプ遺伝型を決定できるかをMatlabソフトウェア(version 7.1,The Math Works Inc., 米国)を使用して分析し、これを用いてチェックした後に組み合わせを決定した。
【0186】
検証の結果、互いに重ならずにディプロタイプ遺伝型を決定できることを確認した。これは本発明で選択したhtSNP組み合わせが互いに同一なものがなく、遺伝型を決定するに当たって不正確な分析が全くないことを示す。
【0187】
実施例4:高速のCYP1A2プロモーター遺伝子変異検索
前記実施例1で確認された韓国人で発見されたCYP1A2遺伝子の17個の単一塩基多型の中で、CYP1A2酵素活性に影響を与えるプロモーター部分の単一塩基多型11個を高速に検索するためにスナップショット分析を実施した。被験者のDNAを鋳型としてPCRを遂行し、増幅された産物をスナップショット分析した。CYP1A2遺伝子のプロモーターは約4,000baseであり、PCRに使用したプライマーは下記の表7の通りである。
【0188】
【表7】

【0189】
PCR産物に対する反応条件は下記の表8の通りである。
【0190】
【表8】

【0191】
前記のように増幅されたPCR産物の反応されていないプライマーとdNTPなどは残っていればスナップショット過程に影響を与えるので、これらを除去するために、混合されたPCR産物5μl当たりExoSAP−IT(USB社)2μlを入れて37℃で30分間反応させた後、80℃で15分間反応させて残っている酵素を非活性化させた。酵素処理された産物は下記の表9に示すプライマーを用いてマルチプレックススナップショット(multiplex SNaPshot)反応物を作ってPCR反応した。マルチプレックススナップショット反応物及びPCR反応条件をそれぞれ表10及び表11に示した。
【0192】
【表9】

【0193】
【表10】

【0194】
【表11】

【0195】
反応が終わった後、[F]ddNTPを除去するためにSNaPshot産物5μlにSAP(USB社)2μlを入れて37℃で30分、80℃で15分間反応させた。反応が全て終わると、反応物0.5μl、Hi−Diホルムアミド(ABI社)9.25μl及びジーンスキャン(GeneScan)−LIZサイズ標準物質(ABI社)0.25μlを混合して95℃で5分間変成させた。その後、3130XL遺伝子分析器(3130XL Genetic Analyzer、 ABI社)で分析し、その結果を図7乃至図14に示した。
【0196】
その結果、図7乃至14に示すように、CYP1A2遺伝子の変異型によりピークの色と位置が異なるように表示されて野生型、異型の対立形質を有する変異型(異型)、同型の対立形質を有する変異型(同型)を容易に識別できることを確認した。従って、本発明による分析方法を通じて時間及び費用の面で効率的かつ容易にCYP1A2遺伝子の変異型を分析できることが分かる。
【0197】
<CYP2A6>
実施例5:韓国人における2A6遺伝子の遺伝型分析
<5−1>2A6遺伝子の増幅
50名の健康な被験者から血液を分離した後、Qiagen社のゲノムDNA分離キットを使用してDNAをそれぞれ分離した。CYP2A6遺伝子は9個のエクソンを含む総遺伝子の長さが約6.9kbである。従って、CYP2A6遺伝子を7個の断片に分けてPCRを遂行した。各PCRに使用したプライマーは下記の表12の通りである。本明細書に記載された塩基配列でA、T、G及びCはそれぞれアデニン、チミン、グアニン及びシトシンを意味する。
【0198】
【表12】

【0199】
各プライマーの位置とPCR産物の大きさは下記の表13の通りである。また、ヌクレオチドの位置はCytochrome P450(CYP) Allele Nomenclature Committee(http://www.cypalleles.ki.se/cyp2a6.htm)の命名法により記載した。
【0200】
【表13】

【0201】
各PCR断片に対する反応条件は下記の表14の通りである。
【0202】
【表14】

【0203】
<5−2>PCR産物の塩基配列分析
前記実施例<5−1>で得られた各PCR産物の塩基配列を自動配列分析器及び配列番号:76乃至89のプライマーを用いて分析した。
【0204】
その後、野生型CYP2A6遺伝子の塩基配列(配列番号:75)と比較した結果、総27個のSNPを発見し、その中で2つは現在まで報告されたことがない新たなSNPである。また、遺伝子欠失による構造分析を通じて3個のSNPを発掘し、総30個のSNPは下記の表15に記載された通りである。
【0205】
【表15】

【0206】
前記の表で「+」はCYP2A6遺伝子が欠失されてCYP2A6遺伝子の一部の残った部分とCYP2A7遺伝子との接合形態(図33、CYP2A6遺伝子の1番から8番エクソンまで全てなくなり、CYP2A7のエクソン9部分末端にCYP2A6のエクソン9番が一部置換された形態)の遺伝子で発見される変異として、数字はCYP2A6遺伝子が完全にあることを仮定して塩基配列に合わせた番号を使用した(CYP2A6遺伝子の欠失であるため、CYP2A6遺伝子のSNPと見ることは困難である)。
【0207】
このような構造でSNPを用いて欠失されたハプロタイプを決定するためには、CYP2A6とCYP2A7のシークエンスが同一な部分内で5’位置の正方向プライマーをデザインし、CYP2A6に特異的でありCYP2A7遺伝子は増幅できないエクソン9部分で逆方向プライマーをデザインし、純粋にCYP2A7遺伝子は増幅されず、完全なCYP2A6遺伝子またはCYP2A6遺伝子が欠失されてCYP2A7遺伝子と接合された形態の遺伝子部分を増幅されるようにできる。
【0208】
増幅されたPCR産物でCYP2A6とCYP2A7に特異的な塩基を選択するが、CYP2A6翻訳開示コドンであるATGを基準にCYP2A6(配列番号:75)の6091C>T塩基例をみれば、次の通りCYP2A6 6091C周辺の部分はCYP2A7(配列番号:104)6521Tの塩基配列周辺と類似することが分かる。このようなサイトが6091だけではなく、CYP2A6配列で5971G及び5983T部分もCYP2A7と差異があることを確認できる。
【0209】
また、前記の表で「*」が意味するところは、国際CYP明明委員会から対立遺伝形質と認められて命名された変異の表示であり、例えば、*11は野生型に比べて224番目アミノ酸がセリンからプロリンに変わる変異を有しているハプロタイプを意味し、国際命名法はhttp://www.cypalleles.ki.se/cyp2a6.htmを従った。
また、前記の表で「♯」は新規変異を意味する。
【0210】
実施例6:CYP2A6遺伝子の遺伝型のハプロタイプ分析
前記実施例5で糾明された27個のCYP2A6遺伝子の変異と3個のCYP2A6欠失標識変異は、その組み合わせに応じてCYP2A6の酵素活性に影響を与える可能性がある。従って、本発明者等は実施例5で確認した変異によるハプロタイプをDYNACOM社のSNPAlyzeプログラムを使用して分析した。通常的にハプロタイプの分析で頻度が低い変異の場合、統計的有意性を確保し難いため、主に5%または10%以上の頻度を有する変異を選別してハプロタイプの分布を予測する。しかしながら、アミノ酸の置換を招く場合には、頻度が低いにもかかわらず、明確な機能性を有しているため、本発明では頻度が高い変異である−48T>G、22C>T、51G>A、1620T>C、1836G>T、及び6354T>Cの6個の変異とアミノ酸の変化を招く13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>Tの8個の変異を用いてハプロタイプ分析を遂行し、遺伝子の欠失がある場合、これを標識できる変異である5971G>A、5983T>G、6091C>T変異を検査変異対象に追加した。総17個の変異を用いてハプロタイプを分析した結果、図15のように総20個の韓国人におけるハプロタイプ分布を得ることができた。
【0211】
実施例7:htSNPの選別及び検証
CYP2A6遺伝子のSNPの組み合わせであるハプロタイプがCYP2A6の酵素活性に影響を与える可能性が多くのハプロタイプで報告されたことがある。このような詳細なハプロタイプの情報は最小マーカで確認することができる。これら最小マーカをhtSNPといい、前記htSNPはそれぞれのハプロタイプの正確な表示のために必要なマーカであり、色々な組み合わせを構成するようになる。この最適化された標識セットであるhtSNP組み合わせを選別するために、前記実施例6で選別された総20個のハプロタイプの塩基配列をSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype/)を用いて分析した。
【0212】
その結果、図16乃至図21に示すhtSNP組み合わせを選別した。図16乃至図21に図示されている、各選別されたhtSNP組み合わせは最適の標識セットとして、「1」は野生型を、「2」は変異型、そして「V」表示は選択されたそれぞれのhtSNPを示す。
【0213】
htSNP分析で各変異の遺伝型が決定されると、その結果から二倍体型を予測することができる。しかしながら、互いに異なるハプロタイプの組み合わせが同一な遺伝型を有する可能性もあるので、htSNP組み合わせの中でディプロタイプ(二倍体型)を考慮する時、互いに重ならずに二倍体型遺伝型を決定できるかをMatlabソフトウェア(version 7.1,The Math Works Inc., 米国)を使用して分析した。
【0214】
検査の結果、本実施例により決定されたhtSNPだけでも互いに重ならずに二倍体型を決定できることを確認した。これは本発明で選択したhtSNP組み合わせが互いに同一なものがなく、遺伝型を決定するに当たって不確実な分析が全くないことを示す。
【0215】
実施例8:高速のCYP2A6遺伝子の機能的変異検索
前記実施例5で確認された韓国人で発見されたCYP2A6遺伝子の27個の変異とCYP2A6遺伝子欠失を標識する3個の変異のうち、機能性を変化させるCYP2A6の遺伝型が遺伝子診断に有用に使用され得る。従って、図16にある変異の中でアミノ酸の変化を起こしているか、または既に機能性が立証されている変異である−48T>G、13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>Tの9個の変異と遺伝子欠失を標識する6091C>T変異とを含む総10個の機能的変異を高速に検索するために、CYP2A6遺伝子の高速遺伝型分析技術の一つであるスナップショット分析を実施した。選別されたhtSNPは図16のhtSNP組み合わせの中で機能性を反映する10個の変異が選択されたものであり、変異の位置は下記の表17の通りである。
【0216】
【表17】

【0217】
具体的に、被験者のDNAを鋳型としてPCRを遂行し、増幅された産物をスナップショット分析した。この時、PCRに使用したプライマーは下記の表18の通りである。
【0218】
この時、CYP2A6_longを増幅するためのプライマーは、CYP2A6の遺伝子全長を増幅するプライマーであるので、CYP2A6遺伝子欠失の場合には適用できず、遺伝子欠失を標識する6091C>Tを判別するためにはCYP2A6*4産物を増幅できるCYP2A6 delFとCYP2A6 delRのプライマー対を利用しなければならない。
【0219】
【表18】

【0220】
PCR産物に対する反応条件は下記の表19の通りである。
【0221】
【表19】

【0222】
前記のように増幅されたPCR産物の反応されていないプライマーとdNTPなどは残っていればスナップショット過程に影響を与えるので、これらを除去するために、混合されたPCR産物5μl当たりExoSAP−IT(USB社)2μlを入れて37℃で30分間反応させた後、80℃で15分間反応させて残っている酵素を非活性化させた。酵素処理された産物は下記の表20に示すプライマーを用いてマルチプレックススナップショット(multiplex SNaPshot)反応物を作ってPCR反応した。マルチプレックススナップショット反応物及びPCR反応条件をそれぞれ表21及び表22に示した。
【0223】
【表20】

【0224】
【表21】

【0225】
【表22】

反応が終わった後に残ったddNTPを除去するために、スナップショット産物10μlにSAP(USB社)1μlを入れて37℃で60分、65℃で15分間反応させた。反応が全て終わると、反応物0.5μl、Hi−Diホルムアミド(ABI社)9.3μl及びジーンスキャン(GeneScan)−LIZサイズ標準物質(ABI社)0.2μlを混合して95℃で5分間変成させた。その後、3100遺伝子分析器(3100 Genetic Analyzer、ABI社)で分析した。その結果を図22乃至30に示す。この遺伝型分析は下記の表23の通りである。
【0226】
【表23】

【0227】
図22乃至30に示すように、各SNPによりピークの色と大きさが一致し、CYP2A6遺伝子の機能的変異型によりピークの色と位置が異なるように表示されて野生型、異型の対立形質を有する変異型(異型)、同型の対立形質を有する変異型(同型)を容易に識別できることを確認した。図22乃至30のグラフでX軸は各プライマーの長さの差異により自動塩基配列分析器でプライマーが移動した距離であり、Y軸は各塩基に含まれた特異的な波長の蛍光物質から放出される蛍光の強さを示す。
【0228】
また、図31及び32は前記図22乃至図30の遺伝子変異以外にCYP2A6遺伝子が欠失された場合を追加的に検査するために、図22乃至図30の遺伝子検査に並行して遂行するスナップショット方法の例示として、図31はCYP2A6遺伝子が相同染色体に正常に存在する場合であり、図32はCYP2A6遺伝子が一つの染色体には存在しないため、一つの遺伝子だけを有する場合である。
【0229】
以上の発明を通じて開発したスナップショット方法により50個の試料を分析し、同一の試料に対して全長塩基配列を分析した結果、遺伝型判読結果が100%一致した。これは本発明方法の再現性が高く、正確であることを示す。
【0230】
従って、本発明による分析方法を通じて時間及び費用の面で効率的かつ容易にCYP2A6遺伝子の機能的変異型を分析できることが分かる
従って、前記方法は韓国人で主に発見される10種のCYP2A6ハプロタイプを決定することによって、これら組み合わせによるCYP2A6遺伝型を同時に高速に分析できる方法であり、韓国人で発見される遺伝的変異を含むので、遺伝型決定の正確度が高い。また、韓国人と遺伝的特性が非常に類似する日本人の場合にもほとんど全ての遺伝型分析が可能であり、既に知られた結果から中国人でも90%以上の範囲でCYP2A6遺伝型を判別できる技術であると思料される。
【0231】
<CYP2D6>
実施例9:韓国人のCYP2D6遺伝子の遺伝型分析
<9−1>ゲノムDNAの分離
174名の韓国人から採取した血液試料からゲノムDNA分離キット(Qiagen社)を用いてゲノムDNAをそれぞれ分離した。
【0232】
<9−2>CYP2D6遺伝子の増幅及び全長塩基配列分析
前記実施例<9−1>で分離された総174個のゲノムDNAサンプルの中で任意に選んだ51個のサンプルを鋳型とし、下記のプライマー対でPCRを遂行してヒトCYP2D6遺伝子を構成する9個のエクソンと1.8kbのプロモーター部位が増幅されるようにした。
【0233】
【表24】

【0234】
PCRは94℃で1分間反応させた後、98℃で10秒、64℃で30秒及び72℃で7分間30サイクルを反復し、最終的に72℃で10分間反応させた。その結果、6,569bpの大きさのPCR産物を収得した(下記の表25参照)。
【0235】
【表25】

【0236】
その後、増幅されたPCR産物を鋳型とし、下記の表26に示す総13種のプライマーを用いて増幅されたCYP2D6遺伝子の塩基配列を分析した。
【0237】
【表26】

【0238】
<9−3>各遺伝型による個別的分析
前記実施例<9−1>から分離した残りの123個のゲノムDNAサンプルについては、下記の方法により東洋人で主に発見される*2A、*5、*2N、*10B、*14B、*18、*21B、*41A、*49、*52、*60変異に対する遺伝型分析を個別的に遂行した。
【0239】
a)CYP2D6*5遺伝型の分析
CYP2D6*5遺伝型を分析するために、下記の表27に記載されたプライマーを使用してPCRを遂行した。PCR反応条件としては、94℃で1分間反応させた後に98℃で10秒、64℃で30秒及び72℃で5分間30サイクルを反復して72℃で10分間反応させた。その結果、野生型の場合、9個のエクソンを含む5.1kbのPCR産物が増幅され、CYP2D6*5変異型の場合、3.5kbのPCR産物が増幅された。
【0240】
【表27】

【0241】
b)CYP2D6*2N遺伝型の分析
CYP2D6*2N遺伝型を分析するために、下記の表28に記載されたプライマーを使用してPCRを遂行した。PCRは94℃で1分間反応させた後に98℃で10秒、64℃で30秒及び72℃で8分間30サイクルを反復して72℃で10分間反応させた。その結果、CYP2D6*2N変異型の場合、7.8kbのPCR産物を収得した。
【0242】
【表28】

【0243】
c)CYP2D6*2及び*41遺伝型の分析
CYP2D6*2遺伝型とCYP2D6*41遺伝型の場合、−1584C>G変異を除いて同一の変異(1235A>G;−740C>T;−678G>A;イントロン1でのCYP2D7への遺伝子転換(gene conversion);1661G>C;2850C>T;4180G>C)を有する。従って、先ずイントロン1でのCYP2D7への遺伝子転換の変異配列を下記の表29に記載されたプライマーを用いてAS−PCR方法(Johanson,Molecular Pharmacology,46:452-459,1994)で分析した。
【0244】
イントロン1でのCYP2D7への遺伝子転換が起こった場合、配列番号129のプライマー9と配列番号125のプライマー10Bを使用してPCR反応を行うことにより増幅産物を得ることができ、正常の場合には配列番号129のプライマー9と配列番号130のプライマー10の組み合わせで反応をした場合にのみ増幅産物が得られる。従って、2個のプライマー組み合わせ、つまり、プライマー9/プライマー10とプライマー9/プライマー10Bの組み合わせでそれぞれ反応して増幅された産物の有無に応じてイントロン1でのCYP2D7への遺伝子転換を確認することができる。
PCRは94℃で5分間反応させた後に94℃で30秒、64℃で30秒及び72℃で30秒間35サイクルを反復して72℃で10分間反応させた。その後、−1584C>G変異を下記の表29に記載されたシーケンシングプライマーを用いてパイロシーケンシングで分析して−1584Gである場合をCYP2D6*2遺伝型に、−1584Cである場合をCYP2D6*41遺伝型に決定した。
【0245】
【表29】

【0246】
d)CYP2D6*10B、*14、*18及び*49遺伝型の分析
CYP2D6*10B、*14、*18及び*49遺伝型はPCR−RFLP方法で分析した(Johanson,Molecular Pharmacology,46:452-459,1994; Wang,Drug Metabolism and Dispososition,27:385-388,1998; 及び Geadigk,Pharmacogenetics,9:669-682,1999)。この時に使用したプライマーは下記の表30の通りであり、実験条件は下記の表31に示す通りである。
【0247】
【表30】

【0248】
【表31】

【0249】
e)CYP2D6*21、*52及び*60遺伝型の分析
CYP2D6*21、*52及び*60遺伝型の分析はPCR−パイロシーケンシング法で分析した。分析に使用したプライマーの配列は下記の表32に示す通りである。
【0250】
【表32】

【0251】
前記実施例<9−2>及び<9−3>で得られたデータをGenBank accession No. M33388で公知のCYP2D6の配列に基づいて比較分析し、各対立因子の頻度を調査した。その結果、韓国人で主に発見されるCYP2D6遺伝子ハプロタイプの種類及び頻度は下記の表33の通りである。
【0252】
【表33】

【0253】
前記の表33でNormalは「正常水準」、Incrは「増加」、Decrは「減少」、Noneは「活性ない」をそれぞれ示す。また、括弧内の表記は分析に使用した標識薬物の略字である: b, bufuralol;d, debrisoquine;dx, dextromethorphan;s, sparteine。
【0254】
韓国人で主に発見される12種の遺伝型を中心に遺伝子を選択してCytochrome P450(CYP) Allele Nomenclature Committee (http://www.cypalleles.ki.se/cyp2d6.htm)を基準として各遺伝型の変異を下記の表34に示した。表34で「1」は野生型を、「2」は変異型をそれぞれ示す。
【0255】
【表34】

【0256】
実施例10:htSNPの選別及び検証
前記実施例9で確認した韓国人で発見される12種のCYP2D6遺伝型を決定するために、表34に示す33個の変異を全て分析することは、時間と費用的な面で効率性が非常に落ちる。従って、詳細なハプロタイプの情報を用いて標識遺伝子変異であるhtSNPを選定して遺伝型を決定すれば経済的な遺伝型判別が可能である。前記htSNPはそれぞれのハプロタイプの正確な表示のために必要なマーカであり、色々な組み合わせを構成するようになる。この最適化された標識セットであるhtSNP組み合わせをSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype/)を用いて選別した。選別されたhtSNP組み合わせの例を図34乃至図39に示し、各選別されたhtSNP組み合わせは最適の標識セットとして、「1」は野生型を、「2」は変異型を示し、「V」表示はhtSNPを示す。
【0257】
その後、探し出した組み合わせの中でディプロタイプ(diplotype)を考慮して互いに重ならずにディプロタイプ遺伝型を決定できるかをMatlabソフトウェア(version 7.1,The Math Works Inc., USA)を使用して分析し、これを用いてチェックした後に組み合わせを決定した。
検証の結果、互いに重ならずにディプロタイプ遺伝型を決定できることを確認した。これは本発明で選択したhtSNP組み合わせが互いに同一なものがなく、遺伝型を決定するに当たって不正確な分析が全くないことを示す。
【0258】
実施例11:SNaPshot分析
前記実施例10で選別されたhtSNP組み合わせを用いてCYP2D6遺伝子の高速遺伝型分析技術の一つであるスナップショット分析を遂行した。このために図34のhtSNP組み合わせを選択した。各htSNPで選別される変異の位置は下記の表35の通りである。下記の表35でhtSNP1乃至htSNP3の場合には該当する複数個の変異の中で一つのSNPだけを分析しても遺伝型判別が可能である。また、htSNP9の場合には9個の塩基(GTGCCCACT)が挿入されて反復される様相を示すようになるので、4125番目塩基から4133番目塩基位置のうちのいずれか一つの塩基位置だけを分析し、野生型遺伝子の塩基配列と比較することによって遺伝型判別が可能である。
【0259】
【表35】

【0260】
前記実施例<9−2>と同様の方法によりCYP2D6遺伝子を増幅して約6.7kbの産物を得た。そして、CYP2D6*5を決定するために配列番号154のプライマーCYP2D6_3(5'−ACCTCTCTGGGCCCTCAGGGA−3')と配列番号123のプライマー3‘2D6*5を用いてCYP−REP−Del部位を増幅し、PCRは94℃で1分間反応させた後に98℃で10秒、64℃で30秒及び72℃で3分間30サイクルを反復し、最終的に72℃で10分間反応させた。その結果、6,569bp大きさのPCR産物を収得した。
【0261】
前記のように増幅されたPCR産物の反応されていないプライマーとdNTPなどは残っていればスナップショット過程に影響を与えるので、これらを除去するために、PCR産物5μl当たりExoSAP−IT(USB Corporation)2μlを入れて37℃で30分間反応させた後、80℃で15分間反応させて残っている酵素を非活性化させた。酵素処理された産物を鋳型3μl(6.7kbのCYP2D6遺伝子2μlと3.5kbのCYP−REP−DEL1μlの混合物)、スナップショットマルチプレックスレディーリアクションミックス(SNaPshot Multiplex Ready Reaction Mix, ABI)1μl、1/2ターム(term)緩衝液(200 mM Tris-HCl,5 mM MgCl2,pH 9)4μl及びPooled SNaPshotプライマーを入れて全体反応物の量を10μlに合わせた後、96℃で10秒、50℃で5秒、60℃で30秒間40サイクル反復してPCRを遂行した。PCR反応した。使用したPooled SNaPshotプライマーの処理濃度を下記の表36に示した。
【0262】
【表36】

【0263】
反応が終わった後に反応物10μlにSAP(USB Corporation)1μlを入れて37℃で1時間、65℃で15分間反応させた。反応が全て終わると、反応物0.5μl、LIZ120(ABI)0.2μl、Hi−Diホルムアミド(ABI)9.3μlを混合して96ウェルプレートに分注した。分注されたサンプルを95℃で2分間反応させた後、3100遺伝子分析器(ABI)で分析した。分析した結果は図40に示す通りである。
【0264】
その結果、図40に示すように、各SNPによりピークの色と大きさが一致し、野生型と変異型が明確に決定されることが分かる。
【0265】
また、CYP2D6重複をスナップショットを用いて分析するために、CYP−REP−Dup部位を配列番号155のDup−F_2(5’−CCTCACCACAGGACTGGCCACC−3’)と配列番号156のDup−R(5’−CACGTGCAGGGCACCTAGAT−3’)を用いたことを除いては前記と同様な方法で増幅して3.3kb大きさのPCR産物を収得した。PCR反応以降、PCR産物から反応して残ったプライマーなどを除去するためにPCR産物5μl当たりExoSAP−IT(USB Corporation)2μlを入れて37℃で30分間反応させた。
【0266】
その後、80℃で15分間反応させて残っているExoSAP−ITを非活性化させた。酵素処理された産物を鋳型3μl、スナップショットマルチプレックスレディーリアクションミックス1μl、1/2ターム緩衝液4μl及び配列番号157のスナップショットプライマー(CYP2D6−5R、5’−CTCGTCACTGGTCAGGGGTC−3’)を入れて全体反応物の量を10μlに合わせた後、前記と同一な条件でスナップショット反応を遂行した後、3100遺伝子分析器で分析してその結果を図41に示した。
【0267】
その結果、図41に示すように、SNPによりピークの色と大きさが一致し、野生型と変異型が明確に決定されることが分かる。
【0268】
このような方法により野生型及び変異遺伝型を含む50個のサンプルに対してシーケンシングを通じた妥当性検査(validation)を遂行した結果、100%一致する結果を得た。この結果は本発明方法が再現性が高く、正確であることを示す。
【0269】
従って、前記方法はこれら組み合わせて高速で、韓国人で主に発見される12種のCYP2D6ハプロタイプを決定し、同時にCYP2D6遺伝型を決定する。韓国人で発見される遺伝的変異を含むので、遺伝型決定における正確度が高い。また、韓国人と遺伝的特性が非常に類似する日本人の場合にもほとんど全ての遺伝型分析が可能であり、既に知られた結果から中国人でも90%以上の範囲でCYP2D6遺伝型を判別できる技術であると思料される。
【0270】
実施例12:遺伝子分析チップを用いた遺伝型分析
<12−1>ZiP Codeチップの製作
1)プローブ製作
ASPE PCR反応に使用するZiPCodeと相補的な塩基配列でデザインし、プローブは3’方向に10bpのヌクレオチド配列(5’−CAG GCC AAGT−3’)をスペーサで挿入してターゲットと交雑反応がよく生じるように誘導した。
【0271】
また、前記スペーサの5’方向に24bpのZiP Codeオリゴ塩基が含まれており、前記プローブ(cZiP Code)の塩基配列は下記の表37に記載された通りである。この時、下線部は10個のスペーサ配列である(図43)。
【0272】
【表37】

【0273】
2)スポッティング及びプローブ固定
チップ製作のための基板はアミンがコーティングされているコーニン(Corning)社のGAPSIIガラススライドを使用した。SMP4XBピンを使用してOmniGgid100スポッターでスポッティングし、スポッティング条件は温度−22℃、湿度−54%とし、27種のプローブはそれぞれ2回反復でスポッティングした。スポッティング後、7,500μJ/cm2のUVを照査してガラススライドにプローブを固定させた。
【0274】
3)ZiP Codeテスト用遺伝型分析チップの構成
CYP2D6遺伝子の9個の遺伝型標識(SNP;前記の表で下線にて表示)を用いて11個の遺伝型(CYP2D6*1、*2、*5、*10B、*14A、*14B、*18、*21、*41、*49、*2N)を検証した。
【0275】
【表38】

【0276】
<12−2>ターゲット製作
1)ロング(Long)PCR
CYP 2D6ゲノムDNAサンプル2μl、1X LA緩衝液、2.5mM MgCl2、0.4mM dNTP、下記の表16の各0.2pmol/μlプライマー、LAタクDNAポリメラーゼ(taq DNA polymerase,TAKARA:cat. No. RR002A)2.5ユニット、3次蒸溜水を入れて50μlに合わせた後、94℃で1分間1回変成させた後、98℃10秒、64℃30秒、72℃6分間30サイクル反応後、72℃で1分間延長反応して増幅させた(図44)。(CYP2D6遺伝子は、*5、*2N対立遺伝子(allele)、及びその他対立遺伝子用として各3種類の1STPCR産物を得た。各条件は前記と同一である。)
【0277】
【表39】

【0278】
前記で得られたロングPCR産物0.5μl、1X amplitaq緩衝液、0.2mM dNTP、各プライマー0.5pmol/μlずつ及びAmpli taq gold(Applied Biosystems : cat. No. N8080242)0.5unitに3次蒸溜水を入れて10μlに合わせた後、4℃で5分間1回変成させた後、94℃45秒、57℃45秒、72℃1分間30サイクル反応後、72℃で1分間延長反応して増幅させた。2ND PCRは多重PCRで遂行し、下記の表40の4個のセットで増幅させた。プライマー配列は表41に記載した通りである。
【0279】
【表40】

【0280】
【表41】

【0281】
3)ASPE(allele specific primer extension)反応
前記で得られた多重PCR産物の各6μl、1X amplitaq緩衝液、Cy5dUTP(ジーンケム)10μM、各ASPEプライマー125nM、 Amplitaq gold(Applied Biosystems:cat. No. N8080242)1ユニット、1X Band doctor(ソルジェント)及び3次蒸溜水を入れて20μlに合わせた後、94℃で5分間1回変成させた後、94℃で30秒、60℃で1分、72℃で1分間30回のサイクルで反応して増幅させた(図45参照)。ASPE反応セット及びプライマー配列は下記の表42及び43に記載した通りである。
【0282】
【表42】

【0283】
【表43】

【0284】
4)PCR精製
ASPE反応で得られた1乃至4セットの産物をpoolingしてQiagen精製キット(Qiagen:ca. no. 28106)を用いて製造会社のマニュアルにより精製し、最終溶出体積を50μlとした。
【0285】
精製された各産物を濃縮器(Speed Vacuum concentrator,BioTron社module 4080C)を用いて体積が1乃至2μl程度残るまで乾燥させた。
【0286】
5)チップ混成化
前混成化(prehybridization)緩衝液(25%ホルムアミド、5X SSC、0.1%SDS、10mg/ml BSA)を42℃で温めた後、チップを前記緩衝液に漬けて42℃で30分以上培養した。前記チップを蒸溜水で1分ずつ3回洗浄した後、チップをコニカル(conical)チューブに入れて800rpmで5分間遠心分離して乾燥させた。
【0287】
その後、混成化緩衝液(25%ホルムアミド、5X SSC、0.1%SDS、0.5mg/mlポリA、25μg/ml Cot−1DNA、10%デキストランスルフェート)を予め42℃で温めた後、前記で得られた乾燥されたサンプルをここに溶かした。よく溶かしたサンプル0.5mlをPCRチューブに移した後、95℃で5分間加熱した。混成化チャンバー(chamber)を準備してチャンバー空間に3Mペーパ切れを入れて3X SSCを20μl程度落とした。加熱されたサンプルを前混成化されたチップ上にローディングした後、チャンバーにチップを入れて組立てた後、42℃で一晩混成化させた。
【0288】
前記チップを予め50℃に温められた2X SSC 0.1%SDS溶液で10分間1回洗浄した後、0.1X SSC溶液にて1分ずつ常温で4回洗浄した。洗浄したチップを直ちにコニカルチューブに入れて800rpmで5分間遠心分離して乾燥させた。
【0289】
6)分析
前記のように準備されたチップをAxonのGenePix 4100Bスキャナーを使用して出力波長は650nm付近でスキャニングし、スキャニングされたイメージはGenePix Pro 6.0 softwareを使用して蛍光信号の強さを分析し、その結果を図46及び下記の表44に示した。
【0290】
【表44】

【0291】
その結果、遺伝子分析チップを用いて分析したCYP2D6遺伝子の変異は配列分析を通じて確認された結果と同一であった。
【0292】
<PXR>
実施例13:韓国人のPXR遺伝子の遺伝型分析
<13−1>PXR遺伝子の増幅
54名の健康な被験者から血液を分離した後、Qiagen社のゲノムDNA分離キットを使用してDNAをそれぞれ分離した。PXR遺伝子の全体塩基配列を自動塩基配列分析器(ABI Genetic Analyzer 3130XL)を用いて分析した結果、現在まで報告された総18個の機能的変異の中で6個を確認した。PXR遺伝子は9個のエクソンを含んで総遺伝子の長さが約38kbである。従って、PXR遺伝子を機能的変異があるエクソンを中心に10個の断片に分けてPCRを遂行した。各PCRに使用したプライマーは下記の表45の通りである。本明細書に記載された塩基配列でA、T、G及びCはそれぞれアデニン、チミン、グアニン及びシトシンを意味する。
【0293】
【表45】

【0294】
各プライマーの位置とPCR産物の大きさは下記の表46の通りである。また、ヌクレオチドの位置は文献[HUMAN MUTATION 11:1. 3 (1998)]の命名法により記載した。
【0295】
【表46】

【0296】
各PCR断片に対する反応条件は下記の表47の通りである。
【0297】
【表47】

【0298】
<13−2>PCR産物の塩基配列分析
前記実施例<13−1>で得られた各PCR産物の塩基配列を自動配列分析器及び配列番号:131乃至150のプライマーを用いて分析した。
【0299】
その後、野生型PXR遺伝子の塩基配列(配列番号:130)と比較した結果、総22個のSNPを発見し、そのうち、6個は現在まで報告された機能的変異18個の一部である。22個のSNPは下記の表48に記載された通りであり、その中で報告された機能的変異は♯で表示した。
【0300】
【表48】

【0301】
前記の表48に示すように、PXRの変異遺伝子は7個がプロモーター地域で現れ、残りは3'UTRとイントロン地域で現れ、アミノ酸置換を見せる変異は現れなかった。
【0302】
実施例14:PXR機能的変異型のハプロタイプ分析
前記実施例13で機能性が研究されたことがある6個のPXR遺伝子の機能的変異はその組み合わせによりPXRの機能性に影響を与える可能性がある。
【0303】
従って、本発明者等は実施例13で確認した変異によるハプロタイプをDYNACOM社のSNPAlyzeプログラムを使用して分析した。その結果、下記の表49に示したように14種以上の1%以上の頻度を有するハプロタイプを確認することができた。
【0304】
【表49】

□:各SNPの変異型
【0305】
実施例15:htSNPの選別及び検証
PXR遺伝子のSNPの組み合わせであるハプロタイプがPXRの活性に影響を与える可能性が多くのハプロタイプで報告されたことがある。このように作られたハプロタイプの詳細な情報は最小マーカで確認できる。これら最小マーカをhtSNPといい、前記htSNPはそれぞれのハプロタイプの正確な表示のために必要なマーカであり、種々の組み合わせを含む。この最適化された標識セットであるhtSNP組み合わせを選別するために、前記実施例14で選別された14個のハプロタイプの塩基配列をSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype)を用いて分析した。
【0306】
その結果、図47に示すようなhtSNP組み合わせを選別した。図47に示されている、各選別されたhtSNP組み合わせは最適の標識セットのうちの一つとして、「1」は野生型を、「2」は変異型、そして「V」表示は選択されたそれぞれのhtSNPを示す。htSNPの選別は図1の組み合わせ以外にも他の組み合わせが可能である。
【0307】
その後、探し出した組み合わせの中でディプロタイプを考慮して互いに重ならずにディプロタイプ遺伝型を決定できるかをMatlabソフトウェア(version 7.1,The Math Works Inc.,米国)を使用して分析し、これを用いてチェックした後に組み合わせを決定した。
【0308】
検証の結果、互いに重ならずにディプロタイプ遺伝型を決定できることを確認した。これは本発明で選択したhtSNP組み合わせが互いに同一なものがなく、遺伝型を決定することに当たって不正確な分析が全くないことを示す。
【0309】
実施例16:高速のPXR遺伝子の機能的変異検索
前記実施例13で確認された韓国人で発見されたPXR遺伝子の6個の機能的変異の中で、PXR機能に影響を与える機能的変異を高速に検索するためにスナップショット分析を実施した。被験者のDNAを鋳型としてPCRを遂行し、増幅された産物をスナップショット分析した。PCRに使用したプライマーは下記の表50の通りである。
【0310】
【表50】

【0311】
PCR産物に対する反応条件は下記の表51の通りである。
【0312】
【表51】

【0313】
前記のように増幅された4種類のPCR産物を同量で混合した後、混合されたPCR産物の反応していないプライマーとdNTPなどは残っていればスナップショット過程に影響を与えるので、これらを除去するために、混合されたPCR産物5μl当たりExoSAP−IT(USB社)2μlを入れて37℃で30分間反応させた後、80℃で15分間反応させて残っている酵素を非活性化させた。酵素処理された産物は下記の表52に示すプライマーを用いてマルチプレックススナップショット(multiplex SNaPshot)反応物を作ってPCR反応した。マルチプレックススナップショット反応物及びPCR反応条件をそれぞれ表53及び表54に示した。
【0314】
【表52】

【0315】
【表53】

【0316】
【表54】

【0317】
反応が終わった後に[F]ddNTPを除去するためにスナップショット産物10μlにSAP(USB社)1μlを入れて37℃で60分、65℃で15分間反応させた。反応が全て終わると、反応物0.5μl、Hi−Diホルムアミド(ABI社)9.3μl及びジーンスキャン(GeneScan)−LIZサイズ標準物質(ABI社)0.2μlを混合して95℃で5分間変成させた。その後、3130XL遺伝子分析器(3130XL Genetic Analyzer, ABI社)で分析した。その結果を図48乃至図50に示した。
【0318】
図48乃至50に示されるように、PXR遺伝子の機能的変異型によりピークの色と位置が異なるように表示され、野生型、異型の対立形質を有する変異型(異型)、同型の対立形質を有する変異型(同型)を容易に識別できることを確認した。従って、本発明による分析方法を通じて時間及び費用の面で効率的かつ容易にPXR遺伝子の機能的変異型を分析できることが分かる。
【0319】
<UGT1A>
実施例17:韓国人由来のUGT1A族遺伝子群の変異遺伝子選別
工程1)遺伝体DNAの分離
50名の韓国人を対象として血液をそれぞれ採取した後、遺伝体DNA分離キット(Qiagen社)を用いて採取された各血液試料から遺伝体DNAを分離した。
工程2)UGT1A族遺伝子群の増幅及び全長塩基配列分析
【0320】
前記工程1で分離された総50個の遺伝体DNA試料をそれぞれ鋳型として使用し、下記の表55に記載された各プライマー対を使用してPCRを遂行することによって、各ヒトUGT1A族遺伝子を増幅した。この時、増幅されたUGT1A族の遺伝子名、位置、使用されたプライマー名、その配列番号、大きさ、及びPCR反応条件は下記の表55にそれぞれ示す通りである。
【0321】
【表55】

【0322】
工程3)各UGT1A族遺伝子の変異型分析
前記工程2で増幅させた各UGT1A族遺伝子の全長配列を対象として公知の3100遺伝子分析器(3130x Genetic Analyzer,Applied Biosystems)を用いて配列を分析し、その結果をそれぞれ野生型UGT1A族遺伝子の塩基配列(GenBank accession No.:NT_005120)と比較した。結果を下記の表56及び57に示した。
【0323】
【表56】

【0324】
【表57】

【0325】
工程17−1)UGT1A族遺伝子の機能的変異型選別
前記工程3で得られた韓国人50名で発見されたUGT1A族遺伝子の多型性に基づき、酵素活性の増加または減少のように機能的に関連したと報告された変異型を選別して下記の表58に示した。この時、UGT1A9でのG766A変異型は工程3では確認されなかったが、日本人で報告された機能的変異型であるので下記の表58に追加した。「truncated protein」は蛋白質が翻訳過程中間に突然変異により非正常的に翻訳終結されることをを意味する。
【0326】
【表58】

【0327】
工程17−2)UGT1A族遺伝子の薬物感受性関連多型性選別
前記工程3で得られた韓国人50名で発見されたUGT1A族遺伝子の多型性に基づき、大腸癌治療用抗癌剤であるイリノテカン(Irinotecan)の代謝に関与すると知られたUGT1A1、UGT1A6及びUGT1A9の多型性を選別して下記の表59に示した。この時、UGT1A9でのG766A変異型は工程3では確認されなかったが、日本人で報告された機能的変異型であるので下記の表59に追加した。
【0328】
【表59】

【0329】
実施例18:UGT1A族遺伝子群の機能的変異型及び薬物感受性関連多型性分析
18−1)UGT1A族遺伝子の機能的変異型分析
前記実施例17の被験者の中で各UGT1A族遺伝子の野生型、異型の対立形質を有する変異型、同型の対立形質を有する変異型を有するヒトからの血液を対象として、次の通り本発明によるヒトUGT1A族遺伝子の機能的変異型を確認した。
【0330】
前記実施例17の工程1及び2と同様な方法によりUGT1A1、UGT1A3、UGT1A4、UGT1A6、UGT1A7及びUGT1A9族遺伝子の配列をそれぞれ増幅した。その後、得られた各UGT1A族遺伝子のPCR産物5μl当りエクソSAP−IT(ExoSAP-ITTM,USB Corporation)2μlずつを加えた後、37℃で30分間反応させて残余プライマーなどを除去した。得られた反応物を80℃で15分間反応させて残っているエクソSAP−ITを非活性化させた後、これを2μlずつ取ってスナップショットマルチプレックスレディーリアクションミックス(SNaPshot Multiplex Ready Reaction Mix, ABI)1μl、ハーフターム溶液(Half term buffer、組成: 200 mM Tris-HCl,5 mM MgCl2,pH 9)4μl及び下記の表60に提示された各スナップショットプライマーと混合してスナップショット反応溶液を得た。この時、全体反応溶液の量は10μlになるようにした。
【0331】
【表60】

【0332】
前記各反応溶液を[96℃で10秒、50℃で5秒、60℃で30秒]の40サイクル反復条件下でPCR反応させ、反応が終わった反応溶液10μlにSAP(shrimp alkaline phosphatase)(USB社)1μlずつを加えて37℃で1時間、65℃で15分間反応させた。これをそれぞれ0.5μlずつ取った後、LIZ120(ABI)0.2μlとHi−Diホルムアミド(ABI)9.3μlと混合して96ウェルプレートに分注した。分注された反応試料を95℃で2分間反応させた後、3130x遺伝子分析器(3130x Genetic Analyzer,Applied Biosystems)で分析し、その結果を図51乃至54に示した。
【0333】
図51乃至54に示されるように、各UGT1A族遺伝子の機能的変異型によりピークの色と位置が異なるように表示され、野生型、異型の対立形質を有する変異型(異型)、同型の対立形質を有する変異型(同型)を容易に識別できることを確認し、得られたピークの大きさと種類が前記実施例17において配列分析で得られた表55の結果と100%一致することを確認した。従って、本発明による分析方法を通じて時間及び費用の面で効率的かつ容易にUGT1A族遺伝子群の機能的変異型を分析できることが分かる。
【0334】
また、UGT1A1族遺伝子の−39insTA遺伝型は単一塩基変異に該当せず、 前記のようなスナップショット分析が不可能であるので、次のようなPCR−パイロシーケンシング法を遂行して変異型を確認した。この時、分析に使用したプライマーの配列を下記の表61に示した。この時、プライマーUGT1A1*28Fの場合、5’末端にビオチンが付着していた(配列番号:202参照)。パイロシーケンシングに使用されたプライマーは文献[Clin Chem.,Jul;49(7):1182-5,2003]を参考にした。
【0335】
具体的には、配列番号202と203を用いて収得したPCR産物を鋳型として配列番号204の塩基配列分析用プライマーを反応させた後、パイロシーケンサー(Pyrosequencing社)を用いて変異有無を確認した。
【0336】
収得したPCR産物にバインディングバッファー37μl(Binding buffer pH 7.6、組成:10 mM Tris-HCI、2 M NaCI、1 mM EDTA、0.1% Tween20)にセファロースビーズ (Streptavidin SepharoseTM High performance:Amersham Bioscience)3μlを入れて混合した後、96ウェルプレートに分注して常温で1,4000rpmで5分間反応させた。そして、下記の配列番号204のプライマー(100pmol)0.3μl当たり100μlアニーリングバッファー(1X annealing buffer pH 7.6、組成:20 mM Tris acetate、2 mM MgAc2)を96ウェルプレートに分注した。反応させたサンプルを真空プレップツール(vacuum Prep Tool)を用いて試料を準備し、90℃で3分間加熱した後、常温で冷却した。冷却プレートに Pyro Gold Reagent kit(Biotage)で提供されている酵素混合物(enzyme mixture)、気質混合物(substrate mixture)、dATP、dCTP、dGTP及びdTTPを入れた後、パイロシーケンサーを用いて変異有無を確認した。
【0337】
【表61】

【0338】
18−2)UGT1A族遺伝子の機能的変異型分析
前記の表60に提示されたプライマーの代わりに、下記の表62に提示されたプライマーを同時に使用したことを除いて、前記18−1でのスナップショット分析と同一な工程を遂行してイリノテカン感受性と関連したUGT1A族多型性を分析し、その結果を図55に示した。この時、表62に提示されたプライマーを5'末端に互いに異なる長さのT反復配列を付けてそれぞれの長さを異なるようにした。
【0339】
【表62】

【0340】
その結果、図55に示したように、UGT1A族のイリノテカン感受性関連の色々な多型性を同時に容易に識別できることを確認し、得られたピークの大きさと種類が前記実施例17で配列分析により得られた表55の結果と100%一致することを確認した。
【産業上の利用可能性】
【0341】
前記で考察した通り、本発明によるCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A族遺伝子群の機能的変異型または薬物感受性関連多型性を分析する方法は、今まで確認されたことのない韓国人のCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A族遺伝子の多型性に基づいて得られた最適の探索セットを用いて時間及び費用の面で効率的にCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A族遺伝子群の機能的変異型またはUGT1A族遺伝子群の薬物感受性関連多型性を容易に確認できる方法であるため、韓国人だけではなく、韓国人と遺伝的特性が類似する日本、中国などのアジア圏人種のCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A族遺伝子の遺伝型分析にも有用に活用され得る。
【技術分野】
【0001】
本発明は、シトクロムP450 1A2(CYP1A2)、2A6(CYP2A6)及び2D6(CYP2D6)、PXR及びUDP−グルクロノシルトランスフェラーゼ1A族(UGT1A)遺伝子の遺伝型分析のためのhtSNP、及びそれを用いた遺伝子分析チップに関し、より詳しくは、ヒトCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子のハプロタイプ分析のためのhtSNPの選別方法、前記htSNPを用いて前記遺伝子の遺伝型を決定する方法及びそのための遺伝子分析チップに関するものである。
【背景技術】
【0002】
個体間の遺伝的多様性は薬物の毒性及び効能において個体間の差異をもたらす。従って、薬物開発の初期段階でこのような薬剤学的に重要な蛋白質の遺伝的多様性に関する効果を考慮するのは、薬物開発の失敗に対する危険性を低めることができる重要な要素である。このような個体間遺伝的多様性を測定する一つの調査集団となるのが“ハプロタイプ(haplotype)”である。ハプロタイプとは、一つの調査集団内に存在する各遺伝子配列の多型性の組み合わせを意味し、これは個別的な多型性よりも遺伝的多様性に関するより正確かつ信頼できる情報を提供する。
【0003】
一方、ヒトシトクロムP450は、薬物、発癌原及び毒素のような外来化学物質とステロイド、脂肪酸及びビタミンのような内部気質の酸化を促進するヘム蛋白質の一員である(Nelson et al.,Pharmacogenetics 6:1-42,1996)。肝を含む腎臓、腸管及び肺のような器官で多様なシトクロムP450の亜型が発見された。
【0004】
ヒトシトクロムP450 1A2(Cytochrome P450 1A2、以下‘CYP1A2’という)はCYP1A1、CYP1B1と共にCYP1族に属する薬物代謝酵素である。CYP1A2は肝で主に生産され、総シトクロムP450酵素量の15%を占める。この酵素はカフェイン(caffeine)、クロザピン(clozapine)、イミパラミン(imiparamine)、プロプラノロール(propranolol)を含む医学的に重要な薬物の代謝に関与する。この酵素は、また17β−エストラジオール(estradiol)、ウロポルフィリノーゲン(uroporphyrinogen)IIIのような内部合成物質とポリサイクリック芳香族炭化水素エポキシ化(polycyclic aromatic hydrocarbon epoxidation)と芳香族/ヘテロサイクリックアミンN−ヒドロキシ化(aromatic/heterocyclic amine N-hydroxylation)のような発癌物質生活性化(carcinogen bioactivation)反応を触媒する(Brosen K.,Clinical Pharmacokinetic, 1995,29(suppl1):20-25;Josephy PD.,Environ. Mol. Mutagen,2001,38:12-18)。
【0005】
また、ヒトシトクロムP450 2A6(Cytochrome P450 2A6、以下‘CYP2A6’という)遺伝子は19番目染色体に位置しており、CYP2A6遺伝子の上流には塩基配列類似性の高い類似遺伝子(pseudogene)であるCYP2A7が位置している。
【0006】
CYP2A6酵素はニコチン(nicotine)をコチニン(cotinine)に変換する機能を有し、ニコチン代謝の約80%に関与する重要な酵素であり、生体内で抗癌剤であるテガフール(tegafur)を活性型である5−フルオロウラシル(5-fluorouracil、5-FU)に変換させる過程に関与する酵素である。前記酵素は肝で主に生産され、肺、大腸、乳房、腎腸及び子宮のような臓器では少量発現する(Drug Metab Dispos.,29(2):91-5,2001;Adv Drug Deliv Rev.,18;54(10):1245-56,2002)。
【0007】
また、ヒトシトクロムP450 2D6(Cytochrome P450 2D6、以下‘CYP2D6’という)遺伝子は22番目染色体に位置しており、CYP2D6遺伝子の一方の面には類似遺伝子(pseudogenes)であるCYP2D7とCYP2D8が位置している。前記遺伝子によりコーディングされる酵素は精神活性薬物(psychoactive drugs)、心血管系薬物、モルヒネ系薬物など、100種類以上の臨床的に重要な薬物を代謝することが知られている。
【0008】
CYP2D6遺伝子によりコーディングされる酵素は、肝で主に生産され、シトクロムP450酵素の総量の約2%を占めるが、30%程度の薬物代謝に関与する重要な酵素である。
【0009】
個体内で前記酵素の活性は非常に多様であり、これらはその活性程度によりそれぞれPM(poor metabolizers)、IM(intermediate metabolizers)、EM(extensive metabolizers)及びUM(ultrarapid metaboilzers)群に分類されている。前記のように酵素の活性がそれぞれ異なるように現れるのは、前記遺伝子の遺伝的多型性による原因もある。CYP1A2の遺伝子は遺伝的多型性を現わすことが知られているが、現在までこの遺伝子のプロモーター、エクソン及びイントロンで24種以上の変異が発見され、これら変異遺伝子の組み合わせであるハプロタイプは2007年6月現在36種類(http://www.cypalleles.ki.se/cyp1a2.htm)、CYP2A6の遺伝型は50種類(http://www.cypalleles.ki.se/cyp2a6.htm)、CYP2D6の遺伝的多型性は約80種類知られており(www.imm.ki.se/cypalleles/cyp2d6.htm)、種族間の明確な差異を示している。しかしながら、このような遺伝子変異とハプロタイプに対して多様な種類が報告されているため、分析時間と費用の効率性を増大するために最小限の単一塩基多型(single nucleotide polymorphism、SNP)を通じて正確なハプロタイプを決定することが重要である。
【0010】
一方、多様な種類の薬物が体内に入って排出されるまで体内では代謝と輸送が起こるが、ここに関与する酵素系としては、シトクロムP450(CYP)と薬物輸送蛋白質がある。この中で薬物の酸化代謝に関与するCYP酵素に対する研究が多く行われており、現在15種類程度のCYP酵素が遺伝的多型性を示すことが報告されており、代表的なものとしてCYP2D6、CYP2C9、CYP3A4、CYP2B6、MDR1及びCYP2C19などがある。遺伝的な多型性はこれら酵素の基質薬物の臨床効果、治療効果及び副作用の発生に影響を与える重要な因子として作用するようになる。これらの中で一部の変異遺伝子は酵素喪失を招いて薬物代謝能が全くない形態で現れ、一部の変異遺伝子は酵素活性度が一部減少する形態で現れることもある。例えば、CYP2D6及びCYP2C9などのような酵素は変異遺伝子により表現型が変わり、比較的に遺伝型と表現型間に高い相関性を示す反面、CYP3A4、CYP2B6及びMDR1などの遺伝子は機能性変異遺伝子の有無から表現型を予測するのに困難がある。
【0011】
ヒトの場合、服用する薬物の50%以上を代謝させるCYP3A4は個体間に多くの活性差異を示し、CYP2B6の場合も個体間の差異が最大270倍程度生じることが知られている。このような個人差にもかかわらず、個人による活性の差異は遺伝型から直ちに予測し難いが、これはこれら遺伝型と表現型の相関性が低い薬物代謝酵素や薬物輸送蛋白質の場合、蛋白質の発現が外部因子により顕著に変わることがあるためである。従って、これら遺伝子の場合、変異遺伝子の有無よりは蛋白質の発現調節が代謝活性の個人差を招くより重要な要素になるだろう。また、酵素の発現誘導により酵素の生産量自体が多くなり、活性が増加する結果を示すことがあるが、これら発現誘導メカニズムは外部物質が薬物受容体と結合して標的遺伝子のプロモーターに結合することによって行われる。このような薬物受容体の代表的なものとして、プレグナンX受容体(Pregnane X receptor;PXR)がある。PXR受容体はNR1I2という遺伝子で発現することが知られている。
【0012】
PXRは個人間発現量の差異が報告されており、興味深い点は受容体の発現量がCYP3A4とCYP2B6のような薬物代謝酵素の発現量と高い相関性を示すということである(Current Drug Metabolism, 2005,6:369-383)。従って、個体間の薬物代謝酵素の発現量差異は変異型蛋白質によるものというよりはPXR遺伝子の発現量や活性差異に起因する。
【0013】
このような脈絡で、最近、個人別PXR遺伝子の遺伝的多型性(polymorphism)研究や発現差異に対する研究が関心を引いているが、アミノ酸の配列に変化がないにもかかわらず、人体でエリスロマイシン呼吸検査法(erythromycin breath test)またはリファムピン(rifampin)によるCYP3A4活性増加などの薬物反応に個人間の差異を招く一部の変異遺伝子が報告されたことがある(Pharmacogenetics,2001,11:555-572)。このようなPXRの変異は標的遺伝子の発現変化に応じた活性変化を招くため、薬物や生体分子の体内動態の差異を招くこともでき、PXRの結合子である併用薬物による薬物相互作用の個人差にも大きく寄与する。
【0014】
一方、UDP−グルクロノシルトランスフェラーゼ(UDP-glucuronosyltransferase;UGT)は生体内で耐因性及び外因性物質にグルクロン酸が接合する反応を触媒する酵素として、フェノール、アルコール、アミン及び脂肪酸化合物などのように生体毒性を有する多様な物質のグルクロン酸接合体を生成させることによって、水溶性物質に転換させて胆汁や尿で排出されるようにする(Parkinson A,Toxicol Pathol.,24:48-57,1996)。
【0015】
このようなUGTは肝細胞の小胞体及び核膜に主に存在することが報告されており、腎腸及び皮膚のような他の組織でもその発現が報告されたことがある。UGT酵素は一次アミノ酸配列間の類似性に基づいて大きくUGT1及びUGT2の亜族に区分することができ、この中でヒトUGT1A族の場合には9種類(UGT1A1、及びUGT1A3乃至UGT1A10)の異性体が報告されており、その中の5種類(UGT1A1、UGT1A3、UGT1A4、UGT1A6、及びUGT1A9)が肝で発現することが知られている。
【0016】
このようなUGT1A族遺伝子は個人毎に遺伝的多型性を有することが知られており、最近までUGT1A族の遺伝的多型性はUGT1A1、及びUGT1A3乃至UGT1A10のそれぞれにより多様な種類が存在することが知られている(http://galien.pha.ulaval.ca/alleles/alleles.html)。UGT1A族遺伝子群の多型性は特に種族間に明確な差異を見せており、このような多型性により酵素の活性が異なるように現れることが確認され、薬物治療などに対する感受性を決定する要因として重要視されている。また、UGT1A1*6とUGT1A1*28はギルバート(Gilbert)症候群と関連があり(Monaghan G,Lancet,347:578-81,1996)、その他にも多様な疾病と関連した多様な機能的な変異型が報告されている。
【0017】
ところで、このような遺伝子変異とハプロタイプは多様な種類が報告されているため、検索方法の効率性を考えざるを得ない。ハプロタイプはArlequin、SNPAlyzeまたはこれらと類似するソフトウェアを用いて分析が可能である。各ハプロタイプに対する遺伝子変異検索を全ての単一塩基多型に対して分析するとすれば、費用及び時間の面で効率性を期待し難い。
【0018】
効率性増大のための方法として、ハプロタイプタグ単一塩基多型(haplotype-tagging SNP;htSNP)選別技術を挙げることができる。htSNP選別技術は、それぞれのハプロタイプの正確な表示のために最小限の標識セットの選択を助けるための方法として、選別された単一塩基多型(SNP)のみを確認する場合、全てのハプロタイプを予測できる。
【0019】
多くの遺伝子の場合に遺伝的多型性の分布は種族による差異を示すことが知られているため、前記遺伝子の場合にも韓国人に頻繁に発生される固有遺伝子変異及びハプロタイプはないか、あるとすれば頻度はどのくらいか、それぞれのハプロタイプに応じたhtSNPの選別はどのようになるか、確認する必要がある。しかしながら、韓国人において前記遺伝子の変異、これによるハプロタイプ及びこれを分析するためのhtSNPに対する研究は非常に不十分であるのが実情である。
【0020】
また、最近西洋人で主に発見されるCYP2D6遺伝子変異を中心にスナップショット(SNaPshot)分析を用いて11個のSNPを分析する方法(Sistonen J et al.,Clin Chem. 2005 Jul;51(7):1291-5)、及びロシュ社やジュリーラブ社のCYP2D6診断チップが報告された。しかしながら、これらはCYP2D6変異遺伝子の組み合わせが西洋人で発見される変異を中心に形成されているため、韓国人を含む東洋人に対するCYP2D6遺伝子変異診断に関する研究は非常に不十分であるのが実情である。
【0021】
そこで、本発明者等は韓国人で主に発見されるヒトCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子の変異を短時間内に正確に確認できる方法を開発するために研究を行い、韓国人で主に発見されるヒトCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子の変異に対するhtSNP選別方法及び前記選別されたhtSNPの有用性を確認することによって本発明を完成した。
【発明の概要】
【発明が解決しようとする課題】
【0022】
従って、本発明の目的は、韓国人で発見されるCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子のハプロタイプを分析できるhtSNPを選別する方法を提供することにある。
また、本発明の他の目的は、前記htSNPを用いてヒトCYP1A2、CYP2A6、CYP2D6、NR1I2(=PXR)及びUGT1A遺伝子の遺伝型を決定する方法を提供することにある。
【0023】
また、本発明のさらに他の目的は、遺伝子分析チップを含むキットを用いてヒトCYP2D6遺伝子の遺伝型を決定する方法を提供することにある。
【課題を解決するための手段】
【0024】
前記のような目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A遺伝子からなる群より選択された遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記(c)工程で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;(e)前記(d)工程で変異が存在すると確認されたPCR産物の塩基配列において、ハプロタイプ(haplotype)を分析する工程;及び(f)前記(e)工程で分析されたハプロタイプの塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析し、htSNPを選別する工程を含む、ヒトCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1Aからなる群より選択される遺伝子のhtSNPを選別する方法を提供する。
【0025】
本発明の他の目的を達成するために、本発明は、(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;(c)前記(b)工程のゲノムDNAの鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G、−163C>A、1514G>A、2159G>A、2321G>C、3613T>C、5347C>T及び5521A>Gからなる群より選択される少なくとも11個のCYP1A2遺伝子の変異の存在有無を調査する工程を含む、ヒトCYP1A2遺伝子の遺伝型を決定する方法を提供する。
【0026】
本発明のさらに他の目的を達成するために、本発明は、(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;(c)前記(b)工程のゲノムDNAを鋳型とし、ヒトCYP1A2遺伝子のプロモーター領域を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G及び163C>Aを含むCYP1A2遺伝子の変異の存在有無を調査する工程を含む、CYP1A2プロモーター遺伝子の変異を検出する方法を提供する。
【0027】
本発明のさらに他の目的を達成するために、本発明は、(a)被験者から生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び(d)前記工程(c)で得られたPCR産物の塩基配列において、−48T>G、13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>T、及び6091C>Tからなる群より選択されるCYP2A6遺伝子変異の存在有無を調査する工程を含む、ヒトCYP2A6遺伝子の遺伝型を決定する方法を提供する。
【0028】
本発明のさらに他の目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D欠失(deletion);及び2D6重複(duplication)を含む少なくとも11個のCYP2D6遺伝子変異の存在有無をSNaPshot技法を用いて調査する工程を含む、ヒトCYP2D6遺伝子の遺伝型を決定する方法を提供する。
【0029】
本発明のさらに他の目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記工程(c)で得られたPCR産物の塩基配列において、−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cからなる群より選択されたPXR遺伝子の遺伝子変異の存在有無を調査する工程を含む、PXR遺伝子の遺伝型を分析する方法を提供する。
【0030】
本発明の他の目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)で採取された試料から核酸を抽出する工程;(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族個別遺伝子増幅する工程;及び(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での−39(TA)6>(TA)7、211G>A、233C>T及び686C>A;UGT1A3での31T>C、133C>T及び140T>C;UGT1A4での31C>T、142T>G及び292C>T;UGT1A6での19T>G、541A>G及び552A>C;UGT1A7での387T>G、391C>A、392G<A、622T>C及び701T>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子群の機能的変異型の存在有無を確認する工程を含む、UGT1A族遺伝子群の機能的変異型を決定する方法を提供する。
【0031】
本発明のさらに他の目的を達成するために、本発明は、(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)で採取された試料から核酸を抽出する工程;(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族遺伝子を増幅する工程;及び(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での211G>A、233C>T及び686C>A;UGT1A6での19T>G、541A>G及び552A>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子変異型の存在有無を確認する工程を含む、イリノテカン(irinotecan)に対する感受性と関連したGT1A族遺伝子の多型性を決定する方法を提供する。
【0032】
本発明のさらに他の目的を達成するために、本発明は、(a)検査しようとする遺伝子を抽出した後、多重(multiplex)PCRを遂行して同定しようとするSNP周辺を含むPCR産物を得る工程;(b)各対立遺伝子(allele)の特異的な塩基を同定できるASPE(allele specific primer extension)プライマーを用いてASPE反応を遂行する工程;(c)前記反応産物を遺伝子チップに混成化させる工程;及び(d)前記チップを分析する工程を含む、遺伝子分析チップを用いてヒトCYP2D6遺伝子の遺伝型を決定する方法を提供する。
【0033】
また、本発明は、CYP2D6遺伝型の判別のためのSNaPshot方法の遺伝型分析用キット及びSNP検査用ジップ・コード(ZiP Code)オリゴ塩基チップを含む、遺伝型分析用チップを提供する。
【0034】
本発明で“生物学的試料”は、被験者の血液、皮膚細胞、粘膜細胞及び毛髪を含み、好ましくは血液であっても良い。
【0035】
本発明で核酸は、DNAまたはRNAであっても良く、好ましくはDNA、より好ましくはゲノム(genomic)DNAであっても良い。
【0036】
以下、本明細書に記載された変異について説明する。
本発明で用語“aN>M”または“NaM”(この時、aは整数、N及びMはそれぞれ独立的にA、C、TまたはGである。)は、遺伝子塩基配列でa番目N塩基がM塩基に置換されたことを意味し、“ainsN”または“adelN”(この時、aは整数、NはA、C、TまたはGである。)は、遺伝子塩基配列でa番目にN塩基がもう一つ挿入され、または欠失されたことを意味する。
【0037】
例えば、“−1584C>T”変異とは、ヒトCYP2D6遺伝子の塩基配列で−1584番目塩基がCからTに置換されたことをいう。
【0038】
“2573insC”変異とは、ヒトCYP2D6遺伝子の塩基配列で2573番目塩基にCが挿入(付加)されたことをいい、“4125〜4133insGTGCCCACT”変異とは、ヒトCYP2D6遺伝子の4125番目塩基から4133番目塩基の位置にGTGCCCACTの9個の塩基が挿入されたことをいう。
【0039】
また、“2D6欠失(deletion)”変異とは、ヒトCYP2D6遺伝子全体が染色体上から欠失されたことをいう。
【0040】
ひいては、本発明で“2D6重複(duplication)”変異とは、ヒトCYP2D6遺伝子が二以上同一の染色体上に重複していることをいう。
【図面の簡単な説明】
【0041】
【図1】CYP1A2遺伝子の塩基配列において、本発明で初めで糾明したCYP1A2遺伝子の1個の変異の位置を示したものである。
【図2】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの一例である。
【図3】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの多の一例である。
【図4】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの他の一例である。
【図5】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの他の一例である。
【図6】本発明で選別されたCYP1A2遺伝子のhtSNP組み合わせの他の一例である。
【図7】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの全ての単一塩基多型が野生型である遺伝子の変異を検索した結果を示す(X軸は各プライマーの分子量による移動程度、Y軸は各ピークの高さである。以下図14まで同様)。
【図8】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−3860G>A(CYP1A2*1C)、−2467delT(CYP1A2*1D)及び163C>A(CYP1A2*1F)の変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(hetero)変異遺伝子の場合の結果を示す。
【図9】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの3860G>A(CYP1A2*1C)、−2467delT(CYP1A2*1D)及び163C>A(CYP1A2*1F)の位置が二本のDNAの全てが変異型を有している同型接合型(homo)変異遺伝子である場合の結果を示す。
【図10】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)及び2808A>Cの位置が異型接合型変異遺伝子である場合の結果を示す。
【図11】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)は同型接合型変異、−2467delT(CYP1A2*1D)、−739T>G(CYP1A2*1E)、−3598G>T、−3113G>A、−2847T>C及び1708T>Cの位置は異型接合型変異遺伝子である場合の結果を示す。
【図12】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)は同型接合型変異、−2467delT(CYP1A2*1D)、−3598G>T及び2847T>Cの位置は異型接合型変異遺伝子である場合の結果を示す。
【図13】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)、−2467delT(CYP1A2*1D)及び3594T>Gの位置は異型接合型変異遺伝子である場合の結果を示す。
【図14】本発明の方法により選別されたCYP1A2遺伝子の機能的変異である、CYP1A2プロモーターの−163C>A(CYP1A2*1F)は同型接合型変異、−3860G>A(CYP1A2*C)、−2467delT(CYP1A2*1D)及び2603insAの位置は異型接合型変異遺伝子である場合の結果を示す。
【図15】本発明でhtSNP組み合わせを選別するために用いたCYP2A6の韓国人でのハプロタイプの種類及び頻度を示す。
【図16】本発明による、機能性変異6種類と機能性が疑われる3種類の変異遺伝子を遺伝子変異検査対象に含むCYP2A6遺伝子のハプロタイプを分析するためのhtSNP組み合わせの選別を例示したものである。
【図17】本発明による、アミノ酸置変異が8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含むCYP2A6遺伝子のハプロタイプを分析するためのhtSNP組み合わせの選別を例示したものである。
【図18】本発明による、アミノ酸置換変異8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含むCYP2A6遺伝子のハプロタイプを分析するための他のhtSNP組み合わせの選別を例示したものである。
【図19】本発明による、アミノ酸置換変異8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含むCYP2A6遺伝子のハプロタイプを分析するための他のhtSNP組み合わせの選別を例示したものである。
【図20】本発明による、アミノ酸置換変異8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含んでCYP2A6遺伝子のハプロタイプを分析するための他のhtSNP組み合わせの選別を例示したものである。
【図21】本発明による、アミノ酸置換変異8種類、CYP2A6遺伝子の欠失を標識する変異3種類及び6個の頻度が高いCYP2A6遺伝子変異を含んでCYP2A6遺伝子のハプロタイプを分析するための他のhtSNP組み合わせの選別を例示したものである。
【図22】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の−48T>G、2134A>G及び6558T>Cの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(hetero)変異遺伝子対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図23】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の567C>Tの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図24】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の6458A>T及び6558T>Cの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図25】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の−48T>G、13G>A及び6558T>Cの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図26】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の3391T>Cの変異位置が一本は変異型、他の一本は欠失されたタイプの異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図27】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の−48T>G及び2134A>Gの変異位置が一本は変異型、他の一本は欠失されたタイプの異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図28】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の−48T>G、6558T>C、及び6600G>Tの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図29】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の6458A>Tの変異位置が一本は変異型、他の一本は欠失されたタイプの異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図30】本発明で選別されたhtSNP組み合わせ及びCYP2A6遺伝子の6558T>C及び6582G>Tの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(Hetero)変異遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図31】CYP2A6遺伝子が相同染色体に正常に存在する遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図32】CYP2A6遺伝子が一つの染色体には存在せず、一つの遺伝子だけを有する遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図33】CYP2A6遺伝子が欠失されてCYP2A6遺伝子の一部の残った部分とCYP2A7遺伝子の接合形態にある遺伝子に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図34】本発明で選別されたCYP2D6のhtSNP組み合わせの例を示す。
【図35】本発明で選別されたCYP2D6のhtSNP組み合わせの他の例を示す。
【図36】本発明で選別されたCYP2D6のhtSNP組み合わせの他の例を示す。
【図37】本発明で選別されたCYP2D6のhtSNP組み合わせの他の例を示す。
【図38】本発明で選別されたCYP2D6のhtSNP組み合わせの例を示す。示したものである。
【図39】本発明で選別されたCYP2D6のhtSNP組み合わせの例を示す。
【図40】本発明で選別されたCYP2D6のhtSNP組み合わせの1種類に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図41】本発明で選別されたCYP2D6のhtSNP組み合わせの1種類に対してスナップショット(SNaPshot)分析を遂行した結果を示す。
【図42】遺伝子分析チップを用いてCYP2D6の遺伝型を分析する過程を示す。
【図43】CYP2D6の遺伝子分析チップ上のプローブを示す。
【図44】ロング(long)PCRを用いたCYP2D6遺伝子の増幅を示す。
【図45】ASPE反応過程を示す。
【図46】実施例12によりCYP2D6遺伝子の変異を分析した結果を示す遺伝子チップである。
【図47】本発明で選別されたPXR遺伝子のhtSNP組み合わせの例を示す。
【図48】本発明の方法により選別された−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cが全て野生型であるPXR遺伝子の機能的変異型を検索した結果を示す(X軸は各プライマーの分子量による移動程度、Y軸は各ピークの高さを示す。)。
【図49】本発明の方法により選別された−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cの変異位置が二本のDNAのうち、一本は変異型、他の一本は野生型を有している異型接合型(hetero)変異遺伝子であるPXR遺伝子の機能的変異型を検索した結果を示す。
【図50】本発明の方法により選別された−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cの位置が二本のDNAの全てが変異型を有している同型接合型(homo)変異遺伝子であるPXR遺伝子の機能的変異型を検索した結果を示す。
【図51】韓国人50名を対象としてUGT1A1(a)及びUGT1A3(b)族遺伝子の機能的変異型を分析した結果を示す(図中、Tはチアミン、Cはシトシン、Gはグアニン、Aはアデニンを意味する。)。
【図52】韓国人50名を対象としてUGT1A4(a)及びUGT1A6(b)族遺伝子の機能的変異型を分析した結果を示す。
【図53】韓国人50名を対象としてUGT1A7族遺伝子の機能的変異型を分析した結果を示す。
【図54】韓国人50名を対象としてUGT1A9族遺伝子の機能的変異型を分析した結果を示す。
【図55】韓国人50名を対象としてUGT1A1、UGT1A6及びUGT1A9族遺伝子のイリノテカン感受性関連多型性を分析した結果であり、(a)は、UGT1A1で211G>A、233C>T及び686C>A;UGT1A6で19T>G、541A>G及び552A>C;及びUGT1A9で726T>G及び766G>Aが全て野生型でC>Aが異型(hetero);UGT1A6で19T>G及び552A>Cが異型、541A>Gが野生型;及びUGT1A9で726T>G及び766G>Aが野生型である場合の、(c)は、UGT1A1で211G>A、233C>T及び686C>Aが野生型;UGT1A6で19T>G、541A>G及び552A>Cが異型;及びUGT1A9で726T>Gが異型、766G>Aが野生型である場合の、(d)は、UGT1A1で211G>A及び233C>Tが異型、686C>Aが野生型;UGT1A6で19T>G、541A>G及び552A>Cが異型;及びUGT1A9で726T>G及び766G>Aが野生型である場合の結果である。
【発明を実施するための形態】
【0042】
本発明の上記した特徴及び/または他の特徴は添付図面を参照した下記の実施態様の説明により明らかになり、また容易に理解できるであろう。
【0043】
1.CYP2A6
本発明は、韓国人CYP1A2遺伝子の変異分析を通じて韓国人において発見されるCYP1A2遺伝子の遺伝型を糾明し、これに基づいてそれぞれのハプロタイプの最適の標識セットであるhtSNPを選別し、その有用性を確認したという点に特徴がある。また、本発明は、ヒトCYP1A2遺伝子の新規なハプロタイプを糾明したという点に特徴がある。
【0044】
本発明によるヒトCYP1A2遺伝子のhtSNPを選別する方法は次の工程を含む:
(a)被験者から生物学的試料を採取する工程;
(b)前記(a)工程で採取された試料から核酸を抽出する工程;
(c)前記(b)工程の核酸を鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;
(d)前記(c)工程で得られたPCR産物の塩基配列を分析して変異の存在有無を確認する工程;及び
(e)前記(d)工程で変異が存在すると確認されたPCR産物の塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析する工程。
【0045】
前記(a)工程で採取された試料から核酸を抽出する方法は、特に限定されず、当業界に公知となった技術または市販されている抽出用キットを使用することができる。例えば、DNAまたはRNA抽出用キットはQiagen(米国)及びStratagene(米国)で購入できる。前記でRNAを抽出して使用する場合には逆転写によりcDNAを製造して使用する。
【0046】
前記(c)工程で前記ヒトCYP1A2遺伝子の断片は、ヒトCYP1A2遺伝子の公知となった変異、例えば単一塩基多型(single nucleotide polymorphism;SNP)を含んでいる断片をいう。前記ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーは、ヒトCYP1A2遺伝子またはその断片の塩基配列に基づいてデザインすることができ、例えば、配列番号2乃至配列番号31のプライマーからなる群より選択されることができるが、これに制限されない。
【0047】
前記(d)工程で変異は、単一塩基多型、遺伝子の欠失及び遺伝子の重複を含むが、これに制限されず、例えば、表5に示す17個の変異を含むことができる。
【0048】
また、塩基配列を分析する方法としては、特に限定されず、当業界に公知となった方法を使用することができる。例えば、その配列部分を直接決定するために、自動塩基配列分析器を使用するか、またはパイロシーケンシング(pyrosequencing)を遂行できる。前記パイロシーケンシングはDNAシーケンシングに利用されることもある公知のSNP分析方法として、DNAが重合される間に放出されるPPi(inorganic pyrophosphate)の光の発現を検出する方法である。前記塩基配列分析は、例えば、配列番号32乃至61のプライマーからなる群より選択されたプライマーを用いて遂行されることができるが、これに制限されない。
【0049】
また、前記(d)工程で変異の存在有無は、野生型CYP1A2遺伝子の塩基配列と比較して確認できる。野生型CYP1A2遺伝子の塩基配列、例えば、配列番号1(GenBank accession No.:NT_010194)の塩基配列、または当業界に公知となったCYP1A2遺伝型の各塩基配列に基づいて比較して確認できる(Drug Metab. Pharmacokinet,2005,20(1):24-33)。
【0050】
前記でハプロタイプの頻度及び種類を予測することは当業界で公知の技術プログラムまたは市販されるプログラムを使用して分析できる。例えば、Haploviewプログラムは無料で配布されるプログラムで利用でき、SNPAlyzeのような商用化されたプログラムを使用することもできる。前記 Haploviewソフトウェアは当業界に公知となっており、好ましくはインターネットウェブサイトである http://www.broad.mit.edu/mpg/haploview を利用することができる。
【0051】
本発明の方法は前記(a)乃至(d)工程を反復する工程を追加的に含むことができる。種族や患者などのようなある特定集団におけるCYP1A2遺伝子の変異様相及びこれに対するハプロタイプを分析しようとする場合には、各CYP1A2遺伝型の頻度を調査して前記集団で多く発見されるCYP1A2遺伝型を選定した後、これを対象として以降の(e)工程を遂行することができる。
【0052】
前記(e)工程では、前記(e)工程で予測されたCYP1A2のハプロタイプ資料をSNPタガーソフトウェアで分析してhtSNPを選別する。前記SNPタガーソフトウェアは当業界に公知となっており、例えば、Genehunter、 Merlin、Allegro、SNPHAP、htSNP finder(PCA based)などが挙げられ、好ましくは、http://www.well.ox.ac.uk/〜xiayi/haplotypeまたは http://slack.ser.man.ac.uk/progs/htsnp.htmlのウェブサイトを利用することができる。
【0053】
このように選別されたhtSNPはディプロタイプ(diplotype)決定時に正確度を高めるために検証され得る。人体の遺伝型は二本の染色体により決定されるので、遺伝型を判読すれば二つのハプロタイプ組み合わせで判定される。しかしながら、複数個のSNPを同時に分析した場合、特定のハプロタイプの組み合わせが他のハプロタイプの組み合わせと同一な変異分析結果を示す可能性がある。従って、本発明により開発された診断法を用いて遺伝型が判読された場合、正確な遺伝型を決定できることが検証されなければならない。このような検証は、Matlab(The Math Works Inc., 米国)プログラムを用いて遺伝子分析結果から正確な遺伝型判読が可能である否かが分析される。
【0054】
本発明の一実施例では、韓国人で発見されるCYP1A2遺伝型に対するhtSNPを選別するために、まず韓国人におけるCYP1A2遺伝子の変異を調査した。その結果、韓国人のCYP1A2遺伝子で総17個の単一塩基多型を発見した(表5参照)。その中の1個の単一塩基多型(−2603insA)は新規なものである。
【0055】
本発明で初めて提供される前記1個の単一塩基多型は二本のDNAのうち、一本は変異型、他の一本は野生型を有していることが示された(図1参照)。
【0056】
本発明の他の実施例では、韓国人で発見される17個の単一塩基多型によるハプロタイプを分析した。その結果、本発明では今まで明らかになったことのないCYP1A2遺伝子に対する韓国人のハプロタイプ(表6参照)及びこれに基づいた遺伝型を糾明した。例えば、表6に記載されたCYP1A2遺伝子のハプロタイプ2(CYP1A2*1L)は、CYP1A2遺伝子の塩基配列において−3860、−2467及び163塩基位置に単一塩基多型がある遺伝型をいうものとして、より具体的には−3860G>A、−2467T>delT(−2467delT)及び163C>Aの単一塩基多型を有することを示す。
【0057】
本発明の他の実施例では、韓国人で発見されたCYP1A2遺伝子の変異による17種のハプロタイプに対する最小マーカであるhtSNPを選別するために、CYP1A2遺伝子の変異を有している塩基配列を用いて分析されたハプロタイプに基づいてSNPタガーソフトウェアを用いて分析した。本発明で選別されたhtSNP組み合わせの例を図2乃至図6に示した。
【0058】
前記方法により本発明で選別されたhtSNP組み合わせは、ヒトCYP1A2遺伝子のハプロタイプを分析することに使用することができる。従って、本発明はヒトCYP1A2遺伝子のハプロタイプを決定する方法を提供する。前記方法は次の工程を含む:
【0059】
(a)被験者から生物学的試料を採取する工程;
(b)前記(a)工程の採取された試料から核酸を抽出する工程;
(c)前記(b)工程の核酸を鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び
(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G、−163C>A、1514G>A、2159G>A、2321G>C、3613T>C、5347C>T及び5521A>Gからなる群より選択されるCYP1A2遺伝子の変異の存在有無を調査する工程。
【0060】
前記(b)工程で核酸を抽出する方法は前記の通りである。
前記ヒトCYP1A2遺伝子の断片は、前記の通り、ヒトCYP1A2遺伝子の公知となった単一塩基多型を含む断片をいう。前記(c)工程で使用され得るプライマーは、これに制限されないが、配列番号2乃至配列番号31からなる群より選択される。
【0061】
前記(d)工程で調査される単一塩基多型は、図4乃至図6に示すhtSNPの中で選択することができ、図4に示す−3860G>A、−3598G>T、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型;図5に示す−3860G>A、−3113G>A、−2808A>C、−2603insA、−2467delT、−739T>G、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型;及び図6に示す−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型または−3860G>A、−3598G>T2321G>C、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査することもできる。
【0062】
このように前記(d)工程で調査される単一塩基多型は、韓国人で発見されるCYP1A2遺伝子変異に関するものであるため、韓国人CYP1A2遺伝子のハプロタイプと遺伝型の決定に非常に特異的である。
【0063】
前記(d)工程で前記PCR産物の塩基配列にCYP1A2遺伝子の単一塩基多型が存在するか否かの調査は、当業界で公知の多型性分析方法を用いて遂行することができる。好ましくはスナップショット分析([Peter M. Vallone,et al.,Int J Legal Med,2004,118:147-157]参照)、電気泳動分析またはこれらの組み合わせ、より好ましくはスナップショット分析を用いて遂行することができる。
【0064】
前記スナップショット分析はSNP位置の隣接部位にアニーリング(annealing)される配列(前記SNP部位は含まない)を有するプライマーとddNTPを用いたPCR反応を通じて遺伝子型を分析する方法である。本発明に使用されるスナップショット分析は、前記工程(c)で調査されるCYP1A2遺伝子のSNPに基づいて公知の方法により設計及び製作したものを使用することができ、SNP位置のすぐそばの塩基が3'末端となり、前記SNP位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたものであれば制限なしに使用されることができ、好ましくは配列番号64乃至74のプライマーからなる群より選択されるプライマーを使用することができる。この時、前記SNP位置の隣接部位にアニーリングされる配列は、約20bpの長さを有することが好ましく、同時に複数個のSNPを決定しようとする場合、各SNPに対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計、例えばT塩基を5個ずつ5'位置にさらに添加し、プライマー間にサイズ差異を作り合成してPCR産物の長さをそれぞれ異なるようにすることができる。このように作られたスナップショットプライマーに各SNPに相補的なddNTPが結合するようになり、これら合成物はSNPにより長さの差異が発生することによって同時に複数個のSNPの決定が可能である。
【0065】
その後、スナップショット分析で判定された遺伝型診断結果が正確であるかを検査するために、遺伝型が分かる他の遺伝子分析法を通じた結果との一致性を調査することができるが、ここで他の遺伝子分析法とは、特別に限定されないが、好ましくは自動塩基配列分析法またはパイロシーケンシング法であっても良い。
【0066】
本発明により韓国人で発見されたCYP1A2遺伝子の変異総17個の中で11個の単一塩基多型はプロモーター領域に位置する。前記11個の単一塩基多型には本発明で初めて糾明した−2603insA変異が含まれる。従って、本発明ではヒトCYP1A2プロモーター遺伝子の変異を分析する方法を提供する。前記方法は次の工程を含む:
【0067】
(a)被験者から生物学的試料を採取する工程;
(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;
(c)前記(b)工程のゲノムDNAを鋳型とし、ヒトCYP1A2遺伝子のプロモーター領域を増幅できるプライマーを用いてPCRを遂行する工程;及び
(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G及び163C>AからなるCYP1A2遺伝子の変異の存在有無を調査する工程。
【0068】
前記(b)工程で核酸を抽出する方法は前記の通りである。
前記(c)工程でヒトCYP1A2遺伝子のプロモーター領域を増幅できるプライマーは、配列番号1の−3860G>Aから−163C>Aまでの単一塩基多型を増幅できるプライマーであれば、制限なしに使用することができ、好ましくは配列番号62及び配列番号63のプライマーを使用することができる。
【0069】
前記(d)工程で前記PCR産物の塩基配列にCYP1A2遺伝子の単一塩基多型が存在するか否かの調査は、当業界に公知となった多型性分析方法を用いて遂行することができる。好ましくはスナップショット分析を用いて遂行することができる。本発明に使用されるスナップショット分析は、前記11個のCYP1A2遺伝子の単一塩基多型に基づいてデザインされたプライマーを用いて遂行することができる。本発明に使用されるスナップショットプライマーは、単一塩基多型を含まない隣接した配列の塩基配列が含まれるようにデザインされたものであれば、制限なしに使用することができる。好ましくは配列番号64乃至配列番号74からなる群より選択される塩基配列を有するプライマーを使用することができる。
【0070】
本発明の他の実施例では、CYP1A2酵素活性に影響を与えるプロモーター部分の11個の単一塩基多型を用いてCYP1A2プロモーター遺伝子の変異をSNaPshot分析により検出した。その結果、本発明の方法によりCYP1A2プロモーター遺伝子の変異を高速に正確に検出できることが確認された(図7乃至図14参照)。
【0071】
2.CYP2A6
本発明は韓国人CYP2A6遺伝子の変異分析を通じて韓国人で主に発見されるCYP2A6遺伝子の遺伝型を糾明し、これに基づいてそれぞれのハプロタイプの最適の標識セットであるhtSNPを選別し、その有用性を確認したという点に特徴がある。
本発明によるヒトCYP2A6遺伝子のhtSNPを選別する方法は次の工程を含む:
【0072】
(a)被験者から生物学的試料を採取する工程;
(b)前記工程(a)の採取された試料から核酸を抽出する工程;
(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;
(d)前記工程(c)で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;
(e)前記工程(d)で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプを分析する工程;及び
(f)前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype/)を用いて分析してhtSNPを選別する工程。
【0073】
前記工程(a)で採取された試料から核酸を抽出する方法は特に限定されず、当業界に公知となった技術または市販されている抽出用キットを使用することができる。例えば、DNAまたはRNA抽出用キットはQiagen(米国)及びStratagene(米国)などで購入できる。前記でRNAを抽出して使用する場合には逆転写によりcDNAを製造して使用する。
【0074】
前記工程(c)で前記ヒトCYP2A6遺伝子の断片は、ヒトCYP2A6遺伝子の公知の変異、例えば単一塩基多型(single nucleotide polymorphism;SNP)を含む断片をいう。ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーはヒトCYP2A6遺伝子またはその断片の塩基配列に基づいてデザインすることができ、例えば、配列番号76乃至89のプライマーからなる群より選択することができるが、これに制限されない。
【0075】
前記工程(d)で確認される変異は、SNP、遺伝子の欠失及び遺伝子の重複を含むが、これに制限されず、例えば、表15に示す30個の変異を含むことができる。
【0076】
また、前記工程(d)で変異の存在有無の確認は当業界に公知となった変異検出方法により遂行されることができ、好ましくは配列分析、電気泳動分析を用いて当業界に公知となっている野生型CYP2A6遺伝子の塩基配列、例えば、配列番号75(GenBank accession No.:NC_000019)の塩基配列、または当業界に公知のCYP2A6遺伝型の各塩基配列に基づいて比較することによって遂行したり、RFLP分析などを用いて野生型CYP2A6遺伝子の制限酵素切断様相と比較することによって遂行することができる。また、CYP2A6遺伝子の欠失または重複変異の場合にはPCR産物の電気泳動分析を通じて確認できる。前記配列分析は自動塩基配列分析器を使用して、またはパイロシーケンシング(pyrosequencing)を用いて遂行できる。
【0077】
前記工程(e)で変異が存在することが確認されたPCR産物の塩基配列中のハプロタイプは、SNPAlyzeやHaplotyper、Arlequinなどのプログラムを使用して分析できる。
【0078】
本発明の方法は、前記工程(a)乃至(e)を反復する工程を追加的に含むことができる。種族や患者などのようなある特定集団におけるCYP2A6遺伝子の変異様相を分析しようとする場合には、各CYP2A6遺伝型の頻度を調査して前記集団で多く発見されるCYP2A6遺伝型を選定した後、これを対象として以降の工程(f)を遂行できる。
【0079】
前記工程(f)では前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアで分析してhtSNPを選別し、htSNPの選別に用いられるソフトウェアとしてはSNPtagger以外にも、HapBlock、LDSelect、Haploview、htSNP、TagIT、tagSNPsなど多様なものが使用され得る。前記SNPtaggerソフトウェアは当業界に公知となっており、好ましくはhttp://www.well.ox.ac.uk/〜xiayi/haplotype/を用いることができる。このように選別されたhtSNPは、正確度を高めディプロタイプ(diplotype)を決定できることが検証された。前記検証はMatlab(The Math Works Inc., 米国)を用いて遂行することができる。
【0080】
本発明の一実施例では、韓国人のCYP2A6遺伝型に対するhtSNPを選別するために、まず韓国人で発見されるCYP2A6遺伝子の変異を調査した。その結果、韓国人のCYP2A6遺伝子で総30個のSNPを発見した(表15参照)。
【0081】
本発明の他の実施例では、前記で選別された30個のSNPのうち、機能性遺伝子変異を含むアミノ酸置換を招く変異8個及び頻度が高い変異6個を含む14個SNPに対してDYNACOM社のSNPAlyzeプログラムを用いてハプロタイプを分析し、総19個の1%以上頻度のハプロタイプを確認することができた。ハプロタイプの分析に用いられるプログラムは、前記SNPAlyzeプログラムに制限されず、Haplotyper(http://www.people.fas.harvard.edu/〜junliu/Haplo/docMain.htm)、Arlequin(http://lgb.unife.ch/arlequin)、イズテック社のSNP Analyzer(http://www.istech21.com/)など、多くの類似ソフトウェアが当業界に公知となっている
【0082】
本発明のさらに他の実施例では、韓国人で主に発見されるCYP2A6遺伝子の遺伝型を容易に決定できる最小マーカであるhtSNPを選別するために、前記19個のハプロタイプと遺伝子欠失の場合を含む20種類のハプロタイプに対する塩基配列及び頻度をSNPタガーソフトウェアを用いて分析してhtSNPを選別し、その例を図16乃至21に示した。
【0083】
前記方法により本発明で選別されたhtSNP組み合わせはヒトCYP2A6遺伝子の遺伝型を分析することに使用され得る。従って、本発明はヒトCYP2A6遺伝子の遺伝型を決定する方法を提供する。前記方法は次の工程を含む:
【0084】
(a)被験者から生物学的試料を採取する工程;
(b)前記工程(a)の採取された試料から核酸を抽出する工程;
(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び
(d)前記工程(c)で得られたPCR産物の塩基配列において、−48T>G、13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>T、及び6091C>Tからなる群より選択されるCYP2A6遺伝子変異の存在有無を調査する工程。
【0085】
前記工程(b)で核酸を抽出する方法は前記の通りである。
前記ヒトCYP2A6遺伝子の断片は、前記の通り、ヒトCYP2A6遺伝子の公知となった変異、例えばSNPを含んでいる断片をいう。前記工程(c)で用いられるプライマーは、これに制限されないが、配列番号90、91、120及び103のプライマーであっても良い。
【0086】
前記工程(d)で調査される変異は、図16乃至図21に示すhtSNPの中で選択され得る。例えば、前記工程(d)では図16に示す−48T>G;22C>T;567C>T;2134A>G;3391T>C;6458A>T;6558T>C;6582G>T;6600G>T;6091C>T、5971G>A及び5983T>Gの中の一つ;及び13G>A;51G>A;1620T>C;及び1836G>Tからなる変異の存在有無を調査でき、
【0087】
図17に示す−48T>G;22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6458A>T;6558T>C;6600G>T;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、
【0088】
図18に示す22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6354T>C;6458A>T;6558T>C;6600G>T;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、
【0089】
図19に示す−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>C;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、
【0090】
図20に示す−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6458A>T;6558T>C;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、
【0091】
図21に示す−48T>G;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>C;6600G>T;及び6091C>T、5971G>A及び5983T>Gの中の一つからなる変異の存在有無を調査でき、好ましくは図16に例示した変異の存在有無を調査できる。
【0092】
図16に例示した変異の中で機能性が立証されるか、機能性が最も有力に予想される変異は、アミノ酸を置換する変異または遺伝子の欠失を招く変異である。従って、図16の変異の中で機能性CYP2A6変異を検出するためには、最も好ましくは、図16の例示変異の中でアミノ酸置換変異や遺伝子欠失変異が調査される。遺伝子欠失の場合、単一塩基多型により判別し難いため、遺伝子欠失を標識できる変異を探さなければならないが、この過程で6091C>T変異を発見した。6091C>T変異は、前記工程(c)で増幅されるPCR産物の中でCYP2A6遺伝子が欠失された染色体で特異的に発見される単一塩基多型であり、遺伝子欠失に対する標識変異として用いることができる。このような機能性の判別目的により選定された変異組み合わせは、−48T>G;13G>A;567C>T;2134A>G;3391T>C;6458A>T;6558T>C;6582G>T;6600G>T;及び6091C>Tの10個の変異である。6091C>T変異の代わりに遺伝子欠失を標識する他の変異として、CYP2A6遺伝子基準に5971G>A及び5983T>Gを用いても良い。
【0093】
前記工程(d)で調査される変異は、韓国人で主に発見されるCYP2A6遺伝子変異に対するものであるので、韓国人のCYP2A6遺伝子のハプロタイプと遺伝型の決定に非常に特異的である。
【0094】
前記工程(d)で前記PCR産物の塩基配列にCYP2A6遺伝子の変異が存在するか否かの調査は、当業界に公知となった多型性分析方法を用いて遂行することができる。スナップショット分析([Peter M. Vallone,et al.,Int J Legal Med,2004,118:147-157]参照)、電気泳動分析またはこれらの組み合わせ、最も好ましくはスナップショット分析が変異を調査するために用いられる。
【0095】
前記スナップショット分析はSNP位置の隣接部位にアニーリング(annealing)される配列(前記SNP部位は含まない)を有するプライマーとddNTPを用いたPCR反応を通じて遺伝子型を分析する方法である。本発明に使用されるスナップショット分析は、前記工程(c)で調査されるCYP2A6遺伝子のSNPに基づいて公知となった方法により設計及び製作したものを使用することができ、SNP位置のすぐそばの塩基が3'末端となり、前記SNP位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたものであれば制限なしに使用されることができ、好ましくは配列番号97乃至102のプライマーからなる群より選択されるプライマーを使用することができる。この時、前記SNP位置の隣接部位にアニーリングされる配列は約20bpの長さを有することが好ましく、同時に複数個のSNPを決定しようとする場合、各SNPに対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計、例えばT塩基を5個ずつ5'位置にさらに添加してプライマー間にサイズ差異を作って合成し、PCR産物の長さをそれぞれ異なるようにすることができる。このように作られたスナップショットプライマーに各SNPに相補的なddNTPが結合するようになり、これら合成物はSNPにより長さの差異が発生することによって同時に複数個のSNPの決定が可能である。
【0096】
その後、スナップショット分析のために増幅されたPCR産物の塩基配列を当業界に公知となった配列分析法で分析でき、特別に限定されないが、好ましくは自動塩基配列分析法であっても良い。
【0097】
例えば、前記工程(c)で図16に示すhtSNP組み合わせを調査する場合には、配列番号92乃至配列番号101からなる群より選択される塩基配列を有するプライマーを使用することができ、好ましくは配列番号92乃至配列番号101のプライマーを全て使用することができるが、これに限定されない。
【0098】
その後、スナップショット分析で増幅されたPCR産物の塩基配列を当業界に公知となった配列分析法で分析でき、特別に限定されないが、好ましくは自動塩基配列分析法であっても良い。
【0099】
本発明のさらに他の実施例では、本発明で選別されたhtSNP組み合わせの有用性を確認した。このために図16に示すhtSNP組み合わせの中で10個の機能性または機能性予想CYP2A6変異を選定してスナップショット分析を実施するために、工程(c)で得られたPCR産物の塩基配列を分析した。その結果、本発明の方法が韓国人で発見されるCYP2A6遺伝型を同時に高速に分析できることを確認した(図22乃至図32参照)。
【0100】
本発明の方法により分析されることができるCYP2A6遺伝子の遺伝型は、−48T>G、13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>T、及び6091C>Tを含み、各遺伝型及びこれによる変異について図22乃至32に示した。例えば、図22を参照して説明すれば、−48T>G、6558T>C、2134A>Gの3個の位置で変異を有しており、残りの7個の位置では野生型を示す遺伝型である。図23は567C>T位置だけで変異を有しており、残りの9個の位置では野生型を有する遺伝型である。また、CYP2A6*4遺伝型は2A6欠失変異を含む遺伝型であり、CYP2A6遺伝子がヒト染色体上で欠失されて酵素生産が全く生じない。CYP2A6遺伝子が欠失された場合、遺伝子の形態はCYP2A6遺伝子の一部とCYP2A7の一部が互いに連結された形態をしているので、この部分を調査して欠失に特異的な変異を探すことができる。
【0101】
3.CYP2D6
本発明は韓国人CYP2D6遺伝子の変異分析を通じて韓国人で主に発見されるCYP2D6遺伝子の遺伝型を糾明し、これに基づいてそれぞれのハプロタイプの最適の標識セットであるhtSNPを選別し、その有用性を確認したという点に特徴がある。
【0102】
本発明によるヒトCYP2D6遺伝子のhtSNPを選別する方法は次の工程を含む:
(a)ヒトから生物学的試料を採取する工程;
(b)前記(a)工程の採取された試料から核酸を抽出する工程;
(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;
(d)前記(c)工程で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;
(e)前記(d)工程で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプ(haplotype)を分析する工程;及び
(f)前記(e)工程で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアを用いて分析してhtSNPを選別する工程。
【0103】
前記(a)工程で採取された試料から核酸を抽出する方法は、特に限定されず、当業界に公知となった技術または市販されている抽出用キットを使用することができる。例えば、DNAまたはRNA抽出用キットは、Qiagen(米国)及びStratagene(米国)で購入できる。前記でRNAを抽出して使用する場合には逆転写によりcDNAを製造して使用する。
【0104】
前記(c)工程で前記ヒトCYP2D6遺伝子の断片は、ヒトCYP2D6遺伝子の公知となった変異、例えば単一塩基多型(single nucleotide polymorphism ;SNP)を含んでいる断片をいう。前記ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーはヒトCYP2D6遺伝子またはその断片の塩基配列に基づいてデザインすることができ、例えば、配列番号106、配列番号107、配列番号121乃至配列番号127、配列番号129乃至配列番号136、配列番号138、配列番号139、配列番号149及び配列番号150からなる群より選択される塩基配列を有するものであっても良いが、これに制限されない。
【0105】
前記(d)工程で確認される変異は、単一塩基多型、遺伝子の欠失及び遺伝子の重複を含むが、これに制限されず、例えば、表34に示す33個の変異を含むことができる。
【0106】
また、前記(d)工程で変異の存在有無の確認は、当業界に公知となった変異検出方法により遂行することができ、好ましくは配列分析、電気泳動分析、RFLP分析などを用いて遂行することができる。前記配列分析は自動塩基配列分析器を使用するかパイロシーケンシング(pyrosequencing)を用いて遂行することができる。前記パイロシーケンシングはDNAシーケンシングに利用されることもある公知のSNP分析方法によりDNAが重合される間に放出されるPPi(inorganic pyrophosphate)の光の発現を検出する方法である。
【0107】
変異の存在有無は野生型CYP2D6遺伝子の塩基配列と比較して確認できる。野生型CYP2D6遺伝子の塩基配列は当業界に公知となっている。例えば、配列番号105(GenBank accession No. AY545216)の塩基配列、または当業界に公知となったCYP2D6遺伝型の各塩基配列または情報に基づいて比較して確認できる(GenBank accession No. M33388,http://www.cypalleles.ki.se/cyp2d6.htm)。または、RFLPを遂行して野生型CYP2D6遺伝子の制限酵素切断様相と比較して確認することもできる。CYP2D6遺伝子の欠失または重複変異の場合にはPCR産物の電気泳動分析を通じて確認できる。
【0108】
前記(d)工程で変異が存在することが確認されたPCR産物の塩基配列中のハプロタイプの分析は全長塩基配列を通じて遂行できる。
【0109】
本発明の方法は前記(a)乃至(e)工程を反復する工程を追加的に含むことができる。種族や患者などのようなある特定集団でCYP2D6遺伝子の変異様相を分析しようとする場合には、各CYP2D6遺伝型の頻度を調査して前記集団で多く発見されるCYP2D6遺伝型を選定した後、これを対象として以下の(f)工程を遂行できる。
【0110】
前記(f)工程では、前記(e)工程で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアで分析してhtSNPを選別する。前記SNPタガーソフトウェアは当業界に公知となっており、例えば Genehunter、Merlin、Allegro、SNPHAP、htSNP finder(PCA based)などが挙げられ、好ましくは、http://www.well.ox.ac.uk/〜xiayi/haplotypeまたは http://slack.ser.man.ac.uk/progs/htsnp.htmlのウェブサイトを利用することができる。
【0111】
このように選別されたhtSNPはディプロタイプ(diplotype)決定時に正確度を高めるために検証され得る。人体の遺伝型は二本の染色体により決定されるので、遺伝型を判読すれば二つのハプロタイプ組み合わせで判定される。しかしながら、複数個のSNPを同時に分析した場合、特定のハプロタイプの組み合わせが他のハプロタイプの組み合わせと同一の変異分析結果を示す可能性がある。従って、本発明により開発された診断法を用いて遺伝型が判読された場合、正確な遺伝型を決定できることが検証されなければならない。このような検証は、Matlab(The Math Works Inc., 米国)プログラムを用いた遺伝子分析結果から正確な遺伝型判読が可能であるか否かを分析することによって遂行される。
【0112】
本発明の一実施例では韓国人で発見されるCYP2D6遺伝型に対するhtSNPを選別するために、まず韓国人で発見されるCYP2D6遺伝子の変異を調査した。その結果、韓国人で主に発見される33個の変異とこれによる12種のハプロタイプ(遺伝型)を糾明した(表34及び35参照)。
【0113】
本発明の他の実施例では韓国人で主に発見されるCYP2D6遺伝型を容易に決定できる最小マーカであるhtSNPを選別するために、12種のCYP2D6遺伝型の塩基配列をSNPtaggerソフトウェアを用いて分析した。本発明で選別されたhtSNP組み合わせの例を図34乃至図39に示した。
【0114】
前記方法により本発明で選別されたhtSNP組み合わせは、ヒトCYP2D6遺伝子の遺伝型を分析することに使用され得る。従って、本発明はヒトCYP2D6遺伝子の遺伝型を決定する方法を提供する。前記方法は次の工程を含む:
【0115】
(a)ヒトから生物学的試料を採取する工程;
(b)前記(a)工程の採取された試料から核酸を抽出する工程;
(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び
(d)前記(c)工程で得られたPCR産物の塩基配列において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複からなる群より選択される一つを含む少なくとも11個のCYP2D6遺伝子変異の存在有無を調査する工程。
【0116】
前記(b)工程で核酸を抽出する方法は前記の通りである。
前記ヒトCYP2D6遺伝子の断片は、前記の通り、ヒトCYP2D6遺伝子の公知となった変異、例えば単一塩基多型を含んでいる断片をいう。前記(c)工程で使用され得るプライマーは、これに制限されないが、配列番号106、配列番号107、配列番号121乃至配列番号127、配列番号129乃至配列番号136、配列番号138、配列番号139、配列番号149及び配列番号150からなる群より選択される塩基配列を有するものであっても良い。
【0117】
前記(d)工程で調査される変異は、図34乃至図39に示すhtSNPの中で選択され得る。前記(d)工程では、図34に示す−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複からなる一つを含む変異の存在有無を調査できる。
【0118】
また、図35に示す−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>C、3877G>A、4388C>T及び4401C>Tからなる群より一つ;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査することもできる。
【0119】
また、図36に示す−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1584C>G;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複から選択される一つを含む変異の存在有無を調査することもできる。
【0120】
また、図37に示す−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;4125〜4133insGTGCCCACT;−1235A>G;1887insTA;2D6欠失;及び2D6重複を含む変異の存在有無を調査することもできる。
【0121】
また、図38に示す−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;1611T>A;1661G>C及び4180G>Cからなる群より一つ;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;−1235A>G;1887insTA;2D6欠失;及び2D6重複から選択される一つを含む変異の存在有無を前記(d)工程で調査することもできる。
【0122】
また、図39に示す−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;1887insTA;2988G>A;4125−4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査することもできる。
【0123】
好ましくは図34に例示した変異の存在有無を調査できる。このように前記(d)工程で調査される変異は、韓国人で主に発見されるCYP2D6遺伝子変異に関するものであるので、韓国人のCYP2D6遺伝子のハプロタイプと遺伝型の決定に非常に特異的である。
【0124】
前記(d)工程で前記PCR産物の塩基配列にCYP2D6遺伝子の変異が存在するか否かの調査は、当業界に公知となった多型性分析方法を用いて遂行できる。好ましくはスナップショット分析([Peter M. Vallone,et al.,Int J Legal Med,2004,118:147-157]参照)、電気泳動分析またはこれらの組み合わせを用いて遂行できる。前記CYP2D6遺伝子の変異が単一塩基多型である場合にはスナップショット分析を利用することができる。
【0125】
前記スナップショット分析は、SNP位置の隣接部位にアニーリング(annealing)される配列(前記SNP部位は含まない)を有するプライマーとddNTPを用いたPCR反応を通じて遺伝子型を分析する方法である。本発明に使用されるスナップショット分析は、前記工程(c)で調査されるCYP2D6遺伝子のSNPに基づいて公知の方法により設計及び製作したものを使用することができ、SNP位置のすぐそばの塩基が3'末端となり、前記SNP位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたものであれば制限なしに使用することができる。この時、前記SNP位置の隣接部位にアニーリングされる配列は約20bpの長さを有することが好ましく、同時に複数個のSNPを決定しようとする場合、各SNPに対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計、例えばT塩基を5個ずつ5'位置にさらに添加してプライマー間のサイズに差を作って合成し、PCR産物の長さをそれぞれ異なるようにすることができる。このように作られたスナップショットプライマーに各SNPに相補的なddNTPが結合するようになり、これら合成物はSNPにより長さの差異が発生することによって同時に複数個のSNPの決定が可能である。
【0126】
例えば、前記(c)工程で図34に示すhtSNP組み合わせを調査する場合には、配列番号141乃至配列番号148、配列番号152及び配列番号153からなる群より選択される塩基配列を有するプライマーを使用することができ、好ましくは配列番号141乃至配列番号148、配列番号152及び配列番号153のプライマーを全て使用することができる。その後、スナップショット分析で増幅されたPCR産物の塩基配列を公知となった配列分析法で分析できる。前記配列分析法としては当業界に公知となった方法であれば制限なしに使用されることができ、好ましくは自動塩基配列分析法であっても良い。
【0127】
本発明のさらに他の実施例では、本発明で選別されたhtSNP組み合わせの有用性を確認した。このために図34に示すhtSNP組み合わせを用いてスナップショット分析を実施した後、得られたPCR産物の塩基配列を分析した。その結果、本発明の方法が韓国人で発見されるCYP2D6遺伝型を同時に高速で分析できることを確認した(図40及び41参照)。
【0128】
本発明の方法により分析することができるCYP2D6遺伝子の遺伝型は、CYP2D6*1A、CYP2D6*2A、CYP2D6*5、CYP2D6*2N、CYP2D6*10B、CYP2D6*14B、CYP2D6*18、CYP2D6*21B、CYP2D6*41A、CYP2D6*49、CYP2D6*52及びCYP2D6*60を含む。各遺伝型及びこれによる変異を表34に示した。表34を参照して説明すれば、例えばCYP2D6*1A遺伝型は野生型であり、CYP2D6*2A遺伝型は野生型CYP2D6遺伝子の塩基配列でSNP1、SNP5、SNP8、SNP9、SNP12−SNP18、SNP21、SNP25及びSNP28位置に変異を含む遺伝型である。また、CYP2D6*5遺伝型は2D6欠失変異を含む遺伝型として、CYP2D6遺伝子がヒト染色体上で完全欠如したものであって酵素生産が全く生じない。CYP2D6*2N遺伝型は2D6重複変異を含む遺伝型として、CYP2D6遺伝子が2個以上同一染色体に存在する。
【0129】
また、本発明は遺伝子分析チップを用いてヒトCYP2D6遺伝子の遺伝型を決定する方法を提供する。前記方法は次の工程を含む:
【0130】
(a)検査しようとする遺伝子を抽出した後、多重(multiplex)PCRを遂行して同定しようとするSNP周辺を含むPCR産物を得る工程;
(b)各対立遺伝子(allele)の特異的な塩基を同定できるASPE(allele specific primer extension)プライマーを用いてASPE反応を遂行する工程;
(c)前記反応産物を遺伝子チップに混成化させる工程;及び
(d)前記チップを分析する工程。
また、本発明はSNP検査用ジップ・コード(ZiP Code)オリゴ塩基チップを含む遺伝型分析用キットを提供する(図42参照)。
【0131】
前記工程(b)で、ASPE反応のためにはそれぞれのSNPに合う一対のプライマーを製作するようになるが、前記ASPEプライマーは多型性を示す塩基(SNP site)を3’末端に含んで対立遺伝子(allele)と特異的に結合するシークエンスで製作され、5’方向には24bpのオリゴヌクレオチドであるZiP Codeが含まれており、前記ZiP Codeはそれぞれの対立遺伝子ごとに異なる配列を有するように製作される。
【0132】
本発明では、論文などを通じて公開された配列及び生命情報学技法を通じて選別され設計された配列のうち、実験的検証を通じて他の試料と相互交差反応が起こらない最適のZiP Code配列を選別し、選別された配列はTM値が61℃±2であり、ZiP Codeの相互間に干渉がないように製作され、ヘアピン二次構造のΔG値が−2以上である配列だけが選択された。
【0133】
前記のように構成されたASPEプライマーを用いてASPE反応を遂行すれば、プライマーの3’末端に該当する対立遺伝子を有している試料だけがプライマーと反応して対立遺伝子特異的延長反応が生じるようになる。この時、シアニン5(Cyanine 5、CY5)蛍光物質が共有結合されたdUTP(Cy5-dUTP)を混合して延長反応を遂行すれば、各対立遺伝子を有している試料だけがCy5蛍光物質を標識するようになる(図45参照)。この時、蛍光物質はCy5に制限されず、Cy3、TAMRA、Texas−Red、Cy3.5、Rhodamin 6G、SyBR Greenなど、当該分野で使用可能な種々のものが代替して使用され得る。
【0134】
本発明の分析チップ上には、前記ZiP Codeと相補的に結合するオリゴヌクレオチドをプローブ(cZip Code)が植えられており、前記のようにZiP Codeプライマーを用いて延長された試料に含まれている各対立遺伝子を同定できるようになる(図43参照)。
【0135】
前記プローブは3’方向に10bpの塩基配列をスペーサで挿入してターゲットと交雑反応がよく生じるように誘導し、例えば、前記スペーサ配列は5’−CAG GCCA AGT−3’であることが好ましい。
【0136】
また、本発明において、前記プローブは配列番号158乃至配列番号184の塩基配列を有することが好ましい。
【0137】
前記工程(c)及び(d)で前記反応産物を遺伝子チップに混成化させ、混成化されたチップを分析する方法は、当業界に公知となった方法により遂行されることができ、一般的なDNAチップスキャナーは如何なるものでも使用することができる。より好ましくは、Axon社のGenePix 4100Bスキャナーを用い、スキャニングされたイメージをGenePix Pro 6.0 softwareを用いて分析できる。
【0138】
本発明の遺伝子分析チップを用いてCYP2D6遺伝子の変異を分析すれば、配列分析を通じて確認された結果と同一の結果を得ることができる。従って、本発明の遺伝子分析チップによれば多様な遺伝子の変異を経済的に容易に分析することができる。
【0139】
4.PXR
本発明の方法は韓国人のPXR遺伝子の変異に基づいて選別された、htSNPを用いてPXR遺伝子の機能的変異型を分析することを特徴とする。
本発明によるヒトPXR遺伝子のhtSNPを選別する方法は次の工程を含む:
【0140】
(a)ヒトから生物学的試料を採取する工程;
(b)前記工程(a)の採取された試料から核酸を抽出する工程;
(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;
(d)前記工程(c)で得られたPCR産物の塩基配列を分析して変異の存在有無を確認する工程;
(e)前記工程(d)で変異が存在することが確認されたPCR産物の塩基配列中のハプロタイプを分析する工程;及び
(f)前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアを用いて分析してhtSNPを選別する工程。
【0141】
前記工程(a)で採取された試料から核酸を抽出する方法は、特別に限定されず、当業界に公知となった技術または市販されている抽出用キットを使用することができる。例えば、DNAまたはRNA抽出用キットは、Qiagen(米国)及びStratagene(米国)などで購入できる。前記でRNAを抽出して使用する場合には逆転写によりcDNAを製造して使用する。
【0142】
前記工程(c)で前記ヒトPXR遺伝子の断片は、ヒトPXR遺伝子の公知となった変異、例えば単一塩基多型(single nucleotide polymorphism;SNP)を含んでいる断片をいう。前記ヒトPXR遺伝子またはその断片を増幅できるプライマーは、ヒトPXR遺伝子またはその断片の塩基配列に基づいてデザインすることができ、例えば、配列番号221乃至240のプライマーからなる群より選択することができるが、これに制限されない。
【0143】
前記工程(d)で確認される変異は、単一塩基多型、遺伝子の欠失及び遺伝子の重複を含むが、これに制限されず、例えば、表48に示す22個の変異を含むことができる。
【0144】
また、前記工程(d)で変異の存在有無の確認は、当業界に公知となった変異検出方法により遂行でき、好ましくは配列分析、電気泳動分析、RFLP分析などを用いて遂行できる。前記配列分析は自動塩基配列分析器を使用するかパイロシーケンシング(pyrosequencing)を用いて遂行できる。
【0145】
また、前記工程(d)で変異の存在有無は、当業界に公知となっている野生型PXR遺伝子の塩基配列、例えば、配列番号2200(GenBank accession No.:NT_005612)の塩基配列、または当業界に公知となったPXR遺伝型の各塩基配列に基づいて比較して確認したり、RFLPを遂行して野生型PXR遺伝子の制限酵素切断様相と比較して確認することもできる。PXR遺伝子の欠失または重複変異の場合にはPCR産物の電気泳動分析を通じて確認できる。
【0146】
前記工程(d)で変異が存在することが確認されたPCR産物の塩基配列でハプロタイプの頻度及び種類を予測することは、当業界に公知となった技術プログラムまたは市販されるプログラムを使用して分析できる。例えば、Haploviewプログラムは無料で配布されるプログラムとして利用でき、SNPAlyzeのような商用化されたプログラムを使用することもできる。前記 Haploviewソフトウェアは当業界に公知となっており、好ましくはインターネットウェブサイトである http://www.broad.mit.edu/mpg/haploviewを利用することができる。
本発明の方法は前記工程(a)乃至(e)を反復する工程を追加的に含むことができる。種族や患者などのようなある特定集団におけるPXR遺伝子の変異様相及びこれに対するハプロタイプを分析しようとする場合には、各PXR遺伝型の頻度を調査して前記集団で多く発見されるPXR遺伝型を選定した後、これを対象として工程(f)を遂行できる。
【0147】
前記工程(f)では前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガーソフトウェアで分析してhtSNPを選別する。前記SNPタガーソフトウェアは当業界に公知となっており、例えば Genehunter、Merlin、Allegro、SNPHAP、htSNP finder(PCA based)などが挙げられ、好ましくは、http://www.well.ox.ac.uk/〜xiayi/haplotypeまたは http://slack.ser.man.ac.uk/progs/htsnp.htmlのウェブサイトを利用することができる。
【0148】
このように選別されたhtSNPはディプロタイプ(diplotyp)決定時に正確度を高めるために検証することができる。人体の遺伝型は二本の染色体により決定されるので、遺伝型は二つのハプロタイプ組み合わせで判読される。しかしながら、複数個のSNPを同時に分析した場合、特定のハプロタイプの組み合わせが他のハプロタイプの組み合わせと同一の変異分析結果を示す可能性がある。従って、本発明により開発された診断法を用いて遺伝型が判読られた場合、正確な遺伝型を決定できることが検証されなければならない。このような検証は、Matlab(The Math Works Inc., 米国)プログラムを用いた遺伝子分析結果から遺伝型が判読される分析によって遂行される。
【0149】
本発明の一実施例では、韓国人のPXR遺伝子の機能的変異型に対するhtSNPを選別するために、まず韓国人でPXR遺伝子の変異を調査した。その結果、韓国人のPXR遺伝子で総22個のSNPを発見した(表48参照)。
本発明の他の実施例では、前記で選別された22個のSNPの中で6個の機能的変異によるハプロタイプをDYNACOM社のSNPAlyzeプログラムを用いて分析し、総14個のハプロタイプを確認した(表49参照)。
【0150】
本発明のさらに他の実施例では、韓国人で発見されたPXR遺伝子の機能的変異型を容易に決定できる最小マーカであるhtSNPを選別するために、前記14個のハプロタイプの塩基配列をSNPタガーソフトウェアを用いて分析してhtSNPを選別した(図47参照)。
【0151】
前記方法で本発明で選別されたhtSNP組み合わせは、ヒトPXR遺伝子の機能的変異型を分析することに使用することができる。従って、本発明はヒトPXR遺伝子の機能的変異型を決定する方法を提供する。前記方法は次の工程を含む:
【0152】
(a)ヒトから生物学的試料を採取する工程;
(b)前記工程(a)の採取された試料から核酸を抽出する工程;
(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び
(d)前記工程(c)で得られたPCR産物の塩基配列において、−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cからなる群より選択されたPXR遺伝子の機能的変異の存在有無を調査する工程。
【0153】
前記工程(b)で核酸を抽出する方法は前記の通りである。
前記ヒトPXR遺伝子の断片は、前記の通り、ヒトPXR遺伝子の公知となった変異、例えばSNPを含む断片をいう。前記工程(c)で使用されるプライマーは、これに制限されないが、配列番号242乃至247のプライマーからなる群より選択され得る。
【0154】
このように前記工程(d)で調査されるSNPは韓国人で発見されるPXRの機能的変異遺伝子に関するものであり、韓国人PXR遺伝子の機能的変異のハプロタイプ及と機能的変異型の決定に非常に特異的である。
【0155】
前記工程(d)で前記PCR産物の塩基配列にPXR遺伝子の変異が存在するか否かの調査は、当業界に公知となった多型性分析方法を用いて遂行できる。好ましくはスナップショット分析([Peter M. Vallone,et al.,Int J Legal Med,2004,118:147-157]参照)、電気泳動分析またはこれらの組み合わせ、より好ましくはスナップショット分析を用いて遂行できる。
【0156】
前記スナップショット分析は、SNP位置の隣接部位にアニーリング(annealing)される配列(前記SNP部位は含まない)を有するプライマーとddNTPを用いたPCR反応を通じて遺伝子型を分析する方法である。本発明に使用されるスナップショット分析は、前記工程(c)で調査されるPXR遺伝子のSNPに基づいて公知の方法により設計及び製作したものを使用することができ、SNP位置のすぐそばの塩基が3'末端となり、前記SNP位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたものであれば制限なしに使用することができ、好ましくは配列番号242乃至2457のプライマーからなる群より選択されるプライマーを使用することができる。この時、前記SNP位置の隣接部位にアニーリングされる配列は約20bpの長さを有することが好ましく、同時に複数個のSNPを決定しようとする場合、各SNPに対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計、例えばT塩基を5個ずつ5'位置にさらに添加してプライマー間にサイズ差異を作って合成し、PCR産物の長さをそれぞれ異なるようにすることができる。このように作られたスナップショットプライマーに各SNPに相補的なddNTPが結合するようになり、これら合成物はSNPにより長さの差異が発生することによって同時に複数個のSNPの決定が可能である。
【0157】
その後、スナップショット分析で判定された遺伝型診断結果が正確であることを検査するために、遺伝型が分かる他の遺伝子分析法を通じた結果との一致性を調査することができる。ここで他の遺伝子分析法は、特に限定されないが、好ましくは自動塩基配列分析法またはパイロシーケンシング法であっても良い。
【0158】
本発明の他の実施例では、本発明で選別されたhtSNP組み合わせの有用性を確認した。このために図47に示すhtSNP組み合わせを用いてスナップショット分析を実施した後、得られたPCR産物の塩基配列を分析した。その結果、本発明の方法が韓国人で発見されるPXR遺伝子の機能的変異型を同時に高速に分析できることを確認した(図48乃至図50参照)。
本発明の方法により分析され得るPXR遺伝子の機能的変異型は−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cを含む。
【0159】
5.UGT1A
本発明によるヒトUGT1A族遺伝子群の機能的変異型を決定する方法は次の工程を含む:
(a)ヒトから生物学的試料を採取する工程;
(b)前記工程(a)で採取された試料から核酸を抽出する工程;
(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族個別遺伝子を増幅する工程;及び
(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での−39(TA)6>(TA)7、211G>A、233C>T及び686C>A;UGT1A3での31T>C、133C>T及び140T>C;UGT1A4での31C>T、142T>G及び292C>T;UGT1A6での19T>G、541A>G及び552A>C;UGT1A7での387T>G、391C>A、392G<A、622T>C及び701T>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子群の機能的変異型の存在有無を確認する工程。
【0160】
また、本発明によるイリノテカン(Irinotecan)に対する感受性と関連したUGT1A族遺伝子の多型性を決定する方法は次の工程を含む:
(a)ヒトから生物学的試料を採取する工程;
(b)前記工程(a)で採取された試料から核酸を抽出する工程;
(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族遺伝子を増幅する工程;及び
(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での211G>A、233C>T及び686C>A;UGT1A6での19T>G、541A>G及び552A>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子変異型の存在有無を確認する工程。
【0161】
本発明の方法は、韓国人で主に発見されるUGT1A族遺伝子群の多型性に基づいて選別された、最適のUGT1A族遺伝子群の機能的変異型または薬物感受性を決定する多型性標識セットを用いることを特徴とするため、既存の方法と比較して韓国人を対象として時間及び費用の面で効率的に分析できる効果を奏する。
【0162】
本発明の工程(a)では、ヒト、好ましくは韓国人、中国人及び日本人などのアジア人、さらに好ましくは韓国人から生物学的試料を採取する。前記生物学的試料は、血液、皮膚細胞、粘膜細胞または毛髪などであり、好ましくは血液であっても良い。
【0163】
本発明の工程(b)では、前記工程(a)で採取された生物学的試料から核酸を抽出する。前記核酸はDNAまたはRNAなどであっても良く、好ましくはDNA、より好ましくはゲノム(genomic)DNAであっても良い。また、採取された試料から核酸を抽出する工程は特に限定されないが、当業界に公知となった技術により遂行したり、市販されている抽出用キット、例えばQiagen社(米国)またはStratagene社(米国)で市販されているDNAまたはRNA抽出用キットを使用して遂行できる。
【0164】
本発明の工程(c)では、前記工程(b)で抽出された核酸を鋳型とし、ヒトUGT1A族の各遺伝子を増幅できるプライマーを使用してUGT1A族遺伝子が増幅される。この時、前記工程(b)で抽出された核酸がRNAである場合には、逆転写を用いてcDNAに転換してこれを鋳型として使用する。前記各プライマーはヒトUGT1A族遺伝子またはその断片の塩基配列に基づいて公知の方法により設計し製作される。
【0165】
本発明の工程(c)で、UGT1A族遺伝子群の機能的変異型を決める方法の場合には、UGT1A1、UGT1A3、UGT1A4、UGT1A6、UGT1A7及びUGT1A9族の各遺伝子を増幅することが好ましく、イリノテカン(Irinotecan)に対する感受性を決定するUGT1A族遺伝子群の多型性を決定する方法の場合には、UGT1A1、UGT1A6及びUGT1A9族の各遺伝子を増幅することが好ましい。
【0166】
本発明の工程(d)では、前記工程(c)で増幅された各UGT1A族遺伝子を使用してUGT1A族遺伝子群の機能的変異型または薬物感受性と関連した多型を分析する。この時、当該分野で公知の多型性分析法を用いて機能的変異型または多型が分析される。例えば、スナップショット(SNaPshot)分析、電気泳動分析、パイロシーケンシング(pyrosequencing)またはこれらの組み合わせが行われる。
【0167】
具体的には、分析するUGT1A遺伝子の変異型が単一塩基多型である場合には、スナップショット分析を遂行することが好ましい。、この時のスナップショット分析では単一塩基多型(SNP)位置の隣接部位にアニーリング(annealing)されるプライマー及びddNTPを用いてPCR反応を遂行する。この時、スナップショット分析で使用されるプライマーは、UGT1A族遺伝子の単一塩基多型に基づいて公知の方法により設計及び製作したものである。例えば、単一塩基多型位置のすぐそばの塩基が3'末端となり、隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されるように設計及び製作される。この時、アニーリングされる配列は約20bpの長さを有することが好ましく、同時に複数個の単一塩基多型を決定しようとする場合は、各単一塩基多型に対するスナップショットプライマーの5'末端T塩基の長さをそれぞれ異なるように設計し、PCR産物の長さをそれぞれ異なるようにする。
【0168】
本発明の一実施様態によれば、UGT1A族遺伝子群の機能的変異型を確認するスナップショット分析では、配列番号:2905乃至314の配列を有するプライマーを使用することができ、UGT1A族遺伝子群のイリノテカン感受性関連多型性を確認するスナップショット分析には配列番号:315乃至322の配列を有するプライマーを使用することができる。
【0169】
一方、スナップショット分析で増幅されたPCR産物の塩基配列を公知の配列分析法で分析することができる。好ましくは自動塩基配列分析法により分析することができるがこれに限定されない。
【0170】
また、分析するUGT1A遺伝子の変異型が単一塩基多型ではない場合(例えば、UGT1A1での−39(TA)6>(TA)7型)にはスナップショット分析の代わりに公知のパイロシーケンシング(pyrosequencing)分析を遂行でき、この時のパイロシーケンシングは、公知の通り、DNAが重合される間に放出されるPPi(無機ピロリン酸塩)の発光程度を測定して分析を遂行できる。本発明の一実施様態によれば、UGT1A1族遺伝子の−39(TA)6>(TA)7型を確認するパイロシーケンシング分析には、配列番号:292乃至294の配列を有するプライマーを使用することができる。
【実施例】
【0171】
以下、本発明を実施例により詳しく説明する。但し、下記の実施例は本発明を例示するものに過ぎず、本発明の内容が下記の実施例に限定されるものではない。
【0172】
<CYP1A2>
実施例1:韓国人におけるCYP1A2遺伝子の遺伝型分析
<1−1>CYP1A2遺伝子の増幅
48名の健康な被験者から血液を分離した後、Qiagen社のゲノムDNA分離キットを使用してDNAをそれぞれ分離した。CYP1A2遺伝子は7個のエクソンを含み総遺伝子の長さが約11kbである。従って、CYP1A2遺伝子を15個の断片に分けてPCRを遂行した。各PCRに使用したプライマーは下記の表1の通りである。本明細書に記載された塩基配列でA、T、G及びCはそれぞれアデニン、チミン、グアニン及びシトシンを意味する。
【0173】
【表1】

【0174】
各プライマーの位置とPCR産物の大きさは下記の表2の通りである。また、ヌクレオチドの位置はCytochrome P450(CYP) Allele Nomenclature Committee(http://www.cypalleles.ki.se/cyp1a2.htm)の命名法により記載した。
【0175】
【表2】

【0176】
各PCR断片に対する反応条件は下記の表3の通りである。
【0177】
【表3】

【0178】
<1−2>PCR産物の塩基配列分析
前記実施例<1−1>で得られた各PCR産物の塩基配列を自動配列分析器を用いて分析した。この時に使用したプライマーは下記の表4の通りである。
【0179】
【表4】

【0180】
前記実施例<1−1>で増幅されたCYP1A2遺伝子の全体塩基配列を自動塩基配列分析器を用いて分析した。その後、野生型CYP1A2遺伝子の塩基配列(配列番号1)と比較した結果、総17個の単一塩基多型を発見し、その結果を下記の表5に示した。その中で単一塩基多型−2603insAは新規なものであることを確認した。
【0181】
【表5】

【0182】
その後、本発明者等は前記新規な1個の単一塩基多型がCYP1A2の一本に位置するか否かと、同一なDNA本に他の遺伝子変異はないか、染色体の他の部分に位置した類似遺伝子から起因したものではないかどうかを調査するために、前記単一塩基多型が発見された変異遺伝子を含む被験者のDNAを鋳型とし、前記配列番号38及び39のプライマーを使用してPCRを遂行し、増幅された産物の配列を前記と同様な方法で分析した。
その結果、前記1個の単一塩基多型は全てプロモーター部位−2603insAに位置した。この変異型の単一塩基多型は二本のDNAのうち、一本は変異型、他の一本は野生型を有していることが示された(図1)。
【0183】
実施例2:CYP1A2変異型のハプロタイプ分析
前記実施例1で糾明された17個のCYP1A2遺伝子の変異は、その組み合わせによりCYP1A2の酵素活性に影響を与える可能性があり、幾つかのハプロタイプに対する酵素活性変異は既に報告されている。従って、本発明者等は実施例1で確認した変異によるハプロタイプをDYNACOM社のSNPAlyzeプログラムを使用して分析した。その結果、下記の表6に示した通り、他種族で発見されない韓国型の新たなハプロタイプが確認された。
【0184】
【表6】

□:各単一塩基多型の変異
【0185】
実施例3:htSNPの選別及び検証
CYP1A2遺伝子の単一塩基多型の組み合わせであるハプロタイプがCYP1A2の酵素活性に影響を与える可能性が多くのハプロタイプで報告されたことがある。このように作られた詳細なハプロタイプの情報は最小マーカで確認することができる。これら最小マーカをhtSNPといい、前記htSNPはそれぞれのハプロタイプの正確な表示のために必要なマーカであり、色々な組み合わせを構成するようになる。この最適化された標識セットであるhtSNP組み合わせをSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype)を用いて選別した。選別されたhtSNP組み合わせの例を図2乃至図6に示し、各選別されたhtSNP組み合わせは最適の標識セットのうちの一つとして、「1」は野生型を、「2」は変異型、そして「V」表示は選択されたそれぞれのhtSNPを示す。htSNPの選別は図2乃至6の組み合わせ以外にも他の組み合わせが可能である。
その後、探し出した組み合わせの中でディプロタイプを考慮して互いに重ならずにディプロタイプ遺伝型を決定できるかをMatlabソフトウェア(version 7.1,The Math Works Inc., 米国)を使用して分析し、これを用いてチェックした後に組み合わせを決定した。
【0186】
検証の結果、互いに重ならずにディプロタイプ遺伝型を決定できることを確認した。これは本発明で選択したhtSNP組み合わせが互いに同一なものがなく、遺伝型を決定するに当たって不正確な分析が全くないことを示す。
【0187】
実施例4:高速のCYP1A2プロモーター遺伝子変異検索
前記実施例1で確認された韓国人で発見されたCYP1A2遺伝子の17個の単一塩基多型の中で、CYP1A2酵素活性に影響を与えるプロモーター部分の単一塩基多型11個を高速に検索するためにスナップショット分析を実施した。被験者のDNAを鋳型としてPCRを遂行し、増幅された産物をスナップショット分析した。CYP1A2遺伝子のプロモーターは約4,000baseであり、PCRに使用したプライマーは下記の表7の通りである。
【0188】
【表7】

【0189】
PCR産物に対する反応条件は下記の表8の通りである。
【0190】
【表8】

【0191】
前記のように増幅されたPCR産物の反応されていないプライマーとdNTPなどは残っていればスナップショット過程に影響を与えるので、これらを除去するために、混合されたPCR産物5μl当たりExoSAP−IT(USB社)2μlを入れて37℃で30分間反応させた後、80℃で15分間反応させて残っている酵素を非活性化させた。酵素処理された産物は下記の表9に示すプライマーを用いてマルチプレックススナップショット(multiplex SNaPshot)反応物を作ってPCR反応した。マルチプレックススナップショット反応物及びPCR反応条件をそれぞれ表10及び表11に示した。
【0192】
【表9】

【0193】
【表10】

【0194】
【表11】

【0195】
反応が終わった後、[F]ddNTPを除去するためにSNaPshot産物5μlにSAP(USB社)2μlを入れて37℃で30分、80℃で15分間反応させた。反応が全て終わると、反応物0.5μl、Hi−Diホルムアミド(ABI社)9.25μl及びジーンスキャン(GeneScan)−LIZサイズ標準物質(ABI社)0.25μlを混合して95℃で5分間変成させた。その後、3130XL遺伝子分析器(3130XL Genetic Analyzer、 ABI社)で分析し、その結果を図7乃至図14に示した。
【0196】
その結果、図7乃至14に示すように、CYP1A2遺伝子の変異型によりピークの色と位置が異なるように表示されて野生型、異型の対立形質を有する変異型(異型)、同型の対立形質を有する変異型(同型)を容易に識別できることを確認した。従って、本発明による分析方法を通じて時間及び費用の面で効率的かつ容易にCYP1A2遺伝子の変異型を分析できることが分かる。
【0197】
<CYP2A6>
実施例5:韓国人における2A6遺伝子の遺伝型分析
<5−1>2A6遺伝子の増幅
50名の健康な被験者から血液を分離した後、Qiagen社のゲノムDNA分離キットを使用してDNAをそれぞれ分離した。CYP2A6遺伝子は9個のエクソンを含む総遺伝子の長さが約6.9kbである。従って、CYP2A6遺伝子を7個の断片に分けてPCRを遂行した。各PCRに使用したプライマーは下記の表12の通りである。本明細書に記載された塩基配列でA、T、G及びCはそれぞれアデニン、チミン、グアニン及びシトシンを意味する。
【0198】
【表12】

【0199】
各プライマーの位置とPCR産物の大きさは下記の表13の通りである。また、ヌクレオチドの位置はCytochrome P450(CYP) Allele Nomenclature Committee(http://www.cypalleles.ki.se/cyp2a6.htm)の命名法により記載した。
【0200】
【表13】

【0201】
各PCR断片に対する反応条件は下記の表14の通りである。
【0202】
【表14】

【0203】
<5−2>PCR産物の塩基配列分析
前記実施例<5−1>で得られた各PCR産物の塩基配列を自動配列分析器及び配列番号:76乃至89のプライマーを用いて分析した。
【0204】
その後、野生型CYP2A6遺伝子の塩基配列(配列番号:75)と比較した結果、総27個のSNPを発見し、その中で2つは現在まで報告されたことがない新たなSNPである。また、遺伝子欠失による構造分析を通じて3個のSNPを発掘し、総30個のSNPは下記の表15に記載された通りである。
【0205】
【表15】

【0206】
前記の表で「+」はCYP2A6遺伝子が欠失されてCYP2A6遺伝子の一部の残った部分とCYP2A7遺伝子との接合形態(図33、CYP2A6遺伝子の1番から8番エクソンまで全てなくなり、CYP2A7のエクソン9部分末端にCYP2A6のエクソン9番が一部置換された形態)の遺伝子で発見される変異として、数字はCYP2A6遺伝子が完全にあることを仮定して塩基配列に合わせた番号を使用した(CYP2A6遺伝子の欠失であるため、CYP2A6遺伝子のSNPと見ることは困難である)。
【0207】
このような構造でSNPを用いて欠失されたハプロタイプを決定するためには、CYP2A6とCYP2A7のシークエンスが同一な部分内で5’位置の正方向プライマーをデザインし、CYP2A6に特異的でありCYP2A7遺伝子は増幅できないエクソン9部分で逆方向プライマーをデザインし、純粋にCYP2A7遺伝子は増幅されず、完全なCYP2A6遺伝子またはCYP2A6遺伝子が欠失されてCYP2A7遺伝子と接合された形態の遺伝子部分を増幅されるようにできる。
【0208】
増幅されたPCR産物でCYP2A6とCYP2A7に特異的な塩基を選択するが、CYP2A6翻訳開示コドンであるATGを基準にCYP2A6(配列番号:75)の6091C>T塩基例をみれば、次の通りCYP2A6 6091C周辺の部分はCYP2A7(配列番号:104)6521Tの塩基配列周辺と類似することが分かる。このようなサイトが6091だけではなく、CYP2A6配列で5971G及び5983T部分もCYP2A7と差異があることを確認できる。
【0209】
また、前記の表で「*」が意味するところは、国際CYP明明委員会から対立遺伝形質と認められて命名された変異の表示であり、例えば、*11は野生型に比べて224番目アミノ酸がセリンからプロリンに変わる変異を有しているハプロタイプを意味し、国際命名法はhttp://www.cypalleles.ki.se/cyp2a6.htmを従った。
また、前記の表で「♯」は新規変異を意味する。
【0210】
実施例6:CYP2A6遺伝子の遺伝型のハプロタイプ分析
前記実施例5で糾明された27個のCYP2A6遺伝子の変異と3個のCYP2A6欠失標識変異は、その組み合わせに応じてCYP2A6の酵素活性に影響を与える可能性がある。従って、本発明者等は実施例5で確認した変異によるハプロタイプをDYNACOM社のSNPAlyzeプログラムを使用して分析した。通常的にハプロタイプの分析で頻度が低い変異の場合、統計的有意性を確保し難いため、主に5%または10%以上の頻度を有する変異を選別してハプロタイプの分布を予測する。しかしながら、アミノ酸の置換を招く場合には、頻度が低いにもかかわらず、明確な機能性を有しているため、本発明では頻度が高い変異である−48T>G、22C>T、51G>A、1620T>C、1836G>T、及び6354T>Cの6個の変異とアミノ酸の変化を招く13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>Tの8個の変異を用いてハプロタイプ分析を遂行し、遺伝子の欠失がある場合、これを標識できる変異である5971G>A、5983T>G、6091C>T変異を検査変異対象に追加した。総17個の変異を用いてハプロタイプを分析した結果、図15のように総20個の韓国人におけるハプロタイプ分布を得ることができた。
【0211】
実施例7:htSNPの選別及び検証
CYP2A6遺伝子のSNPの組み合わせであるハプロタイプがCYP2A6の酵素活性に影響を与える可能性が多くのハプロタイプで報告されたことがある。このような詳細なハプロタイプの情報は最小マーカで確認することができる。これら最小マーカをhtSNPといい、前記htSNPはそれぞれのハプロタイプの正確な表示のために必要なマーカであり、色々な組み合わせを構成するようになる。この最適化された標識セットであるhtSNP組み合わせを選別するために、前記実施例6で選別された総20個のハプロタイプの塩基配列をSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype/)を用いて分析した。
【0212】
その結果、図16乃至図21に示すhtSNP組み合わせを選別した。図16乃至図21に図示されている、各選別されたhtSNP組み合わせは最適の標識セットとして、「1」は野生型を、「2」は変異型、そして「V」表示は選択されたそれぞれのhtSNPを示す。
【0213】
htSNP分析で各変異の遺伝型が決定されると、その結果から二倍体型を予測することができる。しかしながら、互いに異なるハプロタイプの組み合わせが同一な遺伝型を有する可能性もあるので、htSNP組み合わせの中でディプロタイプ(二倍体型)を考慮する時、互いに重ならずに二倍体型遺伝型を決定できるかをMatlabソフトウェア(version 7.1,The Math Works Inc., 米国)を使用して分析した。
【0214】
検査の結果、本実施例により決定されたhtSNPだけでも互いに重ならずに二倍体型を決定できることを確認した。これは本発明で選択したhtSNP組み合わせが互いに同一なものがなく、遺伝型を決定するに当たって不確実な分析が全くないことを示す。
【0215】
実施例8:高速のCYP2A6遺伝子の機能的変異検索
前記実施例5で確認された韓国人で発見されたCYP2A6遺伝子の27個の変異とCYP2A6遺伝子欠失を標識する3個の変異のうち、機能性を変化させるCYP2A6の遺伝型が遺伝子診断に有用に使用され得る。従って、図16にある変異の中でアミノ酸の変化を起こしているか、または既に機能性が立証されている変異である−48T>G、13G>A、567C>T、2134A>G、3391T>C、6458A>T、6558T>C、6582G>T、6600G>Tの9個の変異と遺伝子欠失を標識する6091C>T変異とを含む総10個の機能的変異を高速に検索するために、CYP2A6遺伝子の高速遺伝型分析技術の一つであるスナップショット分析を実施した。選別されたhtSNPは図16のhtSNP組み合わせの中で機能性を反映する10個の変異が選択されたものであり、変異の位置は下記の表17の通りである。
【0216】
【表17】

【0217】
具体的に、被験者のDNAを鋳型としてPCRを遂行し、増幅された産物をスナップショット分析した。この時、PCRに使用したプライマーは下記の表18の通りである。
【0218】
この時、CYP2A6_longを増幅するためのプライマーは、CYP2A6の遺伝子全長を増幅するプライマーであるので、CYP2A6遺伝子欠失の場合には適用できず、遺伝子欠失を標識する6091C>Tを判別するためにはCYP2A6*4産物を増幅できるCYP2A6 delFとCYP2A6 delRのプライマー対を利用しなければならない。
【0219】
【表18】

【0220】
PCR産物に対する反応条件は下記の表19の通りである。
【0221】
【表19】

【0222】
前記のように増幅されたPCR産物の反応されていないプライマーとdNTPなどは残っていればスナップショット過程に影響を与えるので、これらを除去するために、混合されたPCR産物5μl当たりExoSAP−IT(USB社)2μlを入れて37℃で30分間反応させた後、80℃で15分間反応させて残っている酵素を非活性化させた。酵素処理された産物は下記の表20に示すプライマーを用いてマルチプレックススナップショット(multiplex SNaPshot)反応物を作ってPCR反応した。マルチプレックススナップショット反応物及びPCR反応条件をそれぞれ表21及び表22に示した。
【0223】
【表20】

【0224】
【表21】

【0225】
【表22】

反応が終わった後に残ったddNTPを除去するために、スナップショット産物10μlにSAP(USB社)1μlを入れて37℃で60分、65℃で15分間反応させた。反応が全て終わると、反応物0.5μl、Hi−Diホルムアミド(ABI社)9.3μl及びジーンスキャン(GeneScan)−LIZサイズ標準物質(ABI社)0.2μlを混合して95℃で5分間変成させた。その後、3100遺伝子分析器(3100 Genetic Analyzer、ABI社)で分析した。その結果を図22乃至30に示す。この遺伝型分析は下記の表23の通りである。
【0226】
【表23】

【0227】
図22乃至30に示すように、各SNPによりピークの色と大きさが一致し、CYP2A6遺伝子の機能的変異型によりピークの色と位置が異なるように表示されて野生型、異型の対立形質を有する変異型(異型)、同型の対立形質を有する変異型(同型)を容易に識別できることを確認した。図22乃至30のグラフでX軸は各プライマーの長さの差異により自動塩基配列分析器でプライマーが移動した距離であり、Y軸は各塩基に含まれた特異的な波長の蛍光物質から放出される蛍光の強さを示す。
【0228】
また、図31及び32は前記図22乃至図30の遺伝子変異以外にCYP2A6遺伝子が欠失された場合を追加的に検査するために、図22乃至図30の遺伝子検査に並行して遂行するスナップショット方法の例示として、図31はCYP2A6遺伝子が相同染色体に正常に存在する場合であり、図32はCYP2A6遺伝子が一つの染色体には存在しないため、一つの遺伝子だけを有する場合である。
【0229】
以上の発明を通じて開発したスナップショット方法により50個の試料を分析し、同一の試料に対して全長塩基配列を分析した結果、遺伝型判読結果が100%一致した。これは本発明方法の再現性が高く、正確であることを示す。
【0230】
従って、本発明による分析方法を通じて時間及び費用の面で効率的かつ容易にCYP2A6遺伝子の機能的変異型を分析できることが分かる
従って、前記方法は韓国人で主に発見される10種のCYP2A6ハプロタイプを決定することによって、これら組み合わせによるCYP2A6遺伝型を同時に高速に分析できる方法であり、韓国人で発見される遺伝的変異を含むので、遺伝型決定の正確度が高い。また、韓国人と遺伝的特性が非常に類似する日本人の場合にもほとんど全ての遺伝型分析が可能であり、既に知られた結果から中国人でも90%以上の範囲でCYP2A6遺伝型を判別できる技術であると思料される。
【0231】
<CYP2D6>
実施例9:韓国人のCYP2D6遺伝子の遺伝型分析
<9−1>ゲノムDNAの分離
174名の韓国人から採取した血液試料からゲノムDNA分離キット(Qiagen社)を用いてゲノムDNAをそれぞれ分離した。
【0232】
<9−2>CYP2D6遺伝子の増幅及び全長塩基配列分析
前記実施例<9−1>で分離された総174個のゲノムDNAサンプルの中で任意に選んだ51個のサンプルを鋳型とし、下記のプライマー対でPCRを遂行してヒトCYP2D6遺伝子を構成する9個のエクソンと1.8kbのプロモーター部位が増幅されるようにした。
【0233】
【表24】

【0234】
PCRは94℃で1分間反応させた後、98℃で10秒、64℃で30秒及び72℃で7分間30サイクルを反復し、最終的に72℃で10分間反応させた。その結果、6,569bpの大きさのPCR産物を収得した(下記の表25参照)。
【0235】
【表25】

【0236】
その後、増幅されたPCR産物を鋳型とし、下記の表26に示す総13種のプライマーを用いて増幅されたCYP2D6遺伝子の塩基配列を分析した。
【0237】
【表26】

【0238】
<9−3>各遺伝型による個別的分析
前記実施例<9−1>から分離した残りの123個のゲノムDNAサンプルについては、下記の方法により東洋人で主に発見される*2A、*5、*2N、*10B、*14B、*18、*21B、*41A、*49、*52、*60変異に対する遺伝型分析を個別的に遂行した。
【0239】
a)CYP2D6*5遺伝型の分析
CYP2D6*5遺伝型を分析するために、下記の表27に記載されたプライマーを使用してPCRを遂行した。PCR反応条件としては、94℃で1分間反応させた後に98℃で10秒、64℃で30秒及び72℃で5分間30サイクルを反復して72℃で10分間反応させた。その結果、野生型の場合、9個のエクソンを含む5.1kbのPCR産物が増幅され、CYP2D6*5変異型の場合、3.5kbのPCR産物が増幅された。
【0240】
【表27】

【0241】
b)CYP2D6*2N遺伝型の分析
CYP2D6*2N遺伝型を分析するために、下記の表28に記載されたプライマーを使用してPCRを遂行した。PCRは94℃で1分間反応させた後に98℃で10秒、64℃で30秒及び72℃で8分間30サイクルを反復して72℃で10分間反応させた。その結果、CYP2D6*2N変異型の場合、7.8kbのPCR産物を収得した。
【0242】
【表28】

【0243】
c)CYP2D6*2及び*41遺伝型の分析
CYP2D6*2遺伝型とCYP2D6*41遺伝型の場合、−1584C>G変異を除いて同一の変異(1235A>G;−740C>T;−678G>A;イントロン1でのCYP2D7への遺伝子転換(gene conversion);1661G>C;2850C>T;4180G>C)を有する。従って、先ずイントロン1でのCYP2D7への遺伝子転換の変異配列を下記の表29に記載されたプライマーを用いてAS−PCR方法(Johanson,Molecular Pharmacology,46:452-459,1994)で分析した。
【0244】
イントロン1でのCYP2D7への遺伝子転換が起こった場合、配列番号129のプライマー9と配列番号125のプライマー10Bを使用してPCR反応を行うことにより増幅産物を得ることができ、正常の場合には配列番号129のプライマー9と配列番号130のプライマー10の組み合わせで反応をした場合にのみ増幅産物が得られる。従って、2個のプライマー組み合わせ、つまり、プライマー9/プライマー10とプライマー9/プライマー10Bの組み合わせでそれぞれ反応して増幅された産物の有無に応じてイントロン1でのCYP2D7への遺伝子転換を確認することができる。
PCRは94℃で5分間反応させた後に94℃で30秒、64℃で30秒及び72℃で30秒間35サイクルを反復して72℃で10分間反応させた。その後、−1584C>G変異を下記の表29に記載されたシーケンシングプライマーを用いてパイロシーケンシングで分析して−1584Gである場合をCYP2D6*2遺伝型に、−1584Cである場合をCYP2D6*41遺伝型に決定した。
【0245】
【表29】

【0246】
d)CYP2D6*10B、*14、*18及び*49遺伝型の分析
CYP2D6*10B、*14、*18及び*49遺伝型はPCR−RFLP方法で分析した(Johanson,Molecular Pharmacology,46:452-459,1994; Wang,Drug Metabolism and Dispososition,27:385-388,1998; 及び Geadigk,Pharmacogenetics,9:669-682,1999)。この時に使用したプライマーは下記の表30の通りであり、実験条件は下記の表31に示す通りである。
【0247】
【表30】

【0248】
【表31】

【0249】
e)CYP2D6*21、*52及び*60遺伝型の分析
CYP2D6*21、*52及び*60遺伝型の分析はPCR−パイロシーケンシング法で分析した。分析に使用したプライマーの配列は下記の表32に示す通りである。
【0250】
【表32】

【0251】
前記実施例<9−2>及び<9−3>で得られたデータをGenBank accession No. M33388で公知のCYP2D6の配列に基づいて比較分析し、各対立因子の頻度を調査した。その結果、韓国人で主に発見されるCYP2D6遺伝子ハプロタイプの種類及び頻度は下記の表33の通りである。
【0252】
【表33】

【0253】
前記の表33でNormalは「正常水準」、Incrは「増加」、Decrは「減少」、Noneは「活性ない」をそれぞれ示す。また、括弧内の表記は分析に使用した標識薬物の略字である: b, bufuralol;d, debrisoquine;dx, dextromethorphan;s, sparteine。
【0254】
韓国人で主に発見される12種の遺伝型を中心に遺伝子を選択してCytochrome P450(CYP) Allele Nomenclature Committee (http://www.cypalleles.ki.se/cyp2d6.htm)を基準として各遺伝型の変異を下記の表34に示した。表34で「1」は野生型を、「2」は変異型をそれぞれ示す。
【0255】
【表34】

【0256】
実施例10:htSNPの選別及び検証
前記実施例9で確認した韓国人で発見される12種のCYP2D6遺伝型を決定するために、表34に示す33個の変異を全て分析することは、時間と費用的な面で効率性が非常に落ちる。従って、詳細なハプロタイプの情報を用いて標識遺伝子変異であるhtSNPを選定して遺伝型を決定すれば経済的な遺伝型判別が可能である。前記htSNPはそれぞれのハプロタイプの正確な表示のために必要なマーカであり、色々な組み合わせを構成するようになる。この最適化された標識セットであるhtSNP組み合わせをSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype/)を用いて選別した。選別されたhtSNP組み合わせの例を図34乃至図39に示し、各選別されたhtSNP組み合わせは最適の標識セットとして、「1」は野生型を、「2」は変異型を示し、「V」表示はhtSNPを示す。
【0257】
その後、探し出した組み合わせの中でディプロタイプ(diplotype)を考慮して互いに重ならずにディプロタイプ遺伝型を決定できるかをMatlabソフトウェア(version 7.1,The Math Works Inc., USA)を使用して分析し、これを用いてチェックした後に組み合わせを決定した。
検証の結果、互いに重ならずにディプロタイプ遺伝型を決定できることを確認した。これは本発明で選択したhtSNP組み合わせが互いに同一なものがなく、遺伝型を決定するに当たって不正確な分析が全くないことを示す。
【0258】
実施例11:SNaPshot分析
前記実施例10で選別されたhtSNP組み合わせを用いてCYP2D6遺伝子の高速遺伝型分析技術の一つであるスナップショット分析を遂行した。このために図34のhtSNP組み合わせを選択した。各htSNPで選別される変異の位置は下記の表35の通りである。下記の表35でhtSNP1乃至htSNP3の場合には該当する複数個の変異の中で一つのSNPだけを分析しても遺伝型判別が可能である。また、htSNP9の場合には9個の塩基(GTGCCCACT)が挿入されて反復される様相を示すようになるので、4125番目塩基から4133番目塩基位置のうちのいずれか一つの塩基位置だけを分析し、野生型遺伝子の塩基配列と比較することによって遺伝型判別が可能である。
【0259】
【表35】

【0260】
前記実施例<9−2>と同様の方法によりCYP2D6遺伝子を増幅して約6.7kbの産物を得た。そして、CYP2D6*5を決定するために配列番号154のプライマーCYP2D6_3(5'−ACCTCTCTGGGCCCTCAGGGA−3')と配列番号123のプライマー3‘2D6*5を用いてCYP−REP−Del部位を増幅し、PCRは94℃で1分間反応させた後に98℃で10秒、64℃で30秒及び72℃で3分間30サイクルを反復し、最終的に72℃で10分間反応させた。その結果、6,569bp大きさのPCR産物を収得した。
【0261】
前記のように増幅されたPCR産物の反応されていないプライマーとdNTPなどは残っていればスナップショット過程に影響を与えるので、これらを除去するために、PCR産物5μl当たりExoSAP−IT(USB Corporation)2μlを入れて37℃で30分間反応させた後、80℃で15分間反応させて残っている酵素を非活性化させた。酵素処理された産物を鋳型3μl(6.7kbのCYP2D6遺伝子2μlと3.5kbのCYP−REP−DEL1μlの混合物)、スナップショットマルチプレックスレディーリアクションミックス(SNaPshot Multiplex Ready Reaction Mix, ABI)1μl、1/2ターム(term)緩衝液(200 mM Tris-HCl,5 mM MgCl2,pH 9)4μl及びPooled SNaPshotプライマーを入れて全体反応物の量を10μlに合わせた後、96℃で10秒、50℃で5秒、60℃で30秒間40サイクル反復してPCRを遂行した。PCR反応した。使用したPooled SNaPshotプライマーの処理濃度を下記の表36に示した。
【0262】
【表36】

【0263】
反応が終わった後に反応物10μlにSAP(USB Corporation)1μlを入れて37℃で1時間、65℃で15分間反応させた。反応が全て終わると、反応物0.5μl、LIZ120(ABI)0.2μl、Hi−Diホルムアミド(ABI)9.3μlを混合して96ウェルプレートに分注した。分注されたサンプルを95℃で2分間反応させた後、3100遺伝子分析器(ABI)で分析した。分析した結果は図40に示す通りである。
【0264】
その結果、図40に示すように、各SNPによりピークの色と大きさが一致し、野生型と変異型が明確に決定されることが分かる。
【0265】
また、CYP2D6重複をスナップショットを用いて分析するために、CYP−REP−Dup部位を配列番号155のDup−F_2(5’−CCTCACCACAGGACTGGCCACC−3’)と配列番号156のDup−R(5’−CACGTGCAGGGCACCTAGAT−3’)を用いたことを除いては前記と同様な方法で増幅して3.3kb大きさのPCR産物を収得した。PCR反応以降、PCR産物から反応して残ったプライマーなどを除去するためにPCR産物5μl当たりExoSAP−IT(USB Corporation)2μlを入れて37℃で30分間反応させた。
【0266】
その後、80℃で15分間反応させて残っているExoSAP−ITを非活性化させた。酵素処理された産物を鋳型3μl、スナップショットマルチプレックスレディーリアクションミックス1μl、1/2ターム緩衝液4μl及び配列番号157のスナップショットプライマー(CYP2D6−5R、5’−CTCGTCACTGGTCAGGGGTC−3’)を入れて全体反応物の量を10μlに合わせた後、前記と同一な条件でスナップショット反応を遂行した後、3100遺伝子分析器で分析してその結果を図41に示した。
【0267】
その結果、図41に示すように、SNPによりピークの色と大きさが一致し、野生型と変異型が明確に決定されることが分かる。
【0268】
このような方法により野生型及び変異遺伝型を含む50個のサンプルに対してシーケンシングを通じた妥当性検査(validation)を遂行した結果、100%一致する結果を得た。この結果は本発明方法が再現性が高く、正確であることを示す。
【0269】
従って、前記方法はこれら組み合わせて高速で、韓国人で主に発見される12種のCYP2D6ハプロタイプを決定し、同時にCYP2D6遺伝型を決定する。韓国人で発見される遺伝的変異を含むので、遺伝型決定における正確度が高い。また、韓国人と遺伝的特性が非常に類似する日本人の場合にもほとんど全ての遺伝型分析が可能であり、既に知られた結果から中国人でも90%以上の範囲でCYP2D6遺伝型を判別できる技術であると思料される。
【0270】
実施例12:遺伝子分析チップを用いた遺伝型分析
<12−1>ZiP Codeチップの製作
1)プローブ製作
ASPE PCR反応に使用するZiPCodeと相補的な塩基配列でデザインし、プローブは3’方向に10bpのヌクレオチド配列(5’−CAG GCC AAGT−3’)をスペーサで挿入してターゲットと交雑反応がよく生じるように誘導した。
【0271】
また、前記スペーサの5’方向に24bpのZiP Codeオリゴ塩基が含まれており、前記プローブ(cZiP Code)の塩基配列は下記の表37に記載された通りである。この時、下線部は10個のスペーサ配列である(図43)。
【0272】
【表37】

【0273】
2)スポッティング及びプローブ固定
チップ製作のための基板はアミンがコーティングされているコーニン(Corning)社のGAPSIIガラススライドを使用した。SMP4XBピンを使用してOmniGgid100スポッターでスポッティングし、スポッティング条件は温度−22℃、湿度−54%とし、27種のプローブはそれぞれ2回反復でスポッティングした。スポッティング後、7,500μJ/cm2のUVを照査してガラススライドにプローブを固定させた。
【0274】
3)ZiP Codeテスト用遺伝型分析チップの構成
CYP2D6遺伝子の9個の遺伝型標識(SNP;前記の表で下線にて表示)を用いて11個の遺伝型(CYP2D6*1、*2、*5、*10B、*14A、*14B、*18、*21、*41、*49、*2N)を検証した。
【0275】
【表38】

【0276】
<12−2>ターゲット製作
1)ロング(Long)PCR
CYP 2D6ゲノムDNAサンプル2μl、1X LA緩衝液、2.5mM MgCl2、0.4mM dNTP、下記の表16の各0.2pmol/μlプライマー、LAタクDNAポリメラーゼ(taq DNA polymerase,TAKARA:cat. No. RR002A)2.5ユニット、3次蒸溜水を入れて50μlに合わせた後、94℃で1分間1回変成させた後、98℃10秒、64℃30秒、72℃6分間30サイクル反応後、72℃で1分間延長反応して増幅させた(図44)。(CYP2D6遺伝子は、*5、*2N対立遺伝子(allele)、及びその他対立遺伝子用として各3種類の1STPCR産物を得た。各条件は前記と同一である。)
【0277】
【表39】

【0278】
前記で得られたロングPCR産物0.5μl、1X amplitaq緩衝液、0.2mM dNTP、各プライマー0.5pmol/μlずつ及びAmpli taq gold(Applied Biosystems : cat. No. N8080242)0.5unitに3次蒸溜水を入れて10μlに合わせた後、4℃で5分間1回変成させた後、94℃45秒、57℃45秒、72℃1分間30サイクル反応後、72℃で1分間延長反応して増幅させた。2ND PCRは多重PCRで遂行し、下記の表40の4個のセットで増幅させた。プライマー配列は表41に記載した通りである。
【0279】
【表40】

【0280】
【表41】

【0281】
3)ASPE(allele specific primer extension)反応
前記で得られた多重PCR産物の各6μl、1X amplitaq緩衝液、Cy5dUTP(ジーンケム)10μM、各ASPEプライマー125nM、 Amplitaq gold(Applied Biosystems:cat. No. N8080242)1ユニット、1X Band doctor(ソルジェント)及び3次蒸溜水を入れて20μlに合わせた後、94℃で5分間1回変成させた後、94℃で30秒、60℃で1分、72℃で1分間30回のサイクルで反応して増幅させた(図45参照)。ASPE反応セット及びプライマー配列は下記の表42及び43に記載した通りである。
【0282】
【表42】

【0283】
【表43】

【0284】
4)PCR精製
ASPE反応で得られた1乃至4セットの産物をpoolingしてQiagen精製キット(Qiagen:ca. no. 28106)を用いて製造会社のマニュアルにより精製し、最終溶出体積を50μlとした。
【0285】
精製された各産物を濃縮器(Speed Vacuum concentrator,BioTron社module 4080C)を用いて体積が1乃至2μl程度残るまで乾燥させた。
【0286】
5)チップ混成化
前混成化(prehybridization)緩衝液(25%ホルムアミド、5X SSC、0.1%SDS、10mg/ml BSA)を42℃で温めた後、チップを前記緩衝液に漬けて42℃で30分以上培養した。前記チップを蒸溜水で1分ずつ3回洗浄した後、チップをコニカル(conical)チューブに入れて800rpmで5分間遠心分離して乾燥させた。
【0287】
その後、混成化緩衝液(25%ホルムアミド、5X SSC、0.1%SDS、0.5mg/mlポリA、25μg/ml Cot−1DNA、10%デキストランスルフェート)を予め42℃で温めた後、前記で得られた乾燥されたサンプルをここに溶かした。よく溶かしたサンプル0.5mlをPCRチューブに移した後、95℃で5分間加熱した。混成化チャンバー(chamber)を準備してチャンバー空間に3Mペーパ切れを入れて3X SSCを20μl程度落とした。加熱されたサンプルを前混成化されたチップ上にローディングした後、チャンバーにチップを入れて組立てた後、42℃で一晩混成化させた。
【0288】
前記チップを予め50℃に温められた2X SSC 0.1%SDS溶液で10分間1回洗浄した後、0.1X SSC溶液にて1分ずつ常温で4回洗浄した。洗浄したチップを直ちにコニカルチューブに入れて800rpmで5分間遠心分離して乾燥させた。
【0289】
6)分析
前記のように準備されたチップをAxonのGenePix 4100Bスキャナーを使用して出力波長は650nm付近でスキャニングし、スキャニングされたイメージはGenePix Pro 6.0 softwareを使用して蛍光信号の強さを分析し、その結果を図46及び下記の表44に示した。
【0290】
【表44】

【0291】
その結果、遺伝子分析チップを用いて分析したCYP2D6遺伝子の変異は配列分析を通じて確認された結果と同一であった。
【0292】
<PXR>
実施例13:韓国人のPXR遺伝子の遺伝型分析
<13−1>PXR遺伝子の増幅
54名の健康な被験者から血液を分離した後、Qiagen社のゲノムDNA分離キットを使用してDNAをそれぞれ分離した。PXR遺伝子の全体塩基配列を自動塩基配列分析器(ABI Genetic Analyzer 3130XL)を用いて分析した結果、現在まで報告された総18個の機能的変異の中で6個を確認した。PXR遺伝子は9個のエクソンを含んで総遺伝子の長さが約38kbである。従って、PXR遺伝子を機能的変異があるエクソンを中心に10個の断片に分けてPCRを遂行した。各PCRに使用したプライマーは下記の表45の通りである。本明細書に記載された塩基配列でA、T、G及びCはそれぞれアデニン、チミン、グアニン及びシトシンを意味する。
【0293】
【表45】

【0294】
各プライマーの位置とPCR産物の大きさは下記の表46の通りである。また、ヌクレオチドの位置は文献[HUMAN MUTATION 11:1. 3 (1998)]の命名法により記載した。
【0295】
【表46】

【0296】
各PCR断片に対する反応条件は下記の表47の通りである。
【0297】
【表47】

【0298】
<13−2>PCR産物の塩基配列分析
前記実施例<13−1>で得られた各PCR産物の塩基配列を自動配列分析器及び配列番号:131乃至150のプライマーを用いて分析した。
【0299】
その後、野生型PXR遺伝子の塩基配列(配列番号:130)と比較した結果、総22個のSNPを発見し、そのうち、6個は現在まで報告された機能的変異18個の一部である。22個のSNPは下記の表48に記載された通りであり、その中で報告された機能的変異は♯で表示した。
【0300】
【表48】

【0301】
前記の表48に示すように、PXRの変異遺伝子は7個がプロモーター地域で現れ、残りは3'UTRとイントロン地域で現れ、アミノ酸置換を見せる変異は現れなかった。
【0302】
実施例14:PXR機能的変異型のハプロタイプ分析
前記実施例13で機能性が研究されたことがある6個のPXR遺伝子の機能的変異はその組み合わせによりPXRの機能性に影響を与える可能性がある。
【0303】
従って、本発明者等は実施例13で確認した変異によるハプロタイプをDYNACOM社のSNPAlyzeプログラムを使用して分析した。その結果、下記の表49に示したように14種以上の1%以上の頻度を有するハプロタイプを確認することができた。
【0304】
【表49】

□:各SNPの変異型
【0305】
実施例15:htSNPの選別及び検証
PXR遺伝子のSNPの組み合わせであるハプロタイプがPXRの活性に影響を与える可能性が多くのハプロタイプで報告されたことがある。このように作られたハプロタイプの詳細な情報は最小マーカで確認できる。これら最小マーカをhtSNPといい、前記htSNPはそれぞれのハプロタイプの正確な表示のために必要なマーカであり、種々の組み合わせを含む。この最適化された標識セットであるhtSNP組み合わせを選別するために、前記実施例14で選別された14個のハプロタイプの塩基配列をSNPタガーソフトウェア(http://www.well.ox.ac.uk/〜xiayi/haplotype)を用いて分析した。
【0306】
その結果、図47に示すようなhtSNP組み合わせを選別した。図47に示されている、各選別されたhtSNP組み合わせは最適の標識セットのうちの一つとして、「1」は野生型を、「2」は変異型、そして「V」表示は選択されたそれぞれのhtSNPを示す。htSNPの選別は図1の組み合わせ以外にも他の組み合わせが可能である。
【0307】
その後、探し出した組み合わせの中でディプロタイプを考慮して互いに重ならずにディプロタイプ遺伝型を決定できるかをMatlabソフトウェア(version 7.1,The Math Works Inc.,米国)を使用して分析し、これを用いてチェックした後に組み合わせを決定した。
【0308】
検証の結果、互いに重ならずにディプロタイプ遺伝型を決定できることを確認した。これは本発明で選択したhtSNP組み合わせが互いに同一なものがなく、遺伝型を決定することに当たって不正確な分析が全くないことを示す。
【0309】
実施例16:高速のPXR遺伝子の機能的変異検索
前記実施例13で確認された韓国人で発見されたPXR遺伝子の6個の機能的変異の中で、PXR機能に影響を与える機能的変異を高速に検索するためにスナップショット分析を実施した。被験者のDNAを鋳型としてPCRを遂行し、増幅された産物をスナップショット分析した。PCRに使用したプライマーは下記の表50の通りである。
【0310】
【表50】

【0311】
PCR産物に対する反応条件は下記の表51の通りである。
【0312】
【表51】

【0313】
前記のように増幅された4種類のPCR産物を同量で混合した後、混合されたPCR産物の反応していないプライマーとdNTPなどは残っていればスナップショット過程に影響を与えるので、これらを除去するために、混合されたPCR産物5μl当たりExoSAP−IT(USB社)2μlを入れて37℃で30分間反応させた後、80℃で15分間反応させて残っている酵素を非活性化させた。酵素処理された産物は下記の表52に示すプライマーを用いてマルチプレックススナップショット(multiplex SNaPshot)反応物を作ってPCR反応した。マルチプレックススナップショット反応物及びPCR反応条件をそれぞれ表53及び表54に示した。
【0314】
【表52】

【0315】
【表53】

【0316】
【表54】

【0317】
反応が終わった後に[F]ddNTPを除去するためにスナップショット産物10μlにSAP(USB社)1μlを入れて37℃で60分、65℃で15分間反応させた。反応が全て終わると、反応物0.5μl、Hi−Diホルムアミド(ABI社)9.3μl及びジーンスキャン(GeneScan)−LIZサイズ標準物質(ABI社)0.2μlを混合して95℃で5分間変成させた。その後、3130XL遺伝子分析器(3130XL Genetic Analyzer, ABI社)で分析した。その結果を図48乃至図50に示した。
【0318】
図48乃至50に示されるように、PXR遺伝子の機能的変異型によりピークの色と位置が異なるように表示され、野生型、異型の対立形質を有する変異型(異型)、同型の対立形質を有する変異型(同型)を容易に識別できることを確認した。従って、本発明による分析方法を通じて時間及び費用の面で効率的かつ容易にPXR遺伝子の機能的変異型を分析できることが分かる。
【0319】
<UGT1A>
実施例17:韓国人由来のUGT1A族遺伝子群の変異遺伝子選別
工程1)遺伝体DNAの分離
50名の韓国人を対象として血液をそれぞれ採取した後、遺伝体DNA分離キット(Qiagen社)を用いて採取された各血液試料から遺伝体DNAを分離した。
工程2)UGT1A族遺伝子群の増幅及び全長塩基配列分析
【0320】
前記工程1で分離された総50個の遺伝体DNA試料をそれぞれ鋳型として使用し、下記の表55に記載された各プライマー対を使用してPCRを遂行することによって、各ヒトUGT1A族遺伝子を増幅した。この時、増幅されたUGT1A族の遺伝子名、位置、使用されたプライマー名、その配列番号、大きさ、及びPCR反応条件は下記の表55にそれぞれ示す通りである。
【0321】
【表55】

【0322】
工程3)各UGT1A族遺伝子の変異型分析
前記工程2で増幅させた各UGT1A族遺伝子の全長配列を対象として公知の3100遺伝子分析器(3130x Genetic Analyzer,Applied Biosystems)を用いて配列を分析し、その結果をそれぞれ野生型UGT1A族遺伝子の塩基配列(GenBank accession No.:NT_005120)と比較した。結果を下記の表56及び57に示した。
【0323】
【表56】

【0324】
【表57】

【0325】
工程17−1)UGT1A族遺伝子の機能的変異型選別
前記工程3で得られた韓国人50名で発見されたUGT1A族遺伝子の多型性に基づき、酵素活性の増加または減少のように機能的に関連したと報告された変異型を選別して下記の表58に示した。この時、UGT1A9でのG766A変異型は工程3では確認されなかったが、日本人で報告された機能的変異型であるので下記の表58に追加した。「truncated protein」は蛋白質が翻訳過程中間に突然変異により非正常的に翻訳終結されることをを意味する。
【0326】
【表58】

【0327】
工程17−2)UGT1A族遺伝子の薬物感受性関連多型性選別
前記工程3で得られた韓国人50名で発見されたUGT1A族遺伝子の多型性に基づき、大腸癌治療用抗癌剤であるイリノテカン(Irinotecan)の代謝に関与すると知られたUGT1A1、UGT1A6及びUGT1A9の多型性を選別して下記の表59に示した。この時、UGT1A9でのG766A変異型は工程3では確認されなかったが、日本人で報告された機能的変異型であるので下記の表59に追加した。
【0328】
【表59】

【0329】
実施例18:UGT1A族遺伝子群の機能的変異型及び薬物感受性関連多型性分析
18−1)UGT1A族遺伝子の機能的変異型分析
前記実施例17の被験者の中で各UGT1A族遺伝子の野生型、異型の対立形質を有する変異型、同型の対立形質を有する変異型を有するヒトからの血液を対象として、次の通り本発明によるヒトUGT1A族遺伝子の機能的変異型を確認した。
【0330】
前記実施例17の工程1及び2と同様な方法によりUGT1A1、UGT1A3、UGT1A4、UGT1A6、UGT1A7及びUGT1A9族遺伝子の配列をそれぞれ増幅した。その後、得られた各UGT1A族遺伝子のPCR産物5μl当りエクソSAP−IT(ExoSAP-ITTM,USB Corporation)2μlずつを加えた後、37℃で30分間反応させて残余プライマーなどを除去した。得られた反応物を80℃で15分間反応させて残っているエクソSAP−ITを非活性化させた後、これを2μlずつ取ってスナップショットマルチプレックスレディーリアクションミックス(SNaPshot Multiplex Ready Reaction Mix, ABI)1μl、ハーフターム溶液(Half term buffer、組成: 200 mM Tris-HCl,5 mM MgCl2,pH 9)4μl及び下記の表60に提示された各スナップショットプライマーと混合してスナップショット反応溶液を得た。この時、全体反応溶液の量は10μlになるようにした。
【0331】
【表60】

【0332】
前記各反応溶液を[96℃で10秒、50℃で5秒、60℃で30秒]の40サイクル反復条件下でPCR反応させ、反応が終わった反応溶液10μlにSAP(shrimp alkaline phosphatase)(USB社)1μlずつを加えて37℃で1時間、65℃で15分間反応させた。これをそれぞれ0.5μlずつ取った後、LIZ120(ABI)0.2μlとHi−Diホルムアミド(ABI)9.3μlと混合して96ウェルプレートに分注した。分注された反応試料を95℃で2分間反応させた後、3130x遺伝子分析器(3130x Genetic Analyzer,Applied Biosystems)で分析し、その結果を図51乃至54に示した。
【0333】
図51乃至54に示されるように、各UGT1A族遺伝子の機能的変異型によりピークの色と位置が異なるように表示され、野生型、異型の対立形質を有する変異型(異型)、同型の対立形質を有する変異型(同型)を容易に識別できることを確認し、得られたピークの大きさと種類が前記実施例17において配列分析で得られた表55の結果と100%一致することを確認した。従って、本発明による分析方法を通じて時間及び費用の面で効率的かつ容易にUGT1A族遺伝子群の機能的変異型を分析できることが分かる。
【0334】
また、UGT1A1族遺伝子の−39insTA遺伝型は単一塩基変異に該当せず、 前記のようなスナップショット分析が不可能であるので、次のようなPCR−パイロシーケンシング法を遂行して変異型を確認した。この時、分析に使用したプライマーの配列を下記の表61に示した。この時、プライマーUGT1A1*28Fの場合、5’末端にビオチンが付着していた(配列番号:202参照)。パイロシーケンシングに使用されたプライマーは文献[Clin Chem.,Jul;49(7):1182-5,2003]を参考にした。
【0335】
具体的には、配列番号202と203を用いて収得したPCR産物を鋳型として配列番号204の塩基配列分析用プライマーを反応させた後、パイロシーケンサー(Pyrosequencing社)を用いて変異有無を確認した。
【0336】
収得したPCR産物にバインディングバッファー37μl(Binding buffer pH 7.6、組成:10 mM Tris-HCI、2 M NaCI、1 mM EDTA、0.1% Tween20)にセファロースビーズ (Streptavidin SepharoseTM High performance:Amersham Bioscience)3μlを入れて混合した後、96ウェルプレートに分注して常温で1,4000rpmで5分間反応させた。そして、下記の配列番号204のプライマー(100pmol)0.3μl当たり100μlアニーリングバッファー(1X annealing buffer pH 7.6、組成:20 mM Tris acetate、2 mM MgAc2)を96ウェルプレートに分注した。反応させたサンプルを真空プレップツール(vacuum Prep Tool)を用いて試料を準備し、90℃で3分間加熱した後、常温で冷却した。冷却プレートに Pyro Gold Reagent kit(Biotage)で提供されている酵素混合物(enzyme mixture)、気質混合物(substrate mixture)、dATP、dCTP、dGTP及びdTTPを入れた後、パイロシーケンサーを用いて変異有無を確認した。
【0337】
【表61】

【0338】
18−2)UGT1A族遺伝子の機能的変異型分析
前記の表60に提示されたプライマーの代わりに、下記の表62に提示されたプライマーを同時に使用したことを除いて、前記18−1でのスナップショット分析と同一な工程を遂行してイリノテカン感受性と関連したUGT1A族多型性を分析し、その結果を図55に示した。この時、表62に提示されたプライマーを5'末端に互いに異なる長さのT反復配列を付けてそれぞれの長さを異なるようにした。
【0339】
【表62】

【0340】
その結果、図55に示したように、UGT1A族のイリノテカン感受性関連の色々な多型性を同時に容易に識別できることを確認し、得られたピークの大きさと種類が前記実施例17で配列分析により得られた表55の結果と100%一致することを確認した。
【産業上の利用可能性】
【0341】
前記で考察した通り、本発明によるCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A族遺伝子群の機能的変異型または薬物感受性関連多型性を分析する方法は、今まで確認されたことのない韓国人のCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A族遺伝子の多型性に基づいて得られた最適の探索セットを用いて時間及び費用の面で効率的にCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A族遺伝子群の機能的変異型またはUGT1A族遺伝子群の薬物感受性関連多型性を容易に確認できる方法であるため、韓国人だけではなく、韓国人と遺伝的特性が類似する日本、中国などのアジア圏人種のCYP1A2、CYP2A6、CYP2D6、PXR及びUGT1A族遺伝子の遺伝型分析にも有用に活用され得る。
【特許請求の範囲】
【請求項1】
(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記(c)工程で得られたPCR産物の塩基配列を分析して変異の存在有無を確認する工程;及び(e)前記(d)工程で変異が存在することが確認されたPCR産物の塩基配列からハプロタイプを予測した後、SNPタガー(SNPtagger)ソフトウェアを用いて分析する工程を含む、ヒトCYP1A2遺伝子のhtSNPを選別する方法。
【請求項2】
前記(a)工程の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項1に記載の方法。
【請求項3】
前記(c)工程のプライマーが配列番号2乃至配列番号31のプライマーからなる群より選択されるものである請求項1に記載の方法。
【請求項4】
前記(d)工程の変異が単一塩基多型、遺伝子の欠失及び遺伝子の重複からなる群より選択されるものである請求項1に記載の方法。
【請求項5】
前記(d)工程の配列分析が自動塩基配列分析またはパイロシーケンシング法を用いて行われる請求項1に記載の方法。
【請求項6】
前記(d)工程が前記PCR産物の塩基配列を野生型CYP1A2遺伝子の塩基配列と比較して行われる請求項1に記載の方法。
【請求項7】
前記(a)乃至(d)工程を反復する工程を追加的に含む請求項1に記載の方法。
【請求項8】
(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;(c)前記(b)工程のゲノムDNAを鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G、−163C>A、1514G>A、2159G>A、2321G>C、3613T>C、5347C>T及び5521A>Gからなる群より選択される少なくとも11個のCYP1A2遺伝子の変異の存在有無を調査する工程を含む、ヒトCYP1A2遺伝子のハプロタイプを決定する方法。
【請求項9】
前記(c)工程のプライマーが配列番号2乃至配列番号31、配列番号62及び配列番号63のプライマーからなる群より選択されるものである請求項8に記載の方法。
【請求項10】
前記(d)工程において、−3860G>A、−3598G>T、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査する請求項8に記載の方法。
【請求項11】
前記(d)工程において、−3860G>A、−3113G>A、−2808A>C、−2603insA、−2467delT、−739T>G、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査する請求項8に記載の方法。
【請求項12】
前記(d)工程において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査する請求項8に記載の方法。
【請求項13】
前記(d)工程において、−3860G>A、−3598G>T、2321G>C、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査する請求項8に記載の方法。
【請求項14】
前記(d)工程の変異の存在有無の確認がスナップショット分析を用いて行われる請求項8に記載の方法。
【請求項15】
前記スナップショット分析法は、配列番号64乃至配列番号74のプライマーからなる群より選択されたプライマーを用いて行われる請求項14に記載の方法。
【請求項16】
(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;(c)前記(b)工程のゲノムDNAを鋳型とし、ヒトCYP1A2遺伝子のプロモーター領域を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G及び163C>AからなるCYP1A2遺伝子の単一塩基多型の存在有無を調査する工程を含む、CYP1A2プロモーター遺伝子の変異を検出する方法。
【請求項17】
前記(a)工程の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものであることを特徴とする、請求項8または16に記載の方法。
【請求項18】
前記(b)工程のプライマーが配列番号62及び配列番号63の塩基配列を有することを特徴とする、請求項16に記載の方法。
【請求項19】
前記(d)工程の変異の存在有無の確認がスナップショット分析法を用いて行われる請求項16に記載の方法。
【請求項20】
前記スナップショット分析法が、配列番号64乃至配列番号74のプライマーからなる群より選択されたプライマーを用いて行われる請求項19に記載の方法。
【請求項21】
(a)被験者から生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記工程(c)で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;(e)前記工程(d)で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプ(haplotype)を分析する工程;及び(f)前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析してhtSNPを選別する工程を含む、ヒトCYP2A6遺伝子のhtSNPを選別する方法。
【請求項22】
前記工程(a)の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項21に記載の方法。
【請求項23】
前記工程(c)のプライマーが、配列番号76乃至89のプライマーからなる群より選択されるものである請求項21に記載の方法。
【請求項24】
前記工程(d)の変異は、単一塩基多型(SNP)、遺伝子の欠失及び遺伝子の重複からなる群より選択されるものである請求項21に記載の方法。
【請求項25】
前記工程(d)での変異の存在有無の確認がPCR産物の塩基配列を野生型CYP2A6遺伝子の塩基配列と比較して遂行する請求項21に記載の方法。
【請求項26】
前記工程(a)乃至(e)を反復する工程を追加的に含む請求項21に記載の方法。
【請求項27】
(a)被験者から生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び(d)前記工程(c)で得られたPCR産物の塩基配列において、−48T>G;13G>A;567C>T;2134A>G;3391T>C;6458A>T;6558T>C;6582G>T;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含むCYP2A6遺伝子変異の存在有無を調査する工程を含む、ヒトCYP2A6遺伝子の遺伝型を決定する方法。
【請求項28】
前記工程(a)の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項27に記載の方法。
【請求項29】
前記工程(c)のプライマーが配列番号90、91、102及び103のプライマーである請求項27に記載の方法。
【請求項30】
前記工程(d)において、−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>C;6582G>T;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項31】
前記工程(d)において、−48T>G;22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6458A>T;6558T>C;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項32】
前記工程(d)において、22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6354T>C;6458A>T;6558T>C;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項33】
前記工程(d)において、−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>Cを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項34】
前記工程(d)において、−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6458A>T;6558T>C;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項35】
前記工程(d)において、−48T>G;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>C;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項36】
前記工程(d)の変異の存在有無の調査をスナップショット分析を用いて遂行すること請求項27に記載の方法。
【請求項37】
前記スナップショット分析を配列番号92乃至101のプライマーからなる群より選択されたプライマーを用いて遂行する請求項36に記載の方法。
【請求項38】
(a)ヒトから生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記(c)工程で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;(e)前記(d)工程で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプ(haplotype)を分析する工程;及び(f)前記(e)工程で分析されたハプロタイプの塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析してhtSNPを選別する工程を含む、ヒトCYP2D6遺伝子のhtSNPを選別する方法。
【請求項39】
前記(a)工程の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項38に記載の方法。
【請求項40】
前記(c)工程のプライマーが、配列番号106、配列番号107、配列番号121乃至配列番号127、配列番号129乃至配列番号136、配列番号138、配列番号139、配列番号149及び配列番号150からなる群より選択される塩基配列を有するものである請求項38に記載の方法。
【請求項41】
前記(d)工程の変異が、単一塩基多型、遺伝子の欠失及び遺伝子の重複からなる群より選択されるものである請求項38に記載の方法。
【請求項42】
前記(d)工程の変異の存在有無の確認を、配列分析、電気泳動分析及びRFLP分析からなる群より選択される一つを用いて遂行する請求項38に記載の方法。
【請求項43】
前記(a)乃至(e)工程を反復する工程を追加的に含む請求項38に記載の方法。
【請求項44】
(a)ヒトから生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D6欠失(deletion);及び2D6重複(duplication)を含む少なくとも11個のCYP2D6遺伝子変異の存在有無を調査する工程を含む、ヒトCYP2D6遺伝子の遺伝型を決定する方法。
【請求項45】
前記(a)工程の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項44に記載の方法。
【請求項46】
前記(c)工程のプライマーが、配列番号106、配列番号107、配列番号121乃至配列番号127、配列番号129乃至配列番号136、配列番号138、配列番号139、配列番号149及び配列番号150からなる群より選択される塩基配列を有するものである請求項44に記載の方法。
【請求項47】
前記(d)工程において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項48】
前記(d)工程において、−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>C、3877G>A、4388C>T及び4401C>Tからなる群より一つ;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項49】
前記(d)工程において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1584C>G;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項50】
前記(d)工程において、−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;4125〜4133insGTGCCCACT;−1235A>G;1887insTA;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項51】
前記(d)工程において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;1611T>A;1661G>C及び4180G>Cからなる群より一つ;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;−1235A>G;1887insTA;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項52】
前記(d)工程において、−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;1887insTA;2988G>A;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項53】
前記(d)工程の変異の存在有無の調査を、スナップショット(SNaPshot)分析を用いて遂行する請求項44に記載の方法。
【請求項54】
前記スナップショット分析を、単一塩基多型位置の直ぐそばの塩基が3'末端となり、前記単一塩基多型位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたプライマーを用いて遂行する請求項53に記載の方法。
【請求項55】
前記プライマーが、配列番号141乃至配列番号148、配列番号152及び配列番号153からなる群より選択される塩基配列を有する請求項54に記載の方法。
【請求項56】
(a)検査しようとする遺伝子を抽出した後、多重(multiplex)PCRを遂行して同定しようとするSNP周辺を含むPCR産物を得る工程;(b)各対立遺伝子(allele)の特異的な塩基を同定できるASPE(allele specific primer extension)プライマーを用いてASPE反応を遂行する工程;(c)前記反応産物を遺伝子チップに混成化させる工程;及び(d)前記チップを分析する工程を含む、遺伝子分析チップを用いてヒトCYP2D6遺伝子の遺伝型を決定する方法。
【請求項57】
遺伝子チップが配列番号158乃至184の塩基配列を有するプローブを有する請求項56に記載の方法。
【請求項58】
SNP検査用Zip Codeオリゴ塩基チップを含む、CYP2D6遺伝型分析用キット。
【請求項59】
(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記工程(c)で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;(e)前記工程(d)で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプ(haplotype)を分析する工程;及び(f)前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析してhtSNPを選別する工程を含む、ヒトPXR遺伝子の機能的変異のhtSNPを選別する方法。
【請求項60】
前記工程(a)の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項59に記載の方法。
【請求項61】
前記工程(c)のプライマーが配列番号221乃至配列番号240のプライマーからなる群より選択されるものである請求項59に記載の方法。
【請求項62】
前記工程(d)の変異が単一塩基多型、遺伝子の欠失及び遺伝子の重複からなる群より選択されるものである請求項59に記載の方法。
【請求項63】
前記工程(d)が前記PCR産物の塩基配列を野生型PXR遺伝子の塩基配列と比較して行われる請求項59に記載の方法。
【請求項64】
前記工程(a)乃至(e)を反復する工程を追加的に含む請求項59に記載の方法。
【請求項65】
(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記工程(c)で得られたPCR産物の塩基配列において、−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cからなる群より選択されたPXR遺伝子の機能的変異型の存在有無を調査する工程を含む、PXR遺伝子の機能的変異型を分析する方法。
【請求項66】
前記工程(a)の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項65に記載の方法。
【請求項67】
前記工程(c)のプライマーが、配列番号221、222、225、226、235及び239乃至241のプライマーからなる群より選択されるものである請求項65に記載の方法。
【請求項68】
前記工程(d)の機能的変異型の存在有無の確認がスナップショット分析を用いて行われる請求項65に記載の方法。
【請求項69】
前記スナップショット分析法が配列番号242乃至配列番号247のプライマーからなる群より選択されたプライマーを用いて行われる請求項68に記載の方法。
【請求項70】
(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)で採取された試料から核酸を抽出する工程;(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族遺伝子を増幅する工程;及び(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での−39(TA)6>(TA)7、211G>A、233C>T及び686C>A;UGT1A3での31T>C、133C>T及び140T>C;UGT1A4での31C>T、142T>G及び292C>T;UGT1A6での19T>G、541A>G及び552A>C;UGT1A7での387T>G、391C>A、392G<A、622T>C及び701T>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子の機能的変異型の存在有無を確認する工程を含むことを特徴とする、UGT1A族遺伝子群の機能的変異型を決定する方法。
【請求項71】
(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)で採取された試料から核酸を抽出する工程;(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族遺伝子を増幅する工程;及び(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での211G>A、233C>T及び686C>A;UGT1A6での19T>G、541A>G及び552A>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子変異型の存在有無を確認する工程を含むことを特徴とする、イリノテカン (Irinotecan)に対する感受性と関連したUGT1A族遺伝子の多型性を決定する方法。
【請求項72】
前記工程(a)のヒトが韓国人である請求項70または71に記載の方法。
【請求項73】
前記生物学的試料が、血液、皮膚細胞、粘膜細胞または毛髪である請求項70または71に記載の方法。
【請求項74】
工程(b)の前記核酸がDNAまたはRNAである請求項70または71に記載の方法。
【請求項75】
前記核酸が遺伝体(genomic)DNAである請求項74に記載の方法。
【請求項76】
工程(c)のUGT1A族遺伝子が、UGT1A1、UGT1A3、UGT1A4、UGT1A6、UGT1A7及びUGT1A9族からなる群より選択される請求項70に記載の方法。
【請求項77】
工程(c)のUGT1A族遺伝子が、UGT1A1、UGT1A6及びUGT1A9族からなる群より選択される請求項71に記載の方法。
【請求項78】
工程(d)の前記分析が、スナップショット(SNaPshot)分析、電気泳動分析、パイロシーケンシング(pyrosequencing)分析またはこれらの組み合わせを通じて行われる請求項70または71に記載の方法。
【請求項79】
工程(d)の前記分析が、配列番号:295乃至314の配列を有するプライマーを使用するスナップショット分析、または配列番号:292乃至294の配列を有するプライマーを使用するパイロシーケンシング分析を通じて行われる請求項70に記載の方法。
【請求項80】
工程(d)の前記分析が、配列番号:315乃至322の配列を有するプライマーを使用するスナップショット分析を通じて行われる請求項71に記載の方法。
【請求項1】
(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記(c)工程で得られたPCR産物の塩基配列を分析して変異の存在有無を確認する工程;及び(e)前記(d)工程で変異が存在することが確認されたPCR産物の塩基配列からハプロタイプを予測した後、SNPタガー(SNPtagger)ソフトウェアを用いて分析する工程を含む、ヒトCYP1A2遺伝子のhtSNPを選別する方法。
【請求項2】
前記(a)工程の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項1に記載の方法。
【請求項3】
前記(c)工程のプライマーが配列番号2乃至配列番号31のプライマーからなる群より選択されるものである請求項1に記載の方法。
【請求項4】
前記(d)工程の変異が単一塩基多型、遺伝子の欠失及び遺伝子の重複からなる群より選択されるものである請求項1に記載の方法。
【請求項5】
前記(d)工程の配列分析が自動塩基配列分析またはパイロシーケンシング法を用いて行われる請求項1に記載の方法。
【請求項6】
前記(d)工程が前記PCR産物の塩基配列を野生型CYP1A2遺伝子の塩基配列と比較して行われる請求項1に記載の方法。
【請求項7】
前記(a)乃至(d)工程を反復する工程を追加的に含む請求項1に記載の方法。
【請求項8】
(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;(c)前記(b)工程のゲノムDNAを鋳型とし、ヒトCYP1A2遺伝子またはその断片を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G、−163C>A、1514G>A、2159G>A、2321G>C、3613T>C、5347C>T及び5521A>Gからなる群より選択される少なくとも11個のCYP1A2遺伝子の変異の存在有無を調査する工程を含む、ヒトCYP1A2遺伝子のハプロタイプを決定する方法。
【請求項9】
前記(c)工程のプライマーが配列番号2乃至配列番号31、配列番号62及び配列番号63のプライマーからなる群より選択されるものである請求項8に記載の方法。
【請求項10】
前記(d)工程において、−3860G>A、−3598G>T、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査する請求項8に記載の方法。
【請求項11】
前記(d)工程において、−3860G>A、−3113G>A、−2808A>C、−2603insA、−2467delT、−739T>G、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査する請求項8に記載の方法。
【請求項12】
前記(d)工程において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査する請求項8に記載の方法。
【請求項13】
前記(d)工程において、−3860G>A、−3598G>T、2321G>C、−3113G>A、−2808A>C、−2603insA、−2467delT、−163C>A、1514G>A、2159G>A、5347C>T及び5521A>Gの単一塩基多型の存在有無を調査する請求項8に記載の方法。
【請求項14】
前記(d)工程の変異の存在有無の確認がスナップショット分析を用いて行われる請求項8に記載の方法。
【請求項15】
前記スナップショット分析法は、配列番号64乃至配列番号74のプライマーからなる群より選択されたプライマーを用いて行われる請求項14に記載の方法。
【請求項16】
(a)被験者から生物学的試料を採取する工程;(b)前記(a)工程の採取された試料からゲノムDNAを抽出する工程;(c)前記(b)工程のゲノムDNAを鋳型とし、ヒトCYP1A2遺伝子のプロモーター領域を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−3860G>A、−3598G>T、−3594T>G、−3113G>A、−2847T>C、−2808A>C、−2603insA、−2467delT、−1708T>C、−739T>G及び163C>AからなるCYP1A2遺伝子の単一塩基多型の存在有無を調査する工程を含む、CYP1A2プロモーター遺伝子の変異を検出する方法。
【請求項17】
前記(a)工程の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものであることを特徴とする、請求項8または16に記載の方法。
【請求項18】
前記(b)工程のプライマーが配列番号62及び配列番号63の塩基配列を有することを特徴とする、請求項16に記載の方法。
【請求項19】
前記(d)工程の変異の存在有無の確認がスナップショット分析法を用いて行われる請求項16に記載の方法。
【請求項20】
前記スナップショット分析法が、配列番号64乃至配列番号74のプライマーからなる群より選択されたプライマーを用いて行われる請求項19に記載の方法。
【請求項21】
(a)被験者から生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記工程(c)で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;(e)前記工程(d)で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプ(haplotype)を分析する工程;及び(f)前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析してhtSNPを選別する工程を含む、ヒトCYP2A6遺伝子のhtSNPを選別する方法。
【請求項22】
前記工程(a)の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項21に記載の方法。
【請求項23】
前記工程(c)のプライマーが、配列番号76乃至89のプライマーからなる群より選択されるものである請求項21に記載の方法。
【請求項24】
前記工程(d)の変異は、単一塩基多型(SNP)、遺伝子の欠失及び遺伝子の重複からなる群より選択されるものである請求項21に記載の方法。
【請求項25】
前記工程(d)での変異の存在有無の確認がPCR産物の塩基配列を野生型CYP2A6遺伝子の塩基配列と比較して遂行する請求項21に記載の方法。
【請求項26】
前記工程(a)乃至(e)を反復する工程を追加的に含む請求項21に記載の方法。
【請求項27】
(a)被験者から生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトCYP2A6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び(d)前記工程(c)で得られたPCR産物の塩基配列において、−48T>G;13G>A;567C>T;2134A>G;3391T>C;6458A>T;6558T>C;6582G>T;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含むCYP2A6遺伝子変異の存在有無を調査する工程を含む、ヒトCYP2A6遺伝子の遺伝型を決定する方法。
【請求項28】
前記工程(a)の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項27に記載の方法。
【請求項29】
前記工程(c)のプライマーが配列番号90、91、102及び103のプライマーである請求項27に記載の方法。
【請求項30】
前記工程(d)において、−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>C;6582G>T;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項31】
前記工程(d)において、−48T>G;22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6458A>T;6558T>C;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項32】
前記工程(d)において、22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6354T>C;6458A>T;6558T>C;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項33】
前記工程(d)において、−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>Cを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項34】
前記工程(d)において、−48T>G;13G>A;22C>T;51G>A;567C>T;1620T>C;1836G>T;3391T>C;6458A>T;6558T>C;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項35】
前記工程(d)において、−48T>G;22C>T;51G>A;567C>T;1620T>C;1836G>T;2134A>G;3391T>C;6458A>T;6558T>C;6600G>Tを含み;かつ6091C>T、5971G>A及び5983T>Gからなる群より選択される一つを含む変異の存在有無を調査する請求項27に記載の方法。
【請求項36】
前記工程(d)の変異の存在有無の調査をスナップショット分析を用いて遂行すること請求項27に記載の方法。
【請求項37】
前記スナップショット分析を配列番号92乃至101のプライマーからなる群より選択されたプライマーを用いて遂行する請求項36に記載の方法。
【請求項38】
(a)ヒトから生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記(c)工程で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;(e)前記(d)工程で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプ(haplotype)を分析する工程;及び(f)前記(e)工程で分析されたハプロタイプの塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析してhtSNPを選別する工程を含む、ヒトCYP2D6遺伝子のhtSNPを選別する方法。
【請求項39】
前記(a)工程の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項38に記載の方法。
【請求項40】
前記(c)工程のプライマーが、配列番号106、配列番号107、配列番号121乃至配列番号127、配列番号129乃至配列番号136、配列番号138、配列番号139、配列番号149及び配列番号150からなる群より選択される塩基配列を有するものである請求項38に記載の方法。
【請求項41】
前記(d)工程の変異が、単一塩基多型、遺伝子の欠失及び遺伝子の重複からなる群より選択されるものである請求項38に記載の方法。
【請求項42】
前記(d)工程の変異の存在有無の確認を、配列分析、電気泳動分析及びRFLP分析からなる群より選択される一つを用いて遂行する請求項38に記載の方法。
【請求項43】
前記(a)乃至(e)工程を反復する工程を追加的に含む請求項38に記載の方法。
【請求項44】
(a)ヒトから生物学的試料を採取する工程;(b)前記(a)工程の採取された試料から核酸を抽出する工程;(c)前記(b)工程の核酸を鋳型とし、ヒトCYP2D6遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;及び(d)前記(c)工程で得られたPCR産物の塩基配列において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D6欠失(deletion);及び2D6重複(duplication)を含む少なくとも11個のCYP2D6遺伝子変異の存在有無を調査する工程を含む、ヒトCYP2D6遺伝子の遺伝型を決定する方法。
【請求項45】
前記(a)工程の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項44に記載の方法。
【請求項46】
前記(c)工程のプライマーが、配列番号106、配列番号107、配列番号121乃至配列番号127、配列番号129乃至配列番号136、配列番号138、配列番号139、配列番号149及び配列番号150からなる群より選択される塩基配列を有するものである請求項44に記載の方法。
【請求項47】
前記(d)工程において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項48】
前記(d)工程において、−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>C、3877G>A、4388C>T及び4401C>Tからなる群より一つ;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項49】
前記(d)工程において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1584C>G;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;1611T>A;1758G>A;1887insTA;2573insC;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項50】
前記(d)工程において、−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;4125〜4133insGTGCCCACT;−1235A>G;1887insTA;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項51】
前記(d)工程において、−1426C>T、100C>T及び1039C>Tからなる群より一つ;−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;1611T>A;1661G>C及び4180G>Cからなる群より一つ;1758G>A;1887insTA;2573insC;2988G>A;4125〜4133insGTGCCCACT;−1235A>G;1887insTA;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項52】
前記(d)工程において、−1584C>G;−1426C>T、100C>T及び1039C>Tからなる群より一つ;1611T>A;1758G>A;2573insC;−740C>T、−678G>A、214G>C、221C>A、223C>G、227T>C、232G>C、233A>C、245A>G及び2850C>Tからなる群より一つ;−1245insGA、−1028T>C、−377A>G、3877G>A、4388C>T及び4401C>Tからなる群より一つ;1887insTA;2988G>A;4125〜4133insGTGCCCACT;2D6欠失;及び2D6重複を含む変異の存在有無を調査する請求項44に記載の方法。
【請求項53】
前記(d)工程の変異の存在有無の調査を、スナップショット(SNaPshot)分析を用いて遂行する請求項44に記載の方法。
【請求項54】
前記スナップショット分析を、単一塩基多型位置の直ぐそばの塩基が3'末端となり、前記単一塩基多型位置の隣接部位にアニーリングされる配列を含み、5'末端にはT塩基が付加されたプライマーを用いて遂行する請求項53に記載の方法。
【請求項55】
前記プライマーが、配列番号141乃至配列番号148、配列番号152及び配列番号153からなる群より選択される塩基配列を有する請求項54に記載の方法。
【請求項56】
(a)検査しようとする遺伝子を抽出した後、多重(multiplex)PCRを遂行して同定しようとするSNP周辺を含むPCR産物を得る工程;(b)各対立遺伝子(allele)の特異的な塩基を同定できるASPE(allele specific primer extension)プライマーを用いてASPE反応を遂行する工程;(c)前記反応産物を遺伝子チップに混成化させる工程;及び(d)前記チップを分析する工程を含む、遺伝子分析チップを用いてヒトCYP2D6遺伝子の遺伝型を決定する方法。
【請求項57】
遺伝子チップが配列番号158乃至184の塩基配列を有するプローブを有する請求項56に記載の方法。
【請求項58】
SNP検査用Zip Codeオリゴ塩基チップを含む、CYP2D6遺伝型分析用キット。
【請求項59】
(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーでPCRを遂行する工程;(d)前記工程(c)で得られたPCR産物の塩基配列で変異の存在有無を確認する工程;(e)前記工程(d)で変異が存在すると確認されたPCR産物の塩基配列でハプロタイプ(haplotype)を分析する工程;及び(f)前記工程(e)で分析されたハプロタイプの塩基配列をSNPタガー(SNPtagger)ソフトウェアを用いて分析してhtSNPを選別する工程を含む、ヒトPXR遺伝子の機能的変異のhtSNPを選別する方法。
【請求項60】
前記工程(a)の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項59に記載の方法。
【請求項61】
前記工程(c)のプライマーが配列番号221乃至配列番号240のプライマーからなる群より選択されるものである請求項59に記載の方法。
【請求項62】
前記工程(d)の変異が単一塩基多型、遺伝子の欠失及び遺伝子の重複からなる群より選択されるものである請求項59に記載の方法。
【請求項63】
前記工程(d)が前記PCR産物の塩基配列を野生型PXR遺伝子の塩基配列と比較して行われる請求項59に記載の方法。
【請求項64】
前記工程(a)乃至(e)を反復する工程を追加的に含む請求項59に記載の方法。
【請求項65】
(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)の採取された試料から核酸を抽出する工程;(c)前記工程(b)の核酸を鋳型とし、ヒトPXR遺伝子またはその断片を増幅できるプライマーを用いてPCRを遂行する工程;及び(d)前記工程(c)で得られたPCR産物の塩基配列において、−25385C>T、−24113G>A、7635A>G、8055C>T、11156A>C及び11193T>Cからなる群より選択されたPXR遺伝子の機能的変異型の存在有無を調査する工程を含む、PXR遺伝子の機能的変異型を分析する方法。
【請求項66】
前記工程(a)の生物学的試料が、血液、皮膚細胞、粘膜細胞及び毛髪からなる群より選択されるものである請求項65に記載の方法。
【請求項67】
前記工程(c)のプライマーが、配列番号221、222、225、226、235及び239乃至241のプライマーからなる群より選択されるものである請求項65に記載の方法。
【請求項68】
前記工程(d)の機能的変異型の存在有無の確認がスナップショット分析を用いて行われる請求項65に記載の方法。
【請求項69】
前記スナップショット分析法が配列番号242乃至配列番号247のプライマーからなる群より選択されたプライマーを用いて行われる請求項68に記載の方法。
【請求項70】
(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)で採取された試料から核酸を抽出する工程;(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族遺伝子を増幅する工程;及び(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での−39(TA)6>(TA)7、211G>A、233C>T及び686C>A;UGT1A3での31T>C、133C>T及び140T>C;UGT1A4での31C>T、142T>G及び292C>T;UGT1A6での19T>G、541A>G及び552A>C;UGT1A7での387T>G、391C>A、392G<A、622T>C及び701T>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子の機能的変異型の存在有無を確認する工程を含むことを特徴とする、UGT1A族遺伝子群の機能的変異型を決定する方法。
【請求項71】
(a)ヒトから生物学的試料を採取する工程;(b)前記工程(a)で採取された試料から核酸を抽出する工程;(c)前記工程(b)で抽出された核酸を用いてヒトUGT1A族遺伝子を増幅する工程;及び(d)前記工程(c)で増幅された遺伝子の塩基配列を分析し、UGT1A1での211G>A、233C>T及び686C>A;UGT1A6での19T>G、541A>G及び552A>C;及びUGT1A9での−118T9>T10、726T>G及び766G>Aからなる群より選択されたUGT1A族遺伝子変異型の存在有無を確認する工程を含むことを特徴とする、イリノテカン (Irinotecan)に対する感受性と関連したUGT1A族遺伝子の多型性を決定する方法。
【請求項72】
前記工程(a)のヒトが韓国人である請求項70または71に記載の方法。
【請求項73】
前記生物学的試料が、血液、皮膚細胞、粘膜細胞または毛髪である請求項70または71に記載の方法。
【請求項74】
工程(b)の前記核酸がDNAまたはRNAである請求項70または71に記載の方法。
【請求項75】
前記核酸が遺伝体(genomic)DNAである請求項74に記載の方法。
【請求項76】
工程(c)のUGT1A族遺伝子が、UGT1A1、UGT1A3、UGT1A4、UGT1A6、UGT1A7及びUGT1A9族からなる群より選択される請求項70に記載の方法。
【請求項77】
工程(c)のUGT1A族遺伝子が、UGT1A1、UGT1A6及びUGT1A9族からなる群より選択される請求項71に記載の方法。
【請求項78】
工程(d)の前記分析が、スナップショット(SNaPshot)分析、電気泳動分析、パイロシーケンシング(pyrosequencing)分析またはこれらの組み合わせを通じて行われる請求項70または71に記載の方法。
【請求項79】
工程(d)の前記分析が、配列番号:295乃至314の配列を有するプライマーを使用するスナップショット分析、または配列番号:292乃至294の配列を有するプライマーを使用するパイロシーケンシング分析を通じて行われる請求項70に記載の方法。
【請求項80】
工程(d)の前記分析が、配列番号:315乃至322の配列を有するプライマーを使用するスナップショット分析を通じて行われる請求項71に記載の方法。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】


【図16】


【図17】


【図18】


【図19】


【図20】


【図21】


【図22】


【図23】


【図24】


【図25】


【図26】


【図27】


【図28】


【図29】


【図30】


【図31】


【図32】


【図33】


【図34】


【図35】


【図36】


【図37】


【図38】


【図39】


【図40】


【図41】


【図42】


【図43】


【図44】


【図45】


【図46】


【図47】


【図48】


【図49】


【図50】


【図51】


【図52】


【図53】


【図54】


【図55】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】


【図16】


【図17】


【図18】


【図19】


【図20】


【図21】


【図22】


【図23】


【図24】


【図25】


【図26】


【図27】


【図28】


【図29】


【図30】


【図31】


【図32】


【図33】


【図34】


【図35】


【図36】


【図37】


【図38】


【図39】


【図40】


【図41】


【図42】


【図43】


【図44】


【図45】


【図46】


【図47】


【図48】


【図49】


【図50】


【図51】


【図52】


【図53】


【図54】


【図55】


【公表番号】特表2010−502212(P2010−502212A)
【公表日】平成22年1月28日(2010.1.28)
【国際特許分類】
【出願番号】特願2009−527287(P2009−527287)
【出願日】平成19年6月26日(2007.6.26)
【国際出願番号】PCT/KR2007/003102
【国際公開番号】WO2008/032921
【国際公開日】平成20年3月20日(2008.3.20)
【出願人】(509069788)インジェ・ユニバーシティ・インダストリー−アカデミック・コーポレーション・ファウンデーション (3)
【氏名又は名称原語表記】INJE UNIVERSITY INDUSTRY−ACADEMIC COOPERATION FOUNDATION
【Fターム(参考)】
【公表日】平成22年1月28日(2010.1.28)
【国際特許分類】
【出願日】平成19年6月26日(2007.6.26)
【国際出願番号】PCT/KR2007/003102
【国際公開番号】WO2008/032921
【国際公開日】平成20年3月20日(2008.3.20)
【出願人】(509069788)インジェ・ユニバーシティ・インダストリー−アカデミック・コーポレーション・ファウンデーション (3)
【氏名又は名称原語表記】INJE UNIVERSITY INDUSTRY−ACADEMIC COOPERATION FOUNDATION
【Fターム(参考)】
[ Back to top ]

