ジンクフィンガータンパク質によって標的にされる部位の選択および予め選択された部位に結合するジンクフィンガータンパク質を設計する方法
【課題】ジンクフィンガータンパク質によって標的にされるために、標的から最適なサブ配列を選択するための基準および方法を提供する。
【解決手段】配列5’NNGK3’を有する、1つ以上の、いわゆるD可能サブ部位を含むDNAモチーフを有する、1つ以上の標的セグメントを同定する、標的遺伝子部位の選択方法。または、異なる3つの塩基のトリプレットと9塩基部位内のトリプレット部位の3つの潜在的な位置との間の対応規則を使用した、標的遺伝子内の標的セグメントの選択方法。さらに、所定の選択標的部位に結合するジンクフィンガータンパク質を設計する方法。これらの方法は、上記の手順および基準に従って、標的部位の予備選択の後に使用され得る。設計の方法は、予め特徴づけられたジンクフィンガータンパク質の情報を含むデータベースを使用する。
【解決手段】配列5’NNGK3’を有する、1つ以上の、いわゆるD可能サブ部位を含むDNAモチーフを有する、1つ以上の標的セグメントを同定する、標的遺伝子部位の選択方法。または、異なる3つの塩基のトリプレットと9塩基部位内のトリプレット部位の3つの潜在的な位置との間の対応規則を使用した、標的遺伝子内の標的セグメントの選択方法。さらに、所定の選択標的部位に結合するジンクフィンガータンパク質を設計する方法。これらの方法は、上記の手順および基準に従って、標的部位の予備選択の後に使用され得る。設計の方法は、予め特徴づけられたジンクフィンガータンパク質の情報を含むデータベースを使用する。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、生命情報科学およびタンパク質工学の技術分野にある。
【背景技術】
【0002】
ジンクフィンガータンパク質(ZFP)は、配列特異的様式においてDNAと結合し得るタンパク質である。ジンクフィンガーは、最初に、アフリカツメガエル、Xenopus laevisの卵母細胞由来の転写因子TFIIIAにおいて同定された。これらのタンパク質の1つのクラス(C2H 2クラス)を特徴付ける例示的モチーフは、−Cys−(X)2-4−Cys−(X) 12−His−(X)3-5−His(ここでXは、任意のアミノ酸である)である。単一のフィンガードメインは、約30アミノ酸長であり、そしていくつかの構造研究が、これがジンクを通して配位した βターンにおいて2つの不変のヒスチジン残基および2つの不変のシステイン残基を含むαヘリックスを含むことを実証している。これまでに、10,000を超えるジンクフィンガー配列が、数千の公知のまたは推定転写因子において同定されている。ジンクフィンガードメインは、DNA認識のみならず、RNA結合およびタンパク質−タンパク質結合にも関与する。現在推定されていることは、このクラスの分子が全ヒト遺伝子の約2%を構成することである。
【0003】
Zif268のX線結晶構造である、マウス転写因子由来の3つのフィンガードメインは、同族のDNA配列との複合体において解析され、そして各フィンガーは、周期的な循環によって次のフィンガー上に重ね合わされ得ることが示される。この構造は、各フィンガーが、各DNAトリプレット部分部位と接触する各認識ヘリックス上の−1、2、3および6位で側鎖を有し、3塩基対の間隔にわたって独立してDNAと相互作用することを示唆する。Zif268のアミノ末端は、DNA鎖の3 ’末端に位置し、ここで、最も多く接触する。近年の結果は、いくつかのジンクフィンガーが、標的セグメントにおける第4番めの塩基と結合し得ることを示した。ジンクフィンガータンパク質が最も多く接触する鎖が、標的鎖を設計される場合、いくつかのジンクフィンガータンパク質は、標的鎖における3塩基トリプレット、および非標的鎖上の第4番目の塩基と結合する。この第4番目の塩基は、3塩基の部分部位の3 ’末端すぐの塩基に相補的である。
【0004】
Zif268−DNA複合体の構造はまた、ジンクフィンガータンパク質のDNA配列特異性が、各ジンクフィンガー認識ヘリックス上の4つのヘリックスの位置(−1、2、3および6)にアミノ酸置換体を作製することによって変化し得ることが示唆された。この観察を試験するための、ジンクフィンガーコンビナトリアルライブラリーを用いたファージディスプレイ実験が、1994年に一連の論文で公開されていた(非特許文献1;非特許文献2;非特許文献3)。コンビナトリアルライブラリーを、第1のZif268または中間のZif268のいずれかのフィンガーにおける無作為化した側鎖を用いて構築し、次いで、適切なDNA部分部位が、変化したDNAトリプレットによって置換された、変化したZif268結合部位を選択するために使用した。さらに、導入された変異の性質と結合特異性に生じた変化との間の相関は、変化した結合特異性を有するZFPの設計についての置換の規則の部分的なセットを生じた。
【0005】
非特許文献4は、Zif268の各フィンガーが首尾良く無作為化され、そして新しいトリプレット配列と結合するために選択されるファージディスプレイ法の加工を議論している。この論文は、核ホルモン応答エレメントである、p53標的部位およびTATAボックス配列についてのZFPの選択を報告した。
【0006】
多くの論文が、特定の標的部位を調節するためにZFPを生成する試みを報告している。例えば、非特許文献5は、ZFPがbrc−abl癌遺伝子の発現を抑制するように設計する試みを報告している。ZFPが結合する標的セグメントは、brcおよびablをコードする遺伝子を融合する特異的な癌遺伝子の転座によって作製される連結を重複させるように選択された9つの塩基配列5 ’GCA GAA3’GCCであった。この意図は、この標的部位に対して特異的なZFPが、ablまたはbrc構成要素遺伝子に結合することなく癌遺伝子に結合することである。著者らは、ファージディスプレイを使用して、この標的セグメントへの結合について改変体ZFPのミニライブラリーをスクリーニングした。従って、単離された改変体ZFPが、次いで細胞株における安定してトランスフェクトされたbrc−able構築物の発現を抑制することが報告された。
【0007】
非特許文献6は、Zif268由来の2つのフィンガーをOct−1由来のホメオドメインと融合することによって、新規なDNA結合タンパク質を設計する試みを報告した。次いで、このハイブリッドタンパク質は、キメラタンパク質としての発現に関する転写活性因子と融合された。このキメラタンパク質は、その2つの構成要素の部分配列のハイブリッドを示す標的部位と結合すると報告された。次いで、著者らは、プロモーターに作動可能に連結されるルシフェラーゼ、およびそのプロモーターに近接したキメラDNA結合タンパク質についてのハイブリッド部位を含むレポーターベクターを構築した。著者らは、それらのキメラDNA結合タンパク質がルシフェラーゼ遺伝子の発現を活性化し得ることを報告した。
【0008】
非特許文献7は、ペプチドスペーサーを用いてそれぞれ3つのフィンガーを有する2つの構成要素のジンクフィンガータンパク質を連結し、複合ジンクフィンガータンパク質を形成することを報告している。次いで、この複合タンパク質を、転写活性化ドメインとさらに連結した。得られたキメラタンパク質が、2つの構成要素ジンクフィンガータンパク質によって結合された標的セグメントから形成された標的部位に結合することが、報告された。このキメラジンクフィンガータンパク質が、その標的部位がレポーター遺伝子に作動可能に連結されるプロモーターと近接したレポータープラスミドに挿入される場合、そのレポーター遺伝子の転写を活性化し得ることが、さらに報告された。
【0009】
Chooら、特許文献1、特許文献2、および特許文献3(1998)は、HIV Tat遺伝子内の標的部位に結合するジンクフィンガータンパク質の選択を議論している。Chooらはまた、癌遺伝子rasにおける共通した変異の部位を取り囲む標的部位に結合するためのジンクフィンガータンパク質の選択を議論している。従って、ras内の標的部位は、変異の位置によって制約された。
【0010】
上記の研究はどれも、候補遺伝子内の種々の潜在的な標的部位の各利点を系統的に評価するための判定基準を提供しなかった。Rebarら(前出)、Jamiesonら(前出)およびChooら、PNAS.(1994)前出によるファージディスプレイの研究は、全て天然のZif268結合部位、5 ’GCG TGG GCGc3’の変化に集中し、そして予め決められた標的遺伝子に関してなされなかった。Chooら、Nature(1994)、前出の標的部位の選択は、その部位がbrcセグメントとablセグメントとの間の界面を重複する意図によってのみ構築され、そしてこれは種々の潜在的な標的部位の比較を含まなかった。同様に、GreismanおよびPaboは、公知の調節的役割のために特定の標的部位を選択し、そしてあらかじめ選択された標的遺伝子内の種々の潜在的な標的セグメントの相対的な利点を考慮しなかった。同様に、Chooら(1998)、前出のras内の標的部位の選択は、変異の位置によって強制された。Chooら(1998)のHIV Tatにおける標的部位の選択について判定基準は提供されていない。最終的には、Pomerantzら、前出およびLiuら、前出の両方は、複合体ジンクフィンガーについての人工ハイブリッド標的部位を構築し、次いでその標的部位をレポーター構築物に挿入した。
【0011】
【特許文献1】WO98/53058
【特許文献2】WO98/53059
【特許文献3】WO98/53060
【非特許文献1】Rebarら、Science 263、671−673(1994)
【非特許文献2】Jamiesonら、Biochemistry 33、5689−5695(1994)
【非特許文献3】Chooら、PNAS 91、11163−11167(1994)
【非特許文献4】GreismanおよびPabo、Science 275、657−661(1997)
【非特許文献5】Chooら、Nature 372、645(1994)
【非特許文献6】Pomerantzら、Science 267、93−96(1995)
【非特許文献7】Liuら、PNAS 94、5525−5530(1997)
【発明の開示】
【0012】
本発明は、ジンクフィンガータンパク質によって標的化するために標的配列内の標的部位を選択する方法を提供する。いくつかのこのような方法は、ジンクフィンガータンパク質によって標的化される標的核酸を提供する工程、および5 ’NNx aNy bNzc3’を含む標的核酸内の標的部位を産出する工程を含む。(x,a)、(y,b)および(z,c)の少なくとも1つが(G,K)である場合、各(x,a)、(y,b)および(z,c)は、(N,N)または(G,K)である。NおよびKは、IUPAC−IUB多義性コードである。いくつかの方法において、標的核酸内の複数のセグメントが選択され、そして5 ’NNx aNy bNzc3’を含む複数のセグメントのサブセットが産出される。代表的には、この標的核酸は、標的遺伝子を含む。いくつかの方法において、(x,a)、(y,b)および(z,c)の少なくとも2つが(G,K)である。いくつかの方法において、(x,a)、(y,b)および(z,c)の3つ全てが(G,K)である。いくつかの方法は、5 ’NNx aNy bNzc3’を含む遺伝子の第2のセグメントを同定する工程をさらに含む。ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)のうち少なくとも1つが(G,K)であり、そしてNおよびKはIUPAC−IUB多義性コードである。いくつかの方法において、第2のセグメントにおいて、(x,a)、(y,b)および(z,c)のうち少なくとも2つが(G,K)である。いくつかの方法において、少なくとも1つの(x,a)、(y,b)および(z,c)の3つ全てが(G,K)である。いくつかの方法において、第1および第2のセグメントが、標的部位において5塩基より少ない塩基によって分離される。
【0013】
いくつかの方法は、bNz aNyおよびNNxトリプレットにそれぞれ結合する第1、第2および第3のフィンガーを含むジンクフィンガータンパク質を合成する工程をさらに含む。いくつかのこのような方法において、合成工程は、標的セグメント中のNNx aNyおよびbNzトリプレットにそれぞれ結合する3つのジンクフィンガーおよび第2の標的セグメント中のNNx aNyおよびbNzトリプレットにそれぞれ結合する第2の3つのフィンガーを含む、第1のジンクフィンガータンパク質を合成する工程を含む。いくつかの方法において、第1、第2および第3のフィンガーの各々は、独立して選択されるかまたは設計される。いくつかの方法において、フィンガーは、ジンクフィンガータンパク質の指定、フィンガー構成要素のサブ指定、およびジンクフィンガータンパク質によって結合される核酸配列を含むデータベースから設計される。いくつかの方法において、フィンガーは、標的部位に結合する改変体を同定するために標的部位への特異的結合について、ジンクフィンガー結合タンパク質の改変体をスクリーニングすることによって選択される。
【0014】
いくつかの方法は、標的核酸を含むサンプルをジンクフィンガータンパク質と接触させ、それによってジンクフィンガータンパク質が、標的核酸の存在またはその特定の対立遺伝子形態を明らかにする標的部位に結合する工程を、さらに含む。いくつかの方法において、標的核酸を含むサンプルは、ジンクフィンガータンパク質と接触され、それによってこのジンクフィンガータンパク質が標的部位に結合し、それによってその標的核酸の発現を調節する。
【0015】
いくつかの方法において、標的部位はコード領域中に生じる。いくつかの方法において、この標的部位は、プロモーター、エンハンサーまたは転写開始部位内かまたはそれらに近接して存在する。いくつかの方法において、この標的部位は、プロモーター、調節配列、またはその標的核酸内の多型部位の外側に存在する。
【0016】
別の局面において、本発明は、ジンクフィンガータンパク質によって標的化されるポリヌクレオチド内の標的部位を選択するための代替方法を提供する。これらの方法は、ポリヌクレオチド配列を提供する工程、およびそのポリヌクレオチド配列内の潜在的な標的部位を選択する工程を含む;この潜在的な標的部位は、連続した第1の、第2のおよび第3の塩基のトリプレットを、その潜在的な標的部位において第1、第2および第3の位置で含む。次いで、3つの連続するトリプレットの配列においてトリプレットとトリプレットの位置との間の対応規則(correspondince regime)を適用することによって、複数のサブスコアが決定される。ここで、各トリプレットは、第1の、第2のおよび第3の対応する位置を有し、そしてトリプレットとトリプレットの位置との各組み合わせは、特定のサブスコアを有する。次いで、第1の、第2のおよび第3のトリプレットについてのサブスコアを組み合わせることによって潜在的な標的部位についてスコアを算出する。次いで、選択、決定および算出工程は、さらなるスコアを決定するためのさらなる潜在的な標的部位の第1の、第2のおよび第3の位置で、第1の、第2のおよび第3のトリプレットを含むさらなる潜在的な標的部位上で少なくとも1回繰り返される。次いで、そのスコアを有する少なくとも1つの潜在的な標的部位の産出が提供される。いくつかの方法において、最も高いスコアを有する潜在的な標的部位の産出が提供される。いくつかの方法において、最も高いスコアを有するn潜在的な標的部位の産出が提供され、そしてこの方法は、nについての値のユーザー入力を提供する工程をさらに含む。いくつかの方法において、サブスコアは、サブスコアの産物を形成することいよって組み合わされる。いくつかの方法において、対応規則は、64トリプレットを含み、それぞれが第1の、第2のおよび第3の対応する位置、および192サブスコアを有する。
【0017】
いくつかの方法において、対応規則におけるサブスコアは以下によって決定される:トリプレットおよび対応する位置のサブセットのサブスコアとして、第1の値を、対応規則におけるそのトリプレットの対応する位置として、既存のジンクフィンガータンパク質中の同じ位置からのトリプレットに特異的に結合するフィンガーを含む、既存のジンクフィンガータンパク質が存在するトリプレットおよび位置の各々に割り当てる工程;トリプレットおよび対応する位置のサブセットのサブスコアとして第2の値を、対応規則におけるそのトリプレットの対応する位置よりも、既存のジンクフィンガータンパク質中の異なる位置からのトリプレットに特異的に結合するフィンガーを含む、既存のジンクフィンガータンパク質が存在するトリプレットおよび位置の各々に割り当てる工程;ならびに、トリプレットおよびそのトリプレットに特異的に結合するフィンガーを含む既存のジンクタンパク質が存在しない対応する位置のサブセットのサブスコアとして、第3の値を割り当てる工程。
【0018】
いくつかの方法において、状況パラメーターは、少なくとも1つのトリプレットのスケーリングされたサブスコアを与えるために、少なくとも1つの第1の、第2のおよび第3のトリプレットのサブスコアを有する。いくつかの方法において、この状況パラメーターは、標的部位が塩基配列5 ’NNGK3’(ここで、NNGは、少なくとも1つのトリプレットである)を含む場合、サブスコアと組み合わされる。
【0019】
別の局面において、本発明は、ジンクフィンガータンパク質を設計する方法を提供する。このような方法は、複数のジンクフィンガータンパク質(少なくとも第1の、第2のおよび第3のフィンガーを含む各タンパク質)についての指定、ならびに各ジンクフィンガータンパク質の各3つのフィンガーについてのサブ指定を含み;各ジンクフィンガータンパク質について対応する核酸配列であって、各配列は、各ジンクフィンガータンパク質において、それぞれ少なくとも第1の、第2のおよび第3フィンガーによって特異的に結合される、少なくとも第1の、第2のおよび第3のトリプレットを含み、第1の、第2のおよび第3のトリプレットは、第1の、第2のおよび第3のフィンガーが、ジンクフィンガータンパク質において配列される(N末端からC末端)のと同じ個々の順で、核酸配列(3 ’−5’)において配列されている3つのフィンガーの各々について核酸配列を含むデータベースを使用する。標的部位は、ジンクフィンガータンパク質の設計を提供し、この標的部位は、連続した第1の、第2のおよび第3のトリプレットを3 ’−5’の整列で含む。標的部位における第1の、第2のおよび第3のトリプレットについて、データベース中のジンクフィンガータンパク質の第1の、第2のおよび第3のセットは、同定され、第1のセットは、標的部位における第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、第2のセットは、標的部位における第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、第3のセットは、標的部位における第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含む。工程(c)で同定された第1の、第2のおよび第3のセットにおけるジンクフィンガータンパク質の指定およびサブ指定が、次いで出力される。いくつかの方法は、第1のフィンガー(第1のセットからのジンクフィンガータンパク質由来)、第2のフィンガー(第2のセットからのジンクフィンガータンパク質由来)、および第3のフィンガー(第3のセットからのジンクフィンガータンパク質由来)を含む標的部位に結合するジンクフィンガータンパク質を生成する工程をさらに含む。
【0020】
いくつかの方法は、第1の、第2のおよび第3のセットのサブセットを同定する工程をさらに含む。第1のセットのサブセットは、データベース中のジンクフィンガータンパク質の第1のフィンガー位置から標的部位における第1のトリプレットに特異的に結合するフィンガーを含む、ジンクフィンガータンパク質を含む。第2のセットのサブセットは、データベース中のジンクフィンガータンパク質の第2のフィンガー位置から標的部位における第2のトリプレットに特異的に結合するフィンガーを含む、ジンクフィンガータンパク質を含む;第3のセットのサブセットは、データベース中のジンクフィンガータンパク質の第3のフィンガー位置から標的部位における第3のトリプレットに特異的に結合するフィンガーを含む、ジンクフィンガータンパク質を含む。第1の、第2のおよび第3のセットのサブセットの指定およびサブ指定が、出力される。次いで、第1のサブセット由来の第1のフィンガー、第2のサブセット由来の第2のフィンガー、および第3のサブセット由来の第3のフィンガーを含むジンクフィンガータンパク質が、産生される。上記のいくつかの設計方法において、標的部位は、使用者の入力によって提供される。いくつかの方法において、標的部位は、上記の標的部位選択方法の1つによって提供される。
【0021】
本発明は、任意の上記方法を実行するためのコンピュータープログラム製品をさらに提供する。1つのコンピュータープログラム製品は、ジンクフィンガータンパク質による標的化のためのポリヌクレオチド内の標的部位を選択するための方法を実行する。このような製品は、(a)ポリヌクレオチド配列を提供するためのコード;(b)そのポリヌクレオチド配列内の潜在的な標的部位を選択するためのコード;この潜在的な標的部位は、その潜在的な標的部位における第1の、第2のおよび第3の位置で、第1の、第2のおよび第3のトリプレットの塩基を含む;(c)第1の、第2のおよび第3のトリプレットについてのサブスコア、トリプレットとトリプレット位置との間の対応規則から得られるサブスコアの組み合わせから潜在的な標的部位についてのスコアを算出するためのコード、ここで、各トリプレットは、第1の、第2のおよび第3に対応する位置を有し、そして各対応するトリプレットおよび位置は、特定のサブスコアを有する;(d)さらなるスコアを決定するためのさらなる潜在的な標的部位の第1の、第2のおよび第3の位置での第1の、第2のおよび第3のトリプレットを含むさらなる潜在的な標的部位上で、少なくとも1回工程(b)および(c)を繰り返すためのコード;e)そのスコアを有する潜在的な標的部位の少なくとも1つの出力を提供するためのコード;ならびに(f)そのコードを保持するためのコンピューター読取り可能記憶媒体、を含む。
【0022】
本発明はさらに、上記の任意の方法を実行するためのコンピュータシステムを提供する。ジンクフィンガータンパク質による標的化について、ポリヌクレオチド中の標的部位の選択のための1つのこのようなシステムは、以下を含む:(a)メモリ;(b)システムバス;および(c)プロセッサ。このプロセッサは、以下のことをするために作動可能に配置される:(1)ポリヌクレオチド配列を提供するか、または受け入れること;(2)ポリヌクレオチド配列中の潜在的な標的部位を選択すること;この潜在的な標的部位は、この潜在的な標的部位において第1、第2,および第3の位置で、第1、第2,および第3の塩基のトリプレットを含む;(3)これらの第1、第2,および第3のトリプレットについてのサブスコア(subscore)の組合せから、この潜在的な標的部位についてのスコアを計算すること。このサブスコアは、トリプレットとトリプレット位置との間の対応規則から得られる。ここで、各トリプレットは、第1、第2,および第3に対応する位置を有し、そして各対応するトリプレットおよび位置は、特定のサブスコアを有する;(4)さらなる潜在的な標的部位の第1、第2,および第3の位置で第1、第2,および第3のトリプレットを含むさらなる標的部位上で、工程(2)および(3)を少なくとも一回繰り返して、さらなるスコアを決定すること;(5)そのスコアを有する、少なくとも1つの潜在的な標的部位の出力を提供すること。
【0023】
ジンクフィンガータンパク質を生成するためのさらなるコンピュータプログラム製品は、以下を含む:(a)各タンパク質が少なくとも第1、第2および第3のフィンガーを含む、複数のジンクフィンガータンパク質の指定を含むデータベースを提供するためのコード;このジンクフィンガータンパク質の各々の3つのフィンガーの各々のサブ指定(subdesignation);各ジンクフィンガータンパク質に対応する核酸配列であって、各配列は、各ジンクフィンガータンパク質において、それぞれ少なくとも第1、第2および第3のフィンガーによって特異的に結合される、少なくとも第1、第2および第3のトリプレットを含み、これらの第1、第2,および第3のフィンガーと同一のそれぞれの順序においてこの核酸配列(3 ’−5’)において配列される、これらの第1、第2,および第3のトリプレットが、このジンクフィンガータンパク質(N末端からC末端)において配列される、核酸配列;(b)ジンクフィンガータンパク質の設計のために標的部位を提供するためのコードであって、この標的部位は、少なくとも第1、第2,および第3のトリプレットを含む;(c)標的部位の第1、第2,および第3のトリプレットについて、データベース中のジンクフィンガータンパク質の第1、第2,および第3のセットを同定するためのコードであって、この第1のセットは、この標的部位において第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、この第2のセットは、この標的部位において第2のトリプレットに特異的に結合するフィンガーを含み、この第3のセットは、この標的部位において第3のトリプレットに特異的に結合するフィンガーを含む;(d)工程(c)において同定された第1、第2,および第3のセットにおいて、このジンクフィンガータンパク質の指定およびサブ指定を出力するためのコード、ならびに(e)これらのコードを保持するためのコンピュータ読み出し可能な記録媒体。
【0024】
本発明はさらに、ジンクフィンガータンパク質を生成するためのシステムを提供する。このシステムは、以下を含む:(a)メモリ;(b)システムバス;および(c)プロセッサ。このプロセッサは、以下のことをするために作動可能に配置される:(1)各タンパク質が、少なくとも第1、第2,および第3のフィンガーを含む、複数のジンクフィンガータンパク質の指定、このジンクフィンガータンパク質の各々の3つのファンガーの各々のサブ指定を含むデータベース;各ジンクフィンガータンパク質に対応する核酸配列であって、各配列は、各ジンクフィンガータンパク質において、それぞれ少なくとも第1、第2,および第3のフィンガーによって特異的に結合される、少なくとも第1、第2,および第3のトリプレットを含み、第1、第2,および第3のフィンガーと同一のそれぞれの整列においてこの核酸配列(3 ’−5’)において配列される、第1、第2,および第3のトリプレットが、このジンクフィンガータンパク質(N末端からC末端)において配列される、核酸配列を提供すること;(2)ジンクフィンガータンパク質の設計のために標的部位を提供すること(この標的部位は、少なくとも第1、第2,および第3のトリプレットを含む)、(3)第1、第2,および第3のトリプレットについて、データベース中のジンクフィンガータンパク質の第1、第2,および第3のセットを同定すること(この第1のセットは、この標的部位において第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、この第2のセットは、この標的部位において第2のトリプレットに特異的に結合するフィンガーを含み、この第3のセットは、この標的部位において第3のトリプレットに特異的に結合するフィンガーを含む)、;ならびに(4)工程(3)において同定された第1、第2,および第3のセットにおいて、このジンクフィンガータンパク質の指定およびサブ指定を出力すること。
【0025】
(定義)
ジンクフィンガーDNA結合タンパク質は、亜鉛イオンの配位によってタンパク質構造の安定化を生じる結果として、配列特異的様式においてDNAを結合する、タンパク質またはより大きなタンパク質内のセグメントである。用語、ジンクフィンガーDNA結合タンパク質は、しばしば、ジンクフィンガータンパク質またはZFPと省略される。
【0026】
設計されたジンクフィンガータンパク質は、その設計/組成物が、主に合理的基準から得られる、天然に存在しないタンパク質である。設計のための合理的基準は、既存のZFP設計および結合データの情報を格納するデータベースにおける情報処理するための置換の規則およびコンピュータ化されたアルゴリズムの適用を含む。
【0027】
選択されたジンクフィンガータンパク質は、その産物が主に、ファージディスプレイのような経験的プロセスから得られる、天然に見出されないタンパク質である。
【0028】
用語、天然に存在する、とは、人間によって人工的に産生されたものとは異なるような、天然に見出され得る対象物を記載するために使用される。例えば、天然にある供給源から単離され得、そしてこれが研究室の人間によって意図的に改変されていない生物体(ウイルスを含む)に存在するポリペプチドまたはポリヌクレオチド配列は、天然に存在する。一般に、用語、天然に存在する、とは、病的でない(疾患していない)個体において存在するような対象物(例えば、種を象徴するもの)をいう。
【0029】
核酸は、それが別の核酸配列と機能的な関係に置かれる場合、作動可能に連結される。例えば、プロモーターまたはエンハンサーは、コード配列の転写を増加させる場合に、そのコード配列に作動可能に連結される。作動可能に連結するとは、連結されたDNA配列が代表的に連続し、そして2つのタンパク質コード領域を連結することが必要である場合、連続しかつリーディングフレーム中にあることを意味する。しかし、エンハンサーは、一般に数キロベース以上までプロモーターから分離され、そしてイントロン配列が可変の長さであり得る場合に機能するので、いくつかのポリヌクレオチドエレメントは、作動可能に連結され得るが、連続ではない。
【0030】
例えば、ZFPと特定の標的部位との間の特異的結合親和性は、少なくとも1×10 6M-1の結合親和性を意味する。
【0031】
用語、遺伝子の「発現を調節する」、「発現を阻害する」、および「発現を活性化する」とは、遺伝子の転写を活性化または阻害するようなジンクフィンガータンパク質の能力をいう。活性化は、続く転写阻害の防止(すなわち、遺伝子発現の抑制の防止)を含み、そして阻害は、続く転写活性化の阻害(すなわち、遺伝子の活性化の防止)を含む。調節は、標的遺伝子の発現によって直接的または間接的に影響される任意のパラメーターを決定することによってアッセイされ得る。このようなパラメーターとしては、例えば、RNAまたはタンパク質レベルにおける変化、タンパク質活性における変化、産物のレベルにおける変化、下流の遺伝子発現における変化、レポーター遺伝子の転写における変化(ルシフェラーゼ、CAT、 βガラクトシダーゼ、GFP(例えば、MistiliおよびSpector、Nature Biotechnology 15:961〜964(1997)を参照のこと);シグナル伝達、リン酸化および脱リン酸化、レセプターリガンド相互作用、セカンドメッセンジャーの濃度(例えば、cGMP、cAMP、IP3、およびCa 2+)、細胞増殖、新血管形成、インビトロ、インビボ、ならびにエキソビボにおける変化が挙げられる。このような機能的な効果は、例えば、RNAまたはタンパク質レベルの測定、RNA安定性の測定、下流またはレセプター遺伝子発現の同定を、例えば化学発光、蛍光、比色反応、抗体結合、誘導マーカー、リガンド結合アッセイを介して;細胞内のセカンドメッセンジャー(例えば、cGMPおよびイノシトール三リン酸(IP3)における変化;細胞内のカルシウムレベルにおける変化;サイトカイン放出などによる、当業者に公知な任意の手段によって測定され得る。
【0032】
「調節ドメイン」とは、転写調節活性を有するタンパク質またはタンパク質サブ配列をいう。代表的に、調節ドメインは、転写を調節するためにZFPに共有結合的に、または非共有結合的に連結される。あるいは、ZFPは、調節ドメインを有さずに単独で作用し得るか、または転写を調節するために多数の調節ドメインを有して単独で作用し得る。
【0033】
標的部位内のD可能サブ部位は、モチーフ5’NNGK3’を有する。1つ以上のこのようなモチーフを含む標的部位は、時にD可能標的部位と記載される。D可能サブ部位に結合するように適切に設計されたジンクフィンガーは、時にD可能フィンガーといわれる。さらに、少なくとも1つのD可能サブ部位を含む標的部位に結合するように設計され、そして選択された、少なくとも1つのフィンガーを含むジンクフィンガータンパク質は、時にD可能ジンクフィンガータンパク質といわれる。
【発明を実施するための最良の形態】
【0034】
(I.概説)
1つの局面において、本発明は、遺伝子を調節または検出することにおける使用を意図されるジンクフィンガータンパク質の設計のために予め選択された標的部位内の適切なセグメントを選択する方法に関する。潜在的な標的遺伝子のサイズは、約100〜数100,000(数十万)bpまで広範に変化し得る。ジンクフィンガータンパク質は、このような遺伝子内の小さなサブ配列または標的部位に結合し得る。例えば、3つのフィンガーを含むジンクフィンガータンパク質は、代表的に9または10塩基の標的遺伝子に結合する。本発明は、ジンクフィンガータンパク質によって標的化される標的遺伝子から、最適なサブ配列を選択するための基準および方法を提供する。
【0035】
標的部位を選択する方法のいくつかは、1つ以上のいわゆるD可能サブ部位を含むDNAモチーフを有する1つ以上の標的セグメントを同定するために調査される。D可能サブ部位は、以下で詳細に議論されるように特徴的なDNA配列式によって規定される。ジンクフィンガータンパク質は、ジンクフィンガータンパク質の少なくとも1つの成分フィンガーが、フィンガーによって通常結合される3つの塩基サブ部位の外側のさらなる塩基を接触するようなな様式において、このようなモチーフを結合し得る。2つのD可能部位が標的セグメント中に存在する場合、ジンクフィンガータンパク質の2つの成分フィンガーは、標的部位の4つの塩基に各々結合し得る。3つのD可能サブ部位が標的セグメント中に存在する場合、ジンクフィンガータンパク質の3つの成分フィンガーは、標的部位の4つの塩基に各々結合し得る。一般に、少なくとも1つのD可能サブ部位を含む標的部位に結合するジンクフィンガータンパク質は、D可能サブ部位を有さない標的セグメントに結合するジンクフィンガータンパク質よりも、より高い結合親和性を示す。同様に、2つのD可能サブ部位を有する標的部位に結合するジンクフィンガータンパク質は、一般に、1つのD可能サブ部位を有する標的部位に結合するジンクフィンガータンパク質よりも、より高い結合親和性を示し、そして3つのD可能サブ部位を有するジンクフィンガータンパク質は、一般に、2つのD可能サブ部位を有する標的部位に結合するジンクフィンガータンパク質よりも、より高い結合親和性を示す。機構を理解することは本発明を実施するために必要でないが、より高い結合親和性は、ジンクフィンガーと標的セグメント中の3つの塩基との間に可能な相互作用に対して、ジンクフィンガーと標的セグメント中の4つの塩基との間に可能なさらなる相互作用から生じることが考えられる。一般に、D可能サブ部位を有する標的セグメントの高い結合親和性についての可能性は、このサブ部位をジンクフィンガータンパク質の設計のために標的遺伝子内から選択される標的部位にする。なぜなら、より高い結合親和性は、しばしば、標的遺伝子の調節のより大きな程度、および/または標的遺伝子の調節のより大きな特異性を生じるからである。
【0036】
本発明の他の方法は、D可能サブ部位に対するさらなる基準または代替の基準によって標的遺伝子内の標的セグメントを選択することに関する。このような方法における標的セグメントの選択についての主な基準は、3つの塩基の異なるトリプレットと9つの塩基部位内の1つのトリプレットの3つの可能な位置(すなわち、塩基1〜3、4〜6、および7〜9)との間の対応規則の形成において提供される。例示的な対応規則を表1に示す。この対応規則は、標的部位内のトリプレットおよびトリプレット位置の異なる組合せについて異なる値を提供する。標的遺伝子内の潜在的な標的部位は、対応規則から得られるその成分トリプレットについてサブスコアを組合わせることによりこの部位についてのスコアを決定することによって評価される。異なる潜在的な標的部位のスコアは、ジンクフィンガー結合タンパク質の設計のための標的部位として望ましい特定のセグメントを示す、高いスコアと比較される。
【0037】
別の局面において、本発明は、予め選択された標的部位に結合するジンクフィンガータンパク質を設計する方法を提供する。当然のことながら、これらの方法は、上記の手順および基準に従って、標的部位を予め選択した後に使用され得る。設計の方法は、以前に特徴付けられたジンクフィンガータンパク質についての情報を含むデータベースを使用する。この情報は、以前に特徴付けられたジンクフィンガータンパク質、これらの成分フィンガーのアミノ酸配列、およびタンパク質の各フィンガーによって結合されるヌクレオチドトリプレットの名前および他の指定を含む。データベース中の情報は、選択された標的部位に対して特異性を有する新規なジンクフィンガータンパク質において組合せのための以前と異なる設計からのフィンガーの選択を可能にするアルゴリズムを使用して利用される。
【0038】
(II ジンクフィンガータンパク質)
ジンクフィンガータンパク質は、ジンクフィンガー成分から形成される。例えば、ジンクフィンガータンパク質は、1〜37のフィンガーを有し得、一般に2、3、4、5または6つのフィンガーを有する。ジンクフィンガータンパク質は、標的部位(時に標的セグメントといわれる)を認識し、そしてこれに結合する。この標的部位は、標的遺伝子内に比較的小さなサブ配列を示す。ジンクフィンガータンパク質の各成分フィンガーは、標的部位内のサブ部位に結合し得る。このサブ部位は、同一鎖(時に、標的鎖といわれる)上で3つ連続する塩基のトリプレットを全て含む。このサブ部位はまた、標的鎖上の3つ連続する塩基のすぐ3 ’側の塩基の成分である、反対鎖上の第4の塩基を含んでもよいし、含まなくてもよい。多くのジンクフィンガータンパク質において、ジンクフィンガーは、そのトリプレットサブ部位に、同一のジンクフィンガータンパク質中の実質的に独立な他のフィンガーを結合する。従って、多数のフィンガーを含むジンクフィンガータンパク質の結合特異性は、通常ほぼ、その成分フィンガーの特異性の集合体である。例えば、ジンクフィンガータンパク質が、トリプレットXXX、YYY、およびZZZに対して個々に結合する第1、第2、および第3のフィンガーから形成される場合、このジンクフィンガータンパク質の結合特異性は、3 ’XXX YYY ZZZ5’である。
【0039】
N末端からC末端のジンクフィンガータンパク質においてフィンガーの相対的な整列は、標的中の3’から5 ’方向におけるトリプレットの相対的な順序を決定する。例えば、ジンクフィンガータンパク質が上記の第1、第2、および第3のフィンガーをN末端からC末端に含む場合、このジンクフィンガータンパク質は標的セグメント3 ’XXXYYYZZZ5’に結合する。このジンクフィンガータンパク質が別の順序(例えば、第2のフィンガー、第1のフィンガー、第3のフィンガー)でこのフィンガーを含む場合、このジンクフィンガータンパク質は、異なる順列のトリプレット(この例においては、3 ’YYYXXXZZZ5’)を含む標的セグメントに結合する(BergおよびShi、Science 271、1081〜1086(1996)を参照のこと)。しかし、その成分フィンガーの集合体としてジンクフィンガータンパク質の結合特性の評価は、おおよそであるのみである。これは、同一のタンパク質中の多数のフィンガー結合の状況依存性相互作用に起因する。
【0040】
2つ以上のジンクフィンガータンパク質は、標的特異性を有するように連結され得る。この特異性は、成分ジンクフィンガータンパク質の特異性の集合体である(例えば、KimおよびPabo、PNAS 95、2812〜2817(1998)を参照のこと)。例えば、XXX、YYY、およびZZZにそれぞれ結合する第1、第2、および第3の成分フィンガーを有する第1のジンクフィンガータンパク質は、結合特異性AAA、BBB、およびCCCを有する第1、第2、および第3の成分フィンガーを有する第2のジンクフィンガータンパク質に連結し得る。従って、組合せた第1および第2のタンパク質の結合特異性は、3 ’XXXYYYZZZ_AAABBBCCC5’であり、ここで、下線は、短い介入領域(代表的に、任意の型の0〜5の塩基)を示す。この状況において、標的部位は、介入セグメントによって分離される2つの標的セグメントを含むと考えられ得る。
【0041】
連結は、以下の任意のペプチドリンカーを使用して達成され得る。TGEKP(Liuら、1997、前出);(G4S)n(Kimら、PNAS 93、1156〜1160(1996);GGRRGGGS;LRQRDGERP;LRQKDGGGSERP;LRQKD(G3S)2ERP。あるいは、可撓性リンカーは、DNA結合部位およびペプチド自体の両方をモデリングし得るコンピュータプログラムを使用して、またはファージディスプレイ方法によって、合理的に設計され得る。さらなる改変において、非共有結合的な連結は、2つのジンクフィンガータンパク質のヘテロ二量体形成を促進するドメインと2つのジンクフィンガータンパク質とを融合することによって達成され得る。例えば、一方のジンクフィンガータンパク質がfosと融合され得、そして他方はjunと融合され得る(Barbasら、WO95/119431を参照のこと)。
【0042】
2つのジンクフィンガータンパク質の連結は、哺乳動物ゲノム内の独特な結合特異性を与えるために有利である。代表的な哺乳動物二倍体ゲノムは、3 ×109bpからなる。4つのヌクレオチドA、C、G、およびTがランダムに分布すると仮定すると、所定の9bpの配列は、約23,000回存在する。従って、絶対的な特異性を有する9bp標的を認識するZFPは、このゲノム内の約23,000の部位に結合する可能性を有する。18bpの配列は、3.4 ×1010bp中に1回またはランダムなDNA配列中に約1回存在し、これらの複雑性は、哺乳動物ゲノムの複雑性の10倍である。
【0043】
ジンクフィンガータンパク質の成分フィンガーは代表的に、約30アミノ酸を含み、そして以下のモチーフ(N−C)を有する:
【0044】
【数1】

【0045】
単一のβターン中の2つの不変ヒスチジン残基および2つの不変システイン残基は、亜鉛を通じて配位される(例えば、BergおよびShi、Science 271、1081〜1085(1996)を参照のこと)。上記のモチーフは、結合特異性を与えるジンクフィンガーの領域の分野において標準的である番号付けの取り決めを示す。第1の不変His残基の左(N末端側)のアミノ酸は番号+6を割り当てられ、そしてさらに左の他のアミノ酸は、連続的に減じられた数を割り当てられる。 αヘリックスは、残基1で始まり、そして第2の保存ヒスチジンの後の残基まで伸長する。従って、このヘリックス全体は、11残基と13残基との間の可変の長さである。
【0046】
天然に存在しないZFPまたは改変ZFPを設計または選択する工程は、代表的にフレームワーク残基の供給源としての天然ZFPで開始する。この選択または設計の工程は、所望される結合特異性を与えるように非保存位置(すなわち、位置−1〜+6)を規定するために作用する。1つの適切なZFPは、マウス転写因子Zif268のDNA結合ドメインである。このタンパク質のDNA結合ドメインは、以下のアミノ酸配列を有し、そして標的5 ’GCG TGG GCG3’に結合する:
【0047】
【数2】

【0048】
フレームワーク残基の供給源として適切な別の天然のジンクフィンガータンパク質はSp−1である。ジンクフィンガータンパク質の構築のために使用されるSp−1配列は、Sp−1転写因子のアミノ酸531〜624に対応する。この配列は94アミノ酸長である。Sp−1のアミノ酸配列は以下である:
【0049】
【数3】

【0050】
Sp−1は標的部位5’GGG GCG GGG 3’に結合する。
【0051】
Sp−1の代替的な形態であるSp−1コンセンサス配列は、以下のアミノ酸配列を有する:
【0052】
【数4】

【0053】
(小文字はShiおよびBerg、Chemistry and Biology1,83〜89(1995)からのリーダー配列である)。Sp−1コンセンサス配列について最適な結合配列は、5 ’GGGGCGGGG3’である。他の適切なZFPを以下に記載する。
【0054】
いくつかのジンクフィンガータンパク質の合理的な設計を助ける、多数の置換法則が存在する(DesjarlaisおよびBerg、PNAS 90、2256〜2260(1993);ChooおよびKlug、PNAS 91、11163〜11167(1994);DesjarlaisおよびBerg、PNAS 89、7345〜7349(1992);Jamiesonら、上述;Chooら、WO98/53057、WO98/53058;WO98/53059;WO98/53060を参照のこと)。これらの法則の多くは、偏在性転写因子であるSp−1の3つのフィンガードメインの部位特異的変異誘発により支持される(DesjarlaisおよびBerg,1992;1993)。これらの法則の1つは、DNAトリプレット中の5 ’Gが、認識へリックスの6位にアルギニンを組み込んでいるジンクフィンガーにより結合され得ることである。別の置換法則は、サブ部位の中央のGが、ジンクフィンガーの3位にヒスチジン残基を含むことによって認識され得ることである。さらなる置換法則は、トリプレットの中央のAを認識するためにアスパラギンが組み込まれ得、アスパラギン酸、グルタミン酸、セリンまたはスレオニンがトリプレットの中央のCを認識するために組み込まれ得、そして小さい側鎖を有するアミノ酸(例えば、アラニン)がトリプレットの中央のTを認識するために組み込まれ得ることである。さらなる置換法則は、トリプレットサブ部位の3 ’塩基が以下のアミノ酸を認識へリックスの−1位に組み込むことによって認識され得ることである:Gを認識するためにアルギニン、Aを認識するためにグルタミン、Cを認識するためにグルタミン酸(またはアスパラギン酸)、およびTを認識するためにスレオニン。これらの置換法則はジンクフィンガータンパク質の設計のために有用であるが、これらは全ての潜在的な標的部位を考慮に入れるわけではない。さらに、この法則の基となる仮説、すなわち、ジンクフィンガー中の特定のアミノ酸がサブ部位中の特定の塩基への結合の原因であることは、単なる概算である。フィンガー中の近位のアミノ酸間の前後関係依存性の相互作用または複数のアミノ酸の単塩基への結合もしくはその逆は、既存の置換法則により予測される結合特異性の変動を引き起こし得る。
【0055】
ファージディスプレイ技術は、所望の標的特異性を有するジンクフィンガータンパク質を生成する多数の経験的手段を提供する(例えば、Rebar、米国特許第5,789,538号;Chooら、WO96/06166;Barbasら、WO95/19431およびWO98/543111;Jamiesonら、上述を参照のこと)。この方法は合理的設計と組み合わせて、または合理的設計の代替法として使用され得る。この方法は、変異誘発されたジンクフィンガータンパク質の多様なライブラリーの産生、続いて親和性選択方法を使用して所望のDNA結合特性を有するタンパク質を単離することを含む。この方法を使用するために、実験者は、代表的には以下のように進める。最初に、ジンクフィンガータンパク質の遺伝子を変異誘発させて、結合特異性および/または親和性に重要である領域に多様性を導入する。代表的な適用において、これは−1位、+2位、+3位および+6位、ならびにしばしば+1位、+5位、+8位および+10位のような補助(accessory)位置で1つのフィンガーのランダム化を介して達成される。次に、変異誘発された遺伝子は、繊維状ファージの第III遺伝子(これはコートタンパク質であるpIIIをコードする)との融合物としてファージまたはファージミドにベクターにクローニングされる。このジンクフィンガー遺伝子は膜搬出シグナルペプチドをコードする第III遺伝子のセグメントとpIIIの残余の間に挿入され、その結果ジンクフィンガータンパク質はpIIIとのアミノ末端融合物としてか、または成熟したプロセシングされたタンパク質で発現される。ファージミドベクターを使用する場合、変異誘発されたジンクフィンガー遺伝子はまた、pIIIのファージ粒子へのアセンブリのために必要とされるC末端領域を最小限にコードする第III遺伝子の短縮型バージョンに融合され得る。得られるベクターライブラリーはE.coliに形質転換され、そしてその表面上にコートタンパク質pIIIとの融合物として改変型ジンクフィンガータンパク質を発現する繊維状ファージを産生するために使用される。ファージミドベクターが使用される場合、この工程はヘルパーファージでの重感染を必要とする。次いで、ファージライブラリーは標的DNA部位とともにインキュベートされ、そして親和性選択法を使用してバルクファージ(bulk phage)から高親和性で標的と結合するファージを単離する。代表的には、このDNA標的は固相支持体上に固定化されて、次いでこの支持体は最も強固に結合するファージ以外を取り除くために十分な条件下で洗浄される。洗浄後、支持体上に残存する任意のファージが、ジンクフィンガー−DNA結合を破壊する条件下での溶出を介して回収される。回収したファージを使用して新しいE.coliを感染させ、次いでこれを増幅させてファージ粒子の新しいバッチを産生するために使用する。次いで、選択および増幅は、強固に結合するものについてファージプールを富化するに必要な回数だけ反復され、その結果として配列決定方法および/またはスクリーニング方法を使用してこれらは同定され得る。この方法はpIII融合物について例示されるが、類似の原則がZFP改変体をpVIII融合物としてスクリーニングするために使用され得る。
【0056】
ジンクフィンガータンパク質は、しばしば、融合タンパク質として異種ドメインと共に発現される。ZFPへの付加のための共通のドメインとしては、例えば、転写因子ドメイン(アクチベーター、リプレッサー、コアクチベーター、コリプレッサー)、サイレンサー、オンコジーン(例えば、myc、jun、fos、myb、max、mad、rel、ets、bcl、myb、mosファミリーメンバーなど);DNA修復酵素ならびにその関連因子および修飾因子;DNA再配置酵素ならびにその関連因子および修飾因子;クロマチン関連タンパク質およびそれらの修飾因子(例えば、キナーゼ、アセチラーゼおよびデアセチラーゼ);ならびにDNA修飾酵素(例えば、メチルトランスフェラーゼ、トポイソメラーゼ、ヘリカーゼ、リガーゼ、キナーゼ、ホスファターゼ、ポリメラーゼ、エンドヌクレアーゼ)ならびにそれらの関連因子および修飾因子が挙げられる。ZFPが標的遺伝子の発現を抑制するために使用されるべきである場合、ZFPとの融合に好ましいドメインは、ヒトKOX−1タンパク質由来のKRAB抑制ドメインである(Thiesenら、New Biologist 2、363〜374(1990);Margolinら、Proc.Natl.Acad.Sci.USA 91、4509〜4513(1994);Pengueら、Nucl.Acids Res.22:2908〜2914(1994);Witzgallら、Proc.Natl.Acad.Sci.USA 91、4514〜4518(1998))。活性化を達成するために好ましいドメインとしては、HSV VP16活性化ドメイン(例えば、Hagmannら、J.Virol.71、5952〜5962(1997)を参照のこと)、核ホルモンレセプター(例えば、Torchiaら、Curr.Opin.Cell.Biol.10:373〜383(1998)を参照のこと);核因子 κBのp65サブユニット(BitkoおよびBarik、J.Virol.72:5610〜5618(1998)ならびにDoyleおよびHunt、Neuroreport 8:2937〜2942(1997));Liuら、Cancer Gene Ther.5:3〜28(1998))、または人工キメラ機能的ドメイン(例えば、VP64(Seifpalら、EMBO J.11、4961〜4968(1992)))が挙げられる。
【0057】
ポリペプチド化合物(例えば、ZFP)の投与に重要な因子は、このポリペプチドが細胞の原形質膜または細胞内区画(例えば、核)の膜を横切る能力を有することを保証することである。細胞膜は脂質−タンパク質二重層からなり、これは小さな非イオン性の親油性化合物を自由に通し、そして極性化合物、高分子および治療剤または診断剤に対して本質的に不透過性である。しかしながらタンパク質および他の化合物(例えば、リポソーム)が、細胞膜を横切ってZFPのようなポリペプチドをトランスロケーションさせる能力を有することが記載されている。
【0058】
例えば、「膜トランスロケーション(membrane translocation)ポリペプチド」は、膜トランスロケーションキャリアとして作用する能力を有する、両親媒性または疎水性アミノ酸のサブ配列を有する。1つの実施態様において、ホメオドメインタンパク質は細胞膜を横切ってトランスロケーションする能力を有する。ホメオドメインタンパク質であるAntennapediaの最も短いインターナライゼーション可能なペプチドは、タンパク質の第3のへリックス(アミノ酸43位〜58位)であることが見出された(例えば、Prochiantz、Current Opinion in Neurobiology 6:629〜634(1996)を参照のこと)。別のサブ配列である、シグナルペプチドのh(疎水性)ドメインは、類似の細胞膜トランスロケーション特性を有することが見出された(例えば、Linら、J.Biol.Chem.270:14255〜14258(1995)を参照のこと)。
【0059】
ZFPの細胞内への取り込みを容易にするために、本発明のZFPに連結され得るペプチド配列の例としては、以下が挙げられるがこれらに限定されない:HIVのtatタンパク質の11アミノ酸ペプチド;p16タンパク質のアミノ酸84〜103に対応する20残基のペプチド配列(Fahraeusら、Current Biology 6:84(1996)を参照のこと);Antennapediaの60アミノ酸長ホメオドメインの第3へリックス(Derossiら、J.Biol.Chem.269:10444(1994));カポージ線維芽細胞増殖因子(K−FGF)h領域(Linら、上述)のようなシグナルペプチドのh領域;またはHSV由来のVP22トランスロケーションドメイン(ElliotおよびO ’Hare、Cell 88:223〜233(1997))。増強された細胞性取り込みを提供する他の適切な化学的部分もまた、ZFPに化学的に連結され得る。
【0060】
トキシン分子もまた、細胞膜を横切ってポリペプチドを輸送する能力を有する。しばしば、このような分子は少なくとも2つの部分からなる(「二元トキシン」と呼ばれる):トランスロケーションまたは結合ドメインまたはポリペプチド、ならびに別個のトキシンドメインまたはポリペプチド。代表的に、トランスロケーションドメインまたはポリペプチドは細胞性レセプターに結合し、次いでこのトキシンは細胞内に輸送される。いくつかの細菌性トキシン(Clostridium perfringensイオータトキシン、ジフテリアトキシン(DT)、PseudomonasエキソトキシンA(PE)、百日咳トキシン(PT)、Bacillus anthracisトキシンおよび百日咳アデニル酸シクラーゼ(CYA)を含む)は、細胞のサイトゾルへとペプチドを内部融合物またはアミノ末端融合物として送達する試みにおいて使用されてきた(Aroraら、J.Biol.Chem.、268:3334〜3341(1993);Perelleら、Infect.Immun.、61:5147〜5156(1993);Stenmarkら、J.Cell Biol.113:1025〜1032(1991);Donnellyら、PNAS 90:3530〜3534(1993);Carbonettiら、Abstr.Annu.Meet.Am.Soc.Microbiol.95:295(1995);Seboら、Infect.Immun.63:3851〜3857(1995);Klimpelら、PNAS U.S.A.89:10277〜10281(1992);およびNovakら、J.Biol.Chem.267:17186〜17193(1992))。
【0061】
このようなサブ配列は、細胞膜を横切ってZFPをトランスロケーションさせるために使用され得る。ZFPは、便利に、このような配列に融合され得るか、またはこのような配列を用いて誘導体化され得る。代表的には、このトランスロケーション配列は融合タンパク質の一部として提供される。必要に応じて、リンカーはZFPとトランスロケーション配列とを連結させるために使用され得る。任意の適切なリンカー、例えば、ペプチドリンカーが使用され得る。
【0062】
(III.標的遺伝子の選択)
ジンクフィンガータンパク質は、任意の標的ポリヌクレオチド配列の発現を調整するために使用され得る。この配列は、例えば、ゲノム、cDNAまたはRNAあるいは発現された配列タグ(EST)であり得る。代表的には、標的ポリヌクレオチドとしては、遺伝子またはそのフラグメントが挙げられる。用語、遺伝子は、以下を含むように広範に使用される:例えば、エキソン領域、イントロン領域、5 ’UTR、3’UTR、5’隣接配列、3 ’隣接配列、プロモーター、エンハンサー、転写開始部位、リボソーム結合部位、調節部位、ポリアデニル化部位が挙げられる。標的遺伝子は、細胞性、ウイルス性または他の供給源由来(純粋に理論的な配列を含む)のものであり得る。標的遺伝子配列は、データベース(例えば、GenBank)、刊行物から得られ得るか、またはデノボで得られ得る。標的遺伝子としては、感染を停止するために発現の抑制が使用され得る病的なウイルスおよび微生物由来の遺伝子が挙げられる。病原性ウイルスの例としては、肝炎(A型、B型またはC型)、ヘルペスウイルス(例えば、ZVZ、HSV−1、HSV−6、HSV−IIおよびCMV、エプスタイン−バールウイルス)、HIV、エボラ、アデノウイルス、インフルエンザウイルス、フラビウイルス、ECHOウイルス、ライノウイルス、コクサッキーウイルス、コロナウイルス(cornovirus)、RSウイルス、流行性耳下腺炎ウイルス、ロタウイルス、麻疹ウイルス、風疹ウイルス、パルボウイルス、ワクシニアウイルス、HTLVウイルス、デング熱ウイルス、パピローマウイルス、軟属腫ウイルス(molluscum virus)、ポリオウイルス、狂犬病ウイルス、JCウイルスおよびアルボウイルス性脳炎ウイルスが挙げられる。病原性細菌のいくつかの例としては、クラミジア、リケッチア細菌、ミコバクテリア、ブドウ球菌、連鎖球菌(treptoccus)、肺炎球菌、髄膜炎菌および淋菌(conococcus)、クレブシエラ、プロテウス、セラチア、シュードモナス、レジオネラ、ジフテリア、サルモネラ、バシラス、コレラ、破傷風、ボツリヌス中毒、炭疽、ペスト、レプトスピラおよびライム病細菌が挙げられる。
【0063】
標的遺伝子はまた、疾患の原因であるヒトまたは他の哺乳動物由来の遺伝子を含む。いくつかのこのような遺伝子はオンコジーン、腫瘍サプレッサーまたは増殖因子であり、これらはガンの原因である。オンコジーンの例としては、hMSH2(Fishelら、Cell 75、1027〜1038(1993))およびhMLH1(Papadopoulosら、Science 263、1625〜1628(1994))が挙げられる。増殖因子のいくつかの例としは、線維芽細胞増殖因子、血小板由来増殖因子、GM−SCF、VEGF、EPO、Erb−B2およびhGHが挙げられる。他のヒト遺伝子は、被験体を微生物またはウイルスによる感染に罹患し易くすることにより、疾患の原因となる。例えば、CCR5レセプターをコードする遺伝子の特定の対立遺伝子は、被験体をHIVによる感染に罹患し易くする。他のヒト遺伝子(例えば、アミロイド前駆体タンパク質またはApoEをコードする遺伝子)は、他の疾患(例えば、アルツハイマー病)の原因である。
【0064】
標的遺伝子としてはまた、他の供給源が原因である疾患に対する防御機構を提供する、ヒトまたは他の哺乳動物の遺伝子が挙げられる。例えば、腫瘍リプレッサー遺伝子は、ガンに対する防御を提供する。このような遺伝子の発現は望ましく、そしてジンクフィンガータンパク質は発現を活性化するために使用される。
【0065】
標的遺伝子としてはまた、通常発現が止められているかまたは低レベルで発現されている遺伝子が挙げられるが、これらは活性化を通してある個体に存在する別の欠損型遺伝子を置換するために使用され得る。例えば、胎児ヘモグロビン(hemaglobin)遺伝子(これは成体ヒトにおいては通常不活性である)を活性化して、鎌状赤血球貧血を有する個体における欠損型 β−グロビン遺伝子を代用し得る。
【0066】
標的遺伝子としてはまた、その抑制または活性化が植物特性の改善(例えば、改善された作物生産、病害耐性または除草剤耐性)を導く植物遺伝子が挙げられる。例えば、FAD2−1遺伝子の発現の抑制は、オレイン酸の増加ならびにリノール酸およびリノレン酸の減少という利点を生じる。
【0067】
(IV.D可能サブ部位に結合するためのジンクフィンガータンパク質の設計)
(1.方法)
本発明は、標的遺伝子を選択し、そして1〜6(以上)のD可能サブ部位を含む遺伝子内の標的部位を同定する方法を提供する。次いで、予め選択された部位に結合するジンクフィンガータンパク質が合成され得る。標的部位選択のこれらの方法は、標的セグメント中の1つ以上のD可能サブ部位の存在が、D可能サブ部位を欠損する標的セグメントに結合するジンクフィンガータンパク質と比較して、その部位に結合するように選択されたかまたは設計されたジンクフィンガータンパク質により高い結合親和性の可能性を付与するという本発明者らの認識を、一部で前提とする。この洞察を支持する実験的証拠は、実施例2〜9に提供される。
【0068】
D可能サブ部位は、適切に設計された単一のジンクフィンガーが、標的部位の3塩基までよりもむしろ4塩基までに結合することを可能にする標的部位の領域である。このようなジンクフィンガーは、二本鎖標的セグメント(標的鎖)の1つの鎖上の塩基のトリプレットおよび他方の鎖上の第4の塩基に結合する(図2を参照のこと)。単一のジンクフィンガーが、4塩基の標的セグメントに結合することは、標的鎖の配列上およびジンクフィンガーのアミノ酸配列上の両方に束縛を課する。標的鎖内の標的部位は、「D可能」サブ部位モチーフ5 ’NNGK3’を含むべきであり、ここで、NおよびKは、従来のIUPAC−IUB多義性コードである。このような部位に結合するためのジンクフィンガーは、−1位にアルギニン残基および+2位にアスパラギン酸(または次に好ましくはグルタミン酸)を含むべきである。−1位におけるアルギニン残基は、D可能サブ部位中のG残基と相互作用する。ジンクフィンガーの+2位におけるアスパラギン酸(またはグルタミン酸)残基は、D可能サブ部位中のK塩基に相補的な反対鎖の塩基と相互作用する。名前D可能サブ部位を与えるのは、アスパラギン酸(シンボルD)と、反対鎖塩基(第4の塩基)との間の相互作用である。D可能サブ部位式から明らかなように、D可能サブ部位には2つのサブタイプ:5 ’NNGG3’および5’NNGT3 ’がある。前者のサブ部位には、ジンクフィンガーの+2位のアスパラギン酸またはグルタミン酸が、D可能サブ部位に対する反対鎖中のCと相互作用する。後者のサブ部位では、ジンクフィンガーの+2位のアスパラギン酸またはグルタミン酸が、D可能サブ部位に対する反対鎖中のAと相互作用する。一般に、NNGGは、NNGTより好ましい。
【0069】
3つのフィンガーを持つジンクフィンガータンパク質の設計では、標的部位は、その中で、このタンパク質の少なくとも1つのフィンガー、そして好ましくは、2つまたは3つのフィンガーが、標的部位中のD可能サブ部位を結合する能力を有するように選択されるべきである。これは、以下の式を有するより大きな標的遺伝子内から標的部位を選択することにより達成され得る:5 ’NNx aNy bNzc3’、ここでセット(x、a)、(y、b)および(z、c)の各々は、(N、N)または(G、K)のいずれかであり、(x、a)、(y、b)および(z、c)の少なくとも1つは(G、K)であり、そしてNおよびKは、IUPAC−IUB多義性コードである。
【0070】
換言すれば、3つのセット(x、a)、(y、b)および(z、c)の少なくとも1つは、このセットの第1の位置がGであり、そして第2の位置がGまたはTであることを意味するセット(G、K)である。3つのセットのうち(G、K)ではないもの(もしあれば)は、このセットの第1の位置が任意のヌクレオチドにより占められ得、そしてこのセットの第2の位置が任意のヌクレオチドにより占められ得ることを意味する(N、N)である。例として、セット(x、a)は(G、K)であり得、そしてセット(y、b)および(z、c)は、両方とも(N、N)であり得る。
【0071】
式5’NNx aNyb Nzc3’において、NNx aNyおよびbNzのトリプレットは、ジンクフィンガータンパク質中の3つのフィンガーにより結合される標的鎖上の塩基のトリプレットを示す。強調された塩基の相補物は、非標的鎖上で結合する潜在的な第4の塩基の部位である。x、yおよびzのたった1つがGであり、そしてこのGにKが続く場合、標的部位は単一のD可能サブ部位を含む。例えば、xのみがGであり、そしてaがKである場合、この部位は、D可能サブ部位を強調して、以下のように読める:
【0072】
【化1】

【0073】
zではなく、xおよびyの両方がGであり、そしてaおよびbがKである場合、そのときは、標的部位は、以下のように、1つのこのような部位を太字でそして他をイタリックで示して、2つの重複するD可能サブ部位を有する:
【0074】
【化2】

【0075】
x、yおよびzの3つのすべてがGであり、そしてa、bおよびcがKである場合、そのときは、標的セグメントは、以下のように、3つのD可能サブ部位である、以下:
【0076】
【化3】

【0077】
を含み、D可能サブ部位は、太字、イタリックおよび下線で示される。
【0078】
従って、本発明の方法は、標的遺伝子を選択すること、および式5’NNxaNybNzc3 ’に一致する標的部位のための遺伝子の潜在的なサブ配列内を系統的にサーチすることにより行われ、ここで、(x、a)、(y、b)および(z、c)の各々は、(N、N)または(G、K)であり;
(x、a)、(y、b)および(z、c)の少なくとも1つは(G、K)であり、そしてNおよびKは、IUPAC−IUB多義性コードである。
このような方法のいくつかでは、潜在的な標的遺伝子のいずれかの鎖上の10の連続塩基の潜在的なサブ配列毎に、それが上記の式に一致するか否かを決定するため、そしてもしそうであれば、D可能サブ部位がいくつあるか評価される。代表的には、このような比較は、コンピューターにより実施され、そして上記式に一致する標的部位のリストが出力される。必要に応じて、このような標的部位は、D可能サブ部位がいくつあるかに従って、異なるサブセットで出力され得る。
【0079】
改変法では、本発明の方法は、各々が独立に上記の式に一致する第1および第2の標的セグメントを同定する。このような方法における2つの標的セグメントは、束縛されて、標的遺伝子中、互いに隣接するかまたは近接(すなわち、約0〜5塩基内)する。近接する標的セグメントの選択の基礎となる戦略は、第1および第2の標的セグメントにそれぞれ特異的な2つの成分ジンクフィンガータンパク質の連結により形成されるジンクフィンガータンパク質の設計を可能にする。これらの原理は、任意の数の成分フィンガーを持つジンクフィンガータンパク質により結合される標的部位を選択するために拡張され得る。例えば、9フィンガーのタンパク質に適切な標的部位は、各々が上記の式に一致する3つの成分セグメントを有する。
【0080】
上記の方法により同定される標的部位は、他の規準によるさらなる評価を受け得るか、またはこのような部位に特異的なジンクフィンガータンパク質の設計または選択(必要であれば)および産生のために直接用いられ得る。潜在的な標的部位を評価するためのさらなる規準は、遺伝子内の特定領域への近接度である。ジンクフィンガータンパク質が、それ自身に関する細胞遺伝子を抑制するために用いられるべき場合(すなわち、このジンクフィンガータンパク質を、抑制する部分に連結することなく)、最適位置は、転写開始の部位か、または約50bp上流もしくは下流内であるか、あるいは転写複合体の形成(Kim&Pabo、J.Biol.Chem.(1977))を妨害するためか、または必須エンハンサー結合タンパク質と競合するためにエンハンサーエレメント内にあるようである。しかし、ZFPが、KRABリプレッサードメインまたはVP16アクチベータードメインのような機能的ドメインに融合される場合、結合部位の位置の選択は、かなりより柔軟であり、そして既知の調節領域の外側であり得る。例えば、KRABドメインは、KRABが結合する場所から少なくとも3kbまで、プロモーターの転写を抑制し得る。従って、調節配列、または多形性部位のような標的遺伝子を含まないか、または標的遺伝子との重要なセグメントと重複しない標的部位が選択され得る。標的セグメントをさらに評価するためのその他の規準としては、このようなセグメントまたは関連セグメントへのジンクフィンガータンパク質結合の先立つ利用可能性、および/または所定の標的セグメントを結合するために新たなジンクフィンガータンパク質を設計する容易さが挙げられる。選択プロセスにおけるこのような基準の履行は、以下でより詳細に論議される。
【0081】
一旦、標的セグメントが選択されたなら、このセグメントに結合するジンクフィンガータンパク質は、種々のアプローチにより提供され得る。最も簡単なアプローチは、既に標的部位に結合することが知られている現存するコレクションから予め特徴付けられたジンクフィンガータンパク質を提供することである。しかし、多くの例では、このようなジンクフィンガータンパク質は存在しない。代替のアプローチは、現存するジンクフィンガータンパク質および結合特異性のデータベース中の情報を用いて、新たなジンクフィンガータンパク質を設計することである。このアプローチを以下により詳細に記載する。さらなるアプローチは、上記で議論されるような置換規則に基づきジンクフィンガータンパク質を設計することである。なおさらなる代替は、ファージディスプレイのような経験的プロセスにより所定の標的に対して特異性を持つジンクフィンガータンパク質を選択することである。このような方法のいくつかでは、ジンクフィンガータンパク質の各成分フィンガーは、他の成分フィンガーとは独立に設計または選択される。例えば、各フィンガーは、予め存在する異なったZFPから得られ得るか、または各フィンガーは、別のランダム化および選択を受け得る。
【0082】
一旦、ジンクフィンガータンパク質が、所定の標的セグメントに対して、選択、設計、またはそうでなく提供されたなら、ジンクフィンガータンパク質またはコードDNAが合成される。ジンクタンパク質を合成およびジンクタンパク質のコードDNAを発現する例示の方法は以下に記載される。次いで、ジンクフィンガータンパク質またはそれをコードするポリヌクレオチドは、このジンクフィンガータンパク質が結合する標的部位を含む標的遺伝子の発現の調整、または分析に用いられ得る。
【0083】
(2.D可能ジンクフィンガータンパク質)
ジンクフィンガータンパク質は、それが、ポリヌクレオチド配列5’NNGK3 ’である、少なくとも1つのD可能サブ部位の第4の塩基に結合し得るフィンガーを含む場合、D可能として記載される。D可能ジンクフィンガーを設計するための好適なフレームワークは、ヒト野生型Sp−1 DNA結合ドメインである。ヒト転写因子Sp−1の標的は、5’GGG GCG GGG3 ’であり、そしてこのタンパク質のフィンガー1および2は、R−1D+2配置を有する。設計されたZFPは、3つのフィンガーの各々の認識ヘリックス中を除いてSp−1と同一であり得、ここでは、配列は、それらが相互作用するトリプレットの各々を認識するように設計される。マウスZFP Zif268は、部位GCG TGG GCGを結合し、3つすべてのフィンガーにR−1D+2配置を有して、また適切である。
【0084】
D可能サブ部位に結合し得るジンクフィンガータンパク質の設計のためのフレームワーク残基の供給源としての他のジンクフィンガータンパク質は、いくつかの代替供給源からのZFPから得られか、または由来し得る。例えば、ショウジョウバエ Drosophila melanogasterのTTK転写調節タンパク質は、その認識ヘリックスおよびそのDNA部位の両方の配列に関して良く特徴付けられている。このタンパク質は、たった2つのフィンガーを有し、そして6塩基の標的に結合し、そこでフィンガー2が、第1のDNAトリプレットと相互作用し、そしてフィンガー1がこの部位の第2のトリプレットを認識する。この部位は、第1および第2のトリプレットの接合部に存在するGGタイプD可能サブ部位を持つ5 ’AAGGAT3’であり、そしてフィンガー2は、R−1 D+2配列を有する。他の適切なZFPは、単細胞真核生物Sacchromyces cerevisiae中に見出される。ADR遺伝子産物は、ADHプロモーター内に結合することにりADH遺伝子の発現を調節することが知られている。TTKについて上記に記載されるように、ADR ZFP結合ドメインは、2つのフィンガーを有し、そして6塩基の標的TTGGAGに結合する。このフィンガー2認識ヘリックスは、D可能サブ部位を持つ標的部位へのZFP結合に適切なR−1 D+2配列を有する。
【0085】
(IV.対応規則による標的部位の選択)
本発明は、標的遺伝子内から標的部位を選択するためのさらなる、または代替の方法をさらに提供する。これらの方法は、部分的には、個々のフィンガーにより結合される異なる3塩基サブ部位(トリプレット)が、ジンクフィンガータンパク質設計について異なる望ましさを有すること、これらの異なる望ましさが、数値として表現され得ること、および標的部位を含む3つの個々のトリプレットに対する数値が、標的部位に対する全体スコアを与えるために組み合わせられ得るという洞察を前提とする。異なる標的部位の相対的なメリットは、それらの相対的スコアから比較され得る。
【0086】
この方法は、ZFPによる検出または調整のための標的部位をその中で選択することを望む、代表的には遺伝子またはcDNAであるポリヌクレオチド配列を提供することにより実施する。実際、代表的には、ポリヌクレオチド配列の2つの鎖に対して2つの配列を提供するが、単純さのために、この方法は、単一のポリヌクレオチド配列について例示される。このようなポリヌクレオチド配列内から、連続する第1、第2および第3の、塩基のトリプレットを含む少なくとも9塩基の潜在的な標的部位が選択される。これらのトリプレットは、5 ’−3配向における塩基1が塩基1であるとして、第1のトリプレットが部位の塩基7〜9を、第2のトリプレットが部位の塩基4〜6を、そして第3のトリプレットが部位の塩基1〜3を占めるという点で連続している。トリプレットの第1、第2、および第3としてのこの指定は任意であり、そして逆転され得る。しかし、第1のトリプレットが塩基7〜9を、第2のトリプレットが塩基4〜6を、そして第3のトリプレットが塩基1〜3を占めるとして指定することにより、N−C末端配向における3つのフィンガーZFPの第1、第2、および第3のフィンガーは、標的部位の第1、第2および第3のトリプレットに結合する。別の様式で見ると、N末端からC末端へのジンクフィンガータンパク質順序にある、第1、第2および第3のフィンガーは、3 ’−5’配向で並べられた標的部位中の第1、第2および第3のトリプレットにそれぞれ特異的である。
【0087】
次いで、サブスコアを、各トリプレットについて、トリプレットと標的部位内の対応する位置との間の対応規則から決定される。例示の対応規則を表1に提供する。対応規則は、9塩基の標的部位内のその3つの可能な位置にある各トリプレットに対する3つの値を提供するマトリックスである。この表は、64の可能なトリプレットの各々について3つの値を提供する。例えば、潜在的な標的部位5 ’AAA AAG AAC3’を考慮する。AACトリプレットは、標的部位の第1の位置(塩基7〜9)にあり、そして表1から1のサブスコアを割り当てられる。AAGトリプレットは、標的部位の第2の位置(塩基4〜6)にあり、そして8のサブスコアを割り当てられる。AAAトリプレットは、標的部位の第3の位置(塩基1〜3)にあり、そして8のサブスコアを割り当てられる。次いで、潜在的な標的部位中にある3つのトリプレットのサブスコアは、例えば、乗算または加算またはその他の関数により、組み合わせられる。例えば、3つのトリプレットのサブスコアの乗算は、1 ×8×8=64の組み合わせスコアを与える。
【0088】
次いで、このプロセスは、第2の潜在的な標的部位について繰り返される。第2の潜在的な標的部位の3成分トリプレットの各々についてサブスコアが決定され、そして組み合わせスコアが、第2の潜在的な標的部位に対して計算される。次いで、このプロセスは、さらなる潜在的な標的部位について繰り返され得る。必要に応じて、このプロセスは、目的の標的遺伝子のいずれかの鎖中の少なくとも9塩基の可能な連続サブ配列毎に繰り返され得る。目的のすべての可能な標的部位のスコアが決定された場合、スコアが比較される。一般に、高スコアは、ZFPの設計について標的部位の望ましさを示す。高スコアで同定された1つ以上の標的部位が、スコアとともに出力され得る。
【0089】
対応規則における値の指定は、ジンクフィンガータンパク質の設計または選択のために、1つのトリプレットサブ部位を他より望ましくする任意の規準を反映し得る。表1の例示の対応規則における値は、所定のヌクレオチドトリプレットを結合することが知られる先に特徴付けられたZFPの利用可能性を反映する。標的部位の所定の位置における所定のトリプレットについて、所定の位置におけるトリプレットを含む標的セグメントに特異的に結合する、1つ以上の先に特徴付けられたZFPが存在する場合、そのときは、このトリプレットと所定の位置との組み合わせは、スコア10が割り当てられる。所定の位置における所定のトリプレットについて、所定の位置におけるトリプレットを含む標的部位を特異的に結合する、先に特徴付けられたZFPが存在しないが、異なる位置におけるトリプレットに特異的に結合する1つ以上の先に特徴付けられたZFPが存在する場合、そのときは、このトリプレットは、スコア8が割り当てられる。所定のトリプレットおよび所定の位置について、所定の位置または別の位置のいずれかでトリプレットを結合する、先に特徴付けられたZFPが存在しない場合、このトリプレットおよび位置は値1が割り当てられる。
【0090】
これらの値10、8および1は例示に過ぎず、そして他の値が用いられ得る。さらに、より洗練された値の割り当てを用い得、それはまた、他の因子のなかでとりわけ、異なる結合親和性、特異性およびD可能部位の存在を考慮する。このようなスキームでは、強力な結合親和性を有する先のZFPが存在するトリプレットと位置の組み合わせが、代表的には、より低い結合親和性を有する先のZFPが存在するトリプレットと位置との組み合わせより高い値を与える。
【0091】
より大きな配列内の潜在的な標的部位の選択およびスコアの計算は、代表的には、適切にプログラムされたコンピューターにより実施され、それは、1つ以上の潜在的な標的部位を、それらのスコアとともに出力する。必要に応じて、ユーザー入力が、このようなコンピューターに提供され、いくつの潜在的な標的部位が出力されるべきかを特定する。例えば、ユーザーは、最高のスコア出力を持つnの潜在的な標的部位を有するように選択し得る。ここでnは、ユーザーの裁量である。ユーザーはまた、閾値スコアを特定し得、それは、出力されるべき潜在的な標的部位に等しいまたはそれを超過するに違いない。
【0092】
上記の方法の改変法では、潜在的な標的部位は、対応表中の値および1つ以上のD可能サブ部位の存在の両方に基づいて評価され得る。これは、状況パラメーターのユーザー入力により達成され、トリプレットの状況が、D可能サブ部位の存在を示す場合、トリプレットと特定位置との1つ以上の組み合わせに対するスケーリングされたスコアを提供する。例えば、Aが続くトリプレット5 ’NNG3’は、D可能サブ部位を提供しない。しかし、Kが続く5 ’NNG3’は、D可能部位を提供する。ユーザーは、5 ’NNG3’にKが続く場合、5’NNG3 ’トリプレットのサブスコアの値を増大する状況パラメーターを入力することを選択し得る。次いで、このトリプレットに対するスケーリングされたサブスコアを、他のトリプレットに対するサブスコアまたはスケーリングされたサブスコアと組み合わせ、潜在的な標的部位に対する全体スコアを与える。
【0093】
さらなる改変例では、上記の分析を実施するコンピューターは、プログラムされて、互いにそれらの物理的近接度により決定される対で高スコアを受ける特定の標的セグメントを出力する。両方が互いの約5塩基内にある高スコアを受ける対とされた標的セグメントは、各々が3つのフィンガーを有する2つの成分ジンクフィンガータンパク質の連結により形成される6フィンガーのジンクタンパク質の設計に適切な標的である。
【0094】
上記の方法により同定される潜在的な標的部位は、さらなる評価を受け得るか、またはジンクフィンガータンパク質の設計または(必要であれば)選択および産生のために直接用いられ得る。ジンクフィンガータンパク質は、上記のD可能サブ部位を含む潜在的な標的セグメントについて記載された同じ方法を用いて、このような標的部位に対して設計および合成され得る。
【0095】
(V.ZFPのデータベース設計)
本発明は、予め選択された標的部位に対するZFPの設計のための方法を提供する。これらの方法は、上記の標的部位選択の方法と組み合わせるか、または標的部位選択のその他の方法による使用のために適切である。
【0096】
新たなZFPを設計することにおいて、予め特徴付けられたZFPおよびそれらの標的部位中の固有の情報を使用し、それによってデノボ設計または選択のための必要性を最小にすることが一般に有利である。標的部位選択のように、いくつかの因子がこのプロセスに含まれる。標的部位中の各トリプレットサブ部位について、フィンガーが、現存するZFP中で利用可能であるばかりではなく、このようなフィンガーがまた、提案された設計におけるように、この現存するタンパク質中の同じ位置からのそれらの個々のトリプレットサブ部位と接触する場合、設計は促進される。例えば、3つの現存するZFPの対および標的部位を考える:フィンガーF1−F2−F3を持つZFPにより結合される、5 ’GCG TGG GAC3’(ここでF3はGCGと、F2はTGGと、そしてF1はGACと相互作用する)、フィンガーF4−F5−F6を持つZFPにより結合される、5 ’AAG GAG GTG3’、およびフィンガーF7−F8−F9を持つZFPにより結合される5 ’CCG TGA GCA3’、ならびにそれに対してZFPが設計されるべき標的部位5 ’GCG GAG GCA3’である。この状況では、新規タンパク質F7−F5−F3は、この新規タンパク質中の各フィンガーが、それが得られたデータベースタンパク質中でそうであったように、新規タンパク質中で同じ相対的位置中に存在して、5 ’GCG GAG GCA3’に結合する。この設計は、新規ZFP中の各フィンガーの、先のそのZFPの各フィンガーと類似の環境は、このフィンガーが、親におけるように、新規ZFP中で同様の特異性および親和性で結合するらしいことを意味するので有利である。従って、ジンクフィンガータンパク質の結合特徴は、その成分フィンガーの凝集物であるという一般規則は、保持されるようである。
【0097】
新規ジンクフィンガータンパク質はまた、現存するタンパク質で利用可能であるが、設計されるタンパク質中では同じ位置でない、成分フィンガーから設計され得る。例えば、上記の現存するZFP部位対のセットを用いて、タンパク質F3−F7−F5が、配列5 ’GAG GCA GCG3’を結合するように設計され得る。この新規タンパク質において、フィンガーは、それらの個々の親タンパク質中とは異なる位置を占める。所定のフィンガーが、それがZFP中で占めるその位置にかかわらず、そのトリプレット特異性および親和性を保持する近似に対してとはいえ、実際、状況の影響が、フィンガーが異なるジンクフィンガータンパク質中で異なる位置を占める場合、そのトリプレットサブ部位に対するこのフィンガーの特異性および/または親和性における変化を引き起こすようである。従って、先に特徴付けられたZFP中とは異なる位置を占める成分フィンガーから形成されたZFPは、代表的には、なおこの部位に結合するが、特異性または親和性は、時に、予想されるより異なる(代表的にはより低い)。
【0098】
最後に、利用可能である予め存在するフィンガーがないトリプレットを含む予め選択された標的部位には、完全に新規なフィンガーは、規則を基礎にしたアプローチまたはファージディスプレイを用いて設計または選択され得る。
【0099】
本発明は、上記の原理に従って、予め選択された標的部位に対する新規ZFPの設計における現存ZFPについての情報を含むデータベースを系統的に用いる方法を提供する。代表的データベースの構成を表9に示す。代表的には、このデータベースは、予め特徴付けられたZFPのコレクションの各々に対する指定を含む。このZFPは、天然のZFPまたは改変体ZFPであり得る。この指定は、例えば、各ZFPを示す名前またはシンボルであり得る。このデータベースはまた、ZFP中のフィンガーの各々に対するサブ指定を含む。代表的には、このサブ指定は、フィンガー中の選択された位置を占めるアミノ酸残基の形態にある。例えば、表9では、このサブ指定は、従来の番号付けに従って−1位〜+6位を占めるアミノ酸である。このデータベースは、各ジンクフィンガータンパク質により結合される標的核酸セグメントをさらに含む。通常、この核酸セグメントは、3塩基の3つのトリプレットを含む。この塩基の3つのトリプレットは、1つの配列として連結されるか、または別の配列として含められ得る。9塩基の標的部位中の塩基が、5 ’末端から連続的に番号付けされる場合、第1のトリプレットは塩基7〜9を占め、第2のトリプレットは塩基4〜6を占め、そして第3のトリプレットは塩基1〜3を占める。標的セグメント内のトリプレット位置のこの指定に従って、ジンクフィンガータンパク質の第1のフィンガー(すなわち、N−末端に最も近い)は、第1のトリプレットに結合し、第2のフィンガーは、第2のトリプレットに結合し、そして第3のフィンガーは第3のトリプレットに結合する。このデータベースはまた、必須ではないが、その標的部位に対するZFPの結合親和性または解離定数のようなさらなる情報を含み得る。
【0100】
標的部位は、このデータベースを用いてジンクフィンガータンパク質の設計のために提供される。いくつかの方法では、この標的部位は、ユーザー入力により提供される。他の方法では、この標的部位は、上記の標的部位の選択の任意の方法からの出力として提供される。代表的には、この標的部位は、少なくとも3つのトリプレットを形成する少なくとも9つの塩基を含む。この3つの成分トリプレットは、その5塩基 ’を塩基1として割り当てて、標的部位の塩基7〜9、4〜6および1〜3を占める、それぞれ第1、第2および第3のトリプレットと指定される。この標的部位における第1のトリプレットに対して、コンピューターは、このトリプレットに結合するフィンガーを含むジンクフィンガータンパク質についてデータベースを調査する。このコンピューターは、ジンクフィンガータンパク質に関する記録を貯蔵し、そしてそれによって、第1のトリプレットに結合するそれらのフィンガーを同定する。必要に応じて、このコンピューターは、第1のフィンガー位置で標的部位の第1のトリプレットに結合するフィンガーを含むジンクフィンガータンパク質と他の位置における標的部位の第1のトリプレットに結合するフィンガーを含むジンクフィンガータンパク質との間を区別する。そうである場合、このコンピューターは、2つのサブセットのジンクフィンガータンパク質を、別々の記録として蓄える。次いで、このプロセスを、標的部位にある第2のトリプレットについて繰り返す。コンピューターは、第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を同定する。必要に応じて、コンピューターは、現存するジンクフィンガータンパク質の第2の位置から第2のトリプレットを結合するジンクフィンガーまたは異なる位置にある第2のトリプレットを結合するジンクフィンガーとの間を区別する。最後に、コンピューターは、標的部位の第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を同定する。必要に応じて、このコンピューターは、現存するジンクフィンガータンパク質の第3の位置から第3のトリプレットを結合するジンクフィンガーと、または別の位置から第3のトリプレットを結合するジンクフィンガーとの間を区別する。標的セグメント中の、第1、第2および第3のトリプレットの各々に結合するZFPを調査した後、このコンピューターは、同定されたZFPの指定、および第1、第2および第3のトリプレットに結合するフィンガーのサブ指定を出力する。必要に応じて、このコンピューターは、第1のフィンガー位置から第1のトリプレットを結合するZFPのサブセット、および他の位置から第1のトリプレットを結合するZFPのサブセット;および第2のフィンガー位置からおよびその他の位置から第2のトリプレットを結合するZFPの対応するサブセット、および第3のフィンガー位置からおよびその他の位置から第3のトリプレットを結合するZFPの対応するサブセットの別々の出力を提供する。
【0101】
このコンピューターによる情報出力は、予め選択された標的に結合する新規ジンクフィンガータンパク質の設計および合成に用いられ得る。例えば、この出力が、標的の第1のトリプレットを結合するフィンガーXを持つZFP1、標的の第2のトリプレットに結合するフィンガーYを含むZFP2、および標的の第3のトリプレットに結合するフィンガーZを含むZFP3を含む場合、新規ZFPは、この順序(N−末端からC−末端)でフィンガーXYZを含んで合成され得る。このコンピューターが、所定のトリプレットに結合する複数の異なるフィンガーを含む複数の異なるジンクフィンガータンパク質を出力する場合、ユーザーは、フィンガーが、設計されるZFP中におけるのと、データベースタンパク質中の同じ位置からの特定のトリプレット位置に結合するか否かに依存して、フィンガー間を選択し得る。例えば、Xが標的部位中の第1のトリプレットに結合するフィンガーXYZを含むZFP1は、一般に、フィンガーCが標的部位中の第1のトリプレットに結合するフィンガーABCを含むZFP2より好まれる。従って、代表的には、この標的セグメントを結合するために設計されたZFP中の第1のフィンガー位置を占めるために、フィンガーCよりもフィンガーXを用いる。しばしば、コンピュータープログラムは、各々が特定のトリプレットを結合するフィンガーを含む、2つのZFPを同定し、そして各ZFPにおいて、このフィンガーは、それが由来するデータベースタンパク質において、意図された設計ZFP中におけるのと同じ位置を占める。このような場合には、それらの個々の標的に対する結合親和性に基づいて2つのフィンガー間を、しばしば、より高い結合親和性が好適であるとして選択する。必要に応じて、コンピューターはまた、フィンガーにより結合される対応するトリプレットに対する1つ以上のフィンガーに対して提案されるアミノ酸置換の出力を提供する。
【0102】
データベース分析は、3つのフィンガーを有する予め特徴付けられたジンクフィンガータンパク質について主に例示されるが、このようなデータベースは、より少ないかまたはより多い数のフィンガーを持つジンクフィンガータンパク質に関する情報を代わりにまたは付加的に貯蔵し得る。同様に、このようなデータベースは、3つのフィンガーより少ないかまたはより多くを有するジンクフィンガータンパク質の設計に用いられ得る。例えば、本発明のいくつかのデータベースは、2つのフィンガーのみを持つZFPに関する情報およびまたはそれに代わって3つのフィンガーを持つZFPに関する情報を貯蔵する。2つのフィンガーのみを持つZFPは、2つのトリプレットのみを持つ対応する標的部位を有する。2つのフィンガーZFPに関する情報は、上記と本質的に同じ様式で9つの塩基標的部位に結合する3フィンガーZFPの設計に用いられ得る。しかし、2フィンガータンパク質中の2つのフィンガーの相対的位置と、3フィンガージンクフィンガータンパク質中の3つのフィンガーの相対的位置との間には正確な対応はない。この論点は、2つの方法で扱われ得る。第1に、2フィンガータンパク質中のすべてのフィンガーは、3フィンガータンパク質中のフィンガーとは異なる位置を占めるとして効率的に処理され得る。従って、2フィンガータンパク質が、所定のトリプレットに結合するフィンガーを含む場合、コンピューターはこの情報を出力し、そしてこのフィンガーが、データベース2フィンガータンパク質中で、設計される3フィンガータンパク質におけるのと同じ位置に生じないことを示す。あるいは、2フィンガータンパク質中の第1(N末端)のフィンガーは、3フィンガータンパク質中の第1または第2のフィンガーのいずれかと等価であると考慮され得る。2フィンガータンパク質中の第2のフィンガーは、3フィンガータンパク質中の第2または第3のフィンガーのいずれかと等価であると考慮され得る。従って、コンピューターが、それに対してジンクフィンガータンパク質が設計されるべき標的部位中の第1のトリプレットに結合する第1(N末端)のフィンガーを持つ2フィンガータンパク質を同定する場合、コンピューターは、2フィンガータンパク質が、適切なフィンガーと、データベースタンパク質において、設計されるべき3フィンガータンパク質におけるのと同じ位置を供給することを出力し得る。
【0103】
(VII.ZFPの産生)
ZFPポリペプチドおよびこれをコードする核酸は、組換え遺伝学の分野における慣用的な技法を用いて作製され得る。本発明における使用の一般的方法を開示する基礎的テキストは、Samrookら、Molecular Cloning、A Laboratory Manual(第2版,1989);Kriegler、Gene Transfer and Expression:A Laboratory Manual(1990);およびCurrent Protocols in Molecular Biology(Ausubelら編、1994)を含む。さらに、約100塩基より少ない核酸は、The Midland Certified Reagent Company(mcrc@oligos.com)、The Great American Gene Company(http://www.genco.com)、ExpressGen Inc.(www.expressgen.com)、Operon Technologies Inc.(Alameda、CA)のような、任意の種々の市販供給源から注文生産され得る。同様に、ペプチドは、PeptidoGenic(pkim@ccnet.com)、HTI Bio−products、inc.(http://www.htibio.com)、BMA Biomedicals Ltd(U.K.)、Bio.Synthesis、Inc.のような任意の種々の供給源から注文生産され得る。
【0104】
オリゴヌクレオチドは、Beaucage & Caruthers Tetrahedron Letts.22:1859−1862(1981)により最初に記載された固相ホスホルアミダイトトリエステル法に従い、Van Devanterら、Nucleic Acids Res.12:6159−6168(1984)に記載のように、自動化合成器を用いて化学的に合成され得る。オリゴヌクレオチドの精製は、変性ポリアクリルアミドゲル電気泳動によるか、または逆相HPLCによるかのいずれかによる。クローン化遺伝子および合成オリゴヌクレオチドの配列は、クローニング後に、例えば、Wallaceら、Gene 16:21−26(1981)の二本鎖テンプレートを配列決定するための鎖停止方法を用いて確認され得る。
【0105】
代表的には、2つの代替法を用いて、新たに設計されたDNA結合性ペプチドを発現するために必要なコード配列を生成する。1つのプロトコルは、6つの重複オリゴヌクレオチドを利用するPCRを基礎にするアセンブリ手法である(図3)。3つのオリゴヌクレオチド(図3中のオリゴ1、3、および5)は、認識ヘリックスの間のDNA結合ドメインの部分をコードする「ユニバーサル」配列に対応する。代表的には、これらのオリゴヌクレオチドは、すべてのジンクフィンガー構築物について一定のままである。他の3つの「特異的」オリゴヌクレオチド(図3中オリゴ2、4および6)を、認識ヘリックスをコードするために設計する。これらのオリゴヌクレオチドは、認識ヘリックス上の主に位置1、2、3および6における置換を含み、それらを、異なるDNA結合ドメインの各々に対して特異的にする。
【0106】
PCR合成は2段階で実施される。第1に、二本鎖DNAテンプレートを、低温アニーリング工程を伴う4サイクルPCR反応で、6オリゴヌクレオチド(3つのユニバーサル、3つの特異的)を組み合わせることにより生成し、それによってオリゴヌクレオチドをアニーリングし、DNA「足場」を形成する。この足場中のギャップを、高い忠実度の熱安定性ポリメラーゼ(TaqおよびPfuポリメラーゼの組み合わせもまた十分である)によりフィルインする。構築の第2段階では、このジンクフィンガーテンプレートを、シャトルベクター中または直接発現ベクター中へのクローニングのために、いずれかの末端で制限部位を取り込むために設計された外部プライマーにより増幅する。
【0107】
新たに設計されたDNA結合性タンパク質をクローニングする代替法は、所望のZFPの特異的領域をコードする相補的オリゴヌクレオチドをアニーリングすることに依存する。この特定の適用は、オリゴヌクレオチドが、最終の連結工程の前にリン酸化されることを必要とする。通常、これは、アニーリング反応をセットアップする前に実施される。簡単に述べれば、このタンパク質の定常領域をコードする上記「ユニバーサル」オリゴヌクレオチド(上記のオリゴ1、2および3)を、それらの相補的オリゴヌクレオチドとアニールする。さらに、フィンガー認識ヘリックスをコードする上記「特異的」オリゴヌクレオチドを、それらの個々の相補的オリゴヌクレオチドとアニールする。これらの相補的オリゴは、上記で述べたプロトコルでポリメラーゼによって先にフィルインされた領域をフィルインするために設計される。共通オリゴ1およびフィンガー3に相補的なオリゴは加工されて、次の工程で選択されたベクター中へのクローニングにおいて用いられる制限部位に特異的なオーバーハング配列を残す。第2のアセンブリプロトコルは、最初のプロトコルと、次の局面で異なる:新たに設計されたZFPをコードする上記「足場」は、完全に合成DNAからなり、それによって、ポリメラーゼフィルイン工程をなくし、さらに、ベクター中にクローニングされるフラグメントは増幅を必要としない。最後に、配列特異的オーバーハングを残す設計は、挿入フラグメントの制限酵素消化の必要性をなくする。あるいは、ZFP認識ヘリックスへの変化は、従来の部位特異的変異誘発方法を用いて作製され得る。
【0108】
両方のアセンブリ方法は、新たに設計されたZFPをコードする得られるフラグメントが、ベクター中に連結されることを必要とする。最終的に、ZFPをコードする配列は、発現ベクター中にクローニングされる。一般に利用される発現ベクターは、改変pMAL−c2細菌発現ベクター(New England BioLabs)または真核生物発現ベクター、pcDNA(Promega)を含むがこれらに限定されない。最終構築物は、配列分析により確認される。
【0109】
当業者に公知の任意の適切なタンパク質精製方法を用いて、本発明のZFPを精製し得る(Ausubel、前述、Sambrook、前述を参照のこと)。さらに、任意の適切な宿主、例えば、細菌細胞、昆虫細胞、酵母細胞、哺乳動物細胞などを発現のために使用し得る。
【0110】
細菌株JM109における、マルトース結合性タンパク質に融合したジンクフィンガータンパク質(MBP−ZFP)の発現は、アミロースカラム(NEB)による直接精製を可能にする。このジンクフィンガーキメラタンパク質の高い発現レベルは、pMal−c2発現プラスミド中のこのMBP−ZFP融合が、tacプロモーター(NEB)の制御下にあるので、IPTGを用いた誘導により得られ得る。MBP−ZFP融合プラスミドを含む細菌を、10 μM ZnCl2、0.02%グルコース、および50 μg/mlアンピシリンを含む2×YT培地中に接種し、そして37 ℃で振盪する。対数増殖の中央でIPTGを0.3mM添加し、そしてこの培養物を振盪した。3時間後、細菌を遠心分離により回収し、音波処理によるか、またはフレンチプレスセルを通過させることによるかまたはリゾチームの使用により破壊し、そして不溶性物質を、遠心分離により取り除いた。MBP−ZFPタンパク質を、アミロース結合樹脂上に捕獲し、20mM Tris−HCl(pH7.5)、200mM NaCl、5mM DTTおよび50μM ZnCl 2を含む緩衝液で大規模に洗浄し、次いで、本質的に同じ緩衝液中のマルトースで溶出した(精製は、NEBからの標準的なプロトコールに基づく)。精製タンパク質を定量し、そして生化学的分析のために貯蔵した。
【0111】
精製したタンパク質の解離定数(例えば、Kd)は、代表的には、電気泳動移動度シフトアッセイ(EMSA)(BuratowskiおよびChodosh、Current Protocols in Molecular Biology、12.2.1.−12.2.7頁(Ausubel編、1996))を介して特徴付けられる。親和性は、固定量の標識した二本鎖オリゴヌクレオチド標的に対して、精製したタンパク質を力価決定することによって測定される。この標的は、代表的に、天然配列に見出される3bpおよびさらなる定常の隣接配列に隣接する天然の結合部位配列を含む。天然結合部位は、代表的に、3フィンガータンパク質について9bpであり、6フィンガーZFPについては、2 ×9bp+介在塩基である。アニールしたオリゴヌクレオチド標的は、1塩基の5’オーバーハングを有し、これによって、T4ファージポリヌクレオチドキナーゼを用いてその標的の効率よい標識が可能となる。そのアッセイについて、その標的を、1nM以下の濃度で添加し(実際の濃度を、予測解離定数のものから少なくとも10分の1に維持する)、精製したZFPを、種々の濃度で添加し、そしてその反応物を少なくとも45分間平衡化させた。さらに、反応混合物はまた、10mM Tris(pH7.5)、100mM KCl、1mM MgCl2、0.1mM ZnCl 2、5mM DTT、10%グリセロール、0.02% BSAを含む。(註:初期のアッセイにおいては、ポリd(IC)もまた、10〜100 μg/μlで加えた)。
【0112】
平衡化した反応物を、Tris/グリシン緩衝液において45分間前泳動した10%ポリアクリルアミドゲルにロードし、次いで、結合した標識標的および結合しない標識標的を、150Vでの電気泳動によって分離した。(あるいは、4%のポリアクリルアミドスタッカーを含む10%〜20%の勾配Tris−HClゲルが使用され得る)。乾燥させたゲルを、オートラジオグラフィーまたはホスホルイメージによって可視化し、そして見かけ上のKdを、最大の半分の結合を与えるタンパク質濃度を計算することによって決定した。
【0113】
このアッセイはまた、タンパク質調製物における活性画分を決定することを含み得る。活性画分を、化学量論的なゲルシフトによって決定する。ここで、タンパク質を、高い濃度の標的DNAに対して力価決定する。力価決定を、100%、50%および25%の標的で行う(通常、マイクロモル濃度レベル)。
【0114】
(IX.設計したZFPの適用)
特定の標的遺伝子に結合するZFP、およびそれらをコードする核酸を、種々の適用について使用し得る。これらの適用は、ZFPまたはそれをコードする核酸を被験体に投与し、そしてそれを使用して、その被験体内の標的遺伝子の発現を調節する治療方法を含む(同時係属中の出願Townsend & Townsend & Crew 代理人整理番号019496−002200、1998年1月12日出願を参照のこと)。この調節は、例えば、その標的遺伝子が病原性の感染性微生物中、または疾患状態に寄与している患者の内因性遺伝子(例えば、ガン遺伝子またはウイルスレセプター)に存在する場合は、抑制の形態であり得る。あるいは、この調節は、内因性細胞遺伝子の発現の活性化または発現の増加が疾患状態を改善し得る場合は、活性化の形態であり得る。そのような適用について、ZFPまたはより代表的にはそれをコードする核酸は、薬学的に受容可能なキャリアとともに、薬学的組成物として処方される。
【0115】
薬学的に受容可能なキャリアは、投与される特定の組成物、およびその組成物を投与するために使用される特定の方法によって、部分的に決定される(例えば、Remington ’s Pharmaceutical Sciences、第17版、1985を参照のこと)。ZFPは、単独または他の適切な成分との組み合わせで、エアロゾル処方物が作製されて(すなわち、それらは、「噴霧化」され得て)、吸入を介して投与され得る。エアロゾル処方物を、加圧した受容可能な噴霧剤(例えば、ジクロロジフルオロメタン、プロパン、窒素など)中に入れ得る。非経口投与(例えば、静脈内、筋肉内、皮内、および皮下の経路)に適した処方物としては、水性および非水性の、等張性の無菌注射溶液(これは、抗酸化剤、緩衝液、静菌剤、およびその処方物を意図するレシピエントの血液と等張にさせる溶質を含み得る)および懸濁剤、可溶化剤、濃稠化剤、安定化剤および保存剤を含み得る水性および非水性の、無菌懸濁物が挙げられる。組成物は、例えば、静脈注入により、経口的、局所的、腹腔内的、嚢内的または髄腔内的に投与され得る。化合物の処方物は、単位投薬形態または多数回の封入された容器中に(例えば、アンプルまたはバイアル)提示され得る。注射溶液および懸濁液は、以前に記載されている種類の無菌の粉末、顆粒および錠剤から調製され得る。
【0116】
患者に投与される用量は、経時的にその患者における利益のある治療的応答をもたらすに充分であるべきである。この用量は、使用する特定のZFPの効力およびK d、標的細胞およびその患者の状態ならびに処置される患者の体重または表面積によって決定される。用量の大きさもまた、特定の患者における特定の化合物またはベクターの投与に伴う任意の有害な副作用の存在、性質および程度によって決定される。
【0117】
他の適用において、ZFPは、サンプルにおける標的核酸の配列特異的な検出のための診断方法において使用される。例えば、ZFPを使用して、患者のサンプルにおける疾患または表現型と関連する改変体対立遺伝子を検出し得る。例として、ZFPを使用して、mRNAまたはcDNAの複合混合物における特定のmRNA種またはcDNAの存在を検出し得る。さらなる例として、ZFPを使用して、サンプルにおける遺伝子のコピー数を定量し得る。例えば、臨床サンプルにおける1コピーのp53遺伝子の欠失の検出は、ガンに対する感受性の指標である。さらなる例において、ZFPを使用して、臨床サンプルにおける病原性微生物の存在を検出する。これは、検出される微生物内の遺伝子に特異的な1以上のZFPを用いて達成される。診断アッセイを実施するための適切な形式は、ELISAプレートにおいてZFPの固定を可能にするドメインに連結されたZFPを使用する。この固定されたZFPを、結合が生じ得る条件下で標的核酸を含むと疑われるサンプルと接触させる。代表的には、サンプル中の核酸を標識する(例えば、PCR増幅の過程において)。あるいは、標識されていないプローブを、第二の標識したプローブを用いて検出し得る。洗浄後、結合した、標識された核酸を検出する。
【0118】
ZFPはまた、遺伝子発現の表現型および機能を決定するためのアッセイのために使用され得る。遺伝子機能の決定のための現在の方法論は、主に、天然の生物学的設定からの目的の遺伝子の過剰発現または除去(完全にノックアウトすること)にのいずれかおよびその効果を観察することに依存する。観察される表現型の効果は、生物学的系における遺伝子の役割を示す。
【0119】
従来のノックアウト分析に対する遺伝子のZFP媒介調節の1つの利点は、ZFPの発現が低分子のコントロールの下に置かれ得るということである。次いでZFPの発現レベルを制御することによって、ZFPによって制御される遺伝子の発現レベルを制御して、所定の表現型効果または生化学的効果を達成するのに必要とされる発現の抑制または刺激の程度を決定し得る。このアプローチは、薬物開発について特に価値がある。ZFPを低分子の制御下に置くことによって、胚性の致死性および発生補償の問題は、マウスの発生後期においてZFPリプレッサーにスイッチを入れること、および成体動物での効果を観察することによって回避され得る。ZFPによって調節される標的遺伝子を有するトランスジェニックマウスは、その標的遺伝子に対してイントランスの任意の部位にZFPをコードする核酸を組み込むことによって、作製され得る。したがって、相同組換えは、その核酸の組込みには必要ではない。さらに、ZFPがトランス優性であることから、1つの染色体コピーのみが必要であり、それゆえ、機能的ノックアウト動物は、戻し交配することなく作製され得る。
【0120】
(X.コンピュータシステムおよびプログラム)
図4は、本発明を実行するのに適切な代表的なコンピュータシステムを示す。図4は、本発明での使用に適切なコンピュータシステム10の基本的なサブシステムを示す。図4において、コンピュータ10は、バス12を備える。このバスは、中央プロセッサ14、システムメモリ16、入力/出力コントローラ18、外部デバイス(例えば、パラレルポート22を介したプリンタ20)、ディスプレーアダプタ26を介したディスプレースクリーン24、シリアルポート28、キーボード30、固定ディスクドライブ32およびフロッピディスク33Aを作動可能に受け入れるためのフロッピディスクドライブ33)のような主要なサブシステムを相互接続する。多くの他のデバイスが、接続可能である(例えば、I/Oコントローラ18を介したスキャナ60(示さず)、シリアルポート28に接続されたマウス36、またはネットワークインタフェイス40)。他の多くのデバイスまたはサブシステム(示さず)は、類似の様式で接続され得る。また、図4に示されるデバイスの全てが本発明を実行するに存在することは、下記で議論するように必要ではない。これらのデバイスおよびサブシステムは、図4に示すものとは異なる方法において、相互接続され得る。図4に示されるもののようなコンピュータシステムの操作は、当該分野で容易に公知であり、そして本願においては詳細には議論しない。本発明を実行するためのソースコードは、システムメモリ16に作動可能に配置され得るか、または固定ディスク32またはフロッピディスク33Aのような記憶媒体上に格納され得る。
【0121】
図5は、本発明の方法を具現化するに適切な図4の代表的なコンピュータシステム10の例示である。図5は、本発明とともに用いられ得る多くの可能なコンピュータタイプまたは構成のほんの一例を示す。図5は、ディスプレースクリーン24、キャビネット20、キーボード30、スキャナ60、およびマウス36を備えるコンピュータシステム10を示す。マウス36およびキーボード30は、「ユーザー入力デバイス」を例示する。ユーザー入力デバイスの他の例は、タッチスクリーン、ライトペン、トラックボール、データグローブなどである。
【0122】
好ましい実施態様において、システム10は、Pentium(登録商標)クラスに基づくコンピュータを含み、このコンピュータは、Microsoft CorporationによるWindows(登録商標)バージョン3.1、Windows95(登録商標)またはWindows98(登録商標)オペレーティングシステムを実行する。しかし、この方法は、本発明の範囲を逸脱することなく、他のオペレーティングシステムに容易に適用される。
【0123】
マウス36は、ボタン37のような1以上のボタンを有し得る。キャビネット20は、ディスクドライブ33、プロセッサ、記憶媒体などのような慣用のコンピュータ構成要素を収容する。本明細書において使用される場合、「記憶手段」には、コンピュータシステムと接続して使用される任意の記憶デバイスが含まれる(例えば、ディスクドライブ、磁気テープ、半導体メモリ、バブルメモリなど)。キャビネット20は、スキャナ60、外部記憶装置、他のコンピュータまたはさらなる周辺機器のような外部デバイスへコンピュータシステム10を接続するための入力/出力(I/O)インターフェイス18のようなさらなるハードウェアを備え得る。図5は、本発明を具現化するためのシステムのほんの一つのタイプの代表である。他の多くのシステムのタイプおよび構成が、本発明と組み合せた使用について適切である。
【0124】
図6は、ジンクフィンガータンパク質によって標的化するための標的配列内のD可能サブ部位を含む標的部位を選択するための代表的な実施態様における単純化された工程のフローチャート301を示す。工程302において、ジンクフィンガータンパク質によって標的化される標的配列が提供される。次いで、工程303において、その標的配列内の潜在的な標的部位を、評価のために選択する。決定工程304において、潜在的な標的部位を評価して、D可能サブ部位を含むか否かを決定する。そのような標的部位は、以下の式に従う:5 ’NNx aNy bNzc3’、ここで、 (x、a)、(y、b)および(z、c)の各々は、(N,N)または(G,K)であり;
(x、a)、(y、b)および(z、c)の少なくとも1つは、(G,K)であり、そして NおよびKは、IUPAC−IUBの多義性コードである。
【0125】
潜在的な標的部位は、D可能サブ部位を含む場合、潜在的な標的部位は、205における記録として格納される。この方法は、さらなる決定工程306を継続する。さらなる潜在的な標的部位の評価は、ユーザーによって要求される場合、その方法のさらなる反復は、303から出発して実施する。充分な潜在的標的部位が、すでに評価されている場合、工程305において格納された標的部位の記録を工程307において出力する。
【0126】
図7Aは、ジンクフィンガータンパク質によって標的化するためのポリヌクレオチド内の標的部位を選択するための別の代表的な実施態様において単純化された工程のフローチャートを示す。工程402において、ポリヌクレオチド標的配列は、分析のために提供される。次いで、工程404において、そのポリヌクレオチド配列内の潜在的な標的部位が選択される。この潜在的な標的部位は第一、第二および第三の塩基トリプレットを、その潜在的な標的部位における第一、第二および第三の位置にて含む。次いで、工程406において、複数のサブスコアを、トリプレットとトリプレット位との間の対応規則を適用することによって決定する。ここで、各トリプレットは、第一、第二および第三の対応する位置を有し、そして各対応するトリプレットおよび位置には、特定のサブスコアを割り当てる。次に、必要に応じて、決定工程408が存在する。この工程において、そのユーザーは、工程410におけるスケーリング計数を用いて1つ以上のサブスコアをスケーリングするように選択し得る。その後、工程412において、スコアを、第一、第二および第三のトリプレットについて、サブスコアから決定する(適切にスケーリングする)。次いで、決定工程414において、なんらかのさらなる潜在的な標的部位が試験されるべきか否かを決定するためのチェックを行う。そうである場合、工程404での処理を継続する。そうでなければ、工程416において、潜在的な標的部位およびそのスコアの少なくとも1つを出力として提供する。
【0127】
図7Bは、ジンクフィンガータンパク質を産生するための代表的な実施態様における単純化された工程のフローチャートを示す。工程450において、複数のジンクフィンガータンパク質についての指定を含むデータベースが提供される。このデータベースにおける各タンパク質は、少なくとも第一、第二および第三のフィンガーを含む。このデータベースは、そのジンクフィンガータンパク質の各々の3つのフィンガーの各々についてのサブ指定、および各ジンクフィンガータンパク質について対応する核酸配列をさらに含む。各配列は、少なくとも第一、第二および第三のトリプレットを含む。これらのトリプレットは、各ジンクフィンガータンパク質において、それぞれ、少なくとも第一、第二および第三のフィンガーによって、特異的に結合される。この第一、第二、および第三のトリプレットは、それぞれ同じ順序(3 ’−5’)で核酸配列においてある配置を有する。なぜなら、第一、第二および第三のフィンガーは、ジンクフィンガータンパク質において配列されているからである(N末端からC末端)。
【0128】
工程452において、少なくとも第一、第二および第三のトリプレットを含むジンクフィンガータンパク質の設計のための標的部位が提供される。次いで、工程454において、その標的配列において第一のトリプレットに結合するフィンガーを有するジンクフィンガータンパク質の第一のセットが同定される。次に、454において決定されるセットの第一および第二のサブセットを同定する任意の工程456が続く。この第一のサブセットは、そのジンクフィンガータンパク質における第一のフィンガー位から第一のトリプレットを結合するフィンガーを有するジンクフィンガータンパク質を含む。第二のサブセットは、そのジンクフィンガータンパク質において第一のフィンガー位以外からの第一のトリプレットを結合するフィンガーを有するジンクフィンガータンパク質を含む。この方法は、工程458に続く。この工程において、ジンクフィンガータンパク質のさらなるセットが同定され、このセットは、その標的部位における第二のトリプレットに結合するフィンガーを含む。この工程の後に、工程458において同定されるセットの第一および第二のサブセットを同定する任意の工程460が続く。この第一のサブセットは、ジンクフィンガータンパク質内の第二の位置からの第二のトリプレットに結合するジンクフィンガータンパク質を含む。この第二のサブセットは、ジンクフィンガータンパク質の第二の位置以外からの第二のトリプレットを結合するジンクフィンガータンパク質を含む。この方法は、工程462に続く。462において、その標的部位の第三のトリプレットに結合するフィンガーを含む1セットのジンクフィンガータンパク質を同定する。任意の工程464において、工程462において同定されたセットの第一および第二のサブセットを、同定する。第一のサブセットは、ジンクフィンガータンパク質の第三のフィンガー位からの第三のトリプレットに結合するフィンガーを含むジンクフィンガータンパク質を含む。第二のサブセットは、ジンクフィンガータンパク質の第三のフィンガー位以外からの第三のトリプレットに結合するフィンガーを含むジンクフィンガータンパク質を含む。この方法は、工程466に続く。この工程では、工程454、458および462において同定されたジンクフィンガータンパク質のセットが別々に出力される。さらに任意の工程468が存在する。ここで、工程460、464および468において同定されたジンクフィンガータンパク質の第一および第二のサブセットが出力される。
【0129】
図8Aは、Entity Representation Diagram(ERD)へのキーである。このダイヤグラムを使用して、ZFPデータベースの内容を記述する。代表的な表502は、1以上のキー属性504および1以上のキーではない(ノンキー)属性506を含む。代表的な表502は、1以上のレコードを含み、ここで、各レコードは、列挙した属性に対応するフィールドを含む。キーフィールドの内容を一緒にして、個々のレコードを同定する。ERDにおいて、各表は、水平線によって分割された長方形によって表される。その線より上のフィールドまたは属性は、キーであるが、他方、その線より下のフィールドまたは属性は、キーではないフィールドである。同定する関係508は、親の表510のキー属性もまた、子供の表512のキー属性であることを示す。同定しない関係514は、親の表516のキー属性もまた、子供の表518のキーではない属性であることを示す。(FK)が括弧内に現れる場合は、それは、1つの表の属性が、別の表のキー属性であることを示す。同定しない関係および同定する関係の両方について、親の表における1つのレコードは、子供の表における1以上のレコードに対応する。
【0130】
図8Bは、本発明の特定の実施態様に従った代表的なZFPデータベース550を示す。データベース550は、代表的には、予め特徴付けたZFPの収集物の各々についての指定を含み得る。このZFPは、天然のZFPまたは改変体ZFPであり得る。この指定は、例えば、各ZFPを代表する名称またはシンボルであり得る。例えば、図8Bにおけるデータベース550のZFP552は、「ZFP001」と指定される。データベース550はまた、ZFPにおけるフィンガーの各々についてのサブ指定(例えば、サブ指定554、ZFP001 552のフィンガー1)を含む。代表的には、このサブ指定は、フィンガーにおいて選択された位置を占有するアミノ酸残基の形態である。さらに、ZFPは、従来の番号付けに従って−1位から+6位までを占有するアミノ酸であるサブ指定を有する。このデータベースはさらに、各ジンクフィンガータンパク質によって結合される標的核酸セグメントを含み得る。この核酸セグメントは通常、3つの塩基の3つのトリプレットを含む。塩基の3つのトリプレットは、1つの配列として共同で含まれ得るか、または別個の配列として含まれ得る。9塩基の標的部位における塩基が5 ’末端から連続的に番号付けされる場合、第一のトリプレットは、塩基7〜9を占有し、第二のトリプレットは、塩基4〜6を占有し、そして第三のトリプレットは、塩基1〜3を占有する。標的セグメント内のトリプレット位のこの指定に従って、ジンクフィンガータンパク質の第一のフィンガー(すなわち、N末端に最も近い)は、第一のトリプレットに結合し、第二のフィンガーは、第二のトリプレットに結合し、そして第三のフィンガーは第三のトリプレットに結合する。このデータベースはまた、その標的部位についてZFPの結合親和定数または解離定数のようなさらなる情報を含み得る。しかし、そのようなものは必須ではない。さらにデータベース550は、本発明の範囲を逸脱することなく、図8Bに示される以外のZFP、フィンガーおよび核酸の間の他の配置および関係を含み得る。
【実施例】
【0131】
(実施例1:DNAモチーフについての検索プロトコル)
本実施例は、標的セグメントがより長い遺伝子からどのように選択されるかを例示する。検索手順を、検索プロトコルにおける1以上のDNA配列モチーフを特定することを可能にするコンピュータプログラムを用いて実行する。通常の手順は、遺伝子またはcDNAのDNA配列を入力すること、次いで、異なるモチーフについて、最も所望されるものから最も所望されないものまで、複数回配列を検索することである。従って、下記に列挙した例示的プロトコルのなかで、代表的に、プロトコル1をまず実行し、そしてそれが潜在的な標的セグメントの適切な数を得られない場合は、プロトコル2を試行し、そして以下同様とする。
【0132】
プロトコル1は、各々9塩基または10塩基の2つの別個のセグメントから形成された標的部位について標的遺伝子を検索する。2つのセグメントは、0〜3の介入塩基によって分離され得る。各セグメントは、NNGG形態(ボールドで示す)のD可能サブ部位を含む。セグメント内の3塩基のサブ部位の各々は、Gで始まる。この分析によって同定された標的部位は、ZFP設計について直接使用され得るか、またはさらなる分析に供され得、例えば、標的セグメントがさらなるD可能サブ部位を有するものを同定し得る。それぞれ10塩基からなる2つのセグメントから形成された標的部位において、合計で6つのD可能サブ部位が存在し得る。以下の全ての標的部位を、5 ’から3’で示し、そして指定「0,3」は、任意のタイプの0〜3のヌクレオチドが存在し得ることを示す。
【0133】
【数5】

【0134】
プロトコル2は、標的遺伝子内の標的部位を評価するための第二の手順である。この手順は、再度、それぞれ9塩基または10塩基の2つのセグメントから形成される標的部位について検索する。各セグメントは、形態KNGGの少なくとも1つのD可能サブ部位を含む。プロトコル2は、プロトコル1とは、プロトコル2が、3塩基サブ部位がGで始まることを必要としない点において異なる。プロトコル2においてはむしろ、3塩基サブ部位はGまたはTのいずれか(IUBPAC−IUBの多義性コードにおいてはK)で始まる。標的部位は、5 ’から3’で示し、そしてシンボル(0,3)および(0,2)は、それぞれ、0〜3塩基および0〜2塩基の介入セグメントを示す。
【0135】
【数6】

【0136】
プロトコル3は、そのプロトコル3がKNGGまたはKNGTのいずれかのD可能サブ部位を用いて標的部位を選択することを除いて、プロトコル2と同じである。標的部位を、5 ’から3’で示す。
【0137】
【数7】

【0138】
プロトコル4は、上記のプロトコルのいずれよりもより一般的であり、そして標的部位がD可能サブ部位を含むことを必要としない。プロトコル4は、互いに0〜3塩基内に、形態GNN GNN GNNのそれぞれが9塩基である2つのセグメントを同様に必要とする。
【0139】
プロトコル5は、それが、互いに0〜3塩基内で、式5’KNN KNN KNN3 ’の2つの標的セグメントから形成された標的部位について、検索することを除いてプロトコル4と同じである。
【0140】
(実施例2)
本実施例は、少なくとも1つのD可能サブ部位を含む標的セグメントに結合するジンクフィンガータンパク質が、一般的に、そのZFPが+2位にD残基を有する場合、ジンクフィンガータンパク質結合より高い親和性を伴って、D可能サブ部位を欠く標的セグメントへ結合することを例示する。53のZFP(各々は、3つのフィンガーを有する)を、結合親和性またはD可能サブ部位への結合に関係なく収集物から選択した。選択したZFPの解離定数を、いずれかの側に標的配列由来の少なくとも1つの隣接塩基を有するZFPの3つのフィンガーによってそれぞれ結合した3つの連続的なヌクレオチドトリプレットを含む標的セグメントへのZFPの結合によって決定した。すべてのZFPは、ヒトSp1フレームワークを有した。これらの53のZFPの結合親和性を、表2におけるKd値として列挙される任意の4群に分けた。
【0141】
【表2】

【0142】
この分類に従うと、これらのタンパク質の約25%(14/53)のみが、それらのそれぞれの標的について、高親和性(100nM以下のKd)を有した。これらの14のタンパク質のうち、すべてが少なくとも1つのD可能サブ部位をその標的内に有していた。
【0143】
(実施例3)
本発明者らは、ダイズ(Glycine max)FAD2−1 cDNAの配列を、対合した近位の9塩基標的セグメントについて、プロトコル2およびプロトコル3を用いて検索した。5つの標的セグメントを選択し、そして1または2のいずれかのZFPを、その標的の各々に結合するように設計した。選択した標的およびそれぞれの設計したZFPについてのKd値を、表3に示す。D可能サブ部位をボールドで示す。配列を5 ’から3’にむけて示す。
【0144】
【表3】

【0145】
作製した8つのZFPのうち、すべてが高親和性(100nM以下のKd)で、それらの標的に結合した。このことは、9bpの標的内にD可能サブ部位を有する標的を選択することによって、高親和性ZFPを効率よく設計することが可能であることを示す。さらに、2つのD可能サブ部位を有する標的部位へ結合するZFPのすべては、1つのみのD可能サブ部位を有する標的部位へ結合するZFPよりも強力に結合した。
【0146】
(実施例4)
本実施例は、D可能サブ部位が高親和性を付与する証拠をさらに提供する。53の標的セグメントを上記のプロトコル5によって同定した。このプロトコルは、D可能サブ部位が標的部位に存在することを必要としない。53のZFPを、これらのそれぞれの部位に結合するように設計した。33の標的セグメントを、上記のプロトコル3によって同定した。このプロトコルは、D可能サブ部位を必要とする。そして、33のZFPを、これらのそれぞれの部位に結合するように設計した。表4は、異なる手順によって設計されるZFPのKdを比較する。
【0147】
【表4】

【0148】
表4は、プロトコル3によって設計された33のZFPのうち、31が高親和性(100nM未満のKd)を有することを示す。対照的に、プロトコル5によって設計したZFPのうち14のみが高結合親和性を有する。これらのデータは、高親和性ZFP(Kd<100nM)は、検索プロトコルがD可能サブ部位を必要としない場合よりも、その検索プロトコルがD可能サブ部位の基準を含む場合に、標的についてより効率よく設計され得ることを示す。
【0149】
(実施例5)
ZFPの親和性と、標的における1以上のD可能サブ部位の存在との間の関係を、殆どが異なる標的部位に対して特異的な約300の設計したZFPについて分析した。この分析および続く分析において、1つのZFPのみが、1つの標的部位あたりに含まれる。これは最も高い親和性を伴うZFPである。
【0150】
表5および図1は、結合した9塩基の標的部位におけるD可能サブ部位の数およびタイプによって分類された異なるカテゴリーのZFPの平均Kdを示す。表4において、および後の表6、表7および表8において、s.e.m.は、平均の標準誤差を示し、そしてnは試験したタンパク質の数を示す。
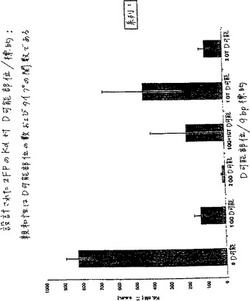
【0151】
【表5】

【0152】
2つのGGタイプのD可能サブ部位を有する標的に対して設計された22のZFPは、平均Kd=15nMを伴い、最高の結合親和性を有する。100nM未満のKdを有する50のZFPのうち、49が、少なくとも1つのD可能サブ部位を有する。この表は、以下の結論を示す:(1)1つのD可能サブ部位を伴う標的部位への結合は、D可能サブ部位を欠く標的部位へのZFPの結合よりもさらに強力に結合する;(2)2つのD可能サブ部位を伴う標的部位へ結合するZFPは、1つのD可能サブ部位を伴う標的部位へ結合するZFPよりもさらに強力に結合する、および(3)GG D可能サブ部位を伴う標的部位を有するZFPは、GT D可能サブ部位を伴う標的部位を有するZFPよりもさらに強力に結合する。
【0153】
(実施例6)
設計したZFPの結合親和性に影響を与える別の因子は、標的部位が、KNN KNN KNNよりむしろGNN GNN GNNの形態を有するか否かである。本実施例は、D可能サブ部位が、高度の結合親和性を、GNN GNN GNNモチーフの状況においてさえも、与えることを示す。この分析のために、本発明者らは、59のZFPの集団を選択した。これらの各々は、形態GNN GNN GNNの異なる標的部位に結合する。表6は、設計されたZFPのKd値を、GNN GNN GNN標的を有するD可能サブ部位の存在の関数として示す。
【0154】
【表6】

【0155】
D可能サブ部位の存在は、その標的がGNN GNN GNNモチーフに適合する場合でさえも、ZFPの結合親和性に強力に影響を与える。
【0156】
(実施例7)
本実施例は、D可能サブ部位の増加した結合親和性を付与するという点に対する効果が、他の残基に対してより高い結合親和性の付与におけるG残基の任意の効果について相加的であるというさらなる証拠を提供する。この分析について、本発明者らは、101のジンクフィンガータンパク質の、本発明者らの収集物からの異なる標的部位への結合を選択し、そして存在するG残基の数によってこれらの標的部位を分類した。これらの標的部位は、2〜8のG残基を、9塩基配列中に含んでいた。表7は、概して、標的部位に存在するG残基が増えれば、その部位についてのZFPの結合親和性も強くなることを示す。
【0157】
【表7】

【0158】
本発明者らは、D可能サブ部位の存在または非存在が設計したZFPの平均Kd値に影響を与えたか否かを問い掛けることによってさらにこれらのデータを分析した。表7からの9塩基標的の各カテゴリーを、D可能サブ部位を含む標的またはそれを含まない標的へと細分した。この分析の結果を表8に示す。
【0159】
【表8】

【0160】
この表は、同じ数のG残基を有するが、異なる数のD可能サブ部位を有する標的部位を比較した場合、D可能サブ部位を含む部位がより高い結合親和性を付与することを示す。4つ以上のGを有する9塩基の標的部位について、平均Kdは、その標的が少なくとも1つのD可能サブ部位を有する場合、約100nM以下である。特に顕著なのは、5つのG残基を有する標的部位間の比較である。D可能サブ部位を欠く5つのそのような標的は、640nMの平均Kdを有した。2つのD可能サブ部位を有する23のそのような標的部位は、98nMの平均Kdを有した。
【0161】
(実施例10:ZFP予測モジュール)
本実施例は、対応規制を用いた、標的遺伝子内の標的セグメントの選択、および選択した標的セグメントに結合するZFPを設計するためのデータベースの使用を例示する。ZFP Prediction Moduleは、(i)目的のDNA配列、(ii)種々のデータ表、(iii)設計パラメータおよび(iv)出力パラメータを入力として採用すること、ならびに目的の配列における潜在的なZFP標的部位のリストおよび各標的部位におけるサブ部位に対して設計されたフィンガーのまとめを出力として提供することによって、部位の選択およびZFP設計プロセスの両方を容易にする。この節は、プログラムの入力、出力およびそのプログラムについてのプロトコルをスコア付けすることを記載する。明確さのために、その記述を部位選択および設計機能へと分割する。
【0162】
(1.目的のDNA領域内の標的部位の選択)
(入力)
1)標的DNA配列
2)9bp標的部位における可能な3塩基対サブ部位およびその3つの可能な位置についてのスコアの各々を列挙するスコア表を、図1に示す。そのスコア表は、実行時にユーザーによって提供され、そしてカスタマイズされ得、そしてジンクフィンガーモチーフのDNA配列優先性についてのユーザーの最新の理解を反映するようにアップデートされ得る。
【0163】
3)既存の高親和性ZFPについて、標的部位、アミノ酸配列および参照データを含む「ZFPデータ表」。この表は、出力パラメータ(ii)が下記において選択される場合にのみそのプログラムのこの部分について必要とされる。ZFPデータ表の例を表9に提供する。
【0164】
4)任意の状況パラメータ−実行時にユーザーによって入力される「『D可能』トリプレットについての増強因子」。このパラメータは、3 ’GまたはTが隣接する任意の「xxG」サブ部位についてのスコアに増強因子を乗じる。
【0165】
5)出力パラメータ(ユーザーによって供給される)は、以下を特定する: i)その出力において含まれる標的部位の数 ii)そのプログラムが、(存在する場合)3つのフィンガータンパク質がすでに設計されているその標的部位を特に強調すべきか否か iii)そのプログラムが、入力標的配列におけるその相対位置に従って、出力標的部位を再整列すべきか否か iv)そのプログラムが、9bp DNA部位の標的化可能な対を強調すべきか否か(隣接し、重複しない、n以下の塩基だけ分離されている部位の対、ここで、nは、代表的には、5、4、3、2または1である)。
【0166】
出力:スコアによって階級付けされた標的DNA配列における潜在的な標的部位のセット。
【0167】
特定されている場合、3フィンガータンパク質がすでに設計されている任意の標的部位のリスト。
【0168】
特定されている場合、入力配列における配置に従って、再整列された出力部位のリスト。
【0169】
特定されている場合、9bpDNA部位のすべての標的化可能な対のリスト。
【0170】
そのプログラムの部位選択部分は、所定の標的DNAフラグメントにおいて、すべての可能な9bp配列に対してスコアを割り当てる。このスコアは、先に設計したジンクフィンガータンパク質からの情報を使用することに基づいて標的化可能性の容易さを反映する。所定の9塩基配列を評価するにおいて、そのプログラムは、まず、その標的を、その成分サブ部位へと分割し、次いで、そのスコア表を参考にして潜在的な標的部位におけるその位置での各サブ部位についてのスコアを入手する。最後に、これにサブ部位スコアを乗じて、9bp標的部位についての全体スコアを得る。例えば、試験配列5 ’AGTGCGCGGTGC3’および表1におけるスコア表を用いると、出力部位(5 ’−3’)およびスコアは以下のとおりである:
【0171】
【数8】

【0172】
本実施例において、最良の標的部位は、5’TGC GCG GTG 3 ’であり、スコアは1000である。このプログラムはまた、反対の(アンチセンスの)鎖における潜在的な標的に対してスコアを割り当てるが、単純さのために、これらの部位は、本実施例において無視する。任意の因子である「『D可能』トリプレットについての増強因子」が、標的部位の評価において、状況因子「D可能接触」を担う上記のスコア付けプロトコルを変更するために提供され得る。この特徴が選択される場合、このプログラムは、サブ部位スコアを割り当てる場合に、引き続きのチェックを実施する。
【0173】
サブ部位が形態xxGのものである場合、次いで隣接塩基(3’側)は、TまたはGである場合、xxGサブ部位のスコアに増強因子を乗じるか、そうでなければ、サブ部位スコアは同じままである。
【0174】
(そのサブ部位が形態xxA、xxCまたはxxTのものである場合、そのスコアもまた、変化しないままである。)
例えば、ユーザーが、1.25という「D可能」トリプレットについての増強因子を入力する場合、上記のスコアは、以下のように調整される:
【0175】
【数9】

【0176】
(この選択肢を使用する場合、このプログラムは、その標的部位のすぐ3’側の塩基(小文字)の同一性を考慮する。最後の部位について、この塩基は、本実施例において規定されておらず、そしてこれは、この位置にてシャープ記号「#」を入れることによって注意が喚起される。
【0177】
その標的DNAにおけるすべての9塩基対配列に対してスコアを割り当てた後、次いで、そのプログラムは、トップスコアを、ユーザーによって決定され、出力された部位の数とともに、印刷する。
【0178】
ユーザーによって特定される場合、そのプログラムはまた、以下を提供する: i.3つのフィンガータンパク質がすでに設計されている任意の標的部位のリスト ii.入力配列における位置に従って、再整列された出力部位のリスト iii.9bp DNA部位のすべての標的可能な対のリスト(隣接し、重複しない、5、3またはそれ未満の塩基だけ分離されている部位の対)。
【0179】
(II.選択した標的部位についてのタンパク質の設計)
入力:そのプログラムの部位選択部分からの部位(または、他の方法によって決定される)既存の高親和性ZFPについて、標的部位、アミノ酸配列および参照データを含む、「ZFPデータ表」。
【0180】
そのプログラムがその出力を以下のいずれかに限定するか否かを特定する出力パラメータ(ユーザーによって供給される): i (存在する場合)その標的部位がその出力における部位と完全に同一であるタンパク質のみ、または ii (存在する場合)その標的部位が3bpサブ部位の2以上にて出力部位に適合するタンパク質のみ。
【0181】
出力:制限(i)も(ii)も存在しない場合: 潜在的な9塩基対標的部位の各々について、ZFPデータ表からのZFPおよびその成分フィンガーの3つのセットの表。これら成分フィンガーは、それぞれ、その標的部位内の3つのトリプレットサブ部位に結合する。各サブ部位について、ZFPのそのセットは、2つのサブセットへと細分され得る。1つのサブセットは、その親のZFPにおける対応するフィンガー位からの所定の位置にてトリプレットを結合するZFPおよびそれらのフィンガーを含む。他のサブセットは、親のZFP内の対応しない位置からの所定の位置にてトリプレットを結合するZFPおよびそのフィンガーを含む。第一のフィンガー位(N−C)は、最初のトリプレット3 ’−5’に対応する。
【0182】
このプログラムのZFP設計部分は、ユーザーが所定の9塩基標的部位におけるサブ部位を結合することが公知のすべてのフィンガーを迅速に再検討することを可能にすることによって設計プロセスを容易にする。上記の例(5 ’TGCGCGGTG3’)からの最適な設計標的、および表9に提供される短いZFPデータ表を与えられると、出力(制限(i)も(ii)も存在しない場合)は以下のとおりである:
【0183】
【数10】

【0184】
「整列した(ordered)」出力は、ZFPデータ表において、TGCサブ部位が標的部位の第三のトリプレットにおけるジンクフィンガーが接触する1つの例が存在することを示す。この場合におけるフィンガーは、ERDHLRTであり、そしてその部位は、5 ’TGCGGGGCA3’である。他の2つのサブ部位−GCGおよびGTGの各々について1つの類似する例もまた存在する。これらの場合におけるフィンガーは、それぞれ、RSDELQRおよびRKDSLVRである。この情報を使用して、3つのフィンガータンパク質F1−RKDSLVR、F2−RSDELQR、F3−ERDHLRTを、標的5 ’TGCGCGGTG3’を結合するための設計として提唱する。
【0185】
「整列していない」出力は、フィンガーが標的の中心サブ部位ではなくGCGサブ部位に接触するZFPデータ表において2つの場合が存在することを示す。むしろ、1つの場合において、GCGは、5 ’末端に接触し、そして他方は3’末端に接触し、そしてこれらの場合において、フィンガー配列は、RSDELTRおよびRSDERKRである。これらは、その標的部位においてGCGを結合するための別の設計である。
【0186】
【表1】

【0187】
【表9】

【0188】
そのようなデータベースにおける包含に適切な、ジンクフィンガータンパク質、それらのフィンガーの配列および結合する標的部位の他の例は、背景の節において引用された参考文献に議論される。
【0189】
上記の発明を、理解の明確化の目的のために、詳細に記載してきたが、特定の改変は、添付の請求の範囲の範囲内で実施され得ることは自明である。本明細書において、引用した全ての刊行物および特許文書は、本明細書において、その全体が、それら各々が個々そのように注記されているのと同程度にまで全ての目的のために参考として援用される。
【図面の簡単な説明】
【0190】
【図1】図1は、ジンクフィンガータンパク質によって結合される標的部位においてサブ部位の存在および数が結合親和性に相関するデータを提供する図を示す。
【図2】図2は、3つのD可能サブ部位を含む標的部位に結合される3つのジンクフィンガータンパク質を示す。
【図3】図3は、設計されたZFPをコードする核酸をアセンブルするプロセスを示す。
【図4】図4は、標的部位の選択およびジンクフィンガータンパク質の設計の方法を実行するためのコンピュータシステムを示す。
【図5】図5は、標的部位の選択およびジンクフィンガータンパク質の設計の方法を実行するためのコンピュータシステムを示す。
【図6】図6は、標的配列内にD可能サブ部位を含む標的部位を選択するための方法のフローチャートを示す。
【図7A】図7Aは、対応規則を使用して標的配列内の標的部位を選択するためのフローチャートを示す。
【図7B】図7Bは、データベースを使用して、所望の標的部位を結合するようにZFPを設計するためのフローチャートを示す。
【図8A】図8Aは、ZFPデータベースのエンティティの代表図を示す。
【図8B】図8Bは、ZFPデータベースの代表例である。
【技術分野】
【0001】
本発明は、生命情報科学およびタンパク質工学の技術分野にある。
【背景技術】
【0002】
ジンクフィンガータンパク質(ZFP)は、配列特異的様式においてDNAと結合し得るタンパク質である。ジンクフィンガーは、最初に、アフリカツメガエル、Xenopus laevisの卵母細胞由来の転写因子TFIIIAにおいて同定された。これらのタンパク質の1つのクラス(C2H 2クラス)を特徴付ける例示的モチーフは、−Cys−(X)2-4−Cys−(X) 12−His−(X)3-5−His(ここでXは、任意のアミノ酸である)である。単一のフィンガードメインは、約30アミノ酸長であり、そしていくつかの構造研究が、これがジンクを通して配位した βターンにおいて2つの不変のヒスチジン残基および2つの不変のシステイン残基を含むαヘリックスを含むことを実証している。これまでに、10,000を超えるジンクフィンガー配列が、数千の公知のまたは推定転写因子において同定されている。ジンクフィンガードメインは、DNA認識のみならず、RNA結合およびタンパク質−タンパク質結合にも関与する。現在推定されていることは、このクラスの分子が全ヒト遺伝子の約2%を構成することである。
【0003】
Zif268のX線結晶構造である、マウス転写因子由来の3つのフィンガードメインは、同族のDNA配列との複合体において解析され、そして各フィンガーは、周期的な循環によって次のフィンガー上に重ね合わされ得ることが示される。この構造は、各フィンガーが、各DNAトリプレット部分部位と接触する各認識ヘリックス上の−1、2、3および6位で側鎖を有し、3塩基対の間隔にわたって独立してDNAと相互作用することを示唆する。Zif268のアミノ末端は、DNA鎖の3 ’末端に位置し、ここで、最も多く接触する。近年の結果は、いくつかのジンクフィンガーが、標的セグメントにおける第4番めの塩基と結合し得ることを示した。ジンクフィンガータンパク質が最も多く接触する鎖が、標的鎖を設計される場合、いくつかのジンクフィンガータンパク質は、標的鎖における3塩基トリプレット、および非標的鎖上の第4番目の塩基と結合する。この第4番目の塩基は、3塩基の部分部位の3 ’末端すぐの塩基に相補的である。
【0004】
Zif268−DNA複合体の構造はまた、ジンクフィンガータンパク質のDNA配列特異性が、各ジンクフィンガー認識ヘリックス上の4つのヘリックスの位置(−1、2、3および6)にアミノ酸置換体を作製することによって変化し得ることが示唆された。この観察を試験するための、ジンクフィンガーコンビナトリアルライブラリーを用いたファージディスプレイ実験が、1994年に一連の論文で公開されていた(非特許文献1;非特許文献2;非特許文献3)。コンビナトリアルライブラリーを、第1のZif268または中間のZif268のいずれかのフィンガーにおける無作為化した側鎖を用いて構築し、次いで、適切なDNA部分部位が、変化したDNAトリプレットによって置換された、変化したZif268結合部位を選択するために使用した。さらに、導入された変異の性質と結合特異性に生じた変化との間の相関は、変化した結合特異性を有するZFPの設計についての置換の規則の部分的なセットを生じた。
【0005】
非特許文献4は、Zif268の各フィンガーが首尾良く無作為化され、そして新しいトリプレット配列と結合するために選択されるファージディスプレイ法の加工を議論している。この論文は、核ホルモン応答エレメントである、p53標的部位およびTATAボックス配列についてのZFPの選択を報告した。
【0006】
多くの論文が、特定の標的部位を調節するためにZFPを生成する試みを報告している。例えば、非特許文献5は、ZFPがbrc−abl癌遺伝子の発現を抑制するように設計する試みを報告している。ZFPが結合する標的セグメントは、brcおよびablをコードする遺伝子を融合する特異的な癌遺伝子の転座によって作製される連結を重複させるように選択された9つの塩基配列5 ’GCA GAA3’GCCであった。この意図は、この標的部位に対して特異的なZFPが、ablまたはbrc構成要素遺伝子に結合することなく癌遺伝子に結合することである。著者らは、ファージディスプレイを使用して、この標的セグメントへの結合について改変体ZFPのミニライブラリーをスクリーニングした。従って、単離された改変体ZFPが、次いで細胞株における安定してトランスフェクトされたbrc−able構築物の発現を抑制することが報告された。
【0007】
非特許文献6は、Zif268由来の2つのフィンガーをOct−1由来のホメオドメインと融合することによって、新規なDNA結合タンパク質を設計する試みを報告した。次いで、このハイブリッドタンパク質は、キメラタンパク質としての発現に関する転写活性因子と融合された。このキメラタンパク質は、その2つの構成要素の部分配列のハイブリッドを示す標的部位と結合すると報告された。次いで、著者らは、プロモーターに作動可能に連結されるルシフェラーゼ、およびそのプロモーターに近接したキメラDNA結合タンパク質についてのハイブリッド部位を含むレポーターベクターを構築した。著者らは、それらのキメラDNA結合タンパク質がルシフェラーゼ遺伝子の発現を活性化し得ることを報告した。
【0008】
非特許文献7は、ペプチドスペーサーを用いてそれぞれ3つのフィンガーを有する2つの構成要素のジンクフィンガータンパク質を連結し、複合ジンクフィンガータンパク質を形成することを報告している。次いで、この複合タンパク質を、転写活性化ドメインとさらに連結した。得られたキメラタンパク質が、2つの構成要素ジンクフィンガータンパク質によって結合された標的セグメントから形成された標的部位に結合することが、報告された。このキメラジンクフィンガータンパク質が、その標的部位がレポーター遺伝子に作動可能に連結されるプロモーターと近接したレポータープラスミドに挿入される場合、そのレポーター遺伝子の転写を活性化し得ることが、さらに報告された。
【0009】
Chooら、特許文献1、特許文献2、および特許文献3(1998)は、HIV Tat遺伝子内の標的部位に結合するジンクフィンガータンパク質の選択を議論している。Chooらはまた、癌遺伝子rasにおける共通した変異の部位を取り囲む標的部位に結合するためのジンクフィンガータンパク質の選択を議論している。従って、ras内の標的部位は、変異の位置によって制約された。
【0010】
上記の研究はどれも、候補遺伝子内の種々の潜在的な標的部位の各利点を系統的に評価するための判定基準を提供しなかった。Rebarら(前出)、Jamiesonら(前出)およびChooら、PNAS.(1994)前出によるファージディスプレイの研究は、全て天然のZif268結合部位、5 ’GCG TGG GCGc3’の変化に集中し、そして予め決められた標的遺伝子に関してなされなかった。Chooら、Nature(1994)、前出の標的部位の選択は、その部位がbrcセグメントとablセグメントとの間の界面を重複する意図によってのみ構築され、そしてこれは種々の潜在的な標的部位の比較を含まなかった。同様に、GreismanおよびPaboは、公知の調節的役割のために特定の標的部位を選択し、そしてあらかじめ選択された標的遺伝子内の種々の潜在的な標的セグメントの相対的な利点を考慮しなかった。同様に、Chooら(1998)、前出のras内の標的部位の選択は、変異の位置によって強制された。Chooら(1998)のHIV Tatにおける標的部位の選択について判定基準は提供されていない。最終的には、Pomerantzら、前出およびLiuら、前出の両方は、複合体ジンクフィンガーについての人工ハイブリッド標的部位を構築し、次いでその標的部位をレポーター構築物に挿入した。
【0011】
【特許文献1】WO98/53058
【特許文献2】WO98/53059
【特許文献3】WO98/53060
【非特許文献1】Rebarら、Science 263、671−673(1994)
【非特許文献2】Jamiesonら、Biochemistry 33、5689−5695(1994)
【非特許文献3】Chooら、PNAS 91、11163−11167(1994)
【非特許文献4】GreismanおよびPabo、Science 275、657−661(1997)
【非特許文献5】Chooら、Nature 372、645(1994)
【非特許文献6】Pomerantzら、Science 267、93−96(1995)
【非特許文献7】Liuら、PNAS 94、5525−5530(1997)
【発明の開示】
【0012】
本発明は、ジンクフィンガータンパク質によって標的化するために標的配列内の標的部位を選択する方法を提供する。いくつかのこのような方法は、ジンクフィンガータンパク質によって標的化される標的核酸を提供する工程、および5 ’NNx aNy bNzc3’を含む標的核酸内の標的部位を産出する工程を含む。(x,a)、(y,b)および(z,c)の少なくとも1つが(G,K)である場合、各(x,a)、(y,b)および(z,c)は、(N,N)または(G,K)である。NおよびKは、IUPAC−IUB多義性コードである。いくつかの方法において、標的核酸内の複数のセグメントが選択され、そして5 ’NNx aNy bNzc3’を含む複数のセグメントのサブセットが産出される。代表的には、この標的核酸は、標的遺伝子を含む。いくつかの方法において、(x,a)、(y,b)および(z,c)の少なくとも2つが(G,K)である。いくつかの方法において、(x,a)、(y,b)および(z,c)の3つ全てが(G,K)である。いくつかの方法は、5 ’NNx aNy bNzc3’を含む遺伝子の第2のセグメントを同定する工程をさらに含む。ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)のうち少なくとも1つが(G,K)であり、そしてNおよびKはIUPAC−IUB多義性コードである。いくつかの方法において、第2のセグメントにおいて、(x,a)、(y,b)および(z,c)のうち少なくとも2つが(G,K)である。いくつかの方法において、少なくとも1つの(x,a)、(y,b)および(z,c)の3つ全てが(G,K)である。いくつかの方法において、第1および第2のセグメントが、標的部位において5塩基より少ない塩基によって分離される。
【0013】
いくつかの方法は、bNz aNyおよびNNxトリプレットにそれぞれ結合する第1、第2および第3のフィンガーを含むジンクフィンガータンパク質を合成する工程をさらに含む。いくつかのこのような方法において、合成工程は、標的セグメント中のNNx aNyおよびbNzトリプレットにそれぞれ結合する3つのジンクフィンガーおよび第2の標的セグメント中のNNx aNyおよびbNzトリプレットにそれぞれ結合する第2の3つのフィンガーを含む、第1のジンクフィンガータンパク質を合成する工程を含む。いくつかの方法において、第1、第2および第3のフィンガーの各々は、独立して選択されるかまたは設計される。いくつかの方法において、フィンガーは、ジンクフィンガータンパク質の指定、フィンガー構成要素のサブ指定、およびジンクフィンガータンパク質によって結合される核酸配列を含むデータベースから設計される。いくつかの方法において、フィンガーは、標的部位に結合する改変体を同定するために標的部位への特異的結合について、ジンクフィンガー結合タンパク質の改変体をスクリーニングすることによって選択される。
【0014】
いくつかの方法は、標的核酸を含むサンプルをジンクフィンガータンパク質と接触させ、それによってジンクフィンガータンパク質が、標的核酸の存在またはその特定の対立遺伝子形態を明らかにする標的部位に結合する工程を、さらに含む。いくつかの方法において、標的核酸を含むサンプルは、ジンクフィンガータンパク質と接触され、それによってこのジンクフィンガータンパク質が標的部位に結合し、それによってその標的核酸の発現を調節する。
【0015】
いくつかの方法において、標的部位はコード領域中に生じる。いくつかの方法において、この標的部位は、プロモーター、エンハンサーまたは転写開始部位内かまたはそれらに近接して存在する。いくつかの方法において、この標的部位は、プロモーター、調節配列、またはその標的核酸内の多型部位の外側に存在する。
【0016】
別の局面において、本発明は、ジンクフィンガータンパク質によって標的化されるポリヌクレオチド内の標的部位を選択するための代替方法を提供する。これらの方法は、ポリヌクレオチド配列を提供する工程、およびそのポリヌクレオチド配列内の潜在的な標的部位を選択する工程を含む;この潜在的な標的部位は、連続した第1の、第2のおよび第3の塩基のトリプレットを、その潜在的な標的部位において第1、第2および第3の位置で含む。次いで、3つの連続するトリプレットの配列においてトリプレットとトリプレットの位置との間の対応規則(correspondince regime)を適用することによって、複数のサブスコアが決定される。ここで、各トリプレットは、第1の、第2のおよび第3の対応する位置を有し、そしてトリプレットとトリプレットの位置との各組み合わせは、特定のサブスコアを有する。次いで、第1の、第2のおよび第3のトリプレットについてのサブスコアを組み合わせることによって潜在的な標的部位についてスコアを算出する。次いで、選択、決定および算出工程は、さらなるスコアを決定するためのさらなる潜在的な標的部位の第1の、第2のおよび第3の位置で、第1の、第2のおよび第3のトリプレットを含むさらなる潜在的な標的部位上で少なくとも1回繰り返される。次いで、そのスコアを有する少なくとも1つの潜在的な標的部位の産出が提供される。いくつかの方法において、最も高いスコアを有する潜在的な標的部位の産出が提供される。いくつかの方法において、最も高いスコアを有するn潜在的な標的部位の産出が提供され、そしてこの方法は、nについての値のユーザー入力を提供する工程をさらに含む。いくつかの方法において、サブスコアは、サブスコアの産物を形成することいよって組み合わされる。いくつかの方法において、対応規則は、64トリプレットを含み、それぞれが第1の、第2のおよび第3の対応する位置、および192サブスコアを有する。
【0017】
いくつかの方法において、対応規則におけるサブスコアは以下によって決定される:トリプレットおよび対応する位置のサブセットのサブスコアとして、第1の値を、対応規則におけるそのトリプレットの対応する位置として、既存のジンクフィンガータンパク質中の同じ位置からのトリプレットに特異的に結合するフィンガーを含む、既存のジンクフィンガータンパク質が存在するトリプレットおよび位置の各々に割り当てる工程;トリプレットおよび対応する位置のサブセットのサブスコアとして第2の値を、対応規則におけるそのトリプレットの対応する位置よりも、既存のジンクフィンガータンパク質中の異なる位置からのトリプレットに特異的に結合するフィンガーを含む、既存のジンクフィンガータンパク質が存在するトリプレットおよび位置の各々に割り当てる工程;ならびに、トリプレットおよびそのトリプレットに特異的に結合するフィンガーを含む既存のジンクタンパク質が存在しない対応する位置のサブセットのサブスコアとして、第3の値を割り当てる工程。
【0018】
いくつかの方法において、状況パラメーターは、少なくとも1つのトリプレットのスケーリングされたサブスコアを与えるために、少なくとも1つの第1の、第2のおよび第3のトリプレットのサブスコアを有する。いくつかの方法において、この状況パラメーターは、標的部位が塩基配列5 ’NNGK3’(ここで、NNGは、少なくとも1つのトリプレットである)を含む場合、サブスコアと組み合わされる。
【0019】
別の局面において、本発明は、ジンクフィンガータンパク質を設計する方法を提供する。このような方法は、複数のジンクフィンガータンパク質(少なくとも第1の、第2のおよび第3のフィンガーを含む各タンパク質)についての指定、ならびに各ジンクフィンガータンパク質の各3つのフィンガーについてのサブ指定を含み;各ジンクフィンガータンパク質について対応する核酸配列であって、各配列は、各ジンクフィンガータンパク質において、それぞれ少なくとも第1の、第2のおよび第3フィンガーによって特異的に結合される、少なくとも第1の、第2のおよび第3のトリプレットを含み、第1の、第2のおよび第3のトリプレットは、第1の、第2のおよび第3のフィンガーが、ジンクフィンガータンパク質において配列される(N末端からC末端)のと同じ個々の順で、核酸配列(3 ’−5’)において配列されている3つのフィンガーの各々について核酸配列を含むデータベースを使用する。標的部位は、ジンクフィンガータンパク質の設計を提供し、この標的部位は、連続した第1の、第2のおよび第3のトリプレットを3 ’−5’の整列で含む。標的部位における第1の、第2のおよび第3のトリプレットについて、データベース中のジンクフィンガータンパク質の第1の、第2のおよび第3のセットは、同定され、第1のセットは、標的部位における第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、第2のセットは、標的部位における第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、第3のセットは、標的部位における第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含む。工程(c)で同定された第1の、第2のおよび第3のセットにおけるジンクフィンガータンパク質の指定およびサブ指定が、次いで出力される。いくつかの方法は、第1のフィンガー(第1のセットからのジンクフィンガータンパク質由来)、第2のフィンガー(第2のセットからのジンクフィンガータンパク質由来)、および第3のフィンガー(第3のセットからのジンクフィンガータンパク質由来)を含む標的部位に結合するジンクフィンガータンパク質を生成する工程をさらに含む。
【0020】
いくつかの方法は、第1の、第2のおよび第3のセットのサブセットを同定する工程をさらに含む。第1のセットのサブセットは、データベース中のジンクフィンガータンパク質の第1のフィンガー位置から標的部位における第1のトリプレットに特異的に結合するフィンガーを含む、ジンクフィンガータンパク質を含む。第2のセットのサブセットは、データベース中のジンクフィンガータンパク質の第2のフィンガー位置から標的部位における第2のトリプレットに特異的に結合するフィンガーを含む、ジンクフィンガータンパク質を含む;第3のセットのサブセットは、データベース中のジンクフィンガータンパク質の第3のフィンガー位置から標的部位における第3のトリプレットに特異的に結合するフィンガーを含む、ジンクフィンガータンパク質を含む。第1の、第2のおよび第3のセットのサブセットの指定およびサブ指定が、出力される。次いで、第1のサブセット由来の第1のフィンガー、第2のサブセット由来の第2のフィンガー、および第3のサブセット由来の第3のフィンガーを含むジンクフィンガータンパク質が、産生される。上記のいくつかの設計方法において、標的部位は、使用者の入力によって提供される。いくつかの方法において、標的部位は、上記の標的部位選択方法の1つによって提供される。
【0021】
本発明は、任意の上記方法を実行するためのコンピュータープログラム製品をさらに提供する。1つのコンピュータープログラム製品は、ジンクフィンガータンパク質による標的化のためのポリヌクレオチド内の標的部位を選択するための方法を実行する。このような製品は、(a)ポリヌクレオチド配列を提供するためのコード;(b)そのポリヌクレオチド配列内の潜在的な標的部位を選択するためのコード;この潜在的な標的部位は、その潜在的な標的部位における第1の、第2のおよび第3の位置で、第1の、第2のおよび第3のトリプレットの塩基を含む;(c)第1の、第2のおよび第3のトリプレットについてのサブスコア、トリプレットとトリプレット位置との間の対応規則から得られるサブスコアの組み合わせから潜在的な標的部位についてのスコアを算出するためのコード、ここで、各トリプレットは、第1の、第2のおよび第3に対応する位置を有し、そして各対応するトリプレットおよび位置は、特定のサブスコアを有する;(d)さらなるスコアを決定するためのさらなる潜在的な標的部位の第1の、第2のおよび第3の位置での第1の、第2のおよび第3のトリプレットを含むさらなる潜在的な標的部位上で、少なくとも1回工程(b)および(c)を繰り返すためのコード;e)そのスコアを有する潜在的な標的部位の少なくとも1つの出力を提供するためのコード;ならびに(f)そのコードを保持するためのコンピューター読取り可能記憶媒体、を含む。
【0022】
本発明はさらに、上記の任意の方法を実行するためのコンピュータシステムを提供する。ジンクフィンガータンパク質による標的化について、ポリヌクレオチド中の標的部位の選択のための1つのこのようなシステムは、以下を含む:(a)メモリ;(b)システムバス;および(c)プロセッサ。このプロセッサは、以下のことをするために作動可能に配置される:(1)ポリヌクレオチド配列を提供するか、または受け入れること;(2)ポリヌクレオチド配列中の潜在的な標的部位を選択すること;この潜在的な標的部位は、この潜在的な標的部位において第1、第2,および第3の位置で、第1、第2,および第3の塩基のトリプレットを含む;(3)これらの第1、第2,および第3のトリプレットについてのサブスコア(subscore)の組合せから、この潜在的な標的部位についてのスコアを計算すること。このサブスコアは、トリプレットとトリプレット位置との間の対応規則から得られる。ここで、各トリプレットは、第1、第2,および第3に対応する位置を有し、そして各対応するトリプレットおよび位置は、特定のサブスコアを有する;(4)さらなる潜在的な標的部位の第1、第2,および第3の位置で第1、第2,および第3のトリプレットを含むさらなる標的部位上で、工程(2)および(3)を少なくとも一回繰り返して、さらなるスコアを決定すること;(5)そのスコアを有する、少なくとも1つの潜在的な標的部位の出力を提供すること。
【0023】
ジンクフィンガータンパク質を生成するためのさらなるコンピュータプログラム製品は、以下を含む:(a)各タンパク質が少なくとも第1、第2および第3のフィンガーを含む、複数のジンクフィンガータンパク質の指定を含むデータベースを提供するためのコード;このジンクフィンガータンパク質の各々の3つのフィンガーの各々のサブ指定(subdesignation);各ジンクフィンガータンパク質に対応する核酸配列であって、各配列は、各ジンクフィンガータンパク質において、それぞれ少なくとも第1、第2および第3のフィンガーによって特異的に結合される、少なくとも第1、第2および第3のトリプレットを含み、これらの第1、第2,および第3のフィンガーと同一のそれぞれの順序においてこの核酸配列(3 ’−5’)において配列される、これらの第1、第2,および第3のトリプレットが、このジンクフィンガータンパク質(N末端からC末端)において配列される、核酸配列;(b)ジンクフィンガータンパク質の設計のために標的部位を提供するためのコードであって、この標的部位は、少なくとも第1、第2,および第3のトリプレットを含む;(c)標的部位の第1、第2,および第3のトリプレットについて、データベース中のジンクフィンガータンパク質の第1、第2,および第3のセットを同定するためのコードであって、この第1のセットは、この標的部位において第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、この第2のセットは、この標的部位において第2のトリプレットに特異的に結合するフィンガーを含み、この第3のセットは、この標的部位において第3のトリプレットに特異的に結合するフィンガーを含む;(d)工程(c)において同定された第1、第2,および第3のセットにおいて、このジンクフィンガータンパク質の指定およびサブ指定を出力するためのコード、ならびに(e)これらのコードを保持するためのコンピュータ読み出し可能な記録媒体。
【0024】
本発明はさらに、ジンクフィンガータンパク質を生成するためのシステムを提供する。このシステムは、以下を含む:(a)メモリ;(b)システムバス;および(c)プロセッサ。このプロセッサは、以下のことをするために作動可能に配置される:(1)各タンパク質が、少なくとも第1、第2,および第3のフィンガーを含む、複数のジンクフィンガータンパク質の指定、このジンクフィンガータンパク質の各々の3つのファンガーの各々のサブ指定を含むデータベース;各ジンクフィンガータンパク質に対応する核酸配列であって、各配列は、各ジンクフィンガータンパク質において、それぞれ少なくとも第1、第2,および第3のフィンガーによって特異的に結合される、少なくとも第1、第2,および第3のトリプレットを含み、第1、第2,および第3のフィンガーと同一のそれぞれの整列においてこの核酸配列(3 ’−5’)において配列される、第1、第2,および第3のトリプレットが、このジンクフィンガータンパク質(N末端からC末端)において配列される、核酸配列を提供すること;(2)ジンクフィンガータンパク質の設計のために標的部位を提供すること(この標的部位は、少なくとも第1、第2,および第3のトリプレットを含む)、(3)第1、第2,および第3のトリプレットについて、データベース中のジンクフィンガータンパク質の第1、第2,および第3のセットを同定すること(この第1のセットは、この標的部位において第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、この第2のセットは、この標的部位において第2のトリプレットに特異的に結合するフィンガーを含み、この第3のセットは、この標的部位において第3のトリプレットに特異的に結合するフィンガーを含む)、;ならびに(4)工程(3)において同定された第1、第2,および第3のセットにおいて、このジンクフィンガータンパク質の指定およびサブ指定を出力すること。
【0025】
(定義)
ジンクフィンガーDNA結合タンパク質は、亜鉛イオンの配位によってタンパク質構造の安定化を生じる結果として、配列特異的様式においてDNAを結合する、タンパク質またはより大きなタンパク質内のセグメントである。用語、ジンクフィンガーDNA結合タンパク質は、しばしば、ジンクフィンガータンパク質またはZFPと省略される。
【0026】
設計されたジンクフィンガータンパク質は、その設計/組成物が、主に合理的基準から得られる、天然に存在しないタンパク質である。設計のための合理的基準は、既存のZFP設計および結合データの情報を格納するデータベースにおける情報処理するための置換の規則およびコンピュータ化されたアルゴリズムの適用を含む。
【0027】
選択されたジンクフィンガータンパク質は、その産物が主に、ファージディスプレイのような経験的プロセスから得られる、天然に見出されないタンパク質である。
【0028】
用語、天然に存在する、とは、人間によって人工的に産生されたものとは異なるような、天然に見出され得る対象物を記載するために使用される。例えば、天然にある供給源から単離され得、そしてこれが研究室の人間によって意図的に改変されていない生物体(ウイルスを含む)に存在するポリペプチドまたはポリヌクレオチド配列は、天然に存在する。一般に、用語、天然に存在する、とは、病的でない(疾患していない)個体において存在するような対象物(例えば、種を象徴するもの)をいう。
【0029】
核酸は、それが別の核酸配列と機能的な関係に置かれる場合、作動可能に連結される。例えば、プロモーターまたはエンハンサーは、コード配列の転写を増加させる場合に、そのコード配列に作動可能に連結される。作動可能に連結するとは、連結されたDNA配列が代表的に連続し、そして2つのタンパク質コード領域を連結することが必要である場合、連続しかつリーディングフレーム中にあることを意味する。しかし、エンハンサーは、一般に数キロベース以上までプロモーターから分離され、そしてイントロン配列が可変の長さであり得る場合に機能するので、いくつかのポリヌクレオチドエレメントは、作動可能に連結され得るが、連続ではない。
【0030】
例えば、ZFPと特定の標的部位との間の特異的結合親和性は、少なくとも1×10 6M-1の結合親和性を意味する。
【0031】
用語、遺伝子の「発現を調節する」、「発現を阻害する」、および「発現を活性化する」とは、遺伝子の転写を活性化または阻害するようなジンクフィンガータンパク質の能力をいう。活性化は、続く転写阻害の防止(すなわち、遺伝子発現の抑制の防止)を含み、そして阻害は、続く転写活性化の阻害(すなわち、遺伝子の活性化の防止)を含む。調節は、標的遺伝子の発現によって直接的または間接的に影響される任意のパラメーターを決定することによってアッセイされ得る。このようなパラメーターとしては、例えば、RNAまたはタンパク質レベルにおける変化、タンパク質活性における変化、産物のレベルにおける変化、下流の遺伝子発現における変化、レポーター遺伝子の転写における変化(ルシフェラーゼ、CAT、 βガラクトシダーゼ、GFP(例えば、MistiliおよびSpector、Nature Biotechnology 15:961〜964(1997)を参照のこと);シグナル伝達、リン酸化および脱リン酸化、レセプターリガンド相互作用、セカンドメッセンジャーの濃度(例えば、cGMP、cAMP、IP3、およびCa 2+)、細胞増殖、新血管形成、インビトロ、インビボ、ならびにエキソビボにおける変化が挙げられる。このような機能的な効果は、例えば、RNAまたはタンパク質レベルの測定、RNA安定性の測定、下流またはレセプター遺伝子発現の同定を、例えば化学発光、蛍光、比色反応、抗体結合、誘導マーカー、リガンド結合アッセイを介して;細胞内のセカンドメッセンジャー(例えば、cGMPおよびイノシトール三リン酸(IP3)における変化;細胞内のカルシウムレベルにおける変化;サイトカイン放出などによる、当業者に公知な任意の手段によって測定され得る。
【0032】
「調節ドメイン」とは、転写調節活性を有するタンパク質またはタンパク質サブ配列をいう。代表的に、調節ドメインは、転写を調節するためにZFPに共有結合的に、または非共有結合的に連結される。あるいは、ZFPは、調節ドメインを有さずに単独で作用し得るか、または転写を調節するために多数の調節ドメインを有して単独で作用し得る。
【0033】
標的部位内のD可能サブ部位は、モチーフ5’NNGK3’を有する。1つ以上のこのようなモチーフを含む標的部位は、時にD可能標的部位と記載される。D可能サブ部位に結合するように適切に設計されたジンクフィンガーは、時にD可能フィンガーといわれる。さらに、少なくとも1つのD可能サブ部位を含む標的部位に結合するように設計され、そして選択された、少なくとも1つのフィンガーを含むジンクフィンガータンパク質は、時にD可能ジンクフィンガータンパク質といわれる。
【発明を実施するための最良の形態】
【0034】
(I.概説)
1つの局面において、本発明は、遺伝子を調節または検出することにおける使用を意図されるジンクフィンガータンパク質の設計のために予め選択された標的部位内の適切なセグメントを選択する方法に関する。潜在的な標的遺伝子のサイズは、約100〜数100,000(数十万)bpまで広範に変化し得る。ジンクフィンガータンパク質は、このような遺伝子内の小さなサブ配列または標的部位に結合し得る。例えば、3つのフィンガーを含むジンクフィンガータンパク質は、代表的に9または10塩基の標的遺伝子に結合する。本発明は、ジンクフィンガータンパク質によって標的化される標的遺伝子から、最適なサブ配列を選択するための基準および方法を提供する。
【0035】
標的部位を選択する方法のいくつかは、1つ以上のいわゆるD可能サブ部位を含むDNAモチーフを有する1つ以上の標的セグメントを同定するために調査される。D可能サブ部位は、以下で詳細に議論されるように特徴的なDNA配列式によって規定される。ジンクフィンガータンパク質は、ジンクフィンガータンパク質の少なくとも1つの成分フィンガーが、フィンガーによって通常結合される3つの塩基サブ部位の外側のさらなる塩基を接触するようなな様式において、このようなモチーフを結合し得る。2つのD可能部位が標的セグメント中に存在する場合、ジンクフィンガータンパク質の2つの成分フィンガーは、標的部位の4つの塩基に各々結合し得る。3つのD可能サブ部位が標的セグメント中に存在する場合、ジンクフィンガータンパク質の3つの成分フィンガーは、標的部位の4つの塩基に各々結合し得る。一般に、少なくとも1つのD可能サブ部位を含む標的部位に結合するジンクフィンガータンパク質は、D可能サブ部位を有さない標的セグメントに結合するジンクフィンガータンパク質よりも、より高い結合親和性を示す。同様に、2つのD可能サブ部位を有する標的部位に結合するジンクフィンガータンパク質は、一般に、1つのD可能サブ部位を有する標的部位に結合するジンクフィンガータンパク質よりも、より高い結合親和性を示し、そして3つのD可能サブ部位を有するジンクフィンガータンパク質は、一般に、2つのD可能サブ部位を有する標的部位に結合するジンクフィンガータンパク質よりも、より高い結合親和性を示す。機構を理解することは本発明を実施するために必要でないが、より高い結合親和性は、ジンクフィンガーと標的セグメント中の3つの塩基との間に可能な相互作用に対して、ジンクフィンガーと標的セグメント中の4つの塩基との間に可能なさらなる相互作用から生じることが考えられる。一般に、D可能サブ部位を有する標的セグメントの高い結合親和性についての可能性は、このサブ部位をジンクフィンガータンパク質の設計のために標的遺伝子内から選択される標的部位にする。なぜなら、より高い結合親和性は、しばしば、標的遺伝子の調節のより大きな程度、および/または標的遺伝子の調節のより大きな特異性を生じるからである。
【0036】
本発明の他の方法は、D可能サブ部位に対するさらなる基準または代替の基準によって標的遺伝子内の標的セグメントを選択することに関する。このような方法における標的セグメントの選択についての主な基準は、3つの塩基の異なるトリプレットと9つの塩基部位内の1つのトリプレットの3つの可能な位置(すなわち、塩基1〜3、4〜6、および7〜9)との間の対応規則の形成において提供される。例示的な対応規則を表1に示す。この対応規則は、標的部位内のトリプレットおよびトリプレット位置の異なる組合せについて異なる値を提供する。標的遺伝子内の潜在的な標的部位は、対応規則から得られるその成分トリプレットについてサブスコアを組合わせることによりこの部位についてのスコアを決定することによって評価される。異なる潜在的な標的部位のスコアは、ジンクフィンガー結合タンパク質の設計のための標的部位として望ましい特定のセグメントを示す、高いスコアと比較される。
【0037】
別の局面において、本発明は、予め選択された標的部位に結合するジンクフィンガータンパク質を設計する方法を提供する。当然のことながら、これらの方法は、上記の手順および基準に従って、標的部位を予め選択した後に使用され得る。設計の方法は、以前に特徴付けられたジンクフィンガータンパク質についての情報を含むデータベースを使用する。この情報は、以前に特徴付けられたジンクフィンガータンパク質、これらの成分フィンガーのアミノ酸配列、およびタンパク質の各フィンガーによって結合されるヌクレオチドトリプレットの名前および他の指定を含む。データベース中の情報は、選択された標的部位に対して特異性を有する新規なジンクフィンガータンパク質において組合せのための以前と異なる設計からのフィンガーの選択を可能にするアルゴリズムを使用して利用される。
【0038】
(II ジンクフィンガータンパク質)
ジンクフィンガータンパク質は、ジンクフィンガー成分から形成される。例えば、ジンクフィンガータンパク質は、1〜37のフィンガーを有し得、一般に2、3、4、5または6つのフィンガーを有する。ジンクフィンガータンパク質は、標的部位(時に標的セグメントといわれる)を認識し、そしてこれに結合する。この標的部位は、標的遺伝子内に比較的小さなサブ配列を示す。ジンクフィンガータンパク質の各成分フィンガーは、標的部位内のサブ部位に結合し得る。このサブ部位は、同一鎖(時に、標的鎖といわれる)上で3つ連続する塩基のトリプレットを全て含む。このサブ部位はまた、標的鎖上の3つ連続する塩基のすぐ3 ’側の塩基の成分である、反対鎖上の第4の塩基を含んでもよいし、含まなくてもよい。多くのジンクフィンガータンパク質において、ジンクフィンガーは、そのトリプレットサブ部位に、同一のジンクフィンガータンパク質中の実質的に独立な他のフィンガーを結合する。従って、多数のフィンガーを含むジンクフィンガータンパク質の結合特異性は、通常ほぼ、その成分フィンガーの特異性の集合体である。例えば、ジンクフィンガータンパク質が、トリプレットXXX、YYY、およびZZZに対して個々に結合する第1、第2、および第3のフィンガーから形成される場合、このジンクフィンガータンパク質の結合特異性は、3 ’XXX YYY ZZZ5’である。
【0039】
N末端からC末端のジンクフィンガータンパク質においてフィンガーの相対的な整列は、標的中の3’から5 ’方向におけるトリプレットの相対的な順序を決定する。例えば、ジンクフィンガータンパク質が上記の第1、第2、および第3のフィンガーをN末端からC末端に含む場合、このジンクフィンガータンパク質は標的セグメント3 ’XXXYYYZZZ5’に結合する。このジンクフィンガータンパク質が別の順序(例えば、第2のフィンガー、第1のフィンガー、第3のフィンガー)でこのフィンガーを含む場合、このジンクフィンガータンパク質は、異なる順列のトリプレット(この例においては、3 ’YYYXXXZZZ5’)を含む標的セグメントに結合する(BergおよびShi、Science 271、1081〜1086(1996)を参照のこと)。しかし、その成分フィンガーの集合体としてジンクフィンガータンパク質の結合特性の評価は、おおよそであるのみである。これは、同一のタンパク質中の多数のフィンガー結合の状況依存性相互作用に起因する。
【0040】
2つ以上のジンクフィンガータンパク質は、標的特異性を有するように連結され得る。この特異性は、成分ジンクフィンガータンパク質の特異性の集合体である(例えば、KimおよびPabo、PNAS 95、2812〜2817(1998)を参照のこと)。例えば、XXX、YYY、およびZZZにそれぞれ結合する第1、第2、および第3の成分フィンガーを有する第1のジンクフィンガータンパク質は、結合特異性AAA、BBB、およびCCCを有する第1、第2、および第3の成分フィンガーを有する第2のジンクフィンガータンパク質に連結し得る。従って、組合せた第1および第2のタンパク質の結合特異性は、3 ’XXXYYYZZZ_AAABBBCCC5’であり、ここで、下線は、短い介入領域(代表的に、任意の型の0〜5の塩基)を示す。この状況において、標的部位は、介入セグメントによって分離される2つの標的セグメントを含むと考えられ得る。
【0041】
連結は、以下の任意のペプチドリンカーを使用して達成され得る。TGEKP(Liuら、1997、前出);(G4S)n(Kimら、PNAS 93、1156〜1160(1996);GGRRGGGS;LRQRDGERP;LRQKDGGGSERP;LRQKD(G3S)2ERP。あるいは、可撓性リンカーは、DNA結合部位およびペプチド自体の両方をモデリングし得るコンピュータプログラムを使用して、またはファージディスプレイ方法によって、合理的に設計され得る。さらなる改変において、非共有結合的な連結は、2つのジンクフィンガータンパク質のヘテロ二量体形成を促進するドメインと2つのジンクフィンガータンパク質とを融合することによって達成され得る。例えば、一方のジンクフィンガータンパク質がfosと融合され得、そして他方はjunと融合され得る(Barbasら、WO95/119431を参照のこと)。
【0042】
2つのジンクフィンガータンパク質の連結は、哺乳動物ゲノム内の独特な結合特異性を与えるために有利である。代表的な哺乳動物二倍体ゲノムは、3 ×109bpからなる。4つのヌクレオチドA、C、G、およびTがランダムに分布すると仮定すると、所定の9bpの配列は、約23,000回存在する。従って、絶対的な特異性を有する9bp標的を認識するZFPは、このゲノム内の約23,000の部位に結合する可能性を有する。18bpの配列は、3.4 ×1010bp中に1回またはランダムなDNA配列中に約1回存在し、これらの複雑性は、哺乳動物ゲノムの複雑性の10倍である。
【0043】
ジンクフィンガータンパク質の成分フィンガーは代表的に、約30アミノ酸を含み、そして以下のモチーフ(N−C)を有する:
【0044】
【数1】

【0045】
単一のβターン中の2つの不変ヒスチジン残基および2つの不変システイン残基は、亜鉛を通じて配位される(例えば、BergおよびShi、Science 271、1081〜1085(1996)を参照のこと)。上記のモチーフは、結合特異性を与えるジンクフィンガーの領域の分野において標準的である番号付けの取り決めを示す。第1の不変His残基の左(N末端側)のアミノ酸は番号+6を割り当てられ、そしてさらに左の他のアミノ酸は、連続的に減じられた数を割り当てられる。 αヘリックスは、残基1で始まり、そして第2の保存ヒスチジンの後の残基まで伸長する。従って、このヘリックス全体は、11残基と13残基との間の可変の長さである。
【0046】
天然に存在しないZFPまたは改変ZFPを設計または選択する工程は、代表的にフレームワーク残基の供給源としての天然ZFPで開始する。この選択または設計の工程は、所望される結合特異性を与えるように非保存位置(すなわち、位置−1〜+6)を規定するために作用する。1つの適切なZFPは、マウス転写因子Zif268のDNA結合ドメインである。このタンパク質のDNA結合ドメインは、以下のアミノ酸配列を有し、そして標的5 ’GCG TGG GCG3’に結合する:
【0047】
【数2】

【0048】
フレームワーク残基の供給源として適切な別の天然のジンクフィンガータンパク質はSp−1である。ジンクフィンガータンパク質の構築のために使用されるSp−1配列は、Sp−1転写因子のアミノ酸531〜624に対応する。この配列は94アミノ酸長である。Sp−1のアミノ酸配列は以下である:
【0049】
【数3】

【0050】
Sp−1は標的部位5’GGG GCG GGG 3’に結合する。
【0051】
Sp−1の代替的な形態であるSp−1コンセンサス配列は、以下のアミノ酸配列を有する:
【0052】
【数4】

【0053】
(小文字はShiおよびBerg、Chemistry and Biology1,83〜89(1995)からのリーダー配列である)。Sp−1コンセンサス配列について最適な結合配列は、5 ’GGGGCGGGG3’である。他の適切なZFPを以下に記載する。
【0054】
いくつかのジンクフィンガータンパク質の合理的な設計を助ける、多数の置換法則が存在する(DesjarlaisおよびBerg、PNAS 90、2256〜2260(1993);ChooおよびKlug、PNAS 91、11163〜11167(1994);DesjarlaisおよびBerg、PNAS 89、7345〜7349(1992);Jamiesonら、上述;Chooら、WO98/53057、WO98/53058;WO98/53059;WO98/53060を参照のこと)。これらの法則の多くは、偏在性転写因子であるSp−1の3つのフィンガードメインの部位特異的変異誘発により支持される(DesjarlaisおよびBerg,1992;1993)。これらの法則の1つは、DNAトリプレット中の5 ’Gが、認識へリックスの6位にアルギニンを組み込んでいるジンクフィンガーにより結合され得ることである。別の置換法則は、サブ部位の中央のGが、ジンクフィンガーの3位にヒスチジン残基を含むことによって認識され得ることである。さらなる置換法則は、トリプレットの中央のAを認識するためにアスパラギンが組み込まれ得、アスパラギン酸、グルタミン酸、セリンまたはスレオニンがトリプレットの中央のCを認識するために組み込まれ得、そして小さい側鎖を有するアミノ酸(例えば、アラニン)がトリプレットの中央のTを認識するために組み込まれ得ることである。さらなる置換法則は、トリプレットサブ部位の3 ’塩基が以下のアミノ酸を認識へリックスの−1位に組み込むことによって認識され得ることである:Gを認識するためにアルギニン、Aを認識するためにグルタミン、Cを認識するためにグルタミン酸(またはアスパラギン酸)、およびTを認識するためにスレオニン。これらの置換法則はジンクフィンガータンパク質の設計のために有用であるが、これらは全ての潜在的な標的部位を考慮に入れるわけではない。さらに、この法則の基となる仮説、すなわち、ジンクフィンガー中の特定のアミノ酸がサブ部位中の特定の塩基への結合の原因であることは、単なる概算である。フィンガー中の近位のアミノ酸間の前後関係依存性の相互作用または複数のアミノ酸の単塩基への結合もしくはその逆は、既存の置換法則により予測される結合特異性の変動を引き起こし得る。
【0055】
ファージディスプレイ技術は、所望の標的特異性を有するジンクフィンガータンパク質を生成する多数の経験的手段を提供する(例えば、Rebar、米国特許第5,789,538号;Chooら、WO96/06166;Barbasら、WO95/19431およびWO98/543111;Jamiesonら、上述を参照のこと)。この方法は合理的設計と組み合わせて、または合理的設計の代替法として使用され得る。この方法は、変異誘発されたジンクフィンガータンパク質の多様なライブラリーの産生、続いて親和性選択方法を使用して所望のDNA結合特性を有するタンパク質を単離することを含む。この方法を使用するために、実験者は、代表的には以下のように進める。最初に、ジンクフィンガータンパク質の遺伝子を変異誘発させて、結合特異性および/または親和性に重要である領域に多様性を導入する。代表的な適用において、これは−1位、+2位、+3位および+6位、ならびにしばしば+1位、+5位、+8位および+10位のような補助(accessory)位置で1つのフィンガーのランダム化を介して達成される。次に、変異誘発された遺伝子は、繊維状ファージの第III遺伝子(これはコートタンパク質であるpIIIをコードする)との融合物としてファージまたはファージミドにベクターにクローニングされる。このジンクフィンガー遺伝子は膜搬出シグナルペプチドをコードする第III遺伝子のセグメントとpIIIの残余の間に挿入され、その結果ジンクフィンガータンパク質はpIIIとのアミノ末端融合物としてか、または成熟したプロセシングされたタンパク質で発現される。ファージミドベクターを使用する場合、変異誘発されたジンクフィンガー遺伝子はまた、pIIIのファージ粒子へのアセンブリのために必要とされるC末端領域を最小限にコードする第III遺伝子の短縮型バージョンに融合され得る。得られるベクターライブラリーはE.coliに形質転換され、そしてその表面上にコートタンパク質pIIIとの融合物として改変型ジンクフィンガータンパク質を発現する繊維状ファージを産生するために使用される。ファージミドベクターが使用される場合、この工程はヘルパーファージでの重感染を必要とする。次いで、ファージライブラリーは標的DNA部位とともにインキュベートされ、そして親和性選択法を使用してバルクファージ(bulk phage)から高親和性で標的と結合するファージを単離する。代表的には、このDNA標的は固相支持体上に固定化されて、次いでこの支持体は最も強固に結合するファージ以外を取り除くために十分な条件下で洗浄される。洗浄後、支持体上に残存する任意のファージが、ジンクフィンガー−DNA結合を破壊する条件下での溶出を介して回収される。回収したファージを使用して新しいE.coliを感染させ、次いでこれを増幅させてファージ粒子の新しいバッチを産生するために使用する。次いで、選択および増幅は、強固に結合するものについてファージプールを富化するに必要な回数だけ反復され、その結果として配列決定方法および/またはスクリーニング方法を使用してこれらは同定され得る。この方法はpIII融合物について例示されるが、類似の原則がZFP改変体をpVIII融合物としてスクリーニングするために使用され得る。
【0056】
ジンクフィンガータンパク質は、しばしば、融合タンパク質として異種ドメインと共に発現される。ZFPへの付加のための共通のドメインとしては、例えば、転写因子ドメイン(アクチベーター、リプレッサー、コアクチベーター、コリプレッサー)、サイレンサー、オンコジーン(例えば、myc、jun、fos、myb、max、mad、rel、ets、bcl、myb、mosファミリーメンバーなど);DNA修復酵素ならびにその関連因子および修飾因子;DNA再配置酵素ならびにその関連因子および修飾因子;クロマチン関連タンパク質およびそれらの修飾因子(例えば、キナーゼ、アセチラーゼおよびデアセチラーゼ);ならびにDNA修飾酵素(例えば、メチルトランスフェラーゼ、トポイソメラーゼ、ヘリカーゼ、リガーゼ、キナーゼ、ホスファターゼ、ポリメラーゼ、エンドヌクレアーゼ)ならびにそれらの関連因子および修飾因子が挙げられる。ZFPが標的遺伝子の発現を抑制するために使用されるべきである場合、ZFPとの融合に好ましいドメインは、ヒトKOX−1タンパク質由来のKRAB抑制ドメインである(Thiesenら、New Biologist 2、363〜374(1990);Margolinら、Proc.Natl.Acad.Sci.USA 91、4509〜4513(1994);Pengueら、Nucl.Acids Res.22:2908〜2914(1994);Witzgallら、Proc.Natl.Acad.Sci.USA 91、4514〜4518(1998))。活性化を達成するために好ましいドメインとしては、HSV VP16活性化ドメイン(例えば、Hagmannら、J.Virol.71、5952〜5962(1997)を参照のこと)、核ホルモンレセプター(例えば、Torchiaら、Curr.Opin.Cell.Biol.10:373〜383(1998)を参照のこと);核因子 κBのp65サブユニット(BitkoおよびBarik、J.Virol.72:5610〜5618(1998)ならびにDoyleおよびHunt、Neuroreport 8:2937〜2942(1997));Liuら、Cancer Gene Ther.5:3〜28(1998))、または人工キメラ機能的ドメイン(例えば、VP64(Seifpalら、EMBO J.11、4961〜4968(1992)))が挙げられる。
【0057】
ポリペプチド化合物(例えば、ZFP)の投与に重要な因子は、このポリペプチドが細胞の原形質膜または細胞内区画(例えば、核)の膜を横切る能力を有することを保証することである。細胞膜は脂質−タンパク質二重層からなり、これは小さな非イオン性の親油性化合物を自由に通し、そして極性化合物、高分子および治療剤または診断剤に対して本質的に不透過性である。しかしながらタンパク質および他の化合物(例えば、リポソーム)が、細胞膜を横切ってZFPのようなポリペプチドをトランスロケーションさせる能力を有することが記載されている。
【0058】
例えば、「膜トランスロケーション(membrane translocation)ポリペプチド」は、膜トランスロケーションキャリアとして作用する能力を有する、両親媒性または疎水性アミノ酸のサブ配列を有する。1つの実施態様において、ホメオドメインタンパク質は細胞膜を横切ってトランスロケーションする能力を有する。ホメオドメインタンパク質であるAntennapediaの最も短いインターナライゼーション可能なペプチドは、タンパク質の第3のへリックス(アミノ酸43位〜58位)であることが見出された(例えば、Prochiantz、Current Opinion in Neurobiology 6:629〜634(1996)を参照のこと)。別のサブ配列である、シグナルペプチドのh(疎水性)ドメインは、類似の細胞膜トランスロケーション特性を有することが見出された(例えば、Linら、J.Biol.Chem.270:14255〜14258(1995)を参照のこと)。
【0059】
ZFPの細胞内への取り込みを容易にするために、本発明のZFPに連結され得るペプチド配列の例としては、以下が挙げられるがこれらに限定されない:HIVのtatタンパク質の11アミノ酸ペプチド;p16タンパク質のアミノ酸84〜103に対応する20残基のペプチド配列(Fahraeusら、Current Biology 6:84(1996)を参照のこと);Antennapediaの60アミノ酸長ホメオドメインの第3へリックス(Derossiら、J.Biol.Chem.269:10444(1994));カポージ線維芽細胞増殖因子(K−FGF)h領域(Linら、上述)のようなシグナルペプチドのh領域;またはHSV由来のVP22トランスロケーションドメイン(ElliotおよびO ’Hare、Cell 88:223〜233(1997))。増強された細胞性取り込みを提供する他の適切な化学的部分もまた、ZFPに化学的に連結され得る。
【0060】
トキシン分子もまた、細胞膜を横切ってポリペプチドを輸送する能力を有する。しばしば、このような分子は少なくとも2つの部分からなる(「二元トキシン」と呼ばれる):トランスロケーションまたは結合ドメインまたはポリペプチド、ならびに別個のトキシンドメインまたはポリペプチド。代表的に、トランスロケーションドメインまたはポリペプチドは細胞性レセプターに結合し、次いでこのトキシンは細胞内に輸送される。いくつかの細菌性トキシン(Clostridium perfringensイオータトキシン、ジフテリアトキシン(DT)、PseudomonasエキソトキシンA(PE)、百日咳トキシン(PT)、Bacillus anthracisトキシンおよび百日咳アデニル酸シクラーゼ(CYA)を含む)は、細胞のサイトゾルへとペプチドを内部融合物またはアミノ末端融合物として送達する試みにおいて使用されてきた(Aroraら、J.Biol.Chem.、268:3334〜3341(1993);Perelleら、Infect.Immun.、61:5147〜5156(1993);Stenmarkら、J.Cell Biol.113:1025〜1032(1991);Donnellyら、PNAS 90:3530〜3534(1993);Carbonettiら、Abstr.Annu.Meet.Am.Soc.Microbiol.95:295(1995);Seboら、Infect.Immun.63:3851〜3857(1995);Klimpelら、PNAS U.S.A.89:10277〜10281(1992);およびNovakら、J.Biol.Chem.267:17186〜17193(1992))。
【0061】
このようなサブ配列は、細胞膜を横切ってZFPをトランスロケーションさせるために使用され得る。ZFPは、便利に、このような配列に融合され得るか、またはこのような配列を用いて誘導体化され得る。代表的には、このトランスロケーション配列は融合タンパク質の一部として提供される。必要に応じて、リンカーはZFPとトランスロケーション配列とを連結させるために使用され得る。任意の適切なリンカー、例えば、ペプチドリンカーが使用され得る。
【0062】
(III.標的遺伝子の選択)
ジンクフィンガータンパク質は、任意の標的ポリヌクレオチド配列の発現を調整するために使用され得る。この配列は、例えば、ゲノム、cDNAまたはRNAあるいは発現された配列タグ(EST)であり得る。代表的には、標的ポリヌクレオチドとしては、遺伝子またはそのフラグメントが挙げられる。用語、遺伝子は、以下を含むように広範に使用される:例えば、エキソン領域、イントロン領域、5 ’UTR、3’UTR、5’隣接配列、3 ’隣接配列、プロモーター、エンハンサー、転写開始部位、リボソーム結合部位、調節部位、ポリアデニル化部位が挙げられる。標的遺伝子は、細胞性、ウイルス性または他の供給源由来(純粋に理論的な配列を含む)のものであり得る。標的遺伝子配列は、データベース(例えば、GenBank)、刊行物から得られ得るか、またはデノボで得られ得る。標的遺伝子としては、感染を停止するために発現の抑制が使用され得る病的なウイルスおよび微生物由来の遺伝子が挙げられる。病原性ウイルスの例としては、肝炎(A型、B型またはC型)、ヘルペスウイルス(例えば、ZVZ、HSV−1、HSV−6、HSV−IIおよびCMV、エプスタイン−バールウイルス)、HIV、エボラ、アデノウイルス、インフルエンザウイルス、フラビウイルス、ECHOウイルス、ライノウイルス、コクサッキーウイルス、コロナウイルス(cornovirus)、RSウイルス、流行性耳下腺炎ウイルス、ロタウイルス、麻疹ウイルス、風疹ウイルス、パルボウイルス、ワクシニアウイルス、HTLVウイルス、デング熱ウイルス、パピローマウイルス、軟属腫ウイルス(molluscum virus)、ポリオウイルス、狂犬病ウイルス、JCウイルスおよびアルボウイルス性脳炎ウイルスが挙げられる。病原性細菌のいくつかの例としては、クラミジア、リケッチア細菌、ミコバクテリア、ブドウ球菌、連鎖球菌(treptoccus)、肺炎球菌、髄膜炎菌および淋菌(conococcus)、クレブシエラ、プロテウス、セラチア、シュードモナス、レジオネラ、ジフテリア、サルモネラ、バシラス、コレラ、破傷風、ボツリヌス中毒、炭疽、ペスト、レプトスピラおよびライム病細菌が挙げられる。
【0063】
標的遺伝子はまた、疾患の原因であるヒトまたは他の哺乳動物由来の遺伝子を含む。いくつかのこのような遺伝子はオンコジーン、腫瘍サプレッサーまたは増殖因子であり、これらはガンの原因である。オンコジーンの例としては、hMSH2(Fishelら、Cell 75、1027〜1038(1993))およびhMLH1(Papadopoulosら、Science 263、1625〜1628(1994))が挙げられる。増殖因子のいくつかの例としは、線維芽細胞増殖因子、血小板由来増殖因子、GM−SCF、VEGF、EPO、Erb−B2およびhGHが挙げられる。他のヒト遺伝子は、被験体を微生物またはウイルスによる感染に罹患し易くすることにより、疾患の原因となる。例えば、CCR5レセプターをコードする遺伝子の特定の対立遺伝子は、被験体をHIVによる感染に罹患し易くする。他のヒト遺伝子(例えば、アミロイド前駆体タンパク質またはApoEをコードする遺伝子)は、他の疾患(例えば、アルツハイマー病)の原因である。
【0064】
標的遺伝子としてはまた、他の供給源が原因である疾患に対する防御機構を提供する、ヒトまたは他の哺乳動物の遺伝子が挙げられる。例えば、腫瘍リプレッサー遺伝子は、ガンに対する防御を提供する。このような遺伝子の発現は望ましく、そしてジンクフィンガータンパク質は発現を活性化するために使用される。
【0065】
標的遺伝子としてはまた、通常発現が止められているかまたは低レベルで発現されている遺伝子が挙げられるが、これらは活性化を通してある個体に存在する別の欠損型遺伝子を置換するために使用され得る。例えば、胎児ヘモグロビン(hemaglobin)遺伝子(これは成体ヒトにおいては通常不活性である)を活性化して、鎌状赤血球貧血を有する個体における欠損型 β−グロビン遺伝子を代用し得る。
【0066】
標的遺伝子としてはまた、その抑制または活性化が植物特性の改善(例えば、改善された作物生産、病害耐性または除草剤耐性)を導く植物遺伝子が挙げられる。例えば、FAD2−1遺伝子の発現の抑制は、オレイン酸の増加ならびにリノール酸およびリノレン酸の減少という利点を生じる。
【0067】
(IV.D可能サブ部位に結合するためのジンクフィンガータンパク質の設計)
(1.方法)
本発明は、標的遺伝子を選択し、そして1〜6(以上)のD可能サブ部位を含む遺伝子内の標的部位を同定する方法を提供する。次いで、予め選択された部位に結合するジンクフィンガータンパク質が合成され得る。標的部位選択のこれらの方法は、標的セグメント中の1つ以上のD可能サブ部位の存在が、D可能サブ部位を欠損する標的セグメントに結合するジンクフィンガータンパク質と比較して、その部位に結合するように選択されたかまたは設計されたジンクフィンガータンパク質により高い結合親和性の可能性を付与するという本発明者らの認識を、一部で前提とする。この洞察を支持する実験的証拠は、実施例2〜9に提供される。
【0068】
D可能サブ部位は、適切に設計された単一のジンクフィンガーが、標的部位の3塩基までよりもむしろ4塩基までに結合することを可能にする標的部位の領域である。このようなジンクフィンガーは、二本鎖標的セグメント(標的鎖)の1つの鎖上の塩基のトリプレットおよび他方の鎖上の第4の塩基に結合する(図2を参照のこと)。単一のジンクフィンガーが、4塩基の標的セグメントに結合することは、標的鎖の配列上およびジンクフィンガーのアミノ酸配列上の両方に束縛を課する。標的鎖内の標的部位は、「D可能」サブ部位モチーフ5 ’NNGK3’を含むべきであり、ここで、NおよびKは、従来のIUPAC−IUB多義性コードである。このような部位に結合するためのジンクフィンガーは、−1位にアルギニン残基および+2位にアスパラギン酸(または次に好ましくはグルタミン酸)を含むべきである。−1位におけるアルギニン残基は、D可能サブ部位中のG残基と相互作用する。ジンクフィンガーの+2位におけるアスパラギン酸(またはグルタミン酸)残基は、D可能サブ部位中のK塩基に相補的な反対鎖の塩基と相互作用する。名前D可能サブ部位を与えるのは、アスパラギン酸(シンボルD)と、反対鎖塩基(第4の塩基)との間の相互作用である。D可能サブ部位式から明らかなように、D可能サブ部位には2つのサブタイプ:5 ’NNGG3’および5’NNGT3 ’がある。前者のサブ部位には、ジンクフィンガーの+2位のアスパラギン酸またはグルタミン酸が、D可能サブ部位に対する反対鎖中のCと相互作用する。後者のサブ部位では、ジンクフィンガーの+2位のアスパラギン酸またはグルタミン酸が、D可能サブ部位に対する反対鎖中のAと相互作用する。一般に、NNGGは、NNGTより好ましい。
【0069】
3つのフィンガーを持つジンクフィンガータンパク質の設計では、標的部位は、その中で、このタンパク質の少なくとも1つのフィンガー、そして好ましくは、2つまたは3つのフィンガーが、標的部位中のD可能サブ部位を結合する能力を有するように選択されるべきである。これは、以下の式を有するより大きな標的遺伝子内から標的部位を選択することにより達成され得る:5 ’NNx aNy bNzc3’、ここでセット(x、a)、(y、b)および(z、c)の各々は、(N、N)または(G、K)のいずれかであり、(x、a)、(y、b)および(z、c)の少なくとも1つは(G、K)であり、そしてNおよびKは、IUPAC−IUB多義性コードである。
【0070】
換言すれば、3つのセット(x、a)、(y、b)および(z、c)の少なくとも1つは、このセットの第1の位置がGであり、そして第2の位置がGまたはTであることを意味するセット(G、K)である。3つのセットのうち(G、K)ではないもの(もしあれば)は、このセットの第1の位置が任意のヌクレオチドにより占められ得、そしてこのセットの第2の位置が任意のヌクレオチドにより占められ得ることを意味する(N、N)である。例として、セット(x、a)は(G、K)であり得、そしてセット(y、b)および(z、c)は、両方とも(N、N)であり得る。
【0071】
式5’NNx aNyb Nzc3’において、NNx aNyおよびbNzのトリプレットは、ジンクフィンガータンパク質中の3つのフィンガーにより結合される標的鎖上の塩基のトリプレットを示す。強調された塩基の相補物は、非標的鎖上で結合する潜在的な第4の塩基の部位である。x、yおよびzのたった1つがGであり、そしてこのGにKが続く場合、標的部位は単一のD可能サブ部位を含む。例えば、xのみがGであり、そしてaがKである場合、この部位は、D可能サブ部位を強調して、以下のように読める:
【0072】
【化1】

【0073】
zではなく、xおよびyの両方がGであり、そしてaおよびbがKである場合、そのときは、標的部位は、以下のように、1つのこのような部位を太字でそして他をイタリックで示して、2つの重複するD可能サブ部位を有する:
【0074】
【化2】

【0075】
x、yおよびzの3つのすべてがGであり、そしてa、bおよびcがKである場合、そのときは、標的セグメントは、以下のように、3つのD可能サブ部位である、以下:
【0076】
【化3】

【0077】
を含み、D可能サブ部位は、太字、イタリックおよび下線で示される。
【0078】
従って、本発明の方法は、標的遺伝子を選択すること、および式5’NNxaNybNzc3 ’に一致する標的部位のための遺伝子の潜在的なサブ配列内を系統的にサーチすることにより行われ、ここで、(x、a)、(y、b)および(z、c)の各々は、(N、N)または(G、K)であり;
(x、a)、(y、b)および(z、c)の少なくとも1つは(G、K)であり、そしてNおよびKは、IUPAC−IUB多義性コードである。
このような方法のいくつかでは、潜在的な標的遺伝子のいずれかの鎖上の10の連続塩基の潜在的なサブ配列毎に、それが上記の式に一致するか否かを決定するため、そしてもしそうであれば、D可能サブ部位がいくつあるか評価される。代表的には、このような比較は、コンピューターにより実施され、そして上記式に一致する標的部位のリストが出力される。必要に応じて、このような標的部位は、D可能サブ部位がいくつあるかに従って、異なるサブセットで出力され得る。
【0079】
改変法では、本発明の方法は、各々が独立に上記の式に一致する第1および第2の標的セグメントを同定する。このような方法における2つの標的セグメントは、束縛されて、標的遺伝子中、互いに隣接するかまたは近接(すなわち、約0〜5塩基内)する。近接する標的セグメントの選択の基礎となる戦略は、第1および第2の標的セグメントにそれぞれ特異的な2つの成分ジンクフィンガータンパク質の連結により形成されるジンクフィンガータンパク質の設計を可能にする。これらの原理は、任意の数の成分フィンガーを持つジンクフィンガータンパク質により結合される標的部位を選択するために拡張され得る。例えば、9フィンガーのタンパク質に適切な標的部位は、各々が上記の式に一致する3つの成分セグメントを有する。
【0080】
上記の方法により同定される標的部位は、他の規準によるさらなる評価を受け得るか、またはこのような部位に特異的なジンクフィンガータンパク質の設計または選択(必要であれば)および産生のために直接用いられ得る。潜在的な標的部位を評価するためのさらなる規準は、遺伝子内の特定領域への近接度である。ジンクフィンガータンパク質が、それ自身に関する細胞遺伝子を抑制するために用いられるべき場合(すなわち、このジンクフィンガータンパク質を、抑制する部分に連結することなく)、最適位置は、転写開始の部位か、または約50bp上流もしくは下流内であるか、あるいは転写複合体の形成(Kim&Pabo、J.Biol.Chem.(1977))を妨害するためか、または必須エンハンサー結合タンパク質と競合するためにエンハンサーエレメント内にあるようである。しかし、ZFPが、KRABリプレッサードメインまたはVP16アクチベータードメインのような機能的ドメインに融合される場合、結合部位の位置の選択は、かなりより柔軟であり、そして既知の調節領域の外側であり得る。例えば、KRABドメインは、KRABが結合する場所から少なくとも3kbまで、プロモーターの転写を抑制し得る。従って、調節配列、または多形性部位のような標的遺伝子を含まないか、または標的遺伝子との重要なセグメントと重複しない標的部位が選択され得る。標的セグメントをさらに評価するためのその他の規準としては、このようなセグメントまたは関連セグメントへのジンクフィンガータンパク質結合の先立つ利用可能性、および/または所定の標的セグメントを結合するために新たなジンクフィンガータンパク質を設計する容易さが挙げられる。選択プロセスにおけるこのような基準の履行は、以下でより詳細に論議される。
【0081】
一旦、標的セグメントが選択されたなら、このセグメントに結合するジンクフィンガータンパク質は、種々のアプローチにより提供され得る。最も簡単なアプローチは、既に標的部位に結合することが知られている現存するコレクションから予め特徴付けられたジンクフィンガータンパク質を提供することである。しかし、多くの例では、このようなジンクフィンガータンパク質は存在しない。代替のアプローチは、現存するジンクフィンガータンパク質および結合特異性のデータベース中の情報を用いて、新たなジンクフィンガータンパク質を設計することである。このアプローチを以下により詳細に記載する。さらなるアプローチは、上記で議論されるような置換規則に基づきジンクフィンガータンパク質を設計することである。なおさらなる代替は、ファージディスプレイのような経験的プロセスにより所定の標的に対して特異性を持つジンクフィンガータンパク質を選択することである。このような方法のいくつかでは、ジンクフィンガータンパク質の各成分フィンガーは、他の成分フィンガーとは独立に設計または選択される。例えば、各フィンガーは、予め存在する異なったZFPから得られ得るか、または各フィンガーは、別のランダム化および選択を受け得る。
【0082】
一旦、ジンクフィンガータンパク質が、所定の標的セグメントに対して、選択、設計、またはそうでなく提供されたなら、ジンクフィンガータンパク質またはコードDNAが合成される。ジンクタンパク質を合成およびジンクタンパク質のコードDNAを発現する例示の方法は以下に記載される。次いで、ジンクフィンガータンパク質またはそれをコードするポリヌクレオチドは、このジンクフィンガータンパク質が結合する標的部位を含む標的遺伝子の発現の調整、または分析に用いられ得る。
【0083】
(2.D可能ジンクフィンガータンパク質)
ジンクフィンガータンパク質は、それが、ポリヌクレオチド配列5’NNGK3 ’である、少なくとも1つのD可能サブ部位の第4の塩基に結合し得るフィンガーを含む場合、D可能として記載される。D可能ジンクフィンガーを設計するための好適なフレームワークは、ヒト野生型Sp−1 DNA結合ドメインである。ヒト転写因子Sp−1の標的は、5’GGG GCG GGG3 ’であり、そしてこのタンパク質のフィンガー1および2は、R−1D+2配置を有する。設計されたZFPは、3つのフィンガーの各々の認識ヘリックス中を除いてSp−1と同一であり得、ここでは、配列は、それらが相互作用するトリプレットの各々を認識するように設計される。マウスZFP Zif268は、部位GCG TGG GCGを結合し、3つすべてのフィンガーにR−1D+2配置を有して、また適切である。
【0084】
D可能サブ部位に結合し得るジンクフィンガータンパク質の設計のためのフレームワーク残基の供給源としての他のジンクフィンガータンパク質は、いくつかの代替供給源からのZFPから得られか、または由来し得る。例えば、ショウジョウバエ Drosophila melanogasterのTTK転写調節タンパク質は、その認識ヘリックスおよびそのDNA部位の両方の配列に関して良く特徴付けられている。このタンパク質は、たった2つのフィンガーを有し、そして6塩基の標的に結合し、そこでフィンガー2が、第1のDNAトリプレットと相互作用し、そしてフィンガー1がこの部位の第2のトリプレットを認識する。この部位は、第1および第2のトリプレットの接合部に存在するGGタイプD可能サブ部位を持つ5 ’AAGGAT3’であり、そしてフィンガー2は、R−1 D+2配列を有する。他の適切なZFPは、単細胞真核生物Sacchromyces cerevisiae中に見出される。ADR遺伝子産物は、ADHプロモーター内に結合することにりADH遺伝子の発現を調節することが知られている。TTKについて上記に記載されるように、ADR ZFP結合ドメインは、2つのフィンガーを有し、そして6塩基の標的TTGGAGに結合する。このフィンガー2認識ヘリックスは、D可能サブ部位を持つ標的部位へのZFP結合に適切なR−1 D+2配列を有する。
【0085】
(IV.対応規則による標的部位の選択)
本発明は、標的遺伝子内から標的部位を選択するためのさらなる、または代替の方法をさらに提供する。これらの方法は、部分的には、個々のフィンガーにより結合される異なる3塩基サブ部位(トリプレット)が、ジンクフィンガータンパク質設計について異なる望ましさを有すること、これらの異なる望ましさが、数値として表現され得ること、および標的部位を含む3つの個々のトリプレットに対する数値が、標的部位に対する全体スコアを与えるために組み合わせられ得るという洞察を前提とする。異なる標的部位の相対的なメリットは、それらの相対的スコアから比較され得る。
【0086】
この方法は、ZFPによる検出または調整のための標的部位をその中で選択することを望む、代表的には遺伝子またはcDNAであるポリヌクレオチド配列を提供することにより実施する。実際、代表的には、ポリヌクレオチド配列の2つの鎖に対して2つの配列を提供するが、単純さのために、この方法は、単一のポリヌクレオチド配列について例示される。このようなポリヌクレオチド配列内から、連続する第1、第2および第3の、塩基のトリプレットを含む少なくとも9塩基の潜在的な標的部位が選択される。これらのトリプレットは、5 ’−3配向における塩基1が塩基1であるとして、第1のトリプレットが部位の塩基7〜9を、第2のトリプレットが部位の塩基4〜6を、そして第3のトリプレットが部位の塩基1〜3を占めるという点で連続している。トリプレットの第1、第2、および第3としてのこの指定は任意であり、そして逆転され得る。しかし、第1のトリプレットが塩基7〜9を、第2のトリプレットが塩基4〜6を、そして第3のトリプレットが塩基1〜3を占めるとして指定することにより、N−C末端配向における3つのフィンガーZFPの第1、第2、および第3のフィンガーは、標的部位の第1、第2および第3のトリプレットに結合する。別の様式で見ると、N末端からC末端へのジンクフィンガータンパク質順序にある、第1、第2および第3のフィンガーは、3 ’−5’配向で並べられた標的部位中の第1、第2および第3のトリプレットにそれぞれ特異的である。
【0087】
次いで、サブスコアを、各トリプレットについて、トリプレットと標的部位内の対応する位置との間の対応規則から決定される。例示の対応規則を表1に提供する。対応規則は、9塩基の標的部位内のその3つの可能な位置にある各トリプレットに対する3つの値を提供するマトリックスである。この表は、64の可能なトリプレットの各々について3つの値を提供する。例えば、潜在的な標的部位5 ’AAA AAG AAC3’を考慮する。AACトリプレットは、標的部位の第1の位置(塩基7〜9)にあり、そして表1から1のサブスコアを割り当てられる。AAGトリプレットは、標的部位の第2の位置(塩基4〜6)にあり、そして8のサブスコアを割り当てられる。AAAトリプレットは、標的部位の第3の位置(塩基1〜3)にあり、そして8のサブスコアを割り当てられる。次いで、潜在的な標的部位中にある3つのトリプレットのサブスコアは、例えば、乗算または加算またはその他の関数により、組み合わせられる。例えば、3つのトリプレットのサブスコアの乗算は、1 ×8×8=64の組み合わせスコアを与える。
【0088】
次いで、このプロセスは、第2の潜在的な標的部位について繰り返される。第2の潜在的な標的部位の3成分トリプレットの各々についてサブスコアが決定され、そして組み合わせスコアが、第2の潜在的な標的部位に対して計算される。次いで、このプロセスは、さらなる潜在的な標的部位について繰り返され得る。必要に応じて、このプロセスは、目的の標的遺伝子のいずれかの鎖中の少なくとも9塩基の可能な連続サブ配列毎に繰り返され得る。目的のすべての可能な標的部位のスコアが決定された場合、スコアが比較される。一般に、高スコアは、ZFPの設計について標的部位の望ましさを示す。高スコアで同定された1つ以上の標的部位が、スコアとともに出力され得る。
【0089】
対応規則における値の指定は、ジンクフィンガータンパク質の設計または選択のために、1つのトリプレットサブ部位を他より望ましくする任意の規準を反映し得る。表1の例示の対応規則における値は、所定のヌクレオチドトリプレットを結合することが知られる先に特徴付けられたZFPの利用可能性を反映する。標的部位の所定の位置における所定のトリプレットについて、所定の位置におけるトリプレットを含む標的セグメントに特異的に結合する、1つ以上の先に特徴付けられたZFPが存在する場合、そのときは、このトリプレットと所定の位置との組み合わせは、スコア10が割り当てられる。所定の位置における所定のトリプレットについて、所定の位置におけるトリプレットを含む標的部位を特異的に結合する、先に特徴付けられたZFPが存在しないが、異なる位置におけるトリプレットに特異的に結合する1つ以上の先に特徴付けられたZFPが存在する場合、そのときは、このトリプレットは、スコア8が割り当てられる。所定のトリプレットおよび所定の位置について、所定の位置または別の位置のいずれかでトリプレットを結合する、先に特徴付けられたZFPが存在しない場合、このトリプレットおよび位置は値1が割り当てられる。
【0090】
これらの値10、8および1は例示に過ぎず、そして他の値が用いられ得る。さらに、より洗練された値の割り当てを用い得、それはまた、他の因子のなかでとりわけ、異なる結合親和性、特異性およびD可能部位の存在を考慮する。このようなスキームでは、強力な結合親和性を有する先のZFPが存在するトリプレットと位置の組み合わせが、代表的には、より低い結合親和性を有する先のZFPが存在するトリプレットと位置との組み合わせより高い値を与える。
【0091】
より大きな配列内の潜在的な標的部位の選択およびスコアの計算は、代表的には、適切にプログラムされたコンピューターにより実施され、それは、1つ以上の潜在的な標的部位を、それらのスコアとともに出力する。必要に応じて、ユーザー入力が、このようなコンピューターに提供され、いくつの潜在的な標的部位が出力されるべきかを特定する。例えば、ユーザーは、最高のスコア出力を持つnの潜在的な標的部位を有するように選択し得る。ここでnは、ユーザーの裁量である。ユーザーはまた、閾値スコアを特定し得、それは、出力されるべき潜在的な標的部位に等しいまたはそれを超過するに違いない。
【0092】
上記の方法の改変法では、潜在的な標的部位は、対応表中の値および1つ以上のD可能サブ部位の存在の両方に基づいて評価され得る。これは、状況パラメーターのユーザー入力により達成され、トリプレットの状況が、D可能サブ部位の存在を示す場合、トリプレットと特定位置との1つ以上の組み合わせに対するスケーリングされたスコアを提供する。例えば、Aが続くトリプレット5 ’NNG3’は、D可能サブ部位を提供しない。しかし、Kが続く5 ’NNG3’は、D可能部位を提供する。ユーザーは、5 ’NNG3’にKが続く場合、5’NNG3 ’トリプレットのサブスコアの値を増大する状況パラメーターを入力することを選択し得る。次いで、このトリプレットに対するスケーリングされたサブスコアを、他のトリプレットに対するサブスコアまたはスケーリングされたサブスコアと組み合わせ、潜在的な標的部位に対する全体スコアを与える。
【0093】
さらなる改変例では、上記の分析を実施するコンピューターは、プログラムされて、互いにそれらの物理的近接度により決定される対で高スコアを受ける特定の標的セグメントを出力する。両方が互いの約5塩基内にある高スコアを受ける対とされた標的セグメントは、各々が3つのフィンガーを有する2つの成分ジンクフィンガータンパク質の連結により形成される6フィンガーのジンクタンパク質の設計に適切な標的である。
【0094】
上記の方法により同定される潜在的な標的部位は、さらなる評価を受け得るか、またはジンクフィンガータンパク質の設計または(必要であれば)選択および産生のために直接用いられ得る。ジンクフィンガータンパク質は、上記のD可能サブ部位を含む潜在的な標的セグメントについて記載された同じ方法を用いて、このような標的部位に対して設計および合成され得る。
【0095】
(V.ZFPのデータベース設計)
本発明は、予め選択された標的部位に対するZFPの設計のための方法を提供する。これらの方法は、上記の標的部位選択の方法と組み合わせるか、または標的部位選択のその他の方法による使用のために適切である。
【0096】
新たなZFPを設計することにおいて、予め特徴付けられたZFPおよびそれらの標的部位中の固有の情報を使用し、それによってデノボ設計または選択のための必要性を最小にすることが一般に有利である。標的部位選択のように、いくつかの因子がこのプロセスに含まれる。標的部位中の各トリプレットサブ部位について、フィンガーが、現存するZFP中で利用可能であるばかりではなく、このようなフィンガーがまた、提案された設計におけるように、この現存するタンパク質中の同じ位置からのそれらの個々のトリプレットサブ部位と接触する場合、設計は促進される。例えば、3つの現存するZFPの対および標的部位を考える:フィンガーF1−F2−F3を持つZFPにより結合される、5 ’GCG TGG GAC3’(ここでF3はGCGと、F2はTGGと、そしてF1はGACと相互作用する)、フィンガーF4−F5−F6を持つZFPにより結合される、5 ’AAG GAG GTG3’、およびフィンガーF7−F8−F9を持つZFPにより結合される5 ’CCG TGA GCA3’、ならびにそれに対してZFPが設計されるべき標的部位5 ’GCG GAG GCA3’である。この状況では、新規タンパク質F7−F5−F3は、この新規タンパク質中の各フィンガーが、それが得られたデータベースタンパク質中でそうであったように、新規タンパク質中で同じ相対的位置中に存在して、5 ’GCG GAG GCA3’に結合する。この設計は、新規ZFP中の各フィンガーの、先のそのZFPの各フィンガーと類似の環境は、このフィンガーが、親におけるように、新規ZFP中で同様の特異性および親和性で結合するらしいことを意味するので有利である。従って、ジンクフィンガータンパク質の結合特徴は、その成分フィンガーの凝集物であるという一般規則は、保持されるようである。
【0097】
新規ジンクフィンガータンパク質はまた、現存するタンパク質で利用可能であるが、設計されるタンパク質中では同じ位置でない、成分フィンガーから設計され得る。例えば、上記の現存するZFP部位対のセットを用いて、タンパク質F3−F7−F5が、配列5 ’GAG GCA GCG3’を結合するように設計され得る。この新規タンパク質において、フィンガーは、それらの個々の親タンパク質中とは異なる位置を占める。所定のフィンガーが、それがZFP中で占めるその位置にかかわらず、そのトリプレット特異性および親和性を保持する近似に対してとはいえ、実際、状況の影響が、フィンガーが異なるジンクフィンガータンパク質中で異なる位置を占める場合、そのトリプレットサブ部位に対するこのフィンガーの特異性および/または親和性における変化を引き起こすようである。従って、先に特徴付けられたZFP中とは異なる位置を占める成分フィンガーから形成されたZFPは、代表的には、なおこの部位に結合するが、特異性または親和性は、時に、予想されるより異なる(代表的にはより低い)。
【0098】
最後に、利用可能である予め存在するフィンガーがないトリプレットを含む予め選択された標的部位には、完全に新規なフィンガーは、規則を基礎にしたアプローチまたはファージディスプレイを用いて設計または選択され得る。
【0099】
本発明は、上記の原理に従って、予め選択された標的部位に対する新規ZFPの設計における現存ZFPについての情報を含むデータベースを系統的に用いる方法を提供する。代表的データベースの構成を表9に示す。代表的には、このデータベースは、予め特徴付けられたZFPのコレクションの各々に対する指定を含む。このZFPは、天然のZFPまたは改変体ZFPであり得る。この指定は、例えば、各ZFPを示す名前またはシンボルであり得る。このデータベースはまた、ZFP中のフィンガーの各々に対するサブ指定を含む。代表的には、このサブ指定は、フィンガー中の選択された位置を占めるアミノ酸残基の形態にある。例えば、表9では、このサブ指定は、従来の番号付けに従って−1位〜+6位を占めるアミノ酸である。このデータベースは、各ジンクフィンガータンパク質により結合される標的核酸セグメントをさらに含む。通常、この核酸セグメントは、3塩基の3つのトリプレットを含む。この塩基の3つのトリプレットは、1つの配列として連結されるか、または別の配列として含められ得る。9塩基の標的部位中の塩基が、5 ’末端から連続的に番号付けされる場合、第1のトリプレットは塩基7〜9を占め、第2のトリプレットは塩基4〜6を占め、そして第3のトリプレットは塩基1〜3を占める。標的セグメント内のトリプレット位置のこの指定に従って、ジンクフィンガータンパク質の第1のフィンガー(すなわち、N−末端に最も近い)は、第1のトリプレットに結合し、第2のフィンガーは、第2のトリプレットに結合し、そして第3のフィンガーは第3のトリプレットに結合する。このデータベースはまた、必須ではないが、その標的部位に対するZFPの結合親和性または解離定数のようなさらなる情報を含み得る。
【0100】
標的部位は、このデータベースを用いてジンクフィンガータンパク質の設計のために提供される。いくつかの方法では、この標的部位は、ユーザー入力により提供される。他の方法では、この標的部位は、上記の標的部位の選択の任意の方法からの出力として提供される。代表的には、この標的部位は、少なくとも3つのトリプレットを形成する少なくとも9つの塩基を含む。この3つの成分トリプレットは、その5塩基 ’を塩基1として割り当てて、標的部位の塩基7〜9、4〜6および1〜3を占める、それぞれ第1、第2および第3のトリプレットと指定される。この標的部位における第1のトリプレットに対して、コンピューターは、このトリプレットに結合するフィンガーを含むジンクフィンガータンパク質についてデータベースを調査する。このコンピューターは、ジンクフィンガータンパク質に関する記録を貯蔵し、そしてそれによって、第1のトリプレットに結合するそれらのフィンガーを同定する。必要に応じて、このコンピューターは、第1のフィンガー位置で標的部位の第1のトリプレットに結合するフィンガーを含むジンクフィンガータンパク質と他の位置における標的部位の第1のトリプレットに結合するフィンガーを含むジンクフィンガータンパク質との間を区別する。そうである場合、このコンピューターは、2つのサブセットのジンクフィンガータンパク質を、別々の記録として蓄える。次いで、このプロセスを、標的部位にある第2のトリプレットについて繰り返す。コンピューターは、第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を同定する。必要に応じて、コンピューターは、現存するジンクフィンガータンパク質の第2の位置から第2のトリプレットを結合するジンクフィンガーまたは異なる位置にある第2のトリプレットを結合するジンクフィンガーとの間を区別する。最後に、コンピューターは、標的部位の第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を同定する。必要に応じて、このコンピューターは、現存するジンクフィンガータンパク質の第3の位置から第3のトリプレットを結合するジンクフィンガーと、または別の位置から第3のトリプレットを結合するジンクフィンガーとの間を区別する。標的セグメント中の、第1、第2および第3のトリプレットの各々に結合するZFPを調査した後、このコンピューターは、同定されたZFPの指定、および第1、第2および第3のトリプレットに結合するフィンガーのサブ指定を出力する。必要に応じて、このコンピューターは、第1のフィンガー位置から第1のトリプレットを結合するZFPのサブセット、および他の位置から第1のトリプレットを結合するZFPのサブセット;および第2のフィンガー位置からおよびその他の位置から第2のトリプレットを結合するZFPの対応するサブセット、および第3のフィンガー位置からおよびその他の位置から第3のトリプレットを結合するZFPの対応するサブセットの別々の出力を提供する。
【0101】
このコンピューターによる情報出力は、予め選択された標的に結合する新規ジンクフィンガータンパク質の設計および合成に用いられ得る。例えば、この出力が、標的の第1のトリプレットを結合するフィンガーXを持つZFP1、標的の第2のトリプレットに結合するフィンガーYを含むZFP2、および標的の第3のトリプレットに結合するフィンガーZを含むZFP3を含む場合、新規ZFPは、この順序(N−末端からC−末端)でフィンガーXYZを含んで合成され得る。このコンピューターが、所定のトリプレットに結合する複数の異なるフィンガーを含む複数の異なるジンクフィンガータンパク質を出力する場合、ユーザーは、フィンガーが、設計されるZFP中におけるのと、データベースタンパク質中の同じ位置からの特定のトリプレット位置に結合するか否かに依存して、フィンガー間を選択し得る。例えば、Xが標的部位中の第1のトリプレットに結合するフィンガーXYZを含むZFP1は、一般に、フィンガーCが標的部位中の第1のトリプレットに結合するフィンガーABCを含むZFP2より好まれる。従って、代表的には、この標的セグメントを結合するために設計されたZFP中の第1のフィンガー位置を占めるために、フィンガーCよりもフィンガーXを用いる。しばしば、コンピュータープログラムは、各々が特定のトリプレットを結合するフィンガーを含む、2つのZFPを同定し、そして各ZFPにおいて、このフィンガーは、それが由来するデータベースタンパク質において、意図された設計ZFP中におけるのと同じ位置を占める。このような場合には、それらの個々の標的に対する結合親和性に基づいて2つのフィンガー間を、しばしば、より高い結合親和性が好適であるとして選択する。必要に応じて、コンピューターはまた、フィンガーにより結合される対応するトリプレットに対する1つ以上のフィンガーに対して提案されるアミノ酸置換の出力を提供する。
【0102】
データベース分析は、3つのフィンガーを有する予め特徴付けられたジンクフィンガータンパク質について主に例示されるが、このようなデータベースは、より少ないかまたはより多い数のフィンガーを持つジンクフィンガータンパク質に関する情報を代わりにまたは付加的に貯蔵し得る。同様に、このようなデータベースは、3つのフィンガーより少ないかまたはより多くを有するジンクフィンガータンパク質の設計に用いられ得る。例えば、本発明のいくつかのデータベースは、2つのフィンガーのみを持つZFPに関する情報およびまたはそれに代わって3つのフィンガーを持つZFPに関する情報を貯蔵する。2つのフィンガーのみを持つZFPは、2つのトリプレットのみを持つ対応する標的部位を有する。2つのフィンガーZFPに関する情報は、上記と本質的に同じ様式で9つの塩基標的部位に結合する3フィンガーZFPの設計に用いられ得る。しかし、2フィンガータンパク質中の2つのフィンガーの相対的位置と、3フィンガージンクフィンガータンパク質中の3つのフィンガーの相対的位置との間には正確な対応はない。この論点は、2つの方法で扱われ得る。第1に、2フィンガータンパク質中のすべてのフィンガーは、3フィンガータンパク質中のフィンガーとは異なる位置を占めるとして効率的に処理され得る。従って、2フィンガータンパク質が、所定のトリプレットに結合するフィンガーを含む場合、コンピューターはこの情報を出力し、そしてこのフィンガーが、データベース2フィンガータンパク質中で、設計される3フィンガータンパク質におけるのと同じ位置に生じないことを示す。あるいは、2フィンガータンパク質中の第1(N末端)のフィンガーは、3フィンガータンパク質中の第1または第2のフィンガーのいずれかと等価であると考慮され得る。2フィンガータンパク質中の第2のフィンガーは、3フィンガータンパク質中の第2または第3のフィンガーのいずれかと等価であると考慮され得る。従って、コンピューターが、それに対してジンクフィンガータンパク質が設計されるべき標的部位中の第1のトリプレットに結合する第1(N末端)のフィンガーを持つ2フィンガータンパク質を同定する場合、コンピューターは、2フィンガータンパク質が、適切なフィンガーと、データベースタンパク質において、設計されるべき3フィンガータンパク質におけるのと同じ位置を供給することを出力し得る。
【0103】
(VII.ZFPの産生)
ZFPポリペプチドおよびこれをコードする核酸は、組換え遺伝学の分野における慣用的な技法を用いて作製され得る。本発明における使用の一般的方法を開示する基礎的テキストは、Samrookら、Molecular Cloning、A Laboratory Manual(第2版,1989);Kriegler、Gene Transfer and Expression:A Laboratory Manual(1990);およびCurrent Protocols in Molecular Biology(Ausubelら編、1994)を含む。さらに、約100塩基より少ない核酸は、The Midland Certified Reagent Company(mcrc@oligos.com)、The Great American Gene Company(http://www.genco.com)、ExpressGen Inc.(www.expressgen.com)、Operon Technologies Inc.(Alameda、CA)のような、任意の種々の市販供給源から注文生産され得る。同様に、ペプチドは、PeptidoGenic(pkim@ccnet.com)、HTI Bio−products、inc.(http://www.htibio.com)、BMA Biomedicals Ltd(U.K.)、Bio.Synthesis、Inc.のような任意の種々の供給源から注文生産され得る。
【0104】
オリゴヌクレオチドは、Beaucage & Caruthers Tetrahedron Letts.22:1859−1862(1981)により最初に記載された固相ホスホルアミダイトトリエステル法に従い、Van Devanterら、Nucleic Acids Res.12:6159−6168(1984)に記載のように、自動化合成器を用いて化学的に合成され得る。オリゴヌクレオチドの精製は、変性ポリアクリルアミドゲル電気泳動によるか、または逆相HPLCによるかのいずれかによる。クローン化遺伝子および合成オリゴヌクレオチドの配列は、クローニング後に、例えば、Wallaceら、Gene 16:21−26(1981)の二本鎖テンプレートを配列決定するための鎖停止方法を用いて確認され得る。
【0105】
代表的には、2つの代替法を用いて、新たに設計されたDNA結合性ペプチドを発現するために必要なコード配列を生成する。1つのプロトコルは、6つの重複オリゴヌクレオチドを利用するPCRを基礎にするアセンブリ手法である(図3)。3つのオリゴヌクレオチド(図3中のオリゴ1、3、および5)は、認識ヘリックスの間のDNA結合ドメインの部分をコードする「ユニバーサル」配列に対応する。代表的には、これらのオリゴヌクレオチドは、すべてのジンクフィンガー構築物について一定のままである。他の3つの「特異的」オリゴヌクレオチド(図3中オリゴ2、4および6)を、認識ヘリックスをコードするために設計する。これらのオリゴヌクレオチドは、認識ヘリックス上の主に位置1、2、3および6における置換を含み、それらを、異なるDNA結合ドメインの各々に対して特異的にする。
【0106】
PCR合成は2段階で実施される。第1に、二本鎖DNAテンプレートを、低温アニーリング工程を伴う4サイクルPCR反応で、6オリゴヌクレオチド(3つのユニバーサル、3つの特異的)を組み合わせることにより生成し、それによってオリゴヌクレオチドをアニーリングし、DNA「足場」を形成する。この足場中のギャップを、高い忠実度の熱安定性ポリメラーゼ(TaqおよびPfuポリメラーゼの組み合わせもまた十分である)によりフィルインする。構築の第2段階では、このジンクフィンガーテンプレートを、シャトルベクター中または直接発現ベクター中へのクローニングのために、いずれかの末端で制限部位を取り込むために設計された外部プライマーにより増幅する。
【0107】
新たに設計されたDNA結合性タンパク質をクローニングする代替法は、所望のZFPの特異的領域をコードする相補的オリゴヌクレオチドをアニーリングすることに依存する。この特定の適用は、オリゴヌクレオチドが、最終の連結工程の前にリン酸化されることを必要とする。通常、これは、アニーリング反応をセットアップする前に実施される。簡単に述べれば、このタンパク質の定常領域をコードする上記「ユニバーサル」オリゴヌクレオチド(上記のオリゴ1、2および3)を、それらの相補的オリゴヌクレオチドとアニールする。さらに、フィンガー認識ヘリックスをコードする上記「特異的」オリゴヌクレオチドを、それらの個々の相補的オリゴヌクレオチドとアニールする。これらの相補的オリゴは、上記で述べたプロトコルでポリメラーゼによって先にフィルインされた領域をフィルインするために設計される。共通オリゴ1およびフィンガー3に相補的なオリゴは加工されて、次の工程で選択されたベクター中へのクローニングにおいて用いられる制限部位に特異的なオーバーハング配列を残す。第2のアセンブリプロトコルは、最初のプロトコルと、次の局面で異なる:新たに設計されたZFPをコードする上記「足場」は、完全に合成DNAからなり、それによって、ポリメラーゼフィルイン工程をなくし、さらに、ベクター中にクローニングされるフラグメントは増幅を必要としない。最後に、配列特異的オーバーハングを残す設計は、挿入フラグメントの制限酵素消化の必要性をなくする。あるいは、ZFP認識ヘリックスへの変化は、従来の部位特異的変異誘発方法を用いて作製され得る。
【0108】
両方のアセンブリ方法は、新たに設計されたZFPをコードする得られるフラグメントが、ベクター中に連結されることを必要とする。最終的に、ZFPをコードする配列は、発現ベクター中にクローニングされる。一般に利用される発現ベクターは、改変pMAL−c2細菌発現ベクター(New England BioLabs)または真核生物発現ベクター、pcDNA(Promega)を含むがこれらに限定されない。最終構築物は、配列分析により確認される。
【0109】
当業者に公知の任意の適切なタンパク質精製方法を用いて、本発明のZFPを精製し得る(Ausubel、前述、Sambrook、前述を参照のこと)。さらに、任意の適切な宿主、例えば、細菌細胞、昆虫細胞、酵母細胞、哺乳動物細胞などを発現のために使用し得る。
【0110】
細菌株JM109における、マルトース結合性タンパク質に融合したジンクフィンガータンパク質(MBP−ZFP)の発現は、アミロースカラム(NEB)による直接精製を可能にする。このジンクフィンガーキメラタンパク質の高い発現レベルは、pMal−c2発現プラスミド中のこのMBP−ZFP融合が、tacプロモーター(NEB)の制御下にあるので、IPTGを用いた誘導により得られ得る。MBP−ZFP融合プラスミドを含む細菌を、10 μM ZnCl2、0.02%グルコース、および50 μg/mlアンピシリンを含む2×YT培地中に接種し、そして37 ℃で振盪する。対数増殖の中央でIPTGを0.3mM添加し、そしてこの培養物を振盪した。3時間後、細菌を遠心分離により回収し、音波処理によるか、またはフレンチプレスセルを通過させることによるかまたはリゾチームの使用により破壊し、そして不溶性物質を、遠心分離により取り除いた。MBP−ZFPタンパク質を、アミロース結合樹脂上に捕獲し、20mM Tris−HCl(pH7.5)、200mM NaCl、5mM DTTおよび50μM ZnCl 2を含む緩衝液で大規模に洗浄し、次いで、本質的に同じ緩衝液中のマルトースで溶出した(精製は、NEBからの標準的なプロトコールに基づく)。精製タンパク質を定量し、そして生化学的分析のために貯蔵した。
【0111】
精製したタンパク質の解離定数(例えば、Kd)は、代表的には、電気泳動移動度シフトアッセイ(EMSA)(BuratowskiおよびChodosh、Current Protocols in Molecular Biology、12.2.1.−12.2.7頁(Ausubel編、1996))を介して特徴付けられる。親和性は、固定量の標識した二本鎖オリゴヌクレオチド標的に対して、精製したタンパク質を力価決定することによって測定される。この標的は、代表的に、天然配列に見出される3bpおよびさらなる定常の隣接配列に隣接する天然の結合部位配列を含む。天然結合部位は、代表的に、3フィンガータンパク質について9bpであり、6フィンガーZFPについては、2 ×9bp+介在塩基である。アニールしたオリゴヌクレオチド標的は、1塩基の5’オーバーハングを有し、これによって、T4ファージポリヌクレオチドキナーゼを用いてその標的の効率よい標識が可能となる。そのアッセイについて、その標的を、1nM以下の濃度で添加し(実際の濃度を、予測解離定数のものから少なくとも10分の1に維持する)、精製したZFPを、種々の濃度で添加し、そしてその反応物を少なくとも45分間平衡化させた。さらに、反応混合物はまた、10mM Tris(pH7.5)、100mM KCl、1mM MgCl2、0.1mM ZnCl 2、5mM DTT、10%グリセロール、0.02% BSAを含む。(註:初期のアッセイにおいては、ポリd(IC)もまた、10〜100 μg/μlで加えた)。
【0112】
平衡化した反応物を、Tris/グリシン緩衝液において45分間前泳動した10%ポリアクリルアミドゲルにロードし、次いで、結合した標識標的および結合しない標識標的を、150Vでの電気泳動によって分離した。(あるいは、4%のポリアクリルアミドスタッカーを含む10%〜20%の勾配Tris−HClゲルが使用され得る)。乾燥させたゲルを、オートラジオグラフィーまたはホスホルイメージによって可視化し、そして見かけ上のKdを、最大の半分の結合を与えるタンパク質濃度を計算することによって決定した。
【0113】
このアッセイはまた、タンパク質調製物における活性画分を決定することを含み得る。活性画分を、化学量論的なゲルシフトによって決定する。ここで、タンパク質を、高い濃度の標的DNAに対して力価決定する。力価決定を、100%、50%および25%の標的で行う(通常、マイクロモル濃度レベル)。
【0114】
(IX.設計したZFPの適用)
特定の標的遺伝子に結合するZFP、およびそれらをコードする核酸を、種々の適用について使用し得る。これらの適用は、ZFPまたはそれをコードする核酸を被験体に投与し、そしてそれを使用して、その被験体内の標的遺伝子の発現を調節する治療方法を含む(同時係属中の出願Townsend & Townsend & Crew 代理人整理番号019496−002200、1998年1月12日出願を参照のこと)。この調節は、例えば、その標的遺伝子が病原性の感染性微生物中、または疾患状態に寄与している患者の内因性遺伝子(例えば、ガン遺伝子またはウイルスレセプター)に存在する場合は、抑制の形態であり得る。あるいは、この調節は、内因性細胞遺伝子の発現の活性化または発現の増加が疾患状態を改善し得る場合は、活性化の形態であり得る。そのような適用について、ZFPまたはより代表的にはそれをコードする核酸は、薬学的に受容可能なキャリアとともに、薬学的組成物として処方される。
【0115】
薬学的に受容可能なキャリアは、投与される特定の組成物、およびその組成物を投与するために使用される特定の方法によって、部分的に決定される(例えば、Remington ’s Pharmaceutical Sciences、第17版、1985を参照のこと)。ZFPは、単独または他の適切な成分との組み合わせで、エアロゾル処方物が作製されて(すなわち、それらは、「噴霧化」され得て)、吸入を介して投与され得る。エアロゾル処方物を、加圧した受容可能な噴霧剤(例えば、ジクロロジフルオロメタン、プロパン、窒素など)中に入れ得る。非経口投与(例えば、静脈内、筋肉内、皮内、および皮下の経路)に適した処方物としては、水性および非水性の、等張性の無菌注射溶液(これは、抗酸化剤、緩衝液、静菌剤、およびその処方物を意図するレシピエントの血液と等張にさせる溶質を含み得る)および懸濁剤、可溶化剤、濃稠化剤、安定化剤および保存剤を含み得る水性および非水性の、無菌懸濁物が挙げられる。組成物は、例えば、静脈注入により、経口的、局所的、腹腔内的、嚢内的または髄腔内的に投与され得る。化合物の処方物は、単位投薬形態または多数回の封入された容器中に(例えば、アンプルまたはバイアル)提示され得る。注射溶液および懸濁液は、以前に記載されている種類の無菌の粉末、顆粒および錠剤から調製され得る。
【0116】
患者に投与される用量は、経時的にその患者における利益のある治療的応答をもたらすに充分であるべきである。この用量は、使用する特定のZFPの効力およびK d、標的細胞およびその患者の状態ならびに処置される患者の体重または表面積によって決定される。用量の大きさもまた、特定の患者における特定の化合物またはベクターの投与に伴う任意の有害な副作用の存在、性質および程度によって決定される。
【0117】
他の適用において、ZFPは、サンプルにおける標的核酸の配列特異的な検出のための診断方法において使用される。例えば、ZFPを使用して、患者のサンプルにおける疾患または表現型と関連する改変体対立遺伝子を検出し得る。例として、ZFPを使用して、mRNAまたはcDNAの複合混合物における特定のmRNA種またはcDNAの存在を検出し得る。さらなる例として、ZFPを使用して、サンプルにおける遺伝子のコピー数を定量し得る。例えば、臨床サンプルにおける1コピーのp53遺伝子の欠失の検出は、ガンに対する感受性の指標である。さらなる例において、ZFPを使用して、臨床サンプルにおける病原性微生物の存在を検出する。これは、検出される微生物内の遺伝子に特異的な1以上のZFPを用いて達成される。診断アッセイを実施するための適切な形式は、ELISAプレートにおいてZFPの固定を可能にするドメインに連結されたZFPを使用する。この固定されたZFPを、結合が生じ得る条件下で標的核酸を含むと疑われるサンプルと接触させる。代表的には、サンプル中の核酸を標識する(例えば、PCR増幅の過程において)。あるいは、標識されていないプローブを、第二の標識したプローブを用いて検出し得る。洗浄後、結合した、標識された核酸を検出する。
【0118】
ZFPはまた、遺伝子発現の表現型および機能を決定するためのアッセイのために使用され得る。遺伝子機能の決定のための現在の方法論は、主に、天然の生物学的設定からの目的の遺伝子の過剰発現または除去(完全にノックアウトすること)にのいずれかおよびその効果を観察することに依存する。観察される表現型の効果は、生物学的系における遺伝子の役割を示す。
【0119】
従来のノックアウト分析に対する遺伝子のZFP媒介調節の1つの利点は、ZFPの発現が低分子のコントロールの下に置かれ得るということである。次いでZFPの発現レベルを制御することによって、ZFPによって制御される遺伝子の発現レベルを制御して、所定の表現型効果または生化学的効果を達成するのに必要とされる発現の抑制または刺激の程度を決定し得る。このアプローチは、薬物開発について特に価値がある。ZFPを低分子の制御下に置くことによって、胚性の致死性および発生補償の問題は、マウスの発生後期においてZFPリプレッサーにスイッチを入れること、および成体動物での効果を観察することによって回避され得る。ZFPによって調節される標的遺伝子を有するトランスジェニックマウスは、その標的遺伝子に対してイントランスの任意の部位にZFPをコードする核酸を組み込むことによって、作製され得る。したがって、相同組換えは、その核酸の組込みには必要ではない。さらに、ZFPがトランス優性であることから、1つの染色体コピーのみが必要であり、それゆえ、機能的ノックアウト動物は、戻し交配することなく作製され得る。
【0120】
(X.コンピュータシステムおよびプログラム)
図4は、本発明を実行するのに適切な代表的なコンピュータシステムを示す。図4は、本発明での使用に適切なコンピュータシステム10の基本的なサブシステムを示す。図4において、コンピュータ10は、バス12を備える。このバスは、中央プロセッサ14、システムメモリ16、入力/出力コントローラ18、外部デバイス(例えば、パラレルポート22を介したプリンタ20)、ディスプレーアダプタ26を介したディスプレースクリーン24、シリアルポート28、キーボード30、固定ディスクドライブ32およびフロッピディスク33Aを作動可能に受け入れるためのフロッピディスクドライブ33)のような主要なサブシステムを相互接続する。多くの他のデバイスが、接続可能である(例えば、I/Oコントローラ18を介したスキャナ60(示さず)、シリアルポート28に接続されたマウス36、またはネットワークインタフェイス40)。他の多くのデバイスまたはサブシステム(示さず)は、類似の様式で接続され得る。また、図4に示されるデバイスの全てが本発明を実行するに存在することは、下記で議論するように必要ではない。これらのデバイスおよびサブシステムは、図4に示すものとは異なる方法において、相互接続され得る。図4に示されるもののようなコンピュータシステムの操作は、当該分野で容易に公知であり、そして本願においては詳細には議論しない。本発明を実行するためのソースコードは、システムメモリ16に作動可能に配置され得るか、または固定ディスク32またはフロッピディスク33Aのような記憶媒体上に格納され得る。
【0121】
図5は、本発明の方法を具現化するに適切な図4の代表的なコンピュータシステム10の例示である。図5は、本発明とともに用いられ得る多くの可能なコンピュータタイプまたは構成のほんの一例を示す。図5は、ディスプレースクリーン24、キャビネット20、キーボード30、スキャナ60、およびマウス36を備えるコンピュータシステム10を示す。マウス36およびキーボード30は、「ユーザー入力デバイス」を例示する。ユーザー入力デバイスの他の例は、タッチスクリーン、ライトペン、トラックボール、データグローブなどである。
【0122】
好ましい実施態様において、システム10は、Pentium(登録商標)クラスに基づくコンピュータを含み、このコンピュータは、Microsoft CorporationによるWindows(登録商標)バージョン3.1、Windows95(登録商標)またはWindows98(登録商標)オペレーティングシステムを実行する。しかし、この方法は、本発明の範囲を逸脱することなく、他のオペレーティングシステムに容易に適用される。
【0123】
マウス36は、ボタン37のような1以上のボタンを有し得る。キャビネット20は、ディスクドライブ33、プロセッサ、記憶媒体などのような慣用のコンピュータ構成要素を収容する。本明細書において使用される場合、「記憶手段」には、コンピュータシステムと接続して使用される任意の記憶デバイスが含まれる(例えば、ディスクドライブ、磁気テープ、半導体メモリ、バブルメモリなど)。キャビネット20は、スキャナ60、外部記憶装置、他のコンピュータまたはさらなる周辺機器のような外部デバイスへコンピュータシステム10を接続するための入力/出力(I/O)インターフェイス18のようなさらなるハードウェアを備え得る。図5は、本発明を具現化するためのシステムのほんの一つのタイプの代表である。他の多くのシステムのタイプおよび構成が、本発明と組み合せた使用について適切である。
【0124】
図6は、ジンクフィンガータンパク質によって標的化するための標的配列内のD可能サブ部位を含む標的部位を選択するための代表的な実施態様における単純化された工程のフローチャート301を示す。工程302において、ジンクフィンガータンパク質によって標的化される標的配列が提供される。次いで、工程303において、その標的配列内の潜在的な標的部位を、評価のために選択する。決定工程304において、潜在的な標的部位を評価して、D可能サブ部位を含むか否かを決定する。そのような標的部位は、以下の式に従う:5 ’NNx aNy bNzc3’、ここで、 (x、a)、(y、b)および(z、c)の各々は、(N,N)または(G,K)であり;
(x、a)、(y、b)および(z、c)の少なくとも1つは、(G,K)であり、そして NおよびKは、IUPAC−IUBの多義性コードである。
【0125】
潜在的な標的部位は、D可能サブ部位を含む場合、潜在的な標的部位は、205における記録として格納される。この方法は、さらなる決定工程306を継続する。さらなる潜在的な標的部位の評価は、ユーザーによって要求される場合、その方法のさらなる反復は、303から出発して実施する。充分な潜在的標的部位が、すでに評価されている場合、工程305において格納された標的部位の記録を工程307において出力する。
【0126】
図7Aは、ジンクフィンガータンパク質によって標的化するためのポリヌクレオチド内の標的部位を選択するための別の代表的な実施態様において単純化された工程のフローチャートを示す。工程402において、ポリヌクレオチド標的配列は、分析のために提供される。次いで、工程404において、そのポリヌクレオチド配列内の潜在的な標的部位が選択される。この潜在的な標的部位は第一、第二および第三の塩基トリプレットを、その潜在的な標的部位における第一、第二および第三の位置にて含む。次いで、工程406において、複数のサブスコアを、トリプレットとトリプレット位との間の対応規則を適用することによって決定する。ここで、各トリプレットは、第一、第二および第三の対応する位置を有し、そして各対応するトリプレットおよび位置には、特定のサブスコアを割り当てる。次に、必要に応じて、決定工程408が存在する。この工程において、そのユーザーは、工程410におけるスケーリング計数を用いて1つ以上のサブスコアをスケーリングするように選択し得る。その後、工程412において、スコアを、第一、第二および第三のトリプレットについて、サブスコアから決定する(適切にスケーリングする)。次いで、決定工程414において、なんらかのさらなる潜在的な標的部位が試験されるべきか否かを決定するためのチェックを行う。そうである場合、工程404での処理を継続する。そうでなければ、工程416において、潜在的な標的部位およびそのスコアの少なくとも1つを出力として提供する。
【0127】
図7Bは、ジンクフィンガータンパク質を産生するための代表的な実施態様における単純化された工程のフローチャートを示す。工程450において、複数のジンクフィンガータンパク質についての指定を含むデータベースが提供される。このデータベースにおける各タンパク質は、少なくとも第一、第二および第三のフィンガーを含む。このデータベースは、そのジンクフィンガータンパク質の各々の3つのフィンガーの各々についてのサブ指定、および各ジンクフィンガータンパク質について対応する核酸配列をさらに含む。各配列は、少なくとも第一、第二および第三のトリプレットを含む。これらのトリプレットは、各ジンクフィンガータンパク質において、それぞれ、少なくとも第一、第二および第三のフィンガーによって、特異的に結合される。この第一、第二、および第三のトリプレットは、それぞれ同じ順序(3 ’−5’)で核酸配列においてある配置を有する。なぜなら、第一、第二および第三のフィンガーは、ジンクフィンガータンパク質において配列されているからである(N末端からC末端)。
【0128】
工程452において、少なくとも第一、第二および第三のトリプレットを含むジンクフィンガータンパク質の設計のための標的部位が提供される。次いで、工程454において、その標的配列において第一のトリプレットに結合するフィンガーを有するジンクフィンガータンパク質の第一のセットが同定される。次に、454において決定されるセットの第一および第二のサブセットを同定する任意の工程456が続く。この第一のサブセットは、そのジンクフィンガータンパク質における第一のフィンガー位から第一のトリプレットを結合するフィンガーを有するジンクフィンガータンパク質を含む。第二のサブセットは、そのジンクフィンガータンパク質において第一のフィンガー位以外からの第一のトリプレットを結合するフィンガーを有するジンクフィンガータンパク質を含む。この方法は、工程458に続く。この工程において、ジンクフィンガータンパク質のさらなるセットが同定され、このセットは、その標的部位における第二のトリプレットに結合するフィンガーを含む。この工程の後に、工程458において同定されるセットの第一および第二のサブセットを同定する任意の工程460が続く。この第一のサブセットは、ジンクフィンガータンパク質内の第二の位置からの第二のトリプレットに結合するジンクフィンガータンパク質を含む。この第二のサブセットは、ジンクフィンガータンパク質の第二の位置以外からの第二のトリプレットを結合するジンクフィンガータンパク質を含む。この方法は、工程462に続く。462において、その標的部位の第三のトリプレットに結合するフィンガーを含む1セットのジンクフィンガータンパク質を同定する。任意の工程464において、工程462において同定されたセットの第一および第二のサブセットを、同定する。第一のサブセットは、ジンクフィンガータンパク質の第三のフィンガー位からの第三のトリプレットに結合するフィンガーを含むジンクフィンガータンパク質を含む。第二のサブセットは、ジンクフィンガータンパク質の第三のフィンガー位以外からの第三のトリプレットに結合するフィンガーを含むジンクフィンガータンパク質を含む。この方法は、工程466に続く。この工程では、工程454、458および462において同定されたジンクフィンガータンパク質のセットが別々に出力される。さらに任意の工程468が存在する。ここで、工程460、464および468において同定されたジンクフィンガータンパク質の第一および第二のサブセットが出力される。
【0129】
図8Aは、Entity Representation Diagram(ERD)へのキーである。このダイヤグラムを使用して、ZFPデータベースの内容を記述する。代表的な表502は、1以上のキー属性504および1以上のキーではない(ノンキー)属性506を含む。代表的な表502は、1以上のレコードを含み、ここで、各レコードは、列挙した属性に対応するフィールドを含む。キーフィールドの内容を一緒にして、個々のレコードを同定する。ERDにおいて、各表は、水平線によって分割された長方形によって表される。その線より上のフィールドまたは属性は、キーであるが、他方、その線より下のフィールドまたは属性は、キーではないフィールドである。同定する関係508は、親の表510のキー属性もまた、子供の表512のキー属性であることを示す。同定しない関係514は、親の表516のキー属性もまた、子供の表518のキーではない属性であることを示す。(FK)が括弧内に現れる場合は、それは、1つの表の属性が、別の表のキー属性であることを示す。同定しない関係および同定する関係の両方について、親の表における1つのレコードは、子供の表における1以上のレコードに対応する。
【0130】
図8Bは、本発明の特定の実施態様に従った代表的なZFPデータベース550を示す。データベース550は、代表的には、予め特徴付けたZFPの収集物の各々についての指定を含み得る。このZFPは、天然のZFPまたは改変体ZFPであり得る。この指定は、例えば、各ZFPを代表する名称またはシンボルであり得る。例えば、図8Bにおけるデータベース550のZFP552は、「ZFP001」と指定される。データベース550はまた、ZFPにおけるフィンガーの各々についてのサブ指定(例えば、サブ指定554、ZFP001 552のフィンガー1)を含む。代表的には、このサブ指定は、フィンガーにおいて選択された位置を占有するアミノ酸残基の形態である。さらに、ZFPは、従来の番号付けに従って−1位から+6位までを占有するアミノ酸であるサブ指定を有する。このデータベースはさらに、各ジンクフィンガータンパク質によって結合される標的核酸セグメントを含み得る。この核酸セグメントは通常、3つの塩基の3つのトリプレットを含む。塩基の3つのトリプレットは、1つの配列として共同で含まれ得るか、または別個の配列として含まれ得る。9塩基の標的部位における塩基が5 ’末端から連続的に番号付けされる場合、第一のトリプレットは、塩基7〜9を占有し、第二のトリプレットは、塩基4〜6を占有し、そして第三のトリプレットは、塩基1〜3を占有する。標的セグメント内のトリプレット位のこの指定に従って、ジンクフィンガータンパク質の第一のフィンガー(すなわち、N末端に最も近い)は、第一のトリプレットに結合し、第二のフィンガーは、第二のトリプレットに結合し、そして第三のフィンガーは第三のトリプレットに結合する。このデータベースはまた、その標的部位についてZFPの結合親和定数または解離定数のようなさらなる情報を含み得る。しかし、そのようなものは必須ではない。さらにデータベース550は、本発明の範囲を逸脱することなく、図8Bに示される以外のZFP、フィンガーおよび核酸の間の他の配置および関係を含み得る。
【実施例】
【0131】
(実施例1:DNAモチーフについての検索プロトコル)
本実施例は、標的セグメントがより長い遺伝子からどのように選択されるかを例示する。検索手順を、検索プロトコルにおける1以上のDNA配列モチーフを特定することを可能にするコンピュータプログラムを用いて実行する。通常の手順は、遺伝子またはcDNAのDNA配列を入力すること、次いで、異なるモチーフについて、最も所望されるものから最も所望されないものまで、複数回配列を検索することである。従って、下記に列挙した例示的プロトコルのなかで、代表的に、プロトコル1をまず実行し、そしてそれが潜在的な標的セグメントの適切な数を得られない場合は、プロトコル2を試行し、そして以下同様とする。
【0132】
プロトコル1は、各々9塩基または10塩基の2つの別個のセグメントから形成された標的部位について標的遺伝子を検索する。2つのセグメントは、0〜3の介入塩基によって分離され得る。各セグメントは、NNGG形態(ボールドで示す)のD可能サブ部位を含む。セグメント内の3塩基のサブ部位の各々は、Gで始まる。この分析によって同定された標的部位は、ZFP設計について直接使用され得るか、またはさらなる分析に供され得、例えば、標的セグメントがさらなるD可能サブ部位を有するものを同定し得る。それぞれ10塩基からなる2つのセグメントから形成された標的部位において、合計で6つのD可能サブ部位が存在し得る。以下の全ての標的部位を、5 ’から3’で示し、そして指定「0,3」は、任意のタイプの0〜3のヌクレオチドが存在し得ることを示す。
【0133】
【数5】

【0134】
プロトコル2は、標的遺伝子内の標的部位を評価するための第二の手順である。この手順は、再度、それぞれ9塩基または10塩基の2つのセグメントから形成される標的部位について検索する。各セグメントは、形態KNGGの少なくとも1つのD可能サブ部位を含む。プロトコル2は、プロトコル1とは、プロトコル2が、3塩基サブ部位がGで始まることを必要としない点において異なる。プロトコル2においてはむしろ、3塩基サブ部位はGまたはTのいずれか(IUBPAC−IUBの多義性コードにおいてはK)で始まる。標的部位は、5 ’から3’で示し、そしてシンボル(0,3)および(0,2)は、それぞれ、0〜3塩基および0〜2塩基の介入セグメントを示す。
【0135】
【数6】

【0136】
プロトコル3は、そのプロトコル3がKNGGまたはKNGTのいずれかのD可能サブ部位を用いて標的部位を選択することを除いて、プロトコル2と同じである。標的部位を、5 ’から3’で示す。
【0137】
【数7】

【0138】
プロトコル4は、上記のプロトコルのいずれよりもより一般的であり、そして標的部位がD可能サブ部位を含むことを必要としない。プロトコル4は、互いに0〜3塩基内に、形態GNN GNN GNNのそれぞれが9塩基である2つのセグメントを同様に必要とする。
【0139】
プロトコル5は、それが、互いに0〜3塩基内で、式5’KNN KNN KNN3 ’の2つの標的セグメントから形成された標的部位について、検索することを除いてプロトコル4と同じである。
【0140】
(実施例2)
本実施例は、少なくとも1つのD可能サブ部位を含む標的セグメントに結合するジンクフィンガータンパク質が、一般的に、そのZFPが+2位にD残基を有する場合、ジンクフィンガータンパク質結合より高い親和性を伴って、D可能サブ部位を欠く標的セグメントへ結合することを例示する。53のZFP(各々は、3つのフィンガーを有する)を、結合親和性またはD可能サブ部位への結合に関係なく収集物から選択した。選択したZFPの解離定数を、いずれかの側に標的配列由来の少なくとも1つの隣接塩基を有するZFPの3つのフィンガーによってそれぞれ結合した3つの連続的なヌクレオチドトリプレットを含む標的セグメントへのZFPの結合によって決定した。すべてのZFPは、ヒトSp1フレームワークを有した。これらの53のZFPの結合親和性を、表2におけるKd値として列挙される任意の4群に分けた。
【0141】
【表2】

【0142】
この分類に従うと、これらのタンパク質の約25%(14/53)のみが、それらのそれぞれの標的について、高親和性(100nM以下のKd)を有した。これらの14のタンパク質のうち、すべてが少なくとも1つのD可能サブ部位をその標的内に有していた。
【0143】
(実施例3)
本発明者らは、ダイズ(Glycine max)FAD2−1 cDNAの配列を、対合した近位の9塩基標的セグメントについて、プロトコル2およびプロトコル3を用いて検索した。5つの標的セグメントを選択し、そして1または2のいずれかのZFPを、その標的の各々に結合するように設計した。選択した標的およびそれぞれの設計したZFPについてのKd値を、表3に示す。D可能サブ部位をボールドで示す。配列を5 ’から3’にむけて示す。
【0144】
【表3】

【0145】
作製した8つのZFPのうち、すべてが高親和性(100nM以下のKd)で、それらの標的に結合した。このことは、9bpの標的内にD可能サブ部位を有する標的を選択することによって、高親和性ZFPを効率よく設計することが可能であることを示す。さらに、2つのD可能サブ部位を有する標的部位へ結合するZFPのすべては、1つのみのD可能サブ部位を有する標的部位へ結合するZFPよりも強力に結合した。
【0146】
(実施例4)
本実施例は、D可能サブ部位が高親和性を付与する証拠をさらに提供する。53の標的セグメントを上記のプロトコル5によって同定した。このプロトコルは、D可能サブ部位が標的部位に存在することを必要としない。53のZFPを、これらのそれぞれの部位に結合するように設計した。33の標的セグメントを、上記のプロトコル3によって同定した。このプロトコルは、D可能サブ部位を必要とする。そして、33のZFPを、これらのそれぞれの部位に結合するように設計した。表4は、異なる手順によって設計されるZFPのKdを比較する。
【0147】
【表4】

【0148】
表4は、プロトコル3によって設計された33のZFPのうち、31が高親和性(100nM未満のKd)を有することを示す。対照的に、プロトコル5によって設計したZFPのうち14のみが高結合親和性を有する。これらのデータは、高親和性ZFP(Kd<100nM)は、検索プロトコルがD可能サブ部位を必要としない場合よりも、その検索プロトコルがD可能サブ部位の基準を含む場合に、標的についてより効率よく設計され得ることを示す。
【0149】
(実施例5)
ZFPの親和性と、標的における1以上のD可能サブ部位の存在との間の関係を、殆どが異なる標的部位に対して特異的な約300の設計したZFPについて分析した。この分析および続く分析において、1つのZFPのみが、1つの標的部位あたりに含まれる。これは最も高い親和性を伴うZFPである。
【0150】
表5および図1は、結合した9塩基の標的部位におけるD可能サブ部位の数およびタイプによって分類された異なるカテゴリーのZFPの平均Kdを示す。表4において、および後の表6、表7および表8において、s.e.m.は、平均の標準誤差を示し、そしてnは試験したタンパク質の数を示す。
【0151】
【表5】

【0152】
2つのGGタイプのD可能サブ部位を有する標的に対して設計された22のZFPは、平均Kd=15nMを伴い、最高の結合親和性を有する。100nM未満のKdを有する50のZFPのうち、49が、少なくとも1つのD可能サブ部位を有する。この表は、以下の結論を示す:(1)1つのD可能サブ部位を伴う標的部位への結合は、D可能サブ部位を欠く標的部位へのZFPの結合よりもさらに強力に結合する;(2)2つのD可能サブ部位を伴う標的部位へ結合するZFPは、1つのD可能サブ部位を伴う標的部位へ結合するZFPよりもさらに強力に結合する、および(3)GG D可能サブ部位を伴う標的部位を有するZFPは、GT D可能サブ部位を伴う標的部位を有するZFPよりもさらに強力に結合する。
【0153】
(実施例6)
設計したZFPの結合親和性に影響を与える別の因子は、標的部位が、KNN KNN KNNよりむしろGNN GNN GNNの形態を有するか否かである。本実施例は、D可能サブ部位が、高度の結合親和性を、GNN GNN GNNモチーフの状況においてさえも、与えることを示す。この分析のために、本発明者らは、59のZFPの集団を選択した。これらの各々は、形態GNN GNN GNNの異なる標的部位に結合する。表6は、設計されたZFPのKd値を、GNN GNN GNN標的を有するD可能サブ部位の存在の関数として示す。
【0154】
【表6】

【0155】
D可能サブ部位の存在は、その標的がGNN GNN GNNモチーフに適合する場合でさえも、ZFPの結合親和性に強力に影響を与える。
【0156】
(実施例7)
本実施例は、D可能サブ部位の増加した結合親和性を付与するという点に対する効果が、他の残基に対してより高い結合親和性の付与におけるG残基の任意の効果について相加的であるというさらなる証拠を提供する。この分析について、本発明者らは、101のジンクフィンガータンパク質の、本発明者らの収集物からの異なる標的部位への結合を選択し、そして存在するG残基の数によってこれらの標的部位を分類した。これらの標的部位は、2〜8のG残基を、9塩基配列中に含んでいた。表7は、概して、標的部位に存在するG残基が増えれば、その部位についてのZFPの結合親和性も強くなることを示す。
【0157】
【表7】

【0158】
本発明者らは、D可能サブ部位の存在または非存在が設計したZFPの平均Kd値に影響を与えたか否かを問い掛けることによってさらにこれらのデータを分析した。表7からの9塩基標的の各カテゴリーを、D可能サブ部位を含む標的またはそれを含まない標的へと細分した。この分析の結果を表8に示す。
【0159】
【表8】

【0160】
この表は、同じ数のG残基を有するが、異なる数のD可能サブ部位を有する標的部位を比較した場合、D可能サブ部位を含む部位がより高い結合親和性を付与することを示す。4つ以上のGを有する9塩基の標的部位について、平均Kdは、その標的が少なくとも1つのD可能サブ部位を有する場合、約100nM以下である。特に顕著なのは、5つのG残基を有する標的部位間の比較である。D可能サブ部位を欠く5つのそのような標的は、640nMの平均Kdを有した。2つのD可能サブ部位を有する23のそのような標的部位は、98nMの平均Kdを有した。
【0161】
(実施例10:ZFP予測モジュール)
本実施例は、対応規制を用いた、標的遺伝子内の標的セグメントの選択、および選択した標的セグメントに結合するZFPを設計するためのデータベースの使用を例示する。ZFP Prediction Moduleは、(i)目的のDNA配列、(ii)種々のデータ表、(iii)設計パラメータおよび(iv)出力パラメータを入力として採用すること、ならびに目的の配列における潜在的なZFP標的部位のリストおよび各標的部位におけるサブ部位に対して設計されたフィンガーのまとめを出力として提供することによって、部位の選択およびZFP設計プロセスの両方を容易にする。この節は、プログラムの入力、出力およびそのプログラムについてのプロトコルをスコア付けすることを記載する。明確さのために、その記述を部位選択および設計機能へと分割する。
【0162】
(1.目的のDNA領域内の標的部位の選択)
(入力)
1)標的DNA配列
2)9bp標的部位における可能な3塩基対サブ部位およびその3つの可能な位置についてのスコアの各々を列挙するスコア表を、図1に示す。そのスコア表は、実行時にユーザーによって提供され、そしてカスタマイズされ得、そしてジンクフィンガーモチーフのDNA配列優先性についてのユーザーの最新の理解を反映するようにアップデートされ得る。
【0163】
3)既存の高親和性ZFPについて、標的部位、アミノ酸配列および参照データを含む「ZFPデータ表」。この表は、出力パラメータ(ii)が下記において選択される場合にのみそのプログラムのこの部分について必要とされる。ZFPデータ表の例を表9に提供する。
【0164】
4)任意の状況パラメータ−実行時にユーザーによって入力される「『D可能』トリプレットについての増強因子」。このパラメータは、3 ’GまたはTが隣接する任意の「xxG」サブ部位についてのスコアに増強因子を乗じる。
【0165】
5)出力パラメータ(ユーザーによって供給される)は、以下を特定する: i)その出力において含まれる標的部位の数 ii)そのプログラムが、(存在する場合)3つのフィンガータンパク質がすでに設計されているその標的部位を特に強調すべきか否か iii)そのプログラムが、入力標的配列におけるその相対位置に従って、出力標的部位を再整列すべきか否か iv)そのプログラムが、9bp DNA部位の標的化可能な対を強調すべきか否か(隣接し、重複しない、n以下の塩基だけ分離されている部位の対、ここで、nは、代表的には、5、4、3、2または1である)。
【0166】
出力:スコアによって階級付けされた標的DNA配列における潜在的な標的部位のセット。
【0167】
特定されている場合、3フィンガータンパク質がすでに設計されている任意の標的部位のリスト。
【0168】
特定されている場合、入力配列における配置に従って、再整列された出力部位のリスト。
【0169】
特定されている場合、9bpDNA部位のすべての標的化可能な対のリスト。
【0170】
そのプログラムの部位選択部分は、所定の標的DNAフラグメントにおいて、すべての可能な9bp配列に対してスコアを割り当てる。このスコアは、先に設計したジンクフィンガータンパク質からの情報を使用することに基づいて標的化可能性の容易さを反映する。所定の9塩基配列を評価するにおいて、そのプログラムは、まず、その標的を、その成分サブ部位へと分割し、次いで、そのスコア表を参考にして潜在的な標的部位におけるその位置での各サブ部位についてのスコアを入手する。最後に、これにサブ部位スコアを乗じて、9bp標的部位についての全体スコアを得る。例えば、試験配列5 ’AGTGCGCGGTGC3’および表1におけるスコア表を用いると、出力部位(5 ’−3’)およびスコアは以下のとおりである:
【0171】
【数8】

【0172】
本実施例において、最良の標的部位は、5’TGC GCG GTG 3 ’であり、スコアは1000である。このプログラムはまた、反対の(アンチセンスの)鎖における潜在的な標的に対してスコアを割り当てるが、単純さのために、これらの部位は、本実施例において無視する。任意の因子である「『D可能』トリプレットについての増強因子」が、標的部位の評価において、状況因子「D可能接触」を担う上記のスコア付けプロトコルを変更するために提供され得る。この特徴が選択される場合、このプログラムは、サブ部位スコアを割り当てる場合に、引き続きのチェックを実施する。
【0173】
サブ部位が形態xxGのものである場合、次いで隣接塩基(3’側)は、TまたはGである場合、xxGサブ部位のスコアに増強因子を乗じるか、そうでなければ、サブ部位スコアは同じままである。
【0174】
(そのサブ部位が形態xxA、xxCまたはxxTのものである場合、そのスコアもまた、変化しないままである。)
例えば、ユーザーが、1.25という「D可能」トリプレットについての増強因子を入力する場合、上記のスコアは、以下のように調整される:
【0175】
【数9】

【0176】
(この選択肢を使用する場合、このプログラムは、その標的部位のすぐ3’側の塩基(小文字)の同一性を考慮する。最後の部位について、この塩基は、本実施例において規定されておらず、そしてこれは、この位置にてシャープ記号「#」を入れることによって注意が喚起される。
【0177】
その標的DNAにおけるすべての9塩基対配列に対してスコアを割り当てた後、次いで、そのプログラムは、トップスコアを、ユーザーによって決定され、出力された部位の数とともに、印刷する。
【0178】
ユーザーによって特定される場合、そのプログラムはまた、以下を提供する: i.3つのフィンガータンパク質がすでに設計されている任意の標的部位のリスト ii.入力配列における位置に従って、再整列された出力部位のリスト iii.9bp DNA部位のすべての標的可能な対のリスト(隣接し、重複しない、5、3またはそれ未満の塩基だけ分離されている部位の対)。
【0179】
(II.選択した標的部位についてのタンパク質の設計)
入力:そのプログラムの部位選択部分からの部位(または、他の方法によって決定される)既存の高親和性ZFPについて、標的部位、アミノ酸配列および参照データを含む、「ZFPデータ表」。
【0180】
そのプログラムがその出力を以下のいずれかに限定するか否かを特定する出力パラメータ(ユーザーによって供給される): i (存在する場合)その標的部位がその出力における部位と完全に同一であるタンパク質のみ、または ii (存在する場合)その標的部位が3bpサブ部位の2以上にて出力部位に適合するタンパク質のみ。
【0181】
出力:制限(i)も(ii)も存在しない場合: 潜在的な9塩基対標的部位の各々について、ZFPデータ表からのZFPおよびその成分フィンガーの3つのセットの表。これら成分フィンガーは、それぞれ、その標的部位内の3つのトリプレットサブ部位に結合する。各サブ部位について、ZFPのそのセットは、2つのサブセットへと細分され得る。1つのサブセットは、その親のZFPにおける対応するフィンガー位からの所定の位置にてトリプレットを結合するZFPおよびそれらのフィンガーを含む。他のサブセットは、親のZFP内の対応しない位置からの所定の位置にてトリプレットを結合するZFPおよびそのフィンガーを含む。第一のフィンガー位(N−C)は、最初のトリプレット3 ’−5’に対応する。
【0182】
このプログラムのZFP設計部分は、ユーザーが所定の9塩基標的部位におけるサブ部位を結合することが公知のすべてのフィンガーを迅速に再検討することを可能にすることによって設計プロセスを容易にする。上記の例(5 ’TGCGCGGTG3’)からの最適な設計標的、および表9に提供される短いZFPデータ表を与えられると、出力(制限(i)も(ii)も存在しない場合)は以下のとおりである:
【0183】
【数10】

【0184】
「整列した(ordered)」出力は、ZFPデータ表において、TGCサブ部位が標的部位の第三のトリプレットにおけるジンクフィンガーが接触する1つの例が存在することを示す。この場合におけるフィンガーは、ERDHLRTであり、そしてその部位は、5 ’TGCGGGGCA3’である。他の2つのサブ部位−GCGおよびGTGの各々について1つの類似する例もまた存在する。これらの場合におけるフィンガーは、それぞれ、RSDELQRおよびRKDSLVRである。この情報を使用して、3つのフィンガータンパク質F1−RKDSLVR、F2−RSDELQR、F3−ERDHLRTを、標的5 ’TGCGCGGTG3’を結合するための設計として提唱する。
【0185】
「整列していない」出力は、フィンガーが標的の中心サブ部位ではなくGCGサブ部位に接触するZFPデータ表において2つの場合が存在することを示す。むしろ、1つの場合において、GCGは、5 ’末端に接触し、そして他方は3’末端に接触し、そしてこれらの場合において、フィンガー配列は、RSDELTRおよびRSDERKRである。これらは、その標的部位においてGCGを結合するための別の設計である。
【0186】
【表1】

【0187】
【表9】

【0188】
そのようなデータベースにおける包含に適切な、ジンクフィンガータンパク質、それらのフィンガーの配列および結合する標的部位の他の例は、背景の節において引用された参考文献に議論される。
【0189】
上記の発明を、理解の明確化の目的のために、詳細に記載してきたが、特定の改変は、添付の請求の範囲の範囲内で実施され得ることは自明である。本明細書において、引用した全ての刊行物および特許文書は、本明細書において、その全体が、それら各々が個々そのように注記されているのと同程度にまで全ての目的のために参考として援用される。
【図面の簡単な説明】
【0190】
【図1】図1は、ジンクフィンガータンパク質によって結合される標的部位においてサブ部位の存在および数が結合親和性に相関するデータを提供する図を示す。
【図2】図2は、3つのD可能サブ部位を含む標的部位に結合される3つのジンクフィンガータンパク質を示す。
【図3】図3は、設計されたZFPをコードする核酸をアセンブルするプロセスを示す。
【図4】図4は、標的部位の選択およびジンクフィンガータンパク質の設計の方法を実行するためのコンピュータシステムを示す。
【図5】図5は、標的部位の選択およびジンクフィンガータンパク質の設計の方法を実行するためのコンピュータシステムを示す。
【図6】図6は、標的配列内にD可能サブ部位を含む標的部位を選択するための方法のフローチャートを示す。
【図7A】図7Aは、対応規則を使用して標的配列内の標的部位を選択するためのフローチャートを示す。
【図7B】図7Bは、データベースを使用して、所望の標的部位を結合するようにZFPを設計するためのフローチャートを示す。
【図8A】図8Aは、ZFPデータベースのエンティティの代表図を示す。
【図8B】図8Bは、ZFPデータベースの代表例である。
【特許請求の範囲】
【請求項1】
ジンクフィンガータンパク質によって標的にされるための標的配列内の標的部位を選択する方法であって、該方法が、以下:
ジンクフィンガータンパク質によって標的にされる標的核酸を提供する工程;
5’NNx aNy bNzc3’を含む標的核酸内の標的部位を出力する工程;
を包含し、
ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、方法。
【請求項2】
請求項1に記載の方法であって、
該方法が前記標的核酸内の複数の潜在的標的部位を選択する工程、および
5’NNx aNy bNzc3’を含む該複数の潜在的標的セグメントのサブセットを出力する工程をさらに包含し、
ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、方法。
【請求項3】
前記標的核酸が標的遺伝子を含む、請求項2に記載の方法。
【請求項4】
(x,a)、(y,b)および(z,c)の少なくとも2つが、(G,K)である、請求項1に記載の方法。
【請求項5】
(x,a)、(y,b)および(z,c)の3つ全てが、(G,K)である、請求項1に記載の方法。
【請求項6】
前記標的部位が、第1および第2の標的セグメントを含み、その各々が、5’NNx aNy bNzc3 ’を含み、そして該方法は、該第2の標的セグメントを選択する工程をさらに包含する、請求項1に記載の方法。
【請求項7】
前記第2のセグメントにおいて、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、請求項6に記載の方法。
【請求項8】
前記第2のセグメントにおいて、(x,a)、(y,b)および(z,c)の少なくとも2つが、(G,K)である、請求項7に記載の方法。
【請求項9】
前記第2のセグメントにおいて、(x,a)、(y,b)および(z,c)の3つ全てが、(G,K)である、請求項8に記載の方法。
【請求項10】
前記第1および第2の標的セグメントが、前記標的部位において5未満の塩基によって隔てられている、請求項10に記載の方法。
【請求項11】
前記第1の標的セグメントが、5’NNN NNN NNG3’を含み、前記第2の標的セグメントが、5 ’KNx aNY bNzc3’を含み、そして該標的部位において該第1および第2の標的セグメントを隔てる塩基がゼロである、請求項10に記載の方法。
【請求項12】
前記標的セグメントにおけるNNx aNyおよびbNzトリプレットにそれぞれ結合する3つのジンクフィンガー、ならびに前記第2の標的セグメントにおけるNNx aNyおよびbNzトリプレットにそれぞれ結合する第2の3つのフィンガーを含む、第1のジンクフィンガータンパク質を合成する工程を包含する、合成工程をさらに包含する、請求項7に記載の方法。
【請求項13】
前記bNz aNyおよびNNxトリプレットにそれぞれ結合する第1、第2および第3のフィンガーを含むジンクフィンガータンパク質を合成する工程をさらに包含する、請求項1に記載の方法。
【請求項14】
前記第1、第2および第3のフィンガーの各々が、独立して、選択または設計される、請求項13に記載の方法。
【請求項15】
フィンガーが、ジンクフィンガータンパク質の設計、フィンガー成分のサブ指定(subdesignation)、および該ジンクフィンガータンパク質が結合した核酸配列を含むデータベースから設計される、請求項13に記載の方法。
【請求項16】
フィンガーが、前記標的部位に特異的に結合するためにジンクフィンガー結合タンパク質の改変体をスクリーニングして、該標的部位に結合する改変体を同定する工程によって選択される、請求項13に記載の方法。
【請求項17】
前記標的核酸を含むサンプルと、ジンクフィンガータンパク質とを接触させ、これによって該ジンクフィンガータンパク質が、該標的核酸またはその特定の対立遺伝子形態の存在を示す前記標的部位に結合する工程をさらに包含する、請求項13に記載の方法。
【請求項18】
前記標的核酸を含むサンプルと、ジンクフィンガータンパク質とを接触させ、これによって該ジンクフィンガータンパク質が前記標的部位に結合し、それによって該標的核酸の発現を調節する工程をさらに包含する、請求項13に記載の方法。
【請求項19】
前記標的部位が、遺伝子のコード領域に存在する、請求項1に記載の方法。
【請求項20】
前記標的部位が、プロモーター、エンハンサー、または転写開始部位の内側またはその近傍に存在する、請求項1に記載の方法。
【請求項21】
前記標的部位が、前記標的核酸内のプロモーター、調節配列、または転写開始部位の外側に存在する、請求項1に記載の方法。
【請求項22】
ジンクフィンガータンパク質によって標的にされるためのポリヌクレオチド内の標的部位を選択する方法であって、該方法が、以下: ポリヌクレオチド配列を提供する工程;
該ポリヌクレオチド配列内の潜在的標的部位を選択する工程であって、該潜在的標的部位が、該潜在的標的部位における第1、第2および第3の位置で、連続する第1、第2および第3の塩基のトリプレットを含む、工程;
トリプレットと、一連の3つの連続するトリプレットにおけるトリプレット位置との間に対応規則を適用することによって複数のサブスコアを決定する工程であって、ここで、該トリプレットの各々が、第1、第2および第3の対応位置を有し、そしてトリプレットおよびトリプレット位置の組み合わせの各々が特定のサブスコアを有する、工程;
該第1、第2および第3のトリプレットのサブスコアを組み合わせることによって、該潜在的標的部位のスコアを算出する工程;
さらなる潜在的標的部位の第1、第2および第3の位置で、第1、第2および第3のトリプレットを含む該さらなる潜在的標的部位上で、少なくとも1回、選択工程、決定工程、および算出工程を繰り返して、さらなるスコアを決定する工程;
そのスコアを有する、少なくとも1つの潜在的標的部位の出力を提供する工程、を包含する、方法。
【請求項23】
出力が、最も高いスコアを有する潜在的標的部位で提供される、請求項22に記載の方法。
【請求項24】
出力が、最も高いスコアを有するn個の潜在的標的部位を提供し、そして該方法が、n個の値のユーザー入力を提供する工程をさらに包含する、請求項22に記載の方法。
【請求項25】
前記サブスコアが前記サブスコアの産物を形成することによって組み合わされる、請求項22に記載の方法。
【請求項26】
前記対応規則が、64のトリプレットを含み、その各々が、第1、第2および第3の対応位置、ならびに192のサブスコアを有する、請求項25に記載の方法。
【請求項27】
前記対応規則におけるサブスコアが、以下:トリプレットおよび対応位置のサブセットのサブスコアとして第1の値を割り当てる工程であって、その中の各々について既存のジンクフィンガータンパク質が存在し、該既存のジンクフィンガータンパク質が、該対応規則において該トリプレットの該対応位置と同じ、該既存のジンクフィンガータンパク質における位置に由来する該トリプレットに特異的に結合するフィンガーを含む、工程;トリプレットおよび対応位置のサブセットのサブスコアとして第2の値を割り当てる工程であって、その中の各々について、既存のジンクフィンガータンパク質が存在し、該既存のジンクフィンガータンパク質が、該対応規則において該トリプレットの該対応位置とは異なる、該既存のジンクフィンガータンパク質位置に由来する該トリプレットに特異的に結合するフィンガーを含む、工程;ならびにトリプレットおよび対応位置のサブセットのサブスコアとして第3の値を割り当てる工程であって、それについて、該トリプレットに特異的に結合するフィンガーを含む既知のジンクタンパク質が存在しない、工程、によって決定される、請求項22に記載の方法。
【請求項28】
前記対応規則が表1に示される、請求項22に記載の方法。
【請求項29】
状況(context)パラメーターと前記第1、第2および第3のトリプレットの少なくとも1つのサブスコアとを組み合わせて、該少なくとも1つのトリプレットのスケーリングされたサブスコアを得る工程をさらに包含する、請求項22に記載の方法。
【請求項30】
前記状況パラメーターが、前記標的部位が塩基配列5’NNGK3’を含み、ここでNNGが前記少なくとも1つのトリプレットである場合の前記サブスコアと組み合わされる、請求項29に記載の方法。
【請求項31】
潜在的標的部位のスコアと組み合わされている状況パラメーターを組み合わせて、スケーリングされたスコアを得る工程をさらに包含する、請求項22に記載の方法。
【請求項32】
請求項31に記載の方法であって、前記状況パラメーターが、潜在的標的部位が5’NNx aNy bNzc3 ’を含む場合の前記スコアと組み合わされ、 ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;
(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、方法。
【請求項33】
(x,a)、(y,b)および(z,c)の1つが、(G,K)である場合に、第1の状況パラメーターが前記スコアと組み合わされ、そして(x,a)、(y,b)および(z,c)の2つが、(G,K)である場合、第2の状況パラメーター該スコアと組み合わされ、そして(x,a)、(y,b)および(z,c)の3つが、(G,K)である場合、第3の状況のパラメーターが入力される、請求項32に記載の方法。
【請求項34】
出力が、潜在的標的部位およびそれらのスコアの少なくとも1つの非重複対を提供し、該対のメンバーがポリヌクレオチドにおいて5以下の塩基によって隔てられている、請求項22に記載の方法。
【請求項35】
ジンクフィンガータンパク質を生成する方法であって、該方法は、以下:
(a)複数のジンクフィンガータンパク質であって、該タンパク質の各々が、少なくとも第1、第2および第3のフィンガーを含むジンクフィンガータンパク質の指定、および該ジンクフィンガータンパク質の各々の3つのフィンガーの各々についてのサブ指定を含むデータベース;該ジンクフィンガータンパク質の各々についての対応核酸配列であって、該配列の各々が、該ジンクフィンガータンパク質の各々において、それぞれ該少なくとも第1、第2および第3のフィンガーによって特異的に結合される、少なくとも第1、第2および第3のトリプレットを含み、該第1、第2および第3のトリプレットは、該ジンクフィンガータンパク質に配列された(arranged)(N末端からC末端)該第1および、第2および第3のフィンガーと同じそれぞれの順番で、該核酸配列において配列されている(3 ’−5’)、対応核酸配列、を提供する工程、
(b)ジンクフィンガータンパク質の設計のための標的部位を提供する工程であって、該標的部位は、連続する第1、第2および第3のトリプレットを3 ’−5’の順で含む、工程、
(c)該標的部位の該第1、第2および第3のトリプレットについて、該データベース中のジンクフィンガータンパク質の第1、第2および第3のセットを同定する工程であって、該第1のセットは、該標的部位における該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位における該第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第3のセットは、該標的部位における該第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含む、工程、
(d)工程(c)において同定された該第1、第2および第3のセットのジンクフィンガータンパク質の指定およびサブ指定を出力する工程、を包含する、方法。
【請求項36】
請求項35に記載の方法であって、
(e)前記第1のセット由来のジンクフィンガータンパク質の第1のフィンガー、前記第2のセット由来のジンクフィンガータンパク質の第2のフィンガー、および前記第3のセット由来のジンクフィンガータンパク質の第3のフィンガーを含む前記標的部位に結合する、ジンクフィンガータンパク質を生成する工程、をさらに包含する、方法。
【請求項37】
前記第1、第2および第3のセットのサブセットを同定する工程をさらに包含する、請求項36に記載の方法であって、
該第1のセットのサブセットが、前記データベース中のジンクフィンガータンパク質の第1のフィンガー位置からの標的部位において第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、
該第2のセットのサブセットが、前記データベース中のジンクフィンガータンパク質の第2のフィンガー位置からの標的部位において第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、
該第3のセットのサブセットが、前記データベース中のジンクフィンガータンパク質の第3のフィンガー位置からの標的部位において第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、
ここで、前記出力工程が、該第1、第2および第3のセットのサブセットの指定およびサブ指定を出力する工程を包含し、そして 前記生成工程が、前記第1のサブセット由来の第1のフィンガー、前記第2のサブセット由来の第2のフィンガー、および前記第3のサブセット由来の第3のフィンガーを含むジンクフィンガータンパク質を生成することを包含する工程である、方法。
【請求項38】
前記出力工程が、前記第1、第2および第3のセットのサブセット、ならびにそれらのそれぞれのサブセットを引いた該第1、第2および第3のセットの指定およびサブ指定を出力する工程を包含する、請求項37に記載の方法。
【請求項39】
前記サブセットの各々が、ヌルのセットである、請求項38に記載の方法。
【請求項40】
前記標的部位がユーザー入力によって提供される、請求項35に記載の方法。
【請求項41】
前記標的部位が、請求項1または請求項22に記載の方法によって提供される、請求項35に記載の方法。
【請求項42】
ジンクフィンガータンパク質を生成する方法であって、該方法は、以下:
(a)以下: 複数のジンクフィンガータンパク質の指定であって、各タンパク質が少なくとも第1および第2のフィンガーを含む、指定;該ジンクフィンガータンパク質の各々のフィンガーの各々についてのサブ指定;および ジンクフィンガータンパク質の各々についての対応核酸配列であって、該配列の各々が、それぞれ該第1および第2のフィンガーによって、特異的に結合される第1および第2のトリプレットを含み、該トリプレットは、該ジンクフィンガータンパク質に配列されている(N末端からC末端)第1および第2のフィンガーとそれぞれ同じ順で核酸配列において配列されている(3 ’−5’)、対応核酸配列、を含むデータベースを提供する工程、
(b)ジンクフィンガータンパク質の設計のための標的部位を提供する工程であって、該標的部位が、該標的部位において3 ’から5’に整列された、連続する第1、および第2のトリプレットを含む、工程;
(c)該標的の該第1および第2のトリプレットについて、該データベースにおいて第1および第2のセットのジンクフィンガータンパク質を同定する工程であって、該第1のセットは、該標的部位において該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位において該第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含む、工程;
(d)工程(c)において同定された第1および第2のセットにおけるジンクフィンガータンパク質の指定およびサブ指定を出力する工程、を包含する、方法。
【請求項43】
ジンクフィンガータンパク質を生成する方法であって、該方法は、以下:
(a)以下: 複数のジンクフィンガータンパク質の指定であって、該タンパク質の各々が少なくとも第1および第2のフィンガーを含む指定;該ジンクフィンガータンパク質の各々のフィンガーの各々についてのサブ指定;および 各ジンクフィンガータンパク質についての対応核酸配列であって、該配列の各々が、それぞれ該第1および第2のフィンガーによって、特異的に結合した第1および第2のトリプレットを含み、該トリプレットは、該ジンクフィンガータンパク質に配列されている(N末端からC末端)第1および第2のフィンガーとそれぞれ同じ順で核酸配列において配列されている(3 ’−5’)、対応核酸配列、を含むデータベースを提供する工程、
(b)ジンクフィンガータンパク質の設計のための標的部位を提供する工程であって、該標的部位が、該標的部位において3 ’から5’に整列された、連続する第1、第2および第3のトリプレットを含む、工程;
(c)該標的部位の該第1および第3のトリプレットについて、該データベースにおいて第1および第2のセットのジンクフィンガータンパク質を同定する工程であって、該第1のセットは、該標的部位において該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位において該第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含む、工程;
(d)工程(c)において同定された第1および第2のセットにおけるジンクフィンガータンパク質の指定およびサブ指定を出力する工程、を包含する、方法。
【請求項44】
ジンクフィンガータンパク質によって標的にされるためのポリヌクレオチド内の標的配列を選択するコンピュータープログラム製品であって、該製品は、以下:
(a)ポリヌクレオチド配列を提供するためのコード;
(b)該ポリヌクレオチド配列内の潜在的標的部位を選択するためのコードであって、該潜在的標的部位は、該潜在的標的部位において第1、第2および第3の位置で、第1、第2および第3のトリプレットの塩基を含む、コード;
(c)該潜在的標的部位のスコアを、該第1、第2および第3のトリプレットのサブスコアの組み合わせから算出するためのコードであって、該サブスコアは、トリプレットとトリプレット位置との間での対応規則から得られ、ここで、該トリプレットの各々は、第1、第2および第3の対応位置を有し、そして対応トリプレットおよび位置の各々は特定のサブスコアを有する、コード;
(d)さらなる潜在的標的部位の第1、第2および第3の位置で、第1、第2および第3のトリプレットを含む、該さらなる潜在的標的部位上で少なくとも1回、工程(b)および(c)を繰り返してさらなるスコアを決定するためのコード;
(e)そのスコアとともに該潜在的標的部位の少なくとも1つの出力を提供するためのコード;
(f)コードを保持するためのコンピューター読みとり可能記憶媒体、を含む、コンピュータープログラム製品。
【請求項45】
状況パラメーターとサブスコアとを組み合わせるためのコードをさらに含む、請求項44に記載のコンピュータープログラム製品。
【請求項46】
ジンクフィンガータンパク質によって標的にされるためのポリヌクレオチド内の標的配列を選択するシステムであって、該システムは、以下:
(a)メモリ;
(b)システムバス;
(c)以下を行うように動作可能に配置されたプロセッサ:
(1)ポリヌクレオチド配列を提供するかまたは受け取り;
(2)該ポリヌクレオチド配列内の潜在的標的部位を選択し、該潜在的標的部位は、該潜在的標的部位に第1、第2および第3の位置で、第1、第2および第3のトリプレットの塩基を含み;
(3)該潜在的標的部位のスコアを、該第1、第2および第3のトリプレットのサブスコアの組み合わせから算出し、該サブスコアは、トリプレットとトリプレット位置との間で対応規則から得られ、ここで、該トリプレットの各々は、第1、第2および第3の対応位置を有し、そして該対応トリプレットおよび位置の各々は、特定のサブスコアを有し;
(4)さらなる潜在的標的部位の第1、第2および第3の部位で、第1、第2および第3のトリプレットを含む、該さらなる潜在的標的部位上で少なくとも1回、工程(2)および(3)を繰り返して、さらなるスコアを決定し;
(5)そのスコアとともに該潜在的標的部位の少なくとも1つの出力を提供する、 プロセッサを備える、システム。
【請求項47】
前記プロセッサが、状況パラメーターとサブスコアとを組み合わせるように動作可能にさらに配置されている、請求項46に記載のシステム。
【請求項48】
ジンクフィンガータンパク質を設計するためのコンピュータープログラム製品であって、該製品は、以下:
(a)以下: 複数のジンクフィンガータンパク質であって、該タンパク質の各々が、少なくとも第1、第2および第3のフィンガーを含むジンクフィンガータンパク質の指定、該ジンクフィンガータンパク質の各々の3つのフィンガーの各々についてのサブ指定;該ジンクフィンガータンパク質の各々についての対応核酸配列であって、該配列の各々が、該ジンクフィンガータンパク質の各々において、それぞれ該少なくとも第1、第2および第3のフィンガーによって特異的に結合される、少なくとも第1、第2および第3のトリプレットを含み、該第1、第2および第3のトリプレットが、該ジンクフィンガータンパク質に配列された(N末端からC末端)該第1および、第2および第3のフィンガーと同じそれぞれの順番で、該核酸配列において配列されている(3 ’−5’)、対応核酸配列、を含むデータベースを提供するためのコード、
(b)ジンクフィンガータンパク質の設計のための標的部位を提供するためのコードであって、該標的部位は、少なくとも第1、第2および第3のトリプレットを含む、コード、 (c)該標的部位の該第1、第2および第3のトリプレットについて、該データベース中のジンクフィンガータンパク質の第1、第2および第3のセットを同定するためのコードであって、該第1のセットは、該標的部位における該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位における該第2のトリプレットに特異的に結合するフィンガーを含み、該第3のセットは、該標的部位における該第3のトリプレットに特異的に結合するフィンガーを含む、コード、
(d)工程(c)において同定された該第1、第2および第3のセットのジンクフィンガータンパク質の指定およびサブ指定を出力するためのコード、
(e)該コードを保持するためのコンピューター読みとり可能記憶媒体、を含む、コンピュータープログラム製品。
【請求項49】
ジンクフィンガータンパク質を設計するためのシステムであって、該システムは、以下:
(a)メモリ;
(b)システムバス;
(c)以下を行うように動作可能に配置されたプロセッサ:
(1)以下: 複数のジンクフィンガータンパク質についての指定であって、該タンパク質の各々が、少なくとも第1、第2および第3のフィンガーを含む指定、 該ジンクフィンガータンパク質の各々の3つのフィンガーの各々についてのサブ指定;該ジンクフィンガータンパク質の各々についての対応核酸配列であって、該配列の各々が、該ジンクフィンガータンパク質の各々において、それぞれ該少なくとも第1、第2および第3のフィンガーによって特異的に結合される、少なくとも第1、第2および第3のトリプレットを含み、該第1、第2および第3のトリプレットが、該ジンクフィンガータンパク質に配列された(N末端からC末端)該第1および、第2および第3のフィンガーと同じそれぞれの順番で、該核酸配列において配列されている(3 ’−5’)、対応核酸配列、を含むデータベースを提供し;
(2)ジンクフィンガータンパク質の設計のための標的部位を提供するかまたは提供され、該標的部位は、少なくとも第1、第2および第3のトリプレットを含み、
(3)該標的部位の該第1、第2および第3のトリプレットについて、該データベースにおいて、該第1のセットは、該標的部位において該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位において該第2のトリプレットに特異的に結合するフィンガーを含み、該第3のセットは、該標的部位において該第3のトリプレットに特異的に結合するフィンガーを含む第1、第2および第3のセットのジンクフィンガータンパク質を同定し;
(4)工程(3)において同定された該第1、第2および第3のセットにおける該ジンクフィンガータンパク質の指定およびサブ指定を出力する、 プロセッサを備える、システム。
【請求項50】
ジンクフィンガータンパク質によって標的化されるための標的配列内の標的部位を選択するためのコンピュータープログラム製品であって、該製品は、以下:
ジンクフィンガータンパク質によって標的にされる標的核酸を提供するコード;
5’NNx aNy bNzc3’を含む標的核酸内の標的部位を出力するためのコードであって;
ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、コード;
ならびに 該コードを保持するためのコンピューター読みとり可能記憶媒体、を含む、コンピュータープログラム製品。
【請求項51】
ジンクフィンガータンパク質によって標的化されるための標的配列内の標的部位を選択するためのシステムであって、該システムは、以下:
(a)メモリ;
(b)システムバス;
(c)以下を行うように動作可能に配置されたプロセッサ: ジンクフィンガータンパク質によって標的にされる標的核酸を提供し;5 ’NNx aNy bNzc3’を含む標的核酸内の標的部位を出力し、
ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、 プロセッサ、を備える、システム。
【請求項1】
ジンクフィンガータンパク質によって標的にされるための標的配列内の標的部位を選択する方法であって、該方法が、以下:
ジンクフィンガータンパク質によって標的にされる標的核酸を提供する工程;
5’NNx aNy bNzc3’を含む標的核酸内の標的部位を出力する工程;
を包含し、
ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、方法。
【請求項2】
請求項1に記載の方法であって、
該方法が前記標的核酸内の複数の潜在的標的部位を選択する工程、および
5’NNx aNy bNzc3’を含む該複数の潜在的標的セグメントのサブセットを出力する工程をさらに包含し、
ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、方法。
【請求項3】
前記標的核酸が標的遺伝子を含む、請求項2に記載の方法。
【請求項4】
(x,a)、(y,b)および(z,c)の少なくとも2つが、(G,K)である、請求項1に記載の方法。
【請求項5】
(x,a)、(y,b)および(z,c)の3つ全てが、(G,K)である、請求項1に記載の方法。
【請求項6】
前記標的部位が、第1および第2の標的セグメントを含み、その各々が、5’NNx aNy bNzc3 ’を含み、そして該方法は、該第2の標的セグメントを選択する工程をさらに包含する、請求項1に記載の方法。
【請求項7】
前記第2のセグメントにおいて、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、請求項6に記載の方法。
【請求項8】
前記第2のセグメントにおいて、(x,a)、(y,b)および(z,c)の少なくとも2つが、(G,K)である、請求項7に記載の方法。
【請求項9】
前記第2のセグメントにおいて、(x,a)、(y,b)および(z,c)の3つ全てが、(G,K)である、請求項8に記載の方法。
【請求項10】
前記第1および第2の標的セグメントが、前記標的部位において5未満の塩基によって隔てられている、請求項10に記載の方法。
【請求項11】
前記第1の標的セグメントが、5’NNN NNN NNG3’を含み、前記第2の標的セグメントが、5 ’KNx aNY bNzc3’を含み、そして該標的部位において該第1および第2の標的セグメントを隔てる塩基がゼロである、請求項10に記載の方法。
【請求項12】
前記標的セグメントにおけるNNx aNyおよびbNzトリプレットにそれぞれ結合する3つのジンクフィンガー、ならびに前記第2の標的セグメントにおけるNNx aNyおよびbNzトリプレットにそれぞれ結合する第2の3つのフィンガーを含む、第1のジンクフィンガータンパク質を合成する工程を包含する、合成工程をさらに包含する、請求項7に記載の方法。
【請求項13】
前記bNz aNyおよびNNxトリプレットにそれぞれ結合する第1、第2および第3のフィンガーを含むジンクフィンガータンパク質を合成する工程をさらに包含する、請求項1に記載の方法。
【請求項14】
前記第1、第2および第3のフィンガーの各々が、独立して、選択または設計される、請求項13に記載の方法。
【請求項15】
フィンガーが、ジンクフィンガータンパク質の設計、フィンガー成分のサブ指定(subdesignation)、および該ジンクフィンガータンパク質が結合した核酸配列を含むデータベースから設計される、請求項13に記載の方法。
【請求項16】
フィンガーが、前記標的部位に特異的に結合するためにジンクフィンガー結合タンパク質の改変体をスクリーニングして、該標的部位に結合する改変体を同定する工程によって選択される、請求項13に記載の方法。
【請求項17】
前記標的核酸を含むサンプルと、ジンクフィンガータンパク質とを接触させ、これによって該ジンクフィンガータンパク質が、該標的核酸またはその特定の対立遺伝子形態の存在を示す前記標的部位に結合する工程をさらに包含する、請求項13に記載の方法。
【請求項18】
前記標的核酸を含むサンプルと、ジンクフィンガータンパク質とを接触させ、これによって該ジンクフィンガータンパク質が前記標的部位に結合し、それによって該標的核酸の発現を調節する工程をさらに包含する、請求項13に記載の方法。
【請求項19】
前記標的部位が、遺伝子のコード領域に存在する、請求項1に記載の方法。
【請求項20】
前記標的部位が、プロモーター、エンハンサー、または転写開始部位の内側またはその近傍に存在する、請求項1に記載の方法。
【請求項21】
前記標的部位が、前記標的核酸内のプロモーター、調節配列、または転写開始部位の外側に存在する、請求項1に記載の方法。
【請求項22】
ジンクフィンガータンパク質によって標的にされるためのポリヌクレオチド内の標的部位を選択する方法であって、該方法が、以下: ポリヌクレオチド配列を提供する工程;
該ポリヌクレオチド配列内の潜在的標的部位を選択する工程であって、該潜在的標的部位が、該潜在的標的部位における第1、第2および第3の位置で、連続する第1、第2および第3の塩基のトリプレットを含む、工程;
トリプレットと、一連の3つの連続するトリプレットにおけるトリプレット位置との間に対応規則を適用することによって複数のサブスコアを決定する工程であって、ここで、該トリプレットの各々が、第1、第2および第3の対応位置を有し、そしてトリプレットおよびトリプレット位置の組み合わせの各々が特定のサブスコアを有する、工程;
該第1、第2および第3のトリプレットのサブスコアを組み合わせることによって、該潜在的標的部位のスコアを算出する工程;
さらなる潜在的標的部位の第1、第2および第3の位置で、第1、第2および第3のトリプレットを含む該さらなる潜在的標的部位上で、少なくとも1回、選択工程、決定工程、および算出工程を繰り返して、さらなるスコアを決定する工程;
そのスコアを有する、少なくとも1つの潜在的標的部位の出力を提供する工程、を包含する、方法。
【請求項23】
出力が、最も高いスコアを有する潜在的標的部位で提供される、請求項22に記載の方法。
【請求項24】
出力が、最も高いスコアを有するn個の潜在的標的部位を提供し、そして該方法が、n個の値のユーザー入力を提供する工程をさらに包含する、請求項22に記載の方法。
【請求項25】
前記サブスコアが前記サブスコアの産物を形成することによって組み合わされる、請求項22に記載の方法。
【請求項26】
前記対応規則が、64のトリプレットを含み、その各々が、第1、第2および第3の対応位置、ならびに192のサブスコアを有する、請求項25に記載の方法。
【請求項27】
前記対応規則におけるサブスコアが、以下:トリプレットおよび対応位置のサブセットのサブスコアとして第1の値を割り当てる工程であって、その中の各々について既存のジンクフィンガータンパク質が存在し、該既存のジンクフィンガータンパク質が、該対応規則において該トリプレットの該対応位置と同じ、該既存のジンクフィンガータンパク質における位置に由来する該トリプレットに特異的に結合するフィンガーを含む、工程;トリプレットおよび対応位置のサブセットのサブスコアとして第2の値を割り当てる工程であって、その中の各々について、既存のジンクフィンガータンパク質が存在し、該既存のジンクフィンガータンパク質が、該対応規則において該トリプレットの該対応位置とは異なる、該既存のジンクフィンガータンパク質位置に由来する該トリプレットに特異的に結合するフィンガーを含む、工程;ならびにトリプレットおよび対応位置のサブセットのサブスコアとして第3の値を割り当てる工程であって、それについて、該トリプレットに特異的に結合するフィンガーを含む既知のジンクタンパク質が存在しない、工程、によって決定される、請求項22に記載の方法。
【請求項28】
前記対応規則が表1に示される、請求項22に記載の方法。
【請求項29】
状況(context)パラメーターと前記第1、第2および第3のトリプレットの少なくとも1つのサブスコアとを組み合わせて、該少なくとも1つのトリプレットのスケーリングされたサブスコアを得る工程をさらに包含する、請求項22に記載の方法。
【請求項30】
前記状況パラメーターが、前記標的部位が塩基配列5’NNGK3’を含み、ここでNNGが前記少なくとも1つのトリプレットである場合の前記サブスコアと組み合わされる、請求項29に記載の方法。
【請求項31】
潜在的標的部位のスコアと組み合わされている状況パラメーターを組み合わせて、スケーリングされたスコアを得る工程をさらに包含する、請求項22に記載の方法。
【請求項32】
請求項31に記載の方法であって、前記状況パラメーターが、潜在的標的部位が5’NNx aNy bNzc3 ’を含む場合の前記スコアと組み合わされ、 ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;
(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、方法。
【請求項33】
(x,a)、(y,b)および(z,c)の1つが、(G,K)である場合に、第1の状況パラメーターが前記スコアと組み合わされ、そして(x,a)、(y,b)および(z,c)の2つが、(G,K)である場合、第2の状況パラメーター該スコアと組み合わされ、そして(x,a)、(y,b)および(z,c)の3つが、(G,K)である場合、第3の状況のパラメーターが入力される、請求項32に記載の方法。
【請求項34】
出力が、潜在的標的部位およびそれらのスコアの少なくとも1つの非重複対を提供し、該対のメンバーがポリヌクレオチドにおいて5以下の塩基によって隔てられている、請求項22に記載の方法。
【請求項35】
ジンクフィンガータンパク質を生成する方法であって、該方法は、以下:
(a)複数のジンクフィンガータンパク質であって、該タンパク質の各々が、少なくとも第1、第2および第3のフィンガーを含むジンクフィンガータンパク質の指定、および該ジンクフィンガータンパク質の各々の3つのフィンガーの各々についてのサブ指定を含むデータベース;該ジンクフィンガータンパク質の各々についての対応核酸配列であって、該配列の各々が、該ジンクフィンガータンパク質の各々において、それぞれ該少なくとも第1、第2および第3のフィンガーによって特異的に結合される、少なくとも第1、第2および第3のトリプレットを含み、該第1、第2および第3のトリプレットは、該ジンクフィンガータンパク質に配列された(arranged)(N末端からC末端)該第1および、第2および第3のフィンガーと同じそれぞれの順番で、該核酸配列において配列されている(3 ’−5’)、対応核酸配列、を提供する工程、
(b)ジンクフィンガータンパク質の設計のための標的部位を提供する工程であって、該標的部位は、連続する第1、第2および第3のトリプレットを3 ’−5’の順で含む、工程、
(c)該標的部位の該第1、第2および第3のトリプレットについて、該データベース中のジンクフィンガータンパク質の第1、第2および第3のセットを同定する工程であって、該第1のセットは、該標的部位における該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位における該第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第3のセットは、該標的部位における該第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含む、工程、
(d)工程(c)において同定された該第1、第2および第3のセットのジンクフィンガータンパク質の指定およびサブ指定を出力する工程、を包含する、方法。
【請求項36】
請求項35に記載の方法であって、
(e)前記第1のセット由来のジンクフィンガータンパク質の第1のフィンガー、前記第2のセット由来のジンクフィンガータンパク質の第2のフィンガー、および前記第3のセット由来のジンクフィンガータンパク質の第3のフィンガーを含む前記標的部位に結合する、ジンクフィンガータンパク質を生成する工程、をさらに包含する、方法。
【請求項37】
前記第1、第2および第3のセットのサブセットを同定する工程をさらに包含する、請求項36に記載の方法であって、
該第1のセットのサブセットが、前記データベース中のジンクフィンガータンパク質の第1のフィンガー位置からの標的部位において第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、
該第2のセットのサブセットが、前記データベース中のジンクフィンガータンパク質の第2のフィンガー位置からの標的部位において第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、
該第3のセットのサブセットが、前記データベース中のジンクフィンガータンパク質の第3のフィンガー位置からの標的部位において第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、
ここで、前記出力工程が、該第1、第2および第3のセットのサブセットの指定およびサブ指定を出力する工程を包含し、そして 前記生成工程が、前記第1のサブセット由来の第1のフィンガー、前記第2のサブセット由来の第2のフィンガー、および前記第3のサブセット由来の第3のフィンガーを含むジンクフィンガータンパク質を生成することを包含する工程である、方法。
【請求項38】
前記出力工程が、前記第1、第2および第3のセットのサブセット、ならびにそれらのそれぞれのサブセットを引いた該第1、第2および第3のセットの指定およびサブ指定を出力する工程を包含する、請求項37に記載の方法。
【請求項39】
前記サブセットの各々が、ヌルのセットである、請求項38に記載の方法。
【請求項40】
前記標的部位がユーザー入力によって提供される、請求項35に記載の方法。
【請求項41】
前記標的部位が、請求項1または請求項22に記載の方法によって提供される、請求項35に記載の方法。
【請求項42】
ジンクフィンガータンパク質を生成する方法であって、該方法は、以下:
(a)以下: 複数のジンクフィンガータンパク質の指定であって、各タンパク質が少なくとも第1および第2のフィンガーを含む、指定;該ジンクフィンガータンパク質の各々のフィンガーの各々についてのサブ指定;および ジンクフィンガータンパク質の各々についての対応核酸配列であって、該配列の各々が、それぞれ該第1および第2のフィンガーによって、特異的に結合される第1および第2のトリプレットを含み、該トリプレットは、該ジンクフィンガータンパク質に配列されている(N末端からC末端)第1および第2のフィンガーとそれぞれ同じ順で核酸配列において配列されている(3 ’−5’)、対応核酸配列、を含むデータベースを提供する工程、
(b)ジンクフィンガータンパク質の設計のための標的部位を提供する工程であって、該標的部位が、該標的部位において3 ’から5’に整列された、連続する第1、および第2のトリプレットを含む、工程;
(c)該標的の該第1および第2のトリプレットについて、該データベースにおいて第1および第2のセットのジンクフィンガータンパク質を同定する工程であって、該第1のセットは、該標的部位において該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位において該第2のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含む、工程;
(d)工程(c)において同定された第1および第2のセットにおけるジンクフィンガータンパク質の指定およびサブ指定を出力する工程、を包含する、方法。
【請求項43】
ジンクフィンガータンパク質を生成する方法であって、該方法は、以下:
(a)以下: 複数のジンクフィンガータンパク質の指定であって、該タンパク質の各々が少なくとも第1および第2のフィンガーを含む指定;該ジンクフィンガータンパク質の各々のフィンガーの各々についてのサブ指定;および 各ジンクフィンガータンパク質についての対応核酸配列であって、該配列の各々が、それぞれ該第1および第2のフィンガーによって、特異的に結合した第1および第2のトリプレットを含み、該トリプレットは、該ジンクフィンガータンパク質に配列されている(N末端からC末端)第1および第2のフィンガーとそれぞれ同じ順で核酸配列において配列されている(3 ’−5’)、対応核酸配列、を含むデータベースを提供する工程、
(b)ジンクフィンガータンパク質の設計のための標的部位を提供する工程であって、該標的部位が、該標的部位において3 ’から5’に整列された、連続する第1、第2および第3のトリプレットを含む、工程;
(c)該標的部位の該第1および第3のトリプレットについて、該データベースにおいて第1および第2のセットのジンクフィンガータンパク質を同定する工程であって、該第1のセットは、該標的部位において該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位において該第3のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含む、工程;
(d)工程(c)において同定された第1および第2のセットにおけるジンクフィンガータンパク質の指定およびサブ指定を出力する工程、を包含する、方法。
【請求項44】
ジンクフィンガータンパク質によって標的にされるためのポリヌクレオチド内の標的配列を選択するコンピュータープログラム製品であって、該製品は、以下:
(a)ポリヌクレオチド配列を提供するためのコード;
(b)該ポリヌクレオチド配列内の潜在的標的部位を選択するためのコードであって、該潜在的標的部位は、該潜在的標的部位において第1、第2および第3の位置で、第1、第2および第3のトリプレットの塩基を含む、コード;
(c)該潜在的標的部位のスコアを、該第1、第2および第3のトリプレットのサブスコアの組み合わせから算出するためのコードであって、該サブスコアは、トリプレットとトリプレット位置との間での対応規則から得られ、ここで、該トリプレットの各々は、第1、第2および第3の対応位置を有し、そして対応トリプレットおよび位置の各々は特定のサブスコアを有する、コード;
(d)さらなる潜在的標的部位の第1、第2および第3の位置で、第1、第2および第3のトリプレットを含む、該さらなる潜在的標的部位上で少なくとも1回、工程(b)および(c)を繰り返してさらなるスコアを決定するためのコード;
(e)そのスコアとともに該潜在的標的部位の少なくとも1つの出力を提供するためのコード;
(f)コードを保持するためのコンピューター読みとり可能記憶媒体、を含む、コンピュータープログラム製品。
【請求項45】
状況パラメーターとサブスコアとを組み合わせるためのコードをさらに含む、請求項44に記載のコンピュータープログラム製品。
【請求項46】
ジンクフィンガータンパク質によって標的にされるためのポリヌクレオチド内の標的配列を選択するシステムであって、該システムは、以下:
(a)メモリ;
(b)システムバス;
(c)以下を行うように動作可能に配置されたプロセッサ:
(1)ポリヌクレオチド配列を提供するかまたは受け取り;
(2)該ポリヌクレオチド配列内の潜在的標的部位を選択し、該潜在的標的部位は、該潜在的標的部位に第1、第2および第3の位置で、第1、第2および第3のトリプレットの塩基を含み;
(3)該潜在的標的部位のスコアを、該第1、第2および第3のトリプレットのサブスコアの組み合わせから算出し、該サブスコアは、トリプレットとトリプレット位置との間で対応規則から得られ、ここで、該トリプレットの各々は、第1、第2および第3の対応位置を有し、そして該対応トリプレットおよび位置の各々は、特定のサブスコアを有し;
(4)さらなる潜在的標的部位の第1、第2および第3の部位で、第1、第2および第3のトリプレットを含む、該さらなる潜在的標的部位上で少なくとも1回、工程(2)および(3)を繰り返して、さらなるスコアを決定し;
(5)そのスコアとともに該潜在的標的部位の少なくとも1つの出力を提供する、 プロセッサを備える、システム。
【請求項47】
前記プロセッサが、状況パラメーターとサブスコアとを組み合わせるように動作可能にさらに配置されている、請求項46に記載のシステム。
【請求項48】
ジンクフィンガータンパク質を設計するためのコンピュータープログラム製品であって、該製品は、以下:
(a)以下: 複数のジンクフィンガータンパク質であって、該タンパク質の各々が、少なくとも第1、第2および第3のフィンガーを含むジンクフィンガータンパク質の指定、該ジンクフィンガータンパク質の各々の3つのフィンガーの各々についてのサブ指定;該ジンクフィンガータンパク質の各々についての対応核酸配列であって、該配列の各々が、該ジンクフィンガータンパク質の各々において、それぞれ該少なくとも第1、第2および第3のフィンガーによって特異的に結合される、少なくとも第1、第2および第3のトリプレットを含み、該第1、第2および第3のトリプレットが、該ジンクフィンガータンパク質に配列された(N末端からC末端)該第1および、第2および第3のフィンガーと同じそれぞれの順番で、該核酸配列において配列されている(3 ’−5’)、対応核酸配列、を含むデータベースを提供するためのコード、
(b)ジンクフィンガータンパク質の設計のための標的部位を提供するためのコードであって、該標的部位は、少なくとも第1、第2および第3のトリプレットを含む、コード、 (c)該標的部位の該第1、第2および第3のトリプレットについて、該データベース中のジンクフィンガータンパク質の第1、第2および第3のセットを同定するためのコードであって、該第1のセットは、該標的部位における該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位における該第2のトリプレットに特異的に結合するフィンガーを含み、該第3のセットは、該標的部位における該第3のトリプレットに特異的に結合するフィンガーを含む、コード、
(d)工程(c)において同定された該第1、第2および第3のセットのジンクフィンガータンパク質の指定およびサブ指定を出力するためのコード、
(e)該コードを保持するためのコンピューター読みとり可能記憶媒体、を含む、コンピュータープログラム製品。
【請求項49】
ジンクフィンガータンパク質を設計するためのシステムであって、該システムは、以下:
(a)メモリ;
(b)システムバス;
(c)以下を行うように動作可能に配置されたプロセッサ:
(1)以下: 複数のジンクフィンガータンパク質についての指定であって、該タンパク質の各々が、少なくとも第1、第2および第3のフィンガーを含む指定、 該ジンクフィンガータンパク質の各々の3つのフィンガーの各々についてのサブ指定;該ジンクフィンガータンパク質の各々についての対応核酸配列であって、該配列の各々が、該ジンクフィンガータンパク質の各々において、それぞれ該少なくとも第1、第2および第3のフィンガーによって特異的に結合される、少なくとも第1、第2および第3のトリプレットを含み、該第1、第2および第3のトリプレットが、該ジンクフィンガータンパク質に配列された(N末端からC末端)該第1および、第2および第3のフィンガーと同じそれぞれの順番で、該核酸配列において配列されている(3 ’−5’)、対応核酸配列、を含むデータベースを提供し;
(2)ジンクフィンガータンパク質の設計のための標的部位を提供するかまたは提供され、該標的部位は、少なくとも第1、第2および第3のトリプレットを含み、
(3)該標的部位の該第1、第2および第3のトリプレットについて、該データベースにおいて、該第1のセットは、該標的部位において該第1のトリプレットに特異的に結合するフィンガーを含むジンクフィンガータンパク質を含み、該第2のセットは、該標的部位において該第2のトリプレットに特異的に結合するフィンガーを含み、該第3のセットは、該標的部位において該第3のトリプレットに特異的に結合するフィンガーを含む第1、第2および第3のセットのジンクフィンガータンパク質を同定し;
(4)工程(3)において同定された該第1、第2および第3のセットにおける該ジンクフィンガータンパク質の指定およびサブ指定を出力する、 プロセッサを備える、システム。
【請求項50】
ジンクフィンガータンパク質によって標的化されるための標的配列内の標的部位を選択するためのコンピュータープログラム製品であって、該製品は、以下:
ジンクフィンガータンパク質によって標的にされる標的核酸を提供するコード;
5’NNx aNy bNzc3’を含む標的核酸内の標的部位を出力するためのコードであって;
ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、コード;
ならびに 該コードを保持するためのコンピューター読みとり可能記憶媒体、を含む、コンピュータープログラム製品。
【請求項51】
ジンクフィンガータンパク質によって標的化されるための標的配列内の標的部位を選択するためのシステムであって、該システムは、以下:
(a)メモリ;
(b)システムバス;
(c)以下を行うように動作可能に配置されたプロセッサ: ジンクフィンガータンパク質によって標的にされる標的核酸を提供し;5 ’NNx aNy bNzc3’を含む標的核酸内の標的部位を出力し、
ここで、(x,a)、(y,b)および(z,c)の各々が、(N,N)または(G,K)であり;(x,a)、(y,b)および(z,c)の少なくとも1つが、(G,K)であり、そして NおよびKがIUPAC−IUB多義性コードである、 プロセッサ、を備える、システム。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7A】


【図7B】


【図8A】


【図8B】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7A】


【図7B】


【図8A】


【図8B】


【公開番号】特開2009−106276(P2009−106276A)
【公開日】平成21年5月21日(2009.5.21)
【国際特許分類】
【出願番号】特願2008−262287(P2008−262287)
【出願日】平成20年10月8日(2008.10.8)
【分割の表示】特願2000−593776(P2000−593776)の分割
【原出願日】平成12年1月6日(2000.1.6)
【出願人】(500426135)サンガモ バイオサイエンシーズ, インコーポレイテッド (6)
【Fターム(参考)】
【公開日】平成21年5月21日(2009.5.21)
【国際特許分類】
【出願日】平成20年10月8日(2008.10.8)
【分割の表示】特願2000−593776(P2000−593776)の分割
【原出願日】平成12年1月6日(2000.1.6)
【出願人】(500426135)サンガモ バイオサイエンシーズ, インコーポレイテッド (6)
【Fターム(参考)】
[ Back to top ]

