トバモウイルス抵抗性遺伝子及びその用途
【課題】トバモウイルス抵抗性L遺伝子をクローニングし、トバモウイルス抵抗性植物の作出のための新規手段を提供すること。
【解決手段】L3抵抗性遺伝子を保有するCapsicum chinenseのBACライブラリーを利用し、L3が2つのBACコンティグ(計5クローン)もしくはその間の未知領域に含まれることを突き止めた。この領域に多数存在する抵抗性遺伝子類似配列のDNA断片をサブクローニングし、配列解析及び機能解析によりL3遺伝子を同定した。さらに、該遺伝子の配列をもとにその他のL遺伝子をも同定した。明らかになったL遺伝子配列自体を利用し、各L遺伝子型を簡便に識別できる方法も確立した。
【解決手段】L3抵抗性遺伝子を保有するCapsicum chinenseのBACライブラリーを利用し、L3が2つのBACコンティグ(計5クローン)もしくはその間の未知領域に含まれることを突き止めた。この領域に多数存在する抵抗性遺伝子類似配列のDNA断片をサブクローニングし、配列解析及び機能解析によりL3遺伝子を同定した。さらに、該遺伝子の配列をもとにその他のL遺伝子をも同定した。明らかになったL遺伝子配列自体を利用し、各L遺伝子型を簡便に識別できる方法も確立した。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、トバモウイルス抵抗性遺伝子及びその用途に関する。
【背景技術】
【0002】
植物のウイルス病には有効な防除薬剤が存在しないため、その防除において抵抗性品種の利用はきわめて重要である。しかしながら、ウイルスは変異を起こしやすく、抵抗性を打ち破る変異体、すなわち抵抗性打破ウイルスの出現が農業生産現場で問題となっている。
【0003】
ピーマン・トウガラシなどのトウガラシ(Capsicum)属植物の生産において、ウイルス病、特にトバモウイルス感染は被害が大きい。トウガラシ属植物におけるトバモウイルス病は土壌伝染性であり、臭化メチルの使用禁止によって土壌消毒が十分に行えない現状にあっては、その防除も困難になっている。国内では主にL3抵抗性遺伝子を持つ抵抗性品種が用いられているが、1998年、国内初のL3抵抗性打破トウガラシ微斑ウイルス(PMMoV)が報告(非特許文献1)されて以降、各地で異なるL3抵抗性打破ウイルスが発見された(非特許文献2)。さらに2006年、L3抵抗性打破PMMoVに対して抵抗性を示すL4抵抗性遺伝子を持つ品種に対しても抵抗性打破性を示す、L4抵抗性打破PMMoVが報告された(非特許文献3)。
【0004】
L抵抗性遺伝子は1980年代からその存在を知られており(非特許文献4および5)、ウイルス抵抗性品種の育成に用いられてきた。L遺伝子には、それぞれ対立遺伝子の関係にあるとされる複数の遺伝子型が存在することが知られており、それら遺伝子とトバモウイルスの病原型の間には階層的な関係が成り立っている。すなわち、タバコモザイクウイルスやトマトモザイクウイルス(ToMV)などのP0病原型に対しては、L1、L2、L3およびL4全てが抵抗性を示すが、パプリカ微斑ウイルス(PaMMV)などのP1病原型に対しては、L1は無効で、L2、L3およびL4だけが抵抗性を示す。さらにPMMoVでは、P1,2、P1,2,3およびP1,2,3,4の各病原型が存在し、P1,2に対してはL3およびL4が、P1,2,3に対してはL4だけが抵抗性を示し、P1,2,3,4に対して抵抗性を示すL遺伝子型は見出されていない。
【0005】
多くのL遺伝子は、温度感受性であることが知られており、28℃以上の恒温条件下ではその抵抗性機能は失われる。一方、L遺伝子の亜型であるL1aは、30℃でもP0病原型トバモウイルスに対して抵抗性を示すこと、すなわち高温機能性を持つことが報告された(非特許文献6)。L1aを含め、これまでに知られているL抵抗性遺伝子は、すべてトバモウイルスの外被タンパク質を認識して抵抗反応を駆動する(非特許文献7)。L3およびL4抵抗性打破PMMoV、すなわちP1,2,3およびP1,2,3,4病原型は、P1,2と比較して外被タンパク質のアミノ酸配列が1ないし2アミノ酸しか違わないことが分かっている。このことは、P1,2,3およびP1,2,3,4病原型がP1,2の変異によって生じたことを示している。L3やL4は温度感受性であるがゆえに、高温下でトバモウイルスの増殖を許容するとともに温度が下がると抵抗反応を駆動するため、外被タンパク質遺伝子の抵抗性打破に関わる変異を引き起こしやすいと考えられる。高温機能性のL1aでは実際、少数のアミノ酸変異による抵抗性打破ウイルス株は出現していない。
【0006】
上記の2つの問題、すなわち効果のある抵抗性遺伝子が存在しないウイルスの出現および抵抗性打破ウイルスが容易に出現してしまう抵抗性遺伝子の性質を解決するために、遺伝子組換えあるいは変異の導入による抵抗性遺伝子の改良が期待される。しかし、L遺伝子についてはこれまでクローニングされておらず、その構造も全く不明であり、その改良の糸口もない状況である。
【0007】
交配育種によって既知のL遺伝子型を品種に導入しようとする時、それぞれを識別できる遺伝子マーカーがあると、選抜が容易になる。通常使用される遺伝子マーカーは、必ずしも注目する形質を規定する遺伝子そのものではないが、目的形質と強く連鎖するものであれば遺伝子マーカーとして有用である。しかしながら、遺伝子そのものを検出しているわけではないため、目的形質と分離する可能性は否定できない。遺伝子がクローン化されており、植物系統間で多型のある場合は、遺伝子そのものを用いた方が確実であるが、L遺伝子は未だクローニングされていないため、遺伝子そのものをマーカーとして用いることもできない。
【0008】
一方、ジャガイモのジャガイモウイルスX抵抗性遺伝子、Rxでは、そのロイシンリッチリピート領域に人為的に変異を導入することによって、野生型Rxが認識できなかったウイルスを認識し、抵抗性を付与できることが報告されている(非特許文献8)。
【0009】
【非特許文献1】Molecular Plant-Microbe Interactions 11, 327-31 (1998)
【非特許文献2】J Gen Plant Pathol 68: 155-162 (2002)
【非特許文献3】Phytopathology 97, 787-93 (2007)
【非特許文献4】Euphytica 29: 433-439 (1980)
【非特許文献5】Capsicum Newslet 3: 47-48 (1982)
【非特許文献6】J Jpn Soc Hort Sci 73: 552-557 (2004)
【非特許文献7】Archives of Virology, 153, 645-650. (2008)
【非特許文献8】Proc Natl Acad Sci U S A 103: 18828-33 (2006)
【発明の開示】
【発明が解決しようとする課題】
【0010】
本発明の目的は、未だクローニングされていないトバモウイルス抵抗性L遺伝子をクローニングし、トバモウイルス抵抗性植物の作出のための新規手段を提供することにある。
【課題を解決するための手段】
【0011】
本願発明者らは、鋭意研究の結果、L3抵抗性遺伝子を保有するCapsicum chinenseのBACライブラリーを利用し、抵抗性遺伝子類似配列及びBAC末端配列に基づくマーカーを開発してライブラリーのスクリーニングを行ない、L3が2つのBACコンティグ(計5クローン)もしくはその間の未知領域に含まれること、その未知領域が約30 kb程度であること、以上の結果を考え合わせるとL3は約400 kbの領域に存在することを突き止めた。また、この領域にトマトのフザリウム抵抗性遺伝子I2に類似の配列が多数存在することを確認した。そして、該5クローンよりI2類似配列を持つDNA断片をサブクローニングし、それらの配列を解析し、候補配列について機能解析を行なうことにより、L3抵抗性遺伝子を同定した。さらに、該遺伝子の配列をもとにその他のL遺伝子をも同定し、これらの遺伝子配列自体を利用して各L遺伝子型を簡便に識別できる方法を確立し、本願発明を完成した。
【0012】
すなわち、本願発明は、配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列と90%以上の相同性を有し、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードするポリヌクレオチドを提供する。また、本発明は、配列表の配列番号2、4、6、8、10、12及び14のいずれかに示されるアミノ酸配列と90%以上の同一性を有し、植物にトバモウイルス抵抗性を付与できるポリペプチドを提供する。さらに、本発明は、上記本発明のポリペプチドをコードするポリヌクレオチドを提供する。さらに、本発明は、上記本発明のポリヌクレオチドを含み、植物にトバモウイルス抵抗性を付与できるポリペプチドを宿主細胞内で発現させることができる組換えベクターを提供する。さらに、本発明は、該組換えベクターを含む細胞を提供する。さらに、本発明は、該組換えベクターにより形質転換された形質転換植物を提供する。さらに、本発明は、該組換えベクターを植物細胞内に導入することを含む、トバモウイルス抵抗性が付与された形質転換植物の作製方法を提供する。さらに、本発明は、配列表の配列番号21に示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチド、及び該ポリヌクレオチドからなるトバモウイルス抵抗性遺伝子増幅用プライマーを提供する。さらに、本発明は、上記本発明のプライマーと、配列表の配列番号22に示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーとからなるトバモウイルス抵抗性遺伝子増幅用プライマーセットを提供する。さらに、本発明は、該プライマーセットを用いた核酸増幅法によって植物細胞より分離した核酸試料から増幅された、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードするポリヌクレオチドを提供する。さらに、本発明は、配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーを用いた核酸増幅法によって、植物細胞より分離した核酸試料からトバモウイルスL抵抗性遺伝子のコード領域の全長又は部分領域を増幅し、次いで該増幅断片の塩基配列を対比することを含む、L抵抗性遺伝子型の識別方法を提供する。
【発明の効果】
【0013】
本発明により、トバモウイルスL抵抗性遺伝子の塩基配列及び該塩基配列にコードされるアミノ酸配列が初めて提供された。本発明のLトバモウイルス抵抗性タンパク質は、これを発現させることによって植物にトバモウイルス抵抗性を付与できる。これは本発明のLトバモウイルス抵抗性遺伝子を植物発現ベクターに挿入し、植物を形質転換することによって達成される。また、本発明のLトバモウイルス抵抗性遺伝子は、本発明のプライマーによってトウガラシ属植物ゲノムDNAもしくはmRNAから増幅可能である。特に、Lトバモウイルス抵抗性遺伝子の各遺伝子型でコード領域の3'末端が保存されていることが本発明により明らかになったため、この保存領域を利用すれば未だ同定されていない新規なL遺伝子も単離することができる。単離したL遺伝子の塩基配列を決定し比較することによって、現時点で未同定のものも含め種々のL遺伝子型を識別できる。遺伝子型の識別は、遺伝子多型を検出する種々の方法(制限断片長多型解析、一塩基多型検出方法等)を利用して行なうことができる。
【発明を実施するための最良の形態】
【0014】
配列番号1〜14に示される塩基配列及びアミノ酸配列は、本願発明者らがトウガラシ属植物から単離したトバモウイルス抵抗性L遺伝子のコード領域の配列である。下記実施例に記載されるように、これらL遺伝子がコードするタンパク質のアミノ酸配列(配列番号2、4、6、8、10、12及び14)は、相同性が相互に非常に高く、97%以上の同一性及び99%以上の類似性を有している。本願発明者らが単離した各L遺伝子、その配列番号及びその由来のトウガラシ属植物名を表1に示す。
【0015】
【表1】

【0016】
トバモウイルスとは、一本鎖(+)RNAをゲノムとするトバモウイルス属の植物ウイルスである。具体例としては以下の表1に示すものが挙げられるが、これらに限定されない。この分野で周知の通り、L遺伝子にはそれぞれ対立遺伝子の関係にあるとされるL1〜L4の遺伝子型が知られており、それら遺伝子型とトバモウイルスの病原型の間には階層的な関係が成り立っている。その階層的関係も合わせて下記表2に示す。なお、L遺伝子には、L1cのように他のL遺伝子型と対立遺伝子の関係にあることが確認されていない亜型も存在する。このような亜型も本発明の範囲に包含される。
【0017】
【表2】

【0018】
本発明のポリヌクレオチドとしては、配列表の配列番号1、3、5、7、9、11及び13に示される塩基配列からなるポリヌクレオチド(以下、本明細書において、これらの塩基配列を便宜上「天然型配列」ということがある。これらにコードされるアミノ酸配列についても同様に「天然型配列」と言うことがある。)の他、これらの塩基配列のうち少数の塩基が置換し、欠失し及び/又は挿入された塩基配列からなるものであって、もとの配列と90%以上、好ましくは95%以上、より好ましくは97%以上の相同性を有する塩基配列からなるポリヌクレオチドも挙げられる。このような変異型の塩基配列からなるポリヌクレオチドであっても、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードする限り、本発明の範囲に包含される。変異型のポリヌクレオチドの好ましい例としては、上記した天然型L遺伝子配列のいずれかにおいて、1ないし数個の塩基が置換し、欠失し又は挿入された塩基配列からなるポリヌクレオチドも挙げられる。コドンには縮重があり、従って上記天然型の塩基配列がコードするアミノ酸配列は、上記天然型の塩基配列のみに限定されない。また、後述するように、コードされるアミノ酸配列にも少数のアミノ酸置換、欠失及び/又は挿入が許容されるため、そのような変異型アミノ酸配列をコードするポリヌクレオチドも本発明のポリヌクレオチドの範囲に包含される。
【0019】
ここで、塩基配列の「相同性」とは、比較すべき2つの塩基配列中の塩基ができるだけ多く一致するように両配列を整列させ、一致した塩基数を全塩基数で除したものを百分率で表したものである。上記整列の際には、必要に応じ、比較する2つの配列の一方又は双方に適宜ギャップを挿入する。このような配列の整列化は、例えばBLAST、FASTA、CLUSTAL W等の周知のプログラムを用いて行なうことができる。ギャップが挿入される場合、上記全塩基数は、1つのギャップを1つの塩基として数えた数となる。このようにして数えた全塩基数が、比較する2つの配列間で異なる場合には、相同性(%)は、長い方の配列の全塩基数で、一致した塩基数を除して算出される。
【0020】
ただし、塩基の変異は、L遺伝子型間で保存性の高い領域以外の領域内に存在することが好ましい。具体的には、例えばコード領域3'末端の33bpの領域以外で変異しているものが好ましい。下記実施例に記載されるように、L遺伝子はトマトのフザリウム抵抗性遺伝子I2の類似配列の中から見出されたが、コード領域3'末端の33bpはL遺伝子型間においてのみ同一となっており、L遺伝子で特異的に保存されている。この領域はL遺伝子の機能に重要である可能性が高い。
【0021】
ポリヌクレオチドとしては、DNAでもRNAでもよいが、安定性の観点からはDNAが好ましい。
【0022】
「植物にトバモウイルス抵抗性を付与できるポリペプチド」とは、植物体内で該ポリペプチドを発現させた場合に該植物がトバモウイルス抵抗性を示すようになることをいう。ポリペプチドが植物にトバモウイルス抵抗性を付与できるか否かは、例えば、トバモウイルスに感受性の植物組織(ベンサミアーナタバコ葉など)中でポリペプチドを発現させ、これに各種病原型のトバモウイルスを感染させて抵抗性反応が生じるか否かを調べることにより確認できる。各種の一過性発現系が周知であり、例えば下記実施例に記載されるように、適当な発現ベクター(pBA等)中にポリペプチドをコードするポリヌクレオチドを組み込み、該組換えベクターをアグロバクテリウムに導入し、ベンサミアーナタバコ葉等のトバモウイルス感受性植物組織に該アグロバクテリウムを注入すれば、植物組織中で容易に該ポリペプチドを発現させることができる。トバモウイルスの感染は、トバモウイルス粒子を常法により葉組織に接種することにより行なうこともできるし、あるいは、上記と同様にアグロバクテリウムを利用してトバモウイルス外被タンパク質を植物組織中で発現させることによって植物組織がウイルスに感染した状態を模倣することもできる。L遺伝子は所定の病原型のトバモウイルスに対して過敏感反応抵抗性(すなわち感染部位における細胞死及びウイルスの局在化)を示すため、ポリペプチドとトバモウイルス外被タンパク質が共存する部位で細胞死が生じた場合に該ポリペプチドは植物にトバモウイルス抵抗性を付与できると判断することができる。
【0023】
L遺伝子の単離により、これらがコードするL抵抗性タンパク質のアミノ酸配列も明らかとなった。上記天然型のL遺伝子がコードするアミノ酸配列は、配列表の配列番号2、4、6、8、10、12及び14に示される通りである。下記実施例に具体的に記載されるように、上記天然型のL遺伝子をトバモウイルス感受性の植物細胞内に導入してタンパク質を発現させると、トバモウイルスに対し抵抗性反応を示すようになる。すなわち、これらのアミノ酸配列からなるタンパク質は、植物にトバモウイルス抵抗性を付与する活性を有する。
【0024】
一般に、生理活性を有するポリペプチドは、そのアミノ酸配列を構成するアミノ酸のうち少数のアミノ酸が置換し、欠失し又は挿入された場合でもその生理活性が維持される場合があることは周知である。したがって、上記した天然型のアミノ酸配列(配列番号2、4、6、8、10、12及び14)のみならず、これら天然型のアミノ酸配列のうち少数のアミノ酸残基が置換され、欠失され、及び/又は挿入された配列からなるポリペプチドであって、植物細胞内で発現させた場合に該植物にトバモウイルス抵抗性を付与できるポリペプチドも本発明の範囲に包含される。このような変異型タンパク質のアミノ酸配列は、具体的には、天然型のアミノ酸配列(配列番号2、4、6、8、10、12及び14)のいずれかとの同一性が90%以上、好ましくは95%以上、より好ましくは97%以上である。なお、アミノ酸配列の「同一性」の定義は、上記した塩基配列の相同性と同様である。また、このような変異型のアミノ酸配列としては、上記天然型のアミノ酸配列において1ないし数個のアミノ酸残基が置換され、欠失され、及び/又は挿入されたアミノ酸配列からなるものも好ましい。
【0025】
ただし、塩基配列の変異と同様に、アミノ酸配列の変異も、複数のL遺伝子型の間で高度に保存されている領域以外の領域で生じていることが好ましい。具体的には、変異型のアミノ酸配列としては、上記したコード領域3'末端の33bpの領域(停止コドンを含む。アミノ酸配列でいえばC末端の10残基の領域。)以外の領域に変異が存在するものが好ましい。
【0026】
なお、天然のタンパク質を構成する20種類のアミノ酸は、低極性側鎖を有する中性アミノ酸(Gly, Ile, Val, Leu, Ala, Met, Pro)、親水性側鎖を有する中性アミノ酸(Asn, Gln, Thr, Ser, Tyr Cys)、酸性アミノ酸(Asp, Glu)、塩基性アミノ酸(Arg, Lys, His)、芳香族アミノ酸(Phe, Tyr, Trp)のように類似の性質を有するものにグループ分けでき、これらの間での置換であればポリペプチドの性質が変化しないことが多いことが知られている。従って、上記した変異型のLタンパク質の好ましい例としては、配列番号2、4、6、8、10、12及び14中のアミノ酸残基がこれら各グループ間で置換されたアミノ酸配列からなるものが挙げられる。アミノ酸配列の類似性でいえば、上記変異型のL抵抗性タンパク質は、天然型のL抵抗性タンパク質のアミノ酸配列との類似性が好ましくは95%以上、より好ましくは97%以上、さらに好ましくは98%以上、さらに好ましくは99%以上、最も好ましくは100%である。
【0027】
上記した本発明のポリヌクレオチドは、核酸合成機を用いて常法により合成することができるし、また、公知の遺伝子工学的手法を用いた常法により容易に調製することができる。上記した天然型のLタンパク質(L1, L1a, L1c, L2, L2b, L3, L4タンパク質)をコードするL遺伝子の塩基配列は、配列番号1、3、5、7、9、11、13に示される通りであるから、例えば、これらの配列情報をもとにプライマーを設計し、L遺伝子を有する植物のゲノムDNA又はcDNAを鋳型としてPCRを行うことで、Lタンパク質をコードするDNAを容易に調製することができる。下記実施例に示されるように、本願発明者らが単離したL遺伝子のコード領域にはイントロンが存在しないため、本発明のポリペプチドをコードするポリヌクレオチドはゲノムDNAからでも容易に調製することができる。PCR自体は周知の常法である。また、各種L遺伝子を有する植物も公知であり、例えば上記した表1に示されるトウガラシ属植物から各種L遺伝子を増幅することができる。増幅したL遺伝子の配列は、適宜塩基配列を決定して本願配列表に記載される塩基配列と比較し、L遺伝子型を確認してもよい。なお、L遺伝子型の確認は、後述する本願発明者らが開発したL遺伝子型検出方法により容易に行なうことができる。
【0028】
変異型の塩基配列からなるポリヌクレオチドは、変異を有する植物個体から上記方法により取得することもできるし、また、市販の核酸合成機を用いて常法により合成することもできる。あるいはまた、この分野で公知の変異導入法を用いて天然型遺伝子配列に変異を導入して調製することもできる。遺伝子に変異を導入する具体的な方法としては、例えば、Kunkel法又は Gapped duplex法等の公知手法又はこれに準ずる方法を挙げることができ、そのためのキットも種々のものが市販されている。そのようなキットの具体例としては、例えば部位特異的突然変異誘発法を利用した変異導入用キット(Mutant-K(TAKARA社製)やMutant-G(TAKARA社製)等)、TAKARA社のLA PCR in vitro Mutagenesis シリーズキット等が挙げられる。
【0029】
本発明のポリペプチドは、ペプチド合成機を用いて常法により合成することもできるし、また公知の遺伝子工学的手法を用いた常法により容易に調製することができる。具体的には、上記の通りに調製したポリヌクレオチドを発現ベクターに組み込み、適当な宿主細胞に導入し、該宿主細胞中でポリペプチドを生産させることにより、目的とするポリペプチドを得ることができる。各アミノ酸をコードするコドンは公知であるから、特定のアミノ酸配列をコードするポリヌクレオチドの塩基配列は容易に特定することができる。また、任意の塩基配列からなるポリヌクレオチドは、核酸合成機を用いた常法により或いは天然型配列に変異を導入することにより容易に調製することができる。従って、任意の変異型L抵抗性タンパク質の調製も容易に行なうことができる。
【0030】
本発明は、上記した本発明のポリヌクレオチドを含み、植物にトバモウイルス抵抗性を付与できるポリペプチドを宿主細胞内で発現することができる組換えベクターも提供する。さらに、該組換えベクターを含む細胞も提供する。該組換えベクターは、例えば上記したように本発明のポリペプチドを調製するために利用することができる。また、植物細胞内で本発明のポリペプチドを発現することのできるベクターに導入した場合には、トバモウイルス抵抗性を付与した形質転換植物の作製に利用することができる。
【0031】
本発明の組換えベクターは、適当なベクターに本発明のポリヌクレオチドとその発現を制御する配列を挿入することによって得ることができる。組換えベクターの調製方法自体は周知の常法である。具体的には、ベクターに本発明のポリヌクレオチドを挿入するには、まず、精製されたポリヌクレオチドを適当な制限酵素で切断し、適当なベクターの制限酵素部位又はマルチクローニングサイトに挿入してベクターに連結する方法などが採用される。ポリヌクレオチドは、コードするポリペプチドが宿主細胞中で発現されるようにベクターに組み込まれることが必要である。そこで、本発明の組換えベクターには、プロモーター、本発明のポリヌクレオチドのほか、所望によりエンハンサーなどのシスエレメント、スプライシングシグナル、ポリA付加シグナル、選択マーカー、リボソーム結合配列(SD配列)などを含有するものを連結することができる。なお、選択マーカーとしては、例えば除草剤耐性遺伝子、カナマイシン耐性遺伝子、ハイグロマイシン耐性遺伝子等が挙げられるが、これらに限定されない。
【0032】
用いるベクター及び宿主細胞の種類は特に限定されず、この分野で公知のいかなるベクター及び宿主細胞を用いることもでき、目的に応じて自由に選択することができる。具体的には、例えば宿主細胞としてはタバコ(Nicotiana tabacum)、トマト(Solanum lycopersicum)、イネ(Oryza sativa)などの植物;大腸菌(Escherichia coli)、バチルス・ズブチリス(Bacillus subtilis)、シュードモナス・プチダ(Pseudomonas putida)、リゾビウム・メリロティ(Rhizobium meliloti)等の細菌;サッカロミセス・セレビシエ(Saccharomyces cerevisiae)、シゾサッカロミセス・ポンベ(Schizosaccharomyces pombe)等の酵母;サル細胞COS-7、Vero、チャイニーズハムスター卵巣細胞(CHO細胞)、マウスL細胞、ヒトGH3、ヒトFL細胞等の動物細胞;あるいはSf9、Sf21等の昆虫細胞が挙げられる。ベクターは種々のものが公知であり、市販品も多く存在する。当業者であれば、宿主細胞の種類に応じて適切なベクターを容易に選択することができる。例えば、形質転換植物の作製に用いる組換えベクターであれば、アグロバクテリウムに導入して用いるTiプラスミド由来のバイナリーベクター等を好ましく用いることができる。
【0033】
植物を宿主とする場合は、本発明の組換えベクターが植物細胞内で核ゲノムに組み込まれることが望ましいが、これに限定されない。また、プロモーターとしては、例えばシロイヌナズナ(Arabidopsis thaliana)アクチン2プロモーター等の植物由来プロモーター、カリフラワーモザイクウイルス(CaMV)の35Sプロモーターなどの植物ウイルス由来プロモーターが挙げられる。ポリアデニル化シグナルとしては、アグロバクテリウムTiプラスミド由来のノパリン合成酵素遺伝子のポリAシグナル、カリフラワーモザイクウイルスのポリAシグナル配列などが挙げられる。宿主として用いる植物の種類は特に限定されず、タバコ、トマト、ピーマン、イネ等の形質転換可能ないずれの植物でも好ましく用いることができる。
【0034】
大腸菌等の細菌を宿主とする場合は、本発明の組換えベクターが該細菌中で自律複製可能であると同時に、プロモーター、リボゾーム結合配列、本発明のポリヌクレオチド、転写終結配列により構成されていることが好ましい。また、プロモーターを制御する遺伝子が含まれていてもよい。大腸菌としては、例えばエッシェリヒア・コリ(Escherichia coli)K12、DH1などが挙げられ、枯草菌としては、例えばバチルス・ズブチリス(Bacillus subtilis)などが挙げられる。プロモーターとしては、大腸菌等の宿主中で発現できるものであればいずれを用いてもよい。例えばtrpプロモーター、lacプロモーター、PLプロモーター、PRプロモーターなどの、大腸菌やファージに由来するプロモーターを用いることができる。tacプロモーターなどのように、人為的に設計改変されたプロモーターを用いてもよい。細菌への組換えベクターの導入方法としては、細菌にDNAを導入する方法であれば特に限定されるものではない。例えばカルシウムイオンを用いる方法(Cohen, S.N. et al. (1972) Proc. Natl. Acad. Sci., USA 69, 2110-2114)、エレクトロポレーション法等が挙げられる。
【0035】
動物細胞を宿主とする場合は、例えば、サル細胞COS-7、Vero、チャイニーズハムスター卵巣細胞(CHO細胞)、マウスL細胞、ラットGH3、ヒトHeLa、FL細胞などを用いることができる。用いるプロモーターとしては、SRαプロモーター、SV40プロモーター、LTRプロモーター、CMVプロモーター等を挙げることができる。ヒトサイトメガロウイルスの初期遺伝子プロモーター等を用いてもよい。動物細胞への組換えベクターの導入方法としては、例えばエレクトロポレーション法、リン酸カルシウム法、リポフェクション法等が挙げられる。
【0036】
昆虫細胞を宿主とする場合は、例えばSchneider 2(S2) 細胞、Sf9細胞、Sf21細胞などを用いることができる。昆虫細胞への組換えベクターの導入方法としては、例えばリン酸カルシウム法、リポフェクション法、エレクトロポレーション法などが挙げられる。
【0037】
また、上記の各宿主細胞への遺伝子導入は、組換えベクターによらない方法、例えばパーティクルガン法なども用いることができる。
【0038】
植物細胞中で発現可能なベクターを用いた本発明の組換えベクターは、トバモウイルス抵抗性が付与された形質転換植物の作製に用いることができる。そのようなベクターとしては、例えば上記したようにアグロバクテリウムのTiプラスミドやRiプラスミド由来のバイナリーベクターが挙げられる。アグロバクテリウムを利用した植物の形質転換方法自体は周知の常法である。具体的には、バイナリーベクターの所定の領域に本発明のポリヌクレオチドを挿入し、得られた組換えベクターをエレクトロポレーション法等によりアグロバクテリウム中に導入し、これを植物細胞と共培養して植物細胞に感染させることで、バイナリーベクター中に組み込んだ本発明のポリヌクレオチドを植物細胞のゲノム中に組み込むことができる。該植物細胞を常法により培養して植物体を分化させれば、トバモウイルス抵抗性が付与された形質転換植物を得ることができる。
【0039】
さらに、本発明により、L遺伝子のコード領域中の3'末端33bpの領域がL遺伝子型間で高度に保存されており、L遺伝子に特異的な配列であることが見出された。この33bpの塩基配列を配列表の配列番号21に示す。本発明は、この配列番号21に示される塩基配列中の部分領域に特異的にハイブリダイズするポリヌクレオチドも提供する(以下、該ポリヌクレオチドを便宜的に「検出用ポリヌクレオチド」と呼ぶ)。該検出用ポリヌクレオチドは、例えばトバモウイルス抵抗性遺伝子増幅用プライマーとして有用であり、各種遺伝子型のL遺伝子の増幅・検出に用いることができる。また、該検出用ポリヌクレオチドは、プローブとしてトバモウイルス抵抗性遺伝子の検出や単離に利用することもできる。
【0040】
L遺伝子はトマトI2遺伝子のホモログとして見出されたものであるが、公知のI2ホモログの多くにおいてコード領域の5'末端側(開始コドンから30bpの領域)が保存された領域となっており、L遺伝子型間でも同様に保存されている。この30bpの領域を配列表の配列番号22に示す。この5'末端側の保存領域(配列番号22)中の部分領域に特異的にハイブリダイズするプライマーと、上記トバモウイルス抵抗性遺伝子増幅用プライマーとからなるプライマーセットは、トバモウイルス抵抗性遺伝子増幅用プライマーセットとして有用であり、該プライマーセットによれば、植物細胞から分離した核酸試料より種々の遺伝子型のL遺伝子を増幅することができる。PCR等の核酸増幅法は周知の常法であり、当業者であれば増幅サイズ等に応じて適宜反応条件を定めることができる。
【0041】
ここで、「特異的にハイブリダイズする」とは、通常のハイブリダイズの条件下において、対象とする部分領域とのみハイブリダイズし、その他の領域とは実質的にハイブリダイズしないという意味である。
【0042】
「通常のハイブリダイズの条件下」とは、通常のPCRのアニーリングやプローブによる検出に用いられる条件下のことをいい、例えば、Taqポリメラーゼを用いたPCRの場合には、50mM KCl、10mM Tris-HCl(pH 8.3〜9.0)、1.5mM MgCl2といった一般的な緩衝液を用いて、54℃〜60℃程度の適当なアニーリング温度で反応を行なうことをいい、また、例えばノーザンハイブリダイゼーションの場合には、5 x SSPE、50%ホルムアミド、5 x Denhardt's solution、0.1〜0.5%SDSといった一般的なハイブリダイゼーション溶液を用いて、42℃〜65℃程度の適当なハイブリダイゼーション温度で反応を行なうことをいう。ただし、適当なアニーリング温度又はハイブリダイゼーション温度は、上記例示に限定されず、プライマー又はプローブとして用いるポリヌクレオチドのTm値及び実験者の経験則に基づいて定められ、当業者であれば容易に定めることができる。
【0043】
「実質的にハイブリダイズしない」とは、全くハイブリダイズしないか、するとしても対象とする部分領域にハイブリダイズする量よりも大幅に少なく、相対的に無視できる程度の微量しかハイブリダイズしないという意味である。そのような条件下で特異的にハイブリダイズするポリヌクレオチドとしては、対象の部分領域の塩基配列と一定以上の相同性を有するポリヌクレオチドが挙げられ、例えば70%以上、好ましくは80%以上、より好ましくは90%以上、さらに好ましくは93%以上、さらに好ましくは95%以上、さらに好ましくは98%以上の相同性を有するポリヌクレオチドが挙げられる。最も好ましくは、該ポリヌクレオチドは、対象の部分領域の塩基配列と同一の塩基配列からなる。相同性の定義は上記と同様である。また、検出用ポリヌクレオチドの末端に対象とハイブリダイズしない領域が含まれていても、プローブの場合には、ハイブリダイズする領域がプローブ全体のおよそ半分以上を占めていれば検出に用いることができるし、また、プライマーの場合には、ハイブリダイズする領域がプライマー全体のおよそ半分以上を占め、かつ3'末端側にあれば、5'末端側に任意の付加配列があっても正常にアニーリングして伸長反応を生じ得るので、検出に用いることができる。例えば、プライマーの5'側に制限酵素認識配列等を付加して用いることはこの分野で周知の常法である(具体例としては、配列番号24に示す塩基配列からなるプライマー等。下記実施例参照。)。そのように、検出用ポリヌクレオチドの末端にハイブリダイズしない領域が含まれている場合において、対象の塩基配列との相同性を算出するときは、ハイブリダイズしない末端の領域は考慮せず、ハイブリダイズする領域のみに着目して算出するものとする。
【0044】
ただし、この分野で周知のように、数塩基程度のミスマッチを含むミスマッチプライマーを用いたPCRにより増幅断片に所望の変異を導入することができる。このような手法は周知の常法であり、後述のPIRA-PCR法においてもミスマッチプライマーが用いられる(具体例としては、配列番号19に示す塩基配列からなるプライマー等。下記実施例参照。)。従って、プライマーの配列としては対象の部分領域の塩基配列と同一のものが必ずしも好ましいわけではなく、目的に応じて適宜ミスマッチを含む塩基配列が選択される。当業者であれば、本願配列表に記載の塩基配列を参照し、そのようなミスマッチプライマーを目的に応じて容易に設計することができる。
【0045】
なお、本発明において、塩基配列中の「部分領域」とは、配列表に示される塩基配列中の一部の領域を言い、好ましくは連続する18塩基以上の領域である。また、本発明において、「配列番号1に示される塩基配列」と言った場合には、配列番号1に実際に示されている塩基配列の他、これと相補的な配列も包含する。従って、例えば「配列番号1に示される塩基配列からなるポリヌクレオチド」と言った場合には、配列番号1に実際に示されている塩基配列からなる一本鎖ポリヌクレオチド、その相補的な塩基配列からなる一本鎖ポリヌクレオチド、及びこれらから成る二本鎖ポリヌクレオチドが包含される。「配列番号21に示される塩基配列中の連続する18塩基以上からなるポリヌクレオチド」と言った場合も同様であり、配列番号21中に実際に示されている塩基配列と相補的な配列中の連続する18塩基以上からなるポリヌクレオチドも包含される。本発明のポリヌクレオチド及び検出用ポリヌクレオチドを調製する場合や、本発明のポリペプチドをコードするポリヌクレオチドを調製する場合には、適宜いずれかの塩基配列を選択することとなるが、当業者であれば容易にその選択をすることができる。
【0046】
検出用ポリヌクレオチドの塩基数は、特異性を確保する観点から、18塩基以上が好ましい。サイズは、プローブとして用いる場合には、好ましくは18塩基以上、さらに好ましくは20塩基以上が好ましく、プライマーとして用いる場合には、18塩基以上が好ましい。検出用ポリヌクレオチドの好ましい例としては、配列番号21に示される塩基配列中の連続する18塩基以上からなるポリヌクレオチドが挙げられ、より具体的には配列番号16に示される塩基配列からなるポリヌクレオチドが挙げられるが、これに限定されない。
【0047】
上記したトバモウイルス抵抗性遺伝子増幅用プライマーの好ましい例としては、配列番号16に示される塩基配列からなるプライマーが挙げられる。また、トバモウイルス抵抗性遺伝子増幅用プライマーセットとして該プライマーと組み合わせて用いられるプライマーのうち好ましい例としては、配列番号22に示される塩基配列中の連続する18塩基以上からなるプライマー、特に配列番号15に示される塩基配列からなるプライマーが挙げられるが、これに限定されない。
【0048】
さらにまた、本発明により各種遺伝子型のL遺伝子配列が同定されたため、植物細胞から得た核酸試料よりL遺伝子中の所望の領域を増幅し、その配列を対比することによってL遺伝子型を識別することが可能となった。すなわち、本発明は、配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーを用いた核酸増幅法によって、植物細胞より分離した核酸試料からトバモウイルスL抵抗性遺伝子のコード領域の全長又は部分領域を増幅し、次いで該増幅断片の塩基配列を対比することを含む、L抵抗性遺伝子型の識別方法をも提供する。
【0049】
核酸試料は、ゲノムDNA、mRNA、cDNAのいずれでもよい。下記実施例に示されるように、L遺伝子のコード領域中にはイントロンが存在しないため、抽出作業及び取扱いの簡便性から核酸試料としてはゲノムDNAが好ましい。
【0050】
核酸増幅法としては、周知の常法であるPCR法を好ましく採用することができる。
【0051】
上記したように、5'末端の保存領域と3'末端の保存領域とにプライマーを設定すれば、種々のL遺伝子型をいずれも増幅できる。従って、このようなプライマーを用いてL遺伝子のコード領域全長を増幅し、増幅断片の塩基配列を対比してもよい。あるいは、各遺伝子型で指標とすべき配列部分(多型部分)を含む部分領域のみを増幅し、その塩基配列を対比してもよい。
【0052】
塩基配列の対比は、増幅断片の塩基配列全長を決定して対比してもよいし、また、各遺伝子型に特徴となる一塩基多型等の多型を指標として対比を行なってもよい。遺伝子多型を検出する方法は公知であり、例えばPIRA(Primer-introduced restriction analysis)-PCR法、インベーダー法等を挙げることができる。また、制限酵素断片長多型解析を用いて遺伝子配列の多型を検出・識別することもできる。PIRA-PCR法とは、ミスマッチプライマーを用いて一塩基多型部位に人為的に制限酵素サイトを導入し、制限酵素による切断の有無を指標として一塩基多型を検出する方法であり、この分野で公知の常法である。
【0053】
例えば、配列番号1に示すL3遺伝子配列中の3070nt〜3982ntの領域内には、L2、L2b、L3、L4及びL1c遺伝子型を識別可能な多型が存在する。この多型は、TaqI切断パターン及びHhaI切断パターンに基づいて識別することができる。この領域内の多型を利用した識別方法は下記実施例に詳述される通りであり、具体的には、配列番号17及び18に示す塩基配列からなるプライマーセットを用いて核酸試料から増幅断片を得て、これをTaqIとHhaIとでそれぞれ切断し、以下の表3に示す消化断片パターンを指標としてL2、L2b、L3、L4及びL1c遺伝子型を識別することができる。
【0054】
【表3】

表中の数値は消化断片のサイズ(bp)を示す
【0055】
また、例えば、配列番号5に示すL1遺伝子配列中の3779ntには、L1のみを他の遺伝子型と区別できる一塩基多型が存在する。該多型は、配列番号19および20に示す配列のプライマーセットを用いたPIRA-PCR法による解析においてXmnI切断部位が生じるか否かによって識別することができる。配列番号19のプライマーにはミスマッチが含まれており、これを用いたPCRによればL1由来の増幅断片にのみXmnI切断部位が生じ、他の遺伝子型(L1a, L1c, L2, L2b, L3, L4)ではXmnI切断部位が生じない。この一塩基多型を利用したPIRA-PCR法による識別方法は下記実施例に詳述される通りであり、具体的には、配列番号19及び20に示す塩基配列からなるプライマーセットを用いて核酸試料から増幅断片を得て、該増幅断片をXmnIで消化し、29bpと117bpの断片に切断されるものをL1と識別することができる。該方法では、増幅効率と識別精度を高める観点から、nested PCRにより増幅断片を得ることが好ましい。その場合、配列番号19及び20に示す塩基配列からなるプライマーセットは第2段階のPCRに用いることができる。第1段階のPCRでは、該プライマーセットにより増幅される領域を含む部分領域を増幅できるものであれば特に限定されないが、例えば配列番号17及び18に示す塩基配列からなるプライマーセットを好ましく用いることができる。また、PIRA-PCR法では、プライマー配列中に標的とは異なる配列を含む、すなわちミスマッチを含むため、増幅に用いる酵素は校正機能のないものを用いる必要があり、例えばTaqポリメラーゼを用いることが好ましい。
【0056】
L1a型については、コード領域全長のXbaI切断パターンによって他の遺伝子型と識別することができる。L1a型では、コード領域全長がXbaIにより2433bp、1080bp、474bpの3つの断片に切断される。他の遺伝子型ではXbaI消化により得られる断片は2つである。
【0057】
上記識別方法を適宜組み合わせれば、各種L遺伝子型を全て識別することができる。例えば、ある核酸試料について、まずTaqI切断パターン及びHhaI切断パターンに基づいてL2、L2b、L3、L4及びL1cのいずれであるかを調べ、不明である場合には上記した3779ntの一塩基多型により又はコード領域全長のXbaI切断パターンによりL1かL1aかを識別することが可能である。
【実施例】
【0058】
以下、実施例に基づき本発明をより具体的に説明する。もっとも、本発明は下記実施例に限定されるものではない。
【0059】
(実施例1)L3抵抗性遺伝子のクローニング
L3遺伝子は、Capsicum chinenseのいくつかのアクセションが保有しているトバモウイルス抵抗性遺伝子である。これまでのマッピングの結果から、L遺伝子座は、トマトの11番染色体とシンテニーを持つカプシクム属植物の11番染色体に座乗していることが知られていた。しかし、これまでに得られていたL3と連鎖する遺伝子マーカーは、L3と離れたものや、あるいは分離しないと報告されていても調査個体数が少なく信頼性の低いものであり、ポジショナルクローニングを可能とする、L3を挟み込む隣接したマーカーは得られていなかった。そこで我々は、抵抗性遺伝子類似配列及びBAC末端配列に基づくマーカーを開発し、L3が2つのBACコンティグ(C. chinense PI159236のゲノムライブラリー由来;計5クローンにより構成)もしくはその間の未知領域に含まれること、その未知領域が約30 kb程度であること、以上の結果を考え合わせるとL3は約400 kbの領域に存在することを突き止めた(図1)(Tomita et al, Theor Appl Genet (2008) 117:1107-1118)。また、この領域にトマトのフザリウム抵抗性遺伝子I2に類似の配列が多数存在することを確認した。
【0060】
以上の結果から、5クローンのBACクローン中に存在するI2類似配列のいずれかがL3である可能性を考え、それら5つのBACクローンを制限酵素EcoRIまたはHindIIIで切断し、ピーマン由来I2類似配列の混合物をプローブとして用いたサザンブロッティング法によって検出し、I2類似配列を持つDNA断片をサブクローニングした。それらの配列を決定したところ、10配列中ただ1つ、I2類似アミノ酸配列をコードしうるものを発見した。この配列から遺伝子の3' 端と考えられた配列およびI2類似配列で保存されている5' 端の配列をPCRプライマーセットとして(それぞれ配列番号16及び15)、この遺伝子候補の全長をBACクローン185CA10から増幅し、その塩基配列を決定した。この遺伝子(PIH7)は、CC-NBS-LRR型の抵抗性遺伝子産物をコードしており、I2とのアミノ酸配列の同一性は71.6%、類似性は92.3%であった。PIH7の塩基配列及びアミノ酸配列をそれぞれ配列表の配列番号1及び2に示す。
【0061】
その後、5つのBACクローンを全て塩基配列決定したところ、上記PIH7遺伝子が、それらBACクローン上のL3がマッピングされた領域中に存在する唯一の抵抗性遺伝子様の遺伝子であることが分かった(図2)。サザンブロッティング法で検出されたI2類似配列のうち、PIH7遺伝子を除く全てが偽遺伝子であった。また、PIH7が存在するBACクローン185CA10上のL3がマッピングされた領域外にPIH7と76.0%の同一性、92.0%の類似性を示すタンパク質をコードする遺伝子が存在した。以上の結果から、PIH7がL3の有力な候補と考えられた。
【0062】
(実施例2)一過性発現によるL3抵抗性遺伝子機能の解析
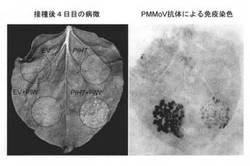
L3の有力な候補遺伝子であるPIH7について、トバモウイルス抵抗性を付与する機能を有すか否か、ベンサミアーナタバコ(Nicotiana benthamiana)における一過性発現系を用いて検討した。制限酵素認識配列を付加したプライマー(配列番号23および24)を用いてBACクローン185CA10からPIH7を増幅したのち、バイナリーベクターpBAのシロイヌナズナのアクチン2遺伝子プロモーターの下流に挿入し(図3)、アグロバクテリウムに形質転換した。これを単独またはPMMoV-IwのアグロインフェクションプラスミドpBTPIWを持つアグロバクテリウムと混合して、ベンサミアーナタバコ葉に注入した。対照実験として、pBTPIWとpBAを持つアグロバクテリウムの混合物、およびpBAを持つアグロバクテリウムを単独で注入した。その結果、注入後3日目からpBAPIH7とpBTPIWの両方を導入した領域で過敏感細胞死様の反応が認められ、注入4日目で細胞死は肉眼で確認できる程度に明確になった(図4)。この領域におけるPMMoV感染の広がりを、ハンマーブロッティング法によってpBYPIWとpBA共導入区と比較したところ、pBAPHI7による明らかなPMMoV感染拡大の抑制が認められた(図4)。このことから、PIH7がL3遺伝子の実体であることが確認された。
【0063】
(実施例3)L3抵抗性遺伝子の対立遺伝子および新規L遺伝子のクローニング
L3のマッピングに際して、マーカーを作製する目的で抵抗性遺伝子様部分配列を多数単離した。その中にL3自体(PIH7)があったが、その配列のホモログをマッピングに用いた交配相手であるCapsicum frutescensから単離したところ、ほとんど多型がなく、マーカー開発には至らなかった。そこで、そのC. frutescens由来の配列が、この植物の持つL遺伝子L2ではないかと考え、同配列を、L1を持つCapsicum annuum Verbeterde Glas、L1aを持つC. annuum KC780、L2様の特性の遺伝子L2bを持つCapsicum baccatumおよびL抵抗性遺伝子を持たないと考えられる感受性のC.annuum昌介のゲノムDNAから増幅した。この際、配列番号27および28に示すプライマーを用いた(PCRの反応条件:94℃20秒、56℃20秒、72℃1分の35サイクル、酵素としてタカラバイオ社のTaKaRa ExTaq DNA polymeraseを使用した)。それらDNA断片の塩基配列を比較したところ、感受性の昌介を除く全ての供試植物が相同性の高い配列を持つことが分かった。そこで、L1、L1a、L2、L2bおよびL3を持つ植物から全RNAを調製し、3'-RACE PCR法によって解析した。この際に用いた遺伝子特異的プライマーは上述の配列で保存されている領域内にデザインした(配列番号29および30;PCRの反応条件:第一段階、配列番号29のプライマー使用、94℃20秒、60℃20秒、72℃2分の35サイクル、第二段階、配列番号30のプライマー使用、94℃20秒、60℃20秒、72℃2分の25サイクル、酵素としてタカラバイオ社のTaKaRa ExTaq DNA polymeraseを使用した)。
【0064】
L遺伝子は、L3遺伝子の構造からコード領域内にイントロンを持たないことが推定されていたが、3'非コード領域内には多くのイントロンがオルタナティブなスプライシングを受け、多様な構造を示した。しかし、コード領域の3'端の配列33塩基対(配列番号21)は、L1、L1a、L2、L2bおよびL3でまったく同一であった(図5)。I2のホモログの多くが保存された5'端(開始コドンから30塩基対)を持つこと、既知のI2ホモログ間で3'端は保存されていないことから、3'端はL遺伝子型間でのみ高度に保存されていると考えられた。そこで、I2ホモログに共通した5'端配列とL遺伝子型間で保存された3'端配列(配列番号15および16)をプライマーセットとし、各種Capsicum属植物のゲノムDNAから遺伝子全域の増幅を試みた(PCRの反応条件:94℃30秒、60℃30秒、68℃4分の45サイクル、酵素としてタカラバイオ社のPrimeSTAR GXL DNA polymeraseを使用した)。
【0065】
その結果、上記5種類のL遺伝子の塩基配列が得られた(L3、配列番号1; L1、配列番号5; L1a、配列番号7; L2、配列番号11; L2b、配列番号13)。また、L4を持つCapsicum chacoensの2つのアクセションから独立に同一の塩基配列(配列番号3)が得られた。さらに、L1様の遺伝子L1cを持つC. chinense KC678からは、得られたL遺伝子の中でL3と最も近縁ではあるが、この遺伝子だけに特異的な6塩基の挿入とその周囲に集中した変異を持つ塩基配列(配列番号9)が得られた。いずれもコード領域にイントロンは存在しなかった。また、L4及びL1cにおいても、コード領域3'端の33bpの配列は同一であった。得られたL遺伝子型間の塩基配列から推定されるアミノ酸配列(L1、配列番号6; L1a、配列番号8; L1c、配列番号10; L2、配列番号12; L2b、配列番号14; L3、配列番号2; L4、配列番号4)の同一性および類似性(%)を下記表4に示す。
【0066】
【表4】

【0067】
以上の結果から、各L遺伝子型は相互に相同性の高い遺伝子であり、それらは全て同じプライマーセット(配列番号15および16)によって増幅できることが分かった。また、L1cのように遺伝分析で他のL遺伝子型の対立遺伝子であることの確かめられていないものも得られたことから、本法によってトウガラシ属野生種や他の属からも新たなトバモウイルス抵抗性遺伝子が発見できると期待される。L3タンパク質のN-およびC-端にHAやcMycなどのエピトープタグを付加した場合、L3の機能が完全に消失した。このことはLタンパク質のN-およびC-端、すなわちL遺伝子の5'および3'端が機能的制約によって高度に保存されていることを示唆しており、Lと類似の機能的なトバモウイルス抵抗性遺伝子は、配列番号15および16のプライマーセットを用いて増幅できると考えられる。
【0068】
(実施例4)L3ホモログの機能解析
上記で得られた各遺伝子型Lタンパク質の機能をL1、L2、L3およびL4について一過性発現系を用いて検討した。各L遺伝子型を、配列番号15および16のプライマーに制限酵素認識部位を付加したプライマーセット(配列番号23および24)を用いて増幅し(PCRの反応条件:94℃30秒、60℃30秒、68℃4分の1サイクルおよび94℃30秒、60℃30秒、68℃4分の44サイクル、酵素としてタカラバイオ社のPrimeSTAR GXL DNA polymeraseを使用した)、発現ベクターpBAに挿入し、アグロバクテリウムを形質転換した。これらを、各病原型トバモウイルスの外被タンパク質(CP)を発現するバイナリーベクターpGT-ToCP(ToMV, P0)、pGT-PaCP(PaMMV, P1)、pGT-PIWCP(PMMoV, P1,2)、pGT-PM13F66V(PMMoV, P1,2,3)、pGT-PM43K50G(PMMoV, P1,2,3)、pGT-PM81S(PMMoV, P1,2,3)、pGT-PM138N(PMMoV, P1,2,3)またはpGT-PM46R85K(PMMoV, P1,2,3,4)を持つアグロバクテリウムと混合し、ベンサミアーナタバコ葉に注入した。その結果を図6に示す。L1はToCP(P0)と共発現させた場合にのみ抵抗反応による細胞死を引き起こした。L2はToCP(P0)またはPaCP(P1)と共発現させた場合に同様の抵抗反応を示した。L3はToCP(P0)、PaCP(P1)及びPIWCP(P1,2)のいずれかと共発現させた場合に同様の抵抗反応を示した。L4はPM46R85(P1,2,3,4)以外のいずれかと共発現させた場合に同様の抵抗反応を示した。以上の結果は、配列番号15および16のプライマーセット並びに配列番号23および24のプライマーセットで増幅した遺伝子がL遺伝子として機能することを示す。また、各遺伝子型Lタンパク質はそれぞれ一種類のポリペプチドで、それぞれが異なる範囲のトバモウイルス外被タンパク質を認識することが示された。
【0069】
(実施例5)L抵抗性遺伝子型の識別
交配育種によって既知のL遺伝子型を品種に導入しようとする時、それぞれを識別できる遺伝子マーカーがあると、選抜が容易になる。通常使用される遺伝子マーカーは、必ずしも注目する形質を規定する遺伝子そのものではないが、目的形質と強く連鎖するものであれば遺伝子マーカーとして有用である。しかしながら、遺伝子そのものではないため、目的形質と分離する可能性は否定できない。従って、遺伝子がクローン化されており、植物系統間で多型のある場合は、遺伝子そのものを用いた方が確実である。そこで、上述の単離した各L遺伝子型を簡便に識別する方法の開発を試みた。
【0070】
全長のコード領域の塩基配列を比較すれば、当然識別は可能である。そこでまず、配列番号15および16のプライマーセットでゲノムDNAから増幅した全長の各L遺伝子型について、制限酵素分析によって識別することを試みた。その結果、L1cとL3との識別、及びL2bとL4との識別ができないことが分かった(表5)。
【0071】
【表5】

【0072】
そこで、L遺伝子の部分配列を配列番号17および18のプライマーセットで増幅し、4ベースカッターで切断する方法を試みた(PCRの反応条件:94℃20秒、58℃20秒、72℃1分の35サイクル、酵素としてタカラバイオ社のTaKaRa ExTaq DNA polymeraseを使用した)。その結果、Taq I切断断片およびHhaI切断断片を比較することによって、L1とL1a以外の遺伝子はこの方法で識別できることが分かった(図7及び表6)。なお、配列番号17及び18のプライマーセットは、配列番号1に示すL3遺伝子配列中の3070nt〜3982ntの領域を増幅するものである。
【0073】
【表6】

【0074】
L1とL1aは、全長増幅産物のXba I切断によって識別可能であるが、全長配列の増幅は効率が高くないため、配列番号17および18のプライマーセットで増幅した断片に含まれる一塩基多型を検出する方法を開発した。配列番号17および18のプライマーセットで増幅した断片を鋳型とし、配列番号19および20を用いて再度増幅し(PCRの反応条件:94℃10秒、60℃10秒、72℃30秒の25サイクル、酵素としてタカラバイオ社のTaKaRa Taq DNA polymeraseを使用した)、その146塩基対の増幅産物を制限酵素Xmn Iで処理すると、L1由来の増幅産物だけが切断されて29および117塩基対になる(図7、PIRA-PCR)。この鎖長の違いを電気泳動で検出し、L1とL1aを識別できる。
【0075】
(実施例6)L遺伝子を用いたウイルス抵抗性植物の育成
トバモウイルス抵抗性植物を遺伝子組換え法によって作製するため、タバコ(Nicotiana tabaccum)SR1をpBAPIH7 (L3)を用いて形質転換した。形質転換したタバコ(T0世代)からRNAを抽出し、RT-PCR法で導入遺伝子の発現を検討した。プライマーセットとしてAtACT2-616FおよびL3C-S19(それぞれ配列番号25及び26)を使用した(PCRの反応条件:94℃30、60℃30、72℃45の25サイクル、酵素としてタカラバイオ社のTaKaRa ExTaq DNA polymeraseを使用した)。PCRの標的領域がイントロンを含むため、ゲノムDNAまたは未スプライシングRNAに由来する約1.2 kbのバンド(genomic fragment)と、スプライシングを受けたRNAに由来する約0.6 kbのバンド(cDNA fragment)が認められた(図8)。
【0076】
導入遺伝子の発現が確認できたT0世代30個体について、ToMV(30μg/ml)を接種し、接種7日後に病徴観察及び接種葉の葉柄のティッシュプリント、並びに接種10日後に非接種上位葉のハンマーブロッティング免疫染色を行ない、接種葉および上位葉におけるウイルスの増殖を検討した。その結果、17個体において明確な抵抗性が認められた。
【0077】
これまでLタンパク質の検出法が確立していないので、抵抗性の程度とLタンパク質の蓄積量の間の関係については不明であるが、高率で抵抗性個体が得られたことから、この方法によってトバモウイルス抵抗性植物を作製できると考えられる。これまで安定形質転換体としてはL3を発現するもののみを作製したが、他のL遺伝子型も一過性発現系で抵抗性遺伝子機能を確認できており、さらにそれらが様々な変異を異なる組み合わせで持っている事実は、L遺伝子型のキメラ形成や点変異導入によってLトバモウイルス抵抗性遺伝子の機能を改良することが可能であることを示す。
【図面の簡単な説明】
【0078】
【図1】L3遺伝子の精密マッピングを説明する図である。A. 連鎖解析の結果を示す。矢印はマーカーの位置とL3の推定上の位置を、数字はそれぞれのマーカーまたは遺伝子間での組換え体の数(3391調査個体中)を示す。B. L3と隣接したマーカーを用いたL3のマッピングを表す。マーカー名およびL3が存在しうる範囲(グレーのバー)を示す。括弧内の数字はマーカー間またはマーカー・遺伝子間の距離(cM)を示す。C. L3領域をカバーするBACコンティグの図である。L3領域を含むBACクローンを黒で、含まないクローンをグレーで示す。それぞれのバーには、BACクローン名および方向性(FおよびRは、ベクター中のそれぞれのM13シークエンスプライマー結合部位に隣接していることを示す)を示す。両向きの矢印はL3が存在しうる範囲を示す。253A1と197AD5をつなぐクローンは、使用したBACライブラリーには含まれなかったが、これらのクローン間の距離は、ストレッチDNAファイバーFISH法によって30 kb程度であることが示された。
【図2】BACコンティグに含まれる各BACクローン上のI2類似配列の位置とオープンリーディングフレーム(ORF)構成を示す。PIH7およびPIH2以外はORFが分断された偽遺伝子であった。
【図3】PIH7を発現するためのバイナリーベクターの模式図を示す。RB, ライトボーダー; nosP, ノパリン合成酵素遺伝子プロモーター; NPT II, ネオマイシンリン酸転移酵素(カナマイシン耐性酵素)遺伝子; nosT, ノパリン合成酵素遺伝子ターミネーター(ポリアデニル化シグナル); AtACT2P, シロイヌナズナアクチン2遺伝子プロモーター; LB, レフトボーダー; Hd, Hind III; Bm, BamH I; Xb, Xba I。
【図4】PIH7の機能解析の結果を示す図である。ベンサミアーナタバコ葉にPIH7挿入バイナリーベクターを持つアグロバクテリウムを注入し、4日後に病徴写真撮影およびハンマーブロッティング免疫染色法によるPMMoVの検出を行った。EV, 空ベクター(pBA); PIH7, pBA-PIH7; PIW, pBTPIW。注入時の菌密度(光学密度, OD)は、PIWが0.001、EV及びPIH7が0.2であり、混合注入時はそれらを1:1で混合した。
【図5】各L遺伝子型の終止コドン付近の塩基配列を示す。各L遺伝子型を持つ植物から抽出したmRNAを3' RACE解析した。3' RACEクローンの終止コドン(網がけ)付近の塩基配列解析結果(生データ)のアラインメントを示す。太線(図上部)で示した領域は全ての遺伝子型で保存されていた。
【図6】各L遺伝子型の機能解析結果を示す。各遺伝子型のL遺伝子コード領域をpBAに挿入したものを持つアグロバクテリウム(OD=0.5)と、トバモウイルスの各病原型の外被タンパク質を発現するプラスミドを持つアグロバクテリウム(OD=1.0)とを等量混合し、ベンサミアーナタバコ葉に注入した。4日後に葉を99.5%エタノールで脱色し、写真撮影した。壊死を起こした部分は脱色されない。
【図7】各L遺伝子型の識別結果を示す。以下の植物から抽出したDNAを鋳型として、配列番号17および18のプライマーセットを用いて増幅したPCR産物をHha IまたはTaq Iで切断後、2%アガロースゲルで分析した。1, C. annuum 昌介;2, C. annuum紫トウガラシ;3, C. annuum Verbeterde Glas;4, C. annuum KC 725;5, C. annuum札幌大長なんばん;6, C. annuum大なつめ;7, C. frutescens Tabasco;8, C. frutescens LS 1839-2-4;9, C. baccatum PI 439381-1-3;10, C. baccatum KC 602;11, C. chinense PI 159236;12, C. chinense PI 152225;13, C. chinense PI 315008;14, C. annuum NDN;15, C. chacoens PI 260429;16, C. chacoens SA 815;17, C. chinense KC 667;18, C. chinense KC 678;19, C. chinense KC 696;20, C. chinense KC 774;21, C. annuum KC 726;22, C. annuum KC 780;23, C. annuum みはた1号;24, C. annuum 昌介とC. chacoens PI 260429のF1。M, 分子量マーカー。
【図8】pBAL3(pBAPIH7)で形質転換したタバコ(T0世代)における導入遺伝子の発現を調べた結果を示す。使用したプライマーセット(AtACT2-616FおよびGSP19)の位置を矢印で示す。
【技術分野】
【0001】
本発明は、トバモウイルス抵抗性遺伝子及びその用途に関する。
【背景技術】
【0002】
植物のウイルス病には有効な防除薬剤が存在しないため、その防除において抵抗性品種の利用はきわめて重要である。しかしながら、ウイルスは変異を起こしやすく、抵抗性を打ち破る変異体、すなわち抵抗性打破ウイルスの出現が農業生産現場で問題となっている。
【0003】
ピーマン・トウガラシなどのトウガラシ(Capsicum)属植物の生産において、ウイルス病、特にトバモウイルス感染は被害が大きい。トウガラシ属植物におけるトバモウイルス病は土壌伝染性であり、臭化メチルの使用禁止によって土壌消毒が十分に行えない現状にあっては、その防除も困難になっている。国内では主にL3抵抗性遺伝子を持つ抵抗性品種が用いられているが、1998年、国内初のL3抵抗性打破トウガラシ微斑ウイルス(PMMoV)が報告(非特許文献1)されて以降、各地で異なるL3抵抗性打破ウイルスが発見された(非特許文献2)。さらに2006年、L3抵抗性打破PMMoVに対して抵抗性を示すL4抵抗性遺伝子を持つ品種に対しても抵抗性打破性を示す、L4抵抗性打破PMMoVが報告された(非特許文献3)。
【0004】
L抵抗性遺伝子は1980年代からその存在を知られており(非特許文献4および5)、ウイルス抵抗性品種の育成に用いられてきた。L遺伝子には、それぞれ対立遺伝子の関係にあるとされる複数の遺伝子型が存在することが知られており、それら遺伝子とトバモウイルスの病原型の間には階層的な関係が成り立っている。すなわち、タバコモザイクウイルスやトマトモザイクウイルス(ToMV)などのP0病原型に対しては、L1、L2、L3およびL4全てが抵抗性を示すが、パプリカ微斑ウイルス(PaMMV)などのP1病原型に対しては、L1は無効で、L2、L3およびL4だけが抵抗性を示す。さらにPMMoVでは、P1,2、P1,2,3およびP1,2,3,4の各病原型が存在し、P1,2に対してはL3およびL4が、P1,2,3に対してはL4だけが抵抗性を示し、P1,2,3,4に対して抵抗性を示すL遺伝子型は見出されていない。
【0005】
多くのL遺伝子は、温度感受性であることが知られており、28℃以上の恒温条件下ではその抵抗性機能は失われる。一方、L遺伝子の亜型であるL1aは、30℃でもP0病原型トバモウイルスに対して抵抗性を示すこと、すなわち高温機能性を持つことが報告された(非特許文献6)。L1aを含め、これまでに知られているL抵抗性遺伝子は、すべてトバモウイルスの外被タンパク質を認識して抵抗反応を駆動する(非特許文献7)。L3およびL4抵抗性打破PMMoV、すなわちP1,2,3およびP1,2,3,4病原型は、P1,2と比較して外被タンパク質のアミノ酸配列が1ないし2アミノ酸しか違わないことが分かっている。このことは、P1,2,3およびP1,2,3,4病原型がP1,2の変異によって生じたことを示している。L3やL4は温度感受性であるがゆえに、高温下でトバモウイルスの増殖を許容するとともに温度が下がると抵抗反応を駆動するため、外被タンパク質遺伝子の抵抗性打破に関わる変異を引き起こしやすいと考えられる。高温機能性のL1aでは実際、少数のアミノ酸変異による抵抗性打破ウイルス株は出現していない。
【0006】
上記の2つの問題、すなわち効果のある抵抗性遺伝子が存在しないウイルスの出現および抵抗性打破ウイルスが容易に出現してしまう抵抗性遺伝子の性質を解決するために、遺伝子組換えあるいは変異の導入による抵抗性遺伝子の改良が期待される。しかし、L遺伝子についてはこれまでクローニングされておらず、その構造も全く不明であり、その改良の糸口もない状況である。
【0007】
交配育種によって既知のL遺伝子型を品種に導入しようとする時、それぞれを識別できる遺伝子マーカーがあると、選抜が容易になる。通常使用される遺伝子マーカーは、必ずしも注目する形質を規定する遺伝子そのものではないが、目的形質と強く連鎖するものであれば遺伝子マーカーとして有用である。しかしながら、遺伝子そのものを検出しているわけではないため、目的形質と分離する可能性は否定できない。遺伝子がクローン化されており、植物系統間で多型のある場合は、遺伝子そのものを用いた方が確実であるが、L遺伝子は未だクローニングされていないため、遺伝子そのものをマーカーとして用いることもできない。
【0008】
一方、ジャガイモのジャガイモウイルスX抵抗性遺伝子、Rxでは、そのロイシンリッチリピート領域に人為的に変異を導入することによって、野生型Rxが認識できなかったウイルスを認識し、抵抗性を付与できることが報告されている(非特許文献8)。
【0009】
【非特許文献1】Molecular Plant-Microbe Interactions 11, 327-31 (1998)
【非特許文献2】J Gen Plant Pathol 68: 155-162 (2002)
【非特許文献3】Phytopathology 97, 787-93 (2007)
【非特許文献4】Euphytica 29: 433-439 (1980)
【非特許文献5】Capsicum Newslet 3: 47-48 (1982)
【非特許文献6】J Jpn Soc Hort Sci 73: 552-557 (2004)
【非特許文献7】Archives of Virology, 153, 645-650. (2008)
【非特許文献8】Proc Natl Acad Sci U S A 103: 18828-33 (2006)
【発明の開示】
【発明が解決しようとする課題】
【0010】
本発明の目的は、未だクローニングされていないトバモウイルス抵抗性L遺伝子をクローニングし、トバモウイルス抵抗性植物の作出のための新規手段を提供することにある。
【課題を解決するための手段】
【0011】
本願発明者らは、鋭意研究の結果、L3抵抗性遺伝子を保有するCapsicum chinenseのBACライブラリーを利用し、抵抗性遺伝子類似配列及びBAC末端配列に基づくマーカーを開発してライブラリーのスクリーニングを行ない、L3が2つのBACコンティグ(計5クローン)もしくはその間の未知領域に含まれること、その未知領域が約30 kb程度であること、以上の結果を考え合わせるとL3は約400 kbの領域に存在することを突き止めた。また、この領域にトマトのフザリウム抵抗性遺伝子I2に類似の配列が多数存在することを確認した。そして、該5クローンよりI2類似配列を持つDNA断片をサブクローニングし、それらの配列を解析し、候補配列について機能解析を行なうことにより、L3抵抗性遺伝子を同定した。さらに、該遺伝子の配列をもとにその他のL遺伝子をも同定し、これらの遺伝子配列自体を利用して各L遺伝子型を簡便に識別できる方法を確立し、本願発明を完成した。
【0012】
すなわち、本願発明は、配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列と90%以上の相同性を有し、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードするポリヌクレオチドを提供する。また、本発明は、配列表の配列番号2、4、6、8、10、12及び14のいずれかに示されるアミノ酸配列と90%以上の同一性を有し、植物にトバモウイルス抵抗性を付与できるポリペプチドを提供する。さらに、本発明は、上記本発明のポリペプチドをコードするポリヌクレオチドを提供する。さらに、本発明は、上記本発明のポリヌクレオチドを含み、植物にトバモウイルス抵抗性を付与できるポリペプチドを宿主細胞内で発現させることができる組換えベクターを提供する。さらに、本発明は、該組換えベクターを含む細胞を提供する。さらに、本発明は、該組換えベクターにより形質転換された形質転換植物を提供する。さらに、本発明は、該組換えベクターを植物細胞内に導入することを含む、トバモウイルス抵抗性が付与された形質転換植物の作製方法を提供する。さらに、本発明は、配列表の配列番号21に示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチド、及び該ポリヌクレオチドからなるトバモウイルス抵抗性遺伝子増幅用プライマーを提供する。さらに、本発明は、上記本発明のプライマーと、配列表の配列番号22に示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーとからなるトバモウイルス抵抗性遺伝子増幅用プライマーセットを提供する。さらに、本発明は、該プライマーセットを用いた核酸増幅法によって植物細胞より分離した核酸試料から増幅された、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードするポリヌクレオチドを提供する。さらに、本発明は、配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーを用いた核酸増幅法によって、植物細胞より分離した核酸試料からトバモウイルスL抵抗性遺伝子のコード領域の全長又は部分領域を増幅し、次いで該増幅断片の塩基配列を対比することを含む、L抵抗性遺伝子型の識別方法を提供する。
【発明の効果】
【0013】
本発明により、トバモウイルスL抵抗性遺伝子の塩基配列及び該塩基配列にコードされるアミノ酸配列が初めて提供された。本発明のLトバモウイルス抵抗性タンパク質は、これを発現させることによって植物にトバモウイルス抵抗性を付与できる。これは本発明のLトバモウイルス抵抗性遺伝子を植物発現ベクターに挿入し、植物を形質転換することによって達成される。また、本発明のLトバモウイルス抵抗性遺伝子は、本発明のプライマーによってトウガラシ属植物ゲノムDNAもしくはmRNAから増幅可能である。特に、Lトバモウイルス抵抗性遺伝子の各遺伝子型でコード領域の3'末端が保存されていることが本発明により明らかになったため、この保存領域を利用すれば未だ同定されていない新規なL遺伝子も単離することができる。単離したL遺伝子の塩基配列を決定し比較することによって、現時点で未同定のものも含め種々のL遺伝子型を識別できる。遺伝子型の識別は、遺伝子多型を検出する種々の方法(制限断片長多型解析、一塩基多型検出方法等)を利用して行なうことができる。
【発明を実施するための最良の形態】
【0014】
配列番号1〜14に示される塩基配列及びアミノ酸配列は、本願発明者らがトウガラシ属植物から単離したトバモウイルス抵抗性L遺伝子のコード領域の配列である。下記実施例に記載されるように、これらL遺伝子がコードするタンパク質のアミノ酸配列(配列番号2、4、6、8、10、12及び14)は、相同性が相互に非常に高く、97%以上の同一性及び99%以上の類似性を有している。本願発明者らが単離した各L遺伝子、その配列番号及びその由来のトウガラシ属植物名を表1に示す。
【0015】
【表1】

【0016】
トバモウイルスとは、一本鎖(+)RNAをゲノムとするトバモウイルス属の植物ウイルスである。具体例としては以下の表1に示すものが挙げられるが、これらに限定されない。この分野で周知の通り、L遺伝子にはそれぞれ対立遺伝子の関係にあるとされるL1〜L4の遺伝子型が知られており、それら遺伝子型とトバモウイルスの病原型の間には階層的な関係が成り立っている。その階層的関係も合わせて下記表2に示す。なお、L遺伝子には、L1cのように他のL遺伝子型と対立遺伝子の関係にあることが確認されていない亜型も存在する。このような亜型も本発明の範囲に包含される。
【0017】
【表2】

【0018】
本発明のポリヌクレオチドとしては、配列表の配列番号1、3、5、7、9、11及び13に示される塩基配列からなるポリヌクレオチド(以下、本明細書において、これらの塩基配列を便宜上「天然型配列」ということがある。これらにコードされるアミノ酸配列についても同様に「天然型配列」と言うことがある。)の他、これらの塩基配列のうち少数の塩基が置換し、欠失し及び/又は挿入された塩基配列からなるものであって、もとの配列と90%以上、好ましくは95%以上、より好ましくは97%以上の相同性を有する塩基配列からなるポリヌクレオチドも挙げられる。このような変異型の塩基配列からなるポリヌクレオチドであっても、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードする限り、本発明の範囲に包含される。変異型のポリヌクレオチドの好ましい例としては、上記した天然型L遺伝子配列のいずれかにおいて、1ないし数個の塩基が置換し、欠失し又は挿入された塩基配列からなるポリヌクレオチドも挙げられる。コドンには縮重があり、従って上記天然型の塩基配列がコードするアミノ酸配列は、上記天然型の塩基配列のみに限定されない。また、後述するように、コードされるアミノ酸配列にも少数のアミノ酸置換、欠失及び/又は挿入が許容されるため、そのような変異型アミノ酸配列をコードするポリヌクレオチドも本発明のポリヌクレオチドの範囲に包含される。
【0019】
ここで、塩基配列の「相同性」とは、比較すべき2つの塩基配列中の塩基ができるだけ多く一致するように両配列を整列させ、一致した塩基数を全塩基数で除したものを百分率で表したものである。上記整列の際には、必要に応じ、比較する2つの配列の一方又は双方に適宜ギャップを挿入する。このような配列の整列化は、例えばBLAST、FASTA、CLUSTAL W等の周知のプログラムを用いて行なうことができる。ギャップが挿入される場合、上記全塩基数は、1つのギャップを1つの塩基として数えた数となる。このようにして数えた全塩基数が、比較する2つの配列間で異なる場合には、相同性(%)は、長い方の配列の全塩基数で、一致した塩基数を除して算出される。
【0020】
ただし、塩基の変異は、L遺伝子型間で保存性の高い領域以外の領域内に存在することが好ましい。具体的には、例えばコード領域3'末端の33bpの領域以外で変異しているものが好ましい。下記実施例に記載されるように、L遺伝子はトマトのフザリウム抵抗性遺伝子I2の類似配列の中から見出されたが、コード領域3'末端の33bpはL遺伝子型間においてのみ同一となっており、L遺伝子で特異的に保存されている。この領域はL遺伝子の機能に重要である可能性が高い。
【0021】
ポリヌクレオチドとしては、DNAでもRNAでもよいが、安定性の観点からはDNAが好ましい。
【0022】
「植物にトバモウイルス抵抗性を付与できるポリペプチド」とは、植物体内で該ポリペプチドを発現させた場合に該植物がトバモウイルス抵抗性を示すようになることをいう。ポリペプチドが植物にトバモウイルス抵抗性を付与できるか否かは、例えば、トバモウイルスに感受性の植物組織(ベンサミアーナタバコ葉など)中でポリペプチドを発現させ、これに各種病原型のトバモウイルスを感染させて抵抗性反応が生じるか否かを調べることにより確認できる。各種の一過性発現系が周知であり、例えば下記実施例に記載されるように、適当な発現ベクター(pBA等)中にポリペプチドをコードするポリヌクレオチドを組み込み、該組換えベクターをアグロバクテリウムに導入し、ベンサミアーナタバコ葉等のトバモウイルス感受性植物組織に該アグロバクテリウムを注入すれば、植物組織中で容易に該ポリペプチドを発現させることができる。トバモウイルスの感染は、トバモウイルス粒子を常法により葉組織に接種することにより行なうこともできるし、あるいは、上記と同様にアグロバクテリウムを利用してトバモウイルス外被タンパク質を植物組織中で発現させることによって植物組織がウイルスに感染した状態を模倣することもできる。L遺伝子は所定の病原型のトバモウイルスに対して過敏感反応抵抗性(すなわち感染部位における細胞死及びウイルスの局在化)を示すため、ポリペプチドとトバモウイルス外被タンパク質が共存する部位で細胞死が生じた場合に該ポリペプチドは植物にトバモウイルス抵抗性を付与できると判断することができる。
【0023】
L遺伝子の単離により、これらがコードするL抵抗性タンパク質のアミノ酸配列も明らかとなった。上記天然型のL遺伝子がコードするアミノ酸配列は、配列表の配列番号2、4、6、8、10、12及び14に示される通りである。下記実施例に具体的に記載されるように、上記天然型のL遺伝子をトバモウイルス感受性の植物細胞内に導入してタンパク質を発現させると、トバモウイルスに対し抵抗性反応を示すようになる。すなわち、これらのアミノ酸配列からなるタンパク質は、植物にトバモウイルス抵抗性を付与する活性を有する。
【0024】
一般に、生理活性を有するポリペプチドは、そのアミノ酸配列を構成するアミノ酸のうち少数のアミノ酸が置換し、欠失し又は挿入された場合でもその生理活性が維持される場合があることは周知である。したがって、上記した天然型のアミノ酸配列(配列番号2、4、6、8、10、12及び14)のみならず、これら天然型のアミノ酸配列のうち少数のアミノ酸残基が置換され、欠失され、及び/又は挿入された配列からなるポリペプチドであって、植物細胞内で発現させた場合に該植物にトバモウイルス抵抗性を付与できるポリペプチドも本発明の範囲に包含される。このような変異型タンパク質のアミノ酸配列は、具体的には、天然型のアミノ酸配列(配列番号2、4、6、8、10、12及び14)のいずれかとの同一性が90%以上、好ましくは95%以上、より好ましくは97%以上である。なお、アミノ酸配列の「同一性」の定義は、上記した塩基配列の相同性と同様である。また、このような変異型のアミノ酸配列としては、上記天然型のアミノ酸配列において1ないし数個のアミノ酸残基が置換され、欠失され、及び/又は挿入されたアミノ酸配列からなるものも好ましい。
【0025】
ただし、塩基配列の変異と同様に、アミノ酸配列の変異も、複数のL遺伝子型の間で高度に保存されている領域以外の領域で生じていることが好ましい。具体的には、変異型のアミノ酸配列としては、上記したコード領域3'末端の33bpの領域(停止コドンを含む。アミノ酸配列でいえばC末端の10残基の領域。)以外の領域に変異が存在するものが好ましい。
【0026】
なお、天然のタンパク質を構成する20種類のアミノ酸は、低極性側鎖を有する中性アミノ酸(Gly, Ile, Val, Leu, Ala, Met, Pro)、親水性側鎖を有する中性アミノ酸(Asn, Gln, Thr, Ser, Tyr Cys)、酸性アミノ酸(Asp, Glu)、塩基性アミノ酸(Arg, Lys, His)、芳香族アミノ酸(Phe, Tyr, Trp)のように類似の性質を有するものにグループ分けでき、これらの間での置換であればポリペプチドの性質が変化しないことが多いことが知られている。従って、上記した変異型のLタンパク質の好ましい例としては、配列番号2、4、6、8、10、12及び14中のアミノ酸残基がこれら各グループ間で置換されたアミノ酸配列からなるものが挙げられる。アミノ酸配列の類似性でいえば、上記変異型のL抵抗性タンパク質は、天然型のL抵抗性タンパク質のアミノ酸配列との類似性が好ましくは95%以上、より好ましくは97%以上、さらに好ましくは98%以上、さらに好ましくは99%以上、最も好ましくは100%である。
【0027】
上記した本発明のポリヌクレオチドは、核酸合成機を用いて常法により合成することができるし、また、公知の遺伝子工学的手法を用いた常法により容易に調製することができる。上記した天然型のLタンパク質(L1, L1a, L1c, L2, L2b, L3, L4タンパク質)をコードするL遺伝子の塩基配列は、配列番号1、3、5、7、9、11、13に示される通りであるから、例えば、これらの配列情報をもとにプライマーを設計し、L遺伝子を有する植物のゲノムDNA又はcDNAを鋳型としてPCRを行うことで、Lタンパク質をコードするDNAを容易に調製することができる。下記実施例に示されるように、本願発明者らが単離したL遺伝子のコード領域にはイントロンが存在しないため、本発明のポリペプチドをコードするポリヌクレオチドはゲノムDNAからでも容易に調製することができる。PCR自体は周知の常法である。また、各種L遺伝子を有する植物も公知であり、例えば上記した表1に示されるトウガラシ属植物から各種L遺伝子を増幅することができる。増幅したL遺伝子の配列は、適宜塩基配列を決定して本願配列表に記載される塩基配列と比較し、L遺伝子型を確認してもよい。なお、L遺伝子型の確認は、後述する本願発明者らが開発したL遺伝子型検出方法により容易に行なうことができる。
【0028】
変異型の塩基配列からなるポリヌクレオチドは、変異を有する植物個体から上記方法により取得することもできるし、また、市販の核酸合成機を用いて常法により合成することもできる。あるいはまた、この分野で公知の変異導入法を用いて天然型遺伝子配列に変異を導入して調製することもできる。遺伝子に変異を導入する具体的な方法としては、例えば、Kunkel法又は Gapped duplex法等の公知手法又はこれに準ずる方法を挙げることができ、そのためのキットも種々のものが市販されている。そのようなキットの具体例としては、例えば部位特異的突然変異誘発法を利用した変異導入用キット(Mutant-K(TAKARA社製)やMutant-G(TAKARA社製)等)、TAKARA社のLA PCR in vitro Mutagenesis シリーズキット等が挙げられる。
【0029】
本発明のポリペプチドは、ペプチド合成機を用いて常法により合成することもできるし、また公知の遺伝子工学的手法を用いた常法により容易に調製することができる。具体的には、上記の通りに調製したポリヌクレオチドを発現ベクターに組み込み、適当な宿主細胞に導入し、該宿主細胞中でポリペプチドを生産させることにより、目的とするポリペプチドを得ることができる。各アミノ酸をコードするコドンは公知であるから、特定のアミノ酸配列をコードするポリヌクレオチドの塩基配列は容易に特定することができる。また、任意の塩基配列からなるポリヌクレオチドは、核酸合成機を用いた常法により或いは天然型配列に変異を導入することにより容易に調製することができる。従って、任意の変異型L抵抗性タンパク質の調製も容易に行なうことができる。
【0030】
本発明は、上記した本発明のポリヌクレオチドを含み、植物にトバモウイルス抵抗性を付与できるポリペプチドを宿主細胞内で発現することができる組換えベクターも提供する。さらに、該組換えベクターを含む細胞も提供する。該組換えベクターは、例えば上記したように本発明のポリペプチドを調製するために利用することができる。また、植物細胞内で本発明のポリペプチドを発現することのできるベクターに導入した場合には、トバモウイルス抵抗性を付与した形質転換植物の作製に利用することができる。
【0031】
本発明の組換えベクターは、適当なベクターに本発明のポリヌクレオチドとその発現を制御する配列を挿入することによって得ることができる。組換えベクターの調製方法自体は周知の常法である。具体的には、ベクターに本発明のポリヌクレオチドを挿入するには、まず、精製されたポリヌクレオチドを適当な制限酵素で切断し、適当なベクターの制限酵素部位又はマルチクローニングサイトに挿入してベクターに連結する方法などが採用される。ポリヌクレオチドは、コードするポリペプチドが宿主細胞中で発現されるようにベクターに組み込まれることが必要である。そこで、本発明の組換えベクターには、プロモーター、本発明のポリヌクレオチドのほか、所望によりエンハンサーなどのシスエレメント、スプライシングシグナル、ポリA付加シグナル、選択マーカー、リボソーム結合配列(SD配列)などを含有するものを連結することができる。なお、選択マーカーとしては、例えば除草剤耐性遺伝子、カナマイシン耐性遺伝子、ハイグロマイシン耐性遺伝子等が挙げられるが、これらに限定されない。
【0032】
用いるベクター及び宿主細胞の種類は特に限定されず、この分野で公知のいかなるベクター及び宿主細胞を用いることもでき、目的に応じて自由に選択することができる。具体的には、例えば宿主細胞としてはタバコ(Nicotiana tabacum)、トマト(Solanum lycopersicum)、イネ(Oryza sativa)などの植物;大腸菌(Escherichia coli)、バチルス・ズブチリス(Bacillus subtilis)、シュードモナス・プチダ(Pseudomonas putida)、リゾビウム・メリロティ(Rhizobium meliloti)等の細菌;サッカロミセス・セレビシエ(Saccharomyces cerevisiae)、シゾサッカロミセス・ポンベ(Schizosaccharomyces pombe)等の酵母;サル細胞COS-7、Vero、チャイニーズハムスター卵巣細胞(CHO細胞)、マウスL細胞、ヒトGH3、ヒトFL細胞等の動物細胞;あるいはSf9、Sf21等の昆虫細胞が挙げられる。ベクターは種々のものが公知であり、市販品も多く存在する。当業者であれば、宿主細胞の種類に応じて適切なベクターを容易に選択することができる。例えば、形質転換植物の作製に用いる組換えベクターであれば、アグロバクテリウムに導入して用いるTiプラスミド由来のバイナリーベクター等を好ましく用いることができる。
【0033】
植物を宿主とする場合は、本発明の組換えベクターが植物細胞内で核ゲノムに組み込まれることが望ましいが、これに限定されない。また、プロモーターとしては、例えばシロイヌナズナ(Arabidopsis thaliana)アクチン2プロモーター等の植物由来プロモーター、カリフラワーモザイクウイルス(CaMV)の35Sプロモーターなどの植物ウイルス由来プロモーターが挙げられる。ポリアデニル化シグナルとしては、アグロバクテリウムTiプラスミド由来のノパリン合成酵素遺伝子のポリAシグナル、カリフラワーモザイクウイルスのポリAシグナル配列などが挙げられる。宿主として用いる植物の種類は特に限定されず、タバコ、トマト、ピーマン、イネ等の形質転換可能ないずれの植物でも好ましく用いることができる。
【0034】
大腸菌等の細菌を宿主とする場合は、本発明の組換えベクターが該細菌中で自律複製可能であると同時に、プロモーター、リボゾーム結合配列、本発明のポリヌクレオチド、転写終結配列により構成されていることが好ましい。また、プロモーターを制御する遺伝子が含まれていてもよい。大腸菌としては、例えばエッシェリヒア・コリ(Escherichia coli)K12、DH1などが挙げられ、枯草菌としては、例えばバチルス・ズブチリス(Bacillus subtilis)などが挙げられる。プロモーターとしては、大腸菌等の宿主中で発現できるものであればいずれを用いてもよい。例えばtrpプロモーター、lacプロモーター、PLプロモーター、PRプロモーターなどの、大腸菌やファージに由来するプロモーターを用いることができる。tacプロモーターなどのように、人為的に設計改変されたプロモーターを用いてもよい。細菌への組換えベクターの導入方法としては、細菌にDNAを導入する方法であれば特に限定されるものではない。例えばカルシウムイオンを用いる方法(Cohen, S.N. et al. (1972) Proc. Natl. Acad. Sci., USA 69, 2110-2114)、エレクトロポレーション法等が挙げられる。
【0035】
動物細胞を宿主とする場合は、例えば、サル細胞COS-7、Vero、チャイニーズハムスター卵巣細胞(CHO細胞)、マウスL細胞、ラットGH3、ヒトHeLa、FL細胞などを用いることができる。用いるプロモーターとしては、SRαプロモーター、SV40プロモーター、LTRプロモーター、CMVプロモーター等を挙げることができる。ヒトサイトメガロウイルスの初期遺伝子プロモーター等を用いてもよい。動物細胞への組換えベクターの導入方法としては、例えばエレクトロポレーション法、リン酸カルシウム法、リポフェクション法等が挙げられる。
【0036】
昆虫細胞を宿主とする場合は、例えばSchneider 2(S2) 細胞、Sf9細胞、Sf21細胞などを用いることができる。昆虫細胞への組換えベクターの導入方法としては、例えばリン酸カルシウム法、リポフェクション法、エレクトロポレーション法などが挙げられる。
【0037】
また、上記の各宿主細胞への遺伝子導入は、組換えベクターによらない方法、例えばパーティクルガン法なども用いることができる。
【0038】
植物細胞中で発現可能なベクターを用いた本発明の組換えベクターは、トバモウイルス抵抗性が付与された形質転換植物の作製に用いることができる。そのようなベクターとしては、例えば上記したようにアグロバクテリウムのTiプラスミドやRiプラスミド由来のバイナリーベクターが挙げられる。アグロバクテリウムを利用した植物の形質転換方法自体は周知の常法である。具体的には、バイナリーベクターの所定の領域に本発明のポリヌクレオチドを挿入し、得られた組換えベクターをエレクトロポレーション法等によりアグロバクテリウム中に導入し、これを植物細胞と共培養して植物細胞に感染させることで、バイナリーベクター中に組み込んだ本発明のポリヌクレオチドを植物細胞のゲノム中に組み込むことができる。該植物細胞を常法により培養して植物体を分化させれば、トバモウイルス抵抗性が付与された形質転換植物を得ることができる。
【0039】
さらに、本発明により、L遺伝子のコード領域中の3'末端33bpの領域がL遺伝子型間で高度に保存されており、L遺伝子に特異的な配列であることが見出された。この33bpの塩基配列を配列表の配列番号21に示す。本発明は、この配列番号21に示される塩基配列中の部分領域に特異的にハイブリダイズするポリヌクレオチドも提供する(以下、該ポリヌクレオチドを便宜的に「検出用ポリヌクレオチド」と呼ぶ)。該検出用ポリヌクレオチドは、例えばトバモウイルス抵抗性遺伝子増幅用プライマーとして有用であり、各種遺伝子型のL遺伝子の増幅・検出に用いることができる。また、該検出用ポリヌクレオチドは、プローブとしてトバモウイルス抵抗性遺伝子の検出や単離に利用することもできる。
【0040】
L遺伝子はトマトI2遺伝子のホモログとして見出されたものであるが、公知のI2ホモログの多くにおいてコード領域の5'末端側(開始コドンから30bpの領域)が保存された領域となっており、L遺伝子型間でも同様に保存されている。この30bpの領域を配列表の配列番号22に示す。この5'末端側の保存領域(配列番号22)中の部分領域に特異的にハイブリダイズするプライマーと、上記トバモウイルス抵抗性遺伝子増幅用プライマーとからなるプライマーセットは、トバモウイルス抵抗性遺伝子増幅用プライマーセットとして有用であり、該プライマーセットによれば、植物細胞から分離した核酸試料より種々の遺伝子型のL遺伝子を増幅することができる。PCR等の核酸増幅法は周知の常法であり、当業者であれば増幅サイズ等に応じて適宜反応条件を定めることができる。
【0041】
ここで、「特異的にハイブリダイズする」とは、通常のハイブリダイズの条件下において、対象とする部分領域とのみハイブリダイズし、その他の領域とは実質的にハイブリダイズしないという意味である。
【0042】
「通常のハイブリダイズの条件下」とは、通常のPCRのアニーリングやプローブによる検出に用いられる条件下のことをいい、例えば、Taqポリメラーゼを用いたPCRの場合には、50mM KCl、10mM Tris-HCl(pH 8.3〜9.0)、1.5mM MgCl2といった一般的な緩衝液を用いて、54℃〜60℃程度の適当なアニーリング温度で反応を行なうことをいい、また、例えばノーザンハイブリダイゼーションの場合には、5 x SSPE、50%ホルムアミド、5 x Denhardt's solution、0.1〜0.5%SDSといった一般的なハイブリダイゼーション溶液を用いて、42℃〜65℃程度の適当なハイブリダイゼーション温度で反応を行なうことをいう。ただし、適当なアニーリング温度又はハイブリダイゼーション温度は、上記例示に限定されず、プライマー又はプローブとして用いるポリヌクレオチドのTm値及び実験者の経験則に基づいて定められ、当業者であれば容易に定めることができる。
【0043】
「実質的にハイブリダイズしない」とは、全くハイブリダイズしないか、するとしても対象とする部分領域にハイブリダイズする量よりも大幅に少なく、相対的に無視できる程度の微量しかハイブリダイズしないという意味である。そのような条件下で特異的にハイブリダイズするポリヌクレオチドとしては、対象の部分領域の塩基配列と一定以上の相同性を有するポリヌクレオチドが挙げられ、例えば70%以上、好ましくは80%以上、より好ましくは90%以上、さらに好ましくは93%以上、さらに好ましくは95%以上、さらに好ましくは98%以上の相同性を有するポリヌクレオチドが挙げられる。最も好ましくは、該ポリヌクレオチドは、対象の部分領域の塩基配列と同一の塩基配列からなる。相同性の定義は上記と同様である。また、検出用ポリヌクレオチドの末端に対象とハイブリダイズしない領域が含まれていても、プローブの場合には、ハイブリダイズする領域がプローブ全体のおよそ半分以上を占めていれば検出に用いることができるし、また、プライマーの場合には、ハイブリダイズする領域がプライマー全体のおよそ半分以上を占め、かつ3'末端側にあれば、5'末端側に任意の付加配列があっても正常にアニーリングして伸長反応を生じ得るので、検出に用いることができる。例えば、プライマーの5'側に制限酵素認識配列等を付加して用いることはこの分野で周知の常法である(具体例としては、配列番号24に示す塩基配列からなるプライマー等。下記実施例参照。)。そのように、検出用ポリヌクレオチドの末端にハイブリダイズしない領域が含まれている場合において、対象の塩基配列との相同性を算出するときは、ハイブリダイズしない末端の領域は考慮せず、ハイブリダイズする領域のみに着目して算出するものとする。
【0044】
ただし、この分野で周知のように、数塩基程度のミスマッチを含むミスマッチプライマーを用いたPCRにより増幅断片に所望の変異を導入することができる。このような手法は周知の常法であり、後述のPIRA-PCR法においてもミスマッチプライマーが用いられる(具体例としては、配列番号19に示す塩基配列からなるプライマー等。下記実施例参照。)。従って、プライマーの配列としては対象の部分領域の塩基配列と同一のものが必ずしも好ましいわけではなく、目的に応じて適宜ミスマッチを含む塩基配列が選択される。当業者であれば、本願配列表に記載の塩基配列を参照し、そのようなミスマッチプライマーを目的に応じて容易に設計することができる。
【0045】
なお、本発明において、塩基配列中の「部分領域」とは、配列表に示される塩基配列中の一部の領域を言い、好ましくは連続する18塩基以上の領域である。また、本発明において、「配列番号1に示される塩基配列」と言った場合には、配列番号1に実際に示されている塩基配列の他、これと相補的な配列も包含する。従って、例えば「配列番号1に示される塩基配列からなるポリヌクレオチド」と言った場合には、配列番号1に実際に示されている塩基配列からなる一本鎖ポリヌクレオチド、その相補的な塩基配列からなる一本鎖ポリヌクレオチド、及びこれらから成る二本鎖ポリヌクレオチドが包含される。「配列番号21に示される塩基配列中の連続する18塩基以上からなるポリヌクレオチド」と言った場合も同様であり、配列番号21中に実際に示されている塩基配列と相補的な配列中の連続する18塩基以上からなるポリヌクレオチドも包含される。本発明のポリヌクレオチド及び検出用ポリヌクレオチドを調製する場合や、本発明のポリペプチドをコードするポリヌクレオチドを調製する場合には、適宜いずれかの塩基配列を選択することとなるが、当業者であれば容易にその選択をすることができる。
【0046】
検出用ポリヌクレオチドの塩基数は、特異性を確保する観点から、18塩基以上が好ましい。サイズは、プローブとして用いる場合には、好ましくは18塩基以上、さらに好ましくは20塩基以上が好ましく、プライマーとして用いる場合には、18塩基以上が好ましい。検出用ポリヌクレオチドの好ましい例としては、配列番号21に示される塩基配列中の連続する18塩基以上からなるポリヌクレオチドが挙げられ、より具体的には配列番号16に示される塩基配列からなるポリヌクレオチドが挙げられるが、これに限定されない。
【0047】
上記したトバモウイルス抵抗性遺伝子増幅用プライマーの好ましい例としては、配列番号16に示される塩基配列からなるプライマーが挙げられる。また、トバモウイルス抵抗性遺伝子増幅用プライマーセットとして該プライマーと組み合わせて用いられるプライマーのうち好ましい例としては、配列番号22に示される塩基配列中の連続する18塩基以上からなるプライマー、特に配列番号15に示される塩基配列からなるプライマーが挙げられるが、これに限定されない。
【0048】
さらにまた、本発明により各種遺伝子型のL遺伝子配列が同定されたため、植物細胞から得た核酸試料よりL遺伝子中の所望の領域を増幅し、その配列を対比することによってL遺伝子型を識別することが可能となった。すなわち、本発明は、配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーを用いた核酸増幅法によって、植物細胞より分離した核酸試料からトバモウイルスL抵抗性遺伝子のコード領域の全長又は部分領域を増幅し、次いで該増幅断片の塩基配列を対比することを含む、L抵抗性遺伝子型の識別方法をも提供する。
【0049】
核酸試料は、ゲノムDNA、mRNA、cDNAのいずれでもよい。下記実施例に示されるように、L遺伝子のコード領域中にはイントロンが存在しないため、抽出作業及び取扱いの簡便性から核酸試料としてはゲノムDNAが好ましい。
【0050】
核酸増幅法としては、周知の常法であるPCR法を好ましく採用することができる。
【0051】
上記したように、5'末端の保存領域と3'末端の保存領域とにプライマーを設定すれば、種々のL遺伝子型をいずれも増幅できる。従って、このようなプライマーを用いてL遺伝子のコード領域全長を増幅し、増幅断片の塩基配列を対比してもよい。あるいは、各遺伝子型で指標とすべき配列部分(多型部分)を含む部分領域のみを増幅し、その塩基配列を対比してもよい。
【0052】
塩基配列の対比は、増幅断片の塩基配列全長を決定して対比してもよいし、また、各遺伝子型に特徴となる一塩基多型等の多型を指標として対比を行なってもよい。遺伝子多型を検出する方法は公知であり、例えばPIRA(Primer-introduced restriction analysis)-PCR法、インベーダー法等を挙げることができる。また、制限酵素断片長多型解析を用いて遺伝子配列の多型を検出・識別することもできる。PIRA-PCR法とは、ミスマッチプライマーを用いて一塩基多型部位に人為的に制限酵素サイトを導入し、制限酵素による切断の有無を指標として一塩基多型を検出する方法であり、この分野で公知の常法である。
【0053】
例えば、配列番号1に示すL3遺伝子配列中の3070nt〜3982ntの領域内には、L2、L2b、L3、L4及びL1c遺伝子型を識別可能な多型が存在する。この多型は、TaqI切断パターン及びHhaI切断パターンに基づいて識別することができる。この領域内の多型を利用した識別方法は下記実施例に詳述される通りであり、具体的には、配列番号17及び18に示す塩基配列からなるプライマーセットを用いて核酸試料から増幅断片を得て、これをTaqIとHhaIとでそれぞれ切断し、以下の表3に示す消化断片パターンを指標としてL2、L2b、L3、L4及びL1c遺伝子型を識別することができる。
【0054】
【表3】

表中の数値は消化断片のサイズ(bp)を示す
【0055】
また、例えば、配列番号5に示すL1遺伝子配列中の3779ntには、L1のみを他の遺伝子型と区別できる一塩基多型が存在する。該多型は、配列番号19および20に示す配列のプライマーセットを用いたPIRA-PCR法による解析においてXmnI切断部位が生じるか否かによって識別することができる。配列番号19のプライマーにはミスマッチが含まれており、これを用いたPCRによればL1由来の増幅断片にのみXmnI切断部位が生じ、他の遺伝子型(L1a, L1c, L2, L2b, L3, L4)ではXmnI切断部位が生じない。この一塩基多型を利用したPIRA-PCR法による識別方法は下記実施例に詳述される通りであり、具体的には、配列番号19及び20に示す塩基配列からなるプライマーセットを用いて核酸試料から増幅断片を得て、該増幅断片をXmnIで消化し、29bpと117bpの断片に切断されるものをL1と識別することができる。該方法では、増幅効率と識別精度を高める観点から、nested PCRにより増幅断片を得ることが好ましい。その場合、配列番号19及び20に示す塩基配列からなるプライマーセットは第2段階のPCRに用いることができる。第1段階のPCRでは、該プライマーセットにより増幅される領域を含む部分領域を増幅できるものであれば特に限定されないが、例えば配列番号17及び18に示す塩基配列からなるプライマーセットを好ましく用いることができる。また、PIRA-PCR法では、プライマー配列中に標的とは異なる配列を含む、すなわちミスマッチを含むため、増幅に用いる酵素は校正機能のないものを用いる必要があり、例えばTaqポリメラーゼを用いることが好ましい。
【0056】
L1a型については、コード領域全長のXbaI切断パターンによって他の遺伝子型と識別することができる。L1a型では、コード領域全長がXbaIにより2433bp、1080bp、474bpの3つの断片に切断される。他の遺伝子型ではXbaI消化により得られる断片は2つである。
【0057】
上記識別方法を適宜組み合わせれば、各種L遺伝子型を全て識別することができる。例えば、ある核酸試料について、まずTaqI切断パターン及びHhaI切断パターンに基づいてL2、L2b、L3、L4及びL1cのいずれであるかを調べ、不明である場合には上記した3779ntの一塩基多型により又はコード領域全長のXbaI切断パターンによりL1かL1aかを識別することが可能である。
【実施例】
【0058】
以下、実施例に基づき本発明をより具体的に説明する。もっとも、本発明は下記実施例に限定されるものではない。
【0059】
(実施例1)L3抵抗性遺伝子のクローニング
L3遺伝子は、Capsicum chinenseのいくつかのアクセションが保有しているトバモウイルス抵抗性遺伝子である。これまでのマッピングの結果から、L遺伝子座は、トマトの11番染色体とシンテニーを持つカプシクム属植物の11番染色体に座乗していることが知られていた。しかし、これまでに得られていたL3と連鎖する遺伝子マーカーは、L3と離れたものや、あるいは分離しないと報告されていても調査個体数が少なく信頼性の低いものであり、ポジショナルクローニングを可能とする、L3を挟み込む隣接したマーカーは得られていなかった。そこで我々は、抵抗性遺伝子類似配列及びBAC末端配列に基づくマーカーを開発し、L3が2つのBACコンティグ(C. chinense PI159236のゲノムライブラリー由来;計5クローンにより構成)もしくはその間の未知領域に含まれること、その未知領域が約30 kb程度であること、以上の結果を考え合わせるとL3は約400 kbの領域に存在することを突き止めた(図1)(Tomita et al, Theor Appl Genet (2008) 117:1107-1118)。また、この領域にトマトのフザリウム抵抗性遺伝子I2に類似の配列が多数存在することを確認した。
【0060】
以上の結果から、5クローンのBACクローン中に存在するI2類似配列のいずれかがL3である可能性を考え、それら5つのBACクローンを制限酵素EcoRIまたはHindIIIで切断し、ピーマン由来I2類似配列の混合物をプローブとして用いたサザンブロッティング法によって検出し、I2類似配列を持つDNA断片をサブクローニングした。それらの配列を決定したところ、10配列中ただ1つ、I2類似アミノ酸配列をコードしうるものを発見した。この配列から遺伝子の3' 端と考えられた配列およびI2類似配列で保存されている5' 端の配列をPCRプライマーセットとして(それぞれ配列番号16及び15)、この遺伝子候補の全長をBACクローン185CA10から増幅し、その塩基配列を決定した。この遺伝子(PIH7)は、CC-NBS-LRR型の抵抗性遺伝子産物をコードしており、I2とのアミノ酸配列の同一性は71.6%、類似性は92.3%であった。PIH7の塩基配列及びアミノ酸配列をそれぞれ配列表の配列番号1及び2に示す。
【0061】
その後、5つのBACクローンを全て塩基配列決定したところ、上記PIH7遺伝子が、それらBACクローン上のL3がマッピングされた領域中に存在する唯一の抵抗性遺伝子様の遺伝子であることが分かった(図2)。サザンブロッティング法で検出されたI2類似配列のうち、PIH7遺伝子を除く全てが偽遺伝子であった。また、PIH7が存在するBACクローン185CA10上のL3がマッピングされた領域外にPIH7と76.0%の同一性、92.0%の類似性を示すタンパク質をコードする遺伝子が存在した。以上の結果から、PIH7がL3の有力な候補と考えられた。
【0062】
(実施例2)一過性発現によるL3抵抗性遺伝子機能の解析
L3の有力な候補遺伝子であるPIH7について、トバモウイルス抵抗性を付与する機能を有すか否か、ベンサミアーナタバコ(Nicotiana benthamiana)における一過性発現系を用いて検討した。制限酵素認識配列を付加したプライマー(配列番号23および24)を用いてBACクローン185CA10からPIH7を増幅したのち、バイナリーベクターpBAのシロイヌナズナのアクチン2遺伝子プロモーターの下流に挿入し(図3)、アグロバクテリウムに形質転換した。これを単独またはPMMoV-IwのアグロインフェクションプラスミドpBTPIWを持つアグロバクテリウムと混合して、ベンサミアーナタバコ葉に注入した。対照実験として、pBTPIWとpBAを持つアグロバクテリウムの混合物、およびpBAを持つアグロバクテリウムを単独で注入した。その結果、注入後3日目からpBAPIH7とpBTPIWの両方を導入した領域で過敏感細胞死様の反応が認められ、注入4日目で細胞死は肉眼で確認できる程度に明確になった(図4)。この領域におけるPMMoV感染の広がりを、ハンマーブロッティング法によってpBYPIWとpBA共導入区と比較したところ、pBAPHI7による明らかなPMMoV感染拡大の抑制が認められた(図4)。このことから、PIH7がL3遺伝子の実体であることが確認された。
【0063】
(実施例3)L3抵抗性遺伝子の対立遺伝子および新規L遺伝子のクローニング
L3のマッピングに際して、マーカーを作製する目的で抵抗性遺伝子様部分配列を多数単離した。その中にL3自体(PIH7)があったが、その配列のホモログをマッピングに用いた交配相手であるCapsicum frutescensから単離したところ、ほとんど多型がなく、マーカー開発には至らなかった。そこで、そのC. frutescens由来の配列が、この植物の持つL遺伝子L2ではないかと考え、同配列を、L1を持つCapsicum annuum Verbeterde Glas、L1aを持つC. annuum KC780、L2様の特性の遺伝子L2bを持つCapsicum baccatumおよびL抵抗性遺伝子を持たないと考えられる感受性のC.annuum昌介のゲノムDNAから増幅した。この際、配列番号27および28に示すプライマーを用いた(PCRの反応条件:94℃20秒、56℃20秒、72℃1分の35サイクル、酵素としてタカラバイオ社のTaKaRa ExTaq DNA polymeraseを使用した)。それらDNA断片の塩基配列を比較したところ、感受性の昌介を除く全ての供試植物が相同性の高い配列を持つことが分かった。そこで、L1、L1a、L2、L2bおよびL3を持つ植物から全RNAを調製し、3'-RACE PCR法によって解析した。この際に用いた遺伝子特異的プライマーは上述の配列で保存されている領域内にデザインした(配列番号29および30;PCRの反応条件:第一段階、配列番号29のプライマー使用、94℃20秒、60℃20秒、72℃2分の35サイクル、第二段階、配列番号30のプライマー使用、94℃20秒、60℃20秒、72℃2分の25サイクル、酵素としてタカラバイオ社のTaKaRa ExTaq DNA polymeraseを使用した)。
【0064】
L遺伝子は、L3遺伝子の構造からコード領域内にイントロンを持たないことが推定されていたが、3'非コード領域内には多くのイントロンがオルタナティブなスプライシングを受け、多様な構造を示した。しかし、コード領域の3'端の配列33塩基対(配列番号21)は、L1、L1a、L2、L2bおよびL3でまったく同一であった(図5)。I2のホモログの多くが保存された5'端(開始コドンから30塩基対)を持つこと、既知のI2ホモログ間で3'端は保存されていないことから、3'端はL遺伝子型間でのみ高度に保存されていると考えられた。そこで、I2ホモログに共通した5'端配列とL遺伝子型間で保存された3'端配列(配列番号15および16)をプライマーセットとし、各種Capsicum属植物のゲノムDNAから遺伝子全域の増幅を試みた(PCRの反応条件:94℃30秒、60℃30秒、68℃4分の45サイクル、酵素としてタカラバイオ社のPrimeSTAR GXL DNA polymeraseを使用した)。
【0065】
その結果、上記5種類のL遺伝子の塩基配列が得られた(L3、配列番号1; L1、配列番号5; L1a、配列番号7; L2、配列番号11; L2b、配列番号13)。また、L4を持つCapsicum chacoensの2つのアクセションから独立に同一の塩基配列(配列番号3)が得られた。さらに、L1様の遺伝子L1cを持つC. chinense KC678からは、得られたL遺伝子の中でL3と最も近縁ではあるが、この遺伝子だけに特異的な6塩基の挿入とその周囲に集中した変異を持つ塩基配列(配列番号9)が得られた。いずれもコード領域にイントロンは存在しなかった。また、L4及びL1cにおいても、コード領域3'端の33bpの配列は同一であった。得られたL遺伝子型間の塩基配列から推定されるアミノ酸配列(L1、配列番号6; L1a、配列番号8; L1c、配列番号10; L2、配列番号12; L2b、配列番号14; L3、配列番号2; L4、配列番号4)の同一性および類似性(%)を下記表4に示す。
【0066】
【表4】

【0067】
以上の結果から、各L遺伝子型は相互に相同性の高い遺伝子であり、それらは全て同じプライマーセット(配列番号15および16)によって増幅できることが分かった。また、L1cのように遺伝分析で他のL遺伝子型の対立遺伝子であることの確かめられていないものも得られたことから、本法によってトウガラシ属野生種や他の属からも新たなトバモウイルス抵抗性遺伝子が発見できると期待される。L3タンパク質のN-およびC-端にHAやcMycなどのエピトープタグを付加した場合、L3の機能が完全に消失した。このことはLタンパク質のN-およびC-端、すなわちL遺伝子の5'および3'端が機能的制約によって高度に保存されていることを示唆しており、Lと類似の機能的なトバモウイルス抵抗性遺伝子は、配列番号15および16のプライマーセットを用いて増幅できると考えられる。
【0068】
(実施例4)L3ホモログの機能解析
上記で得られた各遺伝子型Lタンパク質の機能をL1、L2、L3およびL4について一過性発現系を用いて検討した。各L遺伝子型を、配列番号15および16のプライマーに制限酵素認識部位を付加したプライマーセット(配列番号23および24)を用いて増幅し(PCRの反応条件:94℃30秒、60℃30秒、68℃4分の1サイクルおよび94℃30秒、60℃30秒、68℃4分の44サイクル、酵素としてタカラバイオ社のPrimeSTAR GXL DNA polymeraseを使用した)、発現ベクターpBAに挿入し、アグロバクテリウムを形質転換した。これらを、各病原型トバモウイルスの外被タンパク質(CP)を発現するバイナリーベクターpGT-ToCP(ToMV, P0)、pGT-PaCP(PaMMV, P1)、pGT-PIWCP(PMMoV, P1,2)、pGT-PM13F66V(PMMoV, P1,2,3)、pGT-PM43K50G(PMMoV, P1,2,3)、pGT-PM81S(PMMoV, P1,2,3)、pGT-PM138N(PMMoV, P1,2,3)またはpGT-PM46R85K(PMMoV, P1,2,3,4)を持つアグロバクテリウムと混合し、ベンサミアーナタバコ葉に注入した。その結果を図6に示す。L1はToCP(P0)と共発現させた場合にのみ抵抗反応による細胞死を引き起こした。L2はToCP(P0)またはPaCP(P1)と共発現させた場合に同様の抵抗反応を示した。L3はToCP(P0)、PaCP(P1)及びPIWCP(P1,2)のいずれかと共発現させた場合に同様の抵抗反応を示した。L4はPM46R85(P1,2,3,4)以外のいずれかと共発現させた場合に同様の抵抗反応を示した。以上の結果は、配列番号15および16のプライマーセット並びに配列番号23および24のプライマーセットで増幅した遺伝子がL遺伝子として機能することを示す。また、各遺伝子型Lタンパク質はそれぞれ一種類のポリペプチドで、それぞれが異なる範囲のトバモウイルス外被タンパク質を認識することが示された。
【0069】
(実施例5)L抵抗性遺伝子型の識別
交配育種によって既知のL遺伝子型を品種に導入しようとする時、それぞれを識別できる遺伝子マーカーがあると、選抜が容易になる。通常使用される遺伝子マーカーは、必ずしも注目する形質を規定する遺伝子そのものではないが、目的形質と強く連鎖するものであれば遺伝子マーカーとして有用である。しかしながら、遺伝子そのものではないため、目的形質と分離する可能性は否定できない。従って、遺伝子がクローン化されており、植物系統間で多型のある場合は、遺伝子そのものを用いた方が確実である。そこで、上述の単離した各L遺伝子型を簡便に識別する方法の開発を試みた。
【0070】
全長のコード領域の塩基配列を比較すれば、当然識別は可能である。そこでまず、配列番号15および16のプライマーセットでゲノムDNAから増幅した全長の各L遺伝子型について、制限酵素分析によって識別することを試みた。その結果、L1cとL3との識別、及びL2bとL4との識別ができないことが分かった(表5)。
【0071】
【表5】

【0072】
そこで、L遺伝子の部分配列を配列番号17および18のプライマーセットで増幅し、4ベースカッターで切断する方法を試みた(PCRの反応条件:94℃20秒、58℃20秒、72℃1分の35サイクル、酵素としてタカラバイオ社のTaKaRa ExTaq DNA polymeraseを使用した)。その結果、Taq I切断断片およびHhaI切断断片を比較することによって、L1とL1a以外の遺伝子はこの方法で識別できることが分かった(図7及び表6)。なお、配列番号17及び18のプライマーセットは、配列番号1に示すL3遺伝子配列中の3070nt〜3982ntの領域を増幅するものである。
【0073】
【表6】

【0074】
L1とL1aは、全長増幅産物のXba I切断によって識別可能であるが、全長配列の増幅は効率が高くないため、配列番号17および18のプライマーセットで増幅した断片に含まれる一塩基多型を検出する方法を開発した。配列番号17および18のプライマーセットで増幅した断片を鋳型とし、配列番号19および20を用いて再度増幅し(PCRの反応条件:94℃10秒、60℃10秒、72℃30秒の25サイクル、酵素としてタカラバイオ社のTaKaRa Taq DNA polymeraseを使用した)、その146塩基対の増幅産物を制限酵素Xmn Iで処理すると、L1由来の増幅産物だけが切断されて29および117塩基対になる(図7、PIRA-PCR)。この鎖長の違いを電気泳動で検出し、L1とL1aを識別できる。
【0075】
(実施例6)L遺伝子を用いたウイルス抵抗性植物の育成
トバモウイルス抵抗性植物を遺伝子組換え法によって作製するため、タバコ(Nicotiana tabaccum)SR1をpBAPIH7 (L3)を用いて形質転換した。形質転換したタバコ(T0世代)からRNAを抽出し、RT-PCR法で導入遺伝子の発現を検討した。プライマーセットとしてAtACT2-616FおよびL3C-S19(それぞれ配列番号25及び26)を使用した(PCRの反応条件:94℃30、60℃30、72℃45の25サイクル、酵素としてタカラバイオ社のTaKaRa ExTaq DNA polymeraseを使用した)。PCRの標的領域がイントロンを含むため、ゲノムDNAまたは未スプライシングRNAに由来する約1.2 kbのバンド(genomic fragment)と、スプライシングを受けたRNAに由来する約0.6 kbのバンド(cDNA fragment)が認められた(図8)。
【0076】
導入遺伝子の発現が確認できたT0世代30個体について、ToMV(30μg/ml)を接種し、接種7日後に病徴観察及び接種葉の葉柄のティッシュプリント、並びに接種10日後に非接種上位葉のハンマーブロッティング免疫染色を行ない、接種葉および上位葉におけるウイルスの増殖を検討した。その結果、17個体において明確な抵抗性が認められた。
【0077】
これまでLタンパク質の検出法が確立していないので、抵抗性の程度とLタンパク質の蓄積量の間の関係については不明であるが、高率で抵抗性個体が得られたことから、この方法によってトバモウイルス抵抗性植物を作製できると考えられる。これまで安定形質転換体としてはL3を発現するもののみを作製したが、他のL遺伝子型も一過性発現系で抵抗性遺伝子機能を確認できており、さらにそれらが様々な変異を異なる組み合わせで持っている事実は、L遺伝子型のキメラ形成や点変異導入によってLトバモウイルス抵抗性遺伝子の機能を改良することが可能であることを示す。
【図面の簡単な説明】
【0078】
【図1】L3遺伝子の精密マッピングを説明する図である。A. 連鎖解析の結果を示す。矢印はマーカーの位置とL3の推定上の位置を、数字はそれぞれのマーカーまたは遺伝子間での組換え体の数(3391調査個体中)を示す。B. L3と隣接したマーカーを用いたL3のマッピングを表す。マーカー名およびL3が存在しうる範囲(グレーのバー)を示す。括弧内の数字はマーカー間またはマーカー・遺伝子間の距離(cM)を示す。C. L3領域をカバーするBACコンティグの図である。L3領域を含むBACクローンを黒で、含まないクローンをグレーで示す。それぞれのバーには、BACクローン名および方向性(FおよびRは、ベクター中のそれぞれのM13シークエンスプライマー結合部位に隣接していることを示す)を示す。両向きの矢印はL3が存在しうる範囲を示す。253A1と197AD5をつなぐクローンは、使用したBACライブラリーには含まれなかったが、これらのクローン間の距離は、ストレッチDNAファイバーFISH法によって30 kb程度であることが示された。
【図2】BACコンティグに含まれる各BACクローン上のI2類似配列の位置とオープンリーディングフレーム(ORF)構成を示す。PIH7およびPIH2以外はORFが分断された偽遺伝子であった。
【図3】PIH7を発現するためのバイナリーベクターの模式図を示す。RB, ライトボーダー; nosP, ノパリン合成酵素遺伝子プロモーター; NPT II, ネオマイシンリン酸転移酵素(カナマイシン耐性酵素)遺伝子; nosT, ノパリン合成酵素遺伝子ターミネーター(ポリアデニル化シグナル); AtACT2P, シロイヌナズナアクチン2遺伝子プロモーター; LB, レフトボーダー; Hd, Hind III; Bm, BamH I; Xb, Xba I。
【図4】PIH7の機能解析の結果を示す図である。ベンサミアーナタバコ葉にPIH7挿入バイナリーベクターを持つアグロバクテリウムを注入し、4日後に病徴写真撮影およびハンマーブロッティング免疫染色法によるPMMoVの検出を行った。EV, 空ベクター(pBA); PIH7, pBA-PIH7; PIW, pBTPIW。注入時の菌密度(光学密度, OD)は、PIWが0.001、EV及びPIH7が0.2であり、混合注入時はそれらを1:1で混合した。
【図5】各L遺伝子型の終止コドン付近の塩基配列を示す。各L遺伝子型を持つ植物から抽出したmRNAを3' RACE解析した。3' RACEクローンの終止コドン(網がけ)付近の塩基配列解析結果(生データ)のアラインメントを示す。太線(図上部)で示した領域は全ての遺伝子型で保存されていた。
【図6】各L遺伝子型の機能解析結果を示す。各遺伝子型のL遺伝子コード領域をpBAに挿入したものを持つアグロバクテリウム(OD=0.5)と、トバモウイルスの各病原型の外被タンパク質を発現するプラスミドを持つアグロバクテリウム(OD=1.0)とを等量混合し、ベンサミアーナタバコ葉に注入した。4日後に葉を99.5%エタノールで脱色し、写真撮影した。壊死を起こした部分は脱色されない。
【図7】各L遺伝子型の識別結果を示す。以下の植物から抽出したDNAを鋳型として、配列番号17および18のプライマーセットを用いて増幅したPCR産物をHha IまたはTaq Iで切断後、2%アガロースゲルで分析した。1, C. annuum 昌介;2, C. annuum紫トウガラシ;3, C. annuum Verbeterde Glas;4, C. annuum KC 725;5, C. annuum札幌大長なんばん;6, C. annuum大なつめ;7, C. frutescens Tabasco;8, C. frutescens LS 1839-2-4;9, C. baccatum PI 439381-1-3;10, C. baccatum KC 602;11, C. chinense PI 159236;12, C. chinense PI 152225;13, C. chinense PI 315008;14, C. annuum NDN;15, C. chacoens PI 260429;16, C. chacoens SA 815;17, C. chinense KC 667;18, C. chinense KC 678;19, C. chinense KC 696;20, C. chinense KC 774;21, C. annuum KC 726;22, C. annuum KC 780;23, C. annuum みはた1号;24, C. annuum 昌介とC. chacoens PI 260429のF1。M, 分子量マーカー。
【図8】pBAL3(pBAPIH7)で形質転換したタバコ(T0世代)における導入遺伝子の発現を調べた結果を示す。使用したプライマーセット(AtACT2-616FおよびGSP19)の位置を矢印で示す。
【特許請求の範囲】
【請求項1】
配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列と90%以上の相同性を有し、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードするポリヌクレオチド。
【請求項2】
前記相同性が95%以上である請求項1記載のポリヌクレオチド。
【請求項3】
配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列、又は該塩基配列において1若しくは数個の塩基が置換し、欠失し若しくは挿入された塩基配列からなる請求項2記載のポリヌクレオチド。
【請求項4】
配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列からなる請求項3記載のポリヌクレオチド。
【請求項5】
配列表の配列番号2、4、6、8、10、12及び14のいずれかに示されるアミノ酸配列と90%以上の同一性を有し、植物にトバモウイルス抵抗性を付与できるポリペプチド。
【請求項6】
前記同一性が95%以上である請求項5記載のポリペプチド。
【請求項7】
配列番号2、4、6、8、10、12及び14のいずれかに示されるアミノ酸配列、又は該アミノ酸配列において1若しくは数個のアミノ酸が置換し、欠失し若しくは挿入されたアミノ酸配列からなる請求項6記載のポリペプチド。
【請求項8】
配列番号2、4、6、8、10、12及び14のいずれかに示されるアミノ酸配列から成る請求項7記載のポリペプチド。
【請求項9】
請求項5ないし8のいずれか1項に記載のポリペプチドをコードするポリヌクレオチド。
【請求項10】
請求項1ないし4及び9のいずれか1項に記載のポリヌクレオチドを含み、植物にトバモウイルス抵抗性を付与できるポリペプチドを宿主細胞内で発現させることができる組換えベクター。
【請求項11】
前記宿主が植物である請求項10記載の組換えベクター。
【請求項12】
請求項10又は11記載の組換えベクターを含む細胞。
【請求項13】
請求項11記載の組換えベクターにより形質転換された形質転換植物。
【請求項14】
請求項11記載の組換えベクターを植物細胞内に導入することを含む、トバモウイルス抵抗性が付与された形質転換植物の作製方法。
【請求項15】
配列表の配列番号21に示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチド。
【請求項16】
請求項17記載のポリヌクレオチドからなるトバモウイルス抵抗性遺伝子増幅用プライマー。
【請求項17】
請求項16記載のプライマーと、配列表の配列番号22に示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーとからなるトバモウイルス抵抗性遺伝子増幅用プライマーセット。
【請求項18】
配列番号15に示される塩基配列又は該塩基配列の5'末端側に制限酵素認識配列を付加した塩基配列からなるプライマーと、配列番号16に示される塩基配列又は該塩基配列の5'末端側に制限酵素認識配列を付加した塩基配列からなるプライマーとからなる請求項17記載のプライマーセット。
【請求項19】
請求項17又は18記載のプライマーセットを用いた核酸増幅法によって植物細胞より分離した核酸試料から増幅された、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードするポリヌクレオチド。
【請求項20】
配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーを用いた核酸増幅法によって、植物細胞より分離した核酸試料からトバモウイルスL抵抗性遺伝子のコード領域の全長又は部分領域を増幅し、次いで該増幅断片の塩基配列を対比することを含む、L抵抗性遺伝子型の識別方法。
【請求項21】
前記対比は、一塩基多型解析又は増幅断片の制限断片長多型解析により行なわれる請求項20記載の方法。
【請求項22】
配列番号1に示すL3遺伝子配列中の3070nt〜3982ntの領域内に存在するL遺伝子間の塩基配列の多型をTaqI切断パターン及びHhaI切断パターンに基づいて調べることにより、L2、L2b、L3、L4及びL1c遺伝子型を識別することを含む請求項21記載の方法。
【請求項23】
配列番号17及び18に示す塩基配列からなるプライマーセットを用いてL抵抗性遺伝子のコード領域の一部を増幅し、次いで得られた増幅断片をTaqI及びHhaIで消化し、以下の基準でそれぞれのL遺伝子型を識別する請求項22記載の方法。
L2遺伝子型:TaqI消化断片のサイズ(bp)が470, 244, 185, 14であり、HhaI消化断片のサイズ(bp)が533, 283, 97である
L2b遺伝子型:TaqI消化断片のサイズ(bp)が470, 244, 98, 87, 14であり、HhaIでは消化されない
L3遺伝子型:TaqI消化断片のサイズ(bp)が470, 244, 98, 87, 14であり、HhaI消化断片のサイズ(bp)が533, 283, 97である
L4遺伝子型:TaqI消化断片のサイズ(bp)が470, 244, 98, 87, 14であり、HhaI消化断片のサイズ(bp)が816, 97である
L1c遺伝子型:TaqI消化断片のサイズ(bp)が470, 255, 98, 87, 14であり、HhaI消化断片のサイズ(bp)が636, 283である
【請求項24】
配列番号5に示すL1遺伝子配列中の3779ntに存在する一塩基多型を指標としてL1遺伝子型を識別することを含む請求項21記載の方法。
【請求項25】
配列番号19及び20に示す塩基配列からなるプライマーセットを用いてL抵抗性遺伝子のコード領域の一部を増幅し、次いで得られた増幅断片をXmnIで消化するPIRA-PCR法による解析において、29bpと117bpの断片に切断されるものをL1遺伝子型と識別する請求項24記載の方法。
【請求項26】
コード領域内に存在するL遺伝子間の塩基配列の多型をXbaI切断パターンに基づいて調べることによりL1a遺伝子型を識別することを含む請求項21記載の方法。
【請求項1】
配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列と90%以上の相同性を有し、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードするポリヌクレオチド。
【請求項2】
前記相同性が95%以上である請求項1記載のポリヌクレオチド。
【請求項3】
配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列、又は該塩基配列において1若しくは数個の塩基が置換し、欠失し若しくは挿入された塩基配列からなる請求項2記載のポリヌクレオチド。
【請求項4】
配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列からなる請求項3記載のポリヌクレオチド。
【請求項5】
配列表の配列番号2、4、6、8、10、12及び14のいずれかに示されるアミノ酸配列と90%以上の同一性を有し、植物にトバモウイルス抵抗性を付与できるポリペプチド。
【請求項6】
前記同一性が95%以上である請求項5記載のポリペプチド。
【請求項7】
配列番号2、4、6、8、10、12及び14のいずれかに示されるアミノ酸配列、又は該アミノ酸配列において1若しくは数個のアミノ酸が置換し、欠失し若しくは挿入されたアミノ酸配列からなる請求項6記載のポリペプチド。
【請求項8】
配列番号2、4、6、8、10、12及び14のいずれかに示されるアミノ酸配列から成る請求項7記載のポリペプチド。
【請求項9】
請求項5ないし8のいずれか1項に記載のポリペプチドをコードするポリヌクレオチド。
【請求項10】
請求項1ないし4及び9のいずれか1項に記載のポリヌクレオチドを含み、植物にトバモウイルス抵抗性を付与できるポリペプチドを宿主細胞内で発現させることができる組換えベクター。
【請求項11】
前記宿主が植物である請求項10記載の組換えベクター。
【請求項12】
請求項10又は11記載の組換えベクターを含む細胞。
【請求項13】
請求項11記載の組換えベクターにより形質転換された形質転換植物。
【請求項14】
請求項11記載の組換えベクターを植物細胞内に導入することを含む、トバモウイルス抵抗性が付与された形質転換植物の作製方法。
【請求項15】
配列表の配列番号21に示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチド。
【請求項16】
請求項17記載のポリヌクレオチドからなるトバモウイルス抵抗性遺伝子増幅用プライマー。
【請求項17】
請求項16記載のプライマーと、配列表の配列番号22に示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーとからなるトバモウイルス抵抗性遺伝子増幅用プライマーセット。
【請求項18】
配列番号15に示される塩基配列又は該塩基配列の5'末端側に制限酵素認識配列を付加した塩基配列からなるプライマーと、配列番号16に示される塩基配列又は該塩基配列の5'末端側に制限酵素認識配列を付加した塩基配列からなるプライマーとからなる請求項17記載のプライマーセット。
【請求項19】
請求項17又は18記載のプライマーセットを用いた核酸増幅法によって植物細胞より分離した核酸試料から増幅された、植物にトバモウイルス抵抗性を付与できるポリペプチドをコードするポリヌクレオチド。
【請求項20】
配列表の配列番号1、3、5、7、9、11及び13のいずれかに示される塩基配列中の部分領域と特異的にハイブリダイズするポリヌクレオチドからなるプライマーを用いた核酸増幅法によって、植物細胞より分離した核酸試料からトバモウイルスL抵抗性遺伝子のコード領域の全長又は部分領域を増幅し、次いで該増幅断片の塩基配列を対比することを含む、L抵抗性遺伝子型の識別方法。
【請求項21】
前記対比は、一塩基多型解析又は増幅断片の制限断片長多型解析により行なわれる請求項20記載の方法。
【請求項22】
配列番号1に示すL3遺伝子配列中の3070nt〜3982ntの領域内に存在するL遺伝子間の塩基配列の多型をTaqI切断パターン及びHhaI切断パターンに基づいて調べることにより、L2、L2b、L3、L4及びL1c遺伝子型を識別することを含む請求項21記載の方法。
【請求項23】
配列番号17及び18に示す塩基配列からなるプライマーセットを用いてL抵抗性遺伝子のコード領域の一部を増幅し、次いで得られた増幅断片をTaqI及びHhaIで消化し、以下の基準でそれぞれのL遺伝子型を識別する請求項22記載の方法。
L2遺伝子型:TaqI消化断片のサイズ(bp)が470, 244, 185, 14であり、HhaI消化断片のサイズ(bp)が533, 283, 97である
L2b遺伝子型:TaqI消化断片のサイズ(bp)が470, 244, 98, 87, 14であり、HhaIでは消化されない
L3遺伝子型:TaqI消化断片のサイズ(bp)が470, 244, 98, 87, 14であり、HhaI消化断片のサイズ(bp)が533, 283, 97である
L4遺伝子型:TaqI消化断片のサイズ(bp)が470, 244, 98, 87, 14であり、HhaI消化断片のサイズ(bp)が816, 97である
L1c遺伝子型:TaqI消化断片のサイズ(bp)が470, 255, 98, 87, 14であり、HhaI消化断片のサイズ(bp)が636, 283である
【請求項24】
配列番号5に示すL1遺伝子配列中の3779ntに存在する一塩基多型を指標としてL1遺伝子型を識別することを含む請求項21記載の方法。
【請求項25】
配列番号19及び20に示す塩基配列からなるプライマーセットを用いてL抵抗性遺伝子のコード領域の一部を増幅し、次いで得られた増幅断片をXmnIで消化するPIRA-PCR法による解析において、29bpと117bpの断片に切断されるものをL1遺伝子型と識別する請求項24記載の方法。
【請求項26】
コード領域内に存在するL遺伝子間の塩基配列の多型をXbaI切断パターンに基づいて調べることによりL1a遺伝子型を識別することを含む請求項21記載の方法。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【公開番号】特開2010−124746(P2010−124746A)
【公開日】平成22年6月10日(2010.6.10)
【国際特許分類】
【出願番号】特願2008−302360(P2008−302360)
【出願日】平成20年11月27日(2008.11.27)
【新規性喪失の例外の表示】特許法第30条第1項適用申請有り 平成20年7月29日 インターネットアドレス「http://www.springerlink.com/content/qr271384w3145161/fulltext.pdf(Theoretical and Applied Genetics (2008)Vol.117,No.7,p.1107‐1118)」に発表
【出願人】(390025793)岩手県 (38)
【出願人】(504174180)国立大学法人高知大学 (174)
【Fターム(参考)】
【公開日】平成22年6月10日(2010.6.10)
【国際特許分類】
【出願日】平成20年11月27日(2008.11.27)
【新規性喪失の例外の表示】特許法第30条第1項適用申請有り 平成20年7月29日 インターネットアドレス「http://www.springerlink.com/content/qr271384w3145161/fulltext.pdf(Theoretical and Applied Genetics (2008)Vol.117,No.7,p.1107‐1118)」に発表
【出願人】(390025793)岩手県 (38)
【出願人】(504174180)国立大学法人高知大学 (174)
【Fターム(参考)】
[ Back to top ]

