ネギ群検出プライマー及び検出方法
【課題】食品等の対象物から抽出したDNAを用いて、目視観察では判別の困難なネギ、タマネギ及びワケギのそれぞれを迅速、簡便かつ高感度に区別できるような検出系を構築する。
【解決手段】ネギ(Allium fistulosum)特異プライマー及びタマネギ(Allium cepa)特異プライマーを用いて、PCR法による特定の増幅産物の検出結果からネギ及びタマネギを特異的に検出する。また、これらのプライマーを用いた検出結果からワケギの存在を判断する。
【解決手段】ネギ(Allium fistulosum)特異プライマー及びタマネギ(Allium cepa)特異プライマーを用いて、PCR法による特定の増幅産物の検出結果からネギ及びタマネギを特異的に検出する。また、これらのプライマーを用いた検出結果からワケギの存在を判断する。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、ネギ属の主要な食用種である、ネギ・タマネギ・ワケギを検出対象とし、当該植物が食品原料や製品等に含まれていた場合に、その量が微量であっても高感度で検出することを可能とするネギ群の検出方法、その方法に使用するPCRプライマー等に関するものである。
【背景技術】
【0002】
“ネギ”は加工食品によく用いられる植物の一つである。一方、“ネギ”については、ワケギとその外観が類似しているため、市場において本来の生物種とは違った取り扱われ方をしているものがあり、地域性や食生活の違いによって異なる場合があることが知られている。
【0003】
すなわち、東日本では、白ネギが主流であるが、東日本において『ワケギ』とはワケネギというネギの一品種、もしく博多万能ネギ型の若い葉ネギをいう。遺伝学的分類ではネギ(Allium fistulosum)である。
【0004】
また、西日本では、青ネギ(葉ネギ)が主流であるが、西日本において『ワケギ』とは、ワケギ(Allium wakegi)。分けつ性のネギと、分球性のタマネギとの種間雑種で、アリウム・ワケギの学名が公認されている。ネギと違って種をつくらず、地下の球(鱗茎)で増える。
【0005】
市場において“ネギ”と称されるものについては、上述のように複数種あり、また、使用具材名称が外観や地域性などにより変動する曖昧なものとなっている。すなわち名称と実際の品種が混同されている場合がある。一方、食品の製造メーカにおいては、使用する資材についてこれらの正しい品種を迅速に確認して適切な表記とすることが必要である。
【0006】
加えて、ネギについては加工食品で用いられる場合、乾燥品が用いられる場合が多い。この場合、カットされて小片になる場合が多く、その形態から他の植物種やビニール片等と誤認されることがある。また、これらを確認しようとしても顕微鏡観察や外観のみで「ネギ」と判別することが困難な場合があった。
【0007】
このような観点から、微量のサンプル量であっても迅速に検査できる遺伝子を用いた検査方法を利用できれば便利である。すなわち、対象物からDNAを抽出し、その遺伝子の配列を調べる方法であれば、少ないサンプル量を用いて迅速に検査をすることができる。
【0008】
一方、このような遺伝子の配列を調べる方法として例えば、特許文献1が開示されている。特許文献1は内部転写スペーサー領域1(ITS1)又は内部転写スペーサー領域2(ITS2)をターゲットとし、ITS1又はITS2の各々の両側に存在する領域の塩基配列に基づいて設計された2つの植物ユニバーサルプライマーセットを構成し、そのプライマーセットを用い、異物から調製したDNAを鋳型とした増幅反応を行っている。この方法におけるサンプルの同定法は、増幅された核酸断片の塩基配列決定し、その塩基配列より生物種を特定するものである。
【0009】
【特許文献1】特開2004-337069
【0010】
上記特許文献1の方法によってネギ、ワケギを区別することはできる。しかし、この方法によっては塩基配列を決定するというステップが必要となるため煩雑さを伴う。
【0011】
また、上記のような目的を考えた場合、ネギ、ワケギそれぞれに特異的なプライマーがあればPCRによって迅速に検出することができる。しかし、従来までネギ、ワケギをターゲットとするPCRプライマーは存在しなかった。
【発明の開示】
【発明が解決しようとする課題】
【0012】
そこで、本発明者らは、対象物から抽出したDNAを用いて、迅速且つ簡便にネギ、ワケギのそれぞれを区別できるように、それぞれのネギ属について特異的遺伝子を高感度で検出することができるプライマー及びその検出方法を開発することを課題とした。
【課題を解決するための手段】
【0013】
本発明者らは上記課題の解決のために、ネギ、ワケギのそれぞれに対して特異的なプライマーを作成することを試みた。しかし、これら三種類のうち、ワケギについては種間雑種であり、ネギとタマネギの双方のゲノムを持っている。このような雑種植物においてワケギに特異的なプライマーを見出すことは困難であった。すなわち、ワケギを検出するためのプライマーを作製しようとすると、ネギにも反応してしまうプライマーとなってしまい一対のプライマーセットのみでワケギを特異的に検出することは困難であった。
【0014】
そこで、本発明者らは、視点を変えてタマネギを特異的に検出するプライマーを作成する方法を採用した。そして、ワケギについてはネギとタマネギを特異的に検出するプライマーの両方の結果により判断できるようにした。
【0015】
すなわち、本発明者らはネギ(Allium fistulosum)特異プライマー及びタマネギ(Allium cepa)特異プライマーを作製した。そして、これらのプライマーを利用することでネギ、タマネギ及びワケギを検出できるような検出方法を構築することができた。
【0016】
具体的には、本発明は、以下のPCRプライマー、PCRプライマーセットを提供する。
項1.配列表の配列番号1における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
項2.配列表の配列番号2における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
項3.配列表の配列番号3における塩基番号13〜24の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
項4.配列表の配列番号4における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
項5.請求項1記載のプライマーと請求項2記載のプライマーとからなるPCRプライマーセット。
項6.請求項3記載のプライマーと請求項4記載のプライマーとからなるPCRプライマーセット。
項7.試料からDNAを抽出する工程と、このDNAを鋳型として、請求項5記載のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にネギが存在しているか否かを検出する工程とを含むネギの検出方法。
項8.試料からDNAを抽出する工程と、このDNAを鋳型として、請求項6記載のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にタマネギが存在しているか否かを検出する工程とを含むタマネギの検出方法。
項9.試料からDNAを抽出する工程と、このDNAを鋳型として、請求項5記載のプライマーセットを用いてPCRを行う工程と、請求項6記載のプライマーセットを用いてPCRを行う工程と、前記それぞれのプライマーセットにより増幅されたDNAを検出することにより試料中にワケギが存在しているか否かを検出する工程とを含むワケギの検出方法。
【発明の効果】
【0017】
本発明のプライマーを用いてPCR検査することにより目視観察では判別の困難なネギ、タマネギ及びワケギを迅速に判断することができる。また、異物分析において当該異物がネギ群の由来かどうかを迅速に判断することができる。
【発明を実施するための最良の形態】
【0018】
以下、本発明を詳細に説明する。
ネギ群検出用プライマー
本発明のPCRプライマーセットは、ネギ属のうちネギに対して特徴的な塩基配列を有するもの、タマネギに対して特徴的な塩基配列を有するものである。尚、本発明にいうネギ群とは、ネギ(Allium fistulosum)、タマネギ(Allium cepa)及びワケギ(Allium wakegi)をいうものとする。
【0019】
それぞれのプライマーセットは以下のようにして設計した。候補遺伝子の選定として、NCBI検索によってAllium属のITS領域におけるデータより設計した。ITS領域について以下に説明する。核リボソームDNA(nrDNA)は5'- 18S - ITS1 - 5.8S - ITS2 - 26S -3'を1単位として反復配列の構造をとっている。
【0020】
そのコピー数は原生動物の1コピーからコムギで6400コピーといわれているが、多くの動物では1000コピー以内といわれている。内部転写スペーサー領域1(ITS1)および内部転写スペーサー領域2(ITS2)は、植物種ごとに特異的な非保存領域であり、比較的塩基長の短い塩基配列からなり、これらの両側に隣接する塩基配列、つまりITS1の場合は18SrRNA 及び5.8SrRNA、ITS2の場合5.8SrRNA及び25S 26 S rRNA)は、広く植物全般において保存され、かつ、植物と他の生物種を画する塩基配列の領域を有している。
【0021】
このITS領域はネギ属において種間の差異は見られるが、品種間での差異はまれである。よってネギ種を特異的に検出し、且つ多数存在するネギ品種も網羅できる、という点からITS領域はネギ属特異的プライマーの標的配列として好適であった。
【0022】
具体的には、核リボソームDNAのITS領域(internal Transcribed Spacer region)を抽出し、比較し、ネギ及びタマネギにそれぞれ特異的な塩基配列部分を選定した。さらに、その選定部位からPCRによって検出できる適当な大きさの増幅されたDNA断片を得られる特定のDNA領域を決定し、その領域から目的を達成しうるようなPCR検出プライマーセットを選定後、その性能を評価した。なお、検出対象とするネギの範囲や塩基配列の類似性を考慮して、ネギを検出できるプライマーセット及び、ネギ近縁のタマネギを検出できるプライマーセットの開発を行った。
【0023】
そして、本プライマーセットを用いて、PCR法によってネギ、タマネギの特定領域を増幅させ、増幅されたDNA断片を、アガロースゲル電気泳動して、その電気泳動のパターンより特定のサイズのDNA断片を検出することで、ネギ、タマネギ由来のDNAの存在を判断することができる。さらに、ネギとタマネギの結果からワケギのDNAの存在を判断することができる。また、リアルタイムPCR装置であれば、ゲル電気泳動に加えて、増幅されたDNA断片の融解曲線分析によっても、ネギ属由来のDNAの存在を判断することができる。
【0024】
さらに、他生物種と交差、且つプライマーダイマーを形成しないようプライマー設計した。また、プライマーを併用する場合を考慮し、プライマー使用条件を一律に設定できるように設計した。
【0025】
本発明が提供するプライマーの塩基配列は以下のとおりである。Sはセンスプライマーを表し、ASはアンチセンスプライマーを表す。
配列番号1(ネギS):TTG CGT TGT TTG GAT GGG TTC
配列番号2(ネギAS):CAC TCA CAG TGT AAG TTT ACC
配列番号3(タマネギS):TTG AAG TAA GAT GTA GAG TAG AAC
配列番号4(タマネギAS):ATC ACT CAC AGT ATG TTT ACA
また、上記の各プライマーは、その5´末端側には任意の配列が付加されて全体として最大30塩基のプライマーであってもよい。さらに、それぞれのプライマーについては、3´端から12塩基が鋳型DNAとマッチしていれば検出が可能である。
【0026】
そこで、本発明は、まず第1のプライマーとして、配列表の配列番号1における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマーを提案する。次に第2のプライマーとして、配列表の配列番号2における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマーを提案する。また、これらのプライマーをセットで用いることが好ましい。これらのプライマーをセットで用いることで、ネギ(Allium fistulosum) を検出することができる。
【0027】
次に、本発明は、第3のプライマーとして、配列表の配列番号3における塩基番号13〜24の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマーを提案する。さらに、第4のプライマーとして、配列表の配列番号4における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマーを提案する。また、これらのプライマーをセットで用いることが好ましい。これらのプライマーをセットで用いることで、タマネギ(Allium cepa)を検出することができる。
【0028】
ネギ群の検出方法
・検査対象物
本検出方法における検査対象物は特に限定されないが、具体的には、食品又は異物等が挙げられる。また、飲料等も可能である。具体的には、食品として生又は加熱した原材料や加工食品等が挙げられる。また、本発明はPCR法を用いるため微量のDNAが存在すれば検出可能である。従って微量の食品等があれば検出することができる。
【0029】
・DNAの抽出工程
検査対象物からのDNAの抽出については試料の形態については特に限定されない。一般的な公知のDNA抽出法(厚生労働省医薬局食品保健部長通知、アレルギー物質を含む食品の検査方法について、平成14年11月06日、食発第1106001号)や市販の各種DNA抽出キット[例えば、Nucleon PhytoPure, plant and fungal DNA extraction kits(Amersham Biosciences Corp., USA)、DNA Extraction IsoplantII kit(Nippon Gene Co. Ltd., Japan)、DNeasy Plant Mini Kit(Qiagen GmbH, Hilden, Germany)等]によって、DNAの回収が可能な試料であれば、上記の検出法に適用することができる。
【0030】
・本プライマーセットを用いたPCR工程
本発明のネギ属検出方法は、試料からDNAを抽出する工程と、このDNAを鋳型として上記説明した本発明のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にネギ、タマネギが存在していたかどうかを確認できる。また、ワケギは種間雑種であり、ネギとタマネギの双方のゲノムを持つため、ネギでの結果とタマネギでの結果から判断できる。
【0031】
PCR反応工程では、抽出されたDNAを鋳型として、ネギ属検出用のプライマーセットを用いてPCRを行う。PCR装置としては、例えば、ブロックタイプ、キャピラリータイプの市販の装置を使用できる。尚、迅速にDNAを増幅するためには、キャピラリータイプの装置が好ましい。また、キャピラリータイプのリアルタイムPCR装置を用いれば、増幅産物であるDNA断片の増加をリアルタイムでモニタリングすることができるため、ネギ群DNAの存在の有無を迅速に把握することができる。使用するプライマーの種類と検出可能なネギ群の対応について以下の表1に示す。
【0032】
【表1】

【0033】
・増幅されたDNA断片の検出
PCR増幅産物であるDNA断片の有無の確認は、通常のPCR装置であれば、増幅産物をゲル電気泳動することによって、また、リアルタイムPCR装置であれば、さらに、増幅産物の融解曲線分析を行い、その分析からPCR増幅産物の融解温度(Tm)値を導くことにより、PCR増幅産物が目的の増幅産物であるかどうかを確認できる。具体的には、おのおののプライマーセットにより、ネギ、タマネギのDNAが検出できる。各々の種に対するPCR増幅による理論DNA断片の大きさ及びTmは、概ね、以下の表2のようになる。
【0034】
【表2】

また、被検対象がネギ、タマネギとワケギであった場合に、それぞれのプライマーでどのように検出されるかを表3に示す。尚、表3に示すように、ワケギについては、ネギとタマネギの両方が陽性であった場合に検出されたものと判断する。
【0035】
【表3】

上記3種のネギ属特異プライマーを用いてPCR工程を行えば、ネギのみならず、ネギ近縁のタマネギ、あるいはネギとして使用されているワケギを特異的に識別・検出することができる。
【0036】
・マルチプレックスプライマーを用いた同時鑑定
本発明のネギ検出用プライマー(配列番号1及び配列番号2)及びタマネギ検出用プライマー(配列番号3及び配列番号4)については、これらプライマーを混合して用いることもできる。すなわち、これらのプライマーを混合したタイプを用いて、PCRを行うことにより、被験サンプルにネギ、タマネギ又はワケギのいずれかが存在していたかどうかを判断することができる。
【0037】
尚、PCR反応系においては、それぞれのプライマーの鋳型DNAに対するアニーリングし易さの相違から、タマネギを検出するための配列番号3及び配列番号4のプライマーセットの反応チューブへの添加モル数が、ネギを検出するための配列番号1及び配列番号2のプライマーセットの反応チューブへの添加モル数の3倍程度であることが好ましい。
【0038】
本マルチプレックスによってPCRを行った場合においては、標的サイズの増幅産物の検出は、378 bpの位置にバンドを確認すればネギDNA陽性とみなし、330 bpの位置にバンドを確認すればタマネギDNA陽性とみなし、330 bp且つ378bpの双方の位置にバンドを確認すればワケギDNA陽性とみなす。またバンドがいずれの位置にも確認されない、もしくはその他の位置にバンドが確認された場合、陰性とみなすことができる。
【0039】
・検査対象がワケギとタマネギの混合物であった場合の鑑定
稀な場合であるが、検査対象物がネギとタマネギの混合物であった場合においても本発明のおいてはワケギとの区別が可能である。ネギとタマネギの混合物であるか、又はワケギとの区別方法については、リアルタイムPCR法を用いてそれぞれのDNA量を推定し、その比率によって判別可能となる。すなわち、リアルタイムPCRを用いて指数関数的増幅期が認められるサイクル数を基準とし、元に存在した鋳型DNAの量を比較する。
【0040】
ワケギが検査対象物であった場合はネギ由来DNA量とタマネギ由来DNA量の比が一定になるが、混合物であった場合は、ネギとタマネギの量比が1:1でない限りは、ワケギの場合と同様の比率にはならないため区別が可能となる。
【0041】
具体的な方法としては、リアルタイムPCR法を用いてPCR増幅を行い、指数関数的増幅期のサイクル数を測定する。このサイクル数は、Applied Biosystems製のPCR装置ではCT(Threshold Cycle)値として知られている。また、Roche Diagnostics製のPCR装置ではCP(Crossing Point)値として知られている。
【0042】
指数関数的増幅期のサイクル数(CT値、CP値)は、指数関数的増幅領域の、ある一定の蛍光シグナル:F(Fluorescence)値で補助線を引き、その補助線と増幅曲線との交点から算出したサイクル数である。
【0043】
例えば、Light Cycler(Roche Diagnostics)を使用し、CP値(F=1)を算出する方法であると、本発明のプライマーを使用した場合には、ワケギの場合はCP値比(F=1)(タマネギ/ネギ):1.2前後の一定の数値となるが、そこから外れた場合(ネギの割合が多い場合はCP値比(F=1):1.3以上、タマネギの割合が多い場合はCP(F=1)値比:1.1前後)はネギとタマネギの混合物であることが分かる。
【実施例1】
【0044】
─ネギ属DNA検出用プライマーセットを用いたPCR分析の検出感度の確認─
ネギ、タマネギの各種に対するPCRプライマーの検出感度を確認するために以下の試験を行った。試料とするネギ(Allium fistulosum)、タマネギ(Allium cepa)は自社使用資材を使用した。これらの精製DNAは、各々の試料(フリーズドライ加工されたもの)の一部を70%エタノールで洗浄し、さらに、滅菌水で素早く洗浄後、マルチビーズショッカー(安井器械、大阪)で破砕した試料からDNeasy plant Mini kits(Qiagen GmbH, Germany)を用いて調製したものを使用した。これらのネギ属植物DNA量を測定後、滅菌水を用いて、DNAの濃度を1 ng/μLに調製し、その後1000pg〜0,001pgまで段階希釈をし、各濃度のDNAサンプルを作製した。
【0045】
PCRに供与する2μLのDNA試料液を含む20μL容量の反応液は、タカラバイオ社のSYBR Premix Ex TaqTM (Perfect Real Time)を用いて調製した。PCR反応溶液組成は以下の
表4の配合によった。
【0046】
【表4】

【0047】
PCR反応は以下の条件で行った。使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)で行い、増幅反応条件は以下の通りである。すなわち、ネギ特異プライマーの場合、配列番号1及び配列番号2のプライマーセットを用いた。また、タマネギ特異プライマーの場合、配列番号3及び配列番号4のプライマーセットを用いた。温度条件としては、95℃で1分間のサイクルを一回実施後、20℃/秒の速度で95℃まで昇温後、5秒間同温度で保温、次に20℃/秒の速度で60℃(アニーリング温度)まで降温後、10秒間同温度で保温、さらに10℃/秒の速度で72℃まで昇温後、20秒間同温度で保温する3つのステップからなる増幅サイクルを40回実施した。
【0048】
PCR増幅産物は、SYBR Green I依存性の蛍光量として、各々のサイクルの最終ステップに記録した。増幅サイクル終了後、メルティングカーブは、20℃/秒の速度で97℃まで昇温後、次に20℃/秒の速度で60℃まで降温し、10秒間同温度で保温後、さらに0.2℃/秒の速度で97℃に至るまでのゆるやかな昇温中に、0.2秒毎の蛍光強度を記録することによって得た。同PCR装置のソフトウェアによってメルティングカーブから得られたPCR増幅産物のTm値は、PCRによる特異的な増幅産物の有無を推定するために使用した。さらに、2.0%(wt/vol)アガロースゲル電気泳動によってPCR増幅産物(反応液の2μL)を分離し、分離後のゲルをエチジウムブロマイドで染色後、UV照射下において増幅産物を視覚化することによって、増幅産物の有無を確認した。分子量マーカーとして100bp DNA Ladder(New England BioLabs Inc., USA)を使用した。これらのPCRの結果を表5に記載した。表中の+(プラス)は電気泳動により標的サイズの増幅産物が検出されたことを示し、−(マイナス)は当該増幅産物が検出されなかったことを示す。
【0049】
尚、標的サイズの増幅産物の検出は、ネギ特異プライマーを使用した場合、378 bpの位置にバンドを確認すれば陽性とみなし、バンドがいずれの位置にも確認されない、もしくはその他の位置にバンドが確認された場合、陰性とみなす。また、タマネギ特異プライマーを使用した場合、330 bpの位置にバンドを確認すれば陽性とみなし、バンドがいずれの位置にも確認されない、もしくはその他の位置にバンドが確認された場合、陰性とみなす。ネギ特異プライマーおよびタマネギ特異プライマー双方使用した場合、378 bpの位置にバンドを確認すればネギDNA陽性とみなし、330 bpの位置にバンドを確認すればタマネギDNA陽性とみなし、330 bp且つ378bpの双方の位置にバンドを確認すればワケギDNA陽性とみなす。またバンドがいずれの位置にも確認されない、もしくはその他の位置にバンドが確認された場合、陰性とみなす。以上のような方法により判断した。結果を以下の表5に示す。
【0050】
【表5】

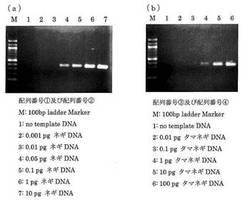
各プライマーの検出限界は、表5に記載してあるようにネギ特異プライマー:0.05 pg、タマネギ特異プライマー:1 pgであった。それぞれの電気泳動図を図1に示す。
【実施例2】
【0051】
─ネギ属DNA検出用プライマーセットの検出特異性の確認─
本発明の検出特異性を検討するために種々の遺伝子を用いて検出特異性を確認した。ヒトの精製DNAは、セマイン社(CeMines, LLC, USA)から購入したものを使用した。
ウシ、ブタ、ニワトリ、マサバ、カツオ、ベニザケ、ブリ、ホタテガイ、タラバガニ、コウイカ、イセエビ、マダコの精製DNAは、商店から購入した各々の試料の(筋)肉質部分を凍結後、マルチビーズショッカー(安井器械、大阪)で破砕した試料からNucleon PhytoPure, plantand fungal DNA extraction kits(Amersham Biosciences Corp., USA)または、DNA Extraction IsoplantII kit(Nippon Gene Co. Ltd., Japan)を用いて調製したものを使用した。
【0052】
コムギ及びコーンDNAは、バイオチェイン・インスティテュート社(BioChain Institute, Inc., USA)から購入したものを使用した。ソバの精製DNAは、商店から購入したそば粉をマルチビーズショッカーで、さらに粉砕した試料からNucleon PhytoPure, plant and fungal DNA extraction kitsを用いて調製したものを使用した。
【0053】
ネギ(元蔵葱、鶏脚葱、黒千本、宏太郎葱、興化香葱、小葱)、タマネギ(白色種)、アサツキは自社資材部から入手し、ネギ(信長葱、浅黄九条、下仁田葱、万能葱)、タマネギ(ペコロス、レッドオニオン)、ワケギ、ダイズ、ゴマ、ホウレンソウ、ニンジン、ゴボウ、アスパラガス、キャベツ、リーキは商店から購入し、イネは国内栽培農家から入手した。これらの精製DNAは、各々の試料の一部を70%エタノールで洗浄し、さらに、滅菌水で素早く洗浄後、マルチビーズショッカー(安井器械、大阪)で破砕した試料からDNeasy plant Mini kits(Qiagen GmbH, Germany)を用いて調製したものを使用した。アスペルギルス・ニガー(Aspergillus niger IFO 9642)、サッカロマイセス・セレビシエ(Saccharomyces cerevisiae IFO 0282)、大腸菌(Escherichia coli JCM 1649T)、及びバチルス・セレウス菌(Bacillus cereus DSM 4312)の精製DNAは、各々の菌株を適切な培地で培養後、その培養液からPuregene Yeast and Gram-Positive DNA Isolation kit(Gentra Systems Inc., USA)を用いて調製したものを使用した。なお、いずれの精製DNAもRNA分解酵素によるRNAの除去操作を実施してある。
【0054】
PCR反応で使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)で行い、増幅反応の条件は増幅サイクルを除いて実施例1と同様である。増幅サイクルは35回行った。これらのPCRの結果を表6に記載した。表中の+(プラス)は当該増幅産物が検出されたことを示し、−(マイナス)は当該増幅産物が検出されなかったことを示す。
【0055】
【表6】

【0056】
ネギ属プライマー2種を用いた他生物種DNAとの特異性試験の結果、非特異的な増幅は見られなかった。ネギ、およびタマネギ特異プライマーを用い、一般的に食用とされているネギ10 品種、タマネギ3品種について特異性試験を行ったところ、試験したすべてのネギ・タマネギ品種において検出可能であった。また、ワケギにおいてもネギ、およびタマネギ特異プライマー双方で検出可能であった。
【実施例3】
【0057】
─ネギ属DNA検出用プライマーセットを用いたPCR分析による市販加工食品中のネギ由来DNAの検出─
ネギ属植物を原材料として使用することを明記されている市販製品に対し、ネギ属プライマー3種を用い、実際に検出することができるかどうかを確認するために実施試験を行った。
【0058】
試料としては、市販加工製品中のネギ属植物を用いた。すなわち、ネギ属植物を原料として使用することを明記されている市販食品を準備し、それら市販製品に含有されているネギ属植物を1〜10 mg(乾燥重量)取り出し、各々の試料を70%エタノールで洗浄し、さらに、滅菌水で素早く洗浄後、マルチビーズショッカー(安井器械、大阪)で破砕した。これらの破砕物から各々のDNAをDNeasy plant Mini kits(Qiagen GmbH, Germany)を用いて抽出した。抽出時には、RNA分解酵素によるRNAの除去操作も実施した。
【0059】
DNA量測定後、DNA濃度を1 ng/μLに調整。濃度1 ng/μL以下のものについては低濃度のため濃度調整を行わなかった。PCR反応で使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)で行い、増幅反応の条件は実施例1と同様である。これらのPCRの結果を表7に記載した。表中の+(プラス)は当該増幅産物が検出されたことを示し、−(マイナス)は当該増幅産物が検出されなかったことを示す。
【0060】
【表7】

【0061】
さらに、食品試料DNAから増幅されたPCR増幅産物を、BigDye Terminator v3.1 Cycle
Sequencing kit(Applied Biosystems社)を用いて、両方向からダイレクトシーケンス後、Applied Biosystems 310 Genetic Analyzer(Applied Biosystems社)によって塩基配列を分析した。分析した塩基配列データをGenBank nucleotide sequence database(BLAST search)もしくは、発明者らが保有するDNAデータベースと比較解析し、その塩基配列の類似性に基づいて、PCR増幅産物由来DNAの生物種を帰属・同定した。以上の結果を総合すると以下のようになった。
【0062】
すなわち、ネギ検出用PCRプライマー(配列番号1及び2からなるPCRプライマーセット)を使用した場合、ネギ及びワケギを使用具材として使用している食品由来DNAにおいてのみ、増幅産物が確認され、この増幅産物のTm値は、約85.8℃であった。他の食品試料由来DNAを使用した場合には、増幅産物は見られなかった。なお、PCR増幅産物の塩基配列解析の結果、市販食品中のネギ由来DNAから増幅されたPCR増幅産物の塩基配列は、ネギのITS領域配列と一致した。すなわち、当該PCRプライマーを用いたPCRは、複数の市販食品において、ネギ非含有食品試料からネギ由来DNAやその他のDNAを検出しないが、ネギ含有食品試料からネギ由来DNAを特異的に検出できることを確認した。
【0063】
タマネギ検出用PCRプライマー(配列番号3及び4からなるPCRプライマーセット)を使用した場合、タマネギ及びワケギを使用具材として使用している食品由来DNAにおいてのみ、増幅産物が確認され、この増幅産物のTm値は、約85.4℃であった。他の食品試料由来DNAを使用した場合には、増幅産物は見られなかった。なお、PCR増幅産物の塩基配列解析の結果、市販食品中のタマネギ由来DNAから増幅されたPCR増幅産物の塩基配列は、タマネギのITS領域配列と一致した。
【実施例4】
【0064】
─マルチプレックスプライマー(ネギ+タマネギ)を用いた同時鑑定─
本発明のプライマー群を用いて、配列番号1〜4のプライマーを入れて、1回のPCRで検査することが可能であることを検証した。
【0065】
配列番号1及び2(ネギ特異プライマー)と配列番号3及び4(タマネギ特異プライマー)についてネギプライマー1に対してタマネギプライマー3の割合で混合して検出試験を行った。試料としては、ネギDNA(1 ng/μL)、タマネギDNA(1 ng/μL)、ワケギDNA(1 ng/μL)を用いて以下の表8の反応組成でPCR反応を行った。
【0066】
【表8】

【0067】
PCR反応で使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)であり行い、増幅反応の条件は増幅サイクル数を35回にした点を除いて実施例1と同様である。電気泳動図を図2に示す。
【0068】
一度にネギ及びワケギの両方のプライマー群(配列番号1〜4)をすべてPCR反応に供したが、ネギについては所定どおり378bpの位置に増幅産物を検出することができた。また、タマネギについても所定どおり330bpの位置の増幅産物を確認することができた。さらに、ワケギについても所定どおり、378bpの位置と330bpの位置の両方に増幅産物を検出することができた。また、目的以外の増幅産物は観察されなかった。
【実施例5】
【0069】
─ネギとタマネギが混在していた場合の鑑定─
検査対象中にネギとタマネギが混在した場合、ワケギと同様に2本のバンドが検出されることになり、増幅産物の有無の違いのみだけではネギとタマネギの混合物であるかワケギであるかを判断することはできない。一方、リアルタイムPCR法を用いてそれぞれのDNA量を推定しその比率によって判別可能かどうかについて実施試験を行った。
【0070】
リアルタイムPCRによって、指数関数的増幅期が認められるサイクル数を基準とし、元に存在した鋳型DNAの量を比較する方法で行った。
【0071】
試料としては、ネギDNA(1 ng/μL)、タマネギDNA(1 ng/μL)、ワケギDNA(1 ng/μL)、を用いて、それぞれを2、4、8倍希釈したサンプルを作製し、ネギDNAとタマネギDNAの混合比を変えて用いた。PCR反応は表9の組成で行った。
【0072】
【表9】

【0073】
PCR反応で使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)で行い、増幅反応の条件は増幅サイクル数を35回にした点を除いて実施例1と同様である。
【0074】
リアルタイムPCRによる解析はLight Cycler(ロシュ・ダイアグノスティックス)にてPCR増幅しCP値(F=1)測定した。
【0075】
尚、指数関数的増幅期のサイクル数(CP値)は、指数関数的増幅領域の、ある一定の蛍光シグナル:F(Fluorescence)値で補助線を引き、その補助線と増幅曲線との交点から算出したサイクル数となる。本実施例ではCP値(F=1)を算出した。結果を表10に示す。
【0076】
【表10】

【0077】
ワケギの場合はCP値比(F=1)(タマネギ/ネギ):1.2前後の一定の数値となるが、そこから外れた場合(ネギの割合が多い場合はCP値比(F=1):1.3以上、タマネギの割合が多い場合はCP(F=1)値比:1.1前後)はネギとタマネギの混合物であることが分かる。このように、ほとんどの場合、ネギとタマネギとの混合物とワケギの場合を区別することができる。
【図面の簡単な説明】
【0078】
【図1】各プライマーの検出限界を示した電気泳動図である。
【図2】マルチプレックスプライマーを用いた同時鑑定の結果を示した電気泳動図である。
【技術分野】
【0001】
本発明は、ネギ属の主要な食用種である、ネギ・タマネギ・ワケギを検出対象とし、当該植物が食品原料や製品等に含まれていた場合に、その量が微量であっても高感度で検出することを可能とするネギ群の検出方法、その方法に使用するPCRプライマー等に関するものである。
【背景技術】
【0002】
“ネギ”は加工食品によく用いられる植物の一つである。一方、“ネギ”については、ワケギとその外観が類似しているため、市場において本来の生物種とは違った取り扱われ方をしているものがあり、地域性や食生活の違いによって異なる場合があることが知られている。
【0003】
すなわち、東日本では、白ネギが主流であるが、東日本において『ワケギ』とはワケネギというネギの一品種、もしく博多万能ネギ型の若い葉ネギをいう。遺伝学的分類ではネギ(Allium fistulosum)である。
【0004】
また、西日本では、青ネギ(葉ネギ)が主流であるが、西日本において『ワケギ』とは、ワケギ(Allium wakegi)。分けつ性のネギと、分球性のタマネギとの種間雑種で、アリウム・ワケギの学名が公認されている。ネギと違って種をつくらず、地下の球(鱗茎)で増える。
【0005】
市場において“ネギ”と称されるものについては、上述のように複数種あり、また、使用具材名称が外観や地域性などにより変動する曖昧なものとなっている。すなわち名称と実際の品種が混同されている場合がある。一方、食品の製造メーカにおいては、使用する資材についてこれらの正しい品種を迅速に確認して適切な表記とすることが必要である。
【0006】
加えて、ネギについては加工食品で用いられる場合、乾燥品が用いられる場合が多い。この場合、カットされて小片になる場合が多く、その形態から他の植物種やビニール片等と誤認されることがある。また、これらを確認しようとしても顕微鏡観察や外観のみで「ネギ」と判別することが困難な場合があった。
【0007】
このような観点から、微量のサンプル量であっても迅速に検査できる遺伝子を用いた検査方法を利用できれば便利である。すなわち、対象物からDNAを抽出し、その遺伝子の配列を調べる方法であれば、少ないサンプル量を用いて迅速に検査をすることができる。
【0008】
一方、このような遺伝子の配列を調べる方法として例えば、特許文献1が開示されている。特許文献1は内部転写スペーサー領域1(ITS1)又は内部転写スペーサー領域2(ITS2)をターゲットとし、ITS1又はITS2の各々の両側に存在する領域の塩基配列に基づいて設計された2つの植物ユニバーサルプライマーセットを構成し、そのプライマーセットを用い、異物から調製したDNAを鋳型とした増幅反応を行っている。この方法におけるサンプルの同定法は、増幅された核酸断片の塩基配列決定し、その塩基配列より生物種を特定するものである。
【0009】
【特許文献1】特開2004-337069
【0010】
上記特許文献1の方法によってネギ、ワケギを区別することはできる。しかし、この方法によっては塩基配列を決定するというステップが必要となるため煩雑さを伴う。
【0011】
また、上記のような目的を考えた場合、ネギ、ワケギそれぞれに特異的なプライマーがあればPCRによって迅速に検出することができる。しかし、従来までネギ、ワケギをターゲットとするPCRプライマーは存在しなかった。
【発明の開示】
【発明が解決しようとする課題】
【0012】
そこで、本発明者らは、対象物から抽出したDNAを用いて、迅速且つ簡便にネギ、ワケギのそれぞれを区別できるように、それぞれのネギ属について特異的遺伝子を高感度で検出することができるプライマー及びその検出方法を開発することを課題とした。
【課題を解決するための手段】
【0013】
本発明者らは上記課題の解決のために、ネギ、ワケギのそれぞれに対して特異的なプライマーを作成することを試みた。しかし、これら三種類のうち、ワケギについては種間雑種であり、ネギとタマネギの双方のゲノムを持っている。このような雑種植物においてワケギに特異的なプライマーを見出すことは困難であった。すなわち、ワケギを検出するためのプライマーを作製しようとすると、ネギにも反応してしまうプライマーとなってしまい一対のプライマーセットのみでワケギを特異的に検出することは困難であった。
【0014】
そこで、本発明者らは、視点を変えてタマネギを特異的に検出するプライマーを作成する方法を採用した。そして、ワケギについてはネギとタマネギを特異的に検出するプライマーの両方の結果により判断できるようにした。
【0015】
すなわち、本発明者らはネギ(Allium fistulosum)特異プライマー及びタマネギ(Allium cepa)特異プライマーを作製した。そして、これらのプライマーを利用することでネギ、タマネギ及びワケギを検出できるような検出方法を構築することができた。
【0016】
具体的には、本発明は、以下のPCRプライマー、PCRプライマーセットを提供する。
項1.配列表の配列番号1における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
項2.配列表の配列番号2における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
項3.配列表の配列番号3における塩基番号13〜24の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
項4.配列表の配列番号4における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
項5.請求項1記載のプライマーと請求項2記載のプライマーとからなるPCRプライマーセット。
項6.請求項3記載のプライマーと請求項4記載のプライマーとからなるPCRプライマーセット。
項7.試料からDNAを抽出する工程と、このDNAを鋳型として、請求項5記載のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にネギが存在しているか否かを検出する工程とを含むネギの検出方法。
項8.試料からDNAを抽出する工程と、このDNAを鋳型として、請求項6記載のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にタマネギが存在しているか否かを検出する工程とを含むタマネギの検出方法。
項9.試料からDNAを抽出する工程と、このDNAを鋳型として、請求項5記載のプライマーセットを用いてPCRを行う工程と、請求項6記載のプライマーセットを用いてPCRを行う工程と、前記それぞれのプライマーセットにより増幅されたDNAを検出することにより試料中にワケギが存在しているか否かを検出する工程とを含むワケギの検出方法。
【発明の効果】
【0017】
本発明のプライマーを用いてPCR検査することにより目視観察では判別の困難なネギ、タマネギ及びワケギを迅速に判断することができる。また、異物分析において当該異物がネギ群の由来かどうかを迅速に判断することができる。
【発明を実施するための最良の形態】
【0018】
以下、本発明を詳細に説明する。
ネギ群検出用プライマー
本発明のPCRプライマーセットは、ネギ属のうちネギに対して特徴的な塩基配列を有するもの、タマネギに対して特徴的な塩基配列を有するものである。尚、本発明にいうネギ群とは、ネギ(Allium fistulosum)、タマネギ(Allium cepa)及びワケギ(Allium wakegi)をいうものとする。
【0019】
それぞれのプライマーセットは以下のようにして設計した。候補遺伝子の選定として、NCBI検索によってAllium属のITS領域におけるデータより設計した。ITS領域について以下に説明する。核リボソームDNA(nrDNA)は5'- 18S - ITS1 - 5.8S - ITS2 - 26S -3'を1単位として反復配列の構造をとっている。
【0020】
そのコピー数は原生動物の1コピーからコムギで6400コピーといわれているが、多くの動物では1000コピー以内といわれている。内部転写スペーサー領域1(ITS1)および内部転写スペーサー領域2(ITS2)は、植物種ごとに特異的な非保存領域であり、比較的塩基長の短い塩基配列からなり、これらの両側に隣接する塩基配列、つまりITS1の場合は18SrRNA 及び5.8SrRNA、ITS2の場合5.8SrRNA及び25S 26 S rRNA)は、広く植物全般において保存され、かつ、植物と他の生物種を画する塩基配列の領域を有している。
【0021】
このITS領域はネギ属において種間の差異は見られるが、品種間での差異はまれである。よってネギ種を特異的に検出し、且つ多数存在するネギ品種も網羅できる、という点からITS領域はネギ属特異的プライマーの標的配列として好適であった。
【0022】
具体的には、核リボソームDNAのITS領域(internal Transcribed Spacer region)を抽出し、比較し、ネギ及びタマネギにそれぞれ特異的な塩基配列部分を選定した。さらに、その選定部位からPCRによって検出できる適当な大きさの増幅されたDNA断片を得られる特定のDNA領域を決定し、その領域から目的を達成しうるようなPCR検出プライマーセットを選定後、その性能を評価した。なお、検出対象とするネギの範囲や塩基配列の類似性を考慮して、ネギを検出できるプライマーセット及び、ネギ近縁のタマネギを検出できるプライマーセットの開発を行った。
【0023】
そして、本プライマーセットを用いて、PCR法によってネギ、タマネギの特定領域を増幅させ、増幅されたDNA断片を、アガロースゲル電気泳動して、その電気泳動のパターンより特定のサイズのDNA断片を検出することで、ネギ、タマネギ由来のDNAの存在を判断することができる。さらに、ネギとタマネギの結果からワケギのDNAの存在を判断することができる。また、リアルタイムPCR装置であれば、ゲル電気泳動に加えて、増幅されたDNA断片の融解曲線分析によっても、ネギ属由来のDNAの存在を判断することができる。
【0024】
さらに、他生物種と交差、且つプライマーダイマーを形成しないようプライマー設計した。また、プライマーを併用する場合を考慮し、プライマー使用条件を一律に設定できるように設計した。
【0025】
本発明が提供するプライマーの塩基配列は以下のとおりである。Sはセンスプライマーを表し、ASはアンチセンスプライマーを表す。
配列番号1(ネギS):TTG CGT TGT TTG GAT GGG TTC
配列番号2(ネギAS):CAC TCA CAG TGT AAG TTT ACC
配列番号3(タマネギS):TTG AAG TAA GAT GTA GAG TAG AAC
配列番号4(タマネギAS):ATC ACT CAC AGT ATG TTT ACA
また、上記の各プライマーは、その5´末端側には任意の配列が付加されて全体として最大30塩基のプライマーであってもよい。さらに、それぞれのプライマーについては、3´端から12塩基が鋳型DNAとマッチしていれば検出が可能である。
【0026】
そこで、本発明は、まず第1のプライマーとして、配列表の配列番号1における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマーを提案する。次に第2のプライマーとして、配列表の配列番号2における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマーを提案する。また、これらのプライマーをセットで用いることが好ましい。これらのプライマーをセットで用いることで、ネギ(Allium fistulosum) を検出することができる。
【0027】
次に、本発明は、第3のプライマーとして、配列表の配列番号3における塩基番号13〜24の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマーを提案する。さらに、第4のプライマーとして、配列表の配列番号4における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマーを提案する。また、これらのプライマーをセットで用いることが好ましい。これらのプライマーをセットで用いることで、タマネギ(Allium cepa)を検出することができる。
【0028】
ネギ群の検出方法
・検査対象物
本検出方法における検査対象物は特に限定されないが、具体的には、食品又は異物等が挙げられる。また、飲料等も可能である。具体的には、食品として生又は加熱した原材料や加工食品等が挙げられる。また、本発明はPCR法を用いるため微量のDNAが存在すれば検出可能である。従って微量の食品等があれば検出することができる。
【0029】
・DNAの抽出工程
検査対象物からのDNAの抽出については試料の形態については特に限定されない。一般的な公知のDNA抽出法(厚生労働省医薬局食品保健部長通知、アレルギー物質を含む食品の検査方法について、平成14年11月06日、食発第1106001号)や市販の各種DNA抽出キット[例えば、Nucleon PhytoPure, plant and fungal DNA extraction kits(Amersham Biosciences Corp., USA)、DNA Extraction IsoplantII kit(Nippon Gene Co. Ltd., Japan)、DNeasy Plant Mini Kit(Qiagen GmbH, Hilden, Germany)等]によって、DNAの回収が可能な試料であれば、上記の検出法に適用することができる。
【0030】
・本プライマーセットを用いたPCR工程
本発明のネギ属検出方法は、試料からDNAを抽出する工程と、このDNAを鋳型として上記説明した本発明のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にネギ、タマネギが存在していたかどうかを確認できる。また、ワケギは種間雑種であり、ネギとタマネギの双方のゲノムを持つため、ネギでの結果とタマネギでの結果から判断できる。
【0031】
PCR反応工程では、抽出されたDNAを鋳型として、ネギ属検出用のプライマーセットを用いてPCRを行う。PCR装置としては、例えば、ブロックタイプ、キャピラリータイプの市販の装置を使用できる。尚、迅速にDNAを増幅するためには、キャピラリータイプの装置が好ましい。また、キャピラリータイプのリアルタイムPCR装置を用いれば、増幅産物であるDNA断片の増加をリアルタイムでモニタリングすることができるため、ネギ群DNAの存在の有無を迅速に把握することができる。使用するプライマーの種類と検出可能なネギ群の対応について以下の表1に示す。
【0032】
【表1】

【0033】
・増幅されたDNA断片の検出
PCR増幅産物であるDNA断片の有無の確認は、通常のPCR装置であれば、増幅産物をゲル電気泳動することによって、また、リアルタイムPCR装置であれば、さらに、増幅産物の融解曲線分析を行い、その分析からPCR増幅産物の融解温度(Tm)値を導くことにより、PCR増幅産物が目的の増幅産物であるかどうかを確認できる。具体的には、おのおののプライマーセットにより、ネギ、タマネギのDNAが検出できる。各々の種に対するPCR増幅による理論DNA断片の大きさ及びTmは、概ね、以下の表2のようになる。
【0034】
【表2】

また、被検対象がネギ、タマネギとワケギであった場合に、それぞれのプライマーでどのように検出されるかを表3に示す。尚、表3に示すように、ワケギについては、ネギとタマネギの両方が陽性であった場合に検出されたものと判断する。
【0035】
【表3】

上記3種のネギ属特異プライマーを用いてPCR工程を行えば、ネギのみならず、ネギ近縁のタマネギ、あるいはネギとして使用されているワケギを特異的に識別・検出することができる。
【0036】
・マルチプレックスプライマーを用いた同時鑑定
本発明のネギ検出用プライマー(配列番号1及び配列番号2)及びタマネギ検出用プライマー(配列番号3及び配列番号4)については、これらプライマーを混合して用いることもできる。すなわち、これらのプライマーを混合したタイプを用いて、PCRを行うことにより、被験サンプルにネギ、タマネギ又はワケギのいずれかが存在していたかどうかを判断することができる。
【0037】
尚、PCR反応系においては、それぞれのプライマーの鋳型DNAに対するアニーリングし易さの相違から、タマネギを検出するための配列番号3及び配列番号4のプライマーセットの反応チューブへの添加モル数が、ネギを検出するための配列番号1及び配列番号2のプライマーセットの反応チューブへの添加モル数の3倍程度であることが好ましい。
【0038】
本マルチプレックスによってPCRを行った場合においては、標的サイズの増幅産物の検出は、378 bpの位置にバンドを確認すればネギDNA陽性とみなし、330 bpの位置にバンドを確認すればタマネギDNA陽性とみなし、330 bp且つ378bpの双方の位置にバンドを確認すればワケギDNA陽性とみなす。またバンドがいずれの位置にも確認されない、もしくはその他の位置にバンドが確認された場合、陰性とみなすことができる。
【0039】
・検査対象がワケギとタマネギの混合物であった場合の鑑定
稀な場合であるが、検査対象物がネギとタマネギの混合物であった場合においても本発明のおいてはワケギとの区別が可能である。ネギとタマネギの混合物であるか、又はワケギとの区別方法については、リアルタイムPCR法を用いてそれぞれのDNA量を推定し、その比率によって判別可能となる。すなわち、リアルタイムPCRを用いて指数関数的増幅期が認められるサイクル数を基準とし、元に存在した鋳型DNAの量を比較する。
【0040】
ワケギが検査対象物であった場合はネギ由来DNA量とタマネギ由来DNA量の比が一定になるが、混合物であった場合は、ネギとタマネギの量比が1:1でない限りは、ワケギの場合と同様の比率にはならないため区別が可能となる。
【0041】
具体的な方法としては、リアルタイムPCR法を用いてPCR増幅を行い、指数関数的増幅期のサイクル数を測定する。このサイクル数は、Applied Biosystems製のPCR装置ではCT(Threshold Cycle)値として知られている。また、Roche Diagnostics製のPCR装置ではCP(Crossing Point)値として知られている。
【0042】
指数関数的増幅期のサイクル数(CT値、CP値)は、指数関数的増幅領域の、ある一定の蛍光シグナル:F(Fluorescence)値で補助線を引き、その補助線と増幅曲線との交点から算出したサイクル数である。
【0043】
例えば、Light Cycler(Roche Diagnostics)を使用し、CP値(F=1)を算出する方法であると、本発明のプライマーを使用した場合には、ワケギの場合はCP値比(F=1)(タマネギ/ネギ):1.2前後の一定の数値となるが、そこから外れた場合(ネギの割合が多い場合はCP値比(F=1):1.3以上、タマネギの割合が多い場合はCP(F=1)値比:1.1前後)はネギとタマネギの混合物であることが分かる。
【実施例1】
【0044】
─ネギ属DNA検出用プライマーセットを用いたPCR分析の検出感度の確認─
ネギ、タマネギの各種に対するPCRプライマーの検出感度を確認するために以下の試験を行った。試料とするネギ(Allium fistulosum)、タマネギ(Allium cepa)は自社使用資材を使用した。これらの精製DNAは、各々の試料(フリーズドライ加工されたもの)の一部を70%エタノールで洗浄し、さらに、滅菌水で素早く洗浄後、マルチビーズショッカー(安井器械、大阪)で破砕した試料からDNeasy plant Mini kits(Qiagen GmbH, Germany)を用いて調製したものを使用した。これらのネギ属植物DNA量を測定後、滅菌水を用いて、DNAの濃度を1 ng/μLに調製し、その後1000pg〜0,001pgまで段階希釈をし、各濃度のDNAサンプルを作製した。
【0045】
PCRに供与する2μLのDNA試料液を含む20μL容量の反応液は、タカラバイオ社のSYBR Premix Ex TaqTM (Perfect Real Time)を用いて調製した。PCR反応溶液組成は以下の
表4の配合によった。
【0046】
【表4】

【0047】
PCR反応は以下の条件で行った。使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)で行い、増幅反応条件は以下の通りである。すなわち、ネギ特異プライマーの場合、配列番号1及び配列番号2のプライマーセットを用いた。また、タマネギ特異プライマーの場合、配列番号3及び配列番号4のプライマーセットを用いた。温度条件としては、95℃で1分間のサイクルを一回実施後、20℃/秒の速度で95℃まで昇温後、5秒間同温度で保温、次に20℃/秒の速度で60℃(アニーリング温度)まで降温後、10秒間同温度で保温、さらに10℃/秒の速度で72℃まで昇温後、20秒間同温度で保温する3つのステップからなる増幅サイクルを40回実施した。
【0048】
PCR増幅産物は、SYBR Green I依存性の蛍光量として、各々のサイクルの最終ステップに記録した。増幅サイクル終了後、メルティングカーブは、20℃/秒の速度で97℃まで昇温後、次に20℃/秒の速度で60℃まで降温し、10秒間同温度で保温後、さらに0.2℃/秒の速度で97℃に至るまでのゆるやかな昇温中に、0.2秒毎の蛍光強度を記録することによって得た。同PCR装置のソフトウェアによってメルティングカーブから得られたPCR増幅産物のTm値は、PCRによる特異的な増幅産物の有無を推定するために使用した。さらに、2.0%(wt/vol)アガロースゲル電気泳動によってPCR増幅産物(反応液の2μL)を分離し、分離後のゲルをエチジウムブロマイドで染色後、UV照射下において増幅産物を視覚化することによって、増幅産物の有無を確認した。分子量マーカーとして100bp DNA Ladder(New England BioLabs Inc., USA)を使用した。これらのPCRの結果を表5に記載した。表中の+(プラス)は電気泳動により標的サイズの増幅産物が検出されたことを示し、−(マイナス)は当該増幅産物が検出されなかったことを示す。
【0049】
尚、標的サイズの増幅産物の検出は、ネギ特異プライマーを使用した場合、378 bpの位置にバンドを確認すれば陽性とみなし、バンドがいずれの位置にも確認されない、もしくはその他の位置にバンドが確認された場合、陰性とみなす。また、タマネギ特異プライマーを使用した場合、330 bpの位置にバンドを確認すれば陽性とみなし、バンドがいずれの位置にも確認されない、もしくはその他の位置にバンドが確認された場合、陰性とみなす。ネギ特異プライマーおよびタマネギ特異プライマー双方使用した場合、378 bpの位置にバンドを確認すればネギDNA陽性とみなし、330 bpの位置にバンドを確認すればタマネギDNA陽性とみなし、330 bp且つ378bpの双方の位置にバンドを確認すればワケギDNA陽性とみなす。またバンドがいずれの位置にも確認されない、もしくはその他の位置にバンドが確認された場合、陰性とみなす。以上のような方法により判断した。結果を以下の表5に示す。
【0050】
【表5】

各プライマーの検出限界は、表5に記載してあるようにネギ特異プライマー:0.05 pg、タマネギ特異プライマー:1 pgであった。それぞれの電気泳動図を図1に示す。
【実施例2】
【0051】
─ネギ属DNA検出用プライマーセットの検出特異性の確認─
本発明の検出特異性を検討するために種々の遺伝子を用いて検出特異性を確認した。ヒトの精製DNAは、セマイン社(CeMines, LLC, USA)から購入したものを使用した。
ウシ、ブタ、ニワトリ、マサバ、カツオ、ベニザケ、ブリ、ホタテガイ、タラバガニ、コウイカ、イセエビ、マダコの精製DNAは、商店から購入した各々の試料の(筋)肉質部分を凍結後、マルチビーズショッカー(安井器械、大阪)で破砕した試料からNucleon PhytoPure, plantand fungal DNA extraction kits(Amersham Biosciences Corp., USA)または、DNA Extraction IsoplantII kit(Nippon Gene Co. Ltd., Japan)を用いて調製したものを使用した。
【0052】
コムギ及びコーンDNAは、バイオチェイン・インスティテュート社(BioChain Institute, Inc., USA)から購入したものを使用した。ソバの精製DNAは、商店から購入したそば粉をマルチビーズショッカーで、さらに粉砕した試料からNucleon PhytoPure, plant and fungal DNA extraction kitsを用いて調製したものを使用した。
【0053】
ネギ(元蔵葱、鶏脚葱、黒千本、宏太郎葱、興化香葱、小葱)、タマネギ(白色種)、アサツキは自社資材部から入手し、ネギ(信長葱、浅黄九条、下仁田葱、万能葱)、タマネギ(ペコロス、レッドオニオン)、ワケギ、ダイズ、ゴマ、ホウレンソウ、ニンジン、ゴボウ、アスパラガス、キャベツ、リーキは商店から購入し、イネは国内栽培農家から入手した。これらの精製DNAは、各々の試料の一部を70%エタノールで洗浄し、さらに、滅菌水で素早く洗浄後、マルチビーズショッカー(安井器械、大阪)で破砕した試料からDNeasy plant Mini kits(Qiagen GmbH, Germany)を用いて調製したものを使用した。アスペルギルス・ニガー(Aspergillus niger IFO 9642)、サッカロマイセス・セレビシエ(Saccharomyces cerevisiae IFO 0282)、大腸菌(Escherichia coli JCM 1649T)、及びバチルス・セレウス菌(Bacillus cereus DSM 4312)の精製DNAは、各々の菌株を適切な培地で培養後、その培養液からPuregene Yeast and Gram-Positive DNA Isolation kit(Gentra Systems Inc., USA)を用いて調製したものを使用した。なお、いずれの精製DNAもRNA分解酵素によるRNAの除去操作を実施してある。
【0054】
PCR反応で使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)で行い、増幅反応の条件は増幅サイクルを除いて実施例1と同様である。増幅サイクルは35回行った。これらのPCRの結果を表6に記載した。表中の+(プラス)は当該増幅産物が検出されたことを示し、−(マイナス)は当該増幅産物が検出されなかったことを示す。
【0055】
【表6】

【0056】
ネギ属プライマー2種を用いた他生物種DNAとの特異性試験の結果、非特異的な増幅は見られなかった。ネギ、およびタマネギ特異プライマーを用い、一般的に食用とされているネギ10 品種、タマネギ3品種について特異性試験を行ったところ、試験したすべてのネギ・タマネギ品種において検出可能であった。また、ワケギにおいてもネギ、およびタマネギ特異プライマー双方で検出可能であった。
【実施例3】
【0057】
─ネギ属DNA検出用プライマーセットを用いたPCR分析による市販加工食品中のネギ由来DNAの検出─
ネギ属植物を原材料として使用することを明記されている市販製品に対し、ネギ属プライマー3種を用い、実際に検出することができるかどうかを確認するために実施試験を行った。
【0058】
試料としては、市販加工製品中のネギ属植物を用いた。すなわち、ネギ属植物を原料として使用することを明記されている市販食品を準備し、それら市販製品に含有されているネギ属植物を1〜10 mg(乾燥重量)取り出し、各々の試料を70%エタノールで洗浄し、さらに、滅菌水で素早く洗浄後、マルチビーズショッカー(安井器械、大阪)で破砕した。これらの破砕物から各々のDNAをDNeasy plant Mini kits(Qiagen GmbH, Germany)を用いて抽出した。抽出時には、RNA分解酵素によるRNAの除去操作も実施した。
【0059】
DNA量測定後、DNA濃度を1 ng/μLに調整。濃度1 ng/μL以下のものについては低濃度のため濃度調整を行わなかった。PCR反応で使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)で行い、増幅反応の条件は実施例1と同様である。これらのPCRの結果を表7に記載した。表中の+(プラス)は当該増幅産物が検出されたことを示し、−(マイナス)は当該増幅産物が検出されなかったことを示す。
【0060】
【表7】

【0061】
さらに、食品試料DNAから増幅されたPCR増幅産物を、BigDye Terminator v3.1 Cycle
Sequencing kit(Applied Biosystems社)を用いて、両方向からダイレクトシーケンス後、Applied Biosystems 310 Genetic Analyzer(Applied Biosystems社)によって塩基配列を分析した。分析した塩基配列データをGenBank nucleotide sequence database(BLAST search)もしくは、発明者らが保有するDNAデータベースと比較解析し、その塩基配列の類似性に基づいて、PCR増幅産物由来DNAの生物種を帰属・同定した。以上の結果を総合すると以下のようになった。
【0062】
すなわち、ネギ検出用PCRプライマー(配列番号1及び2からなるPCRプライマーセット)を使用した場合、ネギ及びワケギを使用具材として使用している食品由来DNAにおいてのみ、増幅産物が確認され、この増幅産物のTm値は、約85.8℃であった。他の食品試料由来DNAを使用した場合には、増幅産物は見られなかった。なお、PCR増幅産物の塩基配列解析の結果、市販食品中のネギ由来DNAから増幅されたPCR増幅産物の塩基配列は、ネギのITS領域配列と一致した。すなわち、当該PCRプライマーを用いたPCRは、複数の市販食品において、ネギ非含有食品試料からネギ由来DNAやその他のDNAを検出しないが、ネギ含有食品試料からネギ由来DNAを特異的に検出できることを確認した。
【0063】
タマネギ検出用PCRプライマー(配列番号3及び4からなるPCRプライマーセット)を使用した場合、タマネギ及びワケギを使用具材として使用している食品由来DNAにおいてのみ、増幅産物が確認され、この増幅産物のTm値は、約85.4℃であった。他の食品試料由来DNAを使用した場合には、増幅産物は見られなかった。なお、PCR増幅産物の塩基配列解析の結果、市販食品中のタマネギ由来DNAから増幅されたPCR増幅産物の塩基配列は、タマネギのITS領域配列と一致した。
【実施例4】
【0064】
─マルチプレックスプライマー(ネギ+タマネギ)を用いた同時鑑定─
本発明のプライマー群を用いて、配列番号1〜4のプライマーを入れて、1回のPCRで検査することが可能であることを検証した。
【0065】
配列番号1及び2(ネギ特異プライマー)と配列番号3及び4(タマネギ特異プライマー)についてネギプライマー1に対してタマネギプライマー3の割合で混合して検出試験を行った。試料としては、ネギDNA(1 ng/μL)、タマネギDNA(1 ng/μL)、ワケギDNA(1 ng/μL)を用いて以下の表8の反応組成でPCR反応を行った。
【0066】
【表8】

【0067】
PCR反応で使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)であり行い、増幅反応の条件は増幅サイクル数を35回にした点を除いて実施例1と同様である。電気泳動図を図2に示す。
【0068】
一度にネギ及びワケギの両方のプライマー群(配列番号1〜4)をすべてPCR反応に供したが、ネギについては所定どおり378bpの位置に増幅産物を検出することができた。また、タマネギについても所定どおり330bpの位置の増幅産物を確認することができた。さらに、ワケギについても所定どおり、378bpの位置と330bpの位置の両方に増幅産物を検出することができた。また、目的以外の増幅産物は観察されなかった。
【実施例5】
【0069】
─ネギとタマネギが混在していた場合の鑑定─
検査対象中にネギとタマネギが混在した場合、ワケギと同様に2本のバンドが検出されることになり、増幅産物の有無の違いのみだけではネギとタマネギの混合物であるかワケギであるかを判断することはできない。一方、リアルタイムPCR法を用いてそれぞれのDNA量を推定しその比率によって判別可能かどうかについて実施試験を行った。
【0070】
リアルタイムPCRによって、指数関数的増幅期が認められるサイクル数を基準とし、元に存在した鋳型DNAの量を比較する方法で行った。
【0071】
試料としては、ネギDNA(1 ng/μL)、タマネギDNA(1 ng/μL)、ワケギDNA(1 ng/μL)、を用いて、それぞれを2、4、8倍希釈したサンプルを作製し、ネギDNAとタマネギDNAの混合比を変えて用いた。PCR反応は表9の組成で行った。
【0072】
【表9】

【0073】
PCR反応で使用した機器はLightCycler(Roche Diagnostics GmbH, Germany社製)で行い、増幅反応の条件は増幅サイクル数を35回にした点を除いて実施例1と同様である。
【0074】
リアルタイムPCRによる解析はLight Cycler(ロシュ・ダイアグノスティックス)にてPCR増幅しCP値(F=1)測定した。
【0075】
尚、指数関数的増幅期のサイクル数(CP値)は、指数関数的増幅領域の、ある一定の蛍光シグナル:F(Fluorescence)値で補助線を引き、その補助線と増幅曲線との交点から算出したサイクル数となる。本実施例ではCP値(F=1)を算出した。結果を表10に示す。
【0076】
【表10】

【0077】
ワケギの場合はCP値比(F=1)(タマネギ/ネギ):1.2前後の一定の数値となるが、そこから外れた場合(ネギの割合が多い場合はCP値比(F=1):1.3以上、タマネギの割合が多い場合はCP(F=1)値比:1.1前後)はネギとタマネギの混合物であることが分かる。このように、ほとんどの場合、ネギとタマネギとの混合物とワケギの場合を区別することができる。
【図面の簡単な説明】
【0078】
【図1】各プライマーの検出限界を示した電気泳動図である。
【図2】マルチプレックスプライマーを用いた同時鑑定の結果を示した電気泳動図である。
【特許請求の範囲】
【請求項1】
配列表の配列番号1における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
【請求項2】
配列表の配列番号2における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
【請求項3】
配列表の配列番号3における塩基番号13〜24の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
【請求項4】
配列表の配列番号4における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
【請求項5】
請求項1記載のプライマーと請求項2記載のプライマーとからなるPCRプライマーセット。
【請求項6】
請求項3記載のプライマーと請求項4記載のプライマーとからなるPCRプライマーセット。
【請求項7】
試料からDNAを抽出する工程と、このDNAを鋳型として、請求項5記載のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にネギが存在しているか否かを検出する工程とを含むネギの検出方法。
【請求項8】
試料からDNAを抽出する工程と、このDNAを鋳型として、請求項6記載のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にタマネギが存在しているか否かを検出する工程とを含むタマネギの検出方法。
【請求項9】
試料からDNAを抽出する工程と、このDNAを鋳型として、請求項5記載のプライマーセットを用いてPCRを行う工程と、請求項6記載のプライマーセットを用いてPCRを行う工程と、前記それぞれのプライマーセットにより増幅されたDNAを検出することにより試料中にワケギが存在しているか否かを検出する工程とを含むワケギの検出方法。
【請求項1】
配列表の配列番号1における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
【請求項2】
配列表の配列番号2における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
【請求項3】
配列表の配列番号3における塩基番号13〜24の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
【請求項4】
配列表の配列番号4における塩基番号10〜21の塩基配列を3´末端側に含む最大30塩基のDNAからなるPCRプライマー
【請求項5】
請求項1記載のプライマーと請求項2記載のプライマーとからなるPCRプライマーセット。
【請求項6】
請求項3記載のプライマーと請求項4記載のプライマーとからなるPCRプライマーセット。
【請求項7】
試料からDNAを抽出する工程と、このDNAを鋳型として、請求項5記載のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にネギが存在しているか否かを検出する工程とを含むネギの検出方法。
【請求項8】
試料からDNAを抽出する工程と、このDNAを鋳型として、請求項6記載のプライマーセットを用いてPCRを行う工程と、増幅されたDNAを検出することにより試料中にタマネギが存在しているか否かを検出する工程とを含むタマネギの検出方法。
【請求項9】
試料からDNAを抽出する工程と、このDNAを鋳型として、請求項5記載のプライマーセットを用いてPCRを行う工程と、請求項6記載のプライマーセットを用いてPCRを行う工程と、前記それぞれのプライマーセットにより増幅されたDNAを検出することにより試料中にワケギが存在しているか否かを検出する工程とを含むワケギの検出方法。
【図1】


【図2】




【図2】


【公開番号】特開2012−34583(P2012−34583A)
【公開日】平成24年2月23日(2012.2.23)
【国際特許分類】
【出願番号】特願2010−174884(P2010−174884)
【出願日】平成22年8月3日(2010.8.3)
【出願人】(000226976)日清食品ホールディングス株式会社 (127)
【Fターム(参考)】
【公開日】平成24年2月23日(2012.2.23)
【国際特許分類】
【出願日】平成22年8月3日(2010.8.3)
【出願人】(000226976)日清食品ホールディングス株式会社 (127)
【Fターム(参考)】
[ Back to top ]

