フルオロキノロン系薬剤耐性菌の検出方法及びそのためのプライマー
【課題】安価な装置を用いて迅速にフルオロキノロン系薬剤耐性菌を検出することができる方法を提供すること。
【解決手段】フルオロキノロン系薬剤耐性菌の検出方法は、被検菌のgyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含む。制限酵素は、GyrA83部位の薬剤耐性変異を調べるためのAciI又はHinfIと、GyrA87部位の薬剤耐性変異を調べるためのTth111Iであり、PCRに用いられる一対のプライマーのうちのリバースプライマーは、AciI切断部位及び/又はHinfI切断部位を有し、かつ、Tth111 I切断部位を与えるミスマッチ部位を有する。
【解決手段】フルオロキノロン系薬剤耐性菌の検出方法は、被検菌のgyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含む。制限酵素は、GyrA83部位の薬剤耐性変異を調べるためのAciI又はHinfIと、GyrA87部位の薬剤耐性変異を調べるためのTth111Iであり、PCRに用いられる一対のプライマーのうちのリバースプライマーは、AciI切断部位及び/又はHinfI切断部位を有し、かつ、Tth111 I切断部位を与えるミスマッチ部位を有する。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、フルオロキノロン系薬剤耐性菌の検出方法及びそのためのプライマーに関する。
【背景技術】
【0002】
臨床現場では、様々な薬剤耐性菌が蔓延しており対策を必要としている。フルオロキノロン系薬は幅広い抗菌スペクトルから汎用されているが、最近耐性菌が出現しており問題視されている。優良な抗菌薬を使用し続けるためにも、耐性菌の監視と適切な抗菌薬使用は重要である。
【0003】
従来、フルオロキノロン系薬剤耐性菌の検出方法としては、被検菌を分離培養し、同定し、薬剤感受性試験を行う方法が行われている。しかしながら、この方法は、結果が出るまでに2〜4日間もかかるという欠点を有する。
【0004】
また、フルオロキノロン系薬剤耐性を付与する遺伝子変異が既に知られているので、フルオロキノロン系薬剤耐性菌を遺伝子検査により検出することも行われている。遺伝子検査法としては、 リアルタイムPCR法やダイレクトシーケンシング法が用いられている。しかしながら、リアルタイムPCR法は、必要とする機器が高価であり、ダイレクトシーケンシング法も高価な機器が必要で、かつ、時間もかかる。
【先行技術文献】
【非特許文献】
【0005】
【非特許文献1】Comparison of Mismatch Amplification Mutation Assay with DNA Sequencing for Characterization of Fluoroquinolone Resistance in Neisseria gonorrhoeae. Z. Sultan, S. Nahar, B. Wretlind, E. Lindback, and M. Rahman. J. Clin. Microb. 2004. 42, 591-594
【非特許文献2】Screening Method for Salmonella enterica Serovar Typhi and Serovar Paratyphi A with Reduced Susceptibility to Fluoroquinolones by PCR-Restriction Fragment Length Polymorphism. K. Hirose, K. Tamura, and H. Watanabe. Microbiol. Immunol. 2003. 47, 161-165.
【発明の概要】
【発明が解決しようとする課題】
【0006】
本発明の目的は、安価な装置を用いて迅速にフルオロキノロン系薬剤耐性菌を検出することができる、フルオロキノロン系薬剤耐性菌の検出方法及びそれに用いられるプライマーを提供することである。
【課題を解決するための手段】
【0007】
本願発明者らは、フルオロキノロン系薬剤耐性変異の有無により切断が起きるか起きないかが異なる制限酵素を用いるPCR-RFLP法によりフルオロキノロン系薬剤耐性変異の有無を検出することができれば、上記本発明の目的を達成できることに想到した。しかしながら、フルオロキノロン系薬剤耐性変異であるGyrA87部位やParC84部位の有無により切断が起きるか起きないかが異なる制限酵素は存在しないことがわかった。そこで、鋭意研究の結果、PCR用のプライマーに人為的にミスマッチ部位を付与することにより、GyrA87部位の変異を制限酵素による切断の有無により検出可能で、かつ、GyrA83部位の変異も制限酵素による切断の有無により検出可能とするPCR用プライマーを創製し、PCR-RFLP法によるGyrA83部位及びGyrA87部位の変異の検出を可能にした。さらに、PCR用のプライマーに人為的にミスマッチ部位を付与することにより、ParC84部位の変異を制限酵素による切断の有無により検出可能で、かつ、ParC80部位の変異も制限酵素による切断の有無により検出可能とするPCR用プライマーを創製し、PCR-RFLP法によるParC80部位及びParC84部位の変異の検出を可能にし、本発明を完成した。
【0008】
すなわち、本発明は、フルオロキノロン系薬剤耐性菌の検出方法であって、被検菌のgyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含み、前記制限酵素が、GyrA83部位の薬剤耐性変異を調べるためのAciI又はHinfIと、GyrA87部位の薬剤耐性変異を調べるためのTth111Iであり、前記PCRに用いられる一対のプライマーのうちのリバースプライマーは、AciI切断部位及び/又はHinfI切断部位を有し、かつ、Tth111 I切断部位を与えるミスマッチ部位を有し、GyrA83部位の薬剤耐性変異を持たない菌はAciI又はHinfIにより切断されるが、GyrA83部位の薬剤耐性変異を持つ菌はAciI又はHinfIにより切断されず、GyrA87部位の薬剤耐性変異を持たない菌はTth111 Iにより切断されるが、GyrA87部位の薬剤耐性変異を持つ菌はTth111 Iにより切断されないことを指標とする、フルオロキノロン系薬剤耐性菌の検出方法を提供する。また、本発明は、この方法に用いられるリバースプライマーをも提供する。
【0009】
さらに、本発明は、フルオロキノロン系薬剤耐性菌の検出方法であって、被検菌のparC遺伝子のParC80部位及びParC84部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含み、前記制限酵素が、ParC80部位の薬剤耐性変異を調べるためのHaeIIと、ParC84部位の薬剤耐性変異を調べるためのXmnIであり、前記PCRに用いられる一対のプライマーのうちのリバースプライマーは、HaeII切断部位を有し、かつ、XmnI切断部位を与えるミスマッチ部位を有し、ParC80部位の薬剤耐性変異を持たない菌はHaeIIにより切断されるが、ParC80部位の薬剤耐性変異を持つ菌はHaeIIにより切断されず、ParC84部位の薬剤耐性変異を持たない菌はXmnIにより切断されるが、ParC84部位の薬剤耐性変異を持つ菌はXmnIにより切断されないことを指標とする、フルオロキノロン系薬剤耐性菌の検出方法を提供する。また、本発明は、この方法に用いられるリバースプライマーをも提供する。
【発明の効果】
【0010】
本願発明により、フルオロキノロン系薬剤耐性菌の検出を、安価な装置を用いて迅速に行うことができるようになった。しかも、本発明の方法によれば、4つの異なる薬剤耐性変異部位の有無を検出可能である。すなわち、本発明の方法を用いることによりどのようなフルオロキノロン系薬剤耐性変異に対しても詳細に検出することができ、細かな薬剤感受性を把握することができる。また、2-3時間で結果を出すことができるため、臨床現場では患者がいる間に最適な抗菌薬使用を施すことができる。さらに、安価な装置を行うことができるので、臨床検査機関のみならず、多くの病院等の臨床現場において検査を実施することが容易となり、検査の普及に貢献できる。
【図面の簡単な説明】
【0011】
【図1】本発明の実施例で行った、gyrA遺伝子についてのPCR-RFLP法の結果を示す電気泳動図である。
【図2】本発明の実施例で行った、parC遺伝子についてのPCR-RFLP法の結果を示す電気泳動図である。
【図3】本発明の方法で用いる各制限酵素の認識配列と切断部位を示す図である。
【発明を実施するための最良の形態】
【0012】
フルオロキノロン系薬剤耐性は、gyrA遺伝子のGyrA83部位及びGyrA87部位並びにparC遺伝子のParC80部位及びParC84部位の変異のそれぞれにより付与されることがわかっている。これらの変異は単独でもフルオロキノロン系薬剤耐性(以下、単に「薬剤耐性」と呼ぶ)を与えるが、重複することによりフルオロキノロン系薬剤耐性が増大する。従って、これらの薬剤耐性変異部位のうち、いくつが変異しているのかを調べることにより、その菌のフルオロキノロン系薬剤耐性の強さも調べることができる。
【0013】
上記の通り、本発明は、PCR-RFLP法により、上記4つの変異を調べることにより、フルオロキノロン系薬剤耐性菌を検出する方法に係る。本発明によれば、上記4つの変異の全てについて有無を調べることができるので、薬剤耐性の強さも調べることができる。
【0014】
本発明の第1の方法では、gyrA遺伝子のGyrA83部位及びGyrA87部位の変異の有無をPCR-RFLPにより調べる。下記実施例で用いたCitrobacter freundii (C. freundii)、Enterobacter cloacae (E. cloacae)、Escherichia coli (E. coli)、Klebsiella pneumoniae (K. pneumoniae)及びSalmonella spp.のgyrA遺伝子の塩基配列を配列番号1、3、5、7及び9にそれぞれ示す。なお、これらの塩基配列は、それぞれ、GenBank Accession No. AF052253、AF052256、X06373、AJ292307、X78977に記載されている。PCR-RFLPでは、被検菌のgyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域をPCRにより増幅する。このPCRに用いられる一対のプライマーのうち、フォワードプライマーは、特に限定されるものではなく、gyrA遺伝子のGyrA83部位よりも上流であって、後述するリバースプライマーと重複しない領域にハイブリダイズし、上記領域を増幅可能なものであればよい。フォワードプライマーは、それがハイブリダイズする鎖と完全に相補的なものが最も好ましいが、1塩基〜2塩基程度のミスマッチ部位を有するものであってもハイブリダイズ及び増幅に支障がない場合が多いので、それらを用いることも可能である。なお、周知のように、プライマーの3'末端にミスマッチがあると増幅が起きないので、3'末端にミスマッチがあるものは使用できない。一方、リバースプライマーは、本発明に特徴的なものであり、AciI切断部位及び/又はHinfI切断部位を有し、かつ、Tth111 I切断部位を与えるミスマッチ部位を有する。なお、AciI切断部位、HinfI切断部位及びTth111 I切断部位の認識配列と切断部位を図3に示す。gyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域にはAciI切断部位(C.freundiiの場合)及び/又はHinfI切断部位(E.cloacae、E.coli、K.pneumoniae及びSalmonella spp.の場合)が存在し、これを利用してGyrA83部位の薬剤耐性変異の有無を調べることが可能である。一方、この領域には、GyrA87部位の薬剤耐性変異の有無を調べることを可能にする制限酵素部位は存在しない。このため、本発明では、リバースプライマーに人為的にミスマッチ部位を導入し、Tth111 I切断部位を創出する。このようなリバースプライマーの例として、5'-TGCGCCATRCGVACGACSGT-3' (R=A or G, V=A,C, or G, S=C or G)を挙げることができる。もっとも、AciI切断部位及び/又はHinfI切断部位並びにTth111 I切断部位を有し、GyrA83部位及びGyrA87部位を含む領域にハイブリダイズするものであれば、これに限定されるものではない。リバースプライマーは、意図的に導入した上記ミスマッチ部位以外は、それがハイブリダイズする鎖と完全に相補的なものが最も好ましいが、さらに1塩基程度のミスマッチ部位を有するものであってもハイブリダイズ及び増幅に支障がない場合が多いので、それらを用いることも可能である。なお、上記の通り、プライマーの3'末端にミスマッチがあると増幅が起きないので、3'末端にミスマッチがあるものは使用できない。各プライマー(フォワードプライマー及びリバースプライマーの両者とも)のサイズは、通常、18nt〜50nt程度であり、より好ましくは20nt〜30nt程度であるので、例えば、上記プライマー(サイズは20nt)よりも長いサイズのものも好ましく用いることができる。
【0015】
PCR増幅産物は、次に、制限酵素処理し、電気泳動にかけて制限酵素断片を分離する。制限酵素としては、GyrA83部位の薬剤耐性変異を調べるためのAciI又はHinfIと、GyrA87部位の薬剤耐性変異を調べるためのTth111 Iを用いる。なお、GyrA83部位の薬剤耐性変異の検査のための前記制限酵素として、C.freundiiについてはAciIを用い、E.cloacae、E.coli、K.pneumoniae又はSalmonella spp.についてはHinfIを用いる。
【0016】
GyrA83部位の薬剤耐性変異を持たない菌はAciI又はHinfIにより切断されるが、GyrA83部位の薬剤耐性変異を持つ菌はAciI又はHinfIにより切断されず、GyrA87部位の薬剤耐性変異を持たない菌はTth111 Iにより切断されるが、GyrA87部位の薬剤耐性変異を持つ菌はTth111 Iにより切断されないので、得られる制限酵素断片長を指標として、被検菌が耐性菌か否か、また、耐性菌の場合には、どの薬剤耐性変異を有するかを調べることができる。
【0017】
本発明の第2の方法では、parC遺伝子のParC80部位及びParC84部位の変異の有無をPCR-RFLPにより調べる。下記実施例で用いたC. freundii、E. cloacae、E. coli、K. pneumoniae及びSalmonella spp.のparC遺伝子の塩基配列を配列番号11、13、15、17及び19にそれぞれ示す。なお、これらの塩基配列は、それぞれ、GenBank Accession No. AF056286、AF302678、M58408、AY034616、M68936に記載されている。PCR-RFLPでは、被検菌のparC遺伝子のParC80部位及びParC84部位を含む領域をPCRにより増幅する。このPCRに用いられる一対のプライマーのうち、フォワードプライマーは、特に限定されるものではなく、parC遺伝子のParC80部位よりも上流であって、後述するリバースプライマーと重複しない領域にハイブリダイズし、上記領域を増幅可能なものであればよい。フォワードプライマーは、それがハイブリダイズする鎖と完全に相補的なものが最も好ましいが、1塩基〜2塩基程度のミスマッチ部位を有するものであってもハイブリダイズ及び増幅に支障がない場合が多いので、それらを用いることも可能である。なお、周知のように、プライマーの3'末端にミスマッチがあると増幅が起きないので、3'末端にミスマッチがあるものは使用できない。一方、リバースプライマーは、本発明に特徴的なものであり、HaeII切断部位を有し、かつ、XmnI切断部位を与えるミスマッチ部位を有する。なお、HaeII及びXmnIの認識配列と切断部位を図3に示す。parC遺伝子のParC80部位及びParC84部位を含む領域にはHaeII切断部位が存在し、これを利用してParC80部位の薬剤耐性変異の有無を調べることが可能である。一方、この領域には、ParC84部位の薬剤耐性変異の有無を調べることを可能にする制限酵素部位は存在しない。このため、本発明では、リバースプライマーに人為的にミスマッチ部位を導入し、XmnI切断部位を創出する。このようなリバースプライマーの例として、5'-GCTGCGCCATCAGSACGAASGC-3'(S=C or G)を挙げることができる。もっとも、HaeII切断部位及びXmnI切断部位を有し、ParC80部位及びParC84部位を含む領域にハイブリダイズするものであれば、これに限定されるものではない。リバースプライマーは、意図的に導入した上記ミスマッチ部位以外は、それがハイブリダイズする鎖と完全に相補的なものが最も好ましいが、さらに1塩基程度のミスマッチ部位を有するものであってもハイブリダイズ及び増幅に支障がない場合が多いので、それらを用いることも可能である。なお、上記の通り、プライマーの3'末端にミスマッチがあると増幅が起きないので、3'末端にミスマッチがあるものは使用できない。各プライマー(フォワードプライマー及びリバースプライマーの両者とも)のサイズは、通常、18nt〜50nt程度であり、より好ましくは20nt〜30nt程度であるので、例えば、上記プライマー(サイズは22nt)よりも長いサイズのものも好ましく用いることができる。
【0018】
PCR増幅産物は、次に、制限酵素処理し、電気泳動にかけて制限酵素断片を分離する。制限酵素としては、ParC80部位の薬剤耐性変異を調べるためのHaeIIと、ParC84部位の薬剤耐性変異を調べるためのXmnIを用いる。
【0019】
ParC80部位の薬剤耐性変異を持たない菌はHaeIIにより切断されるが、ParC80部位の薬剤耐性変異を持つ菌はHaeIIにより切断されず、ParC84部位の薬剤耐性変異を持たない菌はXmnIにより切断されるが、ParC84部位の薬剤耐性変異を持つ菌はXmnIにより切断されないので、得られる制限酵素断片長を指標として、被検菌が耐性菌か否か、また、耐性菌の場合には、どの薬剤耐性変異を有するかを調べることができる。
【0020】
1つの被検菌サンプルについて上記した第1及び第2の方法の両者を行うことにより、上記した4つの薬剤耐性変異の全てを調べることができ、好ましい。
【0021】
以下、本発明を実施例に基づきより具体的に説明する。もっとも、本発明は下記実施例に限定されるものではない。
【実施例】
【0022】
実施例1 腸内細菌(Escherihia coli, Enterobacter cloacae, Klebsiella pneumoniae, Salmonella spp.)に対するミスマッチPCR-RFLP法
【0023】
(対象菌種)
対象となる菌種は次の通りである。Escherihia coli, Enterobacter cloacae, Klebsiella pneumoniae, Salmonella spp. これらはATCC(American Type Culture Collection)株と船橋市立医療センター、茅ケ崎徳州会病院、昭和大学藤が丘病院から分与していただいた臨床分離株、神奈川県衛生研究所から分与していただいた環境由来株から得られた。詳しくは表1の通りである。
【0024】
(対象菌種のDNAの抽出)
各菌種のDNAは寒天培地で培養後、DNeasy Blood & Tissue Kit(Qiagen)を使用してマニュアルに従って抽出した。また、抽出されたDNAはアガロースゲル(1.2%)(gibcoBRL agarose, Parisley, UK)を用いて電気泳動にて確認した。泳動条件は100Vで30分とした。
【0025】
(対象菌種のgyrAとparCのDNA塩基配列の解読)
各菌種のgyrAとparCをPCRにて増幅後DNAシークエンサーABI310にて塩基配列を解読した。PCRはHot-StarTaq DNA polymerase (Qiagen)にてマニュアルに従い行った。この時使用したPCRプライマーは次の通りである。gyrAのQRDRについてはgyrA F 5'-CCAGATGTHCGHGATGG-3'とgyrA R 5'-ACGAAATCAACSGTYTCTTTTTC-3'を用いた。parCのQRDRについてparC F 5'-TTGCCWTTTATYGGKGATGG-3'とparC R 5'-CGCGCWGGCAGCATTTTWGG-3'を用いた。DNA塩基配列の詳細についてはDNA解析ソフトDNAStarを用いて解析した。QRDRのDNA塩基配列について表1に示した。
【0026】
(フルオロキノロン系薬の薬剤感受性試験)
フルオロキノロン系薬の薬剤感受性試験はCLSI法が定める抗菌薬感受性試験微量液体希釈法に従い測定した。薬剤はナリジクス酸(NA)レボフロキサシン(LVFX)とシプロフロキサシン(CPFX)を用いた。詳しくは表1の通りである。NA耐性菌はGyrA83もしくは87に変異が認められた。LVFX、CPFX耐性菌はさらにParC80もしくは84に変異が認められた。GyrA, ParCのQRDRに変異が蓄積することでフルオロキノロン系薬が高度耐性化することが判った。
【0027】
(ミスマッチPCRのプライマー作製)
DNA解析ソフトDNAStarを用いて各菌種の塩基配列の相動性を確認し、各菌種のgyrAとparCのQRDRのDNA塩基配列をそれぞれ表2と表3に示した。gyrAのQRDRであるSer-83とAsp-87を含む領域を増幅できるようにプライマーを設定した。詳細はgyrAについてはSer-83の上流約110bpにforward primer gyrAfwS2 5'-GTWCACCGTCGCGTACTWTA-3'( W=A or T)を設定した。reverse primerはAsp-87のすぐ下流に設定し、gyrArevS 5'-TGCGCCATRCGVACGACSGT-3'(R=A or G, V=A,C, or G, S=C or G)を作製した。この時、PCR産物が制限酵素Tth111I(GACNNNGTC)を認識できるように、該当するプライマー箇所にミスマッチする部位を挿入して作製した。表2のE. coliとC. freundiiの一重下線「t」に対して、プライマー中ではGに置き換えて配置してある。いずれの菌に対してもミスマッチとなる二重下線「a」に対しては、プライマー中ではCに置き換えて配置した。これらのミスマッチ部分をPCRで増幅することでGyrA87以降の配列は次の通りになる。5'-GACACCGTCGTTCGTATGGCGCA-3' このことで、制限酵素Tth111I(GACNNNGTC)が認識できる部位(太字)を創出することができ、制限酵素でGyrA87(GAC)を認識して切断することが可能となる。これらのプライマーにてDNAを増幅すると152 bpの断片を得ることができる。
【0028】
parCについても同様にQRDRであるSer-80とGlu-84を含む領域を増幅できるようにした。詳細はSer-80の上流約60bpにforward primer parCfw6 5'-TTYAARAAATCCGCCCGYACCGT-3'(Y= C or T)を設定した。reverse primerはGlu-84のすぐ下流に設定し、parCrevS3 5'-GCTGCGCCATCAGSACGAASGC-3'を作製した。この時、制限酵素XmnI(GAANNNTTC)が認識できるようにプライマーの一部にミスマッチする部位を挿入して作製した。表3の二重下線「a」と「g」に対して、プライマー中では「G」と「A」に置き換えて配置した。これらのミスマッチ部分をPCRで増幅することでParC84以降の配列は次の通りになる。5'-GAAGCSTTCGTSCTGATGGCGCAGC-3' このことで、制限酵素XmnI(GAANNNTTC)が認識できる部位(太字)を創出することができ、制限酵素でParC84(GAA)を認識して切断することが可能となる。これらのプライマーにてDNAを増幅すると97 bpの断片を得ることができる。
【0029】
PCRによるDNA断片増幅は次の条件で起こった。PCR反応酵素はPlatinum Taq DNA polymerase(invitrogen)を用い、Veriti サーマルサイクラー(アプライドバイオシステムズ)にて温度設定した。gyrAの反応条件は94℃ 2分、(94℃30秒、55℃30秒、72℃30秒)30サイクルとした。parCの反応条件は94℃ 2分、(94℃30秒、52℃30秒、72℃30秒)30サイクルとした。PCR産物はアガロースゲル(1.2%)を用いて電気泳動にて確認した。泳動条件は100Vで30分とした。
【0030】
(ミスマッチPCR-RFLP法)
gyrAとparCともにPCRにて得られた産物を制限酵素で切断した。gyrAについてはGyrA83Sを認識する制限酵素として、C. freundiiについてはAciI(Takara)を、E. coli, E. cloacae, K. pneumoniae, Salmonella spp. についてはHinfI(Takara)を用いた(表2)。GyrA87Dを認識する制限酵素として、いずれもTth111Iを用いた。parCについてはParC80Sを認識する制限酵素としていずれもHaeII(Takara)を用いた(表3)。ParC84Eを認識する制限酵素としてはXmnI(Promega)を用いた。Tth111Iは65℃で、それ以外の制限酵素は37℃で1時間反応させた。反応後それぞれを低分子の核酸を分離することができるMetaphor agarose 4.0%(Takara)を用いて電気泳動して確認した。泳動条件は100Vで40分とした。このようにして求めた結果のうちE. coliのgyrAについて図1に、parCについて図2に示した。GyrAについて感受性株(ATCC8739)はフルオロキノロン系薬に感受性の野生株でGyrA83, 87のDNA塩基配列が変異していないものを示す。NCは制限酵素処理していないものを、83はHinfIで、87はTth111Iで制限酵素反応させたものである。この結果、図1に示されるように、感受性株のgyrAはいずれも制限酵素で切断され、PCR産物のサイズが小さくなっているのが判る。一方、耐性株(206)については制限酵素で切断されずサイズが変わっていない。耐性株については認識部位が変異しているため、制限酵素で認識されず切断されなかったものと思われた。
【0031】
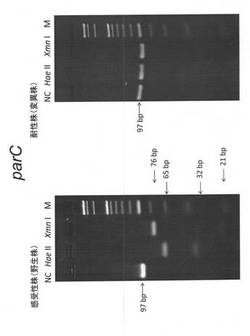
ParCについても同様に感受性株(ATCC8739)と耐性株(206)に対して、ParC80, 84をそれぞれ制限酵素処理した。NCは制限酵素処理していないものを、80はHaeIIで、84はXmnIで制限酵素反応させたものである。この結果、図2に示されるように、感受性株のparCはいずれも制限酵素で切断され、PCR産物のサイズが小さくなっているのが判る。一方、耐性株については制限酵素で切断されずサイズが変わっていない。耐性株については認識部位が変異しているため、制限酵素で認識されず切断されなかったものと思われた。これらのことより表1の対象菌株に対して本法を試したところ、いずれもgyrA及びparCのQRDRの遺伝子変異を判別することができた。GyrA, ParCのQRDRの変異の蓄積はフルオロキノロン系薬の耐性化と相関していることから(表1)、本法によって耐性菌を判別できると考えられた。
【0032】
【表1−1】

【0033】
【表1−2】

【0034】
【表2】

【0035】
【表3】

* R= A or G, Y= C or T
【技術分野】
【0001】
本発明は、フルオロキノロン系薬剤耐性菌の検出方法及びそのためのプライマーに関する。
【背景技術】
【0002】
臨床現場では、様々な薬剤耐性菌が蔓延しており対策を必要としている。フルオロキノロン系薬は幅広い抗菌スペクトルから汎用されているが、最近耐性菌が出現しており問題視されている。優良な抗菌薬を使用し続けるためにも、耐性菌の監視と適切な抗菌薬使用は重要である。
【0003】
従来、フルオロキノロン系薬剤耐性菌の検出方法としては、被検菌を分離培養し、同定し、薬剤感受性試験を行う方法が行われている。しかしながら、この方法は、結果が出るまでに2〜4日間もかかるという欠点を有する。
【0004】
また、フルオロキノロン系薬剤耐性を付与する遺伝子変異が既に知られているので、フルオロキノロン系薬剤耐性菌を遺伝子検査により検出することも行われている。遺伝子検査法としては、 リアルタイムPCR法やダイレクトシーケンシング法が用いられている。しかしながら、リアルタイムPCR法は、必要とする機器が高価であり、ダイレクトシーケンシング法も高価な機器が必要で、かつ、時間もかかる。
【先行技術文献】
【非特許文献】
【0005】
【非特許文献1】Comparison of Mismatch Amplification Mutation Assay with DNA Sequencing for Characterization of Fluoroquinolone Resistance in Neisseria gonorrhoeae. Z. Sultan, S. Nahar, B. Wretlind, E. Lindback, and M. Rahman. J. Clin. Microb. 2004. 42, 591-594
【非特許文献2】Screening Method for Salmonella enterica Serovar Typhi and Serovar Paratyphi A with Reduced Susceptibility to Fluoroquinolones by PCR-Restriction Fragment Length Polymorphism. K. Hirose, K. Tamura, and H. Watanabe. Microbiol. Immunol. 2003. 47, 161-165.
【発明の概要】
【発明が解決しようとする課題】
【0006】
本発明の目的は、安価な装置を用いて迅速にフルオロキノロン系薬剤耐性菌を検出することができる、フルオロキノロン系薬剤耐性菌の検出方法及びそれに用いられるプライマーを提供することである。
【課題を解決するための手段】
【0007】
本願発明者らは、フルオロキノロン系薬剤耐性変異の有無により切断が起きるか起きないかが異なる制限酵素を用いるPCR-RFLP法によりフルオロキノロン系薬剤耐性変異の有無を検出することができれば、上記本発明の目的を達成できることに想到した。しかしながら、フルオロキノロン系薬剤耐性変異であるGyrA87部位やParC84部位の有無により切断が起きるか起きないかが異なる制限酵素は存在しないことがわかった。そこで、鋭意研究の結果、PCR用のプライマーに人為的にミスマッチ部位を付与することにより、GyrA87部位の変異を制限酵素による切断の有無により検出可能で、かつ、GyrA83部位の変異も制限酵素による切断の有無により検出可能とするPCR用プライマーを創製し、PCR-RFLP法によるGyrA83部位及びGyrA87部位の変異の検出を可能にした。さらに、PCR用のプライマーに人為的にミスマッチ部位を付与することにより、ParC84部位の変異を制限酵素による切断の有無により検出可能で、かつ、ParC80部位の変異も制限酵素による切断の有無により検出可能とするPCR用プライマーを創製し、PCR-RFLP法によるParC80部位及びParC84部位の変異の検出を可能にし、本発明を完成した。
【0008】
すなわち、本発明は、フルオロキノロン系薬剤耐性菌の検出方法であって、被検菌のgyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含み、前記制限酵素が、GyrA83部位の薬剤耐性変異を調べるためのAciI又はHinfIと、GyrA87部位の薬剤耐性変異を調べるためのTth111Iであり、前記PCRに用いられる一対のプライマーのうちのリバースプライマーは、AciI切断部位及び/又はHinfI切断部位を有し、かつ、Tth111 I切断部位を与えるミスマッチ部位を有し、GyrA83部位の薬剤耐性変異を持たない菌はAciI又はHinfIにより切断されるが、GyrA83部位の薬剤耐性変異を持つ菌はAciI又はHinfIにより切断されず、GyrA87部位の薬剤耐性変異を持たない菌はTth111 Iにより切断されるが、GyrA87部位の薬剤耐性変異を持つ菌はTth111 Iにより切断されないことを指標とする、フルオロキノロン系薬剤耐性菌の検出方法を提供する。また、本発明は、この方法に用いられるリバースプライマーをも提供する。
【0009】
さらに、本発明は、フルオロキノロン系薬剤耐性菌の検出方法であって、被検菌のparC遺伝子のParC80部位及びParC84部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含み、前記制限酵素が、ParC80部位の薬剤耐性変異を調べるためのHaeIIと、ParC84部位の薬剤耐性変異を調べるためのXmnIであり、前記PCRに用いられる一対のプライマーのうちのリバースプライマーは、HaeII切断部位を有し、かつ、XmnI切断部位を与えるミスマッチ部位を有し、ParC80部位の薬剤耐性変異を持たない菌はHaeIIにより切断されるが、ParC80部位の薬剤耐性変異を持つ菌はHaeIIにより切断されず、ParC84部位の薬剤耐性変異を持たない菌はXmnIにより切断されるが、ParC84部位の薬剤耐性変異を持つ菌はXmnIにより切断されないことを指標とする、フルオロキノロン系薬剤耐性菌の検出方法を提供する。また、本発明は、この方法に用いられるリバースプライマーをも提供する。
【発明の効果】
【0010】
本願発明により、フルオロキノロン系薬剤耐性菌の検出を、安価な装置を用いて迅速に行うことができるようになった。しかも、本発明の方法によれば、4つの異なる薬剤耐性変異部位の有無を検出可能である。すなわち、本発明の方法を用いることによりどのようなフルオロキノロン系薬剤耐性変異に対しても詳細に検出することができ、細かな薬剤感受性を把握することができる。また、2-3時間で結果を出すことができるため、臨床現場では患者がいる間に最適な抗菌薬使用を施すことができる。さらに、安価な装置を行うことができるので、臨床検査機関のみならず、多くの病院等の臨床現場において検査を実施することが容易となり、検査の普及に貢献できる。
【図面の簡単な説明】
【0011】
【図1】本発明の実施例で行った、gyrA遺伝子についてのPCR-RFLP法の結果を示す電気泳動図である。
【図2】本発明の実施例で行った、parC遺伝子についてのPCR-RFLP法の結果を示す電気泳動図である。
【図3】本発明の方法で用いる各制限酵素の認識配列と切断部位を示す図である。
【発明を実施するための最良の形態】
【0012】
フルオロキノロン系薬剤耐性は、gyrA遺伝子のGyrA83部位及びGyrA87部位並びにparC遺伝子のParC80部位及びParC84部位の変異のそれぞれにより付与されることがわかっている。これらの変異は単独でもフルオロキノロン系薬剤耐性(以下、単に「薬剤耐性」と呼ぶ)を与えるが、重複することによりフルオロキノロン系薬剤耐性が増大する。従って、これらの薬剤耐性変異部位のうち、いくつが変異しているのかを調べることにより、その菌のフルオロキノロン系薬剤耐性の強さも調べることができる。
【0013】
上記の通り、本発明は、PCR-RFLP法により、上記4つの変異を調べることにより、フルオロキノロン系薬剤耐性菌を検出する方法に係る。本発明によれば、上記4つの変異の全てについて有無を調べることができるので、薬剤耐性の強さも調べることができる。
【0014】
本発明の第1の方法では、gyrA遺伝子のGyrA83部位及びGyrA87部位の変異の有無をPCR-RFLPにより調べる。下記実施例で用いたCitrobacter freundii (C. freundii)、Enterobacter cloacae (E. cloacae)、Escherichia coli (E. coli)、Klebsiella pneumoniae (K. pneumoniae)及びSalmonella spp.のgyrA遺伝子の塩基配列を配列番号1、3、5、7及び9にそれぞれ示す。なお、これらの塩基配列は、それぞれ、GenBank Accession No. AF052253、AF052256、X06373、AJ292307、X78977に記載されている。PCR-RFLPでは、被検菌のgyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域をPCRにより増幅する。このPCRに用いられる一対のプライマーのうち、フォワードプライマーは、特に限定されるものではなく、gyrA遺伝子のGyrA83部位よりも上流であって、後述するリバースプライマーと重複しない領域にハイブリダイズし、上記領域を増幅可能なものであればよい。フォワードプライマーは、それがハイブリダイズする鎖と完全に相補的なものが最も好ましいが、1塩基〜2塩基程度のミスマッチ部位を有するものであってもハイブリダイズ及び増幅に支障がない場合が多いので、それらを用いることも可能である。なお、周知のように、プライマーの3'末端にミスマッチがあると増幅が起きないので、3'末端にミスマッチがあるものは使用できない。一方、リバースプライマーは、本発明に特徴的なものであり、AciI切断部位及び/又はHinfI切断部位を有し、かつ、Tth111 I切断部位を与えるミスマッチ部位を有する。なお、AciI切断部位、HinfI切断部位及びTth111 I切断部位の認識配列と切断部位を図3に示す。gyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域にはAciI切断部位(C.freundiiの場合)及び/又はHinfI切断部位(E.cloacae、E.coli、K.pneumoniae及びSalmonella spp.の場合)が存在し、これを利用してGyrA83部位の薬剤耐性変異の有無を調べることが可能である。一方、この領域には、GyrA87部位の薬剤耐性変異の有無を調べることを可能にする制限酵素部位は存在しない。このため、本発明では、リバースプライマーに人為的にミスマッチ部位を導入し、Tth111 I切断部位を創出する。このようなリバースプライマーの例として、5'-TGCGCCATRCGVACGACSGT-3' (R=A or G, V=A,C, or G, S=C or G)を挙げることができる。もっとも、AciI切断部位及び/又はHinfI切断部位並びにTth111 I切断部位を有し、GyrA83部位及びGyrA87部位を含む領域にハイブリダイズするものであれば、これに限定されるものではない。リバースプライマーは、意図的に導入した上記ミスマッチ部位以外は、それがハイブリダイズする鎖と完全に相補的なものが最も好ましいが、さらに1塩基程度のミスマッチ部位を有するものであってもハイブリダイズ及び増幅に支障がない場合が多いので、それらを用いることも可能である。なお、上記の通り、プライマーの3'末端にミスマッチがあると増幅が起きないので、3'末端にミスマッチがあるものは使用できない。各プライマー(フォワードプライマー及びリバースプライマーの両者とも)のサイズは、通常、18nt〜50nt程度であり、より好ましくは20nt〜30nt程度であるので、例えば、上記プライマー(サイズは20nt)よりも長いサイズのものも好ましく用いることができる。
【0015】
PCR増幅産物は、次に、制限酵素処理し、電気泳動にかけて制限酵素断片を分離する。制限酵素としては、GyrA83部位の薬剤耐性変異を調べるためのAciI又はHinfIと、GyrA87部位の薬剤耐性変異を調べるためのTth111 Iを用いる。なお、GyrA83部位の薬剤耐性変異の検査のための前記制限酵素として、C.freundiiについてはAciIを用い、E.cloacae、E.coli、K.pneumoniae又はSalmonella spp.についてはHinfIを用いる。
【0016】
GyrA83部位の薬剤耐性変異を持たない菌はAciI又はHinfIにより切断されるが、GyrA83部位の薬剤耐性変異を持つ菌はAciI又はHinfIにより切断されず、GyrA87部位の薬剤耐性変異を持たない菌はTth111 Iにより切断されるが、GyrA87部位の薬剤耐性変異を持つ菌はTth111 Iにより切断されないので、得られる制限酵素断片長を指標として、被検菌が耐性菌か否か、また、耐性菌の場合には、どの薬剤耐性変異を有するかを調べることができる。
【0017】
本発明の第2の方法では、parC遺伝子のParC80部位及びParC84部位の変異の有無をPCR-RFLPにより調べる。下記実施例で用いたC. freundii、E. cloacae、E. coli、K. pneumoniae及びSalmonella spp.のparC遺伝子の塩基配列を配列番号11、13、15、17及び19にそれぞれ示す。なお、これらの塩基配列は、それぞれ、GenBank Accession No. AF056286、AF302678、M58408、AY034616、M68936に記載されている。PCR-RFLPでは、被検菌のparC遺伝子のParC80部位及びParC84部位を含む領域をPCRにより増幅する。このPCRに用いられる一対のプライマーのうち、フォワードプライマーは、特に限定されるものではなく、parC遺伝子のParC80部位よりも上流であって、後述するリバースプライマーと重複しない領域にハイブリダイズし、上記領域を増幅可能なものであればよい。フォワードプライマーは、それがハイブリダイズする鎖と完全に相補的なものが最も好ましいが、1塩基〜2塩基程度のミスマッチ部位を有するものであってもハイブリダイズ及び増幅に支障がない場合が多いので、それらを用いることも可能である。なお、周知のように、プライマーの3'末端にミスマッチがあると増幅が起きないので、3'末端にミスマッチがあるものは使用できない。一方、リバースプライマーは、本発明に特徴的なものであり、HaeII切断部位を有し、かつ、XmnI切断部位を与えるミスマッチ部位を有する。なお、HaeII及びXmnIの認識配列と切断部位を図3に示す。parC遺伝子のParC80部位及びParC84部位を含む領域にはHaeII切断部位が存在し、これを利用してParC80部位の薬剤耐性変異の有無を調べることが可能である。一方、この領域には、ParC84部位の薬剤耐性変異の有無を調べることを可能にする制限酵素部位は存在しない。このため、本発明では、リバースプライマーに人為的にミスマッチ部位を導入し、XmnI切断部位を創出する。このようなリバースプライマーの例として、5'-GCTGCGCCATCAGSACGAASGC-3'(S=C or G)を挙げることができる。もっとも、HaeII切断部位及びXmnI切断部位を有し、ParC80部位及びParC84部位を含む領域にハイブリダイズするものであれば、これに限定されるものではない。リバースプライマーは、意図的に導入した上記ミスマッチ部位以外は、それがハイブリダイズする鎖と完全に相補的なものが最も好ましいが、さらに1塩基程度のミスマッチ部位を有するものであってもハイブリダイズ及び増幅に支障がない場合が多いので、それらを用いることも可能である。なお、上記の通り、プライマーの3'末端にミスマッチがあると増幅が起きないので、3'末端にミスマッチがあるものは使用できない。各プライマー(フォワードプライマー及びリバースプライマーの両者とも)のサイズは、通常、18nt〜50nt程度であり、より好ましくは20nt〜30nt程度であるので、例えば、上記プライマー(サイズは22nt)よりも長いサイズのものも好ましく用いることができる。
【0018】
PCR増幅産物は、次に、制限酵素処理し、電気泳動にかけて制限酵素断片を分離する。制限酵素としては、ParC80部位の薬剤耐性変異を調べるためのHaeIIと、ParC84部位の薬剤耐性変異を調べるためのXmnIを用いる。
【0019】
ParC80部位の薬剤耐性変異を持たない菌はHaeIIにより切断されるが、ParC80部位の薬剤耐性変異を持つ菌はHaeIIにより切断されず、ParC84部位の薬剤耐性変異を持たない菌はXmnIにより切断されるが、ParC84部位の薬剤耐性変異を持つ菌はXmnIにより切断されないので、得られる制限酵素断片長を指標として、被検菌が耐性菌か否か、また、耐性菌の場合には、どの薬剤耐性変異を有するかを調べることができる。
【0020】
1つの被検菌サンプルについて上記した第1及び第2の方法の両者を行うことにより、上記した4つの薬剤耐性変異の全てを調べることができ、好ましい。
【0021】
以下、本発明を実施例に基づきより具体的に説明する。もっとも、本発明は下記実施例に限定されるものではない。
【実施例】
【0022】
実施例1 腸内細菌(Escherihia coli, Enterobacter cloacae, Klebsiella pneumoniae, Salmonella spp.)に対するミスマッチPCR-RFLP法
【0023】
(対象菌種)
対象となる菌種は次の通りである。Escherihia coli, Enterobacter cloacae, Klebsiella pneumoniae, Salmonella spp. これらはATCC(American Type Culture Collection)株と船橋市立医療センター、茅ケ崎徳州会病院、昭和大学藤が丘病院から分与していただいた臨床分離株、神奈川県衛生研究所から分与していただいた環境由来株から得られた。詳しくは表1の通りである。
【0024】
(対象菌種のDNAの抽出)
各菌種のDNAは寒天培地で培養後、DNeasy Blood & Tissue Kit(Qiagen)を使用してマニュアルに従って抽出した。また、抽出されたDNAはアガロースゲル(1.2%)(gibcoBRL agarose, Parisley, UK)を用いて電気泳動にて確認した。泳動条件は100Vで30分とした。
【0025】
(対象菌種のgyrAとparCのDNA塩基配列の解読)
各菌種のgyrAとparCをPCRにて増幅後DNAシークエンサーABI310にて塩基配列を解読した。PCRはHot-StarTaq DNA polymerase (Qiagen)にてマニュアルに従い行った。この時使用したPCRプライマーは次の通りである。gyrAのQRDRについてはgyrA F 5'-CCAGATGTHCGHGATGG-3'とgyrA R 5'-ACGAAATCAACSGTYTCTTTTTC-3'を用いた。parCのQRDRについてparC F 5'-TTGCCWTTTATYGGKGATGG-3'とparC R 5'-CGCGCWGGCAGCATTTTWGG-3'を用いた。DNA塩基配列の詳細についてはDNA解析ソフトDNAStarを用いて解析した。QRDRのDNA塩基配列について表1に示した。
【0026】
(フルオロキノロン系薬の薬剤感受性試験)
フルオロキノロン系薬の薬剤感受性試験はCLSI法が定める抗菌薬感受性試験微量液体希釈法に従い測定した。薬剤はナリジクス酸(NA)レボフロキサシン(LVFX)とシプロフロキサシン(CPFX)を用いた。詳しくは表1の通りである。NA耐性菌はGyrA83もしくは87に変異が認められた。LVFX、CPFX耐性菌はさらにParC80もしくは84に変異が認められた。GyrA, ParCのQRDRに変異が蓄積することでフルオロキノロン系薬が高度耐性化することが判った。
【0027】
(ミスマッチPCRのプライマー作製)
DNA解析ソフトDNAStarを用いて各菌種の塩基配列の相動性を確認し、各菌種のgyrAとparCのQRDRのDNA塩基配列をそれぞれ表2と表3に示した。gyrAのQRDRであるSer-83とAsp-87を含む領域を増幅できるようにプライマーを設定した。詳細はgyrAについてはSer-83の上流約110bpにforward primer gyrAfwS2 5'-GTWCACCGTCGCGTACTWTA-3'( W=A or T)を設定した。reverse primerはAsp-87のすぐ下流に設定し、gyrArevS 5'-TGCGCCATRCGVACGACSGT-3'(R=A or G, V=A,C, or G, S=C or G)を作製した。この時、PCR産物が制限酵素Tth111I(GACNNNGTC)を認識できるように、該当するプライマー箇所にミスマッチする部位を挿入して作製した。表2のE. coliとC. freundiiの一重下線「t」に対して、プライマー中ではGに置き換えて配置してある。いずれの菌に対してもミスマッチとなる二重下線「a」に対しては、プライマー中ではCに置き換えて配置した。これらのミスマッチ部分をPCRで増幅することでGyrA87以降の配列は次の通りになる。5'-GACACCGTCGTTCGTATGGCGCA-3' このことで、制限酵素Tth111I(GACNNNGTC)が認識できる部位(太字)を創出することができ、制限酵素でGyrA87(GAC)を認識して切断することが可能となる。これらのプライマーにてDNAを増幅すると152 bpの断片を得ることができる。
【0028】
parCについても同様にQRDRであるSer-80とGlu-84を含む領域を増幅できるようにした。詳細はSer-80の上流約60bpにforward primer parCfw6 5'-TTYAARAAATCCGCCCGYACCGT-3'(Y= C or T)を設定した。reverse primerはGlu-84のすぐ下流に設定し、parCrevS3 5'-GCTGCGCCATCAGSACGAASGC-3'を作製した。この時、制限酵素XmnI(GAANNNTTC)が認識できるようにプライマーの一部にミスマッチする部位を挿入して作製した。表3の二重下線「a」と「g」に対して、プライマー中では「G」と「A」に置き換えて配置した。これらのミスマッチ部分をPCRで増幅することでParC84以降の配列は次の通りになる。5'-GAAGCSTTCGTSCTGATGGCGCAGC-3' このことで、制限酵素XmnI(GAANNNTTC)が認識できる部位(太字)を創出することができ、制限酵素でParC84(GAA)を認識して切断することが可能となる。これらのプライマーにてDNAを増幅すると97 bpの断片を得ることができる。
【0029】
PCRによるDNA断片増幅は次の条件で起こった。PCR反応酵素はPlatinum Taq DNA polymerase(invitrogen)を用い、Veriti サーマルサイクラー(アプライドバイオシステムズ)にて温度設定した。gyrAの反応条件は94℃ 2分、(94℃30秒、55℃30秒、72℃30秒)30サイクルとした。parCの反応条件は94℃ 2分、(94℃30秒、52℃30秒、72℃30秒)30サイクルとした。PCR産物はアガロースゲル(1.2%)を用いて電気泳動にて確認した。泳動条件は100Vで30分とした。
【0030】
(ミスマッチPCR-RFLP法)
gyrAとparCともにPCRにて得られた産物を制限酵素で切断した。gyrAについてはGyrA83Sを認識する制限酵素として、C. freundiiについてはAciI(Takara)を、E. coli, E. cloacae, K. pneumoniae, Salmonella spp. についてはHinfI(Takara)を用いた(表2)。GyrA87Dを認識する制限酵素として、いずれもTth111Iを用いた。parCについてはParC80Sを認識する制限酵素としていずれもHaeII(Takara)を用いた(表3)。ParC84Eを認識する制限酵素としてはXmnI(Promega)を用いた。Tth111Iは65℃で、それ以外の制限酵素は37℃で1時間反応させた。反応後それぞれを低分子の核酸を分離することができるMetaphor agarose 4.0%(Takara)を用いて電気泳動して確認した。泳動条件は100Vで40分とした。このようにして求めた結果のうちE. coliのgyrAについて図1に、parCについて図2に示した。GyrAについて感受性株(ATCC8739)はフルオロキノロン系薬に感受性の野生株でGyrA83, 87のDNA塩基配列が変異していないものを示す。NCは制限酵素処理していないものを、83はHinfIで、87はTth111Iで制限酵素反応させたものである。この結果、図1に示されるように、感受性株のgyrAはいずれも制限酵素で切断され、PCR産物のサイズが小さくなっているのが判る。一方、耐性株(206)については制限酵素で切断されずサイズが変わっていない。耐性株については認識部位が変異しているため、制限酵素で認識されず切断されなかったものと思われた。
【0031】
ParCについても同様に感受性株(ATCC8739)と耐性株(206)に対して、ParC80, 84をそれぞれ制限酵素処理した。NCは制限酵素処理していないものを、80はHaeIIで、84はXmnIで制限酵素反応させたものである。この結果、図2に示されるように、感受性株のparCはいずれも制限酵素で切断され、PCR産物のサイズが小さくなっているのが判る。一方、耐性株については制限酵素で切断されずサイズが変わっていない。耐性株については認識部位が変異しているため、制限酵素で認識されず切断されなかったものと思われた。これらのことより表1の対象菌株に対して本法を試したところ、いずれもgyrA及びparCのQRDRの遺伝子変異を判別することができた。GyrA, ParCのQRDRの変異の蓄積はフルオロキノロン系薬の耐性化と相関していることから(表1)、本法によって耐性菌を判別できると考えられた。
【0032】
【表1−1】

【0033】
【表1−2】

【0034】
【表2】

【0035】
【表3】

* R= A or G, Y= C or T
【特許請求の範囲】
【請求項1】
フルオロキノン系薬剤耐性菌の検出方法であって、被検菌のgyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含み、前記制限酵素が、GyrA83部位の薬剤耐性変異を調べるためのAci I又はHinf Iと、GyrA87部位の薬剤耐性変異を調べるためのTth111 Iであり、前記PCRに用いられる一対のプライマーのうちのリバースプライマーは、Aci I切断部位及び/又はHinf I切断部位を有し、かつ、Tth111 I切断部位を与えるミスマッチ部位を有し、GyrA83部位の薬剤耐性変異を持たない菌はAci I又はHinf Iにより切断されるが、GyrA83部位の薬剤耐性変異を持つ菌はAci I又はHinf Iにより切断されず、GyrA87部位の薬剤耐性変異を持たない菌はTth111 Iにより切断されるが、GyrA87部位の薬剤耐性変異を持つ菌はTth111 Iにより切断されないことを指標とする、フルオロキノン系薬剤耐性菌の検出方法。
【請求項2】
前記被検菌が、C.freundii、E.cloacae、E.coli、K.pneumoniae又はSalmonella spp.であり、GyrA83部位の薬剤耐性変異の検査のための前記制限酵素として、C.freundiiについてはAci Iを用い、E.cloacae、E.coli、K.pneumoniae又はSalmonella spp.についてはHinf Iを用いる請求項1記載の方法。
【請求項3】
前記リバースプライマーの塩基配列が、5'-TGCGCCATRCGVACGACSGT-3' (R=A or G, V=A,C, or G, S=C or G)である請求項1又は2記載の方法。
【請求項4】
請求項1〜3のいずれか1項に記載の方法に用いられるリバースプライマー。
【請求項5】
フルオロキノン系薬剤耐性菌の検出方法であって、被検菌のparC遺伝子のParC80部位及びParC84部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含み、前記制限酵素が、ParC80部位の薬剤耐性変異を調べるためのHae IIと、ParC84部位の薬剤耐性変異を調べるためのXmn Iであり、前記PCRに用いられる一対のプライマーのうちのリバースプライマーは、Hae II切断部位を有し、かつ、Xmn I切断部位を与えるミスマッチ部位を有し、ParC80部位の薬剤耐性変異を持たない菌はHae IIにより切断されるが、ParC80部位の薬剤耐性変異を持つ菌はHae IIにより切断されず、ParC84部位の薬剤耐性変異を持たない菌はXmn Iにより切断されるが、ParC84部位の薬剤耐性変異を持つ菌はXmn Iにより切断されないことを指標とする、フルオロキノン系薬剤耐性菌の検出方法。
【請求項6】
前記被検菌が、C.freundii、E.cloacae、E.coli、K.pneumoniae又はSalmonella spp.である請求項5記載の方法。
【請求項7】
前記リバースプライマーの塩基配列が、5'-GCTGCGCCATCAGSACGAASGC-3'(S=C or G)である請求項5又は6記載の方法。
【請求項8】
請求項5〜7のいずれか1項に記載の方法に用いられるリバースプライマー。
【請求項9】
1つの被検菌について、請求項1〜3のいずれか1項に記載の方法と請求項5〜7のいずれか1項に記載の方法の両方を行い、GyrA83部位及びGyrA87部位並びにParC80部位及びParC84部位の薬剤耐性変異の有無を調べるフルオロキノン系薬剤耐性菌の検出方法。
【請求項1】
フルオロキノン系薬剤耐性菌の検出方法であって、被検菌のgyrA遺伝子のGyrA83部位及びGyrA87部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含み、前記制限酵素が、GyrA83部位の薬剤耐性変異を調べるためのAci I又はHinf Iと、GyrA87部位の薬剤耐性変異を調べるためのTth111 Iであり、前記PCRに用いられる一対のプライマーのうちのリバースプライマーは、Aci I切断部位及び/又はHinf I切断部位を有し、かつ、Tth111 I切断部位を与えるミスマッチ部位を有し、GyrA83部位の薬剤耐性変異を持たない菌はAci I又はHinf Iにより切断されるが、GyrA83部位の薬剤耐性変異を持つ菌はAci I又はHinf Iにより切断されず、GyrA87部位の薬剤耐性変異を持たない菌はTth111 Iにより切断されるが、GyrA87部位の薬剤耐性変異を持つ菌はTth111 Iにより切断されないことを指標とする、フルオロキノン系薬剤耐性菌の検出方法。
【請求項2】
前記被検菌が、C.freundii、E.cloacae、E.coli、K.pneumoniae又はSalmonella spp.であり、GyrA83部位の薬剤耐性変異の検査のための前記制限酵素として、C.freundiiについてはAci Iを用い、E.cloacae、E.coli、K.pneumoniae又はSalmonella spp.についてはHinf Iを用いる請求項1記載の方法。
【請求項3】
前記リバースプライマーの塩基配列が、5'-TGCGCCATRCGVACGACSGT-3' (R=A or G, V=A,C, or G, S=C or G)である請求項1又は2記載の方法。
【請求項4】
請求項1〜3のいずれか1項に記載の方法に用いられるリバースプライマー。
【請求項5】
フルオロキノン系薬剤耐性菌の検出方法であって、被検菌のparC遺伝子のParC80部位及びParC84部位を含む領域をPCRにより増幅する工程と、増幅産物を制限酵素処理して、得られた制限酵素断片長を調べる工程とを含み、前記制限酵素が、ParC80部位の薬剤耐性変異を調べるためのHae IIと、ParC84部位の薬剤耐性変異を調べるためのXmn Iであり、前記PCRに用いられる一対のプライマーのうちのリバースプライマーは、Hae II切断部位を有し、かつ、Xmn I切断部位を与えるミスマッチ部位を有し、ParC80部位の薬剤耐性変異を持たない菌はHae IIにより切断されるが、ParC80部位の薬剤耐性変異を持つ菌はHae IIにより切断されず、ParC84部位の薬剤耐性変異を持たない菌はXmn Iにより切断されるが、ParC84部位の薬剤耐性変異を持つ菌はXmn Iにより切断されないことを指標とする、フルオロキノン系薬剤耐性菌の検出方法。
【請求項6】
前記被検菌が、C.freundii、E.cloacae、E.coli、K.pneumoniae又はSalmonella spp.である請求項5記載の方法。
【請求項7】
前記リバースプライマーの塩基配列が、5'-GCTGCGCCATCAGSACGAASGC-3'(S=C or G)である請求項5又は6記載の方法。
【請求項8】
請求項5〜7のいずれか1項に記載の方法に用いられるリバースプライマー。
【請求項9】
1つの被検菌について、請求項1〜3のいずれか1項に記載の方法と請求項5〜7のいずれか1項に記載の方法の両方を行い、GyrA83部位及びGyrA87部位並びにParC80部位及びParC84部位の薬剤耐性変異の有無を調べるフルオロキノン系薬剤耐性菌の検出方法。
【図3】


【図1】


【図2】




【図1】


【図2】


【公開番号】特開2012−85582(P2012−85582A)
【公開日】平成24年5月10日(2012.5.10)
【国際特許分類】
【出願番号】特願2010−235602(P2010−235602)
【出願日】平成22年10月20日(2010.10.20)
【新規性喪失の例外の表示】特許法第30条第1項適用申請有り 第59回日本感染症学会東日本地方会学術集会、第57回日本化学療法学会東日本支部総会 合同学会 プログラム・抄録集、第133頁、平成22年9月発行
【出願人】(591243103)財団法人神奈川科学技術アカデミー (271)
【出願人】(598041566)学校法人北里研究所 (180)
【Fターム(参考)】
【公開日】平成24年5月10日(2012.5.10)
【国際特許分類】
【出願日】平成22年10月20日(2010.10.20)
【新規性喪失の例外の表示】特許法第30条第1項適用申請有り 第59回日本感染症学会東日本地方会学術集会、第57回日本化学療法学会東日本支部総会 合同学会 プログラム・抄録集、第133頁、平成22年9月発行
【出願人】(591243103)財団法人神奈川科学技術アカデミー (271)
【出願人】(598041566)学校法人北里研究所 (180)
【Fターム(参考)】
[ Back to top ]

