ブタの椎骨数を支配するVertnin遺伝子、およびその利用
【課題】本発明は、ブタの第7染色体上のVertnin(VRTN)遺伝子上、またはその近傍の多型マーカーを指標とする、ブタの椎骨数増大型遺伝形質の有無の判定方法の提供を課題とする。
【解決手段】 本発明者は、ブタの第7染色体上に、ブタの椎骨数増大と関連する新規なVertnin遺伝子を同定することに成功した。さらに、該遺伝子上もしくはその近傍に存在する多型マーカーを用いることにより、ブタの椎骨数増大型遺伝形質の有無を判定可能であることを見出し、本発明を完成させた。
【解決手段】 本発明者は、ブタの第7染色体上に、ブタの椎骨数増大と関連する新規なVertnin遺伝子を同定することに成功した。さらに、該遺伝子上もしくはその近傍に存在する多型マーカーを用いることにより、ブタの椎骨数増大型遺伝形質の有無を判定可能であることを見出し、本発明を完成させた。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、ブタの第7染色体上のVertnin遺伝子上もしくはその近傍に存在する多型マーカーを指標とするブタの椎骨数増大型遺伝形質の有無の判定方法に関する。
【背景技術】
【0002】
ブタはイノシシを祖先とし、ユーラシア大陸の複数地域で家畜化されたと言われている。19世紀中頃からヨーロッパにおいて、成長・体格などの良い豚を選抜して育種するということが行われるようになり、それらがもとになり現在の商用品種が形成されている。
【0003】
本発明者らはブタの改良を目的に経済形質に関与する遺伝子座の単離を進めてきた。肉質・成長といった経済形質の多くは量的形質でありそれを支配する遺伝子座は量的形質遺伝子座(quantitative trait locus; QTL)と呼ばれる。現在、ブタの様々なQTLについては約110の論文に約1800の報告がある(PigQTLdb, http://www.animalgenome.org/cgi-bin/QTLdb/SS/index)。しかしながら量的形質は複数の遺伝子座に支配され、また環境要因によっても大きく影響を受けるため、QTLの正確なマッピングは難しく、その責任遺伝子を同定すること、また多様性の原因となる多型を同定することは非常に困難である。ブタにおいて遺伝子レベルで解明されたQTLでは肉質(グリコーゲン量)に関与するPRKAG3遺伝子(非特許文献1)、肉量に関与するIGF2遺伝子(非特許文献2)がある。
【0004】
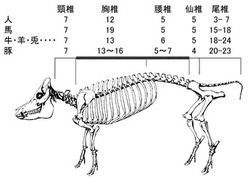
本発明者らは複数のF2実験家系を作製し、成長性、産肉性、肉質などの各種経済形質に関するQTL解析を行った(非特許文献3〜10)。その結果、様々なQTLが検出されたが、その中で胸椎数と腰椎数の和で示される椎骨数(図1)に関与するQTLが複数の家系を通じて2カ所のゲノム領域(第1染色体q腕末端、第7染色体q腕中央)に検出され、その存在は確からしいと判断された(表2(F2実験家系と椎骨数QTL)、図2)。
【0005】
〔表2〕

【0006】
2つの椎骨数QTLにはそれぞれ椎骨数を増大させる対立遺伝子があり、その効果はほぼ等しく、各家系の結果を平均すると対立遺伝子あたり約0.5から0.6個の椎骨数を増大させた。また2つのQTLは互いに独立に働き、すべての対立遺伝子が椎骨数増大型になると平均で約2.3本の椎骨数が増大した(表3(ブタの椎骨数に関する2つのQTLの効果)、図3)。
【0007】
〔表3〕

【0008】
ブタの頸椎は他の哺乳類と同じく7個であるが、胸椎、腰椎の数には多様性があり、胸椎は13個から16個、腰椎は5個から7個とばらついている(非特許文献11)。これらを合計した数(椎骨数)はブタの祖先であるイノシシでは19個であるが、現在の肉用品種では20個から23個であり、このような種内での大きな多様性は、哺乳類ではブタでのみ見られる(図1)。ブタは肉量増大、繁殖性向上のために体が大きくなるように選抜され、その過程で椎骨数が増大したと考えられ、実際、一つの椎骨数の増大により、体長は平均約1.5 cm伸びることが示されている(非特許文献12)。
【0009】
これまでのF2実験家系の解析において、第1染色体q腕末端領域の椎骨数QTLでは、梅山豚、金華豚などのアジア在来種、および日本イノシシの対立遺伝子に増大効果は認められず野生型であり、ランドレース、大ヨークシャー、デュロック、バークシャーといった西洋品種の対立遺伝子のすべてに椎骨数増大効果が認められた(表2、表4(椎骨数QTLの遺伝子型))。
【0010】
〔表4〕

【0011】
第1染色体上の椎骨数QTLについては、椎骨数が増大した西洋品種で固定された約300 kbの領域を発見し、そこに位置する責任遺伝子NR6A1を同定した(非特許文献13)。またそれに関して、特許出願を行った(特許文献1)。
【0012】
第7染色体q腕中央部の椎骨数QTLでは、F2実験家系に用いた西洋品種に由来する一部の対立遺伝子に椎骨数を増大させる効果が認められた(表2、表4)(非特許文献14)。また徳島県の大ヨークシャー種系統豚を用い、その種豚について第7染色体の椎骨数QTLについて半きょうだい解析を行うことにより、QTLがヘテロ型である個体を検出した(図4)(非特許文献14)。これらから現在豚肉生産に用いられている西洋品種においても第7染色体の椎骨数QTLは多様性があることが明らかとなった。
【0013】
なお、本出願の発明に関連する先行技術文献情報を以下に示す。
【先行技術文献】
【特許文献】
【0014】
【特許文献1】特開2006-101871
【非特許文献】
【0015】
【非特許文献1】Milan D., Jeon JT., Looft C., Amarger V., Robic A. et al., 2000 A mutation in PRKAG3 associated with excess glycogen content in pig skeletal muscle. Science. 288: 1248-1251.
【非特許文献2】Van Laere AS., Nguyen M., Braunschweig M., Nezer C., Collette C. et al., 2003 A regulatory mutation in IGF2 causes a major QTL effect on muscle growth in the pig. Nature. 425: 832-836.
【非特許文献3】Wada, Y., T. Akita, T. Awata, T. Furukawa, N. Sugai et al., 2000 Quantitative trait loci (QTL) analysis in a Meishan × Gottingen cross population. Anim. Genet. 31: 376-384.
【非特許文献4】松原英二、湊和之、稲毛優子、楠本宏司、美川智、和田康彦、小林栄治、峰澤満、安江博「イノブタ実験家系における経済形質とマイクロサテライトマーカーとの連鎖解析」日本養豚学会誌、第36巻4号、P189、平成11年11月
【非特許文献5】内藤学、山田渥、内田陽子、稲毛優子、三宅正志、美川智、和田康彦、小林栄治、峰澤満、粟田崇、安江博「梅山豚x大ヨークシャー種実験家系におけるDNAマーカーと経済形質との連鎖解析」日本養豚学会誌、第37巻4号、P182、平成12年10月
【非特許文献6】室伏淳一、河原崎達雄、堀内篤、久松紀子、楠本宏司、美川智、和田康彦、峰澤満、安江博「金華豚・大ヨークシャー種交雑実験家系における産肉性、肉質とDNAマーカーとの連鎖解析」日本養豚学会誌、第35巻2号、P66、平成10年6月
【非特許文献7】加治佐修、犬童政昭、三宅正志、小林栄治、和田康彦、美川智、峰澤満、安江博「バークシャー種とクラウンミニブタ交雑家系のDNAマーカーを用いた連鎖解析」日本養豚学会誌、第35巻2号、P66、平成10年6月
【非特許文献8】新居雅宏、谷史雄、仁木明人、林武司、上西博英、美川智、小林栄治、内田陽子、粟田崇、安江博「日本イノシシと大ヨークシャー種交雑家系におけるQTL解析」第98回日本畜産学会大会講演要旨集、P19、平成13年3月
【非特許文献9】山口倫子、江森格、大澤浩司、内藤昌男、神山佳三、金谷奈保恵、内田陽子、堀内篤、仲沢慶紀、林武司、粟田崇「金華豚・デュロック種交雑家系における経済形質のQTL解析と筋肉内脂肪含量を対象としたマーカー利用選抜」日本養豚学会誌、第40巻4号、P212、平成15年10月
【非特許文献10】堀内篤、知久幹夫、井手華子、金谷奈保恵、内田陽子、山口倫子、仲沢慶紀、林武司、粟田崇「金華豚とデュロック種の交雑家系における肉質に関与するQTLの解析」日本養豚学会誌、第40巻4号、P213、平成15年10月
【非特許文献11】King, J. W. B., and R. C. Roberts, 1960 Carcass length in the bacon pig: its association with vertebrae numbers and prediction from radiographs of the young pig. Anim. Prod. 2: 59-65
【非特許文献12】Berge, S., 1948 Genetical researches on the number of vertebrae in the pigs. J. Anim. Sci. 7: 233-238
【非特許文献13】Mikawa S., Morozumi T., Shimanuki S., Hayashi T., Uenishi H., Domukai M., Okumura N., Awata T. 2007 Fine mapping of a swine quantitative trait locus for number of vertebrae and analysis of an orphan nuclear receptor, germ cell nuclear factor (NR6A1). Genome Research 17:586-593
【非特許文献14】Mikawa S., Hayashi T., Nii M., Shimanuki S., Morozumi T., Awata T., 2005 Two quantitative trait loci on Sus scrofa chromosomes 1 and 7 affecting the number of vertebrae. J Anim Sci. 83:2247-2254.
【発明の概要】
【発明が解決しようとする課題】
【0016】
本発明は、上記状況に鑑みてなされたものであり、その目的は椎骨数増大型遺伝形質を有するブタを判定する方法を提供することにある。より詳しくは、本発明は、ブタの第7染色体上のVertnin(VRTN)遺伝子上、またはその近傍の多型を指標とする、ブタの椎骨数増大型遺伝形質の有無の判定方法の提供を課題とする。
【課題を解決するための手段】
【0017】
F2実験家系を用いた連鎖解析等により絞り込まれた範囲からさらに原因遺伝子の存在する位置を特定するには一般に連鎖不平衡解析が用いられる。家畜は世代間隔が短く、血縁、表形値が記録されるため、現存の動物の形質データを用いて、形質の差と関連する同一祖先由来の共通染色体領域をマッピングする方法(identical by decent解析; IBD解析)が一般的に用いられる。しかしながら量的形質では、形質と遺伝子型との関連性が遺伝病などの質的形質ほど大きくなく、形質値を用いたIBD解析での責任遺伝子の解明は困難であり、これが様々なQTLの責任遺伝子が未同定である原因である。
【0018】
よって本発明者らは、形質値ではなく、半きょうだい解析によって判定したQTL型を用いたIBD解析を試行した。この場合、QTL型と責任遺伝子の遺伝子型は完全に一致する。またQTL型を判定する個体として、系統豚を用いた。これは雄10頭、雌65頭に由来し、7世代の閉鎖群育種により造成された後、雄9頭、雌35頭で維持されているもので、染色体の組み換えの数が多いと期待される。また造成開始時に導入された個体は互いに独立したもので、遺伝的に遠縁にある。つまり同祖染色体領域の組み換えによるマッピングと、独立した個体サンプル間に存在する小さな共通遺伝領域の探索の両方が期待される。
【0019】
具体的には、本発明者らは、大ヨークシャー種系統豚の種豚における椎骨数QTL型を、半きょうだい解析により判定し、高密度に配置したマイクロサテライトマーカーを用いて、QTL型と一致するハプロタイプを有するゲノム領域を検索した。また見いだした領域の近傍においては、これまでにQTL型を判定していた他品種を含むF2実験家系親世代ブタを用いて、さらにQTL型と一致するハプロタイプを有するゲノム領域を絞り込んだ。その結果、約45 kbの領域に到達し、そこに位置する椎骨数を支配する新規遺伝子VRTNを単離した。
【0020】
VRTN遺伝子を含む45 kbの領域において、大ヨークシャー種系統豚を用いて多型検索を行った結果、QTL型と一致する42ヵ所の多型部位が明らかとなった。これら多型から構成されるハプロタイプは、この大ヨークシャー種系統豚では2種であり、またと場サンプル(ランドレース種、大ヨークシャー種、およびデュロック種の交雑種)によるハプロタイプ解析においてもこの2種がメジャーなハプロタイプであった。
【0021】
ヘテロ型のブタ胚においてVRTN遺伝子の発現解析を行った結果、野生型対立遺伝子は交配後8.5日目から10日目の限られた期間に発現しているのに対し、椎骨数増大型対立遺伝子は8日目から14日目まで発現が確認された。この結果から、VRTNの椎骨数増大型対立遺伝子は、発現時期の制御が変化することにより、椎骨数を増大させていると判断される。
【0022】
本発明者らは、ブタの第7染色体上に、ブタの椎骨数増大と関連する新規なVertnin遺伝子を同定することに成功した。さらに、該遺伝子上もしくはその近傍に存在する多型マーカーを用いることにより、ブタの椎骨数増大型遺伝形質の有無を判定可能であることを見出し、本発明を完成させた。
【0023】
即ち本発明は、ブタの第7染色体上のVertnin遺伝子上もしくはその近傍に存在する多型マーカーを指標とするブタの椎骨数増大型遺伝形質の有無の判定方法に関し、より具体的には、
〔1〕 ブタの椎骨数増大型遺伝形質の有無の判定方法であって、ブタの第7染色体上に存在する下記(A)の(1)〜(25)のいずれかに記載の一もしくは複数の多型マーカーを用いて、下記(B)の工程によって判定する方法、
(A)
(1)配列番号:1に記載の塩基配列の40位
(2)配列番号:2に記載の塩基配列の207位
(3)配列番号:3に記載の塩基配列の68位
(4)配列番号:4に記載の塩基配列の47位または101位
(5)配列番号:5に記載の塩基配列の81位
(6)配列番号:6に記載の塩基配列の355位
(7)配列番号:7に記載の塩基配列の87位
(8)配列番号:8に記載の塩基配列の42位
(9)配列番号:9に記載の塩基配列の152位または185位
(10)配列番号:10に記載の塩基配列の82位
(11)配列番号:11に記載の塩基配列の95〜385位
(12)配列番号:12に記載の塩基配列の49位
(13)配列番号:13に記載の塩基配列の31位、116位、335位、または421位
(14)配列番号:14に記載の塩基配列の127位
(15)配列番号:15に記載の塩基配列の78位、154位、173位、または187位、217位、320位、433位
(16)配列番号:16に記載の塩基配列の111位
(17)配列番号:17に記載の塩基配列の71位または192位
(18)配列番号:18に記載の塩基配列の80〜82位
(19)配列番号:19に記載の塩基配列の285位または300〜301位
(20)配列番号:20に記載の塩基配列の55位、65位、75位、または150位
(21)配列番号:21に記載の塩基配列の99位または138位
(22)配列番号:22に記載の塩基配列の75位
(23)配列番号:23に記載の塩基配列の217位
(24)配列番号:24に記載の塩基配列の91位
(25)配列番号:25に記載の塩基配列の136位
(B)上記(A)のそれぞれの多型マーカーにおける塩基種が記載された下記〔表1〕の判定表に基づき、椎骨数増大型遺伝形質の有無を判定する工程
〔表1〕

〔2〕 ブタの第7染色体上の、以下の(a)〜(d)のいずれかに記載のマイクロサテライト配列を検出することを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法、
(a)配列番号:26に記載の塩基配列において、180〜195位または196〜216位のマイクロサテライト配列
(b)配列番号:27に記載の塩基配列において、311〜323位、380〜388位、389〜396位、または400〜406位のマイクロサテライト配列
(c)配列番号:28に記載の塩基配列において、393〜413位のマイクロサテライト配列
(d)配列番号:29に記載の塩基配列において、96〜107位または309〜325位のマイクロサテライト配列
〔3〕 ブタの第7染色体上に存在する前記多型マーカーまたはマイクロサテライト配列を含むDNA領域を増幅し、その増幅産物の多型またはマイクロサテライト配列を検出する工程を含む、〔1〕または〔2〕に記載の方法、
〔4〕 ブタの第7染色体上に存在するVertnin遺伝子の発現量を指標とすることを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法、
を、提供する。
【発明の効果】
【0024】
椎骨数を支配する遺伝子の解明は、椎骨数の制御を可能とすることから、ブタの産肉性、繁殖性の効率的な改良が実現される。特に第7染色体q腕中央部の椎骨数QTLは、豚肉生産に用いられている西洋品種の椎骨数のバラツキの原因であり、この責任遺伝子の解明によりもたらされる情報は、今日の豚肉生産に直接的に利用され、その効率化に大きく貢献することができる。
【図面の簡単な説明】
【0025】
【図1】ヒト、馬、牛・羊・兎、豚における、頸椎、胸椎、腰椎、仙椎、尾椎の数を示す図である。西洋品種(ランドレース、大ヨークシャー、デュロック、バークシャー)の椎骨数は20、21、22、23、アジア在来品種(梅山豚、金華豚)は19、イノシシは19である。
【図2】椎骨数に関するQTL解析(JD家系)の結果を示す図である。第1染色体q腕末端領域と第7染色体q腕中央部に2つの椎骨数に関するQTLが検出された。
【図3】JD家系における2つの椎骨数QTLの効果を示す図である。2つのQTLのそれぞれの遺伝子型を持つF2個体の椎骨数の平均値を示した。異なるアルファベット(a, b, c, d)間で有意差がある。それぞれのQTLは主に相加的効果を持ち、また2つのQTLは独立に働く。
【図4】徳島県大ヨークシャー種系統豚(AY)の種豚を用いた第7染色体上の椎骨数QTLの半きょうだい解析の結果を示す図である。種豚の相同染色体上のQTL領域の子豚への伝達を、マイクロサテライトマーカーを用いて解析した。由来する種豚のQTL領域により2つの群に分け(任意に黒、白で示す。)、それぞれの椎骨数の平均値をプロットした。また子豚の頭数と標準誤差を示した。t検定においてP < 0.01 (**)において種豚のQTLがヘテロ型であると判断した。Z検定は椎骨数増大効果を0.43としてNezerらの方法で行ない、Z <-2.0をホモ型とした。またホモ型種豚それぞれの子豚の椎骨数を、ヘテロ型種豚の椎骨数増大型アリルおよび野生型アリルが伝達した子豚(Q;371頭、q;383頭)の椎骨数と多重検定(Steel検定)し、椎骨数増大型または野生型を判定した。
【図5】第7染色体上の椎骨数QTLの信頼区間(三県合同、JD家系)を示す図である。Bootstrap法(10000回)により、QTLが位置する95%信頼区間を求めた。95%の区間において黒色、それ以外を灰色で示した。95%信頼区間は83 cMから88 cMであった。
【図6】椎骨数QTL近傍領域におけるマイクロサテライトマーカーの開発を示す図である。ヒト遺伝子情報を利用して開発したブタSTSによりBACクローンを単離し、マイクロサテライトマーカーを開発した。開発したマイクロサテライトマーカーは、SGSC(International Swine Genome Sequencing Consortium)によって進行中のブタゲノムドラフト配列と比較し、それらの染色体上の位置を確認した。下線で示したマーカーはブタゲノムドラフト配列に未だ見出されないものを示す。
【図7】徳島県大ヨークシャー種系統豚(AY)の種豚におけるQTLタイプと同祖染色体領域を示す図である。図4においてQTLタイプを判定した種豚の各染色体についてマイクロサテライトマーカーのハプロタイプを解析した。同祖である領域(同じタイプのハプロタイプ)についてそれぞれ同色で示した。椎骨数増大型対立遺伝子Q、野生型対立遺伝子wtが座位する両方の染色体に伝達している領域は赤文字で示した。同祖の染色体領域が、Qおよびwtの両方に伝達していないのはSJ7106からSJ7008の間であった。L、Rは各個体の任意の2つの染色体領域を示す。
【図8】椎骨数QTL近傍領域のゲノム構造解析の結果を示す図である。(A)SGSC(International Swine Genome Sequencing Consortium)により塩基配列が解読されているBACクローン。(B)BACクローンの塩基配列より作成した遺伝子地図。マイクロサテライトマーカーの位置を示した。下線で示したものは、BACクローンの塩基配列より新規に開発した。(C)VRTN遺伝子近傍の多型部位。大ヨークシャー種系統豚におけるQTLと一致する多型部位を示した。
【図9】大ヨークシャー種種豚およびF2実験家系親世代ブタにおけるQTLタイプと同祖染色体領域を示す図である。BACクローンの配列より新規に開発したマイクロサテライトマーカー(下線)を加え、ハプロタイプ解析を行った。L:ランドレース、W:大ヨークシャー、B:バークシャー、D:デュロック、Wb:猪、M:梅山豚、J:金華豚、C:クラウンミニ豚。L、Rは各個体の任意の2つの染色体領域を示す。
【図10】椎骨数QTL近傍領域の多型を用いた椎骨数との関連性解析とハプロタイプブロック解析の結果を示す図である。椎骨数QTL近傍領域に多型マーカーを開発し、と場よりサンプリングした199頭を用いて、それらの遺伝子型と椎骨数との関連性について分散分析を行い、F値をプロットした。またそれらの多型についてハプロタイプブロックを解析した。数字はR2値を示した。
【図11】ヘテロ型ブタ胚を用いたVRTN遺伝子の対立遺伝子間の発現比較解析を示す図である。交配後、8日、8.5日、10日、12日、14日においてヘテロ型(Q/wt)胚を子宮より回収し、トータルRNAを調製した。NV062をRT-PCRにより増幅し、TAクローニングを行った。クローン化した192個のコロニーについて、塩基配列を決定し、NV062の多型パターンの頻度を計測することにより各対立遺伝子の発現量の評価を行った。
【発明を実施するための形態】
【0026】
本発明は、ブタの第7染色体上のVertnin(VRTN)遺伝子上もしくはその近傍に存在する多型マーカーを指標とするブタの椎骨数増大型遺伝形質の有無の判定方法(本明細書において、「本発明の判定方法」と記載する場合あり)を提供する。
【0027】
本発明者らによって見出されたVertnin遺伝子の塩基配列、および該遺伝子によってコードされるタンパク質のアミノ酸配列を、それぞれ、配列番号:88、89へ記載する。
【0028】
本発明において「判定」とは、通常、被検ブタ(本発明の判定方法に供するブタ)が椎骨数野生型遺伝形質を有する、もしくは有さないと判定することを言う。
【0029】
また、椎骨数増大型遺伝形質を有するブタと、その他の豚とを判別すること等をいい、本発明における「判定」の用語は、例えば、「鑑定」、「鑑別」あるいは「判別」、「検査」等と表現してもよい。
【0030】
また、本発明の判定方法は、必ずしもブタ個体を対象として判定する方法に限定されず、例えば、食肉もしくは精肉加工品(例えば、ハム等)、あるいは、被検ブタに由来する生体試料等を対象として、椎骨数増大型遺伝形質の有無を判定する方法も含まれる。
【0031】
本発明において「多型」とは、例えば、一塩基多型(SNP)、欠失変異(deletion)、挿入変異(insertion)、マイクロサテライト等を指す。
【0032】
本発明の判定方法の好ましい態様としては、ブタゲノム中に存在する多型マーカーを用いることを特徴とする方法である。
【0033】
本発明における多型マーカーとは、ブタの椎骨数増大型遺伝形質の有無を判定可能なDNA部位(多型部位)をいう。従って、本発明の方法において多型マーカーを用いるとは、通常、本発明の多型部位におけるDNA配列(塩基種)の違いに基づきブタの椎骨数増大型遺伝形質の有無の判定を行うことをいう。
【0034】
本発明において用いる多型マーカーとしては、具体的には、ブタ第7染色体上に存在する多型マーカーであって、以下の部位の多型マーカー(本明細書において「本発明の多型マーカー」と記載する場合あり)を示すことができる。
(1)配列番号:1に記載の塩基配列の40位
(2)配列番号:2に記載の塩基配列の207位
(3)配列番号:3に記載の塩基配列の68位
(4)配列番号:4に記載の塩基配列の47位または101位
(5)配列番号:5に記載の塩基配列の81位
(6)配列番号:6に記載の塩基配列の355位
(7)配列番号:7に記載の塩基配列の87位
(8)配列番号:8に記載の塩基配列の42位
(9)配列番号:9に記載の塩基配列の152位または185位
(10)配列番号:10に記載の塩基配列の82位
(11)配列番号:11に記載の塩基配列の95〜385位
(12)配列番号:12に記載の塩基配列の49位
(13)配列番号:13に記載の塩基配列の31位、116位、335位、または421位
(14)配列番号:14に記載の塩基配列の127位
(15)配列番号:15に記載の塩基配列の78位、154位、173位、または187位、217位、320位、433位
(16)配列番号:16に記載の塩基配列の111位
(17)配列番号:17に記載の塩基配列の71位または192位
(18)配列番号:18に記載の塩基配列の80〜82位
(19)配列番号:19に記載の塩基配列の285位または300〜301位
(20)配列番号:20に記載の塩基配列の55位、65位、75位、または150位
(21)配列番号:21に記載の塩基配列の99位または138位
(22)配列番号:22に記載の塩基配列の75位
(23)配列番号:23に記載の塩基配列の217位
(24)配列番号:24に記載の塩基配列の91位
(25)配列番号:25に記載の塩基配列の136位
【0035】
本発明の好ましい態様としては、上記多型マーカーにおける多型部位を評価(例えば、多型部位における塩基種(DNA配列)を決定)することにより、ブタの椎骨数増大型遺伝形質の有無を判定する方法である。
【0036】
より具体的には、本発明の判定方法は、ブタゲノム中に存在する上記(1)〜(25)のいずれかに記載の一もしくは複数の多型マーカーを用いて、上記のそれぞれの多型マーカーにおける塩基種(DNA配列)が記載された下記の判定表に基づき、ブタの椎骨数増大型遺伝形質の有無を判定する方法である。
【0037】
〔表1〕

【0038】
上記表1において、delとは欠失変異(deletion)、insとは挿入変異(insertion)を表す。
【0039】
本発明の方法においては、通常、多くの多型マーカーを用いるほど判定精度が向上する。従って、本発明の方法は、必ずしも必須ではないが、複数の多型マーカーを用いてもよい。本発明の方法において用いる多型マーカーは、通常、1もしくは少数(例えば1〜9程度)である。本発明の方法の一つの態様としては、上記多型マーカーを組み合わせて用いる方法であり、即ち、上記(1)〜(25)に記載された複数の多型マーカーからなる群より選択される1もしくは複数の多型マーカーを用いることを特徴とする方法もまた本発明に含まれる。
【0040】
本発明の判定方法の好ましい態様としては、上記(1)〜(25)の多型マーカーにおける多型部位の変異を検出することを特徴とする方法である。
本発明において変異を検出する部位は、好ましくは、上記(1)〜(25)のいずれかに記載の多型マーカーにおける多型部位である。
【0041】
本発明において変異とは、例えば、上記(1)〜(25)の多型マーカーにおける多型部位において、椎骨数増大型(Q)への変異である。
具体的には、被検ブタにおいて、上記(1)〜(25)の多型マーカーにける多型部位が、上記判定表に記載の「増大型Q」である場合に、被検ブタは椎骨数増大型遺伝形質を有すると判定される。
また、本発明の方法は、被検ブタについて上記(1)〜(25)の多型マーカーにおける多型部位が、上記判定表に記載の「増大型Q」でない場合、あるいは、「野生型wt」である場合に被検ブタは椎骨数増大型遺伝形質を有さないと判定される。
【0042】
即ち、本発明の方法の好ましい態様としては、ブタの椎骨数増大型遺伝子形質の有無の判定方法であって、ブタゲノム中に存在する上記(1)〜(25)のいずれかに記載の一もしくは複数の多型マーカーを用いて、当該多型マーカーにおける塩基種(DNA配列)が上記判定表の「増大型Q」でない場合、あるいは「野生型wt」である場合に、被検ブタは椎骨数増大型遺伝子形質を有さないと判定する方法である。
【0043】
また、本発明の好ましい態様としては、ブタの第7染色体上の、以下の(a)〜(d)のいずれかに記載のマイクロサテライト配列を検出することを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法に関する。
(a)配列番号:26に記載の塩基配列において、180〜195位または196〜216位のマイクロサテライト配列
(b)配列番号:27に記載の塩基配列において、311〜323位、380〜388位、389〜396位、または400〜406位のマイクロサテライト配列
(c)配列番号:28に記載の塩基配列において、393〜413位のマイクロサテライト配列
(d)配列番号:29に記載の塩基配列において、96〜107位または309〜325位のマイクロサテライト配列
【0044】
この方法においては、被検ブタの染色体において、上記(a)〜(d)のいずれかのマイクロサテライト配列が検出された場合に、被検ブタは椎骨数増大型遺伝形質を有すると判定される。一方、上記マイクロサテライト配列が検出されない場合に、被検ブタは椎骨数増大型遺伝形質を有さないと判定される。
上記マイクロサテライト配列の詳細を、後述の表6−1〜6−2に記載する。
【0045】
また、後述の実施例において、ブタのVertnin遺伝子の発現が、椎骨数の増大する原因であることが示された。従って、本発明はブタの第7染色体上に存在するVertnin遺伝子の発現量を指標とすることを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法を提供する。ここで遺伝子の発現には、転写および翻訳が含まれる。
【0046】
遺伝子の発現量の検出は、通常、Vertnin遺伝子から転写されたRNAの量を対照と比較することで行われる。このような方法としては、Vertnin遺伝子をコードするポリヌクレオチドにハイブリダイズするプローブを用いたノーザンブロッティング法、またはVertnin遺伝子(ポリヌクレオチド)にハイブリダイズするプライマーを用いたRT-PCR法等を例示することができる。また、Vertnin遺伝子の発現量の検出においては、DNAアレイ(新遺伝子工学ハンドブック、村松正實・山本雅、羊土社、p280-284)を利用することもできる。
【0047】
さらに、Vertnin遺伝子の発現量を、翻訳レベルを評価することによって検出することも可能である。当該検出においては、Vertnin遺伝子から転写・翻訳されたポリペプチドの量を対照と比較することで行われる。このような方法としては、SDSポリアクリルアミド電気泳動法、並びにVertnin(ポリペプチド)に結合する抗体を用いた、ウェスタンブロッティング法、ドットブロッティング法、免疫沈降法、酵素結合免疫測定法(ELISA)、および免疫蛍光法を例示することができる。
【0048】
上記の方法において、対照(野生型)と比較して、Vertnin遺伝子由来のRNAまたはポリペプチドの発現量が上昇していた場合、該遺伝子が由来する個体(被検ブタ)は、椎骨数増大型遺伝形質を有すると判定される。
【0049】
本発明の判定方法において、変異を検出する(多型マーカーにおける多型部位の塩基種(DNA配列)を評価する)方法は、公知の種々の方法を利用することが可能である。即ち、変異を検出可能な方法であれば任意の方法を本発明の判定方法において用いることができる。
【0050】
以下に本発明の多型マーカーにおける多型部位の変異の検出が可能な方法を例示するが、必ずしもこれらの方法に限定されない。当業者であれば、変異の種類や位置等を考慮して、適宜当該変異を検出することが可能である。
【0051】
まず、本発明の多型マーカーにおける多型変異(塩基種)を直接決定する方法が挙げられる。即ち、多型部位を含む塩基配列を直接決定することを特徴とする、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)増幅したDNAの塩基配列を決定する工程
【0052】
この方法においてはまず、被検ブタからDNA試料を調製する。DNA試料は、例えば被検ブタの臓器、または組織、あるいは細胞や血液、口腔粘膜、皮膚、毛等から抽出した染色体DNAを基に調製することができる。
【0053】
本方法においては、次いで、本発明の多型マーカーを含むDNA領域を単離する。該DNAの単離は、本発明の多型マーカーを含むDNAにハイブリダイズするプライマーを用いて、染色体DNAを鋳型としたPCR等によって行うことも可能である。本方法においては、次いで、単離したDNAの塩基配列(多型部位の塩基種)を決定する。単離したDNAの塩基配列の決定は、当業者に公知の方法で行うことができる。
【0054】
本方法においては、次いで、決定したDNAの塩基配列(多型部位の塩基種)を、上記判定表に基づいて、ブタの椎骨数増大型遺伝形質の有無の判定を行う。
【0055】
本発明の判定方法は、上述のように直接被検ブタ由来のDNAの塩基配列を決定する方法以外に、上記多型変異の検出が可能な種々の方法によって行うことができる。
【0056】
例えば、本発明における上記多型変異の検出は、以下の工程を含む方法によっても行うことができる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)増幅したDNAを制限酵素により切断する工程
(d)DNA断片をその大きさに応じて分離する工程
(e)検出されたDNA断片の大きさを対照と比較する工程
【0057】
まず、被検ブタからDNA試料を調製する。次いで、本発明の多型マーカーを含むDNAを増幅する。さらに、増幅したDNAを制限酵素により切断する。次いで、DNA断片をその大きさに応じて分離する。次いで、検出されたDNA断片の大きさを、対照と比較する。
【0058】
このような方法としては、例えば、制限酵素断片長多型(Restriction Fragment Length Polymorphism/RFLP)を利用した方法やPCR−RFLP法等が挙げられる。具体的には、制限酵素の認識部位に変異が存在する場合、制限酵素処理後に生じる断片の大きさが対照と比較して変化する。この変異を含む部分をPCR法によって増幅し、それぞれの制限酵素で処理することによって、これらの変異を電気泳動後のバンドの移動度の差として検出することができる。あるいは、染色体DNAをこれらの制限酵素によって処理し、電気泳動した後、本発明のプローブDNAを用いてサザンブロッティングを行うことにより、変異の有無を検出することができる。用いられる制限酵素は、それぞれの変異に応じて適宜選択することができる。さらに、ゲノムDNAを蛍光ラベルしたプライマーを用いてPCR増幅し、制限酵素切断の後、シークエンサー等のディテクターで変異を検出することも可能である。
【0059】
なお、上記方法において使用する制限酵素は、通常、本発明の多型マーカーを含むDNA配列を認識する酵素である。
制限酵素を利用した上記方法の好ましい態様としては、例えば、後述の表8−1〜8−7に記載されたフォワードプライマーおよびリバースプライマーを用いて増幅されるDNA断片を、制限酵素で処理することによって行うPCR-RFLP法が挙げられる。
【0060】
当業者であれば、上記多型マーカーの多型部位および当該部位の塩基種等の情報を基に、本発明の判定方法において使用可能な適切なプライマーおよび制限酵素を、適宜選択することが可能である。
【0061】
本発明の方法の好ましい態様としては、ブタゲノム中に存在する上記(1)〜(25)のいずれかに記載の多型マーカーを含むDNA領域を増幅し、その増幅産物の多型を検出する工程を含む方法である。
【0062】
上記方法における多型の検出は、好ましくは、増幅産物を制限酵素で処理することによって行う。使用する制限酵素は、対象の多型マーカーの塩基種および周辺DNA領域の塩基配列に関する情報に基づいて、当業者であれば適宜選択することができる。
【0063】
さらに別の方法としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)増幅したDNAを一本鎖に解離させる工程
(d)解離させた一本鎖DNAを非変性ゲル上で分離する工程
(e)分離した一本鎖DNAのゲル上での移動度を対照と比較する工程
【0064】
まず、被検ブタからDNA試料を調製する。次いで、本発明の多型マーカーを含むDNA領域を増幅する。さらに、増幅したDNAを一本鎖DNAに解離させる。次いで、解離させた一本鎖DNAを非変性ゲル上で分離する。分離した一本鎖DNAのゲル上での移動度を対照と比較する。
【0065】
該方法としては、例えばPCR-SSCP(single-strand conformation polymorphism、一本鎖高次構造多型)法(Cloning and polymerase chain reaction-single-strand conformation polymorphism analysis of anonymous Alu repeats on chromosome 11. Genomics. 1992 Jan 1; 12(1): 139-146.、Detection of p53 gene mutations in human brain tumors by single-strand conformation polymorphism analysis of polymerase chain reaction products. Oncogene. 1991 Aug 1; 6(8): 1313-1318.、Multiple fluorescence-based PCR-SSCP analysis with postlabeling. 、PCR Methods Appl. 1995 Apr 1; 4(5): 275-282.)が挙げられる。この方法は操作が比較的簡便であり、また被検試料の量も少なくて済む等の利点を有するため、特に多数のDNA試料をスクリーニングするのに好適である。その原理は次の通りである。二本鎖DNA断片を一本鎖に解離すると、各鎖はその塩基配列に依存した独自の高次構造を形成する。この解離したDNA鎖を、変性剤を含まないポリアクリルアミドゲル中で電気泳動すると、それぞれの高次構造の差に応じて、相補的な同じ鎖長の一本鎖DNAが異なる位置に移動する。一塩基の置換によってもこの一本鎖DNAの高次構造は変化し、ポリアクリルアミドゲル電気泳動において異なる移動度を示す。従って、この移動度の変化を検出することによりDNA断片に欠損等による変異が存在することを検出することができる。
【0066】
具体的には、まず、本発明の多型マーカーを含むDNA領域を増幅する。増幅される範囲としては、通常200〜400 bp程度の長さが好ましい。PCRは、当業者においては反応条件等を適宜選択して行うことができる。PCRの際に、32P等のアイソトープ、蛍光色素、またはビオチン等によって標識したプライマーを用いることにより、増幅DNA産物を標識することができる。あるいはPCR反応液に32P等のアイソトープ、蛍光色素、またはビオチン等によって標識された基質塩基を加えてPCRを行うことにより、増幅DNA産物を標識することも可能である。さらに、PCR反応後にクレノウ酵素等を用いて、32P等のアイソトープ、蛍光色素、またはビオチン等によって標識された基質塩基を、増幅DNA断片に付加することによっても標識を行うことができる。こうして得られた標識DNA断片を、熱を加えること等により変性させ、尿素などの変性剤を含まないポリアクリルアミドゲルによって電気泳動を行う。この際、ポリアクリルアミドゲルに適量(5から10%程度)のグリセロールを添加することにより、DNA断片の分離の条件を改善することができる。また、泳動条件は各DNA断片の性質により変動するが、通常、室温(20から25℃)で行い、好ましい分離が得られないときには4から30℃までの温度で最適の移動度を与える温度の検討を行う。電気泳動後、DNA断片の移動度を、X線フィルムを用いたオートラジオグラフィーや、蛍光を検出するスキャナー等で検出し、解析を行う。移動度に差があるバンドが検出された場合、このバンドを直接ゲルから切り出し、PCRによって再度増幅し、それを直接シークエンシングすることにより、変異の存在を確認することができる。また、標識したDNAを使わない場合においても、電気泳動後のゲルをエチジウムブロマイドや銀染色法などによって染色することによって、バンドを検出することができる。
【0067】
さらに別の方法としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)ヌクレオチドプローブが固定された基板を提供する工程
(d)工程(b)のDNAと工程(c)の基板を接触させる工程
(e)該DNAと該基板に固定されたヌクレオチドプローブとのハイブリダイズの強度を検出する工程
(f)工程(e)で検出された強度を対照と比較する工程
【0068】
まず、被検ブタから調製した本発明の多型マーカーを含むDNA、および該DNAとハイブリダイズするヌクレオチドプローブが固定された基板、を提供する。次いで、該DNAと該基板を接触させる。さらに、基板に固定されたヌクレオチドプローブにハイブリダイズしたDNAを検出することにより、上記「変異」を検出する。
【0069】
このような方法としては、DNAアレイ法が例示できる。本発明の多型マーカーを含むDNA試料の調製は、当業者に周知の方法で行うことができる。該DNA試料の調製の好ましい態様においては、例えば被検ブタの血液、皮膚、口腔粘膜等の組織または細胞から抽出した染色体DNAを基に調製することができる。染色体DNAから本方法のDNA試料を調製するには、例えば、本発明の多型マーカーを含むDNA領域にハイブリダイズするプライマーを用いて、染色体DNAを鋳型としたPCR等によって、本発明の多型マーカーを含むDNAを調製することも可能である。調製したDNA試料には、必要に応じて、当業者に周知の方法によって検出のための標識を施すことができる。
【0070】
本発明において「基板」とは、ヌクレオチドを固定することが可能な板状の材料を意味する。本発明においてヌクレオチドには、オリゴヌクレオチドおよびポリヌクレオチドが含まれる。本発明の基板は、ヌクレオチドを固定することが可能であれば特に制限はないが、一般にDNAアレイ技術で使用される基板を好適に用いることができる。
【0071】
一般にDNAアレイは、高密度に基板にプリントされた何千ものヌクレオチドで構成されている。通常これらのDNAは非透過性(non-porous)の基板の表層にプリントされる。基板の表層は、一般的にはガラスであるが、透過性(porous)の膜、例えばニトロセルロースメンブレムを使用することができる。
【0072】
本発明において、ヌクレオチドの固定(アレイ)方法として、Affymetrix社開発によるオリゴヌクレオチドを基本としたアレイが例示できる。オリゴヌクレオチドのアレイにおいて、オリゴヌクレオチドは通常インサイチュ(in situ)で合成される。例えば、photolithographicの技術(Affymetrix社)、および化学物質を固定させるためのインクジェット(Rosetta Inpharmatics社)技術等によるオリゴヌクレオチドのインサイチュ合成法が既に知られており、いずれの技術も本発明の基板の作製に利用することができる。
【0073】
基板に固定するヌクレオチドプローブは、上記「変異」を検出することができるものであれば、特に制限されない。即ち該プローブは、例えば、本発明の多型マーカーを含むDNAと特異的にハイブリダイズするようなプローブである。特異的なハイブリダイズが可能であれば、ヌクレオチドプローブは、検出する多型マーカーを含むDNAに対し、完全に相補的である必要はない。
【0074】
本発明において基板に結合させるヌクレオチドプローブの長さは、オリゴヌクレオチドを固定する場合は、通常10〜100ベースであり、好ましくは10〜50ベースであり、さらに好ましくは15〜25ベースである。
【0075】
本発明においては、次いで、該DNA試料と該基板を接触させる。本工程により、上記ヌクレオチドプローブに対し、DNA試料をハイブリダイズさせる。ハイブリダイゼーションの反応液および反応条件は、基板に固定するヌクレオチドプローブの長さ等の諸要因により変動しうるが、一般的に当業者に周知の方法により行うことができる。
【0076】
本発明においては、次いで、該DNA試料と基板に固定されたヌクレオチドプローブとのハイブリダイズの有無または強度を検出する。この検出は、例えば、蛍光シグナルをスキャナー等によって読み取ることによって行うことができる。なお、DNAアレイにおいては、一般的にスライドガラスに固定したDNAをプローブといい、一方溶液中のラベルしたDNAをターゲットという。従って、基板に固定された上記ヌクレオチドを、本明細書においてヌクレオチドプローブと記載する。
【0077】
さらに別の方法としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域にハイブリダイズするヌクレオチドとハイブリダイゼーションさせる工程
(c)ハイブリッド形成の程度を検出する工程
(d)工程(c)で検出された程度を対照と比較する工程
【0078】
まず、被検ブタから調製した本発明の多型マーカーを含むDNA、および該DNAとハイブリダイズするヌクレオチドを用意する。次いで、該DNAとヌクレオチドとをハイブリダイゼーションさせる。さらに、ハイブリッド形成の程度を検出し、対照と比較することにより、上記変異を検出する。
【0079】
具体的には、特定位置の欠損のみを検出する目的にはアレル特異的オリゴヌクレオチド(Allele Specific Oligonucleotide/ASO)ハイブリダイゼーション法が利用できる。多型変異が存在すると考えられる塩基配列を含むオリゴヌクレオチドを作製し、これと試料DNAでハイブリダイゼーションを行わせると、変異が存在する場合、ハイブリッド形成の効率が低下する。それをサザンブロット法や、特殊な蛍光試薬がハイブリッドのギャップにインターカレーションすることにより消光する性質を利用した方法、等により検出することができる。
【0080】
またInvader法による検出も可能である。具体的には、本発明の多型マーカーにおける多型部位にハイブリダイズする2種類のオリゴヌクレオチド(アレルオリゴ及びInvaderオリゴ)を用意する。該部位においてInvaderオリゴがアレルオリゴの下に1塩基のみ侵入する。侵入構造を認識する部分を酵素反応で切断することによりアレルオリゴの断片が放出される。過剰なアレルオリゴにより反応が繰り返され、断片の量が増幅される。変異に対応したオリゴヌクレオチドを用い、断片量を検出することにより、変異を検出することができる。
【0081】
またTaqMan probe法による検出も可能である。具体的には、5’末端を特殊な蛍光物質(FAMなど)で、3’末端をクエンチャー物質(TAMRAなど)で修飾した、本発明の多型マーカーを含むDNAにハイブリダイズするオリゴヌクレオチド(TaqManプローブ)の存在下で、本発明の多型マーカーを含むDNAをPCR法等によって増幅する。PCRの伸長反応ステップのときに、鋳型DNAにハイブリダイズしたTaqManプローブが分解されると、蛍光色素がプローブから遊離し、クエンチャーによる抑制が解除されて蛍光が発せられる。異なる変異ごとに異なる蛍光を発するTaqMan probeを用い、蛍光の量を測定することにより、変異を検出することができる。
【0082】
さらに別の方法としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)工程(b)で増幅したDNAを質量分析器にかけ、分子量を測定する工程
(d)工程(c)で測定した分子量を対照と比較する工程
【0083】
まず、被検ブタからDNAを調製し、次いで、本発明の多型マーカーを含むDNA領域を増幅する。次いで、増幅したDNAを質量分析器にかけ、分子量を測定する。次いで、測定した分子量を対照と比較する。このような方法としては、例えば、MALDI-TOF MS法(Trends Biotechnol (2000):18:77-84)等が挙げられる。
【0084】
また、本発明の方法は、PCR-SSP(sequence specific primers)法を用いて実施することが可能である。PCR-SSP法は、特定の配列をもったDNA部位を選択的にPCRによって増幅する方法である。
【0085】
例えば、本発明の多型マーカーにおける「増大型Q」の塩基種(DNA配列)を選択的に増幅し得るプライマーを設定し、PCRを行う。当該PCRの増幅産物の有無により、ブタの椎骨数増大型遺伝形質の有無の判定を行うことが可能である。例えば、当該PCRの増幅産物が検出された場合に、被検ブタは椎骨数増大型遺伝形質を有すると判定される。一方、当該PCRの増幅産物が検出されない場合に、被検ブタは椎骨数増大型遺伝形質を有さないと判定される。
PCR-SSPを行う場合、プライマーは3'末端側に検出したい配列(本発明の多型マーカー)がくるように設計することが好ましい。
【0086】
上記方法の好ましい態様としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーが3'末端側に位置するように設計されたPCR-SSP用プライマーを用いて、当該多型マーカーを含むDNA領域をPCRにて増幅する工程
(c)増幅したDNAを検出する工程
【0087】
また本発明は、ブタ第7染色体上に存在する多型マーカーであって、上記(1)〜(25)のいずれかに記載の多型マーカーを提供する。より具体的には、本発明は、下記(1)〜(25)のいずれかに記載の遺伝マーカーに関する。
(1)配列番号:1に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、40位の多型部位を含み、かつ該多型部位の塩基種がTであることを特徴とする遺伝マーカー
(2)配列番号:2に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、207位の多型部位を含み、かつ該多型部位の塩基種がCであることを特徴とする遺伝マーカー
(3)配列番号:3に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、68位の多型部位を含み、かつ該多型部位の塩基種がAであることを特徴とする遺伝マーカー
(4)配列番号:4に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、47位または101位の多型部位を含み、かつ該多型部位の塩基種がそれぞれAまたはGであることを特徴とする遺伝マーカー
(5)配列番号:5に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、81位の多型部位を含み、かつ該多型部位の塩基種がTであることを特徴とする遺伝マーカー
(6)配列番号:6に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、355位の多型部位を含み、かつ該多型部位の塩基種がTであることを特徴とする遺伝マーカー
(7)配列番号:7に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、87位の多型部位を含み、かつ該多型部位の塩基種がAであることを特徴とする遺伝マーカー
(8)配列番号:8に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、42位の多型部位を含み、かつ該多型部位の塩基種がGであることを特徴とする遺伝マーカー
(9)配列番号:9に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、152位または185位の多型部位を含み、かつ該多型部位の塩基種がそれぞれCまたはCであることを特徴とする遺伝マーカー
(10)配列番号:10に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、82位の多型部位を含み、かつ該多型部位の塩基種がAであることを特徴とする遺伝マーカー
(11)配列番号:11に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、95〜385位の多型部位を含むことを特徴とする遺伝マーカー
(12)配列番号:12に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、49位の多型部位を含み、かつ該多型部位の塩基種がGであることを特徴とする遺伝マーカー
(13)配列番号:13に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、31位、116位、335位、または421位の多型部位を含み、かつ該多型部位の塩基種がそれぞれA、T、C、またはTであることを特徴とする遺伝マーカー
(14)配列番号:14に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、127位の多型部位を含み、かつ該多型部位の塩基種がCであることを特徴とする遺伝マーカー
(15)配列番号:15に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、78位、154位、173位、187位、217位、320位、または433位の多型部位を含み、かつ該多型部位の塩基種がそれぞれ欠失変異(Aの欠失変異)、A、A、G、G、C、またはTであることを特徴とする遺伝マーカー
(16)配列番号:16に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、111位の多型部位を含み、かつ該多型部位の塩基種がAであることを特徴とする遺伝マーカー
(17)配列番号:17に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、71位または192位の多型部位を含み、かつ該多型部位の塩基種がそれぞれTまたはCであることを特徴とする遺伝マーカー
(18)配列番号:18に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、80〜82位の多型部位を含み、かつ該多型部位の塩基種がAAAであることを特徴とする遺伝マーカー
(19)配列番号:19に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、285位または300〜301位の多型部位を含み、かつ該多型部位の塩基種がそれぞれTまたはGAであることを特徴とする遺伝マーカー
(20)配列番号:20に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、55位、65位、75位、または150位の多型部位を含み、かつ該多型部位の塩基種がそれぞれT、T、G、またはGであることを特徴とする遺伝マーカー
(21)配列番号:21に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、99位または138位の多型部位を含み、かつ該多型部位の塩基種がCまたはGであることを特徴とする遺伝マーカー
(22)配列番号:22に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、75位の多型部位を含み、かつ該多型部位の塩基種がGであることを特徴とする遺伝マーカー
(23)配列番号:23に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、217位の多型部位を含み、かつ該多型部位の塩基種がCであることを特徴とする遺伝マーカー
(24)配列番号:24に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、91位の多型部位を含み、かつ該多型部位が欠失変異(Cの欠失変異)であることを特徴とする遺伝マーカー
(25)配列番号:25に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、136位の多型部位を含み、かつ該多型部位の塩基種がCであることを特徴とする遺伝マーカー
【0088】
上記遺伝マーカーの長さは特に制限されないが、例えば、300 bp以下であり、好ましくは100 bp以下であり、より好ましくは50 bp以下であり、さらに好ましくは30もしくは20 bp以下である。
【0089】
なお本明細書において引用された全ての先行技術文献は、参照として本明細書に組み入れられる。
【実施例】
【0090】
以下本発明を実施例により具体的に説明するが、本発明はこれら実施例に制限されるものではない。
【0091】
〔実施例1〕 大ヨークシャー種系統豚を用いたQTLのファインマッピング
(1) 徳島県大ヨークシャー種系統豚(AY)の種豚のQTL型の判定
徳島県大ヨークシャー種系統豚(AY)は、1987年に雄10頭、雌65頭を導入し、以後7世代にわたって閉鎖群で育種された。その間の雄、雌の数は8〜11、および28〜36である。その後、雄9頭、雌35頭で維持された。大ヨークシャー種の種雄豚9頭、種雌豚6頭について、それぞれの子豚(18頭から167頭)の椎骨数を測定し、マイクロサテライトマーカーを用いて半きょうだい解析を行った。QTLはマイクロサテライトマーカーSW252をピークとして検出されており(図2)、その近傍のSW263、S0115とともに種豚と子豚すべてをタイピングし、種豚の2本の染色体上のどちらのQTL領域が子豚に伝達されたかを判別した。各種豚について、2つのQTL領域がそれぞれ伝達された子豚グループ間の椎骨数の平均値の差をt検定により検討し、有意な差が見られた場合は種豚のQTLがヘテロ型であるとした。差が見られなかった場合にはZ検定(Nezer C. 2003 Genetics 165: 277-285)により、Z<-2.0においてホモ型であると判定した。その際、椎骨数増大効果はこれまでのF2実験家系の解析における平均値0.43を用いた。またホモ型種豚の子豚の椎骨数を、ヘテロ型種豚の椎骨数増大型対立遺伝子(Q)および野生型対立遺伝子(wt)が伝達した子豚(Q;371頭、wt;383頭)の椎骨数と多重検定(Steel検定)し、Q/Q型またはwt/wt型を判定した(図4)。その結果、9頭においてQ/wtのヘテロ型であると示された。6頭についてはホモ型であり、5頭がQQ型、1頭がwt/wtであることが示された(図4)。
【0092】
(2) QTL領域におけるマイクロサテライトマーカーの開発
第7染色体の椎骨数QTLの95%信頼区間はQTL express (Seaton G. 2002 Bioinformatics 18: 339-340)を用いて解析した結果、マイクロサテライトマーカーSW263とSW632にはさまれた83 cMから88 cMまでの領域となった(図5)。次にこの領域に新規マイクロサテライトマーカーを開発するため、まずヒトゲノム上の遺伝子配列を用いて相同性検索を行い、相同性のあるブタ配列(主にEST配列)を得た。これらについてPCRプライマーを作製し、PCRを用いたスクリーニングシステムにより得られたBACクローン(Suzuki K. 2000 Anim. Genet. 31: 8-12)からマイクロサテライト配列を単離した。単離したマイクロサテライト配列は、SGSC(International Swine Genome Sequecing Consortium、http://piggenome.org/)によって進行中のブタゲノムドラフト配列と比較し、それらマイクロサテライト配列を有するBACクローンのブタゲノム上の位置をFPC(Finger Print Contig)地図により確認した。39の遺伝子に由来する55個のマイクロサテライトマーカーの開発について表5−1〜5−2(第7染色体上の椎骨数QTL領域に開発したマイクロサテライトマーカー)、図6に示した。
【0093】
〔表5−1〕

【0094】
表5−2は表5−1の続きの表である。
〔表5−2〕

【0095】
(3) 徳島県大ヨークシャー種系統豚(AY)の種豚の各染色体上のマイクロサテライトマーカーのハプロタイプの解析
大ヨークシャー種種豚およびそれらの子豚について上記のマイクロサテライトマーカーのジェノタイピングを行い、種豚におけるハプロタイプを決定した。種豚間においてハプロタイプの比較を行い共通祖先由来(同祖)のゲノム領域、それらの染色体上での組み換え位置を解析した。大ヨークシャー種種豚の各染色体上のマイクロサテライトマーカーのハプロタイプと、QTLの対立遺伝子(Qまたはwt)との関連性を解析した結果、SJ7106、SJ7101、J7008の範囲においてのみ、椎骨数増大型対立遺伝子Q、野生型対立遺伝子wtが座位する両方の染色体に、共通ハプロタイプ(同祖ゲノム領域)が存在しなかった(図7)。よって候補領域はその外側のマイクロサテライトマーカーSJ7088とSJ7040との間の領域となった。
【0096】
(4) QTL領域の遺伝子地図の作成と新規マイクロサテライトマーカーの開発
SGSCにより解読が行われた5つのBACクローン、CH242-31K14、CH242-238O9、CH242-92H3、CH242-154M12、CH242-313I6の塩基配列を用い(図8A)、QTL近傍領域の遺伝子地図を作成した(図8B)。またCH242-92H3、CH242-154M12の塩基配列を用い、新規マイクロサテライト配列を検索し、SJ7099、SJ7103、SJ7107、SJ7113、SJ7114、SJ7121、SJ7126、SJ7136、SJ7139の9つのマイクロサテライトマーカーを開発した。
【0097】
(5) 大ヨークシャー種系統豚の種豚を用いたファインマッピング
上記徳島県大ヨークシャー種系統豚(AY)の種豚を用い、新規マイクロサテライトマーカーSJ7099、SJ7103、SJ7107、SJ7113、SJ7114、SJ7121、SJ7126、SJ7136、SJ7139を加えて解析した結果、同祖ゲノム領域を示す共通ハプロタイプが、椎骨数増大型対立遺伝子Qおよび野生型対立遺伝子wtが座位する両方の染色体に存在しない領域はSJ7121からSJ7008の間であり、また椎骨数増大型Qにおいてハプロタイプが保存された領域はSJ7121からSJ7103の範囲であった(図9)。
【0098】
〔実施例2〕F2実験家系親世代ブタを用いたファインマッピング
9つのF2実験家系の親世代ブタについて、椎骨数QTLの遺伝子型が決定され(表2)、既報(Mikawa S. 2005 J Anim. Sci. 83: 2247)において公開されている。徳島県大ヨークシャー種系統豚により絞り込まれたQTL領域近傍のマイクロサテライトマーカーを用いて、F2実験家系親世代ブタについてもハプロタイプ解析を行い、QTLと一致する領域を検索した。その結果、大ヨークシャー種系統豚において椎骨数増大型対立遺伝子Qに関連して見られたハプロタイプは、F2実験家系の親世代ブタにおいても検出され、F2実験家系親世代ブタにおいて、椎骨数増大型対立遺伝子Qが座位する染色体において保存された範囲は、SJ7088からSJ7114であった(図9)。徳島県大ヨークシャー種系統豚(AY)とF2実験家系親世代ブタの両方の解析をあわせると共通する領域はSJ7121からSJ7114となり、その外側のマーカー、SJ7126からSJ7099にQTL領域を絞り込むことに成功した(表6−1〜6−2(マイクロサテライト配列情報))。これらのマイクロサテライトマーカーからなるハプロタイプにより椎骨数QTL型を判定することが可能である。
【0099】
〔表6−1〕

【0100】
表6−2は表6−1の続きの表である。
〔表6−2〕

【0101】
上記表6−1、6−2において、下線がプライマー、斜体文字および波下線がマイクロサテライト配列を示す。
【0102】
〔実施例3〕QTL近傍領域のハプロタイプブロック解析
椎骨数QTL近傍領域の多型検索を行い、NV101、NV102、orf45-i2、orf45-i4、ALDH6A1-i11、LIN52-i4、LIN52-i5、LIN52-e6、NV103、ABCD4-i15、NV038、NV032、NV004、NV015、NV090、NV025、NV035、NV062、NV067、NV071、NV106、NV108、NV109、NV111、NPC2-i2、ISCA2-i3の多型部位を検出した(図10)。これらについて、と場より無作為に選抜した199頭の肉豚(ランドレース種、大ヨークシャー種、およびデュロック種の交雑豚)のDNAを用いてジェノタイピングを行い、Haploview 4.0 (Barrett et al. 2005 Bioinformatics 21: 263-265)により、ハプロタイプブロックを解析した。その結果、ABCD4-i15からNV067までの、約60 kbが同一ハプロタイプブロックとなった(図10)。このハプロタイプブロックのメジャーなハプロタイプは2種類であり、徳島県大ヨークシャー種系統豚(AY)での、椎骨数増大型対立遺伝子Qおよび野生型対立遺伝子wtにおけるハプロタイプに一致した。また肉豚の椎骨数との関連性はABCD4遺伝子の下流に位置する多型(NV004、NV015、NV090、NV025、NV035、NV062)が周辺の多型部位よりも高いことが確認された(図10)。これらの多型はSJ7126からSJ7099の間に位置する(図8C)。と場サンプルでの遺伝子型による椎骨数増大に対する相加効果は0.53であり、これまでに認められていた効果にほぼ等しかった(表7(と場サンプル(西洋品種)における椎骨数の分布))。また椎骨数増大型対立遺伝子の頻度は、55.5%であった。
【0103】
〔表7〕

【0104】
〔実施例4〕椎骨数を支配する遺伝子の同定
SJ7126からSJ7099の間には機能解明された遺伝子はなく、ヒトにおいてLOC55237、マウスではLOC432677と名付けられたhypothetical proteinをコードする遺伝子座が存在した。ブタにおいてもこの領域の転写産物を単離し、それをコードする遺伝子をVertnin (VRTN)と命名し、椎骨数を支配する遺伝子とした(図8C)。VRTN遺伝子は2つのエキソンからなり(図8C)、698アミノ酸をコードする。また相同性検索の結果、類似した遺伝子は存在しなかった。VRTN遺伝子のcDNA配列および該遺伝子によってコードされるアミノ酸配列をそれぞれ配列番号:88、89に示す。
【0105】
〔実施例5〕ブタVRTN遺伝子の多型解析
VRTN遺伝子を含むSJ7126からSJ7099までの領域について、徳島県大ヨークシャー種系統豚の種豚についてPRE1配列等の繰り返し配列を除いたすべての領域で多型解析を行った結果、図8Cおよび表8−1〜8−7(SNP等配列情報)に示した42個の多型がQTL型と一致した。42個の多型パターンは表9(ブタVRTN遺伝子のハプロタイプ型)に示した。この中ではNV064が非同義置換であり、野生型がGAC(Asp)であるのに対し、増大型ではGGC(Gly)に置換している。
【0106】
〔表8−1〕

【0107】
表8−2は表8−1の続きの表である。
〔表8−2〕

【0108】
表8−3は表8−2の続きの表である。
〔表8−3〕

【0109】
表8−4は表8−3の続きの表である。
〔表8−4〕

【0110】
表8−5は表8−4の続きの表である。
〔表8−5〕

【0111】
表8−6は表8−5の続きの表である。
〔表8−6〕

【0112】
表8−7は表8−6の続きの表である。
〔表8−7〕

【0113】
上記表8−1〜8−7において、四角で囲った文字が多型部位を示す。
【0114】
〔表9〕

【0115】
上記表9において、delとは欠失変異(deletion)、insとは挿入変異(insertion)を表す。
【0116】
〔実施例6〕責任遺伝子のブタ胚での発現解析
VRTN遺伝子のプロモーター領域にQTLと一致する多型が存在することから、対立遺伝子間での発現様式の差異がQTLの原因であることも考えられる。そこでブタVRTN遺伝子の椎骨数増大型対立遺伝子と野生型対立遺伝子のプロモーター領域を、ルシフェラーゼをレポーター遺伝子とするプラスミドに連結し、CGR8細胞、P19細胞を用いて、プロモーター活性を比較したが、有意な差は認められなかった。CGR8細胞はマウスES細胞、P19細胞はマウス胚性腫瘍細胞であり、ともに内因性のマウスVRTN遺伝子を発現している。
【0117】
またブタでの内因性VRTN遺伝子の発現様式を解析するため、ブタ胚でのVRTN遺伝子の対立遺伝子による発現の差異を解析した。VRTN遺伝子が増大型Q/Qである種豚と野生型wt/wtである種豚を交配し、ヘテロ型Q/wtの胚を生産した。発現するmRNAはエキソン上に位置するNV062、NV064、MV065の多型により由来する対立遺伝子を区別することができる。交配後、8日、8.5日、10日、12日、14日において子宮より胚を回収し、VRTN遺伝子についてRT-PCRを行い、NV062を含む領域を増幅し、TOPO TAクローニングキット(インビトロジェン)を用いてクローニングを行った。それぞれクローン化した192個のコロニーについて、塩基配列を決定し、NV062の多型パターンの頻度を計測することにより各対立遺伝子の発現量の評価を行った(図11)。その結果、椎骨数増大型対立遺伝子Qと野生型対立遺伝子wtの相対的頻度は交配後10日においてほぼ等しいが、それ以外では椎骨数増大型が多く発現し、12日、14日では野生型の発現は認められなかった(表10(ブタ胚におけるVRTN遺伝子の発現解析))。椎骨数が増大する原因としてVRTN遺伝子の発現時期の変化の関与が示唆された。
【0118】
〔表10〕

【0119】
これらの結果より、ブタにおいてはVRTN遺伝子が椎骨数を支配することが明らかとなった。VRTN遺伝子の多型情報は育種への利用価値が高い。この遺伝子は改良品種内で椎骨数を1対立遺伝子あたり約0.6個増大させる。椎骨数は屠体長と相関することから、VRTN遺伝子の遺伝子診断は、産肉性、繁殖性(産子数、乳頭数)などの遺伝的改良に広く利用される。またこの遺伝子診断を利用した飼養方法の改善が可能である。またVRTN遺伝子の転写産物、タンパク質を利用して、椎骨数およびそれに関連する各種形質を変化させることが可能である。
【技術分野】
【0001】
本発明は、ブタの第7染色体上のVertnin遺伝子上もしくはその近傍に存在する多型マーカーを指標とするブタの椎骨数増大型遺伝形質の有無の判定方法に関する。
【背景技術】
【0002】
ブタはイノシシを祖先とし、ユーラシア大陸の複数地域で家畜化されたと言われている。19世紀中頃からヨーロッパにおいて、成長・体格などの良い豚を選抜して育種するということが行われるようになり、それらがもとになり現在の商用品種が形成されている。
【0003】
本発明者らはブタの改良を目的に経済形質に関与する遺伝子座の単離を進めてきた。肉質・成長といった経済形質の多くは量的形質でありそれを支配する遺伝子座は量的形質遺伝子座(quantitative trait locus; QTL)と呼ばれる。現在、ブタの様々なQTLについては約110の論文に約1800の報告がある(PigQTLdb, http://www.animalgenome.org/cgi-bin/QTLdb/SS/index)。しかしながら量的形質は複数の遺伝子座に支配され、また環境要因によっても大きく影響を受けるため、QTLの正確なマッピングは難しく、その責任遺伝子を同定すること、また多様性の原因となる多型を同定することは非常に困難である。ブタにおいて遺伝子レベルで解明されたQTLでは肉質(グリコーゲン量)に関与するPRKAG3遺伝子(非特許文献1)、肉量に関与するIGF2遺伝子(非特許文献2)がある。
【0004】
本発明者らは複数のF2実験家系を作製し、成長性、産肉性、肉質などの各種経済形質に関するQTL解析を行った(非特許文献3〜10)。その結果、様々なQTLが検出されたが、その中で胸椎数と腰椎数の和で示される椎骨数(図1)に関与するQTLが複数の家系を通じて2カ所のゲノム領域(第1染色体q腕末端、第7染色体q腕中央)に検出され、その存在は確からしいと判断された(表2(F2実験家系と椎骨数QTL)、図2)。
【0005】
〔表2〕

【0006】
2つの椎骨数QTLにはそれぞれ椎骨数を増大させる対立遺伝子があり、その効果はほぼ等しく、各家系の結果を平均すると対立遺伝子あたり約0.5から0.6個の椎骨数を増大させた。また2つのQTLは互いに独立に働き、すべての対立遺伝子が椎骨数増大型になると平均で約2.3本の椎骨数が増大した(表3(ブタの椎骨数に関する2つのQTLの効果)、図3)。
【0007】
〔表3〕

【0008】
ブタの頸椎は他の哺乳類と同じく7個であるが、胸椎、腰椎の数には多様性があり、胸椎は13個から16個、腰椎は5個から7個とばらついている(非特許文献11)。これらを合計した数(椎骨数)はブタの祖先であるイノシシでは19個であるが、現在の肉用品種では20個から23個であり、このような種内での大きな多様性は、哺乳類ではブタでのみ見られる(図1)。ブタは肉量増大、繁殖性向上のために体が大きくなるように選抜され、その過程で椎骨数が増大したと考えられ、実際、一つの椎骨数の増大により、体長は平均約1.5 cm伸びることが示されている(非特許文献12)。
【0009】
これまでのF2実験家系の解析において、第1染色体q腕末端領域の椎骨数QTLでは、梅山豚、金華豚などのアジア在来種、および日本イノシシの対立遺伝子に増大効果は認められず野生型であり、ランドレース、大ヨークシャー、デュロック、バークシャーといった西洋品種の対立遺伝子のすべてに椎骨数増大効果が認められた(表2、表4(椎骨数QTLの遺伝子型))。
【0010】
〔表4〕

【0011】
第1染色体上の椎骨数QTLについては、椎骨数が増大した西洋品種で固定された約300 kbの領域を発見し、そこに位置する責任遺伝子NR6A1を同定した(非特許文献13)。またそれに関して、特許出願を行った(特許文献1)。
【0012】
第7染色体q腕中央部の椎骨数QTLでは、F2実験家系に用いた西洋品種に由来する一部の対立遺伝子に椎骨数を増大させる効果が認められた(表2、表4)(非特許文献14)。また徳島県の大ヨークシャー種系統豚を用い、その種豚について第7染色体の椎骨数QTLについて半きょうだい解析を行うことにより、QTLがヘテロ型である個体を検出した(図4)(非特許文献14)。これらから現在豚肉生産に用いられている西洋品種においても第7染色体の椎骨数QTLは多様性があることが明らかとなった。
【0013】
なお、本出願の発明に関連する先行技術文献情報を以下に示す。
【先行技術文献】
【特許文献】
【0014】
【特許文献1】特開2006-101871
【非特許文献】
【0015】
【非特許文献1】Milan D., Jeon JT., Looft C., Amarger V., Robic A. et al., 2000 A mutation in PRKAG3 associated with excess glycogen content in pig skeletal muscle. Science. 288: 1248-1251.
【非特許文献2】Van Laere AS., Nguyen M., Braunschweig M., Nezer C., Collette C. et al., 2003 A regulatory mutation in IGF2 causes a major QTL effect on muscle growth in the pig. Nature. 425: 832-836.
【非特許文献3】Wada, Y., T. Akita, T. Awata, T. Furukawa, N. Sugai et al., 2000 Quantitative trait loci (QTL) analysis in a Meishan × Gottingen cross population. Anim. Genet. 31: 376-384.
【非特許文献4】松原英二、湊和之、稲毛優子、楠本宏司、美川智、和田康彦、小林栄治、峰澤満、安江博「イノブタ実験家系における経済形質とマイクロサテライトマーカーとの連鎖解析」日本養豚学会誌、第36巻4号、P189、平成11年11月
【非特許文献5】内藤学、山田渥、内田陽子、稲毛優子、三宅正志、美川智、和田康彦、小林栄治、峰澤満、粟田崇、安江博「梅山豚x大ヨークシャー種実験家系におけるDNAマーカーと経済形質との連鎖解析」日本養豚学会誌、第37巻4号、P182、平成12年10月
【非特許文献6】室伏淳一、河原崎達雄、堀内篤、久松紀子、楠本宏司、美川智、和田康彦、峰澤満、安江博「金華豚・大ヨークシャー種交雑実験家系における産肉性、肉質とDNAマーカーとの連鎖解析」日本養豚学会誌、第35巻2号、P66、平成10年6月
【非特許文献7】加治佐修、犬童政昭、三宅正志、小林栄治、和田康彦、美川智、峰澤満、安江博「バークシャー種とクラウンミニブタ交雑家系のDNAマーカーを用いた連鎖解析」日本養豚学会誌、第35巻2号、P66、平成10年6月
【非特許文献8】新居雅宏、谷史雄、仁木明人、林武司、上西博英、美川智、小林栄治、内田陽子、粟田崇、安江博「日本イノシシと大ヨークシャー種交雑家系におけるQTL解析」第98回日本畜産学会大会講演要旨集、P19、平成13年3月
【非特許文献9】山口倫子、江森格、大澤浩司、内藤昌男、神山佳三、金谷奈保恵、内田陽子、堀内篤、仲沢慶紀、林武司、粟田崇「金華豚・デュロック種交雑家系における経済形質のQTL解析と筋肉内脂肪含量を対象としたマーカー利用選抜」日本養豚学会誌、第40巻4号、P212、平成15年10月
【非特許文献10】堀内篤、知久幹夫、井手華子、金谷奈保恵、内田陽子、山口倫子、仲沢慶紀、林武司、粟田崇「金華豚とデュロック種の交雑家系における肉質に関与するQTLの解析」日本養豚学会誌、第40巻4号、P213、平成15年10月
【非特許文献11】King, J. W. B., and R. C. Roberts, 1960 Carcass length in the bacon pig: its association with vertebrae numbers and prediction from radiographs of the young pig. Anim. Prod. 2: 59-65
【非特許文献12】Berge, S., 1948 Genetical researches on the number of vertebrae in the pigs. J. Anim. Sci. 7: 233-238
【非特許文献13】Mikawa S., Morozumi T., Shimanuki S., Hayashi T., Uenishi H., Domukai M., Okumura N., Awata T. 2007 Fine mapping of a swine quantitative trait locus for number of vertebrae and analysis of an orphan nuclear receptor, germ cell nuclear factor (NR6A1). Genome Research 17:586-593
【非特許文献14】Mikawa S., Hayashi T., Nii M., Shimanuki S., Morozumi T., Awata T., 2005 Two quantitative trait loci on Sus scrofa chromosomes 1 and 7 affecting the number of vertebrae. J Anim Sci. 83:2247-2254.
【発明の概要】
【発明が解決しようとする課題】
【0016】
本発明は、上記状況に鑑みてなされたものであり、その目的は椎骨数増大型遺伝形質を有するブタを判定する方法を提供することにある。より詳しくは、本発明は、ブタの第7染色体上のVertnin(VRTN)遺伝子上、またはその近傍の多型を指標とする、ブタの椎骨数増大型遺伝形質の有無の判定方法の提供を課題とする。
【課題を解決するための手段】
【0017】
F2実験家系を用いた連鎖解析等により絞り込まれた範囲からさらに原因遺伝子の存在する位置を特定するには一般に連鎖不平衡解析が用いられる。家畜は世代間隔が短く、血縁、表形値が記録されるため、現存の動物の形質データを用いて、形質の差と関連する同一祖先由来の共通染色体領域をマッピングする方法(identical by decent解析; IBD解析)が一般的に用いられる。しかしながら量的形質では、形質と遺伝子型との関連性が遺伝病などの質的形質ほど大きくなく、形質値を用いたIBD解析での責任遺伝子の解明は困難であり、これが様々なQTLの責任遺伝子が未同定である原因である。
【0018】
よって本発明者らは、形質値ではなく、半きょうだい解析によって判定したQTL型を用いたIBD解析を試行した。この場合、QTL型と責任遺伝子の遺伝子型は完全に一致する。またQTL型を判定する個体として、系統豚を用いた。これは雄10頭、雌65頭に由来し、7世代の閉鎖群育種により造成された後、雄9頭、雌35頭で維持されているもので、染色体の組み換えの数が多いと期待される。また造成開始時に導入された個体は互いに独立したもので、遺伝的に遠縁にある。つまり同祖染色体領域の組み換えによるマッピングと、独立した個体サンプル間に存在する小さな共通遺伝領域の探索の両方が期待される。
【0019】
具体的には、本発明者らは、大ヨークシャー種系統豚の種豚における椎骨数QTL型を、半きょうだい解析により判定し、高密度に配置したマイクロサテライトマーカーを用いて、QTL型と一致するハプロタイプを有するゲノム領域を検索した。また見いだした領域の近傍においては、これまでにQTL型を判定していた他品種を含むF2実験家系親世代ブタを用いて、さらにQTL型と一致するハプロタイプを有するゲノム領域を絞り込んだ。その結果、約45 kbの領域に到達し、そこに位置する椎骨数を支配する新規遺伝子VRTNを単離した。
【0020】
VRTN遺伝子を含む45 kbの領域において、大ヨークシャー種系統豚を用いて多型検索を行った結果、QTL型と一致する42ヵ所の多型部位が明らかとなった。これら多型から構成されるハプロタイプは、この大ヨークシャー種系統豚では2種であり、またと場サンプル(ランドレース種、大ヨークシャー種、およびデュロック種の交雑種)によるハプロタイプ解析においてもこの2種がメジャーなハプロタイプであった。
【0021】
ヘテロ型のブタ胚においてVRTN遺伝子の発現解析を行った結果、野生型対立遺伝子は交配後8.5日目から10日目の限られた期間に発現しているのに対し、椎骨数増大型対立遺伝子は8日目から14日目まで発現が確認された。この結果から、VRTNの椎骨数増大型対立遺伝子は、発現時期の制御が変化することにより、椎骨数を増大させていると判断される。
【0022】
本発明者らは、ブタの第7染色体上に、ブタの椎骨数増大と関連する新規なVertnin遺伝子を同定することに成功した。さらに、該遺伝子上もしくはその近傍に存在する多型マーカーを用いることにより、ブタの椎骨数増大型遺伝形質の有無を判定可能であることを見出し、本発明を完成させた。
【0023】
即ち本発明は、ブタの第7染色体上のVertnin遺伝子上もしくはその近傍に存在する多型マーカーを指標とするブタの椎骨数増大型遺伝形質の有無の判定方法に関し、より具体的には、
〔1〕 ブタの椎骨数増大型遺伝形質の有無の判定方法であって、ブタの第7染色体上に存在する下記(A)の(1)〜(25)のいずれかに記載の一もしくは複数の多型マーカーを用いて、下記(B)の工程によって判定する方法、
(A)
(1)配列番号:1に記載の塩基配列の40位
(2)配列番号:2に記載の塩基配列の207位
(3)配列番号:3に記載の塩基配列の68位
(4)配列番号:4に記載の塩基配列の47位または101位
(5)配列番号:5に記載の塩基配列の81位
(6)配列番号:6に記載の塩基配列の355位
(7)配列番号:7に記載の塩基配列の87位
(8)配列番号:8に記載の塩基配列の42位
(9)配列番号:9に記載の塩基配列の152位または185位
(10)配列番号:10に記載の塩基配列の82位
(11)配列番号:11に記載の塩基配列の95〜385位
(12)配列番号:12に記載の塩基配列の49位
(13)配列番号:13に記載の塩基配列の31位、116位、335位、または421位
(14)配列番号:14に記載の塩基配列の127位
(15)配列番号:15に記載の塩基配列の78位、154位、173位、または187位、217位、320位、433位
(16)配列番号:16に記載の塩基配列の111位
(17)配列番号:17に記載の塩基配列の71位または192位
(18)配列番号:18に記載の塩基配列の80〜82位
(19)配列番号:19に記載の塩基配列の285位または300〜301位
(20)配列番号:20に記載の塩基配列の55位、65位、75位、または150位
(21)配列番号:21に記載の塩基配列の99位または138位
(22)配列番号:22に記載の塩基配列の75位
(23)配列番号:23に記載の塩基配列の217位
(24)配列番号:24に記載の塩基配列の91位
(25)配列番号:25に記載の塩基配列の136位
(B)上記(A)のそれぞれの多型マーカーにおける塩基種が記載された下記〔表1〕の判定表に基づき、椎骨数増大型遺伝形質の有無を判定する工程
〔表1〕

〔2〕 ブタの第7染色体上の、以下の(a)〜(d)のいずれかに記載のマイクロサテライト配列を検出することを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法、
(a)配列番号:26に記載の塩基配列において、180〜195位または196〜216位のマイクロサテライト配列
(b)配列番号:27に記載の塩基配列において、311〜323位、380〜388位、389〜396位、または400〜406位のマイクロサテライト配列
(c)配列番号:28に記載の塩基配列において、393〜413位のマイクロサテライト配列
(d)配列番号:29に記載の塩基配列において、96〜107位または309〜325位のマイクロサテライト配列
〔3〕 ブタの第7染色体上に存在する前記多型マーカーまたはマイクロサテライト配列を含むDNA領域を増幅し、その増幅産物の多型またはマイクロサテライト配列を検出する工程を含む、〔1〕または〔2〕に記載の方法、
〔4〕 ブタの第7染色体上に存在するVertnin遺伝子の発現量を指標とすることを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法、
を、提供する。
【発明の効果】
【0024】
椎骨数を支配する遺伝子の解明は、椎骨数の制御を可能とすることから、ブタの産肉性、繁殖性の効率的な改良が実現される。特に第7染色体q腕中央部の椎骨数QTLは、豚肉生産に用いられている西洋品種の椎骨数のバラツキの原因であり、この責任遺伝子の解明によりもたらされる情報は、今日の豚肉生産に直接的に利用され、その効率化に大きく貢献することができる。
【図面の簡単な説明】
【0025】
【図1】ヒト、馬、牛・羊・兎、豚における、頸椎、胸椎、腰椎、仙椎、尾椎の数を示す図である。西洋品種(ランドレース、大ヨークシャー、デュロック、バークシャー)の椎骨数は20、21、22、23、アジア在来品種(梅山豚、金華豚)は19、イノシシは19である。
【図2】椎骨数に関するQTL解析(JD家系)の結果を示す図である。第1染色体q腕末端領域と第7染色体q腕中央部に2つの椎骨数に関するQTLが検出された。
【図3】JD家系における2つの椎骨数QTLの効果を示す図である。2つのQTLのそれぞれの遺伝子型を持つF2個体の椎骨数の平均値を示した。異なるアルファベット(a, b, c, d)間で有意差がある。それぞれのQTLは主に相加的効果を持ち、また2つのQTLは独立に働く。
【図4】徳島県大ヨークシャー種系統豚(AY)の種豚を用いた第7染色体上の椎骨数QTLの半きょうだい解析の結果を示す図である。種豚の相同染色体上のQTL領域の子豚への伝達を、マイクロサテライトマーカーを用いて解析した。由来する種豚のQTL領域により2つの群に分け(任意に黒、白で示す。)、それぞれの椎骨数の平均値をプロットした。また子豚の頭数と標準誤差を示した。t検定においてP < 0.01 (**)において種豚のQTLがヘテロ型であると判断した。Z検定は椎骨数増大効果を0.43としてNezerらの方法で行ない、Z <-2.0をホモ型とした。またホモ型種豚それぞれの子豚の椎骨数を、ヘテロ型種豚の椎骨数増大型アリルおよび野生型アリルが伝達した子豚(Q;371頭、q;383頭)の椎骨数と多重検定(Steel検定)し、椎骨数増大型または野生型を判定した。
【図5】第7染色体上の椎骨数QTLの信頼区間(三県合同、JD家系)を示す図である。Bootstrap法(10000回)により、QTLが位置する95%信頼区間を求めた。95%の区間において黒色、それ以外を灰色で示した。95%信頼区間は83 cMから88 cMであった。
【図6】椎骨数QTL近傍領域におけるマイクロサテライトマーカーの開発を示す図である。ヒト遺伝子情報を利用して開発したブタSTSによりBACクローンを単離し、マイクロサテライトマーカーを開発した。開発したマイクロサテライトマーカーは、SGSC(International Swine Genome Sequencing Consortium)によって進行中のブタゲノムドラフト配列と比較し、それらの染色体上の位置を確認した。下線で示したマーカーはブタゲノムドラフト配列に未だ見出されないものを示す。
【図7】徳島県大ヨークシャー種系統豚(AY)の種豚におけるQTLタイプと同祖染色体領域を示す図である。図4においてQTLタイプを判定した種豚の各染色体についてマイクロサテライトマーカーのハプロタイプを解析した。同祖である領域(同じタイプのハプロタイプ)についてそれぞれ同色で示した。椎骨数増大型対立遺伝子Q、野生型対立遺伝子wtが座位する両方の染色体に伝達している領域は赤文字で示した。同祖の染色体領域が、Qおよびwtの両方に伝達していないのはSJ7106からSJ7008の間であった。L、Rは各個体の任意の2つの染色体領域を示す。
【図8】椎骨数QTL近傍領域のゲノム構造解析の結果を示す図である。(A)SGSC(International Swine Genome Sequencing Consortium)により塩基配列が解読されているBACクローン。(B)BACクローンの塩基配列より作成した遺伝子地図。マイクロサテライトマーカーの位置を示した。下線で示したものは、BACクローンの塩基配列より新規に開発した。(C)VRTN遺伝子近傍の多型部位。大ヨークシャー種系統豚におけるQTLと一致する多型部位を示した。
【図9】大ヨークシャー種種豚およびF2実験家系親世代ブタにおけるQTLタイプと同祖染色体領域を示す図である。BACクローンの配列より新規に開発したマイクロサテライトマーカー(下線)を加え、ハプロタイプ解析を行った。L:ランドレース、W:大ヨークシャー、B:バークシャー、D:デュロック、Wb:猪、M:梅山豚、J:金華豚、C:クラウンミニ豚。L、Rは各個体の任意の2つの染色体領域を示す。
【図10】椎骨数QTL近傍領域の多型を用いた椎骨数との関連性解析とハプロタイプブロック解析の結果を示す図である。椎骨数QTL近傍領域に多型マーカーを開発し、と場よりサンプリングした199頭を用いて、それらの遺伝子型と椎骨数との関連性について分散分析を行い、F値をプロットした。またそれらの多型についてハプロタイプブロックを解析した。数字はR2値を示した。
【図11】ヘテロ型ブタ胚を用いたVRTN遺伝子の対立遺伝子間の発現比較解析を示す図である。交配後、8日、8.5日、10日、12日、14日においてヘテロ型(Q/wt)胚を子宮より回収し、トータルRNAを調製した。NV062をRT-PCRにより増幅し、TAクローニングを行った。クローン化した192個のコロニーについて、塩基配列を決定し、NV062の多型パターンの頻度を計測することにより各対立遺伝子の発現量の評価を行った。
【発明を実施するための形態】
【0026】
本発明は、ブタの第7染色体上のVertnin(VRTN)遺伝子上もしくはその近傍に存在する多型マーカーを指標とするブタの椎骨数増大型遺伝形質の有無の判定方法(本明細書において、「本発明の判定方法」と記載する場合あり)を提供する。
【0027】
本発明者らによって見出されたVertnin遺伝子の塩基配列、および該遺伝子によってコードされるタンパク質のアミノ酸配列を、それぞれ、配列番号:88、89へ記載する。
【0028】
本発明において「判定」とは、通常、被検ブタ(本発明の判定方法に供するブタ)が椎骨数野生型遺伝形質を有する、もしくは有さないと判定することを言う。
【0029】
また、椎骨数増大型遺伝形質を有するブタと、その他の豚とを判別すること等をいい、本発明における「判定」の用語は、例えば、「鑑定」、「鑑別」あるいは「判別」、「検査」等と表現してもよい。
【0030】
また、本発明の判定方法は、必ずしもブタ個体を対象として判定する方法に限定されず、例えば、食肉もしくは精肉加工品(例えば、ハム等)、あるいは、被検ブタに由来する生体試料等を対象として、椎骨数増大型遺伝形質の有無を判定する方法も含まれる。
【0031】
本発明において「多型」とは、例えば、一塩基多型(SNP)、欠失変異(deletion)、挿入変異(insertion)、マイクロサテライト等を指す。
【0032】
本発明の判定方法の好ましい態様としては、ブタゲノム中に存在する多型マーカーを用いることを特徴とする方法である。
【0033】
本発明における多型マーカーとは、ブタの椎骨数増大型遺伝形質の有無を判定可能なDNA部位(多型部位)をいう。従って、本発明の方法において多型マーカーを用いるとは、通常、本発明の多型部位におけるDNA配列(塩基種)の違いに基づきブタの椎骨数増大型遺伝形質の有無の判定を行うことをいう。
【0034】
本発明において用いる多型マーカーとしては、具体的には、ブタ第7染色体上に存在する多型マーカーであって、以下の部位の多型マーカー(本明細書において「本発明の多型マーカー」と記載する場合あり)を示すことができる。
(1)配列番号:1に記載の塩基配列の40位
(2)配列番号:2に記載の塩基配列の207位
(3)配列番号:3に記載の塩基配列の68位
(4)配列番号:4に記載の塩基配列の47位または101位
(5)配列番号:5に記載の塩基配列の81位
(6)配列番号:6に記載の塩基配列の355位
(7)配列番号:7に記載の塩基配列の87位
(8)配列番号:8に記載の塩基配列の42位
(9)配列番号:9に記載の塩基配列の152位または185位
(10)配列番号:10に記載の塩基配列の82位
(11)配列番号:11に記載の塩基配列の95〜385位
(12)配列番号:12に記載の塩基配列の49位
(13)配列番号:13に記載の塩基配列の31位、116位、335位、または421位
(14)配列番号:14に記載の塩基配列の127位
(15)配列番号:15に記載の塩基配列の78位、154位、173位、または187位、217位、320位、433位
(16)配列番号:16に記載の塩基配列の111位
(17)配列番号:17に記載の塩基配列の71位または192位
(18)配列番号:18に記載の塩基配列の80〜82位
(19)配列番号:19に記載の塩基配列の285位または300〜301位
(20)配列番号:20に記載の塩基配列の55位、65位、75位、または150位
(21)配列番号:21に記載の塩基配列の99位または138位
(22)配列番号:22に記載の塩基配列の75位
(23)配列番号:23に記載の塩基配列の217位
(24)配列番号:24に記載の塩基配列の91位
(25)配列番号:25に記載の塩基配列の136位
【0035】
本発明の好ましい態様としては、上記多型マーカーにおける多型部位を評価(例えば、多型部位における塩基種(DNA配列)を決定)することにより、ブタの椎骨数増大型遺伝形質の有無を判定する方法である。
【0036】
より具体的には、本発明の判定方法は、ブタゲノム中に存在する上記(1)〜(25)のいずれかに記載の一もしくは複数の多型マーカーを用いて、上記のそれぞれの多型マーカーにおける塩基種(DNA配列)が記載された下記の判定表に基づき、ブタの椎骨数増大型遺伝形質の有無を判定する方法である。
【0037】
〔表1〕

【0038】
上記表1において、delとは欠失変異(deletion)、insとは挿入変異(insertion)を表す。
【0039】
本発明の方法においては、通常、多くの多型マーカーを用いるほど判定精度が向上する。従って、本発明の方法は、必ずしも必須ではないが、複数の多型マーカーを用いてもよい。本発明の方法において用いる多型マーカーは、通常、1もしくは少数(例えば1〜9程度)である。本発明の方法の一つの態様としては、上記多型マーカーを組み合わせて用いる方法であり、即ち、上記(1)〜(25)に記載された複数の多型マーカーからなる群より選択される1もしくは複数の多型マーカーを用いることを特徴とする方法もまた本発明に含まれる。
【0040】
本発明の判定方法の好ましい態様としては、上記(1)〜(25)の多型マーカーにおける多型部位の変異を検出することを特徴とする方法である。
本発明において変異を検出する部位は、好ましくは、上記(1)〜(25)のいずれかに記載の多型マーカーにおける多型部位である。
【0041】
本発明において変異とは、例えば、上記(1)〜(25)の多型マーカーにおける多型部位において、椎骨数増大型(Q)への変異である。
具体的には、被検ブタにおいて、上記(1)〜(25)の多型マーカーにける多型部位が、上記判定表に記載の「増大型Q」である場合に、被検ブタは椎骨数増大型遺伝形質を有すると判定される。
また、本発明の方法は、被検ブタについて上記(1)〜(25)の多型マーカーにおける多型部位が、上記判定表に記載の「増大型Q」でない場合、あるいは、「野生型wt」である場合に被検ブタは椎骨数増大型遺伝形質を有さないと判定される。
【0042】
即ち、本発明の方法の好ましい態様としては、ブタの椎骨数増大型遺伝子形質の有無の判定方法であって、ブタゲノム中に存在する上記(1)〜(25)のいずれかに記載の一もしくは複数の多型マーカーを用いて、当該多型マーカーにおける塩基種(DNA配列)が上記判定表の「増大型Q」でない場合、あるいは「野生型wt」である場合に、被検ブタは椎骨数増大型遺伝子形質を有さないと判定する方法である。
【0043】
また、本発明の好ましい態様としては、ブタの第7染色体上の、以下の(a)〜(d)のいずれかに記載のマイクロサテライト配列を検出することを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法に関する。
(a)配列番号:26に記載の塩基配列において、180〜195位または196〜216位のマイクロサテライト配列
(b)配列番号:27に記載の塩基配列において、311〜323位、380〜388位、389〜396位、または400〜406位のマイクロサテライト配列
(c)配列番号:28に記載の塩基配列において、393〜413位のマイクロサテライト配列
(d)配列番号:29に記載の塩基配列において、96〜107位または309〜325位のマイクロサテライト配列
【0044】
この方法においては、被検ブタの染色体において、上記(a)〜(d)のいずれかのマイクロサテライト配列が検出された場合に、被検ブタは椎骨数増大型遺伝形質を有すると判定される。一方、上記マイクロサテライト配列が検出されない場合に、被検ブタは椎骨数増大型遺伝形質を有さないと判定される。
上記マイクロサテライト配列の詳細を、後述の表6−1〜6−2に記載する。
【0045】
また、後述の実施例において、ブタのVertnin遺伝子の発現が、椎骨数の増大する原因であることが示された。従って、本発明はブタの第7染色体上に存在するVertnin遺伝子の発現量を指標とすることを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法を提供する。ここで遺伝子の発現には、転写および翻訳が含まれる。
【0046】
遺伝子の発現量の検出は、通常、Vertnin遺伝子から転写されたRNAの量を対照と比較することで行われる。このような方法としては、Vertnin遺伝子をコードするポリヌクレオチドにハイブリダイズするプローブを用いたノーザンブロッティング法、またはVertnin遺伝子(ポリヌクレオチド)にハイブリダイズするプライマーを用いたRT-PCR法等を例示することができる。また、Vertnin遺伝子の発現量の検出においては、DNAアレイ(新遺伝子工学ハンドブック、村松正實・山本雅、羊土社、p280-284)を利用することもできる。
【0047】
さらに、Vertnin遺伝子の発現量を、翻訳レベルを評価することによって検出することも可能である。当該検出においては、Vertnin遺伝子から転写・翻訳されたポリペプチドの量を対照と比較することで行われる。このような方法としては、SDSポリアクリルアミド電気泳動法、並びにVertnin(ポリペプチド)に結合する抗体を用いた、ウェスタンブロッティング法、ドットブロッティング法、免疫沈降法、酵素結合免疫測定法(ELISA)、および免疫蛍光法を例示することができる。
【0048】
上記の方法において、対照(野生型)と比較して、Vertnin遺伝子由来のRNAまたはポリペプチドの発現量が上昇していた場合、該遺伝子が由来する個体(被検ブタ)は、椎骨数増大型遺伝形質を有すると判定される。
【0049】
本発明の判定方法において、変異を検出する(多型マーカーにおける多型部位の塩基種(DNA配列)を評価する)方法は、公知の種々の方法を利用することが可能である。即ち、変異を検出可能な方法であれば任意の方法を本発明の判定方法において用いることができる。
【0050】
以下に本発明の多型マーカーにおける多型部位の変異の検出が可能な方法を例示するが、必ずしもこれらの方法に限定されない。当業者であれば、変異の種類や位置等を考慮して、適宜当該変異を検出することが可能である。
【0051】
まず、本発明の多型マーカーにおける多型変異(塩基種)を直接決定する方法が挙げられる。即ち、多型部位を含む塩基配列を直接決定することを特徴とする、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)増幅したDNAの塩基配列を決定する工程
【0052】
この方法においてはまず、被検ブタからDNA試料を調製する。DNA試料は、例えば被検ブタの臓器、または組織、あるいは細胞や血液、口腔粘膜、皮膚、毛等から抽出した染色体DNAを基に調製することができる。
【0053】
本方法においては、次いで、本発明の多型マーカーを含むDNA領域を単離する。該DNAの単離は、本発明の多型マーカーを含むDNAにハイブリダイズするプライマーを用いて、染色体DNAを鋳型としたPCR等によって行うことも可能である。本方法においては、次いで、単離したDNAの塩基配列(多型部位の塩基種)を決定する。単離したDNAの塩基配列の決定は、当業者に公知の方法で行うことができる。
【0054】
本方法においては、次いで、決定したDNAの塩基配列(多型部位の塩基種)を、上記判定表に基づいて、ブタの椎骨数増大型遺伝形質の有無の判定を行う。
【0055】
本発明の判定方法は、上述のように直接被検ブタ由来のDNAの塩基配列を決定する方法以外に、上記多型変異の検出が可能な種々の方法によって行うことができる。
【0056】
例えば、本発明における上記多型変異の検出は、以下の工程を含む方法によっても行うことができる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)増幅したDNAを制限酵素により切断する工程
(d)DNA断片をその大きさに応じて分離する工程
(e)検出されたDNA断片の大きさを対照と比較する工程
【0057】
まず、被検ブタからDNA試料を調製する。次いで、本発明の多型マーカーを含むDNAを増幅する。さらに、増幅したDNAを制限酵素により切断する。次いで、DNA断片をその大きさに応じて分離する。次いで、検出されたDNA断片の大きさを、対照と比較する。
【0058】
このような方法としては、例えば、制限酵素断片長多型(Restriction Fragment Length Polymorphism/RFLP)を利用した方法やPCR−RFLP法等が挙げられる。具体的には、制限酵素の認識部位に変異が存在する場合、制限酵素処理後に生じる断片の大きさが対照と比較して変化する。この変異を含む部分をPCR法によって増幅し、それぞれの制限酵素で処理することによって、これらの変異を電気泳動後のバンドの移動度の差として検出することができる。あるいは、染色体DNAをこれらの制限酵素によって処理し、電気泳動した後、本発明のプローブDNAを用いてサザンブロッティングを行うことにより、変異の有無を検出することができる。用いられる制限酵素は、それぞれの変異に応じて適宜選択することができる。さらに、ゲノムDNAを蛍光ラベルしたプライマーを用いてPCR増幅し、制限酵素切断の後、シークエンサー等のディテクターで変異を検出することも可能である。
【0059】
なお、上記方法において使用する制限酵素は、通常、本発明の多型マーカーを含むDNA配列を認識する酵素である。
制限酵素を利用した上記方法の好ましい態様としては、例えば、後述の表8−1〜8−7に記載されたフォワードプライマーおよびリバースプライマーを用いて増幅されるDNA断片を、制限酵素で処理することによって行うPCR-RFLP法が挙げられる。
【0060】
当業者であれば、上記多型マーカーの多型部位および当該部位の塩基種等の情報を基に、本発明の判定方法において使用可能な適切なプライマーおよび制限酵素を、適宜選択することが可能である。
【0061】
本発明の方法の好ましい態様としては、ブタゲノム中に存在する上記(1)〜(25)のいずれかに記載の多型マーカーを含むDNA領域を増幅し、その増幅産物の多型を検出する工程を含む方法である。
【0062】
上記方法における多型の検出は、好ましくは、増幅産物を制限酵素で処理することによって行う。使用する制限酵素は、対象の多型マーカーの塩基種および周辺DNA領域の塩基配列に関する情報に基づいて、当業者であれば適宜選択することができる。
【0063】
さらに別の方法としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)増幅したDNAを一本鎖に解離させる工程
(d)解離させた一本鎖DNAを非変性ゲル上で分離する工程
(e)分離した一本鎖DNAのゲル上での移動度を対照と比較する工程
【0064】
まず、被検ブタからDNA試料を調製する。次いで、本発明の多型マーカーを含むDNA領域を増幅する。さらに、増幅したDNAを一本鎖DNAに解離させる。次いで、解離させた一本鎖DNAを非変性ゲル上で分離する。分離した一本鎖DNAのゲル上での移動度を対照と比較する。
【0065】
該方法としては、例えばPCR-SSCP(single-strand conformation polymorphism、一本鎖高次構造多型)法(Cloning and polymerase chain reaction-single-strand conformation polymorphism analysis of anonymous Alu repeats on chromosome 11. Genomics. 1992 Jan 1; 12(1): 139-146.、Detection of p53 gene mutations in human brain tumors by single-strand conformation polymorphism analysis of polymerase chain reaction products. Oncogene. 1991 Aug 1; 6(8): 1313-1318.、Multiple fluorescence-based PCR-SSCP analysis with postlabeling. 、PCR Methods Appl. 1995 Apr 1; 4(5): 275-282.)が挙げられる。この方法は操作が比較的簡便であり、また被検試料の量も少なくて済む等の利点を有するため、特に多数のDNA試料をスクリーニングするのに好適である。その原理は次の通りである。二本鎖DNA断片を一本鎖に解離すると、各鎖はその塩基配列に依存した独自の高次構造を形成する。この解離したDNA鎖を、変性剤を含まないポリアクリルアミドゲル中で電気泳動すると、それぞれの高次構造の差に応じて、相補的な同じ鎖長の一本鎖DNAが異なる位置に移動する。一塩基の置換によってもこの一本鎖DNAの高次構造は変化し、ポリアクリルアミドゲル電気泳動において異なる移動度を示す。従って、この移動度の変化を検出することによりDNA断片に欠損等による変異が存在することを検出することができる。
【0066】
具体的には、まず、本発明の多型マーカーを含むDNA領域を増幅する。増幅される範囲としては、通常200〜400 bp程度の長さが好ましい。PCRは、当業者においては反応条件等を適宜選択して行うことができる。PCRの際に、32P等のアイソトープ、蛍光色素、またはビオチン等によって標識したプライマーを用いることにより、増幅DNA産物を標識することができる。あるいはPCR反応液に32P等のアイソトープ、蛍光色素、またはビオチン等によって標識された基質塩基を加えてPCRを行うことにより、増幅DNA産物を標識することも可能である。さらに、PCR反応後にクレノウ酵素等を用いて、32P等のアイソトープ、蛍光色素、またはビオチン等によって標識された基質塩基を、増幅DNA断片に付加することによっても標識を行うことができる。こうして得られた標識DNA断片を、熱を加えること等により変性させ、尿素などの変性剤を含まないポリアクリルアミドゲルによって電気泳動を行う。この際、ポリアクリルアミドゲルに適量(5から10%程度)のグリセロールを添加することにより、DNA断片の分離の条件を改善することができる。また、泳動条件は各DNA断片の性質により変動するが、通常、室温(20から25℃)で行い、好ましい分離が得られないときには4から30℃までの温度で最適の移動度を与える温度の検討を行う。電気泳動後、DNA断片の移動度を、X線フィルムを用いたオートラジオグラフィーや、蛍光を検出するスキャナー等で検出し、解析を行う。移動度に差があるバンドが検出された場合、このバンドを直接ゲルから切り出し、PCRによって再度増幅し、それを直接シークエンシングすることにより、変異の存在を確認することができる。また、標識したDNAを使わない場合においても、電気泳動後のゲルをエチジウムブロマイドや銀染色法などによって染色することによって、バンドを検出することができる。
【0067】
さらに別の方法としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)ヌクレオチドプローブが固定された基板を提供する工程
(d)工程(b)のDNAと工程(c)の基板を接触させる工程
(e)該DNAと該基板に固定されたヌクレオチドプローブとのハイブリダイズの強度を検出する工程
(f)工程(e)で検出された強度を対照と比較する工程
【0068】
まず、被検ブタから調製した本発明の多型マーカーを含むDNA、および該DNAとハイブリダイズするヌクレオチドプローブが固定された基板、を提供する。次いで、該DNAと該基板を接触させる。さらに、基板に固定されたヌクレオチドプローブにハイブリダイズしたDNAを検出することにより、上記「変異」を検出する。
【0069】
このような方法としては、DNAアレイ法が例示できる。本発明の多型マーカーを含むDNA試料の調製は、当業者に周知の方法で行うことができる。該DNA試料の調製の好ましい態様においては、例えば被検ブタの血液、皮膚、口腔粘膜等の組織または細胞から抽出した染色体DNAを基に調製することができる。染色体DNAから本方法のDNA試料を調製するには、例えば、本発明の多型マーカーを含むDNA領域にハイブリダイズするプライマーを用いて、染色体DNAを鋳型としたPCR等によって、本発明の多型マーカーを含むDNAを調製することも可能である。調製したDNA試料には、必要に応じて、当業者に周知の方法によって検出のための標識を施すことができる。
【0070】
本発明において「基板」とは、ヌクレオチドを固定することが可能な板状の材料を意味する。本発明においてヌクレオチドには、オリゴヌクレオチドおよびポリヌクレオチドが含まれる。本発明の基板は、ヌクレオチドを固定することが可能であれば特に制限はないが、一般にDNAアレイ技術で使用される基板を好適に用いることができる。
【0071】
一般にDNAアレイは、高密度に基板にプリントされた何千ものヌクレオチドで構成されている。通常これらのDNAは非透過性(non-porous)の基板の表層にプリントされる。基板の表層は、一般的にはガラスであるが、透過性(porous)の膜、例えばニトロセルロースメンブレムを使用することができる。
【0072】
本発明において、ヌクレオチドの固定(アレイ)方法として、Affymetrix社開発によるオリゴヌクレオチドを基本としたアレイが例示できる。オリゴヌクレオチドのアレイにおいて、オリゴヌクレオチドは通常インサイチュ(in situ)で合成される。例えば、photolithographicの技術(Affymetrix社)、および化学物質を固定させるためのインクジェット(Rosetta Inpharmatics社)技術等によるオリゴヌクレオチドのインサイチュ合成法が既に知られており、いずれの技術も本発明の基板の作製に利用することができる。
【0073】
基板に固定するヌクレオチドプローブは、上記「変異」を検出することができるものであれば、特に制限されない。即ち該プローブは、例えば、本発明の多型マーカーを含むDNAと特異的にハイブリダイズするようなプローブである。特異的なハイブリダイズが可能であれば、ヌクレオチドプローブは、検出する多型マーカーを含むDNAに対し、完全に相補的である必要はない。
【0074】
本発明において基板に結合させるヌクレオチドプローブの長さは、オリゴヌクレオチドを固定する場合は、通常10〜100ベースであり、好ましくは10〜50ベースであり、さらに好ましくは15〜25ベースである。
【0075】
本発明においては、次いで、該DNA試料と該基板を接触させる。本工程により、上記ヌクレオチドプローブに対し、DNA試料をハイブリダイズさせる。ハイブリダイゼーションの反応液および反応条件は、基板に固定するヌクレオチドプローブの長さ等の諸要因により変動しうるが、一般的に当業者に周知の方法により行うことができる。
【0076】
本発明においては、次いで、該DNA試料と基板に固定されたヌクレオチドプローブとのハイブリダイズの有無または強度を検出する。この検出は、例えば、蛍光シグナルをスキャナー等によって読み取ることによって行うことができる。なお、DNAアレイにおいては、一般的にスライドガラスに固定したDNAをプローブといい、一方溶液中のラベルしたDNAをターゲットという。従って、基板に固定された上記ヌクレオチドを、本明細書においてヌクレオチドプローブと記載する。
【0077】
さらに別の方法としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域にハイブリダイズするヌクレオチドとハイブリダイゼーションさせる工程
(c)ハイブリッド形成の程度を検出する工程
(d)工程(c)で検出された程度を対照と比較する工程
【0078】
まず、被検ブタから調製した本発明の多型マーカーを含むDNA、および該DNAとハイブリダイズするヌクレオチドを用意する。次いで、該DNAとヌクレオチドとをハイブリダイゼーションさせる。さらに、ハイブリッド形成の程度を検出し、対照と比較することにより、上記変異を検出する。
【0079】
具体的には、特定位置の欠損のみを検出する目的にはアレル特異的オリゴヌクレオチド(Allele Specific Oligonucleotide/ASO)ハイブリダイゼーション法が利用できる。多型変異が存在すると考えられる塩基配列を含むオリゴヌクレオチドを作製し、これと試料DNAでハイブリダイゼーションを行わせると、変異が存在する場合、ハイブリッド形成の効率が低下する。それをサザンブロット法や、特殊な蛍光試薬がハイブリッドのギャップにインターカレーションすることにより消光する性質を利用した方法、等により検出することができる。
【0080】
またInvader法による検出も可能である。具体的には、本発明の多型マーカーにおける多型部位にハイブリダイズする2種類のオリゴヌクレオチド(アレルオリゴ及びInvaderオリゴ)を用意する。該部位においてInvaderオリゴがアレルオリゴの下に1塩基のみ侵入する。侵入構造を認識する部分を酵素反応で切断することによりアレルオリゴの断片が放出される。過剰なアレルオリゴにより反応が繰り返され、断片の量が増幅される。変異に対応したオリゴヌクレオチドを用い、断片量を検出することにより、変異を検出することができる。
【0081】
またTaqMan probe法による検出も可能である。具体的には、5’末端を特殊な蛍光物質(FAMなど)で、3’末端をクエンチャー物質(TAMRAなど)で修飾した、本発明の多型マーカーを含むDNAにハイブリダイズするオリゴヌクレオチド(TaqManプローブ)の存在下で、本発明の多型マーカーを含むDNAをPCR法等によって増幅する。PCRの伸長反応ステップのときに、鋳型DNAにハイブリダイズしたTaqManプローブが分解されると、蛍光色素がプローブから遊離し、クエンチャーによる抑制が解除されて蛍光が発せられる。異なる変異ごとに異なる蛍光を発するTaqMan probeを用い、蛍光の量を測定することにより、変異を検出することができる。
【0082】
さらに別の方法としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーを含むDNA領域を増幅する工程
(c)工程(b)で増幅したDNAを質量分析器にかけ、分子量を測定する工程
(d)工程(c)で測定した分子量を対照と比較する工程
【0083】
まず、被検ブタからDNAを調製し、次いで、本発明の多型マーカーを含むDNA領域を増幅する。次いで、増幅したDNAを質量分析器にかけ、分子量を測定する。次いで、測定した分子量を対照と比較する。このような方法としては、例えば、MALDI-TOF MS法(Trends Biotechnol (2000):18:77-84)等が挙げられる。
【0084】
また、本発明の方法は、PCR-SSP(sequence specific primers)法を用いて実施することが可能である。PCR-SSP法は、特定の配列をもったDNA部位を選択的にPCRによって増幅する方法である。
【0085】
例えば、本発明の多型マーカーにおける「増大型Q」の塩基種(DNA配列)を選択的に増幅し得るプライマーを設定し、PCRを行う。当該PCRの増幅産物の有無により、ブタの椎骨数増大型遺伝形質の有無の判定を行うことが可能である。例えば、当該PCRの増幅産物が検出された場合に、被検ブタは椎骨数増大型遺伝形質を有すると判定される。一方、当該PCRの増幅産物が検出されない場合に、被検ブタは椎骨数増大型遺伝形質を有さないと判定される。
PCR-SSPを行う場合、プライマーは3'末端側に検出したい配列(本発明の多型マーカー)がくるように設計することが好ましい。
【0086】
上記方法の好ましい態様としては、以下の工程を含む方法が挙げられる。
(a)被検ブタからDNAを調製する工程
(b)本発明の多型マーカーが3'末端側に位置するように設計されたPCR-SSP用プライマーを用いて、当該多型マーカーを含むDNA領域をPCRにて増幅する工程
(c)増幅したDNAを検出する工程
【0087】
また本発明は、ブタ第7染色体上に存在する多型マーカーであって、上記(1)〜(25)のいずれかに記載の多型マーカーを提供する。より具体的には、本発明は、下記(1)〜(25)のいずれかに記載の遺伝マーカーに関する。
(1)配列番号:1に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、40位の多型部位を含み、かつ該多型部位の塩基種がTであることを特徴とする遺伝マーカー
(2)配列番号:2に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、207位の多型部位を含み、かつ該多型部位の塩基種がCであることを特徴とする遺伝マーカー
(3)配列番号:3に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、68位の多型部位を含み、かつ該多型部位の塩基種がAであることを特徴とする遺伝マーカー
(4)配列番号:4に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、47位または101位の多型部位を含み、かつ該多型部位の塩基種がそれぞれAまたはGであることを特徴とする遺伝マーカー
(5)配列番号:5に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、81位の多型部位を含み、かつ該多型部位の塩基種がTであることを特徴とする遺伝マーカー
(6)配列番号:6に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、355位の多型部位を含み、かつ該多型部位の塩基種がTであることを特徴とする遺伝マーカー
(7)配列番号:7に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、87位の多型部位を含み、かつ該多型部位の塩基種がAであることを特徴とする遺伝マーカー
(8)配列番号:8に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、42位の多型部位を含み、かつ該多型部位の塩基種がGであることを特徴とする遺伝マーカー
(9)配列番号:9に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、152位または185位の多型部位を含み、かつ該多型部位の塩基種がそれぞれCまたはCであることを特徴とする遺伝マーカー
(10)配列番号:10に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、82位の多型部位を含み、かつ該多型部位の塩基種がAであることを特徴とする遺伝マーカー
(11)配列番号:11に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、95〜385位の多型部位を含むことを特徴とする遺伝マーカー
(12)配列番号:12に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、49位の多型部位を含み、かつ該多型部位の塩基種がGであることを特徴とする遺伝マーカー
(13)配列番号:13に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、31位、116位、335位、または421位の多型部位を含み、かつ該多型部位の塩基種がそれぞれA、T、C、またはTであることを特徴とする遺伝マーカー
(14)配列番号:14に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、127位の多型部位を含み、かつ該多型部位の塩基種がCであることを特徴とする遺伝マーカー
(15)配列番号:15に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、78位、154位、173位、187位、217位、320位、または433位の多型部位を含み、かつ該多型部位の塩基種がそれぞれ欠失変異(Aの欠失変異)、A、A、G、G、C、またはTであることを特徴とする遺伝マーカー
(16)配列番号:16に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、111位の多型部位を含み、かつ該多型部位の塩基種がAであることを特徴とする遺伝マーカー
(17)配列番号:17に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、71位または192位の多型部位を含み、かつ該多型部位の塩基種がそれぞれTまたはCであることを特徴とする遺伝マーカー
(18)配列番号:18に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、80〜82位の多型部位を含み、かつ該多型部位の塩基種がAAAであることを特徴とする遺伝マーカー
(19)配列番号:19に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、285位または300〜301位の多型部位を含み、かつ該多型部位の塩基種がそれぞれTまたはGAであることを特徴とする遺伝マーカー
(20)配列番号:20に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、55位、65位、75位、または150位の多型部位を含み、かつ該多型部位の塩基種がそれぞれT、T、G、またはGであることを特徴とする遺伝マーカー
(21)配列番号:21に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、99位または138位の多型部位を含み、かつ該多型部位の塩基種がCまたはGであることを特徴とする遺伝マーカー
(22)配列番号:22に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、75位の多型部位を含み、かつ該多型部位の塩基種がGであることを特徴とする遺伝マーカー
(23)配列番号:23に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、217位の多型部位を含み、かつ該多型部位の塩基種がCであることを特徴とする遺伝マーカー
(24)配列番号:24に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、91位の多型部位を含み、かつ該多型部位が欠失変異(Cの欠失変異)であることを特徴とする遺伝マーカー
(25)配列番号:25に記載の塩基配列の全部もしくは部分配列からなる遺伝マーカーであって、136位の多型部位を含み、かつ該多型部位の塩基種がCであることを特徴とする遺伝マーカー
【0088】
上記遺伝マーカーの長さは特に制限されないが、例えば、300 bp以下であり、好ましくは100 bp以下であり、より好ましくは50 bp以下であり、さらに好ましくは30もしくは20 bp以下である。
【0089】
なお本明細書において引用された全ての先行技術文献は、参照として本明細書に組み入れられる。
【実施例】
【0090】
以下本発明を実施例により具体的に説明するが、本発明はこれら実施例に制限されるものではない。
【0091】
〔実施例1〕 大ヨークシャー種系統豚を用いたQTLのファインマッピング
(1) 徳島県大ヨークシャー種系統豚(AY)の種豚のQTL型の判定
徳島県大ヨークシャー種系統豚(AY)は、1987年に雄10頭、雌65頭を導入し、以後7世代にわたって閉鎖群で育種された。その間の雄、雌の数は8〜11、および28〜36である。その後、雄9頭、雌35頭で維持された。大ヨークシャー種の種雄豚9頭、種雌豚6頭について、それぞれの子豚(18頭から167頭)の椎骨数を測定し、マイクロサテライトマーカーを用いて半きょうだい解析を行った。QTLはマイクロサテライトマーカーSW252をピークとして検出されており(図2)、その近傍のSW263、S0115とともに種豚と子豚すべてをタイピングし、種豚の2本の染色体上のどちらのQTL領域が子豚に伝達されたかを判別した。各種豚について、2つのQTL領域がそれぞれ伝達された子豚グループ間の椎骨数の平均値の差をt検定により検討し、有意な差が見られた場合は種豚のQTLがヘテロ型であるとした。差が見られなかった場合にはZ検定(Nezer C. 2003 Genetics 165: 277-285)により、Z<-2.0においてホモ型であると判定した。その際、椎骨数増大効果はこれまでのF2実験家系の解析における平均値0.43を用いた。またホモ型種豚の子豚の椎骨数を、ヘテロ型種豚の椎骨数増大型対立遺伝子(Q)および野生型対立遺伝子(wt)が伝達した子豚(Q;371頭、wt;383頭)の椎骨数と多重検定(Steel検定)し、Q/Q型またはwt/wt型を判定した(図4)。その結果、9頭においてQ/wtのヘテロ型であると示された。6頭についてはホモ型であり、5頭がQQ型、1頭がwt/wtであることが示された(図4)。
【0092】
(2) QTL領域におけるマイクロサテライトマーカーの開発
第7染色体の椎骨数QTLの95%信頼区間はQTL express (Seaton G. 2002 Bioinformatics 18: 339-340)を用いて解析した結果、マイクロサテライトマーカーSW263とSW632にはさまれた83 cMから88 cMまでの領域となった(図5)。次にこの領域に新規マイクロサテライトマーカーを開発するため、まずヒトゲノム上の遺伝子配列を用いて相同性検索を行い、相同性のあるブタ配列(主にEST配列)を得た。これらについてPCRプライマーを作製し、PCRを用いたスクリーニングシステムにより得られたBACクローン(Suzuki K. 2000 Anim. Genet. 31: 8-12)からマイクロサテライト配列を単離した。単離したマイクロサテライト配列は、SGSC(International Swine Genome Sequecing Consortium、http://piggenome.org/)によって進行中のブタゲノムドラフト配列と比較し、それらマイクロサテライト配列を有するBACクローンのブタゲノム上の位置をFPC(Finger Print Contig)地図により確認した。39の遺伝子に由来する55個のマイクロサテライトマーカーの開発について表5−1〜5−2(第7染色体上の椎骨数QTL領域に開発したマイクロサテライトマーカー)、図6に示した。
【0093】
〔表5−1〕

【0094】
表5−2は表5−1の続きの表である。
〔表5−2〕

【0095】
(3) 徳島県大ヨークシャー種系統豚(AY)の種豚の各染色体上のマイクロサテライトマーカーのハプロタイプの解析
大ヨークシャー種種豚およびそれらの子豚について上記のマイクロサテライトマーカーのジェノタイピングを行い、種豚におけるハプロタイプを決定した。種豚間においてハプロタイプの比較を行い共通祖先由来(同祖)のゲノム領域、それらの染色体上での組み換え位置を解析した。大ヨークシャー種種豚の各染色体上のマイクロサテライトマーカーのハプロタイプと、QTLの対立遺伝子(Qまたはwt)との関連性を解析した結果、SJ7106、SJ7101、J7008の範囲においてのみ、椎骨数増大型対立遺伝子Q、野生型対立遺伝子wtが座位する両方の染色体に、共通ハプロタイプ(同祖ゲノム領域)が存在しなかった(図7)。よって候補領域はその外側のマイクロサテライトマーカーSJ7088とSJ7040との間の領域となった。
【0096】
(4) QTL領域の遺伝子地図の作成と新規マイクロサテライトマーカーの開発
SGSCにより解読が行われた5つのBACクローン、CH242-31K14、CH242-238O9、CH242-92H3、CH242-154M12、CH242-313I6の塩基配列を用い(図8A)、QTL近傍領域の遺伝子地図を作成した(図8B)。またCH242-92H3、CH242-154M12の塩基配列を用い、新規マイクロサテライト配列を検索し、SJ7099、SJ7103、SJ7107、SJ7113、SJ7114、SJ7121、SJ7126、SJ7136、SJ7139の9つのマイクロサテライトマーカーを開発した。
【0097】
(5) 大ヨークシャー種系統豚の種豚を用いたファインマッピング
上記徳島県大ヨークシャー種系統豚(AY)の種豚を用い、新規マイクロサテライトマーカーSJ7099、SJ7103、SJ7107、SJ7113、SJ7114、SJ7121、SJ7126、SJ7136、SJ7139を加えて解析した結果、同祖ゲノム領域を示す共通ハプロタイプが、椎骨数増大型対立遺伝子Qおよび野生型対立遺伝子wtが座位する両方の染色体に存在しない領域はSJ7121からSJ7008の間であり、また椎骨数増大型Qにおいてハプロタイプが保存された領域はSJ7121からSJ7103の範囲であった(図9)。
【0098】
〔実施例2〕F2実験家系親世代ブタを用いたファインマッピング
9つのF2実験家系の親世代ブタについて、椎骨数QTLの遺伝子型が決定され(表2)、既報(Mikawa S. 2005 J Anim. Sci. 83: 2247)において公開されている。徳島県大ヨークシャー種系統豚により絞り込まれたQTL領域近傍のマイクロサテライトマーカーを用いて、F2実験家系親世代ブタについてもハプロタイプ解析を行い、QTLと一致する領域を検索した。その結果、大ヨークシャー種系統豚において椎骨数増大型対立遺伝子Qに関連して見られたハプロタイプは、F2実験家系の親世代ブタにおいても検出され、F2実験家系親世代ブタにおいて、椎骨数増大型対立遺伝子Qが座位する染色体において保存された範囲は、SJ7088からSJ7114であった(図9)。徳島県大ヨークシャー種系統豚(AY)とF2実験家系親世代ブタの両方の解析をあわせると共通する領域はSJ7121からSJ7114となり、その外側のマーカー、SJ7126からSJ7099にQTL領域を絞り込むことに成功した(表6−1〜6−2(マイクロサテライト配列情報))。これらのマイクロサテライトマーカーからなるハプロタイプにより椎骨数QTL型を判定することが可能である。
【0099】
〔表6−1〕

【0100】
表6−2は表6−1の続きの表である。
〔表6−2〕

【0101】
上記表6−1、6−2において、下線がプライマー、斜体文字および波下線がマイクロサテライト配列を示す。
【0102】
〔実施例3〕QTL近傍領域のハプロタイプブロック解析
椎骨数QTL近傍領域の多型検索を行い、NV101、NV102、orf45-i2、orf45-i4、ALDH6A1-i11、LIN52-i4、LIN52-i5、LIN52-e6、NV103、ABCD4-i15、NV038、NV032、NV004、NV015、NV090、NV025、NV035、NV062、NV067、NV071、NV106、NV108、NV109、NV111、NPC2-i2、ISCA2-i3の多型部位を検出した(図10)。これらについて、と場より無作為に選抜した199頭の肉豚(ランドレース種、大ヨークシャー種、およびデュロック種の交雑豚)のDNAを用いてジェノタイピングを行い、Haploview 4.0 (Barrett et al. 2005 Bioinformatics 21: 263-265)により、ハプロタイプブロックを解析した。その結果、ABCD4-i15からNV067までの、約60 kbが同一ハプロタイプブロックとなった(図10)。このハプロタイプブロックのメジャーなハプロタイプは2種類であり、徳島県大ヨークシャー種系統豚(AY)での、椎骨数増大型対立遺伝子Qおよび野生型対立遺伝子wtにおけるハプロタイプに一致した。また肉豚の椎骨数との関連性はABCD4遺伝子の下流に位置する多型(NV004、NV015、NV090、NV025、NV035、NV062)が周辺の多型部位よりも高いことが確認された(図10)。これらの多型はSJ7126からSJ7099の間に位置する(図8C)。と場サンプルでの遺伝子型による椎骨数増大に対する相加効果は0.53であり、これまでに認められていた効果にほぼ等しかった(表7(と場サンプル(西洋品種)における椎骨数の分布))。また椎骨数増大型対立遺伝子の頻度は、55.5%であった。
【0103】
〔表7〕

【0104】
〔実施例4〕椎骨数を支配する遺伝子の同定
SJ7126からSJ7099の間には機能解明された遺伝子はなく、ヒトにおいてLOC55237、マウスではLOC432677と名付けられたhypothetical proteinをコードする遺伝子座が存在した。ブタにおいてもこの領域の転写産物を単離し、それをコードする遺伝子をVertnin (VRTN)と命名し、椎骨数を支配する遺伝子とした(図8C)。VRTN遺伝子は2つのエキソンからなり(図8C)、698アミノ酸をコードする。また相同性検索の結果、類似した遺伝子は存在しなかった。VRTN遺伝子のcDNA配列および該遺伝子によってコードされるアミノ酸配列をそれぞれ配列番号:88、89に示す。
【0105】
〔実施例5〕ブタVRTN遺伝子の多型解析
VRTN遺伝子を含むSJ7126からSJ7099までの領域について、徳島県大ヨークシャー種系統豚の種豚についてPRE1配列等の繰り返し配列を除いたすべての領域で多型解析を行った結果、図8Cおよび表8−1〜8−7(SNP等配列情報)に示した42個の多型がQTL型と一致した。42個の多型パターンは表9(ブタVRTN遺伝子のハプロタイプ型)に示した。この中ではNV064が非同義置換であり、野生型がGAC(Asp)であるのに対し、増大型ではGGC(Gly)に置換している。
【0106】
〔表8−1〕

【0107】
表8−2は表8−1の続きの表である。
〔表8−2〕

【0108】
表8−3は表8−2の続きの表である。
〔表8−3〕

【0109】
表8−4は表8−3の続きの表である。
〔表8−4〕

【0110】
表8−5は表8−4の続きの表である。
〔表8−5〕

【0111】
表8−6は表8−5の続きの表である。
〔表8−6〕

【0112】
表8−7は表8−6の続きの表である。
〔表8−7〕

【0113】
上記表8−1〜8−7において、四角で囲った文字が多型部位を示す。
【0114】
〔表9〕

【0115】
上記表9において、delとは欠失変異(deletion)、insとは挿入変異(insertion)を表す。
【0116】
〔実施例6〕責任遺伝子のブタ胚での発現解析
VRTN遺伝子のプロモーター領域にQTLと一致する多型が存在することから、対立遺伝子間での発現様式の差異がQTLの原因であることも考えられる。そこでブタVRTN遺伝子の椎骨数増大型対立遺伝子と野生型対立遺伝子のプロモーター領域を、ルシフェラーゼをレポーター遺伝子とするプラスミドに連結し、CGR8細胞、P19細胞を用いて、プロモーター活性を比較したが、有意な差は認められなかった。CGR8細胞はマウスES細胞、P19細胞はマウス胚性腫瘍細胞であり、ともに内因性のマウスVRTN遺伝子を発現している。
【0117】
またブタでの内因性VRTN遺伝子の発現様式を解析するため、ブタ胚でのVRTN遺伝子の対立遺伝子による発現の差異を解析した。VRTN遺伝子が増大型Q/Qである種豚と野生型wt/wtである種豚を交配し、ヘテロ型Q/wtの胚を生産した。発現するmRNAはエキソン上に位置するNV062、NV064、MV065の多型により由来する対立遺伝子を区別することができる。交配後、8日、8.5日、10日、12日、14日において子宮より胚を回収し、VRTN遺伝子についてRT-PCRを行い、NV062を含む領域を増幅し、TOPO TAクローニングキット(インビトロジェン)を用いてクローニングを行った。それぞれクローン化した192個のコロニーについて、塩基配列を決定し、NV062の多型パターンの頻度を計測することにより各対立遺伝子の発現量の評価を行った(図11)。その結果、椎骨数増大型対立遺伝子Qと野生型対立遺伝子wtの相対的頻度は交配後10日においてほぼ等しいが、それ以外では椎骨数増大型が多く発現し、12日、14日では野生型の発現は認められなかった(表10(ブタ胚におけるVRTN遺伝子の発現解析))。椎骨数が増大する原因としてVRTN遺伝子の発現時期の変化の関与が示唆された。
【0118】
〔表10〕

【0119】
これらの結果より、ブタにおいてはVRTN遺伝子が椎骨数を支配することが明らかとなった。VRTN遺伝子の多型情報は育種への利用価値が高い。この遺伝子は改良品種内で椎骨数を1対立遺伝子あたり約0.6個増大させる。椎骨数は屠体長と相関することから、VRTN遺伝子の遺伝子診断は、産肉性、繁殖性(産子数、乳頭数)などの遺伝的改良に広く利用される。またこの遺伝子診断を利用した飼養方法の改善が可能である。またVRTN遺伝子の転写産物、タンパク質を利用して、椎骨数およびそれに関連する各種形質を変化させることが可能である。
【特許請求の範囲】
【請求項1】
ブタの椎骨数増大型遺伝形質の有無の判定方法であって、ブタの第7染色体上に存在する下記(A)の(1)〜(25)のいずれかに記載の一もしくは複数の多型マーカーを用いて、下記(B)の工程によって判定する方法。
(A)
(1)配列番号:1に記載の塩基配列の40位
(2)配列番号:2に記載の塩基配列の207位
(3)配列番号:3に記載の塩基配列の68位
(4)配列番号:4に記載の塩基配列の47位または101位
(5)配列番号:5に記載の塩基配列の81位
(6)配列番号:6に記載の塩基配列の355位
(7)配列番号:7に記載の塩基配列の87位
(8)配列番号:8に記載の塩基配列の42位
(9)配列番号:9に記載の塩基配列の152位または185位
(10)配列番号:10に記載の塩基配列の82位
(11)配列番号:11に記載の塩基配列の95〜385位
(12)配列番号:12に記載の塩基配列の49位
(13)配列番号:13に記載の塩基配列の31位、116位、335位、または421位
(14)配列番号:14に記載の塩基配列の127位
(15)配列番号:15に記載の塩基配列の78位、154位、173位、または187位、217位、320位、433位
(16)配列番号:16に記載の塩基配列の111位
(17)配列番号:17に記載の塩基配列の71位または192位
(18)配列番号:18に記載の塩基配列の80〜82位
(19)配列番号:19に記載の塩基配列の285位または300〜301位
(20)配列番号:20に記載の塩基配列の55位、65位、75位、または150位
(21)配列番号:21に記載の塩基配列の99位または138位
(22)配列番号:22に記載の塩基配列の75位
(23)配列番号:23に記載の塩基配列の217位
(24)配列番号:24に記載の塩基配列の91位
(25)配列番号:25に記載の塩基配列の136位
(B)上記(A)のそれぞれの多型マーカーにおける塩基種が記載された下記〔表1〕の判定表に基づき、椎骨数増大型遺伝形質の有無を判定する工程
〔表1〕

【請求項2】
ブタの第7染色体上の、以下の(a)〜(d)のいずれかに記載のマイクロサテライト配列を検出することを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法。
(a)配列番号:26に記載の塩基配列において、180〜195位または196〜216位のマイクロサテライト配列
(b)配列番号:27に記載の塩基配列において、311〜323位、380〜388位、389〜396位、または400〜406位のマイクロサテライト配列
(c)配列番号:28に記載の塩基配列において、393〜413位のマイクロサテライト配列
(d)配列番号:29に記載の塩基配列において、96〜107位または309〜325位のマイクロサテライト配列
【請求項3】
ブタの第7染色体上に存在する前記多型マーカーまたはマイクロサテライト配列を含むDNA領域を増幅し、その増幅産物の多型またはマイクロサテライト配列を検出する工程を含む、請求項1または2に記載の方法。
【請求項4】
ブタの第7染色体上に存在するVertnin遺伝子の発現量を指標とすることを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法。
【請求項1】
ブタの椎骨数増大型遺伝形質の有無の判定方法であって、ブタの第7染色体上に存在する下記(A)の(1)〜(25)のいずれかに記載の一もしくは複数の多型マーカーを用いて、下記(B)の工程によって判定する方法。
(A)
(1)配列番号:1に記載の塩基配列の40位
(2)配列番号:2に記載の塩基配列の207位
(3)配列番号:3に記載の塩基配列の68位
(4)配列番号:4に記載の塩基配列の47位または101位
(5)配列番号:5に記載の塩基配列の81位
(6)配列番号:6に記載の塩基配列の355位
(7)配列番号:7に記載の塩基配列の87位
(8)配列番号:8に記載の塩基配列の42位
(9)配列番号:9に記載の塩基配列の152位または185位
(10)配列番号:10に記載の塩基配列の82位
(11)配列番号:11に記載の塩基配列の95〜385位
(12)配列番号:12に記載の塩基配列の49位
(13)配列番号:13に記載の塩基配列の31位、116位、335位、または421位
(14)配列番号:14に記載の塩基配列の127位
(15)配列番号:15に記載の塩基配列の78位、154位、173位、または187位、217位、320位、433位
(16)配列番号:16に記載の塩基配列の111位
(17)配列番号:17に記載の塩基配列の71位または192位
(18)配列番号:18に記載の塩基配列の80〜82位
(19)配列番号:19に記載の塩基配列の285位または300〜301位
(20)配列番号:20に記載の塩基配列の55位、65位、75位、または150位
(21)配列番号:21に記載の塩基配列の99位または138位
(22)配列番号:22に記載の塩基配列の75位
(23)配列番号:23に記載の塩基配列の217位
(24)配列番号:24に記載の塩基配列の91位
(25)配列番号:25に記載の塩基配列の136位
(B)上記(A)のそれぞれの多型マーカーにおける塩基種が記載された下記〔表1〕の判定表に基づき、椎骨数増大型遺伝形質の有無を判定する工程
〔表1〕

【請求項2】
ブタの第7染色体上の、以下の(a)〜(d)のいずれかに記載のマイクロサテライト配列を検出することを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法。
(a)配列番号:26に記載の塩基配列において、180〜195位または196〜216位のマイクロサテライト配列
(b)配列番号:27に記載の塩基配列において、311〜323位、380〜388位、389〜396位、または400〜406位のマイクロサテライト配列
(c)配列番号:28に記載の塩基配列において、393〜413位のマイクロサテライト配列
(d)配列番号:29に記載の塩基配列において、96〜107位または309〜325位のマイクロサテライト配列
【請求項3】
ブタの第7染色体上に存在する前記多型マーカーまたはマイクロサテライト配列を含むDNA領域を増幅し、その増幅産物の多型またはマイクロサテライト配列を検出する工程を含む、請求項1または2に記載の方法。
【請求項4】
ブタの第7染色体上に存在するVertnin遺伝子の発現量を指標とすることを特徴とする、ブタの椎骨数増大型遺伝形質の有無の判定方法。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【公開番号】特開2011−193825(P2011−193825A)
【公開日】平成23年10月6日(2011.10.6)
【国際特許分類】
【出願番号】特願2010−65451(P2010−65451)
【出願日】平成22年3月23日(2010.3.23)
【国等の委託研究の成果に係る記載事項】(出願人による申告)平成18年度農林水産省「有用遺伝子活用のための植物(イネ)・動物ゲノム研究」(ブタ第7染色体の椎骨数QTLの責任遺伝子の同定および椎骨数と関連する多型の検出)、産業技術力強化法第19条の適用を受ける特許出願
【出願人】(501167644)独立行政法人農業生物資源研究所 (200)
【出願人】(592197108)徳島県 (30)
【出願人】(593027587)社団法人農林水産先端技術産業振興センター (7)
【Fターム(参考)】
【公開日】平成23年10月6日(2011.10.6)
【国際特許分類】
【出願日】平成22年3月23日(2010.3.23)
【国等の委託研究の成果に係る記載事項】(出願人による申告)平成18年度農林水産省「有用遺伝子活用のための植物(イネ)・動物ゲノム研究」(ブタ第7染色体の椎骨数QTLの責任遺伝子の同定および椎骨数と関連する多型の検出)、産業技術力強化法第19条の適用を受ける特許出願
【出願人】(501167644)独立行政法人農業生物資源研究所 (200)
【出願人】(592197108)徳島県 (30)
【出願人】(593027587)社団法人農林水産先端技術産業振興センター (7)
【Fターム(参考)】
[ Back to top ]

