変性低密度リポ蛋白質受容体
【目的】 血管内皮細胞の変性低密度リポ蛋白質受容体をコードするDNA配列を提供する。
【構成】 配列番号1、配列番号2、または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列。
【構成】 配列番号1、配列番号2、または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列。
【発明の詳細な説明】
【0001】
【産業上の利用分野】本発明は、哺乳類のリポ蛋白質の受容体、さらに詳しくは哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体に関する。
【0002】
【従来の技術】動脈硬化の進展の過程では、血管内皮細胞の機能変化が初期の段階で重要であることが指摘されている。血管内皮細胞は種々の液性因子を放出し、循環系のホメオスターシスを保っている。この血管内皮細胞の機能は物理的な刺激や、種々の物質により障害を受けるがこの中でも変性低密度リポ蛋白質の一種である酸化低密度リポ蛋白質が最も重要であると考えられている。例えば、血管のトーヌス(tonus) の調節は血管内皮細胞が血管弛緩因子である一酸化窒素を放出することが重要であるが、この一酸化窒素の放出は酸化低密度リポ蛋白質により障害される。
【0003】一方、変性低密度リポ蛋白質は、低密度リポ蛋白質受容体とは異なる受容体を介してマクロファージや血管内皮細胞に取り込まれることが知られていた。マクロファージでは既に構造が決定されている(特表平6−500765号、同6−508604号、特開平3−290184号各公報記載)スカベンジャー受容体を介して変性低密度リポ蛋白質は取り込まれ、マクロファージは動脈硬化巣で特徴的な泡沫細胞へと変化する。血管内皮細胞にはこのスカベンジャー受容体は発現しておらず、別の構造を持った受容体が存在することが予想されていた(荒井秀典、北徹、酸化LDL、代謝28/4、1991参照)。このような点から血管内皮細胞の変性低密度リポ蛋白質受容体の構造、すなわち、アミノ酸配列の解明が重要であるが、これまで全く構造はわかっていなかった。
【0004】
【発明が解決しようとする課題】本発明の目的は、血管内皮細胞の変性低密度リポ蛋白質受容体の構造を解明し、これにより血管内皮細胞の変性低密度リポ蛋白質受容体をコードするDNA配列を提供することである。また、本発明の目的は、変性低密度リポ蛋白質受容体またはその類似体を生産する方法を提供することでもある。さらに、本発明の目的は、変性低密度リポ蛋白質受容体またはその類似体を含む蛋白質組成物を提供することでもある。
【0005】
【課題を解決するための手段】本発明者の研究により、血管内皮細胞の変性低密度リポ蛋白質受容体の構造が明らかとなった。血管内皮細胞の変性低密度リポ蛋白質受容体は、配列番号1、配列番号2または配列番号3に記載のアミノ酸配列を有している。本発明は、哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列を提供するものである。
【0006】上記DNA配列は、天然の哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体遺伝子のオープンリーディングフレームに由来するcDNAクローンであってもよい。また、上記DNA配列は、上記cDNAクローンとハイブリダイズできて、かつ生物学的活性を有する哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードしているDNA配列であってもよい。さらに、以上述べたようなDNA配列について、遺伝暗号の縮重の結果として、同じ変性低密度リポ蛋白質受容体をコードしているDNA配列であってもよい。従って、本発明は、哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域の配列番号1、配列番号2または配列番号3に示すDNA配列(当該塩基配列を有するDNA)、もしくはそのアナログにある。また本発明は、哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域の配列番号1、配列番号2または配列番号3に示すDNA配列を有するcDNAクローン、もしくはそのアナログにある。また、本発明は、42℃、20%(v/v)ホルムアミド水溶液中で、配列番号1、配列番号2または配列番号3に示すDNA配列のcDNAクローンとハイブリダイズでき、かつ哺乳類の血管内皮細胞の変性低密度リポ蛋白質と結合する機能を有する蛋白質(即ち受容体)をコードしているDNA配列(当該塩基配列を有するDNA)にもある。そしてまた、本発明は、配列番号1、配列番号2または配列番号3に示すDNA配列と遺伝的コードの結果として縮重していて、かつ哺乳類の血管内皮細胞の変性低密度リポ蛋白質と結合する機能を有する蛋白質(即ち受容体)をコードしているDNA配列(当該塩基配列を有するDNA)にもある。さらに本発明は、配列番号1、配列番号2または配列番号3に示す塩基配列を有するDNAもしくはそのアナログに対応する変性低密度リポ蛋白質受容体(あるいは配列番号1、配列番号2または配列番号3に示すアミノ酸配列もしくはそのアナログを含む変性低密度リポ蛋白質受容体)の抗体にもある。
【0007】本発明が提供するDNA配列は発現ベクターに組込んで用いることができる。これにより、本発明は、この組換え発現ベクターを宿主細胞に挿入し、宿主細胞を発現を引き起こす条件下で培養することからなる哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を生産する方法も提供する。さらに、本発明は、以上のように生産された生物学的活性を有する哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を含む蛋白質組成物も提供する。このように生産された哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体、その類似体およびそれらを含む蛋白質組成物は、哺乳類の変性低密度リポ蛋白質のアッセイに有用であり、また診断で用いる変性低密度リポ蛋白質受容体に対する抗体を調製する上でも有用である。また、本発明の薬剤が動脈硬化症の疾患の診断に有効であることは、前述した変性低密度リポ蛋白質およびその受容体の生物学的活性から明らかである。
【0008】本明細書において、「変性低密度リポ蛋白質受容体」とは、変性低密度リポ蛋白質に結合することができ、哺乳類の血管内皮細胞の細胞膜蛋白質と同様な本来の立体配置をとる際に、変性低密度リポ蛋白質分子によって与えられる情報を血管内皮細胞内に伝達する役割を持っている蛋白質を意味する。本明細書において用いる場合、この用語は、変性低密度リポ蛋白質に結合するか、あるいは情報伝達活性を有する天然蛋白質の類似物を含んでいる。「変性低密度リポ蛋白質受容体のサブタイプ」とは、変性低密度リポ蛋白質受容体のうちで酸化変性低密度リポ蛋白質あるいはアセチル化変性低密度リポ蛋白質等の各種変性低密度リポ蛋白質に対して、異なる選択性、すなわち薬理学的親和性序列を示す各々の分子をいう。
【0009】本明細書において、DNA配列において用いられる「コードする領域から実質的になる配列」等における『実質的に』の表現は、特定の対象となる配列、例えば変位配列で、本来の配列番号1、配列番号2または配列番号3に示す参照配列の1つ以上の置換、欠失あるいは付加によって変化したものであって、その全体としての作用が、参照配列と比較して不利な機能的相違をもたらさない配列も含むとの趣旨で用いられる。具体的には後述する生物学的活性を有する、さらに具体的には、変性低密度リポ蛋白質と結合可能である範囲内で配列の変化が可能である。そのためには、変性低密度リポ蛋白質と結合する部分をコードする領域については、遺伝暗号の縮重を除き、配列番号1、配列番号2または配列番号3に示す参照配列と同じか、類似のアミノ酸への置換をもたらす程度の変更を加えた配列を有する必要がある。一方、上記以外の部分をコードする領域については、30%以上(好ましくは50%以上、さらに好ましくは80%以上)の相似性があればよい。
【0010】なお、上記の類似のアミノ酸への置換とは、天然のアミノ酸を下記の8群に分類する場合において、各群内におけるアミノ酸相互の置換を意味する。
(1)モノアミノモノカルボン酸Gly Ala Val Leu Ile(2)オキシアミノ酸Ser Thr(3)含硫アミノ酸Cys Met(4)モノアミノジカルボン酸Asp Glu(5)ジアミノモノカルボン酸Lys Arg(6)芳香族環を有するアミノ酸Phe Tyr(7)複素環を有するアミノ酸His Try Pro(8)アミド基を有するアミノ酸Asn Gln類似性を決定する目的においては、参照配列の短縮あるいは内在的欠失は無視してよい。配列の相似性が低い場合でも、同程度の生物学的活性を有し、同等の発現特性を有する配列は、実質的に同等な配列とみなす。
【0011】「生物学的に活性を有する」という用語は、変性低密度リポ蛋白質受容体の特性として用いられる場合、特定の分子が、変性低密度リポ蛋白質を結合可能であることが明らかにされている本発明の態様と充分なアミノ酸配列の類似性を有するか、あるいは混成受容体構成物の構成要素として細胞に変性低密度リポ蛋白質の刺激を伝達するために充分なアミノ酸配列の類似性を変性低密度リポ蛋白質受容体に対して有していることを意味する。変性低密度リポ蛋白質を結合可能であるとは、具体的には、基準とする酸化変性低密度リポ蛋白質に対する親和性(解離定数)が1マイクロモル以下であることを意味する。本発明においては、その親和性が0.1マイクロモル以下であることが好ましく、0.01マイクロモル以下であることがさらに好ましい。生物学的活性は、変性低密度リポ蛋白質の結合に際して、血管内皮細胞内へ変性低密度リポ蛋白質の取り込みを促すことができることを意味する場合もある。
【0012】「DNA配列」は、大きなDNA構成物から分離した断片あるいは構成要素として存在するDNAポリマーを示しており、それらは少なくとも一度は実質的に純粋な形に単離されたDNAを起源とする。すなわち、内在性物質の混入がない形で単離され、かつ同定、操作および標準的な生化学的方法、例えば、クローニングベクターを用いて、配列およびその構成要素である塩基配列が回収可能な程度の量あるいは濃度で存在する。こうした配列は、真核細胞遺伝子に典型的な内在性非翻訳配列(イントロン)によって中断されていないオープンリーディングフレームを持つ形で供給されることが好ましい。しかし、関連した配列を含んだ染色体DNAも用いられることは明らかである。非翻訳DNAは、オープンリーディングフレームの5’あるいは3’にあり、その場合、非翻訳配列はコード領域の操作あるいは発現の妨げとはならない。「組換え発現ベクター」は(a)遺伝子発現における調節の役割を持つ要素、例えばプロモーター或いはエンハンサーのような単独または複数の遺伝的要素、(b)mRNAに転写され、蛋白質に翻訳される構造あるいはコード配列、及び(c)適当な転写及び翻訳開始及び終止配列の集合を含む転写単位からなるプラスミドを示す。酵母の発現系で用いるための構造要素は、翻訳された蛋白質が宿主細胞によって細胞外に分泌されるためのリーダー配列を含んでいることが好ましい。組換え体蛋白質がリーダーあるいは輸送配列を持たずに発現された場合、その蛋白質はN−末端にメチオニン残基を含んでいる可能性がある。続いて、その残基は発現された組換え蛋白質から随意に切断され、最終産物を与える。
【0013】次に、変性低密度リポ蛋白質受容体をコードするcDNAの単離、およびDNA配列の決定について説明する。培養ウシ大動脈内皮細胞から抽出したPoly (A)+ RNAの逆転写によって調製されたcDNAライブラリーから、ウシ変性低密度リポ蛋白質受容体をコードするDNA配列が単離された。このライブラリーは、SV40、I型ヒトT細胞白血病ウイルス由来の調節配列を持った哺乳類発現べクター(pME18S)を用いて、サルのCOS−7細胞に蓄積されたDNA断片よりmRNAを直接発現させることによってスクリーニングした。形質導入されたCOS−7細胞をDiI(1,1'-di-octadecyl-3,3,3',3'-tetramethylindocarbocyanine perchlorate )−酸化低密度リポ蛋白質を含む培地とともにインキュベートし、結合しなかったDiI−酸化低密度リポ蛋白質を除くために洗浄した後、トリプシン処理により細胞を浮遊状態にしてFACS(fluorescence-activated cell sorter)にかけ、DiIの蛍光を測定するとともに蛍光強度の高い細胞を回収した。形質導入細胞からプラスミドを抽出し、大腸菌を形質転換してプラスミドを精製し、再度上記の操作を繰り返した。このようにして4回この操作を繰り返すことにより、単一のクローンで変性低密度リポ蛋白質結合活性を持った表面蛋白質を合成することができた。このクローンは単離され、ウシの変性低密度リポ蛋白質受容体のcDNA配列を決定するために挿入断片の配列が調べられた。
【0014】単離されたcDNAクローンをCOS−7細胞に導入し発現させたところ、この細胞は酸化低密度リポ蛋白質および酸化低密度リポ蛋白質に対する特異的結合活性を獲得し、これらの変性低密度リポ蛋白質は細胞内へと取り込まれた。さらにこのとき未変性の低密度リポ蛋白質に対する結合活性は見られず、変性した低密度リポ蛋白質に特異的な受容体と考えられる。
【0015】以上のように決定された変性低密度リポ蛋白質受容体をコードするDNA配列および相当するアミノ酸配列を配列番号1および配列番号2に示す。DNA配列およびアミノ酸配列について説明する。上記配列番号1及び2に示されるように、変性低密度リポ蛋白質受容体には少なくとも2種類のサブタイプが存在している。配列番号2は、配列番号1の24番目のアミノ酸(Gly) の後に三アミノ酸(Thr Thr Gly) が挿入している以外は、配列番号1と同様である。本明細書において特に断らない場合は、配列番号1を基準に説明する。最初のATG(メチオニンをコードする開始コドン)からTGA(811−813)のストップコドンまで810bpのオープンリーディングフレームが存在し、270アミノ酸残基がコードされている。このcDNAがコードしている変性低密度リポ蛋白質受容体mRNAには過渡的に発現されるサイトカインや成長因子と同様に3’非翻訳領域にAUUUAというmRNAを不安定化する配列が7ヶ所存在する。このポリペプチドには、一ヶ所において26個の疎水性アミノ酸(配列番号1におけるアミノ酸番号31〜56、配列番号2におけるアミノ酸番号34〜59)からなる膜貫通領域と思われる部分があり、膜貫通部分のC末端側に4つの糖鎖付加部位(配列番号1におけるアミノ酸番号69、135、179および208、配列番号2におけるアミノ酸番号72、138、182および211)が見出された。
【0016】次に、ヒト肺より抽出したPoly(A)+RNAの逆転写によって調製されたcDNAライブラリーから、ヒトの変性低密度リポ蛋白質受容体をコードするDNA配列が単離された。このライブラリーのスクリーニングは、ランダムプライマーを用いて[a-32P]dCTP により標識したpBMLR1のXhoI/PstI 切断断片とのプラークハイブリダイゼーション法により行った。ハイブリダイゼーションは50mM Tris-HCl(pH 7.5), 1M NaCl, 1% SDS, 0.2g/l Yeast tRNA 中で55℃で行い、55℃の2xSSC/0.1% SDS中で15分間3回洗浄した後、オートラジオグラフィーにより陽性クローンを同定した。このクローンは単離され、ヒトの変性低密度リポ蛋白質受容体のcDNA配列を決定するために挿入断片の配列が調べられた。この配列は蛋白質をコードする領域の一部のみを含むと考えられた。そこで、ヒト胎盤より抽出したPoly(A)+RNAの逆転写によって調製されたcDNAより、得られた配列を元に5’−RACE(rapid amplification of cDNA end) 法を用いて、蛋白質全体をコードする領域のcDNAを得て、配列が調べられた。配列番号3がヒトの変性低密度リポ蛋白質受容体のDNA配列およびアミノ酸配列である。ヒトの配列は、ウシの配列と同様に、最初のATG(メチオニンをコードする開始コドン)からTGA(811−813)まで、810bpのオープンリーディングフレームが存在し、270アミノ酸残基がコードされている。このcDNAがコードするポリペプチドには、ウシの配列と同様に一ヶ所において27個の疎水性アミノ酸からなる膜貫通領域と思われる部分があり、膜貫通部分のC末端側に4つの糖鎖付加部位(アミノ酸番号69、135、179および206)が見出された。ウシおよびヒトのアミノ酸配列は、共にC型レクチンの細胞外ドメインに持っており、この部分(アミノ酸番号140−270)が変性LDLとの結合に重要と考えられる。従って、アミノ酸番号140−270の部分配列を有するペプチドも、変性LDLとの結合機能を有すると推定できる。
【0017】本発明は、以上述べた変性低密度リポ蛋白質受容体をコードするDNA配列、および上記アミノ酸番号140−270の部分配列をコードするDNA配列を提供する。このDNA配列は、哺乳類、微生物による調節を受ける組換え体転写単位、またはウイルスの転写或いは翻訳調節要素内で発現され得る形で提供されることが好ましい。例えば、微生物内で発現される配列は、イントロンを含まないものである。好ましい態様では、本DNA配列は少なくとも1つ、しかし随意に1つ以上のcDNA配列或いはそれについての複製由来の配列成分からなる。このような配列は、合成オリゴヌクレオチドを組み上げることにより調製されたDNA配列に連結しているか、あるいは隣接している。しかし、主にオリゴヌクレオチドから組み上げられた合成遺伝子は、ここで与えられる配列情報を用いることにより構築することができる。典型的な配列は、前述したものと実質的に同一な配列を含んでいる。コード配列は、N−末端に位置する、例えばヌクレオチド配列上で翻訳フレームと連結したメチオニンを特定するN−末端のATGコドンのような、1つ以上の付加的アミノ酸をコードするコドンを含むことができる。遺伝子コードの縮重のため、同じアミノ酸配列をコードするヌクレオチド配列にも相当の多様性がある。前述した典型的なDNA配列以外の態様には、中等度にストリンジェントな条件(すなわち、42℃、20%(v/v)ホルムアミド存在下)で、典型的なDNA配列にハイブリダイズすることができる配列が含まれる。その他の配列は生物学的な活性を有する変性低密度リポ蛋白質受容体ペプチドをコードする上記の配列に縮重している。
【0018】以上の配列は、微生物もしくはウイルスのオペロン由来の誘導可能な制御要素を含む組み換え転写単位中で発現させることが可能である。本発明はまた、有用な量の精製された変性低密度リポ蛋白質受容体を生産するための発現ベクターを提供する。本発明のベクターは、哺乳類、微生物、酵母、バクテリオファージあるいはウイルス遺伝子由来の調節要素に使用可能な状態に連結された哺乳類の変性低密度リポ蛋白質受容体、あるいは生物学的に等価な類似物をコードする合成あるいはcDNA由来のDNA断片からなることができる。使用できる調節要素は以下に詳細に述べる。適当な細胞系列への形質転換、形質導入あるいは感染によって、このようなベクターは組換え体蛋白質の発現を誘導することができる。哺乳類の変性低密度リポ蛋白質受容体は、適当なプロモーターの調節の下、哺乳類細胞、酵母、細菌、あるいはその他の細胞中で発現することができる。無細胞翻訳系によっても、本発明のDNA構成物由来のmRNAを用いて哺乳類の変性低密度リポ蛋白質受容体を生産することができる。細菌、真菌、酵母および哺乳類細胞宿主に用いるための適当なクローニングおよび発現ベクターは、ポウエル(Pouwel)ら(Cloning Vectors: A Laboratory Manual, エルスビュー社、ニューヨーク州、(1985))によって述べられている。
【0019】種々の哺乳類細胞培養系を組換え体蛋白質の発現に用いることができる。適当な哺乳類宿主細胞系列としては、グルツマン(Gluzman) (Cell, 23, 175 (1981)) により述べられたサル腎臓細胞のCOS−7系列および、例えばC127、3T3、CHO、HeLaおよびBHK細胞系列などのベクターを発現可能な細胞系列が含まれる。哺乳類発現ベクターは、複製開始点、適当なプロモーターおよびエンハンサーのような非転写要素、5’あるいは3’に隣接する非転写配列、また必須のリボソーム結合部位、ポリアデニル化部位、スプライスドナー(splice donor)およびアクセプター部位のような5’或い3’の非翻訳配列、および終止配列等を含んでいる。例えば、SV40複製開始点、初期プロモーター、エンハンサー、スプライスおよびポリアデニル化部位等のSV40ウイルスゲノム由来のDNA配列は、異種DNA配列の発現に必要とされるその他の遺伝的要素を与えるために用いられる。典型的なベクターは、岡山とバーグ(Berg)(Mol.Cell. Biol., 3, 280 (1983)) によって明らかにされた通りに構築することができる。
【0020】C127ネズミ乳腺表皮細胞で、哺乳類受容体cDNAを安定かつ高レベルに発現するために有用な系は、コスマン(Cosman)ら(Molecular Immunol., 23,935 (1986)) により述べられていることに従って構築することができる。酵母の系では、サッカロミセス・セレビシエのようなサッカロミセス属の種を用いることが好ましい。ピキア(Pichia)あるいはクリュベロミセス(Kluyveromyces )等の、他の属の酵母も、組換え体蛋白質の生産株として用いることができる。一般に、有用な酵母ベクターは複製開始点と、例えば大腸菌のアンピシリン耐性Ampr 遺伝子とサッカロミセス・セレビシエのTRP1遺伝子のような、酵母と大腸菌の両方で形質転換に用いることが可能な選択マーカー及び下流の構造遺伝子の転写を誘導する酵母の高発現遺伝子由来のプロモーターを含んでいる。このようなプロモーターは3−ホスホグリセリン酸リン酸化酵素で、α−因子、酸性ホスファターゼおよび熱ショック蛋白質等、高率で発現される遺伝子をコードする酵母の転写単位に由来する。この異種構造配列は、翻訳開始および終止配列並びに好ましくは、翻訳された蛋白質を細胞外の培地中へ分泌することを可能にするリーダー配列とともに適当なフレームに組み上げられる。上記の異質配列は、N−末端の同定可能なペプチドまたは、例えば発現された組換え体産物の安定化を行う、あるいは精製が単純になるような所望の特徴を与える配列を含む融合蛋白質をコードすることも任意に可能である。
【0021】有用な酵母ベクターは、大腸菌内での選択および複製のためにpBR322由来のDNA配列(Ampr 遺伝子と複製開始点)およびグルコースによって抑制されるアルコール脱水素酵素2(ADH2)プロモーターを含む酵母のDNA配列を用いることにより組み上げることができる。ADH2プロモーターはルッセル(Russell )ら(J. Biol. Chem., 258, 2674 (1982)) とベイヤー(Beier )ら(Nature, 300, 724 (1982))によって開示されている。これらのベクターはまた、酵母TRP1遺伝子を選択マーカーとして持ち、酵母2μ複製開始点を含んでいる。酵母リーダー配列、例えば酵母宿主からの異種蛋白質の分泌を導くα−因子のリーダーは、プロモーターと発現される構造遺伝子との間に挿入することができる(U.S. Patent No. 4,546,082 ;Kurianet al., Cell, 30, 933 (1982);およびBittner et al., Proc. Natl. Acad. Sci. USA 81, 983(1984))。リーダー配列はその3’末端付近に、外来の遺伝子とそのリーダー配列の融合を促進するための、一つ以上の有用な制限酵素切断部位を含むように修飾することができる。
【0022】適当な酵母の形質転換法は当業者にはよく知られている;典型的な技法は、ヒネン(Hinnen)ら(Proc. Natl. Acad. Sci. USA 75:1978)によって述べられており、トリプトファン陽性となった形質転換体を、0.67%酵母窒素源、0.5%カザアミノ酸、2%ブドウ糖、10μg/mlアデニン、20μg/mlウラシルからなる選択培地上で選択する。ADH2プロモーターからなるベクターによって形質転換された宿主株は、1%酵母抽出物、2%ペプトン、1%ブドウ糖、80μg/mlアデニン、80μg/mlウラシルを含む栄養豊富な培地で発現のために生育させる。ADH2プロモーターの脱制御は培地中のブドウ糖が消費されてしまったときに起きる。粗酵母上清は濾過によって集められ、続いての精製の前に4℃に保つ。
【0023】細菌で使用する有用な発現ベクターは、哺乳類の変性低密度リポ蛋白質受容体をコードするDNA配列を適当な翻訳開始および終止信号とともに機能を持つプロモーターに読み枠を合わせて挿入することによって構築される。本ベクターは、一つ以上の表現型の選択マーカーと宿主内での増殖を保証する複製開始点を含んでいる。形質転換に適した原核生物宿主には、大腸菌、枯草菌(Bacillus subtilis) 、サルモネラ・ティフィムリウム(Salmonella typhimurium)、およびシュードモナス(Pseudomonas) 属に含まれる様々な種ストレプトマイセス(Streptomyces)、スタフィロコッカス(Staphylococcus)などが含まれるが、他のものも選択の対象として取ることもできる。発現ベクターは、成熟蛋白質のN−末端残基をコードするコドンに近い部位でcDNAを切断することによって都合良く構築される。次いで、コード領域の欠失した部分を「埋め合わせる」ため、或いは発現ベクター中の適当な読み枠や開始メチオニンを特定するコドンとなるようにコード断片を連結するための連結配列を提供するために合成オリゴヌクレオチドを用いることも随時可能である。
【0024】制限的な例としてではなく典型的なものとしては、細菌で用いる有用な発現ベクターは、良く知られたクローニングベクターであるpBR322(ATCC37017)の遺伝的要素からなる市販のプラスミド由来の選択マーカーおよび細菌の複製開始点を含むことができる。こうした市販のベクターは、例えばpKK223−3p(ファルマシア・ファインケミカルズ、ウプサラ、スウェーデン)およびpGEM1(プロジェマ・バイオテック、マジソン、ワイオミング州、米国)などを含んでいる。これらのpBR322の「主鎖」部分は、適当なプロモーターおよび発現される構造配列と結合している。特に有用な細菌の発現系は、λファージのPL プロモーターとc1857熱不安定なリプレッサーを用いている。アメリカン・タイプ・カルチャー・コレクションから得られるλPL 由来のプロモーターを取り入れたプラスミドベクターには、大腸菌株JMB9(ATCC37092)に内在した形のpHUB2プラスミドと大腸菌株RR1(ATCC53082)に内在した形のpPLc28がある。大腸菌内での発現に有用なその他のプロモーターとしては、スツデェー(Studier) ら(J. Mol. Biol. 189:113,1986)によって述べられているT7RNAポリメラーゼのプロモーター、ラウェー(Lauer)(J. Appl. Genet. 1:139-147, 1981)によって記載されておりATCC37121として得られるlacZプロモーター及びマニアチス(Maniatis)(Molecular Coloning;A Laboratory Manual, ColdSpring Harbor Laboratory, 1982.p.412) によって述べられているATCC37138として得られるtacプロモーターがある。
【0025】適当な宿主株を形質転換し適当な細胞濃度までその宿主株を増殖させた後、選択されたプロモーターを適当な方法(例えば、温度シフトあるいは化学的誘導)で脱抑制し、細菌を更に短期間培養する。細胞は普通遠心により集め、物理的あるいは化学的方法で破砕し、その結果生じた粗抽出液をその後の精製のために残しておく。例えば、細胞を最大の通気と激しい撹拌を行なう条件下で、10リットルの培養器中で増殖させる。消泡剤(Antifoam A)を用いるのが好ましい。培養はモット(Mott)ら(Proc. Natl. Acad. Sci. USA 82:88, 1985)によって開示された超誘導培地中30℃で行なうか、あるいはそれに代るものとしては、抗生物質を含んだ培地で培養し、A600 が0.4−0.5に相当する細胞濃度で温度を42℃にあげることによって脱抑制し、次いで温度シフト後、3−6時間が好ましいが、2−20時間の間に細胞を集める。細胞は、初め濾過あるいはその他の方法で濃縮し、次に10.000xgで10分間、4℃で遠心後、直ちに細胞のペレットを凍結する。好ましくは、精製された哺乳類の変性低密度リポ蛋白質受容体あるいは生物学的に等価な類似体は、培養液から精製される本発明の合成遺伝子の組換え体翻訳産物を発現するために適した宿主/ベクター系を培養することによって調製される。
【0026】精製された変性低密度リポ蛋白質受容体を生産する別の方法は、細胞培養上清あるいは抽出液からの精製を含んでいる。この方法では、有用な量の蛋白質を作る細胞系列を用いる。そのような細胞系列からの上清は、例えばアミコン社あるいはミリポア・ファルコン社の限外濾過装置と言った市販の蛋白質濃縮濾紙を用いて随時濃縮することができる。濃縮操作に続き、濃縮液を既に述べたような適当な精製用マトリックスにかける。例えば、適当なアフィニティーマトリックスは、適当な支持体に結合した変性低密度リポ蛋白質受容体あるいはレクチンまたは抗体分子などから成っている。それに代るものとして、例えばジエチルアミノエチル(DEAE)基を持つ担体あるいはマトリックスのような陰イオン交換樹脂が用いられる。マトリックスとしては、アクリルアミド、アガロース、デキストラン、セルロースあるいはその他の蛋白質精製に通常用いられているようなものが使用可能である。また、その代りとして、陽イオン交換樹脂を用いることも可能である。適当な陽イオン交換体としては、スルフォプロピル基あるいはカルボキシメチル基からなる種々の不溶性マトリックスを含んでいるスルフォプロピル基が好ましい。最後に、メチルあるいはその他の脂肪族基を有するシリカゲルなどの疎水性充填剤RP−HPLCを用いた一種以上の逆相系高速液体クロマトグラフィーRP−HPLCを、変性低密度リポ蛋白質受容体組成物を更に精製するために用いることが可能である。上述の精製段階のいくつかあるいは全てを様々に組み合わせることにより、均一な組換え体蛋白質を得ることが可能だろう。
【0027】細菌の培養で生産された組換え体蛋白質は通常細胞ペレットからの抽出を始めとして、一工程以上の濃縮、塩析、水相のイオン交換あるいはゲル濾過クロマトフラフィーの段階を経て単離される。最後に、高速液体クロマログラフィー(HPLC)を最終精製段階として用いることができる。組換え体哺乳類変性低密度リポ蛋白質受容体の発現に用いられた微生物細胞は、凍結−融解の繰り返し、超音波処理、物理的破砕、あるいは細胞溶解試薬を用いるなど、どのような都合の良い方法によっても破壊することができる。哺乳類の変性低密度リポ蛋白質受容体を分泌タンパクとして発現する酵母の発酵は精製を大いに容易にしている。大規模な発酵の結果得られる分泌された組換え体蛋白質はウルダル(Urdal) ら(J. Chromatog. 296:171,1984)によって明らかにされた方法と類似した方法によって精製することが可能である。この引用文献には、組換え体ヒトGM−CSFの精製のための調製用HPLCカラムにおける、二段階の連続した逆相系HPLC処理が述べられている。
【0028】種々の態様において、本発明は内在性物質の混入無しに、実質的に均一で、本来のグリコシル化様式を伴なったあるいは伴なわない組換え体哺乳類変性低密度リポ蛋白質受容体ポリペプチドを提供している。本発明における組換え体である変性低密度リポ蛋白質受容体の蛋白質には、微生物中での発現あるいは微生物によって発現された蛋白質の精製を助けるような適当なペプチドあるいは蛋白質配列と融合された蛋白質として発現した哺乳類の変性低密度リポ蛋白質受容体も本発明により意図されているものである。本発明の蛋白質の生物学的に等価な類似体とは、生物学的活性に不要な末端、あるいは細胞内側の残基または配列が削られた変性低密度リポ蛋白質受容体のような短縮された種々の類似体を含んでいる。ここで用いられている「変異アミノ酸配列」とは、天然の配列から故意に作られた変種のヌクレオチド配列によってコードされるポリペプチドを示している。「変異蛋白質」あるいは「類似体」は、変異アミノ酸配列を含む蛋白質を意味している。「天然の配列」は野生型あるいは天然の形の遺伝子または蛋白質と同一なアミノ酸配列あるいはヌクレオチド配列を示している。
【0029】変性低密度リポ蛋白質受容体の蛋白質を適当なプロテアーゼで切断するか、あるいは、変性低密度リポ蛋白質受容体の一部を組み替えDNAの方法により大腸菌や哺乳動物等に発現させて、可溶化したペプチド断片を得てもよい。そのようなペプチド断片も、前述した定義を有する限り、すなわち、変性低密度リポ蛋白質と結合可能である限り、本発明の範囲に含まれる。受容体を可溶化することにより、可溶化したペプチド断片を変性低密度リポ蛋白質に結合させて、リポ蛋白質を不活性化することが可能になる。すなわち、ペプチド断片を、変性低密度リポ蛋白質を原因とする疾患の治療に用いることが可能である。
【0030】
【実施例】以下、実施例について説明する。以下の実施例において、ウシ大動脈内皮細胞の変性低密度リポ蛋白質受容体のアミノ酸配列およびそれをコードするDNA配列を解明した。このようにして、配列番号1または配列番号2に示される配列が解明された結果、ヒトの哺乳類の内皮細胞の変性低密度リポ蛋白質受容体のアミノ酸配列およびそれをコードするDNA配列(すなわち配列番号3)の解明は、容易に実施することができた。他の哺乳類の内皮細胞の変性低密度リポ蛋白質受容体のアミノ酸配列およびそれをコードするDNA配列の解明も、同様に容易に実施できる。具体的には、他の哺乳類の内皮細胞あるいは適当な臓器より抽出したpoly(A)+ RNAから、配列番号1、配列番号2または配列番号3に示されるDNA配列とハイブリダイズできるものを選択することにより、他の哺乳類の内皮細胞の変性低密度リポ蛋白質受容体をコードするDNAを得ることができる。これは、下記実施例1のように、手掛かりとなる配列が存在していない状況から該当するDNAを選択する場合と異なり、当業者であれば実施が容易である。また、得られた配列の分析も、配列番号1、配列番号2または配列番号3に示されるDNA配列を参照することにより、下記実施例1の場合(参照すべき配列がない状況)よりも、容易に実施できる。なお、以下の実施例では、酸化低密度リポ蛋白質結合に関してアッセイを実施したが、アセチル化低密度リポ蛋白結合に関するアッセイも同様に実施できる。
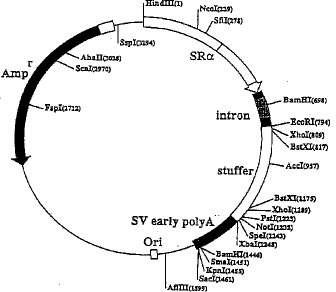
【0031】[実施例1]Chomczynski ら(Biotechniques 15, 532 (1993))と同様の手法を用いて培養ウシ大動脈内皮細胞より抽出した全RNAから単離したPoly(A)+ の付加したmRNAを逆転写することによりcDNAライブラリーを作製した。具体的には、細胞を酸性イソチアン酸グアニジウム/フェノール溶液中で溶かし、溶解物にクロロホルムを加え、水層と有機層を遠心分離した。水層を回収し、アルコール沈澱によりさらに精製した。オリゴdTセルロースクロマトグラフィーによりpoly(A)+ RNAを単離し、二本鎖cDNAは、グブラー(Gubler)とホフマン(Hoffman)(Gene, 25, 263 (1983) )と同様の方法で調製した。具体的には、RNAはオリゴdTまたはランダムオリゴヌクレオチドをプライマーとして用いて逆転写酵素により、cDNAに複製した。cDNAを大腸菌DNAポリメラーゼIとRNaseHと共にインキュベーションすることにより二本鎖にし、その末端はさらにT4 DNAポリメラーゼとインキュベーションすることにより平滑化した。平滑末端を持つこれらのcDNAに、BstXIリンカーを付加した後、セファクリル(sephacryl) S−500HRによるゲル濾過クロマトグラフィーにより短いものを排除した。そして哺乳動物細胞用高発現プラスミドベクターpME18Sに結合させた。このpME18Sの模式図を図1(東京医科歯科大学、丸山博士より入手)に示す。
【0032】pME18SベクターはSV40の複製起点、SV40/ヒトT細胞白血病ウイルスタイプ1プロモーターを含んで創製された3.4kbのプラスミドベクターである。以上のようにしてできたpME18S上のウシ大動脈cDNAライブラリーを大腸菌(E. coli. ElectroMax DH 10B)を形質転換するのに用い、約7×105 のコロニーを得た。これらの組換え体を500mlの2xYT中で37℃にて振とう培養し、プラスミドDNAをCsCl密度勾配遠心にて調製した。Lipofectamine を用いてサルのCOS−7細胞の集密的になっていない(サブコンフルエント)細胞単層にトランスフェクトした。次に導入された配列が過渡的な発現をできるように細胞を3日間培養し、各プレートの細胞の単層を以下のように酸化低密度リポ蛋白質結合に関してアッセイした。
【0033】プレートに、15μgのDiI標識酸化低密度リポ蛋白質を含む10%牛胎児血清(FBS)含有DMEM培地5mlを加え、37℃、5%CO2 下で12時間インキュベートした。次にこの培地を除き、PBS(pH7.4)で2回洗浄後、トリプシン処理により細胞をプレートからはがした。この細胞をFACSにかけ、DiIの蛍光を測定するとともに、蛍光強度の高い細胞を回収した。回収した細胞からプラスミドを抽出し、上記の操作を4回繰り返した。このようにしてライブラリーから約7×105 の組換え体をスクリーンした後、酸化低密度リポ蛋白質受容体の発現を誘導することのできる単一のクローンpBLOX−1が導入されたCOS−7組換え体を得た。図2は、pBLOX−1を導入したCOS−7細胞および導入していない対照のCOS−7細胞のFACSによる蛍光強度分布を示すグラフである。クローンpBLOX−1の挿入断片はBluescriptII SK-プラスミドにサブクローンし、ダイデオキシ法によってDNA配列を決定した(Sanger et al., Proc.Natl. Acad. Sci. USA, 74, 5463(1977))。
【0034】[実施例2]
受容体cDNAを使用したノーザンブロットによる各組織での発現状態前記のような方法でウシの各組織からpoly(A)+RNAを抽出し、それぞれ5μgずつをホルムアルデヒド/1.1%アガロースゲル電気泳動で分離した後、ジーンスクリーンプラス膜(NEN, DuPont) に転写した。1.8kbのcDNA断片をランダムプライム法によってα−32P−dCTPで8×108 c.p.m./mgになるようにラベルしプローブとして用いた。ハイブリダイゼーションは1M塩化ナトリウム/1%SDS、250μg/mlサケ精子DNA溶液中で60℃で行ない、2×SSC/1%SDSにより洗浄した。オートラジオグラフィーは8時間行なった。11のウシ組織から抽出したpoly(A)+RNAに対してノーザンブロットを行なったところ、多量の発現がみられたのは培養内皮細胞と肺であった。
【0035】[実施例3]
CHO−K1細胞による変性低密度リポ蛋白質受容体の発現変性低密度リポ蛋白質受容体cDNA挿入断片を、bsr(blasticidin S-resistance)遺伝子とSV40ウイルス由来のプロモーターを含む発現ベクターpSV2bsrと変性低密度リポ蛋白質受容体発現プラスミドpBLOX−1を用いて、変性低密度リポ蛋白質受容体発現細胞を作成した。CHO(chinese hamster ovary )K1細胞は、10%FBS含有HamF12培地で、サブコンフルエント(subconfluent)に単層培養した。変性低密度リポ蛋白質受容体発現プラスミドpBLOX−1とpSV2bsrをLipofectamine により、CHO−K1細胞にトランスフェクトした。トランスフェクトした細胞を24時間後、約10倍の培養面積にまき直し、さらに24時間待って細胞が充分接着した後、培地を5μgのblasticidin S 含有上記培地に替えた。
【0036】これによって、トランスフェクションされたbsr遺伝子がゲノムに組み込まれた細胞のみが増殖し、コロニーを形成した。充分に成長したコロニーをペニシリンカップ法によりトリプシンにてはがし、12穴プレートにまき直して同じ培地で増殖させた。こうして単離したクローナルな細胞が、実際に変性低密度リポ蛋白質受容体を発現していることを、前記のようにDiI標識変性低密度リポ蛋白質取り込みによる細胞質内蛍光強度上昇を蛍光顕微鏡を用いて観察することにより確認した。蛍光顕微鏡写真を図3に示す。
【0037】[実施例4]
可溶性変性低密度リポ蛋白質受容体の作成変性低密度リポ蛋白質受容体cDNAをPCR法により、プライマーペア(5'-gcggatcctgtgctctcaatagattcgc-3',5'-ggggatcctgatctcataaagaaacag-3')を用いて、細胞外ドメイン(変性低密度リポ蛋白質との結合部分を含む)をコードするDNA配列(配列番号1の塩基配列の160〜813)をBamHI の制限酵素部位を付けて増殖し、BamHI で消化した後、ヒスチジンの6回繰り返し配列をtagとして大腸菌に蛋白を発現するベクターpQE10(Qiagen社製)のBamHI siteに挿入した。このプラスミドを大腸菌株XL-2Blueにトランスフォーム後、2xYT培地にて37℃で振とう培養した。600nmの吸光度が0.6になったところでIPTG(isopropylthio-β-D-galactoside)を1mMの濃度で加え、30℃で20時間さらに培養した。大腸菌を遠心にて回収し、6M塩酸グアニジン、0.1Mリン酸二水素ナトリウム、0.01Mトリス(pH8.0)にて溶解した。不溶物を遠心にて除去後、Ni−NTAアガロース(Qiagen社製)を加え、可溶性変性低密度リポ蛋白質受容体を吸着させた。Ni−NTAアガロースを8M尿素、0.1Mリン酸二水素ナトリウム、0.01Mトリス(pH8.0)、および8M尿素、0.1Mリン酸二水素ナトリウム、0.01Mトリス(pH6.3)にて洗浄後、8M尿素、0.1Mリン酸二水素ナトリウム、0.01Mトリス、0.1M EDTA(pH6.3)にて可溶性変性低密度リポ蛋白質受容体を溶出し、精製した。上記の操作により得られた可溶性変性低密度リポ蛋白質受容体は、SDS−PAGEにて均一な標品であることが示された。なお、tagは、GSTやc−myc等、他のものを用いてもよい。この場合は、精製法もそれに応じて、抗体等を用いた方法を用いることができる。また、適当な哺乳動物発現用ベクターを用いれば、組み替えDNAの操作により、同様に可溶性変性低密度リポ蛋白質受容体を作成することができる。
【0038】[実施例5]
抗LOX−1抗体の作成ポリメラーゼチェーン反応によりウシLOX−1cDNAの細胞外ドメインをコードする領域(アミノ酸61-270)を、BamHIを付加した2つのプライマー5'-ggggatcctgatctcataaagaaacag-3'と5'-gcggatcctgtgctctcaatagattcgc-3'により増幅した。増幅したcDNA断片をBamHIで消化した後、キアゲン(QIAGEN)社のpQE10ベクターのBamHI部位にサブクローンした。細胞外ドメイン蛋白の合成と生成はキアゲン社のQIA express systemを用いて行なった。実際には、上記のプラスミドによりストラタジーン(Stratagene)社の大腸菌株XL−1 blue を形質転換し、2xYT培地にて培養した。蛋白の合成は、イソプロピル・チオ−β−D−ガラクトシドを最終濃度2mMで加えることで誘導した。細胞を遠心分離により回収し、6Mグアニジン・HCl、0.1Mリン酸ナトリウム、および0.01M−Tris・HCl を含むpH8.0の溶液に溶解後、キアゲン社のNi-NTA樹脂カラムにかけてカラムクロマトグラフィーを行なった。次いで、8M尿素、0.1Mリン酸ナトリウム、及び0.01M−Tris・HCl を含むpH6.3の水溶液でカラムを洗浄した後、蛋白質を8M尿素、0.1M−EDTA、0.1Mリン酸ナトリウム、及び0.01M−Tris・HCl を含むpH6.3の溶液を用いて溶出した。バッファーを、アミコン社のCentriprep 10 にてリン酸塩緩衝生理食塩水に交換し、蛋白質を等容量のフロイント・コンプリート・アジュバントでエマルジョンにして、ウサギの肩甲骨脊椎間の皮内に隔週で接種することで免疫を行った。
免疫染色培養ウシ大動脈内皮細胞をSDS−PAGEのサンプルバッファーにて直接溶解し、SDS−PAGEにより分離し、ナイロン膜に転写した。雪印乳業株式会社製のブロックエースにてブロッキングした後、ベクトール(Vector)社の免疫染色キットを用い、ペルオキシダーゼ抱合アビジン・ビオチン複合体による標準的な方法により、上記のウサギから得られた抗血清の染色を行った。
【0039】
【配列表】
【配列番号1】
配列の長さ:1897配列の型:核酸鎖の数:一本鎖トポロジー:直線状配列の種類:DNA、蛋白質ハイポセティカル:Noアンチセンス:No起源生物名:Bos taurus組織の種類:vascular endothelial cells直接の起源ライブラリー名:Bovine aortic endothelial cells cDNA libraryクローン名:pBLOX−1配列の特徴特徴を表わす記号:CDS存在位置:1..813特徴を決定した方法:S特徴を表わす記号:polyA signal存在位置:1825..1830特徴を決定した方法:S特徴を表わす記号:polyA site存在位置:1846..1863特徴を決定した方法:S特徴を表わす記号:3’UTR存在位置:814..1863特徴を決定した方法:S特徴を表わす記号:5’UTR存在位置:−34..−1特徴を決定した方法:S配列-34 GCTT CACTCTCTCA TTCTTGGAAT ACATTTGAAA -1 ATG ACT GTT GAT GAC CCC AAG GGT ATG AAA 30 Met Thr Val Asp Asp Pro Lys Gly Met Lys 5 10 GAT CAA CTT GAT CAG AAG CCA AAT GGC AAG 60 Asp Gln Leu Asp Gln Lys Pro Asn Gly Lys 15 20 ACA GCA AAA GGT TTT GTT TCC TCT TGG AGG 90 Thr Ala Lys Gly Phe Val Ser Ser Trp Arg 25 30 TGG TAC CCT GCT GCT GTG ACT CTA GGG GTC 120 Trp Tyr Pro Ala Ala Val Thr Leu Gly Val 35 40 CTT TGT CTG GGA TTA CTG GTG ACT GTT ATA 150 Leu Cys Leu Gly Leu Leu Val Thr Val Ile 45 50 TTG TTG ATA CTG CAA TTA TCC CAG GTC TCT 180 Leu Leu Ile Leu Gln Leu Ser Gln Val Ser 55 60 GAT CTC ATA AAG AAA CAG CAA GCA AAT ATT 210 Asp Leu Ile Lys Lys Gln Gln Ala Asn Ile 65 70 ACT CAC CAG GAA GAT ATC CTG GAG GGA CAG 240 Thr His Gln Glu Asp Ile Leu Glu Gly Gln 75 80 ATT TTA GCC CAG CGC CGA TCA GAA AAA TCT 270 Ile Leu Ala Gln Arg Arg Ser Glu Lys Ser 85 90 GCC CAG GAG TCA CAG AAG GAA CTC AAA GAA 300 Ala Gln Glu Ser Gln Lys Glu Leu Lys Glu 95 100 ATG ATA GAA ACC CTT GCC CAC AAG CTG GAT 330 Met Ile Glu Thr Leu Ala His Lys Leu Asp 105 110 GAG AAA TCC AAG AAA CTA ATG GAA CTT CAC 360 Glu Lys Ser Lys Lys Leu Met Glu Leu His 115 120 CGC CAG AAC CTG AAT CTC CAA GAA GTT CTG 390 Arg Gln Asn Leu Asn Leu Gln Glu Val Leu 125 130 AAA GAG GCA GCA AAC TAT TCA GGT CCT TGT 420 Lys Glu Ala Ala Asn Tyr Ser Gly Pro Cys 135 140 CCC CAA GAC TGG CTC TGG CAT GAA GAA AAC 450 Pro Gln Asp Trp Leu Trp His Glu Glu Asn 145 150 TGT TAC CAA TTT TCC TCT GGC TCT TTT AAT 480 Cys Tyr Gln Phe Ser Ser Gly Ser Phe Asn 155 160 TGG GAA AAA AGC CAG GAG AAC TGC TTG TCT 510 Trp Glu Lys Ser Gln Glu Asn Cys Leu Ser 165 170 TTG GAT GCC CAC TTG CTG AAG ATT AAT AGC 540 Leu Asp Ala His Leu Leu Lys Ile Asn Ser 175 180 ACA GAT GAA CTG GAA TTC ATC CAG CAA ATG 570 Thr Asp Glu Leu Glu Phe Ile Gln Gln Met 185 190 ATT GCC CAT TCC AGT TTC CCC TTC TGG ATG 600 Ile Ala His Ser Ser Phe Pro Phe Trp Met 195 200 GGG TTG TCA ATG AGG AAA CCC AAT TAC TCG 630 Gly Leu Ser Met Arg Lys Pro Asn Tyr Ser 205 210 TGG CTT TGG GAA GAT GGT ACT CCT TTG ACG 660 Trp Leu Trp Glu Asp Gly Thr Pro Leu Thr 215 220 CCC CAC TTG TTT AGA ATT CAG GGA GCT GTT 690 Pro His Leu Phe Arg Ile Gln Gly Ala Val 225 230 TCC CGT ATG TAT CCT TCA GGG ACC TGT GCA 720 Ser Arg Met Tyr Pro Ser Gly Thr Cys Ala 235 240 TAT ATT CAA AGG GGA ACT GTT TTT GCT GAA 750 Tyr Ile Gln Arg Gly Thr Val Phe Ala Glu 245 250 AAC TGC ATT TTA ACT GCA TTC AGT ATA TGT 780 Asn Cys Ile Leu Thr Ala Phe Ser Ile Cys 255 260 CAA AAG AAG GCG AAT CTA TTG AGA GCA CAG 810 Gln Lys Lys Ala Asn Leu Leu Arg Ala Gln 265 270 TGAATTTGAA GGATCTGGAG GAAAAGAAGG AAACCTTTGA ATTCTCTTCT GGAATTTAAG 870 CTATACTTCA TCACTTAGAT GTAAACCATT AGAGCCCAGG GAAATGCCTG CTACTGGTTG 930 AGTGCAGAAC TCCTTAGCAG AGACTGGCCC AGCTGCCTGG CACCTTGATA GCAAAAGTTG 990 CAATTCCCTC TGTATATTTT TCCCTAACTT GTTCCAAGTC CTCCCCTGCA GGACTTCAGA 1050 GAAGTCAATT TTTCTGTTTC CATTGTTTCT AAGAACTTGT TGCCTAACTC AAGGTCACAG 1110 CATTTTTCTC ACTTTTGTCC TATGCTTTCT TCTAGGCATT GTAGAGTTTT AGATTTTACA 1170 TGGAAATCTA GAACTTATTT TAGATTAATT TCTAAGTGAT ATATGGATGT ATGGAAGTTT 1230 TCTGTTTGTT TTTTGCTTGT GAGTATTCAA TTGTTTTTGC AACATTTGCT GAAAAGACTA 1290 TTCTTCCTTC ACTACATTGC CTTTGCACTG TTGTCAACAA TTATCCATAC ATGCCTGGCT 1350 CTATTTCTGG ATTTTCTATT CCTTTCCATT TATTTATTTA TTATTCTTGG CTTACAACAT 1410 CACCATGATA TTTTGAATTC TATGGTTCTT TAATATATCT TGGAATCACA TGGTAGTAGT 1470 TATTCATTGT TGTTCTTTTT TAGAGTTGTT TGGTTAATCT ATGCTTTTGT ATTTCTGTCT 1530 TAAATTGGCT TGTCCATTTC TAAAAAAACT TGAAATTTTG AATTGCACTG AATCCATACA 1590 TAAATTTAGG GAAAATTGAA TTCTTAAAAA TACTGATTTG TTCAACTCAT GAAAAAGGTG 1650 TATTGCTCTA TTTAGGTATT CCTTATTTTC TTTAAGCAAT GCTTTTTAAT GTTCTTTGTG 1710 TAGATATTGT TAGATTATCA TCATGTATTT CACATTATTT ATGCTACTGT AGATAGTATT 1770 GTTATCATTT GTTGTTCTTA TTTTCAAAGT CTTCTGCTAG TATGTAGAAT TATAATAAAG 1830 TTTGATATTA ATATTAAAAA AAAAAAAAAA AAA 1863
【0040】
【配列番号2】
配列の長さ:1906配列の型:核酸鎖の数:一本鎖トポロジー:直線状配列の種類:DNA、蛋白質ハイポセティカル:Noアンチセンス:No起源生物名:Bos taurus組織の種類:vascular endothelial cells直接の起源ライブラリー名:Bovine aortic endothelial cells cDNA libraryクローン名:pBLOX−1配列の特徴特徴を表わす記号:CDS存在位置:1..822特徴を決定した方法:S特徴を表わす記号:polyA signal存在位置:1834..1839特徴を決定した方法:S特徴を表わす記号:polyA site存在位置:1855..1872特徴を決定した方法:S特徴を表わす記号:3’UTR存在位置:823..1872特徴を決定した方法:S特徴を表わす記号:5’UTR存在位置:−34..−1特徴を決定した方法:S配列-34 GCTT CACTCTCTCA TTCTTGGAAT ACATTTGAAA -1 ATG ACT GTT GAT GAC CCC AAG GGT ATG AAA 30 Met Thr Val Asp Asp Pro Lys Gly Met Lys 5 10 GAT CAA CTT GAT CAG AAG CCA AAT GGC AAG 60 Asp Gln Leu Asp Gln Lys Pro Asn Gly Lys 15 20 ACA GCA AAA GGT ACT ACA GGT TTT GTT TCC 90 Thr Ala Lys Gly Thr Thr Gly Phe Val Ser 25 30 TCT TGG AGG TGG TAC CCT GCT GCT GTG ACT 120 Ser Trp Arg Trp Tyr Pro Ala Ala Val Thr 35 40 CTA GGG GTC CTT TGT CTG GGA TTA CTG GTG 150 Leu Gly Val Leu Cys Leu Gly Leu Leu Val 45 50 ACT GTT ATA TTG TTG ATA CTG CAA TTA TCC 180 Thr Val Ile Leu Leu Ile Leu Gln Leu Ser 55 60 CAG GTC TCT GAT CTC ATA AAG AAA CAG CAA 210 Gln Val Ser Asp Leu Ile Lys Lys Gln Gln 65 70 GCA AAT ATT ACT CAC CAG GAA GAT ATC CTG 240 Ala Asn Ile Thr His Gln Glu Asp Ile Leu 75 80 GAG GGA CAG ATT TTA GCC CAG CGC CGA TCA 270 Glu Gly Gln Ile Leu Ala Gln Arg Arg Ser 85 90 GAA AAA TCT GCC CAG GAG TCA CAG AAG GAA 300 Glu Lys Ser Ala Gln Glu Ser Gln Lys Glu 95 100 CTC AAA GAA ATG ATA GAA ACC CTT GCC CAC 330 Leu Lys Glu Met Ile Glu Thr Leu Ala His 105 110 AAG CTG GAT GAG AAA TCC AAG AAA CTA ATG 360 Lys Leu Asp Glu Lys Ser Lys Lys Leu Met 115 120 GAA CTT CAC CGC CAG AAC CTG AAT CTC CAA 390 Glu Leu His Arg Gln Asn Leu Asn Leu Gln 125 130 GAA GTT CTG AAA GAG GCA GCA AAC TAT TCA 420 Glu Val Leu Lys Glu Ala Ala Asn Tyr Ser 135 140 GGT CCT TGT CCC CAA GAC TGG CTC TGG CAT 450 Gly Pro Cys Pro Gln Asp Trp Leu Trp His 145 150 GAA GAA AAC TGT TAC CAA TTT TCC TCT GGC 480 Glu Glu Asn Cys Tyr Gln Phe Ser Ser Gly 155 160 TCT TTT AAT TGG GAA AAA AGC CAG GAG AAC 510 Ser Phe Asn Trp Glu Lys Ser Gln Glu Asn 165 170 TGC TTG TCT TTG GAT GCC CAC TTG CTG AAG 540 Cys Leu Ser Leu Asp Ala His Leu Leu Lys 175 180 ATT AAT AGC ACA GAT GAA CTG GAA TTC ATC 570 Ile Asn Ser Thr Asp Glu Leu Glu Phe Ile 185 190 CAG CAA ATG ATT GCC CAT TCC AGT TTC CCC 600 Gln Gln Met Ile Ala His Ser Ser Phe Pro 195 200 TTC TGG ATG GGG TTG TCA ATG AGG AAA CCC 630 Phe Trp Met Gly Leu Ser Met Arg Lys Pro 205 210 AAT TAC TCG TGG CTT TGG GAA GAT GGT ACT 660 Asn Tyr Ser Trp Leu Trp Glu Asp Gly Thr 215 220 CCT TTG ACG CCC CAC TTG TTT AGA ATT CAG 690 Pro Leu Thr Pro His Leu Phe Arg Ile Gln 225 230 GGA GCT GTT TCC CGT ATG TAT CCT TCA GGG 720 Gly Ala Val Ser Arg Met Tyr Pro Ser Gly 230 240 ACC TGT GCA TAT ATT CAA AGG GGA ACT GTT 750 Thr Cys Ala Tyr Ile Gln Arg Gly Thr Val 245 250 TTT GCT GAA AAC TGC ATT TTA ACT GCA TTC 780 Phe Ala Glu Asn Cys Ile Leu Thr Ala Phe 255 260 AGT ATA TGT CAA AAG AAG GCG AAT CTA TTG AGA GCA CAG 819 Ser Ile Cys Gln Lys Lys Ala Asn Leu Leu Arg Ala Gln 265 270 TGAATTTGAA GGATCTGGAG GAAAAGAAGG AAACCTTTGA ATTCTCTTCT GGAATTTAAG 879 CTATACTTCA TCACTTAGAT GTAAACCATT AGAGCCCAGG GAAATGCCTG CTACTGGTTG 939 AGTGCAGAAC TCCTTAGCAG AGACTGGCCC AGCTGCCTGG CACCTTGATA GCAAAAGTTG 999 CAATTCCCTC TGTATATTTT TCCCTAACTT GTTCCAAGTC CTCCCCTGCA GGACTTCAGA 1059 GAAGTCAATT TTTCTGTTTC CATTGTTTCT AAGAACTTGT TGCCTAACTC AAGGTCACAG 1119 CATTTTTCTC ACTTTTGTCC TATGCTTTCT TCTAGGCATT GTAGAGTTTT AGATTTTACA 1179 TGGAAATCTA GAACTTATTT TAGATTAATT TCTAAGTGAT ATATGGATGT ATGGAAGTTT 1239 TCTGTTTGTT TTTTGCTTGT GAGTATTCAA TTGTTTTTGC AACATTTGCT GAAAAGACTA 1299 TTCTTCCTTC ACTACATTGC CTTTGCACTG TTGTCAACAA TTATCCATAC ATGCCTGGCT 1359 CTATTTCTGG ATTTTCTATT CCTTTCCATT TATTTATTTA TTATTCTTGG CTTACAACAT 1419 CACCATGATA TTTTGAATTC TATGGTTCTT TAATATATCT TGGAATCACA TGGTAGTAGT 1479 TATTCATTGT TGTTCTTTTT TAGAGTTGTT TGGTTAATCT ATGCTTTTGT ATTTCTGTCT 1539 TAAATTGGCT TGTCCATTTC TAAAAAAACT TGAAATTTTG AATTGCACTG AATCCATACA 1599 TAAATTTAGG GAAAATTGAA TTCTTAAAAA TACTGATTTG TTCAACTCAT GAAAAAGGTG 1659 TATTGCTCTA TTTAGGTATT CCTTATTTTC TTTAAGCAAT GCTTTTTAAT GTTCTTTGTG 1719 TAGATATTGT TAGATTATCA TCATGTATTT CACATTATTT ATGCTACTGT AGATAGTATT 1779 GTTATCATTT GTTGTTCTTA TTTTCAAAGT CTTCTGCTAG TATGTAGAAT TATAATAAAG 1839 TTTGATATTA ATATTAAAAA AAAAAAAAAA AAA 1872
【0041】
【配列番号3】
配列の長さ:1313配列の型:核酸鎖の数:一本鎖トポロジー:直線状配列の種類:DNA、蛋白質ハイポセティカル:Noアンチセンス:No起源生物名:Homo sapiens組織の種類:ヒト肺、胎盤直接の起源ライブラリー名:human lung cDNA library クローン名:λhLOX−1配列の特徴特徴を表わす記号:CDS存在位置:1..822特徴を決定した方法:S特徴を表わす記号:5’UTR存在位置:−60..−1特徴を決定した方法:S特徴を表わす記号:3’UTR存在位置:823..1183特徴を決定した方法:S配列 -126 GGGGCC -121 GCACTAGTGA TTCTGGTTCG GCCCACCTCT GAAGGTTCCA GAATCGATAG TGAATTCGTG -61 ATTTTAGTTT GTTGAAGTTC GTGACTGCTT CACTCTCTCA TTCTTAGCTT GAATTTGGAA -1 ATG ACT TTT GAT GAC CTA AAG ATC CAG ACT 30 Met Thr Phe Asp Asp Leu Lys Ile Gln Thr 5 10 GTG AAG GAC CAG CCT GAT GAG AAG TCA AAT 60 Val Lys Asp Gln Pro Asp Glu Lys Ser Asn 15 20 GGA AAA AAA GCT AAA GGT CTT CAG TTT CTT 90 Gly Lys Lys Ala Lys Gly Leu Gln Phe Leu 25 30 TAC TCT CCA TGG TGG TGC CTG GCT GCT GCG 120 Tyr Ser Pro Trp Trp Cys Leu Ala Ala Ala 35 40 ACT CTA GGG GTC CTT TGC CTG GGA TTA GTA 150 Thr Leu Gly Val Leu Cys Leu Gly Leu Val 45 50 GTG ACC ATT ATG GTG CTG GGC ATG CAA TTA 180 Val Thr Ile Met Val Leu Gly Met Gln Leu 55 60 TCC CAG GTG TCT GAC CTC CTA ACA CAA GAG 210 Ser Gln Val Ser Asp Leu Leu Thr Gln Glu 65 70 CAA GCA AAC CTA ACT CAC CAG AAA AAG AAA 240 Gln Ala Asn Leu Thr His Gln Lys Lys Lys 75 80 CTG GAG GGA CAG ATC TCA GCC CGG CAA CAA 270 Leu Glu Gly Gln Ile Ser Ala Arg Gln Gln 85 90 GCA GAA GAA GCT TCA CAG GAG TCA GAA AAC 300 Ala Glu Glu Ala Ser Gln Glu Ser Glu Asn 95 100 GAA CTC AAG GAA ATG ATA GAA ACC CTT GCT 330 Glu Leu Lys Glu Met Ile Glu Thr Leu Ala 105 110 CGG AAG CTG AAT GAG AAA TCC AAA GAG CAA 360 Arg Lys Leu Asn Glu Lys Ser Lys Glu Gln 115 120 ATG GAA CTT CAC CAC CAG AAT CTG AAT CTC 390 Met Glu Leu His His Gln Asn Leu Asn Leu 125 130 CAA GAA ACA CTG AAG AGA GTA GCA AAT TGT 420 Gln Glu Thr Leu Lys Arg Val Ala Asn Cys 135 140 TCA GCT CCT TGT CCG CAA GAC TGG ATC TGG 450 Ser Ala Pro Cys Pro Gln Asp Trp Ile Trp 145 150 CAT GGA GAA AAC TGT TAC CTA TTT TCC TCG 480 His Gly Glu Asn Cys Tyr Leu Phe Ser Ser 155 160 GGC TCA TTT AAC TGG GAA AAG AGC CAA GAG 510 Gly Ser Phe Asn Trp Glu Lys Ser Gln Glu 165 170 AAG TGC TTG TCT TTG GAT GCC AAG TTG CTG 540 Lys Cys Leu Ser Leu Asp Ala Lys Leu Leu 175 180 AAA ATT AAT AGC ACA GCT GAT CTG GAC TTC 570 Lys Ile Asn Ser Thr Ala Asp Leu Asp Phe 185 190 ATC CAG CAA GCA ATT TCC TAT TCC AGT TTT 600 Ile Gln Gln Ala Ile Ser Tyr Ser Ser Phe 195 200 CCA TTC TGG ATG GGG CTG TCT CGG AGG AAC 630 Pro Phe Trp Met Gly Leu Ser Arg Arg Asn 205 210 CCC AGC TAC CCA TGG CTC TGG GAG GAC GGT 660 Pro Ser Tyr Pro Trp Leu Trp Glu Asp Gly 215 220 TCT CCT TTG ATG CCC CAC TTA TTT AGA GTC 690 Ser Pro Leu Met Pro His Leu Phe Arg Val 225 230 CGA GGC GCT GTC TCC CAG ACA TAC CCT TCA 720 Arg Gly Ala Val Ser Gln Thr Tyr Pro Ser 235 240 GGT ACC TGT GCA TAT ATA CAA CGA GGA GCT 750 Gly Thr Cys Ala Tyr Ile Gln Arg Gly Ala 245 250 GTT TAT GCG GAA AAC TGC ATT TTA GCT GCC 780 Val Tyr Ala Glu Asn Cys Ile Leu Ala Ala 255 260 TTC AGT ATA TGT CAG AAG AAG GCA AAC CTA AGA GCA CAG 819 Phe Ser Ile Cys Gln Lys Lys Ala Asn Leu Arg Ala Gln 265 270 273 TGAATTTGAA GGCTCTGGAA GAAAAGAAAA AAGTCTTTGA GTTTTATTCT GGAATTTAAG 879 CTATTCTTTG TCACTTGGGT GCCAAACATG AGAGCCCAGA AAACTGTCAT TTAGCTGGCT 939 GCAGAACTCC TTTGCAGAAA CTGGGGTTCC AGGTGCCTGG CACCTTTATG TCAACATTTT 999 TGATTCTAGC TATCTGTATT ATTTCACCTA GCTTGTCCCA AGCTTCCCTG CCAGCCTGAA 1059 GTCCATTTTC CCCTTTTTAT TTTAAAATTT GACTCCTCTT CAAGCTTGAA AACCCTCTGA 1119 ACTCAGTCTT CTTTACCTCA TTATCACCTT CCCCTCACAC TCCTAAAATT GCATGAAAGA 1179 CAGACCGGAA TTC 1192
【図面の簡単な説明】
【図1】実施例1で用いた哺乳動物細胞用高発現プラスミドベクターpME18Sの模式図である。
【図2】pBLOX−1を導入したCOS−7細胞および導入していない対照のCOS−7細胞のFACSによる蛍光強度分布を示すグラフである。
【図3】DiI標識変性低密度リポ蛋白質投与による細胞質内蛍光強度上昇を示す蛍光顕微鏡写真である。
【0001】
【産業上の利用分野】本発明は、哺乳類のリポ蛋白質の受容体、さらに詳しくは哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体に関する。
【0002】
【従来の技術】動脈硬化の進展の過程では、血管内皮細胞の機能変化が初期の段階で重要であることが指摘されている。血管内皮細胞は種々の液性因子を放出し、循環系のホメオスターシスを保っている。この血管内皮細胞の機能は物理的な刺激や、種々の物質により障害を受けるがこの中でも変性低密度リポ蛋白質の一種である酸化低密度リポ蛋白質が最も重要であると考えられている。例えば、血管のトーヌス(tonus) の調節は血管内皮細胞が血管弛緩因子である一酸化窒素を放出することが重要であるが、この一酸化窒素の放出は酸化低密度リポ蛋白質により障害される。
【0003】一方、変性低密度リポ蛋白質は、低密度リポ蛋白質受容体とは異なる受容体を介してマクロファージや血管内皮細胞に取り込まれることが知られていた。マクロファージでは既に構造が決定されている(特表平6−500765号、同6−508604号、特開平3−290184号各公報記載)スカベンジャー受容体を介して変性低密度リポ蛋白質は取り込まれ、マクロファージは動脈硬化巣で特徴的な泡沫細胞へと変化する。血管内皮細胞にはこのスカベンジャー受容体は発現しておらず、別の構造を持った受容体が存在することが予想されていた(荒井秀典、北徹、酸化LDL、代謝28/4、1991参照)。このような点から血管内皮細胞の変性低密度リポ蛋白質受容体の構造、すなわち、アミノ酸配列の解明が重要であるが、これまで全く構造はわかっていなかった。
【0004】
【発明が解決しようとする課題】本発明の目的は、血管内皮細胞の変性低密度リポ蛋白質受容体の構造を解明し、これにより血管内皮細胞の変性低密度リポ蛋白質受容体をコードするDNA配列を提供することである。また、本発明の目的は、変性低密度リポ蛋白質受容体またはその類似体を生産する方法を提供することでもある。さらに、本発明の目的は、変性低密度リポ蛋白質受容体またはその類似体を含む蛋白質組成物を提供することでもある。
【0005】
【課題を解決するための手段】本発明者の研究により、血管内皮細胞の変性低密度リポ蛋白質受容体の構造が明らかとなった。血管内皮細胞の変性低密度リポ蛋白質受容体は、配列番号1、配列番号2または配列番号3に記載のアミノ酸配列を有している。本発明は、哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列を提供するものである。
【0006】上記DNA配列は、天然の哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体遺伝子のオープンリーディングフレームに由来するcDNAクローンであってもよい。また、上記DNA配列は、上記cDNAクローンとハイブリダイズできて、かつ生物学的活性を有する哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードしているDNA配列であってもよい。さらに、以上述べたようなDNA配列について、遺伝暗号の縮重の結果として、同じ変性低密度リポ蛋白質受容体をコードしているDNA配列であってもよい。従って、本発明は、哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域の配列番号1、配列番号2または配列番号3に示すDNA配列(当該塩基配列を有するDNA)、もしくはそのアナログにある。また本発明は、哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域の配列番号1、配列番号2または配列番号3に示すDNA配列を有するcDNAクローン、もしくはそのアナログにある。また、本発明は、42℃、20%(v/v)ホルムアミド水溶液中で、配列番号1、配列番号2または配列番号3に示すDNA配列のcDNAクローンとハイブリダイズでき、かつ哺乳類の血管内皮細胞の変性低密度リポ蛋白質と結合する機能を有する蛋白質(即ち受容体)をコードしているDNA配列(当該塩基配列を有するDNA)にもある。そしてまた、本発明は、配列番号1、配列番号2または配列番号3に示すDNA配列と遺伝的コードの結果として縮重していて、かつ哺乳類の血管内皮細胞の変性低密度リポ蛋白質と結合する機能を有する蛋白質(即ち受容体)をコードしているDNA配列(当該塩基配列を有するDNA)にもある。さらに本発明は、配列番号1、配列番号2または配列番号3に示す塩基配列を有するDNAもしくはそのアナログに対応する変性低密度リポ蛋白質受容体(あるいは配列番号1、配列番号2または配列番号3に示すアミノ酸配列もしくはそのアナログを含む変性低密度リポ蛋白質受容体)の抗体にもある。
【0007】本発明が提供するDNA配列は発現ベクターに組込んで用いることができる。これにより、本発明は、この組換え発現ベクターを宿主細胞に挿入し、宿主細胞を発現を引き起こす条件下で培養することからなる哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を生産する方法も提供する。さらに、本発明は、以上のように生産された生物学的活性を有する哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を含む蛋白質組成物も提供する。このように生産された哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体、その類似体およびそれらを含む蛋白質組成物は、哺乳類の変性低密度リポ蛋白質のアッセイに有用であり、また診断で用いる変性低密度リポ蛋白質受容体に対する抗体を調製する上でも有用である。また、本発明の薬剤が動脈硬化症の疾患の診断に有効であることは、前述した変性低密度リポ蛋白質およびその受容体の生物学的活性から明らかである。
【0008】本明細書において、「変性低密度リポ蛋白質受容体」とは、変性低密度リポ蛋白質に結合することができ、哺乳類の血管内皮細胞の細胞膜蛋白質と同様な本来の立体配置をとる際に、変性低密度リポ蛋白質分子によって与えられる情報を血管内皮細胞内に伝達する役割を持っている蛋白質を意味する。本明細書において用いる場合、この用語は、変性低密度リポ蛋白質に結合するか、あるいは情報伝達活性を有する天然蛋白質の類似物を含んでいる。「変性低密度リポ蛋白質受容体のサブタイプ」とは、変性低密度リポ蛋白質受容体のうちで酸化変性低密度リポ蛋白質あるいはアセチル化変性低密度リポ蛋白質等の各種変性低密度リポ蛋白質に対して、異なる選択性、すなわち薬理学的親和性序列を示す各々の分子をいう。
【0009】本明細書において、DNA配列において用いられる「コードする領域から実質的になる配列」等における『実質的に』の表現は、特定の対象となる配列、例えば変位配列で、本来の配列番号1、配列番号2または配列番号3に示す参照配列の1つ以上の置換、欠失あるいは付加によって変化したものであって、その全体としての作用が、参照配列と比較して不利な機能的相違をもたらさない配列も含むとの趣旨で用いられる。具体的には後述する生物学的活性を有する、さらに具体的には、変性低密度リポ蛋白質と結合可能である範囲内で配列の変化が可能である。そのためには、変性低密度リポ蛋白質と結合する部分をコードする領域については、遺伝暗号の縮重を除き、配列番号1、配列番号2または配列番号3に示す参照配列と同じか、類似のアミノ酸への置換をもたらす程度の変更を加えた配列を有する必要がある。一方、上記以外の部分をコードする領域については、30%以上(好ましくは50%以上、さらに好ましくは80%以上)の相似性があればよい。
【0010】なお、上記の類似のアミノ酸への置換とは、天然のアミノ酸を下記の8群に分類する場合において、各群内におけるアミノ酸相互の置換を意味する。
(1)モノアミノモノカルボン酸Gly Ala Val Leu Ile(2)オキシアミノ酸Ser Thr(3)含硫アミノ酸Cys Met(4)モノアミノジカルボン酸Asp Glu(5)ジアミノモノカルボン酸Lys Arg(6)芳香族環を有するアミノ酸Phe Tyr(7)複素環を有するアミノ酸His Try Pro(8)アミド基を有するアミノ酸Asn Gln類似性を決定する目的においては、参照配列の短縮あるいは内在的欠失は無視してよい。配列の相似性が低い場合でも、同程度の生物学的活性を有し、同等の発現特性を有する配列は、実質的に同等な配列とみなす。
【0011】「生物学的に活性を有する」という用語は、変性低密度リポ蛋白質受容体の特性として用いられる場合、特定の分子が、変性低密度リポ蛋白質を結合可能であることが明らかにされている本発明の態様と充分なアミノ酸配列の類似性を有するか、あるいは混成受容体構成物の構成要素として細胞に変性低密度リポ蛋白質の刺激を伝達するために充分なアミノ酸配列の類似性を変性低密度リポ蛋白質受容体に対して有していることを意味する。変性低密度リポ蛋白質を結合可能であるとは、具体的には、基準とする酸化変性低密度リポ蛋白質に対する親和性(解離定数)が1マイクロモル以下であることを意味する。本発明においては、その親和性が0.1マイクロモル以下であることが好ましく、0.01マイクロモル以下であることがさらに好ましい。生物学的活性は、変性低密度リポ蛋白質の結合に際して、血管内皮細胞内へ変性低密度リポ蛋白質の取り込みを促すことができることを意味する場合もある。
【0012】「DNA配列」は、大きなDNA構成物から分離した断片あるいは構成要素として存在するDNAポリマーを示しており、それらは少なくとも一度は実質的に純粋な形に単離されたDNAを起源とする。すなわち、内在性物質の混入がない形で単離され、かつ同定、操作および標準的な生化学的方法、例えば、クローニングベクターを用いて、配列およびその構成要素である塩基配列が回収可能な程度の量あるいは濃度で存在する。こうした配列は、真核細胞遺伝子に典型的な内在性非翻訳配列(イントロン)によって中断されていないオープンリーディングフレームを持つ形で供給されることが好ましい。しかし、関連した配列を含んだ染色体DNAも用いられることは明らかである。非翻訳DNAは、オープンリーディングフレームの5’あるいは3’にあり、その場合、非翻訳配列はコード領域の操作あるいは発現の妨げとはならない。「組換え発現ベクター」は(a)遺伝子発現における調節の役割を持つ要素、例えばプロモーター或いはエンハンサーのような単独または複数の遺伝的要素、(b)mRNAに転写され、蛋白質に翻訳される構造あるいはコード配列、及び(c)適当な転写及び翻訳開始及び終止配列の集合を含む転写単位からなるプラスミドを示す。酵母の発現系で用いるための構造要素は、翻訳された蛋白質が宿主細胞によって細胞外に分泌されるためのリーダー配列を含んでいることが好ましい。組換え体蛋白質がリーダーあるいは輸送配列を持たずに発現された場合、その蛋白質はN−末端にメチオニン残基を含んでいる可能性がある。続いて、その残基は発現された組換え蛋白質から随意に切断され、最終産物を与える。
【0013】次に、変性低密度リポ蛋白質受容体をコードするcDNAの単離、およびDNA配列の決定について説明する。培養ウシ大動脈内皮細胞から抽出したPoly (A)+ RNAの逆転写によって調製されたcDNAライブラリーから、ウシ変性低密度リポ蛋白質受容体をコードするDNA配列が単離された。このライブラリーは、SV40、I型ヒトT細胞白血病ウイルス由来の調節配列を持った哺乳類発現べクター(pME18S)を用いて、サルのCOS−7細胞に蓄積されたDNA断片よりmRNAを直接発現させることによってスクリーニングした。形質導入されたCOS−7細胞をDiI(1,1'-di-octadecyl-3,3,3',3'-tetramethylindocarbocyanine perchlorate )−酸化低密度リポ蛋白質を含む培地とともにインキュベートし、結合しなかったDiI−酸化低密度リポ蛋白質を除くために洗浄した後、トリプシン処理により細胞を浮遊状態にしてFACS(fluorescence-activated cell sorter)にかけ、DiIの蛍光を測定するとともに蛍光強度の高い細胞を回収した。形質導入細胞からプラスミドを抽出し、大腸菌を形質転換してプラスミドを精製し、再度上記の操作を繰り返した。このようにして4回この操作を繰り返すことにより、単一のクローンで変性低密度リポ蛋白質結合活性を持った表面蛋白質を合成することができた。このクローンは単離され、ウシの変性低密度リポ蛋白質受容体のcDNA配列を決定するために挿入断片の配列が調べられた。
【0014】単離されたcDNAクローンをCOS−7細胞に導入し発現させたところ、この細胞は酸化低密度リポ蛋白質および酸化低密度リポ蛋白質に対する特異的結合活性を獲得し、これらの変性低密度リポ蛋白質は細胞内へと取り込まれた。さらにこのとき未変性の低密度リポ蛋白質に対する結合活性は見られず、変性した低密度リポ蛋白質に特異的な受容体と考えられる。
【0015】以上のように決定された変性低密度リポ蛋白質受容体をコードするDNA配列および相当するアミノ酸配列を配列番号1および配列番号2に示す。DNA配列およびアミノ酸配列について説明する。上記配列番号1及び2に示されるように、変性低密度リポ蛋白質受容体には少なくとも2種類のサブタイプが存在している。配列番号2は、配列番号1の24番目のアミノ酸(Gly) の後に三アミノ酸(Thr Thr Gly) が挿入している以外は、配列番号1と同様である。本明細書において特に断らない場合は、配列番号1を基準に説明する。最初のATG(メチオニンをコードする開始コドン)からTGA(811−813)のストップコドンまで810bpのオープンリーディングフレームが存在し、270アミノ酸残基がコードされている。このcDNAがコードしている変性低密度リポ蛋白質受容体mRNAには過渡的に発現されるサイトカインや成長因子と同様に3’非翻訳領域にAUUUAというmRNAを不安定化する配列が7ヶ所存在する。このポリペプチドには、一ヶ所において26個の疎水性アミノ酸(配列番号1におけるアミノ酸番号31〜56、配列番号2におけるアミノ酸番号34〜59)からなる膜貫通領域と思われる部分があり、膜貫通部分のC末端側に4つの糖鎖付加部位(配列番号1におけるアミノ酸番号69、135、179および208、配列番号2におけるアミノ酸番号72、138、182および211)が見出された。
【0016】次に、ヒト肺より抽出したPoly(A)+RNAの逆転写によって調製されたcDNAライブラリーから、ヒトの変性低密度リポ蛋白質受容体をコードするDNA配列が単離された。このライブラリーのスクリーニングは、ランダムプライマーを用いて[a-32P]dCTP により標識したpBMLR1のXhoI/PstI 切断断片とのプラークハイブリダイゼーション法により行った。ハイブリダイゼーションは50mM Tris-HCl(pH 7.5), 1M NaCl, 1% SDS, 0.2g/l Yeast tRNA 中で55℃で行い、55℃の2xSSC/0.1% SDS中で15分間3回洗浄した後、オートラジオグラフィーにより陽性クローンを同定した。このクローンは単離され、ヒトの変性低密度リポ蛋白質受容体のcDNA配列を決定するために挿入断片の配列が調べられた。この配列は蛋白質をコードする領域の一部のみを含むと考えられた。そこで、ヒト胎盤より抽出したPoly(A)+RNAの逆転写によって調製されたcDNAより、得られた配列を元に5’−RACE(rapid amplification of cDNA end) 法を用いて、蛋白質全体をコードする領域のcDNAを得て、配列が調べられた。配列番号3がヒトの変性低密度リポ蛋白質受容体のDNA配列およびアミノ酸配列である。ヒトの配列は、ウシの配列と同様に、最初のATG(メチオニンをコードする開始コドン)からTGA(811−813)まで、810bpのオープンリーディングフレームが存在し、270アミノ酸残基がコードされている。このcDNAがコードするポリペプチドには、ウシの配列と同様に一ヶ所において27個の疎水性アミノ酸からなる膜貫通領域と思われる部分があり、膜貫通部分のC末端側に4つの糖鎖付加部位(アミノ酸番号69、135、179および206)が見出された。ウシおよびヒトのアミノ酸配列は、共にC型レクチンの細胞外ドメインに持っており、この部分(アミノ酸番号140−270)が変性LDLとの結合に重要と考えられる。従って、アミノ酸番号140−270の部分配列を有するペプチドも、変性LDLとの結合機能を有すると推定できる。
【0017】本発明は、以上述べた変性低密度リポ蛋白質受容体をコードするDNA配列、および上記アミノ酸番号140−270の部分配列をコードするDNA配列を提供する。このDNA配列は、哺乳類、微生物による調節を受ける組換え体転写単位、またはウイルスの転写或いは翻訳調節要素内で発現され得る形で提供されることが好ましい。例えば、微生物内で発現される配列は、イントロンを含まないものである。好ましい態様では、本DNA配列は少なくとも1つ、しかし随意に1つ以上のcDNA配列或いはそれについての複製由来の配列成分からなる。このような配列は、合成オリゴヌクレオチドを組み上げることにより調製されたDNA配列に連結しているか、あるいは隣接している。しかし、主にオリゴヌクレオチドから組み上げられた合成遺伝子は、ここで与えられる配列情報を用いることにより構築することができる。典型的な配列は、前述したものと実質的に同一な配列を含んでいる。コード配列は、N−末端に位置する、例えばヌクレオチド配列上で翻訳フレームと連結したメチオニンを特定するN−末端のATGコドンのような、1つ以上の付加的アミノ酸をコードするコドンを含むことができる。遺伝子コードの縮重のため、同じアミノ酸配列をコードするヌクレオチド配列にも相当の多様性がある。前述した典型的なDNA配列以外の態様には、中等度にストリンジェントな条件(すなわち、42℃、20%(v/v)ホルムアミド存在下)で、典型的なDNA配列にハイブリダイズすることができる配列が含まれる。その他の配列は生物学的な活性を有する変性低密度リポ蛋白質受容体ペプチドをコードする上記の配列に縮重している。
【0018】以上の配列は、微生物もしくはウイルスのオペロン由来の誘導可能な制御要素を含む組み換え転写単位中で発現させることが可能である。本発明はまた、有用な量の精製された変性低密度リポ蛋白質受容体を生産するための発現ベクターを提供する。本発明のベクターは、哺乳類、微生物、酵母、バクテリオファージあるいはウイルス遺伝子由来の調節要素に使用可能な状態に連結された哺乳類の変性低密度リポ蛋白質受容体、あるいは生物学的に等価な類似物をコードする合成あるいはcDNA由来のDNA断片からなることができる。使用できる調節要素は以下に詳細に述べる。適当な細胞系列への形質転換、形質導入あるいは感染によって、このようなベクターは組換え体蛋白質の発現を誘導することができる。哺乳類の変性低密度リポ蛋白質受容体は、適当なプロモーターの調節の下、哺乳類細胞、酵母、細菌、あるいはその他の細胞中で発現することができる。無細胞翻訳系によっても、本発明のDNA構成物由来のmRNAを用いて哺乳類の変性低密度リポ蛋白質受容体を生産することができる。細菌、真菌、酵母および哺乳類細胞宿主に用いるための適当なクローニングおよび発現ベクターは、ポウエル(Pouwel)ら(Cloning Vectors: A Laboratory Manual, エルスビュー社、ニューヨーク州、(1985))によって述べられている。
【0019】種々の哺乳類細胞培養系を組換え体蛋白質の発現に用いることができる。適当な哺乳類宿主細胞系列としては、グルツマン(Gluzman) (Cell, 23, 175 (1981)) により述べられたサル腎臓細胞のCOS−7系列および、例えばC127、3T3、CHO、HeLaおよびBHK細胞系列などのベクターを発現可能な細胞系列が含まれる。哺乳類発現ベクターは、複製開始点、適当なプロモーターおよびエンハンサーのような非転写要素、5’あるいは3’に隣接する非転写配列、また必須のリボソーム結合部位、ポリアデニル化部位、スプライスドナー(splice donor)およびアクセプター部位のような5’或い3’の非翻訳配列、および終止配列等を含んでいる。例えば、SV40複製開始点、初期プロモーター、エンハンサー、スプライスおよびポリアデニル化部位等のSV40ウイルスゲノム由来のDNA配列は、異種DNA配列の発現に必要とされるその他の遺伝的要素を与えるために用いられる。典型的なベクターは、岡山とバーグ(Berg)(Mol.Cell. Biol., 3, 280 (1983)) によって明らかにされた通りに構築することができる。
【0020】C127ネズミ乳腺表皮細胞で、哺乳類受容体cDNAを安定かつ高レベルに発現するために有用な系は、コスマン(Cosman)ら(Molecular Immunol., 23,935 (1986)) により述べられていることに従って構築することができる。酵母の系では、サッカロミセス・セレビシエのようなサッカロミセス属の種を用いることが好ましい。ピキア(Pichia)あるいはクリュベロミセス(Kluyveromyces )等の、他の属の酵母も、組換え体蛋白質の生産株として用いることができる。一般に、有用な酵母ベクターは複製開始点と、例えば大腸菌のアンピシリン耐性Ampr 遺伝子とサッカロミセス・セレビシエのTRP1遺伝子のような、酵母と大腸菌の両方で形質転換に用いることが可能な選択マーカー及び下流の構造遺伝子の転写を誘導する酵母の高発現遺伝子由来のプロモーターを含んでいる。このようなプロモーターは3−ホスホグリセリン酸リン酸化酵素で、α−因子、酸性ホスファターゼおよび熱ショック蛋白質等、高率で発現される遺伝子をコードする酵母の転写単位に由来する。この異種構造配列は、翻訳開始および終止配列並びに好ましくは、翻訳された蛋白質を細胞外の培地中へ分泌することを可能にするリーダー配列とともに適当なフレームに組み上げられる。上記の異質配列は、N−末端の同定可能なペプチドまたは、例えば発現された組換え体産物の安定化を行う、あるいは精製が単純になるような所望の特徴を与える配列を含む融合蛋白質をコードすることも任意に可能である。
【0021】有用な酵母ベクターは、大腸菌内での選択および複製のためにpBR322由来のDNA配列(Ampr 遺伝子と複製開始点)およびグルコースによって抑制されるアルコール脱水素酵素2(ADH2)プロモーターを含む酵母のDNA配列を用いることにより組み上げることができる。ADH2プロモーターはルッセル(Russell )ら(J. Biol. Chem., 258, 2674 (1982)) とベイヤー(Beier )ら(Nature, 300, 724 (1982))によって開示されている。これらのベクターはまた、酵母TRP1遺伝子を選択マーカーとして持ち、酵母2μ複製開始点を含んでいる。酵母リーダー配列、例えば酵母宿主からの異種蛋白質の分泌を導くα−因子のリーダーは、プロモーターと発現される構造遺伝子との間に挿入することができる(U.S. Patent No. 4,546,082 ;Kurianet al., Cell, 30, 933 (1982);およびBittner et al., Proc. Natl. Acad. Sci. USA 81, 983(1984))。リーダー配列はその3’末端付近に、外来の遺伝子とそのリーダー配列の融合を促進するための、一つ以上の有用な制限酵素切断部位を含むように修飾することができる。
【0022】適当な酵母の形質転換法は当業者にはよく知られている;典型的な技法は、ヒネン(Hinnen)ら(Proc. Natl. Acad. Sci. USA 75:1978)によって述べられており、トリプトファン陽性となった形質転換体を、0.67%酵母窒素源、0.5%カザアミノ酸、2%ブドウ糖、10μg/mlアデニン、20μg/mlウラシルからなる選択培地上で選択する。ADH2プロモーターからなるベクターによって形質転換された宿主株は、1%酵母抽出物、2%ペプトン、1%ブドウ糖、80μg/mlアデニン、80μg/mlウラシルを含む栄養豊富な培地で発現のために生育させる。ADH2プロモーターの脱制御は培地中のブドウ糖が消費されてしまったときに起きる。粗酵母上清は濾過によって集められ、続いての精製の前に4℃に保つ。
【0023】細菌で使用する有用な発現ベクターは、哺乳類の変性低密度リポ蛋白質受容体をコードするDNA配列を適当な翻訳開始および終止信号とともに機能を持つプロモーターに読み枠を合わせて挿入することによって構築される。本ベクターは、一つ以上の表現型の選択マーカーと宿主内での増殖を保証する複製開始点を含んでいる。形質転換に適した原核生物宿主には、大腸菌、枯草菌(Bacillus subtilis) 、サルモネラ・ティフィムリウム(Salmonella typhimurium)、およびシュードモナス(Pseudomonas) 属に含まれる様々な種ストレプトマイセス(Streptomyces)、スタフィロコッカス(Staphylococcus)などが含まれるが、他のものも選択の対象として取ることもできる。発現ベクターは、成熟蛋白質のN−末端残基をコードするコドンに近い部位でcDNAを切断することによって都合良く構築される。次いで、コード領域の欠失した部分を「埋め合わせる」ため、或いは発現ベクター中の適当な読み枠や開始メチオニンを特定するコドンとなるようにコード断片を連結するための連結配列を提供するために合成オリゴヌクレオチドを用いることも随時可能である。
【0024】制限的な例としてではなく典型的なものとしては、細菌で用いる有用な発現ベクターは、良く知られたクローニングベクターであるpBR322(ATCC37017)の遺伝的要素からなる市販のプラスミド由来の選択マーカーおよび細菌の複製開始点を含むことができる。こうした市販のベクターは、例えばpKK223−3p(ファルマシア・ファインケミカルズ、ウプサラ、スウェーデン)およびpGEM1(プロジェマ・バイオテック、マジソン、ワイオミング州、米国)などを含んでいる。これらのpBR322の「主鎖」部分は、適当なプロモーターおよび発現される構造配列と結合している。特に有用な細菌の発現系は、λファージのPL プロモーターとc1857熱不安定なリプレッサーを用いている。アメリカン・タイプ・カルチャー・コレクションから得られるλPL 由来のプロモーターを取り入れたプラスミドベクターには、大腸菌株JMB9(ATCC37092)に内在した形のpHUB2プラスミドと大腸菌株RR1(ATCC53082)に内在した形のpPLc28がある。大腸菌内での発現に有用なその他のプロモーターとしては、スツデェー(Studier) ら(J. Mol. Biol. 189:113,1986)によって述べられているT7RNAポリメラーゼのプロモーター、ラウェー(Lauer)(J. Appl. Genet. 1:139-147, 1981)によって記載されておりATCC37121として得られるlacZプロモーター及びマニアチス(Maniatis)(Molecular Coloning;A Laboratory Manual, ColdSpring Harbor Laboratory, 1982.p.412) によって述べられているATCC37138として得られるtacプロモーターがある。
【0025】適当な宿主株を形質転換し適当な細胞濃度までその宿主株を増殖させた後、選択されたプロモーターを適当な方法(例えば、温度シフトあるいは化学的誘導)で脱抑制し、細菌を更に短期間培養する。細胞は普通遠心により集め、物理的あるいは化学的方法で破砕し、その結果生じた粗抽出液をその後の精製のために残しておく。例えば、細胞を最大の通気と激しい撹拌を行なう条件下で、10リットルの培養器中で増殖させる。消泡剤(Antifoam A)を用いるのが好ましい。培養はモット(Mott)ら(Proc. Natl. Acad. Sci. USA 82:88, 1985)によって開示された超誘導培地中30℃で行なうか、あるいはそれに代るものとしては、抗生物質を含んだ培地で培養し、A600 が0.4−0.5に相当する細胞濃度で温度を42℃にあげることによって脱抑制し、次いで温度シフト後、3−6時間が好ましいが、2−20時間の間に細胞を集める。細胞は、初め濾過あるいはその他の方法で濃縮し、次に10.000xgで10分間、4℃で遠心後、直ちに細胞のペレットを凍結する。好ましくは、精製された哺乳類の変性低密度リポ蛋白質受容体あるいは生物学的に等価な類似体は、培養液から精製される本発明の合成遺伝子の組換え体翻訳産物を発現するために適した宿主/ベクター系を培養することによって調製される。
【0026】精製された変性低密度リポ蛋白質受容体を生産する別の方法は、細胞培養上清あるいは抽出液からの精製を含んでいる。この方法では、有用な量の蛋白質を作る細胞系列を用いる。そのような細胞系列からの上清は、例えばアミコン社あるいはミリポア・ファルコン社の限外濾過装置と言った市販の蛋白質濃縮濾紙を用いて随時濃縮することができる。濃縮操作に続き、濃縮液を既に述べたような適当な精製用マトリックスにかける。例えば、適当なアフィニティーマトリックスは、適当な支持体に結合した変性低密度リポ蛋白質受容体あるいはレクチンまたは抗体分子などから成っている。それに代るものとして、例えばジエチルアミノエチル(DEAE)基を持つ担体あるいはマトリックスのような陰イオン交換樹脂が用いられる。マトリックスとしては、アクリルアミド、アガロース、デキストラン、セルロースあるいはその他の蛋白質精製に通常用いられているようなものが使用可能である。また、その代りとして、陽イオン交換樹脂を用いることも可能である。適当な陽イオン交換体としては、スルフォプロピル基あるいはカルボキシメチル基からなる種々の不溶性マトリックスを含んでいるスルフォプロピル基が好ましい。最後に、メチルあるいはその他の脂肪族基を有するシリカゲルなどの疎水性充填剤RP−HPLCを用いた一種以上の逆相系高速液体クロマトグラフィーRP−HPLCを、変性低密度リポ蛋白質受容体組成物を更に精製するために用いることが可能である。上述の精製段階のいくつかあるいは全てを様々に組み合わせることにより、均一な組換え体蛋白質を得ることが可能だろう。
【0027】細菌の培養で生産された組換え体蛋白質は通常細胞ペレットからの抽出を始めとして、一工程以上の濃縮、塩析、水相のイオン交換あるいはゲル濾過クロマトフラフィーの段階を経て単離される。最後に、高速液体クロマログラフィー(HPLC)を最終精製段階として用いることができる。組換え体哺乳類変性低密度リポ蛋白質受容体の発現に用いられた微生物細胞は、凍結−融解の繰り返し、超音波処理、物理的破砕、あるいは細胞溶解試薬を用いるなど、どのような都合の良い方法によっても破壊することができる。哺乳類の変性低密度リポ蛋白質受容体を分泌タンパクとして発現する酵母の発酵は精製を大いに容易にしている。大規模な発酵の結果得られる分泌された組換え体蛋白質はウルダル(Urdal) ら(J. Chromatog. 296:171,1984)によって明らかにされた方法と類似した方法によって精製することが可能である。この引用文献には、組換え体ヒトGM−CSFの精製のための調製用HPLCカラムにおける、二段階の連続した逆相系HPLC処理が述べられている。
【0028】種々の態様において、本発明は内在性物質の混入無しに、実質的に均一で、本来のグリコシル化様式を伴なったあるいは伴なわない組換え体哺乳類変性低密度リポ蛋白質受容体ポリペプチドを提供している。本発明における組換え体である変性低密度リポ蛋白質受容体の蛋白質には、微生物中での発現あるいは微生物によって発現された蛋白質の精製を助けるような適当なペプチドあるいは蛋白質配列と融合された蛋白質として発現した哺乳類の変性低密度リポ蛋白質受容体も本発明により意図されているものである。本発明の蛋白質の生物学的に等価な類似体とは、生物学的活性に不要な末端、あるいは細胞内側の残基または配列が削られた変性低密度リポ蛋白質受容体のような短縮された種々の類似体を含んでいる。ここで用いられている「変異アミノ酸配列」とは、天然の配列から故意に作られた変種のヌクレオチド配列によってコードされるポリペプチドを示している。「変異蛋白質」あるいは「類似体」は、変異アミノ酸配列を含む蛋白質を意味している。「天然の配列」は野生型あるいは天然の形の遺伝子または蛋白質と同一なアミノ酸配列あるいはヌクレオチド配列を示している。
【0029】変性低密度リポ蛋白質受容体の蛋白質を適当なプロテアーゼで切断するか、あるいは、変性低密度リポ蛋白質受容体の一部を組み替えDNAの方法により大腸菌や哺乳動物等に発現させて、可溶化したペプチド断片を得てもよい。そのようなペプチド断片も、前述した定義を有する限り、すなわち、変性低密度リポ蛋白質と結合可能である限り、本発明の範囲に含まれる。受容体を可溶化することにより、可溶化したペプチド断片を変性低密度リポ蛋白質に結合させて、リポ蛋白質を不活性化することが可能になる。すなわち、ペプチド断片を、変性低密度リポ蛋白質を原因とする疾患の治療に用いることが可能である。
【0030】
【実施例】以下、実施例について説明する。以下の実施例において、ウシ大動脈内皮細胞の変性低密度リポ蛋白質受容体のアミノ酸配列およびそれをコードするDNA配列を解明した。このようにして、配列番号1または配列番号2に示される配列が解明された結果、ヒトの哺乳類の内皮細胞の変性低密度リポ蛋白質受容体のアミノ酸配列およびそれをコードするDNA配列(すなわち配列番号3)の解明は、容易に実施することができた。他の哺乳類の内皮細胞の変性低密度リポ蛋白質受容体のアミノ酸配列およびそれをコードするDNA配列の解明も、同様に容易に実施できる。具体的には、他の哺乳類の内皮細胞あるいは適当な臓器より抽出したpoly(A)+ RNAから、配列番号1、配列番号2または配列番号3に示されるDNA配列とハイブリダイズできるものを選択することにより、他の哺乳類の内皮細胞の変性低密度リポ蛋白質受容体をコードするDNAを得ることができる。これは、下記実施例1のように、手掛かりとなる配列が存在していない状況から該当するDNAを選択する場合と異なり、当業者であれば実施が容易である。また、得られた配列の分析も、配列番号1、配列番号2または配列番号3に示されるDNA配列を参照することにより、下記実施例1の場合(参照すべき配列がない状況)よりも、容易に実施できる。なお、以下の実施例では、酸化低密度リポ蛋白質結合に関してアッセイを実施したが、アセチル化低密度リポ蛋白結合に関するアッセイも同様に実施できる。
【0031】[実施例1]Chomczynski ら(Biotechniques 15, 532 (1993))と同様の手法を用いて培養ウシ大動脈内皮細胞より抽出した全RNAから単離したPoly(A)+ の付加したmRNAを逆転写することによりcDNAライブラリーを作製した。具体的には、細胞を酸性イソチアン酸グアニジウム/フェノール溶液中で溶かし、溶解物にクロロホルムを加え、水層と有機層を遠心分離した。水層を回収し、アルコール沈澱によりさらに精製した。オリゴdTセルロースクロマトグラフィーによりpoly(A)+ RNAを単離し、二本鎖cDNAは、グブラー(Gubler)とホフマン(Hoffman)(Gene, 25, 263 (1983) )と同様の方法で調製した。具体的には、RNAはオリゴdTまたはランダムオリゴヌクレオチドをプライマーとして用いて逆転写酵素により、cDNAに複製した。cDNAを大腸菌DNAポリメラーゼIとRNaseHと共にインキュベーションすることにより二本鎖にし、その末端はさらにT4 DNAポリメラーゼとインキュベーションすることにより平滑化した。平滑末端を持つこれらのcDNAに、BstXIリンカーを付加した後、セファクリル(sephacryl) S−500HRによるゲル濾過クロマトグラフィーにより短いものを排除した。そして哺乳動物細胞用高発現プラスミドベクターpME18Sに結合させた。このpME18Sの模式図を図1(東京医科歯科大学、丸山博士より入手)に示す。
【0032】pME18SベクターはSV40の複製起点、SV40/ヒトT細胞白血病ウイルスタイプ1プロモーターを含んで創製された3.4kbのプラスミドベクターである。以上のようにしてできたpME18S上のウシ大動脈cDNAライブラリーを大腸菌(E. coli. ElectroMax DH 10B)を形質転換するのに用い、約7×105 のコロニーを得た。これらの組換え体を500mlの2xYT中で37℃にて振とう培養し、プラスミドDNAをCsCl密度勾配遠心にて調製した。Lipofectamine を用いてサルのCOS−7細胞の集密的になっていない(サブコンフルエント)細胞単層にトランスフェクトした。次に導入された配列が過渡的な発現をできるように細胞を3日間培養し、各プレートの細胞の単層を以下のように酸化低密度リポ蛋白質結合に関してアッセイした。
【0033】プレートに、15μgのDiI標識酸化低密度リポ蛋白質を含む10%牛胎児血清(FBS)含有DMEM培地5mlを加え、37℃、5%CO2 下で12時間インキュベートした。次にこの培地を除き、PBS(pH7.4)で2回洗浄後、トリプシン処理により細胞をプレートからはがした。この細胞をFACSにかけ、DiIの蛍光を測定するとともに、蛍光強度の高い細胞を回収した。回収した細胞からプラスミドを抽出し、上記の操作を4回繰り返した。このようにしてライブラリーから約7×105 の組換え体をスクリーンした後、酸化低密度リポ蛋白質受容体の発現を誘導することのできる単一のクローンpBLOX−1が導入されたCOS−7組換え体を得た。図2は、pBLOX−1を導入したCOS−7細胞および導入していない対照のCOS−7細胞のFACSによる蛍光強度分布を示すグラフである。クローンpBLOX−1の挿入断片はBluescriptII SK-プラスミドにサブクローンし、ダイデオキシ法によってDNA配列を決定した(Sanger et al., Proc.Natl. Acad. Sci. USA, 74, 5463(1977))。
【0034】[実施例2]
受容体cDNAを使用したノーザンブロットによる各組織での発現状態前記のような方法でウシの各組織からpoly(A)+RNAを抽出し、それぞれ5μgずつをホルムアルデヒド/1.1%アガロースゲル電気泳動で分離した後、ジーンスクリーンプラス膜(NEN, DuPont) に転写した。1.8kbのcDNA断片をランダムプライム法によってα−32P−dCTPで8×108 c.p.m./mgになるようにラベルしプローブとして用いた。ハイブリダイゼーションは1M塩化ナトリウム/1%SDS、250μg/mlサケ精子DNA溶液中で60℃で行ない、2×SSC/1%SDSにより洗浄した。オートラジオグラフィーは8時間行なった。11のウシ組織から抽出したpoly(A)+RNAに対してノーザンブロットを行なったところ、多量の発現がみられたのは培養内皮細胞と肺であった。
【0035】[実施例3]
CHO−K1細胞による変性低密度リポ蛋白質受容体の発現変性低密度リポ蛋白質受容体cDNA挿入断片を、bsr(blasticidin S-resistance)遺伝子とSV40ウイルス由来のプロモーターを含む発現ベクターpSV2bsrと変性低密度リポ蛋白質受容体発現プラスミドpBLOX−1を用いて、変性低密度リポ蛋白質受容体発現細胞を作成した。CHO(chinese hamster ovary )K1細胞は、10%FBS含有HamF12培地で、サブコンフルエント(subconfluent)に単層培養した。変性低密度リポ蛋白質受容体発現プラスミドpBLOX−1とpSV2bsrをLipofectamine により、CHO−K1細胞にトランスフェクトした。トランスフェクトした細胞を24時間後、約10倍の培養面積にまき直し、さらに24時間待って細胞が充分接着した後、培地を5μgのblasticidin S 含有上記培地に替えた。
【0036】これによって、トランスフェクションされたbsr遺伝子がゲノムに組み込まれた細胞のみが増殖し、コロニーを形成した。充分に成長したコロニーをペニシリンカップ法によりトリプシンにてはがし、12穴プレートにまき直して同じ培地で増殖させた。こうして単離したクローナルな細胞が、実際に変性低密度リポ蛋白質受容体を発現していることを、前記のようにDiI標識変性低密度リポ蛋白質取り込みによる細胞質内蛍光強度上昇を蛍光顕微鏡を用いて観察することにより確認した。蛍光顕微鏡写真を図3に示す。
【0037】[実施例4]
可溶性変性低密度リポ蛋白質受容体の作成変性低密度リポ蛋白質受容体cDNAをPCR法により、プライマーペア(5'-gcggatcctgtgctctcaatagattcgc-3',5'-ggggatcctgatctcataaagaaacag-3')を用いて、細胞外ドメイン(変性低密度リポ蛋白質との結合部分を含む)をコードするDNA配列(配列番号1の塩基配列の160〜813)をBamHI の制限酵素部位を付けて増殖し、BamHI で消化した後、ヒスチジンの6回繰り返し配列をtagとして大腸菌に蛋白を発現するベクターpQE10(Qiagen社製)のBamHI siteに挿入した。このプラスミドを大腸菌株XL-2Blueにトランスフォーム後、2xYT培地にて37℃で振とう培養した。600nmの吸光度が0.6になったところでIPTG(isopropylthio-β-D-galactoside)を1mMの濃度で加え、30℃で20時間さらに培養した。大腸菌を遠心にて回収し、6M塩酸グアニジン、0.1Mリン酸二水素ナトリウム、0.01Mトリス(pH8.0)にて溶解した。不溶物を遠心にて除去後、Ni−NTAアガロース(Qiagen社製)を加え、可溶性変性低密度リポ蛋白質受容体を吸着させた。Ni−NTAアガロースを8M尿素、0.1Mリン酸二水素ナトリウム、0.01Mトリス(pH8.0)、および8M尿素、0.1Mリン酸二水素ナトリウム、0.01Mトリス(pH6.3)にて洗浄後、8M尿素、0.1Mリン酸二水素ナトリウム、0.01Mトリス、0.1M EDTA(pH6.3)にて可溶性変性低密度リポ蛋白質受容体を溶出し、精製した。上記の操作により得られた可溶性変性低密度リポ蛋白質受容体は、SDS−PAGEにて均一な標品であることが示された。なお、tagは、GSTやc−myc等、他のものを用いてもよい。この場合は、精製法もそれに応じて、抗体等を用いた方法を用いることができる。また、適当な哺乳動物発現用ベクターを用いれば、組み替えDNAの操作により、同様に可溶性変性低密度リポ蛋白質受容体を作成することができる。
【0038】[実施例5]
抗LOX−1抗体の作成ポリメラーゼチェーン反応によりウシLOX−1cDNAの細胞外ドメインをコードする領域(アミノ酸61-270)を、BamHIを付加した2つのプライマー5'-ggggatcctgatctcataaagaaacag-3'と5'-gcggatcctgtgctctcaatagattcgc-3'により増幅した。増幅したcDNA断片をBamHIで消化した後、キアゲン(QIAGEN)社のpQE10ベクターのBamHI部位にサブクローンした。細胞外ドメイン蛋白の合成と生成はキアゲン社のQIA express systemを用いて行なった。実際には、上記のプラスミドによりストラタジーン(Stratagene)社の大腸菌株XL−1 blue を形質転換し、2xYT培地にて培養した。蛋白の合成は、イソプロピル・チオ−β−D−ガラクトシドを最終濃度2mMで加えることで誘導した。細胞を遠心分離により回収し、6Mグアニジン・HCl、0.1Mリン酸ナトリウム、および0.01M−Tris・HCl を含むpH8.0の溶液に溶解後、キアゲン社のNi-NTA樹脂カラムにかけてカラムクロマトグラフィーを行なった。次いで、8M尿素、0.1Mリン酸ナトリウム、及び0.01M−Tris・HCl を含むpH6.3の水溶液でカラムを洗浄した後、蛋白質を8M尿素、0.1M−EDTA、0.1Mリン酸ナトリウム、及び0.01M−Tris・HCl を含むpH6.3の溶液を用いて溶出した。バッファーを、アミコン社のCentriprep 10 にてリン酸塩緩衝生理食塩水に交換し、蛋白質を等容量のフロイント・コンプリート・アジュバントでエマルジョンにして、ウサギの肩甲骨脊椎間の皮内に隔週で接種することで免疫を行った。
免疫染色培養ウシ大動脈内皮細胞をSDS−PAGEのサンプルバッファーにて直接溶解し、SDS−PAGEにより分離し、ナイロン膜に転写した。雪印乳業株式会社製のブロックエースにてブロッキングした後、ベクトール(Vector)社の免疫染色キットを用い、ペルオキシダーゼ抱合アビジン・ビオチン複合体による標準的な方法により、上記のウサギから得られた抗血清の染色を行った。
【0039】
【配列表】
【配列番号1】
配列の長さ:1897配列の型:核酸鎖の数:一本鎖トポロジー:直線状配列の種類:DNA、蛋白質ハイポセティカル:Noアンチセンス:No起源生物名:Bos taurus組織の種類:vascular endothelial cells直接の起源ライブラリー名:Bovine aortic endothelial cells cDNA libraryクローン名:pBLOX−1配列の特徴特徴を表わす記号:CDS存在位置:1..813特徴を決定した方法:S特徴を表わす記号:polyA signal存在位置:1825..1830特徴を決定した方法:S特徴を表わす記号:polyA site存在位置:1846..1863特徴を決定した方法:S特徴を表わす記号:3’UTR存在位置:814..1863特徴を決定した方法:S特徴を表わす記号:5’UTR存在位置:−34..−1特徴を決定した方法:S配列-34 GCTT CACTCTCTCA TTCTTGGAAT ACATTTGAAA -1 ATG ACT GTT GAT GAC CCC AAG GGT ATG AAA 30 Met Thr Val Asp Asp Pro Lys Gly Met Lys 5 10 GAT CAA CTT GAT CAG AAG CCA AAT GGC AAG 60 Asp Gln Leu Asp Gln Lys Pro Asn Gly Lys 15 20 ACA GCA AAA GGT TTT GTT TCC TCT TGG AGG 90 Thr Ala Lys Gly Phe Val Ser Ser Trp Arg 25 30 TGG TAC CCT GCT GCT GTG ACT CTA GGG GTC 120 Trp Tyr Pro Ala Ala Val Thr Leu Gly Val 35 40 CTT TGT CTG GGA TTA CTG GTG ACT GTT ATA 150 Leu Cys Leu Gly Leu Leu Val Thr Val Ile 45 50 TTG TTG ATA CTG CAA TTA TCC CAG GTC TCT 180 Leu Leu Ile Leu Gln Leu Ser Gln Val Ser 55 60 GAT CTC ATA AAG AAA CAG CAA GCA AAT ATT 210 Asp Leu Ile Lys Lys Gln Gln Ala Asn Ile 65 70 ACT CAC CAG GAA GAT ATC CTG GAG GGA CAG 240 Thr His Gln Glu Asp Ile Leu Glu Gly Gln 75 80 ATT TTA GCC CAG CGC CGA TCA GAA AAA TCT 270 Ile Leu Ala Gln Arg Arg Ser Glu Lys Ser 85 90 GCC CAG GAG TCA CAG AAG GAA CTC AAA GAA 300 Ala Gln Glu Ser Gln Lys Glu Leu Lys Glu 95 100 ATG ATA GAA ACC CTT GCC CAC AAG CTG GAT 330 Met Ile Glu Thr Leu Ala His Lys Leu Asp 105 110 GAG AAA TCC AAG AAA CTA ATG GAA CTT CAC 360 Glu Lys Ser Lys Lys Leu Met Glu Leu His 115 120 CGC CAG AAC CTG AAT CTC CAA GAA GTT CTG 390 Arg Gln Asn Leu Asn Leu Gln Glu Val Leu 125 130 AAA GAG GCA GCA AAC TAT TCA GGT CCT TGT 420 Lys Glu Ala Ala Asn Tyr Ser Gly Pro Cys 135 140 CCC CAA GAC TGG CTC TGG CAT GAA GAA AAC 450 Pro Gln Asp Trp Leu Trp His Glu Glu Asn 145 150 TGT TAC CAA TTT TCC TCT GGC TCT TTT AAT 480 Cys Tyr Gln Phe Ser Ser Gly Ser Phe Asn 155 160 TGG GAA AAA AGC CAG GAG AAC TGC TTG TCT 510 Trp Glu Lys Ser Gln Glu Asn Cys Leu Ser 165 170 TTG GAT GCC CAC TTG CTG AAG ATT AAT AGC 540 Leu Asp Ala His Leu Leu Lys Ile Asn Ser 175 180 ACA GAT GAA CTG GAA TTC ATC CAG CAA ATG 570 Thr Asp Glu Leu Glu Phe Ile Gln Gln Met 185 190 ATT GCC CAT TCC AGT TTC CCC TTC TGG ATG 600 Ile Ala His Ser Ser Phe Pro Phe Trp Met 195 200 GGG TTG TCA ATG AGG AAA CCC AAT TAC TCG 630 Gly Leu Ser Met Arg Lys Pro Asn Tyr Ser 205 210 TGG CTT TGG GAA GAT GGT ACT CCT TTG ACG 660 Trp Leu Trp Glu Asp Gly Thr Pro Leu Thr 215 220 CCC CAC TTG TTT AGA ATT CAG GGA GCT GTT 690 Pro His Leu Phe Arg Ile Gln Gly Ala Val 225 230 TCC CGT ATG TAT CCT TCA GGG ACC TGT GCA 720 Ser Arg Met Tyr Pro Ser Gly Thr Cys Ala 235 240 TAT ATT CAA AGG GGA ACT GTT TTT GCT GAA 750 Tyr Ile Gln Arg Gly Thr Val Phe Ala Glu 245 250 AAC TGC ATT TTA ACT GCA TTC AGT ATA TGT 780 Asn Cys Ile Leu Thr Ala Phe Ser Ile Cys 255 260 CAA AAG AAG GCG AAT CTA TTG AGA GCA CAG 810 Gln Lys Lys Ala Asn Leu Leu Arg Ala Gln 265 270 TGAATTTGAA GGATCTGGAG GAAAAGAAGG AAACCTTTGA ATTCTCTTCT GGAATTTAAG 870 CTATACTTCA TCACTTAGAT GTAAACCATT AGAGCCCAGG GAAATGCCTG CTACTGGTTG 930 AGTGCAGAAC TCCTTAGCAG AGACTGGCCC AGCTGCCTGG CACCTTGATA GCAAAAGTTG 990 CAATTCCCTC TGTATATTTT TCCCTAACTT GTTCCAAGTC CTCCCCTGCA GGACTTCAGA 1050 GAAGTCAATT TTTCTGTTTC CATTGTTTCT AAGAACTTGT TGCCTAACTC AAGGTCACAG 1110 CATTTTTCTC ACTTTTGTCC TATGCTTTCT TCTAGGCATT GTAGAGTTTT AGATTTTACA 1170 TGGAAATCTA GAACTTATTT TAGATTAATT TCTAAGTGAT ATATGGATGT ATGGAAGTTT 1230 TCTGTTTGTT TTTTGCTTGT GAGTATTCAA TTGTTTTTGC AACATTTGCT GAAAAGACTA 1290 TTCTTCCTTC ACTACATTGC CTTTGCACTG TTGTCAACAA TTATCCATAC ATGCCTGGCT 1350 CTATTTCTGG ATTTTCTATT CCTTTCCATT TATTTATTTA TTATTCTTGG CTTACAACAT 1410 CACCATGATA TTTTGAATTC TATGGTTCTT TAATATATCT TGGAATCACA TGGTAGTAGT 1470 TATTCATTGT TGTTCTTTTT TAGAGTTGTT TGGTTAATCT ATGCTTTTGT ATTTCTGTCT 1530 TAAATTGGCT TGTCCATTTC TAAAAAAACT TGAAATTTTG AATTGCACTG AATCCATACA 1590 TAAATTTAGG GAAAATTGAA TTCTTAAAAA TACTGATTTG TTCAACTCAT GAAAAAGGTG 1650 TATTGCTCTA TTTAGGTATT CCTTATTTTC TTTAAGCAAT GCTTTTTAAT GTTCTTTGTG 1710 TAGATATTGT TAGATTATCA TCATGTATTT CACATTATTT ATGCTACTGT AGATAGTATT 1770 GTTATCATTT GTTGTTCTTA TTTTCAAAGT CTTCTGCTAG TATGTAGAAT TATAATAAAG 1830 TTTGATATTA ATATTAAAAA AAAAAAAAAA AAA 1863
【0040】
【配列番号2】
配列の長さ:1906配列の型:核酸鎖の数:一本鎖トポロジー:直線状配列の種類:DNA、蛋白質ハイポセティカル:Noアンチセンス:No起源生物名:Bos taurus組織の種類:vascular endothelial cells直接の起源ライブラリー名:Bovine aortic endothelial cells cDNA libraryクローン名:pBLOX−1配列の特徴特徴を表わす記号:CDS存在位置:1..822特徴を決定した方法:S特徴を表わす記号:polyA signal存在位置:1834..1839特徴を決定した方法:S特徴を表わす記号:polyA site存在位置:1855..1872特徴を決定した方法:S特徴を表わす記号:3’UTR存在位置:823..1872特徴を決定した方法:S特徴を表わす記号:5’UTR存在位置:−34..−1特徴を決定した方法:S配列-34 GCTT CACTCTCTCA TTCTTGGAAT ACATTTGAAA -1 ATG ACT GTT GAT GAC CCC AAG GGT ATG AAA 30 Met Thr Val Asp Asp Pro Lys Gly Met Lys 5 10 GAT CAA CTT GAT CAG AAG CCA AAT GGC AAG 60 Asp Gln Leu Asp Gln Lys Pro Asn Gly Lys 15 20 ACA GCA AAA GGT ACT ACA GGT TTT GTT TCC 90 Thr Ala Lys Gly Thr Thr Gly Phe Val Ser 25 30 TCT TGG AGG TGG TAC CCT GCT GCT GTG ACT 120 Ser Trp Arg Trp Tyr Pro Ala Ala Val Thr 35 40 CTA GGG GTC CTT TGT CTG GGA TTA CTG GTG 150 Leu Gly Val Leu Cys Leu Gly Leu Leu Val 45 50 ACT GTT ATA TTG TTG ATA CTG CAA TTA TCC 180 Thr Val Ile Leu Leu Ile Leu Gln Leu Ser 55 60 CAG GTC TCT GAT CTC ATA AAG AAA CAG CAA 210 Gln Val Ser Asp Leu Ile Lys Lys Gln Gln 65 70 GCA AAT ATT ACT CAC CAG GAA GAT ATC CTG 240 Ala Asn Ile Thr His Gln Glu Asp Ile Leu 75 80 GAG GGA CAG ATT TTA GCC CAG CGC CGA TCA 270 Glu Gly Gln Ile Leu Ala Gln Arg Arg Ser 85 90 GAA AAA TCT GCC CAG GAG TCA CAG AAG GAA 300 Glu Lys Ser Ala Gln Glu Ser Gln Lys Glu 95 100 CTC AAA GAA ATG ATA GAA ACC CTT GCC CAC 330 Leu Lys Glu Met Ile Glu Thr Leu Ala His 105 110 AAG CTG GAT GAG AAA TCC AAG AAA CTA ATG 360 Lys Leu Asp Glu Lys Ser Lys Lys Leu Met 115 120 GAA CTT CAC CGC CAG AAC CTG AAT CTC CAA 390 Glu Leu His Arg Gln Asn Leu Asn Leu Gln 125 130 GAA GTT CTG AAA GAG GCA GCA AAC TAT TCA 420 Glu Val Leu Lys Glu Ala Ala Asn Tyr Ser 135 140 GGT CCT TGT CCC CAA GAC TGG CTC TGG CAT 450 Gly Pro Cys Pro Gln Asp Trp Leu Trp His 145 150 GAA GAA AAC TGT TAC CAA TTT TCC TCT GGC 480 Glu Glu Asn Cys Tyr Gln Phe Ser Ser Gly 155 160 TCT TTT AAT TGG GAA AAA AGC CAG GAG AAC 510 Ser Phe Asn Trp Glu Lys Ser Gln Glu Asn 165 170 TGC TTG TCT TTG GAT GCC CAC TTG CTG AAG 540 Cys Leu Ser Leu Asp Ala His Leu Leu Lys 175 180 ATT AAT AGC ACA GAT GAA CTG GAA TTC ATC 570 Ile Asn Ser Thr Asp Glu Leu Glu Phe Ile 185 190 CAG CAA ATG ATT GCC CAT TCC AGT TTC CCC 600 Gln Gln Met Ile Ala His Ser Ser Phe Pro 195 200 TTC TGG ATG GGG TTG TCA ATG AGG AAA CCC 630 Phe Trp Met Gly Leu Ser Met Arg Lys Pro 205 210 AAT TAC TCG TGG CTT TGG GAA GAT GGT ACT 660 Asn Tyr Ser Trp Leu Trp Glu Asp Gly Thr 215 220 CCT TTG ACG CCC CAC TTG TTT AGA ATT CAG 690 Pro Leu Thr Pro His Leu Phe Arg Ile Gln 225 230 GGA GCT GTT TCC CGT ATG TAT CCT TCA GGG 720 Gly Ala Val Ser Arg Met Tyr Pro Ser Gly 230 240 ACC TGT GCA TAT ATT CAA AGG GGA ACT GTT 750 Thr Cys Ala Tyr Ile Gln Arg Gly Thr Val 245 250 TTT GCT GAA AAC TGC ATT TTA ACT GCA TTC 780 Phe Ala Glu Asn Cys Ile Leu Thr Ala Phe 255 260 AGT ATA TGT CAA AAG AAG GCG AAT CTA TTG AGA GCA CAG 819 Ser Ile Cys Gln Lys Lys Ala Asn Leu Leu Arg Ala Gln 265 270 TGAATTTGAA GGATCTGGAG GAAAAGAAGG AAACCTTTGA ATTCTCTTCT GGAATTTAAG 879 CTATACTTCA TCACTTAGAT GTAAACCATT AGAGCCCAGG GAAATGCCTG CTACTGGTTG 939 AGTGCAGAAC TCCTTAGCAG AGACTGGCCC AGCTGCCTGG CACCTTGATA GCAAAAGTTG 999 CAATTCCCTC TGTATATTTT TCCCTAACTT GTTCCAAGTC CTCCCCTGCA GGACTTCAGA 1059 GAAGTCAATT TTTCTGTTTC CATTGTTTCT AAGAACTTGT TGCCTAACTC AAGGTCACAG 1119 CATTTTTCTC ACTTTTGTCC TATGCTTTCT TCTAGGCATT GTAGAGTTTT AGATTTTACA 1179 TGGAAATCTA GAACTTATTT TAGATTAATT TCTAAGTGAT ATATGGATGT ATGGAAGTTT 1239 TCTGTTTGTT TTTTGCTTGT GAGTATTCAA TTGTTTTTGC AACATTTGCT GAAAAGACTA 1299 TTCTTCCTTC ACTACATTGC CTTTGCACTG TTGTCAACAA TTATCCATAC ATGCCTGGCT 1359 CTATTTCTGG ATTTTCTATT CCTTTCCATT TATTTATTTA TTATTCTTGG CTTACAACAT 1419 CACCATGATA TTTTGAATTC TATGGTTCTT TAATATATCT TGGAATCACA TGGTAGTAGT 1479 TATTCATTGT TGTTCTTTTT TAGAGTTGTT TGGTTAATCT ATGCTTTTGT ATTTCTGTCT 1539 TAAATTGGCT TGTCCATTTC TAAAAAAACT TGAAATTTTG AATTGCACTG AATCCATACA 1599 TAAATTTAGG GAAAATTGAA TTCTTAAAAA TACTGATTTG TTCAACTCAT GAAAAAGGTG 1659 TATTGCTCTA TTTAGGTATT CCTTATTTTC TTTAAGCAAT GCTTTTTAAT GTTCTTTGTG 1719 TAGATATTGT TAGATTATCA TCATGTATTT CACATTATTT ATGCTACTGT AGATAGTATT 1779 GTTATCATTT GTTGTTCTTA TTTTCAAAGT CTTCTGCTAG TATGTAGAAT TATAATAAAG 1839 TTTGATATTA ATATTAAAAA AAAAAAAAAA AAA 1872
【0041】
【配列番号3】
配列の長さ:1313配列の型:核酸鎖の数:一本鎖トポロジー:直線状配列の種類:DNA、蛋白質ハイポセティカル:Noアンチセンス:No起源生物名:Homo sapiens組織の種類:ヒト肺、胎盤直接の起源ライブラリー名:human lung cDNA library クローン名:λhLOX−1配列の特徴特徴を表わす記号:CDS存在位置:1..822特徴を決定した方法:S特徴を表わす記号:5’UTR存在位置:−60..−1特徴を決定した方法:S特徴を表わす記号:3’UTR存在位置:823..1183特徴を決定した方法:S配列 -126 GGGGCC -121 GCACTAGTGA TTCTGGTTCG GCCCACCTCT GAAGGTTCCA GAATCGATAG TGAATTCGTG -61 ATTTTAGTTT GTTGAAGTTC GTGACTGCTT CACTCTCTCA TTCTTAGCTT GAATTTGGAA -1 ATG ACT TTT GAT GAC CTA AAG ATC CAG ACT 30 Met Thr Phe Asp Asp Leu Lys Ile Gln Thr 5 10 GTG AAG GAC CAG CCT GAT GAG AAG TCA AAT 60 Val Lys Asp Gln Pro Asp Glu Lys Ser Asn 15 20 GGA AAA AAA GCT AAA GGT CTT CAG TTT CTT 90 Gly Lys Lys Ala Lys Gly Leu Gln Phe Leu 25 30 TAC TCT CCA TGG TGG TGC CTG GCT GCT GCG 120 Tyr Ser Pro Trp Trp Cys Leu Ala Ala Ala 35 40 ACT CTA GGG GTC CTT TGC CTG GGA TTA GTA 150 Thr Leu Gly Val Leu Cys Leu Gly Leu Val 45 50 GTG ACC ATT ATG GTG CTG GGC ATG CAA TTA 180 Val Thr Ile Met Val Leu Gly Met Gln Leu 55 60 TCC CAG GTG TCT GAC CTC CTA ACA CAA GAG 210 Ser Gln Val Ser Asp Leu Leu Thr Gln Glu 65 70 CAA GCA AAC CTA ACT CAC CAG AAA AAG AAA 240 Gln Ala Asn Leu Thr His Gln Lys Lys Lys 75 80 CTG GAG GGA CAG ATC TCA GCC CGG CAA CAA 270 Leu Glu Gly Gln Ile Ser Ala Arg Gln Gln 85 90 GCA GAA GAA GCT TCA CAG GAG TCA GAA AAC 300 Ala Glu Glu Ala Ser Gln Glu Ser Glu Asn 95 100 GAA CTC AAG GAA ATG ATA GAA ACC CTT GCT 330 Glu Leu Lys Glu Met Ile Glu Thr Leu Ala 105 110 CGG AAG CTG AAT GAG AAA TCC AAA GAG CAA 360 Arg Lys Leu Asn Glu Lys Ser Lys Glu Gln 115 120 ATG GAA CTT CAC CAC CAG AAT CTG AAT CTC 390 Met Glu Leu His His Gln Asn Leu Asn Leu 125 130 CAA GAA ACA CTG AAG AGA GTA GCA AAT TGT 420 Gln Glu Thr Leu Lys Arg Val Ala Asn Cys 135 140 TCA GCT CCT TGT CCG CAA GAC TGG ATC TGG 450 Ser Ala Pro Cys Pro Gln Asp Trp Ile Trp 145 150 CAT GGA GAA AAC TGT TAC CTA TTT TCC TCG 480 His Gly Glu Asn Cys Tyr Leu Phe Ser Ser 155 160 GGC TCA TTT AAC TGG GAA AAG AGC CAA GAG 510 Gly Ser Phe Asn Trp Glu Lys Ser Gln Glu 165 170 AAG TGC TTG TCT TTG GAT GCC AAG TTG CTG 540 Lys Cys Leu Ser Leu Asp Ala Lys Leu Leu 175 180 AAA ATT AAT AGC ACA GCT GAT CTG GAC TTC 570 Lys Ile Asn Ser Thr Ala Asp Leu Asp Phe 185 190 ATC CAG CAA GCA ATT TCC TAT TCC AGT TTT 600 Ile Gln Gln Ala Ile Ser Tyr Ser Ser Phe 195 200 CCA TTC TGG ATG GGG CTG TCT CGG AGG AAC 630 Pro Phe Trp Met Gly Leu Ser Arg Arg Asn 205 210 CCC AGC TAC CCA TGG CTC TGG GAG GAC GGT 660 Pro Ser Tyr Pro Trp Leu Trp Glu Asp Gly 215 220 TCT CCT TTG ATG CCC CAC TTA TTT AGA GTC 690 Ser Pro Leu Met Pro His Leu Phe Arg Val 225 230 CGA GGC GCT GTC TCC CAG ACA TAC CCT TCA 720 Arg Gly Ala Val Ser Gln Thr Tyr Pro Ser 235 240 GGT ACC TGT GCA TAT ATA CAA CGA GGA GCT 750 Gly Thr Cys Ala Tyr Ile Gln Arg Gly Ala 245 250 GTT TAT GCG GAA AAC TGC ATT TTA GCT GCC 780 Val Tyr Ala Glu Asn Cys Ile Leu Ala Ala 255 260 TTC AGT ATA TGT CAG AAG AAG GCA AAC CTA AGA GCA CAG 819 Phe Ser Ile Cys Gln Lys Lys Ala Asn Leu Arg Ala Gln 265 270 273 TGAATTTGAA GGCTCTGGAA GAAAAGAAAA AAGTCTTTGA GTTTTATTCT GGAATTTAAG 879 CTATTCTTTG TCACTTGGGT GCCAAACATG AGAGCCCAGA AAACTGTCAT TTAGCTGGCT 939 GCAGAACTCC TTTGCAGAAA CTGGGGTTCC AGGTGCCTGG CACCTTTATG TCAACATTTT 999 TGATTCTAGC TATCTGTATT ATTTCACCTA GCTTGTCCCA AGCTTCCCTG CCAGCCTGAA 1059 GTCCATTTTC CCCTTTTTAT TTTAAAATTT GACTCCTCTT CAAGCTTGAA AACCCTCTGA 1119 ACTCAGTCTT CTTTACCTCA TTATCACCTT CCCCTCACAC TCCTAAAATT GCATGAAAGA 1179 CAGACCGGAA TTC 1192
【図面の簡単な説明】
【図1】実施例1で用いた哺乳動物細胞用高発現プラスミドベクターpME18Sの模式図である。
【図2】pBLOX−1を導入したCOS−7細胞および導入していない対照のCOS−7細胞のFACSによる蛍光強度分布を示すグラフである。
【図3】DiI標識変性低密度リポ蛋白質投与による細胞質内蛍光強度上昇を示す蛍光顕微鏡写真である。
【特許請求の範囲】
【請求項1】 配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列。
【請求項2】 配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列を有するcDNAクローン。
【請求項3】 42℃、20%(v/v)ホルムアミド存在下で、配列番号1、配列番号2または配列番号3に示すDNA配列のクローンとハイブリダイズできて、かつ変性低密度リポ蛋白質と結合する機能を有する蛋白質をコードしているDNA配列。
【請求項4】 配列番号1、配列番号2または配列番号3に示すDNA配列と遺伝的コードの結果として縮重していて、かつ変性低密度リポ蛋白質と結合する機能を有する蛋白質をコードしているDNA配列。
【請求項5】 微生物もしくはウイルスのオペロン由来の誘導可能な制御要素を含む組み換え転写単位中で発現させることが可能な、配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体のアミノ酸配列を実質的にコードしている合成遺伝子からなるDNA配列。
【請求項6】 配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列からなる組換え発現ベクター。
【請求項7】 配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列を含む組換え発現ベクターを宿主細胞に挿入し、この細胞を発現を引き起こす条件下で培養することからなる哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を生産する方法。
【請求項8】 組換え細胞培養により生産された、配列番号1、配列番号2または配列番号3に示すアミノ酸配列を有する哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を含むタンパク質組成物。
【請求項9】 配列番号1、配列番号2または配列番号3に示すアミノ酸配列を有する哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を含む、変性低密度リポ蛋白質を検出するための薬剤。
【請求項10】 配列番号1、配列番号2または配列番号3に示すアミノ酸配列の内、アミノ酸番号140から270迄のアミノ酸配列を含むペプチド。
【請求項11】 配列番号1、配列番号2または配列番号3に示すアミノ酸配列のうち、アミノ酸番号140から270迄のアミノ酸配列をコードする領域から実質的になるDNA配列。
【請求項1】 配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列。
【請求項2】 配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列を有するcDNAクローン。
【請求項3】 42℃、20%(v/v)ホルムアミド存在下で、配列番号1、配列番号2または配列番号3に示すDNA配列のクローンとハイブリダイズできて、かつ変性低密度リポ蛋白質と結合する機能を有する蛋白質をコードしているDNA配列。
【請求項4】 配列番号1、配列番号2または配列番号3に示すDNA配列と遺伝的コードの結果として縮重していて、かつ変性低密度リポ蛋白質と結合する機能を有する蛋白質をコードしているDNA配列。
【請求項5】 微生物もしくはウイルスのオペロン由来の誘導可能な制御要素を含む組み換え転写単位中で発現させることが可能な、配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体のアミノ酸配列を実質的にコードしている合成遺伝子からなるDNA配列。
【請求項6】 配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列からなる組換え発現ベクター。
【請求項7】 配列番号1、配列番号2または配列番号3に示す哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体をコードする領域から実質的になるDNA配列を含む組換え発現ベクターを宿主細胞に挿入し、この細胞を発現を引き起こす条件下で培養することからなる哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を生産する方法。
【請求項8】 組換え細胞培養により生産された、配列番号1、配列番号2または配列番号3に示すアミノ酸配列を有する哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を含むタンパク質組成物。
【請求項9】 配列番号1、配列番号2または配列番号3に示すアミノ酸配列を有する哺乳類の血管内皮細胞の変性低密度リポ蛋白質受容体またはその類似体を含む、変性低密度リポ蛋白質を検出するための薬剤。
【請求項10】 配列番号1、配列番号2または配列番号3に示すアミノ酸配列の内、アミノ酸番号140から270迄のアミノ酸配列を含むペプチド。
【請求項11】 配列番号1、配列番号2または配列番号3に示すアミノ酸配列のうち、アミノ酸番号140から270迄のアミノ酸配列をコードする領域から実質的になるDNA配列。
【図1】


【図2】


【図3】




【図2】


【図3】


【公開番号】特開平9−98787
【公開日】平成9年(1997)4月15日
【国際特許分類】
【出願番号】特願平7−334234
【出願日】平成7年(1995)11月30日
【出願人】(000228590)日本ケミファ株式会社 (33)
【公開日】平成9年(1997)4月15日
【国際特許分類】
【出願日】平成7年(1995)11月30日
【出願人】(000228590)日本ケミファ株式会社 (33)
[ Back to top ]

