微生物産生系の再設計法
【課題】ゲノムスケールのネットワークに関連した複雑さに対処する最適化ベースの手順を提供する。
【解決手段】ユニバーサルデータベースの反応にアクセスして、化学量論的バランスを提供するステップ1と、産生宿主内のいくつかの非ネイティブ機能を最小化する少なくとも1つの化学量論的にバランスの取れた経路を、少なくとも部分的に前記反応及び基質に基づいて識別するステップ2と、前記少なくとも1つの化学量論的にバランスの取れた経路を前記宿主に組み込んで、所望の生体内変化を提供するステップ3、4とを含む。
【解決手段】ユニバーサルデータベースの反応にアクセスして、化学量論的バランスを提供するステップ1と、産生宿主内のいくつかの非ネイティブ機能を最小化する少なくとも1つの化学量論的にバランスの取れた経路を、少なくとも部分的に前記反応及び基質に基づいて識別するステップ2と、前記少なくとも1つの化学量論的にバランスの取れた経路を前記宿主に組み込んで、所望の生体内変化を提供するステップ3、4とを含む。
【発明の詳細な説明】
【技術分野】
【0001】
本出願は、それぞれその全体が参照によって本明細書に組み込まれた2002年7月10日出願の米国特許出願第60/395,763号、2002年10月9日出願の米国特許出願60/417,511号及び2003年2月3日出願の米国特許出願第60/444,933号の変更出願である、2003年7月9日出願の米国特許出願第10/616,659号の一部継続出願である。
【0002】
本発明は、米国エネルギー省(助成金番号58855)及び全米科学財団(助成金番号BES0120277)の支援を受けたものである。したがって米国政府は本発明に関して一定の権利を有する。
【0003】
本発明は、反応の追加及び欠失によって経路変更を誘導する計算フレームワークに関する。
【背景技術】
【0004】
生物変換経路の生成は近年、かなりの関心を集めている。この目的に向けた最初の体系的研究は、Seressiotis and Baileyによって実施され(Seressiotis & Bailey, 1988)、彼らは、自身のソフトウェアの開発に人工知能の概念を利用した。これに続いて、反応及び代謝産物に対する化学量論的制約条件を繰返し満たすことに基づくアルゴリズムを利用した、グルコースとアンモニアからのリシン産生に関するケーススタディが、Mavrovouniotis et al.によって実施された(Mavrovouniotis et al., 1990)。より最近になって、原子の追跡(Arita, 2000、Arita, 2004)、酵素機能規則(enzyme function rule)及び熱力学的実現可能性制約条件(thermodynamic feasibility constraint)(Hatzimanikatis et at., 2003)に基づいて新規の生体内変化経路を識別する、エレガントグラフセオレティックコンセプト(elegant graph theoretic concept)(例えば、P-グラフ(Fan et al., 2002)及びk-最短路(shortest path)アルゴリズム(Eppstein, 1994))が創始された。これらの方法の大部分は、比較的に小さな反応データベースにそれらを適用することによって例証されたものである。約5000種の反応からなるKEGGデータベース(Kanehisa et al., 2002)などのゲノムスケールの代謝反応データベースに対するそれらの性能は劇的に低下するであろう。
【0005】
ごく最近になって、(任意の生化学特性に基づく)最小経路コスト(minimum pathway cost)の決定に基づくヒューリスティックアプローチが提案された(McShan et al., 2003)。この方法は、1つの代謝産物から他の代謝産物への変換経路を描くのに非常に成功している。しかし、この方法は、上で論じた他の全ての方法と同様、特定の経路を使用することによって得られる生成物の収率を予測することができない。さらに、これらの方法は大部分、直線的(linear)な生体内変化経路を識別し、全ての代謝産物、特に補因子のバランサビリティを保証しない。
【発明の概要】
【発明が解決しようとする課題】
【0006】
したがって、本発明の主たる目的、特徴又は利点は、ゲノムスケールのネットワークに関連した複雑さに対処する最適化ベースの手順を提供することにある。
【0007】
本発明の他の目的、特徴又は利点は、化学量論的にバランスの取れた分岐及び直線生物変換経路であって、収率の観点及び生成物形成のために必要な宿主内の非ネイティブ反応の数の観点から効率的な生物変換経路を構築するための方法を提供することにある。
【0008】
本発明の他の目的、特徴又は利点は、選択された複数の基質の評価を可能にする方法を提供することにある。
【0009】
本発明の他の目的、特徴又は利点は、産生増大目的を含む生体工学目的を達成する方法を計算的に提案するための方法を提供することにある。
【0010】
本発明の他の目的、特徴又は利点は、遺伝子欠失又は付加の候補を、生物変換経路ネットワークモデルを使用することによって決定することにある。
【0011】
本発明の他の目的、特徴又は利点は、生体工学目的を計算的に達成するための最適化されたフレキシブルでロバストな方法を提供することにある。
【0012】
本発明の他の目的、特徴又は利点は、生体工学目的を計算的に達成するための方法であって、中心的な代謝経路だけでなく、アミノ酸の生合成経路、分解経路などの他の経路をも考慮することができる方法を提供することにある。
【0013】
本発明の他の目的、特徴又は利点は、生体工学目的を計算的に達成するための方法であって、輸送速度、分泌経路又はその他の特性を最適化変数として考慮することができる方法を提供することにある。
【0014】
本発明のこれらの目的、特徴及び利点ならびに/又はその他の目的、特徴及び利点の1つ又は複数は、開示された実施形態の以下の詳細な説明及び添付の請求項を検討することによって明白となろう。
【課題を解決するための手段】
【0015】
本発明は、標的化合物の過剰産生のため、反応の追加及び欠失によって微生物ネットワークの経路変更を誘導することを目的とする「OptStrain」と呼ばれる階層型計算フレームワークを提供する。これらの化合物は例えば、生物燃料電池及び環境応用における電子又は水素から、複雑な前駆分子までに及ぶ。Universalデータベースと呼ばれる生体内変化の包括的なデータベース(5,000超の反応を含む)が編纂され、このデータベースは、複数の生体経路データベース源から反応をダウンロードし、それらの反応にキュレーション(curation)を実施することによって定期的に更新される。次いで、組合せ最適化を使用して、このUniversalデータベースから抽出された非ネイティブ機能の集合を解明し、所望の生成物形成を可能にするために検討中の産生宿主に追加する。続いて、フラックス(flux)を標的生成物からそらす競合機能を識別し、それらを除去して、増殖に結びつけられたより高い生成物収率を保証する。本発明は、化学量論的にバランスの取れた経路(stoichiometrically balanced pathway)を構築し、最大生成物収率要件を課し、最適な基質を特定し、異なる微生物宿主を評価する能力を有する統合計算フレームワークを確立することによって、従来の成果からのかなりの前進を表す。
【0016】
2つの非常に異なる生成物分子に言及することによって、OptStrainの範囲及び有用性を例証する。水素ケーススタディでは、3種の別個の産生宿主に対して2種類の異なる基質を使用して、水素収率を向上させるための反応除去戦略を特定する。対照的に、バニリンスタディでは主に、どの非ネイティブ経路をEscherichia coliに追加する必要があるかを前面に押し出す。要するに、OptStrainは、微生物株の設計に役立つ有用なツールを提供し、より重要には、将来のモデル化の発展を収容する統合フレームワークを確立する。
【0017】
OptStrainプロセスは、それらの全体が参照によって本明細書に組み込まれた7月9日出願の米国特許出願第10/616,659号、2002年7月10日出願の米国特許出願第60/395,763号、2002年10月9日出願の米国特許出願第60/417,511号、及び2003年2月3日出願の米国特許出願第60/444,933号に以前に記載されたOptKnockプロセスを含む。OptKnockプロセスは、バイオテクノロジーにおける最重要課題である化学物質又は生化学物質の産生を最適化するための組換え微生物株の体系的な発展を提供する。ゲノムスケールの代謝モデルの出現は、過剰産生につながる遺伝子操作を提案する計算手順を開発する基礎を築いた。これは、増殖資源(すなわち炭素、酸化還元電位及びエネルギー)に向けての消費が、化学量論のために、所望の生成物の産生を伴うことを保証することによって達成される。具体的には、この計算フレームワークは、仮定された細胞目的(例えばバイオマス形成)を外部から課された化学的産生標的と最大限に結びつける複数の遺伝子欠失組合せを識別する。この入れ子構造は、双対理論によって生み出された変換に基づいて解かれる2層(bilevel)最適化問題を与える。このフレームワークのこの手順は、バイオマス形成を化学的産生と結びつけることによって、過剰産生突然変異体を間接的に発展させるための増殖選択/適応系を提案する。
【0018】
OptKnockはさらに、中心的な代謝ネットワークの遺伝子だけでなく、アミノ酸生合成経路及び分解経路をも含む戦略を含むことができる。遺伝子欠失に加えて、二酸化炭素、アンモニア及び酸素の輸送速度、ならびに主要代謝産物のための分泌経路を、フレームワークの最適化変数として導入することができる。したがって、本発明は、ゲノムスケールのネットワークに関連した複雑さに対処するため、ロバストでフレキシブルである。
【図面の簡単な説明】
【0019】
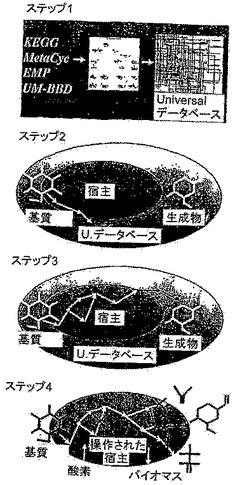
【図1】OptStrain手順を示す図である。ステップ1は、元素的にバランスの取れた反応だけからなるUniversalデータベースを編纂するための反応データベースのキュレーションを含む。ステップ2は、反応の出所に関して一切考慮せずに、基質(例えばグルコース、メタノール、キシロース)から生成物(例えば水素、バニリン)への所望の生体内変化を可能にする経路を識別する。宿主のネイティブ反応と非ネイティブ反応の両方が存在することに留意されたい。ステップ3は、非ネイティブ反応に対する依存を最小化し、ステップ4は、非ネイティブ機能を、微生物宿主の化学量論モデルに組み込み、OptKnock手順を適用して、標的生成物と競合する反応を識別し、排除する。(×)は、欠失反応を指示する。
【図2】さまざまな基質の最大水素収率を重量ベースで示すグラフである。
【図3】水素産生包絡線を、好気的及び嫌気的条件下の野生型E.coliネットワークならびに2反応及び3反応欠失突然変異体ネットワークのバイオマス産生速度の関数として示すグラフである。ベースグルコース取込み速度は10mmol/gDW/hrに固定されている。これらの曲線は、さまざまなバイオマス形成速度において最大及び最小水素産生速度を求めることによって作成されて物である。点Aは、嫌気的条件下の野生型ネットワークの最大バイオマス形成速度における必要な理論水素産生速度を表す。点B及びCはそれぞれ、対応する二酸化炭素輸送速度をOptknockによって提案される値に固定した後の2つの突然変異体ネットワークの最大増殖速度における理論的水素産生速度を識別する。
【図4】野生型(実線)及び突然変異体(破線)Clostridium acetobutylicum代謝ネットワークの、ベースグルコース取込み速度1mmol/gDW/hrでの水素形成限界を示すグラフである。線ABは、野生型ネットワークに対して使用可能な異なる代替最大バイオマス収率解を表す。点Cは、最大増殖速度における突然変異体ネットワークの水素収率を示す。これは、報告されたC.acetobutylicum(45)の実験水素収率(2mol/molグルコース)と対照をなす。
【図5】増補されたE.coli代謝ネットワークの、ベースグルコース取込み速度10mmol/gDW/hrでのバニリン産生包絡線を示すグラフである。点A、B及びCはそれぞれ、1、2及び4反応欠失突然変異体ネットワークに関連した最大増殖点を表す。バイオマス産生の任意の速度でバニリン産生が保証されない野生型ネットワークとは対照的に、突然変異体ネットワークは、高レベルのバイオマス産生を達成するためにかなりのバニリン収率を必要とする。全てのケースで増殖の嫌気的モードが示唆されることに留意されたい。
【図6】Optknockの2層最適化構造を示す図である。内側の問題は、特定の細胞目的(例えばバイオマス収率の最大化、MOMA)の最適化に基づくフラックス配分を実行する。次いで外側の問題が、内側の問題の最適化に対して使用可能な主要反応へのアクセスを制限することによって、生体工学目的(例えば化学的産生)を最大化する。
【発明を実施するための形態】
【0020】
本発明は、反応の追加及び欠失によって経路変更を誘導するための方法及びシステムを提供する。それらの方法は、コンピュータ実現又はコンピュータ援用法、あるいは他の方法で自動化された方法であることが好ましい。本明細書で使用される用語「コンピュータ」は、これに限定されるわけではないが、本明細書に記載の方法を実施するのに適した多数の電子装置を含むものと幅広く解釈しなければならない。さらに、本発明はコンピュータ援用モデル化に関係するため、本発明の範囲は、本明細書に記載された特定の実施形態よりも広いこと、ならびにさまざまな環境及び文脈において本発明を適用して、さまざまな問題に、部分的にはコンピュータ実現に関連した予測可能性によって対処する方法を、当業者が理解するであろうことを、理解されたい。
【0021】
1. OptStrain
システム生物学の基本的な目標は、生体系内の世界にアクセス可能な生体内変化の完全な「パレット(palette)」を解明することである。この目標は、拡大し続ける一連の所望の生体内変化を達成する能力を有する微生物株を構築しようとするバイオテクノロジーにおける継続的な探求目標と平行してある。これらの生体内変化は、単純な前駆化学物質(Nakamura & Whited, 2003、Causey et al., 2004)又はカロテノイドなどの複雑な分子(Misawa et al., 1991)から、生物燃料電池(Liu et al., 2004)又はバッテリー(Bond et al., 2002、Bond et al., 2003)内の電子、及びバイオレメディエーション応用において重金属錯体を沈殿させる能力を有する微生物(Methe et al., 2003、Lovley, 2003、Finneran et al., 2002)に至るまでの生成物に照準を定めている。分子生物学及び組換えDNA技術における最近の進展は、産生微生物株の遺伝子内容及び発現レベルを、直接に、またそれを標的として形にすることができる新しい時代を先導した(Bailey, 1991、Stephanopoulos & Sinskey, 1993)。新しく獲得されたこれらの能力の驚くべき範囲及び多様性、ならびにバイオテクノロジー分野の応用範囲は、株最適化プロジェクトのための最適な遺伝子改変セットを演繹的に識別するためのモデル化及び計算支援が今までにもまして必要であることを暗示している。
【0022】
ゲノムスケール微生物モデルの最近の利用可能性は、株操作(strain engineering)戦略を識別することを目的とした計算法を開発するために必要な経路再構築を提供した(Bailey, 2001)。これらのモデルは、H. pylori(Schilling et al., 2002)、E.coli(Reed et al., 2003、Edwards & Palsson, 2000)、S. cerevisiae(Forster et al., 2003)及び他の微生物(David et al., 2003、Van Dien & Lindstrom, 2002、Valdes et al., 2003)に対してすでに使用可能であり、連続的に洗練された微生物の代謝能力の抽象化を提供する。注釈付きのゲノムからの化学量論モデルの構築を促進する自動プロセス(Segre et al., 2003)は、いくつかの微生物の代謝再構築をさらに加速させそうだ。同時に、個々の反応は、KEGG、EMP、MetaCyc、UM-BBDなどの多くのデータベースの中に収められて(Overbeek et al., 2000、Selkov et al., 1998、Kanehisa et al., 2004、Krieger et al., 2004、Ellis et al., 2003、Karp et al., 2002)、さまざまな種に存在している直接の又は間接的な証拠がある生体内変化の拡大し続ける包括的なコレクションを形成している。すでに数千もの反応が収められているが、特定の生物の代謝再構築(Schilling et al., 2002、Reed et al., 2003、Edwards & Palsson, 2000、Forster et al., 2003)とは異なり、これらの編纂物は、単一の種の反応だけでなく、多くの異なる種の反応を、あまりキュレーションが行われていない状態で含む。これは、現在、微生物モデルの拡大し続けるコレクションが存在し、同時に、非ネイティブ機能のこれまでになく多くの包括的な編纂物が存在することを意味する。この新しく獲得されたおびただしい量のデータは、本明細書の範囲を構成するいくつかの計算上及びモデル化上の課題を最前線に押し出した。具体的には、どうすれば、E.coliなどの既存の産生系に組み込んで、所望の新しい機能をそれらの系に与える適当な経路/遺伝子セットを、さまざまな生物データベースの中にカタログ化された数千の機能の中から体系的に選択することができるのか。続いて、どうすれば、高い生成物収率及び生存能力を保証するために排除する競合機能を識別することができるか。
【0023】
この目標を達成するための既存の戦略及び方法には、基質から標的化合物までの実現可能な全ての生物変換経路を、生化学的変換の所与のリストから探索するデータベース問合せ(Seressiotis & Bailey, 1988、Mavrovouniotis et al., 1990)が含まれる。より最近になって、原子の追跡(Arita, 2000、Arita, 2004)、酵素機能規則及び熱力学的実現可能性制約条件(Hatzimanikatis et at., 2003)に基づいて新規の生体内変化経路を識別する、エレガントグラフセオレティックコンセプト(例えば、P-グラフ(Fan et al., 2002)及びk-最短路アルゴリズム(Eppstein, 1994))が創始された。最近になってさらに、KEGGデータベース(Kanehisa et al., 2004)の中の酵素生化学反応を使用して、基質と代謝産物とをリンクする連結グラフを構築する興味深いヒューリスティック探索法が提案された(McShan et al., 2003)。しかし、これらの方法の大部分は、その代謝ネットワークの残りの部分のバランスが取れていること、ならびに補因子の使用/生成及びエネルギー収支に対する代謝上の要請(imperative)が満たされていることを保証することなく、基質を最終生成物にリンクする直線経路を生成する。
【0024】
本発明は、所望の表現型を与えるための宿主生物への非ネイティブ機能の組込み後に生成される化学量論的にバランスの取れた経路を識別する階層最適化に基づくフレームワークOptStrainを提供する。さまざまな化学量論モデル及び公開されているKEGG(Kanehisa et al., 2004)などのデータベースから一緒にプールされた数千(現在5,734)の反応からなる拡大し続けるアレイの中から、候補代謝経路を識別する。識別された経路は最大収率考慮事項を満たし、基質の選択は最適化変数として取り扱うことができることに留意されたい。それぞれの経路に関連した補因子/エネルギー要件に関する重要な情報を推論し、前述の基準に関して候補経路の比較を可能にする。先に識別した性能目標を満たしながら異種遺伝子に対する依存を連続的に最小化することによって、産生宿主選択を調べる。次いで、識別された非ネイティブ機能を触媒するのに必要な全ての酵素をコードした遺伝子セットを、アイソザイム及び多サブユニット酵素を考慮して、構築することができる。続いて、生化学的産生目的と増殖目的を切り離す競合機能を除去することによって生成物の収率を向上させるために、この増補された宿主ネットワークにおける遺伝子欠失を識別する(Burgard et al., 2003、Pharkya et al., 2003)。生成物分子サイズの点で両極端に位置する2つの異なる生成物分子(すなわち水素とバニリン)について詳細に説明することによって、OptStrafnの幅及び広がりが例示される。簡単には、あるケースでは、計算結果が、既存の株設計及び産生の実際と一致し、他のケースでは、新規の操作戦略を特定する。
【0025】
1.1 材料及び方法
取り組んだ最初の課題は、所望の生体内変化を可能にするために、特定の生物の代謝ネットワーク(例えばE.coli(Reed et al., 2003、Edwards & Palsson, 2000)、S. cerevisiae(Forster et al., 2003)、C. acetobutylicum(Desai et al., 1999、Papoutsakis, 1984)など)にどの機能を追加すべきかを識別する体系的な計算フレームワークの開発である。本発明の発明者らは、ずっと小規模ながら、この目標にすでに貢献した(Burgard & Maranas, 2001)。編纂されたデータベースのサイズが極めて大きく、また、時に相反する複数の目的を同時に満たす必要があることから、本発明者らは、図1に示されたOptStrain手順を開発した。それぞれのステップは、解く必要がある最適化問題の特定の構造及びサイズに起因する異なる計算上の課題を導入する。
【0026】
ステップ1. 本発明者らのUniversalデータベースへの反応の自動ダウンロード及び化学量論的バランスを保証するためのキュレーション。
【0027】
ステップ2. 反応の出所(すなわちネイティブ反応か又は非ネイティブ反応か)に関する制限なしで、選択された所与の基質について、生成物の最大理論収率を計算する。
【0028】
ステップ3. ステップ2で得られた最大理論収率及び最適基質が与えられた場合に検討下の産生宿主内の非ネイティブ機能の数を最小化する、化学量論的にバランスの取れた経路の識別。異なる宿主選択間の比較とともに、収率の最大化と異種遺伝子数の最小化の両方の基準を満たす代替経路が生成される。それぞれの経路に関連した補因子/エネルギー使用に関する情報もこの段階で導き出される。最後に、この段階で、適当な酵素をコードすることによって標的生体内変化の存在を保証する1つ又は複数の遺伝子セットを導き出すことができる。
【0029】
ステップ4. 使用可能な場合に、検討下の産生微生物宿主の化学量論モデルに、識別された非ネイティブ生体内変化を組み込む。次に、これらの増補されたモデルに、OptKnockフレームワークを適用して(Burgard et al., 2003、Pharkya et al., 2003)、代謝ネットワークの接続性を「形にする」ことによりその所望の生成物の産生が増殖の必須の副生物になることを保証する遺伝子欠失を提案する。OptKnockフレームワークについては後にさらに説明する。
【0030】
データベースのキュレーション。OptStrain手順の最初のステップは、さまざまな源から獲得された反応を本発明者らのUniversalデータベースにダウンロードし、それらにキュレーションを実施することから始まる。具体的には、KEGGデータベースには毎月新しい反応が組み込まれるので、本発明者らは、Perl(Brown, 1999)を使用して、KEGGデータベース内の全ての反応を定期的に自動的にダウンロードするカスタマイズされたスクリプトを開発した。次いで、別のスクリプトを使用して、全てのそれぞれの化合物中のそれぞれの元素の原子数を解析する。全ての反応の反応物及び生成物中のそれぞれのタイプの原子の数を計算し、元素的にバランスの取れていない反応を考慮から外す。さらに、化学式中の繰返し単位の数が指定されていない化合物(例えばC25H39N7O17P3S(CH2)nで表されたトランス-2-エノイル-CoA)、又は化学式中のアルキル基Rが指定されていない化合物を、ダウンロードされたセットから除去する。このステップは、ゲノムデータベース中に存在する機能の自動ダウンロード、及びそれに続くそれらの元素バランサビリティの検証を可能にし、組換え標的として使用される大規模な機能セットを形成することを可能にする。
【0031】
本発明は、多数の特定の方法を使用して、反応の持続期間及び/又はキュレーションを自動化することができることを企図する。これらの自動機能は、使用可能な資源、それらの資源へのアクセスのタイプ、及び本発明が実現される特定の環境又は文脈に関連したその他の因子に応じた多数の方法で実行することができる。
【0032】
最大収率の決定。反応セットを決定した後、ある範囲の基質選択からの標的生成物の最大理論収率を、使用される反応の数又は出所に制限を設けることなく決定する第2のステップが実行される。生成物の最大理論収率は、基質の単位取込み速度について、標的代謝産物を産生する全ての反応フラックスから標的代謝産物を消費する全ての反応フラックスを差し引き、これを、これらの反応における標的代謝産物の化学量論係数によって重み付けしたものを最大にすることによって得られる。化学量論的制約条件及び輸送条件に支配されるこの収率の最大化は、フラックスバランス解析(Flux Balance Analysis)フレームワーク(Varna & Palsson, 1994)においてしばしば遭遇する線形計画(Linear Programming:LP)問題(数学的公式化のための支援情報を参照されたい)を与える。LP問題の計算上の扱いやすさからすれば、数千の反応に対してであっても、多数の異なる基質選択を徹底的に調べることができる。
【0033】
この特定の実施形態では、生体工学目的が産生を最大にすることに関係するが、本発明は、他の生体工学目的を使用することができることを企図する。このような場合には、最大収率を決定し又は選択する代わりに、他の適当な目的又は制約条件を使用することができる。
【0034】
宿主生物に対する異種反応の最小数の識別。OptStrainの次のステップでは、最大理論収率についての知識を使用して、特定の宿主生物ネットワークに追加する必要がある非ネイティブ機能の最小数を決定する。数学的には、これは、最初に、関連反応フラックスvjをオン又はオフにするスイッチの働きをする1組の2値変数yjを導入することによって達成される。
【数1】

【0035】
2値変数yjは、反応jが活性である場合に値1をとり、反応jが不活性である場合に値0をとることに留意されたい。この制約条件は、指定された産生宿主にとって異種遺伝子である遺伝子に関連した反応にだけ課せられる。パラメータvjmin及びvjmaxは、その代謝ネットワークの化学量論に従って全てのそれぞれの反応フラックスvjを最小化及び最大化することによって計算される(Burgard & Mamas, 2001)。これは、所望の生成物に対する収率目標を満たしつつ宿主生物ネットワークに追加される遺伝子の最小数を見つけ出す混合整数線形計画(Mixed Integer Linear Programming:MILP)モデルにつながる。後に詳細に論じるこの公式化は、異種遺伝子の必要数と生成物の最大理論収率との間のトレードオフの探求、及び全ての代替最適解の反復的な識別を可能にする。このステップの最終結果は、基質から所望の生成物への効率的な変換のための全ての代替経路のランク付けされたリストを提供する、異なる経路とその対応する遺伝子補足物とからなる1つの集合である。
【0036】
宿主生物の化学量論モデルへの非ネイティブ反応の組込み。適当な宿主生物の識別の後、解析は、識別された非ネイティブ反応の集合によって増補された特定の生物の化学量論モデルに進む。しかし、微生物の代謝は、負わされた選択圧にできるだけ敏感である(例えば競合相手よりも速く成長する)ように準備されているため、微生物産生株に単純に遺伝子を追加するだけで、所望の過剰産生が得られるわけでない。微生物の生存目的は一般に、標的生化学物質の過剰産生と直接に競合する。これを防ぐため、本発明者らは、以前に開発された本発明者らの2層計算フレームワークOptKnock(Burgard et al., 2003、Pharkya et al., 2003)を使用して、一般にバイオマス収率として例示される細胞フィットネス(fitness)目的を関心の生成物の最大収率から除外する全ての機能を排除する。
【0037】
1.2 結果
微生物株最適化の計算結果は、水素及びバニリンの産生に集中する。本開示の恩恵に浴する当業者は、本発明が、本発明を単に例示するにすぎないこれらの特定の生体工学目的に限定されるものではないことを理解するであろう。この水素産生ケーススタディは、最適な産生環境を特定するために複数の基質及び微生物宿主を調べることの重要性、ならびに競合機能を排除する必要性を強調する。対照的に、バニリンケーススタディでは、非ネイティブ反応の最小数を識別することが、株設計の主要な課題であることが示される。メチロトローフMethylobacterium extorquens AM1の代謝経路(Van Dien & Lindstrom, 2002)及びKEGG反応データベース(Kanehisa et al., 2004)を一緒にプールすることによって、両方の例に対して共通の反応データベースを、(ステップ1で)概説されたとおりに構築した。
【0038】
1.2.1 水素産生ケーススタディ
効率的な微生物水素産生戦略は、水素を高い速度で形成する能力を有する最適な基質及び微生物株を選択することを要求する。最初に、本発明者らは、水素、酸素、窒素、イオウ、リン及び炭素に関してバランスの取れたカタログ化された全ての反応(約3,000反応)を組換え候補として使用して、最大収率LP公式(ステップ2)を解いた。OptStrainは、ペントース及びヘキソース、酢酸、乳酸、リンゴ酸、グリセリン、ピルビン酸、コハク酸、メタノールなどのさまざまな基質の選択を考慮したことに留意されたい。メタノール基質に対して得られた最も高い水素収率は、消費基質1gあたり0.126gであった。メタノールの水素と炭素の比は最大4対1なので、これは、驚くべきことではない。相対的に効率的ないくつかの基質の収率の比較を図2に示す。本発明者らは、メタノールの高い収率及びグルコースの使用に伴う有利なコストを動機として、メタノールとグルコースをさらに調べることとした。
【0039】
OptStrain手順の次のステップは、宿主生物内で理論最大収率を達成するための非ネイティブ機能の最小数を決定することを含む。本発明者らは、以下の3つの異なる取込みシナリオを検討した: (i)Escherichia coli(確立された産生系)での基質としてのグルコースの使用、(ii)Clostridium acetobutylicum(知られている水素産生者)でのグルコースの使用、及び(iii)Methylobacterium extorquens(知られているメタノール消費者)内でのメタノールの使用。
【0040】
1.2.1.1 Escherichia coli
MILPフレームワーク(ステップ3で説明)は、基質としてグルコースを使用すると、E.coliは、水素産生のために、非ネイティブ機能を必要としないことを正しく検証した。興味深いことに、水素産生は、陽子を還元して水素を形成するフェレドキシンヒドロゲナーゼ反応(E.C.#1.12.7.2)、又はNADHをNAD+に変換し、陽子会合によって水素を形成する水素デヒドロゲナーゼ反応(E.C.#1.12.1.2)によって可能であった。続いて、E.coli化学量論モデル(Reed et al., 2003)について、最大水素形成の上限及び下限を、好気的条件と嫌気的条件の両方で、ベースグルコース取込み速度10mmol/gDW/hrで、バイオマス形成速度(すなわち増殖速度)の関数として、調べた(図3参照)。注目すべきことに、水素の最大理論収率は好気的条件下のほうが高い。しかし、最大増殖速度で水素が形成され、増殖と結びついた産生モードに至るのは嫌気的条件下だけである(図3の点A参照)。なお、水素産生は、嫌気的条件下でギ酸を水素と二酸化炭素に変換するギ酸水素リアーゼ反応によって起こり、これは現在の実験観察(Nandi & Sengupta., 1998)と一致している。
【0041】
副生物形成を削減するための表現型制限(ステップ4)に移り、本発明者らは、水素産生と直接又は間接に競合する機能をネットワークから除去することによって、野生型E.coliネットワーク(Reed et al.,2003)における水素の産生を高めることができるかどうかを探った。このために、本発明者らは、OptKnockフレームワーク(Burgard et al., 2003、Pharkya et al., 2003)を使用して、水素産生を増殖と結びつける遺伝子欠失戦略を特定した。ここでは識別された戦略のうちの2つに光を当てる。第1の戦略(2反応欠失)は、エノラーゼ(E.C.#4.2.1.11)とグルコース6リン酸デヒドロゲナーゼ(E.C.# 1.1.1.49)の両方を除去する。エノラーゼ反応の除去は、3-ホスホグリセリン酸分岐点に向かう糖分解フラックスをセリン生合成経路に向けることによって、水素形成を強力に促進する。その後、セリンは、1炭素代謝における一連の反応に関与して、10-ホルミルテトラヒドロ葉酸を形成し、それは最終的にギ酸とテトラヒドロ葉酸に変換される。デヒドロゲナーゼ反応の除去は、グルコース6-リン酸フラックスがペントースリン酸経路へ転じることを防ぐ。第2の戦略、3反応欠失ケーススタディは、ATPシンターゼ(E.C.#3.6.3.14)、アルファケトグルタル酸デヒドロゲナーゼ及び酢酸キナーゼ(E.C.#2.7.2.1)の除去を含む。第1の反応の除去は、陽子の利用可能性を高め、他の2つの反応の欠失は、最大炭素フラックスがピルビン酸に向けられることを保証し、これは次いで、ピルビン酸ギ酸リアーゼによってギ酸に変換される。ギ酸は、ギ酸水素リアーゼによって水素と二酸化炭素に異化される。
【0042】
野生型ネットワークと突然変異体ネットワークの両方の増殖速度の関数としての水素産生限界の比較を図3に示す。突然変異体ネットワークの二酸化炭素の輸送速度は、OptKnockによって提案された値に固定され、したがってオペレーショナルインペラティブ(operational imperative)(Pharkya et al., 2003)をセットする。2反応欠失突然変異体の理論水素産生速度は、最大増殖速度(点B)で22.7mmol/gDW/hr(0.025g/gグルコース)であり、3反応欠失突然変異体は、最大増殖速度の低減と引き換えに、最大29.5mmol/gDW/hr(0.033g/gグルコース)を生成する(点C)ことに留意されたい。興味深いことに、両方の突然変異体ネットワークで、最大水素産生は酸素の取込みを要求する。これは、水素形成のためには酸欠が好ましい野生型のケースとは対照的である。注目すべきことに、ギ酸水素リアーゼは無酸素状態でしか誘導されないが、この酵素は好気的環境でも機能することが報告されている(Nandi & Sengupta, 1996)。このことは、これらの結果に基づいて実行されるどの実験的研究においても考慮されなければならない。
【0043】
1.2.1.2 Clostridium acetobutylicum
豊富な文献が、クロストリジウム属の生物を天然の水素産生系として識別している(Nandi & Sengupta, 1998、Katakoka et al., 1997、Chin et al., 2003、Das & Veziroglu, 2001)。フェレドキシンヒドロゲナーゼ(E.C.#1.12.7.2)による陽子の水素への還元が、主たる関連反応である。驚くことではないが、本発明者らは、OptStrain(ステップ3)を使用して、グルコースを基質として使用したClostridium acetobutylicumの水素産生には非ネイティブ反応が必要ない(Papoutsakis & Meyer, 1985)ことを検証した。次に本発明者らは、OptKnockフレームワークを使用して競合機能を賢明に除去することによって、E.coliの場合と同様に、水素産生を高めることができるかどうかを調べた。この目的に、本発明者らは、Papoutsakis及びその共同研究者によって開発されたClostridium acetobutylicumの化学量論モデル(Desai et al., 1999、Papoutsakis, 1984)を使用した。OptKnockは、酢酸形成反応及び酪酸輸送反応の欠失を提案した。
【0044】
後になってみれば、この欠失戦略は、ネットワーク全体のエネルギー収支を考えると合理的である。具体的には、野生型のケースでは、それぞれの酪酸分子の形成及び分泌は、2個のNADH分子の消費を必要とし、したがってネットワークの水素産生能力を低減させる。しかし、酪酸が分泌されず、その代わりにこれがリサイクルされて、アセトン及びブチリルCoAを形成する場合には、ブチリルCoAは、NADHの消費なしで酪酸に再び変換されることができる。この2反応欠失突然変異体は、わずかに低い増殖速度と引き換えに、3.17モル/モルグルコース(0.036g/gグルコース)の理論水素収率を有する(図4の点C)。特筆すべきは、この場合に、バイオマス形成と水素産生がしっかりと結びつけられていることであり、これは、最大増殖速度である範囲の水素形成速度(1.38〜2.96mmol/gDW/hr)が可能である(図4の線AB)野生型ネットワークとは対照的である。実験結果(Nandi & Sengupta, 1998)によれば、嫌気的条件下で、クロストリジウム属は、グルコース1モルあたり最高で2モルの水素しか産生することができない。実際、水素産生に対する酪酸の直接の抑制効果及び増殖を阻害する酢酸の間接効果(Chin et al., 2003)が、観察された低い水素収率の原因であることが報告されている。興味深いことに、提案された反応除去は、これらの抑制ボトルネックを直接に回避する。
【0045】
1.2.1.3 Methylobacterium extorquens AM1
本発明者らは次に、基質をグルコースからメタノールに代え、メタノールを唯一の炭素及びエネルギー源として生存する(Van Dien & Lidstrom, 2002)通性メチロトローフ、Methylobacterium extorquens AM1の水素産生を調べた。この生物は十分に研究されており(Anthony, 1982、Chistoserdova et al., 2004、Chistoserdova et al., 1998、Korotkova et al., 2002、Van Dien et al., 2003)、最近になって、その中心代謝の化学量論モデルが発表された(Van Dien & Lidstrom, 2002)。OptStrainのステップ3を使用して、本発明者らは、水素産生を可能にするためには、M. extorquensの代謝ネットワークに反応を1つ導入すればよいことを識別した。2つの候補は、陽子を水素に還元するヒドロゲナーゼ(E.C.#1.12.7.2)、あるいは以下の変換を触媒するN5,N10-メテニルテトラヒドロメタノプテリンヒドロゲナーゼである。
【0046】
E.C.#1.12.98.2: 5,10-メチレンテトラヒドロメタノプテリン⇔5,10-メテニルテトラヒドロメタノプテリン+H2
メチロトローフの中心代謝経路は、Van Dien & Lidstrom, 2002に抜粋されているように、E.coli及びクロストリジウム属の嫌気性菌には見られるヒドロゲナーゼなどの、陽子を水素に変換する反応を含まないため、追加の反応の必要性が予想される。したがって、本発明者らの知識の及ぶ限りにおいて、Pseudomonas AMI、P. methylica(Nandi & Sengupta, 1998)などのメチロトローフを使用して水素産生を達成した者はいないことは驚くべきことではない。識別された反応の追加は、生成された陽子を水素に変換する機構の欠如を指摘することによって、この結果に対するもっともらしい説明を提供する。
【0047】
1.2.2 バニリン産生ケーススタディ
バニリンは重要な香味及び香気分子である。貯蔵バニラ莢からのその低い収率が、バイオテクノロジーによってバニラを生産する研究に動機を与えた。このケーススタディでは、E.coliでグルコースからバニリンをデノボ(de novo)生産する代謝ネットワーク再設計戦略を識別する。本発明者らは最初に、OptStrainを使用し、水素を除く全ての元素に関してバランスの取れた約4,000の候補反応についてLP最適化問題を解くことによって、グルコースからのバニリンの最大理論収率を、0.63g/gグルコースと決定した(ステップ2)。本発明者らは次に、最大収率を達成するために必要な経路をE.coliに与えるためにE.coliに組み込まなければならない非ネイティブ反応の最小数を3と識別した(ステップ3)。収率と組み込む反応の最小化の両方の最適化基準を満たす、補因子の使用だけが異なる多数の代替経路を識別した。例えば、そのような1つの経路は、以下の3つの非ネイティブ反応を使用する:
(i) E.C.#1.2.1.46: ギ酸+NADH+H+⇔ホルムアルデヒド+NAD++H2O、
(ii) E.C.#1.2.3.12: 3,4-ジヒドロキシ安息香酸(又はプロトカテク酸)+NAD++ H2O+ホルムアルデヒド⇔バニリン酸+O2+NADH、及び
(iii) E.C.#12.1.67: バニリン酸+NADH+H+⇔バニリン+NAD++H2O
興味深いことに、これらのステップは本質的に、Li and Frost(1998)よる実験的研究において、組換えE.coli細胞内でグルコースをバニリンに変換するために使用されたステップと同じであり、このことは、この計算手順によって実際に、適当な操作戦略を発見できることを証明している。しかし、報告された実験による収率0.15g/gグルコースは、ネットワークの最大理論収率(すなわち0.63g/gグルコース)からはほど遠く、向上の余地が相当にあることを示していることに留意されたい。
【0048】
このことは、OptKnockを使用して代謝ネットワークを体系的に簡素化にすることによって(ステップ4)、バニリンのより高い収率に到達することが可能かどうかを調べる動機となる。ここでは、上で識別された3つの機能で増補されたE.coli代謝のゲノムスケールモデルを、OptKnockフレームワークに組み込んで、それらを欠失させることによって増殖とバニリン産生の間の結びつきを強化すると考えられる反応セットを決定する。次に、最高バニリン収率の1、2及び4ノックアウト戦略を、ベースグルコース取込み速度10mmol/gDW/hrに関して論じる。全てのケースで、OptKnockによって、バニリン産生に最も好都合な条件として嫌気的条件が選択される。一般に、OptStrainによって識別される欠失戦略は、OptStrainのステップ4に供給される特定の遺伝子追加戦略に左右されることは強調するに値する。したがって、本発明者らは、より優良であるかもしれない他の欠失戦略が、先に暗に言及した他の候補追加戦略に随伴するかどうかを試験した。バニリンケーススタディに対して、本発明者らは、欠失提案及び最大増殖速度での予想されるバニリン収率は、使用される遺伝子追加戦略の如何にかかわらず非常に類似していることを発見した。
OptStrainによって識別された第1の欠失戦略は、アセトアルデヒドデヒドロゲナーゼ(E.C.# 1.2.1.10)を除去して、アセチルCoAのエタノールへの変換を防ぐことを提案する。このネットワークのバニリン産生は、0.205hr-1の最大バイオマス産生速度で、3.9mmol/gDW/hr、又は仮定されたグルコース取込み速度に基づいて0.33g/gグルコースである。この欠失戦略では、エタノール分泌による炭素の損失を妨げることによって、バニリンの前駆代謝産物、ホスホエノールピルビン酸(PEP)及びエリトロース-4-リン酸(E4P)へフラックスが向け直される。第2の(2反応)欠失戦略は、解糖の上半分を本質的に妨げるグルコース6リン酸イソメラーゼ(E.C.#5.3.1.9)をさらに除去することを含む。これらの欠失によって、ネットワークは、エントナー-ドゥドロフ経路に強く依存して、ピルビン酸及びグリセルアルデヒド-3-リン酸(GAP)を生成し、GAPはさらに解糖の下半分でPEPに変換される。ペントースリン酸経路の非酸性部分を通して生み出されるフルクトース-6-リン酸(F6P)は、その後E4Pに変換される。0.06hr-1への最大増殖速度の低減と引き換えに、バニリン産生は、4.78mmol/gDW/hr又は0.40g/gグルコースに増大する。4反応欠失突然変異体ネットワークでは、増殖速度をあまり犠牲にすることなく、かなり高いバニリン産生レベルが予測される。この戦略では、0.052hr-1の最大増殖速度で、6.79mmol/gDW/hr又は0.57g/gグルコースのバニリン産生が得られる。OptKnockフレームワークは、酢酸キナーゼ(E.C.#2.7.2.1)、ピルビン酸キナーゼ(E.C.#2.7.1.40)、PTS輸送機構及びフルクトース6-リン酸アルドラーゼの欠失を提案する。最初の3つの欠失はPEPからフラックスの漏れを防ぎ、その代わりにそれをバニリン合成に向け直す。フルクトース-6-リン酸アルドラーゼの除去は、GAP及びジヒドロキシアセトン(DHA)へのF6Pの直接変換を防ぐ。F6PとGAPはともに、ペントースリン酸経路の非酸性分枝でE4Pを形成するために使用されることに留意されたい。DHAはさらに反応して、ジヒドロキシアセトンリン酸(DHAP)を形成することができ、そのときにPEP分子を消費する。したがって、フルクトース-6-リン酸アルドラーゼの除去は、バニリン合成のために必要なF6PとPEPの両方の利用を防ぐ。さらに、ネットワークの驚くべきフラックスの再分配は、1炭素代謝の一群の反応を使用して、10-ホルミルテトラヒドロ葉酸を形成し、これは続いてホルムアルデヒドに変換される。
【0049】
図5は、さまざまなバイオマス産生速度でバニリン形成を最大化及び最小化することによって得られた、野生型ネットワーク及び突然変異体ネットワークのバニリン産生包絡線を比較した図である。これらの欠失は、任意の増殖条件下で、高いバニリン産生レベルをネットワークに与える。
【0050】
1.3 議論
本発明のOptStrainフレームワークは、小分子だけでなく複雑な分子を過剰産生させるために、微生物系のゲノムスケール代謝ネットワーク全体を体系的に再構成することを目的とする。本発明者らは、これまでに、さまざまな宿主(すなわちE.coli、C. acetobutylicum、M. extorquens)を使用して、いくつかの異なる生成物(例えば1,3-プロパンジオール、イノシトール、ピルビン酸、電子伝達など)を調べた。上で論じた2つのケーススタディ、水素及びバニリンは、OptStrainが、株の再設計に関連した範囲の課題に対処することができることを示している。同時に、OptStrainフレームワークを用いて得られる結果の妥当性(validity)及び適合性(relevance)は、検討される反応データベース及び微生物代謝モデルの完全性(completeness)及び正確さ(accuracy)のレベルに左右されることを強調することは重要である。本発明者らは、特に水素原子に関してバランスが取れていない反応、及び本発明者らが利用した反応データベースの曖昧な反応方向性の多数の例を識別した。本発明者らの全てのケーススタディでは、それに先立ってダウンロードされた反応の慎重なキュレーションを実施した。炭素に関する反応のバランサビリティを復元することができなかったときはいつでも、その反応を考慮から除外した。本発明者らは、反応データベースの試験及び検証用の自動化ツール(Segre et al., 2003)が使用可能になるにつれて、このステップにかかる時間がより短くなるものと予想している。微生物モデルの代謝経路の純粋に化学量論的な表現は、動力学的障壁(kinetic barrier)及び調節的相互作用(例えばアロステリック制御)を考慮しないことによって、非現実的なフラックス分配につながる可能性がある。これを緩和するため、本発明は、ブール制約条件の形態の調節性情報(Covert & Palsson, 2002)をE.coliの化学量論モデルに組み込むこと、及び必要に応じて動力学的表現(kinetic expression)を使用する(Castellanos et al. 2004、Tomita et al., 1999、Varner & Ramkrishna, 1999)ことを企図する。さらに、本発明は、OptKnockを使用して、反応の欠失だけでなく、さまざまな主要反応ステップのアップ又はダウンレギュレーションをも考慮することを企図する。これらの単純化にもかかわらず、OptStrainはすでに、多くのケースで、微生物宿主再設計に対する有用な洞察を提供しており、より重要には、OptStrainは、将来のモデル化の改良に開いた統合フレームワークを初めて確立した。
【0051】
本発明の方法論を実現する際にはコンピュータが使用されることを理解されたい。本発明は、多数のコンピュータを使用することができること、及び多数のタイプのソフトウェア又はプログラム言語を使用することができることを企図する。さらに、本発明は、生み出されるネットワークの表現を記憶することを提供することを理解されたい。ネットワークの表現は、記憶装置、信号、又は生体工学によって操作された生物中に記憶することができる。
【0052】
1.4 OptStrainの数学的公式化
OptStrain手順を使用することによって生成物収率の向上を可能にする微生物代謝ネットワークの再設計は、複数のタイプの最適化問題の解を必要とする。最初の最適化作業(ステップ2)は、代謝産物の集合N={1,...,N}及び反応の集合M={1,...,M}からなる代謝ネットワークにおける所望の生成物の最大収率を決定することを含む。基質の集合Rから(集合Nの中の)特定の生成物Pへの重量ベースの収率を最大にするための線形計画(LP)問題は、下式によって公式化される。
【数2】

【数3】

上式で、MWiは代謝産物iの分子量、vjは反応jのモルフラックス、Sijは、反応jにおける代謝産物iの化学量論係数である。本発明者らの作業においては、代謝産物の集合Nが約4,800種の代謝産物からなり、反応の集合Mが5,700を超える反応からなる。制約条件(1)の不等式は、分泌のみを考慮し、Rの中の基質以外のネットワークの中の全ての代謝産物の取込みを防ぐ。制約条件(2)は、基質の合計取込みフラックス1グラムに対して結果を基準化する。反応フラックスvjは、不可逆的(すなわちvj≧0 )又は可逆的フラックスであることができ、可逆的である場合には、正又は負の値をとることができる。酸素、二酸化炭素、アンモニア、硫酸、リン酸など増殖に不可欠な化合物の取込みを可能にする反応も存在する。
【0053】
OptStrainのステップ3では、ステップ2で識別された最大収率を満たすのに必要な非ネイティブ反応の最小数を見つける。最初に、検討中の微生物宿主の代謝モデル中に存在しないUniversalデータベース中の反応に、非ネイティブのフラグを立てる。これは、以下の混合整数線形計画(MILP)問題を与える。
【数4】


集合Mnon-nativeは、検討中の宿主の非ネイティブ反応を含み、集合Mの部分集合である。制約条件(1)及び(2)は、生成物収率最大化問題における制約条件(1)及び(2)と同じである。制約条件(3)は、生成物収率が、ステップ2で計算された最大理論収率Yieldtargetを満たすことを保証する。制約条件(4)及び(5)の2値変数yjは、反応をオン又はオフにするスイッチの役目を果たす。yjの値0は、対応するフラックスvjが0であることを強制し、値1は、対応するフラックスvjが、0でない値をとることを可能にする。パラメータvjmin及びvjmaxはそれぞれ、非常に低い値及び非常に高い値をとることができ、又は、制約条件(1〜3)に従って全てのそれぞれの反応フラックスvjを最小化及び最大化することによって計算することができる。
【0054】
最大収率と非ネイティブ反応の最小化の両方の最適化基準を満たす代替経路は、整数切断(integer cut)と呼ばれる追加の制約条件の累積の後のMILP公式の反復解によって得られる。整数切断制約条件は、以前に識別された反応の全ての集合を考慮から除外する。例えば、以前に識別された経路が反応1、2及び3を利用する場合、下式の制約条件は、同じ反応が、その後の解の中で同時に考慮されることを防ぐ: y1+y2+y3≦2。さらなる詳細はBurgard and Maranas (2001)に出ている。
【0055】
OptStrainのステップ4は、非ネイティブ機能で増補されたネットワークから排除する反応を、以前に開発されたOptKnockフレームワーク(Burgard et al., 2003、Pharkya et al., 2003)を使用して識別する。このステップの目的は、ネットワークの表現型の挙動を制限して、増殖と所望の生化学物質の形成とが結びつけられるようにし、それによって副生物の形成を削減することにある。ネットワークに対して使用可能なさまざまなレベルのバイオマス形成速度について、生化学的産生を最大化し、次いで最小化する一連の線形最適化問題を解くことによって、許される標的生成物収率の包絡線をバイオマス収率に対して描く。最適化公式化に関する詳細はPharkya et al., 2003に出ている。以上の最適化問題は全て、IBM RS6000-270ワークステーション上のGAMS(Brooke et al., 1998)モデル化環境を経由してアクセスされるCPLEX 7.0(http://www.ilog.com/products/cplex/)を使用して、数分から数時間で解かれた。
【0056】
2. OptKnock
ゲノムスケールで、したがって体系的に単細胞生物の代謝を調べることができることは、株操作戦略を識別することを目的とする新規の計算法の必要性に動機を与える。本発明は、E.coliでの特定の化合物の過剰産生につながる遺伝子欠失戦略を提案するOptKnockと呼ばれる計算フレームワークを含む。これは、代謝ネットワークの接続性を「形にする」ことにより所望の化学物質の産生が増殖の必須の副生物になることを保証することによって達成される。言い換えると、OptKnockは、細胞増殖を化学的産生から分離する能力を有する代謝反応を識別し、続いてこれを欠失させる。この計算手順は、Palsson及びその共同研究者のin silico E.coliモデル(Edwards & Palsson, 2000)の中に要約されたE.coli代謝ネットワーク全体を同時に考慮することによって、直接的なノックアウト戦略だけでなく、非直感的なノックアウト戦略をも識別するように設計される。このネットワークの複雑さ及び組み込まれた冗長性(例えばこのE.coliモデルは720の反応を包含する)は、候補遺伝子ノックアウト戦略の組合せ的爆発(combinatorial explosion)と戦うために、体系的で効率的な探索法を必要とする。
【0057】
図6に示された入れ子にされた最適化フレームワークは、細胞増殖目的を、外部から課せられた化学的産生目的に最大限に結びつける複数の遺伝子欠失組合せを識別するように作られている。2つの競合する最適戦略(すなわち細胞目的と化学的産生)を含むこの多層最適化構造は、2層最適化問題(Bard, 1998)と呼ばれる。問題公式化の詳細及び線形計画(LP)双対理論を利用したエレガントな解手順については「方法」セクションで説明する。このOptKnock手順を、E.coliでのコハク酸、乳酸及び1,3-プロパンジオール(PDO)産生に適用し、固定グルコース取込み量でのバイオマス収率の最大化を細胞目的として使用する。得られた結果をさらに、代謝調整の最小化(minimization of metabolic adjustment:MOMA)(Segre et al., 2002)を細胞目的として使用して対比させる。OptKnockフレームワークに基づいて、E.coliでのコハク酸、乳酸及びPDO産生の文脈における最も有望な遺伝子ノックアウト戦略及びバイオマス産生に対するそれらの対応する許される化学物質生成の包絡線を識別することが可能である。
【0058】
本発明の好ましい一実施形態は、増殖資源(すなわち炭素、酸化還元電位及びエネルギー)のために必要な代謝産物/化合物に向けての消費が、化学量論により、所望の化学物質の産生を伴わなければならないことを保証することによってE.coliにおける化学的産生につながると考えられる遺伝子欠失戦略を提案する、OptKnockと呼ばれる計算フレームワークを記述する。したがって、所望の生成物の産生は、細胞増殖の必須の副生物になる。具体的には、OptKnockは、代謝ネットワークから除去する反応を特定し、その反応の除去は、識別された機能に関連した遺伝子を欠失させることによって実現することができる。この手順を、E.coli K-12でのコハク酸、乳酸及びPDO産生に基づいて立証した。得られた結果は、文献に発表された株と良好な一致を示す。提案された遺伝子欠失のいくつかは、所望の経路と競合する反応経路を本質的に狭めるので、非常に直接的であるのに対して、他の多くの遺伝子欠失は、E.coliの代謝ネットワークの複雑さ及び組み込まれた冗長性を反映して、最初は非常に非直感的である。コハク酸の場合、OptKnockは、この問題に対する直接の入力としてではなく、細胞目的と化学的産生目的の間の競合の結果として、嫌気的発酵及びホスホトランスフェラーゼグルコース取込み機構の欠失を正確に提案した。乳酸のケーススタディでは、グルコキナーゼベースのグルコース取込み機構が、あるノックアウト戦略に関して乳酸産生とバイオマス産生を分断することが示された。PDOの場合には、エネルギー効率は大幅に悪いにもかかわらず、エントナー-ドゥドロフ経路のほうがEMP解糖よりも有利であることを示す結果が得られた。さらに、これまで一般的であったtpiノックアウトが、PDOの最大収率を低減させることが明白に示され、一方で、15個の反応からなる複雑なネットワークが、PPP経路からTCA回路にフラックスを「漏らす」ことが理論的に可能であり、したがってPDO産生をバイオマス形成から分断することが示された。得られた結果はさらに、細胞目的の選択に関して非常にロバストであるように見えた。
【0059】
本発明は、増殖速度の最大化、ATP産生の最大化、代謝調整の最小化、養分吸収の最小化、酸化還元産生の最小化、ユークリッドノルム(Euclidean norm)の最小化、ならびにこれらの細胞目的及びその他の細胞目的の組合せを含む、多数の細胞目的を企図する。ただしこれらに限定されるわけではない。
【0060】
提案された遺伝子欠失戦略は慎重に解釈しなければならないことに留意することは重要である。例えば、多くの場合、分枝経路の一方の枝の遺伝子の欠失は、もう一方の枝の有意なアップレギュレーションと等価である。さらに、遺伝子欠失の前後のフラックス変化の検査は、どの遺伝子をアップ又はダウンレギュレートするかに関する見通しを提供する。最後に、除去の標的とされる識別された反応の集合をその対応する遺伝子にマップする問題は、一意的に指定されるとは限らない。したがって、アイソザイム及び多機能酵素を考慮した最も経済的な遺伝子セットの注意深い識別を実施する必要がある。
【0061】
OptKnockフレームワークでは、基質取込みフラックス(すなわちグルコース)を10mmol/gDW/hrとすることが好ましい。したがって、全ての報告された化学的産生値及びバイオマス形成値は、予測されたものではないこの仮定の取込みシナリオに基づく。したがって、提案された欠失突然変異体が大幅に低い取込み効率を含むことはよくあることである。しかし、OptKnockは、増殖と化学的産生とが結びつけられた突然変異体を本質的に提案するので、改良された取込み効率、したがって高められた所望の化学的産生特性を有する突然変異体を連続的に発展させる増殖選択系を想像することができる。
【0062】
フラックス配分(flux allocation)を実行する内部最適化問題の純粋に化学量論的な表現の中に調節又は動力学的情報がない場合、OptKnockは、化学的過剰産生のための唯一の機構として任意の遺伝子欠失を識別するために使用される。明らかに、モデルに調節又は動力学的情報が含まれないことは、場合によっては非現実的なフラックス分配を提案しかねない単純化である。調節情報の組込みは、フラックス配分をより適当に解くことによって、提案された遺伝子欠失の質を高めるだけでなく、株改良のための機構として遺伝子欠失とともに調節変更を提案することも可能にする。使用可能な場合には、代替モデル化法(例えばサイバネティック(cybernetic)(Kampala et al., 1984、Ramakrishna et al., 1996、Varner and Ramkrishna, 1999)、代謝制御解析(metabolic control analysis)(Kacser and Bums, 1973、Heinrich and Rapoport, 1974、Hatzimanikatis et al., 1998))の使用を、OptKnockフレームワークの中に組み込んで、遺伝子が欠失した代謝ネットワークの代謝フラックス分配をより正確に推定することができる。とはいえ、このような調節又は動力学的情報がなくても、OptKnockは、株改良のための有用な提案を提供し、より重要には、体系的なフレームワークを確立する。本発明は、代謝及び調節モデル化フレームワークの将来の改良をおのずから企図する。
【0063】
2.1 方法
グルコース基質によって活性化される代謝産物の集合N={1,...,N}及び代謝反応の集合M={1,...,M}を含む定常状態代謝ネットワークに対する総反応フラックスとして定量化される細胞目的の最大化は、数学的には以下のように表現される。
【数5】

上式で、Sijは、反応jにおける代謝産物iの化学量論係数であり、vjは、反応jのフラックスを表し、vglc_uptakeはベースグルコース取込みシナリオ、vatp_mainは、非増殖関連ATP維持要件(non-growth associated ATP maintenance requirement)、vbiomasstargetは、バイオマス産生の最小レベルである。ベクトルvは、内部反応と輸送反応の両方を含む。輸送フラックスの順(すなわち正)方向は、特定の代謝産物の取込みに対応し、逆(すなわち負)方向は代謝産物の分泌に対応する。ホスホトランスフェラーゼ系及びグルコキナーゼを介したグルコースの取込みはそれぞれ、vpts及びvglkによって表される。ネットワークから分泌されるだけの代謝産物の輸送フラックスは、Msecr_onlyの要素である。反応の完全な集合Mは、可逆反応Mrev及び不可逆反応Mirrevに再分割されることにも留意されたい。細胞目的はしばしば、バイオマス形成のために必要な比の生合成前駆物質の消費であると仮定される(Neidhardt and Curtiss, 1996)。これらのフラックスはgDW・hrあたりで報告され、そのためバイオマス形成は、g産生バイオマス/gDW・hr又は1/hrとして表現される。
【0064】
遺伝子欠失のモデル化、したがって反応除去のモデル化は最初に、このフラックスバランス解析フレームワークへの2値変数の組込みを必要とする。(Burgard and Maranas, 2001; Burgard et al., 2001)。これらの2値変数
【数6】

は、反応jが活性の場合、値1をとり、反応jが不活性の場合、値0をとる。以下の制約条件
【数7】

は、変数yjが0に場合にだけ反応フラックスvjが0にセットされることを保証する。あるいは、yjが1のとき、vjは、下限vjminと上限vjmaxの間の任意の値を自由にとることができる。この検討では、vjmin及びvjmaxが、主問題からの制約条件に従う全てのそれぞれの反応フラックスを最小化し、続いて最大化することによって識別される。
【0065】
最適遺伝子/反応ノックアウトの識別は、細胞目的の最適化が、関心の化学物質又は生化学物質の過剰産生に間接的につながるようにアクセスすることができる(yj=1)反応の集合を選択する2層最適化問題の解を必要とする(図6も参照されたい)。バイオマス形成を細胞目的として使用して、これは、以下の2層混合整数最適化問題として数学的に表現される。
【数8】

上式で、Kは、許されるノックアウトの数である。最後の制約条件は、結果として生じるネットワークが最小バイオマス収率vbiomasstargetを満たすことを保証する。
【0066】
フラックス空間の高い次元数(すなわち700を超える反応)及び2つの入れ子にされた最適化問題の存在のため、この2段階最適化問題の直接解は扱いにくい。これを正すため、本発明者らは、あらゆる線形計画問題(主問題)について、その最適目的値が主問題のそれに等しい一意的な最適化問題(双対問題)が存在することを示す、LP双対理論から借用する効率的な解法を開発する。同様の戦略が、Burgard and Maranas, 2003によって、代謝フラックスデータから代謝目的関数を識別/試験するために使用された。OptKnock内部問題に関連した双対問題(Ignizio and Cavalier, 1994)は以下のとおりである。
【数9】

上式で、λistoichは、化学量論的制約条件に関連した双対変数、glcは、グルコース取込み制約条件に関連した双対変数、μjは、主問題中の対応するフラックスvjに対する他の任意の制限に関連した双対変数である。双対変数μjは、OptKnock内部問題中の対応するフラックスが0にセットされている場合、yj=0を強制することによって、無制限の符号を獲得することに留意されたい。パラメータμjmin及びμjmaxは、双対問題の制約条件に従うそれらの値を最小化し、続いて最大化することによって識別される。
【0067】
主問題及び双対問題に対する最適解が境界にある場合、それらの目的関数値は、最適性(optimality)において互いに等しくなければならない。このことは、それらの目的を互いに等しく設定し、それらのそれぞれの制約条件を累積することによって、両方の問題に対するあらゆる最適の解を特徴づけることができることを意味する。したがって、以前に示されたOptKnockに対する2層公式化は、以下の単層(single-level)MILPに変換することができる。
【数10】


上記の公式化の重要な特徴は、問題が実行可能な場合、最適解が常に見つかることである。本発明では、遺伝子ノックアウトの候補が、解糖、TCA回路、ペントースリン酸経路、呼吸及び全てのアナプレロティック反応のうちの全ての反応を含む。ただしこれらに限定されるわけではない。これは、総和(すなわち
【数11】

)に含まれる反応の数を限定することによって達成される。100個の2値変数を含む問題は、IBM RS6000-270ワークステーション上のGAMSモデル化環境を経由してアクセスされるCPLEX 7.0を使用して、数分から数時間で解かれた。しかし、本発明は、使用される特定のタイプのコンピュータ又は環境に依存しないことを理解されたい。本発明の方法に関連した情報を入力し出力することができる任意のタイプのコンピュータ又は環境を使用することができる。さらに、本発明の方法の諸ステップは、多数のタイプのソフトウェアアプリケーション又は言語で実現することができ、本発明はこの点に関して限定されない。他の実施形態及び使用は当業者には明白であり、本発明はこれらの特定の例示的な例に限定されないことを理解されたい。
【実施例】
【0068】
2.2 実施例1
コハク酸及び乳酸産生
それらがある場合に、E.coli K-12 化学量論モデル(Edwards & Nissen, 2000)から除去して、バイオマス最大化がフラックス配分のよい記述子であるときにはいつでも残りのネットワークがコハク酸又は乳酸を産生するようにすることができる反応を識別した。予め指定された量のグルコース(10mmol/gDW/hr)、ならびに無機リン酸塩、酸素、硫酸塩及びアンモニアの制約のない取込み経路を提供して、代謝ネットワークを活性化する。この最適化ステップは、グルコースの取込みのためにホスホトランスフェラーゼ系又はグルコキナーゼ、あるいはその両方の機構を選ぶことができる。酢酸、二酸化炭素、エタノール、ギ酸、乳酸及びコハク酸の分泌経路も使用可能にされる。グルコース取込み速度は固定なので、バイオマス及び生成物収率はそれぞれ、バイオマス及び生成物産生速度と本質的に等しいことに留意されたい。全てのケースで、OptKnock手順は酸素摂取反応を排除し、現在のコハク酸(Zeikus et al., 1999)及び乳酸(Datta et al., 1995)発酵性産生戦略と矛盾しない嫌気的増殖条件を指示した。
【0069】
表Iに、コハク酸過剰産生用の識別された3つの遺伝子ノックアウト戦略(すなわち突然変異体A、B及びC)に概要を示す。突然変異体Aの結果は、ネットワークから2つの反応(すなわちピルビン酸ギ酸リアーゼ及び乳酸デヒドロゲナーゼ)を除去すると、コハク酸産生は、最大バイオマス収率におけるその理論最大値の63%に達することを示唆した。このノックアウト戦略は、Stols and Donnelly (1997)によって、彼らのコハク酸過剰産生E.coli株において使用された戦略と同一である。次に、バイオマス産生に対する許されるコハク酸の包絡線を、野生型E.coliネットワーク及び表Iに示された3つの突然変異体について調べた。コハク酸産生の限界は、突然変異体Aが、バイオマス収率が最大値の80%に近づくまで、コハク酸とバイオマス形成の結合を示さないことを明らかにした。しかし、アセトアルデヒドデヒドロゲナーゼをさらに欠失した突然変異体Bは、ずっと以前に、コハク酸とバイオマス収率との結合を示した。
【0070】
競合する副生物(すなわちエタノール、ギ酸及び乳酸)の産生機構を排除するのではなく、2つのPEP消費反応を不活化することに重点を置いた突然変異体Cでは、より非直感的な戦略が識別された。最初に、ホスホトランスフェラーゼ系を使用不能にして、ネットワークが、グルコースの取込みをグルコキナーゼだけに依存するようにした。次に、ピルビン酸キナーゼを除去し、解糖によって供給されるかなりの量のPEPを排出する能力を有する唯一の中心代謝反応としてPEPカルボキシキナーゼを残した。この戦略は、最大バイオマス収率を達成できると仮定して、理論最大値の88%に近いコハク酸収率を与えた。さらに、達成可能なあらゆるバイオマス収率に対してかなりのコハク酸産生が見られ、コハク酸の最大理論収率は野生株のそれと同じであった。
【0071】
次に、OptKnockフレームワークを適用して、乳酸産生とバイオマス産生とを結びつけるためのノックアウト戦略を識別した。表Iは、識別された3つの遺伝子ノックアウト戦略(すなわち突然変異体A、B及びC)を示す。突然変異体Aは、酢酸及びエタノール産生を妨げることによって、最大バイオマス収率におけるフラックスを乳酸に向け直す。この結果は、adh、pta突然変異体E.coli株が、グルコースを基質として、乳酸を産生することによって嫌気的に増殖することができることを証明する以前の研究(Gupta & Clark, 1989)と矛盾しない。突然変異体Bは、初期解糖反応及び酢酸産生機構の除去を含む代替戦略を提供する。これは、最大バイオマス収率で、その理論的限界の90%の乳酸収率を与える。このネットワークは、乳酸の産生を回避し、その一方でバイオマス形成を最大化することができることにも留意されたい。これは、OptKnockが、に「最悪ケース」代替解を明示的に考慮しないことによる。グルコキナーゼ及びエタノール産生機構をさらに除去した突然変異体Cは、乳酸産生とバイオマス産生の間のよりタイトな結びつきを示したことを理解されたい。
【0072】
表I-OptKnockによって識別されたさまざまな遺伝子ノックアウト戦略のバイオマス及び化学収率。それぞれのノックアウト戦略の反応及び対応する酵素が示されている。最大バイオマス及び対応する化学収率は、供給グルコース10mmol/hr及び細胞1gDWをベースに提供されている。右端の列は、野生型(欠失なし)E.coliネットワークからの代謝フラックスの最小再分配を仮定(MOMA仮定)した同じベースの化学収率を提供する。1,3-プロパンジオールの場合、両方のノックアウト戦略に対してグリセリン分泌が使用不能にされていた。
【表1】

【0073】
2.2 実施例2
1,3-プロパンジオール(PDO)産生
最適な遺伝子ノックアウト戦略を案出することに加えて、OptKnockを使用して、E.coliにおけるPDO産生など、遺伝子の欠失とともに遺伝子の追加が必要な株を設計した。微生物1,3-プロパンジオール(PDO)産生法は、グリセリンを主たる炭素源として利用してすでに開発されているが(Hartlep et al., 2002、Zhu et al., 2002)、単一の微生物でのグルコースからの1,3-プロパンジオールの直接産生は最近、かなりの関心を集めている(Cameron et al., 1998、Biebl et al., 1999、Zeng & Biebl, 2002)。野生型E.coliは、PDO産生に必要な経路を欠いているため、最初に遺伝子追加フレームワークを使用して(Burgard and Maranas, 2001)、E.coliでグルコースからPDOを産生するために必要な追加の反応を識別した。この遺伝子追加フレームワークは、グリセリンホスファターゼによるグリセリン-3Pのグリセリンへの変換、及びその後のグリセリンデヒドラターゼ及び1,3-プロパンジオールオキシドレダクターゼによるグリセリンの1,3-プロパンジオールへの変換を含む、直接的な3反応径路を識別した。次いでこれらの反応をE.coli化学量論モデルに追加し、続いてOptKnock手順を適用した。
【0074】
OptKnockは、PDO産生とバイオマス産生とが結びついた突然変異体は、1反応欠失突然変異体にも、又は2反応欠失突然変異体にもないことを明らかにした。しかし、PDO産生をバイオマス産生に結びつけることができる1つの3欠失ノックアウト戦略及び複数の4欠失ノックアウト戦略が識別された。これらのノックアウト戦略のうちの2つが表Iに示されている。それらの結果は、E.coliネットワークからある主要機能を除去すると、グルコースで増殖するPDO過剰産生突然変異体が得られることを示唆した。具体的には、グリセリンの分解を防ぐ追加のノックアウトとともに2つの解糖反応を除去すると、最大バイオマス収率におけるPDOの理論的最大収率の72%に達する能力を有するネットワークが得られることを、表Iは示している。グリセルアルデヒド-3-リン酸デヒドロゲナーゼ(gapA)ノックアウトは、DuPont社によって、そのPDO過剰産生E.coli株において使用された(Nakamura, 2002)ことに留意されたい。突然変異体Bは、同様のPDO収率及び38%高いバイオマス収率を示す、トリオースリン酸イソメラーゼ(tpi)酵素の除去を含む代替戦略を示した。興味深いことに、最近、トリオースリン酸イソメラーゼ活性を欠く酵母株が、PDOへの主要前駆物質であるグリセリンを、その最大理論収率の80〜90%で産生することが報告された(Compagno et al., 1996)。
【0075】
バイオマス収率を最大化する野生型E.coli、突然変異体A及び突然変異体Bネットワークのフラックス分配を精査すると、驚くことではないが、両突然変異体A及びBで、グリセリンからグリセリンアルデヒドへの更なる変換が途絶されたことが分かる。突然変異体Aでは、解糖の上部及び下部の2つの反応を除去した結果、ペントースリン酸経路及び解糖経路(トリオースリン酸イソメラーゼを除く)がほぼ完全に失活した。それを補うため、エントナー-ドゥドロフ解糖経路が活性化されて、グルコースからのフラックスを、ピルビン酸及びグリセルアルデヒド-3-リン酸(GAP)へ導く。GAPは次いでグリセリンに変換され、グリセリンは次いでPDOに変換される。解糖フラックスの減少に伴って、野生型E.coliネットワークのケースよりも低下したエネルギー需要は、TCA回路フラックスの増大によって満たされる。突然変異体Bに対して提案されたノックアウトは、明確に異なる機構によって、PDOの産生に向けてフラックスを向け直す。ペントースリン酸経路の最初の反応の除去の結果、代謝フラックスは全体が解糖の最初の段階に流れる。フルクトースビスリン酸アルドラーゼ接合部で、フラックスの流れは2つの代謝生成物、PDOに変換されるジヒドロキシアセトンリン酸(DHAP)と、解糖の後半へと続くGAPとに分けられる。トリオースリン酸イソメラーゼ反応の除去は、DHAPとGAPの間の相互変換を防ぐ。興味深いことに、第4のノックアウトは、バイオマス形成と化学的産生の間の結びつきを維持することが予測される。このノックアウトは、一体としてリボース-5-リン酸(R5P)を酢酸及びGAPに変換し、それによって化学的産生から増殖を分断する、15個の反応を含む複雑な経路を通してのフラックスの「漏れ」を防ぐ。
【0076】
次に、表Iに示された2つの突然変異体について、バイオマス収率に対する許されるPDO産生の包絡線を調べる。突然変異体及び本来のE.coliネットワークの産生限界は、バイオマス収率が最大化されることになっている場合には、野生型E.coliネットワークが、PDOを産生する「誘因」を持たないことを示す。一方、多くのバイオマスが形成される場合、遺伝子除去後のネットワークの機能の低減により、両突然変異体A及びBは、かなりの量のPDOを産生しなければならない。突然変異体Aは、バイオマス産生とPDO産生の比を本質的にセットするtpiノックアウトを回避することによって、PDOのより高い最大理論収率によって特徴づけられる。上述の結果は、PDOへの主要中間体としてのグリセリンの使用にかかっている。次に、1,3-プロパンジオール産生のためのグリセリン変換経路の代替経路を利用する可能性を調べた。
【0077】
本出願人らは、3-ヒドロキシプロピオン酸(3-HPA)を生成するマロニルCoAの2段階NADPH依存還元(Menendez et al., 1999、Hugler et al., 2002)を含むChloroflexus aurantiacusにおける経路を識別した。3-HPAを1,3-プロパンジオールに変換する生物機能はないので、3-HPAは次いで、1,3-プロパンジオールに化学的に変換することができる。その最初のステップ(アセチル-CoAカルボキシラーゼ)が炭素固定反応であるため、この経路は、グリセリン経路を通してのPDO産生に優る重要な利点を提供する。したがって、3-HPAの最大理論収率(1.79mmol/mmolグルコース)は、グリセリン変換経路を通してのPDO産生のそれ(1.34mmol/mmolグルコース)よりもかなり高い。3-HPA産生経路追加後のOptKnockフレームワークの適用は、バイオマス形成が3-HPA産生に結びつけられるまでには、より多くのノックアウトが必要であることを明らかにした。最も興味深い戦略の1つは、最適増殖速度におけるその理論最大値の91%の3-HPA産生を与える9つのノックアウトを含む。最初の3つノックアウトは、競合する酢酸、乳酸及びエタノール産生機構の除去を含むので、比較的に直接的であった。さらに、エントナー-ドゥドロフ経路(ホスホグルコン酸デヒドラターゼ又は2-ケト-3-デオキシ-6-ホスホグルコン酸アルドラーゼ)、4つの呼吸反応(すなわちNADHデヒドロゲナーゼI、NADHデヒドロゲナーゼII、グリセリン-3-リン酸デヒドロゲナーゼ及びコハク酸デヒドロゲナーゼ複合体)、及び初期の解糖ステップ(すなわちホスホグルコースイソメラーゼ)が途絶される。この戦略は、最大バイオマス収率を仮定して、グリセリン変換経路を利用した以前に識別された突然変異体よりも69%高い3-HPA収率を与えた。
【0078】
2.3 実施例3
代替細胞目的:代謝調整の最小化
上述の結果は全て、フラックス配分を駆動する細胞目的としてバイオマス収率の最大化を生じさせることによって得られたものである。この仮説は、本質的に、変化する環境条件下でバイオマス収率の最大化を維持するために、代謝ネットワークは任意に変化することができ、かつ/又は調節ループを配線し直すことさえできると仮定する(最大応答)。最近の証拠は、これが時に、複数回の増殖選択サイクルの後に、E.coliのK-12株によって達成されることを示唆している(Ibarra et al., 2002)。このセクションでは、遺伝子欠失後の代謝ネットワークによる近視眼的な(最小)応答を仮定した対照的な仮説(すなわち代謝調整の最小化(MOMA)(Segre et al., 2002))を検討した。具体的には、MOMA仮説は、代謝ネットワークが、遺伝子欠失によっては到達できないその系の本来の定常状態のできるだけ近くにとどまろうとすることを示唆する。この仮説は、遺伝子欠失事象の直後のフラックス配分をより正確に記述することが示された(Segre et al., 2002)。この研究では、MOMA目的を利用して、OptKnockによって識別される突然変異株におけるフラックス分配を予測した。乳酸及びコハク酸シミュレーションに対するベースケースは、嫌気的条件下の最大バイオマス形成であると仮定され、PDOシミュレーションに対するベースケースは、好気的条件下の最大バイオマス形成であった。結果は、表1の最後の列に示されている。全てのケースで、提案された複数の遺伝子ノックアウト戦略は、MOMAケースの化学的産生収率が、最大バイオマス仮説に比べてわずかに低いだけであることを示唆している。これは、OptKnock結果が、細胞目的の選択に関してかなりロバストであることを暗示する。
【0079】
3.0 代替実施形態
本発明の背景を照らし出し、又は実施に関する追加の詳細を提供する刊行物及び明細書において使用された他の資料は、それらの全体が参照によって本明細書に組み込まれる。本発明は、生物の変異、細胞目的の変異、生体工学目的の変異、形成される最適化問題及び使用される解のタイプの変異を含む、多数の変異を企図する。これらの変異、修正又は変更及び/あるいはその他の変異、修正又は変更は、添付の請求項に記載された本発明の趣旨及び範囲から逸脱することなく実施することができる。
【0080】
参考文献
Anthony, C. (1982) The Biochemistry of Methylotrophs (Academic Press.)
Arita, M. (2000) Simulation Practice and Theory 8, 109-125.
Arita, M. (2004) Proc Natl Acad Sci U S A 101, 1543-7.
Badarinarayana, V., Estep, P.W., 3rd, Shendure, J., Edwards, J., Tavazoie, S., Lam, F., Church, G.M. (2001) Nat Biotechnol 19(11): 1060-5.
Bailey, J. E. (1991) Science 252, 1668-75.
Bailey, J. E. (2001) Nat Biotechnol 19, 503-4.
Bard, J. F. 1998. Practical bilevel optimization : algorithms and applications. Dordrecht ; Boston, Kluwer Academic.
Biebl, H., Menzel, K., Zeng, A.P., Deckwer, W.D. (1999) Appl Environ Microbiol 52: 289-297.
Bond, D. R. & Lovley, D. R. (2003) Appl Environ Microbiol 69, 1548-55.
Bond, D. R., Holmes, D. E., Tender, L. M. & Lovley, D. R. (2002) Science 295, 483-5.
Brown, M. (1999) Perl programmer's reference (Osborne/McGraw-Hill, Berkeley, Calif.).
Burgard, A. P. & Maranas, C. D. (2001) Biotechnol Bioeng 74, 364-375.
Burgard, A. P., Pharkya, P. & Maranas, C. D. (2003) Biotechnol Bioeng 84, 647-57.
Burgard, A. P., Maranas, C. D. (2003) Biotechnol Bioeng 82(6): 670-7.
Burgard, A. P., Vaidyaraman, S., Maranas, C. D. (2001) Biotechnol Prog 17: 791-797.
Cameron, D. C., Altaras, N. E., Hoffman, M. L., Shaw, A. J. (1998) Biotechnol Prog 14(1): 116-25.
Castellanos, M., Wilson, D. B. & Shuler, M. L. (2004) Proc Natl Acad Sci U S A 101, 6681-6.
Causey, T. B., Shanmugam, K. T., Yomano, L. P. & Ingram, L. O. (2004) Proc Natl Acad Sci U S A 101, 2235-40.
Chin, H. L., Chen, Z. S. & Chou, C. P. (2003) Biotechnol Prog 19, 383-8.
Chistoserdova, L., Laukel, M., Portais, J. C., Vorholt, J. A. & Lidstrom, M. E. (2004) J Bacteriol 186, 22-8.
Chistoserdova, L., Vorholt, J. A., Thauer, R. K. & Lidstrom, M. E. (1998) Science 281, 99-102.
Compagno, C., Boschi, F., Ranzi, B. M. (1996) Biotechnol Prog 12(5): 591-5.
Covert, M.W., Palsson, B.O. (2002) J Biol Chem 277(31): 28058-64.
Covert, M.W., Schilling, C.H., & Palsson, B.O. (2001) J Theor Biol 213(1): 73-88.
Das, D. & Veziroglu, T. N. (2001) International Journal of Hydrogen Energy 26, 13-28.
Datta, R., Tsai, S., Bonsignore, P., Moon, S., Frank, J. R. (1995) FEMS Microbiol. Rev. 16: 221-231.
David, H., Akesson, M. & Nielsen, J. (2003) Eur J Biochem 270, 4243-53.
Desai, R. P., Nielsen, L. K. & Papoutsakis, E. T. (1999) J Biotechnol 71, 191-205.
Edwards, J. S. & Palsson, B. O. (2000) Proc Natl Acad Sci U S A 97, 5528-33.
Edwards, J. S., Ibarra, R. U., Palsson, B. O. (2001) Nat Biotechnol 19(2): 125-30.
Edwards, J. S., Palsson, B. O. (2000) Proc Natl Acad Sci U S A 97(10): 5528-33.
Ellis, L. B., Hou, B. K., Kang, W. & Wackett, L. P. (2003) Nucleic Acids Res 31, 262-5.
Eppstein, D. (1994) in 35th IEEE Symp. Foundations of Comp. Sci, Santa Fe), pp. 154-165.
Fan, L. T., Bertok, B. & Friedler, F. (2002) Comput Chem 26, 265-92.
Finneran, K. T., Housewright, M. E. & Lovley, D. R. (2002) Environ Microbiol 4, 510-6.
Forster, J., Famili, I., Fu, P. C., Palsson, B., Nielsen, J. (2003) Genome Research 13(2): 244-253.
Forster, J., Famili, I., Fu, P., Palsson, B. O. & Nielsen, J. (2003) Genome Res 13, 244-53.
Gupta, S., Clark, D. P. (1989) J Bacteriol 171(7): 3650-5.
Hartlep, M., Hussmann, W., Prayitno, N., Meynial-Salles, I., Zeng, A. P. (2002) Appl Microbiol Biotechnol 60(1-2): 60-6.
Hatzimanikatis, V., Emmerling, M., Sauer, U., Bailey, J. E. (1998) Biotechnol Bioeng 58(2-3): 154-61.
Hatzimanikatis, V., Li, C., Ionita, J. A. & Broadbelt, L. J. (2003) presented at Biochemical Engineering XIII Conference, Session 2, Boulder, CO.
Heinrich, R., Rapoport, T. A. (1974) Eur. J. Biochem. 41: 89-95.
Hugler, M., Menendez, C., Schagger, H., Fuchs, G. (2002) J Bacteriol 184(9): 2404-10.
Ibarra, R. U., Edwards, J. S., Palsson, B. O. (2002) Nature 420(6912): 186-9.
Ignizio, J.P., Cavalier, T.M. 1994. Linear programming. Englewood Cliffs, N.J., Prentice Hall.
Kacser, H., Burns, J. A. (1973). Symp. Soc. Exp. Biol. 27: 65-104.
Kanehisa, M., Goto, S., Kawashima, S., Okuno, Y. & Hattori, M. (2004) Nucleic Acids Res 32 Database issue, D277-80.
Karp, P. D., Riley, M., Saier, M., Paulsen, I. T., Collado-Vides, J., Paley, S. M., Pellegrini-Toole, A., Bonavides, C. & Gama-Castro, S. (2002) Nucleic Acids Res 30, 56-8.
Kataoka, N., Miya, A. & Kiriyama, K. (1997) Wat. Sci. Tech. 36, 41-47.
Kompala, D. S., Ramkrishna, D., Tsao, G. T. (1984) Biotechnol Bioeng 26(11): 1272-1281.
Korotkova, N., Chistoserdova, L. & Lidstrom, M. E. (2002) J Bacteriol 184, 6174-81.
Krieger, C. J., Zhang, P., Mueller, L. A., Wang, A., Paley, S., Arnaud, M., Pick, J., Rhee, S. Y. & Karp, P. D. (2004) Nucleic Acids Res 32 Database issue, D438-42.
Li, K. & Frost, J. W. (1998) Journal of American Chemical Society 120, 10545-10546.
Liu, H., Ramnarayanan, R. & Logan, B. E. (2004) Environmental Sceince and Technology 38, 2281-2285.
Lovley, D. R. (2003) Nat Rev Microbiol 1, 35-44.
Majewski, R.A., Domach, M. M. (1990) Biotechnol Bioeng 35: 732-738.
Mavrovouniotis, M., Stephanopoulos, G. & Stephanopoulos, G. (1990) Biotechnol Bioeng 36, 1119-1132.
McShan, D. C., Rao, S. & Shah, I. (2003) Bioinformatics 19, 1692-8.
Menendez, C., Bauer, Z., Huber, H., Gad'on, N., Stetter, K.O., Fuchs, G. (1999) J Bacteriol 181(4): 1088-98.
Methe, B. A., Nelson, K. E., Eisen, J. A., Paulsen, I. T., Nelson, W., Heidelberg, J. F., Wu, D., Wu, M., Ward, N., Beanan, M. J., et al. (2003) Science 302, 1967-9.
Misawa, N., Yamano, S. & Ikenaga, H. (1991) Appl Environ Microbiol 57, 1847-9.
Nakamura, C. E. & Whited, G. M. (2003) Curr Opin Biotechnol 14, 454-9.
Nakamura, C.E. 2002. Production of 1,3-Propanediol by E. coli. presented at Metab Eng IV Conf: Tuscany, Italy.
Nandi, R. & Sengupta, S. (1996) Enzyme and microbial tehcnology 19, 20-25.
Nandi, R. & Sengupta, S. (1998) Crit Rev Microbiol 24, 61-84.
Neidhardt, F.C., Curtiss, R. 1996. Escherichia coli and Salmonella : cellular and molecular biology. Washington, D.C., ASM Press.
Overbeek, R., Larsen, N., Pusch, G. D., D'Souza, M., Selkov, E., Jr., Kyrpides, N., Fonstein, M., Maltsev, N. & Selkov, E. (2000) Nucleic Acids Res 28, 123-5.
Papin, J.A., Price, N.D., Wiback, S.J., Fell, D.A., Palsson, B. 2003. Metabolic Pathways in the Post-Genome Era. Trends Biochem Sci, accepted.
Papoutsakis, E. & Meyer, C. (1985) Biotechnol Bioeng 27, 50-66.
Papoutsakis, E. (1984) Biotechnol Bioeng 26, 174-187.
Pharkya, P., Burgard, A. P. & Maranas, C. D. (2003) Biotechnol Bioeng 84, 887-99.
Price, N.D., Papin, J. A., Schilling, C.H., Palsson, B. 2003. Genome-scale Microbial In Silico Models: The Constraints-Based Approach. Trends Biotechnol, accepted.
Ramakrishna, R., Edwards, J. S., McCulloch, A., Palsson, B. O. (2001) Am J Physiol Regul Integr Comp Physiol 280(3): R695-704.
Ramakrishna, R., Ramakrishna, D., Konopka, A. E. (1996). Biotechnol Bioeng 52: 141-151.
Reed, J. L., Vo, T. D., Schilling, C. H. & Palsson, B. O. (2003) Genome Biol 4, R54.
Schilling, C. H., Covert, M. W., Famili, I., Church, G. M., Edwards, J. S. & Palsson, B. O. (2002) J Bacteriol 184, 4582-93.
Schilling, C. H., Covert, M. W., Famili, I., Church, G. M., Edwards, J. S., Palsson, B. O. (2002) J Bacteriol 184(16): 4582-93.
Schilling, C. H., Palsson, B. O. (2000) J Theor Biol 203(3): 249-83.
Segre, D., Vitkup, D., Church, G. M. (2002) Proc Natl Acad Sci U S A 99(23): 15112-7.
Segre, D., Zucker, J., Katz, J., Lin, X., D'Haeseleer, P., Rindone, W. P., Kharchenko, P., Nguyen, D. H., Wright, M. A. & Church, G. M. (2003) Omics 7, 301-16.
Selkov, E., Jr., Grechkin, Y., Mikhailova, N. & Selkov, E. (1998) Nucleic Acids Res 26, 43-5.
Seressiotis, A. & Bailey, J. E. (1988) Biotechnol Bioeng 31, 587-602.
Stephanopoulos, G., Aristidou, A. A., Nielsen, J. 1998. Metabolic engineering : principles and methodologies. San Diego, Academic Press.
Stephanopoulos, G. & Sinskey, A. J. (1993) Trends Biotechnol 11, 392-6.
Stols, L., Donnelly, M. I. (1997) Appl Environ Microbiol 63(7): 2695-701.
Tomita, M., Hashimoto, K., Takahashi, K., Shimizu, T. S., Matsuzaki, Y., Miyoshi, F., Saito, K., Tanida, S., Yugi, K., Venter, J. C., et al. (1999) Bioinformatics 15, 72-84.
Valdes, J., Veloso, F., Jedlicki, E. & Holmes, D. (2003) BMC Genomics 4, 51.
Van Dien, S. J. & Lidstrom, M. E. (2002) Biotechnol Bioeng 78, 296-312.
Van Dien, S. J., Strovas, T. & Lidstrom, M. E. (2003) Biotechnol Bioeng 84, 45-55.
Varma, A., Boesch, B. W., Palsson, B. O. (1993) Appl Environ Microbiol 59(8): 2465-73.
Varma, A, Palsson, B. O. (1993) J. Theor. Biol. 165: 503-522.
Varma, A. & Palsson, B. O. (1994) Bio/Technology 12, 994-998.
Varner, J., Ramkrishna, D. (1999) Biotechnol Prog 15(3): 407-25.
Varner, J. & Ramkrishna, D. (1999) Curr Opin Biotechnol 10, 146-150.
Zeikus, J. G., Jain, M. K., Elankovan, P. (1999) Appl Microbiol Biotechnol 51: 545-552.
Zeng, A. P., Biebl, H. (2002) Adv Biochem Eng Biotechnol 74: 239-59.
Zhu, M. M., Lawman, P. D., Cameron, D. C. (2002) Biotechnol Prog 18(4): 694-9.
【技術分野】
【0001】
本出願は、それぞれその全体が参照によって本明細書に組み込まれた2002年7月10日出願の米国特許出願第60/395,763号、2002年10月9日出願の米国特許出願60/417,511号及び2003年2月3日出願の米国特許出願第60/444,933号の変更出願である、2003年7月9日出願の米国特許出願第10/616,659号の一部継続出願である。
【0002】
本発明は、米国エネルギー省(助成金番号58855)及び全米科学財団(助成金番号BES0120277)の支援を受けたものである。したがって米国政府は本発明に関して一定の権利を有する。
【0003】
本発明は、反応の追加及び欠失によって経路変更を誘導する計算フレームワークに関する。
【背景技術】
【0004】
生物変換経路の生成は近年、かなりの関心を集めている。この目的に向けた最初の体系的研究は、Seressiotis and Baileyによって実施され(Seressiotis & Bailey, 1988)、彼らは、自身のソフトウェアの開発に人工知能の概念を利用した。これに続いて、反応及び代謝産物に対する化学量論的制約条件を繰返し満たすことに基づくアルゴリズムを利用した、グルコースとアンモニアからのリシン産生に関するケーススタディが、Mavrovouniotis et al.によって実施された(Mavrovouniotis et al., 1990)。より最近になって、原子の追跡(Arita, 2000、Arita, 2004)、酵素機能規則(enzyme function rule)及び熱力学的実現可能性制約条件(thermodynamic feasibility constraint)(Hatzimanikatis et at., 2003)に基づいて新規の生体内変化経路を識別する、エレガントグラフセオレティックコンセプト(elegant graph theoretic concept)(例えば、P-グラフ(Fan et al., 2002)及びk-最短路(shortest path)アルゴリズム(Eppstein, 1994))が創始された。これらの方法の大部分は、比較的に小さな反応データベースにそれらを適用することによって例証されたものである。約5000種の反応からなるKEGGデータベース(Kanehisa et al., 2002)などのゲノムスケールの代謝反応データベースに対するそれらの性能は劇的に低下するであろう。
【0005】
ごく最近になって、(任意の生化学特性に基づく)最小経路コスト(minimum pathway cost)の決定に基づくヒューリスティックアプローチが提案された(McShan et al., 2003)。この方法は、1つの代謝産物から他の代謝産物への変換経路を描くのに非常に成功している。しかし、この方法は、上で論じた他の全ての方法と同様、特定の経路を使用することによって得られる生成物の収率を予測することができない。さらに、これらの方法は大部分、直線的(linear)な生体内変化経路を識別し、全ての代謝産物、特に補因子のバランサビリティを保証しない。
【発明の概要】
【発明が解決しようとする課題】
【0006】
したがって、本発明の主たる目的、特徴又は利点は、ゲノムスケールのネットワークに関連した複雑さに対処する最適化ベースの手順を提供することにある。
【0007】
本発明の他の目的、特徴又は利点は、化学量論的にバランスの取れた分岐及び直線生物変換経路であって、収率の観点及び生成物形成のために必要な宿主内の非ネイティブ反応の数の観点から効率的な生物変換経路を構築するための方法を提供することにある。
【0008】
本発明の他の目的、特徴又は利点は、選択された複数の基質の評価を可能にする方法を提供することにある。
【0009】
本発明の他の目的、特徴又は利点は、産生増大目的を含む生体工学目的を達成する方法を計算的に提案するための方法を提供することにある。
【0010】
本発明の他の目的、特徴又は利点は、遺伝子欠失又は付加の候補を、生物変換経路ネットワークモデルを使用することによって決定することにある。
【0011】
本発明の他の目的、特徴又は利点は、生体工学目的を計算的に達成するための最適化されたフレキシブルでロバストな方法を提供することにある。
【0012】
本発明の他の目的、特徴又は利点は、生体工学目的を計算的に達成するための方法であって、中心的な代謝経路だけでなく、アミノ酸の生合成経路、分解経路などの他の経路をも考慮することができる方法を提供することにある。
【0013】
本発明の他の目的、特徴又は利点は、生体工学目的を計算的に達成するための方法であって、輸送速度、分泌経路又はその他の特性を最適化変数として考慮することができる方法を提供することにある。
【0014】
本発明のこれらの目的、特徴及び利点ならびに/又はその他の目的、特徴及び利点の1つ又は複数は、開示された実施形態の以下の詳細な説明及び添付の請求項を検討することによって明白となろう。
【課題を解決するための手段】
【0015】
本発明は、標的化合物の過剰産生のため、反応の追加及び欠失によって微生物ネットワークの経路変更を誘導することを目的とする「OptStrain」と呼ばれる階層型計算フレームワークを提供する。これらの化合物は例えば、生物燃料電池及び環境応用における電子又は水素から、複雑な前駆分子までに及ぶ。Universalデータベースと呼ばれる生体内変化の包括的なデータベース(5,000超の反応を含む)が編纂され、このデータベースは、複数の生体経路データベース源から反応をダウンロードし、それらの反応にキュレーション(curation)を実施することによって定期的に更新される。次いで、組合せ最適化を使用して、このUniversalデータベースから抽出された非ネイティブ機能の集合を解明し、所望の生成物形成を可能にするために検討中の産生宿主に追加する。続いて、フラックス(flux)を標的生成物からそらす競合機能を識別し、それらを除去して、増殖に結びつけられたより高い生成物収率を保証する。本発明は、化学量論的にバランスの取れた経路(stoichiometrically balanced pathway)を構築し、最大生成物収率要件を課し、最適な基質を特定し、異なる微生物宿主を評価する能力を有する統合計算フレームワークを確立することによって、従来の成果からのかなりの前進を表す。
【0016】
2つの非常に異なる生成物分子に言及することによって、OptStrainの範囲及び有用性を例証する。水素ケーススタディでは、3種の別個の産生宿主に対して2種類の異なる基質を使用して、水素収率を向上させるための反応除去戦略を特定する。対照的に、バニリンスタディでは主に、どの非ネイティブ経路をEscherichia coliに追加する必要があるかを前面に押し出す。要するに、OptStrainは、微生物株の設計に役立つ有用なツールを提供し、より重要には、将来のモデル化の発展を収容する統合フレームワークを確立する。
【0017】
OptStrainプロセスは、それらの全体が参照によって本明細書に組み込まれた7月9日出願の米国特許出願第10/616,659号、2002年7月10日出願の米国特許出願第60/395,763号、2002年10月9日出願の米国特許出願第60/417,511号、及び2003年2月3日出願の米国特許出願第60/444,933号に以前に記載されたOptKnockプロセスを含む。OptKnockプロセスは、バイオテクノロジーにおける最重要課題である化学物質又は生化学物質の産生を最適化するための組換え微生物株の体系的な発展を提供する。ゲノムスケールの代謝モデルの出現は、過剰産生につながる遺伝子操作を提案する計算手順を開発する基礎を築いた。これは、増殖資源(すなわち炭素、酸化還元電位及びエネルギー)に向けての消費が、化学量論のために、所望の生成物の産生を伴うことを保証することによって達成される。具体的には、この計算フレームワークは、仮定された細胞目的(例えばバイオマス形成)を外部から課された化学的産生標的と最大限に結びつける複数の遺伝子欠失組合せを識別する。この入れ子構造は、双対理論によって生み出された変換に基づいて解かれる2層(bilevel)最適化問題を与える。このフレームワークのこの手順は、バイオマス形成を化学的産生と結びつけることによって、過剰産生突然変異体を間接的に発展させるための増殖選択/適応系を提案する。
【0018】
OptKnockはさらに、中心的な代謝ネットワークの遺伝子だけでなく、アミノ酸生合成経路及び分解経路をも含む戦略を含むことができる。遺伝子欠失に加えて、二酸化炭素、アンモニア及び酸素の輸送速度、ならびに主要代謝産物のための分泌経路を、フレームワークの最適化変数として導入することができる。したがって、本発明は、ゲノムスケールのネットワークに関連した複雑さに対処するため、ロバストでフレキシブルである。
【図面の簡単な説明】
【0019】
【図1】OptStrain手順を示す図である。ステップ1は、元素的にバランスの取れた反応だけからなるUniversalデータベースを編纂するための反応データベースのキュレーションを含む。ステップ2は、反応の出所に関して一切考慮せずに、基質(例えばグルコース、メタノール、キシロース)から生成物(例えば水素、バニリン)への所望の生体内変化を可能にする経路を識別する。宿主のネイティブ反応と非ネイティブ反応の両方が存在することに留意されたい。ステップ3は、非ネイティブ反応に対する依存を最小化し、ステップ4は、非ネイティブ機能を、微生物宿主の化学量論モデルに組み込み、OptKnock手順を適用して、標的生成物と競合する反応を識別し、排除する。(×)は、欠失反応を指示する。
【図2】さまざまな基質の最大水素収率を重量ベースで示すグラフである。
【図3】水素産生包絡線を、好気的及び嫌気的条件下の野生型E.coliネットワークならびに2反応及び3反応欠失突然変異体ネットワークのバイオマス産生速度の関数として示すグラフである。ベースグルコース取込み速度は10mmol/gDW/hrに固定されている。これらの曲線は、さまざまなバイオマス形成速度において最大及び最小水素産生速度を求めることによって作成されて物である。点Aは、嫌気的条件下の野生型ネットワークの最大バイオマス形成速度における必要な理論水素産生速度を表す。点B及びCはそれぞれ、対応する二酸化炭素輸送速度をOptknockによって提案される値に固定した後の2つの突然変異体ネットワークの最大増殖速度における理論的水素産生速度を識別する。
【図4】野生型(実線)及び突然変異体(破線)Clostridium acetobutylicum代謝ネットワークの、ベースグルコース取込み速度1mmol/gDW/hrでの水素形成限界を示すグラフである。線ABは、野生型ネットワークに対して使用可能な異なる代替最大バイオマス収率解を表す。点Cは、最大増殖速度における突然変異体ネットワークの水素収率を示す。これは、報告されたC.acetobutylicum(45)の実験水素収率(2mol/molグルコース)と対照をなす。
【図5】増補されたE.coli代謝ネットワークの、ベースグルコース取込み速度10mmol/gDW/hrでのバニリン産生包絡線を示すグラフである。点A、B及びCはそれぞれ、1、2及び4反応欠失突然変異体ネットワークに関連した最大増殖点を表す。バイオマス産生の任意の速度でバニリン産生が保証されない野生型ネットワークとは対照的に、突然変異体ネットワークは、高レベルのバイオマス産生を達成するためにかなりのバニリン収率を必要とする。全てのケースで増殖の嫌気的モードが示唆されることに留意されたい。
【図6】Optknockの2層最適化構造を示す図である。内側の問題は、特定の細胞目的(例えばバイオマス収率の最大化、MOMA)の最適化に基づくフラックス配分を実行する。次いで外側の問題が、内側の問題の最適化に対して使用可能な主要反応へのアクセスを制限することによって、生体工学目的(例えば化学的産生)を最大化する。
【発明を実施するための形態】
【0020】
本発明は、反応の追加及び欠失によって経路変更を誘導するための方法及びシステムを提供する。それらの方法は、コンピュータ実現又はコンピュータ援用法、あるいは他の方法で自動化された方法であることが好ましい。本明細書で使用される用語「コンピュータ」は、これに限定されるわけではないが、本明細書に記載の方法を実施するのに適した多数の電子装置を含むものと幅広く解釈しなければならない。さらに、本発明はコンピュータ援用モデル化に関係するため、本発明の範囲は、本明細書に記載された特定の実施形態よりも広いこと、ならびにさまざまな環境及び文脈において本発明を適用して、さまざまな問題に、部分的にはコンピュータ実現に関連した予測可能性によって対処する方法を、当業者が理解するであろうことを、理解されたい。
【0021】
1. OptStrain
システム生物学の基本的な目標は、生体系内の世界にアクセス可能な生体内変化の完全な「パレット(palette)」を解明することである。この目標は、拡大し続ける一連の所望の生体内変化を達成する能力を有する微生物株を構築しようとするバイオテクノロジーにおける継続的な探求目標と平行してある。これらの生体内変化は、単純な前駆化学物質(Nakamura & Whited, 2003、Causey et al., 2004)又はカロテノイドなどの複雑な分子(Misawa et al., 1991)から、生物燃料電池(Liu et al., 2004)又はバッテリー(Bond et al., 2002、Bond et al., 2003)内の電子、及びバイオレメディエーション応用において重金属錯体を沈殿させる能力を有する微生物(Methe et al., 2003、Lovley, 2003、Finneran et al., 2002)に至るまでの生成物に照準を定めている。分子生物学及び組換えDNA技術における最近の進展は、産生微生物株の遺伝子内容及び発現レベルを、直接に、またそれを標的として形にすることができる新しい時代を先導した(Bailey, 1991、Stephanopoulos & Sinskey, 1993)。新しく獲得されたこれらの能力の驚くべき範囲及び多様性、ならびにバイオテクノロジー分野の応用範囲は、株最適化プロジェクトのための最適な遺伝子改変セットを演繹的に識別するためのモデル化及び計算支援が今までにもまして必要であることを暗示している。
【0022】
ゲノムスケール微生物モデルの最近の利用可能性は、株操作(strain engineering)戦略を識別することを目的とした計算法を開発するために必要な経路再構築を提供した(Bailey, 2001)。これらのモデルは、H. pylori(Schilling et al., 2002)、E.coli(Reed et al., 2003、Edwards & Palsson, 2000)、S. cerevisiae(Forster et al., 2003)及び他の微生物(David et al., 2003、Van Dien & Lindstrom, 2002、Valdes et al., 2003)に対してすでに使用可能であり、連続的に洗練された微生物の代謝能力の抽象化を提供する。注釈付きのゲノムからの化学量論モデルの構築を促進する自動プロセス(Segre et al., 2003)は、いくつかの微生物の代謝再構築をさらに加速させそうだ。同時に、個々の反応は、KEGG、EMP、MetaCyc、UM-BBDなどの多くのデータベースの中に収められて(Overbeek et al., 2000、Selkov et al., 1998、Kanehisa et al., 2004、Krieger et al., 2004、Ellis et al., 2003、Karp et al., 2002)、さまざまな種に存在している直接の又は間接的な証拠がある生体内変化の拡大し続ける包括的なコレクションを形成している。すでに数千もの反応が収められているが、特定の生物の代謝再構築(Schilling et al., 2002、Reed et al., 2003、Edwards & Palsson, 2000、Forster et al., 2003)とは異なり、これらの編纂物は、単一の種の反応だけでなく、多くの異なる種の反応を、あまりキュレーションが行われていない状態で含む。これは、現在、微生物モデルの拡大し続けるコレクションが存在し、同時に、非ネイティブ機能のこれまでになく多くの包括的な編纂物が存在することを意味する。この新しく獲得されたおびただしい量のデータは、本明細書の範囲を構成するいくつかの計算上及びモデル化上の課題を最前線に押し出した。具体的には、どうすれば、E.coliなどの既存の産生系に組み込んで、所望の新しい機能をそれらの系に与える適当な経路/遺伝子セットを、さまざまな生物データベースの中にカタログ化された数千の機能の中から体系的に選択することができるのか。続いて、どうすれば、高い生成物収率及び生存能力を保証するために排除する競合機能を識別することができるか。
【0023】
この目標を達成するための既存の戦略及び方法には、基質から標的化合物までの実現可能な全ての生物変換経路を、生化学的変換の所与のリストから探索するデータベース問合せ(Seressiotis & Bailey, 1988、Mavrovouniotis et al., 1990)が含まれる。より最近になって、原子の追跡(Arita, 2000、Arita, 2004)、酵素機能規則及び熱力学的実現可能性制約条件(Hatzimanikatis et at., 2003)に基づいて新規の生体内変化経路を識別する、エレガントグラフセオレティックコンセプト(例えば、P-グラフ(Fan et al., 2002)及びk-最短路アルゴリズム(Eppstein, 1994))が創始された。最近になってさらに、KEGGデータベース(Kanehisa et al., 2004)の中の酵素生化学反応を使用して、基質と代謝産物とをリンクする連結グラフを構築する興味深いヒューリスティック探索法が提案された(McShan et al., 2003)。しかし、これらの方法の大部分は、その代謝ネットワークの残りの部分のバランスが取れていること、ならびに補因子の使用/生成及びエネルギー収支に対する代謝上の要請(imperative)が満たされていることを保証することなく、基質を最終生成物にリンクする直線経路を生成する。
【0024】
本発明は、所望の表現型を与えるための宿主生物への非ネイティブ機能の組込み後に生成される化学量論的にバランスの取れた経路を識別する階層最適化に基づくフレームワークOptStrainを提供する。さまざまな化学量論モデル及び公開されているKEGG(Kanehisa et al., 2004)などのデータベースから一緒にプールされた数千(現在5,734)の反応からなる拡大し続けるアレイの中から、候補代謝経路を識別する。識別された経路は最大収率考慮事項を満たし、基質の選択は最適化変数として取り扱うことができることに留意されたい。それぞれの経路に関連した補因子/エネルギー要件に関する重要な情報を推論し、前述の基準に関して候補経路の比較を可能にする。先に識別した性能目標を満たしながら異種遺伝子に対する依存を連続的に最小化することによって、産生宿主選択を調べる。次いで、識別された非ネイティブ機能を触媒するのに必要な全ての酵素をコードした遺伝子セットを、アイソザイム及び多サブユニット酵素を考慮して、構築することができる。続いて、生化学的産生目的と増殖目的を切り離す競合機能を除去することによって生成物の収率を向上させるために、この増補された宿主ネットワークにおける遺伝子欠失を識別する(Burgard et al., 2003、Pharkya et al., 2003)。生成物分子サイズの点で両極端に位置する2つの異なる生成物分子(すなわち水素とバニリン)について詳細に説明することによって、OptStrafnの幅及び広がりが例示される。簡単には、あるケースでは、計算結果が、既存の株設計及び産生の実際と一致し、他のケースでは、新規の操作戦略を特定する。
【0025】
1.1 材料及び方法
取り組んだ最初の課題は、所望の生体内変化を可能にするために、特定の生物の代謝ネットワーク(例えばE.coli(Reed et al., 2003、Edwards & Palsson, 2000)、S. cerevisiae(Forster et al., 2003)、C. acetobutylicum(Desai et al., 1999、Papoutsakis, 1984)など)にどの機能を追加すべきかを識別する体系的な計算フレームワークの開発である。本発明の発明者らは、ずっと小規模ながら、この目標にすでに貢献した(Burgard & Maranas, 2001)。編纂されたデータベースのサイズが極めて大きく、また、時に相反する複数の目的を同時に満たす必要があることから、本発明者らは、図1に示されたOptStrain手順を開発した。それぞれのステップは、解く必要がある最適化問題の特定の構造及びサイズに起因する異なる計算上の課題を導入する。
【0026】
ステップ1. 本発明者らのUniversalデータベースへの反応の自動ダウンロード及び化学量論的バランスを保証するためのキュレーション。
【0027】
ステップ2. 反応の出所(すなわちネイティブ反応か又は非ネイティブ反応か)に関する制限なしで、選択された所与の基質について、生成物の最大理論収率を計算する。
【0028】
ステップ3. ステップ2で得られた最大理論収率及び最適基質が与えられた場合に検討下の産生宿主内の非ネイティブ機能の数を最小化する、化学量論的にバランスの取れた経路の識別。異なる宿主選択間の比較とともに、収率の最大化と異種遺伝子数の最小化の両方の基準を満たす代替経路が生成される。それぞれの経路に関連した補因子/エネルギー使用に関する情報もこの段階で導き出される。最後に、この段階で、適当な酵素をコードすることによって標的生体内変化の存在を保証する1つ又は複数の遺伝子セットを導き出すことができる。
【0029】
ステップ4. 使用可能な場合に、検討下の産生微生物宿主の化学量論モデルに、識別された非ネイティブ生体内変化を組み込む。次に、これらの増補されたモデルに、OptKnockフレームワークを適用して(Burgard et al., 2003、Pharkya et al., 2003)、代謝ネットワークの接続性を「形にする」ことによりその所望の生成物の産生が増殖の必須の副生物になることを保証する遺伝子欠失を提案する。OptKnockフレームワークについては後にさらに説明する。
【0030】
データベースのキュレーション。OptStrain手順の最初のステップは、さまざまな源から獲得された反応を本発明者らのUniversalデータベースにダウンロードし、それらにキュレーションを実施することから始まる。具体的には、KEGGデータベースには毎月新しい反応が組み込まれるので、本発明者らは、Perl(Brown, 1999)を使用して、KEGGデータベース内の全ての反応を定期的に自動的にダウンロードするカスタマイズされたスクリプトを開発した。次いで、別のスクリプトを使用して、全てのそれぞれの化合物中のそれぞれの元素の原子数を解析する。全ての反応の反応物及び生成物中のそれぞれのタイプの原子の数を計算し、元素的にバランスの取れていない反応を考慮から外す。さらに、化学式中の繰返し単位の数が指定されていない化合物(例えばC25H39N7O17P3S(CH2)nで表されたトランス-2-エノイル-CoA)、又は化学式中のアルキル基Rが指定されていない化合物を、ダウンロードされたセットから除去する。このステップは、ゲノムデータベース中に存在する機能の自動ダウンロード、及びそれに続くそれらの元素バランサビリティの検証を可能にし、組換え標的として使用される大規模な機能セットを形成することを可能にする。
【0031】
本発明は、多数の特定の方法を使用して、反応の持続期間及び/又はキュレーションを自動化することができることを企図する。これらの自動機能は、使用可能な資源、それらの資源へのアクセスのタイプ、及び本発明が実現される特定の環境又は文脈に関連したその他の因子に応じた多数の方法で実行することができる。
【0032】
最大収率の決定。反応セットを決定した後、ある範囲の基質選択からの標的生成物の最大理論収率を、使用される反応の数又は出所に制限を設けることなく決定する第2のステップが実行される。生成物の最大理論収率は、基質の単位取込み速度について、標的代謝産物を産生する全ての反応フラックスから標的代謝産物を消費する全ての反応フラックスを差し引き、これを、これらの反応における標的代謝産物の化学量論係数によって重み付けしたものを最大にすることによって得られる。化学量論的制約条件及び輸送条件に支配されるこの収率の最大化は、フラックスバランス解析(Flux Balance Analysis)フレームワーク(Varna & Palsson, 1994)においてしばしば遭遇する線形計画(Linear Programming:LP)問題(数学的公式化のための支援情報を参照されたい)を与える。LP問題の計算上の扱いやすさからすれば、数千の反応に対してであっても、多数の異なる基質選択を徹底的に調べることができる。
【0033】
この特定の実施形態では、生体工学目的が産生を最大にすることに関係するが、本発明は、他の生体工学目的を使用することができることを企図する。このような場合には、最大収率を決定し又は選択する代わりに、他の適当な目的又は制約条件を使用することができる。
【0034】
宿主生物に対する異種反応の最小数の識別。OptStrainの次のステップでは、最大理論収率についての知識を使用して、特定の宿主生物ネットワークに追加する必要がある非ネイティブ機能の最小数を決定する。数学的には、これは、最初に、関連反応フラックスvjをオン又はオフにするスイッチの働きをする1組の2値変数yjを導入することによって達成される。
【数1】

【0035】
2値変数yjは、反応jが活性である場合に値1をとり、反応jが不活性である場合に値0をとることに留意されたい。この制約条件は、指定された産生宿主にとって異種遺伝子である遺伝子に関連した反応にだけ課せられる。パラメータvjmin及びvjmaxは、その代謝ネットワークの化学量論に従って全てのそれぞれの反応フラックスvjを最小化及び最大化することによって計算される(Burgard & Mamas, 2001)。これは、所望の生成物に対する収率目標を満たしつつ宿主生物ネットワークに追加される遺伝子の最小数を見つけ出す混合整数線形計画(Mixed Integer Linear Programming:MILP)モデルにつながる。後に詳細に論じるこの公式化は、異種遺伝子の必要数と生成物の最大理論収率との間のトレードオフの探求、及び全ての代替最適解の反復的な識別を可能にする。このステップの最終結果は、基質から所望の生成物への効率的な変換のための全ての代替経路のランク付けされたリストを提供する、異なる経路とその対応する遺伝子補足物とからなる1つの集合である。
【0036】
宿主生物の化学量論モデルへの非ネイティブ反応の組込み。適当な宿主生物の識別の後、解析は、識別された非ネイティブ反応の集合によって増補された特定の生物の化学量論モデルに進む。しかし、微生物の代謝は、負わされた選択圧にできるだけ敏感である(例えば競合相手よりも速く成長する)ように準備されているため、微生物産生株に単純に遺伝子を追加するだけで、所望の過剰産生が得られるわけでない。微生物の生存目的は一般に、標的生化学物質の過剰産生と直接に競合する。これを防ぐため、本発明者らは、以前に開発された本発明者らの2層計算フレームワークOptKnock(Burgard et al., 2003、Pharkya et al., 2003)を使用して、一般にバイオマス収率として例示される細胞フィットネス(fitness)目的を関心の生成物の最大収率から除外する全ての機能を排除する。
【0037】
1.2 結果
微生物株最適化の計算結果は、水素及びバニリンの産生に集中する。本開示の恩恵に浴する当業者は、本発明が、本発明を単に例示するにすぎないこれらの特定の生体工学目的に限定されるものではないことを理解するであろう。この水素産生ケーススタディは、最適な産生環境を特定するために複数の基質及び微生物宿主を調べることの重要性、ならびに競合機能を排除する必要性を強調する。対照的に、バニリンケーススタディでは、非ネイティブ反応の最小数を識別することが、株設計の主要な課題であることが示される。メチロトローフMethylobacterium extorquens AM1の代謝経路(Van Dien & Lindstrom, 2002)及びKEGG反応データベース(Kanehisa et al., 2004)を一緒にプールすることによって、両方の例に対して共通の反応データベースを、(ステップ1で)概説されたとおりに構築した。
【0038】
1.2.1 水素産生ケーススタディ
効率的な微生物水素産生戦略は、水素を高い速度で形成する能力を有する最適な基質及び微生物株を選択することを要求する。最初に、本発明者らは、水素、酸素、窒素、イオウ、リン及び炭素に関してバランスの取れたカタログ化された全ての反応(約3,000反応)を組換え候補として使用して、最大収率LP公式(ステップ2)を解いた。OptStrainは、ペントース及びヘキソース、酢酸、乳酸、リンゴ酸、グリセリン、ピルビン酸、コハク酸、メタノールなどのさまざまな基質の選択を考慮したことに留意されたい。メタノール基質に対して得られた最も高い水素収率は、消費基質1gあたり0.126gであった。メタノールの水素と炭素の比は最大4対1なので、これは、驚くべきことではない。相対的に効率的ないくつかの基質の収率の比較を図2に示す。本発明者らは、メタノールの高い収率及びグルコースの使用に伴う有利なコストを動機として、メタノールとグルコースをさらに調べることとした。
【0039】
OptStrain手順の次のステップは、宿主生物内で理論最大収率を達成するための非ネイティブ機能の最小数を決定することを含む。本発明者らは、以下の3つの異なる取込みシナリオを検討した: (i)Escherichia coli(確立された産生系)での基質としてのグルコースの使用、(ii)Clostridium acetobutylicum(知られている水素産生者)でのグルコースの使用、及び(iii)Methylobacterium extorquens(知られているメタノール消費者)内でのメタノールの使用。
【0040】
1.2.1.1 Escherichia coli
MILPフレームワーク(ステップ3で説明)は、基質としてグルコースを使用すると、E.coliは、水素産生のために、非ネイティブ機能を必要としないことを正しく検証した。興味深いことに、水素産生は、陽子を還元して水素を形成するフェレドキシンヒドロゲナーゼ反応(E.C.#1.12.7.2)、又はNADHをNAD+に変換し、陽子会合によって水素を形成する水素デヒドロゲナーゼ反応(E.C.#1.12.1.2)によって可能であった。続いて、E.coli化学量論モデル(Reed et al., 2003)について、最大水素形成の上限及び下限を、好気的条件と嫌気的条件の両方で、ベースグルコース取込み速度10mmol/gDW/hrで、バイオマス形成速度(すなわち増殖速度)の関数として、調べた(図3参照)。注目すべきことに、水素の最大理論収率は好気的条件下のほうが高い。しかし、最大増殖速度で水素が形成され、増殖と結びついた産生モードに至るのは嫌気的条件下だけである(図3の点A参照)。なお、水素産生は、嫌気的条件下でギ酸を水素と二酸化炭素に変換するギ酸水素リアーゼ反応によって起こり、これは現在の実験観察(Nandi & Sengupta., 1998)と一致している。
【0041】
副生物形成を削減するための表現型制限(ステップ4)に移り、本発明者らは、水素産生と直接又は間接に競合する機能をネットワークから除去することによって、野生型E.coliネットワーク(Reed et al.,2003)における水素の産生を高めることができるかどうかを探った。このために、本発明者らは、OptKnockフレームワーク(Burgard et al., 2003、Pharkya et al., 2003)を使用して、水素産生を増殖と結びつける遺伝子欠失戦略を特定した。ここでは識別された戦略のうちの2つに光を当てる。第1の戦略(2反応欠失)は、エノラーゼ(E.C.#4.2.1.11)とグルコース6リン酸デヒドロゲナーゼ(E.C.# 1.1.1.49)の両方を除去する。エノラーゼ反応の除去は、3-ホスホグリセリン酸分岐点に向かう糖分解フラックスをセリン生合成経路に向けることによって、水素形成を強力に促進する。その後、セリンは、1炭素代謝における一連の反応に関与して、10-ホルミルテトラヒドロ葉酸を形成し、それは最終的にギ酸とテトラヒドロ葉酸に変換される。デヒドロゲナーゼ反応の除去は、グルコース6-リン酸フラックスがペントースリン酸経路へ転じることを防ぐ。第2の戦略、3反応欠失ケーススタディは、ATPシンターゼ(E.C.#3.6.3.14)、アルファケトグルタル酸デヒドロゲナーゼ及び酢酸キナーゼ(E.C.#2.7.2.1)の除去を含む。第1の反応の除去は、陽子の利用可能性を高め、他の2つの反応の欠失は、最大炭素フラックスがピルビン酸に向けられることを保証し、これは次いで、ピルビン酸ギ酸リアーゼによってギ酸に変換される。ギ酸は、ギ酸水素リアーゼによって水素と二酸化炭素に異化される。
【0042】
野生型ネットワークと突然変異体ネットワークの両方の増殖速度の関数としての水素産生限界の比較を図3に示す。突然変異体ネットワークの二酸化炭素の輸送速度は、OptKnockによって提案された値に固定され、したがってオペレーショナルインペラティブ(operational imperative)(Pharkya et al., 2003)をセットする。2反応欠失突然変異体の理論水素産生速度は、最大増殖速度(点B)で22.7mmol/gDW/hr(0.025g/gグルコース)であり、3反応欠失突然変異体は、最大増殖速度の低減と引き換えに、最大29.5mmol/gDW/hr(0.033g/gグルコース)を生成する(点C)ことに留意されたい。興味深いことに、両方の突然変異体ネットワークで、最大水素産生は酸素の取込みを要求する。これは、水素形成のためには酸欠が好ましい野生型のケースとは対照的である。注目すべきことに、ギ酸水素リアーゼは無酸素状態でしか誘導されないが、この酵素は好気的環境でも機能することが報告されている(Nandi & Sengupta, 1996)。このことは、これらの結果に基づいて実行されるどの実験的研究においても考慮されなければならない。
【0043】
1.2.1.2 Clostridium acetobutylicum
豊富な文献が、クロストリジウム属の生物を天然の水素産生系として識別している(Nandi & Sengupta, 1998、Katakoka et al., 1997、Chin et al., 2003、Das & Veziroglu, 2001)。フェレドキシンヒドロゲナーゼ(E.C.#1.12.7.2)による陽子の水素への還元が、主たる関連反応である。驚くことではないが、本発明者らは、OptStrain(ステップ3)を使用して、グルコースを基質として使用したClostridium acetobutylicumの水素産生には非ネイティブ反応が必要ない(Papoutsakis & Meyer, 1985)ことを検証した。次に本発明者らは、OptKnockフレームワークを使用して競合機能を賢明に除去することによって、E.coliの場合と同様に、水素産生を高めることができるかどうかを調べた。この目的に、本発明者らは、Papoutsakis及びその共同研究者によって開発されたClostridium acetobutylicumの化学量論モデル(Desai et al., 1999、Papoutsakis, 1984)を使用した。OptKnockは、酢酸形成反応及び酪酸輸送反応の欠失を提案した。
【0044】
後になってみれば、この欠失戦略は、ネットワーク全体のエネルギー収支を考えると合理的である。具体的には、野生型のケースでは、それぞれの酪酸分子の形成及び分泌は、2個のNADH分子の消費を必要とし、したがってネットワークの水素産生能力を低減させる。しかし、酪酸が分泌されず、その代わりにこれがリサイクルされて、アセトン及びブチリルCoAを形成する場合には、ブチリルCoAは、NADHの消費なしで酪酸に再び変換されることができる。この2反応欠失突然変異体は、わずかに低い増殖速度と引き換えに、3.17モル/モルグルコース(0.036g/gグルコース)の理論水素収率を有する(図4の点C)。特筆すべきは、この場合に、バイオマス形成と水素産生がしっかりと結びつけられていることであり、これは、最大増殖速度である範囲の水素形成速度(1.38〜2.96mmol/gDW/hr)が可能である(図4の線AB)野生型ネットワークとは対照的である。実験結果(Nandi & Sengupta, 1998)によれば、嫌気的条件下で、クロストリジウム属は、グルコース1モルあたり最高で2モルの水素しか産生することができない。実際、水素産生に対する酪酸の直接の抑制効果及び増殖を阻害する酢酸の間接効果(Chin et al., 2003)が、観察された低い水素収率の原因であることが報告されている。興味深いことに、提案された反応除去は、これらの抑制ボトルネックを直接に回避する。
【0045】
1.2.1.3 Methylobacterium extorquens AM1
本発明者らは次に、基質をグルコースからメタノールに代え、メタノールを唯一の炭素及びエネルギー源として生存する(Van Dien & Lidstrom, 2002)通性メチロトローフ、Methylobacterium extorquens AM1の水素産生を調べた。この生物は十分に研究されており(Anthony, 1982、Chistoserdova et al., 2004、Chistoserdova et al., 1998、Korotkova et al., 2002、Van Dien et al., 2003)、最近になって、その中心代謝の化学量論モデルが発表された(Van Dien & Lidstrom, 2002)。OptStrainのステップ3を使用して、本発明者らは、水素産生を可能にするためには、M. extorquensの代謝ネットワークに反応を1つ導入すればよいことを識別した。2つの候補は、陽子を水素に還元するヒドロゲナーゼ(E.C.#1.12.7.2)、あるいは以下の変換を触媒するN5,N10-メテニルテトラヒドロメタノプテリンヒドロゲナーゼである。
【0046】
E.C.#1.12.98.2: 5,10-メチレンテトラヒドロメタノプテリン⇔5,10-メテニルテトラヒドロメタノプテリン+H2
メチロトローフの中心代謝経路は、Van Dien & Lidstrom, 2002に抜粋されているように、E.coli及びクロストリジウム属の嫌気性菌には見られるヒドロゲナーゼなどの、陽子を水素に変換する反応を含まないため、追加の反応の必要性が予想される。したがって、本発明者らの知識の及ぶ限りにおいて、Pseudomonas AMI、P. methylica(Nandi & Sengupta, 1998)などのメチロトローフを使用して水素産生を達成した者はいないことは驚くべきことではない。識別された反応の追加は、生成された陽子を水素に変換する機構の欠如を指摘することによって、この結果に対するもっともらしい説明を提供する。
【0047】
1.2.2 バニリン産生ケーススタディ
バニリンは重要な香味及び香気分子である。貯蔵バニラ莢からのその低い収率が、バイオテクノロジーによってバニラを生産する研究に動機を与えた。このケーススタディでは、E.coliでグルコースからバニリンをデノボ(de novo)生産する代謝ネットワーク再設計戦略を識別する。本発明者らは最初に、OptStrainを使用し、水素を除く全ての元素に関してバランスの取れた約4,000の候補反応についてLP最適化問題を解くことによって、グルコースからのバニリンの最大理論収率を、0.63g/gグルコースと決定した(ステップ2)。本発明者らは次に、最大収率を達成するために必要な経路をE.coliに与えるためにE.coliに組み込まなければならない非ネイティブ反応の最小数を3と識別した(ステップ3)。収率と組み込む反応の最小化の両方の最適化基準を満たす、補因子の使用だけが異なる多数の代替経路を識別した。例えば、そのような1つの経路は、以下の3つの非ネイティブ反応を使用する:
(i) E.C.#1.2.1.46: ギ酸+NADH+H+⇔ホルムアルデヒド+NAD++H2O、
(ii) E.C.#1.2.3.12: 3,4-ジヒドロキシ安息香酸(又はプロトカテク酸)+NAD++ H2O+ホルムアルデヒド⇔バニリン酸+O2+NADH、及び
(iii) E.C.#12.1.67: バニリン酸+NADH+H+⇔バニリン+NAD++H2O
興味深いことに、これらのステップは本質的に、Li and Frost(1998)よる実験的研究において、組換えE.coli細胞内でグルコースをバニリンに変換するために使用されたステップと同じであり、このことは、この計算手順によって実際に、適当な操作戦略を発見できることを証明している。しかし、報告された実験による収率0.15g/gグルコースは、ネットワークの最大理論収率(すなわち0.63g/gグルコース)からはほど遠く、向上の余地が相当にあることを示していることに留意されたい。
【0048】
このことは、OptKnockを使用して代謝ネットワークを体系的に簡素化にすることによって(ステップ4)、バニリンのより高い収率に到達することが可能かどうかを調べる動機となる。ここでは、上で識別された3つの機能で増補されたE.coli代謝のゲノムスケールモデルを、OptKnockフレームワークに組み込んで、それらを欠失させることによって増殖とバニリン産生の間の結びつきを強化すると考えられる反応セットを決定する。次に、最高バニリン収率の1、2及び4ノックアウト戦略を、ベースグルコース取込み速度10mmol/gDW/hrに関して論じる。全てのケースで、OptKnockによって、バニリン産生に最も好都合な条件として嫌気的条件が選択される。一般に、OptStrainによって識別される欠失戦略は、OptStrainのステップ4に供給される特定の遺伝子追加戦略に左右されることは強調するに値する。したがって、本発明者らは、より優良であるかもしれない他の欠失戦略が、先に暗に言及した他の候補追加戦略に随伴するかどうかを試験した。バニリンケーススタディに対して、本発明者らは、欠失提案及び最大増殖速度での予想されるバニリン収率は、使用される遺伝子追加戦略の如何にかかわらず非常に類似していることを発見した。
OptStrainによって識別された第1の欠失戦略は、アセトアルデヒドデヒドロゲナーゼ(E.C.# 1.2.1.10)を除去して、アセチルCoAのエタノールへの変換を防ぐことを提案する。このネットワークのバニリン産生は、0.205hr-1の最大バイオマス産生速度で、3.9mmol/gDW/hr、又は仮定されたグルコース取込み速度に基づいて0.33g/gグルコースである。この欠失戦略では、エタノール分泌による炭素の損失を妨げることによって、バニリンの前駆代謝産物、ホスホエノールピルビン酸(PEP)及びエリトロース-4-リン酸(E4P)へフラックスが向け直される。第2の(2反応)欠失戦略は、解糖の上半分を本質的に妨げるグルコース6リン酸イソメラーゼ(E.C.#5.3.1.9)をさらに除去することを含む。これらの欠失によって、ネットワークは、エントナー-ドゥドロフ経路に強く依存して、ピルビン酸及びグリセルアルデヒド-3-リン酸(GAP)を生成し、GAPはさらに解糖の下半分でPEPに変換される。ペントースリン酸経路の非酸性部分を通して生み出されるフルクトース-6-リン酸(F6P)は、その後E4Pに変換される。0.06hr-1への最大増殖速度の低減と引き換えに、バニリン産生は、4.78mmol/gDW/hr又は0.40g/gグルコースに増大する。4反応欠失突然変異体ネットワークでは、増殖速度をあまり犠牲にすることなく、かなり高いバニリン産生レベルが予測される。この戦略では、0.052hr-1の最大増殖速度で、6.79mmol/gDW/hr又は0.57g/gグルコースのバニリン産生が得られる。OptKnockフレームワークは、酢酸キナーゼ(E.C.#2.7.2.1)、ピルビン酸キナーゼ(E.C.#2.7.1.40)、PTS輸送機構及びフルクトース6-リン酸アルドラーゼの欠失を提案する。最初の3つの欠失はPEPからフラックスの漏れを防ぎ、その代わりにそれをバニリン合成に向け直す。フルクトース-6-リン酸アルドラーゼの除去は、GAP及びジヒドロキシアセトン(DHA)へのF6Pの直接変換を防ぐ。F6PとGAPはともに、ペントースリン酸経路の非酸性分枝でE4Pを形成するために使用されることに留意されたい。DHAはさらに反応して、ジヒドロキシアセトンリン酸(DHAP)を形成することができ、そのときにPEP分子を消費する。したがって、フルクトース-6-リン酸アルドラーゼの除去は、バニリン合成のために必要なF6PとPEPの両方の利用を防ぐ。さらに、ネットワークの驚くべきフラックスの再分配は、1炭素代謝の一群の反応を使用して、10-ホルミルテトラヒドロ葉酸を形成し、これは続いてホルムアルデヒドに変換される。
【0049】
図5は、さまざまなバイオマス産生速度でバニリン形成を最大化及び最小化することによって得られた、野生型ネットワーク及び突然変異体ネットワークのバニリン産生包絡線を比較した図である。これらの欠失は、任意の増殖条件下で、高いバニリン産生レベルをネットワークに与える。
【0050】
1.3 議論
本発明のOptStrainフレームワークは、小分子だけでなく複雑な分子を過剰産生させるために、微生物系のゲノムスケール代謝ネットワーク全体を体系的に再構成することを目的とする。本発明者らは、これまでに、さまざまな宿主(すなわちE.coli、C. acetobutylicum、M. extorquens)を使用して、いくつかの異なる生成物(例えば1,3-プロパンジオール、イノシトール、ピルビン酸、電子伝達など)を調べた。上で論じた2つのケーススタディ、水素及びバニリンは、OptStrainが、株の再設計に関連した範囲の課題に対処することができることを示している。同時に、OptStrainフレームワークを用いて得られる結果の妥当性(validity)及び適合性(relevance)は、検討される反応データベース及び微生物代謝モデルの完全性(completeness)及び正確さ(accuracy)のレベルに左右されることを強調することは重要である。本発明者らは、特に水素原子に関してバランスが取れていない反応、及び本発明者らが利用した反応データベースの曖昧な反応方向性の多数の例を識別した。本発明者らの全てのケーススタディでは、それに先立ってダウンロードされた反応の慎重なキュレーションを実施した。炭素に関する反応のバランサビリティを復元することができなかったときはいつでも、その反応を考慮から除外した。本発明者らは、反応データベースの試験及び検証用の自動化ツール(Segre et al., 2003)が使用可能になるにつれて、このステップにかかる時間がより短くなるものと予想している。微生物モデルの代謝経路の純粋に化学量論的な表現は、動力学的障壁(kinetic barrier)及び調節的相互作用(例えばアロステリック制御)を考慮しないことによって、非現実的なフラックス分配につながる可能性がある。これを緩和するため、本発明は、ブール制約条件の形態の調節性情報(Covert & Palsson, 2002)をE.coliの化学量論モデルに組み込むこと、及び必要に応じて動力学的表現(kinetic expression)を使用する(Castellanos et al. 2004、Tomita et al., 1999、Varner & Ramkrishna, 1999)ことを企図する。さらに、本発明は、OptKnockを使用して、反応の欠失だけでなく、さまざまな主要反応ステップのアップ又はダウンレギュレーションをも考慮することを企図する。これらの単純化にもかかわらず、OptStrainはすでに、多くのケースで、微生物宿主再設計に対する有用な洞察を提供しており、より重要には、OptStrainは、将来のモデル化の改良に開いた統合フレームワークを初めて確立した。
【0051】
本発明の方法論を実現する際にはコンピュータが使用されることを理解されたい。本発明は、多数のコンピュータを使用することができること、及び多数のタイプのソフトウェア又はプログラム言語を使用することができることを企図する。さらに、本発明は、生み出されるネットワークの表現を記憶することを提供することを理解されたい。ネットワークの表現は、記憶装置、信号、又は生体工学によって操作された生物中に記憶することができる。
【0052】
1.4 OptStrainの数学的公式化
OptStrain手順を使用することによって生成物収率の向上を可能にする微生物代謝ネットワークの再設計は、複数のタイプの最適化問題の解を必要とする。最初の最適化作業(ステップ2)は、代謝産物の集合N={1,...,N}及び反応の集合M={1,...,M}からなる代謝ネットワークにおける所望の生成物の最大収率を決定することを含む。基質の集合Rから(集合Nの中の)特定の生成物Pへの重量ベースの収率を最大にするための線形計画(LP)問題は、下式によって公式化される。
【数2】

【数3】

上式で、MWiは代謝産物iの分子量、vjは反応jのモルフラックス、Sijは、反応jにおける代謝産物iの化学量論係数である。本発明者らの作業においては、代謝産物の集合Nが約4,800種の代謝産物からなり、反応の集合Mが5,700を超える反応からなる。制約条件(1)の不等式は、分泌のみを考慮し、Rの中の基質以外のネットワークの中の全ての代謝産物の取込みを防ぐ。制約条件(2)は、基質の合計取込みフラックス1グラムに対して結果を基準化する。反応フラックスvjは、不可逆的(すなわちvj≧0 )又は可逆的フラックスであることができ、可逆的である場合には、正又は負の値をとることができる。酸素、二酸化炭素、アンモニア、硫酸、リン酸など増殖に不可欠な化合物の取込みを可能にする反応も存在する。
【0053】
OptStrainのステップ3では、ステップ2で識別された最大収率を満たすのに必要な非ネイティブ反応の最小数を見つける。最初に、検討中の微生物宿主の代謝モデル中に存在しないUniversalデータベース中の反応に、非ネイティブのフラグを立てる。これは、以下の混合整数線形計画(MILP)問題を与える。
【数4】


集合Mnon-nativeは、検討中の宿主の非ネイティブ反応を含み、集合Mの部分集合である。制約条件(1)及び(2)は、生成物収率最大化問題における制約条件(1)及び(2)と同じである。制約条件(3)は、生成物収率が、ステップ2で計算された最大理論収率Yieldtargetを満たすことを保証する。制約条件(4)及び(5)の2値変数yjは、反応をオン又はオフにするスイッチの役目を果たす。yjの値0は、対応するフラックスvjが0であることを強制し、値1は、対応するフラックスvjが、0でない値をとることを可能にする。パラメータvjmin及びvjmaxはそれぞれ、非常に低い値及び非常に高い値をとることができ、又は、制約条件(1〜3)に従って全てのそれぞれの反応フラックスvjを最小化及び最大化することによって計算することができる。
【0054】
最大収率と非ネイティブ反応の最小化の両方の最適化基準を満たす代替経路は、整数切断(integer cut)と呼ばれる追加の制約条件の累積の後のMILP公式の反復解によって得られる。整数切断制約条件は、以前に識別された反応の全ての集合を考慮から除外する。例えば、以前に識別された経路が反応1、2及び3を利用する場合、下式の制約条件は、同じ反応が、その後の解の中で同時に考慮されることを防ぐ: y1+y2+y3≦2。さらなる詳細はBurgard and Maranas (2001)に出ている。
【0055】
OptStrainのステップ4は、非ネイティブ機能で増補されたネットワークから排除する反応を、以前に開発されたOptKnockフレームワーク(Burgard et al., 2003、Pharkya et al., 2003)を使用して識別する。このステップの目的は、ネットワークの表現型の挙動を制限して、増殖と所望の生化学物質の形成とが結びつけられるようにし、それによって副生物の形成を削減することにある。ネットワークに対して使用可能なさまざまなレベルのバイオマス形成速度について、生化学的産生を最大化し、次いで最小化する一連の線形最適化問題を解くことによって、許される標的生成物収率の包絡線をバイオマス収率に対して描く。最適化公式化に関する詳細はPharkya et al., 2003に出ている。以上の最適化問題は全て、IBM RS6000-270ワークステーション上のGAMS(Brooke et al., 1998)モデル化環境を経由してアクセスされるCPLEX 7.0(http://www.ilog.com/products/cplex/)を使用して、数分から数時間で解かれた。
【0056】
2. OptKnock
ゲノムスケールで、したがって体系的に単細胞生物の代謝を調べることができることは、株操作戦略を識別することを目的とする新規の計算法の必要性に動機を与える。本発明は、E.coliでの特定の化合物の過剰産生につながる遺伝子欠失戦略を提案するOptKnockと呼ばれる計算フレームワークを含む。これは、代謝ネットワークの接続性を「形にする」ことにより所望の化学物質の産生が増殖の必須の副生物になることを保証することによって達成される。言い換えると、OptKnockは、細胞増殖を化学的産生から分離する能力を有する代謝反応を識別し、続いてこれを欠失させる。この計算手順は、Palsson及びその共同研究者のin silico E.coliモデル(Edwards & Palsson, 2000)の中に要約されたE.coli代謝ネットワーク全体を同時に考慮することによって、直接的なノックアウト戦略だけでなく、非直感的なノックアウト戦略をも識別するように設計される。このネットワークの複雑さ及び組み込まれた冗長性(例えばこのE.coliモデルは720の反応を包含する)は、候補遺伝子ノックアウト戦略の組合せ的爆発(combinatorial explosion)と戦うために、体系的で効率的な探索法を必要とする。
【0057】
図6に示された入れ子にされた最適化フレームワークは、細胞増殖目的を、外部から課せられた化学的産生目的に最大限に結びつける複数の遺伝子欠失組合せを識別するように作られている。2つの競合する最適戦略(すなわち細胞目的と化学的産生)を含むこの多層最適化構造は、2層最適化問題(Bard, 1998)と呼ばれる。問題公式化の詳細及び線形計画(LP)双対理論を利用したエレガントな解手順については「方法」セクションで説明する。このOptKnock手順を、E.coliでのコハク酸、乳酸及び1,3-プロパンジオール(PDO)産生に適用し、固定グルコース取込み量でのバイオマス収率の最大化を細胞目的として使用する。得られた結果をさらに、代謝調整の最小化(minimization of metabolic adjustment:MOMA)(Segre et al., 2002)を細胞目的として使用して対比させる。OptKnockフレームワークに基づいて、E.coliでのコハク酸、乳酸及びPDO産生の文脈における最も有望な遺伝子ノックアウト戦略及びバイオマス産生に対するそれらの対応する許される化学物質生成の包絡線を識別することが可能である。
【0058】
本発明の好ましい一実施形態は、増殖資源(すなわち炭素、酸化還元電位及びエネルギー)のために必要な代謝産物/化合物に向けての消費が、化学量論により、所望の化学物質の産生を伴わなければならないことを保証することによってE.coliにおける化学的産生につながると考えられる遺伝子欠失戦略を提案する、OptKnockと呼ばれる計算フレームワークを記述する。したがって、所望の生成物の産生は、細胞増殖の必須の副生物になる。具体的には、OptKnockは、代謝ネットワークから除去する反応を特定し、その反応の除去は、識別された機能に関連した遺伝子を欠失させることによって実現することができる。この手順を、E.coli K-12でのコハク酸、乳酸及びPDO産生に基づいて立証した。得られた結果は、文献に発表された株と良好な一致を示す。提案された遺伝子欠失のいくつかは、所望の経路と競合する反応経路を本質的に狭めるので、非常に直接的であるのに対して、他の多くの遺伝子欠失は、E.coliの代謝ネットワークの複雑さ及び組み込まれた冗長性を反映して、最初は非常に非直感的である。コハク酸の場合、OptKnockは、この問題に対する直接の入力としてではなく、細胞目的と化学的産生目的の間の競合の結果として、嫌気的発酵及びホスホトランスフェラーゼグルコース取込み機構の欠失を正確に提案した。乳酸のケーススタディでは、グルコキナーゼベースのグルコース取込み機構が、あるノックアウト戦略に関して乳酸産生とバイオマス産生を分断することが示された。PDOの場合には、エネルギー効率は大幅に悪いにもかかわらず、エントナー-ドゥドロフ経路のほうがEMP解糖よりも有利であることを示す結果が得られた。さらに、これまで一般的であったtpiノックアウトが、PDOの最大収率を低減させることが明白に示され、一方で、15個の反応からなる複雑なネットワークが、PPP経路からTCA回路にフラックスを「漏らす」ことが理論的に可能であり、したがってPDO産生をバイオマス形成から分断することが示された。得られた結果はさらに、細胞目的の選択に関して非常にロバストであるように見えた。
【0059】
本発明は、増殖速度の最大化、ATP産生の最大化、代謝調整の最小化、養分吸収の最小化、酸化還元産生の最小化、ユークリッドノルム(Euclidean norm)の最小化、ならびにこれらの細胞目的及びその他の細胞目的の組合せを含む、多数の細胞目的を企図する。ただしこれらに限定されるわけではない。
【0060】
提案された遺伝子欠失戦略は慎重に解釈しなければならないことに留意することは重要である。例えば、多くの場合、分枝経路の一方の枝の遺伝子の欠失は、もう一方の枝の有意なアップレギュレーションと等価である。さらに、遺伝子欠失の前後のフラックス変化の検査は、どの遺伝子をアップ又はダウンレギュレートするかに関する見通しを提供する。最後に、除去の標的とされる識別された反応の集合をその対応する遺伝子にマップする問題は、一意的に指定されるとは限らない。したがって、アイソザイム及び多機能酵素を考慮した最も経済的な遺伝子セットの注意深い識別を実施する必要がある。
【0061】
OptKnockフレームワークでは、基質取込みフラックス(すなわちグルコース)を10mmol/gDW/hrとすることが好ましい。したがって、全ての報告された化学的産生値及びバイオマス形成値は、予測されたものではないこの仮定の取込みシナリオに基づく。したがって、提案された欠失突然変異体が大幅に低い取込み効率を含むことはよくあることである。しかし、OptKnockは、増殖と化学的産生とが結びつけられた突然変異体を本質的に提案するので、改良された取込み効率、したがって高められた所望の化学的産生特性を有する突然変異体を連続的に発展させる増殖選択系を想像することができる。
【0062】
フラックス配分(flux allocation)を実行する内部最適化問題の純粋に化学量論的な表現の中に調節又は動力学的情報がない場合、OptKnockは、化学的過剰産生のための唯一の機構として任意の遺伝子欠失を識別するために使用される。明らかに、モデルに調節又は動力学的情報が含まれないことは、場合によっては非現実的なフラックス分配を提案しかねない単純化である。調節情報の組込みは、フラックス配分をより適当に解くことによって、提案された遺伝子欠失の質を高めるだけでなく、株改良のための機構として遺伝子欠失とともに調節変更を提案することも可能にする。使用可能な場合には、代替モデル化法(例えばサイバネティック(cybernetic)(Kampala et al., 1984、Ramakrishna et al., 1996、Varner and Ramkrishna, 1999)、代謝制御解析(metabolic control analysis)(Kacser and Bums, 1973、Heinrich and Rapoport, 1974、Hatzimanikatis et al., 1998))の使用を、OptKnockフレームワークの中に組み込んで、遺伝子が欠失した代謝ネットワークの代謝フラックス分配をより正確に推定することができる。とはいえ、このような調節又は動力学的情報がなくても、OptKnockは、株改良のための有用な提案を提供し、より重要には、体系的なフレームワークを確立する。本発明は、代謝及び調節モデル化フレームワークの将来の改良をおのずから企図する。
【0063】
2.1 方法
グルコース基質によって活性化される代謝産物の集合N={1,...,N}及び代謝反応の集合M={1,...,M}を含む定常状態代謝ネットワークに対する総反応フラックスとして定量化される細胞目的の最大化は、数学的には以下のように表現される。
【数5】

上式で、Sijは、反応jにおける代謝産物iの化学量論係数であり、vjは、反応jのフラックスを表し、vglc_uptakeはベースグルコース取込みシナリオ、vatp_mainは、非増殖関連ATP維持要件(non-growth associated ATP maintenance requirement)、vbiomasstargetは、バイオマス産生の最小レベルである。ベクトルvは、内部反応と輸送反応の両方を含む。輸送フラックスの順(すなわち正)方向は、特定の代謝産物の取込みに対応し、逆(すなわち負)方向は代謝産物の分泌に対応する。ホスホトランスフェラーゼ系及びグルコキナーゼを介したグルコースの取込みはそれぞれ、vpts及びvglkによって表される。ネットワークから分泌されるだけの代謝産物の輸送フラックスは、Msecr_onlyの要素である。反応の完全な集合Mは、可逆反応Mrev及び不可逆反応Mirrevに再分割されることにも留意されたい。細胞目的はしばしば、バイオマス形成のために必要な比の生合成前駆物質の消費であると仮定される(Neidhardt and Curtiss, 1996)。これらのフラックスはgDW・hrあたりで報告され、そのためバイオマス形成は、g産生バイオマス/gDW・hr又は1/hrとして表現される。
【0064】
遺伝子欠失のモデル化、したがって反応除去のモデル化は最初に、このフラックスバランス解析フレームワークへの2値変数の組込みを必要とする。(Burgard and Maranas, 2001; Burgard et al., 2001)。これらの2値変数
【数6】

は、反応jが活性の場合、値1をとり、反応jが不活性の場合、値0をとる。以下の制約条件
【数7】

は、変数yjが0に場合にだけ反応フラックスvjが0にセットされることを保証する。あるいは、yjが1のとき、vjは、下限vjminと上限vjmaxの間の任意の値を自由にとることができる。この検討では、vjmin及びvjmaxが、主問題からの制約条件に従う全てのそれぞれの反応フラックスを最小化し、続いて最大化することによって識別される。
【0065】
最適遺伝子/反応ノックアウトの識別は、細胞目的の最適化が、関心の化学物質又は生化学物質の過剰産生に間接的につながるようにアクセスすることができる(yj=1)反応の集合を選択する2層最適化問題の解を必要とする(図6も参照されたい)。バイオマス形成を細胞目的として使用して、これは、以下の2層混合整数最適化問題として数学的に表現される。
【数8】

上式で、Kは、許されるノックアウトの数である。最後の制約条件は、結果として生じるネットワークが最小バイオマス収率vbiomasstargetを満たすことを保証する。
【0066】
フラックス空間の高い次元数(すなわち700を超える反応)及び2つの入れ子にされた最適化問題の存在のため、この2段階最適化問題の直接解は扱いにくい。これを正すため、本発明者らは、あらゆる線形計画問題(主問題)について、その最適目的値が主問題のそれに等しい一意的な最適化問題(双対問題)が存在することを示す、LP双対理論から借用する効率的な解法を開発する。同様の戦略が、Burgard and Maranas, 2003によって、代謝フラックスデータから代謝目的関数を識別/試験するために使用された。OptKnock内部問題に関連した双対問題(Ignizio and Cavalier, 1994)は以下のとおりである。
【数9】

上式で、λistoichは、化学量論的制約条件に関連した双対変数、glcは、グルコース取込み制約条件に関連した双対変数、μjは、主問題中の対応するフラックスvjに対する他の任意の制限に関連した双対変数である。双対変数μjは、OptKnock内部問題中の対応するフラックスが0にセットされている場合、yj=0を強制することによって、無制限の符号を獲得することに留意されたい。パラメータμjmin及びμjmaxは、双対問題の制約条件に従うそれらの値を最小化し、続いて最大化することによって識別される。
【0067】
主問題及び双対問題に対する最適解が境界にある場合、それらの目的関数値は、最適性(optimality)において互いに等しくなければならない。このことは、それらの目的を互いに等しく設定し、それらのそれぞれの制約条件を累積することによって、両方の問題に対するあらゆる最適の解を特徴づけることができることを意味する。したがって、以前に示されたOptKnockに対する2層公式化は、以下の単層(single-level)MILPに変換することができる。
【数10】


上記の公式化の重要な特徴は、問題が実行可能な場合、最適解が常に見つかることである。本発明では、遺伝子ノックアウトの候補が、解糖、TCA回路、ペントースリン酸経路、呼吸及び全てのアナプレロティック反応のうちの全ての反応を含む。ただしこれらに限定されるわけではない。これは、総和(すなわち
【数11】

)に含まれる反応の数を限定することによって達成される。100個の2値変数を含む問題は、IBM RS6000-270ワークステーション上のGAMSモデル化環境を経由してアクセスされるCPLEX 7.0を使用して、数分から数時間で解かれた。しかし、本発明は、使用される特定のタイプのコンピュータ又は環境に依存しないことを理解されたい。本発明の方法に関連した情報を入力し出力することができる任意のタイプのコンピュータ又は環境を使用することができる。さらに、本発明の方法の諸ステップは、多数のタイプのソフトウェアアプリケーション又は言語で実現することができ、本発明はこの点に関して限定されない。他の実施形態及び使用は当業者には明白であり、本発明はこれらの特定の例示的な例に限定されないことを理解されたい。
【実施例】
【0068】
2.2 実施例1
コハク酸及び乳酸産生
それらがある場合に、E.coli K-12 化学量論モデル(Edwards & Nissen, 2000)から除去して、バイオマス最大化がフラックス配分のよい記述子であるときにはいつでも残りのネットワークがコハク酸又は乳酸を産生するようにすることができる反応を識別した。予め指定された量のグルコース(10mmol/gDW/hr)、ならびに無機リン酸塩、酸素、硫酸塩及びアンモニアの制約のない取込み経路を提供して、代謝ネットワークを活性化する。この最適化ステップは、グルコースの取込みのためにホスホトランスフェラーゼ系又はグルコキナーゼ、あるいはその両方の機構を選ぶことができる。酢酸、二酸化炭素、エタノール、ギ酸、乳酸及びコハク酸の分泌経路も使用可能にされる。グルコース取込み速度は固定なので、バイオマス及び生成物収率はそれぞれ、バイオマス及び生成物産生速度と本質的に等しいことに留意されたい。全てのケースで、OptKnock手順は酸素摂取反応を排除し、現在のコハク酸(Zeikus et al., 1999)及び乳酸(Datta et al., 1995)発酵性産生戦略と矛盾しない嫌気的増殖条件を指示した。
【0069】
表Iに、コハク酸過剰産生用の識別された3つの遺伝子ノックアウト戦略(すなわち突然変異体A、B及びC)に概要を示す。突然変異体Aの結果は、ネットワークから2つの反応(すなわちピルビン酸ギ酸リアーゼ及び乳酸デヒドロゲナーゼ)を除去すると、コハク酸産生は、最大バイオマス収率におけるその理論最大値の63%に達することを示唆した。このノックアウト戦略は、Stols and Donnelly (1997)によって、彼らのコハク酸過剰産生E.coli株において使用された戦略と同一である。次に、バイオマス産生に対する許されるコハク酸の包絡線を、野生型E.coliネットワーク及び表Iに示された3つの突然変異体について調べた。コハク酸産生の限界は、突然変異体Aが、バイオマス収率が最大値の80%に近づくまで、コハク酸とバイオマス形成の結合を示さないことを明らかにした。しかし、アセトアルデヒドデヒドロゲナーゼをさらに欠失した突然変異体Bは、ずっと以前に、コハク酸とバイオマス収率との結合を示した。
【0070】
競合する副生物(すなわちエタノール、ギ酸及び乳酸)の産生機構を排除するのではなく、2つのPEP消費反応を不活化することに重点を置いた突然変異体Cでは、より非直感的な戦略が識別された。最初に、ホスホトランスフェラーゼ系を使用不能にして、ネットワークが、グルコースの取込みをグルコキナーゼだけに依存するようにした。次に、ピルビン酸キナーゼを除去し、解糖によって供給されるかなりの量のPEPを排出する能力を有する唯一の中心代謝反応としてPEPカルボキシキナーゼを残した。この戦略は、最大バイオマス収率を達成できると仮定して、理論最大値の88%に近いコハク酸収率を与えた。さらに、達成可能なあらゆるバイオマス収率に対してかなりのコハク酸産生が見られ、コハク酸の最大理論収率は野生株のそれと同じであった。
【0071】
次に、OptKnockフレームワークを適用して、乳酸産生とバイオマス産生とを結びつけるためのノックアウト戦略を識別した。表Iは、識別された3つの遺伝子ノックアウト戦略(すなわち突然変異体A、B及びC)を示す。突然変異体Aは、酢酸及びエタノール産生を妨げることによって、最大バイオマス収率におけるフラックスを乳酸に向け直す。この結果は、adh、pta突然変異体E.coli株が、グルコースを基質として、乳酸を産生することによって嫌気的に増殖することができることを証明する以前の研究(Gupta & Clark, 1989)と矛盾しない。突然変異体Bは、初期解糖反応及び酢酸産生機構の除去を含む代替戦略を提供する。これは、最大バイオマス収率で、その理論的限界の90%の乳酸収率を与える。このネットワークは、乳酸の産生を回避し、その一方でバイオマス形成を最大化することができることにも留意されたい。これは、OptKnockが、に「最悪ケース」代替解を明示的に考慮しないことによる。グルコキナーゼ及びエタノール産生機構をさらに除去した突然変異体Cは、乳酸産生とバイオマス産生の間のよりタイトな結びつきを示したことを理解されたい。
【0072】
表I-OptKnockによって識別されたさまざまな遺伝子ノックアウト戦略のバイオマス及び化学収率。それぞれのノックアウト戦略の反応及び対応する酵素が示されている。最大バイオマス及び対応する化学収率は、供給グルコース10mmol/hr及び細胞1gDWをベースに提供されている。右端の列は、野生型(欠失なし)E.coliネットワークからの代謝フラックスの最小再分配を仮定(MOMA仮定)した同じベースの化学収率を提供する。1,3-プロパンジオールの場合、両方のノックアウト戦略に対してグリセリン分泌が使用不能にされていた。
【表1】

【0073】
2.2 実施例2
1,3-プロパンジオール(PDO)産生
最適な遺伝子ノックアウト戦略を案出することに加えて、OptKnockを使用して、E.coliにおけるPDO産生など、遺伝子の欠失とともに遺伝子の追加が必要な株を設計した。微生物1,3-プロパンジオール(PDO)産生法は、グリセリンを主たる炭素源として利用してすでに開発されているが(Hartlep et al., 2002、Zhu et al., 2002)、単一の微生物でのグルコースからの1,3-プロパンジオールの直接産生は最近、かなりの関心を集めている(Cameron et al., 1998、Biebl et al., 1999、Zeng & Biebl, 2002)。野生型E.coliは、PDO産生に必要な経路を欠いているため、最初に遺伝子追加フレームワークを使用して(Burgard and Maranas, 2001)、E.coliでグルコースからPDOを産生するために必要な追加の反応を識別した。この遺伝子追加フレームワークは、グリセリンホスファターゼによるグリセリン-3Pのグリセリンへの変換、及びその後のグリセリンデヒドラターゼ及び1,3-プロパンジオールオキシドレダクターゼによるグリセリンの1,3-プロパンジオールへの変換を含む、直接的な3反応径路を識別した。次いでこれらの反応をE.coli化学量論モデルに追加し、続いてOptKnock手順を適用した。
【0074】
OptKnockは、PDO産生とバイオマス産生とが結びついた突然変異体は、1反応欠失突然変異体にも、又は2反応欠失突然変異体にもないことを明らかにした。しかし、PDO産生をバイオマス産生に結びつけることができる1つの3欠失ノックアウト戦略及び複数の4欠失ノックアウト戦略が識別された。これらのノックアウト戦略のうちの2つが表Iに示されている。それらの結果は、E.coliネットワークからある主要機能を除去すると、グルコースで増殖するPDO過剰産生突然変異体が得られることを示唆した。具体的には、グリセリンの分解を防ぐ追加のノックアウトとともに2つの解糖反応を除去すると、最大バイオマス収率におけるPDOの理論的最大収率の72%に達する能力を有するネットワークが得られることを、表Iは示している。グリセルアルデヒド-3-リン酸デヒドロゲナーゼ(gapA)ノックアウトは、DuPont社によって、そのPDO過剰産生E.coli株において使用された(Nakamura, 2002)ことに留意されたい。突然変異体Bは、同様のPDO収率及び38%高いバイオマス収率を示す、トリオースリン酸イソメラーゼ(tpi)酵素の除去を含む代替戦略を示した。興味深いことに、最近、トリオースリン酸イソメラーゼ活性を欠く酵母株が、PDOへの主要前駆物質であるグリセリンを、その最大理論収率の80〜90%で産生することが報告された(Compagno et al., 1996)。
【0075】
バイオマス収率を最大化する野生型E.coli、突然変異体A及び突然変異体Bネットワークのフラックス分配を精査すると、驚くことではないが、両突然変異体A及びBで、グリセリンからグリセリンアルデヒドへの更なる変換が途絶されたことが分かる。突然変異体Aでは、解糖の上部及び下部の2つの反応を除去した結果、ペントースリン酸経路及び解糖経路(トリオースリン酸イソメラーゼを除く)がほぼ完全に失活した。それを補うため、エントナー-ドゥドロフ解糖経路が活性化されて、グルコースからのフラックスを、ピルビン酸及びグリセルアルデヒド-3-リン酸(GAP)へ導く。GAPは次いでグリセリンに変換され、グリセリンは次いでPDOに変換される。解糖フラックスの減少に伴って、野生型E.coliネットワークのケースよりも低下したエネルギー需要は、TCA回路フラックスの増大によって満たされる。突然変異体Bに対して提案されたノックアウトは、明確に異なる機構によって、PDOの産生に向けてフラックスを向け直す。ペントースリン酸経路の最初の反応の除去の結果、代謝フラックスは全体が解糖の最初の段階に流れる。フルクトースビスリン酸アルドラーゼ接合部で、フラックスの流れは2つの代謝生成物、PDOに変換されるジヒドロキシアセトンリン酸(DHAP)と、解糖の後半へと続くGAPとに分けられる。トリオースリン酸イソメラーゼ反応の除去は、DHAPとGAPの間の相互変換を防ぐ。興味深いことに、第4のノックアウトは、バイオマス形成と化学的産生の間の結びつきを維持することが予測される。このノックアウトは、一体としてリボース-5-リン酸(R5P)を酢酸及びGAPに変換し、それによって化学的産生から増殖を分断する、15個の反応を含む複雑な経路を通してのフラックスの「漏れ」を防ぐ。
【0076】
次に、表Iに示された2つの突然変異体について、バイオマス収率に対する許されるPDO産生の包絡線を調べる。突然変異体及び本来のE.coliネットワークの産生限界は、バイオマス収率が最大化されることになっている場合には、野生型E.coliネットワークが、PDOを産生する「誘因」を持たないことを示す。一方、多くのバイオマスが形成される場合、遺伝子除去後のネットワークの機能の低減により、両突然変異体A及びBは、かなりの量のPDOを産生しなければならない。突然変異体Aは、バイオマス産生とPDO産生の比を本質的にセットするtpiノックアウトを回避することによって、PDOのより高い最大理論収率によって特徴づけられる。上述の結果は、PDOへの主要中間体としてのグリセリンの使用にかかっている。次に、1,3-プロパンジオール産生のためのグリセリン変換経路の代替経路を利用する可能性を調べた。
【0077】
本出願人らは、3-ヒドロキシプロピオン酸(3-HPA)を生成するマロニルCoAの2段階NADPH依存還元(Menendez et al., 1999、Hugler et al., 2002)を含むChloroflexus aurantiacusにおける経路を識別した。3-HPAを1,3-プロパンジオールに変換する生物機能はないので、3-HPAは次いで、1,3-プロパンジオールに化学的に変換することができる。その最初のステップ(アセチル-CoAカルボキシラーゼ)が炭素固定反応であるため、この経路は、グリセリン経路を通してのPDO産生に優る重要な利点を提供する。したがって、3-HPAの最大理論収率(1.79mmol/mmolグルコース)は、グリセリン変換経路を通してのPDO産生のそれ(1.34mmol/mmolグルコース)よりもかなり高い。3-HPA産生経路追加後のOptKnockフレームワークの適用は、バイオマス形成が3-HPA産生に結びつけられるまでには、より多くのノックアウトが必要であることを明らかにした。最も興味深い戦略の1つは、最適増殖速度におけるその理論最大値の91%の3-HPA産生を与える9つのノックアウトを含む。最初の3つノックアウトは、競合する酢酸、乳酸及びエタノール産生機構の除去を含むので、比較的に直接的であった。さらに、エントナー-ドゥドロフ経路(ホスホグルコン酸デヒドラターゼ又は2-ケト-3-デオキシ-6-ホスホグルコン酸アルドラーゼ)、4つの呼吸反応(すなわちNADHデヒドロゲナーゼI、NADHデヒドロゲナーゼII、グリセリン-3-リン酸デヒドロゲナーゼ及びコハク酸デヒドロゲナーゼ複合体)、及び初期の解糖ステップ(すなわちホスホグルコースイソメラーゼ)が途絶される。この戦略は、最大バイオマス収率を仮定して、グリセリン変換経路を利用した以前に識別された突然変異体よりも69%高い3-HPA収率を与えた。
【0078】
2.3 実施例3
代替細胞目的:代謝調整の最小化
上述の結果は全て、フラックス配分を駆動する細胞目的としてバイオマス収率の最大化を生じさせることによって得られたものである。この仮説は、本質的に、変化する環境条件下でバイオマス収率の最大化を維持するために、代謝ネットワークは任意に変化することができ、かつ/又は調節ループを配線し直すことさえできると仮定する(最大応答)。最近の証拠は、これが時に、複数回の増殖選択サイクルの後に、E.coliのK-12株によって達成されることを示唆している(Ibarra et al., 2002)。このセクションでは、遺伝子欠失後の代謝ネットワークによる近視眼的な(最小)応答を仮定した対照的な仮説(すなわち代謝調整の最小化(MOMA)(Segre et al., 2002))を検討した。具体的には、MOMA仮説は、代謝ネットワークが、遺伝子欠失によっては到達できないその系の本来の定常状態のできるだけ近くにとどまろうとすることを示唆する。この仮説は、遺伝子欠失事象の直後のフラックス配分をより正確に記述することが示された(Segre et al., 2002)。この研究では、MOMA目的を利用して、OptKnockによって識別される突然変異株におけるフラックス分配を予測した。乳酸及びコハク酸シミュレーションに対するベースケースは、嫌気的条件下の最大バイオマス形成であると仮定され、PDOシミュレーションに対するベースケースは、好気的条件下の最大バイオマス形成であった。結果は、表1の最後の列に示されている。全てのケースで、提案された複数の遺伝子ノックアウト戦略は、MOMAケースの化学的産生収率が、最大バイオマス仮説に比べてわずかに低いだけであることを示唆している。これは、OptKnock結果が、細胞目的の選択に関してかなりロバストであることを暗示する。
【0079】
3.0 代替実施形態
本発明の背景を照らし出し、又は実施に関する追加の詳細を提供する刊行物及び明細書において使用された他の資料は、それらの全体が参照によって本明細書に組み込まれる。本発明は、生物の変異、細胞目的の変異、生体工学目的の変異、形成される最適化問題及び使用される解のタイプの変異を含む、多数の変異を企図する。これらの変異、修正又は変更及び/あるいはその他の変異、修正又は変更は、添付の請求項に記載された本発明の趣旨及び範囲から逸脱することなく実施することができる。
【0080】
参考文献
Anthony, C. (1982) The Biochemistry of Methylotrophs (Academic Press.)
Arita, M. (2000) Simulation Practice and Theory 8, 109-125.
Arita, M. (2004) Proc Natl Acad Sci U S A 101, 1543-7.
Badarinarayana, V., Estep, P.W., 3rd, Shendure, J., Edwards, J., Tavazoie, S., Lam, F., Church, G.M. (2001) Nat Biotechnol 19(11): 1060-5.
Bailey, J. E. (1991) Science 252, 1668-75.
Bailey, J. E. (2001) Nat Biotechnol 19, 503-4.
Bard, J. F. 1998. Practical bilevel optimization : algorithms and applications. Dordrecht ; Boston, Kluwer Academic.
Biebl, H., Menzel, K., Zeng, A.P., Deckwer, W.D. (1999) Appl Environ Microbiol 52: 289-297.
Bond, D. R. & Lovley, D. R. (2003) Appl Environ Microbiol 69, 1548-55.
Bond, D. R., Holmes, D. E., Tender, L. M. & Lovley, D. R. (2002) Science 295, 483-5.
Brown, M. (1999) Perl programmer's reference (Osborne/McGraw-Hill, Berkeley, Calif.).
Burgard, A. P. & Maranas, C. D. (2001) Biotechnol Bioeng 74, 364-375.
Burgard, A. P., Pharkya, P. & Maranas, C. D. (2003) Biotechnol Bioeng 84, 647-57.
Burgard, A. P., Maranas, C. D. (2003) Biotechnol Bioeng 82(6): 670-7.
Burgard, A. P., Vaidyaraman, S., Maranas, C. D. (2001) Biotechnol Prog 17: 791-797.
Cameron, D. C., Altaras, N. E., Hoffman, M. L., Shaw, A. J. (1998) Biotechnol Prog 14(1): 116-25.
Castellanos, M., Wilson, D. B. & Shuler, M. L. (2004) Proc Natl Acad Sci U S A 101, 6681-6.
Causey, T. B., Shanmugam, K. T., Yomano, L. P. & Ingram, L. O. (2004) Proc Natl Acad Sci U S A 101, 2235-40.
Chin, H. L., Chen, Z. S. & Chou, C. P. (2003) Biotechnol Prog 19, 383-8.
Chistoserdova, L., Laukel, M., Portais, J. C., Vorholt, J. A. & Lidstrom, M. E. (2004) J Bacteriol 186, 22-8.
Chistoserdova, L., Vorholt, J. A., Thauer, R. K. & Lidstrom, M. E. (1998) Science 281, 99-102.
Compagno, C., Boschi, F., Ranzi, B. M. (1996) Biotechnol Prog 12(5): 591-5.
Covert, M.W., Palsson, B.O. (2002) J Biol Chem 277(31): 28058-64.
Covert, M.W., Schilling, C.H., & Palsson, B.O. (2001) J Theor Biol 213(1): 73-88.
Das, D. & Veziroglu, T. N. (2001) International Journal of Hydrogen Energy 26, 13-28.
Datta, R., Tsai, S., Bonsignore, P., Moon, S., Frank, J. R. (1995) FEMS Microbiol. Rev. 16: 221-231.
David, H., Akesson, M. & Nielsen, J. (2003) Eur J Biochem 270, 4243-53.
Desai, R. P., Nielsen, L. K. & Papoutsakis, E. T. (1999) J Biotechnol 71, 191-205.
Edwards, J. S. & Palsson, B. O. (2000) Proc Natl Acad Sci U S A 97, 5528-33.
Edwards, J. S., Ibarra, R. U., Palsson, B. O. (2001) Nat Biotechnol 19(2): 125-30.
Edwards, J. S., Palsson, B. O. (2000) Proc Natl Acad Sci U S A 97(10): 5528-33.
Ellis, L. B., Hou, B. K., Kang, W. & Wackett, L. P. (2003) Nucleic Acids Res 31, 262-5.
Eppstein, D. (1994) in 35th IEEE Symp. Foundations of Comp. Sci, Santa Fe), pp. 154-165.
Fan, L. T., Bertok, B. & Friedler, F. (2002) Comput Chem 26, 265-92.
Finneran, K. T., Housewright, M. E. & Lovley, D. R. (2002) Environ Microbiol 4, 510-6.
Forster, J., Famili, I., Fu, P. C., Palsson, B., Nielsen, J. (2003) Genome Research 13(2): 244-253.
Forster, J., Famili, I., Fu, P., Palsson, B. O. & Nielsen, J. (2003) Genome Res 13, 244-53.
Gupta, S., Clark, D. P. (1989) J Bacteriol 171(7): 3650-5.
Hartlep, M., Hussmann, W., Prayitno, N., Meynial-Salles, I., Zeng, A. P. (2002) Appl Microbiol Biotechnol 60(1-2): 60-6.
Hatzimanikatis, V., Emmerling, M., Sauer, U., Bailey, J. E. (1998) Biotechnol Bioeng 58(2-3): 154-61.
Hatzimanikatis, V., Li, C., Ionita, J. A. & Broadbelt, L. J. (2003) presented at Biochemical Engineering XIII Conference, Session 2, Boulder, CO.
Heinrich, R., Rapoport, T. A. (1974) Eur. J. Biochem. 41: 89-95.
Hugler, M., Menendez, C., Schagger, H., Fuchs, G. (2002) J Bacteriol 184(9): 2404-10.
Ibarra, R. U., Edwards, J. S., Palsson, B. O. (2002) Nature 420(6912): 186-9.
Ignizio, J.P., Cavalier, T.M. 1994. Linear programming. Englewood Cliffs, N.J., Prentice Hall.
Kacser, H., Burns, J. A. (1973). Symp. Soc. Exp. Biol. 27: 65-104.
Kanehisa, M., Goto, S., Kawashima, S., Okuno, Y. & Hattori, M. (2004) Nucleic Acids Res 32 Database issue, D277-80.
Karp, P. D., Riley, M., Saier, M., Paulsen, I. T., Collado-Vides, J., Paley, S. M., Pellegrini-Toole, A., Bonavides, C. & Gama-Castro, S. (2002) Nucleic Acids Res 30, 56-8.
Kataoka, N., Miya, A. & Kiriyama, K. (1997) Wat. Sci. Tech. 36, 41-47.
Kompala, D. S., Ramkrishna, D., Tsao, G. T. (1984) Biotechnol Bioeng 26(11): 1272-1281.
Korotkova, N., Chistoserdova, L. & Lidstrom, M. E. (2002) J Bacteriol 184, 6174-81.
Krieger, C. J., Zhang, P., Mueller, L. A., Wang, A., Paley, S., Arnaud, M., Pick, J., Rhee, S. Y. & Karp, P. D. (2004) Nucleic Acids Res 32 Database issue, D438-42.
Li, K. & Frost, J. W. (1998) Journal of American Chemical Society 120, 10545-10546.
Liu, H., Ramnarayanan, R. & Logan, B. E. (2004) Environmental Sceince and Technology 38, 2281-2285.
Lovley, D. R. (2003) Nat Rev Microbiol 1, 35-44.
Majewski, R.A., Domach, M. M. (1990) Biotechnol Bioeng 35: 732-738.
Mavrovouniotis, M., Stephanopoulos, G. & Stephanopoulos, G. (1990) Biotechnol Bioeng 36, 1119-1132.
McShan, D. C., Rao, S. & Shah, I. (2003) Bioinformatics 19, 1692-8.
Menendez, C., Bauer, Z., Huber, H., Gad'on, N., Stetter, K.O., Fuchs, G. (1999) J Bacteriol 181(4): 1088-98.
Methe, B. A., Nelson, K. E., Eisen, J. A., Paulsen, I. T., Nelson, W., Heidelberg, J. F., Wu, D., Wu, M., Ward, N., Beanan, M. J., et al. (2003) Science 302, 1967-9.
Misawa, N., Yamano, S. & Ikenaga, H. (1991) Appl Environ Microbiol 57, 1847-9.
Nakamura, C. E. & Whited, G. M. (2003) Curr Opin Biotechnol 14, 454-9.
Nakamura, C.E. 2002. Production of 1,3-Propanediol by E. coli. presented at Metab Eng IV Conf: Tuscany, Italy.
Nandi, R. & Sengupta, S. (1996) Enzyme and microbial tehcnology 19, 20-25.
Nandi, R. & Sengupta, S. (1998) Crit Rev Microbiol 24, 61-84.
Neidhardt, F.C., Curtiss, R. 1996. Escherichia coli and Salmonella : cellular and molecular biology. Washington, D.C., ASM Press.
Overbeek, R., Larsen, N., Pusch, G. D., D'Souza, M., Selkov, E., Jr., Kyrpides, N., Fonstein, M., Maltsev, N. & Selkov, E. (2000) Nucleic Acids Res 28, 123-5.
Papin, J.A., Price, N.D., Wiback, S.J., Fell, D.A., Palsson, B. 2003. Metabolic Pathways in the Post-Genome Era. Trends Biochem Sci, accepted.
Papoutsakis, E. & Meyer, C. (1985) Biotechnol Bioeng 27, 50-66.
Papoutsakis, E. (1984) Biotechnol Bioeng 26, 174-187.
Pharkya, P., Burgard, A. P. & Maranas, C. D. (2003) Biotechnol Bioeng 84, 887-99.
Price, N.D., Papin, J. A., Schilling, C.H., Palsson, B. 2003. Genome-scale Microbial In Silico Models: The Constraints-Based Approach. Trends Biotechnol, accepted.
Ramakrishna, R., Edwards, J. S., McCulloch, A., Palsson, B. O. (2001) Am J Physiol Regul Integr Comp Physiol 280(3): R695-704.
Ramakrishna, R., Ramakrishna, D., Konopka, A. E. (1996). Biotechnol Bioeng 52: 141-151.
Reed, J. L., Vo, T. D., Schilling, C. H. & Palsson, B. O. (2003) Genome Biol 4, R54.
Schilling, C. H., Covert, M. W., Famili, I., Church, G. M., Edwards, J. S. & Palsson, B. O. (2002) J Bacteriol 184, 4582-93.
Schilling, C. H., Covert, M. W., Famili, I., Church, G. M., Edwards, J. S., Palsson, B. O. (2002) J Bacteriol 184(16): 4582-93.
Schilling, C. H., Palsson, B. O. (2000) J Theor Biol 203(3): 249-83.
Segre, D., Vitkup, D., Church, G. M. (2002) Proc Natl Acad Sci U S A 99(23): 15112-7.
Segre, D., Zucker, J., Katz, J., Lin, X., D'Haeseleer, P., Rindone, W. P., Kharchenko, P., Nguyen, D. H., Wright, M. A. & Church, G. M. (2003) Omics 7, 301-16.
Selkov, E., Jr., Grechkin, Y., Mikhailova, N. & Selkov, E. (1998) Nucleic Acids Res 26, 43-5.
Seressiotis, A. & Bailey, J. E. (1988) Biotechnol Bioeng 31, 587-602.
Stephanopoulos, G., Aristidou, A. A., Nielsen, J. 1998. Metabolic engineering : principles and methodologies. San Diego, Academic Press.
Stephanopoulos, G. & Sinskey, A. J. (1993) Trends Biotechnol 11, 392-6.
Stols, L., Donnelly, M. I. (1997) Appl Environ Microbiol 63(7): 2695-701.
Tomita, M., Hashimoto, K., Takahashi, K., Shimizu, T. S., Matsuzaki, Y., Miyoshi, F., Saito, K., Tanida, S., Yugi, K., Venter, J. C., et al. (1999) Bioinformatics 15, 72-84.
Valdes, J., Veloso, F., Jedlicki, E. & Holmes, D. (2003) BMC Genomics 4, 51.
Van Dien, S. J. & Lidstrom, M. E. (2002) Biotechnol Bioeng 78, 296-312.
Van Dien, S. J., Strovas, T. & Lidstrom, M. E. (2003) Biotechnol Bioeng 84, 45-55.
Varma, A., Boesch, B. W., Palsson, B. O. (1993) Appl Environ Microbiol 59(8): 2465-73.
Varma, A, Palsson, B. O. (1993) J. Theor. Biol. 165: 503-522.
Varma, A. & Palsson, B. O. (1994) Bio/Technology 12, 994-998.
Varner, J., Ramkrishna, D. (1999) Biotechnol Prog 15(3): 407-25.
Varner, J. & Ramkrishna, D. (1999) Curr Opin Biotechnol 10, 146-150.
Zeikus, J. G., Jain, M. K., Elankovan, P. (1999) Appl Microbiol Biotechnol 51: 545-552.
Zeng, A. P., Biebl, H. (2002) Adv Biochem Eng Biotechnol 74: 239-59.
Zhu, M. M., Lawman, P. D., Cameron, D. C. (2002) Biotechnol Prog 18(4): 694-9.
【特許請求の範囲】
【請求項1】
宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別するためのコンピュータ援用法であって、
ユニバーサルデータベースの反応にアクセスして、代謝反応に化学量論的バランスを取らせるステップと、
前記ユニバーサルデータベースの反応及び基質に少なくとも部分的に基づいて、少なくとも1つの化学量論的にバランスの取れた経路を識別するステップであって、ここで識別された少なくとも1つの化学量論的にバランスの取れた経路は前記産生宿主内における非ネイティブ機能の数を最小化するものであり、
前記少なくとも1つの化学量論的にバランスの取れた経路の機能を識別することにより、宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別するステップと
を含むコンピュータ援用法。
【請求項2】
前記少なくとも1つの化学量論的にバランスの取れた経路を前記宿主に組み込んで、前記所望の生体内変化を提供するステップを更に含むことを特徴とする請求項1記載のコンピュータ援用法。
【請求項3】
前記少なくとも1つの化学量論的にバランスの取れた経路を識別する前記ステップが、最適化問題を解くステップを含む、請求項1記載のコンピュータ援用法。
【請求項4】
前記最適化問題が、少なくとも1つの細胞目的を生体工学目的に結びつけることによって形成される、請求項3記載のコンピュータ援用法。
【請求項5】
産生宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別するためのコンピュータ援用法であって、
ユニバーサルデータベースの反応にアクセスするステップであって、ここで当該反応は化学量論的にバランスが取られており、
基質に関連した生成物の最大理論収率を計算するステップと、
前記ユニバーサルデータベースの反応、前記基質及び前記生成物の前記最大理論収率に基づいて、少なくとも1つの化学量論的にバランスの取れた経路を識別するステップであって、ここで識別された少なくとも1つの化学量論的にバランスの取れた経路は前記産生宿主内における非ネイティブ機能の数を最小化するものであり、
前記少なくとも1つの化学量論的にバランスの取れた経路の機能を識別することにより、産生宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別するステップと
を含むコンピュータ援用法。
【請求項6】
前記少なくとも1つの化学量論的にバランスの取れた経路を前記産生宿主に組み込んで、前記所望の生体内変化を提供するステップを更に含む、請求項5記載のコンピュータ援用法。
【請求項7】
前記少なくとも1つの化学量論的にバランスの取れた経路を識別する前記ステップが、最適化問題を解くステップを含む、請求項5記載のコンピュータ援用法。
【請求項8】
前記最適化問題が線形計画問題である、請求項7記載のコンピュータ援用法。
【請求項9】
前記最適化問題が混合整数最適化問題である、請求項7記載のコンピュータ援用法。
【請求項10】
前記最適化問題が2層最適化問題である、請求項7記載のコンピュータ援用法。
【請求項11】
前記最適化問題が、少なくとも1つの細胞目的を生体工学目的に結びつける、請求項7記載のコンピュータ援用法。
【請求項12】
前記所望の生体内変化を含むように変更された前記生物特異的な代謝ネットワークを記憶するステップをさらに含む、請求項1記載のコンピュータ援用法。
【請求項13】
産生宿主の所望の生体内変化を可能にするために機能が追加された生物特異的な代謝ネットワークに基づく変更された代謝ネットワークの記憶された表現であって、当該記憶された表現は記憶装置に記憶されており、当該記憶された表現は複数の代謝経路を含み、前記複数の代謝経路が、(a)ユニバーサルデータベースの反応にアクセスして、代謝反応に化学量論的バランスを提供し、(b)基質に関連した生成物の最大理論収率を計算し、(c)前記ユニバーサルデータベースの反応、前記基質及び前記生成物の前記最大理論収率に基づいて、少なくとも1つの化学量論的にバランスの取れた経路を識別し、ここで識別された少なくとも1つの化学量論的にバランスの取れた経路は前記産生宿主内における非ネイティブ機能の数を最小化するものであり、(d)前記少なくとも1つの化学量論的にバランスの取れた経路の機能を識別することにより、産生宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別することによって形成された、少なくとも1つの化学量論的にバランスの取れた経路を含む、記憶された表現。
【請求項14】
産生宿主の所望の生体内変化を可能にするために機能が追加された生物特異的な代謝ネットワークに基づく変更された代謝ネットワークの記憶された表現であって、当該記憶された表現は生体工学によって操作された生物に記憶されており、当該記憶された表現は複数の代謝経路を含み、前記複数の代謝経路が、(a)ユニバーサルデータベースの反応にアクセスして、代謝反応に化学量論的バランスを提供し、(b)基質に関連した生成物の最大理論収率を計算し、(c)前記ユニバーサルデータベースの反応、前記基質及び前記生成物の前記最大理論収率に基づいて、少なくとも1つの化学量論的にバランスの取れた経路を識別し、ここで識別された少なくとも1つの化学量論的にバランスの取れた経路は前記産生宿主内における非ネイティブ機能の数を最小化するものであり、(d)前記少なくとも1つの化学量論的にバランスの取れた経路の機能を識別することにより、産生宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別し、(e) 前記少なくとも1つの化学量論的にバランスの取れた経路を前記産生宿主に組み込んで、前記所望の生体内変化を提供することによって形成された、少なくとも1つの化学量論的にバランスの取れた経路を含む、記憶された表現。
【請求項1】
宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別するためのコンピュータ援用法であって、
ユニバーサルデータベースの反応にアクセスして、代謝反応に化学量論的バランスを取らせるステップと、
前記ユニバーサルデータベースの反応及び基質に少なくとも部分的に基づいて、少なくとも1つの化学量論的にバランスの取れた経路を識別するステップであって、ここで識別された少なくとも1つの化学量論的にバランスの取れた経路は前記産生宿主内における非ネイティブ機能の数を最小化するものであり、
前記少なくとも1つの化学量論的にバランスの取れた経路の機能を識別することにより、宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別するステップと
を含むコンピュータ援用法。
【請求項2】
前記少なくとも1つの化学量論的にバランスの取れた経路を前記宿主に組み込んで、前記所望の生体内変化を提供するステップを更に含むことを特徴とする請求項1記載のコンピュータ援用法。
【請求項3】
前記少なくとも1つの化学量論的にバランスの取れた経路を識別する前記ステップが、最適化問題を解くステップを含む、請求項1記載のコンピュータ援用法。
【請求項4】
前記最適化問題が、少なくとも1つの細胞目的を生体工学目的に結びつけることによって形成される、請求項3記載のコンピュータ援用法。
【請求項5】
産生宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別するためのコンピュータ援用法であって、
ユニバーサルデータベースの反応にアクセスするステップであって、ここで当該反応は化学量論的にバランスが取られており、
基質に関連した生成物の最大理論収率を計算するステップと、
前記ユニバーサルデータベースの反応、前記基質及び前記生成物の前記最大理論収率に基づいて、少なくとも1つの化学量論的にバランスの取れた経路を識別するステップであって、ここで識別された少なくとも1つの化学量論的にバランスの取れた経路は前記産生宿主内における非ネイティブ機能の数を最小化するものであり、
前記少なくとも1つの化学量論的にバランスの取れた経路の機能を識別することにより、産生宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別するステップと
を含むコンピュータ援用法。
【請求項6】
前記少なくとも1つの化学量論的にバランスの取れた経路を前記産生宿主に組み込んで、前記所望の生体内変化を提供するステップを更に含む、請求項5記載のコンピュータ援用法。
【請求項7】
前記少なくとも1つの化学量論的にバランスの取れた経路を識別する前記ステップが、最適化問題を解くステップを含む、請求項5記載のコンピュータ援用法。
【請求項8】
前記最適化問題が線形計画問題である、請求項7記載のコンピュータ援用法。
【請求項9】
前記最適化問題が混合整数最適化問題である、請求項7記載のコンピュータ援用法。
【請求項10】
前記最適化問題が2層最適化問題である、請求項7記載のコンピュータ援用法。
【請求項11】
前記最適化問題が、少なくとも1つの細胞目的を生体工学目的に結びつける、請求項7記載のコンピュータ援用法。
【請求項12】
前記所望の生体内変化を含むように変更された前記生物特異的な代謝ネットワークを記憶するステップをさらに含む、請求項1記載のコンピュータ援用法。
【請求項13】
産生宿主の所望の生体内変化を可能にするために機能が追加された生物特異的な代謝ネットワークに基づく変更された代謝ネットワークの記憶された表現であって、当該記憶された表現は記憶装置に記憶されており、当該記憶された表現は複数の代謝経路を含み、前記複数の代謝経路が、(a)ユニバーサルデータベースの反応にアクセスして、代謝反応に化学量論的バランスを提供し、(b)基質に関連した生成物の最大理論収率を計算し、(c)前記ユニバーサルデータベースの反応、前記基質及び前記生成物の前記最大理論収率に基づいて、少なくとも1つの化学量論的にバランスの取れた経路を識別し、ここで識別された少なくとも1つの化学量論的にバランスの取れた経路は前記産生宿主内における非ネイティブ機能の数を最小化するものであり、(d)前記少なくとも1つの化学量論的にバランスの取れた経路の機能を識別することにより、産生宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別することによって形成された、少なくとも1つの化学量論的にバランスの取れた経路を含む、記憶された表現。
【請求項14】
産生宿主の所望の生体内変化を可能にするために機能が追加された生物特異的な代謝ネットワークに基づく変更された代謝ネットワークの記憶された表現であって、当該記憶された表現は生体工学によって操作された生物に記憶されており、当該記憶された表現は複数の代謝経路を含み、前記複数の代謝経路が、(a)ユニバーサルデータベースの反応にアクセスして、代謝反応に化学量論的バランスを提供し、(b)基質に関連した生成物の最大理論収率を計算し、(c)前記ユニバーサルデータベースの反応、前記基質及び前記生成物の前記最大理論収率に基づいて、少なくとも1つの化学量論的にバランスの取れた経路を識別し、ここで識別された少なくとも1つの化学量論的にバランスの取れた経路は前記産生宿主内における非ネイティブ機能の数を最小化するものであり、(d)前記少なくとも1つの化学量論的にバランスの取れた経路の機能を識別することにより、産生宿主内での所望の生体内変化を可能にするため生物特異的な代謝ネットワークに追加する機能を識別し、(e) 前記少なくとも1つの化学量論的にバランスの取れた経路を前記産生宿主に組み込んで、前記所望の生体内変化を提供することによって形成された、少なくとも1つの化学量論的にバランスの取れた経路を含む、記憶された表現。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】




【図2】


【図3】


【図4】


【図5】


【図6】


【公開番号】特開2012−104129(P2012−104129A)
【公開日】平成24年5月31日(2012.5.31)
【国際特許分類】
【出願番号】特願2011−264256(P2011−264256)
【出願日】平成23年12月2日(2011.12.2)
【分割の表示】特願2007−529791(P2007−529791)の分割
【原出願日】平成16年8月26日(2004.8.26)
【出願人】(502224803)ザ ペン ステイト リサーチ ファウンデーション (6)
【Fターム(参考)】
【公開日】平成24年5月31日(2012.5.31)
【国際特許分類】
【出願日】平成23年12月2日(2011.12.2)
【分割の表示】特願2007−529791(P2007−529791)の分割
【原出願日】平成16年8月26日(2004.8.26)
【出願人】(502224803)ザ ペン ステイト リサーチ ファウンデーション (6)
【Fターム(参考)】
[ Back to top ]

