未知の生体分子と一本鎖核酸の結合プロファイルを生成するための核酸チップ、核酸チップの製造方法、及び核酸チップを利用した未知の生体分子分析方法
本発明は、未知の生体分子と一本鎖核酸の結合プロファイルを生成するための核酸チップ、核酸チップの製造方法、及び核酸チップを利用した未知の生体分子分析方法に関する。本発明の核酸チップは生体試料に含まれた未知生体分子の生物学的意味を分析することに使われる。本発明の核酸チップは、未知の生体分子が含まれている生体試料と無作為塩基配列を持つ無作為一本鎖核酸を反応させて前記未知の生体分子と結合する生体分子結合一本鎖核酸を決定する段階、及び決定された前記生体分子結合一本鎖核酸及び/または前記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸を合成して前記捕捉一本鎖核酸を基板に固着する段階を含む方法から製造する。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、未知の生体分子と一本鎖核酸の結合プロファイルを生成するための核酸チップ、核酸チップの製造方法、及び核酸チップを利用した未知の生体分子分析方法に関する。
【背景技術】
【0002】
生体試料を構成する未知の生体分子を含んだ数多くの生体分子(蛋白質及び炭素化物)のプロファイルを生産する技術は、物理学、生化学、及び生物情報学の発達で広く開発されているが、既存方法と装置は、その使用及び維持費、容易性、正確性、感度、検査時間、及び工程の自動化等に多様な問題があるため、効率的な新しい方法及び装置に対する必要性は非常に高い。
【0003】
生体試料でこれら生体分子の量的な状態を総合化した情報、すなわち生体分子のプロファイル(profile)を生産する方法は、それ自体が窮極的な目標ではなく、目標をなすための一つの手段であるが、微生物、細胞、組織等に有用に使用することができて、医学、獣医学、環境工学、食品工学、農業等広範囲に適用されている。
【0004】
核酸は、ヌクレオチドが共有結合で連結した線形的な重合体であり、ヌクレオチドは小さな有機化合物として、燐酸、糖、及びプリン(アデニン又はグアニン)(purine(adenine、guanine))あるいはピリミジン(シトシン、チミジン、又はウラシル)(pyrimidine(cytosine、thymidine、uracil))から構成される。核酸は一本鎖あるいは二本鎖として存在する。一本鎖核酸は特定の物理的な条件でヌクレオチドの間の水素結合及び相互作用により結合して独特の立体構造を形成するが、こういう立体構造は一本鎖の塩基配列により決定される。
【0005】
一般的に、DNAやRNAのような核酸は、細胞構造や酵素などの活性を有する蛋白質を発現するための情報の貯蔵体である。1982年に、RNAが特定の構造を形成することによって酵素的活性も持っているという報告がなされて以後、核酸の構造的な特性とそれに伴う特定機能に対する多くの報告がなされている。
【0006】
核酸は、4個の塩基の反復で構成されていて、多様性を維持しながら多くの立体構造を形成し、こういう立体構造は特定物質と相互作用をして複合体を形成して安定化される。
【0007】
核酸は、蛋白質を含む分子に対して一つのリガンド(ligand)として作用し、多様な塩基配列の一本鎖核酸から構成されたライブラリーから一定の選別過程と塩基配列決定過程を通して,高い結合力と特異性で特定物質と結合する核酸が選別されている。
【0008】
特定物質と結合する核酸を選別する方法をSELEX (Systematic Evolution of Ligands by EXponential enrichment)といい、選別された核酸をいわゆるアプタマー(aptamer)という(Tuerk C、and Gold L.; Science、249、pp505−510、1990)。
【0009】
SELEXを通して自然状態で核酸と結合する蛋白質や、核酸と結合していない蛋白質のように、多様な生体分子と非常に高い親和力を有して結合できる核酸(アプタマー)が選別されている。
【0010】
既存のSELEXによる核酸選別方法は、特定物質(例えば、蛋白質)に特異的に結合する核酸を選別するに先立ち、優先的に該当蛋白質(特定物質)を大量生産して精製後、生産された蛋白質に一本鎖核酸ライブラリーを反応させ選択と増幅を反復して結合力が強力な核酸(アプタマー)を選定する方法として、核酸を選別するためにはあらかじめ特定物質を確保することが必須であった。このような従来のSELEXを通した核酸選別方法は、生体試料に含まれている数多くの未知の生体分子に対する生物学的意味を持つ集団としての核酸を選別し利用する技術は全く認識していなかった。
【0011】
生体試料である組織、細胞、及び微生物などを構成する未知分子を含んだ生体分子のプロファイルは、物理、化学的な性質を利用して様々な方法から作られており、一般的に、生体分子の分子量あるいはpI値などを利用して電気泳動をすることで生体試料から生体分子の量的な状態である生体分子のプロファイルを確認している。
【0012】
また、プロファイルを分析して有用な生体分子を決定しこれを分離した後、MALDI−TOF(Matrix Assisted Laser Desorption/Ionization−Time Of Flight)などでこれらの構成成分を確認する方法などが行われている。最近では、SELDI−TOF−MS (Surface−enhanced laser desorption/ionization time of flight mass spectrometry)を用いて蛋白質プロファイルに対する多くの研究が進行されている(Adam et al.、Cancer Research、62、3609−3614. 2002; Li et al.、Clinical Chemistry、48、1296−1304. 2002; and Petricoin et al.、The Lancet、359、572−577. 2002)。
【0013】
また、最近では、蛋白質に対する並列高速分析(High Through put Screening)の技法で、蛋白質チップ(Protein chip)あるいはアプタマーチップ(Aptamer chip)(Smith et al.、MolCell Protomics、11−18.2003; and McCauley et al.、Anal Biochem、319(2)、244−250.2003)が開発され、使用されている。
【0014】
蛋白質チップは、抗体の配列状態を知っているため、特定物質の区分及び定量に対する探索及び分析が可能である。蛋白質チップの製作方法では、マイクロアレイヤー(Micro arrayer)を利用して、抗体をスポッティング(spotting)して固定する方法がある。蛋白質チップは、一つのチップでなるべく多くの情報を提供できるように、狭い面積に高密度で多様な抗体が集積されている状態で発生する、非常に弱い信号を感知しなければならない。また、蛋白質に対する生体情報が増えるに従い、その集積度がより一層高まる趨勢で発達しているので、これを定量的に速くて正確に検出する新しい方法が要求されている。
【0015】
現在まで蛋白質チップの分析方法は、主にレーザー誘発蛍光法(laser induced fluorescence)が広く使われていて、電気化学的検出方法などが開発されている。上記の通り、多様な工程により、蛋白質チップを利用した生体試料から特定蛋白質等のプロファイルを生産及び分析する方法が開発されているが高価の装置及び試薬を使用すべく複雑な過程を遂行しなければならないなどの問題があるし、また抗体を製作することのできる分子のみ製作が可能な限界がある。
【0016】
アプタマーチップは、蛋白質チップに抗体を使用することの代わりにアプタマー(核酸)を使用する点が異なり、その他の要素は非常に類似している。
このように、蛋白質チップ及びアプタマーチップを利用した生体試料から生体分子の量的な状態、すなわちプロファイルを生産する方法が開発されているが、高価の装置及び試薬を使用しており、複雑な工程を経なければならないなどの問題がある。特にいままでの蛋白質チップやアプタマーチップは抗体あるいはアプタマーを製作することのできる既に知られている蛋白質のみ制限的に製作が可能な限界がある。
【0017】
生体試料は数百万個の蛋白質から構成されているが、現在知らされている蛋白質の数は数万個に過ぎない実情であるので、生体試料で未知の蛋白質のような未知の生体分子の量的状態、すなわちプロファイルを生産する技術の必要性は非常に高い。
【0018】
生体試料の生体分子を総体的に分析する研究は、医学的に疾病に関連した生体分子のプロファイルを分析することによって、疾病を診断することのできる生体分子、治療成績を分析しえる生体分子、疾病発病及び進行に重要な役割をする生体分子、疾病に対する感受性に関連する生体分子、及び新薬開発の標的分子を究明することに使用することができる。
【発明の概要】
【発明が解決しようとする課題】
【0019】
本発明の目的は、生体試料に含まれている未知の生体分子と一本鎖核酸の結合プロファイルを生成可能な核酸チップを提供することにより、核酸チップを利用して生成した結合プロファイルから未知の生体分子と関連した疾病情報を含んだ各種生物学的意味を分析可能にすることにある。
【課題を解決するための手段】
【0020】
本発明により、一本鎖核酸と未知生体分子の結合プロファイルの生成に使われる核酸チップとその製造方法、及び核酸チップを利用した未知の生体分子分析方法が提供され、本発明にかかる核酸チップは、生体試料に含まれた未知生体分子の生物学的意味を分析するために使われる。
【0021】
本発明による核酸チップの製造方法は、未知の生体分子が含まれている生体試料と無作為塩基配列を持つ無作為一本鎖核酸を反応させて、上記未知の生体分子に結合する生体分子結合一本鎖核酸を決定する第1段階、及び決定された上記生体分子結合一本鎖核酸及び/または上記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸を合成して基板に固着する第2段階を含む。
【0022】
本発明による核酸チップは、生体試料に含まれた未知の生体分子と結合する生体分子結合一本鎖核酸及び/または上記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸が基板に固着されている構成になっている。本発明による核酸チップを利用した未知の生体分子分析方法は、上記本発明による核酸チップを準備する段階;上記核酸チップの上記捕捉一本鎖核酸と同じ塩基配列を持つ一本鎖核酸と上記生体試料の上記未知生体分子を反応させ、生体分子−ターゲット一本鎖核酸複合体を形成し、上記生体分子−ターゲット一本鎖核酸複合体を分離する段階;分離された上記生体分子−ターゲット一本鎖核酸複合体から上記ターゲット一本鎖核酸を分離、増幅及び標識する段階;標識した上記ターゲット一本鎖核酸を上記核酸チップの上記捕捉一本鎖核酸と反応させ上記標識を通し結合プロファイルを獲得する段階;及び獲得された上記プロファイルと既に確保されているプロファイルデータを比較して上記未知の生体分子の生物学的意味を分析する段階;を含む。
【0023】
本発明による核酸チップ及びこれを利用した未知の生体分子分析方法の基本的な原理は、未知の生体分子に結合する一本鎖核酸に相補的な一本鎖核酸(捕捉一本鎖核酸)を核酸チップに固着し、テストの対象になる未知の生体分子とこれに結合する一本鎖核酸を反応させて生体分子−ターゲット一本鎖核酸複合体を形成した後、上記複合体から分離されたターゲット一本鎖核酸と核酸チップの捕捉一本鎖核酸の相補的な結合のプロファイルを獲得してその特性を分析することによって、結果的に未知の生体分子の一本鎖核酸に対する結合プロファイル特性を分析することであるので、核酸チップの捕捉一本鎖核酸として未知の生体分子に結合する一本鎖核酸に相補的に結合する一本鎖核酸を基板に固着すればよいが、未知の生体分子を分析する時、生体分子−ターゲット一本鎖核酸複合体から分離したターゲット一本鎖核酸を増幅する過程で未知の生体分子に結合するターゲット一本鎖核酸に相補的な一本鎖核酸もターゲット一本鎖核酸のプロファイル情報と同一類似の情報を内包した状態で生成されるので、本発明による核酸チップの捕捉一本鎖核酸として未知の生体分子と結合する生体分子結合一本鎖核酸を使用しても同じ目的を達成できて、ひいては生体分子結合一本鎖核酸と、生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を同時に使用しても同じ目的を達成することができる。
【0024】
本発明による核酸チップを利用して未知の生体分子を分析すれば、関連プロファイルデータが蓄積され、これから未知の生体分子の生物学的意味の分析に寄与する特異的な一本鎖核酸が自然に明らかになり、これで未知の生体分子を分析することに意味を持つ一本鎖核酸を発掘することができるようになる。
【0025】
本発明による核酸チップが適用される上記生体試料としては、細菌、真菌類、ウイルス、細胞株、組織などが含まれるし、本発明による核酸チップによりその生物学的意味が分析されることができる生体分子としては蛋白質、炭水化物、脂質、多糖体、糖蛋白、ホルモン、受容体、抗原、抗体及び酵素からなる群から選択された少なくとも一つ以上の生体分子が含まれる。
【0026】
本発明による核酸チップの製造方法の上記1段階は、未知の生体分子が含まれている生体試料と、無作為塩基配列を持つ一本鎖核酸(以下、無作為一本鎖核酸という)を反応させて未知の生体分子と結合する生体分子結合一本鎖核酸を決定する段階である。
【0027】
望ましくは、上記生体分子結合一本鎖核酸を決定する段階は、上記生体試料の上記未知の生体分子に上記無作為一本鎖核酸を反応させて生体分子−一本鎖核酸複合体を製造する段階;上記生体分子−一本鎖核酸複合体を洗浄し、上記生体分子と上記一本鎖核酸の結合力に基づいて一定結合力以上の上記生体分子−一本鎖核酸複合体を選択する段階;選択された上記複合体から上記一本鎖核酸を分離して増幅する段階;及び上記増幅された上記一本鎖核酸をクローニングベクターに挿入して単一クローンを確保して上記一本鎖核酸の塩基配列を決定することによって上記生体分子結合一本鎖核酸を決定する段階;を含む。
【0028】
上記生体分子−一本鎖核酸複合体の選択及び増幅は反復的に多数回実行することができるが、未知の生体分子を含む生体試料は数多くの生体分子から構成されていてこれらの量的な差が非常に多様なので、未知の生体分子に結合する一本鎖核酸の選択と増幅を反復的に行う環形的な方法より選択と増幅を一回行って洗浄過程を強化する線形的な方法で製作するのがより望ましい。
【0029】
上記無作為塩基配列を持つ無作為一本鎖核酸は、下のような無作為の塩基配列を持つ一本鎖DNAオリゴヌクレオチドを利用して二本鎖のDNAを製造した後、生体外転写(in vitro transcription)を通してRNA無作為一本鎖核酸に製造できる。
【0030】
5’−GGGAGAGCGGAAGCGTGCTGGGCC
N40 CATAACCCAGAGGTCGATGGATCCCCCC−3’
(ここでアンダーラインをした塩基配列は核酸ライブラリーの固定された部位で、N40は各位置にA、G、T、C等の塩基が同じ濃度で存在するのを意味する。)
【0031】
PCR(Polymerase Chain Reaction)に利用されるFWプライマー(5’−GGGGGAATTCTAATACGACTCACTATAGGGAGAGCGGAAGCGTGCTGGG−3’:配列番号1)は、前期の塩基配列のアンダーラインをした塩基の5’末端と塩基結合をすることができ、バクテリオファージT7のRNAポリマラーゼのためのプロモーター塩基配列を含んでいる。
【0032】
PCRのREプライマー(5’−GGGGGGATCCATCGACCTCTGGGTTATG−3’:配列番号2)は上記の塩基配列のアンダーラインをした塩基の3’末端と塩基結合をすることができる。
【0033】
望ましくは、本発明の核酸チップ製造方法で生体分子結合一本鎖核酸は2’−F−置換ピリミジンを含むRNA一本鎖核酸であり、生体外転写で合成し精製して準備する。
【0034】
合成された無作為一本鎖核酸は例えば1015塩基配列/1mlの濃度で生体試料に処理して生体分子と30分間反応させることができる。
【0035】
上記生体分子結合一本鎖核酸はRT−PCR(Reverse Transcription−PCR)して得た産物をクローニングベクターに挿入して大腸菌クローンを確保し、その塩基配列を決定できる。
【0036】
この時反応温度はSELEX遂行温度より低い温度で行うことが理想的であり、望ましくは20℃〜37℃の温度で反応を行う。
【0037】
普遍的に蛋白質及び一本鎖核酸を過量処理して生体分子と結合する一本鎖核酸の非特異的な結合を防止し、望ましくは酵母tRNA(yeast tRNA)、サーモンスペルマ(salmon sperm DNA)または人間胎盤DNA(human placental DNA)を使用することができる。
【0038】
本発明による核酸チップの製造方法の上記第2段階は、第1段階で決定された生体分子結合一本鎖核酸及び/または上記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸を合成して、合成された捕捉一本鎖核酸を基板上に固着させて本発明の核酸チップを製作する段階である。
【0039】
捕捉一本鎖核酸は混成化程度に非常に大きい影響を及ぼす核心要素であるので、その塩基配列構成の決定は非常に重要である。本発明の核酸チップに固着されるそれぞれの捕捉一本鎖核酸は特徴的な塩基で構成されて、捕捉一本鎖核酸とターゲット一本鎖核酸の結合体は適切なTm値を維持しなければならない。結合体の混成化程度は蛍光が標識されたターゲット一本鎖核酸により汚染されていない自分のシグナル値を維持できるようにする。
【0040】
したがって捕捉一本鎖核酸の塩基配列は、上記第1段階で無作為一本鎖核酸から選別された生体分子結合一本鎖核酸の塩基配列を基づいて決定される。望ましくは、捕捉一本鎖核酸は16bp〜60bpの塩基配列を持つオリゴヌクレオチドの一本鎖核酸である。
【0041】
また、本発明の核酸チップの製作方法における基板は、ガラス、シリコンなどの無機物やアクリル系やPET(polyethylene terephtalate)、ポリカーボネート(polycarbonate)、ポリスチレン(polystyrene)、ポリプロフィレン(polypropylene)などの高分子物質から製作したものを使用することができて、望ましくはガラスで製作したスライドグラスである。この時、基板はアミン基またはアルデヒド基等でコーティングしたものを使用することができる。例えばGAPS(Gamma Amino Propyl Silane)がコーティングされたガラススライドであるUltra GAPSTM Coated Slide(Corning社)を使用して上記捕捉一本鎖核酸を一定規則で配列固着して核酸チップを製作する。
【0042】
本発明による核酸チップの製造は、マイクロアレイヤーシステム(Micro arrayer system)を使用することができ、それぞれの捕捉一本鎖核酸を緩衝額に溶かして濃度を調節し、この時アレイャー中の湿度は70%〜80%に維持してスポッティング(spotting)を行う。スポッティングされたスライドは湿度維持チャンバー(humidified chamber)で放置した後、UV crosslinkerで焼く(baking)過程を行う。
【0043】
このように本発明による核酸チップは、関連技術分野に広く知られている方法で捕捉一本鎖核酸をガラススライドに固定させた後、スライドをすぐ遠心分離して乾燥させた後、使用する前まで光を遮断した状態で保管する。
【0044】
既に公知されている多様な方法(M.schena; DNAmicro array; apractical approach、Oxford、1999)で捕捉一本鎖核酸が規則的に固着された核酸チップを製作するこてができる。
【0045】
本発明による核酸チップは、同一類似の精密度で未知生体分子の生物学的意味を分析することさえあれば、核酸チップに固着する捕捉一本鎖核酸の数を少なくすることが核酸チップの製作費及び分析の効率性側面で望ましい。
【0046】
かかる理由で、本発明の核酸チップ製造方法は,未知生体分子の生物学的意味分析に対するそれぞれの捕捉一本鎖核酸の寄与度を分析し、未知生体分子の生物学的意味を分析することができる範囲内で、分析された寄与度に基づいて捕捉一本鎖核酸を選別することで、本発明の核酸チップに固着する捕捉一本鎖核酸の数を減少させる段階を更に含むことができる。
【発明の効果】
【0047】
本発明による核酸チップ及びこれを利用した未知の生体分子分析方法によれば、並列高速分析(High Through put Screening)を利用した極めて簡単で低廉で効率的な方法で、微生物、細胞、蛋白質を含む生体試料に含まれた未知の生体分子に対するプロファイルを生産し得るので、医学、獣医学、環境工学、食品工学、農業などに広範囲な分野で未知の生体分子の生物学的意味を分析する道具として応用できるものと期待する。
【0048】
また、本発明による核酸チップ及びこれを利用した生体分子分析方法によれば、未知の生体分子の生物学的意味を分析する過程から、未知の生体分子の生物学的作用を把握し、その構造を決定、究明しえるばかりでなく、生体分子に特異的に結合する特定一本鎖核酸を選別でき、選別された一本鎖核酸を利用して更に精巧な生体分子の作用を把握する道具として利用できる環境を提供する。
【0049】
また、本発明による核酸チップを利用した生体分子分析方法によれば、医学的に疾病に関連した生体分子のプロファイルを分析することによって、疾病を診断する生体分子、治療成績を分析する生体分子、疾病の発病及び進行に重要な役割をする生体分子、疾病の感受性に関連した生体分子、及び新薬開発の標的分子などを非常に低廉で簡便な方法で、しかも效率的に糾明することができる。
【図面の簡単な説明】
【0050】
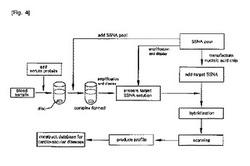
【図1】図1は、本発明の核酸チップの製造方法に係る生体分子結合一本鎖核酸を決定する過程を示す概略図である。
【図2】図2は、本発明に係る核酸チップを利用して未知の生体分子と一本鎖核酸の結合プロファイルを生成する過程を示す概略図である。
【図3】図3は、本発明に係る核酸チップを適用して生成した健康な人(A)と安定狭心症心血管患者(B)血清のプロファイルである。
【図4】図4は、本発明に係る核酸チップを利用して患者の血清試料から生産されたプロファイルを疾患群別でデータベースを構築する過程の順序を示す概略図である。
【図5】図5は、本発明にかかる核酸チップで生成された人間血清蛋白質プロファイルのデータベース及び人工神経網アルゴリズムを利用したプログラムで疾病を診断する過程を示す図面である。
【図6】図6は、本発明に係る核酸チップを利用して生成した人間血清蛋白質プロファイルのデータベース及び人工神経網アルゴリズムを利用したプログラムで心血管疾患を診断した結果を示す図面である。
【図7】図7は本発明に係る核酸チップを利用して生成した人間血清蛋白質プロファイルのデータベース及び人工神経網アルゴリズムを利用したプログラムで肝ガンを診断した結果を示す図面である。
【図8】図8は、本発明に係る核酸チップを利用して生成された人間血清蛋白質プロファイルのデータベース及び人工神経網アルゴリズムを利用したプログラムで、肝ガンの転移を診断した結果を示す図面である。
【図9】図9は、本発明に係る核酸チップを利用して生成した人間血清蛋白質プロファイルのデータベースで心筋梗塞患者の血清に特異的に存在する蛋白質のアミノ酸配列を決定する過程の順序を示す図面である。
【図10】図10は、本発明に係る核酸チップを利用して生成した人間血清蛋白質プロファイルのデータベースで心筋梗塞患者の血清に特異的に存在する蛋白質のアミノ酸配列を決定した図面である。
【図11】図11は、本発明に係る核酸チップを利用して生成した肺ガン細胞株NCI−H1299の表面生体分子プロファイルを生産及び分析して確保した一本鎖核酸が肺ガン細胞株に結合した結果を示す図面である。
【図12】図12は、本発明に係る核酸チップを利用した大腸菌KCTC12006及びサルモネラ菌(Salmonella typhimurium ATCC13311)の表面生体分子プロファイルを生産及び分析して確保した生体分子結合一本鎖核酸の特異性を示す図面である。
【図13】図13は、本発明に係る核酸チップを利用して選別された、大腸菌に特異的に結合する生体分子結合一本鎖核酸とナノ金粒子を利用して大腸菌に汚染した飲食物から汚染程度を測定した結果を示す図面である。
【発明を実施するための形態】
【0051】
以下、貼付図面と実施例を参照して本発明にかかる核酸チップ、核酸チップの製造方法、及び核酸チップを利用した未知の生体分子分析方法を詳細に説明する。以下の具体例は本発明を例示的に説明するものであり本発明の範囲を制限するものに意図しない。
【0052】
[実施例1. 人間血清蛋白質に結合する生体分子結合一本鎖核酸の製造]
図1に示したように、下のような無作為の塩基配列を持つ一本鎖のDNAオリゴヌクレオチドを利用してPCR(Polymerase Chain Reaction)を通し二本鎖のDNAを製造した後、生体外転写を通して一本鎖のRNAライブラリー(無作為一本鎖核酸)を製造した。
【0053】
5’−GGGAGAGCGGAAGCGTGCTGGGCC
N40 CATAACCCAGAGGTCGATGGATCCCCCC−3’
(ここでアンダーラインをした塩基配列は核酸ライブラリーの固定された部位でありN40は各位置にA、G、T、C等の塩基が同じ濃度で存在することを意味する。)
【0054】
PCRに利用される上記配列番号1のFWプライマーは、上の塩基配列のアンダーラインをした塩基の5’末端と塩基結合をすることができ、バクテリオファージT7のRNAポリマラーゼのためのプロモーター塩基配列を含んでいる。PCRに利用される配列番号2のREプライマーは上の塩基配列のアンダーラインをした塩基の3’末端と塩基結合をすることができる。そしてFWとREプライマーは以後のクローニングのために各々EcoRIとBamHI等の制限酵素切断塩基配列を含有している。
【0055】
生体試料と反応する無作為一本鎖核酸は2’−F−置換ピリミジンを含むRNAライブラリーであり、DNA核酸ライブラリー転写体及びPCRプライマーを上記のように設計製作してPCR及び生体外転写方法で準備した。
【0056】
PCRは1,000pmoles一本鎖核酸転写体、2,500pmoles PCRのプライマー対(5P7)を50mM KCl、10mM Tris−Cl(pH8.3)、3mM MgCl2、0.5mM dNTP(dATP、dCTP、dGTP、及びdTTP)、0.1U Taq DNA Polymerase(Perkin−Elmer、Foster City Calif.)でPCRを行った後QIA quick−spin PCR精製コラム(QIAGENInc.、Chatsworth Calif.)で精製した。
【0057】
2’−F−置換ピリミジンを含む無作為一本鎖核酸は生体外転写で合成し精製して準備した。200pmoles二本鎖DNA転写体、40 mM Tris−Cl(pH8.0)、12 mM MgCl2、5 mM DTT、1 mM spermidine、0.002% Triton×−100、4% PEG 8000、5 U T7 RNA Polymerase、そして1 mM ATP、GTP及び3 mM 2’F−CTP、2’F−UTPを37℃で6〜12時間反応させた後、Bio−Spin 6クロマトグラフィーコラム(Bio−Rad Laboratories、Hercules Calif.)で精製した後、UVスペクトロメーターを利用して精製された核酸の量及びその純粋度を検索した。
【0058】
上記で合成した無作為一本鎖核酸の1015塩基配列/1mlの濃度溶液を200pmol/200μlの濃度で選択バッファー(50mM Tris・Cl(pH7.4)、5mM KCl、100mM NaCl、1mM MgCl2、0.1% NaN3)で80℃で10分間加熱した後、氷に10分間放置した。使用した一本鎖核酸の5倍の酵母tRNA(yeast tRNA、Life Technologies社)及び0.2% BSA(bovine serum albumin、Merck社)を添加して反応溶液を準備した。
【0059】
10μlの血清試料を90μlのPBS溶液を添加してナイトロセルロース膜ディスク(nitro cellulose membrane disc)を入れて30分間振りあげながら反応させた。上記で準備したRNA一本鎖核酸を血清試料が付着されたディスクに処理して30分間反応させた。
【0060】
人間血清試料(生体分子)と結合する生体分子結合一本鎖核酸の選定は、一次的に人間血清試料に結合力を見せる一本鎖核酸を対象とし、反応後多様な洗浄バッファーで洗浄過程を反復して一回のみの選択過程で人間血清蛋白質(生体分子)−一本鎖核酸複合体を確保した。
【0061】
生体分子−一本鎖核酸複合体の洗浄バッファーは0〜1×SELEXバッファーまたは0〜500 mM EDTA溶液を使用した。分離された複合体をRT−PCRを行って血清蛋白質に結合するRNA(生体分子結合一本鎖核酸)を指示するDNAプールを増幅製作した。この時、上記の選択と増幅過程を多数回反復して生体分子結合一本鎖核酸を製作することができる。
【0062】
確保されたRT−PCR産物であるDNAをプラスミドにクローニングして単一クローンを確保する過程で、生体分子結合−一本鎖核酸の製作を完了した。これら生体分子に結合する一本鎖核酸の塩基配列は標準方法でプラスミドを分離して行った。
【0063】
生体分子のプロファイルを生成するための本発明に係る核酸チップに使用する捕捉一本鎖核酸の塩基配列は人間血清蛋白質(生体分子)と結合する上記生体分子結合一本鎖核酸の塩基配列及び/または生体分子結合一本鎖核酸に相補的に結合する一本鎖核酸の塩基配列であるので、このために核酸の二次構造をモデリングするMFOLDプログラムを使用して二次構造及び構造体自由エネルギーを確認した後、最も安定度が高い二次構造を有する生体分子結合一本鎖核酸を選定した。
【0064】
[実施例2. 核酸チップの製造]
ガラススライド表面に固着させる捕捉一本鎖核酸として、実施例1から選別された塩基配列を持つ3,000余個μの生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸(オリゴヌクレオチド)を有機合成して(バーイオニア社)準備した。
【0065】
GAPS(Gamma Amino Propyl Silane)がコーティングされたガラススライドであるUltraGAPSTM Coated Slide(Corning社)を使用して捕捉一本鎖核酸が一定規則で固着された核酸チップを製作した。核酸チップの製作はピン(pin)方式であるマイクロアレイヤーシステム(Microarrayer system、GenPak社)を使用したし、スポット間隔(spot spacing)はスポット中心の間(center−to−center)が370μmになるようにした。それぞれの捕捉一本鎖核酸を標準溶液に溶かし濃度を調節した。この時アレイの中の湿度は70%に維持してスポッティング(spotting)を行った。スポッティングされたスライドは湿度維持チャンバー(humidified chamber)で24〜48時間放置した後、UV crosslinkerで焼く(baking)過程を行った。広く知られている方法で捕捉一本鎖核酸をガラススライドに固定させた後、スライドをすぐ遠心分離して乾燥した後、光が遮断された状態で保管した。
【0066】
[実施例3. 人間血清(生体分子)−ターゲット一本鎖核酸複合体の製造及びターゲット一本鎖核酸の製造]
実施例1で選定され核酸チップ製造に使用された生体分子結合一本鎖核酸が挿入されたプラスミドを同量で混合して未知の生体分子を含む生体分子と結合する一本鎖核酸プールの転写体を準備するためのプラスミドプールを準備した。人間血清蛋白質と結合する一本鎖核酸プールは上記プラスミドプール及びPCRプライマーを設計製作してPCR及び生体外転写方法で準備した。
【0067】
準備したプラスミドプールで1pgプラスミドを転写体にして、PCR反応溶液、100pM 5’−プライマー、100pM 3’−プライマー、dNTP混合物(5mM dATP、5mM dCTP、5 mM dGTP、5 mM dTTP) で標準PCR方法で94℃30秒、52℃30秒、72℃20秒の条件で30回サイクルを反復行って二本鎖核酸を合成してQIAquick−spin PCR精製コラム(QIAGEN Inc.、Chatsworth Calif.)で精製した。
【0068】
2’−F−置換ピリミジンを含むRNA分子のターゲット一本鎖核酸を生体外転写で合成し精製して準備した。
【0069】
200 pmoles二本鎖DNA転写体、40 mM Tris−Cl(pH8.0)、12 mM MgCl2、5 mM DTT、1mM spermidine、0.002% TritonX−100、4% PEG 8000、5U T7 RNA Polymerase、そして1 mM ATP、GTP及び3 mM 2’F−CTP、2’F−UTPを37℃で6〜12時間反応させた後、Bio−Spin6クロマトグラフィーコラム(Bio−Rad Laboratories、Hercules Calif.)で精製した。
【0070】
分析に使用した人間血清は健康な人及び安定狭心症である心血管疾患患者の血清であり10μlの血清試料を90μlのPBS溶液を添加してナイトロセルロース膜(Nitrocellulose membrane)ディスクを入れて30分間振りあげながら反応させ試料を準備した。先に製作した100〜400ngのターゲット一本鎖核酸を投入して30分間反応させ生体分子−ターゲット一本鎖核酸複合体を形成した。
【0071】
準備した一本鎖核酸を血清試料が付着されたディスクに処理して30分間反応させた。選択バッファーあるいは50mM EDTAで3回洗浄し反応産物を分離して収穫した。
【0072】
血清蛋白質(生体分子)−ターゲット一本鎖核酸がついたディスクをRT−PCR溶液に処理してRT−PCRを行いながらCy−5で標識されたプライマー(5’−Cy5−CGGAAGCGTGCTGGGCC−3’:配列番号3)を準備した。上記で準備したプラスミドプールで準備したターゲット一本鎖核酸を利用して上記と同一方法でRT−PCRしてCy−3が標識されたプライマー(5’−Cy3−CGGAAGCGTGCTGGGCC−3’)を製作して準備した。上記二つの溶液を同量で混合してターゲット一本鎖核酸を製作した。
【0073】
[実施例4. 核酸チップとターゲット一本鎖核酸の反応]
図2に示したように、上記実施例3で準備したターゲット一本鎖核酸を本発明の核酸チップの捕捉一本鎖核酸に処理して60℃で4〜12時間混成化し42℃で0.1×SSC溶液で洗浄した。このとき混成化溶液は1M塩化ナトリューム、0.3M sodium citrate、0.5% SDSまたは100ug/ml サーモンスペルマDNA、0.2%牛血清アルブミンまたは一本鎖核酸を含む。
【0074】
前混成化が終わったガラススライドに実施例3で準備した溶液を処理して42℃で12時間混成化(Hybridization)を行った後、核酸チップを洗浄溶液で洗浄した。このとき、洗浄溶液の造成は例えば、1×SSC+0.2% SDS、1×SSC+ 0.2% SDS、0.5×SSC+0.2% SDS、0.01×SSC+0.2% SDS順の段階であり各々段階ごとに42℃で30分間行った。
【0075】
[実施例5. 核酸チップのスポット探索及び分析]
上記実施例4の洗浄が終了した後、ガラススライドを遠心分離で乾燥した後、使用した蛍光染料を励起(excitation)するための適切な波長のレーザー(Cy5、635nm)を持つレーザースキャナー(laser scanner、GenePix4000、Axon社)を利用して探索した。蛍光イメージは多重−イメージ−タグされたイメージファイル(multi−image−tagged image file、TIFF)フォーマットで貯蔵した後、適切な画像分析(image analysis)ソフトウェア(GenePix Pro 3.0、Axon社)で分析した。
【0076】
スポット(spot)当たりのシグナル強度(signal intensity、単位:quanta)は周囲背景の基本シグナルを除いたものを使用するが、ここで背景シグナルは特定スポットの周囲にある4個のスポットの周辺シグナルから構成された位置的バックグラウンド(local background)を意味する。スポットが持っている画素(pixel)の90%以上が背景シグナル+2標準偏差(S.D.; standard deviation)を超過するシグナル強度を見せるときデータ分析に含めて、上の条件を充足させることができなければデータに含めないスポット(bad spot)で分類して、分析に含まないことが一般的である。
【0077】
標識効率(Labeling efficient)に伴う偏差(variation)は内部標準(internal standard、IS)のシグナルを利用して平準化(e.g.、Normalized Intensity = Probe Intensity/ IS intensity)し、単一標識(monolabeling)をした場合にはCy5チャンネルのシグナル強度(signal intensity)でその結果を記録し、スポッティング(spotting)が2倍以上になった場合は平均値を使用する。ターゲット一本鎖核酸のシグナル強度(signal intensity、S)はスポットが持つピクセル(pixel) に対してそれぞれのシグナル強度を求めた後これらの中心値(median)を使用する(median value of pixel−by−pixel)。シグナル強度(S)は内部標準(IS)を利用して標識効率にともなう偏差を平準化する。
【0078】
S’(normalized value)=S(Cy5−reference)×(Cy5−IS)。
【0079】
以上の方法でピクセル密度の分析結果と実際試料量の間の関係を糾明してその相関関係を確認することができる。核酸チップ蛍光データをイメージ化し、あらゆるスポットのパターンで生体試料から生体分子プロファイルを確認することができるし、スポット等のパターンを階層クラスタリング(Hierarchical Clustering)及び人工神経網(Artificial Neural Network)などの方法などで分析して使用することができる。
【0080】
スポットの蛍光程度は捕捉一本鎖核酸とターゲット一本鎖核酸が形成する二本鎖の性質によって多様に示される。人間血清蛋白質−(ターゲット)一本鎖核酸複合体の結合強度及び量は、これらの間の特異性及び結合力に従い決定される。
【0081】
人間血清蛋白質−ターゲット一本鎖核酸複合体で起源するターゲット一本鎖核酸と核酸チップに付着された捕捉一本鎖核酸の塩基配列はターゲット一本鎖核酸−捕捉一本鎖核酸の二本鎖安定性に、そして核酸チップ上のターゲット一本鎖核酸の量は蛍光強度に影響を与える。
【0082】
すなわち、蛍光程度はターゲット一本鎖核酸の量を表し、ターゲット一本鎖核酸の量は人間血清蛋白質−ターゲット一本鎖核酸複合体の量を示す。また上記複合体の量は生体試料にある生体分子の量を表す。したがって、スポットの蛍光度からスポットに相応する未知の生体分子を含む生体試料から特定生体分子の量を確認することができる。結局、形成されたスポットの蛍光程度を分析して核酸チップのあらゆるスポットのパターンを確認することによって人間血清で血清蛋白質のプロファイルを確認することができる。
【0083】
すなわち、形成されるスポットが青色−黄色−赤色のカラースペクトラムに見えるのは捕捉一本鎖核酸に結合したCy−3が標識されたターゲット一本鎖核酸とCy−5が標識されたターゲット一本鎖核酸の比率が多様で現れる現状であり、特定スポットのカラースペクトラムの程度は人間血清を構成する特定の血清試料に存在する特定の生体分子(蛋白質)の量を表現するため、核酸チップ上のあらゆるスポットのカラースペクトラムを見せるイメージは特定生体試料で生体分子プロファイルに該当する。
【0084】
すなわち、本発明の生体分子分析方法では特定スポットで捕捉一本鎖核酸と結合して二本鎖を形成するターゲット一本鎖核酸はCy−3が標識されたターゲット一本鎖核酸とCy−5が標識されたターゲット一本鎖核酸から構成されており、前者は一定量として存在するが、後者は人間血清で相応する生体分子に比例して存在する。これに伴い特定のスポットは後者が相対的に少量であれば青色、等しい量であれば黄色、そして過量であれば赤色になる。
【0085】
核酸チップ上からスポットの分析で二本鎖内のターゲット一本鎖核酸の数によって蛍光強度が相異であるし、これは生体分子の数と相関関係がある。従って本発明の核酸チップを構成するあらゆるスポットのカラースペクトラムを反映するイメージは未知の生体分子を含む生体試料の生体分子プロファイルである。
【0086】
以上のテスト結果は図3であり、図示したように捕捉一本鎖核酸が付着されたガラススライドで血清蛋白質に結合するターゲット一本鎖核酸に基づいた捕捉一本鎖核酸が付着された部位であるスポットは、多様な蛍光程度、青色−黄色−赤色のカラースペクトルを形成する。
【0087】
図3の結果から人間血清蛋白質に結合するターゲット一本鎖核酸に基づいた核酸チップを利用して人間血清から血清蛋白質のプロファイルを確認することができ、また健康な人(A)及び安定狭心症心血管疾患者(B)の血清内生体分子のプロファイルが相異することを確認することができる。
【0088】
図4は患者の血液試料から本発明の核酸チップを利用して生産されたプロファイルを疾患群別で構成されたデータベース化する過程に対する順序のフロー図である。図5は生産されたプロファイルを疾患群別で構成されたデータベース及び人工神経網を利用したプログラムで特定の人の血清に対して生体分子プロファイルを生産して疾病を診断するフロー図である。
【0089】
図4に示したように、多くの生体試料から生産されるプロファイルに構築されたデータベースを生物情報学技術で分析して有用な情報を確保することができる。
【0090】
健康な人、安定狭心症、不安定狭心症及び心筋梗塞などの心血管患者の血清プロファイルデータベースを利用してLee2−1(99)という人の血清試料を検査した結果は図6の通りであり、72.5% 10 fold cross validationで健康な人と予測される。
【0091】
心血管疾患診断用データベース構築に使われた血清試料の臨床的な資料は下の表1であり、健康な人37人、安定狭心症36人、不安定狭心症27人及び心筋梗塞患者27人等総127名である。
【0092】
【表1】

【0093】
健康な人と肝ガンの血清プロファイルデータベースを利用してKIMという人の血清試料を検査した結果は図7の通りであり、93.0% 10 fold cross validationで肝ガン患者と予測される。
【0094】
追加的に転移有無を検査するために、健康な人、非転移肝ガン及び転移肝ガンの血清蛋白質プロファイルデータベースを利用してKIMという人の血清試料を検査した結果は、図8の通りであり、76.0% 10 fold cross validationで非転移肝ガン患者と予測される。
【0095】
肝ガン診断用データベース構築に使用した血清試料の臨床的な資料は表2であり、健康な人19人、非転移肝ガン患者72人及び転移性肝ガン患者11人で、肝ガン患者は83名で試料の総数は102例である。
【0096】
【表2】

【0097】
健康な人と心筋梗塞患者の血清プロファイルを比較して心筋梗塞患者に特異的に存在するスポットを決定し、これに相応する一本鎖核酸にビオチン(biotin)を付着してストレプトアビジン(streptavidin)と反応させた後、血清試料と反応させて複合体を分離した後、電気泳動して形成されるバンドを分離して生体分子を同定する過程のフロー図は図9である。選別された一本鎖核酸に結合する蛋白質を分離してMALDI−TOF−TOF機器で蛋白質のアミノ酸配列を決定した図面は図10である。
【0098】
上記の方法で生体試料を構成する生体分子と結合する一本鎖核酸に基づいた一本鎖核酸が付着された本発明の核酸チップを利用して特定の生体試料で生体分子のプロファイルを探索及び分析した。
【0099】
また、医学的に有用な生体試料の生体分子を生産してデータベースを製作した後、特定の人の血清プロファイルを生産し、人工神経網アルゴリズムを利用して製作したプログラムで心血管疾患の有無及びその種類、肝ガン発病の有無及び転移程度を検査した。
【0100】
疾患群別で構成されたデータベースを比較分析して有用なスポットを決定してこれに相応する一本鎖核酸を調剤して相応する蛋白質を分離して同定した。
【0101】
[実施例6. 細胞株の表面生体分子のプロファイル生成及び応用]
(6−1.核酸チップ製作)
上記実施例1及び2の方法で生体試料である肺ガン細胞株NCI−H1299の表面生体分子のプロファイルを生産する本発明の核酸チップを製作した。準備した無作為一本鎖核酸を肺ガン細胞株に反応させた後、洗浄バッファーを使用して細胞株(生体分子)−一本鎖核酸複合体を洗浄して非結合一本鎖核酸及び結合程度に従い一本鎖核酸を解離させた後5,000xgで遠心分離する過程を反復的に処理した後、細胞株−一本鎖核酸複合体を遠心分離方法で分離して肺ガン細胞株の表面生体分子と結合する一本鎖核酸を製作した。
【0102】
上記製作した一本鎖核酸からクローンを選定して塩基配列を決定し分析して1,000余個の生体分子結合一本鎖核酸を選定した。実施例2の方法で選別された1,000余個の生体分子結合一本鎖核酸の相補的な塩基配列からなるオリゴヌクレオチド(捕捉一本鎖核酸)を合成して固体基板上に付着させ本発明の核酸チップを製作した。
【0103】
(6−2. 細胞株の表面生体分子プロファイル生成)
上記実施例6−1の方法で製作した核酸チップを利用して肺ガン細胞株NCI−H1299の表面分子のプロファイルを生産した。肺ガン細胞株と一本鎖核酸プールを反応させた後、細胞株(生体試料)の生体分子−一本鎖核酸複合体を洗浄及び分離した。上記複合体に結びついた一本鎖核酸を実施例3の方法で増幅及び標識した。捕捉一本鎖核酸と標識されたターゲット一本鎖核酸を実施例4の方法で反応させた。上記ターゲット一本鎖核酸に標識された物質の測定は実施例5の方法で行ったし肺ガン細胞株の表面生体分子のプロファイルを確認した。
【0104】
生産されたプロファイルで肺ガン細胞株に強力に結合することと推定される一本鎖核酸を選定してFITCが付着された一本鎖核酸を合成して肺ガン細胞株と反応させた後、蛍光撮影したイメージは図11である。
【0105】
このように本発明の核酸チップを利用して生産された肺ガン細胞株プロファイルを分析して選定した一本鎖核酸が肺ガン細胞株と結合するのを確認した。
【0106】
[実施例7. 大腸菌の表面生体分子のプロファイル生成及び応用]
(7−1.核酸チップ製作)
生体試料である大腸菌KCTC12006と上記実施例1及び2の方法で本発明の核酸チップを製作した。
【0107】
準備した一本鎖核酸と大腸菌を反応させた後、洗浄バッファーを使用して大腸菌(生体分子)−一本鎖核酸複合体を洗浄して非結合一本鎖核酸及び結合程度にしたがい一本鎖核酸を解離させた後、5,000xgで遠心分離する過程を反復的に処理した後、大腸菌−一本鎖核酸複合体を遠心分離方法で分離して大腸菌の表面生体分子と結合する一本鎖核酸を製作した。
【0108】
上記製作した一本鎖核酸からクローンを選定して塩基配列を決定し分析して1000余個の生体分子結合一本鎖核酸を選定した。選別された1,000余個の生体分子結合一本鎖核酸の相補的な塩基配列でオリゴヌクレオチド(捕捉一本鎖核酸)を合成し固体基板上に付着させ本発明に係る核酸チップを製作した。
【0109】
[7−2.大腸菌の表面生体分子プロファイル生成]
上記実施例7−1の方法で製作した核酸チップを利用して大腸菌KCTC12006の表面分子のプロファイルを生産した。大腸菌と一本鎖核酸プールを反応させた後大腸菌−ターゲット一本鎖核酸複合体を洗浄及び分離した。上記複合体に結合されたターゲット一本鎖核酸を実施例3の方法で増幅及び標識した。捕捉一本鎖核酸と標識されたターゲット一本鎖核酸を実施例4の方法で反応させた。上記ターゲット一本鎖核酸に標識された物質の測定は実施例5の方法で行い、大腸菌の表面生体分子のプロファイルを確認した。
【0110】
大腸菌KCTC12006の表面生体分子プロファイルを生産する同一方法でサルモネラ菌(Salmonella typhimurium ATCC13311)の表面生体分子プロファイルを生産してデータベースを構築した後、階層クラスタリング方法に基づいて大腸菌に特異的に結合する一本鎖核酸を選定した。一本鎖核酸の特異性をSPR装備(BIAcore社)で測定した結果は図12のようであり、選別された一本鎖核酸がサルモネラ菌に比べて大腸菌に特異的に結合していることを確認した。
【0111】
大腸菌に結合する一本鎖核酸及びナノ金粒子を利用して大腸菌を測定する方法(例、大韓民国特許出願 No.10−2006−0072480;Patent No.10−828936 参照)を用いて、上記で選別された一本鎖核酸を利用して溶液に大腸菌の有無及び飲食物に大腸菌の汚染程度を測定した結果は図13のようである。飲食物を洗浄したSELEX バッファーに一本鎖核酸を反応させナノ金粒子を入れた後、NaCl溶液を添加して、大腸菌のない溶液は透明な無色に近い青色であり、大腸菌に汚染になった溶液は赤色に変化し、その程度は汚染になった大腸菌数に比例するのを確認した。
【産業上の利用可能性】
【0112】
本発明は未知の生体分子と一本鎖核酸の結合プロファイルを生成するための核酸チップ、核酸チップの製造方法、及び核酸チップを利用した未知の生体分子分析方法に関するもので、並列高速分析(High Throughput Screening)を利用した極めて簡単であり、しかも低廉で効率的な方法として、微生物、細胞、蛋白質を含む生体試料に含まれている未知の生体分子に対するプロファイルを生産することができるので、医学、獣医学、環境工学、食品工学、農業などの広範囲な分野で未知の生体分子の生物学的意味を分析する道具として応用できるものと期待される。
【技術分野】
【0001】
本発明は、未知の生体分子と一本鎖核酸の結合プロファイルを生成するための核酸チップ、核酸チップの製造方法、及び核酸チップを利用した未知の生体分子分析方法に関する。
【背景技術】
【0002】
生体試料を構成する未知の生体分子を含んだ数多くの生体分子(蛋白質及び炭素化物)のプロファイルを生産する技術は、物理学、生化学、及び生物情報学の発達で広く開発されているが、既存方法と装置は、その使用及び維持費、容易性、正確性、感度、検査時間、及び工程の自動化等に多様な問題があるため、効率的な新しい方法及び装置に対する必要性は非常に高い。
【0003】
生体試料でこれら生体分子の量的な状態を総合化した情報、すなわち生体分子のプロファイル(profile)を生産する方法は、それ自体が窮極的な目標ではなく、目標をなすための一つの手段であるが、微生物、細胞、組織等に有用に使用することができて、医学、獣医学、環境工学、食品工学、農業等広範囲に適用されている。
【0004】
核酸は、ヌクレオチドが共有結合で連結した線形的な重合体であり、ヌクレオチドは小さな有機化合物として、燐酸、糖、及びプリン(アデニン又はグアニン)(purine(adenine、guanine))あるいはピリミジン(シトシン、チミジン、又はウラシル)(pyrimidine(cytosine、thymidine、uracil))から構成される。核酸は一本鎖あるいは二本鎖として存在する。一本鎖核酸は特定の物理的な条件でヌクレオチドの間の水素結合及び相互作用により結合して独特の立体構造を形成するが、こういう立体構造は一本鎖の塩基配列により決定される。
【0005】
一般的に、DNAやRNAのような核酸は、細胞構造や酵素などの活性を有する蛋白質を発現するための情報の貯蔵体である。1982年に、RNAが特定の構造を形成することによって酵素的活性も持っているという報告がなされて以後、核酸の構造的な特性とそれに伴う特定機能に対する多くの報告がなされている。
【0006】
核酸は、4個の塩基の反復で構成されていて、多様性を維持しながら多くの立体構造を形成し、こういう立体構造は特定物質と相互作用をして複合体を形成して安定化される。
【0007】
核酸は、蛋白質を含む分子に対して一つのリガンド(ligand)として作用し、多様な塩基配列の一本鎖核酸から構成されたライブラリーから一定の選別過程と塩基配列決定過程を通して,高い結合力と特異性で特定物質と結合する核酸が選別されている。
【0008】
特定物質と結合する核酸を選別する方法をSELEX (Systematic Evolution of Ligands by EXponential enrichment)といい、選別された核酸をいわゆるアプタマー(aptamer)という(Tuerk C、and Gold L.; Science、249、pp505−510、1990)。
【0009】
SELEXを通して自然状態で核酸と結合する蛋白質や、核酸と結合していない蛋白質のように、多様な生体分子と非常に高い親和力を有して結合できる核酸(アプタマー)が選別されている。
【0010】
既存のSELEXによる核酸選別方法は、特定物質(例えば、蛋白質)に特異的に結合する核酸を選別するに先立ち、優先的に該当蛋白質(特定物質)を大量生産して精製後、生産された蛋白質に一本鎖核酸ライブラリーを反応させ選択と増幅を反復して結合力が強力な核酸(アプタマー)を選定する方法として、核酸を選別するためにはあらかじめ特定物質を確保することが必須であった。このような従来のSELEXを通した核酸選別方法は、生体試料に含まれている数多くの未知の生体分子に対する生物学的意味を持つ集団としての核酸を選別し利用する技術は全く認識していなかった。
【0011】
生体試料である組織、細胞、及び微生物などを構成する未知分子を含んだ生体分子のプロファイルは、物理、化学的な性質を利用して様々な方法から作られており、一般的に、生体分子の分子量あるいはpI値などを利用して電気泳動をすることで生体試料から生体分子の量的な状態である生体分子のプロファイルを確認している。
【0012】
また、プロファイルを分析して有用な生体分子を決定しこれを分離した後、MALDI−TOF(Matrix Assisted Laser Desorption/Ionization−Time Of Flight)などでこれらの構成成分を確認する方法などが行われている。最近では、SELDI−TOF−MS (Surface−enhanced laser desorption/ionization time of flight mass spectrometry)を用いて蛋白質プロファイルに対する多くの研究が進行されている(Adam et al.、Cancer Research、62、3609−3614. 2002; Li et al.、Clinical Chemistry、48、1296−1304. 2002; and Petricoin et al.、The Lancet、359、572−577. 2002)。
【0013】
また、最近では、蛋白質に対する並列高速分析(High Through put Screening)の技法で、蛋白質チップ(Protein chip)あるいはアプタマーチップ(Aptamer chip)(Smith et al.、MolCell Protomics、11−18.2003; and McCauley et al.、Anal Biochem、319(2)、244−250.2003)が開発され、使用されている。
【0014】
蛋白質チップは、抗体の配列状態を知っているため、特定物質の区分及び定量に対する探索及び分析が可能である。蛋白質チップの製作方法では、マイクロアレイヤー(Micro arrayer)を利用して、抗体をスポッティング(spotting)して固定する方法がある。蛋白質チップは、一つのチップでなるべく多くの情報を提供できるように、狭い面積に高密度で多様な抗体が集積されている状態で発生する、非常に弱い信号を感知しなければならない。また、蛋白質に対する生体情報が増えるに従い、その集積度がより一層高まる趨勢で発達しているので、これを定量的に速くて正確に検出する新しい方法が要求されている。
【0015】
現在まで蛋白質チップの分析方法は、主にレーザー誘発蛍光法(laser induced fluorescence)が広く使われていて、電気化学的検出方法などが開発されている。上記の通り、多様な工程により、蛋白質チップを利用した生体試料から特定蛋白質等のプロファイルを生産及び分析する方法が開発されているが高価の装置及び試薬を使用すべく複雑な過程を遂行しなければならないなどの問題があるし、また抗体を製作することのできる分子のみ製作が可能な限界がある。
【0016】
アプタマーチップは、蛋白質チップに抗体を使用することの代わりにアプタマー(核酸)を使用する点が異なり、その他の要素は非常に類似している。
このように、蛋白質チップ及びアプタマーチップを利用した生体試料から生体分子の量的な状態、すなわちプロファイルを生産する方法が開発されているが、高価の装置及び試薬を使用しており、複雑な工程を経なければならないなどの問題がある。特にいままでの蛋白質チップやアプタマーチップは抗体あるいはアプタマーを製作することのできる既に知られている蛋白質のみ制限的に製作が可能な限界がある。
【0017】
生体試料は数百万個の蛋白質から構成されているが、現在知らされている蛋白質の数は数万個に過ぎない実情であるので、生体試料で未知の蛋白質のような未知の生体分子の量的状態、すなわちプロファイルを生産する技術の必要性は非常に高い。
【0018】
生体試料の生体分子を総体的に分析する研究は、医学的に疾病に関連した生体分子のプロファイルを分析することによって、疾病を診断することのできる生体分子、治療成績を分析しえる生体分子、疾病発病及び進行に重要な役割をする生体分子、疾病に対する感受性に関連する生体分子、及び新薬開発の標的分子を究明することに使用することができる。
【発明の概要】
【発明が解決しようとする課題】
【0019】
本発明の目的は、生体試料に含まれている未知の生体分子と一本鎖核酸の結合プロファイルを生成可能な核酸チップを提供することにより、核酸チップを利用して生成した結合プロファイルから未知の生体分子と関連した疾病情報を含んだ各種生物学的意味を分析可能にすることにある。
【課題を解決するための手段】
【0020】
本発明により、一本鎖核酸と未知生体分子の結合プロファイルの生成に使われる核酸チップとその製造方法、及び核酸チップを利用した未知の生体分子分析方法が提供され、本発明にかかる核酸チップは、生体試料に含まれた未知生体分子の生物学的意味を分析するために使われる。
【0021】
本発明による核酸チップの製造方法は、未知の生体分子が含まれている生体試料と無作為塩基配列を持つ無作為一本鎖核酸を反応させて、上記未知の生体分子に結合する生体分子結合一本鎖核酸を決定する第1段階、及び決定された上記生体分子結合一本鎖核酸及び/または上記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸を合成して基板に固着する第2段階を含む。
【0022】
本発明による核酸チップは、生体試料に含まれた未知の生体分子と結合する生体分子結合一本鎖核酸及び/または上記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸が基板に固着されている構成になっている。本発明による核酸チップを利用した未知の生体分子分析方法は、上記本発明による核酸チップを準備する段階;上記核酸チップの上記捕捉一本鎖核酸と同じ塩基配列を持つ一本鎖核酸と上記生体試料の上記未知生体分子を反応させ、生体分子−ターゲット一本鎖核酸複合体を形成し、上記生体分子−ターゲット一本鎖核酸複合体を分離する段階;分離された上記生体分子−ターゲット一本鎖核酸複合体から上記ターゲット一本鎖核酸を分離、増幅及び標識する段階;標識した上記ターゲット一本鎖核酸を上記核酸チップの上記捕捉一本鎖核酸と反応させ上記標識を通し結合プロファイルを獲得する段階;及び獲得された上記プロファイルと既に確保されているプロファイルデータを比較して上記未知の生体分子の生物学的意味を分析する段階;を含む。
【0023】
本発明による核酸チップ及びこれを利用した未知の生体分子分析方法の基本的な原理は、未知の生体分子に結合する一本鎖核酸に相補的な一本鎖核酸(捕捉一本鎖核酸)を核酸チップに固着し、テストの対象になる未知の生体分子とこれに結合する一本鎖核酸を反応させて生体分子−ターゲット一本鎖核酸複合体を形成した後、上記複合体から分離されたターゲット一本鎖核酸と核酸チップの捕捉一本鎖核酸の相補的な結合のプロファイルを獲得してその特性を分析することによって、結果的に未知の生体分子の一本鎖核酸に対する結合プロファイル特性を分析することであるので、核酸チップの捕捉一本鎖核酸として未知の生体分子に結合する一本鎖核酸に相補的に結合する一本鎖核酸を基板に固着すればよいが、未知の生体分子を分析する時、生体分子−ターゲット一本鎖核酸複合体から分離したターゲット一本鎖核酸を増幅する過程で未知の生体分子に結合するターゲット一本鎖核酸に相補的な一本鎖核酸もターゲット一本鎖核酸のプロファイル情報と同一類似の情報を内包した状態で生成されるので、本発明による核酸チップの捕捉一本鎖核酸として未知の生体分子と結合する生体分子結合一本鎖核酸を使用しても同じ目的を達成できて、ひいては生体分子結合一本鎖核酸と、生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を同時に使用しても同じ目的を達成することができる。
【0024】
本発明による核酸チップを利用して未知の生体分子を分析すれば、関連プロファイルデータが蓄積され、これから未知の生体分子の生物学的意味の分析に寄与する特異的な一本鎖核酸が自然に明らかになり、これで未知の生体分子を分析することに意味を持つ一本鎖核酸を発掘することができるようになる。
【0025】
本発明による核酸チップが適用される上記生体試料としては、細菌、真菌類、ウイルス、細胞株、組織などが含まれるし、本発明による核酸チップによりその生物学的意味が分析されることができる生体分子としては蛋白質、炭水化物、脂質、多糖体、糖蛋白、ホルモン、受容体、抗原、抗体及び酵素からなる群から選択された少なくとも一つ以上の生体分子が含まれる。
【0026】
本発明による核酸チップの製造方法の上記1段階は、未知の生体分子が含まれている生体試料と、無作為塩基配列を持つ一本鎖核酸(以下、無作為一本鎖核酸という)を反応させて未知の生体分子と結合する生体分子結合一本鎖核酸を決定する段階である。
【0027】
望ましくは、上記生体分子結合一本鎖核酸を決定する段階は、上記生体試料の上記未知の生体分子に上記無作為一本鎖核酸を反応させて生体分子−一本鎖核酸複合体を製造する段階;上記生体分子−一本鎖核酸複合体を洗浄し、上記生体分子と上記一本鎖核酸の結合力に基づいて一定結合力以上の上記生体分子−一本鎖核酸複合体を選択する段階;選択された上記複合体から上記一本鎖核酸を分離して増幅する段階;及び上記増幅された上記一本鎖核酸をクローニングベクターに挿入して単一クローンを確保して上記一本鎖核酸の塩基配列を決定することによって上記生体分子結合一本鎖核酸を決定する段階;を含む。
【0028】
上記生体分子−一本鎖核酸複合体の選択及び増幅は反復的に多数回実行することができるが、未知の生体分子を含む生体試料は数多くの生体分子から構成されていてこれらの量的な差が非常に多様なので、未知の生体分子に結合する一本鎖核酸の選択と増幅を反復的に行う環形的な方法より選択と増幅を一回行って洗浄過程を強化する線形的な方法で製作するのがより望ましい。
【0029】
上記無作為塩基配列を持つ無作為一本鎖核酸は、下のような無作為の塩基配列を持つ一本鎖DNAオリゴヌクレオチドを利用して二本鎖のDNAを製造した後、生体外転写(in vitro transcription)を通してRNA無作為一本鎖核酸に製造できる。
【0030】
5’−GGGAGAGCGGAAGCGTGCTGGGCC
N40 CATAACCCAGAGGTCGATGGATCCCCCC−3’
(ここでアンダーラインをした塩基配列は核酸ライブラリーの固定された部位で、N40は各位置にA、G、T、C等の塩基が同じ濃度で存在するのを意味する。)
【0031】
PCR(Polymerase Chain Reaction)に利用されるFWプライマー(5’−GGGGGAATTCTAATACGACTCACTATAGGGAGAGCGGAAGCGTGCTGGG−3’:配列番号1)は、前期の塩基配列のアンダーラインをした塩基の5’末端と塩基結合をすることができ、バクテリオファージT7のRNAポリマラーゼのためのプロモーター塩基配列を含んでいる。
【0032】
PCRのREプライマー(5’−GGGGGGATCCATCGACCTCTGGGTTATG−3’:配列番号2)は上記の塩基配列のアンダーラインをした塩基の3’末端と塩基結合をすることができる。
【0033】
望ましくは、本発明の核酸チップ製造方法で生体分子結合一本鎖核酸は2’−F−置換ピリミジンを含むRNA一本鎖核酸であり、生体外転写で合成し精製して準備する。
【0034】
合成された無作為一本鎖核酸は例えば1015塩基配列/1mlの濃度で生体試料に処理して生体分子と30分間反応させることができる。
【0035】
上記生体分子結合一本鎖核酸はRT−PCR(Reverse Transcription−PCR)して得た産物をクローニングベクターに挿入して大腸菌クローンを確保し、その塩基配列を決定できる。
【0036】
この時反応温度はSELEX遂行温度より低い温度で行うことが理想的であり、望ましくは20℃〜37℃の温度で反応を行う。
【0037】
普遍的に蛋白質及び一本鎖核酸を過量処理して生体分子と結合する一本鎖核酸の非特異的な結合を防止し、望ましくは酵母tRNA(yeast tRNA)、サーモンスペルマ(salmon sperm DNA)または人間胎盤DNA(human placental DNA)を使用することができる。
【0038】
本発明による核酸チップの製造方法の上記第2段階は、第1段階で決定された生体分子結合一本鎖核酸及び/または上記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸を合成して、合成された捕捉一本鎖核酸を基板上に固着させて本発明の核酸チップを製作する段階である。
【0039】
捕捉一本鎖核酸は混成化程度に非常に大きい影響を及ぼす核心要素であるので、その塩基配列構成の決定は非常に重要である。本発明の核酸チップに固着されるそれぞれの捕捉一本鎖核酸は特徴的な塩基で構成されて、捕捉一本鎖核酸とターゲット一本鎖核酸の結合体は適切なTm値を維持しなければならない。結合体の混成化程度は蛍光が標識されたターゲット一本鎖核酸により汚染されていない自分のシグナル値を維持できるようにする。
【0040】
したがって捕捉一本鎖核酸の塩基配列は、上記第1段階で無作為一本鎖核酸から選別された生体分子結合一本鎖核酸の塩基配列を基づいて決定される。望ましくは、捕捉一本鎖核酸は16bp〜60bpの塩基配列を持つオリゴヌクレオチドの一本鎖核酸である。
【0041】
また、本発明の核酸チップの製作方法における基板は、ガラス、シリコンなどの無機物やアクリル系やPET(polyethylene terephtalate)、ポリカーボネート(polycarbonate)、ポリスチレン(polystyrene)、ポリプロフィレン(polypropylene)などの高分子物質から製作したものを使用することができて、望ましくはガラスで製作したスライドグラスである。この時、基板はアミン基またはアルデヒド基等でコーティングしたものを使用することができる。例えばGAPS(Gamma Amino Propyl Silane)がコーティングされたガラススライドであるUltra GAPSTM Coated Slide(Corning社)を使用して上記捕捉一本鎖核酸を一定規則で配列固着して核酸チップを製作する。
【0042】
本発明による核酸チップの製造は、マイクロアレイヤーシステム(Micro arrayer system)を使用することができ、それぞれの捕捉一本鎖核酸を緩衝額に溶かして濃度を調節し、この時アレイャー中の湿度は70%〜80%に維持してスポッティング(spotting)を行う。スポッティングされたスライドは湿度維持チャンバー(humidified chamber)で放置した後、UV crosslinkerで焼く(baking)過程を行う。
【0043】
このように本発明による核酸チップは、関連技術分野に広く知られている方法で捕捉一本鎖核酸をガラススライドに固定させた後、スライドをすぐ遠心分離して乾燥させた後、使用する前まで光を遮断した状態で保管する。
【0044】
既に公知されている多様な方法(M.schena; DNAmicro array; apractical approach、Oxford、1999)で捕捉一本鎖核酸が規則的に固着された核酸チップを製作するこてができる。
【0045】
本発明による核酸チップは、同一類似の精密度で未知生体分子の生物学的意味を分析することさえあれば、核酸チップに固着する捕捉一本鎖核酸の数を少なくすることが核酸チップの製作費及び分析の効率性側面で望ましい。
【0046】
かかる理由で、本発明の核酸チップ製造方法は,未知生体分子の生物学的意味分析に対するそれぞれの捕捉一本鎖核酸の寄与度を分析し、未知生体分子の生物学的意味を分析することができる範囲内で、分析された寄与度に基づいて捕捉一本鎖核酸を選別することで、本発明の核酸チップに固着する捕捉一本鎖核酸の数を減少させる段階を更に含むことができる。
【発明の効果】
【0047】
本発明による核酸チップ及びこれを利用した未知の生体分子分析方法によれば、並列高速分析(High Through put Screening)を利用した極めて簡単で低廉で効率的な方法で、微生物、細胞、蛋白質を含む生体試料に含まれた未知の生体分子に対するプロファイルを生産し得るので、医学、獣医学、環境工学、食品工学、農業などに広範囲な分野で未知の生体分子の生物学的意味を分析する道具として応用できるものと期待する。
【0048】
また、本発明による核酸チップ及びこれを利用した生体分子分析方法によれば、未知の生体分子の生物学的意味を分析する過程から、未知の生体分子の生物学的作用を把握し、その構造を決定、究明しえるばかりでなく、生体分子に特異的に結合する特定一本鎖核酸を選別でき、選別された一本鎖核酸を利用して更に精巧な生体分子の作用を把握する道具として利用できる環境を提供する。
【0049】
また、本発明による核酸チップを利用した生体分子分析方法によれば、医学的に疾病に関連した生体分子のプロファイルを分析することによって、疾病を診断する生体分子、治療成績を分析する生体分子、疾病の発病及び進行に重要な役割をする生体分子、疾病の感受性に関連した生体分子、及び新薬開発の標的分子などを非常に低廉で簡便な方法で、しかも效率的に糾明することができる。
【図面の簡単な説明】
【0050】
【図1】図1は、本発明の核酸チップの製造方法に係る生体分子結合一本鎖核酸を決定する過程を示す概略図である。
【図2】図2は、本発明に係る核酸チップを利用して未知の生体分子と一本鎖核酸の結合プロファイルを生成する過程を示す概略図である。
【図3】図3は、本発明に係る核酸チップを適用して生成した健康な人(A)と安定狭心症心血管患者(B)血清のプロファイルである。
【図4】図4は、本発明に係る核酸チップを利用して患者の血清試料から生産されたプロファイルを疾患群別でデータベースを構築する過程の順序を示す概略図である。
【図5】図5は、本発明にかかる核酸チップで生成された人間血清蛋白質プロファイルのデータベース及び人工神経網アルゴリズムを利用したプログラムで疾病を診断する過程を示す図面である。
【図6】図6は、本発明に係る核酸チップを利用して生成した人間血清蛋白質プロファイルのデータベース及び人工神経網アルゴリズムを利用したプログラムで心血管疾患を診断した結果を示す図面である。
【図7】図7は本発明に係る核酸チップを利用して生成した人間血清蛋白質プロファイルのデータベース及び人工神経網アルゴリズムを利用したプログラムで肝ガンを診断した結果を示す図面である。
【図8】図8は、本発明に係る核酸チップを利用して生成された人間血清蛋白質プロファイルのデータベース及び人工神経網アルゴリズムを利用したプログラムで、肝ガンの転移を診断した結果を示す図面である。
【図9】図9は、本発明に係る核酸チップを利用して生成した人間血清蛋白質プロファイルのデータベースで心筋梗塞患者の血清に特異的に存在する蛋白質のアミノ酸配列を決定する過程の順序を示す図面である。
【図10】図10は、本発明に係る核酸チップを利用して生成した人間血清蛋白質プロファイルのデータベースで心筋梗塞患者の血清に特異的に存在する蛋白質のアミノ酸配列を決定した図面である。
【図11】図11は、本発明に係る核酸チップを利用して生成した肺ガン細胞株NCI−H1299の表面生体分子プロファイルを生産及び分析して確保した一本鎖核酸が肺ガン細胞株に結合した結果を示す図面である。
【図12】図12は、本発明に係る核酸チップを利用した大腸菌KCTC12006及びサルモネラ菌(Salmonella typhimurium ATCC13311)の表面生体分子プロファイルを生産及び分析して確保した生体分子結合一本鎖核酸の特異性を示す図面である。
【図13】図13は、本発明に係る核酸チップを利用して選別された、大腸菌に特異的に結合する生体分子結合一本鎖核酸とナノ金粒子を利用して大腸菌に汚染した飲食物から汚染程度を測定した結果を示す図面である。
【発明を実施するための形態】
【0051】
以下、貼付図面と実施例を参照して本発明にかかる核酸チップ、核酸チップの製造方法、及び核酸チップを利用した未知の生体分子分析方法を詳細に説明する。以下の具体例は本発明を例示的に説明するものであり本発明の範囲を制限するものに意図しない。
【0052】
[実施例1. 人間血清蛋白質に結合する生体分子結合一本鎖核酸の製造]
図1に示したように、下のような無作為の塩基配列を持つ一本鎖のDNAオリゴヌクレオチドを利用してPCR(Polymerase Chain Reaction)を通し二本鎖のDNAを製造した後、生体外転写を通して一本鎖のRNAライブラリー(無作為一本鎖核酸)を製造した。
【0053】
5’−GGGAGAGCGGAAGCGTGCTGGGCC
N40 CATAACCCAGAGGTCGATGGATCCCCCC−3’
(ここでアンダーラインをした塩基配列は核酸ライブラリーの固定された部位でありN40は各位置にA、G、T、C等の塩基が同じ濃度で存在することを意味する。)
【0054】
PCRに利用される上記配列番号1のFWプライマーは、上の塩基配列のアンダーラインをした塩基の5’末端と塩基結合をすることができ、バクテリオファージT7のRNAポリマラーゼのためのプロモーター塩基配列を含んでいる。PCRに利用される配列番号2のREプライマーは上の塩基配列のアンダーラインをした塩基の3’末端と塩基結合をすることができる。そしてFWとREプライマーは以後のクローニングのために各々EcoRIとBamHI等の制限酵素切断塩基配列を含有している。
【0055】
生体試料と反応する無作為一本鎖核酸は2’−F−置換ピリミジンを含むRNAライブラリーであり、DNA核酸ライブラリー転写体及びPCRプライマーを上記のように設計製作してPCR及び生体外転写方法で準備した。
【0056】
PCRは1,000pmoles一本鎖核酸転写体、2,500pmoles PCRのプライマー対(5P7)を50mM KCl、10mM Tris−Cl(pH8.3)、3mM MgCl2、0.5mM dNTP(dATP、dCTP、dGTP、及びdTTP)、0.1U Taq DNA Polymerase(Perkin−Elmer、Foster City Calif.)でPCRを行った後QIA quick−spin PCR精製コラム(QIAGENInc.、Chatsworth Calif.)で精製した。
【0057】
2’−F−置換ピリミジンを含む無作為一本鎖核酸は生体外転写で合成し精製して準備した。200pmoles二本鎖DNA転写体、40 mM Tris−Cl(pH8.0)、12 mM MgCl2、5 mM DTT、1 mM spermidine、0.002% Triton×−100、4% PEG 8000、5 U T7 RNA Polymerase、そして1 mM ATP、GTP及び3 mM 2’F−CTP、2’F−UTPを37℃で6〜12時間反応させた後、Bio−Spin 6クロマトグラフィーコラム(Bio−Rad Laboratories、Hercules Calif.)で精製した後、UVスペクトロメーターを利用して精製された核酸の量及びその純粋度を検索した。
【0058】
上記で合成した無作為一本鎖核酸の1015塩基配列/1mlの濃度溶液を200pmol/200μlの濃度で選択バッファー(50mM Tris・Cl(pH7.4)、5mM KCl、100mM NaCl、1mM MgCl2、0.1% NaN3)で80℃で10分間加熱した後、氷に10分間放置した。使用した一本鎖核酸の5倍の酵母tRNA(yeast tRNA、Life Technologies社)及び0.2% BSA(bovine serum albumin、Merck社)を添加して反応溶液を準備した。
【0059】
10μlの血清試料を90μlのPBS溶液を添加してナイトロセルロース膜ディスク(nitro cellulose membrane disc)を入れて30分間振りあげながら反応させた。上記で準備したRNA一本鎖核酸を血清試料が付着されたディスクに処理して30分間反応させた。
【0060】
人間血清試料(生体分子)と結合する生体分子結合一本鎖核酸の選定は、一次的に人間血清試料に結合力を見せる一本鎖核酸を対象とし、反応後多様な洗浄バッファーで洗浄過程を反復して一回のみの選択過程で人間血清蛋白質(生体分子)−一本鎖核酸複合体を確保した。
【0061】
生体分子−一本鎖核酸複合体の洗浄バッファーは0〜1×SELEXバッファーまたは0〜500 mM EDTA溶液を使用した。分離された複合体をRT−PCRを行って血清蛋白質に結合するRNA(生体分子結合一本鎖核酸)を指示するDNAプールを増幅製作した。この時、上記の選択と増幅過程を多数回反復して生体分子結合一本鎖核酸を製作することができる。
【0062】
確保されたRT−PCR産物であるDNAをプラスミドにクローニングして単一クローンを確保する過程で、生体分子結合−一本鎖核酸の製作を完了した。これら生体分子に結合する一本鎖核酸の塩基配列は標準方法でプラスミドを分離して行った。
【0063】
生体分子のプロファイルを生成するための本発明に係る核酸チップに使用する捕捉一本鎖核酸の塩基配列は人間血清蛋白質(生体分子)と結合する上記生体分子結合一本鎖核酸の塩基配列及び/または生体分子結合一本鎖核酸に相補的に結合する一本鎖核酸の塩基配列であるので、このために核酸の二次構造をモデリングするMFOLDプログラムを使用して二次構造及び構造体自由エネルギーを確認した後、最も安定度が高い二次構造を有する生体分子結合一本鎖核酸を選定した。
【0064】
[実施例2. 核酸チップの製造]
ガラススライド表面に固着させる捕捉一本鎖核酸として、実施例1から選別された塩基配列を持つ3,000余個μの生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸(オリゴヌクレオチド)を有機合成して(バーイオニア社)準備した。
【0065】
GAPS(Gamma Amino Propyl Silane)がコーティングされたガラススライドであるUltraGAPSTM Coated Slide(Corning社)を使用して捕捉一本鎖核酸が一定規則で固着された核酸チップを製作した。核酸チップの製作はピン(pin)方式であるマイクロアレイヤーシステム(Microarrayer system、GenPak社)を使用したし、スポット間隔(spot spacing)はスポット中心の間(center−to−center)が370μmになるようにした。それぞれの捕捉一本鎖核酸を標準溶液に溶かし濃度を調節した。この時アレイの中の湿度は70%に維持してスポッティング(spotting)を行った。スポッティングされたスライドは湿度維持チャンバー(humidified chamber)で24〜48時間放置した後、UV crosslinkerで焼く(baking)過程を行った。広く知られている方法で捕捉一本鎖核酸をガラススライドに固定させた後、スライドをすぐ遠心分離して乾燥した後、光が遮断された状態で保管した。
【0066】
[実施例3. 人間血清(生体分子)−ターゲット一本鎖核酸複合体の製造及びターゲット一本鎖核酸の製造]
実施例1で選定され核酸チップ製造に使用された生体分子結合一本鎖核酸が挿入されたプラスミドを同量で混合して未知の生体分子を含む生体分子と結合する一本鎖核酸プールの転写体を準備するためのプラスミドプールを準備した。人間血清蛋白質と結合する一本鎖核酸プールは上記プラスミドプール及びPCRプライマーを設計製作してPCR及び生体外転写方法で準備した。
【0067】
準備したプラスミドプールで1pgプラスミドを転写体にして、PCR反応溶液、100pM 5’−プライマー、100pM 3’−プライマー、dNTP混合物(5mM dATP、5mM dCTP、5 mM dGTP、5 mM dTTP) で標準PCR方法で94℃30秒、52℃30秒、72℃20秒の条件で30回サイクルを反復行って二本鎖核酸を合成してQIAquick−spin PCR精製コラム(QIAGEN Inc.、Chatsworth Calif.)で精製した。
【0068】
2’−F−置換ピリミジンを含むRNA分子のターゲット一本鎖核酸を生体外転写で合成し精製して準備した。
【0069】
200 pmoles二本鎖DNA転写体、40 mM Tris−Cl(pH8.0)、12 mM MgCl2、5 mM DTT、1mM spermidine、0.002% TritonX−100、4% PEG 8000、5U T7 RNA Polymerase、そして1 mM ATP、GTP及び3 mM 2’F−CTP、2’F−UTPを37℃で6〜12時間反応させた後、Bio−Spin6クロマトグラフィーコラム(Bio−Rad Laboratories、Hercules Calif.)で精製した。
【0070】
分析に使用した人間血清は健康な人及び安定狭心症である心血管疾患患者の血清であり10μlの血清試料を90μlのPBS溶液を添加してナイトロセルロース膜(Nitrocellulose membrane)ディスクを入れて30分間振りあげながら反応させ試料を準備した。先に製作した100〜400ngのターゲット一本鎖核酸を投入して30分間反応させ生体分子−ターゲット一本鎖核酸複合体を形成した。
【0071】
準備した一本鎖核酸を血清試料が付着されたディスクに処理して30分間反応させた。選択バッファーあるいは50mM EDTAで3回洗浄し反応産物を分離して収穫した。
【0072】
血清蛋白質(生体分子)−ターゲット一本鎖核酸がついたディスクをRT−PCR溶液に処理してRT−PCRを行いながらCy−5で標識されたプライマー(5’−Cy5−CGGAAGCGTGCTGGGCC−3’:配列番号3)を準備した。上記で準備したプラスミドプールで準備したターゲット一本鎖核酸を利用して上記と同一方法でRT−PCRしてCy−3が標識されたプライマー(5’−Cy3−CGGAAGCGTGCTGGGCC−3’)を製作して準備した。上記二つの溶液を同量で混合してターゲット一本鎖核酸を製作した。
【0073】
[実施例4. 核酸チップとターゲット一本鎖核酸の反応]
図2に示したように、上記実施例3で準備したターゲット一本鎖核酸を本発明の核酸チップの捕捉一本鎖核酸に処理して60℃で4〜12時間混成化し42℃で0.1×SSC溶液で洗浄した。このとき混成化溶液は1M塩化ナトリューム、0.3M sodium citrate、0.5% SDSまたは100ug/ml サーモンスペルマDNA、0.2%牛血清アルブミンまたは一本鎖核酸を含む。
【0074】
前混成化が終わったガラススライドに実施例3で準備した溶液を処理して42℃で12時間混成化(Hybridization)を行った後、核酸チップを洗浄溶液で洗浄した。このとき、洗浄溶液の造成は例えば、1×SSC+0.2% SDS、1×SSC+ 0.2% SDS、0.5×SSC+0.2% SDS、0.01×SSC+0.2% SDS順の段階であり各々段階ごとに42℃で30分間行った。
【0075】
[実施例5. 核酸チップのスポット探索及び分析]
上記実施例4の洗浄が終了した後、ガラススライドを遠心分離で乾燥した後、使用した蛍光染料を励起(excitation)するための適切な波長のレーザー(Cy5、635nm)を持つレーザースキャナー(laser scanner、GenePix4000、Axon社)を利用して探索した。蛍光イメージは多重−イメージ−タグされたイメージファイル(multi−image−tagged image file、TIFF)フォーマットで貯蔵した後、適切な画像分析(image analysis)ソフトウェア(GenePix Pro 3.0、Axon社)で分析した。
【0076】
スポット(spot)当たりのシグナル強度(signal intensity、単位:quanta)は周囲背景の基本シグナルを除いたものを使用するが、ここで背景シグナルは特定スポットの周囲にある4個のスポットの周辺シグナルから構成された位置的バックグラウンド(local background)を意味する。スポットが持っている画素(pixel)の90%以上が背景シグナル+2標準偏差(S.D.; standard deviation)を超過するシグナル強度を見せるときデータ分析に含めて、上の条件を充足させることができなければデータに含めないスポット(bad spot)で分類して、分析に含まないことが一般的である。
【0077】
標識効率(Labeling efficient)に伴う偏差(variation)は内部標準(internal standard、IS)のシグナルを利用して平準化(e.g.、Normalized Intensity = Probe Intensity/ IS intensity)し、単一標識(monolabeling)をした場合にはCy5チャンネルのシグナル強度(signal intensity)でその結果を記録し、スポッティング(spotting)が2倍以上になった場合は平均値を使用する。ターゲット一本鎖核酸のシグナル強度(signal intensity、S)はスポットが持つピクセル(pixel) に対してそれぞれのシグナル強度を求めた後これらの中心値(median)を使用する(median value of pixel−by−pixel)。シグナル強度(S)は内部標準(IS)を利用して標識効率にともなう偏差を平準化する。
【0078】
S’(normalized value)=S(Cy5−reference)×(Cy5−IS)。
【0079】
以上の方法でピクセル密度の分析結果と実際試料量の間の関係を糾明してその相関関係を確認することができる。核酸チップ蛍光データをイメージ化し、あらゆるスポットのパターンで生体試料から生体分子プロファイルを確認することができるし、スポット等のパターンを階層クラスタリング(Hierarchical Clustering)及び人工神経網(Artificial Neural Network)などの方法などで分析して使用することができる。
【0080】
スポットの蛍光程度は捕捉一本鎖核酸とターゲット一本鎖核酸が形成する二本鎖の性質によって多様に示される。人間血清蛋白質−(ターゲット)一本鎖核酸複合体の結合強度及び量は、これらの間の特異性及び結合力に従い決定される。
【0081】
人間血清蛋白質−ターゲット一本鎖核酸複合体で起源するターゲット一本鎖核酸と核酸チップに付着された捕捉一本鎖核酸の塩基配列はターゲット一本鎖核酸−捕捉一本鎖核酸の二本鎖安定性に、そして核酸チップ上のターゲット一本鎖核酸の量は蛍光強度に影響を与える。
【0082】
すなわち、蛍光程度はターゲット一本鎖核酸の量を表し、ターゲット一本鎖核酸の量は人間血清蛋白質−ターゲット一本鎖核酸複合体の量を示す。また上記複合体の量は生体試料にある生体分子の量を表す。したがって、スポットの蛍光度からスポットに相応する未知の生体分子を含む生体試料から特定生体分子の量を確認することができる。結局、形成されたスポットの蛍光程度を分析して核酸チップのあらゆるスポットのパターンを確認することによって人間血清で血清蛋白質のプロファイルを確認することができる。
【0083】
すなわち、形成されるスポットが青色−黄色−赤色のカラースペクトラムに見えるのは捕捉一本鎖核酸に結合したCy−3が標識されたターゲット一本鎖核酸とCy−5が標識されたターゲット一本鎖核酸の比率が多様で現れる現状であり、特定スポットのカラースペクトラムの程度は人間血清を構成する特定の血清試料に存在する特定の生体分子(蛋白質)の量を表現するため、核酸チップ上のあらゆるスポットのカラースペクトラムを見せるイメージは特定生体試料で生体分子プロファイルに該当する。
【0084】
すなわち、本発明の生体分子分析方法では特定スポットで捕捉一本鎖核酸と結合して二本鎖を形成するターゲット一本鎖核酸はCy−3が標識されたターゲット一本鎖核酸とCy−5が標識されたターゲット一本鎖核酸から構成されており、前者は一定量として存在するが、後者は人間血清で相応する生体分子に比例して存在する。これに伴い特定のスポットは後者が相対的に少量であれば青色、等しい量であれば黄色、そして過量であれば赤色になる。
【0085】
核酸チップ上からスポットの分析で二本鎖内のターゲット一本鎖核酸の数によって蛍光強度が相異であるし、これは生体分子の数と相関関係がある。従って本発明の核酸チップを構成するあらゆるスポットのカラースペクトラムを反映するイメージは未知の生体分子を含む生体試料の生体分子プロファイルである。
【0086】
以上のテスト結果は図3であり、図示したように捕捉一本鎖核酸が付着されたガラススライドで血清蛋白質に結合するターゲット一本鎖核酸に基づいた捕捉一本鎖核酸が付着された部位であるスポットは、多様な蛍光程度、青色−黄色−赤色のカラースペクトルを形成する。
【0087】
図3の結果から人間血清蛋白質に結合するターゲット一本鎖核酸に基づいた核酸チップを利用して人間血清から血清蛋白質のプロファイルを確認することができ、また健康な人(A)及び安定狭心症心血管疾患者(B)の血清内生体分子のプロファイルが相異することを確認することができる。
【0088】
図4は患者の血液試料から本発明の核酸チップを利用して生産されたプロファイルを疾患群別で構成されたデータベース化する過程に対する順序のフロー図である。図5は生産されたプロファイルを疾患群別で構成されたデータベース及び人工神経網を利用したプログラムで特定の人の血清に対して生体分子プロファイルを生産して疾病を診断するフロー図である。
【0089】
図4に示したように、多くの生体試料から生産されるプロファイルに構築されたデータベースを生物情報学技術で分析して有用な情報を確保することができる。
【0090】
健康な人、安定狭心症、不安定狭心症及び心筋梗塞などの心血管患者の血清プロファイルデータベースを利用してLee2−1(99)という人の血清試料を検査した結果は図6の通りであり、72.5% 10 fold cross validationで健康な人と予測される。
【0091】
心血管疾患診断用データベース構築に使われた血清試料の臨床的な資料は下の表1であり、健康な人37人、安定狭心症36人、不安定狭心症27人及び心筋梗塞患者27人等総127名である。
【0092】
【表1】

【0093】
健康な人と肝ガンの血清プロファイルデータベースを利用してKIMという人の血清試料を検査した結果は図7の通りであり、93.0% 10 fold cross validationで肝ガン患者と予測される。
【0094】
追加的に転移有無を検査するために、健康な人、非転移肝ガン及び転移肝ガンの血清蛋白質プロファイルデータベースを利用してKIMという人の血清試料を検査した結果は、図8の通りであり、76.0% 10 fold cross validationで非転移肝ガン患者と予測される。
【0095】
肝ガン診断用データベース構築に使用した血清試料の臨床的な資料は表2であり、健康な人19人、非転移肝ガン患者72人及び転移性肝ガン患者11人で、肝ガン患者は83名で試料の総数は102例である。
【0096】
【表2】

【0097】
健康な人と心筋梗塞患者の血清プロファイルを比較して心筋梗塞患者に特異的に存在するスポットを決定し、これに相応する一本鎖核酸にビオチン(biotin)を付着してストレプトアビジン(streptavidin)と反応させた後、血清試料と反応させて複合体を分離した後、電気泳動して形成されるバンドを分離して生体分子を同定する過程のフロー図は図9である。選別された一本鎖核酸に結合する蛋白質を分離してMALDI−TOF−TOF機器で蛋白質のアミノ酸配列を決定した図面は図10である。
【0098】
上記の方法で生体試料を構成する生体分子と結合する一本鎖核酸に基づいた一本鎖核酸が付着された本発明の核酸チップを利用して特定の生体試料で生体分子のプロファイルを探索及び分析した。
【0099】
また、医学的に有用な生体試料の生体分子を生産してデータベースを製作した後、特定の人の血清プロファイルを生産し、人工神経網アルゴリズムを利用して製作したプログラムで心血管疾患の有無及びその種類、肝ガン発病の有無及び転移程度を検査した。
【0100】
疾患群別で構成されたデータベースを比較分析して有用なスポットを決定してこれに相応する一本鎖核酸を調剤して相応する蛋白質を分離して同定した。
【0101】
[実施例6. 細胞株の表面生体分子のプロファイル生成及び応用]
(6−1.核酸チップ製作)
上記実施例1及び2の方法で生体試料である肺ガン細胞株NCI−H1299の表面生体分子のプロファイルを生産する本発明の核酸チップを製作した。準備した無作為一本鎖核酸を肺ガン細胞株に反応させた後、洗浄バッファーを使用して細胞株(生体分子)−一本鎖核酸複合体を洗浄して非結合一本鎖核酸及び結合程度に従い一本鎖核酸を解離させた後5,000xgで遠心分離する過程を反復的に処理した後、細胞株−一本鎖核酸複合体を遠心分離方法で分離して肺ガン細胞株の表面生体分子と結合する一本鎖核酸を製作した。
【0102】
上記製作した一本鎖核酸からクローンを選定して塩基配列を決定し分析して1,000余個の生体分子結合一本鎖核酸を選定した。実施例2の方法で選別された1,000余個の生体分子結合一本鎖核酸の相補的な塩基配列からなるオリゴヌクレオチド(捕捉一本鎖核酸)を合成して固体基板上に付着させ本発明の核酸チップを製作した。
【0103】
(6−2. 細胞株の表面生体分子プロファイル生成)
上記実施例6−1の方法で製作した核酸チップを利用して肺ガン細胞株NCI−H1299の表面分子のプロファイルを生産した。肺ガン細胞株と一本鎖核酸プールを反応させた後、細胞株(生体試料)の生体分子−一本鎖核酸複合体を洗浄及び分離した。上記複合体に結びついた一本鎖核酸を実施例3の方法で増幅及び標識した。捕捉一本鎖核酸と標識されたターゲット一本鎖核酸を実施例4の方法で反応させた。上記ターゲット一本鎖核酸に標識された物質の測定は実施例5の方法で行ったし肺ガン細胞株の表面生体分子のプロファイルを確認した。
【0104】
生産されたプロファイルで肺ガン細胞株に強力に結合することと推定される一本鎖核酸を選定してFITCが付着された一本鎖核酸を合成して肺ガン細胞株と反応させた後、蛍光撮影したイメージは図11である。
【0105】
このように本発明の核酸チップを利用して生産された肺ガン細胞株プロファイルを分析して選定した一本鎖核酸が肺ガン細胞株と結合するのを確認した。
【0106】
[実施例7. 大腸菌の表面生体分子のプロファイル生成及び応用]
(7−1.核酸チップ製作)
生体試料である大腸菌KCTC12006と上記実施例1及び2の方法で本発明の核酸チップを製作した。
【0107】
準備した一本鎖核酸と大腸菌を反応させた後、洗浄バッファーを使用して大腸菌(生体分子)−一本鎖核酸複合体を洗浄して非結合一本鎖核酸及び結合程度にしたがい一本鎖核酸を解離させた後、5,000xgで遠心分離する過程を反復的に処理した後、大腸菌−一本鎖核酸複合体を遠心分離方法で分離して大腸菌の表面生体分子と結合する一本鎖核酸を製作した。
【0108】
上記製作した一本鎖核酸からクローンを選定して塩基配列を決定し分析して1000余個の生体分子結合一本鎖核酸を選定した。選別された1,000余個の生体分子結合一本鎖核酸の相補的な塩基配列でオリゴヌクレオチド(捕捉一本鎖核酸)を合成し固体基板上に付着させ本発明に係る核酸チップを製作した。
【0109】
[7−2.大腸菌の表面生体分子プロファイル生成]
上記実施例7−1の方法で製作した核酸チップを利用して大腸菌KCTC12006の表面分子のプロファイルを生産した。大腸菌と一本鎖核酸プールを反応させた後大腸菌−ターゲット一本鎖核酸複合体を洗浄及び分離した。上記複合体に結合されたターゲット一本鎖核酸を実施例3の方法で増幅及び標識した。捕捉一本鎖核酸と標識されたターゲット一本鎖核酸を実施例4の方法で反応させた。上記ターゲット一本鎖核酸に標識された物質の測定は実施例5の方法で行い、大腸菌の表面生体分子のプロファイルを確認した。
【0110】
大腸菌KCTC12006の表面生体分子プロファイルを生産する同一方法でサルモネラ菌(Salmonella typhimurium ATCC13311)の表面生体分子プロファイルを生産してデータベースを構築した後、階層クラスタリング方法に基づいて大腸菌に特異的に結合する一本鎖核酸を選定した。一本鎖核酸の特異性をSPR装備(BIAcore社)で測定した結果は図12のようであり、選別された一本鎖核酸がサルモネラ菌に比べて大腸菌に特異的に結合していることを確認した。
【0111】
大腸菌に結合する一本鎖核酸及びナノ金粒子を利用して大腸菌を測定する方法(例、大韓民国特許出願 No.10−2006−0072480;Patent No.10−828936 参照)を用いて、上記で選別された一本鎖核酸を利用して溶液に大腸菌の有無及び飲食物に大腸菌の汚染程度を測定した結果は図13のようである。飲食物を洗浄したSELEX バッファーに一本鎖核酸を反応させナノ金粒子を入れた後、NaCl溶液を添加して、大腸菌のない溶液は透明な無色に近い青色であり、大腸菌に汚染になった溶液は赤色に変化し、その程度は汚染になった大腸菌数に比例するのを確認した。
【産業上の利用可能性】
【0112】
本発明は未知の生体分子と一本鎖核酸の結合プロファイルを生成するための核酸チップ、核酸チップの製造方法、及び核酸チップを利用した未知の生体分子分析方法に関するもので、並列高速分析(High Throughput Screening)を利用した極めて簡単であり、しかも低廉で効率的な方法として、微生物、細胞、蛋白質を含む生体試料に含まれている未知の生体分子に対するプロファイルを生産することができるので、医学、獣医学、環境工学、食品工学、農業などの広範囲な分野で未知の生体分子の生物学的意味を分析する道具として応用できるものと期待される。
【特許請求の範囲】
【請求項1】
未知の生体分子が含まれている生体試料と無作為塩基配列を持つ無作為一本鎖核酸を反応させて前記未知の生体分子と結合する生体分子結合一本鎖核酸を決定する段階と、
決定された前記生体分子結合一本鎖核酸及び/または前記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸を合成して、合成された前記捕捉一本鎖核酸を基板に固着する段階と、
を含む方法で製造され、
生体試料に含まれた未知生体分子の生物学的意味を分析するための、一本鎖核酸と未知生体分子の結合プロファイルを生成するために使われることを特徴とする、核酸チップの製造方法。
【請求項2】
前記生体分子結合一本鎖核酸を決定する段階は、
前記生体試料の前記未知生体分子に前記無作為一本鎖核酸を反応させて生体分子−一本鎖核酸複合体を製造する段階と、
前記生体分子−一本鎖核酸複合体を洗浄し、前記生体分子と前記一本鎖核酸の結合力の大きさに基づいて一定結合力以上の前記生体分子−一本鎖核酸複合体を選択する段階;
選択された前記複合体から前記一本鎖核酸を分離して増幅する段階と、
前記増幅された前記一本鎖核酸をクローニングベクターに挿入して単一クローンを確保して前記一本鎖核酸の塩基配列を決定することによって前記生体分子結合一本鎖核酸を決定する段階と、
を含むことを特徴とする、請求項1に記載の核酸チップの製造方法。
【請求項3】
前記生体分子−一本鎖核酸複合体等の選択及び増幅を反復的に多数回実行することを特徴とする、請求項2に記載の核酸チップの製造方法。
【請求項4】
前記未知生体分子の生物学的意味分析に対する前記捕捉一本鎖核酸の寄与度に基づいて、前記未知生体分子の生物学的意味を分析することができる範囲内で、前記捕捉一本鎖核酸を選別することによって、前記核酸チップに固着する前記捕捉一本鎖核酸の個数を減らせる段階を更に含むことを特徴とする、請求項1〜3のいずれか1項に記載の核酸チップの製造方法。
【請求項5】
前記生体試料は、細菌、真菌、ウイルス、細胞株及び組織からなる群から選択され;
前記生体分子は蛋白質、炭水化物、地質、多糖体、糖蛋白、ホルモン、受容体、抗原、抗体及び酵素からなる群から選択された少なくとも一つ以上であることを特徴とする、請求項1〜請求項3のいずれかの1項記載の核酸チップの製造方法。
【請求項6】
生体試料に含まれた未知の生体分子と結合する生体分子結合一本鎖核酸、及び/または前記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸が基板に固着される構成からなり、生体試料に含まれた未知の生体分子の生物学的意味を分析するための、生体分子と一本鎖核酸の結合プロファイルを生成するのに使われることを特徴とする、核酸チップ。
【請求項7】
生体試料に含まれた未知の生体分子と結合する生体分子結合一本鎖核酸及び/または前記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸が基板に固着されている核酸チップを準備する段階と、
前記核酸チップの前記捕捉一本鎖核酸と同じ塩基配列を持つ一本鎖核酸と前記生体試料の前記未知生体分子を反応させて生体分子−ターゲット一本鎖核酸複合体を形成し、形成された前記生体分子−ターゲット一本鎖核酸複合体を分離する段階と、
分離された前記生体分子−ターゲット一本鎖核酸複合体から前記ターゲット一本鎖核酸を分離、増幅及び標識する段階と、
標識された前記ターゲット一本鎖核酸を前記核酸チップの前記捕捉一本鎖核酸と反応させて前記標識を通して結合プロファイルを獲得する段階と、
獲得された前記プロファイルと既に確保されているプロファイルデータを比較して前記未知の生体分子の生物学的意味を分析する段階と、
を含むことを特徴とする、核酸チップを利用した未知の生体分子分析方法。
【請求項8】
請求項7に記載の核酸チップを利用した未知の生体分子分析方法を通し得られた蓄積された分析結果から、前記未知の生体分子の生物学的意味の分析に寄与する,特異的な一本鎖核酸を決定する方法。
【請求項1】
未知の生体分子が含まれている生体試料と無作為塩基配列を持つ無作為一本鎖核酸を反応させて前記未知の生体分子と結合する生体分子結合一本鎖核酸を決定する段階と、
決定された前記生体分子結合一本鎖核酸及び/または前記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸を合成して、合成された前記捕捉一本鎖核酸を基板に固着する段階と、
を含む方法で製造され、
生体試料に含まれた未知生体分子の生物学的意味を分析するための、一本鎖核酸と未知生体分子の結合プロファイルを生成するために使われることを特徴とする、核酸チップの製造方法。
【請求項2】
前記生体分子結合一本鎖核酸を決定する段階は、
前記生体試料の前記未知生体分子に前記無作為一本鎖核酸を反応させて生体分子−一本鎖核酸複合体を製造する段階と、
前記生体分子−一本鎖核酸複合体を洗浄し、前記生体分子と前記一本鎖核酸の結合力の大きさに基づいて一定結合力以上の前記生体分子−一本鎖核酸複合体を選択する段階;
選択された前記複合体から前記一本鎖核酸を分離して増幅する段階と、
前記増幅された前記一本鎖核酸をクローニングベクターに挿入して単一クローンを確保して前記一本鎖核酸の塩基配列を決定することによって前記生体分子結合一本鎖核酸を決定する段階と、
を含むことを特徴とする、請求項1に記載の核酸チップの製造方法。
【請求項3】
前記生体分子−一本鎖核酸複合体等の選択及び増幅を反復的に多数回実行することを特徴とする、請求項2に記載の核酸チップの製造方法。
【請求項4】
前記未知生体分子の生物学的意味分析に対する前記捕捉一本鎖核酸の寄与度に基づいて、前記未知生体分子の生物学的意味を分析することができる範囲内で、前記捕捉一本鎖核酸を選別することによって、前記核酸チップに固着する前記捕捉一本鎖核酸の個数を減らせる段階を更に含むことを特徴とする、請求項1〜3のいずれか1項に記載の核酸チップの製造方法。
【請求項5】
前記生体試料は、細菌、真菌、ウイルス、細胞株及び組織からなる群から選択され;
前記生体分子は蛋白質、炭水化物、地質、多糖体、糖蛋白、ホルモン、受容体、抗原、抗体及び酵素からなる群から選択された少なくとも一つ以上であることを特徴とする、請求項1〜請求項3のいずれかの1項記載の核酸チップの製造方法。
【請求項6】
生体試料に含まれた未知の生体分子と結合する生体分子結合一本鎖核酸、及び/または前記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸が基板に固着される構成からなり、生体試料に含まれた未知の生体分子の生物学的意味を分析するための、生体分子と一本鎖核酸の結合プロファイルを生成するのに使われることを特徴とする、核酸チップ。
【請求項7】
生体試料に含まれた未知の生体分子と結合する生体分子結合一本鎖核酸及び/または前記生体分子結合一本鎖核酸に相補的な塩基配列を持つ一本鎖核酸を含む捕捉一本鎖核酸が基板に固着されている核酸チップを準備する段階と、
前記核酸チップの前記捕捉一本鎖核酸と同じ塩基配列を持つ一本鎖核酸と前記生体試料の前記未知生体分子を反応させて生体分子−ターゲット一本鎖核酸複合体を形成し、形成された前記生体分子−ターゲット一本鎖核酸複合体を分離する段階と、
分離された前記生体分子−ターゲット一本鎖核酸複合体から前記ターゲット一本鎖核酸を分離、増幅及び標識する段階と、
標識された前記ターゲット一本鎖核酸を前記核酸チップの前記捕捉一本鎖核酸と反応させて前記標識を通して結合プロファイルを獲得する段階と、
獲得された前記プロファイルと既に確保されているプロファイルデータを比較して前記未知の生体分子の生物学的意味を分析する段階と、
を含むことを特徴とする、核酸チップを利用した未知の生体分子分析方法。
【請求項8】
請求項7に記載の核酸チップを利用した未知の生体分子分析方法を通し得られた蓄積された分析結果から、前記未知の生体分子の生物学的意味の分析に寄与する,特異的な一本鎖核酸を決定する方法。
【図1】


【図2】


【図3(A)】


【図3(B)】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】




【図2】


【図3(A)】


【図3(B)】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【公表番号】特表2010−524459(P2010−524459A)
【公表日】平成22年7月22日(2010.7.22)
【国際特許分類】
【出願番号】特願2010−503952(P2010−503952)
【出願日】平成19年6月11日(2007.6.11)
【国際出願番号】PCT/KR2007/002810
【国際公開番号】WO2008/153228
【国際公開日】平成20年12月18日(2008.12.18)
【出願人】(509286710)コリア テクノロジー インダストリー カンパニー リミテッド (1)
【氏名又は名称原語表記】Korea Technology Industry Co.,Ltd.
【住所又は居所原語表記】309 E&C venture dreamtower 3−cha,197−33,Guro−dong,Guro−gu,Seoul152−719
【Fターム(参考)】
【公表日】平成22年7月22日(2010.7.22)
【国際特許分類】
【出願日】平成19年6月11日(2007.6.11)
【国際出願番号】PCT/KR2007/002810
【国際公開番号】WO2008/153228
【国際公開日】平成20年12月18日(2008.12.18)
【出願人】(509286710)コリア テクノロジー インダストリー カンパニー リミテッド (1)
【氏名又は名称原語表記】Korea Technology Industry Co.,Ltd.
【住所又は居所原語表記】309 E&C venture dreamtower 3−cha,197−33,Guro−dong,Guro−gu,Seoul152−719
【Fターム(参考)】
[ Back to top ]

