核酸配列を修復するための方法およびキット
DNA修復のための方法およびキットが提供される。本明細書中に記載される方法およびキットは、複数のタイプのDNA損傷を修復する。キットには、いずれかの単一の酵素が修復することができるものよりもより多くの種類の損傷を修復するための多数の酵素が含まれていてもよい。損傷DNAの修復には、損傷塩基をDNA鎖から除去する工程、損傷部位でDNAにニックを形成する工程、5'-3'エキソヌクレアーゼ活性を介してニックを翻訳する工程、そしてニックをシールする工程、が含まれてもよい。修復プロセスにおいて使用される酵素は、次いで、熱-不活性化することができ、それにより精製プロセスを回避する。次いで、修復DNAを様々なDNA解析方法を使用して解析することができる。
【発明の詳細な説明】
【技術分野】
【0001】
[0001] 本件出願は、米国特許出願12/169,399(2008年7月8日出願)に基づく優先権を主張し、その開示は、その全体を参照により本明細書中に援用する。
【背景技術】
【0002】
[0002] 本発明は、一般的に法医学的解析に関し、そしてより具体的には、損傷核酸配列の修復に関する。
[0003] 犯罪捜査において、捜査官は、しばしば犯罪現場に残された物的証拠を探して、容疑者と犯罪とを結びつける。例えば、物的証拠には、指紋、毛髪、繊維、などが伝統的に含まれてきた。物的証拠の現在の法医学的解析には、デオキシリボ核酸(DNA)試料またはリボ核酸(RNA)試料を含有する血液またはその他の体液などの証拠の解析が含まれてもよい。法医学の研究室は、そのサンプルの持ち主である人物を特定しようとして、DNAサンプルおよび/またはRNAサンプルを解析する。例えば、刑事上の法医学においては、被検体は、犯罪現場に血液またはDNAを含有するその他の証拠を残す場合がある。DNA証拠の由来を決定することにより、政府は、容疑者を起訴するために、法廷でそのような証拠を使用することができる。
【0003】
[0004] 法医学の研究室で行われるDNA解析は、制御不能な環境条件に曝されたDNAサンプルにより妨げられる可能性がある。環境的に損傷されたサンプルは、標準的な解析方法を反映したものである短縮されたDNAフラグメント(すなわち、DNA中の二本鎖切断部)を含有していてもよく、または一本鎖ニックおよび損傷を有するいくらか無傷のDNAを含有していてもよい。損傷を生じる曝露のいくつかの事例には、失われた塩基および/または脱プリンおよび脱ピリミジン部位(AP部位)を生じる酸への曝露そして熱への曝露;塩基または糖バックボーンを修飾する、電離放射線およびアルキル化剤を含む求電子試薬;塩基の改変を生じる、酸化損傷;そしてシクロブタンダイマーを生成する、UV照射;が含まれる。そのような損傷の事象において、DNAサンプルはしばしば効果的に解析することができず、そのため刑事訴追における証拠として使用することができない。
【発明の概要】
【0004】
[0005] 特定の態様において、5'-3'エキソヌクレアーゼ活性を有する熱に不安定なDNAポリメラーゼおよび熱に不安定なDNAリガーゼを含む、DNA修復キットが提供される。
[0006] その他の態様において、N-グリコシラーゼ活性、AP-リアーゼ活性、5'-3' DNAポリメラーゼ活性、および5'-3'エキソヌクレアーゼニックトランスレーション活性を示す、酵素の組合せを有するDNA修復キットが提供される。DNA酵素の組合せは、熱-不活性化可能なものであってもよい。
【0005】
[0007] DNAグリコシラーゼ、DNAエンドヌクレアーゼ、DNAポリメラーゼI、およびT4 DNAリガーゼを含むDNA修復キットもまた、提供される。
[0008] さらなる態様は、損傷DNAを、DNA修復酵素ブレンドと、第1の温度でインキュベーションして、修復DNAを生成する工程を含む、方法を提供する。DNA修復酵素ブレンドには、5'-3'エキソヌクレアーゼ活性を有する熱に不安定なDNAポリメラーゼおよび熱に不安定なDNAリガーゼが含まれていてもよい。
【0006】
[0009] さらにさらなる態様は、グリコシラーゼを介してDNA鎖上の損傷部位から塩基を除去する工程、エンドヌクレアーゼを介して損傷部位でのDNA鎖にニックを形成する工程、関連する5'-3'エキソヌクレアーゼ活性を有するDNAポリメラーゼを介してDNA鎖の下流に向けてニックを翻訳する工程、そしてニックをリガーゼを用いてシールする工程、を含む方法を提供する。
【図面の簡単な説明】
【0007】
[0010] 本発明のこれらのそしてその他の特徴、観点、そして利点は、図面を通じて同様の特徴は同様の部分を示す、添付する図面を参照して以下の詳細な説明を読む場合に、よりよく理解されるであろう。
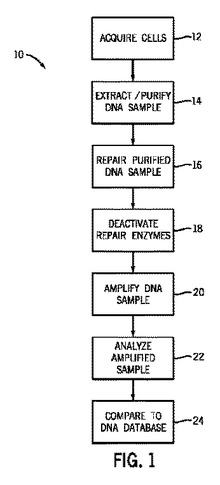
【図1】[0011] 図1は、本発明の技術の観点に従ってDNAを解析するための方法のフローチャートである。
【図2】[0012] 図2は、本発明の技術の観点に従って損傷DNAを修復するための方法の、ダイアグラム的な説明である。
【図3】[0013] 図3は、非損傷DNAを使用して実施したゲルの画像である。
【図4】[0014] 図4は、酸化的損傷DNAを使用して実施したゲルの画像である。
【図5】[0015] 図5は、修復DNAを使用して実施したゲルの画像である。
【図6】[0016] 図6は、UV損傷DNAを使用して実施したゲルの画像である。
【図7】[0017] 図7は、修復DNAを使用して実施したゲルの画像である。
【発明を実施するための形態】
【0008】
[0018] 損傷DNAは、細胞の標準的DNA複製機構に対するブロックであるDNA中の、ヌクレオチド修飾および/または欠損(一本鎖または二本鎖)を含有していてもよい。損傷核酸の修復経路は、4つのカテゴリーに分類することができる:ニックをシールするためのライゲーション;ヌクレオチド修飾の正常状態への直接逆転;鋳型として第2のDNAコピーを使用する組換え修復;そして除去修復。本明細書において記載された方法は、酵素を使用して、1またはそれ以上のこれらの修復技術を行い、損傷DNAを修復することができる。本明細書において記載されるDNA修復についての方法は、例えば、病原性生物の検出、法医学的目的、医学的診断目的、または臨床目的のために、使用することができる。解析用途、診断用途、または予後用途に適用できる方法に一般に関連する態様もまた、提供される。
【0009】
[0019] 図1は、例示的なプロセス10を説明する。この方法では、DNA修復のために本明細書において記載される方法を、損傷DNAを解析する際に効果的に使用することができる。DNAを含有する細胞を、様々な供給源から取得することができる(ブロック12)。DNAを含有することが疑われるサンプルまたはDNAを含有することが知られているサンプルを、様々な供給源、例えば、犯罪現場から採取された血液サンプルまたは精液サンプルなど、から得ることができる。サンプルは、生物学的サンプル、食品サンプルまたは農業サンプル、または環境サンプルであってもよいが、これらには限定されない。この様なサンプルは、生体組織、体液、または滲出物(例えば、血液、血漿、血清または尿、乳、脳脊髄液、胸膜液、リンパ、涙、つば、唾液、糞、肺吸引物、喉ぬぐい液または生殖器ぬぐい液、など);全細胞;細胞画分;または培養物;に由来するものであってもよい。特定の状況において、サンプルを処理して、DNAを放出しおよび/またはDNAを抽出することが好ましい。精製DNAサンプルは、DNAを変性させることなく(例えば、煮沸したりあるいは水酸化カリウムを使用したりせずに)細胞からDNAを穏やかに抽出することにより、取得することができる(ブロック14)。供給源細胞からのDNAの効果的な抽出および精製のためのどの様な方法を使用してもよい。DNA精製のための例示的な方法は、illustra組織および細胞ゲノムPrep Midi Flowキット(カタログNo. 28-9042-73、GE Healthcareより入手可能)を使用してもよい。
【0010】
[0020] 次いで、抽出DNAを、本明細書において記載される方法により修復することができる(ブロック16)。DNA修復のための方法は、以下にさらに詳細に記載される。示されるように、修復方法は、DNAを変性することなく容易に不活性化することができる特定の酵素を使用することができる(ブロック18)。従って、第2の精製工程は、特定の態様において必要とされなくてもよい。次いで、修復DNAサンプルを、等温性増幅技術を使用するなどにより増幅することができ(ブロック20)、サンプル量を増加させそして解析を容易にする。例えば、修復DNAを、illustra GenomiPhi DNA増幅キット(カタログNo. 25-6600、GE Healthcareから入手可能)などの全ゲノム増幅キットを使用して、またはillustra TempliPhiTM増幅キット(カタログNo. 25-6400、GE Healthcareから入手可能)などの循環増幅(circular amplification)キットを使用して、増幅することができる。次いで、様々な解析方法を使用して、DNAサンプルを個体に適合させることができる(ブロック22)。例えば、ポリメラーゼ連鎖反応(PCR)増幅および解析を使用して、塩基対が独自に繰り返すことが知られている13箇所のコア遺伝子座のそれぞれでのミニサテライトの繰り返し数を決定することができる。例えば、増幅断片長多型(AmpFLP)解析、短鎖縦列型リピート(STR)解析、またはY-染色体(Y-STR)解析などの、その他の解析技術を使用することができる。例示的な定量解析技術は、QuantifilerTMヒトDNA定量キット(Part No. 4343895、Applied Biosystemsから入手可能)を使用することができる。解析は、固有のコードを生成し、それを既知のDNAサンプルのデータベース(例えば、連邦捜査局(FBI)により維持管理される統合DNAインデックス・システム(Combined DNA Index System、CODIS))と比較することができる(ブロック24)。
【0011】
[0021] DNA修復(ブロック16)のため、使用することができる別個のクラスのタンパク質酵素が存在する。酵素の第1のクラスは、損傷を直接的に修復する。このクラスには、二本鎖鋳型中の一本鎖ニックをシールするリガーゼ、および特定の光-誘導性損傷の除去を触媒するフォトリアーゼが含まれる。DNA修復のために使用することができる酵素の第2のクラスは、塩基-損傷ヌクレオチドを特定し、そしてヌクレオチドの塩基部分を除去し(例えば、グリコシラーゼ活性)、無傷の糖バックボーンを残すことにより、機能する。糖-のみのバックボーンのこの領域は、脱プリン部位または脱ピリミジン(AP)部位と呼ばれ、そしてそれ自体が複製または増幅に対するブロックである。酵素の次のクラスには、損傷塩基を特定しそして損傷部位の隣のDNAバックボーンにニックを形成するエンドヌクレアーゼが含まれる。In vivoでは、AP部位を、APエンドヌクレアーゼにより除去することができる。いくつかの場合においては、グリコシラーゼ活性は、APエンドヌクレアーゼと関連しており、そして両方の工程が1つの酵素により達成される。この除去の生成物は、AP部位が依然として鎖に付随しているニックが形成されたDNA、またはAP部位が除去された一塩基ギャップのいずれかであってもよい。いずれかの生成物は、複製または増幅に対するブロックである。
【0012】
[0022] 最終的に、ニックトランスレーション酵素もまた、DNA修復のために使用することができる。ニックトランスレーションプロセスにおいて、DNAを複製しかつ同時にブロッキングする一本鎖DNAを除去する活性(例えば、5'-3' DNAポリメラーゼ活性および5'-3'エキソヌクレアーゼ活性)を有するDNAポリメラーゼを使用して、DNA鎖を下流に向けて移動することができ、その前で損傷塩基を本質的に除去する一方で非損傷版を合成する。ニックトランスレーションのプロセスについて、5'-3'エキソヌクレアーゼと機能的に協調する内在性の5'-3'ポリメラーゼ活性を有するいずれかのDNAポリメラーゼを、使用することができる。古典的には、この協調は、E. coli DNAポリメラーゼIを使用して利用可能である;しかしながら、二重鎖DNAの隣接する5'リン酸末端と3'ヒドロキシ末端との間にホスホジエステル結合の形成を触媒する、T4 DNAリガーゼ、T7 DNAリガーゼ、そしてバクテリアDNAリガーゼを含むいずれのDNAリガーゼを使用することができる。
【0013】
[0023] E. coli DNAポリメラーゼIによるニックトランスレーションは、ポリメラーゼ活性によりDNAを追加しつつ、同時的に酵素より早くDNAを除去することにより、達成される。このプロセスの間、DNAの短い部分がニックトランスレーションにより置換された後、ニックトランスレーション酵素が解離し、リガーゼを用いてシールすることができるニックが残される。このプロセスは、DNAポリメラーゼがDNA鎖の下流に向けてニックを翻訳することから、ニックトランスレーションと呼ばれる。ニックトランスレーションのプロセスを使用して、ニックの5'側に位置する損傷DNAを除去することができ、同時に損傷ヌクレオチドを非損傷版により置換することができる。5'-3'エキソヌクレアーゼ活性がこの修復プロセスにおいて好ましいものであるため、クレノーフラグメント(すなわち、DNAポリメラーゼIの大きなフラグメント)をそのような態様における唯一のポリメラーゼとして使用しなくてもよい。
【0014】
[0024] ニックトランスレーションとリガーゼ酵素との両方を同時に使用することにより、短いパッチ修復を完了させることができる。リガーゼなしでニックトランスレーションのみにより、二本鎖欠損を生じる可能性がある。両鎖上に損傷を含有するDNAは、2つの相対するニックトランスレーション部位を含有することができる。これが生じる場合、反応生成物は、二本鎖DNA切断を含有する2つのより短いDNA鎖であってもよいが、それは一般的に望ましくない。ニックトランスレーション反応においてリガーゼが含まれることにより、二本鎖切断を阻害することができる。
【0015】
[0025] いくつかの態様において、修復酵素は、熱に不安定であり、そしてサンプルを40〜80℃に加熱することにより永久的に不活性化することができ、それによりそれらの活性が適切ではないと考えられるその後の取り扱い工程の間、酵素が機能し続けない様にする。タンパク質が温度の上昇に曝される場合、溶解度の喪失または酵素活性の喪失が、極めて狭い範囲で生じる可能性がある。研究されたタンパク質そして加熱の程度に依存して、これらの変化は、可逆的ではない場合がある。タンパク質酵素の三次構造が破壊されるため、疎水性基が、酵素が配置される溶媒に対して曝される。次いで、タンパク質は、できるだけ多くの極性基を溶媒に曝露しつつ、できるだけ多くの疎水性基を被覆することにより、その自由エネルギーを最小化するよう試みることができる。このことはタンパク質が折り畳まれる時に一般的に生じる事象と類似しているが、ずっと高い温度がタンパク質折り畳みを最初に方向付ける短期間の相互作用を大幅に弱める可能性があり、そして得られた構造は天然のタンパク質とは大幅に異なる可能性がある。この様に、多数のタンパク質を高温へ曝露することにより、不可逆的な変性が生じる可能性がある。
【0016】
[0026] 酵素の組合せを使用して、異なる種類のDNA損傷を同時的に修復することができる。例えば、ある酵素は特定の種類の損傷を修復する際に効果的である場合がある一方、別の酵素は異なる損傷を修復するために好ましい。いくつかの酵素を1つのDNA修復キット中に組み合わせることにより、より頑強な修復プロセスを実施することができる。記載される機能を行うために使用することができる例示的な酵素を、それぞれの酵素により修復される損傷、およびそれぞれの酵素により行われる活性とともに、表1に列挙する。
【0017】
【表1】

【0018】
[0027] 図2を参照して、本発明の技術に従う損傷DNAの修復の説明が、記載される。DNA鎖50は、1またはそれ以上の損傷(例えば、増幅-阻害性)部位52および54を有していてもよい。酵素の組合せを使用して、DNA鎖50に対していくつかの修復機能を行うことができる。この組合せには、例えば、グリコシラーゼ(例えば、FPG)および/またはエンドヌクレアーゼ(例えば、エンドヌクレアーゼIV、エンドヌクレアーゼVII)が含まれていてもよく、これにより損傷塩基部分52を除去しそしてAP部位56を残す。組合せには、損傷部位52および54および/またはAP部位56にてDNAにニックを形成するエンドヌクレアーゼ(例えば、T4エンドヌクレアーゼV)がさらに含まれていてもよく、ニック58および60が残る。次いで、組合せ中に含まれるDNAポリメラーゼ(例えば、E. coli DNAポリメラーゼI)は、DNA鎖50の下流方向にニック58および60を翻訳することができ、ニック58および60が翻訳されると同時に損傷DNAを修復する。ニックトランスレーションは、5'-3'エキソヌクレアーゼ活性を介して、達成することができる。最終的に、ニック58および60を、組合せ中に存在するリガーゼ(例えば、T4 DNAリガーゼ)を用いてシールすることができる。
【0019】
[0028] 図2を参照して記載される例示的な技術を、単一混合物または複数混合物中の複数の酵素の組合せを介して、行うことができることを理解すべきである。例示的なDNA修復キットには、表2に記載される酵素混合物が含まれていてもよい。
【0020】
【表2】

【0021】
[0029] 修復バッファ溶液の10×調製物には、例えば、100 mM Tris-HCl、100 mM MgCl2、500 mM NaCl、そして10 mMジチオスレイトールが含まれていてもよく、pH 7.9になる。表2中に記載された混合物中の酵素、ならびにその他の酵素、単一の修復溶液中で損傷DNAに対して同時に適用することができる。例えば、損傷DNAの修復を、表3中に記載される混合物から構成される修復溶液中でのインキュベーションを介して行うことができる。
【0022】
【表3】

【0023】
[0030] 従って、例示的なDNA修復キットは、事前混合された溶液中で、または混合のために利用可能な別個の成分として、表3中に記載されたいくつかのまたは全ての成分を含有することができる。約15μlの損傷DNAを、表3に記載される修復溶液と組み合わせ、そして約20〜42℃にて25〜60分間インキュベーションし、その後42〜75℃でさらに15〜25分間インキュベーションすることができる。最初のインキュベーション期間により損傷DNAの修復を可能にし、一方より高い温度のインキュベーションにより酵素が不活性化する。例示的な態様において、修復酵素の不活性化を、約65℃で約15分間、修復DNAと酵素混合物とをインキュベーションすることにより行うことができる。この高い温度でのインキュベーションは、修復DNAを変性することなく、熱に不安定なDNA修復酵素(リガーゼを含む)を不活性化する。従って、熱に安定なDNAポリメラーゼ(例えば、Bst DNAポリメラーゼ)および/または熱に安定なDNAリガーゼ(例えば、Taq DNAリガーゼ)は好ましくなく、そして、特定の態様において、本発明のプロセスにおいて使用することができない。さらに、特定の酵素または全ての酵素が熱-不活性化可能なものであってもよいため、増幅の前に修復DNAの精製をすることが必要ではなくてもよい。
【0024】
[0031] 表3に記載される修復溶液には、リボアデノシン三リン酸(リボ-ATP)が含まれる一方、この成分は実施する上で除去することができることに注意すべきである。さらに、例示的なキット中の酵素のどれも、機能するためにβ-ニコチンアミドアデニンジヌクレオチド(NAD)を必要とせず、そして従ってその存在は必要ではない。
【0025】
[0032] ひとたび損傷DNAが本明細書で記載される技術に従って修復されると、修復DNAを増幅しそして解析して、例えば、DNAが由来する被検体の同一性を決定することができる。上述した様に、PCR、AmpFLP、STR、Y-STR、などの多数の解析技術を使用することができる。熱-不活性化酵素は、DNAの解析に影響を及ぼさず、そして従って増幅および解析を行う前に修復DNAから除去する必要はない。
【0026】
実験結果
[0033] ゲノムDNAを酸化損傷および紫外線損傷に供し、その後本発明の技術の態様に従って修復した。結果を以下にまとめる。実験において使用するためのゲノムDNAは、HT29細胞株から約80〜160 ng/μlの濃度で抽出した。図3は、非損傷DNAのスライドである。
【0027】
[0034] DNAを損傷するため、9 mM塩化鉄(FeCl3)および18 mM EDTAであるFe-EDTA溶液、約100μlを、0.37 M FeCl3および水中に溶解した0.5 EDTAを使用して調製した。さらに、約1 mlの30 mM過酸化水素(H2O2)溶液を、約3.4μlのストックH2O2(すなわち、約8.8 Mで30%H2O2)を1 mlの水に添加することにより調製した。H2O2溶液を氷上に置いた。
【0028】
[0035] 表4に記載される反応混合物を、チューブ中で調製した。全反応容量は約30μlであった。H2O2溶液を添加して損傷反応を開始した。混合物を、37℃で様々な時間(例えば、20〜100分)インキュベーションし、次いでTEで平衡化したNAP-5脱塩カラム(カタログNo. 17-0853-01、GE Healthcareから入手可能)を使用して脱塩した。次いで、混合物を、1 ml TE中に溶出した。
【0029】
【表4】

【0030】
[0036] 酸化損傷DNAサンプルをFenton反応を使用して調製した。HT-29細胞株から抽出した約130 ng/μlの濃度のゲノムDNA、約2μlを、5μlのFe-EDTA溶液および23μlの二重蒸留水と混合した。約4μlの30mM H2O2溶液を添加して、反応を開始した。反応混合物の8本のチューブを調製し、そして37℃で80分間インキュベーションした。次いで、混合物をNAP-5脱塩カラムを使用して脱塩し、そして1 mlのTE中で溶出し、それにより得られた損傷DNA濃度は約2 ng/μlであった。図4は、酸化損傷DNAのスライドである。
【0031】
[0037] 次いで、酸化損傷DNAを、本発明の技術の態様に従って、単一チューブ内に複数の修復酵素を含有する修復混合物と組み合わせた。修復混合物の成分を、表5にまとめる。修復混合物を、37℃で約30分間、そしてその後65℃で約20分間、インキュベーションした。
【0032】
【表5】

【0033】
[0038] インキュベーションの後、修復DNAを含有する混合物に対して、PCR増幅を行った。PCR増幅を、表6に列挙した成分を使用して行った。PCRビーズは、GE Healthcareから入手可能である(カタログNo. 27-9558-01)。PCR増幅を、95℃で5分間;95℃で30秒間、58.5℃で30秒間、そして72℃で90秒間のサイクルを38サイクル;そして72℃で10分間;で行った。図5は、修復DNAのスライドである。これらの結果に基づいて、本明細書において開示された技術の態様(図5)を使用して修復したDNAは、酸化損傷DNA(図4)よりも、非損傷対照DNA(図3)により近いようである。
【0034】
【表6】

【0035】
[0039] 酸化損傷サンプルに加えて、UV損傷サンプルを調製した。HT-29細胞株から抽出した約130 ng/μlの濃度のゲノムDNA、約100μlを、石英キュベット中に入れ、そして約245 nmおよび4 mW/cm2で5分間、UVCエネルギーに対して曝露した。次いで、UV損傷DNAを、約2 ng/μlにまで希釈した。図6は、UV損傷DNAのスライドである。
【0036】
[0040] 次いで、UV損傷DNAを、本発明の技術の態様に従って、単一チューブ内に複数の修復酵素を含有する修復混合物と組み合わせた。修復混合物の成分は、表7にまとめる。修復混合物を、37℃で約30分間、そしてその後65℃で約20分間、インキュベーションした。
【0037】
【表7】

【0038】
[0041] インキュベーションの後、修復DNAを含有する混合物に対して、上述した様にPCR増幅を行った。図7は、修復DNAのスライドである。これらの結果に基づいて、本明細書において開示された技術の態様(図7)を使用して修復したDNAは、UV損傷DNA(図6)よりも、非損傷対照DNA(図3)により近いようである。
【0039】
[0042] 上述した様に、2 ngのDNA、損傷DNA、または修復DNAを、表8に示すプライマーセットを使用して、上述したPCRにより増幅した。
【0040】
【表8】

【0041】
[0043] 本明細書中では、本発明の特定の態様のみを説明しそして記載したが、多数の修飾および変更が当業者には思い浮かぶであろう。従って、添付の請求の範囲が本発明の真の概念に含まれる全てのそのような修飾および変更を含むことを意図していることを理解すべきである。
【技術分野】
【0001】
[0001] 本件出願は、米国特許出願12/169,399(2008年7月8日出願)に基づく優先権を主張し、その開示は、その全体を参照により本明細書中に援用する。
【背景技術】
【0002】
[0002] 本発明は、一般的に法医学的解析に関し、そしてより具体的には、損傷核酸配列の修復に関する。
[0003] 犯罪捜査において、捜査官は、しばしば犯罪現場に残された物的証拠を探して、容疑者と犯罪とを結びつける。例えば、物的証拠には、指紋、毛髪、繊維、などが伝統的に含まれてきた。物的証拠の現在の法医学的解析には、デオキシリボ核酸(DNA)試料またはリボ核酸(RNA)試料を含有する血液またはその他の体液などの証拠の解析が含まれてもよい。法医学の研究室は、そのサンプルの持ち主である人物を特定しようとして、DNAサンプルおよび/またはRNAサンプルを解析する。例えば、刑事上の法医学においては、被検体は、犯罪現場に血液またはDNAを含有するその他の証拠を残す場合がある。DNA証拠の由来を決定することにより、政府は、容疑者を起訴するために、法廷でそのような証拠を使用することができる。
【0003】
[0004] 法医学の研究室で行われるDNA解析は、制御不能な環境条件に曝されたDNAサンプルにより妨げられる可能性がある。環境的に損傷されたサンプルは、標準的な解析方法を反映したものである短縮されたDNAフラグメント(すなわち、DNA中の二本鎖切断部)を含有していてもよく、または一本鎖ニックおよび損傷を有するいくらか無傷のDNAを含有していてもよい。損傷を生じる曝露のいくつかの事例には、失われた塩基および/または脱プリンおよび脱ピリミジン部位(AP部位)を生じる酸への曝露そして熱への曝露;塩基または糖バックボーンを修飾する、電離放射線およびアルキル化剤を含む求電子試薬;塩基の改変を生じる、酸化損傷;そしてシクロブタンダイマーを生成する、UV照射;が含まれる。そのような損傷の事象において、DNAサンプルはしばしば効果的に解析することができず、そのため刑事訴追における証拠として使用することができない。
【発明の概要】
【0004】
[0005] 特定の態様において、5'-3'エキソヌクレアーゼ活性を有する熱に不安定なDNAポリメラーゼおよび熱に不安定なDNAリガーゼを含む、DNA修復キットが提供される。
[0006] その他の態様において、N-グリコシラーゼ活性、AP-リアーゼ活性、5'-3' DNAポリメラーゼ活性、および5'-3'エキソヌクレアーゼニックトランスレーション活性を示す、酵素の組合せを有するDNA修復キットが提供される。DNA酵素の組合せは、熱-不活性化可能なものであってもよい。
【0005】
[0007] DNAグリコシラーゼ、DNAエンドヌクレアーゼ、DNAポリメラーゼI、およびT4 DNAリガーゼを含むDNA修復キットもまた、提供される。
[0008] さらなる態様は、損傷DNAを、DNA修復酵素ブレンドと、第1の温度でインキュベーションして、修復DNAを生成する工程を含む、方法を提供する。DNA修復酵素ブレンドには、5'-3'エキソヌクレアーゼ活性を有する熱に不安定なDNAポリメラーゼおよび熱に不安定なDNAリガーゼが含まれていてもよい。
【0006】
[0009] さらにさらなる態様は、グリコシラーゼを介してDNA鎖上の損傷部位から塩基を除去する工程、エンドヌクレアーゼを介して損傷部位でのDNA鎖にニックを形成する工程、関連する5'-3'エキソヌクレアーゼ活性を有するDNAポリメラーゼを介してDNA鎖の下流に向けてニックを翻訳する工程、そしてニックをリガーゼを用いてシールする工程、を含む方法を提供する。
【図面の簡単な説明】
【0007】
[0010] 本発明のこれらのそしてその他の特徴、観点、そして利点は、図面を通じて同様の特徴は同様の部分を示す、添付する図面を参照して以下の詳細な説明を読む場合に、よりよく理解されるであろう。
【図1】[0011] 図1は、本発明の技術の観点に従ってDNAを解析するための方法のフローチャートである。
【図2】[0012] 図2は、本発明の技術の観点に従って損傷DNAを修復するための方法の、ダイアグラム的な説明である。
【図3】[0013] 図3は、非損傷DNAを使用して実施したゲルの画像である。
【図4】[0014] 図4は、酸化的損傷DNAを使用して実施したゲルの画像である。
【図5】[0015] 図5は、修復DNAを使用して実施したゲルの画像である。
【図6】[0016] 図6は、UV損傷DNAを使用して実施したゲルの画像である。
【図7】[0017] 図7は、修復DNAを使用して実施したゲルの画像である。
【発明を実施するための形態】
【0008】
[0018] 損傷DNAは、細胞の標準的DNA複製機構に対するブロックであるDNA中の、ヌクレオチド修飾および/または欠損(一本鎖または二本鎖)を含有していてもよい。損傷核酸の修復経路は、4つのカテゴリーに分類することができる:ニックをシールするためのライゲーション;ヌクレオチド修飾の正常状態への直接逆転;鋳型として第2のDNAコピーを使用する組換え修復;そして除去修復。本明細書において記載された方法は、酵素を使用して、1またはそれ以上のこれらの修復技術を行い、損傷DNAを修復することができる。本明細書において記載されるDNA修復についての方法は、例えば、病原性生物の検出、法医学的目的、医学的診断目的、または臨床目的のために、使用することができる。解析用途、診断用途、または予後用途に適用できる方法に一般に関連する態様もまた、提供される。
【0009】
[0019] 図1は、例示的なプロセス10を説明する。この方法では、DNA修復のために本明細書において記載される方法を、損傷DNAを解析する際に効果的に使用することができる。DNAを含有する細胞を、様々な供給源から取得することができる(ブロック12)。DNAを含有することが疑われるサンプルまたはDNAを含有することが知られているサンプルを、様々な供給源、例えば、犯罪現場から採取された血液サンプルまたは精液サンプルなど、から得ることができる。サンプルは、生物学的サンプル、食品サンプルまたは農業サンプル、または環境サンプルであってもよいが、これらには限定されない。この様なサンプルは、生体組織、体液、または滲出物(例えば、血液、血漿、血清または尿、乳、脳脊髄液、胸膜液、リンパ、涙、つば、唾液、糞、肺吸引物、喉ぬぐい液または生殖器ぬぐい液、など);全細胞;細胞画分;または培養物;に由来するものであってもよい。特定の状況において、サンプルを処理して、DNAを放出しおよび/またはDNAを抽出することが好ましい。精製DNAサンプルは、DNAを変性させることなく(例えば、煮沸したりあるいは水酸化カリウムを使用したりせずに)細胞からDNAを穏やかに抽出することにより、取得することができる(ブロック14)。供給源細胞からのDNAの効果的な抽出および精製のためのどの様な方法を使用してもよい。DNA精製のための例示的な方法は、illustra組織および細胞ゲノムPrep Midi Flowキット(カタログNo. 28-9042-73、GE Healthcareより入手可能)を使用してもよい。
【0010】
[0020] 次いで、抽出DNAを、本明細書において記載される方法により修復することができる(ブロック16)。DNA修復のための方法は、以下にさらに詳細に記載される。示されるように、修復方法は、DNAを変性することなく容易に不活性化することができる特定の酵素を使用することができる(ブロック18)。従って、第2の精製工程は、特定の態様において必要とされなくてもよい。次いで、修復DNAサンプルを、等温性増幅技術を使用するなどにより増幅することができ(ブロック20)、サンプル量を増加させそして解析を容易にする。例えば、修復DNAを、illustra GenomiPhi DNA増幅キット(カタログNo. 25-6600、GE Healthcareから入手可能)などの全ゲノム増幅キットを使用して、またはillustra TempliPhiTM増幅キット(カタログNo. 25-6400、GE Healthcareから入手可能)などの循環増幅(circular amplification)キットを使用して、増幅することができる。次いで、様々な解析方法を使用して、DNAサンプルを個体に適合させることができる(ブロック22)。例えば、ポリメラーゼ連鎖反応(PCR)増幅および解析を使用して、塩基対が独自に繰り返すことが知られている13箇所のコア遺伝子座のそれぞれでのミニサテライトの繰り返し数を決定することができる。例えば、増幅断片長多型(AmpFLP)解析、短鎖縦列型リピート(STR)解析、またはY-染色体(Y-STR)解析などの、その他の解析技術を使用することができる。例示的な定量解析技術は、QuantifilerTMヒトDNA定量キット(Part No. 4343895、Applied Biosystemsから入手可能)を使用することができる。解析は、固有のコードを生成し、それを既知のDNAサンプルのデータベース(例えば、連邦捜査局(FBI)により維持管理される統合DNAインデックス・システム(Combined DNA Index System、CODIS))と比較することができる(ブロック24)。
【0011】
[0021] DNA修復(ブロック16)のため、使用することができる別個のクラスのタンパク質酵素が存在する。酵素の第1のクラスは、損傷を直接的に修復する。このクラスには、二本鎖鋳型中の一本鎖ニックをシールするリガーゼ、および特定の光-誘導性損傷の除去を触媒するフォトリアーゼが含まれる。DNA修復のために使用することができる酵素の第2のクラスは、塩基-損傷ヌクレオチドを特定し、そしてヌクレオチドの塩基部分を除去し(例えば、グリコシラーゼ活性)、無傷の糖バックボーンを残すことにより、機能する。糖-のみのバックボーンのこの領域は、脱プリン部位または脱ピリミジン(AP)部位と呼ばれ、そしてそれ自体が複製または増幅に対するブロックである。酵素の次のクラスには、損傷塩基を特定しそして損傷部位の隣のDNAバックボーンにニックを形成するエンドヌクレアーゼが含まれる。In vivoでは、AP部位を、APエンドヌクレアーゼにより除去することができる。いくつかの場合においては、グリコシラーゼ活性は、APエンドヌクレアーゼと関連しており、そして両方の工程が1つの酵素により達成される。この除去の生成物は、AP部位が依然として鎖に付随しているニックが形成されたDNA、またはAP部位が除去された一塩基ギャップのいずれかであってもよい。いずれかの生成物は、複製または増幅に対するブロックである。
【0012】
[0022] 最終的に、ニックトランスレーション酵素もまた、DNA修復のために使用することができる。ニックトランスレーションプロセスにおいて、DNAを複製しかつ同時にブロッキングする一本鎖DNAを除去する活性(例えば、5'-3' DNAポリメラーゼ活性および5'-3'エキソヌクレアーゼ活性)を有するDNAポリメラーゼを使用して、DNA鎖を下流に向けて移動することができ、その前で損傷塩基を本質的に除去する一方で非損傷版を合成する。ニックトランスレーションのプロセスについて、5'-3'エキソヌクレアーゼと機能的に協調する内在性の5'-3'ポリメラーゼ活性を有するいずれかのDNAポリメラーゼを、使用することができる。古典的には、この協調は、E. coli DNAポリメラーゼIを使用して利用可能である;しかしながら、二重鎖DNAの隣接する5'リン酸末端と3'ヒドロキシ末端との間にホスホジエステル結合の形成を触媒する、T4 DNAリガーゼ、T7 DNAリガーゼ、そしてバクテリアDNAリガーゼを含むいずれのDNAリガーゼを使用することができる。
【0013】
[0023] E. coli DNAポリメラーゼIによるニックトランスレーションは、ポリメラーゼ活性によりDNAを追加しつつ、同時的に酵素より早くDNAを除去することにより、達成される。このプロセスの間、DNAの短い部分がニックトランスレーションにより置換された後、ニックトランスレーション酵素が解離し、リガーゼを用いてシールすることができるニックが残される。このプロセスは、DNAポリメラーゼがDNA鎖の下流に向けてニックを翻訳することから、ニックトランスレーションと呼ばれる。ニックトランスレーションのプロセスを使用して、ニックの5'側に位置する損傷DNAを除去することができ、同時に損傷ヌクレオチドを非損傷版により置換することができる。5'-3'エキソヌクレアーゼ活性がこの修復プロセスにおいて好ましいものであるため、クレノーフラグメント(すなわち、DNAポリメラーゼIの大きなフラグメント)をそのような態様における唯一のポリメラーゼとして使用しなくてもよい。
【0014】
[0024] ニックトランスレーションとリガーゼ酵素との両方を同時に使用することにより、短いパッチ修復を完了させることができる。リガーゼなしでニックトランスレーションのみにより、二本鎖欠損を生じる可能性がある。両鎖上に損傷を含有するDNAは、2つの相対するニックトランスレーション部位を含有することができる。これが生じる場合、反応生成物は、二本鎖DNA切断を含有する2つのより短いDNA鎖であってもよいが、それは一般的に望ましくない。ニックトランスレーション反応においてリガーゼが含まれることにより、二本鎖切断を阻害することができる。
【0015】
[0025] いくつかの態様において、修復酵素は、熱に不安定であり、そしてサンプルを40〜80℃に加熱することにより永久的に不活性化することができ、それによりそれらの活性が適切ではないと考えられるその後の取り扱い工程の間、酵素が機能し続けない様にする。タンパク質が温度の上昇に曝される場合、溶解度の喪失または酵素活性の喪失が、極めて狭い範囲で生じる可能性がある。研究されたタンパク質そして加熱の程度に依存して、これらの変化は、可逆的ではない場合がある。タンパク質酵素の三次構造が破壊されるため、疎水性基が、酵素が配置される溶媒に対して曝される。次いで、タンパク質は、できるだけ多くの極性基を溶媒に曝露しつつ、できるだけ多くの疎水性基を被覆することにより、その自由エネルギーを最小化するよう試みることができる。このことはタンパク質が折り畳まれる時に一般的に生じる事象と類似しているが、ずっと高い温度がタンパク質折り畳みを最初に方向付ける短期間の相互作用を大幅に弱める可能性があり、そして得られた構造は天然のタンパク質とは大幅に異なる可能性がある。この様に、多数のタンパク質を高温へ曝露することにより、不可逆的な変性が生じる可能性がある。
【0016】
[0026] 酵素の組合せを使用して、異なる種類のDNA損傷を同時的に修復することができる。例えば、ある酵素は特定の種類の損傷を修復する際に効果的である場合がある一方、別の酵素は異なる損傷を修復するために好ましい。いくつかの酵素を1つのDNA修復キット中に組み合わせることにより、より頑強な修復プロセスを実施することができる。記載される機能を行うために使用することができる例示的な酵素を、それぞれの酵素により修復される損傷、およびそれぞれの酵素により行われる活性とともに、表1に列挙する。
【0017】
【表1】

【0018】
[0027] 図2を参照して、本発明の技術に従う損傷DNAの修復の説明が、記載される。DNA鎖50は、1またはそれ以上の損傷(例えば、増幅-阻害性)部位52および54を有していてもよい。酵素の組合せを使用して、DNA鎖50に対していくつかの修復機能を行うことができる。この組合せには、例えば、グリコシラーゼ(例えば、FPG)および/またはエンドヌクレアーゼ(例えば、エンドヌクレアーゼIV、エンドヌクレアーゼVII)が含まれていてもよく、これにより損傷塩基部分52を除去しそしてAP部位56を残す。組合せには、損傷部位52および54および/またはAP部位56にてDNAにニックを形成するエンドヌクレアーゼ(例えば、T4エンドヌクレアーゼV)がさらに含まれていてもよく、ニック58および60が残る。次いで、組合せ中に含まれるDNAポリメラーゼ(例えば、E. coli DNAポリメラーゼI)は、DNA鎖50の下流方向にニック58および60を翻訳することができ、ニック58および60が翻訳されると同時に損傷DNAを修復する。ニックトランスレーションは、5'-3'エキソヌクレアーゼ活性を介して、達成することができる。最終的に、ニック58および60を、組合せ中に存在するリガーゼ(例えば、T4 DNAリガーゼ)を用いてシールすることができる。
【0019】
[0028] 図2を参照して記載される例示的な技術を、単一混合物または複数混合物中の複数の酵素の組合せを介して、行うことができることを理解すべきである。例示的なDNA修復キットには、表2に記載される酵素混合物が含まれていてもよい。
【0020】
【表2】

【0021】
[0029] 修復バッファ溶液の10×調製物には、例えば、100 mM Tris-HCl、100 mM MgCl2、500 mM NaCl、そして10 mMジチオスレイトールが含まれていてもよく、pH 7.9になる。表2中に記載された混合物中の酵素、ならびにその他の酵素、単一の修復溶液中で損傷DNAに対して同時に適用することができる。例えば、損傷DNAの修復を、表3中に記載される混合物から構成される修復溶液中でのインキュベーションを介して行うことができる。
【0022】
【表3】

【0023】
[0030] 従って、例示的なDNA修復キットは、事前混合された溶液中で、または混合のために利用可能な別個の成分として、表3中に記載されたいくつかのまたは全ての成分を含有することができる。約15μlの損傷DNAを、表3に記載される修復溶液と組み合わせ、そして約20〜42℃にて25〜60分間インキュベーションし、その後42〜75℃でさらに15〜25分間インキュベーションすることができる。最初のインキュベーション期間により損傷DNAの修復を可能にし、一方より高い温度のインキュベーションにより酵素が不活性化する。例示的な態様において、修復酵素の不活性化を、約65℃で約15分間、修復DNAと酵素混合物とをインキュベーションすることにより行うことができる。この高い温度でのインキュベーションは、修復DNAを変性することなく、熱に不安定なDNA修復酵素(リガーゼを含む)を不活性化する。従って、熱に安定なDNAポリメラーゼ(例えば、Bst DNAポリメラーゼ)および/または熱に安定なDNAリガーゼ(例えば、Taq DNAリガーゼ)は好ましくなく、そして、特定の態様において、本発明のプロセスにおいて使用することができない。さらに、特定の酵素または全ての酵素が熱-不活性化可能なものであってもよいため、増幅の前に修復DNAの精製をすることが必要ではなくてもよい。
【0024】
[0031] 表3に記載される修復溶液には、リボアデノシン三リン酸(リボ-ATP)が含まれる一方、この成分は実施する上で除去することができることに注意すべきである。さらに、例示的なキット中の酵素のどれも、機能するためにβ-ニコチンアミドアデニンジヌクレオチド(NAD)を必要とせず、そして従ってその存在は必要ではない。
【0025】
[0032] ひとたび損傷DNAが本明細書で記載される技術に従って修復されると、修復DNAを増幅しそして解析して、例えば、DNAが由来する被検体の同一性を決定することができる。上述した様に、PCR、AmpFLP、STR、Y-STR、などの多数の解析技術を使用することができる。熱-不活性化酵素は、DNAの解析に影響を及ぼさず、そして従って増幅および解析を行う前に修復DNAから除去する必要はない。
【0026】
実験結果
[0033] ゲノムDNAを酸化損傷および紫外線損傷に供し、その後本発明の技術の態様に従って修復した。結果を以下にまとめる。実験において使用するためのゲノムDNAは、HT29細胞株から約80〜160 ng/μlの濃度で抽出した。図3は、非損傷DNAのスライドである。
【0027】
[0034] DNAを損傷するため、9 mM塩化鉄(FeCl3)および18 mM EDTAであるFe-EDTA溶液、約100μlを、0.37 M FeCl3および水中に溶解した0.5 EDTAを使用して調製した。さらに、約1 mlの30 mM過酸化水素(H2O2)溶液を、約3.4μlのストックH2O2(すなわち、約8.8 Mで30%H2O2)を1 mlの水に添加することにより調製した。H2O2溶液を氷上に置いた。
【0028】
[0035] 表4に記載される反応混合物を、チューブ中で調製した。全反応容量は約30μlであった。H2O2溶液を添加して損傷反応を開始した。混合物を、37℃で様々な時間(例えば、20〜100分)インキュベーションし、次いでTEで平衡化したNAP-5脱塩カラム(カタログNo. 17-0853-01、GE Healthcareから入手可能)を使用して脱塩した。次いで、混合物を、1 ml TE中に溶出した。
【0029】
【表4】

【0030】
[0036] 酸化損傷DNAサンプルをFenton反応を使用して調製した。HT-29細胞株から抽出した約130 ng/μlの濃度のゲノムDNA、約2μlを、5μlのFe-EDTA溶液および23μlの二重蒸留水と混合した。約4μlの30mM H2O2溶液を添加して、反応を開始した。反応混合物の8本のチューブを調製し、そして37℃で80分間インキュベーションした。次いで、混合物をNAP-5脱塩カラムを使用して脱塩し、そして1 mlのTE中で溶出し、それにより得られた損傷DNA濃度は約2 ng/μlであった。図4は、酸化損傷DNAのスライドである。
【0031】
[0037] 次いで、酸化損傷DNAを、本発明の技術の態様に従って、単一チューブ内に複数の修復酵素を含有する修復混合物と組み合わせた。修復混合物の成分を、表5にまとめる。修復混合物を、37℃で約30分間、そしてその後65℃で約20分間、インキュベーションした。
【0032】
【表5】

【0033】
[0038] インキュベーションの後、修復DNAを含有する混合物に対して、PCR増幅を行った。PCR増幅を、表6に列挙した成分を使用して行った。PCRビーズは、GE Healthcareから入手可能である(カタログNo. 27-9558-01)。PCR増幅を、95℃で5分間;95℃で30秒間、58.5℃で30秒間、そして72℃で90秒間のサイクルを38サイクル;そして72℃で10分間;で行った。図5は、修復DNAのスライドである。これらの結果に基づいて、本明細書において開示された技術の態様(図5)を使用して修復したDNAは、酸化損傷DNA(図4)よりも、非損傷対照DNA(図3)により近いようである。
【0034】
【表6】

【0035】
[0039] 酸化損傷サンプルに加えて、UV損傷サンプルを調製した。HT-29細胞株から抽出した約130 ng/μlの濃度のゲノムDNA、約100μlを、石英キュベット中に入れ、そして約245 nmおよび4 mW/cm2で5分間、UVCエネルギーに対して曝露した。次いで、UV損傷DNAを、約2 ng/μlにまで希釈した。図6は、UV損傷DNAのスライドである。
【0036】
[0040] 次いで、UV損傷DNAを、本発明の技術の態様に従って、単一チューブ内に複数の修復酵素を含有する修復混合物と組み合わせた。修復混合物の成分は、表7にまとめる。修復混合物を、37℃で約30分間、そしてその後65℃で約20分間、インキュベーションした。
【0037】
【表7】

【0038】
[0041] インキュベーションの後、修復DNAを含有する混合物に対して、上述した様にPCR増幅を行った。図7は、修復DNAのスライドである。これらの結果に基づいて、本明細書において開示された技術の態様(図7)を使用して修復したDNAは、UV損傷DNA(図6)よりも、非損傷対照DNA(図3)により近いようである。
【0039】
[0042] 上述した様に、2 ngのDNA、損傷DNA、または修復DNAを、表8に示すプライマーセットを使用して、上述したPCRにより増幅した。
【0040】
【表8】

【0041】
[0043] 本明細書中では、本発明の特定の態様のみを説明しそして記載したが、多数の修飾および変更が当業者には思い浮かぶであろう。従って、添付の請求の範囲が本発明の真の概念に含まれる全てのそのような修飾および変更を含むことを意図していることを理解すべきである。
【特許請求の範囲】
【請求項1】
5'-3'エキソヌクレアーゼ活性を有する熱に不安定なDNAポリメラーゼ;そして
熱に不安定なDNAリガーゼ;
を含む、DNA修復キット。
【請求項2】
熱に不安定なDNAポリメラーゼおよび熱に不安定なDNAリガーゼが、実質的に、約42℃〜約75℃のあいだの温度でインキュベーションすることにより不活性化される、請求項1に記載のDNA修復キット。
【請求項3】
DNAポリメラーゼがEschericia coliのDNAポリメラーゼIである、請求項1に記載のDNA修復キット。
【請求項4】
少なくとも1種類の追加のDNA修復酵素をさらに含む、請求項1に記載のDNA修復キット。
【請求項5】
8-オキソグアニンDNAグリコシラーゼ、エンドヌクレアーゼIV、エンドヌクレアーゼVIII、またはT4エンドヌクレアーゼVのうちの少なくとも1種類をさらに含む、請求項1に記載のDNA修復キット。
【請求項6】
DNAポリメラーゼおよびDNAリガーゼが、事前混合された単一溶液である、請求項1に記載のDNA修復キット。
【請求項7】
グリコシラーゼ酵素および/またはエンドヌクレアーゼ酵素を含有する溶液をさらに含む、請求項1に記載のDNA修復キット。
【請求項8】
N-グリコシラーゼ活性、AP-リアーゼ活性、5'-3' DNAポリメラーゼ活性、および5'-3'エキソヌクレアーゼニックトランスレーション活性を示し、そして熱-不活性化可能な、酵素の組合せを含む、DNA修復キット。
【請求項9】
DNAグリコシラーゼ;
DNAエンドヌクレアーゼ;
DNAポリメラーゼI;および
T4 DNAリガーゼ;
を含む、DNA修復キット。
【請求項10】
DNAグリコシラーゼが8-オキソグアニンDNAグリコシラーゼを含む、請求項9に記載のDNA修復キット。
【請求項11】
エンドヌクレアーゼが、エンドヌクレアーゼIV、エンドヌクレアーゼVIII、T4エンドヌクレアーゼV、またはこれらの組合せを含む、請求項9に記載のDNA修復キット。
【請求項12】
約7.9のpHを有するバッファ溶液をさらに含む、請求項9に記載のDNA修復キット。
【請求項13】
DNAポリメラーゼIが、5'-3'DNAポリメラーゼ活性および5'-3'エキソヌクレアーゼ活性の両方を含む、請求項9に記載のDNA修復キット。
【請求項14】
損傷DNAを、5'-3'エキソヌクレアーゼ活性を有する熱に不安定なDNAポリメラーゼと熱に不安定なDNAリガーゼとを含むDNA修復酵素ブレンドと、第1の温度でインキュベーションして、修復DNAを生成する工程;
を含む、損傷DNAを修復する方法。
【請求項15】
修復DNAおよびDNA修復酵素ブレンドを、第1の温度よりも高い第2の温度でインキュベーションする工程により、酵素ブレンドを不活性化する工程をさらに含む、請求項14に記載の方法。
【請求項16】
修復DNAを増幅する工程をさらに含む、請求項14に記載の方法。
【請求項17】
修復DNAに対してポリメラーゼ連鎖反応を行う工程をさらに含む、請求項14に記載の方法。
【請求項18】
既知DNAサンプルのデータベースとの比較のために修復DNAを解析する工程をさらに含む、請求項14に記載の方法。
【請求項19】
第2の温度が約42℃〜約75℃の範囲である、請求項14に記載の方法。
【請求項20】
グリコシラーゼを介して、DNA鎖上の損傷部位から塩基を除去する工程;
エンドヌクレアーゼを介して、損傷部位でのDNA鎖にニックを形成する工程;
関連する5'-3'エキソヌクレアーゼ活性を有するDNAポリメラーゼを介して、DNA鎖の下流に向けてニックを翻訳する工程;そして
ニックをリガーゼを用いてシールする工程;
を含む、DNA修復のための方法。
【請求項21】
DNAポリメラーゼおよびリガーゼが熱に不安定である、請求項20に記載の方法。
【請求項22】
DNAポリメラーゼが、5'-3'DNAポリメラーゼ活性および5'-3' DNAエキソヌクレアーゼ活性を両方とも含む、請求項20に記載の方法。
【請求項1】
5'-3'エキソヌクレアーゼ活性を有する熱に不安定なDNAポリメラーゼ;そして
熱に不安定なDNAリガーゼ;
を含む、DNA修復キット。
【請求項2】
熱に不安定なDNAポリメラーゼおよび熱に不安定なDNAリガーゼが、実質的に、約42℃〜約75℃のあいだの温度でインキュベーションすることにより不活性化される、請求項1に記載のDNA修復キット。
【請求項3】
DNAポリメラーゼがEschericia coliのDNAポリメラーゼIである、請求項1に記載のDNA修復キット。
【請求項4】
少なくとも1種類の追加のDNA修復酵素をさらに含む、請求項1に記載のDNA修復キット。
【請求項5】
8-オキソグアニンDNAグリコシラーゼ、エンドヌクレアーゼIV、エンドヌクレアーゼVIII、またはT4エンドヌクレアーゼVのうちの少なくとも1種類をさらに含む、請求項1に記載のDNA修復キット。
【請求項6】
DNAポリメラーゼおよびDNAリガーゼが、事前混合された単一溶液である、請求項1に記載のDNA修復キット。
【請求項7】
グリコシラーゼ酵素および/またはエンドヌクレアーゼ酵素を含有する溶液をさらに含む、請求項1に記載のDNA修復キット。
【請求項8】
N-グリコシラーゼ活性、AP-リアーゼ活性、5'-3' DNAポリメラーゼ活性、および5'-3'エキソヌクレアーゼニックトランスレーション活性を示し、そして熱-不活性化可能な、酵素の組合せを含む、DNA修復キット。
【請求項9】
DNAグリコシラーゼ;
DNAエンドヌクレアーゼ;
DNAポリメラーゼI;および
T4 DNAリガーゼ;
を含む、DNA修復キット。
【請求項10】
DNAグリコシラーゼが8-オキソグアニンDNAグリコシラーゼを含む、請求項9に記載のDNA修復キット。
【請求項11】
エンドヌクレアーゼが、エンドヌクレアーゼIV、エンドヌクレアーゼVIII、T4エンドヌクレアーゼV、またはこれらの組合せを含む、請求項9に記載のDNA修復キット。
【請求項12】
約7.9のpHを有するバッファ溶液をさらに含む、請求項9に記載のDNA修復キット。
【請求項13】
DNAポリメラーゼIが、5'-3'DNAポリメラーゼ活性および5'-3'エキソヌクレアーゼ活性の両方を含む、請求項9に記載のDNA修復キット。
【請求項14】
損傷DNAを、5'-3'エキソヌクレアーゼ活性を有する熱に不安定なDNAポリメラーゼと熱に不安定なDNAリガーゼとを含むDNA修復酵素ブレンドと、第1の温度でインキュベーションして、修復DNAを生成する工程;
を含む、損傷DNAを修復する方法。
【請求項15】
修復DNAおよびDNA修復酵素ブレンドを、第1の温度よりも高い第2の温度でインキュベーションする工程により、酵素ブレンドを不活性化する工程をさらに含む、請求項14に記載の方法。
【請求項16】
修復DNAを増幅する工程をさらに含む、請求項14に記載の方法。
【請求項17】
修復DNAに対してポリメラーゼ連鎖反応を行う工程をさらに含む、請求項14に記載の方法。
【請求項18】
既知DNAサンプルのデータベースとの比較のために修復DNAを解析する工程をさらに含む、請求項14に記載の方法。
【請求項19】
第2の温度が約42℃〜約75℃の範囲である、請求項14に記載の方法。
【請求項20】
グリコシラーゼを介して、DNA鎖上の損傷部位から塩基を除去する工程;
エンドヌクレアーゼを介して、損傷部位でのDNA鎖にニックを形成する工程;
関連する5'-3'エキソヌクレアーゼ活性を有するDNAポリメラーゼを介して、DNA鎖の下流に向けてニックを翻訳する工程;そして
ニックをリガーゼを用いてシールする工程;
を含む、DNA修復のための方法。
【請求項21】
DNAポリメラーゼおよびリガーゼが熱に不安定である、請求項20に記載の方法。
【請求項22】
DNAポリメラーゼが、5'-3'DNAポリメラーゼ活性および5'-3' DNAエキソヌクレアーゼ活性を両方とも含む、請求項20に記載の方法。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【公表番号】特表2011−527577(P2011−527577A)
【公表日】平成23年11月4日(2011.11.4)
【国際特許分類】
【出願番号】特願2011−517506(P2011−517506)
【出願日】平成21年7月7日(2009.7.7)
【国際出願番号】PCT/US2009/049735
【国際公開番号】WO2010/005919
【国際公開日】平成22年1月14日(2010.1.14)
【出願人】(390041542)ゼネラル・エレクトリック・カンパニイ (6,332)
【Fターム(参考)】
【公表日】平成23年11月4日(2011.11.4)
【国際特許分類】
【出願日】平成21年7月7日(2009.7.7)
【国際出願番号】PCT/US2009/049735
【国際公開番号】WO2010/005919
【国際公開日】平成22年1月14日(2010.1.14)
【出願人】(390041542)ゼネラル・エレクトリック・カンパニイ (6,332)
【Fターム(参考)】
[ Back to top ]

