消化管間質腫瘍患者の予後予測のためのキット及び方法
【課題】消化管間質腫瘍(GIST)患者の予後予測のためのキット及び方法を提供する。
【解決手段】GISTをもつ患者の生物学的検体においてREC8遺伝子及びPAX3遺伝子のうちの少なくとも1つの遺伝子のメチル化の有無を測定し、該遺伝子がメチル化されている場合予後不良であり、一方メチル化されていない場合予後良好であると決定することを含む、該患者の予後をインビトロで予測する方法、ならびに、GIST検査用キット。
【解決手段】GISTをもつ患者の生物学的検体においてREC8遺伝子及びPAX3遺伝子のうちの少なくとも1つの遺伝子のメチル化の有無を測定し、該遺伝子がメチル化されている場合予後不良であり、一方メチル化されていない場合予後良好であると決定することを含む、該患者の予後をインビトロで予測する方法、ならびに、GIST検査用キット。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、消化管間質腫瘍患者の予後予測のためのキット及び方法を提供する。
【背景技術】
【0002】
消化管間質腫瘍(以下GIST(gastrointestinal stromal tumor)は、食道、胃、小腸、大腸、直腸などの消化管に発生し、KIT遺伝子やPDGFRα遺伝子の機能獲得性突然変異を高頻度に認める悪性腫瘍であり、間葉系腫瘍のほとんどを占める。GIST患者にはメチル酸イマチニブなどのチロシンキナーゼレセプター活性阻害薬による分子標的療法が有効であるとされているが、GISTには、良性から悪性までの幅広いスペクトルがあり上記の薬剤も一部の腫瘍には有効でないことも知られている。このように、GISTは、高頻度に転移し極めて予後不良な群と、腫瘍の増大が遅く転移をきたさない予後が比較的良好な群に分けることができるが、しかしながら、その両群を規定するメカニズムはほとんど明らかになっていない。
【0003】
さらにまた、患者においてGISTの予後の良・不良を事前に判定することができるならば、上記の分子標的療法を含めた適切な治療計画を組むことが可能になると考えられる。そのための予後予測マーカーとして、 臨床病理像と予後の関係(非特許文献1)や、KITのエクソン11における変異と予後の関係(非特許文献2)が報告されているが、実用化までは至っておらず、治療方針を決定するほどの確度で信頼されうる、予後を予測できる腫瘍マーカーは確立されていない。
【0004】
そのような情況にあって、上記以外に以下のタンパク質又は遺伝子がGISTの予後予測のための腫瘍マーカーの候補として報告されている。
【0005】
特許文献1には、DDX39(UniProt Accession No.O00148)というDEAD box RNA helicaseの一種であるタンパク質の発現がGISTの術後転移症例の原発巣腫瘍組織において、非転移症例の原発巣腫瘍組織よりも有意に発現が高いこと、一方、正常細胞ではほとんど発現していないことが報告されている。
【0006】
特許文献2には、胃を原発巣とするGISTにおいて、ジペプチジルペプチダーゼIV遺伝子が発現していた場合に、その腫瘍が悪性であると判定されうることが記載されている。
【0007】
特許文献3には、フェチンというタンパク質の発現がGIST患者の予後不良と逆相関することが報告されている。
【0008】
非特許文献3には、GIST患者からの検体について多数の遺伝子のプロモーター領域のDNAメチル化の網羅的解析とパイロシークエンス法によるDNAメチル化の定量的解析を行い、悪性度の高いもので異常メチル化を示す遺伝子が存在することが報告されている。しかしながら、DNAメチル化の異常と予後又は悪性度との関係を示す遺伝子の詳細は明らかにされていないし、また、GISTにおいて異常メチル化とは何であるのか、GISTにおいて正常なメチル化とは何であるのか、そのいずれも遺伝子レベルで明確でない。
【0009】
非特許文献4には、ETV1(Ets Variant Gene 1)がすべてのGIST腫瘍及びGIST細胞系統において高度に発現していることが報告されている。
【先行技術文献】
【特許文献】
【0010】
【特許文献1】特開2009-132636号公報
【特許文献2】特開2009-011249号公報
【特許文献3】特開2008-100958号公報
【非特許文献】
【0011】
【非特許文献1】T. Hasegawa et al., Human Pathology, 33(6):669-676 (2002)
【非特許文献2】J. Andersson J. et al., Gastroenterology, 130(6):1573-1581 (2006)
【非特許文献3】第3回エピジェネティクス研究会年会(日本、2009年)(P-38)
【非特許文献4】P. Chi et al., Nature, 467:849-853 (2010)
【発明の概要】
【発明が解決しようとする課題】
【0012】
本発明の目的は、消化管間質腫瘍(GIST)をもつ患者の予後をDNAメチル化異常の検出に基づいて予測する方法を提供することである。
【課題を解決するための手段】
【0013】
本発明者らは、上記の課題を解決するために、GISTをもつ患者の予後を従来法と比べてより精確に判定することを可能にする特定の遺伝子の異常なメチル化が存在することを見出し、本発明を完成させるに至った。
【0014】
したがって、本発明は、以下の特徴を含む。
すなわち、本発明は、第1の態様において、消化管間質腫瘍をもつ患者の生物学的検体においてREC8遺伝子及びPAX3遺伝子のうちの少なくとも1つの遺伝子のメチル化の有無を測定し、該遺伝子がメチル化されている場合予後不良であり、一方メチル化されていない場合予後良好であると決定することを含む、該患者の予後をインビトロで予測する方法を提供する。
その実施形態において、上記方法は、p16遺伝子のメチル化の有無を測定することをさらに含む。
【0015】
REC8遺伝子及び/又はPAX3遺伝子のメチル化の有無と、p16遺伝子のメチル化の有無とを組み合わせることによって上記予後予測の精度をさらに高めることができる。
【0016】
すなわち、好ましくは、本発明ではREC8遺伝子及びPAX3遺伝子の両方の遺伝子のメチル化を測定する。さらに好ましくは、REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化を測定する。
【0017】
別の実施形態において、上記方法における上記遺伝子のメチル化をパイロシークエンス法又はMSP法で測定する。
【0018】
本発明は第2の態様において、REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化の有無を検出するための試薬を含むGIST検査用キットを提供する。
【0019】
その実施形態において、試薬が、REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化領域を特異的に増幅するためのプライマーである。
試薬にはさらに、バイサルファイト試薬及びPCR反応試薬を含むことができる。
【発明の効果】
【0020】
GIST患者の予後を予測するための適当な腫瘍マーカーがない情況のなかで、本発明での特定の遺伝子のメチル化の有無を測定するだけで精度よく予後予測が可能になる。
【図面の簡単な説明】
【0021】
【図1】この図は、GIST患者からの検体(GIST腫瘍組織)における155遺伝子のDNAメチル化の網羅的解析の結果を示す。赤色はメチル化陽性を表わす。
【図2】この図は、REC8遺伝子とPAX3遺伝子のメチル化の有無の測定が予後予測のために有用であることを示す。アレイでの測定結果から、決定木法により同定したREC8遺伝子とPAX3遺伝子は、GIST患者を予後不良群と予後良好群に分けることができることを示す。図中、アレイ上の遺伝子スポットは、GIST腫瘍組織DNAのメチル化(赤シグナル)とコントロール(正常組織)DNAのメチル化(緑シグナル)に由来したシグナルを発するが、これまでのアレイに関する報告(W. Gao et al., Carcinogenesis, 29(10):1901-10 (2008))に基づき、(赤シグナル/緑シグナル)≧2.0のとき、該遺伝子はGIST由来DNAでメチル化陽性であるとし、また(赤シグナル/緑シグナル)<2のとき、該遺伝子はメチル化陰性であると評価し、したがって、REC8≧2.0はREC8メチル化陽性検体を示し、REC8<2&PAX3≧2は、REC8メチル化陰性かつPAX3メチル化陽性検体を示し、REC8<2&PAX3<2はREC8メチル化陰性かつPAX3メチル化陰性検体を示す。
【図3】この図は、GIST検体115例(トレイニングセット40例、検証セット75例)におけるパイロシークエンス法を用いたメチル化解析の結果を示す。トレイニングセットは、図1のDNAメチル化網羅的解析に用いた症例セット、検証セットは、REC8遺伝子、PAX3遺伝子およびp16遺伝子のメチル化の有無の測定が、別の症例群でも予後予測に有用であることを示すために行った解析結果を示している。パイロシークエンス法で計測されるサンプル内の目的とするCpGサイトのメチル化測定値(メチル化レベル)が15%以上を示す場合をメチル化陽性と判断した。この値(15%)はパイロシークエンス法の非特異的な測定値(ノイズ)では通常得られない測定値としての最小値である。図中、「HD」は、homozygous deletion(すなわち、父親由来および母親由来の両アレルが欠失した状態)を表す。
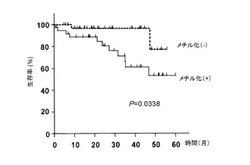
【図4】この図は、REC8遺伝子、PAX3遺伝子及び/又はp16遺伝子のDNAメチル化状態とGIST症例生存期間との関係を示す。REC8遺伝子、PAX3遺伝子及びp16遺伝子のうち1つ以上メチル化が存在する症例では、生存期間が有意に短い(p=0.0338)ことを示す。
【発明を実施するための形態】
【0022】
本発明は、上記のとおり、消化管間質腫瘍をもつ患者の生物学的検体においてREC8遺伝子及びPAX3遺伝子のうちの少なくとも1つの遺伝子のメチル化の有無を測定し、該遺伝子がメチル化されている場合予後不良であり、一方メチル化されていない場合予後良好であると決定することを含む、該患者の予後をインビトロで予測する方法を提供する。
【0023】
以下に、本発明をさらに詳細に説明する。
1.消化管間質腫瘍
消化管間質腫瘍(GIST)は、食道、胃、小腸、大腸、直腸などの消化管の主に壁に発生する間葉系腫瘍であり、かつ、粘膜下腫瘍を構成する腫瘍である。この腫瘍は、増殖因子受容体c-kitの変異が原因であると言われている。
【0024】
この腫瘍は、その患者において予後不良の場合と予後良好の場合に分けられる。予後不良は、腫瘍が局部的浸潤性、破壊的増殖性または 転移性を有する場合を指し、いわゆる悪性かつ再発性である。一方、予後良好は、浸潤性・転移性がなく、すなわち再発性がなく、単なる外科手術で完治する場合を指す。
【0025】
これまで、GISTの診断は、KITタンパク質の発現の有無によって可能であろうと指摘されてきた。また実際には、腫瘍径と腫瘍細胞分裂像数によってリスクの程度が判定されてきた。治療薬については、グリベック(登録商標)以外には効果的な治療薬がないと言われている。
【0026】
2.GIST患者における予後の予測とそれに関連する遺伝子
後述の実施例に記載されるように、本発明者らは、トレイニングセット(40例)でアレイを施行し、そのうち転移・浸潤をきたしている予後不良群(18例)と、細胞分裂像と大きさから判断した予後良好群(低リスク群 9例)の計27例を対象とし、アレイの解析データから予後不良群で特異的にメチル化をしている155遺伝子を選択した(図1)。この155遺伝子を決定木法で解析し、予後不良群と予後良好群を最も的確に区別する2遺伝、すなわちREC8遺伝子とPAX3遺伝子を今回見出した(図2)。これらの遺伝子に、GISTの予後との関係が報告されているヒトp16遺伝子のメチル化を加えて、予後予測の有用性を検討した。トレイニングセットでは中間群13例を、上記27例に加えた40例において、パイロシークエンス法を用いてメチル化定量的解析法を施行した。アレイの結果と同様に予後良好群でメチル化が低頻度であり、予後不良群でメチル化が高頻度であることが確認された(図3(左))。これら遺伝子の有用性をさらに検証するため、新たに別の患者群(検証セット)75例(予後良好群24例、中間群36例、予後不良群15例)を対象にメチル化定量的解析法を行ったところ、これらの遺伝子は、トレイニングセットで得られた結果と同様に、予後不良群で高頻度及び高割合でメチル化されており一方、予後良好群では、特定の遺伝子のメチル化の程度が非常に低く、ほとんど又は全くメチル化されていないことが確認された(図3(右))。さらに生存期間が明らかな69例を対象に生存期間を比較した結果、p16遺伝子、REC8遺伝子、PAX3遺伝子のいずれかでメチル化が陽性の患者群では、メチル化が検出されない患者群と比較して、生存期間が有意に短いことが判明した(図4)。
【0027】
本発明の方法で使用しうる遺伝子は、メチル化の程度に応じて予後不良群、中間群及び予後良好群を適正に判別することを可能にする。
【0028】
これまでGIST症例の中には、中間群に入る症例も多く存在し、この群に属する症例が予後不良なのか、それとも予後良好なのかが不明確であったが、本発明の方法は、この中間群さえも、該群のなかで相対的にメチル化の程度が高い層(高リスク)と、相対的にメチル化の程度が低い層(中リスク)に分類できるほどに高い感度と高い特異度を有している(図3)。
【0029】
本発明の方法では、REC8遺伝子及びPAX3遺伝子のいずれか一方がメチル化されている場合をメチル化陽性であるとした。REC8遺伝子がメチル化陽性である場合、予後不良か又は(悪性の可能性が高い)高リスクに属する。同様に、PAX3遺伝子がメチル化されている場合にも、REC8遺伝子ほどではないにしてもメチル化頻度が高いほど予後不良及び高リスクに属する。特に、これら遺伝子の両方がともにメチル化されている場合は、高い特異度で予後不良又は高リスクに属する。しかし、ときには両遺伝子がメチル化陰性の場合もあり、この結果を修正し、さらに分類の確度を高めるために、本発明の方法ではp16遺伝子等の別の遺伝子のメチル化状態を検査に含めることができる。
【0030】
このように、GISTの悪性化に伴い、REC8遺伝子及び/又はPAX3遺伝子、ならびにp16遺伝子、のメチル化が蓄積する。内視鏡検査等で採取された消化管(胃、小腸、大腸、直腸、食道など)のGIST組織検体に対して上記遺伝子のメチル化解析を行うことによって、GIST患者の予後予測やGISTの悪性度に関する臨床検査への応用が可能になるし、さらにまた、臨床検査の結果を受けてGIST患者に対するより効果的な治療法の選択に寄与することができる。
【0031】
本発明における予後予測のための遺伝子マーカーは、REC8遺伝子及びPAX3遺伝子であり、好ましくは追加的にp16遺伝子も含まれる。これら遺伝子の配列情報は、公知の遺伝子データバンク(米国NCBI等)から入手可能である。
【0032】
ヒトREC8(meiotic recombination REC8 homolog)の塩基配列は、例えばUniprotKB/Swiss-Prot accession No. 095072、GenBank accession No. NM_005132、NM_001048205、NP_005123などとして知られている。REC8遺伝子のプロモーター領域のCpGアイランドの塩基配列を配列番号1として示す。
【0033】
ヒトPAX3 は、転写因子であるpaired box (PAX) ファミリーに属する遺伝子であり、ヒトPAX3タンパク質は、例えば479アミノ酸等を含むが、種々の置換等の変異を含むバリアントからなる。PAX3は、耳、目及び顔の発生において同定され、その変異によってWaardenburg症候群1型及び3型が起きる。本発明では、ヒトPAX3は、バリアントのいずれも包含するものとする。バリアントの例は、GenBank accession No. NM_001127366、NM_181458、NM_000438、NM_181457、NM_18459、NM_013942、NM_181460などである。PAX3遺伝子のプロモーター領域のCpGアイランドの塩基配列を配列番号2として示す。
【0034】
ヒトp16遺伝子のメチル化とGIST症例の予後との関連はこれまで報告されている(R. Ricci et al., Role of p16/INK4a in gastrointestinal stromal tumor progression, Am J Clin Pathol 2004;122:35-43)。ヒトp16遺伝子は、cyclin-dependent kinase inhibitor 2A (melanoma, p16, inhibits CDK4) (CDKN2A)であり、第1エクソンが異なる7つのバリアントからなることが知られており、そのうち少なくとも3つは選択的スプライシングによるものである。本発明では、いずれのバリアントもp16に包含されるものとする。GenBankに登録されたバリアントの例は、NG_007485、NM_058195、NM_000077、NM_001195132、NM_058197などであるが、本発明のp16はそこに記載された塩基配列を含むものとする。p16遺伝子のプロモーター領域のCpGアイランドの塩基配列を配列番号3として示す。
【0035】
ヒト以外の哺乳動物の配列は、GenBank等の遺伝子データベースにアクセスすることにより入手可能である。
【0036】
本明細書中「哺乳動物」又は「患者」には、霊長類、ペット動物、家畜動物、げっ歯類などが含まれ、好ましい哺乳動物はヒトである。
【0037】
3.DNAメチル化の測定
DNAメチル化は、哺乳動物のゲノム上のREC8、PAX3及びp16遺伝子の非翻訳領域、特にプロモーター領域のCpGアイランド配列においてシトシン(C)の5位の炭素が過度にメチル化(hypermethylation)されていることを意味する。ここで、CpGアイランドは、哺乳動物のゲノム配列において非翻訳領域に存在するグアニン(G)シトシン(C)含量が多い領域であり、CpGのpは、CとGの間のホスホジエステル結合を表す。CpGアイランドはCGからなるジヌクレオチドの繰り返し配列を含み、その定義では通常、500塩基対領域内のCpG含有率が55%以上、CpG/GpC比が0.65以上である。
【0038】
REC8遺伝子、PAX3遺伝子及びp16遺伝子のCpGアイランドのhypermetylation領域内でPCR法により約150〜250bpの増幅産物を得ることができるプライマーを設計することができる。
【0039】
これらのヒト遺伝子のプロモーター領域のCpGアイランドの配列の例を以下に示す。
(1) REC8 (下線付き大文字はREC8のエクソン配列(遺伝子の転写領域):エクソン1、エクソン2、エクソン3)(配列番号1)
ctactcaggaggccaagacacgagaatcgcttgaacctgaggggatggggggcagaggatgcagtgagccgagattgccgagatagcgccactgcattccagcctgggcaacagtgagagcctccatctcaaaaaaaaaaaaaaaaaatggacttcttcctcattgacctgtggtggctccaaggccatcaaaaaaaaaaaaaatggacttcttcctcattgacctgtggtggctccaaggccatcatctcacagcctttttttttttttttttttttgagacggagtcttgctcccttgcccaggttggagtggagctgcatgatctcggctcactgcaacctccgcctcccaggttcaagcattctcctgcctcagcctcctgagtagctgggattacaggcgcgtgccaccacgcccagctaacttttgcatttttaatagagacggggtttcaccatattggtcaggctggactcgaactcctaacctcatgatccgcccgcctcggcctcccaaagtgctaggattacaggcgtgagccactgcgcccggcctcatctcacatctttcttcctctgcagcacacgacacgccctagttggaaacaaagtcggagtttgtggattgggggaagggcggggtctaacctcaggtcaggcgccgtgcaaggtacatcttggcacccggaagaggcccagtacagttgcccccgaggtgacccgacctcccctaccaattgaggcgcccttgttgccaggcttgcggcgggggagcggcgggggagcgacggggatgcgctcattggtcaaggaaggggcgcctgttactagaggcgagaaccggagcccattggtcggaacacctcacaatggaccccagcggcgcgcaaaatccttatgattggtttgctggctgcctcgggagaccctgttgccaggatacttggcgttcccgacccgacccccgttccccattggctgtcagggcaaaagccgccATCTAATGAGGAGCGAGGTGCGGTGCCCCGAAGCGCTCGCTTCCCGCGGTGCGATCTAGTCCTGCAGTAGGCGGCCCGGGGCCACACCGCGGCCGCCCAAGCCAGTGCAAGGCCCAGGGGCCTGACATCGCTCCCAGCGCTCGAGGACCGAGGCCTGCTGTGGAGGACACCGTGCTCCCTCGGGACCTGCTCTGGATTCCGGCCCGGACGTCCCCTTGGAGCTCTGCATCTCCAACCTGGAACCCAACCCAGAAGTCTCAAGTTTGACGCATCACGTGGCGTGCGGATCCACTGAGGGTCCACAGAGAGGGGCGCCCATCTCCTGCGTCTCAGTTATCCTGGTAATTGTGTATCTGCCCATTGTTCGTTGCCTCATTAACTTGGCTTTCTAGGTGCACCCACCTTGCCACCAGAGAAGTCCAAATCCTGACTTCTCTCCAAGGTGTTGGGAATTCTGTGCCCTAAAGAATTCCGACTCAGATCCGAACGGGGATCTGGTGGAATCGAGGGTGAAAGACCAGAGGGACAATGTTCTACTATCCCAACGTGCTTCAGCGCCACACCGGCTGCTTTGCCACCATCTGgtaagggcggggcccgttggcgcgcgatggcggacgctgcccgggatcccagcctgacagctccccctccatccccattctcccaccttccccacccacttcagGCTGGCGGCGACTCGCGGCAGCCGGTTGGTGAAGCGCGAATACCTGAGGGTGAATGTGGTGAAAACCTGgtaaggcccagaaaagggaaggagggcctggtgcggggggtgagttaggggatggggtggccaagactgtgggcccactcctggacgcagcggtaatcagggcgcattgttccccagCGAGGAAATCCTCAATTACGTGCTGGTACGAGTGCAACCCCCGCAGCCCGGCCTGCCGCGGCCCCGCTTCTCCCTCTATCTCTCAGCCCAACTTCAGATCGGTGTGATCCGCG
【0040】
(2) PAX3(下線付き大文字はPAX3のエクソン配列(遺伝子の転写領域):エクソン1)(配列番号2)
cgcagcgcgggtccccctcggggccagcagaggcctcggcaccaccagagatgggaagagaaagtggtcgctgttgcccaatcagcgcgtgtctccgccacccgggacggtctacccgtcggccaatcgcagctcagggctcctgaccaagctttgggtaaaagaactaataaatgctcccgagcccggatccccgcACTCGGTGTCACCACAGGAGGAGACTCAGGCAGGCCGCGCTCCAGCCTCACCAGGCTCCCCGGCTCGCCGTGGCTCTCTGAGCCCCCTTTTCAGGGACCCCAGTCGCTGGAACATTTGCCCAGACTCGTACCAAACTTTTCCGCCCTGGGCTCGGGATCCTGGACTCCGGGGCCTCCCCGTCCTCCCCTTTCCCGGGTTCCAGCTCCGGCCTCTGGACTAGGAACCGACAGCCCCCCTCCCCGCGTCCCTCCCTCTCTCTCCAGCCGTTTTGGGGAGGGGCTCTCCACGCTCCGGATAGTTCCCGAGGGTCATCCGCGCCGCACTCGCCTTTCCGTTTCGCCTTCACCTGGATATAATTTCCGAGCGAAGCTGCCCCCAGGATGACCACGCTGGCCGGCGCTGTGCCCAGGATGATGCGGCCGGGCCCGGGGCAGAACTACCCGCGTAGCGGGTTCCCGCTGGAAGgtaagggagggcctcagcgcgccgcctggatcccagggcctgggaccggctgcctcaccccatccccaggctccgcaggctcctttggtgcttccaggaagcccattccctgggcaccccacaccccaagaagcaccagtcgggggcgaggacctactcgatttcctttctgcaaatggagcgcgctgctctctgcaaatcctggcggagctgggcggtcaggcctgcggcgagccggggagacttgtccctgttgttttggaaatgccgcggtggagaaggaggtgaaccggcttctgagcg
【0041】
(3) p16(下線付き大文字はp16のエクソン配列(遺伝子の転写領域):エクソン1)(配列番号3)
cgcggaggaaggaaacggggcgggggcggatttctttttaacagagtgaacgcactcaaacacgcctttgctggcaggcgggggagcgcggctgggagcagggaggccggagggcggtgtggggggcaggtggggaggagcccagtcctccttccttgccaacgctggctctggCGAGGGCTGCTTCCGGCTGGTGCCCCCGGGGGAGACCCAACCTGGGGCGACTTCAGGGGTGCCACATTCGCTAAGTGCTCGGAGTTAATAGCACCTCCTCCGAGCACTCGCTCACGGCGTCCCCTTGCCTGGAAAGATACCGCGGTCCCTCCAGAGGATTTGAGGGACAGGGTCGGAGGGGGCTCTTCCGCCAGCACCGGAGGAAGAAAGAGGAGGGGCTGGCTGGTCACCAGAGGGTGGGGCGGACCGCGTGCGCTCGGCGGCTGCGGAGAGGGGGAGAGCAGGCAGCGGGCGGCGGGGAGCAGCATGGAGCCGGCGGCGGGGAGCAGCATGGAGCCTTCGGCTGACTGGCTGGCCACGGCCGCGGCCCGGGGTCGGGTAGAGGAGGTGCGGGCGCTGCTGGAGGCGGGGGCGCTGCCCAACGCACCGAATAGTTACGGTCGGAGGCCGATCCAGgtgggtagagggtctgcagcgggagcaggggatggcgggcgactctggaggacgaagtttgcaggggaattggaatcaggtagcgcttcgattctccg
【0042】
検体は、患者のGIST腫瘍組織、その組織浸出液などであり、手術時に、あるいは内視鏡検査時に採取することが可能であり、得られた該組織からフェノール/クロロホルム法などの公知の手法によるか又は市販キットを用いてゲノムDNAを抽出する。
【0043】
MCA(methylated CpG island amplification)法とマイクロアレイ(microarray)分析を組み合わせたMCAM解析法を用いた多数の遺伝子プロモーター領域のDNAメチル化の網羅的解析とパイロシークエンス法等によるDNAメチル化の定量的解析により、GISTの悪性度におけるDNA異常メチル化の関与を調べる。MCA産物を階層型クラスター法で解析すると、予後不良群と予後良好群の2つのクラスターを形成する。
【0044】
GISTの悪性度を分類するために、検体採取時にすでに再発・転移/浸潤をきたしているものを予後不良群、転移・再発/浸潤をきたしていないものを予後良好群と定義する。また、予後良好群の症例は、臨床的に予後予測因子とされている腫瘍径と腫瘍細胞の分裂像から、転移・再発/浸潤をきたしていないために予後良好群に分類されるか否かがはっきりしないものを中間群に分類し、中間群には、対象遺伝子のメチル化の程度に応じてメチル化のより高い方から高リスク及び中リスクが含まれる。メチル化の程度の低い、いわゆる低リスクは、明らかに予後良好群に分類される。
【0045】
遺伝子のDNAメチル化を解析する方法には、バイサルファイトシークエンス法、メチル化特異的PCR(MSP; Methylated Specific PCR))法、定量的MSP法、COBRA(Combined Bisulfite Restriction Analaysis)法、パイロシークエンス法などの方法があり、それぞれ解析範囲、感度、精度、必要な機器が大きく異なる。これらは全てバイサルファイト処理によってメチル化していないシトシンのみをウラシルに変換するという原理を利用している(牛島俊和ら編、エピジェネティクス実験プロトコール、羊土社(2008年))。
【0046】
以下にDNAメチル化測定法の例を示す。
(a) パイロシークエンス法:
ポリメラーゼ塩基伸長反応によるルシフェラーゼ発光反応を検出することで塩基配列を解読する。伸長反応の際に生成されるピロリン酸と発光強度が完全に比例関係にあるため高精度な定量解析を行うことができる。この原理により、バイサルファイト(bisulfite)処理を行ったDNAのCpGサイトのT%/C%を解読してDNAメチル化比率を決定する(Alliance Biosystems)。
【0047】
(b) バイサルファイトシークエンス法:
DNAをバイサルファイト処理することによってDNA中のメチル化されていないシトシン(非メチル化シトシン)はウラシルに変換される。一方、メチル化シトシンはバイサルファイト処理で変換されない。それゆえ、このような処理によってメチル化シトシンと非メチル化シトシンを区別できるので、PCR増幅したのち、塩基配列を決定するとか、制限酵素処理などの手法によってDNAメチル化を解析することができる(特表2002-535998号公報)。
【0048】
PCR増幅は、PCRバッファー中、dNTP mixure、プライマー、DNA、耐熱性DNAポリメラーゼの存在下で行う。耐熱性DNAポリメラーゼは、TaKaRa Taq HS(宝酒蔵)などの市販の酵素を使用できる。PCR条件は、例えば95〜98℃、5〜60秒の変性反応、50〜55℃、30〜60秒のアニーリング反応、及び72℃、20秒〜60秒の伸長反応を1サイクルとし、これを30〜40サイクル行う。
【0049】
(c) メチル化特異的PCR(MSP)法:
DNAをバイサルファイト変換し、このDNAを鋳型にして、メチル化DNAを特異的に増幅するプライマーと、非メチル化DNAを特異的に増幅するプライマーを用いて別々にPCRを行い、PCR産物の有無によりDNAメチル化の有無を判定する(特表2002-535998号公報)。
【0050】
(d) 定量的MSP法:
MSP法は、methylation specific PCR技術であり、Na bisulfiteで処理したDNAを、目的遺伝子内部のメチル化領域及び非メチル化領域に特異的な予想配列のプライマーを用いてPCR増幅することを基本としている(上記(c))。
【0051】
複数の蛍光団(fluorophore)を使用することによってリアルタイム定量MSPを行うことが可能であり、この場合メチル化領域特異的プライマーと非メチル化領域特異的プライマーを使用する(T. Swift-Scanlan et al., BioTechniques 40:210-219, 2006)。
【0052】
(e) COBRA法:
バイサルファイト処理後にメチル化DNAと非メチル化DNAに共通のプライマーを用いて目的のDNAをPCR増幅後、メチル化DNAと非メチル化DNAで配列が異なる箇所を認識する制限酵素で処理し、断片をエタノール沈殿して濃縮し、電気泳動でメチル化DNAと非メチル化DNAを識別できる。PCR増幅は、バイサルファイトシークエンス法と同様に行うことができる(Xiong, Z. and Laird, P.W. Nucleic Acids Res. 25(1):2532-2534, 1997)。
【0053】
本発明の方法で使用可能なPAX3遺伝子、REC8遺伝子、及びp16遺伝子のそれぞれのパイロシークエンスプライマーセットの例は、以下のとおりである。ここで、Fはフォワードプライマー、Rはリバースプライマー、bioは5’末端のビオチン修飾であり、これらのそれぞれの遺伝子のプライマーセットのうち1つもしくは2つ以上のセット〜全セットを用いてPCR増幅を行うことができる。seqはパイロシークエンスに用いるシークエンスプライマーを示す。以下に示す塩基配列を順番に配列番号4から配列番号273として表す((注)同一のプライマー配列に対し複数の配列番号が割付けられる場合もある。)。
【0054】
<PAX3遺伝子>
F TAGAGATGGGAAGAGAAAGTGG (配列番号4)
R bio bio-ACCTACCTAAATCTCCTCCTATAA(配列番号5)
seq GGTAAAAGAATTAATAAATG(配列番号6)
F TTAGAGATGGGAAGAGAAAGTGG(配列番号7)
R bio bio-CCTACCTAAATCTCCTCCTATAAT(配列番号8)
seq GGTAAAAGAATTAATAAATG(配列番号9)
F TTAGAGATGGGAAGAGAAAGTGGT(配列番号10)
R bio bio-CCTACCTAAATCTCCTCCTATAA(配列番号11)
seq GGTAAAAGAATTAATAAATG(配列番号12)
F ATTTTTTTTTTTGGGTAAGGGG (配列番号13)
R bio bio-ACCTACCTAAATCTCCTCCTATAA(配列番号14)
seq TAAAAGAATTAATAAATGTT(配列番号15)
F GGTTGTTTTTTTTAGTTTTTAT (配列番号16)
R bio bio-AAAACATTTATTAATTCTTTTACC(配列番号17)
seq GTGGTYGTTGTTGTTTAATT(配列番号18)
F TTTTTAGTAATTGTTATTTTTT (配列番号19)
R bio bio-ACCCAAAACTTAATCAAAAACCCT(配列番号20)
seq GTGGTAGTTGTTGTTTAATT(配列番号21)
F GAGATAGAGAGATAGGAATTTT (配列番号22)
R bio bio-CACTTTCTCTTCCCATCTCTAATA(配列番号23)
seq TTTTTTTTTTTGGGTAAGGG(配列番号24)
F AGAGATGGGAAGAGAAAGTGG(配列番号25)
R bio bio-ATTCCTAATCCAAAAACC (配列番号26)
seq TTGGAATATTTGTTTAGA(配列番号27)
F TATAGGAGGAGATTTAGGTAG(配列番号28)
R bio bio-ATTCCTAATCCAAAAACC(配列番号29)
seq GTTGGAATATTTGTTTAGAT(配列番号30)
F GTTTTGGGGAGGGGTTTTTTA(配列番号31)
R bio bio-TACCCCCAACCCCACCCC (配列番号32)
seq GTTTTTATTTGGATATAATT(配列番号33)
F TTTTTTTTTTTTTYGGGTTTTAGT(配列番号34)
R bio bio-AATCATCCTAAAAACAACTTC(配列番号35)
seq GTTTTTATTTGGATATAATT(配列番号36)
F GATTAGTATTAAATTTTTTAGTTT(配列番号37)
R bio bio-AATCATCCTAAAAACAACTTC(配列番号38)
seq TGGGGAGGGGTTTTTTAAGT(配列番号39)
F GTAGTTTTGGGGAGGGGTTTTTTA(配列番号40)
R bio bio-AATCATCCTAAAAACAACTTC(配列番号41)
seq GTTTTTATTTGGATATAATT(配列番号42)
F bio bio-AATTGTTATTTTTTTGGGGTTGT(配列番号43)
R CACTTTCTCTTCCCATCTCTAATA(配列番号44)
seq CTTCCCATCTCTAATAATAC(配列番号45)
F bio bio-GGGTTGTTTTTTTTAGTTTTTAT(配列番号46)
R AACTTAATCAAAAACCCTAAACTA(配列番号47)
seq CTTCCCATCTCTAATAATAC(配列番号48)
F bio bio-AATTTTTTTTTTTGGGTAAGGGG(配列番号49)
R AAACATTTATTAATTCTTTTACCC(配列番号50)
seq CTTCCCATCTCTAATAATAC(配列番号51)
F bio bio-ATTAGAGATGGGAAGAGAAAGTGG(配列番号52)
R TCTTTTACCCAAAACTTAATCAAA(配列番号53)
seq ACTTAATCAAAAACCCTAAA(配列番号54)
F bio bio-GTATTATTAGAGATGGGAAGAGAA(配列番号55)
R AAACATTTATTAATTCTTTTACCC(配列番号56)
seq ACTTAATCAAAAACCCTAAA (配列番号57)
F bio bio-AAGTGGTYGTTGTTGTTTAATTAG(配列番号58)
R CCTAAATCTCCTCCTATAATAACA(配列番号59)
seq ACTTAATCAAAAACCCTAAA(配列番号60)
F bio bio-GAGTTTTTTTTTTAGGGATTTTAG(配列番号61)
R TAATCATCCTAAAAACAACTTC(配列番号62)
seq AAAATTATATCCAAATAAAA(配列番号63)
F bio bio-GTTTAGGGTTTTTGATTAAGTTT(配列番号64)
R AATCCCTAAAAAAAAAACTCAAA(配列番号65)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号66)
F bio bio-GGGTAAAAGAATTAATAAATGTT(配列番号67)
R ACRAATCTAAACAAATATTCCAA(配列番号68)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号69)
F bio bio-TAGTTTAGGGTTTTTGATTAAGT(配列番号70)
R AATACRAATCTAAACAAATATTC(配列番号71)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号72)
F bio bio-GATTAAGTTTTGGGTAAAAGAAT(配列番号73)
R ACCRAAAAACCTAATAAAACTAA(配列番号74)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号75)
F bio bio-TTTAGGGTTTTTGATTAAGTTTT(配列番号76)
R AAAACCTAATAAAACTAAAACRC(配列番号77)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号78)
F bio bio-AATTGTTATTTTTTTGGGGTTGT(配列番号79)
R CACTTTCTCTTCCCATCTCTAATA(配列番号80)
seq AAACCTCTACTAACC(配列番号81)
F bio bio-TTAATTTTTTTTTTTGGGTAAGG(配列番号82)
R TCTCTTCCCATCTCTAATAATACC(配列番号83)
seq AAACCTCTACTAACC(配列番号84)
F bio bio-GTTTTTAGTAATTGTTATTTTTT(配列番号85)
R AATTAAACAACAACCACCACTTTC(配列番号86)
seq AAACCTCTACTAACC(配列番号87)
F bio bio-GAGATAGAGAGATAGGAATTTTT(配列番号88)
R ACTTTCTCTTCCCATCTCTAATAA(配列番号89)
seq AAACCTCTACTAACC(配列番号90)
F bio bio-GAGATAGAGAGATAGGAATTTTT(配列番号91)
R AACTTAATCAAAAACCCTAAACTA(配列番号92)
seq AAACCTCTACTAACC(配列番号93)
【0055】
<REC8遺伝子>
F TGTTGGTTGTTTYGGGAGATTTT(配列番号94)
R bio bio-TCAAACCCCTAAACCTTACACTAA(配列番号95)
seq ATTTTGTTGTTAGGATATTT(配列番号96)
F TTATGATTGGTTTGTTGGTTGTT(配列番号97)
R bio bio-CCCTAAACCTTACACTAACTTAAA(配列番号98)
seq ATTTTGTTGTTAGGATATTT(配列番号99)
F GGAATATTTTATAATGGATTTTA(配列番号100)
R bio bio-ATATCAAACCCCTAAACCTTACAC(配列番号101)
seq ATTTTGTTGTTAGGATATTT(配列番号102)
F GGAGATTTTGTTGTTAGGATATT(配列番号103)
R bio bio-ACCATATCCTCCACAACAAACCTC(配列番号104)
seq TTATTGGTTGTTAGGGTAAA(配列番号105)
F AGATTTTGTTGTTAGGATATTTG(配列番号106)
R bio bio-AAACCCCTAAACCTTACACTAACT(配列番号107)
seq ATTGGTTGTTAGGGTAAAAG(配列番号108)
F TTTTTTATTGGTTGTTAGGGTAA(配列番号109)
R bio bio-TATCAAACCCCTAAACCTTACACT(配列番号110)
seq ATTGGTTGTTAGGGTAAAAG(配列番号111)
F TTTTYGTTTTTTATTGGTTGTTA(配列番号112)
R bio bio-AAACCTTACACTAACTTAAACRAC(配列番号113)
seq ATTGGTTGTTAGGGTAAAAG(配列番号114)
F TTTTATTGGTTGTTAGGGTAAAA(配列番号115)
R bio bio-CACRATATCCTCCACAACAAACCT(配列番号116)
seq GAYATTTAGTTTTGTAGTAG(配列番号117)
F TATTTTATAATGGATTTTAGAGG(配列番号118)
R bio bio-CCTCCACAACAAACCTCRATCCTC(配列番号119)
seq GYGATTTAGTTTTGTAGTAG(配列番号120)
F AGATTTTGTTGTTAGGATATTTG(配列番号121)
R bio bio-TAAAAATACAAAACTCCAAAAAAA(配列番号122)
seq GTGTAAGGTTTAGGGGTTTG(配列番号123)
F TTTAAGTTAGTGTAAGGTTTAGG(配列番号124)
R bio bio-TCCAAATTAAAAATACAAAACTCC(配列番号125)
seq GTGTAAGGTTTAGGGGTTTG(配列番号126)
F AAGTTAGTGTAAGGTTTAGGGGT(配列番号127)
R bio bio-TTAAAACTTCTAAATTAAATTCCA(配列番号128)
seq TAAGGTTTAGGGGTTTGATA(配列番号129)
F TAGTGTAAGGTTTAGGGGTTTGA(配列番号130)
R bio bio-ACACAAAATTCCCAACACCTTAA(配列番号131)
seq TTTAGAAGTTTTAAGTTTG(配列番号132)
F TTTAAGTTAGTGTAAGGTTTAGG(配列番号133)
R bio bio-TCTCTAATAACAAAATAAATACA(配列番号134)
seq TTTAGAAGTTTTAAGTTTG(配列番号135)
F GAGGTTTGTTGTGGAGGATAT(配列番号136)
R bio bio-CAATAAACAAATACACAATTACC(配列番号137)
seq ATTTAGAAGTTTTAAGTTT(配列番号138)
F TTTTTTGGAGTTTTGTATTTT(配列番号139)
R bio bio-CACAATTACCAAAATAACTAAAA(配列番号140)
seq ATTTAGAAGTTTTAAGTTT(配列番号141)
F TTTTAATTTGGAATTTAATTT(配列番号142)
R bio bio-CCTCTCTATAAACCCTCAATAAA(配列番号143)
seq ATTTAGAAGTTTTAAGTTT(配列番号144)
F TATTTTTAATTTGGAATTTAA(配列番号145)
R bio bio-AATAACTAAAACRCAAAAAATAA(配列番号146)
seq ATTTAGAAGTTTTAAGTTT(配列番号147)
F GTTTTGTATTTTTAATTTGGA(配列番号148)
R bio bio-AAAAAACCAAATTAATAAAACAA(配列番号149)
seq ATTTAGAAGTTTTAAGTTT(配列番号150)
F bio bio-AGTGTAAGGTTTAGGGGTTTGAT(配列番号151)
R CCCAACACCTTAAAAAAAAATCA(配列番号152)
seq TCTATAAACCCTCAATAAAT(配列番号153)
F bio bio-AATTTGGAATTTAATTTAGAAGT(配列番号154)
R TAACAAAATAAATACACCTAAAA(配列番号155)
seq TCTATAAACCCTCAATAAAT(配列番号156)
F bio bio-GGAATTTAATTTAGAAGTTTTAA(配列番号157)
R AACAATAAACAAATACACAATTA(配列番号158)
seq TCTATAAACCCTCAATAAAT(配列番号159)
F bio bio-TGTAAGGTTTAGGGGTTTGATAT(配列番号160)
R CCCCTCTCTATAAACCCTCAATA(配列番号161)
seq AAATACAAAACTCCAAAAAA(配列番号162)
F bio bio-TTATTGGTTGTTAGGGTAAAAGT(配列番号163)
R ATATCAAACCCCTAAACCTTACA(配列番号164)
seq TAAACCTTACACTAACTTAA(配列番号165)
F bio bio-GGGTAAAAGTYGTTATTTAATGA(配列番号166)
R TAAAAATACAAAACTCCAAAAAA(配列番号167)
seq TAAACCTTACACTAACTTAA(配列番号168)
F bio bio-AAGAGGTTTAGTATAGTTGTTTT(配列番号169)
R AATATCCTAACAACAAAATCTCC(配列番号170)
seq ACAAACCAATCATAAAAATT(配列番号171)
F bio bio-TGTAAGGTATATTTTGGTATT(配列番号172)
R AACAACCAACAAACCAATCATAA(配列番号173)
seq ACAAACCAATCATAAAAATT(配列番号174)
F bio bio-GTTTGTGGATTGGGGGAAG(配列番号175)
R AAACAACCAACAAACCAATCATA(配列番号176)
seq CCCTTCCTTAACCAATAAA(配列番号177)
F bio bio-GGGAGATTTTGTTGTTAGGATAT(配列番号178)
R TCAAACCCCTAAACCTTACACTAA(配列番号179)
seq TACCCTAACAACCAATAAA(配列番号180)
F bio bio-TAAGGTATATTTTGGTATT(配列番号181)
R TAAAATCCATTATAAAATATTCC(配列番号182)
seq CCCTTCCTTAACCAATAAA(配列番号183)
【0056】
<p16遺伝子>
F GGGTAGGTGGGGAGGAGTTTAG (配列番号184)
R bio bio-ACCAACCAACCCCTCCTCTTT(配列番号185)
seq GGAGTTAATAGTATTTT(配列番号186)
F GTGTGGGGGGTAGGTGGGGAGG (配列番号187)
R bio bio-CTATCCCTCAAATCCTCTAAA(配列番号188)
seq GGAGTTAATAGTATTTT(配列番号189)
F GGGGGAGATTTAATTTGGGG(配列番号190)
R bio bio-TATCTTTCCAAACAAAAAAAC(配列番号191)
seq GGAGTTAATAGTATTTT(配列番号192)
F GGGGGAGATTTAATTTGGGG(配列番号193)
R bio bio-CAACCAACCCCTCCTCTTTCT(配列番号194)
seq GGAGTTAATAGTATTTT(配列番号195)
F AAAGAGGAGGGGTTGGTTGG(配列番号196)
R bio bio-CTACCTACTCTCCCCCTCTCC(配列番号197)
seq GGTTGGTTATTAGAGGGT(配列番号198)
F GAGGAAGAAAGAGGAGGGGT(配列番号199)
R bio bio-AAAACTCCATACTACTCCCC(配列番号200)
seq TTGGTTATTAGAGGGTGG(配列番号201)
F AAAGAGGAGGGGTTGGTTGG(配列番号202)
R bio bio-ACCCTCTACCCACCTAAATC(配列番203)
seq TGGTTATTAGAGGGTGGG(配列番号204)
F GGGTTGGTTGGTTATTAGAG(配列番号205)
R bio bio-CAAACCCTCTACCCACCTAA(配列番号206)
seq AGAGGGGGAGAGTAGGTA(配列番号207)
F GTTGGTTATTAGAGGGTGGG(配列番号208)
R bio bio-AATCAACCRAAAACTCCATA(配列番号209)
seq AGAGGGGGAGAGTAGGTA(配列番号210)
F AATTAAGGGTTGAGGGGGTAGGG(配列番号211)
R bio bio-AAAAAACTAAACTCCTCCCCACCT(配列番号212)
seq TTTGGTAGTTAGGAAGGTT(配列番号213)
F TTATAATTAGGAAAGAATAGTTT(配列番号214)
R bio bio-TTCACTCTATTAAAAAAAAAT(配列番号215)
seq TTTGGTAGTTAGGAAGGTT(配列番号216)
F TATTTTTTTTATGATATTAAATA(配列番号217)
R bio bio-AATACRTTCACTCTATTAAAA(配列番号218)
seq TTTGGTAGTTAGGAAGGTT(配列番号219)
F GGGTTTTTATAATTAGGAAAGAA(配列番号220)
R bio bio-TTTAAATACRTTCACTCTATT(配列番号221)
seq TTTGGTAGTTAGGAAGGTT(配列番号222)
F ATTTAATTTGGTAGTTAGGAAGG(配列番号223)
R bio bio-TCCCCCRCCTACCAACAAAAA(配列番号224)
seq GGTAGTTAGGAAGGTTGTA(配列番号225)
F ATTTGGTAGTTAGGAAGGTTGTA(配列番号226)
R bio bio-CTCCCCACCTACCCCCCACAC(配列番号227)
seq TTTTTTAATAGAGTGA(配列番号228)
F bio bio-GGGTAGGTGGGGAGGAGTTTA(配列番号229)
R ACCAACCAACCCCTCCTCTTT(配列番号230)
seq CCCTCCTCTTTCTTCCT(配列番号231)
F bio bio-TTTTAGAGGATTTGAGGGATA(配列番号232)
R CCCCACCCTCTAATAACCAAC(配列番号233)
seq CCCTCCTCTTTCTTCCT(配列番号234)
F bio bio-AGAGGATTTGAGGGATAGGGT(配列番号235)
R CTAATAACCAACCAACCCCTC(配列番号236)
seq CCCTCCTCTTTCTTCCT(配列番号237)
F bio bio-TTTTTTTGTTTGGAAAGATAT(配列番号238)
R ATAACCAACCAACCCCTCCTC(配列番号239)
seq CCCTCCTCTTTCTTCCT(配列番号240)
F bio bio-GAAGAAAGAGGAGGGGTTGG(配列番号241)
R ACAAACCCTCTACCCACCTAAA (配列番号242)
seq CTACCTACTCTCCCCCT(配列番号243)
F bio bio-TTGGTTGGTTATTAGAGGGT(配列番号244)
R AAAACTCCATACTACTCCCC(配列番号245)
seq CTACCTACTCTCCCCCT(配列番号246)
F bio bio-AGAGGATTTGAGGGATAGGG(配列番号247)
R CCRACTCCATACTACTCCCC(配列番号248)
seq CTACCTACTCTCCCCCT(配列番号249)
F bio bio-GGTAGTTAGGAAGGTTGTAT(配列番号250)
R CCCCAAATTAAATCTCCCCC(配列番号251)
seq CACCTACCCCCCACA(配列番号252)
F bio bio-TTTTATGATATTAAATATTT(配列番号253)
R ACTAAACTCCTCCCCACCTA(配列番号254)
seq CACCTACCCCCCACA(配列番号255)
F bio bio-GAAGAAGTTATATTTTTTTT(配列番号256)
R AAACCAACRTTAACAAAAAA(配列番号257)
seq CACCTACCCCCCACA(配列番号258)
F bio bio-TGAGGGGGTAGGGGGATATT(配列番号259)
R CAACCTTCCTAACTACCAAATTAA(配列番号260)
seq TTCCTAATTATAAAAACCC(配列番号261)
F bio bio-AATTAAGGGTTGAGGGGGTA(配列番号262)
R TCATAAAAAAAATATAACTTCTTC(配列番号263)
seq TTCCTAATTATAAAAACCC(配列番号264)
F bio bio-TGAGGGGGTAGGGGGATATT(配列番号265)
R AACAAAACTATTCTTTCCTAATTA(配列番号266)
seq TTCCTAATTATAAAAACCC(配列番号267)
F bio bio-GGGTTTTTGATTTAGTGAAT(配列番号268)
R AAAAATATAACTTCTTCTTTTAAT(配列番号269)
seq TTCCTAATTATAAAAACCC(配列番号270)
F bio bio-GTTTTTGAAAATTAAGGGTT(配列番号271)
R AACCTTCCTAACTACCAAATTAAA(配列番号272)
seq TTCCTAATTATAAAAACCC(配列番号273)
【0057】
さらにまた、MSP法を用いても、メチル化陽性の判定は可能である。PAX3遺伝子、REC8遺伝子、及びp16遺伝子のそれぞれのメチル化特異的プライマーセット及び非メチル化特異的プライマーセットの例は、以下のとおりであるが、これらに限定されない。
【0058】
<PAX3遺伝子>
メチル化特異的プライマー (増幅産物の長さ(Product length) 114bp)
forward: TTCGGATAGTTTTCGAGGGTTA(配列番号274)
reverse:CCGACCAACGTAATCATCCT(配列番号275)
メチル化特異的プライマー (Product length 139bp)
forward: AGGGTTATTTGCGTCGTATTC(配列番号276)
reverse:AATAATTCTACCCCGAACCCG(配列番号277)
メチル化特異的プライマー (Product length 103bp)
forward: AGGGTTATTTGCGTCGTATTC(配列番号278)
reverse:AAACACAACGCCGACCAAC(配列番号279)
非メチル化特異的プライマー(増幅産物の長さ121bp)
forward: TTGGATAGTTTTTGAGGGTTATTTG(配列番号280)
reverse:TCCTAAACACAACACCAACCA(配列番号281)
【0059】
<REC8遺伝子>
メチル化特異的プライマー (増幅産物の長さ131bp)
forward: GCGTTTTCGATTCGATTTTC(配列番号282)
reverse:CCGAACCGCCTACTACAAAA(配列番号283)
非メチル化特異的プライマー(Product length 105bp)
forward: TTGGTTGTTAGGGTAAAAGTTGTT(配列番号284)
reverse:CCCCAAACCACCTACTACAAA(配列番号285)
【0060】
<p16遺伝子>
メチル化特異的プライマー (増幅産物の長さ150bp)
forward: TTATTAGAGGGTGGGGCGGATCGC(配列番号292)
reverse:GACCCCGAACCGCGACCGTAA(配列番号293)
非メチル化特異的プライマー(Product length 151bp)
forward: TTATTAGAGGGTGGGGTGGATTGT(配列番号294)
reverse:CAACCCCAAACCACAACCATAA(配列番号295)
(文献:Y. Kondo et al., Mol Cell Biol, 23(1):206-15 (2003))
【0061】
判定:
上記のDNAメチル化測定法を用いて得られた結果に基づいてGIST患者の予後、あるいはGISTの悪性化の程度、を予測することができる。本発明では、食道、胃、小腸、大腸などの消化管のGIST組織についてREC8遺伝子又はPAX3遺伝子あるいはその両方の遺伝子、のメチル化の有無を調べる(又は、検査する)ことによって、そして好ましくはREC8遺伝子及びPAX3遺伝子の両方のメチル化の有無、さらに好ましくはREC8遺伝子及びPAX3遺伝子並びにp16遺伝子のメチル化の有無を調べる(又は検査する)ことによって、上記の予測を行うことができる。
【0062】
予後や悪性度の程度は、予後不良群、中間群及び予後良好群に分類することができる。予後不良群は、転移や再発がある群であり、REC8遺伝子及び/又はPAX3遺伝子、好ましくはREC8遺伝子及びPAX3遺伝子、より好ましくはREC8遺伝子、PAX3遺伝子及びp16遺伝子、が相対的に高頻度でメチル化陽性である。中間群は、今のところ転移や再発はないがそのリスクのある、中リスク群と高リスク群に分類され、上記遺伝子類のメチル化陽性の相対的頻度は予後不良群よりかなり低い。ここで、高リスク群は、中リスク群と比べてメチル化陽性の相対的頻度が高く、予後不良となるリスクを有している。逆に、中リスク群は、高リスク群と比べてメチル化陽性の相対的頻度が低く、予後不良となるリスクがより低い。予後良好群(又は低リスク)では、上記遺伝子類のメチル化陽性頻度は極めて低く、ほとんどメチル化陰性である。
【0063】
ここで、メチル化陽性の頻度は、予後不良群、中間群(高リスクと中リスク)及び予後良好群の間でのメチル化陽性割合の大きさを指し、その割合が大きいほど高頻度であり、割合が小さいほど低頻度である。メチル化陽性と判定されたとき、予後不良群、中間群(高リスクと中リスク)のいずれかに分類される。すなわち、例えばREC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化をGIST検体で調べた場合、3つの遺伝子のうち1つ以上が陽性である場合、そのGISTは感度80%、特異度92%で中間群以上の悪性度を示すこと、また3つの遺伝子のうち1つ以上が陽性である症例群は、3つの遺伝子のいずれにもメチル化が認めない症例群と比較し、生命予後が有意に悪いことを示す(図4)。
【0064】
4.キット
本発明はさらにGISTを検査するためのキットを提供する。
該キットは、REC8遺伝子、PAX3遺伝子及びp16遺伝子のそれぞれの、パイロシークエンス法に使用する少なくとも1つの、好ましくは複数の上記プライマーセット、あるいは、MSP法に使用する少なくとも1つの上記メチル化特異的プライマーセット及び、場合により、非メチル化特異的プライマーセットを含み、これに加えてバイサルファイト試薬、PCR反応試薬ならびに使用説明書などを含むことができる。
【0065】
PAX3遺伝子、REC8遺伝子、及びp16遺伝子のそれぞれのパイロシークエンスプライマーセットの例は、上記の配列番号4から配列番号273までの配列の各セット(但し、ビオチン修飾はあってもよいし、なくてもよい。)であり、本発明のキットには、各遺伝子あたり、少なくとも1つのセットが含まれる。
【0066】
また、MSP法を用いてメチル化陽性の判定を行うための、PAX3遺伝子、REC8遺伝子、及びp16遺伝子のそれぞれのメチル化特異的プライマー及び非メチル化特異的プライマーの例は、PAX3遺伝子について上記の配列番号274から配列番号281の配列の少なくとも1つのセット、REC8遺伝子について配列番号282から配列番号285の配列の少なくとも1つのセット、及びp16遺伝子については、配列番号292から配列番号295の配列の少なくとも1つのセットである。
【0067】
バイサルファイト試薬は非メチル化シトシンをウラシルに変換するための試薬であり、この試薬には、重亜硫酸ナトリウム(Na bisulfite)、スカベンジャー(ハイドロキノン等)などを含むことができる。
PCR反応試薬には、dNTP mixture、PCRバッファー、耐熱性ポリメラーゼなどを含むことができる。
【0068】
上記プライマー及び上記試薬の他に、DNA抽出及び制限消化のための、フェノール/クロロホルム溶液又はフェノール/クロロホルム/イソアミルアルコール溶液、制限酵素などを含むことができる。
使用説明書には、DNAメチル化検査のためのプロトコールなどが記載されうる。
【実施例】
【0069】
以下に実施例によって本発明をさらに具体的に説明するが、本発明の技術的範囲はこれらの実施例によって制限されないものとする。
【0070】
[実施例1]
GISTの臨床検体(トレイニングセット40例)を用いて解析を行った。これらの臨床検体は、胃GIST(23例)、小腸GIST(15例)、直腸GIST(2例)の無作為に抽出した40例である。臨床検体からDNAを抽出した後、制限酵素のイソシゾマー性質を利用した、methylated CpG island amplification (MCA)法を用いて、メチル化しているDNA断片を回収し、カスタムアレイで解析した。
【0071】
GISTはこれまでの診断基準により大きさと細胞分裂像より、転移・再発のリスクを高リスク、中リスク、低リスクに分類される。これらのリスク分類では、既に転移・再発をきたしているGISTは含まれない。既に転移・浸潤をきたしているものを予後不良群(18例)、転移・浸潤がなく低リスクに分類されるものを予後良好群(9例)としてアレイ解析を行った。予後不良群で特異的にメチル化をしている155遺伝子を見出した。(図1)。アレイ解析に用いたアレイは、アジレント社のカスタムアレイ(G4497A)を用いた(文献:Y. Goto et al., Cancer Res, 69(23):9073-82 (2009))。
【0072】
[実施例2]
トレイニングセットでアレイより見出した、予後不良群に高頻度にメチル化する155遺伝子を用いて、決定木法による解析を行った。予後不良群と予後良好群を最も的確に分類する2遺伝、REC8遺伝子、PAX3遺伝子を同定した(図2)。
【0073】
アレイから選び出したREC8遺伝子、PAX3遺伝子の有用性について定量的メチル化法(パイロシークエンス法)を用いてトレイニングセット40例(予後良好群(低リスク群)9例、中間群(中リスク群・高リスク群)13例、予後不良群18例)で確認した(図3)。パイロシークエンス法は、高精度な定量解析能を有した塩基配列の解読装置を用いて行った。臨床検体から抽出したDNAをバイサルファイト処理後、PCR法により目的とする領域(REC8遺伝子、PAX3遺伝子およびp16遺伝子のプロモーター領域)の増幅を表1のプライマーセット(forwardとreverse)を用いて行った。増幅されたPCR産物を精製後、ビオチン(Bio-)標識されたDNA鎖の塩基配列を、シークエンスプライマー(sequence, 表1)を用いてパイロシークエンス法により解読した(文献:S. Colella et al., Biotechniques, 35(1):146-50 (2003))。
【0074】
さらに、REC8遺伝子、PAX3遺伝子の有用性を検証するために、トレイニングセットとは重複しない検証セット75例でメチル化頻度を検討した(図3)。
【0075】
その結果、検証セットGIST 75症例は次のように分類された。
予後良好群(低リスク群)24例
中間群(中リスク群・高リスク群)36例
予後不良群15例
【0076】
これらの症例のREC8遺伝子とPAX3遺伝子に加えて、p16遺伝子のメチル化も解析した。p16遺伝子のメチル化とGIST症例の予後(生存期間)との関連はこれまで報告されている(R. Ricci et al., Role of p16/INK4a in gastrointestinal stromal tumor progression, Am J Clin Pathol 2004;122:35-43)。
【0077】
これらのメチル化様式をパイロシークエンス法を用いて解析し、3遺伝子のうち1遺伝以上にメチル化を認める症例を陽性とした。パイロシークエンス法で計測されるサンプル内の目的とするCpGサイトのメチル化測定値(メチル化レベル)が15%以上を示す場合をメチル化陽性と判断した。この値(15%)はパイロシークエンス法の非特異的な測定値(ノイズ)では通常得られない測定値としての最小値である。
【0078】
上記及び図3でREC8遺伝子、PAX3遺伝子及びP16遺伝子のメチル化解析に用いたプライマー配列等を表1に示す。
【0079】
【表1】

【0080】
その結果、予後良好群、中間群、予後不良群ではそれぞれトレイニングセットでは11%、38%、83%の症例で陽性であり、検証セットではそれぞれ13%、69%、77%の症例で陽性であった。また、中間群においては、中リスクに比較して高リスクで有意に多くの症例で陽性症例が認められた(中リスク43%(9/21)対高リスク75% (21/28); P=0.038)。検証セットで、本アッセイは予後良好群と予後不良群を感度80%、特異度92%で区別した(図3)。
【0081】
[実施例3]
解析症例の中でGIST患者の生存期間が明らかである69症例を対象に、上記と同様のパイロシークエンス法を用いてREC8遺伝子、PAX3遺伝子、p16遺伝子のメチル化状態を測定し、このメチル化結果と生存期間との関係を検討した。
【0082】
その結果、REC8遺伝子、PAX3遺伝子、p16遺伝子のうち1遺伝子でもメチル化している症例は、有意に生存期間が短かった(P=0.0338;図4)。
【産業上の利用可能性】
【0083】
本発明は、GIST患者の予後をDNAメチル化検査によって決定することを可能にするため、治療計画の策定に有用である。
【配列表フリーテキスト】
【0084】
配列番号4〜295:プライマー
【技術分野】
【0001】
本発明は、消化管間質腫瘍患者の予後予測のためのキット及び方法を提供する。
【背景技術】
【0002】
消化管間質腫瘍(以下GIST(gastrointestinal stromal tumor)は、食道、胃、小腸、大腸、直腸などの消化管に発生し、KIT遺伝子やPDGFRα遺伝子の機能獲得性突然変異を高頻度に認める悪性腫瘍であり、間葉系腫瘍のほとんどを占める。GIST患者にはメチル酸イマチニブなどのチロシンキナーゼレセプター活性阻害薬による分子標的療法が有効であるとされているが、GISTには、良性から悪性までの幅広いスペクトルがあり上記の薬剤も一部の腫瘍には有効でないことも知られている。このように、GISTは、高頻度に転移し極めて予後不良な群と、腫瘍の増大が遅く転移をきたさない予後が比較的良好な群に分けることができるが、しかしながら、その両群を規定するメカニズムはほとんど明らかになっていない。
【0003】
さらにまた、患者においてGISTの予後の良・不良を事前に判定することができるならば、上記の分子標的療法を含めた適切な治療計画を組むことが可能になると考えられる。そのための予後予測マーカーとして、 臨床病理像と予後の関係(非特許文献1)や、KITのエクソン11における変異と予後の関係(非特許文献2)が報告されているが、実用化までは至っておらず、治療方針を決定するほどの確度で信頼されうる、予後を予測できる腫瘍マーカーは確立されていない。
【0004】
そのような情況にあって、上記以外に以下のタンパク質又は遺伝子がGISTの予後予測のための腫瘍マーカーの候補として報告されている。
【0005】
特許文献1には、DDX39(UniProt Accession No.O00148)というDEAD box RNA helicaseの一種であるタンパク質の発現がGISTの術後転移症例の原発巣腫瘍組織において、非転移症例の原発巣腫瘍組織よりも有意に発現が高いこと、一方、正常細胞ではほとんど発現していないことが報告されている。
【0006】
特許文献2には、胃を原発巣とするGISTにおいて、ジペプチジルペプチダーゼIV遺伝子が発現していた場合に、その腫瘍が悪性であると判定されうることが記載されている。
【0007】
特許文献3には、フェチンというタンパク質の発現がGIST患者の予後不良と逆相関することが報告されている。
【0008】
非特許文献3には、GIST患者からの検体について多数の遺伝子のプロモーター領域のDNAメチル化の網羅的解析とパイロシークエンス法によるDNAメチル化の定量的解析を行い、悪性度の高いもので異常メチル化を示す遺伝子が存在することが報告されている。しかしながら、DNAメチル化の異常と予後又は悪性度との関係を示す遺伝子の詳細は明らかにされていないし、また、GISTにおいて異常メチル化とは何であるのか、GISTにおいて正常なメチル化とは何であるのか、そのいずれも遺伝子レベルで明確でない。
【0009】
非特許文献4には、ETV1(Ets Variant Gene 1)がすべてのGIST腫瘍及びGIST細胞系統において高度に発現していることが報告されている。
【先行技術文献】
【特許文献】
【0010】
【特許文献1】特開2009-132636号公報
【特許文献2】特開2009-011249号公報
【特許文献3】特開2008-100958号公報
【非特許文献】
【0011】
【非特許文献1】T. Hasegawa et al., Human Pathology, 33(6):669-676 (2002)
【非特許文献2】J. Andersson J. et al., Gastroenterology, 130(6):1573-1581 (2006)
【非特許文献3】第3回エピジェネティクス研究会年会(日本、2009年)(P-38)
【非特許文献4】P. Chi et al., Nature, 467:849-853 (2010)
【発明の概要】
【発明が解決しようとする課題】
【0012】
本発明の目的は、消化管間質腫瘍(GIST)をもつ患者の予後をDNAメチル化異常の検出に基づいて予測する方法を提供することである。
【課題を解決するための手段】
【0013】
本発明者らは、上記の課題を解決するために、GISTをもつ患者の予後を従来法と比べてより精確に判定することを可能にする特定の遺伝子の異常なメチル化が存在することを見出し、本発明を完成させるに至った。
【0014】
したがって、本発明は、以下の特徴を含む。
すなわち、本発明は、第1の態様において、消化管間質腫瘍をもつ患者の生物学的検体においてREC8遺伝子及びPAX3遺伝子のうちの少なくとも1つの遺伝子のメチル化の有無を測定し、該遺伝子がメチル化されている場合予後不良であり、一方メチル化されていない場合予後良好であると決定することを含む、該患者の予後をインビトロで予測する方法を提供する。
その実施形態において、上記方法は、p16遺伝子のメチル化の有無を測定することをさらに含む。
【0015】
REC8遺伝子及び/又はPAX3遺伝子のメチル化の有無と、p16遺伝子のメチル化の有無とを組み合わせることによって上記予後予測の精度をさらに高めることができる。
【0016】
すなわち、好ましくは、本発明ではREC8遺伝子及びPAX3遺伝子の両方の遺伝子のメチル化を測定する。さらに好ましくは、REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化を測定する。
【0017】
別の実施形態において、上記方法における上記遺伝子のメチル化をパイロシークエンス法又はMSP法で測定する。
【0018】
本発明は第2の態様において、REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化の有無を検出するための試薬を含むGIST検査用キットを提供する。
【0019】
その実施形態において、試薬が、REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化領域を特異的に増幅するためのプライマーである。
試薬にはさらに、バイサルファイト試薬及びPCR反応試薬を含むことができる。
【発明の効果】
【0020】
GIST患者の予後を予測するための適当な腫瘍マーカーがない情況のなかで、本発明での特定の遺伝子のメチル化の有無を測定するだけで精度よく予後予測が可能になる。
【図面の簡単な説明】
【0021】
【図1】この図は、GIST患者からの検体(GIST腫瘍組織)における155遺伝子のDNAメチル化の網羅的解析の結果を示す。赤色はメチル化陽性を表わす。
【図2】この図は、REC8遺伝子とPAX3遺伝子のメチル化の有無の測定が予後予測のために有用であることを示す。アレイでの測定結果から、決定木法により同定したREC8遺伝子とPAX3遺伝子は、GIST患者を予後不良群と予後良好群に分けることができることを示す。図中、アレイ上の遺伝子スポットは、GIST腫瘍組織DNAのメチル化(赤シグナル)とコントロール(正常組織)DNAのメチル化(緑シグナル)に由来したシグナルを発するが、これまでのアレイに関する報告(W. Gao et al., Carcinogenesis, 29(10):1901-10 (2008))に基づき、(赤シグナル/緑シグナル)≧2.0のとき、該遺伝子はGIST由来DNAでメチル化陽性であるとし、また(赤シグナル/緑シグナル)<2のとき、該遺伝子はメチル化陰性であると評価し、したがって、REC8≧2.0はREC8メチル化陽性検体を示し、REC8<2&PAX3≧2は、REC8メチル化陰性かつPAX3メチル化陽性検体を示し、REC8<2&PAX3<2はREC8メチル化陰性かつPAX3メチル化陰性検体を示す。
【図3】この図は、GIST検体115例(トレイニングセット40例、検証セット75例)におけるパイロシークエンス法を用いたメチル化解析の結果を示す。トレイニングセットは、図1のDNAメチル化網羅的解析に用いた症例セット、検証セットは、REC8遺伝子、PAX3遺伝子およびp16遺伝子のメチル化の有無の測定が、別の症例群でも予後予測に有用であることを示すために行った解析結果を示している。パイロシークエンス法で計測されるサンプル内の目的とするCpGサイトのメチル化測定値(メチル化レベル)が15%以上を示す場合をメチル化陽性と判断した。この値(15%)はパイロシークエンス法の非特異的な測定値(ノイズ)では通常得られない測定値としての最小値である。図中、「HD」は、homozygous deletion(すなわち、父親由来および母親由来の両アレルが欠失した状態)を表す。
【図4】この図は、REC8遺伝子、PAX3遺伝子及び/又はp16遺伝子のDNAメチル化状態とGIST症例生存期間との関係を示す。REC8遺伝子、PAX3遺伝子及びp16遺伝子のうち1つ以上メチル化が存在する症例では、生存期間が有意に短い(p=0.0338)ことを示す。
【発明を実施するための形態】
【0022】
本発明は、上記のとおり、消化管間質腫瘍をもつ患者の生物学的検体においてREC8遺伝子及びPAX3遺伝子のうちの少なくとも1つの遺伝子のメチル化の有無を測定し、該遺伝子がメチル化されている場合予後不良であり、一方メチル化されていない場合予後良好であると決定することを含む、該患者の予後をインビトロで予測する方法を提供する。
【0023】
以下に、本発明をさらに詳細に説明する。
1.消化管間質腫瘍
消化管間質腫瘍(GIST)は、食道、胃、小腸、大腸、直腸などの消化管の主に壁に発生する間葉系腫瘍であり、かつ、粘膜下腫瘍を構成する腫瘍である。この腫瘍は、増殖因子受容体c-kitの変異が原因であると言われている。
【0024】
この腫瘍は、その患者において予後不良の場合と予後良好の場合に分けられる。予後不良は、腫瘍が局部的浸潤性、破壊的増殖性または 転移性を有する場合を指し、いわゆる悪性かつ再発性である。一方、予後良好は、浸潤性・転移性がなく、すなわち再発性がなく、単なる外科手術で完治する場合を指す。
【0025】
これまで、GISTの診断は、KITタンパク質の発現の有無によって可能であろうと指摘されてきた。また実際には、腫瘍径と腫瘍細胞分裂像数によってリスクの程度が判定されてきた。治療薬については、グリベック(登録商標)以外には効果的な治療薬がないと言われている。
【0026】
2.GIST患者における予後の予測とそれに関連する遺伝子
後述の実施例に記載されるように、本発明者らは、トレイニングセット(40例)でアレイを施行し、そのうち転移・浸潤をきたしている予後不良群(18例)と、細胞分裂像と大きさから判断した予後良好群(低リスク群 9例)の計27例を対象とし、アレイの解析データから予後不良群で特異的にメチル化をしている155遺伝子を選択した(図1)。この155遺伝子を決定木法で解析し、予後不良群と予後良好群を最も的確に区別する2遺伝、すなわちREC8遺伝子とPAX3遺伝子を今回見出した(図2)。これらの遺伝子に、GISTの予後との関係が報告されているヒトp16遺伝子のメチル化を加えて、予後予測の有用性を検討した。トレイニングセットでは中間群13例を、上記27例に加えた40例において、パイロシークエンス法を用いてメチル化定量的解析法を施行した。アレイの結果と同様に予後良好群でメチル化が低頻度であり、予後不良群でメチル化が高頻度であることが確認された(図3(左))。これら遺伝子の有用性をさらに検証するため、新たに別の患者群(検証セット)75例(予後良好群24例、中間群36例、予後不良群15例)を対象にメチル化定量的解析法を行ったところ、これらの遺伝子は、トレイニングセットで得られた結果と同様に、予後不良群で高頻度及び高割合でメチル化されており一方、予後良好群では、特定の遺伝子のメチル化の程度が非常に低く、ほとんど又は全くメチル化されていないことが確認された(図3(右))。さらに生存期間が明らかな69例を対象に生存期間を比較した結果、p16遺伝子、REC8遺伝子、PAX3遺伝子のいずれかでメチル化が陽性の患者群では、メチル化が検出されない患者群と比較して、生存期間が有意に短いことが判明した(図4)。
【0027】
本発明の方法で使用しうる遺伝子は、メチル化の程度に応じて予後不良群、中間群及び予後良好群を適正に判別することを可能にする。
【0028】
これまでGIST症例の中には、中間群に入る症例も多く存在し、この群に属する症例が予後不良なのか、それとも予後良好なのかが不明確であったが、本発明の方法は、この中間群さえも、該群のなかで相対的にメチル化の程度が高い層(高リスク)と、相対的にメチル化の程度が低い層(中リスク)に分類できるほどに高い感度と高い特異度を有している(図3)。
【0029】
本発明の方法では、REC8遺伝子及びPAX3遺伝子のいずれか一方がメチル化されている場合をメチル化陽性であるとした。REC8遺伝子がメチル化陽性である場合、予後不良か又は(悪性の可能性が高い)高リスクに属する。同様に、PAX3遺伝子がメチル化されている場合にも、REC8遺伝子ほどではないにしてもメチル化頻度が高いほど予後不良及び高リスクに属する。特に、これら遺伝子の両方がともにメチル化されている場合は、高い特異度で予後不良又は高リスクに属する。しかし、ときには両遺伝子がメチル化陰性の場合もあり、この結果を修正し、さらに分類の確度を高めるために、本発明の方法ではp16遺伝子等の別の遺伝子のメチル化状態を検査に含めることができる。
【0030】
このように、GISTの悪性化に伴い、REC8遺伝子及び/又はPAX3遺伝子、ならびにp16遺伝子、のメチル化が蓄積する。内視鏡検査等で採取された消化管(胃、小腸、大腸、直腸、食道など)のGIST組織検体に対して上記遺伝子のメチル化解析を行うことによって、GIST患者の予後予測やGISTの悪性度に関する臨床検査への応用が可能になるし、さらにまた、臨床検査の結果を受けてGIST患者に対するより効果的な治療法の選択に寄与することができる。
【0031】
本発明における予後予測のための遺伝子マーカーは、REC8遺伝子及びPAX3遺伝子であり、好ましくは追加的にp16遺伝子も含まれる。これら遺伝子の配列情報は、公知の遺伝子データバンク(米国NCBI等)から入手可能である。
【0032】
ヒトREC8(meiotic recombination REC8 homolog)の塩基配列は、例えばUniprotKB/Swiss-Prot accession No. 095072、GenBank accession No. NM_005132、NM_001048205、NP_005123などとして知られている。REC8遺伝子のプロモーター領域のCpGアイランドの塩基配列を配列番号1として示す。
【0033】
ヒトPAX3 は、転写因子であるpaired box (PAX) ファミリーに属する遺伝子であり、ヒトPAX3タンパク質は、例えば479アミノ酸等を含むが、種々の置換等の変異を含むバリアントからなる。PAX3は、耳、目及び顔の発生において同定され、その変異によってWaardenburg症候群1型及び3型が起きる。本発明では、ヒトPAX3は、バリアントのいずれも包含するものとする。バリアントの例は、GenBank accession No. NM_001127366、NM_181458、NM_000438、NM_181457、NM_18459、NM_013942、NM_181460などである。PAX3遺伝子のプロモーター領域のCpGアイランドの塩基配列を配列番号2として示す。
【0034】
ヒトp16遺伝子のメチル化とGIST症例の予後との関連はこれまで報告されている(R. Ricci et al., Role of p16/INK4a in gastrointestinal stromal tumor progression, Am J Clin Pathol 2004;122:35-43)。ヒトp16遺伝子は、cyclin-dependent kinase inhibitor 2A (melanoma, p16, inhibits CDK4) (CDKN2A)であり、第1エクソンが異なる7つのバリアントからなることが知られており、そのうち少なくとも3つは選択的スプライシングによるものである。本発明では、いずれのバリアントもp16に包含されるものとする。GenBankに登録されたバリアントの例は、NG_007485、NM_058195、NM_000077、NM_001195132、NM_058197などであるが、本発明のp16はそこに記載された塩基配列を含むものとする。p16遺伝子のプロモーター領域のCpGアイランドの塩基配列を配列番号3として示す。
【0035】
ヒト以外の哺乳動物の配列は、GenBank等の遺伝子データベースにアクセスすることにより入手可能である。
【0036】
本明細書中「哺乳動物」又は「患者」には、霊長類、ペット動物、家畜動物、げっ歯類などが含まれ、好ましい哺乳動物はヒトである。
【0037】
3.DNAメチル化の測定
DNAメチル化は、哺乳動物のゲノム上のREC8、PAX3及びp16遺伝子の非翻訳領域、特にプロモーター領域のCpGアイランド配列においてシトシン(C)の5位の炭素が過度にメチル化(hypermethylation)されていることを意味する。ここで、CpGアイランドは、哺乳動物のゲノム配列において非翻訳領域に存在するグアニン(G)シトシン(C)含量が多い領域であり、CpGのpは、CとGの間のホスホジエステル結合を表す。CpGアイランドはCGからなるジヌクレオチドの繰り返し配列を含み、その定義では通常、500塩基対領域内のCpG含有率が55%以上、CpG/GpC比が0.65以上である。
【0038】
REC8遺伝子、PAX3遺伝子及びp16遺伝子のCpGアイランドのhypermetylation領域内でPCR法により約150〜250bpの増幅産物を得ることができるプライマーを設計することができる。
【0039】
これらのヒト遺伝子のプロモーター領域のCpGアイランドの配列の例を以下に示す。
(1) REC8 (下線付き大文字はREC8のエクソン配列(遺伝子の転写領域):エクソン1、エクソン2、エクソン3)(配列番号1)
ctactcaggaggccaagacacgagaatcgcttgaacctgaggggatggggggcagaggatgcagtgagccgagattgccgagatagcgccactgcattccagcctgggcaacagtgagagcctccatctcaaaaaaaaaaaaaaaaaatggacttcttcctcattgacctgtggtggctccaaggccatcaaaaaaaaaaaaaatggacttcttcctcattgacctgtggtggctccaaggccatcatctcacagcctttttttttttttttttttttgagacggagtcttgctcccttgcccaggttggagtggagctgcatgatctcggctcactgcaacctccgcctcccaggttcaagcattctcctgcctcagcctcctgagtagctgggattacaggcgcgtgccaccacgcccagctaacttttgcatttttaatagagacggggtttcaccatattggtcaggctggactcgaactcctaacctcatgatccgcccgcctcggcctcccaaagtgctaggattacaggcgtgagccactgcgcccggcctcatctcacatctttcttcctctgcagcacacgacacgccctagttggaaacaaagtcggagtttgtggattgggggaagggcggggtctaacctcaggtcaggcgccgtgcaaggtacatcttggcacccggaagaggcccagtacagttgcccccgaggtgacccgacctcccctaccaattgaggcgcccttgttgccaggcttgcggcgggggagcggcgggggagcgacggggatgcgctcattggtcaaggaaggggcgcctgttactagaggcgagaaccggagcccattggtcggaacacctcacaatggaccccagcggcgcgcaaaatccttatgattggtttgctggctgcctcgggagaccctgttgccaggatacttggcgttcccgacccgacccccgttccccattggctgtcagggcaaaagccgccATCTAATGAGGAGCGAGGTGCGGTGCCCCGAAGCGCTCGCTTCCCGCGGTGCGATCTAGTCCTGCAGTAGGCGGCCCGGGGCCACACCGCGGCCGCCCAAGCCAGTGCAAGGCCCAGGGGCCTGACATCGCTCCCAGCGCTCGAGGACCGAGGCCTGCTGTGGAGGACACCGTGCTCCCTCGGGACCTGCTCTGGATTCCGGCCCGGACGTCCCCTTGGAGCTCTGCATCTCCAACCTGGAACCCAACCCAGAAGTCTCAAGTTTGACGCATCACGTGGCGTGCGGATCCACTGAGGGTCCACAGAGAGGGGCGCCCATCTCCTGCGTCTCAGTTATCCTGGTAATTGTGTATCTGCCCATTGTTCGTTGCCTCATTAACTTGGCTTTCTAGGTGCACCCACCTTGCCACCAGAGAAGTCCAAATCCTGACTTCTCTCCAAGGTGTTGGGAATTCTGTGCCCTAAAGAATTCCGACTCAGATCCGAACGGGGATCTGGTGGAATCGAGGGTGAAAGACCAGAGGGACAATGTTCTACTATCCCAACGTGCTTCAGCGCCACACCGGCTGCTTTGCCACCATCTGgtaagggcggggcccgttggcgcgcgatggcggacgctgcccgggatcccagcctgacagctccccctccatccccattctcccaccttccccacccacttcagGCTGGCGGCGACTCGCGGCAGCCGGTTGGTGAAGCGCGAATACCTGAGGGTGAATGTGGTGAAAACCTGgtaaggcccagaaaagggaaggagggcctggtgcggggggtgagttaggggatggggtggccaagactgtgggcccactcctggacgcagcggtaatcagggcgcattgttccccagCGAGGAAATCCTCAATTACGTGCTGGTACGAGTGCAACCCCCGCAGCCCGGCCTGCCGCGGCCCCGCTTCTCCCTCTATCTCTCAGCCCAACTTCAGATCGGTGTGATCCGCG
【0040】
(2) PAX3(下線付き大文字はPAX3のエクソン配列(遺伝子の転写領域):エクソン1)(配列番号2)
cgcagcgcgggtccccctcggggccagcagaggcctcggcaccaccagagatgggaagagaaagtggtcgctgttgcccaatcagcgcgtgtctccgccacccgggacggtctacccgtcggccaatcgcagctcagggctcctgaccaagctttgggtaaaagaactaataaatgctcccgagcccggatccccgcACTCGGTGTCACCACAGGAGGAGACTCAGGCAGGCCGCGCTCCAGCCTCACCAGGCTCCCCGGCTCGCCGTGGCTCTCTGAGCCCCCTTTTCAGGGACCCCAGTCGCTGGAACATTTGCCCAGACTCGTACCAAACTTTTCCGCCCTGGGCTCGGGATCCTGGACTCCGGGGCCTCCCCGTCCTCCCCTTTCCCGGGTTCCAGCTCCGGCCTCTGGACTAGGAACCGACAGCCCCCCTCCCCGCGTCCCTCCCTCTCTCTCCAGCCGTTTTGGGGAGGGGCTCTCCACGCTCCGGATAGTTCCCGAGGGTCATCCGCGCCGCACTCGCCTTTCCGTTTCGCCTTCACCTGGATATAATTTCCGAGCGAAGCTGCCCCCAGGATGACCACGCTGGCCGGCGCTGTGCCCAGGATGATGCGGCCGGGCCCGGGGCAGAACTACCCGCGTAGCGGGTTCCCGCTGGAAGgtaagggagggcctcagcgcgccgcctggatcccagggcctgggaccggctgcctcaccccatccccaggctccgcaggctcctttggtgcttccaggaagcccattccctgggcaccccacaccccaagaagcaccagtcgggggcgaggacctactcgatttcctttctgcaaatggagcgcgctgctctctgcaaatcctggcggagctgggcggtcaggcctgcggcgagccggggagacttgtccctgttgttttggaaatgccgcggtggagaaggaggtgaaccggcttctgagcg
【0041】
(3) p16(下線付き大文字はp16のエクソン配列(遺伝子の転写領域):エクソン1)(配列番号3)
cgcggaggaaggaaacggggcgggggcggatttctttttaacagagtgaacgcactcaaacacgcctttgctggcaggcgggggagcgcggctgggagcagggaggccggagggcggtgtggggggcaggtggggaggagcccagtcctccttccttgccaacgctggctctggCGAGGGCTGCTTCCGGCTGGTGCCCCCGGGGGAGACCCAACCTGGGGCGACTTCAGGGGTGCCACATTCGCTAAGTGCTCGGAGTTAATAGCACCTCCTCCGAGCACTCGCTCACGGCGTCCCCTTGCCTGGAAAGATACCGCGGTCCCTCCAGAGGATTTGAGGGACAGGGTCGGAGGGGGCTCTTCCGCCAGCACCGGAGGAAGAAAGAGGAGGGGCTGGCTGGTCACCAGAGGGTGGGGCGGACCGCGTGCGCTCGGCGGCTGCGGAGAGGGGGAGAGCAGGCAGCGGGCGGCGGGGAGCAGCATGGAGCCGGCGGCGGGGAGCAGCATGGAGCCTTCGGCTGACTGGCTGGCCACGGCCGCGGCCCGGGGTCGGGTAGAGGAGGTGCGGGCGCTGCTGGAGGCGGGGGCGCTGCCCAACGCACCGAATAGTTACGGTCGGAGGCCGATCCAGgtgggtagagggtctgcagcgggagcaggggatggcgggcgactctggaggacgaagtttgcaggggaattggaatcaggtagcgcttcgattctccg
【0042】
検体は、患者のGIST腫瘍組織、その組織浸出液などであり、手術時に、あるいは内視鏡検査時に採取することが可能であり、得られた該組織からフェノール/クロロホルム法などの公知の手法によるか又は市販キットを用いてゲノムDNAを抽出する。
【0043】
MCA(methylated CpG island amplification)法とマイクロアレイ(microarray)分析を組み合わせたMCAM解析法を用いた多数の遺伝子プロモーター領域のDNAメチル化の網羅的解析とパイロシークエンス法等によるDNAメチル化の定量的解析により、GISTの悪性度におけるDNA異常メチル化の関与を調べる。MCA産物を階層型クラスター法で解析すると、予後不良群と予後良好群の2つのクラスターを形成する。
【0044】
GISTの悪性度を分類するために、検体採取時にすでに再発・転移/浸潤をきたしているものを予後不良群、転移・再発/浸潤をきたしていないものを予後良好群と定義する。また、予後良好群の症例は、臨床的に予後予測因子とされている腫瘍径と腫瘍細胞の分裂像から、転移・再発/浸潤をきたしていないために予後良好群に分類されるか否かがはっきりしないものを中間群に分類し、中間群には、対象遺伝子のメチル化の程度に応じてメチル化のより高い方から高リスク及び中リスクが含まれる。メチル化の程度の低い、いわゆる低リスクは、明らかに予後良好群に分類される。
【0045】
遺伝子のDNAメチル化を解析する方法には、バイサルファイトシークエンス法、メチル化特異的PCR(MSP; Methylated Specific PCR))法、定量的MSP法、COBRA(Combined Bisulfite Restriction Analaysis)法、パイロシークエンス法などの方法があり、それぞれ解析範囲、感度、精度、必要な機器が大きく異なる。これらは全てバイサルファイト処理によってメチル化していないシトシンのみをウラシルに変換するという原理を利用している(牛島俊和ら編、エピジェネティクス実験プロトコール、羊土社(2008年))。
【0046】
以下にDNAメチル化測定法の例を示す。
(a) パイロシークエンス法:
ポリメラーゼ塩基伸長反応によるルシフェラーゼ発光反応を検出することで塩基配列を解読する。伸長反応の際に生成されるピロリン酸と発光強度が完全に比例関係にあるため高精度な定量解析を行うことができる。この原理により、バイサルファイト(bisulfite)処理を行ったDNAのCpGサイトのT%/C%を解読してDNAメチル化比率を決定する(Alliance Biosystems)。
【0047】
(b) バイサルファイトシークエンス法:
DNAをバイサルファイト処理することによってDNA中のメチル化されていないシトシン(非メチル化シトシン)はウラシルに変換される。一方、メチル化シトシンはバイサルファイト処理で変換されない。それゆえ、このような処理によってメチル化シトシンと非メチル化シトシンを区別できるので、PCR増幅したのち、塩基配列を決定するとか、制限酵素処理などの手法によってDNAメチル化を解析することができる(特表2002-535998号公報)。
【0048】
PCR増幅は、PCRバッファー中、dNTP mixure、プライマー、DNA、耐熱性DNAポリメラーゼの存在下で行う。耐熱性DNAポリメラーゼは、TaKaRa Taq HS(宝酒蔵)などの市販の酵素を使用できる。PCR条件は、例えば95〜98℃、5〜60秒の変性反応、50〜55℃、30〜60秒のアニーリング反応、及び72℃、20秒〜60秒の伸長反応を1サイクルとし、これを30〜40サイクル行う。
【0049】
(c) メチル化特異的PCR(MSP)法:
DNAをバイサルファイト変換し、このDNAを鋳型にして、メチル化DNAを特異的に増幅するプライマーと、非メチル化DNAを特異的に増幅するプライマーを用いて別々にPCRを行い、PCR産物の有無によりDNAメチル化の有無を判定する(特表2002-535998号公報)。
【0050】
(d) 定量的MSP法:
MSP法は、methylation specific PCR技術であり、Na bisulfiteで処理したDNAを、目的遺伝子内部のメチル化領域及び非メチル化領域に特異的な予想配列のプライマーを用いてPCR増幅することを基本としている(上記(c))。
【0051】
複数の蛍光団(fluorophore)を使用することによってリアルタイム定量MSPを行うことが可能であり、この場合メチル化領域特異的プライマーと非メチル化領域特異的プライマーを使用する(T. Swift-Scanlan et al., BioTechniques 40:210-219, 2006)。
【0052】
(e) COBRA法:
バイサルファイト処理後にメチル化DNAと非メチル化DNAに共通のプライマーを用いて目的のDNAをPCR増幅後、メチル化DNAと非メチル化DNAで配列が異なる箇所を認識する制限酵素で処理し、断片をエタノール沈殿して濃縮し、電気泳動でメチル化DNAと非メチル化DNAを識別できる。PCR増幅は、バイサルファイトシークエンス法と同様に行うことができる(Xiong, Z. and Laird, P.W. Nucleic Acids Res. 25(1):2532-2534, 1997)。
【0053】
本発明の方法で使用可能なPAX3遺伝子、REC8遺伝子、及びp16遺伝子のそれぞれのパイロシークエンスプライマーセットの例は、以下のとおりである。ここで、Fはフォワードプライマー、Rはリバースプライマー、bioは5’末端のビオチン修飾であり、これらのそれぞれの遺伝子のプライマーセットのうち1つもしくは2つ以上のセット〜全セットを用いてPCR増幅を行うことができる。seqはパイロシークエンスに用いるシークエンスプライマーを示す。以下に示す塩基配列を順番に配列番号4から配列番号273として表す((注)同一のプライマー配列に対し複数の配列番号が割付けられる場合もある。)。
【0054】
<PAX3遺伝子>
F TAGAGATGGGAAGAGAAAGTGG (配列番号4)
R bio bio-ACCTACCTAAATCTCCTCCTATAA(配列番号5)
seq GGTAAAAGAATTAATAAATG(配列番号6)
F TTAGAGATGGGAAGAGAAAGTGG(配列番号7)
R bio bio-CCTACCTAAATCTCCTCCTATAAT(配列番号8)
seq GGTAAAAGAATTAATAAATG(配列番号9)
F TTAGAGATGGGAAGAGAAAGTGGT(配列番号10)
R bio bio-CCTACCTAAATCTCCTCCTATAA(配列番号11)
seq GGTAAAAGAATTAATAAATG(配列番号12)
F ATTTTTTTTTTTGGGTAAGGGG (配列番号13)
R bio bio-ACCTACCTAAATCTCCTCCTATAA(配列番号14)
seq TAAAAGAATTAATAAATGTT(配列番号15)
F GGTTGTTTTTTTTAGTTTTTAT (配列番号16)
R bio bio-AAAACATTTATTAATTCTTTTACC(配列番号17)
seq GTGGTYGTTGTTGTTTAATT(配列番号18)
F TTTTTAGTAATTGTTATTTTTT (配列番号19)
R bio bio-ACCCAAAACTTAATCAAAAACCCT(配列番号20)
seq GTGGTAGTTGTTGTTTAATT(配列番号21)
F GAGATAGAGAGATAGGAATTTT (配列番号22)
R bio bio-CACTTTCTCTTCCCATCTCTAATA(配列番号23)
seq TTTTTTTTTTTGGGTAAGGG(配列番号24)
F AGAGATGGGAAGAGAAAGTGG(配列番号25)
R bio bio-ATTCCTAATCCAAAAACC (配列番号26)
seq TTGGAATATTTGTTTAGA(配列番号27)
F TATAGGAGGAGATTTAGGTAG(配列番号28)
R bio bio-ATTCCTAATCCAAAAACC(配列番号29)
seq GTTGGAATATTTGTTTAGAT(配列番号30)
F GTTTTGGGGAGGGGTTTTTTA(配列番号31)
R bio bio-TACCCCCAACCCCACCCC (配列番号32)
seq GTTTTTATTTGGATATAATT(配列番号33)
F TTTTTTTTTTTTTYGGGTTTTAGT(配列番号34)
R bio bio-AATCATCCTAAAAACAACTTC(配列番号35)
seq GTTTTTATTTGGATATAATT(配列番号36)
F GATTAGTATTAAATTTTTTAGTTT(配列番号37)
R bio bio-AATCATCCTAAAAACAACTTC(配列番号38)
seq TGGGGAGGGGTTTTTTAAGT(配列番号39)
F GTAGTTTTGGGGAGGGGTTTTTTA(配列番号40)
R bio bio-AATCATCCTAAAAACAACTTC(配列番号41)
seq GTTTTTATTTGGATATAATT(配列番号42)
F bio bio-AATTGTTATTTTTTTGGGGTTGT(配列番号43)
R CACTTTCTCTTCCCATCTCTAATA(配列番号44)
seq CTTCCCATCTCTAATAATAC(配列番号45)
F bio bio-GGGTTGTTTTTTTTAGTTTTTAT(配列番号46)
R AACTTAATCAAAAACCCTAAACTA(配列番号47)
seq CTTCCCATCTCTAATAATAC(配列番号48)
F bio bio-AATTTTTTTTTTTGGGTAAGGGG(配列番号49)
R AAACATTTATTAATTCTTTTACCC(配列番号50)
seq CTTCCCATCTCTAATAATAC(配列番号51)
F bio bio-ATTAGAGATGGGAAGAGAAAGTGG(配列番号52)
R TCTTTTACCCAAAACTTAATCAAA(配列番号53)
seq ACTTAATCAAAAACCCTAAA(配列番号54)
F bio bio-GTATTATTAGAGATGGGAAGAGAA(配列番号55)
R AAACATTTATTAATTCTTTTACCC(配列番号56)
seq ACTTAATCAAAAACCCTAAA (配列番号57)
F bio bio-AAGTGGTYGTTGTTGTTTAATTAG(配列番号58)
R CCTAAATCTCCTCCTATAATAACA(配列番号59)
seq ACTTAATCAAAAACCCTAAA(配列番号60)
F bio bio-GAGTTTTTTTTTTAGGGATTTTAG(配列番号61)
R TAATCATCCTAAAAACAACTTC(配列番号62)
seq AAAATTATATCCAAATAAAA(配列番号63)
F bio bio-GTTTAGGGTTTTTGATTAAGTTT(配列番号64)
R AATCCCTAAAAAAAAAACTCAAA(配列番号65)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号66)
F bio bio-GGGTAAAAGAATTAATAAATGTT(配列番号67)
R ACRAATCTAAACAAATATTCCAA(配列番号68)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号69)
F bio bio-TAGTTTAGGGTTTTTGATTAAGT(配列番号70)
R AATACRAATCTAAACAAATATTC(配列番号71)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号72)
F bio bio-GATTAAGTTTTGGGTAAAAGAAT(配列番号73)
R ACCRAAAAACCTAATAAAACTAA(配列番号74)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号75)
F bio bio-TTTAGGGTTTTTGATTAAGTTTT(配列番号76)
R AAAACCTAATAAAACTAAAACRC(配列番号77)
seq CCTAAATCTCCTCCTATAATAACAC(配列番号78)
F bio bio-AATTGTTATTTTTTTGGGGTTGT(配列番号79)
R CACTTTCTCTTCCCATCTCTAATA(配列番号80)
seq AAACCTCTACTAACC(配列番号81)
F bio bio-TTAATTTTTTTTTTTGGGTAAGG(配列番号82)
R TCTCTTCCCATCTCTAATAATACC(配列番号83)
seq AAACCTCTACTAACC(配列番号84)
F bio bio-GTTTTTAGTAATTGTTATTTTTT(配列番号85)
R AATTAAACAACAACCACCACTTTC(配列番号86)
seq AAACCTCTACTAACC(配列番号87)
F bio bio-GAGATAGAGAGATAGGAATTTTT(配列番号88)
R ACTTTCTCTTCCCATCTCTAATAA(配列番号89)
seq AAACCTCTACTAACC(配列番号90)
F bio bio-GAGATAGAGAGATAGGAATTTTT(配列番号91)
R AACTTAATCAAAAACCCTAAACTA(配列番号92)
seq AAACCTCTACTAACC(配列番号93)
【0055】
<REC8遺伝子>
F TGTTGGTTGTTTYGGGAGATTTT(配列番号94)
R bio bio-TCAAACCCCTAAACCTTACACTAA(配列番号95)
seq ATTTTGTTGTTAGGATATTT(配列番号96)
F TTATGATTGGTTTGTTGGTTGTT(配列番号97)
R bio bio-CCCTAAACCTTACACTAACTTAAA(配列番号98)
seq ATTTTGTTGTTAGGATATTT(配列番号99)
F GGAATATTTTATAATGGATTTTA(配列番号100)
R bio bio-ATATCAAACCCCTAAACCTTACAC(配列番号101)
seq ATTTTGTTGTTAGGATATTT(配列番号102)
F GGAGATTTTGTTGTTAGGATATT(配列番号103)
R bio bio-ACCATATCCTCCACAACAAACCTC(配列番号104)
seq TTATTGGTTGTTAGGGTAAA(配列番号105)
F AGATTTTGTTGTTAGGATATTTG(配列番号106)
R bio bio-AAACCCCTAAACCTTACACTAACT(配列番号107)
seq ATTGGTTGTTAGGGTAAAAG(配列番号108)
F TTTTTTATTGGTTGTTAGGGTAA(配列番号109)
R bio bio-TATCAAACCCCTAAACCTTACACT(配列番号110)
seq ATTGGTTGTTAGGGTAAAAG(配列番号111)
F TTTTYGTTTTTTATTGGTTGTTA(配列番号112)
R bio bio-AAACCTTACACTAACTTAAACRAC(配列番号113)
seq ATTGGTTGTTAGGGTAAAAG(配列番号114)
F TTTTATTGGTTGTTAGGGTAAAA(配列番号115)
R bio bio-CACRATATCCTCCACAACAAACCT(配列番号116)
seq GAYATTTAGTTTTGTAGTAG(配列番号117)
F TATTTTATAATGGATTTTAGAGG(配列番号118)
R bio bio-CCTCCACAACAAACCTCRATCCTC(配列番号119)
seq GYGATTTAGTTTTGTAGTAG(配列番号120)
F AGATTTTGTTGTTAGGATATTTG(配列番号121)
R bio bio-TAAAAATACAAAACTCCAAAAAAA(配列番号122)
seq GTGTAAGGTTTAGGGGTTTG(配列番号123)
F TTTAAGTTAGTGTAAGGTTTAGG(配列番号124)
R bio bio-TCCAAATTAAAAATACAAAACTCC(配列番号125)
seq GTGTAAGGTTTAGGGGTTTG(配列番号126)
F AAGTTAGTGTAAGGTTTAGGGGT(配列番号127)
R bio bio-TTAAAACTTCTAAATTAAATTCCA(配列番号128)
seq TAAGGTTTAGGGGTTTGATA(配列番号129)
F TAGTGTAAGGTTTAGGGGTTTGA(配列番号130)
R bio bio-ACACAAAATTCCCAACACCTTAA(配列番号131)
seq TTTAGAAGTTTTAAGTTTG(配列番号132)
F TTTAAGTTAGTGTAAGGTTTAGG(配列番号133)
R bio bio-TCTCTAATAACAAAATAAATACA(配列番号134)
seq TTTAGAAGTTTTAAGTTTG(配列番号135)
F GAGGTTTGTTGTGGAGGATAT(配列番号136)
R bio bio-CAATAAACAAATACACAATTACC(配列番号137)
seq ATTTAGAAGTTTTAAGTTT(配列番号138)
F TTTTTTGGAGTTTTGTATTTT(配列番号139)
R bio bio-CACAATTACCAAAATAACTAAAA(配列番号140)
seq ATTTAGAAGTTTTAAGTTT(配列番号141)
F TTTTAATTTGGAATTTAATTT(配列番号142)
R bio bio-CCTCTCTATAAACCCTCAATAAA(配列番号143)
seq ATTTAGAAGTTTTAAGTTT(配列番号144)
F TATTTTTAATTTGGAATTTAA(配列番号145)
R bio bio-AATAACTAAAACRCAAAAAATAA(配列番号146)
seq ATTTAGAAGTTTTAAGTTT(配列番号147)
F GTTTTGTATTTTTAATTTGGA(配列番号148)
R bio bio-AAAAAACCAAATTAATAAAACAA(配列番号149)
seq ATTTAGAAGTTTTAAGTTT(配列番号150)
F bio bio-AGTGTAAGGTTTAGGGGTTTGAT(配列番号151)
R CCCAACACCTTAAAAAAAAATCA(配列番号152)
seq TCTATAAACCCTCAATAAAT(配列番号153)
F bio bio-AATTTGGAATTTAATTTAGAAGT(配列番号154)
R TAACAAAATAAATACACCTAAAA(配列番号155)
seq TCTATAAACCCTCAATAAAT(配列番号156)
F bio bio-GGAATTTAATTTAGAAGTTTTAA(配列番号157)
R AACAATAAACAAATACACAATTA(配列番号158)
seq TCTATAAACCCTCAATAAAT(配列番号159)
F bio bio-TGTAAGGTTTAGGGGTTTGATAT(配列番号160)
R CCCCTCTCTATAAACCCTCAATA(配列番号161)
seq AAATACAAAACTCCAAAAAA(配列番号162)
F bio bio-TTATTGGTTGTTAGGGTAAAAGT(配列番号163)
R ATATCAAACCCCTAAACCTTACA(配列番号164)
seq TAAACCTTACACTAACTTAA(配列番号165)
F bio bio-GGGTAAAAGTYGTTATTTAATGA(配列番号166)
R TAAAAATACAAAACTCCAAAAAA(配列番号167)
seq TAAACCTTACACTAACTTAA(配列番号168)
F bio bio-AAGAGGTTTAGTATAGTTGTTTT(配列番号169)
R AATATCCTAACAACAAAATCTCC(配列番号170)
seq ACAAACCAATCATAAAAATT(配列番号171)
F bio bio-TGTAAGGTATATTTTGGTATT(配列番号172)
R AACAACCAACAAACCAATCATAA(配列番号173)
seq ACAAACCAATCATAAAAATT(配列番号174)
F bio bio-GTTTGTGGATTGGGGGAAG(配列番号175)
R AAACAACCAACAAACCAATCATA(配列番号176)
seq CCCTTCCTTAACCAATAAA(配列番号177)
F bio bio-GGGAGATTTTGTTGTTAGGATAT(配列番号178)
R TCAAACCCCTAAACCTTACACTAA(配列番号179)
seq TACCCTAACAACCAATAAA(配列番号180)
F bio bio-TAAGGTATATTTTGGTATT(配列番号181)
R TAAAATCCATTATAAAATATTCC(配列番号182)
seq CCCTTCCTTAACCAATAAA(配列番号183)
【0056】
<p16遺伝子>
F GGGTAGGTGGGGAGGAGTTTAG (配列番号184)
R bio bio-ACCAACCAACCCCTCCTCTTT(配列番号185)
seq GGAGTTAATAGTATTTT(配列番号186)
F GTGTGGGGGGTAGGTGGGGAGG (配列番号187)
R bio bio-CTATCCCTCAAATCCTCTAAA(配列番号188)
seq GGAGTTAATAGTATTTT(配列番号189)
F GGGGGAGATTTAATTTGGGG(配列番号190)
R bio bio-TATCTTTCCAAACAAAAAAAC(配列番号191)
seq GGAGTTAATAGTATTTT(配列番号192)
F GGGGGAGATTTAATTTGGGG(配列番号193)
R bio bio-CAACCAACCCCTCCTCTTTCT(配列番号194)
seq GGAGTTAATAGTATTTT(配列番号195)
F AAAGAGGAGGGGTTGGTTGG(配列番号196)
R bio bio-CTACCTACTCTCCCCCTCTCC(配列番号197)
seq GGTTGGTTATTAGAGGGT(配列番号198)
F GAGGAAGAAAGAGGAGGGGT(配列番号199)
R bio bio-AAAACTCCATACTACTCCCC(配列番号200)
seq TTGGTTATTAGAGGGTGG(配列番号201)
F AAAGAGGAGGGGTTGGTTGG(配列番号202)
R bio bio-ACCCTCTACCCACCTAAATC(配列番203)
seq TGGTTATTAGAGGGTGGG(配列番号204)
F GGGTTGGTTGGTTATTAGAG(配列番号205)
R bio bio-CAAACCCTCTACCCACCTAA(配列番号206)
seq AGAGGGGGAGAGTAGGTA(配列番号207)
F GTTGGTTATTAGAGGGTGGG(配列番号208)
R bio bio-AATCAACCRAAAACTCCATA(配列番号209)
seq AGAGGGGGAGAGTAGGTA(配列番号210)
F AATTAAGGGTTGAGGGGGTAGGG(配列番号211)
R bio bio-AAAAAACTAAACTCCTCCCCACCT(配列番号212)
seq TTTGGTAGTTAGGAAGGTT(配列番号213)
F TTATAATTAGGAAAGAATAGTTT(配列番号214)
R bio bio-TTCACTCTATTAAAAAAAAAT(配列番号215)
seq TTTGGTAGTTAGGAAGGTT(配列番号216)
F TATTTTTTTTATGATATTAAATA(配列番号217)
R bio bio-AATACRTTCACTCTATTAAAA(配列番号218)
seq TTTGGTAGTTAGGAAGGTT(配列番号219)
F GGGTTTTTATAATTAGGAAAGAA(配列番号220)
R bio bio-TTTAAATACRTTCACTCTATT(配列番号221)
seq TTTGGTAGTTAGGAAGGTT(配列番号222)
F ATTTAATTTGGTAGTTAGGAAGG(配列番号223)
R bio bio-TCCCCCRCCTACCAACAAAAA(配列番号224)
seq GGTAGTTAGGAAGGTTGTA(配列番号225)
F ATTTGGTAGTTAGGAAGGTTGTA(配列番号226)
R bio bio-CTCCCCACCTACCCCCCACAC(配列番号227)
seq TTTTTTAATAGAGTGA(配列番号228)
F bio bio-GGGTAGGTGGGGAGGAGTTTA(配列番号229)
R ACCAACCAACCCCTCCTCTTT(配列番号230)
seq CCCTCCTCTTTCTTCCT(配列番号231)
F bio bio-TTTTAGAGGATTTGAGGGATA(配列番号232)
R CCCCACCCTCTAATAACCAAC(配列番号233)
seq CCCTCCTCTTTCTTCCT(配列番号234)
F bio bio-AGAGGATTTGAGGGATAGGGT(配列番号235)
R CTAATAACCAACCAACCCCTC(配列番号236)
seq CCCTCCTCTTTCTTCCT(配列番号237)
F bio bio-TTTTTTTGTTTGGAAAGATAT(配列番号238)
R ATAACCAACCAACCCCTCCTC(配列番号239)
seq CCCTCCTCTTTCTTCCT(配列番号240)
F bio bio-GAAGAAAGAGGAGGGGTTGG(配列番号241)
R ACAAACCCTCTACCCACCTAAA (配列番号242)
seq CTACCTACTCTCCCCCT(配列番号243)
F bio bio-TTGGTTGGTTATTAGAGGGT(配列番号244)
R AAAACTCCATACTACTCCCC(配列番号245)
seq CTACCTACTCTCCCCCT(配列番号246)
F bio bio-AGAGGATTTGAGGGATAGGG(配列番号247)
R CCRACTCCATACTACTCCCC(配列番号248)
seq CTACCTACTCTCCCCCT(配列番号249)
F bio bio-GGTAGTTAGGAAGGTTGTAT(配列番号250)
R CCCCAAATTAAATCTCCCCC(配列番号251)
seq CACCTACCCCCCACA(配列番号252)
F bio bio-TTTTATGATATTAAATATTT(配列番号253)
R ACTAAACTCCTCCCCACCTA(配列番号254)
seq CACCTACCCCCCACA(配列番号255)
F bio bio-GAAGAAGTTATATTTTTTTT(配列番号256)
R AAACCAACRTTAACAAAAAA(配列番号257)
seq CACCTACCCCCCACA(配列番号258)
F bio bio-TGAGGGGGTAGGGGGATATT(配列番号259)
R CAACCTTCCTAACTACCAAATTAA(配列番号260)
seq TTCCTAATTATAAAAACCC(配列番号261)
F bio bio-AATTAAGGGTTGAGGGGGTA(配列番号262)
R TCATAAAAAAAATATAACTTCTTC(配列番号263)
seq TTCCTAATTATAAAAACCC(配列番号264)
F bio bio-TGAGGGGGTAGGGGGATATT(配列番号265)
R AACAAAACTATTCTTTCCTAATTA(配列番号266)
seq TTCCTAATTATAAAAACCC(配列番号267)
F bio bio-GGGTTTTTGATTTAGTGAAT(配列番号268)
R AAAAATATAACTTCTTCTTTTAAT(配列番号269)
seq TTCCTAATTATAAAAACCC(配列番号270)
F bio bio-GTTTTTGAAAATTAAGGGTT(配列番号271)
R AACCTTCCTAACTACCAAATTAAA(配列番号272)
seq TTCCTAATTATAAAAACCC(配列番号273)
【0057】
さらにまた、MSP法を用いても、メチル化陽性の判定は可能である。PAX3遺伝子、REC8遺伝子、及びp16遺伝子のそれぞれのメチル化特異的プライマーセット及び非メチル化特異的プライマーセットの例は、以下のとおりであるが、これらに限定されない。
【0058】
<PAX3遺伝子>
メチル化特異的プライマー (増幅産物の長さ(Product length) 114bp)
forward: TTCGGATAGTTTTCGAGGGTTA(配列番号274)
reverse:CCGACCAACGTAATCATCCT(配列番号275)
メチル化特異的プライマー (Product length 139bp)
forward: AGGGTTATTTGCGTCGTATTC(配列番号276)
reverse:AATAATTCTACCCCGAACCCG(配列番号277)
メチル化特異的プライマー (Product length 103bp)
forward: AGGGTTATTTGCGTCGTATTC(配列番号278)
reverse:AAACACAACGCCGACCAAC(配列番号279)
非メチル化特異的プライマー(増幅産物の長さ121bp)
forward: TTGGATAGTTTTTGAGGGTTATTTG(配列番号280)
reverse:TCCTAAACACAACACCAACCA(配列番号281)
【0059】
<REC8遺伝子>
メチル化特異的プライマー (増幅産物の長さ131bp)
forward: GCGTTTTCGATTCGATTTTC(配列番号282)
reverse:CCGAACCGCCTACTACAAAA(配列番号283)
非メチル化特異的プライマー(Product length 105bp)
forward: TTGGTTGTTAGGGTAAAAGTTGTT(配列番号284)
reverse:CCCCAAACCACCTACTACAAA(配列番号285)
【0060】
<p16遺伝子>
メチル化特異的プライマー (増幅産物の長さ150bp)
forward: TTATTAGAGGGTGGGGCGGATCGC(配列番号292)
reverse:GACCCCGAACCGCGACCGTAA(配列番号293)
非メチル化特異的プライマー(Product length 151bp)
forward: TTATTAGAGGGTGGGGTGGATTGT(配列番号294)
reverse:CAACCCCAAACCACAACCATAA(配列番号295)
(文献:Y. Kondo et al., Mol Cell Biol, 23(1):206-15 (2003))
【0061】
判定:
上記のDNAメチル化測定法を用いて得られた結果に基づいてGIST患者の予後、あるいはGISTの悪性化の程度、を予測することができる。本発明では、食道、胃、小腸、大腸などの消化管のGIST組織についてREC8遺伝子又はPAX3遺伝子あるいはその両方の遺伝子、のメチル化の有無を調べる(又は、検査する)ことによって、そして好ましくはREC8遺伝子及びPAX3遺伝子の両方のメチル化の有無、さらに好ましくはREC8遺伝子及びPAX3遺伝子並びにp16遺伝子のメチル化の有無を調べる(又は検査する)ことによって、上記の予測を行うことができる。
【0062】
予後や悪性度の程度は、予後不良群、中間群及び予後良好群に分類することができる。予後不良群は、転移や再発がある群であり、REC8遺伝子及び/又はPAX3遺伝子、好ましくはREC8遺伝子及びPAX3遺伝子、より好ましくはREC8遺伝子、PAX3遺伝子及びp16遺伝子、が相対的に高頻度でメチル化陽性である。中間群は、今のところ転移や再発はないがそのリスクのある、中リスク群と高リスク群に分類され、上記遺伝子類のメチル化陽性の相対的頻度は予後不良群よりかなり低い。ここで、高リスク群は、中リスク群と比べてメチル化陽性の相対的頻度が高く、予後不良となるリスクを有している。逆に、中リスク群は、高リスク群と比べてメチル化陽性の相対的頻度が低く、予後不良となるリスクがより低い。予後良好群(又は低リスク)では、上記遺伝子類のメチル化陽性頻度は極めて低く、ほとんどメチル化陰性である。
【0063】
ここで、メチル化陽性の頻度は、予後不良群、中間群(高リスクと中リスク)及び予後良好群の間でのメチル化陽性割合の大きさを指し、その割合が大きいほど高頻度であり、割合が小さいほど低頻度である。メチル化陽性と判定されたとき、予後不良群、中間群(高リスクと中リスク)のいずれかに分類される。すなわち、例えばREC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化をGIST検体で調べた場合、3つの遺伝子のうち1つ以上が陽性である場合、そのGISTは感度80%、特異度92%で中間群以上の悪性度を示すこと、また3つの遺伝子のうち1つ以上が陽性である症例群は、3つの遺伝子のいずれにもメチル化が認めない症例群と比較し、生命予後が有意に悪いことを示す(図4)。
【0064】
4.キット
本発明はさらにGISTを検査するためのキットを提供する。
該キットは、REC8遺伝子、PAX3遺伝子及びp16遺伝子のそれぞれの、パイロシークエンス法に使用する少なくとも1つの、好ましくは複数の上記プライマーセット、あるいは、MSP法に使用する少なくとも1つの上記メチル化特異的プライマーセット及び、場合により、非メチル化特異的プライマーセットを含み、これに加えてバイサルファイト試薬、PCR反応試薬ならびに使用説明書などを含むことができる。
【0065】
PAX3遺伝子、REC8遺伝子、及びp16遺伝子のそれぞれのパイロシークエンスプライマーセットの例は、上記の配列番号4から配列番号273までの配列の各セット(但し、ビオチン修飾はあってもよいし、なくてもよい。)であり、本発明のキットには、各遺伝子あたり、少なくとも1つのセットが含まれる。
【0066】
また、MSP法を用いてメチル化陽性の判定を行うための、PAX3遺伝子、REC8遺伝子、及びp16遺伝子のそれぞれのメチル化特異的プライマー及び非メチル化特異的プライマーの例は、PAX3遺伝子について上記の配列番号274から配列番号281の配列の少なくとも1つのセット、REC8遺伝子について配列番号282から配列番号285の配列の少なくとも1つのセット、及びp16遺伝子については、配列番号292から配列番号295の配列の少なくとも1つのセットである。
【0067】
バイサルファイト試薬は非メチル化シトシンをウラシルに変換するための試薬であり、この試薬には、重亜硫酸ナトリウム(Na bisulfite)、スカベンジャー(ハイドロキノン等)などを含むことができる。
PCR反応試薬には、dNTP mixture、PCRバッファー、耐熱性ポリメラーゼなどを含むことができる。
【0068】
上記プライマー及び上記試薬の他に、DNA抽出及び制限消化のための、フェノール/クロロホルム溶液又はフェノール/クロロホルム/イソアミルアルコール溶液、制限酵素などを含むことができる。
使用説明書には、DNAメチル化検査のためのプロトコールなどが記載されうる。
【実施例】
【0069】
以下に実施例によって本発明をさらに具体的に説明するが、本発明の技術的範囲はこれらの実施例によって制限されないものとする。
【0070】
[実施例1]
GISTの臨床検体(トレイニングセット40例)を用いて解析を行った。これらの臨床検体は、胃GIST(23例)、小腸GIST(15例)、直腸GIST(2例)の無作為に抽出した40例である。臨床検体からDNAを抽出した後、制限酵素のイソシゾマー性質を利用した、methylated CpG island amplification (MCA)法を用いて、メチル化しているDNA断片を回収し、カスタムアレイで解析した。
【0071】
GISTはこれまでの診断基準により大きさと細胞分裂像より、転移・再発のリスクを高リスク、中リスク、低リスクに分類される。これらのリスク分類では、既に転移・再発をきたしているGISTは含まれない。既に転移・浸潤をきたしているものを予後不良群(18例)、転移・浸潤がなく低リスクに分類されるものを予後良好群(9例)としてアレイ解析を行った。予後不良群で特異的にメチル化をしている155遺伝子を見出した。(図1)。アレイ解析に用いたアレイは、アジレント社のカスタムアレイ(G4497A)を用いた(文献:Y. Goto et al., Cancer Res, 69(23):9073-82 (2009))。
【0072】
[実施例2]
トレイニングセットでアレイより見出した、予後不良群に高頻度にメチル化する155遺伝子を用いて、決定木法による解析を行った。予後不良群と予後良好群を最も的確に分類する2遺伝、REC8遺伝子、PAX3遺伝子を同定した(図2)。
【0073】
アレイから選び出したREC8遺伝子、PAX3遺伝子の有用性について定量的メチル化法(パイロシークエンス法)を用いてトレイニングセット40例(予後良好群(低リスク群)9例、中間群(中リスク群・高リスク群)13例、予後不良群18例)で確認した(図3)。パイロシークエンス法は、高精度な定量解析能を有した塩基配列の解読装置を用いて行った。臨床検体から抽出したDNAをバイサルファイト処理後、PCR法により目的とする領域(REC8遺伝子、PAX3遺伝子およびp16遺伝子のプロモーター領域)の増幅を表1のプライマーセット(forwardとreverse)を用いて行った。増幅されたPCR産物を精製後、ビオチン(Bio-)標識されたDNA鎖の塩基配列を、シークエンスプライマー(sequence, 表1)を用いてパイロシークエンス法により解読した(文献:S. Colella et al., Biotechniques, 35(1):146-50 (2003))。
【0074】
さらに、REC8遺伝子、PAX3遺伝子の有用性を検証するために、トレイニングセットとは重複しない検証セット75例でメチル化頻度を検討した(図3)。
【0075】
その結果、検証セットGIST 75症例は次のように分類された。
予後良好群(低リスク群)24例
中間群(中リスク群・高リスク群)36例
予後不良群15例
【0076】
これらの症例のREC8遺伝子とPAX3遺伝子に加えて、p16遺伝子のメチル化も解析した。p16遺伝子のメチル化とGIST症例の予後(生存期間)との関連はこれまで報告されている(R. Ricci et al., Role of p16/INK4a in gastrointestinal stromal tumor progression, Am J Clin Pathol 2004;122:35-43)。
【0077】
これらのメチル化様式をパイロシークエンス法を用いて解析し、3遺伝子のうち1遺伝以上にメチル化を認める症例を陽性とした。パイロシークエンス法で計測されるサンプル内の目的とするCpGサイトのメチル化測定値(メチル化レベル)が15%以上を示す場合をメチル化陽性と判断した。この値(15%)はパイロシークエンス法の非特異的な測定値(ノイズ)では通常得られない測定値としての最小値である。
【0078】
上記及び図3でREC8遺伝子、PAX3遺伝子及びP16遺伝子のメチル化解析に用いたプライマー配列等を表1に示す。
【0079】
【表1】

【0080】
その結果、予後良好群、中間群、予後不良群ではそれぞれトレイニングセットでは11%、38%、83%の症例で陽性であり、検証セットではそれぞれ13%、69%、77%の症例で陽性であった。また、中間群においては、中リスクに比較して高リスクで有意に多くの症例で陽性症例が認められた(中リスク43%(9/21)対高リスク75% (21/28); P=0.038)。検証セットで、本アッセイは予後良好群と予後不良群を感度80%、特異度92%で区別した(図3)。
【0081】
[実施例3]
解析症例の中でGIST患者の生存期間が明らかである69症例を対象に、上記と同様のパイロシークエンス法を用いてREC8遺伝子、PAX3遺伝子、p16遺伝子のメチル化状態を測定し、このメチル化結果と生存期間との関係を検討した。
【0082】
その結果、REC8遺伝子、PAX3遺伝子、p16遺伝子のうち1遺伝子でもメチル化している症例は、有意に生存期間が短かった(P=0.0338;図4)。
【産業上の利用可能性】
【0083】
本発明は、GIST患者の予後をDNAメチル化検査によって決定することを可能にするため、治療計画の策定に有用である。
【配列表フリーテキスト】
【0084】
配列番号4〜295:プライマー
【特許請求の範囲】
【請求項1】
消化管間質腫瘍(GIST)をもつ患者の生物学的検体においてREC8遺伝子及びPAX3遺伝子のうちの少なくとも1つの遺伝子のメチル化の有無を測定し、該遺伝子がメチル化されている場合予後不良であり、一方メチル化されていない場合予後良好であると決定することを含む、該患者の予後をインビトロで予測する方法。
【請求項2】
p16遺伝子のメチル化の有無を測定することをさらに含む、請求項1に記載の方法。
【請求項3】
REC8遺伝子及びPAX3遺伝子の両方の遺伝子のメチル化を測定する、請求項1に記載の方法。
【請求項4】
REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化を測定する、請求項1又は2に記載の方法。
【請求項5】
前記遺伝子のメチル化をパイロシークエンス法又はMSP法で測定する、請求項1〜4のいずれか1項に記載の方法。
【請求項6】
REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化の有無を検出するための試薬を含むGIST検査用キット。
【請求項7】
試薬が、REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化領域を特異的に増幅するためのプライマーである、請求項6に記載のキット。
【請求項8】
試薬が、バイサルファイト試薬及びPCR反応試薬をさらに含む、請求項6又は7に記載のキット。
【請求項1】
消化管間質腫瘍(GIST)をもつ患者の生物学的検体においてREC8遺伝子及びPAX3遺伝子のうちの少なくとも1つの遺伝子のメチル化の有無を測定し、該遺伝子がメチル化されている場合予後不良であり、一方メチル化されていない場合予後良好であると決定することを含む、該患者の予後をインビトロで予測する方法。
【請求項2】
p16遺伝子のメチル化の有無を測定することをさらに含む、請求項1に記載の方法。
【請求項3】
REC8遺伝子及びPAX3遺伝子の両方の遺伝子のメチル化を測定する、請求項1に記載の方法。
【請求項4】
REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化を測定する、請求項1又は2に記載の方法。
【請求項5】
前記遺伝子のメチル化をパイロシークエンス法又はMSP法で測定する、請求項1〜4のいずれか1項に記載の方法。
【請求項6】
REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化の有無を検出するための試薬を含むGIST検査用キット。
【請求項7】
試薬が、REC8遺伝子、PAX3遺伝子及びp16遺伝子のメチル化領域を特異的に増幅するためのプライマーである、請求項6に記載のキット。
【請求項8】
試薬が、バイサルファイト試薬及びPCR反応試薬をさらに含む、請求項6又は7に記載のキット。
【図1】


【図2】


【図3】


【図4】




【図2】


【図3】


【図4】


【公開番号】特開2012−223162(P2012−223162A)
【公開日】平成24年11月15日(2012.11.15)
【国際特許分類】
【出願番号】特願2011−95447(P2011−95447)
【出願日】平成23年4月21日(2011.4.21)
【出願人】(304031427)愛知県 (36)
【Fターム(参考)】
【公開日】平成24年11月15日(2012.11.15)
【国際特許分類】
【出願日】平成23年4月21日(2011.4.21)
【出願人】(304031427)愛知県 (36)
【Fターム(参考)】
[ Back to top ]

