生物種類判定方法
【課題】パターン認識を利用した生物種の判定において、あらかじめ定められたどのカテゴリーにも対応しない生物が未知サンプルに含まれている場合に、生物種の生物学的特長に応じて、誤った判定する可能性を低減することができる判定方法を提供すること。
【解決手段】既知生物ごとに固有の判定不能閾値を設けておき、これらの判定不能閾値を利用して、未知サンプルに対応する生物種を判定するか、しないかを決定し、判定をすると決定されたならば判定を行う。
【解決手段】既知生物ごとに固有の判定不能閾値を設けておき、これらの判定不能閾値を利用して、未知サンプルに対応する生物種を判定するか、しないかを決定し、判定をすると決定されたならば判定を行う。
【発明の詳細な説明】
【技術分野】
【0001】
本発明はパターン認識を用いた生物種判定方法に関するものであり、特に、分析する方法としてDNAマイクロアレイを用いた核酸配列解析システムに好適に適用でき、微生物の種類を判定する用途に用いるとその効果を発揮する。
【背景技術】
【0002】
従来、ガラスなどからなる基板上に位置を定めて固定された、プローブと呼ばれる核酸断片を配備したDNAマイクロアレイは未知の核酸サンプルの分析用として広く利用されてきた。このDNAマイクロアレイを用いることで、未知の核酸断片サンプル(未知サンプルと称す)を解析し、未知サンプルがどの生物種に由来するかを判定する方法にも利用されてきた。この方法ではハイブリダイゼーション反応という、核酸の塩基対形成反応が利用される。
【0003】
ハイブリダイゼーション反応とは何かを以下に説明する。生体内でほとんどの場合、DNAは2重らせん構造をしていて、その2本鎖の間の結合は塩基間の水素結合で実現されている。一方、RNAは1本で存在する場合が多い。塩基の種類はDNAの場合はATGCの4種類、RNAの場合はAUGCの4種類であり、それぞれ水素結合ができる塩基対はA-T(U)、G-Cのペアとなっている。ハイブリダイゼーション反応とは、1本鎖状態の2つの核酸分子同士が適切な条件下で反応して、核酸中にある塩基配列を介して1つに結合するこという。
【0004】
このことを踏まえ、従来の生物種を判定する方法を以下に説明する。基板上に固定されたプローブと、そのプローブと塩基対を形成することのできる相補的な塩基配列をもつ核酸断片とは、適切な反応条件のもとでハイブリダイゼーション反応を生じ、プローブと核酸断片が結合することができる。基板上に固定されたプローブが特定の生物に対応した塩基配列であって、かつハイブリダイゼーション反応が生じてプローブと核酸断片とが結合したことを認識できれば、核酸断片に対応する生物種はプローブに対応する生物種と同一であると判定可能である。すなわち未知サンプルに対応する生物種を判定することができる。
【0005】
例えば、核酸断片に蛍光物質を付与することによってハイブリダイゼーション反応が生じたかどうかを光学的に認識することができる。基板上に固定されたプローブから蛍光が生じた場合、ハイブリダイゼーション反応が生じ、プローブと核酸断片との結合体が形成されたと認識できる。この結果に基づいて、核酸断片はプローブに対応する生物種と同一であると判定される。一方、プローブから蛍光が生じなかった場合、ハイブリダイゼーション反応が生じず、プローブと核酸断片との結合体が形成されなかったと認識され、核酸断片はプローブに対応する生物種ではないと判定される。この判定方法を利用すると、ひとつの未知サンプルが与えられた場合、どの生物種に対応するかを一度のハイブリダイゼーション反応で複数種類の生物種に関して判定することができる。すなわち、対応する生物種が既知のプローブを複数準備し、基板上に位置を定めて固定する。そのようにして作成したDNAマイクロアレイに、未知サンプルを適切な反応条件のもとでハイブリダイゼーション反応をさせる。そして、基板上の位置から生物種を特定し、蛍光の有無でその生物種に対応するか否かを、判定する事ができる。つまり、基板上のどの位置のプローブから蛍光が生じるかを確認することによって、未知サンプルの生物種を判定することができる。
【0006】
しかし実際には、未知サンプルとプローブのハイブリダイゼーション反応の結果、ただ一つの生物種に対応するプローブが蛍光を生じるわけではない。多くの場合、たとえ未知サンプルには一種類の生物種しか対応しないことが事前にわかっていても、ハイブリダイゼーション反応させると、生物種を特定するためのプローブとは別に、他のプローブから蛍光が生じる場合がある。これは核酸分子がその中にある塩基配列を介して部分的に他のプローブと結合する場合があるからであり、クロスハイブリダイゼーションと呼ばれる。このクロスハイブリダイゼーションが発生するために、上記のように基板上の位置と蛍光の有無、の二つの情報のみでは未知サンプルに対応する生物種を判定することができない場合が多い。たとえば未知サンプルを、複数種類の生物種に対応するプローブを備えたDNAマイクロアレイとハイブリダイゼーションさせたとき、生物Aと生物Bに対応するプローブから蛍光を生じたとしても、以下の結果を反映している可能性の検討が必要である。
【0007】
すなわち、クロスハイブリダイゼーションの可能性を考慮すると、生物Aのみ未知サンプルに含まれている場合、生物Bのみ含まれる場合、生物Aと生物Bともに含まれる場合、などが考えられ、一意に未知サンプルに含まれる生物種を決定することができない。
【0008】
一般的な傾向として、同じプローブに結合した核酸断片から発生する蛍光強度は、クロスハイブリダイゼーションして部分的にプローブに結合した場合に生じる蛍光強度よりハイブリダイゼーションしてほぼ完全に結合した場合に生じる蛍光強度の方が強い。よって、DNAマイクロアレイを利用して、未知サンプルを解析し、未知サンプルがどの生物種であるかを判定する場合には、プローブの位置情報と蛍光強度に代表されるシグナル強度の情報と、から総合的に生物種を判定する方法を選択するべきである。
【0009】
なお、DNAマイクロアレイと未知サンプルとのハイブリダイゼーション反応後の蛍光強度はプローブ位置によって順序つけられたベクトルデータとして記憶手段に格納して利用することができる。
【0010】
特表2002-533699号公報には、DNAマイクロアレイを利用して未知のサンプルから得られたベクトルデータを解析し、この未知のサンプルから得られたベクトルデータと最も似ている既知のベクトルデータを検索する方法が開示されている。この最も似ている既知のベクトルデータを検索するという情報処理は、パターン認識として知られ、非常に一般的である。パターン認識とは、観測されたパターンを予め定められた複数の「カテゴリー」のうちの一つに対応させる処理である。「カテゴリー」を説明する例として、OCR(Optical Character Recognition)と呼ばれる技術分野では、紙に印刷、または手書きされた文字を一つのパターンとしてパターン認識する。このとき、認識対象を数字としたとすると、「紙に書かれている文字が0から9の数字のどれに一番近いか?」を既知のベクトルデータと照らし合わせて求める。パターン認識の問題において、この認識すべき0から9の10種類の数字が「カテゴリー」である。
【0011】
通常、パターン認識の問題においては、認識すべきカテゴリーの数と種類が事前に定められている。例えば上記の例では、数字といえば0〜9であり、日本語なら3000字程度の漢字、英語のアルファベットなら26文字というようにカテゴリーの数と種類はあらかじめ定められている。
【特許文献1】特表2002-533699号公報
【発明の開示】
【発明が解決しようとする課題】
【0012】
しかしながら、対応する生物種が未知の核酸断片サンプルをつかってDNAマイクロアレイとハイブリダイゼーションさせた結果得られたベクトルデータを使って、パターン認識する場合、カテゴリーが事前に想定されるとは限らない場合が多い。例えば、未知サンプル中にある細菌が存在するかどうかを、DNAマイクロアレイを用いて判定する場合では、あらかじめプローブとして配備する核酸断片に対応する細菌の種類を決める。しかしながら実際に未知サンプル中に存在する生物が、プローブに対応した生物種の中で収まる可能性は低い。なぜなら、先に説明したOCRと呼ばれる技術分野における0〜9までの9種類のカテゴリー、アルファベットAからZの26種類のカテゴリー、または漢字ならば約3000種類のカテゴリーに比べて、生物種全体の種類は圧倒的多数に上る。それゆえ、たとえ判定したい生物種を細菌に限ったとしても、想定すべきカテゴリーが膨大な数となり、あらかじめ全ての種類の細菌に関するカテゴリーを定めることは事実上不可能であるからである。したがって、未知サンプル中に存在すると想定される生物の種類をある程度限定してカテゴリーを定めることが必要である。
【0013】
そのため、OCRなどの文字認識で利用される従来の方法をそのまま生物種の判定に適用するには問題があった。具体的には、未知サンプル内にあらかじめ定められていないカテゴリーの生物が含まれていた場合、この生物を定められたカテゴリーに無理に対応させてしまうといった誤った判定をしてしまうという問題があった。
【0014】
本発明の目的は、パターン認識を利用した生物種の判定において、あらかじめ定められたどのカテゴリーにも対応しない生物が未知サンプルに含まれている場合に、生物種の生物学的特長に応じて、誤った判定する可能性を低減すること目的とする。
【課題を解決するための手段】
【0015】
本発明の生物種判定方法は、生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物の種類を判定する生物種判定方法において、
対応する生物種が判明している複数の既知サンプルを生物種分析方法により分析して、複数の分析データを得る工程と、
既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する工程と、
対応する生物種が未知である未知サンプルを、前記生物種分析法により分析して、該未知サンプルに対応する生物種の特定のための分析データを得る工程と、
前記判定不能閾値に基づいて前記未知サンプルに対応する種類を判定するか、あるいは判定不能であるかを決定する工程と、
判定をすると決定されたならば、前記複数の分析データに基づいて前記未知サンプルの生物種を判定する工程と、
を有する生物種判定方法である。
【0016】
また、本発明の生物種判定方法の他の態様は、生物に由来する物質が含まれていることが想定される物質を生物種分析方法にて分析して、対応する生物の種類を判定する生物種判定方法において、
(1)前記未知サンプルに対する判定結果として想定される生物種を選択する工程と、
(2)前記選択された生物類に属することが判明している複数の個体から得られる既知サンプルの各々から、該生物種に特徴的であって、パターン認識用として使用し得る複数の画像データからなる画像データ群を得る工程と、
(3)前記画像データ群から画像データを選択し、残りの画像データとの関係を用いて判定不能閾値を設定する工程と、
(4)未知サンプルからの画像データを得る工程と、
(5)前記未知サンプルからの画像データを前記判定不能閾値に基づいて、該未知サンプルに対応する生物種を判定するか判定が不能であるかを決定する工程と、
(6)前記(5)で判定を行うことが決定された場合は、前記画像データ群からなる識別辞書を用いて、生物種を判定する工程と、
を有することを特徴とする生物種類判定方法である。
【0017】
本発明の生物種判定のための情報処理装置は、対応する生物種が判明している複数の既知サンプルを生物分析方法により分析して得られた複数の分析データ、及び、該複数の分析データに基づいて設定された判定不能閾値を記憶したメモリと、該メモリに記憶された判定不能閾値に基づいて、未知サンプルに対応する生物種が判定可能か否かを決定し、判定可能と決定した場合には、前記メモリに記憶された複数の分析データに基づいて前記未知サンプルに対応する生物種の判定を行う処理ユニットとから成る生物種判定のための情報処理装置である。
【0018】
本発明の生物種判定のための情報処理装置の他の態様は、生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物種を判定するための情報処理装置において、対応する生物種が判明している複数の既知サンプルを分析して得られる該生物種に特徴的な画像データを入力するための既知サンプル画像データ入力手段と、
未知サンプルを前記既知サンプルと同様に分析して得られる画像データを入力する未知サンプル画像データ入力手段と、
取り込まれた前記画像データを記憶する記憶手段と、
既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する手段と、
未知サンプルからの画像データを前記判定不能閾値にもとづいて判定を行うかまたは判定を行わないかを決定し、判定を行うのであれば、未知サンプルに対応する生物種を判定する生物種判定手段と、
前記判定手段での判定結果を記憶する記憶手段と、
前記記憶手段に記憶された判定結果を出力する出力手段と
を有することを特徴とする生物種判定のための情報処理装置である。
【0019】
本発明の生物種判定のためのプログラムは、未知サンプルに対応する生物種の判定をコンピュータに実行させるためのプログラムであって、
(1)未知サンプルに対する判定結果として想定される生物種に属する複数の異なる個体からの既知サンプルを分析して得られる該想定される生物種に特徴的な画像データに対応する複数の画像データを格納した記憶手段から、これらの複数の既知サンプル画像データを呼び出すステップと、
(2)未知サンプルを前記既知サンプルと同様にして分析して得られる画像データに対応する複数の画像データを格納した記憶手段から、該未知サンプル画像データを読み出すステップと、
(3)前記既知サンプル画像データから1つを選択し、選択された1つと残りの画像データとの関係を用いて判定不能閾値を設定するステップと、
(4)前記判定不能閾値に基づいて前記未知サンプル画像データを処理し、未知サンプルに対応する生物の種類を判定するステップと、
(5)前記判定ステップで得られた判定結果を記憶手段に格納させるステップと、
(6)前記記憶手段に格納された判定結果を出力するステップと
を有することを特徴とする生物種類判定用プログラムである。
【0020】
本発明の生物種判定のための記録媒体は、生物種判定をコンピュータで実行するためのプログラムを読み取り可能に記録した記録媒体であって、該プログラムが上記構成の生物種判定のためのプログラムであることを特徴とする記録媒体である。
【0021】
本発明の生物種判定方法の他の態様は、対応する生物種が判明している複数の既知サンプルを生物分析方法により分析して得られた複数の分析データと、該複数の分析データに基づいて設定された判定不能閾値を用い、前記判定不能閾値に基づいて、未知サンプルに対応する生物種が判定可能か否かを決定した後、判定可能と決定した場合には、前記複数の分析データに基づいて前記未知サンプルに対応する生物種の判定を行う生物種判定方法である。
【発明の効果】
【0022】
本発明によれば、あらかじめ定められたどのカテゴリーにも対応しない生物が未知サンプルに含まれている場合に、判定不能と判断することができるので、適切な生物種判定結果が得られるという効果がある。また、生物種に対応するカテゴリーごとに判定不能の判断を行うためのパラメータを設定することができるので、生物種の生物学的特長に応じた最適な生物種判定結果が得られるという効果がある。
【発明を実施するための最良の形態】
【0023】
本発明にかかる生物種の判定方法は、ベクトルデータを解析して、識別辞書の作成及び判定不能閾値を決定する方法が含まれる。本発明にかかる生物種の判定方法では、最初に生物種が判明している生物から抽出された核酸断片サンプル(既知サンプルと称す)を分析して得られたベクトルデータを外部記憶手段に格納する。この既知サンプルを分析して得られたベクトルデータを辞書のように参照して未知サンプルの生物種を判定するので、外部記憶手段に格納された既知サンプルを分析して得られた生物種判定用のベクトルデータの総体を、識別辞書と称することにする。
【0024】
次に識別辞書として格納されたベクトルデータを用いて、判定不能閾値を設定する。この判定不能閾値の設定方法に関しては後ほど詳述する。設定された判定不能閾値に従って、対応する生物種を判定したい未知サンプルが作成した識別辞書で生物種を判定できるのか、または判定できない(判定不能)のかが判断される。
【0025】
以下、既知サンプルおよび未知サンプルを分析した結果が画像データとして得られる場合について本発明の判定方法を説明する。
【0026】
識別辞書を作成するための既知サンプルの選定に当たっては、まず判定対象としての生物が属すると想定される生物種類が選択される。例えば未知サンプル中に細菌が存在している可能性があって、細菌に関しての生物種判定を行いたい場合、細菌のなかから既知の生物種をあらかじめ選択する。この選択された生物種が、パターン認識を利用した生物種判定方法におけるカテゴリーに相当する。数字やアルファベットに比べ生物種全体の種類は圧倒的多数に上るので、ある程度限定してカテゴリーを定めることが必要であることは既に述べた。
【0027】
次に、選択された各生物種の固体を用意し、各生物種の固体から抽出された核酸断片サンプルを得る。これを既知サンプルとして、既知サンプル及び未知サンプルから画像データを得るための分析方法を選択する。この分析方法は、パターン認識により生物種の判定が可能となる方法から選択される。例えば、DNAマイクロアレイなどを用いて、得られた画像データをベクトルデータとして認識する分析方法が好適に利用できる。
【0028】
DNAマイクロアレイを利用して画像データをどのようにして得るかを説明する。プローブは、生物種ごとに用意されて、前述のように基板上の所定の位置(すなわち、どの生物種に対応するプローブがどの位置に配備されているかがあらかじめ定められた位置)に固定されている。核酸断片に例えば蛍光物質を付与することによって、DNAマイクロアレイと核酸断片とを適切な条件下で反応させたときに、ハイブリダイゼーション反応が生じたかどうかを光学的に認識することができる。
【0029】
本発明では、1つのカテゴリーに対して、1つの既知サンプルから得た画像データから識別辞書を作成するのではなく、各カテゴリーにおいてそれぞれ2つ以上の既知サンプル(同種の生物の異なる2つ以上の個体)から得られる画像データに基づいて判定不能閾値を設定して、生物種類の判定を行なう。
【0030】
なお、判定したい生物が微生物であれば、生物種類として微生物の「種(species)」を選択することができ、その他様々な生物に本発明が適用できることは言うまでもない。
【0031】
以下、図面に基づいて本発明の一例について説明する。
【0032】
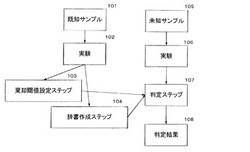
図1は、本発明の生物種類判定方法の一例における処理手順を説明するフローチャートである。この生物種類判定方法は、ある未知サンプル中にターゲットとしての生物種を特定できるこの生物種に由来する物質が存在するか、存在するとすればそれが由来する生物は何の種類に属するかを判定する方法である。生物種類判定方法における棄却とは、ターゲットとして選択した生物種に由来する物質が未知サンプルに存在しないとの判定をすることである。なお、以下においては、微生物などのゲノム解析を用いる生物種類判定を主体として本発明を説明する。しかしながら、例えば、抗原抗体反応を用いた検査システムなどに対しても本発明の技術を適用できる。また、MHCなどの個体識別ゲノム領域などを分析するシステムにも適用してもよい。
【0033】
本発明における未知サンプルの生物種判定処理の流れは、大きくみて、既知サンプルを用いて識別辞書を作成する学習フェーズと未知サンプルを判定する判定フェーズに分かれる。図1において、101から104が学習フェーズで、105から108が判定フェーズである。
【0034】
以下に学習フェーズを説明する。ステップ101では、対応する生物の種類が既知の生物から抽出された核酸断片を含む既知サンプルを用意する。例えば、菌種が特定されている菌のゲノムなどを含む溶液などが既知サンプルに相当する。この既知サンプルを用いて一連のハイブリダイゼーション反応実験102を行ってデータを得る。詳細は後述するが、例えばDNAマイクロアレイを用いた場合、まずPCR反応により既知サンプルに含まれる核酸断片を増幅し、蛍光物質を付与する。その後、DNAマイクロアレイとハイブリダイゼーション反応をして、それぞれのスポットの蛍光強度のデータを画像として認識して外部記憶手段に格納する。この画像データをもとに、判定不能閾値設定ステップ103と、辞書作成ステップ104で、判定不能閾値と識別辞書がそれぞれ作成される。
【0035】
次に判定フェーズを説明する。未知のサンプルを用意し(ステップ105)、ステップ102と全く同じ手順でハイブリダイゼーション反応実験106を実施する。ハイブリダイゼーション反応により得られた画像データと、学習フェーズで得られた判定不能閾値、識別辞書とを照らし合わせることによって、未知サンプルに対して生物種の判定を行う(ステップ107)。その結果判定結果108として、「未知サンプルは生物種Aに対応する」、「未知サンプルには生物種A〜Cに由来する物質が含まれる」、「未知サンプルには生物種A〜Z以外で生物群αに含まれる生物に由来する物質が存在する」、「105の未知サンプルは判定できない(=判定不能)」というような結果が得られる。
【0036】
以上に説明した学習フェーズ中の、特に判定不能閾値の設定方法について異なる2通りの方法を以下に詳しく説明する。
【0037】
まず同種であるが異なる生物個体から得られた既知サンプルを用意し、DNAマイクロアレイとハイブリダイゼーション反応させ画像データを得る。判定不能閾値の設定には、次の方法のいずれかを好ましく用いることができる。
・方法(1)3以上の既知サンプルの画像データから1つを選択して除外し、残りの既知サンプルの画像データから識別辞書を作成し、それを利用して判定不能閾値を設定する方法。
・方法(2)3以上の既知サンプルの画像データから選択した任意の2つの組み合わせの全てについてパターン認識アルゴリズムにより求めた距離を利用して判定不能閾値を設定する方法。
【0038】
まず、上記の方法(1)の判定不能閾値を設定する方法について説明する。この方法の処理手順のフローチャートを図8に示す。まず、未知サンプルに対する生物種判定結果として想定されるn種の異なる生物種(S1〜Sn:n≧2)、すなわちターゲットカテゴリーを選択する(ステップ802)。次に、選択されたターゲットカテゴリーごとに固有の判定不能閾値を得るための処理を行う。次にステップ802でターゲットカテゴリーとして選択されたカテゴリーの既知サンプルを用意して、ハイブリダイゼーションさせた結果、画像データを得ることができる。画像データは、識別辞書を作成するために外部記憶手段に格納される。この画像データの総体を学習データと呼ぶことにする。以下、ターゲットカテゴリーS1に属する生物種を例にあげて、(1)の判定不能閾値を設定する方法を説明する。
【0039】
まず、ターゲットカテゴリーS1に属するm個の個体S1−X(1≦X≦m、m≧3)を用意する。用意した各個体から拡散断片を抽出し、m個(m≧3)の既知サンプルを得る。このm個の既知サンプルとDNAマイクロアレイとを適当な条件下でハイブリダイゼーションさせ、m個(m≧3)(Ps1−1〜Ps1−m)の画像データ群を得る。次に、学習データ分割ステップ803において、これらの画像データから1つを選択して学習データから除去する。次に、1つの画像データを除いた残りのm−1個の学習データ804を用いて、辞書作成ステップ805において識別辞書806を作成する。この辞書作成ステップ805は、採用したパターン認識アルゴリズムに則って作成される。
【0040】
パターン認識による未知パターンの判定には、公知の方法から選択した方法を利用することができる。パターン認識による判定や分類のための方法は、例えば、IEEE Transaction on Pattern Analysis and Machine Learning, Vol. 22, No. 1, January 2000, pp.4-pp.37にある"Statistical Pattern Recognition: A Review"Anil K. Jain, Robert P.W. Duin, and Jianchan Mao. の論文にレビューされている。具体的には、例えばk-Nearest-Neighbor法、分類木、Support Vector Machine、ベイズ識別法、ブースティング法、ニューラルネットなどのパターン認識の技術が利用できる。
【0041】
例えばニューラルネットをパターン認識アルゴリズムとして採用したとすると、ネットワークの重みパラメータ集合が識別辞書として学習される。例えばSupport Vector Machineがパターン認識アルゴリズムとして採用されたとすると、いわゆるSupport Vectorと呼ばれる代表するサンプルベクトルとその重み付けが識別辞書として学習される。本発明において、識別辞書として学習される、もしくは学習する、とは、学習データに基づいて識別辞書を作成することと同義である。
【0042】
次に、学習データから除いた一つの画像データを、識別辞書806を用いて判定する(ステップ808)。ここでは、例えば個体S1−1に対応する画像データPs1−1を学習データから除いたとしよう。この時、注意すべきなのは、識別辞書806はステップ807で除いた1つの画像データを含まない。よって、識別辞書806に対して、ステップ807で除かれた個体S1−1は未知サンプルとなる。識別辞書を用いて判定を行う場合、外部記憶手段に格納されたベクトルデータ同士を比較するために、ユークリッドノルムに代表されるノルムをあらかじめ定義する必要がある。本発明の生物種判定方法にユークリッドノルムを採用した場合については後述されるが、もちろん一般の種々のノルムを採用してもかまわない。以上の工程を経て、判定指数809が得られる(ステップ809)。
【0043】
一般にパターン認識アルゴリズムの判定結果は、数値データとなる。例えば判定確率であったり、類似度であったり、単にベクトルデータ同士の距離であったりする。このように、判定指数809はあらかじめ定義されたノルムを用いて算出された判定結果である数値データを意味する。
【0044】
こうして、m個の画像データのうち、学習データから一つの画像データを除いた学習データをもちいて作成した識別辞書を用いて、ターゲットカテゴリーS1についての一つの判定指数A1−1が得られる。
【0045】
次に、上記の学習データ分割ステップ803で除かれなかったターゲットカテゴリーS1の画像データを新たにS1−X(1≦X≦m、m≧3)の中から選択して同様に除く。ここではS1−2に対応する画像データを選択したとしよう。同様の処理を行ない、ターゲットカテゴリーS1に関する判定指数A1−2を得る。すなわち上記の操作を同様にターゲットカテゴリーS1の各画像データに実行することによりm個の判定指数からなる判定指数集合{A1}を得る。判定指数集合{A1}は、m個の判定指数の元(げん)、A1−1、A−2、・・・A1−m、からなる。
【0046】
こうして得られた判定指数集合{A1}からターゲットカテゴリーS1に関する判定不能閾値を設定することができる。上記ではターゲットカテゴリーS1に関して例にあげて判定指数集合を得る方法を説明した。同様にして最初に選んだn種のターゲットカテゴリーのうち、ターゲットカテゴリーS1以外の他のターゲットカテゴリーに関してもそれぞれ判定指数集合を得る。その結果、n個の判定指数集合を得ることができる。
【0047】
図9に、n個の判定指数集合のうちから一つの判定指数集合を選び、判定指数集合810の分布をヒストグラムで表示した例を示す。判定指数809が類似度を示している場合、判定不能閾値を、例えば、集合の最小値のα倍(α<1)に設定したり、集合の平均値や中央値のβ倍(β>0)にしたりする。逆に、809の判定指数が非類似度を示している場合、判定不能閾値を、例えば、集合の最大値のα倍(α>1)に設定したり、集合の平均値や中央値のβ倍(β>0)にしたりする。判定不能閾値を、判定指数集合に基づいてどのような値に設定するかは、検査対象としての生物の種類、パターン認識を用いる分析方法の種類、目的とする判定精度などに応じてターゲットカテゴリーごとに選択できる。このようにして定めた判定不能閾値の設定が適切かどうかを確認する方法として、選択したターゲットカテゴリーに含まれないことがあらかじめ判明しているサンプルを未知サンプル105として用いる方法がある。図1を用いて前述した未知サンプルの生物種判定処理を実行して、この未知サンプルについて「判定不能である」との結果がでるかどうかを試験して、判定不能閾値の設定が正しいかどうかを確認することができる。
【0048】
つぎに、方法(2)の判定不能閾値を設定する方法を以下に説明する。図10に判定不能閾値を設定する別の例を示す。パターン認識アルゴリズムとしてk-Nearest-Neighbor法(特にk=1)を選び、ノルムとしてユークリッドノルムを採用した場合の判定不能閾値設定方法を以下に説明する。この場合、未知サンプルを分析して得られた画像データから算出された判定指数が非類似度を示すとすると、定められた判定不能閾値より大きい時に「判定不能」という結果がでることになる。判定不能閾値を設定するために、まずひとつのターゲットカテゴリーS1を選び、S1に属する全ての既知サンプルをハイブリダイゼーションさせた結果、画像データを得て外部記憶手段に格納する。この格納された画像データの総体からS1に属する任意の2つの画像データの組み合わせを選択し、この2つの画像データをもとに認識されたプローブ位置によって順序つけられた蛍光強度からなるベクトルデータ同士のユークリッド距離を算出する。つづいて、上記で選ばれなかった2つの画像データの組み合わせを新しく選出し、同様にして、新しく選出された2つの画像データに基づいてユークリッド距離を算出する。このような手順で、S1に属し外部記憶手段に格納された画像データ群に関してそれぞれの組み合わせに基づいてユークリッド距離が算出される。ターゲットカテゴリーに属する既知サンプルが6つ用意された場合を図7に示す。この場合、2つの画像データをもとに算出されるユークリッド距離の個数は6C2=15となる。
【0049】
ターゲットカテゴリーS1に属する全ての画像データの組み合わせに基づいて算出されたユークリッド距離を、判定指数をx軸としてヒストグラムを用いて示したものが図10である。図10は距離の分布に2つの山が存在する。カテゴリーに属するサンプルベクトルが2つの領域に局在することを意味する。このように、ヒストグラムからターゲットカテゴリーS1の性質を確認できるので、各ターゲットカテゴリーにごとに適切な判定不能閾値の設定方法を選ぶことができる。
例えば、この距離集合の平均値や中央値などの統計的代表値をもって判定不能閾値とすることができる。
【0050】
次に、得られた判定不能閾値を、図1を用いて前述した未知サンプルの生物種判定処理を実行して、このようにして定めた判定不能閾値の設定が適切かどうかを、方法(1)と同じ方法で確認する。選択したターゲットカテゴリーに含まれないことがあらかじめ判明しているサンプルを未知サンプル105として用いる。この未知サンプルについて「判定不能である」との結果がでるかどうかを試験して、判定不能閾値の設定が正しいかどうかを確認する。
【0051】
次に、上記の生物種類判定方法に用い得る情報処理装置としてのコンピュータシステム、プログラム、画像認識を用いた分析方法などの各処理について説明する。
【0052】
以上説明した生物種類判定は、予め作成されたプログラムに従ったコンピュータ上での処理により自動化可能である。本発明にかかる生物種類判定のための情報処理装置は、生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物種を判定するための情報処理装置である。この情報装置は以下の少なくとも手段を用いて構成することができる。
(1)対応する生物種が判明している複数の既知サンプルを分析して得られる該生物種に特徴的な画像データを入力するための既知サンプル画像データ入力手段。
(2)未知サンプルを前記既知サンプルと同様に分析して得られる画像データを入力する未知サンプル画像データ入力手段。
(3)取り込まれた前記画像データを記憶する記憶手段。
(4)既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する手段。
(5)未知サンプルからの画像データを前記判定不能閾値にもとづいて処理し、未知サンプルの提供元である生物の種類を判定する生物種類判定手段。
(6)前記判定手段での判定結果を記憶する記憶手段。
(7)前記記憶手段に記憶された判定結果を出力する出力手段。
【0053】
上記の判定不能閾値の設定は、記憶手段に3以上の個体からの画像データを記憶手段に記憶させておき、以下のステップを有するプログラムに基づいて実行されることが好ましい。
(a)3以上の画像データから1つの画像データを選択して除外し、残りの複数の画像データを用いて識別辞書を作成し、得られた識別辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なって3以上の判定指数からなる判定指数集合を得るステップ。
(b)前記判定指数集合から判定不能閾値を設定するステップ。
【0054】
また、上記の判定不能閾値の設定は、次のようにして行われることが好ましい。すなわち、記憶手段に3以上の個体からの画像データを記憶手段に記憶させておく。そして、前記個体が3以上であり、前記記憶手段にこれらの個体からの画像データが記憶されており、前記判定不能閾値の設定が、少なくとも以下の工程を実行するプログラムに基づいて行われることも好ましい。
(A)前記3以上の画像データから選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程。
(B)前記距離集合から判定不能閾値を決定する工程。
【0055】
また、本発明にかかる生物種類判定のためのプログラムは、未知サンプルに対応する生物の種類の判定をコンピュータに実行させるためのプログラムであって、少なくとも以下のステップを実行するためのものである。
(1)未知サンプルに対する判定結果として想定される生物種に属する複数の異なる個体からの既知サンプルを分析して得られる該想定される生物種に特徴的な画像データに対応する複数の画像データを格納した記憶手段から、これらの複数の既知サンプル画像データを呼び出すステップ。
(2)未知サンプルを前記既知サンプルと同様にして分析して得られる画像データに対応する複数の画像データを格納した記憶手段から、該未知サンプル画像データを読み出すステップ。
(3)前記既知サンプル画像データから1つを選択し、選択された1つと残りの画像データとの関係を用いて判定不能閾値を設定するステップ。
(4)前記判定不能閾値に基づいて前記未知サンプル画像データを処理し、未知サンプルに対応する生物の種類を判定するステップ。
(5)前記判定ステップで得られた判定結果を記憶手段に格納させるステップ。
(6)前記記憶手段に格納された判定結果を出力するステップ。
【0056】
上記の判定不能閾値の設定ステップは、個体を3以上とし、記憶手段にこれらの個体からの画像データが記憶を記憶させておき、少なくとも以下のステップによって行うことが好ましい。
(a)前記3以上の画像データから1つの画像データを選択して除外し、残りの複数の画像データを用いて識別辞書を作成し、得られた識別辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なって3以上の判定指数からなる判定指数集合を得るステップ。
(b)前記判定指数集合から判定不能棄却閾値を決定するステップ。
【0057】
また、上記の判定不能閾値の設定ステップは、個体を3以上とし、記憶手段にこれらの個体からの画像データが記憶を記憶させておき、少なくとも以下のステップによって行うことが好ましい。
(A)前記3以上の画像データから選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程。
(B)前記距離集合から判定不能閾値を決定する工程。
【0058】
記憶手段に、多数の既知生物種類のそれぞれにおける判定不能閾値を格納しておき、未知サンプルの種類に応じて、未知試料に含まれる生物由来物質がその存在を示すことが想定される必要数のカテゴリーを選択するステップをプログラムに追加しておくとよい。このことにより、判定不能かどうかを検討するカテゴリー数を効果的に低減でき、より効率の良い判定処理が可能となる。
【0059】
なお、上記のプログラムは、コンピュータシステムの記憶手段中に保持させておいてもよいし、記録媒体に格納して使用者に配布できるようにしてもよい。更には、ネットワークシステムを介して配布できるようにしてもよい。
【0060】
図2に、生物種類判定方法を実行し得るコンピュータシステムを利用した情報処理装置の構成の一例のブロック図を示す。この装置は、外部記憶装置201、中央処理装置(CPU)202、メモリ203、入出力装置204を少なくとも有して構成される。外部記憶装置201には、生物種類判定を行なうための上述した構成のプログラムや、既知サンプル及び未知サンプルを対するハイブリダイゼーション反応を利用した分析の結果としての画像データが保持される。外部記憶装置201には、更に判定不能閾値を用いた決定の結果を保持させる。中央処理装置(CPU)202は、生物種類判定のためのプログラムを実行したり、すべての装置の制御を行なったりする。メモリ203は中央処理装置(CPU)202が使用するプログラム、及びサブルーチンやデータを一時的に記録する。入出力装置204は、ユーザーとのインタラクションを行う。多くの場合、プログラム実行のトリガーはこの入出力装置を介してユーザーが出す。また、ユーザーが結果を見たり、プログラムのパラメータ制御をこの入出力装置を介して行う。
【0061】
図3はDNAマイクロアレイ上のハイブリダイゼーションの様子を示した図である。生体内でほとんどの場合、DNAは2重らせん構造をしていて、その2本鎖の間の結合は塩基間の水素結合で実現されている。一方、RNAは1本で存在する場合が多い。塩基の種類はDNAの場合はATGCの4種類、RNAの場合はAUGCの4種類であり、それぞれ水素結合ができる塩基対はA-T(U)、G-Cのペアとなっている。一般にハイブリダイゼーション反応とは、1本鎖状態の核酸分子同士がその中にある部分塩基配列を介して部分的に結合する状態をいう。図3に示す例では、図中上側の基板にくっついた核酸分子(プローブ)の方が下側のサンプル中にある核酸分子より短い。サンプル中に存在する核酸分子がプローブの塩基配列を含む場合は、このハイブリダイゼーション反応はうまくいき、サンプル中の核酸分子はDNAマイクロアレイにトラップされることとなる。
【0062】
次に、図4を用いてDNAマイクロアレイを用いて画像データを得るための実験手順全般について説明する。401の「サンプル」とは対象としている生物由来物質、例えば核酸(細胞に含まれている状態のものも含む)が含まれている、あるいは含まれていることが想定される液体や個体である。例えば、感染症の原因菌の特定をするために本発明を適用した場合、ヒト、家畜等の動物由来の血液、喀痰、胃液、膣分泌物、口腔内粘液等の体液、尿及び糞便のような排出物、人、動物などから採取した組織片等の細菌などの微生物やそれに由来する物質が存在すると思われるあらゆる物が未知サンプル401の供給元となる。また、食中毒、汚染の対象となる食品、飲料水及び温泉水のような環境中の水等、細菌による汚染が引き起こされる可能性のある媒体が未知サンプルの供給元として用いられることもある。さらに、輸出入時における検疫等の動植物も検体としてその対象となる。既知サンプルの場合には、種類が既知である微生物などから調製されたサンプルである。
【0063】
次に、必要に応じて、402の"生化学的増幅"方法を用いて401のサンプルとしての核酸を増幅する。例えば感染症の原因菌の特定をするために本発明を適用した場合、16s rRNA検出用に設計されたPCR反応用プライマーを用いてPCR法によって対象核酸を増幅したり、或いはPCR増幅物を元にさらにPCR反応等を行なって調整したりする。また、PCR以外のLAMP法などの増幅方法により調整してもよい。
【0064】
その後で、増幅されたサンプル、または401のサンプルそのものに、可視化のために各種標識法により標識する。この標識物質としては、通常Cy3, Cy5, Rodaminなどの蛍光物質が用いられる。また、402の生化学的増幅の実験手順の中で、標識分子を混入することもある。
【0065】
そして、標識分子が付加された核酸を図1における、404のDNAマイクロアレイとハイブリダイゼーション反応(405)を行う。この様子は、図3に示した通りである。例えば、感染症の原因菌の特定をするために本発明を適用した場合、404のDNAマイクロアレイは、菌に特異的なプローブを基板に固定したものとなる。各菌のプローブの設計は、例えば16s rRNAをコーディングしているゲノム部分より、当該菌に対し非常に特異性が高く、十分かつそれぞれのプローブ塩基配列で"出来るだけ"ばらつきのないハイブリダイゼーション感度が期待できるように行う。404のDNAマイクロアレイのプローブを固定する担体(基板)は、ガラス基板、プラスチック基板、シリコンウェハー等の平面基板が考えられる。また、凹凸のある三次元構造体、ビーズのような球状のもの、棒状、紐状、糸状のもの等を用いても、本発明の実景形態、効果には影響ない。
【0066】
通常、基板の表面はプローブDNAの固定化が可能なように処理したものが使用される。特に、表面に化学反応が可能となるように官能基を導入した物は、ハイブリダイゼーション反応の過程でプローブが安定に結合している為に、再現性の点で好ましい形態である。プローブの固定化方法としては、例えば、マレイミド基とチオール(−SH)基との組合わせを用いて基板上にプローブを固定化する方法が挙げられる。即ち、核酸プローブの末端にチオール(−SH)基を結合させておき、固相表面がマレイミド基を有するように処理しておくことで、固相表面に供給された核酸プローブのチオール基と固相表面のマレイミド基とが反応して核酸プローブを固定化する。ガラス基板へのマレイミド基の導入は、まず、ガラス基板にアミノシランカップリング剤を反応させ、次にそのアミノ基とEMCS試薬(N-(6-Maleimidocaproyloxy)succinimide :Dojin社製)との反応により行うことができる。DNAへのSH基の導入は、DNA自動合成機上5'-Thiol-ModifierC6(Glen Research社製)を用いる事により行なうことができる。固定化に利用する官能基の組合わせとしては、上記したチオール基とマレイミド基の組合わせ以外にも、例えばエポキシ基(固相上)とアミノ基(核酸プローブ末端)の組合わせ等が挙げられる。また、各種シランカップリング剤による表面処理も有効であり、該シランカップリング剤により導入された官能基と反応可能な官能基を導入したオリゴヌクレオチドが用いられる。さらに、官能基を有する樹脂をコーティングする方法も利用可能である。
【0067】
ハイブリダイゼーション反応を行った後、404のDNAマイクロアレイの表面を洗浄し、プローブと結合していない核酸を剥がした後で、通常は乾燥し、405の蛍光量を測定する。そして、DNAマイクロアレイの基板に励起光を照射し、蛍光強度を測定した画像(406)が得られる。この画像(406)が画像データとなる。画像データの一例を図6に示した。異なる既知サンプルに対応して、図6の601と602とで異なる画像データ(画像)が得られている。
【0068】
次に、図5を用いて感染症の菌を特定する場合のDNAマイクロアレイの原理を示す。図5で示したDNAマイクロアレイは、例えば、黄色ブドウ球菌を特定する目的で作られている。左の列は、黄色ブドウ球菌野生株由来の処理系列であり、右の列は大腸菌野生株由来の処理系列である。例えば、左は黄色ブドウ球菌に感染した患者の血液を処理する流れで、右は大腸菌に感染した患者の血液を処理する流れだと考えてよい。
【0069】
どちらも基本的には同じ処理を行う。つまり、まず初めに例えば菌感染患者の血液や、痰などからDNAを抽出する。この際に、一般的には、患者の体細胞由来の人間のDNAも含まれる可能性がある。
【0070】
抽出されたDNAが少ない場合、PCR法などの方法で増幅を行う。この際に蛍光物質もしくは蛍光物質を結合させることができる物質を標識として混入させるのが一般的である。増幅をしない場合は、抽出されたDNAを用いて、相補鎖を作りながら蛍光物質もしくは蛍光物質を結合させることができる物質を標識として混入させる、または、そのまま直接抽出されたDNAに蛍光物質もしくは蛍光物質を結合させることができる物質を標識として付加させる。
【0071】
通常、PCR増幅を行う場合、感染症の菌特定目的であれば、いわゆる16s rRNAといわれるリボゾームRNAを構成する塩基配列の部分を増幅するのが一般的である。この場合、左の黄色ブドウ球菌のPCRプライマーと右の大腸菌のPCRプライマーはほとんど同じものを使うこととなる。より具体的には、どんな菌の16s rRNAをコーディングしている部分でも増幅させることができるプライマーセットを用いて、マルチプレックスPCRを行う。
【0072】
黄色ブドウ球菌を判定する目的のために設計されたDNAマイクロアレイが正しく動作するならば、左のハイブリ溶液では、スポットがポジティブに反応し、右のハイブリ溶液では、スポットがネガティブに反応する。これと全く同じように、大腸菌の存在を判定する目的のために設計されたDNAマイクロアレイが正しく動作するならば、次の反応が生じる。すなわち、左のハイブリ溶液(ハイブリダイゼーション用溶液)では、スポットがネガティブに反応し、右のハイブリ溶液(ハイブリダイゼーション用溶液)では、スポットがポジティブに反応する。
【0073】
ポジティブに反応したスポットからの蛍光強度を測定して図4で示すスキャン画像処理を行なうことで画像データを得ることができる。ここで、同じ種類に属する異なる個体からのサンプルを用いて同じ分析条件で画像データを得た場合に、常に同じ蛍光強度が得られれば、それを辞書として用いればよい。しかしながら、実際には、蛍光強度にバラツキが生じるので、未知サンプルからの画像データがこのバラツキの範囲内なのか、あるいはその範囲外で既知のカテゴリーに属さないと判定すべきかどうかの明確な基準を得ることが困難な場合がある。更に、後述の実施例で示すように、プローブによってはクロスハイブリダイゼーションを生じる。そこで本発明では、図8に示すように同一種類に属する多数の異なる個体からのサンプルを用いた識別辞書作成と判定不能閾値の設定より、カテゴリーごとに未知サンプルの判定を行なうかどうかの基準を明確としている。
【実施例】
【0074】
以下、本発明の生物種類判定方法に利用し得る分析データの取得方法の具体例を挙げる。なお、本発明は、以下に述べる感染症の原因菌特定に限ったものではなく、MHCなどの人間の体質判定や、癌などの疾病に関わるDNA、RNAの解析に用いてもよい。
【0075】
実施例1
<プローブDNAの準備>
Enterobacter cloacae菌検出用Probeとして以下に示す核酸配列(I−n)(nは数字)を設計した。具体的には、16s rRNAをコーディングしているゲノム部分より、以下に示したプローブ塩基配列を選んだ。これらのプローブ塩基配列群は、当該菌に対し非常に特異性が高く、十分かつそれぞれのプローブ塩基配列で"出来るだけ"ばらつきのないハイブリダイゼーション感度が期待できるように設計されている。
I-1:CAgAgAgCTTgCTCTCgggTgA
I-2:gggAggAAggTgTTgTggTTAATAAC
I-3:ggTgTTgTggTTAATAACCACAgCAA
I-4:gCggTCTgTCAAgTCggATgTg
I-5:ATTCgAAACTggCAggCTAgAgTCT
I-6:TAACCACAgCAATTgACgTTACCCg
I-7:gCAATTgACgTTACCCgCAgAAgA
上記のプローブは、DNAマイクロアレイに固定するための官能基として、合成後、定法に従って核酸の5'末端にチオール基を導入した。官能基の導入後、精製し、凍結乾燥した。凍結乾燥した内部標準用プローブは、-30℃の冷凍庫に保存した。
【0076】
一方、黄色ブドウ球菌(A−n)、表皮ブドウ球菌(B−n)、大腸菌(C−n)、肺炎桿菌(D−n)、緑膿菌(E−n)、セラチア菌(F−n)、肺炎連鎖球菌(G−n)、インフルエンザ菌(H−n)、及びエンテロコッカス・フェカリス菌(J−n)(nは数字)についても同様な手法により以下に示すプローブセットを設計した。
A-1:gAACCgCATggTTCAAAAgTgAAAgA
A-2:CACTTATAgATggATCCgCgCTgC
A-3:TgCACATCTTgACggTACCTAATCAg
A-4:CCCCTTAgTgCTgCAgCTAACg
A-5:AATACAAAgggCAgCgAAACCgC
A-6:CCggTggAgTAACCTTTTAggAgCT
A-7:TAACCTTTTAggAgCTAgCCgTCgA
A-8:TTTAggAgCTAgCCgTCgAAggT
A-9:TAgCCgTCgAAggTgggACAAAT
B-1:gAACAgACgAggAgCTTgCTCC
B-2:TAgTgAAAgACggTTTTgCTgTCACT
B-3:TAAgTAACTATgCACgTCTTgACggT
B-4:gACCCCTCTAgAgATAgAgTTTTCCC
B-5:AgTAACCATTTggAgCTAgCCgTC
B-6:gAgCTTgCTCCTCTgACgTTAgC
B-7:AgCCggTggAgTAACCATTTgg
C-1:CTCTTgCCATCggATgTgCCCA
C-2:ATACCTTTgCTCATTgACgTTACCCg
C-3:TTTgCTCATTgACgTTACCCgCAg
C-4:ACTggCAAgCTTgAgTCTCgTAgA
C-5:ATACAAAgAgAAgCgACCTCgCg
C-6:CggACCTCATAAAgTgCgTCgTAgT
C-7:gCggggAggAAgggAgTAAAgTTAAT
D-1:TAgCACAgAgAgCTTgCTCTCgg
D-2:TCATgCCATCAgATgTgCCCAgA
D-3:CggggAggAAggCgATAAggTTAAT
D-4:TTCgATTgACgTTACCCgCAgAAgA
D-5:ggTCTgTCAAgTCggATgTgAAATCC
D-6:gCAggCTAgAgTCTTgTAgAgggg
E-1:TgAgggAgAAAgTgggggATCTTC
E-2:TCAgATgAgCCTAggTCggATTAgC
E-3:gAgCTAgAgTACggTAgAgggTgg
E-4:gTACggTAgAgggTggTggAATTTC
E-5:gACCACCTggACTgATACTgACAC
E-6:TggCCTTgACATgCTgAgAACTTTC
E-7:TTAgTTACCAgCACCTCgggTgg
E-8:TAgTCTAACCgCAAgggggACg
F-1:TAgCACAgggAgCTTgCTCCCT
F-2:AggTggTgAgCTTAATACgCTCATC
F-3:TCATCAATTgACgTTACTCgCAgAAg
F-4:ACTgCATTTgAAACTggCAAgCTAgA
F-5:TTATCCTTTgTTgCAgCTTCggCC
F-6:ACTTTCAgCgAggAggAAggTgg
G-1:AgTAgAACgCTgAAggAggAgCTTg
G-2:CTTgCATCACTACCAgATggACCTg
G-3:TgAgAgTggAAAgTTCACACTgTgAC
G-4:gCTgTggCTTAACCATAgTAggCTTT
G-5:AAgCggCTCTCTggCTTgTAACT
G-6:TAgACCCTTTCCggggTTTAgTgC
G-7:gACggCAAgCTAATCTCTTAAAgCCA
H-1:gCTTgggAATCTggCTTATggAgg
H-2:TgCCATAggATgAgCCCAAgTgg
H-3:CTTgggAATgTACTgACgCTCATgTg
H-4:ggATTgggCTTAgAgCTTggTgC
H-5:TACAgAgggAAgCgAAgCTgCg
H-6:ggCgTTTACCACggTATgATTCATgA
H-7:AATgCCTACCAAgCCTgCgATCT
H-8:TATCggAAgATgAAAgTgCgggACT
J-1:TTCTTTCCTCCCgAgTgCTTgCA
J-2:AACACgTgggTAACCTACCCATCAg
J-3:ATggCATAAgAgTgAAAggCgCTT
J-4:gACCCgCggTgCATTAgCTAgT
J-5:ggACgTTAgTAACTgAACgTCCCCT
J-6:CTCAACCggggAgggTCATTgg
J-7:TTggAgggTTTCCgCCCTTCAg
<検体増幅用PCR Primer の準備>
起炎菌検出用の為の16s rRNA核酸(標的核酸)増幅用PCR Primerとして表1に示す核酸配列を設計した。具体的には、16s rRNAをコーディングしているゲノム部分を特異的に増幅するプローブセット、つまり約1500塩基長の16s rRNAコーディング領域の両端部分で、特異的な融解温度をできるだけ揃えたプライマーを設計した。なお、変異株や、ゲノム上に複数存在する16s rRNAコーディング領域も同時に増幅できるように複数種類のプライマーを設計した。
【0077】
【表1】

【0078】
表中に示したPrimerは、合成後、高速液体クロマトグラフィー(HPLC)により精製し、Forward Primer 3種、Reverse Primer 3種を混合し、それぞれのPrimer濃度が、最終濃度10 pmol/μl となるようにTE緩衝液に溶解した。
【0079】
<Enterobacter#cloacae Genome DNA(モデル検体)の抽出>
(微生物の培養 & Genome DNA 抽出の前処理)
まず、Enterobacter cloacae 標準株を、定法に従って培養した。この微生物培養液を1.5ml容量のマイクロチューブに1.0ml(OD600=0.7)採取し、遠心分離で菌体を回収した(8500rpm、5min、4℃)。上精を捨てた後、Enzyme Buffer(50mM Tris-HCl:p.H. 8.0、25mM EDTA)300μlを加え、ミキサーを用いて再縣濁した。再縣濁した菌液は、再度、遠心分離で菌体を回収した(8500rpm、5min、4℃)。上精を捨てた後、回収された菌体に、以下の酵素溶液を加え、ミキサーを用いて再縣濁した。
Lysozyme:50 μl (20 mg/ml in Enzyme Buffer)
N-Acetylmuramidase SG:50 μl (0.2 mg/ml in Enzyme Buffer)
次に、酵素溶液を加え再縣濁した菌液を、37℃のインキュベーター内で30分間静置し、細胞壁の溶解処理を行った。
【0080】
(Genome抽出)
以下に示す微生物のGenome DNA抽出は、核酸精製キット(MagExtractor -Genome-:TOYOBO社製)を用いて行った。具体的には、まず、前処理した微生物縣濁液に溶解・吸着液750μlと磁性ビーズ40μlを加え、チューブミキサーを用いて、10分間激しく攪拌した(ステップ1)。次に、分離用スタンド(Magical Trapper)にマイクロチューブをセットし、30秒間静置して磁性粒子をチューブの壁面に集め、スタンドにセットした状態のまま、上精を捨てた(ステップ2)。次に、洗浄液 900 μl を加え、ミキサーで5sec程度攪拌して再縣濁を行った(ステップ3)。次に、分離用スタンド(Magical Trapper)にマイクロチューブをセットし、30秒間静置して磁性粒子をチューブの壁面に集め、スタンドにセットした状態のまま、上精を捨てた(ステップ4)。ステップ3、4を繰り返して2度目の洗浄(ステップ5)を行った後、70%エタノール 900 μl を加え、ミキサーで5sec程度攪拌して再縣濁した(ステップ6)。次に、分離用スタンド(Magical Trapper)にマイクロチューブをセットし、30秒間静置して磁性粒子をチューブの壁面に集め、スタンドにセットした状態のまま、上精を捨てた(ステップ7)。ステップ6、7を繰り返して70%エタノールによる2度目の洗浄(ステップ8)を行った後、回収された磁性粒子に純水 100 μl を加え、チューブミキサーで10分間攪拌を行った。
【0081】
次に分離用スタンド(Magical Trapper)にマイクロチューブをセットし、30秒間静置して磁性粒子をチューブ壁面に集め、スタンドにセットした状態のまま、上精を新しいチューブに回収した。
【0082】
(回収したGenome DNAの検査)
回収された微生物(Enterobacter cloacae 株)のGenome DNAは、定法に従って、アガロース電気泳動と260/280nmの吸光度測定を行い、その品質(低分子核酸の混入量、分解の程度)と回収量を検定した。本実施例では、約10μgのGenome DNA が回収され、GenomeDNAのデグラデーションやrRNAの混入は認められなかった。回収したGenome DNAは、最終濃度50ng/μlとなるようにTE緩衝液に溶解し、以下の工程に使用した。
【0083】
<DNAマイクロアレイの作製>
[1]ガラス基板の洗浄
合成石英のガラス基板(サイズ:25mmx75mmx1mm、飯山特殊ガラス社製)を耐熱、耐アルカリ のラックに入れ、所定の濃度に調製した超音波洗浄用の洗浄液に浸した。一晩洗浄液中で浸した後、20分間超音波洗浄を行った。続いて基板を取り出し、軽く純水ですすいだ後、超純水中で20分超音波洗浄をおこなった。次に80℃に加熱した1N水酸化ナトリウム水溶液中に10分間基板を浸した。再び純水洗浄と超純水洗浄を行い、DNAチップ用の石英ガラス基板を用意した。
【0084】
[2]表面処理
シランカップリング剤KBM-603(信越シリコーン社製)を、1%の濃度となるように純水中に溶解させ、2時間室温で攪拌した。続いて、先に洗浄したガラス基板をシランカップリング剤水溶液に浸し、20分間室温で放置した。ガラス基板を引き上げ、軽く純水で表面を洗浄した後、窒素ガスを基板の両面に吹き付けて乾燥させた。次に乾燥した基板を120℃に加熱したオーブン中で1時間ベークし、カップリング剤処理を完結させ、基板表面にアミノ基を導入した。次いで同仁化学研究所社製のN-マレイミドカプロイロキシスクシイミドを、ジメチルスルホキシドとエタノールの1:1混合溶媒中に最終濃度が0.3mg/mlとなるように溶解させてEMCS溶液を用意した。なお、N-マレイミドカプロイロキシスクシイミド(N-(6-Maleimidocaproyloxy)succinimido)を以下EMCSと略す。ベークの終了したガラス基板を放冷し、調製したEMCS溶液中に室温で2時間浸した。この処理により、シランカップリング剤によって表面に導入されたアミノ基とEMCSのスクシイミド基が反応し、ガラス基板表面にマレイミド基が導入された。EMCS溶液から引き上げたガラス基板を、先述のMCSを溶解した混合溶媒を用いて洗浄し、さらにエタノールにより洗浄した後、窒素ガス雰囲気下で乾燥させた。
【0085】
[3]プローブDNA
先に作製した微生物検出用プローブを純水に溶解し、それぞれ、最終濃度(インク溶解時)10μMとなるように分注した後、凍結乾燥を行い、水分を除いた。
【0086】
[4]BJプリンターによるDNA吐出、および基板への結合
グリセリン7.5wt%、チオジグリコール7.5wt%、尿素7.5wt%、アセチレノールEH(川研ファインケミカル社製)1.0wt%を含む水溶液を用意した。続いて、先に用意した7種類のプローブ(表1)を上記の混合溶媒に規定濃度なるように溶解した。得られたDNA溶液をバブルジェットプリンター(商品名:BJF-850 キヤノン社製)用インクタンクに充填し、印字ヘッドに装着した。
【0087】
なおここで用いたバブルジェットプリンターは平板への印刷が可能なように改造を施したものである。またこのバブルジェットプリンターは、所定のファイル作成方法に従って印字パターンを入力することにより、約5ピコリットルのDNA溶液を約120マイクロメートルピッチでスポッティングすることが可能となっている。続いて、この改造バブルジェットプリンターを用いて、1枚のガラス基板に対して、印字操作を行い、アレイを作製した。印字が確実に行われていることを確認した後、30分間加湿チャンバー内に静置し、ガラス基板表面のマレイミド基と核酸プローブ末端のチオール基とを反応させた。
【0088】
[5]洗浄
30分間の反応後、100mMのNaClを含む10mMのリン酸緩衝液(pH7.0)により表面に残ったDNA溶液を洗い流し、ガラス基板表面に一本鎖DNAが固定したDNAマイクロアレイを得た。
【0089】
<検体の増幅と標識化(PCR増幅&蛍光標識の取り込み)>
検体となる微生物DNAの増幅、および、標識化反応を以下に示す。
Premix PCR 試薬(TAKARA ExTaq):25μl
Template Genome DNA:2μl (100ng)
Forward Primer mix: 2μl (20pmol/tube each)
Reverse Primer mix: 2μl (20pmol/tube each)
Cy-3 dUTP (1mM): 2μl (2nmol/tube)
H20:17μl
(Total:50μl)
上記組成の反応液を以下のプロトコールに従って、市販のサーマルサイクラーで増幅反応を行った。
(ステップ1)95℃、10 min.
(ステップ2)92℃、45 sec.
(ステップ3)55℃、45 sec.
(ステップ4)72℃、45 sec.
(ステップ5)72℃、10 min.
(ステップ2〜4は35回繰り返した。)
反応終了後、精製用カラム(QIAGEN QIAquick PCR Purification Kit)を用いてPrimerを除去した後、増幅産物の定量を行い、標識化検体とした。
【0090】
<ハイブリダイゼーション>
<DNAマイクロアレイの作製>で作製したDNAマイクロアレイと<検体の増幅と標識化(PCR増幅&蛍光標識の取り込み)>で作製した標識化検体を用いて検出反応を行った。
【0091】
(DNAマイクロアレイのブロッキング)
BSA(牛血清アルブミンFraction V:Sigma社製)を1wt%となるように100mM NaCl / 10mM Phosphate Bufferに溶解した。この溶液に<DNAマイクロアレイの作製>で作製したDNAマイクロアレイを室温で2時間浸し、ブロッキングを行った。ブロッキング終了後、0.1wt%SDS(ドデシル硫酸ナトリウム)を含む2xSSC溶液(NaCl 300mM 、Sodium Citrate (trisodium citrate dihydrate, C6H5Na3・2H2O) 30mM、p.H. 7.0)で洗浄を行った。その後、純水でリンスしてからスピンドライ装置で水切りを行った。
【0092】
(ハイブリダイゼーション)
水切りしたDNAマイクロアレイをハイブリダイゼーション装置(Genomic Solutions Inc. Hybridization Station)にセットし、以下に示すハイブリダイゼーション溶液、条件でハイブリダイゼーション反応を行った。
【0093】
<ハイブリダイゼーション溶液>
6 x SSPE / 10% Form amide / Target (2nd PCR Products 全量)
(6xSSPE: NaCl 900mM、NaH2PO4・H2O 60mM、EDTA 6mM、p.H. 7.4)
<ハイブリダイゼーション条件>
65 ℃、3min→92℃、2min→45℃、3hr→Wash、2xSSC/0.1% SDS、25℃→Wash、2 x SSC、20℃→(Rinse with H2O: Manual)→Spin dry
<微生物の検出(蛍光測定)>
ハイブリダイゼーション反応終了後のDNAマイクロアレイをDNAマイクロアレイ用蛍光検出装置(Axon社製、GenePix 4000B)を用いで蛍光測定を行った。
【0094】
この結果得られた画像データとしての画像の例を図6に示す。なお、図6においてより蛍光強度の強いプローブは、より濃い色で示している。601はDNAマイクロアレイに黄色ブドウ球菌のゲノムを含むサンプルを反応させた画像で、602は大腸菌のゲノムを含むサンプルを反応させた画像の例である。図の左に書いているアルファベットは、プローブ配列のアルファベットである。AからJまでそれぞれ、以下の各菌に特異的に結合するように設計されたプローブである。
(A)黄色ブドウ球菌。
(B)表皮ブドウ球菌。
(C)大腸菌。
(D)肺炎桿菌。
(E)緑膿菌。
(F)セラチア菌。
(G)肺炎連鎖球菌。
(H)インフルエンザ菌。
(I)エンテロバクター・クロアカエ菌。
(J)エンテロコッカス・フェカリス菌。
【0095】
理想的には、601のAの行のプローブだけが蛍光強度が高くなり、かつ、602のCの行のプローブだけが蛍光強度が高くなる。この601の理想的な結果は、図5に示した実験結果の例と同じである。
【0096】
しかし、図6に示すように、実際は理想通りにはならない。つまり、いわゆる"クロスハイブリダイゼーション反応"がおこり、601の場合は、A以外の行のプローブも蛍光強度が強く、また、602の場合は、C以外の行のプローブも蛍光強度が強い。更に、602の場合、Cの行でも蛍光強度の弱いプローブもある。
【0097】
この状況を3つのプローブの系で説明したのが図7である。黄色ブドウ球菌(S. aureus)、表皮ブドウ球菌(S. epiderimidis)、大腸菌(E. coli)の3種類のプローブがあるDNAマイクロアレイを用いてそれぞれの菌について6種類の既知サンプルの実験をしている。一般にプローブがN個ある場合、実験データはN次元のベクトルとなる。図6の場合、プローブが合計72個あるので、72次元のベクトル、図7の場合、プローブが3つあるので、3次元のベクトルが実験データとなる。
【0098】
図7下の図で、3菌それぞれ6種類のサンプル(=合計18個のデータ)を3次元座標にプロットしてある。図に示した通り、3つのプローブが理想的にそれぞれ3つの菌に非常に特異的なプローブである場合、図7下のようにベクトルデータは、それぞれの軸のまわりに集中する。但し、データの揺らぎは存在し、1つの点に集中するわけではない。図7の例でいうと、3菌それぞれのデータ存在範囲の大きさは異なっており、大きい順で大腸菌(E. coli)、表皮ブドウ球菌(S. epiderimidis)、黄色ブドウ球菌(S. aureus)となっている。
【0099】
さらに、各菌ごとに先に示した図1及び図8の方法に従って判定指数集合を導き、判定不能閾値を設定し、設定された判定不能閾値を用いて未知サンプルの供給元である菌の判定を行なうか、あるいは判定しない点を決定することができる。
【0100】
実施例2
以下に肺炎桿菌とセラチア菌のDNAマイクロアレイの実験データを示す。なお、プローブとしては以下の各菌の検出用のものを用いている。
黄色ブドウ球菌(S.aureus)(A−n)。
表皮ブドウ球菌(S.epidermidis)(B−n)。
大腸菌(E.coli)(C−n)、肺炎桿菌(K.pneumoniae)(D−n)。
緑膿菌(P.aeruginosa)(E−n)。
セラチア菌(S.marcescenes)(F−n)。
肺炎連鎖球菌(S.neumoiae)(G−n)。
インフルエンザ菌(H.influenzae)(H−n)。
エンテロバクター・クロアカエ菌(Enterobacter cloacae)(I−n)。
及びエンテロコッカス・フェカリス菌(E.faecelis)(J−n)。
【0101】
なお、上記のカッコ内のnは先に示したn=1〜6である。結局、全プローブ数は10×6=60個となる。
【0102】
まず、肺炎桿菌の10個の異なるサンプルに対するDNAマイクロアレイの実験データを図11〜20に示す。各図において左から右にプローブA−1、A−2、・・・〜J−5、J−6の順で配列されている。図示した通り、DNAマイクロアレイの実験データは、60個の蛍光輝度の値、つまり60次元のベクトルとして得られる。まず、任意のベクトルの間の距離を定義するために、「ベクトルのノルムでベクトルの各要素を割る」正規化を行う。式で記述すると、
【0103】
【数1】

【0104】
式においてベクトルxが元のベクトルで、ベクトルyが正規化後のベクトル、となる。
このように正規化したベクトルはそのノルムが常に1となっている。なお、ここでn次元のベクトルxのノルム(ユークリッドノルム)とは次の式で定義される。
【0105】
【数2】

【0106】
そして、正規化後の2つのベクトル(ベクトルaとベクトルb)間の距離を次の式で定義する。
【0107】
【数3】

【0108】
本実施例では、k-th nearest neighborマッチングアルゴリズムの距離定義を上記のようにする。10個のサンプルの間の任意の1組ずつの距離を計算し、ヒストグラムにしたものを図21に示す。データの数は10C2=45個になる。この図から、肺炎桿菌に上記k-th nearest neighborのアルゴリズムを適用して判定するとすると、その判定不能閾値は最大値である0.057というのが一つの候補になる。少し余裕を持たせて、1.5倍とか2倍の値を使っても良い。
【0109】
次に、セラチア菌の同じく10個のサンプルの実験データを図22〜31に示す。肺炎桿菌で行った正規化、距離計算を用いて、10個のサンプルの任意の2サンプルの距離を計算し、ヒストグラムを取ったのが、図32である。肺炎桿菌のヒストグラムと分布の形状が全く異なるのがわかる。大きく山が2つ存在するということは、10個のベクトルの中で2つのクラスターが存在することが想定される。実際、先に示した10サンプルの蛍光輝度グラフを見ても、大きく分けて2種類のパターンが存在することがわかる。この図から、肺炎桿菌に上記k-th nearest neighborのアルゴリズムを適用して判定する、とすると、その棄却値は1つ目の山の最大値である0.090というのが一つの候補になる。
【図面の簡単な説明】
【0110】
【図1】本発明の生物種類判定方法の一例を示す図である。
【図2】本発明の生物種類判定方法を実行するための情報処理装置の構成を示すブロック図である。
【図3】ハイブリダイゼーション反応を説明する図である。
【図4】DNAマイクロアレイを用いた実験手順を示すである。
【図5】感染症の判定用DNAマイクロアレイの実験手順を示すである。
【図6】ハイブリダイゼーション反応後の蛍光強度からなる画像の一例を示すである。
【図7】ベクトルデータの分布例を示すである。
【図8】判定不能値設定ステップを説明する図である。
【図9】判定指数集合の分布例を示すである。
【図10】同一カテゴリー内の任意の2サンプルの距離集合例を示すである。
【図11】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図12】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図13】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図14】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図15】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図16】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図17】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図18】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図19】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図20】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図21】10個の肺炎桿菌サンプル間の任意の1組ずつの距離に関するヒストグラムである。
【図22】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図23】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図24】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図25】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図26】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図27】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図28】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図29】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図30】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図31】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図32】10個のセラチア菌サンプル間の任意の1組ずつの距離に関するヒストグラムである。
【技術分野】
【0001】
本発明はパターン認識を用いた生物種判定方法に関するものであり、特に、分析する方法としてDNAマイクロアレイを用いた核酸配列解析システムに好適に適用でき、微生物の種類を判定する用途に用いるとその効果を発揮する。
【背景技術】
【0002】
従来、ガラスなどからなる基板上に位置を定めて固定された、プローブと呼ばれる核酸断片を配備したDNAマイクロアレイは未知の核酸サンプルの分析用として広く利用されてきた。このDNAマイクロアレイを用いることで、未知の核酸断片サンプル(未知サンプルと称す)を解析し、未知サンプルがどの生物種に由来するかを判定する方法にも利用されてきた。この方法ではハイブリダイゼーション反応という、核酸の塩基対形成反応が利用される。
【0003】
ハイブリダイゼーション反応とは何かを以下に説明する。生体内でほとんどの場合、DNAは2重らせん構造をしていて、その2本鎖の間の結合は塩基間の水素結合で実現されている。一方、RNAは1本で存在する場合が多い。塩基の種類はDNAの場合はATGCの4種類、RNAの場合はAUGCの4種類であり、それぞれ水素結合ができる塩基対はA-T(U)、G-Cのペアとなっている。ハイブリダイゼーション反応とは、1本鎖状態の2つの核酸分子同士が適切な条件下で反応して、核酸中にある塩基配列を介して1つに結合するこという。
【0004】
このことを踏まえ、従来の生物種を判定する方法を以下に説明する。基板上に固定されたプローブと、そのプローブと塩基対を形成することのできる相補的な塩基配列をもつ核酸断片とは、適切な反応条件のもとでハイブリダイゼーション反応を生じ、プローブと核酸断片が結合することができる。基板上に固定されたプローブが特定の生物に対応した塩基配列であって、かつハイブリダイゼーション反応が生じてプローブと核酸断片とが結合したことを認識できれば、核酸断片に対応する生物種はプローブに対応する生物種と同一であると判定可能である。すなわち未知サンプルに対応する生物種を判定することができる。
【0005】
例えば、核酸断片に蛍光物質を付与することによってハイブリダイゼーション反応が生じたかどうかを光学的に認識することができる。基板上に固定されたプローブから蛍光が生じた場合、ハイブリダイゼーション反応が生じ、プローブと核酸断片との結合体が形成されたと認識できる。この結果に基づいて、核酸断片はプローブに対応する生物種と同一であると判定される。一方、プローブから蛍光が生じなかった場合、ハイブリダイゼーション反応が生じず、プローブと核酸断片との結合体が形成されなかったと認識され、核酸断片はプローブに対応する生物種ではないと判定される。この判定方法を利用すると、ひとつの未知サンプルが与えられた場合、どの生物種に対応するかを一度のハイブリダイゼーション反応で複数種類の生物種に関して判定することができる。すなわち、対応する生物種が既知のプローブを複数準備し、基板上に位置を定めて固定する。そのようにして作成したDNAマイクロアレイに、未知サンプルを適切な反応条件のもとでハイブリダイゼーション反応をさせる。そして、基板上の位置から生物種を特定し、蛍光の有無でその生物種に対応するか否かを、判定する事ができる。つまり、基板上のどの位置のプローブから蛍光が生じるかを確認することによって、未知サンプルの生物種を判定することができる。
【0006】
しかし実際には、未知サンプルとプローブのハイブリダイゼーション反応の結果、ただ一つの生物種に対応するプローブが蛍光を生じるわけではない。多くの場合、たとえ未知サンプルには一種類の生物種しか対応しないことが事前にわかっていても、ハイブリダイゼーション反応させると、生物種を特定するためのプローブとは別に、他のプローブから蛍光が生じる場合がある。これは核酸分子がその中にある塩基配列を介して部分的に他のプローブと結合する場合があるからであり、クロスハイブリダイゼーションと呼ばれる。このクロスハイブリダイゼーションが発生するために、上記のように基板上の位置と蛍光の有無、の二つの情報のみでは未知サンプルに対応する生物種を判定することができない場合が多い。たとえば未知サンプルを、複数種類の生物種に対応するプローブを備えたDNAマイクロアレイとハイブリダイゼーションさせたとき、生物Aと生物Bに対応するプローブから蛍光を生じたとしても、以下の結果を反映している可能性の検討が必要である。
【0007】
すなわち、クロスハイブリダイゼーションの可能性を考慮すると、生物Aのみ未知サンプルに含まれている場合、生物Bのみ含まれる場合、生物Aと生物Bともに含まれる場合、などが考えられ、一意に未知サンプルに含まれる生物種を決定することができない。
【0008】
一般的な傾向として、同じプローブに結合した核酸断片から発生する蛍光強度は、クロスハイブリダイゼーションして部分的にプローブに結合した場合に生じる蛍光強度よりハイブリダイゼーションしてほぼ完全に結合した場合に生じる蛍光強度の方が強い。よって、DNAマイクロアレイを利用して、未知サンプルを解析し、未知サンプルがどの生物種であるかを判定する場合には、プローブの位置情報と蛍光強度に代表されるシグナル強度の情報と、から総合的に生物種を判定する方法を選択するべきである。
【0009】
なお、DNAマイクロアレイと未知サンプルとのハイブリダイゼーション反応後の蛍光強度はプローブ位置によって順序つけられたベクトルデータとして記憶手段に格納して利用することができる。
【0010】
特表2002-533699号公報には、DNAマイクロアレイを利用して未知のサンプルから得られたベクトルデータを解析し、この未知のサンプルから得られたベクトルデータと最も似ている既知のベクトルデータを検索する方法が開示されている。この最も似ている既知のベクトルデータを検索するという情報処理は、パターン認識として知られ、非常に一般的である。パターン認識とは、観測されたパターンを予め定められた複数の「カテゴリー」のうちの一つに対応させる処理である。「カテゴリー」を説明する例として、OCR(Optical Character Recognition)と呼ばれる技術分野では、紙に印刷、または手書きされた文字を一つのパターンとしてパターン認識する。このとき、認識対象を数字としたとすると、「紙に書かれている文字が0から9の数字のどれに一番近いか?」を既知のベクトルデータと照らし合わせて求める。パターン認識の問題において、この認識すべき0から9の10種類の数字が「カテゴリー」である。
【0011】
通常、パターン認識の問題においては、認識すべきカテゴリーの数と種類が事前に定められている。例えば上記の例では、数字といえば0〜9であり、日本語なら3000字程度の漢字、英語のアルファベットなら26文字というようにカテゴリーの数と種類はあらかじめ定められている。
【特許文献1】特表2002-533699号公報
【発明の開示】
【発明が解決しようとする課題】
【0012】
しかしながら、対応する生物種が未知の核酸断片サンプルをつかってDNAマイクロアレイとハイブリダイゼーションさせた結果得られたベクトルデータを使って、パターン認識する場合、カテゴリーが事前に想定されるとは限らない場合が多い。例えば、未知サンプル中にある細菌が存在するかどうかを、DNAマイクロアレイを用いて判定する場合では、あらかじめプローブとして配備する核酸断片に対応する細菌の種類を決める。しかしながら実際に未知サンプル中に存在する生物が、プローブに対応した生物種の中で収まる可能性は低い。なぜなら、先に説明したOCRと呼ばれる技術分野における0〜9までの9種類のカテゴリー、アルファベットAからZの26種類のカテゴリー、または漢字ならば約3000種類のカテゴリーに比べて、生物種全体の種類は圧倒的多数に上る。それゆえ、たとえ判定したい生物種を細菌に限ったとしても、想定すべきカテゴリーが膨大な数となり、あらかじめ全ての種類の細菌に関するカテゴリーを定めることは事実上不可能であるからである。したがって、未知サンプル中に存在すると想定される生物の種類をある程度限定してカテゴリーを定めることが必要である。
【0013】
そのため、OCRなどの文字認識で利用される従来の方法をそのまま生物種の判定に適用するには問題があった。具体的には、未知サンプル内にあらかじめ定められていないカテゴリーの生物が含まれていた場合、この生物を定められたカテゴリーに無理に対応させてしまうといった誤った判定をしてしまうという問題があった。
【0014】
本発明の目的は、パターン認識を利用した生物種の判定において、あらかじめ定められたどのカテゴリーにも対応しない生物が未知サンプルに含まれている場合に、生物種の生物学的特長に応じて、誤った判定する可能性を低減すること目的とする。
【課題を解決するための手段】
【0015】
本発明の生物種判定方法は、生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物の種類を判定する生物種判定方法において、
対応する生物種が判明している複数の既知サンプルを生物種分析方法により分析して、複数の分析データを得る工程と、
既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する工程と、
対応する生物種が未知である未知サンプルを、前記生物種分析法により分析して、該未知サンプルに対応する生物種の特定のための分析データを得る工程と、
前記判定不能閾値に基づいて前記未知サンプルに対応する種類を判定するか、あるいは判定不能であるかを決定する工程と、
判定をすると決定されたならば、前記複数の分析データに基づいて前記未知サンプルの生物種を判定する工程と、
を有する生物種判定方法である。
【0016】
また、本発明の生物種判定方法の他の態様は、生物に由来する物質が含まれていることが想定される物質を生物種分析方法にて分析して、対応する生物の種類を判定する生物種判定方法において、
(1)前記未知サンプルに対する判定結果として想定される生物種を選択する工程と、
(2)前記選択された生物類に属することが判明している複数の個体から得られる既知サンプルの各々から、該生物種に特徴的であって、パターン認識用として使用し得る複数の画像データからなる画像データ群を得る工程と、
(3)前記画像データ群から画像データを選択し、残りの画像データとの関係を用いて判定不能閾値を設定する工程と、
(4)未知サンプルからの画像データを得る工程と、
(5)前記未知サンプルからの画像データを前記判定不能閾値に基づいて、該未知サンプルに対応する生物種を判定するか判定が不能であるかを決定する工程と、
(6)前記(5)で判定を行うことが決定された場合は、前記画像データ群からなる識別辞書を用いて、生物種を判定する工程と、
を有することを特徴とする生物種類判定方法である。
【0017】
本発明の生物種判定のための情報処理装置は、対応する生物種が判明している複数の既知サンプルを生物分析方法により分析して得られた複数の分析データ、及び、該複数の分析データに基づいて設定された判定不能閾値を記憶したメモリと、該メモリに記憶された判定不能閾値に基づいて、未知サンプルに対応する生物種が判定可能か否かを決定し、判定可能と決定した場合には、前記メモリに記憶された複数の分析データに基づいて前記未知サンプルに対応する生物種の判定を行う処理ユニットとから成る生物種判定のための情報処理装置である。
【0018】
本発明の生物種判定のための情報処理装置の他の態様は、生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物種を判定するための情報処理装置において、対応する生物種が判明している複数の既知サンプルを分析して得られる該生物種に特徴的な画像データを入力するための既知サンプル画像データ入力手段と、
未知サンプルを前記既知サンプルと同様に分析して得られる画像データを入力する未知サンプル画像データ入力手段と、
取り込まれた前記画像データを記憶する記憶手段と、
既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する手段と、
未知サンプルからの画像データを前記判定不能閾値にもとづいて判定を行うかまたは判定を行わないかを決定し、判定を行うのであれば、未知サンプルに対応する生物種を判定する生物種判定手段と、
前記判定手段での判定結果を記憶する記憶手段と、
前記記憶手段に記憶された判定結果を出力する出力手段と
を有することを特徴とする生物種判定のための情報処理装置である。
【0019】
本発明の生物種判定のためのプログラムは、未知サンプルに対応する生物種の判定をコンピュータに実行させるためのプログラムであって、
(1)未知サンプルに対する判定結果として想定される生物種に属する複数の異なる個体からの既知サンプルを分析して得られる該想定される生物種に特徴的な画像データに対応する複数の画像データを格納した記憶手段から、これらの複数の既知サンプル画像データを呼び出すステップと、
(2)未知サンプルを前記既知サンプルと同様にして分析して得られる画像データに対応する複数の画像データを格納した記憶手段から、該未知サンプル画像データを読み出すステップと、
(3)前記既知サンプル画像データから1つを選択し、選択された1つと残りの画像データとの関係を用いて判定不能閾値を設定するステップと、
(4)前記判定不能閾値に基づいて前記未知サンプル画像データを処理し、未知サンプルに対応する生物の種類を判定するステップと、
(5)前記判定ステップで得られた判定結果を記憶手段に格納させるステップと、
(6)前記記憶手段に格納された判定結果を出力するステップと
を有することを特徴とする生物種類判定用プログラムである。
【0020】
本発明の生物種判定のための記録媒体は、生物種判定をコンピュータで実行するためのプログラムを読み取り可能に記録した記録媒体であって、該プログラムが上記構成の生物種判定のためのプログラムであることを特徴とする記録媒体である。
【0021】
本発明の生物種判定方法の他の態様は、対応する生物種が判明している複数の既知サンプルを生物分析方法により分析して得られた複数の分析データと、該複数の分析データに基づいて設定された判定不能閾値を用い、前記判定不能閾値に基づいて、未知サンプルに対応する生物種が判定可能か否かを決定した後、判定可能と決定した場合には、前記複数の分析データに基づいて前記未知サンプルに対応する生物種の判定を行う生物種判定方法である。
【発明の効果】
【0022】
本発明によれば、あらかじめ定められたどのカテゴリーにも対応しない生物が未知サンプルに含まれている場合に、判定不能と判断することができるので、適切な生物種判定結果が得られるという効果がある。また、生物種に対応するカテゴリーごとに判定不能の判断を行うためのパラメータを設定することができるので、生物種の生物学的特長に応じた最適な生物種判定結果が得られるという効果がある。
【発明を実施するための最良の形態】
【0023】
本発明にかかる生物種の判定方法は、ベクトルデータを解析して、識別辞書の作成及び判定不能閾値を決定する方法が含まれる。本発明にかかる生物種の判定方法では、最初に生物種が判明している生物から抽出された核酸断片サンプル(既知サンプルと称す)を分析して得られたベクトルデータを外部記憶手段に格納する。この既知サンプルを分析して得られたベクトルデータを辞書のように参照して未知サンプルの生物種を判定するので、外部記憶手段に格納された既知サンプルを分析して得られた生物種判定用のベクトルデータの総体を、識別辞書と称することにする。
【0024】
次に識別辞書として格納されたベクトルデータを用いて、判定不能閾値を設定する。この判定不能閾値の設定方法に関しては後ほど詳述する。設定された判定不能閾値に従って、対応する生物種を判定したい未知サンプルが作成した識別辞書で生物種を判定できるのか、または判定できない(判定不能)のかが判断される。
【0025】
以下、既知サンプルおよび未知サンプルを分析した結果が画像データとして得られる場合について本発明の判定方法を説明する。
【0026】
識別辞書を作成するための既知サンプルの選定に当たっては、まず判定対象としての生物が属すると想定される生物種類が選択される。例えば未知サンプル中に細菌が存在している可能性があって、細菌に関しての生物種判定を行いたい場合、細菌のなかから既知の生物種をあらかじめ選択する。この選択された生物種が、パターン認識を利用した生物種判定方法におけるカテゴリーに相当する。数字やアルファベットに比べ生物種全体の種類は圧倒的多数に上るので、ある程度限定してカテゴリーを定めることが必要であることは既に述べた。
【0027】
次に、選択された各生物種の固体を用意し、各生物種の固体から抽出された核酸断片サンプルを得る。これを既知サンプルとして、既知サンプル及び未知サンプルから画像データを得るための分析方法を選択する。この分析方法は、パターン認識により生物種の判定が可能となる方法から選択される。例えば、DNAマイクロアレイなどを用いて、得られた画像データをベクトルデータとして認識する分析方法が好適に利用できる。
【0028】
DNAマイクロアレイを利用して画像データをどのようにして得るかを説明する。プローブは、生物種ごとに用意されて、前述のように基板上の所定の位置(すなわち、どの生物種に対応するプローブがどの位置に配備されているかがあらかじめ定められた位置)に固定されている。核酸断片に例えば蛍光物質を付与することによって、DNAマイクロアレイと核酸断片とを適切な条件下で反応させたときに、ハイブリダイゼーション反応が生じたかどうかを光学的に認識することができる。
【0029】
本発明では、1つのカテゴリーに対して、1つの既知サンプルから得た画像データから識別辞書を作成するのではなく、各カテゴリーにおいてそれぞれ2つ以上の既知サンプル(同種の生物の異なる2つ以上の個体)から得られる画像データに基づいて判定不能閾値を設定して、生物種類の判定を行なう。
【0030】
なお、判定したい生物が微生物であれば、生物種類として微生物の「種(species)」を選択することができ、その他様々な生物に本発明が適用できることは言うまでもない。
【0031】
以下、図面に基づいて本発明の一例について説明する。
【0032】
図1は、本発明の生物種類判定方法の一例における処理手順を説明するフローチャートである。この生物種類判定方法は、ある未知サンプル中にターゲットとしての生物種を特定できるこの生物種に由来する物質が存在するか、存在するとすればそれが由来する生物は何の種類に属するかを判定する方法である。生物種類判定方法における棄却とは、ターゲットとして選択した生物種に由来する物質が未知サンプルに存在しないとの判定をすることである。なお、以下においては、微生物などのゲノム解析を用いる生物種類判定を主体として本発明を説明する。しかしながら、例えば、抗原抗体反応を用いた検査システムなどに対しても本発明の技術を適用できる。また、MHCなどの個体識別ゲノム領域などを分析するシステムにも適用してもよい。
【0033】
本発明における未知サンプルの生物種判定処理の流れは、大きくみて、既知サンプルを用いて識別辞書を作成する学習フェーズと未知サンプルを判定する判定フェーズに分かれる。図1において、101から104が学習フェーズで、105から108が判定フェーズである。
【0034】
以下に学習フェーズを説明する。ステップ101では、対応する生物の種類が既知の生物から抽出された核酸断片を含む既知サンプルを用意する。例えば、菌種が特定されている菌のゲノムなどを含む溶液などが既知サンプルに相当する。この既知サンプルを用いて一連のハイブリダイゼーション反応実験102を行ってデータを得る。詳細は後述するが、例えばDNAマイクロアレイを用いた場合、まずPCR反応により既知サンプルに含まれる核酸断片を増幅し、蛍光物質を付与する。その後、DNAマイクロアレイとハイブリダイゼーション反応をして、それぞれのスポットの蛍光強度のデータを画像として認識して外部記憶手段に格納する。この画像データをもとに、判定不能閾値設定ステップ103と、辞書作成ステップ104で、判定不能閾値と識別辞書がそれぞれ作成される。
【0035】
次に判定フェーズを説明する。未知のサンプルを用意し(ステップ105)、ステップ102と全く同じ手順でハイブリダイゼーション反応実験106を実施する。ハイブリダイゼーション反応により得られた画像データと、学習フェーズで得られた判定不能閾値、識別辞書とを照らし合わせることによって、未知サンプルに対して生物種の判定を行う(ステップ107)。その結果判定結果108として、「未知サンプルは生物種Aに対応する」、「未知サンプルには生物種A〜Cに由来する物質が含まれる」、「未知サンプルには生物種A〜Z以外で生物群αに含まれる生物に由来する物質が存在する」、「105の未知サンプルは判定できない(=判定不能)」というような結果が得られる。
【0036】
以上に説明した学習フェーズ中の、特に判定不能閾値の設定方法について異なる2通りの方法を以下に詳しく説明する。
【0037】
まず同種であるが異なる生物個体から得られた既知サンプルを用意し、DNAマイクロアレイとハイブリダイゼーション反応させ画像データを得る。判定不能閾値の設定には、次の方法のいずれかを好ましく用いることができる。
・方法(1)3以上の既知サンプルの画像データから1つを選択して除外し、残りの既知サンプルの画像データから識別辞書を作成し、それを利用して判定不能閾値を設定する方法。
・方法(2)3以上の既知サンプルの画像データから選択した任意の2つの組み合わせの全てについてパターン認識アルゴリズムにより求めた距離を利用して判定不能閾値を設定する方法。
【0038】
まず、上記の方法(1)の判定不能閾値を設定する方法について説明する。この方法の処理手順のフローチャートを図8に示す。まず、未知サンプルに対する生物種判定結果として想定されるn種の異なる生物種(S1〜Sn:n≧2)、すなわちターゲットカテゴリーを選択する(ステップ802)。次に、選択されたターゲットカテゴリーごとに固有の判定不能閾値を得るための処理を行う。次にステップ802でターゲットカテゴリーとして選択されたカテゴリーの既知サンプルを用意して、ハイブリダイゼーションさせた結果、画像データを得ることができる。画像データは、識別辞書を作成するために外部記憶手段に格納される。この画像データの総体を学習データと呼ぶことにする。以下、ターゲットカテゴリーS1に属する生物種を例にあげて、(1)の判定不能閾値を設定する方法を説明する。
【0039】
まず、ターゲットカテゴリーS1に属するm個の個体S1−X(1≦X≦m、m≧3)を用意する。用意した各個体から拡散断片を抽出し、m個(m≧3)の既知サンプルを得る。このm個の既知サンプルとDNAマイクロアレイとを適当な条件下でハイブリダイゼーションさせ、m個(m≧3)(Ps1−1〜Ps1−m)の画像データ群を得る。次に、学習データ分割ステップ803において、これらの画像データから1つを選択して学習データから除去する。次に、1つの画像データを除いた残りのm−1個の学習データ804を用いて、辞書作成ステップ805において識別辞書806を作成する。この辞書作成ステップ805は、採用したパターン認識アルゴリズムに則って作成される。
【0040】
パターン認識による未知パターンの判定には、公知の方法から選択した方法を利用することができる。パターン認識による判定や分類のための方法は、例えば、IEEE Transaction on Pattern Analysis and Machine Learning, Vol. 22, No. 1, January 2000, pp.4-pp.37にある"Statistical Pattern Recognition: A Review"Anil K. Jain, Robert P.W. Duin, and Jianchan Mao. の論文にレビューされている。具体的には、例えばk-Nearest-Neighbor法、分類木、Support Vector Machine、ベイズ識別法、ブースティング法、ニューラルネットなどのパターン認識の技術が利用できる。
【0041】
例えばニューラルネットをパターン認識アルゴリズムとして採用したとすると、ネットワークの重みパラメータ集合が識別辞書として学習される。例えばSupport Vector Machineがパターン認識アルゴリズムとして採用されたとすると、いわゆるSupport Vectorと呼ばれる代表するサンプルベクトルとその重み付けが識別辞書として学習される。本発明において、識別辞書として学習される、もしくは学習する、とは、学習データに基づいて識別辞書を作成することと同義である。
【0042】
次に、学習データから除いた一つの画像データを、識別辞書806を用いて判定する(ステップ808)。ここでは、例えば個体S1−1に対応する画像データPs1−1を学習データから除いたとしよう。この時、注意すべきなのは、識別辞書806はステップ807で除いた1つの画像データを含まない。よって、識別辞書806に対して、ステップ807で除かれた個体S1−1は未知サンプルとなる。識別辞書を用いて判定を行う場合、外部記憶手段に格納されたベクトルデータ同士を比較するために、ユークリッドノルムに代表されるノルムをあらかじめ定義する必要がある。本発明の生物種判定方法にユークリッドノルムを採用した場合については後述されるが、もちろん一般の種々のノルムを採用してもかまわない。以上の工程を経て、判定指数809が得られる(ステップ809)。
【0043】
一般にパターン認識アルゴリズムの判定結果は、数値データとなる。例えば判定確率であったり、類似度であったり、単にベクトルデータ同士の距離であったりする。このように、判定指数809はあらかじめ定義されたノルムを用いて算出された判定結果である数値データを意味する。
【0044】
こうして、m個の画像データのうち、学習データから一つの画像データを除いた学習データをもちいて作成した識別辞書を用いて、ターゲットカテゴリーS1についての一つの判定指数A1−1が得られる。
【0045】
次に、上記の学習データ分割ステップ803で除かれなかったターゲットカテゴリーS1の画像データを新たにS1−X(1≦X≦m、m≧3)の中から選択して同様に除く。ここではS1−2に対応する画像データを選択したとしよう。同様の処理を行ない、ターゲットカテゴリーS1に関する判定指数A1−2を得る。すなわち上記の操作を同様にターゲットカテゴリーS1の各画像データに実行することによりm個の判定指数からなる判定指数集合{A1}を得る。判定指数集合{A1}は、m個の判定指数の元(げん)、A1−1、A−2、・・・A1−m、からなる。
【0046】
こうして得られた判定指数集合{A1}からターゲットカテゴリーS1に関する判定不能閾値を設定することができる。上記ではターゲットカテゴリーS1に関して例にあげて判定指数集合を得る方法を説明した。同様にして最初に選んだn種のターゲットカテゴリーのうち、ターゲットカテゴリーS1以外の他のターゲットカテゴリーに関してもそれぞれ判定指数集合を得る。その結果、n個の判定指数集合を得ることができる。
【0047】
図9に、n個の判定指数集合のうちから一つの判定指数集合を選び、判定指数集合810の分布をヒストグラムで表示した例を示す。判定指数809が類似度を示している場合、判定不能閾値を、例えば、集合の最小値のα倍(α<1)に設定したり、集合の平均値や中央値のβ倍(β>0)にしたりする。逆に、809の判定指数が非類似度を示している場合、判定不能閾値を、例えば、集合の最大値のα倍(α>1)に設定したり、集合の平均値や中央値のβ倍(β>0)にしたりする。判定不能閾値を、判定指数集合に基づいてどのような値に設定するかは、検査対象としての生物の種類、パターン認識を用いる分析方法の種類、目的とする判定精度などに応じてターゲットカテゴリーごとに選択できる。このようにして定めた判定不能閾値の設定が適切かどうかを確認する方法として、選択したターゲットカテゴリーに含まれないことがあらかじめ判明しているサンプルを未知サンプル105として用いる方法がある。図1を用いて前述した未知サンプルの生物種判定処理を実行して、この未知サンプルについて「判定不能である」との結果がでるかどうかを試験して、判定不能閾値の設定が正しいかどうかを確認することができる。
【0048】
つぎに、方法(2)の判定不能閾値を設定する方法を以下に説明する。図10に判定不能閾値を設定する別の例を示す。パターン認識アルゴリズムとしてk-Nearest-Neighbor法(特にk=1)を選び、ノルムとしてユークリッドノルムを採用した場合の判定不能閾値設定方法を以下に説明する。この場合、未知サンプルを分析して得られた画像データから算出された判定指数が非類似度を示すとすると、定められた判定不能閾値より大きい時に「判定不能」という結果がでることになる。判定不能閾値を設定するために、まずひとつのターゲットカテゴリーS1を選び、S1に属する全ての既知サンプルをハイブリダイゼーションさせた結果、画像データを得て外部記憶手段に格納する。この格納された画像データの総体からS1に属する任意の2つの画像データの組み合わせを選択し、この2つの画像データをもとに認識されたプローブ位置によって順序つけられた蛍光強度からなるベクトルデータ同士のユークリッド距離を算出する。つづいて、上記で選ばれなかった2つの画像データの組み合わせを新しく選出し、同様にして、新しく選出された2つの画像データに基づいてユークリッド距離を算出する。このような手順で、S1に属し外部記憶手段に格納された画像データ群に関してそれぞれの組み合わせに基づいてユークリッド距離が算出される。ターゲットカテゴリーに属する既知サンプルが6つ用意された場合を図7に示す。この場合、2つの画像データをもとに算出されるユークリッド距離の個数は6C2=15となる。
【0049】
ターゲットカテゴリーS1に属する全ての画像データの組み合わせに基づいて算出されたユークリッド距離を、判定指数をx軸としてヒストグラムを用いて示したものが図10である。図10は距離の分布に2つの山が存在する。カテゴリーに属するサンプルベクトルが2つの領域に局在することを意味する。このように、ヒストグラムからターゲットカテゴリーS1の性質を確認できるので、各ターゲットカテゴリーにごとに適切な判定不能閾値の設定方法を選ぶことができる。
例えば、この距離集合の平均値や中央値などの統計的代表値をもって判定不能閾値とすることができる。
【0050】
次に、得られた判定不能閾値を、図1を用いて前述した未知サンプルの生物種判定処理を実行して、このようにして定めた判定不能閾値の設定が適切かどうかを、方法(1)と同じ方法で確認する。選択したターゲットカテゴリーに含まれないことがあらかじめ判明しているサンプルを未知サンプル105として用いる。この未知サンプルについて「判定不能である」との結果がでるかどうかを試験して、判定不能閾値の設定が正しいかどうかを確認する。
【0051】
次に、上記の生物種類判定方法に用い得る情報処理装置としてのコンピュータシステム、プログラム、画像認識を用いた分析方法などの各処理について説明する。
【0052】
以上説明した生物種類判定は、予め作成されたプログラムに従ったコンピュータ上での処理により自動化可能である。本発明にかかる生物種類判定のための情報処理装置は、生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物種を判定するための情報処理装置である。この情報装置は以下の少なくとも手段を用いて構成することができる。
(1)対応する生物種が判明している複数の既知サンプルを分析して得られる該生物種に特徴的な画像データを入力するための既知サンプル画像データ入力手段。
(2)未知サンプルを前記既知サンプルと同様に分析して得られる画像データを入力する未知サンプル画像データ入力手段。
(3)取り込まれた前記画像データを記憶する記憶手段。
(4)既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する手段。
(5)未知サンプルからの画像データを前記判定不能閾値にもとづいて処理し、未知サンプルの提供元である生物の種類を判定する生物種類判定手段。
(6)前記判定手段での判定結果を記憶する記憶手段。
(7)前記記憶手段に記憶された判定結果を出力する出力手段。
【0053】
上記の判定不能閾値の設定は、記憶手段に3以上の個体からの画像データを記憶手段に記憶させておき、以下のステップを有するプログラムに基づいて実行されることが好ましい。
(a)3以上の画像データから1つの画像データを選択して除外し、残りの複数の画像データを用いて識別辞書を作成し、得られた識別辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なって3以上の判定指数からなる判定指数集合を得るステップ。
(b)前記判定指数集合から判定不能閾値を設定するステップ。
【0054】
また、上記の判定不能閾値の設定は、次のようにして行われることが好ましい。すなわち、記憶手段に3以上の個体からの画像データを記憶手段に記憶させておく。そして、前記個体が3以上であり、前記記憶手段にこれらの個体からの画像データが記憶されており、前記判定不能閾値の設定が、少なくとも以下の工程を実行するプログラムに基づいて行われることも好ましい。
(A)前記3以上の画像データから選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程。
(B)前記距離集合から判定不能閾値を決定する工程。
【0055】
また、本発明にかかる生物種類判定のためのプログラムは、未知サンプルに対応する生物の種類の判定をコンピュータに実行させるためのプログラムであって、少なくとも以下のステップを実行するためのものである。
(1)未知サンプルに対する判定結果として想定される生物種に属する複数の異なる個体からの既知サンプルを分析して得られる該想定される生物種に特徴的な画像データに対応する複数の画像データを格納した記憶手段から、これらの複数の既知サンプル画像データを呼び出すステップ。
(2)未知サンプルを前記既知サンプルと同様にして分析して得られる画像データに対応する複数の画像データを格納した記憶手段から、該未知サンプル画像データを読み出すステップ。
(3)前記既知サンプル画像データから1つを選択し、選択された1つと残りの画像データとの関係を用いて判定不能閾値を設定するステップ。
(4)前記判定不能閾値に基づいて前記未知サンプル画像データを処理し、未知サンプルに対応する生物の種類を判定するステップ。
(5)前記判定ステップで得られた判定結果を記憶手段に格納させるステップ。
(6)前記記憶手段に格納された判定結果を出力するステップ。
【0056】
上記の判定不能閾値の設定ステップは、個体を3以上とし、記憶手段にこれらの個体からの画像データが記憶を記憶させておき、少なくとも以下のステップによって行うことが好ましい。
(a)前記3以上の画像データから1つの画像データを選択して除外し、残りの複数の画像データを用いて識別辞書を作成し、得られた識別辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なって3以上の判定指数からなる判定指数集合を得るステップ。
(b)前記判定指数集合から判定不能棄却閾値を決定するステップ。
【0057】
また、上記の判定不能閾値の設定ステップは、個体を3以上とし、記憶手段にこれらの個体からの画像データが記憶を記憶させておき、少なくとも以下のステップによって行うことが好ましい。
(A)前記3以上の画像データから選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程。
(B)前記距離集合から判定不能閾値を決定する工程。
【0058】
記憶手段に、多数の既知生物種類のそれぞれにおける判定不能閾値を格納しておき、未知サンプルの種類に応じて、未知試料に含まれる生物由来物質がその存在を示すことが想定される必要数のカテゴリーを選択するステップをプログラムに追加しておくとよい。このことにより、判定不能かどうかを検討するカテゴリー数を効果的に低減でき、より効率の良い判定処理が可能となる。
【0059】
なお、上記のプログラムは、コンピュータシステムの記憶手段中に保持させておいてもよいし、記録媒体に格納して使用者に配布できるようにしてもよい。更には、ネットワークシステムを介して配布できるようにしてもよい。
【0060】
図2に、生物種類判定方法を実行し得るコンピュータシステムを利用した情報処理装置の構成の一例のブロック図を示す。この装置は、外部記憶装置201、中央処理装置(CPU)202、メモリ203、入出力装置204を少なくとも有して構成される。外部記憶装置201には、生物種類判定を行なうための上述した構成のプログラムや、既知サンプル及び未知サンプルを対するハイブリダイゼーション反応を利用した分析の結果としての画像データが保持される。外部記憶装置201には、更に判定不能閾値を用いた決定の結果を保持させる。中央処理装置(CPU)202は、生物種類判定のためのプログラムを実行したり、すべての装置の制御を行なったりする。メモリ203は中央処理装置(CPU)202が使用するプログラム、及びサブルーチンやデータを一時的に記録する。入出力装置204は、ユーザーとのインタラクションを行う。多くの場合、プログラム実行のトリガーはこの入出力装置を介してユーザーが出す。また、ユーザーが結果を見たり、プログラムのパラメータ制御をこの入出力装置を介して行う。
【0061】
図3はDNAマイクロアレイ上のハイブリダイゼーションの様子を示した図である。生体内でほとんどの場合、DNAは2重らせん構造をしていて、その2本鎖の間の結合は塩基間の水素結合で実現されている。一方、RNAは1本で存在する場合が多い。塩基の種類はDNAの場合はATGCの4種類、RNAの場合はAUGCの4種類であり、それぞれ水素結合ができる塩基対はA-T(U)、G-Cのペアとなっている。一般にハイブリダイゼーション反応とは、1本鎖状態の核酸分子同士がその中にある部分塩基配列を介して部分的に結合する状態をいう。図3に示す例では、図中上側の基板にくっついた核酸分子(プローブ)の方が下側のサンプル中にある核酸分子より短い。サンプル中に存在する核酸分子がプローブの塩基配列を含む場合は、このハイブリダイゼーション反応はうまくいき、サンプル中の核酸分子はDNAマイクロアレイにトラップされることとなる。
【0062】
次に、図4を用いてDNAマイクロアレイを用いて画像データを得るための実験手順全般について説明する。401の「サンプル」とは対象としている生物由来物質、例えば核酸(細胞に含まれている状態のものも含む)が含まれている、あるいは含まれていることが想定される液体や個体である。例えば、感染症の原因菌の特定をするために本発明を適用した場合、ヒト、家畜等の動物由来の血液、喀痰、胃液、膣分泌物、口腔内粘液等の体液、尿及び糞便のような排出物、人、動物などから採取した組織片等の細菌などの微生物やそれに由来する物質が存在すると思われるあらゆる物が未知サンプル401の供給元となる。また、食中毒、汚染の対象となる食品、飲料水及び温泉水のような環境中の水等、細菌による汚染が引き起こされる可能性のある媒体が未知サンプルの供給元として用いられることもある。さらに、輸出入時における検疫等の動植物も検体としてその対象となる。既知サンプルの場合には、種類が既知である微生物などから調製されたサンプルである。
【0063】
次に、必要に応じて、402の"生化学的増幅"方法を用いて401のサンプルとしての核酸を増幅する。例えば感染症の原因菌の特定をするために本発明を適用した場合、16s rRNA検出用に設計されたPCR反応用プライマーを用いてPCR法によって対象核酸を増幅したり、或いはPCR増幅物を元にさらにPCR反応等を行なって調整したりする。また、PCR以外のLAMP法などの増幅方法により調整してもよい。
【0064】
その後で、増幅されたサンプル、または401のサンプルそのものに、可視化のために各種標識法により標識する。この標識物質としては、通常Cy3, Cy5, Rodaminなどの蛍光物質が用いられる。また、402の生化学的増幅の実験手順の中で、標識分子を混入することもある。
【0065】
そして、標識分子が付加された核酸を図1における、404のDNAマイクロアレイとハイブリダイゼーション反応(405)を行う。この様子は、図3に示した通りである。例えば、感染症の原因菌の特定をするために本発明を適用した場合、404のDNAマイクロアレイは、菌に特異的なプローブを基板に固定したものとなる。各菌のプローブの設計は、例えば16s rRNAをコーディングしているゲノム部分より、当該菌に対し非常に特異性が高く、十分かつそれぞれのプローブ塩基配列で"出来るだけ"ばらつきのないハイブリダイゼーション感度が期待できるように行う。404のDNAマイクロアレイのプローブを固定する担体(基板)は、ガラス基板、プラスチック基板、シリコンウェハー等の平面基板が考えられる。また、凹凸のある三次元構造体、ビーズのような球状のもの、棒状、紐状、糸状のもの等を用いても、本発明の実景形態、効果には影響ない。
【0066】
通常、基板の表面はプローブDNAの固定化が可能なように処理したものが使用される。特に、表面に化学反応が可能となるように官能基を導入した物は、ハイブリダイゼーション反応の過程でプローブが安定に結合している為に、再現性の点で好ましい形態である。プローブの固定化方法としては、例えば、マレイミド基とチオール(−SH)基との組合わせを用いて基板上にプローブを固定化する方法が挙げられる。即ち、核酸プローブの末端にチオール(−SH)基を結合させておき、固相表面がマレイミド基を有するように処理しておくことで、固相表面に供給された核酸プローブのチオール基と固相表面のマレイミド基とが反応して核酸プローブを固定化する。ガラス基板へのマレイミド基の導入は、まず、ガラス基板にアミノシランカップリング剤を反応させ、次にそのアミノ基とEMCS試薬(N-(6-Maleimidocaproyloxy)succinimide :Dojin社製)との反応により行うことができる。DNAへのSH基の導入は、DNA自動合成機上5'-Thiol-ModifierC6(Glen Research社製)を用いる事により行なうことができる。固定化に利用する官能基の組合わせとしては、上記したチオール基とマレイミド基の組合わせ以外にも、例えばエポキシ基(固相上)とアミノ基(核酸プローブ末端)の組合わせ等が挙げられる。また、各種シランカップリング剤による表面処理も有効であり、該シランカップリング剤により導入された官能基と反応可能な官能基を導入したオリゴヌクレオチドが用いられる。さらに、官能基を有する樹脂をコーティングする方法も利用可能である。
【0067】
ハイブリダイゼーション反応を行った後、404のDNAマイクロアレイの表面を洗浄し、プローブと結合していない核酸を剥がした後で、通常は乾燥し、405の蛍光量を測定する。そして、DNAマイクロアレイの基板に励起光を照射し、蛍光強度を測定した画像(406)が得られる。この画像(406)が画像データとなる。画像データの一例を図6に示した。異なる既知サンプルに対応して、図6の601と602とで異なる画像データ(画像)が得られている。
【0068】
次に、図5を用いて感染症の菌を特定する場合のDNAマイクロアレイの原理を示す。図5で示したDNAマイクロアレイは、例えば、黄色ブドウ球菌を特定する目的で作られている。左の列は、黄色ブドウ球菌野生株由来の処理系列であり、右の列は大腸菌野生株由来の処理系列である。例えば、左は黄色ブドウ球菌に感染した患者の血液を処理する流れで、右は大腸菌に感染した患者の血液を処理する流れだと考えてよい。
【0069】
どちらも基本的には同じ処理を行う。つまり、まず初めに例えば菌感染患者の血液や、痰などからDNAを抽出する。この際に、一般的には、患者の体細胞由来の人間のDNAも含まれる可能性がある。
【0070】
抽出されたDNAが少ない場合、PCR法などの方法で増幅を行う。この際に蛍光物質もしくは蛍光物質を結合させることができる物質を標識として混入させるのが一般的である。増幅をしない場合は、抽出されたDNAを用いて、相補鎖を作りながら蛍光物質もしくは蛍光物質を結合させることができる物質を標識として混入させる、または、そのまま直接抽出されたDNAに蛍光物質もしくは蛍光物質を結合させることができる物質を標識として付加させる。
【0071】
通常、PCR増幅を行う場合、感染症の菌特定目的であれば、いわゆる16s rRNAといわれるリボゾームRNAを構成する塩基配列の部分を増幅するのが一般的である。この場合、左の黄色ブドウ球菌のPCRプライマーと右の大腸菌のPCRプライマーはほとんど同じものを使うこととなる。より具体的には、どんな菌の16s rRNAをコーディングしている部分でも増幅させることができるプライマーセットを用いて、マルチプレックスPCRを行う。
【0072】
黄色ブドウ球菌を判定する目的のために設計されたDNAマイクロアレイが正しく動作するならば、左のハイブリ溶液では、スポットがポジティブに反応し、右のハイブリ溶液では、スポットがネガティブに反応する。これと全く同じように、大腸菌の存在を判定する目的のために設計されたDNAマイクロアレイが正しく動作するならば、次の反応が生じる。すなわち、左のハイブリ溶液(ハイブリダイゼーション用溶液)では、スポットがネガティブに反応し、右のハイブリ溶液(ハイブリダイゼーション用溶液)では、スポットがポジティブに反応する。
【0073】
ポジティブに反応したスポットからの蛍光強度を測定して図4で示すスキャン画像処理を行なうことで画像データを得ることができる。ここで、同じ種類に属する異なる個体からのサンプルを用いて同じ分析条件で画像データを得た場合に、常に同じ蛍光強度が得られれば、それを辞書として用いればよい。しかしながら、実際には、蛍光強度にバラツキが生じるので、未知サンプルからの画像データがこのバラツキの範囲内なのか、あるいはその範囲外で既知のカテゴリーに属さないと判定すべきかどうかの明確な基準を得ることが困難な場合がある。更に、後述の実施例で示すように、プローブによってはクロスハイブリダイゼーションを生じる。そこで本発明では、図8に示すように同一種類に属する多数の異なる個体からのサンプルを用いた識別辞書作成と判定不能閾値の設定より、カテゴリーごとに未知サンプルの判定を行なうかどうかの基準を明確としている。
【実施例】
【0074】
以下、本発明の生物種類判定方法に利用し得る分析データの取得方法の具体例を挙げる。なお、本発明は、以下に述べる感染症の原因菌特定に限ったものではなく、MHCなどの人間の体質判定や、癌などの疾病に関わるDNA、RNAの解析に用いてもよい。
【0075】
実施例1
<プローブDNAの準備>
Enterobacter cloacae菌検出用Probeとして以下に示す核酸配列(I−n)(nは数字)を設計した。具体的には、16s rRNAをコーディングしているゲノム部分より、以下に示したプローブ塩基配列を選んだ。これらのプローブ塩基配列群は、当該菌に対し非常に特異性が高く、十分かつそれぞれのプローブ塩基配列で"出来るだけ"ばらつきのないハイブリダイゼーション感度が期待できるように設計されている。
I-1:CAgAgAgCTTgCTCTCgggTgA
I-2:gggAggAAggTgTTgTggTTAATAAC
I-3:ggTgTTgTggTTAATAACCACAgCAA
I-4:gCggTCTgTCAAgTCggATgTg
I-5:ATTCgAAACTggCAggCTAgAgTCT
I-6:TAACCACAgCAATTgACgTTACCCg
I-7:gCAATTgACgTTACCCgCAgAAgA
上記のプローブは、DNAマイクロアレイに固定するための官能基として、合成後、定法に従って核酸の5'末端にチオール基を導入した。官能基の導入後、精製し、凍結乾燥した。凍結乾燥した内部標準用プローブは、-30℃の冷凍庫に保存した。
【0076】
一方、黄色ブドウ球菌(A−n)、表皮ブドウ球菌(B−n)、大腸菌(C−n)、肺炎桿菌(D−n)、緑膿菌(E−n)、セラチア菌(F−n)、肺炎連鎖球菌(G−n)、インフルエンザ菌(H−n)、及びエンテロコッカス・フェカリス菌(J−n)(nは数字)についても同様な手法により以下に示すプローブセットを設計した。
A-1:gAACCgCATggTTCAAAAgTgAAAgA
A-2:CACTTATAgATggATCCgCgCTgC
A-3:TgCACATCTTgACggTACCTAATCAg
A-4:CCCCTTAgTgCTgCAgCTAACg
A-5:AATACAAAgggCAgCgAAACCgC
A-6:CCggTggAgTAACCTTTTAggAgCT
A-7:TAACCTTTTAggAgCTAgCCgTCgA
A-8:TTTAggAgCTAgCCgTCgAAggT
A-9:TAgCCgTCgAAggTgggACAAAT
B-1:gAACAgACgAggAgCTTgCTCC
B-2:TAgTgAAAgACggTTTTgCTgTCACT
B-3:TAAgTAACTATgCACgTCTTgACggT
B-4:gACCCCTCTAgAgATAgAgTTTTCCC
B-5:AgTAACCATTTggAgCTAgCCgTC
B-6:gAgCTTgCTCCTCTgACgTTAgC
B-7:AgCCggTggAgTAACCATTTgg
C-1:CTCTTgCCATCggATgTgCCCA
C-2:ATACCTTTgCTCATTgACgTTACCCg
C-3:TTTgCTCATTgACgTTACCCgCAg
C-4:ACTggCAAgCTTgAgTCTCgTAgA
C-5:ATACAAAgAgAAgCgACCTCgCg
C-6:CggACCTCATAAAgTgCgTCgTAgT
C-7:gCggggAggAAgggAgTAAAgTTAAT
D-1:TAgCACAgAgAgCTTgCTCTCgg
D-2:TCATgCCATCAgATgTgCCCAgA
D-3:CggggAggAAggCgATAAggTTAAT
D-4:TTCgATTgACgTTACCCgCAgAAgA
D-5:ggTCTgTCAAgTCggATgTgAAATCC
D-6:gCAggCTAgAgTCTTgTAgAgggg
E-1:TgAgggAgAAAgTgggggATCTTC
E-2:TCAgATgAgCCTAggTCggATTAgC
E-3:gAgCTAgAgTACggTAgAgggTgg
E-4:gTACggTAgAgggTggTggAATTTC
E-5:gACCACCTggACTgATACTgACAC
E-6:TggCCTTgACATgCTgAgAACTTTC
E-7:TTAgTTACCAgCACCTCgggTgg
E-8:TAgTCTAACCgCAAgggggACg
F-1:TAgCACAgggAgCTTgCTCCCT
F-2:AggTggTgAgCTTAATACgCTCATC
F-3:TCATCAATTgACgTTACTCgCAgAAg
F-4:ACTgCATTTgAAACTggCAAgCTAgA
F-5:TTATCCTTTgTTgCAgCTTCggCC
F-6:ACTTTCAgCgAggAggAAggTgg
G-1:AgTAgAACgCTgAAggAggAgCTTg
G-2:CTTgCATCACTACCAgATggACCTg
G-3:TgAgAgTggAAAgTTCACACTgTgAC
G-4:gCTgTggCTTAACCATAgTAggCTTT
G-5:AAgCggCTCTCTggCTTgTAACT
G-6:TAgACCCTTTCCggggTTTAgTgC
G-7:gACggCAAgCTAATCTCTTAAAgCCA
H-1:gCTTgggAATCTggCTTATggAgg
H-2:TgCCATAggATgAgCCCAAgTgg
H-3:CTTgggAATgTACTgACgCTCATgTg
H-4:ggATTgggCTTAgAgCTTggTgC
H-5:TACAgAgggAAgCgAAgCTgCg
H-6:ggCgTTTACCACggTATgATTCATgA
H-7:AATgCCTACCAAgCCTgCgATCT
H-8:TATCggAAgATgAAAgTgCgggACT
J-1:TTCTTTCCTCCCgAgTgCTTgCA
J-2:AACACgTgggTAACCTACCCATCAg
J-3:ATggCATAAgAgTgAAAggCgCTT
J-4:gACCCgCggTgCATTAgCTAgT
J-5:ggACgTTAgTAACTgAACgTCCCCT
J-6:CTCAACCggggAgggTCATTgg
J-7:TTggAgggTTTCCgCCCTTCAg
<検体増幅用PCR Primer の準備>
起炎菌検出用の為の16s rRNA核酸(標的核酸)増幅用PCR Primerとして表1に示す核酸配列を設計した。具体的には、16s rRNAをコーディングしているゲノム部分を特異的に増幅するプローブセット、つまり約1500塩基長の16s rRNAコーディング領域の両端部分で、特異的な融解温度をできるだけ揃えたプライマーを設計した。なお、変異株や、ゲノム上に複数存在する16s rRNAコーディング領域も同時に増幅できるように複数種類のプライマーを設計した。
【0077】
【表1】

【0078】
表中に示したPrimerは、合成後、高速液体クロマトグラフィー(HPLC)により精製し、Forward Primer 3種、Reverse Primer 3種を混合し、それぞれのPrimer濃度が、最終濃度10 pmol/μl となるようにTE緩衝液に溶解した。
【0079】
<Enterobacter#cloacae Genome DNA(モデル検体)の抽出>
(微生物の培養 & Genome DNA 抽出の前処理)
まず、Enterobacter cloacae 標準株を、定法に従って培養した。この微生物培養液を1.5ml容量のマイクロチューブに1.0ml(OD600=0.7)採取し、遠心分離で菌体を回収した(8500rpm、5min、4℃)。上精を捨てた後、Enzyme Buffer(50mM Tris-HCl:p.H. 8.0、25mM EDTA)300μlを加え、ミキサーを用いて再縣濁した。再縣濁した菌液は、再度、遠心分離で菌体を回収した(8500rpm、5min、4℃)。上精を捨てた後、回収された菌体に、以下の酵素溶液を加え、ミキサーを用いて再縣濁した。
Lysozyme:50 μl (20 mg/ml in Enzyme Buffer)
N-Acetylmuramidase SG:50 μl (0.2 mg/ml in Enzyme Buffer)
次に、酵素溶液を加え再縣濁した菌液を、37℃のインキュベーター内で30分間静置し、細胞壁の溶解処理を行った。
【0080】
(Genome抽出)
以下に示す微生物のGenome DNA抽出は、核酸精製キット(MagExtractor -Genome-:TOYOBO社製)を用いて行った。具体的には、まず、前処理した微生物縣濁液に溶解・吸着液750μlと磁性ビーズ40μlを加え、チューブミキサーを用いて、10分間激しく攪拌した(ステップ1)。次に、分離用スタンド(Magical Trapper)にマイクロチューブをセットし、30秒間静置して磁性粒子をチューブの壁面に集め、スタンドにセットした状態のまま、上精を捨てた(ステップ2)。次に、洗浄液 900 μl を加え、ミキサーで5sec程度攪拌して再縣濁を行った(ステップ3)。次に、分離用スタンド(Magical Trapper)にマイクロチューブをセットし、30秒間静置して磁性粒子をチューブの壁面に集め、スタンドにセットした状態のまま、上精を捨てた(ステップ4)。ステップ3、4を繰り返して2度目の洗浄(ステップ5)を行った後、70%エタノール 900 μl を加え、ミキサーで5sec程度攪拌して再縣濁した(ステップ6)。次に、分離用スタンド(Magical Trapper)にマイクロチューブをセットし、30秒間静置して磁性粒子をチューブの壁面に集め、スタンドにセットした状態のまま、上精を捨てた(ステップ7)。ステップ6、7を繰り返して70%エタノールによる2度目の洗浄(ステップ8)を行った後、回収された磁性粒子に純水 100 μl を加え、チューブミキサーで10分間攪拌を行った。
【0081】
次に分離用スタンド(Magical Trapper)にマイクロチューブをセットし、30秒間静置して磁性粒子をチューブ壁面に集め、スタンドにセットした状態のまま、上精を新しいチューブに回収した。
【0082】
(回収したGenome DNAの検査)
回収された微生物(Enterobacter cloacae 株)のGenome DNAは、定法に従って、アガロース電気泳動と260/280nmの吸光度測定を行い、その品質(低分子核酸の混入量、分解の程度)と回収量を検定した。本実施例では、約10μgのGenome DNA が回収され、GenomeDNAのデグラデーションやrRNAの混入は認められなかった。回収したGenome DNAは、最終濃度50ng/μlとなるようにTE緩衝液に溶解し、以下の工程に使用した。
【0083】
<DNAマイクロアレイの作製>
[1]ガラス基板の洗浄
合成石英のガラス基板(サイズ:25mmx75mmx1mm、飯山特殊ガラス社製)を耐熱、耐アルカリ のラックに入れ、所定の濃度に調製した超音波洗浄用の洗浄液に浸した。一晩洗浄液中で浸した後、20分間超音波洗浄を行った。続いて基板を取り出し、軽く純水ですすいだ後、超純水中で20分超音波洗浄をおこなった。次に80℃に加熱した1N水酸化ナトリウム水溶液中に10分間基板を浸した。再び純水洗浄と超純水洗浄を行い、DNAチップ用の石英ガラス基板を用意した。
【0084】
[2]表面処理
シランカップリング剤KBM-603(信越シリコーン社製)を、1%の濃度となるように純水中に溶解させ、2時間室温で攪拌した。続いて、先に洗浄したガラス基板をシランカップリング剤水溶液に浸し、20分間室温で放置した。ガラス基板を引き上げ、軽く純水で表面を洗浄した後、窒素ガスを基板の両面に吹き付けて乾燥させた。次に乾燥した基板を120℃に加熱したオーブン中で1時間ベークし、カップリング剤処理を完結させ、基板表面にアミノ基を導入した。次いで同仁化学研究所社製のN-マレイミドカプロイロキシスクシイミドを、ジメチルスルホキシドとエタノールの1:1混合溶媒中に最終濃度が0.3mg/mlとなるように溶解させてEMCS溶液を用意した。なお、N-マレイミドカプロイロキシスクシイミド(N-(6-Maleimidocaproyloxy)succinimido)を以下EMCSと略す。ベークの終了したガラス基板を放冷し、調製したEMCS溶液中に室温で2時間浸した。この処理により、シランカップリング剤によって表面に導入されたアミノ基とEMCSのスクシイミド基が反応し、ガラス基板表面にマレイミド基が導入された。EMCS溶液から引き上げたガラス基板を、先述のMCSを溶解した混合溶媒を用いて洗浄し、さらにエタノールにより洗浄した後、窒素ガス雰囲気下で乾燥させた。
【0085】
[3]プローブDNA
先に作製した微生物検出用プローブを純水に溶解し、それぞれ、最終濃度(インク溶解時)10μMとなるように分注した後、凍結乾燥を行い、水分を除いた。
【0086】
[4]BJプリンターによるDNA吐出、および基板への結合
グリセリン7.5wt%、チオジグリコール7.5wt%、尿素7.5wt%、アセチレノールEH(川研ファインケミカル社製)1.0wt%を含む水溶液を用意した。続いて、先に用意した7種類のプローブ(表1)を上記の混合溶媒に規定濃度なるように溶解した。得られたDNA溶液をバブルジェットプリンター(商品名:BJF-850 キヤノン社製)用インクタンクに充填し、印字ヘッドに装着した。
【0087】
なおここで用いたバブルジェットプリンターは平板への印刷が可能なように改造を施したものである。またこのバブルジェットプリンターは、所定のファイル作成方法に従って印字パターンを入力することにより、約5ピコリットルのDNA溶液を約120マイクロメートルピッチでスポッティングすることが可能となっている。続いて、この改造バブルジェットプリンターを用いて、1枚のガラス基板に対して、印字操作を行い、アレイを作製した。印字が確実に行われていることを確認した後、30分間加湿チャンバー内に静置し、ガラス基板表面のマレイミド基と核酸プローブ末端のチオール基とを反応させた。
【0088】
[5]洗浄
30分間の反応後、100mMのNaClを含む10mMのリン酸緩衝液(pH7.0)により表面に残ったDNA溶液を洗い流し、ガラス基板表面に一本鎖DNAが固定したDNAマイクロアレイを得た。
【0089】
<検体の増幅と標識化(PCR増幅&蛍光標識の取り込み)>
検体となる微生物DNAの増幅、および、標識化反応を以下に示す。
Premix PCR 試薬(TAKARA ExTaq):25μl
Template Genome DNA:2μl (100ng)
Forward Primer mix: 2μl (20pmol/tube each)
Reverse Primer mix: 2μl (20pmol/tube each)
Cy-3 dUTP (1mM): 2μl (2nmol/tube)
H20:17μl
(Total:50μl)
上記組成の反応液を以下のプロトコールに従って、市販のサーマルサイクラーで増幅反応を行った。
(ステップ1)95℃、10 min.
(ステップ2)92℃、45 sec.
(ステップ3)55℃、45 sec.
(ステップ4)72℃、45 sec.
(ステップ5)72℃、10 min.
(ステップ2〜4は35回繰り返した。)
反応終了後、精製用カラム(QIAGEN QIAquick PCR Purification Kit)を用いてPrimerを除去した後、増幅産物の定量を行い、標識化検体とした。
【0090】
<ハイブリダイゼーション>
<DNAマイクロアレイの作製>で作製したDNAマイクロアレイと<検体の増幅と標識化(PCR増幅&蛍光標識の取り込み)>で作製した標識化検体を用いて検出反応を行った。
【0091】
(DNAマイクロアレイのブロッキング)
BSA(牛血清アルブミンFraction V:Sigma社製)を1wt%となるように100mM NaCl / 10mM Phosphate Bufferに溶解した。この溶液に<DNAマイクロアレイの作製>で作製したDNAマイクロアレイを室温で2時間浸し、ブロッキングを行った。ブロッキング終了後、0.1wt%SDS(ドデシル硫酸ナトリウム)を含む2xSSC溶液(NaCl 300mM 、Sodium Citrate (trisodium citrate dihydrate, C6H5Na3・2H2O) 30mM、p.H. 7.0)で洗浄を行った。その後、純水でリンスしてからスピンドライ装置で水切りを行った。
【0092】
(ハイブリダイゼーション)
水切りしたDNAマイクロアレイをハイブリダイゼーション装置(Genomic Solutions Inc. Hybridization Station)にセットし、以下に示すハイブリダイゼーション溶液、条件でハイブリダイゼーション反応を行った。
【0093】
<ハイブリダイゼーション溶液>
6 x SSPE / 10% Form amide / Target (2nd PCR Products 全量)
(6xSSPE: NaCl 900mM、NaH2PO4・H2O 60mM、EDTA 6mM、p.H. 7.4)
<ハイブリダイゼーション条件>
65 ℃、3min→92℃、2min→45℃、3hr→Wash、2xSSC/0.1% SDS、25℃→Wash、2 x SSC、20℃→(Rinse with H2O: Manual)→Spin dry
<微生物の検出(蛍光測定)>
ハイブリダイゼーション反応終了後のDNAマイクロアレイをDNAマイクロアレイ用蛍光検出装置(Axon社製、GenePix 4000B)を用いで蛍光測定を行った。
【0094】
この結果得られた画像データとしての画像の例を図6に示す。なお、図6においてより蛍光強度の強いプローブは、より濃い色で示している。601はDNAマイクロアレイに黄色ブドウ球菌のゲノムを含むサンプルを反応させた画像で、602は大腸菌のゲノムを含むサンプルを反応させた画像の例である。図の左に書いているアルファベットは、プローブ配列のアルファベットである。AからJまでそれぞれ、以下の各菌に特異的に結合するように設計されたプローブである。
(A)黄色ブドウ球菌。
(B)表皮ブドウ球菌。
(C)大腸菌。
(D)肺炎桿菌。
(E)緑膿菌。
(F)セラチア菌。
(G)肺炎連鎖球菌。
(H)インフルエンザ菌。
(I)エンテロバクター・クロアカエ菌。
(J)エンテロコッカス・フェカリス菌。
【0095】
理想的には、601のAの行のプローブだけが蛍光強度が高くなり、かつ、602のCの行のプローブだけが蛍光強度が高くなる。この601の理想的な結果は、図5に示した実験結果の例と同じである。
【0096】
しかし、図6に示すように、実際は理想通りにはならない。つまり、いわゆる"クロスハイブリダイゼーション反応"がおこり、601の場合は、A以外の行のプローブも蛍光強度が強く、また、602の場合は、C以外の行のプローブも蛍光強度が強い。更に、602の場合、Cの行でも蛍光強度の弱いプローブもある。
【0097】
この状況を3つのプローブの系で説明したのが図7である。黄色ブドウ球菌(S. aureus)、表皮ブドウ球菌(S. epiderimidis)、大腸菌(E. coli)の3種類のプローブがあるDNAマイクロアレイを用いてそれぞれの菌について6種類の既知サンプルの実験をしている。一般にプローブがN個ある場合、実験データはN次元のベクトルとなる。図6の場合、プローブが合計72個あるので、72次元のベクトル、図7の場合、プローブが3つあるので、3次元のベクトルが実験データとなる。
【0098】
図7下の図で、3菌それぞれ6種類のサンプル(=合計18個のデータ)を3次元座標にプロットしてある。図に示した通り、3つのプローブが理想的にそれぞれ3つの菌に非常に特異的なプローブである場合、図7下のようにベクトルデータは、それぞれの軸のまわりに集中する。但し、データの揺らぎは存在し、1つの点に集中するわけではない。図7の例でいうと、3菌それぞれのデータ存在範囲の大きさは異なっており、大きい順で大腸菌(E. coli)、表皮ブドウ球菌(S. epiderimidis)、黄色ブドウ球菌(S. aureus)となっている。
【0099】
さらに、各菌ごとに先に示した図1及び図8の方法に従って判定指数集合を導き、判定不能閾値を設定し、設定された判定不能閾値を用いて未知サンプルの供給元である菌の判定を行なうか、あるいは判定しない点を決定することができる。
【0100】
実施例2
以下に肺炎桿菌とセラチア菌のDNAマイクロアレイの実験データを示す。なお、プローブとしては以下の各菌の検出用のものを用いている。
黄色ブドウ球菌(S.aureus)(A−n)。
表皮ブドウ球菌(S.epidermidis)(B−n)。
大腸菌(E.coli)(C−n)、肺炎桿菌(K.pneumoniae)(D−n)。
緑膿菌(P.aeruginosa)(E−n)。
セラチア菌(S.marcescenes)(F−n)。
肺炎連鎖球菌(S.neumoiae)(G−n)。
インフルエンザ菌(H.influenzae)(H−n)。
エンテロバクター・クロアカエ菌(Enterobacter cloacae)(I−n)。
及びエンテロコッカス・フェカリス菌(E.faecelis)(J−n)。
【0101】
なお、上記のカッコ内のnは先に示したn=1〜6である。結局、全プローブ数は10×6=60個となる。
【0102】
まず、肺炎桿菌の10個の異なるサンプルに対するDNAマイクロアレイの実験データを図11〜20に示す。各図において左から右にプローブA−1、A−2、・・・〜J−5、J−6の順で配列されている。図示した通り、DNAマイクロアレイの実験データは、60個の蛍光輝度の値、つまり60次元のベクトルとして得られる。まず、任意のベクトルの間の距離を定義するために、「ベクトルのノルムでベクトルの各要素を割る」正規化を行う。式で記述すると、
【0103】
【数1】

【0104】
式においてベクトルxが元のベクトルで、ベクトルyが正規化後のベクトル、となる。
このように正規化したベクトルはそのノルムが常に1となっている。なお、ここでn次元のベクトルxのノルム(ユークリッドノルム)とは次の式で定義される。
【0105】
【数2】

【0106】
そして、正規化後の2つのベクトル(ベクトルaとベクトルb)間の距離を次の式で定義する。
【0107】
【数3】

【0108】
本実施例では、k-th nearest neighborマッチングアルゴリズムの距離定義を上記のようにする。10個のサンプルの間の任意の1組ずつの距離を計算し、ヒストグラムにしたものを図21に示す。データの数は10C2=45個になる。この図から、肺炎桿菌に上記k-th nearest neighborのアルゴリズムを適用して判定するとすると、その判定不能閾値は最大値である0.057というのが一つの候補になる。少し余裕を持たせて、1.5倍とか2倍の値を使っても良い。
【0109】
次に、セラチア菌の同じく10個のサンプルの実験データを図22〜31に示す。肺炎桿菌で行った正規化、距離計算を用いて、10個のサンプルの任意の2サンプルの距離を計算し、ヒストグラムを取ったのが、図32である。肺炎桿菌のヒストグラムと分布の形状が全く異なるのがわかる。大きく山が2つ存在するということは、10個のベクトルの中で2つのクラスターが存在することが想定される。実際、先に示した10サンプルの蛍光輝度グラフを見ても、大きく分けて2種類のパターンが存在することがわかる。この図から、肺炎桿菌に上記k-th nearest neighborのアルゴリズムを適用して判定する、とすると、その棄却値は1つ目の山の最大値である0.090というのが一つの候補になる。
【図面の簡単な説明】
【0110】
【図1】本発明の生物種類判定方法の一例を示す図である。
【図2】本発明の生物種類判定方法を実行するための情報処理装置の構成を示すブロック図である。
【図3】ハイブリダイゼーション反応を説明する図である。
【図4】DNAマイクロアレイを用いた実験手順を示すである。
【図5】感染症の判定用DNAマイクロアレイの実験手順を示すである。
【図6】ハイブリダイゼーション反応後の蛍光強度からなる画像の一例を示すである。
【図7】ベクトルデータの分布例を示すである。
【図8】判定不能値設定ステップを説明する図である。
【図9】判定指数集合の分布例を示すである。
【図10】同一カテゴリー内の任意の2サンプルの距離集合例を示すである。
【図11】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図12】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図13】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図14】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図15】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図16】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図17】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図18】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図19】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図20】肺炎桿菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図21】10個の肺炎桿菌サンプル間の任意の1組ずつの距離に関するヒストグラムである。
【図22】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図23】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図24】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図25】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図26】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図27】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図28】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図29】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図30】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図31】セラチア菌サンプルに対するDNAマイクロアレイの実験データを示す図である。
【図32】10個のセラチア菌サンプル間の任意の1組ずつの距離に関するヒストグラムである。
【特許請求の範囲】
【請求項1】
生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物の種類を判定する生物種判定方法において、
対応する生物種が判明している複数の既知サンプルを生物種分析方法により分析して、複数の分析データを得る工程と、
既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する工程と、
対応する生物種が未知である未知サンプルを、前記生物種分析法により分析して、該未知サンプルに対応する生物種の特定のための分析データを得る工程と、
前記判定不能閾値に基づいて前記未知サンプルに対応する種類を判定するか、あるいは判定不能であるかを決定する工程と、
判定をすると決定されたならば、前記複数の分析データに基づいて前記未知サンプルの生物種を判定する工程と、
を有する生物種判定方法。
【請求項2】
前記判定不能閾値は、一つの生物種における前記既知サンプルから得た記憶手段に格納された複数の分析データの総体から、任意の分析データを除外して、残りの分析データに基づいて学習した識別辞書を作成し、該除外した分析データを該識別辞書に基づいて判定して判定指数を導き、該判定指数に基づいて設定される請求項1に記載の生物種判定方法。
【請求項3】
生物に由来する物質が含まれていることが想定される物質を生物種分析方法にて分析して、対応する生物の種類を判定する生物種判定方法において、
(1)前記未知サンプルに対する判定結果として想定される生物種を選択する工程と、
(2)前記選択された生物類に属することが判明している複数の個体から得られる既知サンプルの各々から、該生物種に特徴的であって、パターン認識用として使用し得る複数の画像データからなる画像データ群を得る工程と、
(3)前記画像データ群から画像データを選択し、残りの画像データとの関係を用いて判定不能閾値を設定する工程と、
(4)未知サンプルからの画像データを得る工程と、
(5)前記未知サンプルからの画像データを前記判定不能閾値に基づいて、該未知サンプルに対応する生物種を判定するか判定が不能であるかを決定する工程と、
(6)前記(5)で判定を行うことが決定された場合は、前記画像データ群からなる識別辞書を用いて、生物種を判定する工程と、
を有することを特徴とする生物種類判定方法。
【請求項4】
前記判定不能閾値の設定が、
(1)前記個体として3以上の異なる個体を選択し、得られた3以上の画像データからなる画像データ群を得る工程と、
(2)前記画像データ群から1つの画像データを選択して除外し、残りの複数の画像データを用いて辞書を作成し、得られた辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なってm個の判定指数からなる判定指数集合を得る工程と、
(3)前記判定指数集合から判定不能閾値を決定する工程と、
を有する方法により行われる請求項3に記載の生物種類判定方法。
【請求項5】
前記判定不能閾値の設定が、
(1)前記個体として3以上の異なる個体を選択し、得られた3以上の画像データからなる画像データ群を得る工程と、
(2)前記画像データ群から選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程と、
(3)前記距離集合から判定不能閾値を決定する工程と、
を有する方法により行われる請求項3に記載の生物種判定方法。
【請求項6】
前記生物種分析方法が、前記選択された生物に特徴的な塩基配列を有する標的核酸と特異的に結合し得るプローブを基板上に位置を定めて固定したプローブ固定担体に、既知または未知のサンプルとしての核酸試料を反応させ、前記基板上に形成された標的核酸とプローブの結合体を光学的に検出して、画像データを得る方法である請求項3記載の生物種判定方法。
【請求項7】
前記結合体の光学な検出が、該結合体に付与した蛍光標識からの蛍光を利用して行なわれる請求項6に記載の判定方法。
【請求項8】
対応する生物種が判明している複数の既知サンプルを生物分析方法により分析して得られた複数の分析データ、及び、該複数の分析データに基づいて設定された判定不能閾値を記憶したメモリと、該メモリに記憶された判定不能閾値に基づいて、未知サンプルに対応する生物種が判定可能か否かを決定し、判定可能と決定した場合には、前記メモリに記憶された複数の分析データに基づいて前記未知サンプルに対応する生物種の判定を行う処理ユニットとから成る生物種判定のための情報処理装置。
【請求項9】
生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物種を判定するための情報処理装置において、対応する生物種が判明している複数の既知サンプルを分析して得られる該生物種に特徴的な画像データを入力するための既知サンプル画像データ入力手段と、
未知サンプルを前記既知サンプルと同様に分析して得られる画像データを入力する未知サンプル画像データ入力手段と、
取り込まれた前記画像データを記憶する記憶手段と、
既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する手段と、
未知サンプルからの画像データを前記判定不能閾値にもとづいて判定を行うかまたは判定を行わないかを決定し、判定を行うのであれば、未知サンプルに対応する生物種を判定する生物種判定手段と、
前記判定手段での判定結果を記憶する記憶手段と、
前記記憶手段に記憶された判定結果を出力する出力手段と
を有することを特徴とする生物種判定のための情報処理装置。
【請求項10】
前記判定不能閾値の設定が、
前記個体が3以上であり、前記記憶手段にこれらの個体からの画像データが記憶されており、
(a)前記3以上の画像データから1つの画像データを選択して除外し、残りの複数の画像データを用いて識別辞書を作成し、得られた識別辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なって3以上の判定指数からなる判定指数集合を得るステップと、
(b)前記判定指数集合から判定不能閾値を設定するステップと、
を有するプログラムに基づいて実行される請求項9に記載の情報処理装置。
【請求項11】
前記判定不能閾値の設定が、前記個体が3以上であり、前記記憶手段にこれらの個体からの画像データが記憶されており、
(A)前記3以上の画像データから選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程と、
(B)前記距離集合から判定不能閾値を決定する工程と、
を有するプログラムに基づいて実行される請求項9に記載の情報処理装置。
【請求項12】
未知サンプルに対応する生物種の判定をコンピュータに実行させるためのプログラムであって、
(1)未知サンプルに対する判定結果として想定される生物種に属する複数の異なる個体からの既知サンプルを分析して得られる該想定される生物種に特徴的な画像データに対応する複数の画像データを格納した記憶手段から、これらの複数の既知サンプル画像データを呼び出すステップと、
(2)未知サンプルを前記既知サンプルと同様にして分析して得られる画像データに対応する複数の画像データを格納した記憶手段から、該未知サンプル画像データを読み出すステップと、
(3)前記既知サンプル画像データから1つを選択し、選択された1つと残りの画像データとの関係を用いて判定不能閾値を設定するステップと、
(4)前記判定不能閾値に基づいて前記未知サンプル画像データを処理し、未知サンプルに対応する生物の種類を判定するステップと、
(5)前記判定ステップで得られた判定結果を記憶手段に格納させるステップと、
(6)前記記憶手段に格納された判定結果を出力するステップと
を有することを特徴とする生物種類判定用プログラム。
【請求項13】
前記判定不能閾値の設定が、
前記個体が3以上であり、前記記憶手段にこれらの個体からの画像データが記憶されており、(a)前記3以上の画像データから1つの画像データを選択して除外し、残りの複数の画像データを用いて識別辞書を作成し、得られた識別辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なって3以上の判定指数からなる判定指数集合を得るステップと、
(b)前記判定指数集合から判定不能閾値を決定するステップと、
を有する請求項10に記載の生物種類判定用プログラム。
【請求項14】
前記判定不能閾値の設定が、
前記個体が3以上のであり、前記記憶手段にこれらの個体からの画像データが記憶されており、(A)前記3以上の画像データから選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程と、
(B)前記距離集合から判定不能閾値を決定する工程と、
を有する請求項11に記載の生物種類判定用プログラム。
【請求項15】
生物種判定をコンピュータで実行するためのプログラムを読み取り可能に記録した記録媒体であって、
前記プログラムが請求項11〜13のいずれかに記載のプログラムである
ことを特徴とする記録媒体。
【請求項16】
対応する生物種が判明している複数の既知サンプルを生物分析方法により分析して得られた複数の分析データと、該複数の分析データに基づいて設定された判定不能閾値を用い、前記判定不能閾値に基づいて、未知サンプルに対応する生物種が判定可能か否かを決定した後、判定可能と決定した場合には、前記複数の分析データに基づいて前記未知サンプルに対応する生物種の判定を行う生物種判定方法。
【請求項1】
生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物の種類を判定する生物種判定方法において、
対応する生物種が判明している複数の既知サンプルを生物種分析方法により分析して、複数の分析データを得る工程と、
既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する工程と、
対応する生物種が未知である未知サンプルを、前記生物種分析法により分析して、該未知サンプルに対応する生物種の特定のための分析データを得る工程と、
前記判定不能閾値に基づいて前記未知サンプルに対応する種類を判定するか、あるいは判定不能であるかを決定する工程と、
判定をすると決定されたならば、前記複数の分析データに基づいて前記未知サンプルの生物種を判定する工程と、
を有する生物種判定方法。
【請求項2】
前記判定不能閾値は、一つの生物種における前記既知サンプルから得た記憶手段に格納された複数の分析データの総体から、任意の分析データを除外して、残りの分析データに基づいて学習した識別辞書を作成し、該除外した分析データを該識別辞書に基づいて判定して判定指数を導き、該判定指数に基づいて設定される請求項1に記載の生物種判定方法。
【請求項3】
生物に由来する物質が含まれていることが想定される物質を生物種分析方法にて分析して、対応する生物の種類を判定する生物種判定方法において、
(1)前記未知サンプルに対する判定結果として想定される生物種を選択する工程と、
(2)前記選択された生物類に属することが判明している複数の個体から得られる既知サンプルの各々から、該生物種に特徴的であって、パターン認識用として使用し得る複数の画像データからなる画像データ群を得る工程と、
(3)前記画像データ群から画像データを選択し、残りの画像データとの関係を用いて判定不能閾値を設定する工程と、
(4)未知サンプルからの画像データを得る工程と、
(5)前記未知サンプルからの画像データを前記判定不能閾値に基づいて、該未知サンプルに対応する生物種を判定するか判定が不能であるかを決定する工程と、
(6)前記(5)で判定を行うことが決定された場合は、前記画像データ群からなる識別辞書を用いて、生物種を判定する工程と、
を有することを特徴とする生物種類判定方法。
【請求項4】
前記判定不能閾値の設定が、
(1)前記個体として3以上の異なる個体を選択し、得られた3以上の画像データからなる画像データ群を得る工程と、
(2)前記画像データ群から1つの画像データを選択して除外し、残りの複数の画像データを用いて辞書を作成し、得られた辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なってm個の判定指数からなる判定指数集合を得る工程と、
(3)前記判定指数集合から判定不能閾値を決定する工程と、
を有する方法により行われる請求項3に記載の生物種類判定方法。
【請求項5】
前記判定不能閾値の設定が、
(1)前記個体として3以上の異なる個体を選択し、得られた3以上の画像データからなる画像データ群を得る工程と、
(2)前記画像データ群から選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程と、
(3)前記距離集合から判定不能閾値を決定する工程と、
を有する方法により行われる請求項3に記載の生物種判定方法。
【請求項6】
前記生物種分析方法が、前記選択された生物に特徴的な塩基配列を有する標的核酸と特異的に結合し得るプローブを基板上に位置を定めて固定したプローブ固定担体に、既知または未知のサンプルとしての核酸試料を反応させ、前記基板上に形成された標的核酸とプローブの結合体を光学的に検出して、画像データを得る方法である請求項3記載の生物種判定方法。
【請求項7】
前記結合体の光学な検出が、該結合体に付与した蛍光標識からの蛍光を利用して行なわれる請求項6に記載の判定方法。
【請求項8】
対応する生物種が判明している複数の既知サンプルを生物分析方法により分析して得られた複数の分析データ、及び、該複数の分析データに基づいて設定された判定不能閾値を記憶したメモリと、該メモリに記憶された判定不能閾値に基づいて、未知サンプルに対応する生物種が判定可能か否かを決定し、判定可能と決定した場合には、前記メモリに記憶された複数の分析データに基づいて前記未知サンプルに対応する生物種の判定を行う処理ユニットとから成る生物種判定のための情報処理装置。
【請求項9】
生物に由来する物質が含まれていることが想定される物質を分析して、対応する生物種を判定するための情報処理装置において、対応する生物種が判明している複数の既知サンプルを分析して得られる該生物種に特徴的な画像データを入力するための既知サンプル画像データ入力手段と、
未知サンプルを前記既知サンプルと同様に分析して得られる画像データを入力する未知サンプル画像データ入力手段と、
取り込まれた前記画像データを記憶する記憶手段と、
既知サンプルから得た該複数の分析データに基づいて、該既知サンプルに対応する生物種に関する判定不能閾値を設定する手段と、
未知サンプルからの画像データを前記判定不能閾値にもとづいて判定を行うかまたは判定を行わないかを決定し、判定を行うのであれば、未知サンプルに対応する生物種を判定する生物種判定手段と、
前記判定手段での判定結果を記憶する記憶手段と、
前記記憶手段に記憶された判定結果を出力する出力手段と
を有することを特徴とする生物種判定のための情報処理装置。
【請求項10】
前記判定不能閾値の設定が、
前記個体が3以上であり、前記記憶手段にこれらの個体からの画像データが記憶されており、
(a)前記3以上の画像データから1つの画像データを選択して除外し、残りの複数の画像データを用いて識別辞書を作成し、得られた識別辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なって3以上の判定指数からなる判定指数集合を得るステップと、
(b)前記判定指数集合から判定不能閾値を設定するステップと、
を有するプログラムに基づいて実行される請求項9に記載の情報処理装置。
【請求項11】
前記判定不能閾値の設定が、前記個体が3以上であり、前記記憶手段にこれらの個体からの画像データが記憶されており、
(A)前記3以上の画像データから選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程と、
(B)前記距離集合から判定不能閾値を決定する工程と、
を有するプログラムに基づいて実行される請求項9に記載の情報処理装置。
【請求項12】
未知サンプルに対応する生物種の判定をコンピュータに実行させるためのプログラムであって、
(1)未知サンプルに対する判定結果として想定される生物種に属する複数の異なる個体からの既知サンプルを分析して得られる該想定される生物種に特徴的な画像データに対応する複数の画像データを格納した記憶手段から、これらの複数の既知サンプル画像データを呼び出すステップと、
(2)未知サンプルを前記既知サンプルと同様にして分析して得られる画像データに対応する複数の画像データを格納した記憶手段から、該未知サンプル画像データを読み出すステップと、
(3)前記既知サンプル画像データから1つを選択し、選択された1つと残りの画像データとの関係を用いて判定不能閾値を設定するステップと、
(4)前記判定不能閾値に基づいて前記未知サンプル画像データを処理し、未知サンプルに対応する生物の種類を判定するステップと、
(5)前記判定ステップで得られた判定結果を記憶手段に格納させるステップと、
(6)前記記憶手段に格納された判定結果を出力するステップと
を有することを特徴とする生物種類判定用プログラム。
【請求項13】
前記判定不能閾値の設定が、
前記個体が3以上であり、前記記憶手段にこれらの個体からの画像データが記憶されており、(a)前記3以上の画像データから1つの画像データを選択して除外し、残りの複数の画像データを用いて識別辞書を作成し、得られた識別辞書に基づいて先に除外した画像データを判定して判定指数を得る処理を、各画像データごとに行なって3以上の判定指数からなる判定指数集合を得るステップと、
(b)前記判定指数集合から判定不能閾値を決定するステップと、
を有する請求項10に記載の生物種類判定用プログラム。
【請求項14】
前記判定不能閾値の設定が、
前記個体が3以上のであり、前記記憶手段にこれらの個体からの画像データが記憶されており、(A)前記3以上の画像データから選択した任意の2つの画像データの全ての組み合せについて、2つの画像データ間の距離を求め、距離集合を得る工程と、
(B)前記距離集合から判定不能閾値を決定する工程と、
を有する請求項11に記載の生物種類判定用プログラム。
【請求項15】
生物種判定をコンピュータで実行するためのプログラムを読み取り可能に記録した記録媒体であって、
前記プログラムが請求項11〜13のいずれかに記載のプログラムである
ことを特徴とする記録媒体。
【請求項16】
対応する生物種が判明している複数の既知サンプルを生物分析方法により分析して得られた複数の分析データと、該複数の分析データに基づいて設定された判定不能閾値を用い、前記判定不能閾値に基づいて、未知サンプルに対応する生物種が判定可能か否かを決定した後、判定可能と決定した場合には、前記複数の分析データに基づいて前記未知サンプルに対応する生物種の判定を行う生物種判定方法。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】


【図16】


【図17】


【図18】


【図19】


【図20】


【図21】


【図22】


【図23】


【図24】


【図25】


【図26】


【図27】


【図28】


【図29】


【図30】


【図31】


【図32】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】


【図16】


【図17】


【図18】


【図19】


【図20】


【図21】


【図22】


【図23】


【図24】


【図25】


【図26】


【図27】


【図28】


【図29】


【図30】


【図31】


【図32】


【公開番号】特開2007−61092(P2007−61092A)
【公開日】平成19年3月15日(2007.3.15)
【国際特許分類】
【出願番号】特願2006−215083(P2006−215083)
【出願日】平成18年8月7日(2006.8.7)
【出願人】(000001007)キヤノン株式会社 (59,756)
【Fターム(参考)】
【公開日】平成19年3月15日(2007.3.15)
【国際特許分類】
【出願日】平成18年8月7日(2006.8.7)
【出願人】(000001007)キヤノン株式会社 (59,756)
【Fターム(参考)】
[ Back to top ]

