糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法及び装置
【課題】糖鎖修飾ペプチドから糖鎖を遊離することなく、糖鎖及びペプチドシーケンス解析を行う。
【解決手段】シアル酸やフコースの脱離が起こりにくい負イオンモードCIDで糖鎖構造解析を行い、正イオンモードECDでペプチドシーケンス解析を行う。
【解決手段】シアル酸やフコースの脱離が起こりにくい負イオンモードCIDで糖鎖構造解析を行い、正イオンモードECDでペプチドシーケンス解析を行う。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、質量分析法を用いた生体関連物質の解析方法や解析システムなどに関し、特に糖鎖修飾タンパク質解析(グライコプロテオミックス)や糖鎖修飾ペプチド解析の方法や解析システムに関する。
【背景技術】
【0002】
タンパク質上に存在する糖鎖は、アスパラギン(N)の側鎖のアミド基がNグリコシル化されたN型糖鎖と、セリン(S)やスレオニン(T)の側鎖のアルコールがグリコシル化されたO型糖鎖に分類される。そして、グライコプロテオミックスでは、糖鎖の種類(構造)、タンパク質の種類、及び、修飾部位の決定が重要視されている。糖鎖修飾タンパク質はトリプシンなどの酵素消化により糖鎖修飾ペプチドにできるので、糖鎖修飾ペプチドのアミノ酸シーケンスが解析できれば、糖鎖修飾タンパク質を決定することができる。しかしながら、実際には、糖鎖の構造の複雑さが解析の障壁になり、困難を極めている。
【0003】
これまで、N型糖鎖で修飾されたペプチドは、酵素消化により糖鎖部分をペプチド部分から遊離させる手法が用いられていた。この手法では、糖鎖は解析できても、元のペプチドやたんぱく質の種類や修飾部位を決定することは困難である。一方、酵素消化により糖鎖を遊離させる際に、ペプチドの修飾部位を同位体標識する方法も考案された。この手法では、タンパク質の種類、及び、修飾部位の決定が可能であるが、構造異性体を含む糖鎖の種類を決定することは困難である。また、O型糖鎖修飾ペプチドの解析においても、糖鎖に化学的な修飾を施す解析方法が検討されているが、ペプチドの解析とは独立しており、関連性が失われる(非特許文献1)。このように、N型糖鎖かO型糖鎖かで、解析手法も異なるのが現状である。
【0004】
一方、糖鎖を遊離させずに、糖鎖修飾タンパク質やペプチドを直接質量分析法により解析する試みも行われている。例えば、特許文献1には、糖鎖修飾ペプチドのプロトン付加あるいはカチオン付加イオンに対して、質量分析装置において衝突誘起解離(CID)によるタンデム質量分析をする解析手法が記載されている。この手法では、MS/MS分析においてペプチド主鎖由来イオンと糖鎖由来イオンが別個のイオンとして検出された場合に、それぞれを別々に更なるタンデム質量分析(MS/MS/MS)することにより、検出イオン情報を単純化させている。その他、糖鎖修飾ペプチドの正イオンに対して、電子捕獲解離(ECD)や赤外多光子解離(IRMPD)、エレクトロントランスファー解離(ETD)をCIDと組み合わせる解析手法が非特許文献2や3に記載されている。また、正イオンのCIDによるタンデム質量分析や負イオンのCIDによるタンデム質量分析データを組み合わせる糖鎖修飾ペプチド解析方法が非特許文献4に記載されている。
【0005】
【特許文献1】特開2005-300420号公報
【非特許文献1】J. Zhang, L. L. Lindsay, J. L. Hedrick. C. B. Lebrilla, Strategy for profiling and structure educidation of mucin-type oligosaccharides by mass spectrometry, Analytical Chemistry Vol. 76 (2004) 5990-6001.
【非特許文献2】K. Hakansson, H. J. Cooper, M. R. Emmett, C. E. Costello, A. G. Marshall, C. L. Nilsson, Electron capture dissociation and infrared multiphoton dissociation MS/MS of an N-glycosylated tryptic peptide to yield complementary sequence information, Analytical Chemistry Vol. 73 (2001) 4530-4536.
【非特許文献3】J. M. Hogan, S. J. Pitteri, P. A. Chrisman, C. A. McLuckey, Complementary structural information from a tryptic N-linked glycopeptide via electron transfer ion/ion reactions and collision-induced-dissociation, Journal of Proteome Research Vol. 4 (2005) 628-632.
【非特許文献4】K. Deguchi, H. Ito, Y. Takegawa, N. Shinji, H. Nakagawa, S. Nishimura, Complementary structural information of positive- and negative-ion MSn spectra of glycopeptides with neutral and sialylated N-glycans, Rapid Communications in Mass Spectrometry Vol. 20 (2006) 741-446.
【発明の開示】
【発明が解決しようとする課題】
【0006】
特許文献1の方法では、分岐構造のある未知の糖鎖の構造を決定することは困難である。非特許文献2や3の解析方法の場合、ECDとETDはほぼ同等の機能を有し、そのタンデム質量分析はペプチドの解析に有利であることが知られるが、修飾部位は解離しないため糖鎖の解析は困難である。一方、CIDやそれと同等の機能を有するIRMPDによるタンデム質量分析では、糖鎖に含まれるシアル酸やフコースは、正イオンモードでは容易に脱離するため、糖鎖構造解析が困難である。非特許文献4の手法では、N型糖鎖が修飾されている場合に解析が可能であるが、O型糖鎖が修飾されている場合には解析が困難である。
【0007】
糖鎖修飾ペプチドの解析では、糖鎖の種類(構造)、タンパク質の種類、及び、修飾部位を決定することが重要である。本発明の目的は、糖鎖修飾ペプチドから糖鎖を遊離させることなく、糖鎖及びペプチドシーケンス解析を行うことにある。
【課題を解決するための手段】
【0008】
本発明は、シアル酸やフコースの脱離が起こりにくい負イオンモードCIDタンデム質量分析(MS/MS、MS/MS/MSなど)により糖鎖構造解析を行い、正イオンモードECDタンデム質量分析(MS/MS)によりペプチドシーケンス解析を行うことにより、上記課題を解決する。
【発明の効果】
【0009】
本発明によると、O型糖鎖修飾ペプチドとN型糖鎖修飾ペプチドとの両方に対して、解析が可能になる。すなわち、同一の解析手順により、糖鎖の種類(構造)、タンパク質の種類、及び、修飾部位を決定することが可能となる。さらに、糖ペプチドから糖鎖を遊離することなく、質量分析装置のみで糖ペプチド構造解析を行うため、解析作業の負荷が低減する。
【発明を実施するための最良の形態】
【0010】
以下、図面を参照して本発明の実施の形態を説明する。
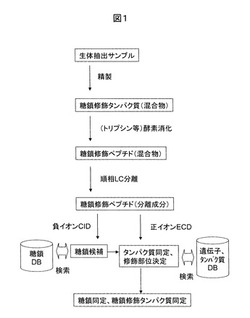
図1に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法の解析手順を示す。血液など、生体から抽出された糖鎖修飾タンパク質(混合物)を解析する場合には、生体抽出サンプルは、予めレクチンなどのアフィニティー精製を実施し、さらにトリプシンなどの酵素消化により、糖鎖修飾ペプチド(混合物)に調製される。
【0011】
糖鎖修飾ペプチド(混合物)は、順相の液体クロマトグラフなどにより分離される。液体クロマトグラフ/質量分析装置(LC/MS)を使用する場合には、分離成分は順次質量分析装置のイオン源に導入される。
【0012】
質量分析装置のイオン分析では、糖鎖修飾ペプチド由来の負イオンを分析し、さらに正イオンを分析する。正負イオン分析モードを高速で切り替え可能な質量分析装置を使用する場合には、1回の分析でデータ取得は完了する。それが困難な質量分析装置を使用する場合には、負イオン分析モードでの分析と正イオン分析モードでの分析の2回の分析を実施する必要がある。
【0013】
負イオン分析モードによる分析では、糖鎖修飾ペプチド由来の負イオンに対し、衝突誘起解離(CID)によるMS/MSを実施する。ここで、MS/MSを実施しても充分なフラグメントイオン情報を持つデータが期待できるイオン強度の前駆イオンがあれば、できるだけ高い荷数のイオンを前駆イオンに選択することが望ましい。得られるMS/MS分析データ(スペクトル)において、豊富なフラグメントイオン情報が期待できるからである。次に、糖鎖修飾ペプチド検出の検知には、MS/MS分析データで検出されるフラグメントイオンの質量を計算し、単糖のグリコシド結合だけで構成される糖鎖イオンで最大の質量を有するイオンの検出の有無を調べる(計算方法は後述する)。そして、糖鎖修飾ペプチド検出の検知が確認されると、単糖のグリコシド結合だけで構成される糖鎖イオンで最大の質量を有するイオンを前駆イオンに選択し、MS/MS/MS分析を実施する。これらの計算は、分析実時間で実施する必要がある。
【0014】
負イオン分析モードによるデータ取得が終了後、糖鎖修飾ペプチドであることが確認された同一成分に対し、イオン源で正イオンを生成する。そして、そのイオンを前駆イオンとする電子捕獲解離(ECD)又は電子移動解離(ETD)によるMS/MSを実施する。糖鎖ペプチドの正イオンは、[M+H]+や[M+2H]2+のようなプロトン化分子だけでなく、[M+Na]+や[M+2Na]2+、[M+Na+H]2+などのカチオン付加したイオンも検出される点に注意が必要である。
【0015】
上記の説明は、LC/MSを用いたオンライン分析の例であるが、LCの分離成分を分画したサンプルを用いたオフライン分析でもデータ取得は可能である。この場合は、負イオンのMS/MS/MS分析における前駆イオン選択に関する計算は分析実時間で実施する必要はない。
【0016】
さて、様々な糖鎖の標準品に対して、予め負イオンのMS/MS分析データやMS/MS/MS分析データを取得しておき、糖鎖のデータベースを作成しておく。このデータベースには、上記データのみならず、対応する前駆イオンの質量(m)や電荷数(z)、あるいは、m/zがセットになって登録される。
【0017】
実際の分析で得られた負イオンMS/MS/MS分析データは、標準品の分析データが登録された上記データベースに対して検索が実施され、分析データのプロファイルマッチングなどにより構造を含む糖鎖(候補)が決定される。ここで重要な点は、別途取得された標準品のデータと分析データとを照合する点であり、マニュアル操作で照合可能ならば標準品のデータが必ずしもデータベース化されている必要はない。
【0018】
一方、正イオン分析データには、通常のプロテオーム解析で利用されるように、遺伝子あるいはタンパク質のデータベースに対してデータベース検索を実施する。ここでは、先に決定された糖鎖(候補)の質量情報をデータベース検索に反映することが重要である。このことにより、タンパク質の同定や糖鎖修飾部位が決定される。さらに、糖鎖が決定される。
【0019】
本手法は、タンパク質を修飾する糖鎖がO型糖鎖でもN型糖鎖でも同一の手順で解析することができるので、未知サンプルの分析にも非常に有効である。
【0020】
図2に、分析データに対する解析手順の具体例を示す。先ず、負イオンのMS/MSやMS/MS/MS分析データを糖鎖のデータベースで検索する(S1)。そして、糖鎖の同定候補を決定する(S2)。この段階では、糖鎖のラフな同定しかできない。即ち、N型糖鎖の場合は、CIDのプロセスにおいてGlcNAc(Nアセチルグルコサミン)やそれにFuc(フコース)が結合した部位が欠落する可能性があるためである。一方、O型では欠落が発生する可能性は極めて低い。こうして決定された候補糖鎖の分子量情報が、次の遺伝子やタンパク質のデータベース検索条件に反映される(S3)。即ち、修飾部位のアミノ酸候補は、N型糖鎖の場合にはアスパラギンだけであり、O型の場合にはセリンとスレオニンである。これらのアミノ酸の分子量情報に糖鎖修飾した場合の質量情報を追加し、正イオンECDデータを呼び出し(S4)、タンパク質や遺伝子のデータベース検索を実施する(S5)。このデータベース検索には、Matrix Science社Mascotなどの検索ソフトが利用可能である。さらに、アミノ酸配列を解読するデノボ解析ソフトも利用可能である。
【0021】
例えば、遺伝子のデータベース検索を実施する場合、遺伝子のデータベースには遺伝子の塩基配列情報と対応するタンパク質情報しか含まれない。そこで、データベース検索ソフトでは、3塩基で1アミノ酸をコードする関係を利用し、アミノ酸配列のデータに変換する。さらに、図1に示すように、トリプシンなどの消化酵素を用いてタンパク質をペプチド化した場合、ペプチドのC末端にあるアミノ酸はリジンかアルギニンなど特定のアミノ酸である。この酵素消化の選択性を利用し、理論上生成されるペプチドのアミノ酸配列を全て計算する。そして、ペプチドのイオンがプロトン付加イオンであり、そのフラグメントイオンはペプチド結合の部位で解離すると仮定すると、アミノ酸の分子量を用いてフラグメントイオンのm/z値が計算でき、仮想的なMS/MSデータを構築することができる。特に、糖鎖修飾されるアミノ酸は、糖鎖が修飾されている分だけ本来の質量より質量が増加するとして計算することができる。上記計算により、発生しうる全てのケースにおけるMS/MSデータが計算でき、実測されたMS/MSデータと相関などの手法による照合を行うことにより、元のタンパク質やペプチドが同定され、さらに、糖鎖修飾部位のアミノ酸を決定することができる。タンパク質のデータベースを用いる場合には、データベースに含まれる情報がタンパク質のアミノ酸配列情報である点が異なるのみで、以降の処理は同様である。
【0022】
実際のデータベース検索の対象となるデータは、先の負イオン分析データに対応した同一糖鎖修飾ペプチドのものである必要があり、ステップ4ではLC保持時間情報や前駆イオン情報を照合し確認することが必要である。ECDあるいはETDの分析データに対し、データベース検索を実施すると、タンパク質の同定、及び、糖鎖修飾部位が決定される(S6)。アスパラギンが修飾部位のアミノ酸である場合には、N型糖鎖修飾部位周辺の特徴であるコンセンサス配列(N-X-S/T)の確認が必要である。ここで、Nはアスパラギン、Xはプロリン以外のアミノ酸、S/Tはセリンあるいはスレオニンである。最後に、修飾糖鎖部位の分子量に基づき、糖鎖候補から糖鎖を決定することができる(S7)。
【0023】
図3に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法で使用する典型的なLC/MSの模式図を示す。先ず、質量分析装置に要求される必要条件について述べる。質量分析装置は、正負イオン分析を実施可能であり、負イオン分析モードでは、CIDによるMS/MS分析ができることが必要であり、MS/MS/MS分析などのMSn分析が可能であることが望ましい。一方、正イオン分析モードでは、ECDあるいはETDによるMS/MS分析ができることが必要である。さらに、質量分析装置は、1秒程度で周期的に正負イオン分析モードが切り替わる機能を有することが望ましい。
【0024】
図3に示す質量分析装置の真空部では、イオンの質量を飛行時間型質量分析計により高精度に決定でき、負イオンのCIDによるMS/MS分析やMS/MS/MS分析は四重極(高周波)イオントラップで実施、正イオンのECDによるMS/MS分析はECD反応セルで実施する構成である。実質的にECD反応セルは反応時間が要求されるため四重極(高周波)イオントラップから構成されることが多いので、同一のセルでCIDとECDを実施しても構わない。このことは、ETDでも同様である。また、高速で正負イオン分析モード切り替えを実施する場合には、特に負イオンのMS/MS/MS分析における前駆イオン選択が重要である。MS/MS分析データに検出されるフラグメントイオン全てを前駆イオンとし、非常に高速でMS/MS/MS分析データを取得することができれば問題ない。しかし、困難が予測される場合には、分析実時間において、単糖のグリコシド結合だけで構成される糖鎖で最大の質量を有するイオンを前駆イオンに選択する機能が必要である。単糖のグリコシド結合だけで構成される糖鎖のイオンのm/zの計算には、以下に示す単糖の分子量を用い、グリコシド結合一箇所あたり分子量が18だけ減少することを考慮し、分子量Mを求める。さらに18だけ分子量を減少させる場合も考慮が必要である。次に、糖鎖ペプチドや糖鎖の負イオンは、[M-H]-あるいは[M-2H]2-で表されるように脱プロトン化分子なので、一価イオンならばm/z=M-1、二価イオンならばm/z=(M-2)/2と決定される。イオンの電荷数は、質量分析データのモノアイソトピックピークと同位体ピークとのm/z差を調べることにより判明する。
Hexose (Galactose, Mannose, Glucose) 180.05
HexNAc (N-acetylglucosamine, N-acetylgalactosamine) 221.08
Deoxyhexose (Fucose, rhamanose) 164.06
Pentose (xylose) 150.04
NeuAc (N-acetyl neuraminic acid) 309.10
NeuGc (N-glycolyl neuraminic acid) 325.09
【0025】
正負切り替えによるオンライン分析を実施しない場合には、順相LCなどの分離成分を分画し、シリンジポンプなどを用いて分画成分をイオン源に導入し、負イオン分析、及び正イオン分析を実施すれば問題ない。以下、具体的なデータを用いて、本発明の解析方法を説明する。
【0026】
[O型糖鎖修飾ペプチドの分析例]
図4に、糖鎖修飾ペプチド由来負イオン(脱プロトン化分子)のCID分析データ例を示す。このデータの前駆イオンは、m/z値が954.9の[M-2H]2-である。ここで、Mは糖鎖修飾ペプチド分子を、Hは水素原子を表し、このイオンは2個プロトンが失われた2価イオンである。この前駆イオンをCIDによりMS/MS分析したデータが図4(a)に示されている。図4(a)の右上に糖鎖修飾部の模式図を示す。ここで、BやCはグリコシド結合の部位で解離したイオンを表す。波線はペプチドを表し、ひし形はNeu5Ac(Nアセチルノイラミン酸)、黒丸はGal(ガラクトース)、四角はGlcNAc(Nアセチルグルコサミン)、三角はFuc(フコース)を示す。また、破線は解離部位を示す。
【0027】
図4(a)で、イオンピークのm/z値が、グリコシド結合の数と18の積を上記単糖の分子量の和から引き、さらに脱プロトン化に対応して1引いたものと一致すれば、そのイオンは糖鎖の脱プロトン化分子[M-H]-であることが判明する。この計算方法を適用すると、グリコシド結合の部分で解離したイオンが多く検出され、単糖のグリコシド結合による糖鎖のイオンで最大の質量のものはm/z値が819.3(=309.1+180.05+221.08+164.06−18×3−1)であるC3であることが分かる。糖鎖の検出が確認されたので、このイオンC3を前駆イオンに選択し、CIDによりMS/MS/MS分析を実施して得られたデータが図4(b)である。また、C3とZ0との質量の和は前駆イオンの質量に対応するため、修飾糖鎖はO型であると期待され、Z0は修飾されないペプチドのイオンであると結論される。
【0028】
さて、糖鎖データベースには、フラグメントイオンのm/z値とそのイオンピークの相対的強度、及び、相対的強度のCID電圧(ゲイン)依存性が情報として登録される。また、CID電圧(ゲイン)を変えて取得されたMS/MSスペクトルが登録されていても問題ない。糖鎖データベース検索で、ヒットしたデータを図5に示す(CID電圧(ゲイン)依存性は省略)。これらは、Sialyl Lewis x とSialyl Lewis aの標準品の負イオンMS/MS分析データである。前者のデータには、m/zが655.2のフラグメントイオンが検出されるのに対し、後者では検出されない点が特徴的である。図4(b)と比較すると、前者のデータの方が高い類似性を示す。そのため、糖鎖構造は前者にFuc(フコース)が結合したものと結論される。ただ、図4(a)に示されるMS/MS分析データだけでも、糖鎖データベースを利用して糖鎖構造を決定することは可能であるが、解析は複雑である。
【0029】
図4に示される負イオン分析データに対応する正イオンに対する、ECDのMS/MS分析データを図6に示す。ECDでは、ペプチド結合の特定の部位で解離し、cイオンやzイオンが検出される点が特徴的である。右上に、糖鎖修飾ペプチドの構造、及び、検出イオンを示す。水平方向に並んだアルファベットはアミノ酸配列を示す。このMS/MS分析データに対し、データベース検索を実施した結果、図に示されるように、アミノ酸配列情報が得られる多数のcイオンやzイオンが検出され、元のタンパク質が同定された。さらに、C末端から7つ目のセリン(S)に糖鎖が修飾されていることもデータベース検索の結果から分かり、修飾糖鎖はO型であると結論される。このデータの前駆イオンはm/zが638.3の[M+3H]3+である。ECDにおける解離でも、前駆イオン強度が一定値以上であり、電荷数が高いイオンが前駆イオンに選択されることが、フラグメントイオン情報の観点から望ましい。なお、ECDのMS/MS分析データでは、前駆イオンの電子捕獲再結合により、電荷数だけが低減したイオン([M+3H]2+、[M+3H]+)も検出される。
【0030】
[N型糖鎖修飾ペプチドの分析例]
別の典型的な糖鎖修飾ペプチドに対する解析手順について、図7、8、9を用いて説明する。図7に、糖鎖修飾ペプチド由来負イオンのCID分析データ例を示す。このデータの前駆イオンは、m/z値が1139.1の[M-2H]2-である。この前駆イオンをCIDによりMS/MS分析したデータを図7(a)に示す。図7(a)の右上に、糖鎖修飾部の模式図を示す。黒丸はGal(ガラクトース)、四角はGlcNAc(Nアセチルグルコサミン)、白丸はMan(マンノース)である。単糖のグリコシド結合による糖鎖のイオンで最大の質量のものはm/zが1418.6(≒180.05×2+221.08×3+180.05×3−18×8−1)であるB5である。なお、この糖鎖はN型であるためであるが、上記計算においてグリコシド結合の数に1を加えた数だけ18を引いている。この例では、検出される糖鎖のイオンの種類が比較的少ない。さて、糖鎖が検出されたので、m/zが1418.6のイオンを前駆イオンに選択し、CIDによりMS/MS/MS分析を実施して得られたデータが図7(b)である。なお、A6は解離したGlcNAcがB5に結合したイオンなので、前駆イオンには選択されない。
【0031】
図8(b)に、図7(b)を糖鎖データベースに対して検索した結果を示す。このデータは、200.4と呼ばれる糖鎖を誘導体化の一種であるPA(ピリジルアミン)化したものである。通常、N型糖鎖の標準品はPA化などの誘導体化が施されている。そのため、図8(b)は図8(a)の右上に示される構造の200.4と呼ばれる糖鎖のMS/MS/MS分析データと説明される。さらに、図7(a)に示されるA6の存在により、元の修飾糖鎖は200.4のペプチド側にGlcNAcが結合したものと考えられる。この糖鎖の分子量を用いて、図9に示すECDのMS/MS分析データに対する遺伝子のデータベース検索を実施することにより、元のタンパク質が同定され、さらに、修飾部位も決定される。さらに、修飾糖鎖の分子量から、元の糖鎖は、先に述べた200.4のペプチド側にGlcNAcが結合した構造のものであることが分かる。さらに、この糖鎖はN型であるため、コンセンサス配列(N-X-S/T)を調べると、N-K-Tとなっており矛盾がないことが確認される。これまで述べたように、本発明の解析手法によれば、修飾糖鎖がO型でもN型でも関係なく、同一の解析手順により糖鎖修飾ペプチドの解析ができる。
【0032】
図10に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSのブロック図を示す。本実施例の装置は、液体クロマトグラフ11、液体クロマトグラフ11によって分離された試料が導入されるイオン源12、イオン源によってイオン化された試料が導入されるECDセル13、CIDセル14、質量分析計15、質量分析されたイオンを検出する検出器16、それらを駆動する電源22〜26、検出器16の検出信号を参照して各電源22〜26及び液体クロマトグラフ11を制御する制御部30、及びデータ処理部40を有する。データ処理部40は、糖鎖解析処理部41、ペプチド解析処理部42及び糖鎖修飾ペプチド同定部43を備える。糖鎖解析処理部41は、検出器16から得られた質量分析データと糖鎖データベース51内のデータを照合して糖鎖の同定候補を決定する。ペプチド解析処理部42は、遺伝子、タンパク質データベース52の例えば塩基配列情報をアミノ酸配列のデータに変換し、それからペプチドのアミノ酸配列を計算した上で、糖鎖候補を考慮して仮想的な質量分析データを構築し、そのデータと検出器16から得られた質量分析データとを照合する。糖鎖修飾ペプチド同定部43は、糖鎖解析処理部41によって得られた候補糖鎖の情報を用いてペプチド解析処理部42による処理に修正を加え、また、糖鎖修飾部位の決定を行う。
【0033】
液体クロマトグラフ11で分離された糖鎖修飾ペプチドは、順次イオン源12に導入され、負又は正イオンに変換される。正負イオン分析モードの切り替えは、制御部30からイオン源12やECDセル13、CIDセル14、質量分析計15、検出器16の駆動電源22〜26に信号が送信されることにより実行される。先ず、負イオン分析では、イオン源12で生成された負イオンは、ECDセル13とCIDセル14を素通りし、質量分析計15で質量分析される。そして、選択された前駆イオンに対し、CIDセル14においてCIDを実施し、生成イオン(フラグメントイオン)が質量分析計15で質量分析されることによりMS/MSデータが取得される。そして、単糖のグリコシド結合による糖鎖由来イオンが検出されると、最大の質量のイオンが前駆イオンに選択されMS/MS/MS分析などが実施される。次に、質量分析装置の分析モードは正イオン分析モードに切り替わり、生成される正イオンに対して質量分析データが取得される。さらに、負イオン分析と同一の成分が前駆イオンに選択され、ECDセル13においてECDが実施され、生成イオン(フラグメントイオン)が質量分析計15で質量分析されることによりMS/MS分析が実施される。もし負イオンのCIDによるMS/MSデータに糖鎖由来イオンが検出されない場合、正イオン分析モードに変更する必要はなく、様々な前駆イオンが検出されれば、それらに対して負イオンのCIDによるMS/MSデータ取得を繰り返す。
【0034】
図11に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSなどにおけるMSの分析フローを示す。先ず、負イオンを生成し、負イオンの質量分析データ(質量スペクトル)を取得する(S11)。次に、検出イオンの中から、MS/MS分析の前駆イオンをイオン強度や電荷数、m/zの範囲に応じて選択する(S12)。次に、選択された前駆イオンに対し、CIDによるMS/MS分析データを取得する(S13)。得られたMS/MS分析データに対して、糖鎖イオンの検出の有無について、実時間で評価を行う(S14)。即ち、単糖の質量和からグリコシド結合の数と18の積を減じ、さらに1を引いたm/zを有する一価イオンの有無を調べる。あるいは、さらに18を減じたm/zと一致する一価イオンの有無を調べる。もし、上記計算結果と一致するイオンが検出されない場合(S15,NO)、ステップ11における負イオンの質量分析データ取得に戻る。上記計算結果と一致するイオンが検出される場合には(S15,YES)、最大の質量を有するイオンを前駆イオンとし、負イオンCIDによるMS/MS/MS分析データを取得する(S16)。
【0035】
その後、質量分析装置の分析モードは負イオン分析モードから正イオン分析モードに切り替わり、正イオンの質量分析データ(質量スペクトル)を取得する(S17)。次に、検出イオンの中から、先の負イオンMS/MS分析の前駆イオンに対応する正イオンのECDによるMS/MS分析の前駆イオンを選択する(S18)。負イオンの前駆イオンは[M-nH]n-(ここで、nは自然数)の脱プロトン化分子であるのに対し、正イオンは[M+mH]m+(ここで、mは自然数)であるプロトン化分子、あるいは、[M+Na]+や[M+2Na]2+、[M+Na+H]2+などのカチオン付加分子である。例えば、負イオンが2価イオンの場合には、質量m=2(m/z)+2である。そして、正イオンが3価のプロトン付加分子である場合には、m/z=(m+3)/3となる。イオンの電荷数はイオンピークのモノアイソトピックイオンと同位体イオンとのm/z差を調べることにより判明する。即ち、2価イオンの場合はm/z差は0.5、3価イオンの場合はm/z差は0.33である。正イオンのECDによるMS/MS分析データを取得すると(S19)、質量分析装置の分析モードは正イオン分析モードから負イオン分析モードに切り替わる(S20)。このような分析フローが実現すると、負イオン分析において、糖鎖由来イオンが検出されない限り、正イオン分析モードに変更されない。そのため、負イオン分析モードで検出される糖鎖のイオンに対して効率的に分析データを取得することができる。
【0036】
なお、ECDセルでの解離反応時間は、CIDの解離反応時間よりも長いことがあるため、負イオンのCIDをCIDセルで実施すると同時に、正イオンのECDをECDセルで実施すると、分析スループットが向上する。この場合、先ず、イオン源で負イオンを生成し、CIDセルで負イオンのMS/MSを実施し、分析データを取得する。次に、糖鎖由来イオンが検出されたら、正イオンをイオン源で生成し、質量分析データを取得することにより前駆イオンを決定し、ECDセルで正イオンの解離反応を実施する。それから、イオン源で負イオンを生成し、CIDセルで負イオンのMS/MS/MSを実施する。最後に、ECDセルで生成される正イオンのフラグメントに対するMS/MSデータを取得し、CIDセルで生成される負イオンのフラグメントに対するMS/MS/MSデータを取得する。なお、IRMPDはCIDと同等の効果を有するので、CIDセルの代わりにIRMPDセルとしても問題ない。ECDとETDも同様の効果を示すため、ECDセルの代わりにETDセルとしても問題ない。以下の説明でも同様である。CIDやIRMPDは断熱的解離の例であり、ECDやETDは非断熱的解離の例である。
【0037】
図10の実施例は、LC/MSだが、オフライン分析においても、質量分析装置の装置制御は同等である。しかし、負イオン分析を行った成分に対し、毎回正イオン分析を行っても問題はない。
【0038】
図12に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるMSの分析フローを示す。この例では、正負イオン分析モードが周期的に切り替わる点が特徴的である。先ず、負イオンを生成し、負イオンの質量分析データ(質量スペクトル)を取得する(S21)。次に、検出イオンの中から、MS/MS分析の前駆イオンをイオン強度や電荷数、m/zの範囲に応じて選択する(S22)。次に、選択された前駆イオンに対し、CIDによるMS/MS分析データを取得する(S23)。得られたMS/MS分析データに対して、糖鎖イオンの検出の有無について、実時間で評価を行う(S24)。即ち、単糖の質量和からグリコシド結合の数と18の積を減じ、さらに1を引いたm/zを有する一価イオンの有無を調べる。あるいは、さらに18を減じたm/zと一致する一価イオンの有無を調べる。もし、ステップ25の判定で上記計算結果と一致するイオンが検出されない場合、一定時間経過後、質量分析装置の分析モードは負イオン分析モードから正イオン分析モードに切り替わり、正イオンの質量分析データ(質量スペクトル)を取得する(S27)。次に、検出イオンの中から、先の負イオンMS/MS分析の前駆イオンに対応する正イオンのECDによるMS/MS分析の前駆イオンを選択する(S28)。そのようなイオンが存在しない場合には、ステップ30にて再度負イオン分析モードに戻るまで待機する。
【0039】
一方、もしステップ25の判定で上記計算結果と一致するイオンが検出される場合、最大の質量を有するイオンを前駆イオンとし、負イオンCIDによるMS/MS/MS分析データを取得する(S26)。その後、質量分析装置の分析モードは負イオン分析モードから正イオン分析モードに切り替わり、正イオンの質量分析データ(質量スペクトル)を取得する(S27)。そして、検出イオンの中から、先の負イオンMS/MS分析の前駆イオンに対応する正イオンのECDによるMS/MS分析の前駆イオンを選択し(S28)、MS/MSデータを取得する(S29)。
【0040】
図13は、ECDセルとCIDセルが同一デバイスの場合の装置構成例を示す。先に述べたように、四重極イオントラップを有する構造のセル、すなわちCID/ECDセル17では、CIDもECDも行うことが原理的に可能である。
【0041】
図14は、ECDの代わりにETDを使用する質量分析装置の例を示す。この例では、CID/ETDセル18を用いている。ECDでは、低エネルギー電子を正イオンに再結合することにより、正イオンの解離を実現する。一方、ETDでは、電子親和力の低い物質の負イオンを正イオンに反応させることにより、負イオンの電子が正イオンに移動し、ECDと同様のプロセスにより解離が実現される。ECDもETDも基本的に四重極イオントラップにおいて捕捉されたイオンに対して反応が実行される。また、図10、13、14に示す質量分析装置では、質量分析計がCIDやECDセルと別に存在するが、これは質量分析計が飛行時間型質量分析計やフーリエ変換イオンサイクロトロン共鳴質量分析計、オービトラップなどのフーリエ変換質量分析計の場合に対応している。そのため、CIDセルやECDセル単独で質量分析も実施できる場合には、独立した質量分析計は不要である。
【図面の簡単な説明】
【0042】
【図1】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法の解析手順を示す図。
【図2】分析データに対する解析手順の詳細を示す図。
【図3】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法で使用する典型的なLC/MSの模式図。
【図4】糖鎖修飾ペプチド由来負イオン(脱プロトン化分子)のCID分析データ例を示す図。
【図5】糖鎖データベース検索で、ヒットしたデータを示す図。
【図6】ECDのMS/MS分析データ例を示す図。
【図7】糖鎖修飾ペプチド由来負イオンのCID分析データ例を示す図。
【図8】糖鎖データベースに対して検索した結果を示す図。
【図9】ECDのMS/MS分析データを示す図。
【図10】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSのブロック図。
【図11】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSなどにおけるMSの分析フローを示す図。
【図12】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるMSの分析フローを示す図。
【図13】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSのブロック図。
【図14】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSのブロック図。
【符号の説明】
【0043】
11:液体クロマトグラフ
12:イオン源
13:ECDセル
14:CIDセル
15:質量分析計
16:検出器
30:制御部
40:データ処理部
41:糖鎖解析処理部
42:ペプチド解析処理部
43:糖鎖修飾ペプチド同定部
51:糖鎖データベース
52:遺伝子、タンパク質データベース
【技術分野】
【0001】
本発明は、質量分析法を用いた生体関連物質の解析方法や解析システムなどに関し、特に糖鎖修飾タンパク質解析(グライコプロテオミックス)や糖鎖修飾ペプチド解析の方法や解析システムに関する。
【背景技術】
【0002】
タンパク質上に存在する糖鎖は、アスパラギン(N)の側鎖のアミド基がNグリコシル化されたN型糖鎖と、セリン(S)やスレオニン(T)の側鎖のアルコールがグリコシル化されたO型糖鎖に分類される。そして、グライコプロテオミックスでは、糖鎖の種類(構造)、タンパク質の種類、及び、修飾部位の決定が重要視されている。糖鎖修飾タンパク質はトリプシンなどの酵素消化により糖鎖修飾ペプチドにできるので、糖鎖修飾ペプチドのアミノ酸シーケンスが解析できれば、糖鎖修飾タンパク質を決定することができる。しかしながら、実際には、糖鎖の構造の複雑さが解析の障壁になり、困難を極めている。
【0003】
これまで、N型糖鎖で修飾されたペプチドは、酵素消化により糖鎖部分をペプチド部分から遊離させる手法が用いられていた。この手法では、糖鎖は解析できても、元のペプチドやたんぱく質の種類や修飾部位を決定することは困難である。一方、酵素消化により糖鎖を遊離させる際に、ペプチドの修飾部位を同位体標識する方法も考案された。この手法では、タンパク質の種類、及び、修飾部位の決定が可能であるが、構造異性体を含む糖鎖の種類を決定することは困難である。また、O型糖鎖修飾ペプチドの解析においても、糖鎖に化学的な修飾を施す解析方法が検討されているが、ペプチドの解析とは独立しており、関連性が失われる(非特許文献1)。このように、N型糖鎖かO型糖鎖かで、解析手法も異なるのが現状である。
【0004】
一方、糖鎖を遊離させずに、糖鎖修飾タンパク質やペプチドを直接質量分析法により解析する試みも行われている。例えば、特許文献1には、糖鎖修飾ペプチドのプロトン付加あるいはカチオン付加イオンに対して、質量分析装置において衝突誘起解離(CID)によるタンデム質量分析をする解析手法が記載されている。この手法では、MS/MS分析においてペプチド主鎖由来イオンと糖鎖由来イオンが別個のイオンとして検出された場合に、それぞれを別々に更なるタンデム質量分析(MS/MS/MS)することにより、検出イオン情報を単純化させている。その他、糖鎖修飾ペプチドの正イオンに対して、電子捕獲解離(ECD)や赤外多光子解離(IRMPD)、エレクトロントランスファー解離(ETD)をCIDと組み合わせる解析手法が非特許文献2や3に記載されている。また、正イオンのCIDによるタンデム質量分析や負イオンのCIDによるタンデム質量分析データを組み合わせる糖鎖修飾ペプチド解析方法が非特許文献4に記載されている。
【0005】
【特許文献1】特開2005-300420号公報
【非特許文献1】J. Zhang, L. L. Lindsay, J. L. Hedrick. C. B. Lebrilla, Strategy for profiling and structure educidation of mucin-type oligosaccharides by mass spectrometry, Analytical Chemistry Vol. 76 (2004) 5990-6001.
【非特許文献2】K. Hakansson, H. J. Cooper, M. R. Emmett, C. E. Costello, A. G. Marshall, C. L. Nilsson, Electron capture dissociation and infrared multiphoton dissociation MS/MS of an N-glycosylated tryptic peptide to yield complementary sequence information, Analytical Chemistry Vol. 73 (2001) 4530-4536.
【非特許文献3】J. M. Hogan, S. J. Pitteri, P. A. Chrisman, C. A. McLuckey, Complementary structural information from a tryptic N-linked glycopeptide via electron transfer ion/ion reactions and collision-induced-dissociation, Journal of Proteome Research Vol. 4 (2005) 628-632.
【非特許文献4】K. Deguchi, H. Ito, Y. Takegawa, N. Shinji, H. Nakagawa, S. Nishimura, Complementary structural information of positive- and negative-ion MSn spectra of glycopeptides with neutral and sialylated N-glycans, Rapid Communications in Mass Spectrometry Vol. 20 (2006) 741-446.
【発明の開示】
【発明が解決しようとする課題】
【0006】
特許文献1の方法では、分岐構造のある未知の糖鎖の構造を決定することは困難である。非特許文献2や3の解析方法の場合、ECDとETDはほぼ同等の機能を有し、そのタンデム質量分析はペプチドの解析に有利であることが知られるが、修飾部位は解離しないため糖鎖の解析は困難である。一方、CIDやそれと同等の機能を有するIRMPDによるタンデム質量分析では、糖鎖に含まれるシアル酸やフコースは、正イオンモードでは容易に脱離するため、糖鎖構造解析が困難である。非特許文献4の手法では、N型糖鎖が修飾されている場合に解析が可能であるが、O型糖鎖が修飾されている場合には解析が困難である。
【0007】
糖鎖修飾ペプチドの解析では、糖鎖の種類(構造)、タンパク質の種類、及び、修飾部位を決定することが重要である。本発明の目的は、糖鎖修飾ペプチドから糖鎖を遊離させることなく、糖鎖及びペプチドシーケンス解析を行うことにある。
【課題を解決するための手段】
【0008】
本発明は、シアル酸やフコースの脱離が起こりにくい負イオンモードCIDタンデム質量分析(MS/MS、MS/MS/MSなど)により糖鎖構造解析を行い、正イオンモードECDタンデム質量分析(MS/MS)によりペプチドシーケンス解析を行うことにより、上記課題を解決する。
【発明の効果】
【0009】
本発明によると、O型糖鎖修飾ペプチドとN型糖鎖修飾ペプチドとの両方に対して、解析が可能になる。すなわち、同一の解析手順により、糖鎖の種類(構造)、タンパク質の種類、及び、修飾部位を決定することが可能となる。さらに、糖ペプチドから糖鎖を遊離することなく、質量分析装置のみで糖ペプチド構造解析を行うため、解析作業の負荷が低減する。
【発明を実施するための最良の形態】
【0010】
以下、図面を参照して本発明の実施の形態を説明する。
図1に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法の解析手順を示す。血液など、生体から抽出された糖鎖修飾タンパク質(混合物)を解析する場合には、生体抽出サンプルは、予めレクチンなどのアフィニティー精製を実施し、さらにトリプシンなどの酵素消化により、糖鎖修飾ペプチド(混合物)に調製される。
【0011】
糖鎖修飾ペプチド(混合物)は、順相の液体クロマトグラフなどにより分離される。液体クロマトグラフ/質量分析装置(LC/MS)を使用する場合には、分離成分は順次質量分析装置のイオン源に導入される。
【0012】
質量分析装置のイオン分析では、糖鎖修飾ペプチド由来の負イオンを分析し、さらに正イオンを分析する。正負イオン分析モードを高速で切り替え可能な質量分析装置を使用する場合には、1回の分析でデータ取得は完了する。それが困難な質量分析装置を使用する場合には、負イオン分析モードでの分析と正イオン分析モードでの分析の2回の分析を実施する必要がある。
【0013】
負イオン分析モードによる分析では、糖鎖修飾ペプチド由来の負イオンに対し、衝突誘起解離(CID)によるMS/MSを実施する。ここで、MS/MSを実施しても充分なフラグメントイオン情報を持つデータが期待できるイオン強度の前駆イオンがあれば、できるだけ高い荷数のイオンを前駆イオンに選択することが望ましい。得られるMS/MS分析データ(スペクトル)において、豊富なフラグメントイオン情報が期待できるからである。次に、糖鎖修飾ペプチド検出の検知には、MS/MS分析データで検出されるフラグメントイオンの質量を計算し、単糖のグリコシド結合だけで構成される糖鎖イオンで最大の質量を有するイオンの検出の有無を調べる(計算方法は後述する)。そして、糖鎖修飾ペプチド検出の検知が確認されると、単糖のグリコシド結合だけで構成される糖鎖イオンで最大の質量を有するイオンを前駆イオンに選択し、MS/MS/MS分析を実施する。これらの計算は、分析実時間で実施する必要がある。
【0014】
負イオン分析モードによるデータ取得が終了後、糖鎖修飾ペプチドであることが確認された同一成分に対し、イオン源で正イオンを生成する。そして、そのイオンを前駆イオンとする電子捕獲解離(ECD)又は電子移動解離(ETD)によるMS/MSを実施する。糖鎖ペプチドの正イオンは、[M+H]+や[M+2H]2+のようなプロトン化分子だけでなく、[M+Na]+や[M+2Na]2+、[M+Na+H]2+などのカチオン付加したイオンも検出される点に注意が必要である。
【0015】
上記の説明は、LC/MSを用いたオンライン分析の例であるが、LCの分離成分を分画したサンプルを用いたオフライン分析でもデータ取得は可能である。この場合は、負イオンのMS/MS/MS分析における前駆イオン選択に関する計算は分析実時間で実施する必要はない。
【0016】
さて、様々な糖鎖の標準品に対して、予め負イオンのMS/MS分析データやMS/MS/MS分析データを取得しておき、糖鎖のデータベースを作成しておく。このデータベースには、上記データのみならず、対応する前駆イオンの質量(m)や電荷数(z)、あるいは、m/zがセットになって登録される。
【0017】
実際の分析で得られた負イオンMS/MS/MS分析データは、標準品の分析データが登録された上記データベースに対して検索が実施され、分析データのプロファイルマッチングなどにより構造を含む糖鎖(候補)が決定される。ここで重要な点は、別途取得された標準品のデータと分析データとを照合する点であり、マニュアル操作で照合可能ならば標準品のデータが必ずしもデータベース化されている必要はない。
【0018】
一方、正イオン分析データには、通常のプロテオーム解析で利用されるように、遺伝子あるいはタンパク質のデータベースに対してデータベース検索を実施する。ここでは、先に決定された糖鎖(候補)の質量情報をデータベース検索に反映することが重要である。このことにより、タンパク質の同定や糖鎖修飾部位が決定される。さらに、糖鎖が決定される。
【0019】
本手法は、タンパク質を修飾する糖鎖がO型糖鎖でもN型糖鎖でも同一の手順で解析することができるので、未知サンプルの分析にも非常に有効である。
【0020】
図2に、分析データに対する解析手順の具体例を示す。先ず、負イオンのMS/MSやMS/MS/MS分析データを糖鎖のデータベースで検索する(S1)。そして、糖鎖の同定候補を決定する(S2)。この段階では、糖鎖のラフな同定しかできない。即ち、N型糖鎖の場合は、CIDのプロセスにおいてGlcNAc(Nアセチルグルコサミン)やそれにFuc(フコース)が結合した部位が欠落する可能性があるためである。一方、O型では欠落が発生する可能性は極めて低い。こうして決定された候補糖鎖の分子量情報が、次の遺伝子やタンパク質のデータベース検索条件に反映される(S3)。即ち、修飾部位のアミノ酸候補は、N型糖鎖の場合にはアスパラギンだけであり、O型の場合にはセリンとスレオニンである。これらのアミノ酸の分子量情報に糖鎖修飾した場合の質量情報を追加し、正イオンECDデータを呼び出し(S4)、タンパク質や遺伝子のデータベース検索を実施する(S5)。このデータベース検索には、Matrix Science社Mascotなどの検索ソフトが利用可能である。さらに、アミノ酸配列を解読するデノボ解析ソフトも利用可能である。
【0021】
例えば、遺伝子のデータベース検索を実施する場合、遺伝子のデータベースには遺伝子の塩基配列情報と対応するタンパク質情報しか含まれない。そこで、データベース検索ソフトでは、3塩基で1アミノ酸をコードする関係を利用し、アミノ酸配列のデータに変換する。さらに、図1に示すように、トリプシンなどの消化酵素を用いてタンパク質をペプチド化した場合、ペプチドのC末端にあるアミノ酸はリジンかアルギニンなど特定のアミノ酸である。この酵素消化の選択性を利用し、理論上生成されるペプチドのアミノ酸配列を全て計算する。そして、ペプチドのイオンがプロトン付加イオンであり、そのフラグメントイオンはペプチド結合の部位で解離すると仮定すると、アミノ酸の分子量を用いてフラグメントイオンのm/z値が計算でき、仮想的なMS/MSデータを構築することができる。特に、糖鎖修飾されるアミノ酸は、糖鎖が修飾されている分だけ本来の質量より質量が増加するとして計算することができる。上記計算により、発生しうる全てのケースにおけるMS/MSデータが計算でき、実測されたMS/MSデータと相関などの手法による照合を行うことにより、元のタンパク質やペプチドが同定され、さらに、糖鎖修飾部位のアミノ酸を決定することができる。タンパク質のデータベースを用いる場合には、データベースに含まれる情報がタンパク質のアミノ酸配列情報である点が異なるのみで、以降の処理は同様である。
【0022】
実際のデータベース検索の対象となるデータは、先の負イオン分析データに対応した同一糖鎖修飾ペプチドのものである必要があり、ステップ4ではLC保持時間情報や前駆イオン情報を照合し確認することが必要である。ECDあるいはETDの分析データに対し、データベース検索を実施すると、タンパク質の同定、及び、糖鎖修飾部位が決定される(S6)。アスパラギンが修飾部位のアミノ酸である場合には、N型糖鎖修飾部位周辺の特徴であるコンセンサス配列(N-X-S/T)の確認が必要である。ここで、Nはアスパラギン、Xはプロリン以外のアミノ酸、S/Tはセリンあるいはスレオニンである。最後に、修飾糖鎖部位の分子量に基づき、糖鎖候補から糖鎖を決定することができる(S7)。
【0023】
図3に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法で使用する典型的なLC/MSの模式図を示す。先ず、質量分析装置に要求される必要条件について述べる。質量分析装置は、正負イオン分析を実施可能であり、負イオン分析モードでは、CIDによるMS/MS分析ができることが必要であり、MS/MS/MS分析などのMSn分析が可能であることが望ましい。一方、正イオン分析モードでは、ECDあるいはETDによるMS/MS分析ができることが必要である。さらに、質量分析装置は、1秒程度で周期的に正負イオン分析モードが切り替わる機能を有することが望ましい。
【0024】
図3に示す質量分析装置の真空部では、イオンの質量を飛行時間型質量分析計により高精度に決定でき、負イオンのCIDによるMS/MS分析やMS/MS/MS分析は四重極(高周波)イオントラップで実施、正イオンのECDによるMS/MS分析はECD反応セルで実施する構成である。実質的にECD反応セルは反応時間が要求されるため四重極(高周波)イオントラップから構成されることが多いので、同一のセルでCIDとECDを実施しても構わない。このことは、ETDでも同様である。また、高速で正負イオン分析モード切り替えを実施する場合には、特に負イオンのMS/MS/MS分析における前駆イオン選択が重要である。MS/MS分析データに検出されるフラグメントイオン全てを前駆イオンとし、非常に高速でMS/MS/MS分析データを取得することができれば問題ない。しかし、困難が予測される場合には、分析実時間において、単糖のグリコシド結合だけで構成される糖鎖で最大の質量を有するイオンを前駆イオンに選択する機能が必要である。単糖のグリコシド結合だけで構成される糖鎖のイオンのm/zの計算には、以下に示す単糖の分子量を用い、グリコシド結合一箇所あたり分子量が18だけ減少することを考慮し、分子量Mを求める。さらに18だけ分子量を減少させる場合も考慮が必要である。次に、糖鎖ペプチドや糖鎖の負イオンは、[M-H]-あるいは[M-2H]2-で表されるように脱プロトン化分子なので、一価イオンならばm/z=M-1、二価イオンならばm/z=(M-2)/2と決定される。イオンの電荷数は、質量分析データのモノアイソトピックピークと同位体ピークとのm/z差を調べることにより判明する。
Hexose (Galactose, Mannose, Glucose) 180.05
HexNAc (N-acetylglucosamine, N-acetylgalactosamine) 221.08
Deoxyhexose (Fucose, rhamanose) 164.06
Pentose (xylose) 150.04
NeuAc (N-acetyl neuraminic acid) 309.10
NeuGc (N-glycolyl neuraminic acid) 325.09
【0025】
正負切り替えによるオンライン分析を実施しない場合には、順相LCなどの分離成分を分画し、シリンジポンプなどを用いて分画成分をイオン源に導入し、負イオン分析、及び正イオン分析を実施すれば問題ない。以下、具体的なデータを用いて、本発明の解析方法を説明する。
【0026】
[O型糖鎖修飾ペプチドの分析例]
図4に、糖鎖修飾ペプチド由来負イオン(脱プロトン化分子)のCID分析データ例を示す。このデータの前駆イオンは、m/z値が954.9の[M-2H]2-である。ここで、Mは糖鎖修飾ペプチド分子を、Hは水素原子を表し、このイオンは2個プロトンが失われた2価イオンである。この前駆イオンをCIDによりMS/MS分析したデータが図4(a)に示されている。図4(a)の右上に糖鎖修飾部の模式図を示す。ここで、BやCはグリコシド結合の部位で解離したイオンを表す。波線はペプチドを表し、ひし形はNeu5Ac(Nアセチルノイラミン酸)、黒丸はGal(ガラクトース)、四角はGlcNAc(Nアセチルグルコサミン)、三角はFuc(フコース)を示す。また、破線は解離部位を示す。
【0027】
図4(a)で、イオンピークのm/z値が、グリコシド結合の数と18の積を上記単糖の分子量の和から引き、さらに脱プロトン化に対応して1引いたものと一致すれば、そのイオンは糖鎖の脱プロトン化分子[M-H]-であることが判明する。この計算方法を適用すると、グリコシド結合の部分で解離したイオンが多く検出され、単糖のグリコシド結合による糖鎖のイオンで最大の質量のものはm/z値が819.3(=309.1+180.05+221.08+164.06−18×3−1)であるC3であることが分かる。糖鎖の検出が確認されたので、このイオンC3を前駆イオンに選択し、CIDによりMS/MS/MS分析を実施して得られたデータが図4(b)である。また、C3とZ0との質量の和は前駆イオンの質量に対応するため、修飾糖鎖はO型であると期待され、Z0は修飾されないペプチドのイオンであると結論される。
【0028】
さて、糖鎖データベースには、フラグメントイオンのm/z値とそのイオンピークの相対的強度、及び、相対的強度のCID電圧(ゲイン)依存性が情報として登録される。また、CID電圧(ゲイン)を変えて取得されたMS/MSスペクトルが登録されていても問題ない。糖鎖データベース検索で、ヒットしたデータを図5に示す(CID電圧(ゲイン)依存性は省略)。これらは、Sialyl Lewis x とSialyl Lewis aの標準品の負イオンMS/MS分析データである。前者のデータには、m/zが655.2のフラグメントイオンが検出されるのに対し、後者では検出されない点が特徴的である。図4(b)と比較すると、前者のデータの方が高い類似性を示す。そのため、糖鎖構造は前者にFuc(フコース)が結合したものと結論される。ただ、図4(a)に示されるMS/MS分析データだけでも、糖鎖データベースを利用して糖鎖構造を決定することは可能であるが、解析は複雑である。
【0029】
図4に示される負イオン分析データに対応する正イオンに対する、ECDのMS/MS分析データを図6に示す。ECDでは、ペプチド結合の特定の部位で解離し、cイオンやzイオンが検出される点が特徴的である。右上に、糖鎖修飾ペプチドの構造、及び、検出イオンを示す。水平方向に並んだアルファベットはアミノ酸配列を示す。このMS/MS分析データに対し、データベース検索を実施した結果、図に示されるように、アミノ酸配列情報が得られる多数のcイオンやzイオンが検出され、元のタンパク質が同定された。さらに、C末端から7つ目のセリン(S)に糖鎖が修飾されていることもデータベース検索の結果から分かり、修飾糖鎖はO型であると結論される。このデータの前駆イオンはm/zが638.3の[M+3H]3+である。ECDにおける解離でも、前駆イオン強度が一定値以上であり、電荷数が高いイオンが前駆イオンに選択されることが、フラグメントイオン情報の観点から望ましい。なお、ECDのMS/MS分析データでは、前駆イオンの電子捕獲再結合により、電荷数だけが低減したイオン([M+3H]2+、[M+3H]+)も検出される。
【0030】
[N型糖鎖修飾ペプチドの分析例]
別の典型的な糖鎖修飾ペプチドに対する解析手順について、図7、8、9を用いて説明する。図7に、糖鎖修飾ペプチド由来負イオンのCID分析データ例を示す。このデータの前駆イオンは、m/z値が1139.1の[M-2H]2-である。この前駆イオンをCIDによりMS/MS分析したデータを図7(a)に示す。図7(a)の右上に、糖鎖修飾部の模式図を示す。黒丸はGal(ガラクトース)、四角はGlcNAc(Nアセチルグルコサミン)、白丸はMan(マンノース)である。単糖のグリコシド結合による糖鎖のイオンで最大の質量のものはm/zが1418.6(≒180.05×2+221.08×3+180.05×3−18×8−1)であるB5である。なお、この糖鎖はN型であるためであるが、上記計算においてグリコシド結合の数に1を加えた数だけ18を引いている。この例では、検出される糖鎖のイオンの種類が比較的少ない。さて、糖鎖が検出されたので、m/zが1418.6のイオンを前駆イオンに選択し、CIDによりMS/MS/MS分析を実施して得られたデータが図7(b)である。なお、A6は解離したGlcNAcがB5に結合したイオンなので、前駆イオンには選択されない。
【0031】
図8(b)に、図7(b)を糖鎖データベースに対して検索した結果を示す。このデータは、200.4と呼ばれる糖鎖を誘導体化の一種であるPA(ピリジルアミン)化したものである。通常、N型糖鎖の標準品はPA化などの誘導体化が施されている。そのため、図8(b)は図8(a)の右上に示される構造の200.4と呼ばれる糖鎖のMS/MS/MS分析データと説明される。さらに、図7(a)に示されるA6の存在により、元の修飾糖鎖は200.4のペプチド側にGlcNAcが結合したものと考えられる。この糖鎖の分子量を用いて、図9に示すECDのMS/MS分析データに対する遺伝子のデータベース検索を実施することにより、元のタンパク質が同定され、さらに、修飾部位も決定される。さらに、修飾糖鎖の分子量から、元の糖鎖は、先に述べた200.4のペプチド側にGlcNAcが結合した構造のものであることが分かる。さらに、この糖鎖はN型であるため、コンセンサス配列(N-X-S/T)を調べると、N-K-Tとなっており矛盾がないことが確認される。これまで述べたように、本発明の解析手法によれば、修飾糖鎖がO型でもN型でも関係なく、同一の解析手順により糖鎖修飾ペプチドの解析ができる。
【0032】
図10に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSのブロック図を示す。本実施例の装置は、液体クロマトグラフ11、液体クロマトグラフ11によって分離された試料が導入されるイオン源12、イオン源によってイオン化された試料が導入されるECDセル13、CIDセル14、質量分析計15、質量分析されたイオンを検出する検出器16、それらを駆動する電源22〜26、検出器16の検出信号を参照して各電源22〜26及び液体クロマトグラフ11を制御する制御部30、及びデータ処理部40を有する。データ処理部40は、糖鎖解析処理部41、ペプチド解析処理部42及び糖鎖修飾ペプチド同定部43を備える。糖鎖解析処理部41は、検出器16から得られた質量分析データと糖鎖データベース51内のデータを照合して糖鎖の同定候補を決定する。ペプチド解析処理部42は、遺伝子、タンパク質データベース52の例えば塩基配列情報をアミノ酸配列のデータに変換し、それからペプチドのアミノ酸配列を計算した上で、糖鎖候補を考慮して仮想的な質量分析データを構築し、そのデータと検出器16から得られた質量分析データとを照合する。糖鎖修飾ペプチド同定部43は、糖鎖解析処理部41によって得られた候補糖鎖の情報を用いてペプチド解析処理部42による処理に修正を加え、また、糖鎖修飾部位の決定を行う。
【0033】
液体クロマトグラフ11で分離された糖鎖修飾ペプチドは、順次イオン源12に導入され、負又は正イオンに変換される。正負イオン分析モードの切り替えは、制御部30からイオン源12やECDセル13、CIDセル14、質量分析計15、検出器16の駆動電源22〜26に信号が送信されることにより実行される。先ず、負イオン分析では、イオン源12で生成された負イオンは、ECDセル13とCIDセル14を素通りし、質量分析計15で質量分析される。そして、選択された前駆イオンに対し、CIDセル14においてCIDを実施し、生成イオン(フラグメントイオン)が質量分析計15で質量分析されることによりMS/MSデータが取得される。そして、単糖のグリコシド結合による糖鎖由来イオンが検出されると、最大の質量のイオンが前駆イオンに選択されMS/MS/MS分析などが実施される。次に、質量分析装置の分析モードは正イオン分析モードに切り替わり、生成される正イオンに対して質量分析データが取得される。さらに、負イオン分析と同一の成分が前駆イオンに選択され、ECDセル13においてECDが実施され、生成イオン(フラグメントイオン)が質量分析計15で質量分析されることによりMS/MS分析が実施される。もし負イオンのCIDによるMS/MSデータに糖鎖由来イオンが検出されない場合、正イオン分析モードに変更する必要はなく、様々な前駆イオンが検出されれば、それらに対して負イオンのCIDによるMS/MSデータ取得を繰り返す。
【0034】
図11に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSなどにおけるMSの分析フローを示す。先ず、負イオンを生成し、負イオンの質量分析データ(質量スペクトル)を取得する(S11)。次に、検出イオンの中から、MS/MS分析の前駆イオンをイオン強度や電荷数、m/zの範囲に応じて選択する(S12)。次に、選択された前駆イオンに対し、CIDによるMS/MS分析データを取得する(S13)。得られたMS/MS分析データに対して、糖鎖イオンの検出の有無について、実時間で評価を行う(S14)。即ち、単糖の質量和からグリコシド結合の数と18の積を減じ、さらに1を引いたm/zを有する一価イオンの有無を調べる。あるいは、さらに18を減じたm/zと一致する一価イオンの有無を調べる。もし、上記計算結果と一致するイオンが検出されない場合(S15,NO)、ステップ11における負イオンの質量分析データ取得に戻る。上記計算結果と一致するイオンが検出される場合には(S15,YES)、最大の質量を有するイオンを前駆イオンとし、負イオンCIDによるMS/MS/MS分析データを取得する(S16)。
【0035】
その後、質量分析装置の分析モードは負イオン分析モードから正イオン分析モードに切り替わり、正イオンの質量分析データ(質量スペクトル)を取得する(S17)。次に、検出イオンの中から、先の負イオンMS/MS分析の前駆イオンに対応する正イオンのECDによるMS/MS分析の前駆イオンを選択する(S18)。負イオンの前駆イオンは[M-nH]n-(ここで、nは自然数)の脱プロトン化分子であるのに対し、正イオンは[M+mH]m+(ここで、mは自然数)であるプロトン化分子、あるいは、[M+Na]+や[M+2Na]2+、[M+Na+H]2+などのカチオン付加分子である。例えば、負イオンが2価イオンの場合には、質量m=2(m/z)+2である。そして、正イオンが3価のプロトン付加分子である場合には、m/z=(m+3)/3となる。イオンの電荷数はイオンピークのモノアイソトピックイオンと同位体イオンとのm/z差を調べることにより判明する。即ち、2価イオンの場合はm/z差は0.5、3価イオンの場合はm/z差は0.33である。正イオンのECDによるMS/MS分析データを取得すると(S19)、質量分析装置の分析モードは正イオン分析モードから負イオン分析モードに切り替わる(S20)。このような分析フローが実現すると、負イオン分析において、糖鎖由来イオンが検出されない限り、正イオン分析モードに変更されない。そのため、負イオン分析モードで検出される糖鎖のイオンに対して効率的に分析データを取得することができる。
【0036】
なお、ECDセルでの解離反応時間は、CIDの解離反応時間よりも長いことがあるため、負イオンのCIDをCIDセルで実施すると同時に、正イオンのECDをECDセルで実施すると、分析スループットが向上する。この場合、先ず、イオン源で負イオンを生成し、CIDセルで負イオンのMS/MSを実施し、分析データを取得する。次に、糖鎖由来イオンが検出されたら、正イオンをイオン源で生成し、質量分析データを取得することにより前駆イオンを決定し、ECDセルで正イオンの解離反応を実施する。それから、イオン源で負イオンを生成し、CIDセルで負イオンのMS/MS/MSを実施する。最後に、ECDセルで生成される正イオンのフラグメントに対するMS/MSデータを取得し、CIDセルで生成される負イオンのフラグメントに対するMS/MS/MSデータを取得する。なお、IRMPDはCIDと同等の効果を有するので、CIDセルの代わりにIRMPDセルとしても問題ない。ECDとETDも同様の効果を示すため、ECDセルの代わりにETDセルとしても問題ない。以下の説明でも同様である。CIDやIRMPDは断熱的解離の例であり、ECDやETDは非断熱的解離の例である。
【0037】
図10の実施例は、LC/MSだが、オフライン分析においても、質量分析装置の装置制御は同等である。しかし、負イオン分析を行った成分に対し、毎回正イオン分析を行っても問題はない。
【0038】
図12に、本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるMSの分析フローを示す。この例では、正負イオン分析モードが周期的に切り替わる点が特徴的である。先ず、負イオンを生成し、負イオンの質量分析データ(質量スペクトル)を取得する(S21)。次に、検出イオンの中から、MS/MS分析の前駆イオンをイオン強度や電荷数、m/zの範囲に応じて選択する(S22)。次に、選択された前駆イオンに対し、CIDによるMS/MS分析データを取得する(S23)。得られたMS/MS分析データに対して、糖鎖イオンの検出の有無について、実時間で評価を行う(S24)。即ち、単糖の質量和からグリコシド結合の数と18の積を減じ、さらに1を引いたm/zを有する一価イオンの有無を調べる。あるいは、さらに18を減じたm/zと一致する一価イオンの有無を調べる。もし、ステップ25の判定で上記計算結果と一致するイオンが検出されない場合、一定時間経過後、質量分析装置の分析モードは負イオン分析モードから正イオン分析モードに切り替わり、正イオンの質量分析データ(質量スペクトル)を取得する(S27)。次に、検出イオンの中から、先の負イオンMS/MS分析の前駆イオンに対応する正イオンのECDによるMS/MS分析の前駆イオンを選択する(S28)。そのようなイオンが存在しない場合には、ステップ30にて再度負イオン分析モードに戻るまで待機する。
【0039】
一方、もしステップ25の判定で上記計算結果と一致するイオンが検出される場合、最大の質量を有するイオンを前駆イオンとし、負イオンCIDによるMS/MS/MS分析データを取得する(S26)。その後、質量分析装置の分析モードは負イオン分析モードから正イオン分析モードに切り替わり、正イオンの質量分析データ(質量スペクトル)を取得する(S27)。そして、検出イオンの中から、先の負イオンMS/MS分析の前駆イオンに対応する正イオンのECDによるMS/MS分析の前駆イオンを選択し(S28)、MS/MSデータを取得する(S29)。
【0040】
図13は、ECDセルとCIDセルが同一デバイスの場合の装置構成例を示す。先に述べたように、四重極イオントラップを有する構造のセル、すなわちCID/ECDセル17では、CIDもECDも行うことが原理的に可能である。
【0041】
図14は、ECDの代わりにETDを使用する質量分析装置の例を示す。この例では、CID/ETDセル18を用いている。ECDでは、低エネルギー電子を正イオンに再結合することにより、正イオンの解離を実現する。一方、ETDでは、電子親和力の低い物質の負イオンを正イオンに反応させることにより、負イオンの電子が正イオンに移動し、ECDと同様のプロセスにより解離が実現される。ECDもETDも基本的に四重極イオントラップにおいて捕捉されたイオンに対して反応が実行される。また、図10、13、14に示す質量分析装置では、質量分析計がCIDやECDセルと別に存在するが、これは質量分析計が飛行時間型質量分析計やフーリエ変換イオンサイクロトロン共鳴質量分析計、オービトラップなどのフーリエ変換質量分析計の場合に対応している。そのため、CIDセルやECDセル単独で質量分析も実施できる場合には、独立した質量分析計は不要である。
【図面の簡単な説明】
【0042】
【図1】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法の解析手順を示す図。
【図2】分析データに対する解析手順の詳細を示す図。
【図3】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法で使用する典型的なLC/MSの模式図。
【図4】糖鎖修飾ペプチド由来負イオン(脱プロトン化分子)のCID分析データ例を示す図。
【図5】糖鎖データベース検索で、ヒットしたデータを示す図。
【図6】ECDのMS/MS分析データ例を示す図。
【図7】糖鎖修飾ペプチド由来負イオンのCID分析データ例を示す図。
【図8】糖鎖データベースに対して検索した結果を示す図。
【図9】ECDのMS/MS分析データを示す図。
【図10】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSのブロック図。
【図11】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSなどにおけるMSの分析フローを示す図。
【図12】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるMSの分析フローを示す図。
【図13】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSのブロック図。
【図14】本発明の一実施例に基づく糖鎖修飾ペプチド構造解析法に用いられるLC/MSのブロック図。
【符号の説明】
【0043】
11:液体クロマトグラフ
12:イオン源
13:ECDセル
14:CIDセル
15:質量分析計
16:検出器
30:制御部
40:データ処理部
41:糖鎖解析処理部
42:ペプチド解析処理部
43:糖鎖修飾ペプチド同定部
51:糖鎖データベース
52:遺伝子、タンパク質データベース
【特許請求の範囲】
【請求項1】
試料を負イオンにイオン化する工程と、
前記負イオンの衝突誘起解離(CID)あるいは赤外多光子解離(IRMPD)によるタンデム質量分析を行う工程と、
試料を正イオンにイオン化する工程と、
前記正イオンの電子捕獲解離(ECD)あるいは電子移動解離(ETD)によるタンデム質量分析を行う工程と、
前記CID又はIRMPDによるタンデム質量分析により取得したスペクトルデータから、該当する糖鎖情報を抽出する工程と、
前記ECD又はETDモードによるタンデム質量分析により取得したスペクトルデータから、該当するタンパク質又はペプチドと修飾部位情報を抽出する工程と、
抽出された前記タンパク質又はペプチドと修飾部位情報により前記試料の同定をする工程と
を有することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項2】
請求項1に記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、
前記CID又はIRMPDによるタンデム質量分析により取得したスペクトルデータから、糖鎖構造に基づくスペクトルパターン情報を格納した第1のデータベースを検索し、該当する糖鎖情報を抽出する工程を有し、
前記ECD又はETDモードにより取得したスペクトルデータから、抽出された前記糖鎖情報を適用して、アミノ酸配列情報を格納した第2のデータベースを検索し、該当するタンパク質又はペプチドと修飾部位情報を抽出する工程を有することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項3】
請求項1に記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、前記CID又はIRMPDによるタンデム質量分析により取得したスペクトルデータを解析することにより、糖鎖修飾ペプチドの検出を確認することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項4】
請求項1に記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、前記糖鎖情報を抽出する工程では、糖鎖の修飾部位毎の候補を情報として抽出することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項5】
請求項1に記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、試料を順相液体クロマトグラフにより分離する工程を有することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項6】
試料の負イオンの質量分析データを取得する工程と、
前記工程で検出された負イオンを前駆イオンとしてCID又はIRMPDによるMS/MS分析データを取得する工程と、
前記MS/MS分析データをもとに糖鎖イオンの検出の有無を判定する工程と、
検出された最大の質量を有する糖鎖イオンを前駆イオンとして負イオンCID又はIRMPDによってMS/MS/MS分析データを取得する工程と、
試料の正イオンの質量分析データを取得する工程と、
前記負イオンMS/MS分析の前駆イオンに対応する正イオンのECD又はETDによるMS/MS分析データを取得する工程と
を有することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項7】
請求項6記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、前記MS/MS/MS分析データを既知の糖鎖のデータベースで検索して糖鎖の候補を決定し、前記正イオンのMS/MS分析データに前記糖鎖の候補の情報を加味してタンパク質あるいは遺伝子のデータベースで検索して試料中のタンパク質又はペプチドの同定と糖鎖修飾部位を決定することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項8】
イオン源と、CIDセルあるいはIRMPDセルと、ECDセルあるいはETDセルと、質量分析計と、前記質量分析計で質量分析されたイオンを検出する検出器と、装置各部を制御する制御部と、前記検出器で検出された質量分析データを処理するデータ処理部とを備え、
前記データ処理部は、前記質量分析データを既知の糖鎖の質量分析データと照合する糖鎖解析処理部と、前記質量分析データを既知の遺伝子あるいはタンパク質のデータと照合するペプチド解析処理部と、前記糖鎖解析処理部の出力と前記ペプチド解析処理部の出力を統合する糖鎖修飾ペプチド同定部とを有し、
試料の負イオンの質量分析データを取得し、検出された負イオンを前駆イオンとしてCID又はIRMPDによるMS/MS分析データを取得し、糖鎖イオンの検出の有無を判定し、糖鎖イオンが検出されたとき最大の質量を有する糖鎖イオンを前駆イオンとして負イオンCID又はIRMPDによってMS/MS/MS分析データを取得し、
次に、試料の正イオンの質量分析データを取得し、前記負イオンMS/MS分析の前駆イオンに対応する正イオンのECD又はETDによるMS/MS分析データを取得することを特徴とする糖鎖修飾タンパク質同定装置。
【請求項1】
試料を負イオンにイオン化する工程と、
前記負イオンの衝突誘起解離(CID)あるいは赤外多光子解離(IRMPD)によるタンデム質量分析を行う工程と、
試料を正イオンにイオン化する工程と、
前記正イオンの電子捕獲解離(ECD)あるいは電子移動解離(ETD)によるタンデム質量分析を行う工程と、
前記CID又はIRMPDによるタンデム質量分析により取得したスペクトルデータから、該当する糖鎖情報を抽出する工程と、
前記ECD又はETDモードによるタンデム質量分析により取得したスペクトルデータから、該当するタンパク質又はペプチドと修飾部位情報を抽出する工程と、
抽出された前記タンパク質又はペプチドと修飾部位情報により前記試料の同定をする工程と
を有することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項2】
請求項1に記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、
前記CID又はIRMPDによるタンデム質量分析により取得したスペクトルデータから、糖鎖構造に基づくスペクトルパターン情報を格納した第1のデータベースを検索し、該当する糖鎖情報を抽出する工程を有し、
前記ECD又はETDモードにより取得したスペクトルデータから、抽出された前記糖鎖情報を適用して、アミノ酸配列情報を格納した第2のデータベースを検索し、該当するタンパク質又はペプチドと修飾部位情報を抽出する工程を有することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項3】
請求項1に記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、前記CID又はIRMPDによるタンデム質量分析により取得したスペクトルデータを解析することにより、糖鎖修飾ペプチドの検出を確認することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項4】
請求項1に記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、前記糖鎖情報を抽出する工程では、糖鎖の修飾部位毎の候補を情報として抽出することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項5】
請求項1に記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、試料を順相液体クロマトグラフにより分離する工程を有することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項6】
試料の負イオンの質量分析データを取得する工程と、
前記工程で検出された負イオンを前駆イオンとしてCID又はIRMPDによるMS/MS分析データを取得する工程と、
前記MS/MS分析データをもとに糖鎖イオンの検出の有無を判定する工程と、
検出された最大の質量を有する糖鎖イオンを前駆イオンとして負イオンCID又はIRMPDによってMS/MS/MS分析データを取得する工程と、
試料の正イオンの質量分析データを取得する工程と、
前記負イオンMS/MS分析の前駆イオンに対応する正イオンのECD又はETDによるMS/MS分析データを取得する工程と
を有することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項7】
請求項6記載の糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法において、前記MS/MS/MS分析データを既知の糖鎖のデータベースで検索して糖鎖の候補を決定し、前記正イオンのMS/MS分析データに前記糖鎖の候補の情報を加味してタンパク質あるいは遺伝子のデータベースで検索して試料中のタンパク質又はペプチドの同定と糖鎖修飾部位を決定することを特徴とする糖鎖修飾タンパク質又は糖鎖修飾ペプチド同定方法。
【請求項8】
イオン源と、CIDセルあるいはIRMPDセルと、ECDセルあるいはETDセルと、質量分析計と、前記質量分析計で質量分析されたイオンを検出する検出器と、装置各部を制御する制御部と、前記検出器で検出された質量分析データを処理するデータ処理部とを備え、
前記データ処理部は、前記質量分析データを既知の糖鎖の質量分析データと照合する糖鎖解析処理部と、前記質量分析データを既知の遺伝子あるいはタンパク質のデータと照合するペプチド解析処理部と、前記糖鎖解析処理部の出力と前記ペプチド解析処理部の出力を統合する糖鎖修飾ペプチド同定部とを有し、
試料の負イオンの質量分析データを取得し、検出された負イオンを前駆イオンとしてCID又はIRMPDによるMS/MS分析データを取得し、糖鎖イオンの検出の有無を判定し、糖鎖イオンが検出されたとき最大の質量を有する糖鎖イオンを前駆イオンとして負イオンCID又はIRMPDによってMS/MS/MS分析データを取得し、
次に、試料の正イオンの質量分析データを取得し、前記負イオンMS/MS分析の前駆イオンに対応する正イオンのECD又はETDによるMS/MS分析データを取得することを特徴とする糖鎖修飾タンパク質同定装置。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【公開番号】特開2008−51552(P2008−51552A)
【公開日】平成20年3月6日(2008.3.6)
【国際特許分類】
【出願番号】特願2006−225715(P2006−225715)
【出願日】平成18年8月22日(2006.8.22)
【出願人】(501387839)株式会社日立ハイテクノロジーズ (4,325)
【Fターム(参考)】
【公開日】平成20年3月6日(2008.3.6)
【国際特許分類】
【出願日】平成18年8月22日(2006.8.22)
【出願人】(501387839)株式会社日立ハイテクノロジーズ (4,325)
【Fターム(参考)】
[ Back to top ]

