胃癌の再発を判定する方法
【課題】簡便かつ精度良く胃癌の再発を判定できる実用的な方法を提供する。
【解決手段】胃癌試料中に含まれる胃癌細胞に特異的なマーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(PCR)により増幅するとともに標識して前記マーカー遺伝子の標識断片を取得するステップを含み、陰性コントロールプローブが固定された位置で測定された標識強度を基準として、マーカー遺伝子検出用プローブが固定された位置で測定された標識強度の結果が、所定の閾値を超えたとき、胃癌試料が陽性であると判定するステップを含む。
【解決手段】胃癌試料中に含まれる胃癌細胞に特異的なマーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(PCR)により増幅するとともに標識して前記マーカー遺伝子の標識断片を取得するステップを含み、陰性コントロールプローブが固定された位置で測定された標識強度を基準として、マーカー遺伝子検出用プローブが固定された位置で測定された標識強度の結果が、所定の閾値を超えたとき、胃癌試料が陽性であると判定するステップを含む。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、胃癌の再発を判定する方法、及び、これに用いる胃癌再発判定用キットに関する。
【背景技術】
【0002】
近年、DNAチップ技術を用いた、細胞診に換わる再発予測法が開発されている。
【0003】
特許文献1には、TFF1、TFF2、FABP1、CK20、MUC2、CEA、TACSTD1、MASPIN、並びにACTBの合計9種類の遺伝子の発現レベルの測定に基づき、胃癌の再発予測を行うことが記載されている。特許文献1の技術によれば、対象の癌患者から採取した試料を利用して対象の遺伝子の一部の領域をPCRにより増幅し、得られたPCR増幅物と、合計9種類のプローブが固相上に配置されているアレイとをハイブリダイゼーションさせて、得られるアレイの結果から、均一で正確な胃癌の再発予測のためのデータ取得が可能になるとされている。
【0004】
また、非特許文献1には、CK20、FABP1、TFF1及びMASPINについてのマイクロアレイアッセイと免疫細胞化学的な結果が非常に一致していたことが記載されている。
【先行技術文献】
【特許文献】
【0005】
【特許文献1】特開2006−223303号公報
【非特許文献】
【0006】
【非特許文献1】Annals of Surgical Oncology,14,1694−1702,2007
【発明の概要】
【発明が解決しようとする課題】
【0007】
しかしながら、上記文献の技術では、カットオフ値を定めるため、検査対象検体と同時に早期胃癌検体を用いた測定を行う必要があった。
【0008】
例えば、特許文献1の技術では、早期胃癌症例39例の最大輝度値を判定値としてこの判定値を上回る輝度値が得られた遺伝子について、「有意な発現があった」と判断している。
【0009】
また、非特許文献1の技術では、早期癌サンプル39例のMAX SD(maximum value plus standard deviation)またはAVG 2SD(average value plus twice or more positive markers)をカットオフ値として設定し、結果として後者が有効であることを示している。
【0010】
したがって、上記文献の技術では、40例近くの早期胃癌の症例を検査ごとに使用しなければ的確な予後再発の判定を行うことが困難であり、実用的な方法ではでないという問題があった。
【0011】
本発明は上記事情に鑑みてなされたものであり、その目的とするところは、検査対象検体と同時に早期胃癌検体を用いた測定を行わなくても、正確に胃癌の再発を判定できる、簡便かつ実用体な方法を提供することにある。
【課題を解決するための手段】
【0012】
本発明によれば、
胃癌試料中に含まれる胃癌細胞に特異的なマーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(Polymerase Chain Reaction,PCR)により増幅するとともに標識して前記マーカー遺伝子の標識断片を取得するステップと、
前記マーカー遺伝子の標識断片と、植物由来の陰性コントロール核酸鎖とを混合させるステップと、
前記マーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、前記陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブとが所定の位置にそれぞれ固定された担体に、前記マーカー遺伝子の前記標識断片と前記陰性コントロール核酸鎖との混合物を接触させて、前記マーカー遺伝子検出用プローブと前記マーカー遺伝子の前記標識断片とをハイブリダイズさせるとともに、前記陰性コントロールプローブと前記陰性コントロール核酸鎖とをハイブリダイズさせるステップと、
ハイブリダイズさせる前記ステップの後に、前記マーカー遺伝子検出用プローブが固定された位置、及び、前記陰性コントロールプローブが固定された位置の標識強度をそれぞれ測定するステップと、
前記陰性コントロールプローブが固定された位置で測定された前記標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置で測定された前記標識強度の結果が所定の閾値を超えたとき、前記胃癌試料が陽性であると判定するステップと、
を含むことを特徴とする胃癌の再発を判定する方法
が提供される。
【0013】
また、本発明によれば、
胃癌細胞に特異的なマーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、陰性コントロールプローブとが所定の位置にそれぞれ固定された担体と、
植物由来の陰性コントロール核酸鎖と、
を備え、
前記陰性コントロールプローブは、前記陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合し、
胃癌試料中に含まれる前記マーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(Polymerase Chain Reaction, PCR)により増幅するとともに標識して、取得された前記マーカー遺伝子の標識断片を前記マーカー遺伝子検出用プローブとハイブリタイズさせるとともに、前記陰性コントロールプローブと前記陰性コントロール核酸鎖とをハイブリダイズさせ、ハイブリダイズ後に測定された前記陰性コントロールプローブが固定された位置の標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置の標識強度の結果が所定の閾値を超えたとき、前記胃癌試料が陽性であると判定することで胃癌の再発を判定するための胃癌再発判定用キット
が提供される。
【0014】
この発明によれば、植物由来の陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブを担体に固定し、マーカー遺伝子検出用プローブと胃癌試料のPCR産物とをハイブリダイゼーションさせるとともに、陰性コントロールプローブと陰性コントロール核酸鎖とをハイブリダイゼーションさせて、マーカー遺伝子検出用プローブが固定された位置で測定された標識強度と陰性コントロールプローブの位置で測定された標識強度との対比の結果を用いて胃癌の予後を判定する。これにより、植物由来の核酸鎖が陰性コントロールとなるため、検査対象検体と早期胃癌検体とを同時に測定することなく、あらかじめ定めた閾値に基づき、胃癌の予後再発の判定を正確に行うことができる。したがって、簡便かつ実用的な胃癌の予後判定が実現可能になる。
【発明の効果】
【0015】
本発明によれば、簡便かつ精度良く胃癌の再発を判定できる実用的な方法が提供される。
【図面の簡単な説明】
【0016】
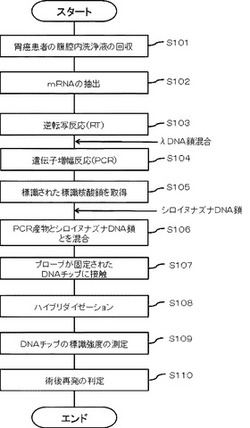
【図1】実施の形態に係る胃癌の再発を判定する方法を説明するフローチャートである。
【図2】実施の形態で用いるDNAマイクロアレイのレイアウトの一例を示す図である。
【発明を実施するための形態】
【0017】
以下、本発明の実施の形態について説明する。
【0018】
図1は、本実施形態の胃癌の再発を判定する方法を説明するフローチャートである。本実施形態は、胃癌試料中に含まれる胃癌細胞に特異的なマーカー遺伝子の少なくとも一部の領域をPCRにより増幅するとともに標識するステップ(S104)と、マーカー遺伝子の標識断片を取得するステップ(S105)と、マーカー遺伝子の標識断片と、植物由来の陰性コントロール核酸鎖とを混合させるステップ(S106)と、マーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブとが所定の位置にそれぞれ固定された担体に、マーカー遺伝子の標識断片と陰性コントロール核酸鎖との混合物を接触させるステップ(S107)と、マーカー遺伝子検出用プローブとマーカー遺伝子の標識断片とをハイブリダイズさせるとともに、陰性コントロールプローブと陰性コントロール核酸鎖とをハイブリダイズさせるステップ(S108)と、S108の後に、マーカー遺伝子検出用プローブが固定された位置、及び、陰性コントロールプローブが固定された位置の標識強度をそれぞれ測定するステップ(S109)と、マーカー遺伝子検出用プローブが固定された位置で測定された標識強度と陰性コントロールプローブが固定された位置で測定された標識強度とを対比し、対比の結果が所定の閾値を超えたとき、胃癌試料が陽性であると判定するステップ(S110)と、を含む。
【0019】
本実施形態において、植物由来の陰性コントロール核酸鎖としては、例えば、シロイヌナズナ、イネ、コムギ、ミヤコグサ、タバコなどの高等植物由来の核酸鎖を用いることができるが、中でもシロイヌナズナ由来の核酸鎖が好ましい。
【0020】
以下、陰性コントロール核酸鎖として、シロイヌナズナ由来のDNA鎖(以下、「シロイヌナズナDNA鎖」ともいう。)を用いる例を挙げて各ステップについて具体的に説明する。
まず、本実施形態の方法を実行するための胃癌再発判定用キットを用意する。このキットは、少なくとも一以上のマーカー遺伝子検出用プローブと、シロイヌナズナDNA鎖を検出する配列番号1で表される塩基配列からなるシロイヌナズナ遺伝子検出用プローブ(以下、「シロイヌナズナプローブ」ともいう。)とが所定の位置にそれぞれ固定された担体と、シロイヌナズナDNA鎖とを備えている。
【0021】
マーカー遺伝子検出用プローブは、好ましくは、6種の腫瘍マーカー遺伝子に対し、それぞれ特異的な配列を有するものである。6種の腫瘍マーカー遺伝子は、具体的には、CEA(がん胎児性抗原)、TFE1(trefoil factor family 1)、FABP1(Fatty acid binding protein 1)、CK20(Cytokeratin. 20)、MUC2(腸型ムチン)及びTACSTD1(tumor−associated calcium signal transducer 1)である。また、マーカー遺伝子を検出するためのプローブとしては、表1に示すものが例示できる。具体的には、配列番号2で表される塩基配列からなるCEA検出用プローブと、配列番号3で表される塩基配列からなるTFF1検出用プローブと、配列番号4で表される塩基配列からなるFABP1検出用プローブと、配列番号5で表される塩基配列からなるCK20検出用プローブと、配列番号6で表される塩基配列からなるMUC2検出用プローブと、配列番号7で表される塩基配列からなるTACSTD1検出用プローブである。
【0022】
【表1】

【0023】
担体としては基板、ビーズ、繊維など様々な担体を用いることができる。基板の形状は板状には限られず、たとえばフィルム状やシート状であってもよい。また、基板は、一つの部材から構成されていてもよいし、複数の部材から構成されていてもよい。担体の材質としては、金属、ガラス、プラスチック、ポリマー、繊維等、を用いることができる。高感度で、かつ、同時に多項目の遺伝子を検出する方法としては、基板に検出用プローブを固定化したDNAマイクロアレイが挙げられる。このDNAマイクロアレイには、基板上の一定の区画内に複数のスポットを設けておき、各スポットにプローブをそれぞれ固定化しておき、マイクロアレイを形成しておくことが好ましい。
【0024】
また、担体としては、高分子物質からなるコーティング層を表面に含む基板を用いてもよい。高分子物質からなるコーティング層を表面に含む基板は、所定の形状に加工された基板の表面に高分子物質を含む液体を塗布し、乾燥することにより得ることができる。また、高分子物質を含む液体中に基板を浸漬し、乾燥してもよい。
【0025】
担体として「高分子物質からなるコーティング層を表面に含む基板」を用いる場合、高分子物質としては、例えば、「リン脂質の親水部を構成するリン酸エステルより誘導される基を有する第一単位とカルボン酸誘導基を有する第二単位とを含む高分子物質」を用いることができる。「リン脂質の親水部を構成するリン酸エステルより誘導される基を有する第一単位とカルボン酸誘導基を有する第二単位とを含む高分子物質」は、例えば、特開2006−187270の段落0033〜0066に記載のものを用いることができるが、中でも、下記一般式(2)で表される、2−メタクリロイルオキシエチルホスホリルコリン(MPC)基を有する第一単量体と、p−ニトロフェニルオキシカルボニルポリエチレングリコールメタクリレート(NPMA)基を有する第二単量体と、ブチルメタリレート(BMA)基を有する第三単量体との共重合体であるpoly(MPC−co−BMA−co−NPMA)(PMBN)が特に好ましい。
【0026】
【化1】

【0027】
ただし、上記一般式(1)において、a、b、およびcは、それぞれ独立して、正の整数であり、aが5〜200、bが10〜500、cが1〜100であることが好ましい。また、上記一般式(2)において、第一〜第三単量体がブロック共重合していてもよいし、これらの単量体がランダムに共重合していてもよい。
【0028】
また、「高分子物質からなるコーティング層を表面に含む基板」を用いる場合、高分子物質からなるコーティング層を形成させる基板としては、プラスチック基板が、形状やサイズの変更に対する柔軟性が確保される上に、ガラス基板のものに比べて安価で提供することができるという観点から好ましい。このようなプラスチック材料としては、表面処理の容易性および量産性の観点から、熱可塑性樹脂を用いることができる。
【0029】
熱可塑性樹脂としては、蛍光発生量の少ないものを用いることができる。蛍光発生量の少ない樹脂を用いることにより、DNA鎖の検出反応におけるバックグランドを低下させることができるため、検出感度をさらに向上させることができる。蛍光発生量の少ない熱可塑性樹脂としては、たとえば、ポリエチレン、ポリプロピレン等の直鎖状ポリオレフィン;環状ポリオレフィン;含フッ素樹脂;等を用いることができる。上記樹脂の中でも、飽和環状ポリオレフィンは、耐熱性、耐薬品性、低蛍光性、透明性および成形性に特に優れるため、光学的な分析に好適であり、基板の材料として好ましく用いられる。
【0030】
ここで、飽和環状ポリオレフィンとは、環状オレフィン構造を有する重合体単独または環状オレフィンとα−オレフィンとの共重合体を水素添加した飽和重合体を指す。前者の例としては、たとえばノルボルネン、ジシクロペンタジエン、テトラシクロドデセンに代表されるノルボルネン系モノマー、及び、これらのアルキル置換体を開環重合して得られる重合体を水素添加して製造される飽和重合体である。後者の共重合体はエチレンやプロピレン、イソプロピレン、1−ブテン、3−メチル−1−ブテン、1−ペンテン、3−メチル−1−ペンテン、1−ヘキセン、1−オクテン等のα−オレフィンと環状オレフィン系モノマーのランダム共重合体を水素添加することにより製造される飽和重合体である。共重合体では、エチレンとの共重合体が最も好ましい。これらの樹脂は単独で用いてもよく、2種類またはそれ以上の共重合体あるいは混合物であってもよい。また、環状オレフィン構造を有する単量体が開環重合して得られる飽和環状ポリオレフィンだけでなく、環状オレフィン構造を有する単量体の付加重合により得られる飽和環状ポリオレフィンを用いることもできる。
【0031】
担体へのシロイヌナズナプローブ及びマーカー遺伝子検出用プローブ(以下、単に「プローブDNA」ともいう。)の固定化方法は、特に限定されないが、担体として、PMBNのコーティング層を表面に含む基板を用いる場合、例えば、PMBNに含まれるp−ニトロフェニルオキシカルボニル基とプローブDNAとを反応させて共有結合を形成させることにより、基板表面でプローブDNAを固定化することができる。
【0032】
プローブDNAを基板上に固定化する際には、プローブDNAを溶解または分散した液体を点着する方法が好ましい。このプローブDNAを溶解または分散した液体は、例えば中性からアルカリ性、例えばpHが7.6以上とすることができる。
【0033】
また、点着後、基板表面に固定化されなかったプローブDNAを除去するため、純水や緩衝液で洗浄してもよい。洗浄後はプローブDNAを固定化した以外の基板表面のp−ニトロフェニルオキシカルボニル基の不活性化処理をアルカリ化合物、あるいは一級アミノ基を有する化合物で行ってもよい。
【0034】
また、基板に固定化するプローブDNAには、p−ニトロフェニルオキシカルボニル基との反応性を高めるため、アミノ基を導入しておくことが好ましい。アミノ基はp−ニトロフェニルオキシカルボニル基との反応性に優れるため、アミノ基が導入されたプローブDNAを用いることにより、効率よくかつ強固に基板の表面上にプローブDNAを固定化することができる。アミノ基の導入位置はプローブDNAの分子鎖末端あるいは側鎖であってもよいが、分子鎖末端に導入されていることが、相補的な鋳型DNA断片とのアニーリングをより一層効率よく行うことができるという観点からは、好ましい。
【0035】
以上により、基板の表面上に表1に示すプローブが固定化されたDNAマイクロアレイを得ることができる。
【0036】
DNAマイクロアレイによる発現解析を行う場合には、より確度の高い判定を行うため、内部コントロールや外部コントロールを用いることが好ましい。本実施形態では、内部コントロールとして、ACTB(ベータアクチン)を用いることができる。また、外部コントロールとしては、陰性コントロールや陽性コントロールを用いることができ、上述のとおり本実施形態では、陰性コントロールとしてシロイヌナズナDNA鎖を用いることができ、陽性コントロールとしてλDNA鎖を用いることができる。ACTBを内部コントロールとして用いる場合は、配列番号8で表される塩基配列からなるACTB検出用プローブを内部コントロールプローブとして担体に固定させることができる。ACTB検出用プローブの固定化もシロイヌナズナプローブやマーカー遺伝子検出用プローブの固定化と同様に行うことができる。
【0037】
胃癌再発判定用キットには、マーカー遺伝子の一部の領域を増幅するためのプライマーセットを備えることが好ましい。こうすることで、審査腹腔鏡時の洗浄液もしくは術中腹腔内洗浄液から抽出したmRNAが少ない場合であっても、増幅させて有効な判定が可能になる。表1で示すプローブによって検出される各マーカー遺伝子のプライマー塩基配列を表2に示す。具体的には、配列番号10、11で表される塩基配列からなるCEA増幅用プライマーセット、配列番号12、13で表される塩基配列からなるTFE1増幅用プライマーセット、配列番号14、15で表される塩基配列からなるFABP1増幅用プライマーセット、配列番号16、17で表される塩基配列からなるCK20プライマーセット、配列番号18、19で表される塩基配列からなるMUC2プライマーセット、及び、配列番号20、21で表される塩基配列からなるTACSTD1プライマーセットが挙げられる。また、内部コントロールとしてACTBを用いる場合は、配列番号22、23で表される塩基配列からなるプライマーセットにより、ACTBの一部の領域を増幅させてもよい。なお、表2には、表1に示すプライマーにより増幅されるPCR産物の塩基長も併せて示す。
【0038】
【表2】

【0039】
このようにして得られる胃癌再発判定用キットを用意した後、研究使用目的に対するインフォームド・コンセントが得られた胃癌患者から腹腔内洗浄液を回収し(S101)、これを遠心して浮遊成分である遊離細胞をペレット状に沈殿させた後、そのペレットからmRNAを抽出する(S102)。mRNAの抽出方法は各種の方法が考案され、多くのメーカーによりキット化されており、本実施形態においてはいずれのキットも使用可能である。
【0040】
ついで、胃癌試料中に含まれるmRNAは増幅させるため、RT−PCRを行う。具体的には、まず、逆転写酵素を用いた逆転写反応(Reverse Transcription、RT)により、抽出したmRNAを鋳型として、例えば、ランダムヘキサマーやオリゴdTプライマー等のプライマーの存在下でデオキシヌクレオチド三リン酸(dNTP)を基質とし、抽出したmRNAに相補的なcDNAを5'→3'方向に合成する。(S103)。
【0041】
その後、得られたcDNAを用いてPCR増幅反応を行う(S104)。具体的には、合成したcDNA、配列番号10〜25で表される塩基配列からなるプライマー、DNAポリメラーゼ及びdNTPと、DNAポリメラーゼが働く至適塩濃度環境を作るためのバッファー溶液とを混合し、市販のPCR装置にセットする。使用するDNAポリメラーゼは、耐熱性を有するものであればよい。
【0042】
その後、(1)cDNAの熱変性(94〜96℃)、(2)配列番号10〜25で表される塩基配列からなるプライマーのアニーリング(55〜60℃)、(3)DNAポリメラーゼによる相補鎖の合成(72〜74℃)の3段階の温度変化を、例えば、20〜30サイクル繰り返し、cDNAを増幅する。
【0043】
ここで、PCR増幅反応は、dNTPとともに、ラベル化したヌクレオチドモノマーを用いて実行する。ラベル方法は、蛍光体、光吸収体、放射性核種(P32)、酵素標識などの方法が挙げられる。
例えば、蛍光ラベル化ヌクレオチドモノマーとしては、dTTPの塩基の3位を蛍光ラベルしたCy3−dUTPが挙げられる。Cy3−dUTPを用いた場合、鋳型DNA断片のアデニン(A)に対応する伸長(プライマー)側の位置にCy3−dUTPが挿入されたPCR産物を得ることができる。
また、放射性核種によるラベル化ヌクレオチドモノマーとしては、P32−ATP、P32−dATPが挙げられる。
また、アミノアリルdUTPを用いてPCR増幅反応をし、反応後に、アミノアリルdUTPを含むPCR産物に対して、Cy3蛍光色素やアミノ基染色試薬(NHS−試薬)を用いて発色させてもよい。
酵素標識の方法においては、ビオチン(biotin)化またはジゴキシゲニン(DIG:ステロイド系天然物)を結合した核酸(例えば、biotin−dUTP、DIG−dUTP)を使用して増幅した後、得られるPCR産物にアルカリフォスファターゼまたはアルカリフォスファターゼ処理しニトロブルーテトラゾリウム(NBT)と5−ブロモ−4−クロロ−3−インドリルリン酸(BCIP)液中で数時間反応させて発色させることもできる。
【0044】
なお、S104では、逆転写反応で得られたcDNAとともに、陽性コントロール核酸鎖をバッファー溶液に混合して、増幅反応を行ってもよい。陽性コントロール核酸鎖としては、微生物由来の核酸鎖を用いることができ、具体的にはバクテリオファージ等のウイルス由来のものが挙げられる。陽性コントロールDNA鎖としてλDNA鎖を用いる場合、表2で示す配列番号24及び25で表される塩基配列からなるプライマーを用いてλDNA鎖の一部を増幅させることができる。この場合、λDNA鎖を検出するため、表1で示す配列番号9で表される塩基配列からなるλDNA検出用プローブをあらかじめ担体に固定しておく。このλDNA検出用プローブは、配列番号24及び25で表される塩基配列からなるプライマーにより増幅されるλDNA鎖の断片と相補的に結合できるように設計されている。
【0045】
このようにして、標識したPCR産物(標識DNA断片)を取得した後(S105)、得られた標識DNA断片とシロイヌナズナDNA鎖とを混合する(S106)。標識DNA断片とシロイヌナズナDNA鎖との混合は、ハイブリダイゼーションバッファー中で行うことが好ましい。ハイブリダイゼーションバッファーは、公知のものを用いることができるが、例えば、SSC又はSSPEと、SDSと、ホルムアミドとの混合液を用いることができる。
【0046】
ついで、混合した標識DNA断片及びシロイヌナズナDNA鎖を、ハイブリダイゼーションバッファー、及び、プローブDNAが固定化されたDNAマイクロアレイとともにハイブリダイゼーション装置にセットし(S107)、例えば、45℃〜60℃で3〜24時間、好ましくは3〜5時間、ハイブリダイゼーションする(S108)。
【0047】
その後、マイクロアレイの洗浄後に、市販のマイクロアレイスキャナーを用いて標識強度を測定する(S109)。S104において、微生物由来のDNA鎖、特に、λDNAを用いてマーカー遺伝子とともにPCR増幅させた場合は、λDNA鎖の標識強度を基準として各マーカー遺伝子の標識強度を測定してもよい。具体的には、λDNA検出用プローブが固定された位置における標識強度が所定の範囲になるように検出感度を制御する。こうすることで、アッセイ間のバラツキを補正することができる。
【0048】
また、得られた標識強度の実測値に種々の補正をかけ、補正された実測値を真の測定値としてもよい。例えば、プローブDNAが固定化されていない担体表面の標識強度をバックグラウンドとして測定結果から差し引いて、測定値としてもよい。
【0049】
その後、得られた測定値に基づき胃癌試料の術後再発の判定を行う(S110)。DNAマイクロアレイによる発現解析を行う場合には、各プローブ、判定対象となるmRNAごとにしかるべき規格化を行い、各サンプル間で比較可能となる調整を行うことで、より確度の高い判定を行うことが出来る。
【0050】
具体的には、各遺伝子の発現量(発現レベル)のカットオフ値を設定し、カットオフ値を上回った遺伝子数をカウントし、定めた数を上回るか下回るかにより判定を行う方法が有効である。そこで、本実施形態では、シロイヌナズナDNA鎖を陰性コントロールとし、これを基準として、カットオフ値(閾値)を導き出す。
【0051】
閾値を導き出す方法は、医療機関において実際の検体を用いた解析により、経験的に、マーカー遺伝子ごとに、所定の閾値を定める。具体的には、臨床の所見やその他の測定値を合わせて所定の閾値が定められる。そして、シロイヌナズナ検出用プローブが固定された基板の位置における標識強度を基準として、各マーカー遺伝子の標識強度の相対値を算出する。各マーカー遺伝子の標識強度の相対値の算出法としては、シロイヌナズナ検出用プローブが固定された基板の位置における標識強度を1として、各マーカー遺伝子の標識強度の相対値を算出する方法や、シロイヌナズナ検出用プローブが固定された基板の位置における標識強度を0として、各マーカー遺伝子の標識強度の相対値を算出する方法が挙げられる。そして、1以上のマーカー遺伝子の標識強度の相対値が閾値を超えた場合に、結果を陽性、すなわち、予後不良(再発する、又は、再発の可能性が大きい)と判定する。一方、すべてのマーカー遺伝子の標識強度の相対値が閾値以下である場合は、結果を陰性、すなわち、予後良好(再発しない、又は、再発の可能性が小さい)と判定する。なお、陽性あるいは陰性の判定手段としては、これに限定されず、設定された閾値を超えたマーカー遺伝子が2以上の所定数あった場合に陽性と判断し、設定された閾値を超えたマーカー遺伝子がこの所定数に満たない場合を陰性と判定してもよいし、ある特定のマーカー遺伝子の標識強度の相対値が閾値を超えた場合に陽性と判断し、この特定のマーカー遺伝子の標識強度の相対値が閾値以下であった場合に陰性と判断してもよい。
【0052】
また、S104において、内部コントロール(ACTB)や陽性コントロール(λDNA)を用いてPCR増幅を実行した場合は、ACTBやλDNAの測定結果も併せて考察することで、S102の抽出、又は、S108のハイブリダイゼーションがそれぞれ良好に進行したかどうかを判別することができる。具体的には、ACTBの結果が陰性であり、かつ、λDNAの結果が陰性である場合は、S102の抽出、及び、S108のハイブリダイゼーションのいずれもが不良であったことがわかる。また、ACTBの結果が陽性である一方、λDNAの結果が陰性であった場合は、S102の抽出は良好に進行したが、S108のハイブリダイゼーションの進行が不良であったことがわかる。ACTBの結果が陰性であり、かつ、λDNAの結果が陽性である場合は、S104のPCRは良好に進行したが、S102の抽出の進行が不良であったことが分かる。
【0053】
なお、本実施形態の判定方法とともに、通常、外科的切除治療症例、審査腹腔鏡症例において必須となっている細胞診の結果に加えて、リンパ節転移の有無、各種の癌マーカーなどの数値を参照し、状況によっては判定方法を使い分けるなど、総合的に胃癌細胞の有無を判断するとより効果的な判定が可能である。
【0054】
つづいて、本実施形態の作用効果について説明する。本実施形態によれば、植物由来の陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブを担体に固定し、マーカー遺伝子検出用プローブと胃癌試料のPCR産物とをハイブリダイゼーションさせるとともに、陰性コントロールプローブと陰性コントロール核酸鎖とをハイブリダイゼーションさせて、マーカー遺伝子検出用プローブが固定された位置で測定された標識強度と陰性コントロールプローブの位置で測定された標識強度との対比の結果を用いて胃癌の予後を判定する。これにより、植物由来の核酸鎖が陰性コントロールとなるため、検査対象検体と早期胃癌検体とを同時に測定することなく、あらかじめ定めた閾値に基づき、胃癌の予後再発の判定を正確に行うことができる。したがって、簡便かつ実用的な胃癌の予後判定が実現可能になる。
【0055】
従来、胃癌の予後判定においては、対象検体と同時に、陰性コントロールとして腹腔内に癌細胞が存在しないものと考えられるIa期の早期癌検体を用いた測定を行い、早期癌検体の結果からカットオフ値を導き出す方法がとられていた。しかしながら、検査ごとに早期癌検体を用意しなければならず、実用性や倫理面で問題があった。
【0056】
一方、本実施形態では、植物由来のDNA鎖を陰性コントロールとして用いて、精度よく胃癌の予後を判定できる。したがって、検査ごとに早期癌検体を用意する必要がなくなり、実用的である。特にシロイヌナズナDNA鎖を陰性コントロールとして用いることで、マーカー遺伝子との反応を無視できるレベルにすることができ、より精度よく胃癌の予後を判定することができる。なお、シロイヌナズナ遺伝子は、交差反応性が低いことが確認されている。また、シロイヌナズナDNA鎖は、入手が容易な点でもメリットがある。
【実施例】
【0057】
以下、具体的な実施例を示し、本発明をさらに詳細に説明する。
【0058】
<実施例1>
(1)プラスチック基板の製造
飽和環状ポリオレフィン樹脂(5−メチル−2−ノルボルネンの開環重合体の水素添加物(MFR(Melt flow rate):21g/10分、水素添加率:実質的に100%、熱変形温度123℃)を用い、射出成形によりスライドガラス形状の基板を得た。得られた基板を上記式(1)で表されるPMBNポリマー(MPC基:BMA基:NPMA基=23:74:3(モル比))の0.3重量%エタノール溶液に浸漬することにより、基板表面にホスホリルコリン基と活性エステル基とを有する高分子物質を導入して、プラスチック基板を得た。なお、MFRは、JIS K 7210:1999法記載の条件に準じて測定したものである。
【0059】
(2)プローブDNA
5'末端がアミノ基で修飾された、胃癌再発に関するマーカー遺伝子6種類の特異配列を有するオリゴDNA鎖、およびコントロール遺伝子3種類を用意した。用意したプローブは表1に記載した9種類の遺伝子に対応したプローブである。各遺伝子に対応したプローブの配列番号は、CEA検出用として配列番号2、TFF1検出用として配列番号3、FABP1検出用として配列番号4、CK20検出用として配列番号5、MUC20検出用として配列番号6、TACSTD1検出用として配列番号7である。また、内部コントロールとして配列番号8(ACTB鎖検出用)、陰性コントロールとして配列番号1(シロイヌナズナDNA鎖検出用)、陽性コントロールとして配列番号9(λDNA鎖検出用)である。
【0060】
(3)基板へのプローブDNA固定
スポッティングバッファー(製品名:×2スポッティング液、住友ベークライト社製)を用いて、本実施例の(2)で用意したプローブDNAを溶解し、10μMのプローブDNA溶液を調製した。この溶液をスポッター(日立ソフトウェアエンジニアリング社製MARKS−I)を用い、300μm径スポットピンで、本実施例の(1)で作製したプラスチック基板(縦75mm×横25mm)の表面上にスポットした。スポットは、各プローブについて、N=5でスポットした。プローブDNAをスポットした基板を、80℃で1時間加熱して、プローブDNAを固定化させた。これによりプラスチック基板のポリマー表面に一本鎖DNAが固定したDNAマイクロアレイを得た。スポットのレイアウトを図2に示す。
【0061】
(4)mRNAの回収
研究使用目的に対するインフォームド・コンセントが得られた胃癌術中患者に対し、通常行われる術式に従って500mLの生理食塩水を用いて腹腔内洗浄を行い、洗浄液を回収した。回収された腹腔内洗浄液のうち病理部に検査検体として提出するのに必要な分量を抜き取った余剰検体を検査対象検体とし、1500rpmで10分間遠心し、上清を除去することによりペレット状になった沈殿成分を腹腔内浮遊細胞として回収した。回収した細胞を培養し、得られた細胞をニッポンジーン社製試薬Isogenに加えてホモジナイズし、少量のクロロホルムを入れ、9420rpmで15分間遠心分離させ、上清を採取した。さらに採取量と等量のイソプロパノールを加え、10分以上インキュベートした後、9420rpmで10分間遠心して、ペレットを回収し、エタノール沈殿(70%)によりmRNAを得た。このmRNAを189症例分用意し、進行癌サンプルとした。
【0062】
(5)逆転写反応
逆転写反応を行うことにより上記抽出したmRNAからcDNAを作製した。
上記抽出したmRNAを1μg/mLに濃度調製し、調製したmRNA1μLと、20μMのランダムヘキサマー(製品名:Random primer、タカラバイオ社製)1μLと、10mMのdNTP(GEヘルスケア社製)1μLと、精製水9μL、逆転写酵素(Invitrogen社製、SuperScriptII)1μL、及び、Rnase Inhibitor(Roch社製)1μLを加え、42℃で50分加熱保持さらに72℃で15分加熱保持させ、一本鎖cDNAを得た。
【0063】
(6)マルチプレックスPCR反応
腹腔内洗浄液から得た一本鎖cDNAを鋳型に検査対象となるマーカー遺伝子のcDNAを増幅するためのPCRを行った。同一反応中に複数のプライマーを用いるマルチプレックスPCR法によりcDNAの増幅を行った。同一のcDNAに対し、プライマーとして表2に示す配列番号10〜25の塩基配列からなるプライマーセットを用いた。
【0064】
表2に示す配列番号10〜25の塩基配列からなるプライマーをDEPC−Treated Water中に各々濃度10μMで含むプライマー溶液を0.5μL、DNAポリメラーゼ(Invitrogen社製AcuuPrime)0.5μL、λDNA鎖(タカラバイオ社製)をPCRバッファー(Invitrogen社製AcuuPrimeのキット内に含まれる)中に溶解させ20μLの反応液を調製した。また、PCR産物の標識のために、アミノアリルdUTP(Ambion社製)0.1μLを基質として、PCRの反応溶液に加えた。
【0065】
調製された反応液について、市販のサーマルサイクラー(製品名:GeneAmp(登録商標)PCRSystem 9700、Applied biosystem社製)を用いて、(i)熱変性95℃15秒、(ii)アニーリング60℃45秒、(iii)DNA鎖の伸長反応72℃3分のヒートサイクルで、25サイクル行い、アミノアリル標識PCR産物を得た。
【0066】
精製は、チップごとにDNA精製キット(キアゲン社製QIA quick)を使用して上記PCR産物の精製を行った。
【0067】
(7)Cy3標識
得られたアミノアリル標識PCR産物に対して、Cy3標識を行った。具体的には、本実施例の(6)で得られた調製ずみのアミノアリル標識PCR産物を蛍光ラベル化試薬(GEヘルスケア社製Cy3 Mono−reactive Dye Pack)を使用してCy3蛍光ラベル化し、さらにDNA精製キット(キアゲン社製QIA quick)を使用して精製を行い、Cy3蛍光ラベル化PCR産物を得た。
【0068】
(8)ハイブリダイゼーション
Cy3蛍光ラベル化PCR産物45μLに対し、バッファー85μLを加え、さらに、シロイヌナズナDNA鎖(SIGMA GENOSYS社製)を加え、ハイブリダイゼーション溶液を得た。得られたハイブリダイゼーション溶液の組成は、以下のとおりである。
[ハイブリダイゼーション溶液]
・3×SSPE
・10%ホルムアミド
・ 0.05%SDS
(3)で作製したDNAマイクロアレイをハイブリダイゼーション装置(Genomic Solutions社製Hyb4)にセットし、上記組成のハイブリダイゼーション溶液を用いてハイブリダイゼーションを行った。具体的には、95℃で5分熱変性させたハイブリダイゼーション溶液を実施例1で作製したDNAマイクロアレイ上に分注し、55℃で4時間反応を行った。ハイブリダイゼーション反応終了後、0.1%SDSを含んだ2×SSC溶液、2×SSC、0.1×SSCの順でマイクロアレイの表面を洗浄して、ハイブリダイゼーション反応を終了した。
【0069】
(9)測定
本実施例(8)で行ったハイブリダイゼーション反応終了後、スピンドライしたDNAマイクロアレイについて、DNAマイクロアレイ用スキャナー(Axon社製GenePix4000B)を用いて、プローブがスポットされた位置ごとに蛍光強度を測定した。測定する際の受光感度(PMT)は、λDNAが固定されたスポットにおける蛍光強度が900から1100となるように調整した。蛍光強度の算出にあたっては、プローブごとに5個の測定値が得られるが、最高値及び最低値を除く中間の3個の測定値を平均化して、プローブごとに平均値を算出し、これを蛍光強度とした。進行癌サンプル全189症例について蛍光強度を測定した。
【0070】
(10)相対値を用いた再発予後判定
得られた蛍光強度の結果について、シロイヌナズナDNA鎖検出用プローブがスポットされた位置の蛍光強度を基準として各プローブが固定された位置における蛍光強度の比を算出した結果を表3−6に示す。また、各マーカー遺伝子の蛍光強度の相対値にそれぞれ閾値を設定し、その閾値を越えた場合には、陽性とし、下回る場合は、陰性とした。この閾値は、一般的なDNAマイクロアレイのカットオフ値の設定が通常バックグランドの3倍であることに基づき設定した。TFF1、FABP1、CK20、及び、MUC2については、CEAやTACSTDに比較して発現量が低く、また癌特有に発現するものであるため、これらの閾値は、CEAやTACSTDの閾値の半分に設定した。具体的には、進行癌サンプルのマーカー遺伝子の閾値は、下記のとおり設定した。
CEA 閾値:3
TFF1 閾値:1.5
FABP1 閾値:1.5
CK20 閾値:1.5
MUC2 閾値:1.5
TACSTD 閾値:3
進行癌サンプル全189症例について陽性と判断されたマーカー遺伝子の数を表3−6に併せて示す。
【0071】
【表3】

【0072】
【表4】

【0073】
【表5】

【0074】
【表6】

【0075】
<実施例2>
実施例1の(1)〜(8)の操作を行った。
ハイブリダイゼーション反応終了後、実施例1の(9)で示すように、プローブがスポットされた位置ごとに蛍光強度を測定した。得られた189症例の蛍光強度について、シロイヌナズナDNA鎖検出用プローブがスポットされた位置の蛍光強度をバックグラウンド値として、各プローブが固定された位置における蛍光強度からシロイヌナズナDNA鎖検出用プローブがスポットされた位置の蛍光強度を差し引いた結果を表7−10に示す。また、各マーカー遺伝子の蛍光強度の相対値にそれぞれ閾値を設定し、その閾値を越えた場合には、陽性とし、下回る場合は、陰性とした。この閾値は、実施例1で説明したように、一般的なDNAマイクロアレイのカットオフ値の設定が通常バックグランドの3倍であることに基づきCEA及びTACSTDの閾値を設定し、TFF1、FABP1、CK20、及び、MUC2については、CEA及びTACSTDの閾値の約半分に設定した。そして、閾値を越えた場合には、陽性とし、下回る場合は、陰性とした。具体的には、進行癌サンプルのマーカー遺伝子の閾値は、下記のとおり設定した。
CEA: 130
TFF1: 70
FABP1: 70
CK20: 70
MUC2: 70
TACSTD1: 130
進行癌サンプル全189症例について陽性と判断されたマーカー遺伝子の数を表7−10に併せて示した。
【0076】
【表7】

【0077】
【表8】

【0078】
【表9】

【0079】
【表10】

【0080】
<比較例1>
(1)DNAマイクロアレイの作製
実施例1の(1)〜(3)の操作にしたがって、DNAマイクロアレイを作製した。ただし、配列番号1で表される塩基配列からなるシロイヌナズナDNA鎖検出用プローブはDNAマイクロアレイに固定しなかった。
(2)進行癌サンプルとDNAマイクロアレイとのハイブリダイゼーション
実施例1の(4)〜(7)の操作を行い、得られた進行癌サンプル189症例のCy3蛍光ラベル化PCR産物を本比較例1の(1)で作製したDNAマイクロアレイにそれぞれハイブリダイズした。ハイブリダイゼーションの条件及び操作は、実施例1の(8)に従った。ただし、Cy3蛍光ラベル化PCR産物にシロイヌナズナDNA鎖は加えなかった。
(3)早期癌サンプルとDNAマイクロアレイとのハイブリダイゼーション
細胞診により早期癌と判定された早期癌細胞25症例について、実施例1の(4)〜(7)で示す方法と同様な操作を行ってCy3蛍光ラベル化PCR産物を用意し、比較例1の(1)で作製したDNAマイクロアレイにハイブリダイズした。この場合も、ハイブリダイゼーションの条件及び操作は、実施例1の(8)に従い、Cy3蛍光ラベル化PCR産物にシロイヌナズナDNA鎖は加えなかった。
(4)測定
ハイブリダイゼーション反応終了後、スピンドライしたDNAマイクロアレイについて、DNAマイクロアレイ用スキャナー(Axon社製GenePix4000B)を用いて、プローブがスポットされた位置ごとに蛍光強度を測定した。測定する際の受光感度(PMT)は、λDNAが固定されたスポットにおける蛍光強度が100から600となるように調整した。蛍光強度の算出にあたっては、プローブごとに5個の測定値が得られるが、最高値及び最低値を除く中間の3個の測定値を平均化して、プローブごとに平均値を算出し、これを蛍光強度とした。全189症例、及び、早期癌サンプル25症例について蛍光強度をそれぞれ測定した。
(5)再発予後判定
早期癌サンプルをハイブリダイズしたDNAマイクロアレイの測定結果25症例を用い、各マーカーについて、平均値+その標準偏差×2をカットオフ値とした。具体的なカットオフ値を以下に示す。
CEA: 56.9
TFF1: 54.0
FABP1: 55.3
CK20: 53.1
MUC2: 49.8
TACSTD1: 85.1
進行癌サンプル189症例について、カットオフ値を超えたマーカー遺伝子を陽性と判断し、カットオフ値以下のマーカー遺伝子は、陰性とした。蛍光強度の結果を表11−14に示す。
【0081】
【表11】

【0082】
【表12】

【0083】
【表13】

【0084】
【表14】

【0085】
<細胞診>
胃癌の術中腹腔内洗浄細胞診ガイドライン(日臨細胞誌44(1);93−97.2001)に基づいて、進行癌サンプル189症例について、細胞診により判定を行った。各症例について回収した腹腔内浮遊細胞をスライドガラス上に載せ、引きガラスを用いて塗抹標本を作製した。速やかに塗抹標本を風乾燥させ、その後、メタノールによって3分間固定させた。ギムザ液と純水を1:2の割合で混合させ、混合液を用いて標本の染色を行った。30分間染色させた後、純水で洗浄し、乾燥させて封じ、標本を完成させた。光学顕微鏡による標本観察で染色された癌細胞を探すことで、細胞診の判定を行った。細胞診では、クラス分類を行い、「1」は、陽性(再発する)とし、「0」は、陰性(再発しない)とした。結果を表19−26に示す。
【0086】
<リンパ節転移>
進行癌サンプル全189症例について、胃癌部とともに外科的に切除した周辺リンパ節の病理診断により、リンパ節転移の有無の診断を行った。結果を表19−26に示す。表19−22では、リンパ節転移がある場合を陽性、ない場合を陰性とした。
【0087】
また、進行癌サンプル全189症例の胃癌患者の術後経過については3年以上、平均4.5年にわたって追跡調査され、再発、及び死亡の有無の情報を得た。表15−18では、生死の結果も併せて示す。「alive」は再発することなく生存、「再発」は再発するが生存、「death」は、再発による死亡を示す。また、表19−22では、各実施例及び各比較例において、陽性と判断されたマーカー遺伝子の数も併せて示す。表19が実施例1の結果であり、表20が実施例2の結果であり、表21が比較例1の結果である。表19−21では再発した症例と死亡した症例数、及び、陽性あるいは陰性と判定された進行癌サンプル中の再発及び死亡症例数の割合(百分率)を各実施例及び各比較例の結果と対応づけて示す。表19−21では、各実施例及び各比較例で陽性マーカー遺伝子が1以上検出されたものを+で示し、全く検出されなかったものを−で示す。
【0088】
【表15】

【0089】
【表16】

【0090】
【表17】

【0091】
【表18】

【0092】
【表19】

【0093】
【表20】

【0094】
【表21】

【0095】
表19−21で示すように、進行癌サンプル189症例中、リンパ節転移が陽性の132症例において、細胞診により検出できなかった98症例中、実施例1,2、比較例1において陽性と判断された各28、27、33症例の再発率、死亡率は、陰性だった各70、71、65症例と比較して、ともに高く予後が悪かった。これら細胞診陰性・マイクロアレイ陽性症例において、実施例1、2の再発率は比較例1に比べて、各15、11ポイント高かった。死亡率では各17、16ポイント高かった。したがって、本発明によれば、早期癌患者の症例を同時に用いた従来の検査法よりも、いっそう精度よく胃癌の予後判定が行えることが明らかとなった。リンパ節転移陰性の57症例についても、症例数は少ないもののマイクロアレイ陽性症例の再発率、死亡率ともに陰性症例に比べて高かった。
【技術分野】
【0001】
本発明は、胃癌の再発を判定する方法、及び、これに用いる胃癌再発判定用キットに関する。
【背景技術】
【0002】
近年、DNAチップ技術を用いた、細胞診に換わる再発予測法が開発されている。
【0003】
特許文献1には、TFF1、TFF2、FABP1、CK20、MUC2、CEA、TACSTD1、MASPIN、並びにACTBの合計9種類の遺伝子の発現レベルの測定に基づき、胃癌の再発予測を行うことが記載されている。特許文献1の技術によれば、対象の癌患者から採取した試料を利用して対象の遺伝子の一部の領域をPCRにより増幅し、得られたPCR増幅物と、合計9種類のプローブが固相上に配置されているアレイとをハイブリダイゼーションさせて、得られるアレイの結果から、均一で正確な胃癌の再発予測のためのデータ取得が可能になるとされている。
【0004】
また、非特許文献1には、CK20、FABP1、TFF1及びMASPINについてのマイクロアレイアッセイと免疫細胞化学的な結果が非常に一致していたことが記載されている。
【先行技術文献】
【特許文献】
【0005】
【特許文献1】特開2006−223303号公報
【非特許文献】
【0006】
【非特許文献1】Annals of Surgical Oncology,14,1694−1702,2007
【発明の概要】
【発明が解決しようとする課題】
【0007】
しかしながら、上記文献の技術では、カットオフ値を定めるため、検査対象検体と同時に早期胃癌検体を用いた測定を行う必要があった。
【0008】
例えば、特許文献1の技術では、早期胃癌症例39例の最大輝度値を判定値としてこの判定値を上回る輝度値が得られた遺伝子について、「有意な発現があった」と判断している。
【0009】
また、非特許文献1の技術では、早期癌サンプル39例のMAX SD(maximum value plus standard deviation)またはAVG 2SD(average value plus twice or more positive markers)をカットオフ値として設定し、結果として後者が有効であることを示している。
【0010】
したがって、上記文献の技術では、40例近くの早期胃癌の症例を検査ごとに使用しなければ的確な予後再発の判定を行うことが困難であり、実用的な方法ではでないという問題があった。
【0011】
本発明は上記事情に鑑みてなされたものであり、その目的とするところは、検査対象検体と同時に早期胃癌検体を用いた測定を行わなくても、正確に胃癌の再発を判定できる、簡便かつ実用体な方法を提供することにある。
【課題を解決するための手段】
【0012】
本発明によれば、
胃癌試料中に含まれる胃癌細胞に特異的なマーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(Polymerase Chain Reaction,PCR)により増幅するとともに標識して前記マーカー遺伝子の標識断片を取得するステップと、
前記マーカー遺伝子の標識断片と、植物由来の陰性コントロール核酸鎖とを混合させるステップと、
前記マーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、前記陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブとが所定の位置にそれぞれ固定された担体に、前記マーカー遺伝子の前記標識断片と前記陰性コントロール核酸鎖との混合物を接触させて、前記マーカー遺伝子検出用プローブと前記マーカー遺伝子の前記標識断片とをハイブリダイズさせるとともに、前記陰性コントロールプローブと前記陰性コントロール核酸鎖とをハイブリダイズさせるステップと、
ハイブリダイズさせる前記ステップの後に、前記マーカー遺伝子検出用プローブが固定された位置、及び、前記陰性コントロールプローブが固定された位置の標識強度をそれぞれ測定するステップと、
前記陰性コントロールプローブが固定された位置で測定された前記標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置で測定された前記標識強度の結果が所定の閾値を超えたとき、前記胃癌試料が陽性であると判定するステップと、
を含むことを特徴とする胃癌の再発を判定する方法
が提供される。
【0013】
また、本発明によれば、
胃癌細胞に特異的なマーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、陰性コントロールプローブとが所定の位置にそれぞれ固定された担体と、
植物由来の陰性コントロール核酸鎖と、
を備え、
前記陰性コントロールプローブは、前記陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合し、
胃癌試料中に含まれる前記マーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(Polymerase Chain Reaction, PCR)により増幅するとともに標識して、取得された前記マーカー遺伝子の標識断片を前記マーカー遺伝子検出用プローブとハイブリタイズさせるとともに、前記陰性コントロールプローブと前記陰性コントロール核酸鎖とをハイブリダイズさせ、ハイブリダイズ後に測定された前記陰性コントロールプローブが固定された位置の標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置の標識強度の結果が所定の閾値を超えたとき、前記胃癌試料が陽性であると判定することで胃癌の再発を判定するための胃癌再発判定用キット
が提供される。
【0014】
この発明によれば、植物由来の陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブを担体に固定し、マーカー遺伝子検出用プローブと胃癌試料のPCR産物とをハイブリダイゼーションさせるとともに、陰性コントロールプローブと陰性コントロール核酸鎖とをハイブリダイゼーションさせて、マーカー遺伝子検出用プローブが固定された位置で測定された標識強度と陰性コントロールプローブの位置で測定された標識強度との対比の結果を用いて胃癌の予後を判定する。これにより、植物由来の核酸鎖が陰性コントロールとなるため、検査対象検体と早期胃癌検体とを同時に測定することなく、あらかじめ定めた閾値に基づき、胃癌の予後再発の判定を正確に行うことができる。したがって、簡便かつ実用的な胃癌の予後判定が実現可能になる。
【発明の効果】
【0015】
本発明によれば、簡便かつ精度良く胃癌の再発を判定できる実用的な方法が提供される。
【図面の簡単な説明】
【0016】
【図1】実施の形態に係る胃癌の再発を判定する方法を説明するフローチャートである。
【図2】実施の形態で用いるDNAマイクロアレイのレイアウトの一例を示す図である。
【発明を実施するための形態】
【0017】
以下、本発明の実施の形態について説明する。
【0018】
図1は、本実施形態の胃癌の再発を判定する方法を説明するフローチャートである。本実施形態は、胃癌試料中に含まれる胃癌細胞に特異的なマーカー遺伝子の少なくとも一部の領域をPCRにより増幅するとともに標識するステップ(S104)と、マーカー遺伝子の標識断片を取得するステップ(S105)と、マーカー遺伝子の標識断片と、植物由来の陰性コントロール核酸鎖とを混合させるステップ(S106)と、マーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブとが所定の位置にそれぞれ固定された担体に、マーカー遺伝子の標識断片と陰性コントロール核酸鎖との混合物を接触させるステップ(S107)と、マーカー遺伝子検出用プローブとマーカー遺伝子の標識断片とをハイブリダイズさせるとともに、陰性コントロールプローブと陰性コントロール核酸鎖とをハイブリダイズさせるステップ(S108)と、S108の後に、マーカー遺伝子検出用プローブが固定された位置、及び、陰性コントロールプローブが固定された位置の標識強度をそれぞれ測定するステップ(S109)と、マーカー遺伝子検出用プローブが固定された位置で測定された標識強度と陰性コントロールプローブが固定された位置で測定された標識強度とを対比し、対比の結果が所定の閾値を超えたとき、胃癌試料が陽性であると判定するステップ(S110)と、を含む。
【0019】
本実施形態において、植物由来の陰性コントロール核酸鎖としては、例えば、シロイヌナズナ、イネ、コムギ、ミヤコグサ、タバコなどの高等植物由来の核酸鎖を用いることができるが、中でもシロイヌナズナ由来の核酸鎖が好ましい。
【0020】
以下、陰性コントロール核酸鎖として、シロイヌナズナ由来のDNA鎖(以下、「シロイヌナズナDNA鎖」ともいう。)を用いる例を挙げて各ステップについて具体的に説明する。
まず、本実施形態の方法を実行するための胃癌再発判定用キットを用意する。このキットは、少なくとも一以上のマーカー遺伝子検出用プローブと、シロイヌナズナDNA鎖を検出する配列番号1で表される塩基配列からなるシロイヌナズナ遺伝子検出用プローブ(以下、「シロイヌナズナプローブ」ともいう。)とが所定の位置にそれぞれ固定された担体と、シロイヌナズナDNA鎖とを備えている。
【0021】
マーカー遺伝子検出用プローブは、好ましくは、6種の腫瘍マーカー遺伝子に対し、それぞれ特異的な配列を有するものである。6種の腫瘍マーカー遺伝子は、具体的には、CEA(がん胎児性抗原)、TFE1(trefoil factor family 1)、FABP1(Fatty acid binding protein 1)、CK20(Cytokeratin. 20)、MUC2(腸型ムチン)及びTACSTD1(tumor−associated calcium signal transducer 1)である。また、マーカー遺伝子を検出するためのプローブとしては、表1に示すものが例示できる。具体的には、配列番号2で表される塩基配列からなるCEA検出用プローブと、配列番号3で表される塩基配列からなるTFF1検出用プローブと、配列番号4で表される塩基配列からなるFABP1検出用プローブと、配列番号5で表される塩基配列からなるCK20検出用プローブと、配列番号6で表される塩基配列からなるMUC2検出用プローブと、配列番号7で表される塩基配列からなるTACSTD1検出用プローブである。
【0022】
【表1】

【0023】
担体としては基板、ビーズ、繊維など様々な担体を用いることができる。基板の形状は板状には限られず、たとえばフィルム状やシート状であってもよい。また、基板は、一つの部材から構成されていてもよいし、複数の部材から構成されていてもよい。担体の材質としては、金属、ガラス、プラスチック、ポリマー、繊維等、を用いることができる。高感度で、かつ、同時に多項目の遺伝子を検出する方法としては、基板に検出用プローブを固定化したDNAマイクロアレイが挙げられる。このDNAマイクロアレイには、基板上の一定の区画内に複数のスポットを設けておき、各スポットにプローブをそれぞれ固定化しておき、マイクロアレイを形成しておくことが好ましい。
【0024】
また、担体としては、高分子物質からなるコーティング層を表面に含む基板を用いてもよい。高分子物質からなるコーティング層を表面に含む基板は、所定の形状に加工された基板の表面に高分子物質を含む液体を塗布し、乾燥することにより得ることができる。また、高分子物質を含む液体中に基板を浸漬し、乾燥してもよい。
【0025】
担体として「高分子物質からなるコーティング層を表面に含む基板」を用いる場合、高分子物質としては、例えば、「リン脂質の親水部を構成するリン酸エステルより誘導される基を有する第一単位とカルボン酸誘導基を有する第二単位とを含む高分子物質」を用いることができる。「リン脂質の親水部を構成するリン酸エステルより誘導される基を有する第一単位とカルボン酸誘導基を有する第二単位とを含む高分子物質」は、例えば、特開2006−187270の段落0033〜0066に記載のものを用いることができるが、中でも、下記一般式(2)で表される、2−メタクリロイルオキシエチルホスホリルコリン(MPC)基を有する第一単量体と、p−ニトロフェニルオキシカルボニルポリエチレングリコールメタクリレート(NPMA)基を有する第二単量体と、ブチルメタリレート(BMA)基を有する第三単量体との共重合体であるpoly(MPC−co−BMA−co−NPMA)(PMBN)が特に好ましい。
【0026】
【化1】

【0027】
ただし、上記一般式(1)において、a、b、およびcは、それぞれ独立して、正の整数であり、aが5〜200、bが10〜500、cが1〜100であることが好ましい。また、上記一般式(2)において、第一〜第三単量体がブロック共重合していてもよいし、これらの単量体がランダムに共重合していてもよい。
【0028】
また、「高分子物質からなるコーティング層を表面に含む基板」を用いる場合、高分子物質からなるコーティング層を形成させる基板としては、プラスチック基板が、形状やサイズの変更に対する柔軟性が確保される上に、ガラス基板のものに比べて安価で提供することができるという観点から好ましい。このようなプラスチック材料としては、表面処理の容易性および量産性の観点から、熱可塑性樹脂を用いることができる。
【0029】
熱可塑性樹脂としては、蛍光発生量の少ないものを用いることができる。蛍光発生量の少ない樹脂を用いることにより、DNA鎖の検出反応におけるバックグランドを低下させることができるため、検出感度をさらに向上させることができる。蛍光発生量の少ない熱可塑性樹脂としては、たとえば、ポリエチレン、ポリプロピレン等の直鎖状ポリオレフィン;環状ポリオレフィン;含フッ素樹脂;等を用いることができる。上記樹脂の中でも、飽和環状ポリオレフィンは、耐熱性、耐薬品性、低蛍光性、透明性および成形性に特に優れるため、光学的な分析に好適であり、基板の材料として好ましく用いられる。
【0030】
ここで、飽和環状ポリオレフィンとは、環状オレフィン構造を有する重合体単独または環状オレフィンとα−オレフィンとの共重合体を水素添加した飽和重合体を指す。前者の例としては、たとえばノルボルネン、ジシクロペンタジエン、テトラシクロドデセンに代表されるノルボルネン系モノマー、及び、これらのアルキル置換体を開環重合して得られる重合体を水素添加して製造される飽和重合体である。後者の共重合体はエチレンやプロピレン、イソプロピレン、1−ブテン、3−メチル−1−ブテン、1−ペンテン、3−メチル−1−ペンテン、1−ヘキセン、1−オクテン等のα−オレフィンと環状オレフィン系モノマーのランダム共重合体を水素添加することにより製造される飽和重合体である。共重合体では、エチレンとの共重合体が最も好ましい。これらの樹脂は単独で用いてもよく、2種類またはそれ以上の共重合体あるいは混合物であってもよい。また、環状オレフィン構造を有する単量体が開環重合して得られる飽和環状ポリオレフィンだけでなく、環状オレフィン構造を有する単量体の付加重合により得られる飽和環状ポリオレフィンを用いることもできる。
【0031】
担体へのシロイヌナズナプローブ及びマーカー遺伝子検出用プローブ(以下、単に「プローブDNA」ともいう。)の固定化方法は、特に限定されないが、担体として、PMBNのコーティング層を表面に含む基板を用いる場合、例えば、PMBNに含まれるp−ニトロフェニルオキシカルボニル基とプローブDNAとを反応させて共有結合を形成させることにより、基板表面でプローブDNAを固定化することができる。
【0032】
プローブDNAを基板上に固定化する際には、プローブDNAを溶解または分散した液体を点着する方法が好ましい。このプローブDNAを溶解または分散した液体は、例えば中性からアルカリ性、例えばpHが7.6以上とすることができる。
【0033】
また、点着後、基板表面に固定化されなかったプローブDNAを除去するため、純水や緩衝液で洗浄してもよい。洗浄後はプローブDNAを固定化した以外の基板表面のp−ニトロフェニルオキシカルボニル基の不活性化処理をアルカリ化合物、あるいは一級アミノ基を有する化合物で行ってもよい。
【0034】
また、基板に固定化するプローブDNAには、p−ニトロフェニルオキシカルボニル基との反応性を高めるため、アミノ基を導入しておくことが好ましい。アミノ基はp−ニトロフェニルオキシカルボニル基との反応性に優れるため、アミノ基が導入されたプローブDNAを用いることにより、効率よくかつ強固に基板の表面上にプローブDNAを固定化することができる。アミノ基の導入位置はプローブDNAの分子鎖末端あるいは側鎖であってもよいが、分子鎖末端に導入されていることが、相補的な鋳型DNA断片とのアニーリングをより一層効率よく行うことができるという観点からは、好ましい。
【0035】
以上により、基板の表面上に表1に示すプローブが固定化されたDNAマイクロアレイを得ることができる。
【0036】
DNAマイクロアレイによる発現解析を行う場合には、より確度の高い判定を行うため、内部コントロールや外部コントロールを用いることが好ましい。本実施形態では、内部コントロールとして、ACTB(ベータアクチン)を用いることができる。また、外部コントロールとしては、陰性コントロールや陽性コントロールを用いることができ、上述のとおり本実施形態では、陰性コントロールとしてシロイヌナズナDNA鎖を用いることができ、陽性コントロールとしてλDNA鎖を用いることができる。ACTBを内部コントロールとして用いる場合は、配列番号8で表される塩基配列からなるACTB検出用プローブを内部コントロールプローブとして担体に固定させることができる。ACTB検出用プローブの固定化もシロイヌナズナプローブやマーカー遺伝子検出用プローブの固定化と同様に行うことができる。
【0037】
胃癌再発判定用キットには、マーカー遺伝子の一部の領域を増幅するためのプライマーセットを備えることが好ましい。こうすることで、審査腹腔鏡時の洗浄液もしくは術中腹腔内洗浄液から抽出したmRNAが少ない場合であっても、増幅させて有効な判定が可能になる。表1で示すプローブによって検出される各マーカー遺伝子のプライマー塩基配列を表2に示す。具体的には、配列番号10、11で表される塩基配列からなるCEA増幅用プライマーセット、配列番号12、13で表される塩基配列からなるTFE1増幅用プライマーセット、配列番号14、15で表される塩基配列からなるFABP1増幅用プライマーセット、配列番号16、17で表される塩基配列からなるCK20プライマーセット、配列番号18、19で表される塩基配列からなるMUC2プライマーセット、及び、配列番号20、21で表される塩基配列からなるTACSTD1プライマーセットが挙げられる。また、内部コントロールとしてACTBを用いる場合は、配列番号22、23で表される塩基配列からなるプライマーセットにより、ACTBの一部の領域を増幅させてもよい。なお、表2には、表1に示すプライマーにより増幅されるPCR産物の塩基長も併せて示す。
【0038】
【表2】

【0039】
このようにして得られる胃癌再発判定用キットを用意した後、研究使用目的に対するインフォームド・コンセントが得られた胃癌患者から腹腔内洗浄液を回収し(S101)、これを遠心して浮遊成分である遊離細胞をペレット状に沈殿させた後、そのペレットからmRNAを抽出する(S102)。mRNAの抽出方法は各種の方法が考案され、多くのメーカーによりキット化されており、本実施形態においてはいずれのキットも使用可能である。
【0040】
ついで、胃癌試料中に含まれるmRNAは増幅させるため、RT−PCRを行う。具体的には、まず、逆転写酵素を用いた逆転写反応(Reverse Transcription、RT)により、抽出したmRNAを鋳型として、例えば、ランダムヘキサマーやオリゴdTプライマー等のプライマーの存在下でデオキシヌクレオチド三リン酸(dNTP)を基質とし、抽出したmRNAに相補的なcDNAを5'→3'方向に合成する。(S103)。
【0041】
その後、得られたcDNAを用いてPCR増幅反応を行う(S104)。具体的には、合成したcDNA、配列番号10〜25で表される塩基配列からなるプライマー、DNAポリメラーゼ及びdNTPと、DNAポリメラーゼが働く至適塩濃度環境を作るためのバッファー溶液とを混合し、市販のPCR装置にセットする。使用するDNAポリメラーゼは、耐熱性を有するものであればよい。
【0042】
その後、(1)cDNAの熱変性(94〜96℃)、(2)配列番号10〜25で表される塩基配列からなるプライマーのアニーリング(55〜60℃)、(3)DNAポリメラーゼによる相補鎖の合成(72〜74℃)の3段階の温度変化を、例えば、20〜30サイクル繰り返し、cDNAを増幅する。
【0043】
ここで、PCR増幅反応は、dNTPとともに、ラベル化したヌクレオチドモノマーを用いて実行する。ラベル方法は、蛍光体、光吸収体、放射性核種(P32)、酵素標識などの方法が挙げられる。
例えば、蛍光ラベル化ヌクレオチドモノマーとしては、dTTPの塩基の3位を蛍光ラベルしたCy3−dUTPが挙げられる。Cy3−dUTPを用いた場合、鋳型DNA断片のアデニン(A)に対応する伸長(プライマー)側の位置にCy3−dUTPが挿入されたPCR産物を得ることができる。
また、放射性核種によるラベル化ヌクレオチドモノマーとしては、P32−ATP、P32−dATPが挙げられる。
また、アミノアリルdUTPを用いてPCR増幅反応をし、反応後に、アミノアリルdUTPを含むPCR産物に対して、Cy3蛍光色素やアミノ基染色試薬(NHS−試薬)を用いて発色させてもよい。
酵素標識の方法においては、ビオチン(biotin)化またはジゴキシゲニン(DIG:ステロイド系天然物)を結合した核酸(例えば、biotin−dUTP、DIG−dUTP)を使用して増幅した後、得られるPCR産物にアルカリフォスファターゼまたはアルカリフォスファターゼ処理しニトロブルーテトラゾリウム(NBT)と5−ブロモ−4−クロロ−3−インドリルリン酸(BCIP)液中で数時間反応させて発色させることもできる。
【0044】
なお、S104では、逆転写反応で得られたcDNAとともに、陽性コントロール核酸鎖をバッファー溶液に混合して、増幅反応を行ってもよい。陽性コントロール核酸鎖としては、微生物由来の核酸鎖を用いることができ、具体的にはバクテリオファージ等のウイルス由来のものが挙げられる。陽性コントロールDNA鎖としてλDNA鎖を用いる場合、表2で示す配列番号24及び25で表される塩基配列からなるプライマーを用いてλDNA鎖の一部を増幅させることができる。この場合、λDNA鎖を検出するため、表1で示す配列番号9で表される塩基配列からなるλDNA検出用プローブをあらかじめ担体に固定しておく。このλDNA検出用プローブは、配列番号24及び25で表される塩基配列からなるプライマーにより増幅されるλDNA鎖の断片と相補的に結合できるように設計されている。
【0045】
このようにして、標識したPCR産物(標識DNA断片)を取得した後(S105)、得られた標識DNA断片とシロイヌナズナDNA鎖とを混合する(S106)。標識DNA断片とシロイヌナズナDNA鎖との混合は、ハイブリダイゼーションバッファー中で行うことが好ましい。ハイブリダイゼーションバッファーは、公知のものを用いることができるが、例えば、SSC又はSSPEと、SDSと、ホルムアミドとの混合液を用いることができる。
【0046】
ついで、混合した標識DNA断片及びシロイヌナズナDNA鎖を、ハイブリダイゼーションバッファー、及び、プローブDNAが固定化されたDNAマイクロアレイとともにハイブリダイゼーション装置にセットし(S107)、例えば、45℃〜60℃で3〜24時間、好ましくは3〜5時間、ハイブリダイゼーションする(S108)。
【0047】
その後、マイクロアレイの洗浄後に、市販のマイクロアレイスキャナーを用いて標識強度を測定する(S109)。S104において、微生物由来のDNA鎖、特に、λDNAを用いてマーカー遺伝子とともにPCR増幅させた場合は、λDNA鎖の標識強度を基準として各マーカー遺伝子の標識強度を測定してもよい。具体的には、λDNA検出用プローブが固定された位置における標識強度が所定の範囲になるように検出感度を制御する。こうすることで、アッセイ間のバラツキを補正することができる。
【0048】
また、得られた標識強度の実測値に種々の補正をかけ、補正された実測値を真の測定値としてもよい。例えば、プローブDNAが固定化されていない担体表面の標識強度をバックグラウンドとして測定結果から差し引いて、測定値としてもよい。
【0049】
その後、得られた測定値に基づき胃癌試料の術後再発の判定を行う(S110)。DNAマイクロアレイによる発現解析を行う場合には、各プローブ、判定対象となるmRNAごとにしかるべき規格化を行い、各サンプル間で比較可能となる調整を行うことで、より確度の高い判定を行うことが出来る。
【0050】
具体的には、各遺伝子の発現量(発現レベル)のカットオフ値を設定し、カットオフ値を上回った遺伝子数をカウントし、定めた数を上回るか下回るかにより判定を行う方法が有効である。そこで、本実施形態では、シロイヌナズナDNA鎖を陰性コントロールとし、これを基準として、カットオフ値(閾値)を導き出す。
【0051】
閾値を導き出す方法は、医療機関において実際の検体を用いた解析により、経験的に、マーカー遺伝子ごとに、所定の閾値を定める。具体的には、臨床の所見やその他の測定値を合わせて所定の閾値が定められる。そして、シロイヌナズナ検出用プローブが固定された基板の位置における標識強度を基準として、各マーカー遺伝子の標識強度の相対値を算出する。各マーカー遺伝子の標識強度の相対値の算出法としては、シロイヌナズナ検出用プローブが固定された基板の位置における標識強度を1として、各マーカー遺伝子の標識強度の相対値を算出する方法や、シロイヌナズナ検出用プローブが固定された基板の位置における標識強度を0として、各マーカー遺伝子の標識強度の相対値を算出する方法が挙げられる。そして、1以上のマーカー遺伝子の標識強度の相対値が閾値を超えた場合に、結果を陽性、すなわち、予後不良(再発する、又は、再発の可能性が大きい)と判定する。一方、すべてのマーカー遺伝子の標識強度の相対値が閾値以下である場合は、結果を陰性、すなわち、予後良好(再発しない、又は、再発の可能性が小さい)と判定する。なお、陽性あるいは陰性の判定手段としては、これに限定されず、設定された閾値を超えたマーカー遺伝子が2以上の所定数あった場合に陽性と判断し、設定された閾値を超えたマーカー遺伝子がこの所定数に満たない場合を陰性と判定してもよいし、ある特定のマーカー遺伝子の標識強度の相対値が閾値を超えた場合に陽性と判断し、この特定のマーカー遺伝子の標識強度の相対値が閾値以下であった場合に陰性と判断してもよい。
【0052】
また、S104において、内部コントロール(ACTB)や陽性コントロール(λDNA)を用いてPCR増幅を実行した場合は、ACTBやλDNAの測定結果も併せて考察することで、S102の抽出、又は、S108のハイブリダイゼーションがそれぞれ良好に進行したかどうかを判別することができる。具体的には、ACTBの結果が陰性であり、かつ、λDNAの結果が陰性である場合は、S102の抽出、及び、S108のハイブリダイゼーションのいずれもが不良であったことがわかる。また、ACTBの結果が陽性である一方、λDNAの結果が陰性であった場合は、S102の抽出は良好に進行したが、S108のハイブリダイゼーションの進行が不良であったことがわかる。ACTBの結果が陰性であり、かつ、λDNAの結果が陽性である場合は、S104のPCRは良好に進行したが、S102の抽出の進行が不良であったことが分かる。
【0053】
なお、本実施形態の判定方法とともに、通常、外科的切除治療症例、審査腹腔鏡症例において必須となっている細胞診の結果に加えて、リンパ節転移の有無、各種の癌マーカーなどの数値を参照し、状況によっては判定方法を使い分けるなど、総合的に胃癌細胞の有無を判断するとより効果的な判定が可能である。
【0054】
つづいて、本実施形態の作用効果について説明する。本実施形態によれば、植物由来の陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブを担体に固定し、マーカー遺伝子検出用プローブと胃癌試料のPCR産物とをハイブリダイゼーションさせるとともに、陰性コントロールプローブと陰性コントロール核酸鎖とをハイブリダイゼーションさせて、マーカー遺伝子検出用プローブが固定された位置で測定された標識強度と陰性コントロールプローブの位置で測定された標識強度との対比の結果を用いて胃癌の予後を判定する。これにより、植物由来の核酸鎖が陰性コントロールとなるため、検査対象検体と早期胃癌検体とを同時に測定することなく、あらかじめ定めた閾値に基づき、胃癌の予後再発の判定を正確に行うことができる。したがって、簡便かつ実用的な胃癌の予後判定が実現可能になる。
【0055】
従来、胃癌の予後判定においては、対象検体と同時に、陰性コントロールとして腹腔内に癌細胞が存在しないものと考えられるIa期の早期癌検体を用いた測定を行い、早期癌検体の結果からカットオフ値を導き出す方法がとられていた。しかしながら、検査ごとに早期癌検体を用意しなければならず、実用性や倫理面で問題があった。
【0056】
一方、本実施形態では、植物由来のDNA鎖を陰性コントロールとして用いて、精度よく胃癌の予後を判定できる。したがって、検査ごとに早期癌検体を用意する必要がなくなり、実用的である。特にシロイヌナズナDNA鎖を陰性コントロールとして用いることで、マーカー遺伝子との反応を無視できるレベルにすることができ、より精度よく胃癌の予後を判定することができる。なお、シロイヌナズナ遺伝子は、交差反応性が低いことが確認されている。また、シロイヌナズナDNA鎖は、入手が容易な点でもメリットがある。
【実施例】
【0057】
以下、具体的な実施例を示し、本発明をさらに詳細に説明する。
【0058】
<実施例1>
(1)プラスチック基板の製造
飽和環状ポリオレフィン樹脂(5−メチル−2−ノルボルネンの開環重合体の水素添加物(MFR(Melt flow rate):21g/10分、水素添加率:実質的に100%、熱変形温度123℃)を用い、射出成形によりスライドガラス形状の基板を得た。得られた基板を上記式(1)で表されるPMBNポリマー(MPC基:BMA基:NPMA基=23:74:3(モル比))の0.3重量%エタノール溶液に浸漬することにより、基板表面にホスホリルコリン基と活性エステル基とを有する高分子物質を導入して、プラスチック基板を得た。なお、MFRは、JIS K 7210:1999法記載の条件に準じて測定したものである。
【0059】
(2)プローブDNA
5'末端がアミノ基で修飾された、胃癌再発に関するマーカー遺伝子6種類の特異配列を有するオリゴDNA鎖、およびコントロール遺伝子3種類を用意した。用意したプローブは表1に記載した9種類の遺伝子に対応したプローブである。各遺伝子に対応したプローブの配列番号は、CEA検出用として配列番号2、TFF1検出用として配列番号3、FABP1検出用として配列番号4、CK20検出用として配列番号5、MUC20検出用として配列番号6、TACSTD1検出用として配列番号7である。また、内部コントロールとして配列番号8(ACTB鎖検出用)、陰性コントロールとして配列番号1(シロイヌナズナDNA鎖検出用)、陽性コントロールとして配列番号9(λDNA鎖検出用)である。
【0060】
(3)基板へのプローブDNA固定
スポッティングバッファー(製品名:×2スポッティング液、住友ベークライト社製)を用いて、本実施例の(2)で用意したプローブDNAを溶解し、10μMのプローブDNA溶液を調製した。この溶液をスポッター(日立ソフトウェアエンジニアリング社製MARKS−I)を用い、300μm径スポットピンで、本実施例の(1)で作製したプラスチック基板(縦75mm×横25mm)の表面上にスポットした。スポットは、各プローブについて、N=5でスポットした。プローブDNAをスポットした基板を、80℃で1時間加熱して、プローブDNAを固定化させた。これによりプラスチック基板のポリマー表面に一本鎖DNAが固定したDNAマイクロアレイを得た。スポットのレイアウトを図2に示す。
【0061】
(4)mRNAの回収
研究使用目的に対するインフォームド・コンセントが得られた胃癌術中患者に対し、通常行われる術式に従って500mLの生理食塩水を用いて腹腔内洗浄を行い、洗浄液を回収した。回収された腹腔内洗浄液のうち病理部に検査検体として提出するのに必要な分量を抜き取った余剰検体を検査対象検体とし、1500rpmで10分間遠心し、上清を除去することによりペレット状になった沈殿成分を腹腔内浮遊細胞として回収した。回収した細胞を培養し、得られた細胞をニッポンジーン社製試薬Isogenに加えてホモジナイズし、少量のクロロホルムを入れ、9420rpmで15分間遠心分離させ、上清を採取した。さらに採取量と等量のイソプロパノールを加え、10分以上インキュベートした後、9420rpmで10分間遠心して、ペレットを回収し、エタノール沈殿(70%)によりmRNAを得た。このmRNAを189症例分用意し、進行癌サンプルとした。
【0062】
(5)逆転写反応
逆転写反応を行うことにより上記抽出したmRNAからcDNAを作製した。
上記抽出したmRNAを1μg/mLに濃度調製し、調製したmRNA1μLと、20μMのランダムヘキサマー(製品名:Random primer、タカラバイオ社製)1μLと、10mMのdNTP(GEヘルスケア社製)1μLと、精製水9μL、逆転写酵素(Invitrogen社製、SuperScriptII)1μL、及び、Rnase Inhibitor(Roch社製)1μLを加え、42℃で50分加熱保持さらに72℃で15分加熱保持させ、一本鎖cDNAを得た。
【0063】
(6)マルチプレックスPCR反応
腹腔内洗浄液から得た一本鎖cDNAを鋳型に検査対象となるマーカー遺伝子のcDNAを増幅するためのPCRを行った。同一反応中に複数のプライマーを用いるマルチプレックスPCR法によりcDNAの増幅を行った。同一のcDNAに対し、プライマーとして表2に示す配列番号10〜25の塩基配列からなるプライマーセットを用いた。
【0064】
表2に示す配列番号10〜25の塩基配列からなるプライマーをDEPC−Treated Water中に各々濃度10μMで含むプライマー溶液を0.5μL、DNAポリメラーゼ(Invitrogen社製AcuuPrime)0.5μL、λDNA鎖(タカラバイオ社製)をPCRバッファー(Invitrogen社製AcuuPrimeのキット内に含まれる)中に溶解させ20μLの反応液を調製した。また、PCR産物の標識のために、アミノアリルdUTP(Ambion社製)0.1μLを基質として、PCRの反応溶液に加えた。
【0065】
調製された反応液について、市販のサーマルサイクラー(製品名:GeneAmp(登録商標)PCRSystem 9700、Applied biosystem社製)を用いて、(i)熱変性95℃15秒、(ii)アニーリング60℃45秒、(iii)DNA鎖の伸長反応72℃3分のヒートサイクルで、25サイクル行い、アミノアリル標識PCR産物を得た。
【0066】
精製は、チップごとにDNA精製キット(キアゲン社製QIA quick)を使用して上記PCR産物の精製を行った。
【0067】
(7)Cy3標識
得られたアミノアリル標識PCR産物に対して、Cy3標識を行った。具体的には、本実施例の(6)で得られた調製ずみのアミノアリル標識PCR産物を蛍光ラベル化試薬(GEヘルスケア社製Cy3 Mono−reactive Dye Pack)を使用してCy3蛍光ラベル化し、さらにDNA精製キット(キアゲン社製QIA quick)を使用して精製を行い、Cy3蛍光ラベル化PCR産物を得た。
【0068】
(8)ハイブリダイゼーション
Cy3蛍光ラベル化PCR産物45μLに対し、バッファー85μLを加え、さらに、シロイヌナズナDNA鎖(SIGMA GENOSYS社製)を加え、ハイブリダイゼーション溶液を得た。得られたハイブリダイゼーション溶液の組成は、以下のとおりである。
[ハイブリダイゼーション溶液]
・3×SSPE
・10%ホルムアミド
・ 0.05%SDS
(3)で作製したDNAマイクロアレイをハイブリダイゼーション装置(Genomic Solutions社製Hyb4)にセットし、上記組成のハイブリダイゼーション溶液を用いてハイブリダイゼーションを行った。具体的には、95℃で5分熱変性させたハイブリダイゼーション溶液を実施例1で作製したDNAマイクロアレイ上に分注し、55℃で4時間反応を行った。ハイブリダイゼーション反応終了後、0.1%SDSを含んだ2×SSC溶液、2×SSC、0.1×SSCの順でマイクロアレイの表面を洗浄して、ハイブリダイゼーション反応を終了した。
【0069】
(9)測定
本実施例(8)で行ったハイブリダイゼーション反応終了後、スピンドライしたDNAマイクロアレイについて、DNAマイクロアレイ用スキャナー(Axon社製GenePix4000B)を用いて、プローブがスポットされた位置ごとに蛍光強度を測定した。測定する際の受光感度(PMT)は、λDNAが固定されたスポットにおける蛍光強度が900から1100となるように調整した。蛍光強度の算出にあたっては、プローブごとに5個の測定値が得られるが、最高値及び最低値を除く中間の3個の測定値を平均化して、プローブごとに平均値を算出し、これを蛍光強度とした。進行癌サンプル全189症例について蛍光強度を測定した。
【0070】
(10)相対値を用いた再発予後判定
得られた蛍光強度の結果について、シロイヌナズナDNA鎖検出用プローブがスポットされた位置の蛍光強度を基準として各プローブが固定された位置における蛍光強度の比を算出した結果を表3−6に示す。また、各マーカー遺伝子の蛍光強度の相対値にそれぞれ閾値を設定し、その閾値を越えた場合には、陽性とし、下回る場合は、陰性とした。この閾値は、一般的なDNAマイクロアレイのカットオフ値の設定が通常バックグランドの3倍であることに基づき設定した。TFF1、FABP1、CK20、及び、MUC2については、CEAやTACSTDに比較して発現量が低く、また癌特有に発現するものであるため、これらの閾値は、CEAやTACSTDの閾値の半分に設定した。具体的には、進行癌サンプルのマーカー遺伝子の閾値は、下記のとおり設定した。
CEA 閾値:3
TFF1 閾値:1.5
FABP1 閾値:1.5
CK20 閾値:1.5
MUC2 閾値:1.5
TACSTD 閾値:3
進行癌サンプル全189症例について陽性と判断されたマーカー遺伝子の数を表3−6に併せて示す。
【0071】
【表3】

【0072】
【表4】

【0073】
【表5】

【0074】
【表6】

【0075】
<実施例2>
実施例1の(1)〜(8)の操作を行った。
ハイブリダイゼーション反応終了後、実施例1の(9)で示すように、プローブがスポットされた位置ごとに蛍光強度を測定した。得られた189症例の蛍光強度について、シロイヌナズナDNA鎖検出用プローブがスポットされた位置の蛍光強度をバックグラウンド値として、各プローブが固定された位置における蛍光強度からシロイヌナズナDNA鎖検出用プローブがスポットされた位置の蛍光強度を差し引いた結果を表7−10に示す。また、各マーカー遺伝子の蛍光強度の相対値にそれぞれ閾値を設定し、その閾値を越えた場合には、陽性とし、下回る場合は、陰性とした。この閾値は、実施例1で説明したように、一般的なDNAマイクロアレイのカットオフ値の設定が通常バックグランドの3倍であることに基づきCEA及びTACSTDの閾値を設定し、TFF1、FABP1、CK20、及び、MUC2については、CEA及びTACSTDの閾値の約半分に設定した。そして、閾値を越えた場合には、陽性とし、下回る場合は、陰性とした。具体的には、進行癌サンプルのマーカー遺伝子の閾値は、下記のとおり設定した。
CEA: 130
TFF1: 70
FABP1: 70
CK20: 70
MUC2: 70
TACSTD1: 130
進行癌サンプル全189症例について陽性と判断されたマーカー遺伝子の数を表7−10に併せて示した。
【0076】
【表7】

【0077】
【表8】

【0078】
【表9】

【0079】
【表10】

【0080】
<比較例1>
(1)DNAマイクロアレイの作製
実施例1の(1)〜(3)の操作にしたがって、DNAマイクロアレイを作製した。ただし、配列番号1で表される塩基配列からなるシロイヌナズナDNA鎖検出用プローブはDNAマイクロアレイに固定しなかった。
(2)進行癌サンプルとDNAマイクロアレイとのハイブリダイゼーション
実施例1の(4)〜(7)の操作を行い、得られた進行癌サンプル189症例のCy3蛍光ラベル化PCR産物を本比較例1の(1)で作製したDNAマイクロアレイにそれぞれハイブリダイズした。ハイブリダイゼーションの条件及び操作は、実施例1の(8)に従った。ただし、Cy3蛍光ラベル化PCR産物にシロイヌナズナDNA鎖は加えなかった。
(3)早期癌サンプルとDNAマイクロアレイとのハイブリダイゼーション
細胞診により早期癌と判定された早期癌細胞25症例について、実施例1の(4)〜(7)で示す方法と同様な操作を行ってCy3蛍光ラベル化PCR産物を用意し、比較例1の(1)で作製したDNAマイクロアレイにハイブリダイズした。この場合も、ハイブリダイゼーションの条件及び操作は、実施例1の(8)に従い、Cy3蛍光ラベル化PCR産物にシロイヌナズナDNA鎖は加えなかった。
(4)測定
ハイブリダイゼーション反応終了後、スピンドライしたDNAマイクロアレイについて、DNAマイクロアレイ用スキャナー(Axon社製GenePix4000B)を用いて、プローブがスポットされた位置ごとに蛍光強度を測定した。測定する際の受光感度(PMT)は、λDNAが固定されたスポットにおける蛍光強度が100から600となるように調整した。蛍光強度の算出にあたっては、プローブごとに5個の測定値が得られるが、最高値及び最低値を除く中間の3個の測定値を平均化して、プローブごとに平均値を算出し、これを蛍光強度とした。全189症例、及び、早期癌サンプル25症例について蛍光強度をそれぞれ測定した。
(5)再発予後判定
早期癌サンプルをハイブリダイズしたDNAマイクロアレイの測定結果25症例を用い、各マーカーについて、平均値+その標準偏差×2をカットオフ値とした。具体的なカットオフ値を以下に示す。
CEA: 56.9
TFF1: 54.0
FABP1: 55.3
CK20: 53.1
MUC2: 49.8
TACSTD1: 85.1
進行癌サンプル189症例について、カットオフ値を超えたマーカー遺伝子を陽性と判断し、カットオフ値以下のマーカー遺伝子は、陰性とした。蛍光強度の結果を表11−14に示す。
【0081】
【表11】

【0082】
【表12】

【0083】
【表13】

【0084】
【表14】

【0085】
<細胞診>
胃癌の術中腹腔内洗浄細胞診ガイドライン(日臨細胞誌44(1);93−97.2001)に基づいて、進行癌サンプル189症例について、細胞診により判定を行った。各症例について回収した腹腔内浮遊細胞をスライドガラス上に載せ、引きガラスを用いて塗抹標本を作製した。速やかに塗抹標本を風乾燥させ、その後、メタノールによって3分間固定させた。ギムザ液と純水を1:2の割合で混合させ、混合液を用いて標本の染色を行った。30分間染色させた後、純水で洗浄し、乾燥させて封じ、標本を完成させた。光学顕微鏡による標本観察で染色された癌細胞を探すことで、細胞診の判定を行った。細胞診では、クラス分類を行い、「1」は、陽性(再発する)とし、「0」は、陰性(再発しない)とした。結果を表19−26に示す。
【0086】
<リンパ節転移>
進行癌サンプル全189症例について、胃癌部とともに外科的に切除した周辺リンパ節の病理診断により、リンパ節転移の有無の診断を行った。結果を表19−26に示す。表19−22では、リンパ節転移がある場合を陽性、ない場合を陰性とした。
【0087】
また、進行癌サンプル全189症例の胃癌患者の術後経過については3年以上、平均4.5年にわたって追跡調査され、再発、及び死亡の有無の情報を得た。表15−18では、生死の結果も併せて示す。「alive」は再発することなく生存、「再発」は再発するが生存、「death」は、再発による死亡を示す。また、表19−22では、各実施例及び各比較例において、陽性と判断されたマーカー遺伝子の数も併せて示す。表19が実施例1の結果であり、表20が実施例2の結果であり、表21が比較例1の結果である。表19−21では再発した症例と死亡した症例数、及び、陽性あるいは陰性と判定された進行癌サンプル中の再発及び死亡症例数の割合(百分率)を各実施例及び各比較例の結果と対応づけて示す。表19−21では、各実施例及び各比較例で陽性マーカー遺伝子が1以上検出されたものを+で示し、全く検出されなかったものを−で示す。
【0088】
【表15】

【0089】
【表16】

【0090】
【表17】

【0091】
【表18】

【0092】
【表19】

【0093】
【表20】

【0094】
【表21】

【0095】
表19−21で示すように、進行癌サンプル189症例中、リンパ節転移が陽性の132症例において、細胞診により検出できなかった98症例中、実施例1,2、比較例1において陽性と判断された各28、27、33症例の再発率、死亡率は、陰性だった各70、71、65症例と比較して、ともに高く予後が悪かった。これら細胞診陰性・マイクロアレイ陽性症例において、実施例1、2の再発率は比較例1に比べて、各15、11ポイント高かった。死亡率では各17、16ポイント高かった。したがって、本発明によれば、早期癌患者の症例を同時に用いた従来の検査法よりも、いっそう精度よく胃癌の予後判定が行えることが明らかとなった。リンパ節転移陰性の57症例についても、症例数は少ないもののマイクロアレイ陽性症例の再発率、死亡率ともに陰性症例に比べて高かった。
【特許請求の範囲】
【請求項1】
胃癌試料中に含まれる胃癌細胞に特異的なマーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(Polymerase Chain Reaction、PCR)により増幅するとともに標識して前記マーカー遺伝子の標識断片を取得するステップと、
前記マーカー遺伝子の標識断片と、植物由来の陰性コントロール核酸鎖とを混合させるステップと、
前記マーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、前記陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブとが所定の位置にそれぞれ固定された担体に、前記マーカー遺伝子の前記標識断片と前記陰性コントロール核酸鎖との混合物を接触させて、前記マーカー遺伝子検出用プローブと前記マーカー遺伝子の前記標識断片とをハイブリダイズさせるとともに、前記陰性コントロールプローブと前記陰性コントロール核酸鎖とをハイブリダイズさせるステップと、
ハイブリダイズさせる前記ステップの後に、前記マーカー遺伝子検出用プローブが固定された位置、及び、前記陰性コントロールプローブが固定された位置の標識強度をそれぞれ測定するステップと、
前記陰性コントロールプローブが固定された位置で測定された前記標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置で測定された前記標識強度の結果が所定の閾値を超えたとき、前記胃癌試料が陽性であると判定するステップと、
を含むことを特徴とする胃癌の再発を判定する方法。
【請求項2】
前記陰性コントロール核酸鎖がシロイヌナズナ由来のDNA鎖であり、
前記陰性コントロールプローブが、配列番号1で表される塩基配列からなるシロイヌナズナ遺伝子検出用プローブである、請求項1に記載の方法。
【請求項3】
前記マーカー遺伝子がCEA、TFF1、FABP1、CK20、MUC20及びTACSTD1であり、
前記担体には、
配列番号2で表される塩基配列からなるCEA検出用プローブと、
配列番号3で表される塩基配列からなるTFF1検出用プローブと、
配列番号4で表される塩基配列からなるFABP1検出用プローブと、
配列番号5で表される塩基配列からなるCK20検出用プローブと、
配列番号6で表される塩基配列からなるMUC2検出用プローブと、
配列番号7で表される塩基配列からなるTACSTD1検出用プローブと、
が前記マーカー遺伝子検出用プローブとしてそれぞれ所定の位置に固定されており、
前記マーカー遺伝子の前記標識断片を取得する前記ステップにおいて、
配列番号10及び11で表される塩基配列からなるCEA増幅用プライマーと、
配列番号12及び13で表される塩基配列からなるTFF1増幅用プライマーと、
配列番号14及び15で表される塩基配列からなるFABP1増幅用プライマーと、
配列番号16及び17で表される塩基配列からなるCK20増幅用プライマーと、
配列番号18及び19で表される塩基配列からなるMUC2増幅用プライマーと、
配列番号20及び21で表される塩基配列からなるTACSTD1増幅用プライマーと、
を用いて前記胃癌試料中に含まれる前記マーカー遺伝子の少なくとも一部の領域を前記ポリメラーゼ連鎖反応により増幅する、請求項1又は2に記載の方法。
【請求項4】
前記マーカー遺伝子の前記標識断片を取得する前記ステップにおいて、前記マーカー遺伝子の少なくとも一部の領域を増幅しながら、微生物由来の陽性コントロール核酸鎖の少なくとも一部の領域を前記ポリメラーゼ連鎖反応により増幅して、前記マーカー遺伝子の標識断片とともに前記陽性コントロール核酸鎖の標識断片を取得し、
ハイブリダイズする前記ステップにおいて、前記陽性コントロコール核酸鎖の前記標識断片と相補的に結合する陽性コントロールプローブがさらに固定された前記担体に、前記陽性コントロール核酸鎖の前記標識断片をさらに含む前記混合物を接触させて、前記陽性コントロールプローブと前記陽性コントロール核酸鎖の前記標識断片とをハイブリダイズし、
前記標識強度を測定する前記ステップは、前記陽性コントロールプローブが固定された位置における標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置、及び、前記陰性コントロールプローブが固定された位置の前記標識強度をそれぞれ測定する、請求項1乃至3いずれか1項に記載の方法。
【請求項5】
前記陽性コントロール核酸鎖がλDNA鎖であり、
前記陽性コントロールプローブが、配列番号9で表される塩基配列からなるλDNA鎖検出用プローブであり、
前記マーカー遺伝子の前記標識断片を取得する前記ステップにおいて、前記マーカー遺伝子の少なくとも一部の領域とともに、配列番号24及び25で表される塩基配列からなるλDNA増幅用プライマーを用いて前記陽性コントロール核酸鎖の少なくとも一部の領域を前記ポリメラーゼ連鎖反応により増幅する、請求項4に記載の方法。
【請求項6】
前記マーカー遺伝子の前記標識断片を取得する前記ステップにおいて、蛍光標識された前記マーカー遺伝子の前記標識断片を取得し、
前記標識強度を測定するステップにおいて、前記マーカー遺伝子検出用プローブが固定された位置、及び、前記陰性コントロールプローブが固定された位置の蛍光強度をそれぞれ測定する、請求項1乃至5いずれか1項に記載の方法。
【請求項7】
胃癌細胞に特異的なマーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、陰性コントロールプローブとが所定の位置にそれぞれ固定された担体と、
植物由来の陰性コントロール核酸鎖と、
を備え、
前記陰性コントロールプローブは、前記陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合し、
胃癌試料中に含まれる前記マーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(Polymerase Chain Reaction, PCR)により増幅するとともに標識して、取得された前記マーカー遺伝子の標識断片を前記マーカー遺伝子検出用プローブとハイブリタイズさせるとともに、前記陰性コントロールプローブと前記陰性コントロール核酸鎖とをハイブリダイズさせ、ハイブリダイズ後に測定された前記陰性コントロールプローブが固定された位置の標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置の標識強度の結果が所定の閾値を超えたとき、前記胃癌試料が陽性であると判定することで胃癌の再発を判定するための胃癌再発判定用キット。
【請求項8】
前記陰性コントロール核酸鎖がシロイヌナズナ由来のDNA鎖であり、
前記陰性コントロールプローブが、配列番号1で表される塩基配列からなるシロイヌナズナ遺伝子検出用プローブである、請求項7に記載の胃癌再発判定用キット。
【請求項9】
前記マーカー遺伝子がCEA、TFF1、FABP1、CK20、MUC20、及びTACSTD1であり、
前記担体には、
配列番号2で表される塩基配列からなるCEA検出用プローブと、
配列番号3で表される塩基配列からなるTFF1検出用プローブと、
配列番号4で表される塩基配列からなるFABP1検出用プローブと、
配列番号5で表される塩基配列からなるCK20検出用プローブと、
配列番号6で表される塩基配列からなるMUC2検出用プローブと、
配列番号7で表される塩基配列からなるTACSTD1検出用プローブと、
が前記マーカー遺伝子検出用プローブとしてそれぞれ所定の位置に固定されており、
前記マーカー遺伝子の一部の領域を増幅するためのプライマーセットとして、
配列番号10及び11で表される塩基配列からなるCEA増幅用プライマーセットと、
配列番号12及び13で表される塩基配列からなるTFF1増幅用プライマーセットと、
配列番号14及び15で表される塩基配列からなるFABP1増幅用プライマーセットと、
配列番号16及び17で表される塩基配列からなるCK20増幅用プライマーセットと、
配列番号18及び19で表される塩基配列からなるMUC20増幅用プライマーセットと、
配列番号20及び21で表される塩基配列からなるTACSTD1増幅用プライマーセットと、
を備える、請求項7又は8に記載の胃癌再発判定用キット。
【請求項10】
微生物由来の遺伝子配列からなる陽性コントロール核酸鎖と、
前記陽性コントロール核酸鎖の少なくとも一部の領域を増幅させる陽性コントロール核酸鎖増幅用プライマーセットと、
をさらに備え、
前記担体には、陽性コントロールプローブが所定の位置に固定されており、
前記陽性コントロールプローブは、前記陽性コントロール核酸鎖増幅用プライマーセットにより増幅される前記陽性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する、請求項9に記載の胃癌再発判定用キット。
【請求項11】
前記陽性コントロール核酸鎖がλDNA鎖であり、
前記陽性コントロールプローブが配列番号9で表される塩基配列からなるλDNA検出用プローブであり、
前記陽性コントロール核酸鎖増幅用プライマーセットが、配列番号24及び25で表される塩基配列からなるλDNA鎖増幅用プライマーである、請求項10に記載の胃癌再発判定用キット。
【請求項1】
胃癌試料中に含まれる胃癌細胞に特異的なマーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(Polymerase Chain Reaction、PCR)により増幅するとともに標識して前記マーカー遺伝子の標識断片を取得するステップと、
前記マーカー遺伝子の標識断片と、植物由来の陰性コントロール核酸鎖とを混合させるステップと、
前記マーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、前記陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する陰性コントロールプローブとが所定の位置にそれぞれ固定された担体に、前記マーカー遺伝子の前記標識断片と前記陰性コントロール核酸鎖との混合物を接触させて、前記マーカー遺伝子検出用プローブと前記マーカー遺伝子の前記標識断片とをハイブリダイズさせるとともに、前記陰性コントロールプローブと前記陰性コントロール核酸鎖とをハイブリダイズさせるステップと、
ハイブリダイズさせる前記ステップの後に、前記マーカー遺伝子検出用プローブが固定された位置、及び、前記陰性コントロールプローブが固定された位置の標識強度をそれぞれ測定するステップと、
前記陰性コントロールプローブが固定された位置で測定された前記標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置で測定された前記標識強度の結果が所定の閾値を超えたとき、前記胃癌試料が陽性であると判定するステップと、
を含むことを特徴とする胃癌の再発を判定する方法。
【請求項2】
前記陰性コントロール核酸鎖がシロイヌナズナ由来のDNA鎖であり、
前記陰性コントロールプローブが、配列番号1で表される塩基配列からなるシロイヌナズナ遺伝子検出用プローブである、請求項1に記載の方法。
【請求項3】
前記マーカー遺伝子がCEA、TFF1、FABP1、CK20、MUC20及びTACSTD1であり、
前記担体には、
配列番号2で表される塩基配列からなるCEA検出用プローブと、
配列番号3で表される塩基配列からなるTFF1検出用プローブと、
配列番号4で表される塩基配列からなるFABP1検出用プローブと、
配列番号5で表される塩基配列からなるCK20検出用プローブと、
配列番号6で表される塩基配列からなるMUC2検出用プローブと、
配列番号7で表される塩基配列からなるTACSTD1検出用プローブと、
が前記マーカー遺伝子検出用プローブとしてそれぞれ所定の位置に固定されており、
前記マーカー遺伝子の前記標識断片を取得する前記ステップにおいて、
配列番号10及び11で表される塩基配列からなるCEA増幅用プライマーと、
配列番号12及び13で表される塩基配列からなるTFF1増幅用プライマーと、
配列番号14及び15で表される塩基配列からなるFABP1増幅用プライマーと、
配列番号16及び17で表される塩基配列からなるCK20増幅用プライマーと、
配列番号18及び19で表される塩基配列からなるMUC2増幅用プライマーと、
配列番号20及び21で表される塩基配列からなるTACSTD1増幅用プライマーと、
を用いて前記胃癌試料中に含まれる前記マーカー遺伝子の少なくとも一部の領域を前記ポリメラーゼ連鎖反応により増幅する、請求項1又は2に記載の方法。
【請求項4】
前記マーカー遺伝子の前記標識断片を取得する前記ステップにおいて、前記マーカー遺伝子の少なくとも一部の領域を増幅しながら、微生物由来の陽性コントロール核酸鎖の少なくとも一部の領域を前記ポリメラーゼ連鎖反応により増幅して、前記マーカー遺伝子の標識断片とともに前記陽性コントロール核酸鎖の標識断片を取得し、
ハイブリダイズする前記ステップにおいて、前記陽性コントロコール核酸鎖の前記標識断片と相補的に結合する陽性コントロールプローブがさらに固定された前記担体に、前記陽性コントロール核酸鎖の前記標識断片をさらに含む前記混合物を接触させて、前記陽性コントロールプローブと前記陽性コントロール核酸鎖の前記標識断片とをハイブリダイズし、
前記標識強度を測定する前記ステップは、前記陽性コントロールプローブが固定された位置における標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置、及び、前記陰性コントロールプローブが固定された位置の前記標識強度をそれぞれ測定する、請求項1乃至3いずれか1項に記載の方法。
【請求項5】
前記陽性コントロール核酸鎖がλDNA鎖であり、
前記陽性コントロールプローブが、配列番号9で表される塩基配列からなるλDNA鎖検出用プローブであり、
前記マーカー遺伝子の前記標識断片を取得する前記ステップにおいて、前記マーカー遺伝子の少なくとも一部の領域とともに、配列番号24及び25で表される塩基配列からなるλDNA増幅用プライマーを用いて前記陽性コントロール核酸鎖の少なくとも一部の領域を前記ポリメラーゼ連鎖反応により増幅する、請求項4に記載の方法。
【請求項6】
前記マーカー遺伝子の前記標識断片を取得する前記ステップにおいて、蛍光標識された前記マーカー遺伝子の前記標識断片を取得し、
前記標識強度を測定するステップにおいて、前記マーカー遺伝子検出用プローブが固定された位置、及び、前記陰性コントロールプローブが固定された位置の蛍光強度をそれぞれ測定する、請求項1乃至5いずれか1項に記載の方法。
【請求項7】
胃癌細胞に特異的なマーカー遺伝子を検出する少なくとも一以上のマーカー遺伝子検出用プローブと、陰性コントロールプローブとが所定の位置にそれぞれ固定された担体と、
植物由来の陰性コントロール核酸鎖と、
を備え、
前記陰性コントロールプローブは、前記陰性コントロール核酸鎖の少なくとも一部の領域に相補的に結合し、
胃癌試料中に含まれる前記マーカー遺伝子の少なくとも一部の領域をポリメラーゼ連鎖反応(Polymerase Chain Reaction, PCR)により増幅するとともに標識して、取得された前記マーカー遺伝子の標識断片を前記マーカー遺伝子検出用プローブとハイブリタイズさせるとともに、前記陰性コントロールプローブと前記陰性コントロール核酸鎖とをハイブリダイズさせ、ハイブリダイズ後に測定された前記陰性コントロールプローブが固定された位置の標識強度を基準として、前記マーカー遺伝子検出用プローブが固定された位置の標識強度の結果が所定の閾値を超えたとき、前記胃癌試料が陽性であると判定することで胃癌の再発を判定するための胃癌再発判定用キット。
【請求項8】
前記陰性コントロール核酸鎖がシロイヌナズナ由来のDNA鎖であり、
前記陰性コントロールプローブが、配列番号1で表される塩基配列からなるシロイヌナズナ遺伝子検出用プローブである、請求項7に記載の胃癌再発判定用キット。
【請求項9】
前記マーカー遺伝子がCEA、TFF1、FABP1、CK20、MUC20、及びTACSTD1であり、
前記担体には、
配列番号2で表される塩基配列からなるCEA検出用プローブと、
配列番号3で表される塩基配列からなるTFF1検出用プローブと、
配列番号4で表される塩基配列からなるFABP1検出用プローブと、
配列番号5で表される塩基配列からなるCK20検出用プローブと、
配列番号6で表される塩基配列からなるMUC2検出用プローブと、
配列番号7で表される塩基配列からなるTACSTD1検出用プローブと、
が前記マーカー遺伝子検出用プローブとしてそれぞれ所定の位置に固定されており、
前記マーカー遺伝子の一部の領域を増幅するためのプライマーセットとして、
配列番号10及び11で表される塩基配列からなるCEA増幅用プライマーセットと、
配列番号12及び13で表される塩基配列からなるTFF1増幅用プライマーセットと、
配列番号14及び15で表される塩基配列からなるFABP1増幅用プライマーセットと、
配列番号16及び17で表される塩基配列からなるCK20増幅用プライマーセットと、
配列番号18及び19で表される塩基配列からなるMUC20増幅用プライマーセットと、
配列番号20及び21で表される塩基配列からなるTACSTD1増幅用プライマーセットと、
を備える、請求項7又は8に記載の胃癌再発判定用キット。
【請求項10】
微生物由来の遺伝子配列からなる陽性コントロール核酸鎖と、
前記陽性コントロール核酸鎖の少なくとも一部の領域を増幅させる陽性コントロール核酸鎖増幅用プライマーセットと、
をさらに備え、
前記担体には、陽性コントロールプローブが所定の位置に固定されており、
前記陽性コントロールプローブは、前記陽性コントロール核酸鎖増幅用プライマーセットにより増幅される前記陽性コントロール核酸鎖の少なくとも一部の領域に相補的に結合する、請求項9に記載の胃癌再発判定用キット。
【請求項11】
前記陽性コントロール核酸鎖がλDNA鎖であり、
前記陽性コントロールプローブが配列番号9で表される塩基配列からなるλDNA検出用プローブであり、
前記陽性コントロール核酸鎖増幅用プライマーセットが、配列番号24及び25で表される塩基配列からなるλDNA鎖増幅用プライマーである、請求項10に記載の胃癌再発判定用キット。
【図1】


【図2】




【図2】


【公開番号】特開2013−102716(P2013−102716A)
【公開日】平成25年5月30日(2013.5.30)
【国際特許分類】
【出願番号】特願2011−247757(P2011−247757)
【出願日】平成23年11月11日(2011.11.11)
【出願人】(510097747)独立行政法人国立がん研究センター (35)
【出願人】(000002141)住友ベークライト株式会社 (2,927)
【Fターム(参考)】
【公開日】平成25年5月30日(2013.5.30)
【国際特許分類】
【出願日】平成23年11月11日(2011.11.11)
【出願人】(510097747)独立行政法人国立がん研究センター (35)
【出願人】(000002141)住友ベークライト株式会社 (2,927)
【Fターム(参考)】
[ Back to top ]

