腸内細菌科に属する微生物内で自律複製可能な新規プラスミド
【課題】 腸内細菌科に属する微生物の育種、改良に有用なプラスミドを提供する。
【解決手段】 パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、特定の配列からなるアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子を含むプラスミド。このプラスミドを用いて、腸内細菌科に属する微生物の形質転換を行うことができる。
【解決手段】 パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、特定の配列からなるアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子を含むプラスミド。このプラスミドを用いて、腸内細菌科に属する微生物の形質転換を行うことができる。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、腸内細菌科に属する微生物内で自律複製可能な新規プラスミド、またプラスミドとプラスミドを導入した微生物の利用法に関する。
【背景技術】
【0002】
現在、エシェリヒア・コリをはじめとする腸内細菌科に属する微生物で、DNA組換え技術による育種改良が進んでいる。DNA組換え技術により微生物の育種改良を行うには、異なる属に属する微生物由来のプラスミドや、高宿主域ベクターを用いることも少なくないが、一般的には類似した性質を持つ微生物由来のプラスミドが用いられる。
【0003】
腸内細菌科に属する微生物由来のプラスミドとしては、エシェリヒア・コリ由来のpBR322、RSF1010 (非特許文献1)、pUC(非特許文献2) 等の小型プラスミドや、サルモネラ属細菌由来のpHCM2(非特許文献3)エルシニア属細菌由来のpCD1(非特許文献4),pMT1 (非特許文献5)らがある。パントエア属細菌が有するプラスミドとして、pSが知られているが(特許文献1)、微生物に導入することにより、酸性条件下での生育を向上させるプラスミドは知られていない。
【特許文献1】WO98/59054号パンフレット
【非特許文献1】Gene vol.75 (2), p271-288, 1989
【非特許文献2】Gene. 1982 Oct;19(3):259-68
【非特許文献3】Plasmid. 2002 May;47(3):159-71.
【非特許文献4】Infect Immun. 1986 Mar;51(3):788-94.
【非特許文献5】J Bacteriol. 2000 Jul;182(14):3924-8.
【発明の開示】
【発明が解決しようとする課題】
【0004】
本発明は、腸内細菌科に属する微生物の育種、改良に有用な新規プラスミドを提供することを課題とする。
【課題を解決するための手段】
【0005】
本発明者らは、パントエア・アナナティスAJ13601(FERM BP- 7207)が新規プラスミドを保持していることを見出し、このプラスミドを同定することに成功し、本発明を完成するに至った。
【0006】
すなわち、本発明は以下のものに関する。
(1)パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子。
(2)(1)に記載の遺伝子を含み、腸内細菌内で自律複製可能なプラスミド。
(3)(2)に記載のプラスミドがさらにマーカー遺伝子を含む、腸内細菌内で自律複製可能なプラスミド。
(4)パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子を含むプラスミドが導入された腸内細菌科に属する微生物。
(5)前記プラスミドが、パントエア・アナナティスFERM BP-7207が保有する約320Kbの大きさを持つプラスミド又はその改変プラスミドである(4)記載の微生物。
(6)前記プラスミドが、図1〜5に示す制限酵素地図を有するpEA320である、(4)又は(5)に記載の微生物。
(7)前記プラスミドが、さらにマーカー遺伝子を含み、腸内細菌科に属する微生物内で自律複製可能なベクターである(4)〜(6)のいずれか1項に記載の微生物。
(8)腸内細菌科に属する微生物が、エシェリヒア属、パントエア属、エンテロバクター属、エルビニア属、クレブシエラ属、セラチア属、サルモネラ属、モルガネラ属に属する細菌からなる群より選択される微生物である、(4)〜(7)のいずれか1項に記載の微生物。
(9)有用物質の生産能を有する(4)〜(8)のいずれか1項に記載の微生物。
(10)(9)に記載の微生物を培地で培養して、有用物質を該培地中又は菌体内に生成蓄積させ、該培地又は菌体より有用物質を採取する、有用物質の製造法。
(11)上記有用物質がL-アミノ酸である(10)に記載の製造法。
(12)(5)に記載の微生物から前記プラスミドを、腸内細菌群に属する微生物に導入することを特徴とする、耐酸性の向上した微生物を製造する方法。
(13)前記耐酸性の向上した微生物が、プラスミドを保有しない菌株と比べてpH3〜5での生育が向上したものであることを特徴とする(12)に記載の方法。
【発明の効果】
【0007】
本発明により、腸内細菌科に属する微生物の育種、改良に有用な新規プラスミドが提供される。本発明のプラスミドは、有用物質の生産菌として使用されている腸内細菌科に属する微生物の育種に利用することができ、微生物の酸性条件下での生育を改善することができる。
【発明を実施するための最良の形態】
【0008】
以下、本発明を詳細に説明する。
<1>本発明の遺伝子及びプラスミド
本発明の遺伝子は、パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子(rep遺伝子)であり、本発明のプラスミドは本発明の遺伝子を含む。
【0009】
本発明の遺伝子は、後述のように、パントエア・アナナティスAJ13601(FERM BP- 7207)が保持するが見出された約320Kbの大きさを持つプラスミドpEA320の解析により同定されたものである。パントエア属細菌、エルビニア属細菌、エンテロバクター属細菌は、γ−プロテオバクテリアに分類される微生物であり、分類学的に非常に近縁である(J Gen Appl Microbiol 1997 Dec;43(6) 355-361、International Journal of Systematic Bacteriology,1993,p162-173、International Journal of Systematic Bacteriology,Oct. 1997,p1061-1067)。近年、16S rRNAの塩基配列解析などにより、エンテロバクター属に属する微生物が、パントエア・アグロメランス(Pantoea agglomerans)又はパントエア・アナナティス(Pantoea ananatis)、パントエア・スチューアルティ(Pantoea stewartii)等に再分類されているものがあり、またエルビニア属に属する微生物がパントエア・アナナス、パントエア・スチューアルティに再分類されているものがある(International Journal of Systematic Bacteriology,Oct. 1997,p1061-1067参照)。
【0010】
また、生理学的にも以下の点で共通している(米国特許6,331,419号、6,197,559号)。
(1)グラム染色性:陰性
(2)酸素に対する挙動:通性嫌気性
(3)オキシダーゼ:ポジティブ
(4)フォゲス−プロスカウエル試験:ポジティブ
(5)メチルレッド試験:ネガティブ
(6)ウレアーゼ:ネガティブ
(7)インドール生成:ポジティブ
(8)TSI培地での硫化水素生成:活性あり
(9)β−ガラクトシダーゼ:ポジティブ
(10)糖資化性:
アラビノース:ポジティブ
シュークロース:ポジティブ
ラクトース:ポジティブ
キシロース:ポジティブ
ソルビトール:ポジティブ
イノシトール:ポジティブ
トレハロース:ポジティブ
マルトース:ポジティブ
メリビオース:ポジティブ
アドニトール:ネガティブ
ラフィノース:ポジティブ
サリシン:ネガティブ
メリビオース:ポジティブ
(11)クエン酸資化性:ポジティブ
(12)アルギニンデヒドラターゼ:ネガティブ
(13)オルニチンデカルボキシラーゼ:ネガティブ
(14)生育pH pH4.5〜7生育良好
【0011】
従って、本発明の遺伝子は、パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物が天然に保有している巨大プラスミド、特に野生株が保有している巨大プラスミドから取得できる。ここで、このような巨大プラスミドを有しているパントエア属細菌の代表的な菌株として、パントエア・アナナティス、パントエア・スチューアルティ(Pantoea stewartii)パントエア・アグロメランスが挙げられる。またエルビニア属細菌の代表的な菌株として、エルビニア・アナナス、エルビニア・ヘルビコーラが挙げられる。また、エンテロバクター属細菌の代表的な菌株として、エンテロバクター・アグロメランス(Enterobacter agglomerans)、エンテロバクター・アエロゲネス(Enterobacter aerogenes)が挙げられる。
【0012】
尚、近年、エンテロバクター・アグロメランスは、16S rRNAの塩基配列解析などにより、パントエア・アグロメランス(Pantoea agglomerans)又はパントエア・アナナティス(Pantoea ananatis)、パントエア・スチューアルティ(Pantoea stewartii)アグロメランス等に再分類されているものがある。本発明においては、腸内細菌科に分類されるものであれば、エンテロバクター属又はパントエア属、あるいはエルビニア属のいずれに属するものから取得される遺伝子あるいはプラスミドであってもよい。パントエア・アナナティスを遺伝子工学的手法を用いて育種する場合には、パントエア・アナナティスAJ13355株(FERM BP−6614)、AJ13356株(FERM BP−6615)、AJ13601株(FERM BP−7207)及びそれらの誘導体を用いることができる。これらの株は、分離された当時はエンテロバクター・アグロメランスと同定され、エンテロバクター・アグロメランスとして寄託されたが、上記のとおり、16S rRNAの塩基配列解析などにより、パントエア・アナナティスに再分類されている。
【0013】
具体的には本発明の遺伝子は、パントエア・アナナティスAJ13601(FERM BP-7207)株から取得可能である(特開2001-333769)。パントエア・アナナティスAJ13601株は経済産業省工業技術院生命工学工業技術研究所(現 独立行政法人産業技術総合研究所特許生物寄
託センター、郵便番号305−8566 茨城県つくば市東1丁目1番地1 中央第6)に受託番号FERM P-17516として1999年8月18日に寄託され、2000年7月6日にブタペスト条約に基づく国際寄託に移管され、受託番号FERM BP-7207が付与されている。
【0014】
また、本発明の遺伝子は、エルビニア・アナナス、エルビニア・ヘルビコーラから取得可能である。エルビニア・アナナスとしては、エルビニア・アナナスATCC33244、エルビニア・ヘルビコーラIAM1595が挙げられる。ATCC33244については、アメリカン・タイプ・カルチャー・コレクション(住所 P.O. Box 1549, Manassas,
VA 20108, United States of America)より分譲を受けることが出来る。すなわち各菌株に対応する登録番号が付与されており、この登録番号を利用して分譲を受けることが出来る(http://www.atcc.org/参照)。各菌株に対応する登録番号は、アメリカン・タイプ・カルチャー・コレクションのカタログに記載されている。IAM1595は、東京大学分子細胞生物学研究所バイオリソーシス研究分野AIMコレクションに保管されていて、登録番号を利用して、分譲を受けることが出来る。欠く菌株に対応する登録番号は、IAMのカタログに記載されている(IAM Catalogue of strain Third Edition,2004)。
【0015】
また、本発明の遺伝子は、エンテロバクター・アグロメランス(Enterobacter agglomerans)、エンテロバクター・アエロゲネス(Enterobacter aerogenes)から取得可能である。エンテロバクター・アグロメランスとしては、ATCC12287、エンテロバクター・アエロゲネスATCC13048が挙げられる。
【0016】
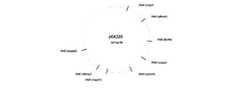
本発明者らは、上記パントエア・アナナティスが約320Kbのプラスミドを有していることを発見、本プラスミドを同定し、pEA320と命名した。これらのプラスミドは、パントエア・アナナティスの細胞中では二本鎖環状DNAとして存在するプラスミドである。pEA320が保持するrep遺伝子の塩基配列を配列番号1に、アミノ酸配列を配列番号2に示した。
【0017】
pEA320を代表的な制限酵素で切断したときに得られる断片の数及び大きさを、表1に示す。さらに、pEM320の制限酵素地図を図1〜図5に示す。
【0018】
【表1】

【0019】
尚、本発明の遺伝子がコードするRepタンパク質とは、配列番号2のアミノ酸配列、またはこのホモログを意味する。本rep遺伝子がコードするRepタンパク質のアミノ酸配列(配列番号2)と公知の腸内細菌群のプラスミドのRepタンパク質のアミノ酸配列について相同性を比較した。Salmonella typhi CT18株の有するクリプティックプラスミ
ドであるpHCM2のRepタンパク質が最も高い相同性を示したが、その値は68.5%と低いことがわかった。このように、本発明の遺伝子と公知の腸内細菌群の持つ遺伝子は、Repタンパク質の相同性によって区別することができる。また本発明のRepタンパク質をコードする遺伝子は、配列番号2のアミノ酸配列全体に対して、70%以上、好ましくは90%以上、より好ましくは95%以上、特に好ましくは97%以上の相同性を有するタンパク質をコードし、プラスミド複製に関与するタンパク質をコードするホモログも含む。ここでの相同性は、遺伝子解析ソフトGenetyx (株式会社ゼネティックスから購入可能)で解析して算出した値であり、アミノ酸の完全一致を示す。また、アミノ酸配列および塩基配列の相同性は、例えばKarlin and AltschulによるアルゴリズムBLAST(Pro. Natl. Acad. Sci. USA, 90, 5873(1993))やFASTA(Methods Enzymol., 183, 63 (1990))を用いて決定することができる。このアルゴリズムBLASTに基づいて、BLASTNやBLASTXとよばれるプログラムが開発されている (http://www.ncbi.nlm.nih.gov参照)。
【0020】
また、パントエア属細菌、エルビニア属細菌、エンテロバクター属細菌の種や菌株によってRepをコードする遺伝子の塩基配列に差異が存在することがあるため、Repタンパク質の機能が変わらない限り、これらのアミノ酸配列において、1若しくは数個のアミノ酸の置換、欠失、挿入、または付加を含むアミノ酸配列を有するものであってもよい。ここで、数個とは、例えば、2〜20個、好ましくは2〜10個、より好ましくは2〜5個を意味する。上記の1若しくは数個のアミノ酸の置換、欠失、挿入、または付加は、プラスミド複製が正常に行われる保存的変異である。保存的変異の代表的なものは、保存的置換であり、保存的置換とみなされる置換としては、AlaからSer又はThrへの置換、ArgからGln、His又はLysへの置換、AsnからGlu、Gln、Lys、His又はAspへの置換、AspからAsn、Glu又はGlnへの置換、CysからSer又はAlaへの置換、GlnからAsn、Glu、Lys、His、Asp又はArgへの置換、GluからAsn、Gln、Lys又はAspへの置換、GlyからProへの置換、HisからAsn、Lys、Gln、Arg又はTyrへの置換、IleからLeu、Met、Val又はPheへの置換、LeuからIle、Met、Val又はPheへの置換、LysからAsn、Glu、Gln、His又はArgへの置換、MetからIle、Leu、Val又はPheへの置換、PheからTrp、Tyr、Met、Ile又はLeuへの置換、SerからThr又はAlaへの置換、ThrからSer又はAlaへの置換、TrpからPhe又はTyrへの置換、TyrからHis、Phe又はTrpへの置換、及び、ValからMet、Ile又はLeuへの置換が挙げられる。
【0021】
またRepタンパク質をコードする遺伝子は、プラスミドを複製する機能をコードする限り、配列番号1からなる塩基配列または同塩基配列から調製され得るプローブとストリンジェントな条件下でハイブリダイズするDNAであってもよい。「ストリンジェントな条件」とは、いわゆる特異的なハイブリッドが形成され、非特異的なハイブリッドが形成されない条件をいう。一例を示せば、相同性が高いDNA同士、例えば70%以上の相同性を有するDNA同士がハイブリダイズし、それより相同性が低いDNA同士がハイブリダイズしない条件、あるいは通常のサザンハイブリダイゼーションの洗いの条件である60℃、1×SSC,0.1%SDS、好ましくは、0.1×SSC、0.1%SDSさらに好ましくは、68℃、0.1×SSC、0.1%SDSに相当する塩濃度、温度で、1回より好ましくは2〜3回洗浄する条件が挙げられる。プローブの長さは、ハイブリダイゼーションの条件により適宜選択されるが、通常には、100bp〜1Kbpである。
【0022】
パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物が天然に保有している本発明のプラスミドはパントエア属細菌、エシェリヒア属細菌をはじめとする腸内細菌科に属する微生物で自律複製できるので、これらのプラスミド又はその自律複製能を維持した改変体のいずれかの位置に、目的遺伝子を挿入し、得られる組換えプラスミドで宿主微生物を形質転換すれば、その外来遺伝子の遺伝情報を宿主微生物内で発現させることが可能となる。
【0023】
本発明のプラスミドを導入可能な腸内細菌科に属する微生物としては、エシェリヒア属
、パントエア属、エンテロバクター属、エルビニア属、クレブシエラ属、セラチア属、サルモネラ属、モルガネラ属に属する細菌が挙げられる。具体的にはNCBI(National Center for Biotechnology Information)データベースに記載されている分類により腸内細菌群に属するものが利用できる。
【0024】
エシェリヒア属細菌としては、具体的にはナイトハルトらの著書(Neidhardt, F.C. et
al., Escherichia coli and Salmonella Typhimurium, American Society for Microbiology, Washington D.C., 1029 table 1) に挙げられるものが利用できる。その中では、例えばエシェリヒア・コリが挙げられる。エシェリヒア・コリとしては具体的には、プロトタイプの野生株K12株由来のエシェリヒア・コリ W3110 (ATCC 27325)、エシェリヒア・コリ MG1655 (ATCC 47076)等が挙げられる。
【0025】
エンテロバクター属細菌としては、エンテロバクター・アグロメランス(Enterobacter
agglomerans)、エンテロバクター・アエロゲネス(Enterobacter aerogenes)等が挙げられる。具体的には、欧州特許出願公開952221号明細書に例示された菌株を使用することが出来る。尚、上述のように、近年、エンテロバクター・アグロメランスは、16S rRNAの塩基配列解析などにより、パントエア・アグロメランス(Pantoea agglomerans)又はパントエア・アナナティス(Pantoea ananatis)、パントエア・スチューアルティ(Pantoea stewartii)等に再分類されているものがある。
【0026】
パントエア属細菌の代表的な菌株として、パントエア・アナナティス、パントエア・スチューアルティ(Pantoea stewartii)パントエア・アグロメランス、パントエア・シトレア(Pantoea citrea)が挙げられる。具体的には、欧州特許出願公開955368号明細書に例示された菌株を使用することが出来る。
【0027】
エルビニア属細菌の代表的な菌株として、エルビニア・アナナス、エルビニア・ヘルビコーラが挙げられ、クレブシエラ属細菌の代表的な菌株として、クレブシエラ・プランティコーラが挙げられる。具体的には、欧州特許出願公開955368号明細書に例示された菌株を使用することが出来る。
【0028】
セラチア属細菌の代表的な菌株としてセラチア・リキュファシエンス、サルモネラ属細菌の代表的菌株としてサルモネラ・ティフィムリウム、モルガネラ属の代表的菌株として、モルガネラ・モルガニーが挙げられる。セラチア属に属する細菌としては、具体的には、欧州特許出願公開952221号明細書に例示された菌株を使用することが出来る。
【0029】
本発明のプラスミドには、本発明の遺伝子を有する限り、パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物が保持する巨大プラスミドの一部、又はその巨大プラスミドもしくはその一部と他のDNA配列からなるプラスミドも包含される。一部とは、プラスミドの自律複製に必須な領域を含む部分を意味する。この態様のプラスミドは、プラスミドの自律複製に必須な領域(複製制御領域)、すなわち複製開始点及び複製に必要な遺伝子を含む領域以外の領域は、その一部又は全部を取り除いても宿主微生物内で複製することができ、しかもその大きさはより小さくなるので、ベクターとして使用するには好ましい。また、本発明のプラスミドに、薬剤耐性遺伝子等のマーカー遺伝子を搭載しておくと、形質転換体中でのマーカー遺伝子の表現型によって、形質転換体の検出が容易となる。前記宿主に使用可能なマーカー遺伝子としては、クロラムフェニコール耐性遺伝子、カナマイシン耐性遺伝子、ストレプトマイシン耐性遺伝子、テトラサイクリン耐性遺伝子、エリスロマイシン耐性遺伝子、アンピシリン耐性遺伝子、スペクチノマイシン耐性遺伝子等が挙げられる。
【0030】
抗生物質耐性遺伝子は、パントエア属細菌、エルビニア属細菌及びエンテロバクター属
細菌からなる群より選択される微生物が天然に保有している巨大プラスミドの複製に必要な領域だけ取り出して連結してもよいし、巨大プラスミドの複製に関与する領域以外の部位に転移させてもよい。抗生物質耐性遺伝子の本プラスミドへの導入方法は、Sambrook, J., Fritsch, E. F., and Maniatis, T., "Molecular Cloning A Laboratory Manual, Second Edition", Cold Spring Harbor Laboratory Press, (1989)等に記載されている当業者によく知られた方法や、DatsenkoとWannerによって開発された「Redドリブンインテグレーション(Red-driven integration)」(Proc. Natl. Acad. Sci. USA, 2000, vol. 97,
No. 12, p6640-6645)と呼ばれる、直鎖状DNAを用いる方法や、特開平2-109985号公報に開示されているように、抗生物質耐性遺伝子をトランスポゾンに搭載してこれを転移させる方法を用いることが出来る。
【0031】
巨大プラスミドの一例であるpEA320自体は、図1〜5に示す制限酵素地図によって特徴付けられるが、本発明のプラスミドはこれらの制限酵素地図を有する必然性はなく、自律複製能に影響のない限り、任意の制限酵素部位を消滅させてもよい。また、本発明のプラスミドは、pEA320が有していない制限酵素部位が導入されていてもよい。
【0032】
上記のようなプラスミドの作製は、従来知られているクローニングベクターや発現ベクター等の作製と同様にして行うことができる。改変体を作製するには、巨大プラスミドの塩基配列が決定されていた方がよい。塩基配列は、ダイデオキシ法等の公知の方法によって決定することができる。
【0033】
本発明のプラスミドに外来遺伝子を挿入するには、これらのプラスミド又は誘導体の制限酵素部位に挿入するのが便利である。制限酵素部位としては、被切断箇所が一箇所であるものが望ましい。外来遺伝子を挿入するには、プラスミドおよび外来遺伝子源となるゲノムDNA等を、それぞれの切断末端の突出部分が同じ配列となるような制限酵素、例えば同じ制限酵素で部分的あるいは完全消化し、これらを適当な条件下で連結すればよい。また、平滑末端で連結してもよい。
【0034】
プラスミドDNAの調製、DNAの切断及び連結、形質転換等の方法は、当業者によく知られている通常の方法を採用することができる。これらの方法は、Sambrook, J., Fritsch, E. F., and Maniatis, T., "Molecular Cloning A Laboratory Manual, Second Edition", Cold Spring Harbor Laboratory Press, (1989)等に記載されている。また、巨大プラスミドの抽出方法としては、J.Bacteriol Mar.1981, Vol.145, No.3, p1365-1373に示す方法が挙げられる。
【0035】
また、本発明のプラスミドは、本発明のプラスミドを保持する微生物から、接合伝達により、他の微生物に導入することができる。
【0036】
プラスミドの接合伝達は、腸内細菌科に属する微生物に通常に適用される系を用いて行うことができる。通常には、oriT、tra遺伝子及びmob遺伝子を有する、又は、有するように改変されたプラスミドを有する供与微生物を準備し、受容微生物に伝達させる。また、導入するプラスミドはoriTを有していれば、Tra及びMobタンパク質の供給により供与微生物から受容微生物に移行できるので、tra遺伝子及び/又はmob遺伝子は、導入するプラスミド上に存在しなくてもよい。すなわち、導入するプラスミドが有さないtra遺伝子及び/又はmob遺伝子を有するヘルパープラスミドを、別途、供与微生物に保持させてもよい。(J Bacteriol. 1995 Aug;177(15):4350-5.参照)
【0037】
以下、本発明のプラスミドの腸内細菌科に属する微生物への導入方法の例について述べる。パントエア属、エルビニア属、エンテロバクター属に属する微生物が天然に保持する本発明のプラスミドは、本来の大きさが非常に大きいので(例えば、pEA320は約320Kb)
、本来の大きさを維持したまま、そのプラスミドを保持する微生物から他微生物に転移するには、接合伝達法を用いることが望ましい。ここで、本発明のプラスミドを天然に保持するパントエア属、エルビニア属、エンテロバクター属に属する微生物を供与菌株、本発明のプラスミドが導入された腸内細菌科に属する微生物を受容菌と便宜的に呼ぶ。
【0038】
接合伝達は、本発明のプラスミドと、接合伝達能を有するヘルパープラスミドを用いて行うことが好ましい。このようにすることによって、本発明のプラスミドに改変が必要な場合、その改変を最小にすることができる。ここで、ヘルパープラスミドは、接合伝達に必要なmob領域、tra領域を有しているプラスミドが好ましい。具体的には、ヘルパープラスミドとしては、pRK2013(CLONTECH社、Biomedal社から入手可能)又はその誘導体が望ましい。(Proc. Natl. Acad. Sci. U.S.A. 76:1648-1652(1979参照)配列はNCCB Plasmids databaseから入手可能である)また、本発明のプラスミドは、受容菌に導入された際に優性選択できるように、受容菌が本来有さないマーカー遺伝子を搭載していることが望ましい。マーカー遺伝子は、上述の通りである。
【0039】
本発明のプラスミドがマーカー遺伝子を有していない場合は、相同組換えなどの通常の方法により、マーカー遺伝子を挿入する。以下、本発明のプラスミドに抗生物質耐性遺伝子を挿入する場合を、例として説明する。挿入位置は、本発明のプラスミドの複製、安定性に影響を及ぼさない箇所であればいずれでもよいが、具体的には、アセトイン−ブタンジオールの生合成系遺伝子budオペロンの近傍領域が用いられる。まず、供与菌の染色体DNAを鋳型にして、変異を含む配列を有するPCRプライマーを用いて、該当箇所を増幅する当業者によく知られたオーバーラップエクステンションPCR法を用いて、変異を含むbud遺伝子を増幅してクローニングすることができる (Urban, A., Neukirchen, S. and Jaeger, K. E., A rapid and efficient method for site-directed mutagenesis using one-step overlap extension PCR. Nucleic Acids Res, 25, 2227-8. (1997).) 。クローニングされた変異型budオペロンをpUT399に搭載し(pUT-Δbud)、λ-pirを有するS17-1λpir株(Biomedal社から入手可能:R. Simon., et al., BIO/TECHNOLOGY NOVEMBER 1983, 784-791 (1983)参照)に導入する。pUT399は、R6Kの複製起点を有するプラスミドであり、接合伝達に必要なmob領域を含むプラスミドであり、pir遺伝子を持たない菌株では複製出来ないプラスミドである。得られた株と供与菌を接合伝達し、供与菌が有する本発明のプラスミド上にプラスミド(pUT-Δbud)がpEA320上に組み込まれた株を取得する。
【0040】
次に、上記のヘルパープラスミド及び抗生物質耐性遺伝子を利用した接合伝達の例を説明する。接合伝達に必要なtra遺伝子を搭載するプラスミドpRK2013にepicentre社のTransposomicsTM&EZ::TNTMを用いてTn5の内部にテトラサイクリン耐性遺伝子を挿入したTn5::Tet遺伝子を有するpRK-Tetプラスミドを構築する。このpRK-Tetを導入したE.coli菌株より、上記のヘルパープラスミドを導入した供与菌への接合伝達によりpRK-Tetを導入する。次にこの pRK-Tetを導入した供与菌と受容菌である腸内細菌科に属する微生物とを接合し、クロラムフェニコール耐性を指標に選択することにより、本発明のプラスミドを有する受容菌を取得することが出来る。
【0041】
<2>本発明のプラスミドの利用方法
(2−1)ベクターとしての利用
腸内細菌科に属する微生物で自律複製できる本発明のプラスミドは、常法によりこれらのプラスミドのいずれかの位置に目的遺伝子を挿入し、得られる組換えプラスミドで宿主微生物を形質転換すれば、その遺伝子の遺伝情報を宿主微生物内で発現させることが可能となる。また、他の宿主での複製に必要な領域を挿入することによって、シャトルベクターとすることができる。
【0042】
宿主微生物としては、腸内細菌群に属し、かつ有用物質を生産可能な微生物であればい
ずれでもよい。有用物質としては、微生物を用いて製造される有用物質であれば、特に限定されないが、例えば、タンパク質、アミノ酸、核酸、ビタミン、糖、有機酸、脂質などを挙げることが出来る。また、外来遺伝子とは、上記有用物質に関連する遺伝子が望ましく、例えば、有用物質であるタンパク質をコードする遺伝子や、アミノ酸、核酸、ビタミン、糖、有機酸、脂質等の生合成系に関与する遺伝子等が挙げられる。
【0043】
例えば、L−アミノ酸としては、L−スレオニン、L−リジン、L−グルタミン酸等が挙げられ、宿主微生物としては、L−アミノ酸生産菌であるエシェリヒア・コリ、パントエア・アナナティス等の腸内細菌が挙げられる。(欧州特許出願公開明細書1078989、米国特許6331419号)
【0044】
例えばL−グルタミン酸生産菌としては、以下の腸内細菌科に属する微生物が利用出来る。
パントエア・アナナティス AJ13601 (FERM BP-7207)
クレブシエラ・プランティコーラ AJ13410株(FERM BP-6617)
パントエア・アナナティス AJ13355 (FERM BP-6614)
エルビニア・ヘルビコーラIAM1595/RSFCPG
(米国特許6,197,559号参照)
クレブシエラ・プランティコーラAJ13399/RSFCPG
(米国特許6,197,559号参照)
セラチア・リクエファシエンス ATCC14460/RSFCPG( 米国特許6,331,419号参照)
エシェリヒア・コリAJ12628(特開平H05-244970号)
エシェリヒア・コリAJ12624(特開平H05-244970号)
【0045】
パントエア・アナナティスAJ13601株は経済産業省工業技術院生命工学工業技術研究所(郵便番号305−8566 茨城県つくば市東1丁目1番3号)に受託番号FERM P-17516として1999年8月18日に寄託され、2000年7月6日にブタペスト条約に基づく国際寄託に移管され、受託番号FERM BP-7207が付与されている。
【0046】
有機酸としては、D−グルコン酸、2−ケト−D−グルコン酸、2、5−ジケト−D−グルコン酸、コハク酸が挙げられ、宿主微生物としては、パントエア属細菌、エルビニア属細菌、エンテロバクター属細菌(国際公開WO98/59504号パンフレット、WO0212468号パンフレット等),エシェリヒア属細菌(WO99/06532号パンフレット等)が使用出来る。(米国特許明細書6,448,061)。また、ビタミンあるいはビタミンの前駆体としては、カロテノイド、パントテン酸、アスコルビン酸が挙げられ、宿主微生物としては、パントエア属細菌、エシェリヒア属細菌が使用出来る。(国際公開第02/072855号パンフレット)
【0047】
また、本発明のプラスミドは、ベクターとして使用するために、外来遺伝子を挿入しやすいようにマルチクローニングサイトのような制限酵素部位を導入してもよいし、目的遺伝子の上流に強力なプロモーター、下流に強力なターミネーターを挿入してもよい。また、タンパク精製に使用するためにアフィニティー精製のための適切な位置にタグを付加したタンパク質を発現させることのできる発現ベクターとして利用してもよい。
【0048】
有用物質は、有用物質の生産能を有する腸内細菌科に属する微生物に、本発明のプラスミドを導入し、培地で培養して、有用物質を該培地中又は菌体内に生成蓄積させ、該培地又は菌体より有用物質を採取することによって取得することが出来る。
【0049】
使用する培地は、微生物を用いた有用物質の発酵生産において従来より用いられてきた
培地を用いることができる。すなわち、炭素源、窒素源、無機イオン及び必要に応じその他の有機成分を含有する通常の培地を用いることができる。ここで、炭素源としては、グルコース、シュクロース、ラクトース、ガラクトース、フラクトースやでんぷんの加水分解物などの糖類、グリセロールやソルビトールなどのアルコール類、フマール酸、クエン酸、コハク酸等の有機酸類を用いることができる。窒素源としては、硫酸アンモニウム、塩化アンモニウム、リン酸アンモニウム等の無機アンモニウム塩、大豆加水分解物などの有機窒素、アンモニアガス、アンモニア水等を用いることができる。有機微量栄養源としては、ビタミンB1、L−ホモセリンなどの要求物質または酵母エキス等を適量含有させることが望ましい。これらの他に、必要に応じて、リン酸カリウム、硫酸マグネシウム、鉄イオン、マンガンイオン等が少量添加される。なお、本発明で用いる培地は、炭素源、窒素源、無機イオン及び必要に応じてその他の有機微量成分を含む培地であれば、天然培地、合成培地のいずれでもよい。
【0050】
培養は好気的条件下で1〜7日間実施するのが通常であり、培養温度は24℃〜37℃、培養中のpHは5〜9が通常である。尚、pH調整には無機あるいは有機の酸性あるいはアルカリ性物質、更にアンモニアガス等を使用することができる。有用物質が菌体外に生成、蓄積した場合には、培養終了後、培養物から菌体などの沈殿物を除去し、通常イオン交換樹脂法、沈殿法その他の公知の方法を組み合わせることにより単離、精製出来る。なお、菌体内に有用物質が蓄積する場合には、例えば菌体を超音波などにより破砕し、遠心分離によって菌体を除去して得られる上清からイオン交換樹脂法などによって、有用物質を回収することができる。
【0051】
また、本発明のプラスミドまたはその誘導体は、腸内細菌科に属する微生物の目的遺伝子を破壊するためのベクターとして用いてもよい。例えば低温での培養温度では複製可能だが、高温では複製出来ないプラスミドになるように配列番号2のアミノ酸配列に変異を導入し、変異遺伝子のDNA断片と、相同な配列を有する腸内細菌科に属する微生物の染色体上に存在する遺伝子との間に相同組換えを生じせしめ、DNA断片が染色体上に組み込まれた腸内細菌科に属する微生物を調製することが出来る。本発明のプラスミドを温度感受性に改変するためには、例えば、DNAをヒドロキシアミンで処理し、低温では自律複製できるが、それよりも高い温度では自律複製できないような変異を有するプラスミドを取得することによって、作成できる。(米国特許6,303,383号参照)
【0052】
(2−2)耐酸性を付与する因子としての利用
パントエア属、エルビニア属又はエンテロバクター属に属する微生物が天然に保持する本発明のプラスミドは、微生物の耐酸性を向上させる効果を有する。本発明のプラスミドは、微生物の耐酸性を向上させる効果を損なわない範囲で改変されている限り、上記ベクターとしての使用の他に、微生物の耐酸性を向上させる因子としても好適である。
【0053】
尚、本発明において、「耐酸性を向上させる」とは、酸性条件下で本発明のプラスミドを導入した微生物を培養した際に、本発明のプラスミドを有さない親株と比べて、生育速度が向上することあるいは生育限界pHが低くなることを意味する。親株としては、本発明のプラスミドを有さない野生株、例えばエシェリヒア・コリMG1655株等が上げられる。またここで培地中のpHは、6以下、好ましくは、5.5以下、より好ましくは4.9以下である。下限pHは、本発明のプラスミドを導入することによって、生育出来るpHであればいずれでもよいが、例えば、pH3以上が挙げられる。
【0054】
また親株との生育を比較する方法としては、親株と本発明のプラスミドを導入した菌株を液体培地で培養した際の培養液の、580〜660nmから選ばれる波長での吸光度を計測し、親株と本発明のプラスミドを導入した菌株の培養液の該吸光度を比較する方法を挙げることが出来る。
【0055】
尚、耐酸性を付与する因子としての利用はベクターとしての利用と兼ねていてもよい。すなわち、目的遺伝子を搭載する本発明のプラスミドを有する菌株を用いると、酸性条件下での生育も向上するし、有用物質生産能も向上する。
【0056】
以下、本発明を、実施例を参照してさらに説明する。ただし、本発明は以下の実施例に限定されない。
【実施例1】
【0057】
pEA320は約320kbと非常に大きく、環状DNAとして単離し、本プラスミドを用いて他の菌株を形質転換することは難しい。また本プラスミド上には薬剤耐性遺伝子やその他優性選択可能なマーカーが存在しないため他菌株へ導入するためには、優性選択可能なマーカー遺伝子を導入する必要がある。そこで、クロラムフェニコールによって、相同組換えが起きたプラスミドを有する株が選択できるように構築したpUT399を用いる手順を採用した。(米国特許出願公開20040180404号明細書)
【0058】
pUT399は以下のようにして構築した。pUT-miniTn5-Cm(Biomedal社より購入可能)を鋳型として、P7(配列番号9)、P8(配列番号10)を用いてR6Kの複製起点とRP4のmob領域をPCRにより増幅した。またpHSG399(TaKaRa社)を鋳型としてクロラムフェニコール耐性遺伝子とマルチクローニングサイトを含む領域をP9(配列番号11)、P10(配列番号12)を用いて増幅した。両者をそれぞれ制限酵素BglIIで処理したのち、T4DNAリガーゼを用いて連結した。
【0059】
pUT399はR6Kの複製起点を持つプラスミドであり、接合伝達に必要なmob領域を含む。またpUT399はpir遺伝子を持たない株では複製できない。従ってpUT399上にpEA320上のある配列を搭載したプラスミドを構築し、接合によりP. ananatisを形質転換しクロラムフェニコールで選択すると、相同領域で組換えを起こしpEA320上にpUT399が導入されたプラスミドを保持するP. ananatisが取得できる。
【0060】
以下に具体的な方法を示す。pEA320は320kbと非常に大きいため、通常のアルカリ-SDS法では単離は難しいが、定法に従った染色体単離の過程で染色体と共に回収される。本実験においてpEA320上の遺伝子としてbudABオペロンの遺伝子を選択した。budAの下流側約370bpおよびbudB遺伝子の上流側約500bpを欠損するように、プライマーP1(配列番号3)、P2(配列番号4)、またはP3(配列番号5)、P4(配列番号6)を合成してAJ13355株(FERM BP-6614)染色体を鋳型として1stPCRを行なった後、プライマーP5(配列番号7)、P6(配列番号8)を用い上記PCR産物を鋳型としてクロスオーバーPCRにより約2.3kbの断片を増幅した。次に得られた増幅断片をpCR2.1(Invitrogen社)に搭載し、このプラスミドのEco RI処理を行なうことにより、Eco RIサイトが付着した挿入断片を得た。最後に、この断片をEco RI処理を施したpUT399に搭載し、S17-1λpir株(Biomedal社より購入可能:R. Simon., et al., BIO/TECHNOLOGY NOVEMBER 1983, 784-791 (1983)参照)を形質転換した。
【0061】
得られた菌株S17-1λpir/pUT-Δbud株とP. ananatis NP106株を接合し、pEA320上の遺伝子領域で相同組換えによりpUT-Δbudが組み込まれたNP106/pEA320-bud::Cm株を取得した。ここでP. ananatis NP106株とは、パントエア・アナナティスAJ13601株(FERM BP-7207株)からプラスミドRSFCPGとpSTVCBを脱落させた株である。パントエア・アナナティスAJ13601(FERM BP-7207)株が有しているプラスミドである。尚、本構築では完全長のbudAB遺伝子と破壊型budAB遺伝子、更にpUT399の全領域がpEA320上に組み込まれている。次に、接合伝達に必要なtra遺伝子を搭載するプラスミドpRK2013(Biomedal社)にepicentre社のTransposomicsTM&EZ::TNTMを用いてTn5::Tet遺伝子を導入したpRK-Tetプラスミドを有するE. coli DH5α株(タカラバイオ社製)より、接合によりpRK-TetプラスミドをNP10
6/pEA320-bud::Cm株に導入した。次にこのNP106/pEA320-bud::Cm, pRK-Tet株とMG1655株を接合し、クロラムフェニコール25mg/Lを含有するM9グルコース寒天培地で選択することによりpEA320が接合伝達されたMG1655, pEA320-bud::Cm株を取得した。
【実施例2】
【0062】
<pEA320を保持するE. coliの酸性における生育>
pEA320を導入したE. coli MG1655株の酸性条件における生育を検討した。
具体的には野生株MG1655をL培地(バクトトリプトン10 g、イーストエキストラクト 5 g、NaCl 5 g、寒天15 gを純水1 Lに含む培地、pH7.0)で、pEA320導入株であるMG1655/pEA320-bud::Cmを、L培地(バクトトリプトン10 g、イーストエキストラクト 5 g、NaCl 5 g、寒天15 gを純水1 Lに含む培地、pH7.0)にクロラムフェニコールを25mg/Lとなるよう添加した培地で前培養し、滅菌水にて2回洗浄した後、30 g/Lのグルタミン酸と5 g/Lのグルコースを含み、pHをアンモニア水にて7.0又は4.5に調整したM9最少培地(硫酸マグネシウム2 mM、リン酸一カリウム3 g、塩化ナトリウム0.5 g 塩化アンモニウム1 g リン酸2ナトリウム6 g を純水1 Lに含む培地)に、OD660nm=0.05となるように植菌し経時のODを測定した。尚、培養・ODの測定にはADVANTEC社製TN1506を用いた。
【0063】
図6に示すとおり、pH7.0の条件においては、ほとんど生育差は見られなかったが、pH4.5の条件では顕著に生育が向上した。すなわち、pEA320が耐酸性を付与するプラスミドであることが本実験により示された。
【0064】
次に本現象を詳細に解析するため、ジャーファーメンターを用いてpHをコントロールした条件で培養を行うこととした。野生株MG1655をL培地(バクトトリプトン10 g、イーストエキストラクト 5 g、NaCl 5 g、寒天15 gを純水1 Lに含む培地、pH7.0)で、pEA320導入株であるMG1655/pEA320-bud::Cmを、L培地(バクトトリプトン10 g、イーストエキストラクト 5 g、NaCl 5 g、寒天15 gを純水1 Lに含む培地、pH7.0)にクロラムフェニコールを25 mg/Lとなるよう添加した培地で前培養し、それぞれプレート1枚を以下に示す組成の培地300 mLを張り込んだジャーファメンターに植菌した。培養は37℃、pH4.9又はpH4.7にて、通気1/1vvm、34℃で酸素濃度が5%以上になるように攪拌制御を行い、培養中のpHは、それぞれ4.9、4.7となるようにアンモニアガスを添加することにより調整を行った。
【0065】
図7に示すとおり、MG1655が生育できないpH4.7、4.9といった条件においてもpEA320を保持するMG1655は生育することが可能であり、pEA320を導入することにより耐酸性が向上することが確認された。
【0066】
【表2】

【図面の簡単な説明】
【0067】
【図1】プラスミドpEA320のNotIの制限酵素地図を示す図。
【図2】プラスミドpEA320のSse8387Iの制限酵素地図を示す図。
【図3】プラスミドpEA320のSwaIの制限酵素地図を示す図。
【図4】プラスミドpEA320のPmeIの制限酵素地図を示す図。
【図5】プラスミドpEA320のAscIの制限酵素地図を示す図。
【図6】プラスミドpEA320を導入したE. coliの中性・酸性条件における生育を示す図。
【図7】プラスミドpEA320を導入したE. coliの酸性条件における生育を示す図。
【技術分野】
【0001】
本発明は、腸内細菌科に属する微生物内で自律複製可能な新規プラスミド、またプラスミドとプラスミドを導入した微生物の利用法に関する。
【背景技術】
【0002】
現在、エシェリヒア・コリをはじめとする腸内細菌科に属する微生物で、DNA組換え技術による育種改良が進んでいる。DNA組換え技術により微生物の育種改良を行うには、異なる属に属する微生物由来のプラスミドや、高宿主域ベクターを用いることも少なくないが、一般的には類似した性質を持つ微生物由来のプラスミドが用いられる。
【0003】
腸内細菌科に属する微生物由来のプラスミドとしては、エシェリヒア・コリ由来のpBR322、RSF1010 (非特許文献1)、pUC(非特許文献2) 等の小型プラスミドや、サルモネラ属細菌由来のpHCM2(非特許文献3)エルシニア属細菌由来のpCD1(非特許文献4),pMT1 (非特許文献5)らがある。パントエア属細菌が有するプラスミドとして、pSが知られているが(特許文献1)、微生物に導入することにより、酸性条件下での生育を向上させるプラスミドは知られていない。
【特許文献1】WO98/59054号パンフレット
【非特許文献1】Gene vol.75 (2), p271-288, 1989
【非特許文献2】Gene. 1982 Oct;19(3):259-68
【非特許文献3】Plasmid. 2002 May;47(3):159-71.
【非特許文献4】Infect Immun. 1986 Mar;51(3):788-94.
【非特許文献5】J Bacteriol. 2000 Jul;182(14):3924-8.
【発明の開示】
【発明が解決しようとする課題】
【0004】
本発明は、腸内細菌科に属する微生物の育種、改良に有用な新規プラスミドを提供することを課題とする。
【課題を解決するための手段】
【0005】
本発明者らは、パントエア・アナナティスAJ13601(FERM BP- 7207)が新規プラスミドを保持していることを見出し、このプラスミドを同定することに成功し、本発明を完成するに至った。
【0006】
すなわち、本発明は以下のものに関する。
(1)パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子。
(2)(1)に記載の遺伝子を含み、腸内細菌内で自律複製可能なプラスミド。
(3)(2)に記載のプラスミドがさらにマーカー遺伝子を含む、腸内細菌内で自律複製可能なプラスミド。
(4)パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子を含むプラスミドが導入された腸内細菌科に属する微生物。
(5)前記プラスミドが、パントエア・アナナティスFERM BP-7207が保有する約320Kbの大きさを持つプラスミド又はその改変プラスミドである(4)記載の微生物。
(6)前記プラスミドが、図1〜5に示す制限酵素地図を有するpEA320である、(4)又は(5)に記載の微生物。
(7)前記プラスミドが、さらにマーカー遺伝子を含み、腸内細菌科に属する微生物内で自律複製可能なベクターである(4)〜(6)のいずれか1項に記載の微生物。
(8)腸内細菌科に属する微生物が、エシェリヒア属、パントエア属、エンテロバクター属、エルビニア属、クレブシエラ属、セラチア属、サルモネラ属、モルガネラ属に属する細菌からなる群より選択される微生物である、(4)〜(7)のいずれか1項に記載の微生物。
(9)有用物質の生産能を有する(4)〜(8)のいずれか1項に記載の微生物。
(10)(9)に記載の微生物を培地で培養して、有用物質を該培地中又は菌体内に生成蓄積させ、該培地又は菌体より有用物質を採取する、有用物質の製造法。
(11)上記有用物質がL-アミノ酸である(10)に記載の製造法。
(12)(5)に記載の微生物から前記プラスミドを、腸内細菌群に属する微生物に導入することを特徴とする、耐酸性の向上した微生物を製造する方法。
(13)前記耐酸性の向上した微生物が、プラスミドを保有しない菌株と比べてpH3〜5での生育が向上したものであることを特徴とする(12)に記載の方法。
【発明の効果】
【0007】
本発明により、腸内細菌科に属する微生物の育種、改良に有用な新規プラスミドが提供される。本発明のプラスミドは、有用物質の生産菌として使用されている腸内細菌科に属する微生物の育種に利用することができ、微生物の酸性条件下での生育を改善することができる。
【発明を実施するための最良の形態】
【0008】
以下、本発明を詳細に説明する。
<1>本発明の遺伝子及びプラスミド
本発明の遺伝子は、パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子(rep遺伝子)であり、本発明のプラスミドは本発明の遺伝子を含む。
【0009】
本発明の遺伝子は、後述のように、パントエア・アナナティスAJ13601(FERM BP- 7207)が保持するが見出された約320Kbの大きさを持つプラスミドpEA320の解析により同定されたものである。パントエア属細菌、エルビニア属細菌、エンテロバクター属細菌は、γ−プロテオバクテリアに分類される微生物であり、分類学的に非常に近縁である(J Gen Appl Microbiol 1997 Dec;43(6) 355-361、International Journal of Systematic Bacteriology,1993,p162-173、International Journal of Systematic Bacteriology,Oct. 1997,p1061-1067)。近年、16S rRNAの塩基配列解析などにより、エンテロバクター属に属する微生物が、パントエア・アグロメランス(Pantoea agglomerans)又はパントエア・アナナティス(Pantoea ananatis)、パントエア・スチューアルティ(Pantoea stewartii)等に再分類されているものがあり、またエルビニア属に属する微生物がパントエア・アナナス、パントエア・スチューアルティに再分類されているものがある(International Journal of Systematic Bacteriology,Oct. 1997,p1061-1067参照)。
【0010】
また、生理学的にも以下の点で共通している(米国特許6,331,419号、6,197,559号)。
(1)グラム染色性:陰性
(2)酸素に対する挙動:通性嫌気性
(3)オキシダーゼ:ポジティブ
(4)フォゲス−プロスカウエル試験:ポジティブ
(5)メチルレッド試験:ネガティブ
(6)ウレアーゼ:ネガティブ
(7)インドール生成:ポジティブ
(8)TSI培地での硫化水素生成:活性あり
(9)β−ガラクトシダーゼ:ポジティブ
(10)糖資化性:
アラビノース:ポジティブ
シュークロース:ポジティブ
ラクトース:ポジティブ
キシロース:ポジティブ
ソルビトール:ポジティブ
イノシトール:ポジティブ
トレハロース:ポジティブ
マルトース:ポジティブ
メリビオース:ポジティブ
アドニトール:ネガティブ
ラフィノース:ポジティブ
サリシン:ネガティブ
メリビオース:ポジティブ
(11)クエン酸資化性:ポジティブ
(12)アルギニンデヒドラターゼ:ネガティブ
(13)オルニチンデカルボキシラーゼ:ネガティブ
(14)生育pH pH4.5〜7生育良好
【0011】
従って、本発明の遺伝子は、パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物が天然に保有している巨大プラスミド、特に野生株が保有している巨大プラスミドから取得できる。ここで、このような巨大プラスミドを有しているパントエア属細菌の代表的な菌株として、パントエア・アナナティス、パントエア・スチューアルティ(Pantoea stewartii)パントエア・アグロメランスが挙げられる。またエルビニア属細菌の代表的な菌株として、エルビニア・アナナス、エルビニア・ヘルビコーラが挙げられる。また、エンテロバクター属細菌の代表的な菌株として、エンテロバクター・アグロメランス(Enterobacter agglomerans)、エンテロバクター・アエロゲネス(Enterobacter aerogenes)が挙げられる。
【0012】
尚、近年、エンテロバクター・アグロメランスは、16S rRNAの塩基配列解析などにより、パントエア・アグロメランス(Pantoea agglomerans)又はパントエア・アナナティス(Pantoea ananatis)、パントエア・スチューアルティ(Pantoea stewartii)アグロメランス等に再分類されているものがある。本発明においては、腸内細菌科に分類されるものであれば、エンテロバクター属又はパントエア属、あるいはエルビニア属のいずれに属するものから取得される遺伝子あるいはプラスミドであってもよい。パントエア・アナナティスを遺伝子工学的手法を用いて育種する場合には、パントエア・アナナティスAJ13355株(FERM BP−6614)、AJ13356株(FERM BP−6615)、AJ13601株(FERM BP−7207)及びそれらの誘導体を用いることができる。これらの株は、分離された当時はエンテロバクター・アグロメランスと同定され、エンテロバクター・アグロメランスとして寄託されたが、上記のとおり、16S rRNAの塩基配列解析などにより、パントエア・アナナティスに再分類されている。
【0013】
具体的には本発明の遺伝子は、パントエア・アナナティスAJ13601(FERM BP-7207)株から取得可能である(特開2001-333769)。パントエア・アナナティスAJ13601株は経済産業省工業技術院生命工学工業技術研究所(現 独立行政法人産業技術総合研究所特許生物寄
託センター、郵便番号305−8566 茨城県つくば市東1丁目1番地1 中央第6)に受託番号FERM P-17516として1999年8月18日に寄託され、2000年7月6日にブタペスト条約に基づく国際寄託に移管され、受託番号FERM BP-7207が付与されている。
【0014】
また、本発明の遺伝子は、エルビニア・アナナス、エルビニア・ヘルビコーラから取得可能である。エルビニア・アナナスとしては、エルビニア・アナナスATCC33244、エルビニア・ヘルビコーラIAM1595が挙げられる。ATCC33244については、アメリカン・タイプ・カルチャー・コレクション(住所 P.O. Box 1549, Manassas,
VA 20108, United States of America)より分譲を受けることが出来る。すなわち各菌株に対応する登録番号が付与されており、この登録番号を利用して分譲を受けることが出来る(http://www.atcc.org/参照)。各菌株に対応する登録番号は、アメリカン・タイプ・カルチャー・コレクションのカタログに記載されている。IAM1595は、東京大学分子細胞生物学研究所バイオリソーシス研究分野AIMコレクションに保管されていて、登録番号を利用して、分譲を受けることが出来る。欠く菌株に対応する登録番号は、IAMのカタログに記載されている(IAM Catalogue of strain Third Edition,2004)。
【0015】
また、本発明の遺伝子は、エンテロバクター・アグロメランス(Enterobacter agglomerans)、エンテロバクター・アエロゲネス(Enterobacter aerogenes)から取得可能である。エンテロバクター・アグロメランスとしては、ATCC12287、エンテロバクター・アエロゲネスATCC13048が挙げられる。
【0016】
本発明者らは、上記パントエア・アナナティスが約320Kbのプラスミドを有していることを発見、本プラスミドを同定し、pEA320と命名した。これらのプラスミドは、パントエア・アナナティスの細胞中では二本鎖環状DNAとして存在するプラスミドである。pEA320が保持するrep遺伝子の塩基配列を配列番号1に、アミノ酸配列を配列番号2に示した。
【0017】
pEA320を代表的な制限酵素で切断したときに得られる断片の数及び大きさを、表1に示す。さらに、pEM320の制限酵素地図を図1〜図5に示す。
【0018】
【表1】

【0019】
尚、本発明の遺伝子がコードするRepタンパク質とは、配列番号2のアミノ酸配列、またはこのホモログを意味する。本rep遺伝子がコードするRepタンパク質のアミノ酸配列(配列番号2)と公知の腸内細菌群のプラスミドのRepタンパク質のアミノ酸配列について相同性を比較した。Salmonella typhi CT18株の有するクリプティックプラスミ
ドであるpHCM2のRepタンパク質が最も高い相同性を示したが、その値は68.5%と低いことがわかった。このように、本発明の遺伝子と公知の腸内細菌群の持つ遺伝子は、Repタンパク質の相同性によって区別することができる。また本発明のRepタンパク質をコードする遺伝子は、配列番号2のアミノ酸配列全体に対して、70%以上、好ましくは90%以上、より好ましくは95%以上、特に好ましくは97%以上の相同性を有するタンパク質をコードし、プラスミド複製に関与するタンパク質をコードするホモログも含む。ここでの相同性は、遺伝子解析ソフトGenetyx (株式会社ゼネティックスから購入可能)で解析して算出した値であり、アミノ酸の完全一致を示す。また、アミノ酸配列および塩基配列の相同性は、例えばKarlin and AltschulによるアルゴリズムBLAST(Pro. Natl. Acad. Sci. USA, 90, 5873(1993))やFASTA(Methods Enzymol., 183, 63 (1990))を用いて決定することができる。このアルゴリズムBLASTに基づいて、BLASTNやBLASTXとよばれるプログラムが開発されている (http://www.ncbi.nlm.nih.gov参照)。
【0020】
また、パントエア属細菌、エルビニア属細菌、エンテロバクター属細菌の種や菌株によってRepをコードする遺伝子の塩基配列に差異が存在することがあるため、Repタンパク質の機能が変わらない限り、これらのアミノ酸配列において、1若しくは数個のアミノ酸の置換、欠失、挿入、または付加を含むアミノ酸配列を有するものであってもよい。ここで、数個とは、例えば、2〜20個、好ましくは2〜10個、より好ましくは2〜5個を意味する。上記の1若しくは数個のアミノ酸の置換、欠失、挿入、または付加は、プラスミド複製が正常に行われる保存的変異である。保存的変異の代表的なものは、保存的置換であり、保存的置換とみなされる置換としては、AlaからSer又はThrへの置換、ArgからGln、His又はLysへの置換、AsnからGlu、Gln、Lys、His又はAspへの置換、AspからAsn、Glu又はGlnへの置換、CysからSer又はAlaへの置換、GlnからAsn、Glu、Lys、His、Asp又はArgへの置換、GluからAsn、Gln、Lys又はAspへの置換、GlyからProへの置換、HisからAsn、Lys、Gln、Arg又はTyrへの置換、IleからLeu、Met、Val又はPheへの置換、LeuからIle、Met、Val又はPheへの置換、LysからAsn、Glu、Gln、His又はArgへの置換、MetからIle、Leu、Val又はPheへの置換、PheからTrp、Tyr、Met、Ile又はLeuへの置換、SerからThr又はAlaへの置換、ThrからSer又はAlaへの置換、TrpからPhe又はTyrへの置換、TyrからHis、Phe又はTrpへの置換、及び、ValからMet、Ile又はLeuへの置換が挙げられる。
【0021】
またRepタンパク質をコードする遺伝子は、プラスミドを複製する機能をコードする限り、配列番号1からなる塩基配列または同塩基配列から調製され得るプローブとストリンジェントな条件下でハイブリダイズするDNAであってもよい。「ストリンジェントな条件」とは、いわゆる特異的なハイブリッドが形成され、非特異的なハイブリッドが形成されない条件をいう。一例を示せば、相同性が高いDNA同士、例えば70%以上の相同性を有するDNA同士がハイブリダイズし、それより相同性が低いDNA同士がハイブリダイズしない条件、あるいは通常のサザンハイブリダイゼーションの洗いの条件である60℃、1×SSC,0.1%SDS、好ましくは、0.1×SSC、0.1%SDSさらに好ましくは、68℃、0.1×SSC、0.1%SDSに相当する塩濃度、温度で、1回より好ましくは2〜3回洗浄する条件が挙げられる。プローブの長さは、ハイブリダイゼーションの条件により適宜選択されるが、通常には、100bp〜1Kbpである。
【0022】
パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物が天然に保有している本発明のプラスミドはパントエア属細菌、エシェリヒア属細菌をはじめとする腸内細菌科に属する微生物で自律複製できるので、これらのプラスミド又はその自律複製能を維持した改変体のいずれかの位置に、目的遺伝子を挿入し、得られる組換えプラスミドで宿主微生物を形質転換すれば、その外来遺伝子の遺伝情報を宿主微生物内で発現させることが可能となる。
【0023】
本発明のプラスミドを導入可能な腸内細菌科に属する微生物としては、エシェリヒア属
、パントエア属、エンテロバクター属、エルビニア属、クレブシエラ属、セラチア属、サルモネラ属、モルガネラ属に属する細菌が挙げられる。具体的にはNCBI(National Center for Biotechnology Information)データベースに記載されている分類により腸内細菌群に属するものが利用できる。
【0024】
エシェリヒア属細菌としては、具体的にはナイトハルトらの著書(Neidhardt, F.C. et
al., Escherichia coli and Salmonella Typhimurium, American Society for Microbiology, Washington D.C., 1029 table 1) に挙げられるものが利用できる。その中では、例えばエシェリヒア・コリが挙げられる。エシェリヒア・コリとしては具体的には、プロトタイプの野生株K12株由来のエシェリヒア・コリ W3110 (ATCC 27325)、エシェリヒア・コリ MG1655 (ATCC 47076)等が挙げられる。
【0025】
エンテロバクター属細菌としては、エンテロバクター・アグロメランス(Enterobacter
agglomerans)、エンテロバクター・アエロゲネス(Enterobacter aerogenes)等が挙げられる。具体的には、欧州特許出願公開952221号明細書に例示された菌株を使用することが出来る。尚、上述のように、近年、エンテロバクター・アグロメランスは、16S rRNAの塩基配列解析などにより、パントエア・アグロメランス(Pantoea agglomerans)又はパントエア・アナナティス(Pantoea ananatis)、パントエア・スチューアルティ(Pantoea stewartii)等に再分類されているものがある。
【0026】
パントエア属細菌の代表的な菌株として、パントエア・アナナティス、パントエア・スチューアルティ(Pantoea stewartii)パントエア・アグロメランス、パントエア・シトレア(Pantoea citrea)が挙げられる。具体的には、欧州特許出願公開955368号明細書に例示された菌株を使用することが出来る。
【0027】
エルビニア属細菌の代表的な菌株として、エルビニア・アナナス、エルビニア・ヘルビコーラが挙げられ、クレブシエラ属細菌の代表的な菌株として、クレブシエラ・プランティコーラが挙げられる。具体的には、欧州特許出願公開955368号明細書に例示された菌株を使用することが出来る。
【0028】
セラチア属細菌の代表的な菌株としてセラチア・リキュファシエンス、サルモネラ属細菌の代表的菌株としてサルモネラ・ティフィムリウム、モルガネラ属の代表的菌株として、モルガネラ・モルガニーが挙げられる。セラチア属に属する細菌としては、具体的には、欧州特許出願公開952221号明細書に例示された菌株を使用することが出来る。
【0029】
本発明のプラスミドには、本発明の遺伝子を有する限り、パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物が保持する巨大プラスミドの一部、又はその巨大プラスミドもしくはその一部と他のDNA配列からなるプラスミドも包含される。一部とは、プラスミドの自律複製に必須な領域を含む部分を意味する。この態様のプラスミドは、プラスミドの自律複製に必須な領域(複製制御領域)、すなわち複製開始点及び複製に必要な遺伝子を含む領域以外の領域は、その一部又は全部を取り除いても宿主微生物内で複製することができ、しかもその大きさはより小さくなるので、ベクターとして使用するには好ましい。また、本発明のプラスミドに、薬剤耐性遺伝子等のマーカー遺伝子を搭載しておくと、形質転換体中でのマーカー遺伝子の表現型によって、形質転換体の検出が容易となる。前記宿主に使用可能なマーカー遺伝子としては、クロラムフェニコール耐性遺伝子、カナマイシン耐性遺伝子、ストレプトマイシン耐性遺伝子、テトラサイクリン耐性遺伝子、エリスロマイシン耐性遺伝子、アンピシリン耐性遺伝子、スペクチノマイシン耐性遺伝子等が挙げられる。
【0030】
抗生物質耐性遺伝子は、パントエア属細菌、エルビニア属細菌及びエンテロバクター属
細菌からなる群より選択される微生物が天然に保有している巨大プラスミドの複製に必要な領域だけ取り出して連結してもよいし、巨大プラスミドの複製に関与する領域以外の部位に転移させてもよい。抗生物質耐性遺伝子の本プラスミドへの導入方法は、Sambrook, J., Fritsch, E. F., and Maniatis, T., "Molecular Cloning A Laboratory Manual, Second Edition", Cold Spring Harbor Laboratory Press, (1989)等に記載されている当業者によく知られた方法や、DatsenkoとWannerによって開発された「Redドリブンインテグレーション(Red-driven integration)」(Proc. Natl. Acad. Sci. USA, 2000, vol. 97,
No. 12, p6640-6645)と呼ばれる、直鎖状DNAを用いる方法や、特開平2-109985号公報に開示されているように、抗生物質耐性遺伝子をトランスポゾンに搭載してこれを転移させる方法を用いることが出来る。
【0031】
巨大プラスミドの一例であるpEA320自体は、図1〜5に示す制限酵素地図によって特徴付けられるが、本発明のプラスミドはこれらの制限酵素地図を有する必然性はなく、自律複製能に影響のない限り、任意の制限酵素部位を消滅させてもよい。また、本発明のプラスミドは、pEA320が有していない制限酵素部位が導入されていてもよい。
【0032】
上記のようなプラスミドの作製は、従来知られているクローニングベクターや発現ベクター等の作製と同様にして行うことができる。改変体を作製するには、巨大プラスミドの塩基配列が決定されていた方がよい。塩基配列は、ダイデオキシ法等の公知の方法によって決定することができる。
【0033】
本発明のプラスミドに外来遺伝子を挿入するには、これらのプラスミド又は誘導体の制限酵素部位に挿入するのが便利である。制限酵素部位としては、被切断箇所が一箇所であるものが望ましい。外来遺伝子を挿入するには、プラスミドおよび外来遺伝子源となるゲノムDNA等を、それぞれの切断末端の突出部分が同じ配列となるような制限酵素、例えば同じ制限酵素で部分的あるいは完全消化し、これらを適当な条件下で連結すればよい。また、平滑末端で連結してもよい。
【0034】
プラスミドDNAの調製、DNAの切断及び連結、形質転換等の方法は、当業者によく知られている通常の方法を採用することができる。これらの方法は、Sambrook, J., Fritsch, E. F., and Maniatis, T., "Molecular Cloning A Laboratory Manual, Second Edition", Cold Spring Harbor Laboratory Press, (1989)等に記載されている。また、巨大プラスミドの抽出方法としては、J.Bacteriol Mar.1981, Vol.145, No.3, p1365-1373に示す方法が挙げられる。
【0035】
また、本発明のプラスミドは、本発明のプラスミドを保持する微生物から、接合伝達により、他の微生物に導入することができる。
【0036】
プラスミドの接合伝達は、腸内細菌科に属する微生物に通常に適用される系を用いて行うことができる。通常には、oriT、tra遺伝子及びmob遺伝子を有する、又は、有するように改変されたプラスミドを有する供与微生物を準備し、受容微生物に伝達させる。また、導入するプラスミドはoriTを有していれば、Tra及びMobタンパク質の供給により供与微生物から受容微生物に移行できるので、tra遺伝子及び/又はmob遺伝子は、導入するプラスミド上に存在しなくてもよい。すなわち、導入するプラスミドが有さないtra遺伝子及び/又はmob遺伝子を有するヘルパープラスミドを、別途、供与微生物に保持させてもよい。(J Bacteriol. 1995 Aug;177(15):4350-5.参照)
【0037】
以下、本発明のプラスミドの腸内細菌科に属する微生物への導入方法の例について述べる。パントエア属、エルビニア属、エンテロバクター属に属する微生物が天然に保持する本発明のプラスミドは、本来の大きさが非常に大きいので(例えば、pEA320は約320Kb)
、本来の大きさを維持したまま、そのプラスミドを保持する微生物から他微生物に転移するには、接合伝達法を用いることが望ましい。ここで、本発明のプラスミドを天然に保持するパントエア属、エルビニア属、エンテロバクター属に属する微生物を供与菌株、本発明のプラスミドが導入された腸内細菌科に属する微生物を受容菌と便宜的に呼ぶ。
【0038】
接合伝達は、本発明のプラスミドと、接合伝達能を有するヘルパープラスミドを用いて行うことが好ましい。このようにすることによって、本発明のプラスミドに改変が必要な場合、その改変を最小にすることができる。ここで、ヘルパープラスミドは、接合伝達に必要なmob領域、tra領域を有しているプラスミドが好ましい。具体的には、ヘルパープラスミドとしては、pRK2013(CLONTECH社、Biomedal社から入手可能)又はその誘導体が望ましい。(Proc. Natl. Acad. Sci. U.S.A. 76:1648-1652(1979参照)配列はNCCB Plasmids databaseから入手可能である)また、本発明のプラスミドは、受容菌に導入された際に優性選択できるように、受容菌が本来有さないマーカー遺伝子を搭載していることが望ましい。マーカー遺伝子は、上述の通りである。
【0039】
本発明のプラスミドがマーカー遺伝子を有していない場合は、相同組換えなどの通常の方法により、マーカー遺伝子を挿入する。以下、本発明のプラスミドに抗生物質耐性遺伝子を挿入する場合を、例として説明する。挿入位置は、本発明のプラスミドの複製、安定性に影響を及ぼさない箇所であればいずれでもよいが、具体的には、アセトイン−ブタンジオールの生合成系遺伝子budオペロンの近傍領域が用いられる。まず、供与菌の染色体DNAを鋳型にして、変異を含む配列を有するPCRプライマーを用いて、該当箇所を増幅する当業者によく知られたオーバーラップエクステンションPCR法を用いて、変異を含むbud遺伝子を増幅してクローニングすることができる (Urban, A., Neukirchen, S. and Jaeger, K. E., A rapid and efficient method for site-directed mutagenesis using one-step overlap extension PCR. Nucleic Acids Res, 25, 2227-8. (1997).) 。クローニングされた変異型budオペロンをpUT399に搭載し(pUT-Δbud)、λ-pirを有するS17-1λpir株(Biomedal社から入手可能:R. Simon., et al., BIO/TECHNOLOGY NOVEMBER 1983, 784-791 (1983)参照)に導入する。pUT399は、R6Kの複製起点を有するプラスミドであり、接合伝達に必要なmob領域を含むプラスミドであり、pir遺伝子を持たない菌株では複製出来ないプラスミドである。得られた株と供与菌を接合伝達し、供与菌が有する本発明のプラスミド上にプラスミド(pUT-Δbud)がpEA320上に組み込まれた株を取得する。
【0040】
次に、上記のヘルパープラスミド及び抗生物質耐性遺伝子を利用した接合伝達の例を説明する。接合伝達に必要なtra遺伝子を搭載するプラスミドpRK2013にepicentre社のTransposomicsTM&EZ::TNTMを用いてTn5の内部にテトラサイクリン耐性遺伝子を挿入したTn5::Tet遺伝子を有するpRK-Tetプラスミドを構築する。このpRK-Tetを導入したE.coli菌株より、上記のヘルパープラスミドを導入した供与菌への接合伝達によりpRK-Tetを導入する。次にこの pRK-Tetを導入した供与菌と受容菌である腸内細菌科に属する微生物とを接合し、クロラムフェニコール耐性を指標に選択することにより、本発明のプラスミドを有する受容菌を取得することが出来る。
【0041】
<2>本発明のプラスミドの利用方法
(2−1)ベクターとしての利用
腸内細菌科に属する微生物で自律複製できる本発明のプラスミドは、常法によりこれらのプラスミドのいずれかの位置に目的遺伝子を挿入し、得られる組換えプラスミドで宿主微生物を形質転換すれば、その遺伝子の遺伝情報を宿主微生物内で発現させることが可能となる。また、他の宿主での複製に必要な領域を挿入することによって、シャトルベクターとすることができる。
【0042】
宿主微生物としては、腸内細菌群に属し、かつ有用物質を生産可能な微生物であればい
ずれでもよい。有用物質としては、微生物を用いて製造される有用物質であれば、特に限定されないが、例えば、タンパク質、アミノ酸、核酸、ビタミン、糖、有機酸、脂質などを挙げることが出来る。また、外来遺伝子とは、上記有用物質に関連する遺伝子が望ましく、例えば、有用物質であるタンパク質をコードする遺伝子や、アミノ酸、核酸、ビタミン、糖、有機酸、脂質等の生合成系に関与する遺伝子等が挙げられる。
【0043】
例えば、L−アミノ酸としては、L−スレオニン、L−リジン、L−グルタミン酸等が挙げられ、宿主微生物としては、L−アミノ酸生産菌であるエシェリヒア・コリ、パントエア・アナナティス等の腸内細菌が挙げられる。(欧州特許出願公開明細書1078989、米国特許6331419号)
【0044】
例えばL−グルタミン酸生産菌としては、以下の腸内細菌科に属する微生物が利用出来る。
パントエア・アナナティス AJ13601 (FERM BP-7207)
クレブシエラ・プランティコーラ AJ13410株(FERM BP-6617)
パントエア・アナナティス AJ13355 (FERM BP-6614)
エルビニア・ヘルビコーラIAM1595/RSFCPG
(米国特許6,197,559号参照)
クレブシエラ・プランティコーラAJ13399/RSFCPG
(米国特許6,197,559号参照)
セラチア・リクエファシエンス ATCC14460/RSFCPG( 米国特許6,331,419号参照)
エシェリヒア・コリAJ12628(特開平H05-244970号)
エシェリヒア・コリAJ12624(特開平H05-244970号)
【0045】
パントエア・アナナティスAJ13601株は経済産業省工業技術院生命工学工業技術研究所(郵便番号305−8566 茨城県つくば市東1丁目1番3号)に受託番号FERM P-17516として1999年8月18日に寄託され、2000年7月6日にブタペスト条約に基づく国際寄託に移管され、受託番号FERM BP-7207が付与されている。
【0046】
有機酸としては、D−グルコン酸、2−ケト−D−グルコン酸、2、5−ジケト−D−グルコン酸、コハク酸が挙げられ、宿主微生物としては、パントエア属細菌、エルビニア属細菌、エンテロバクター属細菌(国際公開WO98/59504号パンフレット、WO0212468号パンフレット等),エシェリヒア属細菌(WO99/06532号パンフレット等)が使用出来る。(米国特許明細書6,448,061)。また、ビタミンあるいはビタミンの前駆体としては、カロテノイド、パントテン酸、アスコルビン酸が挙げられ、宿主微生物としては、パントエア属細菌、エシェリヒア属細菌が使用出来る。(国際公開第02/072855号パンフレット)
【0047】
また、本発明のプラスミドは、ベクターとして使用するために、外来遺伝子を挿入しやすいようにマルチクローニングサイトのような制限酵素部位を導入してもよいし、目的遺伝子の上流に強力なプロモーター、下流に強力なターミネーターを挿入してもよい。また、タンパク精製に使用するためにアフィニティー精製のための適切な位置にタグを付加したタンパク質を発現させることのできる発現ベクターとして利用してもよい。
【0048】
有用物質は、有用物質の生産能を有する腸内細菌科に属する微生物に、本発明のプラスミドを導入し、培地で培養して、有用物質を該培地中又は菌体内に生成蓄積させ、該培地又は菌体より有用物質を採取することによって取得することが出来る。
【0049】
使用する培地は、微生物を用いた有用物質の発酵生産において従来より用いられてきた
培地を用いることができる。すなわち、炭素源、窒素源、無機イオン及び必要に応じその他の有機成分を含有する通常の培地を用いることができる。ここで、炭素源としては、グルコース、シュクロース、ラクトース、ガラクトース、フラクトースやでんぷんの加水分解物などの糖類、グリセロールやソルビトールなどのアルコール類、フマール酸、クエン酸、コハク酸等の有機酸類を用いることができる。窒素源としては、硫酸アンモニウム、塩化アンモニウム、リン酸アンモニウム等の無機アンモニウム塩、大豆加水分解物などの有機窒素、アンモニアガス、アンモニア水等を用いることができる。有機微量栄養源としては、ビタミンB1、L−ホモセリンなどの要求物質または酵母エキス等を適量含有させることが望ましい。これらの他に、必要に応じて、リン酸カリウム、硫酸マグネシウム、鉄イオン、マンガンイオン等が少量添加される。なお、本発明で用いる培地は、炭素源、窒素源、無機イオン及び必要に応じてその他の有機微量成分を含む培地であれば、天然培地、合成培地のいずれでもよい。
【0050】
培養は好気的条件下で1〜7日間実施するのが通常であり、培養温度は24℃〜37℃、培養中のpHは5〜9が通常である。尚、pH調整には無機あるいは有機の酸性あるいはアルカリ性物質、更にアンモニアガス等を使用することができる。有用物質が菌体外に生成、蓄積した場合には、培養終了後、培養物から菌体などの沈殿物を除去し、通常イオン交換樹脂法、沈殿法その他の公知の方法を組み合わせることにより単離、精製出来る。なお、菌体内に有用物質が蓄積する場合には、例えば菌体を超音波などにより破砕し、遠心分離によって菌体を除去して得られる上清からイオン交換樹脂法などによって、有用物質を回収することができる。
【0051】
また、本発明のプラスミドまたはその誘導体は、腸内細菌科に属する微生物の目的遺伝子を破壊するためのベクターとして用いてもよい。例えば低温での培養温度では複製可能だが、高温では複製出来ないプラスミドになるように配列番号2のアミノ酸配列に変異を導入し、変異遺伝子のDNA断片と、相同な配列を有する腸内細菌科に属する微生物の染色体上に存在する遺伝子との間に相同組換えを生じせしめ、DNA断片が染色体上に組み込まれた腸内細菌科に属する微生物を調製することが出来る。本発明のプラスミドを温度感受性に改変するためには、例えば、DNAをヒドロキシアミンで処理し、低温では自律複製できるが、それよりも高い温度では自律複製できないような変異を有するプラスミドを取得することによって、作成できる。(米国特許6,303,383号参照)
【0052】
(2−2)耐酸性を付与する因子としての利用
パントエア属、エルビニア属又はエンテロバクター属に属する微生物が天然に保持する本発明のプラスミドは、微生物の耐酸性を向上させる効果を有する。本発明のプラスミドは、微生物の耐酸性を向上させる効果を損なわない範囲で改変されている限り、上記ベクターとしての使用の他に、微生物の耐酸性を向上させる因子としても好適である。
【0053】
尚、本発明において、「耐酸性を向上させる」とは、酸性条件下で本発明のプラスミドを導入した微生物を培養した際に、本発明のプラスミドを有さない親株と比べて、生育速度が向上することあるいは生育限界pHが低くなることを意味する。親株としては、本発明のプラスミドを有さない野生株、例えばエシェリヒア・コリMG1655株等が上げられる。またここで培地中のpHは、6以下、好ましくは、5.5以下、より好ましくは4.9以下である。下限pHは、本発明のプラスミドを導入することによって、生育出来るpHであればいずれでもよいが、例えば、pH3以上が挙げられる。
【0054】
また親株との生育を比較する方法としては、親株と本発明のプラスミドを導入した菌株を液体培地で培養した際の培養液の、580〜660nmから選ばれる波長での吸光度を計測し、親株と本発明のプラスミドを導入した菌株の培養液の該吸光度を比較する方法を挙げることが出来る。
【0055】
尚、耐酸性を付与する因子としての利用はベクターとしての利用と兼ねていてもよい。すなわち、目的遺伝子を搭載する本発明のプラスミドを有する菌株を用いると、酸性条件下での生育も向上するし、有用物質生産能も向上する。
【0056】
以下、本発明を、実施例を参照してさらに説明する。ただし、本発明は以下の実施例に限定されない。
【実施例1】
【0057】
pEA320は約320kbと非常に大きく、環状DNAとして単離し、本プラスミドを用いて他の菌株を形質転換することは難しい。また本プラスミド上には薬剤耐性遺伝子やその他優性選択可能なマーカーが存在しないため他菌株へ導入するためには、優性選択可能なマーカー遺伝子を導入する必要がある。そこで、クロラムフェニコールによって、相同組換えが起きたプラスミドを有する株が選択できるように構築したpUT399を用いる手順を採用した。(米国特許出願公開20040180404号明細書)
【0058】
pUT399は以下のようにして構築した。pUT-miniTn5-Cm(Biomedal社より購入可能)を鋳型として、P7(配列番号9)、P8(配列番号10)を用いてR6Kの複製起点とRP4のmob領域をPCRにより増幅した。またpHSG399(TaKaRa社)を鋳型としてクロラムフェニコール耐性遺伝子とマルチクローニングサイトを含む領域をP9(配列番号11)、P10(配列番号12)を用いて増幅した。両者をそれぞれ制限酵素BglIIで処理したのち、T4DNAリガーゼを用いて連結した。
【0059】
pUT399はR6Kの複製起点を持つプラスミドであり、接合伝達に必要なmob領域を含む。またpUT399はpir遺伝子を持たない株では複製できない。従ってpUT399上にpEA320上のある配列を搭載したプラスミドを構築し、接合によりP. ananatisを形質転換しクロラムフェニコールで選択すると、相同領域で組換えを起こしpEA320上にpUT399が導入されたプラスミドを保持するP. ananatisが取得できる。
【0060】
以下に具体的な方法を示す。pEA320は320kbと非常に大きいため、通常のアルカリ-SDS法では単離は難しいが、定法に従った染色体単離の過程で染色体と共に回収される。本実験においてpEA320上の遺伝子としてbudABオペロンの遺伝子を選択した。budAの下流側約370bpおよびbudB遺伝子の上流側約500bpを欠損するように、プライマーP1(配列番号3)、P2(配列番号4)、またはP3(配列番号5)、P4(配列番号6)を合成してAJ13355株(FERM BP-6614)染色体を鋳型として1stPCRを行なった後、プライマーP5(配列番号7)、P6(配列番号8)を用い上記PCR産物を鋳型としてクロスオーバーPCRにより約2.3kbの断片を増幅した。次に得られた増幅断片をpCR2.1(Invitrogen社)に搭載し、このプラスミドのEco RI処理を行なうことにより、Eco RIサイトが付着した挿入断片を得た。最後に、この断片をEco RI処理を施したpUT399に搭載し、S17-1λpir株(Biomedal社より購入可能:R. Simon., et al., BIO/TECHNOLOGY NOVEMBER 1983, 784-791 (1983)参照)を形質転換した。
【0061】
得られた菌株S17-1λpir/pUT-Δbud株とP. ananatis NP106株を接合し、pEA320上の遺伝子領域で相同組換えによりpUT-Δbudが組み込まれたNP106/pEA320-bud::Cm株を取得した。ここでP. ananatis NP106株とは、パントエア・アナナティスAJ13601株(FERM BP-7207株)からプラスミドRSFCPGとpSTVCBを脱落させた株である。パントエア・アナナティスAJ13601(FERM BP-7207)株が有しているプラスミドである。尚、本構築では完全長のbudAB遺伝子と破壊型budAB遺伝子、更にpUT399の全領域がpEA320上に組み込まれている。次に、接合伝達に必要なtra遺伝子を搭載するプラスミドpRK2013(Biomedal社)にepicentre社のTransposomicsTM&EZ::TNTMを用いてTn5::Tet遺伝子を導入したpRK-Tetプラスミドを有するE. coli DH5α株(タカラバイオ社製)より、接合によりpRK-TetプラスミドをNP10
6/pEA320-bud::Cm株に導入した。次にこのNP106/pEA320-bud::Cm, pRK-Tet株とMG1655株を接合し、クロラムフェニコール25mg/Lを含有するM9グルコース寒天培地で選択することによりpEA320が接合伝達されたMG1655, pEA320-bud::Cm株を取得した。
【実施例2】
【0062】
<pEA320を保持するE. coliの酸性における生育>
pEA320を導入したE. coli MG1655株の酸性条件における生育を検討した。
具体的には野生株MG1655をL培地(バクトトリプトン10 g、イーストエキストラクト 5 g、NaCl 5 g、寒天15 gを純水1 Lに含む培地、pH7.0)で、pEA320導入株であるMG1655/pEA320-bud::Cmを、L培地(バクトトリプトン10 g、イーストエキストラクト 5 g、NaCl 5 g、寒天15 gを純水1 Lに含む培地、pH7.0)にクロラムフェニコールを25mg/Lとなるよう添加した培地で前培養し、滅菌水にて2回洗浄した後、30 g/Lのグルタミン酸と5 g/Lのグルコースを含み、pHをアンモニア水にて7.0又は4.5に調整したM9最少培地(硫酸マグネシウム2 mM、リン酸一カリウム3 g、塩化ナトリウム0.5 g 塩化アンモニウム1 g リン酸2ナトリウム6 g を純水1 Lに含む培地)に、OD660nm=0.05となるように植菌し経時のODを測定した。尚、培養・ODの測定にはADVANTEC社製TN1506を用いた。
【0063】
図6に示すとおり、pH7.0の条件においては、ほとんど生育差は見られなかったが、pH4.5の条件では顕著に生育が向上した。すなわち、pEA320が耐酸性を付与するプラスミドであることが本実験により示された。
【0064】
次に本現象を詳細に解析するため、ジャーファーメンターを用いてpHをコントロールした条件で培養を行うこととした。野生株MG1655をL培地(バクトトリプトン10 g、イーストエキストラクト 5 g、NaCl 5 g、寒天15 gを純水1 Lに含む培地、pH7.0)で、pEA320導入株であるMG1655/pEA320-bud::Cmを、L培地(バクトトリプトン10 g、イーストエキストラクト 5 g、NaCl 5 g、寒天15 gを純水1 Lに含む培地、pH7.0)にクロラムフェニコールを25 mg/Lとなるよう添加した培地で前培養し、それぞれプレート1枚を以下に示す組成の培地300 mLを張り込んだジャーファメンターに植菌した。培養は37℃、pH4.9又はpH4.7にて、通気1/1vvm、34℃で酸素濃度が5%以上になるように攪拌制御を行い、培養中のpHは、それぞれ4.9、4.7となるようにアンモニアガスを添加することにより調整を行った。
【0065】
図7に示すとおり、MG1655が生育できないpH4.7、4.9といった条件においてもpEA320を保持するMG1655は生育することが可能であり、pEA320を導入することにより耐酸性が向上することが確認された。
【0066】
【表2】

【図面の簡単な説明】
【0067】
【図1】プラスミドpEA320のNotIの制限酵素地図を示す図。
【図2】プラスミドpEA320のSse8387Iの制限酵素地図を示す図。
【図3】プラスミドpEA320のSwaIの制限酵素地図を示す図。
【図4】プラスミドpEA320のPmeIの制限酵素地図を示す図。
【図5】プラスミドpEA320のAscIの制限酵素地図を示す図。
【図6】プラスミドpEA320を導入したE. coliの中性・酸性条件における生育を示す図。
【図7】プラスミドpEA320を導入したE. coliの酸性条件における生育を示す図。
【特許請求の範囲】
【請求項1】
パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子。
【請求項2】
請求項1に記載の遺伝子を含み、腸内細菌内で自律複製可能なプラスミド。
【請求項3】
請求項2に記載のプラスミドがさらにマーカー遺伝子を含む、腸内細菌内で自律複製可能なプラスミド。
【請求項4】
パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子を含むプラスミドが導入された腸内細菌科に属する微生物。
【請求項5】
前記プラスミドが、パントエア・アナナティスFERM BP-7207が保有する約320Kbの大きさを持つプラスミド又はその改変プラスミドである請求項4記載の微生物。
【請求項6】
前記プラスミドが、図1〜5に示す制限酵素地図を有するpEA320である、請求項4又は5に記載の微生物。
【請求項7】
前記プラスミドが、さらにマーカー遺伝子を含み、腸内細菌科に属する微生物内で自律複製可能なベクターである請求項4〜6のいずれか1項に記載の微生物。
【請求項8】
腸内細菌科に属する微生物が、エシェリヒア属、パントエア属、エンテロバクター属、エルビニア属、クレブシエラ属、セラチア属、サルモネラ属、モルガネラ属に属する細菌からなる群より選択される微生物である、請求項4〜7のいずれか1項に記載の微生物。
【請求項9】
有用物質の生産能を有する請求項4〜8のいずれか1項に記載の微生物。
【請求項10】
請求項9に記載の微生物を培地で培養して、有用物質を該培地中又は菌体内に生成蓄積させ、該培地又は菌体より有用物質を採取する、有用物質の製造法。
【請求項11】
上記有用物質がL-アミノ酸である請求項10に記載の製造法。
【請求項12】
請求項5に記載の微生物から前記プラスミドを、腸内細菌群に属する微生物に導入することを特徴とする、耐酸性の向上した微生物を製造する方法。
【請求項13】
前記耐酸性の向上した微生物が、プラスミドを保有しない菌株と比べてpH3〜5での生育が向上したものであることを特徴とする請求項12に記載の方法。
【請求項1】
パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子。
【請求項2】
請求項1に記載の遺伝子を含み、腸内細菌内で自律複製可能なプラスミド。
【請求項3】
請求項2に記載のプラスミドがさらにマーカー遺伝子を含む、腸内細菌内で自律複製可能なプラスミド。
【請求項4】
パントエア属細菌、エルビニア属細菌及びエンテロバクター属細菌からなる群より選択される微生物由来の遺伝子であって、配列番号2に示すアミノ酸配列、又は、同アミノ酸配列と70%以上の相同性を持つアミノ酸配列を有するRepタンパク質をコードする遺伝子を含むプラスミドが導入された腸内細菌科に属する微生物。
【請求項5】
前記プラスミドが、パントエア・アナナティスFERM BP-7207が保有する約320Kbの大きさを持つプラスミド又はその改変プラスミドである請求項4記載の微生物。
【請求項6】
前記プラスミドが、図1〜5に示す制限酵素地図を有するpEA320である、請求項4又は5に記載の微生物。
【請求項7】
前記プラスミドが、さらにマーカー遺伝子を含み、腸内細菌科に属する微生物内で自律複製可能なベクターである請求項4〜6のいずれか1項に記載の微生物。
【請求項8】
腸内細菌科に属する微生物が、エシェリヒア属、パントエア属、エンテロバクター属、エルビニア属、クレブシエラ属、セラチア属、サルモネラ属、モルガネラ属に属する細菌からなる群より選択される微生物である、請求項4〜7のいずれか1項に記載の微生物。
【請求項9】
有用物質の生産能を有する請求項4〜8のいずれか1項に記載の微生物。
【請求項10】
請求項9に記載の微生物を培地で培養して、有用物質を該培地中又は菌体内に生成蓄積させ、該培地又は菌体より有用物質を採取する、有用物質の製造法。
【請求項11】
上記有用物質がL-アミノ酸である請求項10に記載の製造法。
【請求項12】
請求項5に記載の微生物から前記プラスミドを、腸内細菌群に属する微生物に導入することを特徴とする、耐酸性の向上した微生物を製造する方法。
【請求項13】
前記耐酸性の向上した微生物が、プラスミドを保有しない菌株と比べてpH3〜5での生育が向上したものであることを特徴とする請求項12に記載の方法。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【公開番号】特開2006−230311(P2006−230311A)
【公開日】平成18年9月7日(2006.9.7)
【国際特許分類】
【出願番号】特願2005−51163(P2005−51163)
【出願日】平成17年2月25日(2005.2.25)
【出願人】(000000066)味の素株式会社 (887)
【Fターム(参考)】
【公開日】平成18年9月7日(2006.9.7)
【国際特許分類】
【出願日】平成17年2月25日(2005.2.25)
【出願人】(000000066)味の素株式会社 (887)
【Fターム(参考)】
[ Back to top ]

