転写を増大させるためのマトリックス付着領域(MAR)およびその使用
ヒトおよび非ヒト動物由来の単離、精製されたMAR配列が、それらに対応するかまたは基づくヌクレオチド配列として開示される。特に、高い転写および/またはタンパク質生成促進活性を有するMARおよびMARコンストラクトが開示され、かつ例えばタンパク質の高収率の生成を目的とする、かかるMARを同定し、かかるMARコンストラクトを設計し、かつそれらを用いるための方法が開示される。
【発明の詳細な説明】
【技術分野】
【0001】
関連出願の相互参照
本願は、2006年8月23日に出願された米国仮特許出願第60/823,319号明細書および2007年8月3日に出願された米国仮特許出願第60/953,910号明細書の優先権を主張するものであり、それら全体が参照により本明細書中に援用される。
【0002】
本発明は、単離、精製されたヒトおよび非ヒト動物由来のMAR配列に対応するかまたはそれに基づくヌクレオチド配列を含む核酸に関する。これらの核酸は、一般に転写および/またはタンパク質生成促進活性を有する。本発明は、例えば高収率のタンパク質生成を目的としたかかる配列を同定するための方法およびそれらを用いる系にも関する。
【背景技術】
【0003】
本発明を例示し、特に実施についてのさらなる詳細を提供するための本明細書で用いられる特許を含む出版物および他の資料は、参照により本明細書中に援用される。便宜上、出版物は、文章中に完全に記載されない限り、添付の参考文献においてアルファベット順に列挙される。EMBL登録番号AC102666およびEMBL登録番号BH101870およびBH101901に隣接する配列ならびにEMBL登録番号(同義語)126658、23119391、22981746もまたそれら全体が参照により本明細書中に援用される。
【0004】
現在では、真核生物染色体の約50〜100kbのクロマチンループドメインへの組織化のモデルが広く受け入れられている[ボドナー J.W.(Bodnar J.W.)、ブレイン P.(Breyne P.)、ヴァン・モンタグ M.(Van Montagu M.)およびゲイセン G.(Gheysen G.)、ラジン S.V.(Razin S.V.)]。これらのループの外端は、核マトリックス、すなわちRNP(リボ核タンパク質)および他の非ヒストンタンパク質からなるタンパク質網状構造(proteinaceous network)に付着された特定のDNA配列に対応すると考えられる[ボーデ J.(Bode J.)、ベンハム C.(Benham C.)、クノップ A.(Knopp A.)およびミエルケ C.(Mielke C.)]。核マトリックスに付着された染色体DNA配列は、足場付着領域(中期中)またはマトリックス付着領域(間期)の各々に対してSARまたはMARと称される。S/MAR、MAR要素もしくはMAR配列または略してMARは、典型的には300〜3000bp長の多形領域である。哺乳類の核内には約100,000のMARが存在すると推定されている[ボーデ J.(Bode J.)、ステンガート−イベル M.(Stengert−Iber M.)、ケイ V.(Kay V.)、シュラケ T.(Schlake T.)およびディエツ−ファイルステッター A.(Dietz−Pfeilstetter A.)]。
【0005】
クロマチンをループドメインに構造的かつ機能的に分離することにより、MAR要素は、遺伝子発現の複製および調節において重要な役割を果たす、例えば哺乳類の核内での転写の中心部(foci)の連続的な構築および分解を促進すると考えられている。この概念を支持するように多数の間接的証拠がもたらされており、例えば様々な真核ゲノム内でDNAの複製起点がMAR要素内にマッピングされた[アマチ B.(Amati B.)およびガッサー S.M.(Gasser S.M.)(1988年)、アマチ B.(Amati B.)およびガッサー S.M.(Gasser S.M.)(1990年)]。MARは、イントロン内部の非コード遺伝子間領域内[ジロド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)]または転写単位の境界[ガッサー S.M.(Gasser S.M.)およびラエムリ U.K.(Laemmli U.K.);国立バイオテクノロジー情報センター(National Center for Biotechnology Information)]にもほぼ常に見出され、そこでは遍在的でかつ/または組織特異的な転写因子に結合しうる。概して、植物や動物細胞系におけるトランスジェニックの実験では、MAR要素を用い、トランス遺伝子の発現および安定性を増大させることに奏功している[アレン G.C.(Allen G.C.)、スパイカー S.(Spiker S.)、トンプソン W.F.(Thompson W.F.)、ボーデ J.(Bode J.)、シュラケ T.(Schlake T.)、リオス−ラミレス M.(Rios−Ramirez M.)、ミエルケ C.(Mielke C.)、ステンガート M.(Stengart M.)、ケイ V.(Kay V.)およびクレア−ヴィルス D.(Klehr−Wirth D.)、ジロド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)]。例えば、MARを用い、バイオテクノロジーや治療用途に関連する細胞、例えばCHO(チャイニーズハムスター卵巣)細胞において様々な組換えタンパク質の生成が増大している[ジロド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)、キム J.M.(Kim J.M.)、キム J.S.(Kim J.S.)、パーク D.H.(Park D.H.)、カン H.S.(Kang H.S.)、ユーン J.(Yoon J.)、バエク K.(Baek K.)およびユーン Y.(Yoon Y.)、ザーン−ザバル M.(Zahn−Zabal M.)、コブル M.(Kobr M.)、ジロド P.A.(Girod P.A.)、イムホフ M.(Imhof M.)、シャテラード P.(Chatellard P.)、デ・ジーザス M.(De Jesus M.)、ブルム F.(Wurm F.)およびマーモド N.(Mermod N.)](マーモド(Mermod)ら、「Development of stable cell lines for production or regulated expression using matrix attachment regions」、国際公開第02074969号パンフレット、また米国特許出願公開第20030087342号明細書)。
【0006】
MARの機能的活性は、その主要なDNA配列ではなくその構造的特性に関連づけられている。確かにMARではAおよびT含量が高く[ボウリカス T.(Boulikas T.)(1993年)]、いくつかの特定の高次構造的かつ物理化学的な特性、例えば分子の自然曲率、狭い副溝、巻き戻し/不対の高い可能性または変性への感受性が観察されている[ボーデ J.(Bode J.)、シュラケ T.(Schlake T.)、リオス−ラミレス M.(Rios−Ramirez M.)、ミエルケ C.(Mielke C.)、ステンガート M.(Stengart M.)、ケイ V.(Kay V.)およびクレア−ヴィルス D.(Klehr−Wirth D.)、ボウリカス T.(Boulikas T.)(1993年)、ボウリカス T.(Boulikas T.)(1995年)]。事実、まさしくそれらの特性を用いることで、MARがSMAR Scanと称される方法を通じて同定されている。さらに、MARの活性はまた、DNA結合タンパク質、例えば一本鎖および/または曲線状のDNAなどのMAR要素の特定の構造的特徴を認識しうるクロマチン改変酵素および/または転写因子に媒介されうる[ボーデ J.(Bode J.)、ステンガート−イベル M.(Stengert−Iber M.)、ケイ V.(Kay V.)、シュラケ T.(Schlake T.)およびディエツ−ファイルステッター A.(Dietz−Pfeilstetter A.)]。明確なタンパク質の結合部位またはMARコンセンサス配列は全く見出されておらず[ボウリカス T.(Boulikas T.)(1993年)]、それはゲノム配列由来のMARの予測を困難にしている。
【0007】
MARの特定の機能的かつ構造的な特性については記載がなされている一方、一次構造の観点ではほとんど共有されてないことからその同定は困難である。動物のMARが植物核の足場に結合し、その逆もいえるという事実により支持される仮定では、MAR要素が真核ゲノム内で機能的に保存されうる一方[ブレイン P.(Breyne P.)、ヴァン・モンタグ M.(Van Montagu M.)、デピッカー A.(Depicker A.)およびゲイセン G.(Gheysen G.)、ミエルケ C.(Mielke C.)、コーウイ Y.(Kohwi Y.)、コーウイ−シゲマツ T.(Kohwi−Shigematsu T.)およびボーデ J.(Bode J.)]、いかなる特徴がMAR配列、例えば強力なタンパク質を生成する配列をもたらすかについて語れることはほとんどない。また、様々な結果が用いられるアッセイに応じて得られうる[ラジン S.V.(Razin S.V.)、ボウリカス T.(Boulikas T.)(1995年)、ケイ V.(Kay V.)およびボーデ J.(Bode J.)]。真核生物内での大量の予想されたMARおよびゲノムプロジェクトで公表された配列の量を考慮し、MAR DNA配列(SMAR Scan I)または調節タンパク質または転写因子として作用する特定のタンパク質に対する結合部位などの機能的配列(SMAR Scan II)の構造的特徴を検出するためのツール/プログラムが開発された[マーモド(Mermod)らに交付された2007年8月3日に出願された米国仮特許出願第60/953,910号明細書、米国特許出願公開第20070178469号明細書]。かかるプログラムは、DNAベンディング、主溝の深さおよび副溝の幅のポテンシャルに対応する一群のDNA配列の特徴の検出により新規の有望なMAR配列を同定するとともに、特定の転写調節タンパク質に対する結合部位を同定するように設計された。これらのプログラムを用いてヒトゲノムを走査することで、推定上のMAR DNA配列が同定されており、それらのいくつかはCHO細胞に形質移入される発現プラスミドに導入される場合にトランス遺伝子の発現を増大させることが示された(ジロド(Girod)ら、「Identification of S/MAR from genomic sequences with bioinformatics and use to increase protein production in industrial and therapeutic processes」、マーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書]。これは、SMAR Scanプログラムがヒト遺伝要素、つまりタンパク質の合成を増大させるのに用いられうるものを効率的に同定しうることを実証した。これまで実施された機能的スクリーニングがヒトゲノムに限定されていたが、大規模な生成では目的のタンパク質が非ヒト哺乳類細胞内で発現されることが多い。
【発明の概要】
【発明が解決しようとする課題】
【0008】
約1600のMARがヒトゲノム内でSMAR Scanにより同定されており、8つのうち6つが、エンハンサー/プロモーターの上流に配置される場合、CHO細胞内で(例えば緑色蛍光タンパク質(GFP)、抗体および受容体に対する)遺伝子の発現促進を引き起こすことが示された。異所性MAR活性を有することが示されたDNAの長さは2.5kb〜6kbの範囲である。しかし、現段階ではMARの構造的特徴づけの欠如が「デザイナー(designer)」MARの生成を制限している。したがって、MARの改変や設計を可能にするため、MAR、特にMARの機能的かつ/または構造的領域を特徴づける必要性が存在する。
【0009】
これまで実施された機能的スクリーニングはヒトゲノムに限定された。大規模な生成では目的のタンパク質が哺乳類細胞内で発現されることが多いことから、転写および/または遺伝子発現を促進するより強力な天然のMAR、ならびに/あるいはヒトおよび/または非ヒト哺乳類細胞内で強力なタンパク質生成体(producer)の細胞を同定するという必要性も存在する。
【0010】
概して、例えばさらなる天然のMARを同定し、同定されたMARを設計し、かつ/または合成MARを生成することにより、有利な特性を有するMARを同定しかつ/または生成するという必要性が存在する。有利な特性、限定はされないが、転写の促進および/またはタンパク質生成/遺伝子発現の特性;天然MARに対する長さの短縮により、例えば遺伝子工学におけるより多目的の使用が可能になる点;外的刺激物、例えば薬剤の添加時での組織、細胞または器官の特異性および/または誘発性が顕在化する。
【0011】
これらの必要性や以下の開示から明確になる他の必要性のうちの1つもしくは複数に対処するため、推定上のMAR DNA配列を同定するためのマウスゲノムの大規模なバイオインフォーマティクス分析を含むいくつかのアプローチが用いられた。マウスゲノムは、MAR予測ソフトウェアSMAR Scan Iを用いて分析された。新規に同定された齧歯類配列における、培養細胞からの目的の医薬品の組換えタンパク質の生成の改善を媒介する能力について評価された。このため、トランス遺伝子の形質移入アッセイにおいて新規に同定されたMARの転写活性が評価された。
【0012】
さらに、MAR、例えばヒト1_68MARおよびマウスMAR S4について試験した。モジュール、特にMARの特定の構造的/配列特異的モジュールを含むモジュールが同定され、かつこれらのモジュールは有利な特性を有するMARは、例えば配列のリシャッフリング(reshuffling)、欠失および/または複製による設計用に用いられた。モジュールはまた、他の因子、例えば特定の結合部位、特に転写因子結合部位(TFBS)を含む合成ヌクレオチド配列と結合された。
【課題を解決するための手段】
【0013】
本発明は、一実施形態では、少なくとも1つの遺伝子を高レベルに発現させる発現系であって、
目的の遺伝子をコードするヌクレオチド配列に作動可能に連結するためのプロモーターと、
前記発現系で形質転換された細胞内で前記遺伝子の発現を促進するための少なくとも1つの非ヒト哺乳類のMARヌクレオチド配列と、
を含み、ここで前記非ヒト哺乳類のMARヌクレオチド配列は、前記コンストラクトによる前記細胞の形質転換時に約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくはそれ以上、前記遺伝子の発現を増大させる、発現系を対象とする。
【0014】
前記非ヒト哺乳類のMARヌクレオチド配列は、
(i)配列番号3、配列番号10もしくはその機能的断片、または
(ii)(i)の配列のいずれかと約80%、約90%、約95%もしくは約98%の配列同一性を有するヌクレオチド配列
を含むか本質的にそれからなるかまたはそれからなり得る。
【0015】
本発明は、単離、精製された核酸分子であって、
(a)配列番号3もしくは配列番号10またはその機能的断片のヌクレオチド配列、または
(b)(a)の配列と少なくとも約80%、約90%、約95%もしくは約98%の配列同一性を有しかつMAR活性を有するヌクレオチド配列
を含むか本質的にそれからなるかまたはそれからなる、核酸分子も対象とする。
【0016】
本発明は、非ヒト哺乳類のMAR配列を同定するための方法であって、
−少なくとも1つの非ヒト哺乳類の核酸分子、好ましくは非ヒト哺乳類のゲノムもしくはその一部を提供するステップと、
−前記核酸分子に、
−評価されるべき核酸分子に対してウインドウサイズを設定するステップと、
−少なくとも1つもしくは少なくとも2つ、好ましくは3つ、より好ましくは4つもしくはそれより多いMAR関連の特徴を選択するステップと、
−この特徴/これらの特徴を示す配列に対して閾値を設定するステップと、
−これらの閾値を超えるMAR候補ヌクレオチド配列を選択するステップと、
−前記非ヒト哺乳類のMARヌクレオチド配列が、前記非ヒト哺乳類のMARヌクレオチド配列を含む発現系を介するヒトおよび/または非ヒト哺乳類細胞の形質転換時に、遺伝子の発現を約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくはそれ以上、増大させることを確認するステップと、
を含む、MAR配列のための走査手順を施すステップと、
を含む、方法をさらに対象とする。
【0017】
本明細書における特徴は、その値にウインドウ値を乗じることにより、約320〜1320、例えば約420〜約1220、約520〜約1120、約620〜約1020、約720〜約920の乗算値が得られるDNAベンディングの角度である場合があり、本明細書における特徴は、ウインドウ値を乗じることにより約900〜約4000、例えば約1200〜3700、約1500〜約3400、約1800〜約3100、約2100〜約2800の乗算値が得られる主溝の深さ値である場合があり、および/または本明細書における特徴は、ウインドウサイズ値を乗じることにより約500〜約2500、例えば約750〜約2250、約1000〜約2000、約1250〜1750の乗算値が得られる副溝の深さ値である場合がある。
【0018】
本発明は、
(a)(i)同定されたMARの末端領域の少なくとも一部を含む単離ヌクレオチド配列、および
(ii)前記同定されたMARまたは別の同定されたMARの約10%、約15%、約20%、約25%、約30%もしくはそれより多くを含むさらなる単離ヌクレオチド配列、
または
(b)(i)(a)(i)のヌクレオチド配列と約90%、約95%、約96%、約97%、約98%、約99%の配列同一性を有するヌクレオチド配列、および
(ii)(b)(i)のヌクレオチド配列と約70%、約80%、好ましくは約90%、約95%、約96%、約97%、約98%、約99%の配列同一性を有するヌクレオチド配列
を含むMARコンストラクトも対象とする。
【0019】
本発明に記載の他のMARコンストラクトは、同定されたMAR配列もしくはその連続して並ぶ一部の領域を含み、ここでは順序および/または方向が同定されたMAR配列の場合と異なる。
【0020】
さらに、本発明に記載の他のMARコンストラクトは、
(a)(i)同定されたMAR配列の少なくとも1つの単離または合成されたATリッチ領域、または
(ii)(a)(i)のATリッチ領域と少なくとも少なくとも80%、85%、90%、95%、98%もしくは99%の配列同一性を有する少なくとも1つのATリッチ領域
を含むコアヌクレオチド配列と、
(b)(a)の前記ヌクレオチド配列に隣接した少なくとも1つのDNAタンパク質結合部位を含むヌクレオチド配列と、
を含み、ここで前記結合部位は、
(i)さらなる同定されたMAR配列のDNAタンパク質結合部位、
(ii)同定されたMAR配列内で(a)のコアヌクレオチド配列外部に位置する、(a)の同定されたMAR配列のDNAタンパク質結合部位、または
(iii)(a)のコア内に存在するが少なくとも1つのさらなるDNAタンパク質結合部位に隣接する第1のDNAタンパク質結合部位であって、第1のDNAタンパク質結合部位と前記さらなるDNAタンパク質結合部位の少なくとも1つとは(a)のコア内で隣接することがない、第1のDNAタンパク質結合部位、または、
(iv)非MAR配列のDNAタンパク質結合部位
である。
【0021】
本発明は、特定のMARコンストラクトのいずれかを含む発現系、特定の発現系のいずれかを含むキット、ならびに、MARコンストラクト、発現系、細胞、トランスジェニック非ヒト動物、キット、および/または、(1)ヒト病原体タンパク質またはヒト細胞表面タンパク質を認識する抗体などのタンパク質およびエリスロポエチン、インターフェロンまたは他の治療用もしくは診断用タンパク質などのタンパク質の生成、および/または(2)インビトロ、インビボ遺伝子療法、細胞療法または組織再生療法における本明細書中で参照される方法のいずれかの使用も対象とする。
【図面の簡単な説明】
【0022】
【図1】様々なMARの組換え緑色蛍光タンパク質(GFP)の生成に対する効果を示す。
【図2】組換え緑色蛍光タンパク質(GFP)のCHO細胞内での様々なヒトおよびマウスMAR要素の極めて高い生成体の百分率(%M3)に対する効果を示す。
【図3】様々なヒト1_68およびマウスS4 MAR要素の組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。
【図4】マウスMAR要素の組換えモノクローナル抗体の生成に対する効果を示す。
【図5】安定なポリクローナル集団がMARを有しない(MARなしの)場合またはシスに付加されたMAR S4を有する場合にIgG重鎖および軽鎖の発現を駆動するベクターを形質移入されたCHO細胞の集団から生成可能であったことを示す。
【図6】図6Aに、安定な各クローンが(B)におけるMARを有しない(MARなしの)場合またはシスに付加されたMAR S4およびMAR1_68を有する場合にIgG重鎖および軽鎖の発現を駆動するベクターを形質移入されたCHO細胞の集団から限界希釈により生成可能であったことを示す。図6Bに、安定な各クローンが(B)におけるMARを有しない(MARなしの)場合またはシスに付加されたMAR S4およびMAR1_68を有する場合にIgG重鎖および軽鎖の発現を駆動するベクターを形質移入されたCHO細胞の集団から限界希釈により生成可能であったことを示す。
【図7】図7Aに、MARを有しない場合の時間に伴う(2週および26週)遺伝子(GFP)の発現を示す。図7Bに、MARを有する場合の時間に伴う(2週および26週)遺伝子(GFP)の発現を示す。
【図8】図8Aに、はヒト1_68MARの曲げの特徴を示す。図8Bに、ヒト1_68MARの配列の特徴を示す。
【図9】図9Aに、同定された領域のシャッフリングにより得られた異なるMARコンストラクトおよび得られた転写の増大を示す。図9Bに、MARコンストラクト6の曲げパターンを示す。図9Cに、MARコンストラクト6の結合部位などの構造パラメータの詳細を提供する。
【図10】全集団の平均蛍光(Avg Gmean M0)の分析により示された、様々なMAR S4コンストラクトの組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。
【図11】全集団の平均蛍光(Avg Gmean M0)の分析により示された、組換え緑色蛍光タンパク質(GFP)の発現時に得られた様々なMAR S4コンストラクトを示す。
【図12】MATInspectorソフトウェアにより予測された、ヒト1_68MARの有望な転写因子結合部位のマップを示す。
【図13】ATリッチコア(MAR1429〜2880)とプロモーターおよび緑色蛍光タンパク質(GFP)の上流に配置された転写因子における化学合成されたDNA結合部位との構築物から作成された合成MARの活性について試験するのに用いられるプラスミドのマップである。
【図14】図13に記載のように作成された合成MARによる転写促進を図示したものである。
【図15】表5に詳述されるDNA結合部位を含む合成MARによる転写促進を図示したものである。
【発明を実施するための形態】
【0023】
本発明は、非ヒト動物由来の単離、精製されたMAR配列、それら配列を同定する方法およびヒト細胞内および齧歯類細胞などの非ヒト細胞内でのタンパク質の高収率の生成を目的としてそれら配列を用いる系に関する。
【0024】
本発明は、MARコンストラクト、特に促進されたMARコンストラクト、これらのMARコンストラクトを用いる発現系およびキット、ならびにタンパク質の生成、特に大規模生成や治療におけるそれらの使用も対象とする。
【0025】
さらに本発明は、ヒト細胞および非ヒト哺乳類細胞におけるMARコンストラクトを介するタンパク質の高収率の生成を目的とする方法を対象とする。
【0026】
他に特に規定されない限り、本明細書で用いられるあらゆる科学技術用語は、本発明に関係する当業者により一般に理解されている意味と同じ意味を有する。本明細書中に記載の方法および材料とは異なるものが本発明の実施において用いられうるが、典型的で(examplaratory)適切な方法および材料が下記に記載されている。
【0027】
本発明に記載の発現カセットは、少なくとも1つの遺伝子およびこの遺伝子の転写に必要とされる要素を含む核酸である。
【0028】
本発明に記載のプロモーターは、遺伝子の上流に位置する場合に遺伝子の転写を促進するDNAの制限領域である。
【0029】
細胞内での発現、例えば非ヒト哺乳類細胞内での発現は、本発明との関連ではインビトロおよびインビボでの発現を示す。インビトロ発現は、例えばHeLa細胞系またはCHO細胞系などの細胞系内およびインビトロ遺伝子療法において用いられる細胞内での発現を含む。インビボ発現は、トランスジェニックの非ヒト動物における発現や、遺伝子療法でのヒトレシピエントへの細胞の再導入後のインビボ遺伝子療法またはインビトロ遺伝子療法で用いられるヒト細胞内での発現を含む。
【0030】
本発明によると、哺乳類細胞、例えば非ヒト哺乳類細胞は、細胞培養条件下で維持可能である。細胞のこのタイプの非限定例がチャイニーズハムスター卵巣(CHO)細胞である。
【0031】
本発明に記載のMARコンストラクト、MAR要素、MAR配列、S/MARまたは単にMARは、天然の「SAR」または「MAR」と1つもしくは複数の(例えば2つ、3つもしくは4つの)特性を共有しかつ前記MARの作用により任意の遺伝子のタンパク質発現を促進する少なくとも1つの特性を有するヌクレオチド配列である。MARコンストラクトは、MAR活性、特に転写調節、好ましくは促進の活性だけでなく、例えば発現安定性の活性および/または「促進されたMARコンストラクト」としても記載の他の活性を伴う単離および/または精製された核酸としての特徴も有しうる。MARコンストラクトはその主要な基礎である同定されたMARに基づいて定義される場合があり、それ故、MAR S4コンストラクトはそのヌクレオチドの大半(50%以上)がMAR S4に基づくMARコンストラクトである。一般に認められたモデルによると、天然のSARまたはMARは、特定のDNA配列の核マトリックスに対する足場を媒介し、ヘテロクロマチンコアから外部に延在するクロマチンループドメインを生成する。SARまたはMARが任意の明確なコンセンサス配列もしくは認識可能な配列を有することはない一方、それらの最も一貫性のある特徴は全体的にAおよびT含量が高くかつC塩基が一方の鎖で支配的であることのように見える。MARは一般に、場合により鎖の分離を生じさせやすいベント二次構造を形成する傾向を有する。AおよびT含量の高いいくつかの簡単な配列モチーフがSARおよび/またはMAR内に見出されることが多いが、大部分ではそれらの機能的重要性および潜在的な作用様式は未解決のままである。これらは、脊椎動物またはショウジョウバエ(Drosophila)においては、Aボックス、Tボックス、DNA巻き戻しモチーフ(DNA unwinding motif)、SATB1結合部位(Hボックス、A/T/C25)およびコンセンサストポイソメラーゼII部位を含む。
【0032】
本発明に記載のMAR候補またはMAR候補配列は、天然のSARまたはMARと1つもしくは複数、例えば2つ、3つもしくは4つの特性を共有する配列である。
【0033】
本発明に記載の同定されたMARまたは同定されたMAR配列は、単離ヌクレオチド配列であり、かつその天然対応物のタンパク質/遺伝子の発現の十分な促進を可能にするあらゆる領域(「モジュール」または「要素」)を含む点で天然MAR配列に対応する。
【0034】
同定されたMARのモジュール(本明細書中で「領域」、「DNA領域」、「一部」、「ドメイン」とも称される)のすべては、天然MARの能力に対してタンパク質/遺伝子の発現の促進を可能にするのに必要とされる。一般に、単独でMARの十分な活性を発揮することが可能なモジュールは全く存在しない。これらの領域の一部、例えば下記のAT−ジヌクレオチドリッチのベント領域および転写因子結合部位(TFBS)領域は配列特異的である。他の「領域」については、それらの位置、例えば同定されたMAR配列の5’および3’末端領域により特徴づけられる。
【0035】
AT/TA−ジヌクレオチドリッチのベントDNA領域(以後「ATリッチ領域」と称される)は、特にジヌクレオチドATおよびTAの形態で大量のAおよびTを含むベントDNA領域である。好ましい実施形態では、それは隣接する100塩基対のストレッチ上のジヌクレオチドTAの少なくとも10%および/またはジヌクレオチドATの少なくとも12%、好ましくは隣接する100塩基対のストレッチ上(またはATリッチ領域の長さがより短い場合にはより短い各ストレッチ上)のジヌクレオチドTAの少なくとも33%および/またはジヌクレオチドATの少なくとも33%を有する一方、ベント二次構造を有する。しかし、「ATリッチ領域」は、約30ヌクレオチド長かもしくはそれに満たない程度に短い場合があるが、好ましくは約50ヌクレオチド長、約75ヌクレオチド長、約100ヌクレオチド長、約150、約200、約250、約300、約350もしくは約400ヌクレオチド長であるかまたはそれより長いものである。
【0036】
下記に考察されるように、ATリッチ領域は、例えばその比較的高い曲げ角度により、隣接領域、例えば結合部位領域から区別可能である。脊椎動物またはショウジョウバエ(Drosophila)におけるSATB1結合部位(Hボックス、A/T/C25)およびコンセンサスのトポイソメラーゼII部位などの一部の結合部位は比較的高いAおよびTの含量を有することも多い。しかし、一群の結合部位を含む結合部位領域(モジュール)、特にTFBS領域は、領域の曲げパターンの比較により、AおよびT含量の高い結合部位由来のATおよびTAジヌクレオチドリッチ領域(「ATリッチ領域」)から容易に識別可能である。例えばヒトMAR1_68においては、ATリッチ領域が平均で約3.8もしくは約4.0を超える曲度を有しうる一方、TFBS領域は平均で約3.5もしくは約3.3未満の曲度を有しうると思われる。同定されたMARの領域は、代替手段、例えば限定はされないが、本明細書中の他の箇所に記載の相対融解温度によっても確認可能である。しかし、かかる値は、種特異的であることから種間で異なる場合があり、例えばより低い場合がある。したがって、各々のATおよびTAジヌクレオチドリッチ領域は、約3.2〜約3.4もしくは約3.4〜約3.6もしくは約3.6〜約3.8など、より低い曲度を有する場合があり、かつTFBS領域は約2.7未満、約2.9未満、約3.1未満、約3.3未満など、比例的により低い曲度を有する場合がある。SMAR Scan IIでは、各々、より小さいウインドウサイズが当業者により選択されることになる。
【0037】
本発明に記載の同定されたMAR/MAR配列の末端領域は、同定されたMARの少なくとも約5%、約6%、約7%、約8%、約9%もしくは約10%を含む。
【0038】
結合部位またはDNAタンパク質結合部位は、DNA結合タンパク質に結合可能な任意のヌクレオチド配列である。DNA結合タンパク質に対する結合部位は典型的にはTFBSである。TFBSは転写因子に結合可能な任意の配列である。TFBSの供給源は、例えば限定はされないが、ヒトまたはマウスなど任意でありうる。TFBSは設計または合成も可能である。しかし特定の実施形態では、TFBSは、MAR配列内、例えば同一生物、同一種または同一属のMAR配列内に相当物を有する。しかし、TFBSは異なる種または異なる属のMAR配列に由来しうる。MAR配列内に現在既知の相当物を全く有しないTFBSについても本発明の範囲内である。かかるTFBSは、限定はされないが、USF1(上流刺激因子1)またはジンクフィンガータンパク質CTCFに対する結合部位を含みうる。TFBSは、1つ、2つ、3つ、4つ、5つもしくはそれより多くの置換、付加および/または欠失により修飾され、かつ完全または部分的に合成されうる。最適化されたTFBSは、各DNA結合タンパク質に対して最適化された結合親和性を有するTFBSであり、既知の天然対応物を全く有しないことが多く、本発明の範囲内でもある。それらの最適化されたTFBSであれば、天然のTFBSの上記の修飾によるかまたは合成、特に化学合成により生成可能である。本発明の特定の実施形態では、結合部位またはTFBSは、例えば組織特異的な天然か、設計または合成された制御タンパク質または他の天然か、設計または合成されたタンパク質による結合により、MARに組織特異性を与え、それは例えば特定の薬剤および分子に応答しうる。遺伝子および/または細胞療法は、組織特異性およびMARにおける特定の薬剤に特異的に応答する、つまり薬剤により誘発可能な能力から利益を得る典型的な場合である。前者の場合、例えば目的の遺伝子であれば特定の器官内または組織内に限って発現されることになり、後者の場合での発現であれば、例えば特定の薬剤への応答に限って刺激可能である。TFBSが含められうる転写因子の他の非限定例として、例えば、SATB1、NMP4、MEF2、S8、DLX1、FREAC7、BRN2、GATA1/3、TATA、Bright、MSX、AP1、C/EBP、CREBP1、FOX、Freac7、HFH1、HNF3α、Nkx25、POU3F2、Pit1、TTF1、XFD1、AR、C/EBPγ、Cdc5、FOXD3、HFH3、HNF3β、MRF2、Oct1、POU6F1、SRF、V$MTATA_B、XFD2、Bach2、CDP CR3、Cdx2、FOXJ2、HFL、HP1、Myc、PBX、Pax3、TEF、VBP、XFD3、Brn2、COMP1、Evil、FOXP3、GATA4、HFN1、Lhx3、NKX3A、POU1F1、Pax6および/またはTFIIAが挙げられる。
【0039】
結合部位、例えばTFBSは、コアヌクレオチド配列および結合部位の分離距離が約200以下、好ましくは約100ヌクレオチド以下、さらにより好ましくは約50ヌクレオチド以下、さらにより好ましくは約25以下、約15以下、約5ヌクレオチド以下であるかまたは0ヌクレオチドである場合、コアヌクレオチド配列に隣接するといわれる。好ましい実施形態では、結合部位、特にTFBSは、それ自体でTFBSの両側に最大で25ヌクレオチドの短いリンカーまたはアダプターを含む。さらにより好ましい実施形態では、TFBSは、最大で約50ヌクレオチド、最大で約40ヌクレオチドまたは最大で約30ヌクレオチドのオリゴマーの一部である。本発明に記載の一連の結合部位、例えばTFBSは、互いに隣接して配列内に並んだ一連のTFBSである。一連のTFBSは、コアに近いこの一連のTFBSが上で特定された距離を有する場合、コアヌクレオチド配列に隣接するといわれる。結合部位は、結合部位がコアヌクレオチド配列の一部の結合部位でありかつ天然のMAR内の同一位置に相当物を有する場合、「ATリッチ領域」に隣接するといわれる。
【0040】
結合部位は、1つ、2つ、3つ、4つ、5つもしくはそれより多くの置換、付加および/または欠失により修飾されうる。好ましくは、これらの置換、付加および/または欠失は、結合部位が各結合部位のコンセンサス配列に一致するように導入される。
【0041】
種々の促進されたMARコンストラクトは本発明の一部であり、かつ本発明に記載のMARコンストラクトの基礎となりうる天然でありかつ/または同定されたMAR、特にコア核酸配列の基礎となる天然のMARに対して全体的に促進をもたらす特性を有する。かかる特性は、限定はされないが、完全長で天然でありかつ/または同定されたMARに対する長さの短縮、遺伝子発現/転写の促進、発現安定性の促進、組織特異性、誘発性またはそれらの組み合わせを含む。したがって、促進されたMARコンストラクトは、例えば、同定されたMAR配列のヌクレオチド数の約90%未満、好ましくは約80%未満、さらにより好ましくは約70%未満、約60%未満、または約50%未満を含む。MARコンストラクトは、適切な細胞の前記コンストラクトによる形質転換時に遺伝子の発現および/または遺伝子の転写を促進しうる。本発明と関連し、「発現を促進する」か、「遺伝子発現促進活性」を有するか、「タンパク質発現を促進する」かまたはそれに類するMARコンストラクト/MAR(ヌクレオチド)配列についての参照がなされる場合、この「促進」は、例えばかかる配列の不在下である以外は等しい条件下で発現される遺伝子の発現と比較されるものである。促進は、例えば約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくは約15倍、約20倍もしくは約25倍またはそれより高い可能性がある。
【0042】
MARコンストラクトはまた、極めて高産生の細胞の平均百分率を約5倍、約10倍、約15倍もしくはそれ以上に高めうる。したがって、遺伝子の発現を平均的に高める以外に、極めて高発現の細胞の百分率の増加、ならびに安定な(「耐性のある」)コロニーの発生の増加(約100%、約200%、約300%もしくは約400%もしくはそれより高い増加)、および/または発現の変動性の低下(約30%、約40%、約50%もしくはそれ以上のcv(変動係数)の低下)が本発明の範囲内である。
【0043】
MARコンストラクトまたはそれに類するものは「発現の安定性を促進する」場合がある。この「促進」は、例えばMARコンストラクト/MAR配列の不在下である以外は等しい条件下で発現される遺伝子の発現と比較されるものである。安定性の促進では、例えば最大で約5週、10週、20週、25週、30週、35週、40週、45週、もしくは50週後に100%の促進が維持可能である。MARコンストラクトは、例えば筋肉、肝臓、中枢神経系もしくは他の組織に対して特異的である場合があり、および/あるいは、例えば抗生物質、ホルモンおよび/または代謝中間体などの物質の投与時に誘発されうる。
【0044】
MARコンストラクト/MAR配列は、好ましくは目的の遺伝子が作動可能に連結されるかまたは連結可能であるプロモーター領域の上流に挿入されうる。しかし特定の実施形態では、MARコンストラクトが目的の遺伝子/ヌクレオチド酸配列の上流および下流もしくは下流だけに位置することが有利である。シスおよび/またはトランスの双方の他の複数のMAR配列についても本発明の範囲内である。
【0045】
MARコンストラクトまたはMARの領域は、天然の「SAR」または「MAR」もしくはその各領域と1つもしくは複数の(例えば2つ、3つもしくは4つの)特性を共有しかつ前記MARの作用により任意の遺伝子のタンパク質発現を促進する少なくとも1つの特性を有する場合、例えば同定されたMARまたは同定されたMARの領域に基づくといわれる。これらのMARコンストラクトまたはMARの領域は、一般に、本明細書中で提供される用語の定義によると、それらの基礎となる同定されたMARと「実質的同一性」を有する。これらおよび/またはそれらのヌクレオチド配列の修飾にもかかわらず、それらは基礎となる同定されたMARの少なくとも1つの機能性/特性を維持することになる。
【0046】
本発明は、促進されたMARコンストラクトを含むMARコンストラクトの使用も対象とする。これらの使用では、MARコンストラクトは、1つもしくは複数の非MAR後生的遺伝子制御ツール、例えば限定はされないが、ヒストン脱アセチル化酵素(HDAC)などのヒストン修飾因子、遺伝子座制御領域(LCR)などの他のDNA要素、cHS4もしくは抗リプレッサー(antirepressor)要素(例えば安定剤)および抗リプレッサー要素(STARもしくはUCOE因子)などのインシュレーター、またはホットスポット(クワックス T.H.J.(Kwaks T.H.J.)およびオッテ A.P.(Otte A.P.))とも併用可能である。
【0047】
合成は、MAR/MARコンストラクトとの関連で用いられる場合、同定されたMARまたはそれに基づくMARの配列/領域または部分領域における単純なリシャッフリング、複製および/または欠失以上のものを含む設計であるMARを示す。特に、合成MAR/MARコンストラクトは、一般に同定されたMARの1つもしくは複数、好ましくは1つの領域を含むが、特定の実施形態では、場合によっては合成または修飾されるとともに特別に設計され、十分に特徴づけられた要素、例えば単一もしくは一連のTFBSであると思われ、それらは好ましい実施形態では合成的に生成される。これらのデザイナー要素は、多数の実施形態では比較的短く、特にそれらは一般に約300bps長以下、好ましくは約100、約50、約40、約30、約20もしくは約10bps長以下である。特定の実施形態では、これらの要素は多量体化されうる。
【0048】
本発明に記載の非ヒト哺乳類のMARは、少なくとも部分的には非ヒト哺乳類生物のゲノムまたはゲノムの一部を介して確認されるMAR/MAR配列である。これは例えば、限定はされないがマウスゲノムなどの齧歯類ゲノムの分析を介して同定されるMAR/MAR配列を含む。
【0049】
本発明に記載のベクターは、連結されている別の核酸分子を輸送可能な核酸分子である。例えば、プラスミドは1種のベクターであり、レトロウイルスまたはレンチウイルスは別種のベクターである。
【0050】
本発明に記載の形質移入は、例えば限定はされないが、エレクトロポレーション、リポフェクションによるか、ウイルスベクターまたは化学的手段を介する、核酸のレシピエント真核細胞への導入である。
【0051】
本明細書中で用いられる形質転換は、核酸の付加による真核細胞の修飾を示す。例えば、細胞の形質転換は、例えばDNAベクターのエレクトロポレーションを介する導入による細胞への核酸の形質移入を含みうると思われる。しかし、本発明の多数の実施形態では、本発明の促進されたMARを細胞に導入する方法は任意の特定の方法に限定されることはない。
【0052】
転写はDNA鋳型からのRNAの合成を意味する。
【0053】
シスは、同一の核酸分子、例えば限定はされないが同一のベクターまたは染色体上での2種以上の要素(例えばクロマチン因子)の配置を示す。
【0054】
トランスは、2種以上の核酸分子、例えば限定はされないが2種以上のベクターまたは染色体上での2種以上の因子(例えばクロマチン因子)の配置を示す。
【0055】
配列は、シス/トランス位置からその活性を発揮する場合、例えば遺伝子に対してシスおよび/またはトランスで作用するといわれる。
【0056】
本発明に記載のウインドウは、例えばSMAR Scanの方法を実施する間、MARについて評価される塩基対の数を表す。この数は通常、約50bps、約100bps、約200bps、約300bpsである。しかし、400bps、500bps、600bpsもしくはそれより大きいbpsのウインドウも本発明の範囲内である。
【0057】
ヌクレオチド配列またはその断片は、他のヌクレオチド配列(またはその相補鎖)と(適切なヌクレオチドの挿入または欠失により)最適に整列される場合、ヌクレオチド塩基の少なくとも約60%、通常は少なくとも約70%、より通常は少なくとも約80%、好ましくは少なくとも約90%、およびより好ましくは少なくとも約95〜98%のヌクレオチド配列の同一性がある場合に、もう一方と実質的同一性を有する。
【0058】
同一性は、2つのヌクレオチド配列間の配列の、かかる配列、例えば十分かつ完全な配列の2つの鎖間の一致を同一性により判定するときの、関連性の程度を意味する。同一性は容易に計算可能である。2つのヌクレオチド配列間の同一性を測定するには多数の方法が存在する一方で「同一性」という用語は当業者に周知である(「Computational Molecular Biology」、レスク A.M.(Lesk A.M.)編、オクスフォード大学出版会(Oxford University Press)、ニューヨーク(New York)、1988年;「Biocomputing:Informatics and Genome Projects」、スミス D.W.(Smith D.W.)ら、アカデミック・プレス(Academic Press)、ニューヨーク(New York)、1993年;「Computer Analysis of Sequence Data,Part I」、グリフィン A.M.(Griffin A.M.)およびグリフィン H.G.(Griffin H.G.)編、ヒューマナ・プレス(Humana Press)、ニュージャージー(New Jersey)、1994年;「Sequence Analysis in Molecular Biology」、フォン・ハインジェ G.(von Heinje G.)、アカデミック・プレス(Academic Press)、1987年;および「Sequence Analysis Primer」、グリブスコフ M.(Gribskov M.)およびデベルクス J.(Devereux J.)編、エム・ストックトン・プレス(M Stockton Press)、ニューヨーク(New York)、1991年)。2つの配列間の同一性を測定するのに一般に用いられる方法は、限定はされないが、「Guide to Huge Computers」、マーティン J.ビショップ(Martin J.Bishop)編、アカデミック・プレス(Academic Press)、サンディエゴ(San Diego)、1994年およびカリリョ H.(Carillo H.)およびリップマン D.(Lipman D.)、SIAM J Applied Math.48:1073頁(1988年)に開示された方法を含む。同一性を測定するための好ましい方法は、試験対象の2つの配列間の最大の一致が得られるように設計される。かかる方法は、コンピュータプログラム内にコード化される。2つの配列間の同一性を測定するのに好ましいコンピュータプログラムの方法は、限定はされないが、GCG(ジェネティックス・コンピュータ・グループ(Genetics Computer Group)、ウィスコンシン州マディスン(Madison))プログラムパッケージ(デベルクス J.(Devereux J.)ら、Nucleic Acids Research 12(1).387頁(1984年))、BLASTP、BLASTN、FASTA(アルツシュール(Altschul)ら(1990年);アルツシュール(Altschul)ら(1997年))を含む。同一性の測定のため、周知のSmith Watermanアルゴリズムも用いられうる。
【0059】
例として、参照ヌクレオチド配列と例えば少なくとも95%の「同一性」を有するヌクレオチド配列を含む核酸は、核酸のヌクレオチド配列が参照配列と、各参照ヌクレオチド配列の100個のヌクレオチド当たり最大で5つの点突然変異を含みうる点を除いて同一であることを意味する。言い換えると、参照ヌクレオチド配列に対して少なくとも95%同一のヌクレオチド配列を有するヌクレオチドを得るため、参照配列内のヌクレオチドの最大で5%が欠失されるかまたは別のヌクレオチドと置換されうるか、あるいは参照配列内の全ヌクレオチドの最大で5%の数のヌクレオチドが参照配列内に挿入されうる。参照配列のこれらの突然変異は、参照ヌクレオチド配列の5’もしくは3’末端位置またはそれら末端位置間のいずれかの場所で生じ、参照配列内のヌクレオチド内に別々にかまたは参照配列内の1つもしくは複数の隣接基内のいずれかで散在されうる。
【0060】
ヌクレオチド配列の機能的断片もまた本発明の一部である。断片は天然対応物の配列における特にそれらの作用を受ける遺伝子の発現を増大させるという所望の機能を維持する限り機能的であると考えられる。MARまたはMAR領域の断片は、その欠失がMAR/領域の転写促進活性を低下させてもそれを失わせることがない場合にはやはり機能的断片であると考えられる。「完全に機能的な断片」は、断片が他のMAR配列なしに用いられる場合に活性における任意の低下が、たとえ観察されたとしても、統計的に検証不可能である断片である。また、例えば天然MAR、同定されたMAR、MAR領域またはこれらのうちの任意の断片と本明細書で提供される定義に準じる実質的同一性を有する機能的断片は本発明の範囲内に含まれる。
【0061】
本明細書中で詳細に記載のように、特定の実施形態では、モジュールもしくはその一部がリシャッフルされ、複製され、かつ/または欠失が施される。当業者が理解するであろうように、かかる領域のシャッフリングおよび/または複製により、例えば新たな制限部位が生成され、次いでそのように生成されたコンストラクトの新たな制限パターンの生成が可能であり、かつ配列の長さが調節されることになりうる。それらの調節は、限定はされないが、1個、2個、3個、4個、5個、6個、7個、8個、9個、10個、10〜15個、15〜20個、20〜25個、25〜30個、30〜35個、35〜40個のヌクレオチドに作用しうる。これらの調節および他の修飾は本発明の範囲内である。各要素(または領域/モジュール)および/またはそれらの断片の各々と本明細書で提供される定義に準じる実質的同一性を有する、再配列されたMAR、特にリシャッフルされかつ/または複製されたMARの配列は本発明の範囲内である。
【0062】
MAR配列は、植物から哺乳類細胞またはその逆方向に移される可能性があり、異種の宿主細胞内の核マトリックス付着活性を保持することになる[ブレイン P.(Breyne P.)、ヴァン・モンタグ M.(Van Montagu M.)、デピッカー A.(Depicker A.)およびゲイセン G.(Gheysen G.)、ミエルケ C.(Mielke C.)、コーウイ Y.(Kohwi Y.)、コーウイ−シゲマツ T.(Kohwi−Shigematsu T.)およびボーデ J.(Bode J.)]。MARのこの保持がより高等なすべての真核生物において機能すると仮定すると、ある属由来のMAR配列であれば同配列が別の属などに由来する属内で同様に機能することを想定することになる。
【0063】
とはいえ、齧歯類由来のMAR配列であればある意味で組換えタンパク質の生成にとって有利でありうるとの判断から、全マウスゲノムがスクリーニングされ、下記のようなDNA配列の構造的特徴(例えばDNAベンド)を検出するコンピュータプログラム、SMAR Scan Iを用いてMAR候補配列が同定された。
【0064】
下記に考察のように、驚くべきことに、非ヒト、特に齧歯類(ここではマウス)のMAR配列が、例えばCHO細胞内やHeLa細胞などのヒト細胞内において発現促進に関してより強力であることが見出された。さらに一層驚くべきことに、特定の非ヒトMAR配列が、ヒトMAR配列よりも非ヒト細胞、例えばCHO細胞ならびにヒト細胞、例えばHeLa細胞の双方において実質的に優れた働きをすることが見出された。
【0065】
マウス由来の同定された新規のS/MAR DNA配列のいくつかがトランス遺伝子の発現を増大させることが示され、それ故、ヒトMAR配列用に設計されかつそれを用いて試験されたプログラムであるSMAR Scan Iが多数のゲノムの供給源、例えばヒトに加えてマウスに由来するS/MAR要素を同定するための効率的なツールであるという証拠が提供された。しかし重要なことには、より強力なMAR要素がヒトゲノムのスクリーニングよりも齧歯類(例えばマウス)ゲノムのスクリーニングにより同定可能であることが見出された。特に、本発明は、マウスゲノム由来の活性の高いS/MAR要素を用い、種々の細胞内、特にマウスおよびヒト細胞内で組換えタンパク質、例えば医薬品用途を有する組換えタンパク質の生成が増大可能であることを確立している。マウスS/MAR S4は、新規に単離されたマウスMARおよび予めクローニングされたヒトMARにおいて最も強力であることが示された。したがって、本発明は、促進されたンパク質生成を有する非ヒトMARおよび/またはタンパク質発現の安定性を長期にわたり高めるMARを対象とする。
【0066】
SMAR Scan Iは、これらの配列の構造的かつ物理化学的な特徴に基づきMAR候補配列を同定するソフトウェアツールである。本方法についての徹底した考察が他に提供されている(マーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書)。「SMAR Scan」は、本質的には、ジヌクレオチド重み行列に基づいて特性を認識し、DNAの高次構造的かつ生理化学的な特性における理論値を計算するアルゴリズムを含むバイオインフォーマティクスツールを示す。好ましくは、SMAR Scanは可変サイズの走査ウインドウを用い、多種多様な組み合わせでのDNAベンディング、主溝の深さおよび副溝の幅のポテンシャル、融解温度に対応するDNA配列の特徴を評価する。各特徴においては、カットオフまたは閾値が設定される必要がある。プログラムは、所与領域の計算されたスコアが設定されたカットオフ/閾値を超えるごとにヒットを返す。
【0067】
ヒットを扱うため、2つのデータ出力モードが使用可能であり、第1のモード(「プロファイル様(profile−like)」)は、単純に、クエリー配列上のあらゆるヒット位置および選択された異なる基準に対するそれらの対応する値を返す。第2のモードは(「隣接ヒット」と称される)は、いくつかの隣接ヒットの位置およびそれらの対応する配列のみを返す。このモードにおいては、隣接ヒットの最小数は、さらに調節可能なウインドウサイズで設定可能な別のカットオフ/閾値である。例えば4つの理論上の構造的基準におけるデフォルトのカットオフ/閾値を調節するため、例えばSMARt DBから実験的に検証されたMARを用いてもよい。このように、例えばデータベースからのすべてのヒトMAR配列が、4つの基準を有しかつ設定されたカットオフ/閾値を全く有しない「プロファイル様」モードを用いるSMAR Scanで検索され、分析された。これは配列の全位置における各機能の設定を可能にした。次いで、各基準における分布はこれらのデータにより計算された(図1および3を参照、マーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書)。
【0068】
SMAR Scan技術の利用がMAR配列の同定のために好ましいものである一方、当業者は、同程度かまたはやや低めでもある選択性を有するS/MARモチーフの同定を可能にする他のバイオインフォーマティクスツールが本発明との関連で使用可能であることを理解するであろう。好ましくは、設定された閾値またはカットオフ値である特定の値を超えるこれらの特徴を示すMARに関連した特徴のみが正のヒットを生成するかまたは生成するように設定可能であるようにかかるツールを設定可能である。しかし、MARの同定に用いられる多数のバイオインフォーマティクスツールが、マトリックス−結合活性を同定するように設計された。この活性は遺伝子発現を増大させる能力と必ずしも相関するものではない[フィー−バン L.(Phi−Van L.)およびストラトリング W.H.(Stratling W.H.)]。
【0069】
SMAR Scan IはヒトMARの同定を目的に開発されている。したがって、それは既知のヒトMARから収集された構造データを用いて開発された。ヒトに「調節された」SMAR Scan Iプログラムが本発明との関連で用いられ、マウスゲノムのMAR配列についての評価がなされた。しかし、マウスおよびヒトゲノムの塩基組成における差異から、ヒトゲノムを走査するための予め規定された設定を有するSMAR Scanプログラムの使用が抑えられた(マーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書)。したがって、プログラムによる管理可能に回収された候補マウスMAR配列の同定が可能になるまで、明確なウインドウサイズおよび構造パラメータの閾値が試行錯誤により規定される必要があった。それらのいくつかは、試験される場合、例えば各タンパク質をコードする遺伝子を有するベクター上に配置されかつ齧歯類細胞系に導入される場合、結局はタンパク質生成の実質的増大を可能にするMAR配列である「スーパー(super)MAR配列」となった。
【0070】
マウスMAR S4およびマウスMAR S46は、本発明の範囲内である齧歯類MAR配列の例である。単離されたこれらのMAR配列は添付の配列表で配列番号3および配列番号10として示される。しかし、当業者が理解するように、塩基対の挿入、欠失、置換、特に塩基対の挿入、欠失または置換を自ら有しうるこれらや他の非ヒトMARの断片は、野生型配列の特にそれらの作用により遺伝子の発現を増大させるという所望の機能を維持する限り本発明の範囲内である。例えば、MAR配列の転写/遺伝子発現促進活性を低下させてもそれを失わせることがない挿入が、所望の機能、ここではMARの遺伝子発現促進と実質的に干渉することがないと考えられる。同様に、例えば同定されたMARの断片が、同定されたMARに対してやや低下した転写促進活性を有しても転写促進活性を完全に失わせることがない場合、やはり機能的断片であると考えられる。「完全に機能的な断片」は、活性における任意の低下が、観察されたとしても統計的に検証不可能である場合の断片である。本明細書中の他の箇所に詳述のように、天然MARもしくはその断片のヌクレオチド配列と「実質的同一性」を有する配列も本発明の範囲内に含まれる。
【0071】
MARのモジュール性
同定されたMARの分析により、MARがモジュール(または領域)、特に配列特異的モジュールを含むか否かが判定され、それを同定されたMARの設計または合成領域を含むMARを含む合成MARの生成に利用できた。事実、同定されたMARのいくつかの配列特異的モジュールを確認できた。驚くべきことに、特定のモジュールもしくはその一部のシャッフリングおよび/または完全もしくは部分的な複製やさらには欠失により、上記のような促進されたMARがもたらされることが見出された。
【0072】
マウス由来のヒト1_68MARおよびS4 MARは、領域のシャッフリング、欠失および/または複製によるMARコンストラクトを生成するためのモデルとして役立つことになる。しかし、当業者が容易に理解するように、本発明は、任意の同定されたMARの操作およびそれから得られるMARコンストラクトを対象とする。異なる由来のMARを含む異なるMARに対応するのに必要でありうる適切な調節は、当業者の技能の範囲内で十分に可能である。例として、限定はされないが、真核生物、好ましくは哺乳類、特にマウスなどのモデル動物、およびウシ、ブタ、ヒツジなどの経済的重要性のある種ならびにヒトが挙げられる。
【0073】
ヒトMARのモジュール性
ヒト1_68MARは、領域のシャッフリングおよび/または複製によりMARコンストラクトを生成するためのモデルとして役立った。下記のように確認されたモジュールもしくはその一部の使用により、MARコンストラクトが同定されたMAR、例えばヒト1_68MARに基づいて生成された。MARコンストラクトは、特に領域(モジュール)もしくはその一部のシャッフリングおよび/または複製により生成された。
【0074】
1_68MARの例は、同定されたMARの(本明細書中で領域または要素とも称される)モジュールが遺伝子発現の促進を天然のMARの能力に対して可能にするのにすべて必要であったことを示す。MARの完全な活性を単独で発揮できた同定されたモジュールは存在しなかった。驚くべきことに、特定のモジュールのシャッフリングおよび完全もしくは部分的な複製の結果、遺伝子発現のさらなる促進が得られることが見出された。
【0075】
いくつかの非冗長な配列特異的モジュール(領域)が同定された。これらのモジュールは、局所クロマチン構造に作用するように協働する。MARのこの機構は、後生動物の転写の制御にやや類似するものであり、すなわち開始部位から最大で数キロベースにわたり分散されたモジュールの多様な集合が全体として、転写が開始することになる場所を決定づける。
【0076】
同定された配列特異的なモジュールは、特に(1)AおよびT含量の高い領域、例えば対称なATリッチ領域(AおよびTが交互にある)、特に「ATリッチ領域」、および(2)結合部位が豊富な領域、特に、限定はされないがATリッチ領域に分離されたTFBSであった。
【0077】
AおよびT含量の高いベントDNAが一般にプロモーター領域、MARおよびレプリケーターにおいて見出されることが報告されている[アラジェ(Aladjem)およびファニング(Fanning)、2004年]。従来、AおよびT含量の高い配列(上記の「対称」配列および一方の鎖には主にAおよび他方の鎖には主にTを有する配列である「非対称」配列)は、主に二本鎖の開鎖を促進すると考えられた。しかし、これらの領域は幅広い範囲の機能を有しうると思われる。例えば、ラミンB2レプリケーター内のAおよびT含量の高い配列が複製開始点認識複合体(ORC)に結合し[アブドゥラシドバ(Abdurashidova)、ダナイロフ(Danailov)ら、2003年;ステファノヴィッチ(Stefanovic)、スタノイチッチ(Stanojcic)ら、2003年]、インビトロでMcm4/6/7ヘリカーゼの負荷および二本鎖DNAの巻き戻しを促進しうる[ヨウ(You)、イシミ(Ishimi)ら、2003年]。AおよびT含量の高い本質的なベントDNAにおける構造的役割についても検討されている。分裂酵母ORC4の「AT−フックDNA−結合モチーフ」は、移動度の高い集団であるタンパク質HMG−I/Yのそれに類似するものであり、かかる構造的役割を有しうる[ストリック(Strick)およびラエムリ(Laemmli)、1995年;ベル(Bell)、2002年]。V(D)Jの組換えを促進するHMG−I/Y媒介性のDNAベンディングに類似したタンパク質媒介性のベンディング、および真核生物内のエンハンサーおよびプロモーターでの転写複合体の構築および安定化も生じうると思われる[レビン(Levine)およびチアン(Tjian)、2003年]。高いAおよびT含量を有する領域のすべてがベントDNAに対応するわけではない。しかし、それらDNAはベントであり、そうであればヒストンを引き付けてベントDNA全体にヌクレオソームを形成するための「ヒストンマグネット(histone magnet)」として作用可能であり、複製/転写前タンパク質に対するランディングパッド(landing pad)として自由に作用する隣接領域が残る。
【0078】
上記のように、MARは特に「結合部位が豊富な領域」または単に「結合部位領域」(上記(2)を参照)内に他のタンパク質に対する結合部位も有しうる。それらの他のタンパク質は、限定はされないが、1_68MAR内に見出されるようなDNA巻き戻し要素−結合タンパク質(DUE−B)およびHoxタンパク質、SATBI、CEBPなどの転写因子を含みうる。突然変異分析によると、これらの結合部位がMAR機能に寄与することが示される。
【0079】
ヒト1_68MARは、その方向を反転させかつベントDNAを離すことで改善され、プロモーター領域の上流の転写因子結合部位領域のサイズの拡大が可能であった。図9でわかるように、多数のこれらの再配列されたMAR(例えばコンストラクト6)は、MARを有しないコンストラクトに対し(10倍)、また天然のMARを含むコンストラクトに対しても(コンストラクト1および16;約2倍)転写をかなり増大させる。示されるデータは、末端の転写制御因子自体が下流のクロマチンにおける転写開始を制限することも明確に示す。天然MARにおける前方の斜線ボックスとして示される、領域の3’末端に位置する223bpの断片が、コンストラクト11と比べ、コンストラクト7内のこの領域におけるあらゆる活性を保持する。これは、この重要な部分がこの場合にベント領域およびコンストラクト6内のこの因子の残り(ヌクレオチド1〜1425)の5’末端と協働することを示唆している。2つのHMG−I/Y部位がこの末端の近隣に位置することが見出された。コンストラクト2では、2つの同定されたMAR配列が結合し合うことにより発現も増大されることが示される。
【0080】
マウスMARのモジュール性およびサイズの低下
いくつかのMARがS4 MARに基づき構築され(表3)、かつ特徴づけられた(図10)。図10で示されうるように、1600bps長を超える断片における内部の欠失により、MARの活性の低下はあまり生じなかった(S4−1−703_2328−5457)。しかし、プロモーターに隣接する795−bp断片の欠失またはこの配列の同様の長さのルシフェラーゼ遺伝子の断片による置換(S4_1−4661;S4_1−4661−Luc5489)により、この活性の完全な低下が誘発された。
【0081】
非配列特異的モジュール:3’末端のMAR配列の活性
ヒト1_68MARを用いる実験(図9)では、ヒト1−68MARの3’HoxFおよびSATBI結合部位領域の重要性が既に示された。この領域の重要性は、図10に示されるマウスMAR S4を用いる実験によりさらに明示された。図11に示されるように、MAR S4の3’末端配列の活性のさらなる分析のため、MARのこの部分をその一部を取り出すかまたは複製することによってさらに分析した。図11は、様々なMAR S4誘導体の遺伝子発現に対する効果についても示す。興味深いことに、切断された3’末端を有する1つのかかる誘導体(元のMAR S4における4658〜5054対4658〜5457)では、平均で、トランス遺伝子の発現がより長い元のMAR S4配列よりもやや高い(104%対100%)ことが示された。これは、MAR要素のより強力でかつより短い誘導体が得られうることを示している。
【0082】
したがって、本発明は長さが天然対応物よりもかなり短い高活性のMARコンストラクトを含むことから、それらは例えばベクターの設計および転移にとってより都合のよいサイズということになる。
【0083】
特に、同定されたMAR配列のヌクレオチドの数の約90%未満、好ましくは約80%未満、さらにより好ましくは約70%未満、約60%未満もしくは約50%未満を含むMARコンストラクトが本発明の範囲内である。それらのコンストラクトは、好ましくは同定されたMARの3’末端領域、さらにより好ましくは同定されたMAR/MAR配列の3’末端領域の少なくとも約5%、約6%、約7%、約8%、約9%もしくは約10%を含む。しかし、同定されたMARの5’末端領域を有するMARコンストラクトも本発明の範囲内である。
【0084】
合成MAR
ヒト1_68MARの再配列では、特定の実施形態では、単離されたMARの前方の斜線部分の3’末端に位置するHoxリッチ領域の223bpの断片が完全長領域の活性を保持することが示された。これは、本発明の特定の実施形態ではこの部分が他の因子との協働において重要でありうることを示唆している。図12は、MATInspectorソフトウェアによる予測としてのMAR1_68の有望な転写因子結合部位の配列を示す。C/EBP、NMP4、FAST1、SATB1、およびHoxF結合部位の位置が例示されており、それはそれらが5’側(前方の斜線)のフランキング配列内に豊富に存在することを図示している。
【0085】
ヒトMAR1_68内のATリッチのベントDNA領域と転写因子結合部位の間で協働が生じうるという発見により、1つもしくは数箇所の転写因子結合部位に隣接するMAR1−68のATリッチ領域を含むMAR/MARコンストラクトの構築が促進された。図13は、ATリッチ領域およびATリッチ領域の各末端での同定されたMARのTFBSを含むコア(MAR1429〜2880)と緑色蛍光タンパク質(GFP)におけるプロモーターの上流に配置された転写因子のための化学合成されたDNA結合部位との構築物から作成された合成MARの活性についての試験用に用いられるプラスミドのマップを示す。図13は、特に、転写因子結合部位がATリッチドメインとGFPトランス遺伝子の発現を駆動するSV40プロモーターとの間に挿入され、図9で見出された状況が再現されたことを示し、ここでの最も好ましい設定では結合部位を有するMAR部分がプロモーターとベントDNA領域との間に挿入される(コンストラクト6)。表4は、用いられた化学合成されたオリゴヌクレオチドのDNA配列を示す。
【0086】
C/EBP、NMP4、FAST1、SATB1、およびHoxF(Gshとも称される)転写因子に対する結合部位がMAR1−68配列から同定された(図12)。MAR1−68内に存在するこれらの結合部位は変化なしに用いられるか(FAST1、C/EBP、HOXF/Gsh)、またはコンセンサス(すなわち完全な)配列(HoxF、SatB1、NMP4)と比べて1つもしくは2つのミスマッチを有する場合に訂正された。
【0087】
図14からわかるように、ここでの合成結合部位の付加により、ほぼすべての場合、いくつの特定の場合、ATリッチ領域を含むコアMAR配列に対して有意な転写促進がもたらされた。活性ではC/EBPおよびHoxまたはGsh2が最高でSatB1およびFast1が次に高い一方、単一のNMP4部位が検出可能な効果を全く有しなかった。
【0088】
図14は、ATリッチ領域の基礎となる同定されたMARの結合部位に隣接された、ここでのMAR1_68に基づくMAR1429〜2880であるコア配列の挿入により、発現においてあまり改善が得られなかったが、1つもしくは複数の結合部位をさらに含むMARコンストラクが、特にプロモーターの上流以外のATリッチコアの下流に挿入される場合、プロモーターの制御下での遺伝子によるタンパク質の発現/生成においてかなりの促進が得られたという驚異的な結果を示す(ここではM3細胞の%により同定)。
【0089】
好ましい実施形態では、追加の結合部位がプロモーターの上流以外のATリッチコアの下流に存在する一方、例えば限定はされないが、ATリッチ領域の上流、ATリッチ領域内、コアのATリッチ領域の隣接領域または遺伝子の下流における位置といった他の構成も本発明の範囲内である。
【0090】
好ましい実施形態では、合成もしくは単離のいずれかのタンパク質結合部位の特定の組み合わせ、例えば2つの異なるタンパク質結合部位、3つの異なるタンパク質結合部位の組み合わせ、4、5、6、7、8、9、10もしくはそれより多くのタンパク質結合部位の組み合わせが検討される。これらの組み合わせは完全または部分的に多量体化されうる。好ましい実施形態では、こうした組み合わせはHox/GshおよびSATB1を含む。これらの組み合わせまたは多量体化された組み合わせの例えばコアと適切なプロモーターとの間への挿入により、高発現体(high expressor)クローンの発生が、MARコンストラクト/MAR配列を含まないベクターがそれ以外は等しい条件下で用いられる場合での高発現クローンの発生と比較し、約2倍もしくはそれ以上、例えば限定はされないが、約3倍、4倍、5倍、6倍、7倍、8倍、9倍もしくはそれ以上、好ましくは約10倍もしくはそれ以上、さらにより好ましくは、約11倍、12倍、13倍、14倍、15倍、16倍、17倍、18倍、19倍もしくはそれ以上または約20もしくはさらに約25倍もしくは約30倍もしくはそれ以上に高まる可能性がある。
【0091】
要するに、MARコンストラクトはビルディングブロックから構築可能である。これらのビルディングブロックは、同定されたMARもしくはその一部の領域、例えば配列特異的領域、合成ビルディングブロック(その機能性を最適化するための修飾を含む)、例えば一連の化学合成された転写因子結合部位(TFBS)、非MAR配列に由来するかもしくはそれに基づくビルディングブロック、または異なる種もしくは属のMAR配列に由来するかもしくはそれに基づくビルディングブロックを含むかまたはそれに基づくものでありうる。好ましい実施形態では、かかるMARは、表5に示される、TFBS領域に共役されたATリッチ領域または特異的な転写因子DNA結合部位の組み合わせを含む。当業者は、これらの原則が特定の配列または本明細書中に開示される結合部位に限定されず、かつ他の誘導体、相同体または配列の組み合わせも本発明の範囲内であることを理解するであろう。
【0092】
上記のように、本発明のMARコンストラクト、発現系および/またはキットはタンパク質の生成において用いられうる。ここでMARコンストラクトは、プロモーターの制御下での目的のタンパク質、例えばインスリンに対する遺伝子を含むベクター内に含められうる。ベクターは細胞に導入され、細胞は成長される。次いで、このプロセスはインスリンの大規模なバッチ生産のために大規模化される。例えばMARコンストラクトを伴わない場合よりも3〜5倍多いインスリンの大量生産が3週にわたり維持されうる。
【0093】
上記のように、本発明のMARコンストラクト、発現系および/またはキットは、インビトロおよび/またはインビボ遺伝子療法や細胞および組織の置換療法において用いられうる。例えば、インビトロ遺伝子療法では、MARコンストラクトが、プロモーターの制御下での、インビトロ遺伝子療法を必要とする患者において欠損している遺伝子を含むベクター内に含められうる。次いで、MARコンストラクトは、患者の細胞、例えば骨髄細胞に導入される。MARコンストラクトでの形質転換後、骨髄細胞は患者に導入され、目的の遺伝子の発現がMARコンストラクトを伴わない場合よりも5倍高いレベルで先行しうる。それ故、有効量のタンパク質の発現が可能である。インビボ遺伝子療法では、MARコンストラクトを含むベクターが、治療を必要とする患者の細胞に例えば注射で直接導入されうる。
【0094】
同様に、本発明の発現系は、組織再生のための移植または例えば神経変性疾患に対する神経細胞療法において幹細胞に導入されうる。本発明のこの実施形態にて用いられうる幹細胞の非限定例として、任意の年齢の個体の骨髄組織または新生児の臍帯血から得られる造血幹細胞(HSC)および間葉幹細胞(MSC)が挙げられる。幹細胞は、本発明に記載の発現系を用いて形質移入され、奏功した形質転換体が細胞療法もしくは組織再生療法を必要とする患者に移植または再導入されうる。形質転換された幹細胞、例えばNucleofection(登録商標)(Cell Line Solution V(VCA−1003)、ドイツのアマクサ(amaxa GmbH))を入手するため、いくつかの方法が利用可能である。
【0095】
ヒト抗原に結合する抗体を含む多種多様なタンパク質を生成可能なトランスジェニック動物は、公知の方法(例えば限定はされないが、ロンバーグ(Lonberg)らに交付された米国特許第5,770,428号明細書、米国特許第5,569,825号明細書、米国特許第5,545,806号明細書、米国特許第5,625,126号明細書、米国特許第5,625,825号明細書、米国特許第5,633,425号明細書、米国特許第5,661,016号明細書および米国特許第5,789,650号明細書)により生成可能である。発現系およびMARコンストラクトは、例えばトランスジェニックのウシ、ヒツジ、ヤギまたはブタを介した、典型的にはタンパク質の体液(例えば乳)への分泌によるタンパク質の生成において使用可能である。例えば、メーデ(Meade)らに交付された米国特許第5,750,172号明細書を参照のこと。またトランスジェニック動物の生成については、ルボン(Lubon)らに交付された米国特許第6,518,482号明細書を参照のこと。
【実施例】
【0096】
本発明は以下の実施例でさらに説明されることになるが、これは、特許請求の範囲、発明の概要または本明細書中の他の箇所で示される本発明の範囲を限定するものではない。材料、方法、および実施例はあくまで例示目的であり、限定するように意図されていない。当業者は、本明細書で提供される指針に伴い、変更、追加および改善を行うことができ、そのすべては本発明の範囲内である。
【0097】
マウスゲノムのS/MAR予測:SMAR Scan I
NCBI m34マウスの構築物に対応するすべてのマウス染色体配列を集め、SMAR Scan Iで分析した。ストリンジェンシーの低いものと高いもののスクリーニングを3.6°のDNAベンディングの基準および300bpの最小ウインドウサイズにおける閾値または4.2°および100bpの最小ウインドウサイズにおける閾値のいずれかを各々用いて行った。
【0098】
全マウスゲノムのSMAR Scan Iによる低いストリンジェンシーの分析により、全体で622,410bp(全マウスゲノムの0.024%)を表す全部で1496の推定上のS/MAR(候補MAR)が得られた。表1は、各染色体におけるそのサイズ、その遺伝子数、その予測されるMAR(候補MAR)の数、遺伝子当たりのそのMAR密度およびkb単位でのS/MAR間の平均距離を示す。この表は、(MAR当たり遺伝子の密度の平均の約50%を表す標準偏差では)異なる染色体に対する予測されるS/MAR(候補MAR)当たりの遺伝子密度が様々であることを示す。MAR当たりの遺伝子における高い方の密度と低い方の密度との間の倍数の差異は、予測されるMAR(候補MAR)内にそのサイズおよびその遺伝子数に対して極めて豊富に存在する染色体Yを考慮しない場合に6であり、これはこれらのMARの分布における強力かつ想定外のバイアスを示す。表1は、S/MAR間の平均距離(S/MAR当たりのkb)が可変である(標準偏差はS/MAR当たりのkbの平均の38%を表しかつS/MAR当たりのkbの高い方の密度と低い方の密度との間の倍数の差異は8.3である)ことも示す。染色体10、11、XおよびYは、これらの密度の高い標準偏差に有意に寄与する。
【0099】
SMAR Scan Iが元来ヒト配列に対して調整されていることから、最もストリンジェントなパラメータを用いる場合、マウスゲノム配列ではMARはほとんど得られず、それ故、高いストリンジェンシーのスクリーニング(DNAベンディングの基準においては4.2°の閾値)においては、デフォルトのカットオフ値をMARと考えられるような隣接ヒット(contiguous hits)の最小サイズに300bpではなく100bpのウインドウを用いて調節した。マウスゲノムのSMAR Scan Iによる分析によると、DNAベンディングの基準として4.2°を超える値を有する49の「スーパー」MARが予測された。
【0100】
【表1】

【0101】
組換えタンパク質の生成を増大させるための新規に同定されたマウスMARの使用
5つのMAR要素を、SMAR Scanを用いる完全なマウスゲノムの高いストリンジェンシーのスクリーニングで得られた推定上のMAR(候補MAR)から選択した。それらを、オークランド小児病院研究所(Children’s Hospital Oakland Research Institute)(CHORI、http://bacpac.chori.org/)から購入したマウスゲノムDNAの細菌の人工染色体由来のプラスミドベクター内でクローニングした。
【0102】
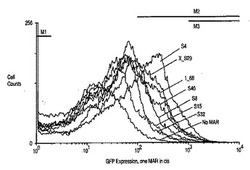
これらの新規に同定されたマウスMARをS4、S8、S15、S32およびS46と名づけた(SMAR Scan Iによる同定の順序による、「スーパー」MARのS1〜S49)。ヒトMARの1_3、1_6、1_9、1_42、1_68、3_S5およびX_S29は予め同定されており、MARの1_68およびX_S29が最も強力なヒト因子である(マーモド(Mermod)ら、「High efficiency gene transfer and expression in mammalian cells by a multiple transfection procedure of MAR sequences」、国際公開第2005/040377号パンフレット、またマーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書も参照)。上記のように、これらのMARを緑色蛍光タンパク質の発現を駆動するSV40プロモーターおよびエンハンサーの上流のpGEGFP対照ベクターに挿入し、これらのプラスミドを培養されたCHO細胞に形質移入した[ジロド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)]。次いで、安定的に形質移入された細胞の全集団内でのトランス遺伝子の発現を蛍光セルソーター(FACS)機器を用いて分析した。図1は、様々なS/MARの組換え緑色蛍光タンパク質(GFP)の生成に対する効果を示す。蛍光活性化セルソーター(FACS(登録商標))により示される、MARを含むかもしくは含まないGFP発現ベクターpGEGFPを形質移入したCHO細胞の集団、および典型的な特性が示される。最も強力なヒトMARの1_68およびX_S29のみがこの図に示される。特性では、GFP蛍光レベルの関数としての細胞数の計数値が示される。2より小さい蛍光値(M1)、または102より大きい相対発光量(M2)もしくは103より大きい相対発光量(M3)を有する細胞小集団M1、M2およびM3を表す水平バーが示されている。
【0103】
図1からわかるように、新規に同定されたすべてのマウスMARで、トランス遺伝子の発現がMARを伴わずにGFP単独で駆動される発現を有意に上回る程に増大し、ここでの「スーパー」マウスMAR S4は示されるすべてのMARの中で最も強力であった。
【0104】
【表2】

【0105】
最も強力なヒトMARの1_68およびX_S29の転写活性を新規に同定されたマウスMARで得られた場合と比較した。5つのマウスMARを最初にGFP発現アッセイを介して試験し、それらがすべてGFPの発現を異なるレベルに増大させることが見出された。マウスMARのS15およびS32は、比較では転写活性が最低のMARであり(GFP単独よりも約2倍の増大)、S8およびS46は中間の活性を示し(3〜4倍の増大)、かつMAR S4は極めて高い転写活性を示した(7倍の増大)。さらに、マウスMAR S4は、この試験における試験対象のあらゆるMARの中で最も強力なものである。ヒトMAR1−68およびマウスMAR S4の間の転写活性の比較によると、全集団(Gmean M0)および高いGFP生成細胞(M2)の平均蛍光が50%増大したことが示される一方、マウスMAR S4の場合、極めて高いGFP生成細胞(M3)の百分率は175%高めであった。マウスMAR S4の場合、GFP蛍光(CV M0)の観点での全集団の同質性は常に1〜2%低く、それは細胞産生性(cell productivity)の安定性の高まりを示すことから有利である。
【0106】
このクローニングの第1ラウンド後、活性が高いMAR要素がマウスゲノムから常に得られうるか否かを判定しようとした。したがって、2つのさらなるマウスMAR(S6およびS10)をクローニングし、特徴づけた。これらの新しいマウスMARをpGEGFP対照ベクターに挿入し、上記のようにFACSで分析した。マウスMAR S10はFACSで分析されたあらゆる異なるパラメータにおいて最高のヒトMARよりも強力であるように見られ、MAR S4とほぼ同程度に転写活性が高く発現全体を増大させるものである。
【0107】
極めて高い生成体について評価するため、M3細胞の百分率をヒトMAR1_68において得られた百分率に正規化した。結果を図2に示す。図2は、様々なヒトおよびマウスのS/MAR要素の、組換え緑色蛍光タンパク質(GFP)の極めて高い生成体の百分率(%M3)に対する効果を示す。指定のMAR要素を有するかまたは有しないGFP発現ベクターを形質移入したCHO細胞の集団を蛍光活性化セルソーター(FACS(登録商標))で分析した。極めて高い生成体の百分率をこの基準において最高のヒトMARで得られた百分率に正規化し、MAR1_68の値を100に設定した。マウスMARのS10およびS4により、各々、ヒトMAR1_68よりも平均で80%および180%といった極めて高い生成体細胞が得られた。全体として、7つのマウスMARと7つのヒトMARとのこの比較から、齧歯類MARを用いたCHO細胞からより高い発現が得られると判断した。
【0108】
異なる細胞種内での新規に同定されたマウスMARにおける効力の評価
S4 MARの効力をCHO細胞内で評価した。さらに、ヒトMAR1−68、マウスMAR S4もしくはMARなしのいずれかを含むEGFP発現ベクターをヒトHeLa細胞内に安定的に形質移入し、EGFP蛍光を分析した。図3は、様々なヒト1_68およびマウスS4 MAR要素の、組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。HeLa細胞の集団に形質移入し、表2に記載のように分析した。HeLa細胞内でのS4および1−68MARの効力の比較においては、S4はいくつかの観点で1_68より優れることが見出され、すなわちS4は高い平均GFP蛍光(平均Gmean M0)とともに中程度の発現および高い発現の範囲内(各々、M1およびM2)でより多くの細胞をもたらし、また発現の変動の低下(平均CV M0)をもたらした。HeLa細胞を用いる極めて高い発現の範囲内(M3)では全く細胞が見出されなかった。
【0109】
マウスMARを用いるモノクローナル抗体の発現促進
マウスMAR、特に最も強力なマウスを用い、医薬品用途のタンパク質の生成が増大するか否かを判定するため、それらをRhesus−Dを認識する免疫グロブリンの重鎖および軽鎖をコードするpMZ37およびpMZ59ベクターに挿入した[ミーシャー S.(Miescher S.)、ザーン−ザバル M.(Zahn−Zabal M.)、デ・ジーザス M.(De Jesus M.)、マウドリ R.(Moudry R.)、フィッシュ I.(Fisch I.)、ヴォーゲル M.(Vogel M.)、コブル M.(Kobr M.)、イムボーデン M.A.(Imboden M.A.)、クラグテン E.(Kragten E.)、ビヒラー J.(Bichler J.)、マーモド N.(Mermod N.)、スタドラー B.C.(Stadler B.C.)、アムシュトゥッツ H.(Amstutz H.)、ブルム F.(Wurm F.)]。上記のように、これらのプラスミドをCHO細胞内に形質移入し、選択および免疫グロブリンアッセイを行った[ジロッド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)]。図4は、S/MAR要素の組換えモノクローナル抗体の生成に対する効果を示す。ここで、CHO細胞に、MARを有しない(MARなし)またはシスで付加されたMAR S4を有するIgG重鎖および軽鎖の発現を駆動する上記のベクターを形質移入した。24、48および72時間後、上清中のIgGの力価を測定した。さらに図5に示されるように、安定なクローンを、MARを有しない(MARなし)またはシスで付加されたMAR S4を有するIgG重鎖および軽鎖の発現を駆動する上記のベクターを形質移入したCHO細胞の集団から生成した。選択後、分泌されたIgGの力価を培地内で測定し、特異的な産生性を細胞計数によりアッセイした。図6(A)は、安定な個々のクローンの、MARを有しない(MARなし)またはシスで付加されたMAR S4を有するIgG重鎖および軽鎖の発現を駆動するベクターを形質移入したCHO細胞の集団からの限界希釈による生成後に得られた結果を示す。選択後、分泌されたIgGの力価を培地内で測定し、特異的な産生性を細胞計数によりアッセイした。また、MAR1_68の場合に得られた結果とMARを含まないクローンの場合に得られた(B)の結果との比較が含まれる。図3〜6で得られ示された結果は、新規に同定されたマウスMAR、特にMAR S4を用い、一時的なトランスフェクタント(図4)内および安定的なトランスフェクタント(図5および6)内で医薬品タンパク質、例えばモノクローナル抗体の生成を増大可能であることを示す。MAR S4を用いる場合、5pg/細胞/日(pcd)程度もしくはそれを超える特定の産生性を有する安定なクローンを数種の候補クローンの分析から容易に同定可能である(図6(A))。実に21の最高のクローンの平均産生性が、MAR S4を用いる場合または用いない場合で各々、7.28±0.78pcd(図6(A))および2.61±1.09pcdであった。これらの結果は、既知のニワトリのリゾチームMARの場合に得られた力価レベル(1.5mg/L未満)またはMARを有しない場合に得られた力価レベル(0.5mg/L未満)に対して際立つものである。特にこれらの結果は、新規に同定されたマウスMARを用いることで限定はされないがモノクローナル抗体などの医薬品用途のタンパク質の生成が増大し、マウスMAR、例えばMAR S4が特に組換えタンパク質の生成において興味深いものになりうることを示す。
【0110】
ヒトMAR1_68の場合の発現の安定性
MAR1_68の使用から、MARを有しないクローンにより生成された遺伝子の発現が一般に無変化であり、MARを有する同じクローンが高レベルの発現を長期間維持するだけでなく無変化の細胞が発現を回復することを実証した。
【0111】
図7は、ジロッド(Girod)ら、2005年に記載のように、MAR1−68を含むpEGFP発現プラスミドのG418抗生物質耐性遺伝子を有するCHO細胞への同時形質移入を示し、安定的に発現された細胞をG418の存在下で3週間かけて選択した。細胞クローンを限界希釈により取得し、9つの個別のクローンにおけるGFP蛍光について分析した。さらなる分析のため、GFPを発現する典型的なクローンを2つの集団の各々から選択し、抗生物質選択の存在下および不在下でさらに最長で26週間培養した。特性が左側に2週間の培養後のGFP蛍光レベル(x軸)およびy軸上に細胞計数を示す一方、右側の特性は26週間培養した細胞から得られたものである。図示のように、MARを含まないクローンでは抗生物質の不在下での26週後におけるGFP蛍光レベルが2週後のレベルに対して低下したことを示す一方、MARを含むクローンでは抗生物質選択の有無にかかわらず26週時のGFP蛍光レベルを維持できたことから、MARを含む発現系は目的の遺伝子の安定な発現にとって有用なものとなる。
【0112】
MARのモジュール性および遺伝子の発現促進との関連
MARの構造分析により、各々が遺伝子の発現促進に寄与するDNA配列領域/モジュールが示された。図8は、1_68MARの構造分析を介して得られた結果を示す。図8(A)は、中央のATリッチ領域がMAR1_68の遺伝子座内でのベントDNAを決定づけることを示す。図8(B)は、このATリッチ領域がMatInspector(カータリウス(Cartharius)、フレック(Frech)ら、2005年)により同定された転写因子結合部位に豊富に存在する領域で囲まれていることを示す。正確には729の有望なTFBSをMAR配列に沿ってMatInspectorにより検出した。図8(B)の下部は、同定された領域に対するコーディングの特性を示す。
【0113】
図9(A)は、1_68MARと、左側に1_68MARの領域もしくは部分を組み込み、かつその領域もしくは部分の順序および/もしくは方向を変化させ、かつ/またはそのかかる領域もしくは部分を複製する異なるMARを示す。右側ではMAR1_68を有するかまたはMARを全く有しない場合に得られた転写の増大の程度とともにコンストラクト1〜16により得られた転写の増大の程度を示す。示されるすべてのMAR配列を、eGFP遺伝子マーカーを駆動するプロモーターの上流に挿入した。矢印は、図8に示される野生型MAR配列と比べての、その領域もしくは断片の方向を示す。ATリッチ領域を囲む配列を、矢印を付けた後方の斜線ボックス(左側)および矢印を付けた前方の斜線ボックス(縮尺不定;右側)として示す。ベント領域を斜交平行線ボックスとして示す。
【0114】
図9(B)は図9.Aにおけるコンストラクト6に対応するMARの曲げパターンを示す。これらの曲げパターンをSMARScanIにより測定した。
【0115】
図9(C)は、MatInspector[カータリウス(Cartharius)、フレック(Frech)ら、2005年]分析の結果を示す。有望な転写因子結合部位(TFBS)をMatInspector[カータリウス(Cartharius)、フレック(Frech)ら、2005年]により同定した。731の有望な部位をMAR配列に沿ってMatInspectorにより検出する。図9(C)最下部のコンストラクト6を図8(B)および図9(A)に対応するコーディングを用いて示す。この図の最下部のコーディングは、図9(A)に示され考察されたものに対応する。
【0116】
図9に示される実験は、単独で完全なMAR活性を示す領域が全く存在しないことを示す。例えば、DNA転写の促進を天然のヒト1_68MARから最大限得るのに、3つの異なる配列(図8)、すなわち複数の転写因子に対する結合部位を有する1189bpのセグメント(すなわちCEBP)(図9A上部、矢印を付けた後方の斜線ボックスとして示される)、本質的には763bpの対称性のある(AおよびTが交互にある)ATリッチ領域により決定づけられるベントDNA(図9A上部、斜交平行線ボックス)、および多数のHoxFおよびSATBI結合部位を含む追加の1648bpのセグメント(図9A上部、矢印を付けた前方の斜線ボックスとして示される)が必要である。
【0117】
図9は、ベントDNAを離してプロモーター領域の上流の転写因子結合部位領域のサイズを拡大することにより、ヒト1_68MARが改善されることを示す。この拡大を行うため、転写因子結合部位(TFBS)領域、ここではATリッチ(配列番号18)領域に隣接するHoxリッチ領域(配列番号19)(以後、矢印を付けた前方の斜線領域である)を、CEBPリッチ領域(配列番号17)(以後、後方の斜線領域でもある(図9))に隣接させた。図9Aの右側に示される、生成された異なるMARコンストラクトの転写促進活性の比較によると、矢印を付けた前方の斜線領域の方向が転写の増大にとって重要であったことが示される(コンストラクト5および6を比較)。示されるデータは、末端の転写制御因子自体が下流のクロマチンにおける転写開始を制限することも明示している。矢印を付けた前方の斜線領域の3’末端に位置する223bpの断片(配列番号20)がコンストラクト7内のこの領域の完全な活性を保持すると仮定すると、この場合、この重要な部分がコンストラクト6内のこの要素のベント領域および残りの5’末端(ヌクレオチド1〜1425)と協働しなければならないことが示唆される。2つのHMG−I/Y部位がこの末端の隣接位置に存在することが見出された。
【0118】
マウスMARのモジュール性およびサイズの縮小
ヒト1_68MARの場合の実験結果に基づき、S4 MARにおけるモジュール、特にその転写活性を担うモジュールについても分析した。またこの分析を、比較的長いS4 MARのサイズの縮小を目標に行った。したがって、いくつかのMARをS4 MARから作成し(表3)、特徴づけた(図10)。図10は、左側に特定のMAR S4コンストラクト、および右側に全集団の平均蛍光(Avg Gmean M0)の分析により示された様々なMAR S4コンストラクトの組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。指定のMARコンストラクトを含むかまたは含まないGFP発現ベクターを形質移入したCHO細胞の集団を、FACScaliburサイトメーター(ベクトン・ディッキンソン(Becton Dickinson))を用いるフローサイトメトリーにより分析した。全集団の平均蛍光をヒトMAR1_68で得られたもの(その値を100に設定)に対して正規化する一方、GFPはMARの不在下での発現を示す。他のMARコンストラクトを完全長の1547bpのS4 MARに対してのそれらの塩基含量に準じて名づけた(表3を参照)。ドット付きボックスは、MAR S4のATリッチのベント領域を示す。S_41−4662−Luc5489は、末端(3’)の795塩基対が除去されかつルシフェラーゼ遺伝子の一部(黒色ボックス)と置換される場合のコンストラクトを示す。興味深いことに、図10からわかるように、1624−bp EcoRI断片がS4 MAR(S4−1−703_2328〜5457)からそのMAR活性の有意な低下を伴うことなく欠失されうることが見出された。しかし、プロモーターに隣接する795−bpの断片の欠失またはこの配列と類似の長さを有するルシフェラーゼ遺伝子の断片(S4_1−4661;S4_1−4661−Luc5489)との置換により、この活性の完全な低下が誘発された。これは、マウスS4 MARの特定の変異体は、長さが短縮する一方で高い活性を示すことで、それが例えばベクターの設計および転移にとってより好都合なサイズになりうることを示す。
【0119】
【表3】

【0120】
3’末端のMAR配列における活性
MAR S4の3’末端配列の活性をさらに分析するため、MARのこの部分についての詳細な分析をその一部の除去または複製を介して行った。図11は、全集団の平均蛍光(Avg Gmean M0)の分析により示された、様々なMAR S4誘導体の組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。CHO細胞の集団を生成させ、上記のようにアッセイした。興味深いことに、切断された3’末端を有する1つのかかる誘導体(元のMAR S4の4658〜5457に対して4658〜5054)では、より長い元のMAR S4配列と比べてトランス遺伝子の発現が平均でわずかに高いことが示された(100%に対して104%)。これは、より強力でかつより短いMAR要素の誘導体が得られうることを示す。
【0121】
合成MAR
図12は、MATInspectorソフトウェアの予測による[1_68MARの]有望な転写因子結合部位のマップを示す。C/EBP、NMP4、FAST1、SATB1、およびHoxF(Gshとも称される)結合部位の位置を例示し、それらが5’側の前方の斜線フランキング配列内に豊富に存在することが図示される。MAR1−68内に存在するこれらの結合部位を変化を加えずに用いるか(FAST1、C/EBP、HOXF/Gsh)またはそれらにコンセンサス(すなわち完全)配列に対して1つもしくは2つのミスマッチを有する場合には訂正した(HoxF、SatB1、NMP4)。
【0122】
ヒトMAR1_68内でのATリッチのベントDNA領域と転写因子結合部位の間で生じうる連携に関する実験結果から、1つもしくはいくつかの転写因子結合部位に隣接するMAR1−68のATリッチ部分を含む合成MARの作成が促進された。図13は、ATリッチ領域を含むコア(MAR1429〜2880)とプロモーターおよび緑色蛍光タンパク質(GFP)の上流に配置された転写因子における化学合成されたDNA結合部位との構築物から作成された合成MARの活性について試験するのに用いられるプラスミドのマップを示す。図13は、転写因子結合部位がATリッチのコアとGFPトランス遺伝子の発現を駆動するSV40プロモーターの間に挿入されたことを示し、図9に見出される状況を再現するものであり、この場合、最も好ましい設定では結合部位を有するMAR部分がプロモーターとベントDNA領域の間に挿入される。表4は、用いた化学合成されたオリゴヌクレオチドのDNA配列を示す。
【0123】
【表4】

【0124】
図14は、図13に記載のように構築された合成MARによる転写促進を示す。挿入された要素は、図示のようにコアに加えて1つもしくはいくつかのタンパク質DNA−結合部位を有する。ATリッチ領域を含むコア配列(ATリッチコア)に加えて1つもしくはいくつかの結合部位を有するプラスミドの形質移入では、結合部位を含めることでATリッチコア単独の場合と比べて転写促進が増大し、かつ活性ではC/EBPおよびHoxまたはGsh2が最高でSatB1およびFast1が次に高い一方、単一のNMP4部位が検出可能な効果を全く有しないことが示された。
【0125】
活性結合部位の異なる混合物についても試験し、相乗効果が観察可能であるか否かを判定した。その実施のため、異なる転写因子に対する結合部位を有するオリゴヌクレオチドの様々な組み合わせをDNAライゲーション反応において混合し、結合部位の正確な順序および配列をDNA配列決定により判定した。得られた組み合わせを表5に示す。
【0126】
【表5】

【0127】
得られたプラスミドを上記のように形質移入により試験した。図15は、表5に示されるDNA結合部位の組み合わせを用いて作成された合成MARによる転写促進を示す。最も活性の高い組み合わせが星印で示され、HoxF/Gsh2またはSatB1部位の存在が示される。図15に示される結果は、この例では合成MARの活性が挿入された結合部位の数に依存しなくても、結合部位の特定の組み合わせが高い促進活性を示す一方、それ以外では活性がないかまたはさらに遺伝子発現が抑制されることを示す。より高い活性を有するコンストラクトがこの場合にHox/Gsh2およびSATB1タンパク質の組み合わせを含み、かつ最も活性の高いコンストラクトはこれらの要素のみからなる。この合成MARの挿入により、発現体クローンの発生が任意のMAR配列を含まないpEGFP対照ベクターに対して約10倍増大した。
【先行技術文献】
【非特許文献】
【0128】
【非特許文献1】Abdurashidova G, DanailovB, et al., "Localization of proteins bound to a replication origin of humanDNA along the cell cycle." EMBO J 22: 4294-4303, 2003.
【非特許文献2】Aladjem, MI and Fanning E.,"The replicon revisited:an old model learns new tricks in metazoan chromosomes." EMBO Rep 5(7):686-91, 2004.
【非特許文献3】Allen GC, SpikerS, Thompson WF, Use of matrix attachment regions (MARs)to minimize transgene silencing, Plant Mol Biol.,43(23):361-376, 2000.
【非特許文献4】Amati B and Gasser SM, ChromosomalARS and CEN elements bind specifically to the yeast nuclear scaffold, Cell,54:967-978, 1988.
【非特許文献5】Amati B and Gasser SM, Drosophilia scaffold-attached regions bind nuclearscaffolds and can function as ARS elements in both budding and fission yeasts,Mol. Cell. Biol., 10:5442-5454, 1990.
【非特許文献6】Bell SP, "The originrecognition complex: from simple origins to complex functions." Genes Dev16: 659-672, 2002.
【非特許文献7】Bode J, SchlakeT, Rios-Ramirez M, Mielke C, StengartM, Kay V and KlehrWirth D, Scaffold/matrix-attachedregions: structural properties creating transcriptionallyactive loci, Structural and Functional Organization of the Nuclear Matrix:International Review of Cytology, 162A:389-453, 1995.
【非特許文献8】Bode J, BenhamC, Knopp A and Mielke C,Transcriptional augmentation: modulation of gene expression byscaffold/matrix-attached regions (S/MAR elements), CritRev Eukaryot Gene Expr,10(1):73-90, 2000.
【非特許文献9】Bode J, Stengert-IberM, Kay V, Schlake T and Dietz-PfeilstetterA, Scaffold/matrix-attached regions: topological switches with multipleregulatory functions, Crit. Rev. Euk.Gene Exp., 6:115-138, 1996.
【非特許文献10】Bodnar JW, A domain model foreukaryotic DNA organization: a molecular basis for cell differentiation andchromosome evolution, J. Ther. Biol., Vol.132:479-507, 1988.
【非特許文献11】Boulikas T, Nature of DNA sequences atthe attachment regions of genes to the nuclear matrix, J. Cell Biochem., 52:14-22, 1993.
【非特許文献12】Boulikas T, Chromatin domains andprediction of MAR sequences. In Structural and Functional Organization of theNuclear Matrix: International Review of Cytology, Academic Press, Orlando, 162A:279-388, 1995.
【非特許文献13】Breyene P, Van MontaguM and Gheyseu G, The role of scaffold attachmentregions in the structural and functional organization of plant chromatin, TransgenicRes., Transgenic Res., 3:195-202, 1994.
【非特許文献14】Breyne P, Van MontaguM, Depicker A and GheysenG, Characterization of a plant scaffold attachment region in a DNA fragmentthat normalizes transgene expression in tobacco,Plant Cell, 4:463-471, 1992.
【非特許文献15】Cartharius, K., K. Frech,et al., MatInspector and beyond: promoter analysisbased on transcription factor binding sites, Bioinformatics 21: 2933-42, 2005.
【非特許文献16】Gasser SM and Laemmli UK, Cohabitation of scaffold binding regions withupstream/enhancer elements of three developmentally regulated genes of D. Melanogaster, Cell, 46:521-530, 1986.
【非特許文献17】Girod PA, Zahn-ZabalM and Mermod N, Use of the chicken lysozyme 5' matrix attachment region to generate highproducer CHO cell lines, Biotechnol. Bioeng., 91(1):1-11, 2005.
【非特許文献18】Kas E and ChaslinLA, Anchorage of the Chinese hamster dihydrofolate reductase gene tothe nuclear scaffold occurs in an intragenic region,J. Mol. Biol., 198:677-692, 1987.
【非特許文献19】Kay V and Bode J, Detection ofscaffold -attached regions (SARs) by in vitrotechniques; activities of these elements in vivo. In Methods in Molecular andCellular Biology: Methods for studying DNA protein interactions: an overview, WileyLiss, NewYork, 5:186-194,1995.
【非特許文献20】Kim JM, Kim JS, Park DH, KangHS, Yoon J, Baek K and Yoon Y, Improved recombinantgene expression in CHO cells using matrix attachment regions, J. Biotechnol., 107(2):95-105, 2004.
【非特許文献21】Kwaks TH, OtteAP, Employing epigenetics to augment the expressionof therapeutic proteins in mammalian cells. Trends Biotechnol.24:137-42, 2006.
【非特許文献22】Labrador, M. and V. G. Corces, Setting the boundaries of chromatin domains andnuclear organization, Cell 111: 151-54, 2002.
【非特許文献23】Levine, M. and R. Tjian, Transcriptional regulation and animal diversity,Nature 424: 147-151, 2003.
【非特許文献24】Mielke C, KohwiY, KohwiShigematsu T and Bode J, Hierarchical bindingof DNA fragments derived from scaffold-attached regions: correlation ofproperties in vitro and function in vivo, Biochemistry, 29:7475-7485, 1990.
【非特許文献25】Miescher S, Zahn-ZabalM, De Jesus M, Moudry R, FischI, Vogel M, Kobr M, ImbodenMA, Kragten E, Bichler J, Mermod N, Stadler BC, Amstutz H, Wurm F, CHO,Expression of a Novel Human Recombinant IgG1 anti-RhD Antibody Isolated by Phage Display, Brit. J. Haematol.,111, 157-166, 2000.
【非特許文献26】National Center for Biotechnology Information(http://www.ncbi.nih.gov).
【非特許文献27】PhiVan L and StratlingWH, Dissection of the ability of the chicken lysozymegene 5' matrix attachment region to stimulate transgeneexpression and to dampen position effects, Biochemistry, 35:10735-10742, 1996.
【非特許文献28】Razin SV, Functional architectureof chromosomal DNA domains, Crit Rev Eukaryot Gene Expr, 6:247-269,1996.
【非特許文献29】Stefanovic D, StanojcicS et al., In vitro protein-DNA interactions at the human laminB2 replication origin, J Biol Chem278: 42737-42743, 2003.
【非特許文献30】Strick R and LaemmliUK, SARs are cis DNA elements of chromosome dynamics: synthesis of a SARrepressor protein, Cell 83(7): 1137-48, 2005.
【非特許文献31】Vogelstein B, Pardoll D and Coffey D, Supercoiledloops and eukaryotic DNA replication, Cell, 22:79-85, 1980.
【非特許文献32】You Z, IshimiY, et al., Thymine-rich single-stranded DNA activates Mcm4/6/7 helicase on Y-fork and bubble-like substrates, EMBO J 22:6148-6160 (2003).
【非特許文献33】Zahn-Zabal M, Kobr M, GirodPA, Imhof M, Chatellard P,de Jesus M, Wurm F and MermodN, Development of stable cell lines for production or regulated expressionusing matrix attachment regions. J Biotechnol, 87(1):29-42, 2001.
【技術分野】
【0001】
関連出願の相互参照
本願は、2006年8月23日に出願された米国仮特許出願第60/823,319号明細書および2007年8月3日に出願された米国仮特許出願第60/953,910号明細書の優先権を主張するものであり、それら全体が参照により本明細書中に援用される。
【0002】
本発明は、単離、精製されたヒトおよび非ヒト動物由来のMAR配列に対応するかまたはそれに基づくヌクレオチド配列を含む核酸に関する。これらの核酸は、一般に転写および/またはタンパク質生成促進活性を有する。本発明は、例えば高収率のタンパク質生成を目的としたかかる配列を同定するための方法およびそれらを用いる系にも関する。
【背景技術】
【0003】
本発明を例示し、特に実施についてのさらなる詳細を提供するための本明細書で用いられる特許を含む出版物および他の資料は、参照により本明細書中に援用される。便宜上、出版物は、文章中に完全に記載されない限り、添付の参考文献においてアルファベット順に列挙される。EMBL登録番号AC102666およびEMBL登録番号BH101870およびBH101901に隣接する配列ならびにEMBL登録番号(同義語)126658、23119391、22981746もまたそれら全体が参照により本明細書中に援用される。
【0004】
現在では、真核生物染色体の約50〜100kbのクロマチンループドメインへの組織化のモデルが広く受け入れられている[ボドナー J.W.(Bodnar J.W.)、ブレイン P.(Breyne P.)、ヴァン・モンタグ M.(Van Montagu M.)およびゲイセン G.(Gheysen G.)、ラジン S.V.(Razin S.V.)]。これらのループの外端は、核マトリックス、すなわちRNP(リボ核タンパク質)および他の非ヒストンタンパク質からなるタンパク質網状構造(proteinaceous network)に付着された特定のDNA配列に対応すると考えられる[ボーデ J.(Bode J.)、ベンハム C.(Benham C.)、クノップ A.(Knopp A.)およびミエルケ C.(Mielke C.)]。核マトリックスに付着された染色体DNA配列は、足場付着領域(中期中)またはマトリックス付着領域(間期)の各々に対してSARまたはMARと称される。S/MAR、MAR要素もしくはMAR配列または略してMARは、典型的には300〜3000bp長の多形領域である。哺乳類の核内には約100,000のMARが存在すると推定されている[ボーデ J.(Bode J.)、ステンガート−イベル M.(Stengert−Iber M.)、ケイ V.(Kay V.)、シュラケ T.(Schlake T.)およびディエツ−ファイルステッター A.(Dietz−Pfeilstetter A.)]。
【0005】
クロマチンをループドメインに構造的かつ機能的に分離することにより、MAR要素は、遺伝子発現の複製および調節において重要な役割を果たす、例えば哺乳類の核内での転写の中心部(foci)の連続的な構築および分解を促進すると考えられている。この概念を支持するように多数の間接的証拠がもたらされており、例えば様々な真核ゲノム内でDNAの複製起点がMAR要素内にマッピングされた[アマチ B.(Amati B.)およびガッサー S.M.(Gasser S.M.)(1988年)、アマチ B.(Amati B.)およびガッサー S.M.(Gasser S.M.)(1990年)]。MARは、イントロン内部の非コード遺伝子間領域内[ジロド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)]または転写単位の境界[ガッサー S.M.(Gasser S.M.)およびラエムリ U.K.(Laemmli U.K.);国立バイオテクノロジー情報センター(National Center for Biotechnology Information)]にもほぼ常に見出され、そこでは遍在的でかつ/または組織特異的な転写因子に結合しうる。概して、植物や動物細胞系におけるトランスジェニックの実験では、MAR要素を用い、トランス遺伝子の発現および安定性を増大させることに奏功している[アレン G.C.(Allen G.C.)、スパイカー S.(Spiker S.)、トンプソン W.F.(Thompson W.F.)、ボーデ J.(Bode J.)、シュラケ T.(Schlake T.)、リオス−ラミレス M.(Rios−Ramirez M.)、ミエルケ C.(Mielke C.)、ステンガート M.(Stengart M.)、ケイ V.(Kay V.)およびクレア−ヴィルス D.(Klehr−Wirth D.)、ジロド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)]。例えば、MARを用い、バイオテクノロジーや治療用途に関連する細胞、例えばCHO(チャイニーズハムスター卵巣)細胞において様々な組換えタンパク質の生成が増大している[ジロド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)、キム J.M.(Kim J.M.)、キム J.S.(Kim J.S.)、パーク D.H.(Park D.H.)、カン H.S.(Kang H.S.)、ユーン J.(Yoon J.)、バエク K.(Baek K.)およびユーン Y.(Yoon Y.)、ザーン−ザバル M.(Zahn−Zabal M.)、コブル M.(Kobr M.)、ジロド P.A.(Girod P.A.)、イムホフ M.(Imhof M.)、シャテラード P.(Chatellard P.)、デ・ジーザス M.(De Jesus M.)、ブルム F.(Wurm F.)およびマーモド N.(Mermod N.)](マーモド(Mermod)ら、「Development of stable cell lines for production or regulated expression using matrix attachment regions」、国際公開第02074969号パンフレット、また米国特許出願公開第20030087342号明細書)。
【0006】
MARの機能的活性は、その主要なDNA配列ではなくその構造的特性に関連づけられている。確かにMARではAおよびT含量が高く[ボウリカス T.(Boulikas T.)(1993年)]、いくつかの特定の高次構造的かつ物理化学的な特性、例えば分子の自然曲率、狭い副溝、巻き戻し/不対の高い可能性または変性への感受性が観察されている[ボーデ J.(Bode J.)、シュラケ T.(Schlake T.)、リオス−ラミレス M.(Rios−Ramirez M.)、ミエルケ C.(Mielke C.)、ステンガート M.(Stengart M.)、ケイ V.(Kay V.)およびクレア−ヴィルス D.(Klehr−Wirth D.)、ボウリカス T.(Boulikas T.)(1993年)、ボウリカス T.(Boulikas T.)(1995年)]。事実、まさしくそれらの特性を用いることで、MARがSMAR Scanと称される方法を通じて同定されている。さらに、MARの活性はまた、DNA結合タンパク質、例えば一本鎖および/または曲線状のDNAなどのMAR要素の特定の構造的特徴を認識しうるクロマチン改変酵素および/または転写因子に媒介されうる[ボーデ J.(Bode J.)、ステンガート−イベル M.(Stengert−Iber M.)、ケイ V.(Kay V.)、シュラケ T.(Schlake T.)およびディエツ−ファイルステッター A.(Dietz−Pfeilstetter A.)]。明確なタンパク質の結合部位またはMARコンセンサス配列は全く見出されておらず[ボウリカス T.(Boulikas T.)(1993年)]、それはゲノム配列由来のMARの予測を困難にしている。
【0007】
MARの特定の機能的かつ構造的な特性については記載がなされている一方、一次構造の観点ではほとんど共有されてないことからその同定は困難である。動物のMARが植物核の足場に結合し、その逆もいえるという事実により支持される仮定では、MAR要素が真核ゲノム内で機能的に保存されうる一方[ブレイン P.(Breyne P.)、ヴァン・モンタグ M.(Van Montagu M.)、デピッカー A.(Depicker A.)およびゲイセン G.(Gheysen G.)、ミエルケ C.(Mielke C.)、コーウイ Y.(Kohwi Y.)、コーウイ−シゲマツ T.(Kohwi−Shigematsu T.)およびボーデ J.(Bode J.)]、いかなる特徴がMAR配列、例えば強力なタンパク質を生成する配列をもたらすかについて語れることはほとんどない。また、様々な結果が用いられるアッセイに応じて得られうる[ラジン S.V.(Razin S.V.)、ボウリカス T.(Boulikas T.)(1995年)、ケイ V.(Kay V.)およびボーデ J.(Bode J.)]。真核生物内での大量の予想されたMARおよびゲノムプロジェクトで公表された配列の量を考慮し、MAR DNA配列(SMAR Scan I)または調節タンパク質または転写因子として作用する特定のタンパク質に対する結合部位などの機能的配列(SMAR Scan II)の構造的特徴を検出するためのツール/プログラムが開発された[マーモド(Mermod)らに交付された2007年8月3日に出願された米国仮特許出願第60/953,910号明細書、米国特許出願公開第20070178469号明細書]。かかるプログラムは、DNAベンディング、主溝の深さおよび副溝の幅のポテンシャルに対応する一群のDNA配列の特徴の検出により新規の有望なMAR配列を同定するとともに、特定の転写調節タンパク質に対する結合部位を同定するように設計された。これらのプログラムを用いてヒトゲノムを走査することで、推定上のMAR DNA配列が同定されており、それらのいくつかはCHO細胞に形質移入される発現プラスミドに導入される場合にトランス遺伝子の発現を増大させることが示された(ジロド(Girod)ら、「Identification of S/MAR from genomic sequences with bioinformatics and use to increase protein production in industrial and therapeutic processes」、マーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書]。これは、SMAR Scanプログラムがヒト遺伝要素、つまりタンパク質の合成を増大させるのに用いられうるものを効率的に同定しうることを実証した。これまで実施された機能的スクリーニングがヒトゲノムに限定されていたが、大規模な生成では目的のタンパク質が非ヒト哺乳類細胞内で発現されることが多い。
【発明の概要】
【発明が解決しようとする課題】
【0008】
約1600のMARがヒトゲノム内でSMAR Scanにより同定されており、8つのうち6つが、エンハンサー/プロモーターの上流に配置される場合、CHO細胞内で(例えば緑色蛍光タンパク質(GFP)、抗体および受容体に対する)遺伝子の発現促進を引き起こすことが示された。異所性MAR活性を有することが示されたDNAの長さは2.5kb〜6kbの範囲である。しかし、現段階ではMARの構造的特徴づけの欠如が「デザイナー(designer)」MARの生成を制限している。したがって、MARの改変や設計を可能にするため、MAR、特にMARの機能的かつ/または構造的領域を特徴づける必要性が存在する。
【0009】
これまで実施された機能的スクリーニングはヒトゲノムに限定された。大規模な生成では目的のタンパク質が哺乳類細胞内で発現されることが多いことから、転写および/または遺伝子発現を促進するより強力な天然のMAR、ならびに/あるいはヒトおよび/または非ヒト哺乳類細胞内で強力なタンパク質生成体(producer)の細胞を同定するという必要性も存在する。
【0010】
概して、例えばさらなる天然のMARを同定し、同定されたMARを設計し、かつ/または合成MARを生成することにより、有利な特性を有するMARを同定しかつ/または生成するという必要性が存在する。有利な特性、限定はされないが、転写の促進および/またはタンパク質生成/遺伝子発現の特性;天然MARに対する長さの短縮により、例えば遺伝子工学におけるより多目的の使用が可能になる点;外的刺激物、例えば薬剤の添加時での組織、細胞または器官の特異性および/または誘発性が顕在化する。
【0011】
これらの必要性や以下の開示から明確になる他の必要性のうちの1つもしくは複数に対処するため、推定上のMAR DNA配列を同定するためのマウスゲノムの大規模なバイオインフォーマティクス分析を含むいくつかのアプローチが用いられた。マウスゲノムは、MAR予測ソフトウェアSMAR Scan Iを用いて分析された。新規に同定された齧歯類配列における、培養細胞からの目的の医薬品の組換えタンパク質の生成の改善を媒介する能力について評価された。このため、トランス遺伝子の形質移入アッセイにおいて新規に同定されたMARの転写活性が評価された。
【0012】
さらに、MAR、例えばヒト1_68MARおよびマウスMAR S4について試験した。モジュール、特にMARの特定の構造的/配列特異的モジュールを含むモジュールが同定され、かつこれらのモジュールは有利な特性を有するMARは、例えば配列のリシャッフリング(reshuffling)、欠失および/または複製による設計用に用いられた。モジュールはまた、他の因子、例えば特定の結合部位、特に転写因子結合部位(TFBS)を含む合成ヌクレオチド配列と結合された。
【課題を解決するための手段】
【0013】
本発明は、一実施形態では、少なくとも1つの遺伝子を高レベルに発現させる発現系であって、
目的の遺伝子をコードするヌクレオチド配列に作動可能に連結するためのプロモーターと、
前記発現系で形質転換された細胞内で前記遺伝子の発現を促進するための少なくとも1つの非ヒト哺乳類のMARヌクレオチド配列と、
を含み、ここで前記非ヒト哺乳類のMARヌクレオチド配列は、前記コンストラクトによる前記細胞の形質転換時に約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくはそれ以上、前記遺伝子の発現を増大させる、発現系を対象とする。
【0014】
前記非ヒト哺乳類のMARヌクレオチド配列は、
(i)配列番号3、配列番号10もしくはその機能的断片、または
(ii)(i)の配列のいずれかと約80%、約90%、約95%もしくは約98%の配列同一性を有するヌクレオチド配列
を含むか本質的にそれからなるかまたはそれからなり得る。
【0015】
本発明は、単離、精製された核酸分子であって、
(a)配列番号3もしくは配列番号10またはその機能的断片のヌクレオチド配列、または
(b)(a)の配列と少なくとも約80%、約90%、約95%もしくは約98%の配列同一性を有しかつMAR活性を有するヌクレオチド配列
を含むか本質的にそれからなるかまたはそれからなる、核酸分子も対象とする。
【0016】
本発明は、非ヒト哺乳類のMAR配列を同定するための方法であって、
−少なくとも1つの非ヒト哺乳類の核酸分子、好ましくは非ヒト哺乳類のゲノムもしくはその一部を提供するステップと、
−前記核酸分子に、
−評価されるべき核酸分子に対してウインドウサイズを設定するステップと、
−少なくとも1つもしくは少なくとも2つ、好ましくは3つ、より好ましくは4つもしくはそれより多いMAR関連の特徴を選択するステップと、
−この特徴/これらの特徴を示す配列に対して閾値を設定するステップと、
−これらの閾値を超えるMAR候補ヌクレオチド配列を選択するステップと、
−前記非ヒト哺乳類のMARヌクレオチド配列が、前記非ヒト哺乳類のMARヌクレオチド配列を含む発現系を介するヒトおよび/または非ヒト哺乳類細胞の形質転換時に、遺伝子の発現を約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくはそれ以上、増大させることを確認するステップと、
を含む、MAR配列のための走査手順を施すステップと、
を含む、方法をさらに対象とする。
【0017】
本明細書における特徴は、その値にウインドウ値を乗じることにより、約320〜1320、例えば約420〜約1220、約520〜約1120、約620〜約1020、約720〜約920の乗算値が得られるDNAベンディングの角度である場合があり、本明細書における特徴は、ウインドウ値を乗じることにより約900〜約4000、例えば約1200〜3700、約1500〜約3400、約1800〜約3100、約2100〜約2800の乗算値が得られる主溝の深さ値である場合があり、および/または本明細書における特徴は、ウインドウサイズ値を乗じることにより約500〜約2500、例えば約750〜約2250、約1000〜約2000、約1250〜1750の乗算値が得られる副溝の深さ値である場合がある。
【0018】
本発明は、
(a)(i)同定されたMARの末端領域の少なくとも一部を含む単離ヌクレオチド配列、および
(ii)前記同定されたMARまたは別の同定されたMARの約10%、約15%、約20%、約25%、約30%もしくはそれより多くを含むさらなる単離ヌクレオチド配列、
または
(b)(i)(a)(i)のヌクレオチド配列と約90%、約95%、約96%、約97%、約98%、約99%の配列同一性を有するヌクレオチド配列、および
(ii)(b)(i)のヌクレオチド配列と約70%、約80%、好ましくは約90%、約95%、約96%、約97%、約98%、約99%の配列同一性を有するヌクレオチド配列
を含むMARコンストラクトも対象とする。
【0019】
本発明に記載の他のMARコンストラクトは、同定されたMAR配列もしくはその連続して並ぶ一部の領域を含み、ここでは順序および/または方向が同定されたMAR配列の場合と異なる。
【0020】
さらに、本発明に記載の他のMARコンストラクトは、
(a)(i)同定されたMAR配列の少なくとも1つの単離または合成されたATリッチ領域、または
(ii)(a)(i)のATリッチ領域と少なくとも少なくとも80%、85%、90%、95%、98%もしくは99%の配列同一性を有する少なくとも1つのATリッチ領域
を含むコアヌクレオチド配列と、
(b)(a)の前記ヌクレオチド配列に隣接した少なくとも1つのDNAタンパク質結合部位を含むヌクレオチド配列と、
を含み、ここで前記結合部位は、
(i)さらなる同定されたMAR配列のDNAタンパク質結合部位、
(ii)同定されたMAR配列内で(a)のコアヌクレオチド配列外部に位置する、(a)の同定されたMAR配列のDNAタンパク質結合部位、または
(iii)(a)のコア内に存在するが少なくとも1つのさらなるDNAタンパク質結合部位に隣接する第1のDNAタンパク質結合部位であって、第1のDNAタンパク質結合部位と前記さらなるDNAタンパク質結合部位の少なくとも1つとは(a)のコア内で隣接することがない、第1のDNAタンパク質結合部位、または、
(iv)非MAR配列のDNAタンパク質結合部位
である。
【0021】
本発明は、特定のMARコンストラクトのいずれかを含む発現系、特定の発現系のいずれかを含むキット、ならびに、MARコンストラクト、発現系、細胞、トランスジェニック非ヒト動物、キット、および/または、(1)ヒト病原体タンパク質またはヒト細胞表面タンパク質を認識する抗体などのタンパク質およびエリスロポエチン、インターフェロンまたは他の治療用もしくは診断用タンパク質などのタンパク質の生成、および/または(2)インビトロ、インビボ遺伝子療法、細胞療法または組織再生療法における本明細書中で参照される方法のいずれかの使用も対象とする。
【図面の簡単な説明】
【0022】
【図1】様々なMARの組換え緑色蛍光タンパク質(GFP)の生成に対する効果を示す。
【図2】組換え緑色蛍光タンパク質(GFP)のCHO細胞内での様々なヒトおよびマウスMAR要素の極めて高い生成体の百分率(%M3)に対する効果を示す。
【図3】様々なヒト1_68およびマウスS4 MAR要素の組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。
【図4】マウスMAR要素の組換えモノクローナル抗体の生成に対する効果を示す。
【図5】安定なポリクローナル集団がMARを有しない(MARなしの)場合またはシスに付加されたMAR S4を有する場合にIgG重鎖および軽鎖の発現を駆動するベクターを形質移入されたCHO細胞の集団から生成可能であったことを示す。
【図6】図6Aに、安定な各クローンが(B)におけるMARを有しない(MARなしの)場合またはシスに付加されたMAR S4およびMAR1_68を有する場合にIgG重鎖および軽鎖の発現を駆動するベクターを形質移入されたCHO細胞の集団から限界希釈により生成可能であったことを示す。図6Bに、安定な各クローンが(B)におけるMARを有しない(MARなしの)場合またはシスに付加されたMAR S4およびMAR1_68を有する場合にIgG重鎖および軽鎖の発現を駆動するベクターを形質移入されたCHO細胞の集団から限界希釈により生成可能であったことを示す。
【図7】図7Aに、MARを有しない場合の時間に伴う(2週および26週)遺伝子(GFP)の発現を示す。図7Bに、MARを有する場合の時間に伴う(2週および26週)遺伝子(GFP)の発現を示す。
【図8】図8Aに、はヒト1_68MARの曲げの特徴を示す。図8Bに、ヒト1_68MARの配列の特徴を示す。
【図9】図9Aに、同定された領域のシャッフリングにより得られた異なるMARコンストラクトおよび得られた転写の増大を示す。図9Bに、MARコンストラクト6の曲げパターンを示す。図9Cに、MARコンストラクト6の結合部位などの構造パラメータの詳細を提供する。
【図10】全集団の平均蛍光(Avg Gmean M0)の分析により示された、様々なMAR S4コンストラクトの組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。
【図11】全集団の平均蛍光(Avg Gmean M0)の分析により示された、組換え緑色蛍光タンパク質(GFP)の発現時に得られた様々なMAR S4コンストラクトを示す。
【図12】MATInspectorソフトウェアにより予測された、ヒト1_68MARの有望な転写因子結合部位のマップを示す。
【図13】ATリッチコア(MAR1429〜2880)とプロモーターおよび緑色蛍光タンパク質(GFP)の上流に配置された転写因子における化学合成されたDNA結合部位との構築物から作成された合成MARの活性について試験するのに用いられるプラスミドのマップである。
【図14】図13に記載のように作成された合成MARによる転写促進を図示したものである。
【図15】表5に詳述されるDNA結合部位を含む合成MARによる転写促進を図示したものである。
【発明を実施するための形態】
【0023】
本発明は、非ヒト動物由来の単離、精製されたMAR配列、それら配列を同定する方法およびヒト細胞内および齧歯類細胞などの非ヒト細胞内でのタンパク質の高収率の生成を目的としてそれら配列を用いる系に関する。
【0024】
本発明は、MARコンストラクト、特に促進されたMARコンストラクト、これらのMARコンストラクトを用いる発現系およびキット、ならびにタンパク質の生成、特に大規模生成や治療におけるそれらの使用も対象とする。
【0025】
さらに本発明は、ヒト細胞および非ヒト哺乳類細胞におけるMARコンストラクトを介するタンパク質の高収率の生成を目的とする方法を対象とする。
【0026】
他に特に規定されない限り、本明細書で用いられるあらゆる科学技術用語は、本発明に関係する当業者により一般に理解されている意味と同じ意味を有する。本明細書中に記載の方法および材料とは異なるものが本発明の実施において用いられうるが、典型的で(examplaratory)適切な方法および材料が下記に記載されている。
【0027】
本発明に記載の発現カセットは、少なくとも1つの遺伝子およびこの遺伝子の転写に必要とされる要素を含む核酸である。
【0028】
本発明に記載のプロモーターは、遺伝子の上流に位置する場合に遺伝子の転写を促進するDNAの制限領域である。
【0029】
細胞内での発現、例えば非ヒト哺乳類細胞内での発現は、本発明との関連ではインビトロおよびインビボでの発現を示す。インビトロ発現は、例えばHeLa細胞系またはCHO細胞系などの細胞系内およびインビトロ遺伝子療法において用いられる細胞内での発現を含む。インビボ発現は、トランスジェニックの非ヒト動物における発現や、遺伝子療法でのヒトレシピエントへの細胞の再導入後のインビボ遺伝子療法またはインビトロ遺伝子療法で用いられるヒト細胞内での発現を含む。
【0030】
本発明によると、哺乳類細胞、例えば非ヒト哺乳類細胞は、細胞培養条件下で維持可能である。細胞のこのタイプの非限定例がチャイニーズハムスター卵巣(CHO)細胞である。
【0031】
本発明に記載のMARコンストラクト、MAR要素、MAR配列、S/MARまたは単にMARは、天然の「SAR」または「MAR」と1つもしくは複数の(例えば2つ、3つもしくは4つの)特性を共有しかつ前記MARの作用により任意の遺伝子のタンパク質発現を促進する少なくとも1つの特性を有するヌクレオチド配列である。MARコンストラクトは、MAR活性、特に転写調節、好ましくは促進の活性だけでなく、例えば発現安定性の活性および/または「促進されたMARコンストラクト」としても記載の他の活性を伴う単離および/または精製された核酸としての特徴も有しうる。MARコンストラクトはその主要な基礎である同定されたMARに基づいて定義される場合があり、それ故、MAR S4コンストラクトはそのヌクレオチドの大半(50%以上)がMAR S4に基づくMARコンストラクトである。一般に認められたモデルによると、天然のSARまたはMARは、特定のDNA配列の核マトリックスに対する足場を媒介し、ヘテロクロマチンコアから外部に延在するクロマチンループドメインを生成する。SARまたはMARが任意の明確なコンセンサス配列もしくは認識可能な配列を有することはない一方、それらの最も一貫性のある特徴は全体的にAおよびT含量が高くかつC塩基が一方の鎖で支配的であることのように見える。MARは一般に、場合により鎖の分離を生じさせやすいベント二次構造を形成する傾向を有する。AおよびT含量の高いいくつかの簡単な配列モチーフがSARおよび/またはMAR内に見出されることが多いが、大部分ではそれらの機能的重要性および潜在的な作用様式は未解決のままである。これらは、脊椎動物またはショウジョウバエ(Drosophila)においては、Aボックス、Tボックス、DNA巻き戻しモチーフ(DNA unwinding motif)、SATB1結合部位(Hボックス、A/T/C25)およびコンセンサストポイソメラーゼII部位を含む。
【0032】
本発明に記載のMAR候補またはMAR候補配列は、天然のSARまたはMARと1つもしくは複数、例えば2つ、3つもしくは4つの特性を共有する配列である。
【0033】
本発明に記載の同定されたMARまたは同定されたMAR配列は、単離ヌクレオチド配列であり、かつその天然対応物のタンパク質/遺伝子の発現の十分な促進を可能にするあらゆる領域(「モジュール」または「要素」)を含む点で天然MAR配列に対応する。
【0034】
同定されたMARのモジュール(本明細書中で「領域」、「DNA領域」、「一部」、「ドメイン」とも称される)のすべては、天然MARの能力に対してタンパク質/遺伝子の発現の促進を可能にするのに必要とされる。一般に、単独でMARの十分な活性を発揮することが可能なモジュールは全く存在しない。これらの領域の一部、例えば下記のAT−ジヌクレオチドリッチのベント領域および転写因子結合部位(TFBS)領域は配列特異的である。他の「領域」については、それらの位置、例えば同定されたMAR配列の5’および3’末端領域により特徴づけられる。
【0035】
AT/TA−ジヌクレオチドリッチのベントDNA領域(以後「ATリッチ領域」と称される)は、特にジヌクレオチドATおよびTAの形態で大量のAおよびTを含むベントDNA領域である。好ましい実施形態では、それは隣接する100塩基対のストレッチ上のジヌクレオチドTAの少なくとも10%および/またはジヌクレオチドATの少なくとも12%、好ましくは隣接する100塩基対のストレッチ上(またはATリッチ領域の長さがより短い場合にはより短い各ストレッチ上)のジヌクレオチドTAの少なくとも33%および/またはジヌクレオチドATの少なくとも33%を有する一方、ベント二次構造を有する。しかし、「ATリッチ領域」は、約30ヌクレオチド長かもしくはそれに満たない程度に短い場合があるが、好ましくは約50ヌクレオチド長、約75ヌクレオチド長、約100ヌクレオチド長、約150、約200、約250、約300、約350もしくは約400ヌクレオチド長であるかまたはそれより長いものである。
【0036】
下記に考察されるように、ATリッチ領域は、例えばその比較的高い曲げ角度により、隣接領域、例えば結合部位領域から区別可能である。脊椎動物またはショウジョウバエ(Drosophila)におけるSATB1結合部位(Hボックス、A/T/C25)およびコンセンサスのトポイソメラーゼII部位などの一部の結合部位は比較的高いAおよびTの含量を有することも多い。しかし、一群の結合部位を含む結合部位領域(モジュール)、特にTFBS領域は、領域の曲げパターンの比較により、AおよびT含量の高い結合部位由来のATおよびTAジヌクレオチドリッチ領域(「ATリッチ領域」)から容易に識別可能である。例えばヒトMAR1_68においては、ATリッチ領域が平均で約3.8もしくは約4.0を超える曲度を有しうる一方、TFBS領域は平均で約3.5もしくは約3.3未満の曲度を有しうると思われる。同定されたMARの領域は、代替手段、例えば限定はされないが、本明細書中の他の箇所に記載の相対融解温度によっても確認可能である。しかし、かかる値は、種特異的であることから種間で異なる場合があり、例えばより低い場合がある。したがって、各々のATおよびTAジヌクレオチドリッチ領域は、約3.2〜約3.4もしくは約3.4〜約3.6もしくは約3.6〜約3.8など、より低い曲度を有する場合があり、かつTFBS領域は約2.7未満、約2.9未満、約3.1未満、約3.3未満など、比例的により低い曲度を有する場合がある。SMAR Scan IIでは、各々、より小さいウインドウサイズが当業者により選択されることになる。
【0037】
本発明に記載の同定されたMAR/MAR配列の末端領域は、同定されたMARの少なくとも約5%、約6%、約7%、約8%、約9%もしくは約10%を含む。
【0038】
結合部位またはDNAタンパク質結合部位は、DNA結合タンパク質に結合可能な任意のヌクレオチド配列である。DNA結合タンパク質に対する結合部位は典型的にはTFBSである。TFBSは転写因子に結合可能な任意の配列である。TFBSの供給源は、例えば限定はされないが、ヒトまたはマウスなど任意でありうる。TFBSは設計または合成も可能である。しかし特定の実施形態では、TFBSは、MAR配列内、例えば同一生物、同一種または同一属のMAR配列内に相当物を有する。しかし、TFBSは異なる種または異なる属のMAR配列に由来しうる。MAR配列内に現在既知の相当物を全く有しないTFBSについても本発明の範囲内である。かかるTFBSは、限定はされないが、USF1(上流刺激因子1)またはジンクフィンガータンパク質CTCFに対する結合部位を含みうる。TFBSは、1つ、2つ、3つ、4つ、5つもしくはそれより多くの置換、付加および/または欠失により修飾され、かつ完全または部分的に合成されうる。最適化されたTFBSは、各DNA結合タンパク質に対して最適化された結合親和性を有するTFBSであり、既知の天然対応物を全く有しないことが多く、本発明の範囲内でもある。それらの最適化されたTFBSであれば、天然のTFBSの上記の修飾によるかまたは合成、特に化学合成により生成可能である。本発明の特定の実施形態では、結合部位またはTFBSは、例えば組織特異的な天然か、設計または合成された制御タンパク質または他の天然か、設計または合成されたタンパク質による結合により、MARに組織特異性を与え、それは例えば特定の薬剤および分子に応答しうる。遺伝子および/または細胞療法は、組織特異性およびMARにおける特定の薬剤に特異的に応答する、つまり薬剤により誘発可能な能力から利益を得る典型的な場合である。前者の場合、例えば目的の遺伝子であれば特定の器官内または組織内に限って発現されることになり、後者の場合での発現であれば、例えば特定の薬剤への応答に限って刺激可能である。TFBSが含められうる転写因子の他の非限定例として、例えば、SATB1、NMP4、MEF2、S8、DLX1、FREAC7、BRN2、GATA1/3、TATA、Bright、MSX、AP1、C/EBP、CREBP1、FOX、Freac7、HFH1、HNF3α、Nkx25、POU3F2、Pit1、TTF1、XFD1、AR、C/EBPγ、Cdc5、FOXD3、HFH3、HNF3β、MRF2、Oct1、POU6F1、SRF、V$MTATA_B、XFD2、Bach2、CDP CR3、Cdx2、FOXJ2、HFL、HP1、Myc、PBX、Pax3、TEF、VBP、XFD3、Brn2、COMP1、Evil、FOXP3、GATA4、HFN1、Lhx3、NKX3A、POU1F1、Pax6および/またはTFIIAが挙げられる。
【0039】
結合部位、例えばTFBSは、コアヌクレオチド配列および結合部位の分離距離が約200以下、好ましくは約100ヌクレオチド以下、さらにより好ましくは約50ヌクレオチド以下、さらにより好ましくは約25以下、約15以下、約5ヌクレオチド以下であるかまたは0ヌクレオチドである場合、コアヌクレオチド配列に隣接するといわれる。好ましい実施形態では、結合部位、特にTFBSは、それ自体でTFBSの両側に最大で25ヌクレオチドの短いリンカーまたはアダプターを含む。さらにより好ましい実施形態では、TFBSは、最大で約50ヌクレオチド、最大で約40ヌクレオチドまたは最大で約30ヌクレオチドのオリゴマーの一部である。本発明に記載の一連の結合部位、例えばTFBSは、互いに隣接して配列内に並んだ一連のTFBSである。一連のTFBSは、コアに近いこの一連のTFBSが上で特定された距離を有する場合、コアヌクレオチド配列に隣接するといわれる。結合部位は、結合部位がコアヌクレオチド配列の一部の結合部位でありかつ天然のMAR内の同一位置に相当物を有する場合、「ATリッチ領域」に隣接するといわれる。
【0040】
結合部位は、1つ、2つ、3つ、4つ、5つもしくはそれより多くの置換、付加および/または欠失により修飾されうる。好ましくは、これらの置換、付加および/または欠失は、結合部位が各結合部位のコンセンサス配列に一致するように導入される。
【0041】
種々の促進されたMARコンストラクトは本発明の一部であり、かつ本発明に記載のMARコンストラクトの基礎となりうる天然でありかつ/または同定されたMAR、特にコア核酸配列の基礎となる天然のMARに対して全体的に促進をもたらす特性を有する。かかる特性は、限定はされないが、完全長で天然でありかつ/または同定されたMARに対する長さの短縮、遺伝子発現/転写の促進、発現安定性の促進、組織特異性、誘発性またはそれらの組み合わせを含む。したがって、促進されたMARコンストラクトは、例えば、同定されたMAR配列のヌクレオチド数の約90%未満、好ましくは約80%未満、さらにより好ましくは約70%未満、約60%未満、または約50%未満を含む。MARコンストラクトは、適切な細胞の前記コンストラクトによる形質転換時に遺伝子の発現および/または遺伝子の転写を促進しうる。本発明と関連し、「発現を促進する」か、「遺伝子発現促進活性」を有するか、「タンパク質発現を促進する」かまたはそれに類するMARコンストラクト/MAR(ヌクレオチド)配列についての参照がなされる場合、この「促進」は、例えばかかる配列の不在下である以外は等しい条件下で発現される遺伝子の発現と比較されるものである。促進は、例えば約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくは約15倍、約20倍もしくは約25倍またはそれより高い可能性がある。
【0042】
MARコンストラクトはまた、極めて高産生の細胞の平均百分率を約5倍、約10倍、約15倍もしくはそれ以上に高めうる。したがって、遺伝子の発現を平均的に高める以外に、極めて高発現の細胞の百分率の増加、ならびに安定な(「耐性のある」)コロニーの発生の増加(約100%、約200%、約300%もしくは約400%もしくはそれより高い増加)、および/または発現の変動性の低下(約30%、約40%、約50%もしくはそれ以上のcv(変動係数)の低下)が本発明の範囲内である。
【0043】
MARコンストラクトまたはそれに類するものは「発現の安定性を促進する」場合がある。この「促進」は、例えばMARコンストラクト/MAR配列の不在下である以外は等しい条件下で発現される遺伝子の発現と比較されるものである。安定性の促進では、例えば最大で約5週、10週、20週、25週、30週、35週、40週、45週、もしくは50週後に100%の促進が維持可能である。MARコンストラクトは、例えば筋肉、肝臓、中枢神経系もしくは他の組織に対して特異的である場合があり、および/あるいは、例えば抗生物質、ホルモンおよび/または代謝中間体などの物質の投与時に誘発されうる。
【0044】
MARコンストラクト/MAR配列は、好ましくは目的の遺伝子が作動可能に連結されるかまたは連結可能であるプロモーター領域の上流に挿入されうる。しかし特定の実施形態では、MARコンストラクトが目的の遺伝子/ヌクレオチド酸配列の上流および下流もしくは下流だけに位置することが有利である。シスおよび/またはトランスの双方の他の複数のMAR配列についても本発明の範囲内である。
【0045】
MARコンストラクトまたはMARの領域は、天然の「SAR」または「MAR」もしくはその各領域と1つもしくは複数の(例えば2つ、3つもしくは4つの)特性を共有しかつ前記MARの作用により任意の遺伝子のタンパク質発現を促進する少なくとも1つの特性を有する場合、例えば同定されたMARまたは同定されたMARの領域に基づくといわれる。これらのMARコンストラクトまたはMARの領域は、一般に、本明細書中で提供される用語の定義によると、それらの基礎となる同定されたMARと「実質的同一性」を有する。これらおよび/またはそれらのヌクレオチド配列の修飾にもかかわらず、それらは基礎となる同定されたMARの少なくとも1つの機能性/特性を維持することになる。
【0046】
本発明は、促進されたMARコンストラクトを含むMARコンストラクトの使用も対象とする。これらの使用では、MARコンストラクトは、1つもしくは複数の非MAR後生的遺伝子制御ツール、例えば限定はされないが、ヒストン脱アセチル化酵素(HDAC)などのヒストン修飾因子、遺伝子座制御領域(LCR)などの他のDNA要素、cHS4もしくは抗リプレッサー(antirepressor)要素(例えば安定剤)および抗リプレッサー要素(STARもしくはUCOE因子)などのインシュレーター、またはホットスポット(クワックス T.H.J.(Kwaks T.H.J.)およびオッテ A.P.(Otte A.P.))とも併用可能である。
【0047】
合成は、MAR/MARコンストラクトとの関連で用いられる場合、同定されたMARまたはそれに基づくMARの配列/領域または部分領域における単純なリシャッフリング、複製および/または欠失以上のものを含む設計であるMARを示す。特に、合成MAR/MARコンストラクトは、一般に同定されたMARの1つもしくは複数、好ましくは1つの領域を含むが、特定の実施形態では、場合によっては合成または修飾されるとともに特別に設計され、十分に特徴づけられた要素、例えば単一もしくは一連のTFBSであると思われ、それらは好ましい実施形態では合成的に生成される。これらのデザイナー要素は、多数の実施形態では比較的短く、特にそれらは一般に約300bps長以下、好ましくは約100、約50、約40、約30、約20もしくは約10bps長以下である。特定の実施形態では、これらの要素は多量体化されうる。
【0048】
本発明に記載の非ヒト哺乳類のMARは、少なくとも部分的には非ヒト哺乳類生物のゲノムまたはゲノムの一部を介して確認されるMAR/MAR配列である。これは例えば、限定はされないがマウスゲノムなどの齧歯類ゲノムの分析を介して同定されるMAR/MAR配列を含む。
【0049】
本発明に記載のベクターは、連結されている別の核酸分子を輸送可能な核酸分子である。例えば、プラスミドは1種のベクターであり、レトロウイルスまたはレンチウイルスは別種のベクターである。
【0050】
本発明に記載の形質移入は、例えば限定はされないが、エレクトロポレーション、リポフェクションによるか、ウイルスベクターまたは化学的手段を介する、核酸のレシピエント真核細胞への導入である。
【0051】
本明細書中で用いられる形質転換は、核酸の付加による真核細胞の修飾を示す。例えば、細胞の形質転換は、例えばDNAベクターのエレクトロポレーションを介する導入による細胞への核酸の形質移入を含みうると思われる。しかし、本発明の多数の実施形態では、本発明の促進されたMARを細胞に導入する方法は任意の特定の方法に限定されることはない。
【0052】
転写はDNA鋳型からのRNAの合成を意味する。
【0053】
シスは、同一の核酸分子、例えば限定はされないが同一のベクターまたは染色体上での2種以上の要素(例えばクロマチン因子)の配置を示す。
【0054】
トランスは、2種以上の核酸分子、例えば限定はされないが2種以上のベクターまたは染色体上での2種以上の因子(例えばクロマチン因子)の配置を示す。
【0055】
配列は、シス/トランス位置からその活性を発揮する場合、例えば遺伝子に対してシスおよび/またはトランスで作用するといわれる。
【0056】
本発明に記載のウインドウは、例えばSMAR Scanの方法を実施する間、MARについて評価される塩基対の数を表す。この数は通常、約50bps、約100bps、約200bps、約300bpsである。しかし、400bps、500bps、600bpsもしくはそれより大きいbpsのウインドウも本発明の範囲内である。
【0057】
ヌクレオチド配列またはその断片は、他のヌクレオチド配列(またはその相補鎖)と(適切なヌクレオチドの挿入または欠失により)最適に整列される場合、ヌクレオチド塩基の少なくとも約60%、通常は少なくとも約70%、より通常は少なくとも約80%、好ましくは少なくとも約90%、およびより好ましくは少なくとも約95〜98%のヌクレオチド配列の同一性がある場合に、もう一方と実質的同一性を有する。
【0058】
同一性は、2つのヌクレオチド配列間の配列の、かかる配列、例えば十分かつ完全な配列の2つの鎖間の一致を同一性により判定するときの、関連性の程度を意味する。同一性は容易に計算可能である。2つのヌクレオチド配列間の同一性を測定するには多数の方法が存在する一方で「同一性」という用語は当業者に周知である(「Computational Molecular Biology」、レスク A.M.(Lesk A.M.)編、オクスフォード大学出版会(Oxford University Press)、ニューヨーク(New York)、1988年;「Biocomputing:Informatics and Genome Projects」、スミス D.W.(Smith D.W.)ら、アカデミック・プレス(Academic Press)、ニューヨーク(New York)、1993年;「Computer Analysis of Sequence Data,Part I」、グリフィン A.M.(Griffin A.M.)およびグリフィン H.G.(Griffin H.G.)編、ヒューマナ・プレス(Humana Press)、ニュージャージー(New Jersey)、1994年;「Sequence Analysis in Molecular Biology」、フォン・ハインジェ G.(von Heinje G.)、アカデミック・プレス(Academic Press)、1987年;および「Sequence Analysis Primer」、グリブスコフ M.(Gribskov M.)およびデベルクス J.(Devereux J.)編、エム・ストックトン・プレス(M Stockton Press)、ニューヨーク(New York)、1991年)。2つの配列間の同一性を測定するのに一般に用いられる方法は、限定はされないが、「Guide to Huge Computers」、マーティン J.ビショップ(Martin J.Bishop)編、アカデミック・プレス(Academic Press)、サンディエゴ(San Diego)、1994年およびカリリョ H.(Carillo H.)およびリップマン D.(Lipman D.)、SIAM J Applied Math.48:1073頁(1988年)に開示された方法を含む。同一性を測定するための好ましい方法は、試験対象の2つの配列間の最大の一致が得られるように設計される。かかる方法は、コンピュータプログラム内にコード化される。2つの配列間の同一性を測定するのに好ましいコンピュータプログラムの方法は、限定はされないが、GCG(ジェネティックス・コンピュータ・グループ(Genetics Computer Group)、ウィスコンシン州マディスン(Madison))プログラムパッケージ(デベルクス J.(Devereux J.)ら、Nucleic Acids Research 12(1).387頁(1984年))、BLASTP、BLASTN、FASTA(アルツシュール(Altschul)ら(1990年);アルツシュール(Altschul)ら(1997年))を含む。同一性の測定のため、周知のSmith Watermanアルゴリズムも用いられうる。
【0059】
例として、参照ヌクレオチド配列と例えば少なくとも95%の「同一性」を有するヌクレオチド配列を含む核酸は、核酸のヌクレオチド配列が参照配列と、各参照ヌクレオチド配列の100個のヌクレオチド当たり最大で5つの点突然変異を含みうる点を除いて同一であることを意味する。言い換えると、参照ヌクレオチド配列に対して少なくとも95%同一のヌクレオチド配列を有するヌクレオチドを得るため、参照配列内のヌクレオチドの最大で5%が欠失されるかまたは別のヌクレオチドと置換されうるか、あるいは参照配列内の全ヌクレオチドの最大で5%の数のヌクレオチドが参照配列内に挿入されうる。参照配列のこれらの突然変異は、参照ヌクレオチド配列の5’もしくは3’末端位置またはそれら末端位置間のいずれかの場所で生じ、参照配列内のヌクレオチド内に別々にかまたは参照配列内の1つもしくは複数の隣接基内のいずれかで散在されうる。
【0060】
ヌクレオチド配列の機能的断片もまた本発明の一部である。断片は天然対応物の配列における特にそれらの作用を受ける遺伝子の発現を増大させるという所望の機能を維持する限り機能的であると考えられる。MARまたはMAR領域の断片は、その欠失がMAR/領域の転写促進活性を低下させてもそれを失わせることがない場合にはやはり機能的断片であると考えられる。「完全に機能的な断片」は、断片が他のMAR配列なしに用いられる場合に活性における任意の低下が、たとえ観察されたとしても、統計的に検証不可能である断片である。また、例えば天然MAR、同定されたMAR、MAR領域またはこれらのうちの任意の断片と本明細書で提供される定義に準じる実質的同一性を有する機能的断片は本発明の範囲内に含まれる。
【0061】
本明細書中で詳細に記載のように、特定の実施形態では、モジュールもしくはその一部がリシャッフルされ、複製され、かつ/または欠失が施される。当業者が理解するであろうように、かかる領域のシャッフリングおよび/または複製により、例えば新たな制限部位が生成され、次いでそのように生成されたコンストラクトの新たな制限パターンの生成が可能であり、かつ配列の長さが調節されることになりうる。それらの調節は、限定はされないが、1個、2個、3個、4個、5個、6個、7個、8個、9個、10個、10〜15個、15〜20個、20〜25個、25〜30個、30〜35個、35〜40個のヌクレオチドに作用しうる。これらの調節および他の修飾は本発明の範囲内である。各要素(または領域/モジュール)および/またはそれらの断片の各々と本明細書で提供される定義に準じる実質的同一性を有する、再配列されたMAR、特にリシャッフルされかつ/または複製されたMARの配列は本発明の範囲内である。
【0062】
MAR配列は、植物から哺乳類細胞またはその逆方向に移される可能性があり、異種の宿主細胞内の核マトリックス付着活性を保持することになる[ブレイン P.(Breyne P.)、ヴァン・モンタグ M.(Van Montagu M.)、デピッカー A.(Depicker A.)およびゲイセン G.(Gheysen G.)、ミエルケ C.(Mielke C.)、コーウイ Y.(Kohwi Y.)、コーウイ−シゲマツ T.(Kohwi−Shigematsu T.)およびボーデ J.(Bode J.)]。MARのこの保持がより高等なすべての真核生物において機能すると仮定すると、ある属由来のMAR配列であれば同配列が別の属などに由来する属内で同様に機能することを想定することになる。
【0063】
とはいえ、齧歯類由来のMAR配列であればある意味で組換えタンパク質の生成にとって有利でありうるとの判断から、全マウスゲノムがスクリーニングされ、下記のようなDNA配列の構造的特徴(例えばDNAベンド)を検出するコンピュータプログラム、SMAR Scan Iを用いてMAR候補配列が同定された。
【0064】
下記に考察のように、驚くべきことに、非ヒト、特に齧歯類(ここではマウス)のMAR配列が、例えばCHO細胞内やHeLa細胞などのヒト細胞内において発現促進に関してより強力であることが見出された。さらに一層驚くべきことに、特定の非ヒトMAR配列が、ヒトMAR配列よりも非ヒト細胞、例えばCHO細胞ならびにヒト細胞、例えばHeLa細胞の双方において実質的に優れた働きをすることが見出された。
【0065】
マウス由来の同定された新規のS/MAR DNA配列のいくつかがトランス遺伝子の発現を増大させることが示され、それ故、ヒトMAR配列用に設計されかつそれを用いて試験されたプログラムであるSMAR Scan Iが多数のゲノムの供給源、例えばヒトに加えてマウスに由来するS/MAR要素を同定するための効率的なツールであるという証拠が提供された。しかし重要なことには、より強力なMAR要素がヒトゲノムのスクリーニングよりも齧歯類(例えばマウス)ゲノムのスクリーニングにより同定可能であることが見出された。特に、本発明は、マウスゲノム由来の活性の高いS/MAR要素を用い、種々の細胞内、特にマウスおよびヒト細胞内で組換えタンパク質、例えば医薬品用途を有する組換えタンパク質の生成が増大可能であることを確立している。マウスS/MAR S4は、新規に単離されたマウスMARおよび予めクローニングされたヒトMARにおいて最も強力であることが示された。したがって、本発明は、促進されたンパク質生成を有する非ヒトMARおよび/またはタンパク質発現の安定性を長期にわたり高めるMARを対象とする。
【0066】
SMAR Scan Iは、これらの配列の構造的かつ物理化学的な特徴に基づきMAR候補配列を同定するソフトウェアツールである。本方法についての徹底した考察が他に提供されている(マーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書)。「SMAR Scan」は、本質的には、ジヌクレオチド重み行列に基づいて特性を認識し、DNAの高次構造的かつ生理化学的な特性における理論値を計算するアルゴリズムを含むバイオインフォーマティクスツールを示す。好ましくは、SMAR Scanは可変サイズの走査ウインドウを用い、多種多様な組み合わせでのDNAベンディング、主溝の深さおよび副溝の幅のポテンシャル、融解温度に対応するDNA配列の特徴を評価する。各特徴においては、カットオフまたは閾値が設定される必要がある。プログラムは、所与領域の計算されたスコアが設定されたカットオフ/閾値を超えるごとにヒットを返す。
【0067】
ヒットを扱うため、2つのデータ出力モードが使用可能であり、第1のモード(「プロファイル様(profile−like)」)は、単純に、クエリー配列上のあらゆるヒット位置および選択された異なる基準に対するそれらの対応する値を返す。第2のモードは(「隣接ヒット」と称される)は、いくつかの隣接ヒットの位置およびそれらの対応する配列のみを返す。このモードにおいては、隣接ヒットの最小数は、さらに調節可能なウインドウサイズで設定可能な別のカットオフ/閾値である。例えば4つの理論上の構造的基準におけるデフォルトのカットオフ/閾値を調節するため、例えばSMARt DBから実験的に検証されたMARを用いてもよい。このように、例えばデータベースからのすべてのヒトMAR配列が、4つの基準を有しかつ設定されたカットオフ/閾値を全く有しない「プロファイル様」モードを用いるSMAR Scanで検索され、分析された。これは配列の全位置における各機能の設定を可能にした。次いで、各基準における分布はこれらのデータにより計算された(図1および3を参照、マーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書)。
【0068】
SMAR Scan技術の利用がMAR配列の同定のために好ましいものである一方、当業者は、同程度かまたはやや低めでもある選択性を有するS/MARモチーフの同定を可能にする他のバイオインフォーマティクスツールが本発明との関連で使用可能であることを理解するであろう。好ましくは、設定された閾値またはカットオフ値である特定の値を超えるこれらの特徴を示すMARに関連した特徴のみが正のヒットを生成するかまたは生成するように設定可能であるようにかかるツールを設定可能である。しかし、MARの同定に用いられる多数のバイオインフォーマティクスツールが、マトリックス−結合活性を同定するように設計された。この活性は遺伝子発現を増大させる能力と必ずしも相関するものではない[フィー−バン L.(Phi−Van L.)およびストラトリング W.H.(Stratling W.H.)]。
【0069】
SMAR Scan IはヒトMARの同定を目的に開発されている。したがって、それは既知のヒトMARから収集された構造データを用いて開発された。ヒトに「調節された」SMAR Scan Iプログラムが本発明との関連で用いられ、マウスゲノムのMAR配列についての評価がなされた。しかし、マウスおよびヒトゲノムの塩基組成における差異から、ヒトゲノムを走査するための予め規定された設定を有するSMAR Scanプログラムの使用が抑えられた(マーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書)。したがって、プログラムによる管理可能に回収された候補マウスMAR配列の同定が可能になるまで、明確なウインドウサイズおよび構造パラメータの閾値が試行錯誤により規定される必要があった。それらのいくつかは、試験される場合、例えば各タンパク質をコードする遺伝子を有するベクター上に配置されかつ齧歯類細胞系に導入される場合、結局はタンパク質生成の実質的増大を可能にするMAR配列である「スーパー(super)MAR配列」となった。
【0070】
マウスMAR S4およびマウスMAR S46は、本発明の範囲内である齧歯類MAR配列の例である。単離されたこれらのMAR配列は添付の配列表で配列番号3および配列番号10として示される。しかし、当業者が理解するように、塩基対の挿入、欠失、置換、特に塩基対の挿入、欠失または置換を自ら有しうるこれらや他の非ヒトMARの断片は、野生型配列の特にそれらの作用により遺伝子の発現を増大させるという所望の機能を維持する限り本発明の範囲内である。例えば、MAR配列の転写/遺伝子発現促進活性を低下させてもそれを失わせることがない挿入が、所望の機能、ここではMARの遺伝子発現促進と実質的に干渉することがないと考えられる。同様に、例えば同定されたMARの断片が、同定されたMARに対してやや低下した転写促進活性を有しても転写促進活性を完全に失わせることがない場合、やはり機能的断片であると考えられる。「完全に機能的な断片」は、活性における任意の低下が、観察されたとしても統計的に検証不可能である場合の断片である。本明細書中の他の箇所に詳述のように、天然MARもしくはその断片のヌクレオチド配列と「実質的同一性」を有する配列も本発明の範囲内に含まれる。
【0071】
MARのモジュール性
同定されたMARの分析により、MARがモジュール(または領域)、特に配列特異的モジュールを含むか否かが判定され、それを同定されたMARの設計または合成領域を含むMARを含む合成MARの生成に利用できた。事実、同定されたMARのいくつかの配列特異的モジュールを確認できた。驚くべきことに、特定のモジュールもしくはその一部のシャッフリングおよび/または完全もしくは部分的な複製やさらには欠失により、上記のような促進されたMARがもたらされることが見出された。
【0072】
マウス由来のヒト1_68MARおよびS4 MARは、領域のシャッフリング、欠失および/または複製によるMARコンストラクトを生成するためのモデルとして役立つことになる。しかし、当業者が容易に理解するように、本発明は、任意の同定されたMARの操作およびそれから得られるMARコンストラクトを対象とする。異なる由来のMARを含む異なるMARに対応するのに必要でありうる適切な調節は、当業者の技能の範囲内で十分に可能である。例として、限定はされないが、真核生物、好ましくは哺乳類、特にマウスなどのモデル動物、およびウシ、ブタ、ヒツジなどの経済的重要性のある種ならびにヒトが挙げられる。
【0073】
ヒトMARのモジュール性
ヒト1_68MARは、領域のシャッフリングおよび/または複製によりMARコンストラクトを生成するためのモデルとして役立った。下記のように確認されたモジュールもしくはその一部の使用により、MARコンストラクトが同定されたMAR、例えばヒト1_68MARに基づいて生成された。MARコンストラクトは、特に領域(モジュール)もしくはその一部のシャッフリングおよび/または複製により生成された。
【0074】
1_68MARの例は、同定されたMARの(本明細書中で領域または要素とも称される)モジュールが遺伝子発現の促進を天然のMARの能力に対して可能にするのにすべて必要であったことを示す。MARの完全な活性を単独で発揮できた同定されたモジュールは存在しなかった。驚くべきことに、特定のモジュールのシャッフリングおよび完全もしくは部分的な複製の結果、遺伝子発現のさらなる促進が得られることが見出された。
【0075】
いくつかの非冗長な配列特異的モジュール(領域)が同定された。これらのモジュールは、局所クロマチン構造に作用するように協働する。MARのこの機構は、後生動物の転写の制御にやや類似するものであり、すなわち開始部位から最大で数キロベースにわたり分散されたモジュールの多様な集合が全体として、転写が開始することになる場所を決定づける。
【0076】
同定された配列特異的なモジュールは、特に(1)AおよびT含量の高い領域、例えば対称なATリッチ領域(AおよびTが交互にある)、特に「ATリッチ領域」、および(2)結合部位が豊富な領域、特に、限定はされないがATリッチ領域に分離されたTFBSであった。
【0077】
AおよびT含量の高いベントDNAが一般にプロモーター領域、MARおよびレプリケーターにおいて見出されることが報告されている[アラジェ(Aladjem)およびファニング(Fanning)、2004年]。従来、AおよびT含量の高い配列(上記の「対称」配列および一方の鎖には主にAおよび他方の鎖には主にTを有する配列である「非対称」配列)は、主に二本鎖の開鎖を促進すると考えられた。しかし、これらの領域は幅広い範囲の機能を有しうると思われる。例えば、ラミンB2レプリケーター内のAおよびT含量の高い配列が複製開始点認識複合体(ORC)に結合し[アブドゥラシドバ(Abdurashidova)、ダナイロフ(Danailov)ら、2003年;ステファノヴィッチ(Stefanovic)、スタノイチッチ(Stanojcic)ら、2003年]、インビトロでMcm4/6/7ヘリカーゼの負荷および二本鎖DNAの巻き戻しを促進しうる[ヨウ(You)、イシミ(Ishimi)ら、2003年]。AおよびT含量の高い本質的なベントDNAにおける構造的役割についても検討されている。分裂酵母ORC4の「AT−フックDNA−結合モチーフ」は、移動度の高い集団であるタンパク質HMG−I/Yのそれに類似するものであり、かかる構造的役割を有しうる[ストリック(Strick)およびラエムリ(Laemmli)、1995年;ベル(Bell)、2002年]。V(D)Jの組換えを促進するHMG−I/Y媒介性のDNAベンディングに類似したタンパク質媒介性のベンディング、および真核生物内のエンハンサーおよびプロモーターでの転写複合体の構築および安定化も生じうると思われる[レビン(Levine)およびチアン(Tjian)、2003年]。高いAおよびT含量を有する領域のすべてがベントDNAに対応するわけではない。しかし、それらDNAはベントであり、そうであればヒストンを引き付けてベントDNA全体にヌクレオソームを形成するための「ヒストンマグネット(histone magnet)」として作用可能であり、複製/転写前タンパク質に対するランディングパッド(landing pad)として自由に作用する隣接領域が残る。
【0078】
上記のように、MARは特に「結合部位が豊富な領域」または単に「結合部位領域」(上記(2)を参照)内に他のタンパク質に対する結合部位も有しうる。それらの他のタンパク質は、限定はされないが、1_68MAR内に見出されるようなDNA巻き戻し要素−結合タンパク質(DUE−B)およびHoxタンパク質、SATBI、CEBPなどの転写因子を含みうる。突然変異分析によると、これらの結合部位がMAR機能に寄与することが示される。
【0079】
ヒト1_68MARは、その方向を反転させかつベントDNAを離すことで改善され、プロモーター領域の上流の転写因子結合部位領域のサイズの拡大が可能であった。図9でわかるように、多数のこれらの再配列されたMAR(例えばコンストラクト6)は、MARを有しないコンストラクトに対し(10倍)、また天然のMARを含むコンストラクトに対しても(コンストラクト1および16;約2倍)転写をかなり増大させる。示されるデータは、末端の転写制御因子自体が下流のクロマチンにおける転写開始を制限することも明確に示す。天然MARにおける前方の斜線ボックスとして示される、領域の3’末端に位置する223bpの断片が、コンストラクト11と比べ、コンストラクト7内のこの領域におけるあらゆる活性を保持する。これは、この重要な部分がこの場合にベント領域およびコンストラクト6内のこの因子の残り(ヌクレオチド1〜1425)の5’末端と協働することを示唆している。2つのHMG−I/Y部位がこの末端の近隣に位置することが見出された。コンストラクト2では、2つの同定されたMAR配列が結合し合うことにより発現も増大されることが示される。
【0080】
マウスMARのモジュール性およびサイズの低下
いくつかのMARがS4 MARに基づき構築され(表3)、かつ特徴づけられた(図10)。図10で示されうるように、1600bps長を超える断片における内部の欠失により、MARの活性の低下はあまり生じなかった(S4−1−703_2328−5457)。しかし、プロモーターに隣接する795−bp断片の欠失またはこの配列の同様の長さのルシフェラーゼ遺伝子の断片による置換(S4_1−4661;S4_1−4661−Luc5489)により、この活性の完全な低下が誘発された。
【0081】
非配列特異的モジュール:3’末端のMAR配列の活性
ヒト1_68MARを用いる実験(図9)では、ヒト1−68MARの3’HoxFおよびSATBI結合部位領域の重要性が既に示された。この領域の重要性は、図10に示されるマウスMAR S4を用いる実験によりさらに明示された。図11に示されるように、MAR S4の3’末端配列の活性のさらなる分析のため、MARのこの部分をその一部を取り出すかまたは複製することによってさらに分析した。図11は、様々なMAR S4誘導体の遺伝子発現に対する効果についても示す。興味深いことに、切断された3’末端を有する1つのかかる誘導体(元のMAR S4における4658〜5054対4658〜5457)では、平均で、トランス遺伝子の発現がより長い元のMAR S4配列よりもやや高い(104%対100%)ことが示された。これは、MAR要素のより強力でかつより短い誘導体が得られうることを示している。
【0082】
したがって、本発明は長さが天然対応物よりもかなり短い高活性のMARコンストラクトを含むことから、それらは例えばベクターの設計および転移にとってより都合のよいサイズということになる。
【0083】
特に、同定されたMAR配列のヌクレオチドの数の約90%未満、好ましくは約80%未満、さらにより好ましくは約70%未満、約60%未満もしくは約50%未満を含むMARコンストラクトが本発明の範囲内である。それらのコンストラクトは、好ましくは同定されたMARの3’末端領域、さらにより好ましくは同定されたMAR/MAR配列の3’末端領域の少なくとも約5%、約6%、約7%、約8%、約9%もしくは約10%を含む。しかし、同定されたMARの5’末端領域を有するMARコンストラクトも本発明の範囲内である。
【0084】
合成MAR
ヒト1_68MARの再配列では、特定の実施形態では、単離されたMARの前方の斜線部分の3’末端に位置するHoxリッチ領域の223bpの断片が完全長領域の活性を保持することが示された。これは、本発明の特定の実施形態ではこの部分が他の因子との協働において重要でありうることを示唆している。図12は、MATInspectorソフトウェアによる予測としてのMAR1_68の有望な転写因子結合部位の配列を示す。C/EBP、NMP4、FAST1、SATB1、およびHoxF結合部位の位置が例示されており、それはそれらが5’側(前方の斜線)のフランキング配列内に豊富に存在することを図示している。
【0085】
ヒトMAR1_68内のATリッチのベントDNA領域と転写因子結合部位の間で協働が生じうるという発見により、1つもしくは数箇所の転写因子結合部位に隣接するMAR1−68のATリッチ領域を含むMAR/MARコンストラクトの構築が促進された。図13は、ATリッチ領域およびATリッチ領域の各末端での同定されたMARのTFBSを含むコア(MAR1429〜2880)と緑色蛍光タンパク質(GFP)におけるプロモーターの上流に配置された転写因子のための化学合成されたDNA結合部位との構築物から作成された合成MARの活性についての試験用に用いられるプラスミドのマップを示す。図13は、特に、転写因子結合部位がATリッチドメインとGFPトランス遺伝子の発現を駆動するSV40プロモーターとの間に挿入され、図9で見出された状況が再現されたことを示し、ここでの最も好ましい設定では結合部位を有するMAR部分がプロモーターとベントDNA領域との間に挿入される(コンストラクト6)。表4は、用いられた化学合成されたオリゴヌクレオチドのDNA配列を示す。
【0086】
C/EBP、NMP4、FAST1、SATB1、およびHoxF(Gshとも称される)転写因子に対する結合部位がMAR1−68配列から同定された(図12)。MAR1−68内に存在するこれらの結合部位は変化なしに用いられるか(FAST1、C/EBP、HOXF/Gsh)、またはコンセンサス(すなわち完全な)配列(HoxF、SatB1、NMP4)と比べて1つもしくは2つのミスマッチを有する場合に訂正された。
【0087】
図14からわかるように、ここでの合成結合部位の付加により、ほぼすべての場合、いくつの特定の場合、ATリッチ領域を含むコアMAR配列に対して有意な転写促進がもたらされた。活性ではC/EBPおよびHoxまたはGsh2が最高でSatB1およびFast1が次に高い一方、単一のNMP4部位が検出可能な効果を全く有しなかった。
【0088】
図14は、ATリッチ領域の基礎となる同定されたMARの結合部位に隣接された、ここでのMAR1_68に基づくMAR1429〜2880であるコア配列の挿入により、発現においてあまり改善が得られなかったが、1つもしくは複数の結合部位をさらに含むMARコンストラクが、特にプロモーターの上流以外のATリッチコアの下流に挿入される場合、プロモーターの制御下での遺伝子によるタンパク質の発現/生成においてかなりの促進が得られたという驚異的な結果を示す(ここではM3細胞の%により同定)。
【0089】
好ましい実施形態では、追加の結合部位がプロモーターの上流以外のATリッチコアの下流に存在する一方、例えば限定はされないが、ATリッチ領域の上流、ATリッチ領域内、コアのATリッチ領域の隣接領域または遺伝子の下流における位置といった他の構成も本発明の範囲内である。
【0090】
好ましい実施形態では、合成もしくは単離のいずれかのタンパク質結合部位の特定の組み合わせ、例えば2つの異なるタンパク質結合部位、3つの異なるタンパク質結合部位の組み合わせ、4、5、6、7、8、9、10もしくはそれより多くのタンパク質結合部位の組み合わせが検討される。これらの組み合わせは完全または部分的に多量体化されうる。好ましい実施形態では、こうした組み合わせはHox/GshおよびSATB1を含む。これらの組み合わせまたは多量体化された組み合わせの例えばコアと適切なプロモーターとの間への挿入により、高発現体(high expressor)クローンの発生が、MARコンストラクト/MAR配列を含まないベクターがそれ以外は等しい条件下で用いられる場合での高発現クローンの発生と比較し、約2倍もしくはそれ以上、例えば限定はされないが、約3倍、4倍、5倍、6倍、7倍、8倍、9倍もしくはそれ以上、好ましくは約10倍もしくはそれ以上、さらにより好ましくは、約11倍、12倍、13倍、14倍、15倍、16倍、17倍、18倍、19倍もしくはそれ以上または約20もしくはさらに約25倍もしくは約30倍もしくはそれ以上に高まる可能性がある。
【0091】
要するに、MARコンストラクトはビルディングブロックから構築可能である。これらのビルディングブロックは、同定されたMARもしくはその一部の領域、例えば配列特異的領域、合成ビルディングブロック(その機能性を最適化するための修飾を含む)、例えば一連の化学合成された転写因子結合部位(TFBS)、非MAR配列に由来するかもしくはそれに基づくビルディングブロック、または異なる種もしくは属のMAR配列に由来するかもしくはそれに基づくビルディングブロックを含むかまたはそれに基づくものでありうる。好ましい実施形態では、かかるMARは、表5に示される、TFBS領域に共役されたATリッチ領域または特異的な転写因子DNA結合部位の組み合わせを含む。当業者は、これらの原則が特定の配列または本明細書中に開示される結合部位に限定されず、かつ他の誘導体、相同体または配列の組み合わせも本発明の範囲内であることを理解するであろう。
【0092】
上記のように、本発明のMARコンストラクト、発現系および/またはキットはタンパク質の生成において用いられうる。ここでMARコンストラクトは、プロモーターの制御下での目的のタンパク質、例えばインスリンに対する遺伝子を含むベクター内に含められうる。ベクターは細胞に導入され、細胞は成長される。次いで、このプロセスはインスリンの大規模なバッチ生産のために大規模化される。例えばMARコンストラクトを伴わない場合よりも3〜5倍多いインスリンの大量生産が3週にわたり維持されうる。
【0093】
上記のように、本発明のMARコンストラクト、発現系および/またはキットは、インビトロおよび/またはインビボ遺伝子療法や細胞および組織の置換療法において用いられうる。例えば、インビトロ遺伝子療法では、MARコンストラクトが、プロモーターの制御下での、インビトロ遺伝子療法を必要とする患者において欠損している遺伝子を含むベクター内に含められうる。次いで、MARコンストラクトは、患者の細胞、例えば骨髄細胞に導入される。MARコンストラクトでの形質転換後、骨髄細胞は患者に導入され、目的の遺伝子の発現がMARコンストラクトを伴わない場合よりも5倍高いレベルで先行しうる。それ故、有効量のタンパク質の発現が可能である。インビボ遺伝子療法では、MARコンストラクトを含むベクターが、治療を必要とする患者の細胞に例えば注射で直接導入されうる。
【0094】
同様に、本発明の発現系は、組織再生のための移植または例えば神経変性疾患に対する神経細胞療法において幹細胞に導入されうる。本発明のこの実施形態にて用いられうる幹細胞の非限定例として、任意の年齢の個体の骨髄組織または新生児の臍帯血から得られる造血幹細胞(HSC)および間葉幹細胞(MSC)が挙げられる。幹細胞は、本発明に記載の発現系を用いて形質移入され、奏功した形質転換体が細胞療法もしくは組織再生療法を必要とする患者に移植または再導入されうる。形質転換された幹細胞、例えばNucleofection(登録商標)(Cell Line Solution V(VCA−1003)、ドイツのアマクサ(amaxa GmbH))を入手するため、いくつかの方法が利用可能である。
【0095】
ヒト抗原に結合する抗体を含む多種多様なタンパク質を生成可能なトランスジェニック動物は、公知の方法(例えば限定はされないが、ロンバーグ(Lonberg)らに交付された米国特許第5,770,428号明細書、米国特許第5,569,825号明細書、米国特許第5,545,806号明細書、米国特許第5,625,126号明細書、米国特許第5,625,825号明細書、米国特許第5,633,425号明細書、米国特許第5,661,016号明細書および米国特許第5,789,650号明細書)により生成可能である。発現系およびMARコンストラクトは、例えばトランスジェニックのウシ、ヒツジ、ヤギまたはブタを介した、典型的にはタンパク質の体液(例えば乳)への分泌によるタンパク質の生成において使用可能である。例えば、メーデ(Meade)らに交付された米国特許第5,750,172号明細書を参照のこと。またトランスジェニック動物の生成については、ルボン(Lubon)らに交付された米国特許第6,518,482号明細書を参照のこと。
【実施例】
【0096】
本発明は以下の実施例でさらに説明されることになるが、これは、特許請求の範囲、発明の概要または本明細書中の他の箇所で示される本発明の範囲を限定するものではない。材料、方法、および実施例はあくまで例示目的であり、限定するように意図されていない。当業者は、本明細書で提供される指針に伴い、変更、追加および改善を行うことができ、そのすべては本発明の範囲内である。
【0097】
マウスゲノムのS/MAR予測:SMAR Scan I
NCBI m34マウスの構築物に対応するすべてのマウス染色体配列を集め、SMAR Scan Iで分析した。ストリンジェンシーの低いものと高いもののスクリーニングを3.6°のDNAベンディングの基準および300bpの最小ウインドウサイズにおける閾値または4.2°および100bpの最小ウインドウサイズにおける閾値のいずれかを各々用いて行った。
【0098】
全マウスゲノムのSMAR Scan Iによる低いストリンジェンシーの分析により、全体で622,410bp(全マウスゲノムの0.024%)を表す全部で1496の推定上のS/MAR(候補MAR)が得られた。表1は、各染色体におけるそのサイズ、その遺伝子数、その予測されるMAR(候補MAR)の数、遺伝子当たりのそのMAR密度およびkb単位でのS/MAR間の平均距離を示す。この表は、(MAR当たり遺伝子の密度の平均の約50%を表す標準偏差では)異なる染色体に対する予測されるS/MAR(候補MAR)当たりの遺伝子密度が様々であることを示す。MAR当たりの遺伝子における高い方の密度と低い方の密度との間の倍数の差異は、予測されるMAR(候補MAR)内にそのサイズおよびその遺伝子数に対して極めて豊富に存在する染色体Yを考慮しない場合に6であり、これはこれらのMARの分布における強力かつ想定外のバイアスを示す。表1は、S/MAR間の平均距離(S/MAR当たりのkb)が可変である(標準偏差はS/MAR当たりのkbの平均の38%を表しかつS/MAR当たりのkbの高い方の密度と低い方の密度との間の倍数の差異は8.3である)ことも示す。染色体10、11、XおよびYは、これらの密度の高い標準偏差に有意に寄与する。
【0099】
SMAR Scan Iが元来ヒト配列に対して調整されていることから、最もストリンジェントなパラメータを用いる場合、マウスゲノム配列ではMARはほとんど得られず、それ故、高いストリンジェンシーのスクリーニング(DNAベンディングの基準においては4.2°の閾値)においては、デフォルトのカットオフ値をMARと考えられるような隣接ヒット(contiguous hits)の最小サイズに300bpではなく100bpのウインドウを用いて調節した。マウスゲノムのSMAR Scan Iによる分析によると、DNAベンディングの基準として4.2°を超える値を有する49の「スーパー」MARが予測された。
【0100】
【表1】

【0101】
組換えタンパク質の生成を増大させるための新規に同定されたマウスMARの使用
5つのMAR要素を、SMAR Scanを用いる完全なマウスゲノムの高いストリンジェンシーのスクリーニングで得られた推定上のMAR(候補MAR)から選択した。それらを、オークランド小児病院研究所(Children’s Hospital Oakland Research Institute)(CHORI、http://bacpac.chori.org/)から購入したマウスゲノムDNAの細菌の人工染色体由来のプラスミドベクター内でクローニングした。
【0102】
これらの新規に同定されたマウスMARをS4、S8、S15、S32およびS46と名づけた(SMAR Scan Iによる同定の順序による、「スーパー」MARのS1〜S49)。ヒトMARの1_3、1_6、1_9、1_42、1_68、3_S5およびX_S29は予め同定されており、MARの1_68およびX_S29が最も強力なヒト因子である(マーモド(Mermod)ら、「High efficiency gene transfer and expression in mammalian cells by a multiple transfection procedure of MAR sequences」、国際公開第2005/040377号パンフレット、またマーモド(Mermod)らに交付された米国特許出願公開第20070178469号明細書も参照)。上記のように、これらのMARを緑色蛍光タンパク質の発現を駆動するSV40プロモーターおよびエンハンサーの上流のpGEGFP対照ベクターに挿入し、これらのプラスミドを培養されたCHO細胞に形質移入した[ジロド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)]。次いで、安定的に形質移入された細胞の全集団内でのトランス遺伝子の発現を蛍光セルソーター(FACS)機器を用いて分析した。図1は、様々なS/MARの組換え緑色蛍光タンパク質(GFP)の生成に対する効果を示す。蛍光活性化セルソーター(FACS(登録商標))により示される、MARを含むかもしくは含まないGFP発現ベクターpGEGFPを形質移入したCHO細胞の集団、および典型的な特性が示される。最も強力なヒトMARの1_68およびX_S29のみがこの図に示される。特性では、GFP蛍光レベルの関数としての細胞数の計数値が示される。2より小さい蛍光値(M1)、または102より大きい相対発光量(M2)もしくは103より大きい相対発光量(M3)を有する細胞小集団M1、M2およびM3を表す水平バーが示されている。
【0103】
図1からわかるように、新規に同定されたすべてのマウスMARで、トランス遺伝子の発現がMARを伴わずにGFP単独で駆動される発現を有意に上回る程に増大し、ここでの「スーパー」マウスMAR S4は示されるすべてのMARの中で最も強力であった。
【0104】
【表2】

【0105】
最も強力なヒトMARの1_68およびX_S29の転写活性を新規に同定されたマウスMARで得られた場合と比較した。5つのマウスMARを最初にGFP発現アッセイを介して試験し、それらがすべてGFPの発現を異なるレベルに増大させることが見出された。マウスMARのS15およびS32は、比較では転写活性が最低のMARであり(GFP単独よりも約2倍の増大)、S8およびS46は中間の活性を示し(3〜4倍の増大)、かつMAR S4は極めて高い転写活性を示した(7倍の増大)。さらに、マウスMAR S4は、この試験における試験対象のあらゆるMARの中で最も強力なものである。ヒトMAR1−68およびマウスMAR S4の間の転写活性の比較によると、全集団(Gmean M0)および高いGFP生成細胞(M2)の平均蛍光が50%増大したことが示される一方、マウスMAR S4の場合、極めて高いGFP生成細胞(M3)の百分率は175%高めであった。マウスMAR S4の場合、GFP蛍光(CV M0)の観点での全集団の同質性は常に1〜2%低く、それは細胞産生性(cell productivity)の安定性の高まりを示すことから有利である。
【0106】
このクローニングの第1ラウンド後、活性が高いMAR要素がマウスゲノムから常に得られうるか否かを判定しようとした。したがって、2つのさらなるマウスMAR(S6およびS10)をクローニングし、特徴づけた。これらの新しいマウスMARをpGEGFP対照ベクターに挿入し、上記のようにFACSで分析した。マウスMAR S10はFACSで分析されたあらゆる異なるパラメータにおいて最高のヒトMARよりも強力であるように見られ、MAR S4とほぼ同程度に転写活性が高く発現全体を増大させるものである。
【0107】
極めて高い生成体について評価するため、M3細胞の百分率をヒトMAR1_68において得られた百分率に正規化した。結果を図2に示す。図2は、様々なヒトおよびマウスのS/MAR要素の、組換え緑色蛍光タンパク質(GFP)の極めて高い生成体の百分率(%M3)に対する効果を示す。指定のMAR要素を有するかまたは有しないGFP発現ベクターを形質移入したCHO細胞の集団を蛍光活性化セルソーター(FACS(登録商標))で分析した。極めて高い生成体の百分率をこの基準において最高のヒトMARで得られた百分率に正規化し、MAR1_68の値を100に設定した。マウスMARのS10およびS4により、各々、ヒトMAR1_68よりも平均で80%および180%といった極めて高い生成体細胞が得られた。全体として、7つのマウスMARと7つのヒトMARとのこの比較から、齧歯類MARを用いたCHO細胞からより高い発現が得られると判断した。
【0108】
異なる細胞種内での新規に同定されたマウスMARにおける効力の評価
S4 MARの効力をCHO細胞内で評価した。さらに、ヒトMAR1−68、マウスMAR S4もしくはMARなしのいずれかを含むEGFP発現ベクターをヒトHeLa細胞内に安定的に形質移入し、EGFP蛍光を分析した。図3は、様々なヒト1_68およびマウスS4 MAR要素の、組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。HeLa細胞の集団に形質移入し、表2に記載のように分析した。HeLa細胞内でのS4および1−68MARの効力の比較においては、S4はいくつかの観点で1_68より優れることが見出され、すなわちS4は高い平均GFP蛍光(平均Gmean M0)とともに中程度の発現および高い発現の範囲内(各々、M1およびM2)でより多くの細胞をもたらし、また発現の変動の低下(平均CV M0)をもたらした。HeLa細胞を用いる極めて高い発現の範囲内(M3)では全く細胞が見出されなかった。
【0109】
マウスMARを用いるモノクローナル抗体の発現促進
マウスMAR、特に最も強力なマウスを用い、医薬品用途のタンパク質の生成が増大するか否かを判定するため、それらをRhesus−Dを認識する免疫グロブリンの重鎖および軽鎖をコードするpMZ37およびpMZ59ベクターに挿入した[ミーシャー S.(Miescher S.)、ザーン−ザバル M.(Zahn−Zabal M.)、デ・ジーザス M.(De Jesus M.)、マウドリ R.(Moudry R.)、フィッシュ I.(Fisch I.)、ヴォーゲル M.(Vogel M.)、コブル M.(Kobr M.)、イムボーデン M.A.(Imboden M.A.)、クラグテン E.(Kragten E.)、ビヒラー J.(Bichler J.)、マーモド N.(Mermod N.)、スタドラー B.C.(Stadler B.C.)、アムシュトゥッツ H.(Amstutz H.)、ブルム F.(Wurm F.)]。上記のように、これらのプラスミドをCHO細胞内に形質移入し、選択および免疫グロブリンアッセイを行った[ジロッド P.A.(Girod P.A.)、ザーン−ザバル M.(Zahn−Zabal M.)およびマーモド N.(Mermod N.)]。図4は、S/MAR要素の組換えモノクローナル抗体の生成に対する効果を示す。ここで、CHO細胞に、MARを有しない(MARなし)またはシスで付加されたMAR S4を有するIgG重鎖および軽鎖の発現を駆動する上記のベクターを形質移入した。24、48および72時間後、上清中のIgGの力価を測定した。さらに図5に示されるように、安定なクローンを、MARを有しない(MARなし)またはシスで付加されたMAR S4を有するIgG重鎖および軽鎖の発現を駆動する上記のベクターを形質移入したCHO細胞の集団から生成した。選択後、分泌されたIgGの力価を培地内で測定し、特異的な産生性を細胞計数によりアッセイした。図6(A)は、安定な個々のクローンの、MARを有しない(MARなし)またはシスで付加されたMAR S4を有するIgG重鎖および軽鎖の発現を駆動するベクターを形質移入したCHO細胞の集団からの限界希釈による生成後に得られた結果を示す。選択後、分泌されたIgGの力価を培地内で測定し、特異的な産生性を細胞計数によりアッセイした。また、MAR1_68の場合に得られた結果とMARを含まないクローンの場合に得られた(B)の結果との比較が含まれる。図3〜6で得られ示された結果は、新規に同定されたマウスMAR、特にMAR S4を用い、一時的なトランスフェクタント(図4)内および安定的なトランスフェクタント(図5および6)内で医薬品タンパク質、例えばモノクローナル抗体の生成を増大可能であることを示す。MAR S4を用いる場合、5pg/細胞/日(pcd)程度もしくはそれを超える特定の産生性を有する安定なクローンを数種の候補クローンの分析から容易に同定可能である(図6(A))。実に21の最高のクローンの平均産生性が、MAR S4を用いる場合または用いない場合で各々、7.28±0.78pcd(図6(A))および2.61±1.09pcdであった。これらの結果は、既知のニワトリのリゾチームMARの場合に得られた力価レベル(1.5mg/L未満)またはMARを有しない場合に得られた力価レベル(0.5mg/L未満)に対して際立つものである。特にこれらの結果は、新規に同定されたマウスMARを用いることで限定はされないがモノクローナル抗体などの医薬品用途のタンパク質の生成が増大し、マウスMAR、例えばMAR S4が特に組換えタンパク質の生成において興味深いものになりうることを示す。
【0110】
ヒトMAR1_68の場合の発現の安定性
MAR1_68の使用から、MARを有しないクローンにより生成された遺伝子の発現が一般に無変化であり、MARを有する同じクローンが高レベルの発現を長期間維持するだけでなく無変化の細胞が発現を回復することを実証した。
【0111】
図7は、ジロッド(Girod)ら、2005年に記載のように、MAR1−68を含むpEGFP発現プラスミドのG418抗生物質耐性遺伝子を有するCHO細胞への同時形質移入を示し、安定的に発現された細胞をG418の存在下で3週間かけて選択した。細胞クローンを限界希釈により取得し、9つの個別のクローンにおけるGFP蛍光について分析した。さらなる分析のため、GFPを発現する典型的なクローンを2つの集団の各々から選択し、抗生物質選択の存在下および不在下でさらに最長で26週間培養した。特性が左側に2週間の培養後のGFP蛍光レベル(x軸)およびy軸上に細胞計数を示す一方、右側の特性は26週間培養した細胞から得られたものである。図示のように、MARを含まないクローンでは抗生物質の不在下での26週後におけるGFP蛍光レベルが2週後のレベルに対して低下したことを示す一方、MARを含むクローンでは抗生物質選択の有無にかかわらず26週時のGFP蛍光レベルを維持できたことから、MARを含む発現系は目的の遺伝子の安定な発現にとって有用なものとなる。
【0112】
MARのモジュール性および遺伝子の発現促進との関連
MARの構造分析により、各々が遺伝子の発現促進に寄与するDNA配列領域/モジュールが示された。図8は、1_68MARの構造分析を介して得られた結果を示す。図8(A)は、中央のATリッチ領域がMAR1_68の遺伝子座内でのベントDNAを決定づけることを示す。図8(B)は、このATリッチ領域がMatInspector(カータリウス(Cartharius)、フレック(Frech)ら、2005年)により同定された転写因子結合部位に豊富に存在する領域で囲まれていることを示す。正確には729の有望なTFBSをMAR配列に沿ってMatInspectorにより検出した。図8(B)の下部は、同定された領域に対するコーディングの特性を示す。
【0113】
図9(A)は、1_68MARと、左側に1_68MARの領域もしくは部分を組み込み、かつその領域もしくは部分の順序および/もしくは方向を変化させ、かつ/またはそのかかる領域もしくは部分を複製する異なるMARを示す。右側ではMAR1_68を有するかまたはMARを全く有しない場合に得られた転写の増大の程度とともにコンストラクト1〜16により得られた転写の増大の程度を示す。示されるすべてのMAR配列を、eGFP遺伝子マーカーを駆動するプロモーターの上流に挿入した。矢印は、図8に示される野生型MAR配列と比べての、その領域もしくは断片の方向を示す。ATリッチ領域を囲む配列を、矢印を付けた後方の斜線ボックス(左側)および矢印を付けた前方の斜線ボックス(縮尺不定;右側)として示す。ベント領域を斜交平行線ボックスとして示す。
【0114】
図9(B)は図9.Aにおけるコンストラクト6に対応するMARの曲げパターンを示す。これらの曲げパターンをSMARScanIにより測定した。
【0115】
図9(C)は、MatInspector[カータリウス(Cartharius)、フレック(Frech)ら、2005年]分析の結果を示す。有望な転写因子結合部位(TFBS)をMatInspector[カータリウス(Cartharius)、フレック(Frech)ら、2005年]により同定した。731の有望な部位をMAR配列に沿ってMatInspectorにより検出する。図9(C)最下部のコンストラクト6を図8(B)および図9(A)に対応するコーディングを用いて示す。この図の最下部のコーディングは、図9(A)に示され考察されたものに対応する。
【0116】
図9に示される実験は、単独で完全なMAR活性を示す領域が全く存在しないことを示す。例えば、DNA転写の促進を天然のヒト1_68MARから最大限得るのに、3つの異なる配列(図8)、すなわち複数の転写因子に対する結合部位を有する1189bpのセグメント(すなわちCEBP)(図9A上部、矢印を付けた後方の斜線ボックスとして示される)、本質的には763bpの対称性のある(AおよびTが交互にある)ATリッチ領域により決定づけられるベントDNA(図9A上部、斜交平行線ボックス)、および多数のHoxFおよびSATBI結合部位を含む追加の1648bpのセグメント(図9A上部、矢印を付けた前方の斜線ボックスとして示される)が必要である。
【0117】
図9は、ベントDNAを離してプロモーター領域の上流の転写因子結合部位領域のサイズを拡大することにより、ヒト1_68MARが改善されることを示す。この拡大を行うため、転写因子結合部位(TFBS)領域、ここではATリッチ(配列番号18)領域に隣接するHoxリッチ領域(配列番号19)(以後、矢印を付けた前方の斜線領域である)を、CEBPリッチ領域(配列番号17)(以後、後方の斜線領域でもある(図9))に隣接させた。図9Aの右側に示される、生成された異なるMARコンストラクトの転写促進活性の比較によると、矢印を付けた前方の斜線領域の方向が転写の増大にとって重要であったことが示される(コンストラクト5および6を比較)。示されるデータは、末端の転写制御因子自体が下流のクロマチンにおける転写開始を制限することも明示している。矢印を付けた前方の斜線領域の3’末端に位置する223bpの断片(配列番号20)がコンストラクト7内のこの領域の完全な活性を保持すると仮定すると、この場合、この重要な部分がコンストラクト6内のこの要素のベント領域および残りの5’末端(ヌクレオチド1〜1425)と協働しなければならないことが示唆される。2つのHMG−I/Y部位がこの末端の隣接位置に存在することが見出された。
【0118】
マウスMARのモジュール性およびサイズの縮小
ヒト1_68MARの場合の実験結果に基づき、S4 MARにおけるモジュール、特にその転写活性を担うモジュールについても分析した。またこの分析を、比較的長いS4 MARのサイズの縮小を目標に行った。したがって、いくつかのMARをS4 MARから作成し(表3)、特徴づけた(図10)。図10は、左側に特定のMAR S4コンストラクト、および右側に全集団の平均蛍光(Avg Gmean M0)の分析により示された様々なMAR S4コンストラクトの組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。指定のMARコンストラクトを含むかまたは含まないGFP発現ベクターを形質移入したCHO細胞の集団を、FACScaliburサイトメーター(ベクトン・ディッキンソン(Becton Dickinson))を用いるフローサイトメトリーにより分析した。全集団の平均蛍光をヒトMAR1_68で得られたもの(その値を100に設定)に対して正規化する一方、GFPはMARの不在下での発現を示す。他のMARコンストラクトを完全長の1547bpのS4 MARに対してのそれらの塩基含量に準じて名づけた(表3を参照)。ドット付きボックスは、MAR S4のATリッチのベント領域を示す。S_41−4662−Luc5489は、末端(3’)の795塩基対が除去されかつルシフェラーゼ遺伝子の一部(黒色ボックス)と置換される場合のコンストラクトを示す。興味深いことに、図10からわかるように、1624−bp EcoRI断片がS4 MAR(S4−1−703_2328〜5457)からそのMAR活性の有意な低下を伴うことなく欠失されうることが見出された。しかし、プロモーターに隣接する795−bpの断片の欠失またはこの配列と類似の長さを有するルシフェラーゼ遺伝子の断片(S4_1−4661;S4_1−4661−Luc5489)との置換により、この活性の完全な低下が誘発された。これは、マウスS4 MARの特定の変異体は、長さが短縮する一方で高い活性を示すことで、それが例えばベクターの設計および転移にとってより好都合なサイズになりうることを示す。
【0119】
【表3】

【0120】
3’末端のMAR配列における活性
MAR S4の3’末端配列の活性をさらに分析するため、MARのこの部分についての詳細な分析をその一部の除去または複製を介して行った。図11は、全集団の平均蛍光(Avg Gmean M0)の分析により示された、様々なMAR S4誘導体の組換え緑色蛍光タンパク質(GFP)の発現に対する効果を示す。CHO細胞の集団を生成させ、上記のようにアッセイした。興味深いことに、切断された3’末端を有する1つのかかる誘導体(元のMAR S4の4658〜5457に対して4658〜5054)では、より長い元のMAR S4配列と比べてトランス遺伝子の発現が平均でわずかに高いことが示された(100%に対して104%)。これは、より強力でかつより短いMAR要素の誘導体が得られうることを示す。
【0121】
合成MAR
図12は、MATInspectorソフトウェアの予測による[1_68MARの]有望な転写因子結合部位のマップを示す。C/EBP、NMP4、FAST1、SATB1、およびHoxF(Gshとも称される)結合部位の位置を例示し、それらが5’側の前方の斜線フランキング配列内に豊富に存在することが図示される。MAR1−68内に存在するこれらの結合部位を変化を加えずに用いるか(FAST1、C/EBP、HOXF/Gsh)またはそれらにコンセンサス(すなわち完全)配列に対して1つもしくは2つのミスマッチを有する場合には訂正した(HoxF、SatB1、NMP4)。
【0122】
ヒトMAR1_68内でのATリッチのベントDNA領域と転写因子結合部位の間で生じうる連携に関する実験結果から、1つもしくはいくつかの転写因子結合部位に隣接するMAR1−68のATリッチ部分を含む合成MARの作成が促進された。図13は、ATリッチ領域を含むコア(MAR1429〜2880)とプロモーターおよび緑色蛍光タンパク質(GFP)の上流に配置された転写因子における化学合成されたDNA結合部位との構築物から作成された合成MARの活性について試験するのに用いられるプラスミドのマップを示す。図13は、転写因子結合部位がATリッチのコアとGFPトランス遺伝子の発現を駆動するSV40プロモーターの間に挿入されたことを示し、図9に見出される状況を再現するものであり、この場合、最も好ましい設定では結合部位を有するMAR部分がプロモーターとベントDNA領域の間に挿入される。表4は、用いた化学合成されたオリゴヌクレオチドのDNA配列を示す。
【0123】
【表4】

【0124】
図14は、図13に記載のように構築された合成MARによる転写促進を示す。挿入された要素は、図示のようにコアに加えて1つもしくはいくつかのタンパク質DNA−結合部位を有する。ATリッチ領域を含むコア配列(ATリッチコア)に加えて1つもしくはいくつかの結合部位を有するプラスミドの形質移入では、結合部位を含めることでATリッチコア単独の場合と比べて転写促進が増大し、かつ活性ではC/EBPおよびHoxまたはGsh2が最高でSatB1およびFast1が次に高い一方、単一のNMP4部位が検出可能な効果を全く有しないことが示された。
【0125】
活性結合部位の異なる混合物についても試験し、相乗効果が観察可能であるか否かを判定した。その実施のため、異なる転写因子に対する結合部位を有するオリゴヌクレオチドの様々な組み合わせをDNAライゲーション反応において混合し、結合部位の正確な順序および配列をDNA配列決定により判定した。得られた組み合わせを表5に示す。
【0126】
【表5】

【0127】
得られたプラスミドを上記のように形質移入により試験した。図15は、表5に示されるDNA結合部位の組み合わせを用いて作成された合成MARによる転写促進を示す。最も活性の高い組み合わせが星印で示され、HoxF/Gsh2またはSatB1部位の存在が示される。図15に示される結果は、この例では合成MARの活性が挿入された結合部位の数に依存しなくても、結合部位の特定の組み合わせが高い促進活性を示す一方、それ以外では活性がないかまたはさらに遺伝子発現が抑制されることを示す。より高い活性を有するコンストラクトがこの場合にHox/Gsh2およびSATB1タンパク質の組み合わせを含み、かつ最も活性の高いコンストラクトはこれらの要素のみからなる。この合成MARの挿入により、発現体クローンの発生が任意のMAR配列を含まないpEGFP対照ベクターに対して約10倍増大した。
【先行技術文献】
【非特許文献】
【0128】
【非特許文献1】Abdurashidova G, DanailovB, et al., "Localization of proteins bound to a replication origin of humanDNA along the cell cycle." EMBO J 22: 4294-4303, 2003.
【非特許文献2】Aladjem, MI and Fanning E.,"The replicon revisited:an old model learns new tricks in metazoan chromosomes." EMBO Rep 5(7):686-91, 2004.
【非特許文献3】Allen GC, SpikerS, Thompson WF, Use of matrix attachment regions (MARs)to minimize transgene silencing, Plant Mol Biol.,43(23):361-376, 2000.
【非特許文献4】Amati B and Gasser SM, ChromosomalARS and CEN elements bind specifically to the yeast nuclear scaffold, Cell,54:967-978, 1988.
【非特許文献5】Amati B and Gasser SM, Drosophilia scaffold-attached regions bind nuclearscaffolds and can function as ARS elements in both budding and fission yeasts,Mol. Cell. Biol., 10:5442-5454, 1990.
【非特許文献6】Bell SP, "The originrecognition complex: from simple origins to complex functions." Genes Dev16: 659-672, 2002.
【非特許文献7】Bode J, SchlakeT, Rios-Ramirez M, Mielke C, StengartM, Kay V and KlehrWirth D, Scaffold/matrix-attachedregions: structural properties creating transcriptionallyactive loci, Structural and Functional Organization of the Nuclear Matrix:International Review of Cytology, 162A:389-453, 1995.
【非特許文献8】Bode J, BenhamC, Knopp A and Mielke C,Transcriptional augmentation: modulation of gene expression byscaffold/matrix-attached regions (S/MAR elements), CritRev Eukaryot Gene Expr,10(1):73-90, 2000.
【非特許文献9】Bode J, Stengert-IberM, Kay V, Schlake T and Dietz-PfeilstetterA, Scaffold/matrix-attached regions: topological switches with multipleregulatory functions, Crit. Rev. Euk.Gene Exp., 6:115-138, 1996.
【非特許文献10】Bodnar JW, A domain model foreukaryotic DNA organization: a molecular basis for cell differentiation andchromosome evolution, J. Ther. Biol., Vol.132:479-507, 1988.
【非特許文献11】Boulikas T, Nature of DNA sequences atthe attachment regions of genes to the nuclear matrix, J. Cell Biochem., 52:14-22, 1993.
【非特許文献12】Boulikas T, Chromatin domains andprediction of MAR sequences. In Structural and Functional Organization of theNuclear Matrix: International Review of Cytology, Academic Press, Orlando, 162A:279-388, 1995.
【非特許文献13】Breyene P, Van MontaguM and Gheyseu G, The role of scaffold attachmentregions in the structural and functional organization of plant chromatin, TransgenicRes., Transgenic Res., 3:195-202, 1994.
【非特許文献14】Breyne P, Van MontaguM, Depicker A and GheysenG, Characterization of a plant scaffold attachment region in a DNA fragmentthat normalizes transgene expression in tobacco,Plant Cell, 4:463-471, 1992.
【非特許文献15】Cartharius, K., K. Frech,et al., MatInspector and beyond: promoter analysisbased on transcription factor binding sites, Bioinformatics 21: 2933-42, 2005.
【非特許文献16】Gasser SM and Laemmli UK, Cohabitation of scaffold binding regions withupstream/enhancer elements of three developmentally regulated genes of D. Melanogaster, Cell, 46:521-530, 1986.
【非特許文献17】Girod PA, Zahn-ZabalM and Mermod N, Use of the chicken lysozyme 5' matrix attachment region to generate highproducer CHO cell lines, Biotechnol. Bioeng., 91(1):1-11, 2005.
【非特許文献18】Kas E and ChaslinLA, Anchorage of the Chinese hamster dihydrofolate reductase gene tothe nuclear scaffold occurs in an intragenic region,J. Mol. Biol., 198:677-692, 1987.
【非特許文献19】Kay V and Bode J, Detection ofscaffold -attached regions (SARs) by in vitrotechniques; activities of these elements in vivo. In Methods in Molecular andCellular Biology: Methods for studying DNA protein interactions: an overview, WileyLiss, NewYork, 5:186-194,1995.
【非特許文献20】Kim JM, Kim JS, Park DH, KangHS, Yoon J, Baek K and Yoon Y, Improved recombinantgene expression in CHO cells using matrix attachment regions, J. Biotechnol., 107(2):95-105, 2004.
【非特許文献21】Kwaks TH, OtteAP, Employing epigenetics to augment the expressionof therapeutic proteins in mammalian cells. Trends Biotechnol.24:137-42, 2006.
【非特許文献22】Labrador, M. and V. G. Corces, Setting the boundaries of chromatin domains andnuclear organization, Cell 111: 151-54, 2002.
【非特許文献23】Levine, M. and R. Tjian, Transcriptional regulation and animal diversity,Nature 424: 147-151, 2003.
【非特許文献24】Mielke C, KohwiY, KohwiShigematsu T and Bode J, Hierarchical bindingof DNA fragments derived from scaffold-attached regions: correlation ofproperties in vitro and function in vivo, Biochemistry, 29:7475-7485, 1990.
【非特許文献25】Miescher S, Zahn-ZabalM, De Jesus M, Moudry R, FischI, Vogel M, Kobr M, ImbodenMA, Kragten E, Bichler J, Mermod N, Stadler BC, Amstutz H, Wurm F, CHO,Expression of a Novel Human Recombinant IgG1 anti-RhD Antibody Isolated by Phage Display, Brit. J. Haematol.,111, 157-166, 2000.
【非特許文献26】National Center for Biotechnology Information(http://www.ncbi.nih.gov).
【非特許文献27】PhiVan L and StratlingWH, Dissection of the ability of the chicken lysozymegene 5' matrix attachment region to stimulate transgeneexpression and to dampen position effects, Biochemistry, 35:10735-10742, 1996.
【非特許文献28】Razin SV, Functional architectureof chromosomal DNA domains, Crit Rev Eukaryot Gene Expr, 6:247-269,1996.
【非特許文献29】Stefanovic D, StanojcicS et al., In vitro protein-DNA interactions at the human laminB2 replication origin, J Biol Chem278: 42737-42743, 2003.
【非特許文献30】Strick R and LaemmliUK, SARs are cis DNA elements of chromosome dynamics: synthesis of a SARrepressor protein, Cell 83(7): 1137-48, 2005.
【非特許文献31】Vogelstein B, Pardoll D and Coffey D, Supercoiledloops and eukaryotic DNA replication, Cell, 22:79-85, 1980.
【非特許文献32】You Z, IshimiY, et al., Thymine-rich single-stranded DNA activates Mcm4/6/7 helicase on Y-fork and bubble-like substrates, EMBO J 22:6148-6160 (2003).
【非特許文献33】Zahn-Zabal M, Kobr M, GirodPA, Imhof M, Chatellard P,de Jesus M, Wurm F and MermodN, Development of stable cell lines for production or regulated expressionusing matrix attachment regions. J Biotechnol, 87(1):29-42, 2001.
【特許請求の範囲】
【請求項1】
少なくとも1つの遺伝子の高レベルの発現のための発現系であって、
目的の遺伝子をコードするヌクレオチド配列を作動可能に連結するためのプロモーターと、
前記発現系で形質転換された細胞内で前記遺伝子の発現を促進するための少なくとも1つの非ヒト哺乳類のMARヌクレオチド配列と、
を含み、
ここで前記非ヒト哺乳類のMARヌクレオチド配列は前記コンストラクトを有する前記細胞の形質転換時に前記遺伝子の発現を約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくはそれ以上に増大させる、発現系。
【請求項2】
前記プロモーターと目的の遺伝子をコードする前記ヌクレオチド配列とを含む発現カセットが前記プロモーターに作動可能に連結される、請求項1に記載の発現系。
【請求項3】
前記少なくとも1つの非ヒト哺乳類のMARヌクレオチド配列が齧歯類のMARヌクレオチド配列、例えばマウスもしくはハムスターのMARヌクレオチド配列である、請求項1および2のいずれか一項に記載の発現系。
【請求項4】
前記非ヒト哺乳類のMARヌクレオチド配列が、
(i)配列番号3、配列番号10もしくはその機能的断片、または
(ii)(i)の配列のいずれかと約80%、約90%、約95%もしくは約98%の配列同一性を有するヌクレオチド配列
を含む、請求項1〜3のいずれか一項に記載の発現系。
【請求項5】
前記遺伝子が、非ヒト哺乳類細胞、例えば齧歯類細胞、特にマウスもしくはハムスター細胞、またはヒト細胞、例えばHeLa細胞において発現される、請求項1〜4のいずれか一項に記載の発現系。
【請求項6】
前記少なくとも1つの非ヒト哺乳類のMARヌクレオチド配列が前記遺伝子に対してシスまたはトランスに作用する、請求項1〜5のいずれか一項に記載の発現系。
【請求項7】
細胞内でのタンパク質の生成を促進するための方法であって、
−ヒトもしくは非ヒト哺乳類細胞を提供するステップと、
−請求項1〜6のいずれか一項に記載の発現系を、遺伝子発現が約2、約3、約4、約5、約6、約7、約8、約9、約10倍もしくはそれ以上に増大するように前記細胞に導入するステップと、
を含む、方法。
【請求項8】
(a)配列番号3もしくは配列番号10もしくはその機能的断片のヌクレオチド配列、または
(b)(a)の配列と少なくとも約80%、約90%、約95%もしくは約98%の配列同一性を有しかつMAR活性を有するヌクレオチド配列
を含む、単離、精製された核酸分子。
【請求項9】
非ヒト哺乳類のMAR配列を同定するための方法であって、
−少なくとも1つの非ヒト哺乳類の核酸分子、好ましくは非ヒト哺乳類のゲノムもしくはその一部を提供するステップと、
−前記核酸分子に、
−評価対象の核酸分子におけるウインドウサイズを設定するステップと、
−少なくとも1つもしくは少なくとも2つ、好ましくは3つ、より好ましくは4つもしくはそれより多いMAR関連の特徴を選択するステップと、
−この特徴/これらの特徴を示す配列に対する閾値を設定するステップと、
−これらの閾値を超えるMAR候補ヌクレオチド配列を選択するステップと、
−前記非ヒト哺乳類のMARヌクレオチド配列が、前記非ヒト哺乳類のMARヌクレオチド配列を含む発現系を介するヒトおよび/または非ヒト哺乳類細胞の形質転換時に、遺伝子の発現を約2、約3、約4、約5、約6、約7、約8、約9、約10倍もしくはそれ以上に増大させることを確認するステップと、
を含む、MAR配列における走査手順を施すステップと、
を含む、方法。
【請求項10】
前記少なくとも1つの特徴が、DNAベンディングの角度、主溝の深さ、副溝の幅、融解温度またはそれらの組み合わせでありうる、請求項9に記載の方法。
【請求項11】
DNAベンディングの角度値が、約3〜約5°(基本的角度°)、好ましくは3.8〜約4.4°を含み、ここでは約3.9°、約4.0°、約4.1°、約4.2°および約4.3°を含む、請求項10に記載の方法。
【請求項12】
主溝の深さ値が約8.9〜約9.3Åでありかつ副溝の幅値が約5.2〜約5.8Åであり、好ましくは前記主溝の深さ値が約9.1Åを含む約9.0〜約9.2Åでありかつ前記副溝の幅値が約5.5Åおよび約5.6Åを含む約5.4〜約5.7Åでありうる、請求項10または11に記載の方法。
【請求項13】
前記融解温度が約55〜約75℃、特に約55〜約62℃であり、ここで約56℃、約57℃、約58℃、約59℃、約60℃および約61℃を含む、請求項10〜12のいずれか一項に記載の方法。
【請求項14】
DNAベンディングの角度値が約4.0〜約5.0°であり、ここで約4.1°、約4.2°、約4.3°、約4.4°、約4.5°、約4.6°、約4.7°、約4.8°および約4.9°を含む、請求項10に記載の方法。
【請求項15】
前記DNAベンディングの角度値が約50bps〜約150bpsの範囲のウインドウ値と組み合わされ、ここで例えば約80bps、約100bpsおよび約120bpsを含む、請求項14に記載の方法。
【請求項16】
前記DNAベンディングの角度値×ウインドウ値が約320〜1320、例えば約420〜約1220、約520〜約1120、約620〜約1020、約720〜約920であり、前記主溝の深さ値×前記ウインドウ値が約900〜約4000、例えば約1200〜3700、約1500〜約3400、約1800〜約3100、約2100〜約2800であり、かつ/または副溝の深さ値×前記ウインドウサイズが約500〜約2500、例えば約750〜約2250、約1000〜約2000、約1250〜1750である、請求項10に記載の方法。
【請求項17】
−実験的に検証されたヒトもしくは非ヒト由来のMARを提供するステップと、
−前記実験的に検証されたヒトもしくは非ヒト由来のMARを用いて前記閾値を判定するステップと、
をさらに含む、請求項9〜16のいずれか一項に記載の方法。
【請求項18】
(a)(i)同定されたMARの末端領域の少なくとも一部を含む単離ヌクレオチド配列、および
(ii)前記同定されたMARまたは別の同定されたMARの約10%、約15%、約20%、約25%、約30%もしくはより多くを含むさらなる単離ヌクレオチド配列、
または
(b)(i)(a)(i)の前記ヌクレオチド配列と約90%、約95%、約96%、約97%、約98%、約99%の配列同一性を有するヌクレオチド配列、および
(ii)(b)(i)の前記ヌクレオチド配列と約70%、約80%、好ましくは約90%、約95%、約96%、約97%、約98%、約99%の配列同一性を有するヌクレオチド配列
を含むMARコンストラクト。
【請求項19】
(a)(ii)における前記ヌクレオチド配列がATリッチ領域を含む、請求項18に記載のMARコンストラクト
【請求項20】
同定されたMAR配列のヌクレオチドの数の約90%未満、好ましくは約80%未満、さらにより好ましくは約70%未満、約60%未満または約50%未満を含む、請求項18または19に記載のMARコンストラクト。
【請求項21】
同定されたMAR配列のヌクレオチドの数のほぼ同数かまたは少なくとも約110%を含む、請求項18〜20のいずれか一項に記載のMARコンストラクト。
【請求項22】
連続配列内の同定されたMAR配列の領域を含み、ここで順序および/または方向が同定されたMAR配列の場合と異なる、MARコンストラクト。
【請求項23】
前記領域が少なくとも1つのATリッチ領域および少なくとも1つの結合部位領域を含む、請求項22に記載のMARコンストラクト。
【請求項24】
場合により前記同定されたMAR配列に由来する、少なくとも1つの結合部位領域の少なくとも一部をさらに含む、請求項22または23に記載のMARコンストラクト。
【請求項25】
前記同定されたMAR配列がヒトまたはマウスのMARである、請求項22〜24に記載のMARコンストラクト。
【請求項26】
前記同定されたMAR配列の前記領域またはその一部が、前記天然のヒト1_68MARまたはマウスMAR S4またはその一部の領域と約70%の配列同一性、約80%の配列同一性、約90%の配列同一性、約95%の配列同一性もしくは約98%の配列同一性を有する、請求項22〜25のいずれか一項に記載のMARコンストラクト。
【請求項27】
前記領域が、各々、天然ヒト1_68MARの1〜1189bps、1190〜1952bpsおよび1953〜3600bpsに対応する、請求項22〜26のいずれか一項に記載のMARコンストラクト。
【請求項28】
前記領域が配列特異的な領域である、請求項22〜27のいずれか一項に記載のMARコンストラクト。
【請求項29】
(a)(i)同定されたMAR配列の少なくとも1つの単離または合成されたATリッチ領域、または
(ii)(a)(i)の前記ATリッチ領域と少なくとも少なくとも80%、85%、90%、95%、98%もしくは99%の配列同一性を有する少なくとも1つのATリッチ領域
を含むコアヌクレオチド配列と、
(b)(a)の前記ヌクレオチド配列に隣接した少なくとも1つのDNAタンパク質結合部位を含むヌクレオチド配列と、
を含み、ここで前記結合部位は、
(i)さらに同定されたMAR配列のDNAタンパク質結合部位、
(ii)前記同定されたMAR配列内で(a)の前記コアヌクレオチド配列外部に位置する、(a)の前記同定されたMAR配列のDNAタンパク質結合部位、または
(iii)(a)の前記コア内に存在するが少なくとも1つのさらなるDNAタンパク質結合部位に隣接する第1のDNAタンパク質結合部位であって、前記第1および前記さらなるDNAタンパク質結合部位の少なくとも1つは(a)の前記コア内で隣接することがない、第1のDNAタンパク質結合部位、または
(iv)非MAR配列のDNAタンパク質結合部位
である、MARコンストラクト。
【請求項30】
プロモーターに作動可能に連結される遺伝子の発現を、前記MARコンストラクトの細胞への導入時に、約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくはそれ以上に促進する、請求項29に記載のMARコンストラクト。
【請求項31】
500ヌクレオチド長未満、好ましくは約250ヌクレオチド長未満、さらにより好ましくは約200未満、約150もしくは約100ヌクレオチド長未満であり、請求項29または30に記載のMARコンストラクト。
【請求項32】
(a)の前記コア核酸配列が、前記同定されたMARの少なくとも1つのTFBSを含み、ここで前記少なくとも1つのTFBSは前記同定されたMAR内の前記ATリッチ領域の一方または両方に隣接する、請求項29〜31のいずれか一項に記載のMARコンストラクト。
【請求項33】
(b)における前記少なくとも1つのDNAタンパク質結合部位がTFBSであり、かつ1つ、2つ、3つ、4つ、5つもしくはより多くの置換、付加および/または欠失により修飾され、かつ/あるいは完全または部分的に合成されている、請求項29〜32のいずれか一項に記載のMARコンストラクト。
【請求項34】
前記ATリッチ領域に隣接する前記TFBSが、1つ、2つ、3つ、4つ、5つもしくはより多くの置換、付加および/または欠失により修飾される、請求項29〜33のいずれか一項に記載のMARコンストラクト。
【請求項35】
前記TFBSが既知の天然相同物を全く有しない最適化されたTFBSである、請求項33または34に記載のMARコンストラクト。
【請求項36】
前記結合部位が、SATB1、NMP4、HOX、HOXF、Gsh、CEBP、Fast1およびSATB1またはこれらの転写因子のうちの2つもしくはそれより多くの組み合わせからなる群から選択される、請求項29〜35のいずれか一項に記載のMARコンストラクト。
【請求項37】
(b)の一連の前記DNAタンパク質結合部位が(a)の前記核酸配列に隣接する、請求項29〜36のいずれか一項に記載のMARコンストラクト。
【請求項38】
前記MARコンストラクトが促進されたMARコンストラクトである、請求項29〜37のいずれか一項に記載のMARコンストラクト。
【請求項39】
上記請求項のいずれか一項に記載のMARコンストラクトの少なくとも1つ、ならびに場合により、プロモーターおよび目的のヌクレオチド配列を前記プロモーターの制御下で導入するための少なくとも1つの制限酵素結合部位を含む、発現系。
【請求項40】
上記請求項のいずれか一項に記載の発現系を含む細胞。
【請求項41】
上記請求項のいずれか一項に記載の発現系を含むトランスジェニックな非ヒト動物。
【請求項42】
上記請求項のいずれか一項に記載の発現系、および
前記発現系の使用方法についての使用説明書
を含むキット。
【請求項43】
遺伝子の発現を促進するための方法であって、
プロモーターおよび上記請求項のいずれか一項に記載のMARコンストラクトの制御下での前記遺伝子を含む発現系を提供するステップと、
前記遺伝子の前記発現が促進されるように細胞に前記発現系を形質移入するステップと、を含む、方法。
【請求項44】
前記発現系が前記遺伝子の発現の安定性をさらに高める、請求項43に記載の方法。
【請求項45】
ヒト病原体タンパク質またはヒト細胞表面タンパク質を認識する抗体などのタンパク質、ならびにエリスロポエチン、インターフェロンまたは他の治療用もしくは診断用タンパク質などのタンパク質の生成における、上記請求項のいずれか一項に記載のMARコンストラクト、発現系、トランスジェニック非ヒト動物、キットおよび/または方法の使用。
【請求項46】
インビトロおよび/またはインビボ遺伝子療法ならびに/あるいは細胞または組織置換療法における、上記請求項のいずれか一項に記載のMARコンストラクト、発現系、細胞、キットおよび/または方法の使用。
【請求項1】
少なくとも1つの遺伝子の高レベルの発現のための発現系であって、
目的の遺伝子をコードするヌクレオチド配列を作動可能に連結するためのプロモーターと、
前記発現系で形質転換された細胞内で前記遺伝子の発現を促進するための少なくとも1つの非ヒト哺乳類のMARヌクレオチド配列と、
を含み、
ここで前記非ヒト哺乳類のMARヌクレオチド配列は前記コンストラクトを有する前記細胞の形質転換時に前記遺伝子の発現を約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくはそれ以上に増大させる、発現系。
【請求項2】
前記プロモーターと目的の遺伝子をコードする前記ヌクレオチド配列とを含む発現カセットが前記プロモーターに作動可能に連結される、請求項1に記載の発現系。
【請求項3】
前記少なくとも1つの非ヒト哺乳類のMARヌクレオチド配列が齧歯類のMARヌクレオチド配列、例えばマウスもしくはハムスターのMARヌクレオチド配列である、請求項1および2のいずれか一項に記載の発現系。
【請求項4】
前記非ヒト哺乳類のMARヌクレオチド配列が、
(i)配列番号3、配列番号10もしくはその機能的断片、または
(ii)(i)の配列のいずれかと約80%、約90%、約95%もしくは約98%の配列同一性を有するヌクレオチド配列
を含む、請求項1〜3のいずれか一項に記載の発現系。
【請求項5】
前記遺伝子が、非ヒト哺乳類細胞、例えば齧歯類細胞、特にマウスもしくはハムスター細胞、またはヒト細胞、例えばHeLa細胞において発現される、請求項1〜4のいずれか一項に記載の発現系。
【請求項6】
前記少なくとも1つの非ヒト哺乳類のMARヌクレオチド配列が前記遺伝子に対してシスまたはトランスに作用する、請求項1〜5のいずれか一項に記載の発現系。
【請求項7】
細胞内でのタンパク質の生成を促進するための方法であって、
−ヒトもしくは非ヒト哺乳類細胞を提供するステップと、
−請求項1〜6のいずれか一項に記載の発現系を、遺伝子発現が約2、約3、約4、約5、約6、約7、約8、約9、約10倍もしくはそれ以上に増大するように前記細胞に導入するステップと、
を含む、方法。
【請求項8】
(a)配列番号3もしくは配列番号10もしくはその機能的断片のヌクレオチド配列、または
(b)(a)の配列と少なくとも約80%、約90%、約95%もしくは約98%の配列同一性を有しかつMAR活性を有するヌクレオチド配列
を含む、単離、精製された核酸分子。
【請求項9】
非ヒト哺乳類のMAR配列を同定するための方法であって、
−少なくとも1つの非ヒト哺乳類の核酸分子、好ましくは非ヒト哺乳類のゲノムもしくはその一部を提供するステップと、
−前記核酸分子に、
−評価対象の核酸分子におけるウインドウサイズを設定するステップと、
−少なくとも1つもしくは少なくとも2つ、好ましくは3つ、より好ましくは4つもしくはそれより多いMAR関連の特徴を選択するステップと、
−この特徴/これらの特徴を示す配列に対する閾値を設定するステップと、
−これらの閾値を超えるMAR候補ヌクレオチド配列を選択するステップと、
−前記非ヒト哺乳類のMARヌクレオチド配列が、前記非ヒト哺乳類のMARヌクレオチド配列を含む発現系を介するヒトおよび/または非ヒト哺乳類細胞の形質転換時に、遺伝子の発現を約2、約3、約4、約5、約6、約7、約8、約9、約10倍もしくはそれ以上に増大させることを確認するステップと、
を含む、MAR配列における走査手順を施すステップと、
を含む、方法。
【請求項10】
前記少なくとも1つの特徴が、DNAベンディングの角度、主溝の深さ、副溝の幅、融解温度またはそれらの組み合わせでありうる、請求項9に記載の方法。
【請求項11】
DNAベンディングの角度値が、約3〜約5°(基本的角度°)、好ましくは3.8〜約4.4°を含み、ここでは約3.9°、約4.0°、約4.1°、約4.2°および約4.3°を含む、請求項10に記載の方法。
【請求項12】
主溝の深さ値が約8.9〜約9.3Åでありかつ副溝の幅値が約5.2〜約5.8Åであり、好ましくは前記主溝の深さ値が約9.1Åを含む約9.0〜約9.2Åでありかつ前記副溝の幅値が約5.5Åおよび約5.6Åを含む約5.4〜約5.7Åでありうる、請求項10または11に記載の方法。
【請求項13】
前記融解温度が約55〜約75℃、特に約55〜約62℃であり、ここで約56℃、約57℃、約58℃、約59℃、約60℃および約61℃を含む、請求項10〜12のいずれか一項に記載の方法。
【請求項14】
DNAベンディングの角度値が約4.0〜約5.0°であり、ここで約4.1°、約4.2°、約4.3°、約4.4°、約4.5°、約4.6°、約4.7°、約4.8°および約4.9°を含む、請求項10に記載の方法。
【請求項15】
前記DNAベンディングの角度値が約50bps〜約150bpsの範囲のウインドウ値と組み合わされ、ここで例えば約80bps、約100bpsおよび約120bpsを含む、請求項14に記載の方法。
【請求項16】
前記DNAベンディングの角度値×ウインドウ値が約320〜1320、例えば約420〜約1220、約520〜約1120、約620〜約1020、約720〜約920であり、前記主溝の深さ値×前記ウインドウ値が約900〜約4000、例えば約1200〜3700、約1500〜約3400、約1800〜約3100、約2100〜約2800であり、かつ/または副溝の深さ値×前記ウインドウサイズが約500〜約2500、例えば約750〜約2250、約1000〜約2000、約1250〜1750である、請求項10に記載の方法。
【請求項17】
−実験的に検証されたヒトもしくは非ヒト由来のMARを提供するステップと、
−前記実験的に検証されたヒトもしくは非ヒト由来のMARを用いて前記閾値を判定するステップと、
をさらに含む、請求項9〜16のいずれか一項に記載の方法。
【請求項18】
(a)(i)同定されたMARの末端領域の少なくとも一部を含む単離ヌクレオチド配列、および
(ii)前記同定されたMARまたは別の同定されたMARの約10%、約15%、約20%、約25%、約30%もしくはより多くを含むさらなる単離ヌクレオチド配列、
または
(b)(i)(a)(i)の前記ヌクレオチド配列と約90%、約95%、約96%、約97%、約98%、約99%の配列同一性を有するヌクレオチド配列、および
(ii)(b)(i)の前記ヌクレオチド配列と約70%、約80%、好ましくは約90%、約95%、約96%、約97%、約98%、約99%の配列同一性を有するヌクレオチド配列
を含むMARコンストラクト。
【請求項19】
(a)(ii)における前記ヌクレオチド配列がATリッチ領域を含む、請求項18に記載のMARコンストラクト
【請求項20】
同定されたMAR配列のヌクレオチドの数の約90%未満、好ましくは約80%未満、さらにより好ましくは約70%未満、約60%未満または約50%未満を含む、請求項18または19に記載のMARコンストラクト。
【請求項21】
同定されたMAR配列のヌクレオチドの数のほぼ同数かまたは少なくとも約110%を含む、請求項18〜20のいずれか一項に記載のMARコンストラクト。
【請求項22】
連続配列内の同定されたMAR配列の領域を含み、ここで順序および/または方向が同定されたMAR配列の場合と異なる、MARコンストラクト。
【請求項23】
前記領域が少なくとも1つのATリッチ領域および少なくとも1つの結合部位領域を含む、請求項22に記載のMARコンストラクト。
【請求項24】
場合により前記同定されたMAR配列に由来する、少なくとも1つの結合部位領域の少なくとも一部をさらに含む、請求項22または23に記載のMARコンストラクト。
【請求項25】
前記同定されたMAR配列がヒトまたはマウスのMARである、請求項22〜24に記載のMARコンストラクト。
【請求項26】
前記同定されたMAR配列の前記領域またはその一部が、前記天然のヒト1_68MARまたはマウスMAR S4またはその一部の領域と約70%の配列同一性、約80%の配列同一性、約90%の配列同一性、約95%の配列同一性もしくは約98%の配列同一性を有する、請求項22〜25のいずれか一項に記載のMARコンストラクト。
【請求項27】
前記領域が、各々、天然ヒト1_68MARの1〜1189bps、1190〜1952bpsおよび1953〜3600bpsに対応する、請求項22〜26のいずれか一項に記載のMARコンストラクト。
【請求項28】
前記領域が配列特異的な領域である、請求項22〜27のいずれか一項に記載のMARコンストラクト。
【請求項29】
(a)(i)同定されたMAR配列の少なくとも1つの単離または合成されたATリッチ領域、または
(ii)(a)(i)の前記ATリッチ領域と少なくとも少なくとも80%、85%、90%、95%、98%もしくは99%の配列同一性を有する少なくとも1つのATリッチ領域
を含むコアヌクレオチド配列と、
(b)(a)の前記ヌクレオチド配列に隣接した少なくとも1つのDNAタンパク質結合部位を含むヌクレオチド配列と、
を含み、ここで前記結合部位は、
(i)さらに同定されたMAR配列のDNAタンパク質結合部位、
(ii)前記同定されたMAR配列内で(a)の前記コアヌクレオチド配列外部に位置する、(a)の前記同定されたMAR配列のDNAタンパク質結合部位、または
(iii)(a)の前記コア内に存在するが少なくとも1つのさらなるDNAタンパク質結合部位に隣接する第1のDNAタンパク質結合部位であって、前記第1および前記さらなるDNAタンパク質結合部位の少なくとも1つは(a)の前記コア内で隣接することがない、第1のDNAタンパク質結合部位、または
(iv)非MAR配列のDNAタンパク質結合部位
である、MARコンストラクト。
【請求項30】
プロモーターに作動可能に連結される遺伝子の発現を、前記MARコンストラクトの細胞への導入時に、約2倍、約3倍、約4倍、約5倍、約6倍、約7倍、約8倍、約9倍、約10倍もしくはそれ以上に促進する、請求項29に記載のMARコンストラクト。
【請求項31】
500ヌクレオチド長未満、好ましくは約250ヌクレオチド長未満、さらにより好ましくは約200未満、約150もしくは約100ヌクレオチド長未満であり、請求項29または30に記載のMARコンストラクト。
【請求項32】
(a)の前記コア核酸配列が、前記同定されたMARの少なくとも1つのTFBSを含み、ここで前記少なくとも1つのTFBSは前記同定されたMAR内の前記ATリッチ領域の一方または両方に隣接する、請求項29〜31のいずれか一項に記載のMARコンストラクト。
【請求項33】
(b)における前記少なくとも1つのDNAタンパク質結合部位がTFBSであり、かつ1つ、2つ、3つ、4つ、5つもしくはより多くの置換、付加および/または欠失により修飾され、かつ/あるいは完全または部分的に合成されている、請求項29〜32のいずれか一項に記載のMARコンストラクト。
【請求項34】
前記ATリッチ領域に隣接する前記TFBSが、1つ、2つ、3つ、4つ、5つもしくはより多くの置換、付加および/または欠失により修飾される、請求項29〜33のいずれか一項に記載のMARコンストラクト。
【請求項35】
前記TFBSが既知の天然相同物を全く有しない最適化されたTFBSである、請求項33または34に記載のMARコンストラクト。
【請求項36】
前記結合部位が、SATB1、NMP4、HOX、HOXF、Gsh、CEBP、Fast1およびSATB1またはこれらの転写因子のうちの2つもしくはそれより多くの組み合わせからなる群から選択される、請求項29〜35のいずれか一項に記載のMARコンストラクト。
【請求項37】
(b)の一連の前記DNAタンパク質結合部位が(a)の前記核酸配列に隣接する、請求項29〜36のいずれか一項に記載のMARコンストラクト。
【請求項38】
前記MARコンストラクトが促進されたMARコンストラクトである、請求項29〜37のいずれか一項に記載のMARコンストラクト。
【請求項39】
上記請求項のいずれか一項に記載のMARコンストラクトの少なくとも1つ、ならびに場合により、プロモーターおよび目的のヌクレオチド配列を前記プロモーターの制御下で導入するための少なくとも1つの制限酵素結合部位を含む、発現系。
【請求項40】
上記請求項のいずれか一項に記載の発現系を含む細胞。
【請求項41】
上記請求項のいずれか一項に記載の発現系を含むトランスジェニックな非ヒト動物。
【請求項42】
上記請求項のいずれか一項に記載の発現系、および
前記発現系の使用方法についての使用説明書
を含むキット。
【請求項43】
遺伝子の発現を促進するための方法であって、
プロモーターおよび上記請求項のいずれか一項に記載のMARコンストラクトの制御下での前記遺伝子を含む発現系を提供するステップと、
前記遺伝子の前記発現が促進されるように細胞に前記発現系を形質移入するステップと、を含む、方法。
【請求項44】
前記発現系が前記遺伝子の発現の安定性をさらに高める、請求項43に記載の方法。
【請求項45】
ヒト病原体タンパク質またはヒト細胞表面タンパク質を認識する抗体などのタンパク質、ならびにエリスロポエチン、インターフェロンまたは他の治療用もしくは診断用タンパク質などのタンパク質の生成における、上記請求項のいずれか一項に記載のMARコンストラクト、発現系、トランスジェニック非ヒト動物、キットおよび/または方法の使用。
【請求項46】
インビトロおよび/またはインビボ遺伝子療法ならびに/あるいは細胞または組織置換療法における、上記請求項のいずれか一項に記載のMARコンストラクト、発現系、細胞、キットおよび/または方法の使用。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】


【公表番号】特表2010−501170(P2010−501170A)
【公表日】平成22年1月21日(2010.1.21)
【国際特許分類】
【出願番号】特願2009−525122(P2009−525122)
【出願日】平成19年8月22日(2007.8.22)
【国際出願番号】PCT/IB2007/002404
【国際公開番号】WO2008/023247
【国際公開日】平成20年2月28日(2008.2.28)
【出願人】(504355653)
【Fターム(参考)】
【公表日】平成22年1月21日(2010.1.21)
【国際特許分類】
【出願日】平成19年8月22日(2007.8.22)
【国際出願番号】PCT/IB2007/002404
【国際公開番号】WO2008/023247
【国際公開日】平成20年2月28日(2008.2.28)
【出願人】(504355653)

【Fターム(参考)】
[ Back to top ]

