遺伝子の発現調節配列
【課題】 NAIP遺伝子のプロモーター配列およびサイレンサー配列に相当するポリヌクレオチドと、このポリヌクレオチドの利用発明を提供する。
【解決手段】 配列番号1の−765番塩基から−416番塩基の塩基配列からなり、遺伝子発現を促進するポリヌクレオチド、配列番号1の−2218番塩基から―766番塩基の塩基配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド、および配列番号1の−421番塩基から296番塩基の塩基配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド、ならびにこれらのポリヌクレオチドを用いた方法発明。
【解決手段】 配列番号1の−765番塩基から−416番塩基の塩基配列からなり、遺伝子発現を促進するポリヌクレオチド、配列番号1の−2218番塩基から―766番塩基の塩基配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド、および配列番号1の−421番塩基から296番塩基の塩基配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド、ならびにこれらのポリヌクレオチドを用いた方法発明。
【発明の詳細な説明】
【0001】
【発明の属する技術分野】この出願の発明は、遺伝子プロモーター配列と遺伝子サイレンサー配列に関するものである。さらに詳しくは、この出願の発明は、主に神経系組織で発現するアポトーシス抑制タンパク質NAIP(Neuronal Apoptosis Inhibitory Protein)をコードする遺伝子のプロモーター配列およびサイレンサー配列に相当するポリヌクレオチドとその利用発明に関するものである。
【0002】
【従来の技術とその課題】アポトーシス(apoptosis)は、プログラムされた細胞死の一種であり、周囲の細胞との接触の欠乏、細胞質の濃縮化、エンドヌクレアーゼの活性に関連したクロマチンの凝縮および核凝縮、核の断片化、膜被包性球状小体化、隣接するマクロファージもしくは上皮細胞などによる球状小体の貪食、またはエンドヌクレアーゼ活性によりDNAのヌクレオソーム単位が180〜200塩基長のDNAに断片化するといった現象が観察され、このような現象が認められるアポプティック体細胞の最終断片が隣接する細胞により貪食される機構として論じられている(例えば、Immunology Today 7:115-119, 1986;Science 245:301-305, 1989 )。
【0003】このアポトーシスを制御する遺伝子としてしては、例えば、1985年に胞性B細胞腫から発見されたガン遺伝子のひとつであるbcl-2遺伝子が知られている。このbcl-2遺伝子は、免疫系や神経性の細胞で高頻度に発現しており、この遺伝子の発現産物はこれら細胞のアポトーシスを抑制することによって、ヒトの免疫機能や神経系機能の恒常性を維持していると考えられている。また、このbcl-2遺伝子は、胎児では特に広範囲には発現していることから、個体発生の際の形態形成にも重要な役割を果たしていると考えられてもいる。
【0004】一方、この出願の発明者等は、家族性の遺伝病である脊髄性筋萎縮症(SpinalMascular Atropy:SMA)の原因遺伝子として、ヒト染色体5q13.1領域より神経細胞アポトーシス抑制蛋白質(Neuronal Apoptosis Inhibitory Protein : NAIP)遺伝子を単離している(Roy et al., Cell 80: 167-178, 1995)。すなわち、このNAIP遺伝子の変異またはコピー数の減少が脊髄ニューロンのアポトーシスを生じさせ、これがSMA発症の原因となると想定されている。また、このNAIP遺伝子を種々の培養細胞に導入し、アポトーシスを誘起させる刺激を細胞に与えたところ、その細胞死が抑制されることが明らかにされ(Liston et al., Nature 379:349-353, 1996 )、NAIPが神経細胞だけではなく、体細胞全体のアポトーシス抑制因子であることが明らかにされている。
【0005】そしてこの出願の発明者等は、NAIPをコードするcDNAを単離し、そのアミノ酸配列を決定して既に特許出願している(特願平9−280831号)。
【0006】
【発明が解決しようとする課題】前記のとおり、NAIPはSMAをはじめとする各種アポトーシス性疾患に関与する蛋白質であることから、NAIP遺伝子の発現に関わる分子の解明は、それら疾患の発症メカニズムの解明、発症の危険性の診断、発症の予防もしくは病態の改善、治療のための医療技術および薬剤の開発等のためにとって極めて重要である。
【0007】この出願の発明は以上のとおりの事情に鑑みてなされたものであり、NAIP遺伝子のプロモーター配列/エンハンサー配列およびサイレンサー配列を提供することを課題としている。
【0008】また、この出願の発明は、これらプロモーター配列/エンハンサー配列およびサイレンサー配列の利用発明を提供することを課題としている。
【0009】
【課題を解決するための手段】この出願は、前記の課題を解決するものとして、以下の(1)から(16)の発明を提供する。
(1) 配列番号1の−765番塩基から−416番塩基の塩基配列、またはその一部配列からなり、遺伝子発現を促進するポリヌクレオチド。
(2) 配列番号1の−482番塩基から−416番塩基の塩基配列からなり、遺伝子プロモーターとして機能するポリヌクレオチド。
(3) 配列番号1の−646番塩基から−531番塩基の塩基配列からなり、遺伝子エンハンサーとして機能するポリヌクレオチド。
(4) 配列番号1の−2218番塩基から−766番塩基の塩基配、またはその一部配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド。
(5) 配列番号1の−421番塩基から296番塩基の塩基配列、またはその一部配列からなり、遺伝子遺伝子サイレンサーとして機能するポリヌクレオチド。
(6) 配列番号1の−421番塩基から−202番塩基の塩基配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド。
(7) 前記(1)または(2)のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
(8) 前記(1)または(2)のポリヌクレオチドおよび前記(4)のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
(9) 前記(1)または(2)のポリヌクレオチドおよび前記(5)はたは(6)のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
(10) 前記(1)または(2)のポリヌクレオチド、前記(4)および(5)または(6)のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
(11) ヒト・アポトーシス抑制タンパク質NAIPの発現促進因子をスクリーニングする方法であって、前記(7)の組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量を指標としてNAIPの発現促進因子を特定することを特徴とする方法。
(12) ヒト・アポトーシス抑制タンパク質NAIPの発現抑制因子をスクリーニングする方法であって、前記(8)から(10)のいずれかの組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量を指標としてNAIPの発現抑制因子を特定することを特徴とする方法。
(13) タンパク質を動物神経組織で発現させる方法であって、前記(1)または(2)のポリヌクレオチドに連結したタンパク質遺伝子動物体内で発現させることを特徴とする方法。
(14) 細胞アポトーシス機能の失調を原因とする疾患の診断方法であって、配列番号1の塩基配列における任意配列領域の変異を検出することを特徴とする方法。
(15) 疾患が、脊髄性筋萎縮症である前記(14)の方法。
(16) 任意配列領域が配列番号1の−765番塩基から−416番塩基の塩基配列、、−2218番塩基から−766番塩基の塩基配列、−421番塩基から296番塩基の塩基配列の少なくとも一つである前記(14)または(15)の方法。
【0010】
【発明の実施の形態】前記発明(1)の遺伝子発現を促進するポリヌクレオチドは、配列番号1の−765番塩基から−416番塩基の塩基配列からなるDNA断片、またはその一部配列からなるDNA断片としてとして提供される。このポリヌクレオチドには、配列番号1の−482番塩基から−416番塩基の塩基配列からなり、遺伝子プロモーターとして機能するポリヌクレオチドと、配列番号1の−646番塩基から−531番塩基の塩基配列からなり、遺伝子エンハンサーとして機能するポリヌクレオチドが含まれている。
【0011】また、前記発明(4)の遺伝子サイレンサーとして機能するポリヌクレオチドは、配列番号1の−2218番塩基から−766番塩基の塩基配列、またはその一部配列からなるDNA断片である。さらに、発明(5)および(6)のポリヌクレオチドは、同じく遺伝子サイレンサーとして機能する配列であって、−421番塩基から296番塩基の塩基配列からなるDNA断片、および−421番塩基から−202番塩基の塩基配列からなるDNA断片、もしくはそれらの一部配列からなるDNA断片である。
【0012】このようなDNA断片は、配列番号1の当該領域の塩基配列に基づいて化学合成することができる。あるいはまた、配列番号1の各々の当該領域の配列に基づいて合成したオリゴヌクレオチドをプローブとして、ヒトゲノムDNAライブラリーからも単離できるし、またはゲノムDNA(鋳型)と合成オリゴヌクレオチド(プライマー)を用いたPCR法等によっても取得することができる。
【0013】前記発明(7)の組換えベクターは、発明(1)または(2)のポリヌクレオチド(少なくともプロモーター配列を含む遺伝子発現促進配列)の下流支配下にレポーター遺伝子を有する組換えベクターである。このような組換えベクターは、例えば、前記のポリヌクレオチドとレポーター遺伝子を連結させてDNA断片を作成し、このDNA断片を公知のベクターDNAに挿入結合させることによって構築することができる。ベクターの種類としては、大腸菌、枯草菌等の原核細胞や、酵母、昆虫細胞、哺乳動物細胞等の真核細胞を宿主とする各種のベクターを用いることができる。また、レポーター遺伝子としては、クロラムフェニコールアセチルトランスフェラーゼ(CAT)遺伝子やルシフェラーゼ遺伝子、lacZ遺伝子等を用いることができる。
【0014】前記発明(8)〜(10)の組換えベクターは、発明(1)または(2)のポリヌクレオチドと発明(4)〜(6)のポリヌクレオチド(サイレンサー配列)を少なくとも1個連結した組換えベクターである。この組換えベクターの場合には、各挿入配列の順番は、「サイレンサー配列」、「遺伝子発現促進配列」、「レポーター」遺伝子の順番であってもよく、あるいは「遺伝子発現促進配列」、「サイレンサー配列」、「レポーター遺伝子」の順番であってもよい。これらの組換えベクターについても、ベクターやレポーター遺伝子は前記発明(7)と同様のものを使用することができる。
【0015】なお、以上の発明(7)〜(10)の組換えベクターの構築においては、レポーター遺伝子を既に保有しているベクターDNAに前記の各ポリヌクレオチドを適宜な部位に挿入するようにしてもよい。
【0016】この出願の前記発明(11)および(12)の方法は、以上のとおりに構築した組換えベクターを用いて、NAIPの発現促進因子や発現抑制因子をスクリーニングする方法である。例えば、発明(11)の方法は、前記(7)の組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量(例えば、CAT活性やルシフェラーゼ活性等)を測定することによって、各候補物質のプロモーター配列への作用(転写活性)を評価する方法である。また、前記(12)の発明は、前記(8)〜(10)の組換えベクターを用いた同様の操作によって、各候補物質のサイレンサー配列への作用(転写抑制活性)を評価する方法である。このようなスクリーニング方法によって特定されたNAIP発現促進因子は、例えば、過剰なアポトーシス性細胞死を原因とする疾患(例えば、SMAやハンチントン病、パーキンソン病、アルツハイマー病、脊髄硬化症、小脳変性疾患等の神経変性疾患、および免疫不全、組織萎縮を伴う各種疾患等)の治療薬成分として有用である。また、NAIP発現抑制因子は、細胞アポトーシスの低下を原因とする疾患(例えば、奇形、自己免疫疾患、ガン等)の治療薬成分として有用である。
【0017】この出願の前記(13)の方法は、例えば、神経組織で作用させることが動物個体の神経系機能・構造の改善等に有用であるにも係わらず、脳血液関門を通過しないために神経系組織に作用させることができないタンパク質を、神経系組織で特異的に発現させる方法である。そのようなタンパク質としては、例えば、インターフェロン、インターロイキン、サイトカイン、プロテアーゼとプロテアーゼインヒビター等である。この発明(13)の方法は、例えば、前記(1)または(2)のポリヌクレオチド(少なくともプロモーター配列を含む遺伝子発現促進配列)の下流支配下に当該タンパク質遺伝子を連結したDNA断片を動物個体内に導入に、当該タンパク質を神経系組織で発現させる方法である。すなわち、発明(1)または(2)のポリヌクレオチドは、神経系組織で発現しているNAIP遺伝子の発現促進配列(プロモーター配列および/またはエンハンサー配列を含む)であり、このポリヌクレオチドの支配によって下流タンパク質遺伝子は神経系組織で優位に発現するようになる。ポリヌクレオチドとタンパク質遺伝子とのDNA断片を動物個体内に導入する方法としては、例えば、遺伝子治療等において用いられているウイルスベクターを使用する方法等を用いることができる。また、動物個体が非ヒト動物の場合には、公知のトランスジェニック動物作成方法等を用いることもできる。
【0018】この出願の前記(14)の方法は、配列番号1に塩基配列を示したNAIP遺伝子5’側配列のいずれかの変異(例えば、配列番号1における1もしくは複数の塩基の欠失、付加、または他の塩基への置換等)を検出することによって、細胞アポトーシス機能の失調を原因とする疾患を診断する方法である。すなわち、NAIP遺伝子の変異がSMAの発症原因の一と推察されている(Roy et al., Cell80:167-178, 1995; Chen et al., Genomics 15:121-127, 1998)、SMA患者におけるNAIP遺伝子変異は、最も症状の重いSMAI型で約60%であり、症状が軽くなるに従って(SMAII〜III型)、NAIP遺伝子欠失変異の出現頻度が低下することが知られている(Roy et al., Cell 80:167-178, 1995; J. Neuro Sci 158(1):43-46, 1998; Arch Dis Child 78(6):531-535, 1998)。このことは、SMAのようなアポトーシス性疾患の原因が、遺伝子そのものの変異だけにあるのではなく、その原因遺伝子の発現を調整する領域の変異にも存在することを示唆する。特にNAIP遺伝子非欠損性のSMA(I型は約40%、II型は約70%、III型は約100%)は、アポトーシス性神経細胞死の抑制メカニズムの失調、すなわちNAIPの発現抑制が発症原因と考えられることから、プロモーター配列の変異がNAIP遺伝子発現を低下させ、結果として脱抑制的な神経細胞アポトーシスがSMAの原因となっていると考えられる。従って、SMA等のアポトーシス性疾患のハイリスク者や、そのような疾患の初期症状を呈する患者の生体試料からDNAを抽出し、配列番号1の−765番塩基から−416番塩基までの塩基配列、−2218番塩基から−766番塩基の塩基配列、−421番塩基から296番塩基の塩基配列に相当するDNA配列を検査することによって、その後の症状の発生や進行を予測することが可能となり、さらには、より適切な治療方法等の選択が可能となる。
【0019】以下、前記発明(1)から(3)のポリヌクレオチド(プロモーター配列/エンハンサー配列)、ならびに前記(4)〜(6)のポリヌクレオチド(サイレンサー配列)を決定した方法について詳しく説明する。
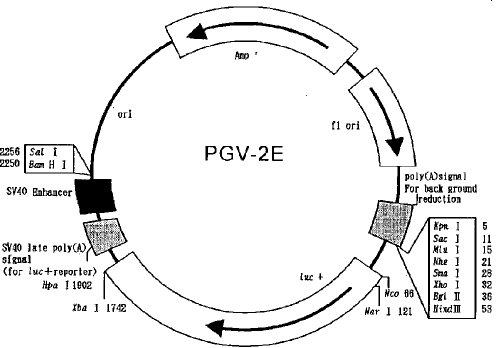
1.ルシフェラーゼアッセイのためのベクター構築配列番号1に塩基配列を示したNAIP遺伝子転写開始領域の欠失変異断片を表1のとおりに作成した。変異断片は、制限酵素による切断、または制限酵素認識部位を含むオリゴヌクレオチドをプライマーとするPCR法により作成した。次いで、両端を制限酵素で調整した各変異断片を、ルシフェラーゼ遺伝子を含むプラスミドベクターPGV−E2(Toyo Ink社製:図1)にサブクローニングした。また、サイレンサーアッセイ用のベクターは、NAIP遺伝子のプロモーター配列の全てとルシフェラーゼ遺伝子を含むベクターpEV2−350に各変異断片をサブクローニングして作成した(図2)。
【0020】
【表1】

【0021】2.ルシフェラーゼアッセイの方法2×10-5個のC33A細胞およびHeLa細胞を35mの培養皿で培養した。C33A細胞は、10%の不活性化ウシ胎児血清(FCS)含有のDulbecco's modifiedMEM low glucose培地で培養した。また、HeLa細胞は10%FCS含有のMEM培地で培養した。次いで、これらの培養細胞に、LipofectAMINE PLUS(Life Technologies社製)を用いて各ベクターをトランスフェクトした。すなわち、各々のプラスミド(C33A細胞に対して1.0μg:HeLa細胞に対して1.5μg)とコントロールベクターpEF−lacZ(0.1μg)を、50μlのOPTI-MEMI製血清培地(Life Technologies社製)と6μlのPLUS試薬を含む同培地50μlとの混合培地に加え、室温で15分間インキュベートした後、4μlのLipofectAMINEを含む同培地100μlと混合し、室温で15分間インキュベートした。次いで、0.8mlのOPTI-MEMI製血清培地を細胞が浸るまで加え、37℃で15時間インキュベートした後、2mlの完全増殖培地を加え、37℃でさらに15時間インキュベートした。培地を2mlの完全増殖培地に代え、37℃でさらに30時間インキュベートした。次に、培養細胞をPicaGene細胞溶解バッファー(Toyo Ink社製)により溶解し、全タンパク質量と、細胞溶解物中のβ−ガラクトシダーゼ発現量を、それぞれタンパク質定量装置(Bio-Rad社製)およびβ−ガラクトシダーゼ定量装置(Promega社製)により測定した。また、細胞溶解物中のルシフェラーゼ活性は、シングルフォトン計測装置Argus-50/CL(Hamamatsu Photonics社製)により測定した。
3.結果結果は図3から図10に示したとおりである。
【0022】図3および4に示したとおり、配列番号1の−765番塩基から−416番塩基までの配列にルシフェラーゼ遺伝子を連結させたベクター(pEV2−SB350)が最も高いルシフェラーゼ活性を示したことから、この配列領域がNAIP遺伝子の発現促進に関係する領域であることが確認された。
【0023】さらに、この配列番号1の−765番塩基から−416番塩基までの配列の様々な欠失変異について、ルシフェラーゼ活性を測定した。結果は図5および6に示したとおりである。すなわち、5'末端が−646番塩基からのDNA断片を含むベクター0.65-pH55-15056と、5'端が−709番塩基からの断片を含むベクター0.65-pH55-14993がpEV2−SB350のように高いルシフェラーゼ活性を示したが、5'端がさらに短くなったクローンのルシフェラーゼ活性は徐々に低下した。また、5'端が−531番塩基からの断片を含むベクター(0.65-pH55-15171)と−482番塩基からの断片を含むベクター(0.65-pH55-15220)ではルシフェラーゼ活性に差は認められなかった。さらに、3'側に−482〜−416番塩基の領域を含まないDNA断片のベクター(0.65-pH55-15231、0.65-pH55-15173、0.65-pH55-15125および0.65-pH55-15070)では有意なルシフェラーゼ活性は得られなかった。これらの結果から、このNAIP遺伝子の発現促進領域は、−482〜−416番塩基までの配列領域が「プロモーター領域(またはコア・プロモーター領域)」であり、−646〜−531番塩基までの領域が「エンハンサー領域(またはエンハンサー様領域)」であることが確認された。
【0024】一方、図3および4に示したとおり、この遺伝子発現促進配列の領域(−765〜−416番塩基までの領域)より5'側の配列を含むベクター(pEV2−BB2.5、pEV2−BB1.65、pEV2−BB0.8、およびpEV2−BB0.65)は、5'側に長くなるに従ってルシフェラーゼ活性が低下した。特に、5'側に長い領域を含むベクター(pEV2−BB2.5およびpEV2−BB1.65)の場合には、プロモーター配列/エンハンサー配列が存在するにもかかわらず、ルシフェラーゼ活性はコントロール(ベクターPVB−E2)と同程度であった。このことから、これらのベクターが含む領域にサイレンサー配列が存在することが示唆された。
【0025】また、プロモーター配列領域より3'側の配列まで含むベクター(pEV2−(A)B3.2、pEV2−(A)B2.35、pEV2−(A)B1.5、pEV2−(A)B1.35、pEV2−SB0.7およびpEV2−(Ba)S0.57)の場合にも、ルシフェラーゼ活性は得られなかった。特に、5'側のサイレンサー配列領域をほとんど含まず、しかもプロモーター配列領域を含むベクター(pEV2−(A)B1.35)においても顕著なルシフェラーゼ活性が得られなかったことから、この3'側の領域にもサイレンサー配列が存在することが示唆された。
【0026】そこで、図7および8に示したように、遺伝子発現促進領域よりも5'側に長い様々な断片を含むベクターを作成し、ルシフェラーゼ活性の抑制をしらべた。その結果、−2218〜−766番塩基までの断片を含むベクター(pEV2−BB1.65)のルシフェラーゼ活性が最も低かった。また、この領域に含まれる−2245〜−1263番塩基までのDNA配列に直接プロモーター配列領域とルシフェラーゼ遺伝子を連結させたベクター1.65pKS16−1020を作成し、そのルシフェラーゼ活性を測定した。その結果、このベクターの場合にも、有意なルシフェラーゼ抑制活性が得られたことから、−2218番塩基から−1263番塩基までの配列領域がNAIP遺伝子のサイレンサー領域の一つであることが確認された。ただし、この−2218番塩基から−1263番塩基までの配列領域のさらに細かい欠失変異を作成し、それぞれのサイレンサー活性を測定した結果、この領域には顕著なコア・サイレンサー領域と認められるような配列領域は存在していなかった。むしろ、完全なサイレンサー活性を発揮するには、pEV2−BB1.65やpEV2−BB2.5におけるような−2218〜−766の領域全てが必要であることが判明した。
【0027】さらに、図9および10に示したように、3’側のサイレンサー配列についても検討した。すなわち、−421番塩基から+296番塩基までの配列領域についてさらに細かい欠失変異ベクター(0.65pX4−15333、−15418、−15500、−15601)を作成し、サイレンサー活性を測定した。その結果、−421番塩基から−202番塩基までの配列領域を含むベクター(0.65pX4−1550)が、プロモーターを持たないベクター(pGV−E2)のルシフェラーゼ活性よりも低いことから、この配列領域がNAIP遺伝子のもう一方のサイレンサー配列であることが確認された。
【0028】以上の結果か、NAIP遺伝子の発現促進領域はは、配列番号1の−765番塩基から−416番塩基までの配列領域に存在し、この領域のは、コア・プロモーター領域(−482〜−416番塩基)とエンハンサー領域(−646〜−531番塩基)が存在することが確認された。また、サイレンサー配列は、配列番号1の−2218番塩基から−766番塩基までの領域と、−421番塩基から+296(特に−202)番塩基までの領域に存在することが確認された。
【0029】
【発明の効果】以上詳しく説明したとおり、この出願によって、NAIP遺伝子のプロモーター配列およびサイレンサー配列に相当するポリヌクレオチドが提供される。
【0030】また、これらのポリヌクレオチドを利用した発明によって、NAIPの発現促進因子や発現抑制因子を特定すること、任意タンパク質を神経系組織で効率よく発現させること、さらには、SMA等のアポトーシス性疾患の診断が可能となる。
【0031】
【配列表】
SEQUENCE LISTING<110> 科学技術振興事業団(Japan Science and Technology Corporation)
<120> 遺伝子プロモーター配列とサイレンサー配列<130> NP99304-YS<170> PatentIn Ver. 2.0<210> 1<211> 3270<212> DNA<213> Homo sapiens<220><221> exon 1<222> (1)..(73)<220><221> exon 2<222> (282)..(370)<400> 1cttcttttta ccacctgtac ttcatttatg gatacgttgt ttatccatgt gtttatcccc -2841aatggacatt gggttgtttc tgattttttg gttattatta tgaataaagt tgctatgaac -2781attattgtat aaatctttgt gtgttcatgt gttttcattt ttcttgggta aatatgtagg -2721aatggaattg ctagattgta tggcaagagt atacttagct ttccatgaca ccagtgaact -2661gtttttccaa agacattgta ttaataccat tttacattcc cacaactaat gtatgagctt -2601ccagttgctc catatcctca actaacagtt gatattgtac aatacaaatg ttaactttta -2541gaatatcttc acaactttag agtagatagt gatttcttag gacacaaaaa ctatcaatca -2481taacaaaaat taaaattgga cttcatcaaa attaaaaact tctttttgaa agacaccatt -2421aagaaaatga aaagatggtt aacagactga gagaatatat gtgcaatgta aatatctaac -2361aaaggactag tatgcaaaat atataaagaa tccttataat ttagtaataa aaaggaaaac -2301acgcactttt taaatgggtc aagaatttaa ttacggccag gtgcagtggc tcacacctgt -2241aatctcagca ctttgggagg ccgaggtggg tggatcacga ggtcaggggt tcgagaccag -2181cctggctaac atggtgaaac cctgtctcta ctaaaaatac aaaaattagc tgggcgtggt -2121ggtgggcgcc tgtaatccca gctactcggg aggctgaggc aggggaatcg cttgaaccca -2061ggaggcggag gttgcagtga gctgagattg tgccactgca ctccagcctg ggcgacagag -2001cgagactctg tcttttaaaa aaaaagaaaa aaatttaatt accttaccaa agaagataca -1941tgaatgacca ataagccatt aaattaaaac ttcagtgagt atacacatat gccctgaagt -1881agctaaactt taaaacactg ccctatgtgt gtgtactttt tagctaaaaa agtgtttgaa -1821aacagtatta ttttgaaact caaaattcca aatcctctct tcaaattagg tagtagtgtc -1761actggaagta aaagagaagt tactttaact gctaaagagt atccactaag aatctacaac -1701aaacatcatg tttaatgctc aaactttaga agaatttata ccaaaagtca agaataagat -1641aggatatcta tcttcattat taagcaacat tatattagag gacctaatca atgcaacaag -1581gcaataaata aaagacaaaa ctgtcataat cagatactac aaacatctaa gaaaattcag -1521ggggatttac agtcatatat taaacagtcc agaaggagtc ctatacatca tcaatgaata -1461ttgagaataa aaaatagttt ttaaaacccc tccaatttat aagggcaaca aaatgtacaa -1401ggtactaaaa taaatgtaat gtgtatttgg gtataaatac agcaacataa tgaaaaggtt -1341tattggggaa aaaattacag ggagaagggt caacaacgtg aagaggttcc cctgccatct -1281ggacttctgg aaaaagacct gctggtctag gcacagctgg atgcaccatg gaaagtggtt -1221ccaataaaca ctggcactgt aacaattgtg tatgttgata caaaaagagg gagccaatga -1161atacttggtc gattatttaa caattgcttc tctgctgaag aaatggcctt gttggactag -1101agcttaactg tggttcacat actaatgctg ctataacagc tagaagcctt actaagaaat -1041ttgctaaacc accaaaaaga ggaaatgaac cttctaggac atctggattg ccttttctta -981aaaatctagt cttggctggg tgctgtggtt cacgcctgta atcccagcat ttggtgaggc -921cgaagtgggc ggatcacgag gtcaagagat cgagaccatc ctggtcaaca tggtgaaacc -861ctgtctctac tgaaaataca aaaattagct gggcgaggtg gcatgtgcct gtagtcccag -801ctgctgggga ggctgaggaa ggagaatcac ttgaacccgg gaggcggggt ttgcagtgag -741ctgagatcac gccattgcac tccagcctgg caacagagca agactcagtc taaaaaaaaa -681aaaaaaaaat ctagtctgta tggcagcaca acatcaggta aaagtacaga ctagccagat -621cactggttaa ccttaacccc tatgtgcttg agtttcctta tctgtaatat ggagatgata -561tagcaatagc tgattttgga ctgttaaagg aattaagtgg acacatgtaa agtgcttaga -501attgtgcctg gcaagtagta ggctgcaata ttgtgatata ataaatatat atatttgatc -441ttcatccagt tcctggcaca gatctccaga aacccttgta atttcctgag tgacaggggt -381gatagaaaca tcttttatta gaatacttgg tcttggttcc tgacacaaga gcttctaaga -321cctttggaat ctccaagtga taagagtgta tgacagtgag ctaactggtg gctgggatcc -261tttagacaac ttcaggatgg gggctatccc ctgaaagact aaggcatgat tagaggtctg -201ggatttgcag ccccacgcct cgacctccag agagggtaaa agggctggcg attgattaac -141caccaattgc cagtgattta gccaatcatg cctaagtgat ggcacctcca ttaaaaaata -81aaccacaggt ttggagagct ttcggtttgg ttaaccccaa ccacatacca agaaggcgat -21gcacctcaaa ctgcatgaag acaaaaggtc ctgtgctcac ctgggaccct tctggacgtt 40gccctgtgta cctcttcgac tgcctgttca tctgtatcct ttataataaa gcagtaaaca 100taagtaaagt ttctgagttc tgtgagccat tataagaaac gatcgaacct gggattttcc 160tttcggaagc cgctctctct cacaagggag agagctgttc tcctttttct tttgcgtgtt 220aaacctccgc tcctaaaccc actcttcgtg tgtatcgtgt ccttaacctt gttggtgcga 280gacgacgaac cccgggtatt gaccccagac aacaatgcca cttcatattg gggacttcgt 340ctgggattcc aaggtgcatt cattgcaaag 370
【図面の簡単な説明】
【図1】ルシフェラーゼ遺伝子を保有するプラスミドベクターPGV−2Eの構造を示す模式図である。
【図2】PGV−2Eにこの発明のポリヌクレオチド(プロモーター配列)を挿入した組換えベクターpEV2−350の構造を示す模式図である。
【図3】ルシフェラーゼアッセイによる発現促進領域の決定に用いた欠失変異ベクターの構成図である。
【図4】図3に示した各ベクターによるトランスフェクタントの相対的ルシフェラーゼ活性を示したグラフである。
【図5】ルシフェラーゼアッセイによる発現促進領域のさらに詳細な分析に用いた欠失変異ベクターの構成図である。
【図6】図5に示した各ベクターによるトランスフェクタントの相対的ルシフェラーゼ活性を示したグラフである。
【図7】ルシフェラーゼアッセイによる5’側サイレンサー領域の決定に用いた欠失変異ベクターの構成図である。
【図8】図7に示した各ベクターによるトランスフェクタントの相対的ルシフェラーゼ活性を示したグラフである。
【図9】ルシフェラーゼアッセイによる3’側サイレンサー領域の決定に用いた欠失変異ベクターの構成図である。
【図10】図9に示した各ベクターによるトランスフェクタントの相対的ルシフェラーゼ活性を示したグラフである。
【0001】
【発明の属する技術分野】この出願の発明は、遺伝子プロモーター配列と遺伝子サイレンサー配列に関するものである。さらに詳しくは、この出願の発明は、主に神経系組織で発現するアポトーシス抑制タンパク質NAIP(Neuronal Apoptosis Inhibitory Protein)をコードする遺伝子のプロモーター配列およびサイレンサー配列に相当するポリヌクレオチドとその利用発明に関するものである。
【0002】
【従来の技術とその課題】アポトーシス(apoptosis)は、プログラムされた細胞死の一種であり、周囲の細胞との接触の欠乏、細胞質の濃縮化、エンドヌクレアーゼの活性に関連したクロマチンの凝縮および核凝縮、核の断片化、膜被包性球状小体化、隣接するマクロファージもしくは上皮細胞などによる球状小体の貪食、またはエンドヌクレアーゼ活性によりDNAのヌクレオソーム単位が180〜200塩基長のDNAに断片化するといった現象が観察され、このような現象が認められるアポプティック体細胞の最終断片が隣接する細胞により貪食される機構として論じられている(例えば、Immunology Today 7:115-119, 1986;Science 245:301-305, 1989 )。
【0003】このアポトーシスを制御する遺伝子としてしては、例えば、1985年に胞性B細胞腫から発見されたガン遺伝子のひとつであるbcl-2遺伝子が知られている。このbcl-2遺伝子は、免疫系や神経性の細胞で高頻度に発現しており、この遺伝子の発現産物はこれら細胞のアポトーシスを抑制することによって、ヒトの免疫機能や神経系機能の恒常性を維持していると考えられている。また、このbcl-2遺伝子は、胎児では特に広範囲には発現していることから、個体発生の際の形態形成にも重要な役割を果たしていると考えられてもいる。
【0004】一方、この出願の発明者等は、家族性の遺伝病である脊髄性筋萎縮症(SpinalMascular Atropy:SMA)の原因遺伝子として、ヒト染色体5q13.1領域より神経細胞アポトーシス抑制蛋白質(Neuronal Apoptosis Inhibitory Protein : NAIP)遺伝子を単離している(Roy et al., Cell 80: 167-178, 1995)。すなわち、このNAIP遺伝子の変異またはコピー数の減少が脊髄ニューロンのアポトーシスを生じさせ、これがSMA発症の原因となると想定されている。また、このNAIP遺伝子を種々の培養細胞に導入し、アポトーシスを誘起させる刺激を細胞に与えたところ、その細胞死が抑制されることが明らかにされ(Liston et al., Nature 379:349-353, 1996 )、NAIPが神経細胞だけではなく、体細胞全体のアポトーシス抑制因子であることが明らかにされている。
【0005】そしてこの出願の発明者等は、NAIPをコードするcDNAを単離し、そのアミノ酸配列を決定して既に特許出願している(特願平9−280831号)。
【0006】
【発明が解決しようとする課題】前記のとおり、NAIPはSMAをはじめとする各種アポトーシス性疾患に関与する蛋白質であることから、NAIP遺伝子の発現に関わる分子の解明は、それら疾患の発症メカニズムの解明、発症の危険性の診断、発症の予防もしくは病態の改善、治療のための医療技術および薬剤の開発等のためにとって極めて重要である。
【0007】この出願の発明は以上のとおりの事情に鑑みてなされたものであり、NAIP遺伝子のプロモーター配列/エンハンサー配列およびサイレンサー配列を提供することを課題としている。
【0008】また、この出願の発明は、これらプロモーター配列/エンハンサー配列およびサイレンサー配列の利用発明を提供することを課題としている。
【0009】
【課題を解決するための手段】この出願は、前記の課題を解決するものとして、以下の(1)から(16)の発明を提供する。
(1) 配列番号1の−765番塩基から−416番塩基の塩基配列、またはその一部配列からなり、遺伝子発現を促進するポリヌクレオチド。
(2) 配列番号1の−482番塩基から−416番塩基の塩基配列からなり、遺伝子プロモーターとして機能するポリヌクレオチド。
(3) 配列番号1の−646番塩基から−531番塩基の塩基配列からなり、遺伝子エンハンサーとして機能するポリヌクレオチド。
(4) 配列番号1の−2218番塩基から−766番塩基の塩基配、またはその一部配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド。
(5) 配列番号1の−421番塩基から296番塩基の塩基配列、またはその一部配列からなり、遺伝子遺伝子サイレンサーとして機能するポリヌクレオチド。
(6) 配列番号1の−421番塩基から−202番塩基の塩基配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド。
(7) 前記(1)または(2)のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
(8) 前記(1)または(2)のポリヌクレオチドおよび前記(4)のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
(9) 前記(1)または(2)のポリヌクレオチドおよび前記(5)はたは(6)のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
(10) 前記(1)または(2)のポリヌクレオチド、前記(4)および(5)または(6)のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
(11) ヒト・アポトーシス抑制タンパク質NAIPの発現促進因子をスクリーニングする方法であって、前記(7)の組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量を指標としてNAIPの発現促進因子を特定することを特徴とする方法。
(12) ヒト・アポトーシス抑制タンパク質NAIPの発現抑制因子をスクリーニングする方法であって、前記(8)から(10)のいずれかの組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量を指標としてNAIPの発現抑制因子を特定することを特徴とする方法。
(13) タンパク質を動物神経組織で発現させる方法であって、前記(1)または(2)のポリヌクレオチドに連結したタンパク質遺伝子動物体内で発現させることを特徴とする方法。
(14) 細胞アポトーシス機能の失調を原因とする疾患の診断方法であって、配列番号1の塩基配列における任意配列領域の変異を検出することを特徴とする方法。
(15) 疾患が、脊髄性筋萎縮症である前記(14)の方法。
(16) 任意配列領域が配列番号1の−765番塩基から−416番塩基の塩基配列、、−2218番塩基から−766番塩基の塩基配列、−421番塩基から296番塩基の塩基配列の少なくとも一つである前記(14)または(15)の方法。
【0010】
【発明の実施の形態】前記発明(1)の遺伝子発現を促進するポリヌクレオチドは、配列番号1の−765番塩基から−416番塩基の塩基配列からなるDNA断片、またはその一部配列からなるDNA断片としてとして提供される。このポリヌクレオチドには、配列番号1の−482番塩基から−416番塩基の塩基配列からなり、遺伝子プロモーターとして機能するポリヌクレオチドと、配列番号1の−646番塩基から−531番塩基の塩基配列からなり、遺伝子エンハンサーとして機能するポリヌクレオチドが含まれている。
【0011】また、前記発明(4)の遺伝子サイレンサーとして機能するポリヌクレオチドは、配列番号1の−2218番塩基から−766番塩基の塩基配列、またはその一部配列からなるDNA断片である。さらに、発明(5)および(6)のポリヌクレオチドは、同じく遺伝子サイレンサーとして機能する配列であって、−421番塩基から296番塩基の塩基配列からなるDNA断片、および−421番塩基から−202番塩基の塩基配列からなるDNA断片、もしくはそれらの一部配列からなるDNA断片である。
【0012】このようなDNA断片は、配列番号1の当該領域の塩基配列に基づいて化学合成することができる。あるいはまた、配列番号1の各々の当該領域の配列に基づいて合成したオリゴヌクレオチドをプローブとして、ヒトゲノムDNAライブラリーからも単離できるし、またはゲノムDNA(鋳型)と合成オリゴヌクレオチド(プライマー)を用いたPCR法等によっても取得することができる。
【0013】前記発明(7)の組換えベクターは、発明(1)または(2)のポリヌクレオチド(少なくともプロモーター配列を含む遺伝子発現促進配列)の下流支配下にレポーター遺伝子を有する組換えベクターである。このような組換えベクターは、例えば、前記のポリヌクレオチドとレポーター遺伝子を連結させてDNA断片を作成し、このDNA断片を公知のベクターDNAに挿入結合させることによって構築することができる。ベクターの種類としては、大腸菌、枯草菌等の原核細胞や、酵母、昆虫細胞、哺乳動物細胞等の真核細胞を宿主とする各種のベクターを用いることができる。また、レポーター遺伝子としては、クロラムフェニコールアセチルトランスフェラーゼ(CAT)遺伝子やルシフェラーゼ遺伝子、lacZ遺伝子等を用いることができる。
【0014】前記発明(8)〜(10)の組換えベクターは、発明(1)または(2)のポリヌクレオチドと発明(4)〜(6)のポリヌクレオチド(サイレンサー配列)を少なくとも1個連結した組換えベクターである。この組換えベクターの場合には、各挿入配列の順番は、「サイレンサー配列」、「遺伝子発現促進配列」、「レポーター」遺伝子の順番であってもよく、あるいは「遺伝子発現促進配列」、「サイレンサー配列」、「レポーター遺伝子」の順番であってもよい。これらの組換えベクターについても、ベクターやレポーター遺伝子は前記発明(7)と同様のものを使用することができる。
【0015】なお、以上の発明(7)〜(10)の組換えベクターの構築においては、レポーター遺伝子を既に保有しているベクターDNAに前記の各ポリヌクレオチドを適宜な部位に挿入するようにしてもよい。
【0016】この出願の前記発明(11)および(12)の方法は、以上のとおりに構築した組換えベクターを用いて、NAIPの発現促進因子や発現抑制因子をスクリーニングする方法である。例えば、発明(11)の方法は、前記(7)の組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量(例えば、CAT活性やルシフェラーゼ活性等)を測定することによって、各候補物質のプロモーター配列への作用(転写活性)を評価する方法である。また、前記(12)の発明は、前記(8)〜(10)の組換えベクターを用いた同様の操作によって、各候補物質のサイレンサー配列への作用(転写抑制活性)を評価する方法である。このようなスクリーニング方法によって特定されたNAIP発現促進因子は、例えば、過剰なアポトーシス性細胞死を原因とする疾患(例えば、SMAやハンチントン病、パーキンソン病、アルツハイマー病、脊髄硬化症、小脳変性疾患等の神経変性疾患、および免疫不全、組織萎縮を伴う各種疾患等)の治療薬成分として有用である。また、NAIP発現抑制因子は、細胞アポトーシスの低下を原因とする疾患(例えば、奇形、自己免疫疾患、ガン等)の治療薬成分として有用である。
【0017】この出願の前記(13)の方法は、例えば、神経組織で作用させることが動物個体の神経系機能・構造の改善等に有用であるにも係わらず、脳血液関門を通過しないために神経系組織に作用させることができないタンパク質を、神経系組織で特異的に発現させる方法である。そのようなタンパク質としては、例えば、インターフェロン、インターロイキン、サイトカイン、プロテアーゼとプロテアーゼインヒビター等である。この発明(13)の方法は、例えば、前記(1)または(2)のポリヌクレオチド(少なくともプロモーター配列を含む遺伝子発現促進配列)の下流支配下に当該タンパク質遺伝子を連結したDNA断片を動物個体内に導入に、当該タンパク質を神経系組織で発現させる方法である。すなわち、発明(1)または(2)のポリヌクレオチドは、神経系組織で発現しているNAIP遺伝子の発現促進配列(プロモーター配列および/またはエンハンサー配列を含む)であり、このポリヌクレオチドの支配によって下流タンパク質遺伝子は神経系組織で優位に発現するようになる。ポリヌクレオチドとタンパク質遺伝子とのDNA断片を動物個体内に導入する方法としては、例えば、遺伝子治療等において用いられているウイルスベクターを使用する方法等を用いることができる。また、動物個体が非ヒト動物の場合には、公知のトランスジェニック動物作成方法等を用いることもできる。
【0018】この出願の前記(14)の方法は、配列番号1に塩基配列を示したNAIP遺伝子5’側配列のいずれかの変異(例えば、配列番号1における1もしくは複数の塩基の欠失、付加、または他の塩基への置換等)を検出することによって、細胞アポトーシス機能の失調を原因とする疾患を診断する方法である。すなわち、NAIP遺伝子の変異がSMAの発症原因の一と推察されている(Roy et al., Cell80:167-178, 1995; Chen et al., Genomics 15:121-127, 1998)、SMA患者におけるNAIP遺伝子変異は、最も症状の重いSMAI型で約60%であり、症状が軽くなるに従って(SMAII〜III型)、NAIP遺伝子欠失変異の出現頻度が低下することが知られている(Roy et al., Cell 80:167-178, 1995; J. Neuro Sci 158(1):43-46, 1998; Arch Dis Child 78(6):531-535, 1998)。このことは、SMAのようなアポトーシス性疾患の原因が、遺伝子そのものの変異だけにあるのではなく、その原因遺伝子の発現を調整する領域の変異にも存在することを示唆する。特にNAIP遺伝子非欠損性のSMA(I型は約40%、II型は約70%、III型は約100%)は、アポトーシス性神経細胞死の抑制メカニズムの失調、すなわちNAIPの発現抑制が発症原因と考えられることから、プロモーター配列の変異がNAIP遺伝子発現を低下させ、結果として脱抑制的な神経細胞アポトーシスがSMAの原因となっていると考えられる。従って、SMA等のアポトーシス性疾患のハイリスク者や、そのような疾患の初期症状を呈する患者の生体試料からDNAを抽出し、配列番号1の−765番塩基から−416番塩基までの塩基配列、−2218番塩基から−766番塩基の塩基配列、−421番塩基から296番塩基の塩基配列に相当するDNA配列を検査することによって、その後の症状の発生や進行を予測することが可能となり、さらには、より適切な治療方法等の選択が可能となる。
【0019】以下、前記発明(1)から(3)のポリヌクレオチド(プロモーター配列/エンハンサー配列)、ならびに前記(4)〜(6)のポリヌクレオチド(サイレンサー配列)を決定した方法について詳しく説明する。
1.ルシフェラーゼアッセイのためのベクター構築配列番号1に塩基配列を示したNAIP遺伝子転写開始領域の欠失変異断片を表1のとおりに作成した。変異断片は、制限酵素による切断、または制限酵素認識部位を含むオリゴヌクレオチドをプライマーとするPCR法により作成した。次いで、両端を制限酵素で調整した各変異断片を、ルシフェラーゼ遺伝子を含むプラスミドベクターPGV−E2(Toyo Ink社製:図1)にサブクローニングした。また、サイレンサーアッセイ用のベクターは、NAIP遺伝子のプロモーター配列の全てとルシフェラーゼ遺伝子を含むベクターpEV2−350に各変異断片をサブクローニングして作成した(図2)。
【0020】
【表1】

【0021】2.ルシフェラーゼアッセイの方法2×10-5個のC33A細胞およびHeLa細胞を35mの培養皿で培養した。C33A細胞は、10%の不活性化ウシ胎児血清(FCS)含有のDulbecco's modifiedMEM low glucose培地で培養した。また、HeLa細胞は10%FCS含有のMEM培地で培養した。次いで、これらの培養細胞に、LipofectAMINE PLUS(Life Technologies社製)を用いて各ベクターをトランスフェクトした。すなわち、各々のプラスミド(C33A細胞に対して1.0μg:HeLa細胞に対して1.5μg)とコントロールベクターpEF−lacZ(0.1μg)を、50μlのOPTI-MEMI製血清培地(Life Technologies社製)と6μlのPLUS試薬を含む同培地50μlとの混合培地に加え、室温で15分間インキュベートした後、4μlのLipofectAMINEを含む同培地100μlと混合し、室温で15分間インキュベートした。次いで、0.8mlのOPTI-MEMI製血清培地を細胞が浸るまで加え、37℃で15時間インキュベートした後、2mlの完全増殖培地を加え、37℃でさらに15時間インキュベートした。培地を2mlの完全増殖培地に代え、37℃でさらに30時間インキュベートした。次に、培養細胞をPicaGene細胞溶解バッファー(Toyo Ink社製)により溶解し、全タンパク質量と、細胞溶解物中のβ−ガラクトシダーゼ発現量を、それぞれタンパク質定量装置(Bio-Rad社製)およびβ−ガラクトシダーゼ定量装置(Promega社製)により測定した。また、細胞溶解物中のルシフェラーゼ活性は、シングルフォトン計測装置Argus-50/CL(Hamamatsu Photonics社製)により測定した。
3.結果結果は図3から図10に示したとおりである。
【0022】図3および4に示したとおり、配列番号1の−765番塩基から−416番塩基までの配列にルシフェラーゼ遺伝子を連結させたベクター(pEV2−SB350)が最も高いルシフェラーゼ活性を示したことから、この配列領域がNAIP遺伝子の発現促進に関係する領域であることが確認された。
【0023】さらに、この配列番号1の−765番塩基から−416番塩基までの配列の様々な欠失変異について、ルシフェラーゼ活性を測定した。結果は図5および6に示したとおりである。すなわち、5'末端が−646番塩基からのDNA断片を含むベクター0.65-pH55-15056と、5'端が−709番塩基からの断片を含むベクター0.65-pH55-14993がpEV2−SB350のように高いルシフェラーゼ活性を示したが、5'端がさらに短くなったクローンのルシフェラーゼ活性は徐々に低下した。また、5'端が−531番塩基からの断片を含むベクター(0.65-pH55-15171)と−482番塩基からの断片を含むベクター(0.65-pH55-15220)ではルシフェラーゼ活性に差は認められなかった。さらに、3'側に−482〜−416番塩基の領域を含まないDNA断片のベクター(0.65-pH55-15231、0.65-pH55-15173、0.65-pH55-15125および0.65-pH55-15070)では有意なルシフェラーゼ活性は得られなかった。これらの結果から、このNAIP遺伝子の発現促進領域は、−482〜−416番塩基までの配列領域が「プロモーター領域(またはコア・プロモーター領域)」であり、−646〜−531番塩基までの領域が「エンハンサー領域(またはエンハンサー様領域)」であることが確認された。
【0024】一方、図3および4に示したとおり、この遺伝子発現促進配列の領域(−765〜−416番塩基までの領域)より5'側の配列を含むベクター(pEV2−BB2.5、pEV2−BB1.65、pEV2−BB0.8、およびpEV2−BB0.65)は、5'側に長くなるに従ってルシフェラーゼ活性が低下した。特に、5'側に長い領域を含むベクター(pEV2−BB2.5およびpEV2−BB1.65)の場合には、プロモーター配列/エンハンサー配列が存在するにもかかわらず、ルシフェラーゼ活性はコントロール(ベクターPVB−E2)と同程度であった。このことから、これらのベクターが含む領域にサイレンサー配列が存在することが示唆された。
【0025】また、プロモーター配列領域より3'側の配列まで含むベクター(pEV2−(A)B3.2、pEV2−(A)B2.35、pEV2−(A)B1.5、pEV2−(A)B1.35、pEV2−SB0.7およびpEV2−(Ba)S0.57)の場合にも、ルシフェラーゼ活性は得られなかった。特に、5'側のサイレンサー配列領域をほとんど含まず、しかもプロモーター配列領域を含むベクター(pEV2−(A)B1.35)においても顕著なルシフェラーゼ活性が得られなかったことから、この3'側の領域にもサイレンサー配列が存在することが示唆された。
【0026】そこで、図7および8に示したように、遺伝子発現促進領域よりも5'側に長い様々な断片を含むベクターを作成し、ルシフェラーゼ活性の抑制をしらべた。その結果、−2218〜−766番塩基までの断片を含むベクター(pEV2−BB1.65)のルシフェラーゼ活性が最も低かった。また、この領域に含まれる−2245〜−1263番塩基までのDNA配列に直接プロモーター配列領域とルシフェラーゼ遺伝子を連結させたベクター1.65pKS16−1020を作成し、そのルシフェラーゼ活性を測定した。その結果、このベクターの場合にも、有意なルシフェラーゼ抑制活性が得られたことから、−2218番塩基から−1263番塩基までの配列領域がNAIP遺伝子のサイレンサー領域の一つであることが確認された。ただし、この−2218番塩基から−1263番塩基までの配列領域のさらに細かい欠失変異を作成し、それぞれのサイレンサー活性を測定した結果、この領域には顕著なコア・サイレンサー領域と認められるような配列領域は存在していなかった。むしろ、完全なサイレンサー活性を発揮するには、pEV2−BB1.65やpEV2−BB2.5におけるような−2218〜−766の領域全てが必要であることが判明した。
【0027】さらに、図9および10に示したように、3’側のサイレンサー配列についても検討した。すなわち、−421番塩基から+296番塩基までの配列領域についてさらに細かい欠失変異ベクター(0.65pX4−15333、−15418、−15500、−15601)を作成し、サイレンサー活性を測定した。その結果、−421番塩基から−202番塩基までの配列領域を含むベクター(0.65pX4−1550)が、プロモーターを持たないベクター(pGV−E2)のルシフェラーゼ活性よりも低いことから、この配列領域がNAIP遺伝子のもう一方のサイレンサー配列であることが確認された。
【0028】以上の結果か、NAIP遺伝子の発現促進領域はは、配列番号1の−765番塩基から−416番塩基までの配列領域に存在し、この領域のは、コア・プロモーター領域(−482〜−416番塩基)とエンハンサー領域(−646〜−531番塩基)が存在することが確認された。また、サイレンサー配列は、配列番号1の−2218番塩基から−766番塩基までの領域と、−421番塩基から+296(特に−202)番塩基までの領域に存在することが確認された。
【0029】
【発明の効果】以上詳しく説明したとおり、この出願によって、NAIP遺伝子のプロモーター配列およびサイレンサー配列に相当するポリヌクレオチドが提供される。
【0030】また、これらのポリヌクレオチドを利用した発明によって、NAIPの発現促進因子や発現抑制因子を特定すること、任意タンパク質を神経系組織で効率よく発現させること、さらには、SMA等のアポトーシス性疾患の診断が可能となる。
【0031】
【配列表】
SEQUENCE LISTING<110> 科学技術振興事業団(Japan Science and Technology Corporation)
<120> 遺伝子プロモーター配列とサイレンサー配列<130> NP99304-YS<170> PatentIn Ver. 2.0<210> 1<211> 3270<212> DNA<213> Homo sapiens<220><221> exon 1<222> (1)..(73)<220><221> exon 2<222> (282)..(370)<400> 1cttcttttta ccacctgtac ttcatttatg gatacgttgt ttatccatgt gtttatcccc -2841aatggacatt gggttgtttc tgattttttg gttattatta tgaataaagt tgctatgaac -2781attattgtat aaatctttgt gtgttcatgt gttttcattt ttcttgggta aatatgtagg -2721aatggaattg ctagattgta tggcaagagt atacttagct ttccatgaca ccagtgaact -2661gtttttccaa agacattgta ttaataccat tttacattcc cacaactaat gtatgagctt -2601ccagttgctc catatcctca actaacagtt gatattgtac aatacaaatg ttaactttta -2541gaatatcttc acaactttag agtagatagt gatttcttag gacacaaaaa ctatcaatca -2481taacaaaaat taaaattgga cttcatcaaa attaaaaact tctttttgaa agacaccatt -2421aagaaaatga aaagatggtt aacagactga gagaatatat gtgcaatgta aatatctaac -2361aaaggactag tatgcaaaat atataaagaa tccttataat ttagtaataa aaaggaaaac -2301acgcactttt taaatgggtc aagaatttaa ttacggccag gtgcagtggc tcacacctgt -2241aatctcagca ctttgggagg ccgaggtggg tggatcacga ggtcaggggt tcgagaccag -2181cctggctaac atggtgaaac cctgtctcta ctaaaaatac aaaaattagc tgggcgtggt -2121ggtgggcgcc tgtaatccca gctactcggg aggctgaggc aggggaatcg cttgaaccca -2061ggaggcggag gttgcagtga gctgagattg tgccactgca ctccagcctg ggcgacagag -2001cgagactctg tcttttaaaa aaaaagaaaa aaatttaatt accttaccaa agaagataca -1941tgaatgacca ataagccatt aaattaaaac ttcagtgagt atacacatat gccctgaagt -1881agctaaactt taaaacactg ccctatgtgt gtgtactttt tagctaaaaa agtgtttgaa -1821aacagtatta ttttgaaact caaaattcca aatcctctct tcaaattagg tagtagtgtc -1761actggaagta aaagagaagt tactttaact gctaaagagt atccactaag aatctacaac -1701aaacatcatg tttaatgctc aaactttaga agaatttata ccaaaagtca agaataagat -1641aggatatcta tcttcattat taagcaacat tatattagag gacctaatca atgcaacaag -1581gcaataaata aaagacaaaa ctgtcataat cagatactac aaacatctaa gaaaattcag -1521ggggatttac agtcatatat taaacagtcc agaaggagtc ctatacatca tcaatgaata -1461ttgagaataa aaaatagttt ttaaaacccc tccaatttat aagggcaaca aaatgtacaa -1401ggtactaaaa taaatgtaat gtgtatttgg gtataaatac agcaacataa tgaaaaggtt -1341tattggggaa aaaattacag ggagaagggt caacaacgtg aagaggttcc cctgccatct -1281ggacttctgg aaaaagacct gctggtctag gcacagctgg atgcaccatg gaaagtggtt -1221ccaataaaca ctggcactgt aacaattgtg tatgttgata caaaaagagg gagccaatga -1161atacttggtc gattatttaa caattgcttc tctgctgaag aaatggcctt gttggactag -1101agcttaactg tggttcacat actaatgctg ctataacagc tagaagcctt actaagaaat -1041ttgctaaacc accaaaaaga ggaaatgaac cttctaggac atctggattg ccttttctta -981aaaatctagt cttggctggg tgctgtggtt cacgcctgta atcccagcat ttggtgaggc -921cgaagtgggc ggatcacgag gtcaagagat cgagaccatc ctggtcaaca tggtgaaacc -861ctgtctctac tgaaaataca aaaattagct gggcgaggtg gcatgtgcct gtagtcccag -801ctgctgggga ggctgaggaa ggagaatcac ttgaacccgg gaggcggggt ttgcagtgag -741ctgagatcac gccattgcac tccagcctgg caacagagca agactcagtc taaaaaaaaa -681aaaaaaaaat ctagtctgta tggcagcaca acatcaggta aaagtacaga ctagccagat -621cactggttaa ccttaacccc tatgtgcttg agtttcctta tctgtaatat ggagatgata -561tagcaatagc tgattttgga ctgttaaagg aattaagtgg acacatgtaa agtgcttaga -501attgtgcctg gcaagtagta ggctgcaata ttgtgatata ataaatatat atatttgatc -441ttcatccagt tcctggcaca gatctccaga aacccttgta atttcctgag tgacaggggt -381gatagaaaca tcttttatta gaatacttgg tcttggttcc tgacacaaga gcttctaaga -321cctttggaat ctccaagtga taagagtgta tgacagtgag ctaactggtg gctgggatcc -261tttagacaac ttcaggatgg gggctatccc ctgaaagact aaggcatgat tagaggtctg -201ggatttgcag ccccacgcct cgacctccag agagggtaaa agggctggcg attgattaac -141caccaattgc cagtgattta gccaatcatg cctaagtgat ggcacctcca ttaaaaaata -81aaccacaggt ttggagagct ttcggtttgg ttaaccccaa ccacatacca agaaggcgat -21gcacctcaaa ctgcatgaag acaaaaggtc ctgtgctcac ctgggaccct tctggacgtt 40gccctgtgta cctcttcgac tgcctgttca tctgtatcct ttataataaa gcagtaaaca 100taagtaaagt ttctgagttc tgtgagccat tataagaaac gatcgaacct gggattttcc 160tttcggaagc cgctctctct cacaagggag agagctgttc tcctttttct tttgcgtgtt 220aaacctccgc tcctaaaccc actcttcgtg tgtatcgtgt ccttaacctt gttggtgcga 280gacgacgaac cccgggtatt gaccccagac aacaatgcca cttcatattg gggacttcgt 340ctgggattcc aaggtgcatt cattgcaaag 370
【図面の簡単な説明】
【図1】ルシフェラーゼ遺伝子を保有するプラスミドベクターPGV−2Eの構造を示す模式図である。
【図2】PGV−2Eにこの発明のポリヌクレオチド(プロモーター配列)を挿入した組換えベクターpEV2−350の構造を示す模式図である。
【図3】ルシフェラーゼアッセイによる発現促進領域の決定に用いた欠失変異ベクターの構成図である。
【図4】図3に示した各ベクターによるトランスフェクタントの相対的ルシフェラーゼ活性を示したグラフである。
【図5】ルシフェラーゼアッセイによる発現促進領域のさらに詳細な分析に用いた欠失変異ベクターの構成図である。
【図6】図5に示した各ベクターによるトランスフェクタントの相対的ルシフェラーゼ活性を示したグラフである。
【図7】ルシフェラーゼアッセイによる5’側サイレンサー領域の決定に用いた欠失変異ベクターの構成図である。
【図8】図7に示した各ベクターによるトランスフェクタントの相対的ルシフェラーゼ活性を示したグラフである。
【図9】ルシフェラーゼアッセイによる3’側サイレンサー領域の決定に用いた欠失変異ベクターの構成図である。
【図10】図9に示した各ベクターによるトランスフェクタントの相対的ルシフェラーゼ活性を示したグラフである。
【特許請求の範囲】
【請求項1】 配列番号1の−765番塩基から−416番塩基の塩基配列、またはその一部配列からなり、遺伝子発現を促進するポリヌクレオチド。
【請求項2】 配列番号1の−482番塩基から−416番塩基の塩基配列からなり、遺伝子プロモーターとして機能するポリヌクレオチド。
【請求項3】 配列番号1の−646番塩基から−531番塩基の塩基配列からなり、遺伝子エンハンサーとして機能するポリヌクレオチド。
【請求項4】 配列番号1の−2218番塩基から−766番塩基の塩基配、またはその一部配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド。
【請求項5】 配列番号1の−421番塩基から296番塩基の塩基配列、またはその一部配列からなり、遺伝子遺伝子サイレンサーとして機能するポリヌクレオチド。
【請求項6】 配列番号1の−421番塩基から−202番塩基の塩基配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド。
【請求項7】 請求項1または2のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
【請求項8】 請求項1または2のポリヌクレオチドおよび請求項4のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
【請求項9】 請求項1または2のポリヌクレオチドおよび請求項5または6のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
【請求項10】 請求項1または2のポリヌクレオチド、請求項4および5または6のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
【請求項11】 ヒト・アポトーシス抑制タンパク質NAIPの発現促進因子をスクリーニングする方法であって、請求項7の組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量を指標としてNAIPの発現促進因子を特定することを特徴とする方法。
【請求項12】 ヒト・アポトーシス抑制タンパク質NAIPの発現抑制因子をスクリーニングする方法であって、請求項8から10のいずれかの組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量を指標としてNAIPの発現抑制因子を特定することを特徴とする方法。
【請求項13】 タンパク質を動物神経組織で発現させる方法であって、請求項1または2のポリヌクレオチドに連結したタンパク質遺伝子動物体内で発現させることを特徴とする方法。
【請求項14】 細胞アポトーシス機能の失調を原因とする疾患の診断方法であって、配列番号1の塩基配列における任意配列領域の変異を検出することを特徴とする方法。
【請求項15】 疾患が、脊髄性筋萎縮症である請求項14の方法。
【請求項16】 任意配列領域が配列番号1の−765番塩基から−416番塩基の塩基配列、−2218番塩基から−766番塩基の塩基配列、−421番塩基から296番塩基の塩基配列の少なくとも一つである請求項14または15の方法。
【請求項1】 配列番号1の−765番塩基から−416番塩基の塩基配列、またはその一部配列からなり、遺伝子発現を促進するポリヌクレオチド。
【請求項2】 配列番号1の−482番塩基から−416番塩基の塩基配列からなり、遺伝子プロモーターとして機能するポリヌクレオチド。
【請求項3】 配列番号1の−646番塩基から−531番塩基の塩基配列からなり、遺伝子エンハンサーとして機能するポリヌクレオチド。
【請求項4】 配列番号1の−2218番塩基から−766番塩基の塩基配、またはその一部配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド。
【請求項5】 配列番号1の−421番塩基から296番塩基の塩基配列、またはその一部配列からなり、遺伝子遺伝子サイレンサーとして機能するポリヌクレオチド。
【請求項6】 配列番号1の−421番塩基から−202番塩基の塩基配列からなり、遺伝子サイレンサーとして機能するポリヌクレオチド。
【請求項7】 請求項1または2のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
【請求項8】 請求項1または2のポリヌクレオチドおよび請求項4のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
【請求項9】 請求項1または2のポリヌクレオチドおよび請求項5または6のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
【請求項10】 請求項1または2のポリヌクレオチド、請求項4および5または6のポリヌクレオチドを連結したレポーター遺伝子を有する組換えベクター。
【請求項11】 ヒト・アポトーシス抑制タンパク質NAIPの発現促進因子をスクリーニングする方法であって、請求項7の組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量を指標としてNAIPの発現促進因子を特定することを特徴とする方法。
【請求項12】 ヒト・アポトーシス抑制タンパク質NAIPの発現抑制因子をスクリーニングする方法であって、請求項8から10のいずれかの組換えベクターをトランスフェクトした細胞の培養培地に候補物質を添加し、レポーター遺伝子の発現量を指標としてNAIPの発現抑制因子を特定することを特徴とする方法。
【請求項13】 タンパク質を動物神経組織で発現させる方法であって、請求項1または2のポリヌクレオチドに連結したタンパク質遺伝子動物体内で発現させることを特徴とする方法。
【請求項14】 細胞アポトーシス機能の失調を原因とする疾患の診断方法であって、配列番号1の塩基配列における任意配列領域の変異を検出することを特徴とする方法。
【請求項15】 疾患が、脊髄性筋萎縮症である請求項14の方法。
【請求項16】 任意配列領域が配列番号1の−765番塩基から−416番塩基の塩基配列、−2218番塩基から−766番塩基の塩基配列、−421番塩基から296番塩基の塩基配列の少なくとも一つである請求項14または15の方法。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図10】


【図9】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図10】


【図9】


【公開番号】特開2001−46064(P2001−46064A)
【公開日】平成13年2月20日(2001.2.20)
【国際特許分類】
【出願番号】特願平11−220394
【出願日】平成11年8月3日(1999.8.3)
【出願人】(396020800)科学技術振興事業団 (35)
【Fターム(参考)】
【公開日】平成13年2月20日(2001.2.20)
【国際特許分類】
【出願日】平成11年8月3日(1999.8.3)
【出願人】(396020800)科学技術振興事業団 (35)
【Fターム(参考)】
[ Back to top ]

