遺伝子絞り込み装置、遺伝子絞り込み方法、及びコンピュータプログラム
【課題】 ある特定の機能や関連性を持った候補遺伝子の中から、より可能性が高い遺伝子を絞り込むことができる遺伝子絞り込み装置、遺伝子絞り込み方法、及びプログラムを提供する。
【解決手段】 本発明の遺伝子絞り込み装置は、記憶手段と、入力手段と、処理手段とを備え、記憶手段は、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスを記憶し、入力手段は、複数の候補遺伝子又は候補タンパク質が情報として入力され、処理手段は、(a)アノテーションを収集する収集処理と、(b)付与の頻度又は数が閾値より高いアノテーションを選択する第1選択処理と、(c)遺伝子を選択する第2選択処理と、を実行する。
【解決手段】 本発明の遺伝子絞り込み装置は、記憶手段と、入力手段と、処理手段とを備え、記憶手段は、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスを記憶し、入力手段は、複数の候補遺伝子又は候補タンパク質が情報として入力され、処理手段は、(a)アノテーションを収集する収集処理と、(b)付与の頻度又は数が閾値より高いアノテーションを選択する第1選択処理と、(c)遺伝子を選択する第2選択処理と、を実行する。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、複数の候補遺伝子から任意の関連性を有する遺伝子を絞り込む遺伝子絞り込み装置、遺伝子絞り込み方法、及びコンピュータプログラムに関する。
【背景技術】
【0002】
近年の分子生物学の発展により、遺伝子やタンパク質についての知見が蓄積している。これらの知見は、遺伝子に関する情報として、NCBI(National Center for Biotechnology
Information)Entrez Geneや、DDBJ(DNA Data Bank of Japan)等、さまざまな公的データベースにより公開されている。
【0003】
これらのデータベースから得られる情報を利用して、遺伝子や遺伝子がコードするタンパク質の機能を予測する技術が開発されている。例えば、アミノ酸配列情報や塩基配列情報を用いてGタンパク共役型受容体を判別する方法(特許文献1及び特許文献2参照)、タンパク質の立体構造情報や、タンパク質に作用する薬剤/化合物の情報等から選択された属性を特徴ベクトルとした教師付き機械学習によりタンパク質相互作用を予測する方法(特許文献3参照)等が挙げられる。
【先行技術文献】
【特許文献】
【0004】
【特許文献1】特開2006−003970号公報
【特許文献2】特開2002−112793号公報
【特許文献3】特開2010−165230号公報
【発明の概要】
【発明が解決しようとする課題】
【0005】
マイクロアレイ等の手法により、特定の機能や関連性を持った候補遺伝子を網羅的に取得することができるようになった。しかし、取得する候補遺伝子の数は大量であり、得られた候補遺伝子の中から、より可能性が高い遺伝子を絞り込むことが必要であった。しかし、公的データベースを利用して容易に遺伝子を絞り込む技術は確立されていなかった。
【0006】
また、従来のデータベースを用いたタンパク質機能予測は、上記の特許文献3にみられるように教師付き学習を伴うものであった。教師付き学習は正例及び負例を必要とするため、正例及び負例が得られない場合には従来の予測法を利用することができなかった。
【課題を解決するための手段】
【0007】
本発明は、複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み装置であり、記憶手段と、入力手段と、処理手段とを備え、記憶手段は、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスを記憶し、入力手段は、複数の候補遺伝子又は候補タンパク質が情報として入力され、処理手段は、(a)記憶手段に記憶されたデータウェアハウスにおいて、入力手段から入力された候補遺伝子又は候補タンパク質に付与されたアノテーションを収集する収集処理と、(b)収集処理で収集されたアノテーションの中から、入力手段から得られた候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択処理と、(c)入力手段から得られた候補遺伝子又は候補タンパク質の中から、選択処理で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択処理と、を実行する。
【0008】
また、本発明の別の遺伝子絞り込み装置では、第1選択処理(b)における、付与の頻度又は数が閾値より高いアノテーションとは、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与されたアノテーションの頻度又は数が、複数の対照遺伝子又は対照タンパク質における該アノテーションの付与の頻度又は数と比較して有意に多いアノテーションをいう。すなわち、第1選択処理(b)は、収集処理で収集されたアノテーションの中から、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与された頻度又は数が、複数の対照遺伝子又は対照タンパク質に付与された頻度又は数と比較して有意に多いアノテーションを1又は複数選択する第1選択処理である。
【0009】
また、本発明の別の遺伝子絞り込み装置は、第1選択処理(b)が、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択する処理である。候補遺伝子群と対照遺伝子群とを比較して、付与数の差が大きいアノテーションを選択することで、遺伝子絞り込みの精度を上げることができる。
【0010】
また、本発明の別の遺伝子絞り込み装置は、データウェアハウスに格納された生物学的情報についてのアノテーションの冗長性を取り除く処理を実行する。また、データウェアハウスに格納された生物学的情報についてのアノテーションの記述の形式を変換する処理を実行する。これらの処理によって、アノテーションの重複を防ぎ、遺伝子又はタンパク質の選択の正確性の向上に寄与することができる。
【0011】
また、本発明の別の遺伝子絞り込み装置は、処理手段が、(a)〜(c)の各処理に加えて、記憶手段に記憶されたデータウェアハウスから、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として入力手段に追加して入力する再入力処理を実行する。相互作用するタンパク質群は同一の機能的役割を果たすことが多く、候補遺伝子又は候補タンパク質の数を増やすことで、遺伝子絞り込みの精度を上げることができる。
【0012】
また、本発明は、コンピュータを用いて複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み方法であり、コンピュータは、入力手段と、処理手段とを備え、(1)入力手段に、複数の候補遺伝子又は候補タンパク質を情報として入力する入力工程と、(2)複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続する接続工程と、(3)処理手段が、記憶手段に記憶されたデータウェアハウスにから、入力工程で入力された候補遺伝子又は候補タンパク質に付与されたアノテーションを収集する収集工程と、(4)処理手段が、収集工程で収集されたアノテーションの中から、入力工程で入力された候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択工程と、(5)処理手段が、前記入力工程で入力された候補遺伝子又は候補タンパク質の中から、前記第1選択工程で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択工程と、を含むことを特徴とする。
【0013】
また、本発明の別の遺伝子絞り込み方法では、第1選択工程(4)において、付与の頻度又は数が閾値より高いアノテーションとは、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与されたアノテーションの頻度又は数が、複数の対照遺伝子又は対照タンパク質における該アノテーションの付与の頻度又は数と比較して有意に多いアノテーションをいう。すなわち、第1選択工程(4)は、収集処理で収集されたアノテーションの中から、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与された頻度又は数が、複数の対照遺伝子又は対照タンパク質に付与された頻度又は数と比較して有意に多いアノテーションを1又は複数選択する第1選択工程(4)である。
【0014】
また、本発明の別の遺伝子絞り込み方法は、データウェアハウスに格納された生物学的情報についてのアノテーションの冗長性を取り除く工程を含む。また、データウェアハウスに格納された生物学的情報についてのアノテーションの記述の形式を変換する工程を含む。これらの工程によって、アノテーションの重複を防ぎ、遺伝子又はタンパク質の選択の正確性の向上に寄与することができる。
【0015】
また、本発明の別の遺伝子絞り込み方法は、第1選択工程(4)が、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択する処理である。また、本発明の別の遺伝絞り込み方法は、処理手段が、記憶手段に記憶されたデータウェアハウスから、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として入力手段に追加して入力する再入力工程を実行する。
【0016】
さらに、本発明は、コンピュータに複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込ませるコンピュータプログラムであり、コンピュータに、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続させる手順と、データウェアハウスから候補遺伝子又は候補タンパク質に付与されたアノテーションを収集させる手順と、コンピュータに、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択させる手順と、コンピュータに、候補遺伝子又は候補タンパク質の中から、選択されたアノテーションが付与された遺伝子又はタンパク質を選択させる手順と、を実行させることを特徴とする。
【0017】
また、本発明の別のコンピュータプログラムでは、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択させる手順において、付与の頻度又は数が閾値より高いアノテーションとは、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与されたアノテーションの頻度又は数が、複数の対照遺伝子又は対照タンパク質における該アノテーションの付与の頻度又は数と比較して有意に多いアノテーションをいう。すなわち、本発明の別のコンピュータプログラムでは、コンピュータに、収集されたアノテーションの中から、複数の候補遺伝子又は候補タンパク質に付与された頻度又は数が、複数の対照遺伝子又は対照タンパク質に付与された頻度又は数と比較して有意に多いアノテーションを1又は複数選択させる手順を実行させる。
【0018】
また、本発明の別のコンピュータプログラムは、コンピュータに、データウェアハウスに格納された生物学的情報についてのアノテーションの冗長性を取り除く手順を実行させる。また、データウェアハウスに格納された生物学的情報についてのアノテーションの記述の形式を変換する手順を実行させる。これらの工程によって、アノテーションの重複を防ぎ、遺伝子又はタンパク質の選択の正確性の向上に寄与することができる。
【0019】
また、本発明の別のコンピュータプログラムは、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質に有意に多く付与されたアノテーションを1又は複数選択させる手順が、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択させる手順である。また、本発明の別のコンピュータプログラムは、コンピュータに、データウェアハウスから、入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として追加して入力させる手順を実行させる。
【0020】
また、本発明のデータウェアハウスに格納される複数の生物学的情報は、遺伝子情報、遺伝子相同性情報、遺伝子多型情報、タンパク質情報、タンパク質間相互作用情報、タンパク質の生物学的機能情報、タンパク質ドメイン情報、タンパク質の立体構造情報、タンパク質発現情報、酵素機能情報、パスウェイ情報、転写因子情報、疾患及び該疾患の原因遺伝子情報、薬剤情報、並びに化合物情報、からなる群から選択される複数の情報である。
【0021】
さらに、本発明の遺伝子絞り込み装置で絞り込む遺伝子の関連性は、疾患の発生についての関連性であり得る。
【発明の効果】
【0022】
本発明は、候補遺伝子から、疾患の発生等の任意の関連性を有する遺伝子を絞り込む装置である。本発明によれば、マイクロアレイ等の網羅的な解析結果から、より可能性が高い遺伝子を容易に絞り込むことができる。本発明では、教師付き学習を伴わなくとも絞り込みが可能なことから、正例及び負例を必要とせず、従来法よりも簡便に絞り込むことができる。
【図面の簡単な説明】
【0023】
【図1】本発明の遺伝子絞り込み装置の一態様である。
【図2】本発明を用いた絞り込みテストの感度及び特異度を示した図である。
【図3】本発明を用いて絞り込まれた遺伝子がC型肝炎の発症に関連性を有するかどうかの実験的検証の結果を示す図である。
【図4】本発明を用いて絞り込まれた遺伝子がC型肝炎の発症に関連性を有するかどうかの実験的検証の結果を示す図である。
【図5】本発明の実施例における各工程、及び各工程におけるデータ配列を示すフローチャート及びデータ構造の概略図である。
【発明を実施するための形態】
【0024】
以下、発明を実施するための形態に従って本発明を詳細に説明するが、本発明はこれらの実施形態に限定されない。
【0025】
本発明では、遺伝子に関する複数の情報を格納したデータウェアハウスを用いて、複数の遺伝子からなる遺伝子群から、任意の関連性を有する遺伝子の候補を絞り込むことができる。任意の関連性とは、遺伝子の持つ機能や性質についての関連性をいう。例えば、本発明によれば、ある疾患の原因となる候補遺伝子群から、より可能性の高い遺伝子を絞り込むことができる。
【0026】
ここで、タンパク質は、遺伝子によってアミノ酸配列が決定され、タンパク質の構造や機能も決定される。したがって、本発明において絞り込みの対象はタンパク質も含む。したがって、本発明は、複数のタンパク質からなる候補タンパク質群から、任意の関連性を有するタンパク質の候補を絞り込むことができる。また、遺伝子情報やタンパク質情報を用いて、遺伝子とタンパク質との情報の相互変換が可能である。
【0027】
本発明では、記憶手段に記憶されたデータウェアハウスを用いて上記の絞り込みを行う。データウェアハウスとは、複数種の情報を格納し、目的に応じて格納された情報から必要な情報を引き出すことが可能なシステムをいう。
【0028】
本発明において用いられるデータウェアハウスには、複数種の生物学的情報、すなわち、遺伝子情報、遺伝子相同性情報、遺伝子多型情報、タンパク質情報、タンパク質間相互作用情報、タンパク質の生物学的機能情報、タンパク質ドメイン情報、タンパク質の立体構造情報、タンパク質発現情報、酵素機能情報、パスウェイ情報、転写因子情報、疾患及び該疾患の原因遺伝子情報、薬剤情報、並びに化合物情報、からなる群から選択される複数を含む情報が格納される。本発明においては、生物学的情報はインターネット経由でデータウェアハウスに格納することがでるため、記憶手段に生物学的情報が記憶されていなくてもよい。
【0029】
上記の生物学的情報の格納には公的データベースを利用することができる。公的データベースとしては、例えば、NCBI(National Center for Biotechnology
Information) Entrez Gene、UCSC (University
of California Santa Cruz) database、DDBJ(DNA Data Bank of Japan)、GeMDBJ、dbSNP、Ensembl、UniProtKB、InterPro、SIFTS、SCOP(Structural Classification of Proteins)、PDB(Protein Data Bank)、PPIview、BioGRID、KEEG(Kyoto
Encyclopedia of Genes and Genomes)、The Gene Ontology、UniProtKB-GOA、OregAnno(The Open Regulatory Annotation database)、AMADEUS、Enzyme Nomenclature Database、OMIM(Online Mendelian Inheritance in Man)、ChEMBL等が挙げられる。また、例示した公的データベース以外でも、実験や文献調査等で得られた生物学的情報を格納することもできる。
【0030】
さらに、データウェアハウスに格納される生物学的情報について詳しく説明する。生物学的情報とは、遺伝子やタンパク質の機能や構造その他特徴を記述したものであり、遺伝子情報、タンパク質情報、タンパク質間相互作用情報、タンパク質の生物学的機能情報、パスウェイ情報、疾患及び該疾患の原因遺伝子情報、タンパク質の立体構造情報、タンパク質発現情報、転写因子情報、並びに、薬剤情報からなる群から選択される1又は複数の情報を含む。
【0031】
遺伝子情報は、遺伝子の名前、シンボル、アクセション番号、核酸の塩基配列、遺伝子がコードするタンパク質の名前、タンパク質ID、タンパク質のアミノ酸配列、ゲノム上での各遺伝子の位置、遺伝子発現情報、及び文献情報からなる群から選択された1又は複数の情報を含む。遺伝子に、上記の遺伝子情報がゲノムアノテーションとして付与される。遺伝子情報は、Entrez GeneやEnsembl等の公的データベースから取得することができる。
【0032】
遺伝子情報を格納することで、例えば、遺伝子名が与えられたときに対応するアクセション番号を取得できる。また、タンパク質名やタンパク質IDが与えられたときに該タンパク質をコードする遺伝子の情報を取得したりする等、遺伝子情報とタンパク質情報との相互変換が可能である。
【0033】
遺伝子相同性情報とは、共通祖先に由来する遺伝子間の関係を示す情報である。特に種分化によって生じた相同な遺伝子(オーソログ)の情報を用いることで、入力された候補遺伝子がモデル動物等ある特定の生物種由来である場合に、より情報の多い別の生物種(例えば、ヒト等)由来の、該候補遺伝子が対応する遺伝子に変換することができる。遺伝子相同性情報は、KEGG Orthology等の公的データベースから取得することができる。
【0034】
遺伝子多型情報とは、人口の約1%以上の頻度で存在する遺伝子配列の変異情報である。遺伝子に対して、該遺伝子について知られている多型がアノテーションとして付与される。遺伝子多型情報は、GeMDBJ、dbSNP等の公的データベースから取得することができる。
【0035】
遺伝子発現情報には、薬物を動物や細胞に暴露して解析された遺伝子発現情報を含み、遺伝子レベルで毒性発現メカニズムの解析や毒性予測を行うことが可能である。化合物を哺乳類の個体や哺乳類細胞へ暴露した際の毒性情報及び遺伝子発現情報は、TG-GATEs(Toxicogenomics
Project-Genomics Assisted Toxicity Evaluation systemの略称である。)、又はその公開版であるOpen TG-GATEs等のデータベースから取得することができる。
【0036】
タンパク質情報は、タンパク質の名前、タンパク質ID、アミノ酸配列、タンパク質をコードする遺伝子の名前、アクセション番号、核酸の塩基配列、タンパク質の機能、及び文献情報からなる群から選択された1又は複数の情報を含む。タンパク質に、上記のタンパク質情報がタンパク質アノテーションとして付与される。タンパク質情報は、UniProtKB等の公的データベースから取得することができる。
【0037】
タンパク質情報を格納することで、例えば、タンパク質名が与えられたときに対応するタンパク質IDを取得できる。また、遺伝子やタンパク質IDが与えられたときに該タンパク質をコードする遺伝子の情報を取得したりする等、遺伝子情報とタンパク質情報との相互変換が可能である。
【0038】
タンパク質間相互作用情報とは、主に酵母2ハイブリッド法等の実験手段によって相互作用が特定されたタンパク質間の関係を示す情報である。相互作用する複数のタンパク質は、同一の機能的役割を果たすことが多いため、入力手段から入力された候補タンパク質と相互作用するタンパク質を収集し、これらを候補遺伝子又はタンパク質に追加して入力することができる。タンパク質間相互作用情報のデータベースでは、タンパク質に対して、該タンパク質と相互作用するタンパク質がアノテーションとして付与される。タンパク質間相互作用情報は、PPIview, BIOGRID等の公的データベースから取得することができる。
【0039】
タンパク質の生物学的機能の情報とは、タンパク質の生体内での機能を示す情報であり、例えば、遺伝子オントロジー(Gene Ontology; GO)を利用することができる。遺伝子オントロジーでは、細胞の構成要素(Cellular Component)、生物学的プロセス(Biological
Process)、及び分子機能(Molecular Function)の3種に分類されるGO Termと呼ばれる用語によりタンパク質が記述される。遺伝子オントロジーのデータベースでは、遺伝子に対し、該遺伝子のGO Termがアノテーションとして付与される。遺伝子オントロジーのデータベースとしては、the Gene Ontology、UniProt KB GOA等が挙げられる。
【0040】
タンパク質ドメイン情報とは、タンパク質を構成するドメイン(進化的に祖先を共有し、共通の立体構造や機能を持つ配列単位)及びその分類からなる情報であり、InterProなどから取得できる。タンパク質に対して、該タンパク質に含まれるドメインがアノテーションとして付与される。
【0041】
タンパク質の立体構造情報とは、タンパク質の三次元立体構造に関する情報であり、タンパク質のリガンド結合部位の体積、構成原子の数、溶媒露出面積、平面性、細長さ、曲率、疎水性度、水素結合供与原子の数、水素結合受容原子の数、リガンド結合部位表面のアミノ酸組成、立体構造ドメインの分類、及びアミノ酸配列に基づくタンパク質情報へのクロスリファレンスからなる群から選択された1又は複数の情報を含む。タンパク質の立体構造情報のデータベースとしては、PDB(Protein Data Bank)、SCOP(Structural Classification of Proteins)、SIFTS等が挙げられる。
【0042】
タンパク質発現情報とは、細胞や組織で発現するタンパク質に関する情報であり、プロテオーム解析により得られた情報等が含まれる。タンパク質発現情報のデータベースとしては、GeMDBJ(Genome Medicine Database of Japan)Proteomics等が挙げられる。
【0043】
酵素機能情報とは、触媒反応に基づく酵素の分類情報であり、例えばThe Enzyme Commission (EC)番号を利用することができる。酵素機能が知られているタンパク質に対し、該酵素機能のEC番号がアノテーションとして付与される。酵素機能情報は、Enzyme database等の公的データベースから取得することができる。
【0044】
パスウェイ情報とは、タンパク質や遺伝子の機能的な関係を示す情報である。パスウェイのデータベースでは、タンパク質や遺伝子が属する代謝や相互作用の一連の流れの情報を得ることができる。同一のパスウェイに属する遺伝子又はタンパク質には、同一のアノテーションが付与される。パスウェイ情報は、KEEG(Kyoto Encyclopedia of Genes and Genomes) Pathwayや、Pathway Interaction Database等のデータベースから得られる。
【0045】
転写因子情報とは、転写因子と該転写因子が作用する遺伝子との関係を示す情報である。転写因子とは、遺伝子の制御領域に特異的に結合し、該遺伝子の発現を制御するタンパク質をいう。転写因子情報のデータベースでは、遺伝子に対し、該遺伝子の発現を制御する転写因子がアノテーションとして付与される。転写因子データベースとしては、OregAnno等が挙げられる。
【0046】
疾患及び該疾患の原因遺伝子の情報とは、遺伝子変異を原因として発生する疾患と、疾患の原因遺伝子を示す情報である。遺伝子に対し、該遺伝子が原因となる疾患がアノテーションとして付与される。疾患及び該疾患の発生の原因遺伝子の情報は、例えば、OMIM(Online Mendelian Inheritance in Man)、Disease Ontology、GWAS(Genome-wide association studies)等のデータベースから得られる。
【0047】
薬剤情報とは、薬剤の一般名、商品名、化学構造、及び該薬剤がターゲットとする遺伝子からなる群から選択される1又は複数の情報を含む。薬剤情報のデータベースでは、遺伝子に対し、該遺伝子をターゲットとする薬剤がアノテーションとして付与される。薬剤情報データベースとしては、DrugBank等が挙げられる。
【0048】
化合物情報とは、特定のタンパク質と相互作用する低分子化合物の解離定数、阻害定数等を含む情報である。タンパク質に対して、該タンパク質と相互作用する低分子化合物がアノテーションとして付与される。化合物情報は、ChEMBL等の公的データベースから取得することができる。
【0049】
本発明では、上記の情報を含む複数の情報がデータウェアハウスに格納されるため、一の遺伝子に対して複数の情報を得ることができる。したがって、一の遺伝子に複数のアノテーションが付与されることになる。
【0050】
また、本発明の別の態様では、上記の情報はデータウェアハウスに蓄積されていなくてもよく、必要な場合に適時データベースから得ることができる。
【0051】
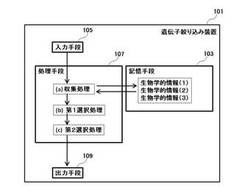
ここで、図1は本発明の遺伝子絞り込み装置の一態様の構成を示すブロック図である。図中の矢印はデータの流れを示している。以下、図面を参照して本発明の遺伝子絞り込み装置をさらに詳細に説明する。遺伝子絞り込み装置101は、上述の生物学的情報が複数格納されたデータウェアハウスを記憶する記憶手段103、入力手段105、処理手段107、及び出力手段109を備える。
【0052】
遺伝子絞り込み装置101の一例はコンピュータであり、遺伝子又はタンパク質を絞りこませるコンピュータプログラムを実行する。遺伝子絞り込み装置101は、好ましくはインターネットにアクセスして生物学的情報に関するデータベースにアクセスすることができる。また、記憶手段103は、遺伝子絞り込み装置101に組み込まれているハードディスクだけでなく、他の装置の記憶手段にネットワークを経由してアクセスすることも含み得る。
【0053】
入力手段105には、複数の候補遺伝子が情報として入力される。遺伝子の入力には、例えば、遺伝子名、遺伝子シンボルや、遺伝子ID、アクセション番号やProtein ID等の遺伝子に固有のIDを利用することができる。
【0054】
また、入力手段105に入力される候補遺伝子は、マイクロアレイ法や酵母2ハイブリッド法等の実験手段によって得ることができる。マイクロアレイ法を用いれば、多数の遺伝子の発現を一度に検出することが可能である。例えば、疾患の有無で発現量に差がある遺伝子を検出することが可能であり、これらは疾患原因候補遺伝子群として、本発明の絞り込みの対象となる。また、酵母2ハイブリッド法は、タンパク質の相互作用の有無を調べる方法であり、あるタンパク質と相互作用するタンパク質をスクリーニングすることができる。例えば、疾患に関連すると知られているタンパク質と相互作用するタンパク質を、酵母2ハイブリッド法を用いてスクリーニングすれば、新規の疾患原因候補遺伝子を得ることができる。
【0055】
また、処理手段107は、記憶手段103に記憶されたデータウェアハウスから、データウェアハウスに格納されたタンパク質間相互作用情報を基に、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として前記入力手段に追加して入力手段105から入力する再入力処理を実行する。タンパク質間相互作用情報のデータベースとしては、PPIview、BioGrid等が挙げられる。
【0056】
さらに、処理手段107は、(a)収集処理、(b)第1選択処理、(c)第2選択処理を実行する。以下、処理手段が実行する各処理(a)〜(c)について詳述する。また、図5には各処理のフローチャートを示す。
【0057】
(a)収集処理
記憶手段103では、遺伝子に対し、データウェアハウスに格納された生物学的情報に応じたアノテーションが付与される。データウェアハウスには複数の生物学的情報が格納されているため、一の遺伝子に複数のアノテーションが付与されることとなる。収集処理とは、入力手段105から入力された候補遺伝子に付与されたアノテーションを記憶手段103から収集する処理である。
【0058】
別の実施態様では、ネットワークを経由して、処理手段107がデータベースにアクセスすることで候補遺伝子に付与されたアノテーションを収集することができる。
【0059】
また、後述する複数の対照遺伝子又は対照タンパク質についても、第1選択処理(b)で比較する場合には、同様に対照群に付与されたアノテーションを収集する処理を行ってもよい。すなわち、本発明における遺伝子絞り込み装置101では、入力手段105は、複数の対照遺伝子又は対照タンパク質が情報として入力され、記憶手段103に記憶されたデータウェアハウスから、入力手段105から入力された対照遺伝子又は対照タンパク質に付与されたアノテーションを収集する収集処理を含みうる。
【0060】
また、ある実施形態では、処理手段107は、データウェアハウスに生物学的情報を格納する際に、収集処理(a)の実行前に、収集処理(a)の実行中に、又は、収集処理(a)の実行後に、アノテーションの冗長性を取り除く処理を行ってもよい。本発明におけるデータウェアハウスには複数の生物学的情報が格納されるため、一の遺伝子又はタンパク質に同一又は類似のアノテーションが重複して付与される場合がある。処理手段107は、冗長性を取り除くこと、すなわち、アノテーションの重複を防ぐことで、遺伝子又はタンパク質の選択の正確性の向上に寄与することができる。
【0061】
また、処理手段107は、データウェアハウスに生物学的情報を格納する際に、収集処理(a)の実行前に、収集処理(a)の実行中に、又は、収集処理(a)の実行後に、ある形式で記述されたアノテーションを、別の形式に変換する処理を行った上で、上記のアノテーションの冗長性を取り除く処理を行うことができる。異なるデータベースから情報を得た場合、同一の情報を示すアノテーションであっても、データベース毎に異なった形式で記述されることがあり得る。したがって、異なった形式で記述されたアノテーションを、共通の形式に変換する工程を行うことで、アノテーションが重複するか否かを判定することができる。
【0062】
例えば、化合物の構造に関するアノテーションでは、InChI等の国際標準規格に相当する記述子に変換することが好ましい。また、各アノテーションを共通の形式に変換することで、アノテーションに含まれる情報を除外することもできる。例えば、化合物の構造に関するアノテーションの中からキラリティに関する情報を除外することで、鏡像異性体や、構造は同一だがキラリティに関する情報を持たない化合物を、同一の化合物として取り扱うことができる。このような情報を除外することで、遺伝子の絞り込みの精度を高めることができる。
【0063】
また、別の実施形態では、処理手段107は、あるアノテーションが他のアノテーションと部分的に重複する場合や、あるアノテーションの記述子が他のアノテーションの記述子と部分的に重複する場合に、重複するアノテーション同士を一のアノテーションとして取り扱うことができる。かかる場合には、例えば、鏡像異性体を同一の化合物としてみなすことができる。
【0064】
さらに別の実施形態では、処理手段107は、複数のアノテーションのハッシュ値を比較することで、これらアノテーションが全体的に又は部分的に重複しているかどうかを判定することができる。特に、アノテーションが構造データを含んでいる場合には、ハッシュ値の比較はアノテーションそのものの比較よりも効率的である。このようなハッシュ値の例として、InChIKeyが挙げられる
【0065】
(b)第1選択処理
さらに、第1選択処理では、収集処理で収集されたアノテーションの中から、候補遺伝子群における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する。付与の頻度又は数についても閾値は、任意の値を既定値として定めておくことができる。例えば、候補遺伝子群に付与された頻度又は数が既定値よりも高いアノテーションを選択してもよく、候補遺伝子群に付与された頻度又は数が多いものから順に既定の数のアノテーションを選択してもよい。
【0066】
また、付与の頻度又は数が閾値より高いか否かは、統計的手法により判定することもできる。例えば、入力手段105から入力された候補遺伝子群における付与の頻度又は数と、対照遺伝子群における付与の頻度又は数とを比較した場合に、候補遺伝子群に多く付与されたか否かを閾値として、候補遺伝子群に多く付与されたアノテーションを、付与の頻度又は数が閾値より高いアノテーションとして選択することができる。
【0067】
また、その場合には、統計的有意差を持って候補遺伝子群に多く付与されたか否かを閾値とすることが好ましい。例えば、候補遺伝子群における付与の頻度又は数と対照遺伝子群における付与の頻度又は数との間に有意差があるかどうかを、フィッシャーの正確確率検定等の統計的有意差検定により検定し、得られたp値が統計的に有意であることの指標になり得る。
【0068】
すなわち、第1選択処理では、収集処理で収集されたアノテーションの中から、候補遺伝子群における付与の頻度又は数が、対照遺伝子群における付与の頻度又は数よりも、有意に多いアノテーションを1又は複数選択する。有意に多いことは、候補遺伝子群における付与の頻度又は数と対照遺伝子群における付与の頻度又は数との間での統計的有意差検定により得られたp値が0.05又は0.01より小さいことにより判定することができる。
【0069】
本発明の別の態様では、付与の頻度又は数が閾値より高いアノテーションとは、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与されたアノテーションの頻度又は数が、複数の対照遺伝子又は対照タンパク質における該アノテーションの付与の頻度又は数と比較して有意に多いアノテーションをいう。すなわち、第1選択処理では、収集処理で収集されたアノテーションの中から、入力手段105から入力された候補遺伝子群に付与された頻度又は数が、対照遺伝子群に付与された頻度又は数と比較して有意に多いアノテーションを1又は複数選択する。対照遺伝子群は限定されないが、例えば、ある生物種が有する遺伝子全体や、ある臓器に発現する遺伝子全体を対照遺伝子群とすることが可能である。
【0070】
具体的には、ヒトの疾患の発症に関連する候補遺伝子を入力した場合には、候補遺伝子に対する全てのアノテーションの付与数のうち、あるアノテーションの付与数が占める割合と、ヒト遺伝子全体に対する全てのアノテーションの付与数のうち、そのアノテーションの付与数が占める割合とを比較して、有意差が認められるかどうかを判定する。有意に多く付与されているか否かの判定には、仮説検定等の公知の統計的手法を用いることができる。
【0071】
第1選択処理では、1又は複数のアノテーションを選択することができる。本処理で選択されるアノテーションが多い程、遺伝子絞り込みの感度(正解の遺伝子を絞り込む確率)は高くなるが、特異度(不正解の遺伝子を絞り込まない確率)は低くなる。一方、選択されるアノテーションが少ない程、遺伝子絞り込みの感度が低くなるが、特異度は高くなる。したがって、絞り込みの対象となる候補遺伝子に応じて、選択するアノテーションの数を変化させればよい。
【0072】
また、第1選択処理では、候補遺伝子群に有意に多く付与されたアノテーションを、多く付与された該アノテーションから順に選択することが好ましい。すなわち、候補遺伝子群と対照遺伝子群との付与の数又は頻度の差が大きいアノテーションから上位いくつかを選択することが好ましい。付与の数又は頻度の差が大きいことは、例えば統計的有意差検定によって得られたp値が小さいこと等を指標にして判定することができる。第1選択処理では、複数の生物学的情報についてのアノテーションを、それぞれの生物学的情報毎に、付与の数又は頻度の差が大きいものから1種乃至10種を選択することが好ましい。
【0073】
(c)第2選択処理
第2選択処理では、入力手段105から入力された候補遺伝子の中から、第1選択処理で選択されたアノテーションが付与された遺伝子を選択する。複数のアノテーションが選択された場合には、該複数のアノテーションのうち1つでも付与された遺伝子を選択してもよく、該複数のアノテーションのうち特定のアノテーションの組み合わせが付与された遺伝子を選択してもよい。
【0074】
上述の第1選択処理により、複数のアノテーションが選択された場合には、選択されたアノテーションの付与数に従って、選択された遺伝子を順位付けすることも可能である。さらに、候補遺伝子群への付与の頻度又は数に従って重み付けをすることができる。候補遺伝子群への付与の頻度又は数と、対照遺伝子群への付与の頻度又は数との差に従ってアノテーションに重み付けを行って、遺伝子を順位付けしてもよい。
【0075】
また、遺伝子絞り込み装置101は、上述の手段に加えて出力手段109を備える。出力手段109は、処理手段107が実行する第2選択処理で選択された遺伝子又はタンパク質を、絞り込まれた遺伝子又はタンパク質として出力する。選択された遺伝子又はタンパク質は、ディスプレイ等の表示装置等や、プリンタ等の印字装置を用いて出力することができる。
【0076】
さらに、本発明では、コンピュータを用いる遺伝子絞り込み方法を提供する。本発明の遺伝絞り込み方法は、コンピュータを用いて複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み方法であって、コンピュータは、入力手段と、処理手段とを備え、(1)入力手段に、複数の候補遺伝子又は候補タンパク質を情報として入力する入力工程と、(2)複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続する接続工程と、(3)処理手段が、記憶手段に記憶されたデータウェアハウスから、入力工程で得られた候補遺伝子又は候補タンパク質に付与されたアノテーションを収集する収集工程と、(4)処理手段が、収集工程で収集されたアノテーションの中から、入力工程で入力された候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択工程と、(5)処理手段が、入力工程で入力された候補遺伝子又は候補タンパク質の中から、第1選択工程で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択工程と、を含むことを特徴とする。データウェアハウスや、データウェアハウスに格納される生物学的情報はインターネット経由で得られるものでよく、アノテーションはインターネット経由でデータウェアハウス又は各データベースから収集してもよい。
【0077】
また、別の遺伝絞り込み方法では、第1選択工程は、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択する工程である。また、記憶手段に記憶されたデータウェアハウスから、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として入力手段に追加して入力する再入力工程を含む。各工程は、上述した説明により明らかである。
【0078】
さらに、本発明は、コンピュータに該遺伝子絞り込み方法を実行させるコンピュータプログラム、及び該コンピュータプログラムが記憶された記憶媒体も提供する。本発明のコンピュータプログラムは、コンピュータに、複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込ませるコンピュータプログラムであって、コンピュータに、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続させる手順と、コンピュータに、データウェアハウスから候補遺伝子又は候補タンパク質に付与されたアノテーションを収集させる手順と、コンピュータに、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択させる手順と、コンピュータに、候補遺伝子又は候補タンパク質の中から、選択されたアノテーションが付与された遺伝子又はタンパク質を選択させる手順と、を実行させることを特徴とする。データウェアハウスや、データウェアハウスに格納される生物学的情報はインターネット経由で得られるものでよく、アノテーションはインターネット経由でデータウェアハウス又は各データベースから収集してもよい。
【0079】
また、別のコンピュータプログラムでは、コンピュータに、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質に有意に多く付与されたアノテーションを、多く付与された該アノテーションから1又は複数選択させる手順を実行させる。また、別のコンピュータプログラムでは、コンピュータに、記憶手段に記憶されたデータウェアハウスから、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集させ、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として入力手段に追加して入力させる手順を実行させる。各手順は、上述した説明により明らかである。
【実施例】
【0080】
さらに実施例を用いて本発明を詳細に説明するが、本発明はこれら実施例に限定されない。
【0081】
実施例1:既知の疾患関連遺伝子を用いた遺伝子絞り込みテスト
本発明を用いて、(1)膵炎、(2)高コレステロール血症、(3)肝硬変、及び(4)子宮頸癌について発生に関連すると知られている遺伝子を用いて遺伝子絞り込みテストを行った。
【0082】
本実施例のデータウェアハウスには、タンパク質の生物学的機能情報、タンパク質のパスウェイ情報、並びに疾患及び該疾患の原因となる遺伝子の情報が格納された。タンパク質の生物学機能情報はthe Gene Ontology、タンパク質のパスウェイ情報はKEEG Pathway、疾患及び該疾患の原因となる遺伝子の情報はOMIMから取得した。
【0083】
それぞれの疾患発生に関連することが既に知られている遺伝子(約30個)を正解遺伝子とし、さらに正解遺伝子の2倍数の遺伝子をランダムに抽出して正解遺伝子に加え、これら遺伝子群を候補遺伝子として入力手段から入力した。入力した遺伝子のリストを表1〜表4に示す。
【0084】
【表1】

【0085】
【表2】

【0086】
【表3】

【0087】
【表4】

【0088】
処理手段は、データウェアハウスに格納された、タンパク質の生物学機能情報(the Gene Ontology)タンパク質のパスウェイ情報(KEEG Pathway)、疾患及び該疾患の原因となる遺伝子の情報(OMIM)に基づき、入力された候補遺伝子に付与されたアノテーションを収集する収集手段を実行した。収集手段で収集された各疾患のアノテーション数(個数及び種類数)は、以下の表5の通りであった。
【0089】
【表5】

【0090】
続いて、処理手段は第1選択処理を実行した。対照遺伝子群は、ヒト遺伝子全体とした。上記収集処理において収集されたアノテーションの中から、候補遺伝子群に対するアノテーション付与の頻度が、対照群であるヒト遺伝子全体に対する付与の頻度と比較して有意に多く含まれるかどうかをp値<0.05を閾値として判定した。収集されたアノテーションのうち、ヒト遺伝子全体に付与されたアノテーションと比較して差が大きいもの上位10種を、差が大きい順(p値が小さい順)に、それぞれ表6〜表9に示す。表中のアノテーションが10種に満たないものは、p値<0.05となるアノテーションが10種存在しなかったことを示している。
【0091】
【表6】

【0092】
【表7】

【0093】
【表8】

【0094】
【表9】

【0095】
第1選択処理では、the Gene Ontology、KEEG Pathway、OMIMのそれぞれのアノテーションを、対照群との差が大きいものから、すなわちp値が小さいものから選択した。選択するアノテーション数は1種〜10種で変化させた。
【0096】
続いて、処理手段で第2選択処理を実行し、第1選択処理で選択されたアノテーションが一つでも付与された遺伝子を選択した。さらに、上記の第2選択処理で選択された遺伝子を、候補遺伝子から絞り込まれた遺伝子として出力手段から出力した。ここで、一例として、(1)膵炎で、第1選択処理でアノテーションをそれぞれ上位1個ずつ選択した場合に、第2選択処理において選択された遺伝子の遺伝子IDを表10に示す。
【0097】
【表10】

【0098】
さらに、上述の遺伝子のランダム抽出から絞り込みまでを合計10回繰り返した。それぞれの疾患の関連遺伝子として出力された遺伝子のうち、正解遺伝子の10回の絞り込みの平均感度及び平均特異度を図2に示す。グラフの横軸は、第1選択処理で選択されたアノテーションの数を示す。いずれの疾患についても、80%程度の感度及び特異度で正解遺伝子を出力することができた。
【0099】
実施例2:新規疾患関連遺伝子を対象とした絞り込み
本発明の遺伝子絞り込み装置を用いてC型肝炎の発生に関連する遺伝子の絞り込みを行った。C型肝炎ウイルス(HCV)Coreタンパク質とヒトのタンパク質であるPA28γとの相互作用がC型肝炎の発症に重要であることは知られていたが(非特許文献1参照)、詳しいメカニズムは不明であり、C型肝炎の発生に関連性を持ったヒト遺伝子は明らかでなかった。そこで、本発明を用いて候補遺伝子の絞り込みを行った。
【0100】
非特許文献1:Moriishi, K. et al., “Critical role of PA28γ
in hepatitis C virus-associated steatogenesis and hepatocarcinogenesis.”P.N.A.S.,
2007.
【0101】
データウェアハウスに格納された情報は、タンパク質間相互作用情報、タンパク質の生物学的機能情報、タンパク質のパスウェイ情報、並びに疾患及び該疾患の原因となる遺伝子の情報とした。タンパク質間の相互作用情報はBioGrid及びPPIview、タンパク質の生物学機能情報はthe Gene Ontology、タンパク質のパスウェイ情報はKEEG Pathway、疾患及び該疾患の原因となる遺伝子の情報はOMIM及びDisease Ontolgyから取得した。
【0102】
酵母2ハイブリッド法により、Human Adult liver library(MoBiTec社製)を用いて、HCVのCore又はNS4Bと相互作用する宿主のタンパク質のスクリーニングを行った。スクリーニングの結果、Coreと相互作用する11種のタンパク質、NS4Bと相互作用する45種のタンパク質が同定された。同定されたタンパク質を表11及び表12に示す。
【0103】
【表11】

【0104】
【表12】

【0105】
上記のタンパク質と相互作用するタンパク質を収集して候補遺伝子に追加した。処理手段は、データウェアハウスに格納されたタンパク質間相互作用情報を用いて、表11に記載のタンパク質と相互作用するタンパク質を196種、表12に記載のタンパク質と相互作用するタンパク質を207種それぞれ収集し、入力手段に追加して入力した。
【0106】
したがって、入力された候補遺伝子群は、(1)HCVのCoreと相互作用するタンパク質及び該タンパク質と相互作用するタンパク質(計:207種)、(2)HCVのNS4Bと相互作用するタンパク質及び該タンパク質と相互作用するタンパク質(計:252種)となった。入力された遺伝子のリストを表13〜15に示す。
【0107】
【表13】

【0108】
【表14】

【0109】
【表15】

【0110】
続いて、処理手段が収集処理を実行し、入力されたタンパク質に付与されたアノテーションを収集した。対照群はヒト遺伝子全体とした。収集したアノテーション数と、収集したアノテーションの中で、ヒト遺伝子全体と比較して候補遺伝子群(1)及び(2)に多く付与され、仮説検定の結果p値が0.05以下となったアノテーション数とを表16に示す。
【0111】
【表16】

【0112】
さらに、処理手段は、上記のアノテーションの中から、p値が小さいもの上位10種を選択する第1選択処理を行い、さらにタンパク質を選択する第2選択処理を行った。第2選択処理では、第1選択処理で選択されたアノテーションが付与され、且つ、タンパク質間相互作用情報でCore又はNS4Bがアノテーションとして付与された(すなわち、Core又はNS4Bに相互作用するタンパク質)ことをタンパク質選択の条件とした。第2選択処理で選択されたタンパク質を表17に示す。
【0113】
【表17】

【0114】
このように絞り込まれた遺伝子がC型肝炎の発生に関連する遺伝子であるかどうかを判定するため、表17に記載の遺伝子の中からSLC25A5及びENO1について実験的に検証を行った。SLC25A5及びENO1が相互作用するタンパク質であるPXNについても検証を行った。SLC25A5、ENO1及びPXNに対するsiRNAを、それぞれHuh7OK1細胞に導入し、24時間後にC型肝炎ウイルスJFH-1株(遺伝子型2a)をHuh7OK1細胞に感染させ、感染後細胞を72時間培養した。培養上清に含まれたウイルスRNAと、細胞中のGAPDHのmRNAとを定量的リアルタイムPCR法を用いて測定した。GAPDHのmRNA量に対するウイルスRNA量を図3に示す。培養上清に含まれたウイルスRNAの量はENO1のノックダウンにより顕著に減少した。また、SLC25A5のノックダウンにより、ウイルスRNAの量が統計上有意(p<0.01)に増加した。しかしながら、PXNのノックダウンでは、ウイルスRNAの量に有意差はみられなかった。
【0115】
また、他のC型肝炎ウイルスの遺伝子型についての影響を調べるため、JFH-1(遺伝子型2a)及びCon-1(遺伝子型1b)から得られたHCVレプリコンを含むHuh-7細胞に、それぞれの遺伝子に対するsiRNAを導入し、定量的リアルタイムPCR法を用いて培養上清のCon-1のウイルスRNA量と細胞中のGAPDHのmRNA量とを測定した。図4にGAPDHのmRNA量に対するCon-1のウイルスRNA量の割合を示す。ENO1及びPXNのノックダウンによりCon-1のHCVの複製が抑制された。
【0116】
以上のように、SLC25A5、ENO1、及びPXNはHCVの複製に関与するタンパク質であることが実験的に明らかとなった。したがって、本発明によって多数の候補遺伝子群から絞り込まれたSLC25A5及びENO1や、SLC25A5及びENO1と相互作用するPXNはC型肝炎の発生に関連性を有することがわかった。
【産業上の利用可能性】
【0117】
本発明の遺伝子絞り込み装置、遺伝子絞り込み装置、及びコンピュータプログラムを利用することで、新規疾患関連遺伝子の発見や、新規薬剤の開発に寄与することができる。
【符号の説明】
【0118】
101:遺伝子絞り込み装置
103:記憶手段
105:入力手段
107:処理手段
109:出力手段
【技術分野】
【0001】
本発明は、複数の候補遺伝子から任意の関連性を有する遺伝子を絞り込む遺伝子絞り込み装置、遺伝子絞り込み方法、及びコンピュータプログラムに関する。
【背景技術】
【0002】
近年の分子生物学の発展により、遺伝子やタンパク質についての知見が蓄積している。これらの知見は、遺伝子に関する情報として、NCBI(National Center for Biotechnology
Information)Entrez Geneや、DDBJ(DNA Data Bank of Japan)等、さまざまな公的データベースにより公開されている。
【0003】
これらのデータベースから得られる情報を利用して、遺伝子や遺伝子がコードするタンパク質の機能を予測する技術が開発されている。例えば、アミノ酸配列情報や塩基配列情報を用いてGタンパク共役型受容体を判別する方法(特許文献1及び特許文献2参照)、タンパク質の立体構造情報や、タンパク質に作用する薬剤/化合物の情報等から選択された属性を特徴ベクトルとした教師付き機械学習によりタンパク質相互作用を予測する方法(特許文献3参照)等が挙げられる。
【先行技術文献】
【特許文献】
【0004】
【特許文献1】特開2006−003970号公報
【特許文献2】特開2002−112793号公報
【特許文献3】特開2010−165230号公報
【発明の概要】
【発明が解決しようとする課題】
【0005】
マイクロアレイ等の手法により、特定の機能や関連性を持った候補遺伝子を網羅的に取得することができるようになった。しかし、取得する候補遺伝子の数は大量であり、得られた候補遺伝子の中から、より可能性が高い遺伝子を絞り込むことが必要であった。しかし、公的データベースを利用して容易に遺伝子を絞り込む技術は確立されていなかった。
【0006】
また、従来のデータベースを用いたタンパク質機能予測は、上記の特許文献3にみられるように教師付き学習を伴うものであった。教師付き学習は正例及び負例を必要とするため、正例及び負例が得られない場合には従来の予測法を利用することができなかった。
【課題を解決するための手段】
【0007】
本発明は、複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み装置であり、記憶手段と、入力手段と、処理手段とを備え、記憶手段は、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスを記憶し、入力手段は、複数の候補遺伝子又は候補タンパク質が情報として入力され、処理手段は、(a)記憶手段に記憶されたデータウェアハウスにおいて、入力手段から入力された候補遺伝子又は候補タンパク質に付与されたアノテーションを収集する収集処理と、(b)収集処理で収集されたアノテーションの中から、入力手段から得られた候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択処理と、(c)入力手段から得られた候補遺伝子又は候補タンパク質の中から、選択処理で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択処理と、を実行する。
【0008】
また、本発明の別の遺伝子絞り込み装置では、第1選択処理(b)における、付与の頻度又は数が閾値より高いアノテーションとは、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与されたアノテーションの頻度又は数が、複数の対照遺伝子又は対照タンパク質における該アノテーションの付与の頻度又は数と比較して有意に多いアノテーションをいう。すなわち、第1選択処理(b)は、収集処理で収集されたアノテーションの中から、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与された頻度又は数が、複数の対照遺伝子又は対照タンパク質に付与された頻度又は数と比較して有意に多いアノテーションを1又は複数選択する第1選択処理である。
【0009】
また、本発明の別の遺伝子絞り込み装置は、第1選択処理(b)が、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択する処理である。候補遺伝子群と対照遺伝子群とを比較して、付与数の差が大きいアノテーションを選択することで、遺伝子絞り込みの精度を上げることができる。
【0010】
また、本発明の別の遺伝子絞り込み装置は、データウェアハウスに格納された生物学的情報についてのアノテーションの冗長性を取り除く処理を実行する。また、データウェアハウスに格納された生物学的情報についてのアノテーションの記述の形式を変換する処理を実行する。これらの処理によって、アノテーションの重複を防ぎ、遺伝子又はタンパク質の選択の正確性の向上に寄与することができる。
【0011】
また、本発明の別の遺伝子絞り込み装置は、処理手段が、(a)〜(c)の各処理に加えて、記憶手段に記憶されたデータウェアハウスから、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として入力手段に追加して入力する再入力処理を実行する。相互作用するタンパク質群は同一の機能的役割を果たすことが多く、候補遺伝子又は候補タンパク質の数を増やすことで、遺伝子絞り込みの精度を上げることができる。
【0012】
また、本発明は、コンピュータを用いて複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み方法であり、コンピュータは、入力手段と、処理手段とを備え、(1)入力手段に、複数の候補遺伝子又は候補タンパク質を情報として入力する入力工程と、(2)複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続する接続工程と、(3)処理手段が、記憶手段に記憶されたデータウェアハウスにから、入力工程で入力された候補遺伝子又は候補タンパク質に付与されたアノテーションを収集する収集工程と、(4)処理手段が、収集工程で収集されたアノテーションの中から、入力工程で入力された候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択工程と、(5)処理手段が、前記入力工程で入力された候補遺伝子又は候補タンパク質の中から、前記第1選択工程で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択工程と、を含むことを特徴とする。
【0013】
また、本発明の別の遺伝子絞り込み方法では、第1選択工程(4)において、付与の頻度又は数が閾値より高いアノテーションとは、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与されたアノテーションの頻度又は数が、複数の対照遺伝子又は対照タンパク質における該アノテーションの付与の頻度又は数と比較して有意に多いアノテーションをいう。すなわち、第1選択工程(4)は、収集処理で収集されたアノテーションの中から、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与された頻度又は数が、複数の対照遺伝子又は対照タンパク質に付与された頻度又は数と比較して有意に多いアノテーションを1又は複数選択する第1選択工程(4)である。
【0014】
また、本発明の別の遺伝子絞り込み方法は、データウェアハウスに格納された生物学的情報についてのアノテーションの冗長性を取り除く工程を含む。また、データウェアハウスに格納された生物学的情報についてのアノテーションの記述の形式を変換する工程を含む。これらの工程によって、アノテーションの重複を防ぎ、遺伝子又はタンパク質の選択の正確性の向上に寄与することができる。
【0015】
また、本発明の別の遺伝子絞り込み方法は、第1選択工程(4)が、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択する処理である。また、本発明の別の遺伝絞り込み方法は、処理手段が、記憶手段に記憶されたデータウェアハウスから、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として入力手段に追加して入力する再入力工程を実行する。
【0016】
さらに、本発明は、コンピュータに複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込ませるコンピュータプログラムであり、コンピュータに、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続させる手順と、データウェアハウスから候補遺伝子又は候補タンパク質に付与されたアノテーションを収集させる手順と、コンピュータに、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択させる手順と、コンピュータに、候補遺伝子又は候補タンパク質の中から、選択されたアノテーションが付与された遺伝子又はタンパク質を選択させる手順と、を実行させることを特徴とする。
【0017】
また、本発明の別のコンピュータプログラムでは、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択させる手順において、付与の頻度又は数が閾値より高いアノテーションとは、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与されたアノテーションの頻度又は数が、複数の対照遺伝子又は対照タンパク質における該アノテーションの付与の頻度又は数と比較して有意に多いアノテーションをいう。すなわち、本発明の別のコンピュータプログラムでは、コンピュータに、収集されたアノテーションの中から、複数の候補遺伝子又は候補タンパク質に付与された頻度又は数が、複数の対照遺伝子又は対照タンパク質に付与された頻度又は数と比較して有意に多いアノテーションを1又は複数選択させる手順を実行させる。
【0018】
また、本発明の別のコンピュータプログラムは、コンピュータに、データウェアハウスに格納された生物学的情報についてのアノテーションの冗長性を取り除く手順を実行させる。また、データウェアハウスに格納された生物学的情報についてのアノテーションの記述の形式を変換する手順を実行させる。これらの工程によって、アノテーションの重複を防ぎ、遺伝子又はタンパク質の選択の正確性の向上に寄与することができる。
【0019】
また、本発明の別のコンピュータプログラムは、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質に有意に多く付与されたアノテーションを1又は複数選択させる手順が、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択させる手順である。また、本発明の別のコンピュータプログラムは、コンピュータに、データウェアハウスから、入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として追加して入力させる手順を実行させる。
【0020】
また、本発明のデータウェアハウスに格納される複数の生物学的情報は、遺伝子情報、遺伝子相同性情報、遺伝子多型情報、タンパク質情報、タンパク質間相互作用情報、タンパク質の生物学的機能情報、タンパク質ドメイン情報、タンパク質の立体構造情報、タンパク質発現情報、酵素機能情報、パスウェイ情報、転写因子情報、疾患及び該疾患の原因遺伝子情報、薬剤情報、並びに化合物情報、からなる群から選択される複数の情報である。
【0021】
さらに、本発明の遺伝子絞り込み装置で絞り込む遺伝子の関連性は、疾患の発生についての関連性であり得る。
【発明の効果】
【0022】
本発明は、候補遺伝子から、疾患の発生等の任意の関連性を有する遺伝子を絞り込む装置である。本発明によれば、マイクロアレイ等の網羅的な解析結果から、より可能性が高い遺伝子を容易に絞り込むことができる。本発明では、教師付き学習を伴わなくとも絞り込みが可能なことから、正例及び負例を必要とせず、従来法よりも簡便に絞り込むことができる。
【図面の簡単な説明】
【0023】
【図1】本発明の遺伝子絞り込み装置の一態様である。
【図2】本発明を用いた絞り込みテストの感度及び特異度を示した図である。
【図3】本発明を用いて絞り込まれた遺伝子がC型肝炎の発症に関連性を有するかどうかの実験的検証の結果を示す図である。
【図4】本発明を用いて絞り込まれた遺伝子がC型肝炎の発症に関連性を有するかどうかの実験的検証の結果を示す図である。
【図5】本発明の実施例における各工程、及び各工程におけるデータ配列を示すフローチャート及びデータ構造の概略図である。
【発明を実施するための形態】
【0024】
以下、発明を実施するための形態に従って本発明を詳細に説明するが、本発明はこれらの実施形態に限定されない。
【0025】
本発明では、遺伝子に関する複数の情報を格納したデータウェアハウスを用いて、複数の遺伝子からなる遺伝子群から、任意の関連性を有する遺伝子の候補を絞り込むことができる。任意の関連性とは、遺伝子の持つ機能や性質についての関連性をいう。例えば、本発明によれば、ある疾患の原因となる候補遺伝子群から、より可能性の高い遺伝子を絞り込むことができる。
【0026】
ここで、タンパク質は、遺伝子によってアミノ酸配列が決定され、タンパク質の構造や機能も決定される。したがって、本発明において絞り込みの対象はタンパク質も含む。したがって、本発明は、複数のタンパク質からなる候補タンパク質群から、任意の関連性を有するタンパク質の候補を絞り込むことができる。また、遺伝子情報やタンパク質情報を用いて、遺伝子とタンパク質との情報の相互変換が可能である。
【0027】
本発明では、記憶手段に記憶されたデータウェアハウスを用いて上記の絞り込みを行う。データウェアハウスとは、複数種の情報を格納し、目的に応じて格納された情報から必要な情報を引き出すことが可能なシステムをいう。
【0028】
本発明において用いられるデータウェアハウスには、複数種の生物学的情報、すなわち、遺伝子情報、遺伝子相同性情報、遺伝子多型情報、タンパク質情報、タンパク質間相互作用情報、タンパク質の生物学的機能情報、タンパク質ドメイン情報、タンパク質の立体構造情報、タンパク質発現情報、酵素機能情報、パスウェイ情報、転写因子情報、疾患及び該疾患の原因遺伝子情報、薬剤情報、並びに化合物情報、からなる群から選択される複数を含む情報が格納される。本発明においては、生物学的情報はインターネット経由でデータウェアハウスに格納することがでるため、記憶手段に生物学的情報が記憶されていなくてもよい。
【0029】
上記の生物学的情報の格納には公的データベースを利用することができる。公的データベースとしては、例えば、NCBI(National Center for Biotechnology
Information) Entrez Gene、UCSC (University
of California Santa Cruz) database、DDBJ(DNA Data Bank of Japan)、GeMDBJ、dbSNP、Ensembl、UniProtKB、InterPro、SIFTS、SCOP(Structural Classification of Proteins)、PDB(Protein Data Bank)、PPIview、BioGRID、KEEG(Kyoto
Encyclopedia of Genes and Genomes)、The Gene Ontology、UniProtKB-GOA、OregAnno(The Open Regulatory Annotation database)、AMADEUS、Enzyme Nomenclature Database、OMIM(Online Mendelian Inheritance in Man)、ChEMBL等が挙げられる。また、例示した公的データベース以外でも、実験や文献調査等で得られた生物学的情報を格納することもできる。
【0030】
さらに、データウェアハウスに格納される生物学的情報について詳しく説明する。生物学的情報とは、遺伝子やタンパク質の機能や構造その他特徴を記述したものであり、遺伝子情報、タンパク質情報、タンパク質間相互作用情報、タンパク質の生物学的機能情報、パスウェイ情報、疾患及び該疾患の原因遺伝子情報、タンパク質の立体構造情報、タンパク質発現情報、転写因子情報、並びに、薬剤情報からなる群から選択される1又は複数の情報を含む。
【0031】
遺伝子情報は、遺伝子の名前、シンボル、アクセション番号、核酸の塩基配列、遺伝子がコードするタンパク質の名前、タンパク質ID、タンパク質のアミノ酸配列、ゲノム上での各遺伝子の位置、遺伝子発現情報、及び文献情報からなる群から選択された1又は複数の情報を含む。遺伝子に、上記の遺伝子情報がゲノムアノテーションとして付与される。遺伝子情報は、Entrez GeneやEnsembl等の公的データベースから取得することができる。
【0032】
遺伝子情報を格納することで、例えば、遺伝子名が与えられたときに対応するアクセション番号を取得できる。また、タンパク質名やタンパク質IDが与えられたときに該タンパク質をコードする遺伝子の情報を取得したりする等、遺伝子情報とタンパク質情報との相互変換が可能である。
【0033】
遺伝子相同性情報とは、共通祖先に由来する遺伝子間の関係を示す情報である。特に種分化によって生じた相同な遺伝子(オーソログ)の情報を用いることで、入力された候補遺伝子がモデル動物等ある特定の生物種由来である場合に、より情報の多い別の生物種(例えば、ヒト等)由来の、該候補遺伝子が対応する遺伝子に変換することができる。遺伝子相同性情報は、KEGG Orthology等の公的データベースから取得することができる。
【0034】
遺伝子多型情報とは、人口の約1%以上の頻度で存在する遺伝子配列の変異情報である。遺伝子に対して、該遺伝子について知られている多型がアノテーションとして付与される。遺伝子多型情報は、GeMDBJ、dbSNP等の公的データベースから取得することができる。
【0035】
遺伝子発現情報には、薬物を動物や細胞に暴露して解析された遺伝子発現情報を含み、遺伝子レベルで毒性発現メカニズムの解析や毒性予測を行うことが可能である。化合物を哺乳類の個体や哺乳類細胞へ暴露した際の毒性情報及び遺伝子発現情報は、TG-GATEs(Toxicogenomics
Project-Genomics Assisted Toxicity Evaluation systemの略称である。)、又はその公開版であるOpen TG-GATEs等のデータベースから取得することができる。
【0036】
タンパク質情報は、タンパク質の名前、タンパク質ID、アミノ酸配列、タンパク質をコードする遺伝子の名前、アクセション番号、核酸の塩基配列、タンパク質の機能、及び文献情報からなる群から選択された1又は複数の情報を含む。タンパク質に、上記のタンパク質情報がタンパク質アノテーションとして付与される。タンパク質情報は、UniProtKB等の公的データベースから取得することができる。
【0037】
タンパク質情報を格納することで、例えば、タンパク質名が与えられたときに対応するタンパク質IDを取得できる。また、遺伝子やタンパク質IDが与えられたときに該タンパク質をコードする遺伝子の情報を取得したりする等、遺伝子情報とタンパク質情報との相互変換が可能である。
【0038】
タンパク質間相互作用情報とは、主に酵母2ハイブリッド法等の実験手段によって相互作用が特定されたタンパク質間の関係を示す情報である。相互作用する複数のタンパク質は、同一の機能的役割を果たすことが多いため、入力手段から入力された候補タンパク質と相互作用するタンパク質を収集し、これらを候補遺伝子又はタンパク質に追加して入力することができる。タンパク質間相互作用情報のデータベースでは、タンパク質に対して、該タンパク質と相互作用するタンパク質がアノテーションとして付与される。タンパク質間相互作用情報は、PPIview, BIOGRID等の公的データベースから取得することができる。
【0039】
タンパク質の生物学的機能の情報とは、タンパク質の生体内での機能を示す情報であり、例えば、遺伝子オントロジー(Gene Ontology; GO)を利用することができる。遺伝子オントロジーでは、細胞の構成要素(Cellular Component)、生物学的プロセス(Biological
Process)、及び分子機能(Molecular Function)の3種に分類されるGO Termと呼ばれる用語によりタンパク質が記述される。遺伝子オントロジーのデータベースでは、遺伝子に対し、該遺伝子のGO Termがアノテーションとして付与される。遺伝子オントロジーのデータベースとしては、the Gene Ontology、UniProt KB GOA等が挙げられる。
【0040】
タンパク質ドメイン情報とは、タンパク質を構成するドメイン(進化的に祖先を共有し、共通の立体構造や機能を持つ配列単位)及びその分類からなる情報であり、InterProなどから取得できる。タンパク質に対して、該タンパク質に含まれるドメインがアノテーションとして付与される。
【0041】
タンパク質の立体構造情報とは、タンパク質の三次元立体構造に関する情報であり、タンパク質のリガンド結合部位の体積、構成原子の数、溶媒露出面積、平面性、細長さ、曲率、疎水性度、水素結合供与原子の数、水素結合受容原子の数、リガンド結合部位表面のアミノ酸組成、立体構造ドメインの分類、及びアミノ酸配列に基づくタンパク質情報へのクロスリファレンスからなる群から選択された1又は複数の情報を含む。タンパク質の立体構造情報のデータベースとしては、PDB(Protein Data Bank)、SCOP(Structural Classification of Proteins)、SIFTS等が挙げられる。
【0042】
タンパク質発現情報とは、細胞や組織で発現するタンパク質に関する情報であり、プロテオーム解析により得られた情報等が含まれる。タンパク質発現情報のデータベースとしては、GeMDBJ(Genome Medicine Database of Japan)Proteomics等が挙げられる。
【0043】
酵素機能情報とは、触媒反応に基づく酵素の分類情報であり、例えばThe Enzyme Commission (EC)番号を利用することができる。酵素機能が知られているタンパク質に対し、該酵素機能のEC番号がアノテーションとして付与される。酵素機能情報は、Enzyme database等の公的データベースから取得することができる。
【0044】
パスウェイ情報とは、タンパク質や遺伝子の機能的な関係を示す情報である。パスウェイのデータベースでは、タンパク質や遺伝子が属する代謝や相互作用の一連の流れの情報を得ることができる。同一のパスウェイに属する遺伝子又はタンパク質には、同一のアノテーションが付与される。パスウェイ情報は、KEEG(Kyoto Encyclopedia of Genes and Genomes) Pathwayや、Pathway Interaction Database等のデータベースから得られる。
【0045】
転写因子情報とは、転写因子と該転写因子が作用する遺伝子との関係を示す情報である。転写因子とは、遺伝子の制御領域に特異的に結合し、該遺伝子の発現を制御するタンパク質をいう。転写因子情報のデータベースでは、遺伝子に対し、該遺伝子の発現を制御する転写因子がアノテーションとして付与される。転写因子データベースとしては、OregAnno等が挙げられる。
【0046】
疾患及び該疾患の原因遺伝子の情報とは、遺伝子変異を原因として発生する疾患と、疾患の原因遺伝子を示す情報である。遺伝子に対し、該遺伝子が原因となる疾患がアノテーションとして付与される。疾患及び該疾患の発生の原因遺伝子の情報は、例えば、OMIM(Online Mendelian Inheritance in Man)、Disease Ontology、GWAS(Genome-wide association studies)等のデータベースから得られる。
【0047】
薬剤情報とは、薬剤の一般名、商品名、化学構造、及び該薬剤がターゲットとする遺伝子からなる群から選択される1又は複数の情報を含む。薬剤情報のデータベースでは、遺伝子に対し、該遺伝子をターゲットとする薬剤がアノテーションとして付与される。薬剤情報データベースとしては、DrugBank等が挙げられる。
【0048】
化合物情報とは、特定のタンパク質と相互作用する低分子化合物の解離定数、阻害定数等を含む情報である。タンパク質に対して、該タンパク質と相互作用する低分子化合物がアノテーションとして付与される。化合物情報は、ChEMBL等の公的データベースから取得することができる。
【0049】
本発明では、上記の情報を含む複数の情報がデータウェアハウスに格納されるため、一の遺伝子に対して複数の情報を得ることができる。したがって、一の遺伝子に複数のアノテーションが付与されることになる。
【0050】
また、本発明の別の態様では、上記の情報はデータウェアハウスに蓄積されていなくてもよく、必要な場合に適時データベースから得ることができる。
【0051】
ここで、図1は本発明の遺伝子絞り込み装置の一態様の構成を示すブロック図である。図中の矢印はデータの流れを示している。以下、図面を参照して本発明の遺伝子絞り込み装置をさらに詳細に説明する。遺伝子絞り込み装置101は、上述の生物学的情報が複数格納されたデータウェアハウスを記憶する記憶手段103、入力手段105、処理手段107、及び出力手段109を備える。
【0052】
遺伝子絞り込み装置101の一例はコンピュータであり、遺伝子又はタンパク質を絞りこませるコンピュータプログラムを実行する。遺伝子絞り込み装置101は、好ましくはインターネットにアクセスして生物学的情報に関するデータベースにアクセスすることができる。また、記憶手段103は、遺伝子絞り込み装置101に組み込まれているハードディスクだけでなく、他の装置の記憶手段にネットワークを経由してアクセスすることも含み得る。
【0053】
入力手段105には、複数の候補遺伝子が情報として入力される。遺伝子の入力には、例えば、遺伝子名、遺伝子シンボルや、遺伝子ID、アクセション番号やProtein ID等の遺伝子に固有のIDを利用することができる。
【0054】
また、入力手段105に入力される候補遺伝子は、マイクロアレイ法や酵母2ハイブリッド法等の実験手段によって得ることができる。マイクロアレイ法を用いれば、多数の遺伝子の発現を一度に検出することが可能である。例えば、疾患の有無で発現量に差がある遺伝子を検出することが可能であり、これらは疾患原因候補遺伝子群として、本発明の絞り込みの対象となる。また、酵母2ハイブリッド法は、タンパク質の相互作用の有無を調べる方法であり、あるタンパク質と相互作用するタンパク質をスクリーニングすることができる。例えば、疾患に関連すると知られているタンパク質と相互作用するタンパク質を、酵母2ハイブリッド法を用いてスクリーニングすれば、新規の疾患原因候補遺伝子を得ることができる。
【0055】
また、処理手段107は、記憶手段103に記憶されたデータウェアハウスから、データウェアハウスに格納されたタンパク質間相互作用情報を基に、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として前記入力手段に追加して入力手段105から入力する再入力処理を実行する。タンパク質間相互作用情報のデータベースとしては、PPIview、BioGrid等が挙げられる。
【0056】
さらに、処理手段107は、(a)収集処理、(b)第1選択処理、(c)第2選択処理を実行する。以下、処理手段が実行する各処理(a)〜(c)について詳述する。また、図5には各処理のフローチャートを示す。
【0057】
(a)収集処理
記憶手段103では、遺伝子に対し、データウェアハウスに格納された生物学的情報に応じたアノテーションが付与される。データウェアハウスには複数の生物学的情報が格納されているため、一の遺伝子に複数のアノテーションが付与されることとなる。収集処理とは、入力手段105から入力された候補遺伝子に付与されたアノテーションを記憶手段103から収集する処理である。
【0058】
別の実施態様では、ネットワークを経由して、処理手段107がデータベースにアクセスすることで候補遺伝子に付与されたアノテーションを収集することができる。
【0059】
また、後述する複数の対照遺伝子又は対照タンパク質についても、第1選択処理(b)で比較する場合には、同様に対照群に付与されたアノテーションを収集する処理を行ってもよい。すなわち、本発明における遺伝子絞り込み装置101では、入力手段105は、複数の対照遺伝子又は対照タンパク質が情報として入力され、記憶手段103に記憶されたデータウェアハウスから、入力手段105から入力された対照遺伝子又は対照タンパク質に付与されたアノテーションを収集する収集処理を含みうる。
【0060】
また、ある実施形態では、処理手段107は、データウェアハウスに生物学的情報を格納する際に、収集処理(a)の実行前に、収集処理(a)の実行中に、又は、収集処理(a)の実行後に、アノテーションの冗長性を取り除く処理を行ってもよい。本発明におけるデータウェアハウスには複数の生物学的情報が格納されるため、一の遺伝子又はタンパク質に同一又は類似のアノテーションが重複して付与される場合がある。処理手段107は、冗長性を取り除くこと、すなわち、アノテーションの重複を防ぐことで、遺伝子又はタンパク質の選択の正確性の向上に寄与することができる。
【0061】
また、処理手段107は、データウェアハウスに生物学的情報を格納する際に、収集処理(a)の実行前に、収集処理(a)の実行中に、又は、収集処理(a)の実行後に、ある形式で記述されたアノテーションを、別の形式に変換する処理を行った上で、上記のアノテーションの冗長性を取り除く処理を行うことができる。異なるデータベースから情報を得た場合、同一の情報を示すアノテーションであっても、データベース毎に異なった形式で記述されることがあり得る。したがって、異なった形式で記述されたアノテーションを、共通の形式に変換する工程を行うことで、アノテーションが重複するか否かを判定することができる。
【0062】
例えば、化合物の構造に関するアノテーションでは、InChI等の国際標準規格に相当する記述子に変換することが好ましい。また、各アノテーションを共通の形式に変換することで、アノテーションに含まれる情報を除外することもできる。例えば、化合物の構造に関するアノテーションの中からキラリティに関する情報を除外することで、鏡像異性体や、構造は同一だがキラリティに関する情報を持たない化合物を、同一の化合物として取り扱うことができる。このような情報を除外することで、遺伝子の絞り込みの精度を高めることができる。
【0063】
また、別の実施形態では、処理手段107は、あるアノテーションが他のアノテーションと部分的に重複する場合や、あるアノテーションの記述子が他のアノテーションの記述子と部分的に重複する場合に、重複するアノテーション同士を一のアノテーションとして取り扱うことができる。かかる場合には、例えば、鏡像異性体を同一の化合物としてみなすことができる。
【0064】
さらに別の実施形態では、処理手段107は、複数のアノテーションのハッシュ値を比較することで、これらアノテーションが全体的に又は部分的に重複しているかどうかを判定することができる。特に、アノテーションが構造データを含んでいる場合には、ハッシュ値の比較はアノテーションそのものの比較よりも効率的である。このようなハッシュ値の例として、InChIKeyが挙げられる
【0065】
(b)第1選択処理
さらに、第1選択処理では、収集処理で収集されたアノテーションの中から、候補遺伝子群における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する。付与の頻度又は数についても閾値は、任意の値を既定値として定めておくことができる。例えば、候補遺伝子群に付与された頻度又は数が既定値よりも高いアノテーションを選択してもよく、候補遺伝子群に付与された頻度又は数が多いものから順に既定の数のアノテーションを選択してもよい。
【0066】
また、付与の頻度又は数が閾値より高いか否かは、統計的手法により判定することもできる。例えば、入力手段105から入力された候補遺伝子群における付与の頻度又は数と、対照遺伝子群における付与の頻度又は数とを比較した場合に、候補遺伝子群に多く付与されたか否かを閾値として、候補遺伝子群に多く付与されたアノテーションを、付与の頻度又は数が閾値より高いアノテーションとして選択することができる。
【0067】
また、その場合には、統計的有意差を持って候補遺伝子群に多く付与されたか否かを閾値とすることが好ましい。例えば、候補遺伝子群における付与の頻度又は数と対照遺伝子群における付与の頻度又は数との間に有意差があるかどうかを、フィッシャーの正確確率検定等の統計的有意差検定により検定し、得られたp値が統計的に有意であることの指標になり得る。
【0068】
すなわち、第1選択処理では、収集処理で収集されたアノテーションの中から、候補遺伝子群における付与の頻度又は数が、対照遺伝子群における付与の頻度又は数よりも、有意に多いアノテーションを1又は複数選択する。有意に多いことは、候補遺伝子群における付与の頻度又は数と対照遺伝子群における付与の頻度又は数との間での統計的有意差検定により得られたp値が0.05又は0.01より小さいことにより判定することができる。
【0069】
本発明の別の態様では、付与の頻度又は数が閾値より高いアノテーションとは、入力手段から得られた複数の候補遺伝子又は候補タンパク質に付与されたアノテーションの頻度又は数が、複数の対照遺伝子又は対照タンパク質における該アノテーションの付与の頻度又は数と比較して有意に多いアノテーションをいう。すなわち、第1選択処理では、収集処理で収集されたアノテーションの中から、入力手段105から入力された候補遺伝子群に付与された頻度又は数が、対照遺伝子群に付与された頻度又は数と比較して有意に多いアノテーションを1又は複数選択する。対照遺伝子群は限定されないが、例えば、ある生物種が有する遺伝子全体や、ある臓器に発現する遺伝子全体を対照遺伝子群とすることが可能である。
【0070】
具体的には、ヒトの疾患の発症に関連する候補遺伝子を入力した場合には、候補遺伝子に対する全てのアノテーションの付与数のうち、あるアノテーションの付与数が占める割合と、ヒト遺伝子全体に対する全てのアノテーションの付与数のうち、そのアノテーションの付与数が占める割合とを比較して、有意差が認められるかどうかを判定する。有意に多く付与されているか否かの判定には、仮説検定等の公知の統計的手法を用いることができる。
【0071】
第1選択処理では、1又は複数のアノテーションを選択することができる。本処理で選択されるアノテーションが多い程、遺伝子絞り込みの感度(正解の遺伝子を絞り込む確率)は高くなるが、特異度(不正解の遺伝子を絞り込まない確率)は低くなる。一方、選択されるアノテーションが少ない程、遺伝子絞り込みの感度が低くなるが、特異度は高くなる。したがって、絞り込みの対象となる候補遺伝子に応じて、選択するアノテーションの数を変化させればよい。
【0072】
また、第1選択処理では、候補遺伝子群に有意に多く付与されたアノテーションを、多く付与された該アノテーションから順に選択することが好ましい。すなわち、候補遺伝子群と対照遺伝子群との付与の数又は頻度の差が大きいアノテーションから上位いくつかを選択することが好ましい。付与の数又は頻度の差が大きいことは、例えば統計的有意差検定によって得られたp値が小さいこと等を指標にして判定することができる。第1選択処理では、複数の生物学的情報についてのアノテーションを、それぞれの生物学的情報毎に、付与の数又は頻度の差が大きいものから1種乃至10種を選択することが好ましい。
【0073】
(c)第2選択処理
第2選択処理では、入力手段105から入力された候補遺伝子の中から、第1選択処理で選択されたアノテーションが付与された遺伝子を選択する。複数のアノテーションが選択された場合には、該複数のアノテーションのうち1つでも付与された遺伝子を選択してもよく、該複数のアノテーションのうち特定のアノテーションの組み合わせが付与された遺伝子を選択してもよい。
【0074】
上述の第1選択処理により、複数のアノテーションが選択された場合には、選択されたアノテーションの付与数に従って、選択された遺伝子を順位付けすることも可能である。さらに、候補遺伝子群への付与の頻度又は数に従って重み付けをすることができる。候補遺伝子群への付与の頻度又は数と、対照遺伝子群への付与の頻度又は数との差に従ってアノテーションに重み付けを行って、遺伝子を順位付けしてもよい。
【0075】
また、遺伝子絞り込み装置101は、上述の手段に加えて出力手段109を備える。出力手段109は、処理手段107が実行する第2選択処理で選択された遺伝子又はタンパク質を、絞り込まれた遺伝子又はタンパク質として出力する。選択された遺伝子又はタンパク質は、ディスプレイ等の表示装置等や、プリンタ等の印字装置を用いて出力することができる。
【0076】
さらに、本発明では、コンピュータを用いる遺伝子絞り込み方法を提供する。本発明の遺伝絞り込み方法は、コンピュータを用いて複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み方法であって、コンピュータは、入力手段と、処理手段とを備え、(1)入力手段に、複数の候補遺伝子又は候補タンパク質を情報として入力する入力工程と、(2)複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続する接続工程と、(3)処理手段が、記憶手段に記憶されたデータウェアハウスから、入力工程で得られた候補遺伝子又は候補タンパク質に付与されたアノテーションを収集する収集工程と、(4)処理手段が、収集工程で収集されたアノテーションの中から、入力工程で入力された候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択工程と、(5)処理手段が、入力工程で入力された候補遺伝子又は候補タンパク質の中から、第1選択工程で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択工程と、を含むことを特徴とする。データウェアハウスや、データウェアハウスに格納される生物学的情報はインターネット経由で得られるものでよく、アノテーションはインターネット経由でデータウェアハウス又は各データベースから収集してもよい。
【0077】
また、別の遺伝絞り込み方法では、第1選択工程は、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択する工程である。また、記憶手段に記憶されたデータウェアハウスから、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として入力手段に追加して入力する再入力工程を含む。各工程は、上述した説明により明らかである。
【0078】
さらに、本発明は、コンピュータに該遺伝子絞り込み方法を実行させるコンピュータプログラム、及び該コンピュータプログラムが記憶された記憶媒体も提供する。本発明のコンピュータプログラムは、コンピュータに、複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込ませるコンピュータプログラムであって、コンピュータに、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続させる手順と、コンピュータに、データウェアハウスから候補遺伝子又は候補タンパク質に付与されたアノテーションを収集させる手順と、コンピュータに、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択させる手順と、コンピュータに、候補遺伝子又は候補タンパク質の中から、選択されたアノテーションが付与された遺伝子又はタンパク質を選択させる手順と、を実行させることを特徴とする。データウェアハウスや、データウェアハウスに格納される生物学的情報はインターネット経由で得られるものでよく、アノテーションはインターネット経由でデータウェアハウス又は各データベースから収集してもよい。
【0079】
また、別のコンピュータプログラムでは、コンピュータに、収集されたアノテーションの中から、候補遺伝子又は候補タンパク質に有意に多く付与されたアノテーションを、多く付与された該アノテーションから1又は複数選択させる手順を実行させる。また、別のコンピュータプログラムでは、コンピュータに、記憶手段に記憶されたデータウェアハウスから、入力手段から入力された候補遺伝子又は候補タンパク質と相互作用する遺伝子又はタンパク質を収集させ、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として入力手段に追加して入力させる手順を実行させる。各手順は、上述した説明により明らかである。
【実施例】
【0080】
さらに実施例を用いて本発明を詳細に説明するが、本発明はこれら実施例に限定されない。
【0081】
実施例1:既知の疾患関連遺伝子を用いた遺伝子絞り込みテスト
本発明を用いて、(1)膵炎、(2)高コレステロール血症、(3)肝硬変、及び(4)子宮頸癌について発生に関連すると知られている遺伝子を用いて遺伝子絞り込みテストを行った。
【0082】
本実施例のデータウェアハウスには、タンパク質の生物学的機能情報、タンパク質のパスウェイ情報、並びに疾患及び該疾患の原因となる遺伝子の情報が格納された。タンパク質の生物学機能情報はthe Gene Ontology、タンパク質のパスウェイ情報はKEEG Pathway、疾患及び該疾患の原因となる遺伝子の情報はOMIMから取得した。
【0083】
それぞれの疾患発生に関連することが既に知られている遺伝子(約30個)を正解遺伝子とし、さらに正解遺伝子の2倍数の遺伝子をランダムに抽出して正解遺伝子に加え、これら遺伝子群を候補遺伝子として入力手段から入力した。入力した遺伝子のリストを表1〜表4に示す。
【0084】
【表1】

【0085】
【表2】

【0086】
【表3】

【0087】
【表4】

【0088】
処理手段は、データウェアハウスに格納された、タンパク質の生物学機能情報(the Gene Ontology)タンパク質のパスウェイ情報(KEEG Pathway)、疾患及び該疾患の原因となる遺伝子の情報(OMIM)に基づき、入力された候補遺伝子に付与されたアノテーションを収集する収集手段を実行した。収集手段で収集された各疾患のアノテーション数(個数及び種類数)は、以下の表5の通りであった。
【0089】
【表5】

【0090】
続いて、処理手段は第1選択処理を実行した。対照遺伝子群は、ヒト遺伝子全体とした。上記収集処理において収集されたアノテーションの中から、候補遺伝子群に対するアノテーション付与の頻度が、対照群であるヒト遺伝子全体に対する付与の頻度と比較して有意に多く含まれるかどうかをp値<0.05を閾値として判定した。収集されたアノテーションのうち、ヒト遺伝子全体に付与されたアノテーションと比較して差が大きいもの上位10種を、差が大きい順(p値が小さい順)に、それぞれ表6〜表9に示す。表中のアノテーションが10種に満たないものは、p値<0.05となるアノテーションが10種存在しなかったことを示している。
【0091】
【表6】

【0092】
【表7】

【0093】
【表8】

【0094】
【表9】

【0095】
第1選択処理では、the Gene Ontology、KEEG Pathway、OMIMのそれぞれのアノテーションを、対照群との差が大きいものから、すなわちp値が小さいものから選択した。選択するアノテーション数は1種〜10種で変化させた。
【0096】
続いて、処理手段で第2選択処理を実行し、第1選択処理で選択されたアノテーションが一つでも付与された遺伝子を選択した。さらに、上記の第2選択処理で選択された遺伝子を、候補遺伝子から絞り込まれた遺伝子として出力手段から出力した。ここで、一例として、(1)膵炎で、第1選択処理でアノテーションをそれぞれ上位1個ずつ選択した場合に、第2選択処理において選択された遺伝子の遺伝子IDを表10に示す。
【0097】
【表10】

【0098】
さらに、上述の遺伝子のランダム抽出から絞り込みまでを合計10回繰り返した。それぞれの疾患の関連遺伝子として出力された遺伝子のうち、正解遺伝子の10回の絞り込みの平均感度及び平均特異度を図2に示す。グラフの横軸は、第1選択処理で選択されたアノテーションの数を示す。いずれの疾患についても、80%程度の感度及び特異度で正解遺伝子を出力することができた。
【0099】
実施例2:新規疾患関連遺伝子を対象とした絞り込み
本発明の遺伝子絞り込み装置を用いてC型肝炎の発生に関連する遺伝子の絞り込みを行った。C型肝炎ウイルス(HCV)Coreタンパク質とヒトのタンパク質であるPA28γとの相互作用がC型肝炎の発症に重要であることは知られていたが(非特許文献1参照)、詳しいメカニズムは不明であり、C型肝炎の発生に関連性を持ったヒト遺伝子は明らかでなかった。そこで、本発明を用いて候補遺伝子の絞り込みを行った。
【0100】
非特許文献1:Moriishi, K. et al., “Critical role of PA28γ
in hepatitis C virus-associated steatogenesis and hepatocarcinogenesis.”P.N.A.S.,
2007.
【0101】
データウェアハウスに格納された情報は、タンパク質間相互作用情報、タンパク質の生物学的機能情報、タンパク質のパスウェイ情報、並びに疾患及び該疾患の原因となる遺伝子の情報とした。タンパク質間の相互作用情報はBioGrid及びPPIview、タンパク質の生物学機能情報はthe Gene Ontology、タンパク質のパスウェイ情報はKEEG Pathway、疾患及び該疾患の原因となる遺伝子の情報はOMIM及びDisease Ontolgyから取得した。
【0102】
酵母2ハイブリッド法により、Human Adult liver library(MoBiTec社製)を用いて、HCVのCore又はNS4Bと相互作用する宿主のタンパク質のスクリーニングを行った。スクリーニングの結果、Coreと相互作用する11種のタンパク質、NS4Bと相互作用する45種のタンパク質が同定された。同定されたタンパク質を表11及び表12に示す。
【0103】
【表11】

【0104】
【表12】

【0105】
上記のタンパク質と相互作用するタンパク質を収集して候補遺伝子に追加した。処理手段は、データウェアハウスに格納されたタンパク質間相互作用情報を用いて、表11に記載のタンパク質と相互作用するタンパク質を196種、表12に記載のタンパク質と相互作用するタンパク質を207種それぞれ収集し、入力手段に追加して入力した。
【0106】
したがって、入力された候補遺伝子群は、(1)HCVのCoreと相互作用するタンパク質及び該タンパク質と相互作用するタンパク質(計:207種)、(2)HCVのNS4Bと相互作用するタンパク質及び該タンパク質と相互作用するタンパク質(計:252種)となった。入力された遺伝子のリストを表13〜15に示す。
【0107】
【表13】

【0108】
【表14】

【0109】
【表15】

【0110】
続いて、処理手段が収集処理を実行し、入力されたタンパク質に付与されたアノテーションを収集した。対照群はヒト遺伝子全体とした。収集したアノテーション数と、収集したアノテーションの中で、ヒト遺伝子全体と比較して候補遺伝子群(1)及び(2)に多く付与され、仮説検定の結果p値が0.05以下となったアノテーション数とを表16に示す。
【0111】
【表16】

【0112】
さらに、処理手段は、上記のアノテーションの中から、p値が小さいもの上位10種を選択する第1選択処理を行い、さらにタンパク質を選択する第2選択処理を行った。第2選択処理では、第1選択処理で選択されたアノテーションが付与され、且つ、タンパク質間相互作用情報でCore又はNS4Bがアノテーションとして付与された(すなわち、Core又はNS4Bに相互作用するタンパク質)ことをタンパク質選択の条件とした。第2選択処理で選択されたタンパク質を表17に示す。
【0113】
【表17】

【0114】
このように絞り込まれた遺伝子がC型肝炎の発生に関連する遺伝子であるかどうかを判定するため、表17に記載の遺伝子の中からSLC25A5及びENO1について実験的に検証を行った。SLC25A5及びENO1が相互作用するタンパク質であるPXNについても検証を行った。SLC25A5、ENO1及びPXNに対するsiRNAを、それぞれHuh7OK1細胞に導入し、24時間後にC型肝炎ウイルスJFH-1株(遺伝子型2a)をHuh7OK1細胞に感染させ、感染後細胞を72時間培養した。培養上清に含まれたウイルスRNAと、細胞中のGAPDHのmRNAとを定量的リアルタイムPCR法を用いて測定した。GAPDHのmRNA量に対するウイルスRNA量を図3に示す。培養上清に含まれたウイルスRNAの量はENO1のノックダウンにより顕著に減少した。また、SLC25A5のノックダウンにより、ウイルスRNAの量が統計上有意(p<0.01)に増加した。しかしながら、PXNのノックダウンでは、ウイルスRNAの量に有意差はみられなかった。
【0115】
また、他のC型肝炎ウイルスの遺伝子型についての影響を調べるため、JFH-1(遺伝子型2a)及びCon-1(遺伝子型1b)から得られたHCVレプリコンを含むHuh-7細胞に、それぞれの遺伝子に対するsiRNAを導入し、定量的リアルタイムPCR法を用いて培養上清のCon-1のウイルスRNA量と細胞中のGAPDHのmRNA量とを測定した。図4にGAPDHのmRNA量に対するCon-1のウイルスRNA量の割合を示す。ENO1及びPXNのノックダウンによりCon-1のHCVの複製が抑制された。
【0116】
以上のように、SLC25A5、ENO1、及びPXNはHCVの複製に関与するタンパク質であることが実験的に明らかとなった。したがって、本発明によって多数の候補遺伝子群から絞り込まれたSLC25A5及びENO1や、SLC25A5及びENO1と相互作用するPXNはC型肝炎の発生に関連性を有することがわかった。
【産業上の利用可能性】
【0117】
本発明の遺伝子絞り込み装置、遺伝子絞り込み装置、及びコンピュータプログラムを利用することで、新規疾患関連遺伝子の発見や、新規薬剤の開発に寄与することができる。
【符号の説明】
【0118】
101:遺伝子絞り込み装置
103:記憶手段
105:入力手段
107:処理手段
109:出力手段
【特許請求の範囲】
【請求項1】
複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み装置であって、
記憶手段と、入力手段と、処理手段とを備え、
前記記憶手段は、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスを記憶し、
前記入力手段は、複数の候補遺伝子又は候補タンパク質が情報として入力され、
前記処理手段は、
(a)前記記憶手段に記憶された前記データウェアハウスから、前記入力手段から入力された前記候補遺伝子又は前記候補タンパク質に付与されたアノテーションを収集する収集処理と、
(b)前記収集処理で収集されたアノテーションの中から、前記入力手段から得られた前記候補遺伝子又は前記候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択処理と、
(c)前記入力手段から得られた前記候補遺伝子又は前記候補タンパク質の中から、前記選択処理で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択処理と、を実行することを特徴とする、遺伝子絞り込み装置
【請求項2】
前記第1選択処理(b)における前記閾値より高いアノテーションは、前記入力手段から得られた前記候補遺伝子又は前記候補タンパク質に付与された頻度又は数が、複数の対照遺伝子又は対照タンパク質に付与された頻度又は数と比較して有意に多いアノテーションである、
請求項1に記載の遺伝子絞り込み装置
【請求項3】
前記第1選択処理(b)が、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択する処理である、
請求項2に記載の遺伝子絞り込み装置
【請求項4】
前記処理手段が、前記(a)〜(c)の各処理に加えて、
前記記憶手段に記憶された前記データウェアハウスから、前記入力手段から入力された前記候補遺伝子又は前記候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として前記入力手段に追加して入力する再入力処理を実行する、
請求項1〜3いずれか一項に記載の遺伝子絞り込み装置
【請求項5】
前記処理手段が、前記生物学的情報についてのアノテーションの冗長性を取り除く処理を実行する、
請求項1〜4いずれか一項に記載の遺伝子絞り込み装置
【請求項6】
前記処理手段が、前記生物学的情報についてのアノテーションの記述の形式を変換する処理を実行する、
請求項1〜5いずれか一項に記載の遺伝子絞り込み装置
【請求項7】
前記データウェアハウスに格納される複数の生物学的情報は、
遺伝子情報、遺伝子相同性情報、遺伝子多型情報、タンパク質情報、タンパク質間相互作用情報、タンパク質の生物学的機能情報、タンパク質ドメイン情報、タンパク質の立体構造情報、タンパク質発現情報、酵素機能情報、パスウェイ情報、転写因子情報、疾患及び該疾患の原因遺伝子情報、薬剤情報、並びに化合物情報からなる群から選択される複数の情報である、
請求項1〜6いずれか一項に記載の遺伝子絞り込み装置
【請求項8】
前記関連性は、疾患の発生についての関連性である、
請求項1〜7いずれか一項に記載の遺伝子絞り込み装置
【請求項9】
コンピュータを用いて複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み方法であって、
前記コンピュータは、入力手段と、処理手段とを備え、
(1)前記入力手段に、複数の候補遺伝子又は候補タンパク質を情報として入力する入力工程と、
(2)複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続する接続工程と、
(3)前記処理手段が、前前記データウェアハウスから、前記入力工程で得られた前記候補遺伝子又は前記候補タンパク質に付与されたアノテーションを収集する収集工程と、
(4)前記処理手段が、前記収集工程で収集されたアノテーションの中から、前記入力工程で入力された前記候補遺伝子又は前記候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択工程と、
(5)前記処理手段が、前記入力工程で入力された前記候補遺伝子又は前記候補タンパク質の中から、前記第1選択工程で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択工程と、
を含むことを特徴とする、遺伝子絞り込み方法
【請求項10】
コンピュータに、複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込ませるコンピュータプログラムであって、
コンピュータに、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続させる手順と、
前記データウェアハウスから、候補遺伝子又は候補タンパク質に付与されたアノテーションを収集させる手順と、
コンピュータに、前記収集されたアノテーションの中から、前記候補遺伝子又は前記候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択させる手順と、
コンピュータに、前記候補遺伝子又は前記候補タンパク質の中から、前記選択されたアノテーションが付与された遺伝子又はタンパク質を選択させる手順と、
を実行させることを特徴とする、コンピュータプログラム
【請求項1】
複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み装置であって、
記憶手段と、入力手段と、処理手段とを備え、
前記記憶手段は、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスを記憶し、
前記入力手段は、複数の候補遺伝子又は候補タンパク質が情報として入力され、
前記処理手段は、
(a)前記記憶手段に記憶された前記データウェアハウスから、前記入力手段から入力された前記候補遺伝子又は前記候補タンパク質に付与されたアノテーションを収集する収集処理と、
(b)前記収集処理で収集されたアノテーションの中から、前記入力手段から得られた前記候補遺伝子又は前記候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択処理と、
(c)前記入力手段から得られた前記候補遺伝子又は前記候補タンパク質の中から、前記選択処理で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択処理と、を実行することを特徴とする、遺伝子絞り込み装置
【請求項2】
前記第1選択処理(b)における前記閾値より高いアノテーションは、前記入力手段から得られた前記候補遺伝子又は前記候補タンパク質に付与された頻度又は数が、複数の対照遺伝子又は対照タンパク質に付与された頻度又は数と比較して有意に多いアノテーションである、
請求項1に記載の遺伝子絞り込み装置
【請求項3】
前記第1選択処理(b)が、有意に多く付与されたアノテーションを、多く付与された該アノテーションから選択する処理である、
請求項2に記載の遺伝子絞り込み装置
【請求項4】
前記処理手段が、前記(a)〜(c)の各処理に加えて、
前記記憶手段に記憶された前記データウェアハウスから、前記入力手段から入力された前記候補遺伝子又は前記候補タンパク質と相互作用する遺伝子又はタンパク質を収集し、該遺伝子又は該タンパク質を候補遺伝子又は候補タンパク質として前記入力手段に追加して入力する再入力処理を実行する、
請求項1〜3いずれか一項に記載の遺伝子絞り込み装置
【請求項5】
前記処理手段が、前記生物学的情報についてのアノテーションの冗長性を取り除く処理を実行する、
請求項1〜4いずれか一項に記載の遺伝子絞り込み装置
【請求項6】
前記処理手段が、前記生物学的情報についてのアノテーションの記述の形式を変換する処理を実行する、
請求項1〜5いずれか一項に記載の遺伝子絞り込み装置
【請求項7】
前記データウェアハウスに格納される複数の生物学的情報は、
遺伝子情報、遺伝子相同性情報、遺伝子多型情報、タンパク質情報、タンパク質間相互作用情報、タンパク質の生物学的機能情報、タンパク質ドメイン情報、タンパク質の立体構造情報、タンパク質発現情報、酵素機能情報、パスウェイ情報、転写因子情報、疾患及び該疾患の原因遺伝子情報、薬剤情報、並びに化合物情報からなる群から選択される複数の情報である、
請求項1〜6いずれか一項に記載の遺伝子絞り込み装置
【請求項8】
前記関連性は、疾患の発生についての関連性である、
請求項1〜7いずれか一項に記載の遺伝子絞り込み装置
【請求項9】
コンピュータを用いて複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込む遺伝子絞り込み方法であって、
前記コンピュータは、入力手段と、処理手段とを備え、
(1)前記入力手段に、複数の候補遺伝子又は候補タンパク質を情報として入力する入力工程と、
(2)複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続する接続工程と、
(3)前記処理手段が、前前記データウェアハウスから、前記入力工程で得られた前記候補遺伝子又は前記候補タンパク質に付与されたアノテーションを収集する収集工程と、
(4)前記処理手段が、前記収集工程で収集されたアノテーションの中から、前記入力工程で入力された前記候補遺伝子又は前記候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択する第1選択工程と、
(5)前記処理手段が、前記入力工程で入力された前記候補遺伝子又は前記候補タンパク質の中から、前記第1選択工程で選択されたアノテーションが付与された遺伝子又はタンパク質を選択する第2選択工程と、
を含むことを特徴とする、遺伝子絞り込み方法
【請求項10】
コンピュータに、複数の候補遺伝子又は候補タンパク質から任意の関連性を有する遺伝子又はタンパク質を絞り込ませるコンピュータプログラムであって、
コンピュータに、複数の生物学的情報が格納され、一の遺伝子又はタンパク質に該情報から得られた複数のアノテーションが付与されたデータウェアハウスに接続させる手順と、
前記データウェアハウスから、候補遺伝子又は候補タンパク質に付与されたアノテーションを収集させる手順と、
コンピュータに、前記収集されたアノテーションの中から、前記候補遺伝子又は前記候補タンパク質における付与の頻度又は数が閾値より高いアノテーションを1又は複数選択させる手順と、
コンピュータに、前記候補遺伝子又は前記候補タンパク質の中から、前記選択されたアノテーションが付与された遺伝子又はタンパク質を選択させる手順と、
を実行させることを特徴とする、コンピュータプログラム
【図1】


【図2】


【図3】


【図4】


【図5】




【図2】


【図3】


【図4】


【図5】


【公開番号】特開2012−69104(P2012−69104A)
【公開日】平成24年4月5日(2012.4.5)
【国際特許分類】
【出願番号】特願2011−184289(P2011−184289)
【出願日】平成23年8月26日(2011.8.26)
【出願人】(505314022)独立行政法人医薬基盤研究所 (17)
【Fターム(参考)】
【公開日】平成24年4月5日(2012.4.5)
【国際特許分類】
【出願日】平成23年8月26日(2011.8.26)
【出願人】(505314022)独立行政法人医薬基盤研究所 (17)
【Fターム(参考)】
[ Back to top ]

