重複ゲノム領域を有する菌
【課題】アスペルギルス属に属する菌の染色体上の任意のゲノム領域を重複させる技術に基づき、従来は取得不能であった新たな形質を持つ麹菌の取得を安定的・計画的に可能にすること。
【解決手段】同一染色体上でタンデムにゲノム重複領域を有すること、更に、ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれていることを特徴とする、アスペルギルス属に属する菌の形質転換菌であって、プロテアーゼ等の醤油製造における必要な多種の酵素群を高生産する菌。
【解決手段】同一染色体上でタンデムにゲノム重複領域を有すること、更に、ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれていることを特徴とする、アスペルギルス属に属する菌の形質転換菌であって、プロテアーゼ等の醤油製造における必要な多種の酵素群を高生産する菌。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、アスペルギルス属に属する形質転換菌であって、重複するゲノム領域を同一染色体上に有する菌に関する。
【背景技術】
【0002】
アスペルギルス・ソーヤ(Aspergillus sojae )及びアスペルギルス・オリゼ(Aspergillus oryzae)等の麹菌は、醤油、酒、味噌などの伝統的な食品の醸造や酵素の生産等のために工業的に広く用いられている、また近年の麹菌(アスペルギルス・オリゼ)の全ゲノム配列の決定やマイクロアレイを用いた網羅的な遺伝子発現解析などの進展に伴い、遺伝子工学的な改変、特に染色体レベルの改変により酵素等の生産性や増殖速度の改良などの効果が期待される糸状菌である。
【0003】
更に、麹菌が生産する酵素は様々な産業に利用されている。例えば、日本の伝統食品である醤油製造においても、麹菌の生産する多様な酵素が利用されている。醤油製造では、麹菌を原料である大豆と小麦に生育させ、多様な酵素を生産させる。ここで麹菌が生産した多様な酵素は、大豆や小麦のタンパク質、糖質、脂質などを分解し、次工程の乳酸発酵、酵母発酵を促す。この過程で、麹菌が原料分解酵素を多量に生産すると、原料利用率や圧搾性が上がり生産性を大幅に向上させることができる。加えて、乳酸発酵、酵母発酵への基質が十分に供給されるため、発酵が適正に行なわれ、醤油の品質は大きく向上する。
【0004】
Aspergillus nidulans, niger, fumigatus, awamoriなどのアスペルギルス属菌が単核の世代を持つのに対し、アスペルギルス・ソーヤ及びアスペルギルス・オリゼ等の麹菌は分生子の状態も含めてその生活環において常に多核の状態を保ち、これまでのところ有性世代が確認されておらず、親細胞から娘細胞への核の分配機構についても解明されていない。そのために、菌株間の交配やRIP(repeat induced mutation)等の手段によって新たな変異株を作成することが出来ず、遺伝学的研究が困難であり、上記のように産業的に極めて有用な菌であるにもかかわらず、遺伝的解析は遅れていた。
【0005】
これらのことから、多種の酵素の生産性の高い等の有用な麹菌を育種することは産業上極めて重要であり、これを目的とした育種が現在までに精力的に行なわれている。このような麹菌の育種方法には、大きく分けて突然変異法と遺伝子組換え法がある。
【0006】
突然変異法にはX線、紫外線、重イオンビーム等の各種変異処理が用いられ、そこから有用な性質を指標としたスクリーニングを行うことにより、様々な酵素活性や、醸造特性等の優れた株が作出されてきた。近年、ゲノム情報を利用してこれらの有用な形質を持つ菌株を解析し、得られた知見により染色体の重複が麹菌に好ましい性質を付与するのに重要であることが判明しており、突然変異処理によって900kb以上の大規模なゲノム領域の重複をもつ麹菌が得られている(特許文献1)。又、最近の研究により、ガンマ線照射により得られた酵母を解析すると、染色体の重複領域の境界領域に高頻度で繰り返し配列が存在することが報告された(非特許文献1)。
【0007】
しかしながら、このような染色体の重複のメカニズムは解明されていない。従って、変異処理を用いた従来の方法では、酵素活性等を指標としてスクリーニングを行った際に、たまたまその活性に関係のある染色体上の領域に重複を持つ菌株が得られるだけで、実際には様々な染色体上の部位でランダムに変異が起こるため、麹菌の染色体上の特定の箇所を狙って重複させることは不可能であった。又、このような変異を利用して得られた染色体の重複株の場合には、それらの間の相同配列間の組換えにより復帰変異(すなわち染色体重複の脱落)が高い頻度で生じることも知られている。
【0008】
一方で、遺伝子組換え法は形質転換を利用して目的のDNAを麹菌に取り込ませることにより育種する方法であり、従来の遺伝子組換え法で取り込ませる遺伝子のサイズは5〜6kbが通常である。しかしながら、上記のような酵素生産性の高い等の有用な麹菌を実際に得るには、目的とする遺伝子領域に加えて、該遺伝子のプロモーター領域、ターミネーター領域、場合によってはスクリーニング時のマーカーとなる遺伝子が取り込ませるDNA領域に含まれている必要があり、これらの領域を併せると通常は10kb以上にも及ぶ大きな領域になることが多い。
【0009】
以上のように、染色体上の任意の位置で比較的大きな領域の重複を起こすことが出来る技術はこれまで存在していない。
【先行技術文献】
【特許文献】
【0010】
【特許文献1】特許第4469014号公報
【非特許文献】
【0011】
【非特許文献1】Argueso et al. (2008) Proc Natl Acad Sci U S A. 105:11845-11850.
【発明の概要】
【発明が解決しようとする課題】
【0012】
上記のとおり、特許文献1に記載されたような変異処理を用いた従来の方法では、実際には様々な染色体上の部位でランダムに変異が起こるため、麹菌の染色体上の特定の箇所を狙って重複させることは不可能である。更に、このような従来方法によって同一染色体上にゲノム重複領域が生じた場合には、その形質転換菌を培養する過程で相同組換えによって復帰変異(すなわち染色体重複の脱落)が生じる。従って、特許文献1に記載の発明では復帰変異しない株が得られているが、これは重複したゲノム領域が元の染色体とは別の染色体に存在している為と考えられる。実際に、特許文献1では、ゲノム重複領域に含まれる遺伝子の活性及び発現量が増加していることは定量的に確認されているが、重複ゲノムが染色体上のどの位置にあるかは確認されていない。
【0013】
本発明は上記の問題点を解決し、麹菌の染色体上の任意のゲノム領域を重複させる技術に基づき、従来は取得不能であった新たな形質を持つ麹菌の取得を安定的・計画的に可能にすることを目的とする。
【課題を解決するための手段】
【0014】
本発明者は、アスペルギルス属に属する菌において、pyrG遺伝子等の形質転換マーカー(選択マーカー)遺伝子の配列を染色体中の任意の領域を挟むように該領域の5’及び3’末端の外側に配する形質転換体を作製することによって、該遺伝子の内部の相同領域で発生した二重鎖切断後の修復機能を介した相同組換えによって染色体の重複が起き、そのような株を該形質転換マーカーに基づき選抜することによって、狙った染色体領域において同一染色体上でのゲノムの一部の領域に重複(ゲノム重複領域を有する)が起きた株(形質転換菌)を作製することが出来ることを見出し、本発明を完成した。
【0015】
即ち、本発明は以下の各態様に係る。
[態様1]
アスペルギルス属に属する菌の形質転換菌であって、同一染色体上でタンデムにゲノム重複領域(重複ゲノム領域)を有することを特徴とする、前記形質転換菌。
[態様2]
ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれていることを特徴とする、態様1記載の形質転換菌。
[態様3]
ゲノム重複領域が十〜数百kbである、態様1又は2記載の形質転換菌。
[態様4]
アスペルギルス属に属する菌がアスペルギルス・オリゼまたはアスペルギルス・ソーヤである、態様1ないし3のいずれか一項に記載の形質転換菌。
[態様5]
ゲノム重複領域がアスペルギルス・オリゼの2番染色体のSC003領域の500kb以上で700kb未満である、態様4記載の形質転換菌。
[態様6]
ゲノム重複領域がアスペルギルス・オリゼの2番染色体のSC003領域におけるAO090003001035〜AO090003001214に挟まれた領域であって、これらの端のORF領域は含まない領域である、態様5記載の形質転換菌。
[態様7]
形質転換マーカー遺伝子が、pyrG、sC及びniaDから成る群から選択される、態様1ないし6のいずれかに記載の形質転換菌。
[態様8]
染色体重複を持たないコントロール株(Ct株)と比べて、プロテアーゼが1.9倍以上、及び/又はα―アミラーゼ活性が1.4倍以上に増加していることを特徴とする、態様1ないし7のいずれか一項に記載の形質転換菌。
[態様9]
遺伝子組換え法によって作製されたものであることを特徴とする、態様1ないし8のいずれか一項に記載の形質転換菌。
[態様10]
態様1ないし9のいずれか一項に記載の形質転換菌を用いて製造される醤油。
【発明の効果】
【0016】
本発明によって、麹菌の同一染色体上において十〜数百kb(例えば、500kb以上で700kb未満)に及ぶ任意の領域の重複を起こさせることが可能となった。このような技術は従来存在しなかった。また本発明は宿主に内在性の遺伝子しか使用しておらず、作出した重複株中にも外来遺伝子が残らないセルフクローニング株であることから、醤油の醸造等、食品製造に使用する微生物(例えば、醤油麹菌)の育種法としても極めて優れた方法である。
【0017】
更に、発明方法によって製造された染色体領域の重複を有する形質転換菌においては、タンデムに並ぶ相同配列間の組換えによりゲノム重複領域が脱落した場合、これらのゲノム重複領域に挟まれた領域に組み込まれているpyrG等の形質転換マーカー遺伝子が一緒に脱落して特定の栄養要求(例えば、ウリジン要求)性となり、その結果、通常の培地では生育不可能であるため、選択圧がかかり、染色体重複の脱落を回避することが可能である。
【図面の簡単な説明】
【0018】
【図1】本発明方法の概略を示す。
【図2】5’ΔpyrGあるいは3’ΔpyrGユニットの作製を示す。
【図3】2番染色体中の染色体重複のtarget領域を示す。
【図4】PCRによる同一染色体上のゲノム領域の確認の結果を示す。
【図5】定量PCRを用いた2番染色体の重複領域における各遺伝子のコピー数を示す。
【発明を実施するための形態】
【0019】
本発明は、アスペルギルス属に属する菌の形質転換菌であって、同一染色体上でタンデムにゲノム重複領域を有することを特徴とする、前記形質転換菌に係る。この形質転換菌の主要な特徴に一つとして、ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれていることを挙げることが出来る。この結果、発明方法の形質転換菌においては、タンデムに並ぶ相同配列間の組換えによりゲノム重複領域が脱落した場合、ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれているpyrG等の形質転換マーカー遺伝子が一緒に脱落して特定の栄養要求(例えば、ウリジン要求)性となり、その結果、通常の培地では生育不可能であるため、選択圧がかかり、染色体重複の脱落を回避することができるのである。
【0020】
本発明の形質転換菌の親株として使用するアスペルギルス属に属する菌としては、アスペルギルス・ソーヤ、アスペルギルス・オリゼ、アスペルギルス・ニガー、アスペルギルス・アワモリ等の任意の菌株が挙げられるが、そのうちアスペルギルス・ソーヤ及びアスペルギルス・オリゼに属する菌株が好ましい。
【0021】
このような菌株としては、例えば、アスペルギルス・ソーヤ 262(FERM P−2188)、アスペルギルス・ソーヤ 2165(FERM P−7280)、アスペルギルス・ソーヤ (ATCC42251)、アスペルギルス・オリゼ(IAM2638)、及びアスペルギルス・オリゼRIB40(NBRC100959)等の公的寄託機関で保存されており当業者には容易に入手可能であるような各菌株等を挙げることができる。
【0022】
外来DNAの染色体への組込みは染色体DNAの二重鎖切断時の修復機構を介して行われることが知られており、このDNA修復機構には相同組換えと非相同組換え(非相同末端結合)の2種類の機構が存在する。相同組換えの場合には外来DNAと相同性のある領域を介して組込みが起こるが、非相同組換えの場合には外来DNAの配列には関係なく染色体上のランダムな位置への組込みが起こり、これら2つの組換え機構は平衡して作用していると考えられている(Ristic et al. Nucl. Acids Res. (2003) 31: 5229-5237)。
【0023】
相同組換え機構の中心をなす遺伝子はrad52グループと呼ばれる一連の遺伝子でその中にrad50、51、52、54、Mre11、XRS2等が含まれる(Kooistra et al. 2004)。相同組換え機構はバクテリアから真核生物まで多くの生物種で存在が確認され、Aspergillus属の実験室株であり単核分生子を持つAspergillus nidulansにおいてもuvsC遺伝子がクローニングされ研究が進められており(van Heemst et al. Mol. Gen. Genet. (1997)254: 654-64)、発現頻度を一定レベル上昇させることで相同組換頻度が向上することが報告されている(Natsume et al. Biosci. Biotechnol. Biochem. (2004) 68: 1649-1656)。
【0024】
一方で、非相同組換え機構は相同組換えとは全く異なる非相同末端結合(Non-Homologous End Joining)によることがに明らかとなっており、この機構の中心になる遺伝子としてはKu70、Ku80、Xrcc4、LIG4、DNAPKcsなどが知られている。Ku70およびKu80はヘテロダイマーとして機能し、ヌクレオチドキナーゼ(XRCC4)およびDNA LigaseVIとともに複合体を形成して、DNA二重鎖切断(DSB)時にその修復のためにDNA末端に結合してNon-Homologous End Joiningを促進することが知られている。(Walker et al. Nature (2001) 412: 607-614)。このKuを介した非相同組換え機構に関しては真核生物でのみ存在が確認されている。
【0025】
本発明の形質転換菌におけるゲノム重複領域は任意に選択することが出来る。その領域の大きさは、好ましくは十〜数百kbである。このようなゲノム重複領域の好適例としては、プロテアーゼ及びα―アミラーゼ等の産業上有用な各種酵素をコードする遺伝子が多数含まれているアスペルギルス・オリゼの2番染色体のSC003領域におけるゲノム領域、例えば、500kb以上で700kb未満のゲノム領域を挙げることができる。特に、ゲノム重複領域として、アスペルギルス・オリゼの2番染色体のSC003領域におけるAO090003001035〜AO090003001214に挟まれた領域であって、これらの端のORF領域は含まない領域が挙げられる。このような形質転換菌の好適例として、以下の実施例に記載されている、A-C-585k株、B-C-517k株、及びB-C4-512k株を挙げることが出来る。
【0026】
本発明の形質転換菌は、染色体重複を持たないコントロール株(Ct株)と比べて、産業上有用な各種酵素の発現量が増加している。例えば、アスペルギルス・オリゼの形質転換菌においてはプロテアーゼ活性が1.8倍以上、好ましくは3.1倍以上、及び/又はα―アミラーゼ活性が1.4倍以上、好ましくは1.6倍以上に増加していることを特徴とする。
【0027】
本発明の形質転換菌において、重複の対象となるゲノム領域は任意に選択することができるので、有用な酵素類をコードする公知の各種領域を重複のターゲット領域とすることによって、醤油の醸造等、食品製造に有用な麹菌(例えば、醤油麹菌)を得ることが出来る。従って、本発明は、このような形質転換菌を用いて製造される醤油等の食品にも係るものである。
【0028】
本発明の形質転換菌の作製方法・手段に特に制限はない。好適方法の一つとして、以下に示すような形質転換体を用いる遺伝子組換え法を挙げることが出来る。
【0029】
即ち、本発明の遺伝子組換え法に使用する形質転換体は、重複を目的とする麹菌染色体上のターゲット領域の5’ 末端又は3’末端のいずれか一方の末端の外側に、コード領域の5’ 末端又は3’末端のいずれか一方の一部が欠損した形質転換マーカー遺伝子が組み込まれ、該ターゲット領域の他方の末端の外側に他方の末端の一部が欠損した形質転換マーカー遺伝子が組み込まれて成り、該ターゲット領域が、夫々、コード領域の5’又は3’末端の一部が欠損した2つの形質転換マーカー遺伝子によって挟み込まれ、且つ、組み込まれた形質転換マーカー遺伝子の欠損部分に接する配列(欠損部分を有する各末端領域配列)が該ターゲット領域とは反対側に位置していることを特徴とする。
【0030】
即ち、重複を目的とするアスペルギルス属に属する菌の染色体上のターゲット領域の5’末端の外側にコード領域の5’末端の一部(適当な長さの塩基配列から成る5’末端領域)が欠損した形質転換マーカー遺伝子を組み込み、一方、ターゲット領域の3’末端の一部(適当な長さの塩基配列から成る3’末端領域)の外側にコード領域の3’末端の一部が欠損した形質転換マーカー遺伝子を組み込むことが出来る。或いは、重複を目的とするアスペルギルス属に属する菌の染色体上のターゲット領域の5’末端の外側にコード領域の3’末端の一部が欠損した形質転換マーカー遺伝子を組み込み、一方、ターゲット領域の3’末端の外側にコード領域の5’末端の一部が欠損した形質転換マーカー遺伝子を組み込んでもよい。
【0031】
いずれの場合でも、重複の対象となるターゲット領域を挟み込んでいる、夫々、コード領域の5’又は3’末端の一部が欠損した2つの形質転換マーカー遺伝子において、欠損した部分に接する配列が該ターゲット領域とは反対側に位置していることが必要である。
【0032】
このような構造上の特徴を有する形質転換体を培養することによって、対応する染色体の間で、夫々、ターゲット領域の5’ 末端又は3’末端の外側に組み込まれた形質転換マーカー遺伝子のコード領域の中間部分に共通して存在する相同配列領域(図1,図2及び図4中で斜線で示した部分)間における二重鎖切断時の修復機構を介する相同組換えが生起し、その結果、該ターゲット領域が重複された菌株が得られる。ここで、「対応する染色体」とは、実施例で示されるように、形質転換体の多核に含まれる相同染色体、例えば、各菌において有用物質(例えば、各種酵素類)をコードする遺伝子が多数含まれている、複数の8及び2番等の染色体を意味する。
【0033】
従って、本発明で使用するアスペルギルス属に属する菌としては、上記のような非相同組換え機構に関与する各種遺伝子が抑制又は欠失している形質転換菌(特開2006−158269号公報)を使用することもできる。
【0034】
又、該形質転換体における該ターゲット領域の5’及び3’末端の外側に組み込まれる形質転換マーカー遺伝子は、相同組換えを効率よく生起するのに十分な長さ、例えば、数百bp以上、数百bp〜数kb程度(例えば、約100bp〜約2kb)の塩基配列がコード領域の中間部分に残るように、そのコード領域の5’末端又は3’末端の一部が欠損していることが好ましい。即ち、重複を目的とするアスペルギルス属に属する菌の染色体のターゲット領域の5’及び3’末端の外側に組み込まれた、コード領域の5’末端又は3’末端の一部が欠損している2つの形質転換マーカー遺伝子のいずれにおいても、元の形質転換マーカー遺伝子のコード領域の中間部分に存在する上記の長さの塩基配列が共通して残っていることが好ましい。従って、形質転換マーカー遺伝子のコード領域において欠損させる5’末端又は3’末端領域の塩基配列の長さは、使用する形質転換マーカー遺伝子の種類及び全長等に応じて、当業者が適宜決めることが出来る。例えば、pyrG遺伝子を使用する場合には、通常、例えば、約0.4kb〜約1.4kbである。又、欠損させる5’末端又は3’末端領域の塩基配列の長さは同じである必要はない。
【0035】
このような形質転換体を培養した場合に、上記の相同組換えによって該ターゲット領域が重複された菌株においては同時に形質転換マーカー遺伝子コード領域の完全長が構築される結果、形質転換マーカー遺伝子に基づく形質によって、該ターゲット領域が重複された菌株を該ターゲット領域が重複されていない(即ち、形質転換マーカー遺伝子コード領域の完全長が構築されていない)菌株から選択すること出来る。
【0036】
本発明で使用する形質転換マーカーに特に制限はないが、ポジティブ選択可能なマーカー遺伝子の代表例として、pyrG、sC及びniaD等を挙げることが出来、これらマーカー遺伝子を使用した際には、夫々の栄養要求性(ウリジン要求、イオウ資化、及び、硝酸資化)を補償する形質によって該ターゲット領域が重複された菌株を選択することが可能となる。
【0037】
尚、これらのマーカー遺伝子は、選択用の薬剤を含む培地で培養することによって、該マーカー遺伝子を含有する菌はそこに含まれている該マーカー遺伝子の発現産物によって選択用の薬剤が細胞毒性物質に変換され細胞死に至らしめることによって、ネガティブ選択にも使用することが出来る。
【0038】
更に、重複を目的とする染色体上のターゲット領域の5’及び3’末端の外側に組み込む形質転換マーカー遺伝子のいずれか一方又はその両方のコード領域の中間部分に存在する相同配列領域に予め、I-sceI、I-ceuI、PI-pspI及びPI-sceI等の当業者に公知の適当な制限酵素認識部位を導入しておくことが出来る。このような制限酵素部位は、相同組換え等の当業者に公知の任意の手段によって導入することが出来る。
【0039】
上記の形質転換体を使用して本発明の形質転換菌を製造する遺伝子組換え法の好適例は以下の通りである。尚、この方法の概略は図1に示したとおりである。即ち、上記方法は、
(1)上記の特徴を有する形質転換体を培養し、
(2)ターゲット領域の5’ 末端及び3’末端の外側に組み込まれた形質転換マーカー遺伝子のコード領域の中間部分に共通して存在する相同配列領域間における二重鎖切断時の修復機構を介した、該形質転換体の対応する染色体の間での相同組換えによって該ターゲット領域が重複された菌株を得、
(3)上記の相同組換えによってコード領域の完全長が構築された該形質転換マーカー遺伝子に基づく形質によって、該ターゲット領域が重複された菌株を選択することから成る。
【0040】
この遺伝子組換え法は、上記の形質転換体を通常の条件で培養することによって、形質転換マーカー遺伝子のコード領域の中間部分に共通して存在する相同配列領域間において二重鎖の切断が適当な頻度で生じた時に働く修復機構を介した相同組換えを利用するものである。上記方法において、形質転換体を当業者に公知の任意の方法で一度プロトプラストの状態にして適当な条件下で一定時間(例えば、数十分間〜1時間程度)保持し、その後、培養することによって相同組換えの効率を高めることが出来る。尚、アスペルギルス・ソーヤ及びアスペルギルス・オリゼ等の麹菌は分生子の状態も含めてその生活環において常に多核の状態を保っているので、対応する染色体の間で上記の相同組換えが容易に起こると考えられる。一方、アスペルギルス属のその他の菌、例えば、Aspergillus nidulans, niger, fumigatus, awamori等は単核の世代を持つが、それらの菌であっても菌糸からであれば多核状態のプロトプラストを容易に得ることができる。
【0041】
更に、コード領域の中間部分に存在する相同配列領域に、予め、I-sceI、I-ceuI、PI-pspI及びPI-sceI等の当業者に公知の適当な制限酵素認識部位が導入された形質転換マーカー遺伝子を使用する場合には、該制限酵素の作用下に形質転換体を培養して相同組換えを生起させることが出来る。具体的には、例えば、プロトプラストの状態にした形質転換体と制限酵素を融合補助剤(例えば、PEG等)の存在下で混合すること(プロトプラストPEG法)によって、制限酵素を該形質転換体に効率よく作用させることが可能である。
【0042】
尚、上記の形質転換体は、本願明細書の実施例に記載されているような当業者の公知の手段を用いて作製することが出来る。又、形質転換体の培養は当業者に公知の適当な条件で実施することが出来る。
【0043】
以下、実施例に基づき本発明を更に詳細に説明するが、本発明の技術的範囲はこれら実施例に限定されるものではない。
【実施例1】
【0044】
以下の通り、麹菌2番染色体重複株の作製を行った。
【0045】
[実験方法]
使用菌株
Aspergillus oryzae RP-1株(ΔpyrG)。Aspergillus oryzae RP-1株はRIB40(=ATCC42149) 由来のpyrG deletion株(Takahashi et al. (2006) Biosci Biotechnol Biochem. 70:135-143)。
【0046】
使用培地
ポリペプトンデキストリン(PD)培地(polypepton 1%, dextrin 2%, KH2PO4 0.5%, NaNO3 0.1%, MgSO4 0.05%, casamino acid 0.1%, pH 6.0)、CzapekDox (CZ) 最小培地、再生培地として1.2M ソルビトール CZを使用した。1.5 mg/ml 5fluoroortic acid(5FOA) (シグマ社) および20mM ウリジンを含むCZ培地プレートをpyrG-(ウリジン要求)株のポジティブセレクション用の培地として使用した。
【0047】
形質転換
150ml容三角フラスコ中の20mM Uridineを含むポリペプトンデキストリン液体培地50mlに分生子を接種し、30℃で約20時間振とう培養を行い、菌体を回収した。回収した菌体を0.7 M KCl bufferで洗浄し、1% Lysing enzyme(シグマ社)を含む0.7M KCl buffer中で30℃、3時間緩やかに振とうし、プロトプラストを調製した。得られたプロトプラストを1.2 Mソルビトール bufferで洗浄した後、プロトプラストPEG法により形質転換を行った。形質転換体の再生は0.5%agarを含む1.2Mソルビトール-CZ培地上で行った。
【0048】
染色体重複株の作製
染色体重複株の作製は、以下の通りに行った。約2x 107/100μl量のプロトプラスト溶液に対し、20μlのPEG溶液を加えた後、氷中に40分保持した後、70μlのPEG溶液を加え、さらに室温で20分保持した後、1.2Mソルビトール-CZ培地プレート上で再生させた。また、I-sceIを使用する場合には、PEG溶液の添加と同時に加えた。CZ培地上で生育の見られた株を染色体重複候補株として以後の解析に使用した。
【0049】
定量PCR(リアルタイムPCR)を用いた染色体中の遺伝子コピー数の比較
定量PCRはMx3005P(アジレント・テクノロジー社)を用いて行った。他の染色体に位置するrad52をノーマライザーとして相対定量法により、2番染色体上のAlp、amyR、prtT、1258Dの各遺伝子のコピー数を親株と染色体重複株との間で比較した。PCRの条件は95℃で10分保持した後、95℃20秒−58℃30秒−72℃30秒を45サイクル繰り返した。PCRにはRad52、Alp、amyR、prtT、1258Dの各遺伝子の増幅にはそれぞれr52U-r52L、amyRU-amyRL、AlpU-AlpL、prtTU-prtTL、1258DU-1258DLの各プライマーを使用した(表1)。
【0050】
【表1】

【0051】
ふすま麹の作製法
麹菌の酵素活性評価は定法に従って行なった。すなわち、80%散水した小麦ふすま5gを150ml容三角フラスコに入れ、121℃、50分滅菌した後、麹菌を2白金耳程度接種し30℃、4日間培養する。培養後、滅菌水100mlを入れ、ゴム栓をして十分に振とうし、4時間室温にて静置した後、No.2の濾紙(アドバンテック社製)で濾過して得られた抽出液を酵素サンプルとした。
【0052】
プロテアーゼ活性の測定法
得られた酵素サンプルを適宜希釈し、「しょうゆ試験法」(財団法人 日本醤油研究所 昭和60年、287ページ)に記載の方法に従って測定した。プロテアーゼ活性は、ふすま麹1g当り1分間に1μモルのチロシンを生成する活性を1U(ユニットまたは単位)として示した。
【0053】
α-アミラーゼ活性の測定法
得られた酵素サンプルを適宜希釈し、α-アミラーゼ測定キット(キッコーマン醸造分析キット、コード60213)を用い、キットのプロトコールに従って測定を行なった。α-アミラーゼ活性は、ふすま麹1g当り1分間に1μモルの2-クロロ-4-ニトロフェノールを遊離する力価を1U(ユニットまたは単位)として示した。
【0054】
[染色体重複株の作製]
染色体の重複は染色体の二重鎖切断修復の過程で生じると考えられているが、そのメカニズムは判っていない。しかし酵母においてガンマ線照射によって取得された変異株の染色体を解析すると染色体の重複が起きた領域の境界付近に繰り返し配列が存在することが報告されている(非特許文献1)ことから、相同領域を持つ5’ΔpyrGおよび3’ΔpyrGで重複ターゲット領域を挟んだコンストラクトを作製し、相同配列内部で二重鎖切断を引き起こすことにより、染色体の重複を作製出来ると考えられる(図1)。
【0055】
そこで、まず、5’ΔpyrGおよび3’ΔpyrGを作製するために、図2に示すような基本ユニットを作製した。5’ΔpyrGあるいは3’ΔpyrGユニットの作製は PCRとライゲーションを用いて行った。pyrGユニットの作製は、当業者に公知の方法を用いて、pyrG全長の転写開始点付近(5’ΔpyrGの場合)あるいはコード領域の3’側付近(3’ΔpyrGの場合)に図2中で斜線で示す相同配列(適当な制限酵素認識部位を含んでいても良い)を組込むことにより行った。各ベクターが染色体上に組込まれた後5FOA耐性株を選択(ネガティブ選択)することにより、それぞれ相同領域での組換えにより内部が切り出された5’ΔpyrGあるいは3’ΔpyrG株が得られる。
【0056】
次に5’ΔpyrGおよび3’ΔpyrGそれぞれのユニットを染色体上のターゲットとする領域に組込むためのベクターを構築した。標的とする領域は図3に示すように、A.oryzaeの2番染色体中のSC003領域のAO090003001003〜AO090003001258の各ORFの領域に対応する部分を含む700 kbの領域(図3、斜線部)のうちのB領域からC4の領域に渡るAO090003001036〜AO090003001212の各ORFの領域に対応する部分を含む512 kbの領域(AO090003001035〜AO090003001214に挟まれた領域であって、これらの端のORF領域は含まない領域)である。本明細書内では以後簡略化のため、各遺伝子名のAO090003000部分を省略し、Bで表現する(例:AO090003000160 → B160)。SC003領域と対応する各遺伝子の配列は独立行政法人製品評価技術基盤機構(NITE)のゲノム解析データベースであるDOGAN(Database Of the Genomes Analyzed at NITE)に基づいている。(http://www.bio.nite.go.jp/dogan/GeneMap?GENOME_ID=ao__G2)。
【0057】
図3中の2番染色体上のB領域に5’ΔpyrGのコンストラクトを組込むためのベクター(pB-5’Δ)とC4領域に3’ΔpyrGのコンストラクトを組込むためのベクター(pC4-3’Δ)の構築を、以下の通り行った。
【0058】
まずプライマーB-UとB-LおよびC4-UとC4-Lを用いてA.oryzaeのゲノムDNAよりPCRによりB領域の近傍およびC4領域の近傍を含む約3 kbの領域を増幅し、ベクターにクローニングした。
【0059】
プライマーB-iUおよびB-iLを用いて増幅したB領域の近傍を含むベクターと、プライマーpyrUおよびpyrLを用いて増幅した5’ΔpyrGユニットとを、In-fusion クローニングキット(Takara社)を用いてライゲーションし、2番染色体上のB領域に5’ΔpyrGのコンストラクトを組込むためのベクター(pB-5’Δ)を構築した。ライゲーションはキットの所定のプロトコールにしたがって行なった。続いてプライマーC4-iUおよびC4-iLを用いて増幅したC4領域を含むベクターと、プライマーpyrUおよびpyrLを用いて増幅した3’ΔpyrGユニットとを、In-fusionクローニングキット(Takara社)を用いてライゲーションし、C4領域に3’ΔpyrGのコンストラクトを組込むためのベクター(pC4-3’Δ)を構築した。このベクターを作製するのに使用したプライマーを表2に示す。
【0060】
【表2】

【0061】
まず2番染色体のB領域に5’ΔpyrGのコンストラクトを組込むためのベクター(pB-5’Δ)をプライマーB-UおよびB-Lを用いて増幅し、そのPCR産物を用いてA.oryzaeのΔpyrG株に対して形質転換を行った。得られた形質転換体をPCRおよびサザンハイブリダイゼーションにより調べた結果、1株で目的部位にベクターが組込まれていることが確認された。次にこの株から分生子を採取し、約1×105を5FOAを含むCZ培地プレート上へ塗布し、得られた5FOA耐性株を解析したところ、B領域に5’ΔpyrGのコンストラクトを持つことが確認された。このB領域に5’ΔpyrGのコンストラクトを持つΔpyrG株を親株として、C4領域(512 kb領域重複用)に3’ΔpyrGのコンストラクトを組込むためのベクター(pC4-3’Δ)をプライマーC4-UおよびC4-Lを用いたPCRにより増幅した後、形質転換を行った。C4領域にベクターが組込まれた株をPCRおよびサザンハイブリダイゼーションを用いて選抜して分生子を回収し、5FOA−CZプレート上で耐性を示すコロニーを選択した結果、重複ターゲット領域中(512 kb領域)の最もセントロメア側であるB領域に5`ΔpyrGが組込まれ、最もテロメア側であるC4領域に3`ΔpyrGが組込まれた株の構築が確認された。こうして2番染色体中の512 kbの領域を重複させるための基本的なコンストラクトを持った株(B-C4-hap株)の構築に成功した(図3)。
【0062】
次に、このB-C4-hap株を親株として、前述の方法により、染色体重複株の取得実験を行った。その結果、再生プレートから1個のpyrG+のコロニーが得られたので、これをB-C4-512k株とした。続いて、得られたpyrG+株(B-C4-512k株)から染色体DNAを抽出し、PCRおよび定量PCR(リアルタイムPCR)により、染色体が重複しているかどうかの確認を試みた。
【0063】
PCRによる染色体重複の確認
まず、PCRにより狙った染色体上の領域の重複が起きているかどうかの確認を行なった。その結果を図4に示す。重複領域のセントロメア側の端に位置し、ベクター(pB-5’Δ)が組込まれているB領域の近傍領域を増やすためのプライマー(B-UとB-L)を用いた場合(図4上)には、コントロール株(親株:B-C4-hap株)、重複株(B-C4-512k株)ともに4.4 kbのバンドの増幅が見られた(図4下、レーン1および2)。また重複領域のテロメア側の端に位置し、ベクター(pC4-3’Δ)が組込まれているC4領域の近傍領域を増やすためのプライマー(C4-UとC4-L)を用いた場合(図4上)にも、コントロール、重複株ともに3.9 kbのバンドの増幅が見られた(図4下、レーン5および6)。一方、プライマーC4-UとB-Lの組合わせでPCRを行った場合(図4上)にはコントロールでは全くバンドの増幅が見られないのに対し、染色体重複株では、はっきりと4.8 kbのバンドの増幅が見られた(レーン3および4)。染色体重複株でのみ見られた4.8kbのバンドは、染色体上の狙った領域が同一染色体上で重複し、図4下に示すような構造が染色体上に存在する場合にのみ得られるバンドであると考えられる。このことから、染色体重複株B-C4-512k株では2番染色体上のB領域からC4領域に渡る512 kbの領域(B1036〜B1212)が同一染色体上で重複していることが示された。
【0064】
リアルタイムPCRによるコピー数の確認
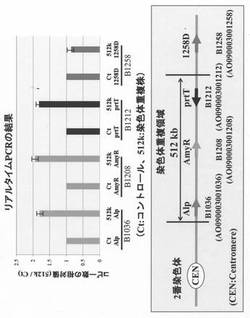
続いてリアルタイムPCR(定量PCR)を用いて2番染色体の重複領域における各遺伝子のコピー数をコントロール株(親株:B-C4-hap株)および染色体重複株(B-C4-512k株)で比較した(図5)。実験では他の染色体に存在し、コピー数が1であるrad52をノーマライザーとして使用し、重複領域内のセントロメア側の端に位置するB1036遺伝子(Alp)とそこから490 kb離れた位置に存在するB1208(amyR)遺伝子およびテロメア側の端に位置するB1212(prtT)遺伝子、そして重複領域外に位置するB1258D遺伝子をターゲットとして、SYBR Greenを用いた相対的定量法により、親株におけるコピー数との比較を行った。図5にはコントロール株(B-C4-hap株)でのコピー数に対する相対値を示す。その結果、染色体重複株であるB-C4-512k株では、染色体重複領域外に位置するB1258Dの相対値は約1程度であったが、重複領域の内部に位置するB1036遺伝子、B1208遺伝子、B1212遺伝子についてはいずれもコントロール株でのコピー数に対して2倍程度の値を示した。これらの結果から染色体重複株(B-C4-512k株)においては、重複領域内に位置する遺伝子のコピー数が2に増幅していることが確認された。これらのPCRおよびリアルタイムPCR(定量PCR)の結果から、染色体重複株(B-C4-512k株)では2番染色体上の狙った領域である512 kbの領域が確かに重複していることが確認された。
【0065】
さらに同様の方法により、図3中のA-D領域間の700 kb、A-C領域間の585 kb、B-C領域間の517 kb、A-C2領域間の573 kb、B-B3領域間の10 kbの重複株を取得するために、A領域に5’ΔpyrGのコンストラクトを、B3領域、C2領域、C領域、D領域にはそれぞれ3’ΔpyrGのコンストラクトを導入するためのベクターを構築した。ベクターの構築には、それぞれ、A領域にはプライマーA-U、A-L、A-iU、A-iLを、B3領域にはプライマーB3-U、B3-L、B3-iU、B3-iLを、C領域にはプライマーC-U、C-L、C-iU、C-iLを、C2領域にはプライマーC2-U、C2-L、C2-iU、C2-iLを、D領域にはプライマーD-U、D-L、D-iU、D-iLを使用し、ΔpyrGユニットとのライゲーションはIn-fusionクローニングキット(Takara社)を用いて行った。得られたベクターを前述の方法と同様にPCRにより増幅した後、それらをA.oryzaeのΔpyrG株に対して形質転換することにより、染色体重複用の親株を作製した後、A-D領域700kbの重複株(A-D-700k株)、A-C領域585kbの重複株(A-C-585k株)、B-C領域517kbの重複株(B-C-517k株)、A-C2領域573kbの重複株(A-C2-573k株)、B-B3領域10kbの重複株(B-B3-10k株)を取得した。これらの重複株の作製に使用したプライマーを表3に示す。これらの菌株に関しても、染色体重複株B-C4-512k株と同様に、2番染色体上の各領域が同一染色体上で重複していることが示された。
【0066】
【表3】

【0067】
活性の測定
2番染色体の重複株については、特許文献1(特許第4469014号「大規模ゲノム重複を保持する麹菌」)で、麹菌の2番染色体上のSC003のAO090003001003〜AO090003001259に対応するゲノム領域の重複株で、プロテアーゼ活性およびアミラーゼ活性の顕著な上昇が見られることが報告されている。本実験で2番染色体を重複させた重複株について、同様の活性上昇が見られるかどうかを調べるために、酵素活性の測定を行なった。酵素活性の比較対照とするために、本実験での方法を用いて2番染色体上のAO090003001003〜AO090003001258(B1003〜B1258)に対応する700 kbの領域を重複させたA-D-700k株を用いた(図3)。さらに同様の方法を用いて作製した重複株であるA-C-585k株、B-C−517k株、A-C2−573k株、B-B2-10k株を用いて酵素活性の測定を行った。
【0068】
プロテアーゼ活性測定の結果
前述の方法によって、ふすま麹より得られた酵素抽出液を用いて全プロテアーゼ活性の測定を行い、染色体重複を持たないコントロール株(Ct株)と重複株とで比較を行なった。その結果を表4に示す。プロテアーゼ活性については、コントロール株(Ct株)の活性に対し、染色体重複株(A-C4-512k株)では約3倍に活性値が上昇していた。また、この値は700 kbの領域の染色体重複株(A-D-700k株)で得られた活性値であるとほぼ同等であった。このことから本発明で作製した512 kbの領域の染色体重複株(B-C4-512k株)では、ふすま培養において、700 kbの領域の染色体株重複(A-D-700k株)と同様のプロテアーゼ活性の上昇が見られることが判明した。また、同様の方法で作製したA-C領域間575kbの重複株(A-C-575k株)、B-C領域間517kbの重複株(B-C-517k株)、においても同様のプロテアーゼ活性の上昇が確認された。一方、B-B3領域間10kbの重複株(B-B3-10k株)およびA-C2領域間の573kbの重複株(A-C2-573k株)においてはプロテアーゼ活性の上昇は2倍弱であった。
【0069】
【表4】

【0070】
α‐アミラーゼ活性測定の結果
続いてα‐アミラーゼ活性についても同一の酵素液を用いて測定を行なった。その結果を表5に示す。その結果、コントロール株(Ct株)の活性値に対し、染色体重複株(B-C4-512k株)での活性値は約1.6倍程度に上昇していることが明らかとなった。この値は700 kbの領域の染色体重複株(A-D-700k株)において得られた値とほぼ同じであった。またB-B3-10k株以外の染色体重複株(A-C-585k株、B-C-517k株、A-C2-573k株)においても同様のアミラーゼ活性の上昇が見られた。このことから本発明で作製した512 kbの領域の染色体重複株(B-C4-512k株)では、ふすま培養において、700 kbの領域の染色体株重複(A-D-700k株)と同様のアミラーゼ活性の上昇が見られることが判明した。
【0071】
【表5】

【0072】
以上の結果より、本実施例で作製した各形質転換菌においては2番染色体の同一染色体上で重複領域が見られた。例えば、B-C4-512k株では2番染色体のB1036-B1212間512 kbの領域が同一染色体上で確かに重複しており、ふすま培養において、B1003-B1258間700 kbの領域の染色体重複株(A-D-700k株)と同様に、染色体重複のない親株に比較して、顕著なプロテアーゼ活性およびアミラーゼ活性の上昇が見られることが判った。また、同様の方法で作製したA-C領域間575kbの重複株(A-C-575k株)、B-C領域間517kbの重複株(B-C-517k株)、においても同等のプロテアーゼ活性の上昇、およびアミラーゼ活性の上昇が確認された。このことから、2番染色体の重複によるプロテアーゼ活性およびアミラーゼ活性の上昇には、B1003-B1258間700 kbの領域より短い領域の重複で十分であることが示された。
【産業上の利用可能性】
【0073】
本発明により、育種の対象である菌株のゲノム情報を利用して実用上有用であると考えられる染色体の領域を特定し、その特定ゲノム領域を同一染色体上でタンデムに重複させた菌株を提供することが可能となり、効率的に分子育種が進められると考えられる。更に、従来知られていなかった全く新たな有用な形質が強化された麹菌が得られるものと期待される。
【技術分野】
【0001】
本発明は、アスペルギルス属に属する形質転換菌であって、重複するゲノム領域を同一染色体上に有する菌に関する。
【背景技術】
【0002】
アスペルギルス・ソーヤ(Aspergillus sojae )及びアスペルギルス・オリゼ(Aspergillus oryzae)等の麹菌は、醤油、酒、味噌などの伝統的な食品の醸造や酵素の生産等のために工業的に広く用いられている、また近年の麹菌(アスペルギルス・オリゼ)の全ゲノム配列の決定やマイクロアレイを用いた網羅的な遺伝子発現解析などの進展に伴い、遺伝子工学的な改変、特に染色体レベルの改変により酵素等の生産性や増殖速度の改良などの効果が期待される糸状菌である。
【0003】
更に、麹菌が生産する酵素は様々な産業に利用されている。例えば、日本の伝統食品である醤油製造においても、麹菌の生産する多様な酵素が利用されている。醤油製造では、麹菌を原料である大豆と小麦に生育させ、多様な酵素を生産させる。ここで麹菌が生産した多様な酵素は、大豆や小麦のタンパク質、糖質、脂質などを分解し、次工程の乳酸発酵、酵母発酵を促す。この過程で、麹菌が原料分解酵素を多量に生産すると、原料利用率や圧搾性が上がり生産性を大幅に向上させることができる。加えて、乳酸発酵、酵母発酵への基質が十分に供給されるため、発酵が適正に行なわれ、醤油の品質は大きく向上する。
【0004】
Aspergillus nidulans, niger, fumigatus, awamoriなどのアスペルギルス属菌が単核の世代を持つのに対し、アスペルギルス・ソーヤ及びアスペルギルス・オリゼ等の麹菌は分生子の状態も含めてその生活環において常に多核の状態を保ち、これまでのところ有性世代が確認されておらず、親細胞から娘細胞への核の分配機構についても解明されていない。そのために、菌株間の交配やRIP(repeat induced mutation)等の手段によって新たな変異株を作成することが出来ず、遺伝学的研究が困難であり、上記のように産業的に極めて有用な菌であるにもかかわらず、遺伝的解析は遅れていた。
【0005】
これらのことから、多種の酵素の生産性の高い等の有用な麹菌を育種することは産業上極めて重要であり、これを目的とした育種が現在までに精力的に行なわれている。このような麹菌の育種方法には、大きく分けて突然変異法と遺伝子組換え法がある。
【0006】
突然変異法にはX線、紫外線、重イオンビーム等の各種変異処理が用いられ、そこから有用な性質を指標としたスクリーニングを行うことにより、様々な酵素活性や、醸造特性等の優れた株が作出されてきた。近年、ゲノム情報を利用してこれらの有用な形質を持つ菌株を解析し、得られた知見により染色体の重複が麹菌に好ましい性質を付与するのに重要であることが判明しており、突然変異処理によって900kb以上の大規模なゲノム領域の重複をもつ麹菌が得られている(特許文献1)。又、最近の研究により、ガンマ線照射により得られた酵母を解析すると、染色体の重複領域の境界領域に高頻度で繰り返し配列が存在することが報告された(非特許文献1)。
【0007】
しかしながら、このような染色体の重複のメカニズムは解明されていない。従って、変異処理を用いた従来の方法では、酵素活性等を指標としてスクリーニングを行った際に、たまたまその活性に関係のある染色体上の領域に重複を持つ菌株が得られるだけで、実際には様々な染色体上の部位でランダムに変異が起こるため、麹菌の染色体上の特定の箇所を狙って重複させることは不可能であった。又、このような変異を利用して得られた染色体の重複株の場合には、それらの間の相同配列間の組換えにより復帰変異(すなわち染色体重複の脱落)が高い頻度で生じることも知られている。
【0008】
一方で、遺伝子組換え法は形質転換を利用して目的のDNAを麹菌に取り込ませることにより育種する方法であり、従来の遺伝子組換え法で取り込ませる遺伝子のサイズは5〜6kbが通常である。しかしながら、上記のような酵素生産性の高い等の有用な麹菌を実際に得るには、目的とする遺伝子領域に加えて、該遺伝子のプロモーター領域、ターミネーター領域、場合によってはスクリーニング時のマーカーとなる遺伝子が取り込ませるDNA領域に含まれている必要があり、これらの領域を併せると通常は10kb以上にも及ぶ大きな領域になることが多い。
【0009】
以上のように、染色体上の任意の位置で比較的大きな領域の重複を起こすことが出来る技術はこれまで存在していない。
【先行技術文献】
【特許文献】
【0010】
【特許文献1】特許第4469014号公報
【非特許文献】
【0011】
【非特許文献1】Argueso et al. (2008) Proc Natl Acad Sci U S A. 105:11845-11850.
【発明の概要】
【発明が解決しようとする課題】
【0012】
上記のとおり、特許文献1に記載されたような変異処理を用いた従来の方法では、実際には様々な染色体上の部位でランダムに変異が起こるため、麹菌の染色体上の特定の箇所を狙って重複させることは不可能である。更に、このような従来方法によって同一染色体上にゲノム重複領域が生じた場合には、その形質転換菌を培養する過程で相同組換えによって復帰変異(すなわち染色体重複の脱落)が生じる。従って、特許文献1に記載の発明では復帰変異しない株が得られているが、これは重複したゲノム領域が元の染色体とは別の染色体に存在している為と考えられる。実際に、特許文献1では、ゲノム重複領域に含まれる遺伝子の活性及び発現量が増加していることは定量的に確認されているが、重複ゲノムが染色体上のどの位置にあるかは確認されていない。
【0013】
本発明は上記の問題点を解決し、麹菌の染色体上の任意のゲノム領域を重複させる技術に基づき、従来は取得不能であった新たな形質を持つ麹菌の取得を安定的・計画的に可能にすることを目的とする。
【課題を解決するための手段】
【0014】
本発明者は、アスペルギルス属に属する菌において、pyrG遺伝子等の形質転換マーカー(選択マーカー)遺伝子の配列を染色体中の任意の領域を挟むように該領域の5’及び3’末端の外側に配する形質転換体を作製することによって、該遺伝子の内部の相同領域で発生した二重鎖切断後の修復機能を介した相同組換えによって染色体の重複が起き、そのような株を該形質転換マーカーに基づき選抜することによって、狙った染色体領域において同一染色体上でのゲノムの一部の領域に重複(ゲノム重複領域を有する)が起きた株(形質転換菌)を作製することが出来ることを見出し、本発明を完成した。
【0015】
即ち、本発明は以下の各態様に係る。
[態様1]
アスペルギルス属に属する菌の形質転換菌であって、同一染色体上でタンデムにゲノム重複領域(重複ゲノム領域)を有することを特徴とする、前記形質転換菌。
[態様2]
ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれていることを特徴とする、態様1記載の形質転換菌。
[態様3]
ゲノム重複領域が十〜数百kbである、態様1又は2記載の形質転換菌。
[態様4]
アスペルギルス属に属する菌がアスペルギルス・オリゼまたはアスペルギルス・ソーヤである、態様1ないし3のいずれか一項に記載の形質転換菌。
[態様5]
ゲノム重複領域がアスペルギルス・オリゼの2番染色体のSC003領域の500kb以上で700kb未満である、態様4記載の形質転換菌。
[態様6]
ゲノム重複領域がアスペルギルス・オリゼの2番染色体のSC003領域におけるAO090003001035〜AO090003001214に挟まれた領域であって、これらの端のORF領域は含まない領域である、態様5記載の形質転換菌。
[態様7]
形質転換マーカー遺伝子が、pyrG、sC及びniaDから成る群から選択される、態様1ないし6のいずれかに記載の形質転換菌。
[態様8]
染色体重複を持たないコントロール株(Ct株)と比べて、プロテアーゼが1.9倍以上、及び/又はα―アミラーゼ活性が1.4倍以上に増加していることを特徴とする、態様1ないし7のいずれか一項に記載の形質転換菌。
[態様9]
遺伝子組換え法によって作製されたものであることを特徴とする、態様1ないし8のいずれか一項に記載の形質転換菌。
[態様10]
態様1ないし9のいずれか一項に記載の形質転換菌を用いて製造される醤油。
【発明の効果】
【0016】
本発明によって、麹菌の同一染色体上において十〜数百kb(例えば、500kb以上で700kb未満)に及ぶ任意の領域の重複を起こさせることが可能となった。このような技術は従来存在しなかった。また本発明は宿主に内在性の遺伝子しか使用しておらず、作出した重複株中にも外来遺伝子が残らないセルフクローニング株であることから、醤油の醸造等、食品製造に使用する微生物(例えば、醤油麹菌)の育種法としても極めて優れた方法である。
【0017】
更に、発明方法によって製造された染色体領域の重複を有する形質転換菌においては、タンデムに並ぶ相同配列間の組換えによりゲノム重複領域が脱落した場合、これらのゲノム重複領域に挟まれた領域に組み込まれているpyrG等の形質転換マーカー遺伝子が一緒に脱落して特定の栄養要求(例えば、ウリジン要求)性となり、その結果、通常の培地では生育不可能であるため、選択圧がかかり、染色体重複の脱落を回避することが可能である。
【図面の簡単な説明】
【0018】
【図1】本発明方法の概略を示す。
【図2】5’ΔpyrGあるいは3’ΔpyrGユニットの作製を示す。
【図3】2番染色体中の染色体重複のtarget領域を示す。
【図4】PCRによる同一染色体上のゲノム領域の確認の結果を示す。
【図5】定量PCRを用いた2番染色体の重複領域における各遺伝子のコピー数を示す。
【発明を実施するための形態】
【0019】
本発明は、アスペルギルス属に属する菌の形質転換菌であって、同一染色体上でタンデムにゲノム重複領域を有することを特徴とする、前記形質転換菌に係る。この形質転換菌の主要な特徴に一つとして、ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれていることを挙げることが出来る。この結果、発明方法の形質転換菌においては、タンデムに並ぶ相同配列間の組換えによりゲノム重複領域が脱落した場合、ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれているpyrG等の形質転換マーカー遺伝子が一緒に脱落して特定の栄養要求(例えば、ウリジン要求)性となり、その結果、通常の培地では生育不可能であるため、選択圧がかかり、染色体重複の脱落を回避することができるのである。
【0020】
本発明の形質転換菌の親株として使用するアスペルギルス属に属する菌としては、アスペルギルス・ソーヤ、アスペルギルス・オリゼ、アスペルギルス・ニガー、アスペルギルス・アワモリ等の任意の菌株が挙げられるが、そのうちアスペルギルス・ソーヤ及びアスペルギルス・オリゼに属する菌株が好ましい。
【0021】
このような菌株としては、例えば、アスペルギルス・ソーヤ 262(FERM P−2188)、アスペルギルス・ソーヤ 2165(FERM P−7280)、アスペルギルス・ソーヤ (ATCC42251)、アスペルギルス・オリゼ(IAM2638)、及びアスペルギルス・オリゼRIB40(NBRC100959)等の公的寄託機関で保存されており当業者には容易に入手可能であるような各菌株等を挙げることができる。
【0022】
外来DNAの染色体への組込みは染色体DNAの二重鎖切断時の修復機構を介して行われることが知られており、このDNA修復機構には相同組換えと非相同組換え(非相同末端結合)の2種類の機構が存在する。相同組換えの場合には外来DNAと相同性のある領域を介して組込みが起こるが、非相同組換えの場合には外来DNAの配列には関係なく染色体上のランダムな位置への組込みが起こり、これら2つの組換え機構は平衡して作用していると考えられている(Ristic et al. Nucl. Acids Res. (2003) 31: 5229-5237)。
【0023】
相同組換え機構の中心をなす遺伝子はrad52グループと呼ばれる一連の遺伝子でその中にrad50、51、52、54、Mre11、XRS2等が含まれる(Kooistra et al. 2004)。相同組換え機構はバクテリアから真核生物まで多くの生物種で存在が確認され、Aspergillus属の実験室株であり単核分生子を持つAspergillus nidulansにおいてもuvsC遺伝子がクローニングされ研究が進められており(van Heemst et al. Mol. Gen. Genet. (1997)254: 654-64)、発現頻度を一定レベル上昇させることで相同組換頻度が向上することが報告されている(Natsume et al. Biosci. Biotechnol. Biochem. (2004) 68: 1649-1656)。
【0024】
一方で、非相同組換え機構は相同組換えとは全く異なる非相同末端結合(Non-Homologous End Joining)によることがに明らかとなっており、この機構の中心になる遺伝子としてはKu70、Ku80、Xrcc4、LIG4、DNAPKcsなどが知られている。Ku70およびKu80はヘテロダイマーとして機能し、ヌクレオチドキナーゼ(XRCC4)およびDNA LigaseVIとともに複合体を形成して、DNA二重鎖切断(DSB)時にその修復のためにDNA末端に結合してNon-Homologous End Joiningを促進することが知られている。(Walker et al. Nature (2001) 412: 607-614)。このKuを介した非相同組換え機構に関しては真核生物でのみ存在が確認されている。
【0025】
本発明の形質転換菌におけるゲノム重複領域は任意に選択することが出来る。その領域の大きさは、好ましくは十〜数百kbである。このようなゲノム重複領域の好適例としては、プロテアーゼ及びα―アミラーゼ等の産業上有用な各種酵素をコードする遺伝子が多数含まれているアスペルギルス・オリゼの2番染色体のSC003領域におけるゲノム領域、例えば、500kb以上で700kb未満のゲノム領域を挙げることができる。特に、ゲノム重複領域として、アスペルギルス・オリゼの2番染色体のSC003領域におけるAO090003001035〜AO090003001214に挟まれた領域であって、これらの端のORF領域は含まない領域が挙げられる。このような形質転換菌の好適例として、以下の実施例に記載されている、A-C-585k株、B-C-517k株、及びB-C4-512k株を挙げることが出来る。
【0026】
本発明の形質転換菌は、染色体重複を持たないコントロール株(Ct株)と比べて、産業上有用な各種酵素の発現量が増加している。例えば、アスペルギルス・オリゼの形質転換菌においてはプロテアーゼ活性が1.8倍以上、好ましくは3.1倍以上、及び/又はα―アミラーゼ活性が1.4倍以上、好ましくは1.6倍以上に増加していることを特徴とする。
【0027】
本発明の形質転換菌において、重複の対象となるゲノム領域は任意に選択することができるので、有用な酵素類をコードする公知の各種領域を重複のターゲット領域とすることによって、醤油の醸造等、食品製造に有用な麹菌(例えば、醤油麹菌)を得ることが出来る。従って、本発明は、このような形質転換菌を用いて製造される醤油等の食品にも係るものである。
【0028】
本発明の形質転換菌の作製方法・手段に特に制限はない。好適方法の一つとして、以下に示すような形質転換体を用いる遺伝子組換え法を挙げることが出来る。
【0029】
即ち、本発明の遺伝子組換え法に使用する形質転換体は、重複を目的とする麹菌染色体上のターゲット領域の5’ 末端又は3’末端のいずれか一方の末端の外側に、コード領域の5’ 末端又は3’末端のいずれか一方の一部が欠損した形質転換マーカー遺伝子が組み込まれ、該ターゲット領域の他方の末端の外側に他方の末端の一部が欠損した形質転換マーカー遺伝子が組み込まれて成り、該ターゲット領域が、夫々、コード領域の5’又は3’末端の一部が欠損した2つの形質転換マーカー遺伝子によって挟み込まれ、且つ、組み込まれた形質転換マーカー遺伝子の欠損部分に接する配列(欠損部分を有する各末端領域配列)が該ターゲット領域とは反対側に位置していることを特徴とする。
【0030】
即ち、重複を目的とするアスペルギルス属に属する菌の染色体上のターゲット領域の5’末端の外側にコード領域の5’末端の一部(適当な長さの塩基配列から成る5’末端領域)が欠損した形質転換マーカー遺伝子を組み込み、一方、ターゲット領域の3’末端の一部(適当な長さの塩基配列から成る3’末端領域)の外側にコード領域の3’末端の一部が欠損した形質転換マーカー遺伝子を組み込むことが出来る。或いは、重複を目的とするアスペルギルス属に属する菌の染色体上のターゲット領域の5’末端の外側にコード領域の3’末端の一部が欠損した形質転換マーカー遺伝子を組み込み、一方、ターゲット領域の3’末端の外側にコード領域の5’末端の一部が欠損した形質転換マーカー遺伝子を組み込んでもよい。
【0031】
いずれの場合でも、重複の対象となるターゲット領域を挟み込んでいる、夫々、コード領域の5’又は3’末端の一部が欠損した2つの形質転換マーカー遺伝子において、欠損した部分に接する配列が該ターゲット領域とは反対側に位置していることが必要である。
【0032】
このような構造上の特徴を有する形質転換体を培養することによって、対応する染色体の間で、夫々、ターゲット領域の5’ 末端又は3’末端の外側に組み込まれた形質転換マーカー遺伝子のコード領域の中間部分に共通して存在する相同配列領域(図1,図2及び図4中で斜線で示した部分)間における二重鎖切断時の修復機構を介する相同組換えが生起し、その結果、該ターゲット領域が重複された菌株が得られる。ここで、「対応する染色体」とは、実施例で示されるように、形質転換体の多核に含まれる相同染色体、例えば、各菌において有用物質(例えば、各種酵素類)をコードする遺伝子が多数含まれている、複数の8及び2番等の染色体を意味する。
【0033】
従って、本発明で使用するアスペルギルス属に属する菌としては、上記のような非相同組換え機構に関与する各種遺伝子が抑制又は欠失している形質転換菌(特開2006−158269号公報)を使用することもできる。
【0034】
又、該形質転換体における該ターゲット領域の5’及び3’末端の外側に組み込まれる形質転換マーカー遺伝子は、相同組換えを効率よく生起するのに十分な長さ、例えば、数百bp以上、数百bp〜数kb程度(例えば、約100bp〜約2kb)の塩基配列がコード領域の中間部分に残るように、そのコード領域の5’末端又は3’末端の一部が欠損していることが好ましい。即ち、重複を目的とするアスペルギルス属に属する菌の染色体のターゲット領域の5’及び3’末端の外側に組み込まれた、コード領域の5’末端又は3’末端の一部が欠損している2つの形質転換マーカー遺伝子のいずれにおいても、元の形質転換マーカー遺伝子のコード領域の中間部分に存在する上記の長さの塩基配列が共通して残っていることが好ましい。従って、形質転換マーカー遺伝子のコード領域において欠損させる5’末端又は3’末端領域の塩基配列の長さは、使用する形質転換マーカー遺伝子の種類及び全長等に応じて、当業者が適宜決めることが出来る。例えば、pyrG遺伝子を使用する場合には、通常、例えば、約0.4kb〜約1.4kbである。又、欠損させる5’末端又は3’末端領域の塩基配列の長さは同じである必要はない。
【0035】
このような形質転換体を培養した場合に、上記の相同組換えによって該ターゲット領域が重複された菌株においては同時に形質転換マーカー遺伝子コード領域の完全長が構築される結果、形質転換マーカー遺伝子に基づく形質によって、該ターゲット領域が重複された菌株を該ターゲット領域が重複されていない(即ち、形質転換マーカー遺伝子コード領域の完全長が構築されていない)菌株から選択すること出来る。
【0036】
本発明で使用する形質転換マーカーに特に制限はないが、ポジティブ選択可能なマーカー遺伝子の代表例として、pyrG、sC及びniaD等を挙げることが出来、これらマーカー遺伝子を使用した際には、夫々の栄養要求性(ウリジン要求、イオウ資化、及び、硝酸資化)を補償する形質によって該ターゲット領域が重複された菌株を選択することが可能となる。
【0037】
尚、これらのマーカー遺伝子は、選択用の薬剤を含む培地で培養することによって、該マーカー遺伝子を含有する菌はそこに含まれている該マーカー遺伝子の発現産物によって選択用の薬剤が細胞毒性物質に変換され細胞死に至らしめることによって、ネガティブ選択にも使用することが出来る。
【0038】
更に、重複を目的とする染色体上のターゲット領域の5’及び3’末端の外側に組み込む形質転換マーカー遺伝子のいずれか一方又はその両方のコード領域の中間部分に存在する相同配列領域に予め、I-sceI、I-ceuI、PI-pspI及びPI-sceI等の当業者に公知の適当な制限酵素認識部位を導入しておくことが出来る。このような制限酵素部位は、相同組換え等の当業者に公知の任意の手段によって導入することが出来る。
【0039】
上記の形質転換体を使用して本発明の形質転換菌を製造する遺伝子組換え法の好適例は以下の通りである。尚、この方法の概略は図1に示したとおりである。即ち、上記方法は、
(1)上記の特徴を有する形質転換体を培養し、
(2)ターゲット領域の5’ 末端及び3’末端の外側に組み込まれた形質転換マーカー遺伝子のコード領域の中間部分に共通して存在する相同配列領域間における二重鎖切断時の修復機構を介した、該形質転換体の対応する染色体の間での相同組換えによって該ターゲット領域が重複された菌株を得、
(3)上記の相同組換えによってコード領域の完全長が構築された該形質転換マーカー遺伝子に基づく形質によって、該ターゲット領域が重複された菌株を選択することから成る。
【0040】
この遺伝子組換え法は、上記の形質転換体を通常の条件で培養することによって、形質転換マーカー遺伝子のコード領域の中間部分に共通して存在する相同配列領域間において二重鎖の切断が適当な頻度で生じた時に働く修復機構を介した相同組換えを利用するものである。上記方法において、形質転換体を当業者に公知の任意の方法で一度プロトプラストの状態にして適当な条件下で一定時間(例えば、数十分間〜1時間程度)保持し、その後、培養することによって相同組換えの効率を高めることが出来る。尚、アスペルギルス・ソーヤ及びアスペルギルス・オリゼ等の麹菌は分生子の状態も含めてその生活環において常に多核の状態を保っているので、対応する染色体の間で上記の相同組換えが容易に起こると考えられる。一方、アスペルギルス属のその他の菌、例えば、Aspergillus nidulans, niger, fumigatus, awamori等は単核の世代を持つが、それらの菌であっても菌糸からであれば多核状態のプロトプラストを容易に得ることができる。
【0041】
更に、コード領域の中間部分に存在する相同配列領域に、予め、I-sceI、I-ceuI、PI-pspI及びPI-sceI等の当業者に公知の適当な制限酵素認識部位が導入された形質転換マーカー遺伝子を使用する場合には、該制限酵素の作用下に形質転換体を培養して相同組換えを生起させることが出来る。具体的には、例えば、プロトプラストの状態にした形質転換体と制限酵素を融合補助剤(例えば、PEG等)の存在下で混合すること(プロトプラストPEG法)によって、制限酵素を該形質転換体に効率よく作用させることが可能である。
【0042】
尚、上記の形質転換体は、本願明細書の実施例に記載されているような当業者の公知の手段を用いて作製することが出来る。又、形質転換体の培養は当業者に公知の適当な条件で実施することが出来る。
【0043】
以下、実施例に基づき本発明を更に詳細に説明するが、本発明の技術的範囲はこれら実施例に限定されるものではない。
【実施例1】
【0044】
以下の通り、麹菌2番染色体重複株の作製を行った。
【0045】
[実験方法]
使用菌株
Aspergillus oryzae RP-1株(ΔpyrG)。Aspergillus oryzae RP-1株はRIB40(=ATCC42149) 由来のpyrG deletion株(Takahashi et al. (2006) Biosci Biotechnol Biochem. 70:135-143)。
【0046】
使用培地
ポリペプトンデキストリン(PD)培地(polypepton 1%, dextrin 2%, KH2PO4 0.5%, NaNO3 0.1%, MgSO4 0.05%, casamino acid 0.1%, pH 6.0)、CzapekDox (CZ) 最小培地、再生培地として1.2M ソルビトール CZを使用した。1.5 mg/ml 5fluoroortic acid(5FOA) (シグマ社) および20mM ウリジンを含むCZ培地プレートをpyrG-(ウリジン要求)株のポジティブセレクション用の培地として使用した。
【0047】
形質転換
150ml容三角フラスコ中の20mM Uridineを含むポリペプトンデキストリン液体培地50mlに分生子を接種し、30℃で約20時間振とう培養を行い、菌体を回収した。回収した菌体を0.7 M KCl bufferで洗浄し、1% Lysing enzyme(シグマ社)を含む0.7M KCl buffer中で30℃、3時間緩やかに振とうし、プロトプラストを調製した。得られたプロトプラストを1.2 Mソルビトール bufferで洗浄した後、プロトプラストPEG法により形質転換を行った。形質転換体の再生は0.5%agarを含む1.2Mソルビトール-CZ培地上で行った。
【0048】
染色体重複株の作製
染色体重複株の作製は、以下の通りに行った。約2x 107/100μl量のプロトプラスト溶液に対し、20μlのPEG溶液を加えた後、氷中に40分保持した後、70μlのPEG溶液を加え、さらに室温で20分保持した後、1.2Mソルビトール-CZ培地プレート上で再生させた。また、I-sceIを使用する場合には、PEG溶液の添加と同時に加えた。CZ培地上で生育の見られた株を染色体重複候補株として以後の解析に使用した。
【0049】
定量PCR(リアルタイムPCR)を用いた染色体中の遺伝子コピー数の比較
定量PCRはMx3005P(アジレント・テクノロジー社)を用いて行った。他の染色体に位置するrad52をノーマライザーとして相対定量法により、2番染色体上のAlp、amyR、prtT、1258Dの各遺伝子のコピー数を親株と染色体重複株との間で比較した。PCRの条件は95℃で10分保持した後、95℃20秒−58℃30秒−72℃30秒を45サイクル繰り返した。PCRにはRad52、Alp、amyR、prtT、1258Dの各遺伝子の増幅にはそれぞれr52U-r52L、amyRU-amyRL、AlpU-AlpL、prtTU-prtTL、1258DU-1258DLの各プライマーを使用した(表1)。
【0050】
【表1】

【0051】
ふすま麹の作製法
麹菌の酵素活性評価は定法に従って行なった。すなわち、80%散水した小麦ふすま5gを150ml容三角フラスコに入れ、121℃、50分滅菌した後、麹菌を2白金耳程度接種し30℃、4日間培養する。培養後、滅菌水100mlを入れ、ゴム栓をして十分に振とうし、4時間室温にて静置した後、No.2の濾紙(アドバンテック社製)で濾過して得られた抽出液を酵素サンプルとした。
【0052】
プロテアーゼ活性の測定法
得られた酵素サンプルを適宜希釈し、「しょうゆ試験法」(財団法人 日本醤油研究所 昭和60年、287ページ)に記載の方法に従って測定した。プロテアーゼ活性は、ふすま麹1g当り1分間に1μモルのチロシンを生成する活性を1U(ユニットまたは単位)として示した。
【0053】
α-アミラーゼ活性の測定法
得られた酵素サンプルを適宜希釈し、α-アミラーゼ測定キット(キッコーマン醸造分析キット、コード60213)を用い、キットのプロトコールに従って測定を行なった。α-アミラーゼ活性は、ふすま麹1g当り1分間に1μモルの2-クロロ-4-ニトロフェノールを遊離する力価を1U(ユニットまたは単位)として示した。
【0054】
[染色体重複株の作製]
染色体の重複は染色体の二重鎖切断修復の過程で生じると考えられているが、そのメカニズムは判っていない。しかし酵母においてガンマ線照射によって取得された変異株の染色体を解析すると染色体の重複が起きた領域の境界付近に繰り返し配列が存在することが報告されている(非特許文献1)ことから、相同領域を持つ5’ΔpyrGおよび3’ΔpyrGで重複ターゲット領域を挟んだコンストラクトを作製し、相同配列内部で二重鎖切断を引き起こすことにより、染色体の重複を作製出来ると考えられる(図1)。
【0055】
そこで、まず、5’ΔpyrGおよび3’ΔpyrGを作製するために、図2に示すような基本ユニットを作製した。5’ΔpyrGあるいは3’ΔpyrGユニットの作製は PCRとライゲーションを用いて行った。pyrGユニットの作製は、当業者に公知の方法を用いて、pyrG全長の転写開始点付近(5’ΔpyrGの場合)あるいはコード領域の3’側付近(3’ΔpyrGの場合)に図2中で斜線で示す相同配列(適当な制限酵素認識部位を含んでいても良い)を組込むことにより行った。各ベクターが染色体上に組込まれた後5FOA耐性株を選択(ネガティブ選択)することにより、それぞれ相同領域での組換えにより内部が切り出された5’ΔpyrGあるいは3’ΔpyrG株が得られる。
【0056】
次に5’ΔpyrGおよび3’ΔpyrGそれぞれのユニットを染色体上のターゲットとする領域に組込むためのベクターを構築した。標的とする領域は図3に示すように、A.oryzaeの2番染色体中のSC003領域のAO090003001003〜AO090003001258の各ORFの領域に対応する部分を含む700 kbの領域(図3、斜線部)のうちのB領域からC4の領域に渡るAO090003001036〜AO090003001212の各ORFの領域に対応する部分を含む512 kbの領域(AO090003001035〜AO090003001214に挟まれた領域であって、これらの端のORF領域は含まない領域)である。本明細書内では以後簡略化のため、各遺伝子名のAO090003000部分を省略し、Bで表現する(例:AO090003000160 → B160)。SC003領域と対応する各遺伝子の配列は独立行政法人製品評価技術基盤機構(NITE)のゲノム解析データベースであるDOGAN(Database Of the Genomes Analyzed at NITE)に基づいている。(http://www.bio.nite.go.jp/dogan/GeneMap?GENOME_ID=ao__G2)。
【0057】
図3中の2番染色体上のB領域に5’ΔpyrGのコンストラクトを組込むためのベクター(pB-5’Δ)とC4領域に3’ΔpyrGのコンストラクトを組込むためのベクター(pC4-3’Δ)の構築を、以下の通り行った。
【0058】
まずプライマーB-UとB-LおよびC4-UとC4-Lを用いてA.oryzaeのゲノムDNAよりPCRによりB領域の近傍およびC4領域の近傍を含む約3 kbの領域を増幅し、ベクターにクローニングした。
【0059】
プライマーB-iUおよびB-iLを用いて増幅したB領域の近傍を含むベクターと、プライマーpyrUおよびpyrLを用いて増幅した5’ΔpyrGユニットとを、In-fusion クローニングキット(Takara社)を用いてライゲーションし、2番染色体上のB領域に5’ΔpyrGのコンストラクトを組込むためのベクター(pB-5’Δ)を構築した。ライゲーションはキットの所定のプロトコールにしたがって行なった。続いてプライマーC4-iUおよびC4-iLを用いて増幅したC4領域を含むベクターと、プライマーpyrUおよびpyrLを用いて増幅した3’ΔpyrGユニットとを、In-fusionクローニングキット(Takara社)を用いてライゲーションし、C4領域に3’ΔpyrGのコンストラクトを組込むためのベクター(pC4-3’Δ)を構築した。このベクターを作製するのに使用したプライマーを表2に示す。
【0060】
【表2】

【0061】
まず2番染色体のB領域に5’ΔpyrGのコンストラクトを組込むためのベクター(pB-5’Δ)をプライマーB-UおよびB-Lを用いて増幅し、そのPCR産物を用いてA.oryzaeのΔpyrG株に対して形質転換を行った。得られた形質転換体をPCRおよびサザンハイブリダイゼーションにより調べた結果、1株で目的部位にベクターが組込まれていることが確認された。次にこの株から分生子を採取し、約1×105を5FOAを含むCZ培地プレート上へ塗布し、得られた5FOA耐性株を解析したところ、B領域に5’ΔpyrGのコンストラクトを持つことが確認された。このB領域に5’ΔpyrGのコンストラクトを持つΔpyrG株を親株として、C4領域(512 kb領域重複用)に3’ΔpyrGのコンストラクトを組込むためのベクター(pC4-3’Δ)をプライマーC4-UおよびC4-Lを用いたPCRにより増幅した後、形質転換を行った。C4領域にベクターが組込まれた株をPCRおよびサザンハイブリダイゼーションを用いて選抜して分生子を回収し、5FOA−CZプレート上で耐性を示すコロニーを選択した結果、重複ターゲット領域中(512 kb領域)の最もセントロメア側であるB領域に5`ΔpyrGが組込まれ、最もテロメア側であるC4領域に3`ΔpyrGが組込まれた株の構築が確認された。こうして2番染色体中の512 kbの領域を重複させるための基本的なコンストラクトを持った株(B-C4-hap株)の構築に成功した(図3)。
【0062】
次に、このB-C4-hap株を親株として、前述の方法により、染色体重複株の取得実験を行った。その結果、再生プレートから1個のpyrG+のコロニーが得られたので、これをB-C4-512k株とした。続いて、得られたpyrG+株(B-C4-512k株)から染色体DNAを抽出し、PCRおよび定量PCR(リアルタイムPCR)により、染色体が重複しているかどうかの確認を試みた。
【0063】
PCRによる染色体重複の確認
まず、PCRにより狙った染色体上の領域の重複が起きているかどうかの確認を行なった。その結果を図4に示す。重複領域のセントロメア側の端に位置し、ベクター(pB-5’Δ)が組込まれているB領域の近傍領域を増やすためのプライマー(B-UとB-L)を用いた場合(図4上)には、コントロール株(親株:B-C4-hap株)、重複株(B-C4-512k株)ともに4.4 kbのバンドの増幅が見られた(図4下、レーン1および2)。また重複領域のテロメア側の端に位置し、ベクター(pC4-3’Δ)が組込まれているC4領域の近傍領域を増やすためのプライマー(C4-UとC4-L)を用いた場合(図4上)にも、コントロール、重複株ともに3.9 kbのバンドの増幅が見られた(図4下、レーン5および6)。一方、プライマーC4-UとB-Lの組合わせでPCRを行った場合(図4上)にはコントロールでは全くバンドの増幅が見られないのに対し、染色体重複株では、はっきりと4.8 kbのバンドの増幅が見られた(レーン3および4)。染色体重複株でのみ見られた4.8kbのバンドは、染色体上の狙った領域が同一染色体上で重複し、図4下に示すような構造が染色体上に存在する場合にのみ得られるバンドであると考えられる。このことから、染色体重複株B-C4-512k株では2番染色体上のB領域からC4領域に渡る512 kbの領域(B1036〜B1212)が同一染色体上で重複していることが示された。
【0064】
リアルタイムPCRによるコピー数の確認
続いてリアルタイムPCR(定量PCR)を用いて2番染色体の重複領域における各遺伝子のコピー数をコントロール株(親株:B-C4-hap株)および染色体重複株(B-C4-512k株)で比較した(図5)。実験では他の染色体に存在し、コピー数が1であるrad52をノーマライザーとして使用し、重複領域内のセントロメア側の端に位置するB1036遺伝子(Alp)とそこから490 kb離れた位置に存在するB1208(amyR)遺伝子およびテロメア側の端に位置するB1212(prtT)遺伝子、そして重複領域外に位置するB1258D遺伝子をターゲットとして、SYBR Greenを用いた相対的定量法により、親株におけるコピー数との比較を行った。図5にはコントロール株(B-C4-hap株)でのコピー数に対する相対値を示す。その結果、染色体重複株であるB-C4-512k株では、染色体重複領域外に位置するB1258Dの相対値は約1程度であったが、重複領域の内部に位置するB1036遺伝子、B1208遺伝子、B1212遺伝子についてはいずれもコントロール株でのコピー数に対して2倍程度の値を示した。これらの結果から染色体重複株(B-C4-512k株)においては、重複領域内に位置する遺伝子のコピー数が2に増幅していることが確認された。これらのPCRおよびリアルタイムPCR(定量PCR)の結果から、染色体重複株(B-C4-512k株)では2番染色体上の狙った領域である512 kbの領域が確かに重複していることが確認された。
【0065】
さらに同様の方法により、図3中のA-D領域間の700 kb、A-C領域間の585 kb、B-C領域間の517 kb、A-C2領域間の573 kb、B-B3領域間の10 kbの重複株を取得するために、A領域に5’ΔpyrGのコンストラクトを、B3領域、C2領域、C領域、D領域にはそれぞれ3’ΔpyrGのコンストラクトを導入するためのベクターを構築した。ベクターの構築には、それぞれ、A領域にはプライマーA-U、A-L、A-iU、A-iLを、B3領域にはプライマーB3-U、B3-L、B3-iU、B3-iLを、C領域にはプライマーC-U、C-L、C-iU、C-iLを、C2領域にはプライマーC2-U、C2-L、C2-iU、C2-iLを、D領域にはプライマーD-U、D-L、D-iU、D-iLを使用し、ΔpyrGユニットとのライゲーションはIn-fusionクローニングキット(Takara社)を用いて行った。得られたベクターを前述の方法と同様にPCRにより増幅した後、それらをA.oryzaeのΔpyrG株に対して形質転換することにより、染色体重複用の親株を作製した後、A-D領域700kbの重複株(A-D-700k株)、A-C領域585kbの重複株(A-C-585k株)、B-C領域517kbの重複株(B-C-517k株)、A-C2領域573kbの重複株(A-C2-573k株)、B-B3領域10kbの重複株(B-B3-10k株)を取得した。これらの重複株の作製に使用したプライマーを表3に示す。これらの菌株に関しても、染色体重複株B-C4-512k株と同様に、2番染色体上の各領域が同一染色体上で重複していることが示された。
【0066】
【表3】

【0067】
活性の測定
2番染色体の重複株については、特許文献1(特許第4469014号「大規模ゲノム重複を保持する麹菌」)で、麹菌の2番染色体上のSC003のAO090003001003〜AO090003001259に対応するゲノム領域の重複株で、プロテアーゼ活性およびアミラーゼ活性の顕著な上昇が見られることが報告されている。本実験で2番染色体を重複させた重複株について、同様の活性上昇が見られるかどうかを調べるために、酵素活性の測定を行なった。酵素活性の比較対照とするために、本実験での方法を用いて2番染色体上のAO090003001003〜AO090003001258(B1003〜B1258)に対応する700 kbの領域を重複させたA-D-700k株を用いた(図3)。さらに同様の方法を用いて作製した重複株であるA-C-585k株、B-C−517k株、A-C2−573k株、B-B2-10k株を用いて酵素活性の測定を行った。
【0068】
プロテアーゼ活性測定の結果
前述の方法によって、ふすま麹より得られた酵素抽出液を用いて全プロテアーゼ活性の測定を行い、染色体重複を持たないコントロール株(Ct株)と重複株とで比較を行なった。その結果を表4に示す。プロテアーゼ活性については、コントロール株(Ct株)の活性に対し、染色体重複株(A-C4-512k株)では約3倍に活性値が上昇していた。また、この値は700 kbの領域の染色体重複株(A-D-700k株)で得られた活性値であるとほぼ同等であった。このことから本発明で作製した512 kbの領域の染色体重複株(B-C4-512k株)では、ふすま培養において、700 kbの領域の染色体株重複(A-D-700k株)と同様のプロテアーゼ活性の上昇が見られることが判明した。また、同様の方法で作製したA-C領域間575kbの重複株(A-C-575k株)、B-C領域間517kbの重複株(B-C-517k株)、においても同様のプロテアーゼ活性の上昇が確認された。一方、B-B3領域間10kbの重複株(B-B3-10k株)およびA-C2領域間の573kbの重複株(A-C2-573k株)においてはプロテアーゼ活性の上昇は2倍弱であった。
【0069】
【表4】

【0070】
α‐アミラーゼ活性測定の結果
続いてα‐アミラーゼ活性についても同一の酵素液を用いて測定を行なった。その結果を表5に示す。その結果、コントロール株(Ct株)の活性値に対し、染色体重複株(B-C4-512k株)での活性値は約1.6倍程度に上昇していることが明らかとなった。この値は700 kbの領域の染色体重複株(A-D-700k株)において得られた値とほぼ同じであった。またB-B3-10k株以外の染色体重複株(A-C-585k株、B-C-517k株、A-C2-573k株)においても同様のアミラーゼ活性の上昇が見られた。このことから本発明で作製した512 kbの領域の染色体重複株(B-C4-512k株)では、ふすま培養において、700 kbの領域の染色体株重複(A-D-700k株)と同様のアミラーゼ活性の上昇が見られることが判明した。
【0071】
【表5】

【0072】
以上の結果より、本実施例で作製した各形質転換菌においては2番染色体の同一染色体上で重複領域が見られた。例えば、B-C4-512k株では2番染色体のB1036-B1212間512 kbの領域が同一染色体上で確かに重複しており、ふすま培養において、B1003-B1258間700 kbの領域の染色体重複株(A-D-700k株)と同様に、染色体重複のない親株に比較して、顕著なプロテアーゼ活性およびアミラーゼ活性の上昇が見られることが判った。また、同様の方法で作製したA-C領域間575kbの重複株(A-C-575k株)、B-C領域間517kbの重複株(B-C-517k株)、においても同等のプロテアーゼ活性の上昇、およびアミラーゼ活性の上昇が確認された。このことから、2番染色体の重複によるプロテアーゼ活性およびアミラーゼ活性の上昇には、B1003-B1258間700 kbの領域より短い領域の重複で十分であることが示された。
【産業上の利用可能性】
【0073】
本発明により、育種の対象である菌株のゲノム情報を利用して実用上有用であると考えられる染色体の領域を特定し、その特定ゲノム領域を同一染色体上でタンデムに重複させた菌株を提供することが可能となり、効率的に分子育種が進められると考えられる。更に、従来知られていなかった全く新たな有用な形質が強化された麹菌が得られるものと期待される。
【特許請求の範囲】
【請求項1】
アスペルギルス属に属する菌の形質転換菌であって、同一染色体上でタンデムにゲノム重複領域を有することを特徴とする、前記形質転換菌。
【請求項2】
ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれていることを特徴とする、請求項1記載の形質転換菌。
【請求項3】
ゲノム重複領域が十〜数百kbである、請求項1又は2記載の形質転換菌。
【請求項4】
アスペルギルス属に属する菌がアスペルギルス・オリゼまたはアスペルギルス・ソーヤである、請求項1ないし3のいずれか一項に記載の形質転換菌。
【請求項5】
ゲノム重複領域がアスペルギルス・オリゼの2番染色体のSC003領域の500kb以上で700kb未満である、請求項4記載の形質転換菌。
【請求項6】
ゲノム重複領域がアスペルギルス・オリゼの2番染色体のSC003領域におけるAO090003001035〜AO090003001214に挟まれた領域であって、これらの端のORF領域は含まない領域である、請求項5記載の形質転換菌。
【請求項7】
形質転換マーカー遺伝子が、pyrG、sC及びniaDから成る群から選択される、請求項1ないし6のいずれかに記載の形質転換菌。
【請求項8】
染色体重複を持たないコントロール株(Ct株)と比べて、プロテアーゼが1.9倍以上、及び/又はα―アミラーゼ活性が1.4倍以上に増加していることを特徴とする、請求項1ないし7のいずれか一項に記載の形質転換菌。
【請求項9】
遺伝子組換え法によって作製されたものであることを特徴とする、請求項1ないし8のいずれか一項に記載の形質転換菌。
【請求項10】
請求項1ないし9のいずれか一項に記載の形質転換菌を用いて製造される醤油。
【請求項1】
アスペルギルス属に属する菌の形質転換菌であって、同一染色体上でタンデムにゲノム重複領域を有することを特徴とする、前記形質転換菌。
【請求項2】
ゲノム重複領域に挟まれた領域に形質転換マーカー遺伝子が組み込まれていることを特徴とする、請求項1記載の形質転換菌。
【請求項3】
ゲノム重複領域が十〜数百kbである、請求項1又は2記載の形質転換菌。
【請求項4】
アスペルギルス属に属する菌がアスペルギルス・オリゼまたはアスペルギルス・ソーヤである、請求項1ないし3のいずれか一項に記載の形質転換菌。
【請求項5】
ゲノム重複領域がアスペルギルス・オリゼの2番染色体のSC003領域の500kb以上で700kb未満である、請求項4記載の形質転換菌。
【請求項6】
ゲノム重複領域がアスペルギルス・オリゼの2番染色体のSC003領域におけるAO090003001035〜AO090003001214に挟まれた領域であって、これらの端のORF領域は含まない領域である、請求項5記載の形質転換菌。
【請求項7】
形質転換マーカー遺伝子が、pyrG、sC及びniaDから成る群から選択される、請求項1ないし6のいずれかに記載の形質転換菌。
【請求項8】
染色体重複を持たないコントロール株(Ct株)と比べて、プロテアーゼが1.9倍以上、及び/又はα―アミラーゼ活性が1.4倍以上に増加していることを特徴とする、請求項1ないし7のいずれか一項に記載の形質転換菌。
【請求項9】
遺伝子組換え法によって作製されたものであることを特徴とする、請求項1ないし8のいずれか一項に記載の形質転換菌。
【請求項10】
請求項1ないし9のいずれか一項に記載の形質転換菌を用いて製造される醤油。
【図1】


【図2】


【図3】


【図4】


【図5】




【図2】


【図3】


【図4】


【図5】


【公開番号】特開2013−34416(P2013−34416A)
【公開日】平成25年2月21日(2013.2.21)
【国際特許分類】
【出願番号】特願2011−171963(P2011−171963)
【出願日】平成23年8月5日(2011.8.5)
【出願人】(000004477)キッコーマン株式会社 (212)
【Fターム(参考)】
【公開日】平成25年2月21日(2013.2.21)
【国際特許分類】
【出願日】平成23年8月5日(2011.8.5)
【出願人】(000004477)キッコーマン株式会社 (212)
【Fターム(参考)】
[ Back to top ]

