麹菌蛋白質加水分解酵素の分泌を増大する組換えベクター
【課題】調味料の食品製造や、消化剤等の医薬品製造、洗剤用の蛋白質加水分解酵素生産等において、固体培地培養及び液体培地培養での麹菌の蛋白質加水分解酵素活性を向上させる。
【解決手段】麹菌蛋白質加水分解酵素の分泌を増大する機能を有する組換えベクター、該ベクターによって形質転換された麹菌により蛋白質加水分解酵素遺伝子発現・分泌が亢進した麹菌、形質転換された麹菌による蛋白質加水分解酵素生産方法等を提供する。
【解決手段】麹菌蛋白質加水分解酵素の分泌を増大する機能を有する組換えベクター、該ベクターによって形質転換された麹菌により蛋白質加水分解酵素遺伝子発現・分泌が亢進した麹菌、形質転換された麹菌による蛋白質加水分解酵素生産方法等を提供する。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する組換えベクター、該ベクターによって形質転換された麹菌により蛋白質加水分解酵素遺伝子発現・分泌が亢進した麹菌、形質転換された麹菌による蛋白質加水分解酵素生産方法等に関する。
【背景技術】
【0002】
アスペルギルス・オリゼ(Aspergillus oryzae)及びアスペルギルス・ソーヤ(Aspergillus sojae)等の麹菌は醤油、酒、味噌などの醸造食品の製造に工業的に用いられている。さらに、近年、他の生物種同様アスペルギルス・オリゼのゲノム配列が明らかとなり(特開2005−176602号公報)、遺伝子の機能的解析の重要性がますます高まってきている。
【0003】
このような麹菌は、様々な酵素類(蛋白質加水分解酵素、アミラーゼ類等)を産生・分泌し、その優れた澱粉糖化能力及び蛋白質分解力等により、醸造食品の製造に幅広く利用されている。
【0004】
従来、特許文献1〜3に記載されているような、麹菌の遺伝子発現を制御する様々な転写因子の解析が行われてきた。
【先行技術文献】
【特許文献】
【0005】
【特許文献1】特開2003−240号公報
【特許文献2】特開2003−70484号公報
【特許文献3】特開2003−235584号公報
【特許文献4】特開2005−176602号公報
【発明の概要】
【発明が解決しようとする課題】
【0006】
本発明は、新たに、麹菌の蛋白質加水分解酵素の分泌を増大させる組換えベクターを提供すること、及び、該組換えベクターを用いることによって固体培地培養及び液体培地培養での蛋白質含有物の分解の効率が向上した麹菌を提供すること等を目的とする。
【課題を解決するための手段】
【0007】
即ち、本発明は、以下の各態様の発明に係るものである。
(1)配列番号1、2、3又は4で表される塩基配列からなるDNAを含む組換えベクター。
(2)配列番号1、2、3又は4で表される塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNAを含む組換えベクター。
(3)配列番号1、2、3又は4で表される塩基配列からなるDNAと80%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNAを含む組換えベクター。
(4)上記1、2又は3記載の組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする麹菌。
(5)上記4記載の麹菌を、固体培地若しくは液体培地で培養して培地中に蛋白質加水分解酵素を分泌させたのち、培地から蛋白質加水分解酵素を採取することを特徴とする、蛋白質加水分解酵素の生産方法。
(6)上記4記載の麹菌を培養して得られる培養物と蛋白質含有原料とを混合し、原料中の蛋白質を分解することを特徴とする、蛋白質分解物の製造方法。
(7)上記4記載の麹菌を培養して得られる培養物とゼラチン含有原料とを混合し、原料中のゼラチンを分解することを特徴とする調味液の製造方法。
(8)以下の(a)、(b)又は(c)のDNA:
(a)配列番号1、2、3又は4で表される塩基配列からなるDNA、
(b)(a)の塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA、
(c)(a)の塩基配列からなるDNAと80%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA。
(9)以下の(a)又は(b)の蛋白質:
(a)配列番号1、2、3又は4で表される塩基配列からなるDNAにコードされるアミノ酸配列からなる蛋白質、
(b)(a)のアミノ酸配列において、1個若しくは数個のアミノ酸が欠失、置換、若しくは付加されたアミノ酸配列からなり、麹菌蛋白質分解酵素の分泌を増大する機能を有する活性を有する蛋白質。
【発明の効果】
【0008】
本発明の組換えベクターを用いることによって、麹菌の蛋白質加水分解酵素の分泌を増大させることが可能になった。該組換えベクターで形質転換麹菌を用いることによって固体培地培養及び液体培地培養での蛋白質含有物の分解の効率を向上させることができる。
【図面の簡単な説明】
【0009】
【図1】本発明の形質転換体(形質転換菌)において、形質転換に用いたプラスミドが形質転換体のゲノム上のniaD遺伝子領域に挿入されていることを示す、サザン解析の結果により得られた電気泳動の写真である。
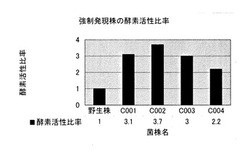
【図2】フスマ培地における、野生株と本発明の形質転換体の蛋白質加水分解酵素活性を比較した結果を示す。
【発明を実施するための形態】
【0010】
本発明の組換えベクターに含まれる配列番号1、2、3又は4で表される塩基配列からなるDNAは麹菌のゲノム由来であり、以下の実施例に具体的に記載されるように、アスペルギルス・オリゼの全ゲノム情報(特開2005−176602号公報)に基づき、相同性検索、モチーフ検索、及び、既知遺伝子のアノテーションや文献情報、モチーフの種類などの情報をもとに、転写調節遺伝子をコードするDNAと推定されたものであり、実施例に示すような、適当なプライマーを用いるPCRにより麹菌のゲノムDNA等から取得することができる。
【0011】
更に、本発明における「配列番号1、2、3又は4で表される塩基配列からなるDNA」には、アミノ酸をコードする領域のみから成る塩基配列(即ち、エクソン部分のみが結合された塩基配列)からなるcDNAも含まれる。このようなcDNAは、本明細書に開示の塩基配列情報に基づき調製した適当なプライマーを用いて、麹菌のmRNAを鋳型としたPCR等により取得することが可能である。又、本発明に関するDNAは当業者に公知の任意の方法によって化学合成することも可能である。
【0012】
さらに、本発明の組換えベクターには、上記DNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNA、及び、上記DNAと約80%以上、好ましくは約95%以上である配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNAが含まれる。
【0013】
ここで、ハイブリダイゼーションは、Molecular cloninng third.ed.(Cold Spring Harbor Lab.Press,2001)に記載の方法等、当業界で公知の方法あるいはそれに準じる方法に従って行うことができる。また、市販のライブラリーを使用する場合、添付の使用説明書に記載の方法に従って行うことができる。
【0014】
ハイブリダイゼーションは、例えば、カレント・プロトコールズ・イン・モレキュラー・バイオロジー(Current protocols in molecular biology(edited by Frederick M. Ausubel et al.,1987)に記載の方法等、当業界で公知の方法あるいはそれに準じる方法に従って行うことができる。また、市販のライブラリーを使用する場合、添付の使用説明書に記載の方法に従って行うことができる。
【0015】
本明細書において、「ストリンジェントな条件下」とは、例えば、温度60℃〜68℃において、ナトリウム濃度150〜900mM、好ましくは600〜900mM、pH6〜8であるような条件を挙げることができる。
【0016】
従って、配列番号1ないし配列番号4で表される塩基配列を含むDNAと相補的な塩基配列からなるDNAとハイブリダイズできるDNAとしては、例えば、該DNAの全塩基配列との相同性の程度が、全体の平均で、約80%以上、好ましくは約95%以上である塩基配列を含有するDNA等を挙げることができる。なお、塩基配列間の同一性は、当業者に公知のアルゴリズム、例えば、Blastを用いて決定することができる。
【0017】
従って、本発明は、配列番号1ないし配列番号4で表される塩基配列を含むDNA、該DNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNA、及び、上記DNAと約80%以上、好ましくは約95%以上である配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA、並びに、配列番号1、2、3又は4で表される塩基配列からなるDNAにコードされるアミノ酸配列からなる蛋白質、及び、これらのアミノ酸配列からなり麹菌蛋白質分解酵素の分泌を増大する機能を有する活性を有する蛋白質にも係るものである。
【0018】
2つの塩基配列又はアミノ酸配列における配列相同性を決定するために、配列は比較に最適な状態に前処理される。例えば、一方の配列にギャップを入れることにより、他方の配列とのアラインメントの最適化を行う。その後、各部位におけるアミノ酸残基又は塩基が比較される。第一の配列におけるある部位に、第二の配列の相当する部位と同じアミノ酸残基又は塩基が存在する場合、それらの配列は、その部位において同一である。2つの配列における配列相同性は、配列間での同一である部位数の全部位(全アミノ酸又は全塩基)数に対する百分率で示される。
【0019】
上記の原理に従い、2つの塩基配列又はアミノ酸配列における配列相同性は、例えば、Karlin及びAltschulのアルゴリズム(Proc.Natl.Acad.Sci.USA 87:2264−2268、1990及びProc.Natl.Acad.Sci.USA 90:5873−5877、1993)により決定される。このようなアルゴリズムを用いたBLASTプログラムやFASTAプログラムは、主に与えられた配列に対し、高い配列相同性を示す配列をデータベース中から検索するために用いられる。これらは、例えば米国National Center for Biotechnology Informationのインターネット上のウェブサイトにおいて利用可能である。
【0020】
上記のような塩基配列の配列相同性又はコードするアミノ酸配列の配列相同性を示すようなDNAは、上記のようにハイブリダイゼーションを指標に得ることもでき、ゲノム塩基配列解析等によって得られた機能未知のDNA群又は公共データベースの中から、本技術分野の研究者か通常用いている方法により、例えば、前述のBLASTソフトウェアを用いた検索により発見することも容易である。さらに、本発明遺伝子は種々の公知の変異導入方法によって得ることもできる。
【0021】
本発明の組換えベクターは、当業者に公知の適当な遺伝子工学的な手段を用いて、上記のDNAをベクター内に連結することにより調製することができる。ベクターとしては、形質導入又は形質転換する宿主微生物のゲノム上の適当な位置に上記のDNAが挿入され、その緒果、親株と比較して、その宿主微生物の蛋白質加水分解酵素分泌能を増大することができるものであれば、その構造及び種類等に、特に、制限はない。例えば、プラスミド、コスミド、ファージ、ウイルス、染色体組み込み型、人工染色体等のベクターを用いることかできる。
【0022】
上記ベクターには、形質転換された細胞を選択することを可能にするための当業者に公知の任意のマーカー遺伝子が含まれていてもよい。マーカー遺伝子としては、例えば、URA3、niaDのような、宿主の栄養要求性を相補する遺伝子又はアンピシリン、カナマイシンあるいはオリゴマイシン等の薬剤に対する抵抗遺伝子等が挙げられる。また、組換えベクターは、宿主細胞中で本発明の遺伝子を発現することのできるプロモーター及びその他の制御配列(例えば、エンハンサー配列、ターミネーター配列、ポリアデニル化配列等)、さらに、目的とするDNAを挿入するためのマルチクローニングを含むことが望ましい。これらの組換えベクターに含まれる各要素は当業者に公知であり、例えば、具体的には、実施例に示されるように、アスペルギルス・オリゼのアミラーゼ遺伝手プロモーター、アスペルギルス・ニーデュランスのアミラーゼターミネーター、マーカー遺伝子としてのアスペルキルス・オリゼのniaD遺伝子を挙げることができる。
【0023】
形質導入又は形質転換は、公知の適当な方法で行うことができる。例えば、プロトプラスト化した後ポリエチレングリコール及び塩化カルシウムを用いる方法(Mol.Gen.Genet.,218,99−104(1989))を用いることかできる。
【0024】
本発明の組換えベクターを形質導入又は形質転換された麹薗は、親株と比較して蛋白質加水分解酵素分泌が増大されたものとなる。本明細書において、「蛋白質加水分解酵秦の分泌を増大する」、又は、「蛋白質加水分解酵素の分泌が増大している」とは、最終的な結果(現象)として、菌体外へ蛋白質加水分解酵素の分泌量(分泌能)が増大していることを意昧し、その原因・機構としては、例えば、蛋白質加水分解酵素の発現量(産生量)自体が増大する場合と、産生された該酵素の菌体外への分泌量(分泌能)が増大している場合等が考えられる。さらに、ここで、「増大する」とは、以下の実施例に記載されているように、ゼラチン又はアゾカゼインを蛋白質として使用する測定方法を用いて、本発明の組換えベクターで形質導入又は形質転換された菌の培地中の蛋白質加水分解酵素量(活性)が、親株と比較して有意に増大することを意味する。
【0025】
従って、該ベクターに含まれる各配列にコードされる蛋白質は、以下の実施例に記載されるように、転写調節因子であると予測されるが、必ずしも蛋白質加水分解酵素の転写調節エレメントに結合し、該蛋白質加水分解酵素の発現を促進させる機能を有するものに限られず、本発明の組換えベクターにより形質導入又は形質転換された麹薗において、発現された蛋白質加水分解酵素の菌体外への分泌を促進させる機能を有するものであっても良い。
【0026】
麹菌とは、アスペルギルス属に属する微生物(菌類)の総称であり、その代表的な例として、既に記載したアスペルギルス・オリゼ(Aspergillus oryzae)及びアスペルギルス・ソーヤ(Aspergillus sojae)に加えて、特に食品・醸造分野で使用される菌としてアスペルギルス・アワモリ(Aspergillus awamori)及びアスペルギルス・ニガー(Aspergillus niger)等を挙げることができる。また、麹菌以外の食品・醸造・化学・医療分野で使用される産業上有用な糸状菌等を用いても実質的に同様の効果を得ることができる。これらの菌は、市販品として、又は、アメリカンタイプカルチャーコレクション(ATCC)等の様々な公的寄託機関から入手することも可能である。
【0027】
また、「蛋白質加水分解酵素」はプロテアーゼとも呼ばれ、蛋白質を主に分解するプロテイナーゼ、又はエンドペプチダーゼ、小ペプチドを分解するペプチダーゼなどを含む総称である。
【0028】
親株と比較して蛋白質加水分解酵素分泌能が増大している本発明の麹菌を当業者に公知の任意の方法で培養することにより、それが分泌する蛋白質分解酵素を生産することができる。例えば、本発明の麹菌を、固体培地若しくは液体培地で培養して培地中に蛋白質加水分解酵素を分泌させたのち、培地から蛋白質加水分解酵素を採取することが可能である。このような生産方法における、培養系・培地の選択、培養温度・時間等の培養条件は、以下の実施例等を参考に、当業者が適宜設定することができる。
【0029】
また、本発明は麹菌を培養して得られる培養物と蛋白質含有原料とを混合し、原料中の蛋白質を分解することによって、蛋白質分解物を製造することができる。この方法で使用される原料に含まれる蛋白質の種類に特に制限はなく、例えば、ゼラチン、コラーゲン及びグルテン等を挙げることができる。例えば、ゼラチン、コラーゲン及びグルテン等含有原料の蛋白質分解物は調味液として有用である。
【0030】
なお、組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする本発明の麹菌の具体例である、C001、C002、C003及びC004は平成17年9月9日付で、独立行政法人産業技術総合研究所特許生物寄託センターに寄託され、受領番号FERM AP−20659、AP−20660、FERM AP−20661及びFERM AP−20662がそれぞれ付与されている。
【0031】
以下、実施例に則して本発明をさらに説明するが、本発明の技術的範囲は、これらに限定されるものでない。
【実施例1】
【0032】
(転写調節因子遺伝子の推定と抽出)
麹菌アスペルギルス・オリゼの全ゲノム情報(特開2005−176602号公報参照)から、次のような方法により、転写調節因子遺伝子をコードすると推定される配列を抽出した。このステップにおいては、詳細な吟味は行わず、転写調節因子遺伝子である可能性のあるものは基本的にリストアップの対象とした。
(1)相同性検索による転写調節因子遺伝子と推定される配列の抽出
麹菌アスペルギルス・オリゼの全ゲノム情報から、遺伝子自動予測ソフトにより遺伝子配列と推定されるDNA配列をすべて抽出し、これらのDNA配列を自動予測遺伝子配列(以下、「自動予測遺伝子配列」という)とした。
【0033】
各々の自動予測遺伝子配列から予測されるその遺伝子産物配列((以下、「自動予測遺伝子産物配列」という)を基礎配列として、既知蛋白質の公共データベース(NCBIによる非重複蛋白質データベースnr)に対して、相同性検索ソフトBLASTを用いて相同性検索を行った。相同性検索の結果得られた配列の機能に関する情報を整理し、その配列に関わる遺伝子機能情報に対してキーワード検索を行い、転写調節因子に関わるキーワードを含む自動予測遺伝子配列を選択した。
(2)転写調節因子の構造に関わるモチーフと推定されるアミノ酸配列をコードするDNA配列の検索
自動予測遺伝子産物配列について、モチーフ検索ソフト(HMMER)を用いてモチーフ(Pfam)検索を行い、転写調節因子に関わるモチーフを含むと推定されるアミノ酸配列をコードする自動予測遺伝子配列を選択した。
(3)既知転写調節因子配列と相同性を有するDNA配列の検索
麹菌や麹菌以外の糸状菌に関する既知の転写調節因子のアミノ酸配列を問い合わせ配列として、今回得られた自動予測遺伝子産物配列に対して、相同性検索ソフトBLASTを用いて相同性検索を行った。同様な問い合わせ配列を用いて、麹菌のゲノムコンティグ配列に対してBLAST(tblast)を用いて相同性検索を行うことにより、自動予測で予測されていなかった遺伝子や、自動予測でDNA結合領域などの保存性の高い領域を欠損した形で予測されてしまっていた遺伝子についても検索可能とした。
【0034】
これらの方法により、自動予測遺伝子配列から合計667個の候補遺伝子(以下、「候補遺伝子」という)を抽出した。
【0035】
(候補遺伝子の絞込みと遺伝子領域のin silico推定)
前項で抽出した転写調節因子遺伝子と思われる候補遺伝子について、順次遺伝子領域の推定を行った。また、推定に先立ち、相同な既知遺伝子のアノテーションや文献情報、モチーフの種類などの情報をもとに、強制発現による解析対象として相応しいか否かを検討し、ヘテロ複合体で機能するものなど、相応しくないものは除外した。
【0036】
遺伝子領域の推定は、主に相同な既知遺伝子との比較により行った。相同な既知遺伝子との比較は、BLAST、FASTA及びALN(Bioinfomatics 2000 16: 190−202)を用いて行った。既知遺伝子との相同性が限定的で5’端が推定できなかった場合は、5’RACEを行うために、相同部分のアラインメント情報を準備した。3’末端の予測が本来の遺伝子領域より短すぎた場合は、転写調節因子のC末側がトランケートされてしまうが、予測が長すぎた場合は、本来のC末端で翻訳は終止すると考えられる。そこで、3’末端が予測できなかった場合は、相同部分からの距離や、隣の遺伝子との位置関係などから、本来の3’末端より十分に下流部分までを強制発現ベクターに組み込む領域とすることにした。
【0037】
このような方法で、前項の抽出の際に既知の遺伝子との相同性が高かったものから順次遺伝子領域の推定を行った。全長がin silicoで推定できたものと5’RACEにより5’末端が明らかにされたもの一部の合計300遺伝子について強制発現株作成ための予測配列を得た。
【実施例2】
【0038】
(発現用プラスミドの作製)
転写調節因子遺伝子を麹菌内で発現させるために用いる発現プラスミドとして、発現ユニットとしてアスペルギルス・オリゼアミラーゼ遺伝子プロモーター及びアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーター、転写調節因子遺伝子挿入用マルチクローニングサイト、マーカー遺伝子としてアスペルギルス・オリゼniaD遺伝子を含むプラスミドpAPTLNを構築した。
【0039】
寒天プレート培地上で生育させた、アスペルギルス・ニドランスIAM2130株の胞子5×105個を掻き取り、滅菌したマイクロチューブに入れた300μlのNuclei Lysis Solutionバッファー(Promega社製)に懸濁した。胞子を懸濁したマイクロチューブに1gのガラスビーズBZ−06(アズワン株式会社製)を加え、tissue lyser(QIAGEN社製)を用いて、25回/秒、3分間処理を2回繰り返した。65℃で15分間加温した後、室温で5分間放置した。1.5μlのRnase Solution(10mg/ml)を加え、37℃で1時間加温した。100μlのProtein Precipitation Solutionを加え20秒間激しく振とうした後、13,500rpmで5分間遠心処理した。遠心後の上清を別のマイクロチューブに移し、350μlのイソプロパノールを加え転倒混和した後、12,000rpmで3分間遠心処理した。得られた沈殿を70%エタノールで洗浄および乾燥処理した後、100μlのTEバッファー〔10mM Tris−HCl(pH7.5),1mM EDTA〕に懸濁しゲノムDNA溶液を得た。得られたゲノムDNAを鋳型として、下記のプライマーを用いてPCRを行いアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーター領域の増幅を行った。
5’−gggtagtcgtacccgatgatgaaac−3’(配列番号5)
5’−agcctaggccgctgcaggcag−3’ (配列番号6)
PCR反応は、TaKaRa LA Taq(タカラバイオ株式会社製)を使用し、PTC−200(MJ Research社製)を用いて行った。反応液の組成は以下のとおりである。
【0040】
(試薬:使用量:終濃度)
TaKaRa LA Taq:0.5μl
10×LA PCR BufferII:5μl:1×
25mM MgCl2:5μl:2.5mM
dNTP Mixture:8μl:それぞれ0.4mM
鋳型DNA(0.5μg):1μl
プライマー:1μl×2種類:それぞれ0.2μM
滅菌水:28.5μl
合計液量:50μl
上記の反応液50μlを0.2ml反応チューブ中で混合してPTC−200にセットし、以下の温度設定によりPCRを行った。
95℃、2分:1サイクル
95℃、30秒 58℃、30秒 72℃、2分:30サイクル
72℃、3分:1サイクル。
【0041】
反応液をエタノール沈澱処理後、沈殿を20μlのTEバッファーに懸濁した。さらに、PstIで消化した後、0.7%アガロースゲルで電気泳動し、目的の増幅産物を切り出した。切り出した増幅産物はゲルエクストラクションキット(QIAGEN社製)を用いて精製し、アスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターを得た。プラスミドpUC19をSmaI及びPstIで処理した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いて約2.7kbのDNA断片を回収した。回収したDNA断片とアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターをライゲーションした後、大腸菌JM109を形質転換し、pUC19のマルチクローニングサイト内にあるSmaI−PstI部位にアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターが挿入されたプラスミドpATを得た。
【0042】
pATをSmaI及びHindIIIで消化した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いてアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターを含むDNA断片を回収した
プラスミドpMAR5(Biosci.Biotech.Biochem.,56(10),1674−1675,1992)をSmaIおよびHindIIIで消化した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いてargB遺伝子およびアスペルギルス・オリゼアミラーゼ遺伝子ターミネーターを除去したDNA断片を回収した。pATおよびpMAR5由来の両DNA断片をライゲーションした後、大腸菌JM109を形質転換し、アスペルギルス・オリゼアミラーゼ遺伝子プロモーターおよびアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターを含むプラスミドpAPTを得た。
【0043】
下記の2種類の合成DNAを含む(各100μM)TEバッファー100μlを5分間煮沸後、室温まで徐冷しマルチクローニングサイトリンカーとした。pAPTをEcoRIおよびSmaIで消化した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いてマルチクローニングサイトを除去したDNA断片を回収した。回収したDNA断片にマルチクローニングサイトリンカーをライゲーションした後、大腸菌JM109を形質転換しプラスミドpAPTLを得た。プラスミドpST14〔(Mol.Gen.Genet(1989)218:99−104)をHindIIIで消化した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いてアスペルギルス・オリゼniaD遺伝子を含むDNA断片を回収した。回収したniaD遺伝子を含むDNA断片をpAPTLのHindIII部位に挿入し、転写調節因子遺伝子発現用プラスミドpAPTLNを得た。
【0044】
(転写調節因子遺伝子発現用組換えベクターの作製)
転写調節因子遺伝子と予測されたDNA配列を基にPCRにより各転写調節因子遺伝子を取得した。まず、アスペルギルス・オリゼRIB40のゲノムDNAを調製した。次に調製したゲノムDNAを鋳型に、予測遺伝子の開始コドン上流100bpのDNA配列及び終始コドン近傍のDNA配列を参考に作製した2種類のプライマーを用いて、PCRにより各転写調節因子遺伝子を取得した。開始コドン上流のDNA配列を参考にプライマーを作製する際には、pAPTLNのマルチクローニングサイト上にある制限酵素認識配列であり、かつ、増幅する転写調節因子遺伝子配列内に認識配列が存在しない制限酵素認識配列を導入した。増幅した転写調節因子遺伝子は作製したプライマーに認識配列を導入した制限酵素で処理した後、pAPTLNのマルチクローニングサイトに挿入し(導入した制限酵素部位とSmaI部位の間)、各転写調節因子遺伝子発現用組換えプラスミドベクターとした。
【0045】
以下、転写調節因子遺伝子発現用プラスミドベクターの作製の一例を示す。
【0046】
前記の方法を用いてアスペルギルス・オリゼRIB40株より調製したゲノムDNAを鋳型として、下記のプライマーを用いてPCRを行い、転写調節因子遺伝子C001(配列番号1)を得た。
5’−atacaggcattctatcgataaaatgtttcc−3’(配列番号7)
5’−gggctacatctgctgttgtagaagttgc−3’ (配列番号8)
PCR反応は、TOYOBO KOD−Plus−DNA Polymerase(TOYOBO社製)を使用し、PTC−200(MJ Research社製)を用いて行った。反応液の組成は以下のとおりである。
【0047】
(試薬:使用量:終濃度)
KOD−Plus−DNA Polymerase:1μl
10×PCR buffer for KOD−Plus−:5μl:1×
25mM MgCl2:2μl:1mM
2mM dNTP Mixture:5μl:それぞれ0.2mM
鋳型DNA(0.2μg):1μl
プライマー:1μl×2種類:それぞれ0.3μM
滅菌水:34μl
合計液量:50μl
上記の反応液50μlを0.2ml反応チューブ中で混合してPTC−200にセットし、以下のような温度設定によりPCRを行った。
94℃、2分:1サイクル
94℃、15秒 58℃、30秒 68℃、4分:30サイクル
68℃、3分:1サイクル。
【0048】
反応液をエタノール沈澱処理後、沈殿を20μlのTEバッファー〔10mM Tris−HCl(pH7.5),1mM EDTA〕に懸濁した。更に、ClaIで消化した後、0.7%アガロースゲルで電気泳動し、目的の増幅産物を切り出した。切り出した増幅産物はゲルエクストラクションキット(QIAGEN社製)を用いて精製した後、pAPTLNのマルチクローニングサイト上のClaI−SmaI部位に挿入し、転写調節因子遺伝子C001発現用プラスミドpC001を得た。
【0049】
同様の方法を用いて転写調節因子遺伝子C002発現用プラスミドpC002、転写調節因子遺伝子C003発現用プラスミドpC003、転写調節因子遺伝子C004発現用プラスミドpC004を得た。
【0050】
転写調節因子遺伝子C002(配列番号2)を増幅する際には、ClaIの代わりにNheI認識配列を導入した下記の2種類のプライマーを用いて増幅、精製後、pAPTLNのマルチクローニングサイト上のClaI−SmaI部位に挿入した。
5’−tcatacaagctagcaaaatggcggaga−3’(配列番号9)
5’−gggctcgataactttttactcccgtgata−3’ (配列番号10)
【0051】
転写調節因子遺伝子C003(配列番号3)を増幅する際には、ClaIおよびSpeI認識配列を導入した下記の2種類のプライマーを用いて増幅、精製後、pAPTLNのマルチクローニングサイト上のClaI−SpeI部位に挿入した。
5’−ccatcgataatattagtatgctgaatga−3’(配列番号11)
5’−ggactagttcaggtctttcgaatgtcagga−3’(配列番号12)
【0052】
転写調節因子遺伝子C004(配列番号4)を増幅する際には、ClaI及びSpeI認識配列を導入した下記の2種類のプライマーを用いて増幅、精製後、pAPTLNのマルチクローニングサイト上のClaI−SpeI部位に挿入した。
5’−ccatcgataagtaaaaggatgttattagat−3’(配列番号13)
5’−ggactagtttaatccgttctcatggccgaa−3’ (配列番号14)
【実施例3】
【0053】
(転写調節因子遺伝子強制発現麹菌の取得)
アスペルギルス・オリゼRIB326株からMol.Gen.Genet(1989)218:99−104記載の方法で取得したniaD欠損株を転写調節因子遺伝子発現用プラスミドpC001、pC002、pC004、pC003で形質転換した。形質転換法は、プロトプラスト化した後ポリエチレングリコール及び塩化カルシウムを用いる方法(Mol.Gen.Genet(1989)218:99−104)によって行った。各プラスミド5μgを用いて形質転換し、最小培地で形質転換体を選択したところ、それぞれ約200個のコロニーが得られた。このうち各プラスミドにつき15コロニーについて最小培地で単分生子分離を繰り返し、形質の安定化を行った。次に、各転写調節因子遺伝子の成熟領域をプローブとしてサザン解析を実施し、それぞれの形質転換体の中から、形質転換に用いたプラスミドが形質転換体のゲノム上のniaD遺伝子領域に挿入されている株を選択し、それぞれの選択株をC001、C002、C003、C004と命名した。サザン解析の結果を図1に示した。
【実施例4】
【0054】
(麹菌の培養方法及び菌体外酵素活性の測定)
(1)ゼラチン培地における麹菌の培養方法とゼラチン分解に関する蛋白質加水分解酵素活性の測定
各形質転換体をゼラチンを含む液体培地で振とう培養し、ゼラチン分解能を確認した。各形質転換体のゼラチン分解能の評価は、ゼラチンが分解されて生じる培地中のグルタミン酸濃度をコントロールと比較することで行った。
【0055】
寒天最小培地上で生育させた各形質転換体の胞子を3mlの滅菌水で掻き取り、150ml容三角フラスコに入れた40mlのゼラチン培地(2% Gelatin、0.1% KH2PO4、0.05% MgSO4、0.05% KCl、0.001% FeSO4・7H2O、0.3%NaNO3、3% Maltose pH6.0)に植菌した(1×107個胞子/40mlゼラチン培地)。30℃、150rpmで115時間振とう培養した後、培養上清中のグルタミン酸量をヤマサ L−グルタミン酸測定キット(ヤマサ醤油株式会社製)を用いて測定した。また、上記のゼラチン分解能を指標として、培養上清の蛋白質加水分解酵素活性を測定した。コントロールとして、実験毎にRIB326株niaD欠損株をpAPTLNで形質転換して得られたniaD+株を培養した。形質転換体の中で最もゼラチン分解能が高いと思われるC004培養上清中のグルタミン酸量及び蛋白質加水分解酵素活性をコントロールに対する相対値として示した。
【0056】
【表1】

【0057】
(2)フスマ培地における麹菌野生株との培養方法と本発明の形質転換体の蛋白質加水分解酵素活性の比較
フスマ:脱イオン水を5:4の比率で混合し、室温で30分間放置した後、150ml容量の三角フラスコに5gずつ分注し、オートクレーブで15分滅菌した。次いで、予め血球計で胞子数を計測しておいた野生株アスペルギルス・オリゼRIB326株や強制発現株の胞子懸濁液を、胞子がフスマ培地1g当たり5×105個になるように接種した。培養は30℃で4日間行い、培養開始後24時間目と48時間目に混合し、その後は培養終了まで静置した。酵素液は、以下のようにして得た。培養終了後のフスマ培地に、50mlの脱イオン水と500μlのトルエンを加え、2時間室温で振とうし、懸濁液をNo.2濾紙(アドバンテック社製)で濾過し濾液を得て、これを酵素液とした。
【0058】
蛋白質加水分解酵素活性は、以下の計算式で示されるように、野生株の酵素活性を1とした時の比率で比較した。酵素活性比率=強制発現株(試料の吸光度-ブランクの吸光度)/野生株(試料の吸光度-ブランクの吸光度)。また、蛋白質加水分解酵素活性測定を下記のようにして行い、試料とブランクの吸光度を求めた。
【0059】
予め30℃に温めた2mlの基質液(1%アゾカゼイン、0.05Mリン酸緩衝液:pH7.0)に酵素液20μlを加えて混合した。次に、これを30℃で20分反応させた後、反応停止液(10%トリクロロ酢酸)2mlを加え、反応を停止した。30℃で20分間放置して、生じた沈殿をアドバンテック社製No.5C濾紙で濾過し、濾液の吸光度(410nm)を測定した。ブランクは、酵素液添加前に反応停止液を加えて、同じ操作を行った。
【0060】
野生株と強制発現株の蛋白質加水分解酵素活性を比較した結果、C002、C001、C003が野生株の3倍の蛋白質加水分解酵素活性を示すことが確認された(図2)。また、C004についても野生株の2.2倍の蛋白質加水分解酵素活性を示した。
【0061】
以上の結果から、C001、C002、C003、C004の各DNA配列を含む本発明の組換えベクターで形質転換された麹菌は麹菌原株に対して著しく蛋白質加水分解酵素分泌が増大していることが判明した。また、液体培地であるゼラチン培地、固体培地であるふすま培地においてそれぞれ蛋白質加水分解酵素活性が増大し、産業上の利用可能性が高いものであった。
【0062】
これらのうち、本発明におけるC001、C002、C003、C004のDNA配列は、配列表1〜4のとおりである(配列中の初めから101番目が開始コドンの先頭塩基、終わりから101番目が終止コドンの最終塩基を示す)。C001は、C2H2型のZinc finger モチーフを持ち、アスペルギルス・ニドランスのsteA遺伝子とアミノ酸レベルで81%同一の相同性を示した。steA遺伝子は、アスペルギルス・ニドランスにおいて、有性生殖に必要とされる遺伝子として報告されている(Vallimら、Mol Microbiol. 36(2):290−301, (2000))。
【0063】
C002は、Zn2−Cys6(Fungal zinc binuclear cluster)型のzinc fingerモチーフを持ち、アスペルギルス・フミガタスのC6 finger domain protein, putativeをコードする機能未知の遺伝子と、アミノ酸レベルで67%同一の相同性を示した。C002と相同性を示す遺伝子のうち、機能が報告されているもので最も高い相同性を示したのは、ネクトリア・ヘマトコッカの、クチナーゼ遺伝子の転写に関わるnaf遺伝子であったが、その相同性は、アミノ酸レベルで31%同一に過ぎなかった。
【0064】
C003は、Zn2−Cys6(Fungal zinc binuclear cluster)型のzinc fingerモチーフを持ち、アスペルギルス・フミガタスのC6 finger domain protein, putativeをコードする機能未知の遺伝子(上記C002と最も高い相同性を示す遺伝子とは別遺伝子)と、アミノ酸レベルで55%同一の相同性を示した。C002と相同性を示す遺伝子のうち、機能が報告されているもので最も高い相同性を示したのは、アスペルギルス・ニドランスの、硝酸同化関連遺伝子の転写に関わるnirA遺伝子であったが、その相同性は、アミノ酸レベルで22%同一に過ぎなかった(以上の相同性は、BLAST(blastp)により、NCBIのnrデータベースを対象とした相同性検索の結果を示した)。
【0065】
C004は、C2H2型のZinc fingerモチーフを持ち、アスペルギルス・ニドランスのamdX遺伝子とアミノ酸レベルで73%同一の相同性を示した。amdX遺伝子は、アスペルギルス・ニドランスにおいて、アセトアミダーゼをコードするamdS遺伝子の転写活性化因子として報告されている(Murphyら、Mol Microbiol. 23(3):591−602, (1997))。
【0066】
従って、これらの従来公知の遺伝子は全て、遺伝子配列、及び相同遺伝子の機能から強制発現により蛋白質加水分解酵素活性を増大させることを何等示唆されるものではない。
【産業上の利用可能性】
【0067】
本発明は、調味料の食品製造や、消化剤等の医薬品製造、洗剤用の蛋白質加水分解酵素生産等において産業上極めて重要でかつ有用なものである。
【技術分野】
【0001】
本発明は、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する組換えベクター、該ベクターによって形質転換された麹菌により蛋白質加水分解酵素遺伝子発現・分泌が亢進した麹菌、形質転換された麹菌による蛋白質加水分解酵素生産方法等に関する。
【背景技術】
【0002】
アスペルギルス・オリゼ(Aspergillus oryzae)及びアスペルギルス・ソーヤ(Aspergillus sojae)等の麹菌は醤油、酒、味噌などの醸造食品の製造に工業的に用いられている。さらに、近年、他の生物種同様アスペルギルス・オリゼのゲノム配列が明らかとなり(特開2005−176602号公報)、遺伝子の機能的解析の重要性がますます高まってきている。
【0003】
このような麹菌は、様々な酵素類(蛋白質加水分解酵素、アミラーゼ類等)を産生・分泌し、その優れた澱粉糖化能力及び蛋白質分解力等により、醸造食品の製造に幅広く利用されている。
【0004】
従来、特許文献1〜3に記載されているような、麹菌の遺伝子発現を制御する様々な転写因子の解析が行われてきた。
【先行技術文献】
【特許文献】
【0005】
【特許文献1】特開2003−240号公報
【特許文献2】特開2003−70484号公報
【特許文献3】特開2003−235584号公報
【特許文献4】特開2005−176602号公報
【発明の概要】
【発明が解決しようとする課題】
【0006】
本発明は、新たに、麹菌の蛋白質加水分解酵素の分泌を増大させる組換えベクターを提供すること、及び、該組換えベクターを用いることによって固体培地培養及び液体培地培養での蛋白質含有物の分解の効率が向上した麹菌を提供すること等を目的とする。
【課題を解決するための手段】
【0007】
即ち、本発明は、以下の各態様の発明に係るものである。
(1)配列番号1、2、3又は4で表される塩基配列からなるDNAを含む組換えベクター。
(2)配列番号1、2、3又は4で表される塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNAを含む組換えベクター。
(3)配列番号1、2、3又は4で表される塩基配列からなるDNAと80%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNAを含む組換えベクター。
(4)上記1、2又は3記載の組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする麹菌。
(5)上記4記載の麹菌を、固体培地若しくは液体培地で培養して培地中に蛋白質加水分解酵素を分泌させたのち、培地から蛋白質加水分解酵素を採取することを特徴とする、蛋白質加水分解酵素の生産方法。
(6)上記4記載の麹菌を培養して得られる培養物と蛋白質含有原料とを混合し、原料中の蛋白質を分解することを特徴とする、蛋白質分解物の製造方法。
(7)上記4記載の麹菌を培養して得られる培養物とゼラチン含有原料とを混合し、原料中のゼラチンを分解することを特徴とする調味液の製造方法。
(8)以下の(a)、(b)又は(c)のDNA:
(a)配列番号1、2、3又は4で表される塩基配列からなるDNA、
(b)(a)の塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA、
(c)(a)の塩基配列からなるDNAと80%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA。
(9)以下の(a)又は(b)の蛋白質:
(a)配列番号1、2、3又は4で表される塩基配列からなるDNAにコードされるアミノ酸配列からなる蛋白質、
(b)(a)のアミノ酸配列において、1個若しくは数個のアミノ酸が欠失、置換、若しくは付加されたアミノ酸配列からなり、麹菌蛋白質分解酵素の分泌を増大する機能を有する活性を有する蛋白質。
【発明の効果】
【0008】
本発明の組換えベクターを用いることによって、麹菌の蛋白質加水分解酵素の分泌を増大させることが可能になった。該組換えベクターで形質転換麹菌を用いることによって固体培地培養及び液体培地培養での蛋白質含有物の分解の効率を向上させることができる。
【図面の簡単な説明】
【0009】
【図1】本発明の形質転換体(形質転換菌)において、形質転換に用いたプラスミドが形質転換体のゲノム上のniaD遺伝子領域に挿入されていることを示す、サザン解析の結果により得られた電気泳動の写真である。
【図2】フスマ培地における、野生株と本発明の形質転換体の蛋白質加水分解酵素活性を比較した結果を示す。
【発明を実施するための形態】
【0010】
本発明の組換えベクターに含まれる配列番号1、2、3又は4で表される塩基配列からなるDNAは麹菌のゲノム由来であり、以下の実施例に具体的に記載されるように、アスペルギルス・オリゼの全ゲノム情報(特開2005−176602号公報)に基づき、相同性検索、モチーフ検索、及び、既知遺伝子のアノテーションや文献情報、モチーフの種類などの情報をもとに、転写調節遺伝子をコードするDNAと推定されたものであり、実施例に示すような、適当なプライマーを用いるPCRにより麹菌のゲノムDNA等から取得することができる。
【0011】
更に、本発明における「配列番号1、2、3又は4で表される塩基配列からなるDNA」には、アミノ酸をコードする領域のみから成る塩基配列(即ち、エクソン部分のみが結合された塩基配列)からなるcDNAも含まれる。このようなcDNAは、本明細書に開示の塩基配列情報に基づき調製した適当なプライマーを用いて、麹菌のmRNAを鋳型としたPCR等により取得することが可能である。又、本発明に関するDNAは当業者に公知の任意の方法によって化学合成することも可能である。
【0012】
さらに、本発明の組換えベクターには、上記DNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNA、及び、上記DNAと約80%以上、好ましくは約95%以上である配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNAが含まれる。
【0013】
ここで、ハイブリダイゼーションは、Molecular cloninng third.ed.(Cold Spring Harbor Lab.Press,2001)に記載の方法等、当業界で公知の方法あるいはそれに準じる方法に従って行うことができる。また、市販のライブラリーを使用する場合、添付の使用説明書に記載の方法に従って行うことができる。
【0014】
ハイブリダイゼーションは、例えば、カレント・プロトコールズ・イン・モレキュラー・バイオロジー(Current protocols in molecular biology(edited by Frederick M. Ausubel et al.,1987)に記載の方法等、当業界で公知の方法あるいはそれに準じる方法に従って行うことができる。また、市販のライブラリーを使用する場合、添付の使用説明書に記載の方法に従って行うことができる。
【0015】
本明細書において、「ストリンジェントな条件下」とは、例えば、温度60℃〜68℃において、ナトリウム濃度150〜900mM、好ましくは600〜900mM、pH6〜8であるような条件を挙げることができる。
【0016】
従って、配列番号1ないし配列番号4で表される塩基配列を含むDNAと相補的な塩基配列からなるDNAとハイブリダイズできるDNAとしては、例えば、該DNAの全塩基配列との相同性の程度が、全体の平均で、約80%以上、好ましくは約95%以上である塩基配列を含有するDNA等を挙げることができる。なお、塩基配列間の同一性は、当業者に公知のアルゴリズム、例えば、Blastを用いて決定することができる。
【0017】
従って、本発明は、配列番号1ないし配列番号4で表される塩基配列を含むDNA、該DNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNA、及び、上記DNAと約80%以上、好ましくは約95%以上である配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA、並びに、配列番号1、2、3又は4で表される塩基配列からなるDNAにコードされるアミノ酸配列からなる蛋白質、及び、これらのアミノ酸配列からなり麹菌蛋白質分解酵素の分泌を増大する機能を有する活性を有する蛋白質にも係るものである。
【0018】
2つの塩基配列又はアミノ酸配列における配列相同性を決定するために、配列は比較に最適な状態に前処理される。例えば、一方の配列にギャップを入れることにより、他方の配列とのアラインメントの最適化を行う。その後、各部位におけるアミノ酸残基又は塩基が比較される。第一の配列におけるある部位に、第二の配列の相当する部位と同じアミノ酸残基又は塩基が存在する場合、それらの配列は、その部位において同一である。2つの配列における配列相同性は、配列間での同一である部位数の全部位(全アミノ酸又は全塩基)数に対する百分率で示される。
【0019】
上記の原理に従い、2つの塩基配列又はアミノ酸配列における配列相同性は、例えば、Karlin及びAltschulのアルゴリズム(Proc.Natl.Acad.Sci.USA 87:2264−2268、1990及びProc.Natl.Acad.Sci.USA 90:5873−5877、1993)により決定される。このようなアルゴリズムを用いたBLASTプログラムやFASTAプログラムは、主に与えられた配列に対し、高い配列相同性を示す配列をデータベース中から検索するために用いられる。これらは、例えば米国National Center for Biotechnology Informationのインターネット上のウェブサイトにおいて利用可能である。
【0020】
上記のような塩基配列の配列相同性又はコードするアミノ酸配列の配列相同性を示すようなDNAは、上記のようにハイブリダイゼーションを指標に得ることもでき、ゲノム塩基配列解析等によって得られた機能未知のDNA群又は公共データベースの中から、本技術分野の研究者か通常用いている方法により、例えば、前述のBLASTソフトウェアを用いた検索により発見することも容易である。さらに、本発明遺伝子は種々の公知の変異導入方法によって得ることもできる。
【0021】
本発明の組換えベクターは、当業者に公知の適当な遺伝子工学的な手段を用いて、上記のDNAをベクター内に連結することにより調製することができる。ベクターとしては、形質導入又は形質転換する宿主微生物のゲノム上の適当な位置に上記のDNAが挿入され、その緒果、親株と比較して、その宿主微生物の蛋白質加水分解酵素分泌能を増大することができるものであれば、その構造及び種類等に、特に、制限はない。例えば、プラスミド、コスミド、ファージ、ウイルス、染色体組み込み型、人工染色体等のベクターを用いることかできる。
【0022】
上記ベクターには、形質転換された細胞を選択することを可能にするための当業者に公知の任意のマーカー遺伝子が含まれていてもよい。マーカー遺伝子としては、例えば、URA3、niaDのような、宿主の栄養要求性を相補する遺伝子又はアンピシリン、カナマイシンあるいはオリゴマイシン等の薬剤に対する抵抗遺伝子等が挙げられる。また、組換えベクターは、宿主細胞中で本発明の遺伝子を発現することのできるプロモーター及びその他の制御配列(例えば、エンハンサー配列、ターミネーター配列、ポリアデニル化配列等)、さらに、目的とするDNAを挿入するためのマルチクローニングを含むことが望ましい。これらの組換えベクターに含まれる各要素は当業者に公知であり、例えば、具体的には、実施例に示されるように、アスペルギルス・オリゼのアミラーゼ遺伝手プロモーター、アスペルギルス・ニーデュランスのアミラーゼターミネーター、マーカー遺伝子としてのアスペルキルス・オリゼのniaD遺伝子を挙げることができる。
【0023】
形質導入又は形質転換は、公知の適当な方法で行うことができる。例えば、プロトプラスト化した後ポリエチレングリコール及び塩化カルシウムを用いる方法(Mol.Gen.Genet.,218,99−104(1989))を用いることかできる。
【0024】
本発明の組換えベクターを形質導入又は形質転換された麹薗は、親株と比較して蛋白質加水分解酵素分泌が増大されたものとなる。本明細書において、「蛋白質加水分解酵秦の分泌を増大する」、又は、「蛋白質加水分解酵素の分泌が増大している」とは、最終的な結果(現象)として、菌体外へ蛋白質加水分解酵素の分泌量(分泌能)が増大していることを意昧し、その原因・機構としては、例えば、蛋白質加水分解酵素の発現量(産生量)自体が増大する場合と、産生された該酵素の菌体外への分泌量(分泌能)が増大している場合等が考えられる。さらに、ここで、「増大する」とは、以下の実施例に記載されているように、ゼラチン又はアゾカゼインを蛋白質として使用する測定方法を用いて、本発明の組換えベクターで形質導入又は形質転換された菌の培地中の蛋白質加水分解酵素量(活性)が、親株と比較して有意に増大することを意味する。
【0025】
従って、該ベクターに含まれる各配列にコードされる蛋白質は、以下の実施例に記載されるように、転写調節因子であると予測されるが、必ずしも蛋白質加水分解酵素の転写調節エレメントに結合し、該蛋白質加水分解酵素の発現を促進させる機能を有するものに限られず、本発明の組換えベクターにより形質導入又は形質転換された麹薗において、発現された蛋白質加水分解酵素の菌体外への分泌を促進させる機能を有するものであっても良い。
【0026】
麹菌とは、アスペルギルス属に属する微生物(菌類)の総称であり、その代表的な例として、既に記載したアスペルギルス・オリゼ(Aspergillus oryzae)及びアスペルギルス・ソーヤ(Aspergillus sojae)に加えて、特に食品・醸造分野で使用される菌としてアスペルギルス・アワモリ(Aspergillus awamori)及びアスペルギルス・ニガー(Aspergillus niger)等を挙げることができる。また、麹菌以外の食品・醸造・化学・医療分野で使用される産業上有用な糸状菌等を用いても実質的に同様の効果を得ることができる。これらの菌は、市販品として、又は、アメリカンタイプカルチャーコレクション(ATCC)等の様々な公的寄託機関から入手することも可能である。
【0027】
また、「蛋白質加水分解酵素」はプロテアーゼとも呼ばれ、蛋白質を主に分解するプロテイナーゼ、又はエンドペプチダーゼ、小ペプチドを分解するペプチダーゼなどを含む総称である。
【0028】
親株と比較して蛋白質加水分解酵素分泌能が増大している本発明の麹菌を当業者に公知の任意の方法で培養することにより、それが分泌する蛋白質分解酵素を生産することができる。例えば、本発明の麹菌を、固体培地若しくは液体培地で培養して培地中に蛋白質加水分解酵素を分泌させたのち、培地から蛋白質加水分解酵素を採取することが可能である。このような生産方法における、培養系・培地の選択、培養温度・時間等の培養条件は、以下の実施例等を参考に、当業者が適宜設定することができる。
【0029】
また、本発明は麹菌を培養して得られる培養物と蛋白質含有原料とを混合し、原料中の蛋白質を分解することによって、蛋白質分解物を製造することができる。この方法で使用される原料に含まれる蛋白質の種類に特に制限はなく、例えば、ゼラチン、コラーゲン及びグルテン等を挙げることができる。例えば、ゼラチン、コラーゲン及びグルテン等含有原料の蛋白質分解物は調味液として有用である。
【0030】
なお、組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする本発明の麹菌の具体例である、C001、C002、C003及びC004は平成17年9月9日付で、独立行政法人産業技術総合研究所特許生物寄託センターに寄託され、受領番号FERM AP−20659、AP−20660、FERM AP−20661及びFERM AP−20662がそれぞれ付与されている。
【0031】
以下、実施例に則して本発明をさらに説明するが、本発明の技術的範囲は、これらに限定されるものでない。
【実施例1】
【0032】
(転写調節因子遺伝子の推定と抽出)
麹菌アスペルギルス・オリゼの全ゲノム情報(特開2005−176602号公報参照)から、次のような方法により、転写調節因子遺伝子をコードすると推定される配列を抽出した。このステップにおいては、詳細な吟味は行わず、転写調節因子遺伝子である可能性のあるものは基本的にリストアップの対象とした。
(1)相同性検索による転写調節因子遺伝子と推定される配列の抽出
麹菌アスペルギルス・オリゼの全ゲノム情報から、遺伝子自動予測ソフトにより遺伝子配列と推定されるDNA配列をすべて抽出し、これらのDNA配列を自動予測遺伝子配列(以下、「自動予測遺伝子配列」という)とした。
【0033】
各々の自動予測遺伝子配列から予測されるその遺伝子産物配列((以下、「自動予測遺伝子産物配列」という)を基礎配列として、既知蛋白質の公共データベース(NCBIによる非重複蛋白質データベースnr)に対して、相同性検索ソフトBLASTを用いて相同性検索を行った。相同性検索の結果得られた配列の機能に関する情報を整理し、その配列に関わる遺伝子機能情報に対してキーワード検索を行い、転写調節因子に関わるキーワードを含む自動予測遺伝子配列を選択した。
(2)転写調節因子の構造に関わるモチーフと推定されるアミノ酸配列をコードするDNA配列の検索
自動予測遺伝子産物配列について、モチーフ検索ソフト(HMMER)を用いてモチーフ(Pfam)検索を行い、転写調節因子に関わるモチーフを含むと推定されるアミノ酸配列をコードする自動予測遺伝子配列を選択した。
(3)既知転写調節因子配列と相同性を有するDNA配列の検索
麹菌や麹菌以外の糸状菌に関する既知の転写調節因子のアミノ酸配列を問い合わせ配列として、今回得られた自動予測遺伝子産物配列に対して、相同性検索ソフトBLASTを用いて相同性検索を行った。同様な問い合わせ配列を用いて、麹菌のゲノムコンティグ配列に対してBLAST(tblast)を用いて相同性検索を行うことにより、自動予測で予測されていなかった遺伝子や、自動予測でDNA結合領域などの保存性の高い領域を欠損した形で予測されてしまっていた遺伝子についても検索可能とした。
【0034】
これらの方法により、自動予測遺伝子配列から合計667個の候補遺伝子(以下、「候補遺伝子」という)を抽出した。
【0035】
(候補遺伝子の絞込みと遺伝子領域のin silico推定)
前項で抽出した転写調節因子遺伝子と思われる候補遺伝子について、順次遺伝子領域の推定を行った。また、推定に先立ち、相同な既知遺伝子のアノテーションや文献情報、モチーフの種類などの情報をもとに、強制発現による解析対象として相応しいか否かを検討し、ヘテロ複合体で機能するものなど、相応しくないものは除外した。
【0036】
遺伝子領域の推定は、主に相同な既知遺伝子との比較により行った。相同な既知遺伝子との比較は、BLAST、FASTA及びALN(Bioinfomatics 2000 16: 190−202)を用いて行った。既知遺伝子との相同性が限定的で5’端が推定できなかった場合は、5’RACEを行うために、相同部分のアラインメント情報を準備した。3’末端の予測が本来の遺伝子領域より短すぎた場合は、転写調節因子のC末側がトランケートされてしまうが、予測が長すぎた場合は、本来のC末端で翻訳は終止すると考えられる。そこで、3’末端が予測できなかった場合は、相同部分からの距離や、隣の遺伝子との位置関係などから、本来の3’末端より十分に下流部分までを強制発現ベクターに組み込む領域とすることにした。
【0037】
このような方法で、前項の抽出の際に既知の遺伝子との相同性が高かったものから順次遺伝子領域の推定を行った。全長がin silicoで推定できたものと5’RACEにより5’末端が明らかにされたもの一部の合計300遺伝子について強制発現株作成ための予測配列を得た。
【実施例2】
【0038】
(発現用プラスミドの作製)
転写調節因子遺伝子を麹菌内で発現させるために用いる発現プラスミドとして、発現ユニットとしてアスペルギルス・オリゼアミラーゼ遺伝子プロモーター及びアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーター、転写調節因子遺伝子挿入用マルチクローニングサイト、マーカー遺伝子としてアスペルギルス・オリゼniaD遺伝子を含むプラスミドpAPTLNを構築した。
【0039】
寒天プレート培地上で生育させた、アスペルギルス・ニドランスIAM2130株の胞子5×105個を掻き取り、滅菌したマイクロチューブに入れた300μlのNuclei Lysis Solutionバッファー(Promega社製)に懸濁した。胞子を懸濁したマイクロチューブに1gのガラスビーズBZ−06(アズワン株式会社製)を加え、tissue lyser(QIAGEN社製)を用いて、25回/秒、3分間処理を2回繰り返した。65℃で15分間加温した後、室温で5分間放置した。1.5μlのRnase Solution(10mg/ml)を加え、37℃で1時間加温した。100μlのProtein Precipitation Solutionを加え20秒間激しく振とうした後、13,500rpmで5分間遠心処理した。遠心後の上清を別のマイクロチューブに移し、350μlのイソプロパノールを加え転倒混和した後、12,000rpmで3分間遠心処理した。得られた沈殿を70%エタノールで洗浄および乾燥処理した後、100μlのTEバッファー〔10mM Tris−HCl(pH7.5),1mM EDTA〕に懸濁しゲノムDNA溶液を得た。得られたゲノムDNAを鋳型として、下記のプライマーを用いてPCRを行いアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーター領域の増幅を行った。
5’−gggtagtcgtacccgatgatgaaac−3’(配列番号5)
5’−agcctaggccgctgcaggcag−3’ (配列番号6)
PCR反応は、TaKaRa LA Taq(タカラバイオ株式会社製)を使用し、PTC−200(MJ Research社製)を用いて行った。反応液の組成は以下のとおりである。
【0040】
(試薬:使用量:終濃度)
TaKaRa LA Taq:0.5μl
10×LA PCR BufferII:5μl:1×
25mM MgCl2:5μl:2.5mM
dNTP Mixture:8μl:それぞれ0.4mM
鋳型DNA(0.5μg):1μl
プライマー:1μl×2種類:それぞれ0.2μM
滅菌水:28.5μl
合計液量:50μl
上記の反応液50μlを0.2ml反応チューブ中で混合してPTC−200にセットし、以下の温度設定によりPCRを行った。
95℃、2分:1サイクル
95℃、30秒 58℃、30秒 72℃、2分:30サイクル
72℃、3分:1サイクル。
【0041】
反応液をエタノール沈澱処理後、沈殿を20μlのTEバッファーに懸濁した。さらに、PstIで消化した後、0.7%アガロースゲルで電気泳動し、目的の増幅産物を切り出した。切り出した増幅産物はゲルエクストラクションキット(QIAGEN社製)を用いて精製し、アスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターを得た。プラスミドpUC19をSmaI及びPstIで処理した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いて約2.7kbのDNA断片を回収した。回収したDNA断片とアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターをライゲーションした後、大腸菌JM109を形質転換し、pUC19のマルチクローニングサイト内にあるSmaI−PstI部位にアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターが挿入されたプラスミドpATを得た。
【0042】
pATをSmaI及びHindIIIで消化した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いてアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターを含むDNA断片を回収した
プラスミドpMAR5(Biosci.Biotech.Biochem.,56(10),1674−1675,1992)をSmaIおよびHindIIIで消化した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いてargB遺伝子およびアスペルギルス・オリゼアミラーゼ遺伝子ターミネーターを除去したDNA断片を回収した。pATおよびpMAR5由来の両DNA断片をライゲーションした後、大腸菌JM109を形質転換し、アスペルギルス・オリゼアミラーゼ遺伝子プロモーターおよびアスペルギルス・ニドランスアミラーゼ遺伝子ターミネーターを含むプラスミドpAPTを得た。
【0043】
下記の2種類の合成DNAを含む(各100μM)TEバッファー100μlを5分間煮沸後、室温まで徐冷しマルチクローニングサイトリンカーとした。pAPTをEcoRIおよびSmaIで消化した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いてマルチクローニングサイトを除去したDNA断片を回収した。回収したDNA断片にマルチクローニングサイトリンカーをライゲーションした後、大腸菌JM109を形質転換しプラスミドpAPTLを得た。プラスミドpST14〔(Mol.Gen.Genet(1989)218:99−104)をHindIIIで消化した後、0.7%アガロースゲルで電気泳動し、ゲルエクストラクションキット(QIAGEN社製)を用いてアスペルギルス・オリゼniaD遺伝子を含むDNA断片を回収した。回収したniaD遺伝子を含むDNA断片をpAPTLのHindIII部位に挿入し、転写調節因子遺伝子発現用プラスミドpAPTLNを得た。
【0044】
(転写調節因子遺伝子発現用組換えベクターの作製)
転写調節因子遺伝子と予測されたDNA配列を基にPCRにより各転写調節因子遺伝子を取得した。まず、アスペルギルス・オリゼRIB40のゲノムDNAを調製した。次に調製したゲノムDNAを鋳型に、予測遺伝子の開始コドン上流100bpのDNA配列及び終始コドン近傍のDNA配列を参考に作製した2種類のプライマーを用いて、PCRにより各転写調節因子遺伝子を取得した。開始コドン上流のDNA配列を参考にプライマーを作製する際には、pAPTLNのマルチクローニングサイト上にある制限酵素認識配列であり、かつ、増幅する転写調節因子遺伝子配列内に認識配列が存在しない制限酵素認識配列を導入した。増幅した転写調節因子遺伝子は作製したプライマーに認識配列を導入した制限酵素で処理した後、pAPTLNのマルチクローニングサイトに挿入し(導入した制限酵素部位とSmaI部位の間)、各転写調節因子遺伝子発現用組換えプラスミドベクターとした。
【0045】
以下、転写調節因子遺伝子発現用プラスミドベクターの作製の一例を示す。
【0046】
前記の方法を用いてアスペルギルス・オリゼRIB40株より調製したゲノムDNAを鋳型として、下記のプライマーを用いてPCRを行い、転写調節因子遺伝子C001(配列番号1)を得た。
5’−atacaggcattctatcgataaaatgtttcc−3’(配列番号7)
5’−gggctacatctgctgttgtagaagttgc−3’ (配列番号8)
PCR反応は、TOYOBO KOD−Plus−DNA Polymerase(TOYOBO社製)を使用し、PTC−200(MJ Research社製)を用いて行った。反応液の組成は以下のとおりである。
【0047】
(試薬:使用量:終濃度)
KOD−Plus−DNA Polymerase:1μl
10×PCR buffer for KOD−Plus−:5μl:1×
25mM MgCl2:2μl:1mM
2mM dNTP Mixture:5μl:それぞれ0.2mM
鋳型DNA(0.2μg):1μl
プライマー:1μl×2種類:それぞれ0.3μM
滅菌水:34μl
合計液量:50μl
上記の反応液50μlを0.2ml反応チューブ中で混合してPTC−200にセットし、以下のような温度設定によりPCRを行った。
94℃、2分:1サイクル
94℃、15秒 58℃、30秒 68℃、4分:30サイクル
68℃、3分:1サイクル。
【0048】
反応液をエタノール沈澱処理後、沈殿を20μlのTEバッファー〔10mM Tris−HCl(pH7.5),1mM EDTA〕に懸濁した。更に、ClaIで消化した後、0.7%アガロースゲルで電気泳動し、目的の増幅産物を切り出した。切り出した増幅産物はゲルエクストラクションキット(QIAGEN社製)を用いて精製した後、pAPTLNのマルチクローニングサイト上のClaI−SmaI部位に挿入し、転写調節因子遺伝子C001発現用プラスミドpC001を得た。
【0049】
同様の方法を用いて転写調節因子遺伝子C002発現用プラスミドpC002、転写調節因子遺伝子C003発現用プラスミドpC003、転写調節因子遺伝子C004発現用プラスミドpC004を得た。
【0050】
転写調節因子遺伝子C002(配列番号2)を増幅する際には、ClaIの代わりにNheI認識配列を導入した下記の2種類のプライマーを用いて増幅、精製後、pAPTLNのマルチクローニングサイト上のClaI−SmaI部位に挿入した。
5’−tcatacaagctagcaaaatggcggaga−3’(配列番号9)
5’−gggctcgataactttttactcccgtgata−3’ (配列番号10)
【0051】
転写調節因子遺伝子C003(配列番号3)を増幅する際には、ClaIおよびSpeI認識配列を導入した下記の2種類のプライマーを用いて増幅、精製後、pAPTLNのマルチクローニングサイト上のClaI−SpeI部位に挿入した。
5’−ccatcgataatattagtatgctgaatga−3’(配列番号11)
5’−ggactagttcaggtctttcgaatgtcagga−3’(配列番号12)
【0052】
転写調節因子遺伝子C004(配列番号4)を増幅する際には、ClaI及びSpeI認識配列を導入した下記の2種類のプライマーを用いて増幅、精製後、pAPTLNのマルチクローニングサイト上のClaI−SpeI部位に挿入した。
5’−ccatcgataagtaaaaggatgttattagat−3’(配列番号13)
5’−ggactagtttaatccgttctcatggccgaa−3’ (配列番号14)
【実施例3】
【0053】
(転写調節因子遺伝子強制発現麹菌の取得)
アスペルギルス・オリゼRIB326株からMol.Gen.Genet(1989)218:99−104記載の方法で取得したniaD欠損株を転写調節因子遺伝子発現用プラスミドpC001、pC002、pC004、pC003で形質転換した。形質転換法は、プロトプラスト化した後ポリエチレングリコール及び塩化カルシウムを用いる方法(Mol.Gen.Genet(1989)218:99−104)によって行った。各プラスミド5μgを用いて形質転換し、最小培地で形質転換体を選択したところ、それぞれ約200個のコロニーが得られた。このうち各プラスミドにつき15コロニーについて最小培地で単分生子分離を繰り返し、形質の安定化を行った。次に、各転写調節因子遺伝子の成熟領域をプローブとしてサザン解析を実施し、それぞれの形質転換体の中から、形質転換に用いたプラスミドが形質転換体のゲノム上のniaD遺伝子領域に挿入されている株を選択し、それぞれの選択株をC001、C002、C003、C004と命名した。サザン解析の結果を図1に示した。
【実施例4】
【0054】
(麹菌の培養方法及び菌体外酵素活性の測定)
(1)ゼラチン培地における麹菌の培養方法とゼラチン分解に関する蛋白質加水分解酵素活性の測定
各形質転換体をゼラチンを含む液体培地で振とう培養し、ゼラチン分解能を確認した。各形質転換体のゼラチン分解能の評価は、ゼラチンが分解されて生じる培地中のグルタミン酸濃度をコントロールと比較することで行った。
【0055】
寒天最小培地上で生育させた各形質転換体の胞子を3mlの滅菌水で掻き取り、150ml容三角フラスコに入れた40mlのゼラチン培地(2% Gelatin、0.1% KH2PO4、0.05% MgSO4、0.05% KCl、0.001% FeSO4・7H2O、0.3%NaNO3、3% Maltose pH6.0)に植菌した(1×107個胞子/40mlゼラチン培地)。30℃、150rpmで115時間振とう培養した後、培養上清中のグルタミン酸量をヤマサ L−グルタミン酸測定キット(ヤマサ醤油株式会社製)を用いて測定した。また、上記のゼラチン分解能を指標として、培養上清の蛋白質加水分解酵素活性を測定した。コントロールとして、実験毎にRIB326株niaD欠損株をpAPTLNで形質転換して得られたniaD+株を培養した。形質転換体の中で最もゼラチン分解能が高いと思われるC004培養上清中のグルタミン酸量及び蛋白質加水分解酵素活性をコントロールに対する相対値として示した。
【0056】
【表1】

【0057】
(2)フスマ培地における麹菌野生株との培養方法と本発明の形質転換体の蛋白質加水分解酵素活性の比較
フスマ:脱イオン水を5:4の比率で混合し、室温で30分間放置した後、150ml容量の三角フラスコに5gずつ分注し、オートクレーブで15分滅菌した。次いで、予め血球計で胞子数を計測しておいた野生株アスペルギルス・オリゼRIB326株や強制発現株の胞子懸濁液を、胞子がフスマ培地1g当たり5×105個になるように接種した。培養は30℃で4日間行い、培養開始後24時間目と48時間目に混合し、その後は培養終了まで静置した。酵素液は、以下のようにして得た。培養終了後のフスマ培地に、50mlの脱イオン水と500μlのトルエンを加え、2時間室温で振とうし、懸濁液をNo.2濾紙(アドバンテック社製)で濾過し濾液を得て、これを酵素液とした。
【0058】
蛋白質加水分解酵素活性は、以下の計算式で示されるように、野生株の酵素活性を1とした時の比率で比較した。酵素活性比率=強制発現株(試料の吸光度-ブランクの吸光度)/野生株(試料の吸光度-ブランクの吸光度)。また、蛋白質加水分解酵素活性測定を下記のようにして行い、試料とブランクの吸光度を求めた。
【0059】
予め30℃に温めた2mlの基質液(1%アゾカゼイン、0.05Mリン酸緩衝液:pH7.0)に酵素液20μlを加えて混合した。次に、これを30℃で20分反応させた後、反応停止液(10%トリクロロ酢酸)2mlを加え、反応を停止した。30℃で20分間放置して、生じた沈殿をアドバンテック社製No.5C濾紙で濾過し、濾液の吸光度(410nm)を測定した。ブランクは、酵素液添加前に反応停止液を加えて、同じ操作を行った。
【0060】
野生株と強制発現株の蛋白質加水分解酵素活性を比較した結果、C002、C001、C003が野生株の3倍の蛋白質加水分解酵素活性を示すことが確認された(図2)。また、C004についても野生株の2.2倍の蛋白質加水分解酵素活性を示した。
【0061】
以上の結果から、C001、C002、C003、C004の各DNA配列を含む本発明の組換えベクターで形質転換された麹菌は麹菌原株に対して著しく蛋白質加水分解酵素分泌が増大していることが判明した。また、液体培地であるゼラチン培地、固体培地であるふすま培地においてそれぞれ蛋白質加水分解酵素活性が増大し、産業上の利用可能性が高いものであった。
【0062】
これらのうち、本発明におけるC001、C002、C003、C004のDNA配列は、配列表1〜4のとおりである(配列中の初めから101番目が開始コドンの先頭塩基、終わりから101番目が終止コドンの最終塩基を示す)。C001は、C2H2型のZinc finger モチーフを持ち、アスペルギルス・ニドランスのsteA遺伝子とアミノ酸レベルで81%同一の相同性を示した。steA遺伝子は、アスペルギルス・ニドランスにおいて、有性生殖に必要とされる遺伝子として報告されている(Vallimら、Mol Microbiol. 36(2):290−301, (2000))。
【0063】
C002は、Zn2−Cys6(Fungal zinc binuclear cluster)型のzinc fingerモチーフを持ち、アスペルギルス・フミガタスのC6 finger domain protein, putativeをコードする機能未知の遺伝子と、アミノ酸レベルで67%同一の相同性を示した。C002と相同性を示す遺伝子のうち、機能が報告されているもので最も高い相同性を示したのは、ネクトリア・ヘマトコッカの、クチナーゼ遺伝子の転写に関わるnaf遺伝子であったが、その相同性は、アミノ酸レベルで31%同一に過ぎなかった。
【0064】
C003は、Zn2−Cys6(Fungal zinc binuclear cluster)型のzinc fingerモチーフを持ち、アスペルギルス・フミガタスのC6 finger domain protein, putativeをコードする機能未知の遺伝子(上記C002と最も高い相同性を示す遺伝子とは別遺伝子)と、アミノ酸レベルで55%同一の相同性を示した。C002と相同性を示す遺伝子のうち、機能が報告されているもので最も高い相同性を示したのは、アスペルギルス・ニドランスの、硝酸同化関連遺伝子の転写に関わるnirA遺伝子であったが、その相同性は、アミノ酸レベルで22%同一に過ぎなかった(以上の相同性は、BLAST(blastp)により、NCBIのnrデータベースを対象とした相同性検索の結果を示した)。
【0065】
C004は、C2H2型のZinc fingerモチーフを持ち、アスペルギルス・ニドランスのamdX遺伝子とアミノ酸レベルで73%同一の相同性を示した。amdX遺伝子は、アスペルギルス・ニドランスにおいて、アセトアミダーゼをコードするamdS遺伝子の転写活性化因子として報告されている(Murphyら、Mol Microbiol. 23(3):591−602, (1997))。
【0066】
従って、これらの従来公知の遺伝子は全て、遺伝子配列、及び相同遺伝子の機能から強制発現により蛋白質加水分解酵素活性を増大させることを何等示唆されるものではない。
【産業上の利用可能性】
【0067】
本発明は、調味料の食品製造や、消化剤等の医薬品製造、洗剤用の蛋白質加水分解酵素生産等において産業上極めて重要でかつ有用なものである。
【特許請求の範囲】
【請求項1】
以下のDNA:
(1)配列番号1で表される塩基配列からなるDNA;
(2)配列番号1で表される塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA;又は
(3)配列番号1で表される塩基配列からなるDNAと95%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA
を含む組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする麹菌を、固体培地若しくは液体培地で培養して培地中に蛋白質加水分解酵素を分泌させたのち、培地から蛋白質加水分解酵素を採取することを特徴とする、蛋白質加水分解酵素の生産方法。
【請求項2】
以下のDNA:
(1)配列番号1で表される塩基配列からなるDNA;
(2)配列番号1で表される塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA;又は
(3)配列番号1で表される塩基配列からなるDNAと95%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA
を含む組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする麹菌を培養して得られる培養物と蛋白質含有原料とを混合し、原料中の蛋白質を分解することを特徴とする、蛋白質分解物の製造方法。
【請求項3】
以下のDNA:
(1)配列番号1で表される塩基配列からなるDNA;
(2)配列番号1で表される塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA;又は
(3)配列番号1で表される塩基配列からなるDNAと95%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA
を含む組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする麹菌を培養して得られる培養物とゼラチン含有原料とを混合し、原料中のゼラチンを分解することを特徴とする調味液の製造方法。
【請求項1】
以下のDNA:
(1)配列番号1で表される塩基配列からなるDNA;
(2)配列番号1で表される塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA;又は
(3)配列番号1で表される塩基配列からなるDNAと95%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA
を含む組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする麹菌を、固体培地若しくは液体培地で培養して培地中に蛋白質加水分解酵素を分泌させたのち、培地から蛋白質加水分解酵素を採取することを特徴とする、蛋白質加水分解酵素の生産方法。
【請求項2】
以下のDNA:
(1)配列番号1で表される塩基配列からなるDNA;
(2)配列番号1で表される塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA;又は
(3)配列番号1で表される塩基配列からなるDNAと95%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA
を含む組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする麹菌を培養して得られる培養物と蛋白質含有原料とを混合し、原料中の蛋白質を分解することを特徴とする、蛋白質分解物の製造方法。
【請求項3】
以下のDNA:
(1)配列番号1で表される塩基配列からなるDNA;
(2)配列番号1で表される塩基配列からなるDNAと相補的な塩基配列からなるDNAとストリンジェントな条件下でハイブリダイズするDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA;又は
(3)配列番号1で表される塩基配列からなるDNAと95%以上の配列相同性を示す塩基配列からなるDNAであって、麹菌蛋白質加水分解酵素の分泌を増大する機能を有する蛋白質をコードするDNA
を含む組換えベクターにより形質導入又は形質転換され、親株と比較して蛋白質加水分解酵素分泌能が増大されていることを特徴とする麹菌を培養して得られる培養物とゼラチン含有原料とを混合し、原料中のゼラチンを分解することを特徴とする調味液の製造方法。
【図1】


【図2】




【図2】


【公開番号】特開2011−92204(P2011−92204A)
【公開日】平成23年5月12日(2011.5.12)
【国際特許分類】
【出願番号】特願2010−291589(P2010−291589)
【出願日】平成22年12月28日(2010.12.28)
【分割の表示】特願2005−278095(P2005−278095)の分割
【原出願日】平成17年9月26日(2005.9.26)
【国等の委託研究の成果に係る記載事項】(出願人による申告)平成15年度、経済産業省、地域申請コンソーシアム研究開発事業(麹菌ゲノム情報による新発酵プロセス開発技術の確立)委託研究、産業再生法第30条の適用を受ける特許出願
【出願人】(000173935)財団法人野田産業科学研究所 (9)
【出願人】(301021533)独立行政法人産業技術総合研究所 (6,529)
【出願人】(000112060)ヒゲタ醤油株式会社 (7)
【出願人】(591014710)千葉県 (49)
【出願人】(000158208)AGCテクノグラス株式会社 (81)
【出願人】(000004477)キッコーマン株式会社 (212)
【Fターム(参考)】
【公開日】平成23年5月12日(2011.5.12)
【国際特許分類】
【出願日】平成22年12月28日(2010.12.28)
【分割の表示】特願2005−278095(P2005−278095)の分割
【原出願日】平成17年9月26日(2005.9.26)
【国等の委託研究の成果に係る記載事項】(出願人による申告)平成15年度、経済産業省、地域申請コンソーシアム研究開発事業(麹菌ゲノム情報による新発酵プロセス開発技術の確立)委託研究、産業再生法第30条の適用を受ける特許出願
【出願人】(000173935)財団法人野田産業科学研究所 (9)
【出願人】(301021533)独立行政法人産業技術総合研究所 (6,529)
【出願人】(000112060)ヒゲタ醤油株式会社 (7)
【出願人】(591014710)千葉県 (49)
【出願人】(000158208)AGCテクノグラス株式会社 (81)
【出願人】(000004477)キッコーマン株式会社 (212)
【Fターム(参考)】
[ Back to top ]

