Bmal1の発現誘導を促進するRORα
【課題】
概日リズムの調節メカニズムのうち、未解明な部分を明らかにすること。
【解決手段】
本発明者らは、RORα(retinoic acid binding−receptor alpha、以下同じ)が、Bmal1の発現誘導を促進すること、及び、低酸素状態において、Bmal1の発現誘導が促進されること、を新規に見出した。このことは、低酸素状態などで、RORαの発現が促進されると、Bmal1の発現誘導が促進され、Bmal1の発現誘導が促進されると、BMAL1とCLOCKとの結合が促進され、Per遺伝子又はCry遺伝子の発現誘導が促進される、という概日リズムの調節メカニズムの存在を強く示唆する。従って、本発明に係る本発明は、時差ぼけ調整剤、抗がん剤として、適用できる可能性がある。
概日リズムの調節メカニズムのうち、未解明な部分を明らかにすること。
【解決手段】
本発明者らは、RORα(retinoic acid binding−receptor alpha、以下同じ)が、Bmal1の発現誘導を促進すること、及び、低酸素状態において、Bmal1の発現誘導が促進されること、を新規に見出した。このことは、低酸素状態などで、RORαの発現が促進されると、Bmal1の発現誘導が促進され、Bmal1の発現誘導が促進されると、BMAL1とCLOCKとの結合が促進され、Per遺伝子又はCry遺伝子の発現誘導が促進される、という概日リズムの調節メカニズムの存在を強く示唆する。従って、本発明に係る本発明は、時差ぼけ調整剤、抗がん剤として、適用できる可能性がある。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、時計遺伝子の作用メカニズム及びその周辺技術に関する。より詳細には、Bmal1(brain−muscle ARNT−like protein 1)の発現誘導を促進するRORα(retinoic acid receptor−related orphan receptor)、並びに抗がん剤、時差ぼけ調整剤、それらの薬剤のスクリーニング方法などに関する。
【背景技術】
【0002】
概日リズム(サーカディアンリズム)は、約24時間を周期とする生物リズムであり、単細胞生物からヒトに至るまで、多くの生物に普遍的に見られる生体内現象である。各生物は、概日リズムの制御に関わる遺伝子を有し、概日リズム制御遺伝子が、概日リズムの調整を行っている。
【0003】
哺乳類では、概日リズム制御遺伝子の発現量が脳視床下部の視交叉上核(SCN)で特に高く、個体レベルでの概日リズム調整の中枢を担っている。一方、生体内のその他の組織(概日リズム末梢組織)においても、概日リズム制御遺伝子は発現しており、概日リズム中枢と同様に、概日リズム制御遺伝子による細胞・組織ごとの概日リズム調整が行われている。
【0004】
哺乳類における概日リズム制御遺伝子としては、Per遺伝子(Per1、Per2、Per3)、Clock遺伝子、Bmal遺伝子(Bmal1、Bmal2、Bmal3)、Cry遺伝子、Rev−erb遺伝子(Rev−erbα、Rev−erbβ)などが知られている。
【0005】
そのうち、BMAL1(Bmal1遺伝子の転写産物、以下同じ)は、CLOCK(Clock遺伝子の転写産物、以下同じ)と二量体を形成し、E−box(Per1遺伝子のプロモーター領域)を介して、Per遺伝子、Cry遺伝子などの発現を活性化することが示唆されている(非特許文献1など参照)。
また、Bmal1遺伝子は、REV−ERB(Rev−erb遺伝子の転写産物、以下同じ)などによって、発現が調節されることが示唆されている(非特許文献2など参照)。
【0006】
ここで、本発明にかかわりのあるタンパク質、RORα(retinoic acid binding−receptor alpha、以下同じ)について、説明する。RORαは、RORA(Rar−related orphan receptor A)などとも呼ばれるタンパク質で、RORα1、RORα4などのスプライシングバリアントが明らかになっている。なお、「スプライシングバリアント」は、同一遺伝子に由来する、アミノ酸配列の一部が異なったタンパク質のことであり、スプライシングの際のつながり方が異なるために生じる。
また、RORαは、低酸素状態において、発現が促進されることが報告されている(非特許文献3参照)。
【非特許文献1】Gekakis, N. et al. “Role of the CLOCK protein in the mammalian circadian mechanism.” Science 280, 1564-9 (1998)
【非特許文献2】Preitner, N. et al. “The orphan nuclear receptor REV-ERBalpha controls circadian transcription within the positive limb of the mammalian circadian clock” Cell 110, 251-60 (2002)
【非特許文献3】Chauvet, C. et al. “Retinoic acid receptor-related orphan receptor (ROR)alpha4 is the predominant isoform of the nuclear receptor RORalpha in the liver and is up-regulated by hypoxia in HepG2 human hepatoma cells.” Biochem J364, 449-56 (2002)
【発明の開示】
【発明が解決しようとする課題】
【0007】
哺乳類における概日リズムの調節メカニズムは、特に、未解明な部分が多い。一方、概日リズムは、睡眠、覚醒だけでなく、体温調節、ホルモン分泌など、生物の恒常性や体内調節機構にも深くかかわっていることが示唆されている。また、がんをはじめとする多くの疾患の発症機構にも関与している可能性がある。
【0008】
そこで、本発明は、概日リズムの調節メカニズムのうち、未解明な部分を明らかにすることを主な目的とする。
【課題を解決するための手段】
【0009】
本発明者らは、RORα(retinoic acid binding−receptor alpha、以下同じ)が、Bmal1の発現誘導を促進すること、及び、低酸素状態において、Bmal1の発現誘導が促進されること、を新規に見出した。
【0010】
そこで、本発明では、Bmal1の発現誘導を促進するRORα、及び、低酸素状態で、Bmal1の発現誘導を促進するRORα、を提供する。
【0011】
本発明は、次のような概日リズムの調節メカニズムの存在を強く示唆する。
低酸素状態などで、RORαの発現が促進されると、Bmal1の発現誘導が促進される。Bmal1の発現誘導が促進されると、BMAL1とCLOCKとの結合が促進され、Per遺伝子及び/又はCry遺伝子の発現誘導が促進される。
【0012】
また、本発明では、RORαが、REV−ERBα又はREV−ERBβと競合的に、Bmal1の発現誘導を促進することを、新規に見出した。従って、本発明は、RORαとREV−ERBの両者がBmal1の発現誘導を調節することにより、概日リズムを調整するメカニズムの存在をも強く示唆する。
【0013】
本発明により、RORαとBmal1のプロモーター領域(RORE)との相互作用が明らかになった。従って、Bmal1の発現リズムの変調などにより、概日リズムの変調が引き起こされた場合、本発明により、概日リズムを調整できる可能性がある。
【0014】
例えば、RORαを抑制する物質は、時差ぼけ調整剤としての適用可能性がある。その他、概日リズムが変調した場合として、例えば、睡眠障害、覚醒障害、不眠症、自律神経失調症、うつ病、老人性痴呆、夜間勤務・交代勤務など生活リズムの不規則化による体調の悪化、自閉症などにおける生活リズムの不規則化による消耗、などが挙げられる。これらの疾患に対しても、本発明は適用可能性がある。
【0015】
加えて、本発明は、抗がん剤として、適用できる可能性がある。
一般に、がん組織では、低酸素状態に陥ることが多い。また、例えば、夜間勤務による看護婦などの乳がん発生率上昇などは、概日リズムの変調が誘因であると指摘されている。
それに対し、本発明では、低酸素状態において、Bmal1の発現誘導が促進されることを明らかにした。従って、RORαを抑制する物質は、抗がん剤として、適用できる可能性がある。
【0016】
その他、本発明は、DNAチップ、プロテインチップ、概日リズムの変調を検出するためのマーカーとしての使用、抗がん剤又は時差ぼけ調整剤のスクリーニング方法などに適用できる可能性がある。
【発明の効果】
【0017】
本発明により、RORαがBmal1の発現誘導をを促進することが明らかになったため、本知見に基づき、概日リズムを調整できる。
【発明を実施するための最良の形態】
【0018】
<本発明に係るRORαについて>
はじめに、本発明に係るRORαについて、以下説明する。
【0019】
本発明に係るRORαは、RORα1、RORα4などのスプライシングバリアントを有する。ヒトにおけるRORα1のアミノ酸配列を配列番号1に、RORα4のアミノ酸配列を配列番号2に記載する。また、マウスにおけるRORα1のアミノ酸配列を配列番号3に、RORα4のアミノ酸配列を配列番号4に記載する。
【0020】
なお、本発明に係るRORαは、RORα1、RORα4、若しくは前記いずれかのタンパク質のアミノ酸配列と相同性を有し、Bmal1のプロモーター領域(RORE)と相互作用する領域を保持するタンパク質である。即ち、例えば、前記いずれかのタンパク質の他のスプライシングバリアントや、前記いずれかのタンパク質のアミノ酸配列中の一部に、置換、欠損、挿入、付加部分が含まれる場合も、本発明に係るRORαに包含される。
【0021】
<本発明に係る概日リズムの調節メカニズムについて>
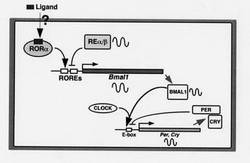
続いて、RORαに関わる概日リズムの調節メカニズムについて、図1を参照にしながら、以下説明する。
【0022】
本発明により、RORαは、Bmal1のプロモーター領域(ROREs)に結合し、Bmal1の発現誘導を促進することが明らかになった。従って、次のような概日リズムの調節メカニズムが存在すると推測される。
【0023】
低酸素状態などにより、RORαの発現が促進されると、RORαは、Bmal1のプロモーター領域(ROREs)に作用し、Bmal1の発現誘導を促進する。発現したBMAL1は、CLOCKと二量体を形成し、E−box(Per遺伝子、Cry遺伝子などのプロモーター領域)を介して、Per遺伝子、Cry遺伝子の発現誘導を促進する。発現したPER(Per遺伝子の転写産物)及びCRY(Cry遺伝子の転写産物)は、概日リズムの形成に直接的に関与する。
【0024】
なお、図中、「REα/β」は、REV−ERBを示す。REV−ERBは、Bmal1のプロモーター領域(ROREs)に、RORαと競合的に結合し、Bmal1の発現を抑制すると考えられている。
【0025】
また、図中、「Ligand」は、RORαの発現を促進又は抑制する物質である。上記の通り、RORαの発現は、概日リズムの調整に深く関わる。従って、その物質(ligand)をスクリーニングすることにより、新規時差ぼけ調整剤、抗がん剤などを探索できる。即ち、本発明に係る概日リズムの調節メカニズムに基づいて、スクリーニング系を構築できる。
【0026】
<本発明に係るRORαを抑制する物質について>
続いて、本発明に係るRORαを抑制する物質(RORαを抑制する物質を少なくとも含有する抗がん剤・時差ぼけ調整剤など)について、以下説明する。
【0027】
後述する実施例において、概日リズムがシフトした場合(明条件の開始時間が変更された場合など)、RORαの発現を抑制することにより、通常よりも早く、新しい概日リズムに同調できる、という結果が得られた。従って、本発明に係るRORαを抑制する物質は、時差ぼけ調整剤としての適用可能性がある。
【0028】
また、本発明では、低酸素状態において、Bmal1の発現誘導が促進されることを明らかにした。一方、上述の通り、がん組織では、低酸素状態に陥ることが知られている。従って、本発明に係るRORαを抑制する物質は、抗がん剤として、適用できる可能性がある。
【0029】
RORαを抑制する物質として、例えば、抗RORα抗体、RORαのコード配列の一部を有するsiRNA、アンチセンス核酸などが挙げられる。
抗RORα抗体は、RORαと特異的に結合することにより、RORαの機能を抑制する。
siRNA(short interfering RNA)は、RNA干渉によって遺伝子発現を抑制する、短い二本鎖RNAである。RORαのコード配列の一部を有するsiRNAは、RORαの発現を抑制するため、RORαの機能を抑制できる。
アンチセンス核酸は、RORαのコード配列の一部を有する核酸で、人工的に合成されたもの、安定化するために修飾・加工などを施したもの、を広く包含する。アンチセンス核酸は、RORαの転写産物(mRNA)と相補的に結合することにより、RORαの発現を抑制するため、RORαの機能を抑制できる。
抗RORα抗体、siRNA、アンチセンス核酸は、いずれも、公知方法により、作製できる。
【0030】
<本発明に係るDNAチップ、プロテインチップについて>
続いて、本発明に係るDNAチップ及びプロテインチップについて、以下説明する。
【0031】
RORαをコードする遺伝子の塩基配列を有する核酸、又は、前記遺伝子の一部分の塩基配列を有する核酸は、DNAチップに適用可能である。DNAチップにそれらの核酸を固定することにより、例えば、血液中や所定の組織中のRORα発現量を検出できるため、概日リズムの変調などの検出に、有用な可能性がある。DNAチップへの核酸の固定は、公知方法により行うことができる。
【0032】
同様に、RORαと特異的に結合する抗体は、プロテインチップに適用可能である。プロテインチップに、この抗体を固定することにより、例えば、血液中や所定の組織中のRORα発現量を検出できるため、概日リズムの変調などの検出に、有用な可能性がある。プロテインチップへの抗体の固定も、公知方法により行うことができる。
【0033】
<本発明に係る概日リズムの変調を検出するためのマーカーとしての使用について>
続いて、本発明に係る概日リズムの変調を検出するためのマーカーとしての使用について、以下説明する。
【0034】
上述の通り、本発明に係るRORαは、概日リズムを調整するメカニズムにおいて、重要な役割を果たしているため、概日リズムの変調を検出するためのマーカーとして使用できる可能性がある。具体的には、例えば、血液中や所定の組織中のRORα量をELISA法(公知方法)などで検出し、RORα発現量の変調を検出することにより、概日リズムの変調を検出できる可能性がある。
【0035】
<本発明に係るスクリーニング方法について>
続いて、本発明に係るスクリーニング方法の一例について、以下説明する。
【0036】
RORαを抑制する物質を探索することにより、スクリーニングを行う場合、例えば、反応系に標的物質を供給した後、RORαとBmal1のプロモーター領域(RORE)との相互作用を検出することにより、目的の物質を探索できる。即ち、反応系に標的物質を供給することにより相互作用が抑制された場合、その標的物質は、抗がん剤又は時差ぼけ調整剤として有効な可能性がある。
【0037】
スクリーニング方法の具体例として、例えば、予め、Bmal1のプロモーター領域(RORE)のオリゴヌクレオチドを、カラム、ビーズなどに固定しておき、RORαと標的物質を供給した後、RORαとBmal1のプロモーター領域(RORE)との相互作用を検出する方法、が考えられる。その場合、相互作用を検出する方法として、例えば、共免疫沈降法、カラム又はビーズを用いたプルダウン法、などを適用できる。
【0038】
なお、本発明に係るスクリーニング方法は、キット化が可能である。その場合、例えば、RORα、Bmal1のプロモーター領域(RORE)のオリゴヌクレオチド、抗RORα抗体、相互作用検出試薬(共免疫沈降法、カラム又はビーズを用いたプルダウン法、ウエスタンブロットなどに用いる試薬)、などを、相互作用検出方法などに応じて適宜組み合わせ、キット化する。その場合、前記タンパク質及びオリゴヌクレオチドは、変異体を用いてもよい。
【実施例1】
【0039】
実施例1及び実施例2では、予備的実験として、Rev−ervα、Rorα、Bmal1の発現パターンを調べた。
【0040】
実施例1では、マウスから複数の組織を採取し、それらの組織における、Rev−ervα、Rorα、Bmal1の発現パターンを調べた。
【0041】
実験手順の概要は次の通りである。
(1)まず、実験に用いたマウスの概日リズムを同調させた。マウスを、2週間、8時から20時まで明条件、20時から翌8時までを暗条件にした部屋で飼育し、マウスの概日リズムを同調させた。
(2)次に、マウスから概日リズム末梢組織として、肝臓、心臓、腎臓、肺、胃を所定の時刻に採取した。なお、各臓器の採取時刻は、概日リズムの同調を行った直後の8時から、4時間ごとに設定した(8時、12時、16時、20時、24時、翌4時)。
(3)次に、採取した各臓器をホモジナイズ後、Promega total SV RNA Isolation Kit(Promega社製)を用いて、各臓器からtotal RNAを抽出した。
(4)次に、定量的リアルタイムRT−PCR(定量的リアルタイム逆転写ポリメラーゼ反応)を行い、Rev−ervα、Rorα、Bmal1の各遺伝子の発現量を測定した。
【0042】
結果を図2に示す。
図中、「liver」は肝臓を、「heart」は心臓を、「kidney」は腎臓を、「lung」は肺を、「stomach」は胃を、それぞれ示す。
各グラフ中、横軸「ZT(Zeitgeber Times)」は、概日時間を示す。また、縦軸「relative mRNA abundance」は、相対的な発現量(発現レベル)を示す。なお、縦軸の発現量の値は、ハウスキーピング遺伝子GAPDH(GlycerAldehyde−3−Phosphate DeHydrogenase)の発現量を1とした場合の相対的な値である。
【実施例2】
【0043】
実施例2では、培養細胞NIH3T3における、Rev−ervα、Rorα、Bmal1の発現パターンを、実施例1と同様の手順で調べた。
【0044】
結果を図3に示す。
図中、横軸は、概日時間を示す。また、縦軸「relative mRNA abundance」は、相対的な発現量(発現レベル)を示す。なお、縦軸の発現量の値は、実施例1と同様、ハウスキーピング遺伝子GAPDH(GlycerAldehyde−3−Phosphate DeHydrogenase)の発現量を1とした場合の相対的な値である。
【0045】
以上、実施例1及び実施例2より、各組織における、Rev−ervα、Rorα、Bmal1の3つの遺伝子の発現パターンを確認できた。
【実施例3】
【0046】
続いて、実施例3及び実施例4は、RORαがBmal1の発現を誘導するかどうかを、ルシフェラーゼアッセイにより調べた実験である。
【0047】
「ルシフェラーゼアッセイ」の概要について、以下説明する。
ルシフェラーゼは、生物発光を触媒する酵素タンパク質である。
例えば、所定の遺伝子のプロモーター領域とルシフェラーゼをコードする遺伝子とを連結させた組換え遺伝子を作製し、その組換え遺伝子を培養細胞に組み込んだ場合、その遺伝子の発現が誘導されると、ルシフェラーゼが発現する。
そこで、ルシフェラーゼによる生物発光を測定し、ルシフェラーゼの発現レベルを取得することにより、その遺伝子の発現誘導レベルを推定することができる。
【0048】
実施例3では、RORファミリータンパク質(RORα1、RORα4、RORβ、RORγ)が、Bmal1の発現を誘導するかどうかを、ルシフェラーゼアッセイにより調べた。
【0049】
実験手順の概要を以下に示す。
(1)まず、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。この組換えベクターは、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する。
(2)また、発現ベクターpcDNA3(Invitrogen社製)に、RORファミリータンパク質をコードするDNA配列を組み込んで、組換えベクターを作製した。
(3)次に、(1)及び(2)で作製した組換えベクターを、共に、培養細胞NIH3T3にトランスフェクションした。次に、トランスフェクションした培養細胞を、単離・継代し、両たんぱく質を共発現させた。そして、トランスフェクションの36時間後に、継代した細胞を集め、溶解させた。
(4)次に、Dual Luciferase Assay System(Promega社製)を用いて、溶解させた細胞溶液を調製した後、発光強度を測定し、Bmal1の発現誘導レベルを取得した。
【0050】
結果を図4に示す。
図中、「BM−Luc」は、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する組換えベクターを、「RORα1」は、RORα1をコードするDNA配列を組み込んだ組換えベクターを、「RORα4」は、RORα4をコードするDNA配列を組み込んだ組換えベクターを、「RORβ」は、RORβをコードするDNA配列を組み込んだ組換えベクターを、「RORγ」は、RORγをコードするDNA配列を組み込んだ組換えベクターを、それぞれ示し、「+」は、それらの組換えベクターを、培養細胞に組み込んだことを表す。
縦軸「relative luc activity」は、発光強度を表す。この値は、「BM−Luc」のみを培養細胞に組み込んだ場合の発光強度を「1」とした場合の相対値である。
【0051】
図4に示す結果は、RORファミリータンパク質、特に、RORα1及びRORα4が、Bmal1の発現を誘導することを強く示唆する。
【実施例4】
【0052】
実施例4では、RORα4の発現レベルを変動させた場合、Bmal1の発現誘導レベルも変動するかどうかについて調べた。
【0053】
実験は、実施例3と同様の方法により行った。
なお、組換えベクターを培養細胞にトランスフェクションする際、RORα4をコードするDNA配列を組み込んだ組換えベクターの添加量を、それぞれ、0(コントロール)、1、5、10、25、50、100ngとした。
【0054】
結果を図5に示す。
図中、「BM−Luc」は、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する組換えベクターを示し、「+」は、組換えベクターを、培養細胞に組み込んだことを表す。
「RORα4」は、RORα4をコードするDNA配列を組み込んだ組換えベクターを示し、数字は、トランスフェクションの際の、組換えベクターの添加量(単位はng)を表す。
縦軸「relative luc activity」は、発光強度を表す。この値は、図4と同様、「BM−Luc」のみを培養細胞に組み込んだ場合の発光強度を「1」とした場合の相対値である。
【0055】
図5の結果は、RORα4の発現量の増大に伴い、Bmal1の発現誘導も促進されることを強く示唆する。従って、この結果は、RORα4の促進がBmal1の発現を誘導することを強く示唆する。
【実施例5】
【0056】
Rev−erb遺伝子は、概日リズム制御遺伝子として知られ、REV−ERBタンパク質は、Bmal1のプロモーター領域(RORE)に結合して、Bmal1の発現を抑制することが知られている。
そこで、実施例5では、RORαタンパク質とREV−ERBタンパク質を共発現させた場合、Bmal1の発現が誘導されるかどうかについて、調べた。
【0057】
実験手順の概要は、以下の通りである。
(1)まず、実施例3などと同様の手順により、Bmal1のプロモーター領域を組み込んだ組換えベクター、RORα1をコードするDNA配列を組み込んだ組換えベクター、Rev−erb遺伝子を組み込んだ組換えベクター、をそれぞれ作製した。
(2)次に、その3つの組み換えベクターを、共に、培養細胞NIH3T3にトランスフェクションし、各タンパク質を共発現させた。
(3)そして、その培養細胞を溶解させた後、Dual Luciferase Assay System(Promega社製)を用いて、溶解させた細胞溶液を調製し、発光強度を測定した。
【0058】
結果を図6に示す。
図中、「BM−Luc」は、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する組換えベクターを示し、「+」は、組換えベクターを、培養細胞に組み込んだことを表す。
「RORα1」は、RORα1をコードするDNA配列を組み込んだ組換えベクターを示し、カッコ書きの数字は、トランスフェクションの際の、組換えベクターの添加量(単位はng)を表す。
「β−Gal」は、前記と同様の手順でβ−ガラクトシダーゼを共発現させた場合を示し、コントロールである。数字は、トランスフェクションの際の、組換えベクターの添加量(単位はng)を表す。
「REα」、「REβ」は、それぞれ、Rev−erbα遺伝子、Rev−erbβ遺伝子を組み込んだ組換えベクターを示し、数字は、トランスフェクションの際の、組換えベクターの添加量(単位はng)を表す。
【0059】
図6の結果は、RORαとREV−ERBタンパク質は、Bmal1のプロモーター領域に競合的に作用することを強く示唆する。
【実施例6】
【0060】
実施例6では、RORαのドミナントネガティブ変異体の存在下で細胞を培養した場合の、Bmal1発現量の概日変化について調べた。
【0061】
実験手順の概要は次の通りである。
まず、実施例3などと同様に、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
次に、培養細胞NIH3Tに、その組み換えベクターをトランスフェクションし、その培養細胞を単離・継代した。
次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
次に、培養液に、RORα1のドミナントネガティブ変異体を加えた後、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
【0062】
結果を図7に示す。
図中、横軸は時間(単位は分)を、縦軸は発光強度(単位はM cpm;Million of Counts Per Minutes)を、それぞれ示す。
「vector」は、ドミナントネガティブ変異体の代わりに空ベクター(pcDNA3)を加えた場合(コントロール)の実験結果であることを示す。
「β−Gal」は、ドミナントネガティブ変異体の代わりにβ−ガラクトシダーゼを加えた場合(コントロール)の実験結果であることを示す。
「hRORα1ΔLBD」は、ヒトRORα1のドミナントネガティブ変異体を加えた場合の実験結果であることを示す。
【0063】
図7に示す通り、RORα1のドミナントネガティブ変異体を加えた場合、Bmal1の転写概日リズムが抑制された。即ち、この結果は、RORαが、bmal1の発現を誘導するだけでなく、Bmal1の転写概日リズムの発現に、重要な役割を果たしていることを強く示唆する。
【実施例7】
【0064】
実施例7及び実施例8では、RORαによるBmal1の発現誘導に関して、RORαの、Bmal1の配列における作用部位を調べた。
【0065】
実施例7では、Bmal1遺伝子の上流領域のDNA配列について、それぞれ長さの異なるものを作製し、実施例3などと同様の手順により、RORαによるBmal1の発現誘導レベルを取得した。
【0066】
実験手順の概要について、以下説明する。
(1)まず、図8に示すDNA配列を有する組換えベクターを、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)を用いて、それぞれ作製した。
図中、「Luciferase」は、その領域が、ルシフェラーゼをコードするDNA配列であることを示し、「Exon1」は、その領域が、Bmal1遺伝子の一番目のエクソン部分の配列を有することを示す。なお、「hBmal1」は、ヒトのBmal1遺伝子を表す。
「RORE1」及び「RORE2」は、その領域が、それぞれ、Bmal1遺伝子のプロモーター領域であることを示す。
「−3465」は、そのDNA配列が、Bmal1遺伝子配列の3465塩基分上流までの配列を有することを示す。他の数字も同様である。
「mt」は、変異体(mutant)を表し、RORE1、RORE2、又は両者の配列の一部分が変異したものであることを示す。
(2)そして、実施例3などと同様の手順により、RORα1とルシフェラーゼを共発現させた後、発光強度を測定し、Bmal1の発現誘導レベルを取得した。
【0067】
結果を図9に示す。
図中、 「RORα1」は、RORα1をコードするDNA配列を組み込んだ組換えベクターを示し、「+」は、その組換えベクターを培養細胞に組み込んだことを、「−」は、組み込んでいないことを、それぞれ表す。
「BM−Luc」は、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する組換えベクターを示す。
「−3465」は、その組換えベクターが、Bmal1遺伝子配列の3465塩基分上流までの配列を含むことを示し、「−829」は、その組換えベクターが、Bmal1遺伝子配列の829塩基分上流までの配列を含むことを示し、「−230」は、その組換えベクターが、Bmal1遺伝子配列の230塩基分上流までの配列を含むことを示す。
また、「RORE1mut/RORE2」はRORE1を欠損させた場合、「RORE1/RORE2mut」はRORE2を欠損させた場合、「RORE1mut/RORE2mut」はRORE1とRORE2の両方を欠損させた場合、を表す。なお、「mut」は、変異体(mutant)を表す。
【実施例8】
【0068】
次に、実施例8では、実施例7で作製した組換えベクター(図8参照)を用いて、実施例6などと同様の手順により、Bmal1発現量の概日変化について調べた。
【0069】
結果を図10に示す。
図中の表示は、図7及び図9と同様である。
【0070】
以上、実施例7(図9)及び実施例8(図10)の結果は、RORαによるBmal1の発現誘導が、Bmal1遺伝子の両方のプロモーター領域(RORE1、RORE2)を必要とすることを示す。従って、RORαは、Bmal1遺伝子の両方のプロモーター領域に作用して、Bmal1の発現を誘導していることを強く示唆する。
【実施例9】
【0071】
実施例9では、プルダウン法により、Bmal1のプロモーター領域(RORE)とRORαとの結合を調べた。
【0072】
実験手順の概要は次の通りである。
(1)まず、所定時刻に、マウスの肝臓を採取後、ホモジナイズし、マウス肝臓の抽出物を得た。
(2)また、Bmal1のプロモーター領域(RORE)の配列を含むオリゴヌクレオチドをビオチン化した。ビオチン化したオリゴヌクレオチドは、ストレプトアヴィジン−セファロースビーズ(Amersham社製)に固定した。
(3)次に、(1)で取得したマウス肝臓抽出物に、(2)のビーズを加え、インキュベートした。
(4)次に、ビーズを回収(プルダウン)した後、抗RORα抗体を用いてウエスタンブロットし、RORαを検出した。
【0073】
以上の手順により、Bmal1のプロモーター領域(RORE)の配列を含むオリゴヌクレオチドとRORα(肝臓抽出物中に存在)が結合しているかどうかを調べることができる。
【0074】
結果を図11に示す。
図中、「no ODN(no oligonucleotide)」は、ビオチンにオリゴヌクレオチドを結合させていない場合(コントロール)を示す。
「BM−ROREs」は、Bmal1のプロモーター領域(RORE)の配列を含むオリゴヌクレオチドをビオチン化した場合の結果を示す。
「BM−mROREs」は、Bmal1のプロモーター領域(RORE)の配列を一部変化させたオリゴヌクレオチド(変異体)をビオチン化した場合の結果を示す。なお、「m」は変異体(mutant)を表す。
「CT(circadian time)」は、概日時間を表す。即ち、マウスから肝臓を採取した時刻を表し、概日時間(明条件の開始時間を0時とした時刻)で示している。
「RORα1」、「RORα4」は、ウエスタンブロットに用いた抗体を示し、バンドの現れる位置を表している。
【0075】
図11に示す通り、Bmal1のプロモーター領域(RORE)の配列を含むオリゴヌクレオチドを用いた場合、「RORα1」、「RORα4」の両方において、バンドが確認できた。それに対し、変異体のオリゴヌクレオチドを用いた場合、バンドが消失した。
【0076】
従って、以上の結果は、RORαが、Bmal1のプロモーター領域(RORE)に結合することにより、Bmal1の発現を誘導することを強く示唆する。
【実施例10】
【0077】
実施例10では、RORα遺伝子を欠損した変異体マウスを用いて、アクトグラムを行い、RORα遺伝子欠損が、マウスの概日リズムに与える影響について、調べた。
【0078】
「アクトグラム」は、マウスの飲食行動を検出することにより、マウスの活動時間を連続的に記録する方法である。
【0079】
図12は、15日間、12時間ごとに明条件と暗条件を繰り返した後、暗条件でフリーラン(自由継続)させた場合のアクトグラムである。
図中、「+/+」は野生型を、「+/sg」はヘテロのRORα遺伝子欠損型を、「sg/sg」はホモのRORα遺伝子欠損型を、それぞれ示す。
「LD」は明条件(light)と暗条件(dark)を繰り返す環境を示し、「DD」は暗条件(dark)を続ける環境、即ち、フリーラン(自由継続)させた場合、を示す。
【0080】
図12に示す通り、飼育環境を24時間暗条件に変更した場合、野生型と比較して、変異体マウスでは、行動時間の開始時刻が早くなった。即ち、概日周期が短くなった。この結果は、RORαが、Bmal1の発現誘導を促進することにより、個体の概日リズムにも影響を与えていることを示唆する。
【0081】
図13は、図12に示した実験結果に基づいて、飼育環境を24時間暗条件に変更した後の、マウスの概日周期の平均値を取得した図である。図13が示す通り、野生型マウスでは、概日周期が、約23.8時間であったのに対し、ホモのRORα遺伝子欠損型マウスでは、概日周期が約23.4時間に減少していた。
【0082】
次に、図14は、15日間、12時間ごとに明条件と暗条件を繰り返した後、暗条件の開始時刻を4時間早め、時差のある状態にした場合のアクトグラムである。
図中の表示は、図12と同様である。
【0083】
図14に示す通り、暗条件の開始時刻を4時間早めた場合、RORα遺伝子欠損型マウスは、野生型マウスよりも早く、新しい環境条件に順応した。
【0084】
図15は、15日間、12時間ごとに明条件と暗条件を繰り返した後、環境条件を、6時間ごとに明条件と暗条件を繰り返す環境に変化させた場合のアクトグラムである。
図中の表示は、図12と同様である。
【0085】
図15に示す通り、野生型マウスでは、環境の変化にかかわらず、概日リズムを維持したのに対し、RORα遺伝子欠損型マウスは、概日リズムを維持したものと、概日リズムが完全に乱れたものの両者が存在した。
【実施例11】
【0086】
実施例11では、RNA干渉により、RORαの発現を抑制した場合、Bmal1の発現誘導も抑制されるかどうか、調べた。
【0087】
実験手順の概要は、次に通りである。
(1)まず、組換えsiRNA発現ベクターを作製した。RORα4をコードする遺伝子配列の第849〜1391塩基の中から、19塩基からなる配列(配列番号5)を選択した。そして、その配列を、siRNA発現ベクターに組み込み、組換え発現ベクターを作製した。
(2)また、実施例3などと同様に、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
(3)次に、培養細胞NIH3Tに、組換えsiRNA発現ベクターとBmal1のプロモーター領域を組み込んだ組換えベクターとを、共に、トランスフェクションした後、その培養細胞を単離・継代した。
(4)次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
(5)次に、培養液に、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
【0088】
結果を図16に示す。
図中、横軸は時間(単位は分)を、縦軸(relative cpm)は発光強度を示す。なお、発光強度は、最初の発光強度のピークを「1」とした時の相対値である。
「vector」は、ドミナントネガティブ変異体の代わりに空ベクター(pcDNA3)を加えた場合(コントロール)の実験結果であることを示す。
「RNAi(control)」は、RORαの配列とは関係のないsiRNAを用いた場合であり、コントロールである。
「RNAi(RORα)」は、RORαをコードする塩基配列の一部を有するsiRNAを用いた場合である。
【0089】
図16に示す通り、RORαをコードする塩基配列の一部を有するsiRNAを用いた場合(図中、一番下のグラフ)、空ベクターを用いた場合(そのグラフの上側の曲線)と比較して、Bmal1の発現誘導の振幅が減少した(そのグラフ中の下側の曲線)。従って、本実験結果は、RNA干渉を用いてRORの発現を抑制した場合、Bmal1の発現誘導も抑制されることを示す。
【実施例12】
【0090】
実施例12及び実施例13では、マウス胚繊維芽細胞(MEF;Mouse Embryonic Fibroblast)を用いて、RORαにBmal1の発現誘導を促進する作用があるかどうか、調べた。
【0091】
実施例12では、マウス胚繊維芽細胞のRORα遺伝子欠損変異体を用いて、Bmal1発現誘導の概日変化を調べた。
【0092】
実験手順の概要は次の通りである。
(1)まず、前記と同様、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
(2)次に、その組換えベクターを、マウス胚繊維芽細胞のRORα遺伝子欠損変異体にトランスフェクションした後、その培養細胞を単離・継代した。
(3)次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
(4)次に、培養液に、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
【0093】
結果を図17に示す。
図中、横軸は時間(単位は分)を、縦軸(relative cpm)は発光強度を示す。なお、発光強度は、最初の発光強度のピークを「1」とした時の相対値である。
「+/+」は野生型を、「+/sg」はヘテロのRORα遺伝子欠損型を、それぞれ示す。
【0094】
図17に示す通り、マウス胚繊維芽細胞のRORα遺伝子欠損変異体では、Bmal1発現誘導の振幅が減少した。
【実施例13】
【0095】
次に、実施例13では、定量的リアルタイムRT−PCRにより、マウス胚繊維芽細胞のRORα遺伝子欠損変異体における、Bmal1発現量の概日変化を調べた。
【0096】
マウス胚繊維芽細胞をホモジナイズ後、Promega total SV RNA Isolation Kit(Promega社製)を用いて、各臓器からtotal RNAを抽出し、定量的リアルタイムRT−PCRにより、Bmal1遺伝子の発現量を測定した。
【0097】
結果を図18に示す。
図中、横軸は、時間(単位は分)を示す。縦軸「relative mRNA abundance」は、相対的な発現量(発現レベル)を示す。なお、縦軸の発現量の値は、実施例1と同様、ハウスキーピング遺伝子GAPDH(GlycerAldehyde−3−Phosphate DeHydrogenase)の発現量を1とした場合の相対的な値である。
「+/+」は野生型を、「+/sg」はヘテロのRORα遺伝子欠損型を、それぞれ示す。
【0098】
図18に示す通り、マウス胚繊維芽細胞のRORα遺伝子欠損変異体では、Bmal1発現量の振幅が減少した。なお、この結果は、図17の結果ともほぼ一致した。
以上、図17及び図18の結果は、細胞レベルでも、RORαがBmal1の発現誘導を促進することを、強く示唆する。
【実施例14】
【0099】
実施例17では、アンチセンス核酸を用いてRORαの発現を抑制した場合、Bmal1の発現誘導も抑制されるかどうか、調べた。
【0100】
実験手順の概要は次の通りである。
(1)まず、アンチセンス核酸発現ベクターを作製した。RORαをコードする遺伝子のうち、所定の領域と同じ塩基配列を有するDNAフラグメントを、pcDNA3に組み込んで、アンチセンス核酸発現ベクターを作製した。
なお、DNAフラグメントとして、RORαをコードする遺伝子の第65〜574塩基までの配列を有するフラグメント、第599〜1122塩基までの配列を有するフラグメント、第849〜1391塩基までの配列を有するフラグメント、の3種類を用いた。
(2)また、実施例3などと同様に、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
(3)次に、培養細胞NIH3Tに、アンチセンス発現ベクターとBmal1のプロモーター領域を組み込んだ組換えベクターとを、コトランスフェクションし、その培養細胞を単離・継代した。
(4)次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
(5)次に、培養液に、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
【0101】
結果を図19に示す。
図中、図中、横軸は時間(単位は分)を、縦軸(relative cpm)は発光強度を示す。なお、発光強度は、最初の発光強度のピークを「1」とした時の相対値である。
「AS(RORα1)」は、アンチセンス核酸として、RORαをコードする遺伝子の第65〜574塩基までの配列を有するフラグメントを用いた場合、「AS(RORα2)」は、アンチセンスとして、第599〜1122塩基までの配列を有するフラグメントを用いた場合、「AS(RORα3)」は、アンチセンスとして、第849〜1391塩基までの配列を有するフラグメントを用いた場合、を示す。
【0102】
図19に示す通り、いずれの場合も、アンチセンス核酸を用いてRORαの発現を抑制することにより、Bmal1の発現誘導の振幅も抑制された。
【実施例15】
【0103】
上述の通り、RORαは低酸素状態において発現が誘導されることが明らかになっている。そこで、実施例15では、低酸素状態において、Bmal1の発現誘導も促進されるかどうかについて調べた。
【0104】
実験手順の概要は次の通りである。
(1)まず、前記と同様、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
(2)次に、その組換えベクターを、マウス胚繊維芽細胞のRORα遺伝子欠損変異体にトランスフェクションし、その培養細胞を単離・継代した。
(3)次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
(4)次に、培養液に、0.01mMCoCl2を加えた後、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
なお、塩化コバルト(CoCl2)を加えた条件下で、細胞を培養することにより、低酸素状態を作り出すことができる。
【0105】
結果を図20に示す。
図中、横軸は時間(単位は分)を、縦軸(relative cpm)は発光強度を示す。なお、発光強度は、最初の発光強度のピークを「1」とした時の相対値である。
「+0.01mMCoCl2」は、培養液に0.01mMCoCl2を加えた場合を、「+0.1mMNaCl」は、培養液に、0.01mMCoCl2の代わりに、0.1mMNaClを加えた場合(コントロール)を、それぞれ示す。
【0106】
図20の結果は、低酸素状態では、Bmal1の発現誘導も促進されることを示す。
【産業上の利用可能性】
【0107】
本発明は、概日リズムの調節メカニズムのうち、未解明な部分を明らかにして点で、有用である。従って、本発明は、概日リズムの調節メカニズムに関与する疾患、例えば、がん、睡眠障害、時差ぼけ、覚醒障害、不眠症、自律神経失調症、うつ病、老人性痴呆などの疾患の予防剤、治療剤として、適用可能性がある。また、DNAチップ、プロテインチップ、概日リズムの変調を検出するためのマーカー、抗がん剤・時差ぼけ調整剤などのスクリーニング方法やスクリーニングキットなどに適用可能である。
【図面の簡単な説明】
【0108】
【図1】概日リズムの調節メカニズムを示す図。
【図2】各組織における、Rev−ervα、Rorα、Bmal1の発現パターンを示す図。
【図3】培養細胞NIH3T3における、Rev−ervα、Rorα、Bmal1の発現パターンを示す図。
【図4】RORαがBmalの発現を誘導したことを示す図。
【図5】RORα4の発現量の増大に伴い、Bmalの発現誘導も促進されることを示す図。
【図6】RORαとREV−ERBタンパク質が、Bmalのプロモーター領域に競合的に作用することを示す図。
【図7】RORαのドミナントネガティブ変異体の存在下で細胞を培養した場合の、Bmal発現量の概日変化を示す図。
【図8】作製した組換えベクターの配列を示す図。
【図9】Bmal1の上流領域の長さをかえた場合における、RORαによるBmalの発現誘導レベルを示す図。
【図10】Bmal1の上流領域の長さをかえた場合における、Bmal発現量の概日変化を示す図。
【図11】プルダウン法による、ウエスタンブロット写真(図面代用写真)。
【図12】15日間、12時間ごとに明条件と暗条件を繰り返した後、暗条件でフリーラン(自由継続)させた場合のアクトグラム。
【図13】15日間、12時間ごとに明条件と暗条件を繰り返した後、飼育環境を24時間暗条件にした場合の、マウスの概日周期を取得した図。
【図14】15日間、12時間ごとに明条件と暗条件を繰り返した後、暗条件の開始時刻を4時間早め、時差のある状態にした場合のアクトグラム。
【図15】15日間、12時間ごとに明条件と暗条件を繰り返した後、環境条件を、6時間ごとに明条件と暗条件を繰り返す環境に変化させた場合のアクトグラム。
【図16】RNA干渉により、RORαの発現を抑制した場合の、Bmalの発現誘導を示す図。
【図17】マウス胚繊維芽細胞のRORα遺伝子欠損変異体を用いた場合の、Bmal発現誘導の概日変化を示す図。
【図18】マウス胚繊維芽細胞のRORα遺伝子欠損変異体を用いた場合における、定量的リアルタイムRT−PCRによる、Bmal発現量の概日変化を示す図。
【図19】アンチセンスを用いてRORαの発現を抑制した場合における、Bmalの発現誘導を示す図。
【図20】低酸素状態において、Bmalの発現誘導が促進されたことを示す図。
【技術分野】
【0001】
本発明は、時計遺伝子の作用メカニズム及びその周辺技術に関する。より詳細には、Bmal1(brain−muscle ARNT−like protein 1)の発現誘導を促進するRORα(retinoic acid receptor−related orphan receptor)、並びに抗がん剤、時差ぼけ調整剤、それらの薬剤のスクリーニング方法などに関する。
【背景技術】
【0002】
概日リズム(サーカディアンリズム)は、約24時間を周期とする生物リズムであり、単細胞生物からヒトに至るまで、多くの生物に普遍的に見られる生体内現象である。各生物は、概日リズムの制御に関わる遺伝子を有し、概日リズム制御遺伝子が、概日リズムの調整を行っている。
【0003】
哺乳類では、概日リズム制御遺伝子の発現量が脳視床下部の視交叉上核(SCN)で特に高く、個体レベルでの概日リズム調整の中枢を担っている。一方、生体内のその他の組織(概日リズム末梢組織)においても、概日リズム制御遺伝子は発現しており、概日リズム中枢と同様に、概日リズム制御遺伝子による細胞・組織ごとの概日リズム調整が行われている。
【0004】
哺乳類における概日リズム制御遺伝子としては、Per遺伝子(Per1、Per2、Per3)、Clock遺伝子、Bmal遺伝子(Bmal1、Bmal2、Bmal3)、Cry遺伝子、Rev−erb遺伝子(Rev−erbα、Rev−erbβ)などが知られている。
【0005】
そのうち、BMAL1(Bmal1遺伝子の転写産物、以下同じ)は、CLOCK(Clock遺伝子の転写産物、以下同じ)と二量体を形成し、E−box(Per1遺伝子のプロモーター領域)を介して、Per遺伝子、Cry遺伝子などの発現を活性化することが示唆されている(非特許文献1など参照)。
また、Bmal1遺伝子は、REV−ERB(Rev−erb遺伝子の転写産物、以下同じ)などによって、発現が調節されることが示唆されている(非特許文献2など参照)。
【0006】
ここで、本発明にかかわりのあるタンパク質、RORα(retinoic acid binding−receptor alpha、以下同じ)について、説明する。RORαは、RORA(Rar−related orphan receptor A)などとも呼ばれるタンパク質で、RORα1、RORα4などのスプライシングバリアントが明らかになっている。なお、「スプライシングバリアント」は、同一遺伝子に由来する、アミノ酸配列の一部が異なったタンパク質のことであり、スプライシングの際のつながり方が異なるために生じる。
また、RORαは、低酸素状態において、発現が促進されることが報告されている(非特許文献3参照)。
【非特許文献1】Gekakis, N. et al. “Role of the CLOCK protein in the mammalian circadian mechanism.” Science 280, 1564-9 (1998)
【非特許文献2】Preitner, N. et al. “The orphan nuclear receptor REV-ERBalpha controls circadian transcription within the positive limb of the mammalian circadian clock” Cell 110, 251-60 (2002)
【非特許文献3】Chauvet, C. et al. “Retinoic acid receptor-related orphan receptor (ROR)alpha4 is the predominant isoform of the nuclear receptor RORalpha in the liver and is up-regulated by hypoxia in HepG2 human hepatoma cells.” Biochem J364, 449-56 (2002)
【発明の開示】
【発明が解決しようとする課題】
【0007】
哺乳類における概日リズムの調節メカニズムは、特に、未解明な部分が多い。一方、概日リズムは、睡眠、覚醒だけでなく、体温調節、ホルモン分泌など、生物の恒常性や体内調節機構にも深くかかわっていることが示唆されている。また、がんをはじめとする多くの疾患の発症機構にも関与している可能性がある。
【0008】
そこで、本発明は、概日リズムの調節メカニズムのうち、未解明な部分を明らかにすることを主な目的とする。
【課題を解決するための手段】
【0009】
本発明者らは、RORα(retinoic acid binding−receptor alpha、以下同じ)が、Bmal1の発現誘導を促進すること、及び、低酸素状態において、Bmal1の発現誘導が促進されること、を新規に見出した。
【0010】
そこで、本発明では、Bmal1の発現誘導を促進するRORα、及び、低酸素状態で、Bmal1の発現誘導を促進するRORα、を提供する。
【0011】
本発明は、次のような概日リズムの調節メカニズムの存在を強く示唆する。
低酸素状態などで、RORαの発現が促進されると、Bmal1の発現誘導が促進される。Bmal1の発現誘導が促進されると、BMAL1とCLOCKとの結合が促進され、Per遺伝子及び/又はCry遺伝子の発現誘導が促進される。
【0012】
また、本発明では、RORαが、REV−ERBα又はREV−ERBβと競合的に、Bmal1の発現誘導を促進することを、新規に見出した。従って、本発明は、RORαとREV−ERBの両者がBmal1の発現誘導を調節することにより、概日リズムを調整するメカニズムの存在をも強く示唆する。
【0013】
本発明により、RORαとBmal1のプロモーター領域(RORE)との相互作用が明らかになった。従って、Bmal1の発現リズムの変調などにより、概日リズムの変調が引き起こされた場合、本発明により、概日リズムを調整できる可能性がある。
【0014】
例えば、RORαを抑制する物質は、時差ぼけ調整剤としての適用可能性がある。その他、概日リズムが変調した場合として、例えば、睡眠障害、覚醒障害、不眠症、自律神経失調症、うつ病、老人性痴呆、夜間勤務・交代勤務など生活リズムの不規則化による体調の悪化、自閉症などにおける生活リズムの不規則化による消耗、などが挙げられる。これらの疾患に対しても、本発明は適用可能性がある。
【0015】
加えて、本発明は、抗がん剤として、適用できる可能性がある。
一般に、がん組織では、低酸素状態に陥ることが多い。また、例えば、夜間勤務による看護婦などの乳がん発生率上昇などは、概日リズムの変調が誘因であると指摘されている。
それに対し、本発明では、低酸素状態において、Bmal1の発現誘導が促進されることを明らかにした。従って、RORαを抑制する物質は、抗がん剤として、適用できる可能性がある。
【0016】
その他、本発明は、DNAチップ、プロテインチップ、概日リズムの変調を検出するためのマーカーとしての使用、抗がん剤又は時差ぼけ調整剤のスクリーニング方法などに適用できる可能性がある。
【発明の効果】
【0017】
本発明により、RORαがBmal1の発現誘導をを促進することが明らかになったため、本知見に基づき、概日リズムを調整できる。
【発明を実施するための最良の形態】
【0018】
<本発明に係るRORαについて>
はじめに、本発明に係るRORαについて、以下説明する。
【0019】
本発明に係るRORαは、RORα1、RORα4などのスプライシングバリアントを有する。ヒトにおけるRORα1のアミノ酸配列を配列番号1に、RORα4のアミノ酸配列を配列番号2に記載する。また、マウスにおけるRORα1のアミノ酸配列を配列番号3に、RORα4のアミノ酸配列を配列番号4に記載する。
【0020】
なお、本発明に係るRORαは、RORα1、RORα4、若しくは前記いずれかのタンパク質のアミノ酸配列と相同性を有し、Bmal1のプロモーター領域(RORE)と相互作用する領域を保持するタンパク質である。即ち、例えば、前記いずれかのタンパク質の他のスプライシングバリアントや、前記いずれかのタンパク質のアミノ酸配列中の一部に、置換、欠損、挿入、付加部分が含まれる場合も、本発明に係るRORαに包含される。
【0021】
<本発明に係る概日リズムの調節メカニズムについて>
続いて、RORαに関わる概日リズムの調節メカニズムについて、図1を参照にしながら、以下説明する。
【0022】
本発明により、RORαは、Bmal1のプロモーター領域(ROREs)に結合し、Bmal1の発現誘導を促進することが明らかになった。従って、次のような概日リズムの調節メカニズムが存在すると推測される。
【0023】
低酸素状態などにより、RORαの発現が促進されると、RORαは、Bmal1のプロモーター領域(ROREs)に作用し、Bmal1の発現誘導を促進する。発現したBMAL1は、CLOCKと二量体を形成し、E−box(Per遺伝子、Cry遺伝子などのプロモーター領域)を介して、Per遺伝子、Cry遺伝子の発現誘導を促進する。発現したPER(Per遺伝子の転写産物)及びCRY(Cry遺伝子の転写産物)は、概日リズムの形成に直接的に関与する。
【0024】
なお、図中、「REα/β」は、REV−ERBを示す。REV−ERBは、Bmal1のプロモーター領域(ROREs)に、RORαと競合的に結合し、Bmal1の発現を抑制すると考えられている。
【0025】
また、図中、「Ligand」は、RORαの発現を促進又は抑制する物質である。上記の通り、RORαの発現は、概日リズムの調整に深く関わる。従って、その物質(ligand)をスクリーニングすることにより、新規時差ぼけ調整剤、抗がん剤などを探索できる。即ち、本発明に係る概日リズムの調節メカニズムに基づいて、スクリーニング系を構築できる。
【0026】
<本発明に係るRORαを抑制する物質について>
続いて、本発明に係るRORαを抑制する物質(RORαを抑制する物質を少なくとも含有する抗がん剤・時差ぼけ調整剤など)について、以下説明する。
【0027】
後述する実施例において、概日リズムがシフトした場合(明条件の開始時間が変更された場合など)、RORαの発現を抑制することにより、通常よりも早く、新しい概日リズムに同調できる、という結果が得られた。従って、本発明に係るRORαを抑制する物質は、時差ぼけ調整剤としての適用可能性がある。
【0028】
また、本発明では、低酸素状態において、Bmal1の発現誘導が促進されることを明らかにした。一方、上述の通り、がん組織では、低酸素状態に陥ることが知られている。従って、本発明に係るRORαを抑制する物質は、抗がん剤として、適用できる可能性がある。
【0029】
RORαを抑制する物質として、例えば、抗RORα抗体、RORαのコード配列の一部を有するsiRNA、アンチセンス核酸などが挙げられる。
抗RORα抗体は、RORαと特異的に結合することにより、RORαの機能を抑制する。
siRNA(short interfering RNA)は、RNA干渉によって遺伝子発現を抑制する、短い二本鎖RNAである。RORαのコード配列の一部を有するsiRNAは、RORαの発現を抑制するため、RORαの機能を抑制できる。
アンチセンス核酸は、RORαのコード配列の一部を有する核酸で、人工的に合成されたもの、安定化するために修飾・加工などを施したもの、を広く包含する。アンチセンス核酸は、RORαの転写産物(mRNA)と相補的に結合することにより、RORαの発現を抑制するため、RORαの機能を抑制できる。
抗RORα抗体、siRNA、アンチセンス核酸は、いずれも、公知方法により、作製できる。
【0030】
<本発明に係るDNAチップ、プロテインチップについて>
続いて、本発明に係るDNAチップ及びプロテインチップについて、以下説明する。
【0031】
RORαをコードする遺伝子の塩基配列を有する核酸、又は、前記遺伝子の一部分の塩基配列を有する核酸は、DNAチップに適用可能である。DNAチップにそれらの核酸を固定することにより、例えば、血液中や所定の組織中のRORα発現量を検出できるため、概日リズムの変調などの検出に、有用な可能性がある。DNAチップへの核酸の固定は、公知方法により行うことができる。
【0032】
同様に、RORαと特異的に結合する抗体は、プロテインチップに適用可能である。プロテインチップに、この抗体を固定することにより、例えば、血液中や所定の組織中のRORα発現量を検出できるため、概日リズムの変調などの検出に、有用な可能性がある。プロテインチップへの抗体の固定も、公知方法により行うことができる。
【0033】
<本発明に係る概日リズムの変調を検出するためのマーカーとしての使用について>
続いて、本発明に係る概日リズムの変調を検出するためのマーカーとしての使用について、以下説明する。
【0034】
上述の通り、本発明に係るRORαは、概日リズムを調整するメカニズムにおいて、重要な役割を果たしているため、概日リズムの変調を検出するためのマーカーとして使用できる可能性がある。具体的には、例えば、血液中や所定の組織中のRORα量をELISA法(公知方法)などで検出し、RORα発現量の変調を検出することにより、概日リズムの変調を検出できる可能性がある。
【0035】
<本発明に係るスクリーニング方法について>
続いて、本発明に係るスクリーニング方法の一例について、以下説明する。
【0036】
RORαを抑制する物質を探索することにより、スクリーニングを行う場合、例えば、反応系に標的物質を供給した後、RORαとBmal1のプロモーター領域(RORE)との相互作用を検出することにより、目的の物質を探索できる。即ち、反応系に標的物質を供給することにより相互作用が抑制された場合、その標的物質は、抗がん剤又は時差ぼけ調整剤として有効な可能性がある。
【0037】
スクリーニング方法の具体例として、例えば、予め、Bmal1のプロモーター領域(RORE)のオリゴヌクレオチドを、カラム、ビーズなどに固定しておき、RORαと標的物質を供給した後、RORαとBmal1のプロモーター領域(RORE)との相互作用を検出する方法、が考えられる。その場合、相互作用を検出する方法として、例えば、共免疫沈降法、カラム又はビーズを用いたプルダウン法、などを適用できる。
【0038】
なお、本発明に係るスクリーニング方法は、キット化が可能である。その場合、例えば、RORα、Bmal1のプロモーター領域(RORE)のオリゴヌクレオチド、抗RORα抗体、相互作用検出試薬(共免疫沈降法、カラム又はビーズを用いたプルダウン法、ウエスタンブロットなどに用いる試薬)、などを、相互作用検出方法などに応じて適宜組み合わせ、キット化する。その場合、前記タンパク質及びオリゴヌクレオチドは、変異体を用いてもよい。
【実施例1】
【0039】
実施例1及び実施例2では、予備的実験として、Rev−ervα、Rorα、Bmal1の発現パターンを調べた。
【0040】
実施例1では、マウスから複数の組織を採取し、それらの組織における、Rev−ervα、Rorα、Bmal1の発現パターンを調べた。
【0041】
実験手順の概要は次の通りである。
(1)まず、実験に用いたマウスの概日リズムを同調させた。マウスを、2週間、8時から20時まで明条件、20時から翌8時までを暗条件にした部屋で飼育し、マウスの概日リズムを同調させた。
(2)次に、マウスから概日リズム末梢組織として、肝臓、心臓、腎臓、肺、胃を所定の時刻に採取した。なお、各臓器の採取時刻は、概日リズムの同調を行った直後の8時から、4時間ごとに設定した(8時、12時、16時、20時、24時、翌4時)。
(3)次に、採取した各臓器をホモジナイズ後、Promega total SV RNA Isolation Kit(Promega社製)を用いて、各臓器からtotal RNAを抽出した。
(4)次に、定量的リアルタイムRT−PCR(定量的リアルタイム逆転写ポリメラーゼ反応)を行い、Rev−ervα、Rorα、Bmal1の各遺伝子の発現量を測定した。
【0042】
結果を図2に示す。
図中、「liver」は肝臓を、「heart」は心臓を、「kidney」は腎臓を、「lung」は肺を、「stomach」は胃を、それぞれ示す。
各グラフ中、横軸「ZT(Zeitgeber Times)」は、概日時間を示す。また、縦軸「relative mRNA abundance」は、相対的な発現量(発現レベル)を示す。なお、縦軸の発現量の値は、ハウスキーピング遺伝子GAPDH(GlycerAldehyde−3−Phosphate DeHydrogenase)の発現量を1とした場合の相対的な値である。
【実施例2】
【0043】
実施例2では、培養細胞NIH3T3における、Rev−ervα、Rorα、Bmal1の発現パターンを、実施例1と同様の手順で調べた。
【0044】
結果を図3に示す。
図中、横軸は、概日時間を示す。また、縦軸「relative mRNA abundance」は、相対的な発現量(発現レベル)を示す。なお、縦軸の発現量の値は、実施例1と同様、ハウスキーピング遺伝子GAPDH(GlycerAldehyde−3−Phosphate DeHydrogenase)の発現量を1とした場合の相対的な値である。
【0045】
以上、実施例1及び実施例2より、各組織における、Rev−ervα、Rorα、Bmal1の3つの遺伝子の発現パターンを確認できた。
【実施例3】
【0046】
続いて、実施例3及び実施例4は、RORαがBmal1の発現を誘導するかどうかを、ルシフェラーゼアッセイにより調べた実験である。
【0047】
「ルシフェラーゼアッセイ」の概要について、以下説明する。
ルシフェラーゼは、生物発光を触媒する酵素タンパク質である。
例えば、所定の遺伝子のプロモーター領域とルシフェラーゼをコードする遺伝子とを連結させた組換え遺伝子を作製し、その組換え遺伝子を培養細胞に組み込んだ場合、その遺伝子の発現が誘導されると、ルシフェラーゼが発現する。
そこで、ルシフェラーゼによる生物発光を測定し、ルシフェラーゼの発現レベルを取得することにより、その遺伝子の発現誘導レベルを推定することができる。
【0048】
実施例3では、RORファミリータンパク質(RORα1、RORα4、RORβ、RORγ)が、Bmal1の発現を誘導するかどうかを、ルシフェラーゼアッセイにより調べた。
【0049】
実験手順の概要を以下に示す。
(1)まず、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。この組換えベクターは、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する。
(2)また、発現ベクターpcDNA3(Invitrogen社製)に、RORファミリータンパク質をコードするDNA配列を組み込んで、組換えベクターを作製した。
(3)次に、(1)及び(2)で作製した組換えベクターを、共に、培養細胞NIH3T3にトランスフェクションした。次に、トランスフェクションした培養細胞を、単離・継代し、両たんぱく質を共発現させた。そして、トランスフェクションの36時間後に、継代した細胞を集め、溶解させた。
(4)次に、Dual Luciferase Assay System(Promega社製)を用いて、溶解させた細胞溶液を調製した後、発光強度を測定し、Bmal1の発現誘導レベルを取得した。
【0050】
結果を図4に示す。
図中、「BM−Luc」は、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する組換えベクターを、「RORα1」は、RORα1をコードするDNA配列を組み込んだ組換えベクターを、「RORα4」は、RORα4をコードするDNA配列を組み込んだ組換えベクターを、「RORβ」は、RORβをコードするDNA配列を組み込んだ組換えベクターを、「RORγ」は、RORγをコードするDNA配列を組み込んだ組換えベクターを、それぞれ示し、「+」は、それらの組換えベクターを、培養細胞に組み込んだことを表す。
縦軸「relative luc activity」は、発光強度を表す。この値は、「BM−Luc」のみを培養細胞に組み込んだ場合の発光強度を「1」とした場合の相対値である。
【0051】
図4に示す結果は、RORファミリータンパク質、特に、RORα1及びRORα4が、Bmal1の発現を誘導することを強く示唆する。
【実施例4】
【0052】
実施例4では、RORα4の発現レベルを変動させた場合、Bmal1の発現誘導レベルも変動するかどうかについて調べた。
【0053】
実験は、実施例3と同様の方法により行った。
なお、組換えベクターを培養細胞にトランスフェクションする際、RORα4をコードするDNA配列を組み込んだ組換えベクターの添加量を、それぞれ、0(コントロール)、1、5、10、25、50、100ngとした。
【0054】
結果を図5に示す。
図中、「BM−Luc」は、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する組換えベクターを示し、「+」は、組換えベクターを、培養細胞に組み込んだことを表す。
「RORα4」は、RORα4をコードするDNA配列を組み込んだ組換えベクターを示し、数字は、トランスフェクションの際の、組換えベクターの添加量(単位はng)を表す。
縦軸「relative luc activity」は、発光強度を表す。この値は、図4と同様、「BM−Luc」のみを培養細胞に組み込んだ場合の発光強度を「1」とした場合の相対値である。
【0055】
図5の結果は、RORα4の発現量の増大に伴い、Bmal1の発現誘導も促進されることを強く示唆する。従って、この結果は、RORα4の促進がBmal1の発現を誘導することを強く示唆する。
【実施例5】
【0056】
Rev−erb遺伝子は、概日リズム制御遺伝子として知られ、REV−ERBタンパク質は、Bmal1のプロモーター領域(RORE)に結合して、Bmal1の発現を抑制することが知られている。
そこで、実施例5では、RORαタンパク質とREV−ERBタンパク質を共発現させた場合、Bmal1の発現が誘導されるかどうかについて、調べた。
【0057】
実験手順の概要は、以下の通りである。
(1)まず、実施例3などと同様の手順により、Bmal1のプロモーター領域を組み込んだ組換えベクター、RORα1をコードするDNA配列を組み込んだ組換えベクター、Rev−erb遺伝子を組み込んだ組換えベクター、をそれぞれ作製した。
(2)次に、その3つの組み換えベクターを、共に、培養細胞NIH3T3にトランスフェクションし、各タンパク質を共発現させた。
(3)そして、その培養細胞を溶解させた後、Dual Luciferase Assay System(Promega社製)を用いて、溶解させた細胞溶液を調製し、発光強度を測定した。
【0058】
結果を図6に示す。
図中、「BM−Luc」は、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する組換えベクターを示し、「+」は、組換えベクターを、培養細胞に組み込んだことを表す。
「RORα1」は、RORα1をコードするDNA配列を組み込んだ組換えベクターを示し、カッコ書きの数字は、トランスフェクションの際の、組換えベクターの添加量(単位はng)を表す。
「β−Gal」は、前記と同様の手順でβ−ガラクトシダーゼを共発現させた場合を示し、コントロールである。数字は、トランスフェクションの際の、組換えベクターの添加量(単位はng)を表す。
「REα」、「REβ」は、それぞれ、Rev−erbα遺伝子、Rev−erbβ遺伝子を組み込んだ組換えベクターを示し、数字は、トランスフェクションの際の、組換えベクターの添加量(単位はng)を表す。
【0059】
図6の結果は、RORαとREV−ERBタンパク質は、Bmal1のプロモーター領域に競合的に作用することを強く示唆する。
【実施例6】
【0060】
実施例6では、RORαのドミナントネガティブ変異体の存在下で細胞を培養した場合の、Bmal1発現量の概日変化について調べた。
【0061】
実験手順の概要は次の通りである。
まず、実施例3などと同様に、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
次に、培養細胞NIH3Tに、その組み換えベクターをトランスフェクションし、その培養細胞を単離・継代した。
次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
次に、培養液に、RORα1のドミナントネガティブ変異体を加えた後、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
【0062】
結果を図7に示す。
図中、横軸は時間(単位は分)を、縦軸は発光強度(単位はM cpm;Million of Counts Per Minutes)を、それぞれ示す。
「vector」は、ドミナントネガティブ変異体の代わりに空ベクター(pcDNA3)を加えた場合(コントロール)の実験結果であることを示す。
「β−Gal」は、ドミナントネガティブ変異体の代わりにβ−ガラクトシダーゼを加えた場合(コントロール)の実験結果であることを示す。
「hRORα1ΔLBD」は、ヒトRORα1のドミナントネガティブ変異体を加えた場合の実験結果であることを示す。
【0063】
図7に示す通り、RORα1のドミナントネガティブ変異体を加えた場合、Bmal1の転写概日リズムが抑制された。即ち、この結果は、RORαが、bmal1の発現を誘導するだけでなく、Bmal1の転写概日リズムの発現に、重要な役割を果たしていることを強く示唆する。
【実施例7】
【0064】
実施例7及び実施例8では、RORαによるBmal1の発現誘導に関して、RORαの、Bmal1の配列における作用部位を調べた。
【0065】
実施例7では、Bmal1遺伝子の上流領域のDNA配列について、それぞれ長さの異なるものを作製し、実施例3などと同様の手順により、RORαによるBmal1の発現誘導レベルを取得した。
【0066】
実験手順の概要について、以下説明する。
(1)まず、図8に示すDNA配列を有する組換えベクターを、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)を用いて、それぞれ作製した。
図中、「Luciferase」は、その領域が、ルシフェラーゼをコードするDNA配列であることを示し、「Exon1」は、その領域が、Bmal1遺伝子の一番目のエクソン部分の配列を有することを示す。なお、「hBmal1」は、ヒトのBmal1遺伝子を表す。
「RORE1」及び「RORE2」は、その領域が、それぞれ、Bmal1遺伝子のプロモーター領域であることを示す。
「−3465」は、そのDNA配列が、Bmal1遺伝子配列の3465塩基分上流までの配列を有することを示す。他の数字も同様である。
「mt」は、変異体(mutant)を表し、RORE1、RORE2、又は両者の配列の一部分が変異したものであることを示す。
(2)そして、実施例3などと同様の手順により、RORα1とルシフェラーゼを共発現させた後、発光強度を測定し、Bmal1の発現誘導レベルを取得した。
【0067】
結果を図9に示す。
図中、 「RORα1」は、RORα1をコードするDNA配列を組み込んだ組換えベクターを示し、「+」は、その組換えベクターを培養細胞に組み込んだことを、「−」は、組み込んでいないことを、それぞれ表す。
「BM−Luc」は、Bmal1のプロモーター領域とルシフェラーゼ遺伝子をコードする領域を有する組換えベクターを示す。
「−3465」は、その組換えベクターが、Bmal1遺伝子配列の3465塩基分上流までの配列を含むことを示し、「−829」は、その組換えベクターが、Bmal1遺伝子配列の829塩基分上流までの配列を含むことを示し、「−230」は、その組換えベクターが、Bmal1遺伝子配列の230塩基分上流までの配列を含むことを示す。
また、「RORE1mut/RORE2」はRORE1を欠損させた場合、「RORE1/RORE2mut」はRORE2を欠損させた場合、「RORE1mut/RORE2mut」はRORE1とRORE2の両方を欠損させた場合、を表す。なお、「mut」は、変異体(mutant)を表す。
【実施例8】
【0068】
次に、実施例8では、実施例7で作製した組換えベクター(図8参照)を用いて、実施例6などと同様の手順により、Bmal1発現量の概日変化について調べた。
【0069】
結果を図10に示す。
図中の表示は、図7及び図9と同様である。
【0070】
以上、実施例7(図9)及び実施例8(図10)の結果は、RORαによるBmal1の発現誘導が、Bmal1遺伝子の両方のプロモーター領域(RORE1、RORE2)を必要とすることを示す。従って、RORαは、Bmal1遺伝子の両方のプロモーター領域に作用して、Bmal1の発現を誘導していることを強く示唆する。
【実施例9】
【0071】
実施例9では、プルダウン法により、Bmal1のプロモーター領域(RORE)とRORαとの結合を調べた。
【0072】
実験手順の概要は次の通りである。
(1)まず、所定時刻に、マウスの肝臓を採取後、ホモジナイズし、マウス肝臓の抽出物を得た。
(2)また、Bmal1のプロモーター領域(RORE)の配列を含むオリゴヌクレオチドをビオチン化した。ビオチン化したオリゴヌクレオチドは、ストレプトアヴィジン−セファロースビーズ(Amersham社製)に固定した。
(3)次に、(1)で取得したマウス肝臓抽出物に、(2)のビーズを加え、インキュベートした。
(4)次に、ビーズを回収(プルダウン)した後、抗RORα抗体を用いてウエスタンブロットし、RORαを検出した。
【0073】
以上の手順により、Bmal1のプロモーター領域(RORE)の配列を含むオリゴヌクレオチドとRORα(肝臓抽出物中に存在)が結合しているかどうかを調べることができる。
【0074】
結果を図11に示す。
図中、「no ODN(no oligonucleotide)」は、ビオチンにオリゴヌクレオチドを結合させていない場合(コントロール)を示す。
「BM−ROREs」は、Bmal1のプロモーター領域(RORE)の配列を含むオリゴヌクレオチドをビオチン化した場合の結果を示す。
「BM−mROREs」は、Bmal1のプロモーター領域(RORE)の配列を一部変化させたオリゴヌクレオチド(変異体)をビオチン化した場合の結果を示す。なお、「m」は変異体(mutant)を表す。
「CT(circadian time)」は、概日時間を表す。即ち、マウスから肝臓を採取した時刻を表し、概日時間(明条件の開始時間を0時とした時刻)で示している。
「RORα1」、「RORα4」は、ウエスタンブロットに用いた抗体を示し、バンドの現れる位置を表している。
【0075】
図11に示す通り、Bmal1のプロモーター領域(RORE)の配列を含むオリゴヌクレオチドを用いた場合、「RORα1」、「RORα4」の両方において、バンドが確認できた。それに対し、変異体のオリゴヌクレオチドを用いた場合、バンドが消失した。
【0076】
従って、以上の結果は、RORαが、Bmal1のプロモーター領域(RORE)に結合することにより、Bmal1の発現を誘導することを強く示唆する。
【実施例10】
【0077】
実施例10では、RORα遺伝子を欠損した変異体マウスを用いて、アクトグラムを行い、RORα遺伝子欠損が、マウスの概日リズムに与える影響について、調べた。
【0078】
「アクトグラム」は、マウスの飲食行動を検出することにより、マウスの活動時間を連続的に記録する方法である。
【0079】
図12は、15日間、12時間ごとに明条件と暗条件を繰り返した後、暗条件でフリーラン(自由継続)させた場合のアクトグラムである。
図中、「+/+」は野生型を、「+/sg」はヘテロのRORα遺伝子欠損型を、「sg/sg」はホモのRORα遺伝子欠損型を、それぞれ示す。
「LD」は明条件(light)と暗条件(dark)を繰り返す環境を示し、「DD」は暗条件(dark)を続ける環境、即ち、フリーラン(自由継続)させた場合、を示す。
【0080】
図12に示す通り、飼育環境を24時間暗条件に変更した場合、野生型と比較して、変異体マウスでは、行動時間の開始時刻が早くなった。即ち、概日周期が短くなった。この結果は、RORαが、Bmal1の発現誘導を促進することにより、個体の概日リズムにも影響を与えていることを示唆する。
【0081】
図13は、図12に示した実験結果に基づいて、飼育環境を24時間暗条件に変更した後の、マウスの概日周期の平均値を取得した図である。図13が示す通り、野生型マウスでは、概日周期が、約23.8時間であったのに対し、ホモのRORα遺伝子欠損型マウスでは、概日周期が約23.4時間に減少していた。
【0082】
次に、図14は、15日間、12時間ごとに明条件と暗条件を繰り返した後、暗条件の開始時刻を4時間早め、時差のある状態にした場合のアクトグラムである。
図中の表示は、図12と同様である。
【0083】
図14に示す通り、暗条件の開始時刻を4時間早めた場合、RORα遺伝子欠損型マウスは、野生型マウスよりも早く、新しい環境条件に順応した。
【0084】
図15は、15日間、12時間ごとに明条件と暗条件を繰り返した後、環境条件を、6時間ごとに明条件と暗条件を繰り返す環境に変化させた場合のアクトグラムである。
図中の表示は、図12と同様である。
【0085】
図15に示す通り、野生型マウスでは、環境の変化にかかわらず、概日リズムを維持したのに対し、RORα遺伝子欠損型マウスは、概日リズムを維持したものと、概日リズムが完全に乱れたものの両者が存在した。
【実施例11】
【0086】
実施例11では、RNA干渉により、RORαの発現を抑制した場合、Bmal1の発現誘導も抑制されるかどうか、調べた。
【0087】
実験手順の概要は、次に通りである。
(1)まず、組換えsiRNA発現ベクターを作製した。RORα4をコードする遺伝子配列の第849〜1391塩基の中から、19塩基からなる配列(配列番号5)を選択した。そして、その配列を、siRNA発現ベクターに組み込み、組換え発現ベクターを作製した。
(2)また、実施例3などと同様に、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
(3)次に、培養細胞NIH3Tに、組換えsiRNA発現ベクターとBmal1のプロモーター領域を組み込んだ組換えベクターとを、共に、トランスフェクションした後、その培養細胞を単離・継代した。
(4)次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
(5)次に、培養液に、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
【0088】
結果を図16に示す。
図中、横軸は時間(単位は分)を、縦軸(relative cpm)は発光強度を示す。なお、発光強度は、最初の発光強度のピークを「1」とした時の相対値である。
「vector」は、ドミナントネガティブ変異体の代わりに空ベクター(pcDNA3)を加えた場合(コントロール)の実験結果であることを示す。
「RNAi(control)」は、RORαの配列とは関係のないsiRNAを用いた場合であり、コントロールである。
「RNAi(RORα)」は、RORαをコードする塩基配列の一部を有するsiRNAを用いた場合である。
【0089】
図16に示す通り、RORαをコードする塩基配列の一部を有するsiRNAを用いた場合(図中、一番下のグラフ)、空ベクターを用いた場合(そのグラフの上側の曲線)と比較して、Bmal1の発現誘導の振幅が減少した(そのグラフ中の下側の曲線)。従って、本実験結果は、RNA干渉を用いてRORの発現を抑制した場合、Bmal1の発現誘導も抑制されることを示す。
【実施例12】
【0090】
実施例12及び実施例13では、マウス胚繊維芽細胞(MEF;Mouse Embryonic Fibroblast)を用いて、RORαにBmal1の発現誘導を促進する作用があるかどうか、調べた。
【0091】
実施例12では、マウス胚繊維芽細胞のRORα遺伝子欠損変異体を用いて、Bmal1発現誘導の概日変化を調べた。
【0092】
実験手順の概要は次の通りである。
(1)まず、前記と同様、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
(2)次に、その組換えベクターを、マウス胚繊維芽細胞のRORα遺伝子欠損変異体にトランスフェクションした後、その培養細胞を単離・継代した。
(3)次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
(4)次に、培養液に、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
【0093】
結果を図17に示す。
図中、横軸は時間(単位は分)を、縦軸(relative cpm)は発光強度を示す。なお、発光強度は、最初の発光強度のピークを「1」とした時の相対値である。
「+/+」は野生型を、「+/sg」はヘテロのRORα遺伝子欠損型を、それぞれ示す。
【0094】
図17に示す通り、マウス胚繊維芽細胞のRORα遺伝子欠損変異体では、Bmal1発現誘導の振幅が減少した。
【実施例13】
【0095】
次に、実施例13では、定量的リアルタイムRT−PCRにより、マウス胚繊維芽細胞のRORα遺伝子欠損変異体における、Bmal1発現量の概日変化を調べた。
【0096】
マウス胚繊維芽細胞をホモジナイズ後、Promega total SV RNA Isolation Kit(Promega社製)を用いて、各臓器からtotal RNAを抽出し、定量的リアルタイムRT−PCRにより、Bmal1遺伝子の発現量を測定した。
【0097】
結果を図18に示す。
図中、横軸は、時間(単位は分)を示す。縦軸「relative mRNA abundance」は、相対的な発現量(発現レベル)を示す。なお、縦軸の発現量の値は、実施例1と同様、ハウスキーピング遺伝子GAPDH(GlycerAldehyde−3−Phosphate DeHydrogenase)の発現量を1とした場合の相対的な値である。
「+/+」は野生型を、「+/sg」はヘテロのRORα遺伝子欠損型を、それぞれ示す。
【0098】
図18に示す通り、マウス胚繊維芽細胞のRORα遺伝子欠損変異体では、Bmal1発現量の振幅が減少した。なお、この結果は、図17の結果ともほぼ一致した。
以上、図17及び図18の結果は、細胞レベルでも、RORαがBmal1の発現誘導を促進することを、強く示唆する。
【実施例14】
【0099】
実施例17では、アンチセンス核酸を用いてRORαの発現を抑制した場合、Bmal1の発現誘導も抑制されるかどうか、調べた。
【0100】
実験手順の概要は次の通りである。
(1)まず、アンチセンス核酸発現ベクターを作製した。RORαをコードする遺伝子のうち、所定の領域と同じ塩基配列を有するDNAフラグメントを、pcDNA3に組み込んで、アンチセンス核酸発現ベクターを作製した。
なお、DNAフラグメントとして、RORαをコードする遺伝子の第65〜574塩基までの配列を有するフラグメント、第599〜1122塩基までの配列を有するフラグメント、第849〜1391塩基までの配列を有するフラグメント、の3種類を用いた。
(2)また、実施例3などと同様に、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
(3)次に、培養細胞NIH3Tに、アンチセンス発現ベクターとBmal1のプロモーター領域を組み込んだ組換えベクターとを、コトランスフェクションし、その培養細胞を単離・継代した。
(4)次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
(5)次に、培養液に、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
【0101】
結果を図19に示す。
図中、図中、横軸は時間(単位は分)を、縦軸(relative cpm)は発光強度を示す。なお、発光強度は、最初の発光強度のピークを「1」とした時の相対値である。
「AS(RORα1)」は、アンチセンス核酸として、RORαをコードする遺伝子の第65〜574塩基までの配列を有するフラグメントを用いた場合、「AS(RORα2)」は、アンチセンスとして、第599〜1122塩基までの配列を有するフラグメントを用いた場合、「AS(RORα3)」は、アンチセンスとして、第849〜1391塩基までの配列を有するフラグメントを用いた場合、を示す。
【0102】
図19に示す通り、いずれの場合も、アンチセンス核酸を用いてRORαの発現を抑制することにより、Bmal1の発現誘導の振幅も抑制された。
【実施例15】
【0103】
上述の通り、RORαは低酸素状態において発現が誘導されることが明らかになっている。そこで、実施例15では、低酸素状態において、Bmal1の発現誘導も促進されるかどうかについて調べた。
【0104】
実験手順の概要は次の通りである。
(1)まず、前記と同様、ルシフェラーゼ発光ベクターpGL3 basic(Promega社製)に、Bmal1のプロモーター領域を組み込んで、組換えベクターを作製した。
(2)次に、その組換えベクターを、マウス胚繊維芽細胞のRORα遺伝子欠損変異体にトランスフェクションし、その培養細胞を単離・継代した。
(3)次に、その培養細胞に血清刺激を加えることにより、概日リズムを発生させた。
(4)次に、培養液に、0.01mMCoCl2を加えた後、15分おきに、ルシフェリン(ルシフェラーゼの基質)を加え、ルシフェラーゼ活性(発光強度)を測定した。そして、その発光強度の概日変化より、Bmal1の転写概日リズムを取得した。
なお、塩化コバルト(CoCl2)を加えた条件下で、細胞を培養することにより、低酸素状態を作り出すことができる。
【0105】
結果を図20に示す。
図中、横軸は時間(単位は分)を、縦軸(relative cpm)は発光強度を示す。なお、発光強度は、最初の発光強度のピークを「1」とした時の相対値である。
「+0.01mMCoCl2」は、培養液に0.01mMCoCl2を加えた場合を、「+0.1mMNaCl」は、培養液に、0.01mMCoCl2の代わりに、0.1mMNaClを加えた場合(コントロール)を、それぞれ示す。
【0106】
図20の結果は、低酸素状態では、Bmal1の発現誘導も促進されることを示す。
【産業上の利用可能性】
【0107】
本発明は、概日リズムの調節メカニズムのうち、未解明な部分を明らかにして点で、有用である。従って、本発明は、概日リズムの調節メカニズムに関与する疾患、例えば、がん、睡眠障害、時差ぼけ、覚醒障害、不眠症、自律神経失調症、うつ病、老人性痴呆などの疾患の予防剤、治療剤として、適用可能性がある。また、DNAチップ、プロテインチップ、概日リズムの変調を検出するためのマーカー、抗がん剤・時差ぼけ調整剤などのスクリーニング方法やスクリーニングキットなどに適用可能である。
【図面の簡単な説明】
【0108】
【図1】概日リズムの調節メカニズムを示す図。
【図2】各組織における、Rev−ervα、Rorα、Bmal1の発現パターンを示す図。
【図3】培養細胞NIH3T3における、Rev−ervα、Rorα、Bmal1の発現パターンを示す図。
【図4】RORαがBmalの発現を誘導したことを示す図。
【図5】RORα4の発現量の増大に伴い、Bmalの発現誘導も促進されることを示す図。
【図6】RORαとREV−ERBタンパク質が、Bmalのプロモーター領域に競合的に作用することを示す図。
【図7】RORαのドミナントネガティブ変異体の存在下で細胞を培養した場合の、Bmal発現量の概日変化を示す図。
【図8】作製した組換えベクターの配列を示す図。
【図9】Bmal1の上流領域の長さをかえた場合における、RORαによるBmalの発現誘導レベルを示す図。
【図10】Bmal1の上流領域の長さをかえた場合における、Bmal発現量の概日変化を示す図。
【図11】プルダウン法による、ウエスタンブロット写真(図面代用写真)。
【図12】15日間、12時間ごとに明条件と暗条件を繰り返した後、暗条件でフリーラン(自由継続)させた場合のアクトグラム。
【図13】15日間、12時間ごとに明条件と暗条件を繰り返した後、飼育環境を24時間暗条件にした場合の、マウスの概日周期を取得した図。
【図14】15日間、12時間ごとに明条件と暗条件を繰り返した後、暗条件の開始時刻を4時間早め、時差のある状態にした場合のアクトグラム。
【図15】15日間、12時間ごとに明条件と暗条件を繰り返した後、環境条件を、6時間ごとに明条件と暗条件を繰り返す環境に変化させた場合のアクトグラム。
【図16】RNA干渉により、RORαの発現を抑制した場合の、Bmalの発現誘導を示す図。
【図17】マウス胚繊維芽細胞のRORα遺伝子欠損変異体を用いた場合の、Bmal発現誘導の概日変化を示す図。
【図18】マウス胚繊維芽細胞のRORα遺伝子欠損変異体を用いた場合における、定量的リアルタイムRT−PCRによる、Bmal発現量の概日変化を示す図。
【図19】アンチセンスを用いてRORαの発現を抑制した場合における、Bmalの発現誘導を示す図。
【図20】低酸素状態において、Bmalの発現誘導が促進されたことを示す図。
【特許請求の範囲】
【請求項1】
Bmal1の発現誘導を促進するRORα。
【請求項2】
低酸素状態で発現が促進されることを特徴とする請求項1記載のRORα。
【請求項3】
REV−ERBα又はREV−ERBβと競合的に作用することを特徴とする請求項1記載のRORα。
【請求項4】
BMAL1とCLOCKとの結合を促進することを特徴とする請求項1記載のRORα。
【請求項5】
Per遺伝子及び/又はCry遺伝子の発現誘導を促進することを特徴とする請求項4記載のRORα。
【請求項6】
請求項1記載のRORαを抑制する物質を少なくとも含有する抗がん剤。
【請求項7】
前記物質は、抗RORα抗体であることを特徴とする請求項6記載の抗がん剤。
【請求項8】
前記物質は、RORαの発現を抑制するsiRNAであることを特徴とする請求項6記載の抗がん剤。
【請求項9】
前記物質は、アンチセンス核酸であることを特徴とする請求項6記載の抗がん剤。
【請求項10】
請求項1記載のRORαを抑制する物質を少なくとも含有する時差ぼけ調整剤。
【請求項11】
前記物質は、抗RORα抗体であることを特徴とする請求項10記載の時差ぼけ調整剤。
【請求項12】
前記物質は、RORαの発現を抑制するsiRNAであることを特徴とする請求項10記載の時差ぼけ調整剤。
【請求項13】
前記物質は、アンチセンス核酸であることを特徴とする請求項10記載の時差ぼけ調整剤。
【請求項14】
請求項1記載のRORαをコードする遺伝子の塩基配列を有する核酸、又は、前記遺伝子の一部分の塩基配列を有する核酸が少なくとも固定されたDNAチップ。
【請求項15】
請求項1記載のRORαと特異的に結合する抗体が少なくとも固定されたプロテインチップ。
【請求項16】
請求項1記載のRORαの、概日リズムの変調を検出するためのマーカーとしての使用。
【請求項17】
請求項1記載のRORαを抑制する物質を探索することによる、抗がん剤のスクリーニング方法。
【請求項18】
請求項1記載のRORαを抑制する物質を探索することによる、時差ぼけ調整剤のスクリーニング方法。
【請求項1】
Bmal1の発現誘導を促進するRORα。
【請求項2】
低酸素状態で発現が促進されることを特徴とする請求項1記載のRORα。
【請求項3】
REV−ERBα又はREV−ERBβと競合的に作用することを特徴とする請求項1記載のRORα。
【請求項4】
BMAL1とCLOCKとの結合を促進することを特徴とする請求項1記載のRORα。
【請求項5】
Per遺伝子及び/又はCry遺伝子の発現誘導を促進することを特徴とする請求項4記載のRORα。
【請求項6】
請求項1記載のRORαを抑制する物質を少なくとも含有する抗がん剤。
【請求項7】
前記物質は、抗RORα抗体であることを特徴とする請求項6記載の抗がん剤。
【請求項8】
前記物質は、RORαの発現を抑制するsiRNAであることを特徴とする請求項6記載の抗がん剤。
【請求項9】
前記物質は、アンチセンス核酸であることを特徴とする請求項6記載の抗がん剤。
【請求項10】
請求項1記載のRORαを抑制する物質を少なくとも含有する時差ぼけ調整剤。
【請求項11】
前記物質は、抗RORα抗体であることを特徴とする請求項10記載の時差ぼけ調整剤。
【請求項12】
前記物質は、RORαの発現を抑制するsiRNAであることを特徴とする請求項10記載の時差ぼけ調整剤。
【請求項13】
前記物質は、アンチセンス核酸であることを特徴とする請求項10記載の時差ぼけ調整剤。
【請求項14】
請求項1記載のRORαをコードする遺伝子の塩基配列を有する核酸、又は、前記遺伝子の一部分の塩基配列を有する核酸が少なくとも固定されたDNAチップ。
【請求項15】
請求項1記載のRORαと特異的に結合する抗体が少なくとも固定されたプロテインチップ。
【請求項16】
請求項1記載のRORαの、概日リズムの変調を検出するためのマーカーとしての使用。
【請求項17】
請求項1記載のRORαを抑制する物質を探索することによる、抗がん剤のスクリーニング方法。
【請求項18】
請求項1記載のRORαを抑制する物質を探索することによる、時差ぼけ調整剤のスクリーニング方法。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】


【図16】


【図17】


【図18】


【図19】


【図20】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11】


【図12】


【図13】


【図14】


【図15】


【図16】


【図17】


【図18】


【図19】


【図20】


【公開番号】特開2006−273760(P2006−273760A)
【公開日】平成18年10月12日(2006.10.12)
【国際特許分類】
【出願番号】特願2005−96459(P2005−96459)
【出願日】平成17年3月29日(2005.3.29)
【出願人】(000002185)ソニー株式会社 (34,172)
【Fターム(参考)】
【公開日】平成18年10月12日(2006.10.12)
【国際特許分類】
【出願日】平成17年3月29日(2005.3.29)
【出願人】(000002185)ソニー株式会社 (34,172)
【Fターム(参考)】
[ Back to top ]

