Cdc25Bとβ−TrCPとの特異的結合を利用した検出系
【課題】本発明は、非遺伝毒性ストレスを与える毒性物質などの、細胞内でJNK1キナーゼやp38キナーゼなどの活性化を誘導する物質をスクリーニングするための簡便な手段を提供すること等を目的とする。
【解決手段】本発明は、Cdc25Bとβ-TrCPとがJNK1キナーゼが誘導される条件下(非遺伝毒性ストレス条件下)においてJNK1キナーゼ活性に依存して相互作用することを利用する。Cdc25Bと第1検出用タンパク質とを融合させた第1融合タンパク質と、β-TrCPと第2検出用タンパク質とを融合させた第2融合タンパク質とを同一細胞で発現させ、被験物質を作用させ、Cdc25Bとβ-TrCPとの相互作用を検出することにより、所望の活性を有する物質をスクリーニングすることができる。
【解決手段】本発明は、Cdc25Bとβ-TrCPとがJNK1キナーゼが誘導される条件下(非遺伝毒性ストレス条件下)においてJNK1キナーゼ活性に依存して相互作用することを利用する。Cdc25Bと第1検出用タンパク質とを融合させた第1融合タンパク質と、β-TrCPと第2検出用タンパク質とを融合させた第2融合タンパク質とを同一細胞で発現させ、被験物質を作用させ、Cdc25Bとβ-TrCPとの相互作用を検出することにより、所望の活性を有する物質をスクリーニングすることができる。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する方法に関する。
【背景技術】
【0002】
非遺伝毒性ストレスを受けた細胞では、JNK1キナーゼやp38キナーゼが誘導されることが知られている。しかしながら従来は、JNK1キナーゼやp38キナーゼが誘導されているか否かを簡便に測定する方法がなかった。また、非遺伝毒性ストレスを与える毒性物質をスクリーニングする方法としては、従来は毒性物質を動物に投与することが行われており、手間と時間がかかるという問題があった。
【0003】
Cdc25Bが非遺伝毒性ストレスにより活性化されるJNK1キナーゼやp38キナーゼによりリン酸化を受け分解されるということは知られていなかった。すなわち、非遺伝毒性ストレスを与える毒性物質が処理された細胞では、Cdc25Bが分解されるということは知られていなかった。
【0004】
一方、Cdc25Bはガン細胞において発現が促進されることが知られている。Cdc25Bを細胞内で減少させる物質は抗ガン剤として有用であると考えられるが、Cdc25Bを減少させる作用を有する物質をスクリーニングする手段は従来存在しなかった。
【0005】
また、Cdc25Bとβ-TrCP1または2とが相互作用することによりCdc25Bがユビキチン化され、Cdc25Bの分解に至ることが知られていた。しかしながら従来知られていた相互作用は、JNK1キナーゼやp38キナーゼの活性に依存しない作用であった。
【発明の開示】
【発明が解決しようとする課題】
【0006】
本発明は、非遺伝毒性ストレスを与える毒性物質などの、細胞内でJNK1キナーゼやp38キナーゼなどの活性化を誘導する物質をスクリーニングするための簡便な手段を提供することを目的とする。
【0007】
本発明はまた、Cdc25BまたはCdc25Aの細胞内での分解を促進する物質をスクリーニングするための簡便な手段を提供することを目的とする。
【0008】
本発明はより一般的には、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を有する物質をスクリーニングするための簡便な手段を提供することを目的とする。
【課題を解決するための手段】
【0009】
本発明者らは、Cdc25Bとβ-TrCPとがJNK1キナーゼが誘導される条件下(非遺伝毒性ストレス条件下)においてJNK1キナーゼ活性に依存して相互作用するという驚くべき知見を得た。更に、JNK1キナーゼ活性に依存してCdc25Bとβ-TrCPとが結合する際には、Cdc25Bの特定の領域(従来知られていた、JNK1キナーゼやp38キナーゼの活性に依存しない結合に関与する箇所とは異なる)が関与することを見出した。これらのことから、Cdc25Bとβ-TrCPとの相互作用の有無によって、JNK1が誘導されているか否か(非遺伝毒性ストレスが負荷されているか否か)を判別することが可能となることに想到するに至った。
【0010】
本発明者らはさらに、緑色蛍光タンパク質のN末端側断片とC末端側断片とをそれぞれCdc25Bとβ-TrCPのどちらかに融合させた2種類の融合タンパク質が、Cdc25Bとβ-TrCPとの相互作用に応じて蛍光を発することを見出した。
【0011】
以上の新規知見に基づいて、本発明者らは以下の発明を完成させるに至った。
(1) 被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する方法であって、
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質とを同一の細胞内で発現させる発現工程と、
発現工程後の細胞に被験物質を作用させる作用工程と、
作用工程後の前記細胞において前記信号を測定し、測定された前記信号を指標にして、前記被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する評価工程とを含む前記方法。
(2) 前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(1)の方法。
(3) 前記因子がJNK1キナーゼ、JNK2キナーゼ、p38キナーゼ、ERK1、ERK2またはCDKキナーゼである、(2)の方法。
(4) Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、(2)または(3)の方法。
(5) 前記評価工程において前記因子を誘導する能力を有すると評価された被験物質を、遺伝毒性ストレス原因物質、非遺伝毒性ストレス原因物質、遺伝毒性ストレス原因物質の働きを軽減する物質、非遺伝毒性ストレス原因物質の働きを軽減する物質、または抗癌物質の候補物質として選抜するスクリーニング工程を更に含む、(2)〜(4)のいずれかの方法。
(6) 前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(1) の方法。
(7) 前記因子がp38キナーゼ、CHK1キナーゼ、CHK2キナーゼまたはMK2キナーゼである、(6)の方法。
(8) 前記評価工程において前記因子を誘導すると評価された被験物質を、遺伝毒性ストレス原因物質、遺伝毒性ストレス原因物質の働きを軽減する物質、または抗癌物質の候補物質として選抜するスクリーニング工程を更に含む、(6)または(7)の方法。
(9) 前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、(1)〜(8)のいずれかの方法。
(10) β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、(9)の方法。
【0012】
(11) 前記発現工程において、前記第1融合タンパク質と前記第2融合タンパク質とが、互いに連結された四成分融合タンパク質として細胞内で発現される、(1)〜(10)のいずれかの方法。
(12) Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質をコードするDNAと、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質をコードするDNAとの組み合わせ。
(13) 前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(12)の組み合わせ。
(14) Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、(13) の組み合わせ。
(15) 前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(12)の組み合わせ。
(16) 前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、(12)〜(15)のいずれかの組み合わせ。
(17) β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、(16) の組み合わせ。
(18) Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質とが互いに連結された四成分融合タンパク質をコードするDNA。
(19) 前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(18)のDNA。
【0013】
(20) Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、(19)のDNA。
(21) 前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(18)のDNA。
(22) 前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、(18)〜(21)のいずれかのDNA。
(23) β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、(22)のDNA。
(24) (12)〜(17)のいずれかにおいて定義される第1融合タンパク質をコードするDNAを含むベクターと、(12)〜(17)のいずれかにおいて定義される前記第2融合タンパク質をコードするDNAを含むベクターとの組み合わせ。
(25) (18)〜(23)のいずれかのDNAを含むベクター。
(26) (24)のベクターの組み合わせにより形質転換された形質転換細胞。
(27) (25)のベクターにより形質転換された形質転換細胞。
(28) (12)〜(23)のいずれかの組み合わせ、またはDNAが導入され(形質転換され)た細胞。
(29) (28) の細胞を体内に含む動物。
(30) (12)〜(17)のいずれかのDNAの組み合わせによりコードされる、第1融合タンパク質と第2融合タンパク質との組み合わせ。
(31) (18)〜(23)いずれかのDNAによりコードされる、四成分融合タンパク質。
【発明の効果】
【0014】
本発明によれば、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を有する物質をスクリーニングするための簡便な手段が提供される。このような物質は、遺伝毒性ストレス原因物質(毒性物質)、非遺伝毒性ストレス原因物質(毒性物質)または抗癌剤として有用である可能性がある。
【発明を実施するための最良の形態】
【0015】
1.第1融合タンパク質
1.1.Cdc25B
本発明に用いるCdc25Bの起源となる動物種は特に限定されない。更に、Cdc25Bとしての活性を有している限り、天然に存在するCdc25B遺伝子およびそれによりコードされるCdc25Bだけでなく、人工合成されたCdc25B遺伝子およびそれによりコードされるCdc25Bもまた、本発明に用いることができる。例えば、哺乳動物(例えばヒト、サル(例えばチンパンジー、カニクイザル)、ウシ、ウマ、マウス、ラット)、鳥類、両生類(例えばアフリカツメガエル)に由来するCdc25Bおよびそれに基づいて人工合成されたCdc25Bを使用することができ、ヒトを含む哺乳動物由来のCdc25Bおよびそれに基づいて人工合成されたCdc25Bが特に好ましい。ヒト由来のCdc25Bには、Cdc25B1、Cdc25B2およびCdc25B3の三種類が知られており、いずれも本発明に使用することができる。配列番号2にはヒト由来Cdc25B1のアミノ酸配列を、配列番号1にはそれをコードする塩基配列を示す。配列番号2のアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるCdc25Bに包含される。
【0016】
種々の動物種のCdc25Bの塩基配列およびはそれから推定されるアミノ酸配列は公知であり、cell division cycle 25 homolog B (S. pombe)、Cdc25B、CDC25B等の名称により各種データベースに登録されている。Cdc25Bの塩基配列の登録番号の具体例は以下に示される。ヒト(Homo sapiens: NM_021873、NM_021872、NM_004358、Z68092、BC006395、BC009953、BC051711、AY892373、M81934)、マウス(Mus musculus: NM_023117、NM_001111075、BC057568、BC002287)、ラット(Rattus norvegicus: D16237、NM_133572)、ウシ(Bos Taurus: XM_866334)、ウマ(Equus caballus: XM_001495948、XM_001495974)、カニクイザル(Macaca fascicularis: AB173014、AB060926)、チンパンジー(Pan troglodytes: XM_001162178、XM_001162284、XM_001162247、XM_001162578、XM_001162531、XM_001162364、XM_001162323、XM_001162134、XM_001162488、XM_001162029、XM_001162625、XM_001162402、XM_001162213、XM_001162666、XM_001162447、XM_514487、XM_001162751、XM_001162706)、アフリカツメガエル(NM_001127885、NM_001127884、AB363841、AB363840)、ニシツメガエル(BC166320、BC125767)。これらの塩基配列がコードするCdc25Bのアミノ酸配列からなるタンパク質だけでなく、これらの塩基配列がコードするCdc25Bのアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるCdc25Bに包含される。
【0017】
本発明では、Cdc25Bは少なくともその一部が使用されればよく、全体を用いる必要はない。Cdc25BのなかでもJNK1依存的にβ-TrCPと相互作用する領域(ヒトCdc25Bの場合は配列番号2の第94〜108番)を含む、全長400アミノ酸以下、特に300アミノ酸以下、さらには200アミノ酸以下の部分配列からなるタンパク質が好ましい。例えば、配列番号2の第1〜175番のアミノ酸配列からなるタンパク質や、配列番号2の第1〜280番のアミノ酸配列からなるタンパク質が好適に使用できる。
【0018】
また、ヒト以外の動物種起源のCdc25Bにおいても、配列番号2の第94〜108番に相当する領域を含む全長400アミノ酸以下、特に300アミノ酸以下、さらには200アミノ酸以下の部分アミノ酸配列からなるタンパク質を好適に使用することができる。ヒトCdc25B1の第94〜108番に相当する領域としては次のアミノ酸配列からなる部分配列が挙げられる:
ヒト(Homo sapiens):DAGLCMDSPSPMDPH(例えば、NM_021873: 108-122、NM_021872: 108-122、NM_004358: 94-108、Z68092: 108-122、BC006395: 108-122、BC009953: 108-122、BC051711: 108-122、 AY892373: 108-122、AY494082中のDAGLCMDSPSPMDPHで示される配列、AF036233中の配列から読み取られるアミノ酸配列中のDAGLCMDSPSPMDPHで示される配列、M81934: 94-108)
チンパンジー(Pan troglodytes):DAGLCMDSPSPMDPH(例えば、XM_001162134: 108-122、XM_001162488: 108-122、XM_001162625: 108-122、XM_001162402: 108-122、XM_001162213: 94-108、XM_001162666: 94-108、XM_001162447: 94-108、XM_514487: 108-122、XM_001162751: 108-122、XM_001162706: 108-122)
カニクイザル(Macaca fascicularis):DAGLCMDSPSPVDPQ(例えば、AB060926: 94-108)
ウシ(Bos Taurus):DAGLCMDSPSPMDPN(例えば、XM_866334: 101-115)
ウマ(Equus caballus):DAGLCMDSPSPMDPQ(例えば、XM_001495948: 91-105、XM_001495974: 101-115)
マウス(Mus musculus):DAGLCMDSPSPVDPQ(例えば、NM_023117: 106-120、NM_001111075: 106-120、BC057568: 106-120)
ラット(Rattus norvegicus):DAGLCMDSPSPMDPQ(例えば、D16237: 106-120、NM_133572: 106-120)
アフリカツメガエル(Xenopus laevis):DSGLCLDSPSPQELE(例えば、NM_001127885: 93-107、NM_001127884: 94-108、AB363841: 93-107、AB363840: 94-108)
ニシツメガエル(例えば、BC166320: 92-106)
【0019】
1.2.Cdc25A
本発明に用いるCdc25Aの起源となる動物種は特に限定されない。更に、Cdc25Aとしての活性を有している限り、天然に存在するCdc25A遺伝子およびそれによりコードされるCdc25Aだけでなく、人工合成されたCdc25A遺伝子およびそれによりコードされるCdc25Aもまた、本発明に用いることができる。例えば、哺乳動物(例えばヒト、サル(例えばチンパンジー、カニクイザル)、ウシ、ウマ、マウス、ラット)、鳥類(例えばニワトリ、ウズラ)、両生類(例えばアフリカツメガエル、ヒキガエル)、魚類(例えばゼブラフィッシュ)に由来のCdc25Aおよびそれに基づいて人工合成されたCdc25Aであれば特に限定されないが、ヒトを含む哺乳動物由来のCdc25Aおよびそれに基づいて人工合成されたCdc25Aが好ましい。
【0020】
種々の動物種のCdc25Aのアミノ酸配列および塩基配列は公知であり、cell division cycle protein 25A、CDC25AまたはCdc25A等の登録名により各種データベースに登録されている。Cdc25Aの塩基配列の登録番号の具体例は以下に示される。ヒト(Homo sapiens: NM_201567、NM_001789、DQ896168、CU675644、CU675643:、AK290554、BC007401、BC018642、AY889759、AY892219、AY137580、AF277722、M81933、DQ892919、DQ890753)、マウス(Mus musculus: U27323、BC046296、AK077991、AK077609、NM_007658)、ラット(Rattus norvegicus: NM_133571、D16236)、ウシ(NM_001101100、BC151493)、ウマ(Equus caballus: XM_001495192)、チンパンジー(Pan troglodytes: XM_001155610、XM_516433)、アフリカツメガエル(Xenopus laevis: D82960、AF090829、BC082625、BC103738、NM_001088487、NM_001096204)、ニシツメガエル(Xenopus tropicalis: BC135487、CR855457、NM_001045598)、ヒキガエル(Bufo gargarizans:AY491052)、ゼブラフィッシュ8Danio rerio: EU921896))。これらの塩基配列がコードするCdc25Aのアミノ酸配列からなるタンパク質だけでなく、これらの塩基配列がコードするこれらのCdc25Aのアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるCdc25Aに包含される。
【0021】
本発明では、Cdc25Aは少なくともその一部が使用されればよく、全体を用いる必要はない。Cdc25Aのなかでもβ-TrCPと相互作用する領域を含む、全長400アミノ酸以下、特に300アミノ酸以下、さらには200アミノ酸以下の部分配列が好ましい。たとえば、第1〜140番のアミノ酸配列からなるタンパク質や、第1〜240番のアミノ酸配列からなるタンパク質が好適に使用できる。
【0022】
1.3.リンカー
第1融合タンパク質では、Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結されている。第1検出用タンパク質については後述する。
【0023】
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質との間には適宜リンカーを介して連結されている。リンカー部分の構造は特に限定されないが、通常は、アミノ酸数が3〜20個程度であり、Cdc25AまたはCdc25Bと第一検出用タンパク質の立体的な動きが阻害されないようにグリシン残基に富む(例えばリンカーを構成するアミノ酸の半数以上がグリシンである)ことが望ましい。
【0024】
またリンカーのほかに、融合タンパク質を作成する際の必要に応じて導入される制限酵素認識配列によりコードされるアミノ酸配列などが、Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質との間に存在していてもよい。
【0025】
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とはどちらがN末端側に位置しても構わない。
【0026】
2.第2融合タンパク質
2.1.β-TrCP1
本発明に用いるβ-TrCP1の起源となる動物種は特に限定されない。更に、β-TrCP1としての活性を有している限り、天然に存在するβ-TrCP1遺伝子およびそれによりコードされるβ-TrCP1だけでなく、人工合成されたβ-TrCP1遺伝子およびそれによりコードされるβ-TrCP1もまた、本発明に用いることができる。例えば、哺乳動物(ヒト、サル(例えばチンパンジー、オラウータン、カニクイザル)、ウシ、マウス、ラット)、鳥類(例えばニワトリ、ウズラ)、両生類(例えばアフリカツメガエル)、ホヤ、キイロショウジョウバエに由来するβ-TrCP1およびそれに基づいて人工合成されたβ-TrCP1を使用することができ、ヒトを含む哺乳動物由来のβ-TrCP1およびそれに基づいて人工合成されたβ-TrCP1が好ましい。配列番号29にはヒト由来β-TrCP1のアミノ酸配列を、配列番号28にはそれをコードする塩基配列を示す。配列番号29のアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるβ-TrCP1に包含される。
【0027】
種々の動物種のβ-TrCP1のアミノ酸配列および塩基配列は公知であり、beta-transducin repeat containing (BTRC、beta-TrCP、β-TrCP、beta-TrCP1)、F-box and WD repeat domain containing 1(Fbw1、Fbw1a、FWD1、FBXW1、FBXW1A)MGC4643等の名称により各種データベースに登録されている。β-TrCP1の塩基配列の登録番号の具体例は以下に示される。
ヒト(Homo sapiens: NM_003939, NM_033637, NM_033637, BD131778, AK313417, AK313353, BC027994, AF129530, Y14153)、マウス(Mus musculus : NM_001037758, NM_009771, AF081887, AF110396, AF112979, AF099932, AK145624, AK145091, AK156660, AK083257, AK052317, AK032221, BC052879)、ラット(Rattus norvegicus : NM_001007148, BC085125)、オラウータン(Pongo abelii : NM_001131502, BC003989, CR859772, CR857777)、ウシ(Bos Taurus: BC123621, NM_001083475)、ウマ(Equus caballus: XM_001499866, XM_001924828)、アフリカツメガエル(Xenopus laevis : NM_001087595, BC079732, M98268)、イヌ(Canis lupus familiaris: XM_856836, XM_856812, XM_543980)、ニシツメガエル(Xenopus tropicalis : NM_001016386)、カニクイザル(Macaca fascicularis: AB071071, AB169034)、チンパンジー(Pan troglodytes: XM_507991)、ショウジョウバエ(Drosophila melanogaster : NM_079706, AF222924, AF222923, AF032878)。これらの塩基配列がコードするβ-TrCP1のアミノ酸配列からなるタンパク質だけでなく、これらの塩基配列がコードするβ-TrCP1のアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるβ-TrCP1に包含される。
【0028】
本発明では、β-TrCP1は少なくともその一部が使用されればよく、全体を用いる必要はない。特に、Cdc25AまたはCdc25Bとの結合後にユビキチン化に関与するF-boxと呼ばれる領域を有していないβ-TrCP1の部分配列を使用することが好ましい。例えば、F-boxと呼ばれる領域の全部または一部が欠損したアミノ酸配列からなるβ-TrCP1や、F-boxと呼ばれる領域の途中のアミノ酸からC末端に至るβ-TrCP1の領域(より好ましくはF-boxと呼ばれる領域および該領域よりもN末端側を欠くβ-TrCP1の領域)に含まれる、全長200アミノ酸以上の部分アミノ酸配列、特に好ましくは全長350アミノ酸以上の部分アミノ酸配列からなるタンパク質が好ましい。例えば、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能であるものが好ましく、なかでも配列番号29の第212番からC末端に至る部分配列が好ましい。F-boxと呼ばれる領域は、上記のデータベースの塩基配列から推定されるアミノ酸配列としては、ヒト、マウス、ラット、オラウータン、ウシ、ウマ、イヌ、カニクイザル、チンパンジーにおいては、TALPARGLDHIAENILSYLDAKSLCAAELVCKEWYRVTSDGMLWKKL、で表わされる。オラウータンにはアミノ酸配列の異なる2種類の遺伝子が登録されており、もうひとつF-box領域は、TALPARGLDHIAENILSYLDAKSLCAAELVCKEWYRVTSDGMLWRKL である。また、アフリカツメガエルとセイヨウツメガエルでは、TALPARGLDHIAENILSYLDAKSLCSAELVCKEWYRVTSDGMLWKKL、ショウジョウバエでは、TLLPIKGLDHIAENILSYLDAESLKSSELVCKEWLRVISEGMLWKKLというアミノ酸配列で表される領域がF-box領域に該当する。
【0029】
2.2.β-TrCP2
本発明に用いるβ-TrCP2の起源となる動物種は特に限定されない。更に、β-TrCP2としての活性を有している限り、天然に存在するβ-TrCP2遺伝子およびそれによりコードされるβ-TrCP2だけでなく、人工合成されたβ-TrCP2遺伝子およびそれによりコードされるβ-TrCP2もまた、本発明に用いることができる。例えば、哺乳動物(例えばヒト、ウシ、ウマ、マウス、ラット)、鳥類(例えばニワトリ、ウズラ)、両生類(例えばアフリカツメガエル)に由来のβ-TrCP2およびそれに基づいて人工合成されたβ-TrCP2であれば特に限定されないが、ヒトを含む哺乳動物由来のβ-TrCP2およびそれに基づいて人工合成されたβ-TrCP2が好ましい。
【0030】
種々の動物種のβ-TrCP2のアミノ酸配列および塩基配列は公知であり、beta-transducin repeat-containing protein 2(BTRC2)、BTRCP2、F-box and WD-40 domain protein 1B(Fbw1B)、FBW11、F-box and WD repeat domain containing 11(F-box and WD repeat domain containing 11 、FBXW11)、HOS等の登録名により各種データベースに登録されている。β-TrCP2の塩基配列の登録番号の具体例は以下に示される。
ヒト(Homo sapiens: NM_033645, NM_012300, NM_033644, DQ894304, DQ891125, AB033281, AB033280, AB033279, AK314999, AB014596, AB383960)、マウス(Mus musculus: NM_134015, BC034261, BC008552, AY038079, AB093260)、チンパンジー(Pan troglodytes : XM_518097)、ニシツメガエル(Xenopus tropicalis: BC170890, BC170892)、ラット(Rattus norvegicus: NM_001106993)、ウシ(Bos Taurus: XM_612428)、ニワトリ (Gallus gallus : NM_001039262)、ゼブラフィッシュ (Danio rerio: NM_201310, BC045356, NM_213504)、ウマ(Equus caballus: XM_001499667)。これらの塩基配列がコードするβ-TrCP2のアミノ酸配列からなるタンパク質だけでなく、これらの塩基配列がコードするβ-TrCP2のアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるβ-TrCP2に包含される。
【0031】
本発明では、β-TrCP2は少なくともその一部が使用されればよく、全体を用いる必要はない。特に、Cdc25AまたはCdc25Bとの結合後にユビキチン化に関与するF-boxと呼ばれる領域を有していないβ-TrCP2の部分配列を使用することが好ましい。例えば、F-boxと呼ばれる領域の全部または一部が欠損したアミノ酸配列からなるβ-TrCP2や、F-boxと呼ばれる領域の途中のアミノ酸からC末端に至るβ-TrCP2の領域(より好ましくはF-boxと呼ばれる領域および該領域よりもN末端側を欠くβ-TrCP2の領域)に含まれる、全長200アミノ酸以上の部分アミノ酸配列、特に好ましくは全長350アミノ酸以上の部分アミノ酸配列からなるタンパク質が好ましい。F-boxと呼ばれる領域は上記のデータベース上の塩基配列から推定されるアミノ酸配列としては、、ヒト、マウス、チンパンジー、ラット、ウシ、ニワトリ、ウマにおいては、TALPEQGLDHIAENILSYLDARSLCAAELVCKEWQRVISEGMLWKKL、ニシツメガエルでは、TALPARGLDHIAENILSYLDARSLCSAELVCKEWYRVTSDGMLWKKL、ゼブラフィッシュでは、TALPARGLDHIAENILSFLDARSLCSAELVCREWQRVISDGMLWKKLというアミノ酸配列であらわされる。
【0032】
2.3.リンカー
第2融合タンパク質ではβ-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結されている。第2検出用タンパク質については後述する。
【0033】
β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質との間には適宜リンカーを介して連結されている。リンカー部分の構造は特に限定されないが、通常は、アミノ酸数が3〜20個程度であり、β-TrCP1またはβ-TrCP2と第二検出用タンパク質の立体的な動きが阻害されないようにグリシン残基に富む(例えばリンカーを構成するアミノ酸の半数以上がグリシンである)ことが望ましい。
【0034】
またリンカーのほかに、融合タンパク質を作成する際の必要に応じて導入される制限酵素認識配列によりコードされるアミノ酸配列などが、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質との間に存在していてもよい。
β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とはどちらがN末端側に位置しても構わない。
【0035】
3.相互作用を識別するための手段
第1検出用タンパク質と第2検出用タンパク質とは、相互作用したときに識別可能な信号を発することができる。
ここで「識別可能な信号」とは好ましくは緑色蛍光、赤色蛍光、黄色蛍光などの蛍光である。
【0036】
このような信号を発する第1検出用タンパク質と第2検出用タンパク質との組合せとしては、緑色蛍光タンパク質(GFP)をはじめ、黄色蛍光タンパク質(YFP)、青色蛍光タンパク質(CFPやBFC)、赤色蛍光タンパク質(RFP)などのGFP類似の蛍光タンパク質のC末端側断片とN末端側断片との組み合わせ(BiFC (bi-molecular fluorescence complementation)法に用いる)や、二種類の全長蛍光タンパク質を用いて蛍光を発光させるFRET(fluorescence resonance energy transfer)法で使用するためのCFPとYFPとの組み合わせや、YFPとRFPとの組み合わせなどの、公知の組み合わせを使用することができる。
【0037】
これらの二種類の検出用タンパク質の組み合わせのなかから、一方を第1検出用タンパク質として、他方を第2検出用タンパクとして用いればよい。
【0038】
4.四成分融合タンパク質
第1融合タンパク質と第2融合タンパク質とは互いに連結された四成分を少なくとも含む融合タンパク質として使用することがより好ましい。
【0039】
第1融合タンパク質と第2融合タンパク質との間にはリンカー部分が介在していることが好ましい。リンカー部分の構造は特に限定されないが、通常は、アミノ酸数が3〜30程度であり、第1融合タンパク質と第2融合タンパク質の立体的な動きが阻害されないようにグリシン残基に富む(例えばリンカーを構成するアミノ酸の半数以上がグリシンである)ことが望ましい。
【0040】
またリンカーのほかに、融合タンパク質を作成する際の必要に応じて導入される制限酵素認識配列によりコードされるアミノ酸配列などが、第1融合タンパク質と第2融合タンパク質との間に存在していてもよい。
第1融合タンパク質と第2融合タンパク質とはどちらがN末端側に配置されてもよい。
【0041】
5.測定に用いる細胞
本発明では、上記の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)を細胞において発現させる。細胞としては特に限定されないがHeLa、Cos7をはじめとし、ベクターにより形質転換が可能であり導入された融合タンパク質が安定に発現されれば、初代培養細胞を含むすべての細胞が使用できる。
【0042】
6.スクリーニング結果の評価
前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である場合には、被験物質が、Cdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価することができる。このような因子としては、JNK1キナーゼ、JNK2キナーゼ、p38キナーゼ(p38α、p38βまたはp38γ)、ERK1、ERK2、CDKキナーゼ(CDK1〜6)が挙げられる。このような因子を誘導する物質は、遺伝毒性ストレス原因物質、非遺伝毒性ストレス原因物質または抗癌物質の候補物質となりうる。更に、遺伝毒性ストレス原因物質の働きを軽減する物質、非遺伝毒性ストレス原因物質の働きを軽減する物質の候補物質ともなりうる。よって、本発明はこれらの物質のスクリーニングのために従来行われていた動物試験に代わる一次スクリーニング手段として有効である。
【0043】
前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である場合には、被験物質が、Cdc25Aの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価することができる。このような因子としては、p38キナーゼ、CHK1キナーゼ、CHK2キナーゼ、MK2キナーゼが挙げられる。このような因子を誘導する物質は、遺伝毒性ストレス原因物質(毒性物質)または抗癌物質の候補物質となりうる。更に、遺伝毒性ストレス原因物質の働きを軽減する物質の候補物質ともなりうる。
【0044】
なお本発明において、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子の「誘導」とは、タンパク質発現量の増大に限らず、当該因子の活性の増強全般を指す。
【0045】
7.ベクター、形質転換細胞
本発明はまた、上述の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)をコードするDNAを含むベクターに関する。このようなベクターとしてはプラスミドベクター、ウイルスベクター等が挙げられるがこれには限定されない。
本発明は更にまた、当該ベクターにより形質転換された形質転換細胞に関する。対象となる細胞としては、上記5に示した細胞が挙げられる。
【0046】
8.ゲノムDNAへの導入および実験動物
本発明では、上述の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)をコードするDNAがゲノム中に取り込まれて永続的にこれらの融合タンパク質が発現可能となった細胞、並びに当該細胞を体内に含む非ヒト実験動物をも提供する。
【0047】
このような細胞の作成方法としては、G418やハイグロマイシンなどの薬剤に対する耐性遺伝子を有するベクターに上述の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)をコードするDNAを組み込み、そのベクターが導入された細胞を薬剤で選択すること(永続的にこれらの融合タンパク質が発現可能となった細胞の分離)や、マウスの受精卵に上述の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)をコードするDNAをマイクロインジェクションすること(当該細胞を体内に含む非ヒト実験動物の作成)が挙げられる。
【0048】
上記細胞を体内に含む非ヒト実験動物としては、マウス、ラット、ブタ、ウシ、ゼブラフィッシュ、メダカが挙げられる。例えば、こうして得られた非ヒト実験動物に、被験物質を接触させ、一定期間経過後に肝臓や皮膚などの組織から細胞を採取し蛍光を測定し、蛍光が観測された場合には該物質は非遺伝毒性ストレス原因物質または抗癌物質であると判断する、などの応用が可能となるであろう。
【0049】
9.キット
上述のDNA(またはDNAの組み合わせ)、ベクター(またはベクターの組み合わせ)および形質転換細胞は、被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価するためのアッセイ用キットの構成要素として利用することが可能である。
【0050】
参考例
Cdc25BはJNK1活性に依存してβ-TrCP1と結合する
Cdc25Bを恒常発現するHeLa細胞を作成し、該細胞を、遺伝毒性ストレス因子であるヒドロキシ尿素(1.5 mM、1時間処理)、アフィディコリン(2.5μg/ml、1時間処理)、エトポシド(10μM、1時間処理)、またはカンプトテシン(2μM、1時間処理)で処理するか、或いは、非遺伝毒性ストレス因子であるアニソマイシン(50 ng/ml、10分処理)または食塩(300mM、1時間処理)で処理した後にCdc25B量を確認した。その結果、非遺伝毒性ストレス因子の処理によって、Cdc25Bの分解が誘導されることが明らかとなった(図1)。
【0051】
更に、阻害剤や強発現実験から、このCdc25B分解促進はJNK経路によることが示された。図2は、β-TrCPを含むSCF (Skp1-Cullin 1-F-box proteinの3種のタンパク質からなる複合体であり、下線で示した頭文字をとって略したものである。F-boxと呼ばれる配列をもつタンパク質群と複合体を形成しタンパク質のユビキチン化を行う複合体の総称) によりCdc25Bの断片がJNK1依存的にユビキチン化されることを示す。
【0052】
JNK1はin vitroでCdc25BのN末端断片(1-175アミノ酸)をリン酸化することが確認された。更に変異体の解析によりSer101がJNKに依存するCdc25Bの分解に必須であることが確認された(図3)。
【0053】
これらの結果から、本発明者らは、非遺伝毒性ストレスの存在下においてJNK/β-TrCPを介したCdc25Bの分解という新しい細胞周期制御機構を明らかにした。図4にはこの新規な機構を模式的に示す。
【0054】
以下の実施例では融合タンパク質の配置順序に関して下記の4通り実施した。しかしながら本発明の範囲はこれらには限定されない。
(1)Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質(実施例では蛍光タンパク質のN末端側断片)とがこの順でN末端側から配置された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質(実施例では蛍光タンパク質のC末端側断片)がこの順でN末端側から配置された第2融合タンパク質との組み合わせ。
(2)Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質(実施例では蛍光タンパク質のN末端側断片)とがこの順でN末端側から配置された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質(実施例では蛍光タンパク質のC末端側断片)がこの順でN末端側から配置された第2融合タンパク質とが、この順でN末端側から配置された四成分融合タンパク質。
(3)第1検出用タンパク質(実施例では蛍光タンパク質のN末端側断片)とCdc25AまたはCdc25Bの少なくとも一部分とがこの順でN末端側から配置された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質(実施例では蛍光タンパク質のC末端側断片)とがこの順でN末端側から配置された第2融合タンパク質との組み合わせ。
(4)第1検出用タンパク質(実施例では蛍光タンパク質のN末端側断片)とCdc25AまたはCdc25Bの少なくとも一部分とがこの順でN末端側から配置された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質(実施例では蛍光タンパク質のC末端側断片)とがこの順でN末端側から配置された第2融合タンパク質とが、この順でN末端側から配置された四成分融合タンパク質。
【実施例】
【0055】
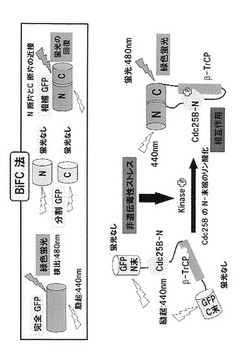
本発明は、新たに見出された上記参考例の知見を利用する発明である。すなわち、Cdc25Bとβ-TrCPとがJNK1が誘導される条件下(非遺伝毒性ストレス条件下)において相互作用することから、Cdc25Bとβ-TrCPとの相互作用の有無によって、JNK1が誘導されているか否か(非遺伝毒性ストレスが負荷されているか否か)を判別することが可能となる。Cdc25Bとβ-TrCPとの相互作用を解析するには、例えばBiFC (bi-molecular fluorescent complementation)法が利用できる。図5には、BiFC法によりJNK1依存性のCdc25Bとβ-TrCPとの相互作用を判別する方法の典型的な例を示す。
【0056】
以下に、非遺伝毒性ストレスの存在下においてBiFC法により蛍光発光が実際に確認された具体例を詳細に説明する。
実験材料と方法
1.融合タンパク質の作成手順概要
1.1.用いたプラスミド
BiFCに用いた、蛍光タンパク質を2分割した断片を発現するプラスミドは、医学生物学研究所(MBL:名古屋市)より購入したキット(商品名:CoralHue Fluo-chase kit, Code No. AM-1100)中の、phmKGN-MNおよびphmKGC-MNを使用した。このキットに用いられている蛍光タンパク質は、サンゴ由来のKusabira-Greenの単量体(mKG)である。
【0057】
Cdc25B及びβ-TrCP1発現プラスミドは、Invitrogen社のpEF6-Myc/Hisプラスミド(Cat.No. V962-20)を基本とし、本発明者らの研究室で作成した。Cdc25BはN-末端にFLAGタグがタンデムに2個付加してあり、またβ-TrCPはN-末端側にあるF-boxと呼ばれる領域を含む211アミノ酸を欠き、その断片のN-末端側にMycタグ配列がやはり2個タンデムに付加されている(F-boxとよばれる領域を欠損させると、基質(Cdc25B)との結合は通常に起きるが、基質の分解に必要なユビキチン化反応が起きなくなるため、Cdc25Bとβ-TrCP1が安定な複合体として検出できることが期待される)(図6〜9)。
【0058】
1.2.Cdc25Bおよびβ-TrCP1断片とmKGタンパク質断片の融合プラスミドの作成
Cdc25Bの2xFLAG配列を含むN-末端側1-175アミノ酸を含む断片をPCRにより増幅し、適当な制限酵素で切断後、同様の制限酵素で切断されたphmKGN-MNプラスミドへ導入した。これにより、FLAG配列を含むCdc25B (N1-175)断片のC-末端側にmKGのN-末端断片(mKGN:168アミノ酸)が融合されたタンパク質の発現されることが予想される(図8プラスミド3)。また、対照としてストレス非依存的にβ-TrCP1との結合が起きる領域を含む1-280アミノ酸断片も、175アミノ酸断片と同様にFLAG配列を含んだ形でmKGNとの融合タンパク質として発現されるようなプラスミドを作成した(図8プラスミド4)。
【0059】
2xMyc配列が融合したF-box欠失型β-TrCP1の場合も同様に、PCRによりアミノ酸をコードする領域全長を増幅し、適当な制限酵素で切断後、やはり同様の制限酵素で切断されたphmKGC-MNへ導入された。これにより、Myc配列を含むβ-TrCP1断片のC-末端側にmKGのC-末端断片(mKGC:50アミノ酸)が融合されたタンパク質が発現されることが予想される(図9プラスミド7)。
【0060】
1.3.2xFLAG-Cdc25B-mKGNと2xMyc-β-TrCP1-mKGCの融合タンパク質発現プラスミドの作成
より効率的なCdc25Bとβ-TrCP1断片の相互作用とそれに続くmKGNとmKGCの再会合を促進するために、それぞれの断片を融合し、1本のポリペプチド(タンパク質)として細胞内で発現させることを計画し、以下の手順によりプラスミドを作成した。
【0061】
まず、2xFLAG-Cdc25B-mKGN断片をPCRにより増幅し、適当な制限酵素で切断後pEF6プラスミドに導入した。さらに、2xFLAG-Cdc25B-mKGN断片が挿入されたpEF6プラスミドに、2xFLAG-Cdc25B-mKGNの下流に位置するように、また、タンパク質としての読み枠が一致するように、PCRにより増幅された2xMyc-β-TrCP1-mKGC断片を挿入した。この結果、細胞内では2xFLAG-Cdc25B-mKGN と2xMyc-β-TrCP1-mKGCの融合タンパク質の発現が予測される。なお、二つのペプチド間には、約15アミノ酸からなるリンカーが存在し、両ペプチドの立体的な動きが阻害されないことが期待される構造となっている(図10プラスミド8)。
【0062】
また、mKGN-Cdc25B(1-175)-2xFLAGというかたちのペプチドを発現させるために、以下のようなプラスミドの作成も行った。まず、mKGN断片をPCRにより増幅し、pEF6プラスミドに導入した。このpEF6プラスミドは、導入断片から発現されるペプチドのC-末端側にFLAG配列が付加されるように遺伝子を組み換えることが可能である。さらに、mKGNと2xFLAGの間に、タンパク質としての読み枠が一致するようにCdc25BのN-末端側175アミノ酸が含まれるDNA断片をPCRにより増幅したのち、制限酵素処理により導入した。このプラスミドからは、mKGN-Cdc25B(1-175)-2xFLAGというペプチドが発現されることが期待される(図8プラスミド5)。
さらに、mKGN-Cdc25B(1-175)-2xFLAG断片と2xMyc-β-TrCP1-mKGC断片の融合タンパク質が発現されるように、上述の方法によりプラスミドを作成した(図10プラスミド9)。
【0063】
2.融合タンパク質の作成手順詳細
上記1において概要を示した融合タンパク質の作成手順について、具体的な配列を参照して詳細に説明する。
【0064】
2.1.Cdc25B(図6)について
ヒトCdc25B1の塩基配列(配列番号1)およびアミノ酸配列(配列番号2)を図11に示す。本実施例では、「Cdc25B」とはヒトCdc25B1を指す。Cdc25Bの第94〜108番のアミノ酸配列(図11一重下線部)が、参考例に示すとおり本発明者らにより新たに特定された、ストレスに依存して活性化されたJNK1によりリン酸化を受けβ-TrCP1との結合に関与するコア配列である。Cdc25Bの第254〜259番のアミノ酸配列(図11二重下線部)が、従来から知られていた、ストレスに関わりなくβ-TrCP1と結合する配列のコア部分である。
【0065】
pEF6/Myc-His Bプラスミド(Invitrogen社製)へ挿入されたCdc25BのcDNAのN-末端側にはFLAGと呼ばれる配列が2個タンデムに結合されている(図6のプラスミド1)。その上流域部分の塩基配列(配列番号3)およびコードされるアミノ酸配列(配列番号4)を図12に示す。
【0066】
2.2.Cdc25B/175NおよびCdc25B/280N(図7)について
2xFLAG-Cdc25B/N175と2xFLAG-Cdc25B/N280は、以下のプライマーを使用し、図6中のプラスミド1を鋳型にしてPCRで必要な断片を増幅後、制限酵素BamHIとXhoIで切断し、2xFLAG-Cdc25B/N175(図7中の断片1)および2xFLAG-Cdc25B/N280(図7中の断片2)を作成した。
【0067】
【表1】

【0068】
Cdc25B/N175-2xFLAGは以下のプライマーを使用し、以下に述べる手順で作成した。
断片3は最終的にCdc25B/N175のC-末端側にFLAG配列が2個タンデムに結合された形の断片4を作成するための中間段階のものとして作成した。使用したプライマーセットは以下のものである。このプライマーセットを使用し、図6中のプラスミド1を鋳型にしてPCR増幅を行い、断片3を得た。
【0069】
【表2】

【0070】
図7中のプラスミド2はInvitrogen社製のpEF6/Myc-His BプラスミドのXbaI-PmeI制限酵素部位に、FLAG配列が2個タンデムに結合したペプチドをコードする図13に示すオリゴヌクレオチドが挿入された改変プラスミド、pEF6-2FLAG/CのEcoRI-XbaI部位に、図7中のPCR増幅断片3を制限酵素EcoRIとXbaIで消化したものを挿入して作成した。
2xFLAG/C断片の塩基配列(配列番号10)およびコードされるアミノ酸配列(配列番号11)を図13に示す。
【0071】
上記手順により得られた図7に示す断片1、2および4の塩基配列(配列番号12、14および16)ならびにコードされるアミノ酸配列(配列番号13、15および17)をそれぞれ図14、15および16に示す。
【0072】
2.3.Cdc25B断片とmKGNの融合タンパク質発現プラスミド(図8)について
2.3.1.2xFLAG-Cdc25B/N175-mKGNおよび2xFLAG-Cdc25B/N280-mKGN発現プラスミド(プラスミド3および4)の作成
MBL社製のphmKGN-MNプラスミドを、図7中の断片1及び2を作成した制限酵素と同じ制限酵素セットで切断し、それぞれ2xFLAG-Cdc25B/N175と2xFLAG-Cdc25B/N280の下流(C末端側)にmKGN断片が融合されたタンパク質を発現する、図8中のプラスミド3及びプラスミド4を作成した。すなわち、図7中の断片1および断片2をBamHI-XhoIのセットで切断し、同様にBamHI-XhoIのセットで切断されたphmKGN-MNプラスミドへ導入し、プラスミド3および4を作成した。
【0073】
プラスミド3中で融合タンパク質2xFLAG-Cdc25B/N175-mKGNをコードする部分の塩基配列(配列番号22)およびコードされるアミノ酸配列(配列番号23)を図18に示す。
【0074】
プラスミド4中で融合タンパク質2xFLAG-Cdc25B/N280-mKGNをコードする部分の塩基配列(配列番号24)およびコードされるアミノ酸配列(配列番号25)を図19に示す。
【0075】
2.3.2.mKGN-Cdc25B/N175-2xFLAG発現プラスミド(プラスミド5)の作成
図8中のプラスミド5は以下の手順で作成した。
(i) phmKGN-MNプラスミドを鋳型にして、表3に示すプライマーセットを用いてPCRによりmKGN断片を増幅した。
(ii) 増幅断片を制限酵素Acc65I-BamHIで切断した。認識配列はそれぞれGGTACCとGGATCCである。
(iii) プラスミド2をAcc65IとBamHIで切断し、(ii)の制限酵素消化断片を導入した。
【0076】
【表3】

【0077】
このようにした作成されたプラスミドのBamHI-EcoRI部位間に図17に示す二本鎖塩基配列(センス鎖を配列番号20とする)のオリゴヌクレオチドを挿入し(図17ではBamHIとEcoRI間に挿入された状態の配列が示されている)、mKGN断片とCdc25B/N175-2xFLAG間にリンカーとなるアミノ酸配列(配列番号21)を導入して、最終的にプラスミド5が作られた。
【0078】
プラスミド5中で融合タンパク質mKGN-Cdc25B/N175-2xFLAGをコードする部分の塩基配列(配列番号26)およびコードされるアミノ酸配列(配列番号27)を図20に示す。
【0079】
2.4.β-TrCP1の全配列について
ヒトβ-TrCP1の塩基配列(配列番号28)およびアミノ酸配列(配列番号29)を図21に示す。以下、本実施例ではβ-TrCP1とはヒトβ-TrCP1を指す。
【0080】
pEF6/Myc-His Bプラスミド(Invitrogen社製)へ挿入されたβ-TrCP1のcDNAのN-末端側にはMycと呼ばれる配列が2個タンデムに結合されている。その上流域部分の塩基配列(配列番号30)およびコードされるアミノ酸配列(配列番号31)を含む配列を図22に示す。
【0081】
2.5.β-TrCP1/Δ211(図9プラスミド6)の作成
β-TrCP1のアミノ酸配列(配列番号29)における第146〜192番の領域は、F-boxと呼ばれる、タンパク質のユビキチン化コンポーネントとの複合体形成に必要な部分である。F-boxを含む、配列番号29の第1〜211番の領域を欠くβ-TrCP1タンパク質断片(β-TrCP1/Δ211)は基質(この場合はCdc25B)との結合はできるが、ユビキチン化をすることができない。このため、β-TrCP1/Δ211を用いれば相互作用されたCdc25Bは分解されずに、反応系中に留まり続けることができる。
【0082】
全長β-TrCP1が図22に示すように導入されたpEF6/Myc-His Bプラスミド(Invitrogen社製)を鋳型とし、以下のプライマーを使用してPCRで断片を増幅後、制限酵素BamHIとEcoRVで消化した。消化された断片を、pEF6/Myc-His Bプラスミド(Invitrogen社製)のBamHI-EcoRV部位に挿入し、Myc配列をN-末端側に2個タンデムに付加しpEF-2xMyc-β-TrCP1/Δ211(図9のプラスミド6)を作成した。
【0083】
【表4】

【0084】
pEF6/Myc-His Bプラスミド(Invitrogen社製)へ挿入されたβ-TrCP1/Δ211のcDNAのN-末端側にはMycと呼ばれる配列が2個タンデムに結合されている。その上流域部分の塩基配列(配列番号34)およびコードされるアミノ酸配列(配列番号35)を含む配列を図23に示す。またプラスミド6におけるβ-TrCP1/Δ211のcDNAの下流部分の塩基配列(配列番号36)およびコードされるアミノ酸配列(配列番号37)を含む配列を図24に示す。
【0085】
2.6.β-TrCP1/Δ211とmKGCの融合タンパク質発現プラスミド(プラスミド7)について
2xMyc-β-TrCP1/Δ211は、表5のプライマーを使用し、図9中のプラスミド6を鋳型にしてPCRで必要な断片を増幅後、制限酵素XhoI-NotIで切断した。その断片を、医学生物学研究所(MBL)より購入したphmKGC-MNプラスミドのXhoI-NotI部位に挿入し、2xMyc-β-TrCP1/Δ211-mKGC(図9中のプラスミド7)を作成した。
【0086】
【表5】

【0087】
プラスミド7中での、タンパク質コード配列(およびその周辺の配列)を図25(核酸配列は配列番号40、コードされるアミノ酸配列は配列番号41)に示す。
【0088】
2.7.2xFLAG-Cdc25B/N175-mKGNと2xMyc-β-TrCP1/Δ211-mKGCの融合タンパク質発現プラスミド(プラスミド8)の作成
2.7.1.2xFLAG-Cdc25B/N175-mKGNのPCR増幅とプラスミドへの導入
図8中のプラスミド3を鋳型にして、表6に示すプライマーセットで2xFLAG-Cdc25B/N175-mKGN断片をPCRにより増幅した。得られた増幅断片を、BamHIとEcoRVで消化・切断した。この増幅断片は、下流のリンカー並びに2xMyc-β-TrCP1/Δ211-mKGCとの融合タンパク質として発現されることとなるため、停止コドンが除かれるようにreverse プライマーが設計されている。
【0089】
【表6】

【0090】
得られた増幅断片をBamHIとEcoRVで消化切断したものの塩基配列(配列番号44)およびコードされるアミノ酸配列(配列番号45)を図26に示す。
【0091】
BamHIとEcoRVで消化切断後の得られた増幅断片を、リンカー導入pEF6プラスミドのBamHI-EcoRV部位(図30)へ導入した。リンカー導入pEF6プラスミドについては、下記2.9において詳述する。
【0092】
2.7.2.2xMyc-β-TrCP1/Δ211-mKGCのPCR増幅とプラスミドへの導入
図9中のプラスミド7を鋳型にして、表7に示すプライマーセットで2xMyc-β-TrCP1/Δ211-mKGC断片を増幅した。
【0093】
【表7】

【0094】
得られた増幅断片をXbaIIとPmeIで消化切断したものの塩基配列(配列番号48)およびコードされるアミノ酸配列(配列番号49)を図27に示す。アミノ酸の読み枠の関係で、XbaI部位の上流に2ヌクレオチドを余分に付加した。
【0095】
XbaIIとPmeIによる消化切断後の増幅断片を、上述の2xFLAG-Cdc25B/N175-mKGN断片を有するpEF6プラスミドのリンカーの下流にあるXbaI-PmeI部位(図30)へ導入して、図10に示すプラスミド8を得た。
【0096】
2.8.mKGN-Cdc25B/N175-2xFLAGと2xMyc-β-TrCP1/Δ211-mKGCの融合タンパク質発現プラスミド(プラスミド9)の作成
導入する遺伝子及びリンカー導入pEF6プラスミド中の制限酵素部位の関係から、本プラスミドの作成は、先に2xMyc-β-TrCP1/Δ211-mKGCをプラスミドに組み込み、得られたプラスミド中のリンカーの上流のEcoRV部位(図30)へmKGN-Cdc25B/N175-2xFLAG断片を導入した。プラスミド8の場合と同様にmKGN-Cdc25B/N175-2xFLAGの増幅断片からは、停止コドンが除かれるようにreverse プライマーが設計されている。
【0097】
2.8.1.2xMyc-β-TrCP1/Δ212-mKGCのPCR増幅とプラスミドへの導入
図9中のプラスミド7を鋳型にして、プラスミド8の作成時に使用したのと同じ表7に示すプライマーセットを用いて2xMyc-β-TrCP1/Δ211-mKGC断片を増幅した。得られた増幅断片をXbaIとPmeIで消化・切断して、図27に示すのと同じ塩基配列(配列番号48)を有する断片を得た。当該断片のリンカー導入pEF6プラスミドのリンカーの下流にあるXbaI-PmeI部位(図30)へ導入した。
【0098】
2.8.2.mKGN-Cdc25B/N175-2xFLAGのPCR増幅とプラスミドへの導入
上記2.3.2で得られたプラスミド5(図8)を鋳型にして、表8に示すプライマーを使用してPCRにより増幅を行って、mKGN-Cdc25B/N175-2xFLAGをコードするDNA断片を得た。
【0099】
【表8】

【0100】
得られた増幅断片をEcoRVで消化切断したものの塩基配列(配列番号52)およびコードされるアミノ酸配列(配列番号53)を図28に示す。
【0101】
EcoRVによる切断後のmKGN-Cdc25B/N175-2xFLAGのDNA断片を、上記2.8.1で得られた、2xMyc-β-TrCP1/Δ211-mKGCが導入されたリンカー導入pEF6プラスミドのリンカーの上流のEcoRV部位(図30)へ導入した。こうして図10に示すプラスミド9を作成した。
【0102】
2.9.リンカー導入pEF6プラスミド
上記2.7および2.8に記載の融合タンパク質の製造において使用したリンカー導入pEF6プラスミドは、pEF6/Myc-His Bプラスミド(Invitrogen社より購入)の制限酵素EcoRIおよびNotI切断部位に、図29(1)に示す二本鎖オリゴヌクレオチド(センス鎖:配列番号54)を挿入することにより作成した。導入されたオリゴヌクレオチドは、図29(2)に示すアミノ酸配列(配列番号55)のポリペプチドをコードする。当該ポリペプチドのうち、第5〜20番の部分がmKGN融合Cdc25B断片とmKGC融合β-TrCP1断片との間を連結するリンカーとして機能すると考えられる。
得られたリンカー導入pEF6プラスミドの、リンカーコード配列の近傍の構造を図30に示す。
【0103】
3.細胞への導入とタンパク質の検出
プラスミドの細胞への導入は、Invitrogen社のLipofectamine2000試薬を用いて行った。導入したプラスミドの組み合わせは以下の通りである(図10)。
1)2xFLAG-Cdc25B(1-175)-mKGN + 2xMyc-β-TrCP1-mKGC(プラスミド3と7)
2)2xFLAG-Cdc25B(1-175)-mKGN-2xMyc-β-TrCP1-mKGC(プラスミド8)
3)mKGN-Cdc25B(1-175)-2xFLAG + 2xMyc-β-TrCP1-mKGC(プラスミド5と7)
4)mKGN-Cdc25B(1-175)-2xFLAG-2xMyc-β-TrCP1-mKGC(プラスミド9)
5)2xFLAG-Cdc25B(1-280)-mKGN + 2xMyc-β-TrCP1-mKGC(プラスミド4と7)
JNK1キナーゼの作用によってCdc25Bとβ-TrCP1の結合が促進されて蛍光が発光することを確認することを目的として、JNK1キナーゼおよびその活性化キナーゼであるMKK7をコードする遺伝子が組み込まれたプラスミドを1)〜4)の組み合わせとともに細胞(HeLa細胞またはCos 7細胞)へ導入した。24時間後に細胞をメタノールで固定し、蛍光顕微鏡により緑色蛍光を観察した。また、比較のために、活性が欠損しているJNK1キナーゼ遺伝子が組み込まれたプラスミドを用いて同様の操作を行った。
【0104】
4.薬剤処理による応答の解析
一過的にプラスミドを導入したHeLa細胞を薬剤(アニソマイシン:50ng/mL、食塩:培養液中の全食塩濃度として250mM、ヒドロキシ尿素:1.5mM、アフィディコリン2.5 μg/ml、エトポシド:2mM)で24時間処理し、細胞をメタノール固定し蛍光顕微鏡により緑色蛍光を観察した。
【0105】
結果
1.蛍光検出に適したプラスミドの選択
図10に記したプラスミドの組み合わせのうちで、JNK1を発現させない場合のノイズが低くかつ、JNK1を発現させた場合のシグナルが高いものを選択した。その結果、4)のタンパク質、即ちmKGN-Cdc25B/N175-2xFLAG-2xMyc-β-TrCP1/Δ211-mKGCを発現するプラスミド(プラスミド9)を使用した場合において高いS/N比が得られた。下の表は、各プラスミドの組み合わせでJNK1に対する応答実験を行った場合の定性的な結果を示す。
【0106】
【表9】

【0107】
図31には、プラスミド9(図10の4))を導入したHeLa細胞およびCos7細胞では、JNK1遺伝子が導入された場合に蛍光が発光したことを示す。なお、写真には示していないが、他のプラスミドを導入した場合にもJNK1遺伝子が導入された細胞では蛍光が確認された。
【0108】
さらに、キナーゼ活性に関して野生型(正常型:図32ではWTと表記)および欠損型(図32ではKDと表記)のJNK1を用いた実験により、緑色蛍光の出現がJNK1のキナーゼ活性に依存していることが明らかとなった(図32では緑色蛍光を緑色で、細胞中で発現しているJNK1タンパク質を赤色で示している。二つの図を重ね合わせると、緑色蛍光を発しておりかつ、JNK1タンパク質が発現されている細胞は、緑+赤で黄色となる(JNK1-KD発現細胞では緑色蛍光が発せられないため、JNK1の発現を示す赤色のみとなる。)。以降の実験は、このタンパク質を発現するプラスミド(プラスミド9)を使用して行った。ただし、表9に示すいずれの組み合わせにおいても明らかにシグナルがノイズよりも高いという結果が得られている。また、JNK非依存的な結合部位を含む5)の組み合わせ、すなわち、Cdc25Bの280アミノ酸断片においても、JNK依存的なシグナルの増強が見られた。
【0109】
2.薬剤処理
DNAに損傷を起こさない薬剤としてアニソマイシン、食塩(胃がんの発がん促進因子)、DNA複製を阻害し鎖切断を誘発するヒドロキシ尿素、アフィディコリン、DNAに二本鎖切断を誘導するエトポシド(抗がん性化学物質)を用いて、プラスミドの有用性を検定した。
【0110】
実験ではプラスミド9を用いた。プラスミド9をHeLa細胞へ導入し、それから6時間後に薬剤を加えた。その結果、薬剤処理18時間後において、すべての例でシグナルの増強が認められた。図33には、アニソマイシンまたは食塩を処理した細胞の結果を示す。図33では、薬剤処理した細胞、特に食塩処理細胞にアポトーシスと思われる細胞死が誘導されたため、細胞数が少なくなっているが、明らかに黄色を示す細胞が出現している。この図においては、発現タンパク質に付加されたFLAG配列を認識する抗体を用いてプラスミドが導入された細胞が示されている。すなわち、タンパク質が発現された細胞が赤色で、そのうちストレス応答によりBiFCが起きた細胞が緑で示されている。よって、両者が一致する細胞は黄色で示される。
【0111】
ヒドロキシ尿素やエトポシドなどのDNA損傷誘発試薬は、短時間処理ではDNA損傷ストレスが主である(図1参照)。しかしながら、図34に示されるように、これらの薬剤を6時間より長時間にわたって連続的に処理したところJNK1活性による蛍光発光が確認された。図34の結果は、DNA損傷ストレスとともにそれとは異なる非遺伝毒性のストレスが細胞にかかり、その結果Cdc25Bの分解を誘発するJNKやp38と呼ばれるストレス応答性キナーゼ群が活性化されたことを反映していると考えられる。
【図面の簡単な説明】
【0112】
【図1】図1は、非遺伝毒性ストレスによりCdc25Bが分解されることを示す。
【図2】図2は、Cdc25Bが、β-TrCP1または2によりJNK1依存的にユビキチン化されることを示す。また、F-boxを欠くβ-TrCP1にはユビキチン可能がないことを示す。
【図3】図3は、Cdc25BのSer101を変異させるとCdc25BはJNKによる分解に対して抵抗性になることを示す。
【図4】図4は、JNK/β-TrCPを介したCdc25Bの分解の機構を模式的に示す。
【図5】図5は、BiFC法によりJNK1依存性のCdc25Bとβ-TrCPとの相互作用を判別する方法の典型的な例を示す。
【図6】図6は、Cdc25B の特性とCdc25B発現プラスミドを示す。
【図7】図7は、PCRによるCdc25B 断片の増幅を示す。
【図8】図8は、Cdc25B 断片とmKGNとの融合タンパク質発現プラスミドを示す。
【図9】図9は、b-TrCP1/D211及び b-TrCP1 /D211 断片とmKGC との融合タンパク質発現プラスミドを示す。
【図10】図10は、検定に用いたプラスミドを示す。
【図11−1】図11は、Cdc25Bの塩基配列(配列番号1)およびアミノ酸配列(配列番号2)を示す。
【図11−2】図11は、Cdc25Bの塩基配列(配列番号1)およびアミノ酸配列(配列番号2)を示す。
【図11−3】図11は、Cdc25Bの塩基配列(配列番号1)およびアミノ酸配列(配列番号2)を示す。
【図12】図12は、プラスミド1の部分配列を示す。
【図13】図13は、2xFLAG/C断片の塩基配列(配列番号10)およびコードされるアミノ酸配列(配列番号11)を示す。
【図14】図14は断片1の塩基配列およびアミノ酸配列を示す。
【図15−1】図15は断片2の塩基配列およびアミノ酸配列を示す。
【図15−2】図15は断片2の塩基配列およびアミノ酸配列を示す。
【図16】図16は断片4の塩基配列およびアミノ酸配列を示す。
【図17】図17は、mKGN断片とCdc25B/N175-2xFLAG間のリンカー用オリゴヌクレオチド配列を示す。
【図18−1】図18は、プラスミド3中で融合タンパク質2xFLAG-Cdc25B/N175-mKGNをコードする部分の塩基配列(配列番号22)およびコードされるアミノ酸配列(配列番号23)を示す。
【図18−2】図18は、プラスミド3中で融合タンパク質2xFLAG-Cdc25B/N175-mKGNをコードする部分の塩基配列(配列番号22)およびコードされるアミノ酸配列(配列番号23)を示す。
【図19−1】図19は、プラスミド4中で融合タンパク質2xFLAG-Cdc25B/N280-mKGNをコードする部分の塩基配列(配列番号24)およびコードされるアミノ酸配列(配列番号25)を示す。
【図19−2】図19は、プラスミド4中で融合タンパク質2xFLAG-Cdc25B/N280-mKGNをコードする部分の塩基配列(配列番号24)およびコードされるアミノ酸配列(配列番号25)を示す。
【図19−3】図19は、プラスミド4中で融合タンパク質2xFLAG-Cdc25B/N280-mKGNをコードする部分の塩基配列(配列番号24)およびコードされるアミノ酸配列(配列番号25)を示す。
【図20−1】図20は、プラスミド5中で融合タンパク質mKGN-Cdc25B/N175-2xFLAGをコードする部分の塩基配列(配列番号26)およびコードされるアミノ酸配列(配列番号27)を示す。
【図20−2】図20は、プラスミド5中で融合タンパク質mKGN-Cdc25B/N175-2xFLAGをコードする部分の塩基配列(配列番号26)およびコードされるアミノ酸配列(配列番号27)を示す。
【図21−1】図21は、β-TrCP1の塩基配列(配列番号28)およびアミノ酸配列(配列番号29)を示す。
【図21−2】図21は、β-TrCP1の塩基配列(配列番号28)およびアミノ酸配列(配列番号29)を示す。
【図21−3】図21は、β-TrCP1の塩基配列(配列番号28)およびアミノ酸配列(配列番号29)を示す。
【図22】図22は、β-TrCP1が導入されたpEF6/Myc-His Bプラスミドの部分配列を示す。
【図23】図23は、β-TrCP1/Δ211が導入されたpEF6/Myc-His Bプラスミドの部分配列を示す。
【図24】図24は、プラスミド6におけるβ-TrCP1/Δ211のcDNAの下流部分の塩基配列(配列番号36)およびコードされるアミノ酸配列(配列番号37)を含む配列を示す。
【図25−1】図25は、プラスミド7中での、タンパク質コード配列(およびその周辺の配列)を示す。
【図25−2】図25は、プラスミド7中での、タンパク質コード配列(およびその周辺の配列)を示す。
【図25−3】図25は、プラスミド7中での、タンパク質コード配列(およびその周辺の配列)を示す。
【図26−1】図26は、2xFLAG-Cdc25B/N175-mKGNの増幅断片をBamHIとEcoRVで消化・切断したものを示す。
【図26−2】図26は、2xFLAG-Cdc25B/N175-mKGNの増幅断片をBamHIとEcoRVで消化・切断したものを示す。
【図27−1】図27は、2xMyc-β-TrCP1/Δ211-mKGCの増幅断片をXbaIIとPmeIで消化・切断したものを示す。
【図27−2】図27は、2xMyc-β-TrCP1/Δ211-mKGCの増幅断片をXbaIIとPmeIで消化・切断したものを示す。
【図27−3】図27は、2xMyc-β-TrCP1/Δ211-mKGCの増幅断片をXbaIIとPmeIで消化・切断したものを示す。
【図28−1】図28は、mKGN-Cdc25B/N175-2xFLAGの増幅断片をEcoRVで消化・切断したものを示す。
【図28−2】図28は、mKGN-Cdc25B/N175-2xFLAGの増幅断片をEcoRVで消化・切断したものを示す。
【図29】図29は、リンカー導入pEF6プラスミドに導入されるリンカー部分の塩基配列およびアミノ酸配列を示す。
【図30】図30は、リンカー導入pEF6プラスミドの、リンカーコード配列の近傍の構造を示す。
【図31】図31は、JNK1依存的に緑色蛍光が出現したことを示す。
【図32】図32は、JNK1依存的に緑色蛍光が出現したことを示す。
【図33】図33は、非遺伝毒性ストレスに応答して緑色蛍光が出現したことを示す。
【図34】図33は、遺伝毒性ストレス因子を加えた場合であっても長時間経過後には緑色蛍光が出現したことを示す。
【配列表フリーテキスト】
【0113】
配列番号3:プラスミド1のFLAG領域
配列番号4:プラスミド1のFLAG領域
配列番号5:プライマー
配列番号6:プライマー
配列番号7:プライマー
配列番号8:プライマー
配列番号9:プライマー
配列番号10:2xFLAG/C断片
配列番号11:2xFLAG/C断片
配列番号12:断片1
配列番号13:断片1
配列番号14:断片2
配列番号15:断片2
配列番号16:断片4
配列番号17:断片4
配列番号18:プライマー
配列番号19:プライマー
配列番号20:mKGN断片とCdc25B/N175-2xFLAG間のリンカー
配列番号21:mKGN断片とCdc25B/N175-2xFLAG間のリンカー
配列番号22:プラスミド3中で2xFLAG-Cdc25B/N175-mKGNをコードする部分
配列番号23:プラスミド3中で2xFLAG-Cdc25B/N175-mKGNをコードする部分
配列番号24:プラスミド4中で2xFLAG-Cdc25B/N280-mKGNをコードする部分
配列番号25:プラスミド4中で2xFLAG-Cdc25B/N280-mKGNをコードする部分
配列番号26:プラスミド5中でmKGN-Cdc25B/N175-2xFLAGをコードする部分
配列番号27:プラスミド5中でmKGN-Cdc25B/N175-2xFLAGをコードする部分
配列番号30:β-TrCP1 が挿入されたpEF6/Myc-His Bプラスミドのβ-TrCP1上流部
配列番号31:β-TrCP1 が挿入されたpEF6/Myc-His Bプラスミドのβ-TrCP1上流部
配列番号32:プライマー
配列番号33:プライマー
配列番号34:プラスミド6のβ-TrCP1/Δ211上流部
配列番号35:プラスミド6のβ-TrCP1/Δ211上流部
配列番号36:プラスミド6のβ-TrCP1/Δ211下流部
配列番号37:プラスミド6のβ-TrCP1/Δ211下流部
配列番号38:プライマー
配列番号39:プライマー
配列番号40:プラスミド7中の2xMyc-β-TrCP1/Δ211-mKGC
配列番号41:プラスミド7中の2xMyc-β-TrCP1/Δ211-mKGC
配列番号42:プライマー
配列番号43:プライマー
配列番号44:2xFLAG-Cdc25B/N175-mKGN
配列番号45:2xFLAG-Cdc25B/N175-mKGN
配列番号46:プライマー
配列番号47:プライマー
配列番号48:2xMyc-β-TrCP1/Δ211-mKGC
配列番号49:2xMyc-β-TrCP1/Δ211-mKGC
配列番号50:プライマー
配列番号51:プライマー
配列番号52:mKGN-Cdc25B/N175-2xFLAG
配列番号53:mKGN-Cdc25B/N175-2xFLAG
配列番号54:リンカーを導入するためのオリゴヌクレオチド
配列番号55:リンカー
配列番号56:リンカー導入pEF6プラスミドのリンカーコード領域
配列番号57:リンカー導入pEF6プラスミドのリンカーコード領域
【技術分野】
【0001】
本発明は、被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する方法に関する。
【背景技術】
【0002】
非遺伝毒性ストレスを受けた細胞では、JNK1キナーゼやp38キナーゼが誘導されることが知られている。しかしながら従来は、JNK1キナーゼやp38キナーゼが誘導されているか否かを簡便に測定する方法がなかった。また、非遺伝毒性ストレスを与える毒性物質をスクリーニングする方法としては、従来は毒性物質を動物に投与することが行われており、手間と時間がかかるという問題があった。
【0003】
Cdc25Bが非遺伝毒性ストレスにより活性化されるJNK1キナーゼやp38キナーゼによりリン酸化を受け分解されるということは知られていなかった。すなわち、非遺伝毒性ストレスを与える毒性物質が処理された細胞では、Cdc25Bが分解されるということは知られていなかった。
【0004】
一方、Cdc25Bはガン細胞において発現が促進されることが知られている。Cdc25Bを細胞内で減少させる物質は抗ガン剤として有用であると考えられるが、Cdc25Bを減少させる作用を有する物質をスクリーニングする手段は従来存在しなかった。
【0005】
また、Cdc25Bとβ-TrCP1または2とが相互作用することによりCdc25Bがユビキチン化され、Cdc25Bの分解に至ることが知られていた。しかしながら従来知られていた相互作用は、JNK1キナーゼやp38キナーゼの活性に依存しない作用であった。
【発明の開示】
【発明が解決しようとする課題】
【0006】
本発明は、非遺伝毒性ストレスを与える毒性物質などの、細胞内でJNK1キナーゼやp38キナーゼなどの活性化を誘導する物質をスクリーニングするための簡便な手段を提供することを目的とする。
【0007】
本発明はまた、Cdc25BまたはCdc25Aの細胞内での分解を促進する物質をスクリーニングするための簡便な手段を提供することを目的とする。
【0008】
本発明はより一般的には、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を有する物質をスクリーニングするための簡便な手段を提供することを目的とする。
【課題を解決するための手段】
【0009】
本発明者らは、Cdc25Bとβ-TrCPとがJNK1キナーゼが誘導される条件下(非遺伝毒性ストレス条件下)においてJNK1キナーゼ活性に依存して相互作用するという驚くべき知見を得た。更に、JNK1キナーゼ活性に依存してCdc25Bとβ-TrCPとが結合する際には、Cdc25Bの特定の領域(従来知られていた、JNK1キナーゼやp38キナーゼの活性に依存しない結合に関与する箇所とは異なる)が関与することを見出した。これらのことから、Cdc25Bとβ-TrCPとの相互作用の有無によって、JNK1が誘導されているか否か(非遺伝毒性ストレスが負荷されているか否か)を判別することが可能となることに想到するに至った。
【0010】
本発明者らはさらに、緑色蛍光タンパク質のN末端側断片とC末端側断片とをそれぞれCdc25Bとβ-TrCPのどちらかに融合させた2種類の融合タンパク質が、Cdc25Bとβ-TrCPとの相互作用に応じて蛍光を発することを見出した。
【0011】
以上の新規知見に基づいて、本発明者らは以下の発明を完成させるに至った。
(1) 被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する方法であって、
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質とを同一の細胞内で発現させる発現工程と、
発現工程後の細胞に被験物質を作用させる作用工程と、
作用工程後の前記細胞において前記信号を測定し、測定された前記信号を指標にして、前記被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する評価工程とを含む前記方法。
(2) 前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(1)の方法。
(3) 前記因子がJNK1キナーゼ、JNK2キナーゼ、p38キナーゼ、ERK1、ERK2またはCDKキナーゼである、(2)の方法。
(4) Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、(2)または(3)の方法。
(5) 前記評価工程において前記因子を誘導する能力を有すると評価された被験物質を、遺伝毒性ストレス原因物質、非遺伝毒性ストレス原因物質、遺伝毒性ストレス原因物質の働きを軽減する物質、非遺伝毒性ストレス原因物質の働きを軽減する物質、または抗癌物質の候補物質として選抜するスクリーニング工程を更に含む、(2)〜(4)のいずれかの方法。
(6) 前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(1) の方法。
(7) 前記因子がp38キナーゼ、CHK1キナーゼ、CHK2キナーゼまたはMK2キナーゼである、(6)の方法。
(8) 前記評価工程において前記因子を誘導すると評価された被験物質を、遺伝毒性ストレス原因物質、遺伝毒性ストレス原因物質の働きを軽減する物質、または抗癌物質の候補物質として選抜するスクリーニング工程を更に含む、(6)または(7)の方法。
(9) 前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、(1)〜(8)のいずれかの方法。
(10) β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、(9)の方法。
【0012】
(11) 前記発現工程において、前記第1融合タンパク質と前記第2融合タンパク質とが、互いに連結された四成分融合タンパク質として細胞内で発現される、(1)〜(10)のいずれかの方法。
(12) Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質をコードするDNAと、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質をコードするDNAとの組み合わせ。
(13) 前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(12)の組み合わせ。
(14) Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、(13) の組み合わせ。
(15) 前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(12)の組み合わせ。
(16) 前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、(12)〜(15)のいずれかの組み合わせ。
(17) β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、(16) の組み合わせ。
(18) Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質とが互いに連結された四成分融合タンパク質をコードするDNA。
(19) 前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(18)のDNA。
【0013】
(20) Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、(19)のDNA。
(21) 前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、(18)のDNA。
(22) 前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、(18)〜(21)のいずれかのDNA。
(23) β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、(22)のDNA。
(24) (12)〜(17)のいずれかにおいて定義される第1融合タンパク質をコードするDNAを含むベクターと、(12)〜(17)のいずれかにおいて定義される前記第2融合タンパク質をコードするDNAを含むベクターとの組み合わせ。
(25) (18)〜(23)のいずれかのDNAを含むベクター。
(26) (24)のベクターの組み合わせにより形質転換された形質転換細胞。
(27) (25)のベクターにより形質転換された形質転換細胞。
(28) (12)〜(23)のいずれかの組み合わせ、またはDNAが導入され(形質転換され)た細胞。
(29) (28) の細胞を体内に含む動物。
(30) (12)〜(17)のいずれかのDNAの組み合わせによりコードされる、第1融合タンパク質と第2融合タンパク質との組み合わせ。
(31) (18)〜(23)いずれかのDNAによりコードされる、四成分融合タンパク質。
【発明の効果】
【0014】
本発明によれば、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を有する物質をスクリーニングするための簡便な手段が提供される。このような物質は、遺伝毒性ストレス原因物質(毒性物質)、非遺伝毒性ストレス原因物質(毒性物質)または抗癌剤として有用である可能性がある。
【発明を実施するための最良の形態】
【0015】
1.第1融合タンパク質
1.1.Cdc25B
本発明に用いるCdc25Bの起源となる動物種は特に限定されない。更に、Cdc25Bとしての活性を有している限り、天然に存在するCdc25B遺伝子およびそれによりコードされるCdc25Bだけでなく、人工合成されたCdc25B遺伝子およびそれによりコードされるCdc25Bもまた、本発明に用いることができる。例えば、哺乳動物(例えばヒト、サル(例えばチンパンジー、カニクイザル)、ウシ、ウマ、マウス、ラット)、鳥類、両生類(例えばアフリカツメガエル)に由来するCdc25Bおよびそれに基づいて人工合成されたCdc25Bを使用することができ、ヒトを含む哺乳動物由来のCdc25Bおよびそれに基づいて人工合成されたCdc25Bが特に好ましい。ヒト由来のCdc25Bには、Cdc25B1、Cdc25B2およびCdc25B3の三種類が知られており、いずれも本発明に使用することができる。配列番号2にはヒト由来Cdc25B1のアミノ酸配列を、配列番号1にはそれをコードする塩基配列を示す。配列番号2のアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるCdc25Bに包含される。
【0016】
種々の動物種のCdc25Bの塩基配列およびはそれから推定されるアミノ酸配列は公知であり、cell division cycle 25 homolog B (S. pombe)、Cdc25B、CDC25B等の名称により各種データベースに登録されている。Cdc25Bの塩基配列の登録番号の具体例は以下に示される。ヒト(Homo sapiens: NM_021873、NM_021872、NM_004358、Z68092、BC006395、BC009953、BC051711、AY892373、M81934)、マウス(Mus musculus: NM_023117、NM_001111075、BC057568、BC002287)、ラット(Rattus norvegicus: D16237、NM_133572)、ウシ(Bos Taurus: XM_866334)、ウマ(Equus caballus: XM_001495948、XM_001495974)、カニクイザル(Macaca fascicularis: AB173014、AB060926)、チンパンジー(Pan troglodytes: XM_001162178、XM_001162284、XM_001162247、XM_001162578、XM_001162531、XM_001162364、XM_001162323、XM_001162134、XM_001162488、XM_001162029、XM_001162625、XM_001162402、XM_001162213、XM_001162666、XM_001162447、XM_514487、XM_001162751、XM_001162706)、アフリカツメガエル(NM_001127885、NM_001127884、AB363841、AB363840)、ニシツメガエル(BC166320、BC125767)。これらの塩基配列がコードするCdc25Bのアミノ酸配列からなるタンパク質だけでなく、これらの塩基配列がコードするCdc25Bのアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるCdc25Bに包含される。
【0017】
本発明では、Cdc25Bは少なくともその一部が使用されればよく、全体を用いる必要はない。Cdc25BのなかでもJNK1依存的にβ-TrCPと相互作用する領域(ヒトCdc25Bの場合は配列番号2の第94〜108番)を含む、全長400アミノ酸以下、特に300アミノ酸以下、さらには200アミノ酸以下の部分配列からなるタンパク質が好ましい。例えば、配列番号2の第1〜175番のアミノ酸配列からなるタンパク質や、配列番号2の第1〜280番のアミノ酸配列からなるタンパク質が好適に使用できる。
【0018】
また、ヒト以外の動物種起源のCdc25Bにおいても、配列番号2の第94〜108番に相当する領域を含む全長400アミノ酸以下、特に300アミノ酸以下、さらには200アミノ酸以下の部分アミノ酸配列からなるタンパク質を好適に使用することができる。ヒトCdc25B1の第94〜108番に相当する領域としては次のアミノ酸配列からなる部分配列が挙げられる:
ヒト(Homo sapiens):DAGLCMDSPSPMDPH(例えば、NM_021873: 108-122、NM_021872: 108-122、NM_004358: 94-108、Z68092: 108-122、BC006395: 108-122、BC009953: 108-122、BC051711: 108-122、 AY892373: 108-122、AY494082中のDAGLCMDSPSPMDPHで示される配列、AF036233中の配列から読み取られるアミノ酸配列中のDAGLCMDSPSPMDPHで示される配列、M81934: 94-108)
チンパンジー(Pan troglodytes):DAGLCMDSPSPMDPH(例えば、XM_001162134: 108-122、XM_001162488: 108-122、XM_001162625: 108-122、XM_001162402: 108-122、XM_001162213: 94-108、XM_001162666: 94-108、XM_001162447: 94-108、XM_514487: 108-122、XM_001162751: 108-122、XM_001162706: 108-122)
カニクイザル(Macaca fascicularis):DAGLCMDSPSPVDPQ(例えば、AB060926: 94-108)
ウシ(Bos Taurus):DAGLCMDSPSPMDPN(例えば、XM_866334: 101-115)
ウマ(Equus caballus):DAGLCMDSPSPMDPQ(例えば、XM_001495948: 91-105、XM_001495974: 101-115)
マウス(Mus musculus):DAGLCMDSPSPVDPQ(例えば、NM_023117: 106-120、NM_001111075: 106-120、BC057568: 106-120)
ラット(Rattus norvegicus):DAGLCMDSPSPMDPQ(例えば、D16237: 106-120、NM_133572: 106-120)
アフリカツメガエル(Xenopus laevis):DSGLCLDSPSPQELE(例えば、NM_001127885: 93-107、NM_001127884: 94-108、AB363841: 93-107、AB363840: 94-108)
ニシツメガエル(例えば、BC166320: 92-106)
【0019】
1.2.Cdc25A
本発明に用いるCdc25Aの起源となる動物種は特に限定されない。更に、Cdc25Aとしての活性を有している限り、天然に存在するCdc25A遺伝子およびそれによりコードされるCdc25Aだけでなく、人工合成されたCdc25A遺伝子およびそれによりコードされるCdc25Aもまた、本発明に用いることができる。例えば、哺乳動物(例えばヒト、サル(例えばチンパンジー、カニクイザル)、ウシ、ウマ、マウス、ラット)、鳥類(例えばニワトリ、ウズラ)、両生類(例えばアフリカツメガエル、ヒキガエル)、魚類(例えばゼブラフィッシュ)に由来のCdc25Aおよびそれに基づいて人工合成されたCdc25Aであれば特に限定されないが、ヒトを含む哺乳動物由来のCdc25Aおよびそれに基づいて人工合成されたCdc25Aが好ましい。
【0020】
種々の動物種のCdc25Aのアミノ酸配列および塩基配列は公知であり、cell division cycle protein 25A、CDC25AまたはCdc25A等の登録名により各種データベースに登録されている。Cdc25Aの塩基配列の登録番号の具体例は以下に示される。ヒト(Homo sapiens: NM_201567、NM_001789、DQ896168、CU675644、CU675643:、AK290554、BC007401、BC018642、AY889759、AY892219、AY137580、AF277722、M81933、DQ892919、DQ890753)、マウス(Mus musculus: U27323、BC046296、AK077991、AK077609、NM_007658)、ラット(Rattus norvegicus: NM_133571、D16236)、ウシ(NM_001101100、BC151493)、ウマ(Equus caballus: XM_001495192)、チンパンジー(Pan troglodytes: XM_001155610、XM_516433)、アフリカツメガエル(Xenopus laevis: D82960、AF090829、BC082625、BC103738、NM_001088487、NM_001096204)、ニシツメガエル(Xenopus tropicalis: BC135487、CR855457、NM_001045598)、ヒキガエル(Bufo gargarizans:AY491052)、ゼブラフィッシュ8Danio rerio: EU921896))。これらの塩基配列がコードするCdc25Aのアミノ酸配列からなるタンパク質だけでなく、これらの塩基配列がコードするこれらのCdc25Aのアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるCdc25Aに包含される。
【0021】
本発明では、Cdc25Aは少なくともその一部が使用されればよく、全体を用いる必要はない。Cdc25Aのなかでもβ-TrCPと相互作用する領域を含む、全長400アミノ酸以下、特に300アミノ酸以下、さらには200アミノ酸以下の部分配列が好ましい。たとえば、第1〜140番のアミノ酸配列からなるタンパク質や、第1〜240番のアミノ酸配列からなるタンパク質が好適に使用できる。
【0022】
1.3.リンカー
第1融合タンパク質では、Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結されている。第1検出用タンパク質については後述する。
【0023】
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質との間には適宜リンカーを介して連結されている。リンカー部分の構造は特に限定されないが、通常は、アミノ酸数が3〜20個程度であり、Cdc25AまたはCdc25Bと第一検出用タンパク質の立体的な動きが阻害されないようにグリシン残基に富む(例えばリンカーを構成するアミノ酸の半数以上がグリシンである)ことが望ましい。
【0024】
またリンカーのほかに、融合タンパク質を作成する際の必要に応じて導入される制限酵素認識配列によりコードされるアミノ酸配列などが、Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質との間に存在していてもよい。
【0025】
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とはどちらがN末端側に位置しても構わない。
【0026】
2.第2融合タンパク質
2.1.β-TrCP1
本発明に用いるβ-TrCP1の起源となる動物種は特に限定されない。更に、β-TrCP1としての活性を有している限り、天然に存在するβ-TrCP1遺伝子およびそれによりコードされるβ-TrCP1だけでなく、人工合成されたβ-TrCP1遺伝子およびそれによりコードされるβ-TrCP1もまた、本発明に用いることができる。例えば、哺乳動物(ヒト、サル(例えばチンパンジー、オラウータン、カニクイザル)、ウシ、マウス、ラット)、鳥類(例えばニワトリ、ウズラ)、両生類(例えばアフリカツメガエル)、ホヤ、キイロショウジョウバエに由来するβ-TrCP1およびそれに基づいて人工合成されたβ-TrCP1を使用することができ、ヒトを含む哺乳動物由来のβ-TrCP1およびそれに基づいて人工合成されたβ-TrCP1が好ましい。配列番号29にはヒト由来β-TrCP1のアミノ酸配列を、配列番号28にはそれをコードする塩基配列を示す。配列番号29のアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるβ-TrCP1に包含される。
【0027】
種々の動物種のβ-TrCP1のアミノ酸配列および塩基配列は公知であり、beta-transducin repeat containing (BTRC、beta-TrCP、β-TrCP、beta-TrCP1)、F-box and WD repeat domain containing 1(Fbw1、Fbw1a、FWD1、FBXW1、FBXW1A)MGC4643等の名称により各種データベースに登録されている。β-TrCP1の塩基配列の登録番号の具体例は以下に示される。
ヒト(Homo sapiens: NM_003939, NM_033637, NM_033637, BD131778, AK313417, AK313353, BC027994, AF129530, Y14153)、マウス(Mus musculus : NM_001037758, NM_009771, AF081887, AF110396, AF112979, AF099932, AK145624, AK145091, AK156660, AK083257, AK052317, AK032221, BC052879)、ラット(Rattus norvegicus : NM_001007148, BC085125)、オラウータン(Pongo abelii : NM_001131502, BC003989, CR859772, CR857777)、ウシ(Bos Taurus: BC123621, NM_001083475)、ウマ(Equus caballus: XM_001499866, XM_001924828)、アフリカツメガエル(Xenopus laevis : NM_001087595, BC079732, M98268)、イヌ(Canis lupus familiaris: XM_856836, XM_856812, XM_543980)、ニシツメガエル(Xenopus tropicalis : NM_001016386)、カニクイザル(Macaca fascicularis: AB071071, AB169034)、チンパンジー(Pan troglodytes: XM_507991)、ショウジョウバエ(Drosophila melanogaster : NM_079706, AF222924, AF222923, AF032878)。これらの塩基配列がコードするβ-TrCP1のアミノ酸配列からなるタンパク質だけでなく、これらの塩基配列がコードするβ-TrCP1のアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるβ-TrCP1に包含される。
【0028】
本発明では、β-TrCP1は少なくともその一部が使用されればよく、全体を用いる必要はない。特に、Cdc25AまたはCdc25Bとの結合後にユビキチン化に関与するF-boxと呼ばれる領域を有していないβ-TrCP1の部分配列を使用することが好ましい。例えば、F-boxと呼ばれる領域の全部または一部が欠損したアミノ酸配列からなるβ-TrCP1や、F-boxと呼ばれる領域の途中のアミノ酸からC末端に至るβ-TrCP1の領域(より好ましくはF-boxと呼ばれる領域および該領域よりもN末端側を欠くβ-TrCP1の領域)に含まれる、全長200アミノ酸以上の部分アミノ酸配列、特に好ましくは全長350アミノ酸以上の部分アミノ酸配列からなるタンパク質が好ましい。例えば、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能であるものが好ましく、なかでも配列番号29の第212番からC末端に至る部分配列が好ましい。F-boxと呼ばれる領域は、上記のデータベースの塩基配列から推定されるアミノ酸配列としては、ヒト、マウス、ラット、オラウータン、ウシ、ウマ、イヌ、カニクイザル、チンパンジーにおいては、TALPARGLDHIAENILSYLDAKSLCAAELVCKEWYRVTSDGMLWKKL、で表わされる。オラウータンにはアミノ酸配列の異なる2種類の遺伝子が登録されており、もうひとつF-box領域は、TALPARGLDHIAENILSYLDAKSLCAAELVCKEWYRVTSDGMLWRKL である。また、アフリカツメガエルとセイヨウツメガエルでは、TALPARGLDHIAENILSYLDAKSLCSAELVCKEWYRVTSDGMLWKKL、ショウジョウバエでは、TLLPIKGLDHIAENILSYLDAESLKSSELVCKEWLRVISEGMLWKKLというアミノ酸配列で表される領域がF-box領域に該当する。
【0029】
2.2.β-TrCP2
本発明に用いるβ-TrCP2の起源となる動物種は特に限定されない。更に、β-TrCP2としての活性を有している限り、天然に存在するβ-TrCP2遺伝子およびそれによりコードされるβ-TrCP2だけでなく、人工合成されたβ-TrCP2遺伝子およびそれによりコードされるβ-TrCP2もまた、本発明に用いることができる。例えば、哺乳動物(例えばヒト、ウシ、ウマ、マウス、ラット)、鳥類(例えばニワトリ、ウズラ)、両生類(例えばアフリカツメガエル)に由来のβ-TrCP2およびそれに基づいて人工合成されたβ-TrCP2であれば特に限定されないが、ヒトを含む哺乳動物由来のβ-TrCP2およびそれに基づいて人工合成されたβ-TrCP2が好ましい。
【0030】
種々の動物種のβ-TrCP2のアミノ酸配列および塩基配列は公知であり、beta-transducin repeat-containing protein 2(BTRC2)、BTRCP2、F-box and WD-40 domain protein 1B(Fbw1B)、FBW11、F-box and WD repeat domain containing 11(F-box and WD repeat domain containing 11 、FBXW11)、HOS等の登録名により各種データベースに登録されている。β-TrCP2の塩基配列の登録番号の具体例は以下に示される。
ヒト(Homo sapiens: NM_033645, NM_012300, NM_033644, DQ894304, DQ891125, AB033281, AB033280, AB033279, AK314999, AB014596, AB383960)、マウス(Mus musculus: NM_134015, BC034261, BC008552, AY038079, AB093260)、チンパンジー(Pan troglodytes : XM_518097)、ニシツメガエル(Xenopus tropicalis: BC170890, BC170892)、ラット(Rattus norvegicus: NM_001106993)、ウシ(Bos Taurus: XM_612428)、ニワトリ (Gallus gallus : NM_001039262)、ゼブラフィッシュ (Danio rerio: NM_201310, BC045356, NM_213504)、ウマ(Equus caballus: XM_001499667)。これらの塩基配列がコードするβ-TrCP2のアミノ酸配列からなるタンパク質だけでなく、これらの塩基配列がコードするβ-TrCP2のアミノ酸配列において1〜数個(例えば1〜10個、好ましくは1〜5個)のアミノ酸が置換、欠失または付加された配列からなるタンパク質もまた本発明におけるβ-TrCP2に包含される。
【0031】
本発明では、β-TrCP2は少なくともその一部が使用されればよく、全体を用いる必要はない。特に、Cdc25AまたはCdc25Bとの結合後にユビキチン化に関与するF-boxと呼ばれる領域を有していないβ-TrCP2の部分配列を使用することが好ましい。例えば、F-boxと呼ばれる領域の全部または一部が欠損したアミノ酸配列からなるβ-TrCP2や、F-boxと呼ばれる領域の途中のアミノ酸からC末端に至るβ-TrCP2の領域(より好ましくはF-boxと呼ばれる領域および該領域よりもN末端側を欠くβ-TrCP2の領域)に含まれる、全長200アミノ酸以上の部分アミノ酸配列、特に好ましくは全長350アミノ酸以上の部分アミノ酸配列からなるタンパク質が好ましい。F-boxと呼ばれる領域は上記のデータベース上の塩基配列から推定されるアミノ酸配列としては、、ヒト、マウス、チンパンジー、ラット、ウシ、ニワトリ、ウマにおいては、TALPEQGLDHIAENILSYLDARSLCAAELVCKEWQRVISEGMLWKKL、ニシツメガエルでは、TALPARGLDHIAENILSYLDARSLCSAELVCKEWYRVTSDGMLWKKL、ゼブラフィッシュでは、TALPARGLDHIAENILSFLDARSLCSAELVCREWQRVISDGMLWKKLというアミノ酸配列であらわされる。
【0032】
2.3.リンカー
第2融合タンパク質ではβ-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結されている。第2検出用タンパク質については後述する。
【0033】
β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質との間には適宜リンカーを介して連結されている。リンカー部分の構造は特に限定されないが、通常は、アミノ酸数が3〜20個程度であり、β-TrCP1またはβ-TrCP2と第二検出用タンパク質の立体的な動きが阻害されないようにグリシン残基に富む(例えばリンカーを構成するアミノ酸の半数以上がグリシンである)ことが望ましい。
【0034】
またリンカーのほかに、融合タンパク質を作成する際の必要に応じて導入される制限酵素認識配列によりコードされるアミノ酸配列などが、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質との間に存在していてもよい。
β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とはどちらがN末端側に位置しても構わない。
【0035】
3.相互作用を識別するための手段
第1検出用タンパク質と第2検出用タンパク質とは、相互作用したときに識別可能な信号を発することができる。
ここで「識別可能な信号」とは好ましくは緑色蛍光、赤色蛍光、黄色蛍光などの蛍光である。
【0036】
このような信号を発する第1検出用タンパク質と第2検出用タンパク質との組合せとしては、緑色蛍光タンパク質(GFP)をはじめ、黄色蛍光タンパク質(YFP)、青色蛍光タンパク質(CFPやBFC)、赤色蛍光タンパク質(RFP)などのGFP類似の蛍光タンパク質のC末端側断片とN末端側断片との組み合わせ(BiFC (bi-molecular fluorescence complementation)法に用いる)や、二種類の全長蛍光タンパク質を用いて蛍光を発光させるFRET(fluorescence resonance energy transfer)法で使用するためのCFPとYFPとの組み合わせや、YFPとRFPとの組み合わせなどの、公知の組み合わせを使用することができる。
【0037】
これらの二種類の検出用タンパク質の組み合わせのなかから、一方を第1検出用タンパク質として、他方を第2検出用タンパクとして用いればよい。
【0038】
4.四成分融合タンパク質
第1融合タンパク質と第2融合タンパク質とは互いに連結された四成分を少なくとも含む融合タンパク質として使用することがより好ましい。
【0039】
第1融合タンパク質と第2融合タンパク質との間にはリンカー部分が介在していることが好ましい。リンカー部分の構造は特に限定されないが、通常は、アミノ酸数が3〜30程度であり、第1融合タンパク質と第2融合タンパク質の立体的な動きが阻害されないようにグリシン残基に富む(例えばリンカーを構成するアミノ酸の半数以上がグリシンである)ことが望ましい。
【0040】
またリンカーのほかに、融合タンパク質を作成する際の必要に応じて導入される制限酵素認識配列によりコードされるアミノ酸配列などが、第1融合タンパク質と第2融合タンパク質との間に存在していてもよい。
第1融合タンパク質と第2融合タンパク質とはどちらがN末端側に配置されてもよい。
【0041】
5.測定に用いる細胞
本発明では、上記の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)を細胞において発現させる。細胞としては特に限定されないがHeLa、Cos7をはじめとし、ベクターにより形質転換が可能であり導入された融合タンパク質が安定に発現されれば、初代培養細胞を含むすべての細胞が使用できる。
【0042】
6.スクリーニング結果の評価
前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である場合には、被験物質が、Cdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価することができる。このような因子としては、JNK1キナーゼ、JNK2キナーゼ、p38キナーゼ(p38α、p38βまたはp38γ)、ERK1、ERK2、CDKキナーゼ(CDK1〜6)が挙げられる。このような因子を誘導する物質は、遺伝毒性ストレス原因物質、非遺伝毒性ストレス原因物質または抗癌物質の候補物質となりうる。更に、遺伝毒性ストレス原因物質の働きを軽減する物質、非遺伝毒性ストレス原因物質の働きを軽減する物質の候補物質ともなりうる。よって、本発明はこれらの物質のスクリーニングのために従来行われていた動物試験に代わる一次スクリーニング手段として有効である。
【0043】
前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である場合には、被験物質が、Cdc25Aの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価することができる。このような因子としては、p38キナーゼ、CHK1キナーゼ、CHK2キナーゼ、MK2キナーゼが挙げられる。このような因子を誘導する物質は、遺伝毒性ストレス原因物質(毒性物質)または抗癌物質の候補物質となりうる。更に、遺伝毒性ストレス原因物質の働きを軽減する物質の候補物質ともなりうる。
【0044】
なお本発明において、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子の「誘導」とは、タンパク質発現量の増大に限らず、当該因子の活性の増強全般を指す。
【0045】
7.ベクター、形質転換細胞
本発明はまた、上述の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)をコードするDNAを含むベクターに関する。このようなベクターとしてはプラスミドベクター、ウイルスベクター等が挙げられるがこれには限定されない。
本発明は更にまた、当該ベクターにより形質転換された形質転換細胞に関する。対象となる細胞としては、上記5に示した細胞が挙げられる。
【0046】
8.ゲノムDNAへの導入および実験動物
本発明では、上述の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)をコードするDNAがゲノム中に取り込まれて永続的にこれらの融合タンパク質が発現可能となった細胞、並びに当該細胞を体内に含む非ヒト実験動物をも提供する。
【0047】
このような細胞の作成方法としては、G418やハイグロマイシンなどの薬剤に対する耐性遺伝子を有するベクターに上述の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)をコードするDNAを組み込み、そのベクターが導入された細胞を薬剤で選択すること(永続的にこれらの融合タンパク質が発現可能となった細胞の分離)や、マウスの受精卵に上述の第1融合タンパク質および第2融合タンパク質(または四成分融合タンパク質)をコードするDNAをマイクロインジェクションすること(当該細胞を体内に含む非ヒト実験動物の作成)が挙げられる。
【0048】
上記細胞を体内に含む非ヒト実験動物としては、マウス、ラット、ブタ、ウシ、ゼブラフィッシュ、メダカが挙げられる。例えば、こうして得られた非ヒト実験動物に、被験物質を接触させ、一定期間経過後に肝臓や皮膚などの組織から細胞を採取し蛍光を測定し、蛍光が観測された場合には該物質は非遺伝毒性ストレス原因物質または抗癌物質であると判断する、などの応用が可能となるであろう。
【0049】
9.キット
上述のDNA(またはDNAの組み合わせ)、ベクター(またはベクターの組み合わせ)および形質転換細胞は、被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価するためのアッセイ用キットの構成要素として利用することが可能である。
【0050】
参考例
Cdc25BはJNK1活性に依存してβ-TrCP1と結合する
Cdc25Bを恒常発現するHeLa細胞を作成し、該細胞を、遺伝毒性ストレス因子であるヒドロキシ尿素(1.5 mM、1時間処理)、アフィディコリン(2.5μg/ml、1時間処理)、エトポシド(10μM、1時間処理)、またはカンプトテシン(2μM、1時間処理)で処理するか、或いは、非遺伝毒性ストレス因子であるアニソマイシン(50 ng/ml、10分処理)または食塩(300mM、1時間処理)で処理した後にCdc25B量を確認した。その結果、非遺伝毒性ストレス因子の処理によって、Cdc25Bの分解が誘導されることが明らかとなった(図1)。
【0051】
更に、阻害剤や強発現実験から、このCdc25B分解促進はJNK経路によることが示された。図2は、β-TrCPを含むSCF (Skp1-Cullin 1-F-box proteinの3種のタンパク質からなる複合体であり、下線で示した頭文字をとって略したものである。F-boxと呼ばれる配列をもつタンパク質群と複合体を形成しタンパク質のユビキチン化を行う複合体の総称) によりCdc25Bの断片がJNK1依存的にユビキチン化されることを示す。
【0052】
JNK1はin vitroでCdc25BのN末端断片(1-175アミノ酸)をリン酸化することが確認された。更に変異体の解析によりSer101がJNKに依存するCdc25Bの分解に必須であることが確認された(図3)。
【0053】
これらの結果から、本発明者らは、非遺伝毒性ストレスの存在下においてJNK/β-TrCPを介したCdc25Bの分解という新しい細胞周期制御機構を明らかにした。図4にはこの新規な機構を模式的に示す。
【0054】
以下の実施例では融合タンパク質の配置順序に関して下記の4通り実施した。しかしながら本発明の範囲はこれらには限定されない。
(1)Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質(実施例では蛍光タンパク質のN末端側断片)とがこの順でN末端側から配置された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質(実施例では蛍光タンパク質のC末端側断片)がこの順でN末端側から配置された第2融合タンパク質との組み合わせ。
(2)Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質(実施例では蛍光タンパク質のN末端側断片)とがこの順でN末端側から配置された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質(実施例では蛍光タンパク質のC末端側断片)がこの順でN末端側から配置された第2融合タンパク質とが、この順でN末端側から配置された四成分融合タンパク質。
(3)第1検出用タンパク質(実施例では蛍光タンパク質のN末端側断片)とCdc25AまたはCdc25Bの少なくとも一部分とがこの順でN末端側から配置された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質(実施例では蛍光タンパク質のC末端側断片)とがこの順でN末端側から配置された第2融合タンパク質との組み合わせ。
(4)第1検出用タンパク質(実施例では蛍光タンパク質のN末端側断片)とCdc25AまたはCdc25Bの少なくとも一部分とがこの順でN末端側から配置された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質(実施例では蛍光タンパク質のC末端側断片)とがこの順でN末端側から配置された第2融合タンパク質とが、この順でN末端側から配置された四成分融合タンパク質。
【実施例】
【0055】
本発明は、新たに見出された上記参考例の知見を利用する発明である。すなわち、Cdc25Bとβ-TrCPとがJNK1が誘導される条件下(非遺伝毒性ストレス条件下)において相互作用することから、Cdc25Bとβ-TrCPとの相互作用の有無によって、JNK1が誘導されているか否か(非遺伝毒性ストレスが負荷されているか否か)を判別することが可能となる。Cdc25Bとβ-TrCPとの相互作用を解析するには、例えばBiFC (bi-molecular fluorescent complementation)法が利用できる。図5には、BiFC法によりJNK1依存性のCdc25Bとβ-TrCPとの相互作用を判別する方法の典型的な例を示す。
【0056】
以下に、非遺伝毒性ストレスの存在下においてBiFC法により蛍光発光が実際に確認された具体例を詳細に説明する。
実験材料と方法
1.融合タンパク質の作成手順概要
1.1.用いたプラスミド
BiFCに用いた、蛍光タンパク質を2分割した断片を発現するプラスミドは、医学生物学研究所(MBL:名古屋市)より購入したキット(商品名:CoralHue Fluo-chase kit, Code No. AM-1100)中の、phmKGN-MNおよびphmKGC-MNを使用した。このキットに用いられている蛍光タンパク質は、サンゴ由来のKusabira-Greenの単量体(mKG)である。
【0057】
Cdc25B及びβ-TrCP1発現プラスミドは、Invitrogen社のpEF6-Myc/Hisプラスミド(Cat.No. V962-20)を基本とし、本発明者らの研究室で作成した。Cdc25BはN-末端にFLAGタグがタンデムに2個付加してあり、またβ-TrCPはN-末端側にあるF-boxと呼ばれる領域を含む211アミノ酸を欠き、その断片のN-末端側にMycタグ配列がやはり2個タンデムに付加されている(F-boxとよばれる領域を欠損させると、基質(Cdc25B)との結合は通常に起きるが、基質の分解に必要なユビキチン化反応が起きなくなるため、Cdc25Bとβ-TrCP1が安定な複合体として検出できることが期待される)(図6〜9)。
【0058】
1.2.Cdc25Bおよびβ-TrCP1断片とmKGタンパク質断片の融合プラスミドの作成
Cdc25Bの2xFLAG配列を含むN-末端側1-175アミノ酸を含む断片をPCRにより増幅し、適当な制限酵素で切断後、同様の制限酵素で切断されたphmKGN-MNプラスミドへ導入した。これにより、FLAG配列を含むCdc25B (N1-175)断片のC-末端側にmKGのN-末端断片(mKGN:168アミノ酸)が融合されたタンパク質の発現されることが予想される(図8プラスミド3)。また、対照としてストレス非依存的にβ-TrCP1との結合が起きる領域を含む1-280アミノ酸断片も、175アミノ酸断片と同様にFLAG配列を含んだ形でmKGNとの融合タンパク質として発現されるようなプラスミドを作成した(図8プラスミド4)。
【0059】
2xMyc配列が融合したF-box欠失型β-TrCP1の場合も同様に、PCRによりアミノ酸をコードする領域全長を増幅し、適当な制限酵素で切断後、やはり同様の制限酵素で切断されたphmKGC-MNへ導入された。これにより、Myc配列を含むβ-TrCP1断片のC-末端側にmKGのC-末端断片(mKGC:50アミノ酸)が融合されたタンパク質が発現されることが予想される(図9プラスミド7)。
【0060】
1.3.2xFLAG-Cdc25B-mKGNと2xMyc-β-TrCP1-mKGCの融合タンパク質発現プラスミドの作成
より効率的なCdc25Bとβ-TrCP1断片の相互作用とそれに続くmKGNとmKGCの再会合を促進するために、それぞれの断片を融合し、1本のポリペプチド(タンパク質)として細胞内で発現させることを計画し、以下の手順によりプラスミドを作成した。
【0061】
まず、2xFLAG-Cdc25B-mKGN断片をPCRにより増幅し、適当な制限酵素で切断後pEF6プラスミドに導入した。さらに、2xFLAG-Cdc25B-mKGN断片が挿入されたpEF6プラスミドに、2xFLAG-Cdc25B-mKGNの下流に位置するように、また、タンパク質としての読み枠が一致するように、PCRにより増幅された2xMyc-β-TrCP1-mKGC断片を挿入した。この結果、細胞内では2xFLAG-Cdc25B-mKGN と2xMyc-β-TrCP1-mKGCの融合タンパク質の発現が予測される。なお、二つのペプチド間には、約15アミノ酸からなるリンカーが存在し、両ペプチドの立体的な動きが阻害されないことが期待される構造となっている(図10プラスミド8)。
【0062】
また、mKGN-Cdc25B(1-175)-2xFLAGというかたちのペプチドを発現させるために、以下のようなプラスミドの作成も行った。まず、mKGN断片をPCRにより増幅し、pEF6プラスミドに導入した。このpEF6プラスミドは、導入断片から発現されるペプチドのC-末端側にFLAG配列が付加されるように遺伝子を組み換えることが可能である。さらに、mKGNと2xFLAGの間に、タンパク質としての読み枠が一致するようにCdc25BのN-末端側175アミノ酸が含まれるDNA断片をPCRにより増幅したのち、制限酵素処理により導入した。このプラスミドからは、mKGN-Cdc25B(1-175)-2xFLAGというペプチドが発現されることが期待される(図8プラスミド5)。
さらに、mKGN-Cdc25B(1-175)-2xFLAG断片と2xMyc-β-TrCP1-mKGC断片の融合タンパク質が発現されるように、上述の方法によりプラスミドを作成した(図10プラスミド9)。
【0063】
2.融合タンパク質の作成手順詳細
上記1において概要を示した融合タンパク質の作成手順について、具体的な配列を参照して詳細に説明する。
【0064】
2.1.Cdc25B(図6)について
ヒトCdc25B1の塩基配列(配列番号1)およびアミノ酸配列(配列番号2)を図11に示す。本実施例では、「Cdc25B」とはヒトCdc25B1を指す。Cdc25Bの第94〜108番のアミノ酸配列(図11一重下線部)が、参考例に示すとおり本発明者らにより新たに特定された、ストレスに依存して活性化されたJNK1によりリン酸化を受けβ-TrCP1との結合に関与するコア配列である。Cdc25Bの第254〜259番のアミノ酸配列(図11二重下線部)が、従来から知られていた、ストレスに関わりなくβ-TrCP1と結合する配列のコア部分である。
【0065】
pEF6/Myc-His Bプラスミド(Invitrogen社製)へ挿入されたCdc25BのcDNAのN-末端側にはFLAGと呼ばれる配列が2個タンデムに結合されている(図6のプラスミド1)。その上流域部分の塩基配列(配列番号3)およびコードされるアミノ酸配列(配列番号4)を図12に示す。
【0066】
2.2.Cdc25B/175NおよびCdc25B/280N(図7)について
2xFLAG-Cdc25B/N175と2xFLAG-Cdc25B/N280は、以下のプライマーを使用し、図6中のプラスミド1を鋳型にしてPCRで必要な断片を増幅後、制限酵素BamHIとXhoIで切断し、2xFLAG-Cdc25B/N175(図7中の断片1)および2xFLAG-Cdc25B/N280(図7中の断片2)を作成した。
【0067】
【表1】

【0068】
Cdc25B/N175-2xFLAGは以下のプライマーを使用し、以下に述べる手順で作成した。
断片3は最終的にCdc25B/N175のC-末端側にFLAG配列が2個タンデムに結合された形の断片4を作成するための中間段階のものとして作成した。使用したプライマーセットは以下のものである。このプライマーセットを使用し、図6中のプラスミド1を鋳型にしてPCR増幅を行い、断片3を得た。
【0069】
【表2】

【0070】
図7中のプラスミド2はInvitrogen社製のpEF6/Myc-His BプラスミドのXbaI-PmeI制限酵素部位に、FLAG配列が2個タンデムに結合したペプチドをコードする図13に示すオリゴヌクレオチドが挿入された改変プラスミド、pEF6-2FLAG/CのEcoRI-XbaI部位に、図7中のPCR増幅断片3を制限酵素EcoRIとXbaIで消化したものを挿入して作成した。
2xFLAG/C断片の塩基配列(配列番号10)およびコードされるアミノ酸配列(配列番号11)を図13に示す。
【0071】
上記手順により得られた図7に示す断片1、2および4の塩基配列(配列番号12、14および16)ならびにコードされるアミノ酸配列(配列番号13、15および17)をそれぞれ図14、15および16に示す。
【0072】
2.3.Cdc25B断片とmKGNの融合タンパク質発現プラスミド(図8)について
2.3.1.2xFLAG-Cdc25B/N175-mKGNおよび2xFLAG-Cdc25B/N280-mKGN発現プラスミド(プラスミド3および4)の作成
MBL社製のphmKGN-MNプラスミドを、図7中の断片1及び2を作成した制限酵素と同じ制限酵素セットで切断し、それぞれ2xFLAG-Cdc25B/N175と2xFLAG-Cdc25B/N280の下流(C末端側)にmKGN断片が融合されたタンパク質を発現する、図8中のプラスミド3及びプラスミド4を作成した。すなわち、図7中の断片1および断片2をBamHI-XhoIのセットで切断し、同様にBamHI-XhoIのセットで切断されたphmKGN-MNプラスミドへ導入し、プラスミド3および4を作成した。
【0073】
プラスミド3中で融合タンパク質2xFLAG-Cdc25B/N175-mKGNをコードする部分の塩基配列(配列番号22)およびコードされるアミノ酸配列(配列番号23)を図18に示す。
【0074】
プラスミド4中で融合タンパク質2xFLAG-Cdc25B/N280-mKGNをコードする部分の塩基配列(配列番号24)およびコードされるアミノ酸配列(配列番号25)を図19に示す。
【0075】
2.3.2.mKGN-Cdc25B/N175-2xFLAG発現プラスミド(プラスミド5)の作成
図8中のプラスミド5は以下の手順で作成した。
(i) phmKGN-MNプラスミドを鋳型にして、表3に示すプライマーセットを用いてPCRによりmKGN断片を増幅した。
(ii) 増幅断片を制限酵素Acc65I-BamHIで切断した。認識配列はそれぞれGGTACCとGGATCCである。
(iii) プラスミド2をAcc65IとBamHIで切断し、(ii)の制限酵素消化断片を導入した。
【0076】
【表3】

【0077】
このようにした作成されたプラスミドのBamHI-EcoRI部位間に図17に示す二本鎖塩基配列(センス鎖を配列番号20とする)のオリゴヌクレオチドを挿入し(図17ではBamHIとEcoRI間に挿入された状態の配列が示されている)、mKGN断片とCdc25B/N175-2xFLAG間にリンカーとなるアミノ酸配列(配列番号21)を導入して、最終的にプラスミド5が作られた。
【0078】
プラスミド5中で融合タンパク質mKGN-Cdc25B/N175-2xFLAGをコードする部分の塩基配列(配列番号26)およびコードされるアミノ酸配列(配列番号27)を図20に示す。
【0079】
2.4.β-TrCP1の全配列について
ヒトβ-TrCP1の塩基配列(配列番号28)およびアミノ酸配列(配列番号29)を図21に示す。以下、本実施例ではβ-TrCP1とはヒトβ-TrCP1を指す。
【0080】
pEF6/Myc-His Bプラスミド(Invitrogen社製)へ挿入されたβ-TrCP1のcDNAのN-末端側にはMycと呼ばれる配列が2個タンデムに結合されている。その上流域部分の塩基配列(配列番号30)およびコードされるアミノ酸配列(配列番号31)を含む配列を図22に示す。
【0081】
2.5.β-TrCP1/Δ211(図9プラスミド6)の作成
β-TrCP1のアミノ酸配列(配列番号29)における第146〜192番の領域は、F-boxと呼ばれる、タンパク質のユビキチン化コンポーネントとの複合体形成に必要な部分である。F-boxを含む、配列番号29の第1〜211番の領域を欠くβ-TrCP1タンパク質断片(β-TrCP1/Δ211)は基質(この場合はCdc25B)との結合はできるが、ユビキチン化をすることができない。このため、β-TrCP1/Δ211を用いれば相互作用されたCdc25Bは分解されずに、反応系中に留まり続けることができる。
【0082】
全長β-TrCP1が図22に示すように導入されたpEF6/Myc-His Bプラスミド(Invitrogen社製)を鋳型とし、以下のプライマーを使用してPCRで断片を増幅後、制限酵素BamHIとEcoRVで消化した。消化された断片を、pEF6/Myc-His Bプラスミド(Invitrogen社製)のBamHI-EcoRV部位に挿入し、Myc配列をN-末端側に2個タンデムに付加しpEF-2xMyc-β-TrCP1/Δ211(図9のプラスミド6)を作成した。
【0083】
【表4】

【0084】
pEF6/Myc-His Bプラスミド(Invitrogen社製)へ挿入されたβ-TrCP1/Δ211のcDNAのN-末端側にはMycと呼ばれる配列が2個タンデムに結合されている。その上流域部分の塩基配列(配列番号34)およびコードされるアミノ酸配列(配列番号35)を含む配列を図23に示す。またプラスミド6におけるβ-TrCP1/Δ211のcDNAの下流部分の塩基配列(配列番号36)およびコードされるアミノ酸配列(配列番号37)を含む配列を図24に示す。
【0085】
2.6.β-TrCP1/Δ211とmKGCの融合タンパク質発現プラスミド(プラスミド7)について
2xMyc-β-TrCP1/Δ211は、表5のプライマーを使用し、図9中のプラスミド6を鋳型にしてPCRで必要な断片を増幅後、制限酵素XhoI-NotIで切断した。その断片を、医学生物学研究所(MBL)より購入したphmKGC-MNプラスミドのXhoI-NotI部位に挿入し、2xMyc-β-TrCP1/Δ211-mKGC(図9中のプラスミド7)を作成した。
【0086】
【表5】

【0087】
プラスミド7中での、タンパク質コード配列(およびその周辺の配列)を図25(核酸配列は配列番号40、コードされるアミノ酸配列は配列番号41)に示す。
【0088】
2.7.2xFLAG-Cdc25B/N175-mKGNと2xMyc-β-TrCP1/Δ211-mKGCの融合タンパク質発現プラスミド(プラスミド8)の作成
2.7.1.2xFLAG-Cdc25B/N175-mKGNのPCR増幅とプラスミドへの導入
図8中のプラスミド3を鋳型にして、表6に示すプライマーセットで2xFLAG-Cdc25B/N175-mKGN断片をPCRにより増幅した。得られた増幅断片を、BamHIとEcoRVで消化・切断した。この増幅断片は、下流のリンカー並びに2xMyc-β-TrCP1/Δ211-mKGCとの融合タンパク質として発現されることとなるため、停止コドンが除かれるようにreverse プライマーが設計されている。
【0089】
【表6】

【0090】
得られた増幅断片をBamHIとEcoRVで消化切断したものの塩基配列(配列番号44)およびコードされるアミノ酸配列(配列番号45)を図26に示す。
【0091】
BamHIとEcoRVで消化切断後の得られた増幅断片を、リンカー導入pEF6プラスミドのBamHI-EcoRV部位(図30)へ導入した。リンカー導入pEF6プラスミドについては、下記2.9において詳述する。
【0092】
2.7.2.2xMyc-β-TrCP1/Δ211-mKGCのPCR増幅とプラスミドへの導入
図9中のプラスミド7を鋳型にして、表7に示すプライマーセットで2xMyc-β-TrCP1/Δ211-mKGC断片を増幅した。
【0093】
【表7】

【0094】
得られた増幅断片をXbaIIとPmeIで消化切断したものの塩基配列(配列番号48)およびコードされるアミノ酸配列(配列番号49)を図27に示す。アミノ酸の読み枠の関係で、XbaI部位の上流に2ヌクレオチドを余分に付加した。
【0095】
XbaIIとPmeIによる消化切断後の増幅断片を、上述の2xFLAG-Cdc25B/N175-mKGN断片を有するpEF6プラスミドのリンカーの下流にあるXbaI-PmeI部位(図30)へ導入して、図10に示すプラスミド8を得た。
【0096】
2.8.mKGN-Cdc25B/N175-2xFLAGと2xMyc-β-TrCP1/Δ211-mKGCの融合タンパク質発現プラスミド(プラスミド9)の作成
導入する遺伝子及びリンカー導入pEF6プラスミド中の制限酵素部位の関係から、本プラスミドの作成は、先に2xMyc-β-TrCP1/Δ211-mKGCをプラスミドに組み込み、得られたプラスミド中のリンカーの上流のEcoRV部位(図30)へmKGN-Cdc25B/N175-2xFLAG断片を導入した。プラスミド8の場合と同様にmKGN-Cdc25B/N175-2xFLAGの増幅断片からは、停止コドンが除かれるようにreverse プライマーが設計されている。
【0097】
2.8.1.2xMyc-β-TrCP1/Δ212-mKGCのPCR増幅とプラスミドへの導入
図9中のプラスミド7を鋳型にして、プラスミド8の作成時に使用したのと同じ表7に示すプライマーセットを用いて2xMyc-β-TrCP1/Δ211-mKGC断片を増幅した。得られた増幅断片をXbaIとPmeIで消化・切断して、図27に示すのと同じ塩基配列(配列番号48)を有する断片を得た。当該断片のリンカー導入pEF6プラスミドのリンカーの下流にあるXbaI-PmeI部位(図30)へ導入した。
【0098】
2.8.2.mKGN-Cdc25B/N175-2xFLAGのPCR増幅とプラスミドへの導入
上記2.3.2で得られたプラスミド5(図8)を鋳型にして、表8に示すプライマーを使用してPCRにより増幅を行って、mKGN-Cdc25B/N175-2xFLAGをコードするDNA断片を得た。
【0099】
【表8】

【0100】
得られた増幅断片をEcoRVで消化切断したものの塩基配列(配列番号52)およびコードされるアミノ酸配列(配列番号53)を図28に示す。
【0101】
EcoRVによる切断後のmKGN-Cdc25B/N175-2xFLAGのDNA断片を、上記2.8.1で得られた、2xMyc-β-TrCP1/Δ211-mKGCが導入されたリンカー導入pEF6プラスミドのリンカーの上流のEcoRV部位(図30)へ導入した。こうして図10に示すプラスミド9を作成した。
【0102】
2.9.リンカー導入pEF6プラスミド
上記2.7および2.8に記載の融合タンパク質の製造において使用したリンカー導入pEF6プラスミドは、pEF6/Myc-His Bプラスミド(Invitrogen社より購入)の制限酵素EcoRIおよびNotI切断部位に、図29(1)に示す二本鎖オリゴヌクレオチド(センス鎖:配列番号54)を挿入することにより作成した。導入されたオリゴヌクレオチドは、図29(2)に示すアミノ酸配列(配列番号55)のポリペプチドをコードする。当該ポリペプチドのうち、第5〜20番の部分がmKGN融合Cdc25B断片とmKGC融合β-TrCP1断片との間を連結するリンカーとして機能すると考えられる。
得られたリンカー導入pEF6プラスミドの、リンカーコード配列の近傍の構造を図30に示す。
【0103】
3.細胞への導入とタンパク質の検出
プラスミドの細胞への導入は、Invitrogen社のLipofectamine2000試薬を用いて行った。導入したプラスミドの組み合わせは以下の通りである(図10)。
1)2xFLAG-Cdc25B(1-175)-mKGN + 2xMyc-β-TrCP1-mKGC(プラスミド3と7)
2)2xFLAG-Cdc25B(1-175)-mKGN-2xMyc-β-TrCP1-mKGC(プラスミド8)
3)mKGN-Cdc25B(1-175)-2xFLAG + 2xMyc-β-TrCP1-mKGC(プラスミド5と7)
4)mKGN-Cdc25B(1-175)-2xFLAG-2xMyc-β-TrCP1-mKGC(プラスミド9)
5)2xFLAG-Cdc25B(1-280)-mKGN + 2xMyc-β-TrCP1-mKGC(プラスミド4と7)
JNK1キナーゼの作用によってCdc25Bとβ-TrCP1の結合が促進されて蛍光が発光することを確認することを目的として、JNK1キナーゼおよびその活性化キナーゼであるMKK7をコードする遺伝子が組み込まれたプラスミドを1)〜4)の組み合わせとともに細胞(HeLa細胞またはCos 7細胞)へ導入した。24時間後に細胞をメタノールで固定し、蛍光顕微鏡により緑色蛍光を観察した。また、比較のために、活性が欠損しているJNK1キナーゼ遺伝子が組み込まれたプラスミドを用いて同様の操作を行った。
【0104】
4.薬剤処理による応答の解析
一過的にプラスミドを導入したHeLa細胞を薬剤(アニソマイシン:50ng/mL、食塩:培養液中の全食塩濃度として250mM、ヒドロキシ尿素:1.5mM、アフィディコリン2.5 μg/ml、エトポシド:2mM)で24時間処理し、細胞をメタノール固定し蛍光顕微鏡により緑色蛍光を観察した。
【0105】
結果
1.蛍光検出に適したプラスミドの選択
図10に記したプラスミドの組み合わせのうちで、JNK1を発現させない場合のノイズが低くかつ、JNK1を発現させた場合のシグナルが高いものを選択した。その結果、4)のタンパク質、即ちmKGN-Cdc25B/N175-2xFLAG-2xMyc-β-TrCP1/Δ211-mKGCを発現するプラスミド(プラスミド9)を使用した場合において高いS/N比が得られた。下の表は、各プラスミドの組み合わせでJNK1に対する応答実験を行った場合の定性的な結果を示す。
【0106】
【表9】

【0107】
図31には、プラスミド9(図10の4))を導入したHeLa細胞およびCos7細胞では、JNK1遺伝子が導入された場合に蛍光が発光したことを示す。なお、写真には示していないが、他のプラスミドを導入した場合にもJNK1遺伝子が導入された細胞では蛍光が確認された。
【0108】
さらに、キナーゼ活性に関して野生型(正常型:図32ではWTと表記)および欠損型(図32ではKDと表記)のJNK1を用いた実験により、緑色蛍光の出現がJNK1のキナーゼ活性に依存していることが明らかとなった(図32では緑色蛍光を緑色で、細胞中で発現しているJNK1タンパク質を赤色で示している。二つの図を重ね合わせると、緑色蛍光を発しておりかつ、JNK1タンパク質が発現されている細胞は、緑+赤で黄色となる(JNK1-KD発現細胞では緑色蛍光が発せられないため、JNK1の発現を示す赤色のみとなる。)。以降の実験は、このタンパク質を発現するプラスミド(プラスミド9)を使用して行った。ただし、表9に示すいずれの組み合わせにおいても明らかにシグナルがノイズよりも高いという結果が得られている。また、JNK非依存的な結合部位を含む5)の組み合わせ、すなわち、Cdc25Bの280アミノ酸断片においても、JNK依存的なシグナルの増強が見られた。
【0109】
2.薬剤処理
DNAに損傷を起こさない薬剤としてアニソマイシン、食塩(胃がんの発がん促進因子)、DNA複製を阻害し鎖切断を誘発するヒドロキシ尿素、アフィディコリン、DNAに二本鎖切断を誘導するエトポシド(抗がん性化学物質)を用いて、プラスミドの有用性を検定した。
【0110】
実験ではプラスミド9を用いた。プラスミド9をHeLa細胞へ導入し、それから6時間後に薬剤を加えた。その結果、薬剤処理18時間後において、すべての例でシグナルの増強が認められた。図33には、アニソマイシンまたは食塩を処理した細胞の結果を示す。図33では、薬剤処理した細胞、特に食塩処理細胞にアポトーシスと思われる細胞死が誘導されたため、細胞数が少なくなっているが、明らかに黄色を示す細胞が出現している。この図においては、発現タンパク質に付加されたFLAG配列を認識する抗体を用いてプラスミドが導入された細胞が示されている。すなわち、タンパク質が発現された細胞が赤色で、そのうちストレス応答によりBiFCが起きた細胞が緑で示されている。よって、両者が一致する細胞は黄色で示される。
【0111】
ヒドロキシ尿素やエトポシドなどのDNA損傷誘発試薬は、短時間処理ではDNA損傷ストレスが主である(図1参照)。しかしながら、図34に示されるように、これらの薬剤を6時間より長時間にわたって連続的に処理したところJNK1活性による蛍光発光が確認された。図34の結果は、DNA損傷ストレスとともにそれとは異なる非遺伝毒性のストレスが細胞にかかり、その結果Cdc25Bの分解を誘発するJNKやp38と呼ばれるストレス応答性キナーゼ群が活性化されたことを反映していると考えられる。
【図面の簡単な説明】
【0112】
【図1】図1は、非遺伝毒性ストレスによりCdc25Bが分解されることを示す。
【図2】図2は、Cdc25Bが、β-TrCP1または2によりJNK1依存的にユビキチン化されることを示す。また、F-boxを欠くβ-TrCP1にはユビキチン可能がないことを示す。
【図3】図3は、Cdc25BのSer101を変異させるとCdc25BはJNKによる分解に対して抵抗性になることを示す。
【図4】図4は、JNK/β-TrCPを介したCdc25Bの分解の機構を模式的に示す。
【図5】図5は、BiFC法によりJNK1依存性のCdc25Bとβ-TrCPとの相互作用を判別する方法の典型的な例を示す。
【図6】図6は、Cdc25B の特性とCdc25B発現プラスミドを示す。
【図7】図7は、PCRによるCdc25B 断片の増幅を示す。
【図8】図8は、Cdc25B 断片とmKGNとの融合タンパク質発現プラスミドを示す。
【図9】図9は、b-TrCP1/D211及び b-TrCP1 /D211 断片とmKGC との融合タンパク質発現プラスミドを示す。
【図10】図10は、検定に用いたプラスミドを示す。
【図11−1】図11は、Cdc25Bの塩基配列(配列番号1)およびアミノ酸配列(配列番号2)を示す。
【図11−2】図11は、Cdc25Bの塩基配列(配列番号1)およびアミノ酸配列(配列番号2)を示す。
【図11−3】図11は、Cdc25Bの塩基配列(配列番号1)およびアミノ酸配列(配列番号2)を示す。
【図12】図12は、プラスミド1の部分配列を示す。
【図13】図13は、2xFLAG/C断片の塩基配列(配列番号10)およびコードされるアミノ酸配列(配列番号11)を示す。
【図14】図14は断片1の塩基配列およびアミノ酸配列を示す。
【図15−1】図15は断片2の塩基配列およびアミノ酸配列を示す。
【図15−2】図15は断片2の塩基配列およびアミノ酸配列を示す。
【図16】図16は断片4の塩基配列およびアミノ酸配列を示す。
【図17】図17は、mKGN断片とCdc25B/N175-2xFLAG間のリンカー用オリゴヌクレオチド配列を示す。
【図18−1】図18は、プラスミド3中で融合タンパク質2xFLAG-Cdc25B/N175-mKGNをコードする部分の塩基配列(配列番号22)およびコードされるアミノ酸配列(配列番号23)を示す。
【図18−2】図18は、プラスミド3中で融合タンパク質2xFLAG-Cdc25B/N175-mKGNをコードする部分の塩基配列(配列番号22)およびコードされるアミノ酸配列(配列番号23)を示す。
【図19−1】図19は、プラスミド4中で融合タンパク質2xFLAG-Cdc25B/N280-mKGNをコードする部分の塩基配列(配列番号24)およびコードされるアミノ酸配列(配列番号25)を示す。
【図19−2】図19は、プラスミド4中で融合タンパク質2xFLAG-Cdc25B/N280-mKGNをコードする部分の塩基配列(配列番号24)およびコードされるアミノ酸配列(配列番号25)を示す。
【図19−3】図19は、プラスミド4中で融合タンパク質2xFLAG-Cdc25B/N280-mKGNをコードする部分の塩基配列(配列番号24)およびコードされるアミノ酸配列(配列番号25)を示す。
【図20−1】図20は、プラスミド5中で融合タンパク質mKGN-Cdc25B/N175-2xFLAGをコードする部分の塩基配列(配列番号26)およびコードされるアミノ酸配列(配列番号27)を示す。
【図20−2】図20は、プラスミド5中で融合タンパク質mKGN-Cdc25B/N175-2xFLAGをコードする部分の塩基配列(配列番号26)およびコードされるアミノ酸配列(配列番号27)を示す。
【図21−1】図21は、β-TrCP1の塩基配列(配列番号28)およびアミノ酸配列(配列番号29)を示す。
【図21−2】図21は、β-TrCP1の塩基配列(配列番号28)およびアミノ酸配列(配列番号29)を示す。
【図21−3】図21は、β-TrCP1の塩基配列(配列番号28)およびアミノ酸配列(配列番号29)を示す。
【図22】図22は、β-TrCP1が導入されたpEF6/Myc-His Bプラスミドの部分配列を示す。
【図23】図23は、β-TrCP1/Δ211が導入されたpEF6/Myc-His Bプラスミドの部分配列を示す。
【図24】図24は、プラスミド6におけるβ-TrCP1/Δ211のcDNAの下流部分の塩基配列(配列番号36)およびコードされるアミノ酸配列(配列番号37)を含む配列を示す。
【図25−1】図25は、プラスミド7中での、タンパク質コード配列(およびその周辺の配列)を示す。
【図25−2】図25は、プラスミド7中での、タンパク質コード配列(およびその周辺の配列)を示す。
【図25−3】図25は、プラスミド7中での、タンパク質コード配列(およびその周辺の配列)を示す。
【図26−1】図26は、2xFLAG-Cdc25B/N175-mKGNの増幅断片をBamHIとEcoRVで消化・切断したものを示す。
【図26−2】図26は、2xFLAG-Cdc25B/N175-mKGNの増幅断片をBamHIとEcoRVで消化・切断したものを示す。
【図27−1】図27は、2xMyc-β-TrCP1/Δ211-mKGCの増幅断片をXbaIIとPmeIで消化・切断したものを示す。
【図27−2】図27は、2xMyc-β-TrCP1/Δ211-mKGCの増幅断片をXbaIIとPmeIで消化・切断したものを示す。
【図27−3】図27は、2xMyc-β-TrCP1/Δ211-mKGCの増幅断片をXbaIIとPmeIで消化・切断したものを示す。
【図28−1】図28は、mKGN-Cdc25B/N175-2xFLAGの増幅断片をEcoRVで消化・切断したものを示す。
【図28−2】図28は、mKGN-Cdc25B/N175-2xFLAGの増幅断片をEcoRVで消化・切断したものを示す。
【図29】図29は、リンカー導入pEF6プラスミドに導入されるリンカー部分の塩基配列およびアミノ酸配列を示す。
【図30】図30は、リンカー導入pEF6プラスミドの、リンカーコード配列の近傍の構造を示す。
【図31】図31は、JNK1依存的に緑色蛍光が出現したことを示す。
【図32】図32は、JNK1依存的に緑色蛍光が出現したことを示す。
【図33】図33は、非遺伝毒性ストレスに応答して緑色蛍光が出現したことを示す。
【図34】図33は、遺伝毒性ストレス因子を加えた場合であっても長時間経過後には緑色蛍光が出現したことを示す。
【配列表フリーテキスト】
【0113】
配列番号3:プラスミド1のFLAG領域
配列番号4:プラスミド1のFLAG領域
配列番号5:プライマー
配列番号6:プライマー
配列番号7:プライマー
配列番号8:プライマー
配列番号9:プライマー
配列番号10:2xFLAG/C断片
配列番号11:2xFLAG/C断片
配列番号12:断片1
配列番号13:断片1
配列番号14:断片2
配列番号15:断片2
配列番号16:断片4
配列番号17:断片4
配列番号18:プライマー
配列番号19:プライマー
配列番号20:mKGN断片とCdc25B/N175-2xFLAG間のリンカー
配列番号21:mKGN断片とCdc25B/N175-2xFLAG間のリンカー
配列番号22:プラスミド3中で2xFLAG-Cdc25B/N175-mKGNをコードする部分
配列番号23:プラスミド3中で2xFLAG-Cdc25B/N175-mKGNをコードする部分
配列番号24:プラスミド4中で2xFLAG-Cdc25B/N280-mKGNをコードする部分
配列番号25:プラスミド4中で2xFLAG-Cdc25B/N280-mKGNをコードする部分
配列番号26:プラスミド5中でmKGN-Cdc25B/N175-2xFLAGをコードする部分
配列番号27:プラスミド5中でmKGN-Cdc25B/N175-2xFLAGをコードする部分
配列番号30:β-TrCP1 が挿入されたpEF6/Myc-His Bプラスミドのβ-TrCP1上流部
配列番号31:β-TrCP1 が挿入されたpEF6/Myc-His Bプラスミドのβ-TrCP1上流部
配列番号32:プライマー
配列番号33:プライマー
配列番号34:プラスミド6のβ-TrCP1/Δ211上流部
配列番号35:プラスミド6のβ-TrCP1/Δ211上流部
配列番号36:プラスミド6のβ-TrCP1/Δ211下流部
配列番号37:プラスミド6のβ-TrCP1/Δ211下流部
配列番号38:プライマー
配列番号39:プライマー
配列番号40:プラスミド7中の2xMyc-β-TrCP1/Δ211-mKGC
配列番号41:プラスミド7中の2xMyc-β-TrCP1/Δ211-mKGC
配列番号42:プライマー
配列番号43:プライマー
配列番号44:2xFLAG-Cdc25B/N175-mKGN
配列番号45:2xFLAG-Cdc25B/N175-mKGN
配列番号46:プライマー
配列番号47:プライマー
配列番号48:2xMyc-β-TrCP1/Δ211-mKGC
配列番号49:2xMyc-β-TrCP1/Δ211-mKGC
配列番号50:プライマー
配列番号51:プライマー
配列番号52:mKGN-Cdc25B/N175-2xFLAG
配列番号53:mKGN-Cdc25B/N175-2xFLAG
配列番号54:リンカーを導入するためのオリゴヌクレオチド
配列番号55:リンカー
配列番号56:リンカー導入pEF6プラスミドのリンカーコード領域
配列番号57:リンカー導入pEF6プラスミドのリンカーコード領域
【特許請求の範囲】
【請求項1】
被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する方法であって、
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質とを同一の細胞内で発現させる発現工程と、
発現工程後の細胞に被験物質を作用させる作用工程と、
作用工程後の前記細胞において前記信号を測定し、測定された前記信号を指標にして、前記被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する評価工程とを含む前記方法。
【請求項2】
前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項1の方法。
【請求項3】
前記因子がJNK1キナーゼ、JNK2キナーゼ、p38キナーゼ、ERK1、ERK2またはCDKキナーゼである、請求項2の方法。
【請求項4】
Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、請求項2または3の方法。
【請求項5】
前記評価工程において前記因子を誘導する能力を有すると評価された被験物質を、遺伝毒性ストレス原因物質、非遺伝毒性ストレス原因物質、遺伝毒性ストレス原因物質の働きを軽減する物質、非遺伝毒性ストレス原因物質の働きを軽減する物質、または抗癌物質の候補物質として選抜するスクリーニング工程を更に含む、請求項2〜4のいずれかの方法。
【請求項6】
前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項1の方法。
【請求項7】
前記因子がp38キナーゼ、CHK1キナーゼ、CHK2キナーゼまたはMK2キナーゼである、請求項6の方法。
【請求項8】
前記評価工程において前記因子を誘導すると評価された被験物質を、遺伝毒性ストレス原因物質、遺伝毒性ストレス原因物質の働きを軽減する物質、または抗癌物質の候補物質として選抜するスクリーニング工程を更に含む、請求項6または7の方法。
【請求項9】
前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、請求項1〜8のいずれかの方法。
【請求項10】
β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、請求項9の方法。
【請求項11】
前記発現工程において、前記第1融合タンパク質と前記第2融合タンパク質とが、互いに連結された四成分融合タンパク質として細胞内で発現される、請求項1〜10のいずれかの方法。
【請求項12】
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質をコードするDNAと、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質をコードするDNAとの組み合わせ。
【請求項13】
前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項12の組み合わせ。
【請求項14】
Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、請求項13の組み合わせ。
【請求項15】
前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項12の組み合わせ。
【請求項16】
前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、請求項12〜15のいずれかの組み合わせ。
【請求項17】
β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、請求項16の組み合わせ。
【請求項18】
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質とが互いに連結された四成分融合タンパク質をコードするDNA。
【請求項19】
前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項18のDNA。
【請求項20】
Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、請求項19のDNA。
【請求項21】
前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項18のDNA。
【請求項22】
前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、請求項18〜21のいずれかのDNA。
【請求項23】
β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、請求項22のDNA。
【請求項24】
請求項12〜17のいずれかにおいて定義される第1融合タンパク質をコードするDNAを含むベクターと、請求項12〜17のいずれかにおいて定義される前記第2融合タンパク質をコードするDNAを含むベクターとの組み合わせ。
【請求項25】
請求項18〜23のいずれかのDNAを含むベクター。
【請求項26】
請求項24のベクターの組み合わせにより形質転換された形質転換細胞。
【請求項27】
請求項25のベクターにより形質転換された形質転換細胞。
【請求項28】
請求項12〜23のいずれかの組み合わせ、またはDNAが導入された細胞。
【請求項29】
請求項28の細胞を体内に含む動物。
【請求項30】
請求項12〜17のいずれかのDNAの組み合わせによりコードされる、第1融合タンパク質と第2融合タンパク質との組み合わせ。
【請求項31】
請求項18〜23いずれかのDNAによりコードされる、四成分融合タンパク質。
【請求項1】
被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する方法であって、
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質とを同一の細胞内で発現させる発現工程と、
発現工程後の細胞に被験物質を作用させる作用工程と、
作用工程後の前記細胞において前記信号を測定し、測定された前記信号を指標にして、前記被験物質が、Cdc25AまたはCdc25Bの少なくとも一部分とβ-TrCP1またはβ-TrCP2の少なくとも一部分との相互作用を惹起する因子を細胞内において誘導する能力を評価する評価工程とを含む前記方法。
【請求項2】
前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項1の方法。
【請求項3】
前記因子がJNK1キナーゼ、JNK2キナーゼ、p38キナーゼ、ERK1、ERK2またはCDKキナーゼである、請求項2の方法。
【請求項4】
Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、請求項2または3の方法。
【請求項5】
前記評価工程において前記因子を誘導する能力を有すると評価された被験物質を、遺伝毒性ストレス原因物質、非遺伝毒性ストレス原因物質、遺伝毒性ストレス原因物質の働きを軽減する物質、非遺伝毒性ストレス原因物質の働きを軽減する物質、または抗癌物質の候補物質として選抜するスクリーニング工程を更に含む、請求項2〜4のいずれかの方法。
【請求項6】
前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項1の方法。
【請求項7】
前記因子がp38キナーゼ、CHK1キナーゼ、CHK2キナーゼまたはMK2キナーゼである、請求項6の方法。
【請求項8】
前記評価工程において前記因子を誘導すると評価された被験物質を、遺伝毒性ストレス原因物質、遺伝毒性ストレス原因物質の働きを軽減する物質、または抗癌物質の候補物質として選抜するスクリーニング工程を更に含む、請求項6または7の方法。
【請求項9】
前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、請求項1〜8のいずれかの方法。
【請求項10】
β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、請求項9の方法。
【請求項11】
前記発現工程において、前記第1融合タンパク質と前記第2融合タンパク質とが、互いに連結された四成分融合タンパク質として細胞内で発現される、請求項1〜10のいずれかの方法。
【請求項12】
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質をコードするDNAと、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質をコードするDNAとの組み合わせ。
【請求項13】
前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項12の組み合わせ。
【請求項14】
Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、請求項13の組み合わせ。
【請求項15】
前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項12の組み合わせ。
【請求項16】
前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、請求項12〜15のいずれかの組み合わせ。
【請求項17】
β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、請求項16の組み合わせ。
【請求項18】
Cdc25AまたはCdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された第1融合タンパク質と、β-TrCP1またはβ-TrCP2の少なくとも一部分と第2検出用タンパク質とが連結された第2融合タンパク質であって、前記第2検出用タンパク質が、前記第1検出用タンパク質と相互作用したときに識別可能な信号を発することができるタンパク質である、前記第2融合タンパク質とが互いに連結された四成分融合タンパク質をコードするDNA。
【請求項19】
前記第1融合タンパク質が、Cdc25Bの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項18のDNA。
【請求項20】
Cdc25Bの少なくとも一部分が、配列番号2で表されるタンパク質の一部分であって、第94〜108番のアミノ酸配列を少なくとも含む、請求項19のDNA。
【請求項21】
前記第1融合タンパク質が、Cdc25Aの少なくとも一部分と第1検出用タンパク質とが連結された融合タンパク質である、請求項18のDNA。
【請求項22】
前記第2融合タンパク質が、β-TrCP1の少なくとも一部分と第2検出用タンパク質とが連結された融合タンパク質である、請求項18〜21のいずれかのDNA。
【請求項23】
β-TrCP1の少なくとも一部分が、配列番号29で表されるタンパク質の一部分であって、第146〜192番のアミノ酸からなる領域の一部または全部を含む領域を含まず、なおかつCdc25AまたはCdc25Bと結合可能である、請求項22のDNA。
【請求項24】
請求項12〜17のいずれかにおいて定義される第1融合タンパク質をコードするDNAを含むベクターと、請求項12〜17のいずれかにおいて定義される前記第2融合タンパク質をコードするDNAを含むベクターとの組み合わせ。
【請求項25】
請求項18〜23のいずれかのDNAを含むベクター。
【請求項26】
請求項24のベクターの組み合わせにより形質転換された形質転換細胞。
【請求項27】
請求項25のベクターにより形質転換された形質転換細胞。
【請求項28】
請求項12〜23のいずれかの組み合わせ、またはDNAが導入された細胞。
【請求項29】
請求項28の細胞を体内に含む動物。
【請求項30】
請求項12〜17のいずれかのDNAの組み合わせによりコードされる、第1融合タンパク質と第2融合タンパク質との組み合わせ。
【請求項31】
請求項18〜23いずれかのDNAによりコードされる、四成分融合タンパク質。
【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11−1】


【図11−2】


【図11−3】


【図12】


【図13】


【図14】


【図15−1】


【図15−2】


【図16】


【図17】


【図18−1】


【図18−2】


【図19−1】


【図19−2】


【図19−3】


【図20−1】


【図20−2】


【図21−1】


【図21−2】


【図21−3】


【図22】


【図23】


【図24】


【図25−1】


【図25−2】


【図25−3】


【図26−1】


【図26−2】


【図27−1】


【図27−2】


【図27−3】


【図28−1】


【図28−2】


【図29】


【図30】


【図31】


【図32】


【図33】


【図34】




【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図9】


【図10】


【図11−1】


【図11−2】


【図11−3】


【図12】


【図13】


【図14】


【図15−1】


【図15−2】


【図16】


【図17】


【図18−1】


【図18−2】


【図19−1】


【図19−2】


【図19−3】


【図20−1】


【図20−2】


【図21−1】


【図21−2】


【図21−3】


【図22】


【図23】


【図24】


【図25−1】


【図25−2】


【図25−3】


【図26−1】


【図26−2】


【図27−1】


【図27−2】


【図27−3】


【図28−1】


【図28−2】


【図29】


【図30】


【図31】


【図32】


【図33】


【図34】


【公開番号】特開2010−124818(P2010−124818A)
【公開日】平成22年6月10日(2010.6.10)
【国際特許分類】
【出願番号】特願2008−306804(P2008−306804)
【出願日】平成20年12月1日(2008.12.1)
【新規性喪失の例外の表示】特許法第30条第1項適用申請有り ▲1▼日本生化学会北陸支部 第26回大会にて発表 刊行物名:日本生化学会北陸支部 第26回大会 要旨集及び総会資料 発行者 :社団法人 日本生化学会北陸支部 開催日 :平成20年5月31日(土) ▲2▼第60回日本細胞生物学会大会にて発表 刊行物名:第60回日本細胞生物学会大会要旨集 発行者 :第60回日本細胞生物学会大会 組織委員会 開催日 :平成20年6月29日(日)〜平成20年7月1日(火) 発表日 :平成20年6月29日(日) 講演番号:1P−056 講演要旨集発行日:平成20年6月5日 ▲3▼第67回 日本癌学会学術総会にて発表 刊行物名:第67回 日本癌学会学術総会記事 発行者 :日本癌学会 開催日 :平成20年10月28日(火)〜30日(木) 発表日 :平成20年10月28日(火) 講演番号:P−1224 講演要旨集発行日:平成20年9月30日 ▲4▼8th International Conference on Protein Phosphatasesにて発表 刊行物名:8th International Conference on Protein Phosphatasesプログラム 発行者 :日本プロテインホスファターゼ研究会 開催日 :平成20年11月12日(水)〜14日(金) 発表日 :平成20年11月12日(水) 講演番号:Session III−4 ▲5▼BMB2008 (第31回日本分子生物学会年会・第81回日本生化学会大会 合同大会)にて発表 刊行物名:BMB2008 (第31回日本分子生物学会年会・第81回日本生化学会大会 合同大会)講演要旨集、及びプログラム 発行者 :BMB2008 (第31回日本分子生物学会年会・第81回日本生化学会大会 合同大会) 開催日 :平成20年12月9日(火)〜12日(金) 発表日 :平成20年12月10日(水)・11日(木) 講演番号:3S22−3、2P−0492(3T11−5) 講演要旨集発行日:平成20年11月20日
【出願人】(504160781)国立大学法人金沢大学 (282)
【Fターム(参考)】
【公開日】平成22年6月10日(2010.6.10)
【国際特許分類】
【出願日】平成20年12月1日(2008.12.1)
【新規性喪失の例外の表示】特許法第30条第1項適用申請有り ▲1▼日本生化学会北陸支部 第26回大会にて発表 刊行物名:日本生化学会北陸支部 第26回大会 要旨集及び総会資料 発行者 :社団法人 日本生化学会北陸支部 開催日 :平成20年5月31日(土) ▲2▼第60回日本細胞生物学会大会にて発表 刊行物名:第60回日本細胞生物学会大会要旨集 発行者 :第60回日本細胞生物学会大会 組織委員会 開催日 :平成20年6月29日(日)〜平成20年7月1日(火) 発表日 :平成20年6月29日(日) 講演番号:1P−056 講演要旨集発行日:平成20年6月5日 ▲3▼第67回 日本癌学会学術総会にて発表 刊行物名:第67回 日本癌学会学術総会記事 発行者 :日本癌学会 開催日 :平成20年10月28日(火)〜30日(木) 発表日 :平成20年10月28日(火) 講演番号:P−1224 講演要旨集発行日:平成20年9月30日 ▲4▼8th International Conference on Protein Phosphatasesにて発表 刊行物名:8th International Conference on Protein Phosphatasesプログラム 発行者 :日本プロテインホスファターゼ研究会 開催日 :平成20年11月12日(水)〜14日(金) 発表日 :平成20年11月12日(水) 講演番号:Session III−4 ▲5▼BMB2008 (第31回日本分子生物学会年会・第81回日本生化学会大会 合同大会)にて発表 刊行物名:BMB2008 (第31回日本分子生物学会年会・第81回日本生化学会大会 合同大会)講演要旨集、及びプログラム 発行者 :BMB2008 (第31回日本分子生物学会年会・第81回日本生化学会大会 合同大会) 開催日 :平成20年12月9日(火)〜12日(金) 発表日 :平成20年12月10日(水)・11日(木) 講演番号:3S22−3、2P−0492(3T11−5) 講演要旨集発行日:平成20年11月20日
【出願人】(504160781)国立大学法人金沢大学 (282)
【Fターム(参考)】
[ Back to top ]

