DNA二本鎖切断損傷の解析装置及び解析方法
【課題】細胞で生じるDNA損傷を迅速かつ定量的に測定できる解析技術を提供する。
【解決手段】DNA解析装置(1)は、DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で撮影された画像を取得する画像取り込み手段(25)と、取得した画像を解析する画像解析手段(15)とを備える。画像解析手段(15)は、時系列で撮影された画像の各々について、各画像に含まれる細胞核の画像の領域を検出し、検出した領域に含まれる、第1の蛍光タンパク質の発光による発光スポットの数をカウントし、さらに、検出した領域に含まれる、第1の蛍光タンパク質の発光による発光領域の面積を求め、時系列で求めた発光スポットのカウント値及び発光領域の面積のデータを解析データとして生成する。
【解決手段】DNA解析装置(1)は、DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で撮影された画像を取得する画像取り込み手段(25)と、取得した画像を解析する画像解析手段(15)とを備える。画像解析手段(15)は、時系列で撮影された画像の各々について、各画像に含まれる細胞核の画像の領域を検出し、検出した領域に含まれる、第1の蛍光タンパク質の発光による発光スポットの数をカウントし、さらに、検出した領域に含まれる、第1の蛍光タンパク質の発光による発光領域の面積を求め、時系列で求めた発光スポットのカウント値及び発光領域の面積のデータを解析データとして生成する。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、細胞のDNA二本鎖切断損傷を定量的に解析する装置及び方法に関する。
【背景技術】
【0002】
がんは我が国の死因の第1位であり、生涯のうちにがんに罹患する可能性は、おおよそ2人に1人とされる。がんは加齢により発症リスクが高まり、高齢化社会の進行にともない罹患者・死者数は今後とも増加していくと推測される。そのような状況で日本におけるがん治療は、これまでの手術療法を中心としたものであったが、今後は化学療法、放射線療法により重きが置かれると考えられている。
【発明の概要】
【発明が解決しようとする課題】
【0003】
がんの放射線治療や化学療法剤の多くはがん細胞のDNAを標的とし、その損傷を引き起こすことによりがん細胞を死滅させる。がん細胞で起こるDNA損傷を生きたまま鋭敏に観察・評価できれば、迅速に新規の抗がん剤のスクリーニングが可能となり、また放射線照射の有効性を評価でき、新規抗がん剤や放射線治療の増感補助薬の開発の迅速化を図ることができる。
【0004】
本発明は、上記のような課題を鑑み、細胞で生じるDNA損傷を迅速かつ定量的に測定できる解析技術を提供することを目的とする。
【課題を解決するための手段】
【0005】
本発明のDNA解析装置は、DNA二本鎖切断損傷を定量的に解析する装置であって、DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で撮影された画像を取得する画像取り込み手段と、取得した画像を解析する画像解析手段とを備える。画像解析手段は、時系列で撮影された画像の各々について、各画像に含まれる細胞核の画像の領域を検出し、検出した領域に含まれる、第1の蛍光タンパク質の発光による発光スポットの数をカウントし、さらに、検出した領域に含まれる、第1の蛍光タンパク質の発光による発光領域の面積を求め、時系列で求めた発光スポットのカウント値及び発光領域の面積のデータを解析データとして生成する。
【0006】
さらに、取得した画像は、第1の蛍光タンパク質に加えて、細胞周期のS期にある細胞を標識する第2の蛍光タンパク質を観察対象の細胞に導入して撮影された画像であってもよい。この場合、画像解析手段は、第1の蛍光タンパク質の発光による発光スポットのカウント及び発光領域の面積の算出に加えて、さらに、第2の蛍光タンパク質の発光による発光スポットの数をカウントする。
【0007】
本発明に係る解析方法は、DNA二本鎖切断損傷を定量的に解析する方法であって、
DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で画像を撮影し、
時系列で撮影された複数の画像の各々について、
各画像に含まれる細胞核の画像の領域を検出し、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光スポットの数をカウントし、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光領域の面積を求め、
時系列で求めた前記発光スポットのカウント値及び前記発光領域の面積のデータを解析データとして生成する。
【発明の効果】
【0008】
本発明によれば、蛍光タンパク質をバイオセンサーとして用いて、細胞核においてDNA損傷が生じた箇所を検出する。具体的には、細胞核を時系列で撮影した画像を解析して、蛍光発光による発光スポットの数、発光領域の面積を求めることで、DNAの損傷の程度を定量的に測定する。これにより、DNAの損傷及び損傷の修復の時間的な変化を定量的にかつ迅速に測定することが可能となる。このようなDNA損傷解析技術を利用することで、がん細胞で起こるDNA損傷を生きたまま鋭敏に観察・評価でき、迅速に新規の抗がん剤のスクリーニングが可能となり、また放射線照射の有効性を評価でき、新規抗がん剤や放射線治療の増感補助薬の開発の迅速化を図ることができる。
【図面の簡単な説明】
【0009】
【図1】DNA二本鎖切断とDNA損傷応答を説明した図
【図2】GFP−53BP1タンパク質の蛍光を利用して、細胞核のDNA損傷の状態を撮影した画像((a)DNA損傷がない場合、(b)DNA損傷がある場合)
【図3】細胞周期を説明した図
【図4】(a)緑色蛍光タンパク質であるGFP−53BP1タンパク質をバイオセンサーとして導入し、細胞核の状態を撮影した画像であり、(b)赤色蛍光タンパク質であるDsRed−PCNAタンパク質をバイオセンサーとして導入し、細胞核の状態を撮影した画像
【図5】細胞にDNA損傷を与えた後のバイオセンサーによる発光状態の時間経過に伴う変化を撮影した画像
【図6】z軸方向における複数の平面でスライスして細胞核の画像データを取得する方法を説明した図
【図7】DNA損傷解析装置の構成を示す図
【図8】顕微鏡により時系列で撮影された細胞画像(画像の一部)の例を示す図
【図9】DNA損傷解析装置の解析処理を示すフローチャート
【図10】画像データにおいて細胞核を特定する処理を説明するための図
【図11】画像データにおける発光スポット数のカウント及び発光領域の面積の算出処理を説明するための図
【図12】DNA損傷解析装置により得られた解析データをグラフ表示した図(抗がん剤として、NCS(Neocarzinostanin)を投与した場合)
【図13】DNA損傷解析装置により得られた解析データをグラフ表示した図(抗がん剤として、CPT(Camptothecin)を投与した場合)
【発明を実施するための形態】
【0010】
以下、添付の図面を参照して本発明の実施の形態を説明する。
【0011】
1.DNA2本鎖切断損傷の検出方法の基本原理
図1を参照し、本実施形態におけるDNA2本鎖切断損傷を検出する方法の基本原理について説明する。
【0012】
通常、がんに対して放射線治療あるいは化学療法を行うと、DNAに損傷が発生する。そのうち最も細胞にダメージを与える損傷がDNA2本鎖切断である。2本鎖切断が起きると、図1に示すように、細胞の中では、「ATM」と呼ばれるタンパク質キナーゼが活性化され、種々のタンパク質が連鎖的に反応を起こし、最終的にDNAが巻き付いているヒストン100にメチル化などの変化が起こり、53BP1タンパク質が集積する。本実施形態では、このようなDNA損傷箇所に53BP1タンパク質が集積するという現象を利用して、DNA2本鎖切断損傷を検出する。
【0013】
ここで、DNA2本鎖切断損傷を検出するためのバイオセンサーについて説明する。バイオセンサーの基本技術として、2008年ノーベル化学賞受賞対象となった緑色蛍光タンパク質GFPを用いている。特に、本発明者は、DNA2本鎖切断損傷に特異的に集積するように53BP1タンパク質に改変を加え、ヒトがん培養細胞内で安定に発現できるGFP−53BP1融合タンパク質(以下「GFP−53BP1タンパク質」という)をバイオセンサーとして用いた。これにより、細胞が生きたままの状態でDNA損傷を蛍光顕微鏡で緑色のスポットとして観察することを可能とした。
【0014】
図1では、便宜上、一つの53BP1しか示していないが、実際は数百の分子が集積する。このため、DNA損傷箇所は、光のスポットとして観察することが出来る。図2(a)は、DNAの損傷がない細胞核の状態を示した画像であり、図2(b)は、8Gy(8Sv)の放射線により損傷が与えられたDNAの細胞核の状態を示した画像である。図2(b)に示すように、8Gy(8Sv)の放射線によりDNAの複数箇所に損傷が発生すると、それらの損傷箇所に集積したGFP−53BP1タンパク質が蛍光発光することにより光スポットとして観察される。
【0015】
さらに、本実施形態では、GFP−53BP1タンパク質に加えて、観察対象の細胞の細胞周期を検出できるセンサータンパク質を一緒に導入している。
【0016】
ここで、細胞周期について説明する。細胞周期とは、増殖・分裂する細胞が一連の過程を経て2つの細胞に分裂する全行程である(図3参照)。細胞周期は、G0期、G1期、S期、G2期、M期の5つの期を有している。分裂する細胞は、G1期、S期、G2期、M期の順に変化する。また、分裂・増殖せずに休止期にある細胞(多くの正常な体細胞)は、G0期に停止している。特に、S期では、活発に増殖・分裂を繰り返すがん細胞のDNA複製が行われており、このS期におけるDNA損傷を検出することは、DNA損傷を誘導する薬剤のスクリーニングにおいてきわめて有用である。
【0017】
具体的には、細胞分裂に際してDNAを複製している細胞周期のS期を標識するPCNA−DsRedタンパク質をセンサーとして観察対象の細胞に導入する。PCNA−DsRedタンパク質は赤色蛍光を示すタンパク質である。これにより、活発に分裂する細胞のS期にある細胞が、赤いスポットを多量に含む細胞として標識される。その結果、がん細胞を観察対象とした場合、がん細胞がS期にあるときに、DNA損傷を引き起こす抗がん剤の候補を迅速にスクリーニングできる。正常細胞は通常分裂を停止して静止状態にあるものが多く、活発に分裂するS期細胞のみで未知の薬剤がDNA損傷を引き起こすことが認識できれば、正常細胞に影響の少ない(副作用の少ない)抗がん剤の発見につながる。
【0018】
図4(a)は、緑色蛍光タンパク質であるGFP−53BP1タンパク質をバイオセンサーとして導入し、細胞核の状態を撮影した画像であり、図4(b)は、図4(a)に示す細胞と同じ細胞について、赤色蛍光タンパク質であるDsRed−PCNAタンパク質をバイオセンサーとして導入し、細胞核の状態を撮影した画像である。図4(b)に示すように、S期にある細胞核の全体において、赤色蛍光タンパク質であるDsRed−PCNAタンパク質による発光スポットが散在していることが分かる。
【0019】
図5は、細胞にDNA損傷を与えた後のバイオセンサーによる発光状態の時間経過に伴う変化を撮影した図である。図5の(a)〜(g)は、それぞれ別々の細胞核の時間変化(損傷前、損傷直後、1分後、2分後、3分後)を示している。ここでは、レーザー照射によりDNA損傷を発生させている。図5の(a)〜(g)より、レーザーが照射されDNA損傷が発生した部分において、時間経過とともに、発光スポットが増加している様子、すなわち、GFP−53BP1が集積している様子が理解できる。このように、損傷を生きたまま観察できることが分かる。
【0020】
本実施形態では、以上のようにGFP−53BP1タンパク質及びDsRed−PCNAタンパク質のような蛍光タンパク質をバイオセンサーとして細胞に加え、その発光状態を撮影し、撮影した画像を解析することによりDNA損傷の状態を検出する。
【0021】
また、細胞は3次元形状を有することから、図6に示すように、z軸方向における複数の平面でスライスした画像データを取得するようにしてもよい。この場合、撮影された画像情報は、細胞の3次元の座標情報に加えて、時間情報、緑/赤の2つの蛍光色情報を含めた5次元の画像情報となる。このような5次元の画像情報を利用することで、3次元的に存在する発光スポットをより精度よく検出でき、結果としてより精度のよい解析が可能となる。
【0022】
2.DNA損傷解析装置の構成
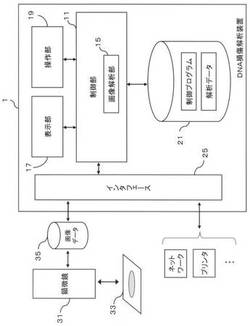
図7に、本実施形態のDNA損傷解析装置の構成の一例を示す。DNA損傷解析装置1は例えばパーソナルコンピュータのような情報処理装置で構成される。DNA損傷解析装置1は、顕微鏡31により撮影されて記録された画像情報を入力し、画像解析することによりDNAの損傷状態を定量的に解析する。
【0023】
DNA損傷解析装置1は、その全体動作を制御する制御部11と、画面表示を行う表示部17と、ユーザが操作を行う操作部19と、データやプログラムを記憶するデータ格納部21とを備える。例えば、表示部17は液晶ディスプレイで構成され、操作部19はキーボードやマウス等である。さらに、DNA損傷解析装置1は、外部機器やネットワークと接続するためのインタフェース25を含む。DNA損傷解析装置1は例えばパーソナルコンピュータ等で実現できる。
【0024】
制御部11は、CPUやMPUからなり、制御プログラムを実行することで後述する解析処理を実現する。制御部11で実行される制御プログラムは、データ格納部21に格納されている。制御プログラムは、通信回線を通じて又は光ディスクや半導体メモリ等の記録媒体を介して提供されてもよい。
【0025】
データ格納部21はデータやプログラムを記憶する手段であり、例えばハードディスク、半導体メモリ等で構成できる。データ格納部21は、制御部11で実行されるプログラムや解析後のデータ等を格納する。
【0026】
顕微鏡31は観察対象33の拡大画像を撮影可能であり、少なくとも2種類の波長の画像を撮影可能である。顕微鏡31は動画像または静止画の撮影が可能である。顕微鏡31により撮影された画像は、所定の記録媒体(ハードディスク装置、光ディスク記録装置、サーバ装置等)35に記録される。なお、記録媒体35は顕微鏡31内部に設けられても良い。または、顕微鏡31により撮影された画像は、インタフェース25を介してDNA損傷解析装置1に入力され、DNA損傷解析装置1内のデータ格納部21に記録されるようにしてもよい。また、顕微鏡31は、フォーカス位置を変更することで、図6に示すような、z方向に異なる位置を有する複数のスライス面上で画像データを撮影することが可能となる。
【0027】
3.DNA損傷解析処理
以上のように構成されるDNA損傷解析装置1によるDNAの損傷状態の解析処理について説明する。
【0028】
3.1 画像撮影
前述の2種の蛍光タンパク質をバイオセンサーとして導入して、顕微鏡31により細胞の画像を生きたまま撮影する。画像撮影中、所定のタイミングで、検査したい薬物を投与する。薬物の投与前から投与後所定時間が経過するまで、細胞の画像を、時間経過を追って撮影し、記録する。なお、以下の説明では、顕微鏡31により、所定の時間間隔毎に、緑色の蛍光を示すGFP−53BP1タンパク質による発光状態を撮影した画像(静止画)と、赤色の蛍光を示すDsRed−PCNAタンパク質による発光状態を撮影した画像(静止画)とが撮影され、記録媒体35に記録されるとする。また、顕微鏡31により、1つのスライス面での画像のみが撮影、記録されるとする。すなわち、顕微鏡31により、緑色の蛍光画像及び赤色の蛍光画像それぞれについて一連の複数枚の静止画が時系列で撮影され、記録媒体35に記録される。なお、本実施形態では、細胞の静止画を撮影したが、動画を撮影し、動画を構成するフレーム画像毎に解析処理を行っても良い。
【0029】
なお、顕微鏡31のフォーカスを調整しながら、図6に示すようにz方向において位置が異なる複数のスライス平面上で画像を撮影してもよい。
【0030】
図8は、顕微鏡31により時系列で撮影された細胞画像(画像の一部)の例を示す図である。図8において、(a1)〜(a4)は、緑色の蛍光を示すGFP−53BP1タンパク質による発光状態を撮影した蛍光画像である。各列(a1)〜(a4)においては上から下に時間が経過し、(a1)、(a2)、(a3)、(a4)の順に時間が経過している。図8において、(b1)〜(b4)は、赤色の蛍光を示すDsRed−PCNAタンパク質による発光状態を撮影した蛍光画像である。各列(b1)〜(b4)においては上から下に時間が経過し、(b1)、(b2)、(b3)、(b4)の順に時間が経過している。
【0031】
3.2 画像解析
顕微鏡31により撮影され、記録された2種の蛍光画像は、インタフェース25を介してDNA損傷解析装置1に入力され、時間経過とともに細胞核で変化する緑色ならびに赤色のスポットが定量解析される。以下、DNA損傷解析装置1による画像解析の詳細を説明する。なお、記録媒体35には、顕微鏡31により撮影された、緑色の蛍光画像及び赤色の蛍光画像それぞれについての一連の複数の静止画が時系列で予め記録されており、DNA損傷解析装置1は記録媒体35から画像を読み出しながら以下の処理を実行する。
【0032】
図9は、DNA損傷解析装置1の画像解析処理を示したフローチャートである。DNA損傷解析装置1は、インタフェース25を介して記録媒体35に記録された画像の中から1つの画像を取り込む(S11)。具体的には、DNA損傷解析装置1は、記録媒体35に記録された画像において時系列で最初の画像を取り込む。
【0033】
制御部11は、取り込んだ画像を画像解析し、画像中の細胞核を特定する(S12)。次に、制御部11は、特定した細胞核について、蛍光発光による発光スポットの数をカウントするとともに、発光領域の面積を求める(S13)。これらの処理の詳細は後述する。なお、発光領域の面積については、GFP−53BP1タンパク質による緑色の蛍光画像に対してのみ求める。
【0034】
制御部11は、発光スポットのカウント数及び発光領域の面積を細胞核と関連づけて解析データとして生成し、データ格納部21に出力して記録する(S14)。画像内の1つの細胞核について、上記の処理(S13、S14)が終了すると、ステップS12に戻り、画像内に他の細胞核が含まれるか否かを検査し、他の細胞核が特定されたときは、発光スポット数のカウント、発光領域の面積の算出等の処理(S13)を行う。
【0035】
取り込んだ画像に含まれる全ての細胞核について、細胞核の特定、発光スポット数のカウント、発光領域の面積の算出(S12〜S14)を実行する(S15)。これらの処理が終了すると、記録媒体35に記録された一連の画像において時系列で次の画像について同様の処理を行い、記録媒体35に記録された全ての画像について上記の処理を行う(S11〜S16)。
【0036】
ここで、本実施形態では、顕微鏡31により、生きている細胞の画像を撮影しているが、生きている細胞は時間とともに除々に移動し、変形する。このため、前回処理された画像(静止画)における細胞核と、今回処理された画像における細胞核とを同定する必要がある。この同定は、例えば、細胞核の形状を考慮することで実現できる。例えば、公知の顔認識技術と同様の技術を用いて、細胞核の形状(特徴)に基づいて時間的に除々に移動、変化する細胞核をトラッキングすることができる。
【0037】
なお、上記の例では、時系列で連続して撮影された一連の画像を連続的に処理(ステップS13)したが、所定の時間間隔毎の画像のみを抽出して処理するようにしてもよい。
【0038】
また、図6に示すように、z方向において位置が異なる複数のスライス平面上で画像が撮影されている場合は、各スライス平面の画像データをそれぞれ解析し、得られた発光スポット数や発光領域の面積の値を合算してもよい。または、複数のスライス平面上の画像の中で最も好適と考えられる1つの画像について上記の処理(ステップS12〜14)を実施するようにしてもよい。
【0039】
3.2.1 細胞核の特定、発光スポット数のカウント、発光領域の面積の算出
図10、図11を参照して、上記の、細胞核の特定処理(ステップS12)、並びに、発光スポット数のカウント及び発光領域の面積の算出処理(ステップS13)の詳細を説明する。これらの処理(ステップS12、S13)は特に制御部11の画像解析部15により実行される。
【0040】
図10(a)は、記録媒体35から取り込まれた一の画像を示す。画像解析部15は、まず、図10(a)に示す画像を、画素値(輝度)に関する所定の閾値に基づき二値化し、図10(b)に示すような二値化画像を生成する。制御部11は、二値化画像において、同じ輝度を含む連続した画素からなる領域51を特定する。次に、画像解析部15は特定した領域51を含む矩形枠55を設定する(図10(c)参照)。そして、画像解析部15は、図10(d)に示すように、撮影された画像上に矩形枠55を設定し、矩形枠55で囲まれる領域の画像を切り出す(図10(e)参照)。
【0041】
上記のようにして切り出された領域に対して、画像解析部15は、発光スポット数のカウント及び発光領域の面積の算出を行う。図11(a)は、上記の方法で切り出された画像である(図10(e)と同様)。画像解析部15は、切り出された画像(図11(a)参照)において、図11(b)に示すように細胞核の領域を特定し、特定した細胞核の背景の画像を削除する。図11(c)は、特定した細胞核の背景の画像が削除された画像を示す。画像解析部15は、細胞核の背景の画像が削除された画像において、所定の画素値(輝度)以上の値を持つ領域(発光スポット)を特定し(図11(d)参照)、その領域の数をカウントすることで、発光スポットの数をカウントする。さらに、画像解析部15は、所定の画素値(輝度)以上の値を持つ画素の数をカウントすることで、発光領域の面積を求める。
【0042】
上記のようにして、細胞核の特定、発光スポット数のカウント及び発光領域の面積の算出処理(ステップS12、S13)が実行される。
【0043】
なお、本実施形態では、発光スポット数及び発光領域の面積の双方を測定している。画像の状況によって、発光スポット数または発光領域の面積が良好に観察できないことがある。例えば、ざらついた画像においては、発光スポット数よりも発光面積の方が良好に測定できる。よって、発光スポット数及び発光領域の面積の双方を測定しておくことで、画像の状況に応じて適宜良好な方のデータを選択することが可能となる。
【0044】
4.解析データのグラフ化
以上のようにして、DNA損傷解析装置1によるDNAの損傷状態の解析処理が実行されることで、細胞核毎に、発光スポットの数及び発光領域の面積のデータを時系列で求めることができる。このようにして求められたデータは、解析データとしてデータ格納部21に格納される。
【0045】
図12及び図13は、DNA損傷解析装置1により以上のようにして得られた解析データをグラフとして表示したものである。図12は、抗がん剤としてNCS(Neocarzinostanin)を投与した際のDNA損傷の状況についての、DNA損傷解析装置1による解析結果をグラフ化した図である。図13は、抗がん剤としてCPT(Camptothecin)を投与した際のDNA損傷の状況についての、DNA損傷解析装置1による解析結果をグラフ化した図である。図中、「DNA損傷の数」は、GFP−53BP1タンパク質による発光スポット数の時間変化をプロットしたものであり、「DNA損傷の占める面積」は、GFP−53BP1タンパク質による発光領域の面積の時間変化をプロットしたものである。また、「S期のDNA複製スポット数」は、DsRed−PCNAタンパク質による発光スポット数の時間変化をプロットしたものである。
【0046】
図12、13において、時間GFP−53BP1タンパク質による発光スポット数の時間変化(実線X)及び発光領域の面積の時間変化(実線Y)を参照すると、DNA損傷やその修復状況の時間的な変化を把握することができる。また、DsRed−PCNAタンパク質による発光スポット数(実線Z)の時間変化を参照することで、細胞周期のS期の範囲を推定することができる。よって、DsRed−PCNAタンパク質による発光スポット数(実線Z)の時間変化とともに、時間GFP−53BP1タンパク質による発光スポット数の時間変化(実線X)及び発光領域の面積の時間変化(実線Y)を観察することで、S期におけるDNA損傷及び損傷の修復状態の時間的変化を把握することができる。
【0047】
5.まとめ
以上のように、本実施形態のDNA損傷解析装置1によれば、細胞核において発生したDNA損傷の程度を定量的に測定することができる。具体的には、DNA損傷部位に集積する蛍光タンパク質をバイオセンサーとして用いて、細胞核においてDNA損傷が生じた箇所を検出する。具体的には、細胞核を撮影した画像を解析して、蛍光発光による発光スポットの数及び発光領域の面積を求めることで、DNAの損傷の程度を定量的に測定する。また、DNA損傷部位に集積する蛍光タンパク質に加えて、細胞周期のS期を標識する蛍光タンパク質をもバイオセンサーとして用いることで、細胞周期のS期を検出する。このような定量的な測定を時系列で行うことにより、DNAの損傷及び損傷の修復の時間的な変化を定量的に測定することが可能となり、生きた細胞におけるDNA損傷の時間的な変化の定量的な観察が可能となる。このようなDNA損傷解析技術を利用することで、がん細胞で起こるDNA損傷を生きたまま鋭敏に観察・評価でき、迅速に新規の抗がん剤のスクリーニングが可能となり、また放射線照射の有効性を評価でき、新規抗がん剤や放射線治療の増感補助薬の開発の迅速化を図ることができる。
【符号の説明】
【0048】
1 DNA損傷解析装置
11 制御部
17 表示部
19 操作部
21 データ格納部
25 インタフェース
31 顕微鏡
33 観察対象
35 記録媒体
【先行技術文献】
【特許文献】
【0049】
【特許文献1】特表2005−502323号公報
【技術分野】
【0001】
本発明は、細胞のDNA二本鎖切断損傷を定量的に解析する装置及び方法に関する。
【背景技術】
【0002】
がんは我が国の死因の第1位であり、生涯のうちにがんに罹患する可能性は、おおよそ2人に1人とされる。がんは加齢により発症リスクが高まり、高齢化社会の進行にともない罹患者・死者数は今後とも増加していくと推測される。そのような状況で日本におけるがん治療は、これまでの手術療法を中心としたものであったが、今後は化学療法、放射線療法により重きが置かれると考えられている。
【発明の概要】
【発明が解決しようとする課題】
【0003】
がんの放射線治療や化学療法剤の多くはがん細胞のDNAを標的とし、その損傷を引き起こすことによりがん細胞を死滅させる。がん細胞で起こるDNA損傷を生きたまま鋭敏に観察・評価できれば、迅速に新規の抗がん剤のスクリーニングが可能となり、また放射線照射の有効性を評価でき、新規抗がん剤や放射線治療の増感補助薬の開発の迅速化を図ることができる。
【0004】
本発明は、上記のような課題を鑑み、細胞で生じるDNA損傷を迅速かつ定量的に測定できる解析技術を提供することを目的とする。
【課題を解決するための手段】
【0005】
本発明のDNA解析装置は、DNA二本鎖切断損傷を定量的に解析する装置であって、DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で撮影された画像を取得する画像取り込み手段と、取得した画像を解析する画像解析手段とを備える。画像解析手段は、時系列で撮影された画像の各々について、各画像に含まれる細胞核の画像の領域を検出し、検出した領域に含まれる、第1の蛍光タンパク質の発光による発光スポットの数をカウントし、さらに、検出した領域に含まれる、第1の蛍光タンパク質の発光による発光領域の面積を求め、時系列で求めた発光スポットのカウント値及び発光領域の面積のデータを解析データとして生成する。
【0006】
さらに、取得した画像は、第1の蛍光タンパク質に加えて、細胞周期のS期にある細胞を標識する第2の蛍光タンパク質を観察対象の細胞に導入して撮影された画像であってもよい。この場合、画像解析手段は、第1の蛍光タンパク質の発光による発光スポットのカウント及び発光領域の面積の算出に加えて、さらに、第2の蛍光タンパク質の発光による発光スポットの数をカウントする。
【0007】
本発明に係る解析方法は、DNA二本鎖切断損傷を定量的に解析する方法であって、
DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で画像を撮影し、
時系列で撮影された複数の画像の各々について、
各画像に含まれる細胞核の画像の領域を検出し、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光スポットの数をカウントし、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光領域の面積を求め、
時系列で求めた前記発光スポットのカウント値及び前記発光領域の面積のデータを解析データとして生成する。
【発明の効果】
【0008】
本発明によれば、蛍光タンパク質をバイオセンサーとして用いて、細胞核においてDNA損傷が生じた箇所を検出する。具体的には、細胞核を時系列で撮影した画像を解析して、蛍光発光による発光スポットの数、発光領域の面積を求めることで、DNAの損傷の程度を定量的に測定する。これにより、DNAの損傷及び損傷の修復の時間的な変化を定量的にかつ迅速に測定することが可能となる。このようなDNA損傷解析技術を利用することで、がん細胞で起こるDNA損傷を生きたまま鋭敏に観察・評価でき、迅速に新規の抗がん剤のスクリーニングが可能となり、また放射線照射の有効性を評価でき、新規抗がん剤や放射線治療の増感補助薬の開発の迅速化を図ることができる。
【図面の簡単な説明】
【0009】
【図1】DNA二本鎖切断とDNA損傷応答を説明した図
【図2】GFP−53BP1タンパク質の蛍光を利用して、細胞核のDNA損傷の状態を撮影した画像((a)DNA損傷がない場合、(b)DNA損傷がある場合)
【図3】細胞周期を説明した図
【図4】(a)緑色蛍光タンパク質であるGFP−53BP1タンパク質をバイオセンサーとして導入し、細胞核の状態を撮影した画像であり、(b)赤色蛍光タンパク質であるDsRed−PCNAタンパク質をバイオセンサーとして導入し、細胞核の状態を撮影した画像
【図5】細胞にDNA損傷を与えた後のバイオセンサーによる発光状態の時間経過に伴う変化を撮影した画像
【図6】z軸方向における複数の平面でスライスして細胞核の画像データを取得する方法を説明した図
【図7】DNA損傷解析装置の構成を示す図
【図8】顕微鏡により時系列で撮影された細胞画像(画像の一部)の例を示す図
【図9】DNA損傷解析装置の解析処理を示すフローチャート
【図10】画像データにおいて細胞核を特定する処理を説明するための図
【図11】画像データにおける発光スポット数のカウント及び発光領域の面積の算出処理を説明するための図
【図12】DNA損傷解析装置により得られた解析データをグラフ表示した図(抗がん剤として、NCS(Neocarzinostanin)を投与した場合)
【図13】DNA損傷解析装置により得られた解析データをグラフ表示した図(抗がん剤として、CPT(Camptothecin)を投与した場合)
【発明を実施するための形態】
【0010】
以下、添付の図面を参照して本発明の実施の形態を説明する。
【0011】
1.DNA2本鎖切断損傷の検出方法の基本原理
図1を参照し、本実施形態におけるDNA2本鎖切断損傷を検出する方法の基本原理について説明する。
【0012】
通常、がんに対して放射線治療あるいは化学療法を行うと、DNAに損傷が発生する。そのうち最も細胞にダメージを与える損傷がDNA2本鎖切断である。2本鎖切断が起きると、図1に示すように、細胞の中では、「ATM」と呼ばれるタンパク質キナーゼが活性化され、種々のタンパク質が連鎖的に反応を起こし、最終的にDNAが巻き付いているヒストン100にメチル化などの変化が起こり、53BP1タンパク質が集積する。本実施形態では、このようなDNA損傷箇所に53BP1タンパク質が集積するという現象を利用して、DNA2本鎖切断損傷を検出する。
【0013】
ここで、DNA2本鎖切断損傷を検出するためのバイオセンサーについて説明する。バイオセンサーの基本技術として、2008年ノーベル化学賞受賞対象となった緑色蛍光タンパク質GFPを用いている。特に、本発明者は、DNA2本鎖切断損傷に特異的に集積するように53BP1タンパク質に改変を加え、ヒトがん培養細胞内で安定に発現できるGFP−53BP1融合タンパク質(以下「GFP−53BP1タンパク質」という)をバイオセンサーとして用いた。これにより、細胞が生きたままの状態でDNA損傷を蛍光顕微鏡で緑色のスポットとして観察することを可能とした。
【0014】
図1では、便宜上、一つの53BP1しか示していないが、実際は数百の分子が集積する。このため、DNA損傷箇所は、光のスポットとして観察することが出来る。図2(a)は、DNAの損傷がない細胞核の状態を示した画像であり、図2(b)は、8Gy(8Sv)の放射線により損傷が与えられたDNAの細胞核の状態を示した画像である。図2(b)に示すように、8Gy(8Sv)の放射線によりDNAの複数箇所に損傷が発生すると、それらの損傷箇所に集積したGFP−53BP1タンパク質が蛍光発光することにより光スポットとして観察される。
【0015】
さらに、本実施形態では、GFP−53BP1タンパク質に加えて、観察対象の細胞の細胞周期を検出できるセンサータンパク質を一緒に導入している。
【0016】
ここで、細胞周期について説明する。細胞周期とは、増殖・分裂する細胞が一連の過程を経て2つの細胞に分裂する全行程である(図3参照)。細胞周期は、G0期、G1期、S期、G2期、M期の5つの期を有している。分裂する細胞は、G1期、S期、G2期、M期の順に変化する。また、分裂・増殖せずに休止期にある細胞(多くの正常な体細胞)は、G0期に停止している。特に、S期では、活発に増殖・分裂を繰り返すがん細胞のDNA複製が行われており、このS期におけるDNA損傷を検出することは、DNA損傷を誘導する薬剤のスクリーニングにおいてきわめて有用である。
【0017】
具体的には、細胞分裂に際してDNAを複製している細胞周期のS期を標識するPCNA−DsRedタンパク質をセンサーとして観察対象の細胞に導入する。PCNA−DsRedタンパク質は赤色蛍光を示すタンパク質である。これにより、活発に分裂する細胞のS期にある細胞が、赤いスポットを多量に含む細胞として標識される。その結果、がん細胞を観察対象とした場合、がん細胞がS期にあるときに、DNA損傷を引き起こす抗がん剤の候補を迅速にスクリーニングできる。正常細胞は通常分裂を停止して静止状態にあるものが多く、活発に分裂するS期細胞のみで未知の薬剤がDNA損傷を引き起こすことが認識できれば、正常細胞に影響の少ない(副作用の少ない)抗がん剤の発見につながる。
【0018】
図4(a)は、緑色蛍光タンパク質であるGFP−53BP1タンパク質をバイオセンサーとして導入し、細胞核の状態を撮影した画像であり、図4(b)は、図4(a)に示す細胞と同じ細胞について、赤色蛍光タンパク質であるDsRed−PCNAタンパク質をバイオセンサーとして導入し、細胞核の状態を撮影した画像である。図4(b)に示すように、S期にある細胞核の全体において、赤色蛍光タンパク質であるDsRed−PCNAタンパク質による発光スポットが散在していることが分かる。
【0019】
図5は、細胞にDNA損傷を与えた後のバイオセンサーによる発光状態の時間経過に伴う変化を撮影した図である。図5の(a)〜(g)は、それぞれ別々の細胞核の時間変化(損傷前、損傷直後、1分後、2分後、3分後)を示している。ここでは、レーザー照射によりDNA損傷を発生させている。図5の(a)〜(g)より、レーザーが照射されDNA損傷が発生した部分において、時間経過とともに、発光スポットが増加している様子、すなわち、GFP−53BP1が集積している様子が理解できる。このように、損傷を生きたまま観察できることが分かる。
【0020】
本実施形態では、以上のようにGFP−53BP1タンパク質及びDsRed−PCNAタンパク質のような蛍光タンパク質をバイオセンサーとして細胞に加え、その発光状態を撮影し、撮影した画像を解析することによりDNA損傷の状態を検出する。
【0021】
また、細胞は3次元形状を有することから、図6に示すように、z軸方向における複数の平面でスライスした画像データを取得するようにしてもよい。この場合、撮影された画像情報は、細胞の3次元の座標情報に加えて、時間情報、緑/赤の2つの蛍光色情報を含めた5次元の画像情報となる。このような5次元の画像情報を利用することで、3次元的に存在する発光スポットをより精度よく検出でき、結果としてより精度のよい解析が可能となる。
【0022】
2.DNA損傷解析装置の構成
図7に、本実施形態のDNA損傷解析装置の構成の一例を示す。DNA損傷解析装置1は例えばパーソナルコンピュータのような情報処理装置で構成される。DNA損傷解析装置1は、顕微鏡31により撮影されて記録された画像情報を入力し、画像解析することによりDNAの損傷状態を定量的に解析する。
【0023】
DNA損傷解析装置1は、その全体動作を制御する制御部11と、画面表示を行う表示部17と、ユーザが操作を行う操作部19と、データやプログラムを記憶するデータ格納部21とを備える。例えば、表示部17は液晶ディスプレイで構成され、操作部19はキーボードやマウス等である。さらに、DNA損傷解析装置1は、外部機器やネットワークと接続するためのインタフェース25を含む。DNA損傷解析装置1は例えばパーソナルコンピュータ等で実現できる。
【0024】
制御部11は、CPUやMPUからなり、制御プログラムを実行することで後述する解析処理を実現する。制御部11で実行される制御プログラムは、データ格納部21に格納されている。制御プログラムは、通信回線を通じて又は光ディスクや半導体メモリ等の記録媒体を介して提供されてもよい。
【0025】
データ格納部21はデータやプログラムを記憶する手段であり、例えばハードディスク、半導体メモリ等で構成できる。データ格納部21は、制御部11で実行されるプログラムや解析後のデータ等を格納する。
【0026】
顕微鏡31は観察対象33の拡大画像を撮影可能であり、少なくとも2種類の波長の画像を撮影可能である。顕微鏡31は動画像または静止画の撮影が可能である。顕微鏡31により撮影された画像は、所定の記録媒体(ハードディスク装置、光ディスク記録装置、サーバ装置等)35に記録される。なお、記録媒体35は顕微鏡31内部に設けられても良い。または、顕微鏡31により撮影された画像は、インタフェース25を介してDNA損傷解析装置1に入力され、DNA損傷解析装置1内のデータ格納部21に記録されるようにしてもよい。また、顕微鏡31は、フォーカス位置を変更することで、図6に示すような、z方向に異なる位置を有する複数のスライス面上で画像データを撮影することが可能となる。
【0027】
3.DNA損傷解析処理
以上のように構成されるDNA損傷解析装置1によるDNAの損傷状態の解析処理について説明する。
【0028】
3.1 画像撮影
前述の2種の蛍光タンパク質をバイオセンサーとして導入して、顕微鏡31により細胞の画像を生きたまま撮影する。画像撮影中、所定のタイミングで、検査したい薬物を投与する。薬物の投与前から投与後所定時間が経過するまで、細胞の画像を、時間経過を追って撮影し、記録する。なお、以下の説明では、顕微鏡31により、所定の時間間隔毎に、緑色の蛍光を示すGFP−53BP1タンパク質による発光状態を撮影した画像(静止画)と、赤色の蛍光を示すDsRed−PCNAタンパク質による発光状態を撮影した画像(静止画)とが撮影され、記録媒体35に記録されるとする。また、顕微鏡31により、1つのスライス面での画像のみが撮影、記録されるとする。すなわち、顕微鏡31により、緑色の蛍光画像及び赤色の蛍光画像それぞれについて一連の複数枚の静止画が時系列で撮影され、記録媒体35に記録される。なお、本実施形態では、細胞の静止画を撮影したが、動画を撮影し、動画を構成するフレーム画像毎に解析処理を行っても良い。
【0029】
なお、顕微鏡31のフォーカスを調整しながら、図6に示すようにz方向において位置が異なる複数のスライス平面上で画像を撮影してもよい。
【0030】
図8は、顕微鏡31により時系列で撮影された細胞画像(画像の一部)の例を示す図である。図8において、(a1)〜(a4)は、緑色の蛍光を示すGFP−53BP1タンパク質による発光状態を撮影した蛍光画像である。各列(a1)〜(a4)においては上から下に時間が経過し、(a1)、(a2)、(a3)、(a4)の順に時間が経過している。図8において、(b1)〜(b4)は、赤色の蛍光を示すDsRed−PCNAタンパク質による発光状態を撮影した蛍光画像である。各列(b1)〜(b4)においては上から下に時間が経過し、(b1)、(b2)、(b3)、(b4)の順に時間が経過している。
【0031】
3.2 画像解析
顕微鏡31により撮影され、記録された2種の蛍光画像は、インタフェース25を介してDNA損傷解析装置1に入力され、時間経過とともに細胞核で変化する緑色ならびに赤色のスポットが定量解析される。以下、DNA損傷解析装置1による画像解析の詳細を説明する。なお、記録媒体35には、顕微鏡31により撮影された、緑色の蛍光画像及び赤色の蛍光画像それぞれについての一連の複数の静止画が時系列で予め記録されており、DNA損傷解析装置1は記録媒体35から画像を読み出しながら以下の処理を実行する。
【0032】
図9は、DNA損傷解析装置1の画像解析処理を示したフローチャートである。DNA損傷解析装置1は、インタフェース25を介して記録媒体35に記録された画像の中から1つの画像を取り込む(S11)。具体的には、DNA損傷解析装置1は、記録媒体35に記録された画像において時系列で最初の画像を取り込む。
【0033】
制御部11は、取り込んだ画像を画像解析し、画像中の細胞核を特定する(S12)。次に、制御部11は、特定した細胞核について、蛍光発光による発光スポットの数をカウントするとともに、発光領域の面積を求める(S13)。これらの処理の詳細は後述する。なお、発光領域の面積については、GFP−53BP1タンパク質による緑色の蛍光画像に対してのみ求める。
【0034】
制御部11は、発光スポットのカウント数及び発光領域の面積を細胞核と関連づけて解析データとして生成し、データ格納部21に出力して記録する(S14)。画像内の1つの細胞核について、上記の処理(S13、S14)が終了すると、ステップS12に戻り、画像内に他の細胞核が含まれるか否かを検査し、他の細胞核が特定されたときは、発光スポット数のカウント、発光領域の面積の算出等の処理(S13)を行う。
【0035】
取り込んだ画像に含まれる全ての細胞核について、細胞核の特定、発光スポット数のカウント、発光領域の面積の算出(S12〜S14)を実行する(S15)。これらの処理が終了すると、記録媒体35に記録された一連の画像において時系列で次の画像について同様の処理を行い、記録媒体35に記録された全ての画像について上記の処理を行う(S11〜S16)。
【0036】
ここで、本実施形態では、顕微鏡31により、生きている細胞の画像を撮影しているが、生きている細胞は時間とともに除々に移動し、変形する。このため、前回処理された画像(静止画)における細胞核と、今回処理された画像における細胞核とを同定する必要がある。この同定は、例えば、細胞核の形状を考慮することで実現できる。例えば、公知の顔認識技術と同様の技術を用いて、細胞核の形状(特徴)に基づいて時間的に除々に移動、変化する細胞核をトラッキングすることができる。
【0037】
なお、上記の例では、時系列で連続して撮影された一連の画像を連続的に処理(ステップS13)したが、所定の時間間隔毎の画像のみを抽出して処理するようにしてもよい。
【0038】
また、図6に示すように、z方向において位置が異なる複数のスライス平面上で画像が撮影されている場合は、各スライス平面の画像データをそれぞれ解析し、得られた発光スポット数や発光領域の面積の値を合算してもよい。または、複数のスライス平面上の画像の中で最も好適と考えられる1つの画像について上記の処理(ステップS12〜14)を実施するようにしてもよい。
【0039】
3.2.1 細胞核の特定、発光スポット数のカウント、発光領域の面積の算出
図10、図11を参照して、上記の、細胞核の特定処理(ステップS12)、並びに、発光スポット数のカウント及び発光領域の面積の算出処理(ステップS13)の詳細を説明する。これらの処理(ステップS12、S13)は特に制御部11の画像解析部15により実行される。
【0040】
図10(a)は、記録媒体35から取り込まれた一の画像を示す。画像解析部15は、まず、図10(a)に示す画像を、画素値(輝度)に関する所定の閾値に基づき二値化し、図10(b)に示すような二値化画像を生成する。制御部11は、二値化画像において、同じ輝度を含む連続した画素からなる領域51を特定する。次に、画像解析部15は特定した領域51を含む矩形枠55を設定する(図10(c)参照)。そして、画像解析部15は、図10(d)に示すように、撮影された画像上に矩形枠55を設定し、矩形枠55で囲まれる領域の画像を切り出す(図10(e)参照)。
【0041】
上記のようにして切り出された領域に対して、画像解析部15は、発光スポット数のカウント及び発光領域の面積の算出を行う。図11(a)は、上記の方法で切り出された画像である(図10(e)と同様)。画像解析部15は、切り出された画像(図11(a)参照)において、図11(b)に示すように細胞核の領域を特定し、特定した細胞核の背景の画像を削除する。図11(c)は、特定した細胞核の背景の画像が削除された画像を示す。画像解析部15は、細胞核の背景の画像が削除された画像において、所定の画素値(輝度)以上の値を持つ領域(発光スポット)を特定し(図11(d)参照)、その領域の数をカウントすることで、発光スポットの数をカウントする。さらに、画像解析部15は、所定の画素値(輝度)以上の値を持つ画素の数をカウントすることで、発光領域の面積を求める。
【0042】
上記のようにして、細胞核の特定、発光スポット数のカウント及び発光領域の面積の算出処理(ステップS12、S13)が実行される。
【0043】
なお、本実施形態では、発光スポット数及び発光領域の面積の双方を測定している。画像の状況によって、発光スポット数または発光領域の面積が良好に観察できないことがある。例えば、ざらついた画像においては、発光スポット数よりも発光面積の方が良好に測定できる。よって、発光スポット数及び発光領域の面積の双方を測定しておくことで、画像の状況に応じて適宜良好な方のデータを選択することが可能となる。
【0044】
4.解析データのグラフ化
以上のようにして、DNA損傷解析装置1によるDNAの損傷状態の解析処理が実行されることで、細胞核毎に、発光スポットの数及び発光領域の面積のデータを時系列で求めることができる。このようにして求められたデータは、解析データとしてデータ格納部21に格納される。
【0045】
図12及び図13は、DNA損傷解析装置1により以上のようにして得られた解析データをグラフとして表示したものである。図12は、抗がん剤としてNCS(Neocarzinostanin)を投与した際のDNA損傷の状況についての、DNA損傷解析装置1による解析結果をグラフ化した図である。図13は、抗がん剤としてCPT(Camptothecin)を投与した際のDNA損傷の状況についての、DNA損傷解析装置1による解析結果をグラフ化した図である。図中、「DNA損傷の数」は、GFP−53BP1タンパク質による発光スポット数の時間変化をプロットしたものであり、「DNA損傷の占める面積」は、GFP−53BP1タンパク質による発光領域の面積の時間変化をプロットしたものである。また、「S期のDNA複製スポット数」は、DsRed−PCNAタンパク質による発光スポット数の時間変化をプロットしたものである。
【0046】
図12、13において、時間GFP−53BP1タンパク質による発光スポット数の時間変化(実線X)及び発光領域の面積の時間変化(実線Y)を参照すると、DNA損傷やその修復状況の時間的な変化を把握することができる。また、DsRed−PCNAタンパク質による発光スポット数(実線Z)の時間変化を参照することで、細胞周期のS期の範囲を推定することができる。よって、DsRed−PCNAタンパク質による発光スポット数(実線Z)の時間変化とともに、時間GFP−53BP1タンパク質による発光スポット数の時間変化(実線X)及び発光領域の面積の時間変化(実線Y)を観察することで、S期におけるDNA損傷及び損傷の修復状態の時間的変化を把握することができる。
【0047】
5.まとめ
以上のように、本実施形態のDNA損傷解析装置1によれば、細胞核において発生したDNA損傷の程度を定量的に測定することができる。具体的には、DNA損傷部位に集積する蛍光タンパク質をバイオセンサーとして用いて、細胞核においてDNA損傷が生じた箇所を検出する。具体的には、細胞核を撮影した画像を解析して、蛍光発光による発光スポットの数及び発光領域の面積を求めることで、DNAの損傷の程度を定量的に測定する。また、DNA損傷部位に集積する蛍光タンパク質に加えて、細胞周期のS期を標識する蛍光タンパク質をもバイオセンサーとして用いることで、細胞周期のS期を検出する。このような定量的な測定を時系列で行うことにより、DNAの損傷及び損傷の修復の時間的な変化を定量的に測定することが可能となり、生きた細胞におけるDNA損傷の時間的な変化の定量的な観察が可能となる。このようなDNA損傷解析技術を利用することで、がん細胞で起こるDNA損傷を生きたまま鋭敏に観察・評価でき、迅速に新規の抗がん剤のスクリーニングが可能となり、また放射線照射の有効性を評価でき、新規抗がん剤や放射線治療の増感補助薬の開発の迅速化を図ることができる。
【符号の説明】
【0048】
1 DNA損傷解析装置
11 制御部
17 表示部
19 操作部
21 データ格納部
25 インタフェース
31 顕微鏡
33 観察対象
35 記録媒体
【先行技術文献】
【特許文献】
【0049】
【特許文献1】特表2005−502323号公報
【特許請求の範囲】
【請求項1】
DNA二本鎖切断損傷を定量的に解析する装置であって、
DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で撮影された画像を取得する画像取り込み手段と、
取得した画像を解析する画像解析手段とを備え、
前記画像解析手段は、時系列で撮影された画像の各々について、
各画像に含まれる細胞核の画像の領域を検出し、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光スポットの数をカウントし、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光領域の面積を求め、
時系列で求めた前記発光スポットのカウント値及び前記発光領域の面積のデータを解析データとして生成する、
解析装置。
【請求項2】
前記画像は、前記第1の蛍光タンパク質に加えて、細胞周期のS期にある細胞を標識する第2の蛍光タンパク質をさらに観察対象の細胞に導入して撮影された画像であり、
前記画像解析手段は、前記第1の蛍光タンパク質の蛍光発光による発光スポットのカウント及び発光領域の面積の算出に加えて、さらに、前記第2の蛍光タンパク質の蛍光発光による発光スポットの数をカウントする、
請求項1記載の解析装置。
【請求項3】
前記第1の蛍光タンパク質と前記第2の蛍光タンパク質とは異なる色の蛍光発光を示す、請求項2記載の解析装置。
【請求項4】
DNA二本鎖切断損傷を定量的に解析するためのプログラムであって、
コンピュータを、
DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で撮影された画像を取得する画像取り込み手段と、
取得した画像を解析する画像解析手段と
して実行させ、
前記画像解析手段は、時系列で撮影された画像の各々について、
各画像に含まれる細胞核の画像の領域を検出し、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光スポットの数をカウントし、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光領域の面積を求め、
時系列で求めた前記発光スポットのカウント値及び前記発光領域の面積のデータを解析データとして生成する、
プログラム。
【請求項5】
前記画像は、前記第1の蛍光タンパク質に加えて、細胞周期のS期にある細胞を標識する第2の蛍光タンパク質をさらに観察対象の細胞に導入して撮影された画像であり、
前記画像解析手段は、前記第1の蛍光タンパク質の蛍光発光による発光スポットのカウント及び発光領域の面積の算出に加えて、さらに、前記第2の蛍光タンパク質の蛍光発光による発光スポットの数をカウントする、
請求項4記載のプログラム。
【請求項6】
DNA二本鎖切断損傷を定量的に解析する方法であって、
DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で画像を撮影し、
時系列で撮影された複数の画像の各々について、
各画像に含まれる細胞核の画像の領域を検出し、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光スポットの数をカウントし、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光領域の面積を求め、
時系列で求めた前記発光スポットのカウント値及び前記発光領域の面積のデータを解析データとして生成する、
解析方法。
【請求項7】
前記第1の蛍光タンパク質に加えて、細胞周期のS期にある細胞を標識する第2の蛍光タンパク質をさらに前記観察対象の細胞に導入して画像を撮影し、
前記第1の蛍光タンパク質の蛍光発光による発光スポットのカウント及び発光領域の面積の算出に加えて、さらに、前記第2の蛍光タンパク質の蛍光発光による発光スポットの数をカウントする、
請求項6記載の解析方法。
【請求項1】
DNA二本鎖切断損傷を定量的に解析する装置であって、
DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で撮影された画像を取得する画像取り込み手段と、
取得した画像を解析する画像解析手段とを備え、
前記画像解析手段は、時系列で撮影された画像の各々について、
各画像に含まれる細胞核の画像の領域を検出し、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光スポットの数をカウントし、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光領域の面積を求め、
時系列で求めた前記発光スポットのカウント値及び前記発光領域の面積のデータを解析データとして生成する、
解析装置。
【請求項2】
前記画像は、前記第1の蛍光タンパク質に加えて、細胞周期のS期にある細胞を標識する第2の蛍光タンパク質をさらに観察対象の細胞に導入して撮影された画像であり、
前記画像解析手段は、前記第1の蛍光タンパク質の蛍光発光による発光スポットのカウント及び発光領域の面積の算出に加えて、さらに、前記第2の蛍光タンパク質の蛍光発光による発光スポットの数をカウントする、
請求項1記載の解析装置。
【請求項3】
前記第1の蛍光タンパク質と前記第2の蛍光タンパク質とは異なる色の蛍光発光を示す、請求項2記載の解析装置。
【請求項4】
DNA二本鎖切断損傷を定量的に解析するためのプログラムであって、
コンピュータを、
DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で撮影された画像を取得する画像取り込み手段と、
取得した画像を解析する画像解析手段と
して実行させ、
前記画像解析手段は、時系列で撮影された画像の各々について、
各画像に含まれる細胞核の画像の領域を検出し、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光スポットの数をカウントし、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光領域の面積を求め、
時系列で求めた前記発光スポットのカウント値及び前記発光領域の面積のデータを解析データとして生成する、
プログラム。
【請求項5】
前記画像は、前記第1の蛍光タンパク質に加えて、細胞周期のS期にある細胞を標識する第2の蛍光タンパク質をさらに観察対象の細胞に導入して撮影された画像であり、
前記画像解析手段は、前記第1の蛍光タンパク質の蛍光発光による発光スポットのカウント及び発光領域の面積の算出に加えて、さらに、前記第2の蛍光タンパク質の蛍光発光による発光スポットの数をカウントする、
請求項4記載のプログラム。
【請求項6】
DNA二本鎖切断損傷を定量的に解析する方法であって、
DNA二本鎖切断箇所に集積する第1の蛍光タンパク質を観察対象の細胞に導入して時系列で画像を撮影し、
時系列で撮影された複数の画像の各々について、
各画像に含まれる細胞核の画像の領域を検出し、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光スポットの数をカウントし、
前記検出した領域に含まれる、前記第1の蛍光タンパク質の蛍光発光による発光領域の面積を求め、
時系列で求めた前記発光スポットのカウント値及び前記発光領域の面積のデータを解析データとして生成する、
解析方法。
【請求項7】
前記第1の蛍光タンパク質に加えて、細胞周期のS期にある細胞を標識する第2の蛍光タンパク質をさらに前記観察対象の細胞に導入して画像を撮影し、
前記第1の蛍光タンパク質の蛍光発光による発光スポットのカウント及び発光領域の面積の算出に加えて、さらに、前記第2の蛍光タンパク質の蛍光発光による発光スポットの数をカウントする、
請求項6記載の解析方法。
【図9】


【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図10】


【図11】


【図12】


【図13】




【図1】


【図2】


【図3】


【図4】


【図5】


【図6】


【図7】


【図8】


【図10】


【図11】


【図12】


【図13】


【公開番号】特開2013−15325(P2013−15325A)
【公開日】平成25年1月24日(2013.1.24)
【国際特許分類】
【出願番号】特願2011−146381(P2011−146381)
【出願日】平成23年6月30日(2011.6.30)
【出願人】(504150461)国立大学法人鳥取大学 (271)
【出願人】(511160099)株式会社eBase Solutions Laboratory (1)
【Fターム(参考)】
【公開日】平成25年1月24日(2013.1.24)
【国際特許分類】
【出願日】平成23年6月30日(2011.6.30)
【出願人】(504150461)国立大学法人鳥取大学 (271)
【出願人】(511160099)株式会社eBase Solutions Laboratory (1)
【Fターム(参考)】
[ Back to top ]

