インスティチュート フォー アドバンスド スタディにより出願された特許
1 - 4 / 4
配列モチーフを同定するための方法、およびその応用
【課題】宿主中のタンパク質産生の最適化方法を提供する。
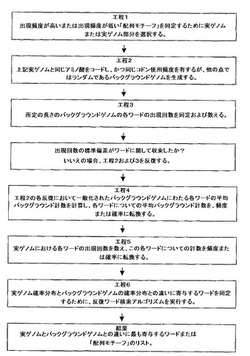
【解決手段】偶然に出現することが予想される配列の頻度と比較して、所定のヌクレオチド配列中で出現頻度が低いもしくは出現頻度が高い配列モチーフ、または他のヌクレオチド配列に存在する配列の頻度と比較して、出現頻度が低いもしくは出現頻度が高い配列モチーフを同定し、これらの配列モチーフの出現に基づいて配列をスコアリングして、出現頻度の低い配列モチーフの数が減少し、出現頻度の高い配列モチーフの数が増加した、タンパク質をコードするヌクレオチド配列を変異させる工程を含む、宿主におけるタンパク質の産生を改善する方法。
(もっと読む)
ウイルス阻害性ヌクレオチド配列およびワクチン
【解決課題】本発明は、レンチウイルスのゲノムにおける阻害性ヌクレオチドシグナル配列または「INS」配列に関する。特に、本発明は全てのウイルスゲノムに存在するAGGモチーフに関する。
【解決手段】AGGモチーフは、例えば、宿主細胞におけるウイルスRNAのレベルを低下させ、またはその低い定常状態レベルを維持し、次いで、ウイルス潜伏を誘導し、および/または維持することによってウイルスに対する阻害効果を有することができる。一つの態様において、本発明は、AGG配列が突然変異されているウイルス核酸を含有する、またはそれから生産されたワクチンを提供する。別の態様において、本発明は、AGGモチーフの機能に影響させるための方法および組成物、およびウイルスゲノムにおける他のINS配列を同定するための方法を提供する。
(もっと読む)
標的バクテリアあるいはファージを特定の属、種若しくは血清型として特定若しくは分類する方法
【課題】宿主−ファージ類似性を捕捉するゲノム指紋についての相対的エントロピーアルゴリズムの提供。
【解決手段】以下のステップにより標的バクテリアあるいはファージを分類する方法を開発した。(a)標的となるゲノム配列を特定するステップ、(b)標的ゲノムから誘導されるランダム化された背景ゲノムを作成するステップ、(c)反復アルゴリズムを実行し、背景ゲノムと標的ゲノム間の相違に影響するオリゴヌクレオチドを特定するステップ、(d)ステップ(C)で選択されたオリゴヌクレオチド配列を知られたバクテリア若しくはファージ配列とを比較するステップ。
(もっと読む)
配列モチーフを同定するための方法、およびその応用
本発明は、偶然に出現することが予想される配列の頻度と比較して、所定のヌクレオチド配列中で出現頻度が低いもしくは出現頻度が高い配列モチーフ、または他のヌクレオチド配列に存在する配列の頻度と比較して、出現頻度が低いもしくは出現頻度が高い配列モチーフを同定するために使用できる方法およびアルゴリズム、ならびにこれらの配列モチーフの出現に基づいて配列をスコアリングする方法に関する。このような配列モチーフは、生物学的に有意義であってよく、例えば、これらは、転写因子結合部位,mRNA安定性/不安定性シグナル、後成的シグナルなどを構成し得る。本発明の方法は、とりわけ、それらの系統発生的関連性によって配列もしくは生物を分類するため、または病原体生物の宿主の可能性を同定するためにもまた使用することができる。本発明の方法は、タンパク質の発現を最適化するためにもまた使用することができる。 (もっと読む)
1 - 4 / 4
[ Back to top ]