弱毒HCMV及びそのワクチンとしての使用
【課題】ヒトサイトメガロウイルス(HCMV)の弱毒化及びワクチンの提供。
【解決手段】HCMVのUL76遺伝子ORF中のIRES配列が不活性化された弱毒HCMVウイルス。前記IRES配列が、IRES以外の別の核酸配列によって置換されている、弱毒HCMVウイルス。該弱毒HCMVウイルスを含む、HCMV感染の予防又は治療用ワクチン。
【解決手段】HCMVのUL76遺伝子ORF中のIRES配列が不活性化された弱毒HCMVウイルス。前記IRES配列が、IRES以外の別の核酸配列によって置換されている、弱毒HCMVウイルス。該弱毒HCMVウイルスを含む、HCMV感染の予防又は治療用ワクチン。
【発明の詳細な説明】
【技術分野】
【0001】
本発明は、弱毒ヒトサイトメガロウイルス(HCMV)及びそのワクチンとしての使用に関する。
【背景技術】
【0002】
ヒトサイトメガロウイルス(HCMV)は、ベータヘルペスウイルスファミリーのプロトタイプメンバーである。多くの個体でHCMV による感染が起こるけれども、その感染は無症候性である。このウイルスは免疫抑制条件下で再活性化されて病原体となり、肺炎、ヘルペス、網膜炎及び胃腸疾患を引き起こす。HCMVはまた、妊娠中に初期感染を起こしたときには、新生児に難聴と精神発達遅滞を引き起こす。HCMVのゲノムのサイズは約240,000bpであり、少なくとも194個の既知のオープンリーディングフレーム(ORF)をもっている(7, 33, 34)。このORFの大部分は、細胞培養中でのウイルス複製に必須ではない。いくつかのORFは有益であるが、それらはウイルスの複製に必要なものではない。しかしながら、ORFの約4分の1に相当する41個のORFは、ウイルスの複製に必要である(47)。
【0003】
ウイルス産生のための感染の間に、HCMV遺伝子群は一時的なカスケードの、指定された前初期(IE)、遅延初期及び後期に発現される。主要なIE(MIE)遺伝子であるUL123/122 (IE1/IE2)は、その後のウイルス遺伝子発現及びウイルス複製の効率において決定的な役割を果たす(17, 18, 22, 29-31)。複製初期のウイルス遺伝子群は、ウイルスDNA複製に必要なタンパク質をコードしている(36)。ウイルスDNA複製後、ウイルス粒子産生のための構造タンパク質をコードする、遅延初期及び後期ウイルス遺伝子群が発現される。これに関連して、MIE遺伝子プロモーターのシス作用性エレメント(crs)配列の一部に突然変異を有し、これによってウイルス複製が不能ではないが抑制されている弱毒化組換えヒトサイトメガロウイルスが特許文献1に開示されている。
【0004】
HCMV UL76遺伝子は、高度に保存されたウイルス粒子関連ヘルペスウイルスタンパク質をコードし、このタンパク質はウイルス複製を増加させることが報告されている(43, 44)。ORFの予測された分子量に相当する38kDaのタンパク質は、感染後(p.i.)2時間の時点でHCMV感染細胞中に検出される。このタンパク質の産生はp.i.24時間の時点で最大に達し、その後、その産生レベルは、ウイルス生活環の後期から同じレベルを維持する。
【0005】
UL77遺伝子は、単純ヘルペスウイルス(HSV) UL25遺伝子の同等物であり、この遺伝子はHCMV複製に必須である(7)。HCMVでは、UL77 ORFの最初のMetは、UL76 ORFの最初のMetと異なるフレーム中に存在する。UL76では明らかな転写終結がないし、また、UL77では転写開始がない(44)。UL78はG-タンパク質共役受容体の特性を有しており(1)、また、UL78 1.7kb mRNAは、HFF細胞中では、p.i.4時間の時点で検出可能になる(32)。
【0006】
5’mRNAキャップは、翻訳開始複合体と結合するために必須である。43S前開始複合体は、それが翻訳のための最初の好適な開始コドン(これは大方、最初のAUGである。)に達するまで、該RNAをスキャニングする。哺乳類のリボソームは一般にバイシストロン性mRNAの下流シストロンを翻訳しないか又は翻訳できない(これに対して、原核生物のシステムではそのような翻訳が偶発的に起こる)。この一般則に2つの例外があり、一方は、下流のシストロンに直接的にリボソームをリクルートすることが可能なインターナルリボソームエントリーサイト(IRES)であり、他方は、下流のAUGコドンで翻訳を再び開始するために5'から3'方向にスキャニングすることである(2, 4-6, 9-11, 25, 26)。
【先行技術文献】
【特許文献】
【0007】
【特許文献1】特開2008-092854号公報
【非特許文献】
【0008】
【非特許文献1】Dunn, W., C. Chou, H. Li, R. Hai, D. Patterson, V. Stolc, H. Zhu, and F. Liu. 2003. Functional profiling of a human cytomegalovirus genome. Proc. Natl. Acad. Sci. U. S. A. 100:14223-14228(文献7)
【非特許文献2】Murphy, E., I. Rigoutsos, T. Shibuya, and T. E. Shenk. 2003. Reevaluation of human cytomegalovirus coding potential. Proc. Natl. Acad. Sci. U. S. A. 100:13585-13590(文献33)
【非特許文献3】Murphy, E., D. Yu, J. Grimwood, J. Schmutz, M. Dickson, M. A. Jarvis, G. Hahn, J. A. Nelson, R. M. Myers, and T. E. Shenk. 2003. Coding potential of laboratory and clinical strains of human cytomegalovirus. Proc. Natl. Acad. Sci. U. S. A. 100:14976-14981(文献34)
【非特許文献4】Yu, D., M. C. Silva, and T. Shenk. 2003. Functional map of human cytomegalovirus AD169 defined by global mutational analysis. Proc. Natl. Acad. Sci. U. S. A. 100:12396-12401(文献47)
【非特許文献5】Isomura, H., and M. F. Stinski. 2003. The human cytomegalovirus major immediate-early enhancer determines the efficiency of immediate-early gene transcription and viral replication in permissive cells at low multiplicity of infection. J. Virol. 77:3602-3614(文献17)
【非特許文献6】Isomura, H., M. F. Stinski, A. Kudoh, T. Daikoku, N. Shirata, and T. Tsurumi. 2005. Two Sp1/Sp3 binding sites in the major immediate-early proximal enhancer of human cytomegalovirus have a significant role in viral replication. J. Virol. 79:9597-9607(文献18)
【非特許文献7】Isomura, H., T. Tsurumi, and M. F. Stinski. 2004. Role of the proximal enhancer of the major immediate-early promoter in human cytomegalovirus replication. J. Virol. 78:12788-12799(文献22)
【非特許文献8】Meier, J. L., M. J. Keller, and J. J. McCoy. 2002. Requirement of multiple cis-acting elements in the human cytomegalovirus major immediate-early distal enhancer for viral gene expression and replication. J. Virol. 76:313-326(文献29)
【非特許文献9】Meier, J. L., and J. A. Pruessner. 2000. The human cytomegalovirus major immediate-early distal enhancer region is required for efficient viral replication and immediate-early gene expression. J. Virol. 74:1602-1613(文献30)
【非特許文献10】Meier, J. L., and M. F. Stinski. 1997. Effect of a modulator deletion on transcription of the human cytomegalovirus major immediate-early genes in infected undifferentiated and differentiated cells. J. Virol. 71:1246-1255(文献31)
【非特許文献11】Pari, G. S., and D. G. Anders. 1993. Eleven loci encoding trans-acting factors are required for transient complementation of human cytomegalovirus oriLyt-dependent DNA replication. J. Virol. 67:6979-6988(文献36)
【非特許文献12】Wang, S. K., C. Y. Duh, and T. T. Chang. 2000. Cloning and identification of regulatory gene UL76 of human cytomegalovirus. J. Gen. Virol. 81:2407-2416(文献43)
【非特許文献13】Wang, S. K., C. Y. Duh, and C. W. Wu. 2004. Human cytomegalovirus UL76 encodes a novel virion-associated protein that is able to inhibit viral replication. J. Virol. 78:9750-9762(文献44)
【非特許文献14】Michel, D., I. Milotic, M. Wagner, B. Vaida, J. Holl, R. Ansorge, and T. Mertens. 2005. The human cytomegalovirus UL78 gene is highly conserved among clinical isolates, but is dispensable for replication in fibroblasts and a renal artery organ-culture system. J. Gen. Virol. 86:297-306(文献32)
【非特許文献15】Biegalke, B. J., and A. P. Geballe. 1990. Translational inhibition by cytomegalovirus transcript leaders. Virology 177:657-667(文献2)
【非特許文献16】Cao, J., and A. P. Geballe. 1996. Coding sequence-dependent ribosomal arrest at termination of translation. Mol. Cell Biol. 16:603-608(文献4)
【非特許文献17】Cao, J., and A. P. Geballe. 1995. Translational inhibition by a human cytomegalovirus upstream open reading frame despite inefficient utilization of its AUG codon. J. Virol. 69:1030-1036(文献5)
【非特許文献18】Degnin, C. R., M. R. Schleiss, J. Cao, and A. P. Geballe. 1993. Translational inhibition mediated by a short upstream open reading frame in the human cytomegalovirus gpUL4 (gp48) transcript. J. Virol. 67:5514-5521(文献6)
【非特許文献19】Geballe, A. P., F. S. Leach, and E. S. Mocarski. 1986. Regulation of cytomegalovirus late gene expression: gamma genes are controlled by posttranscriptional events. J. Virol. 57:864-874(文献9)
【非特許文献20】Geballe, A. P., and E. S. Mocarski. 1988. Translational control of cytomegalovirus gene expression is mediated by upstream AUG codons. J. Virol. 62:3334-3340(文献10)
【非特許文献21】Geballe, A. P., and D. R. Morris. 1994. Initiation codons within 5'-leaders of mRNAs as regulators of translation. Trends Biochem. Sci. 19:159-164(文献11)
【非特許文献22】Kozak, M. 2001. Constraints on reinitiation of translation in mammals. Nucleic Acids Res. 29:5226-5232(文献25)
【非特許文献23】Kozak, M. 1987. Effects of intercistronic length on the efficiency of reinitiation by eucaryotic ribosomes. Mol. Cell Biol. 7:3438-3445(文献26)
【発明の概要】
【発明が解決しようとする課題】
【0009】
今回、本発明者らは、ヒトサイトメガロウイルス(HCMV)のUL76 ORFが、UL77 ORFの翻訳に影響するIRESを含むことを見出した。このUL77はウイルス複製に必須であるので、UL76が、ウイルス複製の理由の一部にUL76中のIRESが関わっていることを今回明らかにした。この知見に基づいてHCMVの弱毒ウイルスを作製することを本発明の目的とした。
【課題を解決するための手段】
【0010】
ヒトサイトメガロウイルス(HCMV)のUL77遺伝子はウイルスDNAパッケージングに特定の役割を果たしており、このパッケージングはウイルス複製にとって重要である。しかし、トリシストロン性転写体は、UL76〜UL78オープンリーディングフレーム (ORF)を含み、一方、UL77は慣用の5’mRNAを有していない。UL77の翻訳が上流配列によって影響を受けるかどうかを決定するために、本発明者らは、UL76 ORFの下流の停止(stop)コドンからATG開始コドンまでを欠失するか或いは挿入することによってUL76を変異させた。組換えウイルスを両方とも用いて、UL77の翻訳レベルの増加が、上流のUL77配列がUL77の発現に影響すること、及び、UL77が、第1シストロンとなるとき、より効率的に発現されることを示すことを今回見出した。本発明者らはまた、UL76 ORFのN末端に融合されたflagエピトープとUL77 ORFのC末端に融合されたHAエピトープをもつ発現プラスミドを構築し、HeLa細胞をトランスフェクトした。UL76 ORF中のフレームシフトにより、UL77の発現が増加したが、一方、UL77のカルボキシ末端へのフレームシフトは影響を与えなかった。このことから、UL76がUL77発現に影響することを確認した。UL76がどのようにUL77発現に影響するかを決定するために、本発明者らは、バイシストロニックなレポータープラスミドを構築し、UL76の上流配列がインターナルリボソームエントリーサイト(IRES)を含むかどうかを測定した。その結果、本発明者らは、今回、UL76中のIRESがピコルナウイルスIRESとほとんど同じ強さの活性をもつことを検出し、UL76遺伝子が、IRESを介して、下流のUL77 ORFの翻訳に影響を及ぼしていることを見出した。本発明は、上記の知見に基づいている。
【0011】
本発明は、要約すると、以下の特徴を含む。
本発明は、第1の態様において、ヒトサイトメガロウイルス(HCMV)のUL76遺伝子ORF中のIRES配列が不活性化されたことを特徴とする弱毒HCMVウイルスを提供する。
【0012】
その実施形態において、上記IRES配列が、IRES以外の別の核酸配列によって置換されている。
【0013】
別の実施形態において、上記別の核酸配列のサイズ(すなわち、ヌクレオチド数)が上記IRES配列のサイズと完全に同じであるか又はほほ同じである。
【0014】
別の実施形態において、上記別の核酸配列が、同じ又は異なるHCMV株のUL30由来である。
【0015】
別の実施形態において、上記HCMVの複製が抑制された弱毒HCMVウイルスである。
【0016】
本発明はまた、第2の態様において、上記の弱毒HCMVウイルスを有効成分として含む、HCMV感染の予防又は治療用ワクチンを提供する。
【0017】
本発明はさらに、第3の態様において、上記の弱毒HCMVウイルスを非ヒト動物に免疫して抗体を作製し、該抗体を回収することを含む、抗HCMV抗体の作製方法を提供する。
【0018】
その実施形態において、上記抗HCMV抗体がヒト抗体である。
【発明の効果】
【0019】
本発明によって、ヒトサイトメガロウイルス(HCMV)の複製が抑制された弱毒ウイルスを提供することができるため、該弱毒ウイルスをワクチンとして使用することができる。
【図面の簡単な説明】
【0020】
【図1−1】この図は、野生型(wt)又は組換えウイルスによる感染後のUL76〜UL78遺伝子転写体の分析結果を示す。(a)は、wt及びRdlUL76+FRTの組換えBAC DNAの図である。UL76は、カナマイシン耐性遺伝子(KanR)で置換され、その後、KanRはFLP仲介組換えによって除かれ、その結果、FRT配列(F)の34bpのみが残った。(b)は、wt及びRdlUL76+Fの親及び組換えBAC DNAのサザンブロット分析の結果を示す。BAC DNAは、制限エンドヌクレアーゼBstB Iで消化され、0.6%アガロースゲルでの電気泳動によって分画され、32P標識プローブとのハイブリダイゼーションにかけられた。標準分子サイズマーカーは塩基対で示されている。レーン1及び3, RdlUL76+F; レーン2及び4, wt; レーン1及び2, UL75プローブ; レーン3及び4, UL76プローブ。
【図1−2】図1の(c)、(d)及び(e): wt又はRdlUL76+Fによる感染後のUL76〜78遺伝子転写体の分析結果を示す。HFFは、約1のMOIで感染され、細胞質RNAが、後述の実施例に記載のように、感染後(p.i)6, 24, 48及び72時間目に回収された。28S及び18S rRNAは、コントロールとして使用され、等量のRNAが負荷(load)された。(c) UL76プローブ、(d) UL77プローブ、(e) UL78プローブ。レーン1及び5, 感染後6時間; レーン2及び6, 感染後24時間; レーン3及び7, 感染後48時間; レーン4及び8, 感染後72時間; レーン1〜4, wt; レーン5〜8, RdlUL76+F。
【図2】この図は、UL77の上流領域が存在する又は存在しないUL77 ORFのN末端に融合されたflagエピトープを有する組換えウイルスを示す。(a)は、後述の実施例の「材料と方法」に記載のように作製された組換えHCMV BAC DNAの図である。(b)は、上記組換えウイルスによるUL77遺伝子転写の定量結果を示す。細胞質RNAは、UL77特異的プライマーとプローブを用いて、後述の実施例の「材料と方法」に記載のように、感染後1日、2日及び3日目にリアルタイムPCRによって分析された。このアッセイを3回行い、平均の標準誤差を決定した。UL77 RNAは、G6PD RNAに対して標準化され、各値は、感染後3日目のRflagUL77 RNAのレベルに対する値とした。(c)は、約1のMOIでRdlUL76flagUL77又はRflagUL77で感染後の表示した時間における前初期pIE72 (UL123), pIE86 (UL122), 初期p52 (UL44), 遅延初期pp65 (UL83), 後期pp28 (UL99)及びflag-pUL77タンパク質のウエスタンブロット分析の結果を示す。レーン1及び2, 感染後1日; レーン3及び4, 感染後2日; レーン5及び6, 感染後3日; レーン7及び8, 感染後4日; レーン1, 3, 5及び7, RdlUL76flagUL77; レーン2, 4, 6及び8, RflagUL77。
【図3】この図は、UL76 ORFにTAG stopコドンが挿入された組換えウイルスを示す。(a)は、後述の実施例の「材料と方法」に記載のように作製された組換えHCMV BAC DNAの図である。小文字は、UL76 ORFにTAG stopコドンを挿入するための変異塩基を示す。(b)は、約1のMOIでRUL76stopflagUL77又はRUL76revertantflagUL77で感染後表示された時間における前初期pIE72 (UL123), pIE86 (UL122), 初期p52 (UL44), 遅延初期pp65 (UL83), 後期pp28 (UL99)及びflag-pUL77タンパク質のウエスタンブロット分析の結果を示す。レーン1及び2, 感染後1日; レーン3及び4, 感染後2日; レーン5及び6, 感染後3日; レーン7及び8, 感染後4日; レーン1, 3, 5及び7, RUL76stopflagUL77; レーン2, 4, 6及び8, RUL76revertantflagUL77。
【図4−1】この図は、UL76中のフレームシフトがUL77発現を増大することを示す。(a)は、フレームシフトを有する又はフレームシフトを有さない、HCMV MIEプロモーターの下流に挿入されたUL76-77配列を含む発現プラスミドの図である。(b)は、UL76及びUL77融合タンパク質のウエスタンブロット分析の結果を示す。HeLa細胞を、フレームシフト変異を有する又はフレームシフト変異を有さないpflagUL76-77HAでトランスフェクトし、感染後48時間目に回収した。レーン1, pCMVflag-frame-shift-UL76-77HA; レーン2, pCMVflagUL76-77HA; レーン3, pCMVflagUL76-77-frame-shift-HA。
【図4−2】図4の(c)は、UL76-77 mRNAのin vitro転写を示す。キャップされたRNAを、後述の実施例の「材料と方法」に記載されるようにin vitroで転写し、その結果得られたRNAを、1%アガロース-ホルムアルデヒドゲル上で分析した。レーン1, RNA分子量マーカー; レーン2, pCMVflagUL76-77HA; レーン3, pCMVflag-frame-shift-UL76-77HA; レーン4, pCMVflagUL76-77-frame-shift-HA。(d)は、キャップしたRNAからの、UL76及びUL77の融合タンパク質のin vitro翻訳を示す。UL76-77 RNAを、後述の実施例の「材料と方法」に記載のようにin vitroで翻訳し、放射性標識したタンパク質をSDS-15% PAGEで分離した。レーン1, pCMVflag-frame-shift-UL76-77HA; レーン2, pCMVflagUL76-77HA; レーン3, pCMVflagUL76-77-frame-shift-HA。
【図5】この図は、発現プラスミド中のUL76 DNA又はその断片が下流のシストロンの翻訳を開始することができることを示す。(a)は、IRES活性を検出するために使用されたバイシストロン性ルシフェラーゼレポーター構築物の図である。構築物pcDNA3.1は、ホタルルシフェラーゼ遺伝子の下流のRenillaルシフェラーゼのコード領域を含んでいる。発現は、トランスフェクション実験においてHCMV MIEプロモーターによって駆動された。UL76のヌクレオチド1-71, 1-116, 1-190, 191-580, 271-580, 451-580及び1-580を含むDNA断片(最も大きい断片はUL77 ORFの上流領域を含む。)は、ベクターのインターシストロニック領域(ICS)内に位置するXho I及びBamH I中に挿入された。該上流領域の580bp断片はまた、アンチセンス配向に挿入された(r 1-580)。(b)は、UL76が仲介するIRES活性を示す。レポータープラスミドを、HeLa細胞内にトランスフェクトし、該細胞を感染48時間後に回収し、ホタル及びRenillaルシフェラーゼ活性を定量した。Renilla対ホタルルシフェラーゼ活性の比を計算した。なお、各トランスフェクションは3連で行われた。
【図6】この図は、IRESがUL76 RNA内に位置することを示す。HeLa細胞を、後述の実施例の「材料と方法」に記載のように、レポータープラスミドからin vitroで転写されたキャップされたRNAでトランスフェクトし、実施例の「材料と方法」に記載のようにトランスフェクション後6時間目に該細胞を回収した。相対的なRenilla ルシフェラーゼ活性(Renilla対ホタルルシフェラーゼ活性の比)をUL76又はその断片について計算し、既知のピコルナウイルスIRESと比較した。なお、各トランスフェクションは3連で行われた。
【図7】UL77の上流領域がUL30 ORFで置換された組換えウイルスの構築を示す。 (a) UL77の上流領域がUL30 ORFで置換された組換えウイルス。組換えウイルスを構築するために、UL30のPCR産物を相同組換えに使用した。(b) 野生型(wt)及び組換えウイルスのサザンブロット分析。ウイルスDNAを制限酵素BstB Iで消化し、1.0%アガロースの電気泳動で分画し、32P-標識プローブによるハイブリダイゼーションにかけた。左に標準分子サイズマーカーを塩基対数で示した。(c) 組換えウイルスに感染させた細胞でのUL77 ORFのN末端に融合させたflagエピトープをもつUL77タンパク質の発現の分析。Flagエピトープをもつ融合タンパク質を検出するために、F3165 (Sigma)を使用した。
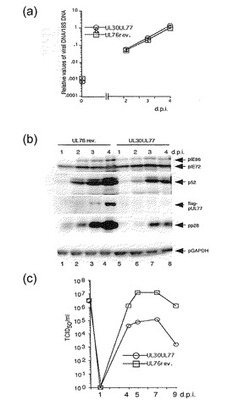
【図8】UL77の上流のUL76遺伝子がUL77遺伝子発現を調節することを示す。(a) 約3のMOIで組換えウイルスでHFF細胞を感染した後のウイルスDNA合成の分析。(b) 約3のMOIで組換えウイルスで感染した後のウイルスタンパク質のウエスタンブロット分析。ウイルスタンパク質は感染1, 2, 3及び4日後(d.p.i.)に分析した。レーン1及び5: 1 d.p.i.; レーン2及び6, 2 d.p.i.; レーン3及び7, 3 d.p.i.; レーン4及び8, 4 d.p.i.; レーン1〜4, RUL76revertantflagUL77; レーン5〜8, RUL30flagUL77。(c) 約3のMOIでの組換えウイルスの増殖曲線。ウイルス力価は、「材料と方法」に記載されるように、50%組織培養感染用量(TCID50)アッセイによって測定された。
【発明を実施するための形態】
【0021】
前述したように、本発明は、ヒトサイトメガロウイルス(HCMV)のUL76中のインターナルリボソームエントリーサイト(IRES)が、例えばピコルナウイルスIRESとほとんど同じ強さの活性をもつことの知見、並びに、UL76遺伝子が、IRESを介して、下流のUL77 ORFの翻訳に影響を及ぼしていることの知見に基づいている。具体的には、UL76中のIRESを不活性化することによってHCMVの複製の抑制、ひいては、HCMVの弱毒化を可能にした。ホルマリンなどの化学物質での処理による弱毒化では、外被(エンベロープ)タンパク質の変性などの問題も生じ兼ねないが、本発明の弱毒ウイルスは天然ウイルスの構造を保持するという利点を有する。
【0022】
<HCMV>
本発明の対象ウイルスであるHCMVは、すべてのウイルス株を包含するものとする。HCMVは、ヘルペスウイルス科βヘルペスウイルス亜科に属し、その直径が約180nmで約230kbpの塩基数からなる二本鎖DNAウイルスである。HCMVの株には公知の株が含まれ、例えばAD169株,Towne株,Major株,BT1943株,Davis株,Merlin株,Toledo株,3157株,6397株,3301株, C87株などが知られている(Dolan, A.ら(2004) J.Gen.Virol. 85:1301-1312; Chee, M.S.ら (1990) Curr.Top.Microbiol.Immunol.154:125-169)。
【0023】
上記のHCMV株のゲノム配列は、例えばGenBank(NCBI,米国)などに蓄積されたデータから入手可能である。例えば、一般によく知られているAD169株及びのTowne株ゲノム配列はそれぞれGenBank Accession Number FJ527563、AY315197として登録されている。
【0024】
HCMVは、ヒト線維芽細胞、例えばヒト包皮線維芽 (HFF)細胞(ATCC CC2509)やヒト胎児線維芽細胞、ヒト内皮細胞、例えばヒト毛細血管内皮細胞(HMVEC)(ATCC CC-2527)やヒト網膜色素内皮細胞(RPE)(ATCC C4000-1)などのヒト細胞に感染することができるので、このような細胞に、HCMV又はそのウイルスベクター、例えばHCMVゲノム配列とBAC(細菌人工染色体)配列から作製されたHCMVBAC、を感染又は導入することによってウイルスDNAを回収することができる(W. Dunnら (2003) Proc. Natl. Acad. Sci. USA, 100(24):14223-14228)。
【0025】
HCMVのUL76タンパク質は、ヘルペスウイルスにおいて高度に保存されたタンパク質ファミリーのメンバーである。UL76ファミリーメンバーは、毒性の決定、細胞溶解性複製、遺伝子発現の調節、アポトーシスの誘導などに関与することが最近指摘され、またウイルス複製の前初期でUL76タンパク質がHCMVの複製を抑制する作用があることが提案されている(V.-K. Siew et al., J. Biomed. Sci., 16:107 (2009);S.-K. Wang et al., J. Virol, 78(18):9750-9762 (2004))。しかし、UL76のウイルス複製抑制に関しては、UL76にウイルス複製に関係するタンパク質が存在するだろうということ以外の発展的な知見は提供されていない。本発明者らは、今回、ウイルス複製に重要であるパケージングに特定の役割をもつUL77 ORFの発現及び翻訳に、UL76遺伝子が、その中に含まれるIRESを介して影響を及ぼしていることを見出した。この知見から、IRESを不活性化することによってウイルス複製を阻害しうることを見いだした。
【0026】
IRES(Internal Ribosome Entry Site)は、一般に、タンパク質合成過程においてmRNA分子の5’末端での翻訳開始の反応に重要な役割をもつヌクレチド配列であるが、RNAウイルスのゲノムでもIRESの存在が確認されており、またDNAウイルスでもIRESの存在が確認されている。本発明では、DNAウイルスであるHCMVのUL76遺伝子配列内に存在するIRES配列が、下流タンパク質コード領域(UL77)の翻訳に関与していると考えられる。
【0027】
UL76中のIRESの不活性化は、以下の「弱毒化」の項で説明するように、上記IRES配列に突然変異を導入する方法などによって行うことができる。好ましい突然変異はIRES配列をIRES以外の別の核酸配列によって置換することである。ここで「IRES以外の」という用語は、UL76 IRESの天然型配列を有しない、かつ翻訳開始などのIRES本来の機能をもたないことを意味している。そして、そのようなIRES以外の別の核酸配列は、例えば、該IRES配列における1個又は複数のヌクレオチドの置換、欠失、付加、挿入、又はそれら変異の組合せなどの突然変異を含む配列からなり、UL76 IRES配列が別の核酸配列に置き換わることによって該IRESが不活性化される。この場合、上記別の核酸配列のサイズ(すなわち、ヌクレオチド数)は、上記IRES配列のサイズと完全に同じか又はほほ同じであることが好ましい。別の核酸配列の例は、非限定的に、HCMVゲノムのなかで非機能性であるようなUL(unique long)配列から選択してもよく、そのようなUL配列は、例えば同じ又は異なるHCMV株由来のUL30配列などである。上記のIRESの不活性化によって、HCMVの複製が抑制された弱毒HCMVウイルスを得ることができる。
【0028】
HCMV UL76及びUL30の配列について、例えば、AD169株(GenBank: FJ527563)及びTowne株(GenBank: FJ616285.1)のUL76及びUL30の配列は、次のとおりである。なお、他のHCMV株のUL76配列及びUL30配列もまた、GenBankなどの公知の遺伝子バンク或いは文献などから入手可能である。
【0029】
・AD169株のUL76の塩基配列(下線はIRES配列を示す。)及びアミノ酸配列:
(塩基配列、配列番号1)
ATGCCGTCCG GGCGTGGGGA CGACGCTGAT TCGACGGGGA ACGCTCTGCG GAGATTGCCT
CACGTGCGTA AGCGGATCGG TAAGCGTAAG CACCTGGACA TCTACCGTCG CCTGTTGCGG
GTCTTTCCCT CGTTTGTGGC CCTCAACCGC CTGTTGGGAG GCCTTTTCCC ACCCGAGCTG
CAAAAGTACC GTCGCCGTCT TTTCATCGAA GTACGATTAA GTCGGCGGAT TCCCGACTGC
GTGTTGGTGT TTTTACCGCC GGACTCTGGG TCGCGCGGCA TCGTGTATTG CTACGTGATT
GAGTTCAAAA CTACGTACTC AGACGCCGAC GATCAGTCCG TGCGGTGGCA CGCCACCCAC
AGCCTGCAGT ACGCCGAGGG CCTGCGCCAG CTCAAGGGCG CCTTGGTGGA CTTTGATTTT
CTGCGTCTGC CGCGCGGTGG CGGTCAAGTC TGGAGCGTAG TGCCCAGTCT GGTTTTTTTT
CAGCAAAAGG CCGATCGCCC ATCTTTTTAC CGGGCTTTTC GTTCGGGCCG TTTCGACTTG
TGTACCGATT CTGTCCTGGA CTATCTGGGA CGGCGTCAGG ATGAGTCTGT TGCACACCTT
TTGGCGGCTA CCCGTCGCCG TCTTCTTCGA ACCGCACGAG GAAAACGTGC TGCGCTGCCC
CGAGCGCGTG CTTCGGCGGT TGCTGGAGGA CGCGGCGGTG ACAATGCGCG GCGGGGGCTG
GCGCGAGGAC GTGCTCATGG ACCGGGTGCG CAAACGGTAT CTGCGTCAGG AGCTCAGGGA
TCTGGGTCAC AGGGTGCAGA CTTACTGCGA GGATCTCGAA GGGCGCGTGT CCGAGGCGGA
GGCGCTGTTG AACCAGCAGT GCGAGCTCGA CGAAGGACCG TCGCCGCGGA CGCTGCTACA
ACCACCGTGT CGTCCGCGTT CTTCGTCCCC AGGGACCGGC GTGGCAGGAG CTTCTGCCGT
CCCACACGGT CTTTATAG
(アミノ酸配列、配列番号2)
MPSGRGDDADSTGNALRRLPHVRKRIGKRKHLDIYRRLLRVFPSFVALNRLLGGLFPPELQKYRRRLFIEVRLSRRIPDCVLVFLPPDSGSRGIVYCYVIEFKTTYSDADDQSVRWHATHSLQYAEGLRQLKGALVDFDFLRLPRGGGQVWSVVPSLVFFQQKADRPSFYRAFRSGRFDLCTDSVLDYLGRRQDESVAHLLAATRRRLLRTARGKRAALPRARASAVAGGRGGDNARRGLARGRAHGPGAQTVSASGAQGSGSQGADLLRGSRRARVRGGGAVEPAVRARRRTVAADAATTTVSSAFFVPRDRRGRSFCRPTRSL
【0030】
・AD169株のUL30の塩基配列及びアミノ酸配列:
(塩基配列、配列番号3)
ATGACGACATCAGCGATGACGGCTCCTGATACGCGCCGGCAGCTGCAGCACGTGGAGACGCTGCGTCGGTTTCTGCGCGGCGACAGCTGCTTTGTGCACGATCTCCGGGGCATGATGGACTATCACGACGGGCTCTCGCGCCGTCAACAGCGTGCCTTTTGCCGCGCGAGTCGCGTGTTGACGGACCCGGAGCCCATCCAGAGCGAAGCGGAGGGGGAGAATAAACAGTTTACGGAGCACACACACAAAGTAGTCTCGTTTTTTATTAAAAGTGTCTTTGTATTTCCCTATCTTGTGTTGCCCAACTGCTGTCAGGTCTCCGTAGATCGCTCCCGGGTGCCCGAGACGGGGGGCAGATGGCTATAA
(アミノ酸配列、配列番号4)
MTTSAMTAPDTRRQLQHVETLRRFLRGDSCFVHDLRGMMDYHDGLSRRQQRAFCRASRVLTDPEPIQSEAEGENKQFTEHTHKVVSFFIKSVFVFPYLVLPNCCQVSVDRSRVPETGGRWL
【0031】
・Towne株のUL76の塩基配列(下線はIRES配列を示す。)及びアミノ酸配列:
(塩基配列、配列番号5)
ATGCCGTCCG GGTGTGGGGA CGACGCTGAT TCGAAGGGGA ACGCTCTGCG GAGATTGCCT
CACGTGCGTA AGCGGATCGG TAAGCGCAAG CACCTGGACA TCTACCGTCG CCTGCTGCGG
GTCTTTCCCT CATTTGTGGC GCTCAACCGC CTGTTGGGAG GCCTTTTCCC ACCCGAGTTG
CAAAAGTACC GTCGCCGTCT TTTCATCGAA GTACGATTAA GTCGGCGGAT TCCCGACTGC
GTGTTGGTGT TTTTACCGCC GGACTCTGGG TCGCGCGGCA TCGTGTATTG CTACGTGATT
GAGTTCAAAA CTACGTACTC AGACGCCGAC GATCAGTCCG TGCGGTGGCA CGCCACCCAC
AGCCTGCAGT ACGCCGAGGG CCTGCGCCAG CTCAAGGGCG CCTTGGTGGA CTTTGATTTT
CTGCGTCTGC CGCGCGGTGG CGGTCAAGTC TGGAGCGTAG TGCCCAGTCT GGTTTTTTTT
CAGCAAAAGG CCGATCGCCC ATCTTTTTAC CGGGCTTTTC GTTCGGGCCG TTTCGACCTG
TGTACCGATT CTGTTCTGGA CTATCTGGGA CGGCGTCAGG ATGAGTCTGT TGCACACCTT
TTGGCGGCTA CCCGTCGCCG TCTTCTTCGA GCCGCACGAG GAAAACGTGC TGCGCTGCCC
CGAGCGCGTG CTTCGGCGGT TGTTGGAGGA CGCGGCGGTG GCAATGCGCG GCGGGGGCTG
GCGCGAGGAC GTGCTCATGG ACCGGGTGCG CAAACGGTAT CTGCGTCAGG AGCTGAGGGA
TCTGGGTCAC AGGGTGCAGA CTTACTGCGA GGATCTCGAA GGGCGCGTGT CCGAGGCGGA
GGCGCTGTTG AACCAGCAGT GCGAGCTCGA CGAAGGACCG TCGCCGCGGA CGCTGCTACA
ACCACCGTGT CGTCCGCGTT CGTCGTCCCC AGGGACCGGC GTGGCAGGAG CTTCCGCCGT
CCCACACGGT CTTTATAG
(アミノ酸配列、配列番号6)
MPSGCGDDADSKGNALRRLPHVRKRIGKRKHLDIYRRLLRVFPSFVALNRLLGGLFPP
ELQKYRRRLFIEVRLSRRIPDCVLVFLPPDSGSRGIVYCYVIEFKTTYSDADDQSVRW
HATHSLQYAEGLRQLKGALVDFDFLRLPRGGGQVWSVVPSLVFFQQKADRPSFYRAFR
SGRFDLCTDSVLDYLGRRQDESVAHLLAATRRRLLRAARGKRAALPRARASAVVGGRG
GGNARRGLARGRAHGPGAQTVSASGAEGSGSQGADLLRGSRRARVRGGGAVEPAVRAR
RRTVAADAATTTVSSAFVVPRDRRGRSFRRPTRSL
【0032】
・Towne株のUL30の塩基配列及びアミノ酸配列:
(塩基配列、配列番号7)
ATGACGACATCGGCGATGACGGCTCCTGATACGCGCCGACAGCTGCAGCACGTGGAGACG
CTGCGTCGGTTTCTGCGCGGCGACAGCTGCTTTGTGCACGATCTCCGGGGCATGATGGAC
TATCACGACGGGCTCTCGCGCCGTCAACAGCGTGCCTTTTGCCGCGCGGGTCGCGTGTTG
ACGGACCCGGAGCCCATCCAGAGCGAGACGGAGGGGGAGAATAAACAGTTTACGGAGCAC
ACACACAAAGTAGTCTCGTTTTTTATTAAAAGTGTCTTTGTATTTCCCTGTCTTGTGTTG
CCCAACTGCTGTCAGGTCTCCGTAGATCGCTCCCGGGTGCCCGAGACGGGGGGCAGATGG
CTATAA
(アミノ酸配列、配列番号8)
MTTSAMTAPDTRRQLQHVETLRRFLRGDSCFVHDLRGMMDYHDGLSRRQQRAFCRAGRV
LTDPEPIQSETEGENKQFTEHTHKVVSFFIKSVFVFPCLVLPNCCQVSVDRSRVPETGG
RWL
【0033】
<弱毒化>
本発明では、HCMV UL76中のIRESを不活性化することによってHCMVを弱毒化する。
本明細書で使用される「弱毒化」なる用語は、ウイルスの毒性(すなわち、感染性および病原性)を減弱することを意味する。
【0034】
IRESの不活性化は、IRESのUL77翻訳開始に関する機能を阻害することを意味し、該不活性化は、IRES配列の突然変異、例えばIRES配列の一部又は全部を別のヌクレオチド配列によって置換、欠失、付加又は挿入することによって行うことができる。好ましくは、IRES配列の全部又はほとんど全部をIRES以外の別の核酸配列によって置換することによってIRESを不活性化することができる。より好ましくは、IRES配列と実質的に同じ長さ(サイズ)の別のヌクレオチド配列である。しかし、IRESのUL77翻訳開始に関する機能を阻害することができるならば、突然変異の度合い(例えば配列同一性の度合い)、突然変異の仕方などに限定されないものとする。
【0035】
本発明の実施形態によれば、HCMVのUL76 IRES以外の核酸配列がHCMV UL30配列によって置換されうる。このUL30配列は、UL76 IRESと同じ又は異なるHCMV株に由来することができる。例えば、弱毒HCMVウイルスがTowne株をベースにする場合、そのUL76 IRES配列をTowne株のUL30配列で置換してもよいし、或いはAD169株などの他の株由来のUL30配列で置換してもよい。このとき、UL30配列のサイズがIRES配列のサイズと実質的に同程度(例えば±5%、好ましくは±3%の範囲内)であることが好ましく、そうすることによってHCMVの複製能がより顕著に低下する。
【0036】
IRESのUL77翻訳開始に関する機能が阻害されることによって、UL77のパケージング機能が阻害され、ひいてはHCMVの複製が抑制される。突然変異の導入によって、組換えHCMVの複製能が約1/10以下、1/50以下、好ましくは1/100以下に抑制される。
【0037】
本発明の組換えHCMVは、野生型HCMVのUL76中のIRES配列の一部又は全部に突然変異を導入したゲノムDNAを、適当なベクターを利用して作製し、これを適当な宿主細胞、例えば上記のヒト線維芽細胞やヒト内皮細胞に感染し、組換えウイルス粒子を回収することを含む手順によって作製することができる。突然変異は、例えば部位特異的突然変異誘発法、PCRを利用した突然変異誘発法、相同組換え法などの公知の方法によって導入しうる。部位特異的突然変異誘発法は、例えば、野生型HCMVのUL76 IRES配列の一部又は全部に突然変異を導入し、それに隣接する野生型と同一の十分な長さの配列を配置したヌクレオチド配列を合成し、クローニングベクターに導入して大腸菌などの細菌中にサブクローニングすること、野生型HCMVゲノムDNAを含む人工染色体ベクター、例えば細菌人工染色体(BAC, PAC)を作製し、ついで、上記ヌクレオチド配列を人工染色体ベクターにアニーリングし、PCRを行ない、目的DNAを増幅 することなどを含む。
【0038】
具体的な方法として、例えば大腸菌内でバクテリオファージλ組換えタンパク質exo、betaおよびgamを発現するための迅速相同組換え系(Ellis, H.M.ら(2001) Proc. Natl.Acad.Sci.USA98:6742-6746)、HCMVのBAC(細菌人工染色体)DNA (Dunn,W ら(2003)Proc.Natl.Acad.Sci.USA100:14223-14228; Lee, E-C.ら(2001)Genomics73:56-65; Borst,E-Mら(1999)J.Virol.73(10):8320-8329)などを使用して突然変異誘発を行うことができる。
【0039】
組換えのためのHCMVの二本鎖DNAは、カナマイシン耐性遺伝子などの薬剤耐性遺伝子と70bp程度の相同ウイルスDNA配列を含み、この耐性遺伝子には、34bpの最小FRT部位をフランキングさせることができる(後述の実施例1参照)。UL76 IRES配列の突然変異体を作製するために、適当なプライマーを使用してPCRによる増幅を行う。PCR条件は、例えば、94℃2分の変性を1サイクル、94℃15秒の変性, 55℃30秒のアニーリング及び72℃1分の伸長を30サイクル、並びに、72℃5分の伸長を1サイクルとするPCRサイクルプログラムからなる。PCR増幅産物をフェノール−クロロホルムで抽出し、95%エタノールで沈澱する。
【0040】
残存する鋳型DNAを除くために、PCR産物を、制限酵素を用いて消化したのち、得られたDNA断片を、HCMV-BAC DNAを含むコンピテント大腸菌中へエレクトロポレーション、リポフェクション、リン酸カルシウム法などの手法、好ましくはエレクトロポレーションを用いて形質転換する。
【0041】
カナマイシン耐性遺伝子を欠失するために、組換えHCMV-BAC DNAを大腸菌に形質転換する。また、リコンビナーゼを発現するプラスミドpCP20(Hahn,W.ら (2003)Virology307:164-177)を、組換えHCMV-BAC DNAを含む大腸菌(DH10B)に形質転換する。カナマイシン耐性遺伝子を含まないHCMV BAC DNAを、アンピシリンおよびクロラムフェニコール含有のLBプレート上で選択する。
【0042】
HFF細胞などの細胞を、リン酸カルシウム沈殿法(Graham,F.L.およびvan der Eb,A.J.(1973)Virology52:456-467)によって、プラスミドpSVpp71の存在中、上記の各組換えBACでトランスフェク ションする。約10日後に、ウイルスプラークが出現し、100%細胞変性作用(CPE)の7日後に、細胞外液を回収し、約1:10に希釈したものと未希釈のものを調製し、HFF細胞などの細胞に感染し、組換えウイルスを回収する。このとき、ウイルスの選択は、IRES配列に目的の変異が導入されていることを配列分析によって確認すること、並びに、HFF細胞などの細胞に導入または感染させて複製が抑制されていることを確認し、また、野生型の表現型と同一または本質的に同一であることを確認することによって行うことができる。
【0043】
<ワクチン>
本発明はさらに、弱毒HCMVを含むことを特徴とする、HCMV感染症を治療または予防するためのワクチンを提供する。
【0044】
ワクチンは、一般に生ワクチンと不活性化ワクチンに大別されるが、特に生ワクチンは安価に製造可能であることや接種回数の少なくて高い効果が期待できるなどの利点とともに毒性復帰のリスクがある。本発明の弱毒HCMVは、パッケージングが阻害されるため顕著にウイルス複製が抑制されたウイルスであるため、安全性の高い生ワクチンとして使用可能であると考えられる。
【0045】
本発明のワクチンは、AIDS患者、臓器移植患者などの免疫抑制患者、胎児、乳幼児、高齢者、重症患者などの抵抗性が低下したヒトに対し、肺炎、肝炎、網 膜炎、胃腸炎などを含む広範な疾患を引き起こす可能性のあるHCMV感染の予防および治療のために使用可能である。HCMVは、血液、尿、唾液などを介して感染するウイルスであり、上記のヒトに感染すると、重症になる場合がある。そのような感染を抑制するために、本発明のワクチンは有効である。
【0046】
本発明のワクチンは、適当な賦形剤、特に希釈剤、例えば滅菌水、生理食塩水、リンゲル液、緩衝液などに処方されうる。また、ブドウ糖、トレハロース、エタノール、グリセリンなどの添加剤を含有させることができる。さらにまた、免疫応答を増強させるためのアジュバント、例えば水酸化アルミニウム(alum)、ムラミルジペプチド、サポニン類、リポポリサッカライド、リポゾーム、消化性ポリマー(Gupta, R.K. et al., Vaccine 13:1263-1276 (1995))などをその有効量で含有させてもよい。さらにまた、安定剤を添加してもよく、安定剤にはゼラチン、ヒト血清アルブミンなどのタンパク質、ポリソルベートなどの界面活性剤、スクロース、トレハロースなどの糖類、メチオニン、リジンなどのアミノ酸類などが含まれる。
【0047】
投与方法は、例えば皮下、筋肉内または皮内注射、鼻腔内などの粘膜投与などを含む。鼻腔内投与の場合、噴霧器による投与が好ましい。投与量は、例えば103〜1011PFUウイルス/kg体重、又は1回あたり10μg〜10mg、の用量であるが、この範囲に制限されない。ワクチンの投与量は、患者などの被験者の状態、年齢、体重、性別、重症度などを考慮に入れて、医師の裁量で判断されるべきである。被験者は、1回又は2回以上の投薬を受容でき、複数回投与の場合、1週間〜1か月以上の間隔、例えば2,3,6,12,18又は24か月間隔で投与しうる。
【0048】
<抗体の作製>
本発明はさらに、上記弱毒HCMVを非ヒト動物に免疫して抗体を作製し、該抗体を回収することを含む、抗HCMV抗体の作製方法を提供する。
【0049】
抗体は、IgA、IgG、IgD、IgE、IgMのいずれのクラスでもよいし、またIgG1、IgG2、IgG3、IgG4、IgA1、IgA2などのいずれのサブクラスでもよい。さらにまた、抗体は、ポリクローナル抗体、モノクローナル抗体、抗体フラグメント、組換え抗体のいずれでもよい。組換え抗体には、例えば単鎖Fv抗体(scFv)、単鎖ダイアボディ(scDb)などが含まれる(浅野竜太郎,生化学77(12):1497-1500(2005))。抗体フラグメントには、例えばFab、F(ab’)2、Fvなどが含まれる。
【0050】
また、抗体には、ヒト抗体、ヒト化抗体、キメラ抗体なども包含され、好ましい抗体はヒト抗体である。
抗体の作製は、慣用の方法で行うことができる。
【0051】
ポリクローナル抗体は、弱毒HCMV粒子を動物に免疫し、抗血清、さらにIgG画分を回収することによって作製できる。
【0052】
モノクローナル抗体は、弱毒HCMV粒子を免疫した動物(例えばマウス、ラットなど)から脾臓を取り出し、脾臓細胞又はリンパ球とミエローマ細胞とを融合させ、該HCMV粒子抗原と特異的に免疫反応する抗体をスクリーニングすることによって作製できる。
【0053】
ヒト抗体は、ヒト抗体産生非ヒト動物(例えばKMマウス、XenoMouse)を用いて作製することができる。あるいは、弱毒HCMV粒子を免疫した動物のゲノムから抗体遺伝子を回収し、重鎖可変領域及び軽鎖可変領域の各CDR1〜CDR3コード配列を決定し、ヒト抗体遺伝子の本来のCDR1〜CDR3コード配列を前記CDR1〜CDR3コード配列と置換することを含む遺伝子組換え技術を用いてヒト抗体を作製することができる。
【0054】
上記の抗体は、ポリエチレングリコールなどの分子によって化学修飾されてもよい。
上記のようにして得られた抗体は、HCMV感染症の治療や予防のために使用することができるし、或いはHCMVの検出のために使用することもできる。ここで、HCMVの検出のために、抗原-抗体反応を利用する手法のいずれも使用できる。そのような手法には、例えば均質法、固相法(例えばサンドイッチ法)などが含まれる。
【実施例】
【0055】
以下に実施例を挙げて本発明をさらに具体的に説明するが、本発明の範囲は、該実施例によって制限されないものとする。
【0056】
[実施例1]
<材料と方法>
細胞及びウイルス
初代ヒト包皮線維芽(HFF)細胞を、従来法に従い(39)、10%牛新生仔血清(Sigma, St. Louis, Mo.)、ペニシリン(100 U/ml)及びストレプトマイシン(100 μg/ml)を補充したEagle最少必須培地(Wako,大阪,日本)中、5%CO2中に37℃に維持した。従来法に従い(17, 31)、HFF細胞での標準プラークアッセイによって、ウイルス力価を測定した。
【0057】
酵素
制限エンドヌクレアーゼは、New England Biolabs Inc.(Beverly, MA.)から購入された。高忠実度のかつ拡大された高忠実度のTaq DNAポリメラーゼはそれぞれInvitrogen及びRoche(Carlsbad, CA.及びMannheim, Germany)から購入された。RNasin及びRNase非含有DNaseはPromega (Madison, WI.)から購入された。これらの酵素は、製造者の説明書に従って使用された。
【0058】
HCMV BAC DNAの突然変異誘発
バクテリオファージλ組換えタンパク質,エクソ(exo),β及びγを発現する大腸菌(D. Court博士(NIH, MD.)から恵与された。)での迅速組換え系を、従来法に従い使用した(8)。HCMV TowneのBAC DNAは、F. Liu 博士(University of California, Berkeley, CA)から恵与された(7)。組換え用の二本鎖DNAは、カナマイシン耐性遺伝子、それに隣接する34bp最小FRT部位(5’-GAAGTTCCTATTCTCTAGAAAGTATAGGAACTTC-3’(配列番号55))(15)、又は、カナマイシン耐性遺伝子とストレプトマイシン感受性遺伝子(RpsLneo, Gene Bridges, Dresden, Germany)及び70bpの相同なウイルスDNA配列を含んでいた。UL76配列の欠失を作るため、次のプライマー対、すなわち、BACdlUL76FRTFKanF: 5’-ATTTAATACGATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCGAAGTTCCTATTCTCTAGAAAGTATAGGAACTTCAACTCAGCAAAAGTTCGATTTATTCAAC-3’(配列番号9); BACdlUL76FRTRKanR: 5’-CCTCGTGCGGTTCGAAGAAGACGGCGACGGGTAGCCGCCAAAAGGTGTGCAACAGACTCATGAAGTTCCTATACTTTCTAGAGAATAGGAACTTCTAATGCTCTGCCAGTGTTACAACCA-3’(配列番号10)を使用した。UL77 ORFとflagエピトープタグ (5’- ATGGACTACAAAGACGATGACGACAAG-3’(配列番号11))のN末端融合体を作製するために、以下に記載のように、逆の手順をとった。RpsLneo遺伝子を、次のプライマー対BACUL76neoF: 5’- CGGGCTTTTCGTTCGGGCCGTTTCGACTTGTGTACCGATTCTGTCCTGGACTATCTGGGACGGCGTCAGGGGCCTGGTGATGATGGCGGGATC-3’(配列番号12); BACUL76neoR: 5’- GCACGTTTTCCTCGTGCGGTTCGAAGAAGACGGCGACGGGTAGCCGCCAAAAGGTGTGCAACAGACTCATTCAGAAGAACTCGTCAAGAAGG-3’(配列番号13)を用いてHCMV BAC DNAに挿入した。カナマイシン耐性遺伝子(KanR)のいずれかの末端に50bpの相同なウイルスDNA配列を有するKanRを、従来法に従いPCRにより増幅した(18, 20, 22)。PCRによるRpsLneo遺伝子の増幅のために、pRpsL-neo(Gene Bridges)を鋳型として用いた。残りの鋳型DNAを除くために、PCR産物を、DpnIによって37℃で1.5時間消化した。
【0059】
従来法に従い逆の手順も行った(46)。簡単に説明すると、50bpの相同なウイルスDNAを含む約500ngの組換え用一本鎖DNA をRpsLneo 遺伝子を用いてHCMV BAC DNAに導入した(19, 21, 46)。RpsLはストレプトマイシン感受性遺伝子であるので、変異型BAC DNAを、Counter Selection Modificationキット(Gene Bridges)を用いてストレプトマイシン耐性の増加に基づき選択した。BACdlUL76flagUL77及びBACUL76flagUL77を構築するために、BACdlUL76flagoligo-2及びBACUL76flagoligo-2 をそれぞれ組換えのために使用した。それらの配列は以下のとおりである。BACdlUL76flagoligo-2: 5’- CGATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCATGGACTACAAAGACGATGACGACAAGAGTCTGTTGCACACCTTTTGGCGGCTACCCGTCGCCGTCTTCTTCGA-3’(配列番号14);BACUL76flagoligo-2: 5’- TTTCGACTTGTGTACCGATTCTGTCCTGGACTATCTGGGACGGCGTCAGGATGGACTACAAAGACGATGACGACAAGAGTCTGTTGCACACCTTTTGGCGGCTACCCGTCGCCGTCTTCTTCGA-3’(配列番号15)
【0060】
flagエピトープを下線で示した。RUL76stopflagUL77及びRUL76revertantflagdUL77を作製するために、上記のとおりに逆手順も用いた。RpsLneo遺伝子を、従来法に従い、以下のプライマー対:BACUL76stopneo+StF: 5’- GATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCATGCCGTCCGGGCGTGGCCTGGTGATGATGGCGGGATC-3’(配列番号16); BACUL76stopneo+StR: 5’- ACCGATCCGCTTACGCACGTGAGGCAATCTCCGCAGAGCGTTCCCCGTCGAATCAGCGTCGTCCTCAGAAGAACTCGTCAAGAAGG -3’(配列番号17)を用いてRflagUL77 BAC DNA中に挿入した。また、以下のオリゴ: BAColigoUL76stop: 5’- AATACGATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCATGCCGTCCGGGCGTtaGGACGACGCTGATTCGACGGGGAACGCTCTGCGGAGATTGCCTCACGTGCGTAAGCGGATCGGTAAGCG-3’(配列番号18); BAColigoUL76revertant: 5’- AATACGATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCATGCCGTCCGGGCGTGGGGACGACGCTGATTCGACGGGGAACGCTCTGCGGAGATTGCCTCACGTGCGTAAGCGGATCGGTAAGCG-3’(配列番号19)を用いて逆選択を行った。小文字は、UL76 ORF中にTAG stopコドンを挿入するための変異塩基を示す。各変異に対して2つの組換えウイルスが単離され、それらは同じウイルスを振舞うことが判明した。
【0061】
カナマイシン耐性遺伝子の除去
カナマイシン耐性遺伝子を欠失させるために、組換えHCMV BAC DNAを、大腸菌DH10B中に形質転換した。プラスミドpCP20 (G. Hahn博士(Max von Pettenkofer Institute, Munich, Germany)から恵与された。)を、組換えHCMV BAC DNAを含むDH10B中に形質転換した。カナマイシンを含まないHCMV BAC DNAを、アンピシリン及びクロラムフェニコール含有のLBプレート上で選択した。
【0062】
PCR分析
組換えBAC DNAを選択するために、PCR分析を以下のプライマー対: UL76detectF: 5’- TACGGGTTACAAAAGTCGCGTCTCTGTCT-3’(配列番号20)及びUL76detectR: 5’ -GCTCGGGGCAGCGCAGCACGTTTT-3’(配列番号21)を用いて行った。PCRサイクルプログラムは、94℃2分の変性を1サイクル、94℃15秒の変性, 55℃30秒のアニーリング及び72℃1分の伸長を30サイクル、並びに、72℃5分の伸長を1サイクルからなった。PCR産物を、pCR 2.1-TOPO TAクローニングベクター (Invitrogen)中にクローニングし、配列決定して組換えと除去を確認した。
【0063】
組換えウイルスの単離
HFF細胞を、2μgのプラスミドpSVpp71の存在下、各組換えBACを5μgもしくは10μgを用いてGrahamとVan der Eb (12)のリン酸カルシウム沈殿法によってトランスフェクトした。10日後、ウイルスプラークが現れた。100%の細胞変性作用(CPE)の7日後に、細胞外液を回収し、HFF細胞の感染のために、1:10に希釈し、又は無希釈とした。100%CPEの5〜7日後に、ウイルス含有細胞外液を、使用するまで、50%牛新生仔血清中-80℃に保存した。
【0064】
プラスミドの構築
プラスミドpCMVflagUL76-77HA, pCMVflag-frame-shift-UL76-77HA及びpCMVflagUL76-77-frame-shift-HAを以下のように構築した。UL76 ORFのN末端に融合したflagエピトープ及びUL77 ORFのC末端に融合したHAエピトープを有するUL76〜UL77領域をpCMVflagUL76-77HAについてプライマー対EcoRIflagUL76F及びXbaIHAUL76R, pCMVflag-frame-shift-UL76-77HAについてプライマー対EcoRIframe-shiftflagUL76及びXbaIHAUL76R、並びにpCMVflagUL76-77-frame-shift-HAについてEcoRIflagUL76F及びXbaIframe-shiftHAUL76Rを用いてHCMV TowneのBAC DNAからPCRによって増幅した。プライマー配列は以下のとおりである:
EcoRIflagUL76F:
5’- GGAATTCATGGACTACAAAGACGATGACGACAAGCCGTCCGGGCGTGGGGACGACG-3’(配列番号22);
EcoRIframe-shiftflagUL76:
5’- GGAATTCATGACTACAAAGACGATGACGACAAGCCGTCCGGGCGTGGGGACGACG-3’(配列番号23);
XbaIHAUL76R:
5’- GCTCTAGATTAAGCGTAATCTGGAACATCGTATGGGTACAACACCGCCACGCTCGGAAGAAACC-3’(配列番号24);
XbaIframe-shiftHAUL76R:
5’-GCTCTAGATTAAGCGTAATCTGGAACATCGTATGGGTACAACACCGCCACGCTCGGAAGAAACCCAACACTA-3’(配列番号25)
【0065】
PCR産物を制限エンドヌクレアーゼEcoR I及びXba Iで消化し、対応の制限エンドヌクレアーゼ部位でプラスミドpcDNA3.1(+) (Invitrogen)にクローン化し、そして配列決定した。プラスミドpCMVFlucRluc は次のように構築された。pSp-luc+NF融合ベクター (Promega)由来のホタルルシフェラーゼを、制限エンドヌクレアーゼEcoR I及びHind IIIで消化し、対応の制限エンドヌクレアーゼ部位でプラスミドpcDNA3.1(+) (Invitrogen)にクローニングし、これをpCMVlucと名付けた。pGL3hRlucベクター(Pomega) 由来のRenilla ルシフェラーゼ遺伝子を、制限エンドヌクレアーゼEcoR I及びApa Iで消化し、 対応の制限エンドヌクレアーゼ部位でpCMVluc中にクローン化した。最後に、インタークローニング部位(ICS)を構築するためのDNA断片を、以下のオリゴDNAを用いて構築した。
MCSsense: 5’- AATTCGGTACCCTCGAGAGATCTGGATCCG-3’(配列番号26)、及び
MCSantisense: 5’- AATTCGGATCCAGATCTCTCGAGGGTACCG-3’(配列番号27)
【0066】
アニーリングされたDNAは、制限エンドヌクレアーゼEcoR Iによって消化されたpCMVFlucRluc中にクローン化された。挿入されたインタークローニング部位は自動ジデオキシヌクレオチド配列決定によって決定された。UL76断片, UL76(71), UL76(116), UL76(190)及びUL76(580)を、以下のプライマー対セット:
XhoIUL76ORFF: 5’- CCGCTCGAGCCGTCCGGGCGTGGGGAC -3’(配列番号28)、及び
BamHIUL76 71R: 5’- CGGGATCCTTACGCACGTGAGGCAATCTCCGC -3’ (配列番号29)、
BamHIUL76 116R: 5’- CGGGATCCAACAGGCGACGGTAGATGTCCAGG -3’ (配列番号30)、
BamHIUL76 190R: 5’- CGGGATCCGGTACTTTTGCAGCTCGGGTGGG -3’ (配列番号31)、又は
BamHIUL76 580R: 5’- CGGGATCCCCTGACGCCGTCCCAGATAGTCC -3’ (配列番号32)
をそれぞれ用いるPCRによってHCMV TowneのBAC DNAから増幅した。同様に、UL76(191-580), UL76(271-580)及びUL76(451-580)を、以下のプライマーセット:
XhoIUL76 191F: 5’- CCGCTCGAGGTCGCCGTCTTTTCATCGAAGTACGATTAAG-3’(配列番号33)、
XhoIUL76 271F: 5’- CCGCTCGAGTCGCGCGGCATCGTGTATTGCTACG-3'(配列番号34)、又は
XhoIUL76 451F: 5’- CCGCTCGAGTGGAGCGTAGTGCCCAGTCTGG-3’(配列番号35)及び
BamHIUL76 580R(上記)
をそれぞれ用いるPCRによって増幅した。rUL76(580)の逆相補体もまた、プライマー対: BamHIUL76F (5’- CGGGATCCCCGTCCGGGCGTGGGGAC -3’ (配列番号36))及びXhoIUL76 580R (5’- CCGCTCGAGCCTGACGCCGTCCCAGATAGTCC -3’ (配列番号37))を用いるPCRによって増幅し、上記のレポータープラスミド中にクローン化した。ポリオウイルスIRES断片を、プライマー対:XhoIpolioIRESF (5’-CCGCTCGAGTTAAAACAGCTCTGGGGTTGTACCCACCCCAG-3’ (配列番号38))及びBamHIpolioIRESR (5’- CGGGATCCTATGATACAATTGTCTGATTGAAATAACTGTTGAAATTG-3’ (配列番号39))を用いてpRluc-wt-luc+ PV IRES (Dr. Nomoto(東京大学)から恵与された。)から増幅し、上記のレポータープラスミド中にクローン化した。
【0067】
サザンブロット分析
NucleoBondキット (Macherey-Nagel Duren, Germany)を用いて組換えBAC DNAを精製し、制限センドヌクレアーゼBstBIで消化し、0.6%アガロースゲル電気泳動(17)及びサザンブロット分析(31)にかけた。UL75プローブは文献に記載されている(20)。UL76 DNAは、プライマー対:UL76F (5’- CCGTCCGGGCGTGGGGACGA-3’ (配列番号40))及びUL76R (5’-CCGTCCCAGATAGTCCAGGACAGA-3’ (配列番号41))を用いるPCRによって上記のHCMV TowneのBAC DNAから増幅された。UL75又はUL76 DNA断片を、MegaprimeDNAラベリングシステム(Amersham Pharmacia Biotech.)を用いて32P-dCTP(Amersham Pharmacia Biotech Piscataway, NJ and PerkinElmer, MA )で標識することによってプローブ(図1参照)を作製した。
【0068】
ノザンブロット分析
20μgの細胞質RNAを、2.2Mホルムアミド含有1%アガロースゲル中の電気泳動にかけたのち、最大強度のHybond N+ (Amersham)にトランスファーした。IE1プローブを用いるノザンブロット分析を文献記載のように行った(31)。UL76ウイルスDNAは上記のように作製した。UL77 及びUL78ウイルスDNAを、プライマー対:UL77F (5’- CGTCTGGCCGACGACGTGAGTCGT-3’ (配列番号42))及びUL77R (5’-CGTCGACTCCAGAGAGAAAGCACGTC-3’ (配列番号43)), 並びに、UL78F (5’-CGTTAGCCTGGTCAACCTGCTGACT-3’ (配列番号44))及びUL78R (5’- ACGATGGAAAGAACCCAGGCAAAGGCG-3’ (配列番号45))をそれぞれ用いるPCRによって上記のHCMV TowneのBAC DNAから増幅した。32P-dCTPで標識することによって放射性標識プローブを得た。
【0069】
ウエスタンブロット分析
感染後、表示の時点で細胞を回収し、PBSで洗浄し、溶解バッファーで処理した(18, 22)。20μgのタンパク質アリコートを各レーンに載せてドデシル硫酸ナトリウム-10%ポリアクリルアミドゲル電気泳動(SDS-PAGE)を行い、そのあと、PVDF膜に転写した。IE1及びIE2によってコードされるpIE72及びpIE86タンパク質、UL44によってコードされるpp52、UL83によってコードされるpp65、並びにUL99によってコードされるpp28を検出するために、一次マウスモノクローナル抗体NEA-9221 (Perkin Elmer, Boston, MA.), M0854 (Dako, Carpinteria, CA.), 023-A (Vitrogen, Watertown, MA)及びCA 004 (EastCoast Bio, North Berwick, ME.)をそれぞれ使用した。flag又はHAエピトープとの融合タンパク質、又は対照としての細胞GAPDH、を検出するために、マウス或いはラットモノクローナル抗体F3165(Sigma, St. Louis, MO.)又は3F10 (Roche)、或いはMAB376 (Chemicon, Temucula, CA.)をそれぞれ使用した。増強された化学発光検出試薬(Enhanced chemiluminescence detection reagents)(Amersham)及び二次西洋ワサビペルオキシダーゼ標識抗マウスIgG抗体(Zymed, San Francisco, Ca.)或いは抗ラット IgG抗体(Jackson ImmunoResearch Laboratories Inc., West Grove, PA)を、製造者の説明書に従って使用した。
【0070】
リアルタイムRT-PCR分析
RNAの検出のために、全細胞RNAを、TRI試薬 (Invitrogen)を用いて表示の時点で精製し、DNase I (Promega)で処理し、製造者の説明書の手順に従って逆転写酵素(RT)(Roche) を用いてcDNAに変換した。第1cDNA鎖を、2μg RNAと250ngランダムプライマー (Roche)から20μlの最終容量で作製した。サンプルを70℃15分加熱して不活性化した。逆転写酵素を含まない対照では、投入したウイルスDNAもプラスミドDNAも検出されず、擬似(mock)対照に類似していた。RT反応は、従来法に従い行った(22)。PLATINUM Quantitative PCR SUPERMIX-UDG カクテル(Invitrogen)を含む25μlの最終容量で増幅を行ったが、各反応混合液には、2μgの第1cDNA鎖, 5mM MgCl2, 500nMの各プライマー、及び250nMの各プローブを含んでいた。HCMV UL77用の正方向及び逆方向プライマー及びレポータープローブ、ホタルルシフェラーゼ、及びRenillaルシフェラーゼは、Primer Express (Applied Biosystems)を用いて以下のように設計した。UL77-566F: 5’- ACGATCCCTTTATCCGCTTTC-3’ (配列番号46); UL77-633R: 5’ - GGCATTCTCGAACATGGTGTT-3’ (配列番号47); UL77 -589プローブ: 5’- FAM- ACCGATTTTCGCGGCGAGGTG(配列番号48)-テトラメチルローダミン (TAMRA)-3’, fireflyluc-1536F: 5’- GCGCGGAGGAGTTGTGTTT -3’ (配列番号49); fireflyluc-1607R: 5’- CTGATTTTTCTTGCGTCGAGTTT -3’ (配列番号50); ホタルluc-1556プローブ: 5’- FAM- TGGACGAAGTACCGAAAGGTCTTACCGG (配列番号51)- テトラメチルローダミン(TAMRA)-3’, renilla luc-300F: 5’- CCTCACCGCTTGGTTCGA-3’ (配列番号52); renilla luc-363R: 5’- CCAGTCGTGGCCCACAA-3’ (配列番号53); renilla-319プローブ: 5’ - FAM- CTGCTGAACCTTCCAAAGAAAATCATC(配列番号54)-テトラメチルローダミン(TAMRA)-3’(日本ジーン研究所,仙台)。熱サイクル条件は、最初50℃で2分、95℃で2分、その後、95℃で15秒及び60℃で1分を40サイクル行うことからなる。相対的なUL77, ホタルルシフェラーゼ又はRenillaルシフェラーゼRNAの定量を、文献記載の標準曲線解析法に従って行った(17, 29)。G6PDプライマー及びプローブ(45)を用いるリアルタイムPCRもまた、投入したRNAに対する内部対照として使用した。リアルタイムRT-PCR分析は3連で行った。各定量のために、ウイルスRNAを、感染3日後のdlUL76flagUL77ウイルスで感染したHFFからの閾値サイクル値に標準化した。Renilla (R)対(F)ルシフェラーゼのmRNA比及びG6PD RNAの定量を、親(IRES-ネガチブ)ベクターでトランスフェクションされたHeLa細胞からの閾値サイクル値に標準化された。親(IRES-ネガチブ)ベクターにより作製されたR:F比は、1.0に設定された。
【0071】
in vitro転写
プラスミドの線状化を、pCMVUL76-77構築体についてはXbaIで、又はルシフェラーゼ構築物についてはApaIで消化することによって行った。ApaI消化後、T4 DNAポリメラーゼ(Promega)を用いてin vitro転写前に3’オーバーハングを除去した。プラスミドを転写して、製造者の説明書(Promega)に従いRiboMAX Large Scale RNA Production System-T7を用いて、キャップされたRNAを作製した。1μgの線状化プラスミドDNAを、 7-メチル-GTP:GTP最終比 2.5:1を含む反応混合液中、37℃3時間転写した。RNAをフェノール−クロロホルム抽出により精製し、DNase I (Promega)で処理し、鋳型DNAを除去した。RNAサイズは、1%アガロース−ホルムアルデヒドゲル上でアリコートを泳動することによって測定された。
【0072】
in vitro翻訳
製造者の説明書に従いRabbit Reticulocyte Lysate System (Promega)を用いて翻訳を行った。反応混合液は、1μlの1mMアミノ酸混合物(メチオニン非含有), 2μlの[35S]メチオニン(Amersham Pharmacia), 1μlのRNasinリボヌクレアーゼ阻害剤, 2μgのキャップされたRNAと35μlのウサギ網状赤血球溶解物, 4μgのキャップされた全長RNAから調製され30℃で90分間インキュベートされた。その後、その5μlを取り出し、10μlのドデシル硫酸ナトリウム(SDS)サンプルバッファーに加え、15% SDS-PAGEによって分離した。
【0073】
ルシフェラーゼアッセイ
すべてのトランスフェクションは、製造者の説明書に従いLipofectamine及びPlus試薬(Invitrogen)を用いて48ウエル上で3連で行われた。HeLa細胞を、1μgの各発現プラスミド又は4μgのキャップされたRNAでトランスフェクトし、その後、48時間又は6時間で回収した。そのあと、細胞溶解物を調製し、製造者の説明書に従いDual-Luciferase(登録商標)Reporter Assay System (Promega)にかけた。データは、3回の独立のトランスフェクション実験の平均である。
【0074】
<結果>
UL77の上流領域の欠失
UL76 ORF内にUL77の転写開始部位が存在する可能性を除外するためにUL76遺伝子を欠失させた組換えウイルスを構築した。野生型(wt)から組換えBAC DNAを選択するために薬剤耐性遺伝子を必要とするため、カナマイシン耐性遺伝子(KanR)を含むようにBAC DNAを構築した。また、 KanRが近隣遺伝子の発現に影響する可能性を排除するために、隣接するFRT配列を有する組換えBAC DNAを構築した。FLP仲介組換え法によってKanRを除去し、その後、FRTの34bpのみがUL77 ORFの前部に残った(図1a)。UL76 ORFは単一のBstB I部位を含むので (図1a)、BAC DNAを、制限エンドヌクレアーゼBstB Iで消化した。ウイルスDNA断片を0.7%アガロースゲルでの電気泳動によって分画し、上記「材料と方法」に記載したように、UL75又はUL76のいずれか一方に対するプローブによるサザンブロットハイブリダイゼーションのために固定化された。UL75プローブを用いて、UL76を欠失したとき、より大きいウイルスDNA断片が検出された(図1b,左パネル)。UL76を含有するDNA断片は、組換えウイルスRdlUL76+Fに対してはUL76プローブで検出されなかったが、野生型に対しては検出された(図1b,右パネル)。PCR分析は、組換えBAC DNA中に組換えを明らかに示した(データ示さず)。DNA配列決定により該組換えを確認した(データを示さず)。
【0075】
ウイルスの単離後、細胞をMOI 約1で感染し、感染後6, 24, 48及び72時間の時点でノザンブロットによりUL76, UL77及びUL78転写体についてアッセイした。RNAの20μgのアリコートを、アガロースゲル電気泳動にかけ、28S及び18SのリボソームRNAの臭化エチジウム染色により、等量のRNAが各レーンに載置されたことを確認した(図1c及び1e)。予測したとおり、UL76が組換えウイルスRdlUL76+Fについて欠失されたとき転写体は存在しなかったが、該転写体は野生型では検出された(図1c)。UL77プローブを用いたとき、野生型(wt)又はRdl76+F ではさらなる転写体は検出されなかった(図1d)。UL78プローブを用いたときには、wt及び組換えウイルスRdlUL76+F中にUL78遺伝子のみに対し検出された転写体が検出された(図1e)。これらの結果は、UL77 ORFがRdlUL76+F中のUL76上流領域を欠失していることを示している。
【0076】
UL76 ORFの発現レベルに影響する
真核生物mRNAの非常に大部分がモノシストロン性であり、すなわち、それらは、単一のタンパク質のみをコードする。UL77の上流配列がUL77遺伝子の転写及び翻訳に影響を及ぼすか否かを決定するために、UL77 ORFの上流領域が存在する、又は存在しない、UL77 ORFのN末端に融合させたflagエピトープを含む組換えHCMV BAC DNAを構築した。図2aに示されるように、はじめに、HCMV BAC DNA中に、RpsLneo遺伝子を有するマーカーカセットを挿入し、ストレプトマイシンに対する感受性を増大させた。カナマイシン耐性に基づいて中間BACクローンを単離した。このクローンの完全性をHind IIIによる消化でチェックし、プライマー対としてUL76detectF及びUL76detectR を用いるPCRによって正しい位置でのマーカーカセットの挿入を確認した(データを示さず)。第2ラウンドの相同組換えでは、マーカーカセット全体が、上記の「材料と方法」に記載されるように、UL77 ORFの上流領域が存在する、又は存在しない、UL77のN末端に融合されたflagエピトープで置換された。従来法に従いストレプトマイシン耐性の増大に基づいて組換え構築物を単離した(19, 21, 46)。N末端にflagエピトープが融合された変異領域を、上記の「材料と方法」に記載されたプライマー対UL76detectF及びUL76detectRを用いるPCRによって増幅した(データ示さず)。Hind IIIによる消化によって、変異BACの完全性をチェックした(データ示さず)。
【0077】
細胞を、MOI約1でRflagUL77又はRdlUL76flagUL77に感染させ、感染後表示の時点で回収した。RflagUL77及びRdlUL76flagUL77のUL77転写体の比率は、感染の2日目と3日目で類似していた(図2b)。このことは、上流配列の欠失がUL77の転写に影響しないことを示している。前初期, 初期, 遅延初期及び後期タンパク質の発現レベルは、MOI約1のRflagUL77とRdlUL76flagUL77では類似していた(図2c)。UL77遺伝子産物の予測された分子量に相当する融合タンパク質が、感染後3日及び4日目に両組換えウイルスで検出されたが、その発現レベルは、RflagUL77 と比べてRdlUL76flagUL77についてより高かった(図2c,レーン5〜8)。たとえRdlUL76flagUL77が、重要なUL77遺伝子産物の発現を高めたとしても、MOI 1でのRflagUL77対RdlUL76flagUL77の増殖の違いは検出されなかった(データを示さず)。UL76遺伝子産物の欠失は、Wangら(43, 44)により提案されたように、ウイルス複製に影響を与えた可能性がある。これらのデータは、UL77の上流の配列がUL77タンパク質発現に対し影響をもつことを示した。UL77の上流配列の欠失により、UL77 ORFガウイルスmRNA中で第1シストロンになったとき、UL77 ORFの発現が増大した。
【0078】
UL77の上流配列の欠失がUL77 mRNAの安定性を高めることは可能である。隠れたスプライス部位又はプロモーターが欠失することも可能である。これらの可能性を除外するために、UL76 ORFのATG開始コドンの下流にTAG stopコドンが挿入された組換えウイルスを構築した。図3aに示すように、RpsLneo遺伝子をもつマーカーカセットをRflagUL77に挿入してRflagUL77Kan+Stを作製した。第2ラウンドの相同組換えのあと、マーカーカセット全体を、UL76 ORFのstopコドンを含む変異オリゴで置換して RUL76stopflagUL77を得た、或いは、復帰突然変異オリゴで置換して、上記の「材料と方法」に記載されたRUL76revertantflagUL77を作製した。変異型BACの完全性は、Hind IIIでの消化によってチェックし(データ示さず)、正しい組換えをPCR産物の配列決定によって確認した。細胞を、MOI約1でRUL76stopflagUL77又はRUL76revertantflagUL77のいずれか一方で感染し、 感染後表示の時点で回収した。前初期(pIE72とpIE86),初期(p52), 遅延初期 (pp65),及び後期(pp28)タンパク質の発現レベルは、RUL76stopflagUL77及びRUL76revertantflagUL77と類似していた(図3b)。UL77 発現レベルは、RUL76revertantflagUL77と比べて、RUL76stopflagUL77についてより高かった(図3b, レーン5〜8)。これらの結果は、UL76配列がUL77 ORF発現に影響することを示した。
【0079】
UL76及びUL77 ORFがin vitroでバイシストロニックなキャップされたRNAから翻訳された
HCMV MIEプロモーターによって駆動されるUL76-77 mRNAが両ウイルスタンパク質を発現することができるかどうかを決定するために、UL76 ORFのN末端に融合されたflagエピトープとUL77 ORFのC末端に融合されたHAエピトープを有するプラスミドを構築した。さらにまた、UL76 ORFのN末端又はUL77 ORFのC末端にフレームシフト変異が挿入されたプラスミドを構築した(図4a)。上記発現プラスミドによるHeLa細胞のトランスフェクション後48時間目に、該細胞を回収した。SDS-ポリアクリルアミドゲル電気泳動により等量のタンパク質を分画し、上記の「材料と方法」に記載されたflag又はHAエピトープに対するモノクローナル抗体を用いたウエスタンブロッティングによって分析した。図4bのレーン2に示されるように、UL76及びUL77融合タンパク質の両方をpCMVflagUL76-77HAでトランスフェクトしたHeLa細胞中で発現させた。UL77融合タンパク質の発現レベルは、UL76 ORFにフレームシフトが挿入されたとき約4倍増加し(図4b、レーン1と2の比較)、UL76配列がUL77 ORFの発現に影響することを確認した。
【0080】
UL77 ORFがバイシストロニックなキャップされたRNAからin vitroで翻訳されうるかどうかを決定するために、上記の「材料と方法」に記載されたようにin vitro 転写によってプラスミドの各々からRNAを調製した。該プラスミドのT7ポリメラーゼ駆動転写によって単一のバイシストロニックなRNAが生じた(図4c)。バイシストロニックなUL76-77 RNAの翻訳によって37 kDaのflag UL76と72 kDaのUL77 HAが検出された(図4d,レーン2)。これらの結果は、UL77 ORFがin vitroでバイシストロニックなキャップされたRNAから翻訳されうることを示した。バイシストロニックなmRNAからのUL77の翻訳は、リボソームの連続的スキャニングか又はUL77の上流のIRESに起因した。
【0081】
UL76 ORF配列は、インターナルリボソームエントリーサイトを含んでいる。通常、リボソームサブユニットは、RNAの5'-末端 m7GpppN cap構造のところでスキャニングを開始したあと、3'方向に直線的に移動するので、uORFの長さは、スキャニングの再開始のための重要なパラメーターである(16)。実際、哺乳類細胞では、スキャニングの再開始は、uORFが短い場合のみ起こる(24, 28)。もしUL77 AUGでのリボソームの開始がUL76中に埋め込まれたIRESによって保証される場合には、再開始は完全な上流シストロンと独立に生じうるだろう。UL76配列がIRESを有するかどうかを決定するために、上記の「材料と方法」に記載されたpCMVFlucRlucを構築した(図5a)。このプラスミドは、第1シストロンとしてのホタルルシフェラーゼ、その後にイントラシストロン配列(ICS)、及び第2シストロンとしてのRenillaルシフェラーゼ遺伝子を有するバイシストロン性mRNAをコードする。ICSに挿入されたIRESは、Renillaルシフェラーゼ遺伝子の翻訳を容易にするだろう。UL76ヌクレオチド(nt)1〜580又はその誘導体を、ICSの部位でpCMVFlucRlucに挿入し、HeLa細胞をトランスフェクトした。そのあと、Renilla (R)ルシフェラーゼ対ホタルルシフェラーゼ(F)の活性比を、上記の「材料と方法」に記載されたように、トランスフェクション後48時間目に細胞溶解物中で測定した。図5bに示されるように、UL76 (1-580)は、Renilla対ホタルルシフェラーゼ活性の比を約4倍増加させた。アンチセンス配向をもつDNA断片(r1-580)は作用しなかった(図5b)。ヌクレオチド1〜116は有意な活性をもたなかったが、ヌクレオチド1〜190は有意な作用を有していた。190〜58のヌクレオチドもまた、有意な作用を有していた(図5b)。これらのデータは、UL76のアミノ末端側半分とカルボキシ末端側半分の両方におけるウイルス配列エレメントが下流シストロンの発現を容易にすることを示した。
【0082】
UL76 (1-580)中の、Renilla ルシフェラーゼ活性に対し作用しうる、隠れたスプライス部位又はプロモーターの可能性を除外するために、上記の「材料と方法」に記載されたようにT7 RNAポリメラーゼを用いてin vitroで構築物を転写し、得られたRNAを、DNaseでDNA鋳型を分解したあと、1%アガロース-ホルムアルデヒドゲルに載置した。プラスミドのT7ポリメラーゼ駆動転写によって単一のバイシストロン性RNAを作製した(データを示さず)。その後、HeLa細胞中にこれらの精製RNAをトランスフェクションすることによってR:F比を調べた。トランスフェクション後6時間目に細胞抽出物をルシフェラーゼ活性についてアッセイし、R:F比を上記と同様に計算した。図6に示すように、キャップされたRNAをもつUL76配列エレメント(1〜580)は、Renilla対ホタルルシフェラーゼ活性の比を約4倍増加させた。アンチセンス配向をもつRNA断片 (r1-580)は効果を有していなかった(図6)。UL76 (1-580)によって仲介されるR:F比はポリオウイルスIRESによって仲介されるものの40.6%であった(図6)。UL76のDNA断片をもつRNA転写体もまた、R:F比を増加させた。RNAのR:F比は、DNAのものと比べて劇的に高かった(図5参照)。この作用の理由は不明であるが、RNAがin vitroで転写されるときには、不十分なキャップ(cap)形成のために、キャップ依存性スキャニングによる第1シストロンの翻訳効率がin vivoデータと比べて低くなったと推定される。2つのアッセイでは、個々の断片はすべて(互いに対して)同等に挙動しないが、全体の傾向は類似していた。in vitroでの二次構造は、in vivoで作られるものと異なるだろうし、IRESへのリボソームの接近能に影響を与えるかもしれない。これらをあわせると、UL76 ORF配列は、下流のUL77 ORFの発現を容易にするインターナルリボソームエントリーサイトを含む。
【0083】
<考察>
IRESは、ピコルナウイルス(23, 37)、ペストウイルス(38)、C型肝炎ウイルス(41)、カポジ肉腫関連ヘルペスウイルス(3, 14, 27)、単純ヘルペスウイルス-1(13)及びサルウイルス40(48)のウイルスmRNA、並びにいくつかの細胞mRNA(35, 40, 42)、中に同定されている。IRES間ではヌクレオチド配列の共通性は小さい。ウイルスIRESは、RNA二次構造の広い領域を有している。RNA二次構造は保存されていないが、リボソームとの結合にとって重要である。UL76配列のRNA二次構造は決定されているが、コンピュータプログラムRNA mfold (http://frontend.bioinfo.rpi.edu/applications/mfold/cgi-bin/rna-form1.cgi)によって、このRNA配列が安定なステムループ構造を含むことが予測された。UL76 (1-580)によって仲介されるIRES活性は、ポリオウイルスIRESは、ポリオウイルスIRESによって仲介された活性の約40%であった。
【0084】
UL76 (RUL76stopflagUL77)中に挿入されたstopコドンをもつ組換えウイルスは、復帰突然変異ウイルス(RUL76revertantflagUL77)と同様に(但し、UL77タンパク質を除く)、前初期、初期及び後期タンパク質を発現した。UL77 ORFの発現レベルは、UL76 ORF中にstopコドンが挿入された組換えウイルスで増加したが、このことは、UL76配列自体がHCMV感染細胞中でUL77 ORFの発現に影響を与えたことを示している。in vitro翻訳アッセイによって、UL77 ORFが再開始によって翻訳されうることが示された。ほとんどの状況において、uORFの存在が下流のAUGで開始を阻害し、uORFの単一の機能が下流のORFの発現をダウンレギュレートすることであるように思われることが多い(11)。上流のUL76 ORFが欠失すると、UL77 ORFの発現が増加する。
【0085】
これまで調べられた多くの細胞IRESは、mRNAの5’UTR内に位置している。これに対して、DNAウイルス内で見出されたすべてのIRESは、上流シストロンのコード配列中に存在する(3, 13, 14, 27, 48)。本発明者らは、UL76 ORF中にIRESを同定した。HCMVは、宿主細胞内の翻訳機構に完全に依存して、複製生産のためのウイルスタンパク質を合成する。5’cap構造からの開始及びリボソームスキャニングが細胞ストレスによって低下する状況下で、UL77タンパク質が翻訳されうる。UL76中のIRESエレメントは、HCMV感染において重要な機能を有しているが、これはUL77ウイルスタンパク質がウイルスDNAの切断及びパッケージングのために必要であるからである。UL76 ORFのタンパク質としての機能は、HCMV感染細胞では不明である。RflagUL77とRdlUL76flagUL77との増殖の差は検出されなかった。Wangら(43, 44)は、UL76がウイルス遺伝子発現を調節することを示した。本発明者ら、UL76もまたcis作用性調節シグナルとして働き、かつ、IRESとして作用し、UL77の発現を容易にすることを提案する。これらのデータは、なぜUL76がウイルス複製を増大するものとして分類されるか(7, 47)という理由を一部説明している。
【0086】
[実施例2]
HFF細胞でのウイルス複製
ウイルス遺伝子欠失変異体の構築
122アミノ酸(図7a)を有するUL30は、ウイルス遺伝子発現全体にほとんど又はまったく影響を及ぼさない可能性がある。本発明者らは、組換えHCMV BAC DNAを構築したが、このDNAは、UL76中のUL77の上流の領域がUL30によって置換されたものである。UL76配列はBstB I部位を含む(図7a)ので、BAC組換えを証明するために、BAC DNAを制限酵素BstB Iで消化した。0.7%アガロースゲルでの電気泳動によってウイルスDNA断片を分画し、「材料と方法」に記載したUL76又はUL77のいずれかに対するプローブによるサザンブロットハイブリダイゼーションのために固定された。UL76を含むDNA断片は、組換えウイルスRdlUL76+FについてはUL76プローブで検出されなかったが、野生型については検出された(図7b、左パネル)。UL77プローブを用いて、UL77の上流領域がUL30で置換されたとき、より大きなウイルスDNA断片が検出された(図7b、右パネル)。PCR分析によって、組換えBAC DNA中の組換えが示された。DNA配列分析によって、前記組換えを確認した。flagエピトープを有するUL77タンパク質が、非常に低いMOIでRUL30flagUL77に感染させた細胞中で発現されるかを調べるために、そしてCPEがほとんどすべての細胞中に現れたとき、細胞を回収し、ウエスタンブロット分析を行って、「材料と方法」に記載したflagエピトープに対する抗体を用いてflagエピトープを有するUL76タンパク質を検出した。図7cに示されるように、RUL30flagUL77に感染させた細胞中にUL77タンパク質を検出することができた。
【0087】
UL30で置換された単離された組換えウイルスについて、複数回のウイルス複製後に高力価のウイルスストックを得ることは難しかった。図8aに示されるように、100% CPEの5〜7日後に単離され及び連続希釈された細胞外液含有ウイルスに感染させた細胞中のウイルスタンパク質の発現を比較したとき、RUL30flagUL77を有する初期タンパク質とIEの発現はRUL76revertantflagUL77の102倍希釈を用いたときの発現とほとんど同様であった。
【0088】
本発明者らは、さらなる分析のために-80℃に保存されていなかった細胞外液含有ウイルスを使用した。-80℃に保存されていなかったウイルス力価を、野生型(wt)のウイルス力価と比較することによって決定した(親ウイルス、図3参照)。ウイルス力価は、「材料と方法」に記載されたGFPフォーカスに基づいた標準プラークアッセイによって測定された。
【0089】
細胞を、約3のMOIでRUL30flagUL77又はRUL76revertantflagUL77のいずれかで感染し、感染後、示された時間に回収した。
【0090】
ウイルスタンパク質の発現を比較した。感染1、2及び3日後(1、2及び3d.p.i.)の、RUL30flagUL77及びRUL76revertantflagUL77を有する、前初期タンパク質(pIE72及びpIE86)、初期タンパク質(p52)及び後期タンパク質(pp28)の発現レベルに有意な差異はなかった(図8c)。RUL30flagUL77を有する初期タンパク質(p52)及び後期タンパク質(pp28)の発現レベルは、4d.p.i.のRUL76revertantflagUL77を有するものより少し低めであった。これに対して、本発明者らは、UL77タンパク質の発現の有意な差を検出することができきた。RUL30flagUL77を有するUL77タンパク質の発現は劇的に低減し、4d.p.i.でさえウエスタンブロット分析で検出することは難しかった(図8c)。このあと、本発明者らは、新しい多数の組換えウイルスを作製し、上に記載したようにウイルス力価を測定し、リアルタイムRT-PCRによってウイルスRNAを分析した。RUL30flagUL77についてUL77転写体の比率は、2d.p.i.でのRUL76revertantflagUL77についてのものより少し高めであったが、その差は2倍未満であった。RUL30flagUL77の転写体の比率は、3d.p.i.でのRUL76revertantflagUL77のものと類似していた(図8d)。RUL30flagUL77を有する各ウイルス転写体の総量は、RUL76revertantflagUL77を有するものより少し高めであったが、検査するIE、初期及び後期転写体のレベルに大きな変化はなかった(図8d)。
【産業上の利用可能性】
【0091】
本発明により、HCMVを予防又は治療するためのワクチンが提供される。
【0092】
参考文献
1. Beisser, P. S., G. Grauls, C. A. Bruggeman, and C. Vink. 1999. Deletion of the R78 G protein-coupled receptor gene from rat cytomegalovirus results in an attenuated, syncytium-inducing mutant strain. J. Virol. 73:7218-7230.
2. Biegalke, B. J., and A. P. Geballe. 1990. Translational inhibition by cytomegalovirus transcript leaders. Virology 177:657-667.
3. Bieleski, L., and S. J. Talbot. 2001. Kaposi's sarcoma-associated herpesvirus vCyclin open reading frame contains an internal ribosome entry site. J. Virol. 75:1864-1869.
4. Cao, J., and A. P. Geballe. 1996. Coding sequence-dependent ribosomal arrest at termination of translation. Mol. Cell Biol. 16:603-608.
5. Cao, J., and A. P. Geballe. 1995. Translational inhibition by a human cytomegalovirus upstream open reading frame despite inefficient utilization of its AUG codon. J. Virol. 69:1030-1036.
6. Degnin, C. R., M. R. Schleiss, J. Cao, and A. P. Geballe. 1993. Translational inhibition mediated by a short upstream open reading frame in the human cytomegalovirus gpUL4 (gp48) transcript. J. Virol. 67:5514-5521.
7. Dunn, W., C. Chou, H. Li, R. Hai, D. Patterson, V. Stolc, H. Zhu, and F. Liu. 2003. Functional profiling of a human cytomegalovirus genome. Proc. Natl. Acad. Sci. U. S. A. 100:14223-14228.
8. Ellis, H. M., D. Yu, T. DiTizio, and D. L. Court. 2001. High efficiency mutagenesis, repair, and engineering of chromosomal DNA using single-stranded oligonucleotides. Proc. Natl. Acad. Sci. U. S. A. 98:6742-6746.
9. Geballe, A. P., F. S. Leach, and E. S. Mocarski. 1986. Regulation of cytomegalovirus late gene expression: gamma genes are controlled by posttranscriptional events. J. Virol. 57:864-874.
10. Geballe, A. P., and E. S. Mocarski. 1988. Translational control of cytomegalovirus gene expression is mediated by upstream AUG codons. J. Virol. 62:3334-3340.
11. Geballe, A. P., and D. R. Morris. 1994. Initiation codons within 5'-leaders of mRNAs as regulators of translation. Trends Biochem. Sci. 19:159-164.
12. Graham, F. L., and A. J. van der Eb. 1973. A new technique for the assay of infectivity of adenovirus 5 DNA. Virology 52:456-467.
13. Griffiths, A., and D. M. Coen. 2005. An unusual internal ribosome entry site in the herpes simplex virus thymidine kinase gene. Proc. Natl. Acad. Sc.i U. S. A. 102:9667-9672.
14. Grundhoff, A., and D. Ganem. 2001. Mechanisms governing expression of the v-FLIP gene of Kaposi's sarcoma-associated herpesvirus. J. Virol. 75:1857-1863.
15. Hahn, G., D. Rose, M. Wagner, S. Rhiel, and M. A. McVoy. 2003. Cloning of the genomes of human cytomegalovirus strains Toledo, TownevarRIT3, and Towne long as BACs and site-directed mutagenesis using a PCR-based technique. Virology 307:164-177.
16. Hwang, W. L., and T. S. Su. 1998. Translational regulation of hepatitis B virus polymerase gene by termination-reinitiation of an upstream minicistron in a length-dependent manner. J. Gen. Virol. 79 ( Pt 9):2181-2189.
17. Isomura, H., and M. F. Stinski. 2003. The human cytomegalovirus major immediate-early enhancer determines the efficiency of immediate-early gene transcription and viral replication in permissive cells at low multiplicity of infection. J. Virol. 77:3602-3614.
18. Isomura, H., M. F. Stinski, A. Kudoh, T. Daikoku, N. Shirata, and T. Tsurumi. 2005. Two Sp1/Sp3 binding sites in the major immediate-early proximal enhancer of human cytomegalovirus have a significant role in viral replication. J. Virol. 79:9597-9607.
19. Isomura, H., M. F. Stinski, A. Kudoh, T. Murata, S. Nakayama, Y. Sato, S. Iwahori, and T. Tsurumi. 2008. Noncanonical TATA sequence in the UL44 late promoter of human cytomegalovirus is required for the accumulation of late viral transcripts. J. Virol. 82:1638-1646.
20. Isomura, H., M. F. Stinski, A. Kudoh, S. Nakayama, S. Iwahori, Y. Sato, and T. Tsurumi. 2007. The late promoter of the human cytomegalovirus viral DNA polymerase processivity factor has an impact on delayed early and late viral gene products but not on viral DNA synthesis. J. Virol. 81:6197-6206.
21. Isomura, H., M. F. Stinski, A. Kudoh, S. Nakayama, T. Murata, Y. Sato, S. Iwahori, and T. Tsurumi. 2008. A cis element between the TATA Box and the transcription start site of the major immediate-early promoter of human cytomegalovirus determines efficiency of viral replication. J. Virol. 82:849-858.
22. Isomura, H., T. Tsurumi, and M. F. Stinski. 2004. Role of the proximal enhancer of the major immediate-early promoter in human cytomegalovirus replication. J. Virol. 78:12788-12799.
23. Jang, S. K., H. G. Krausslich, M. J. Nicklin, G. M. Duke, A. C. Palmenberg, and E. Wimmer. 1988. A segment of the 5' nontranslated region of encephalomyocarditis virus RNA directs internal entry of ribosomes during in vitro translation. J. Virol. 62:2636-2643.
24. Kozak, M. 1995. Adherence to the first-AUG rule when a second AUG codon follows closely upon the first. Proc. Natl. Acad. Sci. U. S. A. 92:2662-2666.
25. Kozak, M. 2001. Constraints on reinitiation of translation in mammals. Nucleic Acids Res. 29:5226-5232.
26. Kozak, M. 1987. Effects of intercistronic length on the efficiency of reinitiation by eucaryotic ribosomes. Mol. Cell Biol. 7:3438-3445.
27. Low, W., M. Harries, H. Ye, M. Q. Du, C. Boshoff, and M. Collins. 2001. Internal ribosome entry site regulates translation of Kaposi's sarcoma-associated herpesvirus FLICE inhibitory protein. J. Virol. 75:2938-2945.
28. Matsuda, D., and T. W. Dreher. 2006. Close spacing of AUG initiation codons confers dicistronic character on a eukaryotic mRNA. RNA 12:1338-1349.
29. Meier, J. L., M. J. Keller, and J. J. McCoy. 2002. Requirement of multiple cis-acting elements in the human cytomegalovirus major immediate-early distal enhancer for viral gene expression and replication. J. Virol. 76:313-326.
30. Meier, J. L., and J. A. Pruessner. 2000. The human cytomegalovirus major immediate-early distal enhancer region is required for efficient viral replication and immediate-early gene expression. J. Virol. 74:1602-1613.
31. Meier, J. L., and M. F. Stinski. 1997. Effect of a modulator deletion on transcription of the human cytomegalovirus major immediate-early genes in infected undifferentiated and differentiated cells. J. Virol. 71:1246-1255.
32. Michel, D., I. Milotic, M. Wagner, B. Vaida, J. Holl, R. Ansorge, and T. Mertens. 2005. The human cytomegalovirus UL78 gene is highly conserved among clinical isolates, but is dispensable for replication in fibroblasts and a renal artery organ-culture system. J. Gen. Virol. 86:297-306.
33. Murphy, E., I. Rigoutsos, T. Shibuya, and T. E. Shenk. 2003. Reevaluation of human cytomegalovirus coding potential. Proc. Natl. Acad. Sci. U. S. A. 100:13585-13590.
34. Murphy, E., D. Yu, J. Grimwood, J. Schmutz, M. Dickson, M. A. Jarvis, G. Hahn, J. A. Nelson, R. M. Myers, and T. E. Shenk. 2003. Coding potential of laboratory and clinical strains of human cytomegalovirus. Proc. Natl. Acad. Sci. U. S. A. 100:14976-14981.
35. Nanbru, C., I. Lafon, S. Audigier, M. C. Gensac, S. Vagner, G. Huez, and A. C. Prats. 1997. Alternative translation of the proto-oncogene c-myc by an internal ribosome entry site. J. Biol. Chem. 272:32061-32066.
36. Pari, G. S., and D. G. Anders. 1993. Eleven loci encoding trans-acting factors are required for transient complementation of human cytomegalovirus oriLyt-dependent DNA replication. J. Virol. 67:6979-6988.
37. Pelletier, J., and N. Sonenberg. 1988. Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA. Nature 334:320-325.
38. Rijnbrand, R., T. van der Straaten, P. A. van Rijn, W. J. Spaan, and P. J. Bredenbeek. 1997. Internal entry of ribosomes is directed by the 5' noncoding region of classical swine fever virus and is dependent on the presence of an RNA pseudoknot upstream of the initiation codon. J. Virol. 71:451-457.
39. Stinski, M. F. 1978. Sequence of protein synthesis in cells infected by human cytomegalovirus: early and late virus-induced polypeptides. J. Virol. 26:686-701.
40. Stoneley, M., F. E. Paulin, J. P. Le Quesne, S. A. Chappell, and A. E. Willis. 1998. C-Myc 5' untranslated region contains an internal ribosome entry segment. Oncogene 16:423-428.
41. Tsukiyama-Kohara, K., N. Iizuka, M. Kohara, and A. Nomoto. 1992. Internal ribosome entry site within hepatitis C virus RNA. J. Virol. 66:1476-1483.
42. Vagner, S., M. C. Gensac, A. Maret, F. Bayard, F. Amalric, H. Prats, and A. C. Prats. 1995. Alternative translation of human fibroblast growth factor 2 mRNA occurs by internal entry of ribosomes. Mol. Cell Biol. 15:35-44.
43. Wang, S. K., C. Y. Duh, and T. T. Chang. 2000. Cloning and identification of regulatory gene UL76 of human cytomegalovirus. J. Gen. Virol. 81:2407-2416.
44. Wang, S. K., C. Y. Duh, and C. W. Wu. 2004. Human cytomegalovirus UL76 encodes a novel virion-associated protein that is able to inhibit viral replication. J. Virol. 78:9750-9762.
45. White, E. A., C. L. Clark, V. Sanchez, and D. H. Spector. 2004. Small internal deletions in the human cytomegalovirus IE2 gene result in nonviable recombinant viruses with differential defects in viral gene expression. J. Virol. 78:1817-1830.
46. White, E. A., C. J. Del Rosario, R. L. Sanders, and D. H. Spector. 2007. The IE2 60-kilodalton and 40-kilodalton proteins are dispensable for human cytomegalovirus replication but are required for efficient delayed early and late gene expression and production of infectious virus. J. Virol. 81:2573-2583.
47. Yu, D., M. C. Silva, and T. Shenk. 2003. Functional map of human cytomegalovirus AD169 defined by global mutational analysis. Proc. Natl. Acad. Sci. U. S. A. 100:12396-12401.
48. Yu, Y., and J. C. Alwine. 2006. 19S late mRNAs of simian virus 40 have an internal ribosome entry site upstream of the virion structural protein 3 coding sequence. J. Virol. 80:6553-6558.
【配列表フリーテキスト】
【0093】
配列番号9〜47:プライマー
配列番号48〜54:プローブ
配列番号55:34bp最小FRT部位
【技術分野】
【0001】
本発明は、弱毒ヒトサイトメガロウイルス(HCMV)及びそのワクチンとしての使用に関する。
【背景技術】
【0002】
ヒトサイトメガロウイルス(HCMV)は、ベータヘルペスウイルスファミリーのプロトタイプメンバーである。多くの個体でHCMV による感染が起こるけれども、その感染は無症候性である。このウイルスは免疫抑制条件下で再活性化されて病原体となり、肺炎、ヘルペス、網膜炎及び胃腸疾患を引き起こす。HCMVはまた、妊娠中に初期感染を起こしたときには、新生児に難聴と精神発達遅滞を引き起こす。HCMVのゲノムのサイズは約240,000bpであり、少なくとも194個の既知のオープンリーディングフレーム(ORF)をもっている(7, 33, 34)。このORFの大部分は、細胞培養中でのウイルス複製に必須ではない。いくつかのORFは有益であるが、それらはウイルスの複製に必要なものではない。しかしながら、ORFの約4分の1に相当する41個のORFは、ウイルスの複製に必要である(47)。
【0003】
ウイルス産生のための感染の間に、HCMV遺伝子群は一時的なカスケードの、指定された前初期(IE)、遅延初期及び後期に発現される。主要なIE(MIE)遺伝子であるUL123/122 (IE1/IE2)は、その後のウイルス遺伝子発現及びウイルス複製の効率において決定的な役割を果たす(17, 18, 22, 29-31)。複製初期のウイルス遺伝子群は、ウイルスDNA複製に必要なタンパク質をコードしている(36)。ウイルスDNA複製後、ウイルス粒子産生のための構造タンパク質をコードする、遅延初期及び後期ウイルス遺伝子群が発現される。これに関連して、MIE遺伝子プロモーターのシス作用性エレメント(crs)配列の一部に突然変異を有し、これによってウイルス複製が不能ではないが抑制されている弱毒化組換えヒトサイトメガロウイルスが特許文献1に開示されている。
【0004】
HCMV UL76遺伝子は、高度に保存されたウイルス粒子関連ヘルペスウイルスタンパク質をコードし、このタンパク質はウイルス複製を増加させることが報告されている(43, 44)。ORFの予測された分子量に相当する38kDaのタンパク質は、感染後(p.i.)2時間の時点でHCMV感染細胞中に検出される。このタンパク質の産生はp.i.24時間の時点で最大に達し、その後、その産生レベルは、ウイルス生活環の後期から同じレベルを維持する。
【0005】
UL77遺伝子は、単純ヘルペスウイルス(HSV) UL25遺伝子の同等物であり、この遺伝子はHCMV複製に必須である(7)。HCMVでは、UL77 ORFの最初のMetは、UL76 ORFの最初のMetと異なるフレーム中に存在する。UL76では明らかな転写終結がないし、また、UL77では転写開始がない(44)。UL78はG-タンパク質共役受容体の特性を有しており(1)、また、UL78 1.7kb mRNAは、HFF細胞中では、p.i.4時間の時点で検出可能になる(32)。
【0006】
5’mRNAキャップは、翻訳開始複合体と結合するために必須である。43S前開始複合体は、それが翻訳のための最初の好適な開始コドン(これは大方、最初のAUGである。)に達するまで、該RNAをスキャニングする。哺乳類のリボソームは一般にバイシストロン性mRNAの下流シストロンを翻訳しないか又は翻訳できない(これに対して、原核生物のシステムではそのような翻訳が偶発的に起こる)。この一般則に2つの例外があり、一方は、下流のシストロンに直接的にリボソームをリクルートすることが可能なインターナルリボソームエントリーサイト(IRES)であり、他方は、下流のAUGコドンで翻訳を再び開始するために5'から3'方向にスキャニングすることである(2, 4-6, 9-11, 25, 26)。
【先行技術文献】
【特許文献】
【0007】
【特許文献1】特開2008-092854号公報
【非特許文献】
【0008】
【非特許文献1】Dunn, W., C. Chou, H. Li, R. Hai, D. Patterson, V. Stolc, H. Zhu, and F. Liu. 2003. Functional profiling of a human cytomegalovirus genome. Proc. Natl. Acad. Sci. U. S. A. 100:14223-14228(文献7)
【非特許文献2】Murphy, E., I. Rigoutsos, T. Shibuya, and T. E. Shenk. 2003. Reevaluation of human cytomegalovirus coding potential. Proc. Natl. Acad. Sci. U. S. A. 100:13585-13590(文献33)
【非特許文献3】Murphy, E., D. Yu, J. Grimwood, J. Schmutz, M. Dickson, M. A. Jarvis, G. Hahn, J. A. Nelson, R. M. Myers, and T. E. Shenk. 2003. Coding potential of laboratory and clinical strains of human cytomegalovirus. Proc. Natl. Acad. Sci. U. S. A. 100:14976-14981(文献34)
【非特許文献4】Yu, D., M. C. Silva, and T. Shenk. 2003. Functional map of human cytomegalovirus AD169 defined by global mutational analysis. Proc. Natl. Acad. Sci. U. S. A. 100:12396-12401(文献47)
【非特許文献5】Isomura, H., and M. F. Stinski. 2003. The human cytomegalovirus major immediate-early enhancer determines the efficiency of immediate-early gene transcription and viral replication in permissive cells at low multiplicity of infection. J. Virol. 77:3602-3614(文献17)
【非特許文献6】Isomura, H., M. F. Stinski, A. Kudoh, T. Daikoku, N. Shirata, and T. Tsurumi. 2005. Two Sp1/Sp3 binding sites in the major immediate-early proximal enhancer of human cytomegalovirus have a significant role in viral replication. J. Virol. 79:9597-9607(文献18)
【非特許文献7】Isomura, H., T. Tsurumi, and M. F. Stinski. 2004. Role of the proximal enhancer of the major immediate-early promoter in human cytomegalovirus replication. J. Virol. 78:12788-12799(文献22)
【非特許文献8】Meier, J. L., M. J. Keller, and J. J. McCoy. 2002. Requirement of multiple cis-acting elements in the human cytomegalovirus major immediate-early distal enhancer for viral gene expression and replication. J. Virol. 76:313-326(文献29)
【非特許文献9】Meier, J. L., and J. A. Pruessner. 2000. The human cytomegalovirus major immediate-early distal enhancer region is required for efficient viral replication and immediate-early gene expression. J. Virol. 74:1602-1613(文献30)
【非特許文献10】Meier, J. L., and M. F. Stinski. 1997. Effect of a modulator deletion on transcription of the human cytomegalovirus major immediate-early genes in infected undifferentiated and differentiated cells. J. Virol. 71:1246-1255(文献31)
【非特許文献11】Pari, G. S., and D. G. Anders. 1993. Eleven loci encoding trans-acting factors are required for transient complementation of human cytomegalovirus oriLyt-dependent DNA replication. J. Virol. 67:6979-6988(文献36)
【非特許文献12】Wang, S. K., C. Y. Duh, and T. T. Chang. 2000. Cloning and identification of regulatory gene UL76 of human cytomegalovirus. J. Gen. Virol. 81:2407-2416(文献43)
【非特許文献13】Wang, S. K., C. Y. Duh, and C. W. Wu. 2004. Human cytomegalovirus UL76 encodes a novel virion-associated protein that is able to inhibit viral replication. J. Virol. 78:9750-9762(文献44)
【非特許文献14】Michel, D., I. Milotic, M. Wagner, B. Vaida, J. Holl, R. Ansorge, and T. Mertens. 2005. The human cytomegalovirus UL78 gene is highly conserved among clinical isolates, but is dispensable for replication in fibroblasts and a renal artery organ-culture system. J. Gen. Virol. 86:297-306(文献32)
【非特許文献15】Biegalke, B. J., and A. P. Geballe. 1990. Translational inhibition by cytomegalovirus transcript leaders. Virology 177:657-667(文献2)
【非特許文献16】Cao, J., and A. P. Geballe. 1996. Coding sequence-dependent ribosomal arrest at termination of translation. Mol. Cell Biol. 16:603-608(文献4)
【非特許文献17】Cao, J., and A. P. Geballe. 1995. Translational inhibition by a human cytomegalovirus upstream open reading frame despite inefficient utilization of its AUG codon. J. Virol. 69:1030-1036(文献5)
【非特許文献18】Degnin, C. R., M. R. Schleiss, J. Cao, and A. P. Geballe. 1993. Translational inhibition mediated by a short upstream open reading frame in the human cytomegalovirus gpUL4 (gp48) transcript. J. Virol. 67:5514-5521(文献6)
【非特許文献19】Geballe, A. P., F. S. Leach, and E. S. Mocarski. 1986. Regulation of cytomegalovirus late gene expression: gamma genes are controlled by posttranscriptional events. J. Virol. 57:864-874(文献9)
【非特許文献20】Geballe, A. P., and E. S. Mocarski. 1988. Translational control of cytomegalovirus gene expression is mediated by upstream AUG codons. J. Virol. 62:3334-3340(文献10)
【非特許文献21】Geballe, A. P., and D. R. Morris. 1994. Initiation codons within 5'-leaders of mRNAs as regulators of translation. Trends Biochem. Sci. 19:159-164(文献11)
【非特許文献22】Kozak, M. 2001. Constraints on reinitiation of translation in mammals. Nucleic Acids Res. 29:5226-5232(文献25)
【非特許文献23】Kozak, M. 1987. Effects of intercistronic length on the efficiency of reinitiation by eucaryotic ribosomes. Mol. Cell Biol. 7:3438-3445(文献26)
【発明の概要】
【発明が解決しようとする課題】
【0009】
今回、本発明者らは、ヒトサイトメガロウイルス(HCMV)のUL76 ORFが、UL77 ORFの翻訳に影響するIRESを含むことを見出した。このUL77はウイルス複製に必須であるので、UL76が、ウイルス複製の理由の一部にUL76中のIRESが関わっていることを今回明らかにした。この知見に基づいてHCMVの弱毒ウイルスを作製することを本発明の目的とした。
【課題を解決するための手段】
【0010】
ヒトサイトメガロウイルス(HCMV)のUL77遺伝子はウイルスDNAパッケージングに特定の役割を果たしており、このパッケージングはウイルス複製にとって重要である。しかし、トリシストロン性転写体は、UL76〜UL78オープンリーディングフレーム (ORF)を含み、一方、UL77は慣用の5’mRNAを有していない。UL77の翻訳が上流配列によって影響を受けるかどうかを決定するために、本発明者らは、UL76 ORFの下流の停止(stop)コドンからATG開始コドンまでを欠失するか或いは挿入することによってUL76を変異させた。組換えウイルスを両方とも用いて、UL77の翻訳レベルの増加が、上流のUL77配列がUL77の発現に影響すること、及び、UL77が、第1シストロンとなるとき、より効率的に発現されることを示すことを今回見出した。本発明者らはまた、UL76 ORFのN末端に融合されたflagエピトープとUL77 ORFのC末端に融合されたHAエピトープをもつ発現プラスミドを構築し、HeLa細胞をトランスフェクトした。UL76 ORF中のフレームシフトにより、UL77の発現が増加したが、一方、UL77のカルボキシ末端へのフレームシフトは影響を与えなかった。このことから、UL76がUL77発現に影響することを確認した。UL76がどのようにUL77発現に影響するかを決定するために、本発明者らは、バイシストロニックなレポータープラスミドを構築し、UL76の上流配列がインターナルリボソームエントリーサイト(IRES)を含むかどうかを測定した。その結果、本発明者らは、今回、UL76中のIRESがピコルナウイルスIRESとほとんど同じ強さの活性をもつことを検出し、UL76遺伝子が、IRESを介して、下流のUL77 ORFの翻訳に影響を及ぼしていることを見出した。本発明は、上記の知見に基づいている。
【0011】
本発明は、要約すると、以下の特徴を含む。
本発明は、第1の態様において、ヒトサイトメガロウイルス(HCMV)のUL76遺伝子ORF中のIRES配列が不活性化されたことを特徴とする弱毒HCMVウイルスを提供する。
【0012】
その実施形態において、上記IRES配列が、IRES以外の別の核酸配列によって置換されている。
【0013】
別の実施形態において、上記別の核酸配列のサイズ(すなわち、ヌクレオチド数)が上記IRES配列のサイズと完全に同じであるか又はほほ同じである。
【0014】
別の実施形態において、上記別の核酸配列が、同じ又は異なるHCMV株のUL30由来である。
【0015】
別の実施形態において、上記HCMVの複製が抑制された弱毒HCMVウイルスである。
【0016】
本発明はまた、第2の態様において、上記の弱毒HCMVウイルスを有効成分として含む、HCMV感染の予防又は治療用ワクチンを提供する。
【0017】
本発明はさらに、第3の態様において、上記の弱毒HCMVウイルスを非ヒト動物に免疫して抗体を作製し、該抗体を回収することを含む、抗HCMV抗体の作製方法を提供する。
【0018】
その実施形態において、上記抗HCMV抗体がヒト抗体である。
【発明の効果】
【0019】
本発明によって、ヒトサイトメガロウイルス(HCMV)の複製が抑制された弱毒ウイルスを提供することができるため、該弱毒ウイルスをワクチンとして使用することができる。
【図面の簡単な説明】
【0020】
【図1−1】この図は、野生型(wt)又は組換えウイルスによる感染後のUL76〜UL78遺伝子転写体の分析結果を示す。(a)は、wt及びRdlUL76+FRTの組換えBAC DNAの図である。UL76は、カナマイシン耐性遺伝子(KanR)で置換され、その後、KanRはFLP仲介組換えによって除かれ、その結果、FRT配列(F)の34bpのみが残った。(b)は、wt及びRdlUL76+Fの親及び組換えBAC DNAのサザンブロット分析の結果を示す。BAC DNAは、制限エンドヌクレアーゼBstB Iで消化され、0.6%アガロースゲルでの電気泳動によって分画され、32P標識プローブとのハイブリダイゼーションにかけられた。標準分子サイズマーカーは塩基対で示されている。レーン1及び3, RdlUL76+F; レーン2及び4, wt; レーン1及び2, UL75プローブ; レーン3及び4, UL76プローブ。
【図1−2】図1の(c)、(d)及び(e): wt又はRdlUL76+Fによる感染後のUL76〜78遺伝子転写体の分析結果を示す。HFFは、約1のMOIで感染され、細胞質RNAが、後述の実施例に記載のように、感染後(p.i)6, 24, 48及び72時間目に回収された。28S及び18S rRNAは、コントロールとして使用され、等量のRNAが負荷(load)された。(c) UL76プローブ、(d) UL77プローブ、(e) UL78プローブ。レーン1及び5, 感染後6時間; レーン2及び6, 感染後24時間; レーン3及び7, 感染後48時間; レーン4及び8, 感染後72時間; レーン1〜4, wt; レーン5〜8, RdlUL76+F。
【図2】この図は、UL77の上流領域が存在する又は存在しないUL77 ORFのN末端に融合されたflagエピトープを有する組換えウイルスを示す。(a)は、後述の実施例の「材料と方法」に記載のように作製された組換えHCMV BAC DNAの図である。(b)は、上記組換えウイルスによるUL77遺伝子転写の定量結果を示す。細胞質RNAは、UL77特異的プライマーとプローブを用いて、後述の実施例の「材料と方法」に記載のように、感染後1日、2日及び3日目にリアルタイムPCRによって分析された。このアッセイを3回行い、平均の標準誤差を決定した。UL77 RNAは、G6PD RNAに対して標準化され、各値は、感染後3日目のRflagUL77 RNAのレベルに対する値とした。(c)は、約1のMOIでRdlUL76flagUL77又はRflagUL77で感染後の表示した時間における前初期pIE72 (UL123), pIE86 (UL122), 初期p52 (UL44), 遅延初期pp65 (UL83), 後期pp28 (UL99)及びflag-pUL77タンパク質のウエスタンブロット分析の結果を示す。レーン1及び2, 感染後1日; レーン3及び4, 感染後2日; レーン5及び6, 感染後3日; レーン7及び8, 感染後4日; レーン1, 3, 5及び7, RdlUL76flagUL77; レーン2, 4, 6及び8, RflagUL77。
【図3】この図は、UL76 ORFにTAG stopコドンが挿入された組換えウイルスを示す。(a)は、後述の実施例の「材料と方法」に記載のように作製された組換えHCMV BAC DNAの図である。小文字は、UL76 ORFにTAG stopコドンを挿入するための変異塩基を示す。(b)は、約1のMOIでRUL76stopflagUL77又はRUL76revertantflagUL77で感染後表示された時間における前初期pIE72 (UL123), pIE86 (UL122), 初期p52 (UL44), 遅延初期pp65 (UL83), 後期pp28 (UL99)及びflag-pUL77タンパク質のウエスタンブロット分析の結果を示す。レーン1及び2, 感染後1日; レーン3及び4, 感染後2日; レーン5及び6, 感染後3日; レーン7及び8, 感染後4日; レーン1, 3, 5及び7, RUL76stopflagUL77; レーン2, 4, 6及び8, RUL76revertantflagUL77。
【図4−1】この図は、UL76中のフレームシフトがUL77発現を増大することを示す。(a)は、フレームシフトを有する又はフレームシフトを有さない、HCMV MIEプロモーターの下流に挿入されたUL76-77配列を含む発現プラスミドの図である。(b)は、UL76及びUL77融合タンパク質のウエスタンブロット分析の結果を示す。HeLa細胞を、フレームシフト変異を有する又はフレームシフト変異を有さないpflagUL76-77HAでトランスフェクトし、感染後48時間目に回収した。レーン1, pCMVflag-frame-shift-UL76-77HA; レーン2, pCMVflagUL76-77HA; レーン3, pCMVflagUL76-77-frame-shift-HA。
【図4−2】図4の(c)は、UL76-77 mRNAのin vitro転写を示す。キャップされたRNAを、後述の実施例の「材料と方法」に記載されるようにin vitroで転写し、その結果得られたRNAを、1%アガロース-ホルムアルデヒドゲル上で分析した。レーン1, RNA分子量マーカー; レーン2, pCMVflagUL76-77HA; レーン3, pCMVflag-frame-shift-UL76-77HA; レーン4, pCMVflagUL76-77-frame-shift-HA。(d)は、キャップしたRNAからの、UL76及びUL77の融合タンパク質のin vitro翻訳を示す。UL76-77 RNAを、後述の実施例の「材料と方法」に記載のようにin vitroで翻訳し、放射性標識したタンパク質をSDS-15% PAGEで分離した。レーン1, pCMVflag-frame-shift-UL76-77HA; レーン2, pCMVflagUL76-77HA; レーン3, pCMVflagUL76-77-frame-shift-HA。
【図5】この図は、発現プラスミド中のUL76 DNA又はその断片が下流のシストロンの翻訳を開始することができることを示す。(a)は、IRES活性を検出するために使用されたバイシストロン性ルシフェラーゼレポーター構築物の図である。構築物pcDNA3.1は、ホタルルシフェラーゼ遺伝子の下流のRenillaルシフェラーゼのコード領域を含んでいる。発現は、トランスフェクション実験においてHCMV MIEプロモーターによって駆動された。UL76のヌクレオチド1-71, 1-116, 1-190, 191-580, 271-580, 451-580及び1-580を含むDNA断片(最も大きい断片はUL77 ORFの上流領域を含む。)は、ベクターのインターシストロニック領域(ICS)内に位置するXho I及びBamH I中に挿入された。該上流領域の580bp断片はまた、アンチセンス配向に挿入された(r 1-580)。(b)は、UL76が仲介するIRES活性を示す。レポータープラスミドを、HeLa細胞内にトランスフェクトし、該細胞を感染48時間後に回収し、ホタル及びRenillaルシフェラーゼ活性を定量した。Renilla対ホタルルシフェラーゼ活性の比を計算した。なお、各トランスフェクションは3連で行われた。
【図6】この図は、IRESがUL76 RNA内に位置することを示す。HeLa細胞を、後述の実施例の「材料と方法」に記載のように、レポータープラスミドからin vitroで転写されたキャップされたRNAでトランスフェクトし、実施例の「材料と方法」に記載のようにトランスフェクション後6時間目に該細胞を回収した。相対的なRenilla ルシフェラーゼ活性(Renilla対ホタルルシフェラーゼ活性の比)をUL76又はその断片について計算し、既知のピコルナウイルスIRESと比較した。なお、各トランスフェクションは3連で行われた。
【図7】UL77の上流領域がUL30 ORFで置換された組換えウイルスの構築を示す。 (a) UL77の上流領域がUL30 ORFで置換された組換えウイルス。組換えウイルスを構築するために、UL30のPCR産物を相同組換えに使用した。(b) 野生型(wt)及び組換えウイルスのサザンブロット分析。ウイルスDNAを制限酵素BstB Iで消化し、1.0%アガロースの電気泳動で分画し、32P-標識プローブによるハイブリダイゼーションにかけた。左に標準分子サイズマーカーを塩基対数で示した。(c) 組換えウイルスに感染させた細胞でのUL77 ORFのN末端に融合させたflagエピトープをもつUL77タンパク質の発現の分析。Flagエピトープをもつ融合タンパク質を検出するために、F3165 (Sigma)を使用した。
【図8】UL77の上流のUL76遺伝子がUL77遺伝子発現を調節することを示す。(a) 約3のMOIで組換えウイルスでHFF細胞を感染した後のウイルスDNA合成の分析。(b) 約3のMOIで組換えウイルスで感染した後のウイルスタンパク質のウエスタンブロット分析。ウイルスタンパク質は感染1, 2, 3及び4日後(d.p.i.)に分析した。レーン1及び5: 1 d.p.i.; レーン2及び6, 2 d.p.i.; レーン3及び7, 3 d.p.i.; レーン4及び8, 4 d.p.i.; レーン1〜4, RUL76revertantflagUL77; レーン5〜8, RUL30flagUL77。(c) 約3のMOIでの組換えウイルスの増殖曲線。ウイルス力価は、「材料と方法」に記載されるように、50%組織培養感染用量(TCID50)アッセイによって測定された。
【発明を実施するための形態】
【0021】
前述したように、本発明は、ヒトサイトメガロウイルス(HCMV)のUL76中のインターナルリボソームエントリーサイト(IRES)が、例えばピコルナウイルスIRESとほとんど同じ強さの活性をもつことの知見、並びに、UL76遺伝子が、IRESを介して、下流のUL77 ORFの翻訳に影響を及ぼしていることの知見に基づいている。具体的には、UL76中のIRESを不活性化することによってHCMVの複製の抑制、ひいては、HCMVの弱毒化を可能にした。ホルマリンなどの化学物質での処理による弱毒化では、外被(エンベロープ)タンパク質の変性などの問題も生じ兼ねないが、本発明の弱毒ウイルスは天然ウイルスの構造を保持するという利点を有する。
【0022】
<HCMV>
本発明の対象ウイルスであるHCMVは、すべてのウイルス株を包含するものとする。HCMVは、ヘルペスウイルス科βヘルペスウイルス亜科に属し、その直径が約180nmで約230kbpの塩基数からなる二本鎖DNAウイルスである。HCMVの株には公知の株が含まれ、例えばAD169株,Towne株,Major株,BT1943株,Davis株,Merlin株,Toledo株,3157株,6397株,3301株, C87株などが知られている(Dolan, A.ら(2004) J.Gen.Virol. 85:1301-1312; Chee, M.S.ら (1990) Curr.Top.Microbiol.Immunol.154:125-169)。
【0023】
上記のHCMV株のゲノム配列は、例えばGenBank(NCBI,米国)などに蓄積されたデータから入手可能である。例えば、一般によく知られているAD169株及びのTowne株ゲノム配列はそれぞれGenBank Accession Number FJ527563、AY315197として登録されている。
【0024】
HCMVは、ヒト線維芽細胞、例えばヒト包皮線維芽 (HFF)細胞(ATCC CC2509)やヒト胎児線維芽細胞、ヒト内皮細胞、例えばヒト毛細血管内皮細胞(HMVEC)(ATCC CC-2527)やヒト網膜色素内皮細胞(RPE)(ATCC C4000-1)などのヒト細胞に感染することができるので、このような細胞に、HCMV又はそのウイルスベクター、例えばHCMVゲノム配列とBAC(細菌人工染色体)配列から作製されたHCMVBAC、を感染又は導入することによってウイルスDNAを回収することができる(W. Dunnら (2003) Proc. Natl. Acad. Sci. USA, 100(24):14223-14228)。
【0025】
HCMVのUL76タンパク質は、ヘルペスウイルスにおいて高度に保存されたタンパク質ファミリーのメンバーである。UL76ファミリーメンバーは、毒性の決定、細胞溶解性複製、遺伝子発現の調節、アポトーシスの誘導などに関与することが最近指摘され、またウイルス複製の前初期でUL76タンパク質がHCMVの複製を抑制する作用があることが提案されている(V.-K. Siew et al., J. Biomed. Sci., 16:107 (2009);S.-K. Wang et al., J. Virol, 78(18):9750-9762 (2004))。しかし、UL76のウイルス複製抑制に関しては、UL76にウイルス複製に関係するタンパク質が存在するだろうということ以外の発展的な知見は提供されていない。本発明者らは、今回、ウイルス複製に重要であるパケージングに特定の役割をもつUL77 ORFの発現及び翻訳に、UL76遺伝子が、その中に含まれるIRESを介して影響を及ぼしていることを見出した。この知見から、IRESを不活性化することによってウイルス複製を阻害しうることを見いだした。
【0026】
IRES(Internal Ribosome Entry Site)は、一般に、タンパク質合成過程においてmRNA分子の5’末端での翻訳開始の反応に重要な役割をもつヌクレチド配列であるが、RNAウイルスのゲノムでもIRESの存在が確認されており、またDNAウイルスでもIRESの存在が確認されている。本発明では、DNAウイルスであるHCMVのUL76遺伝子配列内に存在するIRES配列が、下流タンパク質コード領域(UL77)の翻訳に関与していると考えられる。
【0027】
UL76中のIRESの不活性化は、以下の「弱毒化」の項で説明するように、上記IRES配列に突然変異を導入する方法などによって行うことができる。好ましい突然変異はIRES配列をIRES以外の別の核酸配列によって置換することである。ここで「IRES以外の」という用語は、UL76 IRESの天然型配列を有しない、かつ翻訳開始などのIRES本来の機能をもたないことを意味している。そして、そのようなIRES以外の別の核酸配列は、例えば、該IRES配列における1個又は複数のヌクレオチドの置換、欠失、付加、挿入、又はそれら変異の組合せなどの突然変異を含む配列からなり、UL76 IRES配列が別の核酸配列に置き換わることによって該IRESが不活性化される。この場合、上記別の核酸配列のサイズ(すなわち、ヌクレオチド数)は、上記IRES配列のサイズと完全に同じか又はほほ同じであることが好ましい。別の核酸配列の例は、非限定的に、HCMVゲノムのなかで非機能性であるようなUL(unique long)配列から選択してもよく、そのようなUL配列は、例えば同じ又は異なるHCMV株由来のUL30配列などである。上記のIRESの不活性化によって、HCMVの複製が抑制された弱毒HCMVウイルスを得ることができる。
【0028】
HCMV UL76及びUL30の配列について、例えば、AD169株(GenBank: FJ527563)及びTowne株(GenBank: FJ616285.1)のUL76及びUL30の配列は、次のとおりである。なお、他のHCMV株のUL76配列及びUL30配列もまた、GenBankなどの公知の遺伝子バンク或いは文献などから入手可能である。
【0029】
・AD169株のUL76の塩基配列(下線はIRES配列を示す。)及びアミノ酸配列:
(塩基配列、配列番号1)
ATGCCGTCCG GGCGTGGGGA CGACGCTGAT TCGACGGGGA ACGCTCTGCG GAGATTGCCT
CACGTGCGTA AGCGGATCGG TAAGCGTAAG CACCTGGACA TCTACCGTCG CCTGTTGCGG
GTCTTTCCCT CGTTTGTGGC CCTCAACCGC CTGTTGGGAG GCCTTTTCCC ACCCGAGCTG
CAAAAGTACC GTCGCCGTCT TTTCATCGAA GTACGATTAA GTCGGCGGAT TCCCGACTGC
GTGTTGGTGT TTTTACCGCC GGACTCTGGG TCGCGCGGCA TCGTGTATTG CTACGTGATT
GAGTTCAAAA CTACGTACTC AGACGCCGAC GATCAGTCCG TGCGGTGGCA CGCCACCCAC
AGCCTGCAGT ACGCCGAGGG CCTGCGCCAG CTCAAGGGCG CCTTGGTGGA CTTTGATTTT
CTGCGTCTGC CGCGCGGTGG CGGTCAAGTC TGGAGCGTAG TGCCCAGTCT GGTTTTTTTT
CAGCAAAAGG CCGATCGCCC ATCTTTTTAC CGGGCTTTTC GTTCGGGCCG TTTCGACTTG
TGTACCGATT CTGTCCTGGA CTATCTGGGA CGGCGTCAGG ATGAGTCTGT TGCACACCTT
TTGGCGGCTA CCCGTCGCCG TCTTCTTCGA ACCGCACGAG GAAAACGTGC TGCGCTGCCC
CGAGCGCGTG CTTCGGCGGT TGCTGGAGGA CGCGGCGGTG ACAATGCGCG GCGGGGGCTG
GCGCGAGGAC GTGCTCATGG ACCGGGTGCG CAAACGGTAT CTGCGTCAGG AGCTCAGGGA
TCTGGGTCAC AGGGTGCAGA CTTACTGCGA GGATCTCGAA GGGCGCGTGT CCGAGGCGGA
GGCGCTGTTG AACCAGCAGT GCGAGCTCGA CGAAGGACCG TCGCCGCGGA CGCTGCTACA
ACCACCGTGT CGTCCGCGTT CTTCGTCCCC AGGGACCGGC GTGGCAGGAG CTTCTGCCGT
CCCACACGGT CTTTATAG
(アミノ酸配列、配列番号2)
MPSGRGDDADSTGNALRRLPHVRKRIGKRKHLDIYRRLLRVFPSFVALNRLLGGLFPPELQKYRRRLFIEVRLSRRIPDCVLVFLPPDSGSRGIVYCYVIEFKTTYSDADDQSVRWHATHSLQYAEGLRQLKGALVDFDFLRLPRGGGQVWSVVPSLVFFQQKADRPSFYRAFRSGRFDLCTDSVLDYLGRRQDESVAHLLAATRRRLLRTARGKRAALPRARASAVAGGRGGDNARRGLARGRAHGPGAQTVSASGAQGSGSQGADLLRGSRRARVRGGGAVEPAVRARRRTVAADAATTTVSSAFFVPRDRRGRSFCRPTRSL
【0030】
・AD169株のUL30の塩基配列及びアミノ酸配列:
(塩基配列、配列番号3)
ATGACGACATCAGCGATGACGGCTCCTGATACGCGCCGGCAGCTGCAGCACGTGGAGACGCTGCGTCGGTTTCTGCGCGGCGACAGCTGCTTTGTGCACGATCTCCGGGGCATGATGGACTATCACGACGGGCTCTCGCGCCGTCAACAGCGTGCCTTTTGCCGCGCGAGTCGCGTGTTGACGGACCCGGAGCCCATCCAGAGCGAAGCGGAGGGGGAGAATAAACAGTTTACGGAGCACACACACAAAGTAGTCTCGTTTTTTATTAAAAGTGTCTTTGTATTTCCCTATCTTGTGTTGCCCAACTGCTGTCAGGTCTCCGTAGATCGCTCCCGGGTGCCCGAGACGGGGGGCAGATGGCTATAA
(アミノ酸配列、配列番号4)
MTTSAMTAPDTRRQLQHVETLRRFLRGDSCFVHDLRGMMDYHDGLSRRQQRAFCRASRVLTDPEPIQSEAEGENKQFTEHTHKVVSFFIKSVFVFPYLVLPNCCQVSVDRSRVPETGGRWL
【0031】
・Towne株のUL76の塩基配列(下線はIRES配列を示す。)及びアミノ酸配列:
(塩基配列、配列番号5)
ATGCCGTCCG GGTGTGGGGA CGACGCTGAT TCGAAGGGGA ACGCTCTGCG GAGATTGCCT
CACGTGCGTA AGCGGATCGG TAAGCGCAAG CACCTGGACA TCTACCGTCG CCTGCTGCGG
GTCTTTCCCT CATTTGTGGC GCTCAACCGC CTGTTGGGAG GCCTTTTCCC ACCCGAGTTG
CAAAAGTACC GTCGCCGTCT TTTCATCGAA GTACGATTAA GTCGGCGGAT TCCCGACTGC
GTGTTGGTGT TTTTACCGCC GGACTCTGGG TCGCGCGGCA TCGTGTATTG CTACGTGATT
GAGTTCAAAA CTACGTACTC AGACGCCGAC GATCAGTCCG TGCGGTGGCA CGCCACCCAC
AGCCTGCAGT ACGCCGAGGG CCTGCGCCAG CTCAAGGGCG CCTTGGTGGA CTTTGATTTT
CTGCGTCTGC CGCGCGGTGG CGGTCAAGTC TGGAGCGTAG TGCCCAGTCT GGTTTTTTTT
CAGCAAAAGG CCGATCGCCC ATCTTTTTAC CGGGCTTTTC GTTCGGGCCG TTTCGACCTG
TGTACCGATT CTGTTCTGGA CTATCTGGGA CGGCGTCAGG ATGAGTCTGT TGCACACCTT
TTGGCGGCTA CCCGTCGCCG TCTTCTTCGA GCCGCACGAG GAAAACGTGC TGCGCTGCCC
CGAGCGCGTG CTTCGGCGGT TGTTGGAGGA CGCGGCGGTG GCAATGCGCG GCGGGGGCTG
GCGCGAGGAC GTGCTCATGG ACCGGGTGCG CAAACGGTAT CTGCGTCAGG AGCTGAGGGA
TCTGGGTCAC AGGGTGCAGA CTTACTGCGA GGATCTCGAA GGGCGCGTGT CCGAGGCGGA
GGCGCTGTTG AACCAGCAGT GCGAGCTCGA CGAAGGACCG TCGCCGCGGA CGCTGCTACA
ACCACCGTGT CGTCCGCGTT CGTCGTCCCC AGGGACCGGC GTGGCAGGAG CTTCCGCCGT
CCCACACGGT CTTTATAG
(アミノ酸配列、配列番号6)
MPSGCGDDADSKGNALRRLPHVRKRIGKRKHLDIYRRLLRVFPSFVALNRLLGGLFPP
ELQKYRRRLFIEVRLSRRIPDCVLVFLPPDSGSRGIVYCYVIEFKTTYSDADDQSVRW
HATHSLQYAEGLRQLKGALVDFDFLRLPRGGGQVWSVVPSLVFFQQKADRPSFYRAFR
SGRFDLCTDSVLDYLGRRQDESVAHLLAATRRRLLRAARGKRAALPRARASAVVGGRG
GGNARRGLARGRAHGPGAQTVSASGAEGSGSQGADLLRGSRRARVRGGGAVEPAVRAR
RRTVAADAATTTVSSAFVVPRDRRGRSFRRPTRSL
【0032】
・Towne株のUL30の塩基配列及びアミノ酸配列:
(塩基配列、配列番号7)
ATGACGACATCGGCGATGACGGCTCCTGATACGCGCCGACAGCTGCAGCACGTGGAGACG
CTGCGTCGGTTTCTGCGCGGCGACAGCTGCTTTGTGCACGATCTCCGGGGCATGATGGAC
TATCACGACGGGCTCTCGCGCCGTCAACAGCGTGCCTTTTGCCGCGCGGGTCGCGTGTTG
ACGGACCCGGAGCCCATCCAGAGCGAGACGGAGGGGGAGAATAAACAGTTTACGGAGCAC
ACACACAAAGTAGTCTCGTTTTTTATTAAAAGTGTCTTTGTATTTCCCTGTCTTGTGTTG
CCCAACTGCTGTCAGGTCTCCGTAGATCGCTCCCGGGTGCCCGAGACGGGGGGCAGATGG
CTATAA
(アミノ酸配列、配列番号8)
MTTSAMTAPDTRRQLQHVETLRRFLRGDSCFVHDLRGMMDYHDGLSRRQQRAFCRAGRV
LTDPEPIQSETEGENKQFTEHTHKVVSFFIKSVFVFPCLVLPNCCQVSVDRSRVPETGG
RWL
【0033】
<弱毒化>
本発明では、HCMV UL76中のIRESを不活性化することによってHCMVを弱毒化する。
本明細書で使用される「弱毒化」なる用語は、ウイルスの毒性(すなわち、感染性および病原性)を減弱することを意味する。
【0034】
IRESの不活性化は、IRESのUL77翻訳開始に関する機能を阻害することを意味し、該不活性化は、IRES配列の突然変異、例えばIRES配列の一部又は全部を別のヌクレオチド配列によって置換、欠失、付加又は挿入することによって行うことができる。好ましくは、IRES配列の全部又はほとんど全部をIRES以外の別の核酸配列によって置換することによってIRESを不活性化することができる。より好ましくは、IRES配列と実質的に同じ長さ(サイズ)の別のヌクレオチド配列である。しかし、IRESのUL77翻訳開始に関する機能を阻害することができるならば、突然変異の度合い(例えば配列同一性の度合い)、突然変異の仕方などに限定されないものとする。
【0035】
本発明の実施形態によれば、HCMVのUL76 IRES以外の核酸配列がHCMV UL30配列によって置換されうる。このUL30配列は、UL76 IRESと同じ又は異なるHCMV株に由来することができる。例えば、弱毒HCMVウイルスがTowne株をベースにする場合、そのUL76 IRES配列をTowne株のUL30配列で置換してもよいし、或いはAD169株などの他の株由来のUL30配列で置換してもよい。このとき、UL30配列のサイズがIRES配列のサイズと実質的に同程度(例えば±5%、好ましくは±3%の範囲内)であることが好ましく、そうすることによってHCMVの複製能がより顕著に低下する。
【0036】
IRESのUL77翻訳開始に関する機能が阻害されることによって、UL77のパケージング機能が阻害され、ひいてはHCMVの複製が抑制される。突然変異の導入によって、組換えHCMVの複製能が約1/10以下、1/50以下、好ましくは1/100以下に抑制される。
【0037】
本発明の組換えHCMVは、野生型HCMVのUL76中のIRES配列の一部又は全部に突然変異を導入したゲノムDNAを、適当なベクターを利用して作製し、これを適当な宿主細胞、例えば上記のヒト線維芽細胞やヒト内皮細胞に感染し、組換えウイルス粒子を回収することを含む手順によって作製することができる。突然変異は、例えば部位特異的突然変異誘発法、PCRを利用した突然変異誘発法、相同組換え法などの公知の方法によって導入しうる。部位特異的突然変異誘発法は、例えば、野生型HCMVのUL76 IRES配列の一部又は全部に突然変異を導入し、それに隣接する野生型と同一の十分な長さの配列を配置したヌクレオチド配列を合成し、クローニングベクターに導入して大腸菌などの細菌中にサブクローニングすること、野生型HCMVゲノムDNAを含む人工染色体ベクター、例えば細菌人工染色体(BAC, PAC)を作製し、ついで、上記ヌクレオチド配列を人工染色体ベクターにアニーリングし、PCRを行ない、目的DNAを増幅 することなどを含む。
【0038】
具体的な方法として、例えば大腸菌内でバクテリオファージλ組換えタンパク質exo、betaおよびgamを発現するための迅速相同組換え系(Ellis, H.M.ら(2001) Proc. Natl.Acad.Sci.USA98:6742-6746)、HCMVのBAC(細菌人工染色体)DNA (Dunn,W ら(2003)Proc.Natl.Acad.Sci.USA100:14223-14228; Lee, E-C.ら(2001)Genomics73:56-65; Borst,E-Mら(1999)J.Virol.73(10):8320-8329)などを使用して突然変異誘発を行うことができる。
【0039】
組換えのためのHCMVの二本鎖DNAは、カナマイシン耐性遺伝子などの薬剤耐性遺伝子と70bp程度の相同ウイルスDNA配列を含み、この耐性遺伝子には、34bpの最小FRT部位をフランキングさせることができる(後述の実施例1参照)。UL76 IRES配列の突然変異体を作製するために、適当なプライマーを使用してPCRによる増幅を行う。PCR条件は、例えば、94℃2分の変性を1サイクル、94℃15秒の変性, 55℃30秒のアニーリング及び72℃1分の伸長を30サイクル、並びに、72℃5分の伸長を1サイクルとするPCRサイクルプログラムからなる。PCR増幅産物をフェノール−クロロホルムで抽出し、95%エタノールで沈澱する。
【0040】
残存する鋳型DNAを除くために、PCR産物を、制限酵素を用いて消化したのち、得られたDNA断片を、HCMV-BAC DNAを含むコンピテント大腸菌中へエレクトロポレーション、リポフェクション、リン酸カルシウム法などの手法、好ましくはエレクトロポレーションを用いて形質転換する。
【0041】
カナマイシン耐性遺伝子を欠失するために、組換えHCMV-BAC DNAを大腸菌に形質転換する。また、リコンビナーゼを発現するプラスミドpCP20(Hahn,W.ら (2003)Virology307:164-177)を、組換えHCMV-BAC DNAを含む大腸菌(DH10B)に形質転換する。カナマイシン耐性遺伝子を含まないHCMV BAC DNAを、アンピシリンおよびクロラムフェニコール含有のLBプレート上で選択する。
【0042】
HFF細胞などの細胞を、リン酸カルシウム沈殿法(Graham,F.L.およびvan der Eb,A.J.(1973)Virology52:456-467)によって、プラスミドpSVpp71の存在中、上記の各組換えBACでトランスフェク ションする。約10日後に、ウイルスプラークが出現し、100%細胞変性作用(CPE)の7日後に、細胞外液を回収し、約1:10に希釈したものと未希釈のものを調製し、HFF細胞などの細胞に感染し、組換えウイルスを回収する。このとき、ウイルスの選択は、IRES配列に目的の変異が導入されていることを配列分析によって確認すること、並びに、HFF細胞などの細胞に導入または感染させて複製が抑制されていることを確認し、また、野生型の表現型と同一または本質的に同一であることを確認することによって行うことができる。
【0043】
<ワクチン>
本発明はさらに、弱毒HCMVを含むことを特徴とする、HCMV感染症を治療または予防するためのワクチンを提供する。
【0044】
ワクチンは、一般に生ワクチンと不活性化ワクチンに大別されるが、特に生ワクチンは安価に製造可能であることや接種回数の少なくて高い効果が期待できるなどの利点とともに毒性復帰のリスクがある。本発明の弱毒HCMVは、パッケージングが阻害されるため顕著にウイルス複製が抑制されたウイルスであるため、安全性の高い生ワクチンとして使用可能であると考えられる。
【0045】
本発明のワクチンは、AIDS患者、臓器移植患者などの免疫抑制患者、胎児、乳幼児、高齢者、重症患者などの抵抗性が低下したヒトに対し、肺炎、肝炎、網 膜炎、胃腸炎などを含む広範な疾患を引き起こす可能性のあるHCMV感染の予防および治療のために使用可能である。HCMVは、血液、尿、唾液などを介して感染するウイルスであり、上記のヒトに感染すると、重症になる場合がある。そのような感染を抑制するために、本発明のワクチンは有効である。
【0046】
本発明のワクチンは、適当な賦形剤、特に希釈剤、例えば滅菌水、生理食塩水、リンゲル液、緩衝液などに処方されうる。また、ブドウ糖、トレハロース、エタノール、グリセリンなどの添加剤を含有させることができる。さらにまた、免疫応答を増強させるためのアジュバント、例えば水酸化アルミニウム(alum)、ムラミルジペプチド、サポニン類、リポポリサッカライド、リポゾーム、消化性ポリマー(Gupta, R.K. et al., Vaccine 13:1263-1276 (1995))などをその有効量で含有させてもよい。さらにまた、安定剤を添加してもよく、安定剤にはゼラチン、ヒト血清アルブミンなどのタンパク質、ポリソルベートなどの界面活性剤、スクロース、トレハロースなどの糖類、メチオニン、リジンなどのアミノ酸類などが含まれる。
【0047】
投与方法は、例えば皮下、筋肉内または皮内注射、鼻腔内などの粘膜投与などを含む。鼻腔内投与の場合、噴霧器による投与が好ましい。投与量は、例えば103〜1011PFUウイルス/kg体重、又は1回あたり10μg〜10mg、の用量であるが、この範囲に制限されない。ワクチンの投与量は、患者などの被験者の状態、年齢、体重、性別、重症度などを考慮に入れて、医師の裁量で判断されるべきである。被験者は、1回又は2回以上の投薬を受容でき、複数回投与の場合、1週間〜1か月以上の間隔、例えば2,3,6,12,18又は24か月間隔で投与しうる。
【0048】
<抗体の作製>
本発明はさらに、上記弱毒HCMVを非ヒト動物に免疫して抗体を作製し、該抗体を回収することを含む、抗HCMV抗体の作製方法を提供する。
【0049】
抗体は、IgA、IgG、IgD、IgE、IgMのいずれのクラスでもよいし、またIgG1、IgG2、IgG3、IgG4、IgA1、IgA2などのいずれのサブクラスでもよい。さらにまた、抗体は、ポリクローナル抗体、モノクローナル抗体、抗体フラグメント、組換え抗体のいずれでもよい。組換え抗体には、例えば単鎖Fv抗体(scFv)、単鎖ダイアボディ(scDb)などが含まれる(浅野竜太郎,生化学77(12):1497-1500(2005))。抗体フラグメントには、例えばFab、F(ab’)2、Fvなどが含まれる。
【0050】
また、抗体には、ヒト抗体、ヒト化抗体、キメラ抗体なども包含され、好ましい抗体はヒト抗体である。
抗体の作製は、慣用の方法で行うことができる。
【0051】
ポリクローナル抗体は、弱毒HCMV粒子を動物に免疫し、抗血清、さらにIgG画分を回収することによって作製できる。
【0052】
モノクローナル抗体は、弱毒HCMV粒子を免疫した動物(例えばマウス、ラットなど)から脾臓を取り出し、脾臓細胞又はリンパ球とミエローマ細胞とを融合させ、該HCMV粒子抗原と特異的に免疫反応する抗体をスクリーニングすることによって作製できる。
【0053】
ヒト抗体は、ヒト抗体産生非ヒト動物(例えばKMマウス、XenoMouse)を用いて作製することができる。あるいは、弱毒HCMV粒子を免疫した動物のゲノムから抗体遺伝子を回収し、重鎖可変領域及び軽鎖可変領域の各CDR1〜CDR3コード配列を決定し、ヒト抗体遺伝子の本来のCDR1〜CDR3コード配列を前記CDR1〜CDR3コード配列と置換することを含む遺伝子組換え技術を用いてヒト抗体を作製することができる。
【0054】
上記の抗体は、ポリエチレングリコールなどの分子によって化学修飾されてもよい。
上記のようにして得られた抗体は、HCMV感染症の治療や予防のために使用することができるし、或いはHCMVの検出のために使用することもできる。ここで、HCMVの検出のために、抗原-抗体反応を利用する手法のいずれも使用できる。そのような手法には、例えば均質法、固相法(例えばサンドイッチ法)などが含まれる。
【実施例】
【0055】
以下に実施例を挙げて本発明をさらに具体的に説明するが、本発明の範囲は、該実施例によって制限されないものとする。
【0056】
[実施例1]
<材料と方法>
細胞及びウイルス
初代ヒト包皮線維芽(HFF)細胞を、従来法に従い(39)、10%牛新生仔血清(Sigma, St. Louis, Mo.)、ペニシリン(100 U/ml)及びストレプトマイシン(100 μg/ml)を補充したEagle最少必須培地(Wako,大阪,日本)中、5%CO2中に37℃に維持した。従来法に従い(17, 31)、HFF細胞での標準プラークアッセイによって、ウイルス力価を測定した。
【0057】
酵素
制限エンドヌクレアーゼは、New England Biolabs Inc.(Beverly, MA.)から購入された。高忠実度のかつ拡大された高忠実度のTaq DNAポリメラーゼはそれぞれInvitrogen及びRoche(Carlsbad, CA.及びMannheim, Germany)から購入された。RNasin及びRNase非含有DNaseはPromega (Madison, WI.)から購入された。これらの酵素は、製造者の説明書に従って使用された。
【0058】
HCMV BAC DNAの突然変異誘発
バクテリオファージλ組換えタンパク質,エクソ(exo),β及びγを発現する大腸菌(D. Court博士(NIH, MD.)から恵与された。)での迅速組換え系を、従来法に従い使用した(8)。HCMV TowneのBAC DNAは、F. Liu 博士(University of California, Berkeley, CA)から恵与された(7)。組換え用の二本鎖DNAは、カナマイシン耐性遺伝子、それに隣接する34bp最小FRT部位(5’-GAAGTTCCTATTCTCTAGAAAGTATAGGAACTTC-3’(配列番号55))(15)、又は、カナマイシン耐性遺伝子とストレプトマイシン感受性遺伝子(RpsLneo, Gene Bridges, Dresden, Germany)及び70bpの相同なウイルスDNA配列を含んでいた。UL76配列の欠失を作るため、次のプライマー対、すなわち、BACdlUL76FRTFKanF: 5’-ATTTAATACGATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCGAAGTTCCTATTCTCTAGAAAGTATAGGAACTTCAACTCAGCAAAAGTTCGATTTATTCAAC-3’(配列番号9); BACdlUL76FRTRKanR: 5’-CCTCGTGCGGTTCGAAGAAGACGGCGACGGGTAGCCGCCAAAAGGTGTGCAACAGACTCATGAAGTTCCTATACTTTCTAGAGAATAGGAACTTCTAATGCTCTGCCAGTGTTACAACCA-3’(配列番号10)を使用した。UL77 ORFとflagエピトープタグ (5’- ATGGACTACAAAGACGATGACGACAAG-3’(配列番号11))のN末端融合体を作製するために、以下に記載のように、逆の手順をとった。RpsLneo遺伝子を、次のプライマー対BACUL76neoF: 5’- CGGGCTTTTCGTTCGGGCCGTTTCGACTTGTGTACCGATTCTGTCCTGGACTATCTGGGACGGCGTCAGGGGCCTGGTGATGATGGCGGGATC-3’(配列番号12); BACUL76neoR: 5’- GCACGTTTTCCTCGTGCGGTTCGAAGAAGACGGCGACGGGTAGCCGCCAAAAGGTGTGCAACAGACTCATTCAGAAGAACTCGTCAAGAAGG-3’(配列番号13)を用いてHCMV BAC DNAに挿入した。カナマイシン耐性遺伝子(KanR)のいずれかの末端に50bpの相同なウイルスDNA配列を有するKanRを、従来法に従いPCRにより増幅した(18, 20, 22)。PCRによるRpsLneo遺伝子の増幅のために、pRpsL-neo(Gene Bridges)を鋳型として用いた。残りの鋳型DNAを除くために、PCR産物を、DpnIによって37℃で1.5時間消化した。
【0059】
従来法に従い逆の手順も行った(46)。簡単に説明すると、50bpの相同なウイルスDNAを含む約500ngの組換え用一本鎖DNA をRpsLneo 遺伝子を用いてHCMV BAC DNAに導入した(19, 21, 46)。RpsLはストレプトマイシン感受性遺伝子であるので、変異型BAC DNAを、Counter Selection Modificationキット(Gene Bridges)を用いてストレプトマイシン耐性の増加に基づき選択した。BACdlUL76flagUL77及びBACUL76flagUL77を構築するために、BACdlUL76flagoligo-2及びBACUL76flagoligo-2 をそれぞれ組換えのために使用した。それらの配列は以下のとおりである。BACdlUL76flagoligo-2: 5’- CGATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCATGGACTACAAAGACGATGACGACAAGAGTCTGTTGCACACCTTTTGGCGGCTACCCGTCGCCGTCTTCTTCGA-3’(配列番号14);BACUL76flagoligo-2: 5’- TTTCGACTTGTGTACCGATTCTGTCCTGGACTATCTGGGACGGCGTCAGGATGGACTACAAAGACGATGACGACAAGAGTCTGTTGCACACCTTTTGGCGGCTACCCGTCGCCGTCTTCTTCGA-3’(配列番号15)
【0060】
flagエピトープを下線で示した。RUL76stopflagUL77及びRUL76revertantflagdUL77を作製するために、上記のとおりに逆手順も用いた。RpsLneo遺伝子を、従来法に従い、以下のプライマー対:BACUL76stopneo+StF: 5’- GATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCATGCCGTCCGGGCGTGGCCTGGTGATGATGGCGGGATC-3’(配列番号16); BACUL76stopneo+StR: 5’- ACCGATCCGCTTACGCACGTGAGGCAATCTCCGCAGAGCGTTCCCCGTCGAATCAGCGTCGTCCTCAGAAGAACTCGTCAAGAAGG -3’(配列番号17)を用いてRflagUL77 BAC DNA中に挿入した。また、以下のオリゴ: BAColigoUL76stop: 5’- AATACGATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCATGCCGTCCGGGCGTtaGGACGACGCTGATTCGACGGGGAACGCTCTGCGGAGATTGCCTCACGTGCGTAAGCGGATCGGTAAGCG-3’(配列番号18); BAColigoUL76revertant: 5’- AATACGATGTCATTGGCCGCTGCGAAGGAAGAAGAGGGGACACGCGGGTAAGCCATGCCGTCCGGGCGTGGGGACGACGCTGATTCGACGGGGAACGCTCTGCGGAGATTGCCTCACGTGCGTAAGCGGATCGGTAAGCG-3’(配列番号19)を用いて逆選択を行った。小文字は、UL76 ORF中にTAG stopコドンを挿入するための変異塩基を示す。各変異に対して2つの組換えウイルスが単離され、それらは同じウイルスを振舞うことが判明した。
【0061】
カナマイシン耐性遺伝子の除去
カナマイシン耐性遺伝子を欠失させるために、組換えHCMV BAC DNAを、大腸菌DH10B中に形質転換した。プラスミドpCP20 (G. Hahn博士(Max von Pettenkofer Institute, Munich, Germany)から恵与された。)を、組換えHCMV BAC DNAを含むDH10B中に形質転換した。カナマイシンを含まないHCMV BAC DNAを、アンピシリン及びクロラムフェニコール含有のLBプレート上で選択した。
【0062】
PCR分析
組換えBAC DNAを選択するために、PCR分析を以下のプライマー対: UL76detectF: 5’- TACGGGTTACAAAAGTCGCGTCTCTGTCT-3’(配列番号20)及びUL76detectR: 5’ -GCTCGGGGCAGCGCAGCACGTTTT-3’(配列番号21)を用いて行った。PCRサイクルプログラムは、94℃2分の変性を1サイクル、94℃15秒の変性, 55℃30秒のアニーリング及び72℃1分の伸長を30サイクル、並びに、72℃5分の伸長を1サイクルからなった。PCR産物を、pCR 2.1-TOPO TAクローニングベクター (Invitrogen)中にクローニングし、配列決定して組換えと除去を確認した。
【0063】
組換えウイルスの単離
HFF細胞を、2μgのプラスミドpSVpp71の存在下、各組換えBACを5μgもしくは10μgを用いてGrahamとVan der Eb (12)のリン酸カルシウム沈殿法によってトランスフェクトした。10日後、ウイルスプラークが現れた。100%の細胞変性作用(CPE)の7日後に、細胞外液を回収し、HFF細胞の感染のために、1:10に希釈し、又は無希釈とした。100%CPEの5〜7日後に、ウイルス含有細胞外液を、使用するまで、50%牛新生仔血清中-80℃に保存した。
【0064】
プラスミドの構築
プラスミドpCMVflagUL76-77HA, pCMVflag-frame-shift-UL76-77HA及びpCMVflagUL76-77-frame-shift-HAを以下のように構築した。UL76 ORFのN末端に融合したflagエピトープ及びUL77 ORFのC末端に融合したHAエピトープを有するUL76〜UL77領域をpCMVflagUL76-77HAについてプライマー対EcoRIflagUL76F及びXbaIHAUL76R, pCMVflag-frame-shift-UL76-77HAについてプライマー対EcoRIframe-shiftflagUL76及びXbaIHAUL76R、並びにpCMVflagUL76-77-frame-shift-HAについてEcoRIflagUL76F及びXbaIframe-shiftHAUL76Rを用いてHCMV TowneのBAC DNAからPCRによって増幅した。プライマー配列は以下のとおりである:
EcoRIflagUL76F:
5’- GGAATTCATGGACTACAAAGACGATGACGACAAGCCGTCCGGGCGTGGGGACGACG-3’(配列番号22);
EcoRIframe-shiftflagUL76:
5’- GGAATTCATGACTACAAAGACGATGACGACAAGCCGTCCGGGCGTGGGGACGACG-3’(配列番号23);
XbaIHAUL76R:
5’- GCTCTAGATTAAGCGTAATCTGGAACATCGTATGGGTACAACACCGCCACGCTCGGAAGAAACC-3’(配列番号24);
XbaIframe-shiftHAUL76R:
5’-GCTCTAGATTAAGCGTAATCTGGAACATCGTATGGGTACAACACCGCCACGCTCGGAAGAAACCCAACACTA-3’(配列番号25)
【0065】
PCR産物を制限エンドヌクレアーゼEcoR I及びXba Iで消化し、対応の制限エンドヌクレアーゼ部位でプラスミドpcDNA3.1(+) (Invitrogen)にクローン化し、そして配列決定した。プラスミドpCMVFlucRluc は次のように構築された。pSp-luc+NF融合ベクター (Promega)由来のホタルルシフェラーゼを、制限エンドヌクレアーゼEcoR I及びHind IIIで消化し、対応の制限エンドヌクレアーゼ部位でプラスミドpcDNA3.1(+) (Invitrogen)にクローニングし、これをpCMVlucと名付けた。pGL3hRlucベクター(Pomega) 由来のRenilla ルシフェラーゼ遺伝子を、制限エンドヌクレアーゼEcoR I及びApa Iで消化し、 対応の制限エンドヌクレアーゼ部位でpCMVluc中にクローン化した。最後に、インタークローニング部位(ICS)を構築するためのDNA断片を、以下のオリゴDNAを用いて構築した。
MCSsense: 5’- AATTCGGTACCCTCGAGAGATCTGGATCCG-3’(配列番号26)、及び
MCSantisense: 5’- AATTCGGATCCAGATCTCTCGAGGGTACCG-3’(配列番号27)
【0066】
アニーリングされたDNAは、制限エンドヌクレアーゼEcoR Iによって消化されたpCMVFlucRluc中にクローン化された。挿入されたインタークローニング部位は自動ジデオキシヌクレオチド配列決定によって決定された。UL76断片, UL76(71), UL76(116), UL76(190)及びUL76(580)を、以下のプライマー対セット:
XhoIUL76ORFF: 5’- CCGCTCGAGCCGTCCGGGCGTGGGGAC -3’(配列番号28)、及び
BamHIUL76 71R: 5’- CGGGATCCTTACGCACGTGAGGCAATCTCCGC -3’ (配列番号29)、
BamHIUL76 116R: 5’- CGGGATCCAACAGGCGACGGTAGATGTCCAGG -3’ (配列番号30)、
BamHIUL76 190R: 5’- CGGGATCCGGTACTTTTGCAGCTCGGGTGGG -3’ (配列番号31)、又は
BamHIUL76 580R: 5’- CGGGATCCCCTGACGCCGTCCCAGATAGTCC -3’ (配列番号32)
をそれぞれ用いるPCRによってHCMV TowneのBAC DNAから増幅した。同様に、UL76(191-580), UL76(271-580)及びUL76(451-580)を、以下のプライマーセット:
XhoIUL76 191F: 5’- CCGCTCGAGGTCGCCGTCTTTTCATCGAAGTACGATTAAG-3’(配列番号33)、
XhoIUL76 271F: 5’- CCGCTCGAGTCGCGCGGCATCGTGTATTGCTACG-3'(配列番号34)、又は
XhoIUL76 451F: 5’- CCGCTCGAGTGGAGCGTAGTGCCCAGTCTGG-3’(配列番号35)及び
BamHIUL76 580R(上記)
をそれぞれ用いるPCRによって増幅した。rUL76(580)の逆相補体もまた、プライマー対: BamHIUL76F (5’- CGGGATCCCCGTCCGGGCGTGGGGAC -3’ (配列番号36))及びXhoIUL76 580R (5’- CCGCTCGAGCCTGACGCCGTCCCAGATAGTCC -3’ (配列番号37))を用いるPCRによって増幅し、上記のレポータープラスミド中にクローン化した。ポリオウイルスIRES断片を、プライマー対:XhoIpolioIRESF (5’-CCGCTCGAGTTAAAACAGCTCTGGGGTTGTACCCACCCCAG-3’ (配列番号38))及びBamHIpolioIRESR (5’- CGGGATCCTATGATACAATTGTCTGATTGAAATAACTGTTGAAATTG-3’ (配列番号39))を用いてpRluc-wt-luc+ PV IRES (Dr. Nomoto(東京大学)から恵与された。)から増幅し、上記のレポータープラスミド中にクローン化した。
【0067】
サザンブロット分析
NucleoBondキット (Macherey-Nagel Duren, Germany)を用いて組換えBAC DNAを精製し、制限センドヌクレアーゼBstBIで消化し、0.6%アガロースゲル電気泳動(17)及びサザンブロット分析(31)にかけた。UL75プローブは文献に記載されている(20)。UL76 DNAは、プライマー対:UL76F (5’- CCGTCCGGGCGTGGGGACGA-3’ (配列番号40))及びUL76R (5’-CCGTCCCAGATAGTCCAGGACAGA-3’ (配列番号41))を用いるPCRによって上記のHCMV TowneのBAC DNAから増幅された。UL75又はUL76 DNA断片を、MegaprimeDNAラベリングシステム(Amersham Pharmacia Biotech.)を用いて32P-dCTP(Amersham Pharmacia Biotech Piscataway, NJ and PerkinElmer, MA )で標識することによってプローブ(図1参照)を作製した。
【0068】
ノザンブロット分析
20μgの細胞質RNAを、2.2Mホルムアミド含有1%アガロースゲル中の電気泳動にかけたのち、最大強度のHybond N+ (Amersham)にトランスファーした。IE1プローブを用いるノザンブロット分析を文献記載のように行った(31)。UL76ウイルスDNAは上記のように作製した。UL77 及びUL78ウイルスDNAを、プライマー対:UL77F (5’- CGTCTGGCCGACGACGTGAGTCGT-3’ (配列番号42))及びUL77R (5’-CGTCGACTCCAGAGAGAAAGCACGTC-3’ (配列番号43)), 並びに、UL78F (5’-CGTTAGCCTGGTCAACCTGCTGACT-3’ (配列番号44))及びUL78R (5’- ACGATGGAAAGAACCCAGGCAAAGGCG-3’ (配列番号45))をそれぞれ用いるPCRによって上記のHCMV TowneのBAC DNAから増幅した。32P-dCTPで標識することによって放射性標識プローブを得た。
【0069】
ウエスタンブロット分析
感染後、表示の時点で細胞を回収し、PBSで洗浄し、溶解バッファーで処理した(18, 22)。20μgのタンパク質アリコートを各レーンに載せてドデシル硫酸ナトリウム-10%ポリアクリルアミドゲル電気泳動(SDS-PAGE)を行い、そのあと、PVDF膜に転写した。IE1及びIE2によってコードされるpIE72及びpIE86タンパク質、UL44によってコードされるpp52、UL83によってコードされるpp65、並びにUL99によってコードされるpp28を検出するために、一次マウスモノクローナル抗体NEA-9221 (Perkin Elmer, Boston, MA.), M0854 (Dako, Carpinteria, CA.), 023-A (Vitrogen, Watertown, MA)及びCA 004 (EastCoast Bio, North Berwick, ME.)をそれぞれ使用した。flag又はHAエピトープとの融合タンパク質、又は対照としての細胞GAPDH、を検出するために、マウス或いはラットモノクローナル抗体F3165(Sigma, St. Louis, MO.)又は3F10 (Roche)、或いはMAB376 (Chemicon, Temucula, CA.)をそれぞれ使用した。増強された化学発光検出試薬(Enhanced chemiluminescence detection reagents)(Amersham)及び二次西洋ワサビペルオキシダーゼ標識抗マウスIgG抗体(Zymed, San Francisco, Ca.)或いは抗ラット IgG抗体(Jackson ImmunoResearch Laboratories Inc., West Grove, PA)を、製造者の説明書に従って使用した。
【0070】
リアルタイムRT-PCR分析
RNAの検出のために、全細胞RNAを、TRI試薬 (Invitrogen)を用いて表示の時点で精製し、DNase I (Promega)で処理し、製造者の説明書の手順に従って逆転写酵素(RT)(Roche) を用いてcDNAに変換した。第1cDNA鎖を、2μg RNAと250ngランダムプライマー (Roche)から20μlの最終容量で作製した。サンプルを70℃15分加熱して不活性化した。逆転写酵素を含まない対照では、投入したウイルスDNAもプラスミドDNAも検出されず、擬似(mock)対照に類似していた。RT反応は、従来法に従い行った(22)。PLATINUM Quantitative PCR SUPERMIX-UDG カクテル(Invitrogen)を含む25μlの最終容量で増幅を行ったが、各反応混合液には、2μgの第1cDNA鎖, 5mM MgCl2, 500nMの各プライマー、及び250nMの各プローブを含んでいた。HCMV UL77用の正方向及び逆方向プライマー及びレポータープローブ、ホタルルシフェラーゼ、及びRenillaルシフェラーゼは、Primer Express (Applied Biosystems)を用いて以下のように設計した。UL77-566F: 5’- ACGATCCCTTTATCCGCTTTC-3’ (配列番号46); UL77-633R: 5’ - GGCATTCTCGAACATGGTGTT-3’ (配列番号47); UL77 -589プローブ: 5’- FAM- ACCGATTTTCGCGGCGAGGTG(配列番号48)-テトラメチルローダミン (TAMRA)-3’, fireflyluc-1536F: 5’- GCGCGGAGGAGTTGTGTTT -3’ (配列番号49); fireflyluc-1607R: 5’- CTGATTTTTCTTGCGTCGAGTTT -3’ (配列番号50); ホタルluc-1556プローブ: 5’- FAM- TGGACGAAGTACCGAAAGGTCTTACCGG (配列番号51)- テトラメチルローダミン(TAMRA)-3’, renilla luc-300F: 5’- CCTCACCGCTTGGTTCGA-3’ (配列番号52); renilla luc-363R: 5’- CCAGTCGTGGCCCACAA-3’ (配列番号53); renilla-319プローブ: 5’ - FAM- CTGCTGAACCTTCCAAAGAAAATCATC(配列番号54)-テトラメチルローダミン(TAMRA)-3’(日本ジーン研究所,仙台)。熱サイクル条件は、最初50℃で2分、95℃で2分、その後、95℃で15秒及び60℃で1分を40サイクル行うことからなる。相対的なUL77, ホタルルシフェラーゼ又はRenillaルシフェラーゼRNAの定量を、文献記載の標準曲線解析法に従って行った(17, 29)。G6PDプライマー及びプローブ(45)を用いるリアルタイムPCRもまた、投入したRNAに対する内部対照として使用した。リアルタイムRT-PCR分析は3連で行った。各定量のために、ウイルスRNAを、感染3日後のdlUL76flagUL77ウイルスで感染したHFFからの閾値サイクル値に標準化した。Renilla (R)対(F)ルシフェラーゼのmRNA比及びG6PD RNAの定量を、親(IRES-ネガチブ)ベクターでトランスフェクションされたHeLa細胞からの閾値サイクル値に標準化された。親(IRES-ネガチブ)ベクターにより作製されたR:F比は、1.0に設定された。
【0071】
in vitro転写
プラスミドの線状化を、pCMVUL76-77構築体についてはXbaIで、又はルシフェラーゼ構築物についてはApaIで消化することによって行った。ApaI消化後、T4 DNAポリメラーゼ(Promega)を用いてin vitro転写前に3’オーバーハングを除去した。プラスミドを転写して、製造者の説明書(Promega)に従いRiboMAX Large Scale RNA Production System-T7を用いて、キャップされたRNAを作製した。1μgの線状化プラスミドDNAを、 7-メチル-GTP:GTP最終比 2.5:1を含む反応混合液中、37℃3時間転写した。RNAをフェノール−クロロホルム抽出により精製し、DNase I (Promega)で処理し、鋳型DNAを除去した。RNAサイズは、1%アガロース−ホルムアルデヒドゲル上でアリコートを泳動することによって測定された。
【0072】
in vitro翻訳
製造者の説明書に従いRabbit Reticulocyte Lysate System (Promega)を用いて翻訳を行った。反応混合液は、1μlの1mMアミノ酸混合物(メチオニン非含有), 2μlの[35S]メチオニン(Amersham Pharmacia), 1μlのRNasinリボヌクレアーゼ阻害剤, 2μgのキャップされたRNAと35μlのウサギ網状赤血球溶解物, 4μgのキャップされた全長RNAから調製され30℃で90分間インキュベートされた。その後、その5μlを取り出し、10μlのドデシル硫酸ナトリウム(SDS)サンプルバッファーに加え、15% SDS-PAGEによって分離した。
【0073】
ルシフェラーゼアッセイ
すべてのトランスフェクションは、製造者の説明書に従いLipofectamine及びPlus試薬(Invitrogen)を用いて48ウエル上で3連で行われた。HeLa細胞を、1μgの各発現プラスミド又は4μgのキャップされたRNAでトランスフェクトし、その後、48時間又は6時間で回収した。そのあと、細胞溶解物を調製し、製造者の説明書に従いDual-Luciferase(登録商標)Reporter Assay System (Promega)にかけた。データは、3回の独立のトランスフェクション実験の平均である。
【0074】
<結果>
UL77の上流領域の欠失
UL76 ORF内にUL77の転写開始部位が存在する可能性を除外するためにUL76遺伝子を欠失させた組換えウイルスを構築した。野生型(wt)から組換えBAC DNAを選択するために薬剤耐性遺伝子を必要とするため、カナマイシン耐性遺伝子(KanR)を含むようにBAC DNAを構築した。また、 KanRが近隣遺伝子の発現に影響する可能性を排除するために、隣接するFRT配列を有する組換えBAC DNAを構築した。FLP仲介組換え法によってKanRを除去し、その後、FRTの34bpのみがUL77 ORFの前部に残った(図1a)。UL76 ORFは単一のBstB I部位を含むので (図1a)、BAC DNAを、制限エンドヌクレアーゼBstB Iで消化した。ウイルスDNA断片を0.7%アガロースゲルでの電気泳動によって分画し、上記「材料と方法」に記載したように、UL75又はUL76のいずれか一方に対するプローブによるサザンブロットハイブリダイゼーションのために固定化された。UL75プローブを用いて、UL76を欠失したとき、より大きいウイルスDNA断片が検出された(図1b,左パネル)。UL76を含有するDNA断片は、組換えウイルスRdlUL76+Fに対してはUL76プローブで検出されなかったが、野生型に対しては検出された(図1b,右パネル)。PCR分析は、組換えBAC DNA中に組換えを明らかに示した(データ示さず)。DNA配列決定により該組換えを確認した(データを示さず)。
【0075】
ウイルスの単離後、細胞をMOI 約1で感染し、感染後6, 24, 48及び72時間の時点でノザンブロットによりUL76, UL77及びUL78転写体についてアッセイした。RNAの20μgのアリコートを、アガロースゲル電気泳動にかけ、28S及び18SのリボソームRNAの臭化エチジウム染色により、等量のRNAが各レーンに載置されたことを確認した(図1c及び1e)。予測したとおり、UL76が組換えウイルスRdlUL76+Fについて欠失されたとき転写体は存在しなかったが、該転写体は野生型では検出された(図1c)。UL77プローブを用いたとき、野生型(wt)又はRdl76+F ではさらなる転写体は検出されなかった(図1d)。UL78プローブを用いたときには、wt及び組換えウイルスRdlUL76+F中にUL78遺伝子のみに対し検出された転写体が検出された(図1e)。これらの結果は、UL77 ORFがRdlUL76+F中のUL76上流領域を欠失していることを示している。
【0076】
UL76 ORFの発現レベルに影響する
真核生物mRNAの非常に大部分がモノシストロン性であり、すなわち、それらは、単一のタンパク質のみをコードする。UL77の上流配列がUL77遺伝子の転写及び翻訳に影響を及ぼすか否かを決定するために、UL77 ORFの上流領域が存在する、又は存在しない、UL77 ORFのN末端に融合させたflagエピトープを含む組換えHCMV BAC DNAを構築した。図2aに示されるように、はじめに、HCMV BAC DNA中に、RpsLneo遺伝子を有するマーカーカセットを挿入し、ストレプトマイシンに対する感受性を増大させた。カナマイシン耐性に基づいて中間BACクローンを単離した。このクローンの完全性をHind IIIによる消化でチェックし、プライマー対としてUL76detectF及びUL76detectR を用いるPCRによって正しい位置でのマーカーカセットの挿入を確認した(データを示さず)。第2ラウンドの相同組換えでは、マーカーカセット全体が、上記の「材料と方法」に記載されるように、UL77 ORFの上流領域が存在する、又は存在しない、UL77のN末端に融合されたflagエピトープで置換された。従来法に従いストレプトマイシン耐性の増大に基づいて組換え構築物を単離した(19, 21, 46)。N末端にflagエピトープが融合された変異領域を、上記の「材料と方法」に記載されたプライマー対UL76detectF及びUL76detectRを用いるPCRによって増幅した(データ示さず)。Hind IIIによる消化によって、変異BACの完全性をチェックした(データ示さず)。
【0077】
細胞を、MOI約1でRflagUL77又はRdlUL76flagUL77に感染させ、感染後表示の時点で回収した。RflagUL77及びRdlUL76flagUL77のUL77転写体の比率は、感染の2日目と3日目で類似していた(図2b)。このことは、上流配列の欠失がUL77の転写に影響しないことを示している。前初期, 初期, 遅延初期及び後期タンパク質の発現レベルは、MOI約1のRflagUL77とRdlUL76flagUL77では類似していた(図2c)。UL77遺伝子産物の予測された分子量に相当する融合タンパク質が、感染後3日及び4日目に両組換えウイルスで検出されたが、その発現レベルは、RflagUL77 と比べてRdlUL76flagUL77についてより高かった(図2c,レーン5〜8)。たとえRdlUL76flagUL77が、重要なUL77遺伝子産物の発現を高めたとしても、MOI 1でのRflagUL77対RdlUL76flagUL77の増殖の違いは検出されなかった(データを示さず)。UL76遺伝子産物の欠失は、Wangら(43, 44)により提案されたように、ウイルス複製に影響を与えた可能性がある。これらのデータは、UL77の上流の配列がUL77タンパク質発現に対し影響をもつことを示した。UL77の上流配列の欠失により、UL77 ORFガウイルスmRNA中で第1シストロンになったとき、UL77 ORFの発現が増大した。
【0078】
UL77の上流配列の欠失がUL77 mRNAの安定性を高めることは可能である。隠れたスプライス部位又はプロモーターが欠失することも可能である。これらの可能性を除外するために、UL76 ORFのATG開始コドンの下流にTAG stopコドンが挿入された組換えウイルスを構築した。図3aに示すように、RpsLneo遺伝子をもつマーカーカセットをRflagUL77に挿入してRflagUL77Kan+Stを作製した。第2ラウンドの相同組換えのあと、マーカーカセット全体を、UL76 ORFのstopコドンを含む変異オリゴで置換して RUL76stopflagUL77を得た、或いは、復帰突然変異オリゴで置換して、上記の「材料と方法」に記載されたRUL76revertantflagUL77を作製した。変異型BACの完全性は、Hind IIIでの消化によってチェックし(データ示さず)、正しい組換えをPCR産物の配列決定によって確認した。細胞を、MOI約1でRUL76stopflagUL77又はRUL76revertantflagUL77のいずれか一方で感染し、 感染後表示の時点で回収した。前初期(pIE72とpIE86),初期(p52), 遅延初期 (pp65),及び後期(pp28)タンパク質の発現レベルは、RUL76stopflagUL77及びRUL76revertantflagUL77と類似していた(図3b)。UL77 発現レベルは、RUL76revertantflagUL77と比べて、RUL76stopflagUL77についてより高かった(図3b, レーン5〜8)。これらの結果は、UL76配列がUL77 ORF発現に影響することを示した。
【0079】
UL76及びUL77 ORFがin vitroでバイシストロニックなキャップされたRNAから翻訳された
HCMV MIEプロモーターによって駆動されるUL76-77 mRNAが両ウイルスタンパク質を発現することができるかどうかを決定するために、UL76 ORFのN末端に融合されたflagエピトープとUL77 ORFのC末端に融合されたHAエピトープを有するプラスミドを構築した。さらにまた、UL76 ORFのN末端又はUL77 ORFのC末端にフレームシフト変異が挿入されたプラスミドを構築した(図4a)。上記発現プラスミドによるHeLa細胞のトランスフェクション後48時間目に、該細胞を回収した。SDS-ポリアクリルアミドゲル電気泳動により等量のタンパク質を分画し、上記の「材料と方法」に記載されたflag又はHAエピトープに対するモノクローナル抗体を用いたウエスタンブロッティングによって分析した。図4bのレーン2に示されるように、UL76及びUL77融合タンパク質の両方をpCMVflagUL76-77HAでトランスフェクトしたHeLa細胞中で発現させた。UL77融合タンパク質の発現レベルは、UL76 ORFにフレームシフトが挿入されたとき約4倍増加し(図4b、レーン1と2の比較)、UL76配列がUL77 ORFの発現に影響することを確認した。
【0080】
UL77 ORFがバイシストロニックなキャップされたRNAからin vitroで翻訳されうるかどうかを決定するために、上記の「材料と方法」に記載されたようにin vitro 転写によってプラスミドの各々からRNAを調製した。該プラスミドのT7ポリメラーゼ駆動転写によって単一のバイシストロニックなRNAが生じた(図4c)。バイシストロニックなUL76-77 RNAの翻訳によって37 kDaのflag UL76と72 kDaのUL77 HAが検出された(図4d,レーン2)。これらの結果は、UL77 ORFがin vitroでバイシストロニックなキャップされたRNAから翻訳されうることを示した。バイシストロニックなmRNAからのUL77の翻訳は、リボソームの連続的スキャニングか又はUL77の上流のIRESに起因した。
【0081】
UL76 ORF配列は、インターナルリボソームエントリーサイトを含んでいる。通常、リボソームサブユニットは、RNAの5'-末端 m7GpppN cap構造のところでスキャニングを開始したあと、3'方向に直線的に移動するので、uORFの長さは、スキャニングの再開始のための重要なパラメーターである(16)。実際、哺乳類細胞では、スキャニングの再開始は、uORFが短い場合のみ起こる(24, 28)。もしUL77 AUGでのリボソームの開始がUL76中に埋め込まれたIRESによって保証される場合には、再開始は完全な上流シストロンと独立に生じうるだろう。UL76配列がIRESを有するかどうかを決定するために、上記の「材料と方法」に記載されたpCMVFlucRlucを構築した(図5a)。このプラスミドは、第1シストロンとしてのホタルルシフェラーゼ、その後にイントラシストロン配列(ICS)、及び第2シストロンとしてのRenillaルシフェラーゼ遺伝子を有するバイシストロン性mRNAをコードする。ICSに挿入されたIRESは、Renillaルシフェラーゼ遺伝子の翻訳を容易にするだろう。UL76ヌクレオチド(nt)1〜580又はその誘導体を、ICSの部位でpCMVFlucRlucに挿入し、HeLa細胞をトランスフェクトした。そのあと、Renilla (R)ルシフェラーゼ対ホタルルシフェラーゼ(F)の活性比を、上記の「材料と方法」に記載されたように、トランスフェクション後48時間目に細胞溶解物中で測定した。図5bに示されるように、UL76 (1-580)は、Renilla対ホタルルシフェラーゼ活性の比を約4倍増加させた。アンチセンス配向をもつDNA断片(r1-580)は作用しなかった(図5b)。ヌクレオチド1〜116は有意な活性をもたなかったが、ヌクレオチド1〜190は有意な作用を有していた。190〜58のヌクレオチドもまた、有意な作用を有していた(図5b)。これらのデータは、UL76のアミノ末端側半分とカルボキシ末端側半分の両方におけるウイルス配列エレメントが下流シストロンの発現を容易にすることを示した。
【0082】
UL76 (1-580)中の、Renilla ルシフェラーゼ活性に対し作用しうる、隠れたスプライス部位又はプロモーターの可能性を除外するために、上記の「材料と方法」に記載されたようにT7 RNAポリメラーゼを用いてin vitroで構築物を転写し、得られたRNAを、DNaseでDNA鋳型を分解したあと、1%アガロース-ホルムアルデヒドゲルに載置した。プラスミドのT7ポリメラーゼ駆動転写によって単一のバイシストロン性RNAを作製した(データを示さず)。その後、HeLa細胞中にこれらの精製RNAをトランスフェクションすることによってR:F比を調べた。トランスフェクション後6時間目に細胞抽出物をルシフェラーゼ活性についてアッセイし、R:F比を上記と同様に計算した。図6に示すように、キャップされたRNAをもつUL76配列エレメント(1〜580)は、Renilla対ホタルルシフェラーゼ活性の比を約4倍増加させた。アンチセンス配向をもつRNA断片 (r1-580)は効果を有していなかった(図6)。UL76 (1-580)によって仲介されるR:F比はポリオウイルスIRESによって仲介されるものの40.6%であった(図6)。UL76のDNA断片をもつRNA転写体もまた、R:F比を増加させた。RNAのR:F比は、DNAのものと比べて劇的に高かった(図5参照)。この作用の理由は不明であるが、RNAがin vitroで転写されるときには、不十分なキャップ(cap)形成のために、キャップ依存性スキャニングによる第1シストロンの翻訳効率がin vivoデータと比べて低くなったと推定される。2つのアッセイでは、個々の断片はすべて(互いに対して)同等に挙動しないが、全体の傾向は類似していた。in vitroでの二次構造は、in vivoで作られるものと異なるだろうし、IRESへのリボソームの接近能に影響を与えるかもしれない。これらをあわせると、UL76 ORF配列は、下流のUL77 ORFの発現を容易にするインターナルリボソームエントリーサイトを含む。
【0083】
<考察>
IRESは、ピコルナウイルス(23, 37)、ペストウイルス(38)、C型肝炎ウイルス(41)、カポジ肉腫関連ヘルペスウイルス(3, 14, 27)、単純ヘルペスウイルス-1(13)及びサルウイルス40(48)のウイルスmRNA、並びにいくつかの細胞mRNA(35, 40, 42)、中に同定されている。IRES間ではヌクレオチド配列の共通性は小さい。ウイルスIRESは、RNA二次構造の広い領域を有している。RNA二次構造は保存されていないが、リボソームとの結合にとって重要である。UL76配列のRNA二次構造は決定されているが、コンピュータプログラムRNA mfold (http://frontend.bioinfo.rpi.edu/applications/mfold/cgi-bin/rna-form1.cgi)によって、このRNA配列が安定なステムループ構造を含むことが予測された。UL76 (1-580)によって仲介されるIRES活性は、ポリオウイルスIRESは、ポリオウイルスIRESによって仲介された活性の約40%であった。
【0084】
UL76 (RUL76stopflagUL77)中に挿入されたstopコドンをもつ組換えウイルスは、復帰突然変異ウイルス(RUL76revertantflagUL77)と同様に(但し、UL77タンパク質を除く)、前初期、初期及び後期タンパク質を発現した。UL77 ORFの発現レベルは、UL76 ORF中にstopコドンが挿入された組換えウイルスで増加したが、このことは、UL76配列自体がHCMV感染細胞中でUL77 ORFの発現に影響を与えたことを示している。in vitro翻訳アッセイによって、UL77 ORFが再開始によって翻訳されうることが示された。ほとんどの状況において、uORFの存在が下流のAUGで開始を阻害し、uORFの単一の機能が下流のORFの発現をダウンレギュレートすることであるように思われることが多い(11)。上流のUL76 ORFが欠失すると、UL77 ORFの発現が増加する。
【0085】
これまで調べられた多くの細胞IRESは、mRNAの5’UTR内に位置している。これに対して、DNAウイルス内で見出されたすべてのIRESは、上流シストロンのコード配列中に存在する(3, 13, 14, 27, 48)。本発明者らは、UL76 ORF中にIRESを同定した。HCMVは、宿主細胞内の翻訳機構に完全に依存して、複製生産のためのウイルスタンパク質を合成する。5’cap構造からの開始及びリボソームスキャニングが細胞ストレスによって低下する状況下で、UL77タンパク質が翻訳されうる。UL76中のIRESエレメントは、HCMV感染において重要な機能を有しているが、これはUL77ウイルスタンパク質がウイルスDNAの切断及びパッケージングのために必要であるからである。UL76 ORFのタンパク質としての機能は、HCMV感染細胞では不明である。RflagUL77とRdlUL76flagUL77との増殖の差は検出されなかった。Wangら(43, 44)は、UL76がウイルス遺伝子発現を調節することを示した。本発明者ら、UL76もまたcis作用性調節シグナルとして働き、かつ、IRESとして作用し、UL77の発現を容易にすることを提案する。これらのデータは、なぜUL76がウイルス複製を増大するものとして分類されるか(7, 47)という理由を一部説明している。
【0086】
[実施例2]
HFF細胞でのウイルス複製
ウイルス遺伝子欠失変異体の構築
122アミノ酸(図7a)を有するUL30は、ウイルス遺伝子発現全体にほとんど又はまったく影響を及ぼさない可能性がある。本発明者らは、組換えHCMV BAC DNAを構築したが、このDNAは、UL76中のUL77の上流の領域がUL30によって置換されたものである。UL76配列はBstB I部位を含む(図7a)ので、BAC組換えを証明するために、BAC DNAを制限酵素BstB Iで消化した。0.7%アガロースゲルでの電気泳動によってウイルスDNA断片を分画し、「材料と方法」に記載したUL76又はUL77のいずれかに対するプローブによるサザンブロットハイブリダイゼーションのために固定された。UL76を含むDNA断片は、組換えウイルスRdlUL76+FについてはUL76プローブで検出されなかったが、野生型については検出された(図7b、左パネル)。UL77プローブを用いて、UL77の上流領域がUL30で置換されたとき、より大きなウイルスDNA断片が検出された(図7b、右パネル)。PCR分析によって、組換えBAC DNA中の組換えが示された。DNA配列分析によって、前記組換えを確認した。flagエピトープを有するUL77タンパク質が、非常に低いMOIでRUL30flagUL77に感染させた細胞中で発現されるかを調べるために、そしてCPEがほとんどすべての細胞中に現れたとき、細胞を回収し、ウエスタンブロット分析を行って、「材料と方法」に記載したflagエピトープに対する抗体を用いてflagエピトープを有するUL76タンパク質を検出した。図7cに示されるように、RUL30flagUL77に感染させた細胞中にUL77タンパク質を検出することができた。
【0087】
UL30で置換された単離された組換えウイルスについて、複数回のウイルス複製後に高力価のウイルスストックを得ることは難しかった。図8aに示されるように、100% CPEの5〜7日後に単離され及び連続希釈された細胞外液含有ウイルスに感染させた細胞中のウイルスタンパク質の発現を比較したとき、RUL30flagUL77を有する初期タンパク質とIEの発現はRUL76revertantflagUL77の102倍希釈を用いたときの発現とほとんど同様であった。
【0088】
本発明者らは、さらなる分析のために-80℃に保存されていなかった細胞外液含有ウイルスを使用した。-80℃に保存されていなかったウイルス力価を、野生型(wt)のウイルス力価と比較することによって決定した(親ウイルス、図3参照)。ウイルス力価は、「材料と方法」に記載されたGFPフォーカスに基づいた標準プラークアッセイによって測定された。
【0089】
細胞を、約3のMOIでRUL30flagUL77又はRUL76revertantflagUL77のいずれかで感染し、感染後、示された時間に回収した。
【0090】
ウイルスタンパク質の発現を比較した。感染1、2及び3日後(1、2及び3d.p.i.)の、RUL30flagUL77及びRUL76revertantflagUL77を有する、前初期タンパク質(pIE72及びpIE86)、初期タンパク質(p52)及び後期タンパク質(pp28)の発現レベルに有意な差異はなかった(図8c)。RUL30flagUL77を有する初期タンパク質(p52)及び後期タンパク質(pp28)の発現レベルは、4d.p.i.のRUL76revertantflagUL77を有するものより少し低めであった。これに対して、本発明者らは、UL77タンパク質の発現の有意な差を検出することができきた。RUL30flagUL77を有するUL77タンパク質の発現は劇的に低減し、4d.p.i.でさえウエスタンブロット分析で検出することは難しかった(図8c)。このあと、本発明者らは、新しい多数の組換えウイルスを作製し、上に記載したようにウイルス力価を測定し、リアルタイムRT-PCRによってウイルスRNAを分析した。RUL30flagUL77についてUL77転写体の比率は、2d.p.i.でのRUL76revertantflagUL77についてのものより少し高めであったが、その差は2倍未満であった。RUL30flagUL77の転写体の比率は、3d.p.i.でのRUL76revertantflagUL77のものと類似していた(図8d)。RUL30flagUL77を有する各ウイルス転写体の総量は、RUL76revertantflagUL77を有するものより少し高めであったが、検査するIE、初期及び後期転写体のレベルに大きな変化はなかった(図8d)。
【産業上の利用可能性】
【0091】
本発明により、HCMVを予防又は治療するためのワクチンが提供される。
【0092】
参考文献
1. Beisser, P. S., G. Grauls, C. A. Bruggeman, and C. Vink. 1999. Deletion of the R78 G protein-coupled receptor gene from rat cytomegalovirus results in an attenuated, syncytium-inducing mutant strain. J. Virol. 73:7218-7230.
2. Biegalke, B. J., and A. P. Geballe. 1990. Translational inhibition by cytomegalovirus transcript leaders. Virology 177:657-667.
3. Bieleski, L., and S. J. Talbot. 2001. Kaposi's sarcoma-associated herpesvirus vCyclin open reading frame contains an internal ribosome entry site. J. Virol. 75:1864-1869.
4. Cao, J., and A. P. Geballe. 1996. Coding sequence-dependent ribosomal arrest at termination of translation. Mol. Cell Biol. 16:603-608.
5. Cao, J., and A. P. Geballe. 1995. Translational inhibition by a human cytomegalovirus upstream open reading frame despite inefficient utilization of its AUG codon. J. Virol. 69:1030-1036.
6. Degnin, C. R., M. R. Schleiss, J. Cao, and A. P. Geballe. 1993. Translational inhibition mediated by a short upstream open reading frame in the human cytomegalovirus gpUL4 (gp48) transcript. J. Virol. 67:5514-5521.
7. Dunn, W., C. Chou, H. Li, R. Hai, D. Patterson, V. Stolc, H. Zhu, and F. Liu. 2003. Functional profiling of a human cytomegalovirus genome. Proc. Natl. Acad. Sci. U. S. A. 100:14223-14228.
8. Ellis, H. M., D. Yu, T. DiTizio, and D. L. Court. 2001. High efficiency mutagenesis, repair, and engineering of chromosomal DNA using single-stranded oligonucleotides. Proc. Natl. Acad. Sci. U. S. A. 98:6742-6746.
9. Geballe, A. P., F. S. Leach, and E. S. Mocarski. 1986. Regulation of cytomegalovirus late gene expression: gamma genes are controlled by posttranscriptional events. J. Virol. 57:864-874.
10. Geballe, A. P., and E. S. Mocarski. 1988. Translational control of cytomegalovirus gene expression is mediated by upstream AUG codons. J. Virol. 62:3334-3340.
11. Geballe, A. P., and D. R. Morris. 1994. Initiation codons within 5'-leaders of mRNAs as regulators of translation. Trends Biochem. Sci. 19:159-164.
12. Graham, F. L., and A. J. van der Eb. 1973. A new technique for the assay of infectivity of adenovirus 5 DNA. Virology 52:456-467.
13. Griffiths, A., and D. M. Coen. 2005. An unusual internal ribosome entry site in the herpes simplex virus thymidine kinase gene. Proc. Natl. Acad. Sc.i U. S. A. 102:9667-9672.
14. Grundhoff, A., and D. Ganem. 2001. Mechanisms governing expression of the v-FLIP gene of Kaposi's sarcoma-associated herpesvirus. J. Virol. 75:1857-1863.
15. Hahn, G., D. Rose, M. Wagner, S. Rhiel, and M. A. McVoy. 2003. Cloning of the genomes of human cytomegalovirus strains Toledo, TownevarRIT3, and Towne long as BACs and site-directed mutagenesis using a PCR-based technique. Virology 307:164-177.
16. Hwang, W. L., and T. S. Su. 1998. Translational regulation of hepatitis B virus polymerase gene by termination-reinitiation of an upstream minicistron in a length-dependent manner. J. Gen. Virol. 79 ( Pt 9):2181-2189.
17. Isomura, H., and M. F. Stinski. 2003. The human cytomegalovirus major immediate-early enhancer determines the efficiency of immediate-early gene transcription and viral replication in permissive cells at low multiplicity of infection. J. Virol. 77:3602-3614.
18. Isomura, H., M. F. Stinski, A. Kudoh, T. Daikoku, N. Shirata, and T. Tsurumi. 2005. Two Sp1/Sp3 binding sites in the major immediate-early proximal enhancer of human cytomegalovirus have a significant role in viral replication. J. Virol. 79:9597-9607.
19. Isomura, H., M. F. Stinski, A. Kudoh, T. Murata, S. Nakayama, Y. Sato, S. Iwahori, and T. Tsurumi. 2008. Noncanonical TATA sequence in the UL44 late promoter of human cytomegalovirus is required for the accumulation of late viral transcripts. J. Virol. 82:1638-1646.
20. Isomura, H., M. F. Stinski, A. Kudoh, S. Nakayama, S. Iwahori, Y. Sato, and T. Tsurumi. 2007. The late promoter of the human cytomegalovirus viral DNA polymerase processivity factor has an impact on delayed early and late viral gene products but not on viral DNA synthesis. J. Virol. 81:6197-6206.
21. Isomura, H., M. F. Stinski, A. Kudoh, S. Nakayama, T. Murata, Y. Sato, S. Iwahori, and T. Tsurumi. 2008. A cis element between the TATA Box and the transcription start site of the major immediate-early promoter of human cytomegalovirus determines efficiency of viral replication. J. Virol. 82:849-858.
22. Isomura, H., T. Tsurumi, and M. F. Stinski. 2004. Role of the proximal enhancer of the major immediate-early promoter in human cytomegalovirus replication. J. Virol. 78:12788-12799.
23. Jang, S. K., H. G. Krausslich, M. J. Nicklin, G. M. Duke, A. C. Palmenberg, and E. Wimmer. 1988. A segment of the 5' nontranslated region of encephalomyocarditis virus RNA directs internal entry of ribosomes during in vitro translation. J. Virol. 62:2636-2643.
24. Kozak, M. 1995. Adherence to the first-AUG rule when a second AUG codon follows closely upon the first. Proc. Natl. Acad. Sci. U. S. A. 92:2662-2666.
25. Kozak, M. 2001. Constraints on reinitiation of translation in mammals. Nucleic Acids Res. 29:5226-5232.
26. Kozak, M. 1987. Effects of intercistronic length on the efficiency of reinitiation by eucaryotic ribosomes. Mol. Cell Biol. 7:3438-3445.
27. Low, W., M. Harries, H. Ye, M. Q. Du, C. Boshoff, and M. Collins. 2001. Internal ribosome entry site regulates translation of Kaposi's sarcoma-associated herpesvirus FLICE inhibitory protein. J. Virol. 75:2938-2945.
28. Matsuda, D., and T. W. Dreher. 2006. Close spacing of AUG initiation codons confers dicistronic character on a eukaryotic mRNA. RNA 12:1338-1349.
29. Meier, J. L., M. J. Keller, and J. J. McCoy. 2002. Requirement of multiple cis-acting elements in the human cytomegalovirus major immediate-early distal enhancer for viral gene expression and replication. J. Virol. 76:313-326.
30. Meier, J. L., and J. A. Pruessner. 2000. The human cytomegalovirus major immediate-early distal enhancer region is required for efficient viral replication and immediate-early gene expression. J. Virol. 74:1602-1613.
31. Meier, J. L., and M. F. Stinski. 1997. Effect of a modulator deletion on transcription of the human cytomegalovirus major immediate-early genes in infected undifferentiated and differentiated cells. J. Virol. 71:1246-1255.
32. Michel, D., I. Milotic, M. Wagner, B. Vaida, J. Holl, R. Ansorge, and T. Mertens. 2005. The human cytomegalovirus UL78 gene is highly conserved among clinical isolates, but is dispensable for replication in fibroblasts and a renal artery organ-culture system. J. Gen. Virol. 86:297-306.
33. Murphy, E., I. Rigoutsos, T. Shibuya, and T. E. Shenk. 2003. Reevaluation of human cytomegalovirus coding potential. Proc. Natl. Acad. Sci. U. S. A. 100:13585-13590.
34. Murphy, E., D. Yu, J. Grimwood, J. Schmutz, M. Dickson, M. A. Jarvis, G. Hahn, J. A. Nelson, R. M. Myers, and T. E. Shenk. 2003. Coding potential of laboratory and clinical strains of human cytomegalovirus. Proc. Natl. Acad. Sci. U. S. A. 100:14976-14981.
35. Nanbru, C., I. Lafon, S. Audigier, M. C. Gensac, S. Vagner, G. Huez, and A. C. Prats. 1997. Alternative translation of the proto-oncogene c-myc by an internal ribosome entry site. J. Biol. Chem. 272:32061-32066.
36. Pari, G. S., and D. G. Anders. 1993. Eleven loci encoding trans-acting factors are required for transient complementation of human cytomegalovirus oriLyt-dependent DNA replication. J. Virol. 67:6979-6988.
37. Pelletier, J., and N. Sonenberg. 1988. Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA. Nature 334:320-325.
38. Rijnbrand, R., T. van der Straaten, P. A. van Rijn, W. J. Spaan, and P. J. Bredenbeek. 1997. Internal entry of ribosomes is directed by the 5' noncoding region of classical swine fever virus and is dependent on the presence of an RNA pseudoknot upstream of the initiation codon. J. Virol. 71:451-457.
39. Stinski, M. F. 1978. Sequence of protein synthesis in cells infected by human cytomegalovirus: early and late virus-induced polypeptides. J. Virol. 26:686-701.
40. Stoneley, M., F. E. Paulin, J. P. Le Quesne, S. A. Chappell, and A. E. Willis. 1998. C-Myc 5' untranslated region contains an internal ribosome entry segment. Oncogene 16:423-428.
41. Tsukiyama-Kohara, K., N. Iizuka, M. Kohara, and A. Nomoto. 1992. Internal ribosome entry site within hepatitis C virus RNA. J. Virol. 66:1476-1483.
42. Vagner, S., M. C. Gensac, A. Maret, F. Bayard, F. Amalric, H. Prats, and A. C. Prats. 1995. Alternative translation of human fibroblast growth factor 2 mRNA occurs by internal entry of ribosomes. Mol. Cell Biol. 15:35-44.
43. Wang, S. K., C. Y. Duh, and T. T. Chang. 2000. Cloning and identification of regulatory gene UL76 of human cytomegalovirus. J. Gen. Virol. 81:2407-2416.
44. Wang, S. K., C. Y. Duh, and C. W. Wu. 2004. Human cytomegalovirus UL76 encodes a novel virion-associated protein that is able to inhibit viral replication. J. Virol. 78:9750-9762.
45. White, E. A., C. L. Clark, V. Sanchez, and D. H. Spector. 2004. Small internal deletions in the human cytomegalovirus IE2 gene result in nonviable recombinant viruses with differential defects in viral gene expression. J. Virol. 78:1817-1830.
46. White, E. A., C. J. Del Rosario, R. L. Sanders, and D. H. Spector. 2007. The IE2 60-kilodalton and 40-kilodalton proteins are dispensable for human cytomegalovirus replication but are required for efficient delayed early and late gene expression and production of infectious virus. J. Virol. 81:2573-2583.
47. Yu, D., M. C. Silva, and T. Shenk. 2003. Functional map of human cytomegalovirus AD169 defined by global mutational analysis. Proc. Natl. Acad. Sci. U. S. A. 100:12396-12401.
48. Yu, Y., and J. C. Alwine. 2006. 19S late mRNAs of simian virus 40 have an internal ribosome entry site upstream of the virion structural protein 3 coding sequence. J. Virol. 80:6553-6558.
【配列表フリーテキスト】
【0093】
配列番号9〜47:プライマー
配列番号48〜54:プローブ
配列番号55:34bp最小FRT部位
【特許請求の範囲】
【請求項1】
ヒトサイトメガロウイルス(HCMV)のUL76遺伝子ORF中のIRES配列が不活性化されたことを特徴とする弱毒HCMVウイルス。
【請求項2】
前記IRES配列が、IRES以外の別の核酸配列によって置換されている、請求項1に記載の弱毒HCMVウイルス。
【請求項3】
前記別の核酸配列のサイズが前記IRES配列のサイズと完全に同じであるか又はほほ同じである、請求項2に記載の弱毒HCMVウイルス。
【請求項4】
前記別の核酸配列が、同じ又は異なるHCMV株のUL30由来である、請求項2又は3に記載の弱毒HCMVウイルス。
【請求項5】
前記HCMVの複製が抑制されたものである、請求項1〜4のいずれか1項に記載の弱毒HCMVウイルス。
【請求項6】
請求項1〜5のいずれか1項に記載の弱毒HCMVウイルスを有効成分として含む、HCMV感染の予防又は治療用ワクチン。
【請求項7】
請求項1〜5のいずれか1項に記載の弱毒HCMVウイルスを非ヒト動物に免疫して抗体を作製し、該抗体を回収することを含む、抗HCMV抗体の作製方法。
【請求項8】
前記抗HCMV抗体がヒト抗体である、請求項7に記載の方法。
【請求項1】
ヒトサイトメガロウイルス(HCMV)のUL76遺伝子ORF中のIRES配列が不活性化されたことを特徴とする弱毒HCMVウイルス。
【請求項2】
前記IRES配列が、IRES以外の別の核酸配列によって置換されている、請求項1に記載の弱毒HCMVウイルス。
【請求項3】
前記別の核酸配列のサイズが前記IRES配列のサイズと完全に同じであるか又はほほ同じである、請求項2に記載の弱毒HCMVウイルス。
【請求項4】
前記別の核酸配列が、同じ又は異なるHCMV株のUL30由来である、請求項2又は3に記載の弱毒HCMVウイルス。
【請求項5】
前記HCMVの複製が抑制されたものである、請求項1〜4のいずれか1項に記載の弱毒HCMVウイルス。
【請求項6】
請求項1〜5のいずれか1項に記載の弱毒HCMVウイルスを有効成分として含む、HCMV感染の予防又は治療用ワクチン。
【請求項7】
請求項1〜5のいずれか1項に記載の弱毒HCMVウイルスを非ヒト動物に免疫して抗体を作製し、該抗体を回収することを含む、抗HCMV抗体の作製方法。
【請求項8】
前記抗HCMV抗体がヒト抗体である、請求項7に記載の方法。
【図1−1】


【図1−2】


【図2】


【図3】


【図4−1】


【図4−2】


【図5】


【図6】


【図7】


【図8】




【図1−2】


【図2】


【図3】


【図4−1】


【図4−2】


【図5】


【図6】


【図7】


【図8】


【公開番号】特開2011−177071(P2011−177071A)
【公開日】平成23年9月15日(2011.9.15)
【国際特許分類】
【出願番号】特願2010−42936(P2010−42936)
【出願日】平成22年2月26日(2010.2.26)
【新規性喪失の例外の表示】特許法第30条第1項適用申請有り ▲1▼刊行物名 :第68回 日本癌学会学術総会記事 発行日 :平成21年8月31日 発行者 :日本癌学会 講演番号 :P−0255 公開者 :磯村 寛樹、鶴見 達也 ▲2▼刊行物名 :第57回 日本ウイルス学会学術集会 プログラム・抄録集 発行日 :平成21年10月1日 発行者 :第57回 日本ウイルス学会学術集会 会長 宮村 達男 講演番号 :1P007 公開者 :磯村 寛樹、鶴見 達也
【出願人】(304031427)愛知県 (36)
【Fターム(参考)】
【公開日】平成23年9月15日(2011.9.15)
【国際特許分類】
【出願日】平成22年2月26日(2010.2.26)
【新規性喪失の例外の表示】特許法第30条第1項適用申請有り ▲1▼刊行物名 :第68回 日本癌学会学術総会記事 発行日 :平成21年8月31日 発行者 :日本癌学会 講演番号 :P−0255 公開者 :磯村 寛樹、鶴見 達也 ▲2▼刊行物名 :第57回 日本ウイルス学会学術集会 プログラム・抄録集 発行日 :平成21年10月1日 発行者 :第57回 日本ウイルス学会学術集会 会長 宮村 達男 講演番号 :1P007 公開者 :磯村 寛樹、鶴見 達也
【出願人】(304031427)愛知県 (36)
【Fターム(参考)】
[ Back to top ]

