国際特許分類[C12Q1/34]の内容
化学;冶金 (1,075,549) | 生化学;ビール;酒精;ぶどう酒;酢;微生物学;酵素学;突然変異または遺伝子工学 (115,607) | 酵素または微生物を含む測定または試験方法そのための組成物または試験紙;その組成物を調製する方法;微生物学的または酵素学的方法における状態応答制御 (20,915) | 酵素または微生物を含む測定または試験方法;そのための組成物;そのような組成物の製造方法 (20,907) | 加水分解酵素を含むもの (1,444)
国際特許分類[C12Q1/34]の下位に属する分類
ペプチダーゼまたはプロティナーゼを含むもの (566)
アミラーゼを含むもの (37)
ホスファターゼを含むもの (215)
エステラーゼを含むもの (221)
国際特許分類[C12Q1/34]に分類される特許
351 - 360 / 405
IP3タンパク質結合測定法
結合または連結基を介して2-オキシの位置で検出可能なラベルに接続したIP3の接合体と、IP3受容体の細胞外断片の切断部分を試薬として採用した、試料中のIP3を測定するためのタンパク質結合測定法が提供される。試薬は試料と結合し、検出可能なラベルによってIP3の量を測定する。β-ガラクトシダーゼの酵素ドナー断片または蛍光剤との接合体が特に述べられる。 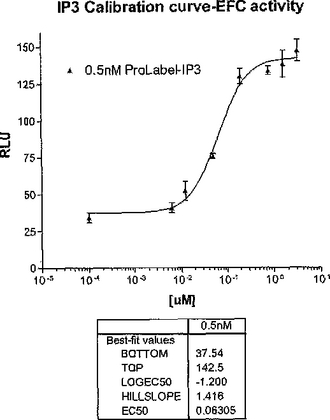 (もっと読む)
(もっと読む)
グリコサミノグリカン分解酵素の活性測定方法
【課題】 グリコサミノグリカン分解酵素の活性を簡便、迅速、安価かつ高感度に、再現性・定量性良く測定する方法を提供すること。
【解決手段】(A)「蛍光物質が結合したグリコサミノグリカン」と検体を接触させ、当該グリコサミノグリカンに検体中のグリコサミノグリカン分解酵素を作用させるステップ、(B)前記作用後のグリコサミノグリカンを蛍光偏光分析法によって検出するステップ、及び(B)で得られた検出結果と、検体中のグリコサミノグリカン分解酵素の活性とを関連づけるステップを少なくとも含む、検体中のグリコサミノグリカン分解酵素の活性測定方法。「蛍光物質が結合したグリコサミノグリカン」を少なくとも含有するグリコサミノグリカン分解酵素の活性測定剤。フルオレセイン−5−チオセミカルバジドとヒアルロン酸とが共有結合していることを特徴とする、ヒアルロン酸誘導体又はその塩。
(もっと読む)
高感受性エンドヌクレアーゼを生産する方法、新規なエンドヌクレアーゼ調製物、及びそれらの使用
本発明は、高感受性を有する組換えエンドヌクレアーゼを生産する方法、当該方法により得られるエンドヌクレアーゼ調製物、及び特にミスマッチの検出のためのそれらの使用に関する。
【配列表】
SEQUENCE LISTING
<110> GENOPLANTE-VALOR
INSTITUT NATIONAL DE LA RECHERCHE AGRONOMIQUE
BENDAHMANE, Abdelhafid
STURBOIS, Benedicte
TRIQUES, Karine
CABOCHE, Michel
<120> METHOD FOR PRODUCING HIGHLY SENSITIVE ENDONUCLEASES, NOVEL
PREPARATIONS OF ENDONUCLEASES AND USES THEREOF.
<130> MJP/bv1516-19
<150> PCT/EP2004/009159
<151> 2004-07-30
<150> PCT/EP2004/009166
<151> 2004-07-30
<160> 25
<170> PatentIn version 3.3
<210> 1
<211> 2640
<212> DNA
<213> Arabidopsis thaliana
<220>
<221> misc#feature
<223> ENDO 1 Gene At1g11190
<400> 1
aaattcgatg aggttgttat agacaagaga agacattttt atacaaaaga gtttatcatt 60
atataagttt caaactttga agatatggca tcggctttta gatcatccac gaggttgatt 120
cttgtattag gtatactgat tttgtgttcg gtttcttctg tccgaagctg gagcaaagaa 180
ggtcatattc ttacttgtag aattgctcag gtaattaagt taatgatcta ttgtttgaag 240
caactatttt ggttattctt gtcttatata tgtattagtg agatatacct acaaattttt 300
aattaggatt gacttttaaa ttgctatacg ttaccatgcc taacatctca tgtagatgat 360
catgaataca aacatgtcta atggcatatc aaattccaag tttttttggt agagatctga 420
gtcatttgac cgttataaga ttcataacaa aagttcgtat gtgtgtgttt ttgtggtgtg 480
accagaatct tttagaagcc ggaccagcac atgtagtaga gaatctgtta ccggattacg 540
tgaaaggaga tttatcagca ttgtgtgtgt ggcctgacca gatccgacat tggtacaagt 600
atcgttggac cagccatctc cattacatcg acactcccga ccaagcctgc tcttacgaat 660
actctagtaa gtcacaaccg agacattttc agataacctt aatccgtttt ctaattatct 720
tgaaccggag ttaaccaaaa aatcaattac aaataccaaa ccggattaaa aacaggggat 780
tgtcatgatc aacatggatt gaaggatatg tgtgtggatg gagcaatcca gaatttcacg 840
tctcagcttc agcattacgg tgaaggaaca tctgatcgta gatgtatgtc atcattttca 900
tttatttcat ataatgatga tatccaaagt gtaactgcgt attttgtatt ttgatgcata 960
acttaagttt ttaaaattat aatatatcct tgttcaatca catagataac atgaccgaag 1020
cccttttgtt cttgtctcat ttcatgggag atattcatca ggtttattac tcatcatcga 1080
ttcatttcac acctccacac atatagctct atttccatgt taaatattta attaacatgg 1140
tttttttttt tttccttaaa aagccgatgc atgtgggatt cacaagtgat gaaggaggaa 1200
acacgataga tttacgttgg tacaaacaca aatccaatct acatcatgta agcttcttct 1260
tttgtctctt tcaactttaa atttcatcat gaaaacaaaa aaaaaattaa cgaaggaaac 1320
aaaatatgta ggtatgggat agagagatca ttctcacggc tctaaaagaa aactacgaca 1380
agaacttgga tcttctccaa gaggatcttg agaagaacat caccaatgta atagacacta 1440
atttattcat attttactat aattttaaga atctttataa tggttatcat atattaggga 1500
ttatggcacg acgatctatc ttcgtggaca gaatgcaacg atcttatcgc ttgtccacac 1560
aagtaagttt taaattactt ggtttaagat tggcttgacg ctcgtttgaa gctagctaca 1620
aattttgata ctttttctgg tccaaaaatc ttacaaagat actgaaaata aaataatagg 1680
ttttaaactt ttaatttatt tggagttgga taggattaag tttcactaac ttccaattca 1740
aagtcaatta atagtagttt accatgatta gtgggttgac taatgtacca tatatattac 1800
cttatatcac atcttatttc cgatgtgaga tttcttatga aacataatta gactcgaacc 1860
ttttgtgttt cgatatatgt agtgtattca tgatcagaat cttattaagt ttacaactga 1920
aaactaaaat attaacatca taattataga ttcttaagta ggttttgttt gggtggagaa 1980
aatatccaat ttcgaataac attatataaa atattgaact aattttaatt gtatacgcag 2040
gtatgcttca gagagtataa agttagcttg taaatgggga tacaaaggcg tcaagtctgg 2100
tgaaacgtta tcaggtacgt tgtttgcttc ttctttttct cgtacgctaa caaaaatatt 2160
taaaaataaa cccgaccaaa tgaagtttaa ttaatcggat taatgatttt taatagtcac 2220
tacttttttt gtgtgggata tatgactgtc taatatataa ttttataaga aagctaaagg 2280
atttgtttaa taatttccga taaataattt tgcagaagaa tatttcaata caaggttgcc 2340
aatagtgatg aagagaatag ttcagggagg agttagacta gccatgatac taaaccgggt 2400
ttttagtgac gatcatgcta ttgctggtgt tgctgccact tgaaccaaac ccgacatacc 2460
ggggcatcaa agcatttgat taagagatta tttgatacat tcacaaaatt aattaaggct 2520
gatcagacat tcttttcttt tagtagcttt atctatgtga cagctaatgc ctgtggactg 2580
ctttgtttag aagtgtttag cattagatca tatgctaatt caatgttatt aattcatcgt 2640
<210> 2
<211> 305
<212> PRT
<213> Arabidopsis thaliana
<220>
<221> misc#feature
<223> Protein ENDO 1 At1g11190
<400> 2
Met Ala Ser Ala Phe Arg Ser Ser Thr Arg Leu Ile Leu Val Leu Gly
1 5 10 15
Ile Leu Ile Leu Cys Ser Val Ser Ser Val Arg Ser Trp Ser Lys Glu
20 25 30
Gly His Ile Leu Thr Cys Arg Ile Ala Gln Asn Leu Leu Glu Ala Gly
35 40 45
Pro Ala His Val Val Glu Asn Leu Leu Pro Asp Tyr Val Lys Gly Asp
50 55 60
Leu Ser Ala Leu Cys Val Trp Pro Asp Gln Ile Arg His Trp Tyr Lys
65 70 75 80
Tyr Arg Trp Thr Ser His Leu His Tyr Ile Asp Thr Pro Asp Gln Ala
85 90 95
Cys Ser Tyr Glu Tyr Ser Arg Asp Cys His Asp Gln His Gly Leu Lys
100 105 110
Asp Met Cys Val Asp Gly Ala Ile Gln Asn Phe Thr Ser Gln Leu Gln
115 120 125
His Tyr Gly Glu Gly Thr Ser Asp Arg Arg Tyr Asn Met Thr Glu Ala
130 135 140
Leu Leu Phe Leu Ser His Phe Met Gly Asp Ile His Gln Pro Met His
145 150 155 160
Val Gly Phe Thr Ser Asp Glu Gly Gly Asn Thr Ile Asp Leu Arg Trp
165 170 175
Tyr Lys His Lys Ser Asn Leu His His Val Trp Asp Arg Glu Ile Ile
180 185 190
Leu Thr Ala Leu Lys Glu Asn Tyr Asp Lys Asn Leu Asp Leu Leu Gln
195 200 205
Glu Asp Leu Glu Lys Asn Ile Thr Asn Gly Leu Trp His Asp Asp Leu
210 215 220
Ser Ser Trp Thr Glu Cys Asn Asp Leu Ile Ala Cys Pro His Lys Tyr
225 230 235 240
Ala Ser Glu Ser Ile Lys Leu Ala Cys Lys Trp Gly Tyr Lys Gly Val
245 250 255
Lys Ser Gly Glu Thr Leu Ser Glu Glu Tyr Phe Asn Thr Arg Leu Pro
260 265 270
Ile Val Met Lys Arg Ile Val Gln Gly Gly Val Arg Leu Ala Met Ile
275 280 285
Leu Asn Arg Val Phe Ser Asp Asp His Ala Ile Ala Gly Val Ala Ala
290 295 300
Thr
305
<210> 3
<211> 28
<212> DNA
<213> Artificial sequence
<220>
<223> Primer 4-960
<400> 3
gtgtttgtcc agtaatagtg tcagcata 28
<210> 4
<211> 26
<212> DNA
<213> Artificial sequence
<220>
<223> Primer 4-721
<400> 4
aggaacctga gaaaagactc gccagc 26
<210> 5
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> CEL N Terminal
<400> 5
tatcgttcta gagggaatga cgcgattata ttctgtgttc 40
<210> 6
<211> 27
<212> DNA
<213> Artificial sequence
<220>
<223> CEL C Terminal
<400> 6
tatctgaatt catgccaaag aatgatc 27
<210> 7
<211> 50
<212> DNA
<213> Artificial sequence
<220>
<223> CEL C terminal 8 His
<400> 7
aattcaatgg tgatggtggt gatggtgatg tgccaaagaa tgatctgcgg 50
<210> 8
<211> 25
<212> DNA
<213> Artificial sequence
<220>
<223> Primer (R21)
<400> 8
gacatatgga ctacagaagc ttggg 25
<210> 9
<211> 25
<212> DNA
<213> Artificial sequence
<220>
<223> Primer (R22)
<400> 9
gttcacgggt cacatcatgc attcc 25
<210> 10
<211> 22
<212> DNA
<213> Artificial sequence
<220>
<223> Primer 4m118
<400> 10
ttggttggac ttcactttga gc 22
<210> 11
<211> 22
<212> DNA
<213> Artificial sequence
<220>
<223> Primer 4m984
<400> 11
cacaacaatc agcaatgaca gc 22
<210> 12
<211> 23
<212> DNA
<213> Artificial sequence
<220>
<223> Primer 4-347
<400> 12
gtgattgctc cacctccgcc acc 23
<210> 13
<211> 30
<212> DNA
<213> Artificial sequence
<220>
<223> Primer 4-134
<400> 13
tacagcgatt gatataatat aaaattatcc 30
<210> 14
<211> 27
<212> DNA
<213> Artificial sequence
<220>
<223> Primer le 2462
<400> 14
tgatattgtc gtgcaatatg atgaaac 27
<210> 15
<211> 25
<212> DNA
<213> Artificial sequence
<220>
<223> Primer le 3082
<400> 15
atacctattt agcccacttg gacac 25
<210> 16
<211> 27
<212> DNA
<213> Artificial sequence
<220>
<223> Forward Primer for ENDO5
<400> 16
aaggatccga aagctctgtg tttcaga 27
<210> 17
<211> 28
<212> DNA
<213> Artificial sequence
<220>
<223> Reverse Primer for ENDO5
<400> 17
ggagttgtta cgtgggttct caaggatc 28
<210> 18
<211> 28
<212> DNA
<213> Artificial sequence
<220>
<223> Forward Primer for ENDO4
<400> 18
ctggatccct gtttttaact ttggaaag 28
<210> 19
<211> 26
<212> DNA
<213> Artificial sequence
<220>
<223> Reverse Primer for ENDO4
<400> 19
ggatgttcaa gtgattctcc tggatc 26
<210> 20
<211> 27
<212> DNA
<213> Artificial sequence
<220>
<223> Forward Primer for ENDO3
<400> 20
aaggatccat tcgacaaact ttgtaac 27
<210> 21
<211> 28
<212> DNA
<213> Artificial sequence
<220>
<223> Reverse Primer for ENDO3
<400> 21
agagtggtct tgggaatatt tatctcag 28
<210> 22
<211> 26
<212> DNA
<213> Artificial sequence
<220>
<223> Forward Primer for ENDO2
<400> 22
acggatccca tttcaaagaa ctctga 26
<210> 23
<211> 26
<212> DNA
<213> Artificial sequence
<220>
<223> Reverse Primer for ENDO2
<400> 23
gaccaatcat tatgctgtaa cttcag 26
<210> 24
<211> 25
<212> DNA
<213> Artificial sequence
<220>
<223> Forward Primer for ENDO1
<400> 24
caggatccaa gtttcaaact tgaag 25
<210> 25
<211> 26
<212> DNA
<213> Artificial sequence
<220>
<223> Reverse Primer for ENDO1
<400> 25
cggtatgtcg ggtttggttc aagtgg 26
(もっと読む)
グルクロン酸残基を含む多糖の分解方法
【課題】 ウロン酸含有多糖を効率的に分解する手段を提供する。
【解決手段】 クロロウイルスCVK2から得られた多糖分解リアーゼであって、グルクロン酸残基間のβ-1,4結合又はα-1,4結合を切断する活性を持つリアーゼを用いて、2以上の連続したグルクロン酸残基を分子中に含む多糖を分解する方法。
(もっと読む)
レビー小体型認知症における分子診断法および治療
本発明は、ユビキチンカルボキシ末端ヒドロラーゼ L1(UCH−L1)のレベルまたはそのユビキチン−リガーゼ活性の変化に関連した、α−シヌクレイン症、詳しくはレビー小体型認知症(DLB)の分子診断法に関する。本発明はまた、UCH−L1レベルまたはUCH−L1の酵素活性を変更することができる化合物の使用にも関する。本発明は、DLBに罹患している患者の診断および治療において適用される。 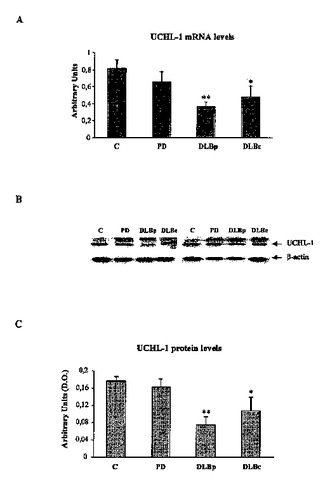 (もっと読む)
(もっと読む)
デンプン合成関連酵素等の機能解析法
【課題】 デンプン合成関連酵素等の機能解析を従来よりも簡便かつ短時間で行うことができる新たな解析手法を提供すること。
【解決手段】 本発明は、非変性の酵素を含む試料をゲル電気泳動後、ゲル内にて酵素反応させ、得られた反応生成物を含むゲル領域を切り出し、このゲル領域内の反応生成物の構造解析を行うことにより、当該酵素の機能を解析する方法である。たとえば、本発明の解析方法によってスターチシンターゼ(SS)の機能解析を行う場合には、Native−PAGE/SS活性染色法で染色されるバンドを含むゲルを切り出し、このゲル内に合成されたポリグルカンの鎖長構造をキャピラリー電気泳動法で決定するといった簡便な手法で、各アイソザイムの機能解析を簡便かつ短時間に行うことができる。
(もっと読む)
化合物同定用の代謝法
本発明は、フレーバーまたは香料としての、それらの前駆体としての、または香料もしくはフレーバーの知覚のモジュレーターとしての、化合物の同定に関する。本方法は、化合物を、鼻、口または気道中で発現される代謝酵素と反応させること、およびその後に、該化合物またはその代謝産物を、フレーバーもしくは香料として、それらの前駆体として、またはそれらの知覚もしくはそれらの対応する手掛かりの知覚のモジュレーターとして、同定することを含む。 (もっと読む)
糖尿病治療剤スクリーニング方法
【課題】新規メカニズムに基づく糖尿病治療剤のスクリーニング方法を提供する。
【解決手段】前記スクリーニング方法は、(1)α/βヒドロラーゼ2を抑制する活性があるか否かを検出する工程、及び(2)α/βヒドロラーゼ2を抑制する活性がある物質を選択する工程を含む。更に、抑制は発現抑制であり、プロモーター活性抑制活性を検出する工程を含む。
(もっと読む)
細胞内修飾の分析
タンパク質の細胞内での状態およびタンパク質修飾を測定するための改善された方法を、酵素断片相補性複合体および標的タンパク質のメンバーからなる代用融合タンパク質を導入することによって提供する。代用融合タンパク質を形質転換された細胞を、環境の変化(たとえば候補薬剤)にさらした後に、細胞を溶解し、好都合にはゲル電気泳動法によって溶解物を分画またはバンドへと分離し、そしてウエスタンブロット法によってタンパク質をメンブレンへとトランスファーする。メンブレン上のバンドを、もう一方の酵素断片相補性複合体および検出可能なシグナルを発する基質を用いて発色させる。本方法は、高い感度を有することと、標的タンパク質の修飾を観察する能力があることが分かった。  (もっと読む)
(もっと読む)
バイオロジカルソイル検出器およびその使用方法
バイオロジカルソイル検出器と、バイオソイルを検出するための方法とが提供される。バイオロジカルソイル検出器は、固体支持部材と、固体支持部材に固定された特異的指標であって、血液の存在に対して敏感な材料を含む特異的指標と、を含み、固体支持体および特異的指標は、表面の接触に用いられた液体との接触を容易にすべく検出器に対して配置されている。バイオソイルを検出するための方法において、この方法は、表面を液体と接触させるステップと、液体が表面と接触した後に、この液体を本願明細書にて記載の検出器の固体支持部材および特異的指標と接触させるステップと、固体支持部材の検出可能な応答を検査して、特異的指標がバイオロジカルソイルと相互作用したか否かを判断するステップと、を含む。 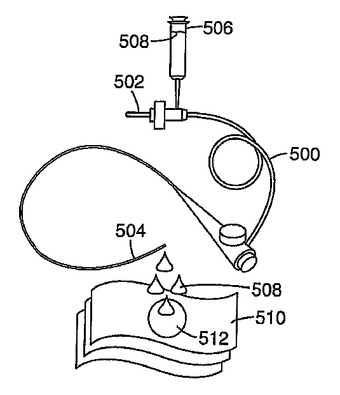 (もっと読む)
(もっと読む)
351 - 360 / 405
[ Back to top ]